Область изобретения
[01] Изобретение относится к области молекулярной биологии и медицины. В частности, изобретение направлено на методы детекции мутаций в образцах ДНК.
Уровень техники
[02] Ранняя диагностика и лечение онкологических заболеваний остаются одними из самых актуальных проблем современной медицины. Выявление таких заболеваний на ранних стадиях и выбор адекватной стратегии лечения позволяют существенно повысить шансы пациентов на выздоровление и снизить показатели смертности от рака.
[03] Основной причиной возникновения злокачественных новообразований в организме человека являются мутации, повреждающие работу важнейших регуляторных генов. Природа этих мутаций определяет вид опухоли, степень их злокачественности и чувствительность опухоли к лекарственным препаратам. Анализ молекул ДНК, несущих мутации опухолевого происхождения (онкомутации), позволяет прогнозировать агрессивность опухолей вероятность появления метастазов, а также рекомендовать выбор стратегии лечения. Вместе с тем онкомутации обладают почти абсолютной специфичностью, что делает их пригодными для оценки лекарственной чувствительности опухоли и детекции приобретенной резистентности. Таким образом, выявление онкомутаций имеет огромное прогностическое и диагностическое значение, что было доказано многолетней практикой (см., например, van der Heijden, M.S., et al. Clin Cancer Res. 2005 Oct 15; 11(20):7508-15; Thomas, R.K., et al. Nat Genet. 2007; 39(3):347-51; Diehl, F., et al. Nat Medicine. 2008; 14(9):985-90; Rizvi, Ν.Α., et al. Clin Cancer Res. 2011; 17(10):3500-6).
[04] Анализ мутаций в «горячих точках» ключевых онкогенов и генов-супрессоров, включая мутации в экзоне 15 гена BRAF, экзоне 7 гена ARAF, экзоне 7 гена RAF1, экзоне 11 гена KIT, экзонах 2, 3, 4 гена KRAS, экзонах 2, 3, 4 гена NRAS, экзонах 2, 3, 4 гена HRAS, экзонах 2, 3, 6 гена MAP2K1, экзонах 4 и 6 гена MAP2K2, экзоне 7 гена MAPK1, экзонах 9 и 20 гена PIK3CA, экзоне 2 гена AKT1, экзоне 5 гена GNAQ, экзоне 5 гена GNA11, экзоне 2 гена RAC1, экзонах 12, 18, 19, 20, 21 гена EGFR, экзонах 8, 18, 19, 20, 21 гена HER2, экзонах 2, 3, 6, 7, 8, 9, 21, 23 гена HER3, экзонах 8, 12, 14, 21 гена HER4, экзонах 5, 6, 7, 8, 9 гена ТР53 и экзоне 3 гена CTNNB1 имеет существенное диагностическое, прогностическое и предиктивное значение при многих видах онкозаболеваний, включая рак легкого, рак молочной железы, рак толстой и прямой кишки, злокачественную меланому кожи, рак щитовидной железы, рак желудка, рак поджелудочной железы и желчных протоков, рак мочевого пузыря, глиальные опухоли голового мозга, саркомы костей и мягких тканей, многие типы лейкозов и лимфом (Vogelstein, В., et al. Science. 2013 Mar 29; 339(6127):1546-58; Watson, I.R., et al. Nat Rev Genet. 2013 Oct; 14(10):703-18; Weinstein, J.N., et al. Nat Genet. 2013 Oct; 45(10):1113-20; Kandoth, C., et al. Nature. 2013 Oct 17; 502(7471):333-9; http:cancer.sanger.ac.uk; http:cancergenome.nih.gov; http:p53.free.fr; и мн. др.) Многие из указанных мутаций могут быть использованы как неинвазивные маркеры опухолевого присутствия, в том числе на ранних стадиях заболевания (Fleischhacker, M., Schmidt, В. Biochim Biophys Acta. 2007; 1775(1):181-232; Chen, Z., et al. PLoS One. 2009; 4(9):e7220; Newman, A.M., et al. Nat Med. 2014 May; 20(5):548-54).
[05] К настоящему моменту разработано значительное количество методов для выявления мутаций в ДНК. Большинство этих методов включают избирательную амплификацию аллелей, несущих мутации или полиморфизмы, в ходе ПЦР. Можно выделить следующие основные группы методов: аллель-специфическую ПЦР (Newton, С.R., et al. Nucleic Acids Res. 1989 Apr 11; 17(7):2503-16; Ruano, G., Kidd, K.K. Nucleic Acids Res. 1989; 17(20):8392; Sommer, S.S., et al. Mayo Clin Proc. 1989; 64(11):1361-72), ПЦР, опосредованную лигированием (Wu, D.Y. and Wallace, R.B. Genomics. 1989; 4(4):560-9; Shi, C., et al. Nat Methods. 2004; 1(2):141-7); корректирующую ПЦР (Zhang, J., et al. Trends Biotechnol. 2005 Feb; 23(2):92-6; Liu, Q., Sommer, S.S. Biotechniques. 2000 Nov; 29(5):1072-6); «холодную» ПЦР (Li, J., et al. Nat Med. 2008; 14(5):579-84; Milbury, C.Α., et al. Nucleic Acids Res. 2011; 39(1):e2); ПЦР, блокирующую дикий тип (Seyama, T., et al. Nucleic Acids Res. 1992; 20(10):2493-6; Orou, Α., et al. Hum Mutat. 1995; 6(2):163-9; Beau-Faller, M., et al. Br J Cancer. 2009; 100(6):985-92).
[06] Существенным недостатком первых двух методов является фактическое внесение в матрицу искомого аллельного варианта в ходе ПЦР. Продукт, полученный с применением этих методов, не может быть проверен никаким дополнительным способом, поскольку истинно положительный и ложноположительный результаты дают абсолютно одинаковую картину.
[07] Метод корректирующей ПЦР основан на реакциях, которые в любых условиях протекают с очень низкой эффективностью; вследствие этого он является не вполне применимым к большинству задач поиска редких мутаций и полиморфизмов, в которых исследуются частично деградированные образцы ДНК, а количество искомых молекул «на старте» может исчисляться единицами.
[08] Методы «холодной» ПЦР и ПЦР, блокирующей дикий тип, основаны на крайне небольших различиях в температуре плавления совершенных и несовершенных дуплексов; вследствие этого для получения воспроизводимых результатов при использовании этих методов необходимо прибегать или к использованию модифицированных олигонуклеотидных зондов (LNA, PNA, Р-HyP-PNA, ZNA и др.), которые дороги и сложны в синтезе, или к особым модификациям приборов для проведения ПЦР, позволяющим трансформировать небольшую разницу в температуре плавления в существенную разницу в эффективности ПЦР (а такие приборы существенно дороже стандартных, и подбор условий ПЦР для них достаточно сложен), или к крайне неудобным режимам проведения ПЦР (60 циклов продолжительностью более 10 минут каждый) (Galbiati, S., et al. Clin Chem. 2011; 57(1):136-8).
[09] Тем не менее, при оптимизации условий перечисленные методы способны обеспечить избирательность порядка 1:100-1:200 (Pao, W., Ladanyi, M. Clin Cancer Res. 2007; 13(17):4954-5; Milbury, C.Α., et al. Clin Chem. 2009; 55(4):632-40) и широко используются для анализа мутаций. Однако для получения образцов ДНК, пригодных для анализа этими методами, необходимо взятие биопсии непосредственно из опухоли, что невозможно на ранних стадиях развития рака. Кроме того, все указанные методы позволяют анализировать только определенные мутации в заранее известных точках, что значительно снижает информативность анализа.
[010] В последние годы было установлено, что фрагменты ДНК, несущие мутации опухолевого происхождения, присутствуют в биологических жидкостях человека даже при минимальном размере опухолевого очага (Diehl, F., et al. Proc Natl Acad Sci USA. 2005; 102(45):16368-73; Fleischhacker, M., Schmidt, В. Biochim Biophys Acta. 2007; 1775(1):181-232; Chen, Z., et al. PLoS One. 2009; 4(9):e7220). Детектирование мутаций в образцах опухолевой ДНК из крови пациентов открывает перспективы для ранней диагностики опухоли и ее рецидивов, мониторинга остаточной болезни и оценки радикальности проведенного лечения.
[011] Однако на пути выявление таких мутаций встают нерешенные до настоящего времени технологические проблемы. Главные из них это: (а) необходимость одновременного анализа панели онкомутаций на фоне крайне малого количество циркулирующей ДНК в биологических жидкостях и (б) наличие в биологических жидкостях немутантой ДНК в соотношении 1000-100000 копий на одну молекулу опухолевого происхождения. Указанные проблемы делают невозможным использование классических методов мутационного анализа, перечисленных выше, так как их разрешающая способность на порядки ниже требуемого уровня (Liu, Q., Sommer, S.S. Hum Mutat. 2004 May; 23(5):426-36; Gilje, В., et al. J Mol Diagn. 2008 Jul; 10(4):325-31; Busby, K., Morris, A.J. Clin Pathol. 2005, 58(4):372-5). Таким образом, существует потребность в методах детекции мутаций, способных выявлять мутантную нуклеиновую кислоту на фоне тысячекратного (и выше) избытка референсной нуклеиновой кислоты.
[012] Предлагаемый в настоящей заявке способ направлен на выявление мутаций в образцах ДНК, где мутантные молекулы находятся в смеси с избытком нормальной (немутированной) ДНК. Метод находит применение, например, для выявления мутаций, анализ которых востребован для диагностики и мониторинга заболеваний, в ДНК опухолевого происхождения, выделенной из биопсийного материала или из биологических жидкостей.
[013] Предлагаемый метод основан на молекулярном баркодировании, последующей амплификации целевых молекул ДНК и их секвенировании высокопроизводительными методами секвенирования, такими как методы секвенирования нового поколения.
[014] Как здесь используется, термины «высокопроизводительное секвенирование», «секвенирование нового поколения» или «NGS» (от next generation sequencing) относятся к группе технологий одновременного секвенирования огромного числа единичных молекул ДНК, в том числе технология 454, технология Illumina (Solexa SBS Technology), технология SoLID и технология IonTorrent (Metzker M.L. Nat Rev Genetics. 2010; 11(1):31-46; Mardis, Ε.R. Annual review of genomics and human genetics. 2008; 9:387-402; Bentley D.R., et al. Nature. 2008; 456(7218):53-59; Rothberg J.M., et al. Nature. 2011; 475(7356):348-352; Drmanac R., et al. Science. 2010; 327(5961):78-81; Eid J., et al. Science. 2009; 323(5910): 133-138; Lam H.Y., et al. Nature biotechnology. 2012; 30(1):78-82; Clark, M.J., et al. Nat Biotech. 2011; 29(10):908-914; Quail M.A., et al. BMC genomics. 2012; 13:341). Для всех этих технологий необходимы этапом пробоподготовки является введение так называемых «сиквенсовых адаптеров» в последовательность ДНК таким образом, что молекула ДНК оказывается фланкирована с одной стороны последовательностью адаптера А, а с другой - последовательностью адаптера Б. Последовательности секвенсовых адаптеров известны специалистам в данной области и предоставляются производителями коммерческих наборов для секвенирования нового поколения.
[015] Близким аналогом предлагаемого метода является метод, предложенный Шмиттом и коллегами (Schmitt, M.W., et al. Proc Natl Acad Sci USA. 2012 Sep 4; 109(36):14508-13). Метод включает этапы синтеза двухцепочечных молекул баркодирующих ДНК-адаптеров, лигирования этих ДНК-адаптеров к молекулам ДНК образца, ПЦР-амплификации с универсальными праймерами, секвенирования нового поколения (NGS) и анализа данных с фильтрацией ошибок ПЦР и NGS. Синтез несущих баркод адаптеров осуществляется с помощью ПЦР на синтетической ДНК-матрице - олигонуклеотиде, содержащем случайную последовательность из 6-24 нуклеотидов.
[016] Заявленный метод отличается от указанного технологией баркодирования: (1) не используется полимеразный синтез двухцепочечных баркодирующих ДНК-адаптеров, что существенно снижает риск появления ошибок внутри самих баркодов; (2) введение баркодов осуществляется с помощью линейной ПЦР, что позволяет существенно увеличить разнообразие библиотеки, кардинально повысить специфичность анализа путем отсечения нецелевых молекул на этапе амплификации, а также сократить время пробоподготовки в случае анализа конечного количества регионов генома.
[017] Другой метод описан в работе Кинде и соавторов (Kinde, I., et al. Proc Nat Acad Sci USA. 2011; 23:9530-35). Метод включает введение синтетических баркодирующих праймеров с помощью ПЦР, дальнейшую ПЦР-амплификацию баркодированных молекул с использованием универсальных праймеров, секвенированиенового поколения (NGS) и анализ данных с фильтрацией ошибок ПЦР и NGS. Для введения баркодов используются 2 цикла ПЦР в присутствии баркодирующего и встречного ему праймеров.
[018] Баркодирование с помощью стандартной ПЦР имеет ряд недостатков. В связи с тем, что баркодированию должны подвергаться только молекулы ДНК исходного образца (но не продукты их амплификации), возможно использование не более двух циклов стандартной ПЦР (а в идеале - один цикл). Таким образом, каждой исходной молекуле соответствует единственная баркодированная копия. Это делает невозможным в дальнейшем различение ошибок полимеразного синтеза от истинных мутаций за счет «фильтрации по количеству». Принцип «фильтрации по количеству» основан на том, что истинные мутации, изначально присутствующие в образце, должны встречаться в «сырых данных» секвенирования значимо чаще, чем случайные замены, возникшие в ходе пробоподготовки и секвенирования. Кроме того, в этом случае не происходит увеличения исходного пула молекул для дальнейшего анализа, что осложняет работу с малыми количествами биологического материала.
[019] Заявленный метод предполагает более эффективную процедуру баркодирования с помощью линейной ПЦР. Использование линейной ПЦР для введения молекулярных баркодов имеет целый ряд принципиальных преимуществ по сравнению со стандартной ПЦР, в частности: 1) снимается ограничение на количество циклов реакции баркодирования, что дает возможность эффективно работать с низкими количествами исходной ДНК, а также выявлять ошибки ДНК-полимеразы, возникающие в процессе реакции баркодирования, путем «фильтрации по количеству»; 2) сохраняется стрэнд-специфичность реакции, что позволяет путем постановки двух реакций (первая - с прямым праймером, вторая - с обратным праймером на тот же регион генома) отличить истинные мутации от результатов повреждения исходной ДНК. Оптимизированные условия экзонуклеазной реакции, используемые в ряде применений заявленного метода, позволяют эффективно гидролизовать неиспользованные праймеры без существенной деградации продуктов линейной ПЦР. Кроме того, заявленный метод, в отличие от описанного Кинде и коллегами, адаптирован под мультиплексный анализ целевых регионов генома.
Сущность изобретения
[020] Настоящее изобретение обеспечивает способ выявления мутаций в целевых последовательностях ДНК (мутантных ДНК), отличающих целевые последовательности анализируемого образца ДНК от референсных последовательностей ДНК.
[021] В некоторых применениях мутантная ДНК содержит одну или несколько точечных мутаций. В некоторых применениях мутантная ДНК содержит одну или несколько делеций одного или нескольких нуклеотидов. В некоторых применениях мутантная ДНК содержит одну или несколько инсерций одного или нескольких нуклеотидов.
[022] Метод настоящего изобретения включает:
(а) молекулярное баркодирование пула целевых молекул ДНК в образце ДНК;
(б) очистку продукта реакции баркодирования от остатков баркодирующего праймера;
(в) амплификацию пула целевых молекул;
(г) введение сиквенсовых адаптеров в продукт ПЦР и секвенирование указанного продукта ПЦР;
(д) идентификацию мутаций в целевых ДНК по данным секвенирования.
[023] Заявленный метод позволяет выявлять мутации в целевой последовательности ДНК в образцах, где содержится как мутантная, так и немутантная ДНК, причем отношение концентрации мутантной ДНК к немутантной может быть менее чем 1:100, например находится в пределах 1:100-1:100000, например, быть менее чем 1:140, быть менее чем 1:300, быть менее чем 1:1000, быть менее чем 1:5000 или быть менее чем 1:10000.
[024] В частности, заявленный метод позволяет выявлять мутации в фрагментах генов, выбранных из SEQ ID No: 1 - SEQ ID No: 62. Число целевых последовательностей, отобранных для анализа, может варьировать в зависимости от целей анализа и включает по крайней мере пять целевых последовательностей, чаще 6, 7 или более последовательностей, например, десять или более последовательностей, чаще семнадцать или более последовательностей, тридцать или более последовательностей, сорок или более последовательностей, пятьдесят или более последовательностей, шестьдесят и более последовательностей, семьдесят или более последовательностей, например, 10, 17, 18, 27, 28, 34, 35, 44, 45 или 62 последовательности.
[025] В преимущественных воплощениях молекулярное баркодирование осуществляется методом мультиплексной линейной ПЦР в присутствии баркодирующих праймеров. Число баркодирующих праймеров, используемых для линейной ПЦР, равно числу последовательностей, отобранных для анализа.
[026] В некоторых воплощениях линейная ПЦР осуществляется в одной пробирке, в некоторых воплощениях линейная ПЦР осуществляется в двух, трех, четырех или более пробирках, каждая из которых содержит свой набор баркодирующих праймеров.
[027] В преимущественных воплощениях очистка от баркодирующего праймера включает обработку реакционной смеси экзонуклеазой I.
[028] Баркодированные молекулы ДНК амплифицируют в мультиплексной ПЦР и продукт амплификации используют для секвенирования. ПЦР осуществляется в том же числе пробирок, что и баркодирование целевых молекул. В преимущественных воплощениях секвенирование продукта амплификации осуществляется методами высокопроизводительного секвенирования.
[029] В некоторых воплощениях введение сиквенсовых адаптеров осуществляется с помощью ПЦР с выходом наружу в присутствии заглубленных внутренних геноспецифических праймеров.
[030] В преимущественных воплощениях для выявления мутаций в данных секвенирования используется анализ потомков каждой целевой молекулы из исходного образца ДНК, где потомки одной молекулы выявляются по последовательности индивидуального баркода.
[031] Также обеспечиваются наборы праймеров для осуществления метода настоящего изобретения для выявления мутаций во фрагментах ДНК, выбранных из группы SEQ ID No: 1 - SEQ ID No: 62.
[032] Также обеспечиваются наборы реагентов для осуществления метода настоящего изобретения для выявления мутаций во фрагментах ДНК, выбранных из группы SEQ ID No: 1 - SEQ ID No: 62. В преимущественных воплощениях наборы включают праймеры для баркодирования и праймеры для амплификации целевых молекул. В некоторых воплощениях наборы также включают праймеры для введения последовательностей сиквенсовых адаптеров в целевые молекулы с помощью заглубленной ПЦР и ПЦР с выходом наружу. В некоторых воплощениях наборы также включают реагентгенты для осуществления ПЦР. В некоторых воплощениях наборы также включают реагентгенты для очистки продуктов реакции баркодирования.
Краткое описание рисунков
[033] Рисунок 1 показывает структуру баркодирующего праймера.
[034] Рисунок 2 иллюстрирует последовательность процедур пробоподготовки преимущественного воплощения метода настоящего изобретения.
[035] Рисунок 3 иллюстрирует различные способы введения сиквенсовых адаптеров.
[036] Рисунок 4 показывает электрофореграммы продуктов двух последовательных раундов ПЦР после реакции баркодирования в присутствии двух праймеров (дорожка 1) и после реакции баркодирования в присутствии одного праймера (дорожка 2). M - маркер длин ДНК 100+ bp DNA Ladder (Евроген, Россия).
[037] Рисунок 5 показывает электрофореграмму продуктов амплифкации образцов BRAF-wt (дорожка 1); BRAF-mut100 (дорожка 2); BRAF-mut1000 (дорожка 3). M - маркер длин ДНК 100+ bp DNA Ladder (Евроген, Россия).
Подробное описание изобретения
[038] Как указано выше, настоящее изобретение направлено на метод и набор для выявления мутаций в образце ДНК.
[039] Метод настоящего изобретения включает
[040] (а) молекулярное баркодирование пула целевых молекул ДНК в образце ДНК;
[041] (б) очистку продукта реакции баркодирования от остатков баркодирующего праймера;
[042] (в) амплификацию пула целевых молекул в мультиплексной ПЦР;
[043] (г) введение сиквенсовых адаптеров в продукт мультиплексной ПЦР и секвенирование указанного продукта мультиплексной ПЦР;
[044] (д) идентификацию мутаций в целевых ДНК по данным секвенирования.
[045] Заявленный метод позволяет выявлять мутации в целевой последовательности ДНК в образцах, где содержится как мутантная, так и немутантная ДНК, причем отношение концентрации мутантной ДНК к немутантной может составлять 1:300-1:100000, например, 1:2000, 1:5000 или 1:10000.
[046] Указанный метод находит применение во многих приложениях, в частности, для детекции соматических мутаций для широкого круга задач в биологии и медицине, при анализе уровня спонтанного мутагенеза и при анализе генетического полиморфизма на пулированных образцах (в частности, в популяционной, эволюционной и экологической генетике). В частности, метод настоящего изобретения находит применение для выявления опухолевого присутствия по анализу внеклеточной ДНК из биологических жидкостей (кровь, моча).
[047] Также обеспечиваются наборы для осуществления метода настоящего изобретения.
[048] В преимущественных воплощениях наборы настоящего изобретения позволяют анализировать мутации во фрагментах генов, выбранных из группы SEQ ID No: 1 - SEQ ID No: 62. Метод настоящего изобретения может быть использован также для анализа любых других мутаций, однако для этого требуется подбор подходящих геноспецифических последовательностей праймеров и условий мультиплексной ПЦР.
[049] Различные термины, относящиеся к биологическим молекулам и методам настоящего изобретения, используются выше и также в описании и в формуле изобретения.
[050] Следует заметить, что, как применяют здесь и в прилагаемой формуле изобретения, формы единственного числа включают объекты-ссылки множественного числа, если контекст явно не требует другого. Так, например, ссылка на «нуклеиновую кислоту» включает множество молекул таких нуклеиновых кислот.
[051] Как здесь используется, молекула нуклеиновой кислоты - это молекула ДНК, такая как геномная ДНК или кДНК молекула. Как здесь используется, термин «кДНК» относится к нуклеиновым кислотам, которые обладают размещением элементов последовательности, найденным в нативных зрелых видах мРНК, где элементы последовательности - это экзоны и 5'- и 3'-некодирующие области.
[052] Как здесь используется, термин «пробоподготовка» относится ко всем манипуляциям, которым подвергаются нуклеиновые кислоты после очистки и до секвенирования.
[053] Как здесь используется, термин «мутант» или «мутантный» относится к молекуле ДНК, последовательность нуклеотидов которой отличается от последовательности референсной ДНК. Кроме того, термин «мутант» здесь относится к любому варианту, который короче или длиннее референсной ДНК. В некоторых применениях отличием является точечная замена. В некоторых применениях отличием является делеция или инсерция одного или нескольких нуклеотидов. Для нужд настоящего изобретения размер делеции или инсерции не превышает 150 нуклеотидов, преимущественно не превышает 100 нуклеотидов. За исключением указанных отличий, последовательности мутантной и референсной ДНК идентичны. В преимущественных применениях последовательность референсной ДНК идентична последовательности ДНК дикого типа человека.
[054] Как здесь используется, термин «горячая точка» относится к позиции в молекуле ДНК, если в этой позиции при определенных условиях часто возникает мутация. Так, по отношению к ключевым онкогенам и генам-супрессорам термин «горячая точка» обозначает позицию мутации, часто возникающей в онкогенезе. По отношению к ошибкам ПЦР термин «горячая точка» обозначает позицию в ДНК-матрице, если в этой позиции преимущественно локализуются ошибки полимеразного синтеза ДНК.
[055] Процент идентичности последовательностей с референсной определяется путем их сравнительного анализа. Алгоритмы для анализа последовательности известны в данной области, такие как BLAST, описанный в: Altschul et al. J. Mol. Biol., 1990, 215, pp. 403-10. Для целей настоящего изобретения сравнение нуклеотидных и аминокислотных последовательностей, производимое с помощью пакета программ Blast, предоставляемого National Center for Biotechnology Information (http:www.ncbi.nlm.nih.gov/blast), с использованием содержащего разрывы выравнивания со стандартными параметрами, может быть использовано для определения уровня идентичности и сходства между нуклеотидными последовательностями.
[056] Как здесь используется, нуклеотидные последовательности «по существу сходны» или «по существу такие же, как референсная последовательность», если нуклеотидные последовательности имеют по крайней мере 85% идентичности друг с другом или с указанной последовательностью внутри выбранного для сравнения региона. Таким образом, по существу сходные последовательности включают те, которые имеют, например, по крайней мере, 85% идентичности, по крайней мере, 90% идентичности, по крайней мере, 95% идентичности или по крайней мере, 96%, 97%, 98% или 99% идентичности. Две последовательности, которые идентичны одна другой, так же по существу сходны.
[057] Как здесь используется, термин «ПЦР» относится к широко известному методу полимеразной цепной реакции (описанному, например, в Ausubel et al., Current Protocols in Molecular Biology, John Wiley and Sons, New York, 1989; Erlich, PCR Technology, Stockton Press, New York, 1989; Innis et al., PCR Protocols: A Guide to Methods and Applications, Academic Press, Harcourt Brace Javanovich, New York, 1990; Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1989; Barnes, W.M. Proc Natl Acad Sd USA, 1994, 91, 2216-2220). Стандартная реакционная смесь для ПЦР включает ДНК-полимеразу, буфер, обеспечивающий оптимальную работу ДНК-полимеразы, хлорид магния, дезоксинуклеотидтрифосфаты, ДНК-матрицу и олигонуклеотидные праймеры.
[058] Реакционная смесь для ПЦР содержит Mg2+-ионы в концентрации 0,1-10 мМ, дезоксирибонуклеозидтрифосфаты (dNTPs), включая АТФ (dATP), ГТФ (dGTP), ЦТФ (dCTP) и ТТФ (dTTP), в конечной концентрации 20-800 мкМ каждого, как правило, 160-600 мкМ, преимущественно 200-300 мкМ. Реакционная смесь для ПЦР может содержать другие компоненты, например, вещества, облегчающие амплификацию GC-богатых участков, антитела, обеспечивающие горячий старт, и т.д. Подобные компоненты хорошо известны специалистам в данной области.
[059] Объем реакционной смеси для ПЦР составляет, как правило, 10-100 мкл, чаще 15-50 мкл, например, 20 мкл или 25 мкл. Количество ДНК матрицы, добавляемой в реакционную смесь для ПЦР, находится в пределах от 10 пг до 1 мкг, как правило, от 100 пкг до 200 нг, например, 2 нг на реакцию, или 5 нг на реакцию, или 10 нг на реакцию, или 50 нг на реакцию, или 100 нг на реакцию.
[060] Как здесь используется, термин «праймер» относится к олигонуклеотидам, рутинно используемым в ПЦР в качестве затравки для ДНК-полимеразы. Как здесь используется, термин «геноспецифический» относится к последовательностям праймеров, способным отжигаться (имеющим «сайт отжига») на целевую нуклеиновую кислоту за счет входящих в его состав последовательностей, комплементарных указанной нуклеиновой кислоте. В качестве праймеров выступают синтетические олигонуклеотиды. Синтетические олигонуклеотиды могут быть приготовлены с помощью фосфорамидитного метода и очищены с помощью методов, хорошо известных в данной области, таких как высокоэффективная жидкостная хроматография (ВЭЖХ), или других методов, как описано, например, в Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd Ed., (1989) Cold Spring Harbor Press, Cold Spring Harbor, NY, или по инструкции, описанной в, например, United States Dept. of HHS, National Institute of Health (NIH) Guidelines for Recombinant DNA Research.
[061] Олигонуклеотидные праймеры, используемые для ПЦР, имеют «сайт отжига» на целевые последовательности, например, на последовательности, введенные в молекулы ДНК в ходе предшествующей пробоподготовки (так называемые универсальные или адаптерные последовательности) или на геноспецифические последовательности целевых молекул. Термин «сайт отжига» в этом контексте означает последовательность нуклеотидов, комплементарную целевой последовательности и служащую затравкой для ДНК-полимеразы.
[062] Способы выбора геноспецифической последовательности для праймера хорошо известны. Как правило, длина геноспецифической последовательности не менее 10, но не более 100 нуклеотидов, чаще 15-30 нуклеотидов. Адаптерная последовательность выбирается таким образом, чтобы минимизировать неспецифическую амплификацию в ходе последующих ПЦР. Длина адаптерной последовательности может варьировать от 10 до 100 нуклеотидов и как правило составляет 15-30 нуклеотидов.
[063] Как здесь используется, термин «целевая нуклеиновая кислота» относится к нуклеиновой кислоте, мутации в которой представляют интерес.
[064] Как здесь используется, термин «выделенный» означает молекулу или клетку, которая находится в среде, отличной от среды, в которой молекула или клетка находится в естественных условиях.
[065] Как здесь используется, термины «опухолевая ДНК», «ДНК опухолевого происхождения» или подобные означают ДНК или кДНК, происходящую из злокачественных опухолевых клеток.
[066] Как здесь используется, термин «онкомутация» означает изменение в последовательности гена (точечную замену, делецию или инсерцию), возникшее в опухолевой ДНК.
[067] Как здесь используется, термин «внеклеточная ДНК» означает ДНК, находящуюся в биологических жидкостях, циркулирующих во внеклеточном пространстве (например, в моче или в плазме крови).
[068] Получение образца ДНК, содержащего целевые молекулы ДНК
[069] Мутантная ДНК может присутствовать в исследуемом образце ДНК, выделенном из биологического материала, или быть добавлена экзогенно.
[070] Различия в нуклеотидной последовательности между высоко сходными нуклеотидными последовательностями мутантной и референсной ДНК (замены, делеции и инсерции) могут представлять однонуклеотидные полиморфизмы, а также мутации в последовательности, которые возникают в процессе нормальной репликации или дупликации, в том числе соматические мутации или мутации de novo.
[071] Другие мутации (замены, делеции и инсерции) могут быть специально рассчитаны и вставлены в последовательность для определенных целей, таких как создание искусственной мутантной ДНК. Такие специальные замены могут быть произведены in vitro с помощью различных технологий мутагенеза или получены в организмах-хозяевах, находящихся в специфических селекционных условиях, которые индуцируют или отбирают эти изменения. Такие специально полученные варианты последовательности могут быть названы «мутантами» или «производными» исходной последовательности.
[072] Мутантные ДНК могут быть получены на матричной нуклеиновой кислоте путем модификации, делеции или добавления одного или более нуклеотидов в матричной последовательности или их комбинации, для получения варианта матричной нуклеиновой кислоты. Модификации, добавления или делеции могут быть выполнены любым способом, известным в данной области (см. например Gustin et al. Biotechniques. 1993. 14:22; Barany Gene. 1985. 37:111-123; Colicelli et al. Mol. Gen. Genet. (1985. 199:537-539; Sambrook et al., Molecular Cloning: A Laboratory Manual, (1989), CSH Press, pp. 15.3-15.108), включая подверженный ошибкам ПЦР (error-prone PCR), shuffling, олигонуклеотид-направленный мутагенез, ПЦР со сборкой, парный ПЦР-мутагенез, мутагенез in vivo, кассетный мутагенез, рекурсивный множественный мутагенез, экспоненциальный множественный мутагенез, сайт-специфический мутагенез, случайный мутагенез, генное реассемблирование (gene reassembly), генный сайт-насыщающий мутагенез (GSSM), искусственную перестройку с лигированием (SLR) или их комбинации. Модификации, добавления или делеции могут быть также выполнены методом, включающим рекомбинацию, рекурсивную рекомбинацию последовательностей, фосфотиоат-модифицированный мутагенез ДНК, мутагенез на урацил-содержащей матрице, мутагенез с двойным пропуском, точечный восстановительный по рассогласованию мутагенез, мутагенез штамма, дефицитного по восстановлениям, химический мутагенез, радиоактивный мутагенез, делеционный мутагенез, рестрикционно-избирательный мутагенез, рестрикционный мутагенез с очисткой, синтез искусственных генов, множественный мутагенез, создание химерных множественных нуклеиновых кислот и их комбинации.
[073] Образец ДНК может быть выделен из биологического материала, например, из культур клеток, тканей, мазков и соскобов, биопсийного материала, биологических жидкостей (кровь, моча, слюна) животных, включая человека. Для выделения геномной ДНК могут быть использованы любые методы, известные в данной области. Например, могут быть использованы методы, описанные Sambrook et al. (Molecular Cloning: A Laboratory Manual, 2nd edition. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, 1989), или коммерчески доступные наборы для выделения ДНК, такие как наборы серий DiaTom или MagNA (ООО «Лаборатория Изоген», Россия) или серии DNEasy (фирма QiaGen, Германия) или их аналоги.
[074] Из биологических объектов может быть так же выделена суммарная РНК или поли(А) фракция РНК. Выделение РНК может быть выполнено любым способом, известным в данной области (например, могут быть использованы коммерчески доступные реагенты для выделения РНК, такие как Trizol (GIBCO/Life Technologies), TRI Reagent (Ambion), или RNeasy kits (QIAGEN) или метод гуанидин тиоцианат-фенол-хлороформной экстракции, описанный Chomczynski & Sacchi, 1987 (Anal. Biochem. 162, 156-159). Образцы РНК могут быть использованы для приготовления образцов кДНК с помощью технологий синтеза, известных в данной области, например, метод Гилбера-Хоффмана (Gubler, U., Hoffman, B.J. Gene. 1983. V. 25. P. 263-269), методы с использованием ДНК-лигазы фага Т4 (Akowitz & Manuelidis Gene. 1989. V. 81. P. 295-306; Lukyanov et al. Biophys. Biochem. Res. Comm. 1997. V. 230, P. 285-288), метод синтеза кДНК со сменой матрицы (Chenchik et al., (1998) Gene Cloning and Analysis by RT-PCR, ed. by Ρ Siebert and J Larrick, BioTechniques Books, Natick, MA, 305-319; Zhu et al. Biotechniques. 2001. 30, 892-897) и другие.
[075] В некоторых применениях образец ДНК является образцом опухолевой или фетальной ДНК или кДНК, присутствующей в биологической жидкости человека или животного в составе циркулирующих опухолевых или фетальных клеток, плазматических микровезикул, комплексов с белками, углеводами и липидами или в свободном виде. Известно, что ДНК опухолевого происхождения содержится в биологических жидкостях в очень малых количествах даже при метастатических опухолях (Diehl, F, et al. Gastroenterology. 2008 Aug; 135(2):489-98). Для обогащения образца такой ДНК биологический материал может быть подвергнут предварительной обработке. Например, может быть использован метод отделения плазмы крови от клеток крови для последующего анализа низкопредставленных мутаций опухолевого или фетального происхождения (как описано, например, в: Lo, Y.M.D., et al. Clin Chem. 1999 Oct; 45(10):1747-51). В другом приложении выделение ДНК или РНК может быть осуществлено из сыворотки крови (как описано, например, Anker, P., et al. Cancer Metastasis Rev. 1999; 18(1):65-73). В некоторых применениях может быть также осуществлено фракционирование клеточного осадка биологической жидкости с целью отделения фракции опухолевых или фетальных клеток путем сепарации с помощью антител на проточных цитометрах (как описано, например, в: Min, A. et al. Clin Exp Metastasis. 2009; 26(7):759-67) или с использованием магнитных частиц (как описано, например, в: Bilkenroth U., et al. Int J Cancer. 2001 May 15; 92(4):577-82), фильтрации через мембрану (как описано, например, в: Takano T., et al. Head Neck. 2008 Aug; 30(8):983-90), центрифугирования в градиенте плотности (как описано, например, в: Rosenberg, R. et al. Cytometry. 2002 Dec 1; 49(4):150-8) или диэлектрофореза (как описано, например, в: Gascoyne, P. et al. Electrophoresis. 2009 Apr; 30(8):1388-98). Может быть осуществлено отделение фракции микровезикул (экзосомы, «виртосомы» и другие), потенциально содержащих нуклеиновые кислоты, из биологических жидкостей с помощью ультрацентрифугирования, ультрафильтрации или сепарации с помощью антител (как описано, например, в: Taylor, D.D., et al. Methods Mol Biol. 2011; 728:235-46; Rood, I.M., et al. Kidney Int. 2010 Oct; 78(8):810-6; Clayton, Α., et al. J Immunol Methods. 2001 Jan 1; 247(1-2):163-74; Gahan, P.В., Stroun, M. Cell Biochem Funct. 2010 Oct; 28(7):529-38). В некоторых применениях с помощью инкубации клеток в буфере, содержащем трипсин и соли ЭДТА, могут быть выделены нуклеиновые кислоты, связанные с поверхностью клеток крови или других биологических жидкостей (как описано, например, Laktionov, P.P., et al. Ann Ν Υ Acad Sci. 2004 Jun; 1022:221-7; Mal'shakova, V.S., et al. Ann Ν Y Acad Sci. 2008 Aug; 1137:47-50). В некоторых применениях может быть осуществлено обогащение препаратов ткани целевыми клетками с помощью лазерной микродиссекции (как описано, например, в: Cha, S., et al. Mol Cell Proteomics. 2010 Nov; 9(11):2529-44; Weier, J.F., et al. Cytogenet Genome Res. 2006; 114(3-4):302-11).
[076] В некоторых применениях обогащение образца опухолевой или фетальной ДНК может быть осуществлено после выделения ДНК из биологических жидкостей. Например, для повышения концентрации фетальной ДНК в образцах ДНК, выделенных из плазмы крови беременных женщин, образец может быть обработан формальдегидом, как предложено Dhallan и соавторами (Dhallan, P., et al. JAMA. 2004. Vol. 291. №9. P. 1114-1119). Также для повышения концентрации фетальной или опухолевой ДНК в образце может быть использовано то, что такая ДНК представлена практически исключительно низкомолекулярными фракциями (Li, Y., et al. Methods Mol Biol. 2008; 444:239-51; Hung, Ε.С, et al. Clin Chem. 2009 Apr; 55(4):715-22; Hahn, T., et al. Clin Chem. 2009 Dec; 55(12):2144-52) и эта фракция может быть отделена от остальных фракций на агарозном электрофорезе или на хроматографической колонке.
[077] В преимущественных применениях образец ДНК для анализа содержит целевую нуклеиновую кислоту (то есть мутантную ДНК, референсную ДНК или их смесь). В некоторых применениях образец нуклеиновых кислот содержит также нецелевые нуклеиновые кислоты, не сходные с целевыми нуклеиновыми кислотами. Как здесь используется, термин «нецелевые» относится к нуклеиновой кислоте, которая не содержит исследуемого нуклеотида и не комплементарна целевой нуклеиновой кислоте.
[078] В преимущественных применениях концентрация нуклеиновых кислот в образце составляет не менее 10 пкг, обычно не менее 500 пкг. Доля целевой нуклеиновой кислоты в образце может колебаться от 0,0000001% до 100% и чаще всего не превышает 50%, преимущественно не превышает 10%, как правило, не превышает 1-2%.
[079] В некоторых применениях образец ДНК содержит избыток референсной целевой последовательности по сравнению с мутантной. Референсная (немутантная) ДНК находится в более чем в 100-кратном избытке, например, более чем в 300-кратном избытке, обычно более чем в 500-кратном избытке, преимущественно более чем в 1000-кратном избытке, например в 10000-кратном избытке. В некоторых применениях референсная ДНК находится более чем в 1 000-кратном избытке, но не более чем в 100000-кратном избытке.
[080] В некоторых воплощениях образец ДНК подвергают фрагментации с целью получения фрагментов ДНК со средней длиной от 50 до 1000 пар нуклеотидов, чаще от 100 до 500 пар нуклеотидов, например 150-300 пар нуклеотидов.
[081] В некоторых воплощениях к образцу ДНК, выделенному из биологического материала, добавляют экзогенную двухцепочечную ДНК из другого организма или искусственно созданную. Амплификация фрагментов этой ДНК в дальнейшем может быть использована как контроль качества амплификации и секвенирования. Например, к образцу ДНК, выделенной из человека, может быть добавлена ДНК беспозвоночных или искусственно полученная последовательность. Примеры таких последовательностей показаны в SEQ ID NOs: 64, 65.
[082] Молекулярное баркодирование пула целевых молекул ДНК
[083] Образец ДНК используется в качестве матрицы для баркодирования пула целевых молекул. Как здесь используется, термин «баркодирование» означает введение в последовательность молекул ДНК последовательности баркода. Как здесь используется, термин «баркод» означает последовательность нуклеотидов, которая позволяет отличить продукт амплификации одной молекулы от продукта амплификации других идентичных или по существу сходных молекул. Как здесь используется, термин «баркод» не относится к индексным последовательностям, которые служат для различения молекул из двух различных образцов ДНК.
[084] Для нужд настоящего изобретения процесс баркодирования заключается во введении случайной последовательности нуклеотидов (random primer) в целевую молекулу ДНК. Длина случайной последовательности может варьировать и обычно составляет 9-20 нуклеотидов, чаще 10-15 нуклеотидов, например 12, 13 или 14 нуклеотидов. Как здесь используется, термин «баркодирующий праймер» означает смесь олигонуклеотидов, содержащих случайную последовательность - баркод. Структура баркодирующего праймера схематически показана на рис.1. На 3'-конце праймер несет геноспецифическую последовательность, комплементарную целевой последовательности ДНК, за которой следует случайная последовательность (баркод). В преимущественных воплощениях за баркодом (на 5'-конце) находится адаптерная последовательность.
[085] Примеры баркодирующих праймеров для целевых последовательностей SEQ ID NOS: 1-62 показаны в SEQ ID Nos: 66-127. Указанные праймеры содержат геноспецифические последовательности, прилегающие к рефернсным последовательностям, показанным в SEQ ID NOS: 1-62. Соответствие праймеров целевым последовательностям показано в Таблице 1.
[086] Для любого специалиста в данной области очевидно, что длина баркода может быть уменьшена или увеличена на 1-4 нуклеотида без существенного влияния на эффективность баркодирования. Также очевидно, что может быть заменена адаптерная последовательность. Варианты адаптерных последовательностей хорошо известны и показаны, например, в базе данных UniVec, предоставляемой National Center for Biotechnology Information, в инструкциях к приборам для секвенирования нового поколения и наборам реагентов для секвенирования нового поколения.
[087] Введение баркода в пул целевых молекул осуществляется в ходе полимеразной цепной реакции (ПЦР) в присутствии баркодирующего праймера в конечной концентрации 0,03-0,3 мкМ, чаще 0,05-0,2 мкМ. Предпочтительным является использование ДНК-полимеразы или смеси ДНК полимераз, отличающейся повышенной точностью синтеза и специфичности затравления синтеза - например, полимеразы, которая содержит дополнительные аминокислотные замены, усиливающие точность синтеза (например, описанные в: Lovmar, L., Syvaenen, А.С. Methods Mol Med. 2005; 114:79-92), такой как, например, Tersus (ЗАО «Евроген», Россия), Phusion (New England Biolabs, США), Kapa Hi-Fi (Kapa Biosystems, ЮАР), TruSeq DNA Polymerase (Illumina, США), KOD DNA Polymerase (TaKaRa Bio, Япония), или подобной.
[088] Баркодирование может осуществляться в ходе стандартной ПЦР, как описано Kinde и соавторами (Proc Nat Acad Sci USΑ. 2011; 23:9530-35). В этом случае реакционная смесь для ПЦР также содержит встречный геноспецифический праймер для каждого баркодирующего праймера. Термины «прямой праймер» и «встречный праймер» используются здесь для обозначения взаиморасположения двух праймеров на молекуле ДНК, но без учета их положения относительно кодирующей цепи ДНК. Примеры встречных геноспецифических праймеров для амплификации референсных последовательностей SEQ ID NOS: 1-62 показаны в SEQ ID NOs: 131-192. Геноспецифические последовательности указанных праймеров отдалены от референсных последовательностей SEQ ID NOs: 1-62 на 10-25 нуклеотидов. Соответствие референсных последовательностей и геноспецифических праймеров приведено в таблице 1.
[089] В качестве альтернативы Заявители разработали оригинальную процедуру баркодирования методом линейной ПЦР в присутствии только баркодирующих праймеров (рис. 2). В ходе такой ПЦР матрицей служит только исходная ДНК, что позволяет увеличить число циклов амплификации до 15, чаще до 5-7. Таким образом, в ходе баркодирования каждой молекуле исходного образца соответствует по крайней мере 5 копий, каждая из которых несет свой баркод.
[090] Это делает возможным дальнейшее выявление ошибок ДНК-полимеразы, происходящих на данном этапе: при достаточном количестве циклов линейной амплификации мутация, присутствующая хотя бы в одной молекуле исходного образца, должна будет присутствовать в нескольких продуктах данной реакции, содержащих разные баркоды (ожидаемое число этих продуктов рассчитывается с учетом эффективности конкретных реакций и числа циклов амплификации). Если число этих продуктов более 3, то вероятность появления столь же частой артефактной мутации крайне невелика, и ошибки ДНК-полимеразы на начальном этапе можно выявлять с помощью «фильтрации по количеству».
[091] Каждый цикл амплификации включает денатурацию при температуре 94-96°C, отжиг праймеров при температуре 50-65°C, элонгацию температуре 72°C. Выбор программы амплификации хорошо известен специалистам в данной области. Температуру отжига выбирают в соответствии с расчетами в программах M-Fold (Zuker, M. Nucleic Acids Res. 2003 Jul 1; 31(13):3406-15), MeltCalc (Schuetz, E., von Ahsen, N. Biotechniques. 1999 Dec; 27(6):1218-22, 1224) или аналогичных и проверяют экспериментально на контрольных образцах ДНК.
[092] В зависимости от числа целевых последовательностей, которые необходимо проанализировать в образце, баркодирование может быть осуществлено в одной пробирке в ходе мультиплекной линейной ПЦР или в нескольких пробирках в ходе мультиплексной линейной ПЦР, где каждая пробирка содержит свой набор баркодирующих праймеров.
[093] В некоторых воплощениях мультиплексной линейной ПЦР различные праймеры для баркодирования присутствуют в реакционной смеси в эквимолярных количествах. В некоторых воплощениях, концентрации некоторых праймеров увеличены или уменьшены (например, в 1,5-3 раза) относительно других таким образом, чтобы обеспечивать сохранение близкое к исходному соотношение концентраций молекул-мишеней по окончании пробоподготовки. Для каждого набора праймеров необходим экспериментальный подбор оптимального отношения концентраций.
[094] В некоторых воплощениях каждая смесь баркодирующих праймеров так же содержит праймеры для баркодирования «внутреннего контроля» - последовательности гена, немутантной для данного образца. Например, в случае использования опухолевой ДНК человека в качестве матрицы внутренним контролем может служить фрагмент гена IDH1, последовательность которого показана в SEQ ID No: 63. Последовательность баркодирующего праймера для этого фрагмента показана в SEQ ID No: 128.
[095] В некоторых воплощениях каждая смесь баркодирующих праймеров также содержит праймеры для баркодирования экзогенно добавленной ДНК. Примеры праймеров для баркодирования фрагментов SEQ ID Nos: 64, 65 показаны в SEQ ID Nos: 129, 130.
[096] Очистка продукта реакции баркодирования от остатков баркодирующего праймера
[097] Очистка продукта баркодирования от остатка праймера призвана исключить участие праймера, содержащего баркод, в дальнейшей амплификации. Для очистки может быть использован любой известный метод очистки ДНК от коротких олигонуклеотидов, включая гель-фильтрационные и энзиматические методы или их комбинацию. В частности, могут быть использованы коммерчески доступные наборы реагентов для очистки ДНК из реакционных смесей, такие как набор Cleanup Standard (Евроген, Россия), DNA Clean & Concentrator-5 (Zymo Research, США) и подобные.
[098] В некоторых применениях для уничтожения остатков баркодирующего праймера может быть произведена обработка реакционной смеси экзонуклеазой I (Exo-I), которая разрушает одноцепочечные ДНК в 3'-5' направлении, высвобождая дезоксирибонуклеозид-5-монофосфаты. Exo-I активна в реакционном буфере для ПЦР и может быть добавлена в реакционную смесь непосредственно после окончания процедуры баркодирования. Как правило, используется Ехо-1 в конечной концентрации от 1 до 3 ед. в мкл, например, 2,4 единицы в мкл.
[099] Обработка Exo-I осуществляется при 37°C. Время обработки может варьировать от 5 мин до 2 часов, чаще в пределах от 7 мин до 1 часа, например 10-15 мин, что достаточно для гидролиза остатков баркодирующего праймера, но не затрагивает целевые последовательности баркодированных молекул ДНК (рис. 2).
[0100] Инактивация Exo-I осуществляется с помощью прогревания при 68-70°C в течение 10-30 мин.
[0101] В некоторых воплощениях продукт реакции после обработки Exo-I может быть переосажден. В некоторых воплощениях продукт реакции после обработки Exo-I может быть очищен с помощью коммерчески доступных наборов реагентов для очистки ДНК из реакционных смесей или любым другим известным методом для очистки ДНК.
[0102] Амплификация пула целевых молекул в мультиплексной ПЦР
[0103] Амплификация пула баркодированных целевых молекул осуществляется в стандартной ПЦР с использованием универсального праймера, содержащего на 3' конце последовательность, по существу сходную с адаптерной последовательностью баркодирующего праймера, и геноспецифического праймера, встречного по отношению к геноспецифической последовательности баркодирующего праймера (рис. 2). Например для амплификации молекул баркодированных с помощью праймеров, выбранных из SEQ ID NOs: 66-130 может быть использован универсальный праймер TS-Uni-short, последовательность которого показана в SEQ ID No: 196 и встречные геноспецифические праймеры, выбранные из SEQ ID NOs: 131-195.
[0104] Число пробирок ПЦР равно числу пробирок использованному для баркодирования. Каждая содержит свой набор геноспецифических праймеров (далее REV-праймеров), встречных использованным на предыдущей стадии баркодирующим праймерам (таблица 1).
[0105] Объем реакционной смеси для ПЦР составляет, как правило, 10-100 мкл, чаще 15-50 мкл, например, 20 мкл или 25 мкл. В качестве матрицы в ПЦР используется очищенный продукт баркодирования.
[0106] Конечная концентрация универсального праймера в реакционной смеси обычно составляет 0,2-0,5 мкМ, например 0,3 мкМ. Конечная концентрация каждого геноспецифического праймера составляет 0,01-0,3 мкМ, чаще 0,03-0,2 мкМ.
[0107] В некоторых воплощениях различные геноспецифические праймеры для мультиплексной ПЦР присутствуют в реакционной смеси в эквимолярных количествах. В некоторых воплощениях, концентрации некоторых праймеров увеличены или уменьшены (например, в 0,5-3 раза) относительно других таким образом, чтобы обеспечивать сохранение близкое к исходному соотношение концентраций молекул-мишеней в амплифицированной образце. Для каждого набора геноспецифических праймеров необходим экспериментальный подбор оптимального отношения концентраций.
[0108] В некоторых воплощениях каждая смесь для ПЦР так же содержит геноспецифические праймеры для амплификации «внутреннего контроля» и/или экзогенно добавленной ДНК. Например для референсных последовательностей показанных в SEQ ID Nos: 63-65 могут быть использованы геноспецифические REV-праймеры, показанные в SEQ ID Nos: 193-195.
[0109] Амплификацию выполняют по следующей программе: 25-90 циклов ПЦР в режиме 1-30 сек - денатурация при температуре 94-96°C, 40 сек - 1 минута - отжиг праймеров при температуре 50-65°C, элонгация в течение 1 минуты при температуре 72°C. Температуру отжига выбирают в соответствии с расчетами в программе M-Fold (Zuker, M. Nucleic Acids Res. 2003 Jul 1; 31(13):3406-15), MeltCalc (Schuetz, E., von Ahsen, N. Biotechniques. 1999 Dec; 27(6):1218-22, 1224) или аналогичных и проверяют экспериментально на контрольных образцах ДНК.
[0110] Введение сиквенсовых адаптеров в продукт мультиплексной ПЦР и секвенирование указанного продукта
[0111] Секвенирование продукта амплификации может быть произведено любым известным методом секвенирования, включая секвенирование по Сэнгеру и высокопроизводительное секвенирование нового поколения.
[0112] Для метода настоящего изобретения сиквенсовые адаптеры для NGS могут быть введены в продукт амплификации разными способами известными специалистам в данной области, включая лигирование или различные варианты ПЦР с праймерами, содержащими последовательности сиквенсовых адаптеров на 3'-конце. Различные способы введения сиквенсовых адаптеров в ходе одной или нескольких ПЦР проиллюстрированы на рис. 3.
[0113] В некоторых воплощениях введение сиквенсовых адаптеров осуществляется с помощью ПЦР с праймерами, содержащими последовательности сиквенсовых адаптеров на «свисающих» 5'-концах. 3'-концевые последовательности праймеров содержат сайты отжига на целевые молекулы, например на геноспецифические последовательности внутри амплифицированных регионов целевых молекул (заглубленный ПЦР) и на адаптерную последовательность баркодирующего праймера.
[0114] Как здесь используется, термин «свисающий конец» или «свисающий хвост» по отношению к праймеру обозначает 5'-концевую последовательность нуклеотидов в праймере, некомплементарную целевым молекулам.
[0115] В некоторых воплощениях введение сиквенсовых адаптеров осуществляется с помощью двух последовательных ПЦР, где в ходе первого ПЦР используются праймеры, содержащие 3'-концевые сайты отжига на целевые молекулы и свисающие концы, содержащие часть последовательности сиквенсового адаптера или любую иную последовательность. Во второй ПЦР используются праймеры, содержащие 3'-концевой сайт отжига на свисающие концы праймеров предыдущей ПЦР и последовательности сиквенсовых адаптеров на 5'-конце.
[0116] В некоторых воплощениях последовательности сиквенсовых адаптеров введены в виде свисающих концов в последовательности праймеров, использованных на стадии мультиплексной ПЦР. В этом случае введение сиквенсовых последовательностей осуществляется сразу на стадии мультиплексной ПЦР.
[0117] В некоторых воплощениях для введения сиквенсовых адаптеров используется «ПЦР с выходом наружу» (Step-Out PCR) (Matz et al. Nucleic Acids Res. 1999; 27(6):1558-1560). Для амплификации используются две смеси праймеров, каждая из которых состоит из (1) «внутреннего» праймера, несущего на 3' конце сайт отжига на целевые последовательности (3'-концевая специфическая последовательность) и свисающий хвост на 5'-конце и (2) «внешнего» праймера, содержащего на 3'-конце последовательность, идентичную последовательности свисающего хвоста «внутреннего» праймера. Концентрации праймеров в смесях подбирают таким образом, чтобы «внутренний» праймер расходовался на первых циклах ПЦР, в ходе которых создаются молекулы, имеющие сайты отжига «внешнего» праймера. На дальнейших циклах ПЦР амплификация пула целевых молекул продолжается с «внешнего» праймера. Например, конечная концентрация каждого внутреннего праймера обычно составляет 0,05-0,3 мкМ, например 0,1 или 0,2 мкМ, а концентрация внешнего праймера превышает ее в 10-15 раз.
[0118] В состав праймеров может быть так же введена индексная последовательность. При амплификации различных образцов используют праймеры, несущие различные индексные последовательности, что впоследствии позволяет отличать данные секвенирования молекул из разных образцов. Длина индексной последовательности обычно составляет 3-10 случайных нуклеотидов, например, 3, 4, 5, 6, 7, 8, 9 или 10 нуклеотидов. Примеры подобных индексных последовательностей (index sequences) доступны на сайтах производителей наборов реагентов для высокопроизводительного секвенирования.
Идентификация мутаций в целевых ДНК по данным секвенирования
[0119] Суммарный уровень ошибок, генерируемых в ходе многоэтапного пробоподготовки и секвенирования составляет не менее 1% (Wei, Ζ., et al. Nucleic Acids Res. 2011 Oct; 39(19):e132; Klein, J.D., et al. PLoS One. 2011; 6(8):e23455). Таким образом, для достоверного выявления редких мутаций (например, онкомутаций) по анализу данных секвенирования требуется отсев ложнопозитивных сигналов - замен нуклеотидов, внесенных в последовательность в результате некорректной работы ДНК-полимеразы на стадиях ПЦР и неправильных прочтений в ходе секвенирования.
[0120] В преимущественных воплощениях отсев ложнопозитивных сигналов включает анализ последовательностей баркодов. Наличие у молекул одинакового баркода означает, что они являются потомками единственной баркодированной молекулы ДНК-матрицы. Потомки одной молекулы должны иметь одинаковый профиль мутаций. Наличие различных вариантов нуклеотидной последовательности среди потомков одной молекулы указывает на то, что или все эти варианты, или все эти варианты, кроме одного, представляют собой недостоверный (чаще - ложнопозитивный) сигнал.
[0121] В некоторых воплощениях отсев ложнопозитивных сигналов также включает «фильтрацию по количеству», что позволяет исключить из рассмотрения потомков баркодированной молекулы, содержащих замену, появившуюся в результате некорректной работы ДНК-полимеразы на стадии баркодирования.
[0122] В некоторых воплощениях отсев ложнопозитивных сигналов также включает отсев ошибок ПЦР с помощью стратегии MIGEC, описанной в работе Shugay и соавт. (Shugay et al. Nat Methods. 2014; 11(6):653-5).
Праймеры для анализа целевых регионов в ключевых онкогенах и генах-суπрессорах человека
[0123] Также обеспечиваются наборы праймеров для реализации метода настоящего изобретения при анализе мутаций в последовательностях генов, показанных в SEQ ID No: 1 - SEQ ID No: 62. По литературным данным, анализ мутаций в указанных регионах является востребованным для выявления и мониторинга ряда онкологических заболеваний, а также имеет высокую прогностическую и предиктивную значимость при выборе стратегии и тактики ведения пациентов с подобными заболеваниями.
[0124] Праймеры для баркодирования и амплификации разделены на группы (таблица 2), каждая из которых может быть использована для одновременного баркодирования и амплификации целевых фрагментов в одной пробирке. Для любого специалиста в данной области очевидно, что некоторые праймеры могут быть исключены из группы, если нет необходимости анализировать какие-то из перечисленных фрагментов. Подобное уменьшение числа праймеров в смеси не влияет на эффективность баркодирования и амплификации оставшихся фрагментов.
[0125] В преимущественных воплощениях, праймеры для мультиплексной ПЦР1 используются вместе со встречным праймером, последовательность которого показана в SEQ ID No: 196.
[0126] Также обеспечиваются праймеры для приготовления смесей для введения сиквенсовых адаптеров с помощью ПЦР с выходом наружу. Смесь 1 включает внутренний праймер, имеющий сайт отжига на адаптерную последовательность баркодирующего праймера, общую для всех баркодирующих праймеров, и «свисающий хвост», и внешний праймер, содержащий последовательность сиквенсового адаптора и последовательность, идентичную «свисающему хвосту» внутреннего праймера. Примеры таких праймеров для секвенирования по технологии Illumina показаны в SEQ ID Nos: 197, 198.
[0127] Смесь 2 включает набор внутренних праймеров, содержащих 5'-концевой «свисающий хвост» и 3'-концевую геноспецифическую последовательность, заглубленную относительно геноспецифических праймеров для мультиплексной ПЦР и прилегающую к референсной последовательности SEQ ID NOs: 1-62 (BRIDGE-праймеры). Смесь 2 также включает внешний праймер, содержащий последовательность «свисающего хвоста» внутреннего праймера на 3'-конце и последовательность сиквенсового адаптора на 5'-конце. После последовательности сиквенсового адаптера (от 5' к 3') указанный внешний праймер может так же содержать индексную последовательность. Примеры внутренних праймеров приведены в SEQ ID NOs: 199-260, примеры внешнего праймера в SEQ ID NO: 264, 265.
[0128] Для любого специалиста в данной области очевидно, что индексная последовательность может быть заменена в таком праймере на любую другую нуклеотидную последовательность такой же или близкой длины. Также индексная последовательность может быть перенесена во внешний праймер смеси 1. Также во внешних праймерах могут быть заменены последовательности сиквенсовых адаптеров в соответствии с требованиями производителей секвенаторов для высокопроизводительного секвенирования. В приведенных примерах показаны сиквенсовые адаптеры для секвенирования на секвенаторах Illumina.
[0129] Внешние и внутренние праймеры также могут быть использованы для введение сиквенсовых адаптеров в ходе двух раундов ПЦР как показано на рис. 3. В этом случае внутренние праймеры используются для первого раунда ПЦР (в концентрации 0,05-0,3 мкМ), а внешние - для второго раунда ПЦР.
[0130] В некоторых воплощениях праймеры в каждой группе смешаны в эквимолярных количествах. Конечная концентрация каждого праймера составляет 0,7-0,15 мкМ, например 0,1 мкМ. В некоторых воплощениях концентрация некоторых праймеров увеличена в 1,5-5 раз по сравнению с остальными. В некоторых воплощениях концентрация некоторых праймеров уменьшена в 1,5-5 раз по сравнению с остальными. Концентрации внешних праймеров в смесях для ПЦР с выходом наружу превышают концентрации внутренних праймеров по крайней мере в 5 раз, чаще в 10 раз, иногда в 15 раз.
[0131] Например, обеспечиваются смеси праймеров для баркодирования А, Б, В и Г (Таблица 2), где по крайней мере вдвое увеличена концентрация баркодирующих праймеров для следующих референсных фрагментов: экзон 4 гена HER4, экзон 12 фрагмент 2 гена HER4, экзон 14 гена HER4, экзон 8 гена HER3, экзон 21 гена HER3, экзон 23 гена HER3, экзон 10 гена PIK3CA, экзон 4 гена KRAS, экзон 2 гена AKT1, экзон 4 гена MAP2K2, экзон 5 гена GNAQ, экзон 2 гена RAC1. Также обеспечиваются смеси REV-праймеров для мультиплексной амплификации баркодированных молекул, где по крайней мере вдвое увеличена концентрация геноспецифических праймеров для амплификации следующих референсных фрагментов: экзон 4 гена HER4, экзон 12 фрагмент 2 гена HER4, экзон 14 гена HER4, экзон 8 гена HER3, экзон 21 гена HER3, экзон 20 фрагмент 1 гена EGFR, экзон 8 фрагмент 2 гена HER4, экзон 5 фрагмент 2 гена ТР53, экзон 23 гена HER3, экзон 10 гена PIK3CA, экзон 4 гена KRAS и по крайней мере в 1,5 раза уменьшена концентрация для амплификации следующих референсных фрагментов: экзон 7 гена ТР53, экзон 18 гена EGFR, экзон 21 гена EGFR, экзон 20 фрагмент 1 гена HER2, экзон 21 фрагмент 1 гена HER2, экзон 9 гена HER3, экзон 3 гена HER3, экзон 11 гена KIT, экзон 15 гена BRAF, экзон 2 гена NRAS, экзон 20 гена PIK3CA, экзон 3 KRAS, экзон 2 и экзон 3 гена HRAS, экзон 3 гена NRAS, экзон 12 гена EGFR, экзон 7 гена RAF1, экзон 2 гена MAP2K1, экзон 6 гена MAP2K2, экзон 5 гена GNAQ, экзон 7 гена MAPK1.
[0132] В преимущественных воплощениях смеси праймеров также содержат праймеры для баркодирования и амплификации «внутреннего контроля», например, экзона 4 гена IDH1 (SEQ ID NOs: 63). Анализ амплификации и данных секвенирования этого гена может служить контролем качества анализа. Примеры таких праймеров показаны в SEQ ID NOs: 128, 193, 261.
[0133] В преимущественных воплощениях смеси праймеров также содержат праймеры для баркодирования и амплификации экзогенно добавляемых последовательностей. Примеры таких праймеров показаны в SEQ ID NOs: 129, 130, 194, 195, 262, 263.
[0134] В некоторых воплощениях смеси праймеров содержат дополнительные праймеры для амплификации иных дополнительных последовательностей, подобранные таким образом, что они не препятствуют амплификации вышеуказанных референсных последовательностей.
[0135] Смеси праймеров могут быть использованы для реализации метода настоящего изобретения, как описано в разделе «Примеры» ниже.
[0136] Смеси баркодирующих праймеров праймеров могут быть использованы для стандартной мультиплексной ПЦР фрагментов ДНК, содержащих референсные регионы SEQ ID NOs: 1-62 вместе с соответствующими им REV-праймерами. Продукты такой ПЦР могут быть использованы в качестве матрицы при для заглубленной мультиплексной ПЦР с BRIDGE-праймерами и TS-Uni-short праймером или баркодирующими праймерами.
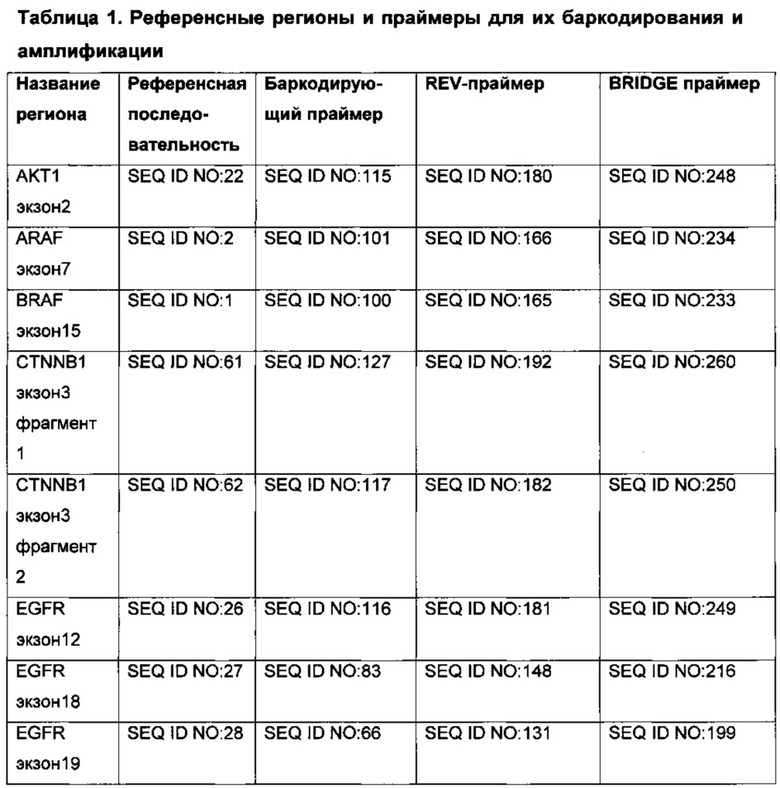
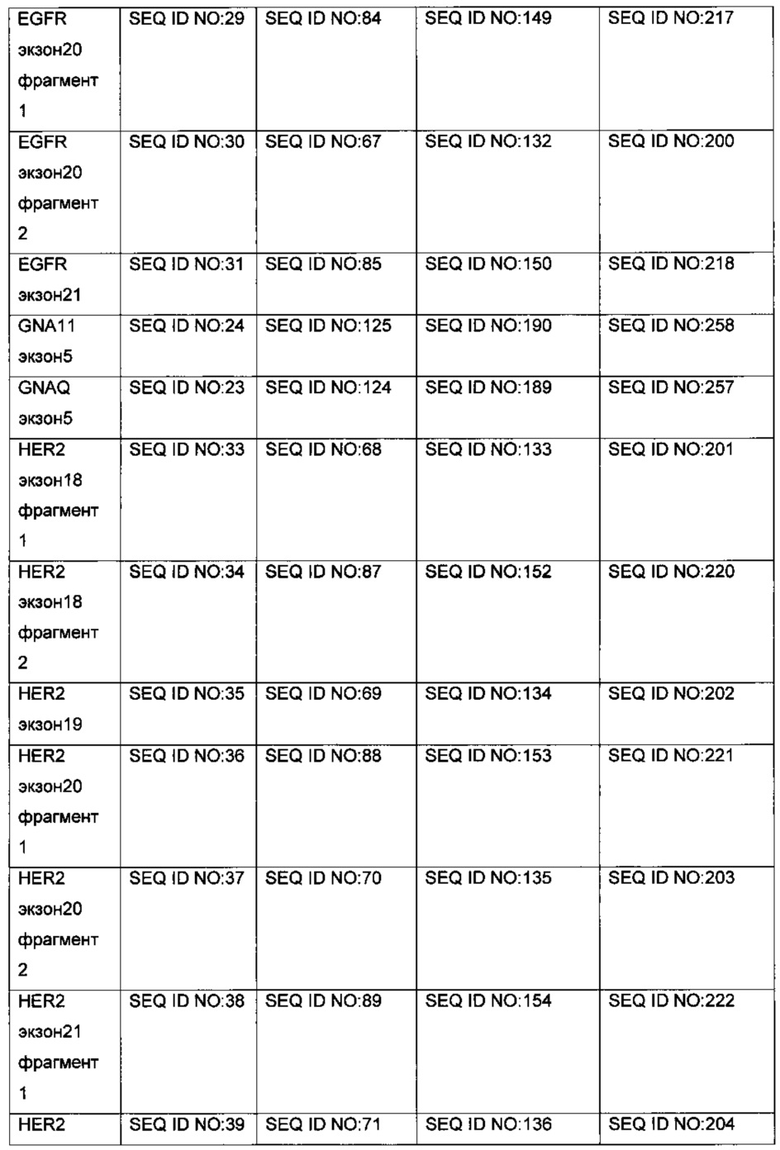
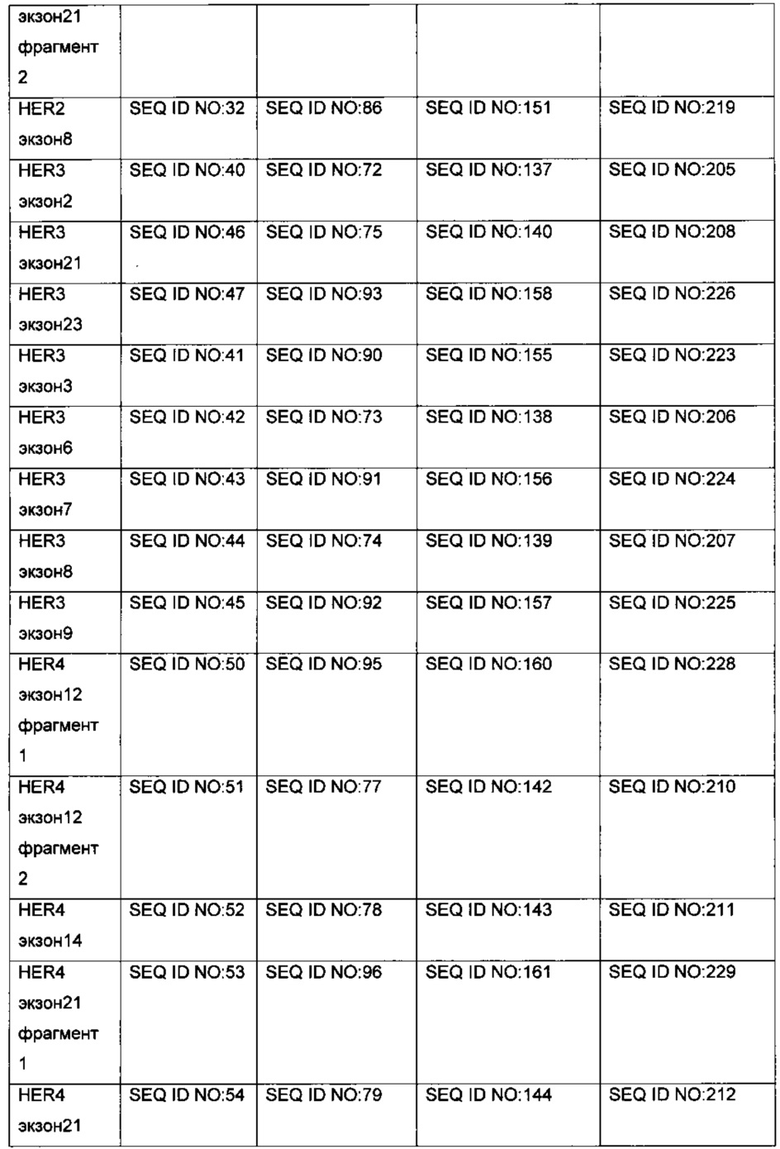
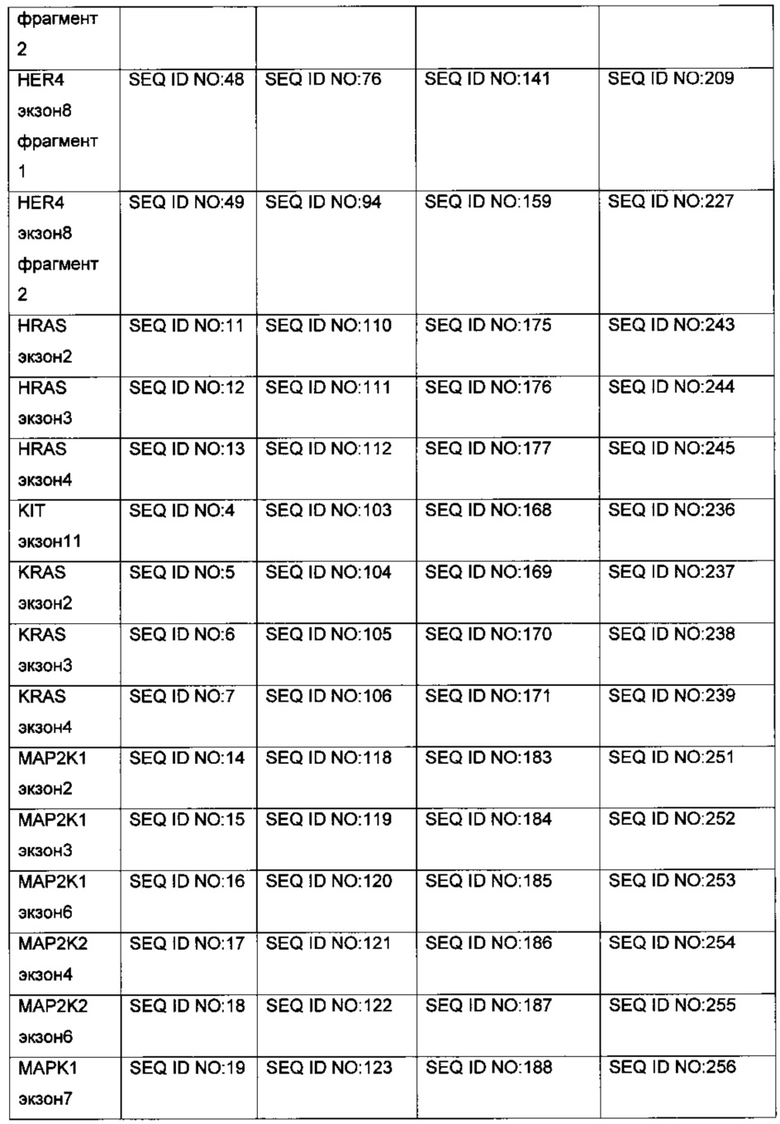



Наборы реагентов
[0137] Также обеспечиваются наборы реагентов для реализации метода настоящего изобретения. В преимущественных воплощениях указанные наборы включают смеси праймеров для баркодирования и мультиплексной амплификации. В некоторых воплощениях наборы также включают праймеры для введения сиквенсовых адаптеров методом ПЦР с выходом наружу. В некоторых воплощениях наборы также включают реагенты для ПЦР. В некоторых воплощениях наборы также включают реагенты для очистки ДНК из реакционных смесей и экзонуклеазу I.
[0138] В преимущественных воплощениях наборы также включают инструкцию по использованию на бумажном или электронном носителе, содержащую протокол пробоподготовки и анализа данных секвенирования.
Способы применения
[0139] Возможные применения изобретения включают все области биологии и медицины, в которых может быть актуально выявление молекул ДНК или РНК с мутациями и полиморфизмами, особенно присутствующих в образцах, содержащих избыток немутированных молекул ДНК или РНК.
[0140] Подобные применения включают в себя, в частности, следующие области, но не исчерпываются ими:
[0141] Детекцию соматических мутаций, прежде всего - неинвазивный скрининг и мониторинг в онкологии, включая раннюю диагностику злокачественных опухолей и их рецидивов, оценку радикальности проведенного лечения, диагностику микрометастазов и «спящих метастазов» и оценку лекарственной чувствительности опухоли без использования биопсии;
[0142] Неинвазивную пренатальную диагностику;
[0143] ДНК-диагностику мутаций de novo и болезней, вызванных химеризмом;
[0144] Мониторинг реципиентов пересаженных органов для своевременного выявления отторжения трансплантата;
[0145] Выявление приобретенной лекарственной устойчивости инфекционных агентов и злокачественных опухолей, в том числе непосредственно в процессе терапии;
[0146] ДНК-диагностику митохондриальных болезней (в большинстве случаев подобные болезни вызываются изменениями в митохондриальном геноме, которые присутствуют далеко не во всех митохондриях - т.н. гетероплазмия);
[0147] Анализ уровня спонтанного мутагенеза для самого широкого круга задач в биологии и медицине;
[0148] Анализ генетического полиморфизма на пулированных образцах (в частности, в популяционной, эволюционной и экологической генетике).
[0149] Следующие примеры предлагаются в качестве иллюстративных, но не ограничивающих.
ПРИМЕРЫ
[0150] Пример 1
[0151] Сравнение эффективности разных способов баркодирования целевых молекул
[0152] Было произведено баркодирование молекул с помощью стандартной ПЦР и линейной ПЦР. Для подготовки реакционных смесей использовали наборы реагентов Tersus PCR kit (Евроген, Россия), согласно инструкции производителя. Было приготовлено две реакционные смеси (объем каждой - 25 мкл): смесь для баркодирования в ПЦР с двумя праймерами содержала 2 мкМ праймера SEQ ID No: 100 и 2 мкМ праймера SEQ ID No: 165, смесь для баркодирования в ПЦР с одним праймером содержала 2 мкМ праймера SEQ ID No: 100. В каждую реакционную смесь было добавлено 10 нг геномной ДНК человека (Clontech, США). Смеси подвергли амплификации в следующем режиме: предварительная денатурация при 95°C в течение 5 мин; 2 цикла: 95°C - 15 сек; 58°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. По окончании реакции обе смеси инкубировали с 3 мкл экзонуклеазы I (New England Biolabs) 1 час при 37°C, прогревали 15 мин при 70°C для инактивации фермента и амплифицировали в присутствии праймеров TS-Uni-short и SEQ ID No: 165 (2 мкМ на реакцию) в ПЦР в следующем режиме: предварительная денатурация при 95°C в течение 5 мин; 28 циклов: 95°C - 15 сек; 62°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. Продукты ПЦР разводили в 40 раз и амплифицировали (1 мкл разведенного продукта на 50 мкл реакционной смеси для ПЦР) в присутствии праймеров TS-Uni-short и SEQ ID NO: 233 с использованием набора реагентов Tersus PCR kit (Евроген, Россия) согласно прилагаемой инструкции. Использовали следующий режим амплификации: предварительная денатурация при 95°C в течение 5 мин; 12 циклов: 95°C - 15 сек; 62°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. Продукты ПЦР разрешали на гель-электрофорезе в агарозе в ТАЕ-буфере с окрашиванием бромистым этидием (Рис. 4). Видно, что в случае баркодирования с помощью линейной ПЦР, целевая амплификация идет более эффективно.
[0153] Пример 2
[0154] Анализ мутаций в гене BRAF
[0155] Для создания экспериментальных образцов был использован коммерческий образец геномной ДНК человека из здоровых доноров мужского пола (Human Male Control Genomic DNA, Promega, кат №G1471) - далее «образец BRAF-wt». Для получения мутантной по BRAF ДНК - геномная ДНК клеточной линии SK-Mel-1, содержащей гетерозиготную мутацию с. 1799 Т→А (p. Val600Glu) в гене BRAF.
[0156] Для выделения ДНК клеточный осадок линии SK-Mel-1 инкубировали с протеиназой K (Amresco, кат №О706) в стандартном буфере (100 мМ Трис-HCl; 50 мМ K3EDTA; 0,5% SDS; pH=8,0). Аликвоты по 1 миллиону клеток инкубировали в 1,2 мл буфера в течение 3 суток при 56°C. ДНК выделяли из лизата с использованием набора реагентов DNA Clean & Concentrator-5 (Zymo Research, кат № D4013) в соответствии с инструкцией производителя, на одно выделение использовали 300 мкл лизата.
[0157] Образцы ДНК подвергали ультразвуковой фрагментации, результаты фрагментации контролировали с помощью гель-электрофореза в агарозе с окрашиванием бромистым этидием. Фрагментацию вели до появления доминирующих фрагментов длиной от 100 до 150 п.о. Концентрацию ДНК измеряли с использованием набора реагентов XY-Detect (Синтол, Россия, кат. № HG-402) и панели ДНК-калибраторов (в концентрации 50, 10 и 1 нг/мкл; Лаборатория Изоген, Россия, кат. № R0010) в соответствии с инструкциями производителей.
[0158] Для получения амплифицированных экспериментальных образцов BRAF-wt ДНК смешивали с ДНК клеточной линии SK-Mel-1 в соотношении 100 к 1 (образец BRAF-mut100) и 1000 к 1 (образец BRAF-mut1000). В качестве контроля использовали образец ДНК BRAF-wt. Мутационный статус гена BRAF в образцах ДНК BRAF-mut100, BRAF-mut1000 и BRAF-wt был подтвержден стандартной и мутационно-специфической ПЦР в режиме реального времени с использованием набора реагентов Insider B-Raf (Евроген, Россия) с последующим секвенированием по Сэнгеру. Было показано что мутантная ДНК находится в 30 и 300-кратном избытке в образцах BRAF-mut100, BRAF-mut1000, соответственно.
[0159] Образцы BRAF-wt ДНК, BRAF-mut100 и BRAF-mut1000 использовали в качестве матрицы для баркодирования целевого фрагмента экзона 15 гена BRAF. Баркодирование с помощью линейной ПЦР, очистку и последующую амплификацию целевого фрагмента осуществляли как описано в Примере 1. Единственное отличие - использовали 3 цикла линейной ПЦР для баркодирования. Продукт ПЦР разводили в 40 раз и 1 мкл разведенного продукта добавляли в 50 мкл реакционной смеси для ПЦР, которую осуществляли в присутствии праймеров SEQ ID NOs: 197 и 233 в режиме предварительная денатурация при 95°C в течение 5 мин; 17 циклов: 95°C - 15 сек; 62°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. Продукты ПЦР разводили в 40 раз и 1 мкл разведенного продукта добавляли в 50 мкл реакционной смеси для ПЦР, которую осуществляли в присутствии праймеров SEQ ID NOs: 198 и одного из 264, 266, 267 (праймеры с различными индексными последовательностями, свой для каждого образца) в режиме предварительная денатурация при 95°C в течение 5 мин; 17 циклов: 95°C - 15 сек; 58°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°С в течение 2 мин. Для контроля эффективности амплификации использовали гель-электрофорез в агарозе в ТАЕ-буфере (40 мМ Трис-ацетат, pH 7,8-8,0; 1 мМ ЭДТА) с окраской бромистым этидием. Результаты гель-электрофореза суммированы на Рис. 4. Продукты ПЦР разных образцов очищали с помощью набора для очистки продуктов ПЦР (Qiagen, Германия), измеряли концентрацию продукта на флуориметре Qubit 2.0 (Life Technologies, США) с помощью набора реагентов Quant-IT dsDNA BR Assay Kit (Life Technologies, США, Cat. ID Q32853) в соответствии с инструкцией производителя и по 700 нг ДНК каждого продукта объединяли и секвенировали на приборе MiSeq v. 2 (Illumina) с использованием набора реагентов MiSeq 2 X 150 Cycle Sequencing Kit v. 2 (Illumina, кат. № MS-102-2002) в соответствии с инструкцией производителя. Были получены парные (двухсторонние) прочтения длиной 150 оснований.
[0160] Результаты массированного секвенирования были автоматически рассортированы по образцам, согласно меткам образцов, и использованы для анализа мутаций в экзоне 15 гена BRAF. На первом этапе анализа данных проводили оценку качества данных по стандартному протоколу с использованием программного обеспечения fastqc с дальнейшей фильтрацией при помощи программного обеспечения flexbar. Далее, результаты секвенирования (индивидуальные прочтения) картировали на референсную последовательность 15-го экзона гена BRAF (SEQ ID NO: 1). В первом случае прочтения картировали с помощью программы Bowtie2 с использованием стандартных параметров. Полученные на данном этапе данные соответствовали стандартно получаемому результату биоинформационного анализа. Во втором случае сначала отфильтровывали прочтения, которые не перекрывали референсную последовательность, или не содержали полноценного баркода. Полученный пул данных группировали в «семейства», внутри которых все молекулы имели одинаковый баркод. Семейства, содержащие один член, отбрасывали как непригодные для статистического анализа. В результате обработки данных с учетом всех требований оказалось отброшено ~20% прочтений. После формирования «семейств» с идентичными баркодами для каждого из них с помощью программы Bowtie2 собирали консенсус с доминирующим вариантом в каждой позиции (доля доминирующего варианта - не менее 95%). Если доминирующего варианта не обнаруживалось в консенсусную последовательность вносился символ «X». Наконец, полученные консенсусные последовательности картировали на референсную последовательность 15-го экзона гена BRAF с помощью программы Bowtie2 с использованием стандартных параметров.
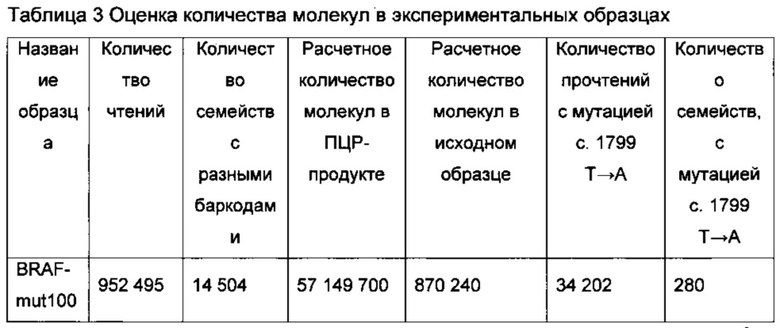
[0161] Результаты выравнивания до и после обработки трансформировали в таблицу, содержащую данные о том, сколько раз каждый вариант встретился в каждой позиции последовательности. Для расчета приблизительного числа молекул целевого фрагмента ДНК в ПЦР-продуктах и в исходных образцах использовали полученные данные о количестве прочтений и семейств с различными баркодами, соответственно, с поправкой на общий объем реакционной смеси: для образцов, использованных для секвенирования: общий объем реакционной смеси был в 60 раз больше объема, использованного для секвенирования (таблица 3).
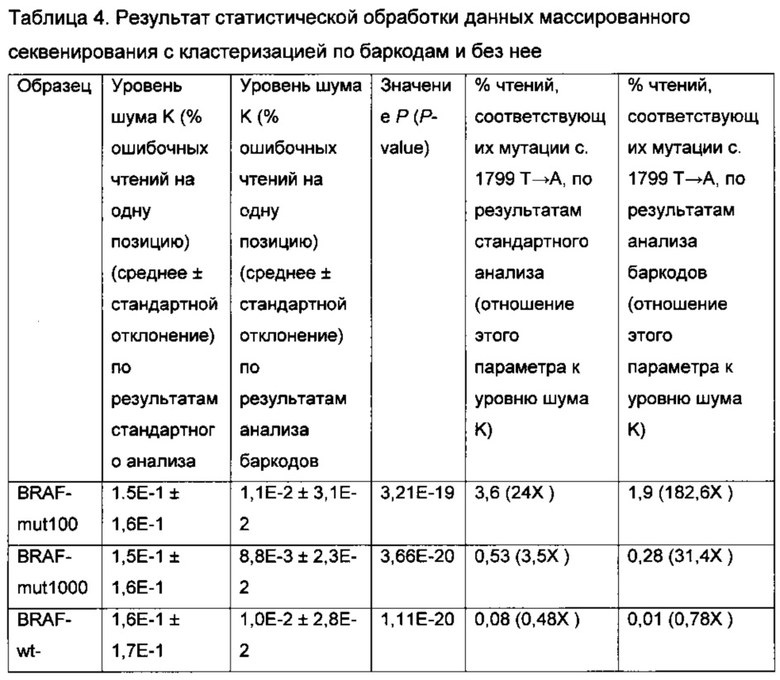
[0162] Для оценки избирательности метода производили проверку наличия в данных по образцам с мутацией «сигнала» (прочтений и семейств, соответствующих мутации с. 1799 Т→А), а в случае наличия этого сигнала производили сравнение его уровня с уровнем «шума» от ошибок ПЦР и секвенирования. Под уровнем шума К понимали отношение числа чтений с неверными вариантами к сумме числа чтений с неверными вариантами и числа чтений с верным вариантом (чтения с символом «N» в обоих случаях не учитывали ни в числителе, ни в знаменателе; чтения с символом «X» в случае выравнивания с предварительной кластеризацией по баркодам учитывали как часть шума, т.е. и в числителе и в знаменателе). Полученные ряды значений параметра K (всего 12 рядов) анализировали с помощью базовых статистических методов. Для каждого ряда вычисляли выборочное среднее и стандартное отклонение выборки. Соответствие распределения значений в каждом ряду нормальному распределению проверяли с помощью критерия Колмогорова-Смирнова. Так как по этому критерию распределение соответствовало нормальному, проводили сравнение средних значений в парах рядов для одного образца с помощью t-критерия Стьюдента (двусторонний, двухвыборочный с неравным отклонением). Статистические операции выполняли с помощью программы Microsoft Excel и надстройки AtteStat (StatSoft, Россия). Результаты статистического анализа суммированы в Таблице 4. Выравнивание с предварительной кластеризацией по баркодам позволило снизить уровень шума приблизительно в 10 раз, причем это различие отличается высоким уровнем статистической значимости (p-value). Параллельно с уменьшением уровня шума увеличивается уровень сигнала, а соответственно, растет и избирательность анализа. Если при стандартном анализе уровень сигнала превышает средний уровень шума всего в 3,5 или 4,6 раз, то после кластеризации по баркодам сигнал превышает средний уровень шума в 31 раз и более. Таким образом, хотя сигнал, соответствующий наличию мутации с. 1799 Т→А, был обнаружен в данных всех образцов, при использовании стандартного выравнивания достоверно отличить сигнал от шума при стандартном выранивании возможно только в образце BRAF-mut100. Иными словами, при стандартном выравнивании достигается уровень избирательности 1:30, но не 1:300.

[0163] Пример 3
[0164] Проверка специфичности праймеров
[0165] Олигонуклеотидные праймеры (таблица 1) были синтезированы на синтезаторе Applied Biosystems ABI 3900. Олигонуклеотиды обессоливали и очищали на ПААГ согласно стандартному протоколу. Для приготовления смесей олигонуклеотидов лиофилизированные праймеры, полученные после синтеза, растворяли стерильной деионизованной водой для молекулярно-биологических работ до конечной концентрации 10 мкМ. Олигонуклеотиды для каждой группы (Таблица 2) смешивали в эквимолярных количествах и использовали для тестовых ПЦР. В каждом случае реакционную смесь объемом 25 мкл, содержащую смесь баркодирующих праймеров (группы А, Б, В или Г) в конечной концентрации 0,2 мкМ каждого, 1Х буфер для полимеразы Tersus (Евроген, Россия), 10 нг геномной ДНК человека (Human Male Control Genomic DNA, Promega, кат № G1471), 200 мкМ каждого нуклеозидтрифосфата и полимеразу Tersus подвергали линейной ПЦР в режиме: предварительная денатурация при 95°C в течение 5 мин; 5 циклов: 95°C - 15 сек; 58°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. Далее в реакционную смесь добавляли 1,5 мкл эндонуклеазы I (New England BioLabs) и инкубировали 45 мин при 37°C. Эндонуклеазу инактивировали 10 мин прогреванием при 68°C и переносили пробирку в лед. Через 2 мин продукты реакции очищали с помощью наборов для выделения ДНК из агарозного геля и реакционных смесей Cleanup Mini (Евроген, Россия), элюировали 11,5 мкл стерильной деионизованной водой для молекулярно-биологических работ. 10 мкл полученного продукта использовали для амплификации в присутствии смеси REV-праймеров для мультиплексной амплификации (группы А, Б, В или Г, соответственно) и праймера TS-Uni-short (конечная концентрация праймеров 0,2 мкМ). Для ПЦР использовали набор реагентов Tersus PCR kit (Евроген, Россия) согласно прилагаемой инструкции. Амплификацию осуществляли в режиме: предварительная денатурация при 95°C в течение 1 мин; 29 циклов: 95°C - 15 сек; 62°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. Продукт ПЦР (конечное разведение в новой реакционной смеси 1:1000) подвергали nested ПЦР в присутствии смеси геноспецифических внутренних праймеров для ПЦР с выходом наружу (смесь 2) и праймера TruSeq Universal long (SEQ ID No: 197) в конечной концентрации 0,2 мкМ в режиме: предварительная денатурация при 95°C в течение 1 мин; 17 циклов ПЦР: 95°C - 15 сек; 58°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин.
[0166] Полученный продукт ПЦР разводили (конечная концентрация в новой реакционной смеси 1:1000) и анализировали специфичность амплификации с использованием индивидуальных пар праймеров (баркодирующий праймер + геноспецифический BRIDGE-праймер) для каждого референсного фрагмента (таблица 1). Для ПЦР использовали набор Encyclo PCR kit (Евроген, Россия) согласно прилагаемой инструкции производителя. Продукты амплификации анализировали при помощи гель-электрофореза в агарозе с ТАЕ-буфером и окрашиванием бромистым этидием. Продукты тестовых ПЦР клонировали в pAL-TA вектор и в каждом случае секвенировали вставки трех отобранных клонов для подтверждения соответствия структуры клонированного фрагмента ожидаемой нуклеотидной последовательности. Секвенирование осуществляли на автоматическом секвенаторе ABI3730xl. Во всех случаях секвенирование подтвердило соответствие структуры продукта амплификации ожидаемой нуклеотидной последовательности.
[0167] Пример 4
[0168] Выявление мутации опухолевого происхождения в ДНК плазмы крови
[0169] Был проведен сравнительный анализ мутаций в трех образцах ДНК от одного и того же пациента со злокачественной меланомой кожи III стадии. В качестве источников ДНК были использованы: а) операционный материал - первичная опухоль - в виде парафинового блока с фиксированной формалином тканью; б) операционный материал - лимфатический узел без признаков метастазирования, но с подозрением на микрометастазы по косвенным признакам («сторожевое» положение узла, увеличенный размер, аномальная структура стромы) - также в виде парафинового блока с фиксированной формалином тканью; в) кровь, забранная у пациента перед операцией.
[0170] Образцы, представленные в виде парафиновых блоков, сперва подвергались морфологическому контролю. По результатам морфологического контроля оба образца были признаны удовлетворяющими критериям для использования в настоящем эксперименте и были использованы для выделения ДНК без дополнительных диссекционных процедур. С каждого из блоков было изготовлено по 4 стеклопрепарата. Использовались предметные стекла, покрытые поли-L-лизином (ApexLab, Москва). На каждое предметное стекло наносилось по 2 среза толщиной 6 мкм и площадью от 0,9 до 1,3 см2. Срезы изготавливались на ротационном микротоме RM5230 (Leica, Германия) с использованием одноразовых лезвий; перед началом работы с каждым новым блоком рабочие поверхности микротома дважды протирались 96%-ным этанолом; первые два среза с каждого блока не использовались (выбрасывались) для исключения кросс-контаминации образцов. Для прикрепления срезов к поли-L-лизиновому покрытию предметные стекла после нанесения срезов прогревались в сухожаровом шкафу при температуре 56°C в течение 3 часов. Покраска и заключение срезов под покровные стекла не производились. Выделение ДНК из прикрепленных срезов производилось с помощью набора реагентов QuickExtract FFPE DNA Extraction Kit (Epicentre, США) в соответствии с инструкцией производителя с небольшими изменениями: по окончании инкубации при +90 С пробирки с раствором ДНК помещали на +4 С на 2 минуты, после чего центрифугировали на микроцентрифуге (СМ-50, BioSan, Латвия) на максимальных оборотах в течение 10 минут. Образовавшееся над раствором кольцо из концентрированного парафина осторожно прокалывали посередине 300-микролитровым одноразовым пластиковым наконечником для автоматического дозатора, после чего через прокол отбирали раствор с помощью одноканального автоматического механического дозатора и аналогичного 200-микролитрового наконечника; отбор раствора производили максимально осторожно, стараясь не захватить осадок на дне пробирки. Полученный раствор ДНК хранили при -20 C до дальнейшего использования.
[0171] Образец крови забирали путем пункции локтевой вены в пробирку типа Vacutainer (Beckton Dickinson, США) объемом 10 мл, содержащую 1,1 мл консерванта на основе K3EDTA. Кровь тщательно перемешивали с консервантом и помещали на +4 C. Не позднее чем через полчаса производили отделение плазмы крови от клеток крови с использованием стандартного протокола (описан, в частности, в Приложении 2 к инструкциям к наборам Insider, ЗАО «Евроген», Москва). Плазму и клетки крови хранили при -80 С до момента выделения ДНК. ДНК из плазмы крови выделяли с использованием набора реагентов QiaAMP Circulating Nucleic Acids Kit (Qiagen, США, cat. ID 55114) в соответствии с инструкцией производителя. Финальную элюцию производили двукратно, каждый раз использовали 11 мкл элюирующего буфера, входящего в состав набора реагентов. Раствор ДНК хранили при -20 С до дальнейшего использования.
[0172] Образцы ДНК подвергали ферментативной фрагментации с помощью набора реагентов dsDNA Fragmentase (New England Biolabs, США) в соответствии с инструкцией производителя, результаты фрагментации контролировали с помощью гель-электрофореза в агарозе с окрашиванием бромистым этидием. Фрагментацию вели до появления доминирующих фрагментов длиной от 100 до 150 п.о. Концентрацию ДНК измеряли с использованием набора реагентов XY-Detect (Синтол, Россия, кат. № HG-402) и панели ДНК-калибраторов (в концентрации 50, 10 и 1 нг/мкл; Лаборатория Изоген, Россия, кат. № R0010) в соответствии с инструкциями производителей.
[0173] Мутационный статус гена BRAF в образцах Τ (ДНК из опухолевой ткани), LN (ДНК из лимфатического узла) и Ρ (ДНК из плазмы крови) был проанализирован стандартной и мутационно-специфической ПЦР в режиме реального времени с использованием набора реагентов Insider B-Raf (Евроген, Россия) с последующим секвенированием по Сэнгеру. Было показано что в образце Τ присутствует мутация с. 1799 Т→А (p. Val600Glu) в 15-м экзоне гена BRAF примерно в 45% копий гена, в образце LN - примерно в 10% копий, а в образце Ρ она не обнаруживалась (предел избирательности анализа для данного образца составил 1:80).
[0174] Образцы Т, LN и Ρ использовали в качестве матрицы для баркодирования целевых фрагментов генов KRAS, NRAS, HRAS, ARAF, BRAF, CRAF, PIK3CA и Akt1. Параллельно через все этапы реакций баркодирования проводились также два контроля - положительный контроль ПЦР (30 нг фрагментированной ДНК из клеточной линии Raji) и отрицательный контроль ПЦР (NTC - no template control - контроль без ДНК). Баркодирование с помощью линейной ПЦР, очистку и последующую амплификацию целевых фрагментов осуществляли как описано в Примере 3. Использовали наборы праймеров, включенные в группу В в Табл. 2, полученные как описано в примере 3 (смешанные в неэквимолярных количествах), и праймеры для амплификации внутреннего контроля - фрагмента гена IDH1. Линейную ПЦР проводили в течение 5 циклов в двух повторностях, после обработки экзонуклеазой и очистки продукта реакции продукты двух повторностей линейной ПЦР объединяли и вносили в реакционную смесь для ПЦР-амплификации, которую проводили в режиме: предварительная денатурация при 95°C в течение 5 мин; 28 циклов: 95°C - 15 сек; 62°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. Продукт ПЦР-амплификации 1 разводили в 40 раз и 1 мкл разведенного продукта добавляли в 50 мкл реакционной смеси для ПЦР с выходом наружу (использовали праймеры с 5-ю различными индексными последовательностями - для трех исследуемых образцов, положительного контроля и отрицательного контроля ПЦР), которую осуществляли в режиме: предварительная денатурация при 95°C в течение 5 мин; 17 циклов: 95°C - 15 сек; 58°C - 20 сек; 72°C - 30 сек; достройка концов молекул при 72°C в течение 2 мин. Для контроля эффективности амплификации использовали гель-электрофорез в агарозе в ТАЕ-буфере (40 мМ Трис-ацетат, pH 7,8-8,0; 1 мМ ЭДТА) с окраской бромистым этидием. Продукты ПЦР разных образцов очищали с помощью набора для очистки продуктов ПЦР (Qiagen, Германия), измеряли концентрацию продукта на флуориметре Qubit 2.0 (Life Technologies, США) с помощью набора реагентов Quant-IT dsDNA BR Assay Kit (Life Technologies, США, Cat. ID Q32853) в соответствии с инструкцией производителя. По 50 нг продуктов амплификации образцов Τ и Raji (положительный контроль ПЦР) и весь объем продуктов амплификации образцов LN, Ρ и NTC (отрицательный контроль ПЦР) объединяли (суммарный вес полученной смеси продуктов ПЦР составил 860 нг) и секвенировали на приборе HiSeq2000 (Illumina) с использованием наборов HiSeq РЕ Cluster Kit v3 cBot (Illumina, кат. № РЕ-301-3001) и HiSeq SBS Kit v.3 (Illumina, кат. № FC-301-2003) в соответствии с инструкцией производителя. Были получены парные (двухсторонние) прочтения длиной 105 оснований.
[0175] Результаты массированного секвенирования были автоматически рассортированы по образцам, согласно индексным последовательностям, с помощью программы CASAVA v.2.3.1 (Illumina) и использованы для анализа мутаций в баркодированных и амплифицированных регионах генома человека. На первом этапе анализа данных проводили оценку качества данных по стандартному протоколу с использованием программного обеспечения fastqc с дальнейшей фильтрацией при помощи программного обеспечения flexbar. Дальнейший анализ проводили с применением стратегии MIGEC (Shugay et al. Nat Meth. 2014; 11(6):653-5). Была проведена оценка достоверности каждого отдельного прочтения, выделение молекулярных баркодов, группировка потомков одной исходной молекулы, сборка консенсусной последовательности внутри каждого «семейства» молекул, выравнивание полученных консенсусных последовательностей на референсные последовательности, вторичная корректировка редких ошибок, случайно оказавшихся доминирующими внутри отдельных «семейств» молекул, и вывод результатов в табличном виде.
[0176] Полученные результаты по экзону 15 гена BRAF представлены в Табл. 5. По другим исследованным регионам не было выявлено ни одного «семейства» с какой-либо мутацией ни в одном из 5 образцов.

[0177] Таким образом, удалось выявить небольшой процент мутантных молекул ДНК опухолевого происхождения в ДНК плазмы крови пациента с опухолью, имеющей соответствующую мутацию, с избирательностью более чем 1:140. Важно также отметить, что низкопредставленные молекулы с мутацией были бы неотличимы от ошибок ПЦР и секвенирования в случае стандартного анализа, а отличить одно от другого оказалось возможно именно благодаря использованию аналитического подхода, опирающегося на предварительную группировку прочтений в «семейства» на основе индивидуальных молекулярных баркодов.
>SEQ ID NO:1
aaatgagatctactgttttcctttacttactacacctcagATATATTTCTTCATGAAGACCTCACAGTAAAAATAGGTGATTTTGGTCTAGCTACAGTGAAATCTCGAT
>SEQ ID NO:2
TCCCCTTCCCTGCCCCAGCCAATGCCCCCCTACAGCGCATCCGCTCCACGTCCACTCCCAACGTCCATATGGTCAGCACCACGGCCCCCATGGACTCCAACCTCATCCAGgttggtgctgtgggggacaatgccggggac
>SEQ ID NO:3
CCTCACGCCTTCACCTTTAACACCTCCAGTCCCTCATCTGAAGGTTCCCTCTCCCAGAGGCAGAGGTCGACATCCACACCTAATGTCCACATGGTCAGCACCACCCTGCCTGTGGAC
>SEQ ID NO:4
acagAAACCCATGTATGAAGTACAGTGGAAGGTTGTTGAGGAGATAAATGGAAACAATTATGTTTACATAGACCCAACACAACTTCCTTATGATCACAAATGGGAGTTTCCCAGAAACAGGCTGAGTTTTGgtcagt
>SEQ ID NO:5
AGCTGGTGGCGTAGGCAAGAGTGCCTTGACGATACAGCTAATTCAGAATCATTTTGTGGACGAATATGATCCAACAATAGAGGtaaatcttgttttaatatgcatattactggt
>SEQ ID NO:6
gtgtttctcccttctcagGATTCCTACAGGAAGCAAGTAGTAATTGATGGAGAAACCTGTCTCTTGGATATTCTCGACACAGCAGGTCAAGAGGAGTACAGTGC
>SEQ ID NO:7
taacttttttttctttcccagAGAACAAATTAAAAGAGTTAAGGACTCTGAAGATGTACCTATGGTCCTAGTAGGAAATAAATGTGATTTGCCTTCTAGAACAGTAGACACAAAACAGGCTCAGGACTTAGCAAGAAGTTATGGAATTCCTTTTATTGAAACATCAGCAAAGACAAGACAGgtaagtaacactgaaataaatacagatctgtt
>SEQ ID NO:8
TGCTGGTGTGAAATGACTGAGTACAAACTGGTGGTGGTTGGAGCAGGTGGTGTTGGGAAAAGCGCACTGACAATCCAGCTAATCCAGAACCACTTTGTAGATGAATATGATCCCACCATAGAGgtgaggcc
>SEQ ID NO:9
CTGTTTGTTGGACATACTGGATACAGCTGGACAAGAAGAGTACAGTGCCATGAGAGACCAATACATGAGGACAGGCGAAGGCTTCCTCTGTGTATTTGCCATCAATAATAG
>SEQ ID NO:10
GTGGGAAACAAGTGTGATTTGCCAACAAGGACAGTTGATACAAAACAAGCCCACGAACTGGCCAAGAGTTACGGGATTCCATTCATTGAAACCTCAGCCAAGACCAGACAGgta
>SEQ ID NO:11
CCCGGGCCGCAGGCCCCTGAGGAGCGATGACGGAATATAAGCTGGTGGTGGTGGGCGCCGGCGGTGTGGGCAAGAGTGCGCTGACCATCCAGCTGATCCAGAACCATTTTGTGGACGAATACGACCC
>SEQ ID NO:12
CCGGAAGCAGGTGGTCATTGATGGGGAGACGTGCCTGTTGGACATCCTGGATACCGCCGGCCAGGAGGAGTACAGCGCCATGCGGGACCAGTACATGCGCACCGGGGAGGGC
>SEQ ID NO:13
GATGACGTGCCCATGGTGCTGGTGGGGAACAAGTGTGACCTGGCTGCACGCACTGTGGAATCTCGGCAGGCTCAGGACCTCGCCCGAAGCTACGGCATCCCCTACATCGAGACCTCGGCCAAGACCCGGCAGg
>SEQ ID NO:14
AACTTGGAGGCCTTGCAGAAGAAGCTGGAGGAGCTAGAGCTTGATGAGCAGCAGCGAAAGCGCCTTGAGGCCTTTCTTACCCAGAAGCAGAAGGTGGGAGAACTGAAGGATGACGACTTTGAGAAGATCAGTGAGCTGGGGGCTGG
>SEQ ID NO:15
CATGTGCTCCATGCAGATACTGATCTCGCCATCGCTGTAGAACGCACCATAGAAGCCCACGATGTACGGAGAGTTGCACTCATGCAGAACCTGCAGCTCCCTTATGATCTGGTTCCGGATTGCGGGTTTGATCTCCAGATGAATTAGct
>SEQ ID NO:16
GTGGGGAGATCAAGCTCTGTGACTTTGGGGTCAGCGGGCAGCTCATCGACTCCATGGCCAACTCCTTCGTGGGCACAAGGTCCTACATGTCGgtatgaacagaagtttccattgcttgagctt
>SEQ ID NO:17
ggccacctgcgcagagcctgcactcactccttgtgtgccctctagGACGGCGGCTCCCTGGACCAGGTGCTGAAAGAGGCCAAGAGGATTCCCGAGGAGATCCTGGGGAAAGTCAGCATCGCGgtgagtccaccg
>SEQ ID NO:18
CCTCCAACATCCTCGTGAACTCTAGAGGGGAGATCAAGCTGTGTGACTTCGGGGTGAGCGGCCAGCTCATCGACTCCATGGCCAACTCCTTCGT
>SEQ ID NO:19
acctttctctttatagCTCTGGACTTATTGGACAAAATGTTGACATTCAACCCACACAAGAGGATTGAAGTAGAACAGGCTCTGGCCCACCCATATCTGGAGCAGTATTACGACCCGAGTGACGAGgtaagagctca
>SEQ ID NO:20
TAAGGGAAAATGACAAAGAACAGCTCAAAGCAATTTCTACACGAGATCCTCTCTCTGAAATCACTGAGCAGGAGAAAGATTTTCTATGGAGTCACAGGTAAGTGCTAAAA
>SEQ ID NO:21
ATTCGAAAGACCCTAGCCTTAGATAAAACTGAGCAAGAGGCTTTGGAGTATTTCATGAAACAAATGAATGATGCACATCATGGTGGCTGGACAACAAAAATGGATTGGATCTTCCACACAATTAAACAGCAT
>SEQ ID NO:22
TCATTCTTGAGGAGGAAGTAGCGTGGCCGCCAGGTCTTGATGTACTCCCctacagacgtgc
>SEQ ID NO:23
aatattttccctaagtttgtaagtagtgctatatttatgttgactttttagttttactttatatgttattaatatgagtattgttaaccttgcagAATGGTCGATGTAGGGGGCCAAAGGTCAGAGAGAAGAAAATGGATACACTGCTTTGAAAATGTCACCTCTATCATGTTTCTAGTAGCGCTTA
>SEQ ID NO:24
ggccttggggcgccaggtggctgagtcctggcgctgtgtcctttcagGATGGTGGATGTGGGGGGCCAGCGGTCGGAGCGGAGGAAGTGGATCCACTGCTTTGAGAACGTGACATCCATCATGTTTCTCGTCGC
>SEQ ID NO:25
accttttttctctttctctttagAGCTGTAGGTAAAACTTGCCTACTGATCAGTTACACAACCAATGCATTTCCTGGAGAATATATCCCTACTGTgtaagtatcttaaattgggaatta
>SEQ ID NO:26
TGATAATTTCAGGAAACAAAAATTTGTGCTATGCAAATACAATAAACTGGAAAAAACTGTTTGGGACCTCCGGTCAGAAAACCAAAATTATAAGCAACAGAGGTGAAAACAGCTGCA
>SEQ ID NO:27
tacCTTATACACCGTGCCGAACGCACCGGAGCCCAGCACTTTGATCTTTTTGAATTCAGTTTCCTTCAAGATCCTCAAGAGAGCTTGGTTGGGAGCTTCTCCACTGGGTGTAAGAGGCTCCACAAGctgggg
>SEQ ID NO:28
gtcttccttctctctctgtcatagGGACTCTGGATCCCAGAAGGTGAGAAAGTTAAAATTCCCGTCGCTATCAAGGAATTAAGAGAAGCAACATCTCCGAAAGCCAACAAGGAAATCCTCGATgtgagttt
>SEQ ID NO:29
gtgcctctccctccctccagGAAGCCTACGTGATGGCCAGCGTGGACAACCCCCACGTGTGCCGCCTGCTGGGCATCTGCCTCACCTCCACCGTGCAGCTCATCACGCAGCTCATGCCCTTCGGCTGCCTCC
>SEQ ID NO:30
ccttccctgattacCTTTGCGATCTGCACACACCAGTTGAGCAGGTACTGGGAGCCAATATTGTCTTTGTGTTCCCGGACATAGTCCAGGAGGCAGCCGAAGGGCATGAGCTGCGTGATGAGCTGC
>SEQ ID NO:31
ccttacTTTGCCTCCTTCTGCATGGTATTCTTTCTCTTCCGCACCCAGCAGTTTGGCCAGCCCAAAATCTGTGATCTTGACATGCTGCGGTGTTTTCACCAGTACGTTCCTGGCTGCCAGGTCGCGGTGCACCAAGCGACGGTCCTCCAAGTAGTTCATGCCctgaaacagagaagaccctgctgtgagggacagatcatcatgggaaga
>SEQ ID NO:32
tccttagACAACTACCTTTCTACGGACGTGGGATCCTGCACCCTCGTCTGCCCCCTGCACAACCAAGAGGTGACAGCAGAGGATGGAACACAGCGGTGTGAGAAGTGCAGCAAGCCCTGTGCCCGAGgtacccactcactgcccccgag
>SEQ ID NO:33
ccccagCTGGTGGAGCCGCTGACACCTAGCGGAGCGATGCCCAACCAGGCGCAGATGCGGATCCTGAAAGAGACGGAGCTGAGGAAGGTGAAGGTGCTTGGATCTGGCGCTTT
>SEQ ID NO:34
TGCTTGGATCTGGCGCTTTTGGCACAGTCTACAAGgtcagggccaggtcctggggtgggcggccccagaggatgggggcggtgcctgg
>SEQ ID NO:35
ctgcccagGGCATCTGGATCCCTGATGGGGAGAATGTGAAAATTCCAGTGGCCATCAAAGTGTTGAGGGAAAACACATCCCCCAAAGCCAACAAAGAAATCTTAGACgtaagcccctccaccctctcctgc
>SEQ ID NO:36
tacccttgtccccagGAAGCATACGTGATGGCTGGTGTGGGCTCCCCATATGTCTCCCGCCTTCTGGGCATCTGCCTGACATCCACGGTGCAGCTGGTGACACAGCTTATG
>SEQ ID NO:37
ATGCCCTATGGCTGCCTCTTAGACCATGTCCGGGAAAACCGCGGACGCCTGGGCTCCCAGGACCTGCTGAACTGGTGTATGCAGATTGCCAAGgtatgcacctg
>SEQ ID NO:38
atgggtgcttcccattccagGGGATGAGCTACCTGGAGGATGTGCGGCTCGTACACAGGGACTTGGCCGCTCGGAACGTGCTGGTCAAGAGTCCCAACCATGTCAAAATTACAGACTTC
>SEQ ID NO:39
AAATTACAGACTTCGGGCTGGCTCGGCTGCTGGACATTGACGAGACAGAGTACCATGCAGATGGGGGCAAGgttaggtgaaggaccaaggagcagaggaggctgg
>SEQ ID NO:40
CGGCGATGCTGAGAACCAATACCAGACACTGTACAAGCTCTACGAGAGGTGTGAGGTGGTGATGGGGAACCTTGAGATTGTGCTCACGGGAC
>SEQ ID NO:41
CTATGTCCTCGTGGCCATGAATGAATTCTCTACTCTACCATTGCCCAACCTCCGCGTGGTGCGAGGGACCCAGGTCTACGATGGGAAGTTTGCCATCTTCGTCATGTTGAACTATAACACCAACTCC
>SEQ ID NO:42
ttctctccttccatagTGACCAAGACCATCTGTGCTCCTCAGTGTAATGGTCACTGCTTTGGGCCCAACCCCAACCAGTGCTGCCATGATGAGTGTGCCGGGGGCTGCTCAGGCCCTCAGGACACAGACTGCTTTgtatg
>SEQ ID NO:43
CCACAGCCTCTTGTCTACAACAAGCTAACTTTCCAGCTGGAACCCAATCCCCACACCAAGTATCAGTATGGAGGAGTTTGTGTAGCCAGCTGTCCCCgtaagtgtctgaggggaagga
>SEQ ID NO:44
tcatctctaatggtgtcctcctcctcttccctagATAACTTTGTGGTGGATCAAACATCCTGTGTCAGGGCCTGTCCTCCTGACAAGATGGAAGTAGATAAAAATGGGCTCAAGATGTGTGAGCCTTGTGGGGGACTATGTCCCAAAGgtgggtag
>SEQ ID NO:45
GGGAACAGGCTCTGGGAGCCGCTTCCAGACTGTGGACTCGAGCAACATTGATGGATTTGTGAACTGCACCAAGATCCTGGGCAACCTGGACTTTCTGATCAC
>SEQ ID NO:46
tacagGGAATGTACTACCTTGAGGAACATGGTATGGTGCATAGAAACCTGGCTGCCCGAAACGTGCTACTCAAGTCACCCAGTCAGGTTCAGGTGGCAGATTTTGGTGTGGCTGACCTGCTGCCTCCTGATGATAAGCAGC
>SEQ ID NO:47
ttcctgcaacagGTGTGACAGTTTGGGAGTTGATGACCTTCGGGGCAGAGCCCTATGCAGGGCTACGATTGGCTGAAGTACCAGACCTGCTAGAGAAGGGGGAGCGGTTGGCACAGCCCCAGATCTGCACAATTGATGTCTACA
>SEQ ID NO:48
ctgaatgttaacattaatgcttattttaccttttttttttttttaatcagATAACTTTGTGGTAGATTCCAGTTCTTGTGTGCGTGCCTGCCCTAGTTCCAAGATGGAAGTAGAAGAAAA
>SEQ ID NO:49
AGATGGAAGTAGAAGAAAATGGGATTAAAATGTGTAAACCTTGCACTGACATTTGCCCAAAAGgtgagtggtagctcagattatttagttagaactgggcctatat
>SEQ ID NO:50
tttctcacttccccctccttagTGGCCTGTCCTTGCTTATCCTCAAGCAACAGGGCATCACCTCTCTACAGTTCCAGTCCCTGAAGGAAATCAGCGCAGGAAACATCTATATTACTGACAACAGCAACC
>SEQ ID NO:51
TATTACTGACAACAGCAACCTGTGTTATTATCATACCATTAACTGGACAACACTCTTCAGCACAATCAACCAGAGAATAGTAATCCGGGACAACAGAAAAGCTGAAAATTGTAgtaagtacattactcagacatttaaaacatagaaacttaaattt
>SEQ ID NO:52
tcctcatgtggctgatgttttcacagTGAATTTCGGGAGTTTGAGAATGGCTCCATCTGTGTGGAGTGTGACCCCCAGTGTGAGAAGATGGAAGATGGCCTCCTCACATGCCATGGACCGgtaag
>SEQ ID NO:53
atatttttcagGGAATGATGTACCTGGAAGAAAGACGACTCGTTCATCGGGATTTGGCAGCCCGTAATGTCTTAGTGAAATCTCCAAACCATGTGAAAATCACAGATTTTGG
>SEQ ID NO:54
TGAAATCTCCAAACCATGTGAAAATCACAGATTTTGGGCTAGCCAGACTCTTGGAAGGAGATGAAAAAGAGTACAATGCTGATGGAGGAAAGgtgggtatcttttt
>SEQ ID NO:55
tcaactctgtctccttcctcttcctacagTACTCCCCTGCCCTCAACAAGATGTTTTGCCAACTGGCCAAGACCTGCCCTGTGCAGCTGTGGGTTGATTCCACACCCCCGCCCGGCACCCGCGTCCGCGCCATGGC
>SEQ ID NO:56
TTCCACACCCCCGCCCGGCACCCGCGTCCGCGCCATGGCCATCTACAAGCAGTCACAGCACATGACGGAGGTTGTGAGGCGCTGCCCCCACCATGAGCGCTGCTCAGATAGCGATGgtgagcagctggggc
>SEQ ID NO:57
ctcactgattgctcttagGTCTGGCCCCTCCTCAGCATCTTATCCGAGTGGAAGGAAATTTGCGTGTGGAGTATTTGGATGACAGAAACACTTTTCGACATAGTGTGGTGGTGCCCTATGAGCCGCCTGAGgtctggtttgcaactggg
>SEQ ID NO:58
ttatctcctagGTTGGCTCTGACTGTACCACCATCCACTACAACTACATGTGTAACAGTTCCTGCATGGGCGGCATGAACCGGAGGCCCATCCTCACCATCATCACACTGGAAGACTCCAGgtcaggagcca
>SEQ ID NO:59
agtagTGGTAATCTACTGGGACGGAACAGCTTTGAGGTGCGTGTTTGTGCCTGTCCTGGGAGAGACCGGCGCACAGAGGAAGAGAATCTCCGCAAGAAAGGGGAGCCTCACCACGAGCTGCCCCCAGGGAGCACTAAGCGAGgtaag
>SEQ ID NO:60
ttgcctctttcctagCACTGCCCAACAACACCAGCTCCTCTCCCCAGCCAAAGAAGAAACCACTGGATGGAGAATATTTCACCCTTCAGgtactaagtctt
>SEQ ID NO:61
atttatagCTGATTTGATGGAGTTGGACATGGCCATGGAACCAGACAGAAAAGCGGCTGTTAGTCACTGGCAGCAACAGTCTTACCTGGACTCTGGAATCCATTCTGGTGCCACTACCACAGCTCCTTC
>SEQ ID NO:62
CACAGCTCCTTCTCTGAGTGGTAAAGGCAATCCTGAGGAAGAGGATGTGGATACCTCCCAAGTCCTGTATGAGTGGGAACAGGGATTTTCTCAGTCCTTCACTCAAGAACAAGTAGCTGGTAAGAGTATTATTTTTCAT
>SEQ ID NO:63
TTGATCCCCATAAGCATGACGACCTATGATGATAGGTTTTACCCATCCACTCACAAGCCGGGGGATATTTTTGCAGATAATGGCTTCTCTGAAGACCGTGCCA
>SEQ ID NO:64
GCAGCCGTCCTGGAGGCTGGTGTCCTGGGTAGCGGTCAGCACGCCCCCGTCTTCGTATGTGGTGACTCTCTCCCATGTGAAGCCCTCGGGGA
>SEQ ID NO:65
AAACACTCGGCTGGGAGGCCTCCACCGAGACCCTGTACCCCGCTGACGGCGGCCTGGAAGGCAGAGCCGACATGGCCCTGAAGCTCGTGGGCGGGGGCCACCTGATCTGCAACTTGA
>SEQ ID NO:66
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCATCTCACAATTGCCAGTTAAC
>SEQ ID NO:67
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTATCTCCCCTCCCCGTATCTC
>SEQ ID NO:68
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCTGACCCACCACCCCCTCA
>SEQ ID NO:69
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGGTCCTTCCTGTCCTCCTA
>SEQ ID NO:70
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCGGAGAGACCTGCAAAGAGCC
>SEQ ID NO:71
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNATGGGCTAGACACCACTCCAC
>SEQ ID NO:72
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGAAGGAGAGGTCGGCATTGT
>SEQ ID NO:73
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGAGCCCTAACAGCCATGCT
>SEQ ID NO:74
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTAGCTGGTGATGTTCCTCCC
>SEQ ID NO:75
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNAATACTTTCTTCCCCTATACC
>SEQ ID NO:76
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTATCTTGTTTTGAGCTTGTTTG
>SEQ ID NO:77
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGTCCAAAGAAGAATGGGAAA
>SEQ ID NO:78
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTTGACGCTTGTTTGCTTACACC
>SEQ ID NO:79
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTTTTCAAGCAAGATTGCTCTC
>SEQ ID NO:80
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTGCTGTGACTGCTTGTAGATG
>SEQ ID NO:81
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNAGGGTCCCCAGGCCTCTGATTC
>SEQ ID NO:82
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTGCTTCTCTTTTCCTATCCTG
>SEQ ID NO:83
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGAGGCCTGTGCCAGGGACCT
>SEQ ID NO:84
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNACCATGCGAAGCCACACTGAC
>SEQ ID NO:85
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNN CTGGCTGACCTAAAGCCACCT
>SEQ ID NO:86
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGACAGGAACTGCAGCTGGC
>SEQ ID NO:87
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGAACTGCCGACCACACCCCT
>SEQ ID NO:88
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGTCTCCCATACCCTCTCAGCG
>SEQ ID NO:89
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGCCCTCCCAGAAGGTCTAC
>SEQ ID NO:90
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTGGATTCGAGAAGTGACAGG
>SEQ ID NO:91
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGATCTACTATTGTTGATCATTGT
>SEQ ID NO:92
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTCTTACATTTGCAGCCTGTGA
>SEQ ID NO:93
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGCCAGTGACTAGTCCATGTC
>SEQ ID NO:94
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCTTAAAAGTGAATTAAAAACCTTGTT
>SEQ ID NO:95
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGAATGCGTTTCATGGTTCCGT
>SEQ ID NO:96
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCTGAATATCATTAAGGAACTTG
>SEQ ID NO:97
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCCAGCCCTGTCGTCTCTCCA
>SEQ ID NO:98
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGCCTCATCTTGGGCCTGTG
>SEQ ID NO:99
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNATTCACTTTTATCACCTTTCC
>SEQ ID NO:100
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCATAATGCTTGCTCTGATAGGA
>SEQ ID NO:101
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNACTGTGACCCGGAGCACT
>SEQ ID NO:102
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTTCTCAGCACAGATATTCTACA
>SEQ ID NO:103
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCTATTTTTCCCTTTCTCCCC
>SEQ ID NO:104
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTATAAACTTGTGGTAGTTGG
>SEQ ID NO:105
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTGCACTGTAATAATCCAGACT
>SEQ ID NO:106
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCAGTGTTACTTACCTGTCTTG
>SEQ ID NO:107
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTTACTGGTTTCCAACAGGTTCT
>SEQ ID NO:108
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNAAGTGGTTATAGATGGTGAAAC
>SEQ ID NO:109
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNACAAATGCTGAAAGCTGTACCA
>SEQ ID NO:110
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGCCAGGCTCACCTCTATAGTG
>SEQ ID NO:111
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGATGGCAAACACACACAGGAA
>SEQ ID NO:112
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGGTGGAGAGCTGCCTCA
>SEQ ID NO:113
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGGAAAATGACAAAGAACAGC
>SEQ ID NO:114
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTCTTTTGATGACATTGCATAC
>SEQ ID NO:115
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGCCAACCCCCCAAATCTGAA
>SEQ ID NO:116
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNAGGAGATAAGTGATGGAGATG
>SEQ ID NO:117
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCATTCTGACTTTCAGTAAGGCA
>SEQ ID NO:118
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTCCCCACTTTGGAACAGGACC
>SEQ ID NO:119
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGCTGAGAGGGTGTCACATAC
>SEQ ID NO:120
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCCTCTCCCTGACCGTACAAG
>SEQ ID NO:121
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGGGCGCGATGTGGGTCTG
>SEQ ID NO:122
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTGCCGCCTCCAGATGTGAAGC
>SEQ ID NO:123
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCTTCTGCTCTCACTACTGCAAA
>SEQ ID NO:124
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNGGATGATCATCGTCATTCAAGAG
>SEQ ID NO:125
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCCGTCCTGGGATTGCAGATTG
>SEQ ID NO:126
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTACATTCATGTTGACTAAGCA
>SEQ ID NO:127
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCTACTAATGCTAATACTGTTTCGT
>SEQ ID NO:128
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCATTATTGCCAACATGACTTAC
>SEQ ID NO:129
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNTGATCTTGACGTTGTAGATGAG
>SEQ ID NO:130
ACACGACGCTCTTCCGATCTNNNNNNNNNNNNNNCGGCCCTGTGATGCAGAAGA
>SEQ ID NO:131
GAGGTTCAGAGCCATGGACC
>SEQ ID NO:132
ACGTGTGCCGCCTGCTGGG
>SEQ ID NO:133
CCTGGCCCTGACCTTGTAGAC
>SEQ ID NO:134
CCCACGCTCTTCTCACTCATA
>SEQ ID NO:135
CATCTGCCTGACATCCACGGT
>SEQ ID NO:136
GGAACGTGCTGGTCAAGAGTC
>SEQ ID NO:137
TCCTGGGACTCTGAATGGC
>SEQ ID NO:138
AATTTCAGAACCCAGGCAATG
>SEQ ID NO:139
GAAAGGCTGTCTCTTTACAACT
>SEQ ID NO:140
CTTTGTGTCTCCTCACCTTG
>SEQ ID NO:141
TTTGGGCAAATGTCAGTGCAAG
>SEQ ID NO:142
CAGTTCCAGTCCCTGAAGGA
>SEQ ID NO:143
TAAATAAGCCAACACACCACAG
>SEQ ID NO:144
GACTCGTTCATCGGGATTTG
>SEQ ID NO:145
GTTGCTTTATCTGTTCACTTG
>SEQ ID NO:146
CACCCGGAGGGCCACTGACAAC
>SEQ ID NO:147
CTCCACCGCTTCTTGTCCT
>SEQ ID NO:148
AGGGCTGAGGTGACCCTTGT
>SEQ ID NO:149
AGCCAATATTGTCTTTGTGTTCC
>SEQ ID NO:150
ATGATCTGTCCCTCACAGCA
>SEQ ID NO:151
CCAGGGTATGTGGCTACATG
>SEQ ID NO:152
CCTGAAAGAGACGGAGCTGAG
>SEQ ID NO:153
TCCCGGACATGGTCTAAGAG
>SEQ ID NO:154
CTCGTCAATGTCCAGCAGCC
>SEQ ID NO:155
GGTGAGCTGAGTCAAGCGG
>SEQ ID NO:156
CGGCACTTCAATGACAGTGGA
>SEQ ID NO:157
GGGAAGGCAGGATCTCTAAC
>SEQ ID NO:158
CAGCAGGTAACTCACACTTGA
>SEQ ID NO:159
GATTCCAGTTCTTGTGTGCGT
>SEQ ID NO:160
GCTGAAGAGTGTTGTCCAGT
>SEQ ID NO:161
CTTTCCTCCATCAGCATTGTA
>SEQ ID NO:162
CTCAACAAGATGTTTTGCCAAC
>SEQ ID NO:163
GGCAAGCAGAGGCTGGGGCAC
>SEQ ID NO:164
ACGGCATTTTGAGTGTTAGAC
>SEQ ID NO:165
CAACTGTTCAAACTGATGGGA
>SEQ ID NO:166
GCCAACAGGGATGGCTCTA
>SEQ ID NO:167
CAAGGTGCCCTATTACCTCA
>SEQ ID NO:168
TACCCAAAAAGGTGACATGG
>SEQ ID NO:169
TGAAAATGGTCAGAGAAACCT
>SEQ ID NO:170
CTCCCCAGTCCTCATGTACT
>SEQ ID NO:171
AAAGAGTTAAGGACTCTGAAGA
>SEQ ID NO:172
CAAGTGAGAGACAGGATCAGG
>SEQ ID NO:173
ACCTGTAGAGGTTAATATCCG
>SEQ ID NO:174
AGGGAGCAGATTAAGCGAGT
>SEQ ID NO:175
GGCAGGTGGGGCAGGAGAC
>SEQ ID NO:176
TCCCTGAGCCTTGTCCTCCT
>SEQ ID NO:177
TCTCGCTTTCCACCTCTCAG
>SEQ ID NO:178
AACATGCTGAGATCAGCCAGAT
>SEQ ID NO:179
AGTGTGGAATCCAGAGTGAGC
>SEQ ID NO:180
TCTGACGGGTAGAGTGTGC
>SEQ ID NO:181
CAAATAAAGGACCCATTAGAACC
>SEQ ID NO:182
CTTACCTGGACTCTGGAATC
>SEQ ID NO:183
AGGCCAGAAGGCTTGTGGG
>SEQ ID NO:184
CCCTCTTTCTTTCATAAAACCTC
>SEQ ID NO:185
TTCTCTTCCCTGCAGATGTCA
>SEQ ID NO:186
GGGAGTGGGGGGACGCATC
>SEQ ID NO:187
CAGCGGGGACTCACAGCCAT
>SEQ ID NO:188
ATCTATGTCCCTGAAGCAGC
>SEQ ID NO:189
CTTACCTCATTGTCTGACTCC
>SEQ ID NO:190
CTCGTTGTCCGACTCCACCA
>SEQ ID NO:191
CATATGAGGATTAAAGATGGCTT
>SEQ ID NO:192
CATCCTCTTCCTCAGGATTGC
>SEQ ID NO:193
ATCACCAAATGGCACCATAC
>SEQ ID NO:194
ACCTTCATCAACCACACCCA
>SEQ ID NO:195
GCATCTTGAGGTTCTTAGCG
>SEQ ID NO:199
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGCCATGGACCCCCACACAG
>SEQ ID NO:200
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCATCTGCCTCACCTCCACCGT
>SEQ ID NO:201
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTTGACCTTGTAGACTGTGCCA
>SEQ ID NO:202
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTCACTCATATCCTCCTCTTT
>SEQ ID NO:203
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGCAGCTGGTGACACAGCTT
>SEQ ID NO:204
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCAAGAGTCCCAACCATGTCA
>SEQ ID NO:205
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTGAATGGCCTGAGTGTGAC
>SEQ ID NO:206
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCCCAGGCAATGGAAAGGGTA
>SEQ ID NO:207
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTTTACAACTTCTTACCATCTC
>SEQ ID NO:208
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTTGGCCTCACTGTATAGCA
>SEQ ID NO:209
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGGTTTACACATTTTAATCCCA
>SEQ ID NO:210
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAATCAGCGCAGGAAACATCTA
>SEQ ID NO:211
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTACACCACAGATGTCTTCAGG
>SEQ ID NO:212
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGGCAGCCCGTAATGTCTTAG
>SEQ ID NO:213
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTTCACTTGTGCCCTGACTT
>SEQ ID NO:214
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTTAACCCCTCCTCCCAGAGAC
>SEQ ID NO:215
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTACCGCTTCTTGTCCTGCTTG
>SEQ ID NO:216
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTCTCTGTGTTCTTGTCCC
>SEQ ID NO:217
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCAGCCGAAGGGCATGAGCT
>SEQ ID NO:218
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCCCTCACAGCAGGGTCTTC
>SEQ ID NO:219
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTGGCTACATGTTCCTGATC
>SEQ ID NO:220
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGAGCTGAGGAAGGTGAAGG
>SEQ ID NO:221
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAAGAGGCAGCCATAGGG
>SEQ ID NO:222
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAGCAGCCGAGCCAGCCC
>SEQ ID NO:223
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTGGCGCAGAGCGTGGCT
>SEQ ID NO:224
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAGCCTGTGTACCTCGCTGT
>SEQ ID NO:225
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTATCTCTAACCATTGAGGCCG
>SEQ ID NO:226
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTCACACTTGACCATCACCA
>SEQ ID NO:227
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCGTGCCTGCCCTAGTTCCA
>SEQ ID NO:228
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAGTTAATGGTATGATAATAACACA
>SEQ ID NO:229
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTTCCAAGAGTCTGGCTAGC
>SEQ ID NO:230
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTGCAGCTGTGGGTTGA
>SEQ ID NO:231
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCCAGTGTGCAGGGTGGCAAG
>SEQ ID NO:232
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCCACTTGATAAGAGGTCCC
>SEQ ID NO:233
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAAACTGATGGGACCCACTCC
>SEQ ID NO:234
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTCTACCCTCTGCCCTGTG
>SEQ ID NO:235
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTATTACCTCAATCATCCTGCT
>SEQ ID NO:236
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCATGGAAAGCCCCTGTTTCAT
>SEQ ID NO:237
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTATCAAAGAATGGTCCTGC
>SEQ ID NO:238
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTCATGTACTGGTCCCTCATT
>SEQ ID NO:239
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGACTCTGAAGATGTACCTATG
>SEQ ID NO:240
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAGGTCAGCGGGCTACCACTG
>SEQ ID NO:241
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTTAATATCCGCAAATGACTTG
>SEQ ID NO:242
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGATGATGTACCTATGGTGCTA
>SEQ ID NO:243
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAGGAGACCCTGTAGGAGGAC
>SEQ ID NO:244
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTCCTCCTGCAGGATTCCTA
>SEQ ID NO:245
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCAAACGGGTGAAGGACTCG
>SEQ ID NO:246
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGAAAAAGAAACAGAGAATCTCCA
>SEQ ID NO:247
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGTTATCTTTTCAGTTCAATGC
>SEQ ID NO:248
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAGAGTGTGCGTGGCTCTCA
>SEQ ID NO:249
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAAACAGAAAGCGGTGACTTAC
>SEQ ID NO:250
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTATCCATTCTGGTGCCACTAC
>SEQ ID NO:251
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTTGAACACCACACCGCCATTG
>SEQ ID NO:252
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCTCTTTCTTCCACCTTTCTCC
>SEQ ID NO:253
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCAACATCCTAGTCAACTCCC
>SEQ ID NO:254
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGACGCATCCGTGGCTTCCC
>SEQ ID NO:255
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCATGTAGGAGCGCGTGCCC
>SEQ ID NO:256
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGAAGCAGCAGCCAGGAACA
>SEQ ID NO:257
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTATATTCACTAAGCGCTACTAGA
>SEQ ID NO:258
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTACTTGGTCGTATTCGCTGAGG
>SEQ ID NO:259
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTAACACAAACAGGTTAATTCCCA
>SEQ ID NO:260
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGGATTGCCTTTACCACTCAGA
>SEQ ID NO:261
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCACCATACGAAATATTCTGGG
>SEQ ID NO:262
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCCCGACTTCTTTAAGCAGTCCT
>SEQ ID NO:263
GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTTCTTGGATCTGTATGTGGTCT
>SEQ ID NO:264
CAAGCAGAAGACGGCATACG AGATTGGTCAGTGACTGGAGTTCAGACGTGT
>SEQ ID NO:265
CAAGCAGAAGACGGCATACGAGAGTGACTGGAGTTCAGACGTGT
>SEQ ID NO:266
CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGT
>SEQ ID NO:267
CAAGCAGAAGACGGCATACGAGATACATCGGTGACTGGAGTTCAGACGTGT
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ АНАЛИЗА СОМАТИЧЕСКИХ МУТАЦИЙ В ГЕНАХ EGFR, KRAS И BRAF С ИСПОЛЬЗОВАНИЕМ LNA-БЛОКИРУЮЩЕЙ МУЛЬТИПЛЕКСНОЙ ПЦР И ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ С ОЛИГОНУКЛЕОТИДНЫМ БИОЛОГИЧЕСКИМ МИКРОЧИПОМ (БИОЧИПОМ) | 2014 |
|
RU2552483C1 |
| СПОСОБ АНАЛИЗА СОМАТИЧЕСКИХ МУТАЦИЙ В ГЕНЕ PI3K С ИСПОЛЬЗОВАНИЕМ LNA-БЛОКИРУЮЩЕЙ МУЛЬТИПЛЕКСНОЙ ПЦР И ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ С ОЛИГОНУКЛЕОТИДНЫМ БИОЛОГИЧЕСКИМ МИКРОЧИПОМ (БИОЧИПОМ) | 2013 |
|
RU2549682C1 |
| Способ анализа соматических мутаций в генах BRAF, NRAS и KIT с использованием LNA-блокирующей мультиплексной ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом) | 2017 |
|
RU2674338C1 |
| ПРЯМОЙ ЗАХВАТ, АМПЛИФИКАЦИЯ И СЕКВЕНИРОВАНИЕ ДНК-МИШЕНИ С ИСПОЛЬЗОВАНИЕМ ИММОБИЛИЗИРОВАННЫХ ПРАЙМЕРОВ | 2011 |
|
RU2565550C2 |
| Способ идентификации мутации в пятом экзоне гена FAD3A, приводящей к снижению содержания линоленовой кислоты в льняном масле | 2022 |
|
RU2806563C1 |
| Способ идентификации мутации во втором экзоне гена FAD3B, приводящей к снижению содержания линоленовой кислоты в льняном масле | 2022 |
|
RU2806561C1 |
| Способ диагностики инфекции Хеликобактер пилори и ее устойчивости к кларитромицину и левофлоксацину | 2022 |
|
RU2806581C1 |
| Способ секвенирования экзонов гена HLA-DQA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2833819C1 |
| ГЕН И ВАРИАЦИИ, СВЯЗАННЫЕ С ФЕНОТИПОМ BM1, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2012 |
|
RU2617958C2 |
| Способ секвенирования экзонов гена HLA-DPA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2837865C1 |


































































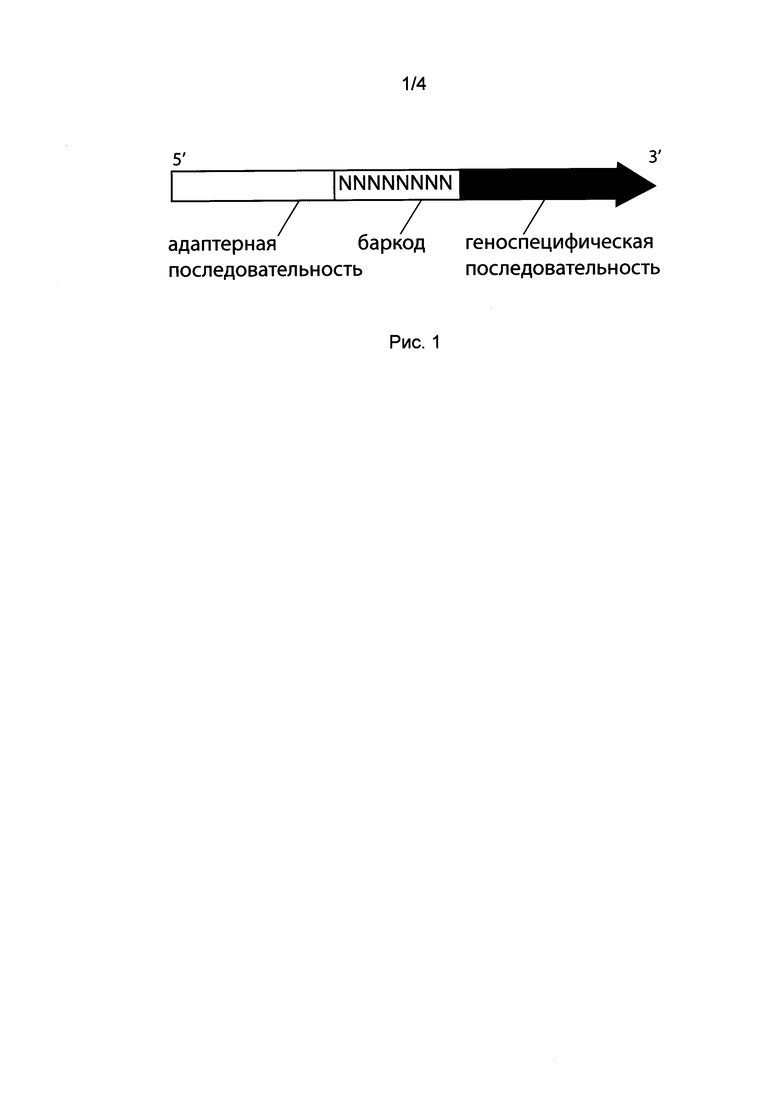
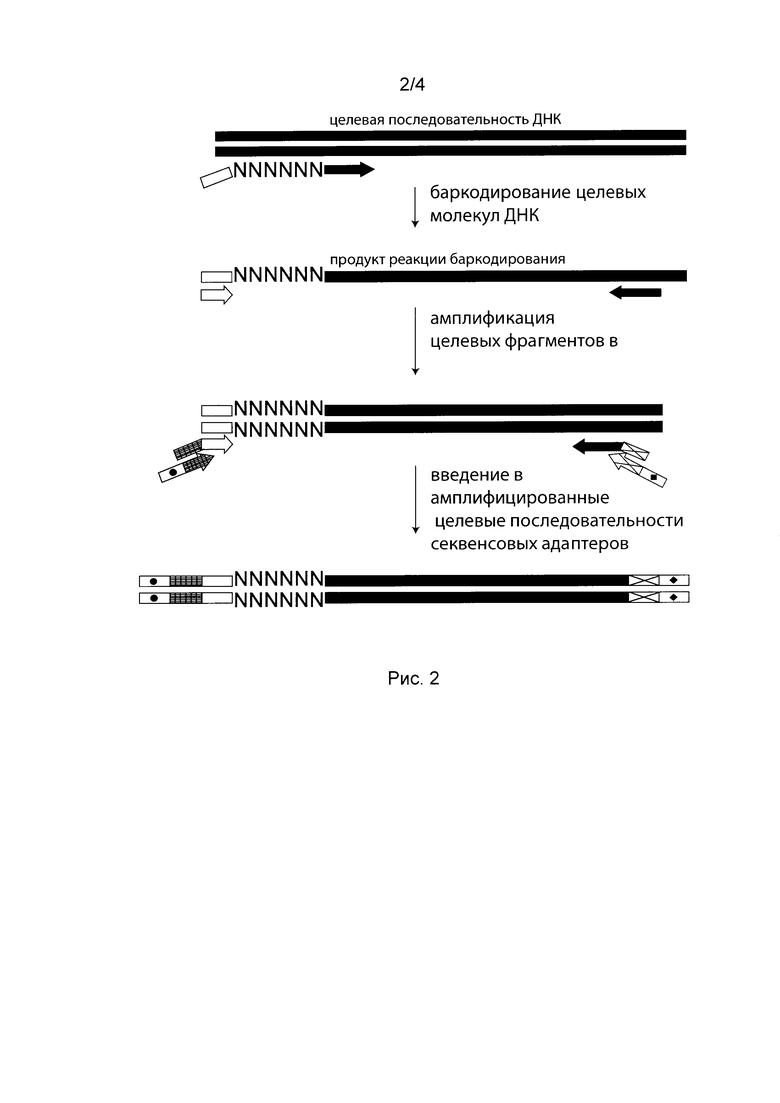
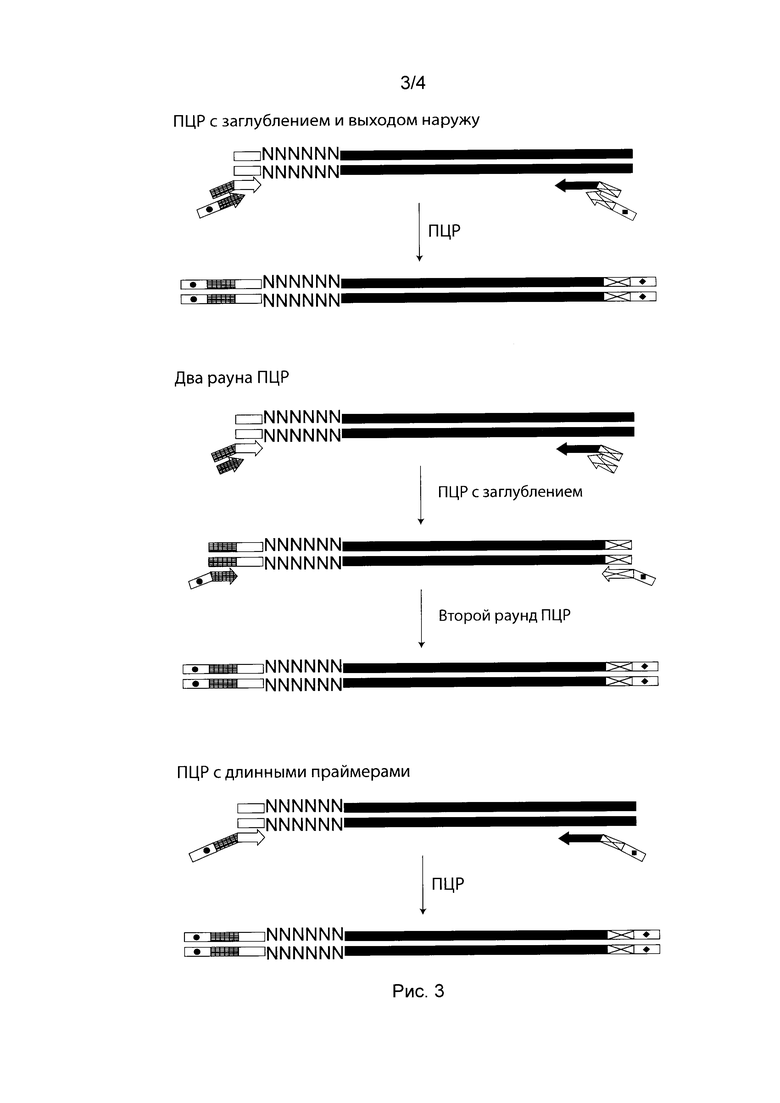
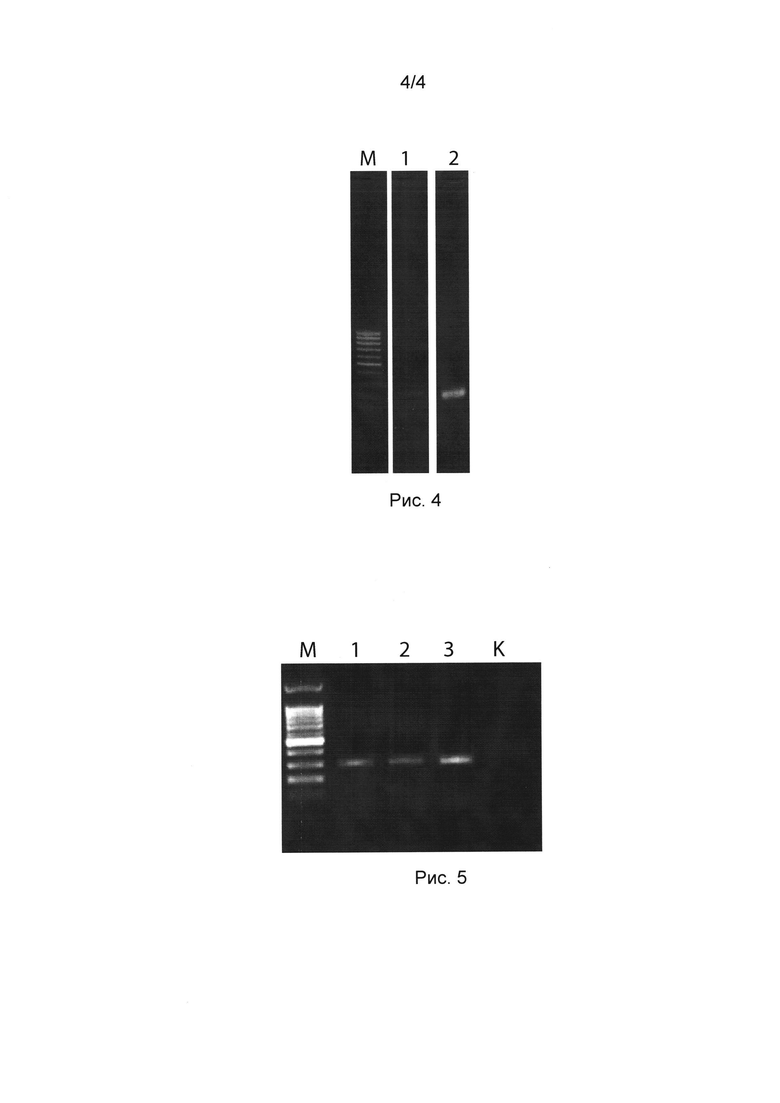
Настоящее изобретение направлено на методы выявления в образце мутаций в ДНК, где мутантная ДНК присутствует на фоне избытка немутантной ДНК. В некоторых применениях немутантная ДНК присутствует в образце в тысячекратном избытке. Метод включает молекулярное баркодирование молекул ДНК, их амплификацию, секвенирование нового поколения и анализ данных секвенирования. 3 н.п. ф-лы, 5 ил., 5 табл., 4 пр.
1. Способ выявления мутаций в целевых последовательностях ДНК, включающий:
(а) молекулярное баркодирование пула целевых молекул ДНК в образце ДНК путем введения в последовательности целевых молекул ДНК случайной последовательности методом мультиплексной линейной ПЦР в присутствии баркодирующих праймеров, несущих на 3'-конце геноспецифические последовательности, комплементарные целевым последовательностям ДНК, за которыми следует случайная последовательность (баркод), и адаптерную последовательность на 5'-конце;
(б) очистку продукта реакции баркодирования от остатков баркодирующего праймера, включающую инкубацию с экзонуклеазой I и дополнительную очистку с помощью гель-фильтрации;
(в) амплификацию пула целевых молекул с помощью мультиплексной ПЦР с праймером, по существу сходным с адаптерной последовательностью баркодирующего праймера, и встречными геноспецифическими праймерами на целевые регионы;
(г) введение сиквенсовых адаптеров в продукт ПЦР с помощью ПЦР с выходом наружу в присутствии заглубленных внутренних геноспецифических праймеров и секвенирование указанного продукта ПЦР;
(д) идентификацию мутаций в целевых ДНК по данным секвенирования.
2. Способ выявления мутаций в целевых последовательностях ДНК, выбранных из SEQ ID No: 1 - SEQ ID No: 62, включающий:
(а) молекулярное баркодирование пула целевых молекул ДНК в образце ДНК путем введения в последовательности целевых молекул ДНК случайной последовательности методом мультиплексной линейной ПЦР в присутствии соответствующих целевым последовательностям баркодирующих праймеров, выбранных из группы SEQ ID NOs: 66-127
(б) очистку продукта реакции баркодирования от остатков баркодирующего праймера, включающую инкубацию с экзонуклеазой I и дополнительную очистку с помощью гель-фильтрации;
(в) амплификацию пула целевых молекул с помощью мультиплексной ПЦР с праймером, по существу сходным с адаптерной последовательностью баркодирующего праймера, и встречными геноспецифическими праймерами на целевые регионы, выбранными из SEQ ID NOs: 131-192;
(г) введение сиквенсовых адаптеров в продукт ПЦР с помощью ПЦР с выходом наружу в присутствии заглубленных внутренних геноспецифических праймеров, выбранных из SEQ ID NOs: 199-260, и секвенирование указанного продукта ПЦР;
(д) идентификацию мутаций в целевых ДНК по данным секвенирования.
3. Набор олигонуклеотидных праймеров для реализации способа по п. 1 или 2, выбранный из групп А, Б, В или Г, показанных в таблице 2, где указанные набор включает набор праймеров для баркодирования, набор REV праймеров для амплификации и набор BRIDGE праймеров для введения секвенсовых адаптеров.
| KINDE I | |||
| et al, "Detection and quantification of rare mutations with massively parallel sequencing", Proc Natl Acad Sci, 2011, v | |||
| Приспособление для останова мюля Dobson аnd Barlow при отработке съема | 1919 |
|
SU108A1 |
| Прибор для равномерного смешения зерна и одновременного отбирания нескольких одинаковых по объему проб | 1921 |
|
SU23A1 |
| Пюпитр для пишущей машины | 1927 |
|
SU9530A1 |
| SCHMITT M., "Detection of ultra-rare mutations by next-generation sequencing", PNAS, 2012, v | |||
| Шкив для канатной передачи | 1920 |
|
SU109A1 |
| Коридорная многокамерная вагонеточная углевыжигательная печь | 1921 |
|
SU36A1 |
| Аппарат для автоматической препарировки сосудов и нервов | 1927 |
|
SU14508A1 |
| WO 2013096802 A1, 27.06.2013 | |||
| WO 2011156529 A2, 15.12.2011 | |||
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ ПОПУЛЯЦИЙ ДРЕВЕСНЫХ ВИДОВ РАСТЕНИЙ | 2012 |
|
RU2505956C2 |
| . | |||
Авторы
Даты
2017-03-16—Публикация
2014-09-17—Подача