Изобретение относится к молекулярной биологии, вирусологии и медицинской биотехнологии и предназначено для экспресс-диагностики ортопоксвирусных инфекций человека и животных на основе мультиплексной ПЦР в реальном времени.
Отказ от вакцинации против натуральной оспы постепенно привел к формированию большой прослойки людей, чувствительных как к вирусу натуральной оспы (VARV), так и другим ортопоксвирусам. Многочисленные сообщения о случаях поражения человека данными вирусами - вспышки заболевания оспой обезьян (MPXV) были зарегистрированы в 2003 г. в США [36], в период 2001-2004 гг. в Демократической Республике Конго [29], в октябре 2005 г. в Судане [15], беспрецедентная для вируса оспы коров (CPXV) вспышка этого заболевания в декабре 2008 - январе 2009 во Франции и Германии [13, 25] - свидетельствуют о все более широком распространении в человеческой популяции ранее относительно неопасных инфекций типа оспы обезьян и оспы коров. Более того, все чаще VARV рассматривают как возможный агент биотеррористических атак [11, 35]. В связи со всем вышесказанным очевидна необходимость в эффективных, высокочувствительных и высокоспецифичных методах быстрой диагностики и видовой идентификации ортопоксвирусов, патогенных для человека.
Классическими методами идентификации ортопоксвирусов являются определение морфологии оспин на хорионаллантоисной оболочке (ХАО) развивающихся куриных эмбрионов (РКЭ) или бляшек на культурах клеток и определение предельной температуры размножения вирусов в этих биологических системах, оценка гемагглютинирующей активности вирусов (как дополнительный тест) [2, 3]. Эти тесты занимают продолжительное время (от 3 до 6 суток) и сопряжены с необходимостью работы с опасными вирусами. Экспресс-диагностика ортопоксвирусов возможна электронномикроскопической визуализацией вирионов, а также при использовании ряда серологических методов. Эти процедуры обычно занимают несколько часов, но они позволяют идентифицировать, как правило, только родовую принадлежность вирусов и их чувствительность не всегда достаточна при анализе клинических образцов [3]. Было показано [17, 23], что рестрикционным анализом вирусных ДНК можно с достаточно высокой надежностью осуществлять видовую идентификацию ортопоксвирусов. Однако для этого необходимо нарабатывать препарат вируса и проводить его очистку, что требует специально оборудованных помещений для работы с особо опасными вирусами и занимает много времени.
Появление метода амплификации фрагментов ДНК в полимеразной цепной реакции (ПЦР) [11] создало предпосылки к разработке вариантов процедур экспресс-идентификации ортопоксвирусов. В настоящее время существуют различные подходы к использованию ПЦР для диагностики ортопоксвирусных инфекций. Наиболее простой вариант - специфичная ПЦР - предусматривает использование праймеров, комплементарных специфической последовательности ДНК, характерной для строго определенного вида ортопоксвируса. Факт наличия четырех патогенных для человека ортопоксвирусов (VARV, MPXV, CPXV и вируса осповакцины - VACV), имеющих различную патогенность и эпидемическую значимость, указывает на целесообразность применения методов индикации с использованием ПЦР, позволяющих одновременно обнаружить и охарактеризовать видовую принадлежность вируса. Для решения этой задачи был разработан одностадийный экспресс-метод идентификации четырех видов ортопоксвирусов, патогенных для человека, на основе мультиплексной ПЦР [1, 33, 4], базирующийся на анализе полиморфизма длины рестрикционных фрагментов (ПДРФ-анализ). Разработанный метод является чувствительным и специфичным, позволяет выявлять и дифференцировать в одной пробе такие патогенные для человека ортопоксвирусы, как вирусы натуральной оспы, оспы обезьян, оспы коров и осповакцины. Аналогичный метод был предложен в Czub с соавторами [7]. Однако методы, базирующиеся на ПДРФ-анализе, достаточно трудоемки и требует навыков интерпретации результатов МПЦР.
Для диагностики патогенных для человека ортопоксвирусов также предложены методы на основе технологии микрочипов [5, 6]. Однако несмотря на все преимущества технологии микрочипов она не получила широкого распространения в практической диагностике.
В настоящее время в практическое здравоохранение внедряется новая технология ПЦР - ПЦР в реальном времени. Важным преимуществом метода по сравнению со стандартной ПЦР является скорость проведения анализа, что достигается в основном исключением суммы процедур, обычно используемых для детекции продуктов амплификации (электрофорез и визуализация), что кроме всего прочего значительно уменьшает риск контаминации и, как следствие, число ложноположительных результатов.
На основе этого метода были разработаны различные модификации методов для детекции ДНК ортопоксвирусов [14, 20, 21, 22, 26, 27, 28].
Наиболее близким аналогом (прототипом) является система ПЦР в реальном времени для обнаружения ортопоксвирусов и одновременной идентификации вируса натуральной оспы [27].
Однако до сих пор с помощью мультиплексной ПЦР в реальном времени не было предложено ни одного технического решения, позволяющего одновременно выявлять и дифференцировать между собой все патогенные для человека виды ортопоксвирусов (VARV, MPXV, CPXV и VACV). Каждая описанная методика позволяет детектировать ДНК ортопоксвирусов и дифференцировать только один из указанных выше видов вирусов. Разработка метода, основанного на мультиплексной ПЦР в реальном времени, даст удобный, точный, быстрый способ обнаружения и дифференциации.
Техническим результатом предлагаемого изобретения является создание средств для проведения одностадийной экспресс-идентификации нескольких патогенных для человека ортопоксвирусов (VARV, MPXV, CPXV и VACV) с помощью мультиплексной ПЦР в реальном времени.
Указанный технический результат достигается тем, что разработан набор видоспецифичных олигонуклеотидных праймеров и соответствующих флуоресцентномеченых зондов, для рутинной идентификации четырех видов ортопоксвирусов, патогенных для человека, при проведении мультиплексной ПЦР в реальном времени:

Данный подход позволит усовершенствовать (адаптировать) технику ПЦР в реальном времени для рутинного использования в диагностике ортопоксвирусных инфекций за счет применения только одной мультиплексной ПЦР (МПЦР) анализа в реальном времени. МПЦР в реальном времени позволит выявлять и дифференцировать вирусный материал четырех патогенных для человека ортопоксвирусов в одной реакционной смеси, не прибегая к использованию дополнительных анализов, тем самым сократив время диагностики, размер финансовых затрат и процент технологических ошибок.
Выбор праймеров ограничивается несколькими критериями, такими как - длина амплификационного продукта не более 250 п.н. и наличие в последовательности продукта видоспецифичных отличий (делеций, вставок, замен) [8].


Предполагаемые пары праймеров и соответствующие зонды в дальнейшем анализировали, используя программу «Oligo» (версия 3.3) [12, 18].
Затем последовательности олигонуклеотидов, рассчитанные для каждого вида ортопоксвирусов, были проверены на наличие гомологии/комплементарности между собой и с последовательностями других поксвирусов, взятых в GenBank. Отсутствие гомологии с другими видами ортопоксвирусов и родами поксвирусов являлось одним из критериев отбора олигонуклеотидов.
Таким образом, для каждого вида ортопоксвирусов была подобрана пара праймеров и соответствующий флуоресцентномеченый зонд, обладающий видовой специфичностью. Последовательности праймеров и соответствующие им расчетные длины амплификационных продуктов представлены в Таблице 1.
Изобретение иллюстрируется следующими графическими материалами.
Фиг.1. Схема организации генома ортопоксвирусов. TIR-обращенные (инвертированные) концевые повторы. Terminal harping loop - обращенная концевая шпилька-последовательность около 100 не полностью спаренных нуклеотидов [2].
Фиг.2. Графическое выравнивание левых (А) и правых (В) концевых видоспецифичных районов генома вирусов оспы коров CPXV-GRI, осповакцины VACV-COP, натуральной оспы VARV-IND и оспы обезьян MPXV-ZAI. Стрелками обозначены направление и размер ОРТ, внутри которых проводили видоспецифичную ПЦР в реальном времени. Над стрелками приведены названия соответствующих ОРТ. Тонкими линиями обозначены районы делеций в ДНК одних вирусов относительно других. Окружностями обозначены концевые шпильки вирусных ДНК.
Фиг.3. Сравнение нуклеотидной последовательности фрагмента ОРТ A38R VARV, штамм India-1967 (подвид variola major) [30] (VAR-IND), с соответствующими районами генома VARV, штаммы Bangladesh-1975 [24] (VAR-BSH), Garcia-1966 [32] (VAR-GAR); MPXV, штаммы: Zaire-96-I-16 [35] (MPV-ZAI), USA_2003_044 [EMBL: DQ011153] (MPV-USA); CPXV, штаммы: Brighton [EMBL: CV32634] (CPV-BRI), GRI-90 [31] (CPV-GRI); VACV, штаммы: Copenhagen [EMBL: M35027] (VAC-COP), Tian-tan [EMBL: AF 095689] (VAC-TT), Ankara [EMBL: U9488] (VAC-ANK); вирус оспы верблюдов, штаммы: M96 [EMBL: AF438165] (CmPV-M96), CMS [EMBL: AY009089] (CmPV-CMS); вирус эктромелии, штамм Moscow (ECT-MOS). Идентичные нуклеотиды в сравниваемых последовательностях вирусного генома по отношению к последовательности ОРТ A38R VARV-IND обозначены точками, делеции - прочерком. Цифры слева и справа от нуклеотидных последовательностей обозначают положение нуклеотидов в анализируемом сегменте ДНК. Полужирным курсивом приведены последовательности олигонуклеотидных праймеров VARV_A38R_upper и VARV_A38R_lower и видоспецифичного для вируса натуральной оспы зонда VARV_A38R_probe.
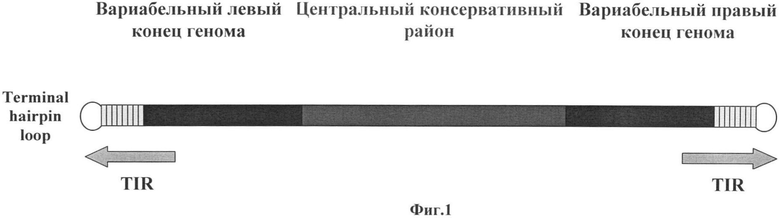
Фиг.4. Графики зависимости флуоресценции от количества циклов в ПЦР в реальном времени. Данные получены на приборе Real-Time PCR System 7500 (“Applied Biosystems”, США) с помощью олигонуклеотидных праймеров (VARV_A38R_upper, VARV_A38R_lower), рассчитанных для района ОРТ A38R VARV Ind, и зонда, специфичного для VARV (VARV_A38R_probe). Анализировали амплификаты, полученные на ДНК 25 штаммов ортопоксвирусов, представленных в Таблице 2. Регистрируется сигнал красителя FAM, конъюгированного с VARV специфичным зондом. Кривые (I) соответствуют положительному результату и отражают уровень накопления продукта в ПЦР реакции для ДНК штаммов VARV, II - для всех остальных видов OPV (см. Таблицу 2) и NTC. Cycle Number - номер цикла. Rn - значение сигнала флюоресценции.
Фиг.5. Сравнение нуклеотидной последовательности фрагмента ОРТ B7R MPXV, штамм Zaire-96-I-16 [34] (MPV-ZAI), с соответствующим районом генома MPXV, штамм USA_2003_044 [EMBL: DQ011153] (MPV-USA); VARV, штаммы: India-1967 (подвид variola major) [30] (VAR-IND), Bangladesh-1975 [25] (VAR-BSH), Garcia-1966 [32] (VAR-GAR); CPXV, штаммы: Brighton [EMBL: CV32634] (CPV-BRI), GRI-90 [31] (CPV-GRI); VACV, штаммы: Copenhagen [EMBL: M35027] (VAC-COP), Tian-tan [EMBL: AF 095689] (VAC-TT), Ankara [EMBL: U9488] (VAC-ANK); вирус оспы верблюдов, штаммы: M96 [EMBL: AF438165] (CmPV-M96), CMS [EMBL: AY009089] (CmPV-CMS); вирус эктромелии, штамм Moscow (ECT-MOS). Идентичные нуклеотиды в сравниваемых последовательностях вирусного генома по отношению к последовательности ОРТ B7R MPXV-ZAI обозначены точками, делеции - прочерком. Цифры слева и справа от нуклеотидных последовательностей обозначают положение нуклеотидов в анализируемом сегменте ДНК. Полужирным курсивом приведены последовательности олигонуклеотидных праймеров MPXV_B7R_upper и MPXV_B7R_lower и видоспецифичного для вируса оспы обезьян зонда MPXV_B7R_probe.
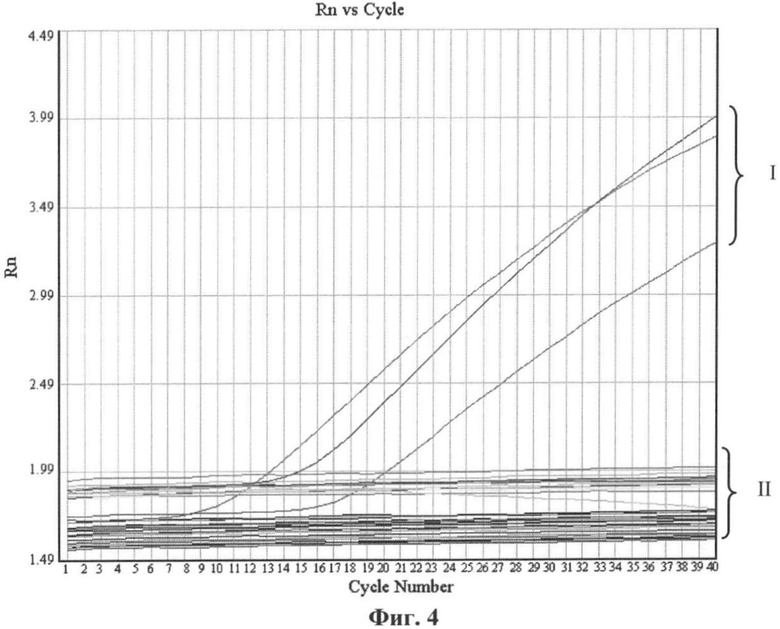
Фиг.6. Графики зависимости флуоресценции от количества циклов в ПЦР в реальном времени. Данные получены на приборе Real-Time PCR System 7500 (“Applied Biosystems”, США) с помощью олигонуклеотидных праймеров (MPXV_B7R_upper, MPXV_B7R_lower), рассчитанных для района ОРТ B7R MPXV-ZAI, и зонда, специфичного для MPXV (MPXV_B7R_probe). Анализировали амплификаты, полученные на ДНК 25 штаммов ортопоксвирусов, представленных в Таблице 2. Регистрируется сигнал красителя TAMRA, конъюгированного с MPXV специфичным зондом. Кривые (I) соответствуют положительному результату и отражают уровень накопления продукта в ПЦР реакции для ДНК штаммов MPXV, II - для всех остальных видов OPV (см. Таблицу 2) и NTC. Cycle Number - номер цикла. Rn - значение сигнала флюоресценции.
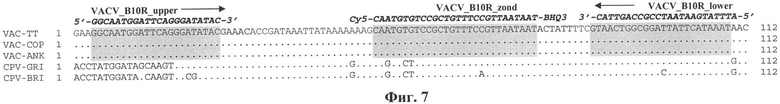
Фиг.7. Сравнение нуклеотидной последовательности фрагмента ОРТ B10R VACV, штамм Tian-tan [EMBL: AF 095689] (VAC-TT), с соответствующим районом генома VACV, штаммы Copenhagen [EMBL: M35027] (VAC-COP) и Ankara [EMBL: U9488] (VAC-ANK); CPXV, штаммы: Brighton [EMBL: CV32634] (CPV-BRI), GRI-90 [31] (CPV-GRI). Идентичные нуклеотиды в сравниваемых последовательностях вирусного генома по отношению к последовательности ОРТ B10R VACV-TT обозначены точками, делеции - прочерком. Цифры слева и справа от нуклеотидных последовательностей обозначают положение нуклеотидов в анализируемом сегменте ДНК. У других представителей рода ортопоксвирусов в данном районе наблюдается делеция. Полужирным курсивом приведены последовательности олигонуклеотидных праймеров VACV_B10R_upper и VACV_B10R_lower и видоспецифичного для вируса оспы обезьян зонда VACV_B10R_probe.
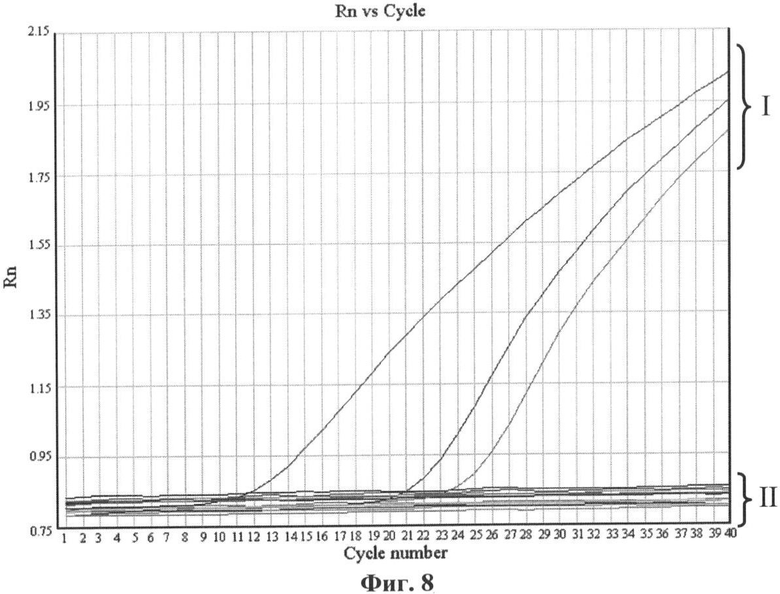
Фиг.8. Графики зависимости флуоресценции от количества циклов в ПЦР в реальном времени. Данные получены на приборе Real-Time PCR System 7500 (“Applied Biosystems”, США) с помощью олигонуклеотидных праймеров (VACV_B10R_upper, VACV_B10R_lower), рассчитанных для района ОРТ B10R VACV-COP, и зонда, специфичного для VACV (VACV_B10R_probe). Анализировали амплификаты, полученные на ДНК 25 штаммов ортопоксвирусов, представленных в Таблице 2. Регистрируется сигнал красителя Cy5, конъюгированного с VACV специфичным зондом. Кривые (I) соответствуют положительному результату и отражают уровень накопления продукта в ПЦР реакции для ДНК штаммов VACV, II - для всех остальных видов OPV (см. Таблицу 2) и NTC. Cycle Number - номер цикла. Rn - значение сигнала флюоресценции.
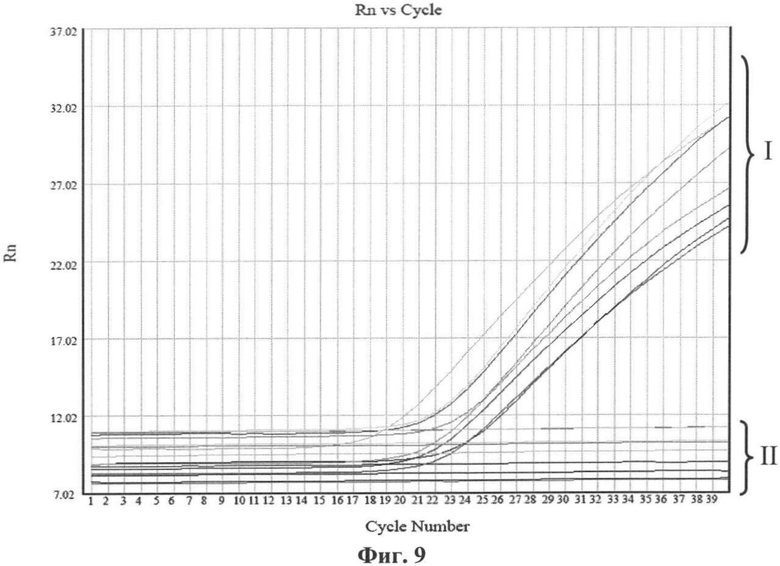
Фиг.9. Графики зависимости флуоресценции от количества циклов в ПЦР в реальном времени. Данные получены на приборе Real-Time PCR System 7500 (“Applied Biosystems”, США) с помощью олигонуклеотидных праймеров (CPXV_D11L_upper, CPXV_D11L_lower), рассчитанных для района ОРТ D11L CPXV GRI-90, и зонда, специфичного для CPXV (CPXV_D11L_probe). Анализировали амплификаты, полученные на ДНК 25 штаммов ортопоксвирусов, представленных в Таблице 2. Регистрируется сигнал красителя JOE, конъюгированного с CPXV специфичным зондом. Кривые (I) соответствуют положительному результату и отражают уровень накопления продукта в ПЦР реакции для ДНК штаммов CPXV, II - для всех остальных видов OPV (см. Таблицу 2) и NTC. Cycle Number - номер цикла. Rn - значение сигнала флюоресценции.
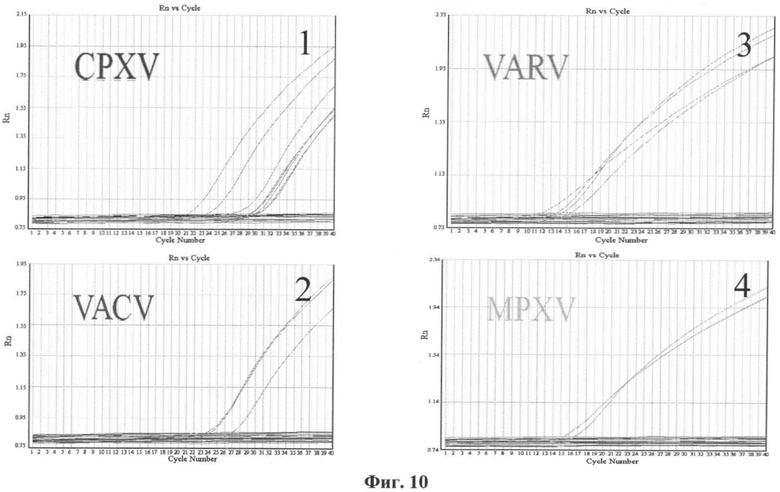
Фиг.10. Графики зависимости флуоресценции от количества циклов в ПЦР в реальном времени. Данные получены на приборе Real-Time PCR System 7500 (“Applied Biosystems”, США) с помощью смеси олигонуклеотидных праймеров (VACV_B10R_upper, VACV_B10R_lower, MPXV_B7R_upper, MPXV_B7R_lower, VARV_A38R_upper, VARV_A38R_lower, CPXV_D11L_upper, CPXV_D11L_lower) и смеси видоспецифичных олигонуклеотидных флуоресцентномеченых зондов (VACV_B10R_probe, MPXV_B7R_probe, VARV_A38R_probe, CPXV_D11L_probe). Результаты амплификации, обозначенные цифрой 1, получены с помощью олигонуклеотидных праймеров (CPXV_D11L_upper, CPXV_D11L_lower), рассчитанных для района ОРТ D11L CPXV GRI-90, и зонда, специфичного для CPXV (CPXV_D11L_probe), регистрируется сигнал красителя JOE, конъюгированного с CPXV специфичным зондом. Положительный сигнал наблюдается только для ДНК CPXV. Результаты амплификации, обозначенные цифрой 2, получены с помощью олигонуклеотидных праймеров (VACV_B10R_upper, VACV_B10R_lower), рассчитанных для района ОРТ B10R VACV-COP, и зонда, специфичного для VACV (VACV_B10R_probe), регистрируется сигнал красителя Cy5, конъюгированного с VACV специфичным зондом. Положительный сигнал наблюдается только для ДНК VACV. Результаты амплификации, обозначенные цифрой 3, получены с помощью олигонуклеотидных праймеров (VARV_A38R_upper, VARV_A38R_lower), рассчитанных для района ОРТ A38R VARV Ind, и зонда, специфичного для VARV (VARV_A38R_probe), регистрируется сигнал красителя FAM, конъюгированного с VARV специфичным зондом. Положительный сигнал наблюдается только для ДНК VARV. Результаты амплификации, обозначенные цифрой 4, получены с помощью олигонуклеотидных праймеров (MPXV_B7R_upper, MPXV_B7R_lower), рассчитанных для района ОРТ B7R MPXV Zai, и зонда, специфичного для MPXV (MPXV_B7R_probe), регистрируется сигнал красителя TAMRA, конъюгированного с MPXV специфичным зондом. Положительный сигнал наблюдается только для ДНК вируса оспы обезьян. Cycle Number - номер цикла. Rn - значение сигнала флуоресценции.
Видоспецифичные различия ортопоксвирусов определяются концевыми вариабельными районами вирусного генома, тогда как центральная часть является высоко консервативной по нуклеотидной последовательности (фиг.1). Поэтому именно в концевых областях и удалось обнаружить потенциальные участки для видоспецифичной амплификации сегментов вирусных ДНК (фиг.2).
Так, для VACV районом для проведения ПЦР была выбрана область открытой рамки трансляции (ОРТ) B10R (по номенклатуре генов вируса осповакцины штамма Copenhagen) на правом вариабельном конце генома ортопоксвирусов. Для MPXV - район ОРТ B7R (по номенклатуре генов вируса оспы обезьян штамма Zaire-96-I-16). Для CPXV - район ОРТ D11L (по номенклатуре генов вируса оспы коров штамма GRI-90) на левом вариабельном конце генома ортопоксвирусов. В случае VARV, был выбран район ОРТ A38R (по номенклатуре генов вируса натуральной оспы штамма India). Область находится в консервативном центральном районе ортопоксвирусного генома и имеет несколько коротких делеций и единичные нуклеотидные замены. Данный факт позволил рассчитать видоспецифичный зонд к VARV (фиг.3). За счет наличия у других видов ортопоксвирусов небольшой вставки в районе выбранных праймеров, а также единичных замен, при проведении ПЦР в реальном времени флуоресцентный сигнал детектируется только для ДНК VARV (фиг.4).
На фиг.5 приведен участок выравнивания нуклеотидной последовательности патогенных для человека ортопоксвирусов со схематическим изображением мест гибридизации праймеров и зонда, подобранных для видоспецифичного выявления MPXV, а на фиг.6 - результаты проведения ПЦР в реальном времени с подобранной парой праймеров и зондом.
В случае VACV выбран район ОРТ B10R, среди патогенных для человека ортопоксвирусов аналогичную последовательность имеет только CPXV. Как видно на фиг.7, область гибридизации зонда VACV_B10R_probe имеет три нуклеотидные замены относительно последовательности ДНК CPXV и при амплификации с рассчитанным видоспецифичным для VACV олигонуклеотидным зондом флуоресцентный сигнал наблюдается только для ДНК VACV (фиг.8).
Район рамки D11L (по номенклатуре генов CPXV GRI-90), выбранный в качестве мишени для олигонуклеотидных праймеров - CPXV_D11L_upper, CPXV_D11L_lower и зонда CPXV_D11L _probe, является уникальным. В своем составе эту последовательность содержит только CPXV, у других патогенных для человека ортопоксвирусов этой нуклеотидной последовательности нет. Результаты проведения ПЦР в реальном времени с этой парой праймеров и зондом представлены на фиг.9.
Рассчитанные олигонуклеотиды были синтезированы на автоматическом синтезаторе ABI 3400 DNA/RNA synthesizer (Applied Biosystems) с использованием стандартной фосфоамидитной процедуры.
Тестирование разработанного набора праймеров и зондов (Таблица 1) проводили на полноразмерной ДНК 22 штаммов различных поксвирусов (Таблица 2). Для этого в МПЦР в реальном времени использовали одновременно все четыре пары праймеров с соответствующими видоспецифичными флуоресцентными зондами (Таблица 1). В состав видоспецифичных зондов входят флуорофоры с неперекрывающимися спектрами излучения, что позволило одновременно определить принадлежность образцов к одному из видов ортопоксвирусов, патогенных для человека: на канале FAM к VARV, на канале TAMRA к MPXV, на канале JOE к CPXV, на канале Cy5 к VACV (фиг.1). Каждый из перечисленных штаммов в Таблице 2 проверяли не менее чем в трех независимых экспериментах для каждой смеси, результаты МПЦР в реальном времени показали высокую специфичность определения.
Для лучшего понимания сущности изобретения ниже следуют примеры его осуществления.
Пример 1. Методика получения набора олигонуклеотидов для дифференциальной диагностики ортопоксвирусов
На основе теоретического изучения доступных нуклеотидных последовательностей ДНК различных штаммов CPXV, VACV, MPXV, VARV были синтезированы праймеры и флуоресцентномеченые олигонуклеотидные зонды для проведения мультиплексной ПЦР в режиме реального времени (Таблица 1).
Отработку условий амплификации проводили при использовании ДНК 22 штаммов различных видов поксвирусов (Таблица 2). В качестве негативных контролей были использованы ДНК вируса миксомы кролика, штамм Lausanne (род Leporipoxvirus), вируса оспы кур, штамм FP9 (род Avipoxvirus), и ДНК таких возбудителей, как вирусы простого герпеса 1-го и 2-го типа, вирусы ветряной оспы (Таблица 2). При анализе материала в МПЦР в реальном времени во всех случаях была обнаружена ДНК ортопоксвирусов, каких-либо посторонних ПЦР-амплификатов выявлено не было. Различные штаммы VARV, MPXV, CPXV и VACV были успешно идентифицированы. Все результаты амплификации соответствовали ожидаемым.
1. Вирусные ДНК выделены из штаммов из коллекции ФГУН ГНЦ ВБ “Вектор”.
2. Вирусные ДНК любезно предоставлены Г.Мейер (Германия).
3. Вирусные ДНК любезно предоставлены Дж.Эспозито (США).
4. Вирусные ДНК любезно предоставлены Г.МакФадден (Канада).
5. Вирусные ДНК любезно предоставлены М.Скиннер (Англия).
6. Вирусные ДНК любезно предоставлены М.А. Суслопаровым, ФГУН ГНЦ ВБ “Вектор”, Кольцово, Россия.
Пример 2. Постановка ПЦР в реальном времени
Состав реакционной смеси
Условия проведения реакции
ПЦР с измерением интенсивности флуоресценции на приборе Real-Time PCR System 7500 (“Applied Biosystems”, США) проводили по следующему алгоритму:
* Измерение флуоресценции проводят при 58°С на каналах FAM, JOE, TAMRA и Cy5.
Пример 3. Анализ результатов
Анализируют результаты амплификации следующих участков ДНК ортопоксвирусов: ОРТ B10R для VACV, OPT D11L для CPXV, OPT A38R для VARV и OPT B7R для MPXV. Накопление продукта амплификации участков ДНК VARV детектируется по каналу детекции флуорофора FAM, накопление продукта амплификации MPXV - по каналу для детекции флуорофора TAMRA, накопление продукта амплификации CPXV - по каналу для детекции флуорофора JOE, накопление продукта амплификации VACV - по каналу для детекции флуорофора Cy5. Рекомендуется результаты, полученные на приборе Real-Time PCR System 7500 (“Applied Biosystems”, США), анализировать при помощи программного обеспечения прибора Real-Time PCR System 7500.
Пример 4. Проведение МПЦР в реальном времени с образцами, содержащими ДНК вируса оспы коров
1. Две группы мышей линии BALB/c, самцов, весом от 18 до 22 г, возраст 5-6 недель, были инфицированы интраназально разными дозами вируса оспы коров (штамм GRI-90): первая - 103 БОЕ/мышь, вторая - 105 БОЕ/мышь. Объем инокулята составил 30 мкл. Контрольной группе вводили аналогичный объем физиологического раствора, на котором готовили разведения вирусной суспензии.
Образцы крови и органов были получены на третьи сутки после заражения. Кровь получали из ретроорбитального синуса мышей, после чего животных умерщвляли путем декомпозиции шейных позвонков и асептически получали образцы внутренних органов мышей: легких, печени, селезенки.
2. Легкие, селезенка или печень гомогенизируют в растворе, содержащем 200 мкл лизирующего буфера (100 мМ Tris-HCl (pH 8.0), 100 мМ ЭДТА, 100 мМ NaCl, 1% SDS) и 20 мкл раствора протеиназы К (10 мг/мл).
3. Инкубируют 10 мин при 56°С.
4. Центрифугируют 7 мин при 18000 g.
5. К супернатанту добавляют 400 мкл смеси фенол/хлороформ (1:1).
6. Перемешивают на Vortex в течение 1 мин, затем центрифугируют материал при 450 g 1 мин.
7. Водную фазу (верхнюю) отбирают в новые пробирки, затем остатки фенола экстрагируют изоамиловым спиртом.
8. Добавляют 1/10 по объему 3 М раствор ацетата натрия (pH 5.5) и два объема этанола (96%).
9. Центрифугируют с последующим удалением водной фазы.
10. Пробирку переворачивают вверх дном и высушивают осадок при комнатной температуре в течение 20 минут.
11. Осадок ДНК растворяют в 15 мкл воды.
12. Со всеми образцами ДНК была проведена МПЦР в реальном времени. Все образцы оказались положительными, для всех наблюдались сигналы в МПЦР в реальном времени.
Таким образом, впервые разработан набор, позволяющий в одну стадию мультиплексной полимеразной цепной реакции детектировать и дифференцировать все четыре вида ортопоксвирусов, патогенных для человека: вирусы натуральной оспы, оспы обезьян, оспы коров и осповакцины в реальном времени. Основными преимуществами заявляемого набора являются:
- высокая надежность метода, благодаря избирательному выявлению уникальных последовательностей четырех патогенных для человека ортопоксвирусов;
- быстрота проведения анализа, благодаря исключению ряда процедур, необходимых для регистрации продуктов амплификации в классической ПЦР;
- простота выполнения методики, исключающая необходимость привлечения высококвалифицированного персонала;
- относительная дешевизна метода, благодаря возможности индикации и идентификации 4 патогенных для человека ортопоксвирусов одновременно.








Изобретение относится к молекулярной биологии, вирусологии и медицинской биотехнологии. Набор содержит видоспецифичные олигонуклеотидные пары праймеров и соответствующие флуоресцентномеченые зонды для проведения одностадийной экспресс-идентификации нескольких патогенных для человека ортопоксвирусов (VARV, MPXV, CPXV и VACV) с помощью мультиплексной ПЦР в реальном времени. Изобретение предназначено для экспресс-диагностики ортопоксвирусных инфекций человека и животных на основе мультиплексной ПЦР в реальном времени. 10 ил., 2 табл.
Набор олигонуклеотидных праймеров и флуоресцентномеченых зондов для видоспецифичной экспресс-идентификации ортопоксвирусов на основе мультиплексной ПЦР в реальном времени, включающий:
последовательность, видоспецифичную для вируса осповакцины
5'→3' 5' ggcaatggattcagggatatac 3'
3'←5' 5' atttatgaataatccgccagttac 3'
Cy5-5' caatgtgtccgctgtttccgttaataat 3'-BHQ3;
последовательность, видоспецифичную для вируса оспы коров
5'→3' 5' aaaactctccactttccatcttct 3'
3'←5' 5' gcattcagatacggatactgattc 3'
JOE-5' ccacaatcaggatctgtaaagcgagc 3'-BHQ1;
последовательность, видоспецифичную для вируса натуральной оспы
5'→3' 5' tctgtactatgtgttaaaagattctacaa 3'
3'←5' 5' aatgtatctgttatagtcagcataccc 3'
Cy5-5' cgttgatggacaccacgtttgttatta 3'-BHQ3;
последовательность, видоспецифичную для вируса оспы обезьян
5'→3' 5' acgtgttaaacaatgggtgatg 3'
3'←5' 5' aacatttccatgaatcgtagtcc 3'
TAMRA-5' tgaatgaatgcgatactgtatgtgtggg 3'-BHQ2.
| OLSON V.A | |||
| et al., "Real-time PCR system for detection of orthopoxviruses and simultaneous identification of smallpox virus", J Clin Microbiol | |||
| Способ приготовления мыла | 1923 |
|
SU2004A1 |
| US 7491493 A1, 08.03.2007 | |||
| НАБОР ОЛИГОНУКЛЕОТИДОВ-ПРАЙМЕРОВ, БИОЧИП И СПОСОБ ВИДОВОЙ ИДЕНТИФИКАЦИИ ОРТОПОКС- И ГЕРПЕСВИРУСОВ, ПАТОГЕННЫХ ДЛЯ ЧЕЛОВЕКА | 2006 |
|
RU2318875C1 |
Авторы
Даты
2011-08-27—Публикация
2010-04-30—Подача