Изобретение относится к молекулярной биологии, генетической инженерии, медицине и предназначено для экспресс-диагностики отопоксвирусов, патогенных для человека и дискриминации их от герпесвирусов, вызывающих сходные клинические проявления. Предлагаемый способ позволяет определять различные типы ортопоксвирусов и основан на проведении одноэтапной полимеразной цепной реакции (ПЦР) с последующей гибридизацией полученных флуоресцентно меченых ампликонов с ДНК-микрочипом, содержащим дискриминирующие ортопокс- и герпесвирус-гибридизационные зонды.
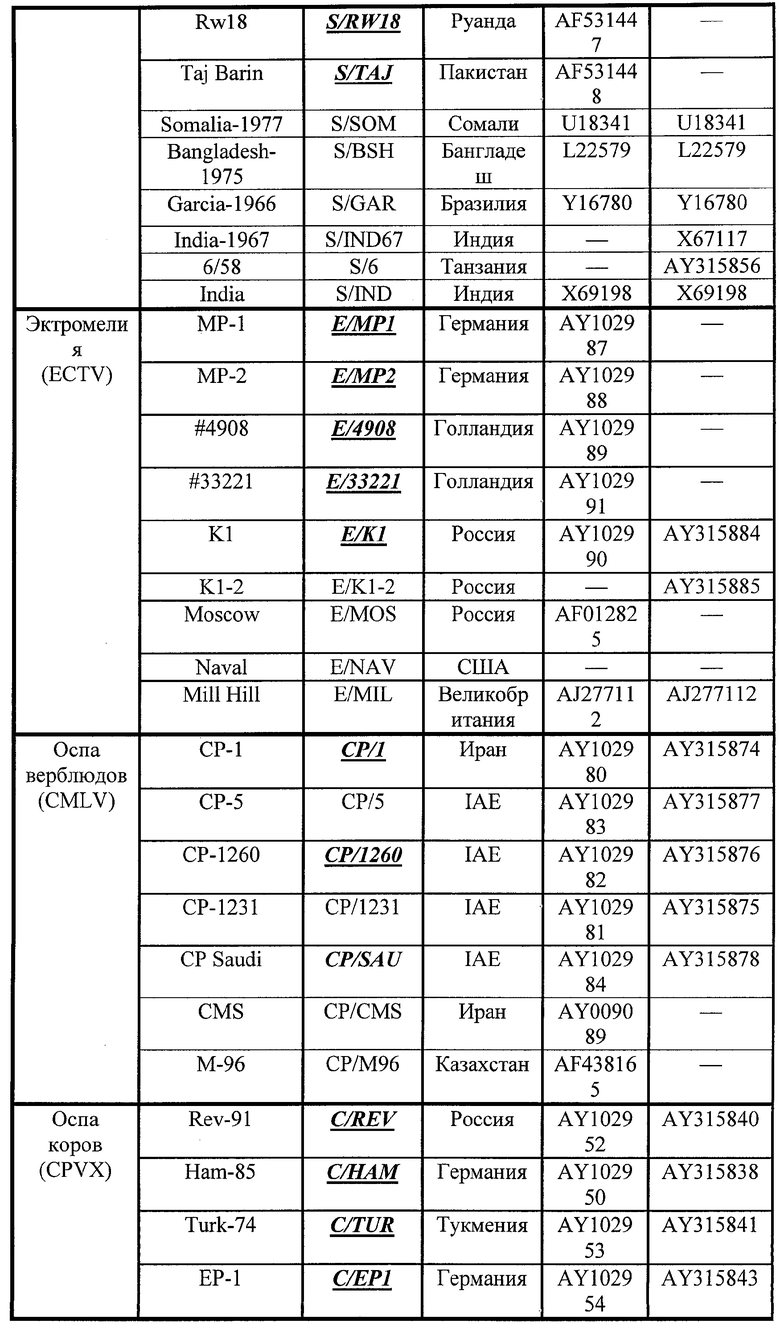
Использование этого способа позволяет определять патогенный для человека тип ортопоквируса и дискриминировать его от герпесвирусов 1, 2 и 3 типов.
Из девяти видов вирусов рода Orthopoxvirus семейства Poxviridae четыре - вирус натуральной оспы (VARV), вирус оспы обезьян (MPXV), вирус оспы коров (CPXV) и вирус осповакцины (VACV) - являются патогенными для человека, вызывая заболевания различной степени тяжести. Натуральная оспа, характеризовавшаяся ранее массовыми эпидемиями с высоким процентом летальных исходов среди заболевших людей в ходе кампании, проведенной Всемирной Организацией здравоохранения (ВОЗ), была ликвидирована к 1977 г. В последующий период массовая вакцинация против натуральной оспы была прекращена, и к настоящему моменту огромная часть населения земного шара оказалась полностью не защищена как от вируса натуральной оспы, так и от других патогенных ортопоксвирусов.
Несмотря на то, что по данным ВОЗ лишь две лаборатории в мире официально владеют образцами вируса натуральной оспы, полностью нет уверенности в отсутствии неучтенных образцов, а высокая вероятность передачи от человека к человеку и большой процент летальных исходов при заражении позволяют рассматривать VARV как потенциальное оружие биотеррористов. Периодические вспышки заболеваний, вызванных MPXV, заставляют разрабатывать и совершенствовать надежные методы лабораторной диагностики ортопоксвирусов.
В настоящее время для видовой идентификации ортопоксвирусов применяется ряд методов, среди которых можно указать следующие.
Классическим способом лабораторной диагностики ортопоксвирусных инфекций является реакция торможения гемагглютинации (РТГА) [1]. Способ обладает низкой чувствительностью. Для получения окончательного ответа требуется повторное взятие крови у больного через 5-6 суток. Следует учесть, что в зависимости от качества материала кожных поражений и срока болезни, отрицательный результат (низкие титры гемагглютинина) не исключает наличия инфекции.
Другим классическим приемом лабораторной диагностики является метод выделения вируса на хорионаллантоисной оболочке развивающихся куриных эмбрионов. С помощью данного подхода возможно осуществлять дифференциацию ортопоксвирусов между собой [2, 3]. Способ обладает высокой чувствительностью, но, несмотря на это, недостатком его является необходимость работы с высокопатогенным вирусным материалом. Кроме того, время исследования составляет от 3 до 6 суток.
Известен способ обнаружения оспенного антигена методом иммунофлуоресценции [4, 5], эффективность которого зависит от вида исследуемого материала. Достоверность результатов исследования материала из пустул и корок с использованием этого способа не велика, поскольку в анализируемых пробах присутствует примесь лейкоцитов, обладающих аутофлуоресценцией.
Применение методов иммунной электронной микроскопии [6, 7] и иммуноферментного анализа для видовой идентификации ортопоксвирусов [5, 8] требует наличия видоспецифичных моноклональных антител для каждого вида ортопоксвирусов. Возможность их применения показана лишь на весьма ограниченном количестве ортопоксвирусов. К тому же, применение таких методов для видовой диагностики ограничено ввиду близкого антигенного родства представителей Orthopoxvirus.
Метод амплификации фрагментов ДНК в полимеразной цепной реакции (ПЦР) позволяет извлекать специфические фрагменты ДНК из следовых количеств анализируемого генетического материала в течение короткого времени. Методы, основанные на ПЦР, с последующим анализом длины амплифицированного фрагмента [9] или с последующим рестрикционным анализом [10] в применении к идентификации ортопоксвирусов позволяют проводить детекцию генетического материала указанных вирусов непосредственно в клинических материалах, для которой не требуется наработки тестируемого вируса, что особенно важно в случае высокопатогенных штаммов.
В настоящее время наиболее перспективным является метод микрочипового анализа с использованием олигонуклеотидных зондов.
Известен ускоренный способ видовой идентификации шести видов (и двух подвидов) вирусов рода Orthopoxvirus [11, 12, прототип]. В качестве мишени для дифференциальной диагностики в данном тесте выбран ген CrmB, кодирующий вирусный аналог клеточного рецептора фактора некроза опухолей. Метод основан на двустадийной ПЦР с получением флуоресцентно меченных красителем TexasRed фрагментов ДНК с последующей гибридизацией на микрочипе, содержащем набор из 14 видоспецифичных олигонуклеотидных зондов. Другими объектами этого же изобретения являются биочип, используемый в способе видовой идентификации ортопоксвирусов, представляющий собой акриламидную гелевую микроматрицу на стеклянной подложке, на которой иммобилизованы типирующие олигонуклеотиды, и собственно набор дискриминирующих олигонуклеотидов, предназначенных для иммобилизации на биочипе и характеризующихся комплементарностью к видам орпопоксвирусов.
Недостатком описанного способа является то, что используемый в этом случае микрочип способен работать с ограниченными по длине амплифицируемыми фрагментами (˜100 нуклеотидов), что вызвано затрудненностью проникновения больших ампликонов в гелевые ячейки чипа. Это значительно ограничивает длину вариабельных участков генома ортопоксвирусов, пригодных для выбора видоспецифичных зондов. Кроме того, этот способ и биочип с набором типирующих олигонуклеотидов не позволяет диагностировать герпесвирусы, вызывающие схожие с ортопоксвирусами клинические проявления.
Известен также способ диагностики [13], основанный на использовании микрочипа, представляющего собой стеклянную подложку с ковалентно присоединенными к ней гибридизационными зондами. Гибридизационные зонды выбраны из последовательности гена B29R, кодирующего вирусный ингибитор СС-хемокинов. Этот способ диагностики также не способен диагностировать герпесвирусы, вызывающие схожие с ортопоксвирусами клинические проявления, и дискриминировать их от последних.
В настоящее время не описаны биочипы с иммобилизованными на них типирующими олигонуклеотидами, обеспечивающими видовую идентификацию ортопоксвирусов, патогенных для человека, и одновременно дискриминацию их от герпесвирусов, вызывающих сходные клинические проявления.
Технической задачей изобретения является создание олигонуклеотидного микрочипа, позволяющего с высокой точностью (специфичностью) определить как наличие ортопоксвируса, осуществить его видовую идентификацию, так и диагностировать наличие герпесвирусов 1, 2 и 3 типов, а также создание способа экспресс-диагностики ортопоксвирусов, патогенных для человека, и дискриминации их от герпесвирусов, вызывающих сходные клинические проявления.
Техническим результатом, достигаемым настоящим изобретением, является определение типа ортопоксвируса либо герпесвируса при проведении одноэтапной ПЦР с последующей гибридизацией флуоресцентно меченых ампликонов с ДНК-микрочипом, содержащим дискриминирующие ортопокс- и герпесвирус-гибридизационные зонды.
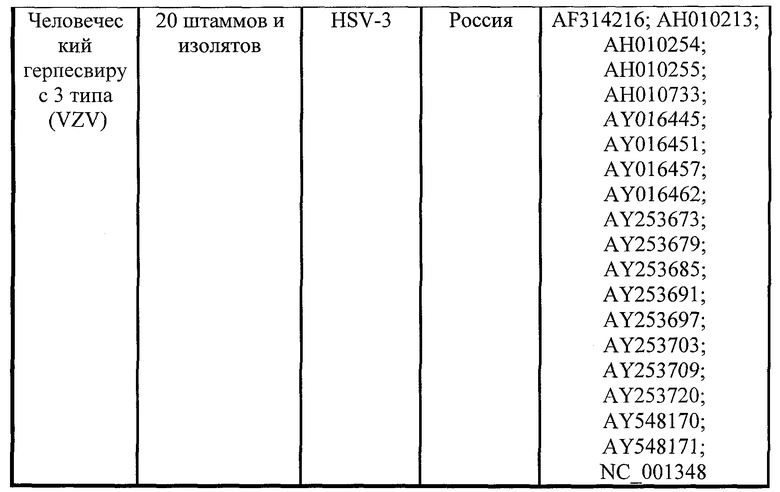
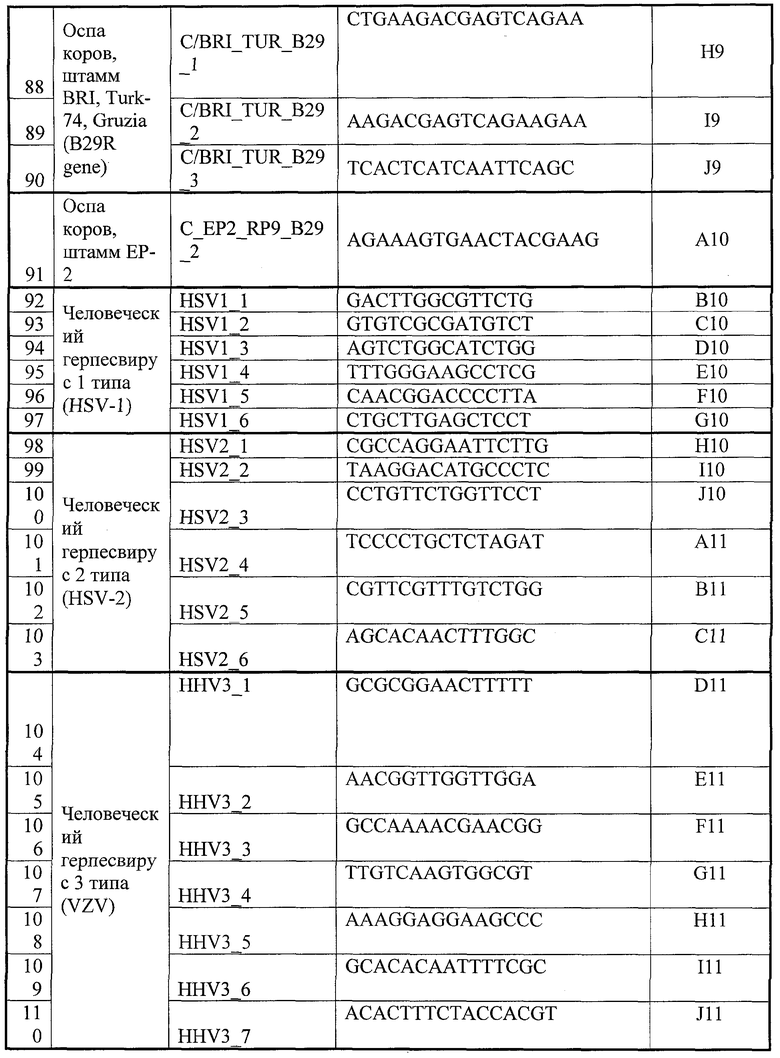
Поставленная задача решается путем дизайна гибридизационных зондов для микрочипа к двум генам-мишеням, кодирующим хемокин (ген B29R)- и α/β-интерферонсвязывающий белок (ген B19R). Дизайн проведен при анализе существенно большего набора ДНК-последовательностей штаммов ортопоксвирусов по сравнению с подходом, описанным в [12, 13], - 86 последовательностей для первого гена и 72 последовательности для второго. Отбор видоспецифичных зондов для HSV-1 выполнен по последовательностям генов US4 и US5, кодирующих гликопротеины «G» (192 штамма и изолята); гена US4 (гликопротеин «G-2») для HSV-2 (17 штаммов и изолятов); гена ORF31 (гликопротеин «B») для VZV (20 штаммов и изолятов).
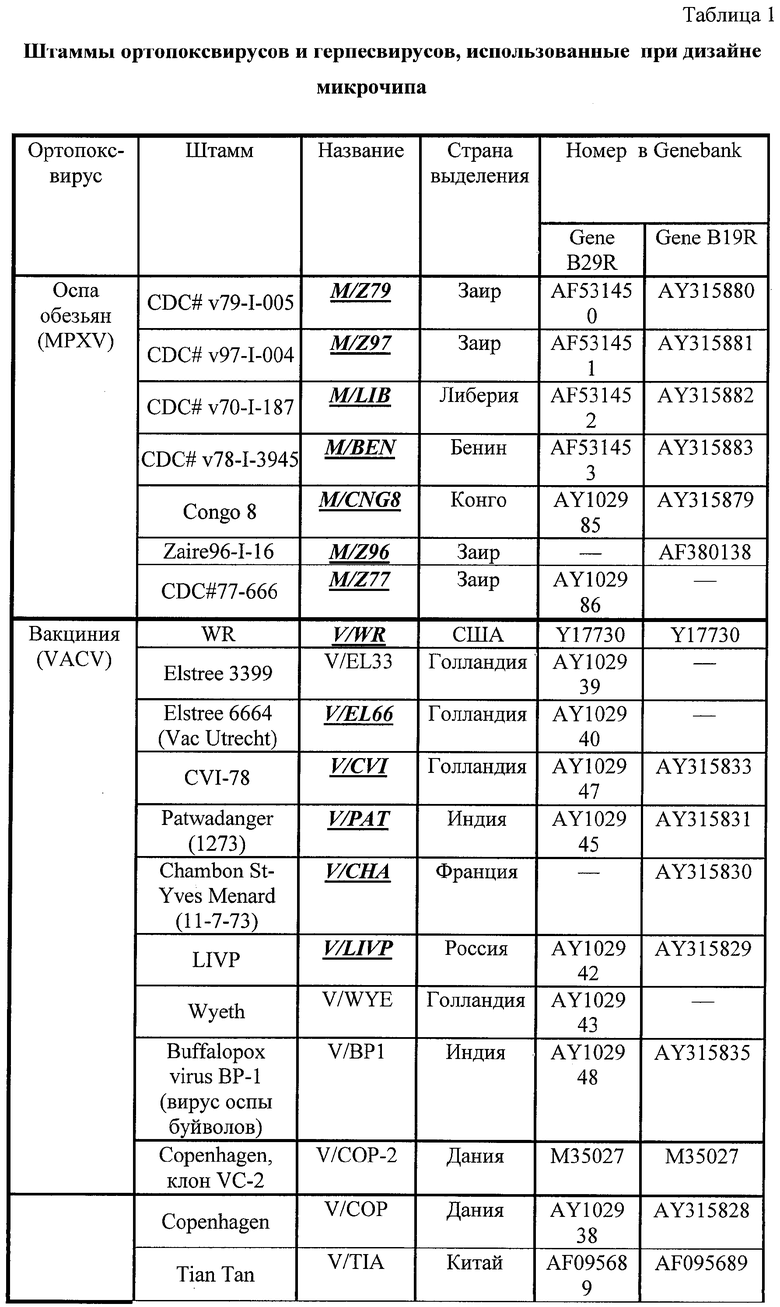
Виды и штаммы ортопоксвирусов, последовательности ДНК которых были использованы при дизайне микрочипа, приведены в Таблице 1. Выполненный филогенетический анализ генов B29R и B19R обнаружил весьма хорошую видовую кластеризацию проанализированных ортопоксвирусов и указал на возможность нахождения нуклеотидных последовательностей, пригодных для их видовой дискриминации.
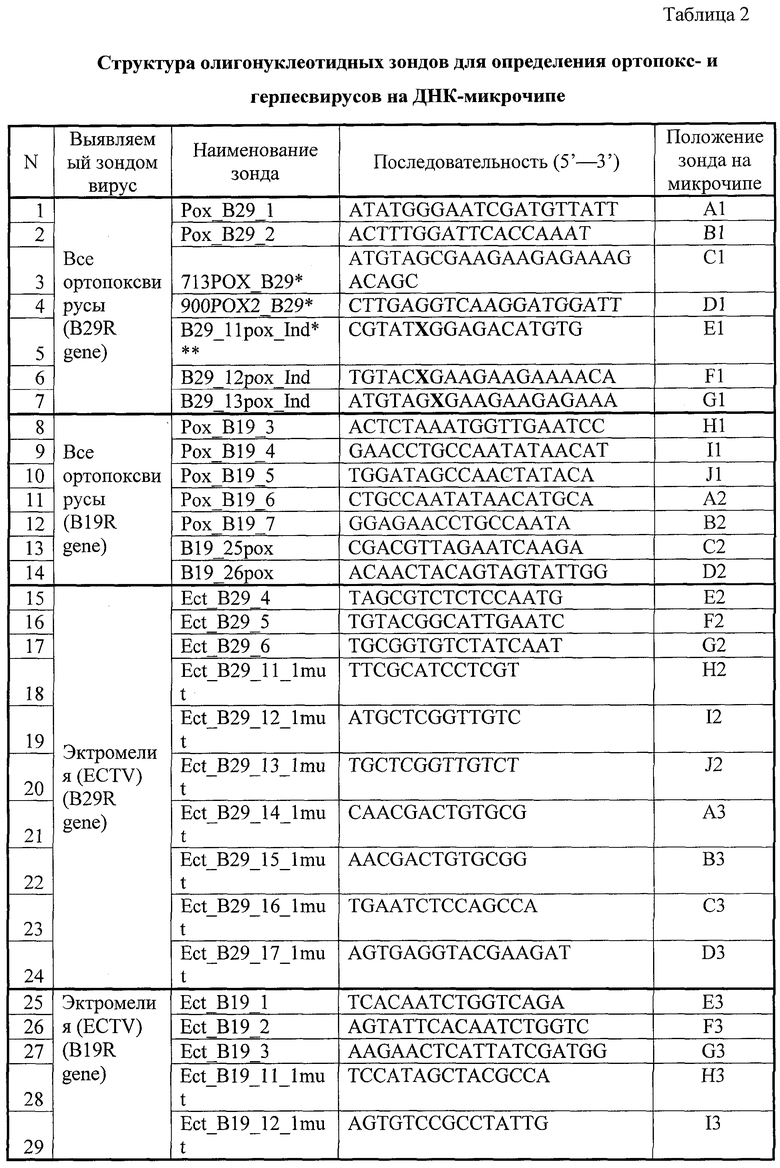
Анализ последовательностей ДНК штаммов ортопоксвирусов, представленных в Таблице 1, показал, что число родоспецифичных зондов (общих для всех рассмотренных штаммов данного вида) ограничено, но они существуют для обоих генов (см. Табл.2).
Для определения видовой принадлежности вируса оспы отбирались два типа зондов. Отбор олигонуклеотидных зондов выполнен по специально разработанной авторами компьютерной программе, согласно которой первый тип олигонуклеотидных последовательностей должен быть общим для данного вида и иметь комплементарный сайт в другом виде только при введении двух замен в любом месте олигонуклеотидного зонда; второй тип может иметь комплементарный сайт связывания при введении одной замены, но такая замена должна находиться не ближе, чем 5 оснований с 3′- или 5′-конца зонда. Длины используемых зондов варьируются от 13 до 21 нуклеотида и имеют теоретическую температуру плавления (Tm) в диапазоне 44-48°С, которая рассчитана по методу «ближайших соседей» для концентрации олигонуклеотида 1.5×10-6 M и [Na+]=50 mM.
Выбранные зонды однозначно определяют все виды ортопоксвирусов, патогенных для человека - VARV, MPXV, CMLV, VACV.
Анализ набора последовательностей показал, что для VACV общие зонды, полностью дискриминирующие этот вид по гену B29R, отсутствуют (Табл.2). Задача по диагностике VACV решается с помощью использования набора зондов, специфичных к известным последовательностям VACV.
Использование в качестве гена-мишени B19R позволяет избежать недостатков микрочипа, разработанного ранее только по данным для гена B29R [13], и повысить достоверность результатов определения. Во-первых, повышается количество родоспецифичных зондов (в дополнение к 7 зондам по гену B29R добавляется 7 зондов по гену B19R), а также набор видоспецифичных зондов для VARV, MPXV, CMLV, ECTV. Вместе с тем, общие для всех штаммов CPXV зонды не удается найти по обоим генам. Тем не менее, олигонуклеотидные последовательности по гену B19R позволяют идентифицировать видовую принадлежность CPXV более полно (см. Табл.2). Это достигается за счет разбиения вида CPXV на 2 группы - «CPXV-A» и «CPXV-B». Группа «CPXV-A» разбивается на две подгруппы «CPXV-A1» и «CPXV-A2». Первая подгруппа включает в себя 3 штамма (С/BRI, C/GRU, C/TUR) и определяется зондами Cpv_B19_1, Cpv_B19_2, Cpv_B19_3, Cpv_B19_4 (Табл. 2). Вторая подгруппа состоит из девяти штаммов (С/BRI, C/GRU, C/TUR, C/EP267, C/EP3 С/EP2, C/EXP, C/RP9, OPV-88) и дискриминируется тремя зондами (Cpv_B19_5, Cpv_B19_6, Cpv_B19_7). Зонды Cpv_B19_4 и Cpv_B19_5 позволяют определить штамм С/OPV-88, а определение штамма С/EP2 возможно только по зонду Cpv_B19_5. Группа «CPXV-B» содержит 9 штаммов (C/HAM, C/PUM, C/REV, C/EP1, C/EP4, C/EP6, C/EP7, C/EP8, C/RP1) и определяется зондами Cpv_B19_6 и Cpv_B19_7 (Табл.2).
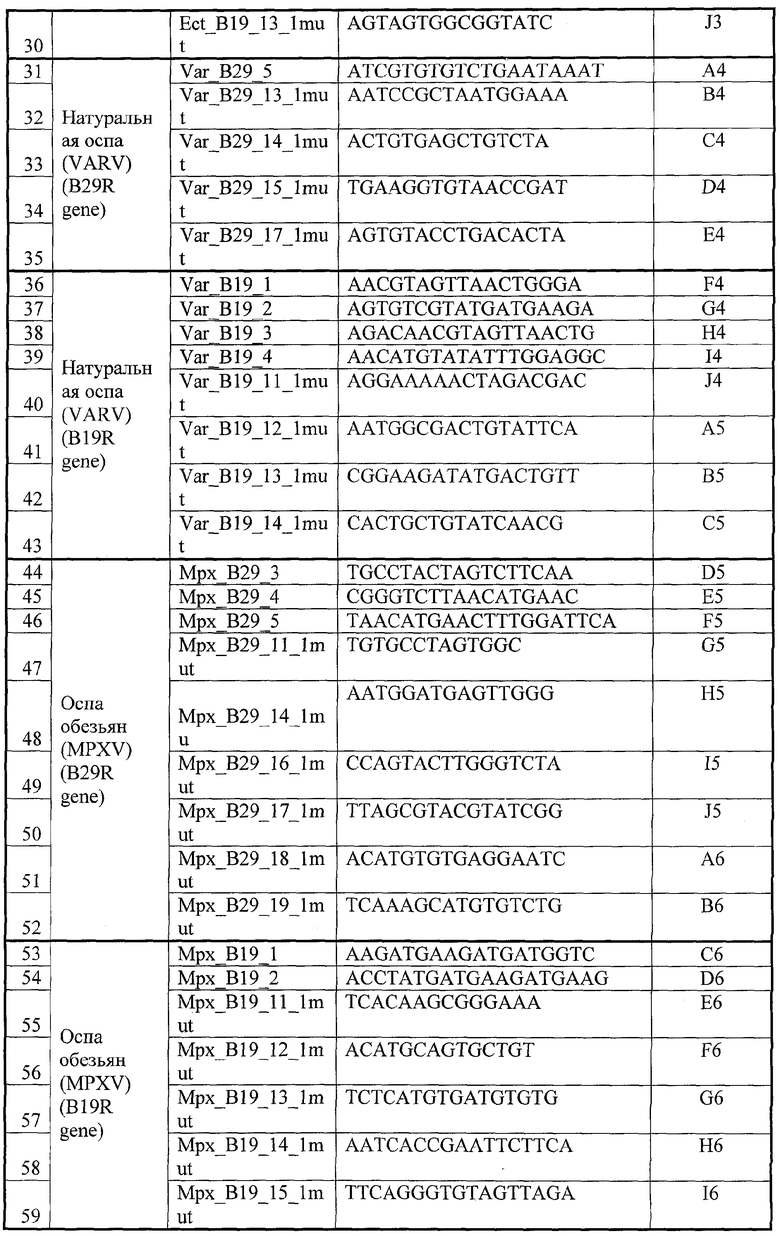
Типирующие олигонуклеотиды в количестве 110 для дифференциальной диагностики ортопоксвирусов и дискриминации от них герпесвирусов 1, 2 и 3 типов на биологическом микрочипе приведены в Таблице 2.
Принципиальная схема дифференциальной диагностики ортопоксвирусов на биочипе включает следующие стадии:
1. Из природного образца (ткани животных или человека) выделяют суммарную ДНК.
2. С помощью праймеров, специфичных к B29R и B19R генам различных видов ортопоксвирусов и генам US4, US5 и ORF31 для герпесвирусов, получают преимущественно одноцепочечную ДНК путем проведения ПЦР в асимметричном режиме.
3. Полученные ампликоны метят путем введения флуоресцентной метки в их состав.
4. На следующей стадии проводят гибридизацию полученных ампликонов с дискриминирующими зондами, входящими в состав разработанного микрочипа.
5. Анализ результатов гибридизации проводят с помощью сканера микрочипов и программы Scanarray Express.
6. Определение видовой принадлежности ортопокс- и герпесвирусов проводится путем сравнения экспериментальной и теоретической картин гибридизации.
Последовательности олигонуклеотидных праймеров, использованных для амплификации участков обоих генов ортопоксвирусов и генов герпесвирусов, приведены в Таблице 3.
Праймеры выбраны таким образом, чтобы либо амплифицировать все виды поксвирусов (универсальные праймеры), либо только один из их видов (видоспецифичные праймеры).
В случае ортопоксвирусов длина нарабатываемых ампликонов составляет ˜1100 нуклеотидов для гена В29R и ˜1300 нуклеотидов для гена В19R, а для герпесвирусов HSV-1, HSV-2, VZV соответственно 275, 335 и 310 нуклеотидов.
Преимуществом разработанного микрочипа является возможность использования при гибридизации ампликонов большого размера, поскольку в отличие от гелевых микрочипов прототипа дискриминирующие олигонуклеотиды пришиты непосредственно к стеклянной поверхности микрочипа. Поэтому в этом случае нет никаких затруднений, связанных с диффузией крупных ампликонов внутрь гелевой ячейки микрочипа.
Изобретение иллюстрируется следующими графическими материалами.
Фиг.1. Структура микрочипа для диагностики ортопокс- и герпесвирусов.
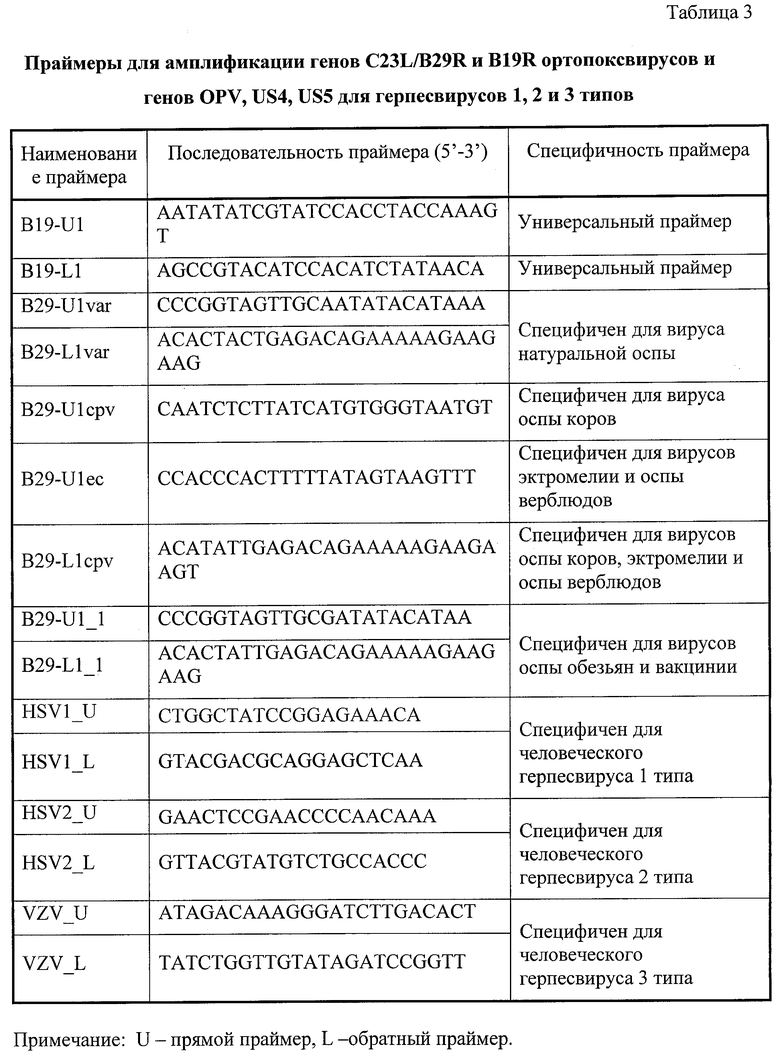
Фиг.2. Результаты анализа ортопокс- и герпесвирусов с помощью микрочипа.
Для лучшего понимания сущности изобретения ниже приведены примеры его конкретного выполненния.
Пример 1. Изготовление микрочипов для диагностики ортопокс- и герпесвирусов.
Печать микрочипов осуществляют на стеклянных альдегидных слайдах методом контактной печати на споттере SpotArrayTM («Perkin Elmer») одновременно четырьмя пинами №7 («Perkin Elmer») со стандартизованным расстоянием между ними 9 мм. В итоге каждый слайд содержит 8 эквивалентных микромассивов зондов, пригодных для одновременной гибридизации 8 образцов. Средний диаметр спотов (ячеек микрочипа с иммобилизованными зондами) составляет ˜280 μm, расстояние между спотами - 380 μm. Логический размер итогового микромассива, включающего все 110 зондов, составляет 10 colomns×11 rows, геометрический размер - 5.9×6.3 мм. Дизайн микрочипа представлен на Фиг.1, а в Таблице 2 показаны позиции соответствующих зондов. Зонды располагаются на микрочипе без повторов, последовательно согласно перечню, приведенному в Таблице 2, и сгруппированы в подмножества соответствующей специфичности. Состав смеси для печатания чипа включает: 70 мкМ олигонуклеотидного зонда, 10 мкМ «нормировочного» олигонуклеотида (QC) с последовательностью: 5′-H2N(CH2)6-TTGGCAGAAGCTATGAAACGATATGGG-3′ (Tm=58°C), индифферентной по отношению ко всем последовательностям ОПВ, HSV и зондов, приведенных в Таблицах 1 и 2. Раствор олигонуклеотидных зондов для печатания чипа готовится в смеси ДМСО:H2O (1:1). Ковалентную иммобилизацию олигонуклеотидов на альдегидном стекле осуществляют путем восстановления образовавшегося на стадии печатания основания Шиффа.
Пример 2. Получение флуоресцентных ампликонов для анализа
Для получения преимущественно одноцепочечной ДНК проводят ПЦР в асимметричном режиме. Состав реакционной смеси (50 мкл): буфер для ПЦР «Qiagen» (1.5 мМ MgCl2, KCl, (NH4)2SO4,Трис-HCl, pH 8.7), MgCl2 - 1.5 мМ,0.2 мМ каждого dNTP, 80 нМ прямого праймера, 800 нМ обратного праймера, 1.25 ед. Hot Start Taq-полимеразы («Qiagen») и 1 мкл раствора вирусной ДНК. ПЦР проводят в термоциклере TGradient («Biometra»), используя следующий протокол: 95°C - 15 мин, затем 35 циклов: 94°C - 30 с, 52-56°C - 30 с, 72°C - 1 мин 30 с, заключительная инкубация 72°C - 10 мин. Контроль за наработкой ампликона осуществляют электрофорезом в 1% агарозном геле с окрашиванием этидий бромидом. Введение флуоресцентной метки осуществляют с помощью цианинового красителя «MicromaxTM ASAP Cy3 labeling reagent» («Perkin Elmer»).
Образцы одноцепочечной ДНК, полученной в ходе асимметричной ПЦР, предварительно очищают на колонке QIAquick («Qiagen») по протоколу производителя, добавляют по 1 мкл 1 мМ раствора цианинового красителя «MicromaxTM ASAP Cy3 labeling reagent» («Perkin Elmer») и выдерживают 1 час при 85°С. Флуоресцентно-меченые ампликоны очищают от избытка красителя на колонках Sentrisep, высушивают досуха на вакуумной центрифуге при 60°С и используют для гибридизации.
Пример 3. Гибридизация анализируемых ампликонов.
Гибридизацию флуоресцентно-меченых ампликонов проводят в гибридизационной камере фирмы ArrayIt, Sunnyvale. Высушенный образец ампликона растворяют в 2.5 мкл воды, добавляют 2.5 мкл 2x гибридизационного буфера (6×SSC, 5(раствор Денхардта, 0.1% SDS и 0.3 мкл 10 мкМ раствора модифицированного олигонуклеотида, меченого цианиновым красителем Cy5 по 5′ концу с последовательностью, комплементарной QC: 5′-Cy5-CONH(CH2)6-CCCATATCGTTTCATAGCTTCTGCC-3′ (QC-Cy5, Tm=56°C). Гибридизацию осуществляют при температуре 45°С в течение 40 мин.
Пример 4. Анализ результатов гибридизации.
Сканирование слайда проводят на сканере ScanArray Express 2.0 (Perkin Elmer) последовательно с использованием двух возбуждающих лазеров с длинами волн λ1=543 нм для Cy3 и λ2=633 нм для Cy5. В последнем случае наблюдают флуоресцентное свечение всех спотов вследствие гибридизации QC и QC-Cy5 (Фиг.1). Изображение, полученное по каналу возбуждения флуоресценции с длиной волны λ2, служит показателем качества печати чипа, а интегральная величина флуоресценции каждого спота рассматривается в качестве нормировочной для флуоресценции того же спота, измеренной по каналу возбуждения с λ1. Анализ изображения осуществляют с использованием программы Scanarray Express, входящих в математическое обеспечение сканера. Для i-го спота нормированную флуоресценцию рассчитывают по формуле:
Ii=Ii(Cy3)/Ii(Cy5),
где Ii(Cy3)=Fi(Cy3)-Bi(Cy3), Fi(Cy3) - интегральная интенсивность флуоресценции в пределах спота, Bi(Cy3) - средняя фоновая интенсивность флуоресценции в окружении спота. Аналогичным образом рассчитывают величину Ii(Cy5). Величины Fi и Bi рассчитываются в вариантах "median spot» и «mean spot". Последний вариант расчета оказался более удобным для количественной обработки изображения, полученного по каналу λ2 при повышенной интенсивности фона.
Определение видовой принадлежности ортопокс- и герпесвирусов проводится путем сравнения экспериментальной и теоретической картин гибридизации.
Результаты анализа различных ортопокс- и герпесвирусов приведены на Фиг.2.
Источники информации
1. Марцевич Г.Р., Радюк С.Н., Рыжов К.А. и др. // Вопр. вирусол. - 1996 - №5 - стр.195-197.
2. Downie, A.W., Dumbell, K.R. // J.Path. Bact. - 1947 - v.59 - №1-2 - p.189-198.
3. Маренникова С.С., Щелкунов С.Н. // Патогенные для человека ортопоксвирусы. // КМК Scientific Press Ltd - Москва - 1998 - стр.386.
4. Авакян А.А., Альтштейн А.Д., Кириллова Ф.М., Быковский А.Ф. // Вопр. вирусол. - 1981 - №2 - стр.196-203.
5. Мальцева Н.Н. // Экспресс-диагностика заболеваний, вызванных ортопокс - и некоторыми герпес вирусами // 1980 - Дисс.Докт. - М.
6. Маренникова С.С., Янова Н.Н., Жукова О.А. // Ж. Микробиол. - 1990 - вып.8 - стр.57-62.
7. Marennikova, S.S., Nagieva, F.G., Matsevich, G.R., Shelulhina, E.M. and all. // Acta Virol. - 1988 - v.32 - p.19-26.
8. Маренникова С.С., Мацевич Г.Р., Хабахпашева Н.А. и др. // Вопр. Вирусол. - 1986 - №6 - стр.689-690.
9. Патент РФ №2230791 // кл. С12Q 1/68, опубл. 2004 г.
10. Roop, S.L., Jin, Q., Knight, J.C., Massung, R.F. & Esposito, J.J. // Journal of Clinical Microbiology - 1995 - v.33 - p.2069-2076.
11. Lapa S., Mikheev M., Shchelkunov S., Mikhailovich V., Sobolev A., Blinov V., Babkin I., Guskov A., Sokunova E., Zasedatelev A., Sandakhchiev L., Mirzabekov A. // J. Clin. Microbiol. - 2002. Vol.-40. - P.753-757.
12. Международная заявка WO №03046221, кл. С07Н 21/00, опубл. 2003 г.
13. Laassri M., Chizhikov V., Mikheev M., Shchelkunov S., Chumakov K. // J. Virol. Methods. - 2003. - Vol.112. - P.67-78.





Изобретение относится к молекулярной биологии, генетической инженерии, медицине. Предлагаемый способ позволяет определять различные типы ортопоксвирусов и основан на проведении одноэтапной ПЦР с последующей гибридизацией полученных флуоресцентно меченых ампликонов с ДНК-микрочипом, содержащим дискриминирующие ортопокс- и герпесвирус-гибридизационные зонды. Изобретение предназначено для экспресс-диагностики ортопоксвирусов, патогенных для человека и дискриминации их от герпесвирусов, вызывающих сходные клинические проявления. 3 н.п. ф-лы, 2 ил., 3 табл.
| LAASSRI M., CHIZHIKOV V., et al | |||
| J Virol Methods | |||
| Способ и приспособление для нагревания хлебопекарных камер | 1923 |
|
SU2003A1 |
| FITZQIBBON J.E., SAQRIPANTI J.L | |||
| J Virol Methods | |||
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
| Способ обработки целлюлозных материалов, с целью тонкого измельчения или переведения в коллоидальный раствор | 1923 |
|
SU2005A1 |
| RYABININ V.A., et al | |||
| J Med Virol | |||
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
Авторы
Даты
2008-03-10—Публикация
2006-04-20—Подача