Изобретение относится к области биотехнологии, в частности к генетической инженерии, и может быть использовано в ветеринарии и медицине для выявления генетического материала вируса бешенства в полевых и клинических образцах.
Изобретение представляет собой продукт химического синтеза олигодезоксирибонуклеотидной природы, предназначенный для индикации РНК лиссавирусов в патологическом и клиническом материале. При помощи этого набора диагностических праймеров возможно экспресс-определение наличия генетического материала вирусов бешенства, принадлежащих к генотипу 1.
Бешенство - острое инфекционное заболевание центральной нервной системы, поражающее всех теплокровных, включая и человека. Это заболевание вызывается вирусом бешенства (Rabies virus). Представителем рода Lyssavirus, семейства Rhabdoviridae. Род Lyssavirus объединяет семь генотипов. Генотип 1 представлен классическими штаммами вируса бешенства (rabies viras), которые циркулируют во всем мире. Генотипы 2-7 включают лиссаподобные rabies-related (non-rabies) вирусы: Lagos bat virus (генотип 2), Mokola virus (генотип 3), Davenhage virus (генотип 4), European bat lyssavirus 1 (EBLV1) и 2 (EBLV2) (генотипы 5 и 6 соответственно) и Australian bat lyssavirus (ABL) (генотип 7). Вирус бешенства (генотип 1) поддерживается в природе межвидовой передачей практически повсеместно (кроме Австралии и некоторых островов) среди представителей Carnivora и Microchiroptera.
Геном представлен единой 1-спиральной линейной молекулой минус-РНК, состоит из 11932 нуклеотидов. Вирионная РНК рабдовирусов не обладает инфекционностью. В вирионах рабдовирусов обнаружено 5 полипептидов (гликопротеин, матриксный белок М, нуклеопротеин N, фосфопротеин NS, обратная транскриптаза L (РНК-зависимая РНК-полимераза)), 3 из которых (L,N,S,N) связаны с нуклеокапсидом, а 2 (G,M) входят в состав липопротеидной оболочки. Белок G гликолизирован, образует на поверхности вириона выступы, индуцирует синтез вируснейтрализующих антител и обеспечивает развитие иммунитета. Белки нуклеокапсида N и NS имеют группоспецифические антигенные детерминанты. Белки нуклеокапсида L и NS являются компонентами транскриптазы. Гены расположены в следующем порядке: 3′-N-NS-M-G-L-5′ (Tordo Ν., Poch О., Ermine Α., Keith. G., Rougeon F. Completion of the rabies virus genome sequence determination: highly conserved domains among the L (polymerase) proteins of unsegmented negative-strand RNA viruses. Virology. 1988:165 (2). 565-76; Tordo, N., Poch, O., Ermine, A., Keith, G. and Rougeon, F. Walking along the rabies genome: is the large G-L intergenic region a remnant gene? Proc. Natl. Acad. Sci. U.S.A. 1986: 83 (11), 3914-18; Tordo Ν., Roch О. Structure of rabies virus. Rabies. Boston: Kluwer Academic Publishers. 1988: 25-45; Tordo Ν., Sacramento D., Bourhy H. The polymerase chain reaction (PCR) technique for diagnosis, typng and epidemiological studies of rabies/ In: Kaplan M.M., Koprowski H., Meslin F.-X. Laboratory techniques in rabies Eds.WHO. Geneva. 1996:157-70) [1-4]·
До настоящего времени наиболее чувствительным и достоверным методом диагностики бешенства в России является классическая биопроба на белых мышах с последующей идентификацией антигена вируса бешенства методом флуоресцирующих антител (МФА) (Государственный стандарт СССР 26075. Животные сельскохозяйственные. Методы лабораторной диагностики бешенства. Введ. 9.01.1984. М.: Государственный комитет СССР по стандартам; 1984) [5].
Недостатками биопробы являются продолжительность исследования (в отрицательных случаях до 30 суток), потенциальная опасность выноса возбудителя болезни, а также невозможность исследования разложившегося материала. Кроме того, постановка биопробы неэкономична, требует особого виварного помещения и обслуживающего персонала (Хисматуллина Н.А., Юсупов Р.Х., Селимов М.А., Янбарисова С.Р. Разработка экспресс-методов иммунологического мониторинга при бешенстве. Вопросы вирусологии. 2001; 5:45-8) [6].
Известен диагностический набор праймеров на консервативную область гена нуклеопротеина фиксированного штамма вируса бешенства CVS, циркулирующего на территории Новосибирской области среди диких и домашних животных, в полевых и клинических образцах, имеющих следующую структуру:
Rabl34F 5′-ATCGT(A,G)GA(T,C)CAATATGAGTACAAGTA-3′ (26 п.н.)
Rabl292R 5′-C(A,G,C,T)TCCATTCATGATTCG-3′ (20 п.н.)
Rab299F 5′-GCAATGCAGTTCTTTGAGGG-3′ (20 п.н.)
Rab857R 5′-TATCTCTTCTTCAAAGTTCTT-3′ (21 п.н.) (патент RU №2340673, МПК C12N 15/00, опубл. 10.12.2008 г.) [7].
Однако для более полной характеристики и расширения диапазона исследований эпизоотических изолятов и вакцинных штаммов вируса бешенства необходим дальнейший поиск синтетических олигонуклеотидных праймеров на ген гликопротеина.
Известно, что одним из широко используемых методов детекции РНК вируса бешенства является обратно-транскриптазная цепная реакция (ОТ-ПЦР) (Nadin-Davis S.A., Yuang W., Wandeler A.I. The design of strain-specific polymerase chain reactions for discrimination of theraccon rabies virus strain from indigenous rabies viruses of Ontario. J. Virol. Methods. 1996 Mar; 57 (1): 1-14) [8]. С помощью ПЦР диагноз можно поставить за 5 часов. Кроме того, применение автоматического секвенирования позволяет получить характеристику изолятов в течение 16 часов.
В большинстве случаев ОТ-ПЦР применяют для штаммовой дифференциации вируса бешенства (Nadin-Davis S.A., Yuang W., Wandeler A.I. The design of strain-specific polymerase chain reactions for discrimination of theraccon rabies virus strain from indigenous rabies viruses of Ontario. J. Virol. Methods. 1996 Mar; 57 (1): 1-14; Wakeley P.R., Johnson N., McElhinney L.M. et al. Development of a real-time, TaqMan reverse transcription-PCR assay for detection and differentiation of Lyssavirus genotypes 1,5 and 6. J.Clin.Microbiol.2005; 43(6): 2786-92) [8, 9]. Кроме того, возможно применение ОТ-ПЦР для прижизненного обнаружения вирусной РНК в слюне, спинномозговой жидкости инфицированных животных и в биоптатах слюнной железы (Crepin P., Andru L., Rotivel Y. et al. Intravitam diagnosis of human rabies by PCR using saliva and cerebrospinal fluid. J. Clin. Microbiol. 1998; 36 (4): 1117-21; Nagaraj T. Kumar S., Dudley J., Nei M. Tamura K. MEGA: biologist-centricsoltware for evolutionary analysis of DNA and protein seqvences. http://Brif.Bioinform.2008; 9:299-306; Kumar S., Dudley J., Nei M. Tamura K. MEGA: biologist-centricsoltware for evolutionary analesis of DNA and protein seqvences. http://Brif.Bioinform.2008; 9:299-306) [9- 12].
Наиболее приемлемым с точки зрения описанных недостатков биопробы и принятым за прототип является способ выявления РНК вируса бешенства, основанный на ОТ-ГТДР, включающей выделение РНК вируса из вируссодержащей суспензии, синтез олигонуклеотидных праймеров на ген нуклеопротеина, амплификацию РНК вируса в ОТ-ПЦР, специфическую идентификацию продуктов ОТ-ПЦР с помощью дот-блот анализа (Tordo Ν., Sacramento D., Bourhy Η. The polymerase chain reaction (PCR) technique for diagnosis, typng and epidemiological studies of rabies/ In: Kaplan M.M., Koprowski H., Meslin F.-X. Laboratory techniques in rabies Eds. WHO. Geneva, 1996: 157-70) [4]. В указанной работе способ выделения РНК основан на фенольно-хлороформном методе, позволяющий выделять тотальную РНК без посторонних примесей (фосфолипиды гол. мозга и др.), оказывающие ингибирующее действие на ОТ-ПЦР. Вместе с тем, для выделения высококачественной РНК фенольно-хлорормным методом требуется соблюдение низкотемпературных условий. В то же время часто используемый сорбентный метод выделения РНК прост в исполнении и не требует соблюдения особых температурных условий.
Однако данный метод не позволяет получать качественные нативные образцы РНК. В связи с этим чувствительность одностадийной ОТ-ПЦР значительно снижается. Кроме того, количество вирусного материала в исследуемых образцах может быть ниже детектируемого ПЦР вследствие несоблюдения условий хранения или транспортировки клинического материала и др.
Задача изобретения - расширение арсенала олигонуклеотидных праймеров и способов выявления вируса бешенства.
Поставленная задача решается тем, что в известном способе синтезируются синтетические олигонуклеотидные праймеры для выявления РНК вируса бешенства, отличающиеся тем, что праймеры имеют нуклеотидные последовательности:
Наружные праймеры для первого раунда амплификации:
Внутренние праймеры для второго раунда амплификации:
Задача решается также тем, что в способе выявления РНК вируса бешенства, включающем проведение ОТ-ПЦР с олигонуклеотидными праймерами, согласно изобретению наружные праймеры для первого раунда амплификации имеют нуклеотидные последовательности:
Внутренние праймеры для второго раунда амплификации имеют следующие последовательности:
Поставленная задача решается также тем, что в способе выявления РНК вируса бешенства, включающем проведение ОТ-ПЦР с олигонуклеотидными параметрами, согласно изобретению, праймеры имеют нуклеотидные последовательности:
fp_850_gp_rabv 5′-TTAGACTTATGGATGGAACATGGGT-3′,
rp_850_gp_rabv 5′-AGTGACTGACACCTCCCTCCCT-3′,
fp_350_gp_rabv 5′-TCAGACGAAATTGAGCACCTTGT-3′,
rp_350_gp_rabv 5′-ACCTCCCCCCAACTCTTAAACA-3′ и синтезированы на консервативный ген гликопротеина, а ОТ-ПЦР проводят в два раунда, при этом в случае положительной реакции синтезируется фрагмент, соответствующий размеру в первом раунде 755 п.н., во втором раунде - 259 п.н.
Изобретение иллюстрируется следующими примерами.
Пример 1. Получение синтетических олигонуклеотидных праймеров.
Конструирование праймеров осуществляют путем сравнения нуклеотидных последовательностей различных штаммов лиссавирусов, депонированных в международной базе данных GeneBank (http:ncbi.nlm.nih.gov/GeneBank/GeneBankSearch.html) при помощи пакета программного обеспечения «Vector NTI 9.1».
Для этого были рассчитаны и синтезированы олигонуклеотидные праймеры на район гена гликопротеина (ЗАО «Синтол»).
Окончательный выбор праймеров основан на следующих критериях: высокий индекс сходства фрагмента и РНК различных штаммов вируса бешенства, высокая температура отжига (GC-метод), большая длина консенсусов. Химический синтез праймеров осуществляют амидофосфидным методом на автоматическом синтезаторе ASM-102U. Концентрацию синтетических олигонуклеотидных праймеров в маточном растворе определяют спектрофотометрическим методом.
Характеристика набора праймеров и участка амплифицируемой геномной РНК. Праймеры фланкируют консервативный участок гена гликопротеина вируса бешенства, кДНК который не имеет полиндромных повторов нуклеотидов и не образует выраженных вторичных структур, не имеет протяженных G-C участков. Для пар праймеров расчетная температура плавления была близкой и составила Tm=58°C.
Таким образом, были выбраны синтетические олигонуклеотидные праймеры, комплементарные консервативной области генома вируса бешенства района гена гликопротеина:
fp_850_gp_rabv 5′-TTAGACTTATGGATGGAACATGGGT-3′,
rp_850_gp_rabv 5′-AGTGACTGACACCTCCCTCCCT-3′,
fp_350_gp_rabv 5′-TCAGACGAAATTGAGCACCTTGT-3′,
rp_350_gp_rabv 5′-АССТССССССААСТСТТАААСА-3′.
Пример 2. Способ выявления РНК вируса бешенства с помощью набора синтетических олигонуклеотидных праймеров в обратно-транскриптазной полимеразной цепной реакции (ОТ-ПЦР).
Способ осуществляется в несколько этапов.
Этап 1. Выделение РНК вируса бешенства.
Обеззараженные в соответствии с ГОСТом 26075 [5] пробы антигена вируса бешенства мозгового происхождения (10%-ные суспензии на физ. растворе) и клинического материала отбирали по 100 мкл. Выделение РНК из образцов головного мозга животных и клинического материала осуществляют стандартным способом с использованием коммерческого набора «РИБО-сорб», производства ФГУН «Центральный НИИ эпидемиологии» Роспотребнадзора, выпускаемый фирмой ООО «ИнтерЛабСервис», Россия.
Этап 2. Проведение реакции обратной транскрипции для получения к-ДНК.
Осуществляют стандартным способом с использованием коммерческого набора «Реверта-L» производства того же института, выпускаемый ООО «ИнтерЛабСервис», Россия.
В пробирку, содержащую 10 мкл реакционной смеси: буфер для ОТ и 6 мкл ревертазы из набора «Реверта-L», добавляют 10 мкл РНК-пробы, осторожно перемешивают и инкубируют в термостате при 37°C в течение 60 минут. Затем добавляют 20 мкл ДНК-буфера, тщательно перемешивают и используют для постановки ОТ-ПЦР.
Этап 3. Амплификация участка к-ДНК вируса бешенства, кодирующего ген гликопротеина.
Полимеразная цепная реакция.
Предварительно готовится ПЦР-смесь-1 и ПЦР-смесь-1а и разливается по 5 мкл в микропробирки объемом 0,6 мл, сверху заливается воском. ПЦР смесь-1 состоит из раствора праймеров в концентрации 1пкмоль/мкл каждого и смеси трифосфатов до конечной концентрации 0,2 тМ каждого. Приготовленные пробирки хранятся при +4°C. В отдельной микропробирке смешивают ПЦР смесь-2 непосредственно перед использованием: 10×ПЦР буфера (60 mM Tris-HCl, 2,5 mM MgCl2, 25 mM KCl, 10 mM 2-меркаптоэтанола, 0,1% Тритон Х-100) из расчета по 1 мкл и по 9 мкл деионизированной стерильной воды на пробу (количество проб + 2 контроля + 1) и добавляют Tag F-ДНК-полимеразу с активностью 5 ед/мкл до конечной концентрации 0,5 ед/мкл.
Отбирают необходимое количество пробирок с ПЦР-смесью-1. На поверхность воска вносится по 10 мкл ПЦР-смеси-2. Сверху добавляется по капле минерального масла для ПЦР (примерно 25 мкл). В подготовленные для ПЦР пробирки под масло или на масло вносят по 10 мкл исследуемых кДНК. Ставят контрольные реакции амплификации: отрицательный контрольный образец - в пробирку вносят 10 мкл ТЕ-буфера; в положительный контрольный образец - в пробирку вносят 10 мкл кДНК рабического вируса.
Температурный режим проведения ОТ-ПЦР. Программа амплификации состоит из температурных режимов, представленных в таблице 1.

Этап 4. Определение размера продуктов диагностической ПНР.
Продукты ОТ-ПЦР анализируют методом электрофореза в 1,7%-ном агарозном геле в стандартном трис-боратном буфере, pH 8,0, по стандартной методике. Результаты электрофореза учитывают, просматривая гель в ультрафиолетовом свете с длиной волны 254 нм на приборе «Трансиллюминатор». Маркер молекулярного веса Fermentas 100-3000 пар оснований.
Результат ОТ-ПЦР считают положительным, если продукт ОТ-ПЦР визуализируется в виде светящегося фрагмента, соответствующего 755 п.н. Проведение 2 этапа амплификации проводят аналогично 1 этапу с применением ПЦР-смеси-1а и в качестве матрицы используют продукты амплификации 1 этапа. Детекцию продуктов ПЦР-амплификации проводят методом электрофореза в агарозном геле аналогично вышеописанному. В положительных пробах визуализируется паттерн 259 п. н.
Пример 3. Определение чувствительности ПЦР.
Для определения чувствительности реакции контрольный вирус бешенства, производственный штамм «Овечий» ГНКИ титруют методом 10-кратных разведений до 107LD50/мл, каждое разведение подвергают исследованию в ПЦР. Чувствительность разработанной ОТ-ПЦР составляет 1,3 lg LD50/мл.
Пример 4. Определение специфичности.
Результаты опытов по определению специфичности реакции представлены в таблице 2.

Из данных таблицы 2 следует, что положительная реакция имеет место в двух раундах предлагаемого способа с применением внешних и внутренних праймеров к гену гликопротеина рабического вируса, с к-ДНК эпизоотических изолятов вируса бешенства (№№13991, 3001, 329, 925, 2228, 139), стандартного вируса бешенства - CVS, производственного штамма «Овечий» ГНКИ и вакцинных - «Внуково-32», «Щелково-51» и ERA 0/333 вируса бешенства. В то же время показана отрицательная реакция с отрицательными контролями - ДНК из мозговой ткани здоровых животных (лисицы, кошки, собаки, овцы и кролика), а также гетерологичными контролями - ДНК различных микроорганизмов, что свидетельствует о специфичности предлагаемого способа с использованием разработанного набора олигонуклеотидных праймеров к гену гликопротеина вируса бешенства.
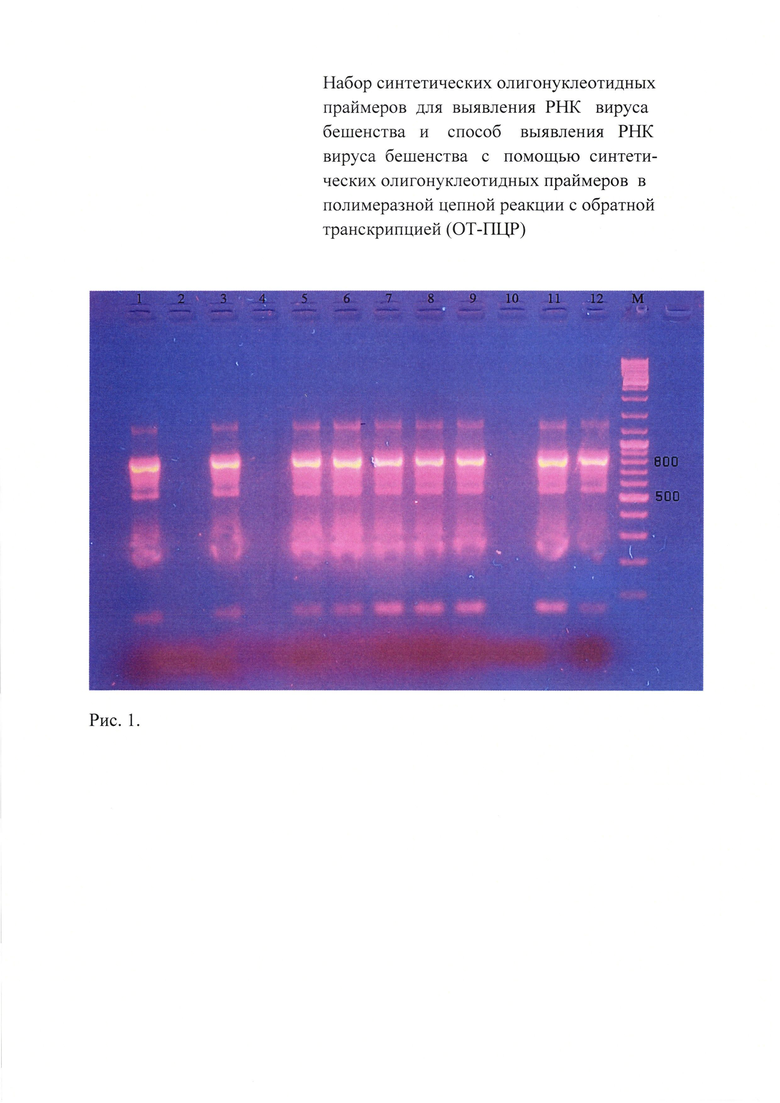
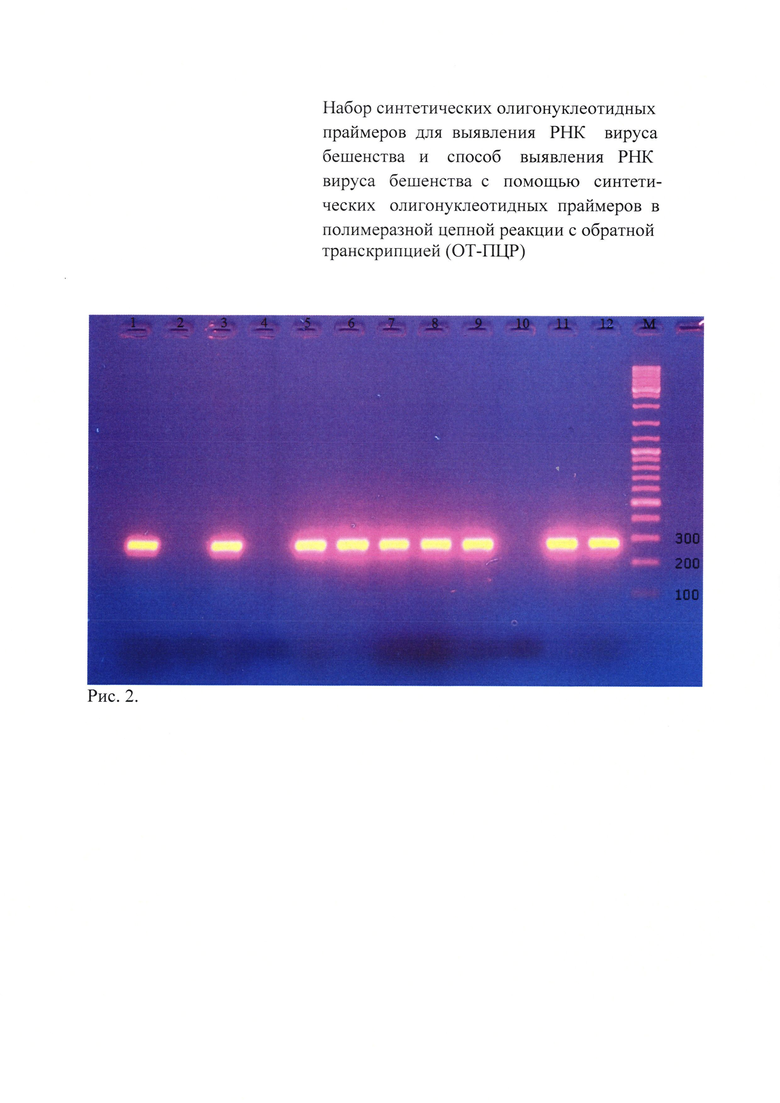
Результаты электрофореграмм продуктов первого и второго этапов амплификации выборочно представлены на рисунках 1 и 2.
На рисунке 1 показана электрофореграмма продуктов первого этапа амплификации (праймеры 755 п.н.), где:
1 - слезная жидкость больного с клиническими проявлениями гидрофобии;
2 - слезная жидкость здорового человека;
3 - слюна от больного с клиническими проявлениями гидрофобии;
4 - слюна здорового человека;
5 - эпизоотический штамм №36, выделенный в РТ;
6 - эпизоотический штамм №40, выделенный в РТ;
7 - эпизоотический штамм №258, выделенный в РТ;
8 - эпизоотический штамм №5359, выделенный в РТ;
9 - эпизоотический штамм №36, выделенный в Смоленской обл.;
10 - отрицательный контрольный образец;
11 - вирус бешенства, штамм «Овечий» ГНКИ;
12 - стандартный вирус бешенства, штамм CVS;
M - маркер молекулярного веса Fermentas 100-3000 пар оснований.
На рисунке 2 показана электрофореграмма продуктов второго этапа амплификации (праймеры 259 п.н.), где:
1 - слезная жидкость больного с клиническими проявлениями гидрофобии;
2 - слезная жидкость здорового человека;
3 - слюна от больного с клиническими проявлениями гидрофобии;
4 - слюна здорового человека;
5 - эпизоотический штамм №36, выделенный в РТ;
6 - эпизоотический штамм №40, выделенный в РТ;
7 - эпизоотический штамм №258, выделенный в РТ;
8 - эпизоотический штамм №5359, выделенный в РТ;
9 - эпизоотический штамм №36, выделенный в Смоленской обл.;
10 - отрицательный контрольный образец;
11 - вирус бешенства, штамм «Овечий» ГНКИ;
12 - стандартный вирус бешенства, штамм CVS;
M - маркер молекулярного веса Fermentas 100-3000 пар оснований.
Из данных рисунка 1 видно, что исследуемые пробы: слезная жидкость и слюна больного с клиническими признаками гидрофобии, эпизоотические изоляты (№№36,40,258 и 5359), выделенные в РТ, и изолят №36, выделенный в Смоленской области, а также производственный штамм «Овечий» ГНКИ вируса бешенства и стандартный вирус бешенства, штамм CVS, идентифицируются с помощью внешних праймеров fp_850_gp_rabv и rp_850_gp_rabv на ген гликопротеина вируса бешенства, амплифицирующие участок гена гликопротеина в 755 п.н. В то же время реакция отсутствовала с пробами слюны и слезной жидкости от здорового человека и в отрицательном контрольном образце, что свидетельствует о специфичности предлагаемого способа.
Из данных рисунка 2 видно, что исследуемые пробы: слезная жидкость и слюна больного с клиническими признаками гидрофобии, эпизоотические изоляты (№№36, 40, 258 и 5359), выделенные в РТ, и изолят №36, выделенный в Смоленской области, а также производственный штамм «Овечий» ГНКИ вируса бешенства и стандартный вирус бешенства, штамм CVS, идентифицируются с помощью внутренних праймеров fp_350_gp_rabv и rp_350_gp_rabv на ген гликопротеина вируса бешенства, амплифицирующие участок гена гликопротеина в 259 п.н. В то же время реакция отсутствовала с пробами слюны и слезной жидкости от здорового человека и в отрицательном контрольном образце, что свидетельствует о специфичности метода ОТ-ПЦР.
Таким образом, предлагаемый способ обладает специфичностью при выявлении РНК штаммов и изолятов вируса бешенства. Указанный способ позволяет выявлять РНК вируса бешенства в исследуемых образцах (патологический материал от больных бешенством животных, культуральных жидкостях, вакцинных штаммов вируса бешенства, клиническом материале - слезная жидкость и слюна больного гидрофобией). Апробация праймеров осуществлена с использованием изолятов вируса бешенства (11 проб), выделенных от различных видов диких и домашних животных на территории Республики Татарстан и Смоленской области Российской Федерации, 3-х вакцинных, производственного и стандартного штаммов вируса бешенства, а также клинического материала (2 пробы). Показано, что применение разработанных праймеров для индикации РНК вируса бешенства в патологическом и клиническом материале обеспечивает синтез фрагментов ДНК рассчитанных размеров (внешнего 755 п.о.) и (внутреннего 259 п.о.) в условиях предлагаемой ОТ-ПЦР.
Преимущества разработанного метода перед классической биопробой на белых мышах представлены в таблице 3.
Таблица 3. Сравнительная оценка эффективности биопробы на белых мышах и предлагаемого ОТ-ПЦР при исследовании образцов на бешенство
Таким образом, предлагаемый способ позволяет проводить эффективное выявление РНК штаммов и изолятов вируса бешенства в патологическом и клиническом материале, а также сократить сроки проведения исследований до 6 часов, снизить себестоимость диагностики в 9,8 раза, трудозатраты - в 40 раз.
Литература
1. Tordo Ν., Poch Ο., Ermine Α., Keith. G., Rougeon F. Completion of the rabies virus genome sequence determination: highly conserved domains among the L (polymerase) proteins of unsegmented negative-strand RNA viruses. Virology. 1988:165(2): 565-76.
2. Tordo, N., Poch, O., Ermine, A., Keith, G. and Rougeon, F. Walking along the rabies genome: is the large G-L intergenic region a remnant gene? Proc. Natl. Acad. Sci. U.S.A. 1986: 83 (11), 3914-18.
3. Tordo Ν., Roch О. Structure of rabies virus. Rabies. Boston: Kluwer Academic Publishers. 1988: 25-45.
4. Tordo N., Sacramento D., Bourhy H. The polymerase chain reaction (PCR) technique for diagnosis, typng and epidemiological studies of rabies/ In: Kaplan M.M., Koprowski H., Meslin F.-X. Laboratory techniques in rabies Eds. WHO. Geneva. 1996:157-70.
5. Государственный стандарт СССР 26075. Животные сельскохозяйственные. Методы лабораторной диагностики бешенства. Введ. 9.01.1984. М.: Государственный комитет СССР по стандартам; 1984.
6. Хисматуллина Н.А., Юсупов Р.Х., Селимов М.А., Янбарисова С.Р. Разработка экспресс-методов иммунологического мониторинга при бешенстве. Вопросы вирусологии. 2001; 5: 45-8.
7. Патент RU №2340673, МПК C12N 15/00, опубл. 10.12.2008 г.
8. Nadin-Davis S.A., Yuang W., Wandeler A.I. The design of strain-specific polymerase chain reactions for discrimination of the raccon rabies virus strain from indigenous rabies viruses of Ontario. J. Virol. Methods. 1996 Mar; 57 (1): 1-14.
9. Wakeley P.R., Johnson N., McElhinney L.M. et al. Development of a real-time, TaqMan reverse transcription-PCR assay for detection and differentiation of Lissavirus genotypes 1,5 and 6. J.Clin.Microbiol.2005; 43(6): 2786-92.
10. Crepin P., Andru L., Rotivel Y. et al. Intravital diagnosis of human rabies by PCR using saliva and cerebrospinal fluid. J. Clin. Microbiol. 1998; 36 (4): 1117-21.
11. Nagaraj T., Kumar S., Dudley J., Nei M,, Tamura K. MEGA: biologist-centricsoltware for evolutionary analysis of DNA and protein seqvences. http://Brif.Bioinform.2008; 9:299-306.
12. Kumar S., Dudley J., Nei M., Tamura K. MEGA: biologist-centricsoltware for evolutionary analysis of DNA and protein seqvences. http://Brif.Bioinform.2008; 9:299-306.
Изобретение относится к области биотехнологии, а именно к способу выявления РНК вируса бешенства и набору, который используется в данном способе. Способ включает проведение ОТ-ПЦР с олигонуклеотидными праймерами. Праймеры имеют следующие нуклеотидные последовательности: fp_850_gp_rabv 5' TTAGACTTATGGATGGAACATGGGT 3', rp_850_gp_rabv 5' AGTGACTGACACCTCCCTCCCT 3', fp_350_gp_rabv 5'TCAGACGAAATTGAGCACCTTGT3', rp_350_gp_rabv 5'ACCTCCCCCCAACTCTTAAACA3'. ОТ-ПЦР проводят в два раунда, при этом в случае положительной реакции синтезируется фрагмент, соответствующий размеру в первом раунде - 755 п.н., во втором - размеру 259 п.н. Предложенное изобретение позволяет проводить выявление РНК штаммов и изолятов вируса бешенства различного происхождения на ранних этапах клинического проявления, а также снизить себестоимость диагностики. 2 н.п. ф-лы, 2 ил., 3 табл., 4 пр.
1. Набор синтетических олигонуклеотидных праймеров для выявления РНК вируса бешенства, отличающийся тем, что праймеры имеют нуклеотидные последовательности:
fp_850_gp_rabv 5' TTAGACTTATGGATGGAACATGGGT 3',
rp_850_gp_rabv 5' AGTGACTGACACCTCCCTCCCT 3',
fp_350_gp_rabv 5' TCAGACGAAATTGAGCACCTTGT 3',
rp_350_gp_rabv 5' ACCTCCCCCCAACTCTTAAACA3'.
2. Способ выявления РНК вируса бешенства, включающий проведение ОТ-ПЦР с олигонуклеотидными праймерами, отличающийся тем, что праймеры имеют нуклеотидные последовательности:
fp_850_gp_rabv 5' TTAGACTTATGGATGGAACATGGGT 3',
rp_850_gp_rabv 5' AGTGACTGACACCTCCCTCCCT 3',
fp_350_gp_rabv 5' TCAGACGAAATTGAGCACCTTGT3',
rp_350_gp_rabv 5' ACCTCCCCCCAACTCTTAAACA3'
и синтезированы на консервативный ген гликопротеина, а ОТ-ПЦР проводят в два раунда, при этом в случае положительной реакции синтезируется фрагмент, соответствующий размеру в первом раунде - 755 п.н., во втором - размеру 259 п.н.
| SATO G | |||
| ET AL., Rapid discrimination of rabies viruses isolated from various host species in Brazil by multiplex reverse transcription-polymerase chain reaction, J Clin Virol., 2005, v.33, no.4, p | |||
| Тепловой измеритель силы тока | 1921 |
|
SU267A1 |
| GUPTA P.K | |||
| ET AL., Preliminary report on a single-tube, non-interrupted reverse transcription-polymerase chain reaction for the detection of | |||
Авторы
Даты
2016-02-10—Публикация
2014-10-02—Подача