ПРИТЯЗАНИЕ НА ПРИОРИТЕТ
Настоящая заявка претендует на приоритет предварительной заявки на патент США с порядковым номером 61/410783, поданной 5 ноября 2010.
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящая заявка относится к устойчивости растений к заболеваниям. В некоторых вариантах осуществления, изобретение относится к устойчивости сои к соевой цистообразующей нематоде (SCN). В отдельных вариантах осуществления, настоящее изобретение относится к композициям и способам выявления признака устойчивости организма к SCN, например, к молекулярным маркерам, которые тесно сцеплены с устойчивостью к SCN. Другие варианты осуществления относятся к композициям и способам введения в организм хозяина признака устойчивости к SCN, например, с применением молекулярных маркеров, тесно сцепленных с устойчивостью к SCN.
ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
Соя, т.е. Glycine max, является одной из основных сельскохозяйственных культур с экономической точки зрения, и ее выращивают по всему миру в качестве важнейшего источника растительного масла и белка. Растущая потребность в рационе питания с низким содержанием холестерина и высоким содержанием пищевых волокон повысила важность соевых бобов в качестве пищевого сырья. К настоящему времени в Соединенных Штатах было внедрено более 10000 сортов сои, ограниченное число которых образует генетическую основу культурных сортов, разработанных в результате осуществления программ гибридизации и селекции. Johnson and Bernard, The Soybean, Norman Ed., Academic Press, N.Y., pp.1-73, 1963.
Соевая цистообразующая нематода (SCN, Heterodera glycines (HG) Ichinohe) является наиболее опасным вредителем сои, который наносит наибольший урон как в Соединенных Штатах, так и в большинстве других крупнейших стран-производителей сои в мире. Согласно оценке, снижения урожая в Соединенных Штатах из-за поражения SCN в 2003 и 2004 годах составило примерно от 2,9 до 3,4 миллиона тонн, что соответствует ежегодному убытку примерно 1,5 миллиарда долларов. Wrather et al. (2001); Wrather and Koenning (2006). SCN-устойчивый фенотип является очень сложным признаком, который управляется большим количеством генов, как рецессивных, так и доминантных. Concibido et al. (2004). Выявление устойчивого к SCN фенотипа является продолжительным, дорогостоящим и трудоемким процессом.
Заражение SCN вызывает различные симптомы, которые могут включать хлороз листьев и стеблей, отмирание корней, уменьшение урожайности, ослабление роста корней и побегов. Симптомы заражения SCN, проявляющиеся на поверхностной части растений, не являются характерными только для заражения SCN, и их можно перепутать с недостатком питательных веществ, в особенности с дефицитом железа, стрессом от недостатка воды, поражением гербицидами или другими болезнями. Первым признаком заражения является появление групп растений с желтеющими листьями и отставанием в росте. Кроме того, обсуждаемый патоген трудно определить на корнях, поскольку ослабленная корневая система также является обычным симптомом стресса или заболевания растений. Взрослые самки и цисты SCN имеют длину примерно 1/32 дюйма, что позволяет видеть их без увеличительных приборов. Регистрация наличия взрослых самок и цист на корнях является единственным точным путем обнаружения и диагностирования заражения данного поля SCN.
Присутствие SCN, как правило, не является очевидным на первом этапе заражения почвы. Плотность популяции SCN в почве должна увеличиваться до тех пор, пока она не станет достаточной для проявления симптомов на надземной части растений или уменьшения продуктивности. Увеличение плотности популяции до значительной величины может занять несколько лет. Таким образом, текущий урон от SCN является результатом заражения, которое возрастало в течение нескольких лет. Хотя соя является основным хозяином SCN, другие бобовые растения, как например, зеленая фасоль, обыкновенная фасоль, зерновая фасоль, красная фасоль, лимская фасоль, золотистая фасоль, кустовая фасоль, фасоль адзуки, горох огородный и вигна китайская, также могут служить хозяевами SCN. Жизненный цикл SCN насчитывает тридцать дней. Поэтому за один сезон роста растений развивается несколько поколений паразитов. Кроме того, яйца SCN могут сохраняться в почве в течение нескольких лет до выведения паразитов.
В прошлом популяции SCN относили к определенной «расе», сравнивая их воспроизводство на наборе из четырех линий зародышевой плазмы (генетического материала) сои с воспроизводством на стандартном культурном сорте сои, неустойчивом к SCN. В наиболее широко применяемой классификации рас идентифицировано 16 рас SCN. Отнесение к той или иной расе давало возможность нематологам и специалистам по разведению сои делиться информацией о способности определенных популяций SCN воспроизводиться на различных сортах сои, которые содержат те или иные гены устойчивости к SCN.
В 2003 году вместо теста для определения расы был разработан тест на определение типа HG. Этот новый тест включает семь источников устойчивости (линий генетического материала), и его результаты представляют в виде процентных долей, показывающих прирост популяции нематод из образца почвы на каждой из этих семи линий. Этот тест показывает, какой из источников устойчивости мог бы оказаться подходящим для данного конкретного поля, проходящего тестирование, а какой оказывается неподходящим. Поскольку генетические источники устойчивости в настоящее время ограничены коммерчески доступными сортами сои, важно чередовать эти «источники устойчивости» для того чтобы отсрочить появление опасной популяции SCN.
Вскоре после обнаружения SCN в Соединенных Штатах, были выявлены источники устойчивости к SCN. Ross and Brim (1957) Plant Dis. Rep. 41:923-4. Некоторые линии, например, Peking и PI 88788 были быстро включены в селекционные программы. Peking нашел широкое применение в качестве источника устойчивости, благодаря отсутствию свойств, нежелательных с агрономической точки зрения, причем первым выведенным культурным сортом, устойчивым к SCN, стал Pickett. Установление того факта, что некоторые популяции SCN способны заражать устойчивые сорта сои привело к расширению поисков дополнительных источников устойчивости к SCN. PI 88788 появился как популярный источник устойчивости к расам SCN 3 и 4, несмотря на то, что индекс его устойчивости к цистам расы 4 был более 10% (но менее 20%), и Peking и его производные стали популярным источником устойчивости к расам 1 и 3. Впоследствии был выявлен сорт PI 437654, обладающий устойчивостью ко всем известным расам, и его устойчивость к SCN путем обратного скрещивания была передана сорту Forrest. В настоящее время известны более 130 PIs, обладающих устойчивостью к SCN. PI 209332 и PI 90763 являются другими примерами SCN-устойчивых селекционных линий сои. Не все сорта одного и того же источника устойчивости обладают сравнимой продуктивностью, и, кроме того, они неодинаковым образом реагируют на SCN.
Устойчивые сорта сои являются наиболее эффективным средством борьбы с SCN. Плотность SCN обычно уменьшается при возделывании устойчивых сортов сои, поскольку большинство неполовозрелых (ювенильных) особей SCN не способны питаться и развиваться на корнях устойчивых сортов. Однако на любом инфицированном поле в природных условиях небольшое количество ювенильных особей SCN (<1%) будут обладать способностью к воспроизводству на имеющихся в настоящее время устойчивых сортах. Количество ювенильных особей SCN, которые способны воспроизводиться на устойчивых сортах сои, может возрастать, если повторно выращивать один и тот же устойчивый сорт. В конце концов популяция SCN может приобрести способность воспроизводиться на устойчивом сорте столь же хорошо, как и на неустойчивом сорте, если SCN-устойчивый сорт постоянно выращивать на зараженном поле. К счастью, количество ювенильных особей SCN, которые способны к воспроизведению на устойчивых сортах, снижается, при выращивании неустойчивых сортов сои, поскольку эти нематоды не могут в достаточной степени конкурировать в почве за источники пищи с другими ювенильными особями SCN, которые не способны питаться устойчивыми сортами.
Считается, что раса 3 SCN является наиболее важной расой в штатах-производителях сои среднего запада. Значительные усилия были посвящены исследованию генетики и достижению устойчивости к SCN расы 3. Хотя и Peking, и PI 88788 устойчивы к расе 3 SCN, классические генетические исследования позволили предположить, что устойчивость в расе 3 вызвана разными генами. Rao-Arelli and Anand (1988) Crop Sci. 28:650-2. Устойчивость к расе 3, по-видимому, управляется тремя или четырьмя различными генами. Там же; см. также Mansur et al., (1993) Crop Sci. 33:1249-53. Одним из основных QTL (локусов количественных признаков), который локализован на группе сцепления G, является rhg1. Concibidio et al. (1996) Theor.Appl.Genet. 93:234-41. Другие QTLs локализованы на группах сцепления A2, C1, M, D, J, L25, L26 и K. Там же; патент США 5491081. QTLs, определяющие устойчивость к SCN, проявляют себя в зависимости от расы, по крайней мере с учетом различных пропорций общего изменения фенотипа в отношении различных рас SCN. Concibido et al. (1997) Crop.Sci. 37:258-64. Однако, локус rhg1 на группе сцепления G может быть необходим для проявления устойчивости к любой из выявленных рас SCN. Но см. Qui et al. (1999) Theor. Appl. Genet. 98:356-64.
Маркеры, которые сцеплены с признаками устойчивости к SCN, включают RFLPs (полиморфизмы длин рестрикционных фрагментов), SSRs (простые повторяющиеся последовательности) и SNPs (одиночные нуклеотидные полиморфизмы). Маркеры SNP, идентифицированные в настоящем изобретении, могут применяться для генотипирования устойчивости к SCN с целью содействия программе выведения устойчивых сортов. Использование раскрытых в настоящей заявке SNP маркеров для осуществления генотипирования устойчивости к SCN при содействии селекционной программе обеспечивает: экономию средств и времени, ранний отбор желаемого потомства и более точное и быстрое продвижение на рынок SCN-устойчивых сортов сои.
СОДЕРЖАНИЕ ИЗОБРЕТЕНИЯ
Молекулярные маркеры, которые сцеплены с SCN-устойчивым фенотипом, могут применяться для облегчения селекции с использованием маркеров для получения признака устойчивости к SCN у сои. Селекция с использованием маркеров обеспечивает значительные преимущества с точки зрения времени, стоимости и трудозатрат по сравнению с SCN-фенотипированием. В настоящем изобретении было неожиданно обнаружено, что среди 15 SNP маркеров, выявленных внутри или вблизи от областей QTL в геноме сои, ответственных за устойчивость к SCN, которые были полиморфными в генотипах родителей, только три оказались сцеплены с признаком устойчивости к SCN. Далее, три этих SNP-маркера приносят значительную пользу при селекции SCN-устойчивых сортов сои с использованием маркеров.
В настоящей заявке описаны содержащиеся в нуклеиновых кислотах молекулярные маркеры, которые сцеплены (например, сцеплены, тесно сцеплены или исключительно тесно сцеплены) с SCN-устойчивым фенотипом. В конкретных примерах эти молекулярные маркеры могут являться SNP-маркерами. Кроме того, в настоящей заявке описаны способы применения содержащихся в нуклеиновых кислотах молекулярных маркеров, которые сцеплены с SCN-устойчивым фенотипом, например, и не ограничиваясь этим, для выявления растений с SCN-устойчивым фенотипом, для введения SCN-устойчивого фенотипа в генотипы новых растений (например, путем селекции с использованием маркеров или генетической трансформации) и для культивирования растений, с высокой вероятностью имеющих SCN-устойчивый фенотип.
Кроме того, описаны средства для введения SCN-устойчивого фенотипа в растения сои, и средства для идентификации растений, имеющих SCN-устойчивый фенотип. В некоторых примерах, средство для введения SCN-устойчивого фенотипа в растения сои может представлять собой маркер, который сцеплен (например, сцеплен, тесно сцеплен или исключительно тесно сцеплен) с SCN-устойчивым фенотипом. В некоторых примерах, средство идентификации растений, имеющих SCN-устойчивый фенотип, может представлять собой зонд, который специфично гибридизуется с маркером, который сцеплен (например, сцеплен, тесно сцеплен или исключительно тесно сцеплен) с SCN-устойчивым фенотипом.
Кроме того, в настоящем изобретении описаны растения и растительные материалы, которые получены из растений, имеющих SCN-устойчивый фенотип, выявленный с помощью молекулярных маркеров, описанных в настоящей заявке. Так, например, описаны растения сои, которые получены селекцией с применением одного или нескольких молекулярных маркеров, которые сцеплены с SCN-устойчивым фенотипом.
КРАТКОЕ ОПИСАНИЕ ИЛЛЮСТРАТИВНОГО МАТЕРИАЛА
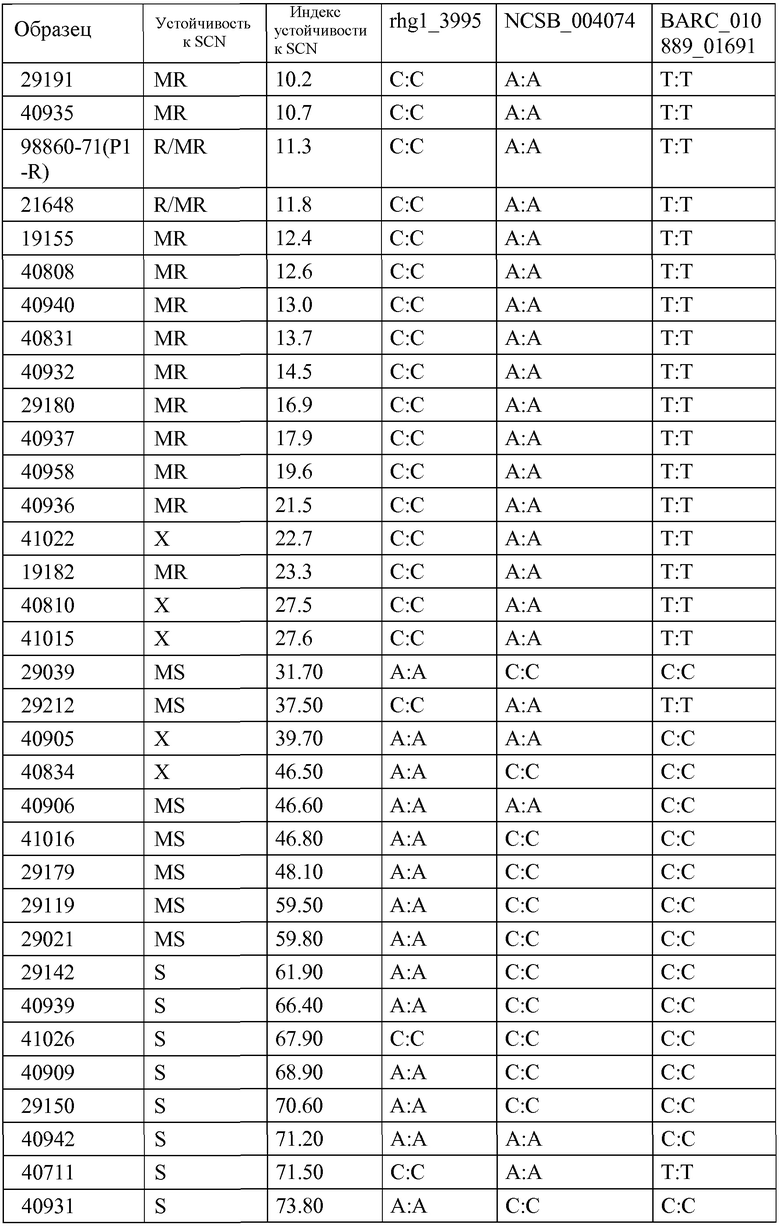
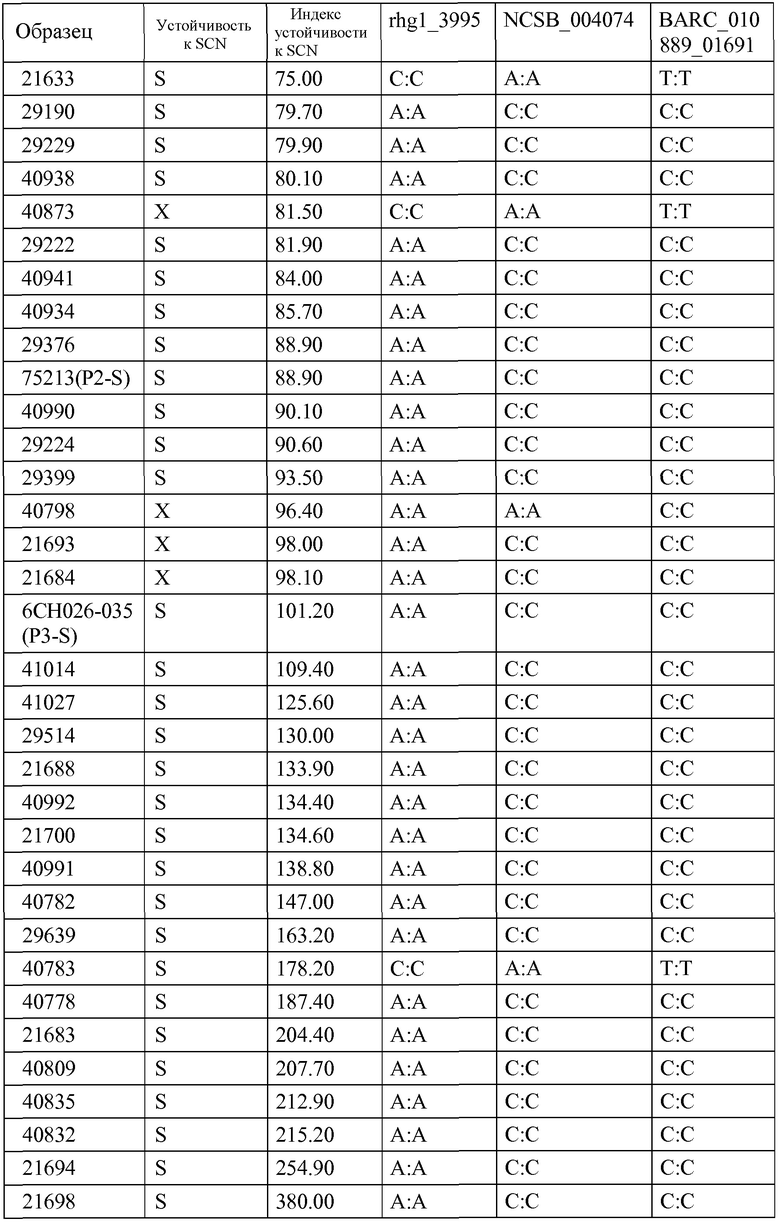
На Фиг. 1a и 1b приведен список QTLs, связанных с устойчивостью к SCN, которые приведены в литературе по SCN.
На Фиг.2 приведен геном сои, включая хромосомы и группы сцепления (LGs).
На Фиг.3 представлена хромосома сои 18 (группа сцепления G), а также QTLs и интервалы QTL, связанные с устойчивостью к SCN, и локализованные в них SNPs.
На Фиг.4 представлена хромосома сои 8 (группа сцепления A2), а также QTLs и интервалы QTL, связанные с устойчивостью к SCN, и локализованные в них SNPs.
На Фиг.5 представлена хромосома сои 11 (группа сцепления B1), а также QTLs и интервалы QTL, связанные с устойчивостью к SCN, и локализованные в них SNPs.
На Фиг.6 представлена хромосома сои 20 (группа сцепления I), а также QTLs и интервалы QTL, связанные с устойчивостью к SCN, и локализованные в них SNPs.
На Фиг.7 изображены кластеры из 24 культурных сортов или родительских линий сои, устойчивых или неустойчивых к SCN, на четырех локусах SNP. Кроме того представлена таблица, в которой показаны 24 культурных сорта сои и использованное SCN-картирование родителей. В приведенной таблице первая строка примеров и два последних примера во второй строке неустойчивы к SCN (зеленые), а первые 10 примеров во второй строке устойчивы к SCN (желтые). Три последних образца во втором ряду являлись родительскими линиями двух популяций для SCN-картирования.
На Фиг.8 показаны кластеры из 96 линий на трех локусах SNPs, которые демонстрируют косегрегацию с признаком устойчивости к SCN.
На Фиг.9 показано распределение индексов устойчивости к SCN, присвоенных популяциям для картирования. На гистограмме отражен диапазон от 0,01 до 3,8 со средним значением 0,63 и медианным значением 0,465.
СПИСКИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Последовательности нуклеиновых кислот, приведенные в приложенных к заявке списках последовательностей, показаны с применением стандартных буквенных сокращений для нуклеотидных оснований, которые определены в 37 C.F.R. § 1.822. Для каждой из последовательностей нуклеиновых кислот показана только одна нить, но предполагается, что комплементарная нить включена посредством ссылки на показанную нить. В приложенных списках последовательностей:
SEQ ID NO:1 демонстрирует последовательность праймера, применяемого в анализе с помощью конкурентной аллель-специфичной системы генотипирования PCR SNP KBiosciences (KASParTM), которая специфична для аллели rhg1-3995: GAAGGTGACCAAGTTCATGCTGGAATT ATGTTGGGTTTTTTTTCTTTCTGT.
SEQ ID NO:2 демонстрирует последовательность второго праймера, используемого в анализе KASParTM, которая специфична для аллели rhg1-3995: GAAGGTCGGAGTCAACGGATTGAATTATGTTGGGTTTT TTTTCTTTCTGG.
SEQ ID NO:3 демонстрирует последовательность общего обратного праймера, используемого в анализе KASParTM для rhg1-3995: GCCCAGAAAAAAGGGATAAATAACGGATA.
SEQ ID NO:4 демонстрирует последовательность праймера, используемого в анализе KASParTM, которая специфична для аллели NCSB_004074: GAAGGTGACCAAGTTCATGCTATTATGTTGTAACACAAATTTGCA CCTCAT.
SEQ ID NO:5 демонстрирует последовательность второго праймера, используемого в анализе KASParTM, которая специфична для аллели NCSB_004074: GAAGGTCGGAGTCAACGGATTATGTTGTAACACAAATTTGCACCTCAG.
SEQ ID NO:6 демонстрирует последовательность общего обратного праймера, используемого в анализе KASParTM, для NCSB_004074: CTATACAACTAAATCGTAATTCCATTGTAT.
SEQ ID NO:7 демонстрирует последовательность праймера, используемого в анализе KASParTM, которая специфична для аллели BARC_010889-01691: GAAGGTGACCAAGTTCATGCTGAAAAAATAAAATTGATCATCACATATGGTTAG.
SEQ ID NO:8 демонстрирует последовательность второго праймера, используемого в анализе KASParTM, которая специфична для аллели BARC_010889-01691: GAAGGTCGGAGTCAACGGATTGAAAAAATAAAATTGATCATCACATATGGTTAA.
SEQ ID NO:9 демонстрирует последовательность общего обратного праймера, используемого в анализе KASParTM для BARC_010889-01691: TAAGTGAGGGCAATGTATTAGTATYAAGTA.
SEQ ID NO:10 демонстрирует последовательность маркера NCSB_004074: CACGATTTTGTTGTGTTACATAAATTACTATACAACTAAATCGTAATTCCA TTGTATTAC [А/С] TGAGGTGCAAATTTGTGTTACAACATAATTGTAATTTTA TTGTACGATAAAAACTATAAC.
SEQ ID NO:11 демонстрирует последовательность маркера BARC_010889-01691:
CTCTTCACACCTTTAAGGAAGTTAGTACCATTCCACTATTCAAGTATTTTTT
TTAATTCAAAATTATTAAGTGAGGGCAATGTATTAGTATNAAGTA[C/T]TA
ACCATATGTGATGATCAATTTTATTTTTTCATGGCTTTGTCGAAAGTAACA
TTATATTGTGGTTTTAAATGAAAATCTGTGATTTGCAT.
SEQ ID NO:12 демонстрирует последовательность маркера rhg1-3995: TCTGATAACTATGACAGCATCTTCCAAGATAATGACTTCCAAGTTCCAACA
CTGGCTCTGTACATTTGAACTAATTTTATATCATTTATCTATTGTGATTGAA
ATATAAAATTGAAGTGATGTGAACAATACAAATCACATCTTGAATTAAAA
TATCTAACAACTGGAACAAATAAGAGGCCCAGAAAAAAGGGATAAATAA
CGGATAACAAG[A/C]CAGAAAGAAAAAAAACCCAACATAATTCCAACTTC
AAAATTCACTCAATAAAAAGTTTAACATGTAAATTTACTTGGAAACAAAA
CTCATAACCAATAATAATAATAATAAAAGAAATCAGTTTTATAGCATTAA
TTTGGGATGCTCTGCTTGTATGCAAATGGCACAACCTTACCCTCAAGATTG
CAAAACACAGATGAGTAACAGATGCAATGTGAATCAATAAAAAGTATTGT
TGCGTTGTTGATGACACAACCTTACTCATAAAAAATGCAT.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
I. Общее описание некоторых вариантов осуществления изобретения
Конкретные варианты осуществления изобретения включают три типовых SNP маркера (rhg1-3995, BARC_010889_01691 и NCSB_004074), которые демонстрируют косегрегацию с признаком устойчивости к цистообразующей соевой нематоде (SCN) в 96 исследованных линиях растений сои. Маркеры, которые проявляют косегрегацию с устойчивостью к SCN, сцеплены с этим признаком и, следовательно, могут быть полезны для селекции и выведения новых сортов с использованием этих маркеров. Кроме того, в заявке раскрыта стратегия, применяемая для выявления этих трех типовых SNP-маркеров, сцепленных с устойчивостью к SCN. Определены положения этих трех типовых SNP маркеров на физической карте генома Glycine max. С применением трех типовых маркеров, описанных в настоящей заявке, был разработан специфичный анализ, использующий конкурентную аллель-специфичную систему PCR SNP генотипирования KBiosciences (KASParTM), для быстрого и точного выявления растений, несущих признак устойчивости к SCN. Хотя варианты осуществления настоящего изобретения описаны применительно к трем типовым SNP маркерам, сцепленным с устойчивостью к SCN, специалист в данной области поймет, что используя методики, описанные в настоящей заявке, можно выявить дополнительные эквивалентные маркеры. SNP маркеры, сцепленные с устойчивостью к SCN, можно применять, например, в SCN-генотипировании, для выбора SCN-устойчивых индивидуальных растений из выведенных популяций сои.
Устойчивость к соевой цистообразующей нематоде (SCN) является очень сложным признаком. Заражение SCN может быть вызвано одной или несколькими различными расами Heterodera glycines, причем устойчивость к каждой из них может потребовать наличия различных генов устойчивости, локализованных на различных группах сцепления. Смотрите таблицу 1. Все три маркера, приведенных в таблице 1, локализованы на группе сцепления G. Считается, что ген(гены) устойчивости к SCN в группе сцепления G несут ответственность за устойчивость к расам 3 и 14.
Описанная в настоящей заявке стратегия применяется для выявления маркеров в других группах сцепления (например, A2, B1 и I), которые сцеплены с устойчивостью к SCN. Таким образом, изобретение относится также к способам идентификации таких маркеров. Разработанная общая стратегия применяется также для картирования других интересующих признаков. Эта стратегия является более эффективной, чем традиционные стратегии картирования, и может найти применение, в частности, в программах молекулярной селекции.
II. Термины
Популяция для картирования: в настоящем описании термин «популяция для картирования» может относиться к популяции растений, использованной для составления генетических карт. Популяции для картирования, как правило, получают в результате управляемого скрещивания родительских генотипов. Решения по поводу выбора родительских растений и плана скрещивания для получения популяции для картирования, а также типа используемых маркеров, зависят от гена, который предполагается картировать, доступности маркеров и молекулярной карты. Предки растений в популяции для картирования должны обладать достаточным разнообразием в отношении интересующего признака (признаков), как на уровне последовательностей нуклеиновых кислот, так и на уровне фенотипа. Разнообразие родительских последовательностей нуклеиновых кислот используется для того, чтобы следить за явлениями рекомбинации у растений популяции для картирования. Доступность информативных полиморфных маркеров зависит от количества вариантов последовательностей нуклеиновых кислот.
Обратное (возвратное) скрещивание: способы обратного скрещивания могут применяться для введения в растения определенной последовательности нуклеиновой кислоты. Методика обратного скрещивания в течение нескольких десятилетий широко применяется для придания растениям новых признаков. N.Jensen, Ed., Plant Breeding Methodology, John Wiley & Sons, Inc., 1988. В типовой методике обратного скрещивания исходный сорт интересующего растения (рекуррентного родителя) скрещивают со вторым сортом (не рекуррентным родителем), несущей интересующий ген, который необходимо передать. Полученное от этого скрещивания потомство вновь скрещивают с рекуррентным родителем и повторяют этот процесс до тех пор, пока не получают растение, в котором воспроизводятся практически все желаемые морфологические и физиологические характеристики рекуррентного растения, наряду с генами, перенесенными из не рекуррентного родителя.
Конкурентная аллель-специфичная система PCR SNP генотипирования KBiosciences (KASParTM): KASParTM представляет собой коммерчески доступную гомогенную флуоресцентную систему для определения SNP генотипов (KBiosciences Ltd., Hoddesdon, UK). Анализ с применением системы KASParTM включает SNP-специфичную «аналитическую смесь», которая содержит три немеченых праймера, и «реакционную смесь», которая содержит все остальные необходимые компоненты, например, универсальную флуоресцентную систему регистрации результатов. Кроме этих смесей, пользователю системы необходим, в числе прочего, планшет-ридер, способный регистрировать FRET (резонансный перенос энергии флуоресценции), микротитровальный планшет (планшеты) и образцы ДНК, в которых содержится примерно 5 нг/л ДНК.
Типовая методика анализа KASParTM включает стадии: разработки аллель-специфичного праймера (например, с использованием PrimerPickerTM, т.е. бесплатного сервиса, который доступен через Интернет на веб-сайте KBiosciences), приготовления реакционной смеси, включающей аллель-специфичные праймеры, смешивание этой реакционной смеси с образцами ДНК в микротитровальном планшете, термоциклирование, считывание планшетов на флуоресцентном планшет-ридере, построение графиков и обсчет данных по флуоресценции. Данные для всех образцов наносят на один двумерный график, где оси x- и y- соответствуют значениям флуоресценции групп FAM и VIC. Образцы, имеющие один и тот же кластер генотипов SNP, находятся на графике вместе (т.е. A/A, A/a и a/a). Более подробную техническую информацию о системе KASPar, включая руководство по решению типичных проблем, можно получить у KBiosciences Ltd. (например, KASPar SNP Genotyping System Reagent Manual (Руководство по реагентам системы SNP генотипирования KASPar)).
Сцепленные, тесно сцепленные и исключительно тесно сцепленные: в настоящем описании сцепление между генами и маркерами может относиться к явлению, при котором гены или маркеры на хромосоме демонстрируют определенную вероятность совместно передаваться индивидуумам в следующем поколении. Чем ближе друг к другу находятся два гена или маркера, тем ближе к единице становится эта вероятность. Таким образом, термин «сцепленный» может относиться к одному или нескольким генам или маркерам, которые передаются по наследству вместе с другим геном с вероятностью более 0,5 (которая ожидается от независимого распределения генов, если маркеры/гены локализованы на различных хромосомах). Если наличие гена вносит вклад в фенотип индивидуума, можно сказать, что маркеры, которые сцеплены с этим геном, сцеплены с фенотипом. Таким образом, термин «сцепленный» может относиться к взаимосвязи между маркером и геном или между маркером и фенотипом.
Поскольку близость двух генов или маркеров на хромосоме непосредственно связана с вероятностью совместной передачи этих генов или маркеров индивидуумам следующего поколения, термин «сцепленный» может также относиться в настоящей заявке к одному или нескольким генам или маркерам, которые расположены в пределах примерно 2,0 Mb (MegaBase, млн.пар оснований) друг от друга на одной и той же хромосоме.
Таким образом, «сцепленные» гены или маркеры могут быть отделены друг от друга примерно 2,1 Mb, 2,00 Mb, примерно 1,95 Mb, примерно 1,90 Mb, примерно 1,85 Mb, примерно 1,80 Mb, примерно 1,75 Mb, примерно 1,70 Mb, примерно 1,65 Mb, примерно 1,60 Mb, примерно 1,55 Mb, примерно 1,50 Mb, примерно 1,45 Mb, примерно 1,40 Mb, примерно 1,35 Mb, примерно 1,30 Mb, примерно 1,25 Mb, примерно 1,20 Mb, примерно 1,15 Mb, примерно 1,10 Mb, примерно 1,05 Mb, примерно 1,00 Mb, примерно 0,95 Mb, примерно 0,90 Mb, примерно 0,85 Mb, примерно 0,80 Mb, примерно 0,75 Mb, примерно 0,70 Mb, примерно 0,65 Mb, примерно 0,60 Mb, примерно 0,55 Mb, примерно 0,50 Mb, примерно 0,45 Mb, примерно 0,40 Mb, примерно 0,35 Mb, примерно 0,30 Mb, примерно 0,25 Mb, примерно 0,20 Mb, примерно 0,15 Mb, примерно 0,10 Mb, примерно 0,05 Mb, примерно 0,025 Mb и примерно 0,01 Mb. Конкретные примеры маркеров, которые «сцеплены» с SCN-устойчивым фенотипом у растений сои, включают нуклеотидные последовательности на хромосоме 18 генома сои.
Термин «тесно сцепленные» в настоящем описании может относиться к одному или нескольким генам или маркерам, которые расположены в пределах примерно 0,5 Mb друг от друга на одной и той же хромосоме. Таким образом, два «тесно сцепленных» гена или маркера могут быть отделены друг от друга примерно 0,6 Mb, примерно 0,55 Mb, 0,5 Mb, примерно 0,45 Mb, примерно 0,4 Mb, примерно 0,35 Mb, примерно 0,3 Mb, примерно 0,25 Mb, примерно 0,2 Mb, примерно 0,15 Mb, примерно 0,1 Mb и примерно 0,05 Mb.
Термин «исключительно тесно сцепленные» в настоящем описании может относиться к одному или нескольким генам или маркерам, которые расположены в пределах примерно 100 Kb друг от друга на одной и той же хромосоме. Таким образом, два «исключительно тесно сцепленных» гена или маркера могут быть отделены друг от друга примерно 125 Kb, примерно 120 Kb, примерно 115 Kb, примерно 110 Kb, примерно 105 Kb, 100 Kb, примерно 95 Kb, примерно 90 Kb, примерно 85 Kb, примерно 80 Kb, примерно 75 Kb, примерно 70 Kb, примерно 65 Kb, примерно 60 Kb, примерно 55 Kb, примерно 50 Kb, примерно 45 Kb, примерно 40 Kb, примерно 35 Kb, примерно 30 Kb, примерно 25 Kb, примерно 20 Kb, примерно 15 Kb, примерно 10 Kb, примерно 5 Kb и примерно 1 Kb.
Конкретные примеры маркеров, которые «исключительно тесно сцеплены» с SCN-устойчивым фенотипом в растениях сои, включают rhg1-3995, BARC_010889_01691 и NCSB_004074.
В связи с изложенным выше, следует понять, что маркеры, сцепленные с конкретным геном или фенотипом, включают те маркеры, которые тесно сцеплены, и те маркеры, которые исключительно тесно сцеплены с этим геном или фенотипом. Сцепленные, тесно сцепленные и исключительно тесно сцепленные генетические маркеры SCN-устойчивого фенотипа могут найти применение в программах селекции с использованием маркеров для выявления SCN-устойчивых сортов сои и для воспроизведения этого признака в других сортах сои и придания им устойчивости к SCN.
Локус: в настоящем описании термин «локус» относится к положению на геноме, которое соответствует определенной характеристике (например, признаку). SNP-локус определяется с помощью зонда, который гибридизуется с ДНК, находящейся в этом локусе.
Маркер: в настоящем описании термин «маркер» относится к гену или нуклеотидной последовательности, которые могут применяться для идентификации растений, имеющих конкретную аллель. Маркер можно описать как изменение в данном локусе генома. Генетический маркер может являться короткой ДНК-последовательностью, например, последовательностью, окружающей изменение в одной паре оснований (одиночный нуклеотидный полиморфизм или «SNP») или длинной ДНК-последовательностью, например, микросателлитом/простой повторяющейся последовательностью («SSR»). Термин «маркерная аллель» относится к варианту маркера, который присутствует у конкретного индивидуума.
Термин «маркер» в настоящем описании может относиться к клонированному сегменту хромосомной ДНК сои (например, сегменту, включающему rhg1-3995, BARC_010889_01691 или NCSB_004074) и может также или в качестве альтернативы относиться к молекуле ДНК, которая комплементарна клонированному сегменту хромосомной ДНК сои (например, ДНК, комплементарной сегменту, включающему rhg1-3995, BARC_010889_01691 или NCSB_004074).
В некоторых вариантах осуществления, присутствие маркера в растении может быть обнаружено с помощью зонда, представляющего собой нуклеиновую кислоту. Зонд может являться молекулой ДНК или молекулой РНК. РНК-зонды можно синтезировать способами, известными в технике, например, используя молекулу ДНК в качестве матрицы. Зонд может содержать часть или всю нуклеотидную последовательность маркера и, кроме того, соседнюю нуклеотидную последовательность из генома растения. Это именуется в настоящей заявке «зондом, включающим соседнюю последовательность». Эта дополнительная соседняя нуклеотидная последовательность именуется «расположенной выше» или «расположенной ниже» исходного маркера, в зависимости от того, находится ли соседняя нуклеотидная последовательность хромосомы растения на 5’ или 3’ стороне от исходного маркера, как это обычно подразумевается. Как понятно рядовому специалисту в данной области техники, способ получения дополнительной соседней нуклеотидной последовательности для включения в маркер можно повторять практически неограниченно (ограничением является только длина хромосомы), выявляя тем самым дополнительные маркеры на хромосоме. Все описанные выше маркеры могут применяться в некоторых вариантах осуществления настоящего изобретения.
Олигонуклеотидную последовательность зонда можно получать синтетически или с помощью клонирования. Подходящие векторы для клонирования хорошо известны специалисту в данной области техники. Олигонуклеотидный зонд может быть меченым или не меченым. Существует целый ряд методик для введения меток в молекулы нуклеиновых кислот, в том числе, например, но не ограничиваясь этим, введение радиоактивной метки с помощью ник-трансляции, случайное праймирование, наращивание цепи с помощью терминальной дезокситрансферазы и т.п., где используемые нуклеотиды помечены, например, радиоактивными атомами 32P. Другие подходящие метки включают, например, и не ограничиваясь этим: флуорофоры (например, FAM и VIC), ферменты, субстраты ферментов, кофакторы ферментов, ингибиторы ферментов и т.п. В качестве альтернативы, вместо применения метки, которая дает измеримый сигнал, сама по себе, либо в сочетании с другими реакционноспособными агентами, можно использовать лиганды, с которыми связываются рецепторы, причем эти рецепторы являются мечеными (например, перечисленными выше метками) и обеспечивают получение измеримых сигналов, либо сами по себе, либо в сочетании с другими реагентами. Смотрите, например, Leary et al., (1983) Proc.Natl.Acad.Sci. USA 80:4045-9.
Зонд может содержать нуклеотидную последовательность, которая не является соседней по отношению к первоначальному маркеру, и такой зонд именуется в настоящей заявке «зондом, включающим несвязанную последовательность». Несвязанная последовательность такого зонда располагается на геноме достаточно близко к последовательности первоначального маркера, так что зонд, включающий несвязанную последовательность, генетически сцеплен с тем же самым геном или признаком (например, с устойчивостью к SCN). Например, в некоторых вариантах осуществления изобретения, зонд, включающий несвязанную последовательность, расположен в пределах 500 kb, 450 kb, 400 kb, 350 kb, 300 kb, 250 kb, 200 kb, 150 kb, 125 kb, 100 kb, 0,9 kb, 0,8 kb, 0,7 kb, 0,6 kb, 0,5 kb, 0,4 kb, 0,3 kb, 0,2 kb или 0,1 kb от первоначального маркера на геноме сои.
Зонд может являться точной копией маркера, который необходимо обнаружить. Кроме того, зонд может являться молекулой нуклеиновой кислоты, включающей или состоящей из нуклеотидной последовательности, которая практически идентична клонированному сегменту хромосомной ДНК интересующего организма (например, сои). В настоящей заявке термин «практически идентичный» может относиться к нуклеотидным последовательностям, которые идентичны более чем на 85%. Например, практически идентичная нуклеотидная последовательность может быть идентична исходной последовательности на 85,5%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 99,5%.
Зонд может также представлять собой молекулу нуклеиновой кислоты, которая «способна специфично гибридизоваться» или «специфично комплементарна» точной копии маркера, который необходимо обнаружить («ДНК мишени»). Термины «способна специфично гибридизоваться» и «специфично комплементарна» указывают на степень комплементарности, достаточную для того, чтобы между молекулой нуклеиновой кислоты и ДНК мишенью возникало устойчивое и специфичное связывание. Молекула нуклеиновой кислоты не должна быть на 100% комплементарна целевой последовательности, для того, чтобы была возможна специфичная гибридизация. Молекулы нуклеиновых кислот способны к специфичной гибридизации, если имеется достаточная степень комплементарности для того, чтобы избежать неспецифичного связывания нуклеиновой кислоты с нецелевыми последовательностями в условиях, когда желательно специфичное связывание, например, в жестких условиях гибридизации.
Условия гибридизации, означающие ту или иную конкретную степень жесткости, будут меняться в зависимости от природы выбранной методики гибридизации, а также от структуры и длины гибридизующихся последовательностей нуклеиновых кислот. Жесткость условий гибридизации, главным образом, будут определять температура гибридизации и ионная сила (в частности, концентрация Na+ и/или Mg++) буфера для гибридизации, хотя время промывания также оказывает влияние на жесткость условий. Рядовому специалисту в данной области техники известны способы расчета условий гибридизации, требующихся для достижения конкретной степени жесткости, и эти способы обсуждаются, например, в Sambrook et al., (ed.) Molecular Cloning: A Laboratory Manual, 2nd ed., vol. 1-3, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 1989, главы 9 и 11; и Hames and Higgins (eds.) Nucleic Acid Hybridization, IRL Press, Oxford, 1985. Более подробные инструкции и указания, относящиеся к гибридизации нуклеиновых кислот, можно найти, например, в Tijssen, "Overview of principles of hybridization и the strategy of nucleic acid probe assays," in Laboratory Techniques в Biochemistry и Molecular Biology - Hybridization with Nucleic Acid Probes, Part I, Chapter 2, Elsevier, NY, 1993; и Ausubel et al., Eds., Current Protocols в Molecular Biology, Chapter 2, Greene Publishing and Wiley-Interscience, NY, 1995.
В настоящем описании, термин «жесткие условия» охватывает условия, в которых гибридизация будет происходить только в том случае, если имеется менее 50% несоответствий между гибридизующейся молекулой и ДНК мишенью. «Жесткие условия» включают дополнительные отдельные уровни жесткости. Так, например, в настоящей заявке условия «умеренной жесткости» представляют собой такие условия, в которых не будут гибридизоваться молекулы с более чем 50% несоответствием последовательностей; условия «высокой жесткости» представляют собой такие условия, в которых не будут гибридизоваться молекулы с более чем 20% несоответствием последовательностей; и условия «очень высокой жесткости» представляют собой такие условия, в которых не будут гибридизоваться молекулы с более чем 10% несоответствием последовательностей.
Приведенные ниже условия являются типовыми, не ограничивающими условиями проведения гибридизации.
Очень высокая жесткость (позволяет обнаружить последовательности с идентичностью не менее 90%): Гибридизация в 5х буфере SSC при 65°C в течение 16 часов; двукратное промывание 2х буфером SSC при комнатной температуре в течение 15 минут каждое; и двукратное промывание 0,5х буфером SSC при 65°C в течение 20 минут каждое.
Высокая жесткость (позволяет обнаружить последовательности с идентичностью не менее 80%): Гибридизация в 5х-6х буфере SSC при 65-70°C в течение 16-20 часов; двукратное промывание 2х буфером SSC при комнатной температуре в течение 5-20 минут каждое; и двукратное промывание 1х буфером SSC при 55-70°C в течение 30 минут каждое.
Умеренная жесткость (позволяет обнаружить последовательности с идентичностью не менее 50%): Гибридизация в 6х буфере SSC при температуре от комнатной до 55°C в течение 16-20 часов; как минимум двукратное промывание 2х-3х буфером SSC при температуре от комнатной до 55°C в течение 20-30 минут каждое.
Что касается всех зондов, обсуждавшихся выше, зонд может включать дополнительные последовательности нуклеиновых кислот, например, промоторы, сигналы транскрипции и/или векторные последовательности. Любые из обсуждавшихся выше зондов могут применяться для определения дополнительных маркеров, которые тесно сцеплены с генами, определяющими устойчивость к SCN, и выявленные таким образом маркеры могут быть эквивалентны типовым маркерам, указанным в настоящем описании и, следовательно, входят в объем настоящего изобретения.
Селекция с использованием маркера: в настоящем описании, термин «селекция с использованием маркера» может относиться к подходу к селекции, направленному на получение одного или нескольких сложных признаков (например, устойчивости к SCN). В современной практике селекционеры растений пытаются выявить легко обнаруживаемые признаки, например, цвет цветков, внешний вид оболочки семян или варианты изоферментов, которые сцеплены с признаками, желаемыми с агрономической точки зрения. Затем специалисты по селекции при отборе растений руководствуются этим агрономическим признаком, осуществляя выбор по легко обнаруживаемому признаку. Однако существует очень небольшое число таких генетически сцепленных взаимосвязей, которые можно применять при селекции растений.
Селекция с использованием маркера обеспечивает эффективный с точки зрения экономии времени и затрат способ улучшения сортов растений. Некоторые примеры применения селекции с использованием маркера включают применение изоферментных маркеров. Смотрите, например, Tanksley and Orton, eds. (1983) Izozymes in Plant Breeding and Genetics, Amsterdam: Elsevier. Одним из примеров является изоферментный маркер, сцепленный с геном устойчивости к нематодным вредителям у томатов. Эта устойчивость, управляемая геном, обозначаемым Mi, локализована на хромосоме 6 томатов, и она очень тесно сцеплена с Aps1, т.е. изоферментом кислой фосфатазой. Применение изоферментного маркера Aps1 для косвенного выбора гена Mi, обеспечивает определенные преимущества при выделении необходимых растений из популяции, поскольку изофермент можно однозначно определить с помощью стандартных электрофоретических методик; содержание изоферментного маркера можно определить в тканях сеянцев, что избавляет от необходимости выращивать растения до взрослого состояния; и кодоминантность аллелей изоферментного маркера позволяет различить гомозиготные и гетерозиготные растения. Смотрите Rick (1983) в Tanksley and Orton выше.
Локус количественных признаков: в настоящей заявке термин «локус количественных признаков» (QTL) может относиться к отрезкам ДНК, которые были идентифицированы как вероятные ДНК-последовательности (например, гены, не кодирующие последовательности и/или межгенные последовательности), которые обусловливают количественный признак или фенотип, меняющийся по своей величине, который можно приписать взаимодействию между двумя или несколькими ДНК-последовательностями (например, генами, не кодирующими последовательностями и/или межгенными последовательностями) или продуктами их экспрессии, и их окружения. Локусы количественных признаков (QTLs) могут быть идентифицированы на молекулярном уровне, что способствует созданию карты областей генома, которые содержат последовательности, вовлеченные в задание того или иного количественного признака.
Термин «интервал QTL» в настоящем описании может относиться к отрезкам ДНК, которые сцеплены с генами, обуславливающими QTL признак. Интервал QTL обычно, но не обязательно, больше, чем сам QTL. Интервал QTL может включать отрезки ДНК, находящиеся в 5’ и/или 3’ направлении относительно QTL.
Идентичность последовательностей: термин «идентичность последовательностей» или «идентичность» в настоящей заявке в контексте двух последовательностей нуклеиновых кислот или полипептидных последовательностей, может относиться к остаткам в этих двух последовательностях, которые являются одинаковыми, при совмещении последовательностей, которое позволяет добиться максимального соответствия в заданном окне сравнения.
Термин «процентная доля идентичности последовательностей» может относиться к величине, определенной путем сравнения двух оптимально совмещенных последовательностей (например, последовательностей нуклеиновых кислот) в окне сравнения, где фрагмент последовательности в окне сравнения может включать вставки или делеции (т.е. гэпы) по отношению к последовательности сравнения (которая не включает вставки или делеции) для оптимального совмещения двух упомянутых последовательностей. Эту процентную долю вычисляют путем определения числа положений, где в обеих последовательностях присутствуют идентичные нуклеотиды или аминокислотные остатки, с получением количества совпадающих положений, деления количества совпадающих положений на общее количество положений в окне сравнения и умножения полученного результата на 100, что дает процентную долю идентичности последовательностей.
Методики совмещения последовательностей для их сравнения хорошо известны в технике. Различные программы и алгоритмы для совмещения описаны, например, в: Smith и Waterman (1981) Adv. Appl. Math. 2:482; Needleman и Wunsch (1970) J. Mol. ВЫ. 48:443; Pearson и Lipman (1988) Proc. Natl. Acad. Sci. U.S.A. 85:2444; Higgins и Sharp (1988) Gene 1Ъ:2Ъ1-АА; Higgins и Sharp (1989) CABIOS 5:151-3; Corpet et al. (1988) Nucleic Acids Res. 16:10881-90; Huang et al. (1992) Сотр. Appl. Biosci. 8:155-65; Pearson et al. (1994) Methods Mol. Biol. 24:307-31; Tatiana et al. (1999) FEMS Microbiol. Lett. 174:247-50. Подробное рассмотрение методик совмещения последовательностей и вычисления гомологичности можно найти, например, в Altschul et al. (1990) J.Mol.Biol.215:403-10.
Разработанное National Center for Biotechnology Information (Национальным центром биотехнологической информации, NCBI) семейство программ Basic Local Alignment Search Tool (BLASTTM; Altschul et al. (1990)) доступно из нескольких источников, включая National Center for Biotechnology Information (Bethesda, MD), а также из Интернета, для использования в связке с несколькими программами анализа последовательностей. Описание того, как определить идентичность последовательностей с помощью этих программ доступно в Интернете в разделе “help” («помощи») для BLASTTM. Для сравнения последовательностей нуклеиновых кислот можно применять функцию “Blast 2 sequences” программы BLASTTM (Blastn), используя стандартную матрицу BLOSUM62 с установленными по умолчанию параметрами. Последовательности нуклеиновых кислот, обладающие большим сходством с эталонной последовательностью, покажут увеличение процентной доли идентичности при оценке данным способом.
Одиночный нуклеотидный полиморфизм: в настоящем описании термин «одиночный нуклеотидный полиморфизм» (SNP) может относиться к варианту последовательности ДНК, возникающему, когда один нуклеотид в геноме (или другой общей последовательности) отличается у двух особей одного вида или в парных хромосомах одного индивидуума. В популяции SNPs могут определяться частотой минорной аллели, которая является самой низкой частотой аллелей, в локусе, наблюдаемой у данной конкретной популяции. Эта частота будет именно меньшей из двух частот аллелей для одиночных нуклеотидных полиморфизмов. Ожидается, что различные популяции будут демонстрировать по крайней мере незначительные различия частот аллелей. Конкретные популяции могут демонстрировать значительные различия частот аллелей. В некоторых примерах, маркеры, сцепленные с устойчивостью к SCN, являются SNP-маркерами.
SNPs могут находиться в пределах кодирующих последовательностей генов, не кодирующих областей генов или межгенных областях между генами. SNPs, находящиеся в кодирующих последовательностях, не будут обязательно изменять аминокислотную последовательность образующегося белка из-за отклонения генетического кода. SNP, при котором обе формы ведут к образованию одной и той же полипептидной последовательности, именуют «синонимичными» (иногда они именуются молчащими мутациями). Если образуются различные полипептидные последовательности, то такой полиморфизм именуется «не синонимичным». Не синонимичное изменение может быть либо миссенс-мутацией, либо нонсенс-мутацией, где миссенс-мутация приводит к другому аминокислотному остатку, а нонсенс-мутация приводит к появлению кодона преждевременной остановки трансляции. SNPs, которые не находятся в областях, кодирующих белки, все же могут иметь определенные последствия для сплайсинга генов, связывания факторов транскрипции или последовательности не кодирующей РНК. SNPs обычно бывают биаллельными, и поэтому они легко выявляются у растений и животных. Sachidanandam (2001) Nature 409:928-33.
Признак или фенотип: термины «признак» или «фенотип» в настоящей заявке являются взаимозаменяемыми. Для целей настоящего изобретения, особый интерес представляет признак устойчивости к SCN.
III. Идентификация маркеров, связанных с интересующим признаком, основанная на исследовании QTL
A. Краткий обзор
В некоторых вариантах осуществления, определяют положение признака (например, устойчивости к SCN) на генетической карте, применяя стратегию, которая отличается от традиционного подхода к картированию. Например, картирование признака можно осуществить согласно стратегии, которую ради удобства можно описать, как включающую четыре стадии. На первой стадии можно определить целевые области QTL-интервалов, которые соответствуют картируемому признаку. На второй стадии могут быть выбраны маркеры (например, SNP-маркеры), которые локализованы в пределах или вблизи от выявленных QTL-интервалов целевого генома (например, генома сои). На третьей стадии могут быть разработаны специфичные праймеры, которые облегчают генотипирование индивидуальных субъектов с точки зрения выбранных маркеров. В конкретных примерах, специфичные праймеры разрабатываются для применения в системе генотипирования KASParTM. На четвертой стадии может быть осуществлен скрининг популяций, которые демонстрируют расщепление по искомому признаку, с применением специфичных праймеров, для выявления тех маркеров, которые сцеплены с признаком.
B. Маркеры, сцепленные с интересующим признаком, и их идентификация
Определение целевых областей интервалов QTL и идентификация маркеров
QTLs можно определять с применением любых методик, имеющихся в распоряжении специалистов в данной области техники. Например, физическое положение QTL, который соответствует конкретному интересующему признаку, на первом этапе может быть определено с учетом расположения генов, о которых известно, что они вносят вклад в конкретный признак. В некоторых вариантах осуществления, гены устойчивости к SCN могут быть выявлены, как минимум, в четырех областях на хромосомах 8, 11, 18 и 20, соответственно. Смотрите, например, Concibido et al. (1996) Theor. Appl. Genet. 93:234-41, Concibido et al. (1997) Crop Sci. 37:258-64, Meksem et al. (1999) Theor. Appl. Genet. 99:1131-42, Qui et al. (1999) Theor. Appl. Genet. 98:356-64, Meksem et al. (2001) Mol. Breeding 7:63-71, Li et al. (2009) Mol. Breeding 24:63-76, Wu et al. (2009) Theor. Appl. Genet. 118:1093-105; патенты США 5491081, 6096944, 6162967, 6271437, 6284948, 6300541, 6538175, 7154021, 7485770; U.S.S.N.s 20020129402, 20020144310, 20030005491, 20030135881, 20060225150, 20060253919, 20080072352 и 20090100537; а также международные публикации PCT №№ WO 1995020669A2, WO 2001051627A2 и WO 2008153804A2. В некоторых вариантах осуществления, первоначально идентифицированные QTLs группируют или разделяют, получая менее сложный или обширный перечень QTLs, которые могут иметь те же самые или другие границы внутри генома, что и границы первоначально идентифицированных QTLs.
В некоторых вариантах осуществления, может быть выбрана область ДНК, которая, вероятно, содержит маркеры, сцепленные с признаком, определяемым QTL. Эта область может именоваться интервалом QTL. Например, интервал QTL может представлять собой область ДНК, которая включает QTL и дополнительную геномную ДНК, которая находится рядом с QTL в любом из направлений 5’ и 3’, или в обоих направлениях. В некоторых вариантах осуществлениях, интервал QTL может иметь протяженность примерно 4 Mb, примерно 3,5 Mb, примерно 3 Mb, примерно 2,5 Mb, примерно 2 Mb, примерно 1,5 Mb или примерно 1 Mb.
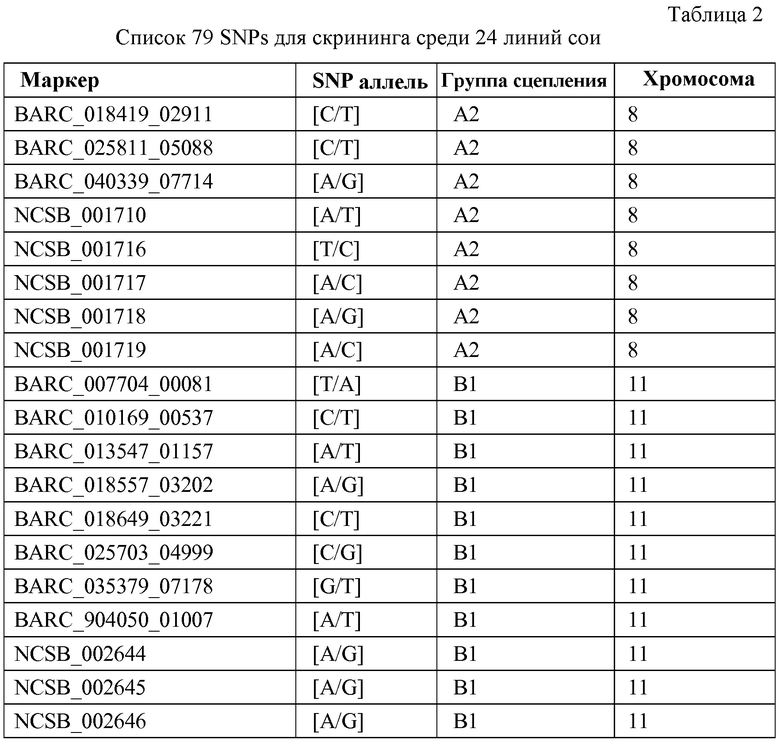
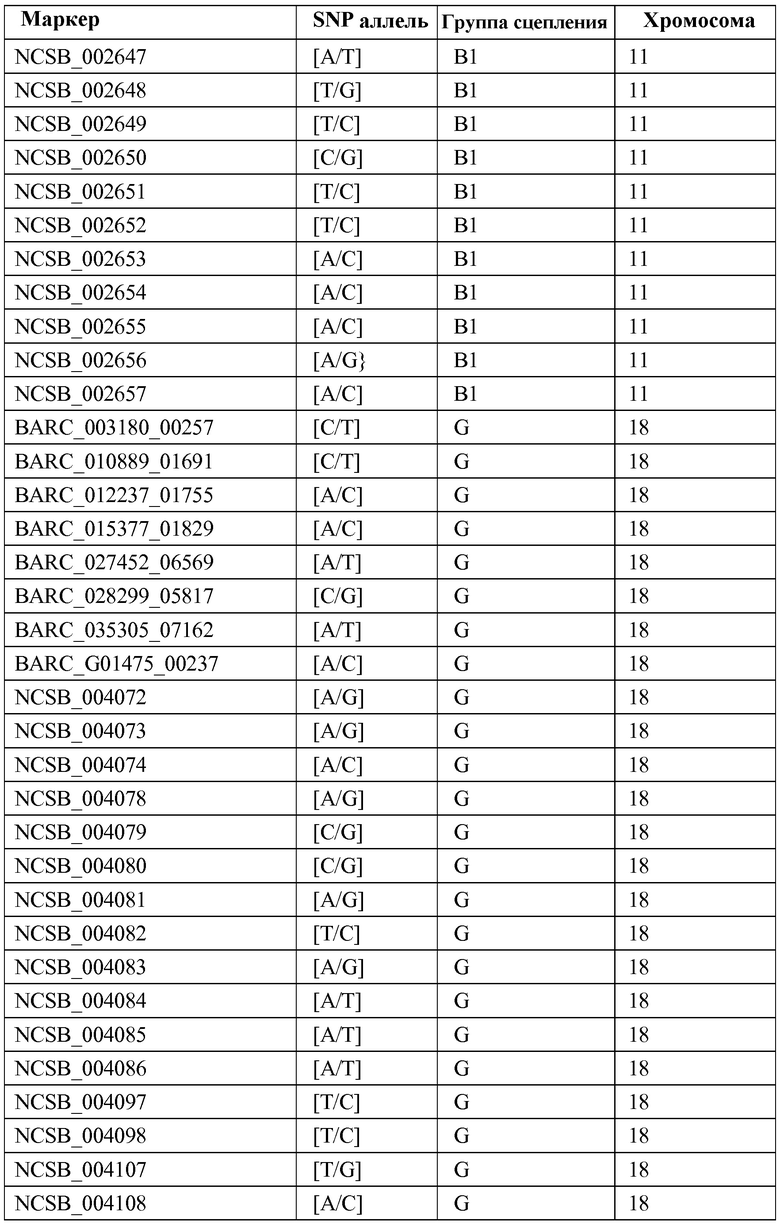
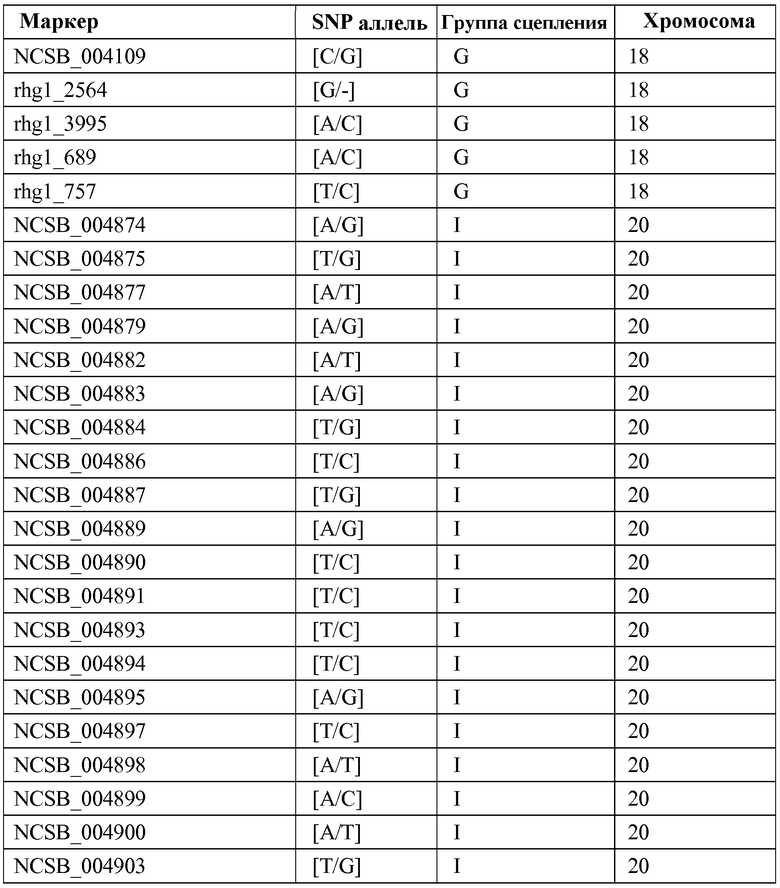
В конкретных вариантах осуществления, можно исследовать целевой геном для выявления маркеров, которые физически локализованы, внутри, около или между QTLs и интервалами QTL. Если для целевого генома имеется справочная карта, включающая места расположения известных маркеров, для идентификации маркеров можно использовать эту справочную карту. Последовательности нуклеиновых кислот целевого генома можно также исследовать, например, с помощью программного обеспечения, например, BLASTTM. В некоторых вариантах осуществления, могут быть идентифицированы SNP-маркеры. В некоторых вариантах осуществления могут быть идентифицированы маркеры, которые физически локализованы внутри, около или между QTLs и интервалами QTL генома сои, которые соответствуют признаку устойчивости к SCN. В конкретных примерах, выявленные SNP-маркеры, которые физически локализованы внутри, около или между QTLs и интервалами QTL генома сои, которые соответствуют признаку устойчивости к SCN, могут быть выбраны из группы, состоящей из маркеров, перечисленных в таблице 2.
В других вариантах осуществления, из выявленных маркеров могут быть выбраны определенные маркеры, которые физически локализованы внутри, около или между QTLs и интервалами QTL, которые соответствуют интересующему признаку, причем эти маркеры являются полиморфными в родительских линиях из которых будет получена популяция для картирования. Полиморфизм данного маркера в родительских линиях непосредственно связан с остаточными явлениями рекомбинации в популяции для картирования, полученной из родительских линий.
В конкретных вариантах осуществления, в родительских линиях сои выбирают полиморфные маркеры для проведения скрининга SCN-устойчивых популяций для картирования, с целью определения какой из полиморфных маркеров, если таковой имеется, сцеплен с признаком устойчивости к SCN. Эти маркеры могут расщепляться таким образом, что одна аллель SNP-маркера появляется исключительно у SCN-устойчивых индивидуумов, а другая аллель SNP-маркера появляется исключительно у SCN-неустойчивых индивидуумов. Популяции для картирования можно генерировать скрещиванием одного сорта, который является устойчивым к SCN, с другим сортом, который является неустойчивым к SCN. В различных вариантах осуществления, популяция для картирования может включать примерно 10, примерно 20, примерно 30, примерно 40, примерно 50, примерно 60, примерно 70, примерно 80, примерно 90, примерно 95, примерно 100, примерно 150, примерно 200, примерно 250, примерно 300, примерно 350, примерно 400, примерно 450, примерно 500 или более индивидуумов. В некоторых вариантах осуществления, SCN-устойчивую зародышевую плазму 98860-71 можно скрещивать с одной или несколькими SCN-неустойчивыми зародышевыми плазмами (например, 75213 и 6CH026-035), с целью создания популяций для картирования.
В некоторых вариантах осуществления, полиморфные маркеры могут являться одиночными нуклеотидными полиморфизмами (SNPs), сцепленными с интересующим признаком, или находящимися в гене или QTL, соответствующем интересующему признаку устойчивости к SCN. Эти SNP-маркеры можно обнаружить путем секвенирования области, содержащей этот ген или QTL, с использованием любых методик секвенирования ДНК, известных в технике, включая, но не ограничиваясь этим, секвенирование по Сэнгеру или высокопроизводительное секвенирование (секвенирование «нового поколения»), которые дают возможность осуществить чтение коротких или длинных последовательностей в интересующей области. В таких вариантах осуществления, если генотипирование с помощью секвенирования применяется для обнаружения SNP маркеров, праймеры, соответствующие фланкирующим последовательностям, области, содержащей SNPs в интересующем гене или QTL, могут применяться в качестве секвенирующих реагентов, для определения последовательности нуклеотидов в интересующем регионе. В этих вариантах осуществления, если различные генотипы используются для секвенирования интересующей области для обнаружения приведенных в настоящей заявке примеров SNPs, в дополнение к этим SNPs могут быть выявлены другие SNPs. В этих вариантах осуществления SNPs, приведенные в настоящей заявке в качестве примеров, сами по себе (индивидуальные SNPs) или в комбинации с другими SNPs, связанными с приведенными последовательностями (гаплотипы), могут применяться с целью дифференцирования генотипов для селекции растений с использованием маркеров по интересующей устойчивости к SCN.
Разработка праймера и скрининг сцепления
Могут быть разработаны олигонуклеотидные зонды (например, праймеры) для специфичного обнаружения маркеров, которые физически локализованы внутри, около или между QTLs и интервалами QTL, которые соответствуют интересующему признаку. Как правило, может быть разработан олигонуклеотидный зонд, который специфично гибридизуется только с одной аллелью маркера. В некоторых вариантах осуществления, для обнаружения SNP маркера осуществляют разработку двух олигонуклеотидных зондов, так чтобы каждый из них специфично гибридизовался с SNP-аллелью, с которой другой зонд не способен специфично гибридизоваться. Как понятно специалисту в данной области техники, длину или состав олигонуклеотидных зондов для конкретного маркера можно менять в соответствии с хорошо известными принципами, не делая этот зонд неспецифичным для одной аллели маркера.
В некоторых вариантах осуществления, олигонуклеотидные зонды могут являться праймерами. В конкретных вариантах осуществления, праймеры могут быть разработаны таким образом, чтобы подходить для обнаружения маркеров в системе генотипирования KASParTM. В конкретных вариантах осуществления, праймеры могут быть разработаны таким образом, чтобы подходить для обнаружения маркеров, сцепленных с SCN-устойчивым фенотипом у сои, с применением системы генотипирования KASParTM. В этих и других вариантах осуществления, система обнаружения может обеспечивать высокопроизводительный и удобный формат генотипирования индивидуальных растений в популяции для картирования, который может существенно облегчить идентификацию индивидуумов, несущих конкретный ген или признак, и, кроме того, может значительно облегчить реализацию или выполнение программы селекции с использованием маркеров.
В конкретных вариантах осуществления, олигонуклеотидные зонды могут являться праймерами, разработанными для обнаружения маркеров в системе генотипирования TAQMAN®. В этой методике используются праймеры, специфичные для маркера, расположенного в ближайшей окрестности гена устойчивости к SCN, и зонды с флуоресцентной меткой, содержащие одиночный нуклеотидный полиморфизм (SNP). SNP-зонд, связанный с устойчивостью к SCN, метят флуоресцентным красителем, например, FAM, в то время, как зонд, реагирующий на неустойчивость к SCN, метят другим флуоресцентным красителем, например VIC. Данные анализируют с точки зрения присутствия или отсутствия сигнала флуоресцентного красителя. Эта система обнаружения способна обеспечить высокопроизводительный и удобный формат, например, одновременное обнаружение сигналов генотипируемых индивидуумов в популяции для картирования, что может существенно облегчить выявление индивидуумов, несущих конкретный ген или признак и, кроме того, может значительно облегчить реализацию или выполнение программы селекции с применением маркеров.
В качестве эквивалентов любым маркерам, приведенным в настоящей заявке в качестве примера (например, маркерам, перечисленным в таблице 3, таким как, например, rhg1-3995, BARC_010889_01691 и NCSB_004074), можно выявить дополнительные маркеры, например, путем определения частоты рекомбинации между показанным маркером и дополнительным маркером. Это определение можно выполнить с применением способа ортогональных контрастов, основанного на методике Mather (1931), The Measurement of Linkage in Heredity, Methuen & Co., London, с последующим применением критерия максимального правдоподобия для определения частоты рекомбинаций. Allard (1956) Hilgardia 24:235-78. Если значение частоты рекомбинаций меньше или равно 0,10 (т.е. 10%), то дополнительный маркер считается эквивалентным данному, показанному в заявке, маркеру для целей применения в способах по настоящему изобретению.
В различных вариантах осуществления настоящего изобретения, можно выявить маркеры, которые сцеплены со всеми без исключения генами устойчивости к SCN. Кроме того, в различных вариантах осуществления изобретения могут быть выявлены маркеры, которые регулируют все без исключения локусы, вносящие вклад в устойчивость ко всем расам SCN HG.
Средством обеспечения устойчивости сои к SCN может являться аллель, содержащая SNP-маркер, причем обнаружение такой аллели, содержащей SNP-маркер, у растения сои, принадлежащего к зародышевой плазме 98860-71 или полученного из нее, обеспечивает, по крайней мере, достаточную долю уверенности в том, что растение, содержащее такую последовательность нуклеиновой кислоты, обладает SCN-устойчивым фенотипом. В некоторых примерах, средством обеспечения устойчивости растений сои к SCN является маркер, выбранный из группы, состоящей из маркеров, перечисленных в таблице 3. В конкретных примерах, средством обеспечения устойчивости растений сои к SCN является маркер, выбранный из группы, состоящей из rhg1-3995, BARC_010889_01691 и NCSB_004074.
Средством идентификации растений сои, имеющих SCN-устойчивый фенотип, может являться молекула, которая при добавлении к образцу, полученному из растения сои, принадлежащего к зародышевой плазме 98860-71, или полученного из нее, и имеющего SCN-устойчивый фенотип, обеспечит получение обнаружимого сигнала, причем это средство не даст такого сигнала при добавлении к образцу, полученному из растения сои, принадлежащего к зародышевой плазме 98860-71, или полученного из нее, которое не обладает SCN-устойчивым фенотипом. Специфичная гибридизация нуклеиновых кислот является обнаружимым сигналом, и, следовательно, зонд, являющийся нуклеиновой кислотой, который специфично гибридизуется с аллелью, несущей SNP-маркер, который сцеплен с SCN-устойчивым фенотипом, может являться средством идентификации растений сои, имеющих SCN-устойчивый фенотип. В некоторых примерах, средство идентификации растений сои, имеющих SCN-устойчивый фенотип, представляет собой зонд, который специфично гибридизуется с маркером, сцепленным с SCN-устойчивым фенотипом.
C. Способы применения маркеров, сцепленных с интересующим признаком
Способы, включающие применение молекулярных маркеров, содержащихся в нуклеиновых кислотах, которые сцеплены с интересующим признаком (например, устойчивостью растений сои к SCN) для выявления растений, обладающих интересующим признаком, могут обеспечивать экономию денежных средств селекционерами растений, поскольку эти способы могут устранять необходимость фенотипирования индивидуальных растений, полученных в ходе селекции (например, при скрещивании сортов сои, обладающих устойчивостью к SCN, с неустойчивыми сортами растений).
В конкретных вариантах осуществления, маркеры, сцепленные с устойчивостью к SCN у растений сои, могут применяться для переноса сегмента(ов) ДНК, который содержит одну или несколько детерминант устойчивости к SCN. В конкретных вариантах осуществления, эти маркеры могут быть выбраны из группы, включающей маркеры, перечисленные в таблице 3, а также маркеры, являющиеся их эквивалентами. В некоторых вариантах осуществления, маркер может быть выбран из группы, состоящей из rhg1-3995, BARC_010889_01691 и NCSB_004074. В некоторых вариантах осуществления, способ применения маркеров, сцепленных с устойчивостью к SCN у растений сои, для переноса сегмента(ов) ДНК, которые содержат одну или несколько детерминант устойчивости к SCN, может включать анализ геномной ДНК двух родительских растений с применением зондов, которые специфично гибридизуются с маркерами, сцепленными с SCN-устойчивым фенотипом; половое скрещивание генотипов двух родительских растений для получения популяции потомства и анализ этого потомства на наличие маркеров, сцепленных с SCN-устойчивым фенотипом; обратное скрещивание потомства, которое содержит маркеры, сцепленные с SCN-устойчивым фенотипом, с реципиентным генотипом для получения первой популяции, являющейся результатом обратного скрещивания, и затем продолжение программы обратного скрещивания до получения конечного потомства, которое включает любой желаемый признак (признаки), демонстрируемые родительским генотипом, наряду с SCN-устойчивым фенотипом. В конкретных вариантах осуществления, индивидуальных потомков, полученных на каждой стадии скрещивания и обратного скрещивания, отбирают с помощью анализа на SCN-маркеры в каждом поколении. В некоторых вариантах осуществления, анализ геномной ДНК двух родительских растений с помощью зондов, которые специфично гибридизуются с маркерами, сцепленными с SCN-устойчивым фенотипом, выявляет, что одно из родительских растений содержит меньшее количество или не содержит сцепленных маркеров, с которыми специфично гибридизуются зонды. В некоторых вариантах осуществления, индивидуальных потомков, полученных при каждом скрещивании и/или обратном скрещивании, отбирают по вариантам последовательностей индивидуальных растений.
В некоторых вариантах осуществления, маркеры, сцепленные с SCN-устойчивым фенотипом, могут применяться для введения одной или нескольких детерминант устойчивости к SCN в растение (например, сои) путем генетической трансформации. В конкретных вариантах осуществления, маркеры могут быть выбраны из группы, включающей маркеры, перечисленные в таблице 3, а также маркеры, которые являются их эквивалентами. В некоторых вариантах осуществления, способ введения одной или нескольких детерминант устойчивости к SCN в растение путем генетической рекомбинации может включать исследование геномной ДНК растения (например, сои) с помощью зондов, которые способны специфично гибридизоваться с маркерами, сцепленными с SCN-устойчивым фенотипом, для идентификации одной или нескольких детерминант устойчивости растения к SCN; изоляцию сегмента геномной ДНК растения, включающего маркеры, сцепленные с SCN-устойчивым фенотипом, например, путем извлечения геномной ДНК и расщепления этой геномной ДНК одной или несколькими рестрикционными эндонуклеазами; необязательную амплификацию изолированного сегмента ДНК; введение изолированного сегмента ДНК в клетку или ткань растения хозяина; и исследование ДНК растения хозяина с помощью зондов, которые способны специфично гибридизоваться с маркерами, сцепленными с SCN-устойчивым фенотипом, для идентификации одной или нескольких детерминант устойчивости к SCN у растения-хозяина. В конкретных вариантах осуществления, изолированный сегмент ДНК можно ввести в растение-хозяина таким образом, чтобы он устойчиво интегрировался в геном растения-хозяина.
В некоторых вариантах осуществления, маркеры, которые сцеплены с SCN-устойчивым фенотипом, могут применяться для введения одной или нескольких детерминант устойчивости к SCN в другие организмы, например, растения. В конкретных вариантах осуществления, эти маркеры могут быть выбраны из группы маркеров, перечисленных в таблице 3, а также маркеров, которые являются их эквивалентами. В некоторых вариантах осуществления, способ введения одной или нескольких детерминант устойчивости к SCN в организм, отличный от сои, может включать исследование геномной ДНК растения (например, сои) с помощью зондов, которые способны специфично гибридизоваться с маркерами, сцепленными с SCN-устойчивым фенотипом, для идентификации одной или нескольких детерминант устойчивости растения к SCN; изоляцию сегмента геномной ДНК растения, включающего одну или несколько детерминант устойчивости к SCN, например, путем извлечения геномной ДНК и расщепления этой геномной ДНК одной или несколькими рестрикционными эндонуклеазами; необязательную амплификацию изолированного сегмента ДНК; введение изолированного сегмента ДНК в организм, отличный от сои; и исследование ДНК организма, отличного от сои, с помощью зондов, которые способны специфично гибридизоваться с маркерами, сцепленными с SCN-устойчивым фенотипом, для идентификации одной или нескольких детерминант устойчивости к SCN у этого организма. В других вариантах осуществления, изолированный сегмент ДНК можно ввести в организм таким образом, чтобы он устойчиво интегрировался в геном этого организма.
В некоторых вариантах осуществления, маркеры, которые сцеплены с SCN-устойчивым фенотипом, могут применяться для выявления растения с одной или несколькими детерминантами устойчивости к SCN. В некоторых вариантах осуществления, растение может быть растением сои. Например, это растение, может быть растением сои, полученным из зародышевой плазмы 98860-71. В конкретных вариантах осуществления, из растения могут быть извлечены молекулы нуклеиновых кислот (например, геномная ДНК или мРНК). Извлеченные молекулы нуклеиновых кислот затем можно ввести в контакт с одним или несколькими зондами, которые способны специфично гибридизоваться с маркерами, сцепленными с SCN-устойчивым фенотипом. Специфичная гибридизация одного или нескольких зондов с извлеченными молекулами нуклеиновых кислот, служит признаком наличия одной или нескольких детерминант устойчивости растения к SCN.
В некоторых вариантах осуществления, могут одновременно применяться маркеры, которые сцеплены с несколькими детерминантами устойчивости к SCN. В других вариантах осуществления, могут применяться маркеры, которые сцеплены только с одной детерминантой устойчивости к SCN. В конкретных вариантах осуществления, могут одновременно применяться маркеры, которые сцеплены устойчивостью к одной или нескольким конкретным расам SCN HG (например, расе 1, расе 2, расе 3, расе 5 и расе 14). Например, одновременно может применяться несколько маркеров, которые сцеплены с устойчивостью к различным расам SCN HG.
Следующие примеры приведены для иллюстрации некоторых конкретных характеристик и/или вариантов осуществления изобретения. Эти примеры не следует истолковывать, как ограничение объема изобретения конкретными описанными характеристиками или вариантами осуществления.
ПРИМЕРЫ
Пример 1: Материалы и методики
Для выявления маркеров, сцепленных с SCN-устойчивым фенотипом, в качестве родительских растений для SCN-картирования использовали 24 культурных сорта сои. Из этих культурных сортов, 14 не обладали устойчивостью к SCN, а именно: 75110, 75155, 75163, 99630, 99726, 95895-755RU, 99345-31, 75192, 75209, 75159, Essex, Williams82, 75213 и 6CH026-035. 10 представителей культурных сортов обладали устойчивостью к SCN, а именно: Maverick, Peking, PI209332, PI437654, 99811, 99294, Forrest, PI88788, PI437654 и 98860-71.
Биологический тест на устойчивость к SCN: биологические тесты на устойчивость к SCN проводили с целью получения информации о фенотипе популяции для картирования, полученной скрещиванием SCN-устойчивого сорта 98860-71 с SCN-неустойчивыми сортами 75213 и 6CH026-035. Информация о фенотипе использованной для картирования популяции приведена в таблице 3. В технике не имеется единого подхода к классификации уровней устойчивости сортов сои к SCN. Поэтому уровни устойчивости классифицировали согласно «индексам устойчивости к SCN»: индекс устойчивости к SCN 0-10=R (устойчивы); индекс устойчивости к SCN 10,1-29,9=MR (средняя устойчивость); индекс устойчивости к SCN 30,0-59,9=MS (средняя неустойчивость); и 60+=S (неустойчивы). Значения индексов устойчивости к SCN определяли путем сравнения тестируемых линий с известными неустойчивыми и устойчивыми линиями. Значения индексов были основаны непосредственно на легкости заражения SCN, наблюдаемой для данного образца. Например, если на каждом из 9 растений тестируемой линии обнаруживали 10 цист, а на каждом из 9 растений сорта Williams (неустойчивых) обнаруживалось 100 цист, тестируемой линии присваивали индекс 10%. За конечное значение индекса принимали среднее значение индексов для 9 растений.
Реакции в системе генотипирования KASParTM: праймеры для системы KASParTM разрабатывали с помощью средства разработки PrimerPickerTM в системе KLIMSTM (KBioscience Laboratory Management System), вводя в программу ДНК-последовательности с SNPs. Для каждой SNP-последовательности разрабатывали три праймера, а именно A1 (аллель-специфичный праймер 1), A2 (аллель-специфичный праймер 2) и C (общий обратный праймер), с учетом реагентов, применяемых в системе KASParTM. Аналитическую смесь каждой реакции KASParTM готовили, как указано в v2.0 системы SNP-генотипирования KASParTM. Конечный объем реакционной смеси составлял 5,0 мкл, в том числе 1 мкл образца ДНК (5 нг/мкл), 2,5 мкл 2Х реакционной смеси, 0,06875 аналитической смеси, 0,04 мкл 50 мМ MgCl2 и 1,39125 мкл dd (деионизированной дистилляцией) H2O. Анализ проводили в 384-луночном формате. При анализе использовали параметры термоциклов в соответствии с инструкцией производителя: 94°C в течение 15 минут; 20 циклов: 94°C в течение 10 секунд, 57°C в течение 5 секунд и 72°C в течение 10 секунд; и 22 цикла: 94°C в течение 10 секунд, 57°C в течение 20 секунд и 72°C в течение 40 секунд. Планшеты с PCR центрифугировали и определяли интенсивности аллель-специфичных сигналов групп FAM и VIC на спектрофотометре (Tecan GENiosTM, Männedorf, Switzerland) при комнатной температуре. Данные непосредственно вводили в систему KLIMSTM, и анализировали с ее помощью, используя Kluster CallerTM.
Пример 2: Определение физического положения QTLs и интервалов QTL, которые связаны с генами устойчивости к SCN
QTLs, которые связаны с устойчивостью к SCN, на первом этапе выявляли путем изучения литературы по SCN. Выявленные на первом этапе QTLs, связанные с устойчивостью SCN и обнаруженные в соответствующей литературе, приведены на Фиг.1a и 1b.
Из списка QTLs, которые были выявлены на первом этапе в литературе по SCN, были определены несколько отчетливо выраженных интервалов QTL, которые связаны с устойчивостью к различным расам SCN, исходя из карты генома сои. См., например, фиг.2. Например, определяли интервалы QTL на группе сцепления (LG) G, которые показаны на фиг.3; интервалы QTL на LG A2, которые показаны на фиг.4; интервалы QTL на LG B1, которые показаны на фиг.5; и интервалы QTL на LG I, которые показаны на фиг.6. В Таблице 2 приведены характерные примеры QTLs и соответствующие определенные интервалы QTL, которые связаны с устойчивостью к различным расам SCN.
Пример 3: Выявление SNP-маркеров, которые физически локализованы внутри/около/между QTLs и интервалами QTL, которые сцеплены с генами устойчивости к SCN
Используя программу BLASTTM, исследовали геном сои для выявления SNP маркеров, которые физически локализованы внутри, около или между интервалами QTL, определенными в предыдущем примере. Было выдвинуто предположение, что некоторые из этих SNP-маркеров могут быть сцеплены с SCN-устойчивым фенотипом. Для первичного скрининга выбирали в общей сложности 79 SNP-маркеров, используя 24 линии сои (14 неустойчивых к SCN и 10 устойчивых к SCN), чтобы определить, какие из этих SNP-маркеров, если таковые существуют, сцеплены с SCN-устойчивым фенотипом. 25 из 79 маркеров были локализованы на LG G, 12 маркеров были локализованы на LG A2, 22 маркера были локализованы на LG B1, и 20 маркеров были локализованы на LG I. Все 79 выбранных маркеров приведены в таблице 2.
Пример 4: Разработка методики анализа KASParTM
На первом этапе проводили скрининг 79 SNP-маркеров в 24 родительских линиях с использованием системы генотипирования KASParTM. 75 из 79 исследованных SNP маркеров получили подтверждение.
Получил подтверждение 21 SNP-маркер на LG G (Gm 18): NCSB_004072, BARC_015277_01929, NCSB_004073, NCSB_004074, NCSB_004075, NCSB_004076, NCSB_004077, NCSB_004078, NCSB_004079, NCSB_004080, NCSB_004081, NCSB_004082, NCSB_004083, NCSB_004084, NCSB_004085, NCSB_004086, NCSB_004097, NCSB_004098, NCSB_004107, NCSB_004108 и NCSB_004109.
Получили подтверждение 12 SNP-маркеров на LG A2 (Gm 08): BARC_025911_05089, BARC_019419_02921, NCSB_001710, NCSB_001711, NCSB_001712, NCSB_001713, NCSB_001714, NCSB_001715, NCSB_001716, NCSB_001717, NCSB_001718 и NCSB_001719.
Получили подтверждение 22 SNP-маркера на LG B1 (Gm 11): NCSB_002644, NCSB_002645, NCSB_002646, NCSB_002647, NCSB_002648, NCSB_002649, NCSB_002650, NCSB_002651, NCSB_002652, NCSB_002653, NCSB_002654, NCSB_002655, NCSB_002656, NCSB_002657, BARC_007704_00091, BARC_010269_00537, BARC_904050_01007, BARC_019557_03202, BARC_018649_03221, BARC_025703_04999, BARC_013547_01157 и BARC_035379_07178.
Получили подтверждение 20 SNP-маркеров на LG I (Gm 20): NCSB_004974, NCSB_004975, NCSB_004977, NCSB_004979, NCSB_004882, NCSB_004883, NCSB_004884, NCSB_004886, NCSB_004887, NCSB_004889, NCSB_004880, NCSB_004882, NCSB_004883, NCSB_004884, NCSB_004885, NCSB_004887, NCSB_004888, NCSB_004899, NCSB_004900 и NCSB_004903.
Предварительный скрининг выявил, что 44 SNP-маркера являются полиморфными среди 24 родительских линий. Было выбрано 24 полиморфных маркера (NCSB_001716 (LG A2), NCSB_002645 (B1), NCSB_002646 (B1), NCSB_002648 (B1), NCSB_002651 (B1), NCSB_002652 (B1), NCSB_002654 (B1), NCSB_002656 (B1), BARC_013547_01157 (B1), NCSB_004073 (G), NCSB_004074 (G), NCSB_004078 (G), NCSB_004080 (G), NCSB_004084 (G), NCSB_004085 (G), NCSB_004097 (G), NCSB_004109 (G), BARC_012237_01755 (G), rgh1-689 (G), rgh1-757 (G), rgh1-2564 (G), rgh1-3995 (G) и NCSB_004900 (I)) для дальнейшего исследования сцепления в популяциях для картирования, полученных скрещиванием SCN-устойчивого сорта сои 98860-71 с SCN-неустойчивыми сортами 75213 и 6CH026-035. На фиг.7 показаны типичные данные генотипирования, полученные с помощью системы KASParTM, для четырех представителей полиморфных маркеров.
15 SNP-маркеров оказались полиморфными между SCN-устойчивыми и SCN-неустойчивыми родительскими растениями. Затем осуществляли скрининг этих 15 полиморфных SNP-маркеров среди 93 индивидуальных растений в популяции для картирования.
Из этих пятнадцати SNP-маркеров, которые были полиморфными между родительскими линиями, и скрининг которых проводили у индивидуальных растений популяции для картирования, только три SNPs продемонстрировали косегрегацию с признаком устойчивости к SCN, а именно: NCSB_004074, BARC_010889_01691 и rhg1-3995. Фиг.8. Последовательности праймеров в системе KASParTM, которые применялись для генотипирования индивидуальных растений с целью обнаружения этих трех маркеров, приведены в таблице 3.


Если использовать в качестве эталона последовательность геномной нуклеиновой кислоты культурного сорта сои Williams 82, то BARC_010889_01691 локализован на хромосоме 18 в положении 1674511 bp; NCSB_004074 локализован на хромосоме 18 в положении 1663671 bp; и rgh1-3995 локализован на хромосоме 18 в положении 1714741 bp. Все три сцепленных маркера (NCSB_004074, BARC_010889_01691 и rgh1-3995) локализованы либо в локусе rhg1 (rgh1-3995) или вблизи него на группе сцепления G (BARC_010889_01691 и NCSB_004074).
Для устойчивых фенотипов и фенотипов со средней устойчивостью, все три генотипа со сцепленными маркерами соответствовали фенотипу. Что касается неустойчивых линий, BARC_010889_01691 имел 5 случаев несоответствия с фенотипами, NSCB_004074 имел 9 случаев несоответствия, и rhg1-3995 имел 6 случаев несоответствия. Смотрите таблицу 4.
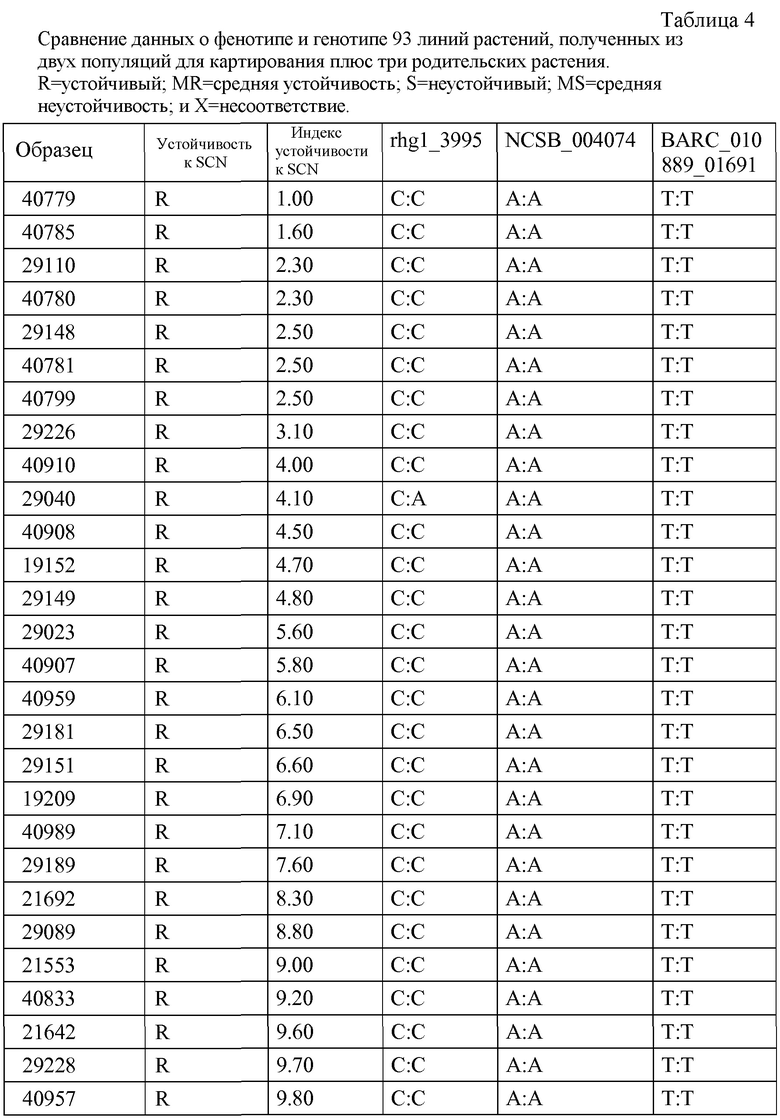
Если с помощью трех указанных маркеров идентифицированы какие-либо неустойчивые генотипы, относительное число ложно-негативных заключений составляет 0%. Другими словами, используя три этих маркера, можно с идеальной точностью выявить неустойчивый к SCN фенотип. Кроме того, можно предсказать SCN-устойчивый генотип с относительным числом ложно-положительных заключений примерно 10-18% (частное от деления 5 или 9 на 51, т.е. общее число неустойчивых образцов). Таким образом, можно ожидать, что только 5-9% из выявленных SCN-устойчивых генотипов продемонстрируют SCN-неустойчивый фенотип.
Пример 5: SNP-маркеры в LG A2, LG B1 и LG I, которые сцеплены с SCN-устойчивым фенотипом
С помощью BLASTTM исследовали геном сои с целью поиска SNP-маркеров, которые физически локализованы внутри, около или между интервалами QTL, связанными с устойчивостью к SCN, на группах сцепления A2, B1 и I. Список SNP-маркеров получен с помощью поиска в программе BLASTTM. Для предварительного скрининга выбирали большое количество SNP-маркеров, используя SCN- неустойчивые и SCN-устойчивые линии сои, чтобы определить, какие из этих SNP-маркеров, если таковые вообще существуют, в группах сцепления A2, B1 и I сцеплены с SCN-устойчивым фенотипом.
Предварительный скрининг выбранных SNP-маркеров в родительских линиях осуществляли с помощью системы генотипирования KASParTM. Подтверждали значимость множества выбранных SNP-маркеров, и определяли, что подмножество выбранных маркеров является полиморфным между родительскими линиями. Как минимум один из этих полиморфных SNP-маркеров использовали для исследования сцепления с устойчивостью к SCN с помощью популяции для картирования, полученной скрещиванием SCN-устойчивого сорта сои с одним или несколькими SCN-неустойчивыми сортами сои. Проводили скрининг одного или нескольких из этих полиморфных SNP-маркеров у индивидуумов в популяции для картирования.
SNPs, которые проявляли косегрегацию с признаком устойчивости к SCN у индивидуумов популяции для картирования, идентифицировали в качестве маркеров на группах сцепления A2, B1 и I, которые сцеплены с устойчивостью к SCN в SCN-устойчивом родительском сорте. Генотипы, которые включали сцепленные маркеры, соответствовали фенотипам, наблюдавшимся у индивидуумов популяции для картирования.
Пример 6: SNP-маркеры, сцепленные с SCN-устойчивым фенотипом, в зародышевой плазме JTN-5109
Выбирали большое количество SNP-маркеров, которые физически локализованы внутри, около или между интервалами QTL, связанными с устойчивостью к SCN (например, SNP-маркеры, выбранные из группы маркеров, перечисленных в таблице 3) для предварительного скрининга, используя SCN-устойчивый сорт сои JTN-5109 и SCN-неустойчивые линии сои, чтобы определить, какие из этих SNP-маркеров, если таковые имеются, сцеплены с SCN-устойчивым фенотипом сорта сои JTN-5109.
Предварительный скрининг выбранных SNP-маркеров в родительских линиях осуществляли с помощью системы генотипирования KASParTM. Подтверждали значимость множества выбранных SNP-маркеров, и определяли, что подмножество выбранных маркеров является полиморфным между сортом сои JTN-5109 и SCN-неустойчивыми родительскими линиями. Как минимум один из этих полиморфных SNP-маркеров использовали для исследования сцепления с устойчивостью к SCN в популяции для картирования, полученной скрещиванием сорта сои JTN-5109 с одним или несколькими SCN-неустойчивыми сортами сои. Проводили скрининг одного или нескольких из этих полиморфных SNP-маркеров у индивидуумов в популяции для картирования.
SNPs, которые проявляли косегрегацию с признаком устойчивости к SCN у индивидуумов популяции для картирования, идентифицировали в качестве маркеров, которые сцеплены с устойчивостью сорта сои JTN-5109 к SCN. Генотипы, которые включали сцепленные маркеры, соответствовали фенотипам, наблюдавшимся у индивидуумов популяции для картирования.
Пример 7: SNP-маркеры, которые сцеплены с фенотипом, устойчивым к SCN рас, отличных от расы 3
Получали популяции для картирования конкретно для рас HG, отличающихся от расы 3, путем скрещивания SCN-устойчивых сортов сои, выбранных из группы, состоящей из PI 88788, Peking, PI 437654, PI 90763, PI 438489B, PI 89772, PI209332, PUSCN14, Hartwig, Forrest и Pyramid, с одним или несколькими SCN-неустойчивыми сортами сои.
С помощью BLASTTM исследовали геном сои с целью поиска SNP-маркеров, которые физически локализованы внутри, около или между интервалами QTL, связанными с SCN-устойчивостью по отношению к конкретным расам HG. Список SNP-маркеров получали с помощью поиска в программе BLASTTM. Для предварительного скрининга выбирали большое количество SNP-маркеров, используя SCN-неустойчивые и SCN-устойчивые линии сои, чтобы определить, какие из этих SNP-маркеров, если они вообще существуют, сцеплены с фенотипом, устойчивым по отношению к конкретным расам HG.
Предварительный скрининг выбранных SNP-маркеров в родительских линиях осуществляли с помощью системы генотипирования KASParTM. Подтверждали значимость множества выбранных SNP-маркеров, и определяли, что подмножество выбранных маркеров является полиморфным между родительскими линиями. Как минимум один из этих полиморфных SNP-маркеров использовали для исследования связи с устойчивостью к конкретным расам SCN в популяции для картирования, полученной скрещиванием SCN-устойчивого сорта сои с одним или несколькими SCN-неустойчивыми сортами сои. Проводили скрининг одного или нескольких из этих полиморфных SNP-маркеров у индивидуумов в популяции для картирования.
SNPs, которые проявляли косегрегацию с признаком устойчивости к SCN у индивидуумов популяции для картирования, идентифицировали в качестве маркеров, которые сцеплены с SCN- устойчивостью в родительском сорте, устойчивом к конкретным расам HG. Генотипы, которые включали сцепленные маркеры, соответствовали фенотипам, наблюдавшимся у индивидуумов популяции для картирования.
| название | год | авторы | номер документа |
|---|---|---|---|
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ НИЗКОГО СОДЕРЖАНИЯ ПАЛЬМИТИНОВОЙ КИСЛОТЫ В ПОДСОЛНЕЧНИКЕ (HELIANTHUS ANNUS) И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2013 |
|
RU2670517C2 |
| ГЕНЕТИЧЕСКИЙ ЛОКУС, АССОЦИИРОВАННЫЙ С ГНИЛЬЮ КОРНЯ И СТЕБЛЯ, ОБУСЛОВЛЕННОЙ PHYTOPHTHORA, У СОИ | 2016 |
|
RU2748688C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM4 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2718584C2 |
| SNP-МАРКЕРЫ И ОТБОР В ОТНОШЕНИИ НИЗКОГО СОДЕРЖАНИЯ КЛЕТЧАТКИ В ПРЕДСТАВИТЕЛЯХ РОДА BRASSICA | 2019 |
|
RU2834673C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| КАНОЛА HO/LL С УСТОЙЧИВОСТЬЮ К ЗАБОЛЕВАНИЮ КИЛОЙ КРЕСТОЦВЕТНЫХ | 2012 |
|
RU2711934C2 |
| КАНОЛА HO/LL С УСТОЙЧИВОСТЬЮ К ЗАБОЛЕВАНИЮ КИЛОЙ КРЕСТОЦВЕТНЫХ | 2012 |
|
RU2618846C2 |
| ГЕНЕТИЧЕСКИЕ ЛОКУСЫ, СВЯЗАННЫЕ С УСТОЙЧИВОСТЬЮ К ПЫЛЬНОЙ ГОЛОВНЕ У МАИСА | 2009 |
|
RU2562864C2 |
| ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ (CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2011 |
|
RU2603005C2 |



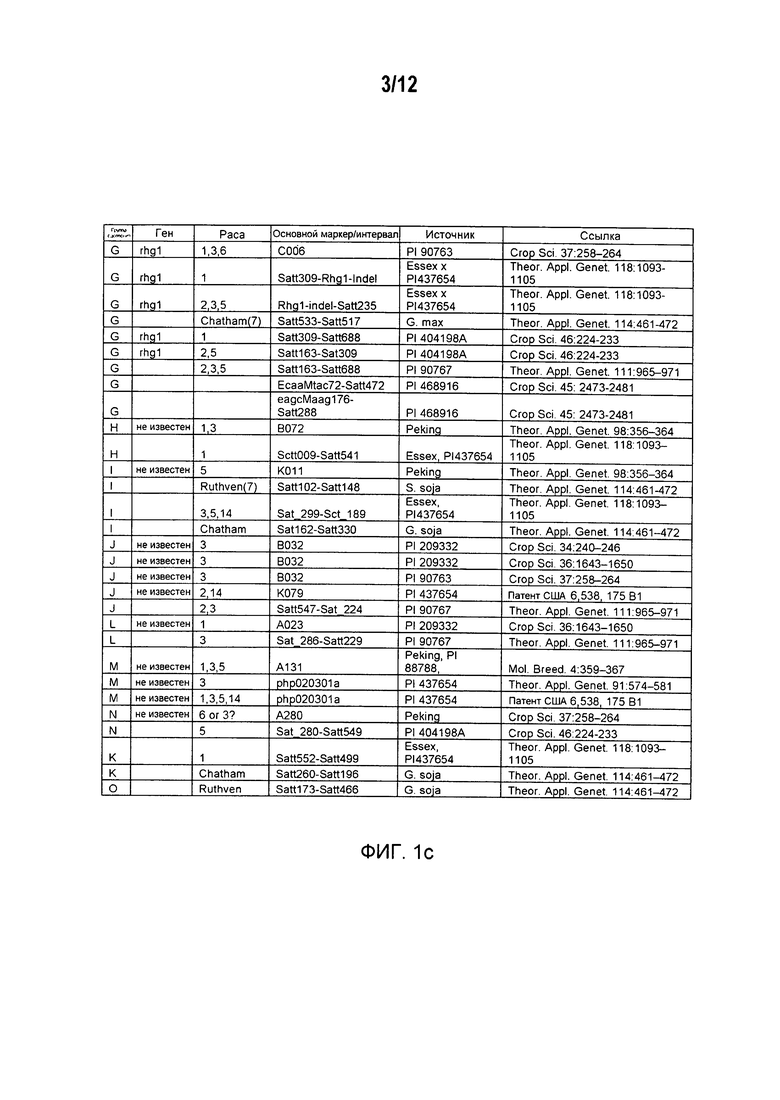
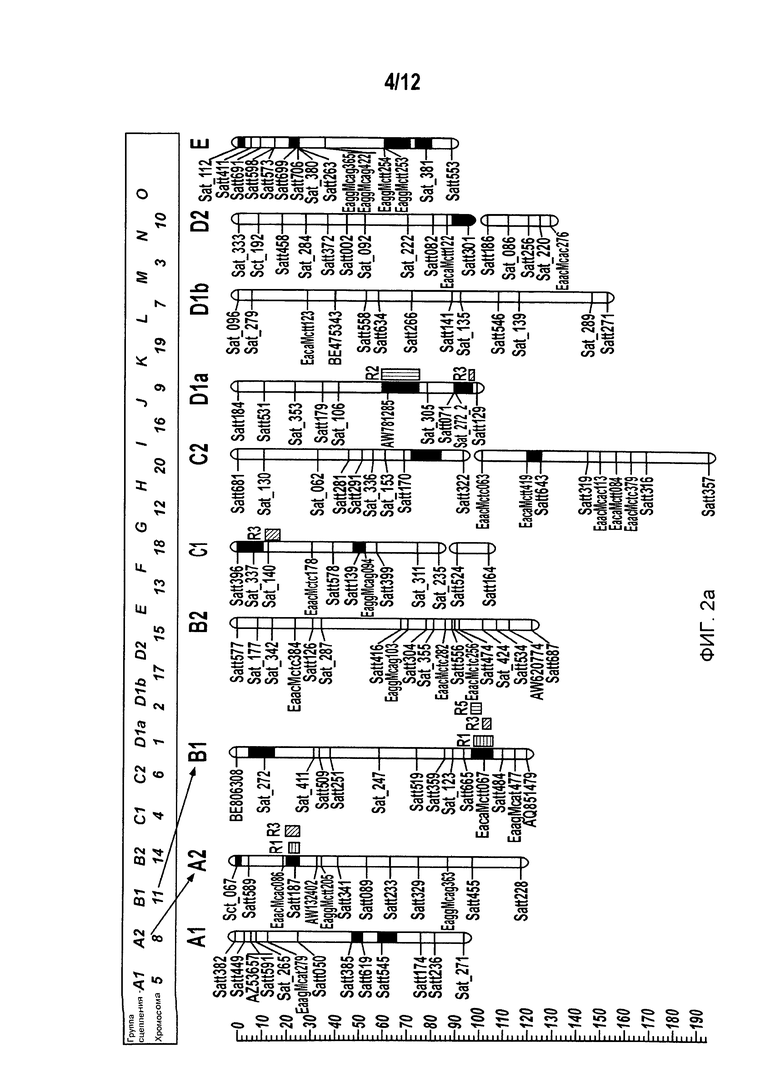
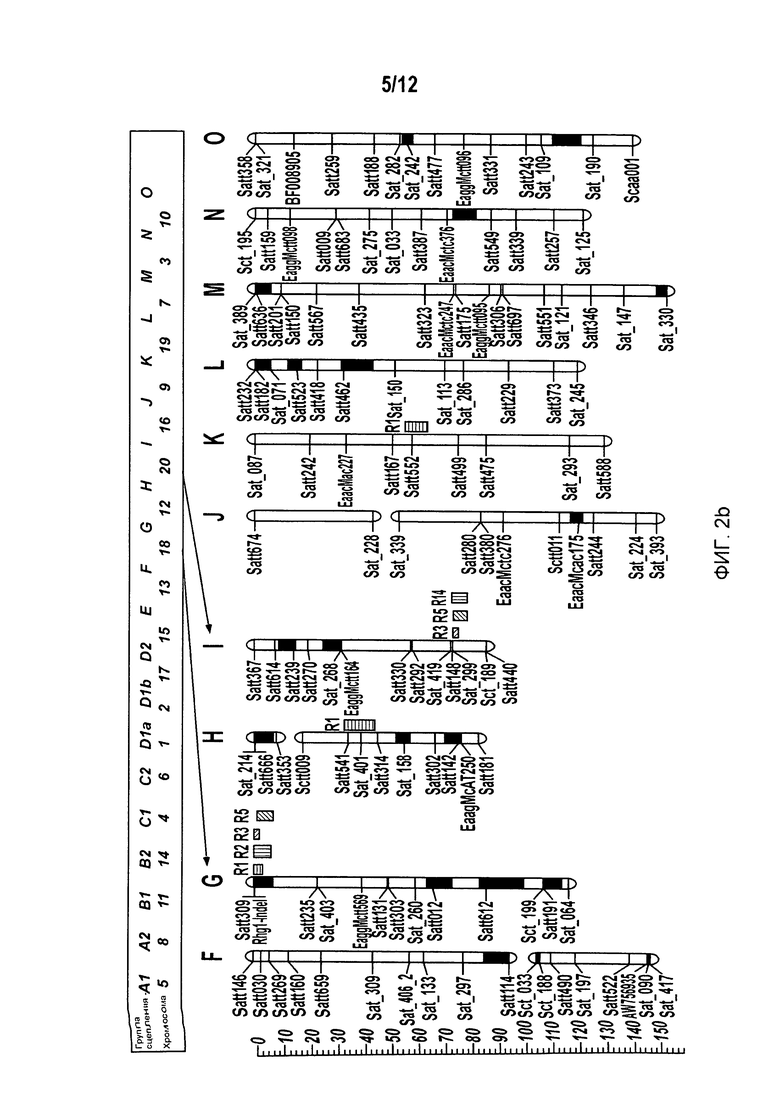
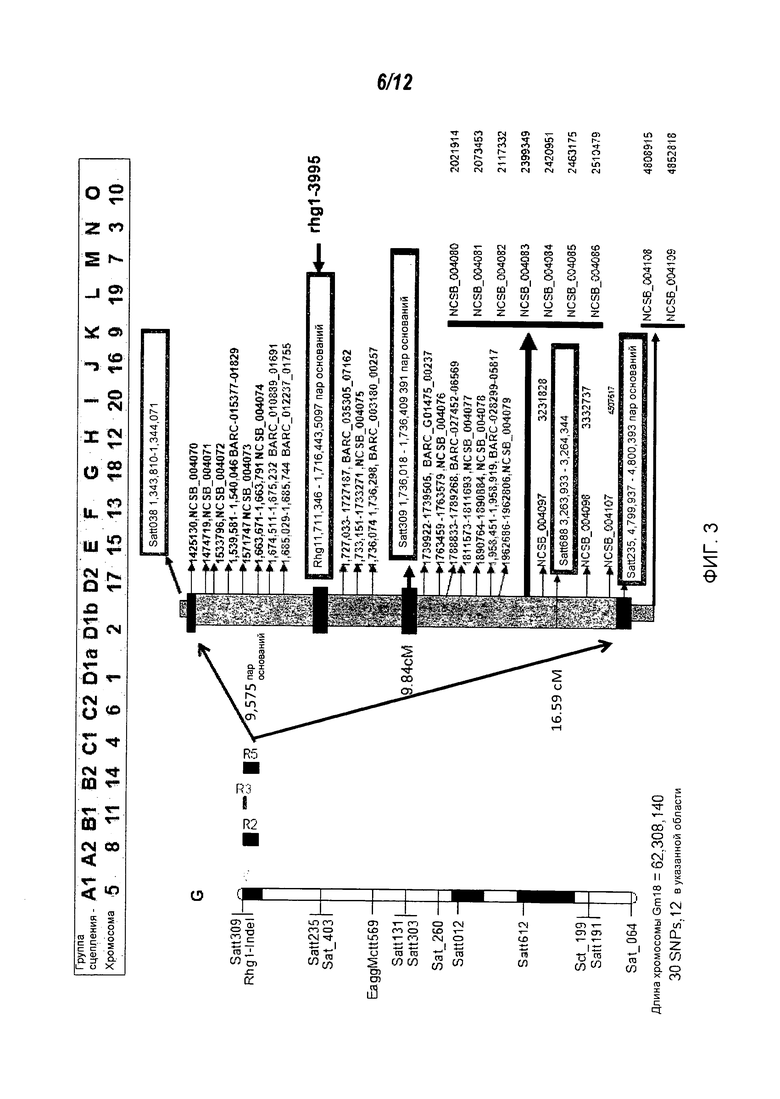
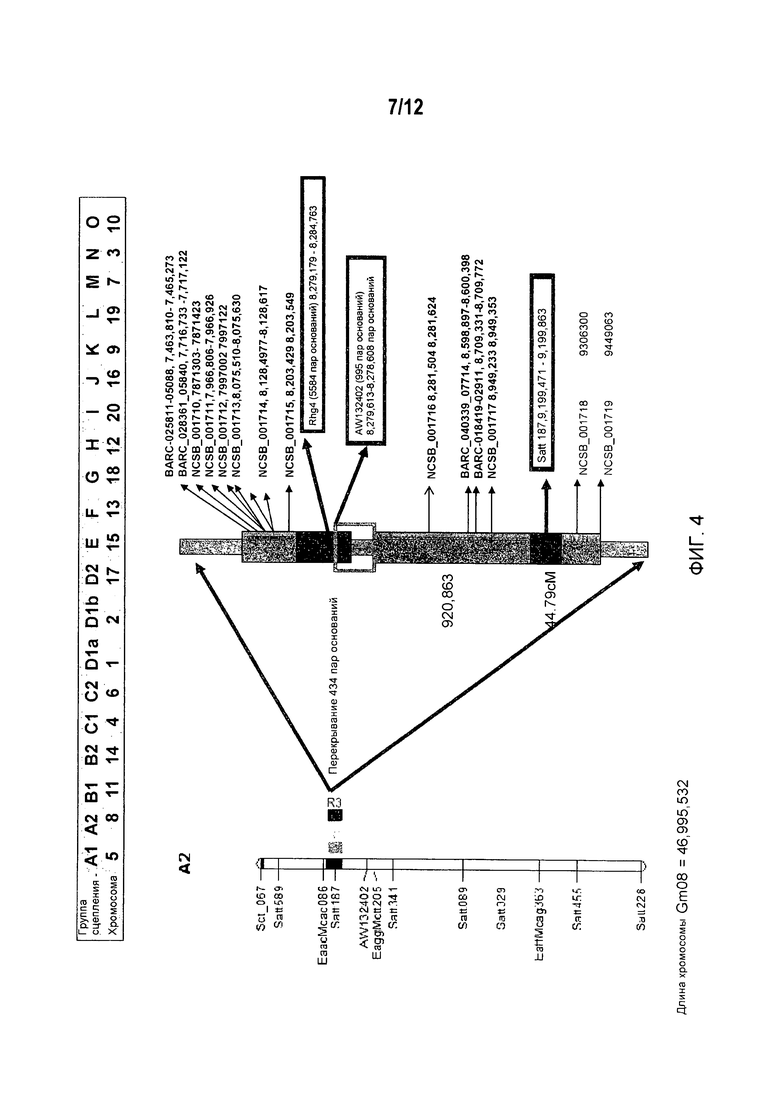

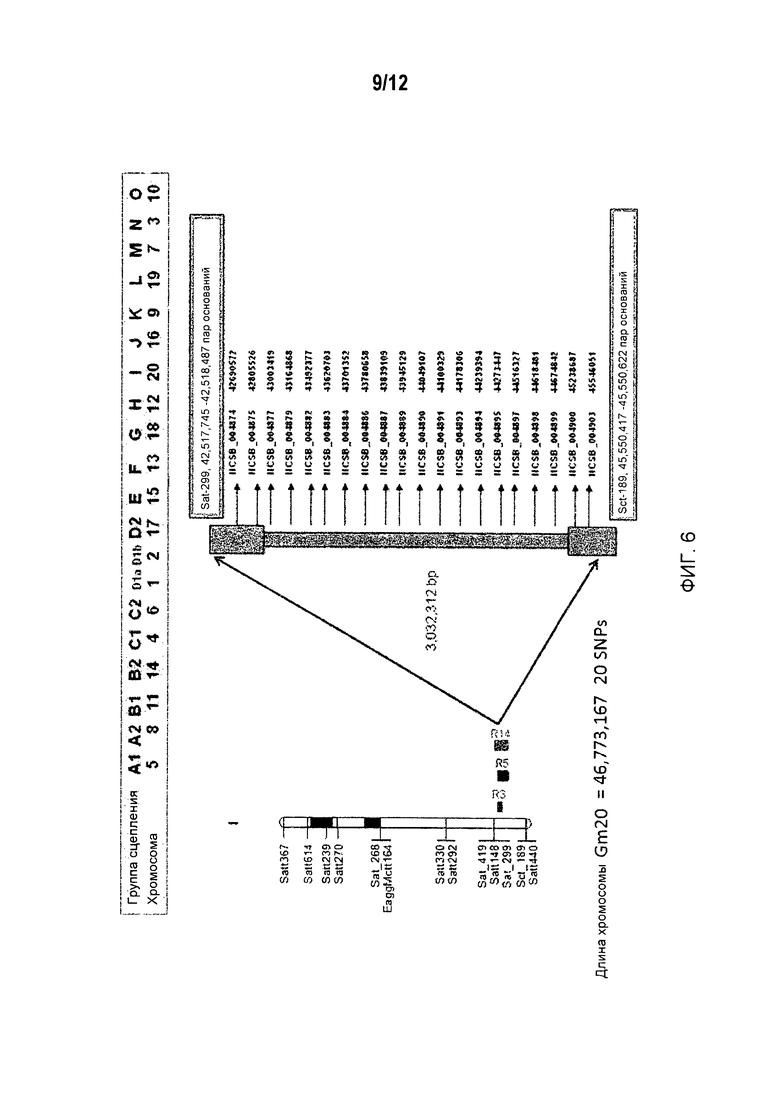
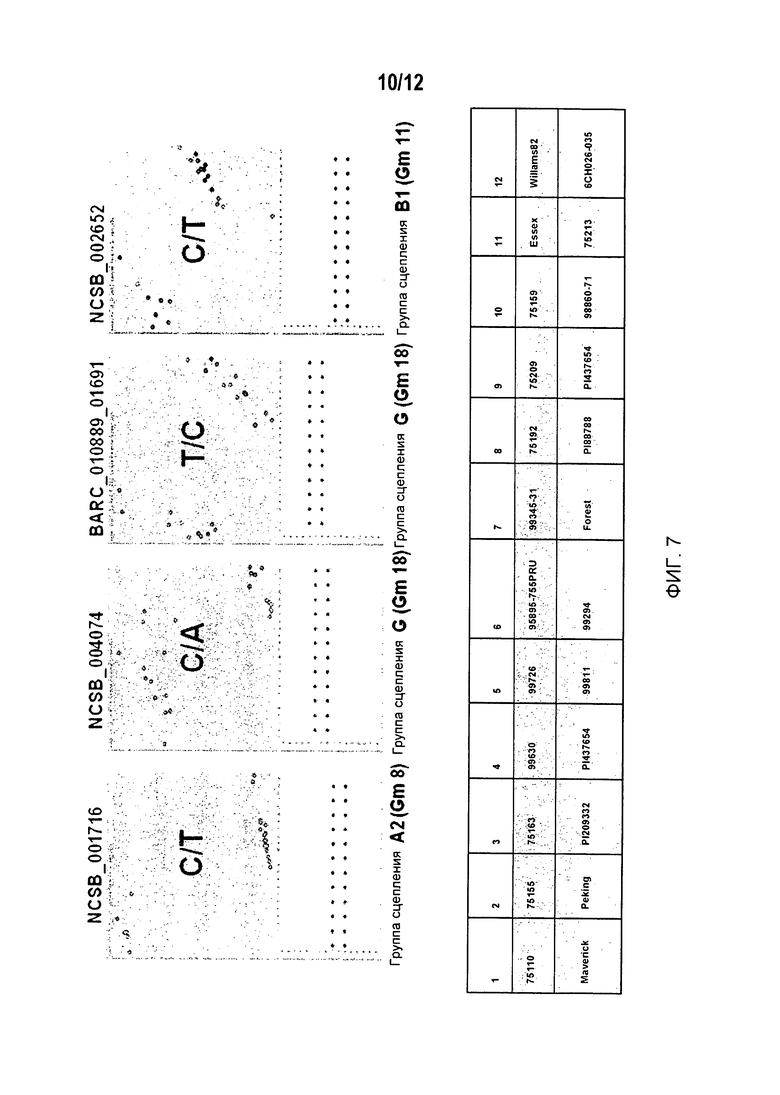

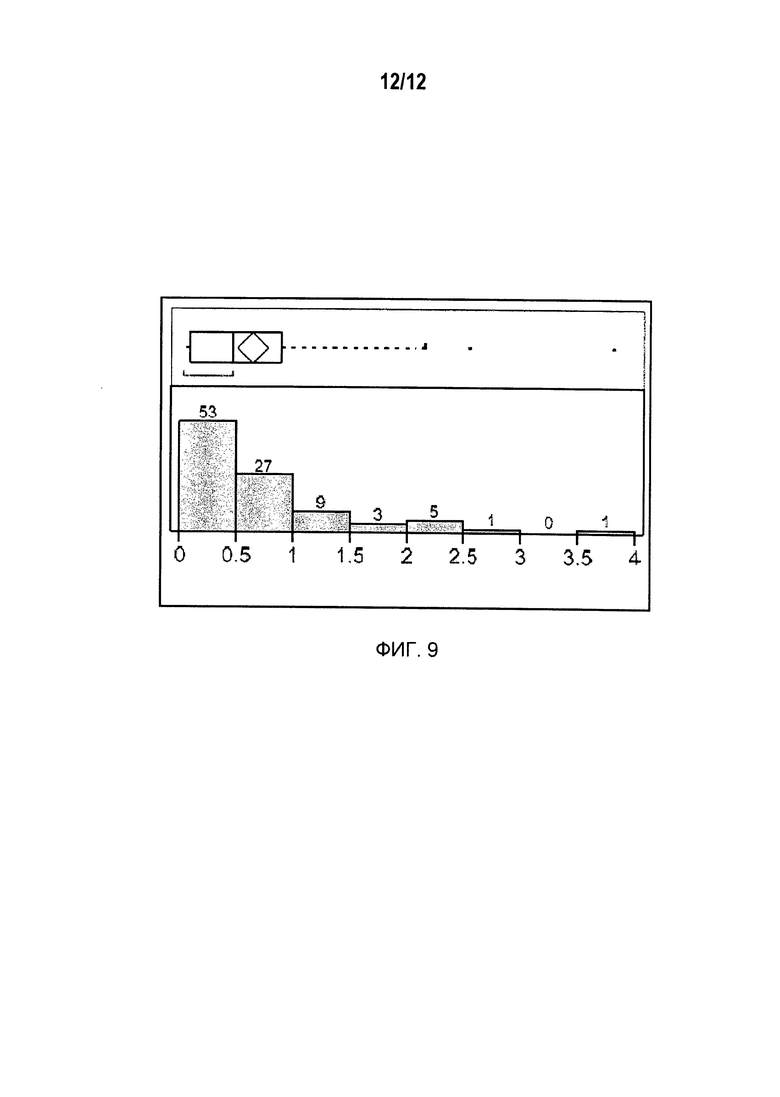
Изобретение относится к области биохимии, в частности к способу идентификации растения, включающему по крайней мере одну детерминанту устойчивости сорта растения сои к соевой цистообразующей нематоде (SCN), а также к способу получения SCN-устойчивого растения сои, представляющего собой интерес сорта, включающему стадии скрещивания растения сои, имеющего признак устойчивости к SCN, с растением сои SCN-неустойчивого сорта, представляющий интерес, применение маркер-вспомогательной селекции, а также размножение растений сои F1 с получением SCN-устойчивого растения сои. Также раскрыт способ придания растению сои устойчивости к SCN, способ переноса как минимум одной детерминанты устойчивости к SCN в сорт сои, а также способ введения как минимум одной детерминанты устойчивости к SCN сорта сои в растение-хозяина с помощью генетической трансформации, где каждый способ предусматривает исследование геномной ДНК растения для обнаружения маркера, сцепленным с SCN-устойчивым фенотипом сорта сои. Изобретение позволяет эффективно идентифицировать растение, включающеее по крайней мере одну детерминанту устойчивости сорта растения сои к SCN. 5 н. и 16 з.п. ф-лы, 9 ил., 4 табл., 7 пр.
1. Способ идентификации растения, включающего по крайней мере одну детерминанту устойчивости сорта растения сои к соевой цистообразующей нематоде (SCN), где указанный способ включает:
изоляцию геномных молекул нуклеиновой кислоты из растения; и
скрининг изолированных молекул нуклеиновой кислоты для обнаружения маркера, сцепленного с SCN-устойчивым фенотипом данного сорта сои, где скрининг для обнаружения маркера включает
определение стабильного и специфического связывания олигонуклеотидного зонда с последовательностью SEQ ID NO: 10, включающей адениновый нуклеотид (А) в положении 61 последовательности SEQ ID NO: 10, где связывание олигонуклеотидного зонда является специфическим, если олигонуклеотидный зонд не связывается стабильно с последовательностью SEQ ID NO: 10, включающей цитозиновый нуклеотид (С) в положении 61 последовательности SEQ ID No: 10, или
определение стабильного и специфического связывания олигонуклеотидного зонда с последовательностью SEQ ID NO: 11, включающей тиминовый нуклеотид (Т) в положении 98 последовательности SEQ ID NO: 11, где связывание олигонуклеотидного зонда является специфическим, если олигонуклеотидный зонд не связывается стабильно с последовательностью SEQ ID NO: 11, включающей цитозиновый нуклеотид (С) в положении 98 последовательности SEQ ID NO: 11, где стабильное и специфическое связывание олигонуклеотидного(ых) зонда(ов) указывает на присутствие маркера, и
где присутствие маркера является признаком наличия, как минимум, одной детерминанты устойчивости к SCN у сорта сои.
2. Способ по п. 1, где скрининг изолированных молекул нуклеиновой кислоты для обнаружения маркера, сцепленного с SCN-устойчивым фенотипом сорта сои, осуществляют с помощью конкурентной аллель-специфичной полимеразной цепной реакции.
3. Способ по п. 1, где SCN-устойчивый фенотип означает устойчивость к SCN расы 3.
4. Способ по п. 2, где олигонуклеотидный зонд, применяемый в конкурентной аллель-специфичной полимеразной цепной реакции для скрининга изолированных молекул нуклеиновой кислоты для обнаружения маркера, сцепленного с SCN-устойчивым фенотипом, является SEQ ID NO: 4 или SEQ ID NO: 7.
5. Способ по п. 1, дополнительно включающий определение генотипа растения для обнаружения маркера, сцепленного с SCN-устойчивым фенотипом сорта сои.
6. Способ получения SCN-устойчивого растения сои, представляющего собой интерес сорта, где указанный способ включает:
скрещивание растения сои, имеющего признак устойчивости к SCN, с растением сои SCN-неустойчивого сорта, представляющий интерес;
применение маркер-вспомогательной селекции для
идентификации растения сои F1, содержащего маркер, сцепленный с SCN-устойчивым фенотипом растения сои, имеющего признак SCN-устойчивости, где маркер выбирают из группы, состоящей из аденинового нуклеотида (А) в положении 61 последовательности SEQ ID NO: 10, и
тиминового нуклеотида (Т) в положении 98 последовательности SEQ ID NO: 11,
где растение сои F1 обладает какими-либо желаемыми признаками интересующего сорта сои; и
размножение идентифицированных растений сои F1 с получением SCN-устойчивого растения сои, представляющего собой интерес сорта.
7. Способ по п. 6, где маркер-вспомогательную селекцию осуществляют с помощью конкурентной аллель-специфичной полимеразной цепной реакции.
8. Способ по п. 6, где устойчивость к SCN является устойчивостью к SCN расы 3.
9. Способ по п. 7, где присутствие маркера определяют с помощью конкурентной аллель-специфичной полимеразной цепной реакции по стабильному и специфическому связыванию олигонуклеотидного зонда, выбранного из группы, состоящей из SEQ ID NO: 4 и SEQ ID NO: 7, с геномной молекулой нуклеиновой кислоты, выделенной из растения сои F1.
10. Способ придания растению сои устойчивости к SCN, где указанный способ включает введение маркера, сцепленного с SCN-устойчивым фенотипом сои, в SCN-неустойчивое растение сои, где маркер, сцепленный с SCN-устойчивым фенотипом сои, выбран из группы, состоящей из
аденинового нуклеотида (А) в положении 61 последовательности SEQ ID NO: 10, и тиминового нуклеотида (Т) в положении 98 последовательности SEQ ID NO: 11.
11. Способ переноса как минимум одной детерминанты устойчивости к SCN в сорт сои, где указанный способ включает:
(a) исследование геномной ДНК первого растения с донорным генотипом и ДНК второго растения с реципиентным генотипом, с использованием олигонуклеотидного зонда, который способен специфично гибридизоваться с маркером, сцепленным с SCN-устойчивым фенотипом сорта сои, где маркер, сцепленный с SCN-устойчивым фенотипом сорта сои, выбран из группы, состоящей из
аденинового нуклеотида (А) в положении 61 последовательности SEQ ID NO: 10, и
тиминового нуклеотида (Т) в положении 98 последовательности SEQ ID NO: 11;
(b) половое скрещивание генотипов двух родительских растений с получением популяции потомства;
(c) исследование популяции потомства на наличие маркера, который сцеплен с SCN-устойчивым фенотипом данного сорта сои;
(d) обратное скрещивание индивидуального растения из популяции потомства, которое содержит упомянутый маркер, сцепленный с SCN-устойчивым фенотипом сорта сои, с реципиентным генотипом для получения популяции следующего поколения;
(e) определение, содержит ли индивидуальное растение из популяции следующего поколения желаемый признак из реципиентного генотипа и маркер, который сцеплен с SCN-устойчивым фенотипом сорта сои; и
(f) если ни одно из растений популяции следующего поколения
не содержит желаемого признака из реципиентного генотипа и маркера, который сцеплен с SCN-устойчивым фенотипом данного сорта сои, повторение стадий (d) и (е), вплоть до выявления индивидуального растения, которое содержит желаемый признак из реципиентного генотипа и маркер, который сцеплен с SCN-устойчивым фенотипом сорта сои.
12. Способ по п. 11, где индивидуальных потомков, полученных на каждой стадии скрещивания и обратного скрещивания, отбирают с помощью анализа на наличие маркеров устойчивости к SCN в каждом поколении.
13. Способ введения как минимум одной детерминанты устойчивости к SCN сорта сои в растение-хозяина с помощью генетической трансформации, где указанный способ включает:
применение олигонуклеотидного зонда, который способен специфично гибридизоваться с маркером, сцепленным с SCN-устойчивым фенотипом сорта сои, для исследования геномной ДНК донорного растения с донорным генотипом и геномной ДНК растения-хозяина с реципиентным генотипом, где маркер, сцепленный с SCN-устойчивым фенотипом сорта сои, выбран из группы, состоящей из
полинуклеотида SEQ ID NO: 10, содержащего адениновый нуклеотид (А) в положении 61; и
полинуклеотида SEQ ID NO: 11, содержащего тиминовый нуклеотид (Т) в положении 98;
изоляцию сегмента геномной ДНК донорного растения, который специфично гибридизуется с олигонуклеотидным зондом, который способен специфично гибридизоваться с маркером, сцепленным с SCN-устойчивым фенотипом сорта сои;
введение изолированного сегмента геномной ДНК в растение-хозяина; и
анализ ДНК растения-хозяина с помощью олигонуклетидного зонда, который способен специфично гибридизоваться с маркером, сцепленным с SCN-устойчивым фенотипом сорта сои, для идентификации в растении-хозяине как минимум одной детерминанты устойчивости сорта сои к SCN.
14. Способ по п. 13, где изолированный сегмент ДНК устойчиво интегрирован в геном растения-хозяина.
15. Способ по п. 14, где растение-хозяин является растением семейства бобовых.
16. Способ по п. 15, где растение-хозяин является растением, выбранным из группы, состоящей из сои, зеленой фасоли, обыкновенной фасоли, зерновой фасоли, красной фасоли, лимской фасоли, золотистой фасоли, кустовой фасоли, фасоли адзуки, гороха огородного и вигны китайской.
17. Способ по п. 11, где олигонуклеотидным зондом являются последовательность SEQ ID NO: 4 или SEQ ID NO: 7.
18. Способ по п. 1, где скрининг на обнаружение маркера дополнительно включает:
определение стабильного и специфического связывания олигонуклеотидного зонда с последовательностью SEQ ID NO: 12, включающей цитозиновый нуклеотид (С) в положении 214 последовательности SEQ ID NO: 12, где связывание олигонуклеотидного зонда является специфическим, если олигонуклеотидный зонд не связывается стабильно с последовательностью SEQ ID NO: 12, включающей адениновый нуклеотид в положении 214 последовательности SEQ ID NO: 12.
19. Способ по п. 6, где маркер-вспомогательную селекцию применяют для идентификации в растении сои F1 дополнительного маркера, сцепленного с SCN-устойчивым фенотипом растения сои, имеющего признак SCN-устойчивости, где дополнительным маркером является цитозиновый нуклеотид (С) в положении 214 последовательности SEQ ID NO: 12.
20. Способ по п. 11, где стадии (а) и (с) дополнительно включают исследование растения(й) с применением олигонуклеотидного зонда, который способен специфично гибридизоваться с маркером, сцепленным с SCN-устойчивым фенотипом сорта сои, где маркером является цитозиновый нуклеотид (С) в положении 214 последовательности SEQ ID NO: 12.
21. Способ по п. 13, дополнительно включающий:
применение олигонуклеотидного зонда, который способен специфично гибридизоваться с дополнительным маркером, сцепленным с SCN-устойчивым фенотипом сорта сои, для исследования геномной ДНК донорного растения и геномной ДНК растения-хозяина, где дополнительным маркером является цитозиновый нуклеотид (С) в положении 214 последовательности SEQ ID NO: 12; и
исследование ДНК растения-хозяина с применением олигонуклеотидного зонда, который способен специфично гибридизоваться с дополнительным маркером, сцепленным с SCN-устойчивым фенотипом сорта сои, для идентификации в растении-хозяине как минимум одной детерминанты устойчивости сорта сои к SCN.
| US2003005491 A1, 02.01.2003 | |||
| US2006253919 A1, 09.11.2006 | |||
| US6096944 A, 01.08.2000 | |||
| РАСТЕНИЕ И РАСТИТЕЛЬНАЯ КЛЕТКА, ТРАНСФОРМИРОВАННЫЕ ХИМЕРНЫМ ГЕНОМ | 2001 |
|
RU2275426C1 |
Авторы
Даты
2017-05-30—Публикация
2011-11-02—Подача