Изобретение относится к области медицины, в частности к неврологии и молекулярной биологии, и может быть использовано для выявления полиморфизмов в генах LINGO1, LINGO2 и SLC1A2, ассоциациированных с мультифакториальным заболеванием - эссенциальным тремором посредством проведения медико-генетического анализа одновременно нескольких однонуклеотидных полиморфизмов (SNP) в этих генах.
Эссенциальный тремор является наиболее распространенным двигательным заболеванием нервной системы, нейродегенеративная природа которого доказывается рядом последних работ. Распространенность заболевания по некоторым данным может достигать 13%. Эссенциальный тремор представляет собой неуклонно прогрессирующее заболевание, приводящее к значительным трудностям в профессиональной деятельности, а затем и в самообслуживании. На молекулярно-генетическом уровне показана высокая гетерогенность этого заболевания, однако до настоящего времени, несмотря на бурное развитие молекулярно-генетических технологий, генетическая структура эссенциального тремора комплексно не определена и непосредственная ДНК-диагностика болезни пока не проводится.
Для эссенциального тремора характерна длительная доклиническая стадия, на которой возможно проведение профилактических мероприятий, предотвращающих их развитие. В свете вышеперечисленного диагностика предрасположенности к развитию такого нейродегенеративного заболевания имеет социальную значимость.
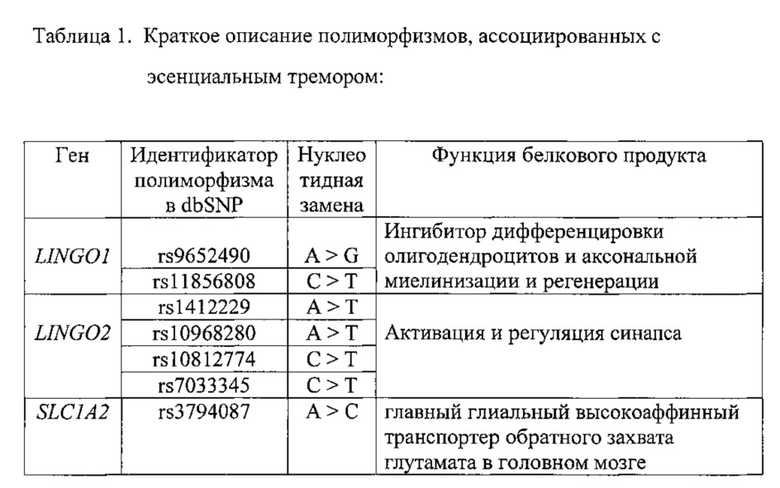
На сегодняшний день не выявлено патогенных мутаций приводящих к эссенциальному тремору, считается что комплекс генетических факторов риска приводит к развитию заболевания. При отборе генетических факторов риска развития эссенциального тремора, мы основывались на анализе результатов работ по выявлению ассоциаций между генетическими вариантами полиморфизмов и развитием заболевания. Так мета-анализ двух исследований полногеномного скрининга ассоциаций (genome-wide association study, GWAS), позволяющих осуществлять тотальный скрининг вариабельных однонуклеотидных участков генома, выявил высокую ассоциацию полиморфизмов в генах SLC1A2 (rs3794087) и LINGO1 (rs9652490), а также в меньшей мере полиморфизма rs11856808 в гене LINGO1 с эссенциальным тремором (Kuhlenbaumer G., Hopflner F. et al. Genetics of essential tremor // Neurology 2014; 18: 1000-1007). В целом ряде работ по изучению ассоциации полиморфизма rs9652490 в гене LINGO1 с эссенциальным тремором в различных европейских популяциях было показано статистически значимое увеличение риска развития заболевания связанное с наличием этого поиморфизма (Thier S, Lorenz D, Nothnagel M, et al. LINGO1 polymorphisms are associated with essential tremor in Europeans. Mov Disord 2010; 25: 709-715; Vilarino-Guell C, Ross OA, Wider C, et al. LINGO1 rs9652490 is associated with essential tremor and Parkinson disease. Parkinsonism Relat Disord2010; 16: 109-111; Clark LN, Park N, Kisselev S, Rios E, Lee JH, Louis ED. Replication of the LINGO1 gene association with essential tremor in a North American population. Eur J Hum Genet 2010; 18: 838-843). Надо отметить, что в китайской популяции не было вывлено достоверной ассоциации полиморфизма rs9652490 в гене LINGO1 с эссенциальным тремором (Zuo X, Jiang Н, Guo JF, et al. Screening for two SNPs of LINGO1 gene in patients with essential tremor or sporadic Parkinson's disease in Chinese population. Neurosci Lett 2010; 481: 69-72), что подтверждает гипотезу существования популяционно-специфичных различий и усиливает значимость расширенных популяционных исследований.
Мультицентровое исследование популяций Северной Америки помимо ассоциации выше перечисленных полиморфизмов с риском развития эссенциального тремора, выявило еще ряд полиморфизмов в гене LINGO2, влияющих в той или иной степени на развитие заболевания (Vilarino-Guell С, Wider С, Ross OA, et al. LINGO1 and LINGO2 variants are associated with essential tremor and Parkinson disease. Neurogenetics 2010; 11: 401-408).
Анализ результатов полногеномных исследований и собственные исследования, позволили отобрать семь однонуклеотидных полиморфизмов: rs9652490, rs11856808 в гене LINGO1 (15q24); rs1412229, rs10968280, rs10812774, rs7033345 в гене LINGO2 (9p21) и rs3794087 в гене SLC1A2 (11p13) (см. таблицу 1), проявивших наибольшую генетическую ассоциацию с эссенциальным тремором и разработать набор реагентов для выявления полиморфизмов в генах LINGO1, LINGO2 и SLC1A2 (гены-кандидаты), который в дальнейшем позволит провести мутационный скрининг эссенциального тремора, а так же ляжет в основу ДНК-диагностики этого заболевания.
Наиболее экономичным, быстрым и доступным методом анализа полиморфизмов (SNP) в настоящее время является ПЦР в «режиме реального времени», однако в обычном формате, на современных амплификаторах (термоциклерах) можно проанализировать в мультиплексе только два полиморфизма, так как имеется только четыре оптических канала детекции флуоресцентного сигнала и следовательно можно использовать только четыре флуоресцентно-меченных зонда, соответственно два для дикого аллеля и два для мутантного (для двух полиморфизмов). Примененная нами модификация позволяет анализировать до восьми полиморфизмов в мультиплексном формате, за счет разных температур плавления ампликонов с зондами, меченными одной и той же флуоресцентной меткой, а именно на одном оптическом канале, без потери специфичности и чувствительности можно детектировать сразу четыре ампликона. В разработанном нами наборе анализируется семь полиморфизмов.
Технический результат заявленного изобретения заключается в высокой специфичности набора и способа выявления полиморфизмов в генах LINGO1, LINGO2 и SLC1A2 за счет использования оригинальных олигонуклеотидных праймеров и флуоресцентно-меченых зондов, а также оригинальных условий и протокола проведения полимеразной цепной реакции и последующего плавления продуктов реакции с флуоресцентно-меченными зондами. И в ускорении и удешевлении идентификации олигонуклеотидных полиморфизмов за счет возможности в мультиплексном формате в одной постановке сразу оценивать до восьми однонуклеотидных полиморфных маркеров (SNP).
Технический результат достигается тем, что создан набор реагентов для выявления генетического полиморфизма в полиморфных точках генов LINGO1 (rs9652490, rs11856808), LINGO2 (rs10968280, rs7033345, rs1412229, rs10812774) и SLC1A2 (rs3794087), определяющих генетическую ассоциацию с эссенциальным тремором, который включает следующие компоненты: 50 мМ KCl, 15 мМ Tris-HCl с рН 8,8, 0,5% глицерол, 0,1% Tween 20, 6,25 мМ MgCl2, 250 мкМ дезоксинуклеотиды (dNTP) и 0,2 ед/мкл термостабильной ДНК-полимеразы с вытесняющей активностью, при этом все компоненты для проведения мультиплексной полимеразной цепной реакции за исключением ДНК-полимеразы смешаны в одном объеме вместе с 7 парами олигонуклеотидных праймеров, обладающих активностью прямого и обратного праймеров в полимеразной цепной реакции - SEQ ID №: 2, 3, 5, 6, 8, 9, 11, 12, 14, 15, 17, 18, 20, 21 в концентрациях, обеспечивающих ассиметричное накопление цепей ДНК, и 7 олигонуклеотидными зондами, меченными флуоресцентными метками на 5'-конце и гасителями на 3'-конце: зонд SEQ ID №1 с меткой FAM и гасителем RTQ1; зонды SEQ ID №4 и 19 с меткой Су5 и гасителем BHQ2; зонд SEQ ID №7 с меткой R6G и гасителем BHQ2; зонды SEQ ID №10 и 16 с меткой ROX и гасителем BHQ2; зонд SEQ ID №13 с меткой R6G и гасителем BHQ1.
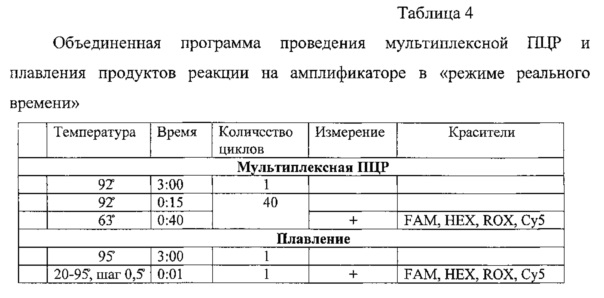
Технический результат достигается также за счет способа выявления генетического полиморфизма в полиморфных точках генов LINGO1 (rs9652490, rs11856808), LINGO2 (rs10968280, rs7033345, rs1412229, rs 10812774) и SLC1A2 (rs3794087), определяющих генетическую ассоциацию с эссенциальным тремором, включающий выделение ДНК, проведение полимеразной цепной реакции, путем начального плавления и ряда циклов амплификации, состоящих из денатурации ДНК, отжига и синтеза, при этом проводят мультиплексную полимеразную цепную реакцию с использованием 7-ми пар олигопраймеров и 7-ми олигонуклеотидных флуоресцентно-меченных зондов, охарактеризованных в п. 1, при этом концентрации прямых и обратных праймеров подобраны так, чтобы происходило ассиметричное накопление одной из цепей ДНК комплементарной соответствующему детектирующему флуоресцентно-меченному зонду, при следующих условиях: начальное плавление ДНК проводят в течение 3 минут при температуре 92°C, далее проводят 40 циклов амплификации, включающие денатурацию при 92°C в течение 15 секунд, отжиг и синтез при температуре 63°C в течение 40 секунд, причем амплификацию проводят с термостабильной полимеразой с вытесняющей активностью, затем осуществляют плавление продуктов полимеразной цепной реакции с олигонуклеотидными флуоресцентно-меченными зондами в температурном диапазоне от 20 до 95°C с шагом 0,5°C и временем задержки на каждом шаге в 1 секунду, при этом на каждом шаге проводят измерение флуоресцентного сигнала по каналам детекции всех меток FAM, R6G, ROX, Су5 для построения кривых плавления продукта и идентификации олигонуклеотидных полиморфизмов, основанную на изменении температуры плавления продукта ПЦР в зависимости от того какой аллельный вариант полиморфизма присутствует в образце ДНК.
При создании набора особое значение придавали выбору праймеров. Так как для создания высокоспецифичных мультиплексных систем требуется сочетание праймеров с определенными характеристиками (отсутствие внутренних вторичных структур).
Олигонуклеотидные праймеры и зонды выбраны с помощью программы «Primer3» и проверены на специфичность с помощью программы «Primer-BLAST».
При подборе праймеров, для повышения специфичности ПНР были соблюдены следующие критерии:
- размер праймера соответствовал 16-25 нуклеотидам;
- температура отжига всех праймеров была одинакова (58-62°C)
- разница в температуре плавления праймеров составляла не более 2 градусов;
- область отжига праймеров находилась вне зон полиморфизмов;
- суммарное количество GC-оснований соответствовало 50-60%;
- для улучшения качества отжига последние несколько нуклеотидов 3'-конца
праймера содержали GC-основания;
- отсутствовала внутренняя вторичная структура (праймеры не должны быть само- и взаимнокомплиментарными);
- отсутствовали комплементарности между 3'-концами (чтобы не образовывались праймер-димеры).
Для идентификации каждого однонуклеотидного полиморфизма были выбраны два праймера (прямой и обратный) и один флуоресцентно-меченный зонд (см. таблица 2). 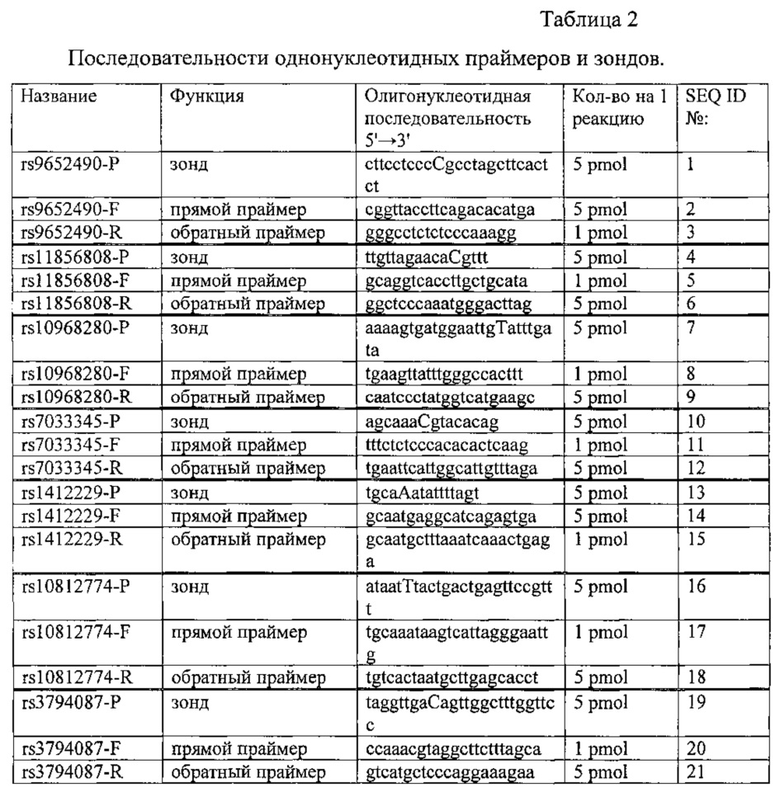
Кроме того набор содержал следующие реагенты: 50 мМ КСl, 15 мМ Tris-HCl с рН 8,8, 0,5% глицерол, 0,1% Tween 20, 6,25 мМ MgCl2, 250 мкМ дезоксинуклеотиды (dNTP) и 0,2 ед/мкл термостабильной ДНК-полимеразы с вытесняющей активностью.
Причем все реагенты для проведения мультиплексной полимеразной цепной реакции за исключением ДНК-полимеразы были смешаны в одном объеме вместе с 7-ю парами олигонуклеотидных праймеров - SEQ ID №: 2, 3, 5, 6, 8, 9, 11, 12, 14, 15, 17, 18, 20, 21 в концентрациях, обеспечивающих ассиметричное накопление цепей ДНК, и 7-ю олигонуклеотидными зондами, меченными флуоресцентными метками на 5'-конце и гасителями на 3'-конце: зонд SEQ ID №1 с меткой FAM и гасителем RTQ1; зонды SEQ ID №4 и 19 с меткой Су5 и гасителем BHQ2; зонд SEQ ID №7 с меткой R6G и гасителем BHQ2; зонды SEQ ID №10 и 16 с меткой ROX и гасителем BHQ2; зонд SEQ ID №13 с меткой R6G и гасителем BHQ1.
Способ выявления полиморфизмов в генах LINGO1, LINGO2 и SLC1A2, определяющих генетическую ассоциацию с эссенциальным тремором состоит из 4 этапов и осуществляется следующим образом.
Этап I. Выделение ДНК по стандартному протоколу.
Этап II. Проведение мультиплексной полимеразной цепной реакции с использованием 7-ми пар олигонуклеотидных праймеров и 7-ю олигонуклеотидными флуоресцентно-меченными зондами в 25 мкл смеси для ПЦР (см. таблица 3).
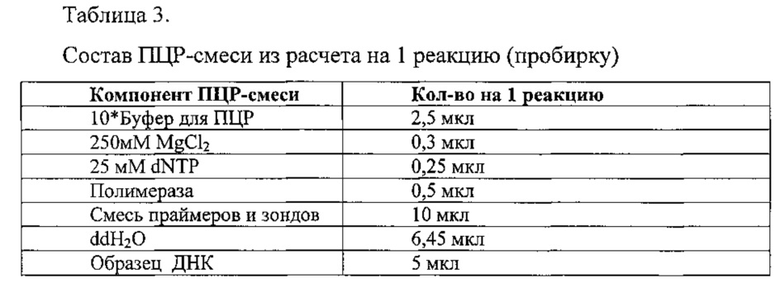
Концентрации олигонуклеотидных праймеров (см. таблица 2) подобраны так, чтобы происходило ассиметричное накопление одной из цепей ДНК (комплементарной детектирующим флуоресцентно-меченным зондам). При этом начальную денатурацию (плавление) ДНК проводят в течение 3 минут при температуре 92°C. Далее проводят 40 циклов амплификации, включающие денатурацию при 92°C в течение 15 секунд, отжиг и синтез при температуре 63°C в течение 40 секунд. Для этапа амплификации используется термостабильная полимераза с вытесняющей активностью, что позволяет избежать разрушения олигонуклеотидных зондов.
Этап III. Плавление продукта полимеразной цепной реакции, полученного на этапе II, с олигонуклеотидными зондами, которые мечены флуорофорами и гасителями флуоресценции. Проводится плавление в температурном диапазоне от 20 до 95°C с шагом 0,5°C и временем задержки на каждом шаге 1 секунда. При этом на каждом шаге проводят измерение флуоресцентного сигнала по каналам детекции красителей FAM, R6G, ROX, Су5 для построения кривых плавления продукта. Этапы II и III могут быть совмещены в одной программе (см. таблица 4).
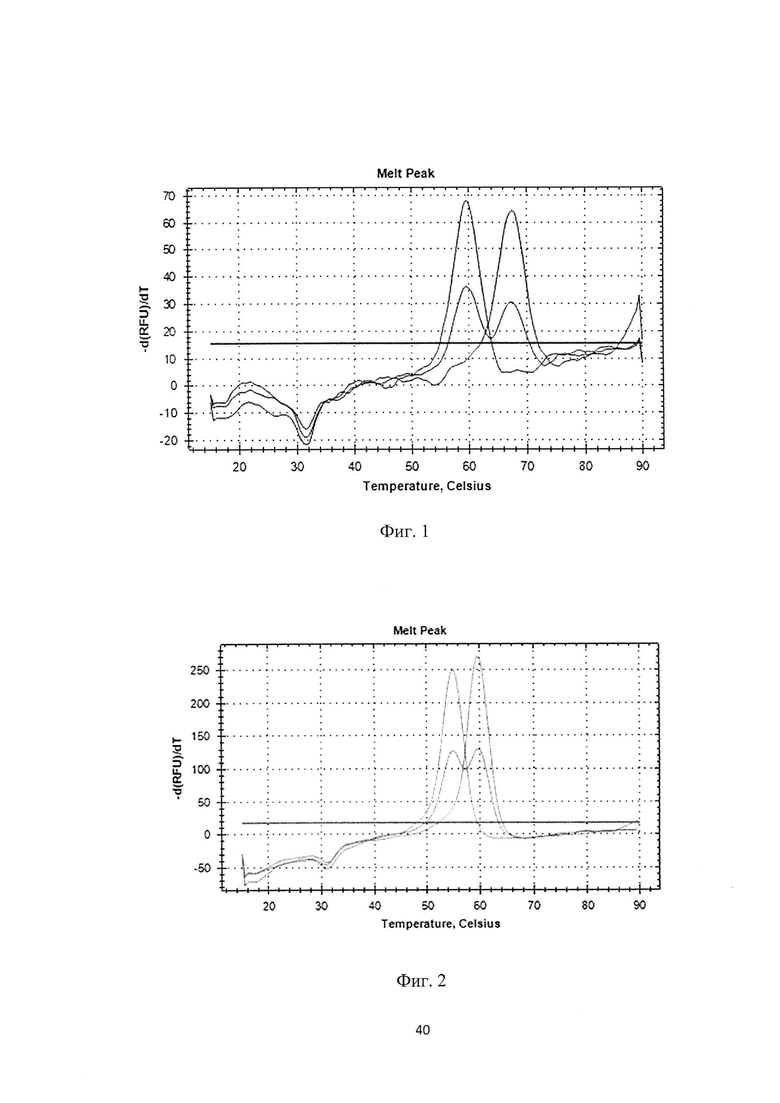
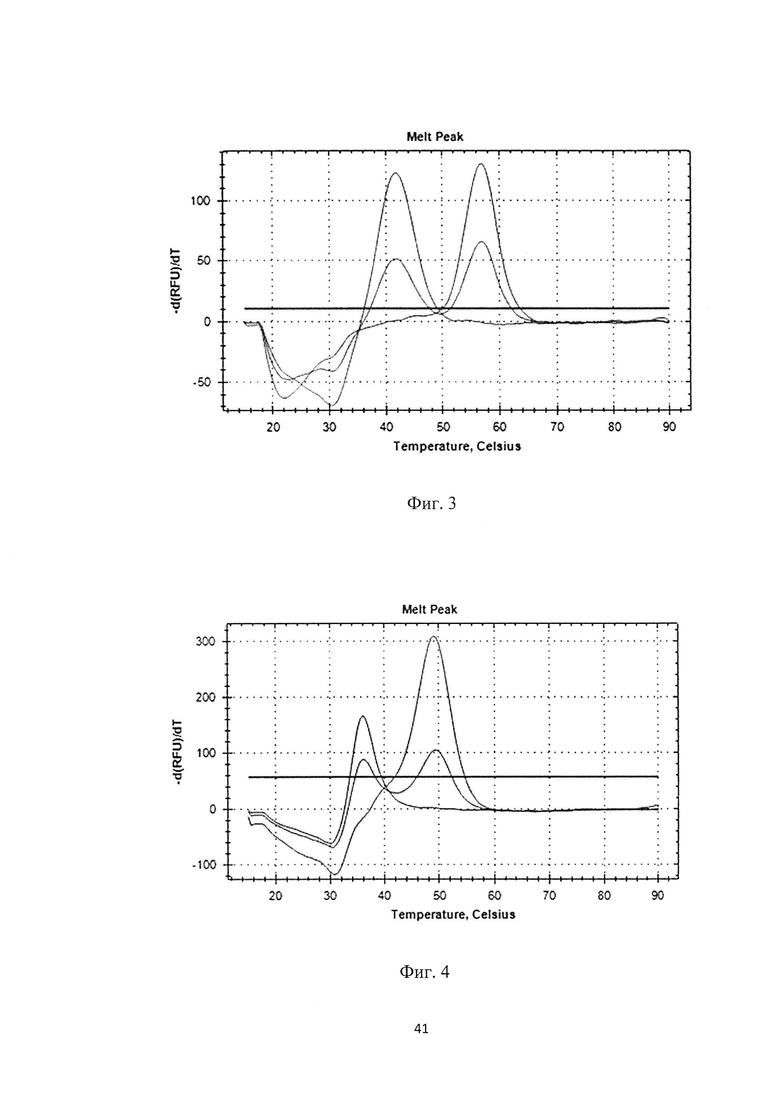
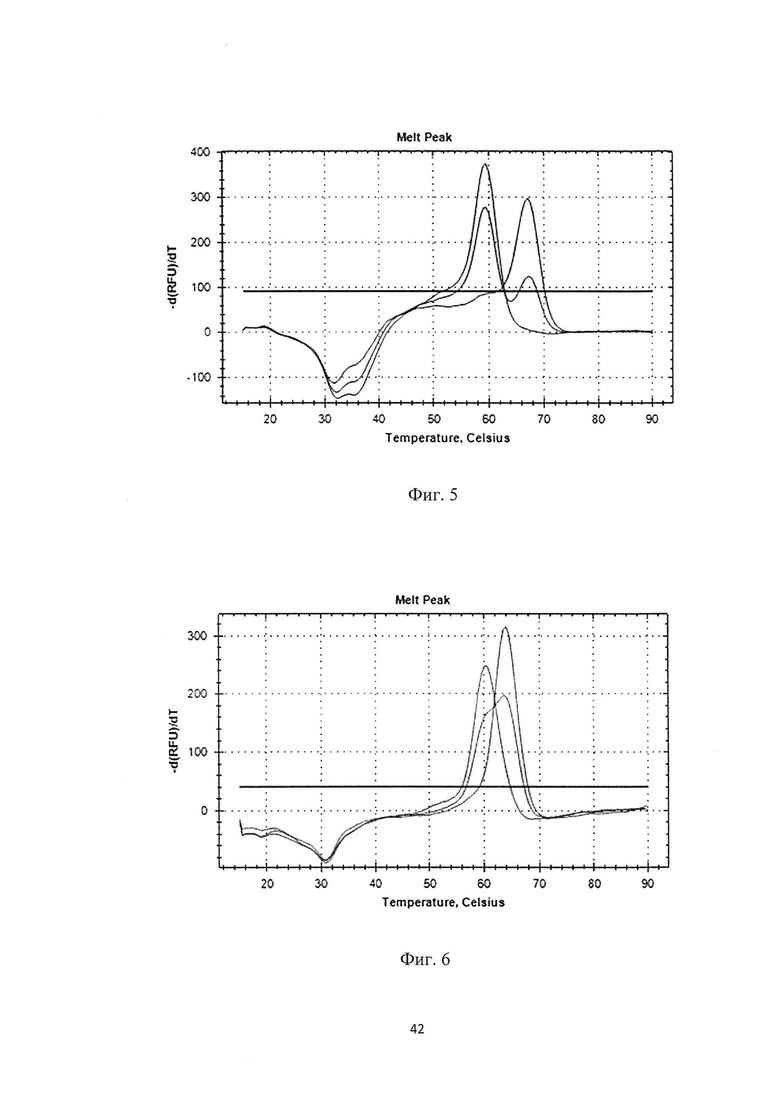
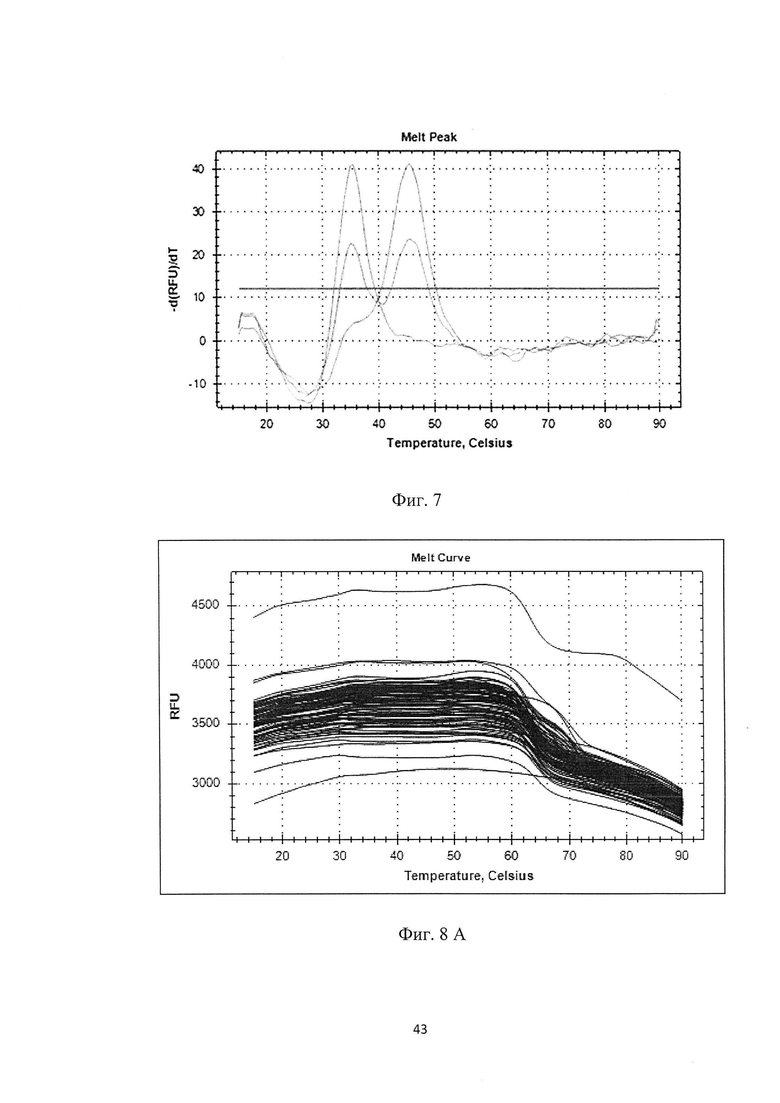
Этап VI. Анализ полученных кривых плавления по заданному протоколу (см. фиг. 1-7).
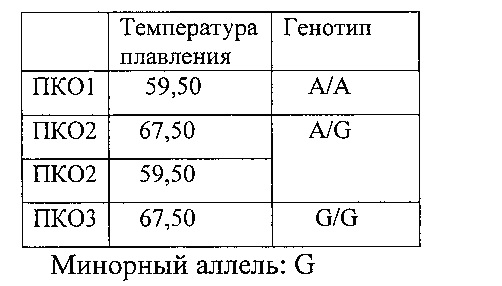
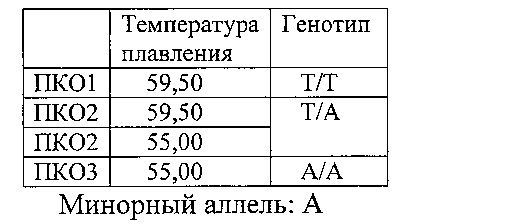
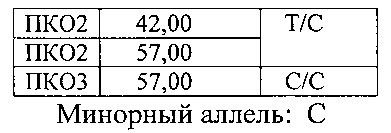
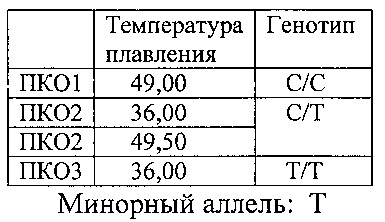
Идентификация полиморфизма для каждого SNP основана на изменении температуры плавления продукта ПЦР в зависимости от того какой аллельный вариант полиморфизма присутствует в образце ДНК. При анализе кривых плавления нужно руководствоваться величинами температур плавления трех положительных контролей (ПКО): ПКО1 - гомозигота дикого аллеля; ПКО2 - гетерозигота; ПКО3-гомозигота минорного аллеля, для каждого полиморфизма, на соответствующем из четырех каналов (FAM, HEX, ROX, Су5):
Канал FAM: rs9652490 (LINGO1) (см. фиг. 1)
(FAM)-cttcctcccCgcctagcttcactct-(RTQ 1)
Канал HEX: rs10968280 (LINGO2) (см. фиг. 2)
(R6G)-aaaagtgatggaattgTatttgata-(BHQ2)
Канал ROX: rs7033345 (LINGO2) (см. фиг. 3)
(ROX)-agcaaaCgtacacag-(BHQ2)

Канал Су5: rs11856808 (LINGO1) (см. фиг. 4)
(Cy5)-ttgttagaacaCgttt-(BHQ2)
Канал Су5: rs3794087 (SLC1A2) (см. фиг. 5)
(Cy5)-taggttgaCagttggctttggttcc-(BHQ2)
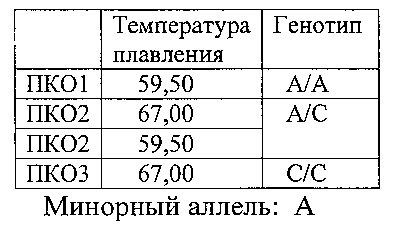
Канал ROX: rs10812774 (LINGO2) (см. фиг. 6)
(ROX)-ataatTtactgactgagttccgttt-(BHQ2)
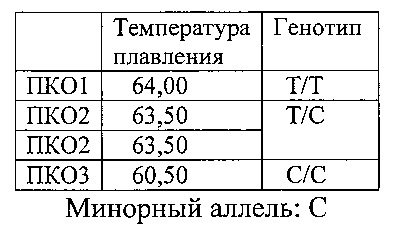
Канал HEX: rs1412229 (LINGO2) (см. фиг. 7)
(R6G)-tgcaAatattttagt-(BHQ1)
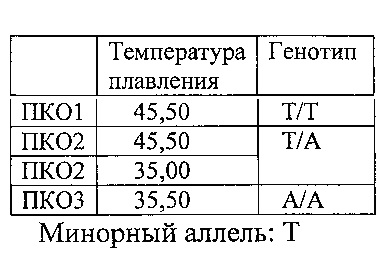
В результате проведенного исследования установлено, что выбранные праймеры и зонды для выявления генетического полиморфизма в полиморфных точках генов LINGO1 (rs9652490, rs11856808), LINGO2 (rs10968280, rs7033345, rs1412229, rs10812774) и SLC1A2 (rs3794087), определяющих генетическую ассоциацию с эссенциальным тремором методом мультиплексной полимеразной цепной реакции с гибридизационно-флуоресцентным учетом результатов обладают высокой специфичностью и обеспечивают ускорение и удешевление идентификации олигонуклеотидных полиморфизмов за счет возможности в мультиплексном формате в одной постановке сразу оценивать до восьми однонуклеотидных полиморфных маркеров (SNP).
Пример.
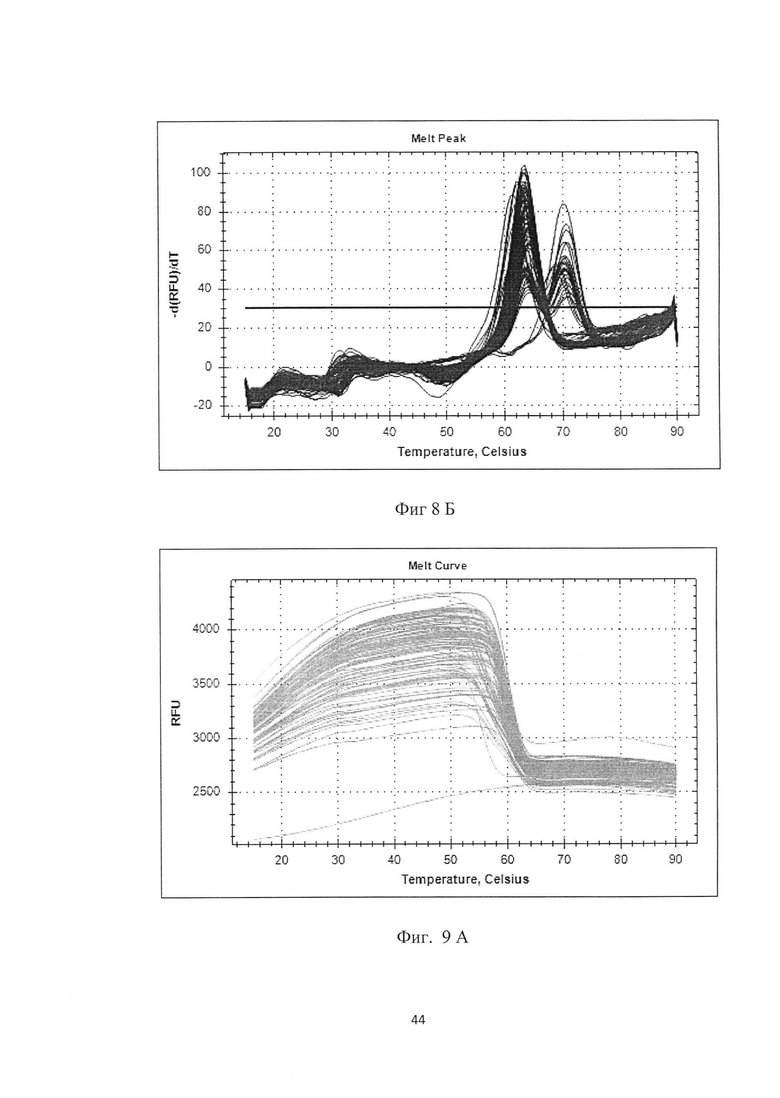
Из 56 образцов цельной крови от пациентов с диагнозом эссенциальный тремор и из 36 образцов цельной крови от пациентов с диагнозом болезнь Паркинсона (группа сравнения) была выделена геномная ДНК с помощью набора для выделения Wizard Genomic DNA Purification Kit (Promega USA). С помощью разработанного набора реагентов проведена ПЦР-наработка ампликонов в мультиплексном формате с последующим плавлением продукта реакции на амплификаторе CFX-96 "REAL TIME" (Bio-Rad США), с помощью объединенной программы. Анализ полученных кривых проведен с помощью программного обеспечения Bio-Rad CFX Manager 3.0 и представлен на фигурах 8-14 и в таблицах 5-11.
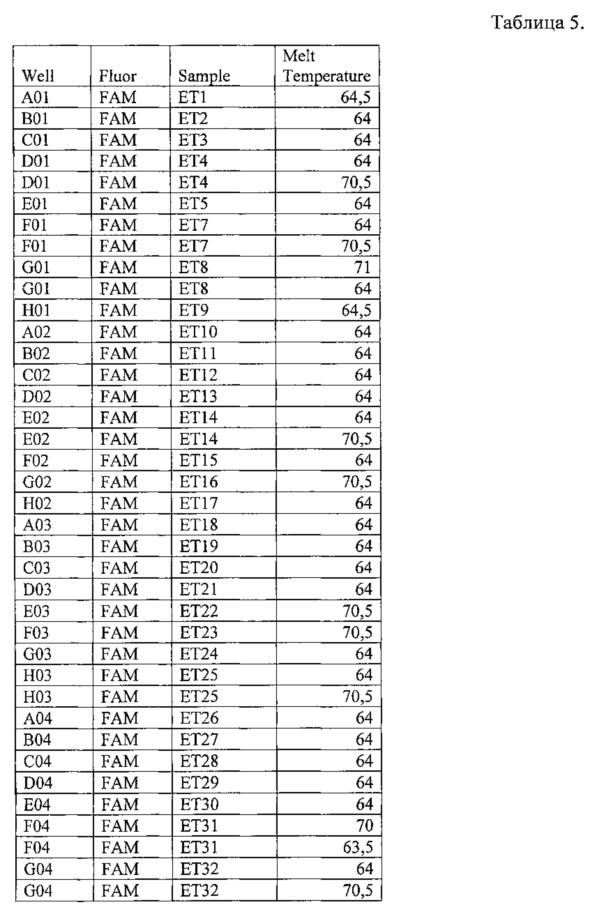
На фигуре 8А и Б и таблице 5 представлен анализ кривых плавления для rs9652490 LINGO1 (канал FAM). На фигуре А показаны кривые плавления (по вертикальной оси - интенсивность флуоресценции, по горизонтальной оси температура), на фигуре Б-дифференциальный анализ кривых плавления (по вертикальной оси - производная кривой интенсивности флуоресценции, по горизонтальной оси температура).
В таблице 5 представлены результаты анализа кривых плавления для rs9652490 LINGO1.
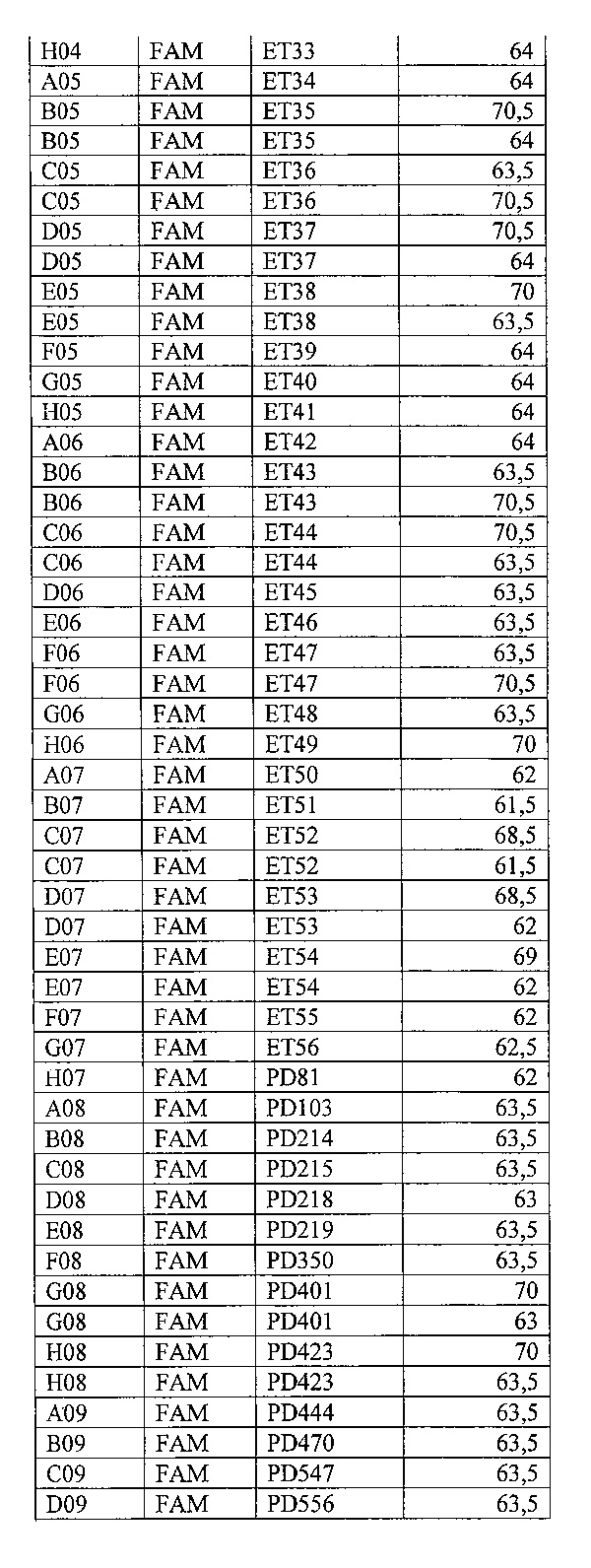
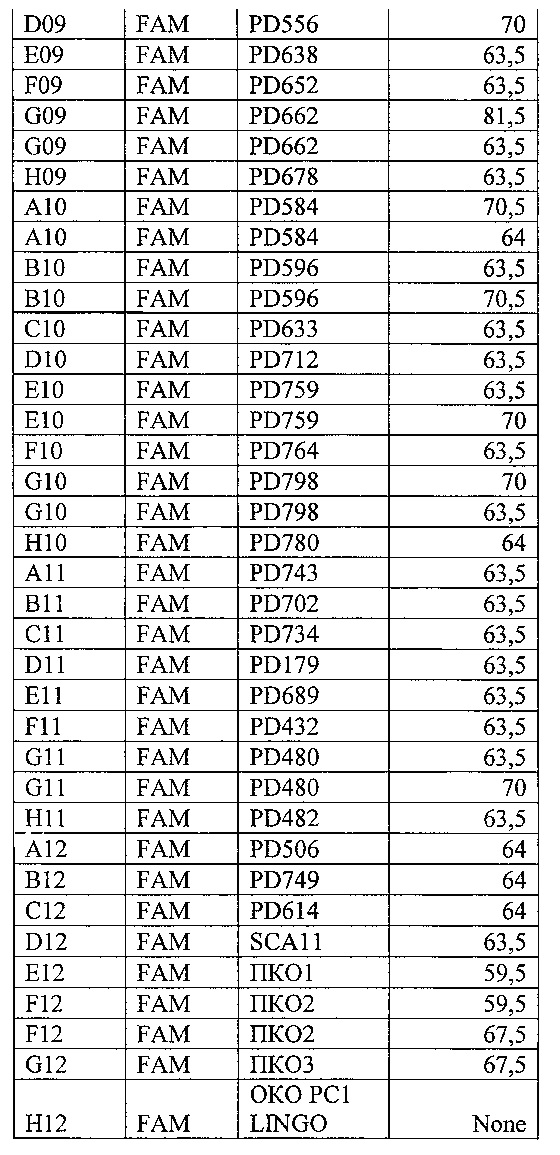
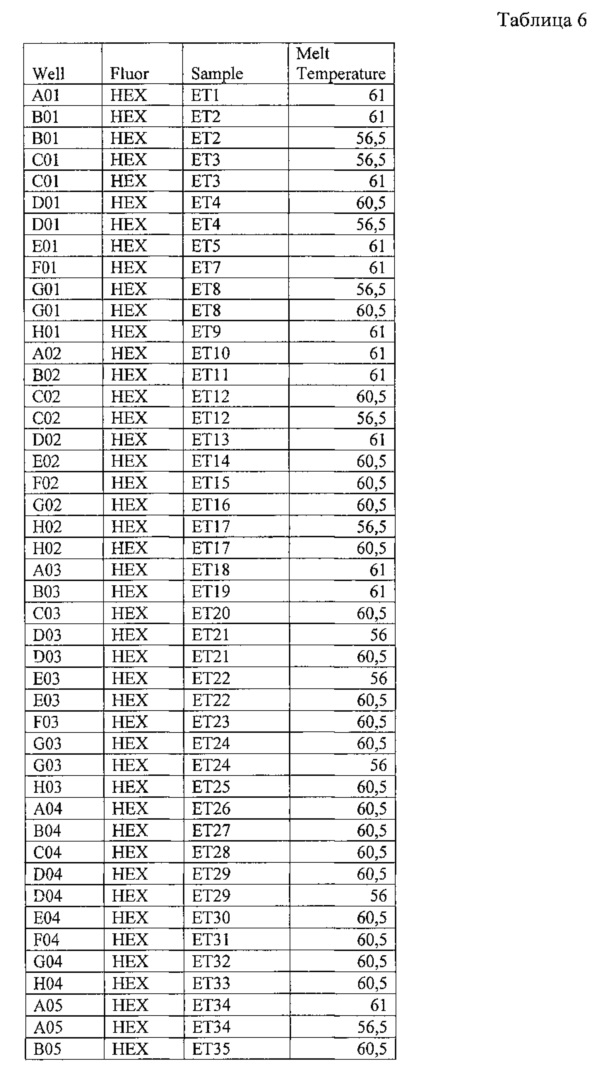
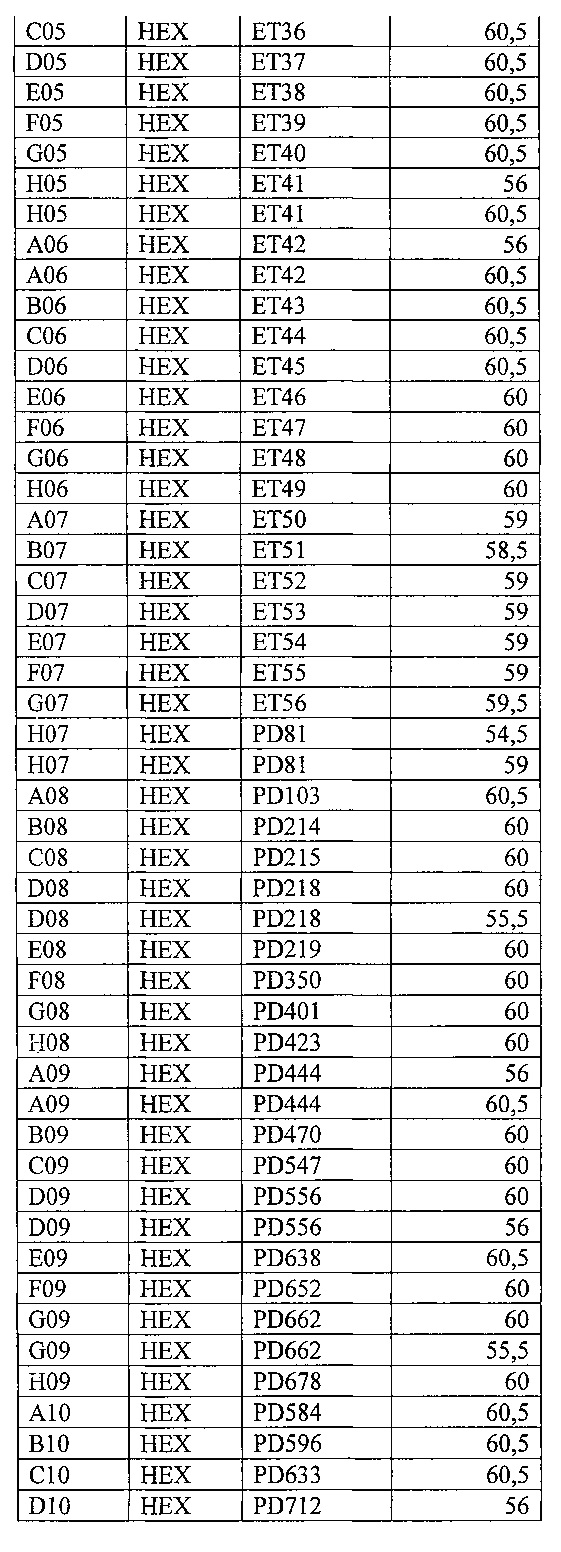
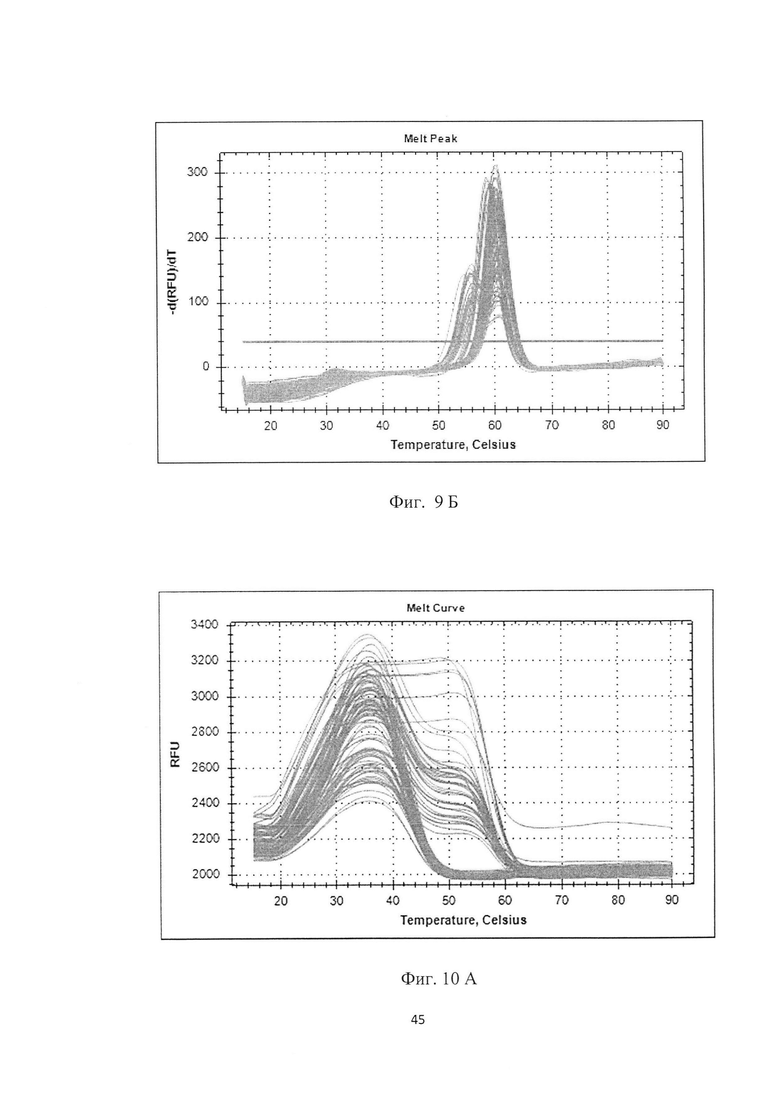
На фигуре 9А, Б и таблице 6 представлен анализ кривых плавления для rs10968280 LINGO2 (канал HEX). На фигуре А - показаны кривые плавления (по вертикальной оси - интенсивность флуоресценции, по горизонтальной оси температура), на фигуре Б - дифференциальный анализ кривых плавления.
В таблице 6 представлены результаты анализа кривых плавления для rs10968280 LINGO2.
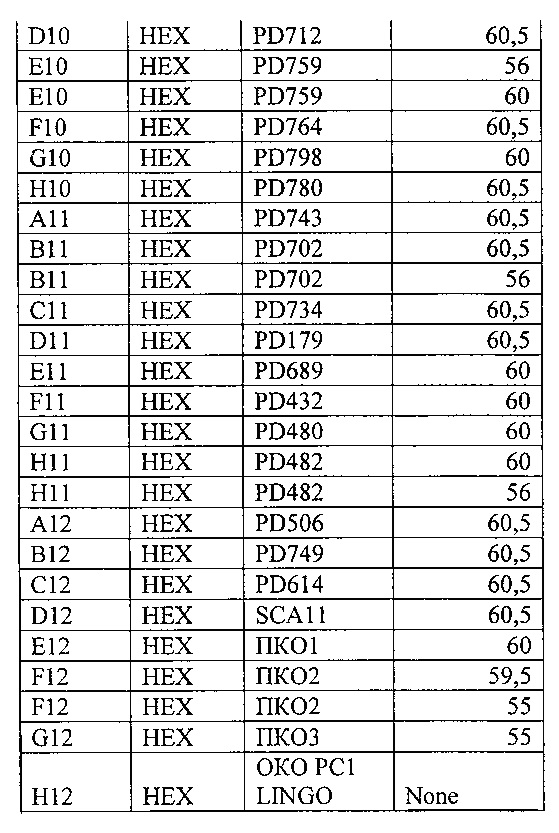
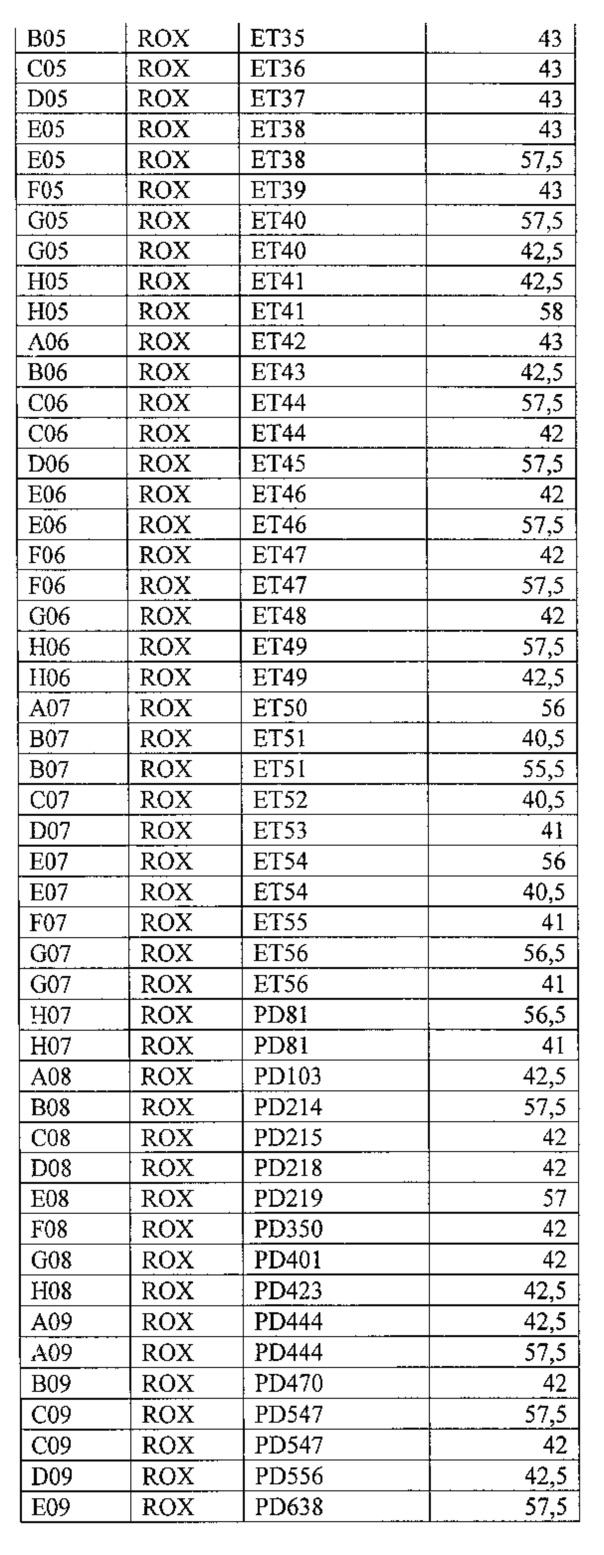
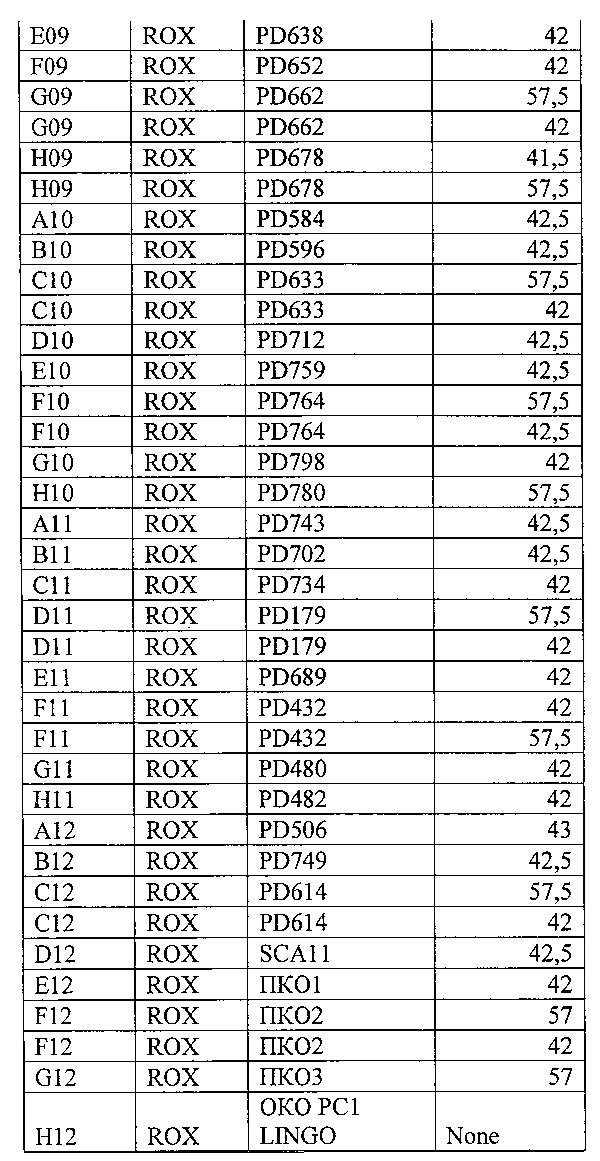
На фигуре 10 А, Б ив таблице 7 представлен анализ кривых плавления для rs7033345 LINGO2 (канал ROX). На фигуре А показаны кривые плавления (по вертикальной оси - интенсивность флуоресценции, по горизонтальной оси температура), на фигуре Б-дифференциальный анализ кривых плавления.
В таблице 7 представлены результаты анализа кривых плавления для rs7033345 LINGO2.

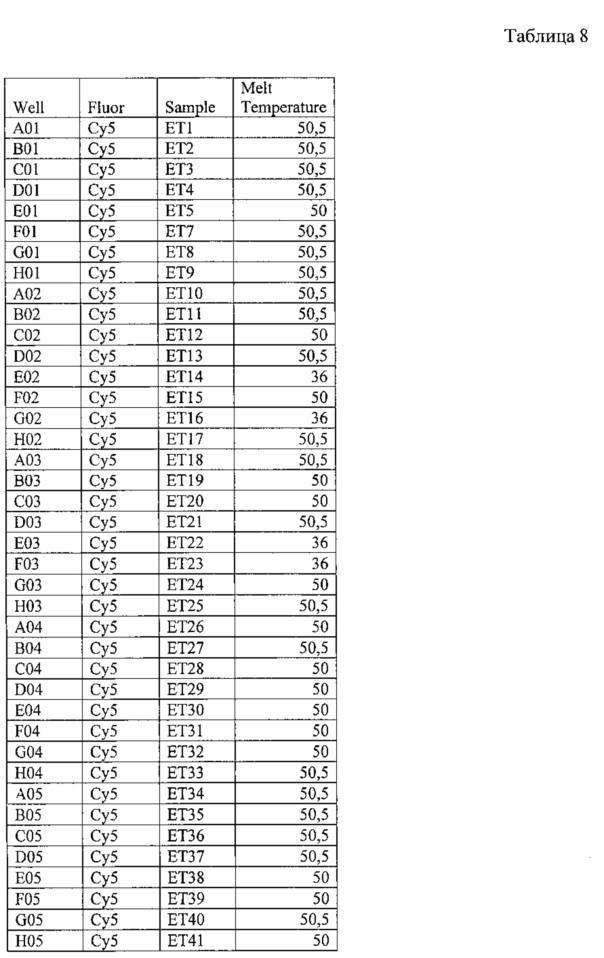
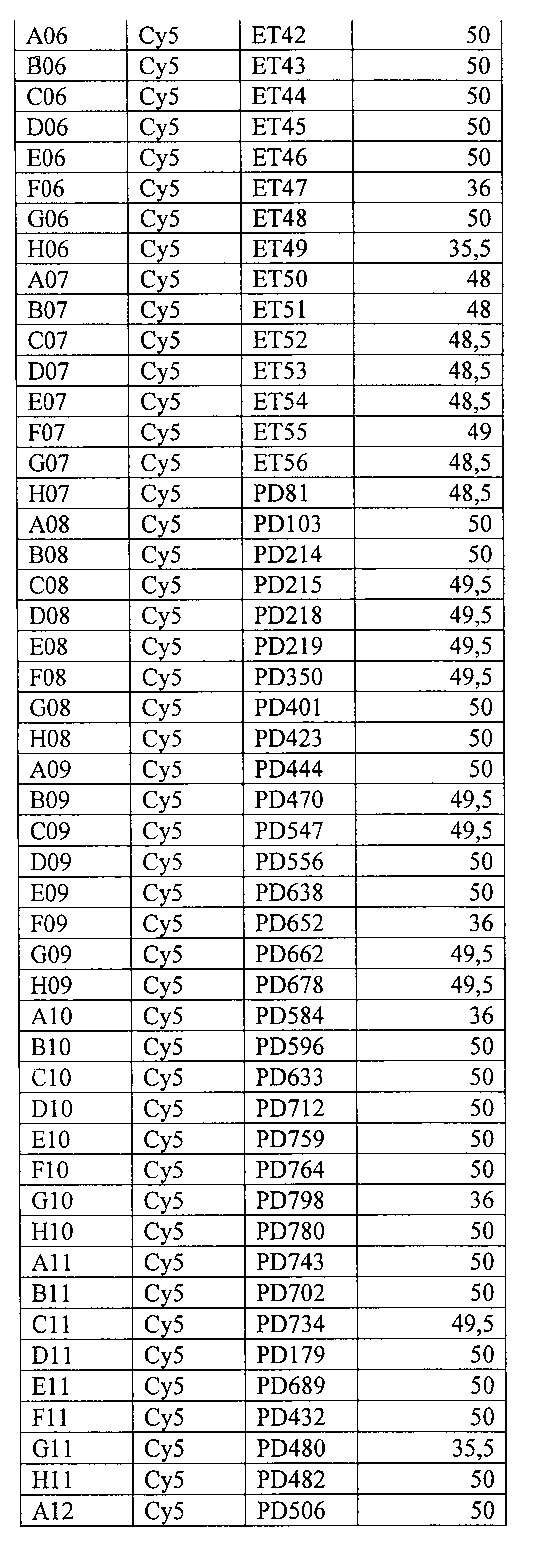
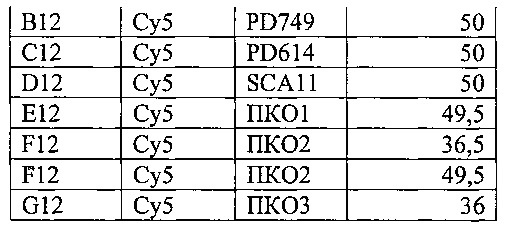
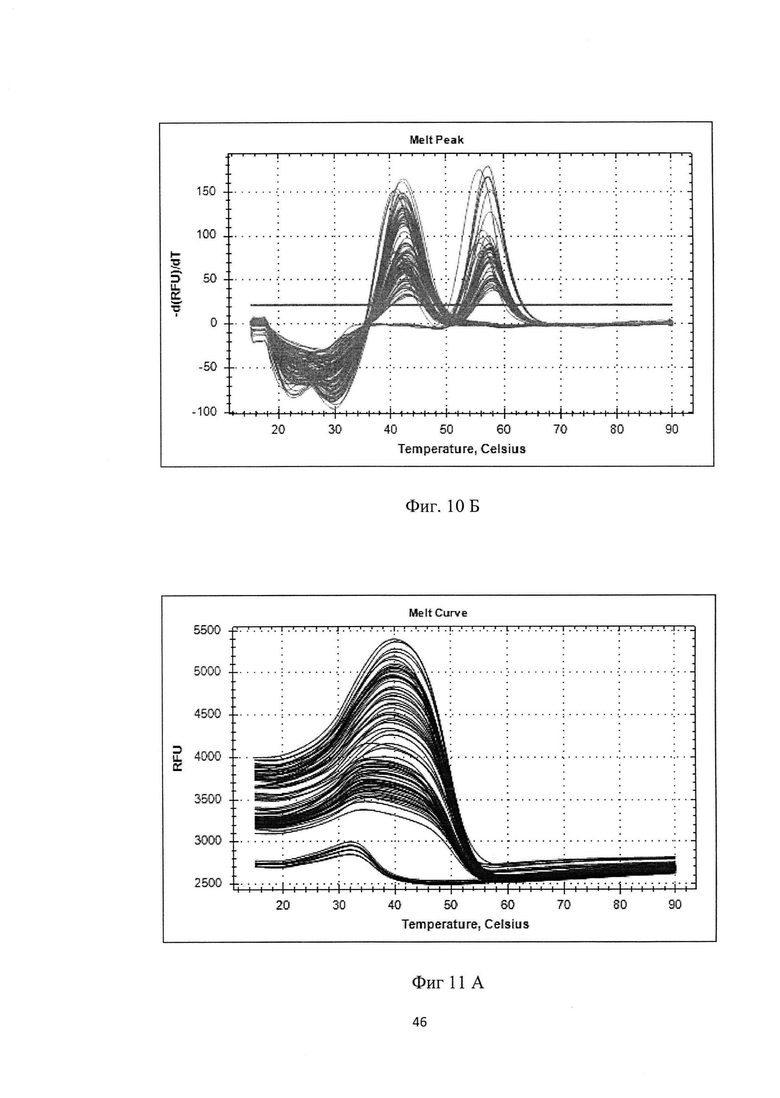
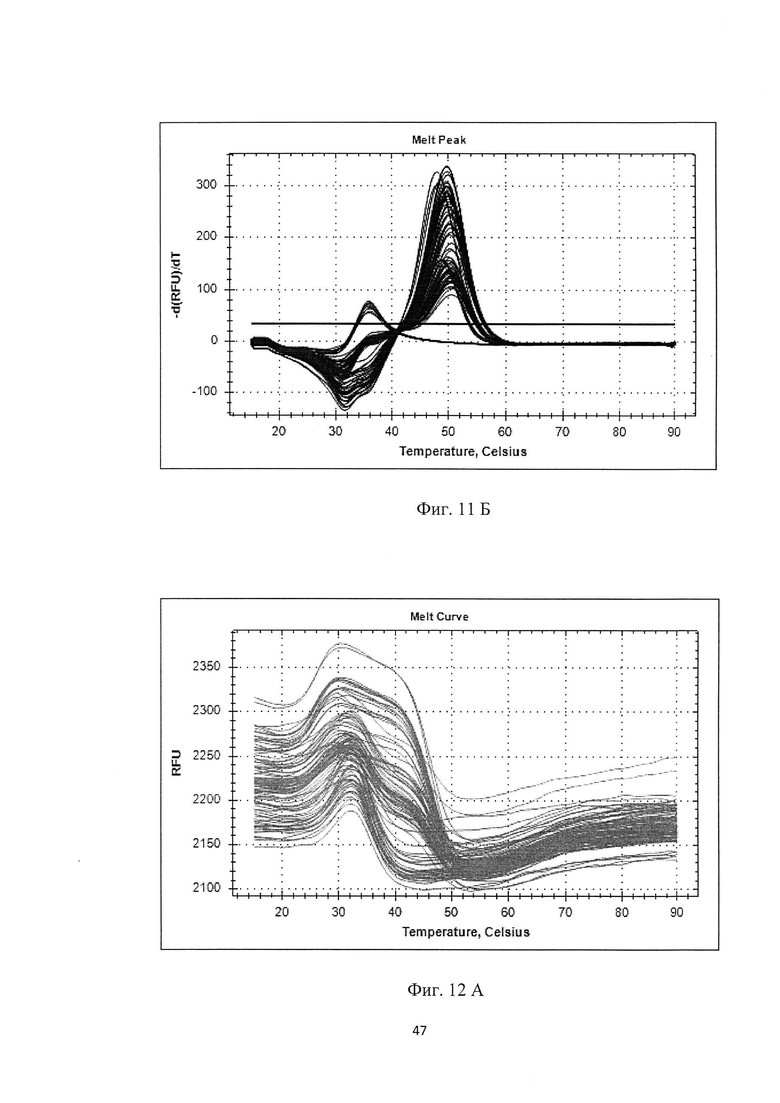
На фигуре 11А, Б и таблице 8 представлен анализ кривых плавления для rs11856808 LINGO1 (канал Су5). На фигуре А показаны кривые плавления (по вертикальной оси - интенсивность флуоресценции, по горизонтальной оси температура), на фигуре Б - дифференциальный анализ кривых плавления.
В таблице 8 представлены результаты анализа кривых плавления для rs1 1856808 LINGO1.
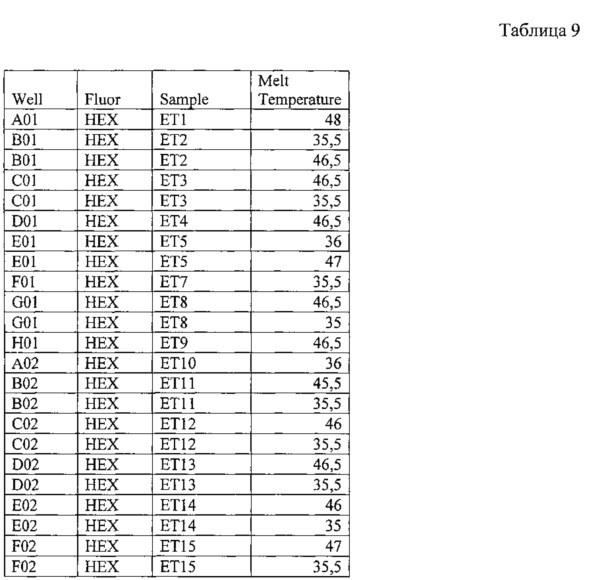
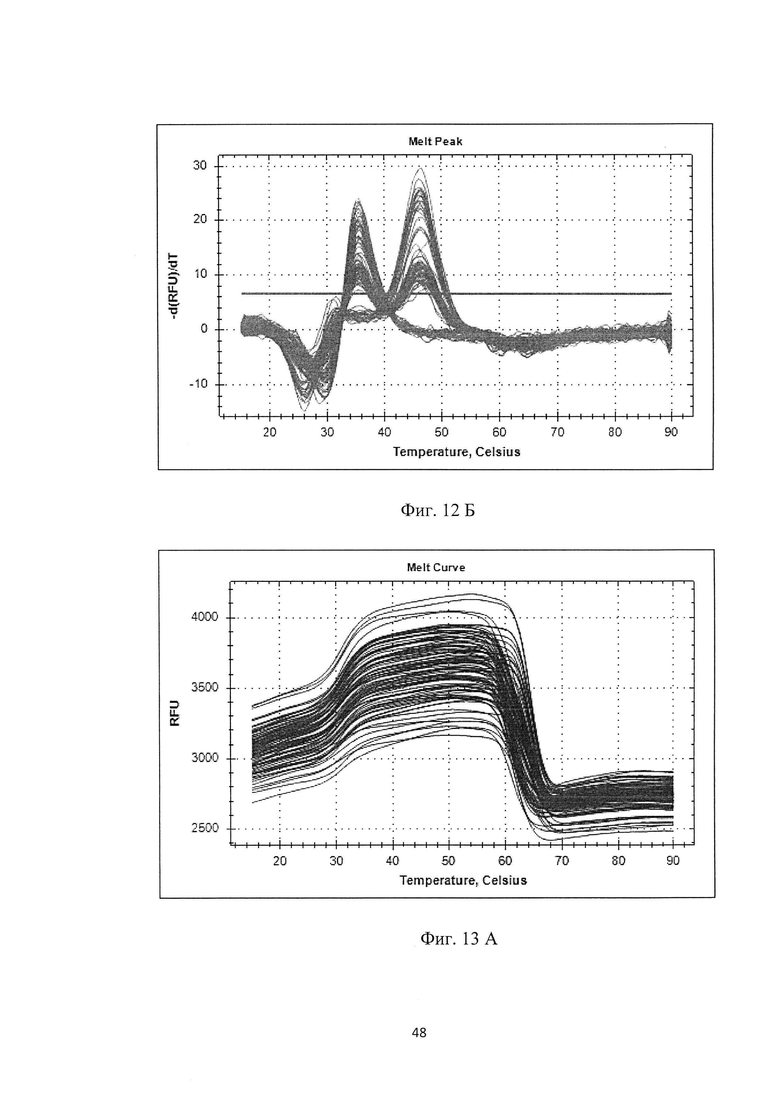
На фигуре 12А, Б и таблице 9 представлен анализ кривых плавления для rs1412229 LINGO2 (канал HEX). На фигуре А показаны кривые плавления (по вертикальной оси - интенсивность флуоресценции, по горизонтальной оси температура), на фигуре Б-дифференциальный анализ кривых плавления.
В таблице 9 представлены результаты анализа кривых плавления для rs1412229 LINGO2.
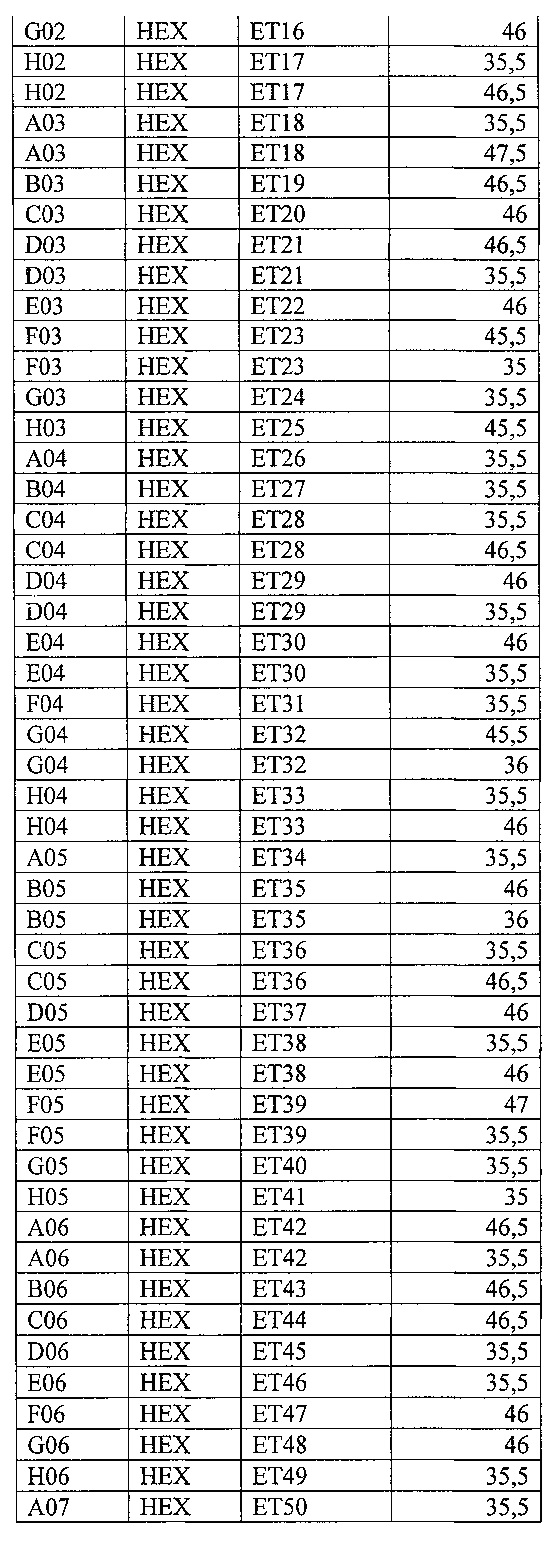
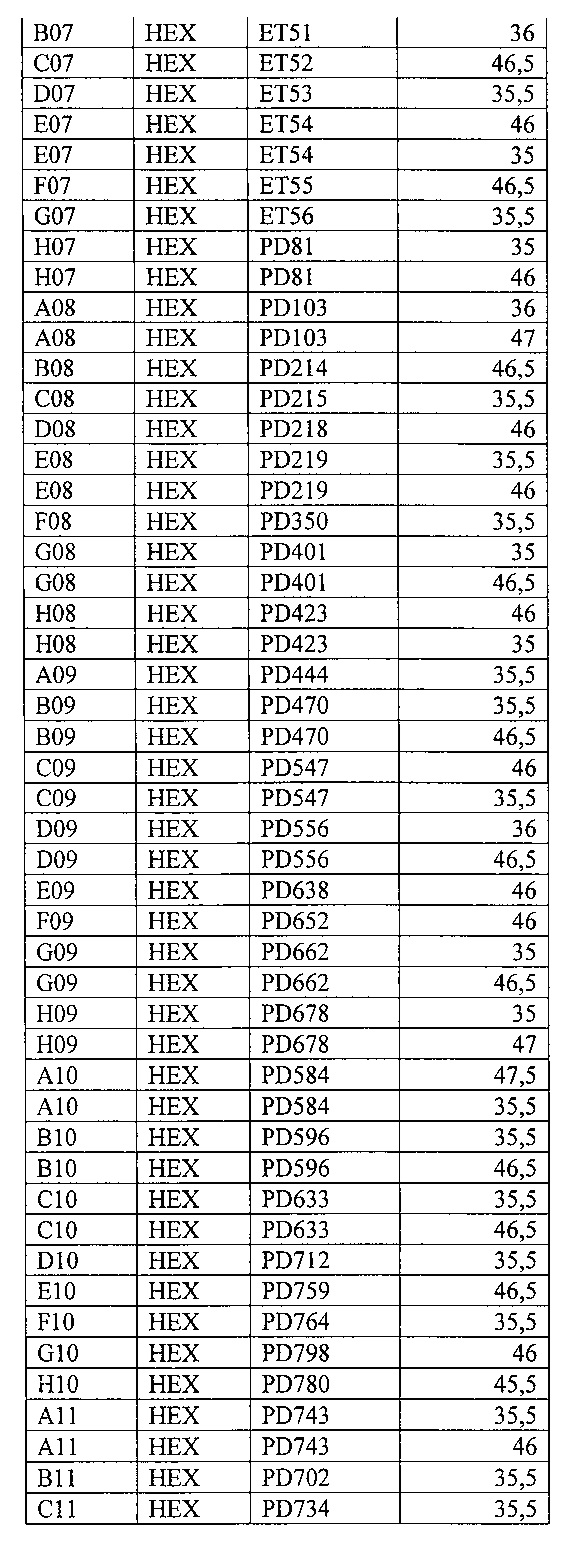
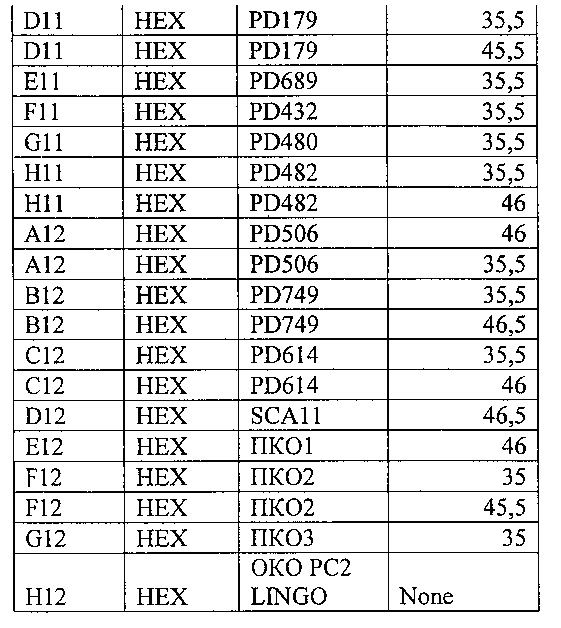
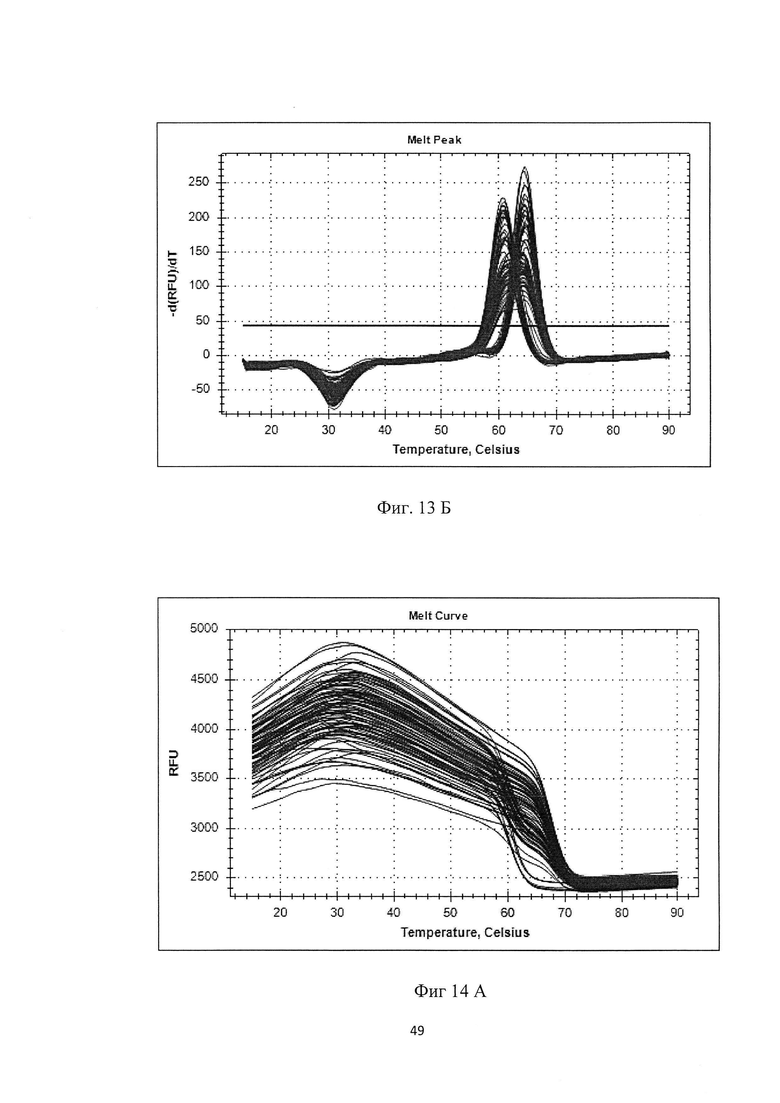
На фигуре 13А, Б и в таблице 10 представлен анализ кривых плавления для rs10812774 LINGO2 (канал ROX). На фигуре А показаны кривые плавления (по вертикальной оси - интенсивность флуоресценции, по горизонтальной оси температура), на фигуре Б - дифференциальный анализ кривых плавления.
В таблице 10 представлены результаты анализа кривых плавления для rs10812774 LINGO2.
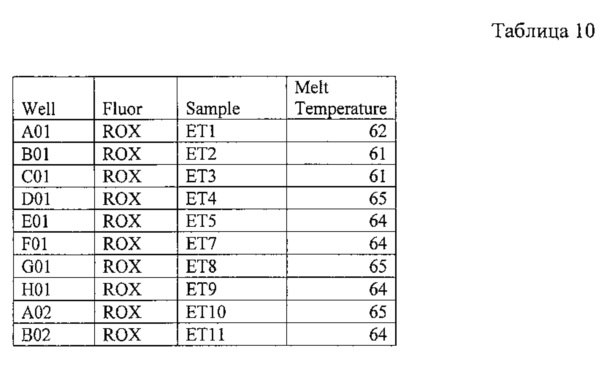
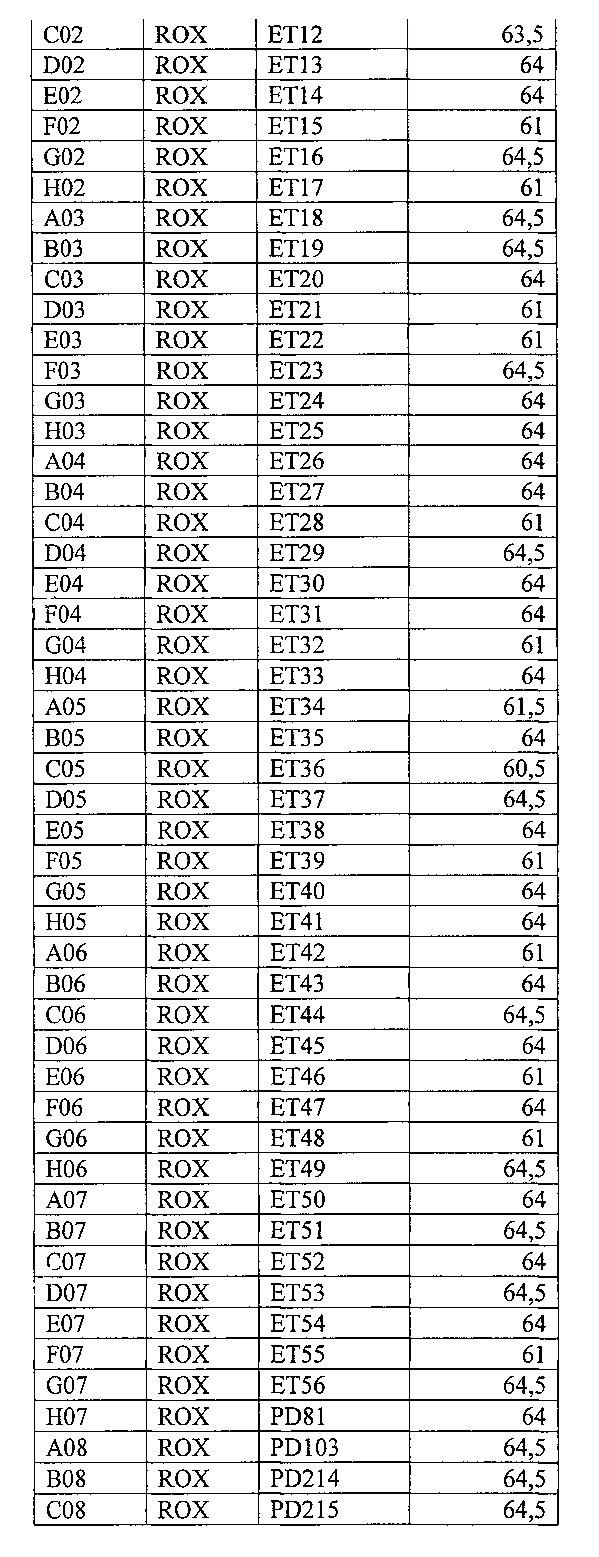
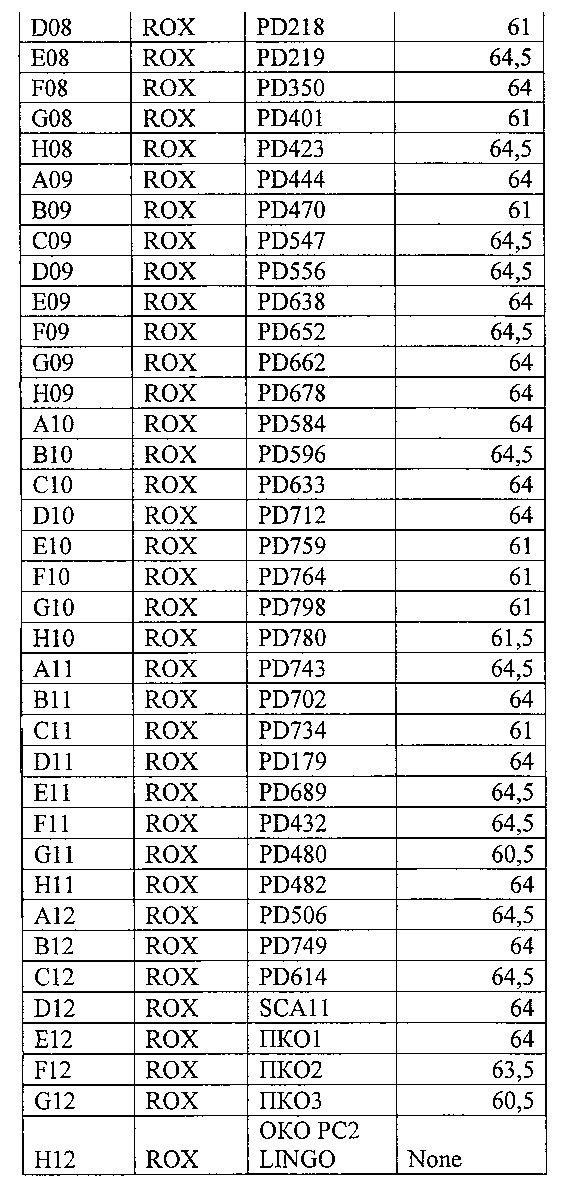
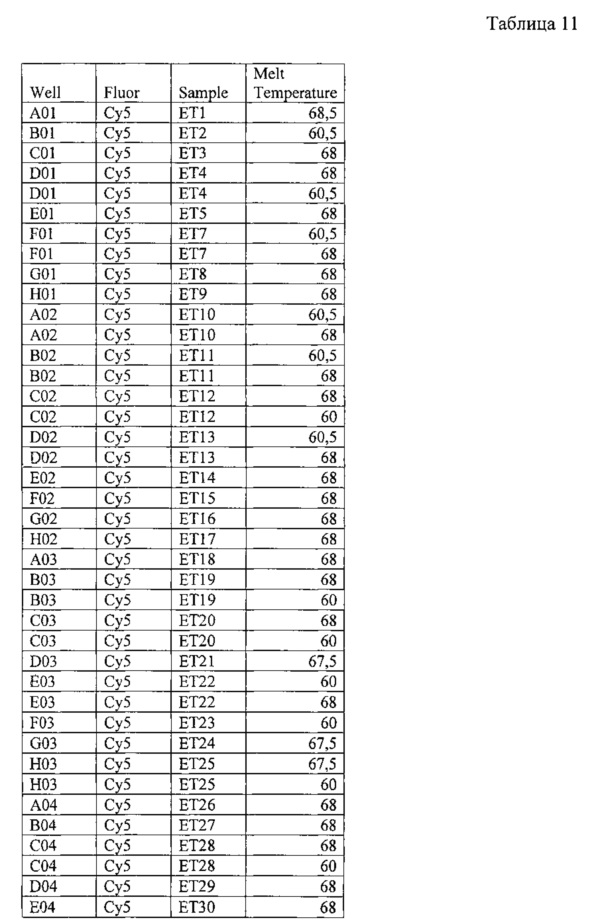
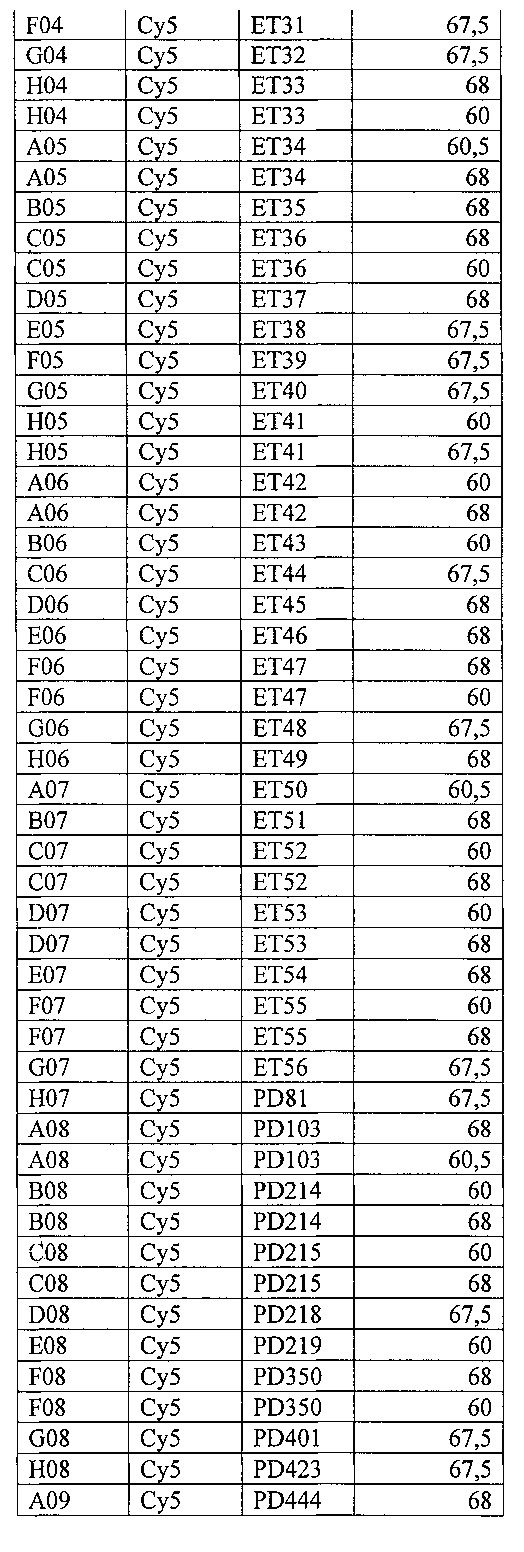
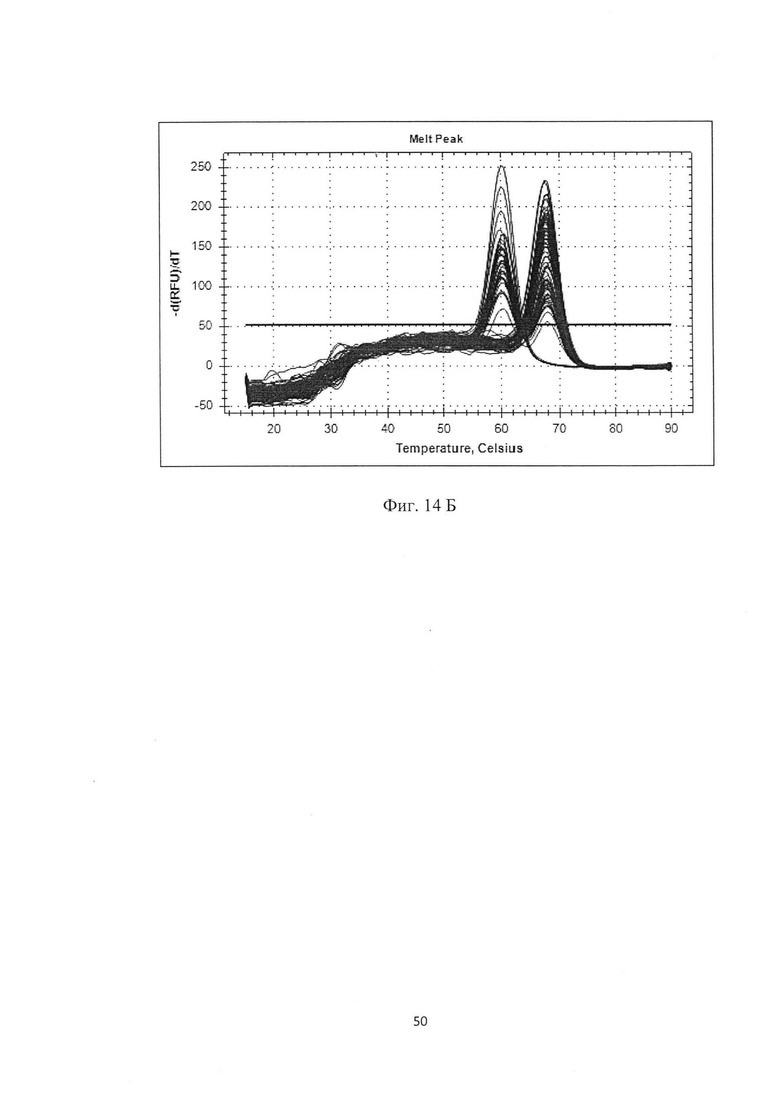
На фигуре 14А, Б и в таблице 11 представлен анализ кривых плавления для rs3794087 SLC1A2 (канал Су5). На фигуре А показаны кривые плавления (по вертикальной оси - интенсивность флуоресценции, по горизонтальной оси температура), на фигуре Б - дифференциальный анализ кривых плавления.
В таблице 11 представлены результаты анализа кривых плавления для rs3794087 SLC1A2.
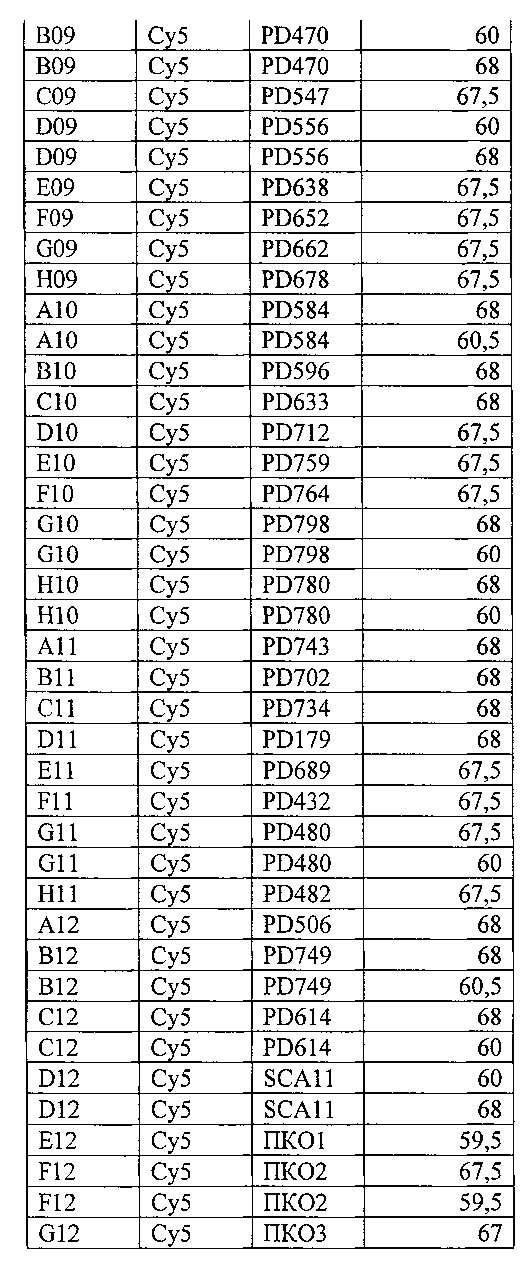
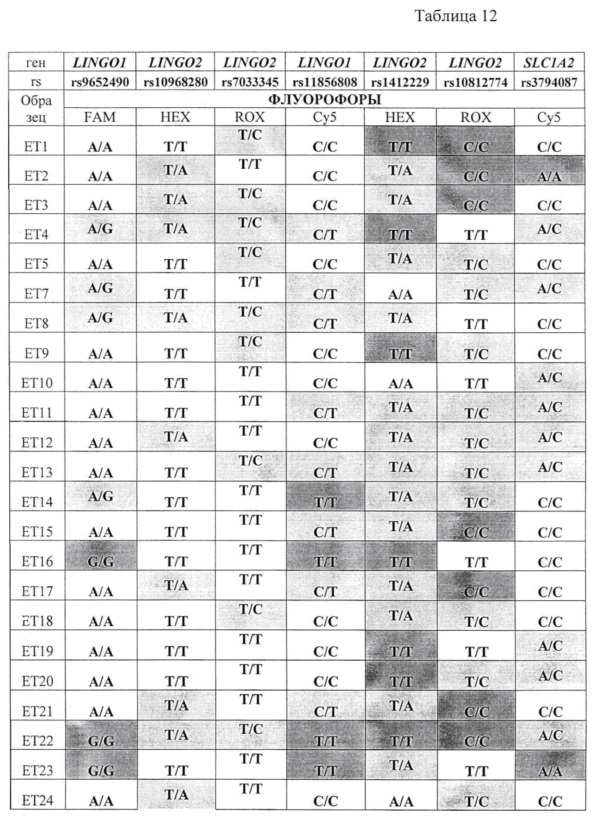
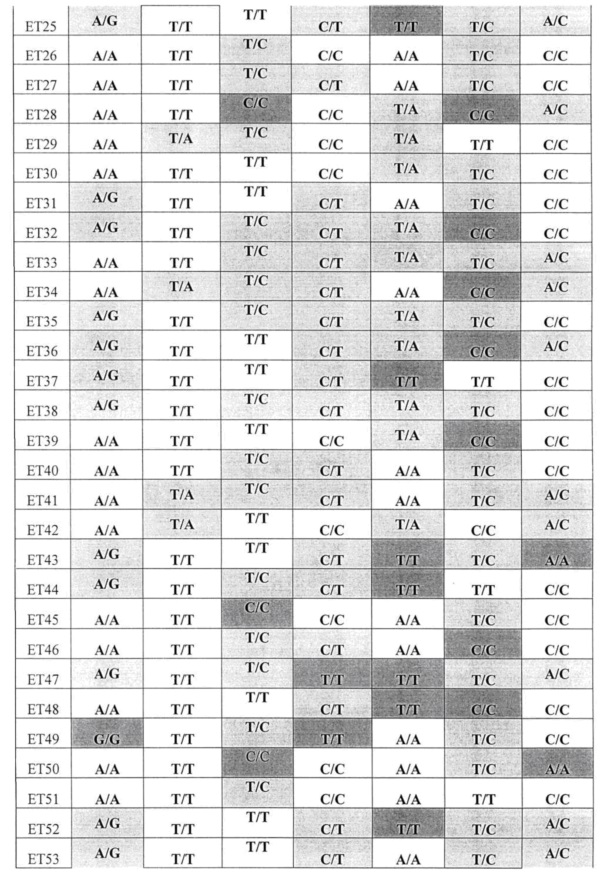
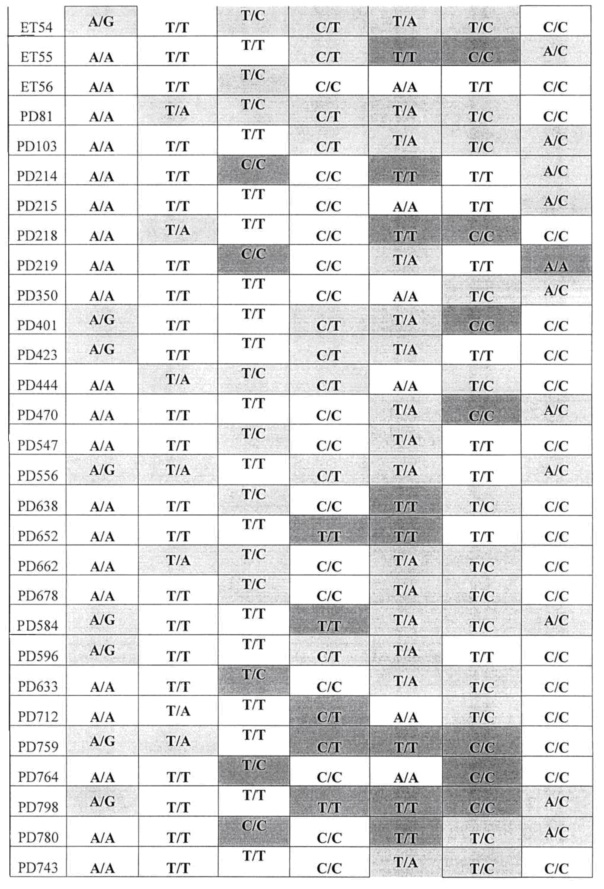
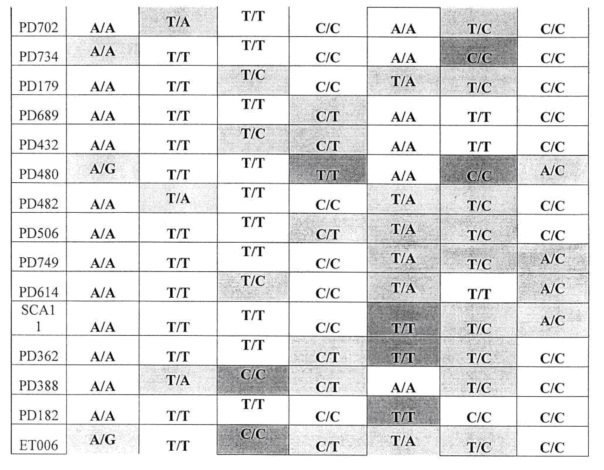
В таблице 12 приведены суммированные результаты анализа 92 образцов ДНК по 7 полиморфизмам (белый фон - гомозиготы дикого типа; светло серый фон - гетерозиготы; темно серый фон - гомозиготы мутантного типа)
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР РЕАГЕНТОВ ДЛЯ ВЫЯВЛЕНИЯ И ИДЕНТИФИКАЦИИ ДНК ВОЗБУДИТЕЛЕЙ БРУЦЕЛЛЕЗА МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ В РЕАЛЬНОМ ВРЕМЕНИ "ОМ-СКРИН-БРУЦЕЛЛЕЗ-РВ" | 2018 |
|
RU2715333C1 |
| СПОСОБ ДЕТЕКЦИИ СПЕЦИФИЧЕСКИХ НУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ И НУКЛЕОТИДНЫХ ЗАМЕН С ПОМОЩЬЮ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ С ЭФФЕКТОМ ГАШЕНИЯ ФЛУОРЕСЦЕНЦИИ ЗОНДА ПРАЙМЕРОМ | 2010 |
|
RU2451086C1 |
| НАБОР И СПОСОБ ДЛЯ УСКОРЕННОЙ ИДЕНТИФИКАЦИИ ЧУМНОГО МИКРОБА С ОДНОВРЕМЕННОЙ ДИФФЕРЕНЦИАЦИЕЙ ВИРУЛЕНТНЫХ И АВИРУЛЕНТНЫХ ШТАММОВ Y.PESTIS, ОПРЕДЕЛЕНИЕМ ИХ ПЛАЗМИДНОГО ПРОФИЛЯ | 2011 |
|
RU2473701C1 |
| Способ диагностики варианта SOPH c.5741G>A в гене NBAS | 2024 |
|
RU2819985C1 |
| Набор для определения копийности гена АРР в геноме человека | 2021 |
|
RU2789799C1 |
| Способ генотипирования полиморфного локуса rs7986407 (A>G) гена FOXO1 у человека методом ПЦР в режиме "реального времени" с применением аллель-специфических флуоресцентных зондов | 2024 |
|
RU2826734C1 |
| Набор для определения CCR5delta32 мутации в геноме человека | 2020 |
|
RU2748998C1 |
| Способ генотипирования полиморфного локуса rs547025 (T>C) гена SIRT3 у человека методом ПЦР в режиме "реального времени" с применением аллель-специфических флуоресцентных зондов | 2024 |
|
RU2835194C1 |
| Способ генотипирования полиморфного локуса rs58415480 (C>G) гена SYNE1 у человека методом ПЦР в режиме "реального времени" с применением аллель-специфических флуоресцентных зондов | 2024 |
|
RU2828032C1 |
| Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов и способ выявления ДНК возбудителей сапа и мелиоидоза методом ПЦР с детекцией продукта в режиме реального времени | 2019 |
|
RU2738358C1 |
Предложенная группа изобретений относится к области медицины, в частности к неврологии и молекулярной биологии. Предложены способ и набор реагентов для выявления генетического полиморфизма в полиморфных точках rs9652490 LINGO1 (A>G), rs11856808 LINGO1 (C>Т), rs10968280 LINGO2 (А>Т), rs7033345 LINGO2 (C>Т), rs1412229 LINGO2 (А>Т), rs10812774 LINGO2 (C>Т) и rs3794087 SLC1A2 (А>С), определяющих генетическую ассоциацию с эссенциальным тремором. Предложенная группа изобретений обеспечивает высокую специфичность и сокращение сроков идентификации олигонуклеотидных полиморфизмов за счет возможности в мультиплексном формате в одной постановке оценивать семь однонуклеотидных полиморфных маркеров. 2 н. и 1 з.п. ф-лы, 21 ил., 19 табл., 1 пр.
1. Набор реагентов для выявления генетического полиморфизма в полиморфных точках: rs9652490 в гене LINGO1 (A>G), rs11856808 в гене LINGO1 (C>Т), rs10968280 в гене LINGO2 (А>Т), rs7033345 в гене LINGO2 (C>Т), rs1412229 в гене LINGO2 (А>Т), rs10812774 в гене LINGO2 (C>Т) и rs3794087 в гене SLC1A2 (А>С), определяющих генетическую ассоциацию с эссенциальным тремором, включающий компоненты: 50 мМ KCl, 15 мМ Tris-HCl с рН 8,8, 0,5% глицерол, 0,1% Tween 20, 6,25 мМ MgCl2, 250 мкМ дезоксинуклеотиды (dNTP) и 0,2 ед/мкл термостабильную ДНК-полимеразу с вытесняющей активностью, при этом все компоненты для проведения мультиплексной полимеразной цепной реакции за исключением ДНК-полимеразы смешаны в одном объеме вместе с 7 парами олигонуклеотидных праймеров, обладающих активностью прямого и обратного праймеров в полимеразной цепной реакции - SEQ ID №: 2, 3, 5, 6, 8, 9, 11, 12, 14, 15, 17, 18, 20, 21 в концентрациях, обеспечивающих ассиметричное накопление цепей ДНК, и 7 олигонуклеотидными зондами, меченными флуоресцентными метками на 5'-конце и гасителями на 3'-конце: зонд SEQ ID №1 с меткой FAM и гасителем RTQ1; зонды SEQ ID №4 и 19 с меткой Су5 и гасителем BHQ2; зонд SEQ ID №7 с меткой R6G и гасителем BHQ2; зонды SEQ ID №10 и 16 с меткой ROX и гасителем BHQ2; зонд SEQ ID №13 с меткой R6G и гасителем BHQ1.
2. Набор по п. 1, отличающийся тем, что олигонуклеотидный флуоресцентно-меченный зонд, прямой и обратный праймеры используют на одну реакцию в следующих количественных соотношениях в pmol: зонд SEQ ID №1, прямой праймер SEQ ID №:2, обратный праймер SEQ ID №:3 - 5:5:1 соответственно, зонд SEQ ID №13, прямой праймер SEQ ID №:14, обратный праймер SEQ ID №:15 - 5:5:1 соответственно; зонд SEQ ID №4, прямой праймер SEQ ID №:5, обратный праймер SEQ ID №:6 - 5:1:5 соответственно, зонд SEQ ID №7, прямой праймер SEQ ID №:8, обратный праймер SEQ ID №:9 - 5:1:5 соответственно, зонд SEQ ID №10, прямой праймер SEQ ID №:11, обратный праймер SEQ ID №:12 - 5:1:5 соответственно, зонд SEQ ID №16, прямой праймер SEQ ID №:17, обратный праймер SEQ ID №:18 - 5:1:5 соответственно и зонд SEQ ID №19, прямой праймер SEQ ID №:20, обратный праймер SEQ ID №:21 - 5:1:5 соответственно.
3. Способ выявления генетического полиморфизма в полиморфных точках: rs9652490 в гене LINGO1 (A>G), rs11856808 в гене LINGO1 (C>Т), rs10968280 в гене LINGO2 (А>Т), rs7033345 в гене LINGO2 (C>Т), rs1412229 в гене LINGO2 (А>Т), rs10812774 в гене LINGO2 (C>Т) и rs3794087 в гене SLC1A2 (А>С), определяющих генетическую ассоциацию с эссенциальным тремором, включающий выделение ДНК, проведение полимеразной цепной реакции путем начального плавления и ряда циклов амплификации, состоящих из денатурации ДНК, отжига и синтеза, отличающийся тем, что проводят мультиплексную полимеразную цепную реакцию с использованием набора реагентов по п. 1, при этом концентрации прямых и обратных праймеров подобраны так, чтобы происходило ассиметричное накопление одной из цепей ДНК комплементарной соответствующему детектирующему флуоресцентно-меченному зонду, при следующих условиях: начальное плавление ДНК проводят в течение 3 минут при температуре 92°С, далее проводят 40 циклов амплификации, включающих денатурацию при 92°С в течение 15 секунд, отжиг и синтез при температуре 63°С в течение 40 секунд, причем амплификацию проводят с термостабильной полимеразой с вытесняющей активностью, затем осуществляют плавление продуктов полимеразной цепной реакции, с соответствующими олигонуклеотидными флуоресцентно-меченными зондами, в температурном диапазоне от 20 до 95°С с шагом 0,5°С и временем задержки на каждом шаге в 1 секунду, при этом на каждом шаге проводят измерение флуоресцентного сигнала по каналам детекции всех меток FAM, R6G, ROX, Су5 для построения кривых плавления продукта и идентификации олигонуклеотидных полиморфизмов, основанную на изменении температуры плавления продукта ПЦР в зависимости от того, какой аллельный вариант полиморфизма присутствует в образце ДНК, а именно для полиморфизма rs9652490 в гене LINGO1: АА, AG или CG; для полиморфизма rs11856808 в гене LINGO1: СС, СТ или ТТ; для полиморфизма rs10968280 в гене LINGO2: ТТ, ТА или АА, для полиморфизма rs7033345 в гене LINGO2: ТТ, ТС или СС; для полиморфизма rs1412229 в гене LINGO2: АА, ТА или ТТ, для полиморфизма rs10812774 в гене LINGO2: ТТ, ТС или СС, для полиморфизма rs3794087 в гене SLC1A2: СС, АС или АА, где в случае выявления аллеля G rs9652490 в гене LINGO1 (A>G), A rs10968280 в гене LINGO2 (А>Т), С rs7033345 в гене LINGO2 (C>Т), Т rs11856808 в гене LINGO1 (C>Т), А rs3794087 в гене SLC1A2 (А>С), С rs10812774 в гене LINGO2 (C>Т) и Т rs1412229 в гене LINGO2 (А>Т) определяют генетическую ассоциацию с эссенциальным тремором.
| WO 2005105839 A2, 10.11.2005VILARINO-GUELL C | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Neurogenetics | |||
| Приспособление для суммирования отрезков прямых линий | 1923 |
|
SU2010A1 |
| Приспособление для суммирования отрезков прямых линий | 1923 |
|
SU2010A1 |
| Найдено из Интернет: URL: https://www.ncbi.nlm.nih.gov/pubmed/20369371KUHLENBAUMER G | |||
| et al | |||
| Genetics of essential tremor: meta-analysis and review | |||
| Neurology | |||
| Способ защиты переносных электрических установок от опасностей, связанных с заземлением одной из фаз | 1924 |
|
SU2014A1 |
| Способ защиты переносных электрических установок от опасностей, связанных с заземлением одной из фаз | 1924 |
|
SU2014A1 |
Авторы
Даты
2017-09-25—Публикация
2015-12-04—Подача