Область техники, к которой относится изобретение
Изобретение относится к области молекулярной биологии, генетики и фармакогеномики и обеспечивает способ идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, с использованием биологических микрочипов.
Уровень техники
В настоящее время существует большой арсенал методов анализа генетических полиморфизмов. Условно можно выделить следующие подходы:
1) Секвенирование целевых фрагментов, несущих полиморфный участок. Секвенирование по Сенгеру является эталонным и наиболее точным методом выявления генетических полиморфизмов. Метод заключается в проведении полимеразной цепной реакции (ПЦР) в присутствии флуоресцентно-меченных дидезоксинуклеозидтрифосфатов (ддНТФ) и последующем разделении фрагментов, синтезированных в ходе реакции, методом электрофореза с детекцией флуоресцентного сигнала на выходе. Преимуществом данного метода является высокая точность и чувствительность, основным недостатком - высокая стоимость оборудования (Yuan Z.J., Zhou W.W., Liu W., et al. Association of GSTP1 and RRM1 polymorphisms with the response and toxicity of gemcitabine-cisplatin combination chemotherapy in Chinese patients with non-small cell lung cancer. Asian Рас.J. Cancer. Prev. 2015, v. 16 (10), p. 4347-4351; Zhou F., Yu Z., Jiang Т., et al. Genetic polymorphisms of GSTP1 and XRCC1: prediction of clinical outcome of platinum-based chemotherapy in advanced non-small cell lung cancer (NSCLC) patients. Swiss Med Wkly. 2011 v. 141, p. 13275-13284).
2) Методы, использующие стратегию элонгации праймера (primer extension). Среди этих методов выделяют методы с использованием общего праймера для детекции обеих аллелей и методы с использованием специфичных праймеров для каждой из аллелей.
В методах элонгации общего праймера (common primer extension, CPE) праймер подбирается таким образом, чтобы его 3'-конец отжигался в позиции непосредственно перед сайтом однонуклеотидного полиморфизма (ОНП). В процессе реакции праймер достраивается нуклеотидом, комплементарным соответствующему аллельному варианту ОНП (Sokolov В.Р. Primer extension technique for the detection of single nucleotide in genomic DNA. Nucleic. Acids. Res. 1990, v. 18 (12), p. 3671). Встроившийся нуклеотид определяется MALDI-TOF масс-спектрометрией (Ross P., Hall L., Smirnov I., Haff L. High level multiplex genotyping by MALDI-TOF mass spectrometry. Nat. Biotechnol. 1998, v. 16 (13), p. 1347-1351; Haff L.A., Smirnov LP. Single-nucleotide polymorphism identification assays using a thermostable DNA polymerase and delayed extraction MALDI-TOF mass spectrometry. Genome. Res. 1997, v. 7 (4), p. 378-388). В качестве встраиваемых нуклеотидов используется ддНТФ, что приводит к элонгации праймера на один нуклеотид (single base extension, SBE). После проведения реакции продукты детектируются с помощью масс-спектрометрии MALDI-TOF. Недостатком метода SBE является низкая разрешающая способность при анализе А/Т гетерозиготных генотипов. Улучшенный вариант метода SBE с использованием модифицированных ддНТФ представлен в работе (Fei Z., Опо Т., Smith L.M. MALDI-TOF mass spectrometric typing of single nucleotide polymorphisms with mass-tagged ddNTPs. Nucleic. Acids. Res. 1998, v. 26 (11), p. 2827-2828). В методе MassEXTEND (Sequenom, США) для элонгации праймеров используется смесь дНТФ и ддНТФ. Такой подход увеличивает разницу в массе у продуктов, соответствующих разным аллельным вариантам, что повышает точность метода. Высокопроизводительный автоматизированный вариант этого метода реализован на платформе MassARRAY (Sequenom, США) (Yang Z., Fang X., Pei X., Li H. Polymorphisms in the ERCC1 and XPF genes and risk of breast cancer in a Chinese population. Genet. Test. Mol. Biomarkers. 2013, v. 17 (9), p. 700-706). В методе, представленном в работе (Berlin К., Gut I.G. Analysis of negatively 'charge tagged' DNA by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid. Commun. Mass. Spectrom. 1999, v. 13 (17), p. 1739-1743), используются праймеры, модифицированные по 3'-концу фосфотионатом, а в качестве нуклеотидов для элонгации используется a-S-ддНТФ. В результате реакции нарабатываются "меченные зарядом" фрагменты ДНК. Благодаря такому подходу не требуется предварительная очистка продуктов реакции перед проведением масс-спектрометрии, а также увеличивается разрешающая способность анализа. Общим недостатком методов с детекцией с применением масс-спектрометрии MALDI-TOF является использование дорогостоящего оборудования и необходимость в высококвалифицированном персонале, что ограничивает их использование в клинической практике.
В методах элонгации общего праймера с детекцией посредством флуоресценции в качестве терминирующих нуклеотидов используют флуоресцентно-меченные ддНТФ. Эти методики реализованы в нескольких форматах: гомогенная реакция и детекция; детекция на твердой фазе; реакция на твердой фазе. В методе SNaPshot (Applied Biosystems, США) используется стандартная ПЦР с элонгацией праймера на один флуоресцентно-меченный ддНТФ, продукты которой затем разделяются и детектируются капиллярным электрофорезом (Le Hellard S., Ballereau S.J., Visscher P.M., et al. SNP genotyping on pooled DNAs: comparison of genotyping technologies and a semi-automated method for data storage and analysis. Nucleic. Acids. Res. 2002, v. 30 (15), p. 74-84; Ryu J.S., Hong Y.C., Han H.S., et al. Association between polymorphisms of ERCC1 and XPD and survival in non-small-cell lung cancer patients treated with cisplatin combination chemotherapy. Lung. Cancer. 2004, v. 44 (3), p. 311-316). В работе (Fan J.B., Chen X., Halushka M.K., et al. Parallel genotyping of human SNPs using generic high-density oligonucleotide tag arrays. Genome. Res. 2000, v. 10 (6), p. 853-860) используются праймеры с адаптерами в гомогенной реакции, а продукты элонгации праймеров гибридизуются с иммобилизованными на твердой подложке олигонуклеотидами, комплементарными адаптерам. Определение генотипа осуществляется посредством детекции флуоресцентного сигнала на подложке. Реакция на твердой фазе используется в методе элонгации олигонуклеотидов, прикрепленных к подложке (arrayed primer extension, APEX). Продукты ПЦР целевых фрагментов генов гибридизуются с олигонуклеотидами и достраиваются флуоресцентно-меченными ддНТФ (Pullat J., Metspalu A. Arrayed primer extension reaction for genotyping on oligonucleotide microarray. Methods. Mol. Biol. 2008, v. 444, p. 161-167). В работе (Shapero M.H., Leuther K.K., Nguyen A., et al. SNP genotyping by multiplexed solid-phase amplification and fluorescent minisequencing. Genome. Res. 2001, v. 11 (11), p. 1926-1934) ПЦР и однонуклеотидная элонгация праймера осуществляются непосредственно на подложке с использованием одного и того же набора праймеров. Преимуществом реакций на твердой фазе является отсутствие межпраймерных взаимодействий, что увеличивает чувствительность и специфичность анализа. К основным недостаткам подходов с детекцией или реакцией на твердой фазе относится высокая стоимость расходных материалов и оборудования. Недостатком методов, выполняемых в жидкой фазе, является невозможность одновременной идентификации нескольких мишеней.
В методах с использованием специфических праймеров для каждой из аллеллей применяются праймеры, отличающиеся только последним нуклеотидом на 3'-конце. Их элонгация происходит в том случае, если их 3'-концевой нуклеотид является комплементарным к нуклеотиду в сайте ОНП, что позволяет дискриминировать аллельные варианты полиморфизмов. К этой группе методов относятся различные модификации аллель-специфичной ПЦР (Allele-specific PCR, AS-PCR). Комбинация аллель-специфичной ПЦР с ПЦР в режиме реального времени с последующим разделением продуктов с помощью капиллярного электрофореза представлена в работе (Medintz I., Wong W.W., Berti L., et al. High-performance multiplex SNP analysis of three hemochromatosis-related mutations with capillary array electrophoresis microplates. Genome. Res. 2001, v. 11 (3), p. 413-421). В методе элонгации аллель-специфичных праймеров (Allele-specific primer extension, ASPE) используются предварительно амплифицированные целевые фрагменты, на которых происходит элонгация праймера с включением флуоресцентной метки, вносимой в реакционную смесь (Ugozzoli L., Wahlqvist J.M., Ehsani A., et al. Detection of specific alleles by using allele-specific primer extension followed by capture on solid support. Genet. Anal. Tech. Appl. 1992, v. 9 (4), p. 107-112). В работе (Bortolin S., Black M., Modi H., et al. Analytical validation of the tag-it high-throughput microsphere-based universal array genotyping platform: application to the multiplex detection of a panel of thrombophilia-associated single-nucleotide polymorphisms. Clin. Chem. 2004, v. 50 (11), p. 2028-2036) для элонгации используются праймеры с адаптерами, которые достраиваются флуоресцентно-меченными дНТФ. После гибридизации продуктов реакции с олигонуклеотидами, иммобилизованными на микросферах и комплементарными адаптерам праймеров, проводится их проточная цитометрия. Недостатком методов, основанных на аллель-специфичной ПЦР, является ограничение по количеству одновременно определяемых полиморфизмов - как правило, в таких методах определяется не более 3-5 мишеней, и сниженная, близкая к субъективной, разрешающая способность при интерпретации результатов.
3) Методы, основанные на проведении гибридизации. В этих методах используется различие в температурах плавления совершенных и несовершенных дуплексов между мишенью, несущей сайт ОНП, и детектирующими олигонуклеотидами (зондами), что позволяет идентифицировать аллельные варианты полиморфизмов. Дискриминация аллельных вариантов в методах, основанных на гибридизации, зависит от различных факторов, в том числе: длины и последовательности зонда; расположения сайта ОНП в зонде; условий гибридизации.
Методы, основанные на гибридизации, используются в высокопроизводительных платформах на основе микрочипов. В технологии микрочипов GeneChip (Affymetrix, США) используются аллель-специфичные зонды длиной 25 оснований, которые синтезируются непосредственно на подложке методом фотолитографии. Целевые фрагменты генов, несущие ОНП, амплифицируются с помощью ПЦР, расщепляются на более короткие фрагменты, метятся биотиновыми метками и гибридизуются на микрочипе. Визуализация результатов гибридизации осуществляется с помощью флуоресцентного маркирования красителем со стрептавидином. На каждом микрочипе может быть расположено несколько миллионов различных олигонуклеотидных зондов, что позволяет определять 104-105 ОНП в рамках одного анализа (Matsuzaki Н., Dong S., Loi Н., et al. Genotyping over 100,000 SNPs on a pair of oligonucleotide arrays. Nat Methods. 2004, v. 1 (2), p. 109-111). Недостатком данного метода является высокая цена расходных материалов и сложность интерпретации полученных флуоресцентных изображений, что драматически ограничивает применение данного метода в клинической практике.
Альтернативный подход с использованием олигонуклеотидных микрочипов представлен применением микрочипов низкой плотности. Методы с их использованием отличаются способом нанесения олигонуклеотидов, типом подложки и процедурой детекции результатов гибридизации. Одним из вариантов методов, относящихся к этой группе, является двухстадийная мультиплексная ПЦР с последующей гибридизацией продуктов реакции на гидрогелевых микрочипах низкой плотности. На первой стадии проводится ПЦР с равными концентрациями прямого и обратного праймеров для наработки фрагментов генов, несущих анализируемые ОНП. На второй стадии в качестве матрицы для ПЦР используется продукт первого этапа, но в реакционную смесь добавляется флуоресцентно-меченный дНТФ. При этом следует заметить, что для наработки одноцепочечного фрагмента ДНК, необходимого для гибридизации, в реакции используется неравная концентрация прямого и обратного праймеров (асимметричная ПЦР). Гибридизацию одноцепочечного фрагмента ДНК проводят на гидрогелевых микрочипах. Гидрогелевые микрочипы представляют собой пластиковые подложки с иммобилизованными ячейками гидрогеля. В каждой ячейке ковалентно закреплены специфические олигонуклеотидные зонды, с которыми гибридизуются продукты второй стадии ПЦР (Heydarov R., Titov S., Abramov M., Timofeev E., Mikhailovich V. Hydrogel microarray for detection of polymorphisms in the UGT1A1, DP YD, GSTP1 and ABCB1 genes. Cancer. Biomark. 2017, v. 18 (3), p. 265-272). Модификацией данного метода является использование в качестве зондов олигонуклеотидов с замкнутыми нуклеотидами (locked nucleic acid, LNA). Дуплексы таких олигонуклеотидов обладают повышенной термостабильностью и обеспечивают существенно более высокую дискриминацию сигналов гибридизации (Fesenko Е.Е., Heydarov R.N., Stepanova E.V., et al. Microarray with LNA-probes for genotyping of polymorphic variants of Gilbert's syndrome gene UGT1 Al(TA)n. Clin. Chem. Lab. Med. 2013, v. 51 (6), p. 1177-1184). Представленный метод является ближайшим аналогом, уступающим по диагностическим характеристикам, и прототипом предлагаемого в данном изобретении способа. К преимуществам вышеописанного метода относятся: невысокая цена расходных материалов, относительная простота проведения анализа и интерпретации результатов. Основным недостатком - малое (по сравнению с предлагаемым способом) количество одновременно идентифицируемых ОНП.
Одним из широко используемых методов для определения ОНП является технология TaqMan (Applied Biosystems, США), которая сочетает гибридизацию, применение 5'-экзонуклеазной активности полимеразы с последующей флуоресцентной детекцией. В этом методе используются четыре олигонуклеотида: два аллель-специфичных зонда, которые отличаются по позиции, соответствующей сайту ОНП, и два ПЦР-праймера, фланкирующие сайт ОНП. Аллель-специфичные зонды несут флуоресцентную метку на одном конце и тушитель флуоресценции на другом конце. В непрореагирующем свернутом петлей состоянии из-за близкого расположения метки и тушителя такие зонды не флуоресцируют. Для детекции ОНП методом TaqMan используется полимераза, обладающая 5'-экзонуклеазной активностью. В процессе элонгации праймеров, полимераза отщепляет флуоресцентно-меченный нуклеотид только с зонда, который полностью комплементарен последовательности целевого участка, что приводит к накоплению флуоресцентного сигнала за счет его выхода в раствор. По накоплению флуоресцентного сигнала определяется аллельный вариант анализируемой мишени (Ludovini V., Floriani I., Pistola L., et al. Association of cytidine deaminase and xeroderma pigmentosum group D polymorphisms with response, toxicity, and survival in cisplatin/gemcitabine-treated advanced non-small cell lung cancer patients. J. Thorac. Oncol. 2011, v. 6 (12), p. 2018-2026; Lv H., Han Т., Shi X., et al. Genetic polymorphism of GSTP1 and ERCC1 correlated with response to platinum-based chemotherapy in non-small cell lung cancer. Med. Oncol. 2014, v. 31 (8), p. 86-93). К недостаткам данного метода можно отнести низкую масштабируемость и необходимость дорогостоящего оборудования для проведения анализа.
Еще одним методом, относящимся к данной группе, является динамическая аллель-специфичная гибридизация. Метод заключается в анализе скорости денатурации дуплексов зонда и мишени в режиме реального времени за счет фиксации уровня флуоресценции. На подготовительном этапе проводится амплификация целевого фрагмента, несущего ОНП, с праймерами, один из которых модифицирован биотином, что позволяет связать одноцепочечный продукт ПЦР с микросферами, покрытыми стрептавидином. После этого проводится гибридизация с аллель-специфичными зондами в присутствии интеркалирующего флуоресцентного красителя. Далее повышается температура и фиксируется скорость денатурации дуплексов. Кривая плавления имеет различную форму для совершенных и несовершенных дуплексов, и по форме этой кривой определяется аллельный вариант анализируемого ОНП (Howell W.M., Jobs М., Gyllensten U., Brookes A J. Dynamic allele-specific hybridization. A new method for scoring single nucleotide polymorphisms. Nat. Biotechnol. 1999, v. 17 (1), p. 87-88). К недостаткам метода относится его относительная трудоемкость и сложности с одновременным определением нескольких ОНП.
4) Методы, основанные на лигазной реакции (Ligase detection Reaction, LDR). В этих методах применяют высокоспецифичные ферменты - лигазы, которые сшивают одноцепочечный разрыв между прилегающими олигонуклеотидными зондами, связанными с целевым фрагментом в сайте ОНП. В реакции используются 3 зонда, два из которых являются аллель-специфичными, а третий зонд является общим. Продукты лигазной реакции в дальнейшем детектируются с помощью электрофореза. В большинстве методов, основанных на лигазной реакции, используются аллель-специфичные олигонуклеотидные зонды 3'-конецевой нуклеотид которых непосредственно комплементарен сайту ОНП, так как лигазы наиболее чувствительны к неспаренным нуклеотидам на 3'-конце (von Keyserling Н., Bergmann Т., Schuetz М., et al. Analysis of 4 single-nucleotide polymorphisms in relation to cervical dysplasia and cancer development using a high-throughput ligation-detection reaction procedure. Int. J. Gynecol. Cancer. 2011, v. 21 (9), p. 1664-1671.). Несмотря на высокую специфичность данного метода (что можно отнести к его преимуществам), его существенным недостатком является сложность выбора зондов при одновременной детекции нескольких ОНП. 5) Методы, основанные на ферментативном расщеплении. Эти методы основаны на способности определенных ферментов расщеплять ДНК при распознавании специфических последовательностей и вторичных структур.
К этой группе методов относится анализ полиморфизма длин рестрикционных фрагментов (restriction fragment length polymorphism, RFLP). Эндонуклеазы рестрикции, используемые в этом методе, подбирают таким образом, чтобы они были специфичны к сайту ОНП. После предварительной амплификации целевых участков и их очистки, проводится расщепление ПЦР продукта и разделение полученных фрагментов путем гель-электрофореза или блот-гибридизации. К преимуществам данного метода относится простота анализа и интерпретации результатов, но существенным недостатком является ограниченность в выборе мишеней для генотипирования, в связи с необходимостью определенных рестриктаз, специфичных последовательности сайта ОНП (Liu J.Y., Liu Q.M., Li L.R. Association of GSTP1 and XRCC1 gene polymorphisms with clinical outcomes of patients with advanced non-small cell lung cancer. Genet. Mol. Res. 2015, v. 14 (3), p. 10331-10337; Shi Z.H., Shi G.Y., Liu L.G. Polymorphisms in ERCC1 and XPF gene and response to chemotherapy and overall survival of non-small cell lung cancer. Int. J. Clin. Exp. Pathol. 2015, v. 8(3), p. 3132-3137).
Другим примером этих методов является инвазивное расщепление олигонуклеотидного зонда (Invader assay). При этом подходе используется специальная Flap-эндонуклеаза, способная распознавать заходящие друг на друга участки ДНК. В реакции принимают участие 3 олигонуклеотидных зонда, два - аллель-специфичных детектирующих, третий - инвазивный зонд. Инвазивный зонд комплементарен 3'-участку сайта ОНП, на конце этого зонда находится не комплементарный ОНП нуклеотид, а аллель-специфичные зонды к 5'-участку и несут на своем 5'-конце сигнальный фрагмент. Удаление сигнального фрагмента Flap-эндонуклеазой происходит только в случае, когда формируется совершенный дуплекс между участком с ОНП и специфичной частью детектирующего зонда. Детекция результатов анализа осуществляется путем регистрации флуоресценции отщепленного сигнального фрагмента. Общим недостатком методов, с применением ферментативного расщепления, является невозможность точной идентификации ОНП (Olivier М. The Invader assay for SNP genotyping. Mutat. Res. 2005, v. 573 (1-2), p. 103-110).
6) Методы, основанные на физических свойствах молекул ДНК.
Анализ конформации одноцепочечных фрагментов ДНК (Single-strand conformation polymorphism, SSCP). Анализ данным методом включает предварительную амплификацию полиморфных участков с последующей их денатурацией и разделением одноцепочечных фрагментов гель-электрофорезом. Подвижность одноцепочечных фрагментов при электрофорезе зависит от их нуклеотидного состава, который определяет вторичную и третичную структуру, что позволяет выявлять аллельные варианты полиморфизмов. Преимущество данного метода заключает в простоте проведения анализа и невысокой стоимости, главный недостаток - низкая воспроизводимость результатов, слабая маштабируемость и неспособность однозначно идентифицировать тип ОНП (Akhtar S., Mahjabeen I., Akram Z., Kayani M.A. CYP1A1 and GSTP1 gene variations in breast cancer: a systematic review and case-control study. Fam. Cancer. 2016, v. 15 (2), p. 201-214).
Кроме указанной литературы имеется ряд патентов и заявок на патенты, описывающих различные способы обнаружения полиморфизмов в одном или нескольких генах из списка: GSTP1 (полиморфизм rs 1695), АВСВ1 (полиморфизм rs 1045642), MTHFR (полиморфизм rs 1801133), FCGR3A (полиморфизм rs 396991), SOD2 (полиморфизм rs 4880), ТР53 (полиморфизм rs 1042522), ERCC1 (полиморфизмы rs 11615, rs 3212986), ХРС (полиморфизм rs 2228001), EGF (полиморфизм rs 4444903), UGT1A1 (полиморфизм rs 8175347), DPYD (полиморфизмы rs 3918290, rs 2297595, rs 67376798), XRCC1 (полиморфизм rs 25487), CYP2D6 (полиморфизм rs 3892097), ТРМТ (полиморфизмы rs 1142345, rs 1800462, rs 1800584, rs 1800460).
Патенты и заявки на патенты, описывающие методы обнаружение полиморфизмов в одном гене:
- способ, описанный в заявке на патент CN 104988226 A (от 2015-10-21), обеспечивает определение полиморфизма rs 1695 в гене GSTP1 с использованием метода SNaPshot;
- способ, описанный в заявке на патент CN 106755352 A (от 2017-05-31), обеспечивает определение полиморфизма rs 1045642 в гене АВСВ1 с использованием аллель-специфичной ПЦР в режиме реального времени с флуоресцентной детекцией продукта;
- способ, описанный в заявке на патент CN 102851365 A (от 2012-08-31), обеспечивает определение полиморфизма rs 1801133 MTHFR с использованием метода аллель-специфичной ПЦР с детекцией продукта посредством электрофореза в агарозном геле;
- способ, описанный в заявке на патент CN 107227363 А (от 2017-10-03), обеспечивает определение полиморфизма rs 11615 в гене ERCC1 с использованием аллель-специфичной ПЦР, в качестве специфичных праймеров используются пептидо-нуклеиновые олигонуклеотиды;
- способ, описанный в заявке на патент CN 108103160 А (от 2018-06-01), обеспечивает определение полиморфизма rs 2228001 в гене ХРС с использованием масс-спектрометрии продуктов амплификации;
- способ, описанный в патенте US 6472157 B2 (от 2002-10-29), обеспечивает определение полиморфизма rs 8175347 в гене UGT1A1 с использованием ПЦР с последующим разделением продуктов реакции методом электрофореза в полиакриламидном геле;
- способ, описанный в заявке на патент CN 107058608 A (от 2017-08-18), обеспечивает определение полиморфизма rs 3918290 в гене DPYD с использованием аллель-специфичной ПЦР в режиме реального времени с флуоресцентной детекцией продукта;
- способ, описанный в заявке на патент CN 104988225 А (от 2015-10-21), обеспечивает определение полиморфизма rs 25487 XRCC1 с использованием метода SNaPshot;
- способ, описанный в заявке на патент CN 103131776 A (от 2013-06-05), обеспечивает определение полиморфизма rs 1142345 в гене ТРМТ посредством ПЦР с последующим секвенированием продуктов реакции;
- способ, описанный в заявке на патент CN 105506097 A (от 2016-04-20), обеспечивает определение полиморфизма rs 1142345 в гене ТРМТ rs 1800462 с использованием метода SNaPshot.
Очевидным существенным недостатком вышеперечисленных способов является возможность обнаружения только одного ОНП. Способы, заявленные в CN 108103160 А и CN 103131776 A, требуют использования дорогостоящего оборудования.
Патенты и заявки на патенты, описывающие методы одновременного обнаружения нескольких полиморфизмов в разных генах:
- способ, описанный в заявке на патент CN 104988224 A (от 2015-10-21), обеспечивает детекцию полиморфизмов в генах XRCC1 (rs 25487), ERCC1 (rs 11615) и GSTP1 (rs 1695) с использованием метода SNaPshot.
- способ, описанный в заявке на патент CN 107663537 A (от 2018-02-06), позволяет выявлять ОНП в 10 генах, в том числе в GSTP1 (rs 1695), MTHFR (rs 1801133), XRCC1 (rs 25487), ERCC1 (rs 11615), DPYD (rs 3918290) посредством мультиплексной ПЦР на твердой фазе, с последующим секвенированием отдельных ампликонов;
- способ, описанный в заявке на патент CN 101275160 А (от 2008-10-01), позволяет идентифицировать 48 ОНП в 45-ти генах, в том числе в ERCC1 (rs 11615), MTHFR (rs 1801133), XRCC1 (rs 25487), GSTP1 (rs 1695) путем проведения ПЦР в режиме реального времени с использованием TaqMan-зондов;
Основным недостатком перечисленных способов является высокая стоимость оборудования и сложность интерпретации результатов анализа.
Раскрытие сущности изобретения
Предлагаемое изобретение обеспечивает способ идентификации генетических полиморфизмов в генах, продукты которых влияют на метаболизм противоопухолевых препаратов, используемых в химиотерапии злокачественных новообразований, с использованием биологических микрочипов, представляющих собой подложку с гелевыми элементами, содержащими иммобилизованные специфичные олигонуклеотидные зонды. Изобретением обеспечиваются: биочип, используемый в способе, набор олигонуклеотидных зондов, применяемых для изготовления биочипа, а также праймеры, необходимые для проведения мультиплексной полимеразной цепной реакции. Способ выгодно отличается от способов, известных из приведенного Уровня техники, тем, что позволяет одновременно (в рамках одного анализа) с высокой специфичностью осуществлять идентификацию 20 клинически значимых полиморфизмов в 14 генах. Способ лишен недостатков, указанных выше при рассмотрении каждого метода, не требует дорогостоящего оборудования и материалов.
Анализ полиморфизмов в генах, участвующих в метаболизме противоопухолевых препаратов, важен для оценки рисков проявления токсических эффектов химиотерапии, подборе индивидуального режима лечения и в прогнозировании его исхода. Способ может использоваться перед назначением курса химиотерапии для повышения эффективности лечения онкологических больных, для коррекции протоколов терапии, в качестве инструмента для анализа популяции с целью разработки новых протоколов и рекомендаций по их применению.
Процедура определения полиморфизмов в предлагаемом способе основана на проведении мультиплексной полимеразной цепной реакции (ПЦР) с использованием в качестве матрицы ДНК, выделенной из периферической крови пациента, с последующей гибридизацией полученного одноцепочечного флуоресцентно-меченного ПЦР-продукта на биологическом микрочипе. Биологический микрочип представляет собой подложку с упорядоченно расположенными гидрогелевыми элементами, содержащими ковалентно иммобилизованные олигонуклеотидные зонды, которые обеспечивают гибридизацию со специфическими участками генов, несущих полиморфные нуклеотидные позиции.
Детекция результатов гибридизации проводится флуоресцентным методом с помощью возбуждения встроившихся в ПЦР-продукт флуоресцентных меток после его гибридизации с иммобилизованными на микрочипе специфическими зондами. Интерпретация результатов осуществляется сравнительным анализом флуоресцентных сигналов в ячейках, в которых прошла гибридизация меченой мишени с иммобилизованными зондами.
Предлагаемый способ включает: а) обеспечение биочипа, представляющего собой подложку с упорядоченными гелевыми элементами, в которых иммобилизованы олигонуклеотидные зонды, относящиеся к следующим группам:
- группа зондов, последовательности которых представлены SEQ ID NO 1-4 (Таблица 1), для идентификации полиморфизма rs 1695 (аллели А и G) в гене GSTP1;
- группа зондов, последовательности которых представлены SEQ ID NO 5, 6 (Таблица 1), для идентификации полиморфизма rs 1045642 (аллели Т и С) в гене АВСВ1;
- группа зондов, последовательности которых представлены SEQ ID NO 7, 8 (Таблица 1), для идентификации полиморфизма rs 1801133 (аллели Т и С) в гене MTHFR;
- группа зондов, последовательности которых представлены SEQ ID NO 9, 10 (Таблица 1), для идентификации полиморфизма rs 396991 (аллели G и Т) в гене FCGR3A;
- группа зондов, последовательности которых представлены SEQ ID NO 11-14 (Таблица 1), для идентификации полиморфизма rs 4880 (аллели С и Т) в гене SOD2;
- группа зондов, последовательности которых представлены SEQ ID NO 15-18 (Таблица 1), для идентификации полиморфизма rs 1042522 (аллели С и G) в гене ТР53;
- группа зондов, последовательности которых представлены SEQ ID NO 19-26 (Таблица 1), для идентификации полиморфизмов rs 11615 (аллели С и Т), rs 3212986 (аллели G и Т) в reHeERCC1;
- группа зондов, последовательности которых представлены SEQ ID NO 27-30 (Таблица 1), для идентификации полиморфизма rs 2228001 (аллели А и С) в гене ХРС;
- группа зондов, последовательности которых представлены SEQ ID NO 31-32 (Таблица 1), для идентификации полиморфизма rs 4444903 (аллели А и G) в гене EGF;
- группа зондов, последовательности которых представлены SEQ ID NO 33-36 (Таблица 1), для идентификации полиморфизма rs 8175347 (аллели ТА5, ТА6, ТА7 и ТА8) в гене UGT1A1;
- группа зондов, последовательности которых представлены SEQ ID NO 37-46 (Таблица 1), для идентификации полиморфизмов rs 3918290 (аллели А и G), rs 2297595 (аллели С и Т), rs 67376798 (аллели А и Т) в гене DPYD;
- группа зондов, последовательности которых представлены SEQ ID NO 47-50 (Таблица 1), для идентификации полиморфизма rs 25487 (аллели А и G) в гене XRCC1;
- группа зондов, последовательности которых представлены SEQ ID NO 51-52 (Таблица 1), для идентификации полиморфизма rs 3892097 (аллели А и G) в гене CYP2D6;
- группа зондов, последовательности которых представлены SEQ ID NO 61-66 (Таблица 1), для идентификации полиморфизмов rs 1142345 (аллели А и G), rs 1800462 (аллели G и С), rs 1800584 (аллели А и G), rs 1800460 (аллели G и А) в гене ТРМТ;
б) проведение полимеразной цепной реакции для амплификации целевых фрагментов генов с использованием набора праймеров и включением в процессе реакции флуоресцентной метки для получения преимущественно одноцепочечных флуоресцентно-меченных продуктов;
в) гибридизацию продуктов ПЦР, полученных на стадии (б), на биочипе, полученном на стадии (а)
г) регистрацию и интерпретацию результатов гибридизации на биочипе, проведенной на стадии (в), путем сравнения интенсивности флуоресценции сигналов в пределах каждой из групп ячеек, в которых образовались совершенные и несовершенные гибридизапионные дуплексы.
Для анализа по предлагаемому способу используется ДНК, выделенная из крови человека. ПЦР с набором праймеров (Таблица 2) на стадии (б), в результате которой осуществляется получение одноцепочечных целевых фрагментов анализируемых генов и их флуоресцентное маркирование за счет встраивания флуоресцентного красителя в продукт реакции.
Регистрацию результатов на стадии (г) проводят с помощью прибора-анализатора флуоресценции, оснащенного специализированным программным обеспечением, что позволяет выполнять программную автоматическую обработку интенсивностей сигналов с последующей интерпретацией результатов.
Изобретение обеспечивает биочип, используемый в способе идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, и представляющий собой подложку с гелевыми элементами, с набором иммобилизованых зондов.
Изобретением предлагается набор олигонуклеотидных зондов, для получения биочипа, используемого в способе идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, причем зонды имеют последовательности SEQ ID NO 1-66 (Таблица 1).
Изобретением предлагается набор олигонуклеотидных праймеров для проведения ПЦР, причем праймеры имеют последовательности SEQ ID NO 67-108 (Таблица 2).
Далее изобретение будет раскрыто подробнее со ссылками на фигуры и примеры, которые приводятся исключительно с целью иллюстрации и пояснения сущности заявленного изобретения, но которые не предназначены для ограничения объема притязаний.
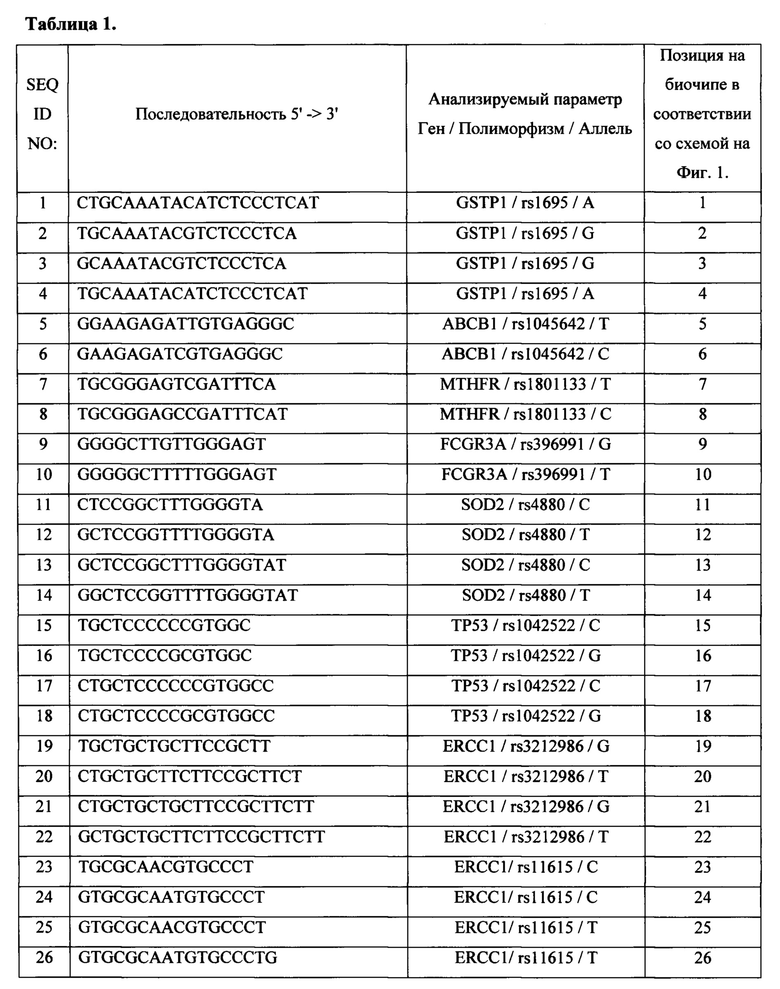
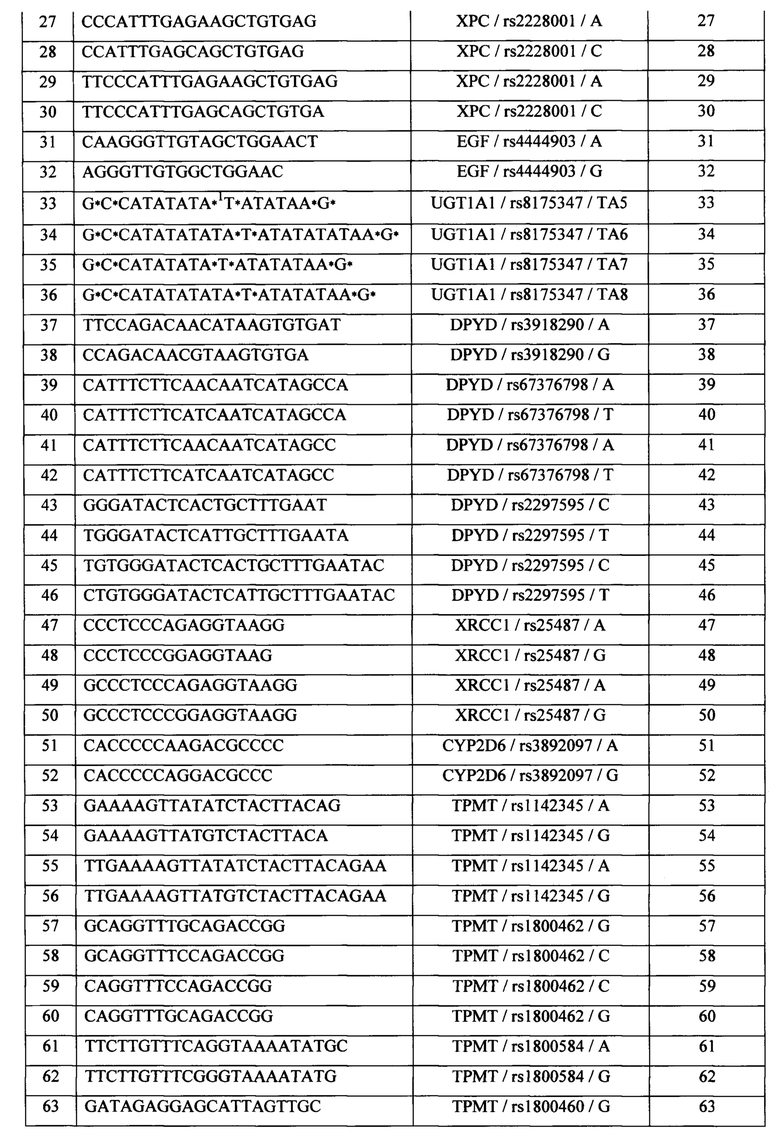
Краткое описание таблиц и фигур Таблица 1. Характеристика олигонуклеотидных зондов, иммобилизованных в ячейках биочипа, для идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов.
Таблица 2. Перечень олигонуклеотидных праймеров используемых в способе идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов.
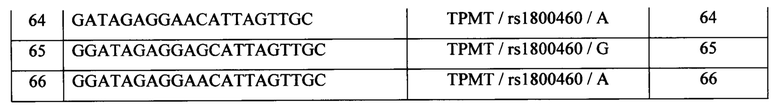
Фиг. 1. представляет схему размещения олигонуклеотидных зондов на биочипе для идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов.
Позиция 0 - элементы, не содержащие иммобилизованные зондов (фоновые ячейки); позиция М - элементы, содержащие флуоресцентный маркер;
позиции 1 - 66 - элементы, содержащие иммобилизованные олигонуклеотидные зонды (последовательности зондов приведены в Таблице 1).
Фиг. 2. представляет флуоресцентное изображение (гибридизационную картину) биочипа и отчет программы после проведения анализа образца ДНК №1.
Фиг. 3. представляет флуоресцентное изображение (гибридизационную картину) биочипа и отчет программы после проведения анализа образца ДНК №2.
Фиг. 4. представляет флуоресцентное изображение (гибридизационную картину) биочипа и отчет программы после проведения анализа образца ДНК №3.
Фиг. 5. представляет флуоресцентное изображение (гибридизационную картину) биочипа и отчет программы после проведения анализа образца ДНК №4.
Осуществление изобретения
Целью настоящего изобретения является создание способа многопараметрического экспресс-анализа генетических полиморфизмов в генах, продукты которых участвуют в метаболизме препаратов, применяемых для химиотерапии онкологических заболеваний.
Идентификация генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, включает:
- идентификацию полиморфизма rs 1695 (аллели А и G) в гене GSTP1;
- идентификацию полиморфизма rs 1045642 (аллели Т и С) в гене АВСВ1;
- идентификацию полиморфизма rs 1801133 (аллели Т и С) в гене MTHFR;
- идентификацию полиморфизма rs 396991 (аллели G и Т) в гене FCGR3A;
- идентификацию полиморфизма rs 4880 (аллели С и Т) в гене SOD2;
- идентификацию полиморфизма rs 1042522 (аллели С и G) в гене ТР53;
- идентификацию полиморфизмов rs 11615 (аллели С и Т), rs 3212986 (аллели G и Т) в гене ERCC1;
- идентификацию полиморфизма rs 2228001 (аллели А и С) в гене ХРС;
- идентификацию полиморфизма rs 4444903 (аллели А и G) в гене EGF;
- идентификацию полиморфизма rs 8175347 (аллели ТА5, ТА6, ТА7 и ТА8) в гене UGT1A1;
- идентификацию полиморфизмов rs 3918290 (аллели А и G), rs 2297595 (аллели С и Т), rs 67376798 (аллели А и Т) в гене DPYD;
- идентификацию полиморфизма rs 25487 (аллели А и G) в гене XRCC1;
- идентификацию полиморфизма rs 3892097 (аллели А и G) в гене CYP2D6;
- идентификацию полиморфизмов rs 1142345 (аллели А и G), rs 1800462 (аллели G и С), rs 1800584 (аллели А и G), rs 1800460 (аллели G и А) в гене ТРМТ;
Одновременный многопараметрический анализ ДНК достигается при использовании биологических микрочипов - массивов элементов, содержащие олигонуклеотидные зонды. Заявленный способ заключается в обеспечении биочипа с соответствующими иммобилизованными специфичными олигонуклеотидными зондами, проведении ПЦР с использованием олигонуклеотидных праймеров, комплементарных фрагментам целевых участков генов, с получением преимущественно одноцепочечных флуоресцентно-меченных фрагментов ДНК, гибридизацию флуоресцентно-меченных продуктов на биочипе, регистрацию и интерпретацию результатов гибридизации.
Биологический микрочип, используемый в предлагаемом способе, представляет собой массив трехмерных гидрогелевых элементов, расположенных на твердой подложке и содержащих ковалентно иммобилизованные олигонуклеотидные зонды. Элементы массива - микрокапли заданного объема, как правило, диаметром от 80 до 300 мкм, расстояние между каплями составляет, как правило, 150-500 мкм. Микрочипы изготавливают методом сополимеризационной иммобилизации по технологии, разработанной в ИМБ РАН (патент US 7,846,656 В2, публикация "Composition for polymerising immobilisation of biological molecules and method for producing said composition", Rubina A.Y., Pan'kov S.V., Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production, Anal. Biochem. 2004, v. 325, p. 92-106).
При изготовлении биочипа полимеризационную смесь, содержащую гелеобразующие мономеры и подлежащие иммобилизации олигонуклеотидные зонды, наносят на подложку в виде массива микрокапель с помощью автоматического микродозатора (робота). Полимеризация геля с одновременной ковалентной иммобилизацией зондов происходит под действием УФ-излучения. Изготовление биочипа на основе гидрогелей для идентификации и анализа РНК вируса гриппа описано в Примере 1.
На Фиг. 1 представлена схема биочипа, гидрогелевые элементы которого содержат иммобилизованные олигонуклеотидные зонды с последовательностями SEQ ID NO 1-66 (позиции 1-66). Кроме того, на биочипе находятся элементы, не содержащие иммобилизованные соединения, для измерения фоновых сигналов (позиция 0), а также элементы, содержащие флуоресцентный маркер (позиция М), для правильного позиционирования биочипа при получении флуоресцентного изображения и захвата изображения программным обеспечением биочип-анализатора. При гибридизации флуоресцентно-меченного продукта с иммобилизованными на чипе олигонуклетидными зондами образуется стабильный комплекс с наиболее комплементарным зондом, что приводит к появлению значимого флуоресцентного сигнала в соответствующем элементе биочипа. Сравнительный анализ флуоресцентных сигналов позволяет сделать вывод о характеристике соответствующего полиморфизма (гомозигота по дикой/мутантной аллели; гетерозигота).
Для проведения анализа по данному способу используется ДНК, выделенная из крови, полученной от человека. Выделение ДНК осуществляют любым из известных в данной области способов или с использованием коммерчески доступных наборов реагентов и автоматических роботизированных станций, например, набор реагентов «Проба-ГС-ГЕНЕТИКА» ООО «ДНК-Технология», Россия, QIAamp DNA Blood Mini Kit, Qiagen, Германия, PureLink Genomic DNA Mini Kit, Thermo Scientific, США.
На следующем этапе проведения анализа по данному способу полученную ДНК используют в качестве матрицы для проведения ПЦР с использованием олигонуклеотидных праймеров, комплементарных соответствующим фрагментам целевых участков генов, смеси четырех дезоксинуклеозидтрифосфатов, флуоресцентно-меченного дезоксинуклеозидтрифосфата, с получением продуктов амплификации в виде преимущественно одноцепочечных флуоресцентно-меченных фрагментов ДНК. Смесь дезоксинуклеозидтрифосфатов содержит дАТФ, дГТФ, дЦТФ и дТТФ; вместо дТТФ можно использовать дУТФ или смесь дТТФ с дУТФ в любом соотношении. Флуоресцентную метку может нести любой из указанных дезоксинуклеозидтрифосфатов.
В качестве флуоресцентной метки может быть использован любой флуоресцентный краситель, который может быть включен в молекулу дезоксинуклеозидтрифосфата таким образом, чтобы не препятствовать проведению ПЦР и последующей гибридизации. Например, флуоресцентный краситель может быть присоединен к 5'-концу аминоаллильного производного дУТФ. Примеры красителей включают красители флуоресцеинового (TAMRA, ROX, JOE), родаминового (Texas Red), полиметинового (Су3, Су5, Су5.5, Су7) рядов, но не ограничиваются ими.
После проведения ПЦР полученные продукты гибридизуют на биочипе, содержащем иммобилизованные олигонуклеотидные зонды, комплементарные анализируемым последовательностям. Гибридизацию проводят в растворе, содержащем буферный компонент для поддержания рН, соль для создания ионной силы и хаотропный (дестабилизирующий водородные связи) агент, в герметичной гибридизационной камере. В качестве хаотропного агента могут быть использованы, например, гуанидин тиоцианат, мочевина, формамид и т.п. Иммобилизованные на биочипе дискриминирующие олигонуклеотиды, заявленные в настоящем изобретении, имеют температуры плавления в интервале от 42 до 44°С, что позволяет проводить гибридизацию при 37°С в присутствии хаотропного агента.
Фрагменты ДНК образуют совершенные гибридизационные дуплексы только с соответствующими, полностью комплементарными олигонуклеотидами. Дискриминацию совершенных и несовершенных дуплексов выполняют путем сравнения интенсивностей флуоресценции ячеек микрочипа, в которых образовались дуплексы. Интенсивность сигнала в ячейках, в которой образовались совершенные гибридизационные дуплексы выше, чем в ячейках, где образовались несовершенные дуплексы. Регистрацию и интерпретацию результатов гибридизации на биочипе проводят путем сравнения интенсивности флуоресценции сигналов в пределах каждой из групп ячеек, в которых образовались совершенные и несовершенные гибридизационные дуплексы.
Регистрация результатов гибридизации на микрочипах может быть осуществлена с помощью любых приборов-анализаторов флуоресценции, оснащенного специализированным программным обеспечением, позволяющим использовать программную автоматическую обработку интенсивностей сигналов с последующей интерпретацией результатов, например, с помощью коммерческих сканирующих устройств GenePix 4000 В (Axon Instruments, США), 'GenePix Pro', 'Acuity' (Axon Instruments, США), портативноного биочип-анализатора: Комплекса универсального аппаратно-программного (УАПК) для анализа биологических микрочипов (ООО «БИОЧИП-ИМБ», Россия).
Проведение анализа: идентификация генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, с использованием биологических микрочипов, - по данному способу описано в Примере 2. Результаты анализа представлены на Фиг. 3-6.
Данным изобретением также предлагается биочип для идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, представляющий собой подложку с гидрогелевыми элементами, в каждом из которых иммобилизован набор специфических олигонуклеотидных зондов. Пример биочипа представлен на Фиг. 1 и 2, примеры результатов анализа с его использованием представлены на Фиг. 3-6.
Нуклеотидные последовательности (SEQ ID NO 1-66) зондов для иммобилизации в гидрогелевых элементах микрочипа, являющегося предметом изобретения, представлены в Таблице 1.
Далее изобретение проиллюстрировано примерами, которые предназначены для обеспечения лучшего понимания сущности изобретения, и которые не должны рассматриваться как ограничивающие данное изобретение.
Пример 1. Изготовление биочипа на основе гидрогелей для идентификации полиморфизмов, влияющих на метаболизм противоопухолевых препаратов. Олигонуклеотидные зонды с последовательностями SEQ ID NO 1-66 синтезировали на автоматическом синтезаторе 394 DNA/RNA synthesizer (Applied Biosystems, США).
Олигонуклетиды для иммобилизации в гидрогелевых ячейках биочипа содержали спейсер со свободной аминогруппой 3'-Amino-Modifier С7 CPG 500 (Glen Research, США). Изготовление биочипов производили в соответствии с процедурой, описанной ранее (Rubina A.Y., Pan'kov S.V. et al. Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal. Biochem., 2004, v. 325, p. 92-106).
Полимеризационные смеси, содержащие смесь гелеобразующих мономеров (производные метакриламида, метилен-бис-акриламида, стабилизаторы и др.) и иммобилизуемые соединения (олигонуклеотиды или флуоресцентный краситель IMD 504), помещали в ячейки 384-луночного планшета. С использованием робота QArray (Genetix, Великобритания) смеси из лунок планшета переносили в виде микрокапель на поверхность пластиковых подложек. Полимеризация геля с одновременной ковалентной иммобилизацией олигонуклеотидов в структуре геля происходила под действием УФ-излучения (УФ-лампа Sylvania GTE F15T8350 BL, Великобритания), 40 мин, 20°С, в токе азота. После полимеризации подложки с массивами гидрогелевых ячеек (микрокапель) промывали 0.1 М фосфатным буфером, водой и высушивали в беспылевой атмосфере.
Полученные биочипы представляли собой совокупность полусферических ячеек диаметром 100 мкм, расстояние между ячейками (периодом) - 300 мкм. Структура биочипа представлена на Фиг. 1, 2.
Микрочип содержит 76 гидрогелевых элементов. Элементы 1-66 (Фиг. 1) содержат иммобилизованные олигонуклеотидные зонды с последовательностями SEQ ID NO 1-66; указанными в Таблице 1;
ячейки 1-4 содержат олигонуклеотидные зонды (SEQ ID NO 1-4), соответствующие фрагменту целевой последовательности гена GSTP1 для определения аллельных вариантов полиморфизма rs 1695 (аллели А и G). Ячейки 1, 4 содержат зонды специфичные к аллели А, ячейки 2, 3 - к аллели G;
ячейки 5, 6 содержат олигонуклеотидные зонды (SEQ ID NO 5, 6), соответствующие фрагменту целевой последовательности гена АВСВ1 для определения аллельных вариантов полиморфизма rs 1045642 (аллели Т и С). Ячейка 5 содержит зонды специфичные к аллели Т, ячейка 6 - к аллели С;
ячейки 7, 8 содержат олигонуклеотидные зонды (SEQ ID NO 7, 8), соответствующие фрагменту целевой последовательности гена MTHFR для определения аллельных вариантов полиморфизма rs 1801133 (аллели Т и С). Ячейка 7 содержит зонды специфичные к аллели Т, ячейка 8 - к аллели С;
ячейки 9, 10 содержат олигонуклеотидные зонды (SEQ ID NO 9, 10), соответствующие фрагменту целевой последовательности гена FCGR3A для определения аллельных вариантов полиморфизма rs 396991 (аллели G и Т). Ячейка 9 содержит зонды специфичные к аллели G, ячейка 10 - к аллели Т;
ячейки 11-14 содержат олигонуклеотидные зонды (SEQ ID NO 11-14), соответствующие фрагменту целевой последовательности гена SOD2 для определения аллельных вариантов полиморфизма rs 4880 (аллели С и Т). Ячейки 11, 13 содержат зонды специфичные к аллели С, ячейки 12, 14 - к аллели Т;
ячейки 15-18 содержат олигонуклеотидные зонды (SEQ ID NO 15-18), соответствующие фрагменту целевой последовательности гена ТР53 для определения аллельных вариантов полиморфизма rs 1042522 (аллели С и G). Ячейки 15, 17 содержат зонды специфичные к аллели С, ячейки 16, 18 - к аллели G;
ячейки 19-26 содержат олигонуклеотидные зонды (SEQ ID NO 19-26), соответствующие фрагменту целевой последовательности гена ERCC1 для определения аллельных вариантов полиморфизмов rs 11615 (аллели С и Т), rs 3212986 (аллели G и Т). Ячейки 23, 24 содержат зонды специфичные к аллели С, ячейки 25, 26 - к аллели Т (полиморфизм rs 11615). Ячейки 19, 21 содержат зонды специфичные к аллели G, ячейки 20, 22 - к аллели Т (полиморфизм rs 3212986);
ячейки 27-30 содержат олигонуклеотидные зонды (SEQ ID NO 27-30), соответствующие фрагменту целевой последовательности гена ХРС для определения аллельных вариантов полиморфизма rs 2228001 (аллели А и С). Ячейки 27, 29 содержат зонды специфичные к аллели А, ячейки 28, 30 - к аллели С;
ячейки 31, 32 содержат олигонуклеотидные зонды (SEQ ID NO 31, 32), соответствующие фрагменту целевой последовательности гена EGF для определения аллельных вариантов полиморфизма rs 4444903 (аллели А и G). Ячейка 31 содержит зонды специфичные к аллели А, ячейка 32 - к аллели G;
ячейки 33-36 содержат LNA-модифицированные олигонуклеотидные зонды (SEQ ID NO 33-36), соответствующие фрагменту целевой последовательности гена UGT1A1 для определения аллельных вариантов полиморфизма rs 8175347 (аллели ТА5, ТА6, ТА7 и ТА8); Ячейка 33 содержит зонды специфичные к аллели ТА5, ячейка 34 - к ТА6, ячейка 35 - к ТА7, ячейка 36 - к ТА8;
ячейки 37-46 содержат олигонуклеотидные зонды (SEQ ID NO 37-46), соответствующие фрагменту целевой последовательности гена DPYD для определения аллельных вариантов полиморфизмов rs 3918290 (аллели А и G), rs 2297595 (аллели С и Т), rs 67376798 (аллели А и Т). Ячейка 37 содержит зонды специфичные к аллели А, ячейка 38 - к аллели G (полиморфизм rs 3918290). Ячейки 43, 45 содержат зонды специфичные к аллели С, ячейки 44, 46 - к аллели Т (полиморфизм rs 2297595). Ячейки 39, 51 содержат зонды специфичные к аллели А, ячейки 40, 42 - к аллели Т (полиморфизм rs 67376798);
ячейки 47-50 содержат олигонуклеотидные зонды (SEQ ID NO 47-50), соответствующие фрагменту целевой последовательности гена XRCC1, для определения аллельных вариантов полиморфизма rs 25487 (аллели А и G). Ячейки 47, 49 содержат зонды специфичные к аллели А, ячейки 48, 50 - к аллели G;
ячейки 51, 52 содержат олигонуклеотидные зонды (SEQ ID NO 51, 52), соответствующие фрагменту целевой последовательности гена CYP2D6, для определения аллельных вариантов полиморфизма rs 3892097 (аллели А и G). Ячейка 51 содержит зонды специфичные к аллели А, ячейка 52 - к аллели G;
ячейки 53-66 содержат олигонуклеотидные зонды (SEQ ID NO 53-66), соответствующие фрагменту целевой последовательности гена ТРМТ, для определения аллельных вариантов полиморфизмов rs 1142345 (аллели А и G), rs 1800462 (аллели G и С), rs 1800584 (аллели А и G), rs 1800460 (аллели G и А). Ячейки 53, 55 содержат зонды специфичные к аллели А, ячейки 40, 42 - к аллели G (полиморфизм rs 1142345). Ячейки 57, 59 содержат зонды специфичные к аллели G, ячейки 58, 60 - к аллели С (полиморфизм rs 1800462). Ячейка 61 содержит зонды специфичные к аллели А, ячейка 62 - к аллели G (полиморфизм rs 1800584). Ячейки 63, 65 содержат зонды специфичные к аллели G, ячейки 64, 66 - к аллели А (полиморфизм rs 1800460).
В состав чипа также входят маркерные точки, содержащие иммобилизованный флуоресцентный краситель IMD 504 (ООО «БИОЧИП», Москва) (элементы 1 на Фиг. 1) для правильного позиционирования биочипа, выполняемого компьютерной программой при анализе флуоресцентного изображения после проведения гибридизации, и ячейки, не содержащие иммобилизованные соединения (элементы 0 на Фиг. 1), необходимые для вычисления фонового значения интенсивности флуоресценции.
Пример 2. Процедура проведения анализа: идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов с использованием биологического микрочипа.
Проведение анализа включало стадии: подготовки образцов ДНК; ПЦР-амплификации с получением одноцепочечных флуоресцентно-меченных фрагментов ДНК; гибридизации амплифицированных флуоресцентно-меченных продуктов на биочипах; регистрации и интерпретации результатов гибридизации,
а) Подготовка образцов ДНК
Взятие цельной периферической крови проводили в вакуумные пластиковые пробирки типа Vacuette объемом 10 мл с добавленной в качестве антикоагулянта динатриевой соли этилендиаминтетраацетата (ЭДТА) в конечной концентрации 2,0 мг/мл. Для перемешивания крови с антикоагулянтом после взятия материала пробирку переворачивали 2-3 раза.
Образцы ДНК выделяли из периферической крови с использование набора «Проба-ГС-ГЕНЕТИКА» ООО «ДНК-Технология», Россия.
1) Готовили смесь лизирующего раствора с сорбентом, смешивая в отдельной пробирке:
- 150 * (N+1) мкл лизирующего раствора;
- 20 * (N+1) мкл предварительно ресуспендированного сорбента;
где N+1 - количество анализируемых образцов с учетом «K-» с запасом на 1 образец;
2) в пробирки объемом 1.5 мл вносили по 170 мкл полученной смеси;
3) в каждую из пробирок со смесью (кроме пробирки «K-») добавляли по 100 мкл периферической крови;
4) в пробирку «K-» добавляли 100 мкл стерильного физиологического раствора;
5) Плотно закрытые пробирки встряхивали на вортексе в течение 3-5 с;
6) инкубировали пробирки в термостате в течение 10 мин при +50°С;
7) центрифугировали пробирки при 13000 об/мин в течение 1 мин;
8) не задевая осадок, полностью удаляли надосадочную жидкость (отдельным наконечником из каждой пробирки);
9) добавляли к осадку 400 мкл промывочного раствора №1 и встряхивали пробирки на вортексе в течение 3-5 с;
10) центрифугировали пробирки при 13000 об/мин в течение 1 мин;
11) не задевая осадок, полностью удаляли надосадочную жидкость (отдельным наконечником из каждой пробирки);
12) добавляли к осадку 200 мкл промывочного раствора №2 и встряхивали пробирки на вортексе в течение 3-5 с, повторяли п.п. 10, 11;
13) повторяли п. 12 один раз;
14) открыв крышки пробирок, высушивали осадок при +50°С в течение 5 мин;
15) добавляли к осадку 300 мкл элюирующего раствора и встряхивали пробирки на вортексе в течение 5-10 с;
16) инкубировали пробирки при +50°С в течение 5 мин;
17) центрифугировали пробирки при 13000 об/мин в течение 1 мин. и переносили надосадочную жидкость в новую пробирку.
Полученные образцы ДНК использовали в качестве матрицы для проведения ПЦР
б) Подготовка праймеров для проведения ПЦР
При выборе последовательностей праймеров для проведения ПЦР использовали базы данных нуклеотидных последовательностей, известные специалистам в данной области, например, база данных http://www.ncbi.nlm.nih.gov/Genbank/index.html. Специфичность праймеров проверяли, например, с помощью программного обеспечения, использующего поиск в базах нуклеотидных последовательностей по алгоритму BLAST (например, www.ncbi.nlm.nih.gov/BLAST).
Перечень используемых в данном примере праймеров приведен в Таблице 2. Праймеры синтезировали на автоматическом синтезаторе 394 DNA/RNA synthesizer (Applied Biosystems, США). В качестве флуоресцентной метки использовали краситель Imd 500- dUTP №49, d-UTP-конъюгат (ООО «БИОЧИП-ИМБ»), максимум поглощения при 645 нм, максимум флуоресценции при 670 нм. Введение метки проводили в соответствии с рекомендациями производителя.
в) ПЦР для амплификации целевых фрагментов генов
Готовили реакционную смесь для проведения ПЦР, внося в пробирку следующие реагенты (из расчета на 1 реакцию):
- вода деионизованная 21,5 мкл;
- ПЦР-буфер (10 × кратная смесь, содержащая 100 тМ трис-HCl, 500 мМ KCl; 15 мМ MgCl2) 3 мкл;
- дНТФ (водный раствор дезоксинуклеозидтрифосфатов, содержащий по 2 ммоль/л дАТФ, дГТФ, дУТФ, дЦТФ) 3 мкл;
- смесь праймеров 1 мкл;
- Taq-полимераза, активность 5 Ед./мкл 0,5 мкл.
К 29 мкл смеси добавляли 1 мкл ДНК, полученной после этапа выделения ДНК. Для отрицательного контрольного образца вместо матрицы вносили деионизованную стерильную воду. Пробирки помещали в ДНК-амплификатор С1000, «Biorad», США и проводили ПЦР по следующей программе: предварительная денатурация ДНК: +95°С, 1 цикл 10 мин; 39 циклов ПЦР по следующему температурно-временному режиму: +95°С - 30 с, +70°С - 30 с, +72°С - 30 с; 42 цикла ПЦР по следующему температурно-временному режиму: +95°С - 30 с, +54°С - 30 с, +72°С - 30 с; финальная элонгация при +72°С - 5 мин.
г) Гибридизация флуоресцентно-меченных продуктов на биочипах
К 27 мкл реакционной смеси, полученной после ПЦР, добавляли концентрированный раствор гибридизационного буфера так, чтобы конечная концентрация компонентов составляла:
- гуанидин тиоцианат - 1М,
- HEPES - 50 мМ (рН 7,5),
- ЭДТА - 5 мМ.
30 мкл полученной смеси помещали в гибридизационную камеру микрочипа через отверстие в камере. Биочипы помещали в термостат на +37°С и инкубировали в течение 5 ч. По окончании гибридизации удаляли гибридизационные камеры, и биочипы трижды промывали дистиллированной водой, прогретой до +37°С, в течение 30 с и высушивали,
д) Регистрация и интерпретация результатов гибридизации
Результаты гибридизации регистрировали с помощью универсального аппаратно-программного комплекса (УАПК) для анализа изображений диагностических микрочипов с использованием специализированного программного обеспечения "ImageWare" (ООО «БИОЧИП-ИМБ»).
Флуоресцентные изображения биочипов после проведения гибридизации приведены на Фиг. 3-6.
Интерпретация результатов гибридизации на биочипе осуществляется по алгоритму, реализованному в модуле программного обеспечения, путем сравнения интенсивности флуоресценции сигналов в пределах каждой из групп ячеек, в которых образовались совершенные и несовершенные гибридизационные дуплексы.
Пример 3. Идентификация генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов. Для проведения анализа использовали биологические микрочипы, полученные, как описано в Примере 1. Процедура анализа описана в Примере 2. Флуоресцентные изображения (гибридизационные картины) и информация, приведенная в отчетах программы, представлены на Фигурах 3-6.
Правильность определения аллельных вариантов полиморфизмов в анализируемых генах по предлагаемому способу во всех случаях проверяли независимым методом секвенирования нуклеотидных последовательностей целевых фрагментов генов. Секвенирование проводилось на автоматическом секвенаторе 3130xL Applied Biosystems (США) с помощью коммерческого набора для проведения секвенирующих реакций «BigDye Terminator v3.1 Ready Reaction Cycle Sequencing Kit». В качестве секвенирующих праймеров применялись соответствующие праймеры, используемые для проведения ПЦР.
Знак «*» после нуклеотида означает, что данный нуклеотид является LNA-модифицированным.


| название | год | авторы | номер документа |
|---|---|---|---|
| Способ определения полиморфных маркеров в генах CYP2C19 и CYP2D6 для определения индивидуальной чувствительности к антидепрессантам | 2018 |
|
RU2716589C1 |
| СПОСОБ АНАЛИЗА ГЕНЕТИЧЕСКОГО ПОЛИМОРФИЗМА, ОПРЕДЕЛЯЮЩЕГО ПРЕДРАСПОЛОЖЕННОСТЬ К ОНКОЛОГИЧЕСКИМ ЗАБОЛЕВАНИЯМ И ИНДИВИДУАЛЬНУЮ ЧУВСТВИТЕЛЬНОСТЬ К ФАРМАЦЕВТИЧЕСКИМ ПРЕПАРАТАМ С ИСПОЛЬЗОВАНИЕМ ОЛИГОНУКЛЕОТИДНОГО БИОЛОГИЧЕСКОГО МИКРОЧИПА (БИОЧИПА) | 2005 |
|
RU2303634C2 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ЗОНДОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ПОЛИМОРФНЫХ ЛОКУСОВ ДНК, АССОЦИИРОВАННЫХ С РИСКОМ РАЗВИТИЯ СПОРАДИЧЕСКОЙ ФОРМЫ БОЛЕЗНИ АЛЬЦГЕЙМЕРА В РОССИЙСКИХ ПОПУЛЯЦИЯХ | 2014 |
|
RU2600874C2 |
| Способ анализа полиморфных маркеров в генах метаболизма лекарственных препаратов и генах иммунного ответа при терапии острых лейкозов у детей | 2016 |
|
RU2643333C1 |
| Способ анализа полиморфных маркеров в генах VKORC1, CYP4F2, CYP2C9, CYP2C19, ABCB1, ITGB3 для определения индивидуальной чувствительности к противосвертывающим препаратам | 2018 |
|
RU2689400C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ГЕНЕТИЧЕСКИХ ФАКТОРОВ РИСКА РАЗВИТИЯ ДЕМЕНЦИЙ АЛЬЦГЕЙМЕРОВСКОГО ТИПА НА ОСНОВЕ ГИДРОГЕЛЕВОГО МАТРИЧНОГО БИОЧИПА | 2022 |
|
RU2795795C1 |
| БИОЛОГИЧЕСКИЙ МИКРОЧИП С НАБОРОМ ПРАЙМЕРОВ ДЛЯ АНАЛИЗА ПОЛИМОРФИЗМА В ГЕНАХ AB0, HLA-DQA1, AMEL, DARC, NAT2 | 2014 |
|
RU2582216C2 |
| СПОСОБ АНАЛИЗА ГЕНЕТИЧЕСКОГО ПОЛИМОРФИЗМА ДЛЯ ОПРЕДЕЛЕНИЯ ПРЕДРАСПОЛОЖЕННОСТИ К ШИЗОФРЕНИИ И АЛКОГОЛИЗМУ | 2012 |
|
RU2565036C2 |
| Способ анализа терминальных мутаций в генах BRCA1, BRCA2, ATM и PALB2 с использованием мультиплексной ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом) | 2020 |
|
RU2729360C1 |
| Способ определения генетических маркеров для оценки полигенного риска развития гормон-позитивного подтипа рака молочной железы | 2020 |
|
RU2753002C1 |




Изобретение относится к биотехнологии и может быть использовано в медицине. Изобретение касается способа выявления нуклеотидных замен в генах, влияющих на метаболизм химиотерапевтических препаратов. Изобретение включает биологический микрочип (биочип), набор олигонуклеотидных зондов и праймеров для осуществления способа. Идентификация полиморфизмов осуществляется путем амплификации фрагментов генов с полиморфными локусами с использованием набора праймеров и последующей гибридизацией продуктов амплификации на биочипе с иммобилизованными олигонуклеотидными зондами. 3 н. и 2 з.п. ф-лы, 5 ил., 2 табл., 2 пр.
1. Способ идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, включающий:
а) обеспечение биочипа, представляющего собой подложку с гелевыми элементами, в каждом из которых иммобилизован специфический олигонуклеотидный зонд, относящийся к одной из следующих групп:
- группа зондов, последовательности которых представлены SEQ ID NO 1-4, для идентификации полиморфизма rs1695 (аллели А и G) в гене GSTP1;
- группа зондов, последовательности которых представлены SEQ ID NO 5, 6, для идентификации полиморфизма rs1045642 (аллели Т и С) в гене АВСВ1;
- группа зондов, последовательности которых представлены SEQ ID NO 7, 8, для идентификации полиморфизма rs1801133 (аллели Т и С) в гене MTHFR;
- группа зондов, последовательности которых представлены SEQ ID NO 9, 10, для идентификации полиморфизма rs396991 (аллели G и Т) в гене FCGR3A;
- группа зондов, последовательности которых представлены SEQ ID NO 11-14, для идентификации полиморфизма rs4880 (аллели С и Т) в гене SOD2;
- группа зондов, последовательности которых представлены SEQ ID NO 15-18, для идентификации полиморфизма rs1042522 (аллели С и G) в гене ТР53;
- группа зондов, последовательности которых представлены SEQ ID NO 19-26, для идентификации полиморфизмов rs11615 (аллели С и Т), rs3212986 (аллели G и Т) в гене ERCC1;
- группа зондов, последовательности которых представлены SEQ ID NO 27-30, для идентификации полиморфизма rs2228001 (аллели А и С) в гене ХРС;
- группа зондов, последовательности которых представлены SEQ ID NO 31-32, для идентификации полиморфизма rs4444903 (аллели А и G) в гене EGF;
- группа зондов, последовательности которых представлены SEQ ID NO 33-36, для идентификации полиморфизма rs8175347 (аллели ТА5, ТА6, ТА7 и ТА8) в гене UGT1A1;
- группа зондов, последовательности которых представлены SEQ ID NO 37-46, для идентификации полиморфизмов rs3918290 (аллели А и G), rs2297595 (аллели С и Т), rs67376798 (аллели А и Т) в гене DP YD;
- группа зондов, последовательности которых представлены SEQ ID NO 47-50, для идентификации полиморфизма rs25487 (аллели А и G) в гене XRCC1;
- группа зондов, последовательности которых представлены SEQ ID NO 51-52, для идентификации полиморфизма rs3892097 (аллели А и G) в гене CYP2D6;
- группа зондов, последовательности которых представлены SEQ ID NO 61-66, для идентификации полиморфизмов rs1142345 (аллели А и G), rs1800462 (аллели G и С), rs1800584 (аллели А и G), rs1800460 (аллели G и А) в гене ТРМТ;
б) набор олигонуклеотидных праймеров, комплементарных целевым фрагментам генов GSTP1, АВСВ1, MTHFR, FCGR3A, SOD2, ТР53, ERCC1, ХРС, EGF, UGT1A1, DPYD, XRCC1, CYP2D6, ТРМТ, обеспечивающих получение продуктов амплификации в виде преимущественно одноцепочечных флуоресцентно-меченных фрагментов ДНК;
в) гибридизацию амплифицированных флуоресцентно-меченных продуктов, полученных на стадии (б), на биочипе, полученном на стадии (а);
г) регистрацию и интерпретацию результатов гибридизации на биочипе, проведенной на стадии (в), путем сравнения интенсивности флуоресценции сигналов в пределах каждой из групп ячеек, в которых образовались совершенные и несовершенные гибридизационные дуплексы.
2. Способ по п. 1, характеризующийся тем, что для анализа используется ДНК, выделенная из периферической крови человека.
3. Способ по п. 1, характеризующийся тем, что регистрацию результатов стадии (г) проводят с помощью прибора-анализатора флуоресценции, оснащенного специализированным для данного способа программным обеспечением, что позволяет использовать программную автоматическую обработку интенсивностей сигналов с последующей интерпретацией результатов.
4. Биочип, используемый в способе идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, представляющий собой подложку с гелевыми элементами, в каждом из которых иммобилизован специфический зонд, причем последовательности зондов представлены SEQ ID NO 1-66.
5. Набор олигонуклеотидных зондов, используемый для получения биочипа, в способе идентификации генетических полиморфизмов, влияющих на метаболизм противоопухолевых препаратов, причем зонды имеют последовательности SEQ ID NO 1-66.
| Глотов А | |||
| С., Наседкина Т.В | |||
| СОЗДАНИЕ БИОЧИПА ДЛЯ АНАЛИЗА ПОЛИМОРФИЗМА В ГЕНАХ СИСТЕМЫ БИОТРАНСФОРМАЦИИ, Молекулярная биология, 2005 г., том 39, номер 3, стр | |||
| Способ включения усилителя в трансляцию | 1923 |
|
SU403A1 |
| Yun Kyung Jung, Jungkyu Kim, Microfluidic Linear Hydrogel Array for Multiplexed Single Nucleotide Polymorphism (SNP) Detection, Analytical chemistry, 2015, Vol | |||
| Торфодобывающая машина с вращающимся измельчающим орудием | 1922 |
|
SU87A1 |
| КОНТРОЛЬНАЯ ДОСКА С ПОДВИЖНЫМИ КУБИКАМИ ДЛЯ ПЛАНИРОВАНИЯ И УЧЕТА ПРОИЗВОДСТВА | 1925 |
|
SU3165A1 |
Авторы
Даты
2019-08-12—Публикация
2018-08-07—Подача