Область техники, к которой относится изобретение
Изобретение относится к области генетики, молекулярной биологии, судебной медицины и криминалистики и касается инструмента для анализа полиморфизма в генах АВО, HLA-DQA1, AMEL, DARC, NAT2 с помощью технологии гидрогелевых ДНК-микрочипов (биочипов).
Уровень техники
Известен ряд способов генетической идентификации личности, использующих различные инструменты для анализа уникальности нуклеотидной последовательности ДНК каждого человека.
1. Анализ полиморфизма длины амплифицированных фрагментов ПДАФ-анализ. Основан на определении длины полиморфных локусов ДНК, содержащих тандемные повторы. Количество повторов внутри такого локуса варьирует у разных людей. Анализ 12-16 подобных локусов позволяет получить генетическую характеристику, являющуюся уникальной в пределах популяции планеты. Метод требует амплификации ДНК в полимеразной цепной реакции, с последующим гель-электрофорезом. Преимуществом метода является высокая дискриминирующая способность, а недостатками - трудоемкость, высокие требования к квалификации персонала, дорогостоящее оборудование для капиллярного гель-электрофореза (ДНК-секвенаторы), в связи со значительной длиной STR-локусов (STR - short tandem repeats; локусы, содержащие короткие тандемные повторы) большое влияние на результат имеет степень деградации геномной ДНК (высокодеградированная ДНК нередко встречается в экспертно-криминалистической работе).
2. Анализ полиморфизма длины рестриктазных фрагментов ДНК (ПДРФ - анализ). В результате мутаций в геномной ДНК возникают новые или утрачиваются ранее существовавшие сайты рестрикции - места воздействия рестриктаз, расщепляющих цепь ДНК. Это, в свою очередь, обусловливает изменение длины получающихся рестриктазных фрагментов ДНК. Распределение фрагментов по длинам выявляется с помощью блот-гибридизации. Однако большинство полиморфных локусов имеют только два варианта: дикий тип/мутация, т.е. являются диаллельными. Ценность диморфных маркеров невелика, т.к. у многих людей может оказаться один и тот же вариант. Эта особенность, наряду с высокой требовательностью к количеству и сохранности исследуемой ДНК, являются основными недостатками метода, приведшими к его исчезновению из криминалистической практики [Joseph Т, Kusumakumary P, Chacko Р, Abraham A, Radhakrishna Pillai M. Genetic polymorphism of CYP1A1, CYP2D6, GSTM1 and GSTT1 and susceptibility to acute lymphoblastic leukaemia in Indian children. Pediatr Blood Cancer. 2004 Oct; 43(5): 539-41].
3. Секвенирование амплифицированных фрагментов ДНК, т.е. определение индивидуального генетического кода в заданном фрагменте ДНК. Самый точный и доказательный метод анализа индивидуальных генетических вариаций. Преимуществом является высокая точность и чувствительность метода, а недостатком - чрезвычайно высокая стоимость оборудования и анализа, что исключает широкое внедрение метода в экспертную практику как минимум в ближайшее десятилетие.
4. Выявление однонуклеотидного полиморфизма методом аллель-специфичной ПЦР (полимеразной цепной реакции). Аллельные варианты различаются за счет того, что 3′ концевой нуклеотид одного из праймеров в процессе ПЦР гибридизуется непосредственно с позицией SNP (т.е. если этот праймер комплементарен последовательности ДНК, то происходит наработка продукта), метод достаточно сложен, т.к. большой степени зависит от условий реакции и правильно выбранной последовательности праймера. Велико количество ложноположительных результатов. Кроме того, невозможно данным методом анализировать более 3-5 мутаций в одном анализе. Несмотря на все приведенные недостатки, при грамотно подобранных условиях, анализ SNP занимает всего 3-4 часа [Murata M, Shiraishi Т, Fukutome К, Watanabe M, Nagao M, Kubota Y, Ito H, Kawamura J, Yatani R. Cytochrome P4501A1 and glutathione S-transferase M1 genotypes as risk factors for prostate cancer in Japan. Jpn J Clin Oncol. 1998 Nov; 28(11):657-60].
5. Выявление однонуклеотидного полиморфизма методом гибридизации амплифицированного исследуемого образца с олигонуклеотидной матрицей. Данный метод предполагает предварительную мультиплексную амплификацию исследуемых фрагментов ДНК с использованием флюоресцентно меченных праймеров. В результате добавления избытка меченного праймера ПЦР достигается образование большого количества преимущественно меченного одноцепочечного продукта. Этот продукт гибридизуют с олигонуклеотидными зондами, иммобилизованными в определенном месте твердой подложки, либо в гелевых микрокаплях, закрепленных на подложке (такая матрица ДНК-зондов носит название. биологического микрочипа или биочипа). Если последовательность анализируемой ДНК полностью комплементарна последовательность зонда, то образуется стабильный дуплекс (регистрируется флюоресцентный сигнал), если искомого фрагмента нет, или же в нем находится некомплементарное основание, то стабильного дуплекса не образуется (сигнал не наблюдается). Простота метода, низкая стоимость и возможность использования флюоресцентной метки позволяет легко автоматизировать процесс. Однако данный метод жестко связан с мультиплексной ПЦР, а ее возможности ограничены. А именно в ходе одной реакции могут быть амплифицированы лишь 4-5 локусов ДНК. При таких ограничениях используемые в данный момент для криминалистических нужд системы SNP в силу своей диаллельности не будут иметь достаточной дискриминационной силы при использовании их на платформе биочипов [Wen SY, Wang H, Sun OJ, Wang SQ. Rapid detection of the known SNPs of CYP2C9 using oligonucleotide microarray. World J Gastroenterol. 2003 Jun; 9(6):1342-6]. Пример реализации данного подхода описан в следующей работе [Наседкина Т.В., Фесенко Д.О., Митяева О.Н., Лысое Ю.П., Барский В.Е., Заседателев А.С. (2007). Способ генетической идентификации личности на основе анализа однонуклеотидного полиморфизма генома человека с использованием олигонуклеотидного биологического микрочипа. Патент PCT/RU/2007/000016, международная заявка № WO 2008088236], где предлагается инструмент, выполняющий выборочный анализ трех генов и позволяющий разделить всю популяцию на 1350 групп.
Ближайшим аналогом настоящего изобретения является инструмент, описанный в п.5. К существенным недостаткам способа можно отнести его низкую дискриминационную силу (1/1350), обусловленную малым количеством анализируемых локусов. Практический недостаток низкой дискриминационной силы заключается в снижении возможностей идентификации подозреваемого, особенно при анализе относительно замкнутых поселений, где генотипы жителей весьма гомологичны, либо при очень масштабных поисковых работах. Решением этой проблемы является создание инструмента с расширенным числом анализируемых маркеров, позволяющих повысить дискриминационную силу до 1/36450, что и предлагается в данном изобретении.
Раскрытие изобретения
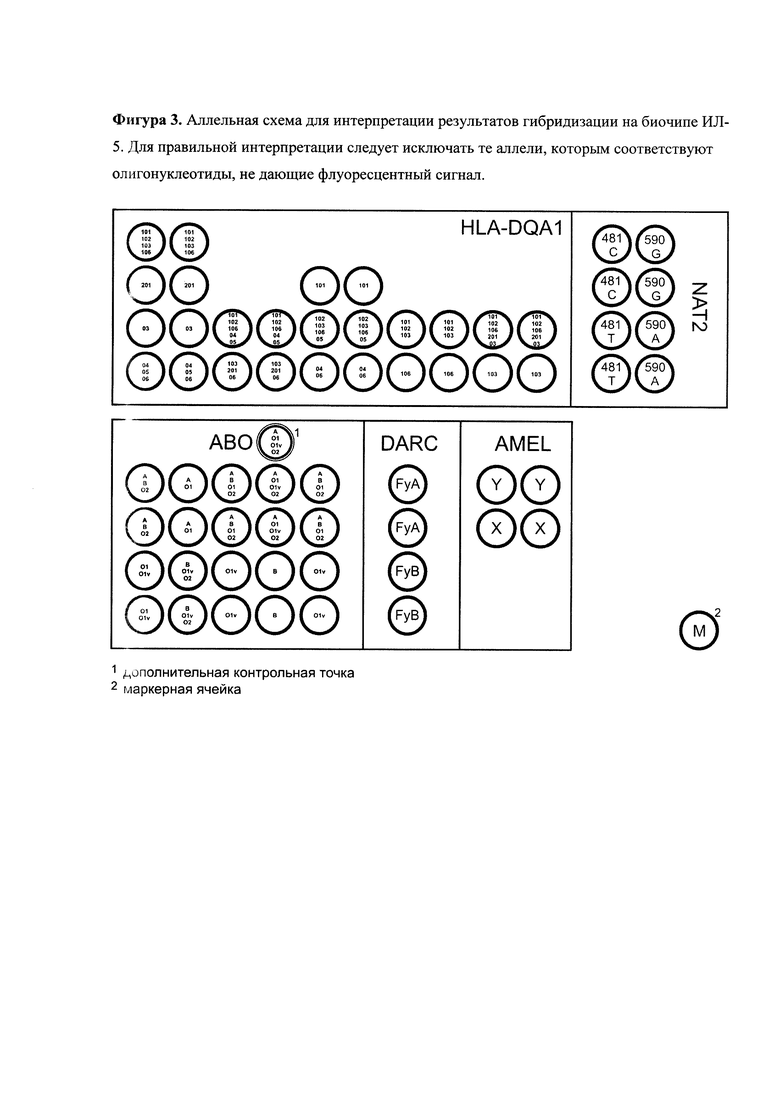
Сущность изобретения заключается в создании биочипа (под условным названием «ИЛ-5») в ячейках которого иммобилизованы оригинальные дифференцирующие олигонуклеотиды, а также в создании набора ПЦР-праймеров для амплификации ДНК пяти локусов: DARC (ген группы крови Duffy), ABO, NAT2, HLA-DQA1 и AMEL. Дифференцирующие олигонуклеотиды подобраны таким образом, чтобы специфично связываться с целевой аллелью, а праймеры для мультиплексной ПЦР подобраны таким образом, чтобы равномерно нарабатывать все исследуемые локусы. Применение биочипа ИЛ-5 позволит разделить человеческую популяцию на 36450 групп.
Дифференцирующие олигонуклеотиды (SEQ ID NO:1-31) иммобилизуются в ячейках гидогелевого микрочипа, как описано в патенте [Мирзабеков А. Д., Рубина А.Ю., Паньков С. В., Перов А.Н., Чупеева В.В. Композиция для иммобилизации биологических макромолекул в гидрогелях, способ приготовления композиции, биочип, способ проведения ПЦР на биочипе. RU 2206575 С2] в концентрации 100-10000 пкмоль на 1 мкл геля в зависимости от задач исследователя. Схема расположения ячеек может быть произвольной, мы использовали схему, приведенную на Фиг.1. Проведение анализа с использованием предлагаемого изобретения выполняется аналогично способу, описанному в работе [Наседкина Т.В., Фесенко Д.О., Митяева О.Н., Лысое Ю.П., Барский В.Е., Заседателев А.С. (2007). Способ генетической идентификации личности на основе анализа однонуклеотидного полиморфизма генома человека с использованием олигонуклеотидного биологического микрочипа. Патент PCT/RU/2007/000016, международная заявка № WO 2008088236]. По итогам гибридизации и отмывки анализируется полученная флуоресцентная картина, на основании чего делается вывод о генотипе в исследуемом образце. Пример гибридизационной картины приведен на Фиг.2.
Характеристики анализируемых локусов: высокополиморфные локусы ABO и HLA-DQA1 в сумме обеспечивают разделение популяции на 675 групп, локус NAT2 - еще на 9, DARC - на 3, AMEL на 2. В сумме, 5 локусов обеспечивают разделение на 36450 групп. Иначе говоря, существует 36450 индивидуальных профиля свечения ячеек биочипа. Данные, полученные с помощью настоящего изобретения, могут быть использованы для получения поисковых сведений (пол, группа крови), сужения круга подозреваемых лиц и в качестве предварительного этапа верификации принадлежности исследуемой ДНК подозреваемому лицу.
В качестве экспертного образца могут быть использованы образцы ДНК, выделенные из крови, слюны, волосяной луковицы, отпечатка пальца, содержащего частички эпителия и других ДНК-содержащих материалов.
Основным аспектом данного изобретения является биочип, содержащий набор иммобилизованных дифференцирующих олигонуклеотидов, последовательность которых приведена в Табл.1 и Перечне последовательностей (SEQ ID NO:1-31).
Другим аспектом изобретения является набор праймеров для амплификации фрагментов генов, используемый для получения исследуемых флуоресцентно меченных локусов в требуемом количестве. Последовательность праймеров приведена в Табл.2 и Перечне последовательностей (SEQ ID NO:31-56).
Осуществление изобретения
Задача настоящего изобретения состоит в создании биологического микрочипа, позволяющего определять генотип в пяти локусах генома человека: DARC, АВО, NAT2, HLA-DQA1 и AMEL. Важнейшей задачей при осуществлении данного изобретения является обеспечение такой структуры дифференцирующих олигонуклеотидных зондов, чтобы происходило их связывание только с полностью комплементарными мишенями, обеспечивая яркий флуоресцентный сигнал в соответствующих им ячейках биочипа. В то же время неспецифическое связывание должно быть сведено к минимуму, чтобы исключить ложноположительное «срабатывание» ячеек. В связи с тем, что область полиморфного сайта консервативна (за исключением полиморфного нуклеотида), парные зонды (анализирующие один однонуклеотидный полиморфный сайт) отличаются только по центральному нуклеотиду и идентичны по флангам. В такой ситуации необходимо варьировать длину фланкирующих областей, чтобы обеспечить высокое специфическое и низкое неспецифическое связывание исследуемой мишени. Т.к. на данный момент отсутствуют точные термодинамические методики расчета структуры зонда, выбор структуры осуществляется эмпирически, последовательным подбором и экспериментальной проверкой. В ходе этой работы были выбраны такие структуры, и они приведены в Табл.1 и Перечне последовательностей (SEQ ID NO:1-31).
Для обеспечения сбалансированной эффективной амплификации исследуемых образцов необходимо выбрать структуру праймеров мультиплексной ПЦР. Праймеры должны быть выбраны таким образом, чтобы их отжиг на мишени происходил при одинаковой для всех температуре (60-65°С), должны отсутствовать высокоэнергетические внутренние шпильки и дуплексы между всеми праймерами. Ампликоны всех локусов должны варьировать по длине в пределах 250-600 п.н. для первого раунда. Решение этих задач требует не только точных расчетов, но и длительной экспериментальной проверки и коррекции. В результате проведенной работы был выбран оптимальный состав и структура ПЦР-праймеров.
Далее приведем последовательность анализа с использованием данного изобретения. Образец ДНК используют как матрицу в мультиплексной «гнездовой» двухэтапной ПЦР. На втором этапе ПЦР получают флуоресцентно меченный амплифицированный фрагмент. Меченый ПЦР-продукт используют для дальнейшей гибридизации на биочипе, содержащем иммобилизованные олигонуклеотиды, представляющие собой участки последовательностей генов, комплементарных как последовательности "дикого типа", так и вариабельной последовательности. Анализируемый фрагмент ДНК образует совершенные гибридизационные дуплексы только с соответствующими полностью комплементарными ему олигонуклеотидами. Со всеми остальными олигонуклеотидами анализируемый фрагмент ДНК будет образовывать несовершенные дуплексы, стабильность которых существенно ниже стабильности совершенных дуплексов. Дискриминацию совершенных и несовершенных дуплексов проводят после отмывки биочипа, сравнивая интенсивности флуоресценции соответствующих ячеек биочипа. Интенсивность сигнала в случае образования совершенного дуплекса выше, чем в случае несовершенного. Используя схему расположения олигонуклеотидов на биочипе, определяют, какие варианты присутствуют в том или ином образце.
Полимеразная цепная реакция может быть проведена с использованием любого вида термостабильной полимеразы (Taq-полимераза, фрагмент Стоффеля, Tth-полимераза, Tfl-полимераза, Pfu-полимераза, Vent-полимераза, DeepVent-полимераза и т.п. без ограничения, которые коммерчески доступны от большого числа производителей), работающей в соответствующем буфере. Для построения новой цепи в буфер добавляется смесь дНТФ (дАТФ, дГТФ, дЦТФ, дТТФ) в принятых концентрациях, но вместо дТТФ может быть использован дУТФ. Для проведения ПЦР могут быть использованы готовые коммерчески доступные наборы, содержащие все необходимые компоненты за исключением праймеров.
В качестве праймеров для проведения мультиплексной «гнездовой» двухстадийной полимеразной цепной реакции используют праймеры SEQ ID NO:32-56, приведенные в Перечне последовательностей, а также в таблице 2. Существуют разнообразные химические подходы к синтезу олигонуклеотидных праймеров, например фосфодиэфирный метод, гидрофосфорильный метод, и т.д. но наибольшее распространение в настоящее время имеет фосфорамидитный метод. Синтез праймеров осуществляют, используя автоматические ДНК/РНК синтезаторы, например производства фирмы Applied Biosystems (США).
Мечение ПЦР-продукта на втором этапе ПЦР проводят с помощью праймеров, несущих флуоресцентную метку на 5′-конце, либо включением меченых трифосфатов в растущую цепь ампликона. В качестве флуоресцентной метки может быть использован любой флуорохром (например, без ограничения, FITC, Texas red, Cy-3, Cy-5 и т.д.), а также биотин.
На второй стадии мультиплексной «гнездовой» двухэтапной ПЦР праймеры, входящие в состав одной пары, используются в неравных количествах. Это позволяет получать преимущественно одноцепочечный ампликон, способный к гибридизации с ДКН-зондами. Перед постановкой гибридизации ампликон денатурируют путем прогрева готовой гибридизационной смеси при 95°С в течение 5 мин с последующим быстрым охлаждением во льду. Гибридизация может быть проведена в любом известном специалисту в данной области гибридизационном буфере, например в SSPE-буфере или гуанидиновом буфере. Типичное время гибридизации - 8-12 ч при 37°С. Отмывка может быть проведена в любом известном в данной области техники буфере с добавлением соли (SSC, SSPE и т.п.) или в деионизованной воде, но за более короткое время (J.F. Sambrook and D.W. Russell, 2001, "Molecular cloning: a laboratory manual" Cold Spring Harbor Laboratory Press).
Регистрация гибридизационной картины может быть произведена с помощью любой детектирующей системы, распознающей флуоресцентный сигнал (флуоресцентный микроскоп с ПЗС-камерой, лазерный сканер или портативный анализатор биочипов, доступные коммерчески от большого числа производителей, например портативный анализатор биочипов, производимый серийно ООО «Биочип-ИМБ», и снабженной регистрирующим устройством (ПЗС-камерой, видеокамерой, фотокамерой)). Анализ изображения может быть произведен как визуально, так и в автоматическом режиме с использованием специальной программы.
При изготовлении микрочипа могут быть использованы олигонуклеотиды, несущие по 5′- или 3′-концу активную группу, обеспечивающую иммобилизацию. Существуют разнообразные химические подходы к синтезу олигонуклеотидов, например фосфодиэфирный метод, гидрофосфорильный метод и т.д., но наибольшее распространение в настоящее время имеет фосфорамидитный метод. Синтез олигонуклеотидов осуществляют, используя автоматические ДНК/РНК синтезаторы, например производства фирмы Applied Biosystems (США). Возможно использование широкого спектра групп для модификации иммобилизуемых олигонуклеотидов, таких как аминогруппа, тиол, гидразид, акриламидная группа, бензальдегид, тиофосфат и т.п. (Seliger H., Hinz М., Нарр Е. "Arrays of immobilized oligonucleotides - contributions to nucleic acids technology" Current Pharmaceutical Biotechnology, 2003, 4, 379-395). Модификация олигонуклеотида, имеющая целью введение активной группы, пригодной для последующей иммобилизации олигонуклеотида на биочипе, может быть осуществлена как в автоматическом режиме при синтезе с использованием широкого спектра модификаторов, которые коммерчески доступны, например, от Glen Research (USA), так и постсинтетически в ручном режиме.
Биочипы могут быть изготовлены путем создания на поверхности стеклянной подложки матрицы из ячеек полиакриламидного геля, активации ячеек и ковалентной иммобилизации в ячейках модифицированных олигонуклеотидов, несущих активные группы (Mirzabekov А. & Kolchinsky A. MAGIChip: Properties and applications in genomic studies. (2002) In Genomic Technologies: Present and Future. Eds. Galas D.J., S.J. McCormack. Caister Academic Press, pp.163-196). Также биочипы могут быть изготовлены методом химически индуцируемой или фотоиндуцируемой сополимеризации олигонуклеотида в акриламидном геле, как описано ранее (Vasiliskov, V., Timofeev E., Surzhikov S., Drobyshev, A., Shick, V. & Mirzabekov, A. 1999. Fabrication of microarray of gel-immobilized compounds on a chip by copolymerization. BioTechnique, 27, 592-606; Мирзабеков А.Д., Рубина А.Ю., Паньков С.В., Чернов Б.К. Патент на изобретение №2175972 «Способ иммобилизации олигонуклеотидов, содержащих непредельные группы, в полимерных гидрогелях при формировании микрочипа». Приоритет от 28.12.1999). Также биочипы могут быть изготовлены любыми другими известными специалисту в данной области способами (Seliger H., Hinz М., Нарр Е. "Arrays of immobilized oligonucleotides - contributions to nucleic acids technology" Current Pharmaceutical Biotechnology, 2003, 4, 379-395), а в качестве подложки помимо стекла может быть использован другой материал (пластик, металл, гибкие мембраны и т.д.). Также для изготовления микрочипов могут быть использованы активированные подложки, коммерчески доступные от различных производителей (см., например, Ramakrishnan R, Domanus D, Lublinsky A, Nguyen A, Domanus М, Prokhorova A, Gieser L, Touma E, Lockner R, Tata М, Zhu X, Patterson М, Shippy R, Sendera TJ, Mazumder A. 2002. An assessment of Motorola CodeLink microarray performance for gene expression profiling applications. Nucleic Acids Res. 30: e30).
Для изготовления биочипа настоящего изобретения используют набор олигонуклеотидов SEQ ID NO:1-31, приведенных в Перечне последовательностей, а также Табл.1. В качестве контроля прохождения гибридизации в настоящем изобретении используют образец контрольной ДНК. Расположение конкретных олигонуклеотидных зондов на биочипе может варьировать и определяется только удобством для интерпретации результатов гибридизации.
С помощью биочипа можно анализировать 9 групп аллелей гена HLA-DQA1: группа 101 объединяет аллели 0101XX, 0104ХХ, 0105, 0107; группа 102 - 0102ХХ; группа 103 - 0103; группа 106 - 0106; группа 02-0201; группа 03 - 03ХХХХ, группа 04 - 04ХХХХ; группа 05 - 05ХХХХ; группа 06 - 06ХХХХ. Для простоты в дальнейшем группы аллелей 101, 102, 103, 106, 02, 03, 04, 05, 06 будем называть просто аллелями. Биочип позволяет анализировать четыре полиморфных участка во втором экзоне гена HLA-DQA1. На чипе каждый полиморфный участок представлен столбцом пар ячеек. Первая пара столбцов имеет 4 строки, вторая пара столбцов - 2 строки, третья пара столбцов - 3 строки, четвертая пара столбцов - 2 строки, пятая пара столбцов - 2 строки. Первый полиморфный участок расположен между 203 и 234 позициями. Первый вариант первого участка (первая строка первой пары столбцов) отвечает за 101, 102, 103, 106 аллели, второй вариант первого полиморфного участка (вторая строка первой пары столбцов) отвечает за 02 аллель, третий вариант первого полиморфного участка (третья строка первой пары столбцов) отвечает за 03 аллель, четвертый вариант первого полиморфного участка (четвертая строка первой пары столбцов) отвечает за 04, 05, 06 аллели. Второй полиморфный участок расположен между 134 и 152 позициями. Первый вариант второго полиморфного участка (первая строка второй пары столбцов) отвечает за 101, 102, 106, 04, 05 аллели, второй вариант второго полиморфного участка (вторая строка второй пары столбцов) отвечает за 103, 02, 06 аллели. Третий полиморфный участок расположен между 161 и 188 позициями. Первый вариант третьего полиморфного участка (первая строка третьей пары столбцов) отвечает за 101 аллель, второй вариант третьего полиморфного участка (вторая строка третьей пары столбцов) отвечает за 102, 103, 106, 04, 05 аллели, третий вариант третьего полиморфного участка (третья строка третьей пары столбцов) отвечает за 04 и 06 аллели. Четвертый полиморфный участок расположен между 191 и 207 позициями. Первый вариант четвертого полиморфного участка (первая строка четвертой пары столбцов) отвечает за 101, 102, 103 аллели, второй вариант четвертого полиморфного участка (вторая строка четвертого столбца) отвечает за 106 аллель. Анализируя совокупность четырех полиморфных участков можно определить генотип. Первый вариант пятого полиморфного участка (верхняя строка пятой пары столбцов) отвечает за 101, 102, 106, 02 и 03 аллели. Второй вариант этого участка (нижняя строка пятой пары столбцов) - за аллель 103. Таким образом, проанализировав профиль светящихся ячеек, можно однозначно определить генотип образца.
В гене АВ0 можно выявить 5 полиморфных вариантов гена АВ0, сочетание которых дает 15 групп. Локус АВ0 является высокополиморфным и к настоящему времени опубликованы по нашим подсчетам около полутора сотен его аллелей. Подавляющее большинство из них встречаются крайне редко. Поэтому при выборе аллелей мы руководствовались, в первую очередь, частотой их встречаемости у белых европейцев, генетически наиболее близких к российской популяции. Для анализа были выбраны следующие группы аллелей: А, В, Ol, Olv и О2, детерминирующие серологическую группу крови у подавляющего большинства белых европейцев. Сочетание анализируемых SNP позволяет определить аллели гена АВО, присутствующие в образце (табл.3). В каждой из выбранных групп аллелей могут быть выделены аллели, различающиеся по другим SNP, но эти варианты редки и в подавляющем большинстве случаев серологически равнозначны между собой.
Ген амелогенина расположен на половой хромосоме и позволяет определять пол индивидуума. AMELX характеризуется трех буквенной делецией (позиции 5878-5880). На биочипе иммобилизованы два олигонуклеотида (AMELX и AMELY), позволяющие анализировать наличие или отсутствие делеции.
В гене DARC анализируется позиция 125G/A, определяющая аллели FyA и FyB группы крови Duffy, соответственно. Анализ по этому гену дает разделение на 3 группы.
В гене NAT2 анализируются две SNP: 481С/Т и 590G/A. Анализ этих точек дает разделение на 9 групп.
Всего разработанный биочип позволяет анализировать 28 групп аллелей в пяти генах (Фиг. 3).
Далее изобретение иллюстрируется примерами, которые показывают применение способа экспресс-анализа генетического полиморфизма. Следует, однако, понимать, что приводимые примеры служат исключительно для иллюстрации и не предназначены для ограничения объема притязаний, выраженных в формуле изобретения. На основании настоящего описания специалист в данной области сможет легко предложить свои варианты и модификации осуществления изобретения, не отходя от общей концепции настоящего изобретения и без привлечения собственной изобретательской деятельности, так что должно быть понятно, что такие варианты и модификации также будут входить в объем притязаний настоящего изобретения.
Пример 1. Олигонуклеотидный биочип для определения генетического полиморфизма в генах DARC, ABO, NAT2, HLA-DQA1 и AMEL.
Олигонуклеотиды для иммобилизации на микрочипе синтезируют на автоматическом синтезаторе 394 DNA/RNA Synthesizer (Applied Biosystems, США) с использованием стандартной фосфоамидитной процедуры. 5′ или 3′-конец олигонуклеотидов содержит спейсер со свободной аминогруппой, который вводят в состав олигонуклеотида при синтезе путем использования 3′-Amino-Modifier С7 CPG 500 (Glen Research, USA). Присоединение флуоресцентной метки к олигонуклеотидам по свободной аминогруппе осуществляют в соответствии с рекомендациями производителя. Олигонуклеотиды, меченные цианиновым красителем Су-3, очищают ВЭЖХ от олигонуклеотидов, не включивших флуоресцентный краситель, на колонке «Hypersil ODS 5 мкм, 4,6×250 мм» («SypeIco Int», США).
Биочип изготовляют методом сополимеризации олигонуклеотида в акриламидном геле, как описано ранее (Патент на изобретение № 2175972 «Способ иммобилизации олигонуклеотидов, содержащих непредельные группы, в полимерных гидрогелях при формировании микрочипа» Мирзабеков А.Д., Рубина А.Ю., Паньков С.В., Чернов Б.К. Приоритет от 28.12.1999). Биочип содержит 31 тип иммобилизованных олигонуклеотидов (SEQ ID NO:1-31), список которых представлен также в Таблице 1. Ячейки наносят согласно схеме на Фиг.1.
Пример 2. Амплификация фрагментов гена АВО методом двухэтапной «гнездовой» мультиплексной ПЦР с целью наработки флуоресцентно меченого фрагмента гена.
Из слюны испытуемых была выделена ДНК набором Qiagen. Для мультиплексной наработки всех анализируемых генов использовали ПЦР-праймеры SEQ ID NO:32-56. ПЦР проводили на приборе MiniCycler (MJ Research, Inc., USA) в объеме 25 мкл реакционной смеси, составом: для 1-го этапа: 1х буфер (67 мМ Трис-HCl, рН 8,6, 166 мМ (NH4)2SO4, 0,01% Тритон Х-100), 1,5 мМ MgCl2 0,2 мМ каждого из dNTP («Силекс», Россия), 5 рМ каждого из 4 праймеров (SEQ ID NO:32-43), 1 мкл геномной ДНК и 1,5 ед. акт. Taq-полимеразы («Силекс», Россия). ДНК денатурировали 5 мин при 94°С и проводили в первом этапе 35 циклов амплификации (94°С - 30 с, 60°С - 30 с, 72°С - 1 мин), затем 7 мин при 72°С. Смесь второго этапа ПЦР отличалась составом и концентрацией праймеров: верхние праймеры (SEQ ID NO:44, 46, 48, 50, 51, 53, 55) брали в концентрации 5 пкмоль/мкл, а нижние (SEQ ID NO:45, 47, 49, 52, 54, 56) - 50 пкмоль/мкл. В смесь добавляли 2 мкл продукта из первого этапа и амплифицировали по схеме: 3,5 мин 94°С, 34 цикла (94°С - 30 с, 62°С - 30 с, 72°С - 1 мин), 7 мин при 72°С. Нижние праймеры 2-го этапа были предварительно помечены по 5′ концу флуоресцентным красителем Cy5 (возб./исп.: 640/657 нм).
Пример 3. Гибридизация меченого продукта на биочипе.
Содержимое всех пробирок, полученных на втором этапе Примера 2, используют для гибридизации на биочипе в буфере следующего состава: 25% формамид (Gibco BRL), 5×SSPE. Готовую гибридизационную смесь перед гибридизацией денатурируют при 95°С в течение 5 мин, охлаждают во льду, 2 мин, и наносят на биочип в гибридизационную камеру объемом 20-40 мкл (Sigma, USA). Гибридизацию проводят в течение 10-14 ч при температуре 37°С. Отмывку проводят в буфере 1×SSPE при комнатной температуре в течение 10 мин.
Пример 4. Регистрация и интерпретация результатов гибридизации.
Регистрацию гибридизационной картины производят с помощью портативного анализатора биочипов, снабженного ПЗС-камерой, производимого ООО «Биочип-ИМБ». При визуальном анализе изображения интерпретацию результатов проводят, как в примере 2. Описание алгоритма автоматического анализа изображения с помощью программы ImageWare™ выходит за рамки настоящего изобретения.
Определим генотип по гибридизационной картине, представленной на Фигуре 2. Первый полиморфный участок HLA-DQA1: положительный сигнал наблюдается в первой и второй строках, следовательно, одна аллель из группы 101, 102, 103, 106, а вторая аллель 201. Далее второй полиморфный участок: положительный сигнал наблюдается в обеих строках, следовательно, присутствие 103 аллели исключаем. Третий полиморфный участок: положительный сигнал наблюдается во второй строке, следовательно, исключаем аллель 101. Четвертый полиморфный участок: положительный сигнал наблюдается в первой строке, следовательно, исключаем 106 аллель. В результате для рассматриваемого образца получаем генотип HLA-DQA1: 102/201.
Рассмотрим область биочипа, ответственную за генотипирование гена АВО. Сопоставим результаты гибридизации с данными из таблицы 3: Отсутствие сигнала в нижней паре первого столбца исключает аллели Ol и Olv. Т.к. образец является гетерозиготой по 297 позиции, следовательно, в нем точно присутствует аллель А и одна из двух: В или 02. Третий столбец не позволяет сделать вывод о второй аллели, а четвертый столбец исключает аллель В (т.к. нижняя пара не флуоресцирует). Остаются аллели А и 02.
Ген амелогенина представлен одной аллелью (Фиг.2): AMELX, из чего можно заключить, что образец принадлежит женщине.
Ген DARC представлен гетерозиготой: FyA/FyB.
Ген NAT2: 481 С/Т, 590G/G.


Изобретение относится к области биохимии. Заявлен набор дифференцирующих олигонуклеотидов для анализа полиморфизма в генах АВ0, HLA-DQA1, AMEL, DARC, NAT2 с помощью технологии гидрогелевых ДНК-микрочипов (биочипов). Биочип обеспечивает возможность определить нуклеотидный состав в четырнадцати полиморфных позициях, что, в свою очередь, позволяет установить генотип в вышеуказанных генах. Также представлен набор ПЦР-праймеров для амплификации вышеуказанных локусов. Изобретение позволяет повысить дискриминационный потенциал биочипа (36450 генотипичеких групп). 3 н.п. ф-лы, 3 ил., 3 табл., 4 пр.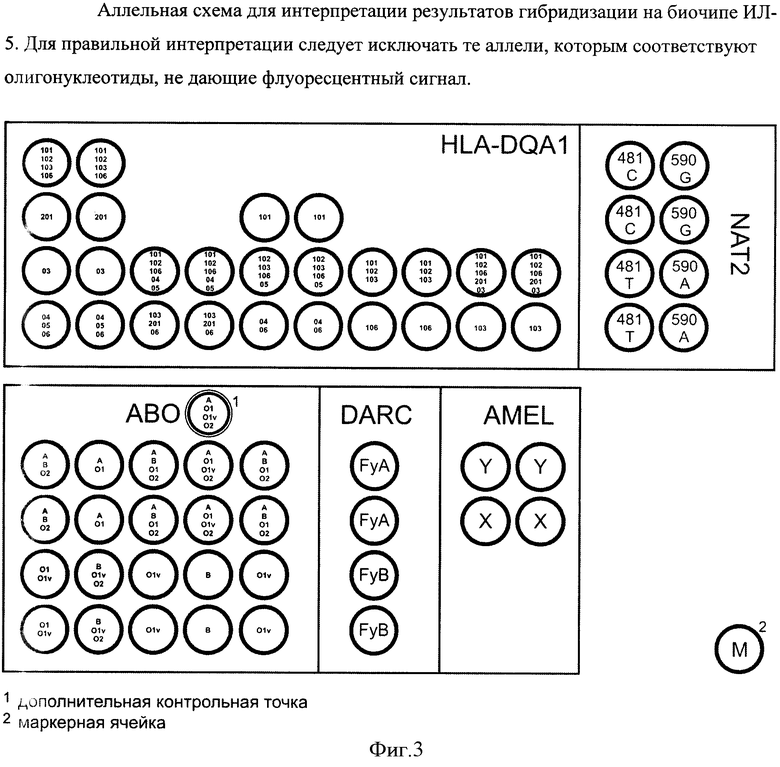
1. Набор дифференцирующих олигонуклеотидов, последовательность которых определена в Перечне последовательностей, для анализа полиморфизма генов: АВ0 (SEQ ID NO: 14-23), HLA-DQA1 (SEQ ID NO: 1-13), AMEL (SEQ ID NO: 26-27), DARC (SEQ ID NO: 24-25) и NAT2 (SEQ ID NO: 28-31), позволяющий идентифицировать аллели А, В, О1, О1v и О2 гена АВ0, группы аллелей 101, 102, 103, 106, 02, 03, 04, 05 и 06 гена HLA-DQA1, аллели AMELX и AMELY гена AMEL, аллели FyA и FyB гена DARC и аллели, определяемые полиморфизмами в 481С/Т и 590G/A гена NAT2.
2. Набор праймеров для амплификации локусов АВ0, HLA-DQA1, AMEL, DARC и NAT2, последовательность которых определена в Перечне последовательностей (SEQ ID NO: 32-56).
3. Биочип для анализа полиморфизма генов АВ0, HLA-DQA1, AMEL, DARC и NAT2, содержащий набор олигонуклеотидов по п.1 и подложку, на которой иммобилизованы указанные олигонуклеотиды.
| FESENKO D.O | |||
| et al., Preparing of single-stranded DNA in single-stage PCR with low-melt excess primer for hybridization on biochips, Molecular Biology, 2011, vol.45, no.2, pp.237-240 | |||
| FESENKO D.O | |||
| et al., HLA-DQA1, AB0, and AMEL genotyping of biological material with biochips, Molecular Biology, 2010, vol.44, no.3, pp.401-406 | |||
| ГРЯДУНОВ Д.А | |||
| и др., |
Авторы
Даты
2016-04-20—Публикация
2014-03-13—Подача