Изобретение относится к ветеринарной микробиологии, в частности к методам определения видовой принадлежности мяса с помощью полимеразной цепной реакции.
Известен набор для определения видовой принадлежности тканей кур и свиней методом полимеразной цепной реакции содержащий комплект реагентов для экстракции ДНК из клинического материала, продуктов питания и кормов для животных; комплект реагентов для амплификации ДНК Gallus gallus и Sus scrofa; комплект реагентов для электрофоретической детекции продуктов амплификации в агарозном геле ((Инструкция по применению тест-системы «ЧИС» для определения видовой принадлежности тканей кур и свиней методом полимеразной цепной реакции, организация-производитель - ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, г. Москва https://docviewer.yandex.ru).
Также известен тест-система, включающий пластиковые флаконы и пробирки, термостабильный фермент Tag-полимеразу, буфер для постановки реакции, смесь четырех дезоксинуклеотидтрифосфатов, специфичных для участка генома ДНК нескольких видов мяса олигонуклеотидных праймеров, зондов, внутренний контрольный образец в виде суспензии бактериофага и положительных контрольных образцов - содержащих фрагменты геномов ДНК нескольких видов мяса, (Сорокина М.Ю. Автореферат диссертации по ветеринарии на тему Разработка тест-системы для определения видовой принадлежности мясных ингредиентов в кормах методом полимеразной цепной реакции, Москва, 2004 - прототип).
Однако известный набор используется для полимеразной цепной реакции с электрофоретической детекцией продуктов амплификации в агарозном геле с использованием специфичных для участка генома ДНК баранины и говядины олигонуклеотидных праймеров, нуклеотидная последовательность которых непосредственно читается по электрофореграмме. Длина фрагмента, который может быть расшифрован этим методом, ограничивается разрешающей способностью метода гель-электрофореза, что влияет на точность диагностирования видовой принадлежности мяса в продуктах питания и кормах для животных. Кроме того, известный набор предназначается только для идентификации генома ДНК баранины (Ovis) и говядины (Bos).
Техническим результатом является расширение функциональных возможностей и повышение точности идентификации видовой принадлежности тканей кур свиней.
Технический результат достигается тем, что в тест-системе для определения видовой принадлежности тканей кур и свиней в продовольственном сырье, кормах и пищевых продуктах, включающем пластиковые флаконы и пробирки, термостабильный фермент Tag-полимеразу, буфер для постановки реакции, смесь четырех дезоксинуклеотидтрифосфатов, специфичных для участка генома ДНК нескольких видов мяса олигонуклеотидных праймеров, зондов, внутренний контрольный образец в виде суспензии бактериофага и положительных контрольных образцов - содержащих фрагменты геномов ДНК нескольких видов мяса, согласно изобретению в качестве генома ДНК мяса используют ткани курицы (Gallus gallus) и свиньи (Sus scrofa), при этом для внутреннего контрольного образца используют суспензию бактериофага Т4 с концентрацией 5×103 копий нуклеотидных последовательностей на 1 мкл, а для положительного контрольного образца - смесь содержащую фрагменты геномов тканей курицы (Gallus gallus) и свиньи (Sus scrofa) и бактериофага Т4 взятых в соотношении 1:1:1 со следующими нуклеотидными последовательностями:
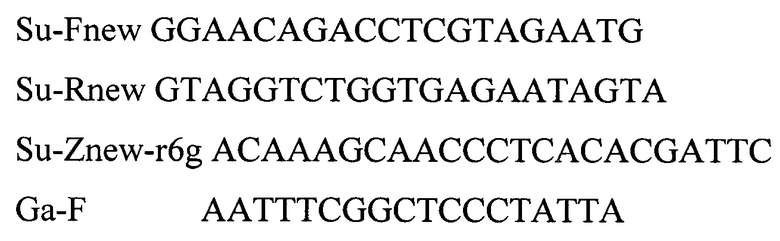
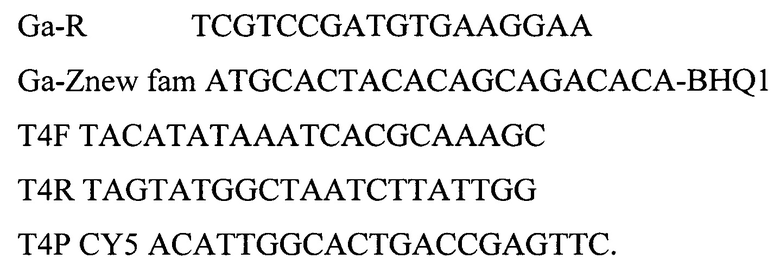
Новизна заявляемого технического решения заключается в том, что обеспечивается возможность идентификации видовой принадлежности мяса птицы и свинины с помощью полимеразной цепной реакции (ПНР) с флуоресцентной детекцией в режиме реального времени, что в свою очередь позволяет с высокой точностью определить наличие их ингредиентов в продовольственном сырье, кормах и пищевых продуктах.
Признаки, отличающие заявляемое техническое решение от прототипа, направлены на достижение технического результата и не выявлены при изучении данной и смежной областей науки и техники и, следовательно, соответствуют критерию «изобретательский уровень».
Заявляемый способ рекомендовано использовать в специализированных ветеринарных, санитарно-эпидемиологических, животноводческих, сельскохозяйственных предприятиях, что соответствует критерию «промышленная применимость».
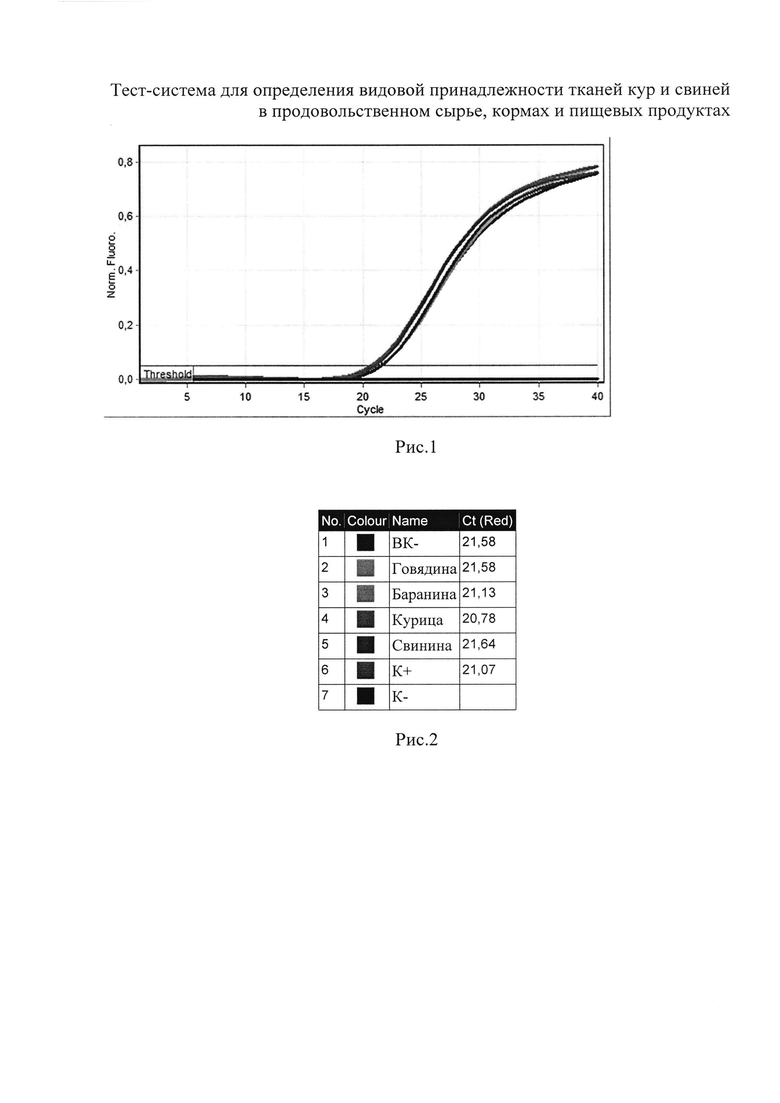
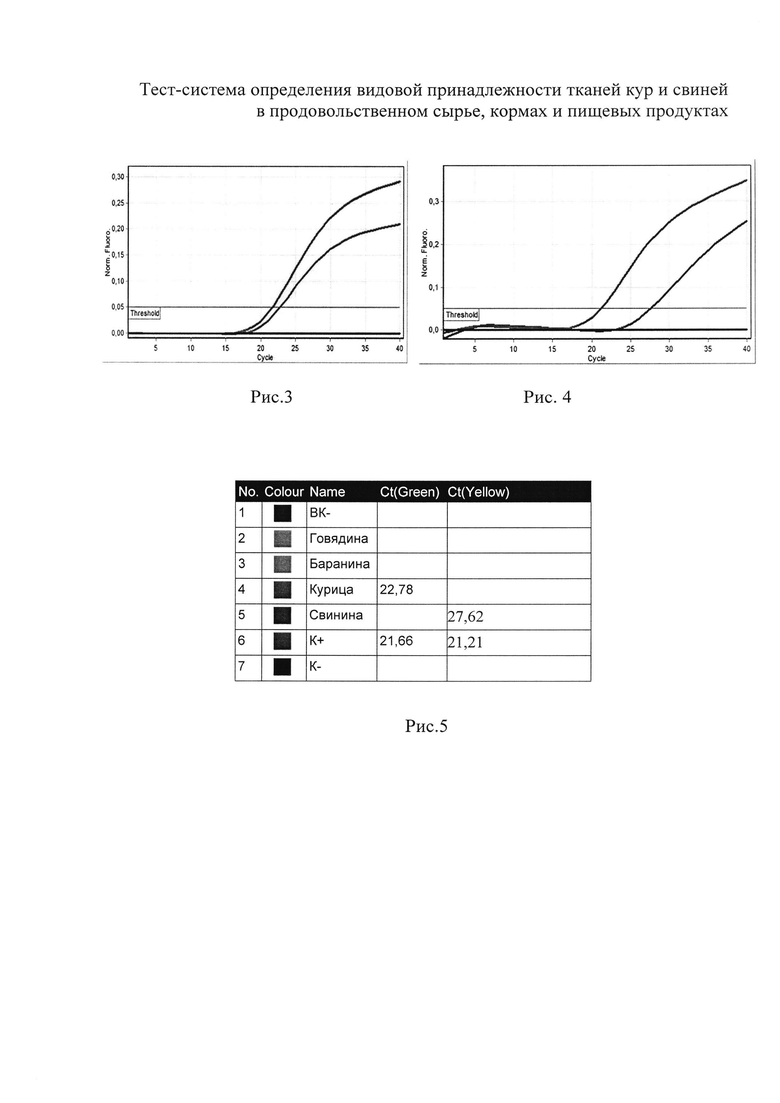
Сущность изобретения поясняется чертежом, где на рисунках 1-5 представлены скриншоты с дисплея прибора Rotor-Gene Q: рис. 1 - представлен график канала Су5 для внутреннего контрольного образца (ВКО); рис. 2 - таблица количественных данных для Cycling A.Red (ВКО); рис. 3 - представлен график канала JOE/Yellow для специфического сигнала для тканей свиньи (Sus scrofa); рис. 4 - представлен график канала FAM/Green - тканей курицы (Gallus gallus); рис. 5 - таблица количественных данных для Cycling A.Yellow (Sus scrofa) и A. Green (Gallus gallus).
Примеры конкретного применения теста-системы для определения видовой принадлежности тканей кур и свиней в продовольственном сырье, кормах и пищевых продуктах.
Для исследования кормов, продовольственного сырья и пищевых продуктов на содержание ДНК в тканях курицы (Gallus gallus) и свиньи (Sus scrofa) для проведения полимеразной цепной реакции с флуоресцентной детекцией с применением термоциклера типа Rotor-Gene Q при соответствующих температурно-временных режимах амплификации используют тест-систему включающий пластиковые флаконы и пробирки, термостабильный фермент Tag-полимеразу, буфер для постановки реакции, смесь четырех дезоксинуклеотидтрифосфатов, специфичных для участка генома ДНК ткани курицы (Gallus gallus) и свиньи (Sus scrofa), при этом для внутреннего контрольного образца используют суспензию бактериофага Т4 с концентрацией 5×103 копий нуклеотидных последовательностей на 1 мкл, а для положительного контрольного образца - смесь содержащую фрагменты геномов тканей курицы (Gallus gallus) и свиньи (Sus scrofa) и бактериофага Т4 взятых в соотношении 1:1:1 со следующими нуклеотидными последовательностями:
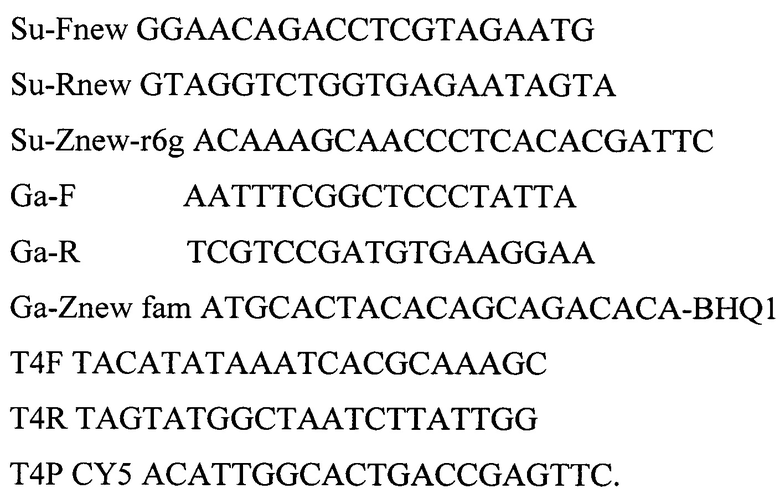
Для повышения точности идентификации мяса для внутреннего контрольного образца используют суспензию бактериофага Т4 с концентрацией 5×103 копий нуклеотидных последовательностей на 1 мкл, если концентрация копий нуклеотидных последовательностей отклоняется в большую или меньшую сторону, то наблюдаются повторности сомнительных образцов. Для положительного контрольного образца используют смесь содержащую фрагменты геномов тканей курицы (Gallus gallus), тканей свиньи (Sus scrofa) и бактериофага Т4 взятых в соотношении 1:1:1 со следующими нуклеотидными последовательностями:
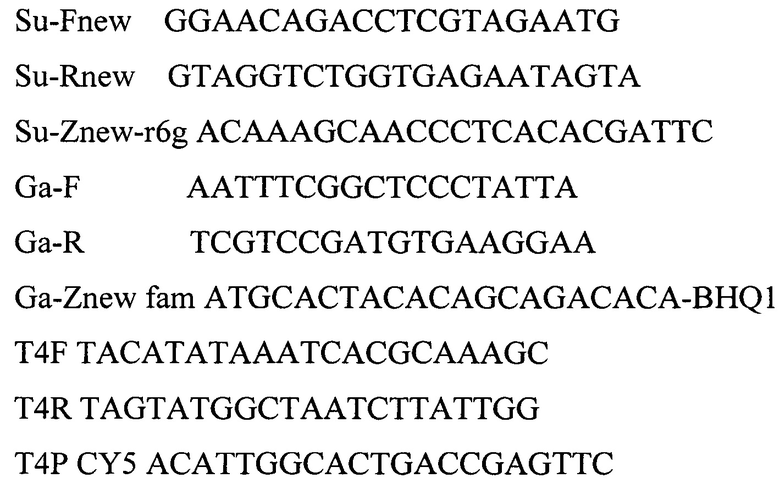
Использование для разных видов контроля различные формы материала бактериофага Т4: суспензии и фрагмента генома со специфическими к нему праймерами и зондом обусловлено тем, что это позволяет контролировать корректное прохождение реакции в каждой пробирки, а также контролируется этап выделения ДНК из образцов.
При конструировании праймеров и зонда основными требованиями были: степень гомологии (комплементарность) с выбранным участком гена; отсутствие самокоплементарных участков внутри олигонуклеотидов и комплементарности друг другу, чтобы не допускать возникновения устойчивых вторичных структур (димеров); близость значений температуры отжига праймеров.
Конструирование специфических праймеров и зонда осуществляли с помощью компьютерных программ на основании анализа нуклеотидных последовательностей референтных штаммов и изолятов, опубликованных на ресурсе GenBank и подбора условий для проведения ПЦР в реальном времени с применением разработанных праймеров и зонда, несущего флуорофор и тушитель, и комплементарного части амплифицируемого со специфическими праймерами фрагмента.
Праймеры, специфичные для тканей курицы (Gallus gallus), тканей свиньи (Sus scrofa) спроектированы с использованием Primer Express Software v3.0 (Applied Biosystems) и исследованы с использованием BLAST, чтобы подтвердить их специфичность. Для детекции продуктов амплификации подобраны олигонуклеотидные флуоресцентно-меченные зонды Su-Znew-r6g (комплементарный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров Su-Fnew и Su-Rnew) и Ga-Znew fam (комплементарный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров Ga-F и Ga-). Зонды были помечены красителями FAM HEX. Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР. Ни одна из выбранных последовательностей не обнаружена в геноме любых видов растений и животных, которые потенциально встречаются вблизи тех, которые определены в кормах и пищевых продуктах.
В качестве внутреннего контроля использовался бактериофаг Т4, имеющий геномную ДНК порядка 169-170 тысяч пар нуклеотидов (Enterobacteria phage Т4Т, complete genome GenBank: HM137666.1). В результате анализа был выбран участок между 400 и 500 нуклеотидами, содержащий уникальные нуклеотидные последовательности, рассчитаны первичные структуры олигонуклеотидных праймеров, фланкирующих выбранный участок генома. Праймеры были спроектированы с использованием Primer Express Software v3.0 (Applied Biosystems) и исследованы с использованием BLAST, чтобы подтвердить их специфичность.
Для детекции продуктов амплификации подобран олигонуклеотидный флуоресцентно-меченный зонд Т4Р, комплементарный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров T4F и T4R. Зонд был помечен красителем Су5. Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР.
Для подтверждения эффективности тест-системы были использованы сухие корма в виде рыбной и мясной муки; сырые и термически обработанные мясные продукты, т.е. мясные полуфабрикаты.
От пробы плотной консистенции отбирают на исследование общую пробу весом 10-50 г. Гранулированную или консервированную продукцию перед исследованием (10-20 г) растирают в ступке до гомогенного состояния.
Лабораторные пробы (20-40 мг) отбирают на исследование в одноразовые микропробирки вместимостью 1,5 мл в двух повторах. Отобранные лабораторные пробы направляют на выделения ДНК.
Исследование проводят с помощью набора реагентов
«ПЦР - СВИНИНА-КУРИЦА - ФАКТОР». Набор состоит из комплекта реагентов для проведения мультиплексной ПЦР (комплект №1) и комплекта контрольных образцов (комплект №2). Набор выпускается в двух вариантах: 1) Для анализа 55 образцов (включая контрольные образцы)
2) Для анализа 110 образцов (включая контрольные образцы).
Наборы используют в соответствии с инструкцией по применению набора реагентов «ПЦР- СВИНИНА-КУРИЦА - ФАКТОР» для определения видовой принадлежности тканей кур и свиней методом полимеразной цепной реакции (ПЦР) с флуоресцентной детекцией в режиме реального времени ТУ 21.10.60-139-51062356-2017, http://www.vetfaktor.ru/.
Состав набора приведен в Таблицах 1 и 2.
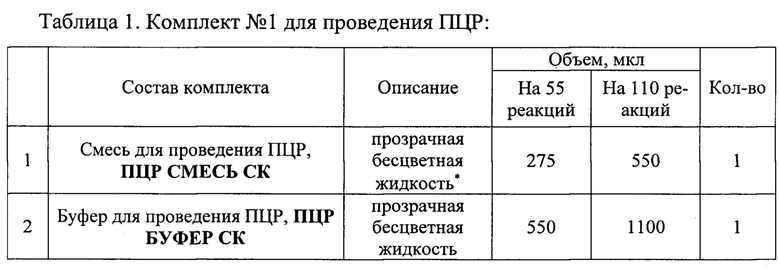
* Возможна легкая опалесценция
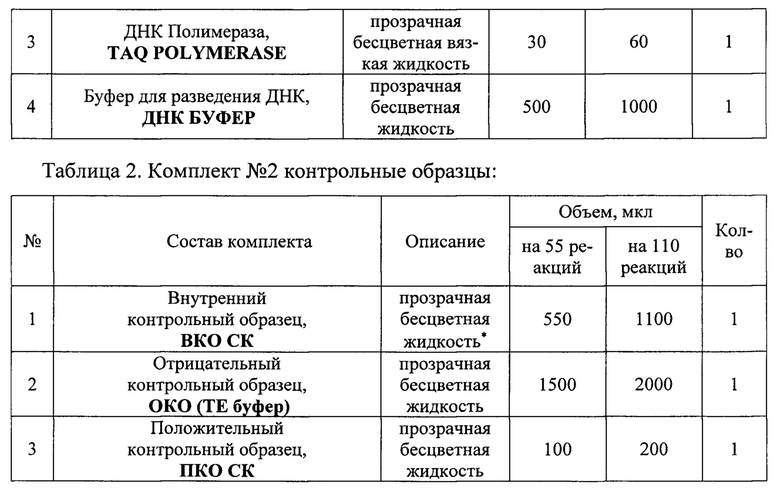
Исследования состоит из трех этапов:
- экстракция нуклеиновая кислота (НК);
- проведение реакции ПЦР РВ;
- учет результатов анализа.
Для экстракции (выделение) НК из исследуемых проб отбирают необходимое количество одноразовых пробирок объемом 1,5 мл, включая отрицательный контроль выделения. Во все пробирки с исследуемыми образцами, включая пробирку для отрицательный контрольный образец (ОКО), вносят по 10 мкл внутренний контрольный образец (ВКО) для ткани курицы и свиньи (СК) в качестве которого используют суспензию бактериофага Т4 с концентрацией 5×103 копий нуклеотидных последовательностей на 1 мкл. Следующий этап это подготовка образцов к проведению ПЦР. Общий объем реакционной смеси - 25 мкл, объем ДНК-пробы - 10 мкл.
Успешное прохождение реакции контролируют использованием положительный контрольный образец (ПКО) СК, ВКО СК и ДНК буфера. В качестве ПКО используют смесь содержащую фрагменты геномов тканей курицы (Gallus gallus), тканей свиньи (Sus scrofa) и бактериофага T4 взятых в соотношении 1:1:1.
В отдельной пробирке смешивают компоненты набора из расчета на каждую реакцию:
5 мкл ПЦР СМЕСЬ СК;
10 мкл ПЦР БУФЕР СК;
0,5 мкл TAQ POLYMERASE
Перемешивают смесь на вортексе и сбрасывают капли кратковременным центрифугированием. Отбирают необходимое количество пробирок для амплификации ДНК исследуемых и контрольных проб. Вносят по 15 мкл приготовленной реакционной смеси. Помещают подготовленные для проведения ПЦР пробирки в ячейки амплификатора и используют программное обеспечение прибора. Далее проводят ПЦР РВ с флуоресцентной детекцией.
Параметры температурно-временного режима амплификации на приборе «Rotor-Gene Q» представлены в таблице 3.

Интерпретация результатов анализа.
Полученные данные - кривые накопления флуоресцентного сигнала анализируются с помощью программного обеспечения используемого прибора для проведения ПЦР в соответствии с инструкцией производителя к прибору и в соответствии с Приложениями 1, 2 и 3 Инструкции.
Учет результатов ПЦР РВ проводится по наличию или отсутствию пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию или отсутствию значения порогового цикла «Ct» для исследуемого образца).
Результат считается достоверным в случае корректного прохождения положительных и отрицательных контролей амплификации и экстракции ДНК в соответствии с таблицей 4.
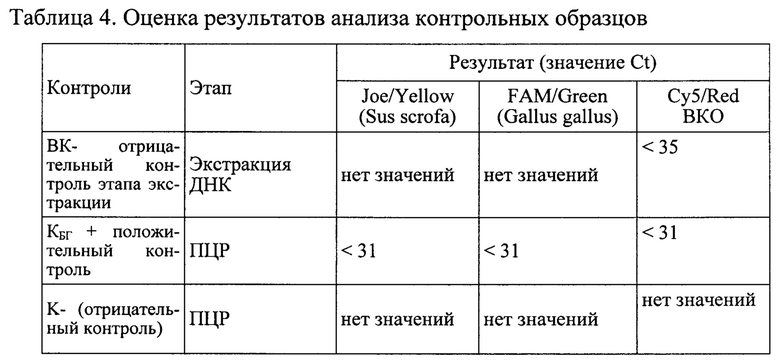
Появление любого значения Ct в таблице 4 результатов для отрицательного контроля этапа экстракции ВК- на каналах FAM/Green и JOE/Yellow и для отрицательного контроля этапа ПЦР К- на любом из каналов свидетельствует о наличии контаминации реактивов или образцов. В этом случае результаты анализа для всех проб считаются недействительными. Требуется повторить анализ всех проб, а также предпринять меры по выявлению и ликвидации источника контаминации.
Образцы, для которых значение Ct по каналу Cy5/Red отсутствует или превышает 35 цикл (и при этом не получен положительный результат на каналах JOE/Yellow и/или FAM/Green) требуют повторного проведения исследования с этапа экстракции ДНК. Задержка в значениях пороговых циклов для исследуемых образцов указывает на присутствие ингибиторов в пробе(ах) или на ошибки при экстракции ДНК или при постановке реакции ПЦР РВ (рис. 1, 2).
В образце обнаружена ДНК ткани свиньи (Sus scrofa), если наблюдается экспоненциальный рост сигнала на канале JOE/Yellow, при этом значения Ct контрольных образцов находятся в пределах нормы (Табл. 4, рис. 3, 5).
В образце обнаружена ДНК ткани курицы (Gallus gallus), если наблюдается экспоненциальный рост сигнала на канале FAM/Green, при этом значения Ct контрольных образцов находятся в пределах нормы (Табл. 4, рис. 4, 5).
Если для исследуемого образца по каналам JOE/Yellow и/или FAM/Green значение Ct определяется позднее 37 цикла при корректном прохождении положительных и отрицательных контролей, образец исследуется повторно с этапа экстракция ДНК. Если при повторной постановке Ct более 37 результат считается отрицательным (содержание целевой ДНК ниже предела обнаружения метода).
Образец считается отрицательным (ДНК ткани свиньи (Sus scrofa) и/или ткани курицы (Gallus gallus) если не определяется значение Ct (не наблюдается рост специфического сигнала) на канале FAM/Green и/или JOE/Yellow при этом значения Ct контрольных образцов находятся в пределах нормы (Табл. 4, рис. 3,4,5), а значение Ct по каналу Cy5/Red менее 35.
Для исследуемых образцов (сухой корм и мясные полуфабрикаты) предел точности содержания ткани курицы и свиньи представлен в таблице 5.
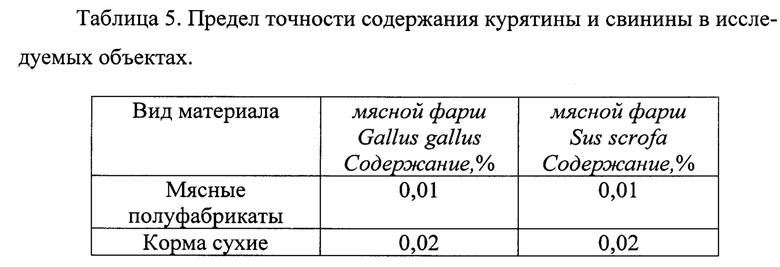
Для доказательства эффективности использования ПЦР с флуоресцентной детекцией в режиме реального времени проводился сравнительный анализ чувствительности заявляемого с прототипом, в котором использовался метод ПЦР с электрофоретической детекцией. Оказалось чувствительность ПЦР с флуоресцентной детекцией при обнаружении примеси тканей курицы и свиныны в кормах в 10 раз выше, чем ПЦР с электрофоретической детекцией.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ определения видовой принадлежности тканей кур и свиней в продовольственном сырье, кормах и пищевых продуктах | 2018 |
|
RU2700479C1 |
| Тест-система для идентификации видовой принадлежности баранины и говядины в продовольственном сырье, кормах и пищевых продуктах | 2018 |
|
RU2702858C1 |
| Способ идентификации ДНК ткани собаки домашней (Canis lupus familiaris) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2728612C1 |
| Способ выявления ДНК ткани домашнего осла (Equus asinus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726248C1 |
| Способ идентификации видовой принадлежности тканей крыс и мышей в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2742952C1 |
| Способ идентификации ДНК ткани ежа обыкновенного (Erinaceus europaeus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726433C1 |
| Способ идентификации ДНК ткани медведя (Ursus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726427C1 |
| Тест-система для выявления ДНК ткани домашнего осла (Equus asinus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726555C1 |
| Способ определения ДНК ткани дятла (Picidae) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2714287C1 |
| Тест-система для идентификации ДНК ткани собаки домашней (Canis lupus familiaris) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2728382C1 |
Изобретение относится к области биотехнологии. Изобретение представляет собой тест-систему для определения видовой принадлежности тканей кур и свиней в продовольственном сырье, кормах и пищевых продуктах в тест-системе для определения видовой принадлежности тканей кур и свиней в продовольственном сырье, кормах и пищевых продуктах, включающем пластиковые флаконы и пробирки, термостабильный фермент Tag-полимеразу, буфер для постановки реакции, смесь четырех дезоксинуклеотидтрифосфатов, специфичных для участка генома ДНК нескольких видов мяса олигонуклеотидных праймеров, зондов, внутренний контрольный образец в виде суспензии бактериофага и положительных контрольных образцов - содержащих фрагменты геномов ДНК нескольких видов мяса, согласно изобретению в качестве генома ДНК мяса используют ткани курицы (Gallus gallus) и свиньи (Sus scrofa), при этом для внутреннего контрольного образца используют суспензию бактериофага Т4 с концентрацией 5×103 копий нуклеотидных последовательностей на 1 мкл, а для положительного контрольного образца – смесь, содержащую фрагменты геномов тканей курицы ({Gallus gallus) и свиньи (Sus scrofa) и бактериофага Т4, взятых в соотношении 1:1:1. Изобретение позволяет расширить функциональные возможности и повысить точность идентификации видовой принадлежности тканей кур свиней. 5 ил., 5 табл.
Тест-система для определения видовой принадлежности тканей кур и свиней в продовольственном сырье, кормах и пищевых продуктах, включающий пластиковые флаконы и пробирки, термостабильный фермент Tag-полимеразу, буфер для постановки реакции, смесь четырех дезоксинуклеотидтрифосфатов, специфичных для участка генома ДНК нескольких видов мяса олигонуклеотидных праймеров, зондов, внутренний контрольный образец в виде суспензии бактериофага и положительных контрольных образцов - содержащих фрагменты геномов ДНК нескольких видов мяса, отличающийся тем, что в качестве генома ДНК мяса используют ткани курицы (Gallus gallus) и свиньи (Sus scrofa), при этом для внутреннего контрольного образца используют суспензию бактериофага Т4 с концентрацией 5×103 копий нуклеотидных последовательностей на 1 мкл, а для положительного контрольного образца – смесь, содержащую фрагменты геномов тканей курицы (Gallus gallus) и свиньи (Sus scrofa) и бактериофага Т4, взятых в соотношении 1:1:1 со следующими нуклеотидными последовательностями:
Su-Fnew GGAACAGACCTCGTAGAATG
Su-Rnew GTAGGTCTGGTGAGAATAGTA
Su-Znew-r6g ACAAAGCAACCCTCACACGATTC
Ga-F AATTTCGGCTCCCTATTA
Ga-R TCGTCCGATGTGAAGGAA
Ga-Znew fam ATGCACTACACAGCAGACACA-BHQ1
T4F TACATATAAATCACGCAAAGC
T4R TAGTATGGCTAATCTTATTGG
T4P CY5 ACATTGGCACTGACCGAGTTC.
| СОРОКИНА М.Ю., Разработка тест-системы для определения видовой принадлежности мясных ингредиентов в кормах методом полимеразной цепной реакции, автореферат диссертации, Москва, 2004, весь документ | |||
| T | |||
| Kozlova, Safety and quality control raw meat and meat products in Russia, Russian Journal of Agricultural and Socio-Economic Science, N 5 (5), 2012, p.33-38, найдено в Интернет 08.09.2019, адрес сайта https:docviewer.yandex.ru/view/0/?*. |
Авторы
Даты
2019-09-17—Публикация
2018-10-01—Подача