Изобретение относится к сельскому хозяйству и биотехнологии и может быть использовано в генетике и селекции зерновых культур.
В настоящее время селекция ячменя направлена на получение сортов с повышенным уровнем урожайности. Одним из факторов повышения продуктивности и качества зерна является выведение сортов, устойчивых к различным болезням. Темно-бурая пятнистость относится к основным болезням, поражающим ячмень в условиях Америки, Европы и России (1). Болезнь вызывается патогеном Cochliobolus sativus и эпифитотии ее влекут за собой до 40% потерь урожая. Создание сортов с генетической устойчивостью к данному патогену является одним из элементов защиты культуры ячменя.
Локусы сопротивляемости в данному патогену начали определять с 1995 года и в настоящее время они обнаружены на всех хромосомах ячменя (2). Возбудитель опасен для районов с теплым, влажным климатом. Одним из ключевых факторов распространения болезни является длительный период (16 и более часов), когда листья увлажнены, то есть находятся в постоянном соприкосновении с влагой. Было установлено, что наиболее высокая устойчивость к темно-бурой пятнистости встречается у сортов из коллекции NSGC (National Small Grain Collection), происходящих из северной части США, а умеренная устойчивость к темно-бурой пятнистости наблюдается у взрослых растений, происходящих из Восточной Азии (3). Используя образец CI-7117-77 из североамериканской коллекции, была выведена линия NDB112, устойчивая к темно-бурой пятнистости (4). Прочная стойкость этой линии использовалась в ячмене более 40 лет. Устойчивые сорта Cree, Manker, Morex и Robust были разработаны и распространены в производстве ячменя в северных штатах США в 1990 году (5). Однако в настоящее время появились более вирулентные штаммы патогена, преодолевающие устойчивость, обеспечиваемую генами от линии NDB112. В настоящее время известно о 9 сортах ярового ячменя, созданных в селекционных центрах Сибирского Федерального округа, обладающих устойчивостью как минимум к 2 различным изолятам С. sativus (2).
Разработка сортов ячменя, устойчивых к темно-бурой пятнистости, актуальна для многих регионов Российской Федерации, где вред, причиняемый этим заболеванием, достигает (в среднем) 10-30% (6).
Классическая селекция основана на поиске и размножении растений, содержащих в себе гены хозяйственно ценных локусов. Также очень важно для селекции количество таких признаков в одном генотипе и создание на их основе линий, обладающих всем комплексом необходимых состояний признаков. Затем путем нескольких последовательных скрещиваний и бэккросов создаются чистые линии, гомозиготные по генам, контролирующим развитие искомых признаков. Следующий шаг - это проверка полученных линий растений в полевых условиях. Получение сортов с заданными свойствами методами классической селекции обычно занимает более десяти лет и является дорогостоящим процессом, поскольку хозяйственно-ценные признаки обычно контролируются большим количеством генов, а потому для нахождения растений с заданными признаками требуется мониторинг тысяч растений в популяциях после каждого скрещивания (7). В конце прошлого столетия традиционные методы селекции были дополнены использованием ДНК-маркеров.
С помощью ДНК-маркеров возможно контролировать аллельные варианты генов, ответственных за хозяйственно ценные признаки. ДНК-маркеры, ассоциированные с хозяйственно-ценным признаком, способствуют более динамичному селекционному процессу, поскольку более просты в использовании, чем, например, фенотипический анализ и более экономичны, чем, например, оценка фитопатологической устойчивости в лаборатории. Время и ресурсы экономит также то, что селекцию с использованием ДНК-маркеров можно проводить на стадии проростков, и отбирать нужные генотипы среди селекционных гибридов (8).
ДНК-маркеры позволяют на любой стадии селекционного процесса осуществить мониторинг, что позволяет выбирать формы удачным сочетанием генов, снижая, таким образом, число растений, которые будут анализироваться в последующем поколении.
Основным недостатком известных ДНК-маркеров является то, что каждый ДНК-маркер сцеплен только с одним локусом. Поскольку гены могут находиться под полигенным контролем, то может потребоваться большое количество маркеров, однако они могут быть меньше, чем количество локусов, участвующих в развитии. Кроме этого, локус, сцепленный с маркером, может проявлять себя по-разному при разном воздействии окружающей среды в зависимости от генетического фона и может не подходить для некоторых популяций (9).
Методом, который учитывает недостатки единичных ДНК-маркеров, с развитием высокопроизводительных технологий секвенирования, стала геномная селекция (10). Принцип геномной селекции заключается в определении маркером количественных признаков с использованием полногеномного генотипирования. Для этого маркеры исследуются на одной популяции, и их валидация проходит на другой. Таким образом, оцениваются все вероятные ошибки (эффекты), связанные с разработкой маркера. В итоге с использованием данного метода возможно комплексное изучение сложных признаков с более высокой точностью.
Известны SSR маркеры (simple sequence repeats, простые повторяющиеся последовательности), разработанные и использующиеся в селекционных программах Австралии: AWBMS22, Bamg183, Bmac167, Вmас30, Вmас96, Bmag353, Bmag359, Bmag375, Bmag692, Ebmac824, Ebmac827, GMS3 и HVM3, которые используются в селекции ячменя. Однако среди них нет маркера, ассоциированного с болезнью темно-бурая пятнистость (11). Известные SSR маркеры не определяют у сортов ячменя устойчивость к темно-бурой пятнистости.
С 1996 года начали разрабатывать ДНК-маркеры класса RAPD (random amplified polymorphic DNA, случайно модифицированная полиморфная ДНК) на сопротивляемость к Cochliobolus sativus. Методика создана на основе амплификации случайных сегментов ДНК с одиночными праймерами произвольной нуклеотидной последовательности. Эффективным методом, определяющим ассоциацию между маркером RAPD и признаком, является анализ ДНК от контрастных форм растений.
Известны ДНК-маркеры класса RAPD: UBC391, UBC428M, UBC428F и UBC335, определяющие устойчивость одновременно как к корневым гнилям, так и к темно-бурой пятнистости. Эти болезни вызываются патогеном Cochliobolus sativus, однако фенотипические эффекты данных ассоциаций были небольшими.
Недостатками известных RAPD маркеров является то, что эти маркеры обладают средним уровнем видового полиморфизма и нет возможности автоматизации процесса анализа. Новые технологии на основе высокопроизводительного ДНК секвенирования позволяют проводить анализ по десяткам тысяч однонуклеотидных полиморфизмов (single nucleotide polymorphism, SNP). Соответственно, получение SNP-маркера позволит автоматизировать процесс и кроме того данные маркеры обладают высоким уровнем внутривидового полиморфизма.
Наиболее близким к заявляемому маркеру - прототипом, является ДНК-маркер UBC335, представляющий собой единичный короткий праймер, имеющий нуклеотидную последовательность TGGACCACCC, предназначенный для селекции сортов ярового ячменя, устойчивых к темно-бурой пятнистости. Данный маркер используется с применением технологии RAPD, позволяет использовать простое оборудование, однако не дает данных о местоположении маркера на генетическом материале, что не позволяет быстро и точно отобрать устойчивые сорта ячменя (12).
Задачей предлагаемого изобретения является выявление ДНК-маркера, позволяющего быстро и точно отбирать растения ярового ячменя, устойчивые к патогену Cochliolobus sativus, вызывающему у ячменя темно-бурую пятнистость.
Техническим результатом изобретения является повышение точности и сокращение времени отбора сортов ярового ячменя, устойчивых к темно-бурой пятнистости.
Поставленная задача достигается предлагаемым ДНК-маркером, представляющим собой специфические олигонуклеотидные праймеры, позволяющие амплифицировать последовательность ДНК ячменя и выявлять аллель на хромосоме 3, ассоциированный с устойчивостью к патогену С. sativus ячменя, имеющие нуклеотидные последовательности:
SEQ ID NO 1: 5'-CCTTTCCTGATTCGTGCTCTTTGG-3'
SEQ ID NO 2: 5'-CCTGTAAAAAGACTTATATTTAGT-3'.
Для выявления маркера при помощи ассоциативного анализа был проведен полногеномный анализ ассоциаций (GWAS, genome wide association) 94 сортов и линий ярового ячменя на устойчивость к темно-бурой пятнистости. Была проведена фитопатологическая оценка проростков коллекции в лаборатории ВИЗР на устойчивость к темно-бурой пятнистости. ДНК данных сортов были генотипированы с помощью чипа 50K Illumine Infinum iSelect (компания Trait Genetics, Germany), который содержал 44040 маркеров.
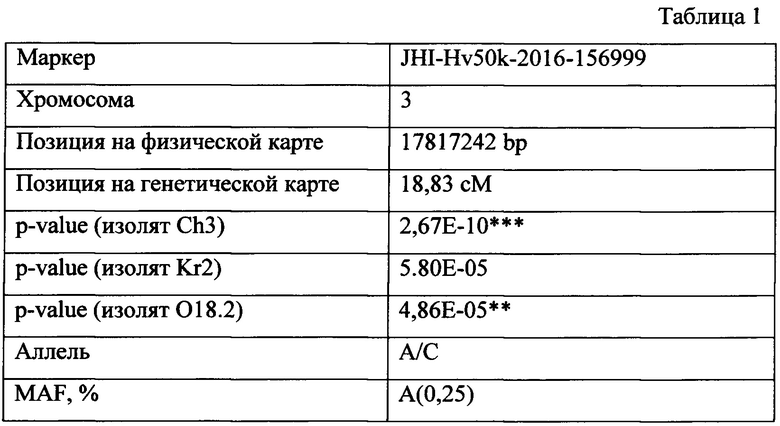
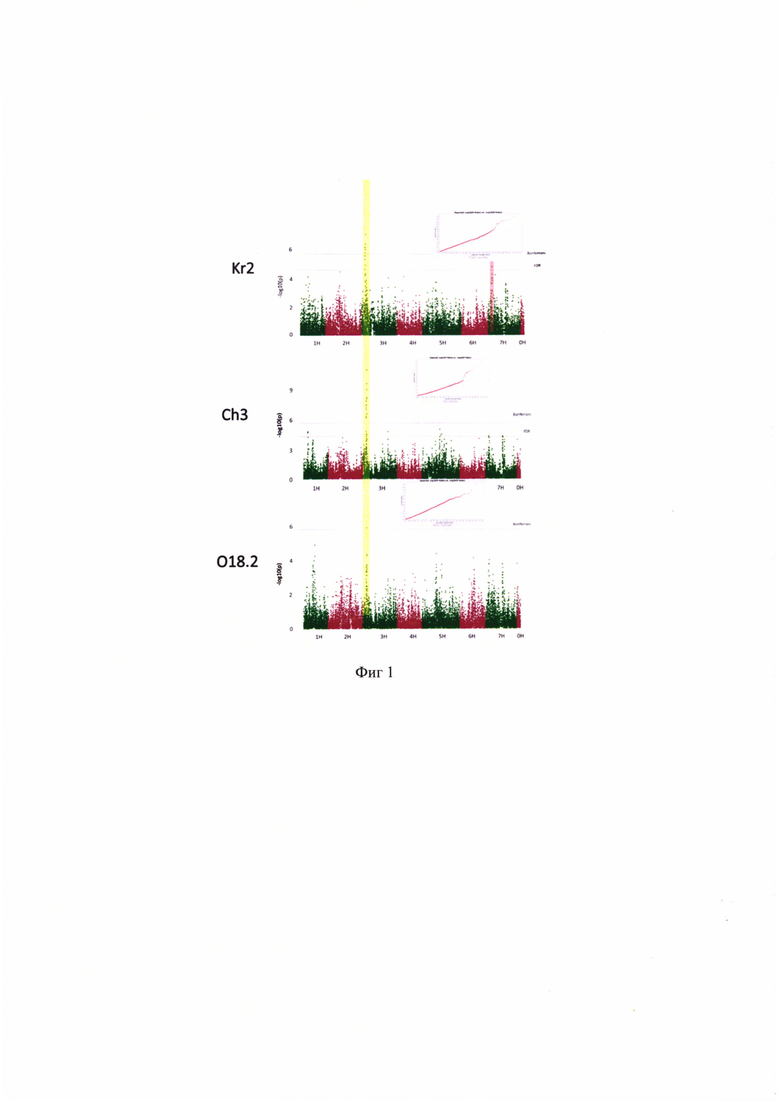
Устойчивость коллекции к патогену С. sativus исследовали с помощью трех изолятов (Ch3, Kr2 и O18.2). Ассоциативный анализ проводился с использованием обобщенной линейной модели (general linear model, GLM) с учетом структуры популяции. Для определения значимости ассоциаций было использовано два критерия: (1) поправка Бонферрони, которая была определена как деление статистического уровня значимости (0.05) на общее число испытаний, в нашем случае на количество маркеров (27,319) и составило 1,83*10-6, и (2) критерий Бенджамини-Хохберга (13) (false discovery rate, FDR), который рассчитывался индивидуально.
На фиг. 1 показано, что с помощью модели GLM+PCA (обобщенная модель с учетом популяционной структуры) выявлен локус для всех изолятов патогена С. sativus на хромосоме 3.
В итоге был отобран маркер с условным названием JHI-Hv50k-2016-156999. Данные ассоциативного анализа по выбранному маркеру представлены в Таблице 1, где *** - уровень Бонферрони, ** -уровень FDR, * - уровень "Suggestive". Из таблицы 1 видно, что заявляемый маркер показал себя значимым к двум изолятам (Ch3 и O18.2), а для третьего он находился на уровне Suggestive, то есть значение p-value было значимым, но не превышало порогового значения. Это все позволило рассматривать его как биологический ДНК-маркер, позволяющий амплифицировать последовательность ДНК ячменя и выявлять аллель на хромосоме 3, ассоциированный с устойчивостью к патогену С. sativus ячменя.
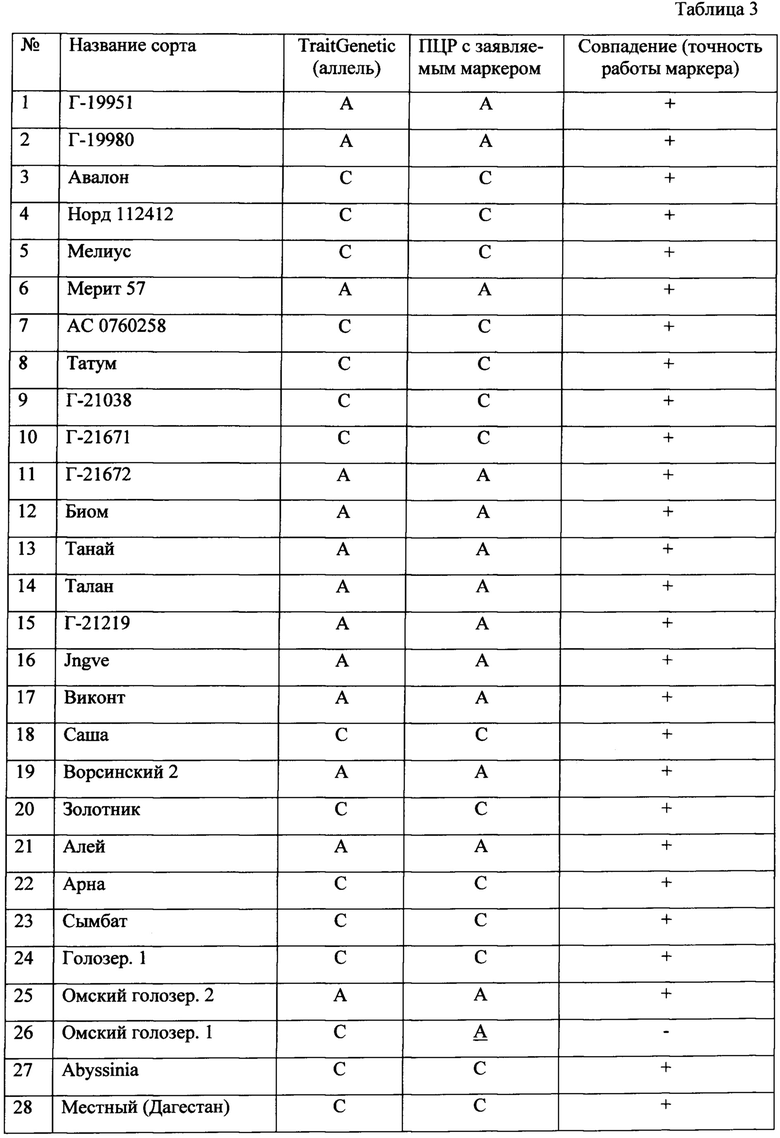
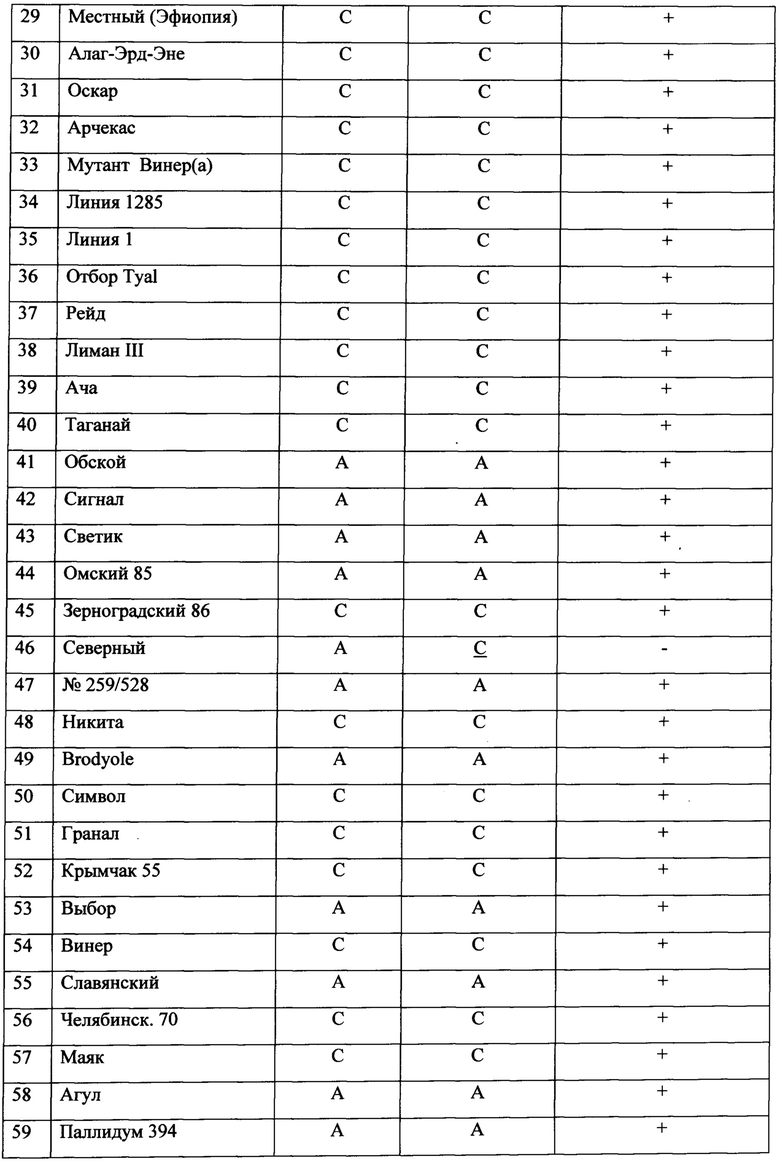
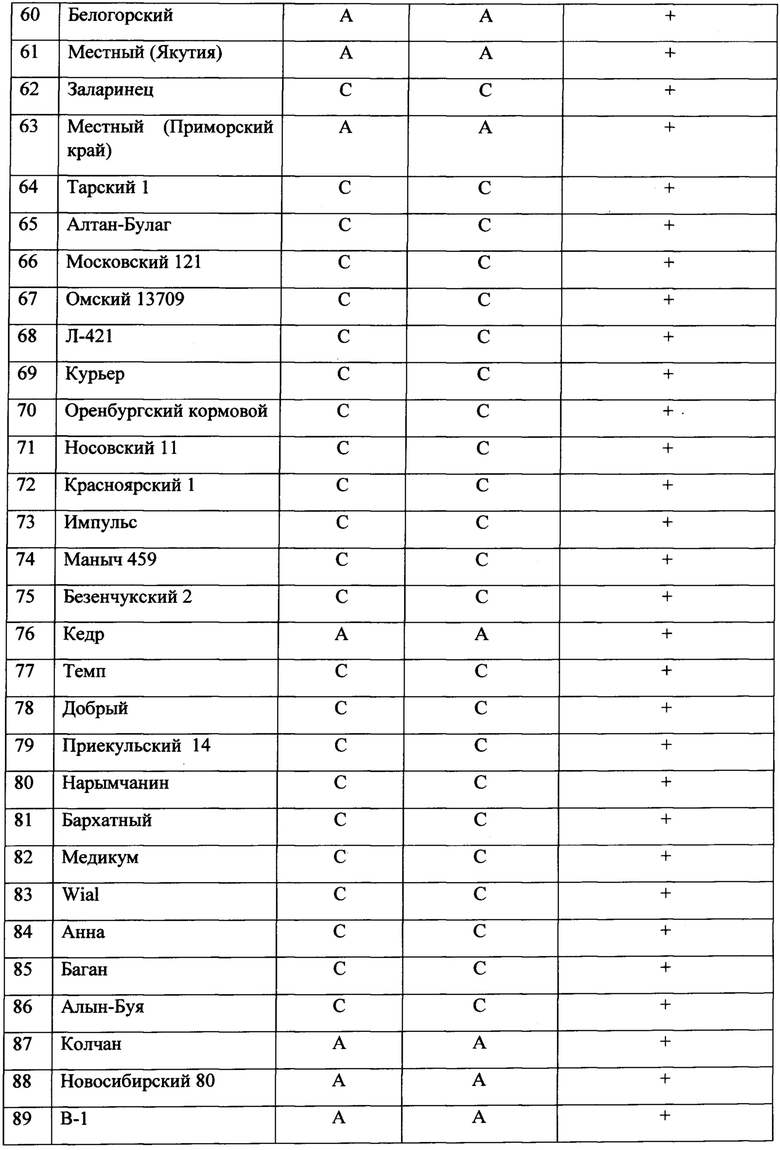
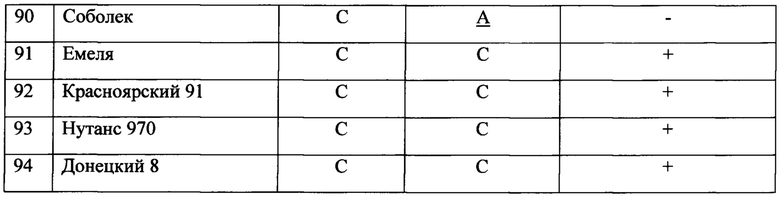
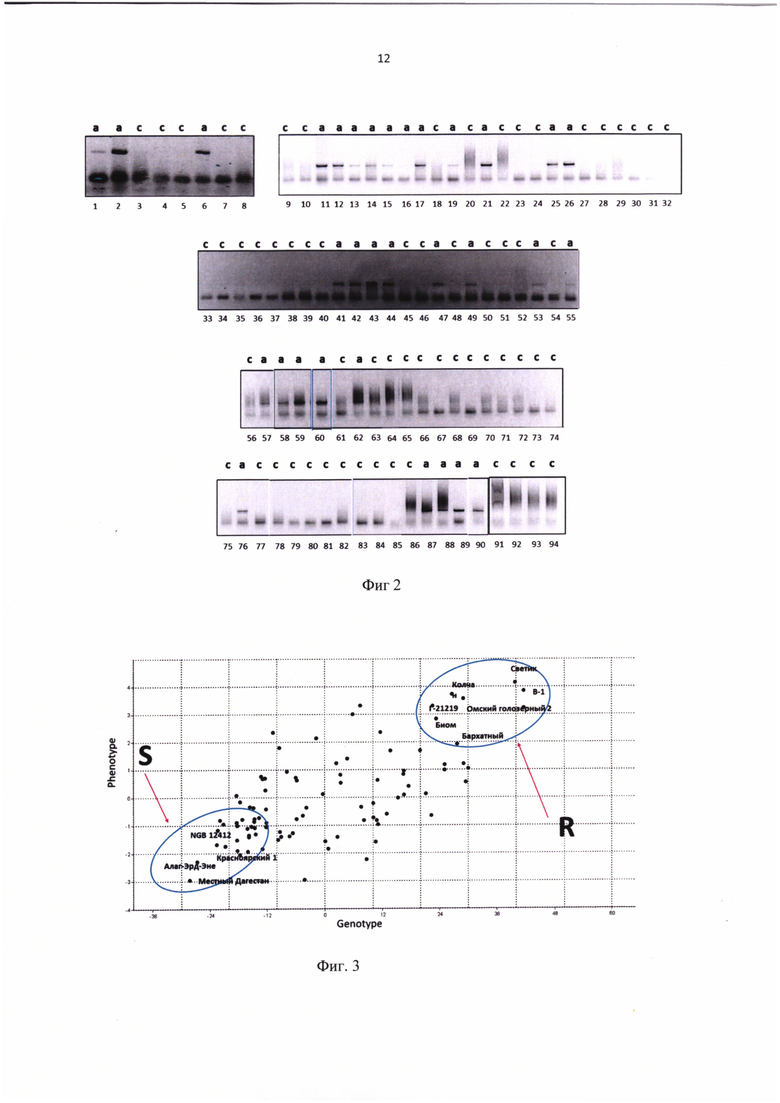
Были подобраны праймеры, с помощью которых была проведена полимеразная цепная реакция (ПЦР) 94 сортов. Условия ПЦР представлены в таблице 2. Результаты для ПЦР-анализа маркера с геномной ДНК исследуемой коллекции приведены на фиг 2. На фиг. 2 видны различия на электрофорезе в случае присутствия С аллеля и в случае присутствия А аллеля. Наличие фрагмента ДНК, длиной ~200 bp указывает на присутствие аллеля А в геноме сорта ячменя, а отсутствие фрагмента ДНК на электрофорезе указывает на присутствие аллеля С. Также эти сорта были исследованы с помощью ДНК-маркера JHI-Hv50k-2016-156999. Из 94 образцов 3 варианта не соответствуют таблице генотипирования, полученной в Trait Genetics (выделены подчеркиванием). Геномная ДНК всех 94 сортов была проверена с помощью предлагаемого ДНК-маркера JHI-Hv50k-2016-156999. В таблице 3 представлены результаты сравнения нуклеотидов, полученных методом генотипирования в Trait Genomics и нуклеотидов, полученных с помощью предлагаемого ДНК-маркера JHI-Hv50k-2016-156999. Данные совпали в 98% (нуклеотиды по 92 сортам из 94 совпали).
Пример 1.
Для идентификации маркера, ассоциированного с устойчивостью, собирают растительный материал - по 5-8 листьев с разных растений ячменя одного сорта. Наилучшим материалом являются молодые листья растения.
ДНК было выделено при использовании набора DNeasy Plant Mini (Qiagen, CA, USA) согласно протоколу производителя. Концентрация и чистота тестируемых образцов была определена с помощью гель электрофореза и с использованием аппарата Nanodrop 2000.
ДНК всех 94 сортов была генотипирована с использованием SNP-чипа 50k Illumina Infinum iSelect в компании TraitGenetics GmbH (Gatersleben, Германия).
К фенотипическим данным (фитопатологическая оценка болезни темно-бурая пятнистость) и генотипическим данным полученным из Trait Genetics был применен PLS анализ. В результате была построена корреляционная линия между данными по фенотипу и генотипу. При рассмотрении устойчивости сортов к различным изолятам патогена на стадии проростков и взрослых растений оказалось, что сорта, проявившие в основном устойчивость, собираются на одном конце корреляционной кривой (8 сортов: Биом, Г-21219, Омский голозерный 2, Светик, Северный, Бархатный, Колчан и В-1). А сорта, восприимчивые к патогену на всех испытаниях (29 сортов: Abyssinia, Алаг-Эрд-Эне, Арна, Авалон, Голозерный 1, Ильмен, Jngve, Красноярский 1, Курьер, Линия 1, Мелиус, Мерит 57, Местный (Дагестан), Местный (Эфиопия), Московский 121, Мутант 68, NGB 122412, Носовский 11, Нутанс 970, Омский голозерный 1, Оренбургский кормовой, Приекульский 14, Рейд, Саша, Сымбат, Таганай, Татум, Темп, Виконт и Мутант Винера) - на другом конце корреляционной прямой. Как результат, были выявлены два кластера: устойчивых и восприимчивых сортов к темно-бурой пятнистости, что показано на фиг 3. Устойчивый кластер обозначен буквой R, восприимчивый кластер обозначен буквой S.
С использованием ДНК-маркера JHI-Hv50k-2016-156999 в 6 сортах из 8 устойчивых обнаружено присутствие аллеля А, что соответствует 75% и в 26 сортах из 29 восприимчивых обнаружено присутствие аллеля С (таблица 3), что соответствует 90%. Соответственно, точность данного маркера составляет не менее 75%.
Пример 2.
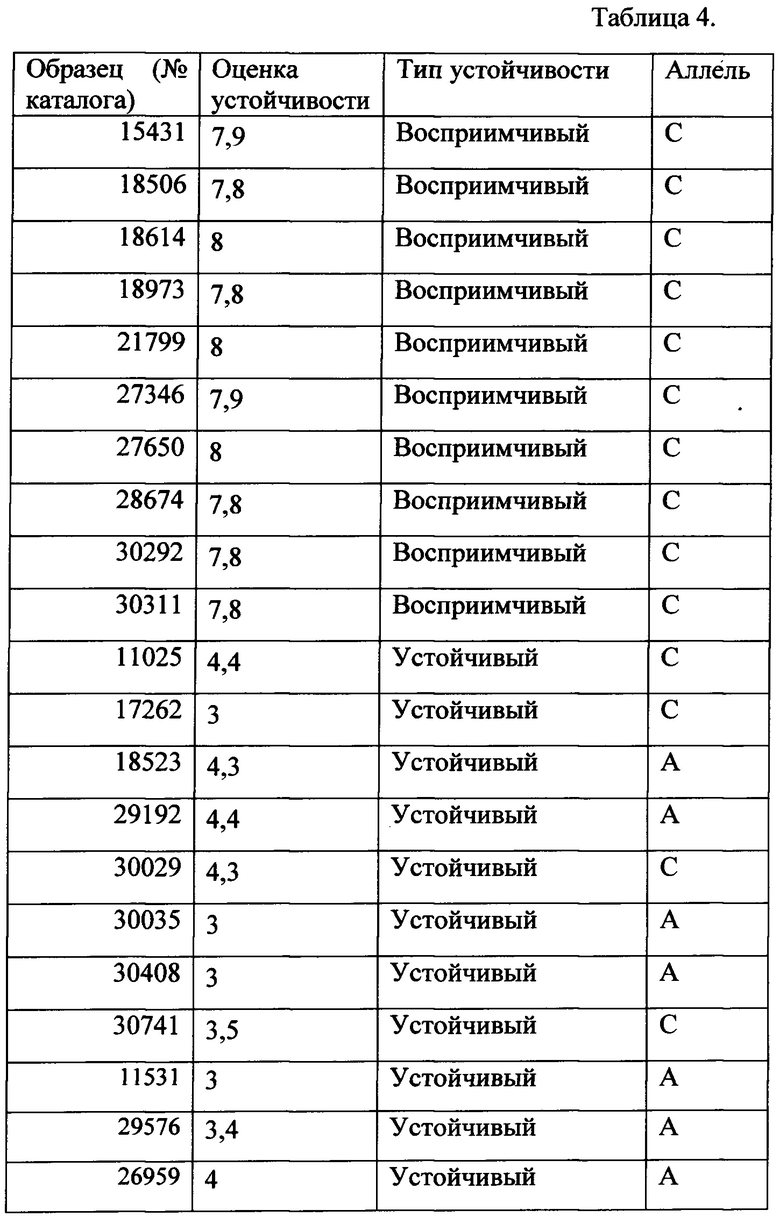
Валидация маркера была проведена на независимой выборке, состоявшей из 22 сортов, описания которых представлены в таблице 4. Выборку составили неродственные сорта из коллекции ВИЗР. 11 сортов были восприимчивые к патогену и 11 сортов были устойчивыми к патогену по маркеру.
Выделение ДНК из проростков.
Проростки нужных сортов выращивают в чашке Петри в течение 5-6 дней. Далее 2-3 кусочка листа (примерно 0,5×2 см) помещают в пробирку Eppendorf объемом 1,5 мл. Добавляют 200 мкл свежего буфера для экстракции (100 mM Tris-HCl, рН 7,5-8,0, 500 mM NaCl, 50 mM EDTA, 1,25% SDS, 0,38% Na2S2O5), нагретого до 600С. Листья тщательно измельчают на гомогенизаторе. Далее добавляют еще 500 мкл экстракционного буфера, перемешивают и инкубируют на водяной бане при 60°С в течение 30-40 мин. После добавляют 700 мкл смеси хлороформ-изоамилового спирта (24:1) и после перемешивания центрифугируют 25 мин при 10000-12000 об/мин при комнатной температуре. Верхнюю фракцию ~500 мкл отбирают, к верхней фракции добавляют 1,4 мл 96% холодного этанола, слегка перемешивают и центрифугируют 10000-12000 об/мин. Спирт сливают и осадок промывают 70% этанолом не менее 3 раз, просушивают и растворяют в 20-25 мкл 50 мкл бидистиллированной стерильной воды.
Оценка качества и количества экстрагированной ДНК.
Оценку качества и количества экстрагированной ДНК производят путем проведения аналитического электрофореза. После определения концентрации и чистоты ДНК концентрацию ДНК в пробах стандартизируют путем разведения в стерильной бидистиллированной воде до 100 мкг/мкл.
Полимеразная цепная реакция (ПЦР).
ПЦР проводили в реакционной смеси объемом 20 мкл, содержащей 5 мкл ДНК, 1.8 мМ MgCl2 (или 1.5, 1.2 мМ MgCl2), 6 мкл H2О, по 0.2 мМ каждого дНТФ, по 1 мкМ прямого и обратного специфичных праймеров, 1 ед. ДНК-полимеразы Taq, в термоциклере БИС в режиме: SSR55 программа: преденатурация - 2 минуты при 94°С; денатурация - 1 минута при 94°С; отжиг матрицы с праймерами - 1 минута при 50, 55 или 60°С; полимеризация - 2 минуты при 72°С; число циклов - 45 (или 35); достраивание ПЦР-фрагментов - 15 минут при 72°С.
Результаты ПЦР представлены на фиг. 4 и показывают, что во всех 10 восприимчивых сортах присутствует аллель С и в 7 из 11 (63%) устойчивых сортов присутствует аллель А. Полученные данные доказывают, что заявляемый ДНК-маркер позволяет с высокой точностью идентифицировать растения, обладающие устойчивостью к темно-бурой пятнистости.
Предлагаемый ДНК-маркер позволяет вести отбор растений, содержащих ген устойчивости к патогену Cochliobolus sativus без проведения полевых тестов и сокращает время создания сортов ярового ячменя, устойчивых к темно-бурой пятнистости.
Список литературы.
1. Novakazi F, Afanasenko O.S., Lashina N.M., Platz G.J., Snowdon R., Loskutov I.B., Ordon F., "Genome-wide association studies in a barley (Hordeum vulgare) diversity set reveal a limited number of loci for resistance to spot blotch (Bipolaris sorokiniana)," Plant Breed., no. October, pp. 1-15, 2019, doi: 10.1111/pbr.12792.
2. Bykova I.V., Lashina N.M., Efimov V.M., Afanasenko O.S., Khlestkina E.K., "Identification of 50 K Illumina-chip SNPs associated with resistance to spot blotch in barley," BMC Plant Biol, vol. 17, no. 2, p.250, 2017, doi: 10.1186/s12870-017-1198-9.
3. Bonman J.M., Bockelman H.E., Jackson L.F., Steffenson B.J., "Disease and insect resistance in cultivated barley accessions from the USDA National Small Grains Collection," Crop Set, vol. 45, no. 4, pp. 1271-1280, 2005, doi: 10.2135/cropsci2004.0546.
4. Wilcoxson R.D., Rasmusson D.C., Miles M.R., "Development of barley resistant to spot blotch and genetics of resistance," Plant Dis., vol. 74, pp. 207-210, 1990.
5. Roy J.K., Smith K.P., Muehlbauer G.J., Chao S., Close T.J., Steffenson B.J., "Association mapping of spot blotch resistance in wild barley," Mol. Breed., vol. 26, no. 2, pp. 243-256, 2010, doi: 10.1007/s11032-010-9402-8.
6. Radyukevich T.N., Bondareva L.M., Kartasheva L.I., "Assessment of new collection samples of barley accoding to commercially valuable traits in the conditions of northwest of Russia," Perm Agrar. J., vol. 4, no. 24, pp. 76-82, 2018, doi: 10.1134/S1064229316050033.16.
7. Witcombe J.R. and Virk D.S., "Number of crosses and population size for participatory and classical plant breeding," Euphytica, vol. 122, no. 3, pp. 451-462, 2001, doi: 10.1023/A:1017524122821.
8. Morris M., Dreher K., Ribaut J.M., Khairallah M., "Money matters (II): Costs of maize inbred line conversion schemes at CIMMYT using conventional and marker-assisted selection," Mol. Breed, vol. 11, no. 3, pp. 235-247, 2003, doi: 10.1023/A:1022872604743.
9. Liao C.Y., Wu P., Ни В., Yi K.K., "Effects of genetic background and environment on QTLs and epistasis for rice (Oryza sativa L.) panicle number," Theor. Appl. Genet., vol. 103, no. 1, pp. 104-111,2001, doi: 10.1007/s001220000528.
10. Meuwissen T.H.E., Hayes B.J., Goddard M.E., "Prediction of total genetic value using genome-wide dense marker maps," Genetics, vol. 157, no. 4, pp. 1819-1829, 2001.
11. Karakousis A., Barr A.R., Chalmers K.J., Ablett G.A., Holton T.A., Henry R.J., Lim P., Langridge P., "Potential of SSR markers for plant breeding and variety identification in Australian barley germplasm," Aust. J. Agric. Res., vol. 54, no. 11-12, pp. 1197-1210, 2003, doi: 10.1071/ar02178.
12. Kutcher H.R., Bailey K.L., Rossnagel B.G., Legge W.G., "Identification of RAPD markers for common root rot and spot blotch (Cochliobolus sativus) resistance in barley," Genome, vol. 39, no. 1, pp. 206-215,1996, doi: 10.1139/g96-027.
13. Benjamini Y. and Hochberg Y., "Controlling the False Discovery Rate: A Practical and Powerful Approach to Multiple Testing," J.R. Stat. Soc. Ser. B, vol. 57, no. 1, pp. 289-300,1995, doi: 10.2307/2346101.

Перечень последовательностей
<110> Федеральное государственное бюджетное научное учреждение «Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения Российской академии наук» (ИЦиГ СО РАН)
<120> ДНК-маркер для селекции сортов ярового ячменя, устойчивых к темно-бурой пятнистости
<160> Номер SEQ ID NO 1
<210> 1
<211> 24
<212> ДНК
<213> Искусственная последовательность
<223> Олигонуклеотидный праймер
<400> 1
CCTTTCCTGATTCGTGCTCTTTGG
<160> Номер SEQ ID NO 2
<210> 2
<211> 24
<212> ДНК
<213> Искусственная последовательность
<223> Олигонуклеотидный праймер
<400> 2
CCTGTAAAAAGACTTATATTTAGT
| название | год | авторы | номер документа |
|---|---|---|---|
| Набор ДНК-маркеров для отбора ячменя с повышенным содержанием антоцианов в перикарпе зерновки | 2022 |
|
RU2797678C1 |
| Способ отбора линий яровой мягкой пшеницы с повышенным содержанием антоцианов в зерне | 2021 |
|
RU2762804C1 |
| Внутригенный ДНК-маркер для отбора пшеницы с повышенным содержанием антоцианов в перикарпе зерновки | 2021 |
|
RU2774444C1 |
| CAPS-маркер для отбора гибридов ячменя, не накапливающих проантоцианидины в зерне | 2023 |
|
RU2809369C1 |
| СПОСОБ СОЗДАНИЯ ЛИНИЙ ОЗИМОЙ МЯГКОЙ ПШЕНИЦЫ С КОМПЛЕКСНОЙ УСТОЙЧИВОСТЬЮ К БУРОЙ И СТЕБЛЕВОЙ РЖАВЧИНЕ И МУЧНИСТОЙ РОСЕ | 2015 |
|
RU2598275C1 |
| Способ получения линий яровой мягкой пшеницы с укороченным сроком колошения | 2019 |
|
RU2710729C1 |
| РАСТЕНИЯ BRASSICA OLERACEA С УСТОЙЧИВОСТЬЮ К MYCOSPHAERELLA BRASSICICOLA | 2007 |
|
RU2438298C2 |
| РАСТЕНИЯ ОГУРЦА, УСТОЙЧИВЫЕ К ЗАБОЛЕВАНИЯМ | 2006 |
|
RU2418405C2 |
| МОЛЕКУЛЯРНЫЙ МАРКЕР FR_ER1 И ЕГО ИСПОЛЬЗОВАНИЕ ДЛЯ СЕЛЕКЦИИ НОВЫХ СОРТОВ ГОРОХА, УСТОЙЧИВЫХ К МУЧНИСТОЙ РОСЕ | 2013 |
|
RU2593691C2 |
| СПОСОБ ПОВЫШЕНИЯ УСТОЙЧИВОСТИ РАСТЕНИЙ ЯРОВОГО ЯЧМЕНЯ К ПАТОГЕННОЙ ИНФЕКЦИИ | 2021 |
|
RU2771487C1 |

Изобретение относится к области биотехнологии. Изобретение представляет собой ДНК-маркер для селекции сортов ярового ячменя, устойчивых к темно-бурой пятнистости, представляющий собой пару олигонуклеотидных праймеров, имеющих нуклеотидную последовательность SEQ ID NO 1: 5'-CCTTTCCTGATTCGTGCTCTTTGG-3' и SEQ ID NO 2: 5'-CCTGTAAAAAGACTTATATTTAGT-3'. Изобретение позволяет повысить точность и сократить время отбора сортов ярового ячменя, устойчивых к темно-бурой пятнистости. 4 ил., 4 табл., 2 пр.
ДНК-маркер для селекции сортов ярового ячменя, устойчивых к темно-бурой пятнистости, представляющий собой олигонуклеотидные праймеры, имеющие нуклеотидную последовательность SEQ ID NO 1: 5'-CCTTTCCTGATTCGTGCTCTTTGG-3' и SEQ ID NO 2: 5'-CCTGTAAAAAGACTTATATTTAGT-3'.
| KUTCHER H.R | |||
| et al | |||
| Identification of RAPD markers for common root rot and spot blotch (Cochliobolus sativus) resistance in barley, Genome, vol | |||
| Машина для изготовления проволочных гвоздей | 1922 |
|
SU39A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Гидравлический способ добычи торфа | 1916 |
|
SU206A1 |
| BENJAMINI Y | |||
| et al | |||
| Controlling the False Discovery Rate: A Practical and Powerful Approach to Multiple Testing, J.R | |||
| Stat | |||
| Soc | |||
| Ser | |||
| B, vol | |||
| Способ получения на волокне оливково-зеленой окраски путем образования никелевого лака азокрасителя | 1920 |
|
SU57A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
Авторы
Даты
2021-01-14—Публикация
2020-06-16—Подача