Изобретение относится к сельскому хозяйству и биотехнологии и может быть использовано для отбора ячменя с повышенным содержанием антоцианов в перикарпе зерновки.
Антоцианы относятся к группе вторичных метаболитов растений. Помимо важных функций, связанных с адаптацией растений к условиям окружающей среды (1), эти соединения обладают широкой биологической активностью и в настоящее время рассматриваются в качестве ингредиентов функционального питания человека (2). На сегодняшний день наблюдается устойчивый интерес к включению антоциановых соединений в ежедневный рацион, в том числе в составе хлебобулочных изделий (3, 4). Особое внимание в этом аспекте занимает селекция новых сортов злаков с повышенным содержанием антоцианов в зерне (5, 6).
У ячменя (Hordeum vulgare, 2n=2х=14, НН) известны образцы с фиолетовой окраской зерна, обусловленной биосинтезом антоциановых соединений в перикарпе зерновки. Такие образцы распространены преимущественно в высокогорных и предгорных районах Эфиопии и Эритреи, Малой Азии, Сирии, Палестины, Месопотамии и Закавказья (7). Синтез антоцианов в перикарпе зерна ячменя контролируется двумя комплементарно взаимодействующими генами - Ant1 и Ant2 (8, 9). Ген Ant1 был картирован на коротком плече хромосомы 7Н; он кодирует регуляторный фактор с доменом R2R3-MYB и помимо окраски зерна также определяет антоциановую окраску вегетативных органов растений (9). Ген Ant2 (синоним Pre2, Red lemma and pericarp 2), картированный на длинном плече хромосомы 2Н, кодирует транскрипционный фактор с регуляторным доменом bHLH. Также как и ген Ant1, ген Ant2, помимо окраски зерна, необходим для активации синтеза антоцианов в вегетативных органах.
Классический селекционный подход для получения фиолетовозерных сортов ячменя включает в себя скрещивание доноров доминантных аллелей генов, контролирующих окраску зерна, с рекуррентными сортами/линиями, зерно которых не имеет окраски, и последующий отбор растений с окрашенным зерном с проверкой стабильности наследования окраски зерна по потомкам. Поскольку пигменты, определяющие фиолетовую окраску зерна, накапливаются в имеющем материнское происхождение перикарпе зерновки и фенотип полученного после скрещивания растения возможно оценить только после его выращивания до стадии созревания зерна, описанный подход является длительным и трудозатратным (10, 11).
Использование внутригенных ДНК-маркеров, продукты амплификации геномной ДНК с помощью которых отличаются по длине между родительскими формами, позволяет проводить отбор в гибридной популяции нужных генотипов уже на стадии проростков, не дожидаясь созревания зерна растений, что может существенно сократить время, снизить трудозатраты и повысить точность отбора. Разработка таких маркеров на основе нуклеотидных последовательностей целевых генов является актуальной задачей селекции растений.
Наиболее близким к заявляемому - прототипом, является набор ДНК-маркеров, разработанных к полиморфным участкам генов Ant1 и Ant1, выявленных при сравнении нуклеотидных последовательностей этих генов у сорта «Bowman» и полученной на его основе фиолетовозерной почти-изогенной линии PLP (Purple Lemma Pericarp; синонимы: BW648, i:BWAnt1Ant2; каталожный номер в генбанке Nordgen: NGB22213), содержащей доминантные аллели генов Ant1 и Ant2 (12). Набор ДНК-маркеров включает ДНК-маркер Ant1.F/Ant1.R и ДНК-маркер Ant2.F/Ant2.R.
ДНК-маркер Ant1.F/Ant1.R представляет собой прямой Ant1.F: 5'-GTTTGCC AAAGGTCTATGTGA-3' и обратный Ant1.R: 5'-ATGAAATTCAGGAAGGTCGT-3' олигонуклеотидные праймеры, места отжига которых фланкируют инсерцию 25 нуклеотидов, выявленную в 3'-некодирующем районе гена Ant1 у фиолетовозерной почти-изогенной линии PLP по сравнению с ее рекурентным сортом «Bowman».
ДНК-маркер Ant2.F/Ant2.R представляет собой прямой Ant2.F: 5'-GCCGTGTGTTTCCTTAGTT-3' и обратный Ant2.R: 5'-CGAGCCAACAACAAGCGAGAC-3'олигонуклеотидные праймеры, места отжига которых фланкируют делецию 179 нуклеотидов, выявленную в промоторе гена Ant2 у фиолетовозерной почти-изогенной линии PLP по сравнению с ее рекуррентным сортом «Bowman». При амплификации ДНК сорта «Bowman» и линии PLP с помощью описанных ДНК-маркеров образуются ПЦР-продукты разной длины, соответствующие разным аллелям генов Ant1 и Ant2, благодаря чему можно определить происхождение аллелей этих генов у гибридов ячменя, полученных в результате скрещивания сорта «Bowman» и линии PLP.
Основным недостатком известного набора ДНК-маркеров является его ограниченная функциональная возможность, связанная с тем, что ДНК-маркеры были разработаны для выявления аллелей генов Ant1 и Ant2 непосредственно у сорта «Bowman» и линии PLP и их использование для отбора фиолетовозерных образцов ограничено лишь гибридными комбинациями сортов, отличающихся наличием описанных делеций в генах Ant1 и Ant2.
Задачей предлагаемого изобретения является разработка набора ДНК-маркеров, позволяющего быстро и эффективно отбирать растения ячменя, содержащие доминантные аллели генов Ant1 и Ant2 в гомозиготном состоянии, на основе любой гибридной комбинации.
Техническим результатом изобретения является расширение функциональных возможностей способа, повышение точности и сокращение времени отбора гибридов ячменя с повышенным содержанием антоцианов в зерне.
Для разработки таких ДНК-маркеров был проведен поиск нуклеотидных последовательностей аллелей генов Ant1 и Ant2 в базе данных пангеномных последовательностей ячменя Galaxy (https://galaxy-web.ipk-gatersleben.de/) по гомологии с ранее выделенными нуклеотидными последовательностями этих генов (номер в базе данных NCBI для генов Ant1: КР265976 и Ant2; КХ035100). Сравнение найденных последовательностей друг с другом, а в случае гена Ant2 - с нуклеотидной последовательностью уникального аллеля, характерного для фиолетовозерных образцов, включая фиолетовозерную почти изогенную линию PLP, позволило выявить отличия между аллелями этих генов и разработать олигонуклеотидные праймеры к их полиморфным участкам. Структура разработанных праймеров, условия амплификации, а также ожидаемые длины ПЦР-фрагментов представлены в таблице 1.
Нуклеотидная последовательность данных праймеров представлена в перечне последовательностей.
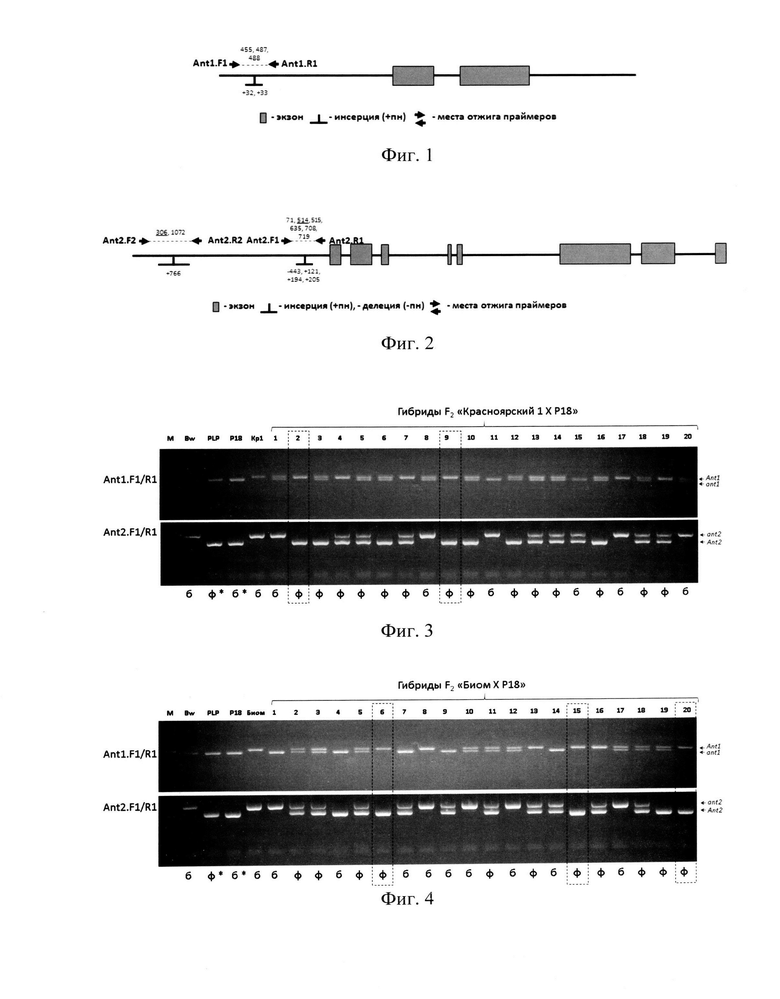
На фиг. 1 схематически представлена структурная организация гена Ant1 и места отжига разработанного маркера Ant1.F1/Ant1.Rl(SEQ ID NO 1, 2), которые фланкируют районы локализации инсерций/делеций, выявленных в 5'-некодирующем районе гена Ant1 с помощью сравнения аллелей этого гена, найденных в базе данных пангеномных последовательностей ячменя. Ожидаемая длина ПЦР-продуктов, амплифицируемых с помощью ДНК-маркера Ant1.F1/Ant1.Rl составляет 455, 487, либо 488 пар нуклеотидов (п.н.).
На фиг. 2 схематически представлена структурная организация гена Ant2 и места отжига ДНК-маркеров Ant2.F1/Ant2.R1 (SEQ ID NO 3, 4), фланкирующие район локализации инсерций/делеций, выявленных на основе сравнения аллелей гена Ant2 из базы данных пангеномных последовательностей ячменя с уникальным аллелем гена Ant2, характерным для фиолетовозерных образцов ячменя. Размер амплифицируемого с помощью праймеров Ant2.F1/Ant2.R1 ПЦР-продукта у фиолетовозерного образца составляет 514 п.н. (подчеркнут на фиг. 3), тогда как у белозерных образцов может составлять 71, 515, 635, 708, 719 п.н.
В частном случае, при отсутствии между родительскими образцами полиморфизма длины ПЦР-продукта, амплифицируемого с помощью праймеров 1, рекомендуется использовать пару праймеров Ant2.F2/Ant2.R2 (SEQ ID NO 5, 6), которая на матрице ДНК фиолетовозерного образца дает ПЦР-продукт длиной 306 п. н. (подчеркнут на фиг. 3), а на матрице ДНК белозерных образцов ячменя - 1072 п. н. ПЦР-продукты разной длины, получаемые с помощью разработанных праймеров на матрице ДНК родительских форм, соответствуют разным аллелями генов Ant1 и Ant2, благодаря чему возможно определить происхождение каждого из аллелей этих генов у гибридов ячменя.
Для отбора фиолетовозерных гибридов ячменя, полученных в результате скрещивания белозерных сортов/линий ячменя, характеризующихся либо наличием антоциановой пигментации основания растения и стебля, свидетельствующей о наличие доминантного аллеля гена Ant1 в геноме этого сорта/линии, либо ее отсутствием, наиболее вероятно обусловленным наличием рецессивных аллелей гена Ant1, с донором доминантных аллелей генов Ant1, Ant2, на первом этапе работы проводят выделение ДНК из молодых листьев родительских форм и гибридов второго поколения F2, полученных в результате их скрещивания. Для этого 2-3 кусочка листа (примерно 0,5×2 см) помещают в пробирку Eppendorf объемом 1,5 мл. Добавляют 200 мкл свежего буфера для экстракции (100 mM Tris-HCl, рН 7,5-8,0, 500 mМ NaCl, 50 mМ EDTA, 1,25% SDS, 0,38% Na2S2O5), нагретого до 60°С. Листья тщательно измельчают на гомогенизаторе. Далее добавляют 500 мкл буфера для экстракции, перемешивают и инкубируют на водяной бане при 60°С в течение 30-40 мин. После чего добавляют 700 мкл смеси хлороформ-изоамилового спирта (24:1) и после перемешивания центрифугируют 25 мин при 10000-12000 об/мин при комнатной температуре. К отобранной верхней фракции (-500 мкл) добавляют 1,4 мл 96% холодного этанола, слегка перемешивают и центрифугируют 10000-12000 об/мин. Осадок промывают 70% этанолом не менее 3 раз, просушивают и растворяют в 20-25 мкл бидистиллированной стерильной воды. Оценку качества и количества экстрагированной ДНК производят путем проведения аналитического электрофореза. После определения концентрации и чистоты ДНК концентрацию ДНК в пробах стандартизуют путем разведения в стерильной бидистиллированной воде до 100 мкг/мкл.
Выделенную ДНК родительских форм используют в качестве матрицы в ГТЦР, которую проводят в реакционной смеси объемом 20 мкл, содержащей 5 мкл ДНК, 1,8 мМ MgCl2, 6 мкл Н2О, по 0,2 мМ каждого дНТФ, по 1 мкМ прямого и обратного специфичных праймеров, 1 ед. ДНК-полимеразы Tag, в амплификаторе в режиме TOCHDOWN в случае маркеров Ant2.F1/Ant2.R1 и Ant2.F2/Ant2.R2 (2 мин при 94°; 13 циклов: 15 сек при 94°С, 30 сек при 65°С, опускаясь на 0,7°С каждый цикл до 56°С, 45 сек при 72°С; 24 цикла: 15 сек при 94°С, 30 сек при 56°С, 30 сек при 72°С; 5 мин при 72°С), либо 55GRAD - в случае маркера Ant1.F1/Ant1.R1 (2 мин при 94°С; 40 циклов: 15 сек при 94°С, 30 сек при 55°С, 45 сек при 72°С, 5 мин при 72°С). Результаты анализируются с помощью электрофоретического разделения продуктов ПЦР в 2% агарозном геле.
Пару праймеров, амплифицирующих у родительских форм участки генов Ant1 и Ant1 разной длины, далее используют для генотипирования ДНК гибридов. Среди гибридов отбирают те растения, которые несут доминантные аллели анализируемых генов в гомозиготном состоянии. При этом доминантные аллели гена Ant1 могут быть унаследованы как от фиолетовозерного родителя, так и от белозерного сорта/линии, если он/она имеет антоциановую окраску основания растения и стебля; доминантные аллели гена Ant2 могут быть унаследованы только от фиолетовозерного образца.
Расширение возможностей заявляемого способа обусловлено использованием для разработки ДНК-маркеров пангеномных нуклеотидных последовательностей ячменя, которые включают в себя геномные последовательности 20 сортов/линий ячменя, охватывающих максимально возможное генетическое разнообразие ячменя (13), а также нуклеотидной последовательности уникального аллеля гена Ant2, характерного для фиолетовозерных образцов. Использование этих последовательностей не только гарантировало выявление максимально доступного для этих генов полиморфизма, но также позволило разработать ДНК-маркеры к часто встречающимся в популяциях ячменя инсерциям/делециям, которые легко детектируются с помощью рутинных методов молекулярной биологии, таких как ПЦР и электрофорез, доступных в любой молекулярно-биологической лаборатории.
Изобретение иллюстрируется следующими примерами.
Пример 1.
Предлагаемый набор ДНК-маркеров был использован для отбора фиолетовозерных гибридов ячменя, полученных после скрещивания белозерного сорта «Красноярский 1» с белозерной линией Р18. Сорт «Красноярский 1» характеризуется наличием рецессивных аллелей гена Ant2 и антоциановой пигментацией основания растения, обусловленной наличием доминантных аллелей гена Ant1. Линия Р18 была получена на основе фиолетовозерной почти изогенной линии PLP, но, в отличие от родительской линии, не имеет окраски основания растения и зерна, поскольку несет рецессивные аллели гена Ant1, однако имеет доминантные аллели гена Ant2 (12).
Полученные после скрещивания гибридные семена второго поколения F2, а также семена родительских форм высаживали в ванну гидропонной теплицы ИЦиГ СО РАН (Новосибирск, Россия). Из молодых листьев проводили выделение ДНК, которую далее генотипировали с помощью ПЦР с использованием разработанных ДНК-маркеров к генам Ant1 и Ant2. Результаты электрофоретического анализа продуктов ПЦР в 2% агарозном геле представлены на фиг. 3. ПЦР-продукт, полученный с помощью ДНК-маркера Ant1.F1/Ant1.R1 у сорта «Красноярский 1» (Kpl) имеет длину 488 п. н., у линии Р18, а также у линии PLP и сорта «Bowman» (Bw), на основе которых была получена линия Р18 - 455 п.н. Поскольку сорт «Красноярский 1» имеет окраску основания растения, а линия Р18 и сорт «Bowman» - нет, то можно заключить, что выявляемые с помощью этих праймеров аллели генов Ant1 являются, соответственно, доминантным и рецессивным (отмечены рядом со стрелками как «Ant1» и «antl»). ПЦР-продукты, получаемые с помощью ДНК-маркера Ant2.F1/Ant2.R1 также отличаются по длине между родительскими формами: доминантный аллель гена Ant2, выявляемый у линий PLP и Р18, имеет длину 514 п. н., а рецессивный аллель ant2 сорта «Красноярский 1» - 719 п. н. (отмечены рядом со стрелками как «Ant2» и «ant2»). Все гибриды, несущие доминантные аллели генов Ant1 и Ant2 в гомозиготном или гетерозиготном состоянии характеризовались фиолетовой окраской зерна. Такие образцы отмечены на фиг. 3 буквой «ф». Наличие рецессивного аллеля одного из генов в гомозиготном состоянии обуславливало отсутствие антоцианов в зерне и белый цвет зерна; такие образцы отмечены буквой «б». Среди проанализированных растений были отобраны два растения под номерами 2 и 9, характеризовавшиеся наличием доминантных аллелей гена Ant1 и Ant2, унаследованных от сорта «Красноярский 1» и линии Р18, соответственно, в гомозиготном состоянии. Полученные с отобранных растений зерна были высажены для размножения в ванну гидропонной теплицы с автоматическим поливом в ИЦиГ СО РАН, в третьем поколении F3 анализируемые растения дали однородное гомозиготное потомство, характеризующееся фиолетовой окраской зерна. Следует обратить внимание на то, что линия Р18, отмеченная на фиг. 3 совместно с линий PLP звездочкой, в отличие от линии PLP, не имеет окраски зерна, поскольку несет рецессивный аллель гена Ant1, унаследованный от сорта «Bowman». При этом рецессивные аллели Ant1 линии Р18 и сорта «Bowman» не отличаются по длине ПЦР-продукта, амплифицруемого с помощью используемых праймеров, от доминантного аллеля гена Ant1 линии PLP.
Пример 2.
Использование разработанного набора ДНК-маркеров для отбора линий ячменя с повышенным содержанием антоцианов в зерне проводили аналогично примеру 1, за исключением того, что в качестве родительского сорта использовали белозерный красностебельный сорт «Биом», характеризующийся наличием рецессивного аллеля гена Ant2 и интенсивной антоциановой пигментацией основания растения, обусловленной наличием доминантных аллелей гена Ant1. ДНК родительских форм и полученных гибридов поколения F2 анализировали с помощью набора ДНК-маркеров Ant1.F1/Ant1.R1 и Ant2.F1/Ant2.R1. ГЩР, электрофорез и анализ полученных результатов проводили так же, как описано в примере 1.
Результаты электрофоретического анализа полученных продуктов ПЦР представлены на фиг. 4. Все гибриды, несущие доминантные аллели генов Ant1 и Ant2, унаследованные, соответственно, от сорта «Биом» и линии Р18, в гомозиготном или гетерозиготном состоянии характеризовались фиолетовой окраской зерна. Из двадцати гибридных растений поколения F2 у трех из них, а именно, у образцов под номерами 6, 15, 20, присутствовали доминантные аллели генов Ant1 и Ant2 в гомозиготном состоянии, унаследованные от сорта «Биом» и линии PLP, соответственно. Собранное с таких растений зерно имело фиолетовую окраску и в следующих поколениях давало гомогенное нерасщепляемое по цвету зерна потомство.
Таким образом, разработанный набор ДНК-маркеров может быть применен для отбора гибридов ячменя с повышенным содержанием антоцианов в перикарпе зерновки на основе как белозерных красностебельных, так и белозерных сортов/линий ячменя, не имеющих антоциановой окраски стебля и основания растения.
Использование предлагаемых ДНК-маркеров позволит существенно расширить функциональные возможности способа, снизить трудозатраты, повысить точность и скорость отбора фиолетовозерных образцов ячменя.
Источники информации
1. Landi М., Tattini М., Gould K.S. Multiple functional roles of anthocyanins in plant-environment interactions. Environmental and Experimental Botany. 2015; 119:4-17.
2. Юдина P.C., Гордеева Е.И., Шоева О.Ю., Тихонова М.А., Хлесткина Е.К. Антоцианы как компоненты функционального питания. Вавиповский журнал генетики и селекции. 2021; 25(2): 178-189.
3. Bellido G.G., Beta Т. Anthocyanin composition and oxygen radical scavenging capacity (ORAC) of milled and pearled purple, black, and common barley. Journal of Agricultural and Food Chemistry. 2009; 57:1022-1028.
4.  M., Romero M.-P.,
M., Romero M.-P.,  A., Puig E., Romagosa I., Moralejo M. Bioactive compounds and antioxidant capacity in pearling fractions of hulled, partially hull-less and hull-less food barley genotypes. Foods. 2021;10:565.
A., Puig E., Romagosa I., Moralejo M. Bioactive compounds and antioxidant capacity in pearling fractions of hulled, partially hull-less and hull-less food barley genotypes. Foods. 2021;10:565.
5. Loskutov I.G., Khlestkina E.K. Wheat, barley, and oat breeding for health benefit components in grain. Plants. 2021; 10:86.
6. Shvachko N.A., Loskutov I.G., Semilet T.V., Popov V.S., Kovaleva O.N., Konarev A.V. Bioactive components in oat and barley grain as a promising breeding trend for functional food production. Molecules. 2021; 26:2260.
7. Вавилов Н.И. Центры происхождения культурных растений. В кн.: Н.И.Вавилов. Избранные произведения: В 2 т. Т. 1. Ленинград: Наука; 1967. С. 88-202.
8. Zhang X-W., Jiang Q-T., Wei Y-M., Liu C. Inheritance analysis and mapping of quantitative trait loci (QTL) controlling individual anthocyanin compounds in purple barley (Hordeum vulgar e L.) grains. PLoS ONE. 2017; 12(8):e0183704.
9. Шоева О.Ю., Стрыгина K.B., Хлесткина E.K. Гены, контролирующие синтез флавоноидных и меланиновых пигментов ячменя. Вавиловский журнал генетики и селекции. 2018; 22(3):333-342.
10. Gordeeva E.I., Shoeva O.Y., Khlestkina E.K. Marker-assisted development of bread wheat near-isogenic lines carrying various combinations of purple pericarp (Pp) alleles. Euphytica. 2015; 203:46-476.
11. Gordeeva E., Shamanin V., Shoeva O., Kukoeva Т., Morgounov A., Khlestkina E. The strategy for marker-assisted breeding of anthocyanin-rich spring bread wheat {Triticum aestivum L.) cultivars in Western Siberia. Agronomy. 2020; 10:1603.
12. Gordeeva E.I., Glagoleva A.Y., Kukoeva T.V., Khlestkina E.K., Shoeva O.Y. Purple-grained barley (Hordeum vulgare L.): marker-assisted development of NILs for investigating peculiarities of the anthocyanin biosynthesis regulatory network. BMC Plant Biology. 2019; 19(Suppl 1):52.
13. Jayakodi M., Padmarasu S., Haberer G. et al. The barley pan-genome reveals the hidden legacy of mutation breeding. Nature. 2020. 588; 284-289.
Набор ДНК-маркеров для отбора ячменя с повышенным содержанием
антоцианов в перикарпе зерновки

--->
Перечень последовательностей
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Ant1,Ant2.xml"
softwareName="WIPO Sequence" softwareVersion="2.0.0"
productionDate="2022-08-15">
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное научное учреждение Федеральный исследовательский центр
«Институт цитологии и генетики Сибирского отделения Российской
академии наук» (ИЦиГ СО РАН)</ApplicantName>
<ApplicantNameLatin>Institute of Cytology and Genetics, Siberian
Branch of Russian Academy of Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru"> Набор ДНК-маркеров к генам Ant1 и
Ant2 для отбора гибридов ячменя с повышенным содержанием антоцианов в
зерне</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
SEQID NO 1
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcggcttgatttgtttcata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
SEQID NO 2
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttaaatggcgaggtaaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
SEQID NO 3
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctggaacacacgtacaaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
SEQID NO 4
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttgagctatggagaccaagaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
SEQID NO 5
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattcatgtcggtccagaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
SEQID NO 6
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagaggaactgcttcgttaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Внутригенный ДНК-маркер для отбора пшеницы с повышенным содержанием антоцианов в перикарпе зерновки | 2021 |
|
RU2774444C1 |
| Способ отбора гибридов мягкой пшеницы с голубой окраской зерновки | 2022 |
|
RU2790186C1 |
| Способ отбора линий яровой мягкой пшеницы с повышенным содержанием антоцианов в зерне | 2021 |
|
RU2762804C1 |
| CAPS-маркер для отбора гибридов ячменя, не накапливающих проантоцианидины в зерне | 2023 |
|
RU2809369C1 |
| Способ диагностики аллельного состояния гена Vi4, контролирующего фиолетовую окраску зерна у ржи | 2021 |
|
RU2787110C1 |
| Система внутригенных ДНК-маркеров для детекции в растениях подсолнечника доминантного и рецессивного аллелей гена HaOr7, контролирующего устойчивость к расе G заразихи (Orobanche cumana Wallr.) | 2023 |
|
RU2830955C1 |
| МОЛЕКУЛЯРНЫЙ МАРКЕР FR_ER1 И ЕГО ИСПОЛЬЗОВАНИЕ ДЛЯ СЕЛЕКЦИИ НОВЫХ СОРТОВ ГОРОХА, УСТОЙЧИВЫХ К МУЧНИСТОЙ РОСЕ | 2013 |
|
RU2593691C2 |
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| Способ определения филогенетической принадлежности штаммов Yersinia pestis основного подвида методом аллель-специфической ПЦР в режиме реального времени | 2022 |
|
RU2799415C1 |
| Способ получения линий яровой мягкой пшеницы с укороченным сроком колошения | 2019 |
|
RU2710729C1 |
Изобретение относится к сельскому хозяйству и биотехнологии и может быть использовано в генетике и селекции ячменя. Предложен набор ДНК-маркеров для отбора гибридов ячменя с повышенным содержанием антоцианов в перикарпе зерновки, синтез которых контролируется генами Ant1 и Ant2. Предлагаемый набор содержит ДНК маркеры Ant1.F1/Ant1.R1, Ant2.F1/Ant2.R1 и Ant2.F2/Ant2.R2, имеющие следующие нуклеотидные последовательности Ant 1.F1: 5'-GCGGCTTGATTTGTTTCАТА-3', Ant1.R1: 5'-TTTAAATGGCGAGGTAAGGT-3', Ant2.F 1: 5'-GCTGGAACACACGTACAAGA-3', Ant2.R1: 5'-CTTTGAGCTATGGAGACC AAGAA-3', Ant2.F2: 5'-CATTCATGTCGGTCCAGAAC-3', Ant2.R2: 5'-CAGAGGAACTGCTTCGTTAAA-3'. Изобретение позволяет повысить точность и скорость отбора растений ячменя с повышенным содержанием антоцианов в перикарпе зерновки. 4 ил., 1 табл., 2 пр.
Набор ДНК-маркеров для отбора ячменя с повышенным содержанием антоцианов в перикарпе зерновки, каждый из которых представляет собой пару синтетических олигонуклеотидных праймеров для выявления аллелей генов Ant1 и Ant2, отличающийся тем, что содержит ДНК маркеры Ant1.F1/Ant1.R1, Ant2.F1/Ant2.R1 и Ant2.F2/Ant2.R2, имеющие следующие нуклеотидные последовательности SEQ ID NO 1 для Ant1.F1, SEQ ID NO 2 для Ant1.R1, SEQ ID NO 3 для Ant2.F1, SEQ ID NO 4 для Ant2.R1, SEQ ID NO 5 для Ant2.F2 и SEQ ID NO 6 для Ant2.R2, позволяющие амплифицировать последовательность ДНК ячменя и выявлять доминантные аллели генов Ant1 и Ant2 в гомозиготном состоянии.
| ELENA I | |||
| Gordeeva, et al., Purple-grained barley (Hordeum vulgare L.): marker-assisted development of NILs for investigating peculiarities of the anthocyanin biosynthesis regulatory network, BMC Plant Biology 2019, 19(Suppl 1):52, c | |||
| Устройство для выпрямления многофазного тока | 1923 |
|
SU50A1 |
| ДНК-маркер для селекции сортов ярового ячменя, устойчивых к темно-бурой пятнистости | 2020 |
|
RU2740404C1 |
| АНДРЕЕВА Е.А, и др., Локализация и состав антоцианов в зерновках ржи с разной | |||
Авторы
Даты
2023-06-07—Публикация
2022-09-06—Подача