Изобретение относится к сельскому хозяйству и биотехнологии и может быть использовано для отбора пшеницы с повышенным содержанием антоцианов в зерне.
Антоцианы - растительные пигменты, положительно влияющие на здоровье человека, в настоящее время рассматриваются как безвредные природные соединения для производства функциональных продуктов питания и для нужд медицины, обладая антиоксидантными, противовоспалительными, гипогликемическими, антимутагенными, антидиабетическими, противораковыми и нейропротекторными свойствами (1). Кроме ягод и фруктов, антоцианы могут накапливаться в оболочках зерна пшеницы, использование которых в качестве сырья для производства функциональных продуктов питания и экстракции антоцианов, позволяет не только получить высококачественные продукты питания, но и снизить отходы мукомольного производства. В связи с этим, создание сортов пшеницы с повышенным содержанием антоциановых соединений в зерне является актуальной задачей, отражающей общемировую тенденцию (2).
Биосинтез и накопление антоциановых пигментов происходит в перикарпе зерновки пшеницы под контролем двух комплементарно взаимодействующих генов Рр-1 и Рр3, картированных в хромосомах 7 гомеологической группы и в хромосоме 2А, соответственно (3). В отличие от генов Рр-1, доминантные аллели которых широко представлены у диплоидных, тетраплоидных и гексаплоидных видов пшениц (4-6), доминантный аллель гена Рр3 известен у единственного вида тетраплоидной пшеницы Т. aephiopicum (7). С помощью классической гибридизации и отбора доминантный аллель гена Рр3 был перенесен в геном мягкой пшеницы от пшеницы эфиопской (8).
Классический селекционный подход, основанный на скрещивании доноров генов, контролирующих окраску зерна, с рекуррентными сортами/линиями, с последующим отбором растений с окрашенным зерном является длительным и трудозатратным процессом. В первую очередь это связано с тем, что пигменты, определяющие фиолетовую окраску зерна, накапливаются в перикарпе, имеющем материнское происхождение, и фенотип полученного после скрещивания растения, возможно оценить только после его выращивания до стадии созревания зерна с последующей проверкой гомозиготности окраски зерновок у потомков (9, 10).
Использование ДНК-маркеров, основанных на методе полимеразной цепной реакции, тесно сцепленных с геном, контролирующим антоциановую окраску зерна, или внутригенных, способных отличить различные аллели целевого гена, позволяет проводить отбор нужных генотипов уже на стадии проростков, не дожидаясь созревания зерна растений, что может существенно сократить время, снизить трудозатраты, а в случае внутривенных маркеров и повысить точность отбора линий пшеницы с фиолетовой окраской зерна. Разработка таких маркеров на основе знаний о структурной организации целевых генов является актуальной задачей селекции растений.
Благодаря секвенированию и расшифровке первичной последовательности ДНК гена Рр3 у мягкой пшеницы было выявлено шесть 261-нуклеотидных тандемных повтора в промоторе доминантного аллеля, тогда как лишь один такой повтор был обнаружен в рецессивном аллеле белозерных сортов пшеницы (7).
Наиболее близким к заявляемому - прототипом, является ДНК-маркер 2APR01 для выявления аллелей гена Рр3, который позволяет отличить доминантный и рецессивный аллели этого гена между собой (11). ДНК-маркер представляет собой два специфических олигонуклеотидных праймера: F: 5'-AAGAAATGACACTTCGCCACAA-3' (прямой) и R: 5'-GGTGGGCTCTTCCTGGCAT-3' (обратный), места отжига которых фланкируют повторенные участки промотора. В результате амплификации геномной ДНК фиолетовозерных (имеющих шесть тандемных повтора в промоторе гена Рр3) и белозерных (имеющих один повтор) образцов пшеницы образуются ПЦР-продукты, отличающиеся по длине. В случае доминантного аллеля гена Рр3, обозначаемого в (11) как ТаРрb1а, длина ПЦР-продукта составляет 3417, а в случае рецессивного TaPpb1b - 2143 пар нуклеотидов.
Основным недостатком известного ДНК-маркера является большая длина амплифицируемого им ПЦР-продукта, который в случае доминантного аллеля составляет 3417, а в случае рецессивного - 2143 пар нуклеотидов. Для получения ПЦР-продуктов такой длины требуется высокоэффективная полимераза, не всегда доступная исследователю, что существенно ограничивает применимость ДНК-маркера 2APR01 в селекционном процессе.
Задачей предлагаемого изобретения является разработка внутригенного ДНК-маркера, который позволит идентифицировать доминантные и рецессивные аллели гена Рр3 в гибридах пшеницы на ранних стадиях развития растений с использованием рутинных методов молекулярной биологии, в том числе любых доступных исследователю ДНК-пол имераз.
Техническим результатом изобретения является повышение точности и сокращение времени отбора пшеницы с повышенным содержанием антоцианов в перикарпе зерновки.
Поставленная задача достигается предлагаемым ДНК-маркером, представляющим собой пару специфических олигонуклеотидных праймеров, позволяющих амплифицировать последовательность ДНК пшеницы и выявлять доминантные и рецессивные аллели гена Рр3 одновременно:
Pp3_Fd: 5'-TAGTGCCGTCTAACTGGTGA-3'
Pp3_Rd: 5'-ACGACGCCTAAGGAAACAC-3'
Нуклеотидная последовательность данных праймеров представлена в перечне последовательностей.
Для разработки указанных маркеров было проведено выравнивание нуклеотидных последовательностей доминантного и рецессивного аллелей гена Рр3, ТаРрb1а (номер в базе данных NCBI: MG066455) и TaPpb1b (номер в базе данных NCBI: MG066456), соответственно, которые были найдены в базе данных нуклеотидных последовательностей NCBI (https://www.ncbi.nlm.nih.gov/) по гомологии с выделенной ранее нуклеотидной последовательностью гена Рр3 (номер в базе данных NCBI: KJ747954; 12).
На фиг.1 схематически показаны отличия в структурной организации промоторов гена Рр3 между доминантным и рецессивным аллелями - TaPpb1a и TaPpb1b, соответственно. Разной штриховкой показаны повторы и его части, обнаруженные в промоторе доминантного аллеля TaPpb1a по сравнению с рецессивным аллелем TaPpb1b. Как видно фиг.1, доминантный аллель имеет пять полных 261-нуклеотидных повтора и два усеченных фрагмента этого повтора, один из которых составляет 27, а другой - 205 пар нуклеотидов (п.н.). Рецессивный аллель имеет лишь один 261-нуклеотидный повтор.
Специфические олигонуклеотидные праймеры были сконструированы таким образом, что прямой праймер Pp3_Fd отжигался на область промотора до начала повторенных участков как у доминантного, так и рецессивного аллеля, а обратный праймер Pp3_Rd - на 261-нуклеотидный повтор. За счет наличия усеченных фрагментов в 27 и 205 нуклеотидов ПНР-продукты, получаемые с помощью разработанных праймеров отличались по длине между доминантным и рецессивным аллелями и составили 398 и 429 пар нуклеотидов, соответственно. При этом у доминантного аллеля при использовании эффективных ДНК-полимераз можно амплифицировать пять дополнительных ПЦР-фрагмента, отличающихся друг от друга на 261 нуклеотид: 659, 920, 1181, 1442, 1703 нуклеотида.
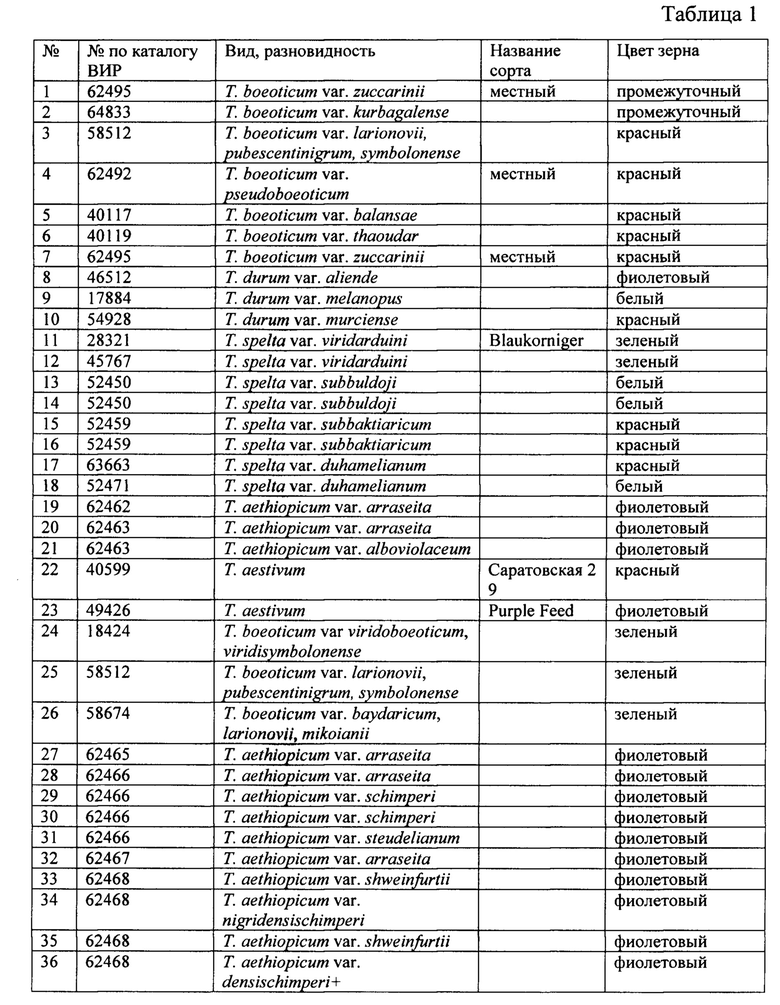
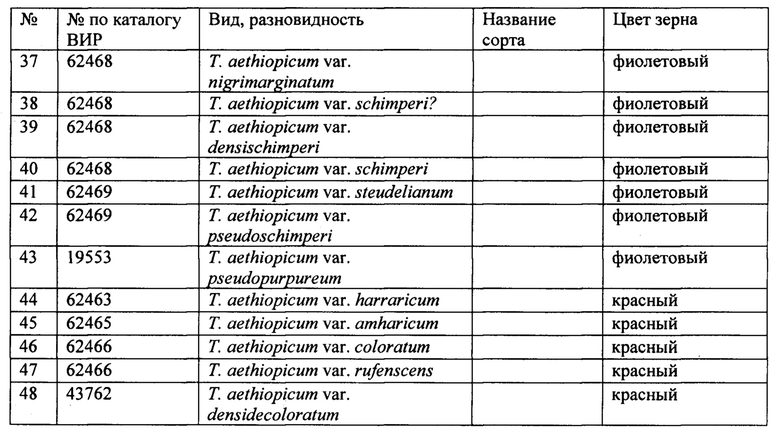
Разработанный ДНК-маркер был протестирован на 48 образцах различных видов пшеницы, окраска зерна которых указана в таблице 1.
На фиг.2 представлен результат электрофоретического анализа амплификации геномной ДНК этих образцов с использованием разработанного ДНК-маркера (Рр3-diagnostic). Как видно на фиг.2, ДНК-маркер показал 100% соответствие наличия доминантного аллеля гена Рр3 и фиолетовой окраски зерна у образцов Т. aethiopicum (образцы под номерами 19-21, 27-43), Т. durum (образец под номером 8) и Т. aestivum (образец под номером 23), рецессивный аллель гена был выявлен у образцов этих видов без антоциановой окраски зерна (Т. aethiopicum: 44-48; Т. durum: 9-10; Т. aestivum: 22), а также у семи проанализированных образцов Т. spelta (образцы под номерами 12-18), один из которых имел зеленую окраску зерна, три - белую и три - красную. У Т. boeoticum (образцы под номерами 1-7 и 24-26) праймеры к гену Рр3 не амплифицировали ПНР-продукты, что свидетельствует об отсутствии гомологии в зоне отжига праймера.
Представленные данные указывают на то, что разработанный ДНК-маркер может быть использован для отбора растений с доминантным аллелем гена Рр3 как среди образцов гексаплоидной (Т. aestivum, Т. spelta), так и тетраплоидной (Т. aethiopicum, Т. durum) пшеницы, но не у диплоидной пшеницы Т. boeoticum.
Изобретение иллюстрируется следующими примерами.
Пример 1
Предлагаемый ДНК-маркер Рр3-diagnostic был использован для отбора целевых растений в поколении F2 при создании новых линий яровой мягкой пшеницы с повышенным содержанием антоцианов в перикарпе зерновки после скрещивания белозерного красностебельного сорта «Эритросперум 4-16», несущего доминантный аллель гена Pp-D1 и рецессивный аллель гена Рр3 (13), с фиолетовозерной линией i:C29PpD1Pp3P, созданной на основе сорта «Саратовская 29» и несущей интрогрессированные участки хромосом 2А и 7D от сорта «Purple» с доминантными аллелями генов Рр3 и Pp-D1, соответственно.
Полученные после скрещивания семена второго поколения F2 высаживали в ванну гидропонной теплицы ИЦиГ СО РАН (Новосибирск, Россия) в осеннюю вегетацию 2020 года. Из молодых листьев всех высаженных растений выделяли ДНК. Для этого 2-3 кусочка листа (примерно 0,5×2 см) помещали в пробирку Eppendorf объемом 1,5 мл. Добавляли 200 мкл свежего буфера для экстракции (100 mM Tris-HCl, рН 7,5-8,0, 500 mM NaCl, 50 mM EDTA, 1,25% SDS, 0,38% Na2S2O5), нагретого до 60°С. Листья тщательно измельчали на гомогенизаторе. Далее добавляли 500 мкл буфера для экстракции, перемешивали и инкубировали на водяной бане при 60°С в течение 30-40 мин. После чего добавляли 700 мкл смеси хлороформ-изоамилового спирта (24:1) и после перемешивания центрифугировали 25 мин при 10000-12000 об/мин при комнатной температуре. К отобранной верхней фракции (-500 мкл) добавляли 1,4 мл 96% холодного этанола, слегка перемешивали и центрифугировали 10000-12000 об/мин. Осадок промывали 70% этанолом не менее 3 раз, просушивали и растворяли в 20-25 мкл бидистиллированной стерильной воды. Оценку качества и количества экстрагированной ДНК производили путем проведения аналитического электрофореза. После определения концентрации и чистоты ДНК концентрацию ДНК в пробах стандартизовали путем разведения в стерильной бидистиллированной воде до 100 мкг/мкл.
Выделенная ДНК использовалась в качестве матрицы в ПЦР, которую проводили в реакционной смеси объемом 20 мкл, содержащей 5 мкл ДНК, 1,8 мМ MgCl2, 6 мкл Н2О, по 0,2 мМ каждого дНТФ, по 1 мкМ прямого и обратного специфичных праймеров, 1 ед. ДНК-полимеразы Taq, в амплификаторе в режиме TOCHDOWN: 13 циклов: преденатурация - 2 минуты при 94°С, денатурация - 15 секунд при 94°С, отжиг матрицы с праймерами - 30 секунд при 65°С с понижением на 0.7°С/цикл), полимеризация - 45 сек при 72°С; число циклов - 24: денатурация - 15 секунд при 94°С, отжиг матрицы с праймерами - 30 секунд при 56°С, полимеризация - 30 секунд при 72°С; достраивание ПЦР-фрагментов - 5 минут при 72°С.
Результаты электрофоретического анализа продуктов ПНР в 2% агарозном геле представлены на фиг.3. Стрелками указан фрагмент ДНК длиной 398 пар нуклеотидов, свидетельствующий о наличии доминантных аллелей гена Рр3, присутствующих в геноме растений под номерами 1-3, 5, 6, 9 и 21 в гомозиготном состоянии.
Отобранные растения имели генотип Pp-D1Pp-D1Рр3Рр3 самоопылялись до поколения F3 в весеннюю вегетацию 2021 в теплице ИЦиГ. Полученные гибридные линии с фиолетовой окраской зерновок на основе сорта «Эритросперум 4-16» были переданы для полевого тестирования в селекционный питомник Омского ГАУ им. П.А. Столыпина (Омск, Россия).
Пример 2
Разработанный ДНК-маркер был использован для отбора линии яровой мягкой пшеницы с повышенным содержанием антоцианов в зерне аналогично примеру 1, за исключением того, что в качестве родительского сорта использовали белозерный красностебельный сорт «Терция». В результате ПЦР-анализа с использованием ДНК-маркера Рр3-diagnostic геномной ДНК, выделенной из пятидесяти индивидуальных гибридов второго поколения F2, полученных в результате скрещивания сорта «Терция» с донорной линией i:C29PpD1Pp3Р были выявлены четырнадцать растений, у которых на электрофореграмме присутствовал единичный ПЦР-продукт длиной 398 пар нуклеотидов, соответствующий доминантном аллелю гена Рр3. Отобранные растения имели генотип Pp-D1Pp-D1Рр3Рр3, самоопылялись до поколения F3 в весеннюю вегетацию 2021 в теплице ИЦиГ. Полученные в результате гибридные линии имели фиолетовую окраску зерновок и были переданы для полевого тестирования в селекционный питомник Омского ГАУ им. П.А. Столыпина (Омск, Россия).
Пример 3
Разработанный ДНК-маркер был использован для отбора линии яровой мягкой пшеницы с повышенным содержанием антоцианов в зерне аналогично примеру 1, за исключением того, что в качестве родительского сорта использовали белозерный красностебельный сорт «Сибирская 21». В результате ПЦР-анализа с использованием Д НК-маркера Рр3-diagnostic геномной ДНК, выделенной из пятидесяти гибридов второго поколения F2, полученных в результате скрещивания сорта «Сибирская 21» с донорной линией i:C29PpD1Pp3Р были выявлены двенадцать растений, у которых на электрофореграмме присутствовал единичный ПЦР-продукт длиной 398 пар нуклеотидов, соответствующий доминантному аллелю гена Рр3. Отобранные растения имели генотип Pp-D1Pp-D1Рр3Рр3, самоопылялись до поколения F3 в весеннюю вегетацию 2021 в теплице ИЦиГ. Полученные растения имели фиолетовую окраску зерновок и являются перспективными для дальнейшей селекции фиолетовозерных сортов пшеницы для Сибирского региона.
Таким образом, разработанный ДНК-маркер (Рр3-diagnostic) может быть применен для отбора линий мягкой пшеницы с повышенным содержанием антоцианов в перикарпе зерновки на основе белозерных красностебельных сортов пшеницы.
Использование предлагаемого ДНК-маркера позволит существенно снизить трудозатраты, повысить точность и скорость отбора по сравнению с отбором линий по фенотипу или с помощью сцепленных с целевым геном микросателлитных ДНК-маркеров.
Источники информации
1. Юдина Р.С., Гордеева Е.И., Шоева О.Ю., Тихонова М.А., Хлесткина Е.К. Антоцианы как компоненты функционального питания. Вавиловский журнал генетики и селекции. 2021; 25(2): 178-189.
2. Loskutov I.G., Khlestkina Е.K. Wheat, barley, and oat breeding for health benefit components in grain. Plants. 2021; 10:86.
3. Khlestkina E.K. Genes determining the coloration of different organs in wheat. Russian Journal of Genetics: Applied Research. 2013; 3(1):54-65.
4. Himi E., Taketa S. Isolation of candidate genes for the barley Antl and wheat Rc genes controlling anthocyanin pigmentation in different vegetative tissues. Mol. Genet. Genomics. 2015;290(4):1287-1298.
5. Ye G.J., Wei L., Chen W.J., Zhang В., Liu B.L., Zhang H.G. Frame-shift mutation causes the function loss of TaMYB-A1 regulating anthocyanin biosynthesis in Triticum aestivum. Cereal Res. Commun. 45; 1: 35-46.
6. Cao D., Fan J., Xi X., Zong Y., Wang D., Zhang H., Liu B. Transcriptome analysis identifies key genes responsible for red coleoptiles in Triticum monococcum. Molecules. 2019;24:932.
7. Zong Y., Xi X., Li S., Chen W., Zhang В., Liu D., Liu В., Wang D., Zhang H. Allelic variation and transcriptional isoforms of wheat TaMYC1 gene regulating anthocyanin synthesis in pericarp.Front. Plant Sci. 2017;8:1645.
8. Zeven A.C. Wheats with purple and blue grains: a review. Euphytica. 1991;56:243-258.
9. Gordeeva E.I., Shoeva O.Y., Khlestkina E.K. Marker-assisted development of bread wheat near-isogenic lines carrying various combinations of purple pericarp (Pp) alleles. Euphytica. 2015;203:469-476.
10. Gordeeva E., Shamanin V., Shoeva O., Kukoeva Т., Morgounov A., Khlestkina E. The strategy for marker-assisted breeding of anthocyanin-rich spring bread wheat (Triticum aestivum L.) cultivars in Western Siberia. Agronomy. 2020; 10:1603.
11. Jiang W., Liu Т., Nan W., Jeewani D.C., Niu Y., Li C, Wang Y., Shi X., Wang C, Wang J., Li Y., Gao X., Wang Z. Two transcription factors TaPpm1 and TaPpb1 co-regulate anthocyanin biosynthesis in purple pericarps of wheat. J. Exp. Botany. 2018;69(10):2555-2567.
12. Shoeva O.Y., Gordeeva E.I., Khlestkina E.K. The regulation of anthocyanin synthesis in the wheat pericarp. Molecules. 2014;19(12):20266-20279.
13. Shamanin V., Salina E., Wanyera R., Zelenskiy Y., Olivera P., Morgounov A. Genetic diversity of spring wheat from Kazakhstan and Russia for resistance to stem rust Ug99. Euphytica. 2016; 212(2):287-296.
Перечень последовательностей
<110> Федеральное государственное бюджетное научное учреждение «Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения Российской академии наук» (ИЦиГ СО РАН)
<120> Внутригенный ДНК-маркер для отбора пшеницы с повышенным содержанием антоцианов в перикарпе зерновки
<160> Номер SEQ ID NO 1
<210> 1
<211> 20
<212> ДНК
<213> Искусственная последовательность
<223> Олигонуклеотидный праймер
<400> 1
TAGTGCCGTCTAACTGGTGA
<160> Номер SEQ ID NO 2
<210> 2
<211> 20
<212> ДНК
<213> Искусственная последовательность
<223> Олигонуклеотидный праймер
<400> 2
ACGACGCCTAAGGAAACAC
| название | год | авторы | номер документа |
|---|---|---|---|
| Набор ДНК-маркеров для отбора ячменя с повышенным содержанием антоцианов в перикарпе зерновки | 2022 |
|
RU2797678C1 |
| Способ отбора гибридов мягкой пшеницы с голубой окраской зерновки | 2022 |
|
RU2790186C1 |
| Способ отбора линий яровой мягкой пшеницы с повышенным содержанием антоцианов в зерне | 2021 |
|
RU2762804C1 |
| CAPS-маркер для отбора гибридов ячменя, не накапливающих проантоцианидины в зерне | 2023 |
|
RU2809369C1 |
| Способ диагностики аллельного состояния гена Vi4, контролирующего фиолетовую окраску зерна у ржи | 2021 |
|
RU2787110C1 |
| Способ отбора константных безостых форм мягкой пшеницы | 2021 |
|
RU2768393C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772577C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772575C2 |
| Система внутригенных ДНК-маркеров для детекции в растениях подсолнечника доминантного и рецессивного аллелей гена HaOr7, контролирующего устойчивость к расе G заразихи (Orobanche cumana Wallr.) | 2023 |
|
RU2830955C1 |
| Молекула РНК-проводника sgRNA для внесения мутаций в консервативный участок промоторной области гена PPD-D1 мягкой пшеницы с применением системы редактирования генома CRISPR/Cas9 | 2024 |
|
RU2822358C1 |

Изобретение относится к сельскому хозяйству и биотехнологии и может быть использовано в генетике и селекции пшеницы. Предложен внутригенный ДНК-маркер для отбора сортов пшеницы с повышенным содержанием антоцианов в перикарпе зерновки, синтез которых контролируется генами Рр3 и Рр-1. ДНК-маркер представляет собой олигонуклеотидные праймеры, имеющие нуклеотидную последовательность SEQ ID NO 1: 5'-TAGTGCCGTCTAACTGGTGA-3' и SEQ ID NO 2: 5'-ACGACGCCTAAGGAAACAC-3'. Изобретение позволяет повысить точность и скорость отбора растений пшеницы с повышенным содержанием антоцианов в перикарпе зерновки. 3 ил., 1 табл., 3 пр.
Внутригенный ДНК-маркер для отбора пшеницы с повышенным содержанием антоцианов в перикарпе зерновки, представляющий собой два специфических олигонуклеотидных праймера для выявления аллелей гена Рр3, отличающийся тем, что праймеры имеют нуклеотидную последовательность SEQ ID NO 1: 5'-TAGTGCCGTCTAACTGGTGA-3' и SEQ ID NO 2: 5'-ACGACGCCTAAGGAAACAC-3' и позволяют амплифицировать последовательность ДНК пшеницы и выявлять доминантные и рецессивные аллели гена Рр3 одновременно.
| JIANG W | |||
| et al., Two transcription factors TaPpm1 and TaPpb1 co-regulate anthocyanin biosynthesis in purple pericarps of wheat., J | |||
| Exp | |||
| Botany | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| ZONG Y | |||
| et al., Wang D., Zhang H | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Plant Sci | |||
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
Авторы
Даты
2022-06-21—Публикация
2021-11-29—Подача