Данные о родственных заявках
Данная Заявка на Патент заявляет приоритет Предварительной Заявки на Патент США No. 61/844,844 от 10 июля 2013 года, которая включается в настоящее изобретение во всей своей полноте путем отсылки для любых целей.
Утверждение интересов правительства
Настоящее изобретение было сделано при поддержке Правительства США в виде гранта No. P50 HG005550 Национального Института Здоровья (NIH) и гранта DE-FG02-02ER63445 от Министерства Энергетики США. Правительство США обладает определенными правами на настоящее изобретение.
Уровень техники
CRISPR-Cas-системы бактерий и архей основаны на коротких направляющих РНК, работающих в комплексе с Cas-белками, которые обеспечивают направленную деградации комплементарных последовательностей, присутствующих в попавшей в клетку чужеродной нуклеиновой кислоты. Смотри: Deltcheva, E. et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471, 602-607 (2011); Gasiunas, G., Barrangou, R., Horvath, P. & Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America 109, E2579-2586 (2012); Jinek, M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012); Sapranauskas, R. et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic acids research 39, 9275-9282 (2011); и Bhaya, D., Davison, M. & Barrangou, R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation. Annual review of genetics 45, 273-297 (2011). Проведенная недавно in vitro реконструкция CRISPR системы типа II S. pyogenes показала, что наличие crРНК ("CRISPR RNA"), слитой с нормально транс-кодируемой tracrРНК ("trans-activating CRISPR RNA"), является достаточным для направления белка Cas9 к специфической последовательности и расщепления целевых последовательностей ДНК, комплементарных crРНК. Экспрессия направляющей РНК (gРНК), гомологичной целевому сайту, приводит к рекрутированию белка Cas9 и к деградации им целевой ДНК. Смотри: H. Deveau et al, Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. Journal of Bacteriology 190, 1390 (Feb, 2008).
Раскрытие изобретения
Аспекты настоящего изобретения касаются комплекса, состоящего из направляющей РНК, ДНК-связывающего белка и целевой последовательности двухцепочечной ДНК. Согласно определенным аспектам, ДНК-связывающие белки в рамках настоящего изобретения включают белок, который образует комплекс с направляющей РНК, где направляющая РНК направляет комплекс к последовательности двухцепочечной ДНК, где комплекс связывается с данной последовательностью ДНК. Этот аспект настоящего изобретения может рассматриваться как ко-локализация РНК и ДНК-связывающего белка с или на двухцепочечной ДНК. Таким образом, комплекс ДНК-связывающий белок-направляющая РНК может применяться для того, чтобы локализовать транскрипционный регуляторный белок или домен на целевой ДНК так, чтобы регулировать экспрессию целевой ДНК.
Согласно одному аспекту, два, или более, или множество ортогональных РНК-направляемых ДНК-связывающих белков, или набор ортогональных РНК-направляемых ДНК-связывающих белков, может применяться для того, чтобы одновременно и независимо регулировать гены в ДНК в клетке. Согласно одному аспекту, два, или более, или множество ортогональных РНК-направляемых ДНК-связывающих белков, или набор ортогональных РНК-направляемых ДНК-связывающих белков, может применяться для того, чтобы одновременно и независимо редактировать гены в ДНК в клетке. Необходимо понимать, что там, где делается ссылка на ДНК-связывающий белок или РНК-направляемые ДНК-связывающие белки, такая ссылка включает ортогональный ДНК-связывающий белок или ортогональный РНК-направляемый ДНК-связывающий белок. Такие ортогональные ДНК-связывающие белки или ортогональные РНК-направляемые ДНК-связывающие белки могут обладать нуклеазной активностью, могут обладать никазной активностью или могут не иметь нуклеазной активности.
Согласно определенным аспектам, предоставляется способ модулирования экспрессии целевой нуклеиновой кислоты в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей одну или более РНК (рибонуклеиновых кислот), комплементарных к ДНК (дезоксирибонуклеиновой кислоте), где ДНК включает целевую нуклеиновую кислоту; введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок, который связывается с ДНК и направляется одной или более РНК; введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей транскрипционный регуляторный белок или домен, где в результате экспрессируется одна или более РНК, РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок и транскрипционный регуляторный белок или домен, где одна или более РНК, РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок и транскрипционный регуляторный белок или домен ко-локализуются на ДНК и где транскрипционный регуляторный белок или домен регулирует экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок, дополнительно кодирует транскрипционный регуляторный белок или домен, слитый с РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или более РНК, дополнительно кодирует мишень для РНК-связывающего домена, и чужеродная нуклеиновая кислота, кодирующая транскрипционный регуляторный белок или домен, дополнительно кодирует РНК-связывающий домен, слитый с транскрипционным регуляторным белком или доменом.
Согласно одному аспекту, клетка представляет собой эукариотическую клетку. Согласно одному аспекту, клетка представляет собой клетку дрожжей, клетку растения или клетку животного. Согласно одному аспекту, клетка представляет собой клетку млекопитающего.
Согласно одному аспекту, РНК включает от примерно 10 до примерно 500 нуклеотидов. Согласно одному аспекту, РНК включает от примерно 20 до примерно 100 нуклеотидов.
Согласно одному аспекту, транскрипционный регуляторный белок или домен представляет собой активатор транскрипции. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты для того, чтобы лечить заболевание или состояние, причиняющее ущерб здоровью. Согласно одному аспекту, целевая нуклеиновая кислота ассоциирована с заболеванием или состоянием, причиняющим ущерб здоровью. Согласно одному аспекту, транскрипционный регуляторный белок или домен представляет собой репрессор транскрипции. Согласно одному аспекту, транскрипционный регуляторный белок или домен снижает уровень экспрессии целевой нуклеиновой кислоты. Согласно одному аспекту, транскрипционный регуляторный белок или домен снижает уровень экспрессии целевой нуклеиновой кислоты для того, чтобы лечить заболевание или состояние, причиняющее ущерб здоровью. Согласно одному аспекту, целевая нуклеиновая кислота ассоциирована с заболеванием или состоянием, причиняющим ущерб здоровью.
Согласно одному аспекту, одна или более РНК представляет собой направляющую РНК. Согласно одному аспекту, одна или более РНК представляет собой слитую tracrРНК-crРНК.
Согласно одному аспекту, ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
Согласно определенным аспектам, предоставляется способ модулирования экспрессии целевой нуклеиновой кислоты в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей одну или более РНК (рибонуклеиновые кислоты), комплементарную к ДНК (дезоксирибонуклеиновой кислоте), где ДНК включает целевую нуклеиновую кислоту, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей РНК-направляемые не обладающие нуклеазной активностью ДНК-связывающие белки CRISPR-системы Типа II, которые связываются с ДНК и которые направляются одной или более РНК, введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей транскрипционный регуляторный белок или домен, где экспрессируются одна или более РНК, РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок CRISPR-системы Типа II и транскрипционный регуляторный белок или домен, где одна или более РНК, РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок CRISPR-системы Типа II и транскрипционный регуляторный белок или домен ко-локализуются на ДНК, и где транскрипционный регуляторный белок или домен регулирует экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок CRISPR-системы Типа II, дополнительно кодирует транскрипционный регуляторный белок или домен, слитый с РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком CRISPR-системы Типа II. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или более РНК, дополнительно кодирует мишень для РНК-связывающего домена, и чужеродная нуклеиновая кислота, кодирующая транскрипционный регуляторный белок или домен, дополнительно кодирует РНК-связывающий домен, слитый с транскрипционным регуляторным белком или доменом.
Согласно одному аспекту, клетка представляет собой эукариотическую клетку. Согласно одному аспекту, клетка представляет собой клетку дрожжей, клетку растения или клетку животного. Согласно одному аспекту, клетка представляет собой клетку млекопитающего.
Согласно одному аспекту, РНК включает от примерно 10 до примерно 500 нуклеотидов. Согласно одному аспекту, РНК включает от примерно 20 до примерно 100 нуклеотидов.
Согласно одному аспекту, транскрипционный регуляторный белок или домен представляет собой активатор транскрипции. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты для того, чтобы лечить заболевание или состояние, причиняющее ущерб здоровью. Согласно одному аспекту, целевая нуклеиновая кислота ассоциирована с заболеванием или состоянием, причиняющим ущерб здоровью.
Согласно одному аспекту, одна или более РНК представляет собой направляющую РНК. Согласно одному аспекту, одна или более РНК представляет собой слитую tracrРНК-crРНК.
Согласно одному аспекту, ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
Согласно определенным аспектам, предоставляется способ модулирования экспрессии целевой нуклеиновой кислоты в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей одну или более РНК (рибонуклеиновые кислоты), комплементарную к ДНК (дезоксирибонуклеиновой кислоте), где ДНК включает целевую нуклеиновую кислоту, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей не обладающий нуклеазной активностью белок Cas9, который связывается с ДНК и направляется одной или более РНК, введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей транскрипционный регуляторный белок или домен, где экспрессируются одна или более РНК, не обладающий нуклеазной активностью белок Cas9 и транскрипционный регуляторный белок или домен, где одна или более РНК, не обладающий нуклеазной активностью белок Cas9 и транскрипционный регуляторный белок или домен ко-локализуются на ДНК и где транскрипционный регуляторный белок или домен регулирует экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая не обладающий нуклеазной активностью белок Cas9, дополнительно кодирует транскрипционный регуляторный белок или домен, слитый с не обладающим нуклеазной активностью белком Cas9. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или более РНК, дополнительно кодирует мишень для РНК-связывающего домена, и чужеродная нуклеиновая кислота, кодирующая транскрипционный регуляторный белок или домен, дополнительно кодирует РНК-связывающий домен, слитый с транскрипционным регуляторным белком или доменом.
Согласно одному аспекту, клетка представляет собой эукариотическую клетку. Согласно одному аспекту, клетка представляет собой клетку дрожжей, клетку растения или клетку животного. Согласно одному аспекту, клетка представляет собой клетку млекопитающего.
Согласно одному аспекту, РНК включает от примерно 10 до примерно 500 нуклеотидов. Согласно одному аспекту, РНК включает от примерно 20 до примерно 100 нуклеотидов.
Согласно одному аспекту, транскрипционный регуляторный белок или домен представляет собой активатор транскрипции. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты для того, чтобы лечить заболевание или состояние, причиняющее ущерб здоровью. Согласно одному аспекту, целевая нуклеиновая кислота ассоциирована с заболеванием или состоянием, причиняющим ущерб здоровью.
Согласно одному аспекту, одна или более РНК представляет собой направляющую РНК. Согласно одному аспекту, одна или более РНК представляет собой слитую tracrРНК-crРНК.
Согласно одному аспекту, ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
Согласно одному аспекту, предоставляется клетка, которая включает первую чужеродную нуклеиновую кислоту, кодирующую одну или более РНК, комплементарную к ДНК, где ДНК включает целевую нуклеиновую кислоту, вторую чужеродную нуклеиновую кислоту, кодирующую РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок, и третью чужеродную нуклеиновую кислоту, кодирующую транскрипционный регуляторный белок или домен, где одна или более РНК, РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок и транскрипционный регуляторный белок или домен являются членами комплекса, который ко-локализуется на целевой нуклеиновой кислоте.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок, дополнительно кодирует транскрипционный регуляторный белок или домен, слитый с РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или более РНК, дополнительно кодирует мишень для РНК-связывающего домена, и чужеродная нуклеиновая кислота, кодирующая транскрипционный регуляторный белок или домен, дополнительно кодирует РНК-связывающий домен, слитый с транскрипционным регуляторным белком или доменом.
Согласно одному аспекту, клетка представляет собой эукариотическую клетку. Согласно одному аспекту, клетка представляет собой клетку дрожжей, клетку растения или клетку животного. Согласно одному аспекту, клетка представляет собой клетку млекопитающего.
Согласно одному аспекту, РНК включает от примерно 10 до примерно 500 нуклеотидов. Согласно одному аспекту, РНК включает от примерно 20 до примерно 100 нуклеотидов.
Согласно одному аспекту, транскрипционный регуляторный белок или домен представляет собой активатор транскрипции. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, транскрипционный регуляторный белок или домен увеличивает экспрессию целевой нуклеиновой кислоты для того, чтобы лечить заболевание или состояние, причиняющее ущерб здоровью. Согласно одному аспекту, целевая нуклеиновая кислота ассоциирована с заболеванием или состоянием, причиняющим ущерб здоровью.
Согласно одному аспекту, одна или более РНК представляет собой направляющую РНК. Согласно одному аспекту, одна или более РНК представляет собой слитую tracrРНК-crРНК.
Согласно одному аспекту, ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
Согласно определенным аспектам, РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок представляет собой РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок CRISPR-системы Типа II. Согласно определенным аспектам, РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок представляет собой не обладающий нуклеазной активностью белок Cas9.
Согласно одному аспекту, предоставляется способ изменения целевой нуклеиновой кислоты ДНК в клетке, который включает введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, где каждая РНК является комплементарной к расположенным рядом сайтам в целевой нуклеиновой кислоте ДНК, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью, который может представлять собой ортогональный РНК-направляемый ДНК-связывающий белок с никазной активностью, и который направляется двумя или более РНК, где экспрессируются две или более РНК и, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью и где, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью ко-локализуется с двумя или более РНК на целевой нуклеиновой кислоте ДНК и расщепляет одну цепь целевой нуклеиновой кислоты ДНК, обеспечивая образование двух или более смежных одноцепочечных разрывов.
Согласно одному аспекту, предоставляется способ изменения целевой нуклеиновой кислоты ДНК в клетке, который включает введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, где каждая РНК является комплементарной к расположенным рядом сайтам в целевой нуклеиновой кислоте ДНК, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью CRISPR-системы Типа II, который направляется двумя или более РНК, где экспрессируются две или более РНК и, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью CRISPR-системы Типа II и где, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью CRISPR-системы Типа II ко-локализуется с двумя или более РНК на целевой нуклеиновой кислоте ДНК и расщепляет одну цепь целевой нуклеиновой кислоты ДНК, обеспечивая образование двух или более смежных одноцепочечных разрывов.
Согласно одному аспекту, предоставляется способ изменения целевой нуклеиновой кислоты ДНК в клетке, который включает введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, где каждая РНК является комплементарной к расположенным рядом сайтам в целевой нуклеиновой кислоте ДНК, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей, по меньшей мере, один белок Cas9 с никазной активностью, имеющий один неактивный нуклеазный домен, который направляется двумя или более РНК, где экспрессируются две или более РНК и, по меньшей мере, один белок Cas9 с никазной активностью и где, по меньшей мере, один белок Cas9 с никазной активностью ко-локализуется с двумя или более РНК на целевой нуклеиновой кислоте ДНК и расщепляет одну цепь целевой нуклеиновой кислоты ДНК, обеспечивая образование двух или более смежных одноцепочечных разрывов.
Согласно способам изменения целевой нуклеиновой кислоты ДНК, два или более смежных одноцепочечных разрыва находятся на одной и той же цепи двухцепочечной ДНК. Согласно одному аспекту, два или более смежных одноцепочечных разрыва находятся на одной и той же цепи двухцепочечной ДНК и приводят к гомологичной рекомбинации. Согласно одному аспекту, два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК. Согласно одному аспекту, два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и создают двухцепочечные разрывы. Согласно одному аспекту, два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к негомологичному сшиванию концов. Согласно одному аспекту, два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и являются смещенными относительно друг друга. Согласно одному аспекту, два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и являются смещенными относительно друг друга и создают двухцепочечные разрывы. Согласно одному аспекту, два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и являются смещенными относительно друг друга и создают двухцепочечные разрывы, что приводит к негомологичному сшиванию концов. Согласно одному аспекту, способ дополнительно включает введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей последовательность донорной нуклеиновой кислоты, где два или более одноцепочечных разрыва приводят к гомологичной рекомбинации целевой нуклеиновой кислоты с последовательностью донорной нуклеиновой кислоты.
Согласно одному аспекту, предоставляется способ изменения целевой нуклеиновой кислоты ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, где каждая РНК является комплементарной к расположенным рядом сайтам в целевой нуклеиновой кислоте ДНК, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью, и который направляется двумя или более РНК, где экспрессируются две или более РНК и, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью и где, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью ко-локализуется с двумя или более РНК на целевой нуклеиновой кислоте ДНК и расщепляет одну цепь целевой нуклеиновой кислоты ДНК, обеспечивая образование двух или более смежных одноцепочечных разрывов, и где два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, предотвращая, таким образом, экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, предоставляется способ изменения целевой нуклеиновой кислоты ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, где каждая РНК является комплементарной к расположенным рядом сайтам в целевой нуклеиновой кислоте ДНК, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью CRISPR-системы Типа II, и который направляется двумя или более РНК, где экспрессируются две или более РНК и, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью CRISPR-системы Типа II и где, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью CRISPR-системы Типа II ко-локализуется с двумя или более РНК на целевой нуклеиновой кислоте ДНК и расщепляет одну цепь целевой нуклеиновой кислоты ДНК, обеспечивая образование двух или более смежных одноцепочечных разрывов, и где два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, предотвращая, таким образом, экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, предоставляется способ изменения целевой нуклеиновой кислоты ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, где каждая РНК является комплементарной к расположенным рядом сайтам в целевой нуклеиновой кислоте ДНК, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей, по меньшей мере, один белок Cas9 с никазной активностью, имеющий один неактивный нуклеазный домен, и который направляется двумя или более РНК, где экспрессируются две или более РНК и, по меньшей мере, один белок Cas9 с никазной активностью, и где, по меньшей мере, один белок Cas9 с никазной активностью ко-локализуется с двумя или более РНК на целевой нуклеиновой кислоте ДНК и расщепляет одну цепь целевой нуклеиновой кислоты ДНК, обеспечивая образование двух или более смежных одноцепочечных разрывов, и где два или более смежных одноцепочечных разрыва находятся на разных цепях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, предотвращая, таким образом, экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, предоставляется клетка, включающая первую чужеродную нуклеиновую кислоту, кодирующую две или более РНК, где каждая РНК является комплементарной к расположенным рядом сайтам в целевой нуклеиновой кислоте ДНК, и вторую чужеродную нуклеиновую кислоту, кодирующую, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью, и где две или более РНК и, по меньшей мере, один РНК-направляемый ДНК-связывающий белок с никазной активностью являются членами комплекса, который ко-локализуется на целевой нуклеиновой кислоте ДНК.
Согласно одному аспекту, РНК-направляемый ДНК-связывающий белок с никазной активностью представляет собой РНК-направляемый ДНК-связывающий белок с никазной активностью CRISPR-системы Типа II. Согласно одному аспекту, РНК-направляемый ДНК-связывающий белок с никазной активностью представляет собой белок Cas9 с никазной активностью, имеющий один неактивный нуклеазный домен.
Согласно одному аспекту, клетка представляет собой эукариотическую клетку. Согласно одному аспекту, клетка представляет собой клетку дрожжей, клетку растения или клетку животного. Согласно одному аспекту, клетка представляет собой клетку млекопитающего.
Согласно одному аспекту, РНК включает от примерно 10 до примерно 500 нуклеотидов. Согласно одному аспекту, РНК включает от примерно 20 до примерно 100 нуклеотидов.
Согласно одному аспекту, целевая нуклеиновая кислота ассоциирована с заболеванием или состоянием, причиняющим ущерб здоровью.
Согласно одному аспекту, две или более РНК представляют собой направляющие РНК. Согласно одному аспекту, две или более РНК представляют собой слитые tracrРНК-crРНК.
Согласно одному аспекту, целевая нуклеиновая кислота ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
Согласно одному аспекту, способы могут включать одновременное применение ортогональных РНК-направляемых ДНК-связывающих белков с никазной активностью, ортогональных РНК-направляемых ДНК-связывающих белков с нуклеазной активностью, ортогональных РНК-направляемых не обладающих нуклеазной активностью ДНК-связывающих белков. Таким образом, в одной и той же клетке может быть проведено изменение трансляции за счет изменений в ДНК, вносимых с помощью одноцепочечных или двухцепочечных разрывов. Кроме того, в клетку можно ввести одну, или более, или множество экзогенных донорных нуклеиновых кислот с использованием методов введения нуклеиновых кислот в клетки, известных специалистам в данной области техники, таких как электропорация, и в ДНК клетки могут быть введены одна, или более, или множество экзогенных донорных нуклеиновых кислот путем рекомбинации, такой как гомологичная рекомбинация, или других методов, известных специалистам в данной области техники. Таким образом, применение множества описанных здесь ортогональных РНК-направляемых ДНК-связывающих белков позволяет изменить одну клетку путем введения в ДНК одноцепочечных или двухцепочечных разрывов, позволяет ввести в ДНК клетки донорные нуклеиновые кислоты и позволяет регулировать транскрипцию генов.
Другие особенности и преимущества конкретных воплощений настоящего изобретения станут более понятными из описания осуществления приведенных в качестве примера воплощений, приведенных Фигур и Формулы изобретения.
Краткое описание чертежей
Вышеупомянутые и другие особенности и преимущества конкретных воплощений настоящего изобретения будут более понятны из приведенного ниже детального описания иллюстративных воплощений, которое дается в сопровождении соответствующих Фигур, в которых:
Фигура 1A и Фигура 1B представляют собой схематическое изображение РНК-направляемой активации транскрипции. Фигура 1C представляет собой дизайн репортерной конструкции. На Фигуре 1D приведены данные, демонстрирующие, что слитые Cas9N-VP64 обеспечивают РНК-направляемую активацию транскрипции, что можно видеть по результатам как сортировки клеток с активацией флуоресценции (fluorescence-activated cell sorting, FACS), так и иммунофлуоресцентного анализа (immunofluorescence assays, IF). На Фигуре 1E приведены данные, полученные с использованием FACS и IF, которые показывают, что слитые конструкции демонстрируют специфичную к последовательности gРНК (направляющей РНК) активацию транскрипции репортерных конструкций в присутствии Cas9N, MS2-VP64 и gРНК, несущих подходящие MS2 аптамерные сайты связывания. Фигура 1F приводит данные, демонстрирующие индукцию транскрипции индивидуальными gРНК и множественными gРНК.
Фигура 2A описывает методологию для оценки перспективы направленной доставки с помощью комплексов Cas9-gРНК и TALE (transcription activator-like effectors). Фигура 2B показывает данные, демонстрирующие, что комплекс Cas9-gРНК в среднем является устойчивым к 1-3 мутациям в целевых последовательностях. Фигура 2С показывает данные, демонстрирующие, что комплекс Cas9-gРНК в значительной степени нечувствителен к точечным мутациям, за исключением мутаций, локализованных в последовательности мотива, расположенного рядом с протоспейсером (protospacer adjacent motif, РАМ). Фигура 2D показывает данные графика heat plot, демонстрирующие, что введение 2 неправильных оснований достоверно нарушает активность комплекса Cas9-gРНК. Фигура 2Е показывает данные, демонстрирующие, что 18-мерный TALE в среднем является устойчивым к 1-2 мутациям в последовательностях-мишенях. Фигура 2F показывает данные, демонстрирующие, что 18-мерный TALE, как и комплексы Cas9-gРНК, в значительной степени нечувствителен к точечным мутациям в его целевой последовательности. Фигура 2D показывает данные графика heat plot, демонстрирующие, что введение 2 неправильных оснований достоверно нарушает активность 18-мерного TALE.
Фигура 3A показывает в виде схемы дизайн направляющей РНК. Фигура 3B представляет данные, показывающие процент негомологичного сшивания концов для смещенных одноцепочечных разрывов, приводящих к образованию выступающих 5'-концов и для смещенных одноцепочечных разрывов, приводящих к образованию выступающих 3'-концов. Фигура 3C представляет данные, показывающие процент направленной доставки в случае смещенных одноцепочечных разрывов, приводящих к образованию выступающих 5'-концов и в случае смещенных одноцепочечных разрывов, приводящих к образованию выступающих 3'-концов.
Фигура 4A представляет собой схематическое изображение аминокислотных остатков, участвующих в координации металла в RuvC PDB ID: 4EP4 (синий) положение D7 (левый рисунок), схематическое изображение HNH эндонуклеазных доменов из PDB IDs: 3M7K (оранжевый) и 4H9D (голубой), включая координируемый ион Mg (серая сфера) и ДНК из 3M7K (пурпурная) (средний рисунок), и список анализируемых мутаций (правый рисунок). Фигура 4B представляет данные, показывающие не обнаруживаемую нуклеазную активность для мутантов Cas9 m3 и m4, а также их соответствующих слитых конструкций с VP64. Фигура 4C представляет собой анализ данных Фигуры 4B с более высоким разрешением.
Фигура 5A представляет собой схему определения гомологичной рекомбинации для измерения активности Cas9-gРНК. Фигура 5B показывает направляющие РНК со случайными вставками в последовательность и процент гомологичной рекомбинации.
Фигура 6A представляет собой схематическое изображение направляющих РНК для гена OCT4. Фигура 6B показывает активацию транскрипции для конструкции промотор - репортерный ген люциферазы. Фигура 6C показывает активацию транскрипции посредством количественной ПЦР эндогенных генов.
Фигура 7A представляет собой схематическое изображение направляющих РНК для гена REX1. Фигура 7B показывает активацию транскрипции для конструкции промотор-репортерный ген люциферазы. Фигура 7C показывает активацию транскрипции посредством qPCR эндогенных генов.
Фигура 8A представляет в виде диаграммы схему проведения анализа с высоким уровнем специфичности для расчета нормализованных уровней экспрессии. Фигура 8B представляет данные распределения процента сайтов связывания в зависимости от числа несовпадений оснований, сделанных в смещенной библиотеке конструкций (biased construct library). Слева: Теоретическое распределение. Справа: Распределение, полученное с использованием реальной библиотеки конструкций TALE. Фигура 8С представляет данные распределения процента подсчета тагов, агрегированных с сайтами связывания, в зависимости от числа несовпадений. Слева: Распределение, полученное для образца положительного контроля. Справа: Распределение, полученное для образца, в котором был индуцирован неконтрольный TALE.
Фигура 9A представляет данные для анализа перспективы направленной доставки комплекса Cas9-gРНК, показывающие, что комплекс является устойчивым к 1-3 мутациям в целевой последовательности. Фигура 9В представляет данные для анализа перспективы направленной доставки комплекса Cas9-gРНК, показывающие его нечувствительность к точечным мутациям, за исключением мутаций, локализованных в последовательности РАМ. Фигура 9С показывает данные графика heat plot для анализа перспективы направленной доставки комплекса Cas9-gРНК, демонстрирующие, что введение 2 неправильных оснований достоверно нарушает активность комплекса. Фигура 9D представляет данные опосредованного нуклеазой определения HR, подтверждающие, что предсказанным РАМ для Cas9 из S. pyogenes является NGG и также NAG.
Фигура 10A представляет данные опосредованного нуклеазой определения HR, подтверждающие, что 18-мерные TALE устойчивы к множественным мутациям в их целевых последовательностях. Фигура 10B Фигура 9A представляет данные анализа перспективы направленной доставки TALE трех разных размеров (18-мерный, 14-мерный и 10-мерный). Фигура 10С представляет данные для 10-мерных TALE, показывающие разрешение практически на уровне несовпадения одного основания. Фигура 10D представляет данные графика heat plot для 10-мерных TALE, показывающие разрешение практически на уровне несовпадения одного основания.
Фигура 11A представляет сконструированные направляющие РНК. Фигура 11B представляет процент негомологичного сшивания концов для разных направляющих РНК.
Фигуры 12A-12F показывают сравнение и характеристику предположительно ортогональных белков Cas9. Фигура 12A: последовательности повторов SP, ST1, NM и TD. Основания выделены цветом, чтобы указать степень консервативности. Фигура 12B: плазмиды, использованные для характеристики белков Cas9 в E. coli. Фигура 12C: Содержание функциональных РАМ в библиотеке снижается вследствие разрезания Cas9, когда спейсер и протоспейсер совпадают. Фигура 12D: Cas9 не разрезает ДНК, когда направляющий плазмидный спейсер и протоспейсер библиотеки не совпадают. Фигура 12D: Нефункциональные РАМ никогда не разрезаются или их содержание не снижается. Фигура 12F: Схема проведения селекции для идентификации РАМ. Клетки, экспрессирующие белок Cas9 и одну или две спейсер-содержащие направляющие плазмиды, были трансформированы одной или двумя библиотеками с соответствующими протоспейсерами, после чего была проведена селекция на антибиотике. Оставшиеся нерасщепленными плазмиды были подвергнуты глубокому секвенированию. Опосредованное Cas9 уменьшение количества РАМ было количественно измерено путем сравнения относительного содержания каждой последовательности в случае совпадающих по сравнению с несовпадающими библиотеками протоспейсеров.
Фигуры 13A-13F показывают уменьшение содержания функциональных смежных с протоспейсером мотивов (protospacer-adjacent-motifs, PAM) в библиотеках за счет действия белков Cas9. Логарифм частоты каждого основания в каждом положении для совпадающих пар спейсер-протоспейсер отложен относительно контрольных условий, в которых спейсер и протоспейсер не совпадают. Результаты отражают среднее значение истощения библиотек NM (Фигура 13 A), ST1 (Фигура 13B) и TD (Фигура 13C) на основании двух разных последовательностей протоспейсера (Фигура 13D). Уменьшение содержания специфических последовательностей для каждого протоспейсера отложено по отдельности для каждого белка Cas9 (Фигуры 13E-13F).
Фигуры 14A-14B показывают репрессию транскрипции, опосредованную NM. Фигура 14A: Для количественной оценки репрессии использовалась репортерная плазмида. Фигура 14B: Нормализованная флуоресценция клеток для совпадающих и несовпадающих пар спейсер-протоспейсер. Величина разброса показывает стандартное отклонение для пяти повторов.
Фигура 15 показывает ортогональное распознавание crРНК в E. coli. В клетки со всеми комбинациями Cas9 и crРНК были введены плазмиды, несущие совпадающий или несовпадающий протоспейсер и подходящий PAM. Было высеяно достаточное количество клеток для надежного получения колоний для совпадающих пар спейсер и протоспейсер и подсчета общего числа колоний, используемых для расчета уменьшения содержания (во сколько раз).
Фигуры 16A-16B показывают Cas9-опосредуемое редактирование генов в клетках человека. Фигура 16A: Для количественной оценки эффективности редактирования гена использовалось определение гомологичной рекомбинации. Опосредуемые Cas9 двухцепочечные разрывы в протоспейсере стимулируют восстановление нарушенной кассеты зеленого флуоресцентного белка (GFP) с использованием донорной матрицы, что приводит к появлению клеток с интактным GFP. Для того, чтобы предоставить корректные РАМ для каждого белка Cas9, использовались три разные матрицы. Количество флуоресцирующих клеток подсчитывалось с помощью проточной цитометрии. Фигура 16B: Результаты сортировки клеток для NM, ST1 и TD в комбинации с соответствующими каждой их них sgРНК. Последовательность протоспейсера и PAM для каждого белка Cas9 показана над каждым из графиков. Эффективность восстановления приведена в верхнем правом углу каждого графика.
Фигуры 17A- 17B показывают активацию транскрипции в клетках человека. Фигура 17A: Репортерные конструкции для активации транскрипции содержали tdTomato под минимальным промотором. Последовательности протоспейсера и PAM были помещены перед минимальным промотором. Связывание с протоспейсерами не обладающих нуклеазной активностью слитых белков Cas9-VP64 приводило к активации транскрипции и усилению флуоресценции.
Фигура 17B: Клетки, трансфецированные всеми комбинациями активатора Cas9 и sgРНК и визуализация флуоресценции tdTomato. Активация траскрипции наблюдалась только тогда, когда каждый Cas9 был в паре со своей собственной sgРНК.
Осуществление изобретения
Приводимые здесь для подтверждения ссылки даны в виде надстрочных символов. Необходимо понимать, что такая надстрочная ссылка относится к публикации, которая полностью поддерживает определенное утверждение.
Системы CRISPR-Cas бактерий и архей обеспечивают приобретенный иммунитет путем включения фрагментов вирусной или плазмидной ДНК в локусы CRISPR и использования транскрибируемых crРНК для направления нуклеаз с целью деградации гомологичных последовательностей1,2. CRISPR-системы Типа II представляют собой тройной комплекс Cas9-нуклеазы с crРНК и tracrРНК (trans-activating crРНК), который связывается с и расщепляет протоспейсерные последовательности двухцепочечной ДНК, которые совпадают со спейсерной crРНК и также содержат короткий смежный с протоспейсером мотив (protospacer-adjacent motif, PAM) 3, 4. Сливание crРНК и tracrРНК обеспечивает получение единой направляющей РНК (sgРНК), которая является достаточной для направленной доставки Cas94.
Поскольку белок Cas9 является РНК-направляемой нуклеазой и никазой, он был адаптирован для направленного редактирования генов5-9 и селекции10 в разных организмах. Хотя способность к успешной трансформации таких подходов является спорной, не обладающие нуклеазной активностью варианты Cas9 являются пригодными для регуляторных целей, поскольку они способны локализовать белки и РНК практически в любом месте последовательностей двухцепочечных ДНК, обеспечивая бесконечное разнообразие для контролирования биологических систем11-17. Начиная с направленной репрессии генов через промотор и подавление 5'-нетранслируемых областей (5'-UTR-obstruction) у бактерий18, опосредуемая Cas9 регуляция распространилась на активацию транскрипции с помощью вовлечения VP6419 в клетках человека. Согласно определенным аспектам, описанные здесь ДНК-связывающие белки, включая ортогональные РНК-направляемые ДНК-связывающие белки, такие как ортогональные белки Cas9, могут применяться как активаторы транскрипции, репрессоры, флуоресцентные белковые метки, хромосомные метки и другие многочисленные инструменты, известные специалистам в данной области техники. Согласно этому аспекту, применение ортогонального белка Cas9 позволяет обеспечить генетическую модификацию с использованием любого или всех из транскрипционных активаторов, репрессоров, флуоресцентных белковых меток, хромосомных меток и других многочисленных инструментов, известных специалистам в данной области техники. Таким образом, аспект настоящего изобретения направлен на применение ортогональных белков Cas9 для множественной РНК-направляемой активации транскрипции, репрессии транскрипции и редактирования генов.
Воплощения настоящего изобретения направлены на характеристику и демонстрацию ортогональности (независимости) между множеством белков Cas9 в бактериях и клетках человека. Такие ортогональные РНК-направляемые ДНК-связывающие белки могут применяться во множестве или одновременно и независимо обеспечивать регуляцию транскрипции, мечение или редактирование множества генов в ДНК индивидуальных клеток.
Согласно одному аспекту, идентифицировано множество ортогональных белков Cas9 в пределах одного семейства CRISPR-систем. Хотя они и являются родственными, показательные белки Cas9 из S. pyogenes, N. meningitidis, S. thermophilus и T. denticola имеют размер от 3,25 до 4,6 т.п.о. в длину и распознают абсолютно разные последовательности PAM.
Воплощения настоящего изобретения основаны на применении ДНК-связывающих белков для того, чтобы ко-локализовать транскрипционные регуляторные белки или домены на ДНК таким образом, чтобы обеспечить регуляцию целевой нуклеиновой кислоты. Такие ДНК-связывающие белки хорошо известны специалистам в данной области техники для связывания с ДНК для разных целей. Такие ДНК-связывающие белки могут быть природными белками. Такие ДНК-связывающие белки, входящие в рамки настоящего изобретения, включают такие, которые могут направляться РНК, которая называется здесь направляющей РНК. Согласно этому аспекту, направляющая РНК и РНК-направляемый ДНК-связывающий белок формируют комплекс, который ко-локализуется на ДНК. Согласно определенным аспектам, ДНК-связывающий белок может представлять собой не обладающий нуклеазной активностью ДНК-связывающий белок. Согласно этому аспекту, не обладающий нуклеазной активностью ДНК-связывающий белок может быть получен путем изменения или модификации ДНК-связывающего белка, обладающего нуклеазной активностью. Такие ДНК-связывающие белки, обладающие нуклеазной активностью, известны специалистам в данной области техники, и включают природные ДНК-связывающие белки, обладающие нуклеазной активностью, такие как белки Cas9, присутствующие, например, в CRISPR-системах Типа II. Такие белки Cas9 и CRISPR-системы Типа II хорошо изучены в данной области техники. Смотри: Makarova et al., Nature Reviews, Microbiology, Vol. 9, June 2011, pp. 467-477, включая всю дополнительную информацию, которая включается в настоящее изобретение путем отсылки во всей своей полноте.
Согласно определенному аспекту, предоставляются способы для идентификации двух, или более, или множества, или набора ортогональных ДНК-связывающих белков, таких как ортогональные РНК-направляемые ДНК-связывающие белки, таких как ортогональные РНК-направляемые ДНК-связывающие белки CRISPR-системы Типа II, таких как ортогональные белки Cas9, каждый из которых может обладать или не обладать нуклеазной активностью. Согласно определенным аспектам, два, или более, или множество, или набор ортогональных ДНК-связывающих белков может применяться с соответствующими направляющими РНК для того, чтобы одновременно и независимо регулировать гены или редактировать нуклеиновые кислоты в клетке. Согласно определенным аспектам, в клетку могут быть введены нуклеиновые кислоты, которые кодируют два, или более, или множество, или набор ортогональных ДНК-связывающих белков, соответствующие направляющие РНК, и два, или более, или множество, или набор соответствующих транскрипционных регуляторов или доменов. Таким образом, в одной и той же клетке можно параллельно выбрать много генов в качестве мишени для регуляции или редактирования. Методы редактирования геномной ДНК хорошо известны специалистам в данной области техники.
Показательные ДНК-связывающие белки, обладающие нуклеазной активностью, обеспечивают разрыв одной цепи или разрезаний двух цепей двухцепочечной ДНК. Такая нуклеазная активность может обеспечиваться ДНК-связывающим белком, имеющим одну или более полипептидных последовательностей, обладающих нуклеазной активностью. Такие показательные ДНК-связывающие белки могут иметь два отдельных нуклеазных домена, каждый из которых отвечает за двухцепочечный разрыв или одноцепочечный разрыв определенной цепи двухцепочечной ДНК. Показательные полипептидные последовательности, обладающие нуклеазной активностью, известны специалистам в данной области техники, и включают родственный McrA-HNH-нуклеазе домен и RuvC-подобный нуклеазный домен. Таким образом, показательными ДНК-связывающими белками являются такие белки, которые в природе содержат один или более родственных McrA-HNH-нуклеазе доменов и RuvC-подобный нуклеазный домен. Согласно определенным аспектам, ДНК-связывающий белок является измененным или модифицированным другим способом для того, чтобы инактивировать нуклеазную активность. Такое изменение или модификация включают изменение одной или более аминокислот для инактивации нуклеазной активности или нуклеазного домена. Такая модификация включает удаление полипептидной последовательности или полипептидных последовательностей, обладающих нуклеазной активностью, то есть, нуклеазного домена, так, что полипептидная последовательность или полипептидные последовательности, обладающие нуклеазной активностью, то есть, нуклеазный домен, удаляются из ДНК-связывающего белка. Другие модификации, обеспечивающие инактивацию нуклеазной активности, станут понятны специалисту в данной области техники из воплощения настоящего изобретения. Таким образом, не обладающий нуклеазной активностью ДНК-связывающий белок включает полипептидные последовательности, модифицированные для инактивации нуклеазной активности, или это белок с удаленной полипептидной последовательностью или последовательностями для инактивации нуклеазной активности. Не обладающий нуклеазной активностью ДНК-связывающий белок сохраняет способность связываться с ДНК, даже хотя его нуклеазная активность инактивирована. Таким образом, ДНК-связывающий белок включает полипептидную последовательность или последовательности, необходимые для связывания ДНК, но может не содержать одной, или более, или всех нуклеазных последовательностей, проявляющих нуклеазную активность. Таким образом, ДНК-связывающий белок включает полипептидную последовательность или последовательности, необходимые для связывания ДНК, но может содержать одну, или более, или все нуклеазные последовательностей, проявляющие нуклеазную активность, в инактивированном виде.
Согласно одному аспекту, ДНК-связывающий белок, имеющий два или более нуклеазных домена, может быть модифицирован или изменен таким образом, чтобы инактивировать все, кроме одного, нуклеазные домены. Такой модифицированный или измененный ДНК-связывающий белок обозначается как ДНК-связывающий белок с никазной активностью, чтобы указать, что такой ДНК-связывающий белок разрезает или расщепляет только одну цепь двухцепочечной ДНК. Когда ДНК-связывающий белок с никазной активностью направляется к ДНК с помощью направляющей РНК, он обозначается как РНК-направляемый ДНК-связывающий белок с никазной активностью.
Показательный ДНК-связывающий белок представляет собой РНК-направляемый ДНК-связывающий белок CRISPR-системы Типа II, который не обладает нуклеазной активностью. Показательный ДНК-связывающий белок представляет собой не обладающий нуклеазной активностью белок Cas9. Показательный ДНК-связывающий белок представляет собой белок Cas9 с никазной активностью.
В бактерии S. pyogenes Cas9 делает двухцепочечный разрыв с тупыми концами на 3 пары оснований выше смежного с протоспейсером мотива (РАМ) в ходе процесса, который обеспечивается двумя каталитическими доменами белка: HNH-доменом, который расщепляет комплементарную цепь ДНК, и RuvC-подобным доменом, который расщепляет не комплементарную цепь. Смотри: Jinke et al., Science 337, 816-821 (2012), которая включена в настоящее изобретение путем отсылки во всей своей полноте. Известно, что белки Cas9 присутствуют во многих CRISPR-системах Типа II, включая таковые, которые были указаны в дополнительной информации к статье Makarova et al., Nature Reviews, Microbiology, Vol. 9, June 2011, pp. 467-477: Methanococcus maripaludis C7; Corynebacterium diphtheriae; Corynebacterium efficiens YS-314; Corynebacterium glutamicum ATCC 13032 Kitasato; Corynebacterium glutamicum ATCC 13032 Bielefeld; Corynebacterium glutamicum R; Corynebacterium kroppenstedtii DSM 44385; Mycobacterium abscessus ATCC 19977; Nocardia farcinica IFM10152; Rhodococcus erythropolis PR4; Rhodococcus jostii RHA1; Rhodococcus opacus B4 uid36573; Acidothermus cellulolyticus 11B; Arthrobacter chlorophenolicus A6; Kribbella flavida DSM 17836 uid43465; Thermomonospora curvata DSM 43183; Bifidobacterium dentium Bdl; Bifidobacterium longum DJO10A; Slackia heliotrinireducens DSM 20476; Persephonella marina EX HI; Bacteroides fragilis NCTC 9434; Capnocytophaga ochracea DSM 7271; Flavobacterium psychrophilum JIP02 86; Akkermansia muciniphila ATCC BAA 835; Roseiflexus castenholzii DSM 13941; Roseiflexus RS1; Synechocystis PCC6803; Elusimicrobium minutum Peil91; некультивируемая бактерия группы Termite 1 филотипа Rs D17; Fibrobacter succinogenes S85; Bacillus cereus ATCC 10987; Listeria innocua; Lactobacillus casei; Lactobacillus rhamnosus GG; Lactobacillus salivarius UCC118; Streptococcus agalactiae A909; Streptococcus agalactiae NEM316; Streptococcus agalactiae 2603; Streptococcus dysgalactiae equisimilis GGS 124; Streptococcus equi zooepidemicus MGCS10565; Streptococcus gallolyticus UCN34 uid46061; Streptococcus gordonii Challis subst CH1; Streptococcus mutans N 2025 uid46353; Streptococcus mutans; Streptococcus pyogenes Ml GAS; Streptococcus pyogenes MGAS5005; Streptococcus pyogenes MGAS2096; Streptococcus pyogenes MGAS9429; Streptococcus pyogenes MGAS10270; Streptococcus pyogenes MGAS6180; Streptococcus pyogenes MGAS315; Streptococcus pyogenes SSI-1; Streptococcus pyogenes MGAS10750; Streptococcus pyogenes NZ131; Streptococcus thermophiles CNRZ1066; Streptococcus thermophiles LMD-9; Streptococcus thermophiles LMG 18311; Clostridium botulinum A3 Loch Maree; Clostridium botulinum B Eklund 17B; Clostridium botulinum Ba4 657; Clostridium botulinum F Langeland; Clostridium cellulolyticum H10; Finegoldia magna ATCC 29328; Eubacterium rectale ATCC 33656; Mycoplasma gallisepticum; Mycoplasma mobile 163K; Mycoplasma penetrans; Mycoplasma synoviae 53; Streptobacillus moniliformis DSM 12112; Bradyrhizobium BTAil; Nitrobacter hamburgensis X14; Rhodopseudomonas palustris BisB18; Rhodopseudomonas palustris BisB5; Parvibaculum lavamentivorans DS-1; Dinoroseobacter shibae DFL 12; Gluconacetobacter diazotrophicus Pal 5 FAPERJ; Gluconacetobacter diazotrophicus Pal 5 JGI; Azospirillum B510 uid46085; Rhodospirillum rubrum ATCC 11170; Diaphorobacter TPSY uid29975; Verminephrobacter eiseniae EF01-2; Neisseria meningitides 053442; Neisseria meningitides alphal4; Neisseria meningitides Z2491; Desulfovibrio salexigens DSM 2638; Campylobacter jejuni doylei 269 97; Campylobacter jejuni 81116; Campylobacter jejuni; Campylobacter lari RM2100; Helicobacter hepaticus; Wolinella succinogenes; Tolumonas auensis DSM 9187; Pseudoalteromonas atlantica T6c; Shewanella pealeana ATCC 700345; Legionella pneumophila Paris; Actinobacillus succinogenes 130Z; Pasteurella multocida; Francisella tularensis novicida U112; Francisella tularensis holarctica; Francisella tularensis FSC 198; Francisella tularensis; Francisella tularensis WY96-3418; и Treponema denticola ATCC 35405. Таким образом, аспекты настоящего изобретения направлены на белок Cas9, присутствующий в CRISPR-системе Типа II, который был сделан не обладающим нуклеазной активностью или который был сделан обладающим никазной активностью, как здесь описано.
Белок Cas9 мог встречаться в литературе специалисту в данной области техники как белок Csnl. Последовательность белка Cas9 из S. pyogenes, который использовался в приведенных здесь экспериментах, приведена ниже. Смотри статью: Deltcheva et al., Nature 471, 602-607 (2011), которая включается в настоящее изобретение путем отсылки во всей своей полноте.
MDKKYSIGLDIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIRRNLIGALLFDSGETAE
ATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFG
NIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSD
VDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGN
LIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAI
LLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYA
GYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELH
AILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEE
VVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFL
SGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKI
IKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWG
RLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSL
HEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRER
MKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDH
IVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNL
TKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKS
KLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRK
MIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDF
ATVRKVLSMPQVNVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVA
YSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPK
YSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVE
QHKHYLEIIEQISEFSKRVILADANLDKVLSAYNKHRDKΡIREQAENIIHLFTLTNLGA
PAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD
Согласно определенным аспектам описанных здесь способов РНК-направляемой регуляции генома, Cas9 является измененным для снижения, значительного снижения или полного устранения нуклеазной активности. Таким Cas9 может быть ортогональный Cas9, когда предусматривается применение более чем одного белка Cas9. В этом контексте, в описанных здесь способах могут применяться два, или более, или множество, или набор ортогональных белков Cas9. Согласно одному аспекту, нуклеазная активность Cas9 снижена, существенно снижена или полностью устранена путем изменения RuvC-нуклеазного домена или HNH-нуклеазного домена. Согласно одному аспекту, инактивирован RuvC-нуклеазный домен. Согласно одному аспекту, инактивирован HNH-нуклеазный домен. Согласно одному аспекту, инактивированы RuvC-нуклеазный домен и HNH-нуклеазный домен. Согласно дополнительному аспекту, предоставляются белки Cas9, в которых инактивированы RuvC-нуклеазный домен и HNH-нуклеазный домен. Согласно дополнительному аспекту, предоставляются не обладающие нуклеазной активностью белки Cas9, в которых инактивированы RuvC-нуклеазный домен и HNH-нуклеазный домен. Согласно дополнительному аспекту, предоставляется Cas9 с никазной активностью, в котором инактивирован либо RuvC-нуклеазный домен, либо HNH-нуклеазный домен, тогда как оставшийся нуклеазный домен обладает нуклеазной активностью. В таком случае расщепляется или разрезается только одна цепь двухцепочечной ДНК.
Согласно дополнительному аспекту, предоставляются не обладающие нуклеазной активностью белки Cas9, в которых одна или более аминокислот в Cas9 изменены или удалены другими способами для получения не обладающих нуклеазной активностью белков Cas9. Согласно одному аспекту, данные аминокислоты включают D10 и H840. Смотри: Jinke et al., Science 337, 816-821 (2012). Согласно дополнительному аспекту, данные аминокислоты включают D839 и N863. Согласно одному аспекту, одна, или более, или все аминокислоты из D10, H840, D839 и H863 заменены на аминокислоту, которая снижает, существенно устраняет или устраняет нуклеазную активность. Согласно одному аспекту, одна, или более, или все аминокислоты из D10, H840, D839 и H863 заменены на аланин. Согласно одному аспекту, белок Cas9, в котором одна, или более, или все аминокислоты из D10, H840, D839 и H863 заменены на аминокислоту, которая снижает, существенно устраняет или устраняет нуклеазную активность, такую как аланин, обозначается как не обладающий нуклеазной активностью Cas9 или Cas9N, который проявляет сниженную или не обладает нуклеазной активностью, или нуклеазная активность отсутствует или практически отсутствует на определяемых уровнях. Согласно этому аспекту, нуклеазная активность Cas9N может быть неопределяемой с использованием известных методов определения, то есть ниже уровня чувствительности известных методов определения.
Согласно одному аспекту, не обладающий нуклеазной активностью белок Cas9 включает его гомологи и ортологи, которые сохраняют способность белка связываться с ДНК и быть направляемыми РНК. Согласно одному аспекту, не обладающий нуклеазной активностью белок Cas9 включает последовательность, которая установлена для природного белка Cas9 из S. рyogenes, и содержит одну, или более или все аминокислоты D10, H840, D839 и H863, замененные на аланин, и белковые последовательности, гомологичные ему, по меньшей мере, на 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 98% или 99%, и является ДНК-связывающим белком, таким как РНК-направляемый ДНК-связывающий белок.
Согласно одному аспекту, не обладающий нуклеазной активностью белок Cas9 включает последовательность, которая установлена для природного белка Cas9 из S. рyogenes, за исключением белковой последовательности RuvC-нуклеазного домена и HNH-нуклеазного домена, а также белковые последовательности, гомологичные ему, по меньшей мере, на 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 98% или 99%, и является ДНК-связывающим белком, таким как РНК-направляемый ДНК-связывающий белок. Таким образом, аспекты настоящего изобретения включают белковую последовательность, ответственную за связывание с ДНК, например, за ко-локализацию с направляющей РНК и связывание с ДНК, и гомологичные им белковые последовательности, и не обязательно включает белковые последовательности RuvC-нуклеазного домена и HNH-нуклеазного домена (до той степени, которая не является необходимой для связывания с ДНК), и эти домены могут быть либо инактивированы, либо удалены из белковой последовательности природного белка Cas9 для получения не обладающего нуклеазной активностью белка Cas9.
Для целей настоящего изобретения Фигура 4A показывает металл-координирующие аминокислотные остатки в известной структуре белка, гомологичного Cas9. Остатки помечены на основании их положения в последовательности Cas9. Слева: структура RuvC, PDB ID: 4EP4 (синий цвет), положение D7, которое соответствует положению D10 в последовательности Cas9, выделено в положении, участвующем в координации иона Mg. В центре: Структуры HNH-эндонуклеазных доменов из PDB IDs: 3M7K (оранжевый) и 4H9D (голубой), включающие координируемый ион Mg (серая сфера) и ДНК из 3M7K (пурпурный). Остатки D92 и N113 и 4H9D положения D53 и N77 в 3M7K, которые имеют гомологию последовательности с аминокислотами Cas9 D839 и N863, показаны в виде палочек. Справа: Список полученных мутантов, проанализированных на нуклеазную активность: Cas9 дикого типа, Cas9m1, в котором D10 заменен на аланин; Cas9m2, в котором D10 заменен на аланин и H840 заменен на аланин; Cas9m3, в котором D10 заменен на аланин, H840 заменен на аланин и D839 заменен на аланин; и Cas9m4, в котором D10 заменен на аланин, H840 заменен на аланин, D839 заменен на аланин и N863 заменен на аланин.
Как показано на Фигуре 4B, мутанты Cas9 m3 и m4 и также их соответствующие слитые конструкции с VP64 демонстрируют не обнаруживаемую нуклеазную активность, что показано путем глубокого секвенирования в целевых локусах. Графики показывают зависимость частоты мутаций от положения в геноме, где красные линии отмечают мишень gРНК. Фигура 4C представляет собой данные Фигуры 4B, проанализированные с более высоким разрешением, и подтверждает, что вид мутаций (mutation landscape) дает сравнимый профиль с не модифицированными локусами.
Согласно одному аспекту, предоставляется сконструированная Cas9-gРНК-система, которая обеспечивает РНК-направляемую регуляцию генома в клетках человека путем привязывания транскрипционных активаторных доменов либо к не обладающему нуклеазной активностью белку Cas9, либо к гидовым РНК. Согласно одному аспекту настоящего изобретения, один или более транскрипционных регуляторных белков или доменов (эти термины в используемом здесь значении являются взаимозаменяемыми) слиты или соединены другим способом с не обладающим нуклеазной активностью белком Cas9 или одной, или более направляющими РНК (gРНК). Транскрипционные регуляторные домены соответствуют целевым локусам. Таким образом, аспекты настоящего изобретения включают способы и материалы для локализации транскрипционных регуляторных доменов в целевых локусах путем слияния, соединения или объединения таких доменов либо с Cas9N, либо с gРНК.
Согласно одному аспекту, предоставляется слитый Cas9N белок, способный к активации транскрипции. Согласно одному аспекту, активаторный домен VP64 (смотри статью: Zhang et al., Nature Biotechnology 29, 149-153 (2011), которая включается в настоящее изобретение путем отсылки во всей своей полноте) объединяется, сливается, связывается или прикрепляется другим способом к С-концу Cas9N. Согласно одному способу, транскрипционный регуляторный домен доставляется к конкретному сайту целевой геномной ДНК с помощью Cas9N-белка. Согласно одному способу, Cas9N, слитый с транскрипционным регуляторным доменом, доставляется в клетку с одной или более направляющими РНК. Cas9N, слитый с транскрипционным регуляторным доменом, связывается с, или около, целевой геномной ДНК. Одна или более направляющие РНК связываются с, или около, целевой геномной ДНК. Транскрипционный регуляторный домен регулирует экспрессию целевого гена. Согласно специфическому аспекту, слитый белок Cas9N-VP64 активирует транскрипцию репортерных конструкций, когда комбинируется с направляющими РНК, направляющими последовательности к промотору, демонстрируя, таким образом, РНК-направляемую активацию транскрипции.
Согласно одному аспекту, предоставляется слитый с gРНК белок, способный активировать транскрипцию Согласно одному аспекту, VP64 активаторный домен объединяется, сливается, связывается или прикрепляется другим способом к gРНК. Согласно одному способу, транскрипционный регуляторный домен доставляется к конкретному сайту целевой геномной ДНК с помощью gРНК. Согласно одному способу, gРНК, слитая с транскрипционным регуляторным доменом, доставляется в клетку вместе с белком Cas9N. Cas9N связывается с, или около, целевой геномной ДНК. Одна или более направляющие РНК, слитая с транскрипционным регуляторным белком или доменом, связывается с, или около, целевой геномной ДНК. Транскрипционный регуляторный домен регулирует экспрессию целевого гена. Согласно специфическому аспекту, Cas9N-белок и gРНК, слитые с транскрипционным регуляторным доменом, активирует транскрипцию репортерных конструкций, демонстрируя, таким образом, РНК-направляемую активацию транскрипции.
Были сконструированы gРНК-метки, способные обеспечивать регуляцию транскрипции, для идентификации участков gРНК, устойчивых к модификациям путем введения случайных последовательностей, в gРНК и последующего измерения функциональной активности Cas9. Было установлено, что gРНК, имеющие вставки случайных последовательностей либо в 5'-конце участка crРНК, либо в 3'-конце участка tracrРНК химерной gРНК, сохраняли функциональную активность, тогда как вставки в каркасную часть tracrРНК химерной gРНК приводили к потере функциональной активности. Смотри Фигуры 5A-5B, показывающие устойчивость gРНК к случайным вставкам оснований. Фигура 5A представляет собой схему определения гомологичной рекомбинации (HR) для измерения активности Cas9-gРНК. Как показано в Фигуре 5B, gРНК, имеющие вставки случайных последовательностей либо в 5'-конце участка crРНК, либо в 3'-конце участка tracrРНК химерной gРНК, сохраняют функциональную активность, тогда как вставки в каркасную часть tracrРНК химерной gРНК приводят к потере функциональной активности. Положения вставок в последовательности gРНК выделены красным цветом нуклеотидов. Не желая быть связанным с конкретной научной теорией, можно предположить, что увеличенная активность при случайных вставках оснований в 5'-конец может быть следствием увеличенного времени полужизни более длинных gРНК. Для присоединения VP64 к gРНК, к 3'-концу gРНК были прикреплены две копии белка оболочки бактериофага MS2, связывающего торчащие шпильки РНК. Смотри статью: Fusco et al., Current Biology: CB13, 161-167 (2003), которая включается в настоящее изобретение путем отсылки во всей своей полноте. Такие химерные gРНК экспрессировались вместе с Cas9N и слитым белком MS2-VP64. Специфичная к определенной последовательности активация транскрипции репортерных конструкций наблюдалась в присутствии всех 3 компонентов.
Фигура 1A представляет собой схему РНК-направляемой активации транскрипции. Как показано на Фигуре 1A, для создания Cas9N-слитого белка, способного к активации транскрипции, VP64-активационный домен был прикреплен прямо к С-концу белка Cas9N. Как показано на Фигуре 1В, для создания конструкций gРНК, способных к активации транскрипции, к 3'-концу gРНК были прикреплены две копии белка оболочки бактериофага MS2, связывающего торчащие шпильки РНК. Такие химерные gРНК экспрессировались вместе с Cas9N и слитым белком MS2-VP64. На Фигуре 1C представлен дизайн репортерных конструкций, которые использовались для измерения активации транскрипции. Две репортерные конструкции содержали разные сайты-мишени для gРНК, но имели общий контрольный сайт мишени для TALE-TF. Как показано на Фигуре 1D, слитые белки Cas9N-VP64 демонстрируют РНК-направляемую активацию транскрипции, что было определено как при сортировке клеток с активацией флуоресценции (FACS), так и при проведении иммунофлуоресцентного анализа (IF). Более конкретно, тогда как контрольный TALE-TF активировал оба репортера, слитой белок Cas9N-VP64 активировал репортеры специфически в зависимости от последовательности gРНК. Как показано на Фигуре 1Е, активация транскрипции репортерных конструкций, специфичная к последовательности gРНК, наблюдается только в присутствии всех 3 компонентов: Cas9N, MS2-VP64 и gРНК, несущей подходящие аптамерные сайты связывания MS2, что видно по данным как FACS, так и IF.
Согласно определенным аспектам, предоставляются способы для регуляции эндогенных генов с применением Cas9N, одной или более gРНК и транскрипционного регуляторного белка или домена. Согласно одному аспекту, эндогенным геном может быть любой желаемый ген, называемый здесь целевым геном. Согласно одному показательному аспекту, целевые гены для регуляции включали ZFP42 (REX1) и POU5F1 (OCT4), которые оба представляют собой строго регулируемые гены, участвующие в поддержании плюрипотентности. Как показано на Фигуре 1F, для гена REXl были сконструированы десять gРНК, мишенями которых является отрезок ДНК размером ~5 тысяч пар оснований, расположенный перед сайтом начала транскрипции (сайты, гиперчувствительные к ДНКазе, выделены зеленым цветом). Активацию транскрипции определяли с использованием либо репортерной конструкции промотор - люцифераза (смотри статью: Takahashi et al., Cell 131 861-872 (2007), которая включается в настоящее изобретение путем отсылки во всей своей полноте), либо прямо с помощью количественной ПЦР эндогенных генов.
Фигуры 6A-6D показывают РНК-направленную регуляцию OCT4 с применением Cas9N-VP64. Как показано на Фигуре 6A, для гена OCT4 были сконструированы 21 gРНК, мишенями которых является отрезок ДНК размером ~5 тысяч пар оснований, расположенный перед сайтом начала транскрипции. Сайты, гиперчувствительные к ДНКазе, выделены зеленым цветом. Фигура 6B показывает активацию транскрипции с использованием репортерной конструкции промотор-люцифераза. Фигура 6С показывает активацию транскрипции прямо с помощью количественной ПЦР эндогенных генов. Тогда как введение индивидуальных gРНК умеренно стимулировало транскрипцию, использование множества разных gРНК давало синергический эффект и обеспечивало мощную многократную активацию транскрипции.
Фигуры 7A-7C показывают РНК-направленную регуляцию REX1 с применением Cas9N, MS2-VP64 и gРНК+2X-MS2-аптамеров. Как показано на Фигуре 7A, для гена REX1 были сконструированы 10 gРНК, мишенями которых является отрезок ДНК размером ~5 тысяч пар оснований, расположенный перед сайтом начала транскрипции. Сайты, гиперчувствительные к ДНКазе, выделены зеленым цветом. Фигура 7B показывает активацию транскрипции с использованием репортерной конструкции промотор-люцифераза. Фигура 7С показывает активацию транскрипции прямо с помощью количественной ПЦР эндогенных генов. Тогда как введение индивидуальных gРНК умеренно стимулировало транскрипцию, введение множества разных gРНК давало синергический эффект и обеспечивало мощную многократную активацию транскрипции. В одном аспекте, отсутствие аптамеров 2X-MS2 в gРНК не приводит к активации транскрипции. Смотри статьи: Maeder et al., Nature Methods 10, 243-245 (2013) и Perez-Pinera et al., Nature Methods 10, 239-242 (2013), которые обе включаются в настоящее изобретение путем отсылки во всей своей полноте.
Таким образом, способы настоящего изобретения направлены на применение множества направляющих РНК с белком Cas9N и транскрипционным регуляторным белком или доменом для регуляции экспрессии целевого гена.
Эффективными оказались оба способа с применением Cas9 и химерных gРНК, причем первый способ был в 1,5-2 раза более эффективным. Вероятно, такие различия связаны с необходимостью формирования комплексов, состоящих из двух компонентов, а не из трех компонентов. Однако подход с применением химерных gРНК в принципе обеспечивает применение разных эффекторных доменов в разных gРНК, поскольку каждая gРНК использует разную взаимодействующую пару РНК-белок. Смотри статью: Karyer-Bibens et al., Biology of Cell / Under the Auspices of the European Cell Biology Organization 100, 125-138 (2008), которая включается в настоящее изобретение путем отсылки во всей своей полноте. Согласно одному аспекту настоящего изобретения, разные целевые гены могут регулироваться с применением специфической направляющей РНК и модифицированного белка Cas9N, то есть, с применением одного и того же или сходных белков Cas9N для разных целевых генов. Согласно одному аспекту, предоставляются способы множественной регуляции генов с применением одного и того же или сходных белков Cas9N.
Способы настоящего изобретения также направлены на редактирование целевых генов с применением описанных здесь белков Cas9N и направляющих РНК с целью множественной генетической и эпигенетической модификации клеток человека. На основе направленной доставки Cas9-gРНК (смотри статью: Jiang et al., Nature Biotechnology 31, 233-239 (2013), которая включается в настоящее изобретение путем отсылки во всей своей полноте) предоставляются способы для углубленного исследования сродства Cas9 к очень большому набору вариаций целевых последовательностей. Таким образом, аспекты настоящего изобретения предоставляют способ прямого высокопроизводительного анализа направленной доставки Cas9 в клетках человека, который лишен проблем, связанных с токсичностью разрывов двухцепочечной ДНК и мутагенной репарации, которые возникают при тестировании специфичности с использованием нативного Cas9 с нуклеазной активностью.
Дополнительные аспекты настоящего изобретения направлены на применение ДНК-связывающих белков или систем в целом для регуляции транскрипции целевого гена. Специалист в данной области техники легко подберет показательные ДНК-связывающие системы на основе настоящего изобретения. Такие ДНК-связывающие системы не должны иметь никакой нуклеазной активности, которую имеет нативный белок Cas9. Таким образом, такие ДНК-связывающие системы не обязательно должны иметь инактивированную нуклеазную активность. Одной показательной ДНК-связывающей системой является TALE. Согласно одному аспекту, специфичность TALE была оценена с использованием методологии, показанной на Фигуре 2A. Создается библиотека конструкций, в которой каждый элемент библиотеки включает флуоресцентный белок dTomato под минимальным промотором. После сайта начала транскрипции m вставляется случайная последовательность (транскрипционный таг) из 24 пар оснований (A/C/G), а перед промотором помещаются два TF-связывающий сайта: один представляет собой константную последовательность ДНК, общую для всех элементов библиотеки, тогда как второй представляет собой вариабельную структуру, которая создает «настаиваемую» библиотеку сайтов связывания, сконструированных для охватывания большой коллекции последовательностей, представляющих собой много комбинаций мутаций за пределами целевой последовательности программируемого направленного на ДНК комплекса, который был создан для связывания с ДНК. Это достигается с использованием вырожденных олигонуклеотидов, сконструированных таким образом, чтобы обеспечить такую частоту встречаемости нуклеотидов в каждом положении, чтобы нуклеотид целевой последовательности появлялся с частотой 79%, а все другие нуклеотиды с частотой 7%. Смотри статью: Patwardhan et al., Nature Biotechnology 30, 265-270 (2012), которая включается в настоящее изобретение путем отсылки во всей своей полноте. Репортерная библиотека затем секвенируется для выявления ассоциаций между транскрипционными тагами dTomato размером 24 пары оснований и их соответствующими «смещенными» целевыми сайтами в элементе библиотеки. Большое разнообразие транскрипционных тагов обеспечивает уверенность в том, что совпадение тагов между различными мишенями будет встречаться крайне редко, принимая во внимание, что «смещенность» конструкций целевых мишеней означает, что сайты с несколькими мутациями будут связываться с большим числом тагов, чем сайты с большим количеством мутаций. Вслед за этим, транскрипция репортерных генов dTomato стимулируется либо контрольным TF, сконструированным для связывания с общим сайтом ЛНК, либо целевым TF, который был сконструирован для связывания с целевым сайтом. Количество каждого экспрессированного транскрипционного тага измеряется в каждом образце с помощью секвенирования РНК в стимулированных клетках, с последующим обратным картированием с их соответствующими сайтами связывания с использованием построенной ранее таблицы связывания. Ожидается, что контрольный TF стимулирует всех членов библиотеки в равной степени, поскольку его сайт связывания является общим для всех элементов библиотеки, тогда как направленный TF, как ожидается, будет смещать распределение экспрессируемых членов в пользу тех, к которым он предпочтительно направляется. Такое предположение используется на стадии 5 для расчета нормализованного уровня экспрессии для каждого сайта связывания путем деления значений, полученных для направленного TF, на значения, полученные для контрольного TF.
Как показано на Фигуре 2B, характер связывания с мишенью комплекса Cas9-gРНК показывает, что он в среднем нечувствителен к 1-3 мутациям в своей целевой последовательности. Как показано на Фигуре 2С, комплекс Cas9-gРНК также в значительной степени нечувствителен к точечным мутациям, за исключением мутаций, локализованных в РАМ последовательности. Примечательно, что эти данные показывают, что предсказанным РАМ для Cas9 из S. pyogenes является не только NGG, но также и NAG. Как показано на Фигуре 2D, введение 2 неправильных оснований достоверно нарушает активность комплекса Cas9-gРНК, однако только если они локализованы в области 8-10 оснований от 3'-конца целевой последовательности gРНК (на графике heat plot положения оснований в целевой последовательности обозначены цифрами 1-23, начиная с 5'-конца).
Устойчивость к мутациям другого широко используемого для редактирования генома инструмента, доменов TALE, определялась с использованием описанного здесь способа определения специфичности. Как показано на Фигуре 2E, результаты по сбоям в направленном действии TALE показывают, что он в среднем устойчив к 1-2 мутациям в целевой последовательности, и не может активировать большинство вариантов с 3 неверными основаниями в его целевых последовательностях. Как показано на Фигуре 2F, 18-мерный TALE, как и комплексы Cas9-gРНК, в значительной степени является устойчивым к несовпадению одного основания в его последовательности-мишени. Как показано на Фигуре 2G, введение 2 несовпадающих оснований достоверно нарушает активность 18-мерного TALE. Активность TALE более чувствительна к несовпадению оснований вблизи 5'-конца его целевой последовательности (на графике heat plot положения оснований в целевой последовательности обозначены цифрами 1-18, начиная с 5'-конца).
Результаты были подтверждены в экспериментах по направленному действию с определением нуклеазной активности, результаты которых отражены на Фигурах 10A-10D, где оценивался характер направленной доставки TALE разных размеров. Как показано на Фигуре 10А, с использованием измерения опосредуемой нуклеазой гомологичной рекомбинации (HR), было подтверждено, что 18-мерные TALE устойчивы к множественным мутациям в их целевых последовательностях. Как показано на Фигуре 10В, с использованием подхода, описанного в Фигуре 2А, был проанализирован характер направленной доставки TALE трех разных размеров (18-мерного, 14-мерного и 10-мерного). Более короткие TALE (14-мерный и 10-мерный) являются прогрессивно более специфичными по своему направленному действию, но их активность снижена примерно на порядок. Как показано на Фигурах 10C и 10D, 10-мерные TALE показывают разрешение практически на уровне несовпадения одного основания, теряя почти всю активность в отношении мишеней, содержащих 2 несовпадающих основания (на графике heat plot положения оснований в целевой последовательности обозначены цифрами 1-10, начиная с 5'-конца). В совокупности эти данные показывают, что конструирование более коротких TALE может обеспечить более высокую специфичность в применениях для редактирования генома, тогда как необходимость в Fokl-димеризации в применениях TALE с нуклеазной активностью является важной для того, чтобы избежать ошибок в адресовании. Смотри статьи: Kim et al., Proceedings of the National Academy of Sciences of the United States of America 93, 1156- 160 (1996) и Pattanayak et al., Nature Methods 8, 765-770 (2011), каждая из которых включается в настоящее изобретение путем отсылки во всей своей полноте.
Фигуры 8A-8C показывают схему проведения анализа с высоким уровнем специфичности для расчета нормализованных уровней экспрессии, которая проиллюстрирована примерами из экспериментальных данных. Как показано на Фигуре 8А, создаются библиотеки конструкций со смещенным распределением последовательностей сайта связывания и тагами со случайной последовательностью размером 24 пары оснований, которые затем встраиваются в транскрипты репортерного гена (вверху). Транскрибируемые таги являются высоко вырожденными, поэтому они должны картировать связывающие последовательности для Cas9 или TALE по типу многие-к-одному (many-to-one). Библиотеки конструкций секвенируются (третий уровень, слева), чтобы выяснить, какие таги встречаются вместе с сайтами связывания, в результате чего создается таблица ассоциации сайтов связывания с транскрибируемыми тагами (четвертый уровень, слева). Множественные библиотеки конструкций, созданные для разных сайтов связывания, могут секвенироваться одновременно с использованием библиотеки баркодов (выделены светло голубым и светло желтым цветами, уровни 1-4, слева). Библиотека конструкций затем трансфецируется в клеточную популяцию, и набор разных транскрипционных факторов Cas9/gРНК или TALE индуцируется в образцах из этой клеточной популяции (второй уровень, справа). Один образец всегда индуцируется фиксированным активатором TALE, мишенью которого является последовательность фиксированного сайта связывания в конструкции (верхний уровень, зеленый квадратик); этот образец выступает в качестве положительного контроля (выделенный зеленым цветом образец, также отмеченный значком «+»). С репортерных молекул мРНК в индуцированных образцах создается кДНК, которая затем секвенируется и анализируется для подсчета количества тагов для каждого тага в образце (третий и четвертый уровни, справа). Как и в случае секвенирования библиотеки конструкций, множественные образцы, включая положительный контроль, секвенируются и анализируются вместе на основе введенных баркодов образцов. Светло красным цветом здесь выделен один не-контрольный образец, который был просеквенирован и проанализирован с положительным контролем (выделен зеленым цветом). Поскольку при каждом прочтении выявляются только транскрибируемые таги, а не сайты связывания конструкций, далее используется таблица ассоциации сайтов связывания и тагов, полученная при секвенировании библиотеки конструкций, для того, чтобы подсчитать общее количество тагов, проэкспрессированных с каждого сайта связывания в каждом образце (пятый уровень). Подсчеты для каждого образца не-положительного контроля затем переводятся в нормализованные уровни экспрессии для каждого сайта связывания путем деления их числа на число, полученное в образце положительного контроля. Примеры графиков зависимости нормализованных уровней экспрессии от числа несовпадающих оснований показаны на Фигурах 2B и 2E, и на Фигуре 9A и Фигуре 10B. В данной схеме проведения не показаны несколько уровней отфильтровывания ложных тагов, тагов, не связанных с библиотекой конструкций, и тагов, которые очевидно являются общими для многих сайтов связывания. Фигура 8B показывает пример распределения процента сайтов связывания в зависимости от числа несовпадений, сделанных в смещенной библиотеке конструкций. Слева: Теоретическое распределение. Справа: Распределение, полученной с использованием реальной библиотеки конструкций TALE. Фигура 8С показывает пример распределения процента подсчета тагов, агрегированных с сайтами связывания, в зависимости от числа несовпадений. Слева: Распределение, полученное от образца положительного контроля. Справа: Распределение, полученное от образца, в котором был индуцирован неконтрольный TALE. Поскольку используемый в качестве положительного контроля TALE связывается с фиксированным сайтом в конструкции, распределение количества агрегированных тагов точно отражает распределение сайтов связывания на Фигуре 8B, тогда как распределение сдвинуто влево для образца неконтрольного TALE, поскольку сайты с меньшим количеством несовпадающих оснований индуцируют более высокие уровни экспрессии. Внизу: Расчет относительного увеличения между ними путем деления количественных значений, полученных для направленного TF, на таковые для контрольного TF, показывает средний уровень экспрессии в зависимости от количества мутаций в целевом сайте.
Эти результаты были дополнительно подтверждены данными по специфичности, полученными с использованием разных комплексов Cas9-gРНК. Как показано на Фигуре 9A, разные комплексы Cas9-gРНК устойчивы к 1-3 мутациям в их целевой последовательности. Как показано на Фигуре 9В, комплекс Cas9-gРНК также в значительной степени нечувствителен к точечным мутациям, за исключением мутаций, локализованных в РАМ последовательности. Однако, как показано на Фигуре 9С, введение 2 неправильных оснований достоверно нарушает активность комплекса (на графике heat plot положения оснований в целевой последовательности обозначены цифрами 1-23, начиная с 5'-конца). Как показано на Фигуре 9D, с использованием опосредованного нуклеазой определения HR подтверждено, что предсказанным PAM для S. pyogenes Cas9 является NGG, а также NAG.
Согласно определенным аспектам, специфичность связывания увеличивается согласно описанным здесь способам. Поскольку синергизм между множественными комплексами является особенностью активации целевых генов Cas9N-VP64, применения в регуляции транскрипции Cas9N по своей природе обеспечивают высокую специфичность, так как единичные события связывания с нецелевой последовательностью должны иметь минимальный эффект. Согласно одному аспекту, в способах редактирования генома применяются сдвинутые одноцепочечные разрывы. Абсолютное большинство одноцепочечных разрывов редко приводит к событиям NHEJ (смотри статью: Certo et al., Nature Methods 8, 671-676 (2011), которая включается в настоящее изобретение путем отсылки во всей своей полноте), минимизируя, таким образом, эффекты одноцепочечных разрывов в нецелевых последовательностях. В отличие от этого, индуцирование сдвинутых одноцепочечных разрывов для создания разрывов двухцепочечной ДНК (DSB) является высокоэффективным способом индукции нарушения генов. Согласно определенным аспектам, выступающие 5'-концы создают более значительные события NHEJ по сравнению с выступающими 3'-концами. Аналогично этому, выступающие 3'-концы являются предпочтительными для событий HR по сравнению с NHEJ, хотя общее число событий HR достоверно ниже, чем в случае образования выступающих 5'-концов. Таким образом, предоставляются способы применения одноцепочечных разрывов для гомологичной рекомбинации и сдвинутых одноцепочечных разрывов для создания двухцепочечных разрывов с целью минимизирования эффектов направленной на нецелевые последовательности активности Cas9-gРНК.
Фигуры 3A-3C показывают множественные сдвинутые одноцепочечные разрывы и способы снижения связывания направляющих РНК с нецелевыми последовательностями. Как показано на Фигуре 3A, для одновременного измерения событий HR и NHEJ при введении направленных одноцепочечных или двухцепочечных разрывов использовался репортер «светофорного» типа. События расщепления ДНК, происходящие по пути HDR, восстанавливают последовательность зеленого флуоресцентного белка (GFP), тогда как мутагенный случай NHEJ вызывает сдвиги рамки, выводя GFP из рамки считывания и помещая расположенную ниже последовательность mCherry в рамку считывания. Для проведения анализа было сконструировано 14 gРНК, покрывающих участок ДНК в 200 пар оснований: 7, комплементарных к смысловой цепи (U1-7), и 7, комплементарных к антисмысловой цепи (D1-7). При использовании мутанта Cas9D10A, который делает одноцепочечный разрыв в комплементарной цепи, были взяты разные двухсторонние комбинации gРНК для получения ряда запрограммированных разрывов с выступающими 5'- или 3'-концами (указаны сайты одноцепочечных разрывов для этих 14 gРНК). Как показано на Фигуре 3B, индукция сдвинутых одноцепочечных разрывов для создания двухцепочечных разрывов (DSB) является высокоэффективной для индукции нарушения гена. Примечательно, что сдвинутые одноцепочечные разрывы, приводящие к выступающим 5'-концам, дают большее число событий NHEJ, в отличие от выступающих 3'-концов. Как показано на Фигуре 3С, создание выступающих 3'-концов также обеспечивает преимущество событий HR над NHEJ, но общее число событий HR достоверно ниже, чем в случае образования выступающих 5'-концов.
Фигуры 11A-11B посвящены NHEJ, опосредуемым Cas9D10A с никазной активностью. Как показано на Фигуре 11А, для анализа событий NHEJ при введении направленных одноцепочечных или двухцепочечных разрывов использовался репортер «светофорного» типа. Вкратце, при введении в ДНК двухцепочечных разрывов, если разрыв восстанавливается по пути мутагенного NHEJ, GFP оказывается вне рамки считывания, а расположенная ниже последовательность mCherry попадает в рамку, что приводит к красной флуоресценции. Было сконструировано 14 gРНК, покрывающих участок ДНК в 200 пар оснований: 7, комплементарных к смысловой цепи (U1-7), и 7, комплементарных к антисмысловой цепи (Dl-7). Как показано на Фигуре 11В, было обнаружено, что в отличие от Cas9 дикого типа, который приводит к разывам DSB и значительным NHEJ среди всех мишеней, большинство одноцепочечных разрывов (с использованием мутанта Cas9D10A) редко приводит к событиям NHEJ. Все 14 сайтов расположены в пределах непрерывного участка ДНК размером 200 пар оснований, причем была обнаружена более чем 10-кратная разница в эффективности направленной доставки.
Согласно определенным аспектам, предоставляются описанные здесь способы модулирования экспрессии целевой нуклеиновой кислоты в клетке, которые включают введение одной или более, двух или более, или множества чужеродных нуклеиновых кислот в клетку. Чужеродные нуклеиновые кислоты, введенные в клетку, кодируют направляющую РНК или направляющие РНК, не обладающий нуклеазной активностью белок Cas9 или белки и транскрипционный регуляторный белок или домен. Взятые вместе, направляющая РНК, не обладающий нуклеазной активностью белок Cas9 и транскрипционный регуляторный белок или домен образуют комплекс, который здесь называется комплексом ко-локализации, так что специалист в данной области техники будет понимать, что направляющая РНК, не обладающий нуклеазной активностью белок Cas9 и транскрипционный регуляторный белок или домен связываются с ДНК и регулируют экспрессию целевой нуклеиновой кислоты. Согласно определенным дополнительным аспектам, чужеродные нуклеиновые кислоты, введенные в клетку, кодируют направляющую РНК или направляющие РНК и белок Cas9 с никазной активностью. Взятые вместе, направляющая РНК и белок Cas9 с никазной активностью образуют комплекс, который здесь называется комплексом ко-локализации, так что специалист в данной области техники будет понимать, что направляющая РНК и белок Cas9 с никазной активностью связываются с ДНК и делают одноцепочечный разрыв целевой нуклеиновой кислоты.
Клетки согласно настоящему изобретению включают любую клетку, в которую могут быть введены и экспрессированы чужеродные нуклеиновые кислоты, как здесь описано. Необходимо понимать, что описанные здесь основные концепции настоящего изобретения не ограничиваются типом клеток. Клетки согласно настоящему изобретению включают эукариотические клетки, прокариотические клетки, клетки животных, клетки растений, клетки грибов, клетки архей, клетки эубактерий и тому подобное. Клетки включают эукариотические клетки, такие как клетки дрожжей, клетки растений и клетки животных. Предпочтительно клетки включают клетки млекопитающих. Дополнительно клетки включают любые клетки, в которых было бы предпочтительно или желательно регулировать целевую нуклеиновую кислоту. Такие клетки могут включать таковые, которые являются дефицитными по экспрессии определенного белка, что приводит к заболеванию или вредному для здоровья состоянию. Такие заболевания или состояния, причиняющие ущерб здоровью, хорошо известны специалистам в данной области техники. Согласно настоящему изобретению, нуклеиновая кислота, ответственная за экспрессию конкретного белка, может быть мишенью описанных здесь способов и мишенью транскрипционного активатора, что приводит к увеличению транскрипции целевой нуклеиновой кислоты и соответствующей экспрессии конкретного белка. Таким образом, описанные здесь способы предоставляют терапевтическое лечение.
Целевые нуклеиновые кислоты включают любую последовательность нуклеиновой кислоты, в отношении которой описанный здесь ко-локализующийся комплекс может быть пригодным с целью либо регуляции, либо внесения одноцепочечного разрыва. Целевые нуклеиновые кислоты включают гены. Для целей настоящего изобретения ДНК, такая как двухцепочечная ДНК, может включать целевую нуклеиновую кислоту, а ко-локализующийся комплекс может связываться или другим способом ко-локализоваться на ДНК, или поблизости, или рядом с целевой нуклеиновой кислотой таким образом, при котором ко-локализующийся комплекс может оказывать желаемый эффект на целевую нуклеиновую кислоту. Такие целевые нуклеиновые кислоты могут включать эндогенные (или встречающиеся в природе) нуклеиновые кислоты и экзогенные (или чужеродные) нуклеиновые кислоты. Специалист в данной области техники на основе настоящего изобретения сможет легко идентифицировать или сконструировать направляющие РНК и белки Cas9, которые будут ко-локализоваться на ДНК, включая целевую нуклеиновую кислоту. Специалист в данной области техники также сможет идентифицировать транскрипционные регуляторные белки или домены, которые аналогичным образом будут ко-локализоваться на ДНК, включая целевую нуклеиновую кислоту. ДНК включает геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
Чужеродные нуклеиновые кислоты (то есть такие, которые не являются частью имеющейся в клетке композиции природных нуклеиновых кислот) могут быть введены в клетку с использованием любого метода, известного специалистам в данной области техники и применяемого для такого введения. Такие методы включают трансфекцию, трансдукцию, вирусную трансдукцию, микроинъекцию, липофекцию, нуклеофекцию, бомбардировку наночастицами, трансформацию, конъюгацию и тому подобное. Специалист в данной области техники легко поймет и адаптирует такие методы, используя широко известные и легкодоступные литературные источники.
Транскрипционные регуляторные белки или домены, которые представляют собой активаторы транскрипции, включают VP16 и VP64 и другие, широко известные и легкодоступные специалистам в данной области техники на основе настоящего изобретения.
Заболеваниями и состояниями, причиняющими ущерб здоровью, являются такие, которые характеризуются ненормальным снижением экспрессии конкретного белка. Такие заболевания и состояния, причиняющие ущерб здоровью, могут лечиться путем увеличения экспрессии конкретного белка. Таким образом, предоставляются способы лечения заболевания и состояния, вредного для здоровья, в которых описанный здесь комплекс ко-локализации ассоциируется или связывается другим способом с ДНК, включая целевую нуклеиновую кислоту, и транскрипционный активатор, входящий в состав комплекса ко-локализации, увеличивает экспрессию целевой нуклеиновой кислоты. Например, активация PRDM16 и других генов, стимулирующих дифференцировку бурого жира и повышенное метаболическое потребление, может применяться для лечения метаболического синдрома или ожирения. Активация противовоспалительных генов является пригодной для лечения аутоиммунных и сердечно-сосудистых заболеваний. Активация генов-супрессоров опухолей является пригодной для лечения рака. Специалист в данной области техники легко идентифицирует такие заболевания и состояния, причиняющие ущерб здоровью, на основе настоящего изобретения.
Приведенные ниже примеры даны для иллюстрации настоящего изобретения. Эти примеры не должны рассматриваться как ограничивающие рамки настоящего изобретения, так как эти и другие эквивалентные воплощения становятся очевидными при рассмотрении Осуществления настоящего изобретения, Фигур и Формулы изобретения.
ПРИМЕР I
Мутанты Cas9
Был проведен поиск последовательностей, гомологичных белку Cas9 с известной структурой, с целью поиска подходящих мутаций в Cas9, которые бы устранили природную активность его доменов RuvC и HNH. С использованием HHpred (world wide website toolkit.tuebingen.mpg.de/hhpred) и последовательности полноразмерного Cas9 был проведен поиск в полной базе данных Protein Data Bank (на январь 2013). Это поиск выдал две разные эндонуклеазы, которые имели значительную гомологию последовательности с HNH доменом Cas9; PaсI и предполагаемая эндонуклеаза (PDB IDs: 3M7K и 4H9D соответственно). В этих белках был проведен поиск аминокислотных остатков, участвующих в координации иона магния. Соответствующие остатки затем были идентифицированы в Cas9 после выравнивания последовательностей. В каждой структуре были идентифицированы два боковых радикала, участвующих в координации Mg, которые были соотнесены с аминокислотами этого же типа в Cas9. Это были аминокислоты D92 и N113 в 3M7K, и D53 и N77 в 4H9D. Эти остатки соответствовали D839 и N863 в Cas9. Также сообщалось, что мутации в Pad с заменой остатков D92 и N113 на аланин лишали нуклеазу каталитической активности. На основании этого анализа в Cas9 были сделаны мутации D839A и N863A. Кроме того, HНpred также предсказал гомологию между Cas9 и N-концом Thermus thermophilus RuvC (PDB ID: 4EP4). Это выравнивание последовательностей объяснило известный ранее факт, почему мутация D10A устраняет функциональную активность RuvC домена в Cas9. Для подтверждения того, что это пригодная мутация, провели определение металлсвязывающих остатков, как описано выше. В 4EP4 остаток D7 участвует в координации иона магния. Это положение имеет гомологию последовательности с остатком D10 в Cas9, подтверждая, что такая мутация будет устранять связывание металла и, следовательно, каталитическую активность RuvC домена Cas9.
ПРИМЕР II
Конструирование плазмид
Мутанты Cas9 были созданы с использованием набора Quikchange kit (Agilent technologies). Конструкции для экспрессии целевых gРНК были либо (1) прямо выстроены как индивидуальные блоки (gBlocks) из IDT и клонированы в вектор pCR-BluntII-TOPO (Invitrogen); либо (2) синтезированы по заказу в Genewiz; либо (3) собраны с использованием набора олигонуклеотидов Gibson в вектор для клонирования gРНК (плазмида #41824). Векторы для определения репортеров HR, включающие «сломанный» GFP, были сконструированы путем сливания полученной ПЦР последовательности GFP, несущей стоп-кодон, и подходящего фрагмента, собранных вместе в лентивекторе EGIP из Addgene (плазмида #26777). Эти лентивекторы затем использовались для создания стабильных линий-репортеров GFP. Используемые в данном исследовании TALEN были сконструированы с использованием стандартных протоколов. Смотри статью: See Sanjana et al., Nature Protocols 7, 171-192 (2012), которая включается в настоящее изобретение путем отсылки во всей своей полноте. Cas9N и слитые MS2-VP64 были получены с использованием стандартных протоколов для процедуры слияния последовательностей при ПЦР. Конструкции промотора люциферазы для OCT4 и REX1 были получены от Addgene (плазмида #17221 и плазмида #17222).
ПРИМЕР III
Культура клеток и трансфекции
Клетки HEK 293T культивировали в среде Дульбекко в модификации Игла (DMEM, Invitrogen) в присутствии высокой концентрации глюкозы с добавлением 10% фетальной сыворотки теленка (FBS, Invitrogen), пенициллина/стрептомицина (pen/strep, Invitrogen), и заменимых аминокислот (NEAA, Invitrogen). Клетки поддерживали при 37°C и 5% CO2 во влажном инкубаторе.
Трансфекции для определения нуклеазной активности проводили следующим образом: 0,4 × 106 клеток трансфецировали 2 мкг плазмиды Cas9, 2 мкг gРНК и/или 2 мкг ДНК донорной плазмиды с использованием Lipofectamine 2000 согласно инструкции производителя. Клетки собирали через 3 дня после трансфекции и либо анализировали методом FACS, либо экстрагировали геномную ДНК из ~1 × 106 клеток с использованием набора DNAeasy kit (Qiagen) для прямого определения геномных разрывов. Для этого проводили ПЦР для амплификации целевого участка геномной ДНК, полученной из клеток, после чего ампликоны глубоко севкенировали на секвенаторе MiSeq Personal Sequencer (Illumina) с покрытием >200000 прочтений. Данные секвенирования анализировали для оценки эффективности NHEJ.
Для трансфекций с последующим определением активации транскрипции 0,4 × 106 клеток трансфецировали (1) 2 мкг плазмиды Cas9N-VP64, 2 мкг gРНК и/или 0,25 мкг репортерной конструкции; или (2) 2 мкг плазмиды Cas9N, 2 мкг MS2-VP64, 2 мкг gРНК-2XMS2-аптамера и/или 0,25 мкг репортерной конструкции. Клетки собирали через 24-48 часов после трансфекции и либо анализировали методами FACS или иммунофлуоресценции, либо экстрагировали из них суммарную РНК, которую анализировали методом ПЦР в реальном времени. При этом использовали стандартные зонды taqman для OCT4 и REX1 с нормализацией по ГАФДГ для каждого образца.
Для трансфекций с последующим определением активации транскрипции для оценки профиля специфичности комплексов Cas9- gРНК и TALE: 0,4 × 106 клеток трансфецировали (1) 2 мкг плазмиды Cas9N-VP64, 2 мкг gРНК и/или 0,25 мкг репортерной библиотеки; или (2) 2 мкг плазмиды TALE-TF и 0,25 мкг репортерной библиотеки; или (3) 2 мкг контрольной TF плазмиды и 0,25 мкг репортерной библиотеки. Клетки собирали через 24 часа после трансфекции (чтобы избежать стимуляции репортеров, находящихся на уровне насыщения). Экстракцию суммарной РНК проводили с использованием набора RNAeasy-plus kit (Qiagen), и проводили стандартную ПЦР в реальном времени с использованием Superscript-III (Invitrogen). Библиотеки для следующего поколения секвенирования создавали посредством адресной ПЦР-амплификации тагов-транскриптов.
ПРИМЕР IV
Вычислительный анализ и анализ последовательностей для расчета уровней экспрессии репортеров Cas9-TF и TALE-TF
Многоуровневая схема этого процесса показана на Фигуре 8A, а здесь приводятся дополнительные детали. Для деталей состава библиотеки конструкций смотри Фигуры 8A (уровень 1) и 8B.
Секвенирование: В экспериментах с Cas9 последовательности кДНК библиотеки конструкций (Фигура 8 A, уровень 3, слева) и репортерного гена (Фигура 8A, уровень 3, справа) были получены как результаты секвенирования перекрывающихся спаренных концов размером 150 пар оснований на секвенаторе Illumina MiSeq, тогда как в экспериментах с TALE соответствующие последовательности были получены как результаты секвенирования неперекрывающихся спаренных концов размером 51 пара оснований на секвенаторе Illumina HiSeq.
Анализ последовательности библиотеки конструкций: Выравнивание: В экспериментах с Cas9 использовалась программа novoalign V2.07.17 (world wide website novocraft.com/main/index/php) для выравнивания спаренных прочтений с набором референсных последовательностей размером 250 пар оснований, которые соответствовали конструкциям из 234 пар оснований, фланкированных парами баркодов библиотеки размером 8 пар оснований (смотри Фигуру 8A, 3 уровень, слева). В референсных последовательностях, используемых в программе novoalign, вырожденные участки сайта связывания Cas9 размером 23 пары оснований и вырожденные участки транскриптов тагов размером 24 пары оснований (смотри Фигуру 8A, первый уровень) обозначались буквами N, тогда как баркоды библиотеки конструкций были указаны точно. В экспериментах с TALE использовалась такая же процедура, за исключением того, что референсные последовательности имели в длину 203 пары оснований, а участки вырожденных сайтов связывания имели в длину 18 пар оснований, а не 23 пары оснований. Проверка достоверности: Результаты, полученные в программе Novoalign для сравниваемых файлов, для левого и правого прочтений для каждого из парных прочтений, индивидуально выравнивались по референсным последовательностям. Только парные прочтения, в которых оба прочтения уникально выравнивались с референсной последовательностью, подвергались дополнительной проверке достоверности, и только те парные прочтения, которые удовлетворяли всем этим условиям, сохранялись. Условия достоверности включали: (i) каждый из двух баркодов библиотеки конструкций должен совпадать, по меньшей мере, по 4 положениям с баркодом референсной последовательности, и эти два баркода должны быть парой баркодов для одной и той же библиотеки конструкций, (ii) Все основания, которые выравнивались по N участкам референсной последовательности, должны были определяться программой novoalign как A, C, G или T. Обратите внимание, что ни в экспериментах с Cas9, ни в экспериментах с TALE, левые и правые прочтения не перекрывались в референсном N участке, так что возможность неоднозначных определений этих оснований N программой novoalign не возникала, (iii) Подобным образом, в этих участках не должно появляться вставок или делеций, даваемых программой novoalign. (iv) В участках транскриционных тагов не должны появляться основания Т (поскольку эти случайные последовательности создавались только из A, C и G). Пары прочтений, для которых любое одно из этих условий не соблюдалось, отбрасывались и объединялись в файл отклоненных парных прочтений. Такая оценка достоверности проводилась с использованием алгоритма custom perl scripts.
Анализ последовательности кДНК индуцируемого образца репортерного гена: Выравнивание: Для анализа совпадения перекрывающихся парных прочтений с общим сегментом размером 79 пар оснований сначала использовалась программа SeqPrep (загружена с сайта world wide website github.com/jstjohn/SeqPrep), после чего использовалась программа novoalign (указанная выше версия) для выравнивания этих общих сегментов размером 79 пар оснований как непарных одиночных прочтений с набором референсных последовательностей (смотри Фигуру 8A, 3 уровень, справа), в которых (как при секвенировании библиотеки конструкций) вырожденный транскрипционный таг размером 24 пары оснований был указан основаниями N, тогда как баркоды образцов указывались точно. Участки кДНК для обоих TALE и Cas9 соответствовали одинаковым участкам кДНК размером 63 пары оснований, фланкированным парой последовательностей баркодов образца размером 8 пар оснований. Проверка достоверности: Применялись те же условия, что и при секвенировании библиотеки конструкций (смотри выше), за исключением следующего: (а) В данном случае, поскольку проводился предварительный SeqPrep анализ совпадения парных прочтений, проверка достоверности не должна была отфильтровывать уникальные выравнивания обоих прочтений в паре прочтений, а только уникальные выравнивания совпадающих прочтений. (b) В прочтениях последовательности кДНК появлялись только транскрипционные таги, поэтому система проверки достоверности применяла только эти участки тагов из референсных последовательностей, и не делала этого для отдельного участка сайта связывания.
Создание таблицы ассоциаций сайтов связывания и транскрипционного тага: Для создания этих таблиц применялся алгоритм Custom perl с использованием прошедших проверку последовательностей библиотеки конструкций (Фигура 8A, 4ый уровень, слева). Хотя последовательности тагов размером 24 пары оснований, состоящие из оснований A, C и G, должны были быть уникальными в библиотеке конструкций (вероятность совпадения = ~2,8e-11), предварительный анализ соответствия сайтов связывания и тагов показал, что не незначительная фракция последовательностей тагов оказалась общей для многих последовательностей сайтов связывания, что, вероятно, могло произойти в результате комбинации ошибок в секвенировании связывающих последовательностей, либо ошибок при синтезе олигонуклетидов, которые использовались для создания библиотек конструкций. Помимо наличия общих тагов, таги, для которых была обнаружена взаимосвязь с сайтами связывания в прошедших проверку парах, могли также быть найдены и в прочитанных парах библиотек конструкций, которые не прошли проверку и оказались отвергнутыми, если это не было ясно, вследствие несовпадения баркодов с той библиотекой конструкций, к которой они могли бы принадлежать. Наконец, последовательности тагов сами могли содержать ошибки последовательности. Чтобы разобраться с такими источниками ошибок, таги были разделены на категории по трем признакам: (i) безопасные и небезопасные, где термин небезопасные означает, что таг может быть найден в файле отвергнутых пар прочтений библиотеки конструкций; (ii) общие и не общие, где термин общие означает, что было обнаружено, что данный таг ассоциирован с последовательностями множества сайтов связывания, и (iii) 2+ и только 1, где 2+ означает, что данный таг появлялся, по меньшей мере, дважды среди прошедших проверку последовательностей библиотеки конструкций и, предположительно, менее вероятно содержит ошибки последовательности. Комбинирование этих трех критериев привело к получению 8 классов тагов, ассоциированных с каждым сайтом связывания, самый надежный (но самый малочисленный) класс, включающий только безопасные, не общие и 2+ таги; и самый ненадежный (но самый многочисленный) класс, включающий все таги, независимо от их безопасности, общности или числа появлений.
Расчет нормализованных уровней экспрессии: Алгоритм Custom perl был использован для проведения шагов, указанных на Фигуре 8A, уровни 5-6. В первую очередь, подсчеты тагов для каждого индуцированного образца были собраны для каждого сайта связывания с использованием таблицы соответствия сайта связывания и транскрипционного тага, которая была рассчитана ранее для библиотеки конструкций (смотри Фигуру 8С). Затем для каждого образца объединенное число подсчитанных тагов для каждого сайта связывания было поделено на объединенное число подсчитанных тагов для образца положительного контроля, чтобы получить нормализованные уровни экспрессии. Дополнительные ограничения, важные для таких расчетов, включали:
1. Для каждого образца среди прошедших проверку достоверности последовательностей кДНК генов было обнаружено некоторое количество «новых» тагов, которые не могли быть обнаружены в таблице ассоциации сайта связывания и транскрипционного тага. Эти таги не учитывались в дальнейших расчетах.
2. Объединение числа подсчитанных тагов, как описано выше, было сделано для каждого из описанных выше восьми классов тагов и сведено в таблицу ассоциации сайта связывания и транскрипционного тага. Поскольку сайты связывания в библиотеках конструкций были смещенными для получения последовательностей, часто подобных центральной последовательности, но последовательности с увеличивающимся числом несовпадений возникали редко, сайты связывания с несколькими несовпадающими основаниями обычно давали большие количества объединенных подсчетов тагов, тогда как сайты связывания с большим числом несовпадений давали более низкие количества объединенных подсчетов. Таким образом, хотя использование класса наиболее безопасных тагов обычно было желательным, оценка сайтов связывания с двумя или более несовпадениями может основываться на меньших количествах тагов на сайт связывания, делая подсчеты и соотношения менее статистически надежными, даже если сами таги были более надежными. В таких случаях в расчетах использовались все таги. Некоторым подтверждением правомочности такого предположения является тот факт, что количество отдельных объединенных подсчетов тагов для n несовпадающих положений растет с числом комбинаций несовпадающих положений (равно величине (Ln) 3n), и таким образом, драматически увеличивается с n; таким образом, средние значения для объединенного подсчета тагов для разного числа n несовпадений (показано на Фигурах 2B, 2E, и на Фигурах 9A, 10B) основаны на статистически очень большом наборе объединенных подсчетов тагов при n ≥ 2.
3. Наконец, сайт связывания, встроенный в библиотеки конструкций TALE, состоял из 18 пар оснований, ассоциации тагов определялись на основании этих последовательностей из 18 пар оснований, но некоторые эксперименты были проведены с использованием TALE, запрограммированных на связывание с центральной частью из 14 пар оснований или 10 пар оснований в пределах участков конструкций сайтов связывания из 18 пар оснований. При расчетах уровней экспрессии для этих TALE, таги объединялись с сайтами связывания на основании соответствующих участков сайтов связывания размером 18 пар оснований в таблицы соответствий, тогда как несовпадения сайтов связывания за пределами этого участка в расчет не принимались.
ПРИМЕР V
Конструирование векторов и штаммов
Последовательности Cas9 из S. thermophilus, N. meningitidis и T. denticola были получены из NCBI, оптимизированы по кодонам для экспрессии в клетках человека с использованием JCAT (world wide website jcat.de)27 и модифицированы для облегчения синтеза ДНК и экспрессии в E. coli. Блоки gBlocks размером 500 пар оснований (Integrated DNA Technologies, Coralville IA) были объединены путем иерархической перекрывающейся ПЦР и изотермической сборки24. Полученные полноразмерные продукты были субклонированы в векторы для экспрессии в бактериях и клетках человека. Кассеты не обладающего нуклеазной активностью Cas9 (NM: D16A D587A H588A N611A, SP: D10A D839A H840A N863A, ST1: D9A D598A H599A N622A, TD: D13A D878A H879A N902A) были сконструированы из этих матриц с использованием стандартных методов.
ПРИМЕР VI
Бактериальные плазмиды
Cas9 экспрессировали в бактериях из каркаса плазмиды cloDF13/aadA с использованием конститутивного промотора средней силы proC. Кассеты tracrРНК, включающие промоторы и терминаторы из нативных бактериальных локусов, были синтезированы как блоки gBlocks и встроены после кодирующей последовательности Cas9 в каждый вектор для надежной продукции tracrРНК. Когда ожидалось, что кассета tracrРНК дополнительно содержит промотор в противоположной ориентации, был вставлен терминатор lambda t1 для предотвращения интерференции с транскрипцией cas9. Бактериальные целевые плазмиды были созданы на основе каркаса плазмиды pl5A/cat с сильным промотором J23100, за которым находилась одна из двух последовательностей спейсера размером 20 пар оснований (Фигура 13D), функционирующего с использованием SP, как было показано ранее. Сразу за последовательностями спейсера находились последовательности одного из трех повторов из 36 пар оснований, показанных на Фигуре 12A. Репортерный вектор желтого флуоресцентного белка YFP быт сделан на основе каркаса pSC101/kan с GFP под управлением промотора pR и T7 g10 RBS, находящимся перед кодирующей последовательностью EYFP, с протоспейсером 1 и AAAAGATT PAM, вставленными в некодирующую цепь в 5' UTR (нетранслируемый участок). Субстратные плазмиды для тестирования ортогональности в бактериях были идентичны плазмидам библиотеки (смотри ниже), но содержали следующие PAM: GAAGGGTT (NM), GGGAGGTT (SP), GAAGAATT (ST1), AAAAAGGG (TD).
ПРИМЕР VII
Векторы для клеток млекопитающих
Векторы для экспрессии Cas9 в клетках млекопитающих были созданы на основе pcDNA3.3-TOPO с присоединенными к С-концу сигналами ядерной локализации (NLS) SV40. Для каждого Cas9 sgРНК были сконструированы путем выравнивания повторов crРНК с tracrРНК и слияния 5'-повтора crРНК с 3'-концом tracrРНК, чтобы обеспечить образование стабильной шпильки для взаимодействия с Cas925. Экспрессионные конструкции sgРНК получали клонированием блоков gBlocks размером 455 пар оснований в каркас вектора pCR-BluntII-TOPO. Спейсеры были идентичны таковым, которые использовались в предыдущей работе8. Лентивекторы для определения HR «сломанного» GFP-репортера были модифицированы по сравнению с описанными ранее путем включения подходящих последовательностей PAM для каждого Cas9 и применялись для получения стабильных GFP-репортерных линий.
Были сконструированы РНК-направляемые активаторы транскрипции, которые состояли из не обладающих нуклеазной активностью белков Cas9, слитых с VP64 активатором, и соответствующих репортерных конструкций, несущих ген tdTomato под минимальным промотором.
ПРИМЕР VIII
Создание и трансформация библиотек
Библиотеки протоспейсеров были сконструированы путем амплификации вектора pZE21 (ExpressSys, Ruelzheim, Germany) с использованием праймеров (IDT, Coralville, IA), кодирующих одну из двух последовательностей протоспейсеров, за которыми находились 8 случайных оснований, и собирались стандартными изотермическими методами24. Готовые библиотеки сначала трансформировались в клетки NEBTurbo (New England Biolabs, Ipswich MA), что давало >1E8 клонов на одну библиотеку согласно данным посевов с разведением, и были очищены с помощью Midiprep (Qiagen, Carlsbad CA). Электрокомпетентные клетки NEBTurbo, содержащие экспрессионную плазмиду Cas9 (DS-NMcas, DS-ST1cas или DS-TDcas) и направляющую плазмиду (PM-NM!sp1, PM-NM!sp2, PM-ST1!sp1, PM- ST1!sp2, PM-TD!sp1 или PM-TD!sp2) были трансформированы 200 нг каждой библиотеки и выдерживались 2 часа при 37°C до разведения средой, содержащей спектиномицин (50 мкг/мл), хлорамфеникол (30 мкг/мл) и канамицин (50 мкг/мл). Серийные разведения высевали для определения размера библиотек после трансформации. Размер всех библиотек превышал ~1E7 клонов, что указывает на полное покрытие 65536 случайных последовательностей PAM.
ПРИМЕР IX
Высокопроизводительное секвенирование
ДНК библиотек собирали на центрифугируемых спин-колонках (Qiagen, Carlsbad CA) через 12 часов селекции на антибиотиках. Интактные РАМ амплифицировали с праймерами к баркодам и получали последовательности по данным прочтения перекрывающихся на 25 пар оснований спаренных концов на секвенаторе Illumina MiSeq. Секвенирование MiSeq давало 18411704 общих прочтения или 9205852 прочтений спаренных концов со средним показателем качества >34 для каждой библиотеки. Прочтения спаренных концов совмещали и отбирали на безупречное соответствие друг другу, их протоспейсеру и каркасу плазмиды. Оставшиеся 7652454 совмещенных, но не прошедших отбора прочтений, обрезали для удаления последовательностей каркаса плазмиды и протоспейсера, после чего использовали для создания матриц «веса» положения основания для каждой библиотеки РАМ. Комбинация каждой библиотеки получала, по меньшей мере, 450000 прочтений высокого качества.
ПРИМЕР X
Обработка последовательностей
Для расчета уровня снижения количества каждого предполагаемого PAM были использованы два подхода для отбора данных. Программа patternProp (usage: python patternProp.py [PAM] file.fastq) дает число и фракцию прочтений, совпадающих с каждым производным по 1 основанию для указанного РАМ. Программа patternProp3 дает соотношение фракции прочтений, совпадающих с каждым производным по 1 основанию, и общего числа прочтений для данной библиотеки. Для идентификации минимального уровня снижения количества среди всех производных по 1 основанию, и, следовательно, для классификации РАМ, применялась сводная таблица соотношений детальных уровней снижения количества каждого рассчитанного PAM.
ПРИМЕР XI
Определение репрессии и ортогональности в бактериях
Опосредуемая Cas9 репрессия была измерена путем трансформации экспрессионной плазмиды NM и репортерной плазмиды YFP с каждой из двух соответствующих направляющих плазмид. Колонии с совпадающими и несовпадающими спейсером и протоспейсером собирали и выращивали в 96-луночных планшетах. Флуоресценцию при 495/528 нм и поглощение при 600 нм измеряли с использованием ридера для микропланшетов Synergy Neo (BioTek, Winooski VT).
Тесты на ортогональность проводили с помощью получения электрокомпетентных клеток NEBTurbo, несущих все комбинации Cas9 и направляющих плазмид, и трансформирования их совпадающими или несовпадающими субстратными плазмидами, несущими подходящий РАМ для каждого Cas9. Было высеяно достаточное количество клеток и разведений для того, чтобы убедиться в том, что, по меньшей мере, несколько колоний появлялось даже для комбинаций, содержащих корректный Cas9 + направляющая плазмида + совпадающий протоспейсер, которые обычно возникают в результате мутационной инактивации Cas9 или crРНК. Колонии подсчитывались, и для каждой из них был рассчитан fold depletion.
ПРИМЕР XII
Культура клеток и трансфекции
Клетки HEK 293T культивировали в среде Дульбекко в модификации Игла (DMEM, Invitrogen) с высоким содержанием глюкозы и с добавлением 10% фетальной сыворотки теленка (FBS, Invitrogen), пенициллина/стрептомицина (pen/strep, Invitrogen) и заменимых аминокислот (NEAA, Invitrogen). Клетки содержали при 37°C и 5% CO2 во влажном инкубаторе.
Трансфекции для определения нуклеазной активности проводили следующим образом: 0,4 × 106 клеток трансфецировали 2 мкг плазмиды Cas9, 2 мкг gРНК и/или 2 мкг ДНК донорной плазмиды с использованием Lipofectamine 2000 согласно инструкции производителя. Клетки собирали через 3 дня после трансфекции и либо анализировали методом FACS, либо экстрагировали геномную ДНК из ~1 × 106 клеток с использованием набора DNAeasy kit (Qiagen) для прямого определения геномных разрывов.
Для трансфекций с последующим определением активации транскрипции: 0,4 × 106 клеток трансфецировали 2 мкг плазмиды Cas9N-VP64, 2 мкг gРНК и/или 0,25 мкг репортерной конструкции. Клетки собирали через 24-48 часов после трансфекции и анализировали методами FACS или иммунофлуоресценции, или проводили экстракцию суммарной РНК с последующим анализом методом ПЦР в реальном времени.
ПРИМЕР XIII
Отбор потенциальных ортогональных белков Cas9
Связывание РНК белками Cas9 и специфичность sgРНК первично определяли на основании последовательности повтора из 36 пар оснований в crРНК. Известные гены Cas9 проверяли на высоко дивергентные повторы в их прилегающих локусах CRISPR. Были отобраны CRISPR1 белки Cas9 из Streptococcus pyogenes и Streptococcus thermophilus (SP и ST1)6, 22 и два дополнительных белка Cas9 из Neisseria meningitidis (NM) и Treponema denticola (TD), в чьих локусах имелись повторы, которые отличались, по меньшей мере, на 13 нуклеотидов друг от друга и от таковых для SP и ST1 (Фигура 12A).
ПРИМЕР XIV
Характеристика PAM
Белки Cas9 связываются только с последовательностями двухцепочечной dsДНК, фланкированными 3'-последовательностью РАМ, специфической для интересующего Cas9. Из четырех вариантов Cas9, только SP имеет экспериментально охарактеризованный РАМ, тогда как ST1 PAM и, совсем недавно, NM PAM были «вычислены» с помощью методов биоинформатики. SP легко направляется благодаря его короткому PAM в виде NGG10, тогда как направленная доставка ST1 и NM не такая легкая, поскольку их РАМ имеют вид NNAGAAW и NNNNGATT соответственно22, 23. Биоинформационные подходы предъявляют более жесткие требования к в РАМ для активности Cas9, чем, как известно из эмпирических данных, необходимо для расщепления мишени после стадии взаимодействия со спейсером. Поскольку последовательность РАМ является наиболее частой мишенью мутаций у бактериофагов, избыточные требования ко взаимодействию с РАМ должны были бы снижать устойчивость к ним бактерий. Подход на основе библиотек был адаптирован для всесторонней характеристики этих последовательностей у бактерий с использованием высокопроизводительного секвенирования.
Гены, кодирующие ST1, NM, и TD, были собраны из синтетических фрагментов и клонированы в бактериальные экспрессионные плазмиды вместе с их соответствующими tracrРНК (Фигура 12В). Было выбрано два SP-функциональных спейсера для включения в шесть направляющих плазмид. Каждая направляющая плазмида кодирует конститутивно экспрессируемую crРНК с одним из двух спейсеров, за которыми располагается последовательность повтора из 36 пар оснований, специфическая для белка Cas9 (Фигура 12В). Методом ПЦП были созданы и собраны плазмидные библиотеки, содержащие один из двух протоспейсеров, за которыми располагались все возможные последовательности РАМ из 8 пар оснований24. Каждая библиотека была введена методом электропорации в клетки E. сoli, несущие экспрессионные и направляющие плазмиды Cas9, так что в итоге получилось 12 комбинаций белка Cas9, спейсера и протоспейсера. Выжившие плазмиды из библиотек были селективно амплифицированы путем ПЦР по баркодам и просеквенированы с помощью MiSeq, чтобы отделить функциональные последовательности РАМ, содержание которых снижается, только когда спейсер и протоспейсер совпадают (Фигуры 12C-12D), от нефункциональных РАМ, содержание которых никогда не снижается (Фигуры 12D-12E). Чтобы графически обозначить важность каждого нуклеотида в каждом положении, были построены графики логарифма относительной частоты каждого основания для совпадающих пар спейсер-протоспейсер относительно соответствующего случая несовпадения (Фигуры 13A-13F).
NM и ST1 распознают РАМ, которые являются менее жесткими и более сложными, чем ранее было предсказано с помощью биоинформатики, что позволяет предполагать, что требования для взаимодействия со спейсером являются более жесткими, чем таковые для расщепления эффекторного участка. NM в первую очередь требует присутствия единственного нуклеотида G в пятом положении от 3'-конца протоспейсера (Фигура 13А), тогда как ST1 и TD каждый требуют, по меньшей мере, трех специфических оснований (Фигуры 5В-С). Результаты сортировки по положению позволяют количественно оценить снижение содержания последовательности любого РАМ из каждой библиотеки протоспейсеров (Фигуры 13D-13F). Все три фермента расщепляют протоспейсер 2 более эффективно, чем протоспейсер 1, почти со всеми РАМ, при этом ST1 демонстрирует примерно 10-кратное различие. Однако наблюдается также значительная РАМ-зависимая вариация в таком взаимодействии. Например, NM расщеплял протоспейсеры 1 и 2 примерно одинаково, когда за ними были расположены последовательности, совпадающие с TNNNGNNN, но был в 10 раз более активным в расщеплении протоспейсера 2, когда РАМ совпадал с ANNNGNNN.
Результаты показывают сложность определения единственного приемлемого РАМ для данного белка Cas9. Уровень активности зависит не только от последовательности протоспейсера, но специфические комбинации не самых важных оснований РАМ могут значительно снижать активность, даже когда выполняются требования к самым важным основаниям. Изначально РАМ были идентифицированы нами как такие последовательности, содержание которых снижалось в среднем более, чем в 100 раз при более низкой активности протоспейсера 1, и более, чем в 50 раз для всех производных с одним дополнительно фиксированным основанием (Таблица 1, обычный шрифт). Хотя такие уровни предположительно являются достаточными для защиты от мишеней у бактерий, определенные комбинации вредных мутаций значительно снижают активность. Например, NM снижает содержание последовательностей, совпадающих с NCCAGGTN только в 4 раза. Более жесткий порог, требующий более чем 500-кратного снижения содержания совпадающих последовательностей и более чем 200-кратного снижения содержания производных по 1 основанию, был установлен для приложений, требующих высокого сродства (Таблица 1, жирный шрифт).
Таблица 1
ПРИМЕР XV
Регуляция транскрипции у бактерий
Было установлено, что не обладающий нуклеазной активностью вариант SP подавляет целевые гены у бактерий с эффективностью, которая зависит от положения направляемого протоспейсера и PAM18. Поскольку PAM для NM встречается более часто, чем таковой для SP, не обладающая нуклеазной активностью версия может сходным образом обеспечивать направленную репрессию. Участвующие в катализе аминокислотные остатки в нуклеазных доменах RuvC и HNH были идентифицированы на основании гомологии последовательностей и инактивированы для получения предположительно не обладающего нуклеазной активностью NM. Для создания подходящего репортера, протоспейсер 1 с пригодным РАМ был вставлен в нематричную цепь в области 5'-UTR репортерной плазмиды YFP (Фигура 14А). Эти конструкции были ко-трансформированы в E. coli вместе с каждой из двух NM-направляющих плазмид, используемых ранее, и было проведено сравнение их флуоресценции. Клетки с совпадающими спейсером и протоспейсером демонстрировали в 22 раза более слабую флуоресценцию по сравнению с соответствующим несовпадающим случаем (Фигура 14В). Эти результаты позволяют предполагать, что NM может функционировать как легко направляемый репрессор для контроля транскрипции у бактерий, значительно увеличивая число эндогенных генов, которые могут подвергаться Cas9-опосредуемой репрессии.
ПРИМЕР XVI
Ортогональность у бактерий
Был выбран набор белков Cas9, которые характеризовались несовпадающими последовательностями своих crРНК повторов. Для подтверждения того, что они действительно являются ортогональными, каждая экспрессионная плазмида Cas9 была ко-трансформирована со всеми четырьмя направляющими плазмидами, содержащими спейсер 2. Эти клетки были трансформированы субстратными плазмидами, содержащими либо протоспейсер 1, либо протоспейсер 2, и подходящий РАМ. Уменьшение содержания плазмид наблюдалось исключительно в тех случаях, когда каждый Cas9 находился в паре со своей собственной crРНК, демонстрируя, что все четыре конструкции являются в бактериях действительно ортогональными (Фигура 15).
ПРИМЕР XVII
Редактирование генома в клетках человека
Эти варианты Cas9 затем применялись для редактирования генома в клетках человека. Были сконструированы одиночные направляющие РНК (sgРНК) из соответствующих crРНК и tracrРНК для NM и ST1, двух более мелких ортологов Cas9, путем анализа комплементарности участков между crРНК и tracrРНК25 и слияния двух последовательностей для образования петель-шпилек в различных слитых конструкциях, аналогичных таковым в sgРНК, созданных для SP. В некоторых случаях, когда несколько последовательно расположенных урацилов вызывали терминацию полимеразы Pol III в системе экспрессии, были созданы разнообразные мутанты по одному основанию. Всегда включалась полная 3'-последовательность tracrРНК, поскольку, как известно, укороченные варианты являются нежелательными8. Все sgРНК были проанализированы на их активность вместе с соответствующими им белками Cas9 с использованием описанного ранее определения гомологичной рекомбинации в клетках линии 2938. Вкратце, для каждого белка Cas9 была создана репортерная линия с интегрированным в геном нефлуоресцирующим белком GFP, где кодирующая последовательность GFP была нарушена путем вставки, кодирующей стоп-кодон и последовательность протоспейсера с функциональным РАМ. Репортерные линии трансфецировали экспрессионными векторами, кодирующими белок Cas9 и соответствующую sgРНК вместе с репарирующим донором, способным восстановить флуоресценцию в случае индуцированной нуклеазой гомологичной рекомбинации (Фигура 16А). Опосредованное ST1 и NM редактирование наблюдалось на уровнях, сравнимых с таковыми, индуцированными SP. ST1 sgРНК с 5 последовательными урацилами функционировала эффективно, подтверждая, что терминация полимеразы Pol III не происходит на уровнях, достаточных для подавления активности. Полноразмерные слитые crРНК-tracrРНК были активны во всех случаях и являются пригодными для дизайна химерных sgРНК. Оба фермента NM и ST1 были способны эффективно редактировать гены в клетках человека с использованием химерных направляющих РНК.
ПРИМЕР XVIII
Ортогональность Cas9 в клетках млекопитающих
После открытия высоко эффективных sgРНК для активности NM и ST1 в клетках человека, было проверено, что ни один из трех белков не может направляться с помощью sgРНК других белков. Для измерения сравнительной эффективности NM, SP и ST1 в комбинации с каждой из этих трех sgРНК был использован такой же способ анализа гомологичной рекомбинации. Было установлено, что все три белка Cas9 являются полностью ортогональными по отношению друг к другу, демонстрируя, что они могут направляться к разным и неперекрывающимся наборам последовательностей в одной и той же клетке (Фигура 16В). Для выяснения значения sgРНК и PAM в ортогональной направленной доставке было протестировано множество расположенных ниже последовательностей РАМ с SP и ST1 и соответствующих им sgРНК. Как совпадающая sgРНК, так и правильный РАМ являются необходимыми для активности, а ортогональность почти полностью определяется специфическим сродством sgРНК к соответствующему Cas9.
ПРИМЕР XIX
Активация транскрипции в клетках человека
NM и ST1 опосредуют активацию транскрипции в клетках человека. Гены не обладающих нуклеазной активностью NM и ST1 были слиты с активаторным доменом VP64 на их С-конце для получения предполагаемых РНК-направляемых активаторов, созданных по типу активатора SP. Репортерные конструкции доля активации состояли из протоспейсера с подходящим РАМ, встроенным перед кодирующим участком tdTomato. Были ко-трансфецированы векторы, экспрессирующие РНК-направляемый транскрипционный активатор, sgРНК и подходящий репортер, после чего степень активации транскрипции измеряли методом FACS (Фигура 17А). В каждом случае наблюдалась мощная активация транскрипции всеми тремя вариантами Cas9 (Фигура 17В). Каждый активатор Cas9 стимулировал транскрипцию только тогда, когда находился в паре со своей соответствующей sgРНК.
ПРИМЕР XX
Обсуждение
Использование двух разных протоспейсеров для всесторонней характеристики РАМ позволило провести исследование комплексного характера управляющего значения протоспейсера и распознавания РАМ. Разные эффективности расщепления протоспейсеров демонстрировали устойчивую тенденцию среди разных белков Cas9, хотя величина различий между ортологами значительно варьировала. Это позволяет предполагать, что зависимые от последовательности различия в образовании или стабилизации D-петли определяют базальную направляющую активность каждого протоспейсера, но определенную роль также играют дополнительные факторы, зависящие от Cas9 или повторов. Подобным образом многочисленные факторы делают невозможными попытки описать распознавание РАМ с помощью одного мотива последовательности. Индивидуальные основания, расположенные рядом с первичными детерминантами распознавания РАМ, могут комбинироваться таким образом, что это резко снижает общую активность. Действительно, похоже, что определенные РАМ нелинейно взаимодействуют со спейсером или протоспейсером, что определяет общую активность. Кроме того, разные уровни сродства могут быть необходимы для определенных активностей в разных типах клеток. И наконец, экспериментально идентифицированные РАМ требуют меньшего количества оснований, чем таковые, предсказанные по данным биоинформационного анализа, позволяя предполагать, что требования к связыванию спейсера отличаются от таковых для расщепления эффекторного участка.
Это различие наиболее значительно для белка Cas9 из Neisseria meningitidis, который предъявляет гораздо меньше требований к PAM и по сравнению с биоинформационными предсказаниями, и по сравнению с широко используемым сейчас Cas9 из S. pyogenes. Это открытие значительно расширяет число последовательностей, которые могут быть хорошими мишенями для белка Cas9. Размер его гена составляет 3,25 тысяч пар оснований, что на 850 пар оснований меньше размера гена SP, что является серьезным преимуществом, когда возможности доставки генов являются ограниченными. Особенно важно, что NM и ST1 оба достаточно малы для встраивания в вектор AAV для терапевтических применений, при этом NM может представлять собой более подходящую отправную точку для попыток направленной эволюции с целью изменения распознавания или специфичности PAM.
Приведенные ниже ссылки включаются в настоящее изобретение путем отсылки во всей своей полноте для любых целей.
ЛИТЕРАТУРА
1. Bhaya, D., Davison, M. & Barrangou, R. CRISPR-Cas systems in bacteria and archaea:
versatile small RNAs for adaptive defense and regulation. Annual review of genetics 45, 273-297 (2011).
2. Wiedenheft, B., Sternberg, S.H. & Doudna, J.A. RNA-guided genetic silencing systems in bacteria and archaea. Nature 482, 331-338 (2012).
3. Gasiunas, G., Barrangou, R., Horvath, P. & Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America 109, E2579-2586 (2012).
4. Jinek, M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012).
5. Cho, S.W., Kim, S., Kim, J.M. & Kim, J.S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature biotechnology 31, 230-232 (2013).
6. Cong, L. et al. Multiplex genome engineering using CRISPR/Cas systems. Science 339, 819- 823 (2013).
7. Ding, Q. et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell stem cell 12, 393-394 (2013).
8. Mali, P. et al. RNA-guided human genome engineering via Cas9. Science 339, 823-826 (2013).
9. Wang, H. et al. One-Step Generation of Mice Carrying Mutations in Multiple Genes by CRISPR/Cas-Mediated Genome Engineering. Cell 153, 910-918 (2013).
10. Jiang, W., Bikard, D., Cox, D., Zhang, F. & Marraffini, L.A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature biotechnology 31 , 233-239 (2013).
11. Boch, J. et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science 326, 1509- 1512 (2009).
12. Gaj, T., Gersbach, C.A. & Barbas, C.F., 3rd ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in biotechnology (2013).
13. Hockemeyer, D. et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nature biotechnology 27, 851-857 (2009).
14. Kim, Y.G., Cha, J. & Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proceedings of the National Academy of Sciences of the United States of America 93, 1 156- 1160 (1996).
15. Moscou, M.J. & Bogdanove, A.J. A simple cipher governs DNA recognition by TAL effectors. Science 326, 1501 (2009).
16. Porteus, M.H. & Carroll, D. Gene targeting using zinc finger nucleases. Nature biotechnology 23, 967-973 (2005).
17. Urnov, F.D. et al. Highly efficient endogenous human gene correction using designed zinc- finger nucleases. Nature 435, 646-651 (2005).
18. Qi, L.S. et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell 152, 1173-1183 (2013).
19. Beerli, R.R., Dreier, B. & Barbas, C.F., 3rd Positive and negative regulation of endogenous genes by designed transcription factors. Proceedings of the National Academy of Sciences of the United States of America 97, 1495-1500 (2000).
20. Podgornaia, A.I. & Laub, M.T. Determinants of specificity in two-component signal transduction. Current opinion in microbiology 16, 156-162 (2013).
21. Purnick, P.E. & Weiss, R. The second wave of synthetic biology: from modules to systems. Nature reviews. Molecular cell biology 10, 410-422 (2009).
22. Horvath, P. et al. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus. Journal of bacteriology 190, 1401-1412 (2008).
23. Zhang, Y. et al. Processing-Independent CRISPR RNAs Limit Natural Transformation in Neisseria meningitidis. Molecular cell 50, 488-503 (2013).
24. Gibson, D.G. et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature methods 6, 343-345 (2009).
25. Deltcheva, E. et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471, 602-607 (2011).
26. Bondy-Denomy, J., Pawluk, A., Maxwell, K.L. & Davidson, A.R. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system. Nature 493, 429-432 (2013).
27. Grote, A. et al. JCat: a novel tool to adapt codon usage of a target gene to its potential expression host. Nucleic acids research 33, W526-531 (2005).
| название | год | авторы | номер документа |
|---|---|---|---|
| ОРТОГОНАЛЬНЫЕ БЕЛКИ CAS9 ДЛЯ РНК-НАПРАВЛЯЕМОЙ РЕГУЛЯЦИИ И РЕДАКТИРОВАНИЯ ГЕНОВ | 2014 |
|
RU2704981C2 |
| ОРТОГОНАЛЬНЫЕ БЕЛКИ CAS9 ДЛЯ РНК-НАПРАВЛЯЕМОЙ РЕГУЛЯЦИИ И РЕДАКТИРОВАНИЯ ГЕНОВ | 2021 |
|
RU2771583C1 |
| МУЛЬТИПЛЕКСНАЯ ГЕНОМНАЯ ИНЖЕНЕРИЯ, НАПРАВЛЯЕМАЯ РНК | 2014 |
|
RU2764637C2 |
| НАПРАВЛЯЕМАЯ РНК РЕГУЛЯЦИЯ ТРАНСКРИПЦИИ | 2014 |
|
RU2690935C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ЛЕЧЕНИЯ БОЛЕЗНИ ХАНТИНГТОНА | 2015 |
|
RU2691102C2 |
| СИСТЕМЫ CRISPR-CAS И СПОСОБЫ ИЗМЕНЕНИЯ ЭКСПРЕССИИ ПРОДУКТОВ ГЕНОВ | 2013 |
|
RU2687451C1 |
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2699523C2 |
| НАПРАВЛЯЕМАЯ РНК РЕГУЛЯЦИЯ ТРАНСКРИПЦИИ | 2014 |
|
RU2756865C2 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2701662C2 |
| МУЛЬТИПЛЕКСНАЯ ГЕНОМНАЯ ИНЖЕНЕРИЯ, НАПРАВЛЯЕМАЯ РНК | 2014 |
|
RU2706562C2 |
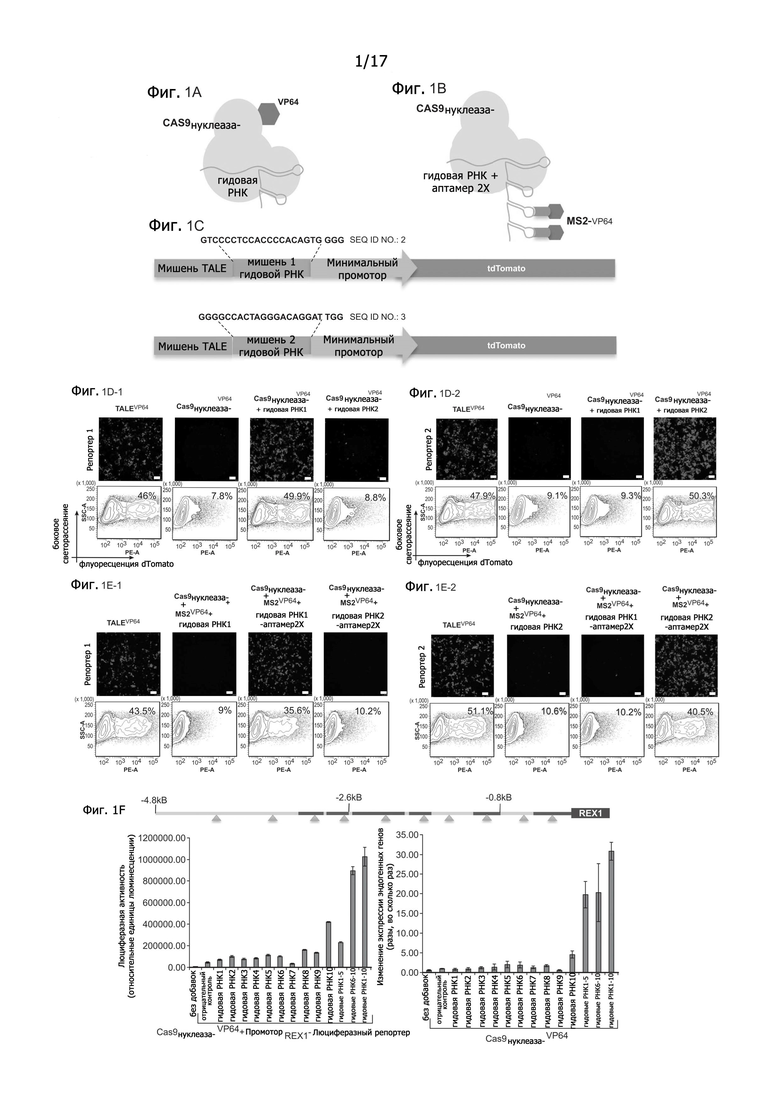
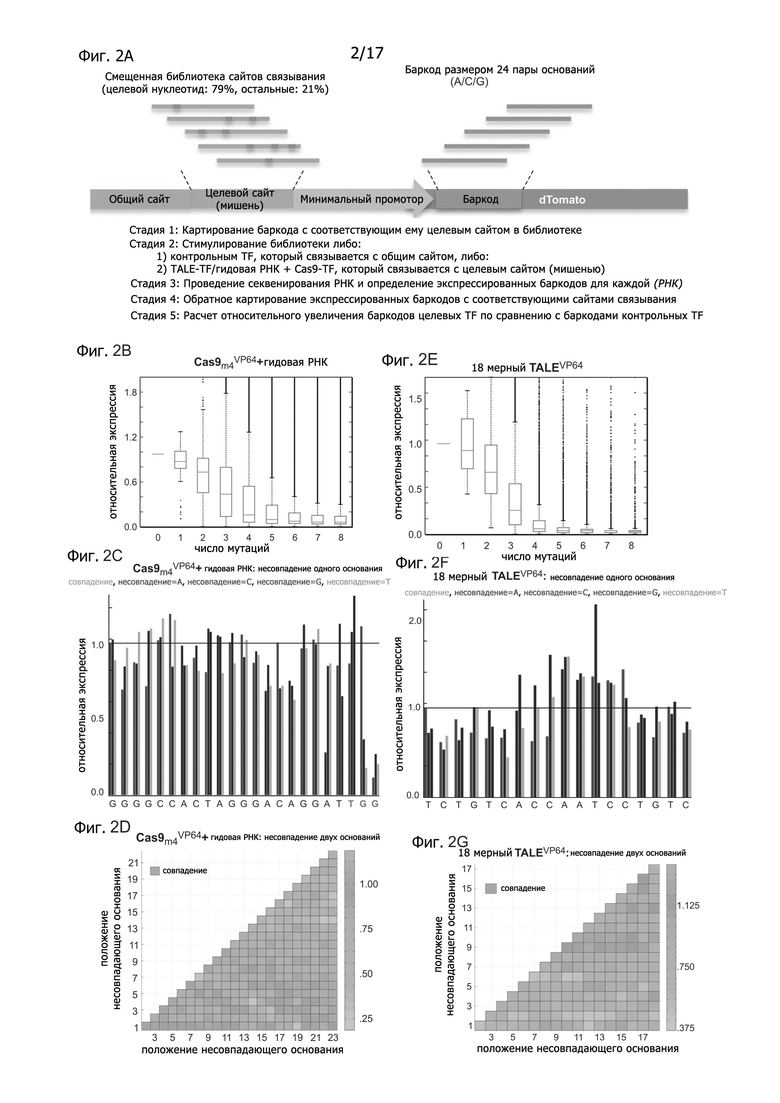
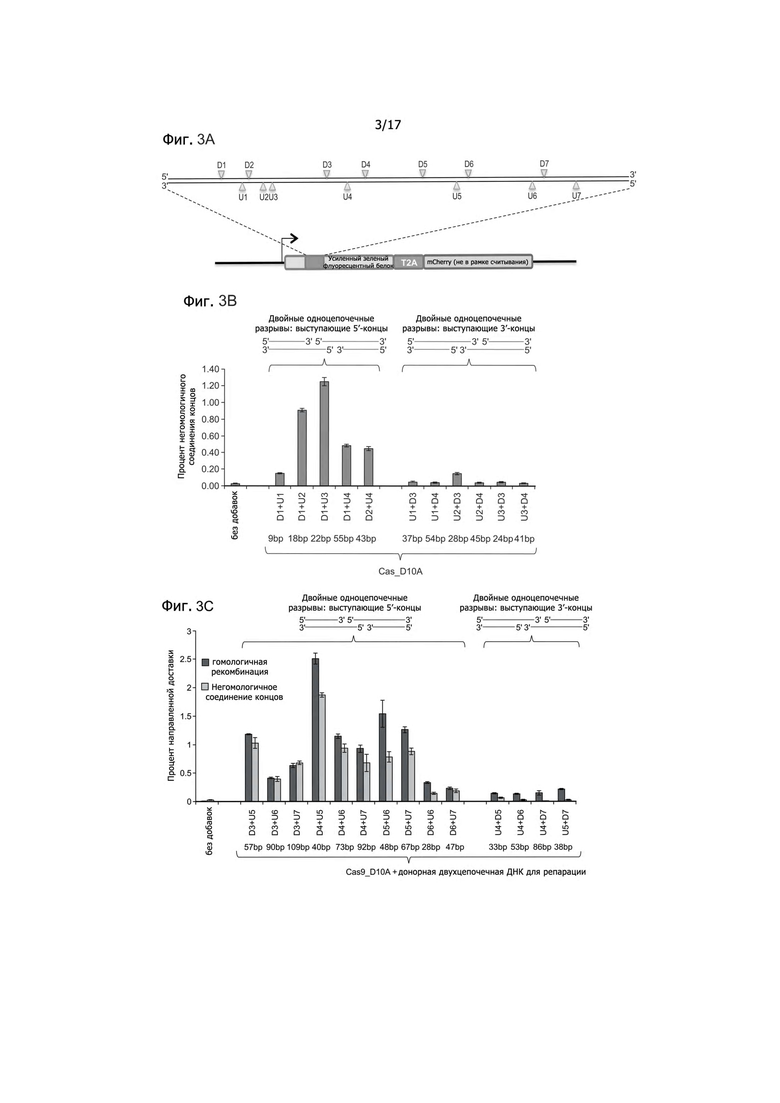
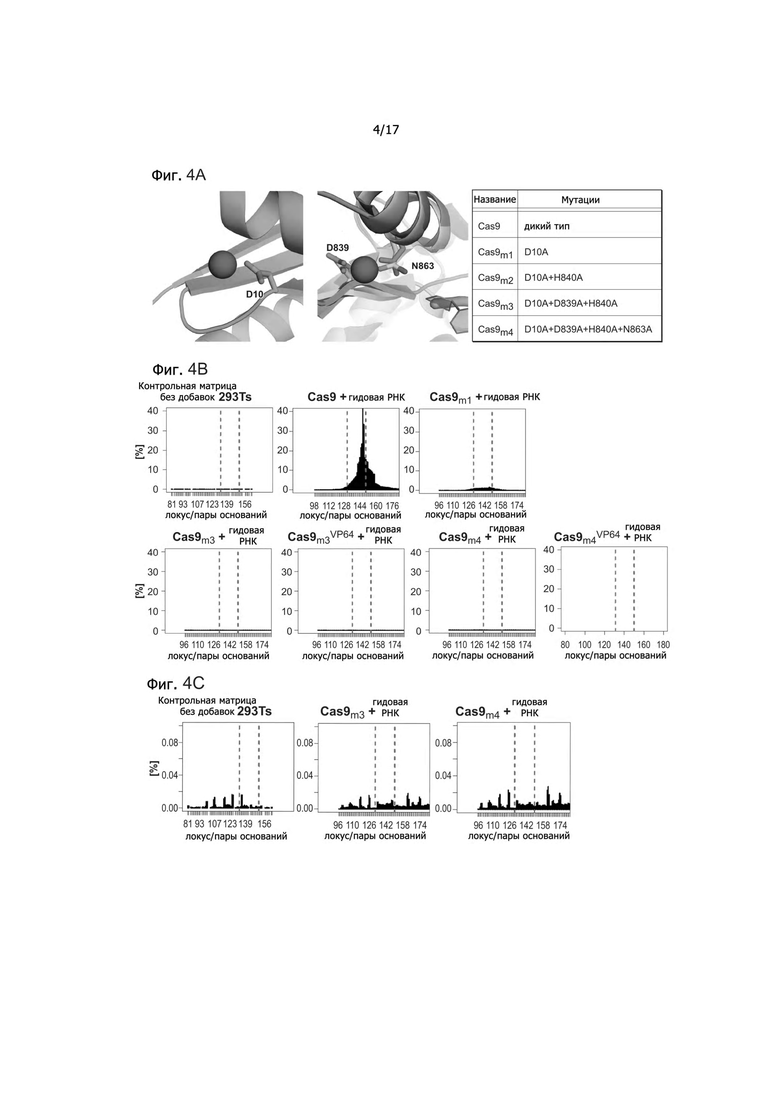
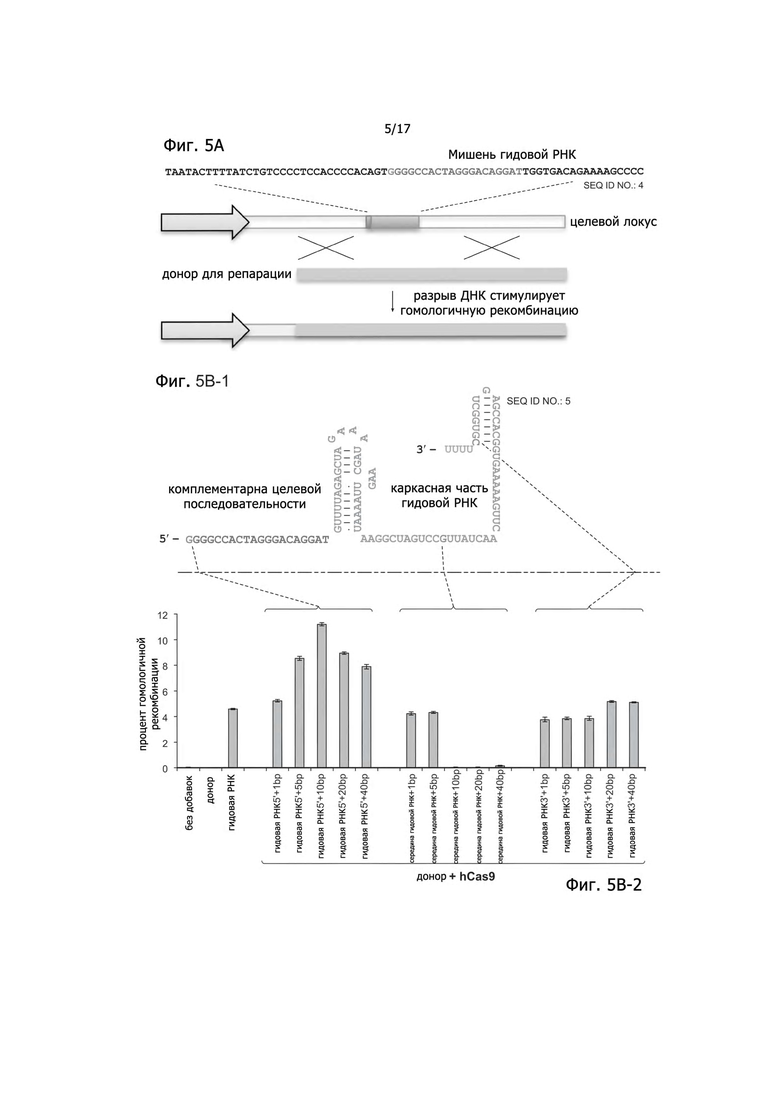
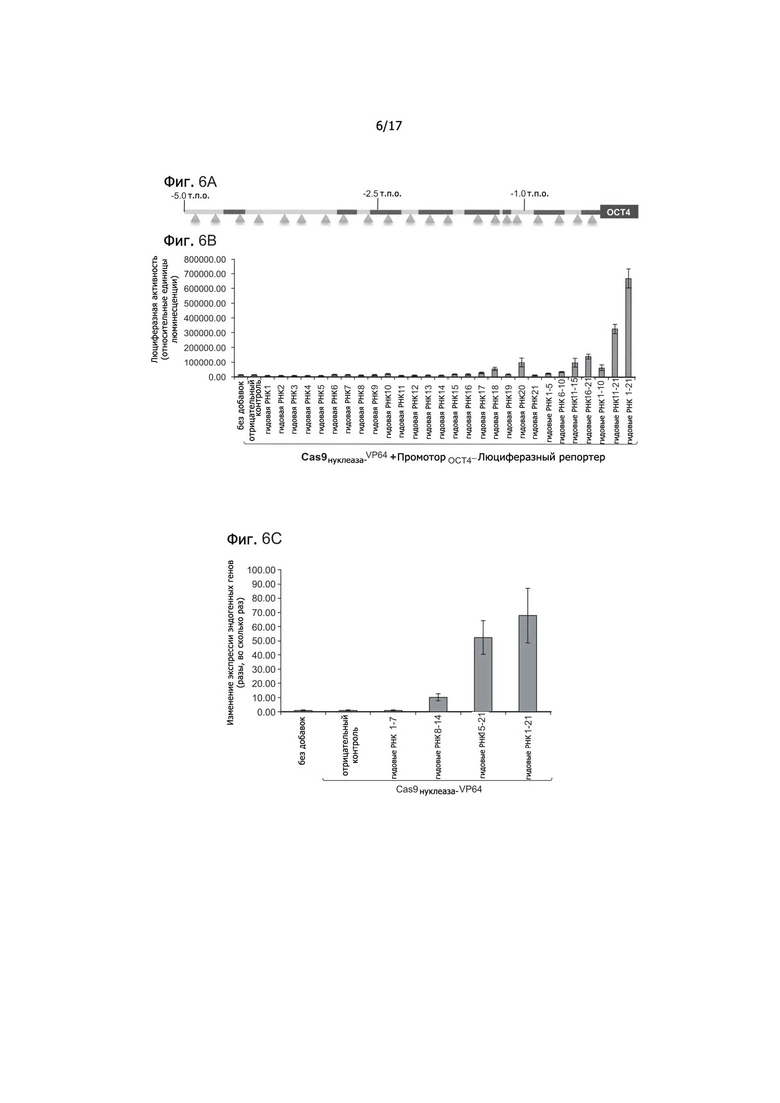
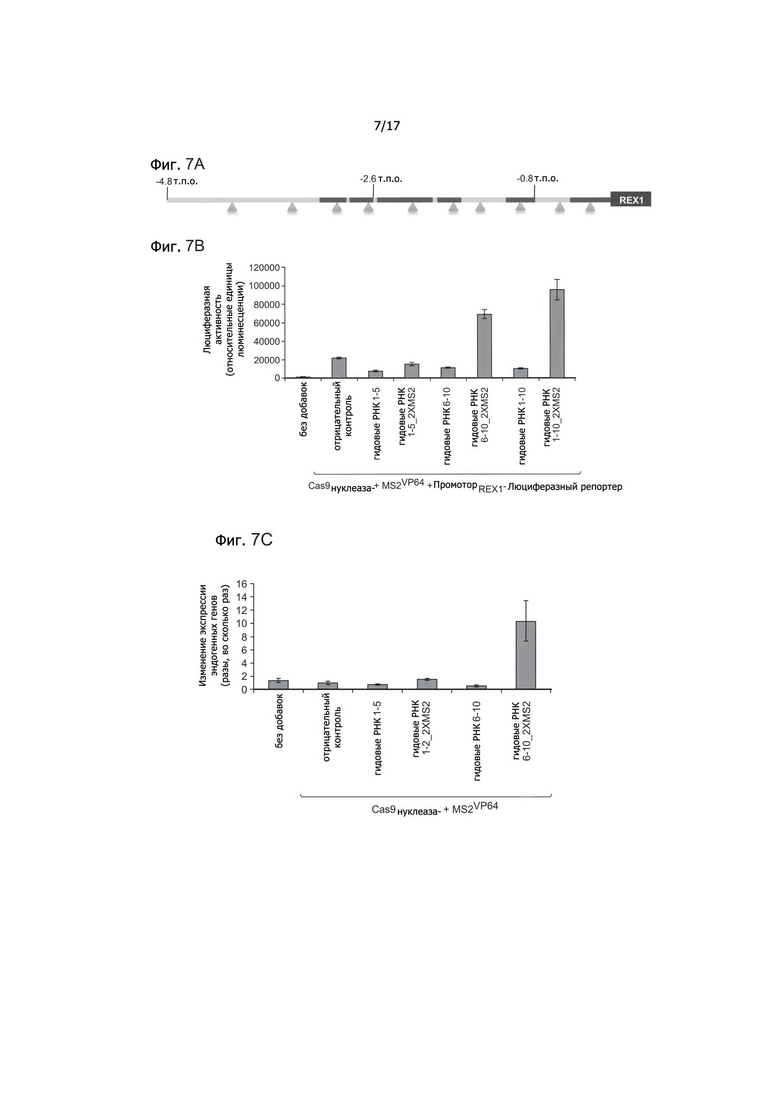

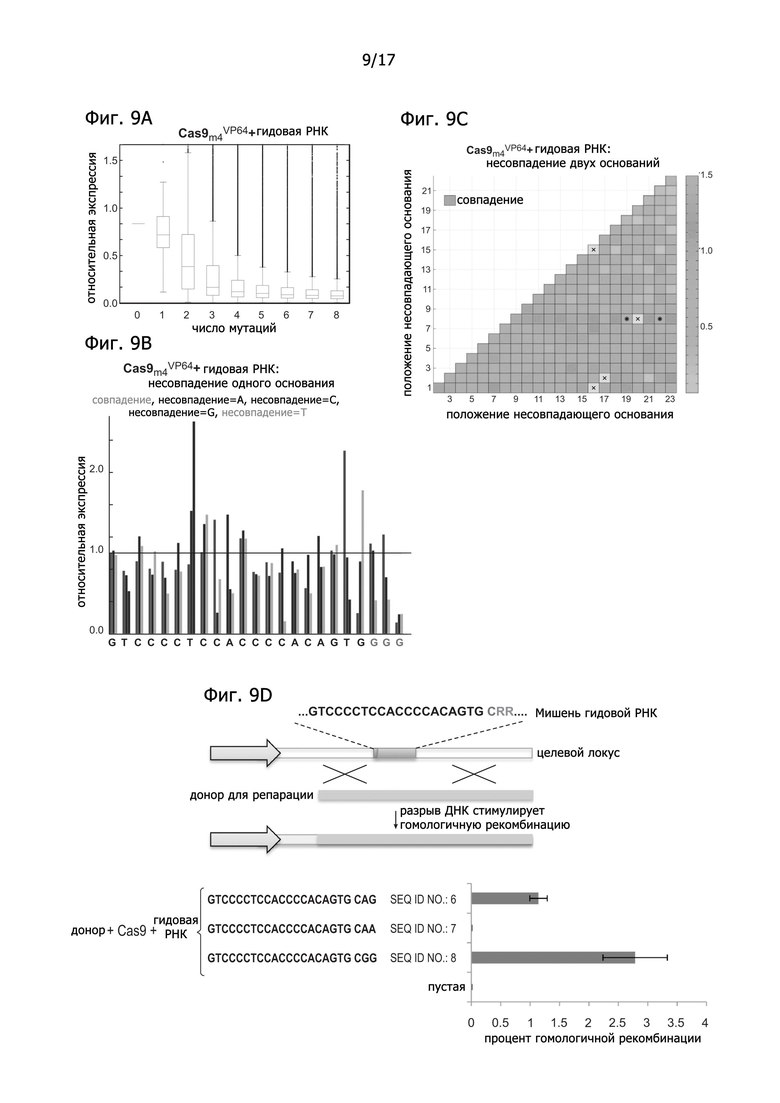
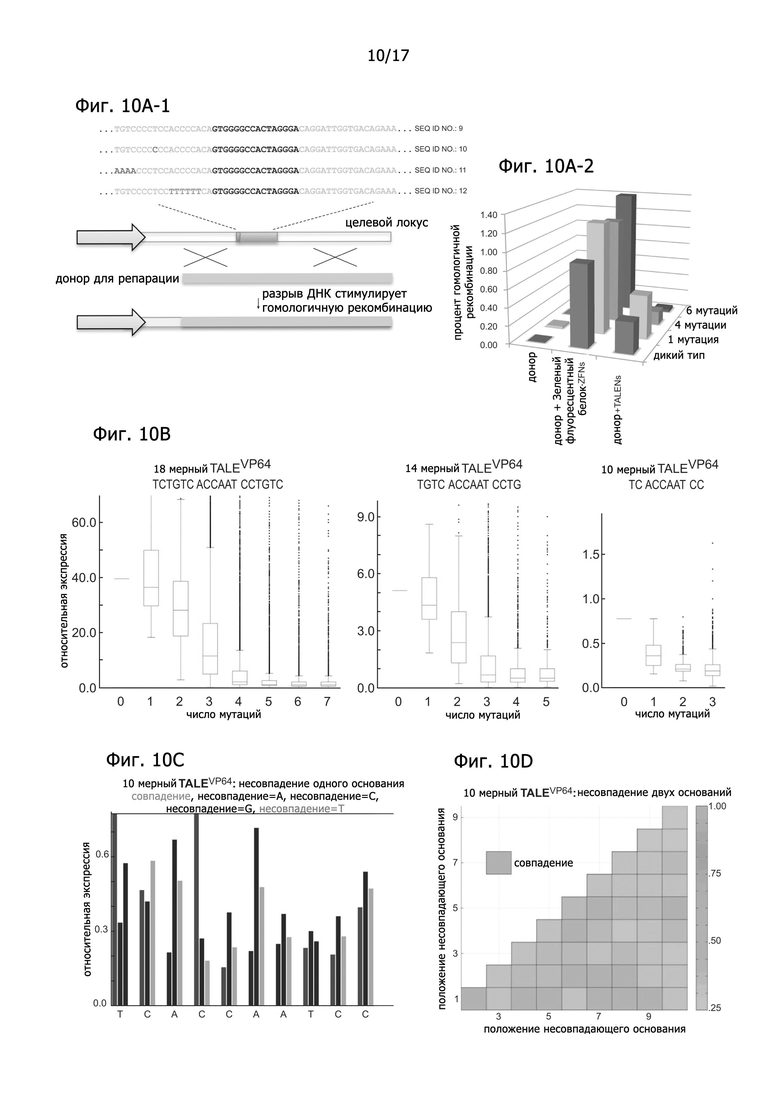

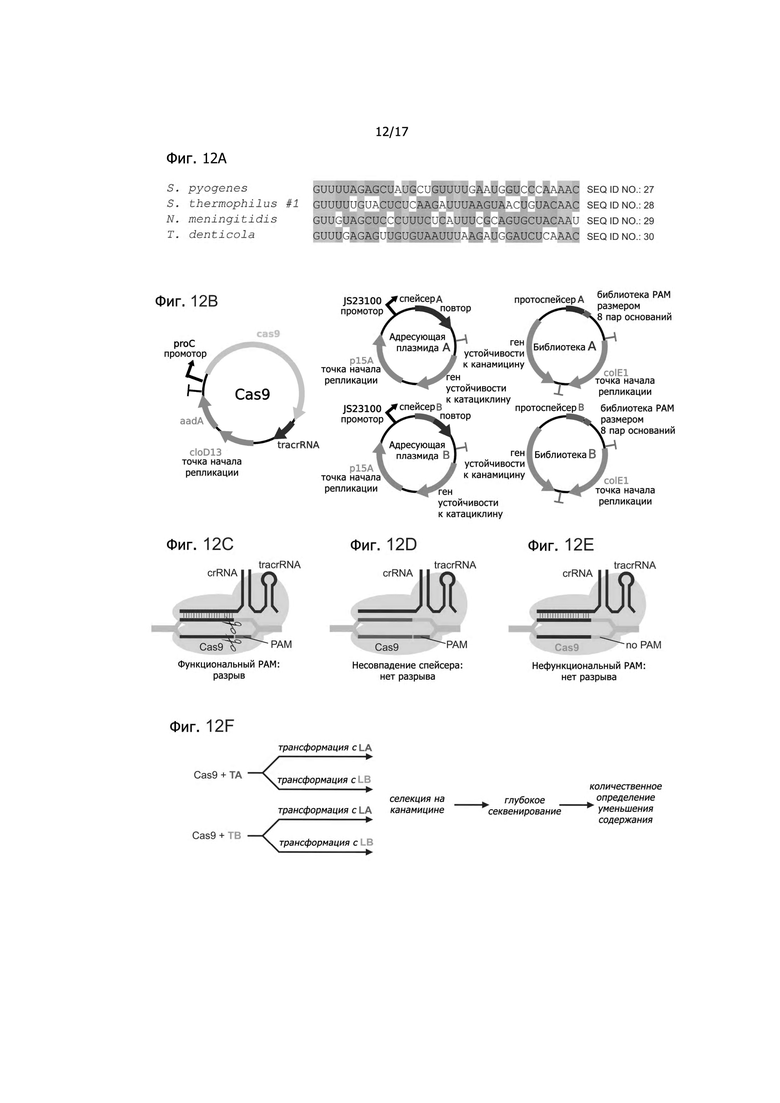
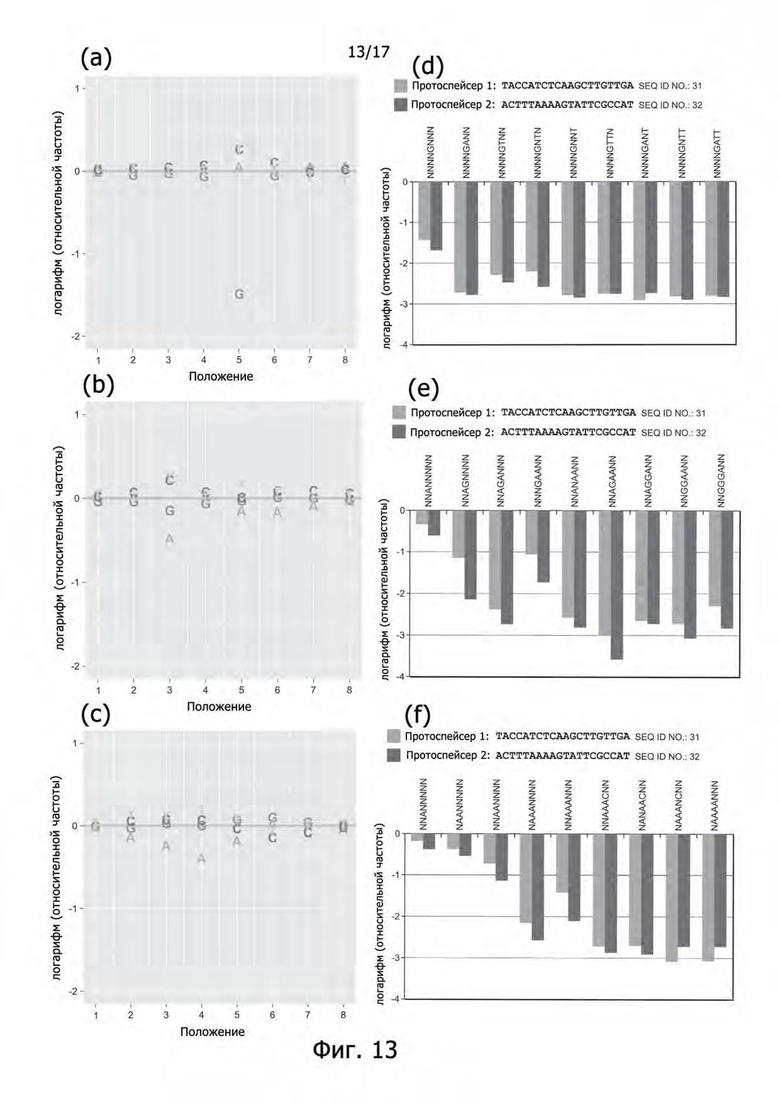
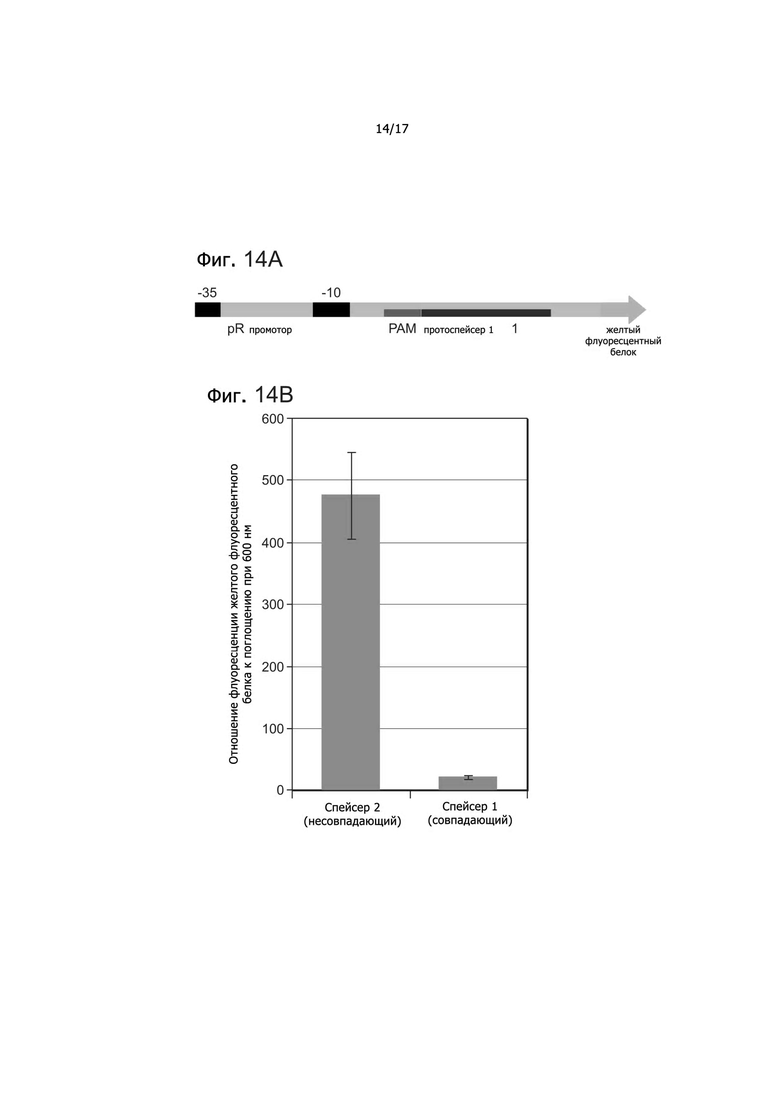
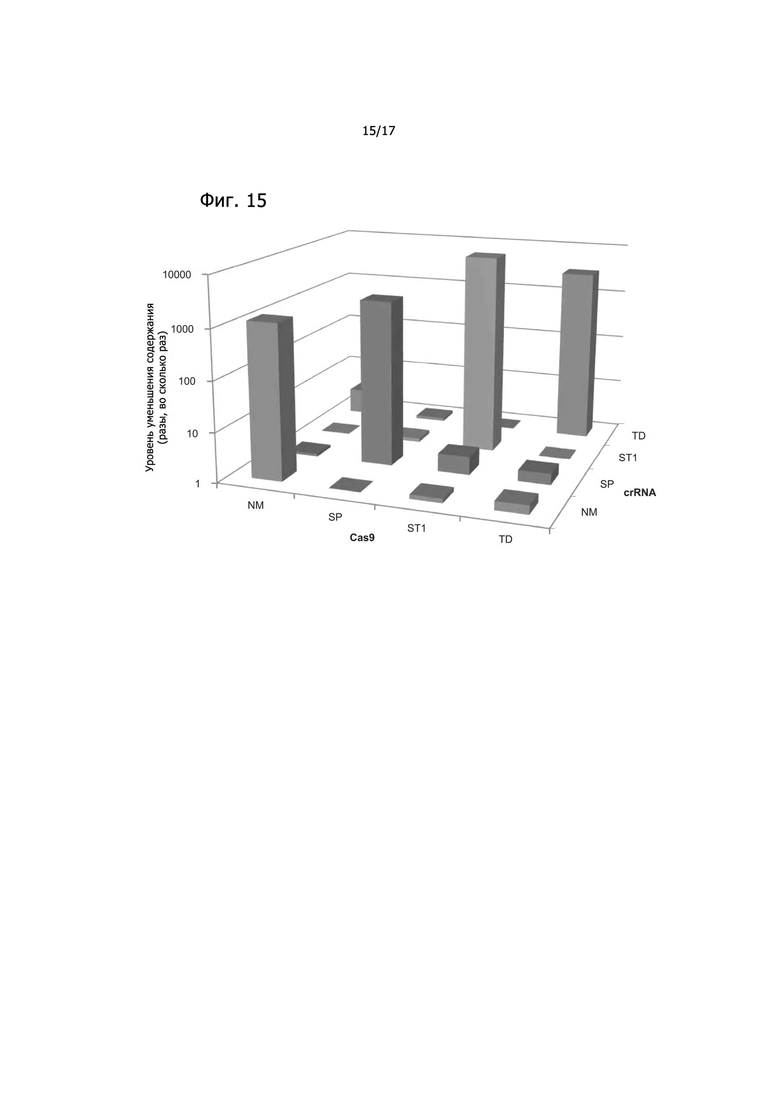
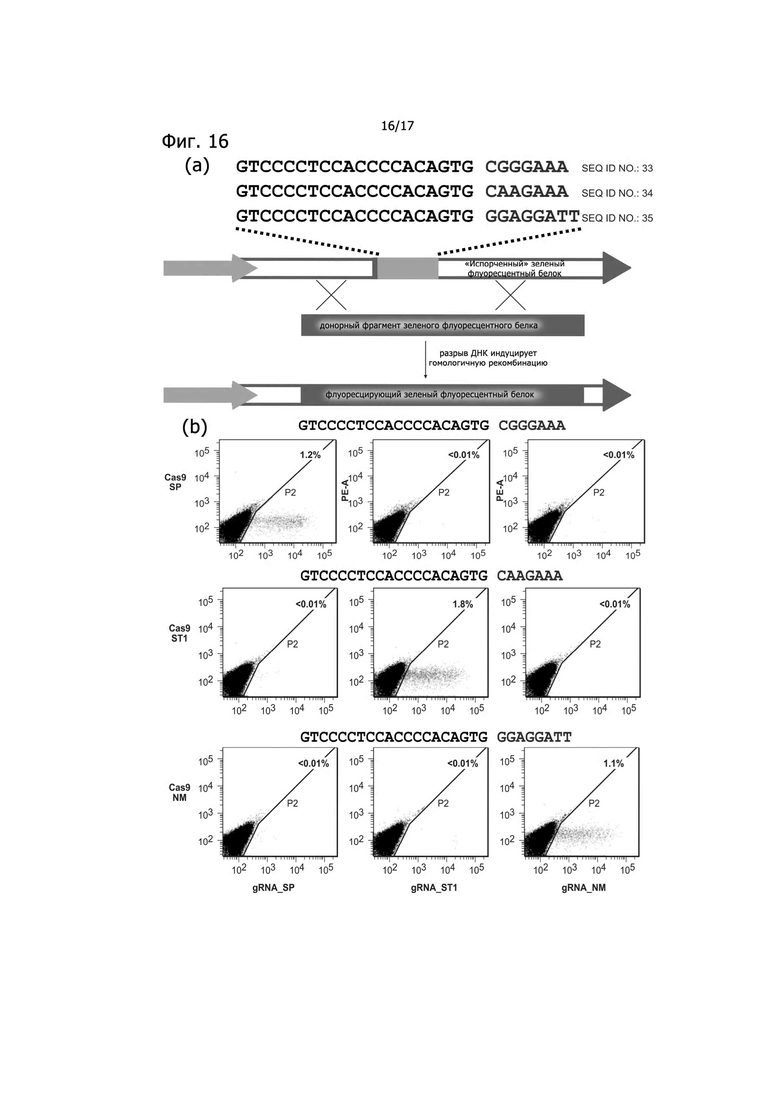
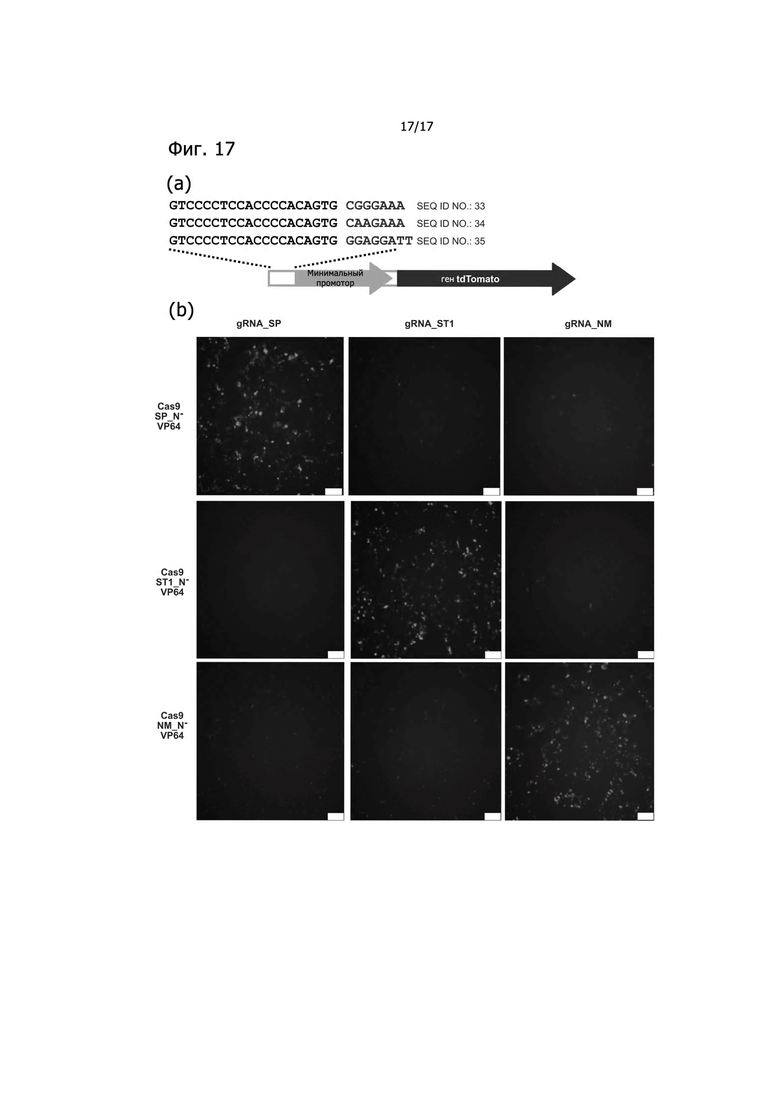
Изобретение относится к области биотехнологии, в частности к способу модулирования экспрессии двух или более целевых генов в эукариотической клетке, предусматривающему использование ортогональных РНК-направляемых не обладающих нуклеазной активностью ДНК-связывающих белков CRISPR-системы Типа II, которые связываются с двумя или более целевыми генами и направляются двумя или более направляющими РНК. Также раскрыты способы изменения одного или более целевых генов в эукариотической клетке, также предусматривающие использование ортогональных РНК-направляемых не обладающих нуклеазной активностью ДНК-связывающих белков CRISPR-системы Типа II. Изобретение также относится к эукариотической клетке, экспрессирующей гены, которые модулируются вышеуказанными способами. Изобретение позволяет эффективно модулировать экспрессию двух или более целевых генов в эукариотической клетке. 6 н. и 44 з.п. ф-лы, 17 ил., 1 табл., 20 пр.
1. Способ модулирования экспрессии двух или более целевых генов в эукариотической клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более направляющих РНК, комплементарных к двум или более целевым генам,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей два или более ортогональных РНК-направляемых не обладающих нуклеазной активностью ДНК-связывающих белков CRISPR-системы Типа II, которые соответственно связываются с двумя или более целевыми генами и направляются двумя или более направляющими РНК,
введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей два или более транскрипционных регуляторных белка,
где экспрессируются направляющая РНК, ортогональные РНК-направляемые не обладающие нуклеазной активностью ДНК-связывающие белки и транскрипционные регуляторные белки,
где транскрипционные регуляторные белки связываются или иным образом соединяются с двумя или несколькими ортогональными РНК-направляемыми не обладающими нуклеазной активностью ДНК-связывающими белками системы CRISPR Типа II или двумя или несколькими направляющими РНК,
где формируется два или более ко-локализованных комплекса между направляющей РНК, ортогональным РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком, транскрипционным регуляторным белком и целевым геном, и где транскрипционный регуляторный белок регулирует экспрессию целевого гена,
и где клетка не является клеткой зародышевой линии человека или эмбриональной клеткой человека.
2. Способ по п. 1, где чужеродная нуклеиновая кислота, кодирующая ортогональный РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок, дополнительно кодирует транскрипционный регуляторный белок, слитый с ортогональным РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком.
3. Способ по п. 1, где чужеродная нуклеиновая кислота, кодирующая две или более направляющих РНК, дополнительно кодирует мишень РНК-связывающего домена, и чужеродная нуклеиновая кислота, кодирующая транскрипционный регуляторный белок, дополнительно кодирует РНК-связывающий домен, слитый с транскрипционным регуляторным белком.
4. Способ по п. 1, где эукариотическая клетка является клеткой человека.
5. Способ по п. 1, где эукариотическая клетка представляет собой клетку дрожжей, клетку растения или клетку животного.
6. Способ по п. 1, где направляющая РНК включает от примерно 10 до примерно 500 нуклеотидов.
7. Способ по п. 1, где направляющая РНК включает от примерно 20 до примерно 100 нуклеотидов.
8. Способ по п. 1, где транскрипционный регуляторный белок представляет собой активатор транскрипции.
9. Способ по п. 1, где транскрипционный регуляторный белок увеличивает экспрессию целевого гена.
10. Способ по п. 1, где транскрипционный регуляторный белок увеличивает экспрессию целевого гена для лечения заболевания или вредного для здоровья состояния.
11. Способ по п. 1, где транскрипционный регуляторный белок представляет собой репрессор транскрипции.
12. Способ по п. 1, где транскрипционный регуляторный белок снижает уровень экспрессии целевого гена.
13. Способ по п. 1, где транскрипционный регуляторный белок снижает уровень экспрессии целевого гена для лечения заболевания или вредного для здоровья состояния.
14. Способ по п. 1, где целевой ген ассоциирован с заболеванием или состоянием, причиняющим ущерб здоровью.
15. Способ по п. 1, где каждая из двух или более направляющих РНК представляет собой слитую tracrРНК-crРНК.
16. Способ по п. 1, где ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
17. Способ по п. 1, где происходит активация первого целевого гена и репрессия второго целевого гена.
18. Способ по п. 1, где первое множество целевых генов активируется и второе множество целевых генов репрессируется.
19. Способ по п. 1, где происходит активация первых одного или более целевых генов и репрессия второго одного или более целевых генов.
20. Способ по п. 1, где РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок представляет собой ортогональный не обладающий нуклеазной активностью белок Cas9.
21. Способ изменения одного или более целевых генов в эукариотической клетке одновременно с регуляцией экспрессии одного или более целевых генов в данной эукариотической клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более направляющих РНК, где, по меньшей мере, одна направляющая РНК является комплементарной к одному или более целевым генам, которые должны быть изменены, и, по меньшей мере, одна направляющая РНК является комплементарной к одному или более целевым генам, которые должны регулироваться,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей 1) ортогональный РНК-направляемый белок с никазной активностью CRISPR-системы Типа II, имеющий один неактивный нуклеазный домен и направляемый одной или более направляющих РНК, комплементарными к одной или более целевым генам, которые должны быть изменены, и 2) ортогональный РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок CRISPR-системы Типа II, который направляется одной или более РНК, комплементарными к целевым генам, которые должны быть отрегулированы,
введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей один или более транскрипционных регуляторных белка,
где экспрессируются направляющая РНК, ортогональный РНК-направляемый белок с никазной активностью, ортогональный РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок и один или несколько транскрипционных регуляторных белка,
где транскрипционные регуляторные белки связываются или иным образом соединяются с двумя или несколькими ортогональными РНК-направляемыми не обладающими нуклеазной активностью ДНК-связывающими белками системы CRISPR Типа II или двумя или несколькими направляющими РНК, комплементарными генам-мишеням, которые должны быть отрегулированы,
где формируется, по меньшей мере, один ко-локализованный комплекс между направляющей РНК, ортогональным РНК-направляемым белком с никазной активностью и целевым геном, который должен быть изменен, и где белок с никазной активностью расщепляет одну цепь целевого гена, который должен быть изменен,
где формируется, по меньшей мере, один ко-локализованный комплекс между направляющей РНК, ортогональным РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком, транскрипционным регуляторным белком и целевым геном, который должен регулироваться, и где транскрипционный регуляторный белок регулирует экспрессию целевого гена, который должен регулироваться,
и где клетка не является клеткой зародышевой линии человека или эмбриональной клеткой человека.
22. Способ изменения одного или более целевых генов в эукариотической клетке одновременно с регуляцией экспрессии одного или более целевых генов в данной эукариотической клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более направляющих РНК, где, по меньшей мере, одна направляющая РНК является комплементарной к одному или более целевым генам, которые должны быть изменены, и, по меньшей мере, одна направляющая РНК является комплементарной к одному или более целевым генам, которые должны регулироваться,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей 1) ортогональный РНК-направляемый белок с нуклеазной активностью CRISPR-системы Типа II, который направляется одной или более направляющими РНК, комплементарными к одному или более целевым генам, которые должны быть изменены, и 2) ортогональный РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок CRISPR-системы Типа II, который направляется одной или более направляющих РНК, комплементарных к целевым генам, которые должны регулироваться,
введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей один или более транскрипционных регуляторных белка,
где экспрессируются направляющая РНК, ортогональный РНК-направляемый белок с нуклеазной активностью, ортогональный РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок и один или несколько транскрипционных регуляторных белка,
где транскрипционные регуляторные белки связаны или иным образом соединены с двумя или несколькими ортогональными РНК-направляемыми не обладающими нуклеазной активностью ДНК-связывающими белками системы CRISPR типа II или двумя или несколькими направляющими РНК, комплементарными с целевыми генами, которые должны регулироваться,
где формируется, по меньшей мере, один ко-локализованный комплекс между направляющей РНК, ортогональным РНК-направляемым белком с нуклеазной активностью и целевым геном, который должен быть изменен, и где нуклеаза расщепляет целевой ген, который должен быть изменен,
где формируется, по меньшей мере, один ко-локализованный комплекс между направляющей РНК, ортогональным РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком, транскрипционным регуляторным белком и целевым геном, который должен регулироваться, и где транскрипционный регуляторный белок регулирует экспрессию целевого гена, который должен регулироваться,
и где клетка не является клеткой зародышевой линии человека или эмбриональной клеткой человека.
23. Эукариотическая клетка, которая экспрессирует два или несколько целевых генов, экспрессия которых модулируется, включающая:
первую чужеродную нуклеиновую кислоту, кодирующую две или более направляющих РНК, комплементарных к двум или более соответствующим целевым генам,
вторую чужеродную нуклеиновую кислоту, кодирующую два или более ортогональных РНК-направляемых не обладающих нуклеазной активностью ДНК-связывающих белка CRISPR-системы Типа II, и
третью чужеродную нуклеиновую кислоту, кодирующую два или более транскрипционных регуляторных белка, где каждая направляющая РНК, ортогональный РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок и транскрипционный регуляторный белок являются членами ко-локализованного комплекса для соответствующего целевого гена,
где транскрипционные регуляторные белки связаны или иным образом соединены с двумя или несколькими ортогональными РНК-направляемыми не обладающими нуклеазной активностью ДНК-связывающими белками системы CRISPR типа II или двумя или несколькими направляющими РНК,
и где клетка не является клеткой зародышевой линии человека или эмбриональной клеткой человека.
24. Эукариотическая клетка по п. 23, где чужеродная нуклеиновая кислота, кодирующая ортогональный РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок, дополнительно кодирует транскрипционный регуляторный белок, слитый с ортогональным РНК-направляемым не обладающим нуклеазной активностью ДНК-связывающим белком.
25. Эукариотическая клетка по п. 23, где чужеродная нуклеиновая кислота, кодирующая одну или более направляющих РНК, дополнительно кодирует мишень РНК-связывающего домена и чужеродная нуклеиновая кислота, кодирующая транскрипционный регуляторный белок, дополнительно кодирует РНК-связывающий домен, слитый с транскрипционным регуляторным белком.
26. Эукариотическая клетка по п. 23, где эукариотическая клетка является клеткой человека.
27. Эукариотическая клетка по п. 23, где эукариотическая клетка представляет собой клетку дрожжей, клетку растения или клетку животного.
28. Эукариотическая клетка по п. 23, где направляющая РНК включает от примерно 10 до примерно 500 нуклеотидов.
29. Эукариотическая клетка по п. 23, где направляющая РНК включает от примерно 20 до примерно 100 нуклеотидов.
30. Эукариотическая клетка по п. 23, где транскрипционный регуляторный белок представляет собой активатор транскрипции.
31. Эукариотическая клетка по п. 23, где транскрипционный регуляторный белок увеличивает экспрессию целевого гена.
32. Эукариотическая клетка по п. 23, где транскрипционный регуляторный белок увеличивает экспрессию целевого гена для лечения заболевания или вредного для здоровья состояния.
33. Эукариотическая клетка по п. 23, где целевой ген ассоциирован с заболеванием или состоянием, причиняющим ущерб здоровью.
34. Эукариотическая клетка по п. 23, где каждая из двух или более направляющих РНК представляет собой слитую tracrРНК-crРНК.
35. Эукариотическая клетка по п. 23, где ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
36. Эукариотическая клетка по п. 23, где РНК-направляемый не обладающий нуклеазной активностью ДНК-связывающий белок представляет собой ортогональный не обладающий нуклеазной активностью белок Cas9.
37. Способ изменения двух или более целевых генов в эукариотической клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более направляющих РНК, комплементарных к двум или более целевым генам,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей два или более ортогональных РНК-направляемых ДНК-связывающих белков с никазной активностью CRISPR-системы Типа II, которые соответственно связываются с двумя или более целевыми генами и направляются двумя или более направляющими РНК,
где экспрессируются направляющая РНК и ортогональные РНК-направляемые ДНК-связывающие белки с никазной активностью,
где формируется два или более ко-локализованных комплекса между направляющей РНК, РНК-направляемым ДНК-связывающим белком с никазной активностью и целевым геном, и
где два или более ортогональных РНК-направляемых ДНК-связывающих белков с никазной активностью разрезают одну цепь у двух или более целевых генов,
и где клетка не является клеткой зародышевой линии человека или эмбриональной клеткой человека.
38. Способ по п. 37, где эукариотическая клетка является клеткой человека.
39. Способ по п. 37, где эукариотическая клетка представляет собой клетку дрожжей, клетку растения или клетку животного.
40. Способ по п. 37, где направляющая РНК включает от примерно 10 до примерно 500 нуклеотидов.
41. Способ по п. 37, где направляющая РНК включает от примерно 20 до примерно 100 нуклеотидов.
42. Способ по п. 37, где каждая из двух или более направляющих РНК представляет собой слитую tracrРНК-crРНК.
43. Способ по п. 37, где ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
44. Способ изменения двух или более целевых генов в эукариотической клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более направляющих РНК, комплементарных к двум или более целевым нуклеиновым кислотам,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей два или более ортогональных РНК-направляемых ДНК-связывающих белков CRISPR-системы Типа II, которые соответственно связываются с двумя или более целевыми генами и направляются двумя или более направляющими РНК,
где экспрессируются направляющая РНК и ортогональные РНК-направляемые ДНК-связывающие белки,
где формируется два или более ко-локализованных комплекса между направляющей РНК, РНК-направляемым ДНК-связывающим белком и целевым геном, и
где два или более РНК-направляемых ДНК-связывающих белков разрезают два или более целевые гена,
и где клетка не является клеткой зародышевой линии человека или эмбриональной клеткой человека.
45. Способ по п. 44, где эукариотическая клетка является клеткой человека.
46. Способ по п. 44, где эукариотическая клетка представляет собой клетку дрожжей, клетку растения или клетку животного.
47. Способ по п. 44, где направляющая РНК включает от примерно 10 до примерно 500 нуклеотидов.
48. Способ по п. 44, где направляющая РНК включает от примерно 20 до примерно 100 нуклеотидов.
49. Способ по п. 44, где каждая из двух или более направляющих РНК представляет собой слитую tracrРНК-crРНК.
50. Способ по п. 44, где ДНК представляет собой геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
| QI LEI S., et al., Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression, Cell, 2013, Vol.152, N.5, pp | |||
| Двухпроводная автоматическая тормозная система с сжатым воздухом | 1924 |
|
SU1173A1 |
| JINEK M | |||
| et al | |||
| A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity Science, 2012, 337(6096), pp.816-821 | |||
| US2010076057 A1, 25.03.2010 | |||
| TAM J.P | |||
| et al., Orthogonal | |||
Авторы
Даты
2021-05-25—Публикация
2014-07-08—Подача