Данные о родственных заявках
Настоящая заявка претендует на приоритет по предварительной заявке на патент США No. 61/830,787, поданной 4 июня 2013 г., которая включена сюда путем ссылки во всей полноте на все случаи.
Заявление о правительственных интересах
Настоящее изобретение было совершено при поддержке правительства по гранту No. Р50 HG005550 от Национальных институтов здравоохранения и DE-FG02-02ER63445 от Министерства энергетики США. Правительство имеет определенные права на это изобретение.
Уровень техники
Бактериальные и архейные системы CRISPR-Cas зависят от коротких направляющих РНК, которые в комплексе с белками Сas направляют деградацию комплементарных последовательностей, присутствующих в проникающей чужеродной нуклеиновой кислоте. См. Deltcheva Е. et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNAse III. Nature 471, 602-607 (2011); Gasiunas G, Barrangou R., Horvath P. & Siksnys V. Cas9-rRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the USA 109, E2579-2586 (2012); Jinek M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012); Sapranauskas R. et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Research 39, 9275-9282 (2011); и Bhaya D., Davison M. & Barrangou R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation. Annual Review of Genetics 45, 273-297 (2011). Недавняя реконструкция системы CRISPR S. pyogenes II-го типа in vitro показала, что crPHK ("CRISPR-РНК"), слитая с обычной транс-кодируемой tracr-РНК ("транс-активирующая CRISPR-РНК"), достаточна для того, чтобы направить белок Cas9 на специфичное к последовательности расщепление целевых последовательностей ДНК, соответствующих этой cr-РНК. Экспрессия направляющей РНК (gRNA, гидРНК), гомологичной сайту мишени, приводит к привлечению Cas9 и деградации ДНК мишени. См. Н. Deveau et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. Journal of Bacteriology 190, 1390 (Feb, 2008).
Сущность изобретения
Различные аспекты настоящего изложения касаются комплекса из направляющей РНК, ДНК-связывающего белка и последовательности двухцепочечной ДНК-мишени. Согласно некоторым аспектам, ДНК-связывающие белки в рамках настоящего изобретения включают белок, образующий комплекс с направляющей РНК, причем направляющая РНК направляет комплекс на последовательность двухцепочечной ДНК. при этом комплекс связывается с этой последовательностью ДНК. Этот аспект настоящего изобретения может быть назван совместной локализацией РНК и ДНК-связывающего белка на или с двухцепочечной ДНК. Таким образом, комплекс ДНК-связывающего белка с направляющей РНК можно использовать для локализации регулирующего транскрипцию белка или домена на ДНК мишени с тем, чтобы регулировать экспрессию целевой ДНК.
Согласно некоторым аспектам, предусмотрен способ модулирования экспрессии целевой нуклеиновой кислоты в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей одну или несколько РНК (рибонуклеиновых кислот), комплементарных ДНК (дезоксирибонуклеиновой кислоте), причем ДНК включает целевую нуклеиновую кислоту, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей РНК-направляемый безнуклеазный ДНК-связывающий белок, который связывается с ДНК и направляется одной или несколькими РНК, введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей регулирующий транскрипцию белок или домен, причем одна или несколько РНК, РНК-направляемый безнуклеазный ДНК-связывающий белок и регулирующий транскрипцию белок или домен экспрессируются, при этом одна или несколько РНК, РНК-направляемый безнуклеазный ДНК-связывающий белок и регулирующий транскрипцию белок или домен совместно локализуются на ДНК, а регулирующий транскрипцию белок или домен регулирует экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая РНК-направляемый безнуклеазный ДНК-связывающий белок, также кодирует регулирующий транскрипцию белок или домен, слитый с РНК-направляемым безнуклеазным ДНК-связывающим белком. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или несколько РНК, также кодирует мишень РНК-связывающего домена, а чужеродная нуклеиновая кислота, кодирующая регулирующий транскрипцию белок или домен, также кодирует РНК-связывающий домен, слитый с регулирующим транскрипцию белком или доменом.
Согласно одному аспекту, клетка представлена эукариотической клеткой. Согласно одному аспекту, клетка представлена клеткой дрожжей, клеткой растений или клеткой животных. Согласно одному аспекту, клетка представлена клеткой млекопитающих.
Согласно одному аспекту, РНК содержит от 10 до 500 нуклеотидов. Согласно одному аспекту, РНК содержит от 20 до 100 нуклеотидов.
Согласно одному аспекту, регулирующий транскрипцию белок или домен является активатором транскрипции. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты для лечения заболевания или болезненного состояния. Согласно одному аспекту, целевая нуклеиновая кислота связана с заболеванием или болезненным состоянием.
Согласно одному аспекту, одна или несколько РНК представлены направляющей РНК. Согласно одному аспекту, одна или несколько РНК представляют собой слияния tracr-РНК и cr-РНК. Согласно одному аспекту, направляющая РНК включает в себя последовательность спейсера и последовательность, гибридизующуюся с tracr (tracr mate). Направляющая РНК также может включать в себя последовательность tracr, часть которой гибридизуется с последовательностью tracr mate. Направляющая РНК также может включать в себя последовательность линкерной нуклеиновой кислоты, которая связывает последовательность tracr mate и последовательность tracr, что приводит к слиянию tracr-РНК и cr-РНК. Последовательность спейсера связывается с целевой ДНК, как-то посредством гибридизации.
Согласно одному аспекту, направляющая РНК включает в себя усеченную последовательность спейсера. Согласно одному аспекту, направляющая РНК включает в себя усеченную последовательность спейсера, укороченную на 1 основание на 5'-конце последовательности спейсера. Согласно одному аспекту, направляющая РНК включает в себя усеченную последовательность спейсера, укороченную на 2 основания на 5'-конце последовательности спейсера. Согласно одному аспекту, направляющая РНК включает в себя усеченную последовательность спейсера, укороченную на 3 основания на 5'-конце последовательности спейсера. Согласно одному аспекту, направляющая РНК включает в себя усеченную последовательность спейсера, укороченную на 4 основания на 5'-конце последовательности спейсера. Соответственно, последовательность спейсера может быть укорочена на 1-4 основания на 5'-конце последовательности спейсера.
Согласно некоторым воплощениям, последовательность спейсера может содержать от 16 до 20 нуклеотидов, гибридизующихся с последовательностью целевой нуклеиновой кислоты. Согласно некоторым воплощениям, последовательность спейсера может содержать примерно 20 нуклеотидов, гибридизующихся с последовательностью целевой нуклеиновой кислоты.
Согласно некоторым аспектам, последовательность линкерной нуклеиновой кислоты может содержать от 4 до 6 нуклеотидов.
Согласно некоторым аспектам, последовательность tracr может содержать от 60 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 64 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 65 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 66 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 67 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 68 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 69 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 70 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 80 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 90 до 500 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 100 до 500 нуклеотидов.
Согласно некоторым аспектам, последовательность tracr может содержать от 60 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 64 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 65 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 66 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 67 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 68 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 69 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 70 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 80 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 90 до 200 нуклеотидов. Согласно некоторым аспектам, последовательность tracr может содержать от 100 до 200 нуклеотидов.
Типичная направляющая РНК представлена на фиг. 5В.
Согласно одному аспекту, ДНК представлена геномной ДНК, митохондриальной ДНК, вирусной ДНК либо экзогенной ДНК.
Согласно некоторым аспектам, предусмотрен способ модулирования экспрессии целевой нуклеиновой кислоты в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей одну или несколько РНК (рибонуклеиновых кислот), комплементарных ДНК (дезоксирибонуклеиновой кислоте), причем ДНК включает целевую нуклеиновую кислоту, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей РНК-направляемый безнуклеазный ДНК-связывающий белок из системы CRISP II типа, который связывается с ДНК и направляется одной или несколькими РНК, введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей регулирующий транскрипцию белок или домен, причем одна или несколько РНК, РНК-направляемый безнуклеазный ДНК-связывающий белок и регулирующий транскрипцию белок или домен экспрессируются, при этом одна или несколько РНК, РНК-направляемый безнуклеазный ДНК-связывающий белок и регулирующий транскрипцию белок или домен совместно локализуются на ДНК, а регулирующий транскрипцию белок или домен регулирует экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая РНК-направляемый безнуклеазный ДНК-связывающий белок из системы CRISP II типа, также кодирует регулирующий транскрипцию белок или домен, слитый с РНК-направляемым безнуклеазным ДНК-связывающим белком из системы CRISP II типа. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или несколько РНК, также кодирует мишень РНК-связывающего домена, а чужеродная нуклеиновая кислота, кодирующая регулирующий транскрипцию белок или домен, также кодирует РНК-связывающий домен, слитый с регулирующим транскрипцию белком или доменом.
Согласно одному аспекту, клетка представлена эукариотической клеткой. Согласно одному аспекту, клетка представлена клеткой дрожжей, клеткой растений или клеткой животных. Согласно одному аспекту, клетка представлена клеткой млекопитающих.
Согласно одному аспекту, РНК содержит от 10 до 500 нуклеотидов. Согласно одному аспекту, РНК содержит от 20 до 100 нуклеотидов.
Согласно одному аспекту, регулирующий транскрипцию белок или домен является активатором транскрипции. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты для лечения заболевания или болезненного состояния. Согласно одному аспекту, целевая нуклеиновая кислота связана с заболеванием или болезненным состоянием.
Согласно одному аспекту, одна или несколько РНК представлены направляющей РНК. Согласно одному аспекту, одна или несколько РНК представляют собой слияния tracr-РНК и cr-РНК.
Согласно одному аспекту, ДНК представлена геномной ДНК, митохондриальной ДНК, вирусной ДНК либо экзогенной ДНК.
Согласно одному аспекту, предусмотрен способ модулирования экспрессии целевой нуклеиновой кислоты в клетке, предусматривающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей одну или несколько РНК (рибонуклеиновых кислот), комплементарных ДНК (дезоксирибонуклеиновой кислоте), причем ДНК включает целевую нуклеиновую кислоту, введение второй чужеродной нуклеиновой кислоты, кодирующей безнуклеазный белок Cas9, который связывается с ДНК и направляется одной или несколькими РНК, и введение в клетку третей чужеродной нуклеиновой кислоты, кодирующей регулирующий транскрипцию белок или домен, при этом одна или несколько РНК, безнуклеазный белок Cas9, и регулирующий транскрипцию белок или домен экспрессируются, и регулирующий транскрипцию белок или домен совместно локализуются на ДНК, а регулирующий транскрипцию белок или домен регулирует экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая безнуклеазный белок Cas9, также кодирует регулирующий транскрипцию белок или домен, слитый с безнуклеазным белком Cas9. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или несколько РНК, также кодирует мишень РНК-связывающего домена, а чужеродная нуклеиновая кислота, кодирующая регулирующий транскрипцию белок или домен, также кодирует РНК-связывающий домен, слитый с регулирующим транскрипцию белком или доменом.
Согласно одному аспекту, клетка представлена эукариотической клеткой. Согласно одному аспекту, клетка представлена клеткой дрожжей, клеткой растений или клеткой животных. Согласно одному аспекту, клетка представлена клеткой млекопитающих.
Согласно одному аспекту, РНК содержит от 10 до 500 нуклеотидов. Согласно одному аспекту, РНК содержит от 20 до 100 нуклеотидов.
Согласно одному аспекту, регулирующий транскрипцию белок или домен является активатором транскрипции. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты для лечения заболевания или болезненного состояния. Согласно одному аспекту, целевая нуклеиновая кислота связана с заболеванием или болезненным состоянием.
Согласно одному аспекту, одна или несколько РНК представлены направляющей РНК. Согласно одному аспекту, одна или несколько РНК представляют собой слияния tracr-РНК и cr-РНК.
Согласно одному аспекту, ДНК представлена геномной ДНК, митохондриальной ДНК, вирусной ДНК либо экзогенной ДНК.
Согласно одному аспекту, предусмотрена клетка, содержащая первую чужеродную нуклеиновую кислоту, кодирующую одну или несколько РНК, комплементарных ДНК, причем ДНК включает целевую нуклеиновую кислоту, вторую чужеродную нуклеиновую кислоту, кодирующую РНК-направляемый безнуклеазный ДНК-связывающий белок, и третью чужеродную нуклеиновую кислоту, кодирующую регулирующий транскрипцию белок или домен, при этом одна или несколько РНК, РНК-направляемый безнуклеазный ДНК-связывающий белок и регулирующий транскрипцию белок или домен входят в состав комплекса совместной локализации для целевой нуклеиновой кислоты.
Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая РНК-направляемый безнуклеазный ДНК-связывающий белок, также кодирует регулирующий транскрипцию белок или домен, слитый с РНК-направляемым безнуклеазным ДНК-связывающим белком. Согласно одному аспекту, чужеродная нуклеиновая кислота, кодирующая одну или несколько РНК, также кодирует мишень РНК-связывающего домена, а чужеродная нуклеиновая кислота, кодирующая регулирующий транскрипцию белок или домен, также кодирует РНК-связывающий домен, слитый с регулирующим транскрипцию белком или доменом.
Согласно одному аспекту, клетка представлена эукариотической клеткой. Согласно одному аспекту, клетка представлена клеткой дрожжей, клеткой растений или клеткой животных. Согласно одному аспекту, клетка представлена клеткой млекопитающих.
Согласно одному аспекту, РНК содержит от 10 до 500 нуклеотидов. Согласно одному аспекту, РНК содержит от 20 до 100 нуклеотидов.
Согласно одному аспекту, регулирующий транскрипцию белок или домен является активатором транскрипции. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты. Согласно одному аспекту, регулирующий транскрипцию белок или домен усиливает экспрессию целевой нуклеиновой кислоты для лечения заболевания или болезненного состояния. Согласно одному аспекту, целевая нуклеиновая кислота связана с заболеванием или болезненным состоянием.
Согласно одному аспекту, одна или несколько РНК представлены направляющей РНК. Согласно одному аспекту, одна или несколько РНК представляют собой слияния tracr-РНК и cr-РНК.
Согласно одному аспекту, ДНК представлена геномной ДНК, митохондриальной ДНК, вирусной ДНК либо экзогенной ДНК.
Согласно некоторым аспектам, РНК-направляемый безнуклеазный ДНК-связывающий белок представлен РНК-направляемым безнуклеазным ДНК-связывающим белком системы CRISPR II-го типа. Согласно некоторым аспектам, РНК-направляемый безнуклеазный ДНК-связывающий белок представляет собой безнуклеазный белок Cas9.
Согласно некоторым аспектам, предусмотрен способ изменения целевой нуклеиновой кислоты входящей в состав ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к области ДНК прилегающей к целевой нуклеиновой кислоте, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу, который направляется двумя или несколькими РНК, причем эти две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза экспрессируются, при этом по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрывов (nicks).
Согласно одному аспекту, предусмотрен способ изменения целевой нуклеиновой кислоты входящей в состав ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к области ДНК прилегающей к целевой нуклеиновой кислоте, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу системы CRISPR II-го типа, который направляется двумя или несколькими РНК, причем эти две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза системы CRISPR II-го типа экспрессируются, при этом по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза системы CRISPR II-го типа локализуется совместно с двумя или несколькими РНК на целевой нуклеиновой кислоте - ДНК и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрывов (nicks).
Согласно одному аспекту, предусмотрен способ изменения целевой нуклеиновой кислоты входящей в состав ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к области ДНК прилегающей к целевой нуклеиновой кислоте, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один белок-никазу Cas9 с одним неактивным нуклеазным доменом, который направляется двумя или несколькими РНК, причем эти две или несколько РНК и по меньшей мере один белок-никаза Cas9 экспрессируются, при этом по меньшей мере один белок-никаза Cas9 локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрывов (nicks).
В соответствии со способами изменения целевой нуклеиновой кислоты входящей в состав ДНК, два или несколько соседних одноцепочечных разрывов находятся на одной той же нити двухцепочечной ДНК. Согласно одному аспекту, два или несколько соседних одноцепочечных разрывов находятся на одной и той же нити двухцепочечной ДНК, что приводит к гомологичной рекомбинации. Согласно одному аспекту, два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК. Согласно одному аспекту, два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы. Согласно одному аспекту, два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к негомологичному соединению концов. Согласно одному аспекту, два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК и смещены относительно друг друга. Согласно одному аспекту, два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК, смещены относительно друг друга и создают двухцепочечные разрывы. Согласно одному аспекту, два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК, смещены относительно друг друга и создают двухцепочечные разрывы, что приводит к негомологичному соединению концов. Согласно одному аспекту, способ дополнительно включает введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей последовательность донорной нуклеиновой кислоты, при этом два или несколько одноцепочечных разрывов приводят к гомологичной рекомбинации целевой нуклеиновой кислоты с последовательностью донорной нуклеиновой кислоты.
Согласно одному аспекту, предусмотрен способ изменения целевой нуклеиновой кислоты входящей в состав ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к области ДНК прилегающей к целевой нуклеиновой кислоте, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу, который направляется двумя или несколькими РНК, причем эти две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза экспрессируются, при этом по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрывов, причем эти два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, тем самым предотвращая экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, предусмотрен способ изменения целевой нуклеиновой кислоты входящей в состав ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к области ДНК прилегающей к целевой нуклеиновой кислоте, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу системы CRISPR II-го типа, который направляется двумя или несколькими РНК, причем эти две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза системы CRISPR II-го типа экспрессируются, при этом по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза системы CRISPR II-го типа локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрывов, причем эти два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, тем самым предотвращая экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, предусмотрен способ изменения целевой нуклеиновой кислоты входящей в состав ДНК в клетке, включающий введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к области ДНК прилегающей к целевой нуклеиновой кислоте, введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один белок-никазу Cas9 с одним неактивным нуклеазным доменом, который направляется двумя или несколькими РНК, причем эти две или несколько РНК и по меньшей мере один белок-никаза Cas9 экспрессируются, при этом по меньшей мере один белок-никаза Cas9 локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрывов, причем эти два или несколько соседних одноцепочечных разрывов находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, тем самым предотвращая экспрессию целевой нуклеиновой кислоты.
Согласно одному аспекту, предусмотрены клетки, содержащие первую чужеродную нуклеиновую кислоту, кодирующую две или несколько РНК, причем каждая РНК комплементарна к области ДНК прилегающей к целевой нуклеиновой кислоте, и вторую чужеродную нуклеиновую кислоту, кодирующую по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу, при этом две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза входят в состав комплекса совместной локализации для ДНК целевой нуклеиновой кислоты.
Согласно одному аспекту, РНК-направляемый ДНК-связывающий белок-никаза представлен РНК-направляемым ДНК-связывающим белком никазы из системы CRISPR II-го типа. Согласно одному аспекту, РНК-направляемый ДНК-связывающий белок-никаза представлен белком никазы Cas9 с одним неактивным нуклеазным доменом.
Согласно одному аспекту, клетка представлена эукариотической клеткой. Согласно одному аспекту, клетка представлена клеткой дрожжей, клеткой растений или клеткой животных. Согласно одному аспекту, клетка представлена клеткой млекопитающих.
Согласно одному аспекту, РНК содержит от 10 до 500 нуклеотидов. Согласно одному аспекту, РНК содержит от 20 до 100 нуклеотидов.
Согласно одному аспекту, целевая нуклеиновая кислота связана с заболеванием или болезненным состоянием.
Согласно одному аспекту, две или несколько РНК представлены направляющей РНК. Согласно одному аспекту, две или несколько РНК представляют собой слияния tracr-РНК и cr-РНК.
Согласно одному аспекту, ДНК представлена геномной ДНК, митохондриальной ДНК, вирусной ДНК либо экзогенной ДНК.
Другие признаки и преимущества определенных воплощений настоящего изобретения станут более понятными из нижеследующего описания воплощений и их чертежей, а также из формулы изобретения.
Краткое описание фигур
Патент или патентная заявка содержит рисунки, выполненные в цвете. Копии этого патента или публикации патентной заявки с цветными рисунками будут предоставляться Ведомством по запросу при оплате необходимой пошлины. Вышеизложенные и другие признаки и преимущества настоящих воплощений станут более понятными из нижеследующего подробного описания воплощений вместе с прилагаемыми рисунками.
На фиг. 1А и фиг. 1В представлены схемы РНК-направляемой активации транскрипции. На фиг. 1С представлено схематическое изображение конструкции-репортера. На фиг. 1D (1D-1 и 1D-2) представлены данные, свидетельствующие, что слитые белки Cas9N-VP64 проявляют РНК-направляемую активацию транскрипции при измерении методами сортировки клеток с активируемой флуоресценцией(FACS) и иммунофлуоресценции (IF). На фиг. 1E (1Е-1 и 1Е-2) представлены данные оценки методом FACS и IF, свидетельствующие о специфичной к последовательности гидРНК активации транскрипции репортерными конструкциями в присутствии Cas9N, VP64 MS2 и гидРНК, несущей соответствующие сайты связывания аптамера MS2. На фиг. 1F представлены данные, свидетельствующие об индукции транскрипции под действием индивидуальных гидРНК и множественных гидРНК.
На фиг. 2А представлена методология оценки ландшафта нацеливания у комплексов Cas9-гидРНК и TALEs. На фиг. 2В представлены данные, показывающие, что комплекс Cas9-гидРНК допускает в среднем 1-3 мутации в последовательностях своих мишеней. На фиг. 2С представлены данные, показывающие, что комплекс Cas9-гидРНК почти нечувствителен к точечным мутациям, за исключением тех, которые локализуются в последовательности РАМ. На фиг. 2D представлены данные в виде термограммы (heat-plot), показывающие, что введение несоответствия по 2 основаниям существенно ухудшает активность комплекса Cas9-гидРНК. На фиг. 2Е представлены данные, показывающие, что 18-мер TALE допускает в среднем 1-2 мутации в последовательности своей мишени. На фиг. 2F приведены данные, показывающие, что 18-мер TALE, аналогично комплексам Cas9-гидРНК, почти нечувствителен к несоответствию по 1 основанию у своей мишени. На фиг. 2G представлены данные в виде термограммы, показывающие, что введение несоответствия по 2 основаниям существенно ухудшает активность 18-мера TALE.
На фиг. 3А представлено схематическое изображение структуры направляющих РНК. На фиг. 3В представлены данные, показывающие частоту негомологичного соединения концов для смещенных одноцепочечных разрывов, образующих (неспаренные) свисающие 5'-концы, и смещенных одноцепочечных разрывов, образующих свисающие 3'-концы. На фиг. 3С представлены данные, показывающие частоту в % нацеливания для смещенных одноцепочечных разрывов, образующих свисающие 5'-концы, и смещенных одноцепочечных разрывов, образующих свисающие 3'-концы.
На фиг. 4А схематически представлен координирующий металл остаток в RuvC из PDB ID: 4EP4 (синий) в положении D7 (слева), схема эндонуклеазных доменов HNH из PDB IDs: 3M7K (оранжевый) и 4H9D (голубой), включая координированный ион Mg (серый шар) и ДНК из 3M7K (фиолетовая) (посредине), и список проанализированых мутантов (справа). На фиг. 4В представлены данные, показывающие отсутствие заметной нуклеазной активности у мутантов Cas9 m3 и m4, а также соответствующих слияний их с VP64. На фиг. 4С представлен вид данных из фиг. 4В при более высоком разрешении.
На фиг. 5А представлена схема метода гомологичной рекомбинации для определения активности Cas9-гидРНК. На фиг. 5В (5В-1 и 5-В2) представлены направляющие РНК со вставками случайных последовательностей и частота гомологичной рекомбинации.
На фиг. 6А представлена схема направляющих РНК для гена ОСТ4. На фиг. 6В представлена активация транскрипции для конструкции репортера промотор-люцифераза. На фиг. 6С представлена активация транскрипции по данным количественного метода ПЦР эндогенных генов.
На фиг. 7А представлена схема направляющих РНК для гена REX1. На фиг. 7В представлена активация транскрипции для конструкции репортера промотор-люцифераза. На фиг. 7С представлена активация транскрипции по данным количественного метода ПЦР эндогенных генов.
На фиг. 8А схематически представлена блок-схема анализа специфичности высокого уровня для расчета нормированных уровней экспрессии. На фиг. 8В представлены данные по распределению сайтов связывания в зависимости от числа несоответствий, генерируемых в смещенной библиотеке конструкций. Слева: теоретическое распределение. Справа: фактическое распределение в реальной библиотеке конструкций TALE. На фиг. 8С представлены данные о распределении тегов, агрегированных с сайтами связывания, в зависимости от числа несоответствий. Слева: фактическое распределение в положительном контрольном образце. Справа: фактическое распределение в образце, в котором был индуцирован не контрольный TALE.
На фиг. 9А представлены данные по анализу ландшафта нацеливания у комплекса Cas9-гидРНК, свидетельствующие о допустимости 1-3 мутаций в последовательности его мишени. На фиг. 9В представлены данные по анализу ландшафта нацеливания у комплекса Cas9-гидРНК, свидетельствующие о допустимости точечных мутаций, за исключением тех, которые локализованы в последовательности РАМ. На фиг. 9С представлены данные в виде термограммы по анализу ландшафта нацеливания у комплекса Cas9-гидРНК, показывающие, что введение несоответствия по 2 основаниям существенно ухудшает активность. На фиг. 9D представлены данные анализа опосредованной нуклеазой HR, подтверждающие, что предполагаемый РАМ для Cas9 S. pyogenes представлен не только NGG, но и NAG.
На фиг. 10А (10А-1 и 10А-2) представлены данные анализа опосредованной нуклеазой гомологичной рекомбинации, подтверждающие, что 18-меры TALE допускают множественные мутации в последовательности своей мишени. На фиг. 10В представлены данные по анализу ландшафта нацеливания у TALEs 3 разных размеров (18-мера, 14-мера и 10-мера). На фиг. 10С представлены данные для 10-мера TALE, показывающие несоответствия с разрешением почти в 1 основание. На фиг. 10D представлены данные в виде термограммы t для 10-мера TALE, показывающие несоответствия с разрешением почти в 1 основание.
На фиг. 11А представлены разработанные направляющие РНК. На фиг. 11В представлена частота негомологичного соединения концов для различных направляющих РНК.
На фиг. 12А изображен ген Sox2. На фиг. 12В изображен ген Nanog.
На фигурах 13A-13F изображен ландшафт нацеливания двух дополнительных комплексов Cas9-гидРНК.
На фиг. 14А оттображен профиль специфичности двух гидРНК (дикого типа (SEQ ID NO: 88) и мутантов (SEQ ID NOs: 89-90). Различия в последовательности выделены красным цветом. На фигурах 14В и 14С показано, что данный анализ был специфичен для гидРНК, которую оценивали (другое изображение данных с фигуры 13D).
На фигурах 15A-15D изображена гидРНК2 (Фигуры 15А и 15В (15В-1 и 15В-2)) и гидРНК3 (фигуры 15С и 15D(15D-1 и 15D-2)), которые несут одиночные мутации или мутации по два основания (выделенные красным) в последовательности спейсера по сравнению с мишенью. Представлены последовательности SEQ ID NO: 91-131.
На фигурах 16A-16D представлен нуклеазный анализ двух независимых гидРНК, где анализировали гидРНК1 (фигуры 16А и 16В (16В-1 и 16В-2)) и гидРНК3 (фигуры 16С и 16D (16D-1 и 16D-2)), которые усечены с 5' конца своего спейсера. Представлены последовательности SEQ ID NOs: 66, 185-186 и 133-140.
На фигурах 17А-17В изображен опосредованный нуклеазой анализ HR, где показано, что РАМ у S. pyogenes Cas9 представлена NGG а также NAG. Представлены последовательности SEQ ID NOs: 67-69 и 141.
На фигурах 18А-18В изображен опосредованный нуклеазой анализ HR, где было подтверждено, что 18-мер TALE невосприимчив в множественным мутациям в своей последовательности-мишени. Представлены последовательности SEQ ID NOs: 70-73.
На фигурах 19А, 19В (19В-1 и 19В-2) и 19С (19С-1 и 19С-2) показано сравнение специфичности мономера TALE против специфичности белка TALE. Представлены последовательности SEQ ID NOs: 142-150.
На фигурах 20А-20В приведены данные, касающиеся смещенных одноцепочечных разрывов. Представлены последовательности SEQ ID NOs: 151-158.
На фигурах 21А-21С приведены данные, касающиеся смещенных одноцепочечных разрывов и профилей NHEJ. Представлены последовательности SEQ ID NOs: 159-184 и 187.
Раскрытие сущности изобретения
Воплощения настоящего изобретения основываются на использовании ДНК-связывающих белков для совместной локализации регулирующих транскрипцию белков или доменов на ДНК таким образом, чтобы управлять целевой нуклеиновой кислотой. Такие ДНК-связывающие белки хорошо известны специалистам в данной области и их используют для связывания ДНК в различных целях. Такие ДНК-связывающие белки могут быть природного происхождения. ДНК-связывающие белки, входящие в объем настоящего изобретения, включают такие, которые могут направляться РНК, именуемой здесь направляющей РНК. Согласно этому аспекту, направляющая РНК и РНК-направляемый ДНК-связывающий белок образуют совместно локализуемый комплекс на ДНК. Согласно некоторым аспектам, ДНК-связывающий белок может представлять собой безнуклеазный ДНК-связывающий белок. Согласно этому аспекту, безнуклеазный ДНК-связывающий белок может быть результатом изменения или модификации ДНК-связывающего белка, обладающего нуклеазной активностью. Такие ДНК-связывающие белки, обладающие нуклеазной активностью, известны специалистам и включают ДНК-связывающие белки природного происхождения, обладающие нуклеазной активностью, как-то белки Cas9, присутствующие, к примеру, в системах CRISPR II-го типа. Такие белки Cas9 и системы CRISPR II-го типа хорошо описаны в данной области. Напр., см. Makarova et al., Nature Reviews, Microbiology, Vol. 9, June 2011, pp. 467-477, которая включена сюда путем ссылки во всей полноте, включая всю дополнительную информацию.
Типичные ДНК-связывающие белки, обладающие нуклеазной активностью, функционируют для надрезания или разрезания двухцепочечной ДНК. Такая нуклеазная активность может быть следствием того, что ДНК-связывающий белок содержит одну или несколько полипептидных последовательностей, проявляющих нуклеазную активность. Такие типичные ДНК-связывающие белки могут содержать два отдельных нуклеазных домена, причем каждый домен отвечает за разрезание или надрезание определенной нити двухцепочечной ДНК. Типичные полипептидные последовательности, обладающие нуклеазной активностью, известны специалистам и включают нуклеазный домен MCRA-HNH и нуклеазный домен типа RuvC. Соответственно, примерами ДНК-связывающих белков служат те, которые по своей природе содержат один или несколько нуклеазных доменов MCRA-HNH и нуклеазных доменов типа RuvC. Согласно некоторым аспектам, ДНК-связывающий белок подвергается модификации или иным изменениям для инактивации нуклеазной активности. Такие изменения или модификации включают изменения одной или нескольких аминокислот для инактивации нуклеазной активности или нуклеазного домена. Такие модификации включают удаление полипептидной последовательности или полипептидных последовательностей, проявляющих нуклеазную активность, т.е. нуклеазного домена, с тем чтобы в ДНК-связывающем белке отсутствовала полипептидная последовательность или полипептидные последовательности, проявляющие нуклеазную активность, т.е. нуклеазный домен. Другие модификации для инактивации нуклеазной активности станут понятными специалистам в данной области на основе настоящего описания. Соответственно, безнуклеазный ДНК-связывающий белок включает полипептидные последовательности, подвергнутые модификации для инактивации нуклеазной активности, или же удаление полипептидной последовательности или последовательностей для инактивации нуклеазной активности. Безнуклеазный ДНК-связывающий белок сохраняет способность к связыванию с ДНК, даже если нуклеазная активность была инактивирована. Соответственно, ДНК-связывающий белок включает полипептидную последовательность или последовательности, необходимые для связывания с ДНК, но может отсутствовать одна или несколько или же все нуклеазные последовательности, проявляющие нуклеазную активность. Соответственно, ДНК-связывающий белок включает полипептидную последовательность или последовательности, необходимые для связывания с ДНК, но у одной или нескольких или же всех нуклеазных последовательностей может быть инактивирована нуклеазная активность.
Согласно одному аспекту, ДНК-связывающий белок, содержащий два или несколько нуклеазных доменов, может быть модифицирован или изменен так, чтобы инактивировать все нуклеазные домены, кроме одного. Такой модифицированный или измененный ДНК-связывающий белок именуется ДНК-связывающим белком-никазой, если этот ДНК-связывающий белок разрезает или надрезает только одну нить двухцепочечной ДНК. А если он направляется на ДНК с помощью РНК, то такой ДНК-связывающий белок-никаза именуется РНК-направляемым ДНК-связывающим белком-никазой.
Типичным ДНК-связывающим белком является РНК-направляемый ДНК-связывающий белок системы CRISPR II-го типа, у которого отсутствует нуклеазная активность. Типичным ДНК-связывающим белком является безнуклеазный белок Cas9. Типичным ДНК-связывающим белком является белок-никаза Cas9.
У S. pyogenes Cas9 создает двухцепочечный разрыв с тупыми концами за 3 п. о. до примыкающего к протоспейсеру мотива (РАМ) с помощью процесса, опосредованного двумя каталитическими доменами в этом белке: доменом HNH, который расщепляет комплементарную нить ДНК, и доменом типа RuvC, который расщепляет некомплементарную нить. См. Jinke et al., Science 337, 816-821 (2012), включенную сюда путем ссылки во всей полноте. Белки Cas9, как известно, существуют во многих системах CRISPR II-го типа, включая следующие, приведенные в дополнительной информации к Makarova et al., Nature Reviews, Microbiology, Vol. 9, June 2011, pp. 467-477: Methanococcus maripaludis C7; Corynebacterium diphtheriae; Corynebacterium efficiens YS-314; Corynebacterium glutamicum ATCC 13032 Kitasato; Corynebacterium glutamicum ATCC 13032 Bielefeld; Corynebacterium glutamicum R; Corynebacterium kroppenstedtii DSM 44385; Mycobacterium abscessus ATCC 19977; Nocardia farcinica IFM10152; Rhodococcus erythropolis PR4; Rhodococcus jostii RHA1; Rhodococcus opacus B4 uid36573; Acidothermus cellulolyticus 11B; Arthrobacter chlorophenolicus A6; Kribbella flavida DSM 17836 uid43465; Thermomonospora curvata DSM 43183; Bifidobacterium dentium Bdl; Bifidobacterium longum DJO10A; Slackia heliotrinireducens DSM 20476; Persephonella marina EX HI; Bacteroides fragilis NCTC 9434; Capnocytophaga ochracea DSM 7271; Flavobacterium psychrophilum JIP02 86; Akkermansia muciniphila ATCC BAA 835; Roseiflexus castenholzii DSM 13941; Roseiflexus RSI; Synechocystis PCC6803; Elusimicrobium minutum Peil91; uncultured Termite group 1 bacterium phylotype Rs D17; Fibrobacter succinogenes S85; Bacillus cereus ATCC 10987; Listeria innocua; Lactobacillus casei; Lactobacillus rhamnosus GG; Lactobacillus salivarius UCC118; Streptococcus agalactiae A909; Streptococcus agalactiae NEM316; Streptococcus agalactiae 2603; Streptococcus dysgalactiae equisimilis GGS 124; Streptococcus equi zooepidemicus MGCS10565; Streptococcus gallolyticus UCN34 uid46061; Streptococcus gordonii Challis subst. CHI; Streptococcus mutans NN2025 uid46353; Streptococcus mutans; Streptococcus pyogenes Ml GAS; Streptococcus pyogenes MGAS5005; Streptococcus pyogenes MGAS2096; Streptococcus pyogenes MGAS9429; Streptococcus pyogenes MGAS 10270; Streptococcus pyogenes MGAS6180; Streptococcus pyogenes MGAS315; Streptococcus pyogenes SSI-1; Streptococcus pyogenes MGAS10750; Streptococcus pyogenes NZ131; Streptococcus thermophiles CNRZ1066; Streptococcus thermophiles LMD-9; Streptococcus thermophiles LMG 18311; Clostridium botulinum A3 Loch Maree; Clostridium botulinum В Eklund 17B; Clostridium botulinum Ba4 657; Clostridium botulinum F Langeland; Clostridium cellulolyticum H10; Finegoldia magna ATCC 29328; Eubacterium rectale ATCC 33656; Mycoplasma gallisepticum; Mycoplasma mobile 163K; Mycoplasma penetrans; Mycoplasma synoviae 53; Streptobacillus moniliformis DSM 12112; Bradyrhizobium BTAil; Nitrobacter hamburgensis XI4; Rhodopseudomonas palustris BisB18; Rhodopseudomonas palustris BisB5; Parvibaculum lavamentivorans DS-1; Dinoroseobacter shibae DFL 12; Gluconacetobacter diazotrophicus Pal 5 FAPERJ; Gluconacetobacter diazotrophicus Pal 5 JGI; Azospirillum B510 uid46085; Rhodospirillum rubrum ATCC 11170; Diaphorobacter TPSY uid29975; Verminephrobacter eiseniae EF01-2; Neisseria meningitides 053442; Neisseria meningitides alphal4; Neisseria meningitides Z2491; Desulfovibrio salexigens DSM 2638; Campylobacter jejuni doylei 269 97; Campylobacter jejuni 81116; Campylobacter jejuni; Campylobacter lari RM2100; Helicobacter hepaticus; Wolinella succinogenes; Tolumonas auensis DSM 9187; Pseudoalteromonas atlantica T6c; Shewanella pealeana ATCC 700345; Legionella pneumophila Paris; Actinobacillus succinogenes 130Z; Pasteurella multocida; Francisella tularensis novicida U112; Francisella tularensis holarctica; Francisella tularensis FSC 198; Francisella tularensis tularensis; Francisella tularensis WY96-3418; и Treponema denticola ATCC 35405. Соответственно, аспекты настоящего изобретения направлены на белок Cas9, присутствующий в системе CRISPR II-го типа, который стал безнуклеазным или который стал никазой, как описано здесь.
Белок Cas9 может упоминаться специалистами в литературе как Csnl. Последовательность белка Cas9 S. pyogenes, который является предметом описанных здесь экспериментов, представлена ниже. См. Deltcheva et al., Nature 471, 602-607 (2011), которая включена сюда путем ссылки во всей полноте.


Согласно некоторым аспектам описанных здесь способов РНК-направляемой регуляции генома, Cas9 подвергается изменениям для снижения, существенного снижения или устранения нуклеазной активности. Согласно одному аспекту, нуклеазная активность Cas9 уменьшается, существенно уменьшается или инактивируется путем изменения нуклеазного домена RuvC или нуклеазного домена HNH. Согласно одному аспекту, инактивируется нуклеазный домен RuvC. Согласно одному аспекту, инактивируется нуклеазный домен HNH. Согласно одному аспекту, инактивируется нуклеазный домен RuvC и нуклеазный домен HNH. Согласно другому аспекту, предусматриваются белки Cas9, у которых инактивирован нуклеазный домен RuvC и нуклеазный домен HNH. Согласно другому аспекту, предусматриваются безнуклеазные белки Cas9, у которых инактивирован нуклеазный домен RuvC и нуклеазный домен HNH. Согласно другому аспекту, предусматривается никаза Cas9, у которой инактивирован либо нуклеазный домен RuvC, либо нуклеазный домен HNH, при этом оставшийся нуклеазный домен остается активным для нуклеазной активности. Таким образом, разрезается или надрезается только одна нить двухцепочечной ДНК.
Согласно другому аспекту, предусматриваются безнуклеазные белки Cas9, у которых одна или несколько аминокислот у Cas9 изменяются или же удаляются для получения безнуклеазных белков Cas9. Согласно одному аспекту, аминокислоты включают D10 и Н840. См. Jinke et al., Science 337, 816-821 (2012). Согласно другому аспекту, аминокислоты включают D839 и N863. Согласно одному аспекту, одна или несколько или все аминокислоты D10, Н840, Н863 и D839 заменяются такой аминокислотой, которая снижает, существенно снижает или устраняет нуклеазную активность. Согласно одному аспекту, одна или несколько или все аминокислоты D10, Н840, Н863 и D839 заменяются на аланин. Согласно одному аспекту, белок Cas9, у которого одна или несколько или все аминокислоты D10, Н840, D839 и Н863 заменены такой аминокислотой, которая снижает, существенно снижает или устраняет нуклеазную активность, типа аланина, именуется безнуклеазным Cas9 или Cas9N и проявляет сниженную или отсутствующую нуклеазную активность, или же нуклеазная активность отсутствует или практически отсутствует на уровне предела обнаружения. Согласно этому аспекту, нуклеазная активность у Cas9N может не обнаруживаться известными методами, т.е. она ниже предела обнаружения известными методами.
Согласно одному аспекту, безнуклеазный белок Cas9 охватывает и его гомологи и ортологи, которые сохраняют способность белка к связыванию с ДНК и способность направляться РНК. Согласно одному аспекту, безнуклеазный белок Cas9 включает последовательность, приведенную для природного Cas9 из S. pyogenes, у которой одна или несколько или все аминокислоты D10, Н840, Н863 и D839 заменены на аланин, а также последовательности белков, которые по меньшей мере на 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 98% или 99% гомологичны этой последовательности и являются ДНК-связывающими белками типа РНК-направляемых ДНК-связывающих белков.
Согласно одному аспекту, безнуклеазный белок Cas9 включает последовательность, приведенную для природного Cas9 из S. pyogenes, за исключением последовательности нуклеазного домена RuvC и нуклеазного домена HNH, а также последовательности белков, которые по меньшей мере на 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 98% или 99% гомологичны этой последовательности и являются ДНК-связывающими белками типа РНК-направляемых ДНК-связывающих белков. Таким образом, аспекты настоящего изобретения включают последовательность белка, отвечающую за связывание с ДНК, к примеру, для совместной локализации с направляющей РНК и связывание с ДНК, и белковые последовательности, гомологичные ей, но не обязательно включают белковые последовательности нуклеазного домена RuvC и нуклеазного домена HNH (если только они не нужны для связывания ДНК), поскольку эти домены могут быть инактивированы или удалены из последовательности природного белка Cas9 для получения безнуклеазного белка Cas9.
В целях настоящего изобретения, на фиг. 4А представлены координирующие металл остатки в структурах известных белков, гомологичных Cas9. Остатки пронумерованы согласно их положению в последовательности Cas9. Слева: структура RuvC, PDB ID: 4ЕР4 (синий), положение D7, которое соответствует D10 в последовательности Cas9, выделено в положениях, координирующих ион Mg. Посредине: структуры эндонуклеазных доменов HNH у PDB ID: 3M7K (оранжевый) и 4H9D (голубой), включая скоординированный ион Mg (серый шар) и ДНК из 3M7K (фиолетовый). Остатки D92 и N113 в 3M7K и позиции D53 и N77 в 4H9D, которые гомологичны по последовательности аминокислотам D839 и N863 в Cas9, представлены в виде палочек. Справа: список мутантов, полученных и проанализированных на нуклеазную активность: Cas9 дикого типа; Cas9m1, у которого D10 заменен на аланин; Cas9m2, у которого D10 и Н840 заменены на аланин; Cas9m3, У которого D10, Н840 и D839 заменены на аланин; и Cas9m4, у которого D10, Н840, D839 и N863 заменены на аланин.
Как видно из фиг. 4В, мутанты Cas9: m3 и m4, а также соответствующие им слияния с VP64 не проявляли заметной нуклеазной активности по результатам глубокого секвенирования локуса мишени. На графиках представлена частота мутаций в зависимости от положения в геноме, а красными линиями отмечена мишень гидРНК. На фиг. 4С представлены данные из фиг. 4В при более высоком разрешении, которые подтверждают, что мутационный ландшафт проявляет сравнимый с немодифицированными локусами профиль.
Согласно одному аспекту, предусмотрена сконструированная система Cas9-гидРНК, которая позволяет осуществлять РНК-направляемую регуляцию генома в клетках человека путем привязывания доменов активации транскрипциио к безнуклеазному Cas9 либо к направляющей РНК. Согласно одному аспекту настоящего изобретения, один или несколько регулирующих транскрипцию белков или доменов (данные термины применяются взаимозаменяемо) присоединяются или иным образом соединяются с безнуклеазным Cas9 либо с одной или несколькими направляющими РНК (гидРНК). Регулирующие транскрипцию домены соответствуют локусам мишени. Соответственно, аспекты настоящего изобретения включают способы и материалы для локализации регулирующих транскрипцию доменов на локусах мишенях путем слияния, соединения или присоединения таких доменов к Cas9N либо к гидРНК.
Согласно одному аспекту, предусмотрен слитый с Cas9N белок, способный активировать транскрипцию. Согласно одному аспекту, к С-концу Cas9N присоединяется, сливается, соединяется или иным образом прикрепляется домен активации VP64 (см. Zhang et al., Nature Biotechnology 29,149-153 (2011), которая включена сюда путем ссылки во всей полноте). Согласно одному способу, регулирующий транскрипцию домен доставляется на сайт целевой геномной ДНК белком Cas9N. Согласно одному способу, слитый с регулирующим транскрипцию доменом Cas9N поступает внутрь клетки вместе с одной или несколькими направляющими РНК. Cas9N со слитым с ним регулирующим транскрипцию доменом связывается на или возле целевой геномной ДНК. Одна или несколько направляющих РНК связываются на или возле целевой геномной ДНК. Регулирующий транскрипцию домен регулирует экспрессию гена-мишени. Согласно одному конкретному аспекту, слитый белок Cas9N-VP64 в сочетании с гидРНК, воздействующей на последовательности вблизи от промотора, активируют транскрипцию репортерных конструкций, тем самым проявляется РНК-направляемая активация транскрипции.
Согласно одному аспекту, предусмотрен слитый с гидРНК белок, способный активировать транскрипцию. Согласно одному аспекту, к гидРНК присоединяется, сливается, соединяется или иным образом прикрепляется домен активации VP64. Согласно одному способу, регулирующий транскрипцию домен попадает на сайт целевой геномной ДНК при помощи гидРНК. Согласно одному способу, соединенная с регулирующим транскрипцию доменом гидРНК поступает внутрь клетки вместе с белком Cas9N. Cas9N связывается на или возле целевой геномной ДНК. Одна или несколько направляющих РНК со слитым с ними регулирующим транскрипцию белком или доменом связываются на или возле целевой геномной ДНК. Регулирующий транскрипцию домен регулирует экспрессию гена-мишени. Согласно одному конкретному аспекту, белок Cas9N-VP64 и гидРНК, соединенная с регулирующим транскрипцию доменом, активируют транскрипцию репортерных конструкций, тем самым проявляется РНК-направляемая активация транскрипции.
Были сконструированы составные гидРНК, способные регулировать транскрипцию, путем определения тех участков гидРНК, которые допускают изменения, путем вставки случайных последовательностей в гидРНК и анализа функции Cas9. Направляющие РНК, несущие вставки случайных последовательностей либо на 5'-конце участка cr-РНК, либо на 3'-конце участка tracr-РНК в химерной гидРНК, сохраняют функциональность, тогда как вставки в каркасный участок tracr-РНК химерной гидРНК ведут к потере функции. См. фиг. 5А-В, на которой суммированы данные по устойчивости гидРНК к случайным вставкам оснований. На фиг. 5А представлена схема метода гомологичной рекомбинации (HR) для определения активности Cas9-гидРНК. Как видно из фиг. 5В, направляющие РНК, несущие вставки случайных последовательностей либо на 5'-конце участка cr-РНК, либо на 3'-конце участка tracr-РНК в химерной гидРНК, сохраняют функциональность, тогда как вставки в каркасный участок tracr-РНК химерной гидРНК ведут к потере функции. Точки вставки в последовательность гидРНК обозначены в виде красных нуклеотидов. Не придерживаясь какой-либо научной теории, повышение активности при случайных вставках оснований на 5'-конце может быть обусловлено увеличением периода полужизни у более длинной гидРНК.
Для присоединения VP64 к гидРНК пришивали две копии области стебель-петля РНК, связывающей белок оболочки бактериофага MS2, к 3'-концу гидРНК. См. Fusco et al., Current Biology: CB13, 161-167 (2003), которая включена сюда путем ссылки во всей полноте. Эти химерные гидРНК экспрессировали вместе со слитым с Cas9N белком VP64 MS2. В присутствии всех 3 компонентов наблюдалась активация специфичной к последовательности транскрипции из репортерных конструкций.
На фиг. 1А представлена схема РНК-направляемой активации транскрипции. Как видно из фиг. 1А, для получения слитого с Cas9N белка, способного активировать транскрипцию, к С-концу Cas9N непосредственно пришивали домен активации VP64. Как видно из фиг. 1В, для получения составной гидРНК, способной активировать транскрипцию, к 3'-концу гидРНК пришивали две копии области стебель-петля РНК, связывающей белок оболочки бактериофага MS2. Эти химерные гидРНК экспрессировали вместе со слитым с Cas9N белком VP64 MS2. На фиг. 1С представлена схема репортерных конструкций, используемых для анализа активации транскрипции. Два репортера несут разные сайты-мишени для гидРНК и одинаковый контрольный сайт-мишень TALE-TF. Как видно из фиг 1D, слитый белок Cas9N-VP64 проявляет РНК-направляемую активацию транскрипции при измерении методами сортировки клеток с активируемой флуоресценцией (FACS) и иммунофлуоресценции (IF). В то время, как контрольный TALE-TF активирует оба репортера, слитый белок Cas9N-VP64 активирует репортеры специфичным к последовательности гидРНК образом. Как видно из фиг. 1Е, специфичная к последовательности гидРНК активация транскрипции у репортерных конструкций наблюдалась методами FACS и IF только в присутствии всех 3 компонентов: Cas9N, VP64 MS2 и гидРНК, несущей соответствующие сайты связывания аптамера MS2.
Согласно некоторым аспектам, предусмотрены способы регуляции эндогенных генов с помощью Cas9N, одной или нескольких гидРНК и регулирующего транскрипцию белка или домена. Согласно одному аспекту, эндогенным геном может быть любой желательный ген, именуемый здесь геном-мишенью. Согласно одному типичному аспекту, подлежащие регуляции гены включают ZFP42 (REX1),и POU5F1 (OUT4), которые оба являются жестко регулируемыми генами, участвующими в поддержании плюрипотентности. Как видно из фиг. 1F, для гена REX1 было разработано 10 гидРНК, направленных на отрезок ДНК в ~5 т.п.о. перед сайтом инициации транскрипции (сверхчувствительные к ДНКазе сайты выделены зеленым цветом). Активацию транскрипции анализировали либо с помощью репортерной конструкции промотор-люцифераза (см. Takahashi et al., Cell, 131: 861-872 (2007), которая включена сюда путем ссылки во всей полноте), либо непосредственно методом кПЦР эндогенных генов.
Фиг. 6А-С касается РНК-направляемой регуляции ОСТ4 с помощью Cas9N-VP64. Как видно из фиг. 6А, для гена ОСТ4 было разработано 21 гидРНК, направленных на отрезок ДНК в ~5 т.п.о. перед сайтом инициации транскрипции. Сверхчувствительные к ДНКазе сайты выделены зеленым цветом. На фиг. 6В представлена активация транскрипции с помощью репортерной конструкции промотор-люцифераза. На фиг. 6С представлена активация транскрипции анализируемая непосредственно методом кПЦР эндогенных генов. В то время, как введение индивидуальных гидРНК умеренно стимулировало транскрипцию, несколько гидРНК действовали синергически, вызывая устойчивую многократную активацию транскрипции.
Фиг. 7А-С касается РНК-направляемой регуляции REX1 с помощью Cas9N, VP64 MS2 и гидРНК + аптамер 2X-MS2. Как видно из фиг. 7А, для гена REX1 было разработано 10 гидРНК, направленных на отрезок ДНК в ~5 т.п.о. перед сайтом инициации транскрипции. Сверхчувствительные к ДНКазе сайты выделены зеленым цветом. На фиг. 7В представлена активация транскрипции с помощью репортерной конструкции промотор-люцифераза. На фиг. 7С представлена активация транскрипции непосредственно методом кПЦР эндогенных генов. В то время, как введение индивидуальных гидРНК умеренно стимулировало транскрипцию, несколько гидРНК действовали синергически, вызывая устойчивую многократную активацию транскрипции. В одном аспекте, отсутствие аптамеров 2X-MS2 на гидРНК не вызывает активации транскрипции. См. Maeder et al., Nature Methods 10, 243-245 (2013); и Perez-Pinera et al., Nature Methods 10, 239-242 (2013); каждая из которых включена сюда путем ссылки во всей полноте.
Соответственно, способы направлены на использование множественных направляющих РНК с белком Cas9N и регулирующим транскрипцию белком или доменом для регуляции экспрессии гена-мишени.
Оба подхода и с Cas9, и с пришиванием гидРНК оказались эффективными, причем первый из них проявляет в ~1,5-2 раза большую активность. Это различие, вероятно, связано с необходимостью сборки 2-компонентного, в отличие от 3-компонентного комплекса. Тем не менее, метод пришивания гидРНК в принципе позволяет рекрутировать различные эффекторные домены различными гидРНК, если только каждая из гидРНК использует другую взаимодействующую пару РНК-белок. См. Karyer-Bibens et al. Biology of the Cell / Under the Auspices of the European Cell Biology Organization 100, 125-138 (2008), которая включена сюда путем ссылки во всей полноте. Согласно одному аспекту настоящего изобретения, различные гены-мишени можно регулировать с помощью специфичных направляющих РНК и общего белка Cas9N, т.е. одного и того же или близкого белка Cas9N для различных генов-мишеней. Согласно одному аспекту, предусмотрены способы мультиплексной регуляции генов с помощью одного и того же или близкого белка Cas9N.
Способы настоящего изобретения также направлены на редактирование генов-мишеней с помощью описанных здесь белков Cas9N и направляющих РНК, чтобы обеспечить мультиплексную генетическую и эпигенетическую инженерию клеток человека. Поскольку таргетинг Cas9-гидРНК является спорным вопросом (см. Jiang et al., Nature Biotechnology 31, 233-239 (2013), которая включена сюда путем ссылки во всей полноте), то предусмотрены способы углубленного изучения сродства Cas9 для очень большого ряда вариантов последовательностей мишеней. Соответственно, аспекты настоящего изобретения обеспечивают прямое высокопроизводительное изучение нацеливания Cas9 в клетках человека, при этом избегая осложнений, вызванных токсичностью разрезанной дцДНК и репарацией мутагенных повреждений, возникающих при тестировании на специфичность с помощью нативного Cas9, обладающего нуклеазной активностью.
Другие аспекты настоящего изобретения направлены на использование ДНК-связывающих белков или систем вообще для регуляции транскрипции генов-мишеней. Специалист в данной области легко сможет установить типичные ДНК-связывающие системы, исходя из настоящего описания. Такие ДНК-связывающие системы могут и не обладать такой нуклеазной активностью, как у природного белка Cas9. Соответственно, у таких ДНК-связывающих систем и не нужно инактивировать нуклеазную активность. Одной из типичных ДНК-связывающих систем является TALE. В качестве инструмента редактирования генома обычно используются димеры TALE-FokI, а для регулирования генома очень эффективными оказались слияния TALE-VP64. Согласно одному аспекту, специфичность TALE оценивали по методологии, представленной на фиг. 2А. Создавали библиотеку конструкций, в которой каждый элемент библиотеки содержит минимальный промотор для экспрессии флуоресцентного белка dTomato. После сайта инициации транскрипции m вставлен рандомизованный тег транскрипта в 24 п.о. (A/C/G), а перед промотором располагаются два TF-связывающих сайта: один - это постоянная последовательность ДНК, общая для всех элементов библиотеки, а второй - переменная, несущая "смещенную" библиотеку сайтов связывания, которая разработана так, чтобы она охватывала большую коллекцию последовательностей, содержащую много комбинаций мутаций относительно целевой последовательности, с которой должен связываться программируемый комплекс нацеливания на ДНК. Это осуществляется с помощью вырожденных олигонуклеотидов, составленных так, чтобы нуклеотиды в каждом положении находились с определенной частотой, а именно целевой нуклеотид в последовательности встречался с частотой 79%, а каждый из остальных нуклеотидов - с частотой 7%. См. Patwardhan et al., Nature Biotechnology 30, 265-270 (2012), которая включена сюда путем ссылки во всей полноте. Затем библиотеку репортеров секвенировали, чтобы выявить связь между метками транскрипта dTomato в 24 п.о. и соответствующим "смещенным" целевым сайтом элемента библиотеки. Большое разнообразие меток транскриптов гарантирует, что общие метки между различными мишенями будут встречаться крайне редко, а "смещенность" целевых последовательностей означает, что сайты с небольшим числом мутаций будут связаны с большим количеством меток, чем сайты с большим числом мутаций. Далее стимулируют транскрипцию генов репортеров dTomato либо контрольным TF, который связывается с общим сайтом ДНК, или целевым TF, который должен связываться с целевым сайтом. В каждом образце измеряют содержание каждой экспрессированной метки транскрипта путем секвестрования РНК у стимулированных клеток, которое затем сопоставляют с соответствующими сайтами связывания с помощью установленной ранее таблицы соответствия. Как ожидается, контрольный TF будет стимулировать всех представителей библиотеки одинаково, так как его сайт связывания является общим для всех элементов библиотеки, тогда как целевой TF должен сдвинуть распределение экспрессируемых элементов в сторону тех, на которые он преимущественно воздействует. Это предположение используется на стадии 5 при вычислении нормализованного уровня экспрессии для каждого сайта связывания путем деления уровня метки, полученного для целевого TF, на уровень, полученный для контрольного TF.
Как видно из фиг. 2В, ландшафт нацеливания у комплекса Cas9-гидРНК свидетельствует, что он допускает в среднем 1-3 мутации в последовательностях своих мишеней. Как видно из фиг. 2С, комплекс Cas9-гидРНК также почти нечувствителен к точечным мутациям, за исключением тех, которые локализуются в последовательности РАМ. А именно, эти данные показывают, что предполагаемый РАМ для Cas9 S. pyogenes представлен не только NGG, но и NAG. Как видно из фиг. 2D, введение несоответствия по 2 основаниям существенно ухудшает активность комплекса Cas9-гидРНК, но только тогда, когда они локализуются на 8-10 оснований ближе к 3'-концу последовательности мишени гидРНК (на термограмме позиции в последовательности мишени отмечены как 1-23, начиная с 5'-конца).
Мутационную толерантность у другого широко используемого инструмента для редактирования генома, доменов TALE, определяли описанным здесь методом анализа транскрипционной специфичности. Как видно из фиг. 2Е, данные по взаимодействию TALE с мишенью для 18-мера TALE показывают, что он допускает в среднем 1-2 мутации в последовательности своей мишени и не способен активировать большую часть вариантов с несоответствием по 3 основаниям у своих мишеней. Как видно из фиг. 2F, 18-мер TALE, аналогично комплексам Cas9-гидРНК, почти нечувствителен к несоответствию по 1 основанию у своей мишени. Как видно из фиг. 2G, введение несоответствия по 2 основаниям существенно ухудшает активность 18-мера TALE. Активность TALE более чувствительна к несоответствиям ближе к 5'-концу последовательности своей мишени (на графике термограмме позиции в последовательности мишени отмечены как 1-18, начиная с 5'-конца).
Эти результаты были подтверждены нуклеазным методом в целенаправленных экспериментах, которые являются предметом фиг. 10А-С, направленных на изучение ландшафта нацеливания у TALEs различного размера. Как видно из фиг. 10А, методом анализа опосредованной нуклеазой HR было подтверждено, что 18-меры TALE допускают множественные мутации в последовательности своих мишеней. Как видно из фиг. 10В, анализировали ландшафт нацеливания у TALEs 3 разных размеров (18-мера, 14-мера и 10-мера) с помощью методики, описанной на фиг. 2. Более короткие TALEs (14-мер и 10-мер) более специфичны с точки зрения нацеливания, но также снижается активность почти на порядок. Как видно из фиг. 10С и 10D, 10-мер TALE проявляет разрешение несоответствий почти в одно основание, теряя почти всю активность в отношении мишеней, несущих 2 несоответствия (на графике термограмме позиции в последовательности мишени отмечены как 1-10, начиная с 5-конца). В целом эти данные означают, что разработка более коротких TALEs может давать более высокую специфичность в применении к геномной инженерии, тогда как при нуклеазном применении TALEs возникает необходимость в димеризации Fokl, чтобы избежать эффекта промашки. См. Kim et al., Proceedings of the National Academy of Sciences of the USA 93, 1156-1160 (1996); и Pattanayak et al., Nature Methods 8, 765-770 (2011); каждая из которых включена сюда путем ссылки во всей полноте.
Фиг. 8А-С касается последовательности операций при анализе специфичности высокого уровня для расчета нормированных уровней экспрессии, которая проиллюстрирована примерами из экспериментальных данных. Как видно из фиг. 8А, создаются библиотеки конструкций со смещенным распределением последовательностей сайтов связывания и рандомизованными последовательностями тегов в 24 п.о., которые будут включены в транскрипты генов-репортеров (сверху). Транскрибируемые теги сильно вырождены с тем, чтобы они соответствовали связывающим последовательностям Cas9 или TALE по принципу многие-к-одному. Библиотеки конструкций секвенируют (3-й уровень, слева), чтобы установить, какие теги встречаются вместе с сайтами связывания, получая таблицу соответствия между сайтами связывания и транскрибируемыми тегами (4-й уровень, слева). Можно одновременно секвенировать несколько библиотек конструкций, построенных для различных сайтов связывания, используя библиотечные штрихкоды (обозначенные здесь светло-голубым и светло-желтым цветом; уровни 1-4, слева). Библиотеки конструкций затем трансфецируют в популяции клеток и в образцах популяций индуцируют комплект различных Cas9/гидРНК или факторов транскрипции TALEs (2-й уровень, справа). Один образец всегда индуцируется с помощью фиксированного активатора TALE, нацеленного на фиксированную последовательность сайта связывания в данной конструкции (верхний уровень, зеленая рамка); этот образец служит в качестве положительного контроля (зеленый образец, также указан знаком +). Затем секвенируют и анализируют кДНК, полученные из молекул-репортеров мРНК в индуцированных образцах, чтобы получить число тегов для каждого тега в образце (3-й и 4-й уровень, справа). Как и при секвенировании библиотек конструкций, секвенируют и анализируют вместе несколько образцов, включая положительный контроль, прибавляя к ним штрихкоды образцов. Здесь светло-красным цветом обозначен один не контрольный образец, который секвенировали и анализировали вместе с положительным контролем (зеленый). Поскольку при каждом секвенировании проявляются только транскрибируемые теги, а не сайты связывания у конструкций, то затем для подсчета общего числа тегов, экспрессированных из каждого сайта связывания в каждом образце (5-й уровень), используется таблица соответствия между сайтами связывания и тегами, полученная при секвенировании библиотек конструкций. Затем числа для каждого образца без положительного контроля преобразуются в нормированные уровни экспрессии для каждого сайта связывания путем деления их на числа, полученные в образце положительного контроля. Примеры графиков нормированных уровней экспрессии от количества несоответствий представлены на фиг. 2В и 2Е и на фиг. 9А и фиг. 10В. На этой общей блок-схеме не представлено несколько уровней фильтрования для ошибочных тегов, для не связанных с библиотекой конструкций тегов и для тегов, явно связанных с несколькими сайтами связывания. На фиг. 8В представлен пример по распределению процента сайтов связывания в зависимости от числа несоответствий, генерируемых в смещенной библиотеке конструкций. Слева: теоретическое распределение. Справа: фактическое распределение в реальной библиотеке конструкций TALE. На фиг. 8С представлен пример по распределению процента тегов, агрегированных с сайтами связывания, в зависимости от числа несоответствий. Слева: фактическое распределение в положительном контрольном образце. Справа: фактическое распределение в образце, в котором был индуцирован не контрольный TALE. Поскольку положительный контрольный TALE связывается с фиксированным сайтом в конструкции, то распределение числа агрегированных тегов точно отражает распределение сайтов связывания на фиг. 8В, тогда как в не контрольном образце TALE распределение сдвигается влево, потому что сайты с меньшим числом несоответствий индуцируют более высокие уровни экспрессии. Внизу: вычисление относительного соотношения между ними путем деления числа тегов, полученного для целевого TF, на число, полученное для контрольного TF, дает средний уровень экспрессии в зависимости от количества мутаций в целевом сайте (мишени).
Эти результаты также подтверждаются данными по специфичности, полученными с использованием другого комплекса Cas9-гидРНК. Как видно из фиг. 9А, другой комплекс Cas9-гидРНК допускает 1-3 мутации в последовательности своей мишени. Как видно из фиг. 9В, этот комплекс Cas9-гидРНК также почти нечувствителен к точечным мутациям, за исключением тех, которые локализованы в последовательности РАМ. Как видно из фиг. 9С, введение несоответствия по 2 основаниям существенно ухудшает активность (на термограмме позиции в последовательности мишени отмечены как 1-23, начиная с 5'-конца). Как видно из фиг. 9D, методом анализа опосредованной нуклеазой HR подтверждается, что предполагаемый РАМ для Cas9 S. pyogenes представлен не только NGG, но и NAG.
Согласно некоторым аспектам, специфичность связывания повышается в соответствии с описанными здесь способами. Поскольку синергия между несколькими комплексами является фактором при активации генов мишени под действием Cas9N-VP64, то регуляция транскрипции с применением Cas9N, естественно, будет весьма специфичной, так как отдельные случаи связывания вне мишени должны иметь минимальное влияние. Согласно одному аспекту, в способах редактирования генома используются смещенные надрезы (off-set nicks). Большая часть одноцепочечных разрывов редко приводит к случаям NHEJ (см. Certo et al., Nature Methods 8, 671-676 (2011), которая включена сюда путем ссылки во всей полноте), тем самым сводя к минимуму эффекты надреза вне мишени. Напротив, использование смещенных одноцепочечных разрывов для получения двухцепочечных разрывов (DSBs) очень эффективно вызывает разрушение гена. Согласно некоторым аспектам, свисающие 5'-концы вызывают более значительные события NHEJ, чем свисающие 3'-концы. Точно так же, свисающие 3'-концы благоприятствуют событиям HR перед NHEJ, хотя общее количество случаев HR существенно ниже, чем при образовании свисающих 5'-концов. Соответственно, предусмотрены способы использования одноцепочечных разрывов для гомологичной рекомбинации и смещенных одноцепочечных разрывов для получения двухцепочечных разрывов, чтобы свести к минимуму эффекты активности Cas9-гидРНК вне мишени.
Фиг. 3А-С касается мультиплексного применения смещенных одноцепочечных разрывов и способов уменьшения связывания вне мишени с помощью направляющих РНК. Как видно из фиг. 3А, для одновременного анализа событий HR и NHEJ после введения прицельных одноцепочечных разрывов или разрывов использовали репортер "светофор". При репарации расщепленной ДНК по механизму HDR (направляемая гомологией репарация) восстанавливается последовательность GFP, тогда как мутагенное NHEJ вызывает сдвиг рамки считывания, при этом GFP выходит из рамки считывания, а нижележащая последовательность mCherry попадает в рамку. Для анализа составляли 14 гидРНК, охватывающих отрезок ДНК в 200 п.о.: 7 для смысловой нити (U1-7) и 7 для антисмысловой (D1-7). С помощью мутанта Cas9D10A, который надрезает комплементарную нить, использовали различные двусторонние комбинации гидРНК для получения целого ряда запрограммированных свисающих 5'- или 3'-концов (отмечены сайты надреза для всех 14 гидРНК). Как видно из фиг. 3В, использование смещенных одноцепочечных разрывов для создания двухцепочечных разрывов (DSBs) очень эффективно вызывает разрушение гена. А именно, смещенные надрезы, образующие свисающие 5'-концы, дают больше случаев NHEJ, чем свисающие 3'-концы. Как видно из фиг. 3С, образование свисающих 3'-концов благоприятствует преобладанию HR перед NHEJ, но общее количество случаев HR существенно ниже, чем при образовании свисающих 5'-концов.
Фиг. 11А-В касается NHEJ, опосредованного никазой Cas9D10A. Как видно из фиг. 11А, для анализа событий NHEJ после введения прицельных одноцепочечных разрывов или двухцепочечных разрывов использовали репортер "светофор". Вкратце, если репарация разрывов при расщеплении ДНК идет по механизму мутагенного NHEJ. то OFF при трансляции выходит из рамки считывания, а нижележащая последовательность mCherry попадает в рамку, издавая красную флуоресценцию. Составляли 14 гидРНК, охватывающих отрезок ДНК в 200 п. о.: 7 для смысловой нити (U1-7) и 7 для антисмысловой (D1-7). Как видно из фиг. 11В, оказалось, что, в отличие от Cas9 дикого типа, который образует DSBs и дает хорошее NHEJ по всем мишеням, большинство одноцепочечных разрывов (с помощью мутанта Cas9D10A) редко приводят к событиям NHEJ. Все 14 сайтов располагаются на непрерывном отрезке ДНК в 200 п.о., причем наблюдались более чем 10-кратные различия по эффективности нацеливания.
Согласно некоторым аспектам, здесь описаны способы модулирования экспрессии целевой нуклеиновой кислоты в клетках, которые включают введение в клетки одной или нескольких, двух или нескольких либо множества чужеродных нуклеиновых кислот. Чужеродные нуклеиновые кислоты, введенные в клетки, кодируют направляющую РНК или направляющие РНК, безнуклеазный белок или белки Cas9 и регулирующий транскрипцию белок или домен. Направляющая РНК, безнуклеазный белок Cas9 и регулирующий транскрипцию белок или домен все вместе именуются комплексом совместной локализации, как этот термин понимается специалистами в данной области, в той степени, что направляющая РНК, безнуклеазный белок Cas9 и регулирующий транскрипцию белок или домен связываются с ДНК и регулируют экспрессию целевой нуклеиновой кислоты. Согласно некоторым другим аспектам, чужеродные нуклеиновые кислоты, введенные в клетки, кодируют направляющую РНК или направляющые РНК и белок никазы Cas9. Направляющая РНК и белок никазы Cas9 все вместе именуются комплексом совместной локализации, как этот термин понимается специалистами в данной области, в той степени, что направляющая РНК и белок никазы Cas9 связываются с ДНК и делают надрезы целевой нуклеиновой кислоты.
Клетки по настоящему изобретению включают любые клетки, в которые можно вводить и экспрессировать чужеродные нуклеиновые кислоты, как описано здесь. Следует иметь в виду, что основные концепции настоящего изобретения, описанного здесь, не ограничиваются типом клеток. Клетки по настоящему изобретению включают эукариотические клетки, прокариотические клетки, клетки животных, растительные клетки, грибковые клетки, архейные клетки, эубактериальные клетки и др. Клетки включают такие эукариотические клетки, как дрожжевые клетки, растительные клетки и клетки животных. Предпочтительными клетками являются клетки млекопитающих. Кроме того, клетки включают такие клетки, у которых была бы выгодна или желательна регуляция целевой нуклеиновой кислоты. Такие клетки могут включать клетки, которые дефектны по экспрессии определенного белка, что ведет к заболеванию или болезненному состоянию. Такие заболевания или болезненные состояния хорошо известны специалистам. В соответствии с настоящим изобретением, на нуклеиновую кислоту, отвечающую за экспрессию определенного белка, можно воздействовать описанными здесь способами и активатором транскрипции, что ведет к повышающей регуляции целевой нуклеиновой кислоты и экспрессии соответствующего конкретного белка. Таким образом, описанные здесь способы обеспечивают терапевтическое лечение.
Целевые нуклеиновые кислоты включают такие последовательности нуклеиновой кислоты, для регуляции или надреза которых может применяться комплекс совместной локализации, как описано здесь. Целевые нуклеиновые кислоты включают гены. В целях настоящего изобретения ДНК, как-то двухцепочечная ДНК, может включать в себя целевую нуклеиновую кислоту, а комплекс совместной локализации может связываться или иным образом совместно локализироваться на ДНК на или вблизи или возле целевой нуклеиновой кислоты, причем таким образом, чтобы комплекс совместной локализации мог оказывать желательный эффект на целевую нуклеиновую кислоту. Такие целевые нуклеиновые кислоты могут включать в себя эндогенные (или природные) нуклеиновые кислоты и экзогенные (или чужеродные) нуклеиновые кислоты. Исходя из настоящего изобретения, специалист легко сможет установить или разработать такие направляющые РНК и белки Cas9, которые совместно локализуются на ДНК, включая целевую нуклеиновую кислоту. Также специалист легко сможет установить регулирующие транскрипцию белки или домены, которые точно так же совместно локализуются на ДНК, включая целевую нуклеиновую кислоту. ДНК означает геномную ДНК, митохондриальную ДНК, вирусную ДНК или экзогенную ДНК.
Чужеродные нуклеиновые кислоты (то есть те, что не входят в состав природных нуклеиновых кислот в клетке) можно вводить в клетки любым методом, известным специалистам для такого введения. Такие способы включают трансфекцию, трансдукцию, вирусную трансдукцию, микроинъекцию, липофекцию, нуклеофекцию, бомбардировку наночастицами, трансформацию, конъюгацию и др. Специалисты смогут легко понять и адаптировать такие методы, используя легко определимые литературные источники.
Регулирующие транскрипцию белки или домены, которые являются активаторами транскрипции, включают VP16 и VP64 и другие, легко определяемые специалистами в данной области на основании настоящего описания.
Заболевания или болезненные состояния представляет собой те, которые характеризуются аномальной потерей экспрессии определенного белка. Такие заболевания или болезненные состояния можно лечить посредством повышающей регуляции конкретного белка. Соответственно, предусмотрены способы лечения заболеваний или болезненных состояний, где совместная локализация комплекс, в которых описанный здесь комплекс совместной локализации связывается или иным образом ассоциируется с ДНК, включая целевую нуклеиновую кислоту, а активатор транскрипции из комплекса совместной локализации усиливает экспрессию целевой нуклеиновой кислоты. Например, повышающая регуляция PRDM16 и других генов, вызывающих дифференцировку и усиление метаболизма бурого жира, может применяться для лечения метаболического синдрома или ожирения. Активация противовоспалительных генов применима при аутоиммунных и сердечно-сосудистых заболеваниях. Активация генов-супрессоров опухолей применима при лечении рака. Специалисты в данной области смогут легко установить такие заболевания и болезненные состояния на основании настоящего описания.
Следующие примеры приводятся в качестве репрезентативных для настоящего изобретению. Эти примеры не должны истолковываться как ограничивающие объем настоящего изобретения, поскольку эти и другие эквивалентные воплощения станут очевидными в свете настоящего описания, рисунков и прилагаемой формулы изобретения.
ПРИМЕРЫ
Пример I. Мутанты Cas9
Проводили поиск последовательностей, гомологичных Cas9 с известной структурой, для выявления таких возможных мутаций у Cas9, которые могли бы устранить естественную активность его доменов RuvC и HNH. Используя HHpred (адрес в интернете: toolkit.tuebingen.mpg.de/hhpred), вводили полную последовательность Cas9 для поиска по всей базе данных Protein Data Bank (января 2013 г.). Этот поиск выдал две разные эндонуклеазы HNH со значительной гомологией последовательности к домену HNH у Cas9; а именно Pad и предполагаемые эндонуклеазы (PDB IDs: 3M7K и 4H9D, соответственно). Эти белки изучали, чтобы найти остатки, участвующие в координировании иона магния. Затем соответствующие остатки были идентифицированы при выравнивании их последовательностей с Cas9. В каждой структуре были идентифицированы две координирующие Mg боковые цепи, которые выравнивались с аминокислотами одного и того же типа у Cas9. Это D92 и N113 у 3M7K и D53 и N77 у 4H9D. Эти остатки соответствуют D839 и N863 у Cas9. Также сообщалось, что у Pad мутации остатков D92 и N113 на аланин делают нуклеазу каталитически дефектной. Мутации D839A и N863A у Cas9 были сделаны на основе этого анализа. Кроме того, HHpred также предсказывает гомологию между Cas9 и N-концом RuvC Thermus thermophilus (PDB ID: 4EP4). Это выравнивание последовательностей охватывает приведенную ранее мутацию D10A, которая устраняет функцию домена RuvC у Cas9. Чтобы проверить, что это правильная мутация, определяли связывающие металл остатки, как и ранее. У 4ЕР4 остаток D7 помогает координировать ион магния. Это положение в последовательности обладает гомологией, соответствующей D10 у Cas9, подтверждая, что эта мутация способствует устранению связывания металла, а тем самым и каталитической активности домена RuvC у Cas9.
Пример II. Конструирование плазмид
Мутантов Cas9 получали с помощью набора QuikChange (Agilent Technologies). Конструкции, экспрессирующие целевые гидРНК, либо (1) заказывали непосредственно в виде индивидуальных gBlocks у фирмы ГОТ и клонировали в вектор pCR-BluntII-TOPO (Invitrogen); либо (2) синтезировали по заказу на фирме Genewiz; либо (3) собирали из олигонуклеотидов методом Gibson assembly в клонирующий вектор для гидРНК (плазмида #41824). Векторы для анализа репортера HR с разорванным GFP конструировали путем слияния методом ПЦР-сборки (assembly PCR) последовательности GFP, несущей стоп-кодон, а соответствующие фрагменты собирали в лентивектор EGIP фирмы Addgene (плазмида #26777). Эти лентивекторы затем использовали для получения стабильных линий репортера GFP. TALE-нуклеазы (TALENs), используемые в данном исследовании, конструировали по стандартным методикам. См. Sanjana et al., Nature Protocols 7, 171-192 (2012), которая включена сюда путем ссылки во всей полноте. Слияние Cas9N с VP64 из MS2 проводили по стандартным методикам слияния методом ПЦР. Конструкции промотор-люцифераза для ОСТ4 и REX1 получали из фирмы Addgene (плазмида # 17221 и плазмида #17222).
Пример III. Культивирование и трансфекция клеток
Клетки НЕК 293Т культивировали в модифицированной Дюльбекко среде Игла (DMEM, Invitrogen) с высоким содержанием глюкозы и с добавлением 10% фетальной бычьей сыворотки (FBS, Invitrogen), пенициллина/стрептомицина (pen/strep, Invitrogen) и заменимых аминокислот (NEAA, Invitrogen). Клетки содержали при 37°С и 5% СО2 в увлажненном инкубаторе.
Трансфекции для нуклеазных методов проводили следующим образом: 0,4×106 клеток трансфецировали 2 мкг плазмиды Cas9, 2 мкг гидРНК и/или 2 мкг плазмиды с донорской ДНК с помощью Lipofectamine 2000 в соответствии с методикой производителя. Через 3 дня после трансфекции клетки собирали и либо анализировали методом FACS, либо экстрагировали геномную ДНК из ~1×106 клеток с помощью набора DNAeasy (Qiagen) для непосредственного анализа геномных разрывов. Для этого проводили ПЦР для амплификации целевого участка, используя геномную ДНК, полученную из клеток, а ампликоны подвергали глубокому секвенированию на MiSeq Personal Sequencer (Illumina) с охватом >200000 раз. Данные по секвенированию подвергали анализу для оценки эффективности NHEJ.
Трансфекции для анализа активации транскрипции: 0,4×106 клеток трансфецировали (1) 2 мкг плазмиды Cas9N-VP64, 2 мкг гидРНК и/или 0,25 мкг репортерной конструкции; или (2) 2 мкг плазмиды Cas9N, 2 мкг VP64 MS2, 2 мкг гидРНК-аптамер 2X-MS2 и/или 0,25 мкг репортерной конструкции. Через 24-48 ч после трансфекции клетки собирали и анализировали методом FACS или методом иммунофлуоресценции либо экстрагировали из них тотальную РНК, а затем анализировали методом ОТ-ПЦР. При этом использовали стандартные зонды TaqMan фирмы Invitrogen для ОСТ4 и REX1, а нормирование для каждого образца выполняли по GAPDH.
Трансфекции для анализа активации транскрипции на профиль специфичности комплексов Cas9-гидРНК и TALEs: 0,4×106 клеток трансфецировали (1) 2 мкг плазмиды Cas9N-VP64, 2 мкг гидРНК и 0,25 мкг репортерной библиотеки; или (2) 2 мкг плазмиды TALE-TF и 0,25 мкг репортерной библиотеки; или (3) 2 мкг плазмиды контроль-TF и 0,25 мкг репортерной библиотеки. Клетки собирали через 24 ч после трансфекции (чтобы избежать стимуляции репортеров, находящихся в режиме насыщения). Экстрагирование тотальной РНК проводили с помощью набора RNAeasy-plus (Qiagen), а стандартный ОТ-ПЦР выполняли с помощью Superscript-III (Invitrogen). Библиотеки для секвенирования следующего поколения получали методом прицельной ПЦР-амплификации транскриптов с тегами.
Пример IV. Вычисления и анализ последовательности для расчета уровней экспрессии репортеров Cas9-TF и TALE-TF
На фиг. 8А схематически представлена блок-схема высокого уровня для этого процесса, а здесь приводятся дополнительные подробности. Подробнее насчет состава библиотек конструкций см. фиг. 8А (уровень 1) и 8В.
Секвенирование. Для экспериментов с Cas9, последовательности из библиотеки конструкций (фиг. 8А, уровень 3, слева) и последовательности кДНК генов-репортеров (фиг. 8А, уровень 3, справа) получали в виде перекрывающихся парных конечных прочтений в 150 п.о. на приборе Illumina MiSeq, тогда как для экспериментов с TALE соответствующие последовательности получали в виде неперекрывающихся парных конечных прочтений в 51 п.о. на приборе Illumina HiSeq.
Обработка последовательностей из библиотеки конструкций. Выравнивание. Для экспериментов с Cas9, для выравнивания парных прочтений с комплектом контрольных последовательностей в 250 п.о., что соответствует 234 п.о. из конструкций, фланкированных парами штрихкодов библиотеки по 8 п.о., использовали Novoalign V2.07.17 (сайт в интернете: novocraft.com/main/index/php) (см. фиг. 8А, 3-й уровень, слева). У контрольных последовательностей, вводимых в Novoalign, вырожденные участки сайта связывания Cas9 в 23 п.о. и вырожденные участки тегов транскриптов в 24 п.о. (фиг. 8А, первый уровень) приводятся как Ns, а штрихкоды библиотеки конструкций приводятся в явном виде. Для экспериментов с TALE использовали те же методики, за исключением того, что контрольные последовательности были длиной в 203 п.о., а вырожденные участки сайта связывания были длиной в 18 п.о., а не 23 п.о. Проверка достоверности. На выходе из Novoalign получали файлы, в которых прочтения слева и справа для каждой пары прочтений индивидуально выравнивались с контрольными последовательностями. Только те пары прочтений, в которых оба прочтения выравнивались с эталонной последовательностью, подвергали дополнительным условиям проверки, и сохраняли только те пары прочтений, которые удовлетворяли всем этим условиям. Условия достоверности включали: (i) каждый из двух штрихкодов библиотеки конструкций должен выравниваться по крайней мере по 4 позициям со штрихкодом контрольной последовательности, и оба штрихкода должны выравниваться с парой штрихкодов для той же библиотеки конструкций; (ii) все основания, выравнивающиеся с N-участками контрольной последовательности, должны быть обозначены Novoalign как А, С, G или Т. Обратите внимание, что ни для экспериментов с Cas9, ни с TALE, прочтения слева и справа не перекрывались в контрольном N-участке, так что возможность неоднозначного обозначения Novoalign этих N-оснований не возникала; (iii) точно так же, в этих участках не должны появляться выявляемые Novoalign вставки или делеции; (iv) не должно быть Т в участках тегов транскриптов (так как эти случайные последовательности создавались только из А, С и G). Пары прочтений, у которых любое из этих условий нарушалось, собирали в файл отклоненных пар прочтений. Эти проверки на достоверность выполнялись при помощи пользовательских скриптов Perl.
Обработка последовательностей кДНК гена репортера в индуцированных образцах. Выравнивание. Сначала использовали SeqPrep (загруженный из сайта в интернете github.com/jstjohn/SeqPrep) для слияния перекрывающихся пар прочтений в один общий сегмент в 79 п.о., после чего использовали Novoalign (версия выше) для выравнивания этих общих сегментов по 79 п.о. в виде единого неспаренного прочтения с комплектом контрольных последовательностей (см. фиг. 8А, 3-й уровень, слева), в котором (как и при секвенировании библиотеки конструкций) вырожденные участки тегов транскриптов в 24 п.о. приводятся как Ns, а штрихкоды библиотеки конструкций приводятся в явном виде. И для TALE, и для Cas9 участки последовательностей кДНК соответствовали одним и тем же участкам кДНК в 63 п.о., фланкированным парами последовательностей штрихкодов образца по 8 п.о.
Проверка применимости. Применяли те же условия достоверности, что и при секвенировании библиотеки конструкций (см. выше), за исключением того, что: (а) вследствие предварительного слияния SeqPrep пар прочтений, при проверке на достоверность не нужны были фильтры на однозначное выравнивание обоих прочтений из одной пары прочтений, а только на однозначное выравнивание слившихся прочтений; (b) при прочтении последовательностей кДНК встречались только теги транскриптов, поэтому проверка на достоверность проводилась только по этим участкам тегов из контрольных последовательностей, а не по отдельным участкам сайтов связывания.
Составление таблицы соответствия между сайтами связывания и тегами транскриптов. Для составления этих таблиц по проверенным последовательностям библиотеки конструкций (фиг. 8А, 4-й уровень, слева) использовали пользовательский скрипт Perl. Хотя последовательности тегов в 24 п.о., состоящие из оснований А, Си G, должны быть практически уникальными по всей библиотеке конструкций (вероятность общности =~2,8×10-11), однако в самом начале анализ соответствия между сайтами связывания и тегами показал, что довольно значительная доля последовательностей тегов на самом деле является общей для нескольких последовательностей сайтов связывания, что может быть вызвано в основном сочетанием ошибок в последовательности сайтов связывания или ошибок в синтезе олигонуклеотидов, используемых для получения библиотек конструкций. Наряду с совместным использованием тегов, теги, оказавшиеся связанными с сайтами связывания в проверенных парах прочтений, также могли оказаться в файле отклоненных пар прочтений библиотеки конструкций, если не было ясно, из-за несоответствия штрихкодов, из какой библиотеки конструкций они могут быть. Наконец, сами последовательности тегов могут содержать ошибки в последовательности. Чтобы разобраться с этими источниками ошибок, теги классифицировали по трем атрибутам: (i) надежный или ненадежный, где ненадежный означает то, что тег может находиться в файле отклоненных пар прочтений библиотеки конструкций; (ii) совместный или не совместный, где совместный означает то, что тег оказался связан с последовательностями нескольких сайтов связывания; и (iii) 2+ или только 1, где 2+ означает то, что тег встречался по крайней мере дважды среди проверенных последовательностей библиотеки конструкций и поэтому считается, что он вряд ли содержит ошибки последовательности. Сочетание этих трех критериев дает 8 классов тегов, связанных с каждым сайтом связывания, причем самый надежный (но наименее распространенный) класс включает только теги типа надежный, не совместный, 2+; а наименее надежный (но самый распространенный) класс включает все теги, независимо от надежности, совместного использования или встречаемости.
Расчет нормированных уровней экспрессии. Для выполнения операций, указанных на фиг. 8А, уровни 5-6, использовали пользовательский код Perl. Во-первых, для каждого сайта связывания суммировали число тегов, полученное для каждого индуцированного образца, используя таблицу соответствия между сайтами связывания и тегами транскриптов, составленную ранее для библиотеки конструкций (см. фиг. 8С). Затем для каждого образца суммарное число тегов по каждому сайту связывания делили на суммарное число тегов для положительного контрольного образца, получая нормированные уровни экспрессии. Дополнительные соображения, касающиеся этих расчетов, включают:
1). У каждого образца среди проверенных на достоверность последовательностей кДНК генов встречалось подмножество "новых" тегов, которые не встречались в таблице соответствия между сайтами связывания и тегами транскриптов. Эти теги не учитывали при последующих вычислениях.
2). Суммирование числа тегов, описанное выше, проводили для каждого из описанных выше 8 классов тегов в таблице соответствия между сайтами связывания и тегами транскриптов. Поскольку сайты связывания в библиотеках конструкций часто были "смещенными", чтобы получить последовательности, близкие к центральной последовательности, а последовательности с большим количеством несоответствий встречались редко, то сайты связывания с немногими несоответствиями при суммировании обычно давали большое число тегов, тогда как сайты связывания со многими несоответствиями давали меньшее число тегов. Поэтому, хотя вообще-то было желательно использовать самый надежный класс тегов, однако оценка сайтов связывания с двумя и более несоответствиями могла бы основываться на небольшом числе тегов на 1 сайт связывания, что сделает надежные числа и соотношения менее достоверными статистически, даже если сами теги и были бы более надежными. В таких случаях использовали все теги. Некоторая компенсация за это вытекает из того факта, что количество отдельных суммарных чисел тегов для n позиций с несоответствиями возрастает вместе с количеством комбинаций этих позиций (равным 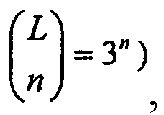 которое резко возрастает с увеличением n; поэтому средние значения суммарного числа тегов для различного количества n несоответствий (приведенные на фиг. 2В, 2Е и на фиг. 9А и 10В) основываются на статистически очень большой совокупности суммарных чисел тегов для n≥2.
которое резко возрастает с увеличением n; поэтому средние значения суммарного числа тегов для различного количества n несоответствий (приведенные на фиг. 2В, 2Е и на фиг. 9А и 10В) основываются на статистически очень большой совокупности суммарных чисел тегов для n≥2.
3). Наконец, сайт связывания, встроенный в библиотеки конструкций TALE, составлял 18 п.о., и таблица соответствия тегов составлялась на основе этих последовательностей в 18 п.о., но некоторые эксперименты проводились с TALEs, запрограммированными на связывание с центральными участками в 14 п. о. или 10 п. о. в пределах сайта связывания в 18 п.о. у конструкций. При вычислении уровней экспрессии для этих TALEs, теги суммировали по сайтам связывания, исходя из соответствующих участков сайтов связывания в 18 п.о. в таблице соответствия, при этом несоответствия у сайтов связывания за пределами этого участка не учитывали.
Пример V. РНК-направляемая регуляция SOX2 и NANOG с помощью Cas9N-VP64 Описанный здесь подход с пришиванием s(одиночная)-гидРНК (модифицированная аптамером одиночная направляющая РНК) позволяет рекрутировать различные эффекторные домены при помощи различных sгидРНК, если только каждая одиночная-гидРНК использует другую взаимодействующую пару РНК-белок, что позволяет мультиплексную регуляцию генов с помощью одного и того же белка Cas9N. Для генов SOX2 (фиг. 12А) и NANOG (фиг. 12В) составляли 10 гидРНК, нацеленных на отрезок ДНК в ~1 т.п.о. впереди от сайта инициации транскрипции. Гиперчувствительные к ДНКазе сайты выделены зеленым цветом. Определяли активация транскрипции методом кПЦР эндогенных генов. В обоих случаях, в то время как введение отдельных гидРНК умеренно стимулировало транскрипцию, несколько гидРНК действовали синергически, вызывая сильную многократную активацию транскрипции. Данные в виде среднего ±SEM (n=3). Как видно из фиг. 12А-В, два других гена, SOX2 и NANOG, тоже регулировались с помощью sгидPHКs, нацеленных на отрезок ДНК в ~1 т.п.о. перед промотором. Нацеленные проксимально к сайту инициации транскрипции одиночные-гидРНК вызывали сильную активацию генов.
Пример VI. Оценка ландшафта нацеливания у комплексов Cas9-гидРНК
Используя подход, представленный на фиг. 2, анализировали ландшафт нацеливания у двух дополнительных комплексов Cas9-гидРНК (фиг. 13А-С и фиг. 13D-F). Эти две гидРНК имеют очень разные профили специфичности, причем гидРНК2 допускает до 2-3 несоответствий, а гидРНК3-только 1. Эти аспекты отражены на графиках с несоответствием и по одному основанию (фиг. 13В, 13Е), и по двум основаниям (фиг. 13С, 13F). На фиг. 13С и 13F неспаренные пары оснований, для которых было недостаточно данных для расчета нормированного уровня экспрессии, обозначены как серые блоки, содержащие "×", тогда как, чтобы улучшить отображение данных, неспаренные пары, у которых нормированные уровни экспрессии представляют собой выбросы, превышающие верхнюю часть цветовой гаммы, обозначены как желтые блоки, содержащие звездочки "*". Символы статистической значимости: *** для р<0,0005/n, ** для р<0,005/n, * для р<0,05/n, n.s. (не значимо) для р≥0,05/n, где n - количество сравнений (см. табл. 2).
Пример VII. Проверка специфичности в анализе репортеров
Как видно из фиг. 14А-С, данные по специфичности получали с использованием двух разных комплексов sгидPHК:Cas9. Было подтверждено, что анализ является специфичным для исследуемых sгидРНК, поскольку соответствующая мутантная гидРНК была неспособна стимулировать репортерную библиотеку. Фиг. 14А. Профиль специфичности двух гидРНК (дикого типа и мутанта; различия в последовательностях выделены красным цветом) оценивали с помощью репортерной библиотеки, созданной по последовательности мишени гидРНК дикого типа. Фиг. 14 В. Было подтверждено, что данный анализ является специфичным для исследуемой гидРНК оценивается (данные взяты из графика на фиг. 13D), поскольку соответствующая мутантная гидРНК была неспособна стимулировать репортерную библиотеку. Символы статистической значимости: *** для р<0,0005/n, ** для р<0,005/n, * для р<0,05/n, n.s. (не значимо) для р≥0,05/n, где n - количество сравнений (см. табл. 2). Различные гидРНК могут иметь разные профили специфичности (фиг. 13А, 13D), в частности, гидРНК2 допускает до 3 несоответствий, а гидРНК3-только 1. Наибольшая чувствительность к несоответствиям приходится на 3'-конец спейсера, хотя несоответствия в других позициях также оказывали влияние на активность.
Пример VIII. Проверка, несоответствия в гидРНК по одному и двум основаниям
Как видно из фиг. 15A-D, прицельные эксперименты подтверждают, что при несоответствии по одному основанию в пределах 12 п.о. от 3'-конца спейсера в исследуемых sгидРНК сохраняется заметная успешность нацеливания. Однако несоответствия по 2 основаниям в этом участке приводят к значительной потере активности. Используя нуклеазный метод, исследовали 2 независимые гидРНК: гидРНК2 (фиг. 15А-В) и гидРНК3 (фиг. 15C-D), несущие несоответствия по одному или двум основаниям (выделены красным цветом) в последовательности спейсера в отношении мишени. Было подтверждено, что при несоответствии по одному основанию в пределах 12 п.о. от 3'-конца спейсера в исследуемых гидРНК сохраняется заметная успешность нацеливания, однако несоответствия по 2 основаниям в этом участке приводят к быстрой потере активности. Эти результаты также подчеркивают различия в профилях специфичности между различными гидРНК в соответствии с результатами на фиг. 13. Данные в виде среднего ±SEM (n=3).
Пример IX. Проверка, 5'-усечения гидРНК
Как видно из фиг. 16A-D, усечения в 5'-части спейсера позволяют сохранить активность одниночной-гидРНК. Используя нуклеазный метод, исследовали 2 независимые гидРНК: гидРНК 1 (фиг. 16А-В) и гидРНК 3 (фиг. 16C-D), несущие усечения на 5-конце спейсера. Как оказалось, 5'-усечения в 1-3 п. о. хорошо переносятся, но более крупные делеции приводят к потере активности. Данные в виде среднего ±SEM (n=3).
Пример X. Проверка, РАМ у S. pyogenes
Как видно из фиг. 17А-В, используя метод опосредованной нуклеазой HR, было подтверждено, что РАМ для Cas9 S. pyogenes представлен не только NGG, но и NAG. Данные в виде среднего ±SEM (n=3). Согласно дополнительному исследованию, просканировали созданный набор примерно из 190 тысяч мишеней Cas9 в экзонах человека, у которых не было других NGG-мишеней с общими последними 13 нуклеотидами в целевой последовательности, на наличие альтернативных NAG-сайтов или NGG-сайтов с несоответствием в первых 13 нт. Как оказалось, только 0,4% не имеют таких альтернативных мишеней.
Пример XI. Проверка, мутации TALE
Используя метод опосредованной нуклеазой HR (фиг. 18А-В), подтвердили, что 18-меры TALE допускают множественные мутации в последовательности своей мишени. Как видно из фиг. 18А-В, некоторые мутации посреди мишени приводят к повышению активности TALE, как установлено в прицельных экспериментах нуклеазным методом.
Пример XII. Специфичность мономеров TALE или специфичность белка TALE
Чтобы выяснить роль индивидуальных вариабельных би-остатков из повторов (repeat variable diresidue, RVD), подтвердили, что выбор RVDs вносит вклад в специфичность к основаниям, но специфичность TALE также является функцией энергии связывания всего белка в целом. На фиг. 19А-С представлено сравнение специфичности мономеров TALE со специфичностью всего белка TALE. Фиг. 19А. Используя модификацию подхода, описанного на фиг. 2, анализировали ландшафт нацеливания у двух 14-меров TALE-TF, несущих последовательный набор из 6 NI-повторов или 6 NH-повторов. При таком подходе создается редуцированная библиотека репортеров, несущих вырожденную последовательность 6-мера посредине, которая используется для анализа специфичности TALE-TF. Фиг. 19В-С. В обоих случаях оказалось, что ожидаемая последовательность мишени является обогащенной (т.е. она несет 6 А для NI-повторов и 6 G для NH-повторов). Каждый из этих TALEs все еще допускает 1-2 несоответствия в последовательности центрального 6-мера мишени. Хотя выбор мономеров действительно вносит вклад в специфичность к основаниям, но специфичность TALE также является функцией энергии связывания всего белка в целом. Согласно одному аспекту, короткие сконструированные TALEs или TALEs, несущие сочетания мономеров с высоким и низким сродством, дают большую специфичность в применении к генной инженерии, а димеризация FokI в применении к нуклеазе позволяет еще больше уменьшить эффекты промашки при использовании коротких TALEs.
Пример XIII. Смещенные надрезы, нативный локус
На фиг. 20А-В представлены данные, касающиеся смещенных одноцепочечных разрывов (off-set nicking). В контексте редактирования генома смещенные надрезы создаются для получения двухцепочечных разрывов (DSBs). Большая часть надрезов не вызывает опосредованных негомологичным соединением концов (NHEJ) вставок или делеций (indels), поэтому при образовании смещенных одноцепочечных разрывов отдельные случаи одноцепочечных разрывов вне мишени, скорее всего, будут давать очень низкий уровень вставок или делеций (indels). Образование смещенных одноцепочечных разрывов для получения DSBs эффективно вызывает разрушение генов как во встроенных локусах репортеров, так и в нативном геномном локусе AAVS1. Фиг. 20А. Нативный локус AAVS1, на который нацелены 8 гидРНК, охватывающих отрезок ДНК в 200 п. о.: 4 на смысловую нить (s1-4) и 4 на антисмысловую нить (as1-4). Используя мутанта Cas9D10A, который надрезает комплементарную нить, создавали различные двусторонние комбинации гидРНК, чтобы получить целый ряд запрограммированных свисающих 5'- или 3'-концов. Фиг. 20В. Используя метод на основе секвенирования по Сэнгеру, оказалось, что в то время, как одиночные гидРНК не вызывают заметных случаев NHEJ, создание смещенных одноцепочечных разрывов для получения DSBs очень эффективно вызывает разрушение генов. А именно, смещенные надрезы, дающие свисающие 5'-концы, дают больше случаев NHEJ, чем свисающие 3'-концы. Количество клонов для секвенирования по Сэнгеру приведено над столбиками, а прогнозируемая длина свисающих концов указана под соответствующими надписями на оси х.
Пример XIV. Смещенные надрезы, профили NHEJ
Фиг. 21А-С касается смещенных одноцепочечных разрывов и профилей NHEJ. Представлены репрезентативные результаты секвенирования по Сэнгеру для трех различных комбинаций смещенных одноцепочечных разрывов, а положения таргетинговых гидРНК выделены рамками. Далее, в соответствии со стандартной моделью опосредованной гомологичной рекомбинацией (HR) репарации, создание свисающих 5'-концов посредством смещенных одноцепочечных разрывов вызывает гораздо больше случаев NHEJ, чем у свисающих 3'-концов (фиг. 3В). Наряду со стимуляцией NHEJ, при создании свисающих 5'-концов наблюдалась сильная индукция HR. Создание свисающих 3'-концов не вызывало улучшения степени HR (фиг. 3С).
Пример XV. Мишени гидРНК для регуляции эндогенных генов
В табл. 1 приведены мишени в промоторах REX1, ОСТ4, SOX2 и NANOG, использовавшиеся в экспериментах по опосредованной Cas9-гидРНК активации.


Пример XVI. Сводка по статистическому анализу данных по специфичности Cas9-гидРНКиТАLЕ
Таблица 2(a). р-значения для сравнения нормированных уровней экспрессии при связывании активаторов TALE и Cas9-VP64 с последовательностями мишеней с определенным количеством мутаций в сайте мишени. Нормированные уровни экспрессии приведены в виде ящичковых диаграмм на фигурах, указанных в столбце "Фигура", где рамочками представлено распределение этих уровней по числу несоответствий у сайта мишени, р-значения рассчитывали по t-критерию для каждой последовательной пары чисел несоответствий в каждой ящичковой диаграмме, причем t-критерии были либо для одной выборки, либо для двух выборок (см. Методы). Статистическую значимость определяли по скорректированным по Бонферрони пороговым р-значениям, причем коррекция основывалась на количестве сравнений в пределах каждой ящичковой диаграммы. Символы статистической значимости: *** для р<0,0005/n, ** для р<0,005/n, * для р<0,05/n, n.s. (не значимо) для р≥0,05/n, где n - количество сравнений.
Таблица 2(b). Статистические характеристики области "ядра" на фиг. 2D. Величина log10(значения р) показывает степень разделения между уровнями экспрессии при связывании Cas9N-VP64 + гидРНК с последовательностями мишеней с двумя мутациями для тех пар позиций, которые мутированы в пределах предполагаемой области ядра на 3'-конце сайта мишени в 20 п. о. по сравнению со всеми остальными парами позиций. Наибольшее разделение, засвидетельствованное наибольшими величинами -log10(значения р) (выделены выше), приходится на последние 8-9 п.о. сайта мишени. Эти позиции можно интерпретировать как означающие начало области ядра этого сайта мишени. См. раздел "Статистическая характеристика области ядра" в Методах насчет информации о том, как рассчитывали р-значения.
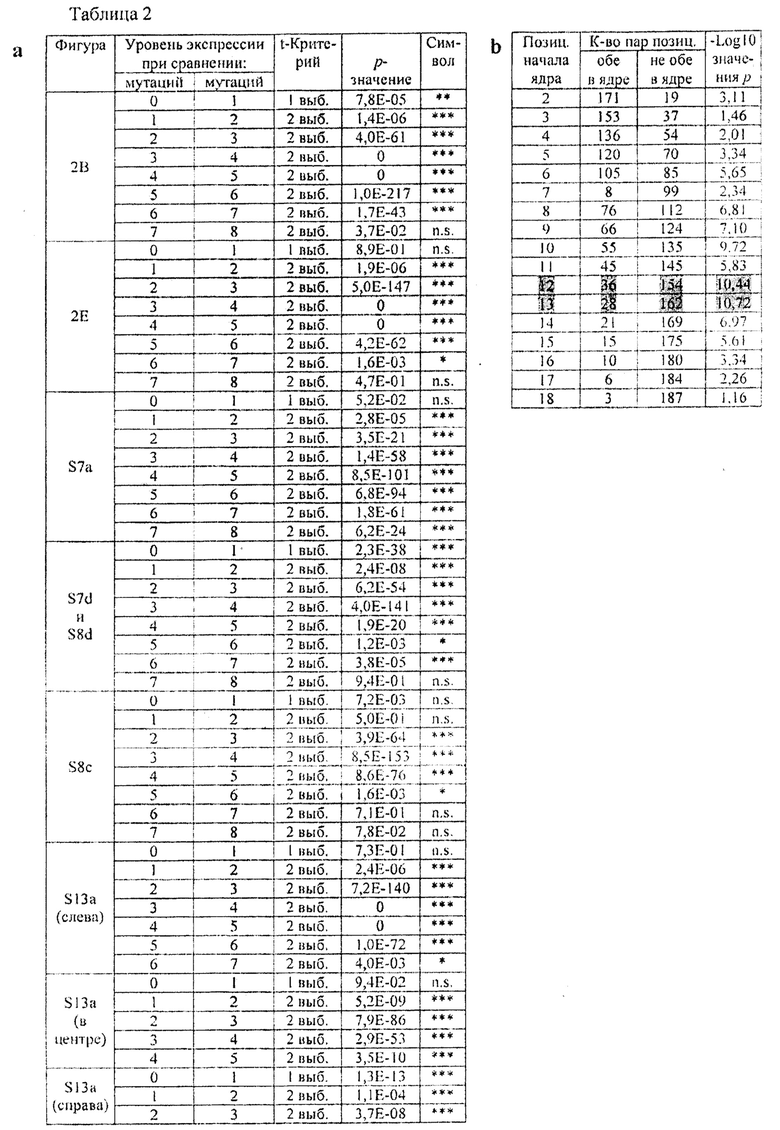
Пример XVII. Последовательности белков и РНК в примерах
А. Последовательности конструкций активаторов Cas9N--VP64 на основе мутанта m4, представленные ниже. Были созданы 3 версии в формате слитых белков Cas9m4VP64 и Cas9m4VP64N, проявляющих самую высокую активность. Также были составлены соответствующие векторы для мутантов m3 и m2 (фиг. 4А) (выделены домены NLS и VP64).
>Cas9m4VP64













В. Последовательности конструкций MS2-активаторов и соответствующего базового вектора для гидРНК с доменами аптамера 2Х MS2, представленные ниже (выделены домены NLS, VP64, спейсер для гидРНК и область стебелек-петля MS2-связывающей РНК). Были созданы 3 версии активаторов в формате слитого белка MS2VP64N, проявляющие самую высокую активность.
>MS2VP64N


С. Последовательности репортеров активации транскрипции по флуоресценции dTomato, представленные ниже (выделены последовательности мишени контрольного TF IScel, мишеней для гидРНК, промотора minCMV и тега FLAG + dTomato).
>TF-репортер 1


D. Общий формат библиотек репортеров, используемых для анализа специфичности TALE и Cas9-гидРНК, которые представлены ниже (выделены последовательности мишени для контрольного TF IScel, сайта мишени для гидРНК/TALE (23 п.о. для гидРНК и 18 п.о. для TALE), промотора minCMV, "штрихкода" РНК и dTomato).
>Библиотеки репортеров для специфичности

--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQUENCE LISTING
<110> President and Fellows of Harvard College
<120> RNA-Guided Transcriptional Regulation
<130> 010498.00503
<140> PCT/US14/040868
<141> 2014-06-04
<150> US 61/830787
<151> 2013-06-04
<160> 184
<170> PatentIn version 3.5
<210> 1
<211> 1368
<212> PRT
<213> Streptococcus pyogenes
<400> 1
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 2
<211> 4332
<212> DNA
<213> Artificial
<220>
<223> VP64-activator construct
<400> 2
gccaccatgg acaagaagta ctccattggg ctcgctatcg gcacaaacag cgtcggctgg 60
gccgtcatta cggacgagta caaggtgccg agcaaaaaat tcaaagttct gggcaatacc 120
gatcgccaca gcataaagaa gaacctcatt ggcgccctcc tgttcgactc cggggagacg 180
gccgaagcca cgcggctcaa aagaacagca cggcgcagat atacccgcag aaagaatcgg 240
atctgctacc tgcaggagat ctttagtaat gagatggcta aggtggatga ctctttcttc 300
cataggctgg aggagtcctt tttggtggag gaggataaaa agcacgagcg ccacccaatc 360
tttggcaata tcgtggacga ggtggcgtac catgaaaagt acccaaccat atatcatctg 420
aggaagaagc ttgtagacag tactgataag gctgacttgc ggttgatcta tctcgcgctg 480
gcgcatatga tcaaatttcg gggacacttc ctcatcgagg gggacctgaa cccagacaac 540
agcgatgtcg acaaactctt tatccaactg gttcagactt acaatcagct tttcgaagag 600
aacccgatca acgcatccgg agttgacgcc aaagcaatcc tgagcgctag gctgtccaaa 660
tcccggcggc tcgaaaacct catcgcacag ctccctgggg agaagaagaa cggcctgttt 720
ggtaatctta tcgccctgtc actcgggctg acccccaact ttaaatctaa cttcgacctg 780
gccgaagatg ccaagcttca actgagcaaa gacacctacg atgatgatct cgacaatctg 840
ctggcccaga tcggcgacca gtacgcagac ctttttttgg cggcaaagaa cctgtcagac 900
gccattctgc tgagtgatat tctgcgagtg aacacggaga tcaccaaagc tccgctgagc 960
gctagtatga tcaagcgcta tgatgagcac caccaagact tgactttgct gaaggccctt 1020
gtcagacagc aactgcctga gaagtacaag gaaattttct tcgatcagtc taaaaatggc 1080
tacgccggat acattgacgg cggagcaagc caggaggaat tttacaaatt tattaagccc 1140
atcttggaaa aaatggacgg caccgaggag ctgctggtaa agcttaacag agaagatctg 1200
ttgcgcaaac agcgcacttt cgacaatgga agcatccccc accagattca cctgggcgaa 1260
ctgcacgcta tcctcaggcg gcaagaggat ttctacccct ttttgaaaga taacagggaa 1320
aagattgaga aaatcctcac atttcggata ccctactatg taggccccct cgcccgggga 1380
aattccagat tcgcgtggat gactcgcaaa tcagaagaga ccatcactcc ctggaacttc 1440
gaggaagtcg tggataaggg ggcctctgcc cagtccttca tcgaaaggat gactaacttt 1500
gataaaaatc tgcctaacga aaaggtgctt cctaaacact ctctgctgta cgagtacttc 1560
acagtttata acgagctcac caaggtcaaa tacgtcacag aagggatgag aaagccagca 1620
ttcctgtctg gagagcagaa gaaagctatc gtggacctcc tcttcaagac gaaccggaaa 1680
gttaccgtga aacagctcaa agaagactat ttcaaaaaga ttgaatgttt cgactctgtt 1740
gaaatcagcg gagtggagga tcgcttcaac gcatccctgg gaacgtatca cgatctcctg 1800
aaaatcatta aagacaagga cttcctggac aatgaggaga acgaggacat tcttgaggac 1860
attgtcctca cccttacgtt gtttgaagat agggagatga ttgaagaacg cttgaaaact 1920
tacgctcatc tcttcgacga caaagtcatg aaacagctca agaggcgccg atatacagga 1980
tgggggcggc tgtcaagaaa actgatcaat gggatccgag acaagcagag tggaaagaca 2040
atcctggatt ttcttaagtc cgatggattt gccaaccgga acttcatgca gttgatccat 2100
gatgactctc tcacctttaa ggaggacatc cagaaagcac aagtttctgg ccagggggac 2160
agtcttcacg agcacatcgc taatcttgca ggtagcccag ctatcaaaaa gggaatactg 2220
cagaccgtta aggtcgtgga tgaactcgtc aaagtaatgg gaaggcataa gcccgagaat 2280
atcgttatcg agatggcccg agagaaccaa actacccaga agggacagaa gaacagtagg 2340
gaaaggatga agaggattga agagggtata aaagaactgg ggtcccaaat ccttaaggaa 2400
cacccagttg aaaacaccca gcttcagaat gagaagctct acctgtacta cctgcagaac 2460
ggcagggaca tgtacgtgga tcaggaactg gacatcaatc ggctctccga ctacgacgtg 2520
gctgctatcg tgccccagtc ttttctcaaa gatgattcta ttgataataa agtgttgaca 2580
agatccgata aagctagagg gaagagtgat aacgtcccct cagaagaagt tgtcaagaaa 2640
atgaaaaatt attggcggca gctgctgaac gccaaactga tcacacaacg gaagttcgat 2700
aatctgacta aggctgaacg aggtggcctg tctgagttgg ataaagccgg cttcatcaaa 2760
aggcagcttg ttgagacacg ccagatcacc aagcacgtgg cccaaattct cgattcacgc 2820
atgaacacca agtacgatga aaatgacaaa ctgattcgag aggtgaaagt tattactctg 2880
aagtctaagc tggtctcaga tttcagaaag gactttcagt tttataaggt gagagagatc 2940
aacaattacc accatgcgca tgatgcctac ctgaatgcag tggtaggcac tgcacttatc 3000
aaaaaatatc ccaagcttga atctgaattt gtttacggag actataaagt gtacgatgtt 3060
aggaaaatga tcgcaaagtc tgagcaggaa ataggcaagg ccaccgctaa gtacttcttt 3120
tacagcaata ttatgaattt tttcaagacc gagattacac tggccaatgg agagattcgg 3180
aagcgaccac ttatcgaaac aaacggagaa acaggagaaa tcgtgtggga caagggtagg 3240
gatttcgcga cagtccggaa ggtcctgtcc atgccgcagg tgaacatcgt taaaaagacc 3300
gaagtacaga ccggaggctt ctccaaggaa agtatcctcc cgaaaaggaa cagcgacaag 3360
ctgatcgcac gcaaaaaaga ttgggacccc aagaaatacg gcggattcga ttctcctaca 3420
gtcgcttaca gtgtactggt tgtggccaaa gtggagaaag ggaagtctaa aaaactcaaa 3480
agcgtcaagg aactgctggg catcacaatc atggagcgat caagcttcga aaaaaacccc 3540
atcgactttc tcgaggcgaa aggatataaa gaggtcaaaa aagacctcat cattaagctt 3600
cccaagtact ctctctttga gcttgaaaac ggccggaaac gaatgctcgc tagtgcgggc 3660
gagctgcaga aaggtaacga gctggcactg ccctctaaat acgttaattt cttgtatctg 3720
gccagccact atgaaaagct caaagggtct cccgaagata atgagcagaa gcagctgttc 3780
gtggaacaac acaaacacta ccttgatgag atcatcgagc aaataagcga attctccaaa 3840
agagtgatcc tcgccgacgc taacctcgat aaggtgcttt ctgcttacaa taagcacagg 3900
gataagccca tcagggagca ggcagaaaac attatccact tgtttactct gaccaacttg 3960
ggcgcgcctg cagccttcaa gtacttcgac accaccatag acagaaagcg gtacacctct 4020
acaaaggagg tcctggacgc cacactgatt catcagtcaa ttacggggct ctatgaaaca 4080
agaatcgacc tctctcagct cggtggagac agcagggctg accccaagaa gaagaggaag 4140
gtggaggcca gcggttccgg acgggctgac gcattggacg attttgatct ggatatgctg 4200
ggaagtgacg ccctcgatga ttttgacctt gacatgcttg gttcggatgc ccttgatgac 4260
tttgacctcg acatgctcgg cagtgacgcc cttgatgatt tcgacctgga catgctgatt 4320
aactctagat ga 4332
<210> 3
<211> 4365
<212> DNA
<213> Artificial
<220>
<223> VP64-activator construct
<400> 3
gccaccatgc ccaagaagaa gaggaaggtg ggaaggggga tggacaagaa gtactccatt 60
gggctcgcta tcggcacaaa cagcgtcggc tgggccgtca ttacggacga gtacaaggtg 120
ccgagcaaaa aattcaaagt tctgggcaat accgatcgcc acagcataaa gaagaacctc 180
attggcgccc tcctgttcga ctccggggag acggccgaag ccacgcggct caaaagaaca 240
gcacggcgca gatatacccg cagaaagaat cggatctgct acctgcagga gatctttagt 300
aatgagatgg ctaaggtgga tgactctttc ttccataggc tggaggagtc ctttttggtg 360
gaggaggata aaaagcacga gcgccaccca atctttggca atatcgtgga cgaggtggcg 420
taccatgaaa agtacccaac catatatcat ctgaggaaga agcttgtaga cagtactgat 480
aaggctgact tgcggttgat ctatctcgcg ctggcgcata tgatcaaatt tcggggacac 540
ttcctcatcg agggggacct gaacccagac aacagcgatg tcgacaaact ctttatccaa 600
ctggttcaga cttacaatca gcttttcgaa gagaacccga tcaacgcatc cggagttgac 660
gccaaagcaa tcctgagcgc taggctgtcc aaatcccggc ggctcgaaaa cctcatcgca 720
cagctccctg gggagaagaa gaacggcctg tttggtaatc ttatcgccct gtcactcggg 780
ctgaccccca actttaaatc taacttcgac ctggccgaag atgccaagct tcaactgagc 840
aaagacacct acgatgatga tctcgacaat ctgctggccc agatcggcga ccagtacgca 900
gacctttttt tggcggcaaa gaacctgtca gacgccattc tgctgagtga tattctgcga 960
gtgaacacgg agatcaccaa agctccgctg agcgctagta tgatcaagcg ctatgatgag 1020
caccaccaag acttgacttt gctgaaggcc cttgtcagac agcaactgcc tgagaagtac 1080
aaggaaattt tcttcgatca gtctaaaaat ggctacgccg gatacattga cggcggagca 1140
agccaggagg aattttacaa atttattaag cccatcttgg aaaaaatgga cggcaccgag 1200
gagctgctgg taaagcttaa cagagaagat ctgttgcgca aacagcgcac tttcgacaat 1260
ggaagcatcc cccaccagat tcacctgggc gaactgcacg ctatcctcag gcggcaagag 1320
gatttctacc cctttttgaa agataacagg gaaaagattg agaaaatcct cacatttcgg 1380
ataccctact atgtaggccc cctcgcccgg ggaaattcca gattcgcgtg gatgactcgc 1440
aaatcagaag agaccatcac tccctggaac ttcgaggaag tcgtggataa gggggcctct 1500
gcccagtcct tcatcgaaag gatgactaac tttgataaaa atctgcctaa cgaaaaggtg 1560
cttcctaaac actctctgct gtacgagtac ttcacagttt ataacgagct caccaaggtc 1620
aaatacgtca cagaagggat gagaaagcca gcattcctgt ctggagagca gaagaaagct 1680
atcgtggacc tcctcttcaa gacgaaccgg aaagttaccg tgaaacagct caaagaagac 1740
tatttcaaaa agattgaatg tttcgactct gttgaaatca gcggagtgga ggatcgcttc 1800
aacgcatccc tgggaacgta tcacgatctc ctgaaaatca ttaaagacaa ggacttcctg 1860
gacaatgagg agaacgagga cattcttgag gacattgtcc tcacccttac gttgtttgaa 1920
gatagggaga tgattgaaga acgcttgaaa acttacgctc atctcttcga cgacaaagtc 1980
atgaaacagc tcaagaggcg ccgatataca ggatgggggc ggctgtcaag aaaactgatc 2040
aatgggatcc gagacaagca gagtggaaag acaatcctgg attttcttaa gtccgatgga 2100
tttgccaacc ggaacttcat gcagttgatc catgatgact ctctcacctt taaggaggac 2160
atccagaaag cacaagtttc tggccagggg gacagtcttc acgagcacat cgctaatctt 2220
gcaggtagcc cagctatcaa aaagggaata ctgcagaccg ttaaggtcgt ggatgaactc 2280
gtcaaagtaa tgggaaggca taagcccgag aatatcgtta tcgagatggc ccgagagaac 2340
caaactaccc agaagggaca gaagaacagt agggaaagga tgaagaggat tgaagagggt 2400
ataaaagaac tggggtccca aatccttaag gaacacccag ttgaaaacac ccagcttcag 2460
aatgagaagc tctacctgta ctacctgcag aacggcaggg acatgtacgt ggatcaggaa 2520
ctggacatca atcggctctc cgactacgac gtggctgcta tcgtgcccca gtcttttctc 2580
aaagatgatt ctattgataa taaagtgttg acaagatccg ataaagctag agggaagagt 2640
gataacgtcc cctcagaaga agttgtcaag aaaatgaaaa attattggcg gcagctgctg 2700
aacgccaaac tgatcacaca acggaagttc gataatctga ctaaggctga acgaggtggc 2760
ctgtctgagt tggataaagc cggcttcatc aaaaggcagc ttgttgagac acgccagatc 2820
accaagcacg tggcccaaat tctcgattca cgcatgaaca ccaagtacga tgaaaatgac 2880
aaactgattc gagaggtgaa agttattact ctgaagtcta agctggtctc agatttcaga 2940
aaggactttc agttttataa ggtgagagag atcaacaatt accaccatgc gcatgatgcc 3000
tacctgaatg cagtggtagg cactgcactt atcaaaaaat atcccaagct tgaatctgaa 3060
tttgtttacg gagactataa agtgtacgat gttaggaaaa tgatcgcaaa gtctgagcag 3120
gaaataggca aggccaccgc taagtacttc ttttacagca atattatgaa ttttttcaag 3180
accgagatta cactggccaa tggagagatt cggaagcgac cacttatcga aacaaacgga 3240
gaaacaggag aaatcgtgtg ggacaagggt agggatttcg cgacagtccg gaaggtcctg 3300
tccatgccgc aggtgaacat cgttaaaaag accgaagtac agaccggagg cttctccaag 3360
gaaagtatcc tcccgaaaag gaacagcgac aagctgatcg cacgcaaaaa agattgggac 3420
cccaagaaat acggcggatt cgattctcct acagtcgctt acagtgtact ggttgtggcc 3480
aaagtggaga aagggaagtc taaaaaactc aaaagcgtca aggaactgct gggcatcaca 3540
atcatggagc gatcaagctt cgaaaaaaac cccatcgact ttctcgaggc gaaaggatat 3600
aaagaggtca aaaaagacct catcattaag cttcccaagt actctctctt tgagcttgaa 3660
aacggccgga aacgaatgct cgctagtgcg ggcgagctgc agaaaggtaa cgagctggca 3720
ctgccctcta aatacgttaa tttcttgtat ctggccagcc actatgaaaa gctcaaaggg 3780
tctcccgaag ataatgagca gaagcagctg ttcgtggaac aacacaaaca ctaccttgat 3840
gagatcatcg agcaaataag cgaattctcc aaaagagtga tcctcgccga cgctaacctc 3900
gataaggtgc tttctgctta caataagcac agggataagc ccatcaggga gcaggcagaa 3960
aacattatcc acttgtttac tctgaccaac ttgggcgcgc ctgcagcctt caagtacttc 4020
gacaccacca tagacagaaa gcggtacacc tctacaaagg aggtcctgga cgccacactg 4080
attcatcagt caattacggg gctctatgaa acaagaatcg acctctctca gctcggtgga 4140
gacagcaggg ctgaccccaa gaagaagagg aaggtggagg ccagcggttc cggacgggct 4200
gacgcattgg acgattttga tctggatatg ctgggaagtg acgccctcga tgattttgac 4260
cttgacatgc ttggttcgga tgcccttgat gactttgacc tcgacatgct cggcagtgac 4320
gcccttgatg atttcgacct ggacatgctg attaactcta gatga 4365
<210> 4
<211> 4425
<212> DNA
<213> Artificial
<220>
<223> VP64-activator construct
<400> 4
gccaccatgg acaagaagta ctccattggg ctcgctatcg gcacaaacag cgtcggctgg 60
gccgtcatta cggacgagta caaggtgccg agcaaaaaat tcaaagttct gggcaatacc 120
gatcgccaca gcataaagaa gaacctcatt ggcgccctcc tgttcgactc cggggagacg 180
gccgaagcca cgcggctcaa aagaacagca cggcgcagat atacccgcag aaagaatcgg 240
atctgctacc tgcaggagat ctttagtaat gagatggcta aggtggatga ctctttcttc 300
cataggctgg aggagtcctt tttggtggag gaggataaaa agcacgagcg ccacccaatc 360
tttggcaata tcgtggacga ggtggcgtac catgaaaagt acccaaccat atatcatctg 420
aggaagaagc ttgtagacag tactgataag gctgacttgc ggttgatcta tctcgcgctg 480
gcgcatatga tcaaatttcg gggacacttc ctcatcgagg gggacctgaa cccagacaac 540
agcgatgtcg acaaactctt tatccaactg gttcagactt acaatcagct tttcgaagag 600
aacccgatca acgcatccgg agttgacgcc aaagcaatcc tgagcgctag gctgtccaaa 660
tcccggcggc tcgaaaacct catcgcacag ctccctgggg agaagaagaa cggcctgttt 720
ggtaatctta tcgccctgtc actcgggctg acccccaact ttaaatctaa cttcgacctg 780
gccgaagatg ccaagcttca actgagcaaa gacacctacg atgatgatct cgacaatctg 840
ctggcccaga tcggcgacca gtacgcagac ctttttttgg cggcaaagaa cctgtcagac 900
gccattctgc tgagtgatat tctgcgagtg aacacggaga tcaccaaagc tccgctgagc 960
gctagtatga tcaagcgcta tgatgagcac caccaagact tgactttgct gaaggccctt 1020
gtcagacagc aactgcctga gaagtacaag gaaattttct tcgatcagtc taaaaatggc 1080
tacgccggat acattgacgg cggagcaagc caggaggaat tttacaaatt tattaagccc 1140
atcttggaaa aaatggacgg caccgaggag ctgctggtaa agcttaacag agaagatctg 1200
ttgcgcaaac agcgcacttt cgacaatgga agcatccccc accagattca cctgggcgaa 1260
ctgcacgcta tcctcaggcg gcaagaggat ttctacccct ttttgaaaga taacagggaa 1320
aagattgaga aaatcctcac atttcggata ccctactatg taggccccct cgcccgggga 1380
aattccagat tcgcgtggat gactcgcaaa tcagaagaga ccatcactcc ctggaacttc 1440
gaggaagtcg tggataaggg ggcctctgcc cagtccttca tcgaaaggat gactaacttt 1500
gataaaaatc tgcctaacga aaaggtgctt cctaaacact ctctgctgta cgagtacttc 1560
acagtttata acgagctcac caaggtcaaa tacgtcacag aagggatgag aaagccagca 1620
ttcctgtctg gagagcagaa gaaagctatc gtggacctcc tcttcaagac gaaccggaaa 1680
gttaccgtga aacagctcaa agaagactat ttcaaaaaga ttgaatgttt cgactctgtt 1740
gaaatcagcg gagtggagga tcgcttcaac gcatccctgg gaacgtatca cgatctcctg 1800
aaaatcatta aagacaagga cttcctggac aatgaggaga acgaggacat tcttgaggac 1860
attgtcctca cccttacgtt gtttgaagat agggagatga ttgaagaacg cttgaaaact 1920
tacgctcatc tcttcgacga caaagtcatg aaacagctca agaggcgccg atatacagga 1980
tgggggcggc tgtcaagaaa actgatcaat gggatccgag acaagcagag tggaaagaca 2040
atcctggatt ttcttaagtc cgatggattt gccaaccgga acttcatgca gttgatccat 2100
gatgactctc tcacctttaa ggaggacatc cagaaagcac aagtttctgg ccagggggac 2160
agtcttcacg agcacatcgc taatcttgca ggtagcccag ctatcaaaaa gggaatactg 2220
cagaccgtta aggtcgtgga tgaactcgtc aaagtaatgg gaaggcataa gcccgagaat 2280
atcgttatcg agatggcccg agagaaccaa actacccaga agggacagaa gaacagtagg 2340
gaaaggatga agaggattga agagggtata aaagaactgg ggtcccaaat ccttaaggaa 2400
cacccagttg aaaacaccca gcttcagaat gagaagctct acctgtacta cctgcagaac 2460
ggcagggaca tgtacgtgga tcaggaactg gacatcaatc ggctctccga ctacgacgtg 2520
gctgctatcg tgccccagtc ttttctcaaa gatgattcta ttgataataa agtgttgaca 2580
agatccgata aagctagagg gaagagtgat aacgtcccct cagaagaagt tgtcaagaaa 2640
atgaaaaatt attggcggca gctgctgaac gccaaactga tcacacaacg gaagttcgat 2700
aatctgacta aggctgaacg aggtggcctg tctgagttgg ataaagccgg cttcatcaaa 2760
aggcagcttg ttgagacacg ccagatcacc aagcacgtgg cccaaattct cgattcacgc 2820
atgaacacca agtacgatga aaatgacaaa ctgattcgag aggtgaaagt tattactctg 2880
aagtctaagc tggtctcaga tttcagaaag gactttcagt tttataaggt gagagagatc 2940
aacaattacc accatgcgca tgatgcctac ctgaatgcag tggtaggcac tgcacttatc 3000
aaaaaatatc ccaagcttga atctgaattt gtttacggag actataaagt gtacgatgtt 3060
aggaaaatga tcgcaaagtc tgagcaggaa ataggcaagg ccaccgctaa gtacttcttt 3120
tacagcaata ttatgaattt tttcaagacc gagattacac tggccaatgg agagattcgg 3180
aagcgaccac ttatcgaaac aaacggagaa acaggagaaa tcgtgtggga caagggtagg 3240
gatttcgcga cagtccggaa ggtcctgtcc atgccgcagg tgaacatcgt taaaaagacc 3300
gaagtacaga ccggaggctt ctccaaggaa agtatcctcc cgaaaaggaa cagcgacaag 3360
ctgatcgcac gcaaaaaaga ttgggacccc aagaaatacg gcggattcga ttctcctaca 3420
gtcgcttaca gtgtactggt tgtggccaaa gtggagaaag ggaagtctaa aaaactcaaa 3480
agcgtcaagg aactgctggg catcacaatc atggagcgat caagcttcga aaaaaacccc 3540
atcgactttc tcgaggcgaa aggatataaa gaggtcaaaa aagacctcat cattaagctt 3600
cccaagtact ctctctttga gcttgaaaac ggccggaaac gaatgctcgc tagtgcgggc 3660
gagctgcaga aaggtaacga gctggcactg ccctctaaat acgttaattt cttgtatctg 3720
gccagccact atgaaaagct caaagggtct cccgaagata atgagcagaa gcagctgttc 3780
gtggaacaac acaaacacta ccttgatgag atcatcgagc aaataagcga attctccaaa 3840
agagtgatcc tcgccgacgc taacctcgat aaggtgcttt ctgcttacaa taagcacagg 3900
gataagccca tcagggagca ggcagaaaac attatccact tgtttactct gaccaacttg 3960
ggcgcgcctg cagccttcaa gtacttcgac accaccatag acagaaagcg gtacacctct 4020
acaaaggagg tcctggacgc cacactgatt catcagtcaa ttacggggct ctatgaaaca 4080
agaatcgacc tctctcagct cggtggagac agcagggctg accccaagaa gaagaggaag 4140
gtggaggcca gcggttccgg acgggctgac gcattggacg attttgatct ggatatgctg 4200
ggaagtgacg ccctcgatga ttttgacctt gacatgcttg gttcggatgc ccttgatgac 4260
tttgacctcg acatgctcgg cagtgacgcc cttgatgatt tcgacctgga catgctgatt 4320
aactctagag cggccgcaga tccaaaaaag aagagaaagg tagatccaaa aaagaagaga 4380
aaggtagatc caaaaaagaa gagaaaggta gatacggccg catag 4425
<210> 5
<211> 587
<212> DNA
<213> Artificial
<220>
<223> MS2-activator construct
<400> 5
ccaccatggg acctaagaaa aagaggaagg tggcggccgc ttctagaatg gcttctaact 60
ttactcagtt cgttctcgtc gacaatggcg gaactggcga cgtgactgtc gccccaagca 120
acttcgctaa cgggatcgct gaatggatca gctctaactc gcgttcacag gcttacaaag 180
taacctgtag cgttcgtcag agctctgcgc agaatcgcaa atacaccatc aaagtcgagg 240
tgcctaaagg cgcctggcgt tcgtacttaa atatggaact aaccattcca attttcgcca 300
cgaattccga ctgcgagctt attgttaagg caatgcaagg tctcctaaaa gatggaaacc 360
cgattccctc agcaatcgca gcaaactccg gcatctacga ggccagcggt tccggacggg 420
ctgacgcatt ggacgatttt gatctggata tgctgggaag tgacgccctc gatgattttg 480
accttgacat gcttggttcg gatgcccttg atgactttga cctcgacatg ctcggcagtg 540
acgcccttga tgatttcgac ctggacatgc tgattaactc tagatga 587
<210> 6
<211> 681
<212> DNA
<213> Artificial
<220>
<223> MS2-activator construct
<400> 6
gccaccatgg gacctaagaa aaagaggaag gtggcggccg cttctagaat ggcttctaac 60
tttactcagt tcgttctcgt cgacaatggc ggaactggcg acgtgactgt cgccccaagc 120
aacttcgcta acgggatcgc tgaatggatc agctctaact cgcgttcaca ggcttacaaa 180
gtaacctgta gcgttcgtca gagctctgcg cagaatcgca aatacaccat caaagtcgag 240
gtgcctaaag gcgcctggcg ttcgtactta aatatggaac taaccattcc aattttcgcc 300
acgaattccg actgcgagct tattgttaag gcaatgcaag gtctcctaaa agatggaaac 360
ccgattccct cagcaatcgc agcaaactcc ggcatctacg aggccagcgg ttccggacgg 420
gctgacgcat tggacgattt tgatctggat atgctgggaa gtgacgccct cgatgatttt 480
gaccttgaca tgcttggttc ggatgccctt gatgactttg acctcgacat gctcggcagt 540
gacgcccttg atgatttcga cctggacatg ctgattaact ctagagcggc cgcagatcca 600
aaaaagaaga gaaaggtaga tccaaaaaag aagagaaagg tagatccaaa aaagaagaga 660
aaggtagata cggccgcata g 681
<210> 7
<211> 557
<212> DNA
<213> Artificial
<220>
<223> MS2-activator construct
<220>
<221> misc_feature
<222> (320)..(339)
<223> wherein N is G, A, T or C
<400> 7
tgtacaaaaa agcaggcttt aaaggaacca attcagtcga ctggatccgg taccaaggtc 60
gggcaggaag agggcctatt tcccatgatt ccttcatatt tgcatatacg atacaaggct 120
gttagagaga taattagaat taatttgact gtaaacacaa agatattagt acaaaatacg 180
tgacgtagaa agtaataatt tcttgggtag tttgcagttt taaaattatg ttttaaaatg 240
gactatcata tgcttaccgt aacttgaaag tatttcgatt tcttggcttt atatatcttg 300
tggaaaggac gaaacaccgn nnnnnnnnnn nnnnnnnnng ttttagagct agaaatagca 360
agttaaaata aggctagtcc gttatcaact tgaaaaagtg gcaccgagtc ggtgctctgc 420
aggtcgactc tagaaaacat gaggatcacc catgtctgca gtattcccgg gttcattaga 480
tcctaaggta cctaattgcc tagaaaacat gaggatcacc catgtctgca ggtcgactct 540
agaaattttt tctagac 557
<210> 8
<211> 882
<212> DNA
<213> Artificial
<220>
<223> Activation reporter construct
<400> 8
tagggataac agggtaatag tgtcccctcc accccacagt ggggcgaggt aggcgtgtac 60
ggtgggaggc ctatataagc agagctcgtt tagtgaaccg tcagatcgcc tggagaattc 120
gccaccatgg actacaagga tgacgacgat aaaacttccg gtggcggact gggttccacc 180
gtgagcaagg gcgaggaggt catcaaagag ttcatgcgct tcaaggtgcg catggagggc 240
tccatgaacg gccacgagtt cgagatcgag ggcgagggcg agggccgccc ctacgagggc 300
acccagaccg ccaagctgaa ggtgaccaag ggcggccccc tgcccttcgc ctgggacatc 360
ctgtcccccc agttcatgta cggctccaag gcgtacgtga agcaccccgc cgacatcccc 420
gattacaaga agctgtcctt ccccgagggc ttcaagtggg agcgcgtgat gaacttcgag 480
gacggcggtc tggtgaccgt gacccaggac tcctccctgc aggacggcac gctgatctac 540
aaggtgaaga tgcgcggcac caacttcccc cccgacggcc ccgtaatgca gaagaagacc 600
atgggctggg aggcctccac cgagcgcctg tacccccgcg acggcgtgct gaagggcgag 660
atccaccagg ccctgaagct gaaggacggc ggccactacc tggtggagtt caagaccatc 720
tacatggcca agaagcccgt gcaactgccc ggctactact acgtggacac caagctggac 780
atcacctccc acaacgagga ctacaccatc gtggaacagt acgagcgctc cgagggccgc 840
caccacctgt tcctgtacgg catggacgag ctgtacaagt aa 882
<210> 9
<211> 882
<212> DNA
<213> Artificial
<220>
<223> Activation reporter construct
<400> 9
tagggataac agggtaatag tggggccact agggacagga ttggcgaggt aggcgtgtac 60
ggtgggaggc ctatataagc agagctcgtt tagtgaaccg tcagatcgcc tggagaattc 120
gccaccatgg actacaagga tgacgacgat aaaacttccg gtggcggact gggttccacc 180
gtgagcaagg gcgaggaggt catcaaagag ttcatgcgct tcaaggtgcg catggagggc 240
tccatgaacg gccacgagtt cgagatcgag ggcgagggcg agggccgccc ctacgagggc 300
acccagaccg ccaagctgaa ggtgaccaag ggcggccccc tgcccttcgc ctgggacatc 360
ctgtcccccc agttcatgta cggctccaag gcgtacgtga agcaccccgc cgacatcccc 420
gattacaaga agctgtcctt ccccgagggc ttcaagtggg agcgcgtgat gaacttcgag 480
gacggcggtc tggtgaccgt gacccaggac tcctccctgc aggacggcac gctgatctac 540
aaggtgaaga tgcgcggcac caacttcccc cccgacggcc ccgtaatgca gaagaagacc 600
atgggctggg aggcctccac cgagcgcctg tacccccgcg acggcgtgct gaagggcgag 660
atccaccagg ccctgaagct gaaggacggc ggccactacc tggtggagtt caagaccatc 720
tacatggcca agaagcccgt gcaactgccc ggctactact acgtggacac caagctggac 780
atcacctccc acaacgagga ctacaccatc gtggaacagt acgagcgctc cgagggccgc 840
caccacctgt tcctgtacgg catggacgag ctgtacaagt aa 882
<210> 10
<211> 912
<212> DNA
<213> Artificial
<220>
<223> Specificity reporter library
<220>
<221> misc_feature
<222> (22)..(44)
<223> wherein N is G, A, T or C
<220>
<221> misc_feature
<222> (154)..(177)
<223> wherein N is G, A, T or C
<400> 10
tagggataac agggtaatag tnnnnnnnnn nnnnnnnnnn nnnncgaggt aggcgtgtac 60
ggtgggaggc ctatataagc agagctcgtt tagtgaaccg tcagatcgcc tggagaattc 120
gccaccatgg actacaagga tgacgacgat aaannnnnnn nnnnnnnnnn nnnnnnnact 180
tccggtggcg gactgggttc caccgtgagc aagggcgagg aggtcatcaa agagttcatg 240
cgcttcaagg tgcgcatgga gggctccatg aacggccacg agttcgagat cgagggcgag 300
ggcgagggcc gcccctacga gggcacccag accgccaagc tgaaggtgac caagggcggc 360
cccctgccct tcgcctggga catcctgtcc ccccagttca tgtacggctc caaggcgtac 420
gtgaagcacc ccgccgacat ccccgattac aagaagctgt ccttccccga gggcttcaag 480
tgggagcgcg tgatgaactt cgaggacggc ggtctggtga ccgtgaccca ggactcctcc 540
ctgcaggacg gcacgctgat ctacaaggtg aagatgcgcg gcaccaactt cccccccgac 600
ggccccgtaa tgcagaagaa gaccatgggc tgggaggcct ccaccgagcg cctgtacccc 660
cgcgacggcg tgctgaaggg cgagatccac caggccctga agctgaagga cggcggccac 720
tacctggtgg agttcaagac catctacatg gccaagaagc ccgtgcaact gcccggctac 780
tactacgtgg acaccaagct ggacatcacc tcccacaacg aggactacac catcgtggaa 840
cagtacgagc gctccgaggg ccgccaccac ctgttcctgt acggcatgga cgagctgtac 900
aagtaagaat tc 912
<210> 11
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 11
ctggcggatc actcgcggtt agg 23
<210> 12
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 12
cctcggcctc caaaagtgct agg 23
<210> 13
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 13
acgctgattc ctgcagatca ggg 23
<210> 14
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 14
ccaggaatac gtatccacca ggg 23
<210> 15
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 15
gccacaccca agcgatcaaa tgg 23
<210> 16
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 16
aaataataca ttctaaggta agg 23
<210> 17
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 17
gctactgggg aggctgaggc agg 23
<210> 18
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 18
tagcaataca gtcacattaa tgg 23
<210> 19
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 19
ctcatgtgat ccccccgtct cgg 23
<210> 20
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 20
ccgggcagag agtgaacgcg cgg 23
<210> 21
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 21
ttccttccct ctcccgtgct tgg 23
<210> 22
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 22
tctctgcaaa gcccctggag agg 23
<210> 23
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 23
aatgcagttg ccgagtgcag tgg 23
<210> 24
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 24
cctcagcctc ctaaagtgct ggg 23
<210> 25
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 25
gagtccaaat cctctttact agg 23
<210> 26
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 26
gagtgtctgg atttgggata agg 23
<210> 27
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 27
cagcacctca tctcccagtg agg 23
<210> 28
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 28
tctaaaaccc agggaatcat ggg 23
<210> 29
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 29
cacaaggcag ccagggatcc agg 23
<210> 30
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 30
gatggcaagc tgagaaacac tgg 23
<210> 31
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 31
tgaaatgcac gcatacaatt agg 23
<210> 32
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 32
ccagtccaga cctggccttc tgg 23
<210> 33
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 33
cccagaaaaa cagaccctga agg 23
<210> 34
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 34
aagggttgag cacttgttta ggg 23
<210> 35
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 35
atgtctgagt tttggttgag agg 23
<210> 36
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 36
ggtcccttga aggggaagta ggg 23
<210> 37
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 37
tggcagtcta ctcttgaaga tgg 23
<210> 38
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 38
ggcacagtgc cagaggtctg tgg 23
<210> 39
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 39
taaaaataaa aaaactaaca ggg 23
<210> 40
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 40
tctgtggggg acctgcactg agg 23
<210> 41
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 41
ggccagaggt caaggctagt ggg 23
<210> 42
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 42
cacgaccgaa acccttctta cgg 23
<210> 43
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 43
gttgaatgaa gacagtctag tgg 23
<210> 44
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 44
taagaacaga gcaagttacg tgg 23
<210> 45
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 45
tgtaaggtaa gagaggagag cgg 23
<210> 46
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 46
tgacacacca actcctgcac tgg 23
<210> 47
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 47
tttacccact tccttcgaaa agg 23
<210> 48
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 48
gtggctggca ggctggctct ggg 23
<210> 49
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 49
ctcccccggc ctcccccgcg cgg 23
<210> 50
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 50
caaaacccgg cagcgaggct ggg 23
<210> 51
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 51
aggagccgcc gcgcgctgat tgg 23
<210> 52
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 52
cacacacacc cacacgagat ggg 23
<210> 53
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 53
gaagaagcta aagagccaga ggg 23
<210> 54
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 54
atgagaattt caataacctc agg 23
<210> 55
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 55
tcccgctctg ttgcccaggc tgg 23
<210> 56
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 56
cagacaccca ccaccatgcg tgg 23
<210> 57
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 57
tcccaattta ctgggattac agg 23
<210> 58
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 58
tgatttaaaa gttggaaacg tgg 23
<210> 59
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 59
tctagttccc cacctagtct ggg 23
<210> 60
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 60
gattaactga gaattcacaa ggg 23
<210> 61
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target probe
<400> 61
cgccaggagg ggtgggtcta agg 23
<210> 62
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Reporter construct
<400> 62
gtcccctcca ccccacagtg ggg 23
<210> 63
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Reporter construct
<400> 63
ggggccacta gggacaggat tgg 23
<210> 64
<211> 71
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 64
taatactttt atctgtcccc tccaccccac agtggggcca ctagggacag gattggtgac 60
agaaaagccc c 71
<210> 65
<211> 20
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 65
ggggccacta gggacaggat 20
<210> 66
<211> 80
<212> RNA
<213> Artificial
<220>
<223> Guide RNA
<400> 66
guuuuagagc uagaaauagc aaguuaaaau aaggcuagcu uguuaucaac uugaaaaagu 60
ggcaccgagu cggugcuuuu 80
<210> 67
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 67
gtcccctcca ccccacagtg cag 23
<210> 68
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 68
gtcccctcca ccccacagtg caa 23
<210> 69
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 69
gtcccctcca ccccacagtg cgg 23
<210> 70
<211> 52
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 70
tgtcccctcc accccacagt ggggccacta gggacaggat tggtgacaga aa 52
<210> 71
<211> 52
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 71
tgtccccccc accccacagt ggggccacta gggacaggat tggtgacaga aa 52
<210> 72
<211> 52
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 72
aaaaccctcc accccacagt ggggccacta gggacaggat tggtgacaga aa 52
<210> 73
<211> 52
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 73
tgtcccctcc ttttttcagt ggggccacta gggacaggat tggtgacaga aa 52
<210> 74
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 74
caccggggtg gtgcccatcc tgg 23
<210> 75
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 75
ggtgcccatc ctggtcgagc tgg 23
<210> 76
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 76
cccatcctgg tcgagctgga cgg 23
<210> 77
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 77
ggccacaagt tcagcgtgtc cgg 23
<210> 78
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 78
cgcaaataag agctcaccta cgg 23
<210> 79
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 79
ctgaagttca tctgcaccac cgg 23
<210> 80
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 80
ccggcaagct gcccgtgccc tgg 23
<210> 81
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 81
gaccaggatg ggcaccaccc cgg 23
<210> 82
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 82
gccgtccagc tcgaccagga tgg 23
<210> 83
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 83
ggccggacac gctgaacttg tgg 23
<210> 84
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 84
taacagggta atgtcgaggc cgg 23
<210> 85
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 85
aggtgagctc ttatttgcgt agg 23
<210> 86
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 86
cttcagggtc agcttgccgt agg 23
<210> 87
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 87
gggcacgggc agcttgccgg tgg 23
<210> 88
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 88
gagatgatcg ccccttcttc tgg 23
<210> 89
<211> 20
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 89
gagatgatcg ccccttcttc 20
<210> 90
<211> 20
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 90
gtgatgaccg gccgttcttc 20
<210> 91
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 91
gtcccctcca ccccacagtg ggg 23
<210> 92
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 92
gagatgatcg cccgttcttc tgg 23
<210> 93
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 93
guccccucca ccccacagug 20
<210> 94
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 94
guccccucca ccccacaguc 20
<210> 95
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 95
guccccucca ccccacagag 20
<210> 96
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 96
guccccucca ccccacacug 20
<210> 97
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 97
guccccucca ccccacugug 20
<210> 98
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 98
guccccucca ccccagagug 20
<210> 99
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 99
guccccucca ccccucagug 20
<210> 100
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 100
guccccucca cccgacagug 20
<210> 101
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 101
guccccucca ccgcacagug 20
<210> 102
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 102
guccccucca cgccacagug 20
<210> 103
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 103
guccccucca gcccacagug 20
<210> 104
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 104
guccccuccu ccccacagug 20
<210> 105
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 105
guccccucga ccccacagug 20
<210> 106
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 106
guccccucca ccccacagac 20
<210> 107
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 107
guccccucca ccccacucug 20
<210> 108
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 108
guccccucca ccccugagug 20
<210> 109
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 109
guccccucca ccggacagug 20
<210> 110
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 110
guccccucca ggccacagug 20
<210> 111
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 111
guccccucgu ccccacagug 20
<210> 112
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 112
ggggccacta gggacaggat ggg 23
<210> 113
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 113
gagaugaucg ccccuucuuc 20
<210> 114
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 114
gagaugaucg ccccuucuug 20
<210> 115
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 115
gagaugaucg ccccuucuac 20
<210> 116
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 116
gagaugaucg ccccuucauc 20
<210> 117
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 117
gagaugaucg ccccuuguuc 20
<210> 118
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 118
gagaugaucg ccccuacuuc 20
<210> 119
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 119
gagaugaucg ccccaucuuc 20
<210> 120
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 120
gagaugaucg cccguucuuc 20
<210> 121
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 121
gagaugaucg ccgcuucuuc 20
<210> 122
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 122
gagaugaucg cgccuucuuc 20
<210> 123
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 123
gagaugaucg gcccuucuuc 20
<210> 124
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 124
gagaugaucc ccccuucuuc 20
<210> 125
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 125
gagaugaugg ccccuucuuc 20
<210> 126
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 126
gagaugaucg ccccuucuag 20
<210> 127
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 127
gagaugaucg ccccuugauc 20
<210> 128
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 128
gagaugaucg ccccaacuuc 20
<210> 129
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 129
gagaugaucg ccgguucuuc 20
<210> 130
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 130
gagaugaucg ggccuucuuc 20
<210> 131
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 131
gagaugaugc ccccuucuuc 20
<210> 132
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 132
gagatgatcg ccccttcttc tgg 23
<210> 133
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 133
ggggccacua gggacaggau 20
<210> 134
<211> 19
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 134
gggccacuag ggacaggau 19
<210> 135
<211> 18
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 135
ggccacuagg gacaggau 18
<210> 136
<211> 17
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 136
gccacuaggg acaggau 17
<210> 137
<211> 20
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 137
gagaugaucg ccccuucuuc 20
<210> 138
<211> 18
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 138
gaugaucgcc ccuucuuc 18
<210> 139
<211> 15
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 139
gaucgccccu ucuuc 15
<210> 140
<211> 11
<212> RNA
<213> Artificial
<220>
<223> RNA target sequence
<400> 140
gccccuucuu c 11
<210> 141
<211> 21
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 141
gtcccctcca ccccacagtg c 21
<210> 142
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<220>
<221> misc_feature
<222> (5)..(10)
<223> wherein N id G, A, T or C
<400> 142
tgtcnnnnnn accc 14
<210> 143
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 143
tgtcaaaaaa accc 14
<210> 144
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 144
tgtcgggggg accc 14
<210> 145
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 145
tgtcaaaaaa accc 14
<210> 146
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 146
tgtcgggggg accc 14
<210> 147
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 147
tgtccccccc accc 14
<210> 148
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 148
tgtctttttt accc 14
<210> 149
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 149
tgtccccccc accc 14
<210> 150
<211> 14
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 150
tgtctttttt accc 14
<210> 151
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 151
ggatcctgtg tccccgagct ggg 23
<210> 152
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 152
gttaatgtgg ctctggttct ggg 23
<210> 153
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 153
ggggccacta gggacaggat tgg 23
<210> 154
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 154
cttcctagtc tcctgatatt ggg 23
<210> 155
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 155
tggtcccagc tcggggacac agg 23
<210> 156
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 156
agaaccagag ccacattaac cgg 23
<210> 157
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 157
gtcaccaatc ctgtccctag tgg 23
<210> 158
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 158
agacccaata tcaggagact agg 23
<210> 159
<211> 75
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 159
gggatcctgt gtccccgagc tgggaccacc ttatattccc agggccggtt aatgtggctc 60
tggttctggg tactt 75
<210> 160
<211> 69
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 160
gggatcctgt gtccccgagc tgggaccacc ttatattccc agggccggtt aatgtggttc 60
tgggtactt 69
<210> 161
<211> 113
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 161
gggatcctgt gtccccgagc tgggaccacc ttatattccc agggcagggc cggttggacc 60
accttatatt cccagggcag ggccggttaa tgtggctctg gttctgggta ctt 113
<210> 162
<211> 34
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 162
gggatcctgt gtccccgtct ggttctgggt actt 34
<210> 163
<211> 47
<212> DNA
<213> Artificial
<220>
<223> Target oligoncleotide sequence
<400> 163
gggatcctgt gtccccgagc tgggaccacc ttatattctg ggtactt 47
<210> 164
<211> 17
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 164
gggatcctgt ggtactt 17
<210> 165
<211> 93
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 165
agggccggtt aatgtggctc tggttctggg tacttttatc tgtcccctcc accccacagt 60
ggggccacta gggacaggat tggtgacaga aaa 93
<210> 166
<211> 83
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 166
agggccggtt aatgaatgtg gctctggttc tgggtacttt tatctgtccc ctccacccca 60
cagtggggcc actagacaga aaa 83
<210> 167
<211> 76
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 167
agggccggtt aatgtggctc tggttctggg tacttttatc tgtcccccag tggggccact 60
gattggtgac agaaaa 76
<210> 168
<211> 29
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 168
agggccggtt caggattggt gacagaaaa 29
<210> 169
<211> 34
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 169
agggccggtt aatgtggcga ttggtgacag aaaa 34
<210> 170
<211> 63
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 170
agggccggtt aatgtggctc tggttctggg tacttttatc tgtccccgat tggtgacaga 60
aaa 63
<210> 171
<211> 84
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 171
agggccggtt aatgtggctc tggttctggg tacttttatc tgtcccctcc accccacagt 60
ggggacagga ttggtgacag aaaa 84
<210> 172
<211> 27
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 172
agggccggtt aatgtggtga cagaaaa 27
<210> 173
<211> 105
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 173
agggccggtt aatgtggctc tggttctggg tacttttatc tgtcccctcc accccagggg 60
acagtctgtc ccctccaccc cagggacagg attggtgaca gaaaa 105
<210> 174
<211> 80
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 174
agggccggtt aatgtggctc tggttctggg tacttttatc tgtcccctcc accactaggg 60
acaggattgg tgacagaaaa 80
<210> 175
<211> 53
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 175
cccacagtgg ggccactagg gacaggattg gtgacagaaa agccccatac ccc 53
<210> 176
<211> 22
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 176
cccacagtgg ggccactacc cc 22
<210> 177
<211> 96
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 177
cccacagtgg ggccactagt agaaaagccc catccttagg cctcccccat ccttaggcct 60
cctccttcct agtctcctga tattgggtct aacccc 96
<210> 178
<211> 94
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 178
cccacagtgg ggccactagg gacaggattg gtgacagaaa agccccatcc ttaggcctcc 60
tccttcctag tctcctgata ttgggtctaa cccc 94
<210> 179
<211> 62
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 179
cccacagtgg ggccaccctt aggcctcctc cttcctagtc tcctgatatt gggtctaacc 60
cc 62
<210> 180
<211> 38
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 180
cccacagtgg ggccactagt gatattgggt ctaacccc 38
<210> 181
<211> 94
<212> DNA
<213> Artificial
<220>
<223> target oligonucleotide sequence
<400> 181
cccacagtgg ggccactagg gacaggattg gtgacaaaaa agccccatcc ttacgcctcc 60
tccttcctag tctcctgata ttgggtctaa cccc 94
<210> 182
<211> 65
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 182
cccacagtgg ggccactagg gacaggcctc ctccttccta gtctcctgat attgggtcta 60
acccc 65
<210> 183
<211> 102
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 183
cccacagtgg ggccactagg gacaggggga caggattggt gacagaaaag ccccatcctt 60
aggcctcctc cttcctagtc tcctgatatt gggtctaacc cc 102
<210> 184
<211> 76
<212> DNA
<213> Artificial
<220>
<223> Target oligonucleotide sequence
<400> 184
cccacaggat tggtgacaga aaagccccat ccttaggcct cctccttcct agtctcctga 60
tattgggtct aacccc 76
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ГЕНОМНАЯ ИНЖЕНЕРИЯ | 2014 |
|
RU2764757C2 |
| ГЕНОМНАЯ ИНЖЕНЕРИЯ | 2021 |
|
RU2812848C2 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМА CRISPR/CAS9 II ТИПА И ЕЕ ПРИМЕНЕНИЕ | 2022 |
|
RU2794774C1 |
| ДОСТАВКА, КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБОВ И КОМПОЗИЦИЙ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ И ПРИМЕНЕНИЯ В ТЕРАПИИ | 2013 |
|
RU2721275C2 |
| КОНСТРУИРОВАНИЕ СИСТЕМ, СПОСОБЫ И ОПТИМИЗИРОВАННЫЕ НАПРАВЛЯЮЩИЕ КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796017C2 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
| НОВЫЕ ФЕРМЕНТЫ CRISPR И СИСТЕМЫ | 2016 |
|
RU2771826C2 |
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2766685C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| МУТАЦИИ ФЕРМЕНТА CRISPR, УМЕНЬШАЮЩИЕ НЕЦЕЛЕВЫЕ ЭФФЕКТЫ | 2016 |
|
RU2752834C2 |
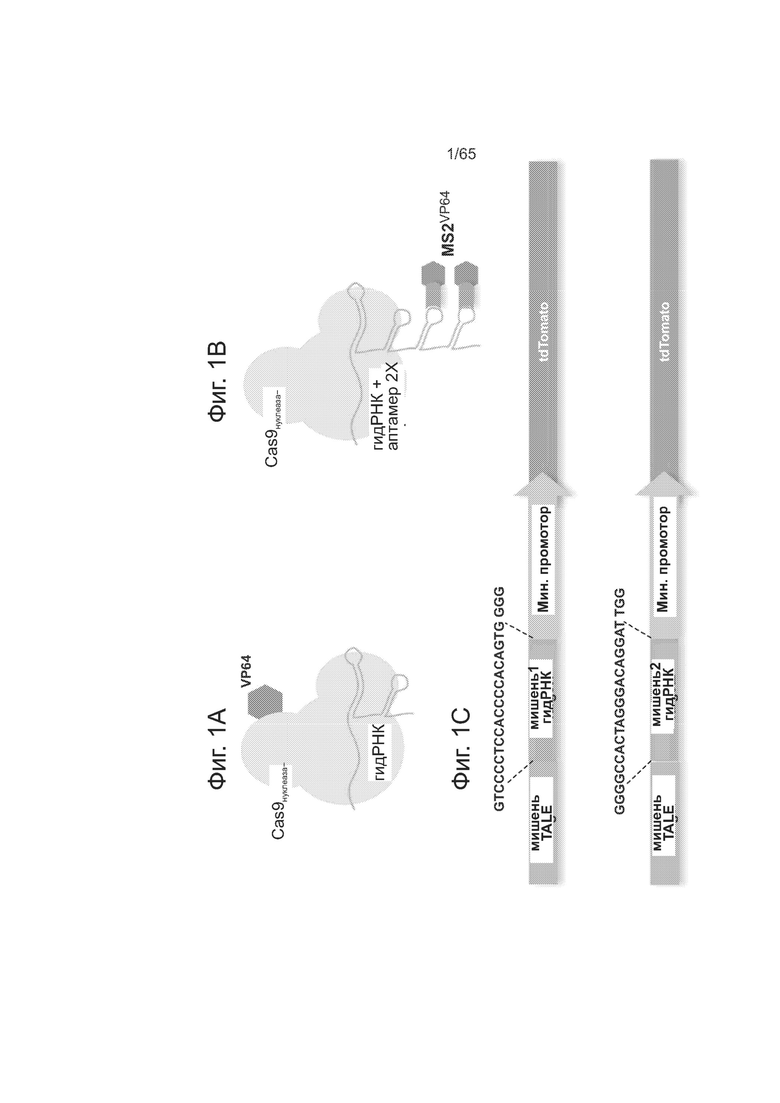
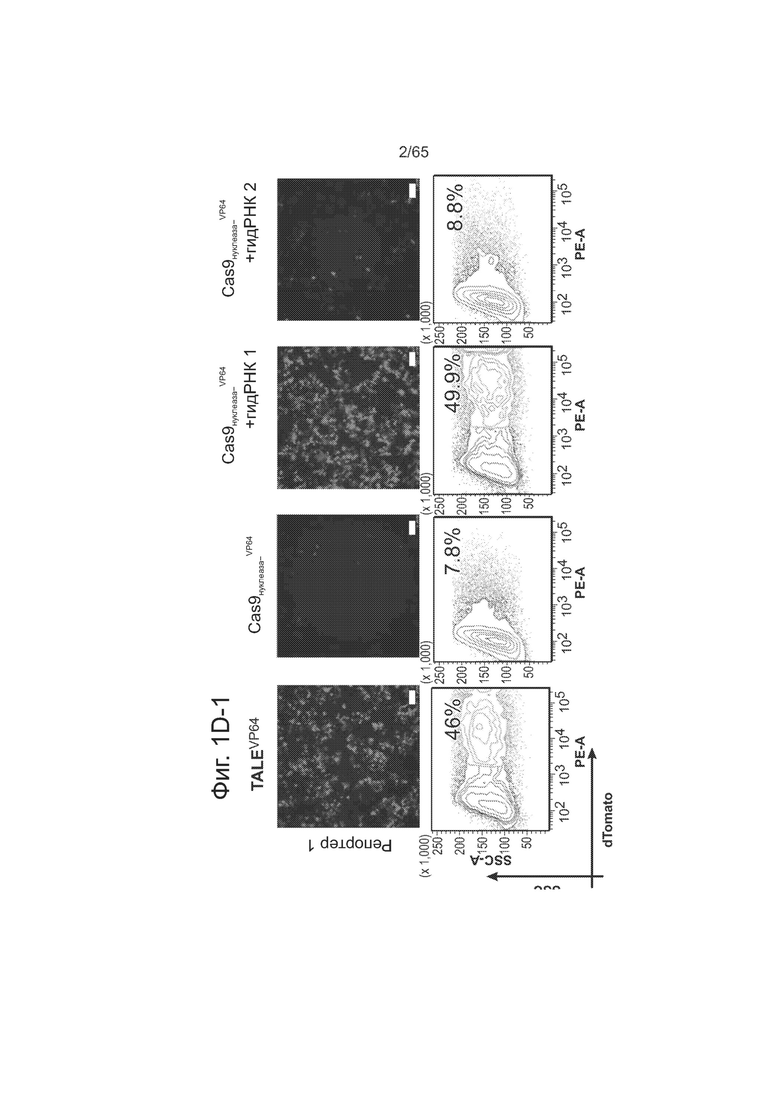
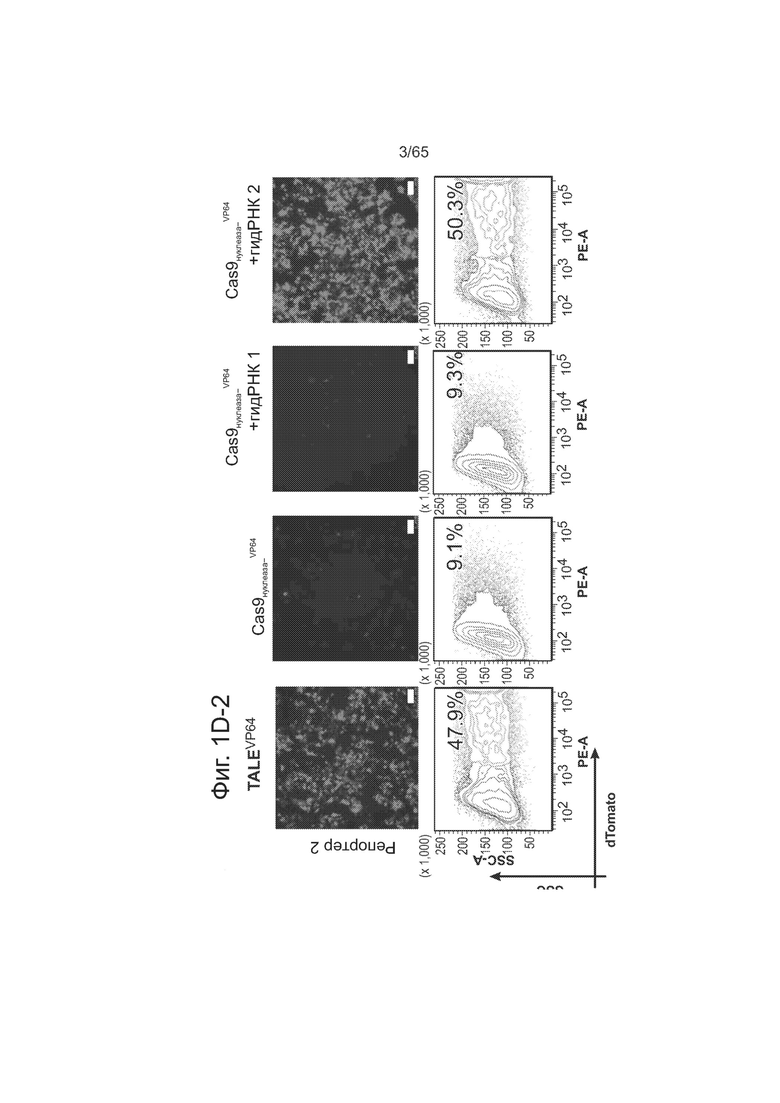
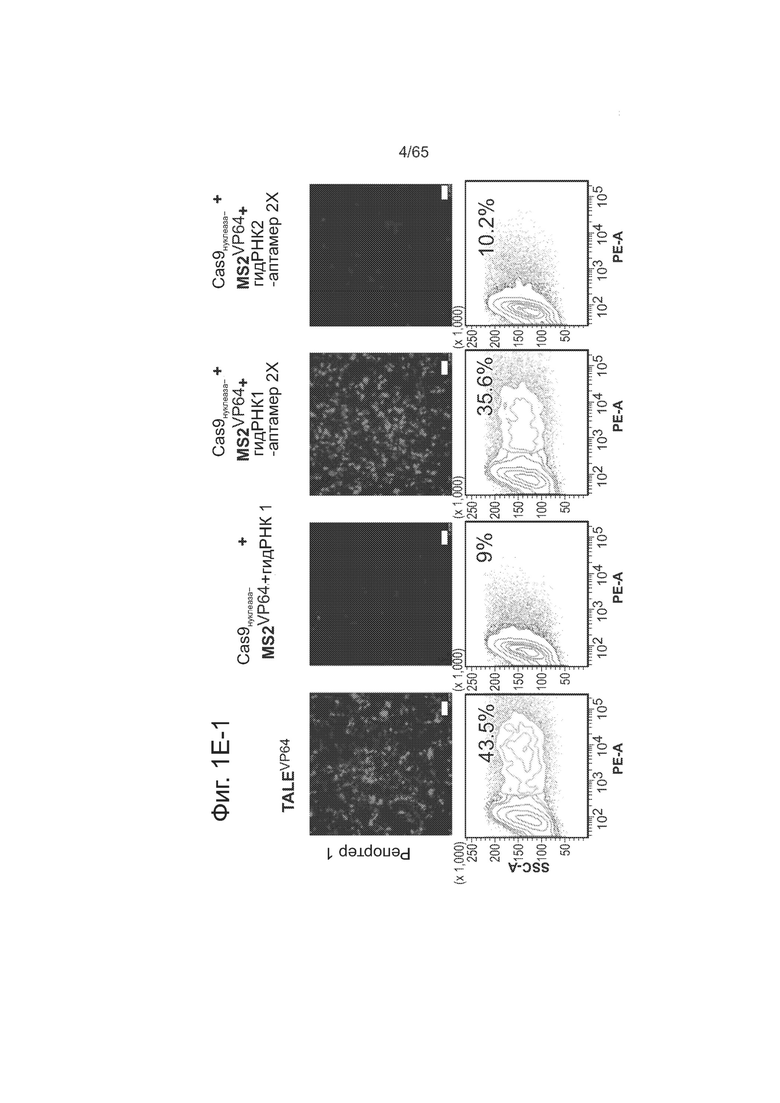
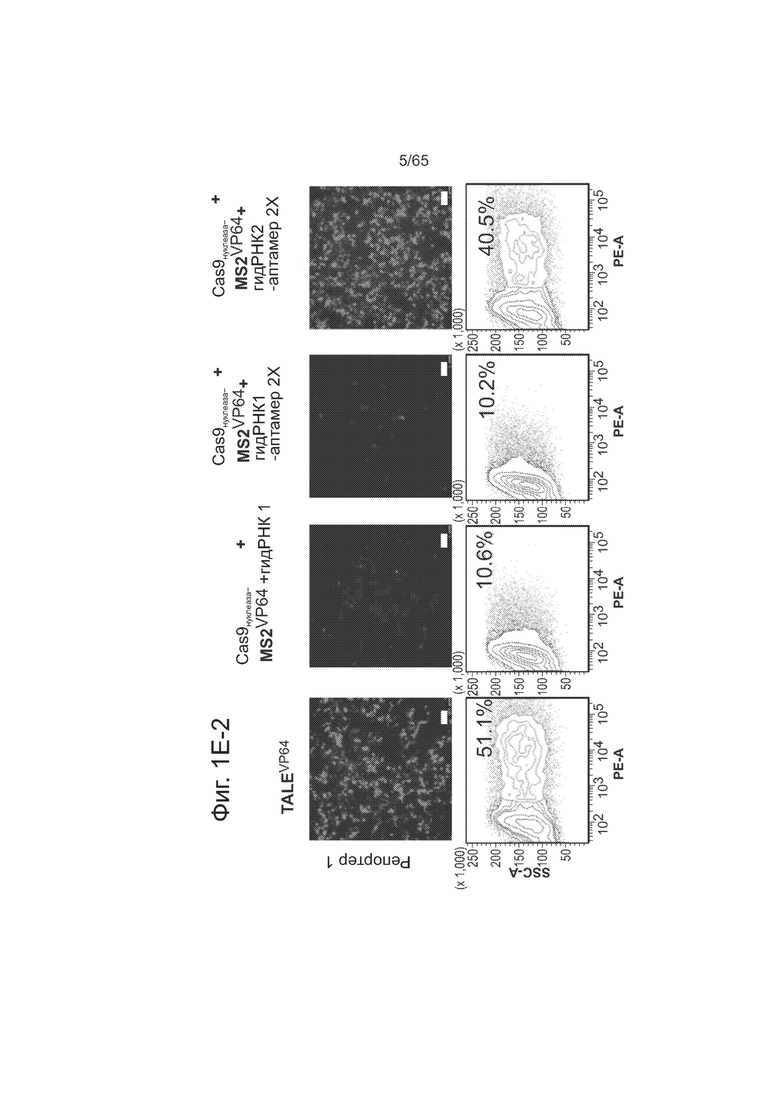


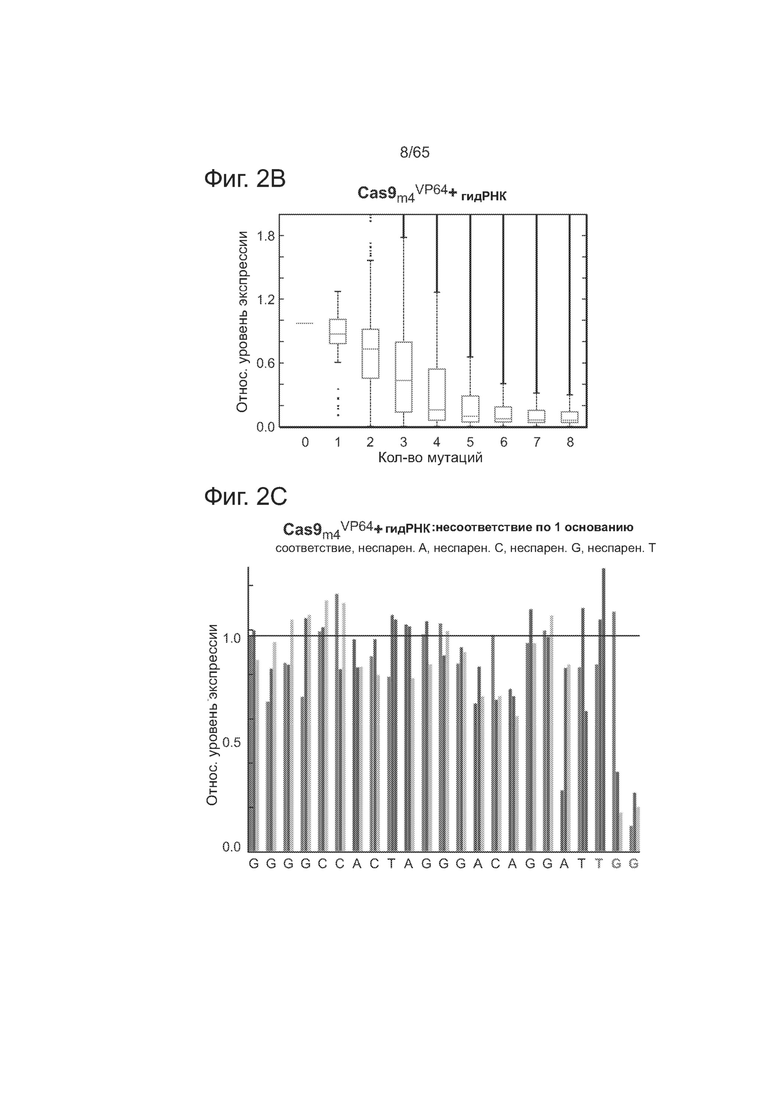
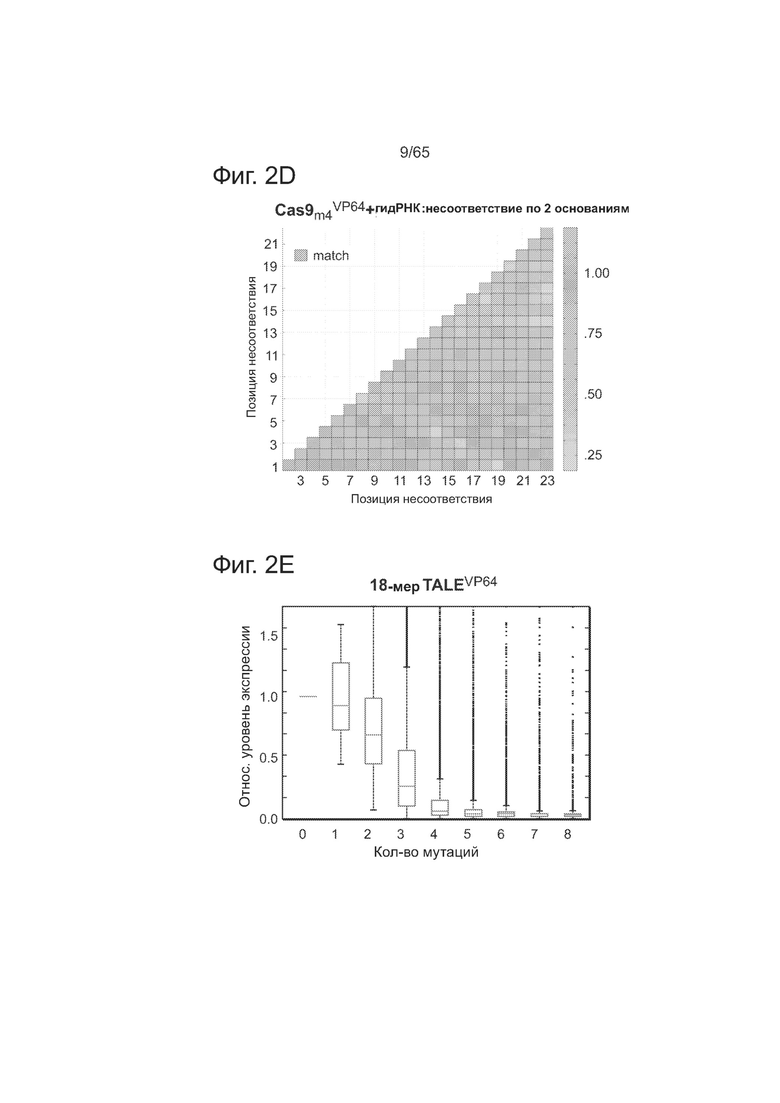
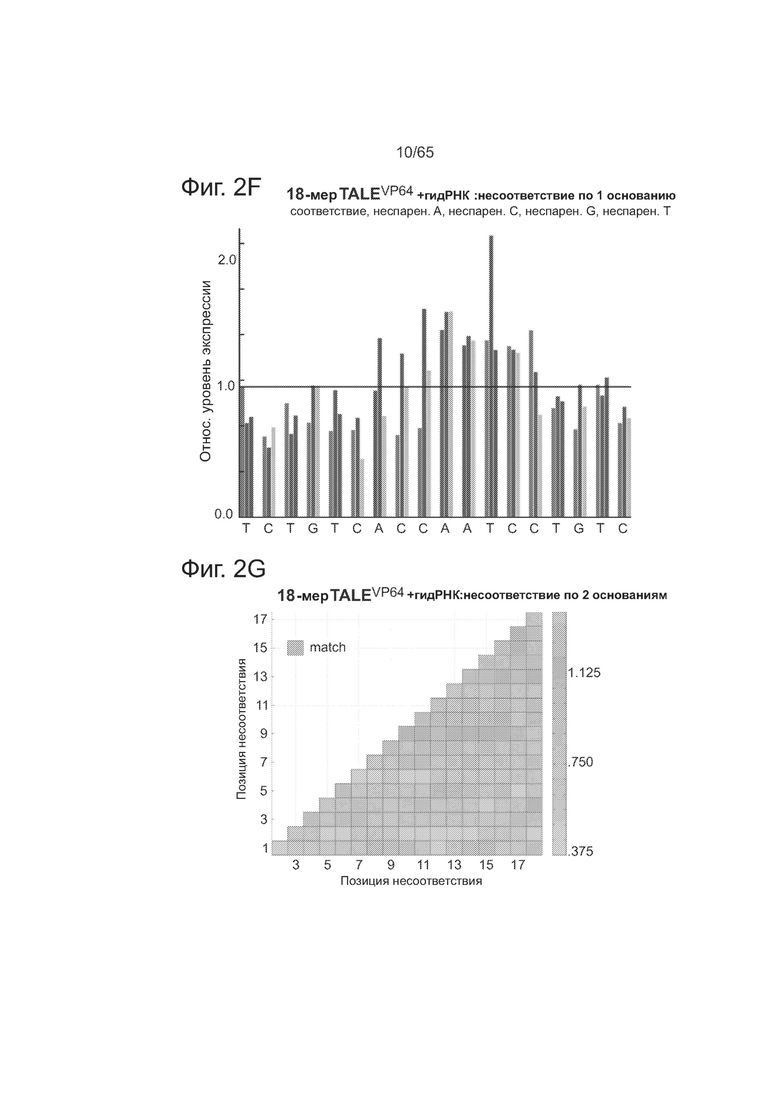
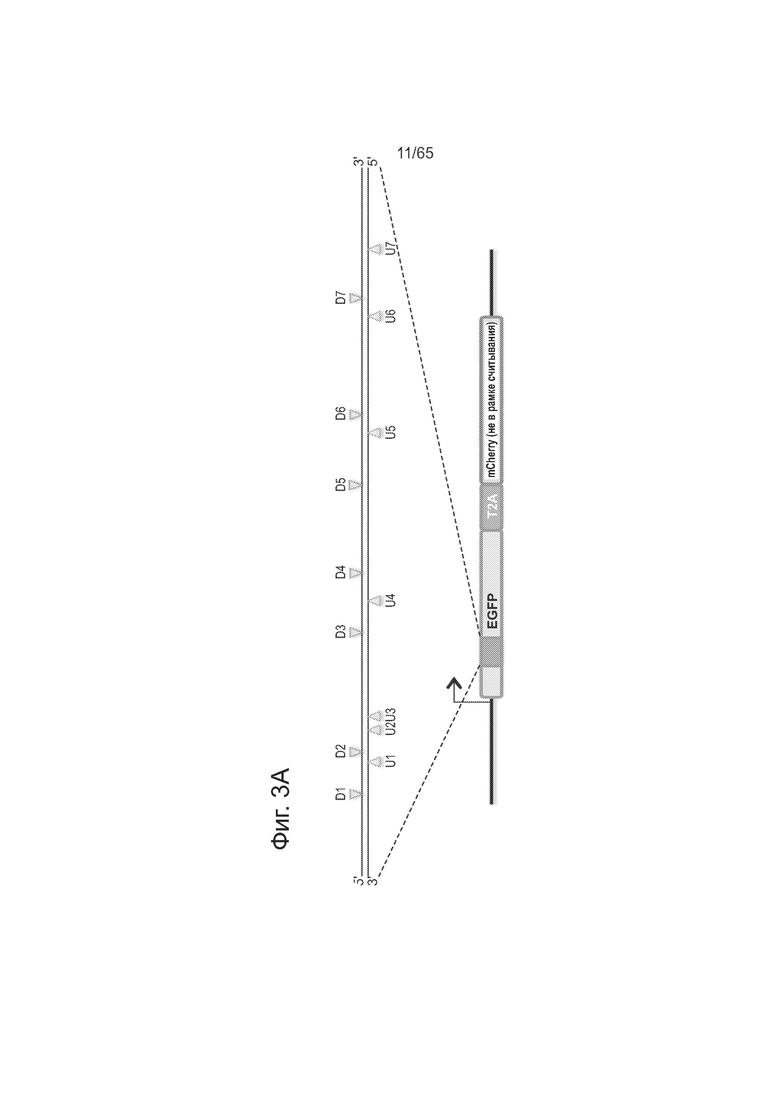

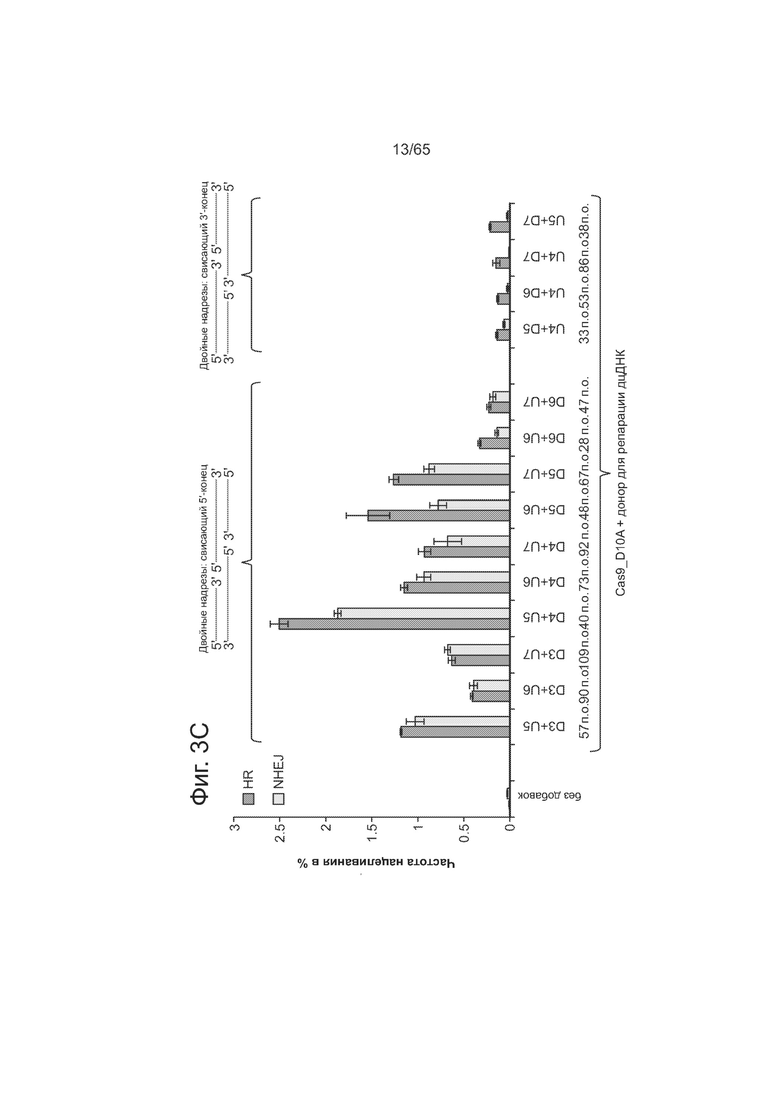

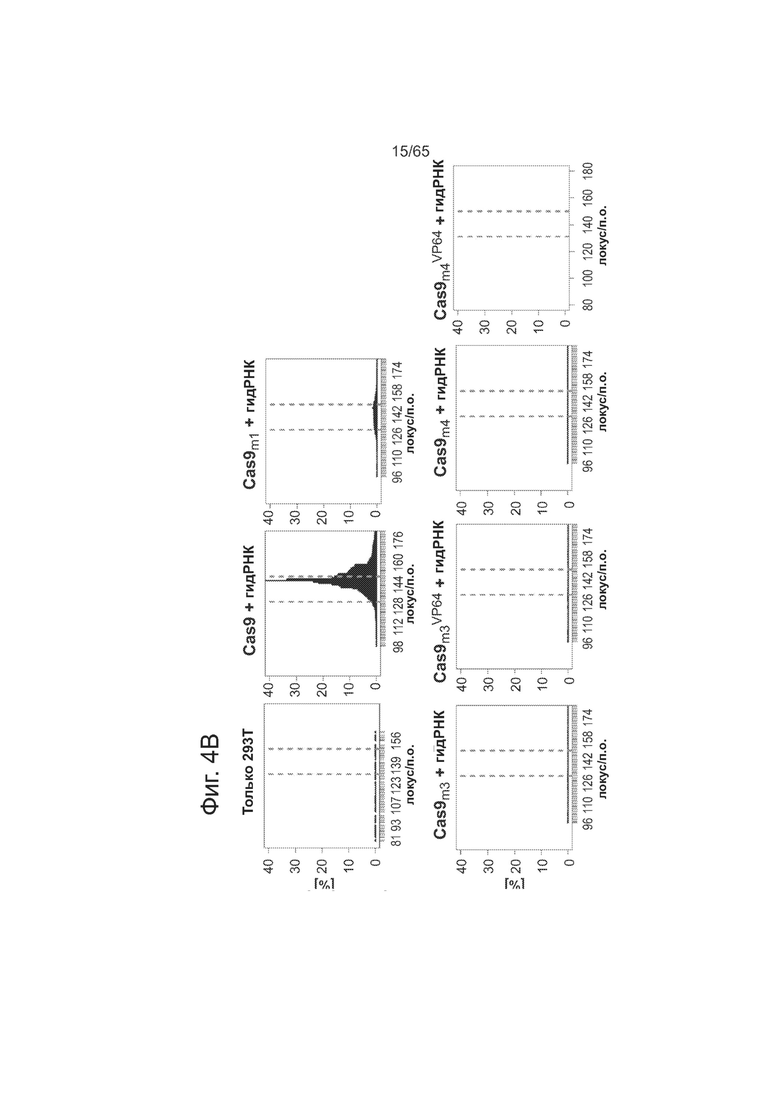
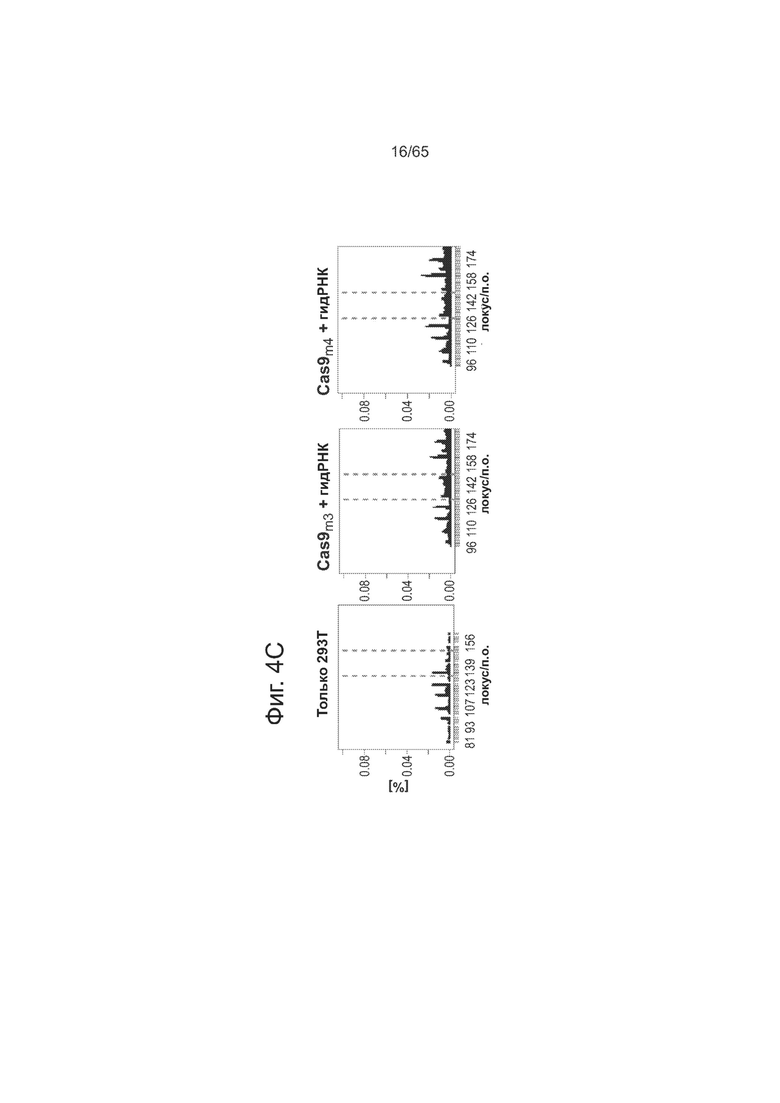
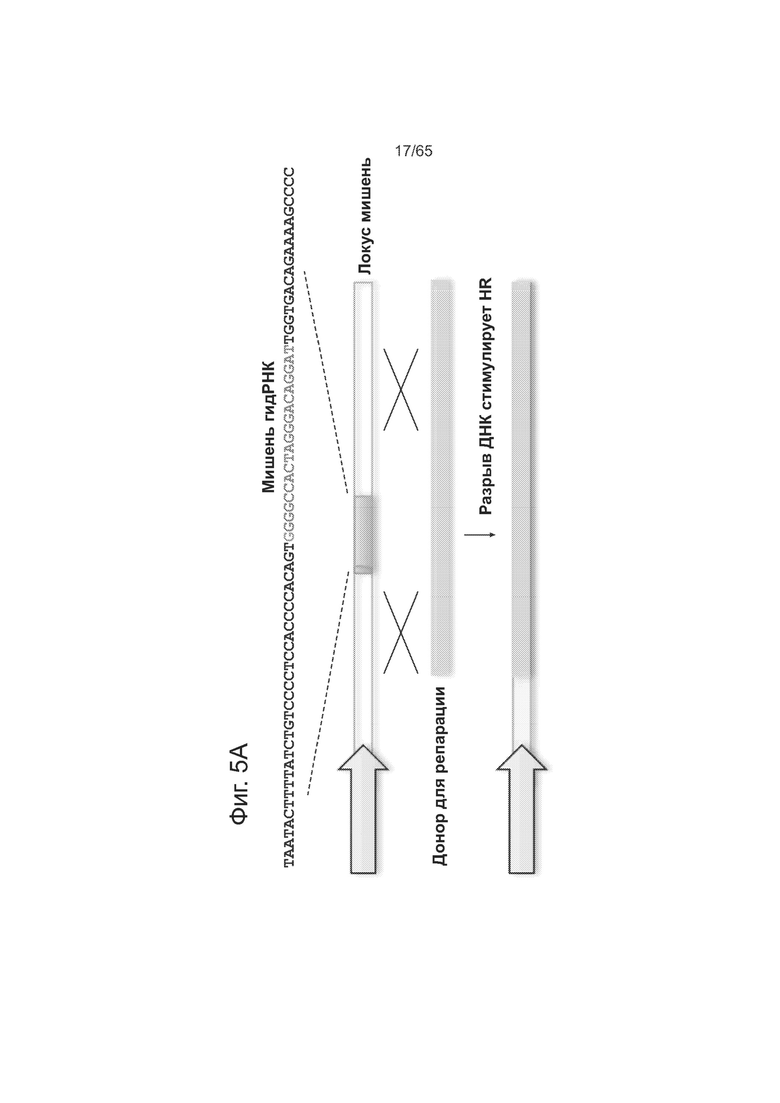
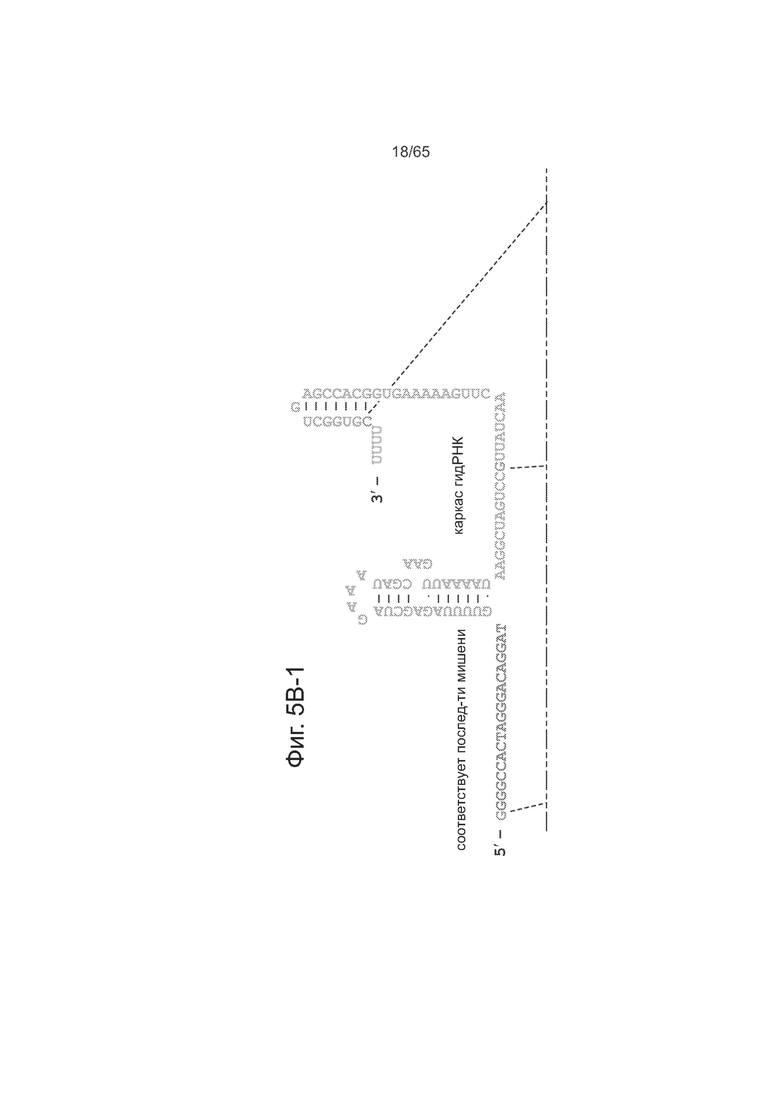
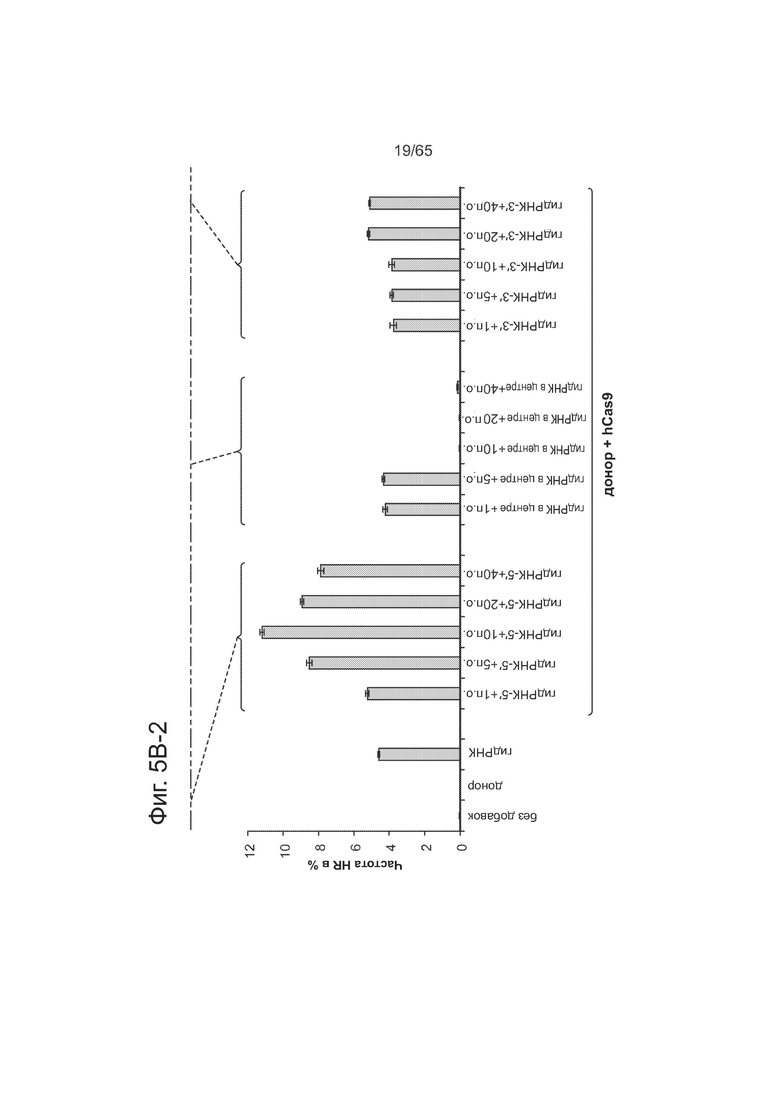

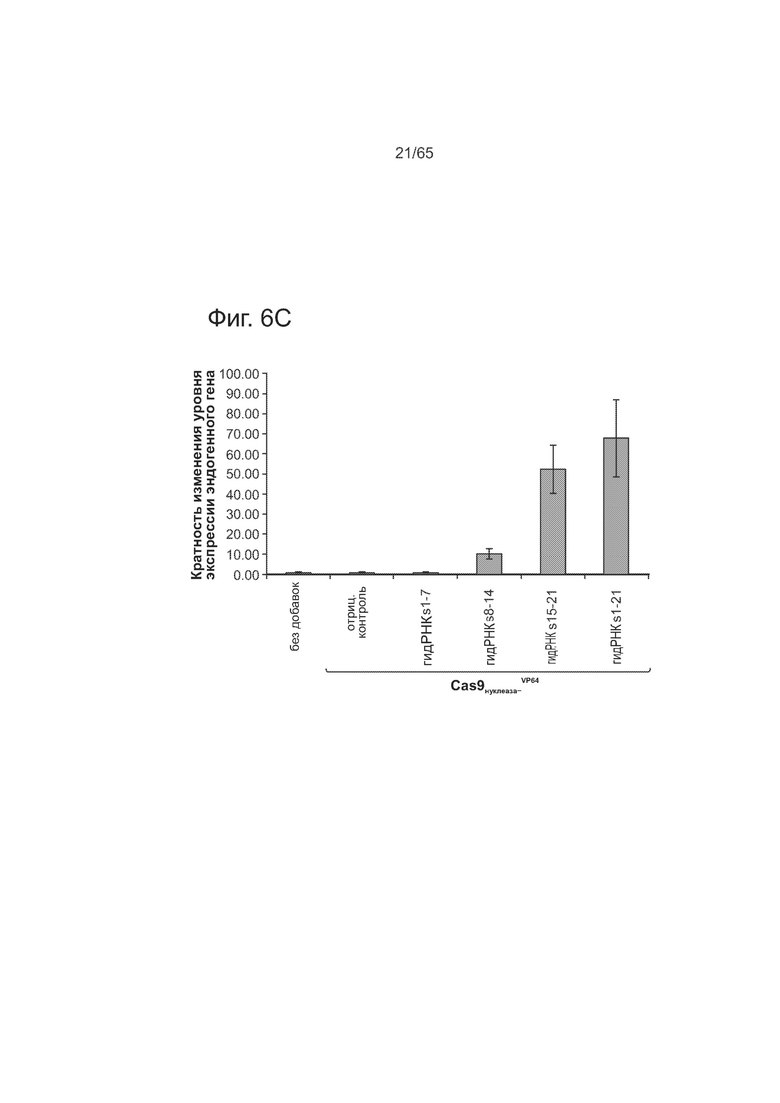
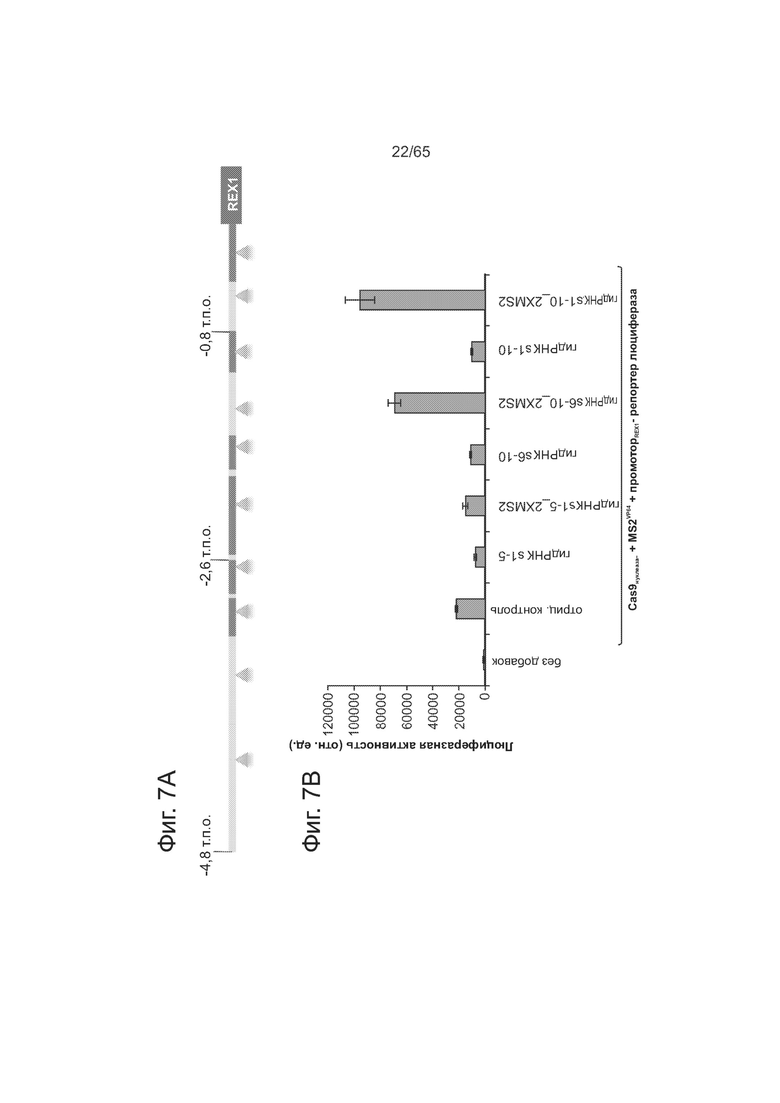
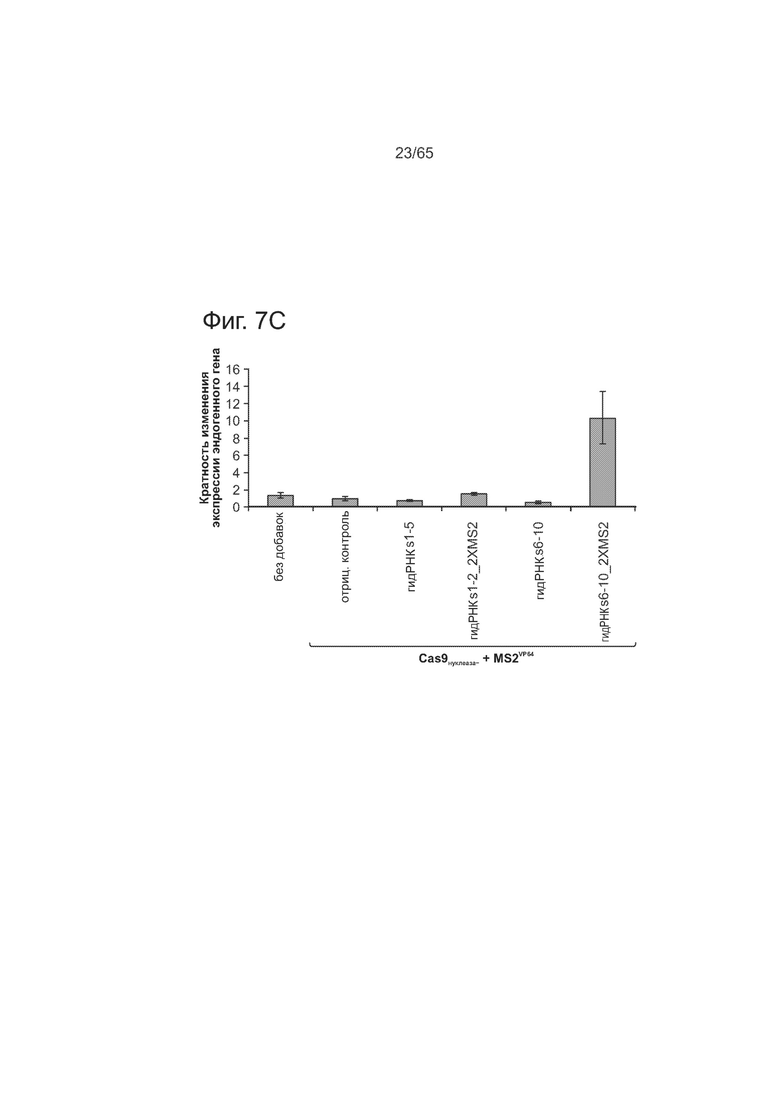
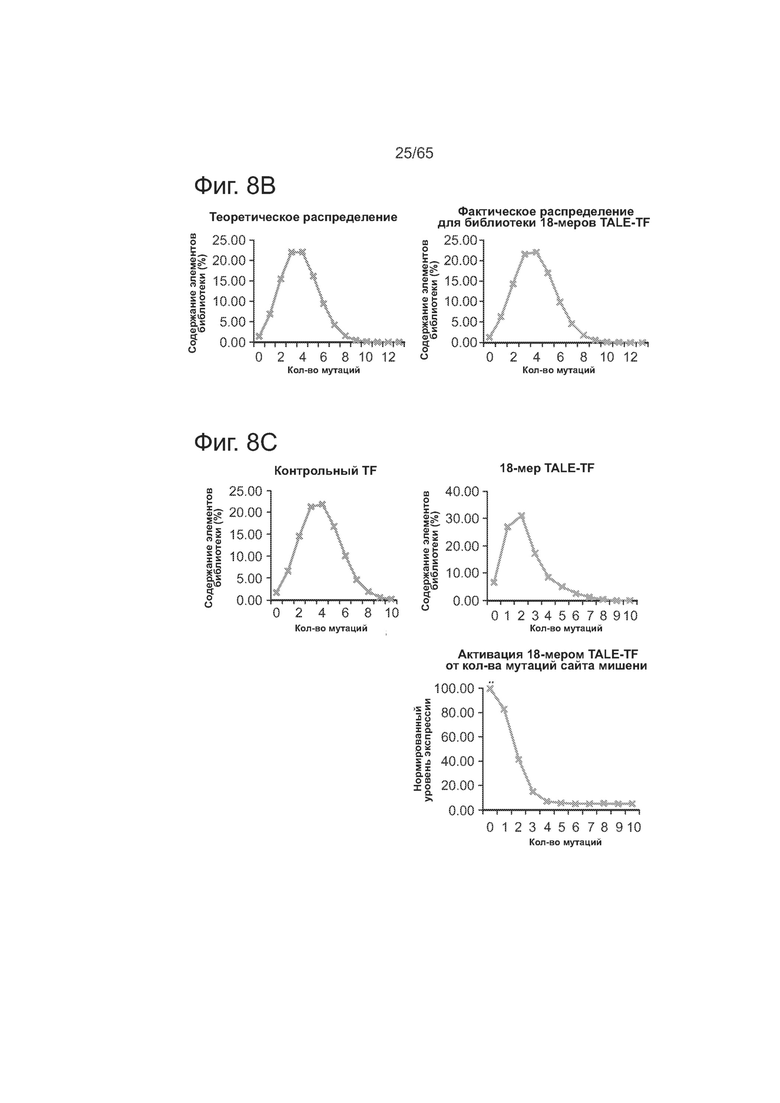
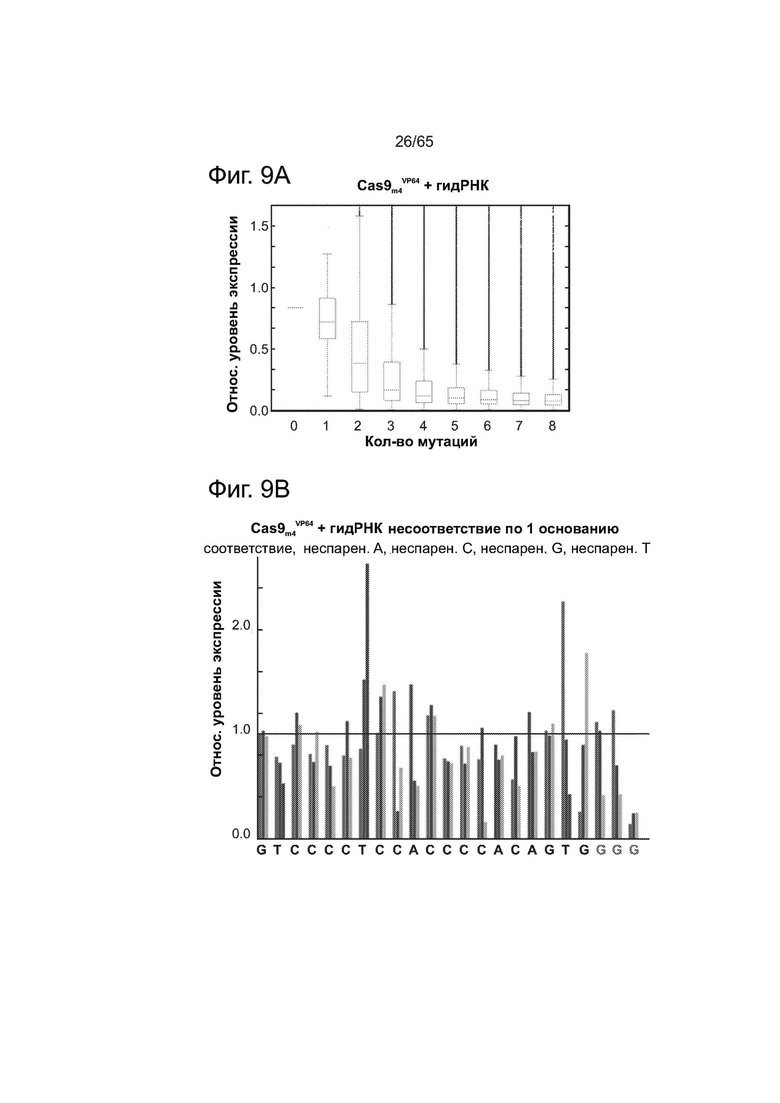
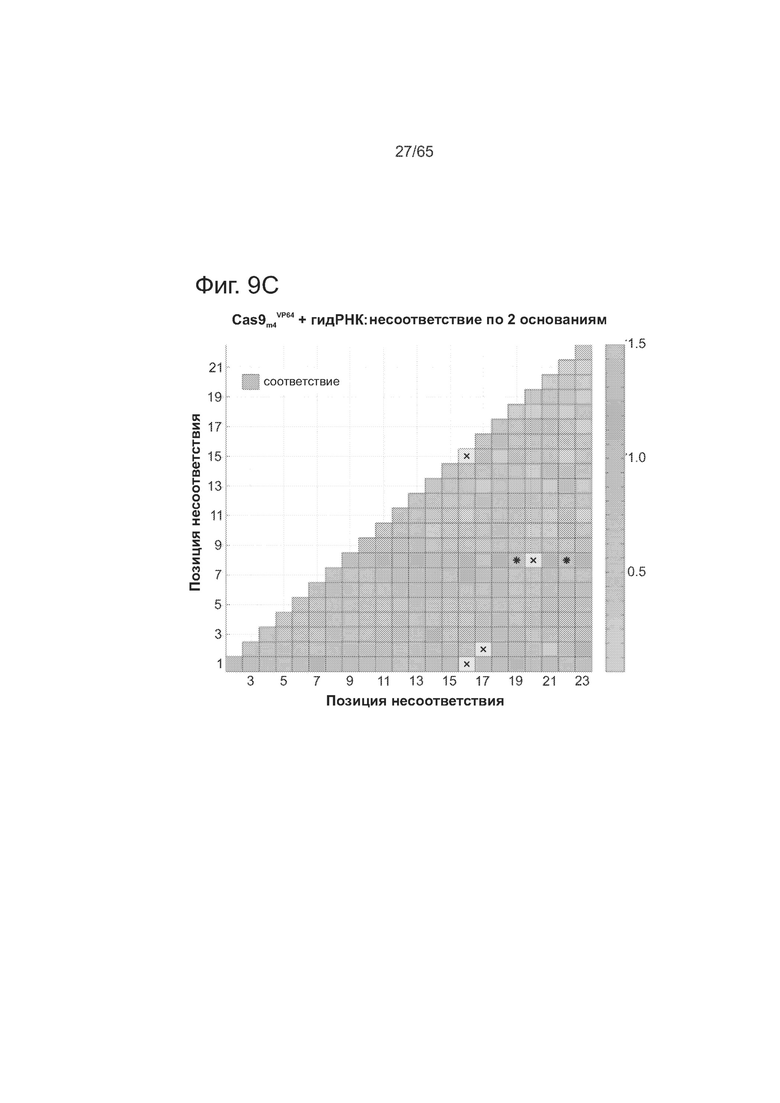
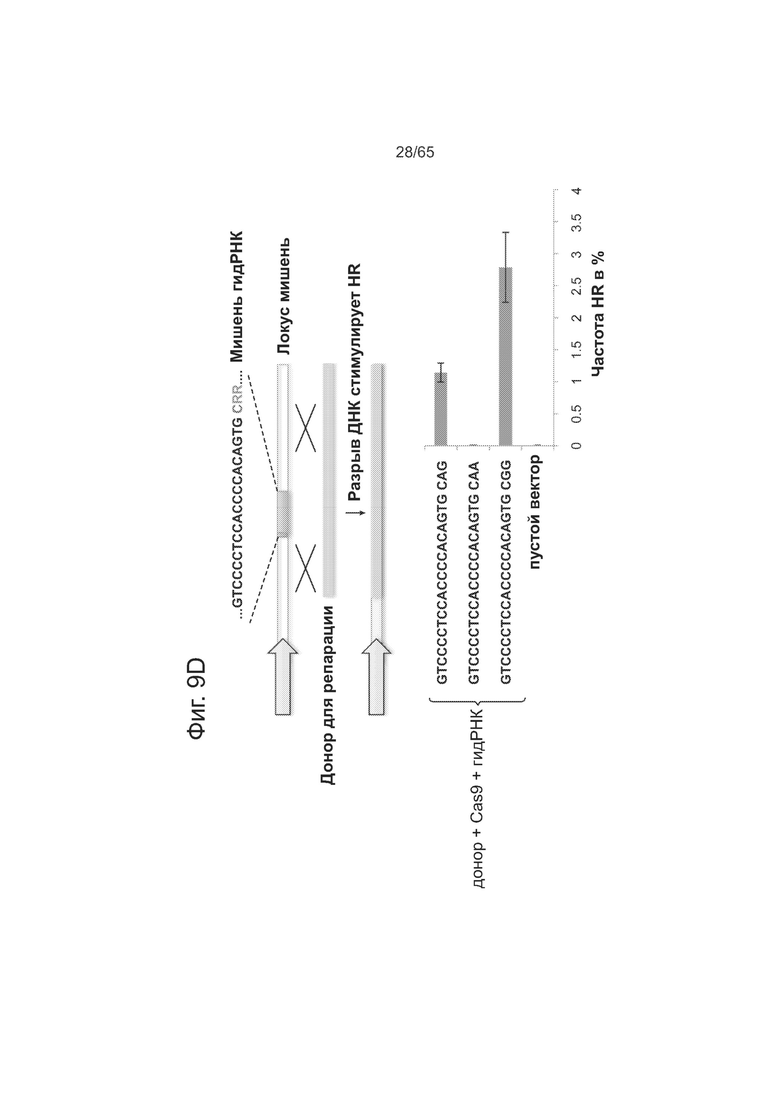
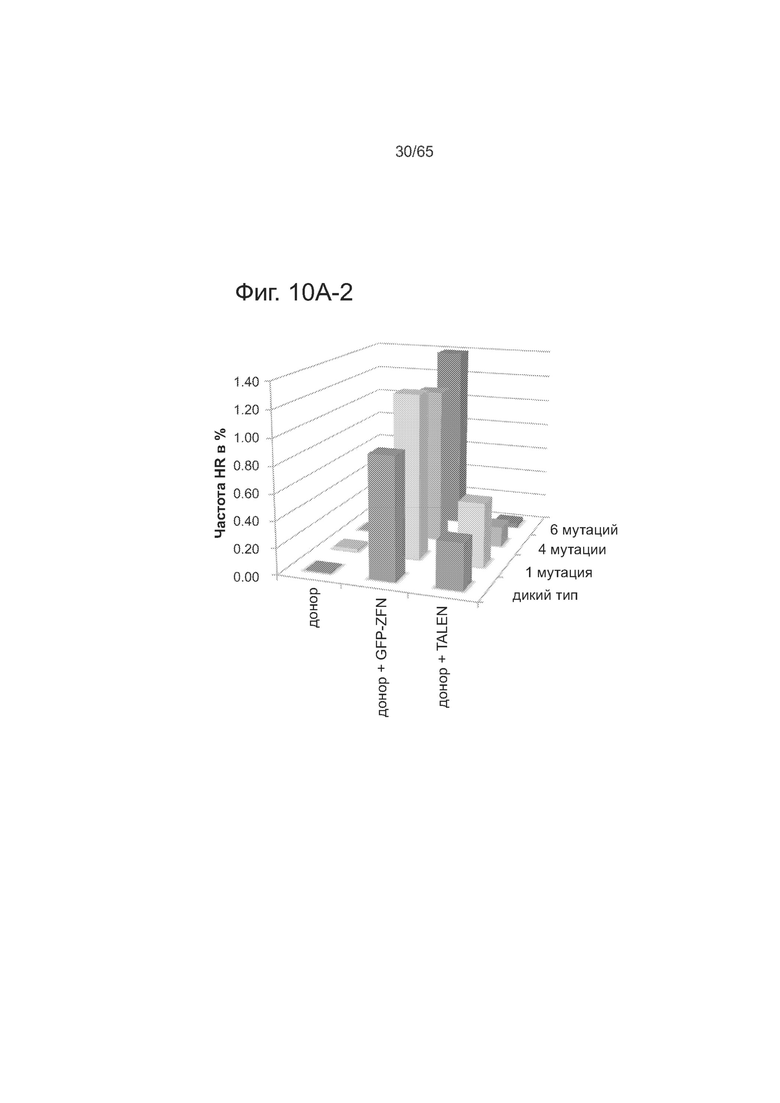
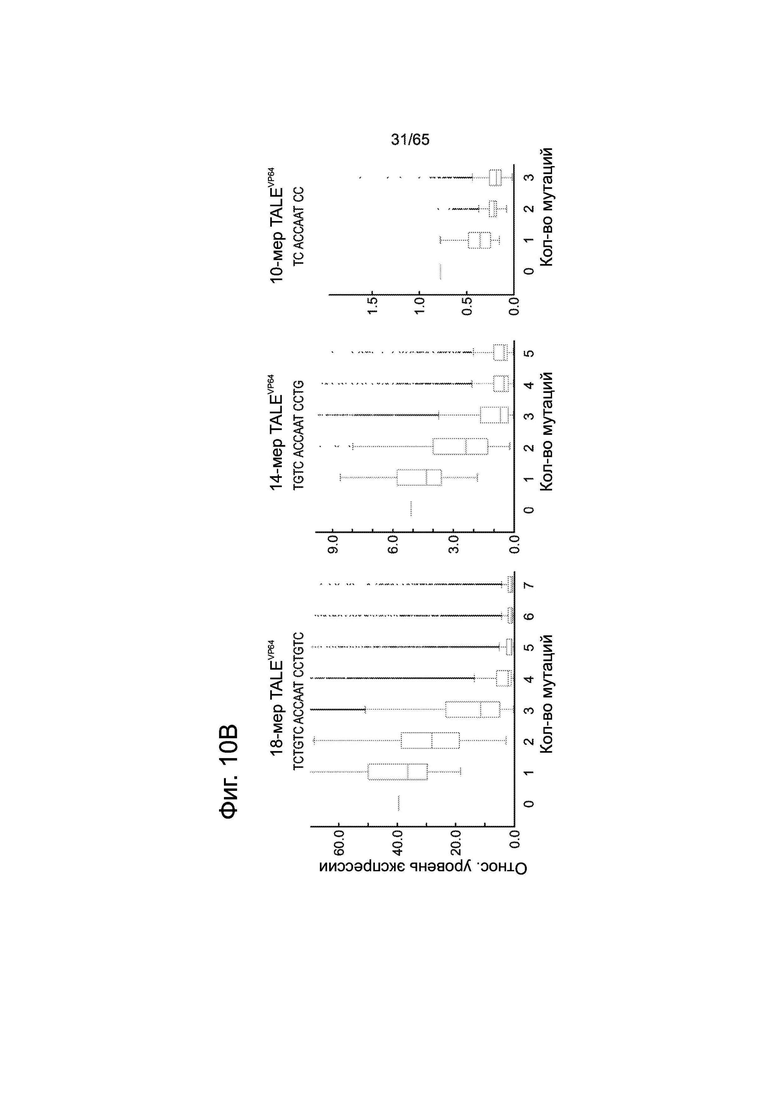
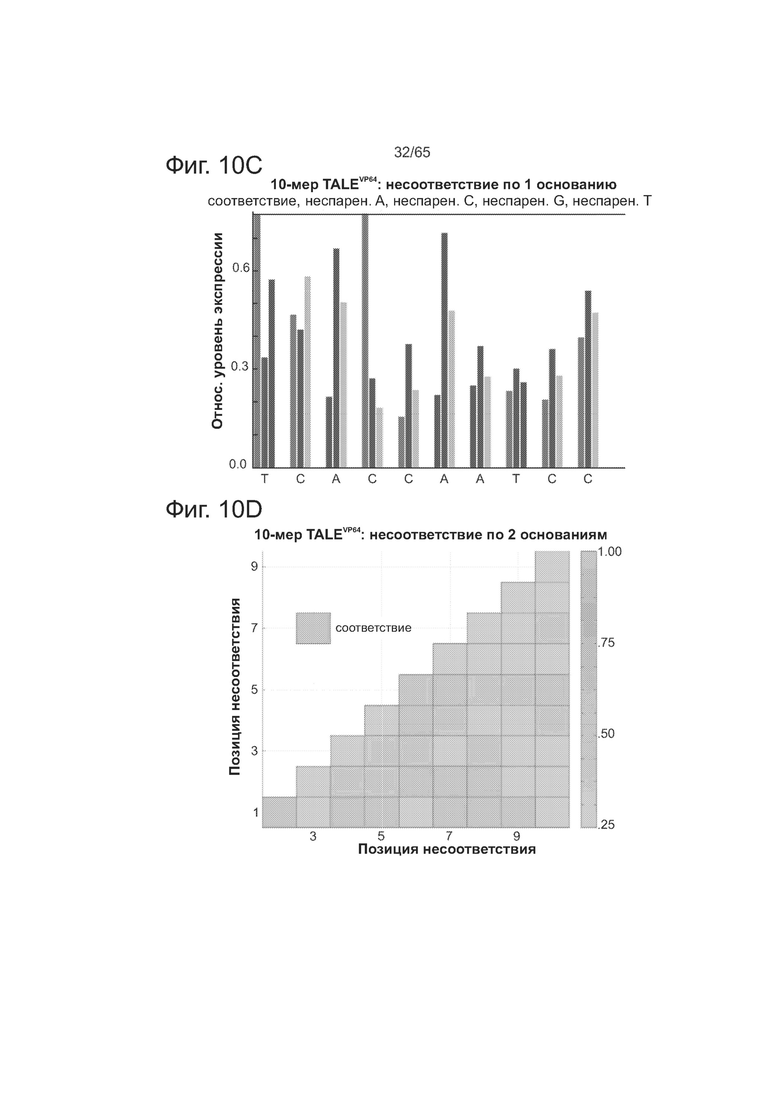
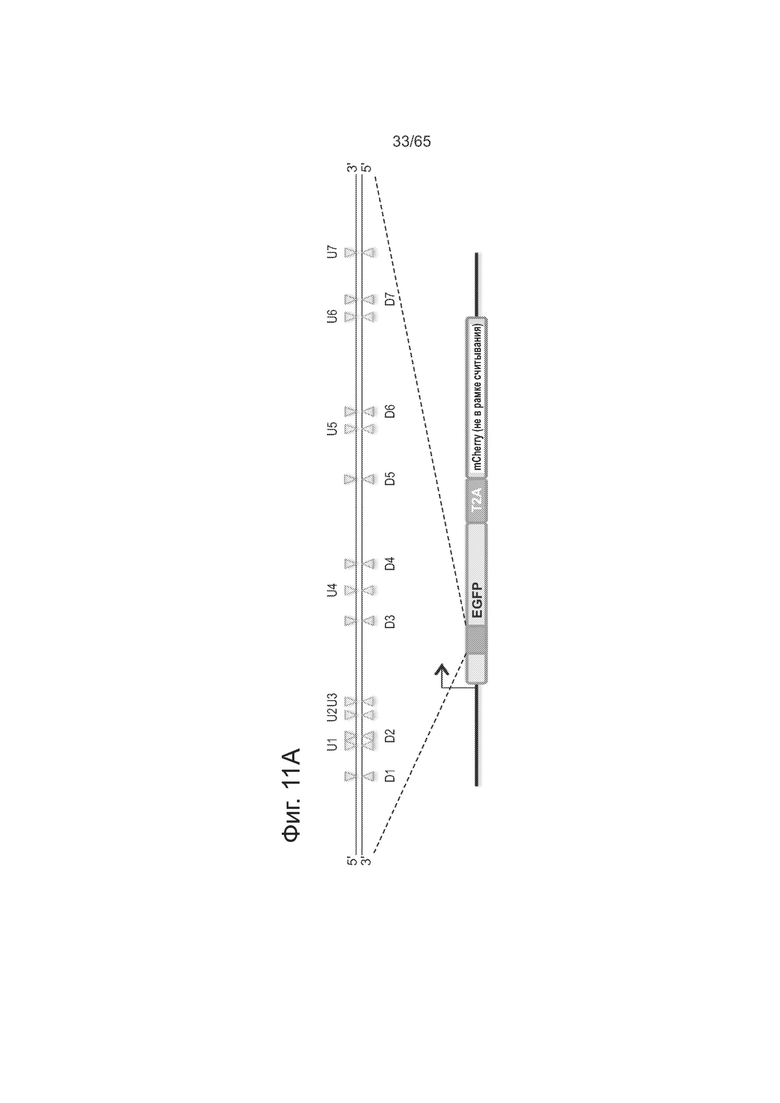
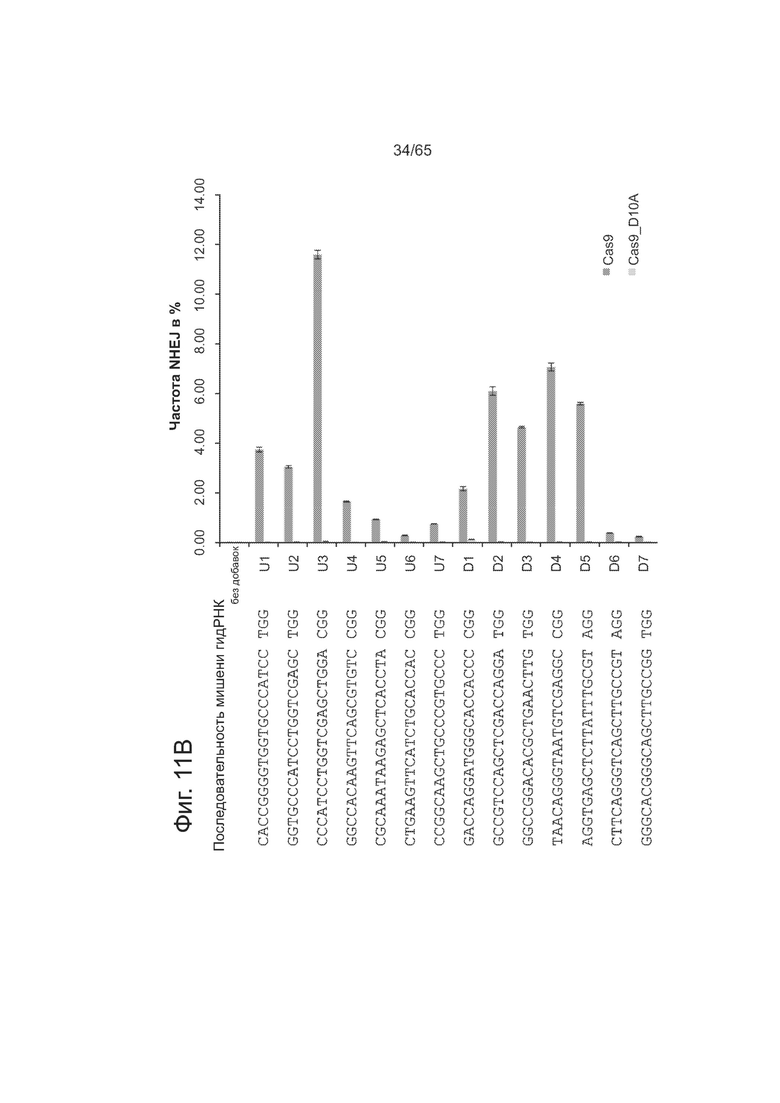
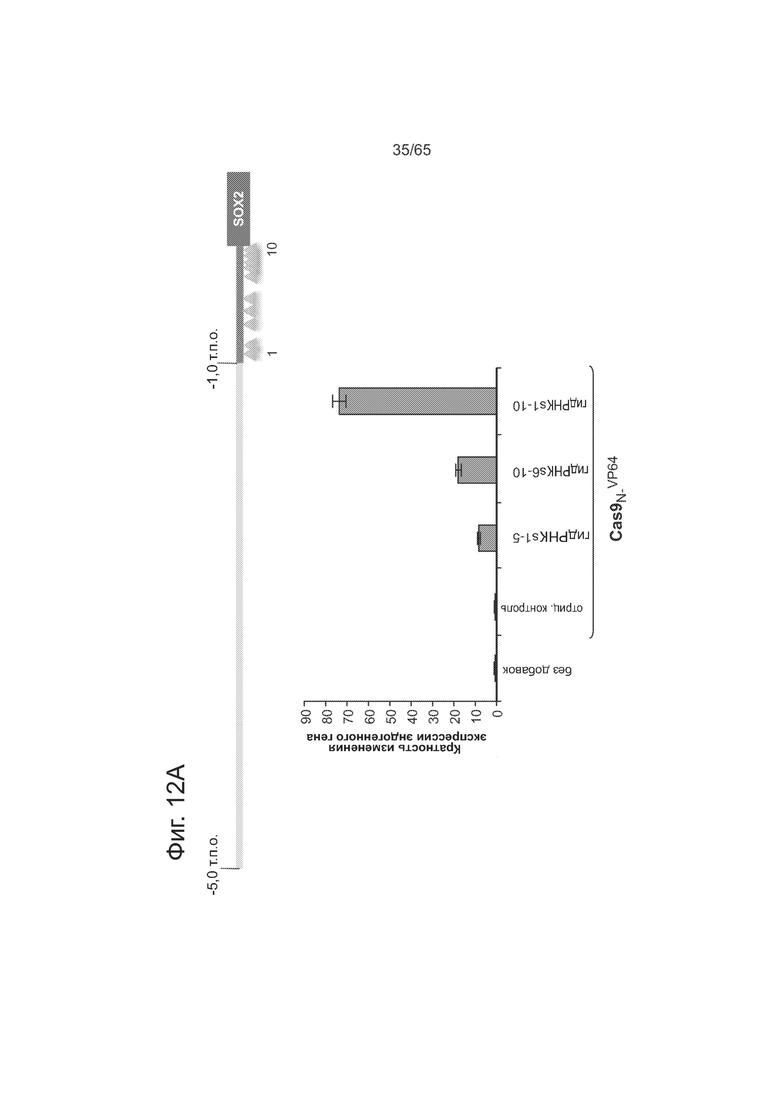

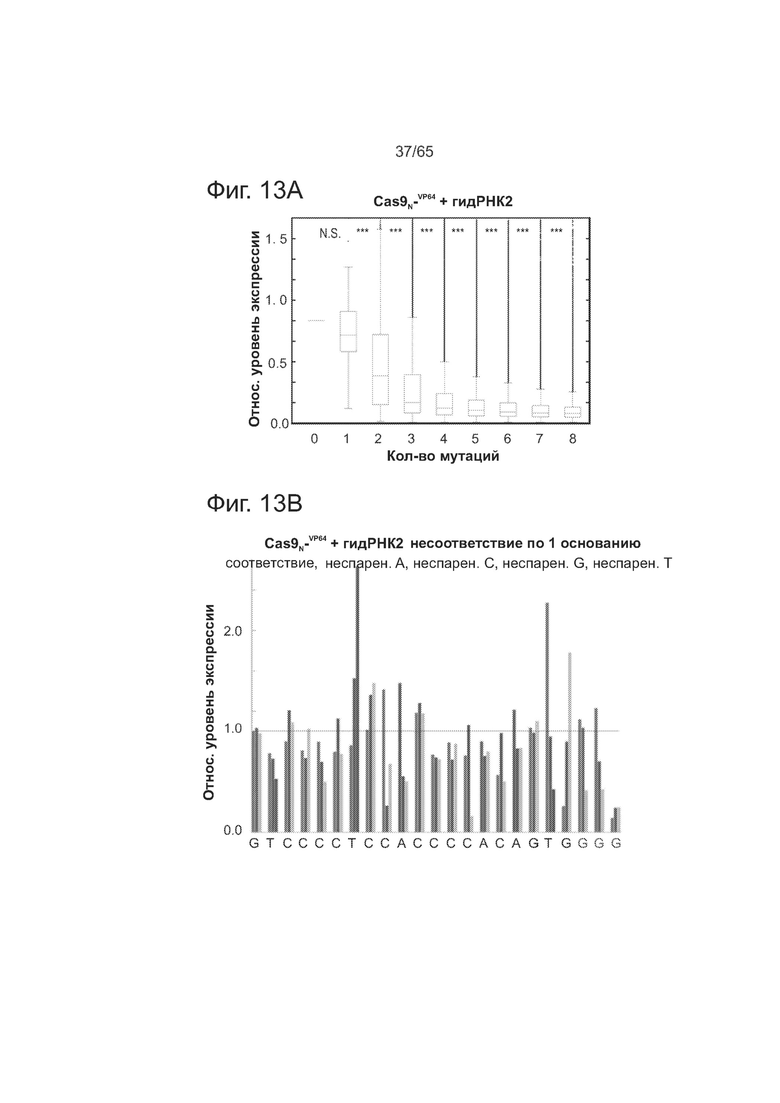
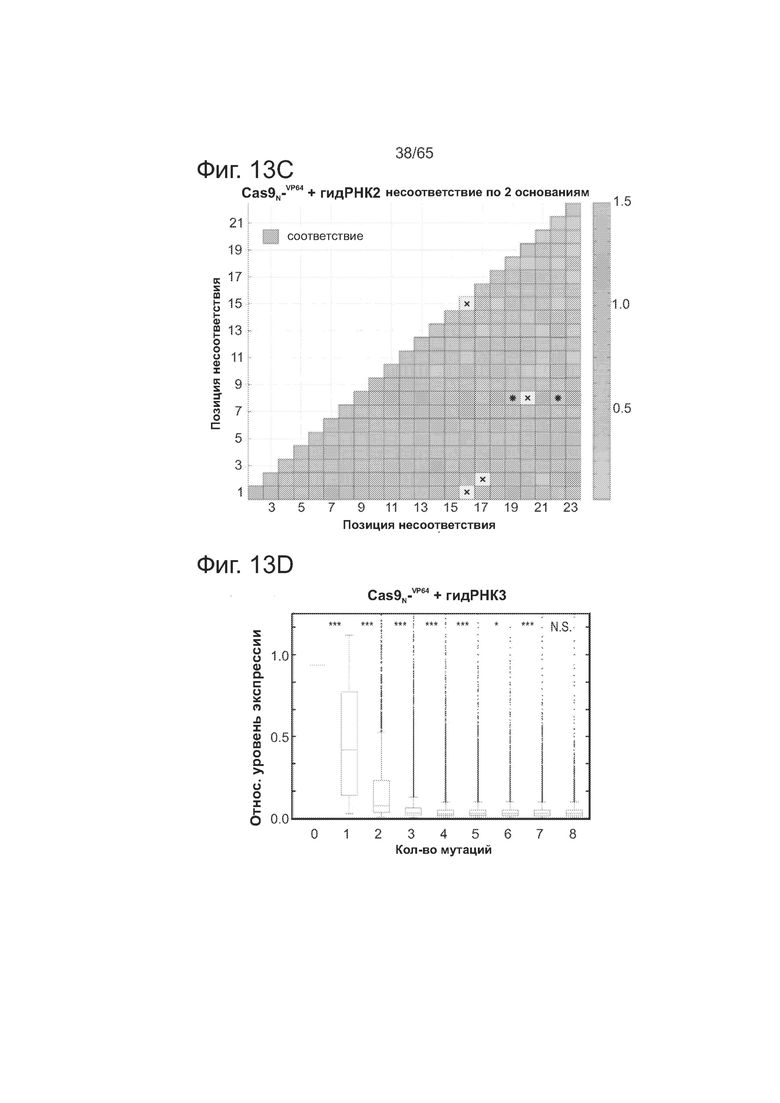
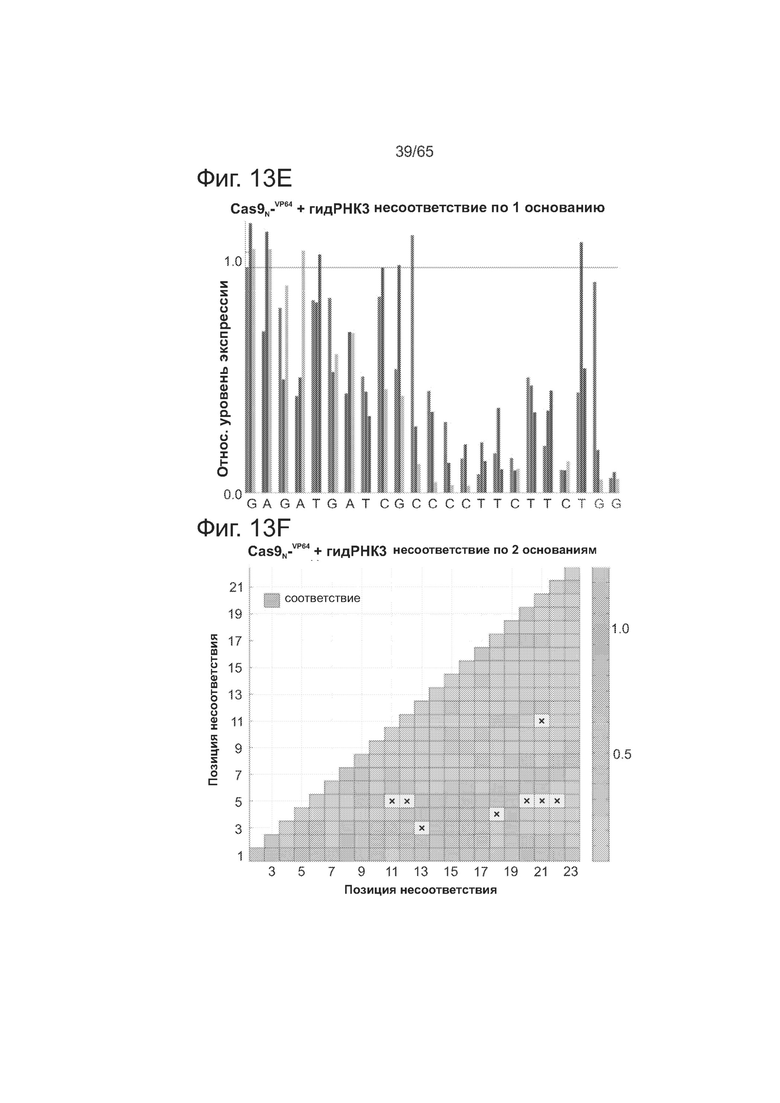
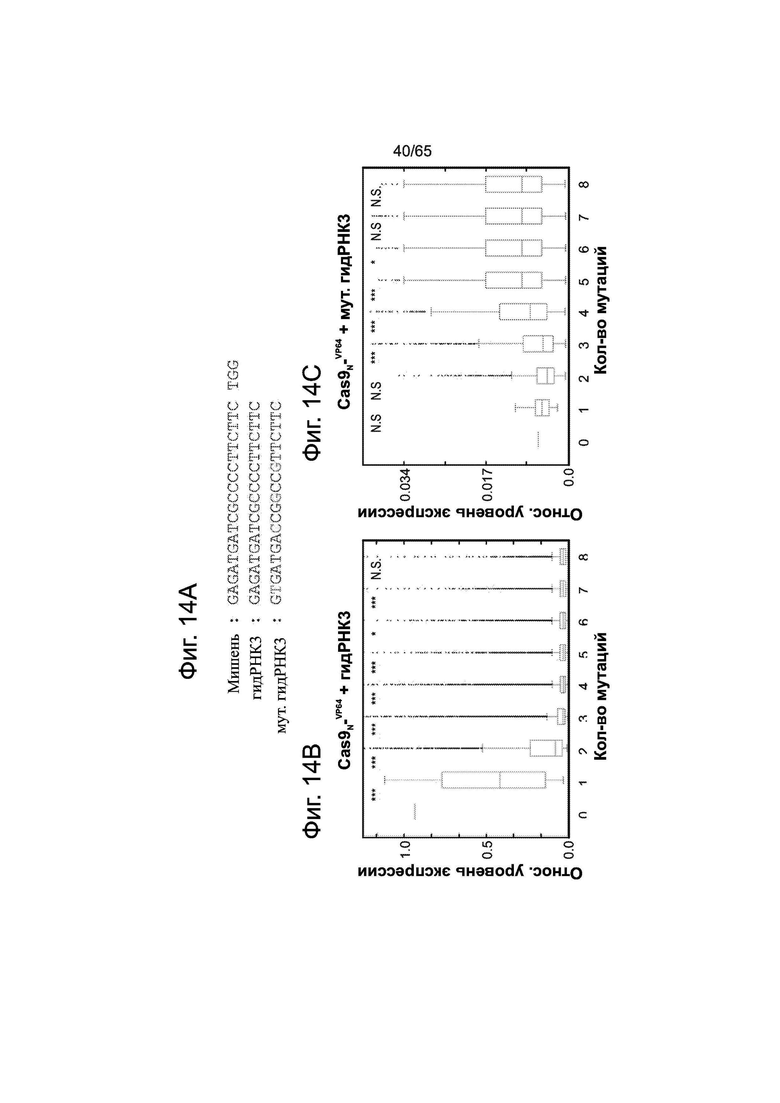
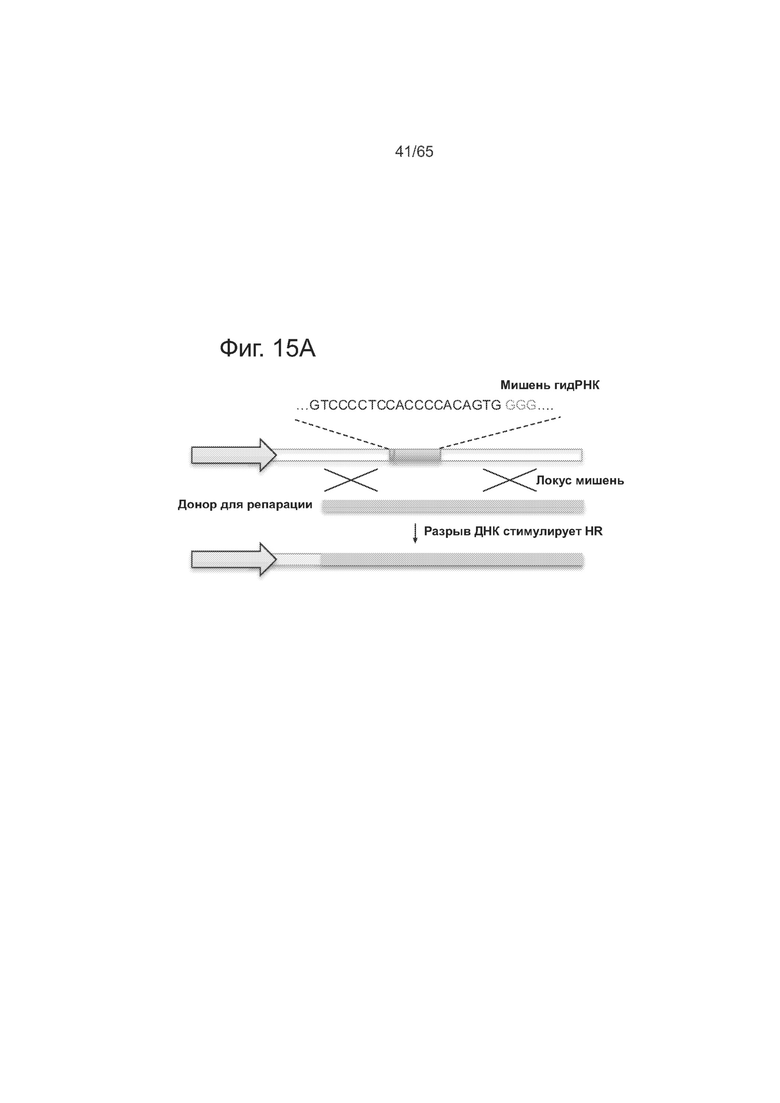
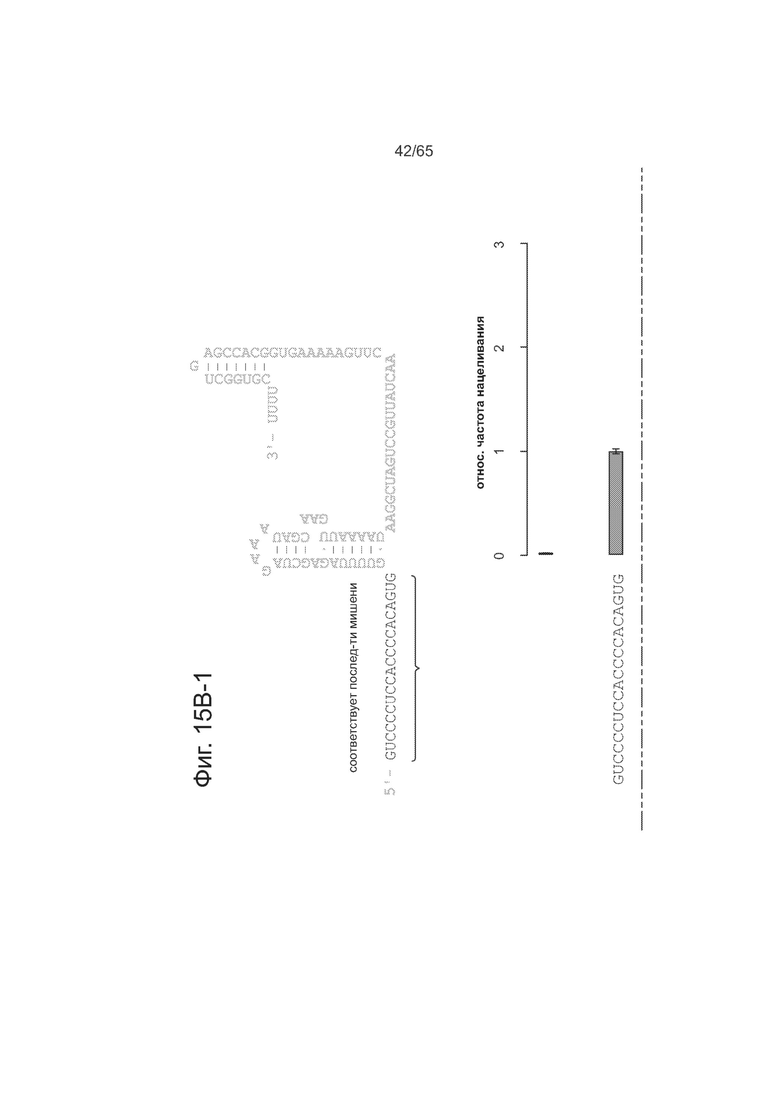
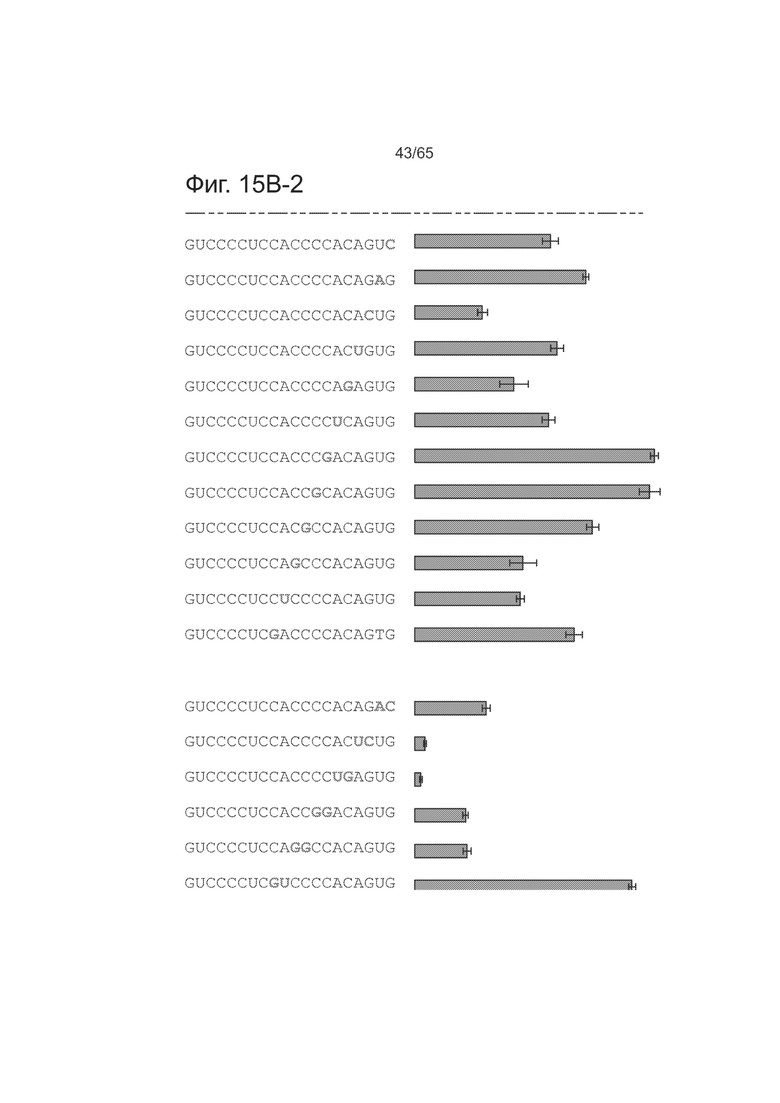
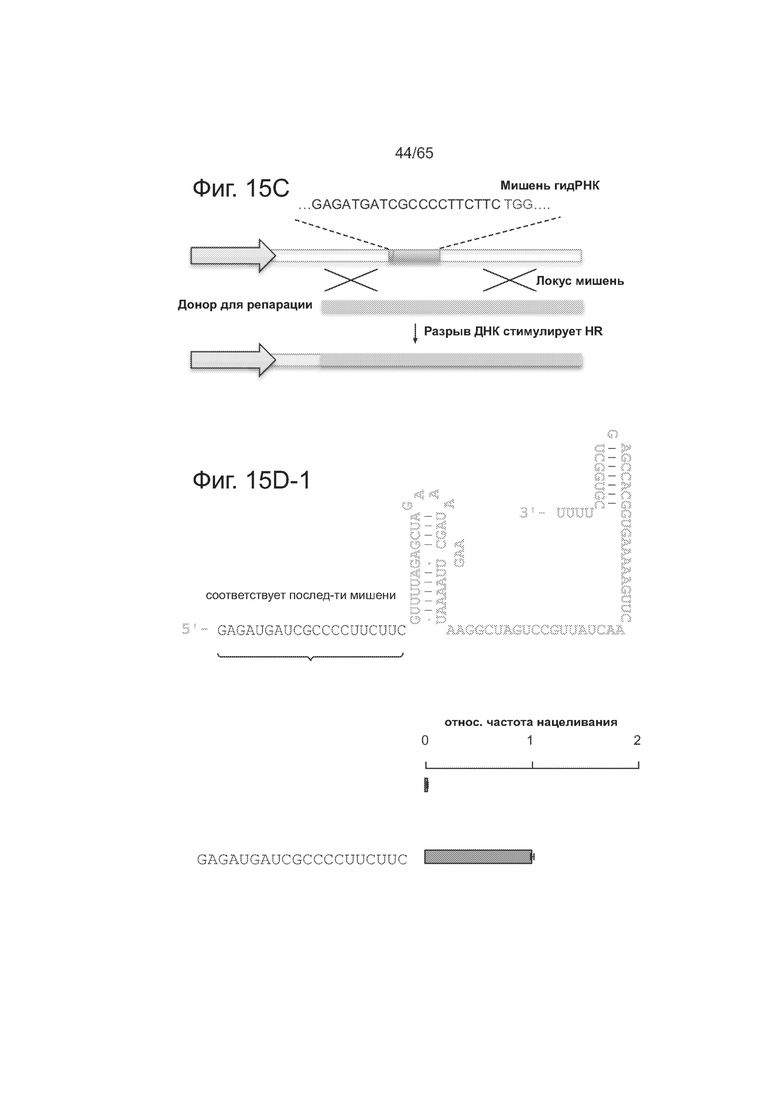

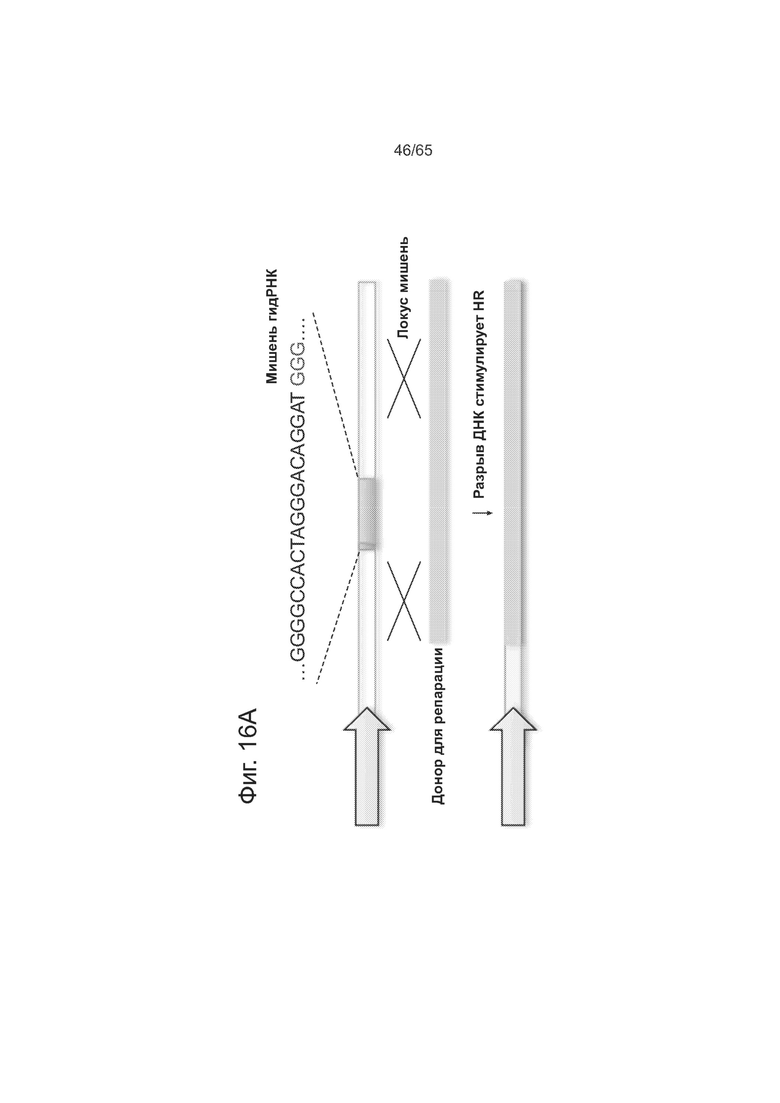

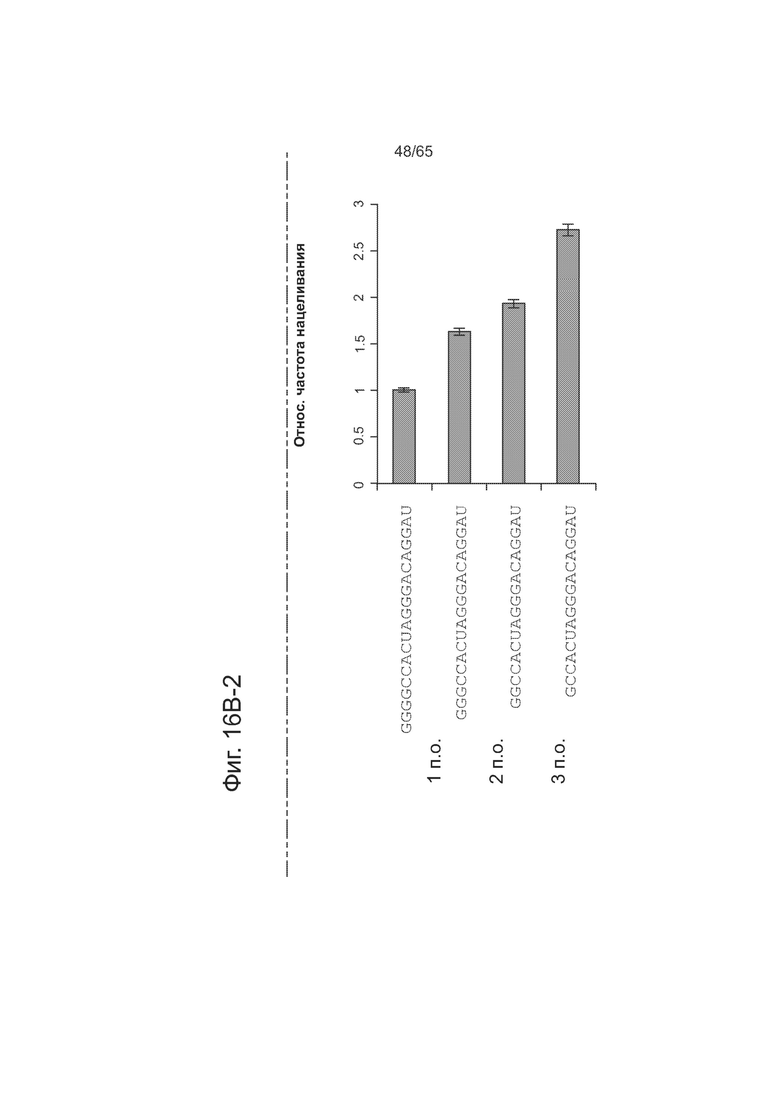
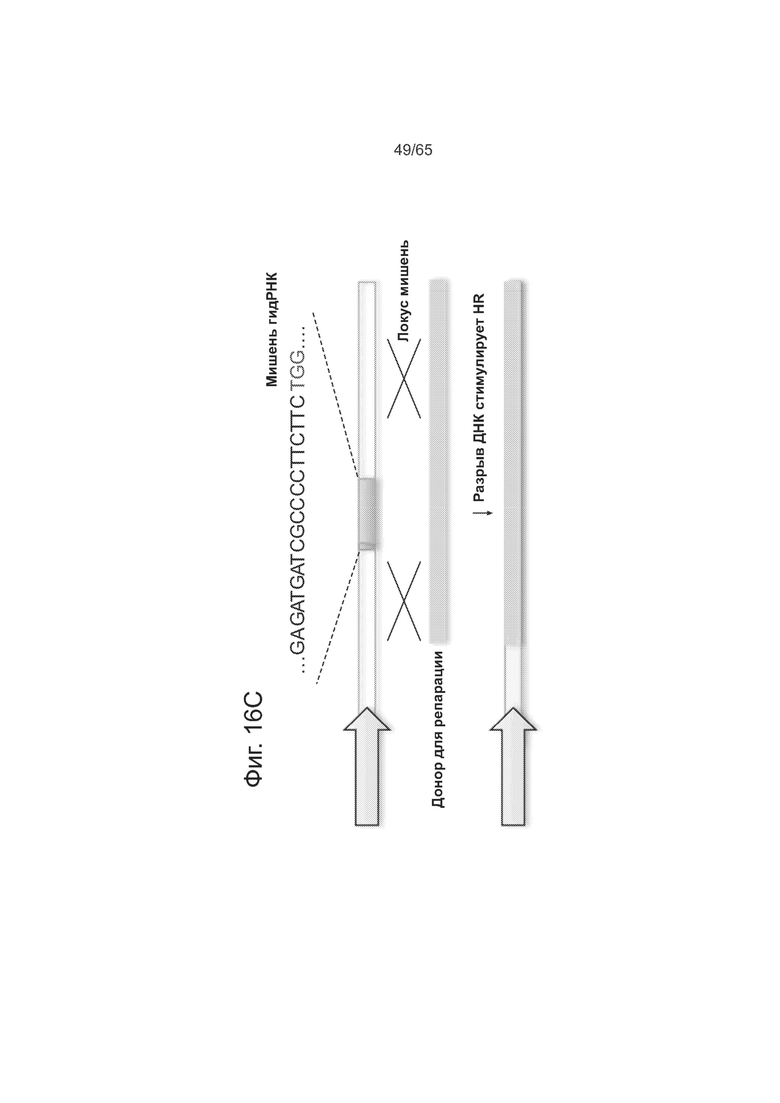

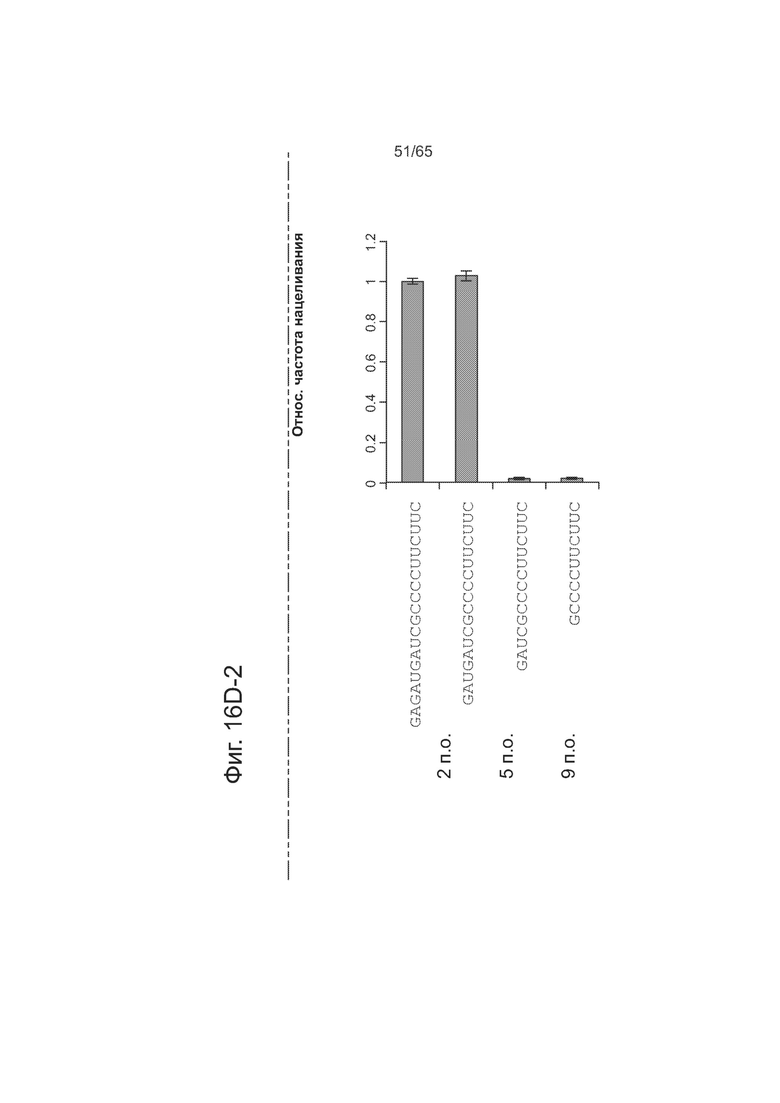
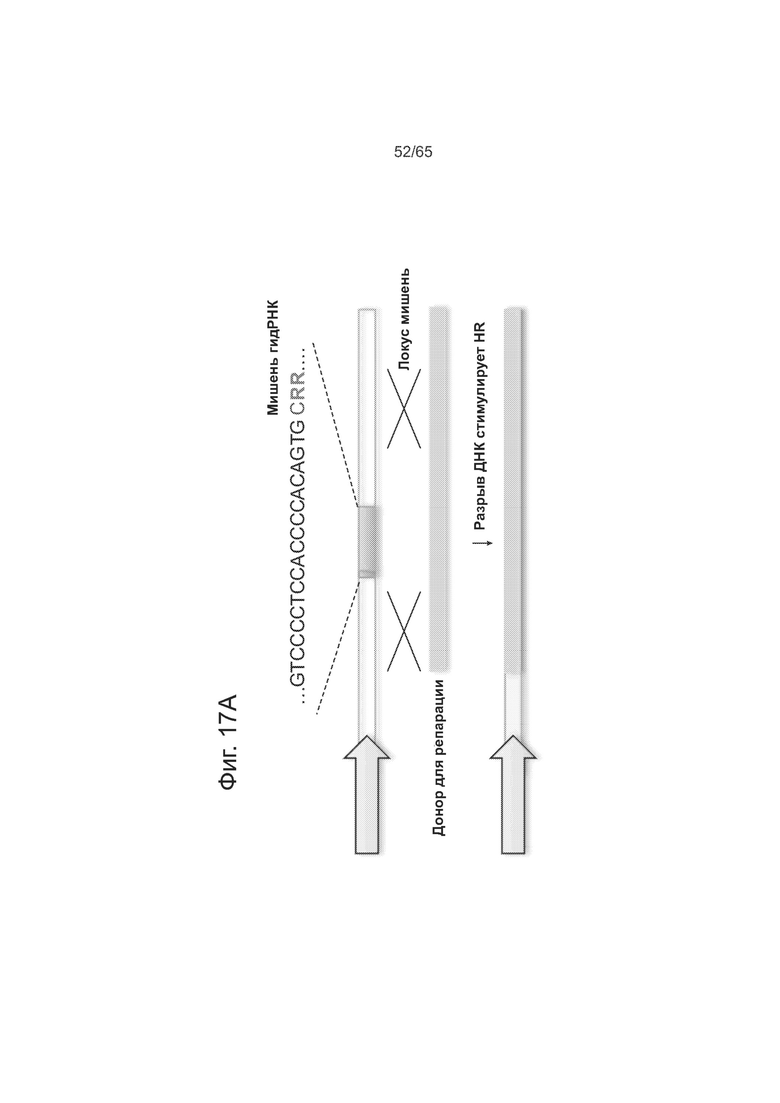
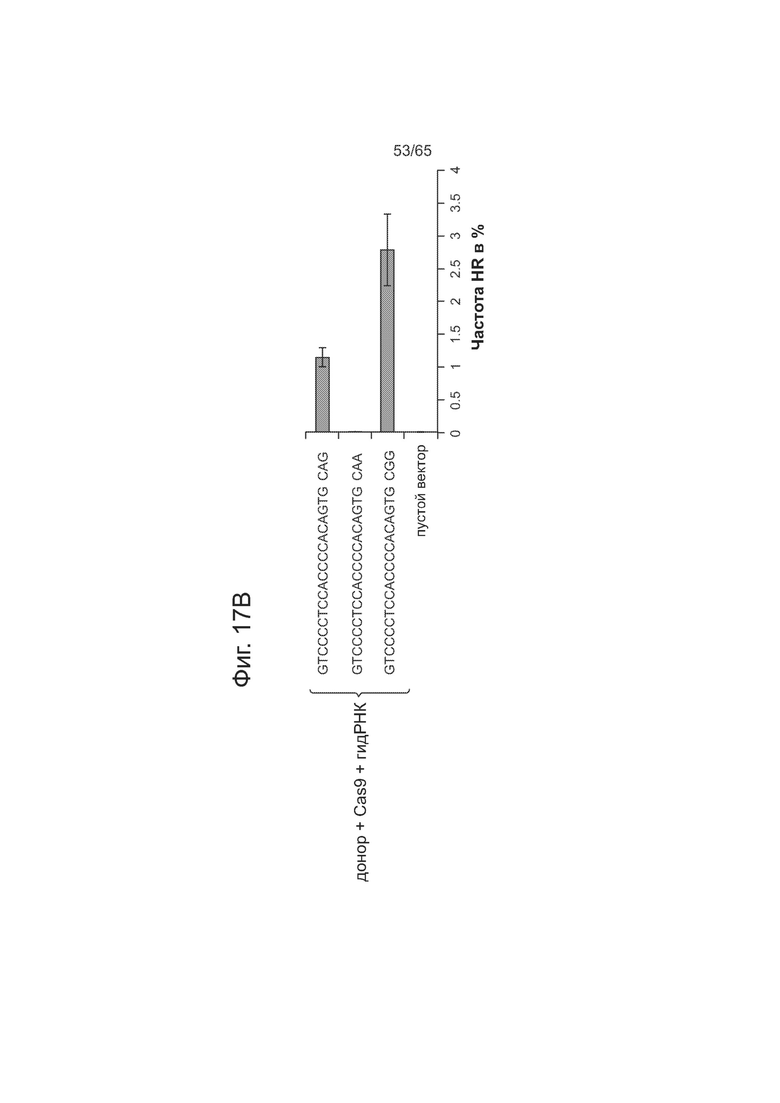

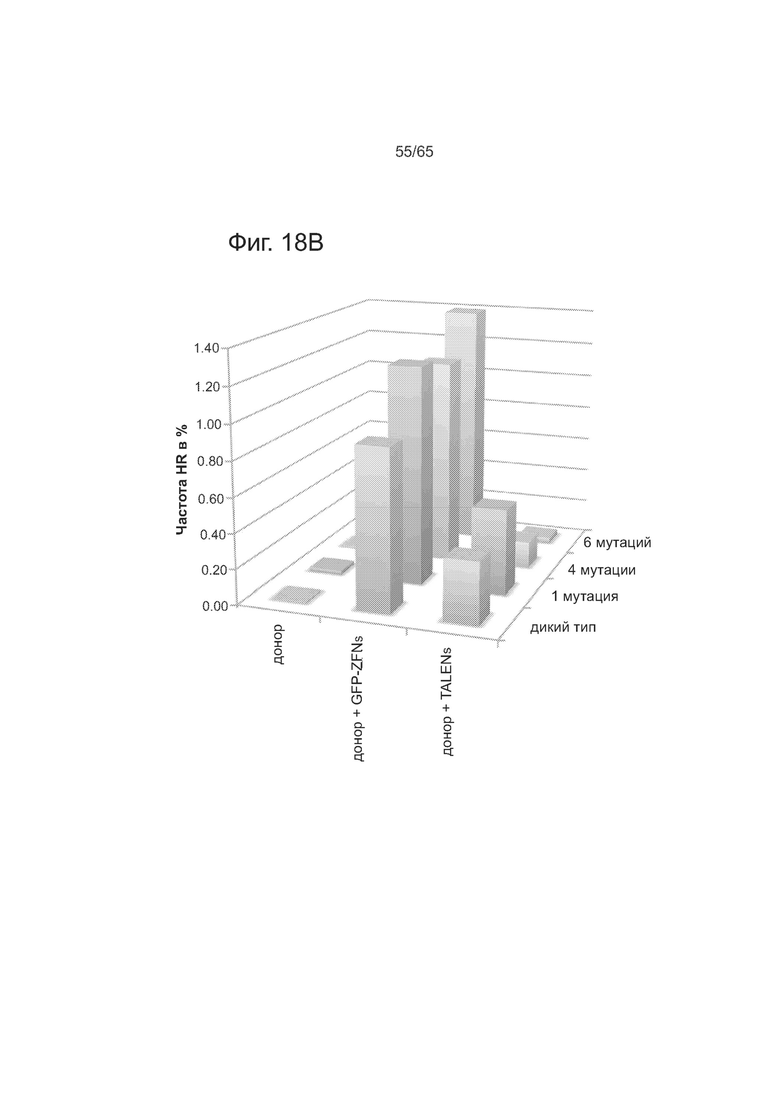

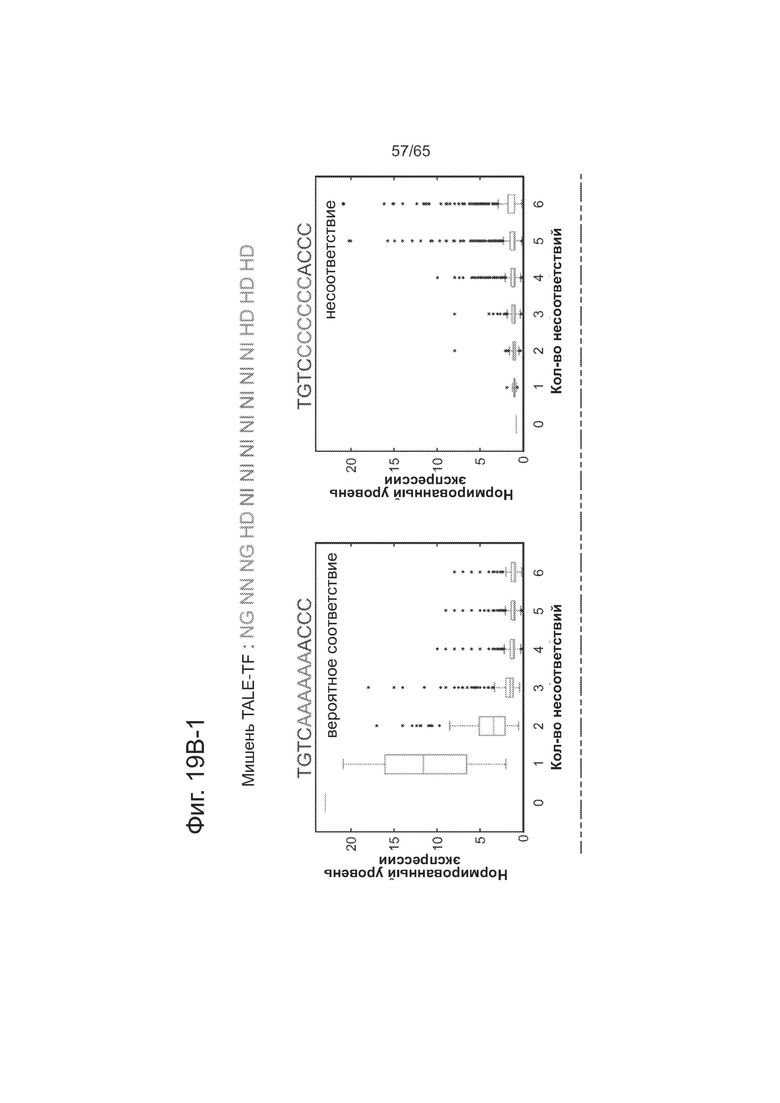

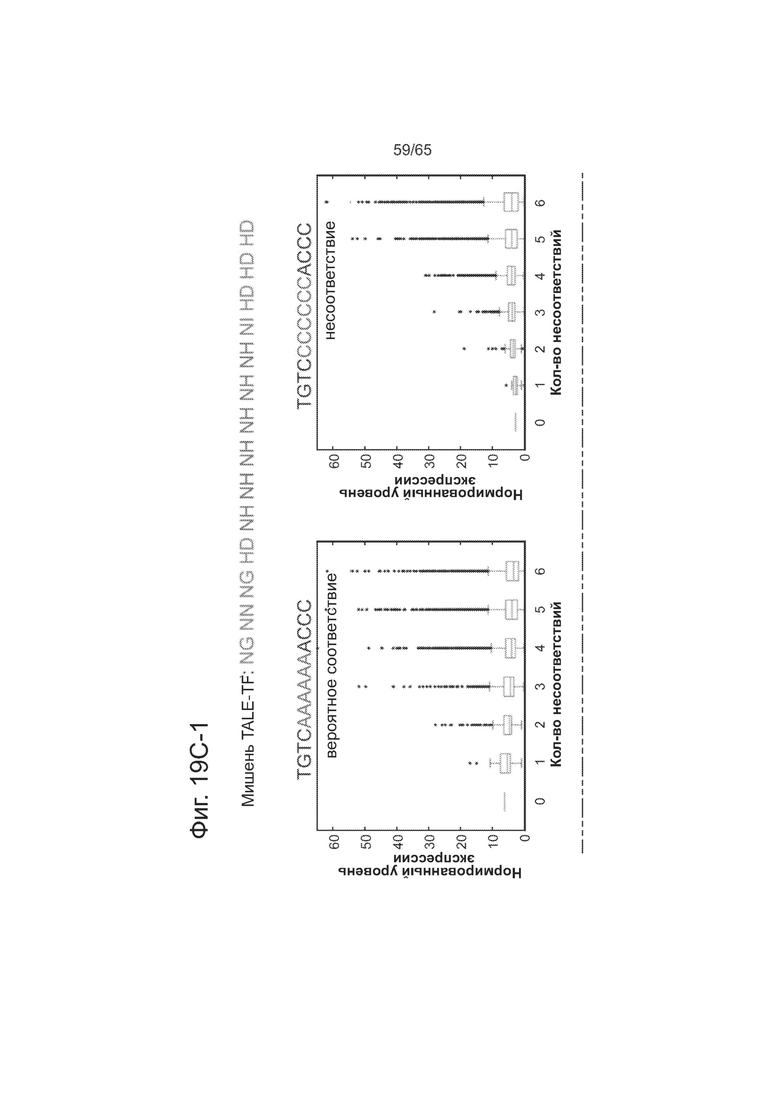
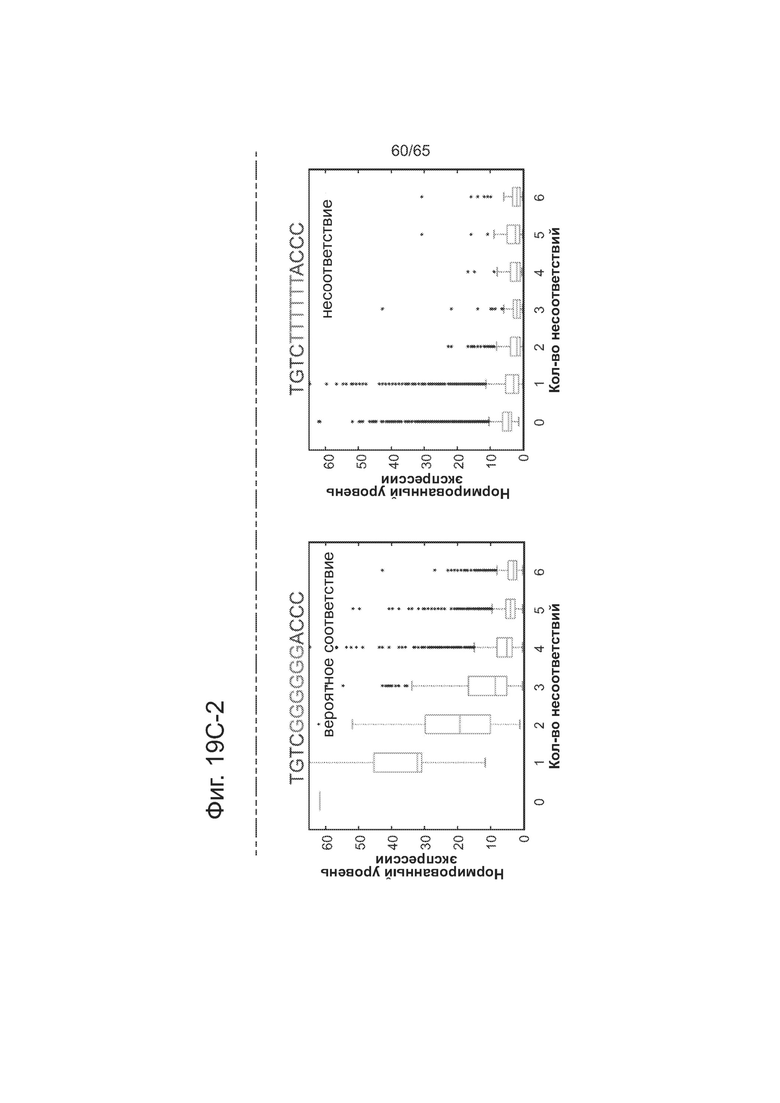
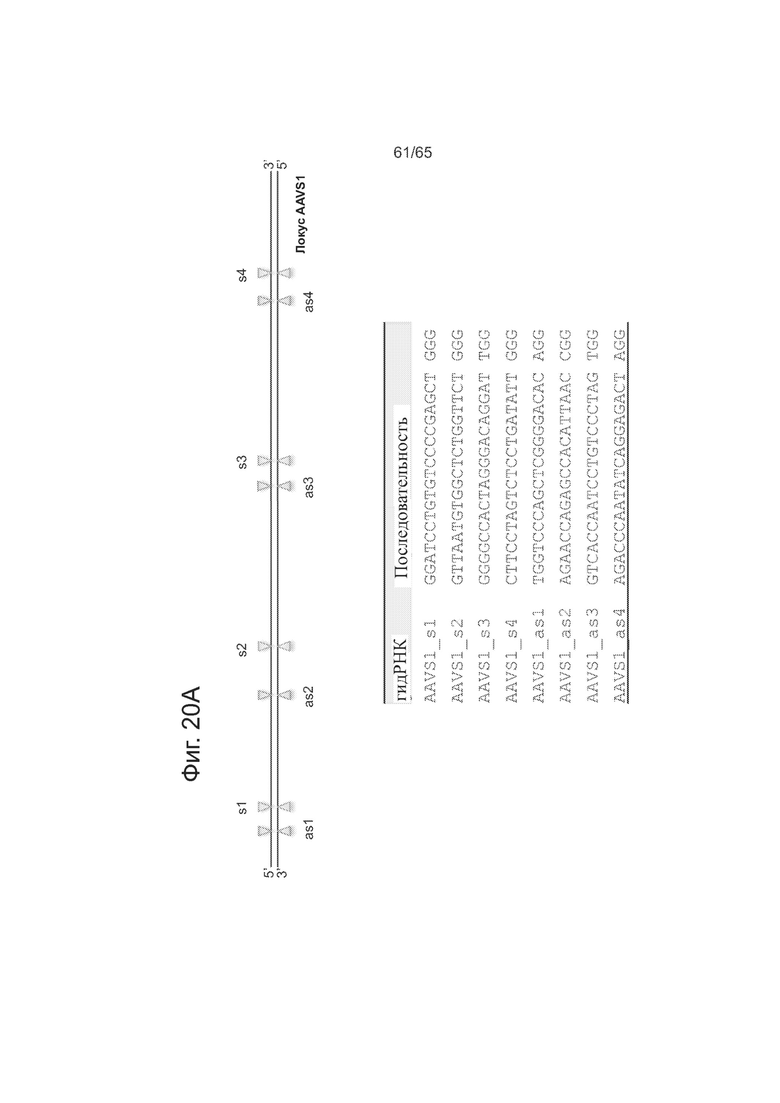
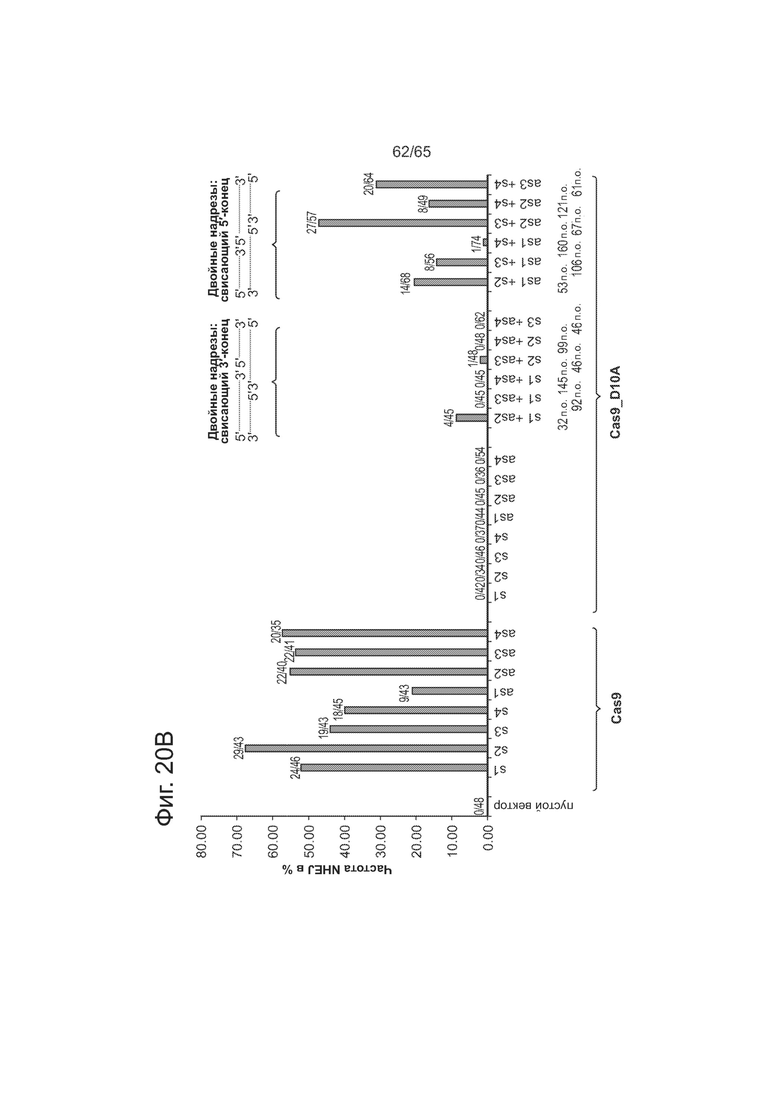
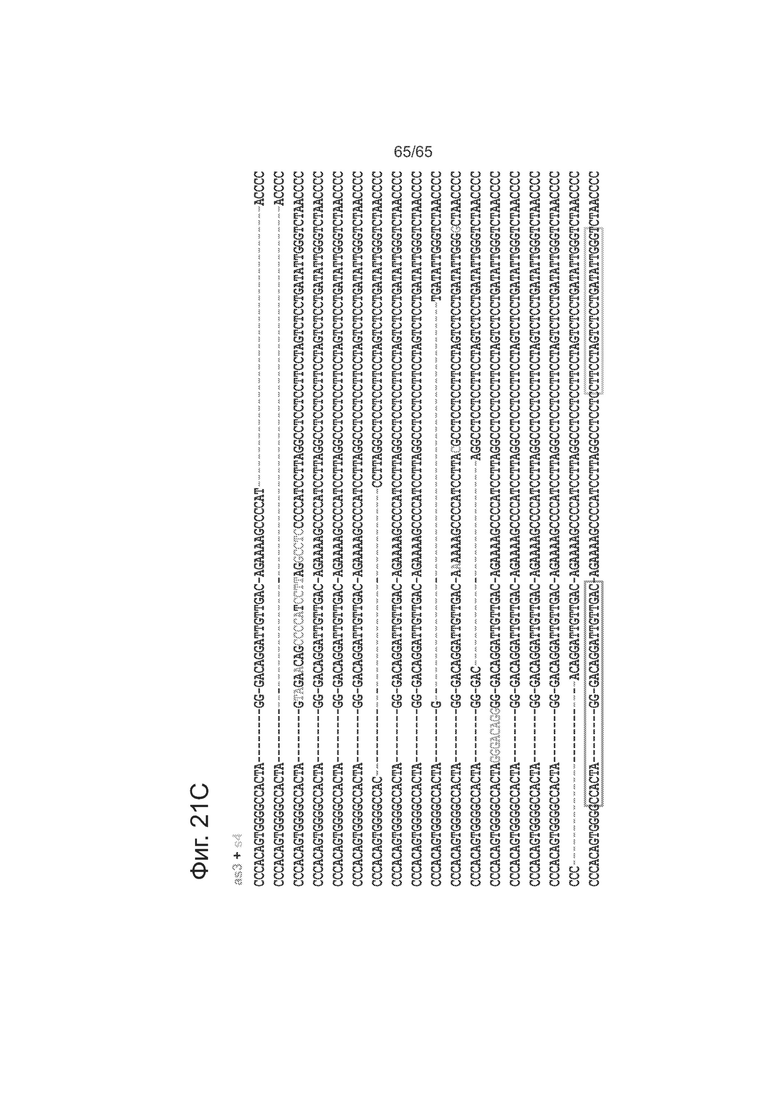
Изобретение относится к области биотехнологии, в частности к способам изменения ДНК целевой нуклеиновой кислоты в клетке, предусматривающим введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, причем каждая РНК комплементарна к сайту, прилегающему к ДНК целевой нуклеиновой кислоты, и введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один белок-никазу Cas9 с одним неактивным нуклеазным доменом или ДНК-связывающий белок-никазу из системы CRISPR типа II. Также раскрыты клетки для направляемого РНК основанного на Cas редактирования генома. Изобретение позволяет эффективно осуществлять изменение ДНК целевой нуклеиновой кислоты в клетке. 6 н. и 34 з.п. ф-лы, 21 ил., 2 табл., 17 пр.
1. Способ изменения ДНК целевой нуклеиновой кислоты в клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, причем каждая РНК комплементарна к сайту, прилегающему к ДНК целевой нуклеиновой кислоты,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один белок-никазу Cas9 с одним неактивным нуклеазным доменом, который направляется двумя или более РНК,
причем эти две или более РНК и по меньшей мере один белок-никаза Cas9 экспрессируются, при этом по меньшей мере один белок-никаза Cas9 локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрыва,
где клетка не является клеткой зародышевой линии человека.
2. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на одной и той же нити двухцепочечной ДНК.
3. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на одной и той же нити двухцепочечной ДНК, что приводит к гомологичной рекомбинации.
4. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК.
5. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы.
6. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к негомологичному соединению концов.
7. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и смещены относительно друг друга.
8. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК, смещены относительно друг друга и создают двухцепочечные разрывы.
9. Способ по п. 1, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК, смещены относительно друг друга и создают двухцепочечные разрывы, что приводит к негомологичному соединению концов.
10. Способ по п. 1, дополнительно включающий введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей последовательность донорной нуклеиновой кислоты, при этом два или несколько одноцепочечных разрыва приводят к гомологичной рекомбинации целевой нуклеиновой кислоты с последовательностью донорной нуклеиновой кислоты.
11. Клетка для направляемого РНК основанного на Cas редактирования генома, где клетка содержит:
первую чужеродную нуклеиновую кислоту, кодирующую две или более РНК, причем каждая РНК комплементарна к сайту, прилегающему к ДНК целевой нуклеиновой кислоты, и
вторую чужеродную нуклеиновую кислоту, кодирующую по меньшей мере один белок-никазу Cas9 с одним неактивным нуклеазным доменом, причем две или несколько РНК и по меньшей мере один белок-никаза Cas9 входят в состав комплекса совместной локализации для ДНК целевой нуклеиновой кислоты.
12. Клетка по п. 11, при этом клетка является эукариотической клеткой.
13. Клетка по п. 11, при этом клетка является клеткой дрожжей, клеткой растений или клеткой животных.
14. Клетка по п. 11, при этом РНК содержит от 10 до 500 нуклеотидов.
15. Клетка по п. 11, при этом РНК содержит от 20 до 100 нуклеотидов.
16. Клетка по п. 11, при этом целевая нуклеиновая кислота связана с заболеванием или болезненным состоянием.
17. Клетка по п. 11, при этом одна или несколько РНК представлены направляющей РНК.
18. Клетка по п. 11, при этом две или несколько РНК представляют собой слияния tracr-РНК и cr-РНК.
19. Клетка по п. 11, при этом ДНК целевой нуклеиновой кислоты представлена геномной ДНК, митохондриальной ДНК, вирусной ДНК либо экзогенной ДНК.
20. Способ изменения ДНК целевой нуклеиновой кислоты в клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к сайту, прилегающему к ДНК целевой нуклеиновой кислоты,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один белок-никазу Cas9 с одним неактивным нуклеазным доменом, который направляется двумя или несколькими РНК,
причем эти две или несколько РНК и по меньшей мере один белок-никаза Cas9 экспрессируются, при этом по меньшей мере один белок-никаза Cas9 локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрыва,
причем эти два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, тем самым предотвращая экспрессию целевой нуклеиновой кислоты,
где клетка не является клеткой зародышевой линии человека.
21. Способ изменения ДНК целевой нуклеиновой кислоты в клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или несколько РНК, причем каждая РНК комплементарна к сайту, прилегающему к ДНК целевой нуклеиновой кислоты,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу из системы CRISPR типа II,
причем эти две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза экспрессируются, при этом по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрыва,
где клетка не является клеткой зародышевой линии человека.
22. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на одной и той же нити двухцепочечной ДНК.
23. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на одной и той же нити двухцепочечной ДНК, что приводит к гомологичной рекомбинации.
24. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК.
25. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы.
26. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к негомологичному соединению концов.
27. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и смещены относительно друг друга.
28. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК, смещены относительно друг друга и создают двухцепочечные разрывы.
29. Способ по п. 21, при этом два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК, смещены относительно друг друга и создают двухцепочечные разрывы, что приводит к негомологичному соединению концов.
30. Способ по п. 21, дополнительно включающий введение в клетку третьей чужеродной нуклеиновой кислоты, кодирующей последовательность донорной нуклеиновой кислоты, при этом два или несколько одноцепочечных разрыва приводят к гомологичной рекомбинации целевой нуклеиновой кислоты с последовательностью донорной нуклеиновой кислоты.
31. Клетка для направляемого РНК основанного на Cas редактирования генома, где клетка содержит:
первую чужеродную нуклеиновую кислоту, кодирующую две или несколько РНК, причем каждая РНК комплементарна к сайту, прилегающему к ДНК целевой нуклеиновой кислоты, и
вторую чужеродную нуклеиновую кислоту, кодирующую по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу из системы CRISPR типа II, причем две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза входят в состав комплекса совместной локализации для ДНК целевой нуклеиновой кислоты.
32. Клетка по п. 31, при этом клетка является эукариотической клеткой.
33. Клетка по п. 31, при этом клетка является клеткой дрожжей, клеткой растений или клеткой животных.
34. Клетка по п. 31, при этом РНК содержит от 10 до 500 нуклеотидов.
35. Клетка по п. 31, при этом РНК содержит от 20 до 100 нуклеотидов.
36. Клетка по п. 31, при этом целевая нуклеиновая кислота связана с заболеванием или болезненным состоянием.
37. Клетка по п. 31, при этом две или несколько РНК представлены направляющей РНК.
38. Клетка по п. 31, при этом две или несколько РНК представляют собой слияния tracr-РНК и cr-РНК.
39. Клетка по п. 31, при этом ДНК целевой нуклеиновой кислоты представлена геномной ДНК, митохондриальной ДНК, вирусной ДНК либо экзогенной ДНК.
40. Способ изменения ДНК целевой нуклеиновой кислоты в клетке, включающий:
введение в клетку первой чужеродной нуклеиновой кислоты, кодирующей две или более РНК, причем каждая РНК комплементарна к сайту, прилегающему к ДНК целевой нуклеиновой кислоты,
введение в клетку второй чужеродной нуклеиновой кислоты, кодирующей по меньшей мере один РНК-направляемый ДНК-связывающий белок-никазу из системы CRISPR типа II,
причем эти две или несколько РНК и по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза экспрессируются, при этом по меньшей мере один РНК-направляемый ДНК-связывающий белок-никаза локализуется совместно с двумя или несколькими РНК на ДНК целевой нуклеиновой кислоты и надрезает ДНК целевой нуклеиновой кислоты, в результате чего образуются два или несколько соседних одноцепочечных разрыва,
причем эти два или несколько соседних одноцепочечных разрыва находятся на разных нитях двухцепочечной ДНК и создают двухцепочечные разрывы, что приводит к фрагментации целевой нуклеиновой кислоты, тем самым предотвращая экспрессию целевой нуклеиновой кислоты,
где клетка не является клеткой зародышевой линии человека.
| JINEK M | |||
| et al., A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity, Science, 2012, 337(6096): 816-821 | |||
| MALI P | |||
| et al., RNA-guided human genome engineering via Cas9, Science, 15 February 2013, 339(6121): 823-826 & Supplementary materials | |||
| US2010076057 A1, 25.03.2010 | |||
| DICARLO J.E | |||
| et al., Genome engineering in |
Авторы
Даты
2021-10-06—Публикация
2014-06-04—Подача