Изобретение относится к медицине и молекулярной биологии, а именно к методам молекулярно-генетического выявления РНК коронавируса SARS-CoV-2, а также количественной оценки вирусной нагрузки в аутопсийном, биопсийном и операционном материале при патологоанатомических исследованиях методом количественной полимеразной цепной реакции в реальном времени (кПЦР РВ).
Пандемия новой коронавирусной болезни 2019 года (COVID-19), вызванная новым коронавирусом SARS-CoV-2, стала причиной смерти более 2 миллионов людей по всему миру (https://coronavirus.jhu.edu/map.html). Основной причиной смерти пациентов с COVID-19 является острый респираторный дистресс синдром сочетающийся с мультиорганной недостаточностью [1], зачастую обусловленной внелегочной диссеминацией и непосредственным поражающим действием коронавируса SARS-CoV-2 на ткани жизненно важных органов [2]. Подтверждением тому является данные молекулярно-генетических исследований по выявлению РНК коронавируса SARS-CoV-2 в тканях различных органов как живых [3, 4], так и умерших от COVID-19 пациентов [5-7].
Однако, установление причинно-следственных связей патогенеза фатальных поражений жизненно важных органов, приведших к смерти пациентов с COVID-19, требует не только выявления инфицированности, но и количественной оценки вирусной нагрузки SARS-CoV-2 в тканях пораженных органов.
Тем не менее, для широкого применения молекулярно-генетических методов исследования РНК SARS-CoV-2 в тканях различных органов имеется ряд технических препятствий, связанных не только с соблюдением требований СП 1.3.3118-13 «Безопасность работы с микроорганизмами 1-11 групп патогенности (опасности)», но и отсутствием адаптированных способов количественной полимеразной цепной реакции в реальном времени (кПЦР РВ), позволяющей выявить и провести количественную оценку вирусной нагрузки SARS-CoV-2 в тканях различных органов.
В связи с этим разработка набора синтетических олигодезоксирибонуклеотидов и флуоресцентных зондов, а также способа количественной оценки вирусной нагрузки SARS-CoV-2 в тканях различных органов становится актуальной задачей молекулярно-генетической и патологоанатомической диагностики COVID-19.
Согласно Российским временным методическим рекомендациям по профилактике, диагностике и лечению новой коронавирусной инфекции (COVID-19) 9 версии от 26.10.2020 года, только образцы тканей, фиксированные в 10% нейтральном забуференном растворе формалина не менее 24 часов, могут считаться биологически безопасными [8].
Известен способ выявления коронавируса SARS-CoV-2 в образцах, фиксированных в формалине и заключенных в парафин тканей человека методом ОТ-ПЦР РВ, описанный в работах Tian и соавторов [9].
Известен способ, описанный в статье Sekulic и соавторов «Молекулярное определение инфекции SARS-CoV-2 в образцах FFPE и гистопатологические данные в случаях летального исхода SARS-CoV-2» [10].
Известен способ, описанный в статье Puelles и соавторов, посвященной исследованию мультиорганного и почечного тропизма коронавируса SARS-CoV-2 по данным посмертных исследований пациентов с COVID-19 [11].
Известен способ, описанный в статье Adachi и соавторов [12].
Вышеописанные способы анализа РНК коронавируса SARS-CoV-2 методом ПЦР РВ имеет ряд недостатков, заключающихся в нормирование образцов по концентрации общей РНК (где также присутствует человеческая мРНК с неизвестной пропорцией вирусной РНК), измеряемой спектрофотометрическим методом (также имеющий свои погрешности в измерениях, особенно в низких концентрациях). Еще одним недостатком является оценка результатов по значению порога циклов ПЦР РВ, что в случае с низкой концентрацией РНК SARS-CoV-2, может быть больше 40 циклов и может неправильно оцениваться как ложноотрицательный результат.
В 2020 году в РФ выданы несколько патентов на изобретение наборов олигонуклеотидов и флюоресцентных зондов, способов и тест систем для выявления РНК коронавируса SARS-CoV-2:
1. РФ №2731390 от 02.09.2020 года "Тест-система и способ для выявления РНК коронавируса SARS-CoV-2, вируса-возбудителя коронавирусного заболевания 2019 COVID-19, методом полимеразной цепной реакции в режиме реального времени (варианты)";
2. РФ №2733665 от 06.10.2020 года «Набор олигодезоксирибонуклетидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавирусов человека SARS и 2019-nCoV методом ОТ-ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени»;
3. РФ №2734300 от 14.10.2020 года «Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавируса человека 2019-nCoV методом ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени».
Однако запатентованные в РФ способы и тест системы имеют свои ограничения в техническом использовании, заключающиеся в аналитических характеристиках предлагаемых способов, т.е., все они является только "качественными" и не позволяют оценить вирусную нагрузку в исследуемом образце. Кроме того, совмещение ОТ реакции с ПЦР в одной пробирке, способствуют накоплению димеров используемых праймеров и снижению эффективности прохождения ПЦР [13-15]. Также, предложенные праймеры на мРНК генов GAPDH и АСТВ выбраны не совсем удачно, так как перекрывает и последовательности ДНК, которые всегда присутствует в качестве примеси в растворах РНК.
Наиболее близким к заявляемому способу является патент КНР CN111057797 от 24.04.2020 "Праймер, зонд, тест-набор и способ флуоресцентного количественного определения в реальном времени нового коронавируса 2019-nCoV". Предложенный способ рекомендован как для детекции, так и количественной оценки РНК коронавируса 2019-nCoV в смывах альвеолярной жидкости, мазках их носоглотки, в крови, сыворотке, фекалии и тканях пациентов с COVID-19. Тем не менее, согласно данным NCBI Blast (https://blast.ncbi.nlm.nih.gov/Blast.cgi) праймеры и зонд (5'-AGATTTGGACCTGCGAGCG-3'; 5'-gagcggctgtctccacaagt-3'; 5' HEXttctgacctgaaggctctgcgcg-3') подобрана на мРНК (NM_001104546.1 и NM_006413.4) человеческой рибонуклеазы. Из-за специфической функции фермента рибонуклеазы, по данным "The Human Protein Atlas" экспрессия гена RPP30 в различны тканях широко варьируется: от отсутствия до высокой (https://www.proteinatlas.org/ENSG00000148688-RPP30/tissue). Поэтому количественные данные копии РНК коронавируса 2019-nCoV относительно количества мРНК RPP30 в различных тканях являются неверными.
Кроме того, интерпретация вирусной нагрузки SARS-CoV-2 с привязкой значениям порогового цикла графической кривой ПЦР РВ является ошибочной и чревато получением "ложноотрицательных" результатов при низких концентрациях РНК коронавируса SARS-CoV-2 в исходном материале. Привязка числовых значений копии вируса к объему жидкости также невозможно из-за различной гигроскопичности используемых тупферов и отсутствия стандартизации техники забора биоматериала. Наиболее оптимальным в данном случае является привязка вирусной нагрузки к какому-либо эндогенному калибратору, например к числу копий мРНК клеток хозяина.
Таким образом, целью настоящего изобретения является разработка набора синтетических олигонуклеотидных праймеров и флюоресцентных зондов и способа количественной оценки вирусной нагрузки коронавируса SARS-CoV-2 в тканях различных органов методом кПЦР РВ.
Техническими задачами, решаемыми настоящим изобретением, являются:
1. Создание набора синтетических олигонуклеотидных праймеров и флюоресцентных зондов, необходимых для флуоресцентно-гибридизационной детекции РНК коронавируса SARS-CoV-2 в тканях различных органов;
2. Разработка способа количественной оценки вирусной нагрузки SARS-CoV-2 в тканях различных органов, основанного на измерении истинной копии РНК SARS-CoV-2 относительно мРНК контрольного гена домашнего хозяйства.
Техническим решением первой задачи стала разработка предлагаемого набора оригинальных синтетических олигонуклеотидных праймеров и флуоресцентных зондов для гена ORF1ab SARS-CoV-2 (https://www.ncbi.nlm.nih.gov/nuccore/NC_045512)
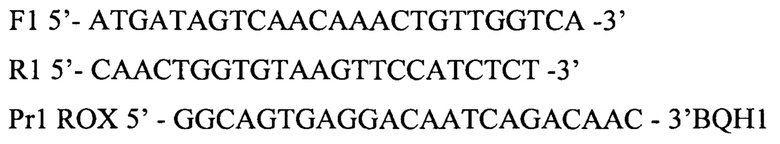
И ABL1 (https://www.ncbi.nlm.nih.gov/nuccore/NM_005157.6),
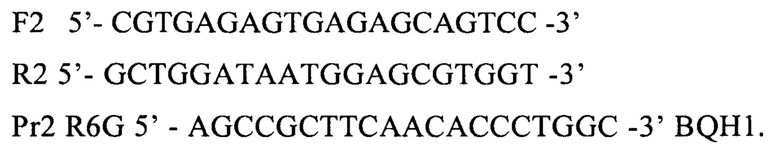
Решение второй технической задачи осуществлено в несколько этапов:
1. Разработан дополнительный набор синтетических олигонуклеотидов для получения ампликонов:
- длиной 406 нуклеотидов, охватывающий F1 и R1 участков гена ORF1ab SARS-CoV-2:
SARS-CoV-2 isolate Wuhan-Hu-1, NCBI Reference Seq. NC_045512.2
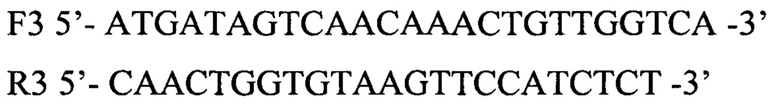
- и 301 нуклеотид, охватывающий комплементарные F2 и R2 праймерам участки ABL1:
Homo sapiens ABL proto-oncogene 1. NCBI Reference Seq. NM_005157.6

2. Полученные ампликоны путем генно-инженерной технологии вставлены в плазмидный вектор pGEM®-T Easy ("Promega" USA) и, после трансфекция и культивирования компетентных клеток, выделена плазмидная ДНК, согласно инструкции производителя. Схематическая конструкция плазмидного вектора pGEM®-T Easy [17] представлена в рисунке 1.
3. Подготовлены серийные разведения плазмидной ДНК с участком генома SARS-CoV-2 с концентрацией 100000, 10000, 1000, 100 и 50 копий в 10 мкл.
Также подготовлены серийные разведения плазмидной ДНК с участком кДНК ABL1 с концентрацией 60000, 6000, 500 и 70 копий в 10 мкл.
Количественная оценка вирусной нагрузка коронавируса SARS-CoV-2 в тканях различных органов производится относительно 100 копий контрольного гена ABL1 с использованием следующей формулы:
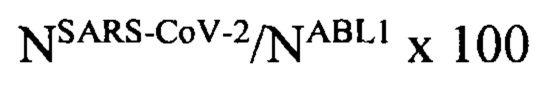
где NSARS-CoV-2 - число копий РНК SARS-CoV-2, NABL1 - число копий кДНК ABL1.
Результат анализа интерпретируется как число копий РНК SARS-CoV-2 на 100 копий кДНК ABL1.
Технический результат изобретения состоит в том, что предлагается новый способ количественной оценки вирусной нагрузки на основе измерения истинного числа копий кДНК коронавируса SARS-CoV-2 и ABL1, рассчитываемое по стандартной кривой серийных разведений копий плазмид pGEM®-T Easy со вставкой участков генома коронавируса SARS-CoV-2 и ABL1, методом количественной ПЦР РВ. Количественная оценка выражается в числе копий кДНК SARS-CoV-2 относительно 100 копий кДНК ABL1.
Сущность изобретения пояснена в следующих рисунках:
Рисунок 1. Схематическая структура плазмиды pGEM®-T Easy
Рисунок 2. Стандартная кривая ПЦР РВ и пороговые циклы разных концентраций копии ДНК плазмиды pGEM®-T Easy со встроенным участком кДНК SARS-CoV-2 содержащей 100000, 10000, 1000, 100 и 50 копий в 10 мкл раствора.
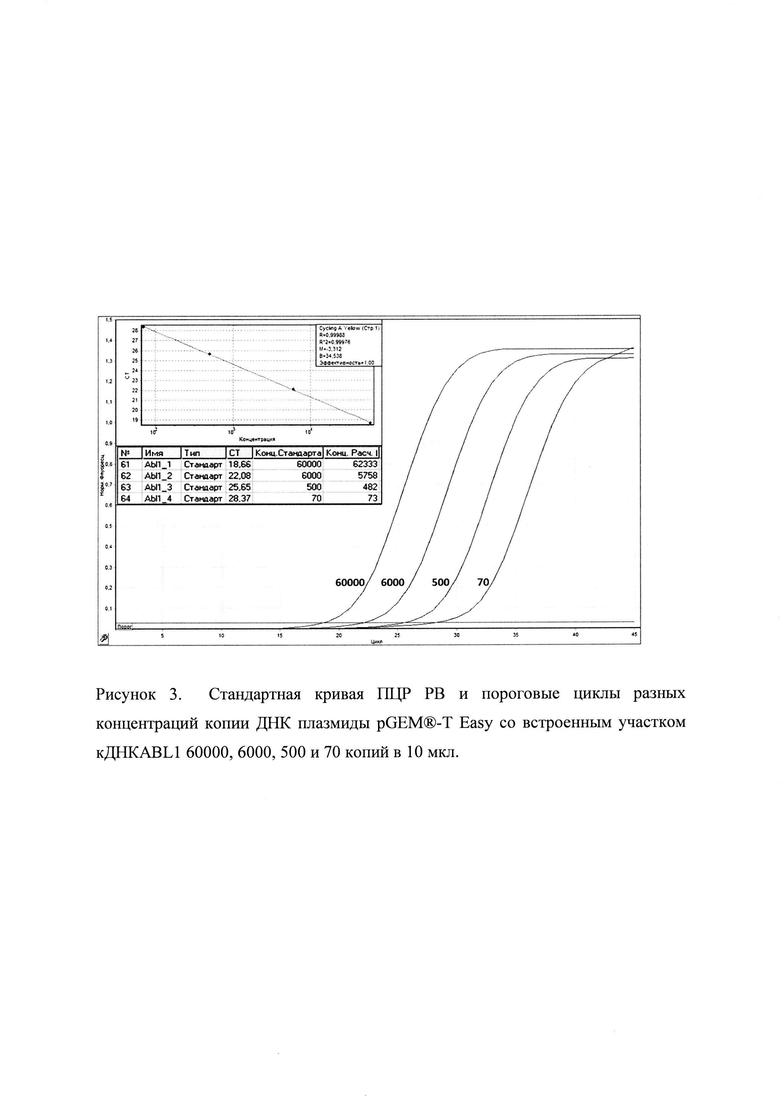
Рисунок 3. Стандартная кривая ПЦР РВ и пороговые циклы разных концентраций копии ДНК плазмиды pGEM®-T Easy со встроенным участком кДНК ABL1 60000, 6000, 500 и 70 копий в 10 мкл.
Рисунок 4. Число пороговых циклов и копий кДНК ABL1 в тканях различных органов пациента №16 с COVID-19.
Рисунок 5. Число пороговых циклов и копий кДНК коронавируса SARS-CoV-2 в тканях различных органов пациента №16 с COVID-19.
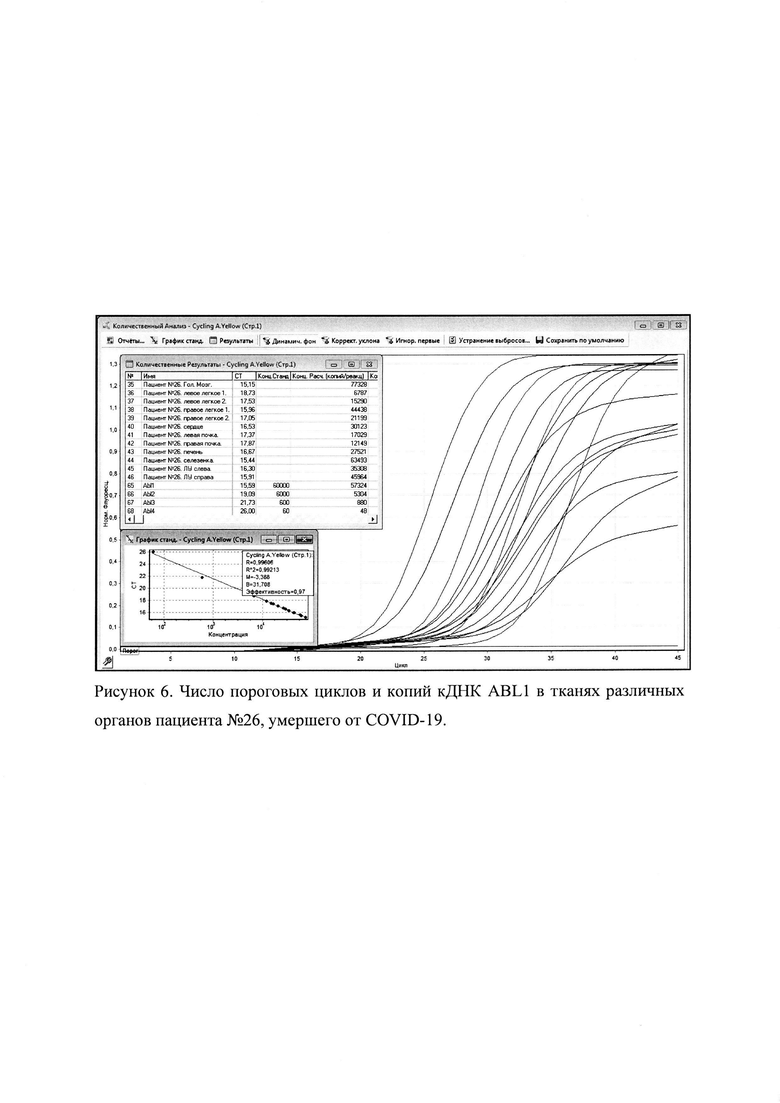
Рисунок 6. Число пороговых циклов и копий к ДНК ABL1 в тканях различных органов пациента №26, умершего от COVID-19.
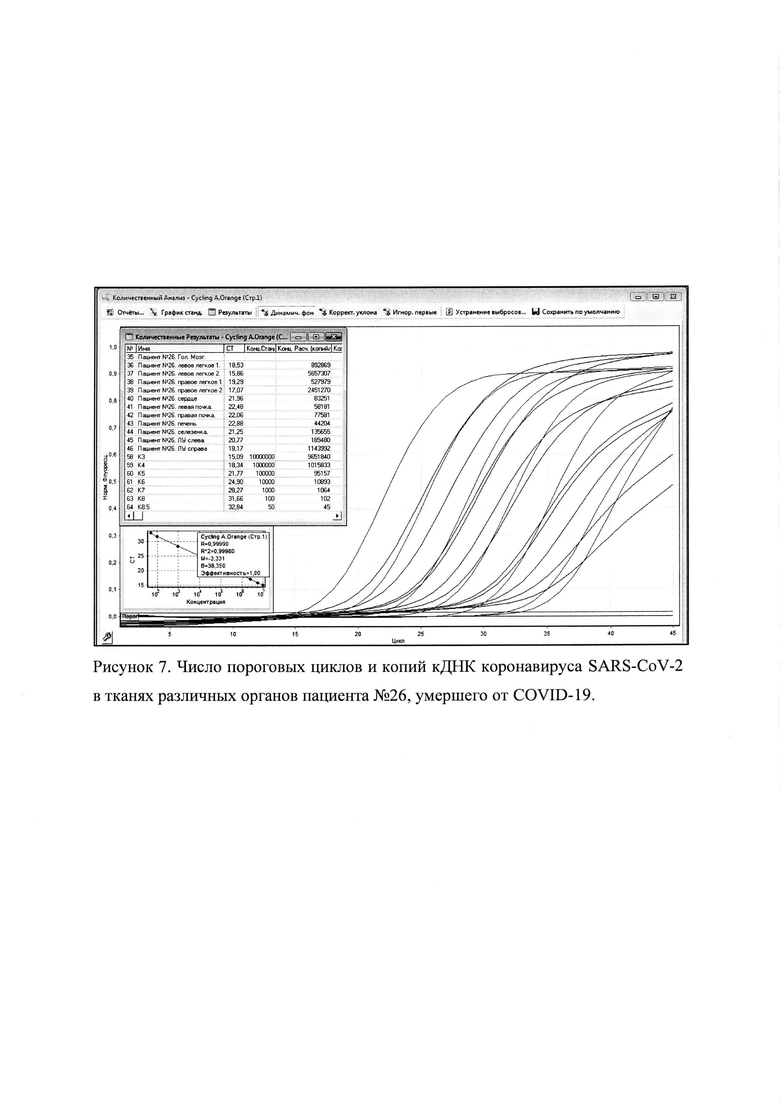
Рисунок 7. Число пороговых циклов и копий кДНК коронавируса SARS-CoV-2 в тканях различных органов пациента №26, умершего от COVID-19.
Также сущность изобретения пояснена следующими таблицами, где:
Таблица 1. Состав реакционной смеси для количественной ПЦР РВ
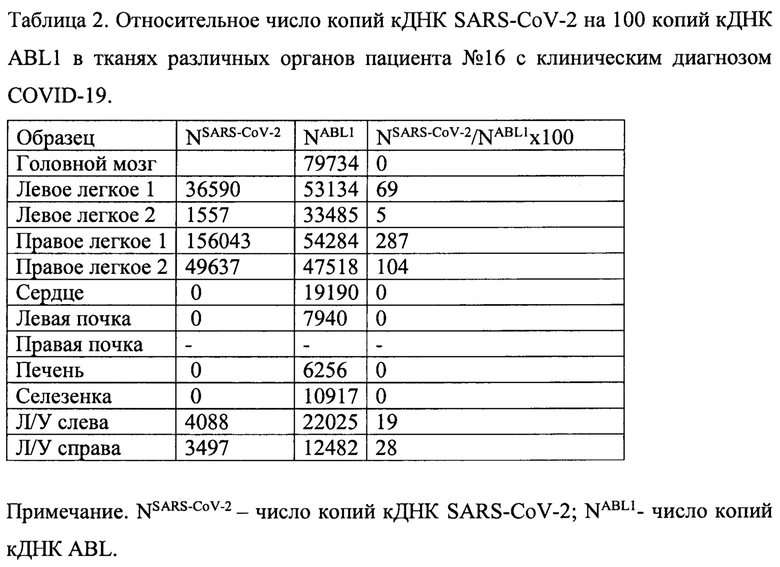
Таблица 2. Относительное число копий кДНК SARS-CoV-2 на 100 копий ABL1 в тканях различных органов пациента №16 с клиническим диагнозом COVID-19.
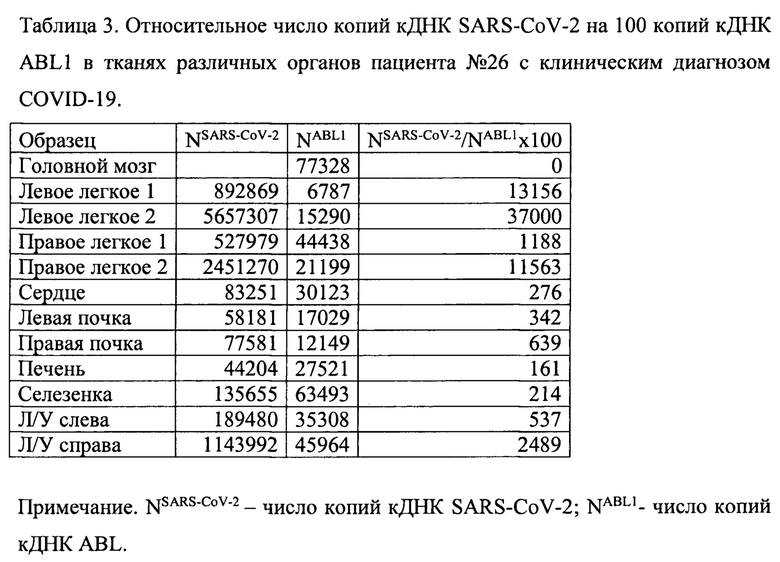
Таблица 3. Относительное число копий кДНК SARS-CoV-2 на 100 копий кДНК ABL1 в тканях различных органов пациента №26 с клиническим диагнозом COVID-19.
Сущность группы изобретений пояснена примерами конкретной реализации, которые не ограничивают объем изобретения.
Пример 1. Количественная ПЦР РВ с образцами серийных разведений плазмидной ДНК со вставкой участка кДНК SARS-CoV-2 и ABL1.
Количественную ПЦР РВ проводили в смеси реагентов с общим объемом 25 мкл, из них: реакционная смесь для проведения ПЦР-РВ с Taq ДНК-полимеразой и ингибирующими активность фермента антителами 2,5х («Синтол» Россия) -10 мкл, MgCl2 25 mM -1 мкл, Смесь олигонуклеотидов (F1, R1, F2, R2 по 10 пкмоль/2 мкл) - 2 мкл, Смесь меченых зондов (Pr1 и Pr2 по 5 пкмоль/2 мкл) -2 мкл. В качестве матрицы использованы образцы плазмидной ДНК с участком генома SARS-CoV-2 (с концентрацией 100.000, 10.000, 1000, 100 и 50 копий) и ABL1 (с концентрацией 60.000, 6000, 500 и 70 копий) в объеме 10 мкл. Состав и количество реагентов для кПЦР РВ представлены в таблице 1.
Способ кПЦР РВ проводили в амплификаторе "Rotor-Gene Q" («Qiagen», USA) в температурном режиме: 95°С - 600 с, 50 циклов 95°С - 15 с, 60°С - 60 с. Результаты флуоресцентной гибридизации кДНК отражается в оранжевом (SARS-CoV-2) и желтом (ABL1) канале амплификатора. Измерение истинной копии ДНК плазмиды со вставкой SARS-CoV-2 и ABL1 осуществляется специальной программой, для количественных исследований установленной в амплификатор.
Стандартная кривая ПЦР РВ и пороговые циклы разных концентраций копии ДНК плазмиды pGEM®-T Easy с различными (100000, 10000, 1000, 100 и 50) копиями SARS-CoV-2 и ABL1 (60000, 6000, 500 и 70) в 10 мкл, представлены на рисунке 2 и 3, соответственно. Как видно, из рисунков, коэффициент корреляции серийных разведений ДНК плазмиды со вставкой SARS-CoV-2 (оранжевый канал) и ABL1 (желтый канал) была равной R2 = 0,99906 и R2 = 0,99976, а эффективность показала 1,00 (100%). Предлагаемый способ обладает чувствительностью выявлять не менее 50 копий SARS-CoV-2 в исследуемом образце.
Пример 2. Количественная оценка вирусной нагрузки SARS-CoV-2 в тканях различных органов пациента №16, умершего с клиническим диагнозом COVID-19.
Из фиксированных в 10%-ном забуференном формалине и заключенных в парафиновые блоки тканей головного мозга, двух зон обоих легких, сердца, обеих почек, печени и селезенки, а также от правых и левых перибронхиальньгх лимфатических узлов были сделаны 8-10 срезов толщиной 3 мкм для молекулярно-генетического исследования коронавируса SARS-CoV-2.
Выделение РНК проводилось с использованием набора Pure Link™ FFPE ("Thermo Fisher Scientific", США), согласно инструкции производителя. В зависимости от размеров осадка, РНК была разведена в 15-30 мкл очищенной от РНКаз и ДНКаз воде. Для получения кДНК в объеме 20 мкл, проведена реакция обратной транскрипции с использованием 10 мкл раствора РНК и набора реагентов «Реверта-L» (компания «Интерлабсервис» Россия), в соответствие с инструкцией производителя. Для проведения кПЦР РВ была собрана смесь реактивов и исследуемой кДНК общим объемом 25 мкл, представленная в таблице 1.
В отдельные пробирки с реакционной смесью были внесены по 10 мкл положительных и отрицательных контрольных образцов (ПКО и ОКО), а также калибраторы SARS-CoV-2 и ABL1. кПЦР РВ проводили в амплификаторе "Rotor-Gene Q" («Qiagen», USA) в температурном режиме: 95°С - 600 с, 50 циклов 95°С - 15 с, 60°С - 60 с.
Значения пороговых циклов, коэффициент корреляции калибровочных разведений и эффективность прохождения ПЦР РВ представлены на рисунке 4-5.
Коэффициент корреляции по оранжевым (копии SARS-CoV-2) и желтым (ABL1) каналам составил R2 = 0,99963 и R2 = 0,99505, а эффективность была равна 1,00 и 1,06, соответственно.
Название исследованных органов и число копий кДНК SARS-CoV-2 и ABL1, а также копий кДНК SARS-CoV-2 относительно 100 копий кДНК ABL1, представлены в таблице 2.
Результаты количественной оценки вирусной нагрузки SARS-CoV-2 в легочной ткани пациента №16, показал, от 5 до 287 копий кДНК SARS-COV-2 на 100 копий ABL1. В левом и правом перибронхиальном лимфатическом узле значение вирусной нагрузки SARS-CoV-2 составил 19 и 28 копий кДНК SARS-CoV-2 на 100 ABL1. РНК коронавируса SARS-CoV-2 в образцах ткани сердца, почек, печени, селезенки и головного мозга не обнаружена.
Пример 3. Количественная оценка вирусной нагрузки SARS-CoV-2 в тканях различных органов пациента №26, умершего с клиническим диагнозом COVID-19.
Из фиксированных в 10%-ном забуференном формалине и заключенных в парафиновые блоки тканей головного мозга, двух зон обоих легких, сердца, обеих почек, печени и селезенки, а также от правых и левых перибронхиальных лимфатических узлов были сделаны 8-10 срезов толщиной 3 мкм для молекулярно-генетического исследования коронавируса SARS-CoV-2.
Выделение РНК проводилось с использованием набора Pure Link™ FFPE ("Thermo Fisher Scientific", США), согласно инструкции производителя. В зависимости от размеров осадка, РНК была разведена в 15-30 мкл очищенной от РНКаз и ДНКаз воде. Для получения кДНК в объеме 20 мкл, проведена реакция обратной транскрипции с использованием 10 мкл раствора РНК и набора реагентов «Реверта-L» (компания «Интерлабсервис» Россия), в соответствие с инструкцией производителя. Для проведения кПЦР РВ была собрана смесь реактивов и исследуемой кДНК общим объемом 25 мкл, представленная в таблице 1.
В отдельные пробирки с реакционной смесью были внесены по 10 мкл положительных и отрицательных контрольных образцов (ПКО и ОКО), а также калибраторы SARS-CoV-2 и ABL1. кПЦР РВ проводили в амплификаторе "Rotor-Gene Q" («Qiagen», USA) в температурном режиме: 95°С - 600 с, 50 циклов 95°С - 15 с, 60°С - 60 с.
Значения пороговых циклов, коэффициент корреляции калибровочных разведений и эффективность прохождения ПЦР РВ представлены на рисунке 6-7.
Коэффициент корреляции по оранжевым (копии SARS-CoV-2) и желтым (ABL1) каналам составил R2 = 0,99980 и R2 = 0,99213, а эффективность была равна 1,00 и 0,97, соответственно.
Название исследованных органов и число копий кДНК SARS-CoV-2 и ABL1, а также копий кДНК SARS-CoV-2 относительно 100 копий кДНК ABL1, представлены в таблице 3.
Результаты количественной оценки вирусной нагрузки SARS-CoV-2 в легочной ткани пациента №26 показал от 1188 до 37000, в ткани сердца - 276, почек - 342-639, печени - 161, селезенки - 214, и лимфатических узлов - 537-2489 копий кДНК SARS-CoV-2 на 100 копий ABL1. РНК коронавируса SARS-CoV-2 в образце ткани головного мозга не обнаружена.
Таким образом, предложенный набор синтетических олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов позволяют осуществить флуоресцентно-гибридизационную детекцию кДНК коронавируса SARS-CoV-2 и ABL1, присутствующие в исследуемом образце при анализе методом ПЦР РВ.
Серийные разведения плазмидной ДНК, содержащие участки кДНК SARS-CoV-2 и ABL1 с высокой корреляцией линейных разведений, равной R2≥0,99, позволит создание кривой ПЦР РВ, относительно которой производится расчет истинных копий кДНК SARS-CoV-2 с чувствительностью не менее 50 копий в исследуемом образце.
Предложенный способ количественной оценки вирусной нагрузки методом кПЦР РВ способствует не только детекции, но и измерению истинной копии РНК SARS-CoV-2 в исследуемых образцах тканей различных органов с использованием следующей формулы:

где NSARS-CoV-2 - число копий РНК SARS-CoV-2, NABL1 - число копий кДНК человеческой ABL1.
Результат анализа интерпретируется как число копий РНК SARS-CoV-2 относительно 100 копий кДНК ABL1.
Литературные источники:
1. Chen N, Zhou М, Dong X, et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study. Lancet. 2020; 395(10223):507-513. doi: 10.1016/S0140-673 6(20)30211-7
2. Rieg S, von Cube M, Kalbhenn J, et al. COVID-19 in-hospital mortality and mode of death in a dynamic and non-restricted tertiary care model in Germany. PLoS One. 2020;15(11):e0242127. Published 2020 Nov 12. doi:10.1371/joumal.pone.0242127
3. Guerini- Rocco E, Taormina SV, Vacirca D, et al. J Clin Pathol 2020; 73:754-757
4. Maria Casagrande, Antonia Fitzek, Klaus Puschel, Ganna Aleshcheva, Heinz-Peter Schultheiss, Laura Bemeking, Martin S. Spitzer & Maximilian Schultheiss (2020) Detection of SARS-CoV-2 in Human Retinal Biopsies of Deceased COVID-19 Patients, Ocular Immunology and Inflammation, 28:5, 721-725, DOI: 10.1080/09273948.2020.1770301
5. Hanley В, Naresh KN, Roufosse C, et al. Histopathological findings and viral tropism in UK patients with severe fatal COVID-19: a post-mortem study. Lancet Microbe. 2020; 1(6):e245-e253. doi:10.1016/S2666-5247(20)30115-4
6. Sekulic M, Harper H, Nezami BG, et al. Molecular Detection of SARS-CoV-2 Infection in FFPE Samples and Histopathologic Findings in Fatal SARS-CoV-2 Cases. Am J Clin Pathol. 2020; 154(2):190-200. doi:10.1093/ajcp/aqaa091
7. Bradley ВТ, Maioli H, Johnston R, et al. Histopathology and ultrastructural findings of fatal COVID-19 infections in Washington State: a case series [published correction appears in Lancet. 2020 Aug 1; 396(10247):312]. Lancet. 2020; 396(10247):320-332. doi:10.1016/S0140-6736(20)31305-2
8. Временные методические рекомендации «Профилактика, диагностика и лечение новой коронавирусной инфекции (COVID-19)». Версия 9 (26.10.2020) https://static-0.minzdrav.gov.ru/system/attachments/attaches/000/052/550/original/%D0%9C%D0%A0_COVID-19_%28v9%29.pdf?1603788097
9. Tian S, Xiong Y, Liu H, Niu L, Guo J, Liao M, Xiao SY. Pathological study of the 2019 novel coronavirus disease (COVID-19) through postmortem core biopsies. Mod Pathol. 2020 Jun; 33(6):1007-1014. doi: 10.1038/s41379-020-0536-x. Epub 2020 Apr 14. PMID: 32291399; PMCID: PMC7156231
10. Sekulic M, Harper H, Nezami BG, Shen DL, Sekulic SP, Koeth AT, Harding CV, Gilmore H, Sadri N. MolecularDetectionof SARS-CoV-2 Infectionin FFPE SamplesandHistopathologicFindingsinFatal SARS-CoV-2 Cases. Am J Clin Pathol. 2020 Jul 7; 154(2): 190-200. doi: 10.1093/ajcp/aqaa091. PMID: 32451533; PMCID: PMC7314275
11. Puelles VG, Liitgehetmann M, Lindenmeyer MT, etal. Multiorgan and Renal Tropism of SARS-CoV-2. N Engl J Med. 2020; 383(6):590-592. doi:10.1056/NEJMc2011400
12. Adachi T, Chong JM, Nakajima N, etal. Clinicopathologic and Immunohistochemical Findings from Autopsy of Patient with COVID-19, Japan. EmergInfectDis. 2020; 26(9):2157-2161. doi:10.3201/eid2609.201353
13. Vandesompele, J., De Preter, K., Pattyn, F. et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol3, research0034.1 (2002). https://doi.org/10.1186/gb-2002-3-7-research0034
14. Wong ML, Medrano JF. Real-time PCR for mRNA quantitation. Biotechniques. 2005 Jul; 39(1):75-85. doi: 10.2144/05391RV01. PMID: 16060372
15. Bustin SA. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. J Mol Endocrinol. 2002 Aug; 29(1):23-39. doi: 10.1677/jme.0.0290023.PMID: 12200227

| название | год | авторы | номер документа |
|---|---|---|---|
| Набор олигонуклеотидов и способ мультиплексной полимеразной цепной реакции в режиме реального времени для выявления РНК SARS-CoV-2 | 2021 |
|
RU2752902C1 |
| Тест-система и способ для выявления РНК коронавируса SARS-COV-2, вируса-возбудителя коронавирусного заболевания 2019 COVID-19, методом полимеразной цепной реакции в режиме реального времени (Варианты) | 2020 |
|
RU2731390C1 |
| Способ выявления и количественного определения вируса SARS-COV-2 с помощью цифровой капельной ПЦР | 2024 |
|
RU2839700C1 |
| Набор олигодезоксирибонуклетидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавирусов человека SARS и 2019-nCoV методом ОТ-ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2020 |
|
RU2733665C1 |
| Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации РНК коронавируса человека SARS-CoV-2 методом изотермической ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2021 |
|
RU2778855C1 |
| Набор для выявления вируса SARS-CoV методом ОТ-ПЦР в реальном времени | 2020 |
|
RU2744198C1 |
| Набор реагентов для выявления РНК вируса SARS-CoV-2 методом прямой полимеразной цепной реакции в режиме реального времени | 2022 |
|
RU2795939C2 |
| Набор реагентов для выявления РНК вируса SARS-CoV-2, возбудителя нового коронавирусного заболевания COVID-2019, методом обратной транскрипции-полимеразной цепной реакции в реальном времени | 2020 |
|
RU2732608C1 |
| Олигонуклеотиды для определения мутации S:L452R SARS-CoV-2 | 2022 |
|
RU2795018C1 |
| Олигонуклеотиды для определения мутации S:delHV69-70 SARS-CoV-2 | 2022 |
|
RU2795014C1 |




Изобретение относится к медицине и молекулярной биологии, а именно к методам молекулярно-генетического выявления РНК коронавируса SARS-CoV-2, а также количественной оценки вирусной нагрузки в аутопсийном, биопсийном и операционном материале при патологоанатомических исследованиях методом количественной полимеразной цепной реакции в реальном времени (кПЦР РВ). Описан набор праймеров и зондов для идентификации кДНК коронавируса. Также описан способ измерения вирусной нагрузки SARS-CoV-2 в тканях различных органов методом количественной полимеразной цепной реакции в реальном времени, который основан на выявлении числа копий кДНК SARS-CoV-2 относительно 100 копий кДНК ABL1. Технический результат - повышение достоверности количественной оценки. Серийные разведения плазмидной ДНК pGEM®)-T Easy, содержащие участки кДНК SARS-CoV-2 и ABL1, позволяют выявить с высокой корреляцией линейных разведений, равной R2≥0,99. Разработанные олигонуклеотидные праймеры и флуоресцентно-меченые зонды могут быть использованы для детекции и количественной оценки вирусной нагрузки SARS-CoV-2 в тканях различных органов. 2 н.п. ф-лы, 7 ил., 3 табл., 3 пр.
1. Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации кДНК коронавируса SARS-CoV-2 (F1, R1, Pr1) и ABL1 (F2, R2, Pr2) методом ПЦР РВ со следующей нуклеотидной последовательностью:
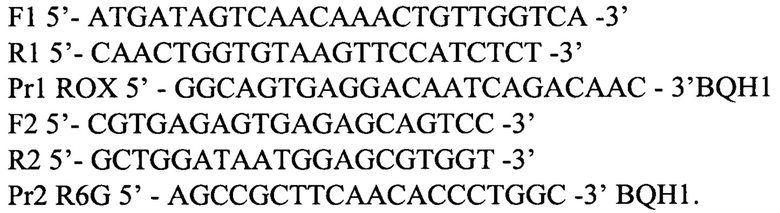
2. Способ количественной оценки вирусной нагрузки SARS-CoV-2 в различных тканях пациентов с COVID-19 с использованием набора олигонуклеотидных праймеров и флуоресцентно-меченых зондов по п.1, включающий в себя этапы выделения РНК, получения кДНК и проведения количественной ПЦР РВ, отличающийся тем, что истинная копия кДНК SARS-CoV-2 и ABL1 вычисляется относительно стандартной кривой, построенной на основе серийных разведений плазмидного вектора pGEM®-T Easy со встроенным участком кДНК SARS-CoV-2 и участком кДНК ABL1, с корреляцией R2≥0,99, вирусная нагрузка выражается числом копий кДНК SARS-CoV-2 на 100 копий референсного гена ABL1 по формуле:
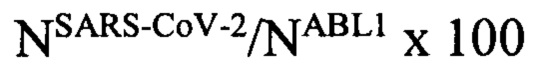 ,
,
где NSARS-CoV-2 - число копий кДНК SARS-CoV-2,
NABL1 - число копий кДНК человеческой ABL1,
с чувствительностью не ниже 50 копий кДНК SARS-CoV-2 в исследуемом образце тканей различных органов.
| Набор реагентов для выявления РНК вируса SARS-CoV-2, возбудителя нового коронавирусного заболевания COVID-2019, методом обратной транскрипции-полимеразной цепной реакции в реальном времени | 2020 |
|
RU2732608C1 |
| НАБОР СИНТЕТИЧЕСКИХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ ВЫЯВЛЕНИЯ РНК КОРОНАВИРУСА | 2020 |
|
RU2720713C9 |
| CN 111139317 A, 12.05.2020 | |||
| Puelles V.G., Liitgehetmann M., Lindenmeyer M.T | |||
| et al | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| N Engl J Med | |||
| Способ восстановления спиралей из вольфрамовой проволоки для электрических ламп накаливания, наполненных газом | 1924 |
|
SU2020A1 |
Авторы
Даты
2021-12-07—Публикация
2021-02-05—Подача