Изобретение относится к биотехнологии, а именно к генетической инженерии, и может быть использовано в ветеринарной вирусологии для диагностики инфекционных заболеваний сельскохозяйственных животных, в частности, коронавирусной инфекции крупного рогатого скота, а также для идентификации вирусов, выявленных у крупного рогатого скота.
Возбудитель коронавирусной инфекции крупного рогатого скота (Bovine Coronavirus (BCoV)) является РНК-содержащим вирусом, относящимся к виду Betacoronavirus 1, роду Betacoronavirus семейства Coronaviridae [Taxonomy browser (Betacoronavirus 1). www.ncbi.nlm.nih.gov.]. Вирион имеет оболочку, диаметром 120 нм, а геном представлен одноцепочечной (+) РНК размером 27-32 Кб.
К виду Betacoronavirus 1 кроме BCoV относятся Canine respiratory coronavirus, Dromedary camel coronavirus HKU23, Equine coronavirus, Human coronavirus OC43 (HCoV-OC43), Porcine hemagglutinating encephalomyelitis virus, Yak coronavirus (https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Tree&id=694003&lvl=3&lin=f&keep=l&srchmode=l&unlock). На основании изучения биологических, антигенных и генетических характеристик было предложено классифицировать эти вирусы как варианты по диапазону хозяев, а не как отдельные виды вирусов (Bovine-like coronaviruses in domestic and wild ruminants. Amer HM. Anim Health Res Rev. 2018 Dec; 19(2): 113-124).
Геном вируса кодирует пять структурных белков: нуклеокапсид (N), трансмембранный белок (М), гемагглютининэстераза (НЕ), шип (S) и малые мембранные (Е) белки. Шип S состоит из двух субъединиц (S1 и S2) и играет важную роль во взаимодействии вируса с клеткой хозяина. Ген нуклеокапсида N представляет наиболее консервативную область генома и считается лучшей мишенью для обнаружения геномной РНК.
Инфекции BCoV связаны с диарейным синдромом у новорожденных телят, инфекциями дыхательных путей у молочных телят и откормочного скота, а также зимней дизентерией у взрослых животных. Несмотря на некоторые различия в гене шипа (S) между кишечными и респираторными изолятами, антигенные или генетические маркеры, позволяющие различать их между собой, до сих пор не выявлены.
Геном BCoV на 98% совпадает с геномом штамма вируса HCoV-ОС43, выделенного от человека и вызывающего 10-15% всех сезонных респираторных инфекции у людей. Недавние исследования показали, что BCoV и HCoV-OC43 имеют общего предка, a HCoV-OC43 выделяли от крупного рогатого скота при респираторных болезнях, которые протекали в легкой форме. Также есть сообщения о выделении BCoV от человека при кишечной форме, от собак и лошадей - при респираторном заболевании (Lim, Y.X.; Ng, Y.L.; Tam, J.P.; Liu, D.X. Human Coronaviruses: A Review of Virus-Host Interactions. Diseases. 2016; 4: 26).
Таким образом, при проведении диагностических мероприятий для определения этиологии вспышек респираторной инфекции у крупного рогатого скота в конкретном хозяйстве, помимо выявления BCoV, необходимо также идентифицировать HCoV-OC43, в короткие сроки, наиболее экономичным и эффективным способом. Однако существующие в настоящее время методы диагностики не позволяют этого сделать.
Широко используется на практике иммунохроматографический тест для качественного выявления коронавируса крупного рогатого скота (D. Klein, A. Kern, G. Lapan, V. Benetka, K. Mostl, A. Hassl, W. Baumgartner. Evaluation of rapid assays for the detection of bovine coronavirus, rotavirus A and Cryptosporidium parvum in faecal samples of calves. Vet J. 2009 Dec; 182(3):484-6). При высокой специфичности (95,7%) этот метод обладает низкой эффективностью (65%), применяется только для выявления вируса в пробах кала и не способен идентифицировать различные генетические его варианты.
До настоящего времени основным способом выявления коронавирусов и их идентификации является выделение вируса в культуре клеток с последующим типированием с помощью специфических сывороток. Выделение BCoV в культуре клеток затруднено ввиду его чрезвычайной лабильности и возможно только на ранней стадии инфекции (Сюрин В.Н., Самуйленко А.Я., Соловьев Б.В., Фомина Н.В. Вирусные болезни животных. - М.: ВНИТИБП. - 1998. - 928 с.). Таким образом, основным недостатком выделения BCoV в культуре клеток и определения его варианта является длительность осуществления процедуры до одного и более месяца.
Известен способ секвенирования всего генома вируса. Для этого необходимо выделить вирус в культуре клеток, провести амплификацию с 6 парами праймеров (1F TGCATCCCGCTTCACTGATCTCTT, 1R CACCTACAGGCACAAACCTGTTAGTG, 2F GCACTTGATGATGA-TGCACGAACC, 2R GGTGGTTGGTAAAGC АС АТС ACT АСС, 3F GCCACGCTTTATTTCCCTTGG-GAGAT, 3R GAACACCACGTGTAGCTGCTAT-AC, 4F GAGGAGCAGGATGAAA-TTTACGCC, 4R ACATCCTGACCCT-CTTTACGCACA, 5F GACGTGTGTTGGAA-GCACGTCATTTG, 5R GTAAAAACACCTGCAGGTTGAGGC, 6F GTGCGAGTACCCA-CATACGATTTG, 6R CTGGGTGGTAACTTAACATGCTGG) для синтеза 6 перекрывающихся фрагментов генома вируса и провести их секвенирование (Genomic Characterization and Phylogenetic Classification of Bovine Coronaviruses Through Whole Genome Sequence Analysis. Suzuki T, Otake Y, Uchimoto S, Hasebe A, Goto Y. Viruses. 2020 Feb 6; 12 (2): 183).
Однако этот способ очень длительный, дорогостоящий и не подходит для рутинной лабораторной диагностики заболевания.
Наиболее приемлемым с точки зрения описанных недостатков и принятым за прототип является способ, основанный на применении полу гнездовой ПЦР и последующего секвенирования ампликонов. Для обнаружения BCoV проводят полу гнездовую ОТ-ПЦР с использованием видоспецифических праймеров на ген, кодирующий протеин нуклеокапсида N. На первом этапе реакцию проводят с праймерами BCoV2 (5'AGAGCTCAAYCCAAGCAA ACTGY 3') и BCoV2 (5'AG CAGACCTTCCTGAGCCTTCAAT 3'), а на втором этапе с праймерами BCoV1l (5'AGAGCTCAAYCCAAGCAA ACTGY 3') и BCoV3 (5'TCAATRTCGGTGCC ATACTGGTCT 3') с последующим секвенированием ампликонов для определения их последовательности и построения филогенетического дерева. (Genetic diversity of BCoV in Brazilian cattle herds. de Mira Fernandes A, Brandao PE, Dos Santos Lima M, de Souza Nunes Martins M, da Silva TG, da Silva Cardoso Pinto V, de Paula LT, Vicente MES, Okuda LH, Pituco EM. Vet Med Sci. 2018 Apr 24; 4(3): 183-189).
К недостаткам данного способа можно отнести то, что реакция проводится в электрофоретическом формате и требуется длительное время для постановки диагноза.
Технической задачей изобретения являлась разработка высокоспецифичного и чувствительного способа на основе полимеразной цепной реакции (ПЦР) с использованием гибридизационно-флуоресцентной детекции в режиме реального времени, позволяющего выявлять и проводить типирование BCoV и HCoV-OC43 в пробах биологического материала различного происхождения.
Сущность изобретения заключается в том, что предложены синтетические олигонуклеотидные праймеры и зонды для выявления РНК BCoV и HCoV-OC43, согласно изобретению, синтетические олигонуклеотидные праймеры и зонды имеют нуклеотидные последовательности: SEQ ID NO: 1-7.
Сущность изобретения заключается также в том, что в способе выявления и идентификация коронавирусов у крупного рогатого скота, включающем выделение РНК из проб образцов культур клеток, эмбриональных сывороток и проб биоматериала от крупного рогатого скота, проведение обратной транскрипции и полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени, согласно изобретения, используют синтетические олигонуклеотидные праймеры и зонды SEQ ID NO: 1 - 7, исследование каждой пробы проводят методом мультиплексной ПЦР для одновременного выявления штаммов и изолятов вируса BCoV и HCoV-OC43.
Изобретение иллюстрируется следующими примерами.
Пример 1. Получение синтетических олигонуклеотидных праймеров
Поиск новых синтетических олигонуклеотидных праймеров и зондов осуществляют на основе фрагментов гена нуклеокапсида N вариантов BCoV и HCoV-OC43, представленных в базе данных GenBank (http://www.ncbi.nlm.nih.gov/GenBank/GenBankSearch.html), при помощи пакета программного обеспечения «Lasergen». Полученные последовательности нескольких пар праймеров и зондов дополнительно тестируют на специфичность с помощью моделирования ПЦР в программе "Vector NTI Suite" с последовательностями генома других представителей семейства Coronaviridae.
Окончательный выбор праймеров и зондов основывается на следующих критериях: высокий индекс сходства фрагмента и РНК различных штаммов BCoV и HCoV-OC43, высокая температура отжига (GC-метод), большая длина консенсусов. Данные праймеры и зонд должны быть комплементарны только специфическим вариантам.
Химический синтез праймеров и зондов осуществляют амидофосфитным методом на автоматическом синтезаторе ASM-102U. Концентрацию синтетических олигонуклеотидных праймеров и зондов в маточном растворе определяют спектрометрическим методом.
Характеристики выбранных праймеров и зондов представлены в таблице 1.

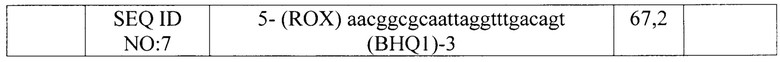
Таким образом, были выбраны синтетические олигонуклеотидные праймеры и зонды, комплементарные высоко консервативной области генома вирусов BCoV и HCoV-OC43 района гена протеина нуклеокапсида N.
Пример 2. Способ выявления РНК и типирования вариантов коронавируса крупного рогатого скота
Способ осуществляется в несколько этапов.
Этап 1. Выделение РНК вируса
Выделение РНК осуществляют стандартным способом с использованием коммерческого набора «Рибо - сорб» производства ФГУН «Центральный НИИ эпидемиологии» Роспотребнадзора.
Этап 2. Проведение реакции обратной транскрипции для получения кДНК.
Осуществляют стандартным способом с использованием коммерческого набора «Реверта-L» производства того же института.
В пробирку, содержащую 9,5 мкл реакционной смеси: буфер для ОТ (50 mM Tris-HCl [рН 8.3], 3 мМ MgCl2, 75 mM KCl, 10 мМ DTT), 0,1 мМ dNTP, 0,1 мкг праймера для ОТ) и 0,5 мкл ревертазы из набора «Реверта-L», добавляют 10 мкл РНК-пробы, осторожно перемешивают и помещают в термостат 37°С на 30 минут. Затем добавляют 20 мкл ДНК-буфера, тщательно перемешивают и используют для постановки ПЦР.
Этап 3. Постановка полимеразной цепной реакции
Полимеразная цепная реакция. Состав реакционной смеси: ПЦР-буфер (60 mM Tris-HCl [рН 8.5], 1,5 мМ MgCl2, 25 mM KCl, 10 мМ 2-меркаптэтанола, 0,1% Тритон Х-100), 0,2 мМ dNTP, по 0,2 мкг каждого праймера, по 0,1 мкг каждого зонда, 1.25 U Taq-ДНК-полимеразы, 5 мкл кДНК.
Температурный режим проведения ПЦР: 95°С - 5 мин - 1 цикл; 95°С - 15 сек, 60°С - 60 сек - 55 циклов.
Измерение флуоресценции осуществляют при температуре 60°С на канале FAM для BCoV и на канале ROX для HCoV-OC43. Положительными считают образцы со значением Ct не превышающим 45.
Пример 3. Оценка чувствительности и специфичности выявление РНК вариантов вирусов BCoV и HCoV-OC43 в пробах биологического материала, полученного от больных и инфицированных животных и референтных штаммах
Для оценки контрольный штамм вируса BCoV «КМИЭВ-1» с титром 106,5 ТЦД50/см3 титруют методом 10-кратных разведений на физиологическом растворе до разведения 10-7 степени, каждое разведение подвергают исследованию в ПЦР.
Для исследования на наличие РНК вирусов BCoV и HCoV-OC43 используют пробы носовых выделений, смывы со слизистой носа, соскобы со слизистой трахеи, трахеальную и бронхиальную слизь, пробы легких и бронхов, пробы кала, пробы кишечника, лимфоидные органы, кровь.
Жидкости и пробы слизистых выделений, кала отбирают в объеме не менее 1 мл, из органов вырезают кусочки размером 1x1x1 см3.
Культуральную жидкость, пробы штаммов и изолятов BCoV, HCoV-ОС43, пробы носовых и глазных выделений, трахеальной и бронхиальной слизи, крови используют для выделения РНК без предварительной подготовки.
Из проб органов, тканей предварительно готовят 10% суспензии на стерильном физиологическом растворе, смесь переносят в пластиковые пробирки емкостью 1,5 мл, центрифугируют на центрифуге при 10x103 об/мин в течение 5 мин. Для выделения РНК используют 100 мкл осветленной надосадочной жидкости, полученной после центрифугирования.
Густые пробы носовых выделений, слизи и кал разводят стерильным физиологическим раствором (примерно 1:5), тщательно перемешивают, центрифугируют при 10x103 об/мин в течение 5 мин и 100 мкл осветленной надосадочной жидкости используют для выделения РНК.
Дальнейшая процедура согласно примеру 2 и 3.
Чувствительность реакции при тестировании десятикратных разведений контрольного штамма BCoV «КМИЭВ-1» с заранее определенной концентрацией, равной 106,5ТЦД50/см3, составила 100,5ТЦД50/см3.
С использованием разработанного набора исследовали 1508 проб биоматериала от 116 животных. BCoV выявили в 178 пробах от 30 животных, что составило 11,8% от общего числа исследованных проб, а HCoV-OC43 в 12 пробах, что составило 0,8% от числа исследованных проб.
Для подтверждения специфичности полученных фрагментов РНК определяли их нуклеотидную последовательность с использованием набора BigDye 3.1 (Applied Biosystems, США) с последующей очисткой на сефадексе G-50 superfine. Секвенирование ПЦР-фрагментов проводили по обеим цепям ДНК. Расшифровку первичных данных секвенирования (хроматограмм) проводили с помощью программы Sequencher v.4.0.5 (Gene Codes Corporation, США).
Анализ нуклеотидных последовательностей синтезируемых фрагментов проводили методами выравнивания с опубликованными последовательностями других штаммов короновируса крупного рогатого скота с помощью программы BioEdit 7.0.0. Построение дендрограмм проводили при помощи метода максимальной эволюции в программе MEGA 7. Для оценки достоверности топологии использовали бутстрэп-тест (1000 реплик) (Рисунок 1).
Результаты секвенирования и филогенетического анализа показали принадлежность выявленных участков генома BCoV и HCoV-OC43 и подтвердили достоверность результатов, полученных с помощью разработанного нами метода.
Таким образом, предлагаемый способ обладает высокой чувствительностью, специфичностью и эффективностью при выявлении и идентификации BCoV и HCoV-OC43 в пробах биологического материала от животных.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления возбудителей респираторных инфекций крупного рогатого скота: BPIV, BRSV, BHV-4, BCoV, BVDV-1, BVDV-2, BVDV-3, на основе мультиплексной полимеразной цепной реакции (ПЦР) | 2022 |
|
RU2798286C1 |
| Тест-система для обнаружения генома возбудителя коронавирусной инфекции у крупного рогатого скота с помощью мультиплексной полимеразной цепной реакции с флуоресцентной детекцией в режиме реального времени | 2018 |
|
RU2694499C1 |
| Набор реагентов для выявления РНК вируса SARS-CoV-2, возбудителя нового коронавирусного заболевания COVID-2019, методом обратной транскрипции-полимеразной цепной реакции в реальном времени | 2020 |
|
RU2732608C1 |
| Способ выявления генома возбудителя коронавирусной инфекции у крупного рогатого скота | 2018 |
|
RU2694558C1 |
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавируса человека 2019-nCoV методом ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2020 |
|
RU2734300C1 |
| Набор олигонуклеотидов и способ мультиплексной полимеразной цепной реакции в режиме реального времени для выявления РНК SARS-CoV-2 | 2021 |
|
RU2752902C1 |
| НАБОР ОЛИГОДЕЗОКСИРИБОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО МЕЧЕНЫХ ЗОНДОВ ДЛЯ ИДЕНТИФИКАЦИИ РНК КОРОНАВИРУСОВ ВИДОВ 229Е, NL63, ОС43, HKU1 МЕТОДОМ ГИБРИДИЗАЦИОННО-ФЛУОРЕСЦЕНТНОЙ ОБРАТНО-ТРАНСКРИПТАЗНОЙ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2011 |
|
RU2473702C1 |
| Набор олигодезоксирибонуклетидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавирусов человека SARS и 2019-nCoV методом ОТ-ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2020 |
|
RU2733665C1 |
| Тест-система для выявления SARS-CoV-2 линии Омикрон методом одношаговой полимеразной цепной реакции с обратной транскрипцией | 2021 |
|
RU2772362C1 |
| Набор реагентов для выявления РНК вируса SARS-CoV-2 методом прямой полимеразной цепной реакции в режиме реального времени | 2022 |
|
RU2795939C2 |

Изобретение относится к области биотехнологии. Предложены синтетические олигонуклеотидные праймеры и зонды и способ выявления и идентификации биологических вариантов коронавирусов крупного рогатого скота с помощью синтетических олигонуклеотидных праймеров и зондов на основе полимеразной цепной реакции (ПЦР) с использованием гибридизационно-флуоресцентной детекции в режиме реального времени. Предложенный способ включает выделение РНК, проведение обратной транскрипции, исследование каждой пробы проводят методом мультиплексной ПЦР для одновременного выявления двух вариантов вируса BCoV и HCoV-OC43 по гену протеина нуклеокапсида N. Способ обладает высокой чувствительностью, специфичностью и эффективностью при выявлении и идентификации BCoV и HCoV-OC43 в пробах биологического материала от животных. 2 н.п. ф-лы, 1 табл., 1 ил.
1. Набор для выявления и идентификации коронавирусов BCoV и HCoV-OC43 у крупного рогатого скота, согласно изобретению, синтетические олигонуклеотидные праймеры и зонды имеют нуклеотидные последовательности: SEQ ID NO: 1-7.
2. Способ выявления и идентификации коронавирусов BCoV и HCoV-ОС43 у крупного рогатого скота, включающий выделение РНК из проб образцов культур клеток, эмбриональных сывороток и проб биоматериала от крупного рогатого скота, проведение обратной транскрипции и полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени, отличающийся тем, что используют синтетические олигонуклеотидные праймеры и зонды SEQ ID N0:1 - 7, исследование каждой пробы проводят методом мультиплексной ПЦР для одновременного выявления штаммов и изолятов двух вариантов вируса BCoV и HCoV-OC43.
| DE MIRA FERNANDES A | |||
| et al | |||
| Genetic diversity of BCoV in Brazilian cattle herds, Vet Med Sci, 2018, Apr 24 (3), p | |||
| Переносная мусоросжигательная печь-снеготаялка | 1920 |
|
SU183A1 |
| SUZUKI T | |||
| et al | |||
| Genomic characterization and Phylogenetic classification of Bovine coronaviruses through whole genome sequence analysis, Viruses, 2020 Feb 6, 12 (2), p.183 | |||
| КУДРЯВЦЕВ В.А | |||
| Использование ПЦР и секвенирования | |||
Авторы
Даты
2022-03-15—Публикация
2021-07-08—Подача