Изобретение относится к биотехнологии, а именно к генетической инженерии, и может быть использовано в ветеринарии для диагностики инфекционных заболеваний сельскохозяйственных животных, в частности комплекса респираторных инфекций крупного рогатого скота, а также для идентификации вирусов, выявленных у крупного рогатого скота.
Возбудители респираторного комплекса (РК) крупного рогатого скота (КРС) оказывают серьезное влияние на производство животноводческой продукции во всем мире. Экономические потери от РК включают гибель и снижение массы тела животных, затраты на средства терапии и профилактики, снижение производительности животноводства. Чаще болеет молодняк, чем взрослые животные.
В развитии РК принимают участие несколько возбудителей вирусной и бактериальной природы, при этом вирусы играют наиболее важную роль, запуская весь каскад реакций, приводящих к поражению респираторных органов у животных и созданию благоприятных условий для размножения патогенных бактерий.
По результатам вирусологических и серологических диагностических исследований установлено, что наиболее значимыми возбудителями РК КРС по частоте выявления у животных являются: вирус вирусной диареи - болезни слизистых оболочек КРС 1-3 генотипов (Bovine viral diarrhea virus, BVDV1, BVDV2, BVDV3), вирус репираторно-синцитиальной инфекции КРС (Bovine respiratory syncytial virus, BRSV), вирус инфекционного ринотрахеита КРС (Bovine herpesvirus-1, BHV-1), коронавирус КРС (Bovine coronavirus, BCV), вирус парагриппа-3 КРС (Bovine parainfluenza virus 3, BPIV), вирус герпеса 4-го типа КРС (Bovine herpesvirus-4, BHV-4) (Fulton R.W. Bovine respiratory disease research (1983-2009). Anim. Health Res. Rev. 2009; 10: 131-139). Другие вирусы КРС имеют меньшее значение и не способны самостоятельно вызывать поражение респираторных органов.
Ключом к успешной профилактике или лечению РК является раннее выявление возбудителей и установление их роли в развитии болезни в максимально короткие сроки, наиболее экономичным и эффективным способом. Однако существующие в настоящее время способы диагностики не позволяют этого сделать максимально эффективно.
Наиболее точным способом постановки диагноза на вирусную инфекцию является выделение вируса в чувствительной культуре клеток и его идентификация с помощью специфичных иммунных сывороток или молекулярно-генетическими методами (Сюрин В.Н., Самуйленко А.Я., Соловьев Б.В., Фомина Н.В. Вирусные болезни животных. М.: ВНИТИБП, 1998, 928 с.). Главным недостатком этого способа является длительность постановки диагноза (не менее 3-х недель), высокая стоимость, а также то, что эффективность данного способа во многом зависит от стадии заболевания, сроков отбора проб биоматериала и соблюдения всех требований их доставки, так как вирусы можно выделить в культуре клеток не на всех стадиях развития инфекционной болезни.
На практике широкое применение получили способы диагностики, основанные на выявлении фрагментов геномов возбудителей с помощью полимеразной цепной реакции (ПЦР). К настоящему времени разработаны ПНР для диагностики и генотипирования каждого из респираторных вирусов крупного рогатого скота в отдельности, как методом гель-электрофореза, так и в режиме реального времени.
Главная трудность и ограничение применения мультиплексной ПЦР заключаются в том, что отдельные праймеры и зонды в мультиплексной реакции могут взаимодействовать между собой, а также конкурировать за реактивы во время самой реакции (Kalle E., Kubista M., Rensing С. Multi-template polymerase chain reaction. Biomol. Detect. Quantif. 2014; 2: 11-29; Parker J., Fowler N., Walmsley M.L., Schmidt Т., Scharrer J., Kowaleski J., Grimes Т., Hoyos S., Chen J. Analytical sensitivity comparison between sin-gleplex real-time PCR and a multiplex PCR platform for detecting respiratory viruses. PLoS One. 2015; 10). Поэтому некоторые авторы предпочитают использовать отдельные реакции для каждого возбудителя, но проводить их при одинаковых температурных режимах. Так, известен способ постановки мультиплексной ПЦР на 16 возбудителей (10 вирусов и 6 бактерий), вызывающих болезни КРС. Он предусматривает проведение реакций на каждый возбудитель отдельно при одинаковых режимах (Kishimoto M.,  , Rahpaya S.S., Hasebe A., Otsu K., Sugimura S., Kobayashi S., Komatsu N., Nagai M, Omatsu T. Development of a one-run real-time PCR detection system for pathogens associated with bovine respiratory disease complex. J. Vet. Med. Sci. 2017; 79: 517-523). Однако этот способ достаточно трудоемкий, поскольку для исследования одной пробы необходимо проводить 16 отдельных реакций для выявления всех возбудителей с отдельными положительными и отрицательными контролями для каждого из них. Поэтому данный способ не подходит для рутинной лабораторной диагностики заболеваний.
, Rahpaya S.S., Hasebe A., Otsu K., Sugimura S., Kobayashi S., Komatsu N., Nagai M, Omatsu T. Development of a one-run real-time PCR detection system for pathogens associated with bovine respiratory disease complex. J. Vet. Med. Sci. 2017; 79: 517-523). Однако этот способ достаточно трудоемкий, поскольку для исследования одной пробы необходимо проводить 16 отдельных реакций для выявления всех возбудителей с отдельными положительными и отрицательными контролями для каждого из них. Поэтому данный способ не подходит для рутинной лабораторной диагностики заболеваний.
Наиболее приемлемым с точки зрения описанных недостатков и принятым за прототип является способ, основанный на мультиплексной ПЦР для одновременного выявления пяти вирусов (BPI3, BCoV, BRSV, BoHV-1 и BVDV) в одной пробе. Для обнаружения возбудителей после выделения РНК/ДНК из пробы, сначала проводят отдельно реакцию обратной транскрипции для получения кДНК, а затем уже проводят ПЦР. Учет результатов амплификации проводят на пяти каналах, отдельно для каждого выявляемого вируса. Авторы не публикуют ни последовательности праймеров и зондов, ни гены возбудителей, на которые они были синтезированы. Эффективность выявления возбудителей в мультиплексной ПЦР не отличалась от эффективности отдельной ПЦР на каждый из вирусов (Pansri P, Katholm J, Krogh KM, et al. Evaluation of novel multiplex qPCR assays for diagnosis of pathogens associated with the bovine respiratory disease complex. Vet J. 2020; 256:105425. doi:10.1016/j.tvjl.2020.105425).
К недостаткам данного способа можно отнести то, что реакция проводится в два этапа и выявляются только 5 вирусов.
Технической задачей изобретения являлась разработка высокоспецифичного и чувствительного способа на основе мультиплексной полимеразной цепной реакции (ПЦР) с использованием гибридизационно-флуоресцентной детекции в режиме реального времени, позволяющей выявлять одновременно восемь возбудителей респираторного комплекса КРС без проведения отдельной реакции обратной транскрипции и с минимальными затратами времени и реактивов.
Поставленная задача решается тем, что в способе выявления возбудителей респираторных инфекций крупного рогатого скота: BPIV, BRSV, BHV-1, BHV-4, BCoV, BVDV-1, BVDV-2, BVDV-3, на основе мультиплексной полимеразной цепной реакции (ПЦР), включающем выделение РНК/ДНК из проб биоматериала от крупного рогатого скота и проведение полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени, согласно изобретения, используют синтетические олигонуклеотидные праймеры и зонды SEQ ID N0: 1-28, обратная транскрипция для синтеза кДНК проводится непосредственно в ходе мультиплексной ПЦР, а исследование каждой пробы проводят в двух независимых реакциях для одновременного выявления восьми вирусов в одной пробе.
Изобретение иллюстрируется следующими примерами.
Пример 1. Оптимизация реакции и проверка аналитической чувствительности праймеров и зондов
Для идентификации возбудителей методом мультиплексной ПЦР были использованы синтетические олигонуклеотидные праймеры и зонды, опубликованные нами ранее (Патент РФ №2731716, 08.09.2020; Патент РФ №2700750, 19.09.2019; Патент РФ №2766344, 15.03.2022; Патент РФ №2768753, 24.03.2022; Глотов А.Г., Глотова Т.П., Нефедченко А.В., Котенева С.В. Респираторные болезни у импортного скота в период адаптации на молочных комплексах // Ветеринария. 2022. №2. С. 3-8).
С использованием программы Vector NTI 9.0.0 (InforMax) они были проверены на комплементарность друг другу и отсутствие перекрестных реакций. Характеристики выбранных праймеров и зондов представлены в таблице 1.
Так как приборы для ПЦР в режиме реального времени имеют только 5 каналов детекции, поэтому все праймеры и зонды разделили на две реакции, для выявления в каждой 4-х возбудителей. Пятый канал используется для выявления гена glyceraldehyde-3-phosphate dehydrogenase (GAPDH) КРС в качестве контроля ПЦР.

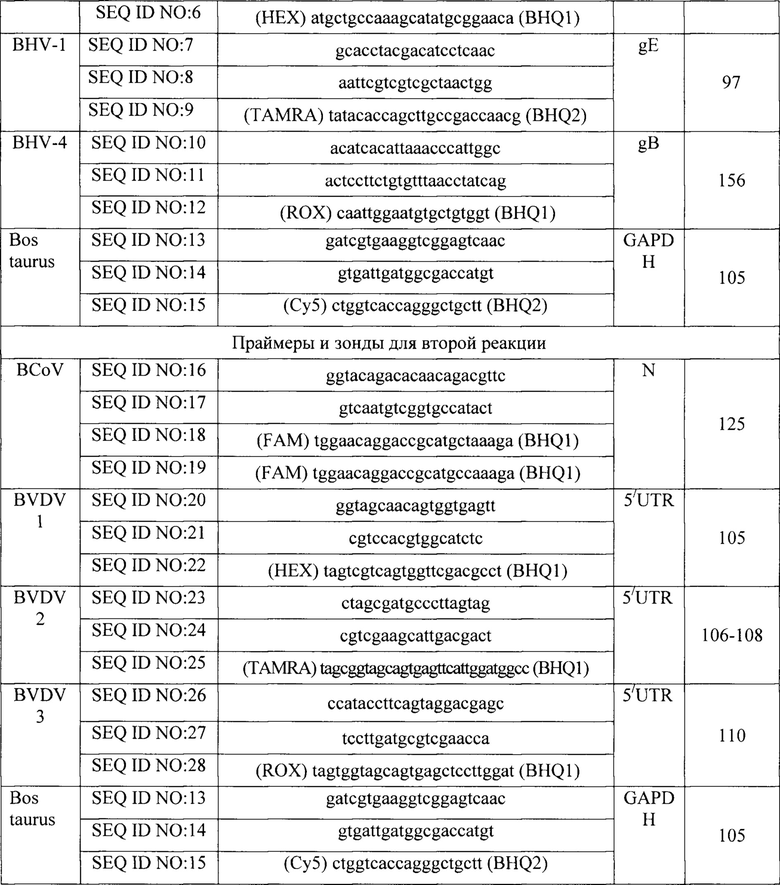
Для контроля амплификации были получены положительные контрольные образцы (ПКО) методом молекулярной трансформации компетентных бактериальных клеток Escherichia coli плазмидами pCR2.1, содержащими специфические ДНК вставки соответствующих детектируемых участков геномов каждого возбудителя.
Для подтверждения специфичности полученных фрагментов определяли их нуклеотидную последовательность, для чего использовали набор реагентов BigDye® Terminator v3.1 Cycle Sequencing Kits (Applied Biosystems, США). Продукты секвенирующей реакции анализировали методом капиллярного электрофореза в автоматическом секвенаторе ABI PRISM® 3130xl (Applied Biosystems/Hitachi, Япония). Полученные нуклеотидные последовательности сравнивали с последовательностями базы данных NCBI BLAST (http://www.ncbi.nlm.nih.gov/). Все фрагменты ДНК, необходимые для получения ПКО, являлись целевыми и соответствовали участкам геномов анализируемых возбудителей.
Условия проведения амплификации оптимизировали по следующим параметрам: концентрация ионов магния в реакционной смеси; концентрация праймеров и зондов в реакционной смеси; температура отжига праймеров.
Оптимизированный состав реакционной смеси включал следующие компоненты: Набор БиоМастер ОТ-ПЦР - РВ (2×) (ООО «ООО "БИОЛАБМИКС", Россия) - конечная концентрация 1×; смесь праймеров - конечная концентрация каждого 0,15 μM; смесь зондов - конечная концентрация каждого 0,2 μM; Объем РНК/ДНК - 3 мкл; вода для ПЦР. Общий объем реакционной смеси составлял 30 мкл.
Концентрацию плазмидной ДНК определяли с использованием набора реагентов Quant-iT dsDNA, HS (Invitrogen, США) и флуориметра QUBIT (Invitrogen, США) и выражали в геномных эквивалентах (ГЭ) в 1 мкл (ГЭ/мкл).
Пересчет концентрации ДНК в количество копий произведен в программе-конвертере http://molbiol.ru/scripts/01_07.html по формулам:
m[g]=Q[mol]×Mwолиг[kDa]×103
m[g]=Q[mol]×<Mw>[Da]×L[kb]×103
N[штук]=Q[mol]×NΑ
c[M]×V[L]=Q[mol], где
m[g] - вес нуклеиновой кислоты;
Mwолиг[kDa] - молекулярный вес олигонуклеотида.
<Mw>[Da] - средний молекулярный вес одного/пары оснований.
base = 324.5 Da
base pair = 649 Da
ribo base=340.5 Da
Q[mol] - количество нукл. кислоты;
N[копий] - количество молекул нукл. кислоты;
с[М] - молярная концентрация нукл. кислоты;
V[L] - объем, в котором растворена нукл. кислота;
L[kb] - длина нукл. кислоты;
NA - Число Авогадро = 6.022045×1023[1/моль].
Аналитическую чувствительность метода для каждого вируса определяли постановкой ПЦР в режиме реального времени, где в качестве исследуемых проб использовали 10-кратные разведения положительных контрольных образцов, при этом оценку проводили как отдельно для каждой пары праймеры - зонд (в моноварианте), так и в окончательном варианте в мультиплексном формате. Результаты представлены в таблице 2.
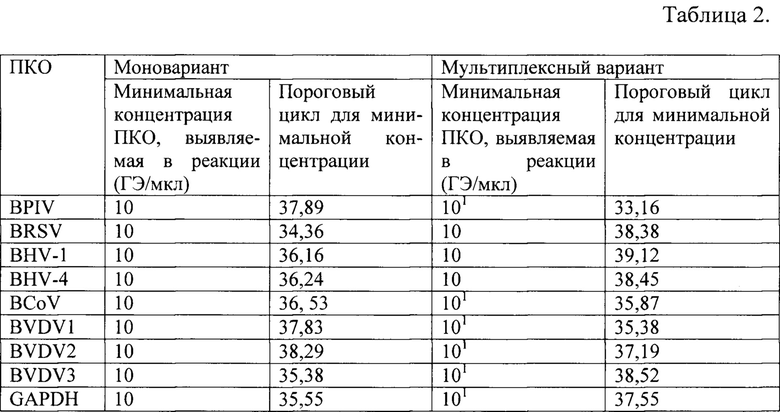
Таким образом, предложенные для мультиплексного формата реакционные смеси, включающие смеси праймеров и зондов, представленные в таблице 1, оказались эффективными. Практически не наблюдалось конкурентного связывания и перекрестных реакций между отдельными парами праймеров и зондов. Минимальное количество ПКО, детектируемое с применением наших праймеров и зондов после оптимизации условий проведения реакции, выраженное в ГЭ (геномных эквивалентах) в 1 мкл ПКО, составило от 10 до 101 ГЭ для разных ПКО.
Пример 2. Способ выявления возбудителей респираторного комплекса крупного рогатого скота на основе мультиплексной полимеразной цепной реакции
Способ осуществляется в три этапа.
Этап 1. Выделение РНК/ДНК из исследуемого материала
Выделение РНК/ДНК осуществляют стандартным способом с использованием коммерческого набора «Рибо-сорб» производства ФГУН «Центральный НИИ эпидемиологии» Роспотребнадзора.
Этап 2. Постановка полимеразной цепной реакции с обратной транскрипцией
ПЦР в режиме реального времени проводили в реакционной смеси следующего состава: Набор БиоМастер ОТ-ПЦР - РВ (2×) (ООО «ООО "БИОЛАБМИКС", Россия) - конечная концентрация 1×; смесь праймеров - конечная концентрация каждого 0,15 μM; смесь зондов - конечная концентрация каждого 0,2 μM; Объем РНК/ДНК - 3 мкл; вода для ПЦР. Общий объем реакционной смеси составлял 30 мкл.
Температурный режим проведения ПЦР: 50°С - 30 мин - 1 цикл; 95°С - 5 мин - 1 цикл; 95°С - 15 сек., 60°С - 60 сек. - 55 циклов.
Измерение флуоресценции осуществляют при температуре 60°С на всех каналах.
Положительными считают образцы со значением порогового цикла (cycle threshold, Ct), не превышающим 40.
Этап 3. Анализ результатов полимеразной цепной реакции
Анализ результатов поводят с помощью программного обеспечения, используемого прибора для проведения ПЦР с детекцией в режиме «реального времени». Анализируют кривые накопления флуоресцентного сигнала по пяти каналам FAM, JOE/HEX, TAMRA/Су3, ROX (Texas Red), Су5. Устанавливают уровень пороговой линии выше шумовых сигналов и так чтобы отсечь все кривые с не экспоненциальным подъемом и рассчитывают Ct для каждой пробы.
Учет результатов ПЦР в первой и второй реакциях начинают с анализа амплификации на канале Су5 (ген GAPDH КРС). Результат считают достоверным только в случае прохождения амплификации на данном канале для всех проб биологического материала, полученных от КРС или для штаммов вирусов КРС. Для выявляемых вирусов КРС учет результатов проводят в соответствии с таблицей 3.
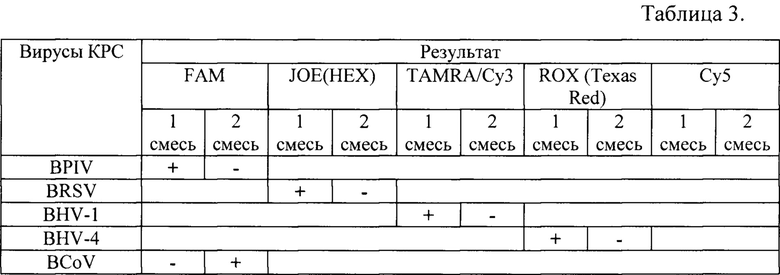
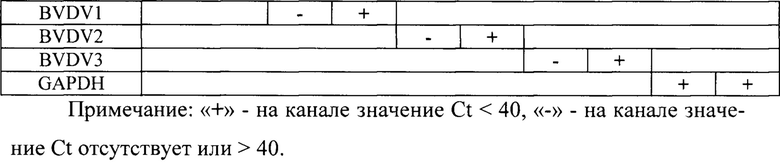
Результат исследования пробы биоматериала считают отрицательным на исследуемые вирусы, если на канале Су5 для него значение Ct не превышает 40, а на остальных каналах значение Ct отсутствует или превышает 40. Результат исследования пробы биоматериала, полученного от крупного рогатого скота, или штамма вируса считают сомнительным, если на канале Су5 значение Ct отсутствует или превышает 40. В этом случае необходимо повторить реакцию.
Пример 3. Оценка специфичности выявления анализируемых вирусов
Для оценки специфичности реакции исследовали контрольные штаммы вирусов: BVDV 1 «Oregon C24V» с титром 104,5 ТЦД50/см3, BVDV 2 «Изолят БЛ» с титром 103,5 ТЦД50/см3, BVDV 3 «Изолят ЦТ», BCoV «КМИЭВ-1» с титром 106,5 ТЦД50/см3, BRSV «РСВ №3» с титром 104 ТЦД50/мл, BHV-4 «Movar»» с титром 105,5 ТЦД50/см3, BHV-1 «Оренбург» с титром 107,5 ТЦД50/см3, вакцины Бови-шилд Голд FP5, в первом компоненте содержащей вирусы BHV-1, BVDV1, BVDV2, BRSV, BPVI, а во втором - штаммы лептоспир, и вакцину Кэтлмастер Голд FP5 L5, первый компонент которой содержит вирусы BHV-1, BRSV, BPVI, а второй компонент - штаммы лептоспир и вирусы BVDV1, BVDV2.
В качестве контроля применяли родственные штаммы вирусов КРС, свиней, культуры клеток, а также сыворотку крови КРС, свиней, собак, человека.
Выделение ДНК/РНК и постановку ПЦР проводили, как описано в примере №2. Результаты представлены в таблице 4.
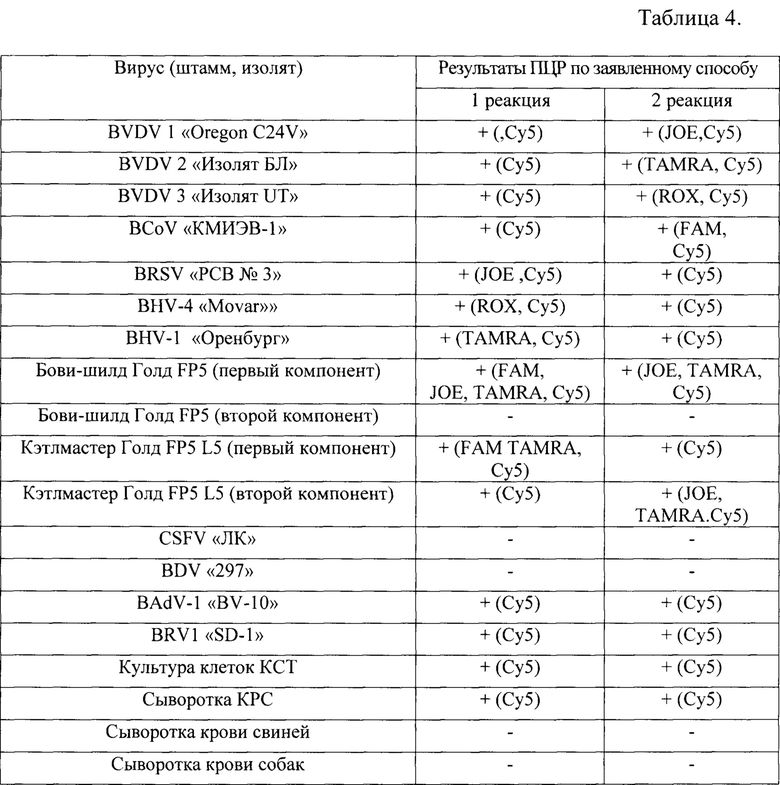

Таким образом, заявленный способ обладает высокой специфичностью при выявлении ДНК и РНК исследуемых вирусов.
Пример 4. Выявление вирусов респираторного комплекса крупного рогатого скота в пробах биологического материала, полученных от больных и инфицированных животных
Для исследования на наличие РНК/ДНК вирусов используют различные пробы биологического материала: смывы со слизистой носа, слизистую оболочку трахеи, легкие, лимфатические узлы, миндалины. Также могут быть отобраны для исследования кровь, носовые выделения, слизистая бронхов, трахеальная и бронхиальная слизь, а также другие органы с учетом особенностей патогенетического воздействия конкретного вируса.
Пробы слизистых выделений и смывы отбирают в объеме не менее 1 мл, из органов вырезают кусочки размером 1×1×1 см3.
Из проб органов предварительно готовят 10% суспензию на стерильном физиологическом растворе, смесь переносят в пластиковые пробирки емкостью 1,5 мл, центрифугируют при 10×103 об./мин. в течение 5 минут. Для выделения РНК используют 100 мкл осветленной надосадочной жидкости, полученной после центрифугирования.
Густые пробы носовых выделений и слизи разводят стерильным физиологическим раствором (примерно 1:5), тщательно перемешивают, центрифугируют при 10×103 об./мин. в течение 5 минут и 100 мкл осветленной надосадочной жидкости используют для выделения РНК.
Выделение РНК/ДНК и постановку ПЦР проводят согласно примеру 2.
С использованием разработанного способа исследовали 115 проб биологического материала от 23 телят в возрасте 2-4-х месяцев. Результаты опытов по определению эффективности разработанного способа при исследовании проб биоматериала от крупного рогатого скота представлены в таблице 5.
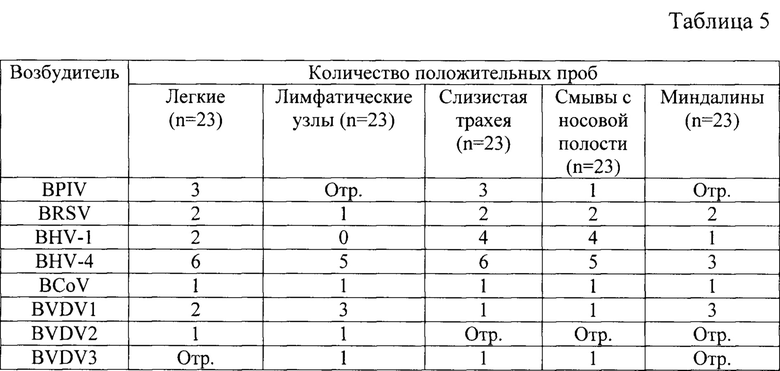
По заявленному способу количество положительных проб составило 56,5%. В исследованных пробах внутренних органов выявили все анализируемые возбудители респираторного комплекса КРС, чаще всего выявляли BHV-4 и BHV-1, реже BVDV2 и BVDV3.
Для подтверждения специфичности полученных фрагментов ДНК определяли их нуклеотидную последовательность как описано в примере 2. Результаты секвенирования показали, что все полученные ампликоны соответствуют исследованным участкам генов вирусов BPIV, BRSV, BHV-1, BHV-4, BCoV, BVDV1, BVDV2, BVDV3.
Таким образом, предлагаемый способ обладает высокой чувствительностью, специфичностью и эффективностью при выявлении ДНК и РНК вирусов респираторного комплекса крупного рогатого скота в пробах биологического материала от больных животных.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Последовательность
нуклеотидов к заявке 2022114969 20(031379).xml" softwareName="WIPO
Sequence" softwareVersion="2.1.2" productionDate="2022-08-16">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022114969/20(031379)</ApplicationNumberText>
<FilingDate>2022-06-01</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>2022114969/20(031379)</ApplicantFileReference
>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022114969/20(031379)</ApplicationNumberText>
<FilingDate>2022-06-01</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Сибирский федеральный научный центр
агробиотехнологий Российской академии наук (СФНЦА
РАН)</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Institution of Science
Siberian Federal Scientific Center for Agricultural Technology of the
Russian Academy of Sciences (SFNCA RAS)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ выявления возбудителей
респираторного комплекса крупного ро-гатого скота на основе
мультиплексной полимеразной цепной реакции (ПЦР)</InventionTitle>
<SequenceTotalQuantity>29</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine respirovirus
3</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggacatcaataggctcaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine respirovirus
3</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctacacccatcgcataactcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine respirovirus
3</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caccaggcaactatcccgctctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine
orthopneumovirus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgctgcaggactaggtataatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine
orthopneumovirus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acactgtaattgatgaccccattctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine
orthopneumovirus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgctgccaaagcatatgcggaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine alphaherpesvirus
1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcacctacgacatcctcaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine alphaherpesvirus
1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aattcgtcgtcgctaactgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine alphaherpesvirus
2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatacaccagcttgccgaccaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine gammaherpesvirus
4</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acatcacattaaacccattggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine gammaherpesvirus
4</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actccttctgtgtttaacctatcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bovine gammaherpesvirus
4</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caattggaatgtgctgtggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bos taurus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatcgtgaaggtcggagtcaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bos taurus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgattgatggcgaccatgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bos taurus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggtcaccagggctgctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Betacoronavirus 1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtacagacacaacagacgttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Betacoronavirus 1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcaatgtcggtgccatact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Betacoronavirus 1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaacaggaccgcatgctaaaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Betacoronavirus 1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaacaggaccgcatgccaaaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtagcaacagtggtgagtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtccacgtggcatctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagtcgtcagtggttcgacgcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus B</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctagcgatgcccttagtag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus B</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtcgaagcattgacgact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus B</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagcggtagcagtgagttcattggatggcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus H</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccataccttcagtaggacgagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus H</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccttgatgcgtcgaacca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pestivirus H</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagtggtagcagtgagctccttggat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР ВЫСОКОСПЕЦИФИЧНЫХ ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ЗОНДОВ ДЛЯ ДЕТЕКЦИИ И ДИФФЕРЕНЦИАЦИИ ВИРУСОВ АЧС, КЧС И ВД | 2024 |
|
RU2828887C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНЫХ ЗОНДОВ ДЛЯ ИДЕНТИФИКАЦИИ И ДИФФЕРЕНЦИАЦИИ ДНК ГЕРПЕСВИРУСОВ КРУПНОГО РОГАТОГО СКОТА 1 И 5 ТИПОВ | 2024 |
|
RU2837794C1 |
| Тест-система для выявления ДНК возбудителя контагиозной плевропневмонии крупного рогатого скота методом полимеразной цепной реакции в режиме реального времени | 2023 |
|
RU2821042C1 |
| Набор для дифференциации пестивирусов крупного рогатого скота и способ дифференциации пестивирусов крупного рогатого скота | 2019 |
|
RU2731716C1 |
| Набор реагентов для обнаружения возбудителей клещевых нейроинфекций | 2022 |
|
RU2800261C1 |
| Набор олигонуклеотидных праймеров и зондов и способ выявления пестивируса Н крупного рогатого скота | 2019 |
|
RU2728342C1 |
| Набор синтетических олигонуклеотидных праймеров и зондов для выявления вируса респираторно-синцитиальной инфекции крупного рогатого скота и гена GAPDH крупного рогатого скота и способ выявления РНК вируса респираторно-синцитиальной инфекции крупного рогатого скота | 2020 |
|
RU2768753C2 |
| Набор олигонуклеотидных праймеров и зонда для выявления вируса герпеса крупного рогатого скота 4-го типа (BHV-4) в пробах биоматериала | 2018 |
|
RU2700750C1 |
| Способ диагностики инвазивного кандидоза и видовой идентификации его основных возбудителей методом ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2022 |
|
RU2809386C1 |
| Тест-система и способ обнаружения специфических фрагментов нуклеиновых кислот 16 патогенов с использованием изотермической реакции амплификации | 2023 |
|
RU2810751C1 |
Изобретение относится к биотехнологии, а именно к генетической инженерии. Предложен способ выявления возбудителей респираторных инфекций крупного рогатого скота: BPIV, BRSV, BHV-1, BHV-4, BCoV, BVDV-1, BVDV-2, BVDV-3, на основе мультиплексной полимеразной цепной реакции (ПЦР). Предложенный способ включает выделение РНК/ДНК из проб биологического материала от крупного рогатого скота и проведение полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени для одновременного выявления вирусов BVDV1, BVDV2, BVDV3, BRSV, BHV-1, BHV-4 BPI-3, BCoV, вызывающих респираторные болезни крупного рогатого скота. При этом обратная транскрипция для синтеза кДНК проводится непосредственно в ходе мультиплексной ПЦР и исследование каждой пробы проводят в двух независимых реакциях для одновременного выявления восьми вирусов в одной пробе. Способ может быть использован в ветеринарной вирусологии для диагностики инфекционных заболеваний сельскохозяйственных животных, в частности инфекционного ринотрахеита, вирусной диареи - болезни слизистых оболочек, респираторно-синцитиальной инфекции, коронавирусной инфекции, герпесвирусной инфекции 4 типа, парагриппа-3 крупного рогатого скота. 5 табл., 4 пр.
Способ выявления возбудителей респираторных инфекций крупного рогатого скота: BPIV, BRSV, BHV-1, BHV-4, BCoV, BVDV-1, BVDV-2, BVDV-3, на основе мультиплексной полимеразной цепной реакции (ПЦР), включающий выделение РНК/ДНК из проб биоматериала от крупного рогатого скота и проведение полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени, отличающийся тем, что используют синтетические олигонуклеотидные праймеры и зонды SEQ ID NO: 1-28, обратная транскрипция для синтеза кДНК проводится непосредственно в ходе мультиплексной ПЦР, а исследование каждой пробы проводят в двух независимых реакциях для одновременного выявления восьми вирусов в одной пробе.
Авторы
Даты
2023-06-21—Публикация
2022-06-01—Подача