Изобретение относится к области биотехнологии, а именно к генетической инженерии, и касается получения генететической конструкции для экспрессии в дрожжах Pichia pastoris - продуцента прохимозина быка, путем введения в геном штамма-реципиент синтетического гена, кодирующего вариант препрохимозина с модифицированной сигнальной последовательностью секреции в составе рекомбинантной плазмидной ДНК и разработки способа микробиологического синтеза прохимозина. Изобретение предназначено для использования в пищевой промышленности для получения молокосвертывающих ферментов.
Ферментативная коагуляция молока под действием молокосвертывающих ферментов, таких как химозин и пепсин, представляет собой один из наиболее важных процессов при приготовлении сыров. В ходе данного 2-х ступенчатого процесса химозин или пепсин атакуют κ-казеин, что приводит к дестабилизации казеиновых мицелл, которые затем агрегируют с образованием творожистого сгустка.
Химозин (сычужный фермент, ренин, EC3.4.23.4) - аспарагиновая протеаза, которая вырабатывается в слизистой оболочке четвертого отдела желудка молочных телят [1] обладает высокой молокосвертывающей активностью при низкой протеолитической активности, что обуславливает его широкое использование в молочной промышленности для производства сыра [2].
Вновь синтезируемый в клетках слизистой желудка химозин находится в виде ферментативно неактивного пре-прохимозина. В процессе экскреции N-концевой сигнальный пептид отщепляется с образованием прохимозина, содержащего про-фрагмент. Прохимозин находится, по-существу, в неактивной форме фермента, который, однако, становится активным при кислотных условиях, превращаясь в активный химозин посредством аутокаталитического удаления про-фрагмента. Эта активация осуществляется in vivo в полости желудка при соответствующих условиях pH или in vitro при кислотных условиях [1] [3].
Структурные и функциональные характеристики пре-прохимозина, прохимозина и химозина подробно исследованы. Пре-часть молекулы бычьего пре-прохимозина содержит 16 аминокислотных остатков, про-часть содержит 42 аминокислотных остатка. Активный бычий химозин содержит 323 аминокислотных остатка и представляет собой смесь двух форм, A и B, обе являются активными [3].
Фермент содержит два каталитически активных остатка аспарагиновой кислоты Asp32 и Asp215, которые катализируют селективное разрушение пептидной связи между остатками Phe105 и Met106 в составе каппа-казеина. Это сайт-специфическое расщепление приводит к дестабилизации мицелл казеина и индуцирует свертывание молока [4].
Получение рекомбинантного химозина быка с использованием микробных систем экспрессии, таких как мицелиальные грибы и дрожжи дает многочисленные преимущества по сравнению с экстракцией нативного химозина из желудка молочных телят, в частности благодаря возможности наработки гомогенного продукта в стандартизованном биопроцессе [2,5-7].
Одной из наиболее привлекательных систем гетерологичной экспрессии для продукции химозина быка и многих других практически-важных белков являются метилотрофные дрожжи P. pastoris и Hansenula polymorpha. В последние десятилетия эти микроорганизмы стали, несомненно, самой популярной и широко используемой среди низших эукариот «клеточной биофабрикой» для экспрессии рекомбинантных белков для фундаментальных и прикладных исследований. Согласно веб-платформе www.pichia.com, в этих эукариотических микроорганизмах было получено более 5000 различных рекомбинантных белков [8] с рекордными уровнями продукции (выход продукта - до 35 г с литра, в некоторых случаях). Популярность этих дрожжей обусловлена такими факторами, как доступность простых и надежных процедур высокоплотного культивирования, наличие строго регулируемых и необычайно сильных промоторов, развитой способностью этих дрожжей к секреции чужеродных белков и к осуществлению ряда характерных для эукариот пост-трансляционных модификаций, простотой генетических манипуляций, способностью к утилизации дешевых углеродных субстратов, развитой пероксисомальной системой ( [9,10]. Если ранние разработки в основном были посвящены получению технических ферментов [11], то последующее присвоение статуса GRAS положило начало широкому использованию этих дрожжей как «платформы» для получения белков медицинского назначения и ферментов для пищевой промышленности [12]. Также P. pastoris - наиболее удачный хоязин для получения целого ряда «трудных» для экспрессии белков, таких, например, как различные мембранные белки, гликопротеины [13,14]. Поэтому, в настоящее время P. pastoris принято считать «золотым стандартом» для получения на регулярной основе рекомбинантных белков для лабораторных либо промышленных целей [15].
Химозины самых различных видов млекопитающих, в том числе буйвола [16], верблюда [17], яка [18], козы [19], были успешно экспрессированы в клетках P. pastoris.
Тем не менее, из уровня техники следует, что уровень продукции рекомбинантных химозинов в P. pastoris относительно невелик по сравнению с использованными для его коммерческой продукции рекомбинантными штаммами Aspergillus [20] , и требует оптимизации.
Известно, что различия в частотах использования кодонов между кодирующими последовательностями природного гетерологичного гена и клеткой-хозяином могут снижать скорость трансляции, негативно влияя на уровни экспрессии целевых белков [21]. Также эти различия могут приводить к ошибкам в процессах посттрансляционной модификации белков, снижая стабильность белков. Оптимизацию кодонового состава проводят путем «ретрансляции» кодируюшей последовательности исходного природного гена, заменяя редкие для клетки хозяина на те, которые встречаются с высокой частотой. Таком образом проектируют ген, кодирующий белок с идентичной аминокислотной последовательностью, нуклеотидная последовательность которого лучше соответствует клетке хозяину [22]. При создании штаммов P. pastoris - продуцентов химозина, оптимизация кодонового состава позволяет в 10 раз повысить уровень продукции этого белка в клетках дрожжей, поскольку природный ген химозина быка значительно отличается по кодоновому составу от высоко-экспрессируемых генов P. pastoris [23].
Возможность эффективного получения целевого белка в секретируемом виде наряду с низким уровнем эндогенного белкового экспорта является важным технологическим преимуществом P. pastoris [24]. Наряду с природными сигнальными последовательностями секреции, ряд гетерологичных сигнальных пептидов успешно используют для секреторной экспрессии в P. pastoris [25,26]. Однако наиболее часто для этих целей используют ppMFα1 - пре-просегмент полового феромона дрожжей альфа-фактора [27]. Лидерная последовательность ppMFα1 состоит из 19-аминокислотной пре-последовательности за которой следует 67-аминокислотный просегмент с тремя сайтами гликозилирования и LysArg сайтом процессинга для Kex2 эндопептидазы [28]. Процессинг секреторной сигнальной последовательности происходит в 3 этапа: сначала пре-последовательность удаляется сигнальными пептидазами в эндоплазматическом ретикулуме, затем Kex2 эндопептидаза разрезает участок просегмента между аминокислотными остатками лизина и аргинина, а затем пептидаза Ste13 быстро удаляет остатки Glu-Ala в цистернах Гольджи [29]. Такой «двучастный» сигнальный пептид оказался достаточно эффекктивным для секреции множества чужеродных белков, однако уровень секреции варьирует достаточно значительно [30], что стимулирует попытки для увеличения эффективности секреции [10,26,31].
Одна из стратегий усиления эффективности секреции состоит в модификации сигнального пептида. Из уровня техники известно, что модифицированный ppMFα1 с делецией 3-го альфа-спирального сегмента более эффективно направляет секрецию рекомбинантных липазы Candida Antarctica и пероксидазы хрена в клетках P. pastoris [32]. Также известно, что оптимизация кодонового состава ppMFα1 с учетом наиболее часто встречающихся кодонов для P. pastoris позволяет в несколько раз повысить эффективность секреции липазы Candida Antarctica [33] .
Конструирование векторов для экспрессии гена прохимозина быка с таким вариантом сигнального пептида ранее не проводилось, также из уровня техники неизвестно, позволяет ли использование такой комбинации повысить эффективность экспрессии и секреции рекомбинантного химозина по сравнению с использованием «нативной» сигнальной последовательности.
Известен способ получения рекомбинантного proCYM с использованием рекомбинатного штамма P. pastoris, модифицированного плазмидой ppic9K::PROCHY, содержащей нативный ген прохимозина быка под контролем промотора AOX1 и сигнального пептида ppMFα1 [34]. При выращивании в колбах, наилучший из отобранных клонов обеспечивал продукцию прохимозина на уровне 4,8 Ед./мл за 120 часов культивирования при выходе биомассы 18 г сухих дрожжей с литра.
Известен также способ микробиологического синтеза прохимозина c использованием рекомбинантных штаммов P. pastoris, содержащих синтетический ген proCYM под контролем вектора pPICZα [35] с высоким выходом. Однако в данном источнике отсутствует информация о структуре синтетического гена proCYM, поэтому данный способ не подлежит сравнению с заявляемым методом.
Задача заявляемой группы изобретений - расширение арсенала способов микробиологического синтеза proCYM.
Данная цель в настоящем изобретении достигнута за счет оптимизации процессов транскрипции целевого гена CYM, трансляции синтезируемой мРНК CYM и секреции предшественника proCYM.
Для этого:
a) получен синтетический ген, кодирующий пробелок химозина быка. Кодоновый состав гена соответствует часто используемым кодонам клеток P. pastoris;
б) сконструирован ген варианта предшественника химозина белка с модифицированной сигнальной последовательностью ppMFα1;
в) сконструирована рекомбинантная ДНК, содержащая ген прохимозина в составе вектора экспрессии для P. pastoris и обеспечивающая высокий уровень метанол-индуцируемого синтеза и секреции прохимозина в клетках этих дрожжей;
г) в результате трансформации экспрессирующей плазмидой клеток штамма P. pastoris GS115 получен рекомбинантный штамм этих дрожжей, характеризующийся высоким уровнем индуцируемого синтеза и стабильной продукций proCYM. При культивировании заявляемым способом данный штамм позволяет получать целевой фермент с выходом более чем в 3 раза превышающим продукцию фермента, полученного с использованием способа - ближайшего аналога.
Процесс конструирования заявляемого штамма состоит из следующих этапов:
- дизайна и получения синтетического гена BtCYMsyn (SEQ ID No 1) кодирующего модифицированный предшественник proCYM;
- конструирования гена MaBtCYMsyn, кодирующий вариант предшественника химозина белка с модифицированной N-концевой сигнальной последовательностью ppMF1D (SEQ ID No 15);
- конструирования плазмидной ДНК pSF001, содержащей ген MaBtCYMsyn в составе вектора pVR2;
- конструирования рекомбинантного штамма дрожжей P. pastoris/pSF001, способного синтезировать proCYM;
- разработки способа микробиологического синтеза прохимозина на основе рекомбинантного штамма дрожжей P. pastoris/рSF001, сочетающего высокий уровень синтеза proCYM и способностью к его секреции.
Выбор модифицированной последовательности пре-прообласти предшественника феромона дрожжей альфа-фактора (SEQ ID No 12) в качестве сигнального пептида обусловлен тем, что такая последовательность обеспечивает в 1,4-2 раза более эффективную секрецию целевых белков по сравнению с последовательностью ppMFa1 «дикого типа» [36] .
Выбор метода получения гена BtCYMsyn с помощью олигонуклеотидного синтеза обусловлен тем, что такой подход позволяет оптимизировать кодирующую последовательность гена для наиболее эффективной трансляции и стабильности мРНК BtCYM в клетках P. pastoris, что должно приводить к значительному повышению выхода рекомбинантного proCYM.
Выбор плазмиды pVR2 в качестве основы для создания вектора экспрессии BtCYM состоит в том, что данный вектор, в отличии от известных коммерческих векторов (pPICZA,B,C, pPICZaA,B,C, pPIC9K) содержит близкую к нативной последовательность участка перед АУГ кодоном промотора АОХ1, что должно повышать эффективность трансляции целевой мРНК, а также помимо маркера устойчивости к зеоцину содержит дополнительный селективный маркер устойчивости к ампициллину, что упрощает и удешевляет процедуры конструирования векторов экспрессии на его основе [37].
Этап 1. Дизайн и конструирование синтетического гена MaBtCYMsyn.
Дизайн нуклеотидной последовательности BtCYMsyn разрабатывают на основании аминокислотной последовательности preproCYM (Номер доступа Genbank NP_851337.1). МРНК preproCYM (Номер доступа Genbank NM_180994.2) кодирует предшественник preproCYM с собственным N-концевым сигнальным пептидом, продоменом. Для обеспечения эффективной секреции proCYM в клетках дрожжей проводят замену собственного сигнального пептида preproCYM на модифицированную последовательность ppMFa1, которую состыковывают с кодирующей последовательностью proCYM.
Кодирующую последовательность гена BtCYMsyn проектируют с учетом данных о предпочтении кодонов у активно экспрессирующихся генов P. pastoris, их GC составе, попутно избегая участки, способные формировать обширные вторичные структуры мРНК, сайты преждевременного полиаденилирования и терминации трансляции, удаляя часто встречающиеся сайт рестрикции, в том числе сайты XhoI и NdeI, которыми затем фланкируют последовательность для последующего встраивания в плазмидную конструкцию. Полученную последовательность используют для синтеза ДНК BtCYM по заказу от компании TWIST Bioscience. Полученный синтетический фрагмент ДНК гена BtCYMsyn объединяют с помощью рекомбинантной ПЦР с другим синтетическим фрагментом, кодирующим вариант последовательности сигнального пептида ppMFa1.
Этап 2. Конструирование интегративной плазмиды pSF002, содержащей ген MaBtCYMsyn под контролем промотора AOX1.
Плазмиду pSF002 конструируют путем клонирования, полученного на этапе 1 фрагмента ДНК, кодирующего ген MaBtCYMsyn в вектор pVR2, разработанный для интеграции в штаммы Pichia pastoris (his 4, mut+) и предназначенный для метанол-индуцируемой экспрессии целевых белков [38]. Вектор pVR2 размером 6251 пары оснований содержит промотор АОХ1; терминатор гена CYC1 S. cerevisiae; «кассету» экспрессии гена Zeo, обеспечивающего устойчивость штаммов Е. coli и P. pastoris к зеоцину; включающую кодирующую область гена St. griseus, промотор гена TEF1 P. pastoris, терминатор гена CYC1 дрожжей, ген Amp, обеспечивающий устойчивость к ампициллину штаммов E. coli, репликон pUC.
Плазмида pSF002 размером 6106 пар оснований, наряду с генами вектора pVR2, содержит кодирующую область гена MaBtCYMsyn прохимозина быка под контролем промотора АОХ1 (Рис. 1).
Этап 3. Конструирование рекомбинантного штамма дрожжей P. pastoris, способного синтезировать секретируемый прохимозин быка, кодируемый геном MaBtCYMsyn.
В качестве штамма-реципиента используют штамм P. pastoris GS115. Компетентные клетки данного штамма трансформируют плазмидой pSF002. В результате получают рекомбинантный штамм P. pastoris, способный синтезировать и секретировать прохимозин быка.
Заявляемый штамм P. pastoris имеет следующие морфологические и физиолого-биохимические характеристики:
Морфологические признаки: при культивировании при температуре 28°С в течение 48 часов на агаризованной среде YPD следующего состава (мас.%: пептон - 2, дрожжевой экстракт - 1, глюкоза - 2, агар - 2, вода - остальное) формируются колонии светло-бежевого цвета с ровным краем, матовой поверхностью, линзовидным профилем и пастообразной консистенцией. При росте в жидкой среде YPD при 28°С в течение 24 ч культивирования - жидкость мутная, осадок белый, коагуляции не наблюдается, пристеночных пленок культура не образует. Клетки имеют овальную форму, 3-4 мкм в диаметре; почкуются; почкование истинное, многостороннее; истинного мицелия не образуют.
Физиолого-биохимические признаки: штамм является факультативным анаэробом с температурой роста - 20-33°C (оптимум - 28°C) и рН среды культивирования - 4,8-7,4 (оптимум - 6,0). В качестве источников углерода штамм может использовать глюкозу, глицерин, метанол, олеат, сорбитол, рамнозу. Не утилизирует галактозу, ксилозу, арабинозу. В качестве источников азота штамм может использовать аминокислоты, сернокислый аммоний, азотнокислый аммоний. Штамм P. pastoris синтезирует рекомбинантный секретируемый прохимозин быка.
Пример 1. Способ микробиологического синтеза прохимозина. Посевной материал, представляющий собой клетки рекомбинантного штамма-продуцента, подготавливают путем инкубации в течение 15-24 часов при температуре 29°С на среде YPD при постоянной аэрации на термостатируемой качалке (250 об/мин). Затем выросшую культуру переносят в соотношении 1:200 (по объему) в среду BMMY (мас.%: пептон - 2, дрожжевой экстракт - 1, метанол - 0,5, фосфатный буфер рН=6.6 - до 100 mM, вода - остальное) до ОД600=1.
Процесс биосинтеза ведут в колбах Эрленмейера, содержащих 20 мл среды BMMY, в течение 96 часов в ротационном шейкере-термостате (200-250 об/мин), при температуре 28°C. Каждые 24 часа проводят индукцию метанолом, путем асептического добавления 50% раствора метанола в пробирки, до конечной концентрации 0,5%. По истечении 96 часов биомассу отделяют центрифугированием. Наличие рекомбинантного прохимозина в культуральной среде определяют при энзиматического теста после активации прохимозина в составе супернатанта путем кислотной обработки. Уровень продукции прохимозина выражают в международных казеиновых единицах (IMCU/мл, МКЕ/мл). Уровень синтеза прохимозина заявляемым способом составляет не менее 15 МКЕ/мл культуральной жидкости.
Изобретение проиллюстрировано следующими фигурами графических изображений
Фиг. 1. - физическая и генетическая карты плазмиды pSF002. Обозначены положения гена MaBtCYM, индикаторных сайтов рестрикции, участков промотора AOX1, терминатора tTEF1 область начала репликации pBR322 (pBR322 origin), генов устойчивости к зеоцину, ампициллину.
Фиг. 2. - Сравнение нуклеотидных последовательностей природного гена WT_CYM и заявляемого гена BtCYM.
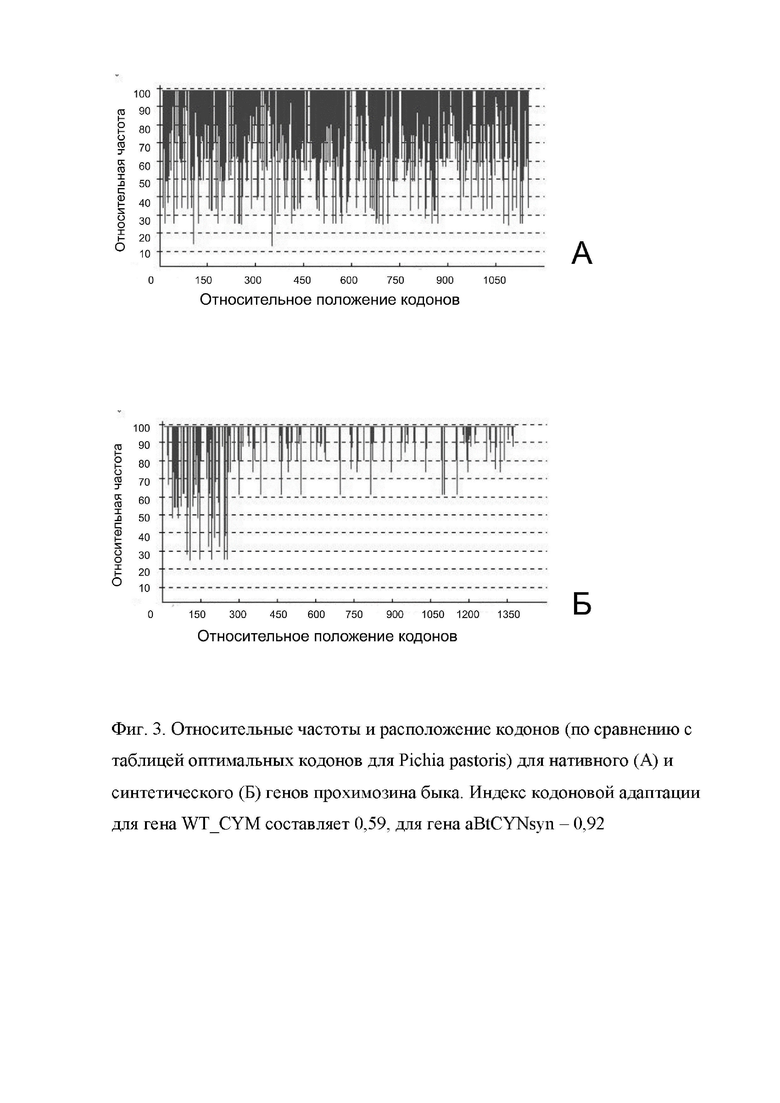
Фиг. 3. - Распределение частот использования кодонов вдоль кодирующих последовательностей природного и синтетического генов BtCYM.
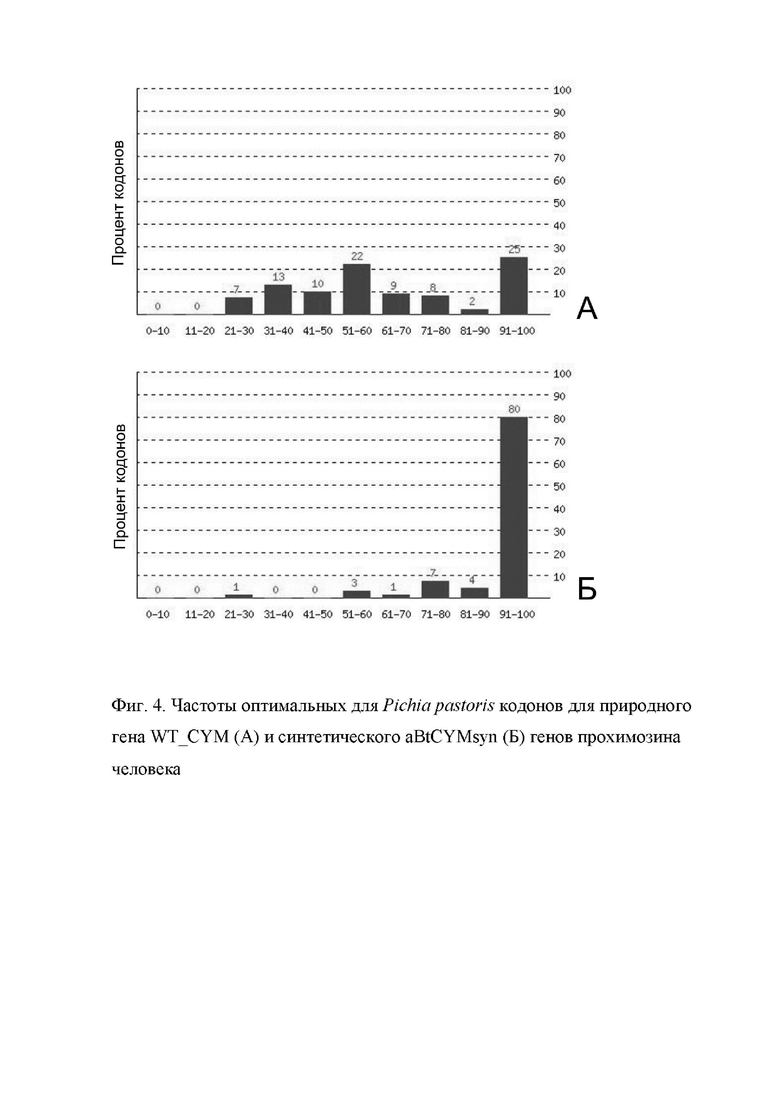
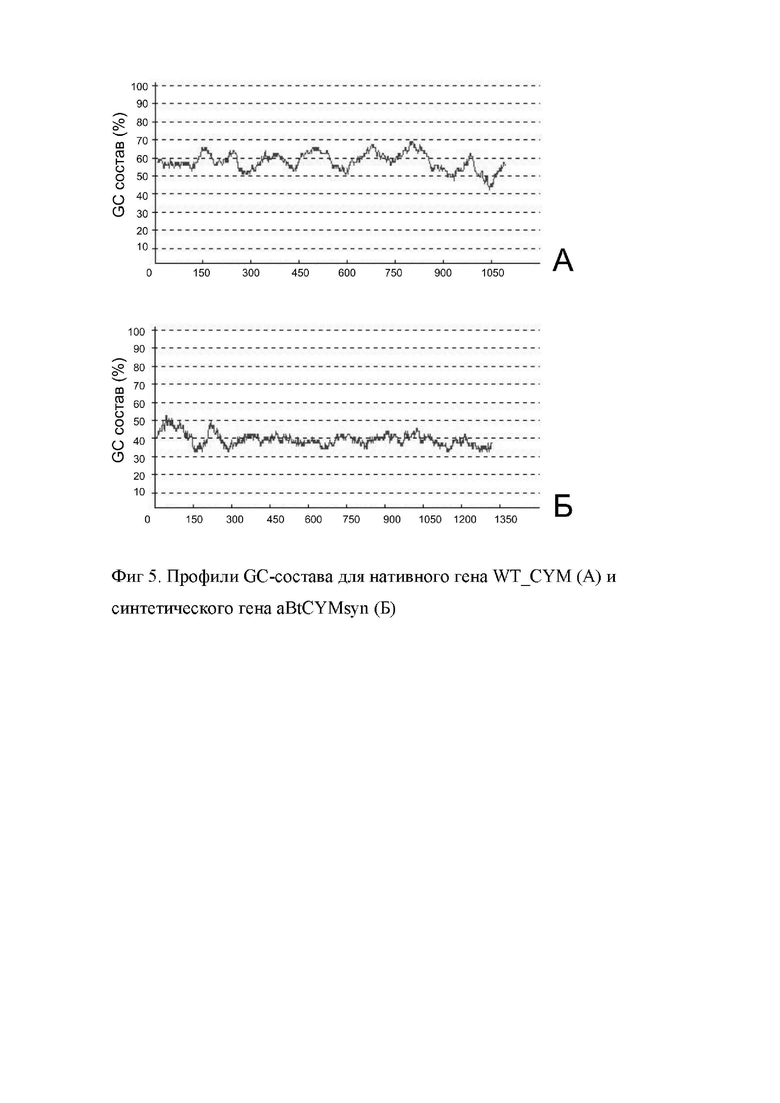
Фиг. 4. - Распределение GC-богатых участков.
Фиг. 5. - Выравнивание N-концевых аминокислотных последовательностей предшественника CYM c нативным сигнальным пептидом ppMFα1 и варианта предшественника CYM с модифицированным сигнальным пептидом ppMFα1.
Фиг. 6. - Динамика накопления активности CYM (столбцы, основная ось Y) и прироста биомассы (график, дополнительная ось Y) штаммом P. pastoris /SF002.
При осуществлении изобретения, помимо методов, подробно раскрытых в нижеследующих примерах, используют хорошо известные специалистам методики, описанные в руководствах по молекулярной биологии и генетической инженерии [28].
Пример 2. Дизайн синтетического гена аналога proCYM
Синтетический ген BtCYMsyn получают путем оптимизации кодирующей последовательности нативного гена СYM для синтеза в P. pastoris. При дизайне аминокислотной последовательности proCYM в качестве последовательности каталитически-активного домена используют участок последовательности proCYM, включающий 42-аминокислотную прочасть и часть, соответствующую зрелому химозину. На основании выбранной аминокислотной последовательности (SEQ ID No 2) c помощью программы Eugene [22] проводят подбор последовательности ДНК кодирующей области гена с учетом наиболее часто встречающихся кодонов для Pichia pastoris, отсутствия нежелательных элементов вторичных структур, криптических стоп-кодонов и участков полиаденилирования, удаляя также нежелательные рестрикционные сайты XhoI и NdeI. Степень гомологии синтетической последовательности BtCYMSyn и природной последовательности CYM составляет 60% (Фиг. 6).
По ряду критериев спроектированная последовательность (SEQ ID No1) должна обеспечивать более эффективную экспресссию proCYM в дрожжах по сравнению с природной последовательностью.
Частота встречаемости оптимальных для Pichia pastoris в последовательности BtCYMsyn выше, чем в природной последовательности, и, соответственно, индекс адаптации кодонов для P. pastoris для последовательности BtCYMsyn составляет 0,92, а для природной последовательности – 0,61. Синтетическая последовательность характеризуется пониженным GC-содержанием, что должно снижать вероятность образования нежелательных вторичных структур мРНК, препятствующих эффективной трансляции.
Полученную последовательность используют для синтеза ДНК BtCYMsyn по заказу на фирме TWIST Bioscence путем сборки по Гибсону (g-блок).
Пример 3. Конструирование плазмиды рSF001 для экспрессии для экспрессии и секреции proCYM в P. pastoris под контролем нативного сигнального пептида ppMFα1.
Для получения вектора, содержащего вариант гена BtCYMsyn, кодирующий preproCYM c нативным сигнальным пептидом ppMFa1 под контролем промотора AOX1, объединяют кодирующую последовательность proCYM c сигнальной последовательностью ppMFα1 и полученный модифицированный ген встраивают под контроль промотора AOX1 вектора pVR2. Все стандартные генно-инженерные и микробиологические манипуляции проводят по известным методикам [39].
В качестве источника ppMFα1 используют плазмидную ДНК pPICZ(alpha)A(https://www.snapgene.com/resources/plasmid-files/?set=yeast_plasmids&plasmid=pPICZ(alpha)_A). Объединение кодирующей последовательности ppMFα1 с кодирующей последовательностью BtCYMsyn проводят с использованием метода «рекомбинантной» ПЦР. Для этого сначала на матрице на матрице g-блока BtCYMsyn получают - фрагмент №1, а на матрице плазмиды pPICZ(alpha)A получают ПЦР фрагмент №2.
Фрагмент №1, размером 1200 пн, получают с использованием праймеров CYM_F (SEQ ID №3) и СYM_R (SEQ ID №4). Праймер CYM_F комплементарен N-концевой кодируюшей последовательности proCYM гена BtCYMsyn, а праймер CYM_R - C-концевой последовательности этого гена и фланкирован сайтом SalI.
Фрагмент №2, размером 226 пн, получают с использованием праймеров MFa1F (SEQ ID №5)/ MFa1R (SEQ ID №6). Праймер MFa1F специфичен к N-концевой кодирующей последовательности ppMFa1 плазмиды pPICZ(alpha)A, а праймер MFa1R, соответственно, к C-концевой последовательности ppMFa1 (по комплементарной цепи).
Полученные с использованием высокоточной ДНК полимеразы Q5 (“New England Biolabs Inc”) фрагменты №1 и №2 выделяют из агарозного геля и используют в качестве матрицы в ПЦР реакции, которую проводят, как описано ниже:
Смесь для ПЦР (50 мкл):
25 мкл 2-кратной готовой смест для полимеразы Q5 (“New England Biolabs Inc”);
1 мкл Фрагмента 1 (20 нг/мкл);
1 мкл Фрагмента 2 (20 нг/мкл)
4 мкл 5 мкМ праймера Mfa1_F
4 мкл 5 мкМ праймера CYM_R;
15 мкл деионизованной воды
Условия проведения ПЦР (30 циклов): 98°, 3' (денатурация), 98°, 20''; 60°, 30''; 72°,1' (амплификация).
После амплификации 5 мкл ПЦР смеси анализируют электрофорезом в 1% агарозном геле и выявляют гомогенный фрагмент размером около 1,3 тпн. Фрагмент выделяют из геля, гидролизуют рестриктазами NdeI/SalI и клонируют в NdeI/XhoI вектор pVR2. Отбор клонов с нужной вставкой среди полученных ампициллин-устойчивых трансформантов штамма DH10B проводят методом ПЦР скрининга по критерию образования фрагмента размером пн с праймерами pAOX1 (SEQ ID No 7, специфичен к промотору AOX1) и ChySeq (SEQ ID No 8, специфичен к центральной части кодирующей последовательности BtCYM) Полученные «положительные» клоны секвенируют и отбирают плазмиду с вариантом гена BtCYMsyn (aBtCYMsyn,SEQ ID No 9), в котором кодирующая последовательность BtCYM слита с кодирующей последовательностью ppMFα1 и не содержит неспецифических мутаций, обусловленных ошибками ПЦР. Плазмиду, выделенную из отобранного клона, обозначают как pSF001.
Пример 4. Конструирование вектора pSF002 для экспрессии и секреции proCYM в P. pastoris под контролем модифицированного сигнального пептида ppMFα1.
Для получения вектора, содержащего вариант гена BtCYMsyn, кодирующий preproCYM c модифицированным сигнальным пептидом ppMFα1 под контролем промотора AOX1, объединяют кодирующую последовательность proCYM c сигнальной последовательностью ppMFα1D.
В качестве источника ppMFα1D используют полученный от компании TWIST Bioscience сборкой по Гибсону синтетический фрагмент ДНК mFαD (SEQ ID No 11). Объединение коидирующей последовательности ppMFα1D с кодирующей последовательностью BtCYMsyn проводят с использованием метода «рекомбинантной» ПЦР как описано в примере 2. На матрице фрагмента mFαD получают ПЦР фрагмент №3 размером 250 пн с использованием праймеров MFa1DF (SEQ ID No 13)/ MFa1DR (SEQ ID No 14) и объединяют с фрагментом №1 гена BtCYMsyn (пример 1) методом рекомбинатной ПЦР с использованием праймеров MFa1DF/CYM_R. Полученный фрагмент ДНК выделяют, гидролизуют рестриктазами NdeI/SalI и клонируют в NdeI/XhoI вектор pVR2. Отбор клонов с нужной вставкой среди полученных ампициллин-устойчивых трансформантов штамма DH10B проводят методом ПЦР скрининга как описано в примере 2. Полученные «положительные» клоны секвенируют и отбирают плазмиду с вариантом гена BtCYMsyn (MaBtCYMsyn,SEQ ID No 15), в котором кодирующая последовательность BtCYM слита с кодирующей последовательностью ppMFa1D и не содержит неспецифических мутаций, обусловленных ошибками ПЦР. В результате получают плазмиду рSF002 размером 61 п.о., включающую ген MaBtCYMsyn, кодирующий аналог proCYM (Фиг. 1).
Пример 5. Получение рекомбинантного штамма дрожжей P. pastoris GS115/pSF001 - продуцента рекомбинантного proCYM.
В качестве штамма-реципиента используют штамм P. pastoris GS115. Трансформацию проводят с использованием 5 мкг линеаризованной плазмидной ДНК, для получения которой исходную плазмиду обрабатывают эндонуклеазой рестрикции PmeI, сайт узнавания которой расположен в промоторе гена AOXI.
Культуру дрожжей штамма Pichia pastoris GS115 выращивают на среде YPD, в аэробных условиях (250 об/мин) до оптической плотности 1-2 единицы. Осадок клеток, полученный из 100 мл культуры отделяют центрифугированием при 2000 g в течение 5 минут, ресуспендируют в 10 мл буфера, содержащего 1 мМ дитиотреитол и 100 мМ HEPES, pH 8,0 затем инкубируют на шейкере в течение 30 минут при комнатной температуре и после двукратной промывки холодной деионизированной водой ресуспендируют в 1 мл 1 М сорбитола. Суспензию разделяют на аликвоты по 40 мкл. Электропорацию проводят на приборе GenePulser Xcell (Biorad) в кюветах с зазором 2 мм при 2000 В, 25 мкФ, 600 Ом. После электропорации быстро добавляют 1 мл холодной среды YPD, инкубируют 2 часа при температуре 30°С и высевают на селективную среду для отбора трансформантов. Отбор трансформантов проводят на среде YPDS/Zeo, содержащей 1% дрожжевой экстракт, 2% пептон, 2% глюкозу, 1М сорбитол, 2% бактоагар, антибиотик Зеоцин (100 мкг/мл). В результате трансформации должна происходить интеграция экспрессирующей кассеты, состоящей из гена устойчивости к зеоцину и гена BtCYMsyn, слитого с сигнальным пептидом ppMFa1 в хромосомный локус AOX1 путем гомологичной рекомбинации. Таким образом, трансформанты, выросшие на агаризованной среде с селективным антибиотиком зеоцином, генетически стабильны как в присутствии, так и в отсутствие в среде культивирования селективного антибиотика.
Скрининг отобранных через 3-5 дней инкубации при 30°С трансформантов на способность к синтезу и секреции proCYM путем культивирования в 96-луночном глубоком планшете (объем лунок по 2 мл, круглодонная форма, Сellstar) следующим образом: в лунки добавляют по 300 мкл среды BMGY, стерильной зубочисткой переносят часть колонии в лунки, при этом перекалывая этой же зубочисткой колонии на 2 чашки с YPDZeo параллельно. Плашки инкубируют при 30°С на качалке, 650 об/мин в течение 60 часов, планшет при этом накрывают 2-мя слоями стерильной марли. Добавляют по 250 мкл среды ВММY, инкубируют далее в течение 12 часов. Добавляют по 50 мкл среды ВММY через каждые последующие 24 часа. На третьи сутки индукции клетки осаждают центрифугированием при 2500xg. Супернатанты (500 мкл) переносят в чистые 96-луночные глубокие планшеты. Супернатанты полученных культур анализируют на способность к секреции proCYM. Для этого 20 мкл супернатантов отобранных с помощью многоканальной пипетки переносят в лунки стандартного 96 луночного круглодонного планшета с помощью многоканальной пипетки. Далее в каждую лунку добавляют по 5 мкл 0,5 М раствора HCl и инкубируют 2 часа при комнатной температуре. Далее к лункам добавляют по 5 мкл 0,4 М раствора NaOH и 20 мкл 0,12 М раствора ацетат натрия, рН5,5, инкубируют 2 часа при комнатной температуре.
Далее к лункам добавляют по 50 мкл прогретого до 37°С раствора 26% сухого молока в 5,5 мМ CaCl2, планшеты инкубируют при 37 градусах 5 мин, центрифугируют при 2500xg, 5 мин.
Супернатанты стряхивают, анализируют визуально объемы осадка в лунках. Отбирают штаммы, образующие осадки максимального объема.
Отобранные штаммы используют для идентификации штамма-продуцента, обладающего максимальной продуктивностью в условиях культивирования в колбах. Посевной материал выращивают путем инкубации клеток отобранных трансформантов в течение 15-24 часов при температуре 29°С на среде YPD при постоянной аэрации (250 об/мин). Подготовленный посевной материал (200 мкл) используют для инокуляции 20 мл среды YPgM и ведут процесс биосинтеза в аэробных условиях при температуре 28°С в течение 96 часов, проводя индукцию метанолом.
Уровень продукции proCYM определяют путем кислотной активации аликвот супернатантов культуральной среды. Удельную казеингидролизующую активность выражают в казеиновых единицах фермента на мл культуры штамма-продуцента при помощи метода, описанного в работе [34].
Как видно из представленных данных (Фиг. 5), общая активность фермента достигает максимума через 96 часов инкубации; при этом в отобранном таким образом наилучшем штамме P. pastoris GS115/pSF001 она составляет 12 МКЕ/мл.
Пример 6. Получение рекомбинантного штамма дрожжей P. pastoris GS115/pSF001 - продуцента рекомбинантного proCYM.
Получение устойчивых к зеоцину трансформантов штамма P. pastoris GS115 (his 4, mut+) линеаризованной по сайту PmeI плазмидой pNC002, скрининг трансформантов, обладающих максимальной продуктивностью, отбор продуцентов, способных к наиболее высокому уровню синтеза и секреции proCYM при культивировании в колбах проводят как описано в примере 3.
Уровень продукции proCYM в отобранном таким образом штамме P. pastoris GS115/pSF002 составляет 15 МКЕ/мл, что на 20% выше чем в контрольном штамме P. pastoris GS115/pSF001
Таким образом, получен рекомбинантный штамм дрожжей P. pastoris GS115/pSF002, способный к биосинтезу секретируемого прохимозина быка, кодируемого синтетическим геном предшественника химозина с модифицированной сигнальной последовательностью секреции дрожжевого феромона альфа-фактора. При культивировании заявляемым способом уровень синтеза proCYM составляет 15 МКЕ/мл, что превосходит способ-ближайший аналог более чем в 2 раза.
Источники литературы:
1. Szecsi P.B., Harboe M. Chapter 5 - Chymosin // Handbook of Proteolytic Enzymes (Third Edition). Third Edit / ed. Rawlings N.D., Salvesen G. Academic Press, 2013. P. 37-42.
2. Kumar A. et al. Chymosin and other milk coagulants: Sources and biotechnological interventions // Crit. Rev. Biotechnol. 2010. Vol. 30, № 4. P. 243-258.
3. BARKHOLT PEDERSEN V., ASBÆK CHRISTENSEN K., FOLTMANN B. Investigations on the Activation of Bovine Prochymosin // Eur. J. Biochem. 1979. Vol. 94, № 2. P. 573-580.
4. Mellor J. et al. Efficient synthesis of enzymatically active calf chymosin in Saccharomyces cerevisiae. // Gene. 1983. Vol. 24, № 1. P. 1-14.
5. Almeida C.M., Simões I. Cardoon-based rennets for cheese production // Applied Microbiology and Biotechnology. Springer Verlag, 2018. Vol. 102, № 11. P. 4675-4686.
6. Vallejo J.A. et al. Short communication: A comparative analysis of recombinant chymosins. // J. Dairy Sci. 2012. Vol. 95, № 2. P. 609-613.
7. Ogel Z.B. Microbial production of recombinant rennet: Recent developments // Microbial Cultures and Enzymes in Dairy Technology. IGI Global, 2018. P. 222-233.
8. No Title [Electronic resource]. URL: https://www.validogen.com/pichia-pastoris/overview.
9. Ahmad M. et al. Protein expression in Pichia pastoris: Recent achievements and perspectives for heterologous protein production // Applied Microbiology and Biotechnology. 2014.
10. Gasser B. et al. Pichia pastoris: protein production host and model organism for biomedical research. // Future Microbiol. 2013. Vol. 8, № 2. P. 191-208.
11. Cregg J.M. et al. Recombinant Protein Expression in Pichia pastoris // Mol. Biotechnol. 2000. Vol. 16, № 1. P. 23-52.
12. McNeil B. et al. Microbial Production of Food Ingredients, Enzymes and Nutraceuticals // Microbial Production of Food Ingredients, Enzymes and Nutraceuticals. 2013.
13. Singh S. et al. Large-scale production of membrane proteins in Pichia pastoris: The production of g protein-coupled receptors as a case study // Methods Mol. Biol. 2012.
14. Laukens B., De Visscher C., Callewaert N. Engineering yeast for producing human glycoproteins: where are we now? // Future Microbiol. England, 2015. Vol. 10, № 1. P. 21-34.
15. Bill R.M. Playing catch-up with escherichia coli: Using yeast to increase success rates in recombinant protein production experiments // Front. Microbiol. 2014.
16. Tyagi A. et al. Expression of buffalo chymosin in Pichia pastoris for application in mozzarella cheese // LWT - Food Sci. Technol. 2017.
17. Wang N. et al. Expression and characterization of camel chymosin in Pichia pastoris // Protein Expr. Purif. 2015.
18. Ersöz F., İnan M. Large-scale production of yak (Bos grunniens) chymosin A in Pichia pastoris // Protein Expr. Purif. 2019.
19. Tyagi A. et al. Functional expression of recombinant goat chymosin in Pichia pastoris bioreactor cultures: A commercially viable alternate // LWT - Food Sci. Technol. 2016.
20. Dunn-Coleman N.S. et al. Commercial Levels of Chymosin Production by Aspergillus // Bio/Technology. 1991. Vol. 9, № 10. P. 976-981.
21. Parret A.H., Besir H., Meijers R. Critical reflections on synthetic gene design for recombinant protein expression // Curr. Opin. Struct. Biol. 2016. Vol. 38. P. 155-162.
22. Gaspar P. et al. EuGene: maximizing synthetic gene design for heterologous expression // Bioinformatics. 2012. Vol. 28, № 20. P. 2683-2684.
23. Espinoza-Molina J.A. et al. Codon Optimization of the “Bos Taurus Chymosin” Gene for the Production of Recombinant Chymosin in Pichia pastoris // Mol. Biotechnol. 2016.
24. Damasceno L.M., Huang C.J., Batt C.A. Protein secretion in Pichia pastoris and advances in protein production // Applied Microbiology and Biotechnology. 2012. Vol. 93, № 1. P. 31-39.
25. Liang S. et al. Endogenous signal peptides efficiently mediate the secretion of recombinant proteins in Pichia pastoris. // Biotechnol. Lett. Netherlands, 2013. Vol. 35, № 1. P. 97-105.
26. Puxbaum V., Mattanovich D., Gasser B. Quo vadis? The challenges of recombinant protein folding and secretion in Pichia pastoris // Applied Microbiology and Biotechnology. 2015.
27. Brake A.J. et al. Alpha-factor-directed synthesis and secretion of mature foreign proteins in Saccharomyces cerevisiae. // Proc. Natl. Acad. Sci. U. S. A. 1984. Vol. 81, № 15. P. 4642-4646.
28. Dancourt J., Barlowe C. Protein sorting receptors in the early secretory pathway. // Annu. Rev. Biochem. United States, 2010. Vol. 79. P. 777-802.
29. Fuller R.S., Sterne R.E., Thorner J. Enzymes required for yeast prohormone processing. // Annu. Rev. Physiol. United States, 1988. Vol. 50. P. 345–362.
30. Delic M. et al. The secretory pathway: exploring yeast diversity. // FEMS Microbiol. Rev. 2013. Vol. 37, № 6. P. 872–914.
31. Yang Z., Zhang Z. Engineering strategies for enhanced production of protein and bio-products in Pichia pastoris: A review // Biotechnology Advances. 2018.
32. Lin-Cereghino G.P. et al. The effect of α-mating factor secretion signal mutations on recombinant protein expression in Pichia pastoris // Gene. 2013. Vol. 519, № 2. P. 311-317.
33. Ahn J. et al. Codon optimization of Saccharomyces cerevisiae mating factor alpha prepro-leader to improve recombinant protein production in Pichia pastoris // Biotechnol. Lett. Springer Netherlands, 2016. Vol. 38, № 12. P. 2137-2143.
34. Noseda D.G. et al. Cloning, expression and optimized production in a bioreactor of bovine chymosin B in Pichia (Komagataella) pastoris under AOX1 promoter // Protein Expr. Purif. 2013.
35. Espinoza-Molina J.A. et al. Codon Optimization of the “Bos Taurus Chymosin” Gene for the Production of Recombinant Chymosin in Pichia pastoris. // Mol. Biotechnol. 2016. Vol. 58, № 10. P. 657-664.
36. Lin-Cereghino G.P. et al. The effect of α-mating factor secretion signal mutations on recombinant protein expression in Pichia pastoris. // Gene. NIH Public Access, 2013. Vol. 519, № 2. P. 311-317.
37. Redo V.A., Novikova E.K., Eldarov M.A. Expression of modified oxidase of D-aminoacids of Trigonopsis variabilis in methylotrophic yeasts Pichia pastoris // Appl. Biochem. Microbiol. 2011. Vol. 47, № 1.
38. Редо В.А.., Новикова Е.К., Эльдаров М.А. Экспрессия модифицированной оксидазы D-аминокислот Trigonopsis variabilis в метилотрофных дрожжах Pichia pastoris // Прикладная биохимия и микробиология. 2011. Vol. 47, № 1. P. 39-45.
39. Sambrook J. Molecular cloning: a laboratory manual. Third edition. Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press, [2001] ©2001.
--->
Аминокислотные последовательности
<110> FRC Biotechnology RAS
<120> Рекомбинантная ДНК, кодирующая вариант предшественника химозина
быка (BtCYM) с модифицированной сигнальной последовательностью
секреции препроальфафактора дрожжей Saccharmomyces cerevisiae,
рекомбинантная плазмида pCF002, обеспечивающая синтез BtCYM в
клетках Pichia pastoris и штамм Pichia pastoris GS115 /p SF002 -
продуцент рекомбинантного BtCYM
<130> 001
<160> 16
<170> PatentIn version 3.5
<210> 1
<211> 1107
<212> DNA
<213> Artificial Sequence
<220>
<223> Синтетический ген прохимозина быка
<220>
<221> CDS
<222> (1)..(1095)
<400> 1
gct gaa att act aga atc cca ttg tat aag ggt aaa tct ttg aga aag 48
Ala Glu Ile Thr Arg Ile Pro Leu Tyr Lys Gly Lys Ser Leu Arg Lys
1 5 10 15
gct ttg aaa gaa cat ggt ttg ttg gag gat ttc ttg caa aag caa caa 96
Ala Leu Lys Glu His Gly Leu Leu Glu Asp Phe Leu Gln Lys Gln Gln
20 25 30
tac ggt atc tct tct aag tac tct ggt ttc ggt gaa gtt gct tct gtt 144
Tyr Gly Ile Ser Ser Lys Tyr Ser Gly Phe Gly Glu Val Ala Ser Val
35 40 45
cca ttg act aac tac ttg gat tct caa tac ttc ggt aaa atc tac ttg 192
Pro Leu Thr Asn Tyr Leu Asp Ser Gln Tyr Phe Gly Lys Ile Tyr Leu
50 55 60
ggt act cca cct caa gag ttt act gtt ttg ttc gat act ggt tct tct 240
Gly Thr Pro Pro Gln Glu Phe Thr Val Leu Phe Asp Thr Gly Ser Ser
65 70 75 80
gat ttc tgg gtt cct tct atc tac tgt aag tct aac gct tgt aag aac 288
Asp Phe Trp Val Pro Ser Ile Tyr Cys Lys Ser Asn Ala Cys Lys Asn
85 90 95
cat caa aga ttc gat cca aga aag tct tct act ttc caa aac ttg ggt 336
His Gln Arg Phe Asp Pro Arg Lys Ser Ser Thr Phe Gln Asn Leu Gly
100 105 110
aaa cct ttg tct att cac tat ggt act ggt tct atg caa ggt att ttg 384
Lys Pro Leu Ser Ile His Tyr Gly Thr Gly Ser Met Gln Gly Ile Leu
115 120 125
ggt tac gat act gtt act gtt tct aac atc gtt gat att caa caa act 432
Gly Tyr Asp Thr Val Thr Val Ser Asn Ile Val Asp Ile Gln Gln Thr
130 135 140
gtt ggt ttg tct act caa gaa cct gga gat gtt ttt act tac gct gag 480
Val Gly Leu Ser Thr Gln Glu Pro Gly Asp Val Phe Thr Tyr Ala Glu
145 150 155 160
ttc gat ggt att ttg ggt atg gct tat cca tct ttg gct tct gaa tac 528
Phe Asp Gly Ile Leu Gly Met Ala Tyr Pro Ser Leu Ala Ser Glu Tyr
165 170 175
tct atc cct gtt ttc gat aac atg atg aac aga cat ttg gtt gct caa 576
Ser Ile Pro Val Phe Asp Asn Met Met Asn Arg His Leu Val Ala Gln
180 185 190
gat ttg ttc tct gtt tac atg gat aga aac ggt caa gag tct atg ttg 624
Asp Leu Phe Ser Val Tyr Met Asp Arg Asn Gly Gln Glu Ser Met Leu
195 200 205
act ttg ggt gct att gat cca tct tac tat act ggt tct ttg cac tgg 672
Thr Leu Gly Ala Ile Asp Pro Ser Tyr Tyr Thr Gly Ser Leu His Trp
210 215 220
gtt cct gtt act gtt caa caa tac tgg caa ttc act gtt gat tct gtt 720
Val Pro Val Thr Val Gln Gln Tyr Trp Gln Phe Thr Val Asp Ser Val
225 230 235 240
act att tct ggt gtt gtt gtt gct tgt gag ggt ggt tgt caa gct att 768
Thr Ile Ser Gly Val Val Val Ala Cys Glu Gly Gly Cys Gln Ala Ile
245 250 255
ttg gat act ggt act tct aag ttg gtt ggt cca tct tct gat atc ttg 816
Leu Asp Thr Gly Thr Ser Lys Leu Val Gly Pro Ser Ser Asp Ile Leu
260 265 270
aac atc caa caa gct att ggt gct act caa aac caa tac ggt gaa ttt 864
Asn Ile Gln Gln Ala Ile Gly Ala Thr Gln Asn Gln Tyr Gly Glu Phe
275 280 285
gat atc gat tgt gat aac ttg tct tac atg cct act gtt gtt ttc gag 912
Asp Ile Asp Cys Asp Asn Leu Ser Tyr Met Pro Thr Val Val Phe Glu
290 295 300
att aat ggt aaa atg tac cca ttg act cct tct gct tac act tct caa 960
Ile Asn Gly Lys Met Tyr Pro Leu Thr Pro Ser Ala Tyr Thr Ser Gln
305 310 315 320
gat caa ggt ttt tgt act tct ggt ttc caa tct gaa aat cac tct caa 1008
Asp Gln Gly Phe Cys Thr Ser Gly Phe Gln Ser Glu Asn His Ser Gln
325 330 335
aag tgg att ttg gga gat gtt ttt att aga gag tac tac tct gtt ttc 1056
Lys Trp Ile Leu Gly Asp Val Phe Ile Arg Glu Tyr Tyr Ser Val Phe
340 345 350
gat aga gct aac aat ttg gtt ggt ttg gct aaa gct att taagtcgaca ac 1107
Asp Arg Ala Asn Asn Leu Val Gly Leu Ala Lys Ala Ile
355 360 365
<210> 2
<211> 365
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 2
Ala Glu Ile Thr Arg Ile Pro Leu Tyr Lys Gly Lys Ser Leu Arg Lys
1 5 10 15
Ala Leu Lys Glu His Gly Leu Leu Glu Asp Phe Leu Gln Lys Gln Gln
20 25 30
Tyr Gly Ile Ser Ser Lys Tyr Ser Gly Phe Gly Glu Val Ala Ser Val
35 40 45
Pro Leu Thr Asn Tyr Leu Asp Ser Gln Tyr Phe Gly Lys Ile Tyr Leu
50 55 60
Gly Thr Pro Pro Gln Glu Phe Thr Val Leu Phe Asp Thr Gly Ser Ser
65 70 75 80
Asp Phe Trp Val Pro Ser Ile Tyr Cys Lys Ser Asn Ala Cys Lys Asn
85 90 95
His Gln Arg Phe Asp Pro Arg Lys Ser Ser Thr Phe Gln Asn Leu Gly
100 105 110
Lys Pro Leu Ser Ile His Tyr Gly Thr Gly Ser Met Gln Gly Ile Leu
115 120 125
Gly Tyr Asp Thr Val Thr Val Ser Asn Ile Val Asp Ile Gln Gln Thr
130 135 140
Val Gly Leu Ser Thr Gln Glu Pro Gly Asp Val Phe Thr Tyr Ala Glu
145 150 155 160
Phe Asp Gly Ile Leu Gly Met Ala Tyr Pro Ser Leu Ala Ser Glu Tyr
165 170 175
Ser Ile Pro Val Phe Asp Asn Met Met Asn Arg His Leu Val Ala Gln
180 185 190
Asp Leu Phe Ser Val Tyr Met Asp Arg Asn Gly Gln Glu Ser Met Leu
195 200 205
Thr Leu Gly Ala Ile Asp Pro Ser Tyr Tyr Thr Gly Ser Leu His Trp
210 215 220
Val Pro Val Thr Val Gln Gln Tyr Trp Gln Phe Thr Val Asp Ser Val
225 230 235 240
Thr Ile Ser Gly Val Val Val Ala Cys Glu Gly Gly Cys Gln Ala Ile
245 250 255
Leu Asp Thr Gly Thr Ser Lys Leu Val Gly Pro Ser Ser Asp Ile Leu
260 265 270
Asn Ile Gln Gln Ala Ile Gly Ala Thr Gln Asn Gln Tyr Gly Glu Phe
275 280 285
Asp Ile Asp Cys Asp Asn Leu Ser Tyr Met Pro Thr Val Val Phe Glu
290 295 300
Ile Asn Gly Lys Met Tyr Pro Leu Thr Pro Ser Ala Tyr Thr Ser Gln
305 310 315 320
Asp Gln Gly Phe Cys Thr Ser Gly Phe Gln Ser Glu Asn His Ser Gln
325 330 335
Lys Trp Ile Leu Gly Asp Val Phe Ile Arg Glu Tyr Tyr Ser Val Phe
340 345 350
Asp Arg Ala Asn Asn Leu Val Gly Leu Ala Lys Ala Ile
355 360 365
<210> 3
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Прямой праймер для амплификации кодирующей последовательности
гена SynCYM
<400> 3
gctgaaatta ctagaatccc a 21
<210> 4
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Обратный праймер для амплификации кодирующей последовательности
гена SynCYM
<400> 4
gttgtcgact taaatagctt tagccaaacc aacca 35
<210> 5
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер для ПЦР-сборки гена aBtCYMsyn
<400> 5
catatgagat ttccttcaat ttttactg 28
<210> 6
<211> 38
<212> DNA
<213> Saccharomyces cerevisiae
<400> 6
gtaatttcag cgaattcagc ttcagcctct cttttctc 38
<210> 7
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Секвенирующий праймер промотор АОХ1
<400> 7
gactggttcc aattgacaag c 21
<210> 8
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер для секвенирования гена BtCYM
<400> 8
gaacccagtg caaagaacca 20
<210> 9
<211> 1374
<212> DNA
<213> artificial sequence
<220>
<223> Synthetic Bos taurus prochymosin gene cloned in pPicZa vector
<220>
<221> CDS
<222> (1)..(1368)
<220>
<221> sig_peptide
<222> (1)..(267)
<223> Native MFa preproregion
<220>
<221> mat_peptide
<222> (268)..(1368)
<223> Prochmosin
<400> 9
atg aga ttt cct tca att ttt act gct gtt tta ttc gca gca tcc tcc 48
Met Arg Phe Pro Ser Ile Phe Thr Ala Val Leu Phe Ala Ala Ser Ser
-85 -80 -75
gca tta gct gct cca gtc aac act aca aca gaa gat gaa acg gca caa 96
Ala Leu Ala Ala Pro Val Asn Thr Thr Thr Glu Asp Glu Thr Ala Gln
-70 -65 -60
att ccg gct gaa gct gtc atc ggt tac tca gat tta gaa ggg gat ttc 144
Ile Pro Ala Glu Ala Val Ile Gly Tyr Ser Asp Leu Glu Gly Asp Phe
-55 -50 -45
gat gtt gct gtt ttg cca ttt tcc aac agc aca aat aac ggg tta ttg 192
Asp Val Ala Val Leu Pro Phe Ser Asn Ser Thr Asn Asn Gly Leu Leu
-40 -35 -30
ttt ata aat act act att gcc agc att gct gct aaa gaa gaa ggg gta 240
Phe Ile Asn Thr Thr Ile Ala Ser Ile Ala Ala Lys Glu Glu Gly Val
-25 -20 -15 -10
tct ctc gag aaa aga gag gct gaa gct gaa ttc gct gaa att act aga 288
Ser Leu Glu Lys Arg Glu Ala Glu Ala Glu Phe Ala Glu Ile Thr Arg
-5 -1 1 5
atc cca ttg tat aag ggt aaa tct ttg aga aag gct ttg aaa gaa cat 336
Ile Pro Leu Tyr Lys Gly Lys Ser Leu Arg Lys Ala Leu Lys Glu His
10 15 20
ggt ttg ttg gag gat ttc ttg caa aag caa caa tac ggt atc tct tct 384
Gly Leu Leu Glu Asp Phe Leu Gln Lys Gln Gln Tyr Gly Ile Ser Ser
25 30 35
aag tac tct ggt ttc ggt gaa gtt gct tct gtt cca ttg act aac tac 432
Lys Tyr Ser Gly Phe Gly Glu Val Ala Ser Val Pro Leu Thr Asn Tyr
40 45 50 55
ttg gat tct caa tac ttc ggt aaa atc tac ttg ggt act cca cct caa 480
Leu Asp Ser Gln Tyr Phe Gly Lys Ile Tyr Leu Gly Thr Pro Pro Gln
60 65 70
gag ttt act gtt ttg ttc gat act ggt tct tct gat ttc tgg gtt cct 528
Glu Phe Thr Val Leu Phe Asp Thr Gly Ser Ser Asp Phe Trp Val Pro
75 80 85
tct atc tac tgt aag tct aac gct tgt aag aac cat caa aga ttc gat 576
Ser Ile Tyr Cys Lys Ser Asn Ala Cys Lys Asn His Gln Arg Phe Asp
90 95 100
cca aga aag tct tct act ttc caa aac ttg ggt aaa cct ttg tct att 624
Pro Arg Lys Ser Ser Thr Phe Gln Asn Leu Gly Lys Pro Leu Ser Ile
105 110 115
cac tat ggt act ggt tct atg caa ggt att ttg ggt tac gat act gtt 672
His Tyr Gly Thr Gly Ser Met Gln Gly Ile Leu Gly Tyr Asp Thr Val
120 125 130 135
act gtt tct aac atc gtt gat att caa caa act gtt ggt ttg tct act 720
Thr Val Ser Asn Ile Val Asp Ile Gln Gln Thr Val Gly Leu Ser Thr
140 145 150
caa gaa cct gga gat gtt ttt act tac gct gag ttc gat ggt att ttg 768
Gln Glu Pro Gly Asp Val Phe Thr Tyr Ala Glu Phe Asp Gly Ile Leu
155 160 165
ggt atg gct tat cca tct ttg gct tct gaa tac tct atc cct gtt ttc 816
Gly Met Ala Tyr Pro Ser Leu Ala Ser Glu Tyr Ser Ile Pro Val Phe
170 175 180
gat aac atg atg aac aga cat ttg gtt gct caa gat ttg ttc tct gtt 864
Asp Asn Met Met Asn Arg His Leu Val Ala Gln Asp Leu Phe Ser Val
185 190 195
tac atg gat aga aac ggt caa gag tct atg ttg act ttg ggt gct att 912
Tyr Met Asp Arg Asn Gly Gln Glu Ser Met Leu Thr Leu Gly Ala Ile
200 205 210 215
gat cca tct tac tat act ggt tct ttg cac tgg gtt cct gtt act gtt 960
Asp Pro Ser Tyr Tyr Thr Gly Ser Leu His Trp Val Pro Val Thr Val
220 225 230
caa caa tac tgg caa ttc act gtt gat tct gtt act att tct ggt gtt 1008
Gln Gln Tyr Trp Gln Phe Thr Val Asp Ser Val Thr Ile Ser Gly Val
235 240 245
gtt gtt gct tgt gag ggt ggt tgt caa gct att ttg gat act ggt act 1056
Val Val Ala Cys Glu Gly Gly Cys Gln Ala Ile Leu Asp Thr Gly Thr
250 255 260
tct aag ttg gtt ggt cca tct tct gat atc ttg aac atc caa caa gct 1104
Ser Lys Leu Val Gly Pro Ser Ser Asp Ile Leu Asn Ile Gln Gln Ala
265 270 275
att ggt gct act caa aac caa tac ggt gaa ttt gat atc gat tgt gat 1152
Ile Gly Ala Thr Gln Asn Gln Tyr Gly Glu Phe Asp Ile Asp Cys Asp
280 285 290 295
aac ttg tct tac atg cct act gtt gtt ttc gag att aat ggt aaa atg 1200
Asn Leu Ser Tyr Met Pro Thr Val Val Phe Glu Ile Asn Gly Lys Met
300 305 310
tac cca ttg act cct tct gct tac act tct caa gat caa ggt ttt tgt 1248
Tyr Pro Leu Thr Pro Ser Ala Tyr Thr Ser Gln Asp Gln Gly Phe Cys
315 320 325
act tct ggt ttc caa tct gaa aat cac tct caa aag tgg att ttg gga 1296
Thr Ser Gly Phe Gln Ser Glu Asn His Ser Gln Lys Trp Ile Leu Gly
330 335 340
gat gtt ttt att aga gag tac tac tct gtt ttc gat aga gct aac aat 1344
Asp Val Phe Ile Arg Glu Tyr Tyr Ser Val Phe Asp Arg Ala Asn Asn
345 350 355
ttg gtt ggt ttg gct aaa gct att taatag 1374
Leu Val Gly Leu Ala Lys Ala Ile
360 365
<210> 10
<211> 456
<212> PRT
<213> artificial sequence
<220>
<223> Synthetic Construct
<400> 10
Met Arg Phe Pro Ser Ile Phe Thr Ala Val Leu Phe Ala Ala Ser Ser
-85 -80 -75
Ala Leu Ala Ala Pro Val Asn Thr Thr Thr Glu Asp Glu Thr Ala Gln
-70 -65 -60
Ile Pro Ala Glu Ala Val Ile Gly Tyr Ser Asp Leu Glu Gly Asp Phe
-55 -50 -45
Asp Val Ala Val Leu Pro Phe Ser Asn Ser Thr Asn Asn Gly Leu Leu
-40 -35 -30
Phe Ile Asn Thr Thr Ile Ala Ser Ile Ala Ala Lys Glu Glu Gly Val
-25 -20 -15 -10
Ser Leu Glu Lys Arg Glu Ala Glu Ala Glu Phe Ala Glu Ile Thr Arg
-5 -1 1 5
Ile Pro Leu Tyr Lys Gly Lys Ser Leu Arg Lys Ala Leu Lys Glu His
10 15 20
Gly Leu Leu Glu Asp Phe Leu Gln Lys Gln Gln Tyr Gly Ile Ser Ser
25 30 35
Lys Tyr Ser Gly Phe Gly Glu Val Ala Ser Val Pro Leu Thr Asn Tyr
40 45 50 55
Leu Asp Ser Gln Tyr Phe Gly Lys Ile Tyr Leu Gly Thr Pro Pro Gln
60 65 70
Glu Phe Thr Val Leu Phe Asp Thr Gly Ser Ser Asp Phe Trp Val Pro
75 80 85
Ser Ile Tyr Cys Lys Ser Asn Ala Cys Lys Asn His Gln Arg Phe Asp
90 95 100
Pro Arg Lys Ser Ser Thr Phe Gln Asn Leu Gly Lys Pro Leu Ser Ile
105 110 115
His Tyr Gly Thr Gly Ser Met Gln Gly Ile Leu Gly Tyr Asp Thr Val
120 125 130 135
Thr Val Ser Asn Ile Val Asp Ile Gln Gln Thr Val Gly Leu Ser Thr
140 145 150
Gln Glu Pro Gly Asp Val Phe Thr Tyr Ala Glu Phe Asp Gly Ile Leu
155 160 165
Gly Met Ala Tyr Pro Ser Leu Ala Ser Glu Tyr Ser Ile Pro Val Phe
170 175 180
Asp Asn Met Met Asn Arg His Leu Val Ala Gln Asp Leu Phe Ser Val
185 190 195
Tyr Met Asp Arg Asn Gly Gln Glu Ser Met Leu Thr Leu Gly Ala Ile
200 205 210 215
Asp Pro Ser Tyr Tyr Thr Gly Ser Leu His Trp Val Pro Val Thr Val
220 225 230
Gln Gln Tyr Trp Gln Phe Thr Val Asp Ser Val Thr Ile Ser Gly Val
235 240 245
Val Val Ala Cys Glu Gly Gly Cys Gln Ala Ile Leu Asp Thr Gly Thr
250 255 260
Ser Lys Leu Val Gly Pro Ser Ser Asp Ile Leu Asn Ile Gln Gln Ala
265 270 275
Ile Gly Ala Thr Gln Asn Gln Tyr Gly Glu Phe Asp Ile Asp Cys Asp
280 285 290 295
Asn Leu Ser Tyr Met Pro Thr Val Val Phe Glu Ile Asn Gly Lys Met
300 305 310
Tyr Pro Leu Thr Pro Ser Ala Tyr Thr Ser Gln Asp Gln Gly Phe Cys
315 320 325
Thr Ser Gly Phe Gln Ser Glu Asn His Ser Gln Lys Trp Ile Leu Gly
330 335 340
Asp Val Phe Ile Arg Glu Tyr Tyr Ser Val Phe Asp Arg Ala Asn Asn
345 350 355
Leu Val Gly Leu Ala Lys Ala Ile
360 365
<210> 11
<211> 219
<212> DNA
<213> Saccharomyces cerevisiae
<220>
<221> CDS
<222> (1)..(219)
<400> 11
atg aga ttc cca tct atc ttt act gcc gtt ttg ttt gct gct tcc tct 48
Met Arg Phe Pro Ser Ile Phe Thr Ala Val Leu Phe Ala Ala Ser Ser
1 5 10 15
gct ttg gca gct cca gtt aac aca act act gag gat gaa act gct caa 96
Ala Leu Ala Ala Pro Val Asn Thr Thr Thr Glu Asp Glu Thr Ala Gln
20 25 30
att cct gct gag gca gtt att ggt tac tct gat ttg gaa ggt gac ttt 144
Ile Pro Ala Glu Ala Val Ile Gly Tyr Ser Asp Leu Glu Gly Asp Phe
35 40 45
gat gtt gct gtc ttg cct ttc tct gcc tct att gct gcc aag gaa gag 192
Asp Val Ala Val Leu Pro Phe Ser Ala Ser Ile Ala Ala Lys Glu Glu
50 55 60
ggt gtt tct ttg gac aag aga gag gct 219
Gly Val Ser Leu Asp Lys Arg Glu Ala
65 70
<210> 12
<211> 73
<212> PRT
<213> Saccharomyces cerevisiae
<400> 12
Met Arg Phe Pro Ser Ile Phe Thr Ala Val Leu Phe Ala Ala Ser Ser
1 5 10 15
Ala Leu Ala Ala Pro Val Asn Thr Thr Thr Glu Asp Glu Thr Ala Gln
20 25 30
Ile Pro Ala Glu Ala Val Ile Gly Tyr Ser Asp Leu Glu Gly Asp Phe
35 40 45
Asp Val Ala Val Leu Pro Phe Ser Ala Ser Ile Ala Ala Lys Glu Glu
50 55 60
Gly Val Ser Leu Asp Lys Arg Glu Ala
65 70
<210> 13
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Прямой праймер для ПЦР-амплификации последовательности
модифицированного сигнального пептида ppMFa1
<400> 13
gatacatatg agattcccat ctatctttac 30
<210> 14
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> Обратный праймер для ПЦР-амплификации последовательности
модифицированного сигнального пептида ppMFa1
<400> 14
tgggattcta gtaatttcag cttcagcctc tctcttgtcc aaaga 45
<210> 15
<211> 1332
<212> DNA
<213> Artificial Sequence
<220>
<223> Синтетический ген химозина быка с оптимизированной для Pichia
pastoris кодирующей последовательностью
<220>
<221> CDS
<222> (4)..(1320)
<223> Препрохимозин
<220>
<221> sig_peptide
<222> (4)..(225)
<223> Модифицированный сигрнальный пептид препроальфафактора
<220>
<221> misc_feature
<222> (226)..(1320)
<223> Прохимозин
<400> 15
cat atg aga ttc cca tct atc ttt act gcc gtt ttg ttt gct gct tcc 48
Met Arg Phe Pro Ser Ile Phe Thr Ala Val Leu Phe Ala Ala Ser
1 5 10 15
tct gct ttg gca gct cca gtt aac aca act act gag gat gaa act gct 96
Ser Ala Leu Ala Ala Pro Val Asn Thr Thr Thr Glu Asp Glu Thr Ala
20 25 30
caa att cct gct gag gca gtt att ggt tac tct gat ttg gaa ggt gac 144
Gln Ile Pro Ala Glu Ala Val Ile Gly Tyr Ser Asp Leu Glu Gly Asp
35 40 45
ttt gat gtt gct gtc ttg cct ttc tct gcc tct att gct gcc aag gaa 192
Phe Asp Val Ala Val Leu Pro Phe Ser Ala Ser Ile Ala Ala Lys Glu
50 55 60
gag ggt gtt tct ttg gac aag aga gag gct gaa gct gaa att act aga 240
Glu Gly Val Ser Leu Asp Lys Arg Glu Ala Glu Ala Glu Ile Thr Arg
65 70 75
atc cca ttg tat aag ggt aaa tct ttg aga aag gct ttg aaa gaa cat 288
Ile Pro Leu Tyr Lys Gly Lys Ser Leu Arg Lys Ala Leu Lys Glu His
80 85 90 95
ggt ttg ttg gag gat ttc ttg caa aag caa caa tac ggt atc tct tct 336
Gly Leu Leu Glu Asp Phe Leu Gln Lys Gln Gln Tyr Gly Ile Ser Ser
100 105 110
aag tac tct ggt ttc ggt gaa gtt gct tct gtt cca ttg act aac tac 384
Lys Tyr Ser Gly Phe Gly Glu Val Ala Ser Val Pro Leu Thr Asn Tyr
115 120 125
ttg gat tct caa tac ttc ggt aaa atc tac ttg ggt act cca cct caa 432
Leu Asp Ser Gln Tyr Phe Gly Lys Ile Tyr Leu Gly Thr Pro Pro Gln
130 135 140
gag ttt act gtt ttg ttc gat act ggt tct tct gat ttc tgg gtt cct 480
Glu Phe Thr Val Leu Phe Asp Thr Gly Ser Ser Asp Phe Trp Val Pro
145 150 155
tct atc tac tgt aag tct aac gct tgt aag aac cat caa aga ttc gat 528
Ser Ile Tyr Cys Lys Ser Asn Ala Cys Lys Asn His Gln Arg Phe Asp
160 165 170 175
cca aga aag tct tct act ttc caa aac ttg ggt aaa cct ttg tct att 576
Pro Arg Lys Ser Ser Thr Phe Gln Asn Leu Gly Lys Pro Leu Ser Ile
180 185 190
cac tat ggt act ggt tct atg caa ggt att ttg ggt tac gat act gtt 624
His Tyr Gly Thr Gly Ser Met Gln Gly Ile Leu Gly Tyr Asp Thr Val
195 200 205
act gtt tct aac atc gtt gat att caa caa act gtt ggt ttg tct act 672
Thr Val Ser Asn Ile Val Asp Ile Gln Gln Thr Val Gly Leu Ser Thr
210 215 220
caa gaa cct gga gat gtt ttt act tac gct gag ttc gat ggt att ttg 720
Gln Glu Pro Gly Asp Val Phe Thr Tyr Ala Glu Phe Asp Gly Ile Leu
225 230 235
ggt atg gct tat cca tct ttg gct tct gaa tac tct atc cct gtt ttc 768
Gly Met Ala Tyr Pro Ser Leu Ala Ser Glu Tyr Ser Ile Pro Val Phe
240 245 250 255
gat aac atg atg aac aga cat ttg gtt gct caa gat ttg ttc tct gtt 816
Asp Asn Met Met Asn Arg His Leu Val Ala Gln Asp Leu Phe Ser Val
260 265 270
tac atg gat aga aac ggt caa gag tct atg ttg act ttg ggt gct att 864
Tyr Met Asp Arg Asn Gly Gln Glu Ser Met Leu Thr Leu Gly Ala Ile
275 280 285
gat cca tct tac tat act ggt tct ttg cac tgg gtt cct gtt act gtt 912
Asp Pro Ser Tyr Tyr Thr Gly Ser Leu His Trp Val Pro Val Thr Val
290 295 300
caa caa tac tgg caa ttc act gtt gat tct gtt act att tct ggt gtt 960
Gln Gln Tyr Trp Gln Phe Thr Val Asp Ser Val Thr Ile Ser Gly Val
305 310 315
gtt gtt gct tgt gag ggt ggt tgt caa gct att ttg gat act ggt act 1008
Val Val Ala Cys Glu Gly Gly Cys Gln Ala Ile Leu Asp Thr Gly Thr
320 325 330 335
tct aag ttg gtt ggt cca tct tct gat atc ttg aac atc caa caa gct 1056
Ser Lys Leu Val Gly Pro Ser Ser Asp Ile Leu Asn Ile Gln Gln Ala
340 345 350
att ggt gct act caa aac caa tac ggt gaa ttt gat atc gat tgt gat 1104
Ile Gly Ala Thr Gln Asn Gln Tyr Gly Glu Phe Asp Ile Asp Cys Asp
355 360 365
aac ttg tct tac atg cct act gtt gtt ttc gag att aat ggt aaa atg 1152
Asn Leu Ser Tyr Met Pro Thr Val Val Phe Glu Ile Asn Gly Lys Met
370 375 380
tac cca ttg act cct tct gct tac act tct caa gat caa ggt ttt tgt 1200
Tyr Pro Leu Thr Pro Ser Ala Tyr Thr Ser Gln Asp Gln Gly Phe Cys
385 390 395
act tct ggt ttc caa tct gaa aat cac tct caa aag tgg att ttg gga 1248
Thr Ser Gly Phe Gln Ser Glu Asn His Ser Gln Lys Trp Ile Leu Gly
400 405 410 415
gat gtt ttt att aga gag tac tac tct gtt ttc gat aga gct aac aat 1296
Asp Val Phe Ile Arg Glu Tyr Tyr Ser Val Phe Asp Arg Ala Asn Asn
420 425 430
ttg gtt ggt ttg gct aaa gct att taagtcgaca ac 1332
Leu Val Gly Leu Ala Lys Ala Ile
435
<210> 16
<211> 439
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 16
Met Arg Phe Pro Ser Ile Phe Thr Ala Val Leu Phe Ala Ala Ser Ser
1 5 10 15
Ala Leu Ala Ala Pro Val Asn Thr Thr Thr Glu Asp Glu Thr Ala Gln
20 25 30
Ile Pro Ala Glu Ala Val Ile Gly Tyr Ser Asp Leu Glu Gly Asp Phe
35 40 45
Asp Val Ala Val Leu Pro Phe Ser Ala Ser Ile Ala Ala Lys Glu Glu
50 55 60
Gly Val Ser Leu Asp Lys Arg Glu Ala Glu Ala Glu Ile Thr Arg Ile
65 70 75 80
Pro Leu Tyr Lys Gly Lys Ser Leu Arg Lys Ala Leu Lys Glu His Gly
85 90 95
Leu Leu Glu Asp Phe Leu Gln Lys Gln Gln Tyr Gly Ile Ser Ser Lys
100 105 110
Tyr Ser Gly Phe Gly Glu Val Ala Ser Val Pro Leu Thr Asn Tyr Leu
115 120 125
Asp Ser Gln Tyr Phe Gly Lys Ile Tyr Leu Gly Thr Pro Pro Gln Glu
130 135 140
Phe Thr Val Leu Phe Asp Thr Gly Ser Ser Asp Phe Trp Val Pro Ser
145 150 155 160
Ile Tyr Cys Lys Ser Asn Ala Cys Lys Asn His Gln Arg Phe Asp Pro
165 170 175
Arg Lys Ser Ser Thr Phe Gln Asn Leu Gly Lys Pro Leu Ser Ile His
180 185 190
Tyr Gly Thr Gly Ser Met Gln Gly Ile Leu Gly Tyr Asp Thr Val Thr
195 200 205
Val Ser Asn Ile Val Asp Ile Gln Gln Thr Val Gly Leu Ser Thr Gln
210 215 220
Glu Pro Gly Asp Val Phe Thr Tyr Ala Glu Phe Asp Gly Ile Leu Gly
225 230 235 240
Met Ala Tyr Pro Ser Leu Ala Ser Glu Tyr Ser Ile Pro Val Phe Asp
245 250 255
Asn Met Met Asn Arg His Leu Val Ala Gln Asp Leu Phe Ser Val Tyr
260 265 270
Met Asp Arg Asn Gly Gln Glu Ser Met Leu Thr Leu Gly Ala Ile Asp
275 280 285
Pro Ser Tyr Tyr Thr Gly Ser Leu His Trp Val Pro Val Thr Val Gln
290 295 300
Gln Tyr Trp Gln Phe Thr Val Asp Ser Val Thr Ile Ser Gly Val Val
305 310 315 320
Val Ala Cys Glu Gly Gly Cys Gln Ala Ile Leu Asp Thr Gly Thr Ser
325 330 335
Lys Leu Val Gly Pro Ser Ser Asp Ile Leu Asn Ile Gln Gln Ala Ile
340 345 350
Gly Ala Thr Gln Asn Gln Tyr Gly Glu Phe Asp Ile Asp Cys Asp Asn
355 360 365
Leu Ser Tyr Met Pro Thr Val Val Phe Glu Ile Asn Gly Lys Met Tyr
370 375 380
Pro Leu Thr Pro Ser Ala Tyr Thr Ser Gln Asp Gln Gly Phe Cys Thr
385 390 395 400
Ser Gly Phe Gln Ser Glu Asn His Ser Gln Lys Trp Ile Leu Gly Asp
405 410 415
Val Phe Ile Arg Glu Tyr Tyr Ser Val Phe Asp Arg Ala Asn Asn Leu
420 425 430
Val Gly Leu Ala Lys Ala Ile
435
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ ДЛЯ ЛЕЧЕНИЯ И/ИЛИ ПРОФИЛАКТИКИ ЗЛОКАЧЕСТВЕННОЙ ОПУХОЛИ | 2019 |
|
RU2781542C2 |
| МАТЕРИАЛЫ И МЕТОДЫ, ИСПОЛЬЗУЕМЫЕ ДЛЯ ЛЕЧЕНИЯ РЕСПИРАТОРНЫХ ЗАБОЛЕВАНИЙ У СОБАК | 2020 |
|
RU2811752C2 |
| ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ ДЛЯ ЛЕЧЕНИЯ И/ИЛИ ПРЕДУПРЕЖДЕНИЯ ЗЛОКАЧЕСТВЕННОЙ ОПУХОЛИ | 2017 |
|
RU2766586C2 |
| ВИРУС ГРИППА, СПОСОБНЫЙ ИНФИЦИРОВАТЬ СОБАЧЬИХ, И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2802222C2 |
| Способ получения секретируемой полностью функциональной фосфолипазы А2 в дрожжах Saccharomyces cerevisiae, белок-предшественник для осуществления этого способа (варианты) | 2019 |
|
RU2728240C1 |
| МУКОАДГЕЗИВНЫЙ МИКРООРГАНИЗМ | 2017 |
|
RU2762940C2 |
| Рекомбинантная плазмидная ДНК, кодирующая химерный интерферон alpha2b, рекомбинантный штамм дрожжей P. pastoris X33 - продуцент химерного интерферона alpha2b и способ получения указанного белка | 2020 |
|
RU2764787C1 |
| ПАРВОВИРУС СВИНЕЙ | 2015 |
|
RU2817872C2 |
| СПОСОБ ПРОДУЦИРОВАНИЯ ПОЧЕЧНЫХ КЛЕТОК-ПРЕДШЕСТВЕННИКОВ | 2016 |
|
RU2730861C2 |
| ПРОДУКТЫ И СПОСОБЫ ДЛЯ УХОДА ЗА ПОЛОСТЬЮ РТА, СОДЕРЖАЩИЕ HLP | 2016 |
|
RU2759134C2 |
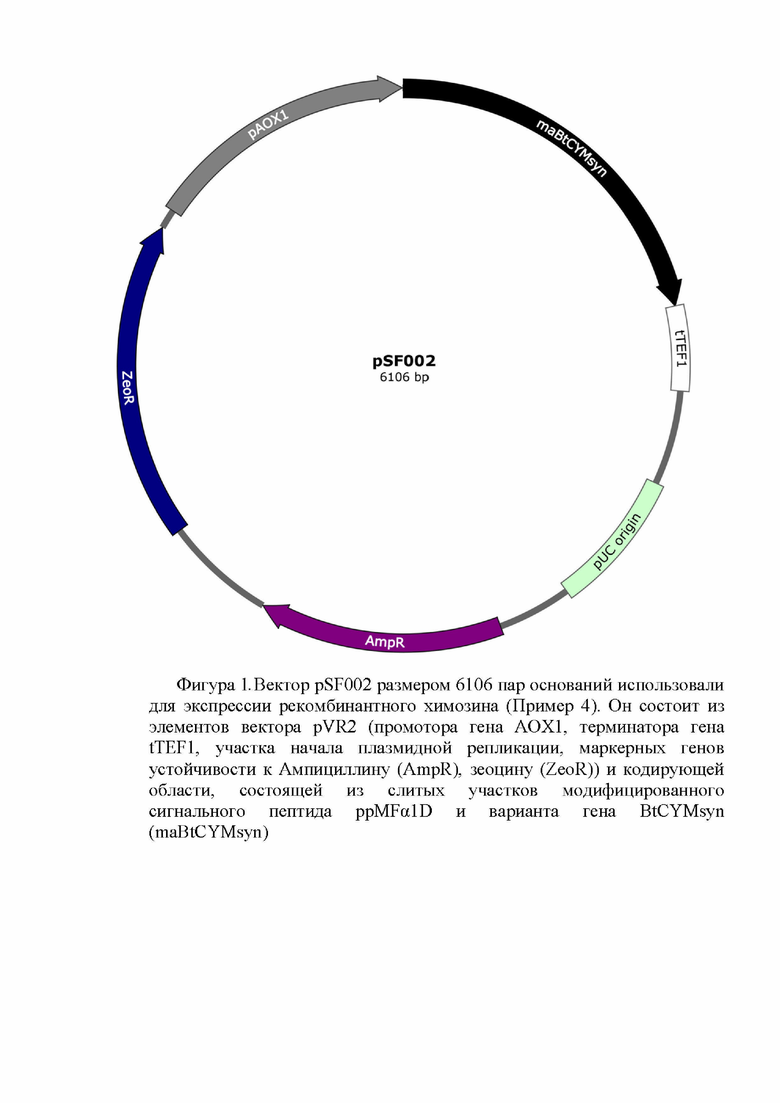






Изобретение относится к биотехнологии, а именно к рекомбинантной плазмиде, обеспечивающей биосинтез и секрецию прохимозина. Предложена рекомбинантная плазмида pSF002, обеспечивающая биосинтез и секрецию прохимозина с SEQ ID NO: 1, имеющая размер 6106 п.о. и состоящая из следующих элементов: а) AOX1 promoter - 5'-концевая область промотора алкоголь оксидазы; б) Alpha factor signal peptide - участок, кодирующий N-концевой сигнальный пептид ppMFα1D с SEQ ID NO: 12; в) последовательность гена прохимозина быка, оптимизированная для экспрессии в Pichia pastoris; г) tTEF1 transcription terminator - терминатор транскрипции; д) pUC origin - бактериальная точка начала репликации pUC; е) Amp(R) - ген Amp, обеспечивающий устойчивость к ампициллину; ж) Zeo(R) - ген BleoR устойчивости к селективному антибиотику зеоцину. Изобретение обеспечивает эффективный синтез целевого белка. 6 ил., 6 пр.
Рекомбинантная плазмида pSF002, обеспечивающая биосинтез и секрецию прохимозина с SEQ ID NO: 1, имеющая размер 6106 п.о. и состоящая из следующих элементов:
а) AOX1 promoter - 5'-концевая область промотора алкоголь оксидазы;
б) Alpha factor signal peptide - участок, кодирующий N-концевой сигнальный пептид ppMFα1D с SEQ ID NO: 12;
в) последовательность гена прохимозина быка, оптимизированная для экспрессии в Pichia pastoris;
г) tTEF1 transcription terminator - терминатор транскрипции;
д) pUC origin - бактериальная точка начала репликации pUC;
е) Amp(R) - ген Amp, обеспечивающий устойчивость к ампициллину;
ж) Zeo(R) - ген BleoR устойчивости к селективному антибиотику зеоцину.
| NOSEDA D.G | |||
| ET AL | |||
| Cloning, expression and optimized production in a bioreactor of bovine chymosin B in Pichia (Komagataella) pastoris under AOX1 promoter | |||
| Protein Expr Purif | |||
| Многоступенчатая активно-реактивная турбина | 1924 |
|
SU2013A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| ESPINOZA-MOLINA J.A | |||
| ET AL | |||
| Codon Optimization of the "Bos Taurus Chymosin" Gene for the Production of Recombinant | |||
Авторы
Даты
2022-09-06—Публикация
2020-10-28—Подача