[001] По настоящей заявке испрашивается приоритет в соответствии с предварительной заявкой на патент США № 62/400372, поданной 27 сентября 2016 г.; № 62/508885, поданной 19 мая 2017 г., и № 62/557566, поданной 12 сентября 2017 г., каждая из которых полностью включена в настоящий документ посредством ссылки.
Уровень техники
1. Область техники
[002] Настоящее изобретение в целом относится к областям микробиологии, иммунологии и медицины. В частности, оно касается применения микробиома для повышения эффективности блокадной терапии иммунных контрольных точек.
2. Описание предшествующего уровня техники
[003] В течение последнего десятилетия были достигнуты значительные успехи в лечении меланомы посредством применения таргетной терапии и иммунотерапии. В частности, применение ингибиторов иммунных контрольных точек показало огромную перспективу, что привело к одобрению FDA нескольких средств, блокирующих иммуномодулирующие молекулы на поверхности Т-лимфоцитов (например, антитело к CTLA-4, ипилимумаб, и антитела к PD-1, ниволумаб, пембролизумаб). Важно отметить, что лечение с помощью блокады иммунных контрольных точек может приводить к длительным долгосрочным полным ответам, хотя общие показатели ответов являются невысокими (т.е. 15% с блокадой CTLA-4 и 30-40% с блокадой PD-1).
[004] Однако ингибиторы иммунных контрольных точек могут быть связаны с существенной токсичностью, и польза может быть только у подгруппы пациентов. Предпринимаются усилия, чтобы лучше понять различия в ответах на блокаду иммунных контрольных точек, однако остается неясным, что способствует этому усиленному ответу у этих пациентов, и существует острая необходимость в определении действенных стратегий для улучшения ответов на терапию у всех пациентов.
[005] Растет понимание роли микробиома хозяина в ответах на лечение рака, и исследования показывают, что бактерии, присутствующие в опухоли и кишечнике, могут влиять на терапевтические ответы. Растет понимание роли микробиома желудочно-кишечного тракта в формировании иммунных реакций в норме и патологии. Однако существует значительный разрыв в переходных знаниях, и существует неудовлетворенная потребность в терапевтических стратегиях для усиления реакции на блокаду иммунных контрольных точек при меланоме и других формах рака.
Сущность изобретения
[006] Согласно одному варианту осуществления настоящее раскрытие относится к композиции, содержащей по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae. Согласно другим вариантам осуществления композиция содержит по меньшей мере две выделенные или очищенные популяции бактерий, принадлежащие к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae. Согласно определенным вариантам осуществления композиция представляет собой живой бактериальный продукт, живой биотерапевтический продукт или пробиотическую композицию. Согласно еще другим вариантам осуществления по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий представлены в виде бактериальных спор. Согласно другому варианту осуществления по меньшей мере одна популяция бактерий принадлежит к семейству Clostridiales XII и/или к семейству Clostridiales XIII. Согласно некоторым аспектам композиция содержит по меньшей мере две выделенные или очищенные популяции бактерий, принадлежащих к семейству Ruminococcaceae и/или к семейству Clostridiaceae. Согласно другим вариантам осуществления композиция содержит по меньшей мере одну популяцию, принадлежащую к семейству Ruminococcaceae, и по меньшей мере одну популяцию, принадлежащую к семейству Clostridiaceae. Согласно некоторым аспектам две популяции бактерий, принадлежащих к семейству Ruminococcaceae, далее определяются как популяции бактерий, принадлежащих к роду Ruminococcus. Согласно определенным аспектам по меньшей мере две выделенные или очищенные популяции бактерий, принадлежащих к семейству Ruminococcaceae, дополнительно определяются как популяции бактерий, принадлежащих к роду Faecalibacterium. Согласно определенным аспектам популяция бактерий, принадлежащих к роду Faecalibacterium, далее определяется как популяция бактерий, принадлежащих к виду Faecalibacterium prausnitzii. Согласно определенным аспектам популяция бактерий, принадлежащих к роду Ruminococcus, далее определяется как популяция бактерий, принадлежащих к виду Ruminococcus bromii. Согласно некоторым аспектам по меньшей мере две выделенные или очищенные популяции бактерий, принадлежащих к семейству Micrococcaceae, дополнительно определяются как популяции бактерий, принадлежащих к роду Rothia. Согласно дополнительным аспектам композиция дополнительно содержит популяцию бактерий, принадлежащих к виду Porphyromonas pasteri, виду Clostridium hungatei, виду Phascolarctobacterium faecium, роду Peptoniphilus и/или классу Mollicutes. Согласно определенным аспектам композиция не содержит популяции бактерий, относящихся к порядку Bacteroidales.
[007] Конкретные варианты осуществления настоящего изобретения относятся к способу предотвращения рака у субъекта, предусматривающему введение субъекту композиции по вариантам осуществления. Например, согласно некоторым аспектам представлен способ предотвращения рака у субъекта с риском развития рака (например, меланомы) или лечения рака у субъекта, характеризующегося наличием опухоли, предусматривающий введение субъекту композиции, содержащей по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из класса Clostridia, класса Mollicutes, порядка Clostridiales, семейства Ruminococcaceae и/или рода Faecalibacterium, причем введение композиции приводит к увеличению CD8+ T-лимфоцитов в опухоли. Согласно конкретным вариантам осуществления Т-лимфоциты представляют собой цитотоксические Т-лимфоциты. Согласно еще другим вариантам осуществления способ представляет собой способ лечения рака у субъекта, предусматривающий введение композиции, содержащей по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из класса Clostridia, класса Mollicutes, порядка Clostridiales, семейства Ruminococcaceae и/или рода Faecalibacterium, причем введение композиции приводит к увеличению эффекторных CD4+, CD8+ Т-лимфоцитов, моноцитов и/или миелоидных дендритных клеток в системном кровообращении или периферической крови субъекта. Согласно некоторым вариантам осуществления этот способ представляет собой способ лечения рака у субъекта, предусматривающий введение композиции, содержащей по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из класса Clostridia, класса Mollicutes, порядка Clostridiales, семейства Ruminococcaceae и/или рода Faecalibacterium и/или Ruminococcus, причем введение композиции приводит к уменьшению количества В-клеток, регуляторных Т-клеток и/или миелоидных клеток-супрессоров в системном кровообращении или периферической крови субъекта. Согласно другим аспектам способ представляет собой способ лечения рака у субъекта, характеризующегося наличием опухоли, предусматривающий введение композиции, содержащей по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из класса Clostridia, класса Mollicutes, порядка Clostridiales, семейства Ruminococcaceae и/или рода Faecalibacterium, причем введение композиции субъекту приводит к увеличению экспрессии CD3, CD8, PD1, FoxP3, гранзима B и/или PD-L1 в опухолевом иммунном инфильтрате. Согласно еще другим аспектам способ представляет собой способ лечения рака у субъекта, характеризующегося наличием опухоли, предусматривающий введение композиции, содержащей по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из класса Clostridia, класса Mollicutes, порядка Clostridiales, семейства Ruminococcaceae и/или рода Faecalibacterium, причем введение композиции субъекту приводит к снижению экспрессии RORγT в опухолевом иммунном инфильтрате. Также описаны способы лечения опухоли у субъекта с диагнозом рак или подозрением на наличие рака, предусматривающие введение композиции, содержащей по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из класса Clostridia, класса Mollicutes, порядка Clostridiales, семейства Ruminococcaceae и/или рода Faecalibacterium, причем введение композиции субъекту приводит к увеличению содержания клеток CD45+, CD3+/CD20+/CD56+, CD68+ и/или HLA-DR+ в опухоли. Согласно некоторым аспектам композицию вариантов осуществления вводят в количестве, достаточном для увеличения содержания врожденных эффекторных клеток у субъекта. Согласно другим аспектам введение композиции субъекту приводит к увеличению содержания врожденных эффекторных клеток у субъекта. Например, введение композиции может увеличить содержание врожденных эффекторных клеток, такие как клетки CD45+CD11b+Ly6G+. Согласно некоторым аспектам композицию вариантов осуществления вводят в количестве, достаточном для снижения содержания супрессорных миелоидных клеток у субъекта. Согласно дополнительным аспектам введение композиции субъекту приводит к снижению содержания супрессорных миелоидных клеток у субъекта. Например, введение композиции может снизить содержание супрессорных миелоидных клеток, таких как клетки CD45+CD11b+CD11c+. Согласно конкретным вариантам осуществления композиция содержит бактерии Faecalibacterium prausnitzii.
[008] Согласно другому варианту осуществления представлен способ лечения рака у субъекта, предусматривающий введение терапевтически эффективного количества ингибитора иммунных контрольных точек указанному субъекту, причем установлено, что субъект характеризуется благоприятным микробиологическим профилем в микробиоме кишечника. Согласно некоторым аспектам благоприятный микробиологический профиль дополнительно определяется как наличие одной или более бактериальных популяций пробиотических или живых бактериальных композиций продуктов по вариантам осуществления. Согласно дополнительному варианту осуществления представлен способ прогнозирования ответа (например, прогнозирования выживаемости) на ингибитор иммунных контрольных точек у пациента, характеризующегося наличием рака, предусматривающий обнаружение микробиологического профиля в образце, полученном от указанного пациента, причем если микробиологический профиль содержит одну или более бактериальных популяциях пробиотических или живых бактериальных композиций продуктов по вариантам осуществления, ответ является благоприятным. Согласно конкретным вариантам осуществления пациенту вводят ингибитор иммунных контрольных точек, если прогнозируют, что у пациента будет благоприятный ответ на ингибитор иммунных контрольных точек. Согласно определенным вариантам осуществления благоприятный микробиологический профиль представляет собой благоприятный микробиологический профиль кишечника.
[009] Согласно некоторым вариантам осуществления по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий принадлежат к одному или более видам, подвидам или бактериальным штаммам, выбранным из группы, состоящей из видов, представленных в таблице 1 с индексом преимущественной представленности (ei) более чем 0,5, 0,6, 0,7, 0,8 или 0,9. Согласно конкретным вариантам осуществления по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий выбирают из группы, состоящей из видов, представленных в таблице 1, с «ei» равным 1.
[0010] Согласно определенным аспектам по меньшей мере одна выделенная или очищенная популяция бактерия или по меньшей мере две выделенные или очищенные популяции бактерий принадлежат к виду, подвиду или бактериальным штаммам, идентифицированным по идентификаторам таксономии NCBI, выбранным из группы, состоящей из следующих идентификаторов таксономии NCBI: 717959, 587, 758823, 649756, 44749, 671218, 1264, 1122135, 853, 484018, 46503, 54565, 290052, 216931, 575978, 433321, 1796646, 213810, 228924, 290054, 1509, 1462919, 29375, 337097, 1298596, 487174, 642492, 1735, 1297424, 742766, 46680, 132925, 411467, 1318465, 1852367, 1841857, 169679, 1175296, 259063, 172901, 39488, 57172, 28118, 166486, 28133, 1529, 694434, 1007096, 84030, 56774, 102148, 626947, 216933, 1348613, 1472417, 100176, 824, 1471761, 1297617, 288966, 1317125, 28197, 358743, 264639, 1265, 1335, 66219, 69473, 115117, 341220, 1732, 873513, 396504, 1796619, 45851, 2741, 105841, 86332, 1349822, 84037, 180311, 54291, 1217282, 762984, 1185412, 154046, 663278, 1543, 398512, 69825, 1841867, 1535, 1510, 84026, 1502, 1619234, 39497, 1544, 29343, 649762, 332095, 536633, 1033731, 574930, 742818, 177412, 1121308, 419208, 1673717, 55779, 28117, 626937, 180332, 1776382, 40519, 34062, 40518, 74426, 1216062, 293826, 850, 645466, 474960, 36835, 115544, 1515, 88431, 216932, 1417852, 39492, 1583, 420247, 118967, 169435, 37658, 138595, 31971, 100886, 1197717, 234908, 537007, 319644, 168384, 915173, 95159, 1816678, 626940, 501571, 1796620, 888727, 1147123, 376806, 1274356, 1267, 39495, 404403, 1348, 253314, 258515, 33033, 1118061, 357276, 214851, 320502, 217731, 246787, 29371, 649764, 901, 29374, 33043, 39778, 682400, 871665, 160404, 745368, 408, 1584, 333367, 47246, 1096246, 53342, 438033, 351091, 1796622, 1776384, 817, 48256, 720554, 500632, 36849, 301302, 879970, 655811, 264463, 1532, 285, 995, 242750, 29539, 1432052, 622312, 1796636, 1337051, 328814, 28446, 1492, 820, 39496, 52786, 1549, 1796618, 582, 46507, 109327, 1531, 1382, 33039, 311460, 230143, 216935, 539, 35519, 1681, 328813, 214853, 89014, 1121115, 1585974, 29466, 1363, 292800, 270498, 214856, 142877, 133926, 209880, 179628, 1121102, 105612, 1796615, 39777, 29353, 1579, 163665, 53443, 261299, 1302, 1150298, 938289, 358742, 471875, 938278, 1796613, 1118057, 1077144, 1737, 218205, 1121298, 684066, 433659, 52699, 204516, 706562, 253257, 328812, 1280, 147802, 58134, 1335613, 891, 585394, 1582, 235931, 308994, 1589, 1682, 1736, 28129, 178001, 551788, 2051, 856, 118562, 101070, 515619, 40215, 187979, 82979, 29363, 1776391, 1285191, 84112, 157688, 38304, 36850, 341694, 287, 75612, 818, 371674, 338188, 88164, 588581, 676965, 546271, 1236512, 178338, 862517, 157687, 158, 51048, 1583331, 529, 888745, 394340, 40545, 855, 553973, 938293, 93063, 708634, 179995, 1351, 476652, 1464038, 555088, 237576, 879566, 1852371, 742727, 1377, 35830, 997353, 218538, 83771, 1605, 28111, 131109, 46609, 690567, 46206, 155615, 51616, 40542, 203, 294, 1034346, 156456, 80866, 554406, 796942, 1002367, 29347, 796944, 61592, 487175, 1050201, 762948, 137732, 1211819, 1019, 272548, 1717, 384636, 216940, 2087, 45634, 466107, 1689, 47678, 575, 979627, 840, 1660, 1236517, 617123, 546, 28135, 82171, 483, 501496, 99656, 1379, 84032, 39483, 1107316, 584, 28124, 1033744, 657309, 536441, 76123, 1118060, 89152, 76122, 303, 1541, 507751, 515620, 38302, 53419, 726, 40324, 1796610, 988946, 1852370, 1017, 1168289, 76936, 94869, 1161098, 215580, 1125779, 327575, 549, 1450648 и 478. Согласно конкретным аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий тесно связаны с видами, подвидами или бактериальными штаммами, идентифицированными с помощью идентификаторов таксономии NCBI, перечисленных выше. Например, согласно некоторым аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий относятся к видам, подвидам или штаммам, содержащим нуклеотидную последовательность 16S рибосомальной РНК (рРНК), которая по меньшей мере на 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична нуклеотидной последовательностью 16S рРНК одной из бактерий, перечисленных выше (т.е. бактерий набора 1 из таблицы 1), или бактерий, перечисленных в таблице 1 и имеющих значение ei более чем 0,5 или равное 1.
[0011] Согласно еще другим аспектам по меньшей мере одна выделенная или очищенная популяция бактерия или по меньшей мере две выделенные или очищенные популяции бактерий принадлежат к виду, подвиду или бактериальным штаммам, выбранным из группы, состоящей из Bacteroides coagulans, Clostridium aldenense, Clostridium aldrichii, Clostridium alkalicellulosi, Clostridium amygdalinum, Clostridium asparagiforme, Clostridium cellulosi, Clostridium citroniae, Clostridium clariflavum DSM 19732, Clostridium clostridioforme, Clostridium colinum, Clostridium fimetarium, Clostridium hiranonis, Clostridium hungatei, Clostridium hylemonae DSM 15053, Clostridium indolis, Clostridium lactatifermentans, Clostridium leptum, Clostridium methylpentosum, Clostridium oroticum, Clostridium papyrosolvens DSM 2782, Clostridium populeti, Clostridium propionicum, Clostridium saccharolyticum, Clostridium scindens, Clostridium sporosphaeroides, Clostridium stercorarium, Clostridium straminisolvens, Clostridium sufflavum, Clostridium termitidis, Clostridium thermosuccinogenes, Clostridium viride, Clostridium xylanolyticum, Desulfotomaculum guttoideum, Eubacterium rectale ATCC 33656, Eubacterium dolichum, Eubacterium eligens ATCC 27750, Eubacterium hallii, Eubacterium infirmum, Eubacterium siraeum, Eubacterium tenue, Ruminococcus torques, Acetanaerobacterium elongatum, Acetatifactor muris, Acetivibrio cellulolyticus, Acetivibrio ethanolgignens, Acholeplasma brassicae 0502, Acholeplasma parvum, Acholeplasma vituli, Acinetobacter junii, Actinobacillus porcinus, Actinomyces bowdenii, Actinomyces dentalis, Actinomyces odontolyticus, Acutalibacter muris, Aerococcus viridans, Aeromicrobium fastidiosum, Alistipes finegoldii, Alistipes obesi, Alistipes onderdonkii, Alistipes putredinis, Alistipes shahii, Alistipes shahii WAL 8301, Alistipes timonensis JC136, Alkalibacter saccharofermentans, Alkaliphilus metalliredigens QYMF, Allisonella histaminiformans, Allobaculum stercoricanis DSM 13633, Alloprevotella rava, Alloprevotella tannerae, Anaerobacterium chartisolvens, Anaerobiospirillum thomasii, Anaerobium acetethylicum, Anaerococcus octavius NCTC 9810, Anaerococcus provenciensis, Anaerococcus vaginalis ATCC 51170, Anaerocolumna jejuensis, Anaerofilum agile, Anaerofustis stercorihominis, Anaeroglobus geminatus, Anaeromassilibacillus senegalensis, Anaeroplasma abactoclasticum, Anaerorhabdus furcosa, Anaerosporobacter mobilis, Anaerostipes butyraticus, Anaerostipes caccae, Anaerostipes hadrus, Anaerotruncus colihominis, Anaerovorax odorimutans, Anoxybacillus rupiensis, Aquabacterium limnoticum, Arcobacter butzleri, Arthrospira platensis, Asaccharobacter celatus, Atopobium parvulum, Bacteroides caccae, Bacteroides caecimuris, Bacteroides cellulosilyticus, Bacteroides clarus YIT 12056, Bacteroides dorei, Bacteroides eggerthii, Bacteroides finegoldii, Bacteroides fragilis, Bacteroides gallinarum, Bacteroides massiliensis, Bacteroides oleiciplenus YIT 12058, Bacteroides plebeius DSM 17135, Bacteroides rodentium JCM 16496, Bacteroides thetaiotaomicron, Bacteroides uniformis, Bacteroides xylanisolvens XB1A, Bacteroides xylanolyticus, Barnesiella intestinihominis, Beduini massiliensis, Bifidobacterium bifidum, Bifidobacterium dentium, Bifidobacterium longum subsp. infantis, Blautia caecimuris, Blautia coccoides, Blautia faecis, Blautia glucerasea, Blautia hansenii DSM 20583, Blautia hydrogenotrophica, Blautia luti, Blautia luti DSM 14534, Blautia wexlerae DSM 19850, Budvicia aquatica, Butyricicoccus pullicaecorum, Butyricimonas paravirosa, Butyrivibrio crossotus, Caldicoprobacter oshimai, Caloramator coolhaasii, Caloramator proteoclasticus, Caloramator quimbayensis, Campylobacter gracilis, Campylobacter rectus, Campylobacter ureolyticus DSM 20703, Capnocytophaga gingivalis, Capnocytophaga leadbetteri, Capnocytophaga sputigena, Casaltella massiliensis, Catabacter hongkongensis, Catenibacterium mitsuokai, Christensenella minuta, Christensenella timonensis, Chryseobacterium taklimakanense, Citrobacter freundii, Cloacibacillus porcorum, Clostridioides difficile ATCC 9689 = DSM 1296, Clostridium amylolyticum, Clostridium bowmanii, Clostridium butyricum, Clostridium cadaveris, Clostridium colicanis, Clostridium gasigenes, Clostridium lentocellum DSM 5427, Clostridium oceanicum, Clostridium oryzae, Clostridium paraputrificum, Clostridium pascui, Clostridium perfringens, Clostridium quinii, Clostridium saccharobutylicum, Clostridium sporogenes, Clostridium ventriculi, Collinsella aerofaciens, Comamonas testosteroni, Coprobacter fastidiosus NSB1, Coprococcus eutactus, Corynebacterium diphtheriae, Corynebacterium durum, Corynebacterium mycetoides, Corynebacterium pyruviciproducens ATCC BAA-1742, Corynebacterium tuberculostearicum, Culturomica massiliensis, Cuneatibacter caecimuris, Defluviitalea saccharophila, Delftia acidovorans, Desulfitobacterium chlororespirans, Desulfitobacterium metallireducens, Desulfosporosinus acididurans, Desulfotomaculum halophilum, Desulfotomaculum intricatum, Desulfotomaculum tongense, Desulfovibrio desulfuricans subsp. desulfuricans, Desulfovibrio idahonensis, Desulfovibrio litoralis, Desulfovibrio piger, Desulfovibrio simplex, Desulfovibrio zosterae, Desulfuromonas acetoxidans, Dethiobacter alkaliphilus AHT 1, Dethiosulfatibacter aminovorans, Dialister invisus, Dialister propionicifaciens, Dielma fastidiosa, Dietzia alimentaria 72, Dorea longicatena, Dysgonomonas gadei ATCC BAA-286, Dysgonomonas mossii, Eggerthella lenta, Eikenella corrodens, Eisenbergiella tayi, Emergencia timonensis, Enorma massiliensis phI, Enterococcus faecalis, Enterorhabdus muris, Ethanoligenens harbinense YUAN-3, Eubacterium coprostanoligenes, Eubacterium limosum, Eubacterium oxidoreducens, Eubacterium sulci ATCC 35585, Eubacterium uniforme, Eubacterium ventriosum, Eubacterium xylanophilum, Extibacter muris, Ezakiella peruensis, Faecalibacterium prausnitzii, Faecalicoccus acidiformans, Faecalitalea cylindroides, Filifactor villosus, Flavonifractor plautii, Flintibacter butyricus, Frisingicoccus caecimuris, Fucophilus fucoidanolyticus, Fusicatenibacter saccharivorans, Fusobacterium mortiferum, Fusobacterium nucleatum subsp. vincentii, Fusobacterium simiae, Fusobacterium varium, Garciella nitratireducens, Gemella haemolysans, Gemmiger formicilis, Gordonibacter urolithinfaciens, Gracilibacter thermotolerans JW/YJL-S1, Granulicatella elegans, Guggenheimella bovis, Haemophilus haemolyticus, Helicobacter typhlonius, Hespellia stercorisuis, Holdemanella biformis, Holdemania massiliensis AP2, Howardella ureilytica, Hungatella effluvii, Hungatella hathewayi, Hydrogenoanaerobacterium saccharovorans, Ihubacter massiliensis, Intestinibacter bartlettii, Intestinimonas butyriciproducens, Irregularibacter muris, Kiloniella laminariae DSM 19542, Kroppenstedtia guangzhouensis, Lachnoanaerobaculum orale, Lachnoanaerobaculum umeaense, Lachnoclostridium phytofermentans, Lactobacillus acidophilus, Lactobacillus algidus, Lactobacillus animalis, Lactobacillus casei, Lactobacillus delbrueckii, Lactobacillus fornicalis, Lactobacillus iners, Lactobacillus pentosus, Lactobacillus rogosae, Lactococcus garvieae, Lactonifactor longoviformis, Leptotrichia buccalis, Leptotrichia hofstadii, Leptotrichia hongkongensis, Leptotrichia wadei, Leuconostoc inhae, Levyella massiliensis, Loriellopsis cavernicola, Lutispora thermophila, Marinilabilia salmonicolor JCM 21150, Marvinbryantia formatexigens, Mesoplasma photuris, Methanobrevibacter smithii ATCC 35061, Methanomassiliicoccus luminyensis B10, Methylobacterium extorquens, Mitsuokella jalaludinii, Mobilitalea sibirica, Mobiluncus curtisii, Mogibacterium pumilum, Mogibacterium timidum, Moorella glycerini, Moorella humiferrea, Moraxella nonliquefaciens, Moraxella osloensis, Morganella morganii, Moryella indoligenes, Muribaculum intestinale, Murimonas intestini, Natranaerovirga pectinivora, Neglecta timonensis, Neisseria cinerea, Neisseria oralis, Nocardioides mesophilus, Novibacillus thermophilus, Ochrobactrum anthropi, Odoribacter splanchnicus, Olsenella profusa, Olsenella uli, Oribacterium asaccharolyticum ACB7, Oribacterium sinus, Oscillibacter ruminantium GH1, Oscillibacter valericigenes, Oxobacter pfennigii, Pantoea agglomerans, Papillibacter cinnamivorans, Parabacteroides faecis, Parabacteroides goldsteinii, Parabacteroides gordonii, Parabacteroides merdae, Parasporobacterium paucivorans, Parasutterella excrementihominis, Parasutterella secunda, Parvimonas micra, Peptococcus niger, Peptoniphilus duerdenii ATCC BAA-1640, Peptoniphilus grossensis ph5, Peptoniphilus koenoeneniae, Peptoniphilus senegalensis JC140, Peptostreptococcus stomatis, Phascolarctobacterium succinatutens, Phocea massiliensis, Pontibacter indicus, Porphyromonas bennonis, Porphyromonas endodontalis, Porphyromonas pasteri, Prevotella bergensis, Prevotella buccae ATCC 33574, Prevotella denticola, Prevotella enoeca, Prevotella fusca JCM 17724, Prevotella loescheii, Prevotella nigrescens, Prevotella oris, Prevotella pallens ATCC 700821, Prevotella stercorea DSM 18206, Prevotellamassilia timonensis, Propionispira arcuata, Proteus mirabilis, Providencia rettgeri, Pseudobacteroides cellulosolvens ATCC 35603 = DSM 2933, Pseudobutyrivibrio ruminis, Pseudoflavonifractor capillosus ATCC 29799, Pseudomonas aeruginosa, Pseudomonas fluorescens, Pseudomonas mandelii, Pseudomonas nitroreducens, Pseudomonas putida, Raoultella ornithinolytica, Raoultella planticola, Raoultibacter massiliensis, Robinsoniella peoriensis, Romboutsia timonensis, Roseburia faecis, Roseburia hominis A2-183, Roseburia intestinalis, Roseburia inulinivorans DSM 16841, Rothia dentocariosa ATCC 17931, Ruminiclostridium thermocellum, Ruminococcus albus, Ruminococcus bromii, Ruminococcus callidus, Ruminococcus champanellensis 18P13 = JCM 17042, Ruminococcus faecis JCM 15917, Ruminococcus flavefaciens, Ruminococcus gauvreauii, Ruminococcus lactaris ATCC 29176, Rummeliibacillus pycnus, Saccharofermentans acetigenes, Scardovia wiggsiae, Schlegelella thermodepolymerans, Sedimentibacter hongkongensis, Selenomonas sputigena ATCC 35185, Slackia exigua ATCC 700122, Slackia piriformis YIT 12062, Solitalea canadensis, Solobacterium moorei, Sphingomonas aquatilis, Spiroplasma alleghenense, Spiroplasma chinense, Spiroplasma chrysopicola, Spiroplasma culicicola, Spiroplasma lampyridicola, Sporobacter termitidis, Staphylococcus aureus, Stenotrophomonas maltophilia, Stomatobaculum longum, Streptococcus agalactiae ATCC 13813, Streptococcus cristatus, Streptococcus equinus, Streptococcus gordonii, Streptococcus lactarius, Streptococcus parauberis, Subdoligranulum variabile, Succinivibrio dextrinosolvens, Sutterella stercoricanis, Sutterella wadsworthensis, Syntrophococcus sucromutans, Syntrophomonas zehnderi OL-4, Terrisporobacter mayombei, Thermoleophilum album, Treponema denticola, Treponema socranskii, Tyzzerella nexilis DSM 1787, Vallitalea guaymasensis, Vallitalea pronyensis, Vampirovibrio chlorellavorus, Veillonella atypica, Veillonella denticariosi, Veillonella dispar, Veillonella parvula, Victivallis vadensis, Vulcanibacillus modesticaldus and Weissella confusa.
[0012] Согласно некоторым аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции относятся к видам бактерий, выбранным из видов в таблице 2, обозначенных статусом ответа отвечающего на лечение (R). Согласно еще дополнительным аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий относятся к видам, подвидам или штаммам, содержащим нуклеотидную последовательность 16S рибосомальной РНК (рРНК), которая по меньшей мере на 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична нуклеотидной последовательности 16S рРНК бактерий, выбранных из видов, представленных в таблице 2, обозначенных статусом ответа отвечающего на лечение (R). Согласно конкретным аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий содержит нуклеотидную последовательность 16S рибосомальной РНК (рРНК), которая по меньшей мере на 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична нуклеотидной последовательности 16S рРНК бактерий, выбранных из группы, состоящей из видов, представленных в таблице 2, которые характеризуются статусом ответа отвечающего на лечение (R) и нескорректированным р-значением менее чем 0,1, 0,09, 0,08, 0,07, 0,06, 0,05, 0,04, 0,03, 0,02 или 0,01.
[0013] Согласно некоторым аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий содержит нуклеотидную последовательность 16S рибосомальной РНК (рРНК), которая по меньшей мере на 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична нуклеотидной последовательности 16S рРНК бактерий, выбранных из группы, состоящей из видов, представленных в таблице 1, которые характеризуются статусом ответа отвечающего на лечение (R) и нескорректированным р-значением менее чем 0,1, 0,09, 0,08, 0,07, 0,06, 0,05, 0,04, 0,03, 0,02 или 0,01. Согласно конкретным вариантам осуществления по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий представляют собой виды, подвиды или бактериальные штаммы, содержащие последовательность гена 16S рРНК, которая по меньшей мере на 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1-876.
[0014] Согласно некоторым аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции относятся к видам бактерий, выбранным из видов в таблице 2, обозначенным статусом ответа отвечающего на лечение (R). Согласно конкретным аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий относятся к видам бактерий, выбранным из группы, состоящей из видов в таблице 2, обозначенных статусом ответа отвечающего на лечение (R) и характеризующихся нескорректированным p-значением менее чем 0,1, 0,09, 0,08, 0,07, 0,06, 0,05, 0,04, 0,03, 0,02 или 0,01. Согласно другим аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий относятся к видам, подвидам или штаммам, содержащим нуклеотидные последовательности, которые по меньшей мере на 60%, 65%, 70%, 75%, 80%, 90%, 95%, 96%, 97%, 98%, 99% или 100% идентичны последовательностям группы совместно представленных генов (CAG) (смотрите таблицу 2А), выбранной из группы, состоящей из SEQ ID NO: 877-926, SEQ ID NO: 927-976, SEQ ID NO: 977-1026, SEQ ID NO: 1027-1076, SEQ ID NO: 1077-1126, SEQ ID NO: 1127-1176, SEQ ID NO: 1177-1226, SEQ ID NO: 1227-1276, SEQ ID NO: 1277-1326, SEQ ID NO: 1327-1376, SEQ ID NO: 1377-1426, SEQ ID NO: 1427-1476, SEQ ID NO: 1477-1526, SEQ ID NO: 1527-1576, SEQ ID NO: 1577-1626, SEQ ID NO: 1627-1676, SEQ ID NO: 1677-1726, SEQ ID NO: 1727-1776, SEQ ID NO: 1777-1826, SEQ ID NO: 1827-1876, SEQ ID NO: 1877-1926, SEQ ID NO: 1927-1976, SEQ ID NO: 1977-2026, SEQ ID NO: 2027-2076, SEQ ID NO: 2077-2126, SEQ ID NO: 2127-2176, SEQ ID NO: 2177-2226, SEQ ID NO: 2227-2276, SEQ ID NO: 2277-2326, SEQ ID NO: 2327-2376, SEQ ID NO: 2377-2426, SEQ ID NO: 2427-2476, SEQ ID NO: 2477-2526, SEQ ID NO: 2527-2576, SEQ ID NO: 2577-2626 and SEQ ID NO: 2627-2676.
[0015] Согласно определенным аспектам по меньшей мере одну выделенную или очищенную популяцию бактерий или две популяции бактерий выбирают из группы, состоящей из видов, подвидов или штаммов, содержащих нуклеотидные последовательности с идентичностью по меньшей мере 29% по отношению к SEQ ID NO: 877-926, идентичностью по меньшей мере 16,5% по отношению к SEQ ID NO: 927-976, идентичностью по меньшей мере 48,5% по отношению к SEQ ID NO: 977-1026, идентичностью по меньшей мере 28% по отношению к SEQ ID NO: 1027-1076, идентичностью по меньшей мере 93,5% по отношению к SEQ ID NO: 1077-1126, идентичностью по меньшей мере 99,5% по отношению к SEQ ID NO: 1127-1176, идентичностью по меньшей мере 99,5% по отношению к SEQ ID NO: 1177-1226, идентичностью по меньшей мере 99% по отношению к SEQ ID NO: 1227-1276, идентичностью 100% по отношению к SEQ ID NO: 1277-1326, идентичностью по меньшей мере 21,5% по отношению к SEQ ID NO: 1327-1376, идентичностью 100% по отношению к SEQ ID NO: 1377-1426, идентичностью по меньшей мере 97% по отношению к SEQ ID NO: 1427-1476, идентичностью по меньшей мере 55,5% по отношению к SEQ ID NO: 1477-1526, идентичностью 100% по отношению к SEQ ID NO: 1527-1576, идентичностью по меньшей мере 34% по отношению к SEQ ID NO: 1577-1626, идентичностью по меньшей мере 14% по отношению к SEQ ID NO: 1627-1676, идентичностью 100% по отношению к SEQ ID NO: 1677-1726, идентичностью по меньшей мере 93% по отношению к SEQ ID NO: 1727-1776, идентичностью 100% по отношению к SEQ ID NO: 1777-1826, идентичностью по меньшей мере 45% по отношению к SEQ ID NO: 1827-1876, идентичностью по меньшей мере 99% по отношению к SEQ ID NO: 1877-1926, идентичностью по меньшей мере 74% по отношению к SEQ ID NO: 1927-1976, идентичностью 100% по отношению к SEQ ID NO: 1977-2026, идентичностью 100% по отношению к SEQ ID NO: 2027-2076, идентичностью по меньшей мере 20% по отношению к SEQ ID NO: 2077-2126, идентичностью по меньшей мере 84% по отношению к SEQ ID NO: 2127-2176, идентичностью по меньшей мере 35,5% по отношению к SEQ ID NO: 2177-2226, идентичностью по меньшей мере 32,5% по отношению к SEQ ID NO: 2227-2276, идентичностью по меньшей мере 70% по отношению к SEQ ID NO: 2277-2326, идентичностью 100% по отношению к SEQ ID NO: 2327-2376, идентичностью по меньшей мере 70,5% по отношению к SEQ ID NO: 2377-2426, идентичностью по меньшей мере 99,5% по отношению к SEQ ID NO: 2427-2476, идентичностью по меньшей мере 68,5% по отношению к SEQ ID NO: 2477-2526, идентичностью 100% по отношению к SEQ ID NO: 2527-2576, идентичностью по меньшей мере 97,5% по отношению к SEQ ID NO: 2577-2626 или идентичностью 100% по отношению к SEQ ID NO: 2627-2676.
[0016] Согласно определенным аспектам по меньшей мере одну выделенную или очищенную популяцию бактерий или две популяции бактерий выбирают из группы, состоящей из видов, подвидов или штаммов, содержащих нуклеотидные последовательности с идентичностью по меньшей мере 29% по отношению к генам CAG:74 Faecalibacterium sp., соответствующим SEQ ID NO: 877-926, идентичностью по меньшей мере 16,5% по отношению к генам NK3B98 Clostridiales bacterium, соответствующим SEQ ID NO: 927-976, идентичностью по меньшей мере 48,5% по отношению к генам 4_3_54A2FAA Subdoligranulum sp., соответствующим SEQ ID NO: 977-1026, идентичностью по меньшей мере 28% по отношению к генам CAG:74 Faecalibacterium sp., соответствующим генам, соответствующим SEQ ID NO: 1027-1076, идентичностью по меньшей мере 93,5% по отношению к генам CAG:155 Oscillibacter sp., соответствующим SEQ ID NO: 1077-1126, идентичностью по меньшей мере 99,5% по отношению к генам CAG:7 Clostridium sp., соответствующим SEQ ID NO: 1127-1176, идентичностью по меньшей мере 99,5% по отношению к генам CAG:86 Eubacterium sp., соответствующим SEQ ID NO: 1177-1226, идентичностью по меньшей мере 99% по отношению к генам CAG:176 Firmicutes bacterium, соответствующим SEQ ID NO: 1227-1276, идентичностью 100% по отношению к генам CAG:344 Akkermansia sp., соответствующим SEQ ID NO: 1277-1326, идентичностью по меньшей мере 21,5% по отношению к генам CAG:74 Faecalibacterium sp., соответствующим SEQ ID NO: 1327-1376, идентичностью 100% по отношению к генам DSM 20438 = JCM 1200 = LMG 10505 Bifidobacterium pseudocatenulatum, соответствующим SEQ ID NO: 1377-1426, идентичностью по меньшей мере 97% по отношению к генам JCC Clostridium sp., соответствующим SEQ ID NO: 1427-1476, идентичностью по меньшей мере 55,5% по отношению к генам SL3/3 Faecalibacterium prausnitzii, соответствующим SEQ ID NO: 1477-1526, идентичностью 100% по отношению к генам CAG:242 Clostridium sp., соответствующим SEQ ID NO: 1527-1576, идентичностью по меньшей мере 34% по отношению к генам CAG:226 Clostridium sp., соответствующим SEQ ID NO: 1577-1626, идентичностью по меньшей мере 14% по отношению к генам CAG:382 Ruminococcus sp., соответствующим SEQ ID NO: 1627-1676, идентичностью 100% по отношению к генам S17 Bifidobacterium bifidum, соответствующим SEQ ID NO: 1677-1726, идентичностью по меньшей мере 93% по отношению к генам CAG:309 Roseburia sp., соответствующим SEQ ID NO: 1727-1776, идентичностью 100% по отношению к генам JC136 Alistipes timonensis, соответствующим SEQ ID NO: 1777-1826, идентичностью по меньшей мере 45% по отношению к генам CAG:103 Firmicutes bacterium, соответствующим SEQ ID NO: 1827-1876, идентичностью по меньшей мере 99% по отношению к генам JC50 Alistipes senegalensis, соответствующим SEQ ID NO: 1877-1926, идентичностью по меньшей мере 74% по отношению к генам CAG:176 Firmicutes bacterium, соответствующим SEQ ID NO: 1927-1976, идентичностью 100% по отношению к генам DSM 3989 Holdemanella biformis, соответствующим SEQ ID NO: 1977-2026, идентичностью 100% по отношению к генам CAG:314 Subdoligranulum sp., соответствующим SEQ ID NO: 2027-2076, идентичностью по меньшей мере 20% по отношению к генам CAG:226 Clostridium sp., соответствующим SEQ ID NO: 2077-2126, идентичностью по меньшей мере 84% по отношению к генам CAG:124 Firmicutes bacterium, соответствующим SEQ ID NO: 2127-2176, идентичностью по меньшей мере 35,5% по отношению к генам Intestinimonas butyriciproducens, соответствующим SEQ ID NO: 2177-2226, идентичностью по меньшей мере 32,5% по отношению к генам CAG:226 Clostridium sp., соответствующим SEQ ID NO: 2227-2276, идентичностью по меньшей мере 70% по отношению к генам CAG:124 Firmicutes bacterium, соответствующим SEQ ID NO: 2277-2326, идентичностью 100% по отношению к генам L2-6 Faecalibacterium prausnitzii, соответствующим SEQ ID NO: 2327-2376, идентичностью по меньшей мере 70,5% по отношению к генам D16 Ruminococcaceae bacterium, соответствующим SEQ ID NO: 2377-2426, идентичностью по меньшей мере 99,5% по отношению к генам DSM 1552 Clostridium spiroforme, соответствующим SEQ ID NO: 2427-2476, идентичностью по меньшей мере 68,5% по отношению к генам Intestinimonas butyriciproducens, соответствующим SEQ ID NO: 2477-2526, идентичностью 100% по отношению к генам CAG:207 Phascolarctobacterium sp., соответствующим SEQ ID NO: 2527-2576, идентичностью по меньшей мере 97,5% по отношению к генам L2-6 Faecalibacterium prausnitzii, соответствующим SEQ ID NO: 2577-2626, или идентичностью 100% по отношению к генам ATCC 15912 Streptococcus parasanguinis, соответствующим SEQ ID NO: 2627-2676.
[0017] Согласно некоторым аспектам бактерии лиофилизируют или сушат посредством замораживания. Согласно конкретным аспектам композицию составляют для пероральной доставки. Например, композиция, составленная для пероральной доставки, представляет собой таблетку или капсулу. Согласно конкретным аспектам таблетка или капсула содержит устойчивое к кислоте энтеросолюбильное покрытие. Согласно определенным аспектам композицию, содержащую по меньшей мере одну выделенную или очищенную популяцию бактерий или по меньшей мере две выделенные или очищенные популяции бактерий, составляют для ректального введения посредством колоноскопии, сигмоидоскопии с помощью назогастральной трубки или клизмы. Согласно некоторым аспектам композицию лиофилизируют или замораживают. Согласно определенным аспектам композицию можно повторно составлять для конечной доставки в виде жидкости, суспензии, геля, гельтаба, полутвердого вещества, таблетки, саше, пастилки, капсулы или в виде энтерального состава. Согласно некоторым аспектам композицию составляют для многократного введения. Согласно некоторым аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий содержат ген устойчивости к антибиотику. Согласно некоторым аспектам по меньшей мере одна выделенная или очищенная популяция бактерий или по меньшей мере две выделенные или очищенные популяции бактерий представляют собой производящую короткоцепочечные жирные кислоты популяцию бактерий. Согласно некоторым аспектам производящая короткоцепочечные жирные кислоты популяция бактерий представляет собой производящую бутират популяцию бактерий. Согласно конкретным аспектам по меньшей мере один ингибитор иммунных контрольных точек вводят внутривенно, а производящую бутират популяцию бактерий вводят перорально.
[0018] Варианты осуществления настоящего изобретения предусматривают способ лечения рака у субъекта, предусматривающий введение терапевтически эффективного количества короткоцепочечной жирной кислоты, такой как бутират, и/или производящей короткоцепочечные жирные кислоты бактериальной популяции, такой как производящая бутират бактериальная популяция, указанному субъекту, причем субъекту вводили ингибитор иммунных контрольных точек. Согласно некоторым аспектам способ дополнительно предусматривает введение по меньшей мере одного ингибитора иммунных контрольных точек. Согласно определенным аспектам вводят более одного ингибитора контрольных точек. Согласно некоторым аспектам способ дополнительно предусматривает введение пребиотика или пробиотика.
[0019] Согласно некоторым аспектам производящая короткоцепочечные жирные кислоты бактериальная популяция содержит бактерии, содержащие ген устойчивости к антибиотикам. Согласно некоторым аспектам производящая бутират бактериальная популяция содержит бактерии, содержащие ген устойчивости к антибиотикам. Согласно некоторым вариантам осуществления способ дополнительно предусматривает стадию введения такой устойчивой к антибиотикам производящей короткоцепочечные жирные кислоты бактериальной популяции, например, такой производящей бутират устойчивой к антибиотикам бактериальной популяции, субъекту, характеризующемуся наличием рака. Согласно некоторым вариантам осуществления способ дополнительно предусматривает введение антибиотика, к которому устойчива производящая короткоцепочечные жирные кислоты устойчивая к антибиотику бактериальная популяция, такая как производящая бутират устойчивая к антибиотику бактериальная популяция, причем ген устойчивости к антибиотику придает устойчивость к антибиотику.
[0020] Согласно некоторым аспектам производящая бутират бактериальная популяция содержит один или более видов бактерий порядка Clostridiales. Согласно определенным аспектам один или более видов бактерий относятся к семейству Ruminococcaceae, Christensenellaceae, Clostridiaceae или Coriobacteriaceae. Согласно конкретным аспектам один или более видов бактерий выбирают из группы, состоящей из Faecalibacterium prausnitzii, Ruminococcus albus, Ruminococcus bromii, Ruminococcus callidus, Ruminococcus flavefaciens, Ruminococcus champanellensis, Ruminococcus faecis, Ruminococcus gauvreauii, Ruminococcus gnavus, Ruminococcus hansenii, Ruminococcus hydrogenotrophicus, Ruminococcus lactaris, Ruminococcus luti, Ruminococcus obeum, Ruminococcus palustris, Ruminococcus pasteurii, Ruminococcus productus, Ruminococcus schinkii, Ruminococcus torques, Subdoligranulum variabile, Butyrivibrio fibrisolvens, Roseburia intestinalis, Anaerostipes caccae, Blautia obeum, Eubacterium nodatum и Eubacterium oxidoreducens. Согласно одному конкретному аспекту один или более видов бактерий представляют собой Faecalibacterium prausnitzii. Согласно конкретным аспектам производящая бутират бактериальная популяция не содержит виды бактерий семейства Prevotellaceae или порядка Bacteriodales.
[0021] Согласно некоторым аспектам введение бутирата предусматривает введение пролекарства или соли бутирата. Согласно конкретным аспектам введение бутирата предусматривает введение бутирата натрия, бутирата аргинина, этилбутириллактата, трибутирина, 4-фенилбутирата, пивалоилоксиметилбутирата (AN-9) или бутилидендибутирата (AN-10).
[0022] Согласно некоторым аспектам бутират или производящую бутират бактериальную популяцию вводят перорально, ректально, с помощью колоноскопии, сигмоидоскопии, клизмы или путем прямой инъекции. Согласно конкретным аспектам по меньшей мере один ингибитор иммунных контрольных точек вводят внутривенно, а бутират и/или производящую бутират бактериальную популяцию вводят перорально.
[0023] Согласно некоторым аспектам по меньшей мере один ингибитор контрольных точек выбирают из ингибитора CTLA-4, PD-1, PD-L1, PD-L2, LAG-3, BTLA, B7H3, B7H4, TIM3, KIR или A2aR. Согласно определенным аспектам по меньшей мере один ингибитор иммунных контрольных точек представляет собой антагониста, связывающегося с осью белка запрограммированной клеточной смерти 1 (PD-1) человека. Согласно некоторым аспектам связывающегося с осью PD-1 антагониста выбирают из группы, состоящей из связывающегося с PD-1 антагониста, связывающегося с PDL1 антагониста и связывающегося с PDL2 антагониста. Согласно определенным аспектам связывающийся с осью PD-1 антагонист представляет собой связывающийся с PD-1 антагонист. Согласно некоторым аспектам связывающийся с PD-1 антагонист ингибирует связывание PD-1 с PDL1 и/или PDL2. Согласно конкретным аспектам связывающийся с PD-1 антагонист представляет собой моноклональное антитело или его антигенсвязывающий фрагмент. Согласно конкретным аспектам связывающийся с PD-1 антагонист представляет собой ниволумаб, пембролизумаб, пидиллизумаб, KEYTRUDA®, AMP-514, REGN2810, CT-011, BMS 936559, MPDL328OA или AMP-224. Согласно некоторым аспектам по меньшей мере один ингибитор иммунных контрольных точек представляет собой антитело к CTLA-4. Согласно конкретным аспектам антитело к CTLA-4 представляет собой тремелимумаб, YERVOY® или ипилимумаб. Согласно определенным аспектам по меньшей мере один ингибитор иммунных контрольных точек представляет собой антитело к иммуноглобулиноподобному рецептору клеток-киллеров (KIR). Согласно некоторым аспектам антитело к KIR представляет собой лирилумаб.
[0024] Согласно определенным аспектам рак представляет собой рак кожи, такой как базально-клеточный рак кожи, плоскоклеточный рак кожи или меланому. Согласно другим аспектам рак кожи представляет собой рак кожи, выбранный из группы, состоящей из возвышающейся дерматофибросаркомы, карциномы из клеток Меркеля, саркомы Капоши, кератоакантомы, опухолей веретенообразных клеток, карцином сальных желез, микрокистозной карциномы придатков, болезни Педжета молочной железы, атипичной фиброзной опухоли, атипичной миомы, фиброзной опухоли и ангиосаркомы. Согласно определенным аспектам меланома представляет собой метастатическую меланому. Согласно другим аспектам меланома представляет собой злокачественное лентиго, меланому типа злокачественного лентиго, поверхностную распространяющуюся меланому, нодулярную меланому, акральную лентигиозную меланому или десмопластическую меланому.
[0025] Согласно некоторым аспектам способ дополнительно предусматривает введение по меньшей мере одного дополнительного противоракового лечения. Согласно некоторым аспектам по меньшей мере одно дополнительное противораковое лечение представляет собой хирургическую терапию, химиотерапию, лучевую терапию, гормональную терапию, иммунотерапию, низкомолекулярную терапию, терапию ингибиторами рецепторной киназы, антиангиогенную терапию, цитокиновую терапию, криотерапию или биологическую терапию. Согласно некоторым аспектам биологическая терапия представляет собой моноклональное антитело, siRNA, miRNA, антисмысловой олигонуклеотид, рибозим или генную терапию.
[0026] Согласно некоторым аспектам по меньшей мере один ингибитор иммунных контрольных точек и/или по меньшей мере одно дополнительное противоопухолевое лечение вводят внутриопухолево, внутриартериально, внутривенно, внутрисосудисто, внутриплеврально, внутрибрюшинно, интратрахеально, интратекально, внутримышечно, эндоскопически, внутрилегочно, чрескожно, подкожно, регионарно, стереотаксически, орально или путем прямой инъекции или перфузии. Согласно конкретным аспектам по меньшей мере один ингибитор иммунных контрольных точек вводят внутривенно, а бутират и/или производящую бутират бактериальную популяцию вводят перорально.
[0027] Согласно другому варианту осуществления представлен способ лечения рака у субъекта, предусматривающий введение терапевтически эффективного количества ингибитора иммунных контрольных точек указанному субъекту, причем установлено, что субъект характеризуется благоприятным микробиологическим профилем в микробиоме кишечника. Согласно некоторым аспектам благоприятный микробиологический профиль дополнительно определяется как характеризующийся: (а) высоким альфа-разнообразием микробиома кишечника; (b) большой численностью производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, в микробиоме кишечника; (c) одной или более (например, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более) бактериями, выбранными из группы, состоящей из видов, представленных в таблице 1, с индексом преимущественной представленности (ei), превышающим 0,5, 0,6, 0,7, 0,8 или 0,9 или равным 1 в микробиоме кишечника; (d) одним или более (например, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более) видами бактерий в таблице 2, обозначенных статусом ответа отвечающего на лечение (R) в микробиоме кишечника; и/или (e) кластерами, центрированными вокруг R-центроида с помощью бета-разнообразия (например, взвешенные расстояния unifrac).
[0028] Согласно некоторым аспектам благоприятный микробиологический профиль дополнительно определяется как наличие или высокая численность бактерий типа Firmicutes, класса Clostridia, порядка Clostridiales, семейства Ruminococcaceae, рода Ruminococcus, рода Faecalibacterium, рода Hydrogenoanaerobacterium, типа Actinobacteria, класса Coriobacteriia, порядка Coriobacteriales, семейства Coriobacteriaceae, домена Archaea, типа Cyanobacteria, типа Euryarchaeota или семейства Christensenellaceae. Согласно определенным аспектам благоприятный микробиологический профиль дополнительно определяется как отсутствие или низкая численность бактерий вида Escherichia coli, вида Anerotruncus colihominis, рода Dialister, семейства Veillonellaceae, типа Bacteroidetes, класса Bacteroidia, порядка Bacteroidales или семейства Prevotellaceae. Согласно конкретным аспектам благоприятные микробиологические профили определяются как наличие или высокая численность бактерий класса Clostridiales и отсутствие или низкая численность бактерий порядка Bacteroidales. Согласно некоторым аспектам благоприятный микробиологический профиль дополнительно определяется как высокая численность производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии. Согласно определенным аспектам производящие бутират бактерии содержат один или более видов из рода Ruminococcus или Faecalibacterium.
[0029] Согласно некоторым аспектам субъекта определяют как характеризующегося благоприятным микробиологическим профилем или благоприятным микробиомом кишечника путем анализа микробиома в образце пациента. Согласно определенным аспектам образец пациента представляет собой фекальный или буккальный образец. Согласно некоторым аспектам анализ предусматривает выполнение 16S рибосомного секвенирования и/или метагеномики секвенирования всего генома.
[0030] Согласно дополнительному варианту осуществления представлен способ прогнозирования ответа (например, выживаемости пациента) на ингибитор иммунных контрольных точек у пациента, характеризующегося наличием рака, предусматривающий обнаружение микробиологического профиля в образце, полученном от указанного пациента, причем если микробиологический профиль содержит: (а) высокое альфа-разнообразие; (b) большую численность производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии; (c) одну или более (например, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более) бактерий, выбранных из группы, состоящей из видов, представленных в таблице 1, с индексом преимущественной представленности (ei), превышающим 0,5, 0,6, 0,7, 0,8 или 0,9 или равным 1; (d) один или более (например, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более) видов бактерий в таблице 2, обозначенных статусом ответа отвечающего на лечение (R); (e) низкую численность Bacteriodales и/или (f) отдельные кластеры с помощью взвешенных расстояний unifrac бета-разнообразия, то у пациента прогнозируется благоприятный ответ на ингибитор иммунных контрольных точек. Согласно конкретным вариантам осуществления пациенту вводят ингибитор иммунных контрольных точек, если прогнозируют, что пациент будет характеризоваться благоприятным ответом на ингибитор иммунных контрольных точек. Согласно определенным вариантам осуществления пациенту вводят второй ингибитор иммунных контрольных точек. Согласно определенным вариантам осуществления благоприятный микробиологический профиль представляет собой благоприятный микробиологический профиль кишечника.
[0031] Согласно определенным аспектам рак представляет собой рак кожи, такой как базально-клеточный рак кожи, плоскоклеточный рак кожи или меланому. Согласно другим аспектам рак кожи представляет собой рак кожи, выбранный из группы, состоящей из возвышающейся дерматофибросаркомы, карциномы из клеток Меркеля, саркомы Капоши, кератоакантомы, опухолей веретенообразных клеток, карцином сальных желез, микрокистозной карциномы придатков, болезни Педжета молочной железы, атипичной фиброзной опухоли, атипичной миомы, фиброзной опухоли и ангиосаркомы. Согласно другим аспектам меланома представляет собой злокачественное лентиго, меланому типа злокачественного лентиго, поверхностную распространяющуюся меланому, нодулярную меланому, акральную лентигиозную меланому или десмопластическую меланому. Согласно конкретным аспектам ингибитор иммунных контрольных точек представляет собой моноклональное антитело к PD1 или моноклональное антитело к CTLA4.
[0032] Согласно некоторым аспектам производящие короткоцепочечные жирные кислоты бактерии, такие как производящая бутират бактериальная популяция, содержат один или более видов бактерий порядка Clostridiales. Согласно определенным аспектам один или более видов принадлежат к семейству Ruminococcaceae, Christensenellaceae, Clostridiaceae или Coriobacteriacease. Согласно конкретным аспектам один или более видов выбраны из группы, состоящей из Faecalibacterium prausnitzii, Ruminococcus albus, Ruminococcus bromii, Ruminococcus callidus, Ruminococcus flavefaciens, Ruminococcus champanellensis, Ruminococcus faecis, Ruminococcus gauvreauii, Ruminococcus gnavus, Ruminococcus hansenii, Ruminococcus hydrogenotrophicus, Ruminococcus lactaris, Ruminococcus luti, Ruminococcus obeum, Ruminococcus palustris , Ruminococcus pasteurii, Ruminococcus productus, Ruminococcus schinkii, Ruminococcus torques, Subdoligranulum variabile, Butyrivibrio fibrisolvens, Roseburia intestinalis, Anaerostipes caccae, Blautia obeum, Eubacterium nodatum и Eubacterium oxidoreducens. Согласно определенным аспектам один или более видов представляют собой Faecalibacterium prausnitzii.
[00303] Согласно дополнительным аспектам способ дополнительно предусматривает введение ингибитора иммунных контрольных точек субъекту, у которого прогнозируется благоприятный ответ на ингибитор иммунных контрольных точек. Согласно некоторым аспектам ингибитор иммунных контрольных точек представляет собой моноклональное антитело к PD1 или моноклональное антитело к CTLA4.
[0034] Согласно некоторым аспектам способ дополнительно предусматривает введение по меньшей мере одного дополнительного противоракового лечения. Согласно определенным аспектам по меньшей мере одно дополнительное противораковое лечение представляет собой хирургическую терапию, химиотерапию, лучевую терапию, гормональную терапию, иммунотерапию, низкомолекулярную терапию, терапию ингибиторами рецепторной киназы, антиангиогенную терапию, цитокиновую терапию, криотерапию или биологическую терапию. Согласно конкретным аспектам по меньшей мере одно дополнительное противораковое лечение представляет собой короткоцепочечную жирную кислоту, такую как бутират, и/или производящую короткоцепочечную жирную кислоту бактериальную популяцию, такую как производящая бутират бактериальная популяция. Согласно конкретным аспектам по меньшей мере одно противораковое лечение представляет собой композицию по вариантам осуществления. Согласно некоторым аспектам способ дополнительно предусматривает введение пребиотика или пробиотика.
[0035] Согласно другому варианту осуществления представлен способ прогнозирования ответа на ингибитор иммунных контрольных точек у пациента, характеризующегося наличием рака, предусматривающий обнаружение микробиологического профиля в образце, полученном от указанного пациента, причем, если микробиологический профиль характеризуется: (a) низкой численностью производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии; (b) одним или более видами бактерий в таблице 2, обозначенными статусом ответа не отвечающего на лечение (NR); (с) низким альфа-разнообразием и/или (d) большим количеством порядка Bacteriodales, тогда прогнозируется у пациента отсутствие благоприятного ответа на ингибитор иммунных контрольных точек. Согласно дополнительным аспектам способ дополнительно предусматривает введение пациенту композиции пробиотического или живого бактериального продукта согласно вариантам осуществления, если прогнозируется, что пациент не будет характеризоваться благоприятным ответом на ингибитор иммунных контрольных точек. Согласно еще одним аспектам пациенту, у которого прогнозируется отсутствие положительного ответа на ингибитор иммунных контрольных точек, вводят ингибитор иммунных контрольных точек после введения композиции пребиотика или живого бактериального продукта по вариантам осуществления.
[0036] Согласно дополнительным аспектам способ дополнительно предусматривает введение по меньшей мере одного дополнительного неиммунного противоракового лечения ингибитором контрольных точек субъекту, у которого прогнозируется отсутствие благоприятного ответа на ингибитор иммунных контрольных точек.
[0037] Согласно дополнительным аспектам способ предусматривает введение субъекту по меньшей мере одного противоракового лечения. Согласно некоторым аспектам по меньшей мере одно противораковое лечение представляет собой хирургическую терапию, химиотерапию, лучевую терапию, гормональную терапию, иммунотерапию, низкомолекулярную терапию, терапию ингибиторами рецепторной киназы, антиангиогенную терапию, цитокиновую терапию, криотерапию, ингибитор иммунных контрольных точек, второй ингибитор иммунных контрольных точек или биологическую терапию. Согласно конкретным аспектам по меньшей мере одно дополнительное противораковое лечение представляет собой короткоцепочечную жирную кислоту, такую как бутират, и/или производящую короткоцепочечную жирную кислоту бактериальную популяцию, такую как производящую бутират бактериальную популяцию. Согласно некоторым аспектам противораковая терапия представляет собой пребиотик или пробиотик. Согласно конкретным аспектам пробиотик представляет собой пробиотическую композицию по вариантам осуществления.
[0038] Согласно определенным аспектам рак представляет собой рак кожи, такой как базально-клеточный рак кожи, плоскоклеточный рак кожи или меланому. Согласно другим аспектам рак кожи представляет собой рак кожи, выбранный из группы, состоящей из возвышающейся дерматофибросаркомы, карциномы из клеток Меркеля, саркомы Капоши, кератоакантомы, опухолей веретенообразных клеток, карцином сальных желез, микрокистозной карциномы придатков, болезни Педжета молочной железы, атипичной фиброксантомы, лейомиосаркомы и ангиосаркомы. Согласно другим аспектам меланома представляет собой злокачественное лентиго, меланому типа злокачественного лентиго, поверхностную распространяющуюся меланому, нодулярную меланому, акральную лентигиозную меланому или десмопластическую меланому.
[0039] Согласно некоторым аспектам, если микробиологический профиль содержит один или более видов бактерий в таблице 2, обозначенных статусом ответа не отвечающего на лечение (NR) или большим количеством порядка Bacteriodales, то прогнозируется, что пациент не будет характеризоваться благоприятным ответом на ингибитор иммунных контрольных точек. Согласно некоторым аспектам, если микробиологический профиль содержит 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более видов бактерий в таблице 2, обозначенных статусом ответа не отвечающего на лечение (NR), то прогнозируется, что пациент не будет характеризоваться благоприятным ответом на ингибитор иммунных контрольных точек. Согласно дополнительным аспектам способ предусматривает введение пациенту композиции пробиотического или живого бактериального продукта по вариантам осуществления.
[0040] Другие цели, особенности и преимущества настоящего изобретения станут очевидными из следующего подробного описания. Однако следует понимать, что подробное описание и конкретные примеры, хотя и указывают предпочтительные варианты осуществления настоящего изобретения, даны только в качестве иллюстрации, поскольку различные изменения и модификации в пределах сущности и объема настоящего изобретения станут очевидными для специалистов в настоящей области техники из этого подробного описания.
Краткое описание чертежей
[0041] Следующие графические материалы составляют часть настоящего описания и включены для дополнительной демонстрации определенных аспектов настоящего изобретения. Настоящее изобретение может быть лучше понято при обращении к одному или более из этих графических материалов в сочетании с подробным описанием конкретных вариантов осуществления, представленных в настоящем документе.
[0042] Фиг. 1A-G: Увеличение разнообразия микробиома кишечника связано с усилением ответов на блокаду PD-1 у пациентов с метастатической меланомой. (А) Схема сбора и анализа образцов. (B) График в виде составных прямоугольников филогенетической композиции общих бактериальных таксонов (численность >0,1%) на уровне порядка в оральных (n=109, вверху) и фекальных (n=53, внизу) образцах с помощью секвенирования 16S рРНК. (C) Двудольная сетевая диаграмма соответствующих оральных и фекальных образцов от 48 пациентов, получавших анти-PD-1. Края соединяют OTU уровня вида с узлами образца, в которых они обнаружены. (D) Обратные формы индекса разнообразия Симпсона для микробиома кишечника у R (n=30) и NR (n=13) по сравнению со способом лечения анти-PD-1 по критерию Манна-Уитни (MW). (E) Филогенетическая композиции 39 фекальных образцов на уровне семейства (численность >0,1%) на исходном уровне. Группы с высоким (>11,63, n=13), промежуточным (7,46-11,63, n=13) и низким (<7,46, n=13) разнообразием определяли с использованием обратной формы индекса Симпсона. (F) График Каплана-Мейера (КМ) выживания без прогрессирования (PFS) по фекальному разнообразию; высокий (средний PFS не определен), промежуточный (средний PFS=232 дня) и низкий (средний PFS=188 дней). Высокое по сравнению с промежуточным разнообразие (HR=3,60, 95% C.I. = 1,02-12,74) и высокое по сравнению с низким (низкое HR=3,57, 95% C.I. = 1,02-12,52) по одномерной модели Кокса. *р<0,05, **р<0,01. (G) Анализ главных координат фекальных образцов (n=43) по ответу с использованием взвешенных расстояний UniFrac.
[0043] Фиг. 2A-F. Различия в композиции микробиома кишечника связаны с ответами на блокаду PD-1. (A) Карта интенсивности для численности OTU в R (n=30) и NR (n=13). Столбцы обозначают пациентов, а ряды обозначают виды бактерий, сгруппированные по их преимущественной представленности у R по сравнению с NR в 3 набора. (B) Филогенетическая композиция OTU в каждом наборе на уровне порядка. (C) Таксономическая кладограмма от LEfSe, показывающая различия в фекальных таксонах. Размер точки пропорционален распространенности таксона. (D) Показатели LDA, рассчитанные для избирательно многочисленных таксонов в фекальных микробиомах R и NR, как указано. Длина указывает на величину эффекта, связанную с таксоном. р=0,05 для критерия Крускала-Уоллиса; оценка LDA >3. (E) Избирательно многочисленные бактерии кишечника в R против NR по тесту MW (с поправкой на FDR) во всех таксономических уровнях. (F) Парные сравнения численности видов бактерий, идентифицированных посредством метагеномного WGS в 25 фекальных образцах: R (n=14), NR (n=11). *р<0,05, **р<0,01.
[0044] Фиг. 3A-F: Численность crOTU в микробиоме кишечника представляет собой прогностический фактор ответа на блокаду PD-1. (A) Иерархическая кластеризация без обучения путем полной связи численности crOTU в 43 фекальных образцах. (B) Ассоциация кластеров crOTU с ответом на анти-PD-1 с помощью точного теста Фишера. Кластер 1 crOTU (n=14: R=14, NR=0); кластер 2 crOTU (n=29: R=16, NR=13). (C) График Каплана-Мейера PFS по кластеру crOTU. Кластер 1 crOTU (медиана PFS не определена), кластер 2 crOTU (медиана PFS=242 дня). (D) Избирательно многочисленные фекальные таксоны в кластере 1 crOTU по сравнению с кластером 2 crOTU, с помощью теста MW (с поправкой на FDR) на всех таксономических уровнях. (E) PFS у пациентов с высокой (n=19, медиана PFS не определена) или низкой (n=20, медиана PFS=242 дня) численностью F. prausnitzii (вверху) или высокой (n=20, медиана PFS=188 дней ) или низкой (n=19, медиана PFS=393 дня) численностью Bacteroidales (внизу). (F) Неконтролируемая иерархическая кластеризация численности классов путей, выведенная из путей MetaCyc, предсказанных в 28 фекальных образцах от 25 пациентов (R=14, NR=11). Обычный шрифт: пути биосинтеза, жирный шрифт: пути разрушения. *р <0,05.
[0045] Фиг. 4A-G: Благоприятный микробиом кишечника связан с системным противоопухолевым иммунитетом. (A) Количественная оценка посредством IHC CD8+ инфильтрата при предварительном лечении в количествах/мм2 у R (n=15) и NR (n=6) с помощью одностороннего теста MW. *р=0,04. (B) Карта интенсивностей попарной корреляции рангов Спирмена для существенно различных таксонов в фекальных образцах (n=15) на исходном уровне и плотности CD3, CD8, PD-1, FoxP3, GzmB и RORγT в единицах/мм2 и PD-L1 по H-критерию в соответствующих опухолях. (C) Одномерная линейная регрессия между количеством CD8+/мм2 в опухоли по сравнению с численностью Faecalibacterium (незакрашенные кружки и пунктирная линия; r2=0,42, p=0,0067) и Bacteroidales (закрашенные кружки и сплошная линия; r2=0,056, p=0,38) в кишечнике (D) Карта интенсивностей попарной корреляции рангов Спирмена между существенно различными фекальными таксонами и частотой CD4+ эффекторных Т-клеток, CD8+ Т-клеток, миелоидных дендритных клеток, моноцитов, В-клеток, Treg и MDSC с помощью проточной цитометрии в периферической крови на исходном уровне. (E) Мультиплексная IHC, показывающая репрезентативные изображения, и (F) частота иммунных клеток, лимфоидных клеток, миелоидных клеток и MHC II у пациентов с высокой численностью Faecalibacterium или высокой численностью Bacteroidales в кишечнике. (G) Предлагаемый механизм действия микробиома кишечника на опухолевый иммунитет в благоприятных и неблагоприятных условиях.
[0046] Фиг. 5A-B. Не наблюдается различий в мутационном ландшафте R и NR при блокаде PD-1. (A) Количество мутаций на мегабазу и ландшафт драйверных мутаций в опухолях пациентов с соответствующими образцами фекального микробиома (n=7R против 3NR). (B) Общая нагрузка несмысловыми мутациями в доступных опухолях (n=8R против 4NR, p=0,683), согласно двустороннему критерию Манна-Уитни (MW).
[0047] Фиг. 6: Различия в структуре сообществ между оральным и фекальным микробиомами. Двусторонняя сетевая диаграмма операционных таксономических единиц (OTU) полученных из бактериальных 16S рРНК, из 109 буккальных и 53 фекальных образцов. Края соединяют OTU на уровне видов (ромбы) с узлами оральных образцов (незакрашенные кружки) и фекальных образцов (закрашенные кружки), в которых они обнаружены.
[0048] Фиг. 7A-C: Разнообразие фекального микробиома увеличено у R при анти-PD-1 терапии. Сравнение показателей альфа-разнообразия у R (n=30, незакрашенные кружки) и NR (n=13, закрашенные кружки) с использованием индексов (A) Шеннона, (B) Симпсона и (C) Chao1 с помощью двустороннего теста MW. *р<0,05, **р<0,01.
[0049] Фиг. 8A-D: Не наблюдается различий в разнообразии орального микробиома между R и NR при анти-PD-1 терапии. Сравнение показателей альфа-разнообразия у R (n=54, незакрашенные кружки) и NR (n=32, закрашенные кружки) с использованием (A) обратной формы индекса Симпсона (p=0,107), (B) индекса Шеннона (p=0,139), (C) индекса Симпсона (p=0,136) и (D) индекса Chao1 (p=0,826) по двустороннему тесту MW.
[0050] Фиг. 9A-B: Высокое разнообразие фекального микробиома связано с более длительной PFS. Микробиота кишечника на исходном уровне и при последующем курсе лечения по субъектам (n=39). (A) Горизонтальные столбцы представляют оценки альфа-разнообразия, измеренные по обратной форме индекса Симпсона у каждого пациента. (B) Графики временной шкалы, показывающие прошедшие дни терапии. х = прогрессировал, о = не прогрессировал при последующем наблюдении.
[0051] Фиг. 10A-D: Разнообразие орального микробиома не связано с PFS. Оральная микробиота и последующее лечение пациента (n=86). (A) Блочные столбцы представляют филогенетическую композицию каждого образца на уровне семейства на исходном уровне. Все пациенты были классифицированы на группы с высоким (>6,17), промежуточным (3,26-6,17) и низким (<3,26) разнообразием, как указано, на основе обратной формы индекса Симпсона. (B) График Каплана-Мейера выживаемости без прогрессирования посредством тертилей орального разнообразия: высокое (n=29, медиана PFS=279 дней), промежуточное (n=28, медиана PFS не определена), низкое (n=29, медиана PFS=348 дней), как указано. Высокое по сравнению с промежуточным, р=0,34; высокое против низкого, р=0,54 по логарифмическому критерию. (C) Горизонтальные столбцы представляют собой оценки альфа-разнообразия, измеренные по обратной форме индекса Симпсона. (D) Графики временной шкалы, показывающие прошедшие дни терапии. х = прогрессировал, о = не прогрессировал при последующем наблюдении.
[0052] Фиг. 11A-D: Пороговые значения для показателей индекса преимущественной представленности (ei) и относительной численности для OTU в 86 образцах орального микробиома и 43 образцах фекального микробиома. Распределение показателей преимущественной представленности для бактериальных OTU на уровне видов в (A) образцах фекального микробиома и (B) образцах орального микробиома по наборам. Границы для каждого набора указаны. Распределение log10 относительной численности видов в (C) образцах фекального микробиома и (D) образцах орального микробиома. Диапазон для каждой категории численности указан.
[0053] Фиг. 12A-B: Нет существенных различий в OTU орального микробиома между R и NR при анти-PD-1 терапии по показателю индекса преимущественной представленности (ei). (A) Карта интенсивности численности видов у R (n=52) и NR (n=34), как указано, с помощью набора бактериальных OTU, основанных на показателях ei. Каждый столбец обозначает пациента, а каждый ряд обозначает бактериальную OTU. Указаны высокая, средняя и низкая. (B) Филогенетическая композиция бактериальных OTU в каждом наборе на уровне порядка.
[0054] Фиг. 13A-B: Сравнение высокоразмерных классов с использованием LEfSe выявляет увеличенную численность Bacteroidales в оральном микробиоме NR при анти-PD-1 терапии. (A) Таксономическая кладограмма от LEfSe, показывающая различия в оральных таксонах. Таксоны, характеризующиеся преимущественной представленностью у R и NR, соотносятся с размером точки, пропорциональной численности таксона. (B) Гистограмма показателей LDA, рассчитанная для избирательно многочисленных таксонов между оральными микробиомами R и NR, где длина столбца указывает на величину эффекта, связанную с таксоном. р=0,05 для критерия Крускала-Уоллиса; показатель LDA >3.
[0055] Фиг. 14А-С: Разнообразие и композиция микробиома кишечника стабильны во времени. (A) Альфа-разнообразие микробиома кишечника по обратной форме индекса Симпсона с течением времени у 3 пациентов (R) со сборами за различные периоды. (B) Анализ основных компонентов с использованием невзвешенных расстояний UniFrac. (C) Блочные столбцы, показывающие композицию микробиома кишечника у пациентов с течением времени на уровне порядка.
[0056] Фиг. 15A-B: Кластеризация по относительной численности OTU не показывает никакой связи с ответом на анти-PD-1 терапию. Иерархическая кластеризация без обучения посредством полной связи с использованием евклидовых расстояний, основанных на численности OTU в (A) 43 образцах фекального микробиома и (B) 86 образцах орального микробиома. Каждый столбец представляет собой уникальный образец микробиома, тогда как каждый ряд представляет собой уникальную OTU.
[0057] Фиг. 16A-B: Кластеры, основанные на численностях crOTU орального микробиома, не связаны с ответом на блокаду PD-1. (A) Иерархическая кластеризация без обучения посредством полной связи 86 образцов орального микробиома на основе численности crOTU. (B) Сравнение кластеров по ответу, показывающее кластер 1 crOTU (n=11, R=9 и NR=2) и кластер 2 (n=75, R=45 и NR=30). р=0,20 по двустороннему точному критерию Фишера.
[0058] Фиг. 17: Метаболические профили, основанные на KEGG-ортологах, различаются в микробиоме кишечника R против NR при PD-1 блокаде. Иерархическая кластеризация без обучения общих функциональных путей (обнаружена по меньшей мере в 20 образцах) в 28 фекальных образцах, полученных от 25 пациентов (n=14R и 11NR) в соответствии с относительными численностями KEGG-ортолога.
[0059] Фиг. 18A-F: Отвечающие на блокаду PD-1 пациенты представляют обогащенный опухолевый иммунный инфильтрат на исходном уровне. Иммуногистохимическое количественное определение и репрезентативные изображения при 40-кратном увеличении (A) CD3, (B) PD-1, (C) FoxP3, (D) GzmB, (E) PD-L1 и (F) RORγT в виде количества/мм2 или H-показателя у отвечающих (R) и не отвечающих (NR) на анти-PD-1 пациентов.
[0060] Фиг. 19: Пациенты с высокой численностью Faecalibacterium представляют благоприятный противоопухолевый иммунный инфильтрат перед анти-PD-1 терапией. Карта интенсивности корреляции рангов Спирмена для GzmB, CD3, CD8, PD-1, FoxP3, RORγT по количеству/мм2, PD-L1 по H-показателю посредством IHC и численности всех родов семейства Ruminococcaceae в фекальном микробиоме (n=15). Указаны положительная корреляция, отрицательная корреляция и отсутствие корреляции.
[0061] Фиг. 20A-F: Численность Faecalibacterium и Bacteroidales в фекальном микробиоме характеризуется четкими ассоциациями с опухолевым иммунным инфильтратом до блокады PD-1. Линейная регрессия между численностью Faecalibacterium, численностью Bacteroidales и плотностью по количеству/мм2 или по H-показателю (A) CD3, (B) GzmB, (C) PD-1, (D) PD-L1, (E) FoxP3 и (F) RORγT по IHC в опухолях пациентов, которых лечили анти-PD-1, на исходном уровне. Линии показывают регрессию для Faecalibacterium (тонкая линия, значения нормальным шрифтом) и Bacteroidales (жирная линия, значения жирным шрифтом) со связанным r2 и p-значениями.
[0062] Фиг. 21: Стратегия гейтирования для анализа посредством проточной цитометрии периферической крови у пациентов, получавших анти-PD-1 терапию. PBMC на исходном уровне у пациентов, получавших анти-PD-1, анализировали путем гейтирования CD19+ B-клеток, CD3+CD8+ T-клеток, CD3+CD4+ T-клеток, (CD3+CD4+FoxP3+ регуляторных и CD3+CD4+FoxP3- эффекторов), моноцитов (на основе CD14/HLA-DR) и MDSC (CD3-CD19-HLADRCD33+CD11b+).
[0063] Фиг. 22A-D: У пациентов с высокой численностью Faecalibacterium наблюдается профиль периферических цитокинов, благоприятный для ответа на блокаду PD-1, на исходном уровне и усиленный ответ цитокинов в течение курса терапии. (A) Карта интенсивности ранговой корреляции Спирмена между численностью Clostridiales, Faecalibacterium, Ruminococcaceae и Bacteroidales и периферической концентрацией цитокинов в пг/мл с помощью мультиплексного анализа с использованием шариков. Указана положительная корреляция, отрицательная корреляция и отсутствие корреляции. Изменение производства цитокинов в сыворотке отвечающих (n=2) и не отвечающих (n=2) на анти-PD-1 терапию пациентов для (B) IP-10 (p=0,042 и p=0,344, соответственно), (C) MIP-1β (p=0,043 и p=0,898, соответственно) и (D) IL-17A (p=0,072 и p=0,862, соответственно) в кратности изменения от исходного уровня с помощью парного t-критерия отношения.
[0064] Фиг. 23: Стратегия гейтирования для миелоидной мультиплексной IHC в опухолях пациентов, подвергаемых лечению блокадой PD-1, на исходном уровне. Стратегия гейтирования миелоидной мультиплексной иммуногистохимии, показывающая иммунные клетки (CD45+), лимфоидные клетки (CD45+CD3+CD20+CD56+), миелоидные клетки (CD45+CD3-CD20-CD56-), тучные клетки (CD45+CD3-CD20-CD56-HLADR-Триптаза+), гранулоциты (CD45+CD3-CD20-CD56-HLADRCD66b+), M1 опухолеассоциированные макрофаги (CD45+CD3-CD20-CD56-HLADR+CSF1R+CD163-), M2 опухолеассоциированные макрофаги (CD45+CD3-CD20-CD56-HLADR+CSF1R+CD163+), зрелые дендритные клетки (CD45+CD3-CD20-CD56-HLADR+CSF1R-DCSIGN-) и незрелые дендритные клетки (CD45+CD3-CD20-CD56-HLADR+CSF1R-DCSIGN+).
[0065] Фиг. 24A-C: Высокая численность Faecalibacterium на исходном уровне связана с повышенным иммунным инфильтратом до блокады PD-1. (A) Мультиплексная иммуногистохимия, показывающая типичное окрашивание миелоидных иммунных клеток при 40-кратном увеличении. (B) Количественная оценка CD45, CD3/CD20/CD56, CD68, CD66b, триптазы, HLA-DR, CD163 и DC-SIGN в виде количества/мм2. (C) Количественное определение миелоидных клеток, лимфоидных клеток, тучных клеток, гранулоцитов, макрофагов, M1 и M2 опухолеассоциированных макрофагов, незрелых дендритных клеток и зрелых дендритных клеток, в процентах от общего количества CD45+ иммунных клеток у пациентов с высокой численностью Faecalibacterium (n=2) или высокой численностью Bacteroidales (n=2).
[0066] Фиг. 25A-B. Трансплантация фекальной микробиоты (FMT) благоприятного микробиома кишечника безмикробным (GF) мышам уменьшает рост опухоли. (A) Экспериментальный дизайн эксперимента FMT1 на безмикробных (GF) мышах. Время указывается в днях (D) относительно дня введения опухоли (8×10-5 опухолевых клеток). (B) Разница в размере опухолей, имплантированных отвечающим на лечение (R)-FMT и не отвечающим на лечение (NR)-FMT мышам или контрольным мышам. Объемы опухолей на 14-й день после имплантации опухоли наносят на график, причем каждое значение представляет одну мышь.
[0067] Фиг. 26A-C: Благоприятный FMT стимулирует врожденный эффектор и уменьшает инфильтрацию миелоидного супрессора в селезенке мышей GF. (A) Количественная оценка посредством проточной цитометрии, показывающая частоту CD45+ иммунных клеток у R-FMT (R), NR-FMT (NR) и контрольных мышей (C) в селезенке. (B) Количественная оценка посредством проточной цитометрии, показывающая частоту CD45+CD11b+Ly6G+ врожденных эффекторных клеток у R-FMT (R), NR-FMT (NR) и контрольных мышей (C) в селезенке. (C) Количественная оценка посредством проточной цитометрии, показывающая частоту CD45+CD11b+CD11c+ супрессивных клеток у R-FMT (R), NR-FMT (NR) и контрольных мышей (C) в селезенке.
[0068] Фиг. 27A-C: Благоприятный FMT увеличивает CD45+ и CD8+ в кишечнике и опухоли мышей GF. Репрезентативное иммунофлуоресцентное окрашивание опухоли (A) и кишечника (B) из контроля (слева), NR-FMT (в центре) и R-FMT (справа) в опухоли мышей GF после FMT для CD45, CD8 и ядер (DAPI). (C) Количественная оценка плотности CD8+ в опухоли (вверху) R-FMT (n=2, медиана=433,5 клеток/HPF в 12 областях), NR-FMT (NR-FMT n=2, медиана=325 клеток/HPF в 12 областях) и контрольных мышей (n=2, медиана=412 клеток/HPF в 9 областях). p=0,30 (R-FMT против контроля) и кишечник (внизу) (R-FMT n=2, медиана=67 клеток/HPF в 7 областях, NR-FMT n=2, медиана=24 клетки/HPF в 5 областях , контроль n=2, медиана=47 клеток/HPF в 10 областях). р=0,17 (R-FMT против контроля).
[0069] Фиг. 28A-C: FMT благоприятного микробиома кишечника у мышей GF уменьшает рост опухоли и усиливает ответ на терапию α-PD-L1. (А) Экспериментальный дизайн эксперимента FMT2 у безмикробных мышей (GF). Время указывается в днях (D) относительно дня введения опухоли (2,5×10-5 опухолевых клеток). (B) Различие в размерах опухолей, имплантированных мышам R-FMT (R, квадраты) и NR-FMT (NR, треугольники) или контрольным мышам (круги). Объемы опухолей на 14-й день после имплантации опухоли наносят на график, причем каждое значение представляет одну мышь. (C) Кривые роста опухоли для каждой мыши GF, подвергнутых лечению α-PD-L1 (3×100 мкг внутрибрюшинно каждые 3 дня) R-FMT (квадраты, n=2, средний объем опухоли=403,7 мм3), NR-FMT (треугольники, n=2, средний объем опухоли=2301 мм3) и контрольные (круги, n=2, средний объем опухоли=771,35 мм3). p=0,20 (R-FMT против NR-FMT), p=0,33 (NR-FMT против контроля посредством двустороннего теста MW). Пунктирная черная линия обозначает отсечение размера опухоли для лечения α-PD-L1 (500 мм3).
[0070] Фиг. 29A-E: Усиленный терапевтический ответ при благоприятном FMT коррелирует с увеличенным врожденным эффектором и уменьшенной инфильтрацией миелоидного супрессора в опухолях у мышей GF. (A) Количественная оценка с помощью проточной цитометрии, показывающая частоту инфильтрирующих опухоль CD45+ иммунных клеток у R-FMT, NR-FMT и контрольных мышей, как указано. (B) Репрезентативные графики проточной цитометрии CD45+CD11b+Ly6G+ врожденных эффекторных клеток и (D) CD11b+CD11c+ супрессорных миелоидных клеток у контрольных (слева), NR-FMT (в центре) и R-FMT (справа) мышей. (C) Количественная оценка посредством проточной цитометрии, показывающая частоту CD45+CD11b+Ly6G+ врожденных эффекторных клеток и (E) инфильтрирующих опухоль CD45+CD11b+CD11c+ супрессивных клеток у R-FMT, NR-FMT и контрольных мышей, как указано.
[0071] Фиг. 30A-D: Мыши GF, получающие FMT от NR-донора, характеризуются высокоактивированным компартментом Th17. (A) Репрезентативные изображения окрашивания IHC для ядерного рецептора-сироты гамма t (RORγT), связанного с ретиноевой кислотой, на опухолях мышей R-FMT (справа), NR-FMT (в центре) и контрольных (слева) мышей. Стрелки указывают на RORγT-положительные клетки. (B) Количественная оценка IHC, показывающая количество клеток RORγT+ Th17 у R-FMT (R), NR-FMT (NR) и контрольных мышей (C) в опухоли в виде количества/мм2. (C) Количественная оценка посредством проточной цитометрии, показывающая частоту CD4+ FoxP3+ регуляторных Т-клеток у R-FMT (R), NR-FMT (NR) и контрольных мышей (C) в селезенке. (D) Количественная оценка посредством проточной цитометрии, показывающая частоту CD4+IL17+Th17 клеток у R-FMT (R), NR-FMT (NR) и контрольных мышей (C) в селезенке.
[0072] Фиг. 31A-B: Активация PD-L1 в опухолевом микроокружении у мышей, получающих R-FMT по сравнению с NR-FMT, с помощью масс-цитометрии (CyTOF). (A) График t-SNE общего количества живых клеток (слева), выделенных из опухолей, полученных от контрольных, NR- и R-колонизированных мышей, как указано, с помощью масс-цитометрии. График t-SNE общего количества живых клеток с наложением экспрессии CD45 (в центре) и PD-L1 (справа). Равные количества клеток отображаются из каждой группы. (B) (вверху слева) график t-SNE всех CD45+ клеток, выделенных из опухолей, полученных от контрольных мышей, NR-FMT и R-FMT, как указано, посредством CyTOF. (вверху справа) Графики плотности всех CD45+ клеток, выделенных из опухолей, полученных из указанных экспериментальных групп. (внизу) график t-SNE всех клеток CD45+ с наложением экспрессии указанных маркеров.
[0073] Фиг. 32A-C: FMT от другого R-донора у мышей GF подтверждает влияние благоприятного микробиома кишечника на рост опухоли. (А) Экспериментальный дизайн эксперимента FMT2 у безмикробных мышей (GF). Время указывается в днях (D) относительно дня введения опухоли (2,5×10-5 опухолевых клеток). (B) Различие в размере опухолей, имплантированных мышам R-FMT (квадраты) и NR-FMT (треугольники) или контрольным мышам (круги). Объемы опухолей на 14-й день после имплантации опухоли наносят на график, причем каждое значение представляет одну мышь. (C) Кривые роста опухоли для каждой мыши GF от R-FMT (квадрат, объем опухоли=414,3 мм3), NR-FMT (треугольник, объем опухоли=1909,1 мм3) и контрольных (кружок, n=3, средний объем опухоли=1049,3 мм3) мышей.
[0074] Фиг. 33A-33F: Генетически идентичные мыши C57/BL6 из Jackson и Taconic демонстрируют избирательный рост опухоли (раньше у Jackson) (A), выживание (выше у Taconic) (B-C) и композицию микробиома (Taconic с одиночным размещением в верхнем правом углу; Jackson с одиночным размещением в нижнем правом углу) (D) после имплантации опухолей мышиной меланомы (BRAF-мутант, PTEN-ноль). Совместное размещение мышей Taconic и Jackson привело к сходному разрастанию опухоли (C), и при анализе основных координат (D) наблюдалось увеличение сходства микробиомов. Избирательная многочисленность на уровне рода наблюдалась у мышей с одиночным размещением из Jackson и Taconic, но никаких различий не наблюдалось после совместного размещения (E). Пероральное введение бутирата значительно замедляло разрастание опухоли у мышей, которым имплантировали опухоли меланомы (F).
[0075] Фиг. 34A-C: 16S анализ фекальных образцов от доноров R и NR и безмикробных мышей-реципиентов. Сравнение относительной численности (A) Faecalibacterium, (B) Ruminococcaceae и (C) Bacteroidales на 14 день после инъекции опухоли. Представлены данные 2 независимых экспериментов. **р<0,01.
Подробное описание изобретения
[0076] Огромные успехи в лечении рака были достигнуты благодаря применению молекулярно-направленной терапии и иммунотерапии, однако ответы являются изменчивыми и не всегда долговременными. Лечение ингибиторами иммунных контрольных точек связано с частотой ответа 15-40% у пациентов с распространенной меланомой, и в настоящее время предпринимаются усилия по определению стратегий для усиления ответов на терапию ингибитором контрольных точек. Таким образом, способы улучшения терапевтических ответов, а также увеличения числа отвечающих на лечение пациентов крайне необходимы.
[0077] Настоящее раскрытие преодолевает проблемы с современными технологиями, предоставляя способы модуляции микробиома для улучшения иммунного ответа на рак и терапевтического ответа на ингибиторы иммунных контрольных точек у онкологических пациентов. В исследованиях по настоящему изобретению использовалась большая группа пациентов с метастатической меланомой, проходивших системное лечение (n=233), подгруппа которых подвергалась лечению иммунотерапией на основе PD-1 (n=112). Образцы орального и кишечного микробиома были охарактеризованы у этих пациентов с помощью секвенирования гена 16S рРНК и метагеномного секвенирования всего генома. В этих анализах наблюдались значительные различия в разнообразии и композиции микробиома кишечника у отвечающих на блокаду по сравнению с не отвечающими на блокаду иммунных контрольных точек (например, на терапию на основе PD-1) пациентами со значительно более высоким разнообразием и повышенной численностью специфических бактерий (например, в пределах порядка Clostridiales и семейства Ruminococcaceae) в микробиоме кишечника отвечающих на лечение пациентов по сравнению с не отвечающими. В частности, было обнаружено, что вид Faecalibacterium prausnitzii является более многочисленным среди отвечающих на лечение пациентов. Известно, что эти бактерии производят короткоцепочечные жирные кислоты, такие как бутират, которые помогают поддерживать целостность специфических клеток в кишечнике (то есть энтероцитов) и могут повышать иммунитет.
[0078] Интересно отметить, что у не отвечающих на терапию пациентов было отмечено низкое содержание этих бактерий и значительно более высокое содержание бактерий порядка Bacteroidales, что было показано в некоторых исследованиях для подавления системных иммунных ответов. Метагеномный анализ с помощью секвенирования всего генома способом дробовика был выполнен в подгруппе этих пациентов, чтобы подтвердить эти результаты, и дополнительно продемонстрировал различия в метаболических процессах в бактериях отвечающих и не отвечающих на лечение пациентов. Кроме того, было продемонстрировано, что модуляция кишечного микробиома совместным размещением мышей Taconic и Jackson и пероральным введением короткоцепочечных жирных кислот (например, бутирата) приводила к замедленному росту опухоли у мышей с менее благоприятным микробиомом кишечника (мыши Jackson). Эти результаты исследований на людях и мышах имеют потенциально далеко идущие последствия для усиления ответов на блокаду иммунных контрольных точек посредством модуляции кишечного микробиома.
[0079] Важно, что настоящие исследования показывают, что пациенты с «благоприятным» микробиомом кишечника (с большим разнообразием и высокой относительной численностью бактерий порядка Clostridiales и/или семейства Ruminococcaceae) характеризуются усиленными системными и противоопухолевыми иммунными ответами, опосредованными усиленной презентацией антигена на уровне лимфатического узла и опухоли, а также сохраненной эффекторной функцией Т-клеток на периферии и микроокружении опухоли. Напротив, у пациентов с «неблагоприятным» микробиомом кишечника (с низким разнообразием и высокой относительной численностью бактерий порядка Bacteroidales) наблюдаются нарушения системного и противоопухолевого иммунного ответа, опосредованные ограниченной внутриопухолевой инфильтрацией как лимфоидных, так и миелоидных элементов, ослабленная способность к презентации антигена и ориентация на иммунорегуляторные клеточные и гуморальные элементы на периферии, в том числе Treg и MDSC.
[0080] Дальнейшие исследования также были предприняты в модельной системе меланомы мыши. Эти исследования показали, что мыши, получавшие трансплантацию фекальной микробиоты от отвечавшей на лечение популяции, характеризовались сниженным ростом опухоли и повышенным ответом на анти-PDL1 терапию. Кроме того, мыши, которые получали трансплантацию микробиологической популяции отвечавших на лечение пациентов, характеризовались более высоким процентом врожденных эффекторных клеток (экспрессирующих CD45+CD11b+Ly6G+) и более низкой частотой супрессорных миелоидных клеток (экспрессирующих CD11b+CD11c+) в селезенке, а также увеличенным количеством CD45+ иммунных и CD8+ Т-клеток в кишечнике. Эти данные подчеркивают возможность параллельной модуляции микробиома кишечника для значительного повышения эффективности блокады контрольных точек, что требует быстрой оценки в клинических испытаниях.
[0081] На основании этих результатов в настоящем документе представлены способы лечения и диагностики рака. В одном способе короткоцепочечные жирные кислоты, такие как бутират, и/или производящую короткоцепочечные жирные кислоты бактериальную популяцию, таких как производящие бутират бактерии, вводят пациентам во время лечения с помощью блокады иммунных контрольных точек для усиления терапевтических ответов. В настоящем документе также представлены способы применения разнообразия и композиции микробиома кишечника в качестве прогностического биомаркера для идентификации пациентов, которые будут характеризоваться благоприятным ответом на блокаду иммунных контрольных точек.
I. Определения
[0082] Используемый в настоящем документе термин «по существу свободный» в терминах определенного компонента используется в настоящем документе для обозначения того, что ни один из указанных компонентов не был целенаправленно составлен в композицию и/или присутствует только в качестве загрязняющего вещества или в следовых количествах. Таким образом, общее количество указанного компонента в результате любого непреднамеренного загрязнения композиции значительно ниже 0,01%. Наиболее предпочтительной является композиция, в которой никакое количество указанного компонента не может быть обнаружено стандартными аналитическими способами.
[0083] Используемое в настоящем документе единственное число может означать один или более чем один.
Использование термина «или» в формуле изобретения используется для обозначения «и/или», если явно не указано, что оно относится только к альтернативам или альтернативы являются взаимоисключающими, хотя раскрытие поддерживает определение, которое относится только к альтернативам и «и/или». Используемый в настоящем документе термин «другой» может означать по меньшей мере второй или более.
[0085] Во всей настоящей заявке термин «приблизительно» используется для указания того, что значение включает в себя внутреннее изменение ошибки для устройства, способ, используемый для определения значения, или изменение, которое существует среди субъектов исследования.
[0086] Фраза «эффективное количество» или «терапевтически эффективное количество», или «достаточное количество» означает дозировку лекарственного средства или средства, достаточную для получения желаемого результата. Желаемым результатом может быть уменьшение размера опухоли, уменьшение скорости роста раковых клеток, уменьшение метастазирования, увеличение CD8+ Т-лимфоцитов в опухоли или опухолевом иммунном инфильтрате, увеличение CD45+, CD3+/CD20+/CD56+, CD68+ и/или HLA-DR+ клеток в опухоли, увеличение экспрессии CD3, CD8, PD1, FoxP3, гранзима B и/или PD-L1 в опухолевом иммунном инфильтрате, снижение экспрессии RORγT в опухолевом иммунном инфильтрате, увеличение эффекторных CD4+, CD8+ Т, моноцитов и/или миелоидных дендритных клеток в системном кровообращении или периферической крови, уменьшение количества В-клеток, регуляторных Т-клеток и/или миелоидных клеток-супрессоров в системном кровообращении или периферической крови субъекта или любая комбинация вышеперечисленного.
[0087] Термин «опухолевая клетка» или «раковая клетка» обозначает клетку, которая демонстрирует неприемлемую, нерегулируемую пролиферацию. «Человеческая» опухоль состоит из клеток, которые содержат человеческие хромосомы. Такие опухоли включают в себя опухоли у пациента-человека и опухоли, возникающие в результате введения отличному от человека животному-хозяину злокачественной клеточной линии, имеющей человеческие хромосомы.
[0088] Используемый в настоящем документе термин «антитело» относится к иммуноглобулину, его производным, которые сохраняют специфическую способность к связыванию, и белкам, содержащим домен связывания, который гомологичен или в значительной степени гомологичен домену, связывающему иммуноглобулин. Эти белки могут быть получены из природных источников или частично или полностью получены синтетическим путем. Антитело может быть моноклональным или поликлональным. Антитело может быть представителем любого класса иммуноглобулинов, включая в себя любой из классов человека: IgG, IgM, IgA, IgD и IgE. Антитела, используемые в описанных в настоящем документе способах и композициях, как правило, являются производными класса IgG. Термин антитело также относится к антигенсвязывающим фрагментам антител. Примеры таких фрагментов антител включают в себя, без ограничения, фрагменты Fab, Fab', F(ab')2, scFv, Fv, диатела dsFv и Fd. Фрагменты антител могут быть получены любым способом. Например, фрагмент антитела может быть получен ферментативно или химически путем фрагментации интактного антитела, он может быть получен рекомбинантно из гена, кодирующего частичную последовательность антитела, или он может быть полностью или частично синтезирован. Фрагмент антитела может необязательно представлять собой одноцепочечный фрагмент антитела. Альтернативно, фрагмент может содержать множество цепей, которые связаны вместе, например, дисульфидными связями. Фрагмент также может необязательно представлять собой мультимолекулярный комплекс. Фрагмент функционального антитела сохраняет способность связывать свой родственный антиген с сопоставимой с полным антителом аффинностью.
[0089] Используемый в настоящем документе термин «моноклональное антитело» относится к антителу, полученному из популяции по существу гомогенных антител, например, отдельные антитела, составляющие популяцию, являются идентичными, за исключением возможных мутаций, например, встречающихся в природе мутаций, которые могут присутствовать в незначительных количествах. Таким образом, модификатор «моноклональный» указывает на характер антитела как не являющегося смесью отдельных антител. Согласно определенным вариантам осуществления такое моноклональное антитело, как правило, включает в себя антитело, содержащее полипептидную последовательность, которая связывает мишень, причем связывающуюся с мишенью полипептидную последовательность получали способом, который предусматривает выбор единственной связывающейся с мишенью полипептидной последовательности из множества полипептидных последовательностей. Например, процесс отбора может представлять собой отбор уникального клона из множества клонов, таких как пул гибридомных клонов, фаговых клонов или рекомбинантных клонов ДНК. Следует понимать, что выбранная связывающаяся с мишенью последовательность может быть дополнительно изменена, например, для улучшения аффинности к мишени, для гуманизации связывающейся с мишенью последовательности, для улучшения ее производства в клеточной культуре, для снижения ее иммуногенности in vivo, для создания мультиспецифического антитела и т. д., и что антитело, содержащее измененную связывающуюся с мишенью последовательность также представляет собой моноклональное антитело по настоящему изобретению. В отличие от препаратов поликлональных антител, которые, как правило, включают в себя несколько разных антител, направленных против разных детерминант (эпитопов), каждое моноклональное антитело препарата моноклональных антител направлено против одной детерминанты на антигене. В дополнение к специфичности препараты моноклональных антител характеризуются тем преимуществом, что они, как правило, не загрязнены другими иммуноглобулинами.
[0090] Фразы «фармацевтическая композиция» или «фармакологически приемлемая композиция» относятся к молекулярным веществам и композициям, которые не вызывают нежелательную, аллергическую или другую неблагоприятную реакцию при введении животному, такому как человек, в зависимости от ситуации. Приготовление фармацевтической композиции, содержащей антитело или дополнительный активный ингредиент, будет известно специалистам в настоящей области техники в свете настоящего раскрытия. Более того, для введения животным (например, человеку) следует понимать, что препараты должны соответствовать стандартам стерильности, пирогенности, общей безопасности и чистоты, как того требует Управление биологических стандартов FDA.
[0091] Используемый в настоящем документе термин «фармацевтически приемлемый носитель» включает в себя любые и все водные растворители (например, воду, спиртовые/водные растворы, солевые растворы, парентеральные носители, такие как хлорид натрия и декстроза Рингера), неводные растворители (например, пропиленгликоль, полиэтиленгликоль, растительное масло и инъецируемые органические сложные эфиры, такие как этилолеат), дисперсионные среды, покрытия, поверхностно-активные вещества, антиоксиданты, консерванты (например, антибактериальные или противогрибковые средства, антиоксиданты, хелатирующие средства и инертные газы), изотонические средства, замедляющие абсорбцию средства, соли, лекарственные средства, стабилизаторы лекарственных средств, гели, связующие вещества, вспомогательные вещества, дезинтеграторы, смазывающие вещества, подсластители, ароматизаторы, красители, пополнители жидкости и питательных веществ, такие как материалы и их комбинации, как известно специалисту в настоящей области техники. pH и точную концентрацию различных компонентов в фармацевтической композиции можно регулировать в соответствии с хорошо известными параметрами.
[0092] Термин «однократная доза» или «дозировка» относится к физически дискретным единицам, подходящим для применения у субъекта, причем каждая единица содержит заданное количество терапевтической композиции, рассчитанной для получения желаемых ответов, обсуждаемых в настоящем документе, в связи с ее введением, т.е. соответствующим путем и схемой лечения. Количество, которое следует вводить, как в зависимости от количества воздействий, так и от однократной дозы, зависит от желаемого эффекта. Фактическое дозированное количество вводимой пациенту или субъекту композиции по настоящему изобретению может определяться физическими и физиологическими факторами, такими как масса тела, возраст, состояние здоровья и пол субъекта, тип подвергаемого лечению заболевания, степень проникновения заболевания, предыдущие или параллельные терапевтические вмешательства, идиопатия пациента, путь введения, а также активность, стабильность и токсичность конкретного терапевтического вещества. Например, доза может также составлять от приблизительно 1 мкг/кг/масса тела до приблизительно 1000 мг/кг/масса тела (этот такой диапазон включает в себя промежуточные дозы) или более на введение, и любая конкретная доза, полученная в ней. В неограничивающих примерах диапазон, полученный из чисел, перечисленных в настоящем документе, составляет от приблизительно 5 мкг/кг/масса тела до приблизительно 100 мг/кг/масса тела, от приблизительно 5 мкг/кг/масса тела до приблизительно 500 мг/кг/масса тела и т.д. В любом случае, ответственный за введение специалист будет определять концентрацию активного ингредиента(ов) в композиции и соответствующую дозу(ы) для отдельного субъекта.
[0093] «Противораковое» средство способно отрицательно влиять на раковые клетки/опухоли у субъекта, например, способствуя уничтожению раковых клеток, индуцируя апоптоз в раковых клетках, уменьшая скорость роста раковых клеток, уменьшая частоту или число метастазов, уменьшая размеры опухоли, ингибируя рост опухоли, уменьшая кровоснабжение опухоли или раковых клеток, стимулируя иммунный ответ против раковых клеток или опухоли, предотвращая или ингибируя прогрессирование рака или увеличивая продолжительность жизни субъект с раком.
[0094] Термин «иммунная контрольная точка» относится к компоненту иммунной системы, который обеспечивает ингибирующие сигналы для своих компонентов, чтобы регулировать иммунные реакции. Известные белки иммунных контрольных точек включают в себя CTLA-4, PD-1 и его лиганды PD-L1 и PD-L2, а также LAG-3, BTLA, B7H3, B7H4, TIM3, KIR. В настоящей области техники признано, что пути, включающие в себя LAG3, BTLA, B7H3, B7H4, TIM3 и KIR, представляют собой пути иммунных контрольных точек, сходные с CTLA-4- и PD-1-зависимыми путями (смотрите, например, Pardoll, 2012, Nature Rev Cancer 12:252-264; Mellman et al., 2011, Nature 480:480-489).
[0095] Термин «антагонист связывания оси PD-1» относится к молекуле, которая ингибирует взаимодействие партнера по связыванию оси PD-1 с одним или более его партнерами по связыванию, таким образом удаляя дисфункцию Т-клеток, возникающую в результате передачи сигналов на ось передачи сигналов PD-1, в результате происходит восстановление или усиление функции Т-клеток (например, пролиферация, производство цитокинов, уничтожение клеток-мишеней). Термин «ось PD-1» относится к любому компоненту иммунной контрольной точки PD-1 (например, PD-1, PD-L1 и PD-L2). Используемый в настоящем документе антагонист связывания оси PD-1 включает в себя антагониста связывания PD-1, антагониста связывания PD-L1 и антагониста связывания PD-L2.
[0096] Термин «антагонист связывания PD-1» относится к молекуле, которая уменьшает, блокирует, ингибирует, отменяет или мешает передаче сигнала, возникающей в результате взаимодействия PD-1 с одним или более его партнерами по связыванию, такими как PD-L1 и/или PD-L2. Антагонист связывания PD-1 может представлять собой молекулу, которая ингибирует связывание PD-1 с одним или более его партнерами по связыванию. Согласно конкретному аспекту антагонист связывания PD-1 ингибирует связывание PD-1 с PD-L1 и/или PD-L2. Например, антагонисты связывания PD-1 включают в себя антитела к PD-1, их антигенсвязывающие фрагменты, иммуноадгезины, слитые белки, олигопептиды и другие молекулы, которые уменьшают, блокируют, ингибируют, отменяют или препятствуют передаче сигнала, возникающей в результате взаимодействия PD-1 с PD-L1 и/или PD-L2. Иллюстративный антагонист связывания PD-1 представляет собой антитело к PD-1. Например, антагонистом связывания PD-1 является MDX-1106 (ниволумаб), MK-3475 (пембролизумаб), CT-011 (пидилизумаб) или AMP-224.
[0097] Термин «антагонист связывания PD-L1» относится к молекуле, которая уменьшает, блокирует, ингибирует, отменяет или мешает передаче сигнала, возникающей в результате взаимодействия PD-L1 с одним или более его партнерами по связыванию, такими как PD-1 или B7-1. Например, антагонист связывания PD-L1 представляет собой молекулу, которая ингибирует связывание PD-L1 с его партнерами по связыванию. Согласно конкретному аспекту антагонист связывания PD-L1 ингибирует связывание PD-L1 с PD-1 и/или B7-1. Антагонисты связывания PD-L1 могут включать в себя антитела к PD-L1, их антигенсвязывающие фрагменты, иммуноадгезины, слитые белки, олигопептиды и другие молекулы, которые уменьшают, блокируют, ингибируют, отменяют или препятствуют передаче сигнала, возникающей в результате взаимодействия PD-L1 с одним или более его партнерами по связыванию, такими как PD-1 или B7-1. Например, антагонист связывания PD-L1 уменьшает отрицательный костимуляторный сигнал, опосредованный белками клеточной поверхности или через них, экспрессируемый на Т-лимфоцитах, опосредованный передачей сигналов через PD-L1, таким образов приводя к тому, что дисфункциональные Т-клетки становятся менее дисфункциональными (например, усиливая эффекторные ответы для распознавания антигена). В одном примере антагонист связывания PD-L1 представляет собой антитело к PD-L1. Антитело к PD-L1 может представлять собой YW243.55.S70, MDX-1105, MPDL3280A или MEDI4736.
[0098] Термин «антагонист связывания PD-L2» относится к молекуле, которая уменьшает, блокирует, ингибирует, отменяет или мешает передаче сигнала, возникающей в результате взаимодействия PD-L2 с одним или более его партнерами по связыванию, такими как PD-1. Связывающий PD-L2 антагонист может представлять собой молекулу, которая ингибирует связывание PD-L2 с одним или более его партнерами по связыванию. Например, антагонист связывания PD-L2 ингибирует связывание PD-L2 с PD-1, таким как антагонисты PD-L2, включая в себя антитела к PD-L2, их антигенсвязывающие фрагменты, иммуноадгезины, слитые белки, олигопептиды и другие молекулы, которые уменьшают, блокируют, ингибируют, отменяют или мешают передаче сигнала, возникающей в результате взаимодействия PD-L2 с одним или более партнерами по связыванию, такими как PD-1.
[0099] Термин "ингибитор иммунных контрольных точек" относится к любому соединению, ингибирующему функцию белка иммунных контрольных точек. Ингибирование предусматривает снижение функции и полную блокаду. В частности, белок иммунных контрольных точек представляет собой белок иммунных контрольных точек человека. Таким образом, ингибитор белка иммунных контрольных точек, в частности, является ингибитором белка иммунных контрольных точек человека.
[00100] «Субъект» и «пациент» относятся к человеку или отличным от человека животным, например, приматам, млекопитающим и позвоночным животным. Согласно конкретным вариантам осуществления субъектом является человек.
[00101] Используемые в настоящем документе термины «лечить», «лечение» или «улучшение» при использовании в отношении заболевания, нарушения или медицинского состояния, относятся к терапевтическому лечению состояния, причем объектом является обращение вспять, облегчение, улучшение, подавление, замедление или остановка прогрессирования или тяжести симптома или состояния. Термин «лечение» включает в себя уменьшение или облегчение по меньшей мере одного неблагоприятного воздействия или симптома состояния. Лечение, как правило, «эффективно», если один или более симптомов или клинических маркеров уменьшаются. Альтернативно, лечение является «эффективным», если прогрессирование состояния уменьшается или останавливается. То есть «лечение» включает в себя не только улучшение симптомов или маркеров, но также прекращение или по меньшей мере замедление прогрессирования или ухудшения симптомов, которые можно ожидать в отсутствие лечения. Благоприятные или желательные клинические результаты включают в себя, без ограничения, ослабление одного или более симптомов, уменьшение степени дефицита, стабилизированное (т.е. не ухудшающееся) состояние опухоли или злокачественного новообразования, задержку или замедление роста опухоли и/или метастазирования и увеличение продолжительности жизни по сравнению с ожидаемым при отсутствии лечения.
[00102] «Микробиота кишечника» или «микробиом кишечника» обозначает популяцию микроорганизмов, живущих в кишечнике субъекта.
[00103] Термин «альфа разнообразие» представляет собой меру разнообразия внутри образца и относится к паттернам распределения и сборки всей микробиоты в образцах и рассчитывается как скалярное значение для каждого образца. «Бета разнообразие» представляет собой термин для разнообразия между образцами, который включает в себя сравнение образцов с каждым из них, что обеспечивает меру расстояния или различия между каждой парой образцов.
[00104] Термин «относительное количество», который также может быть обозначен как «относительная численность», определяется как число бактерий определенного таксономического уровня (от типа до вида) в виде процента от общего количества бактерий этого уровня в биологическом образце. Эта относительная численность может быть оценена, например, путем измерения процентного содержания последовательностей генов 16S рРНК, присутствующих в образце, которые относятся к этим бактериям. Его можно измерить любым подходящим способом, известным специалисту в настоящей области техники, таким как пиросеквенирование 454 и количественная ПЦР этих специфических бактериальных маркеров гена 16S рРНК или количественная ПЦР конкретного гена.
[00105] В настоящем тексте «хорошо отвечающий на лечение пациент», также называемый «отвечающий на лечение пациент» или «реагирующий на лечение пациент» или другими словами, пациент, который «получает пользу» от этого лечения, относится к пациенту, который характеризуется наличием рака и который показывает или покажет клинически значимое облегчение заболевания после получения этого лечения. И наоборот, «плохо отвечающий на лечение пациент» или «не отвечающий на лечение пациент» представляет собой пациента, который не демонстрирует или не продемонстрирует клинически значимого облегчения рака после получения этого лечения. Сниженный ответ на лечение может быть оценен в соответствии со стандартами, признанными в настоящей области техники, такими как критерии иммунного ответа (irRC), критерии ВОЗ или RECIST.
[00106] Термин «выделенный» охватывает бактерию или другую сущность или вещество, которое было (1) отделено по меньшей мере от некоторых компонентов, с которыми оно было связано при первоначальном производстве (в природе или в экспериментальных условиях), и/или (2) произведено, приготовлено, очищено и/или изготовлено руками человека. Выделенные бактерии могут быть отделены от по меньшей мере приблизительно 10%, приблизительно 20%, приблизительно 30%, приблизительно 40%, приблизительно 50%, приблизительно 60%, приблизительно 70%, приблизительно 80%, приблизительно 90% или более других компонентов, с которыми они изначально были связаны. Согласно некоторым вариантам осуществления выделенные бактерии характеризуются чистотой более чем приблизительно на 80%, приблизительно на 85%, приблизительно на 90%, приблизительно на 91%, приблизительно на 92%, приблизительно на 93%, приблизительно на 94%, приблизительно на 95%, приблизительно на 96%, приблизительно на 97%, приблизительно на 98%, приблизительно на 99% или более чем на 99%. Как используется в настоящем документе вещество является «чистым», если оно по существу не содержит других компонентов.
[00107] Термины «очищать», «очистка» и «очищенный» относятся к бактерии или другому материалу, который был отделен по меньшей мере от некоторых компонентов, с которыми он был связан, либо при первоначальном производстве, либо при получении (например, при природой или в экспериментальной обстановке), или в любое время после его первоначального производства. Бактерия или бактериальная популяция может считаться очищенной, если она выделена во время или после производства, например, из материала или среды, содержащей бактерию или бактериальную популяцию, и очищенная бактерия или бактериальная популяция может содержать другие материалы до приблизительно 10%, приблизительно 20%, приблизительно 30%, приблизительно 40%, приблизительно 50%, приблизительно 60%, приблизительно 70%, приблизительно 80%, приблизительно 90% или более чем приблизительно 90% и все еще считаться «выделенной». Согласно некоторым вариантам осуществления очищенные бактерии и бактериальные популяции характеризуются чистотой более чем приблизительно 80%, приблизительно 85%, приблизительно 90%, приблизительно 91%, приблизительно 92%, приблизительно 93%, приблизительно 94%, приблизительно 95%, приблизительно 96%, приблизительно 97%, приблизительно 98% чистота приблизительно 99% или более чем приблизительно 99%. В случае представленных в настоящем документе бактериальных композиций один или более типов бактерий, присутствующих в композиции, могут быть независимо очищены от одной или более других бактерий, произведенных и/или присутствующих в материале или среде, содержащей тип бактерий. Бактериальные композиции и их бактериальные компоненты, как правило, очищают от остаточных продуктов среды обитания.
II. Очищенная бактериальная популяция
[00108] Варианты осуществления настоящего изобретения относятся к композициям, содержащим короткоцепочечные жирные кислоты, таким как композиции, содержащие бутират, и очищенным бактериальным популяциям (например, бактериальным популяциям, содержащим короткоцепочечные жирные кислоты, таким как производящие бутират бактериальные популяции), для лечение рака, например, у субъекта, которому вводят или вводили ингибитор иммунных контрольных точек. Согласно некоторым вариантам осуществления субъекту вводят пребиотик и/или пробиотик для обогащения производящими бутират бактериями. Согласно определенным аспектам субъект подвергается диетическим изменениям для обогащения производящими бутират бактериями.
[00109] Согласно определенным вариантам осуществления настоящее раскрытие относится к пробиотическим композициям и живым бактериальным продуктам, которые содержат бактериальные популяции, благоприятные для ответа на терапию иммунных контрольных точек. Пробиотическая композиция может содержать бактерии типа Firmicutes. Бактериальная популяция может принадлежать к классу Clostridia, конкретно к порядку Clostridales, или одна или более бактериальных популяций могут принадлежать к семейству Clostridiaceae, Ruminococcaceae (например, конкретно к роду Ruminococcus или роду Faecalibacterium), Micrococcaceae (например, конкретно к роду Rothia), Lachnospiraceae и/или Veilonellaceae. Согласно дополнительным аспектам бактериальная популяция может принадлежать к типу Tenericutes, в частности к классу Mollicutes. Бактерии могут принадлежать к роду Peptoniphilus, особенно к видам P. asaccharolyticus, P. gorbachii, P. harei, P. ivorii, P. lacrimalis и/или P. olsenii. Другие иллюстративные бактериальные популяции для пробиотической композиции могут включать в себя бактериальные популяции, которые принадлежат к роду Porphyromonas, в частности к виду Porphyromonas pasteri, виду Clostridium hungatei, роду Phascolarctobacterium или виду Phascolarctobacterium faecium.
[00110] Например, бактериальные популяции рода Ruminococcus могут включать в себя бактерии вида Ruminococcus albus, Ruminococcus bromii, Ruminococcus callidus, Ruminococcus flavefaciens, Ruminococcus champanellensis, Ruminococcus faecis, Ruminococcus gauvreauii, Ruminococcus gnavus, Ruminococcus hansenii, Ruminococcus hydrogenotrophicus, Ruminococcus lactaris, Ruminococcus luti , Ruminococcus obeum, Ruminococcus palustris , Ruminococcus pasteurii, Ruminococcus productus, Ruminococcus schinkii и/или Ruminococcus torques. Бактериальные популяции рода Faecalibacterium могут включать в себя бактерии вида Faecalibacterium prausnitzii.
[00111] Иллюстративные бактериальные популяции рода Rothia могут включать в себя бактерии видов R. aeria, R. amarae, R. dentocariosa, R. endophytica, R. mucilaginosa, R. nasimurium и/или R. terrae.
[00112] Иллюстративные бактериальные популяции для включения в пробиотическую композицию включают в себя бактериальные популяции, которые принадлежат к типу Firmicutes, классу Clostridia, семейству Ruminococcaceae, виду Faecalibacterium prausnitzii, роду Ruminococcus, виду Porphyromonas pasteri, семейству Veilonellaceae, виду Colostridium hungatei, роду Phascolarctobacterium, виду Phascolarctobacterium faecium, роду Peptoniphilus, семейству Micrococcaceae, классу Mollicutes и/или роду Rothia.
[00113] Согласно конкретным аспектам пробиотическая композиция или живой бактериальный продукт не содержит бактериальные популяции порядка Bacteroidales, такие как род Bacteroides, в частности, виды B. thetaiotaomicron, B. fragilis, B. vulgatus, B. distasonis, B. ovatus, B. stercoris, B. merda, B. uniformis, B. eggerithii или B. caccae. В частности, пробиотическая композиция не содержит бактериальные популяции рода Gardnerella или видов Collinsella stercoris, Desulfovibrio alaskensis, Bacteroides mediterraneensis, Prevotella histicola или Gardnerella vaginalis.
ria
ceae
ceae
ceae
ceae
ria
ceae
bia
biales
riales
ceae
teria
rales
ceae
cales
ceae
terium
rium faecium
teria
teria
rales
ceae
bacteria
rales
ceae
cetaceae
bacteria
rales
ceae
teria
teria
teria
teria
teria
teria
ceae
teria
nales
ceae
tales
teria
nales
ceae
ceae
teria
teria
ceae
ceae
ceae
teria
tor
teria
tales
ceae
coccales
coccus
teria
ван. Нет данных
[00114] Таблица 2: Различия видов фекальных бактерий, полученных из WGS, по статусу ответа на лечение.
[00115] В таблице 2А показаны бактериальные гены, используемые для характеристики групп совместно представленных генов бактерий (CAG), и соответствующая SEQ ID NO для каждого гена в представляющих интерес бактериях. Каждая из перечисленных групп и генов CAG доступна во всемирной паутине по адресу meta.genomics.cn/meta/dataTools и включена в настоящий документ посредством ссылки.
[00116] Настоящее раскрытие также относится к фармацевтической композиции, содержащей одну или более описанных выше микробиологических культур. Таким образом, виды бактерий присутствуют в дозированной форме в виде живых бактерий, в высушенной, лиофилизированной или споролированной форме. Это может быть предпочтительно адаптировано для подходящего введения; например, в форме таблеток или порошка, потенциально с энтеросолюбильным покрытием, для перорального лечения.
[00117] Согласно конкретным аспектам композицию составляют для перорального введения. Пероральное введение может быть достигнуто с использованием жевательного состава, растворяющегося состава, капсулированного/покрытого оболочкой состава, многослойной пастилки (для разделения активных ингредиентов и/или активных ингредиентов и наполнителей), состава с медленным высвобождением/с замедленным высвобождением или другого подходящего состава, известного специалистам в настоящей области техники. Хотя в настоящем документе используется слово «таблетка», состав может принимать различные физические формы, которые, как правило, могут упоминаться другими терминами, такими как пастилка, пилюля, капсула или т.п.
[00118] Хотя композиции по настоящему изобретению предпочтительно составляют для перорального введения, однако могут быть использованы другие пути введения, включая в себя, без ограничения, подкожный, внутримышечный, внутрикожный, трансдермальный, внутриглазной, внутрибрюшинный, слизистый, вагинальный, ректальный и внутривенный.
[00119] Желаемая доза композиции по настоящему изобретению может быть представлена в виде нескольких (например, двух, трех, четырех, пяти, шести или более) поддоз, вводимых с соответствующими интервалами в течение дня.
[00120] Согласно одному аспекту раскрытая композиция может быть приготовлена в виде капсулы. Капсула (т.е. носитель) может быть полой, как правило, представлять собой цилиндрическую капсулу, образованную из различных веществ, таких как желатин, целлюлоза, углевод или т.п.
[00121] Согласно другому аспекту раскрытая композиция может быть приготовлена в виде суппозитория. Суппозиторий может включать в себя, без ограничения, бактерии и один или более носителей, таких как полиэтиленгликоль, аравийская камедь, ацетилированные моноглицериды, карнаубский воск, фталат ацетата целлюлозы, кукурузный крахмал, дибутилфталат, докузат натрия, желатин, глицерин, оксиды железа, каолин, лактоза, стеарат магния, метилпарабен, фармацевтическая глазурь, повидон, пропилпарабен, бензоат натрия, сорбитанмонолеат, тальк, сахароза, диоксид титана, белый воск и красители.
[00122] Согласно некоторым аспектам раскрытый пробиотик может быть приготовлен в виде таблетки. Таблетка может включать в себя бактерии и один или более средств для изготовления таблеток (т.е. носителей), таких как двухосновный фосфат кальция, стеариновая кислота, кроскармеллоза, диоксид кремния, целлюлоза и целлюлозное покрытие. Таблетки могут быть получены с использованием процесса прямого прессования, хотя специалисты в настоящей области техники поймут, что для получения таблеток могут использоваться различные техники.
[00123] Согласно другим аспектам раскрытый пробиотик может быть составлен в виде еды или напитка или, альтернативно, в качестве добавки к еде или напитку, причем соответствующее количество бактерий добавляют в еду или напиток для превращения еды или напитка в носитель.
[00124] Пробиотические композиции по настоящему изобретению могут дополнительно содержать один или более пребиотиков, известных в настоящей области техники, таких как лактит, инулин или их комбинацию.
[00125] Согласно некоторым вариантам осуществления композиции по вариантам осуществления содержат один или более видов бактерий, которые производят короткоцепочечные жирные кислоты. В частности, виды бактерий производят бутират. Например, бактериальная популяция может содержать один или более видов бактерий порядка Clostridiales. Бактерии Clostridiales могут быть в основном в форме спор. Согласно определенным аспектам бактериальный вид относится к семейству Ruminococcaceae, Christensenellaceae, Clostridiaceae или Coriobacteriacease. Согласно некоторым вариантам осуществления бактерии Clostridiales содержат первое семейство и второе семейство. Согласно некоторым вариантам осуществления первое семейство выбирают из группы, состоящей из Ruminococcaceae, Christensenellaceae, Clostridiaceae и Coriobacteriacease, а второе семейство не является идентичным первому семейству. Примеры видов бактерий включают в себя, без ограничения, Faecalibacterium prausnitzii, Ruminococcus albus, Ruminococcus bromii, Ruminococcus callidus, Ruminococcus flavefaciens, Ruminococcus champanellensis, Ruminococcus faecis, Ruminococcus gauvreauii, Ruminococcus gnavus, Ruminococcus hansenii, Ruminococcus hydrogenotrophicus, Ruminococcus lactaris, Ruminococcus luti , Ruminococcus obeum, Ruminococcus palustris , Ruminococcus pasteurii, Ruminococcus productus, Ruminococcus schinkii, Ruminococcus torques, Subdoligranulum variabile, Butyrivibrio fibrisolvens, Roseburia intestinalis, Anaerostipes caccae, Blautia obeum, Eubacterium nodatum и Eubacterium oxidoreducens. Согласно определенным аспектам бактериальным видом является Faecalibacterium prausnitzii. Согласно определенным вариантам осуществления бактериальная популяция не содержит бактериальные виды класса Bacteroidia или семейства Prevotellaceae.
[00126] Согласно некоторым вариантам осуществления бактериальная популяция содержит бактерии порядка Clostridiales в количестве, эффективном или достаточном для производства одного или более метаболитов, способных усиливать терапию иммунных контрольных точек у субъекта. Согласно некоторым вариантам осуществления один или более метаболитов содержат короткоцепочечную жирную кислоту. Согласно определенным аспектам короткоцепочечная жирная кислота представляет собой бутират. Согласно некоторым вариантам осуществления бактерии порядка Clostridiales производят одну или более короткоцепочечных жирных кислот (например, бутират) в эффективном количестве, чтобы увеличить локальную концентрацию короткоцепочечной жирной кислоты в 2 раза, 4 раза, 5 раз, 10 раз, 50 раз, 100 раз, 1000 раз или более 1000 раз.
[00127] Согласно некоторым вариантам осуществления пробиотическая композиция может дополнительно содержать еду или пищевую добавку, эффективную для стимуляции роста бактерий порядка Clostridiales, присутствующих в желудочно-кишечном тракте субъекта. Согласно некоторым вариантам осуществления пищевая добавка производится бактерией, связанной со здоровым кишечным микробиомом человека. Согласно некоторым вариантам осуществления пищевая добавка производится бактериями порядка Clostridiales. Согласно определенным вариантам осуществления пищевая добавка содержит короткоцепочечную жирную кислоту. Согласно некоторым вариантам осуществления короткоцепочечную жирную кислоту выбирают из бутирата, пропионата или их комбинации. Согласно конкретным вариантам осуществления короткоцепочечная жирная кислота представляет собой бутират.
[00128] Соответственно, определенные варианты осуществления настоящего раскрытия касаются введения бутиратных пролекарств или солей субъекту, которому вводили или в настоящее время вводят ингибитор иммунных контрольных точек. Например, бутират может представлять собой бутират натрия, бутират аргинина, этилбутириллактат, трибутирин, 4-фенилбутират, AN-9 или AN-10. Пролекарства и соли бутирата описаны в публикации международной заявки WO 96/15660 и в патенте США № 5763488, раскрытия которых включены в настоящий документ посредством ссылки. Другие перорально доступные пролекарства и соли бутирата, которые могут вводиться, включают в себя, без ограничения, изобутирамид, 1-октилбутират, ортонитробензилбутират, монобутират-3-моноацетон глюкозу, монобутират-1-моноацетон маннозу, монобутират ксилит, изобрутид, изобрутид, 4-фенилбутират и 4-фенилацетат. Каждое из этих соединений высвобождает бутират или аналог бутирата в кровоток при введении. Одна или более изоформ бутирата могут включать в себя бутилбутират, амилбутират, изобутилбутират, бензилбутират, а-метилбензилбутират, гексилбутират, гептилбутират, пеннетилбутират, метилбутират и 2-гидрокси-3-метилбутановую кислоту.
[00129] Согласно другим вариантам осуществления настоящее раскрытие относится к способам получения профиля микробиома, предусматривающему стадии: i) получения образца, полученного от субъекта (например, человека), ii) выделения одного или более видов бактерий из образца, iii) выделение одной или более нуклеиновых кислот по меньшей мере из одного вида бактерий, iv) секвенирование выделенных нуклеиновых кислот и v) сравнение секвенированных нуклеиновых кислот с эталонной последовательностью нуклеиновой кислоты. При выполнении способов, требующих генотипирования, можно использовать любой анализ генотипирования. Например, это может быть сделано путем секвенирования рибосомной субъединицы 16S или 23S или путем метагеномного секвенирования ДНК способом дробовика, связанного с метатранскриптомикой. Биологический образец может быть выбран из группы, содержащей: цельную кровь, плазму крови, мочу, слезы, сперму, слюну, слизистую оболочку щеки, интерстициальную жидкость, лимфатическую жидкость, менингеальную жидкость, амниотическую жидкость, железистую жидкость, мокроту, кал, пот, слизистую, вагинальный секрет, спинномозговую жидкость, волосы, кожу, фекальный материал, экссудат раны, гомогенат раны и раневую жидкость. Согласно определенным аспектам образец представляет собой фекальный материал или буккальный образец.
[00130] Согласно некоторым вариантам осуществления профиль микробиома идентифицируют как благоприятный для терапии иммунных контрольных точек. Благоприятный микробиологический профиль будет характеризоваться высокой относительной численностью одного или более видов бактерий из типа Firmicutes, класса Clostridia, порядка Clostridiales, семейства Ruminococcaceae, рода Ruminococcus, рода Hydrogenoanaerobacterium, рода Faecalibacterium, типа Actinobacteria, класса Coriobacteriia, порядка Coriaobacteriales, семейства Coriobacteriaceae, домена Archaea, типа Cyanobacteria, типа Euryarchaeota или семейства Christensenellaceae. Благоприятный микробиологический профиль будет характеризоваться низкой относительной численностью бактерий из рода Dialister, семейства Veillonellaceae, типа Bacteroidetes, класса Bacteroida, порядка Bacteroidales или семейства Prevotellaceae. Соответственно, благоприятный микробиологический профиль будет характеризоваться более высокой относительной численностью одного или более видов бактерий из типа Firmicutes, класса Clostridia, порядка Clostridiales, семейства Ruminococcaceae, рода Ruminococcus, рода Hydrogenoanaerobacterium, типа Actinobacteria, класса Coriobacteria, порядка Coriaobacteriales, семейства Coriobacteriaceae, домена Archaea, типа Cyanobacteria, типа Euryarchaeota или семейства Christensenellaceae, и будет характеризоваться сниженной численностью одного или более видов бактерий из рода Dialister, семейства Veillonellaceae, типа Bacteroidetes, класса Bacteroida, порядка Bacteroidales и/или семейства Prevotellaceae.
III. Блокада иммунных контрольных точек
[00131] Настоящее раскрытие представляет способы повышения эффективности блокады иммунных контрольных точек путем модуляции микробиома субъекта, например, путем введения короткоцепочечной жирной кислоты, такой как бутират, и/или композиции, содержащей одну или более популяций бактерий, производящих короткоцепочечные жирные кислоты, такие как бактерии, производящие бутират. Иммунные контрольные точки либо увеличивают сигнал (например, костимулирующие молекулы), либо понижают сигнал. Ингибирующей иммунные контрольные точки молекулы, на которые может быть нацелена блокада иммунных контрольных точек, включают в себя аденозиновый рецептор A2A (A2AR), B7-H3 (также известный как CD276), аттенюатор B- и T-лимфоцитов (BTLA), связанный с T-лимфоцитами цитотоксический белок 4 (CTLA-4, также известный как CD152), индоламин-2,3-диоксигеназу (IDO), иммуноглобулин клеток-киллеров (KIR), ген активации 3 лимфоцитов (LAG3), белок запрограммированной клеточной смерти 1 (PD-1), домен иммуноглобулинов и муциновый домен 3 Т-клеток (TIM-3) и V-домен Ig-супрессора активации Т-клеток (VISTA). В частности, ингибиторы иммунных контрольных точек нацеливают на ось PD-1 и/или CTLA-4.
[00132] Ингибиторы иммунных контрольных точек могут представлять собой лекарственные средства, такие как небольшие молекулы, рекомбинантные формы лиганда или рецепторов, или антитела, такие как антитела человека (например, Международная патентная публикация № WO2015016718; Pardoll, Nat Rev Cancer, 12(4): 252-64, 2012; оба включены в настоящий документ посредством ссылки). Могут быть использованы известные ингибиторы белков иммунных контрольных точек или их аналогов, в частности, могут использоваться химерные, гуманизированные или человеческие формы антител. Как известно специалисту в настоящей области техники, для определенных антител, указанных в настоящем раскрытии, могут использоваться альтернативные и/или эквивалентные названия. Такие альтернативные и/или эквивалентные названия являются взаимозаменяемыми в контексте настоящего изобретения. Например, известно, что ламбролизумаб также известен под альтернативными и эквивалентными названиями МК-3475 и пембролизумаб.
[00133] Предполагается, что может быть использован любой из ингибиторов иммунных контрольных точек, которые известны в настоящей области техники как стимулирующие иммунные ответы. Это включает в себя ингибиторы, которые прямо или опосредованно стимулируют или усиливают антиген-специфические Т-лимфоциты. Эти ингибиторы иммунных контрольных точек включают в себя, без ограничения, средства, нацеленные на белки иммунных контрольных точек и пути, включающие в себя PD-L2, LAG3, BTLA, B7H4 и TIM3. Например, известные в настоящей области техники ингибиторы LAG3 включают в себя растворимый LAG3 (IMP321 или LAG3-Ig, раскрытый в WO2009044273), а также мышиные или гуманизированные антитела, блокирующие человеческий LAG3 (например, IMP701, раскрытый в WO2008132601), или полностью человеческие антитела, блокирующие человеческий LAG3 (такие как раскрытые в ЕР 2320940). Другой пример представлен применением блокирующих средств в отношении BTLA, включая в себя, без ограничения, антитела, блокирующие взаимодействие BTLA человека с его лигандом (таким как 4C7, раскрытый в WO2011014438). Еще один пример представлен применением средств, нейтрализующих B7H4, включая в себя без ограничения антитела к человеческому B7H4 (раскрытые в WO 2013025779 и в WO2013067492) или растворимые рекомбинантные формы B7H4 (такие как раскрытые в US20120177645). Еще один пример представлен средствами, нейтрализующими B7-H3, включая в себя, без ограничения, антитела, нейтрализующие B7-H3 человека (например, MGA271, раскрытый как BRCA84D, и производные в US 20120294796). Еще один пример представлен средствами, нацеленными на TIM3, включая в себя, без ограничения, антитела, нацеленные на человеческий TIM3 (например, как описано в WO 2013006490 A2, или к TIM3 человека, блокирующее антитело F38-2E2, раскрытое Jones et al., J Exp Med. 2008; 205(12):2763-79).
A. Антагонисты оси PD-1
[00134] Дисфункция или толерантность Т-клеток происходит одновременно с индуцированной и устойчивой экспрессией ингибирующего рецептора, полипептида запрограммированной клеточной смерти 1 (PD-1). Таким образом, терапевтический таргетинг PD-1 и другие молекулы, которые передают сигналы посредством взаимодействий с PD-1, такие как лиганд 1 белка запрограммированной клеточной смерти (PD-L1) и лиганд 2 белка запрограммированной клеточной смерти (PD-L2), представлен в настоящем документе. PD-L1 сверхэкспрессируется при многих раковых заболеваниях и часто ассоциируется с плохим прогнозом (Okazaki T et al., Intern. Immun. 2007 19(7):813). Таким образом, в настоящем изобретении представлены улучшенные способы лечения рака путем ингибирования взаимодействия PD-L1/PD-1 в сочетании с модуляцией микробиома.
[00135] Например, антагонисты связывания оси PD-1 включают в себя антагонист связывания PD-1, антагонист связывания PDL1 и антагонист связывания PDL2. Альтернативные названия для «PD-1» включают в себя CD279 и SLEB2. Альтернативные названия для «PDL1» включают в себя B7-H1, B7-4, CD274 и B7-H. Альтернативные названия для «PDL2» включают в себя B7-DC, Btdc и CD273. Согласно некоторым вариантам осуществления PD-1, PDL1 и PDL2 представляют собой человеческие PD-1, PDL1 и PDL2.
[00136] Согласно некоторым вариантам осуществления антагонист связывания PD-1 представляет собой молекулу, которая ингибирует связывание PD-1 с его партнерами по связыванию лиганда. Согласно конкретному аспекту партнерами по связыванию лиганда PD-1 являются PDL1 и/или PDL2. Согласно другому варианту осуществления антагонист связывания PDL1 представляет собой молекулу, которая ингибирует связывание PDL1 с его партнерами по связыванию. Согласно конкретному аспекту партнерами по связыванию PDL1 являются PD-1 и/или B7-1. Согласно другому варианту осуществления антагонист связывания PDL2 представляет собой молекулу, которая ингибирует связывание PDL2 с его партнерами по связыванию. Согласно конкретному аспекту партнером по связыванию PDL2 является PD-1. Антагонист может представлять собой антитело, его антигенсвязывающий фрагмент, иммуноадгезин, слитый белок или олигопептид. Иллюстративные антитела описаны в патентах США № US8735553, US8354509 и US8008449, все они включены в настоящий документ посредством ссылки. Другие антагонисты оси PD-1 для применения в способах, представленных в настоящем документе, известны в настоящей области техники, такие как описанные в заявке на патент США № US20140294898, US2014022021 и US20110008369, все из которых включены в настоящий документ посредством ссылки.
[00137] Согласно некоторым вариантам осуществления антагонист связывания PD-1 представляет собой антитело к PD-1 (например, человеческое антитело, гуманизированное антитело или химерное антитело). Согласно некоторым вариантам осуществления антитело к PD-1 выбирают из группы, состоящей из ниволумаба, пембролизумаба и CT-011. Согласно некоторым вариантам осуществления антагонист связывания PD-1 представляет собой иммуноадгезин (например, иммуноадгезин, содержащий внеклеточную или PD-1-связывающую часть PDL1 или PDL2, слитую с константной областью (например, область Fc последовательности иммуноглобулина)). Согласно некоторым вариантам осуществления антагонистом связывания PD-1 является AMP-224. Ниволумаб, также известный как MDX-1106-04, MDX-1106, ONO-4538, BMS-936558 и OPDIVO®, представляет собой антитело к PD-1, описанное в WO2006/121168. Пембролизумаб, также известный как MK-3475, Merck 3475, ламбролизумаб, KEYTRUDA® и SCH-900475, представляет собой антитело к PD-1, описанное в WO2009/114335. CT-011, также известный как hBAT или hBAT-1, представляет собой антитело к PD-1, описанное в WO2009/101611. AMP-224, также известный как B7-DCIg, является растворимым рецептором слияния PDL2-Fc, описанным в WO2010/027827 и WO2011/066342. Дополнительные антагонисты связывания PD-1 включают в себя пидилизумаб, также известный как CT-011, MEDI0680, также известный как AMP-514, и REGN2810.
[00138] Согласно некоторым вариантам осуществления ингибитор иммунных контрольных точек представляет собой антагонист PD-L1, такой как дурвалумаб, также известный как MEDI4736, атезолизумаб, также известный как MPDL3280A, или авелумаб, также известный как MSB00010118C. Согласно определенным аспектам ингибитор иммунных контрольных точек представляет собой антагонист PD-L2, такой как rHIgM12B7. Согласно некоторым аспектам ингибитор иммунных контрольных точек представляет собой антагонист LAG-3, такой как, без ограничения, IMP321 и BMS-986016. Ингибитор иммунных контрольных точек может представлять собой антагонист рецептора аденозина A2a (A2aR), такой как PBF-509.
[00139] Согласно некоторым вариантам осуществления антитело, описанное в настоящем документе (такое как антитело к PD-1, антитело к PDL1 или антитело к PDL2), дополнительно содержит константную область человека или мыши. Согласно еще одному аспекту константную область человека выбирают из группы, состоящей из IgG1, IgG2, IgG2, IgG3 и IgG4. Согласно еще одному конкретному аспекту константная область человека представляет собой IgG1. Согласно еще одному дополнительному аспекту константную область мыши выбирают из группы, состоящей из IgG1, IgG2A, IgG2B и IgG3. Согласно еще одному конкретному аспекту антитело обладает сниженной или минимальной эффекторной функцией. Согласно еще одному конкретному аспекту минимальная эффекторная функция представляет собой результат производства в прокариотических клетках. Согласно еще одному конкретному аспекту минимальная эффекторная функция представляет собой результат «эффекторной Fc-мутации» или агликозилирования.
[00140] Соответственно, используемое в настоящем документе антитело может быть агликозилированным. Гликозилирование антител, как правило, является либо N-связанным, либо O-связанным. N-связанное относится к присоединению углеводной части к боковой цепи остатка аспарагина. Трипептидные последовательности аспарагин-Х-серин и аспарагин-Х-треонин, где Х представляет собой любую аминокислоту, кроме пролина, представляют собой последовательности распознавания для ферментативного присоединения углеводного фрагмента к боковой цепи аспарагина. Таким образом, присутствие любой из этих трипептидных последовательностей в полипептиде создает потенциальный сайт гликозилирования. О-связанное гликозилирование относится к присоединению одного из сахаров N-ацетилгалактозамина, галактозы или ксилозы к гидроксиаминокислоте, чаще всего серину или треонину, хотя также можно использовать 5-гидроксипролин или 5-гидроксилизин. Удаление сайтов гликозилирования с образованием антитела удобно осуществлять путем изменения аминокислотной последовательности таким образом, чтобы удалить одну из описанных выше трипептидных последовательностей (для N-связанных сайтов гликозилирования). Изменение может быть осуществлено путем замены остатка аспарагина, серина или треонина в сайте гликозилирования другим аминокислотным остатком (например, глицин, аланин или консервативная замена).
[00141] Антитело или его антигенсвязывающий фрагмент могут быть получены с использованием способов, известных в настоящей области техники, например, посредством процесса, предусматривающего культивирование клетки-хозяина, содержащей нуклеиновую кислоту, кодирующую любое из ранее описанных антител к PDL1, к PD-1 или к PDL2 или антигенсвязывающий фрагмент в форме, подходящей для экспрессии, в условиях, подходящих для производства такого антитела или фрагмента, и выделения антитела или фрагмента.
B. CTLA-4
[00142] Другой иммунной контрольной точкой, на которую можно ориентироваться в способах, представленных в настоящем документе, является ассоциированный с Т-лимфоцитами цитотоксический белок 4 (CTLA-4), также известный как CD152. Полная последовательность кДНК CTLA-4 человека характеризуется регистрационным номером Genbank L15006. CTLA-4 обнаружен на поверхности Т-клеток и действует как выключатель, когда он связан с CD80 или CD86 на поверхности антигенпрезентирующих клеток. CTLA4 является представителем суперсемейства иммуноглобулинов, который экспрессируется на поверхности хелперных Т-клеток и передает ингибирующий сигнал Т-клеткам. CTLA4 похож на костимулирующий белок Т-клеток, CD28, и обе молекулы связываются с CD80 и CD86, также называемыми B7-1 и B7-2, соответственно, на антигенпрезентирующих клетках. CTLA4 передает ингибирующий сигнал Т-клеткам, тогда как CD28 передает стимулирующий сигнал. Внутриклеточный CTLA4 также обнаружен в регуляторных Т-клетках и может быть важен для их функции. Активация Т-клеток через рецептор Т-клеток и CD28 приводит к повышенной экспрессии CTLA-4, ингибиторного рецептора для молекул B7.
[00143] Согласно некоторым вариантам осуществления ингибитор иммунных контрольных точек представляет собой антитело к CTLA-4 (например, человеческое антитело, гуманизированное антитело или химерное антитело), его антигенсвязывающий фрагмент, иммуноадгезин, слитый белок или олигопептид.
[00144] Антитела к CTLA-4 человека (или полученные из них домены VH и/или VL), подходящие для применения в настоящих способах, могут быть получены с использованием способов, хорошо известных в настоящей области техники. В качестве альтернативы могут применяться антитела к CTLA-4, признанные в настоящей области техники. Например, антитела к CTLA-4 раскрыты в: US 8119129, WO 01/14424, WO 98/42752; WO 00/37504 (CP 675 206, также известный как тремелимумаб; ранее тицилимумаб), патент США № 6207156; Hurwitz et al., 1998; могут быть использованы в способах, раскрытых в настоящем документе. Идеи каждой из вышеуказанных публикаций тем самым включены посредством ссылки. Антитела, которые конкурируют с любым из этих известных в настоящей области техники антител за связывание с CTLA-4, также могут быть использованы. Например, гуманизированное антитело к CTLA-4 описано в международной заявке на патент № WO2001014424, WO2000037504 и в патенте США № US8017114; все включено в настоящий документ посредством ссылки.
[00145] Иллюстративным антителом к CTLA-4 является ипилимумаб (также известный как 10D1, MDX-010, MDX-101 и Yervoy®) или его антигенсвязывающие фрагменты и их варианты (смотрите, например, WO0 1/14424). Согласно другим вариантам осуществления антитело содержит CDR или VR тяжелой и легкой цепи ипилимумаба. Соответственно, согласно одному варианту осуществления антитело содержит домены CDR1, CDR2 и CDR3 области VH ипилимумаба и домены CDR1, CDR2 и CDR3 области VL ипилимумаба. Согласно другому варианту осуществления антитело конкурирует за связывание и/или связывается с тем же эпитопом на CTLA-4, что и вышеуказанные антитела. Согласно другому варианту осуществления антитело характеризуется идентичностью аминокислотной последовательности вариабельной области, составляющей по меньшей мере приблизительно 90% по отношению к вышеуказанным антителам (например, идентичностью вариабельной области по меньшей мере приблизительно 90%, 95% или 99% по отношению к ипилимумабу).
[00146] Другие молекулы для модуляции CTLA-4 включают в себя растворимые лиганды и рецепторы CTLA-4, такие как описаны в патентах США № US5844905, US5885796 и в международных заявках на патент WO1995001994 и WO1998042752; все включены в настоящий документ посредством ссылки, и иммуноадгезины, такие как описанные в патенте США № US8329867, включены в настоящий документ посредством ссылки.
C. Иммуноглобулиноподобный рецептор клеток-киллеров (KIR)
[00147] Другим ингибитором иммунных контрольных точек для применения в настоящем изобретении является антитело к KIR. Антитела к KIR человека (или полученные из них домены VH/VL), подходящие для применения в настоящих способах, могут быть получены с использованием способов, хорошо известных в настоящей области техники.
[00148] В качестве альтернативы можно применять известные антитела к KIR. Антитело к KIR может быть перекрестно-реактивным с множественными ингибирующими рецепторами KIR и усиливать цитотоксичность NK-клеток, несущих один или более из этих рецепторов. Например, антитело к KIR может связываться с каждым из KIR2D2DL1, KIR2DL2 и KIR2DL3 и усиливать активность NK-клеток путем снижения, нейтрализации и/или реверсирования ингибирования цитотоксичности NK-клеток, опосредованного любым или всеми из этих KIR. Согласно некоторым аспектам антитело к KIR не связывается с KIR2DS4 и/или KIR2DS3. Например, могут быть использованы моноклональные антитела 1-7F9 (также известные как IPH2101), 14F1, 1-6F1 и 1-6F5, описанные в WO 2006/003179, идеи которых включены в настоящий документ посредством ссылки. Антитела, которые конкурируют с любым из этих известных в настоящей области техники антител за связывание с KIR, также могут быть использованы. Дополнительные признанные в настоящей области техники антитела к KIR, которые можно использовать, включают в себя, например, те, которые раскрыты в WO 2005/003168, WO 2005/009465, WO 2006/072625, WO 2006/072626, WO 2007/042573, WO 2008/084106, WO 2010/065939, WO 2012/071411 и WO 2012/160448.
[00149] Иллюстративным антителом к KIR является лирилумаб (также называемый BMS-986015 или IPH2102). Согласно другим вариантам осуществления антитело к KIR содержит определяющие комплементарность области тяжелой и легкой цепей (CDR), или вариабельные области (VR) лирилумаба. Соответственно, согласно одному варианту осуществления антитело содержит домены CDR1, CDR2 и CDR3 вариабельной области (VH) тяжелой цепи лирилумаба и домены CDR1, CDR2 и CDR3 вариабельной области (VL) легкой цепи лирилумаба. Согласно другому варианту осуществления антитело характеризуется идентичностью аминокислотной последовательности вариабельной области, составляющей по меньшей мере приблизительно 90% по отношению к лирилумабу.
IV. Способы лечения
[00150] В настоящем документе представлены способы лечения или задержки прогрессирования рака у индивидуума, предусматривающие введение индивидууму эффективного или достаточного количества короткоцепочечной жирной кислоты, такой как бутират, и/или популяции производящих короткоцепочечные жирные кислоты бактерий, такие как производящие бутират бактерии, субъекту, который получал или в настоящее время получает терапию иммунных контрольных точек. В настоящем документе также представлены способы выбора субъектов, которые будут благоприятно реагировать на терапию иммунных контрольных точек, путем оценки микробиологического профиля субъекта и введения ингибитора иммунных контрольных точек субъекту, который идентифицирован как имеющий благоприятный микробиологический профиль.
[00151] Согласно некоторым вариантам осуществления лечение приводит к устойчивому ответу у индивидуума после прекращения лечения. Способы, описанные в настоящем документе, могут найти применение при лечении состояний, когда желательна повышенная иммуногенность, таких как повышение иммуногенности опухоли для лечения рака. В настоящем документе также представлены способы усиления иммунной функции, такие как у индивидуума, характеризующегося наличием рака, предусматривающие введение индивидууму эффективного количества ингибитора иммунных контрольных точек (например, антагониста, связывающего ось PD-1 и/или антитела CTLA-4) и короткоцепочечной жирной кислоты, такой как бутират, и/или популяции производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии. Согласно некоторым вариантам осуществления индивидуумом является человек.
[00152] Примеры видов рака, предполагаемых для лечения, включают в себя рак легких, рак головы и шеи, рак молочной железы, рак поджелудочной железы, рак предстательной железы, рак почки, рак кости, рак яичка, рак шейки матки, рак желудочно-кишечного тракта, лимфомы, предопухолевые поражения в легких, рак толстой кишки, меланому, метастатическую меланому, базально-клеточный рак кожи, плоскоклеточный рак кожи, возвышающуюся дерматофибросаркому, карциному из клеток Меркеля, саркому Капоши, кератоакантому, опухоли веретенообразных клеток, карциномы сальных желез, микрокистозную карциному придатков, болезнь Педжета молочной железы, атипичную фиброзную опухоль, лейомиосаркому и ангиосаркому, злокачественное лентиго, меланому типа злокачественного лентиго, поверхностную распространяющуюся меланому, нодулярную меланому, акральную лентигиозную меланому или десмопластическую меланому и рак мочевого пузыря.
[00153] Согласно некоторым вариантам осуществления индивидуум характеризуется наличием рака, который является устойчивым (было продемонстрировано, что он устойчив) к одной или более противораковым терапиям. Согласно некоторым вариантам осуществления устойчивость к противораковой терапии включает в себя рецидив рака или рефрактерный рак. Рецидив может относиться к повторному появлению рака в первоначальном или новом сайте после лечения. Согласно некоторым вариантам осуществления устойчивость к противораковой терапии включает в себя прогрессирование рака во время лечения противораковой терапией. Согласно некоторым вариантам осуществления рак находится на ранней стадии или на поздней стадии.
[00154] У индивидуума может быть рак, который экспрессирует (как было показано, например, в диагностическом тесте) биомаркер PD-L1. Согласно некоторым вариантам осуществления рак пациента экспрессирует низкий биомаркер PD-L1. Согласно некоторым вариантам осуществления рак пациента экспрессирует высокий биомаркер PD-L1. Биомаркер PD-L1 можно обнаружить в образце с использованием способа, выбранного из группы, состоящей из FACS, вестерн-блоттинга, ELISA, иммунопреципитации, иммуногистохимии, иммунофлуоресценции, радиоиммуноанализа, дот-блоттинга, способов иммунодетекции, ВЭЖХ, поверхностного плазмонного резонанса, оптической спектроскопии, масс-спектрометрии, ВЭЖХ, кПЦР, ОФ-кПЦР, мультиплексной кПЦР или ОФ-КПЦР, RNA-seq, анализ микрочипов, способ SAGE, MassARRAY и FISH и их комбинации.
[00155] Согласно некоторым вариантам осуществления способов по настоящему изобретению рак характеризуется низкими уровнями инфильтрации Т-клеток. Согласно некоторым вариантам осуществления рак не имеет обнаруживаемого инфильтрата Т-клеток. Согласно некоторым вариантам осуществления рак представляет собой неиммуногенный рак (например, неиммуногенный колоректальный рак и/или рак яичников). Без ограничения какой-либо теорией, комбинированное лечение может увеличивать примирование, активацию, пролиферацию и/или инфильтрацию Т-клеток (например, CD4+ Т-клеток, CD8+ Т-клеток, Т-клеток памяти) по сравнению с предшествующим введением комбинации.
A. Введение
[00156] Терапия, представленная в настоящем документе, предусматривает введение ингибитора иммунных контрольных точек, пребиотической или пробиотической композиции, содержащей короткоцепочечную жирную кислоту, такую как бутират, и/или популяции производящих короткоцепочечную жирную кислоту бактерий, таких как производящие бутират бактерии. Терапия может проводиться любым подходящим способом, известным в настоящей области техники. Например, ингибитор иммунных контрольных точек (например, антагонист, связывающий ось PD-1 и/или антитела CTLA-4) и короткоцепочечную жирную кислоту, такую как бутират, и/или популяцию производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, можно вводить последовательно (в разное время) или одновременно (в одно и то же время). Согласно некоторым вариантам осуществления один или более ингибиторов иммунных контрольных точек находятся в отдельной композиции в качестве пробиотической терапии. Согласно некоторым вариантам осуществления ингибитор иммунных контрольных точек находится в той же композиции, что и пробиотическая композиция.
[00157] Согласно предпочтительному варианту осуществления пробиотическую бактериальную композицию составляют для перорального введения. Специалисту в настоящей области техники известно множество формул, которые могут включать живые или убитые микроорганизмы и которые могут быть представлены в виде пищевых добавок (например, пилюль, таблеток и т.п.) или в качестве функциональных пищевых продуктов, таких как напитки или ферментированные йогурты.
[00158] Один или более ингибиторов иммунных контрольных точек и короткоцепочечные жирные кислоты, такие как бутират, и/или популяции производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, могут быть введены одним и тем же путем введения или различными путями введения. Согласно некоторым вариантам осуществления ингибитор иммунных контрольных точек вводят внутривенно, внутримышечно, подкожно, местно, перорально, трансдермально, внутрибрюшинно, внутриорбитально, путем имплантации, ингаляцией, интратекально, интравентрикулярно или интраназально. Согласно некоторым вариантам осуществления короткоцепочечную жирную кислоту, такую как бутират, и/или популяцию производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, вводят внутривенно, внутримышечно, подкожно, местно, перорально, трансдермально, внутрибрюшинно, внутриорбитально, имплантацией, ингаляцией, интратекально, интравентрикулярно или интраназально. Согласно конкретным аспектам ингибитор иммунных контрольных точек вводят внутривенно, а короткоцепочечную жирную кислоту, такую как бутират, и/или популяцию производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, вводят перорально. Эффективное количество ингибитора иммунных контрольных точек и короткоцепочечной жирной кислоты, такой как бутират, и/или популяции производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, можно вводить для профилактики или лечения заболевания. Подходящая доза ингибитора иммунных контрольных точек и/или короткоцепочечной жирной кислоты, такой как бутират, и/или популяции производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, может быть определена на основе типа подвергаемого лечению заболевания, тяжести и течения заболевания, клинического состояния индивидуума, истории болезни индивидуума и ответа на лечение, а также усмотрения лечащего врача.
[00159] Например, терапевтически эффективное или достаточное количество ингибитора иммунных контрольных точек, такого как антитело и/или короткоцепочечная жирная кислота, такая как бутират, которое вводят человеку, будет в диапазоне от приблизительно 0,01 до приблизительно 50 мг/кг массы тела пациента, в один или более приемов. Согласно некоторым вариантам осуществления используемое антитело составляет от приблизительно 0,01 до приблизительно 45 мг/кг, от приблизительно 0,01 до приблизительно 40 мг/кг, от приблизительно 0,01 до приблизительно 35 мг/кг, от приблизительно 0,01 до приблизительно 30 мг/кг, от приблизительно 0,01 до приблизительно 25 мг/кг, от приблизительно 0,01 до приблизительно 20 мг/кг, от приблизительно 0,01 до приблизительно 15 мг/кг, от приблизительно 0,01 до приблизительно 10 мг/кг, от приблизительно 0,01 до приблизительно 5 мг/кг или от приблизительно 0,01 до приблизительно 1 мг/кг, вводимых ежедневно, например. Согласно некоторым вариантам осуществления антитело вводят в дозе 15 мг/кг. Тем не менее, другие схемы дозирования могут быть применимы. Согласно одному варианту осуществления описанное в настоящем документе антитело к PDL1 вводят человеку в дозе приблизительно 100 мг, приблизительно 200 мг, приблизительно 300 мг, приблизительно 400 мг, приблизительно 500 мг, приблизительно 600 мг, приблизительно 700 мг, приблизительно 800 мг, приблизительно 900 мг, приблизительно 1000 мг, приблизительно 1100 мг, приблизительно 1200 мг, приблизительно 1300 мг или приблизительно 1400 мг в 1-й день 21-дневных циклов. Доза может быть введена в виде одной дозы или в виде нескольких доз (например, 2 или 3 дозы), таких как инфузии. Прогресс этой терапии легко контролируется обычными способами.
[00160] Например, терапевтически эффективное или достаточное количество каждой по меньшей мере из одной выделенной или очищенной популяции бактерий или каждой по меньшей мере из двух выделенных или очищенных популяций бактерий пробиотических или живых бактериальных композиций продуктов по вариантам осуществления, которое вводится человеку будет составлять по меньшей мере приблизительно 1×103 колониеобразующих единиц (КОЕ) бактерий или по меньшей мере приблизительно 1×104 (КОЕ). Согласно некоторым вариантам осуществления однократная доза будет содержать приблизительно 1×104, 1×105, 1×106, 1×107, 1×108, 1×109, 1×1010, 1×1011, 1×1012, 1×1013, 1×1014, 1×1015 или более чем 1×1015 КОЕ бактерий. Согласно конкретным вариантам осуществления бактерии представлены в форме спор или в виде спорулированных бактерий. Согласно конкретным вариантам осуществления концентрация спор каждой выделенной или очищенной популяции бактерий, например, каждого вида, подвида или штамма, составляет 1×104, 1×105, 1×106, 1×107, 1×108, 1×109, 1×1010, 1×1011, 1×1012, 1×1013, 1×1014, 1×1015 или более чем 1×1015 жизнеспособных бактериальных спор на грамм композиции или на введенную дозу.
[00161] Внутриопухолевая инъекция или инъекция в сосудистую сеть опухоли специально предусмотрена для дискретных, солидных, доступных опухолей. Местное, регионарное или системное введение также может быть уместным. Для опухолей >4 см вводимый объем будет приблизительно 4-10 мл (в частности, 10 мл), тогда как для опухолей <4 см будет использоваться объем приблизительно 1-3 мл (в частности, 3 мл). Многократные инъекции, доставляемые в виде однократной дозы, составляют от приблизительно 0,1 до приблизительно 0,5 мл объема. Например, аденовирусные частицы могут преимущественно контактировать путем введения нескольких инъекций в опухоль.
[00162] Схемы лечения также могут варьировать и часто зависят от типа опухоли, локализации опухоли, прогрессирования заболевания, а также состояния здоровья и возраста пациента. Очевидно, что определенные типы опухолей потребуют более агрессивного лечения, в то же время, некоторые пациенты не могут терпеть больше протоколов. Лечащий врач будет лучше всего подходить для принятия таких решений на основе известной эффективности и токсичности (если таковая имеется) терапевтических составов.
[00163] Согласно некоторым вариантам осуществления подвергаемая лечению опухоль может не быть, по меньшей мере на начальной стадии, способной к резекции. Лечение терапевтическими вирусными конструкциями может повысить резектабельность опухоли из-за сморщивания по краям или путем удаления определенных особенно инвазивных участков. После лечения возможна резекция. Дополнительные способы лечения после резекции будут использоваться для устранения микроскопических остаточных заболеваний в месте опухоли.
[00164] Лечение может включать в себя различные «однократные дозы». Однократная доза определяется как содержащая заданное количество терапевтической композиции. Количество, которое должно быть введено, и конкретный путь и состав, находятся в пределах компетенции определения специалистов в области медицины. Однократная доза не должна вводиться в виде однократной инъекции, но может предусматривать непрерывную инфузию в течение установленного периода времени.
B. Дополнительные противораковые терапии
[00165] Согласно некоторым вариантам осуществления ингибитор иммунных контрольных точек, композицию, содержащую короткоцепочечную жирную кислоту, такую как бутират, и/или композицию, содержащую популяцию производящих короткоцепочечные жирные кислоты бактерий, таких как производящие бутират бактерии, в настоящем документе можно вводить в комбинации по меньшей мере с одним дополнительным терапевтическим средством. Дополнительная терапия может представлять собой терапию рака, такую как лучевая терапия, хирургия, химиотерапия, генная терапия, ДНК-терапия, вирусная терапия, РНК-терапия, иммунотерапия, трансплантация костного мозга, нанотерапия, терапия моноклональными антителами или комбинация вышеперечисленного. Дополнительная терапия может быть в форме адъювантной или неоадъювантной терапии.
[00166] Согласно некоторым вариантам осуществления дополнительная терапия рака представляет собой введение низкомолекулярного ферментативного ингибитора или антиметастатического средства. Согласно некоторым вариантам осуществления дополнительная терапия представляет собой введение средств, ограничивающих побочные эффекты (например, средств, предназначенных для уменьшения возникновения и/или серьезности побочных эффектов лечения, таких как средства против тошноты и т.д.). Согласно некоторым вариантам осуществления дополнительная терапия рака представляет собой лучевую терапию. Согласно некоторым вариантам осуществления дополнительная терапия рака представляет собой хирургическое вмешательство. Согласно некоторым вариантам осуществления дополнительная терапия рака представляет собой комбинацию лучевой терапии и хирургического вмешательства. Согласно некоторым вариантам осуществления дополнительная терапия рака представляет собой гамма-облучение. Согласно некоторым вариантам осуществления дополнительная терапия рака представляет собой терапию, нацеленную на путь PBK/AKT/mTOR, ингибитор HSP90, ингибитор тубулина, ингибитор апоптоза и/или химиопрофилактическое средство. Дополнительная терапия рака может представлять собой одно или более химиотерапевтических средств, известных в настоящей области техники.
[00167] Также могут быть использованы различные комбинации. Для приведенного ниже примера популяция ингибиторов контрольных точек, бутират и/или производящая бутират бактериальная популяция представляет собой «А», а дополнительная терапия рака - «В»:
A/B/A B/A/B B/B/A A/A/B A/B/B B/A/A A/B/B/B B/A/B/B
B/B/B/A B/B/A/B A/A/B/B A/B/A/B A/B/B/A B/B/A/A
B/A/B/A B/A/A/B A/A/A/B B/A/A/A A/B/A/A A/A/B/A
[00168] Введение любого соединения или терапии по настоящим вариантам осуществления пациенту будет следовать общим протоколам введения таких соединений, принимая во внимание токсичность средств, если таковые имеются. Следовательно, согласно некоторым вариантам осуществления существует стадия мониторинга токсичности, которая относится к комбинированной терапии.
1. Химиотерапия
[00169] В соответствии с настоящими вариантами осуществления может использоваться широкий спектр химиотерапевтических средств. Термин «химиотерапия» относится к применению лекарств для лечения рака. «Химиотерапевтическое средство» используется для обозначения соединения или композиции, которые вводят при лечении рака. Эти средства или лекарственные средства классифицируются по способу их активности в клетке, например, влияют ли они на клеточный цикл и на какой стадии. Альтернативно, средство может быть охарактеризовано на основании его способности непосредственно сшивать ДНК, интеркалировать в ДНК или индуцировать хромосомные и митотические аберрации, влияя на синтез нуклеиновой кислоты.
[00170] Примеры химиотерапевтических средств включают в себя такие алкилирующие средства, как тиотепа и циклосфосфамид; такие алкилсульфонаты, как бусульфан, импросульфан и пипосульфан; такие азиридины, как бензодопа, карбоквон, метуредопа и уредопа; этиленимины и метиламеламины, включая в себя алтретамин, триэтиленмеламин, триэтиленфосфорамид, триэтилентиофосфорамид и триметилоломеламин; ацетогенины (особенно булатацин и булатацинон); камптотецин (включая в себя синтетический аналог топотекан); бриостатин; каллистатин; CC-1065 (включая в себя его синтетические аналоги адозелезин, карзелезин и бизелезин); криптофицины (особенно криптофицин 1 и криптофицин 8); доластатин; дуокармицин (включая в себя синтетические аналоги, KW-2189 и CB1-TM1); элейтеробин; панкратистатин; саркодиктин; спонгистатин; такие азотистые иприты, как хлорамбуцил, хлорнафазин, холофосфамид, эстрамустин, ифосфамид, мехлоретамин, гидрохлорид окиси мехлорэтамина, мелфалан, новембихин, фенестерин, преднимустин, трофосфамид и урациловый иприт; такие нитрозомочевины, как кармустин, хлорозотоцин, фотемустин, ломустин, нимустин и ранимнустин; такие антибиотики, как энедииновые антибиотики (например, калихеамицин, особенно калихеамицин гамма lI и калихеамицин омега I1); динемицин, включая в себя динемицин А; такие бисфосфонаты, как клодронат; эсперамицин; а также неокарциностатиновый хромофор и родственные хромопротеиновые энедииновые антибактериальные хромофоры, аклациномизины, актиномицин, аутрамицин, азасерин, блеомицины, кактиномицин, карабицин, карминомицин, карзинофилин, хромомицины, дактиномицин, даунорубицин, деторубицин, 6-диазо-5-оксо-L-норлейцин, доксорубицин (включая в себя морфолинодоксорубицин, цианоморфолинодоксорубицин, 2-пирролинодоксорубициндоксорубицин и дезоксидоксорубицин), эпирубицин, эзорубицин, идарубицин, марцелломицин, такие митомицины, как митомицин C, микофеноловая кислота, ногаларницин, оливомицины, пепломицин, потфиромицин, пуромицин, квеламицин, родорубицин, стрептонигрин, стрептозоцин, туберцидин, убенимекс, зиностатин и зорубицин; такие антиметаболиты, как метотрексат и 5-фторурацил (5-FU); такие аналоги фолиевой кислоты, как деноптерин, птероптерин и триметрексат; такие аналоги пурина, как флударабин, 6-меркаптопурин, тиамиприн и тиогуанин; такие аналоги пиримидина, как анцитабин, азацитидин, 6-азауридин, кармофур, цитарабин, дидезоксиуридин, доксифлуридин, эноцитабин и флоксуридин; такие андрогены, как калустерон, дромостанолон пропионат, эпитиостанол, мепитиостан и тестолактон; такие антиадреналовые средства, как митотан и трилостан; такой пополнитель фолиевой кислоты, как фролиновая кислота; ацеглатон; альдофосфамидный гликозид; аминолевулиновая кислота; энилурацил; амсакрин; бестрабуцил; бизантрен; эдатраксат; дефофамин; демеколцин; диазиквон; элформитин; эллиптиний ацетат; эпотилон; этоглюцид; нитрат галлия; гидроксимочевина; лентинан; лонидаинин; такие майтанзиноиды, как майтанзин и ансамитоцины; митогуазон; митоксантрон; мопиданмол; нитраэрин; пентостатин; фенамет; пирарубицин; лозоксантрон; подофиллиновая кислота; 2-этилгидразид; прокарбазин; ПСК полисахаридный комплекс; разоксан; ризоксин; сизофиран; спирогерманий; тенуазоновая кислота; триазиквон; 2,2',2”-трихлортриэтиламин; трихотецены (особенно токсин Т-2, верракурин А, риридин А и ангидин); уретан; виндезин; дакарбазин; манномустин; митобронитол; митолактол; пипоброман; гацитозин; арабинозид («Ara-C»); циклофосфамид; таксоиды, например, паклитаксел и доцетаксел гемцитабин; 6-тиогуанин; меркаптопурин; такие координационные комплексы платины, как цисплатин, оксалиплатин и карбоплатин; винбластин; платина; этопозид (VP-16); ифосфамид; митоксантрон; винкристин; винорелбин; новантрон; тенипозид; эдатрексат; дауномицин; аминоптерин; кселода; ибандронат; иринотекан (например, CPT-11); ингибитор топоизомеразы RFS 2000; дифторметиллорнитин (ДМФО); такие ретиноиды, как ретиноевая кислота; капецитабин; карбоплатин, прокарбазин, пликомицин, гемцитабиен, навельбин, ингибиторы фарнезил-протеин-трансферазы, трансплатина и фармацевтически приемлемые соли, кислоты или производные любого из вышеперечисленного.
2. Лучевая терапия
[00171] Другие факторы, которые вызывают повреждение ДНК и широко используются, включают в себя так называемые γ-лучи, рентгеновские лучи и/или направленную доставку радиоизотопов в опухолевые клетки. Также рассматриваются другие формы повреждающих ДНК факторов, такие как микроволны, облучение протонным пучком (патенты США 5760395 и 4870287) и УФ-облучение. Скорее всего, все эти факторы влияют на широкий спектр повреждений на ДНК, предшественников ДНК, репликации и репарации ДНК, а также на сборку и поддержание хромосом. Диапазон доз для рентгеновских лучей варьирует от суточных доз 50-200 рентген в течение продолжительных периодов времени (от 3 до 4 недель) до разовых доз 2000-6000 рентген. Диапазоны доз для радиоизотопов варьируют в широких пределах и зависят от периода полураспада изотопа, силы и типа испускаемого излучения и поглощения опухолевыми клетками.
3. Иммунотерапия
[00172] Специалисту в настоящей области техники будет понятно, что иммунотерапию можно использовать в комбинации или в сочетании со способами, описанными в настоящем документе. В контексте лечения рака иммунотерапевтические средства, как правило, основаны на применении иммунных эффекторных клеток и молекул для нацеливания и уничтожения раковых клеток. Ритуксимаб (RITUXAN®) является примером иммунотерапии. Иммунный эффектор может представлять собой, например, антитело, специфическое для маркера на поверхности опухолевой клетки. Одно антитело может служить эффектором терапии или может привлекать другие клетки для фактического уничтожения клеток. Антитело также может быть конъюгировано с лекарственным средством или токсином (химиотерапевтическое средство, радионуклид, цепь рицина А, токсин холеры, токсин коклюша и т.д.) и служить в качестве нацеливающего средства. Альтернативно, эффектор может представлять собой лимфоцит, несущий поверхностную молекулу, которая взаимодействует, прямо или опосредованно, с мишенью опухолевых клеток. Различные эффекторные клетки включают в себя цитотоксические Т-клетки и NK-клетки.
[00173] Конъюгаты антитело-лекарственное средство появились в качестве прорывного подхода к разработке противоопухолевых терапевтических средств. Конъюгаты антитело-лекарственное средство (ADC) содержат моноклональные антитела (MAb), которые ковалентно связаны с лекарственными средствами, уничтожающими клетки. Этот подход сочетает высокую специфичность MAb против их антигенных мишеней с высокоэффективными цитотоксическими лекарственными средствами, в результате чего получают «вооруженные» MAb, которые доставляют молекулярный груз (лекарственное средство) в опухолевые клетки с повышенным уровнем антигена. Целевая доставка лекарственного средства также минимизирует его воздействие на нормальные ткани, что приводит к снижению токсичности и улучшению терапевтического индекса. Одобрение FDA получили два лекарственных средства ADC, ADCETRIS® (брентуксимаб ведотин) в 2011 г. и KADCYLA® (трастузумаб эмтанзин или T-DM1) в 2013 г. В настоящее время на различных стадиях клинических испытаний для лечения рака имеется более 30 кандидатов в лекарственные средства ADC. По мере того, как разработка антител и оптимизация линкера-молекулярного груза становятся все более и более зрелыми, обнаружение и разработка новых ADC все больше зависят от идентификации и проверки новых мишеней, подходящих для этого подхода, и создания целевых MAb. Двумя критериями для мишеней ADC являются повышенный/высокий уровень экспрессии в опухолевых клетках и надежная интернализация.
[00174] Согласно одному аспекту иммунотерапии опухолевая клетка должна нести некоторый маркер, который поддается нацеливанию, т.е. не присутствует на большинстве других клеток. Существует множество опухолевых маркеров, и любой из них может быть подходящим для нацеливания в контексте настоящих вариантов осуществления. Общие опухолевые маркеры включают в себя CD20, карциноэмбриональный антиген, тирозиназу (p97), gp68, TAG-72, HMFG, сиалированный антиген Lewis, MucA, MucB, PLAP, рецептор ламинина, erb B и p155. Альтернативным аспектом иммунотерапии является сочетание противоопухолевых эффектов с иммуностимулирующими эффектами. Иммуностимулирующие молекулы также существуют, включая в себя такие цитокины, как IL-2, IL-4, IL-12, GM-CSF, гамма-IFN, такие хемокины, как MIP-1, MCP-1, IL-8, и такие факторы роста, как лиганд FLT3.
4. Хирургическое лечение
[00175] Приблизительно 60% больных раком будут подвергаться хирургическому вмешательству определенного типа, которое включает в себя профилактическую, диагностическую хирургию или хирургическое стадирование, лечебную и паллиативную хирургию. Лечебная хирургия включает в себя резекцию, при которой вся или часть злокачественной ткани физически удаляется, иссекается и/или разрушается и может использоваться в сочетании с другими способами лечения, такими как лечение по настоящему изобретению, химиотерапия, лучевая терапия, гормональная терапия, генная терапия, иммунотерапия и/или альтернативные способы лечения. Резекция опухоли относится к физическому удалению по меньшей мере части опухоли. В дополнение к резекции опухоли хирургическое лечение включает в себя лазерную хирургию, криохирургию, электрохирургию и микроскопически контролируемую хирургию (операция Мохса).
[00176] При удалении части или всех раковых клеток, тканей или опухоли в теле может образовываться полость. Лечение может быть достигнуто перфузией, прямой инъекцией или местным применением области с дополнительной противораковой терапией. Такое лечение может повторяться, например, каждые 1, 2, 3, 4, 5, 6 или 7 дней или каждые 1, 2, 3, 4 и 5 недель, или каждые 1, 2, 3, 4, 5 6, 7, 8, 9, 10, 11 или 12 месяцев. Эти способы лечения могут также варьировать.
5. Другие средства
[00177] Предполагается, что другие средства могут применяться в сочетании с некоторыми аспектами настоящих вариантов осуществления для улучшения терапевтической эффективности лечения. Эти дополнительные средства включают в себя средства, которые влияют на активацию рецепторов клеточной поверхности и GAP-соединений, цитостатические и дифференцирующие средства, ингибиторы клеточной адгезии, средства, которые повышают чувствительность гиперпролиферативных клеток к апоптическим индукторам или другие биологические средства. Увеличение межклеточной передачи сигналов за счет увеличения числа GAP-соединений увеличит антигиперпролиферативные эффекты на соседнюю популяцию гиперпролиферативных клеток. Согласно другим вариантам осуществления цитостатические или дифференцирующие средства могут применяться в сочетании с некоторыми аспектами настоящих вариантов осуществления для улучшения антигиперпролиферативной эффективности лечения. Предполагается, что ингибиторы клеточной адгезии улучшают эффективность настоящих вариантов осуществления. Примерами ингибиторов клеточной адгезии являются ингибиторы фокальной адгезионной киназы (FAK) и ловастатин. Кроме того, предполагается, что другие средства, которые повышают чувствительность гиперпролиферативной клетки к апоптозу, такие как антитело c225, можно применять в сочетании с некоторыми аспектами настоящих вариантов осуществления для улучшения эффективности лечения.
V. Примеры
[00178] Следующие примеры включены для демонстрации предпочтительных вариантов осуществления настоящего изобретения. Специалистам в настоящей области техники должно быть понятно, что способы, раскрытые в следующих примерах, представляют собой способы, открытые автором настоящего изобретения для эффективного функционирования при практическом применении настоящего изобретения, и, таким образом, могут рассматриваться как составляющие предпочтительные способы его применения. Однако специалистам в настоящей области техники в свете настоящего раскрытия должно быть понятно, что согласно конкретным вариантам осуществления, которые раскрыты, могут быть сделаны многие изменения и все же получен подобный или аналогичный результат без отклонения от сущности и объема настоящего изобретения.
Пример 1 - Характеристика микробиома пациентов с меланомой
[00179] Чтобы лучше понять роль микробиома в ответе на блокаду иммунных контрольных точек у больных раком, образцы микробиома проспективно отбирали у пациентов с метастатической меланомой, переходящих на лечение с помощью блокады PD-1 (n=112 пациентов). Образцы орального (буккального) и кишечного (фекального) микробиома собирали в начале лечения, а биоптаты опухолей и образцы крови отбирали, когда это было возможно, для оценки геномных изменений, а также плотности и фенотипа подгрупп инфильтрирующих и циркулирующих в опухоли иммунных клеток (фиг. 1А). Таксономическое профилирование с помощью секвенирования гена 16S рРНК выполняли на всех доступных оральных и кишечных образцах, с метагеномным секвенированием всего генома (WGS) на подгруппе. Пациентов классифицировали как отвечающие на лечение (R) или не отвечающие на лечение (NR) на основе анализа изображений по критериям RECIST 1.1 (Schwartz et al., 2016). Следует отметить, что пациенты в группах R по сравнению с NR были относительно схожи по возрасту, полу, первичному типу, предшествующей терапии, параллельной терапии и LDH сыворотки (таблица 3). Частота специфических драйверных мутаций меланомы и общая мутационная нагрузка также были одинаковыми между группами (фиг. 5) (17).
[00180] Таблица 3: Таблица характеристик пациентов по типу образца микробиома.
*p-значения рассчитаны по критерию суммы рангов Уилкоксона (возраст), критерию хи-квадрата (пол, предыдущая контрольная точка) и точному критерию Фишера (все остальные)
**другие включают в себя акральные, слизистые, неизвестные первичные типы
***комбинации включают в себя: абраксан, урелуманб, крем Aldera
****1 пропущенное значение от 1 R, который не имел исходного прочтения LDH
[00181] Ландшафт оральной и фекальной микробиоты впервые оценивали у пациентов с метастатической меланомой с помощью секвенирования 16S (области V4), отмечая, что оба сообщества были относительно разнообразными, с высокой численностью Lactobacillales в оральном микробиоме и Bacteroidales в фекальном микробиоме (фиг. 1В). Анализ двудольных сетей (Muegge et al., 2011) продемонстрировал четкое разделение структуры сообщества между оральным и фекальным микробиомами с точки зрения как сопоставленных, так и совокупных образцов (фиг. 1C и 6), предполагая, что эти сообщества различны.
[00182] Потеря разнообразия (дисбиоз) связана с хроническими заболеваниями (Turnbaugh et al., 2008; Qin et al., 2010) и раком (Garrett et al., 2015; Segre et al., 2015; Drewes et al., 2016), а также связана с плохими результатами при некоторых формах лечения рака, включая трансплантацию аллогенных стволовых клеток (Taur et al., 2014). На основании этих данных проводили исследование разнообразия орального и кишечного микробиомов у пациентов с блокадой PD-1, и было обнаружено, что разнообразие кишечного микробиома было значительно выше у R по сравнению с NR с использованием нескольких показателей (р=0,009, фиг. 1D и 7). Никаких существенных различий не наблюдалось в оральном микробиоме (р=0,11, фиг. 8). Отношение разнообразия к выживаемости без прогрессирования заболевания (PFS) исследовали в когорте, демонстрируя, что у пациентов с высоким разнообразием фекального микробиома была значительно более длительная PFS по сравнению с пациентами с промежуточным или низким разнообразием (p=0,021 и 0,041, соответственно; фиг.1Е-F и 9). При сравнении разнообразия орального микробиома не было отмечено различий в PFS (фиг. 10A-D).
[00183] Композиционные различия в микробиоме могут также влиять на развитие рака и ответ на терапию ((Sivan et al., 2015; Iida et al., 2013; Viaud et al., 2013; Vetizou et al., 2015), таким образом, также пытались определить, существуют ли различия в компонентах микробиоты в оральном или кишечном микробиомах R и NR при блокаде PD-1. Чтобы исследовать это, рассчитывали индекс преимущественной представленности (ei) операционных таксономических единиц (OTU) и проводили сравнение R с NR, демонстрирующее, что различные наборы бактерий были связаны с ответом на терапию анти-PD-1 с преимущественной представленностью Clostridiales у R и Bacteroidales у NR в микробиоме кишечника (р<0,001, фиг. 2A-B и 11). Не отмечалось существенных различий в преимущественной представленности в оральном микробиоме R по сравнению с NR (фиг. 12A-B). Для дальнейшего изучения этих результатов проводили сравнения классов с высокой размерностью с помощью линейного дискриминантного анализа величины эффекта (LEfSe) (Segata et al., 2011), который снова продемонстрировал избирательно многочисленные бактерии в фекальном микробиоме R по сравнению с NR при блокаде PD-1 с преимущественной представленностью Clostridiales/Ruminococcaceae у R и преимущественной представленностью Bacteroidales у NR (фиг. 2C-D). Никаких существенных различий в оральном микробиоме между R и NR не наблюдалось, за исключением более высоких Bacteroidales у NR при блокаде PD-1 (фиг. 13A-B). Затем проводили парные сравнения по ответу для бактериальных таксонов на всех уровнях. В дополнение к подтверждению предыдущих таксономических различий, эти анализы идентифицировали значительную преимущественную представленность у R вида Faecalibacterium prausnitzii (фиг. 2E, таблица 4). Метагеномный WGS дополнительно выявил преимущественную представленность видов Faecalibacterium в дополнение к другим, включая в себя Akkermansia у R, тогда как NR характеризовались преимущественной представленностью Bacteroides thetaiotaomicron, Escherichia coli и Anaerotruncus colihominis (фиг. 2F, таблица 4). Следует отметить, что была продемонстрирована относительная стабильность кишечного микробиома во времени в ограниченном количестве продолжительно исследованных образцов (фиг. 14A-C).
[00184] На основании этих выводов затем был задан вопрос, может ли бактериальная композиция и численность в кишечном и/или оральном микробиомах предсказать ответ на блокаду PD-1 в когорте изобретателей. Для этого все идентифицированные OTU группировали в кластеры связанных OTU (crOTU) с помощью построения филогенетического дерева по данным выравнивания последовательностей (Peled et al., 2017). Этот способ предусматривает сравнение численности различных потенциальных групп бактерий на основе сходства последовательностей 16S и помогает устранить редкое распределение численности OTU, наблюдаемое в отсутствие этого подхода (фиг. 15A-B). Затем выполняли неконтролируемую иерархическую кластеризацию численности crOTU в кишечном и оральном микробиомах без ввода данных ответа. Результаты показали, что пациенты разделились на 2 отдельных кластера, причем один кластер (кластер 1) полностью состоял из R, а другой (кластер 2) состоял из смеси R и NR (p=0,02) с преимущественной представленностью Clostridiales в кластере 1 и Bacteroidales в кластере 2 (фиг. 3A-B). Затем оценивали PFS в каждом из этих кластеров, демонстрируя значительно более короткое время до прогрессирования на блокаде PD-1 среди пациентов в кластере 1 по сравнению с пациентами в кластере 2 (р=0,02) (фиг. 3C). Чтобы лучше понять композиционные различия в этих кластерах, проводили попарные сравнения кишечной микробиоты, и идентифицировали паттерн, очень похожий на тот, который наблюдается при кластеризации по ответу, с преимущественной представленностью Clostridiales/Ruminococcaceae в кластере 1 и преимущественной представленностью Bacteroidales в кластере 2 (фиг. 3D, таблица 5). Анализ crOTU в оральном микробиоме не выявил явной связи с ответом на лечение (фиг. 16A-B).
[00185] Чтобы исследовать связь специфических бактериальных таксонов и ответа на лечение, авторы настоящего изобретения сравнивали PFS с анти-PD-1 терапией, так как она связана с «лучшими результатами», последовательно наблюдаемыми в анализах (F. prausnitzii у R и Bacteroidales у NR), разделяя пациентов на высокие и низкие категории на основе средней относительной численности этих таксонов в кишечном микробиоме. Пациенты с высокой численностью F. prausnitzii характеризовались значительно более длительной PFS по сравнению с пациентами с низкой численностью (p=0,03). Наоборот, у пациентов с высокой численностью Bacteroidales наблюдалась более короткая PFS по сравнению с пациентами с низкой численностью (p=0,05) (фиг. 3E). Это согласуется с недавно опубликованными данными в небольшой когорте пациентов на блокаде CTLA-4, где пациенты с более высокой численностью Faecalibacterium характеризовались более длительной PFS по сравнению с пациентами с более высокой численностью Bacteroides в микробиоме кишечника (Chaput et al., 2017)). Кроме того, в когорте изобретателей проводили анализ пропорциональных рисков Кокса, демонстрирующий, что наиболее сильными прогностическими факторами ответа на блокаду PD-1 были альфа-разнообразие (HR=3,94; 95% C.I.=1,02-12,52), численность F.prausnitzii (HR=2,92; 95% C.I.=1,08-7,89) и кластеры crOTU (HR=3,80; 95% C.I.=1,09-13,21) в фекальном микробиоме. Эти эффекты оставались значительными в многофакторном анализе после корректировки в отношении предшествующего лечения иммунотерапией (таблица 6).
[00186] Таблица 4: Парные сравнения бактериальных таксонов между R и NR с помощью 2-стороннего теста MW на каждом уровне таксономии.
[00187] Таблица 5: Парные сравнения бактериальных таксонов, полученных из 16S, между сообществом типа 1 crOTU и сообществом типа 2 crOTU с помощью двухстороннего критерия Манна-Уитни в пределах каждого уровня таксономии.
aFDR p-значения корректируются в пределах каждого уровня таксономии
[00188] Затем была предпринята попытка понять механизм, посредством которого кишечный микробиом может влиять на ответ на анти-PD-1 терапию, и сначала проводили функциональное профилирование генома образцов кишечного микробиома с помощью метагеномного WGS у R против NR до терапии. Специфические для организма попадания генов относили к ортологии Энциклопедии генов и геномов Института химических исследований в Киото (KEGG) (KO), и на основании этих аннотаций метагеномы для каждого образца реконструировали в метаболические пути с использованием иерархии MetaCyc классификаций путей (Caspi et al., 2008; Kanehisa et al., 2000). Неконтролируемая иерархическая кластеризация по относительной численности как КО, так и прогнозируемых путей определила три группы образцов пациентов с частотой ответов 72,7%, 57,1% и 42,9%. Сравнение численностей функций генов в этих группах показало изменения метаболических функций, причем анаболические функции преобладают у R, включая в себя биосинтез аминокислот (фиг. 3F), что может способствовать иммунитету хозяина (Blacher et al., 2017), тогда как катаболические функции преобладают у NR (фиг. 3F, 17, таблица 7).
[00189] В доклинических моделях имеются четкие доказательства того, что избирательная многочисленность микробиома кишечника может влиять на терапевтические ответы на блокаду PD-1 на уровне микроокружения опухоли (Sivan et al., 2015), таким образом, детально исследовали связь между микробиотой кишечника и системными и противоопухолевыми иммунными ответами в когорте пациентов на блокаде PD-1. Чтобы сделать это, опухолеассоциированные иммунные инфильтраты сравнивали с помощью многопараметрической IHC и наблюдали более высокую плотность CD8+ T-лимфоцитов в исходных образцах R по сравнению с NR (p=0,04), что согласуется с предыдущими сообщениями (фиг. 4A и 18A-F) (Tumeh et al., 2014; Chen et al., 2016). Затем проводили парные сравнения с использованием корреляции рангов Спирмена между специфическими бактериальными таксонами, обогащающими микробиом кишечника R и NR, и иммунными маркерами в микроокружении опухоли, демонстрируя сильную положительную корреляцию между численностью Ruminococcaceae/Faecalibacterium в кишечнике и инфильтратом цитотоксических Т-клеток в опухоли и сильную отрицательную корреляцию с Bacteroidales (фиг. 4B-C и 19-20). Анализ системных иммунных ответов с помощью проточной цитометрии и анализов цитокинов показал, что у пациентов с высокой численностью Ruminococcaceae в кишечнике было более высокое содержание эффекторных CD4+ и CD8+ T-клеток в системном кровообращении с сохраненным цитокиновым ответом на блокаду PD-1, тогда как у пациентов с более высокой численностью Bacteroidales в микробиоме кишечника было более высокое содержание регуляторных Т-клеток (Treg) и миелоидных супрессорных клеток (MDSC) в системном кровообращении со сниженным цитокиновым ответом (фиг. 4D и 21-22). Чтобы лучше понять влияние композиционных различий в микробиоме кишечника на процессинг и презентацию антигена в микроокружении опухоли, проводили мультиплексную IHC, нацеленную на миелоидный компартмент (Tsujikawa et al., 2017). В этих исследованиях пациенты с высокой численностью Faecalibacterium в кишечном микробиоме характеризовались более высокой плотностью иммунных клеток и маркеров процессинга и презентации антигена по сравнению с пациентами с высокой численностью Bacteroidales (фиг. 4E-F и 23-24), предполагая возможный механизм, посредством которого микробиом кишечника может модулировать противоопухолевые иммунные ответы (Sivan et al., 2015).
[00190] С учетом этих выводов в контексте опубликованной литературы предположили, чтобы микробиом кишечника модулирует ответы на лечение анти-PD-1 терапией (фиг. 4G). В частности, предположили, что пациенты с «благоприятным» микробиомом кишечника (с высоким разнообразием и численностью Ruminococcaceae/Faecalibacterium) характеризовались усиленными системными и противоопухолевыми иммунными ответами, опосредованными усиленной презентацией антигена на уровне лимфатического узла и опухоли, а также сохраненной эффекторной функцией Т-клеток на периферии и микроокружение опухоли. Напротив, пациенты с «неблагоприятным» микробиомом кишечника (с низким разнообразием и высокой относительной численностью Bacteroidales) характеризовались нарушенными системными и противоопухолевыми иммунными ответами, опосредованными ограниченной внутриопухолевой инфильтрацией как лимфоидных, так и миелоидных элементов, ослабленной способностью к презентации антигена и перекосом к иммунорегуляторным клеточным и гуморальным элементам на периферии, включая в себя Treg и MDSC. Эти результаты подчеркивают возможность параллельной модуляции микробиома кишечника для значительного повышения эффективности блокады контрольных точек.
[00191] Таблица 6: Одномерные и многомерные модели пропорциональных рисков Кокса для выживаемости без прогрессирования заболевания в группе фекального анализа.
(95% C.I.)
(95% C.I.)a
b Определение конечной модели посредством передового отбора и подхода лучших подмножеств. Альфа-разнообразие включали в качестве двоичной переменной на основе медианной точки разделения.
c повышенное содержание LDH: превышение верхнего предела нормы (618 МЕ/мл), все образцы оценивали в общей лаборатории
d плотность CD8+ на основе образцов от 15 пациентов с доступной исходной опухолью
[00192] Таблица 7: Парное сравнение класса пути MetaCyc по ответу
[00193] Таблица 8: Индивидуальные значения и сводная статистика метаболических реконструкций.
Пример 2. Материалы и способы.
[00194] Когорта пациентов: в настоящее исследование включали исходную когорту из 112 пациентов с метастатической меланомой. Эти пациенты проходили курс лечения посредством анти-PD1 блокады иммунных контрольных точек в онкологическом центре MD Anderson при Университете Техаса (Юта) в период с апреля 2015 г. по март 2016 г. и подписали добровольное информированное согласие на сбор и анализ образцов опухолей, крови и микробиомов при одобренных институциональным наблюдательным советом (IRB) протоколах. Пациентов, у которых была диагностирована увеальная меланома (n=10), или которые получали анти-PD1 в комбинации со средствами целенаправленного воздействия или с терапией адоптивным переносом Т-клеток (n=8), или у которых ответ не мог быть определен (n=6), исключали из этого анализа. Три исследователя независимо друг от друга изучали электронные медицинские карты, чтобы определить группу клинического ответа и задокументировать другие клинические параметры (таблица 3). Первичный исход клинического ответа (отвечающий на лечение) (R) определяли рентгенографическим подтверждением полного ответа (CR), частичного ответа (PR) или стабильного заболевания (SD) по критериям RECIST 1.1 в течение по меньшей мере 6 месяцев. Отсутствие клинического ответа (не отвечающий на лечение) (NR) определяли по прогрессированию заболевания (PD) при серийном КТ-сканировании или клинической пользе, продолжавшейся менее 6 месяцев (минимальная польза).
[00195] Сбор образцов микробиома. Буккальные образцы собирали во время обычных визитов в клинику перед лечением с использованием тампона для сбора образцов Catch-All (Epicenter, Madison, WI). Всем пациентам также предоставляли амбулаторный набор OMNIgene GUT (OMR-200) (наборы для сбора образцов кала DNA Genotek и их попросили вернуться). Важно отметить, что этот набор помогает поддерживать стабильность профиля микробиома при комнатной температуре до 60 дней. Все образцы замораживали при температуре -80°С до выделения и анализа ДНК.
[00196] Конечная когорта состояла из буккальных образцов, собранных от 87 пациентов, из которых 52 были R и 34 были NR, и фекальных образцов, собранных от 43 пациентов (из которых 30 были R и 13 были NR). Все, кроме 2 буккальных и 2 фекальных образцов, были собраны в начале исследования. Их включали в качестве исходного суррогата как анализ субпопуляции для пролонгированных образцов, которые не показали изменений после лечебного вмешательства в этой когорте.
[00197] Сбор образцов опухоли и крови. Доступные образцы опухолей (n=23) в согласованные моменты времени до лечения получали из Департамента онкологического центра MD Anderson архива патологии и институционального банка тканей. После того, как образцы прошли проверку качества на процент жизнеспособности опухоли у патолога MD Anderson, авторы настоящего изобретения включили 17 образцов из R и 6 из NR. Образцы крови, собранные и сохраненные для исследования (протоколы ранее перечислены) на исходном уровне (n=11), также были запрошены для включения в исследование, давая образцы из 8 R и 3 NR.
[00198] Экстракция ДНК и бактериальное секвенирование 16S. Подготовка и секвенирование выполняли в сотрудничестве с Центром исследований метагеномики и микробиомов (CMMR) при Медицинском колледже Бейлора. Способы секвенирования генов 16S рРНК адаптировали на основе способов, разработанных для проекта микробиома человека NIH (Структура исследования микробиома человека, 2012).
[00199] Вкратце, бактериальную геномную ДНК экстрагировали с использованием набора для выделения ДНК MO BIO PowerSoil (MO BIO Laboratories, США). Область V4 rDNA 16S амплифицировали с помощью ПЦР и секвенировали на платформе MiSeq (Illumina, Inc, Сан-Диего, Калифорния) с использованием протокола парных прочтений 2×250 п.н., что дает парные прочтения, которые практически полностью перекрываются. Праймеры, используемые для амплификации, содержат адаптеры для секвенирования MiSeq и штрих-коды на одном конце, позволяющие объединять и напрямую секвенировать продукты ПЦР (Caporaso et al., 2012).
[00200] Отфильтрованные по качеству последовательности с идентичностью >97% группировали в контейнеры, известные как операционные таксономические единицы (OTU), с использованием сортировки OTU с открытой ссылкой (Edgar, 2010; Rognes et al., 2016; Caporaso et al., 2010), и классифицировали на уровне вида по базе данных последовательности рибосомальной РНК 16S NCBI с использованием пакета ncbi-blast + 2.5.0. Филогенетическую классификацию получали из базы данных таксономии NCBI. Относительную численность каждой OTU определяли для всех образцов. Таксономическую классификацию подтверждали с использованием баз данных Greengenes, SILVA и RDP.
[00201] Филогенетическое дерево строили эмпирически с использованием алгоритма FastTree (Price et al., 2010) в программном пакете QIIME, как описано ранее (Peled et al., 2016). Вкратце, все узлы дерева рассматривали как кластеры связанных OTU (crOTU), где численность каждого crOTU представляла собой сумму численностей его представителей OTU. Деревья строили на основе выравнивания последовательностей всех наблюдаемых OTU как орального 1152 (97,5 % из 1182 OTU), так и кишечного 1434 (98,6% из 1455 OTU) микробиомов. Полученные деревья crOTU орального и кишечного микробиомов содержали 1152 и 1434 узла, соответственно.
[00202] Таксономическое альфа-разнообразие оценивали с использованием обратной формы индекса Симпсона. Пределы разрежения устанавливали на основе наименьшего числа прочтений во всех проанализированных оральных (13000) и фекальных образцах (8000), где D = 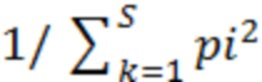 , где pi представляет собой долю всех видов S, который состоит из вида i. Поскольку он отражает дисперсию таксономического распределения численности, его часто считают одним из наиболее значимых и надежных показателей разнообразия (Shannon et al., 2013).
, где pi представляет собой долю всех видов S, который состоит из вида i. Поскольку он отражает дисперсию таксономического распределения численности, его часто считают одним из наиболее значимых и надежных показателей разнообразия (Shannon et al., 2013).
[00203] Двудольная сеть для сравнения и сопоставления оральной и кишечной микробиоты: Двудольную сеть строили с использованием сценария make_biparitite_network.py в QIIME с использованием параметров по умолчанию (Caporaso et al., 2010), а затем визуализировали в Cytoscape с использованием встроенного в среду spring макета со взвешенными ребрами. Создавали 2 сети с использованием всех буккальных и фекальных образцов (фиг. 6) и с использованием только парных образцов (фиг. 1А), когда оба образца получали от одних и тех же пациентов.
[00204] Индекс преимущественной представленности для визуализации различий в оральном и кишечном микробиоме между R и NR: Индекс репрезентации OTU (ri): используется для количественной оценки репрезентативности каждого вида в R; (riR) и NR (riNR), как доля образцов в каждой группе, которые характеризовались ненулевым содержанием для определенного вида. Значения ri варьировали от 0 (OTU не обнаружена ни в одном образце в группе) до 1 (OTU обнаружена во всех образцах в группе).
[00205] Индекс преимущественной представленности OTU (ei): использовали для количественной оценки и сравнения преимущественной представленности каждой OTU в R против NR, где ei=(riR - riNR)/(riR + riNR). Этот индекс характеризуется значениями в диапазоне от -1 до +1. Когда вид идентифицирован во всех образцах R, но не в образцах NR, ei=+1. Противоположное верно для ei=-1.
[00206] Показатели ei распределения использовали для классификации всех видов на 3 набора: набор 1 - избирательно преимущественно представленный в R, набор 2 - обнаруженный в обеих группах, и набор 3 - избирательно преимущественно представленный в NR. В каждом наборе все OTU сортировали по численности и затем визуализировали в виде карты интенсивности преобразованных в log(10) численностей OTU во всех образцах, представленных в виде столбцов. Пороговые значения для численностей OTU (низкие, средние и высокие) получали из распределения численностей для всех OTU.
[00207] Статистическая оценка биомаркеров с использованием LEfSe. Способ LEfSe анализа сначала сравнивает численность всех бактериальных кладов между R и NR в оральном и кишечном микробиомах с использованием критерия Крускала-Уоллиса с предварительно определенным значением α 0,05. Значительно отличающиеся векторы, представляющие собой результат сравнения численностей (например, относительной численности Faecalibacterium) между группами, используют в качестве входных данных для линейного дискриминантного анализа (LDA), который дает величину эффекта (фиг. 2B). Основное преимущество LEfSe перед традиционными статистическими тестами заключается в том, что в дополнение к p-значению создается величина эффекта. Это позволяет сортировать результаты множества тестов по величине разницы между группами. В случае иерархически организованных бактериальных клад, возможно, отсутствует корреляция между значениями p и величинами эффекта из-за различий в количестве гипотез, рассматриваемых на разных уровнях, поскольку необходимо провести большее количество сравнений на уровне рода и вида, по сравнению с уровнями типа и класса.
[00208] Метагеномное секвенирование целого генома (WGS): Это также выполняли в сотрудничестве с CMMR и Metagenopolis (MGP). Вкратце, данные метагеномного секвенирования обеспечивают разрешение бактерий на уровне видов, а также почти полное геномное содержание коллекции микробов в конкретном образце, также называемое пангеномом (глубина секвенирования напрямую связана с количеством пангенома, которое покрыто в конкретном наборе данных).
[00209] Для секвенирования целого генома (WGS) используют ту же экстрагированную бактериальную геномную ДНК, которую использовали для композиционного анализа гена 16S рРНК. Однако WGS-секвенирование обеспечивает более высокую глубину секвенирования за счет использования более мощной платформы секвенирования. Отдельные библиотеки конструировали из каждого образца и загружали в платформу HiSeq (Illumina) и секвенировали с использованием протокола парных прочтений 2x100 п.н. Процесс качественной фильтрации, выравнивания и демультиплексирования осуществляли собственным конвейером, разработанным путем сборки общедоступных инструментов, таких как Casava v1.8.3 (Illumina) для генерации fastqs, Trim Galore и cutadapt для адаптера и выравнивания качества, и PRINSEQ для демультиплексирования образцов.
[00210] Анализ кишечной микробиоты проводили с использованием количественного метагеномного конвейера, разработанного в MGP. Такой подход позволяет проводить анализ микробиоты на уровне генов и видов. Прочтения высокого качества отбирали и очищали для устранения возможных загрязнений как прочтения человека. Их картировали и подсчитывали с использованием каталога генов MetaHIT hs_9.9M (Li et al., 2014) с использованием METEOR Studio в собственном конвейере с использованием двухэтапной процедуры: сначала с использованием однозначного картирования прочтений, затем причисления распределенных прочтений (картирование разных генов из каталога) в соответствии с соотношением их картирования с использованием уникальных прочтений. Картирование выполняли с использованием >95%-ного порога идентичности, чтобы учесть изменчивость гена и отсутствие избыточного характера каталога.
[00211] После стадии сокращения в 14M прочтений (для корректировки различной глубины секвенирования) и нормализации (RPKM) получали матрицу профиля частоты гена, которую использовали в качестве эталона для выполнения анализа с использованием MetaOMineR, набора пакетов R разработанный в MGP и посвященный анализу больших количественных наборов метагеномных данных.
[00212] Каталог генов hs_9.9M кластеризовали в 1438 MGS (MetaGenomic Species), группы из >500 генов, которые совместно изменяются в численности среди сотен образцов и, таким образом, принадлежат к одним и тем же микробиологическим видам (Nielson et al., 2014). Таксономическую аннотацию MGS выполняли с использованием гомологии его генов с ранее секвенированными организмами (с использованием blastN против банков данных nt и wgs). Сигнал MGS среди образцов рассчитывали как средний или медианный сигнал 50 маркерных генов. Матрицу профиля частоты MGS конструировали с использованием сигналов средних значений MGS и после нормализации (сумма частоты MGS образца = 1).
[00213] Прочтения, чьи геномные координаты перекрываются с известными ортологами KEGG, сводили в таблицу, и модули KEGG рассчитывали поэтапно и определяли как полные, если присутствовали 65% стадий реакции для каждого обнаруженного вида и для метагенома. Пути конструировали для каждого таксона и метагенома путем расчета минимального набора, установленного через MinPath, в результате присутствия генных ортологов.
[00214] Базы данных метагенома Pathway (PGDB): Базы данных получали для каждого образца WGS с использованием программы PathoLogic из программного обеспечения Pathway Tools [PMID: 26454094]. Входные данные для программы получали с использованием предсказанных функций генов на основе ортологии KEGG и их назначения таксономии в метагеномах. Таким образом, если одна и та же функция (группа KO) характеризовалась несколькими таксономическими аннотациями, каждую из аннотаций рассматривали как отдельный ген. Определение группы KO в KEGG использовали в качестве функции гена, а название группы KO, если доступно, использовали в качестве названия гена. Номера EC присваивали гену в соответствии с аннотациями группы KO по номерам в KEGG. Метаболические реконструкции делали в автоматическом режиме, как описано в руководстве Pathway Tools с опцией -tip для автоматического прогнозирования транспортных реакций. Значение DOMAIN ‘TAX-2 (Bacteria)’ использовали в качестве класса организмов для PGDB, а значение CODON-TABLE было равно 1. Полученные PGDB обобщали и сравнивали с использованием Pathway Tools. Полученные PGDB доступны по запросу.
[00215] Статистический анализ. Альфа-разнообразие сравнивали между R и NR, используя критерий суммы рангов Уилкоксона или критерий Манна-Уитни (MW). Всех пациентов классифицировали на группы с высоким, промежуточным или низким разнообразием в зависимости от тертилей распределения. Попарные сравнения таксономической численности как по ответу, так и по кластеру проводили с использованием теста MW. На каждом уровне (тип, класс, порядок, семейство, род и вид) исключали таксоны низкой численности (<0,1%) и низкой дисперсии (<0,001). Корректировки для множественных сравнений выполняли с использованием способа уровня ложноположительных обнаружений при альфа-уровне 0,05. Величину эффекта оценивали для каждого таксона как U/√n, где U - критерий значимости для теста MW, а n - общий размер выборки (n=43) (Fritz et al., 2012), и строили вулканные диаграммы для log10 (значения p, скорректированные с помощью FDR) по оси y и величины эффекта, скорректированные по медиане, по оси x. Кроме того, пациентов также классифицировали как характеризующиеся высокой или низкой численностью Faecalibacterium prausnitzii или Bacteroidales на основании медианной численности этих таксонов в кишечном микробиоме. Оценки Каплана-Мейера оценивали для каждой группы и сравнивали с использованием логрангового критерия. Отношения рисков оценивали с использованием модели пропорциональных рисков Кокса.
[00216] В общем, тест MW использовали для сравнения двоичных выходных переменных (R против NR), а корреляционный тест Спирмена использовали для сравнения непрерывных случайных переменных. Кроме того, использовали точный критерий Фишера, когда сравнивали пропорции между двоичными переменными. Проверку гипотез проводили с использованием как односторонних, так и двусторонних тестов в зависимости от ситуации на уровне значимости 95%. Все анализы проводили в R и GraphPad Prism (La Jolla, CA).
[00217] Иммуногистохимия. Вкратце, срезы (толщиной 4 мкм) готовили из фиксированных в формалине погруженных в парафин тканей (FFPE). Наличие опухоли было подтверждено патологом на предметных стеклах, окрашенных гематоксилином и эозином (H&E). Затем предметные стекла окрашивали с использованием автоматического красителя для стекол Leica Bond RX (Leica Biosystems, Buffalo Grove, IL) для CD3 (n=17) (DAKO, Santa Clara, CA, 1:100), CD8 (n=21) (Thermo Scientific , Waltham, MA, 1:100), PD-1 (n=16) (Abcam, Cambridge, UK, 1:250), PD-L1 (n=15) (1:100, Cell Signaling, Danvers, MA) , GzmB (n=17), RORγT (n=14) (1:800, EMD Millipore, Billerica, MA), FoxP3 (n=16) (1:50, BioLegend, San Diego, CA) и контрастно окрашивали с помощью гематоксилина. Затем окрашенные стекла сканировали с использованием автоматического сканера слайдов Aperio (Leica), а плотность иммунного инфильтрата количественно определяли в опухолевых областях с использованием модифицированной версии алгоритма «Nuclear v9» по умолчанию и выражали в виде положительных значений/мм2 для CD3, CD8, PD-1, FoxP3 и RORγT, а также как H-показатель для PD-L1, который учитывает процент положительных клеток, умноженный на их интенсивность по шкале от 1 до 3 для показателя между 1-300.
[00218] Проточную цитометрию проводили на мононуклеарных клетках периферической крови (РВМС). РВМС окрашивали CD3 (UCHT1, BioLegend), CD4 (SK3, eBioscience, Thermo Scientific), CD8 (RPAT8, BD Biosciences, Mississauga, Canada), FoxP3 (PCH101, eBioscience), CD127 (HIL-7R-M21, BD Biosciences), CD19 (HIB-19, BioLegend), CD14 (61D3, eBioscience), HLA-DR (L243, BD Biosciences), CD33 (WM53, BD Biosciences), CD56 (NCAM1, BD Biosciences) и CD11b (ICRF44, BD Biosciences) и сбор данных выполняли на проточном цитометре Fortessa (BD Biosciences). Анализ проводили с использованием FlowJo версии 10 (Tree Star Inc., Ashland, OR).
[00219] Мультиплексная иммуногистохимия. Последовательную миелоидную мультиплексную иммуногистохимию с 12 маркерами проводили, как описано ранее (Tsujikawa, Cell Reports, 2017). Вкратце, срезы FFPE подвергали последовательным циклам окрашивания, сканирования и удаления красителя с использованием AEC в качестве хромагена и сканировали с помощью Aperio Slide Scanner (Leica). После окрашивания и сканирования гематоксилином на наличие ядер, CD68, триптазы, CSF1R, DC-SIGN, CD66b, CD83, CD163, HLA-DR, PD-L1, CD3/CD20/CD56 и CD45. CD45-положительные области всех изображений затем извлекали с использованием ImageScope, выравнивали, накладывали и сегментировали с использованием CellProfiler и слоев, псевдоокрашенных для анализа и количественного определения с использованием FCS Express.
[00220] Мультиплексирование цитокинов: 41 уровень цитокинов в плазме оценивали с использованием мультиплексного анализа с использованием шариков (Bio-Rad, Hercules, CA). Количественное определение цитокинов, хемокинов и растворимых медиаторов включало в себя IL-1b, IL-1ra, IL-2, IL-4, IL-5, IL-6, IL-7, IL-8, IL-9, IL-10, IL- 12 (p70), IL-13, IL-15, IL-17, эотаксин, основной FGF, G-CSF, GM-CSF, IFN-g, IP-10, MIP-1a, PDGF-bb, MIP-1b, RANTES, TNF-a, VEGF, IL-2ra, HGF, TRAIL, IL-17A, IL-17F, IL-23, SDF1/CXCL12, CCL22, MCP-1/CCL2, Gro-a/CXCL1, ENA78/CXCL5, EGF, TGF-b1, TGF-b2 и TGF-b3.
Пример 3 - Модуляция кишечного микробиома усиливает противоопухолевые ответы в мышиной модели меланомы
[00221] Затем предприняли попытка использовать информацию, полученную в результате исследований на людях, для проверки гипотезы о том, что модуляция кишечного микробиома может усиливать противоопухолевые ответы в мышиной модели меланомы. Проводили исследования, чтобы определить, может ли модуляция кишечного микробиома для обогащения бактериями, производящими короткоцепочечные жирные кислоты, усиливать противоопухолевые ответы.
[00222] В этих исследованиях использовали модель опухоли мыши с общей драйверной мутацией, которая обнаруживается при меланоме (BRAF). Клетки имплантировали генетически идентичным мышам (C57BL6), приобретенным у 2 разных поставщиков (лаборатории Taconic farms против Jackson), которые значительно различаются по своему микробиому (с преимущественной представленностью производящими короткоцепочечные жирные кислоты бактериями у мышей Taconic). Опухолевые клетки BRAF (V600E)/Pten-/- имплантировали мышам Taconic и Jackson, и наблюдали существенные различия в росте опухоли (фиг. 33A) и выживаемости (фиг. 33B) у этих генетически идентичных мышей с различными микробиомами. Интересно, что модуляция кишечного микробиома путем совместного размещения Taconic и Jackson (фиг. 33C) (поскольку мыши, как правило, являются копрофагами) или посредством фекального трансплантата устраняет эти различия, предполагая, что модуляция кишечного микробиома может изменить рост опухоли у BRAF-мутантной опухолевой модели. Поскольку мыши являются копрафагами, совместное содержание приводит к появлению объединенного микробиома, который извлекается из обоих отдельных микробиомов. Секвенирование 16S проводили на мышах Taconic и Jackson с отдельным или общим содержанием, демонстрируя различные микробиомы у мышей с отдельным или общим содержанием (фиг. 33D и E), подтверждая модуляцию микробиома кишечника у этих мышей.
[00223] На основании данных, полученных на людях, было проверено, может ли пероральное введение бутирата (короткоцепочечной жирной кислоты) усиливать противоопухолевые ответы, обеспечивая адекватный субстрат для облегчения благоприятного кишечного микробиома. В этих исследованиях бутират вводили перорально мышам Taconic и Jackson. Лечение бутиратом было связано с усиленным противоопухолевым ответом в этих исследованиях (фиг. 33F), дополнительно предполагая, что модуляция кишечного микробиома может усиливать противоопухолевые ответы.
[00224] Таким образом, эти исследования предоставляют новые данные, касающиеся разнообразия и композиции орального и кишечного микробиома у пациентов с метастатической меланомой при системной терапии и, что важно, демонстрируют наличие различных бактериальных «сигнатур» у отвечающих на лечение пациентов по сравнению с пациентами, не отвечающими на блокаду иммунных контрольных точек (в частности, терапию на основе PD-1).
Пример 4 - Трансплантация фекальной микробиоты
[00225] Трансплантация фекальной микробиоты (FMT) благоприятного микробиома кишечника безмикробным мышам (GF) уменьшает рост опухоли (FMT1): Для изучения причинной связи между «благоприятным» микробиомом кишечника и ответом на блокаду иммунных контрольных точек, эксперименты по трансплантации фекального микробиома (FMT) проводили на безмикробных мышах-реципиентах (GF). Использовали хорошо разработанную сингенную модель инъецируемой мышиной меланомы, обусловленной онкогенной экспрессией BRAFV600E и делецией PTEN (клетки BP). В первом эксперименте (FMT1) пытались определить, может ли FMT оказывать какое-либо влияние на рост опухоли. Мышам GF (n=3 на группу) трансплантировали пероральным зондом стул от отвечающих (группа R-FMT) или от не отвечающих (группа NR-FMT) на терапию анти-PD-1. Мышам контрольной группы трансплантировали только PBS. Через две недели после FMT каждой мыши подкожно инъецировали 8×105 клеток BP, и рост опухоли проверяли два раза в неделю (фиг. 25A). Кровь и фекальные комочки собирали в разные моменты времени в течение эксперимента.
[00226] Результаты FMT1 продемонстрировали значительную задержку роста опухоли к 14 дню в группе R-FMT по сравнению с мышами, которым трансплантировали стул от NR при анти-PD-1 (p=0,04, фиг. 1B).
[00227] Затем системное воздействие FMT на иммунную систему исследовали с использованием анализа многоцветной клеточной сортировки с активацией флуоресценции (FACS). Примечательно, что мыши, получавшие R-FMT, характеризовались более высоким процентом врожденных эффекторных клеток (экспрессирующих CD45+CD11b+Ly6G+) и более низкой частотой супрессорных миелоидных клеток (экспрессирующих CD11b+CD11c+) в селезенке по сравнению с мышами в группе NR-FMT (фиг. 26). Эти данные позволяют предположить, что специфические субпопуляции в компартменте врожденного иммунитета играют роль в противоопухолевом ответе, вызванном трансплантацией благоприятной кишечной микробиоты у мышей-реципиентов GF.
[00228] Этот вывод дополнительно подтверждали количественной конфокальной визуализацией на срезах кишечника и опухоли от мышей, которые получали FMT. Действительно, опухоли мышей, получавших R-FMT, характеризовались более высокой плотностью CD45+ и CD8+ T-клеток, чем мыши, получавшие NR-FMT (фиг. 27A и верхняя панель 27C). Кроме того, FMT от R локально увеличивала количество CD45+ иммунных и CD8+ T-клеток в кишечнике по сравнению с NR-FMT (фиг. 27B и нижняя панель 27C).
[00229] Кроме того, таксономическая характеристика с использованием секвенирования 16S выявила резкие различия в кишечном микробиоме безмикробных мышей до и после трансплантации фекальной микробиоты (FMT) с существенным увеличением таксонов Bacteroidales и Clostridiales. Как и ожидалось, контрольные мыши, получавшие PBS, обладали заметно отличающимися таксонами в своем кишечном микробиоме по сравнению с мышами, которые получали стул от отвечающих (R-FMT) или не отвечающих (NR-FMT) на терапию анти-PD1 мышей. Кроме того, кишечные микробиомы мышей, получавших R-FMT и NR-FMT, оставались довольно стабильными во времени после трансплантации.
[00230] FMT благоприятного кишечного микробиома у мышей GF уменьшает рост опухоли и усиливает ответ на терапию α-PD-L1 (FMT2): во втором эксперименте FMT (FMT2) спрашивали, может ли микробиом от пациента R усиливать ответ на иммунотерапию при трансплантации на мышиной модели меланомы. Чтобы ответить на этот вопрос, мышам GF трансплантировали через желудочный зонд стул от отвечающих (n=2, группа R-FMT) или от не отвечающих (n=3, группа NR-FMT) на терапию анти-PD-1. Мышам контрольной группы (n=2) трансплантировали только PBS. Через две недели после FMT каждой мыши инъецировали 8×105 клеток BP, и рост опухоли проверяли два раза в неделю. Когда объем опухоли достигал 250-500 мм3, мышей подвергали воздействию антителом к PD-L1, вводимым внутрибрюшинной инъекцией (фиг. 28A).
[00231] Результаты FMT2 продемонстрировали значительную задержку роста опухоли к 14 дню в группе R-FMT по сравнению с мышами у мышей с трансплантированным стулом от NR при анти-PD-1 (p=0,04, фиг. 28B). Важно, что мыши, которым трансплантировали R-FMT, также демонстрировали улучшенные ответы на терапию анти-PD-L1 (фиг. 28C) по сравнению с мышами, которым трансплантировали стул от NR (NR-FMT).
[00232] Затем определяли механизм, посредством которого кишечный микробиом может влиять на системные и противоопухолевые иммунные ответы. FACS-анализ опухолей и иммунных инфильтратов селезенки показал, что мыши, получающие R-FMT, характеризовались более высоким процентом инфильтрирующих опухоль CD45+ миелоидных клеток, по сравнению с мышами в группе NR-FMT (фиг. 29A). В частности, более высокий процент врожденных эффекторных клеток (экспрессирующих CD45+CD11b+Ly6G+) и более низкая частота супрессорных миелоидных клеток (экспрессирующих CD11b+CD11c+) обнаруживались в опухолевых иммунных инфильтратах от R-FMT по сравнению с NR-FMT (фиг. 29B-E). Эти данные коррелировали специфические субпопуляции врожденного иммунного компартмента с противоопухолевым ответом, индуцированным FMT от R пациента, как на периферическом (как показано на FMT1), так и на опухолевом уровне.
[00233] Увеличение частоты RORγT+ Th17 клеток в опухоли также обнаруживали у мышей NR-FMT (фиг. 30A и B), в соответствии с наблюдениями, сделанными в опухолях от пациентов, которые не смогли ответить на блокаду PD-1. Кроме того, мыши, получающие NR-FMT, также продемонстрировали более высокое содержание регуляторных CD4+ FOXP3+ T-клеток (фиг. 30C) и CD4+ IL-17+ (фиг. 31D) в селезенке, что свидетельствует о нарушении иммунного ответа хозяина.
[00234] Анализ масс-цитометрии (CyTOF) с использованием уменьшения размера t-SNE проводили на опухолях от мышей и продемонстрировали активацию PD-L1 в опухолевом микроокружении у мышей, получающих R-FMT, по сравнению с NR-FMT (фиг. 31A), предполагая развитие «горячего» опухолевого микроокружения. Анализ CyTOF также подтвердил, что различные миелоидные субпопуляции преимущественно инфильтрируют опухоли у R-FMT или не отвечающих NR-FMT (фиг. 31B).
[00235] Продолжительный отбор образцов и характеристика микробиома фекальных комочков из эксперимента FMT2 показали результаты, аналогичные FMT1, с относительной стабильностью в кишечном микробиоме во времени.
[00236] Стабильность микробиома у безмикробных мышей после приживления FMT. Секвенирование 16S проводили на фекальных комочках за различные периоды, собранных у безмикробных мышей, которые получали стул от пациента, который отвечал на блокаду PD-1, по сравнению с пациентом, который этого не делал. Изучение филогенетических таксонов на уровне порядка и сравнение исходных комочков с теми, которые были собраны через две недели после завершения FMT, выявили успешное приживление. Более того, постоянство самых многочисленных порядков во времени свидетельствовало о том, что приживленный микробиом был стабилен в течение всего эксперимента.
[00237] Основываясь на гипотезах о том, что определенные бактерии во флоре мышей R-FMT помогают замедлять рост опухоли, и что определенные бактерии во флоре мышей NR-FMT помогают стимулировать рост опухоли, авторы настоящего изобретения сравнили OTU, которые присутствовали только либо у мышей R-FMT, либо у мышей NR-FMT (или также отсутствовали у контрольных мышей). Это выполняли на репрезентативных мышах из каждой группы из 2 экспериментов FMT. Авторы настоящего изобретения также рассмотрели OTU, которые показали последовательные результаты переноса в обоих экспериментах FMT. Этот анализ показал, что OTU, классифицируемые как Acetanaerobacterium elongatum, Alistipes timonensis, Anaerocolumna jejuensis, Anaerocolumna xylanovorans, Bacteroides fragilis, Bacteroides nordii, Bacteroides stercoris, Blautia faecis, Blautia glucerasea, Blautia hansenii, Blautia obeum, Blautia schinkii, Caproiciproducens galactitolivorans, Christensenella minuta, Clostridium aldenense, Clostridium alkalicellulosi, Clostridium amygdalinum, Clostridium oroticum, Clostridium polysaccharolyticum, Clostridium xylanolyticum, Coprobacillus cateniformis, Emergencia timonensis, Eubacterium hallii, Extibacter muris, Faecalibacterium prausnitzii, Ihubacter massiliensis, Neglecta timonensis, Novibacillus thermophilus, Oscillibacter ruminantium, Papillibacter cinnamivorans, Parabacteroides johnsonii, Parasporobacterium paucivorans, Peptococcus niger, Pseudoflavonifractor capillosus, Robinsoniella peoriensis, Ruminococcus gauvreauii, Ruminococcus gnavus, Ruminococcus torques и Slackia piriformis обнаруживали только у мышей R-FMT в обоих экспериментах.
[00238] FMT от различных R и NR доноров безмикробным мышам GF подтверждает связь между микробиотой R и сниженным ростом опухоли (FMT3). Для подтверждения того факта, что «благоприятный» кишечный микробиом сдерживает рост опухоли при трансплантации мышам GF, проводили эксперимент, аналогичный FMT1, с использованием стула от разных пациентов R и NR. В этом третьем эксперименте (FMT3) мышам GF трансплантировали путем орального зондирования стул от одного отвечающего (n=1, группа R-FMT) или от одного не отвечающего (n=1, группа NR-FMT) на терапию анти-PD-1 пациента. Мышам контрольной группы трансплантировали только PBS (n=3, контрольная группа). Через две недели после FMT каждой мыши подкожно инъецировали 2,5×105 клеток BP, и рост опухоли проверяли два раза в неделю (фиг. 32A). Кровь и фекальные комочки собирали в разные моменты времени во время эксперимента.
[00239] Результаты, полученные с помощью FMT3, подтвердили значительную задержку роста опухоли к 14 дню в группе R-FMT по сравнению с мышами с трансплантированным стулом от NR при анти-PD-1 (фиг. 32B). Важно, что эта разница сохранялась с течением времени до конечной точки (фиг. 32C).
Пример 5 - Способы трансплантации фекальной микробиоты
[00240] Трансплантация фекальной микробиоты (FMT). Все исследования на животных были одобрены Институциональным комитетом по уходу за животными и их использованию, UT в Онкологическом центре MD Anderson в соответствии с Руководством по уходу и использованию лабораторных животных. Безмикробных мышей B6 для исследований на мышах приобретали в гнотобиотическом отделении Медицинского колледжа Бейлора (Хьюстон). Всех мышей перевозили в специализированных автоклавных транспортировочных клетках и содержали в отделении для мышей в Онкологическом центре MD Anderson. Все клетки, бутылки с пробками и питьевая вода для животных перед использованием подвергались автоклавированию. Пищу и подстилку подвергали двойному облучению и проверяли на стерильность перед применением в эксперименте. В пределах каждой категории лечения контрольная группа мышей получала только предварительно восстановленный PBS. Все остальные мыши из экспериментальных групп получали FMT от донора R или донора NR, причем каждый образец донора доставлялся одной, двум или трем мышам. 200 мкл очищенного супернатанта из 0,1 г/мкл фекальной суспензии человека получали с использованием сита с размером пор 100 мкм и вводили мышам в количестве 3 доз в течение 1 недели с последующим перерывом в 1 неделю для установления микробиома. Мышам затем вводили клеточную линию сингенных опухолей BP (день 14), а животных подвергали воздействию моноклональным антителом к PD-L1 (очищенная функциональная композиция с низкой эндотоксичностью, B7-H1, CD274, Leinco Technologies Inc.), как только опухоли достигали ~250-500 мм3. Оценивали рост опухолей/выживаемость. Фекальные образцы, кровь, селезенку и опухоли собирали и обрабатывали для дальнейшего анализа.
[00241] Проточная цитометрия опухоли и селезенки мыши: Опухоли выделяли и измельчали на мелкие кусочки и расщепляли в течение 1 часа в RPMI, содержащей коллагеназу A (2 мг/мл; Roche, номер по каталогу 11088793001) и ДНКазу I (40 единиц/мл; Sigma-Aldrich, номер по каталогу D5025) с перемешиванием при температуре 37°C. Суспензии клеток пропускали через клеточный фильтр, промывали в 2% RPMI, дополненном 2 ммоль/л EDTA и ресуспендировали в буфере FACS (PBS, содержащий 2% инактивированной нагреванием FBS с добавкой 2 мМ EDTA). Селезенки разбивали в буфере FACS и эритроциты лизировали инкубацией в буфере ACK (Gibco) в течение 2 минут при комнатной температуре. Затем осадок промывали и ресуспендировали в буфере FACS. Для анализа маркеров клеточной поверхности использовали следующие антитела: CD45 (30-F11, BD Biosciences), CD11b (M1/70, eBioscience), CD11c (HL3, BD Pharmigen), Ly6G (RB6-8C5, eBioscience), Ly6C (AL-21, BD Bioscience), F4/80 (BM8, eBioscience). Клетки метили красителем на жизнеспособность LIVE/DEAD (Life Technologies), и выборки собирали с помощью проточной цитометрии LSR Fortessa X20 (BD). Дублеты выделяли и исключали путем построения графика зависимости площади FSC от высоты FSC и данные анализировали с использованием программного обеспечения FlowJo (Tree Star).
[00242] Иммунофлуоресценция на образцах FFPE. Ксенотрансплантатные опухоли, кишечник мыши и селезенки собирали, фиксировали сначала в забуференном 10% формалине (4 часа при комнатной температуре), затем переключали на 70% этанол и хранили при температуре 4°С. Ткани погружали в парафин, и срезы толщиной 5 мкм устанавливали на положительно заряженных предметных стеклах. Ткани депарафинизировали и извлекали антиген в цитратном буфере с pH 6,0 (Dako), используя микроволновую печь. Срезы блокировали в блокирующем буфере (5% козья сыворотка/0,3% BSA/0,01% тритон в PBS) с последующей инкубацией с первичными антителами в течение ночи при температуре 4°C. Срезы промывали и затем инкубировали с Alexa-конъюгированным вторичным антителом (1:500, Molecular Probes) в течение 1 часа при комнатной температуре. Покровные стекла промывали 3 раза в PBS/0,01% Triton и затем инкубировали в течение 15 минут при комнатной температуре в красителе Hoechst (1:5000, Invitrogen). После 3 промываний в PBS образцы закрепляли в заливочной среде ProLog Diamond (Molecular Probe). Изображения получали с использованием конфокального микроскопа Nikon A1R+, оснащенного четырехпроводной лазерной системой и объективом 20×.
[00243] CyTOF: опухоли диссоциировали вручную, расщепляли с использованием либразы TL (Roche) и ДНКазы I в течение 30 минут при температуре 37°C и пропускали через фильтр с ячейками 70 мкм. Затем образцы центрифугировали с использованием прерывистого градиента среды Histopaque 1119 (Sigma-Aldrich) и RMPI. Суспензии отдельных клеток размером до 2,5×106 клеток на образец блокировали Fc-рецептором и окрашивали смесью поверхностных антител в течение 30 минут при температуре 4°С. Антитела, конъюгированные с металлом, приобретали у Fluidigm или конъюгировали с использованием наборов для мечения полимерных антител X8 в соответствии с протоколом производителя (Fluidigm). Образцы окрашивали с использованием 2,5 мкМ 194Pt-цисплатина (Fluidigm) в течение 1 минуты и дважды промывали 2% FCS PBS. Клетки кодировали с использованием способа с использованием подхода к штриховому кодированию посредством палладиевой масс-метки в соответствии с протоколом изготовителя (Fluidigm) и объединяли после двух промывок с 2% FCS PBS. Затем клетки фиксировали и пермеабилизировали с использованием набора для окрашивания фактора транскрипции FoxP3 в соответствии с протоколом производителя (eBioscience). Затем образцы окрашивали с использованием смеси антител к внутриклеточным мишеням в течение 30 минут при комнатной температуре. Образцы дважды промывали 2% FCS PBS, а затем инкубировали в течение ночи в растворе 1,6% PFA/100 нМ иридий/PBS перед сбором данных с использованием масс-цитометра Helios (Fluidigm).
[00244] Данные масс-цитометрии нормировали с помощью шариков, а прочтение штрих-кодов проводили с использованием программного обеспечения Fluidigm. Все живые и CD45+ клетки вручную гейтировали с использованием FlowJo. Анализ t-SNE проводили на всех живых и CD45+ клетках с использованием пакета Cyt в Matlab. Данные подвергали арксинус-преобразованию с использованием коэффициента 4 и случайным образом понижали выборку до 50000 событий на выборку перед анализом t-SNE. Затем получали графики t-SNE для каждой экспериментальной группы путем слияния выборок из каждой группы и отображения равного количества случайных пониженных выборок (50000) из каждой группы лечения.
[00245] Таблица 9: Панель CyTOF.
[00246] Все способы, раскрытые и заявленные в настоящем документе, могут быть выполнены и осуществлены без чрезмерных экспериментов в свете настоящего раскрытия. Хотя композиции и способы по настоящему изобретению были описаны в терминах предпочтительных вариантов осуществления, специалистам в настоящей области техники должно быть очевидно, что к способам и на стадиях или в последовательности стадий описанного способа могут применяться изменения описанного в настоящем документе способа, не отступая от концепции, сущности и объема настоящего изобретения. Более конкретно, будет очевидно, что определенные средства, которые являются как химически, так и физиологически родственными, могут заменить описанные в настоящем документе средства, при этом будут достигнуты такие же или аналогичные результаты. Все такие аналогичные заменители и модификации, очевидные для специалистов в настоящей области техники, считаются находящимися в пределах сущности, объема и концепции настоящего изобретения, как определено в прилагаемой формуле изобретения.
ССЫЛКИ
Следующие ссылки в той мере, в которой они предоставляют иллюстративные процедурные или иные подробности, дополняющие изложенные в настоящем документе, специально включены в настоящий документ посредством ссылки.
A framework for human microbiome research. Nature 486, 215-221, 2012.
Caporaso et al., The ISME journal 6, 1621-1624, 2012.
Caspi et al., Nucleic Acids Research 36, D623-D631, 2008.
Chen et al., Cancer Discovery 2016.
Cooper et al., Cancer Immunology Research 2015.
Fritz et al., Journal of experimental psychology: General 141, 2, 2012.
Hurwitz et al., Proc Natl Acad Sci USA 95(17): 10067-10071, 1998.
Публикация Международной заявки на патент № WO00/37504
Публикация Международной заявки на патент № WO01/14424
Публикация Международной заявки на патент № WO01/14424
Публикация Международной заявки на патент № WO1995/001994
Публикация Международной заявки на патент № WO1998/042752
Публикация Международной заявки на патент № WO2000/037504
Публикация Международной заявки на патент № WO2001/014424
Публикация Международной заявки на патент № WO2005/003168
Публикация Международной заявки на патент № WO2005/009465
Публикация Международной заявки на патент № WO2006/00317
Публикация Международной заявки на патент № WO2006/072625
Публикация Международной заявки на патент № WO2006/072626
Публикация Международной заявки на патент № WO2006/121168
Публикация Международной заявки на патент № WO2007/042573
Публикация Международной заявки на патент № WO2008/084106
Публикация Международной заявки на патент № WO2008132601
Публикация Международной заявки на патент № WO2009/101611
Публикация Международной заявки на патент № WO2009/114335
Публикация Международной заявки на патент № WO2009044273
Публикация Международной заявки на патент № WO2010/027827
Публикация Международной заявки на патент № WO2010/065939
Публикация Международной заявки на патент № WO2011/0008369
Публикация Международной заявки на патент № WO2011/014438
Публикация Международной заявки на патент № WO2011/066342
Публикация Международной заявки на патент № WO2012/071411
Публикация Международной заявки на патент № WO2012/160448
Публикация Международной заявки на патент № WO2013/006490
Публикация Международной заявки на патент № WO2013/025779
Публикация Международной заявки на патент № WO2013/067492
Публикация Международной заявки на патент № WO2014/022021
Публикация Международной заявки на патент № WO2015/016718
Публикация Международной заявки на патент № WO96/15660
Публикация Международной заявки на патент № WO98/42752
Jones et al., J Exp Med. 205(12):2763-79, 2008.
Kanehisa et al., Nucleic Acids Res 28, 27-30, 2000.
Li et al., Nat Biotech 32, 834-841, 2014.
Mellman et al., Nature 480:480- 489, 2011.
Muegge et al., Science 332, 970-974, 2011.
Nielsen et al., Nat Biotech 32, 822-828, 2014.
Okazaki T et al., Intern. Immun. 19(7):813, 2007.
Pardoll, Nature Rev Cancer 12:252-264, 2012.
Публикация патента № EP 2320940
Peled et al., Journal of Clinical Oncology 0, JCO.2016.2070.3348.
Price et al., PLOS ONE 5, e9490, 2010.
Qin et al., Nature 464, 59-65, 2010.
Schwartz et al., RECIST 1.1. European Journal of Cancer 62, 132-137, 2016.
Segata et al., Genome Biology 12, R60, 2011.
Shannon et al., BMC Bioinformatics 14, 217 10.1186/1471-2105-14-217, 2013.
Sivan et al., Science (New York, N.Y.) 350, 1084-1089, 2015.
Structure, function and diversity of the healthy human microbiome. Nature 486, 207-214, 2012.
Taur et al., Blood 124, 1174-1182, 2014.
Tsujikawa et al., Cell reports, 2017.
Tumeh et al., Nature 515, 568-571, 2014.
Turnbaugh et al., Nature 457, 480-484, 2009.
Патент США № 4870287
Патент США № 5760395
Патент США № 5763488
Патент США № 5885796
Патент США № 5844905
Патент США № 6207156
Патент США № 8008449
Патент США № 8017114
Патент США № 8119129
Патент США № 8329867
Патент США № 8354509
Патент США № 8735553
Публикация патента США № 2012/0177645
Публикация патента США № 2012/0294796
Публикация патента США № 2014/0294898
Vetizou et al., Science (New York, N.Y.) 350, 1079-1084, 2015.
| название | год | авторы | номер документа |
|---|---|---|---|
| Кормовая добавка для жвачных животных | 2022 |
|
RU2794794C1 |
| КОМПОЗИЦИЯ ФЕКАЛЬНОЙ МИКРОБИОТЫ ДЛЯ ПРИМЕНЕНИЯ ДЛЯ УМЕНЬШЕНИЯ ИНДУЦИРОВАННОГО ЛЕЧЕНИЕМ ВОСПАЛЕНИЯ | 2019 |
|
RU2816462C2 |
| Кормовая добавка для крупного рогатого скота, улучшающая деструкцию структурных углеводов в рубце | 2023 |
|
RU2804123C1 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ УЛУЧШЕНИЯ ПОПУЛЯЦИИ КИШЕЧНОЙ МИКРОБИОТЫ И ИХ ОСУЩЕСТВЛЕНИЯ | 2013 |
|
RU2631595C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ ИНДУКЦИИ CD8+ T-КЛЕТОК | 2017 |
|
RU2761873C2 |
| СИСТЕМА ДЕТЕКЦИИ НАИБОЛЕЕ ЗНАЧИМЫХ ПРОКАРИОТИЧЕСКИХ ПРЕДСТАВИТЕЛЕЙ МИКРОБИОТЫ КИШЕЧНИКА ЧЕЛОВЕКА НА ОСНОВЕ ПЦР ПАНЕЛИ | 2017 |
|
RU2680268C1 |
| КОМПОЗИЦИИ И СПОСОБЫ | 2014 |
|
RU2664479C2 |
| МАНИПУЛЯЦИЯ МЕТАБОЛИЗМОМ ТРИПТАМИНОВ | 2018 |
|
RU2794244C2 |
| ВСПОМОГАТЕЛЬНЫЙ СПОСОБ ДИАГНОСТИКИ ДЛЯ ПРИМЕНЕНИЯ В ЛЕЧЕНИИ СИНДРОМА РАЗДРАЖЕННОГО КИШЕЧНИКА С ПРИМЕНЕНИЕМ КОРРЕКЦИОННОЙ ДИЕТЫ ИЛИ ТРАНСПЛАНТАЦИИ ФЕКАЛЬНОЙ МИКРОБИОТЫ | 2017 |
|
RU2762266C1 |
| КОРМОВАЯ ДОБАВКА ДЛЯ КРУПНОГО РОГАТОГО СКОТА | 2020 |
|
RU2744381C1 |
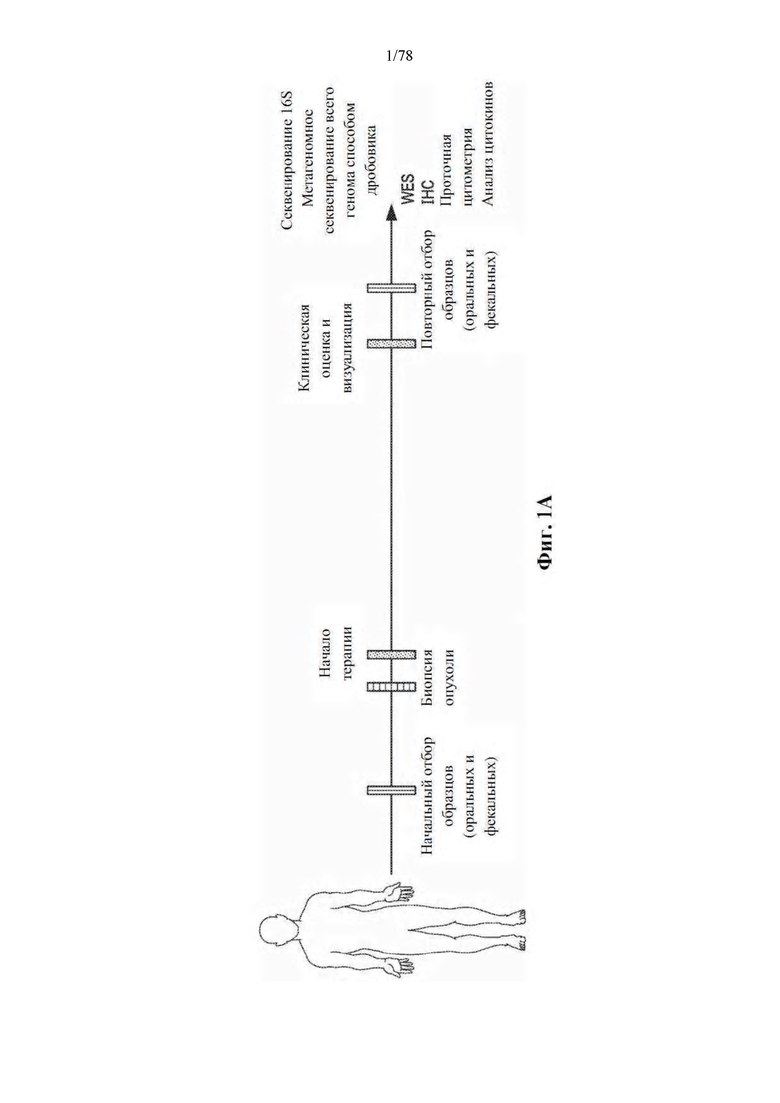
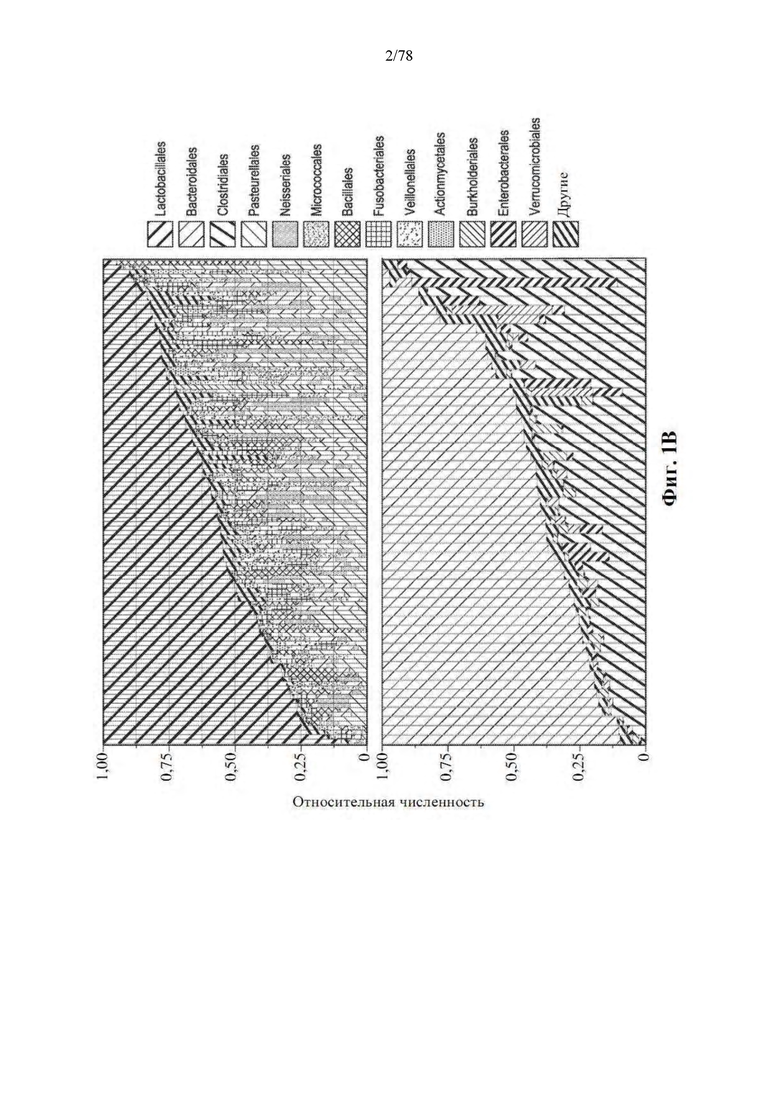

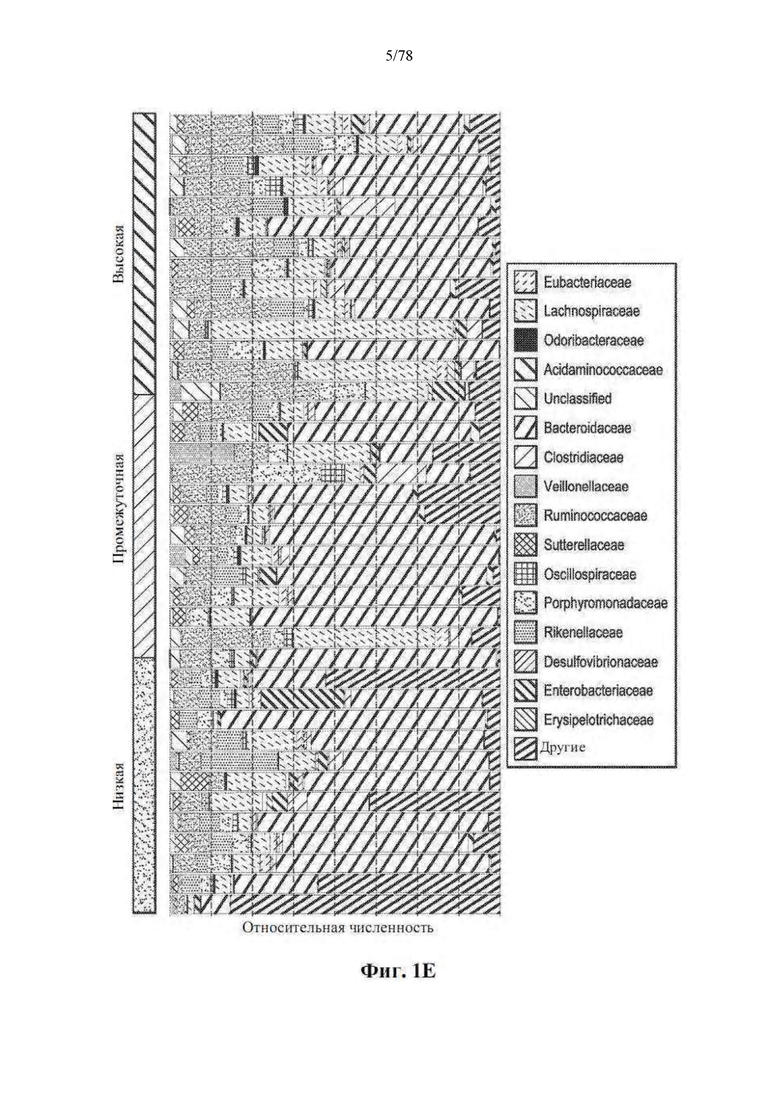
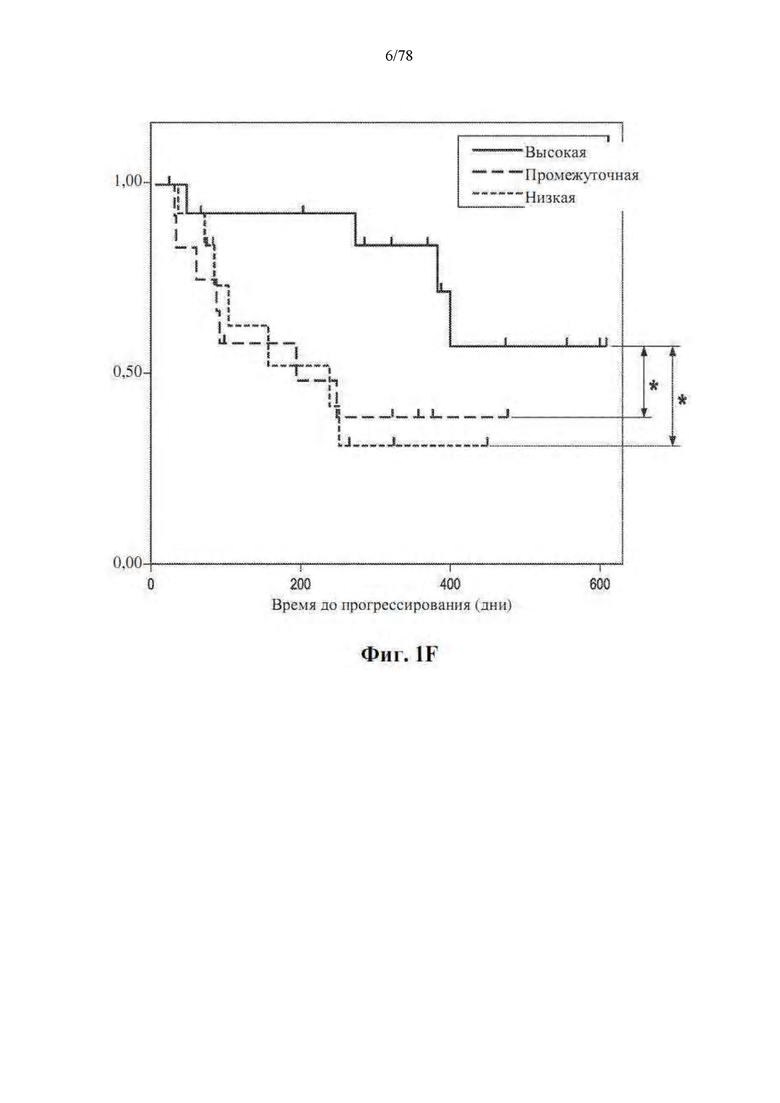
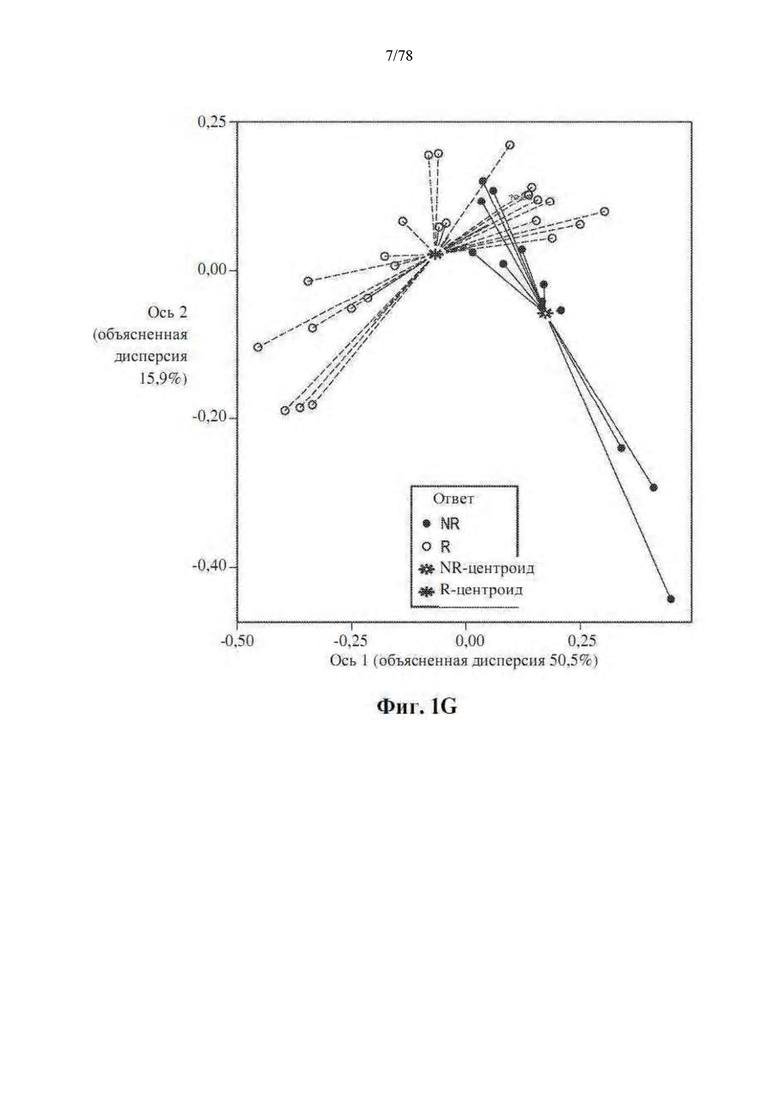
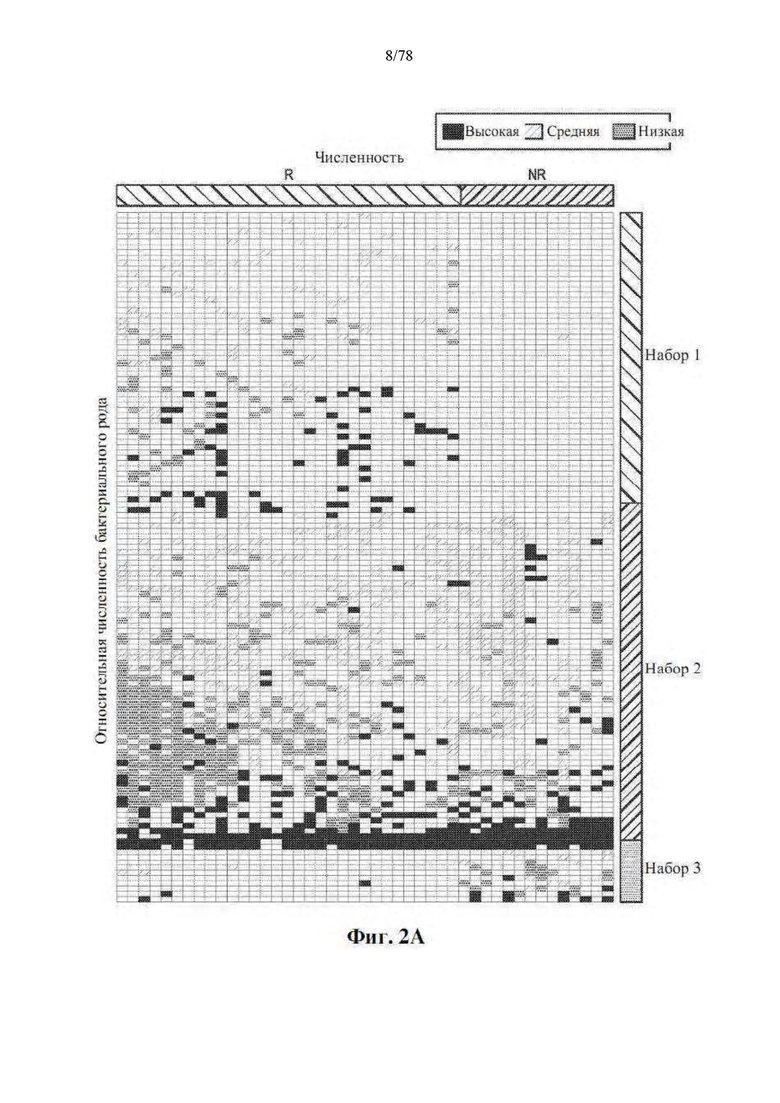
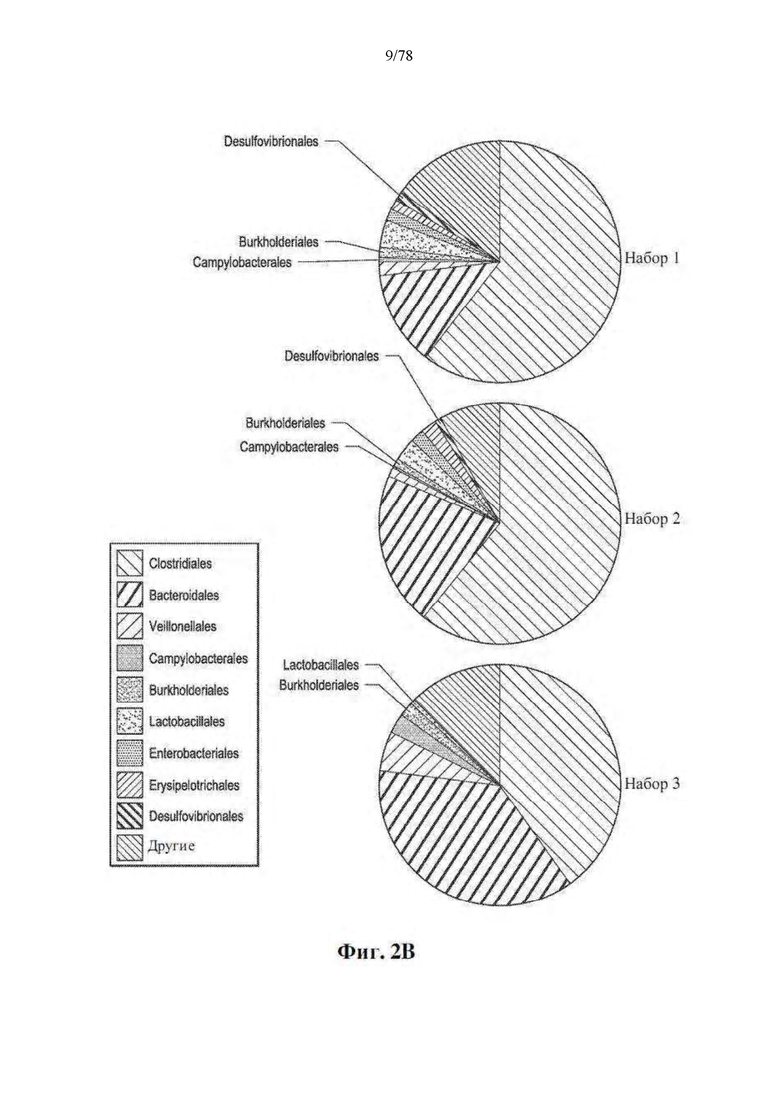
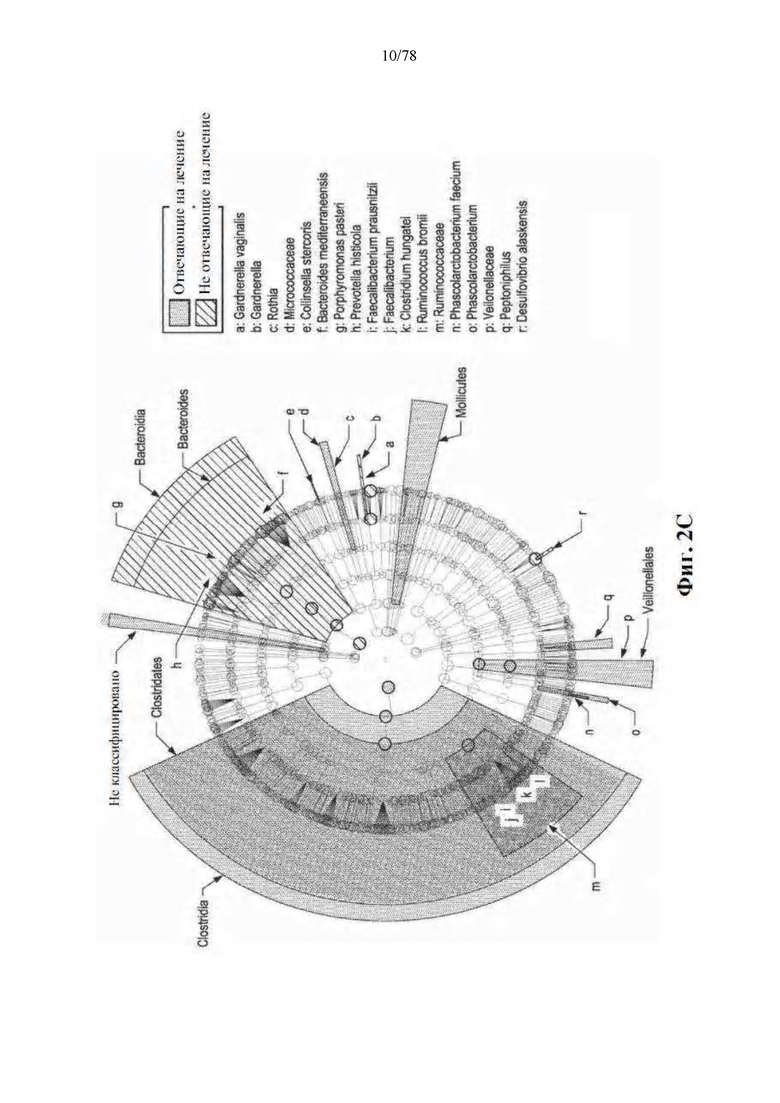
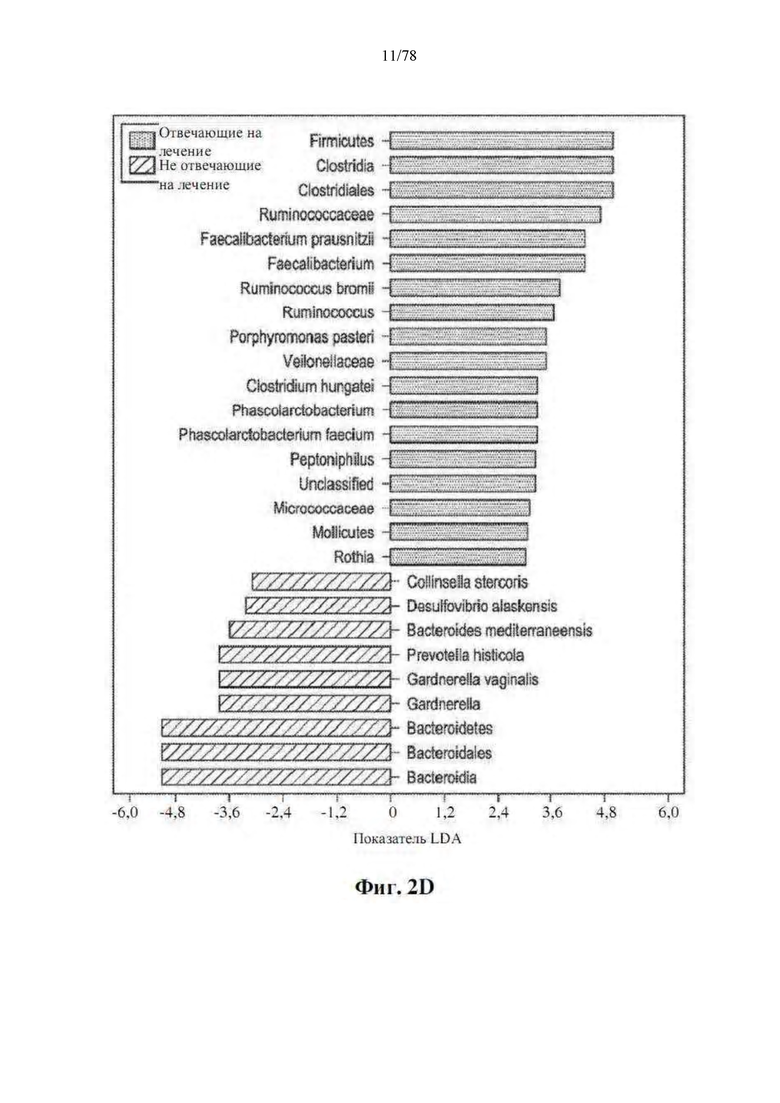
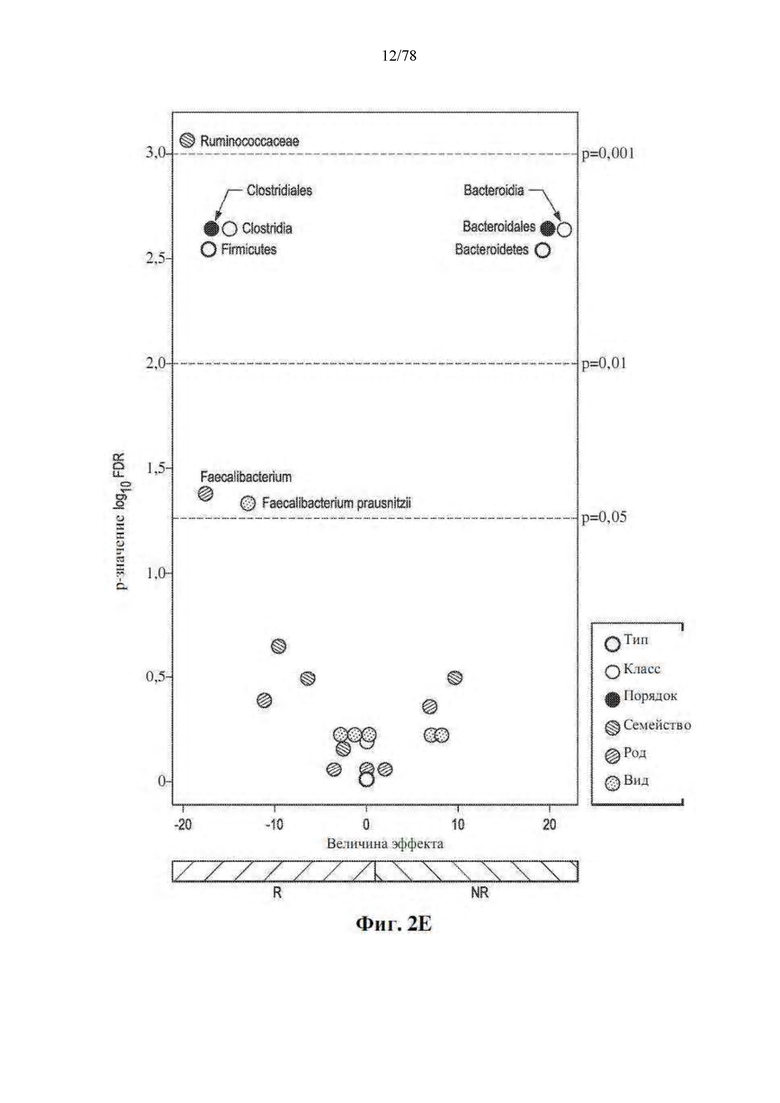


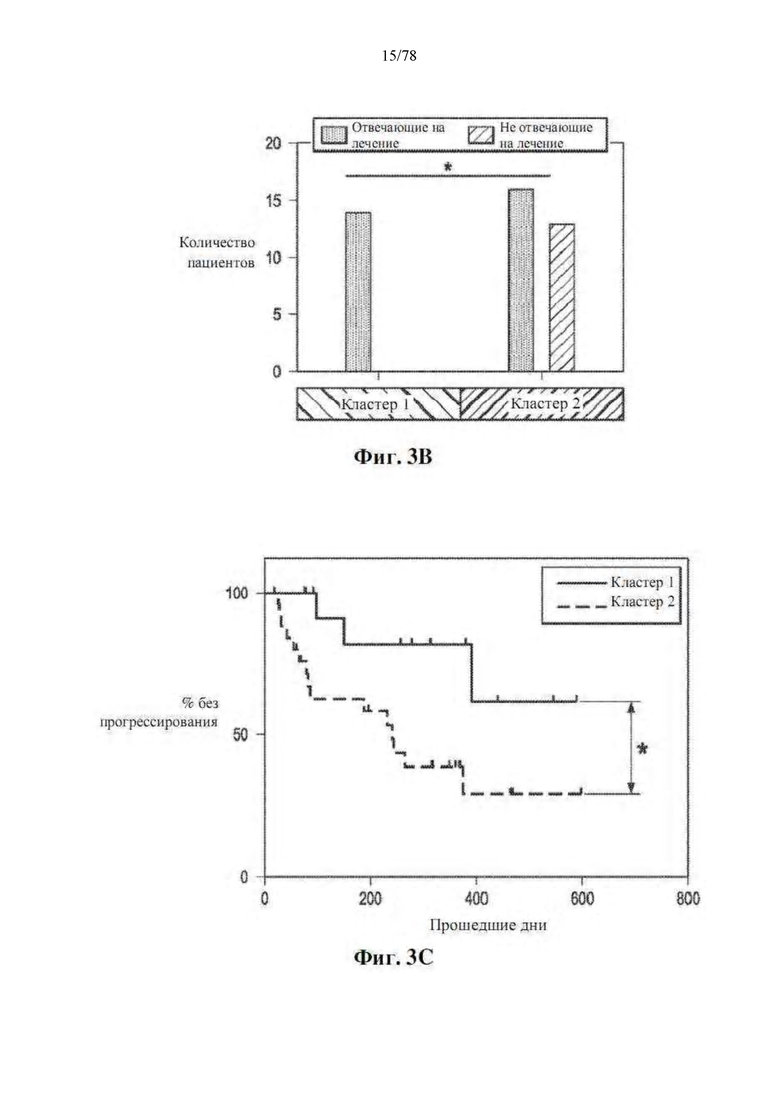
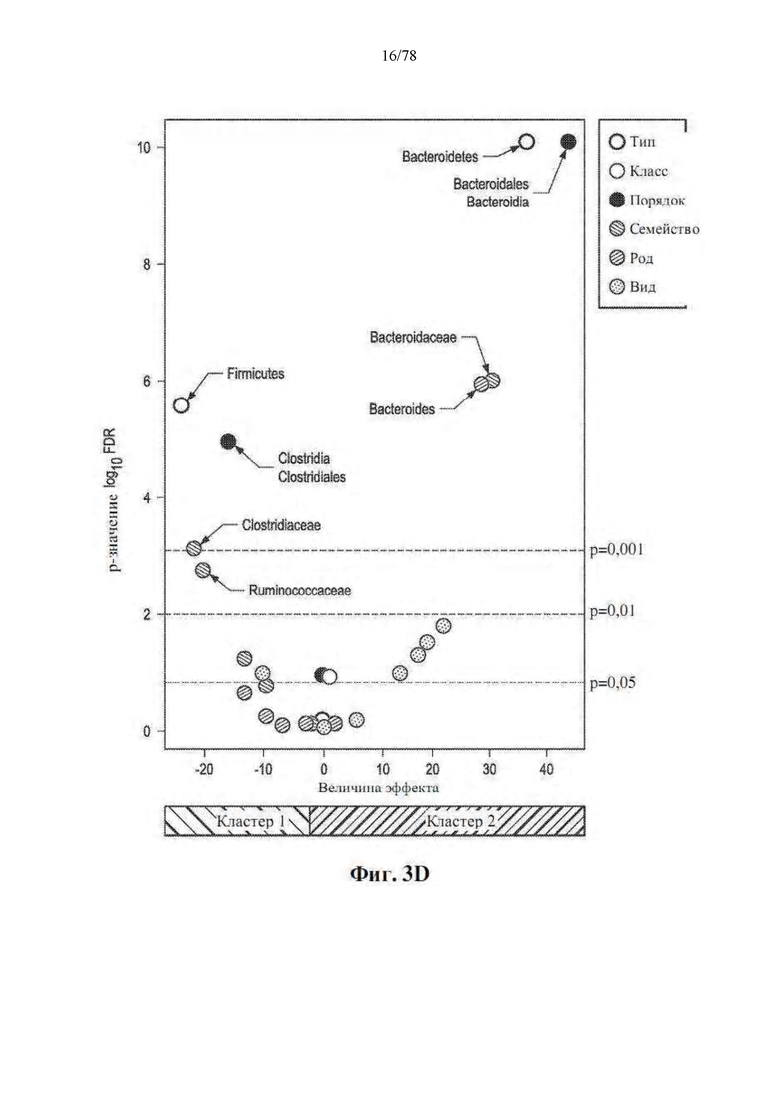
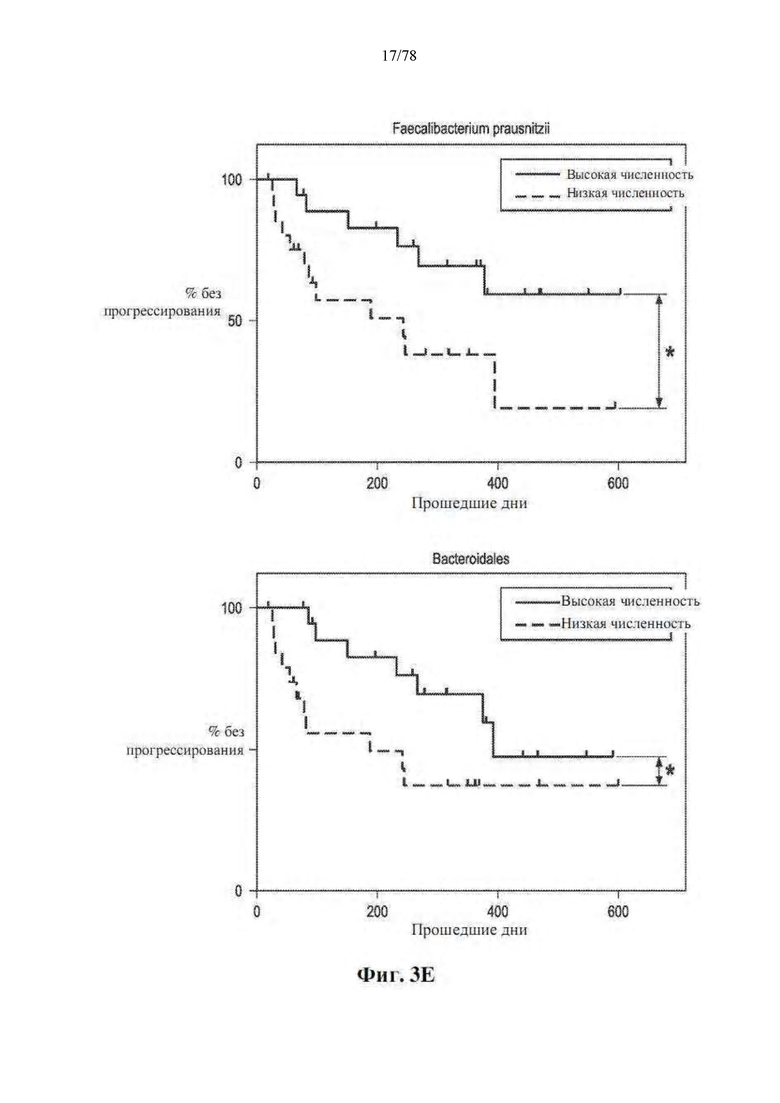

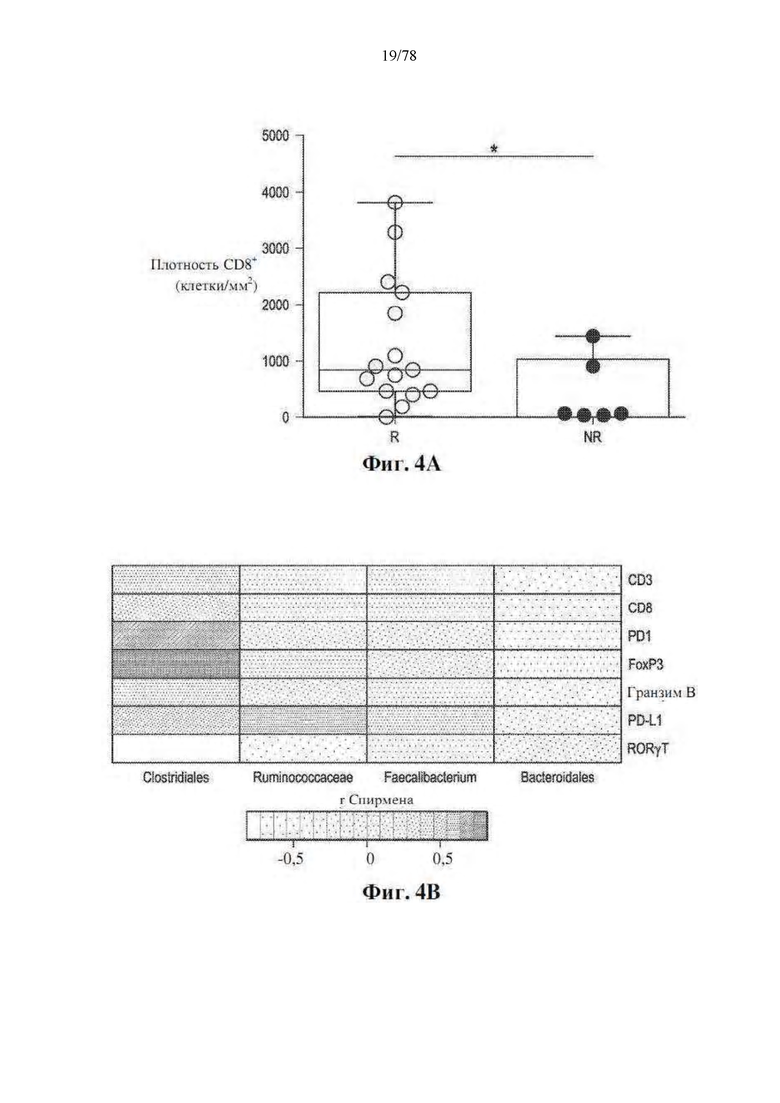
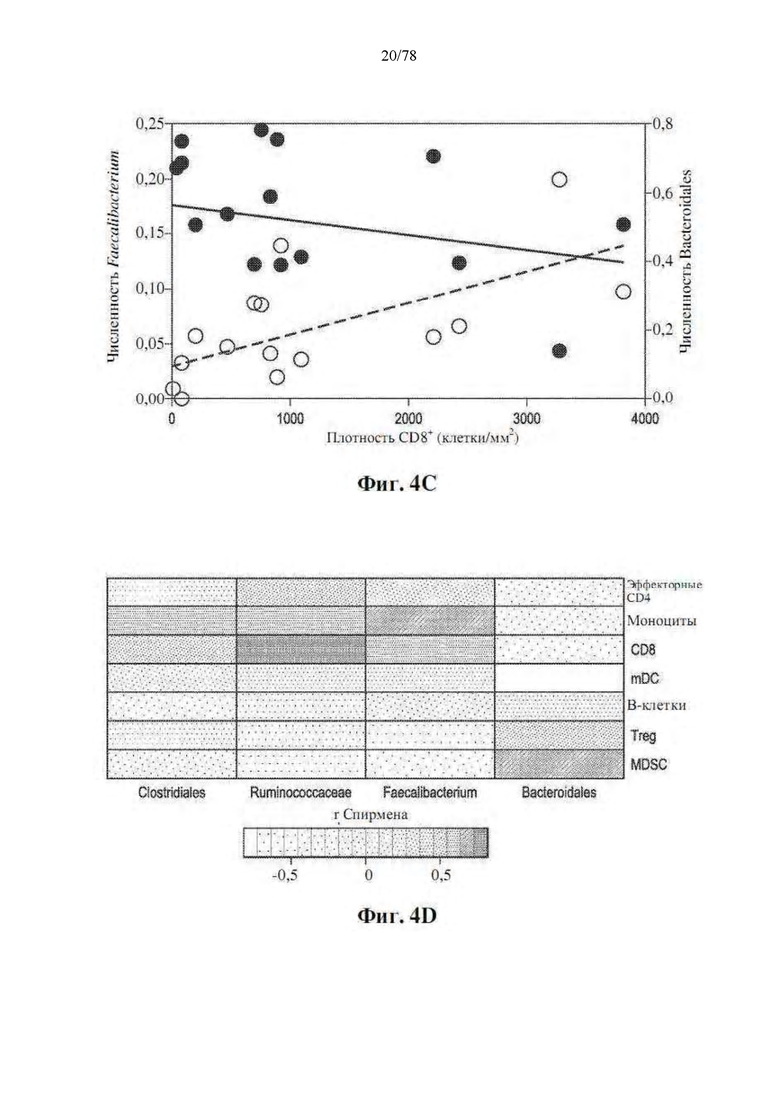

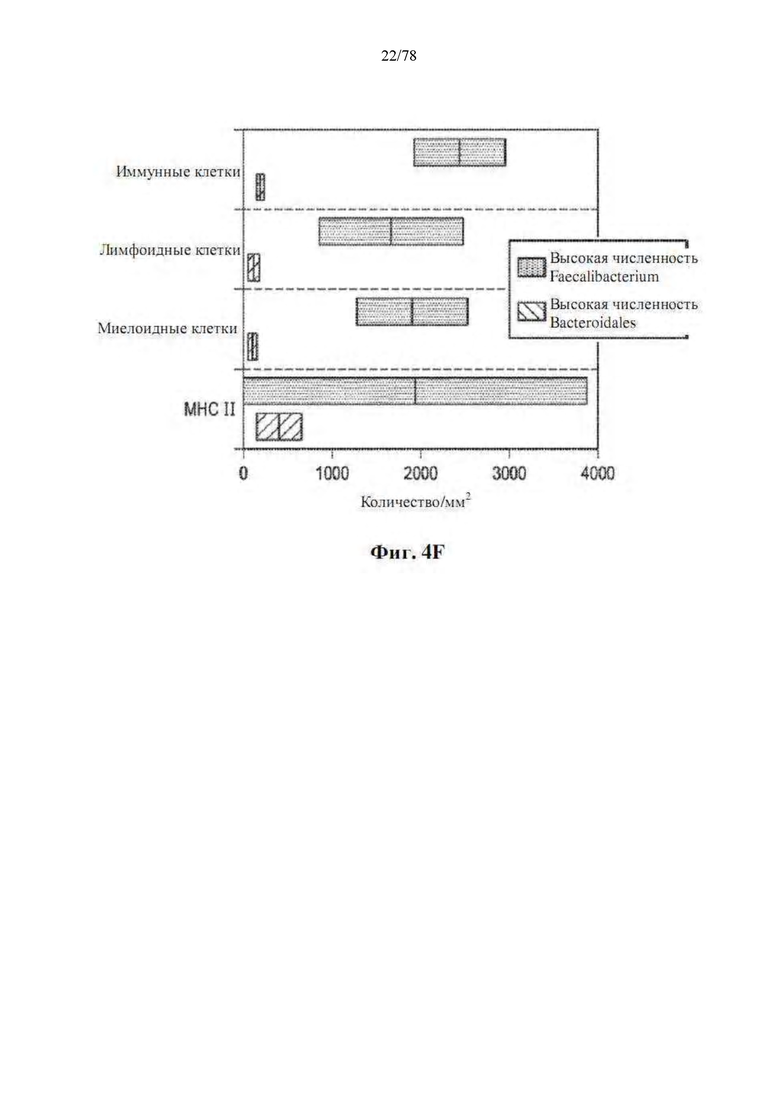
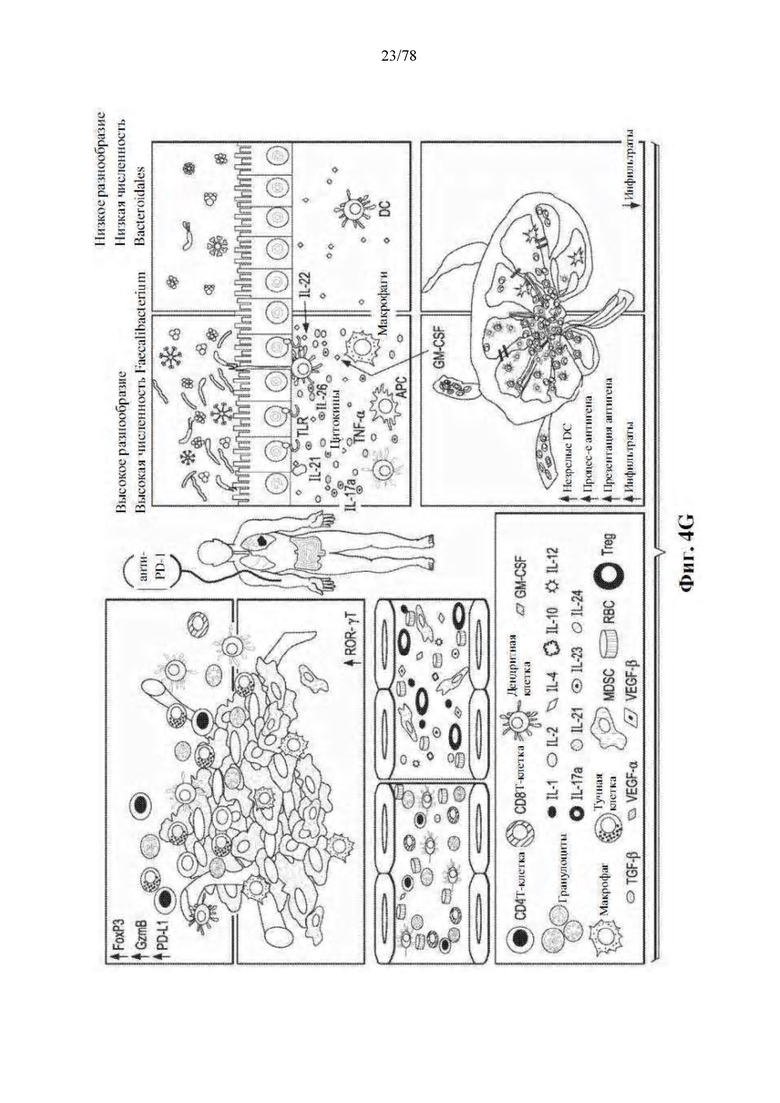
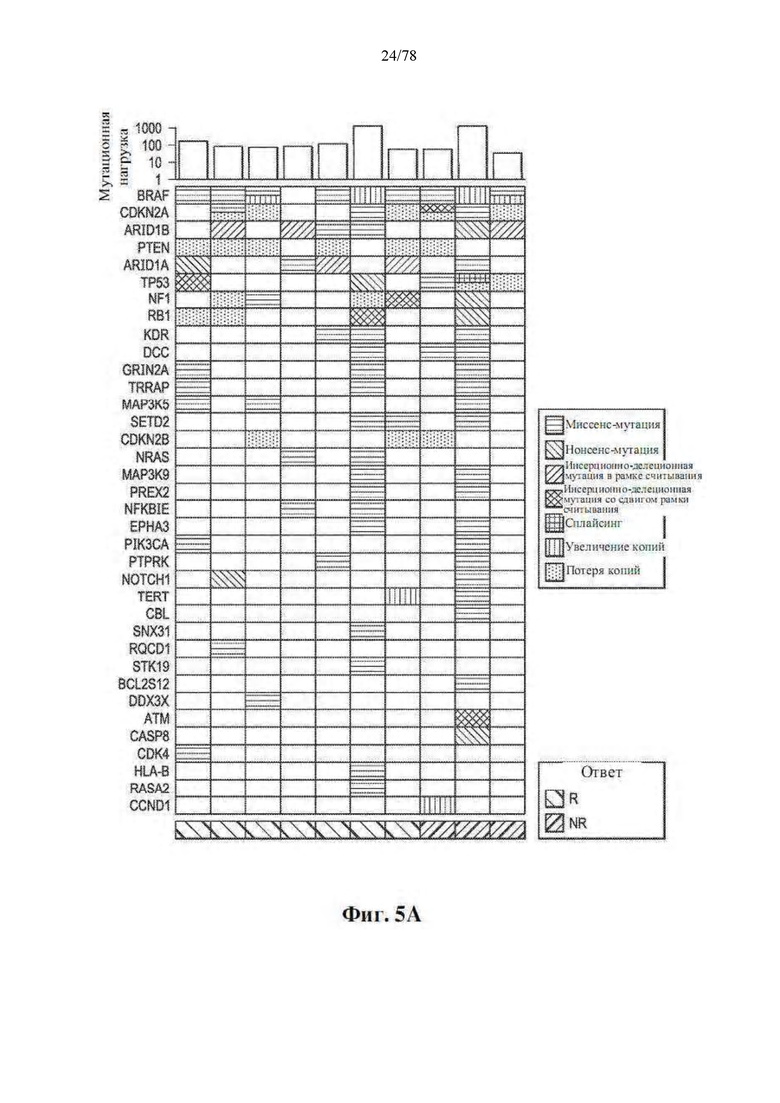
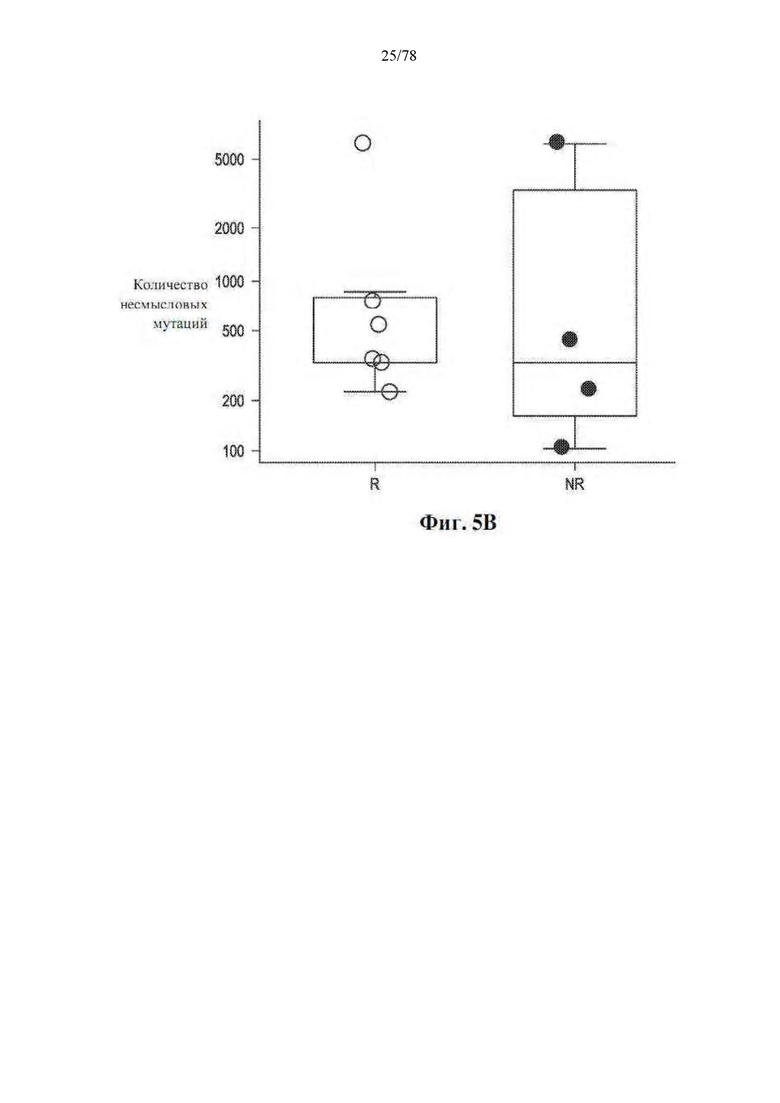

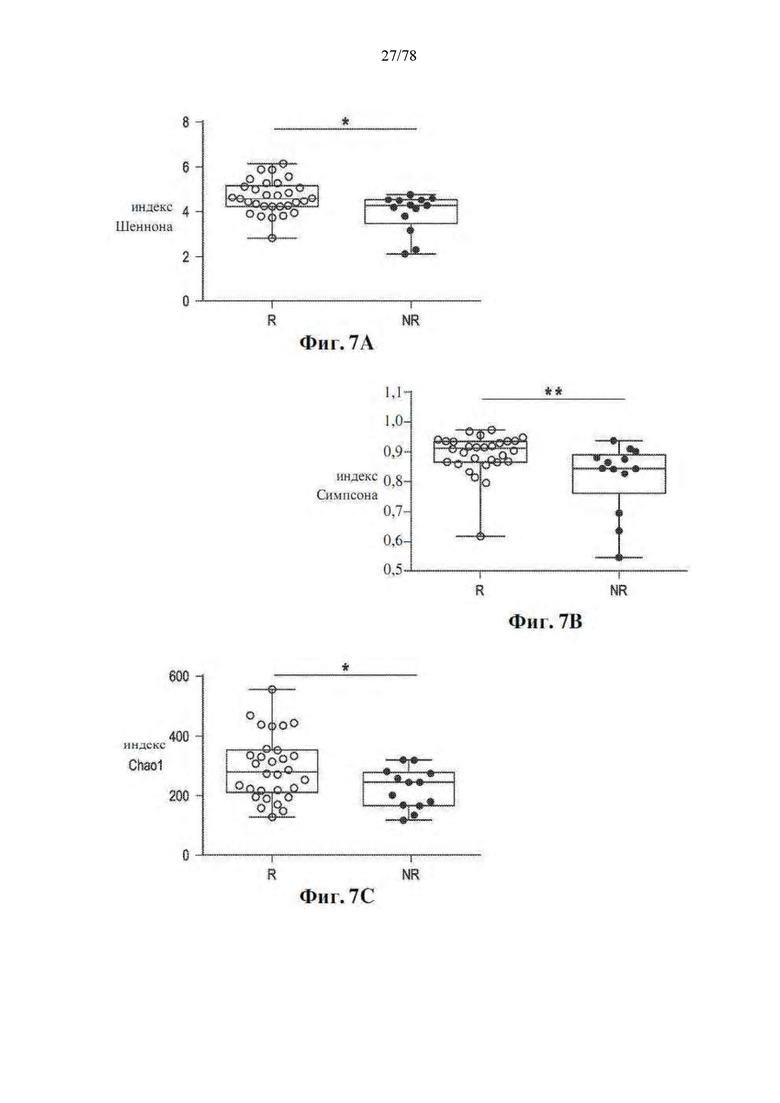

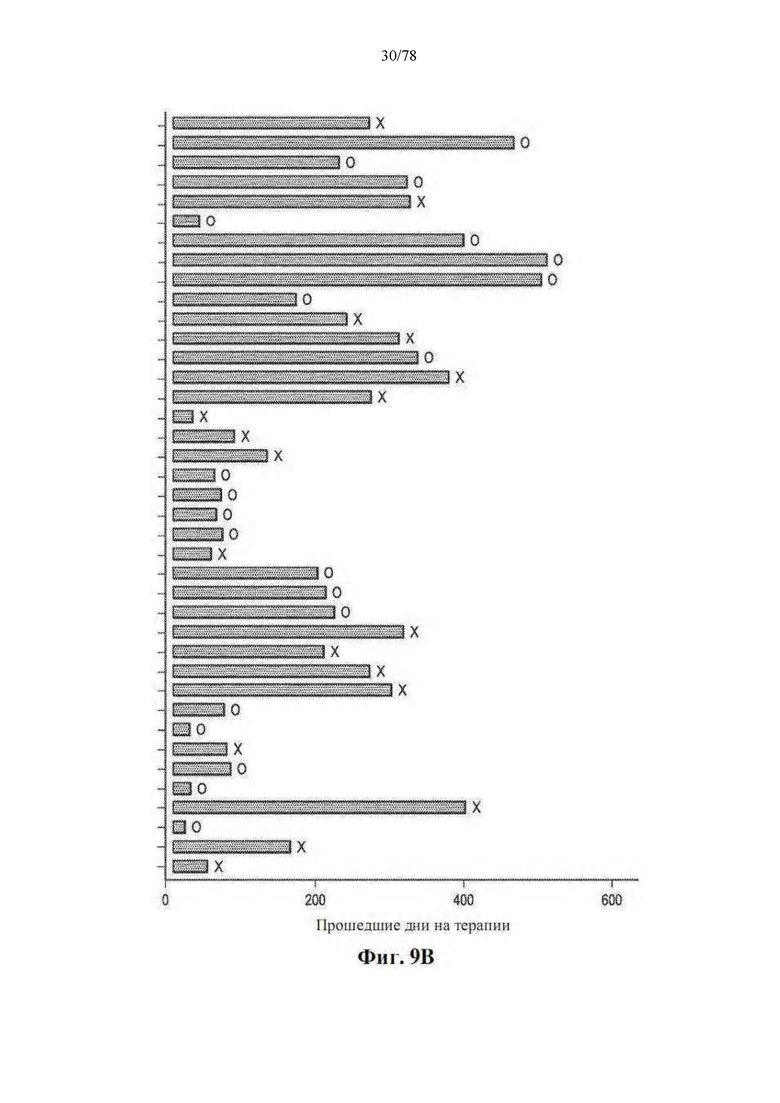
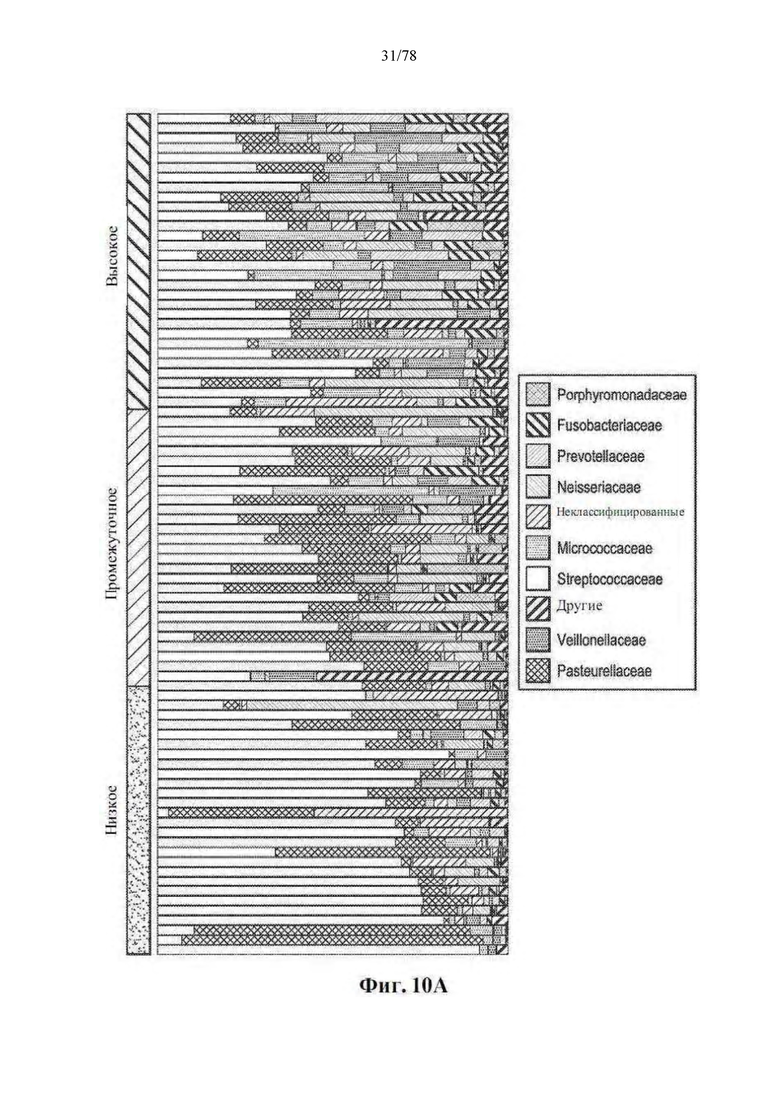
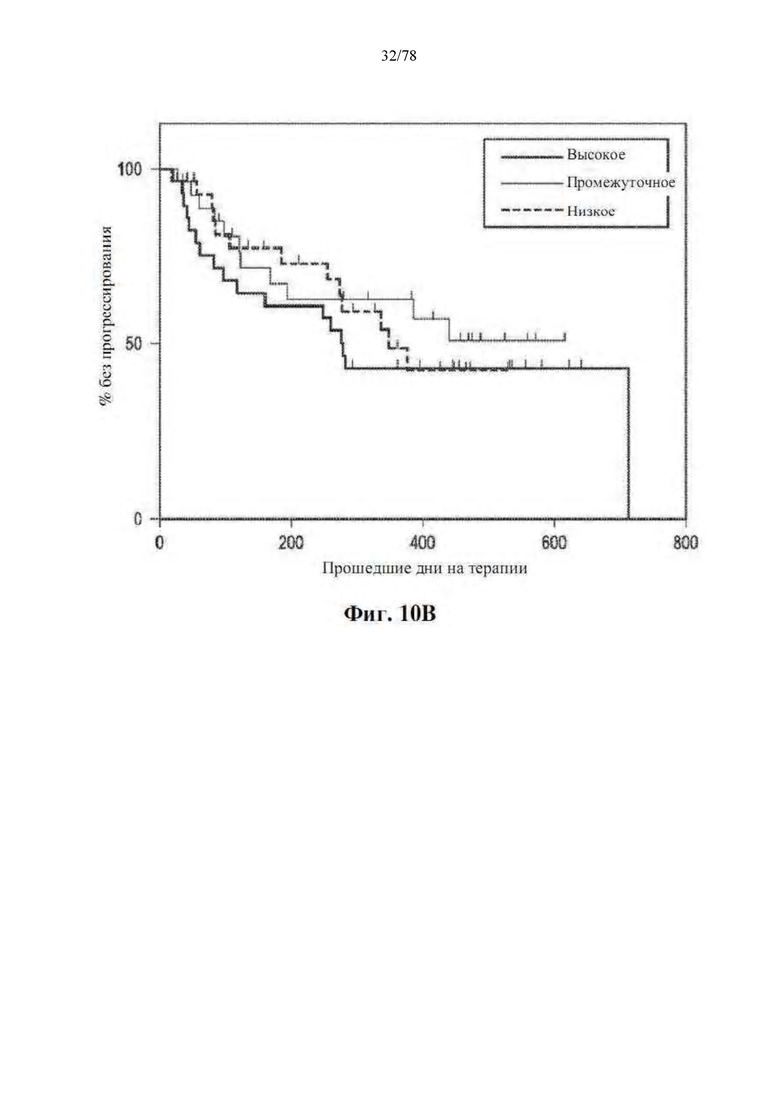
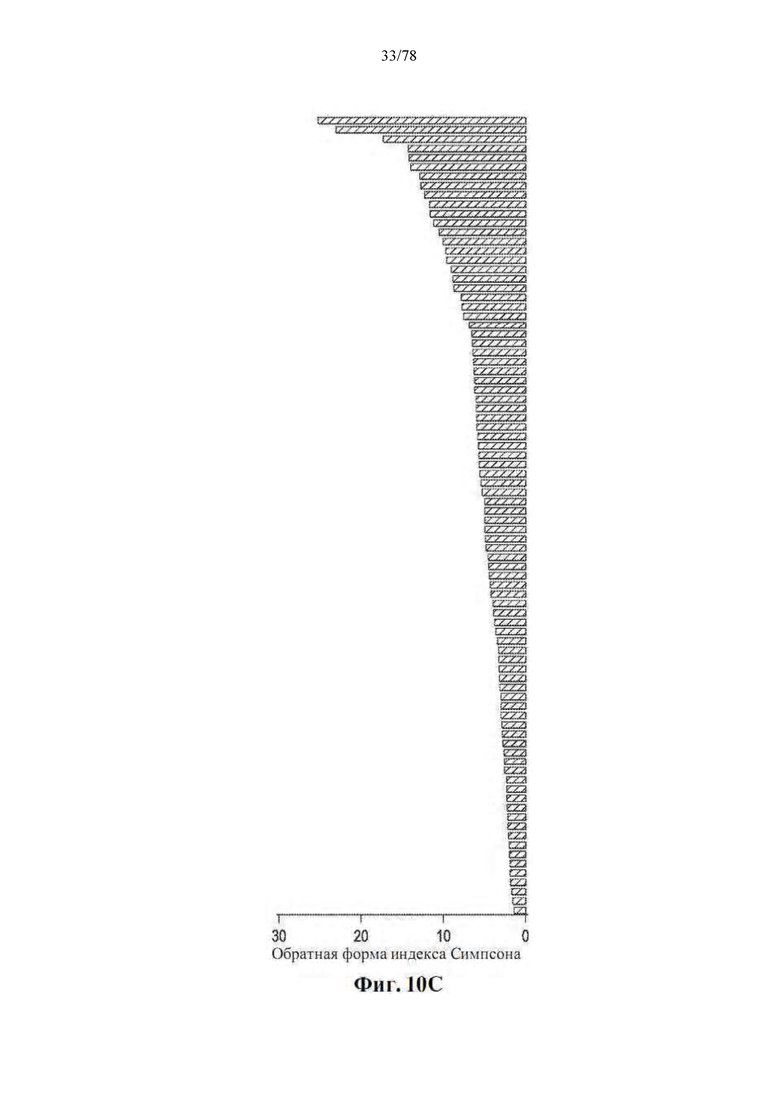

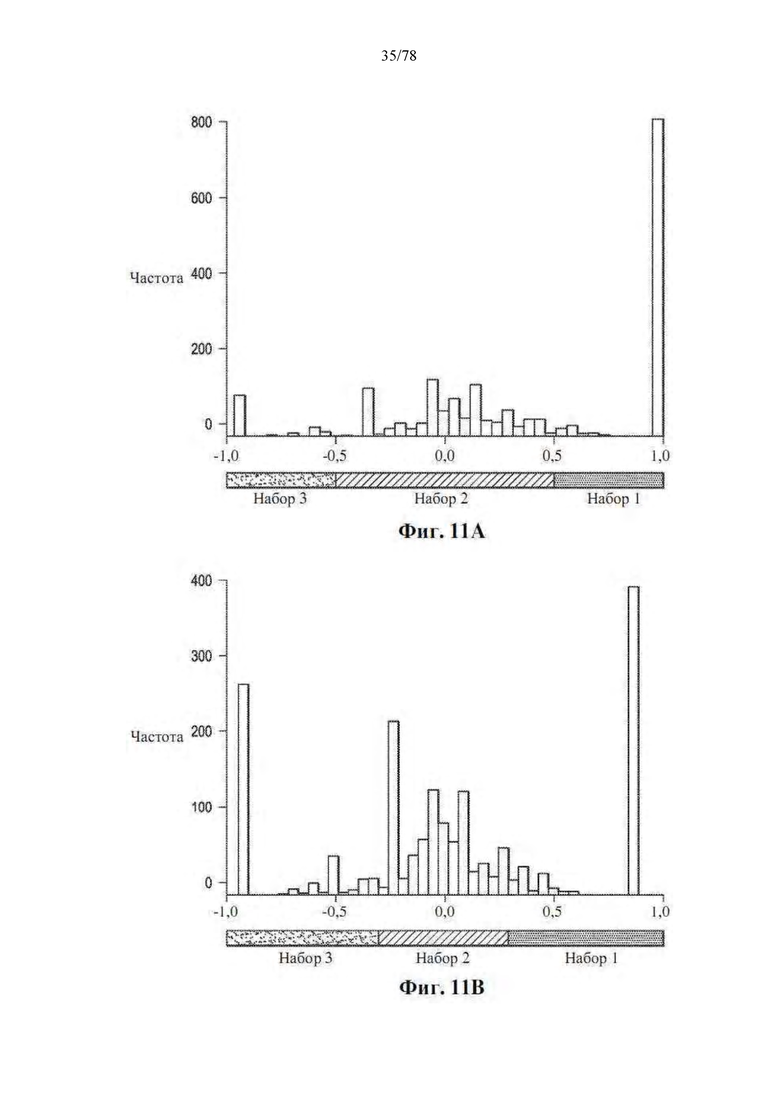
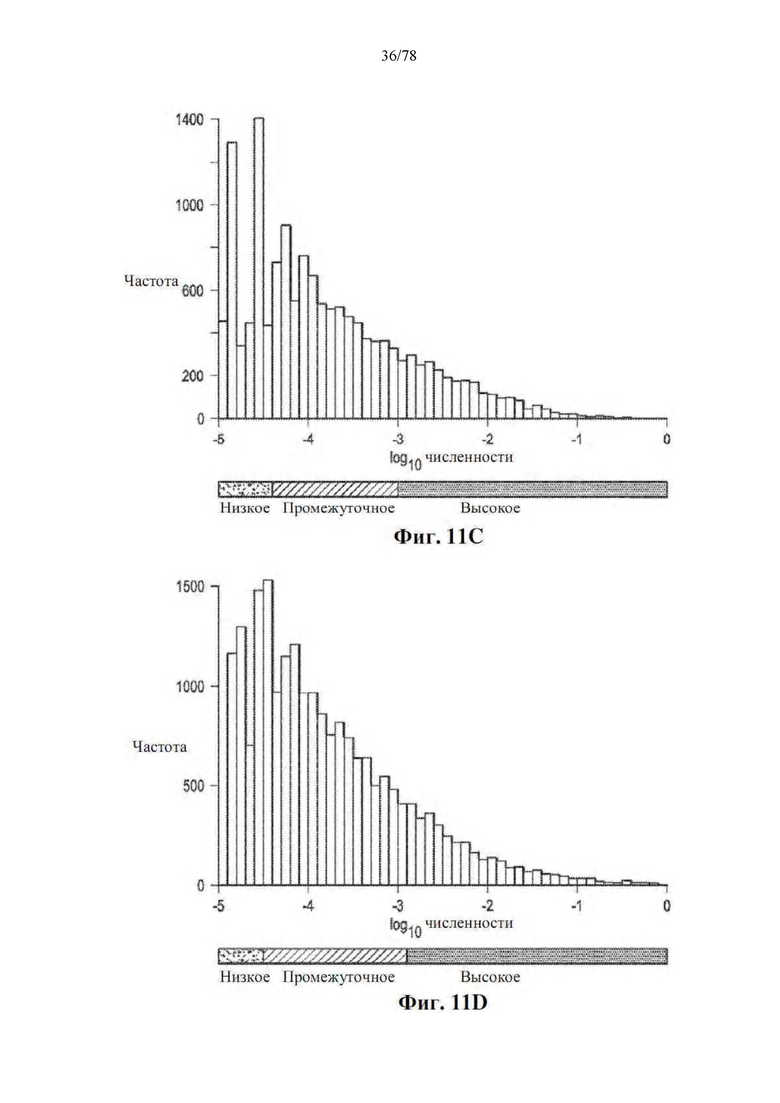

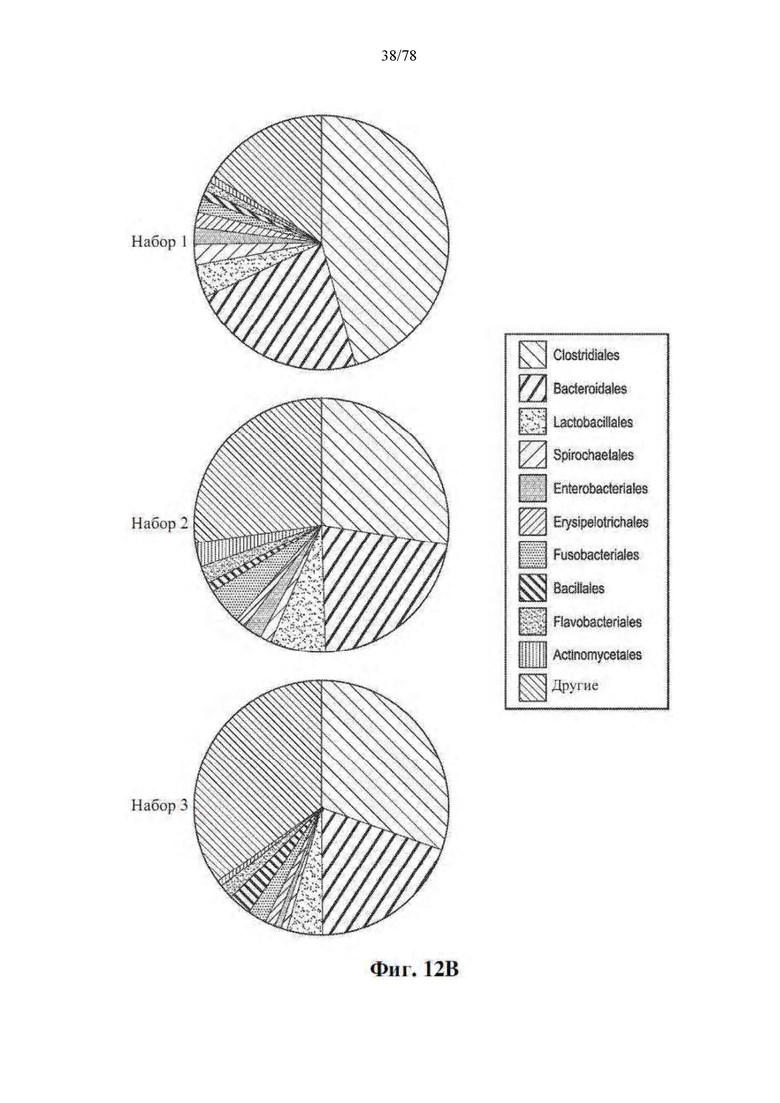
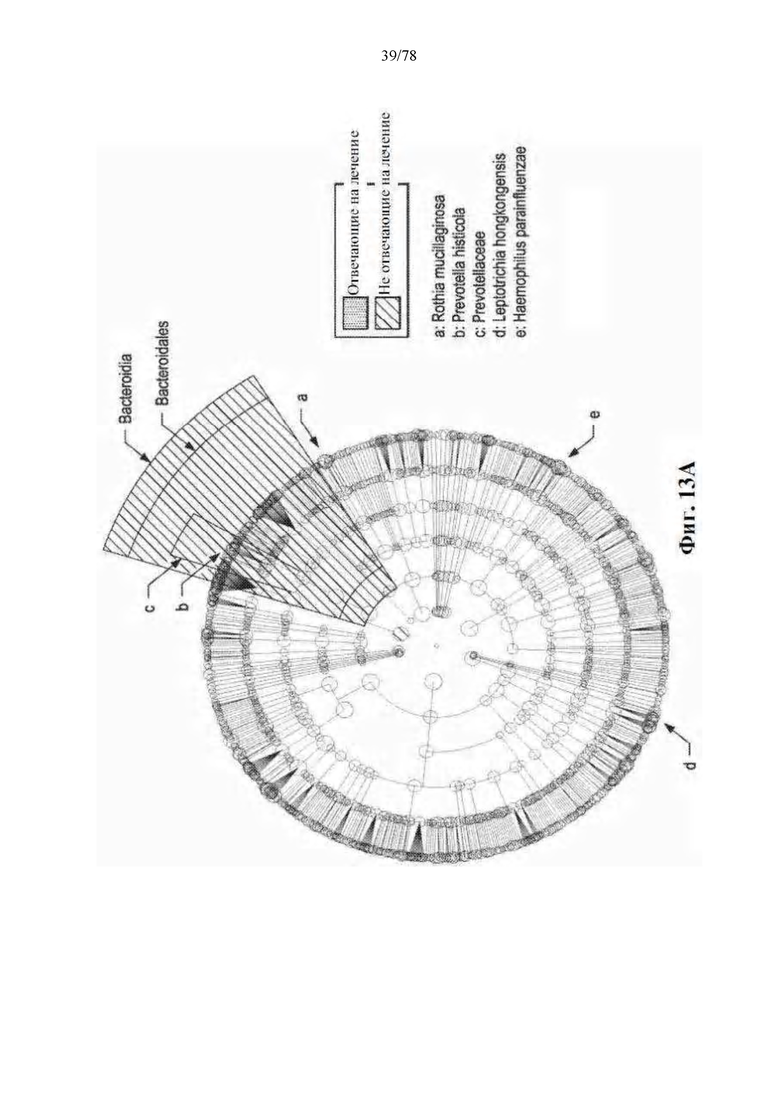
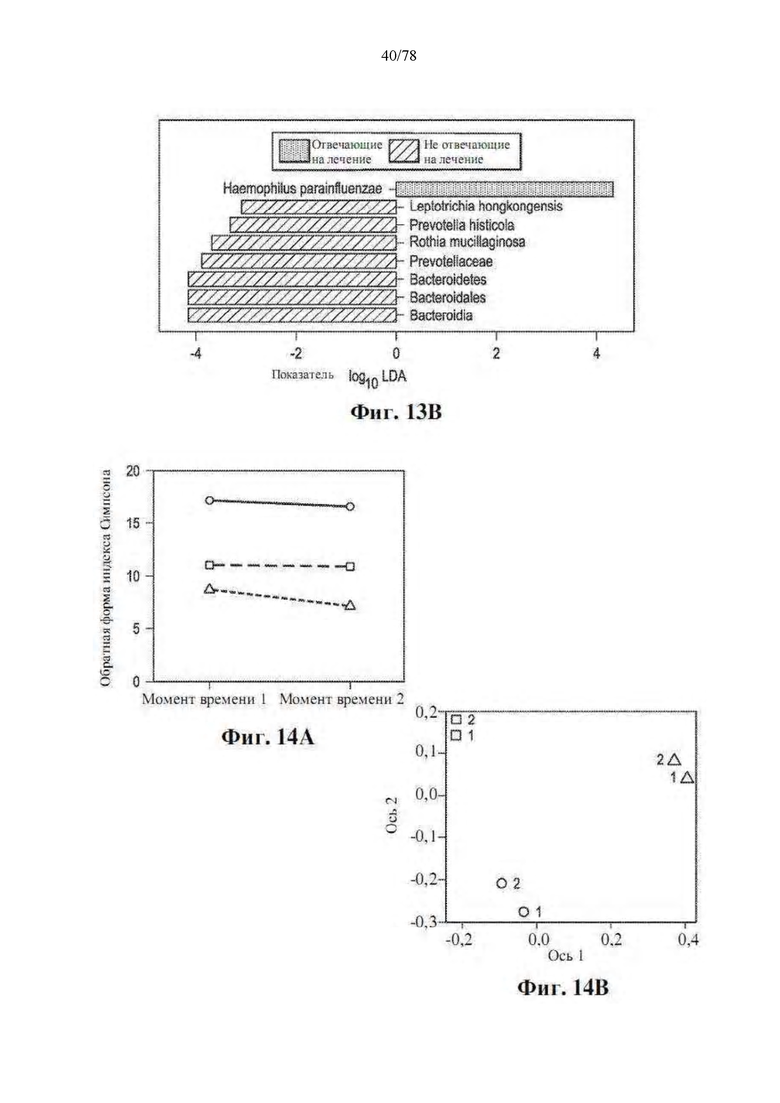
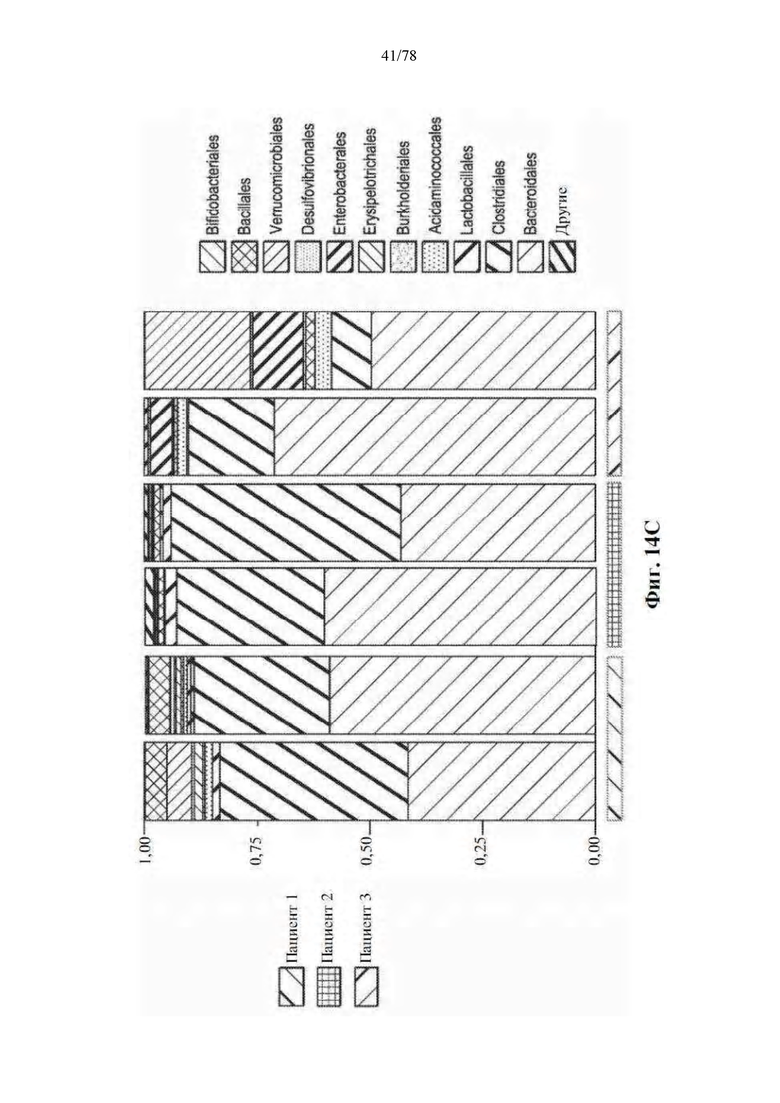
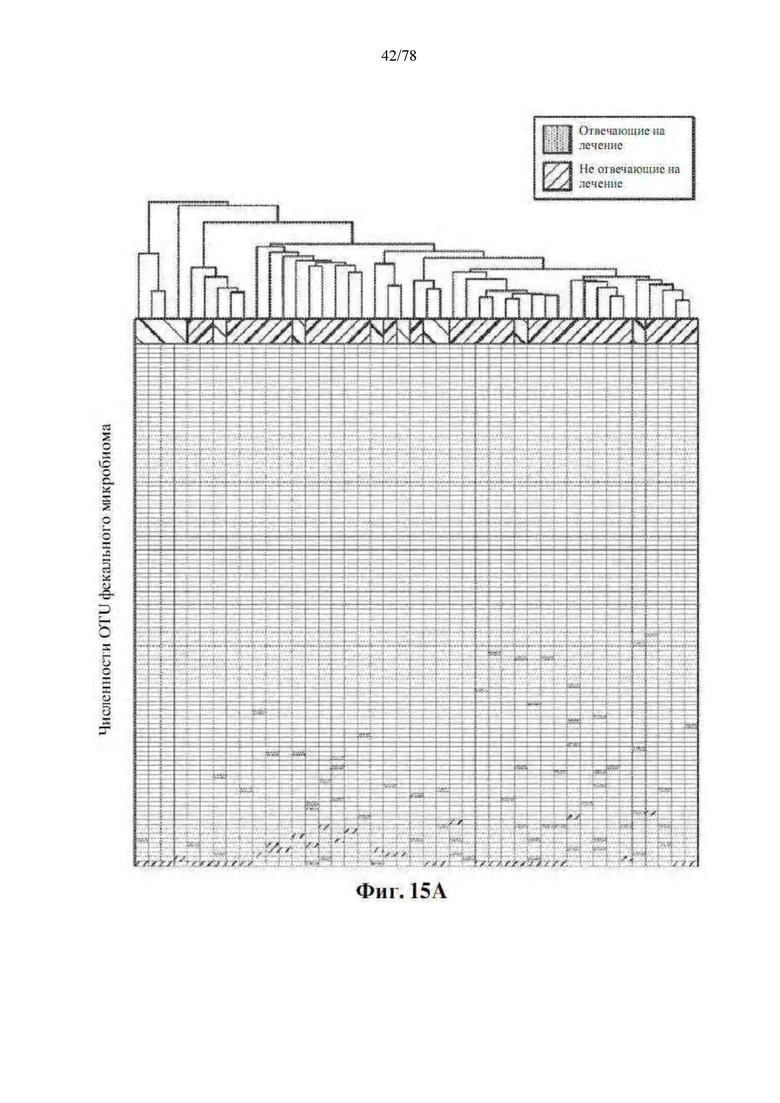

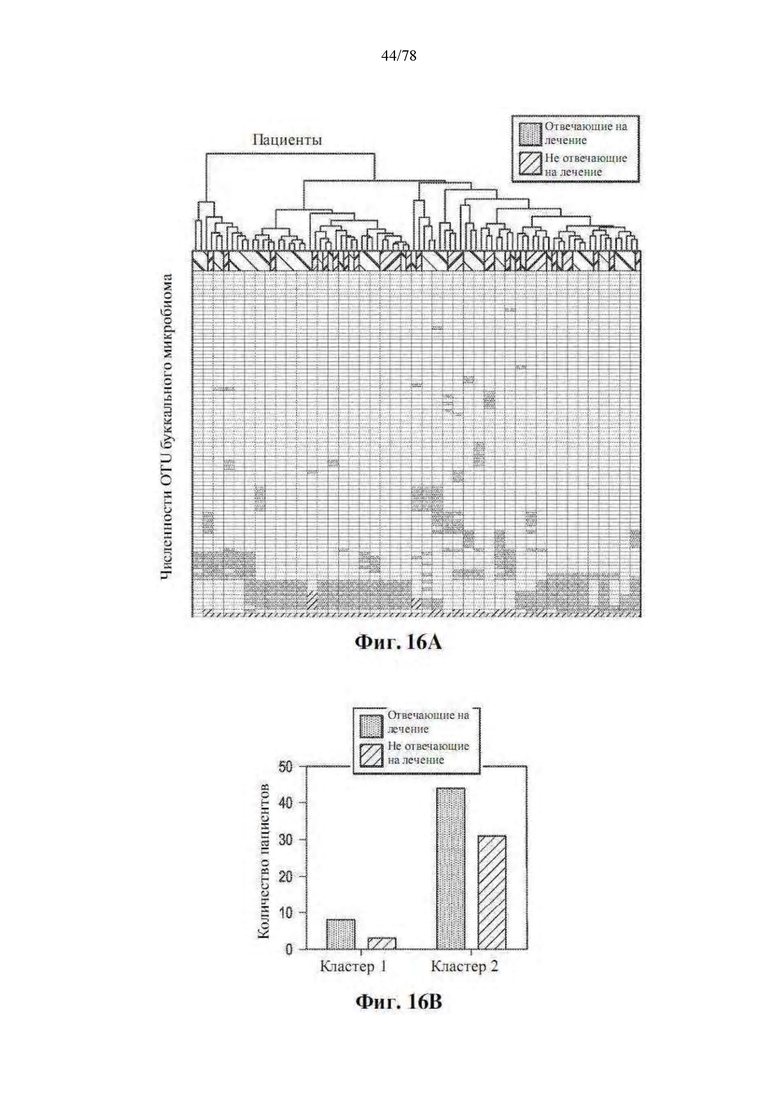
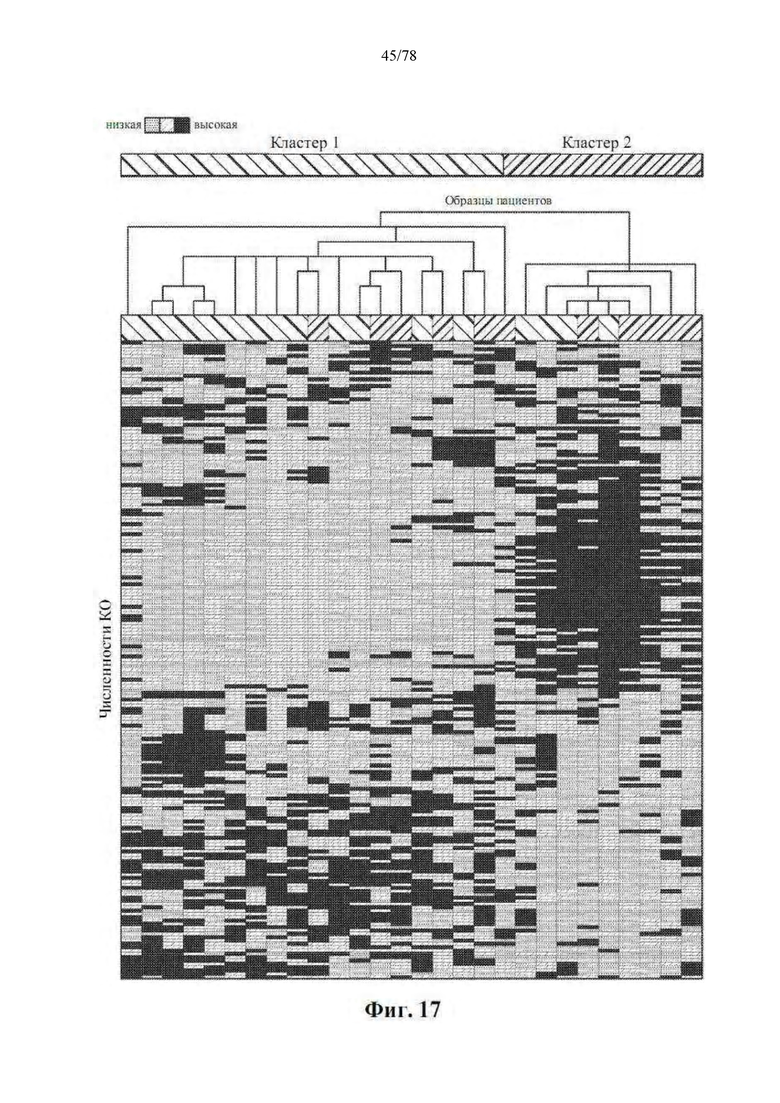
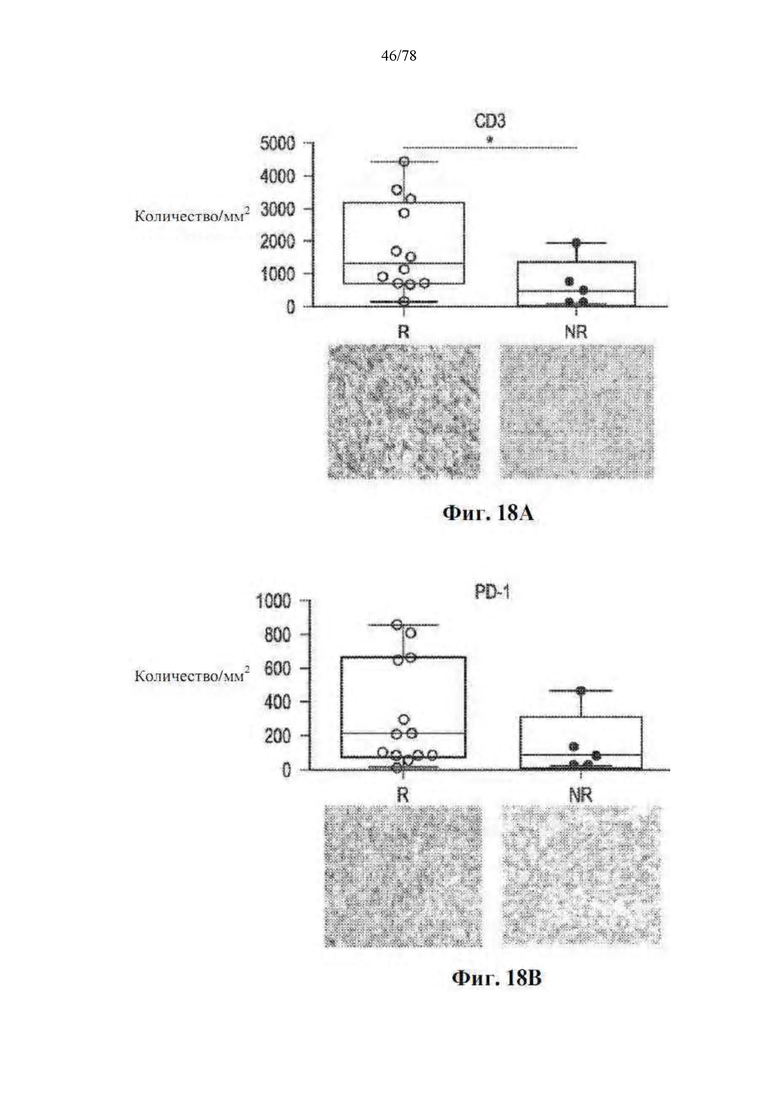
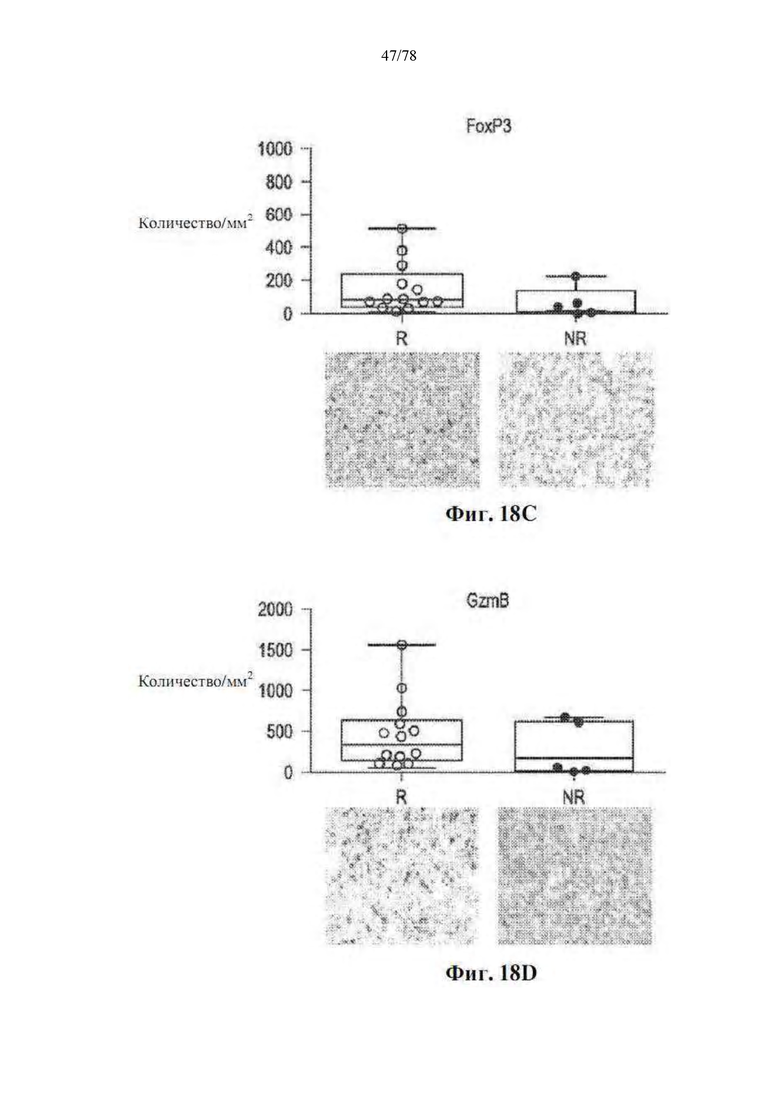
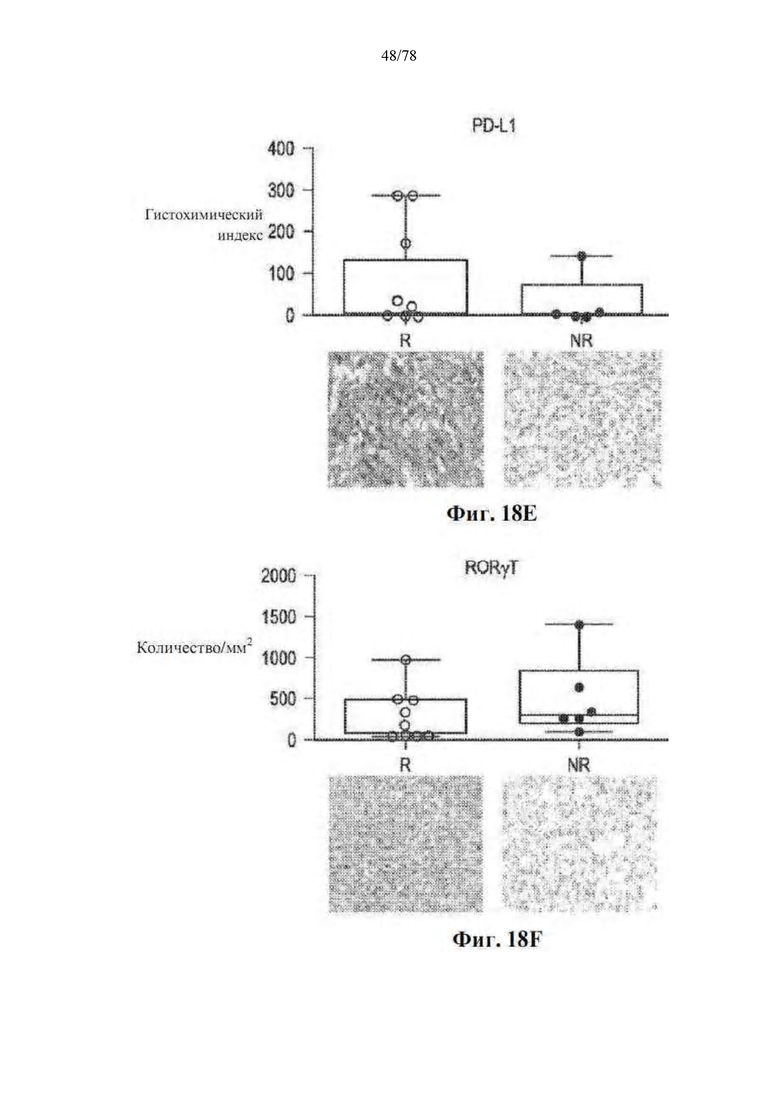
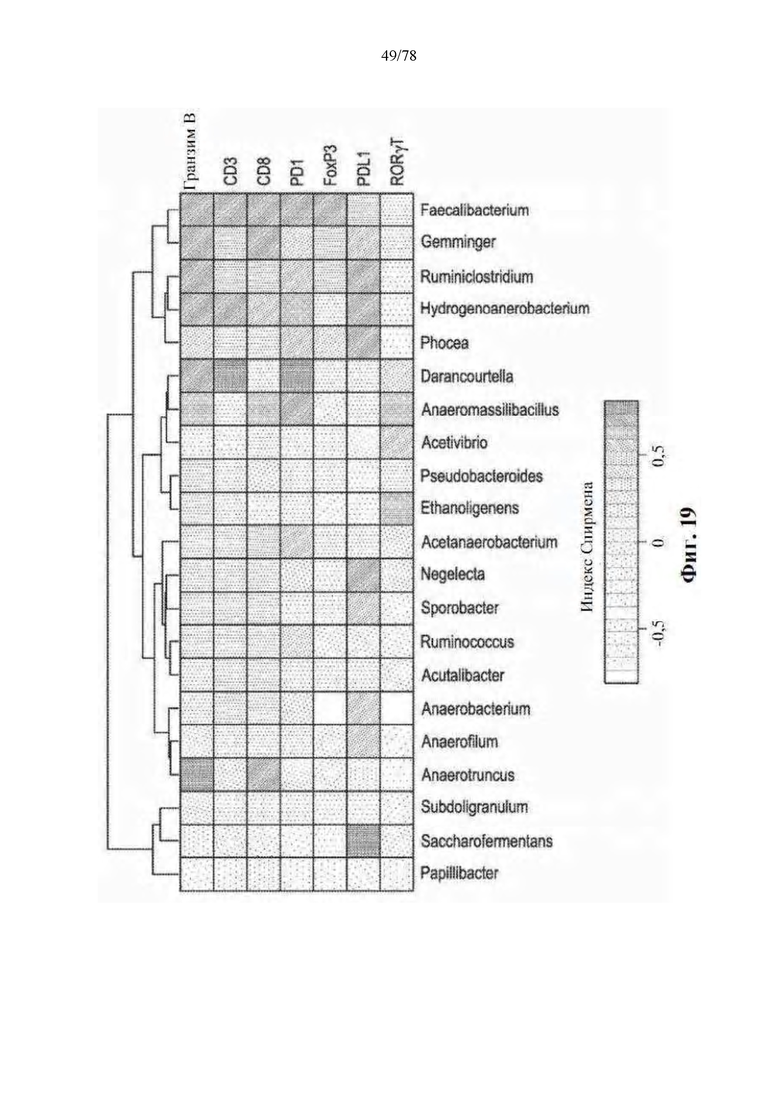
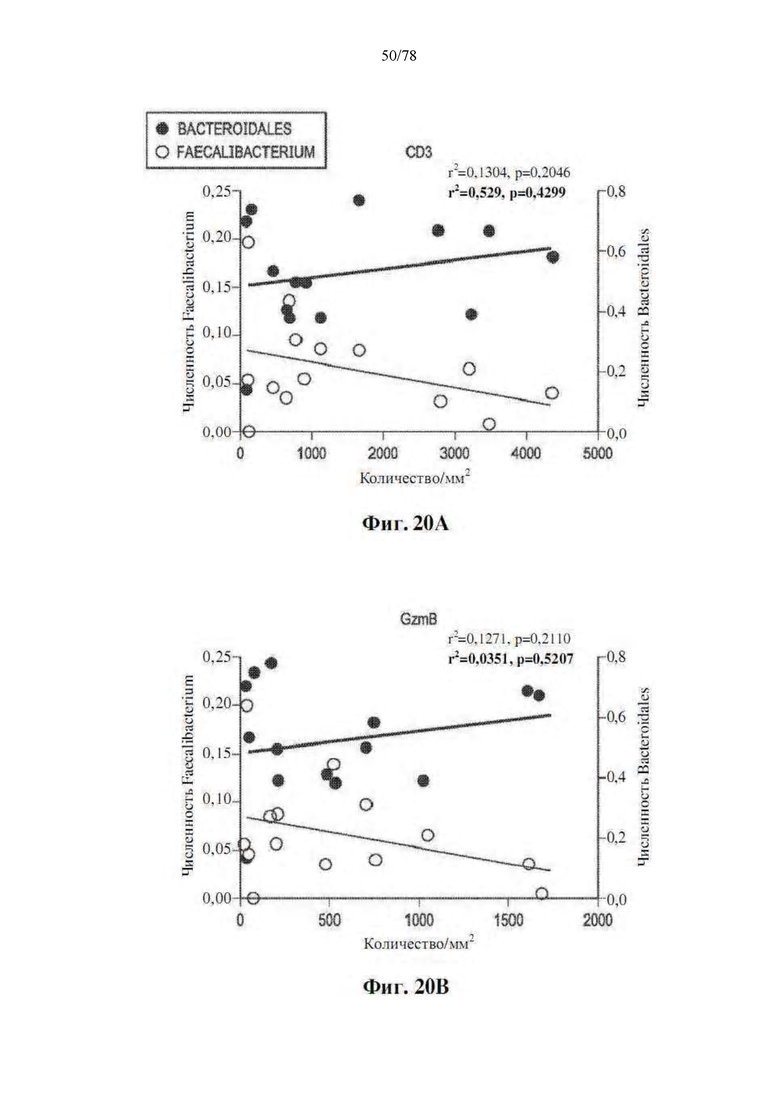

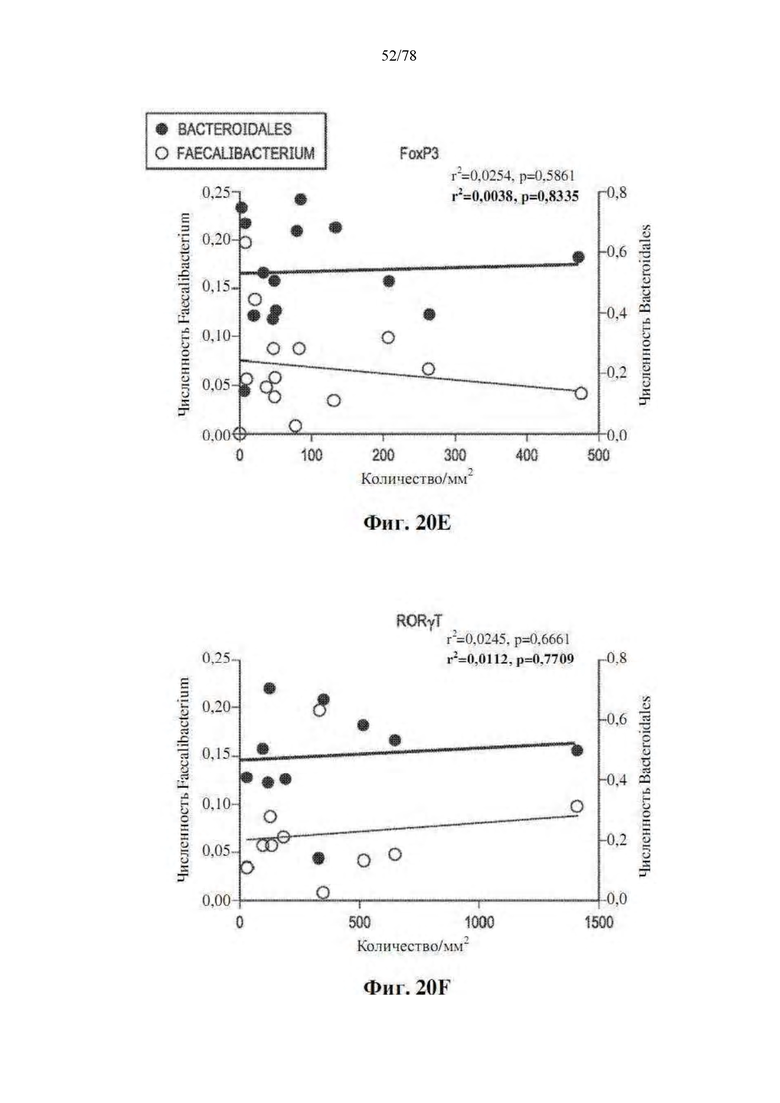
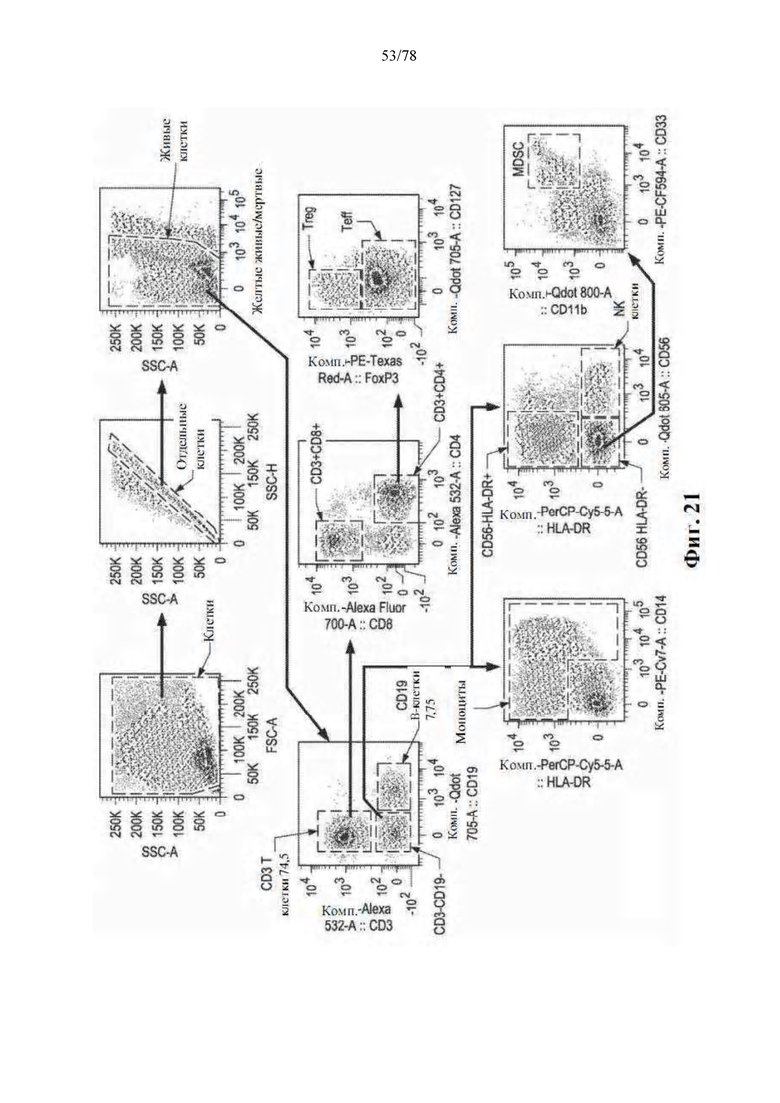

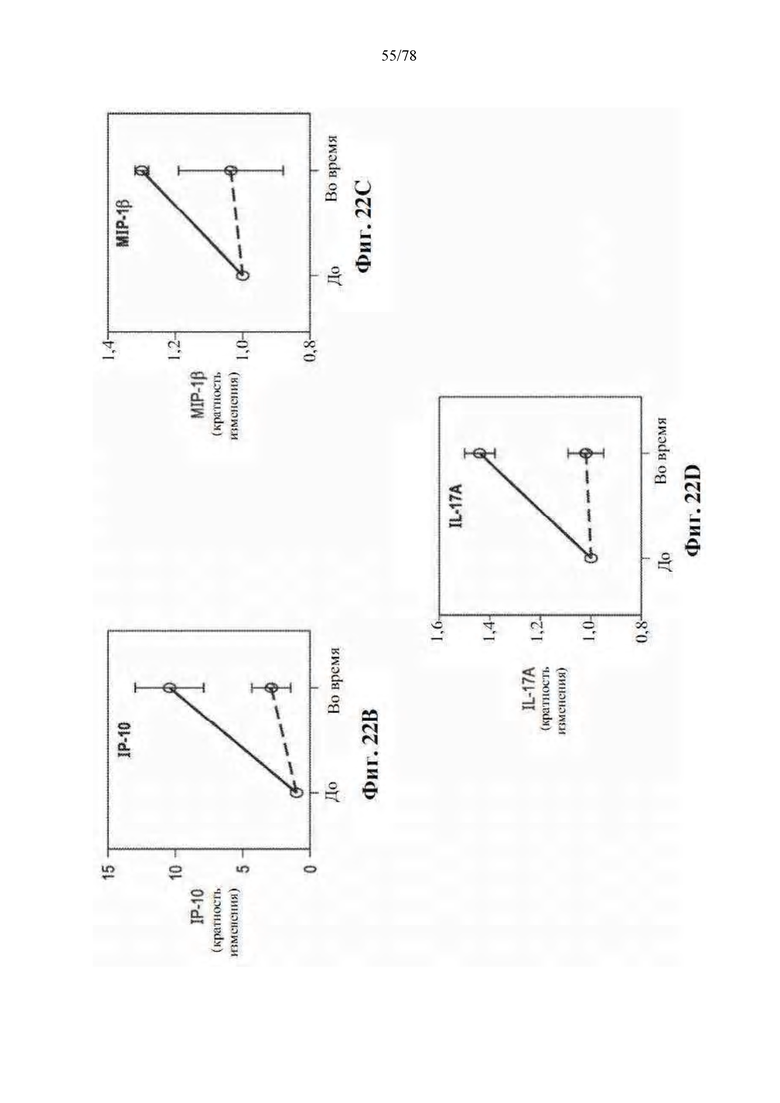
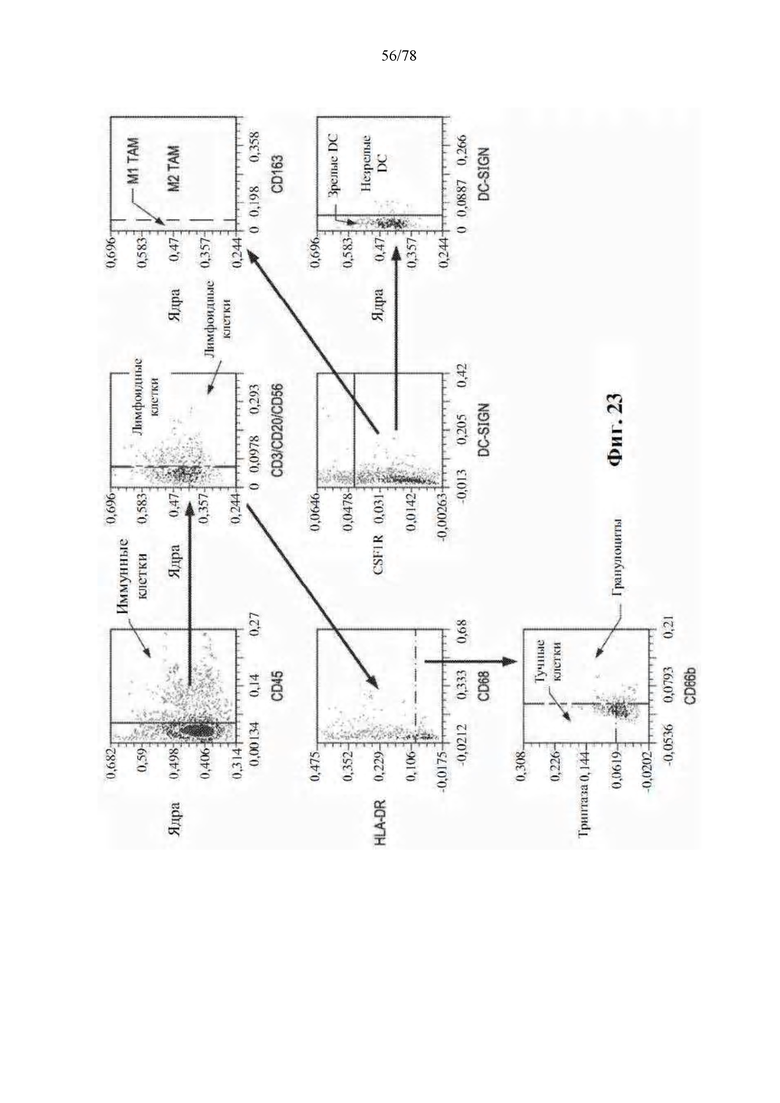

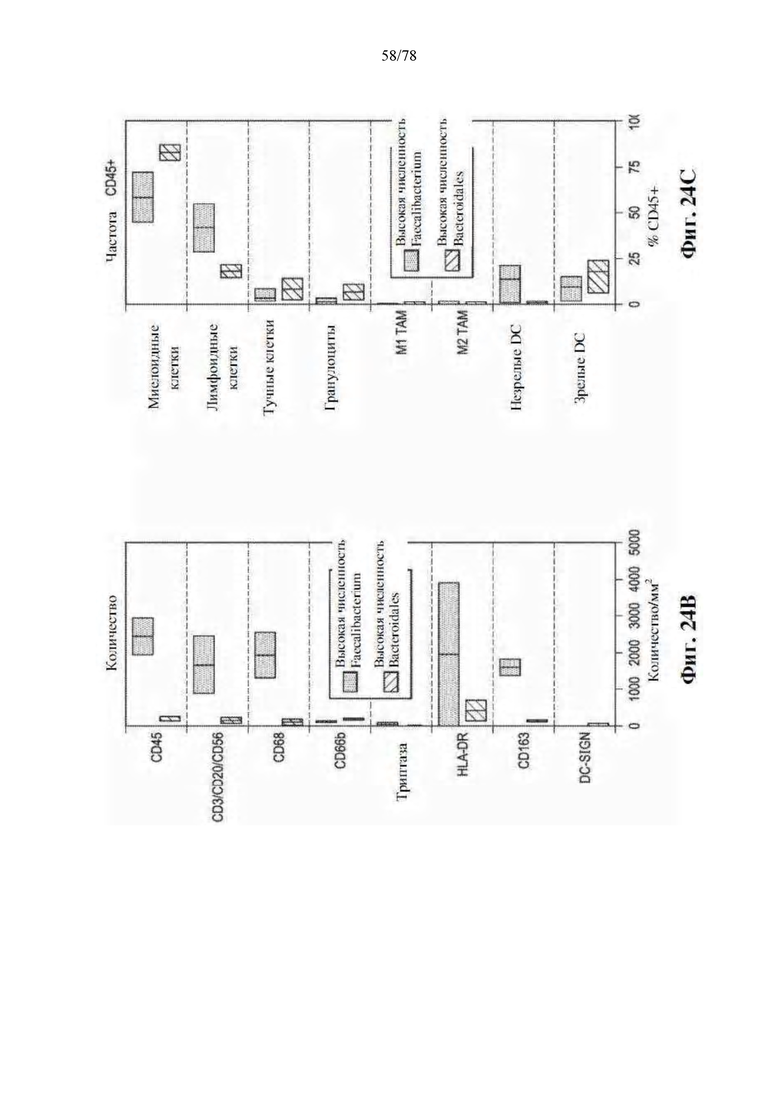
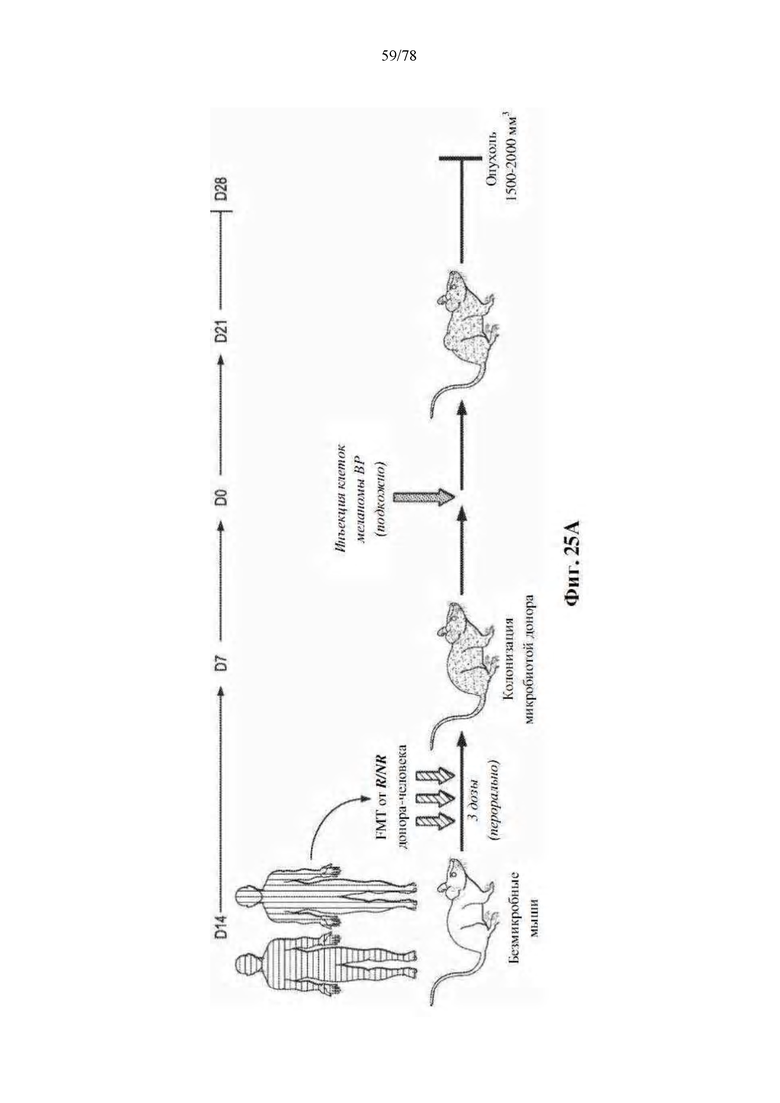
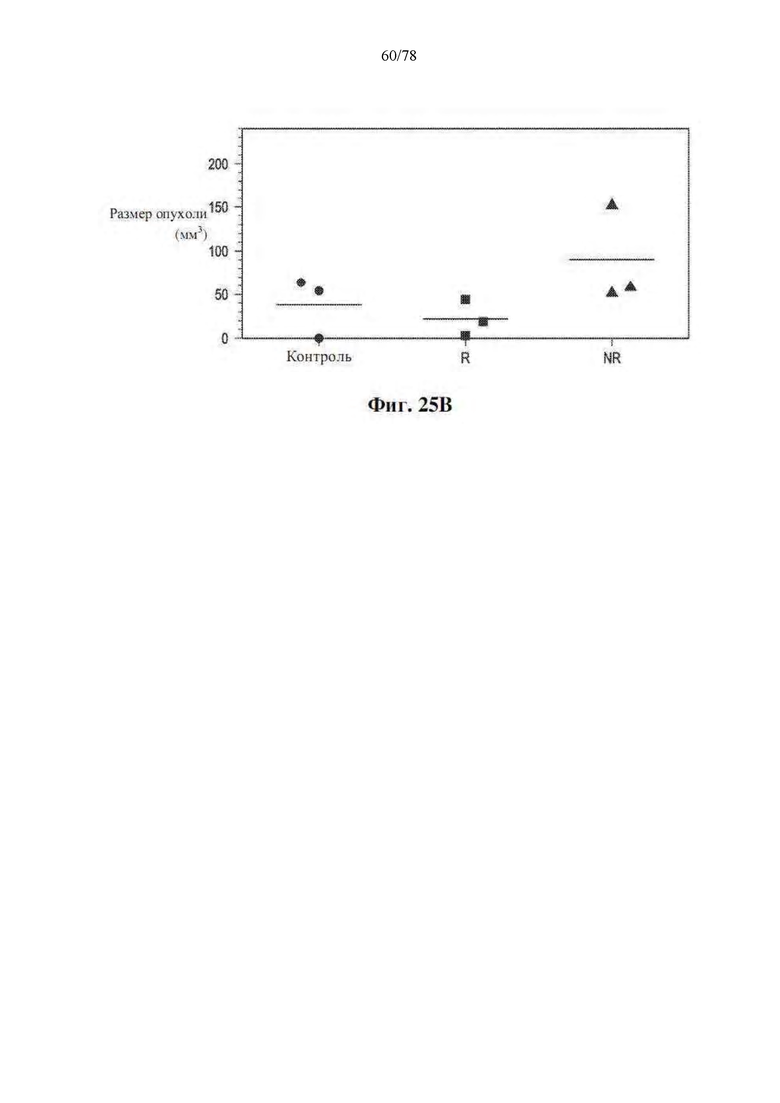



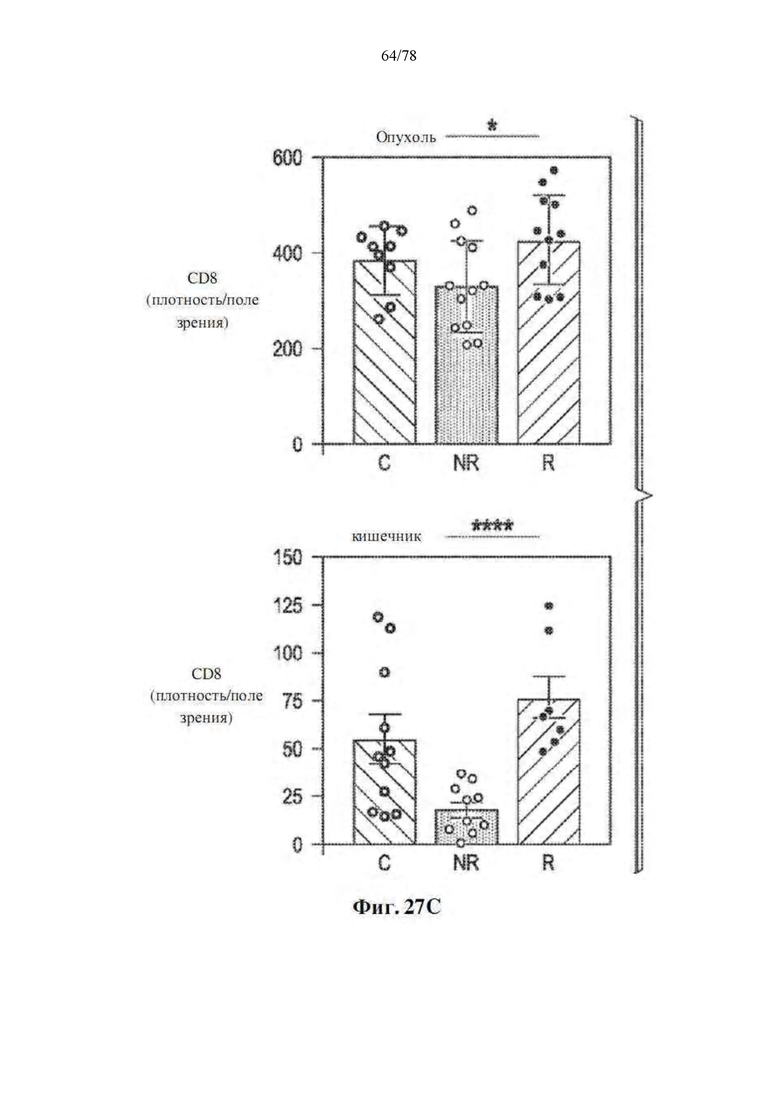
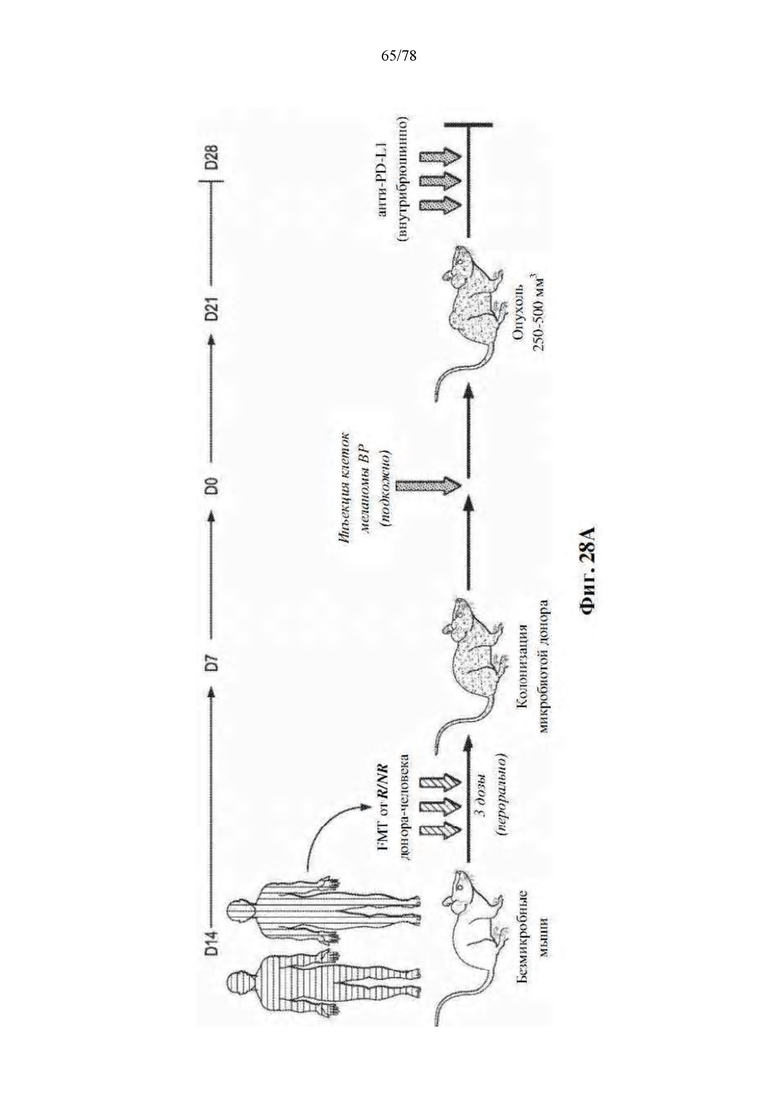
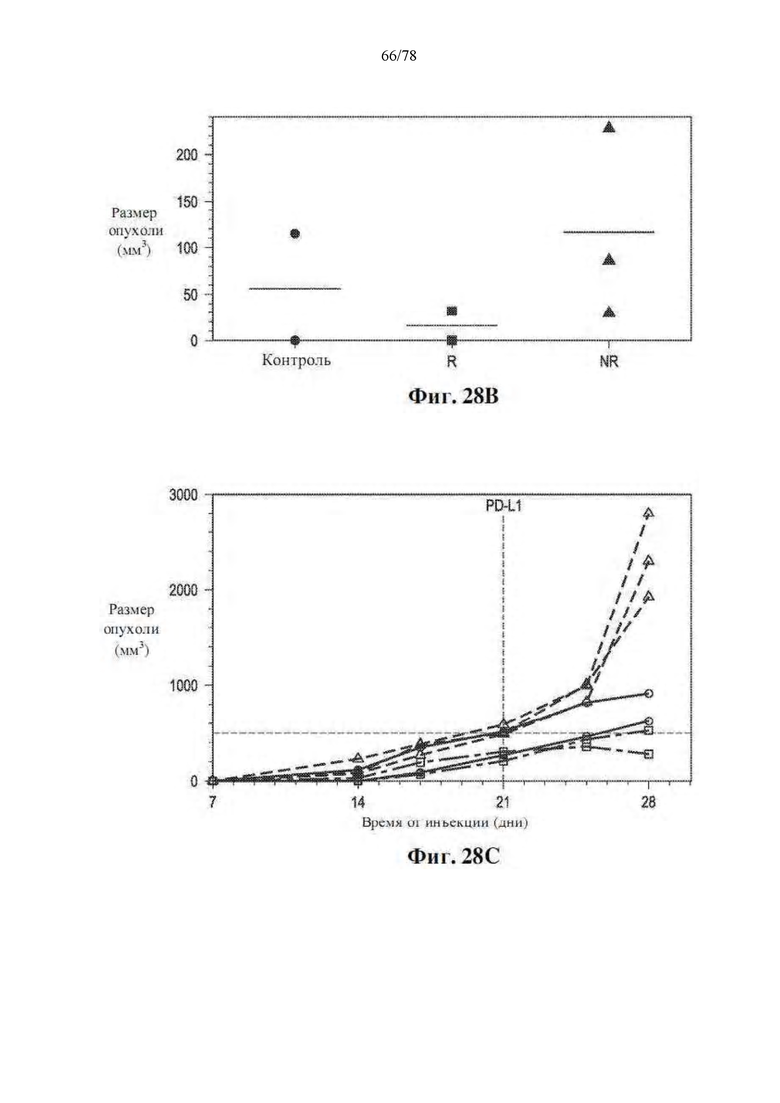
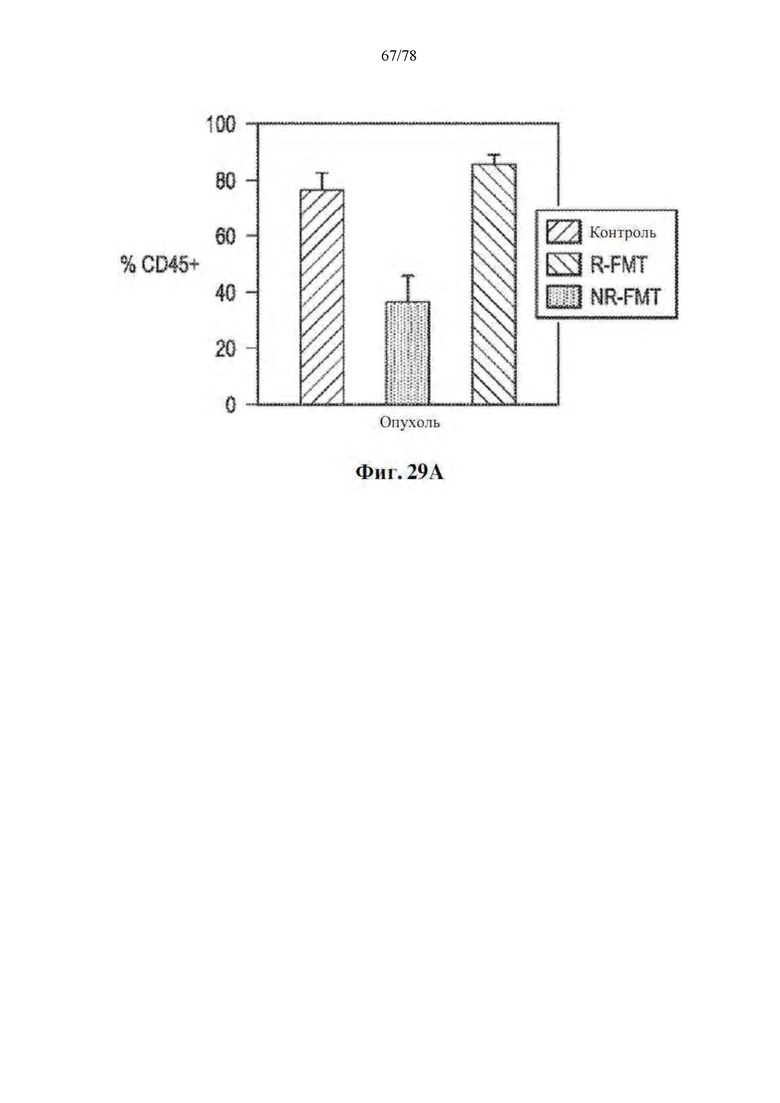
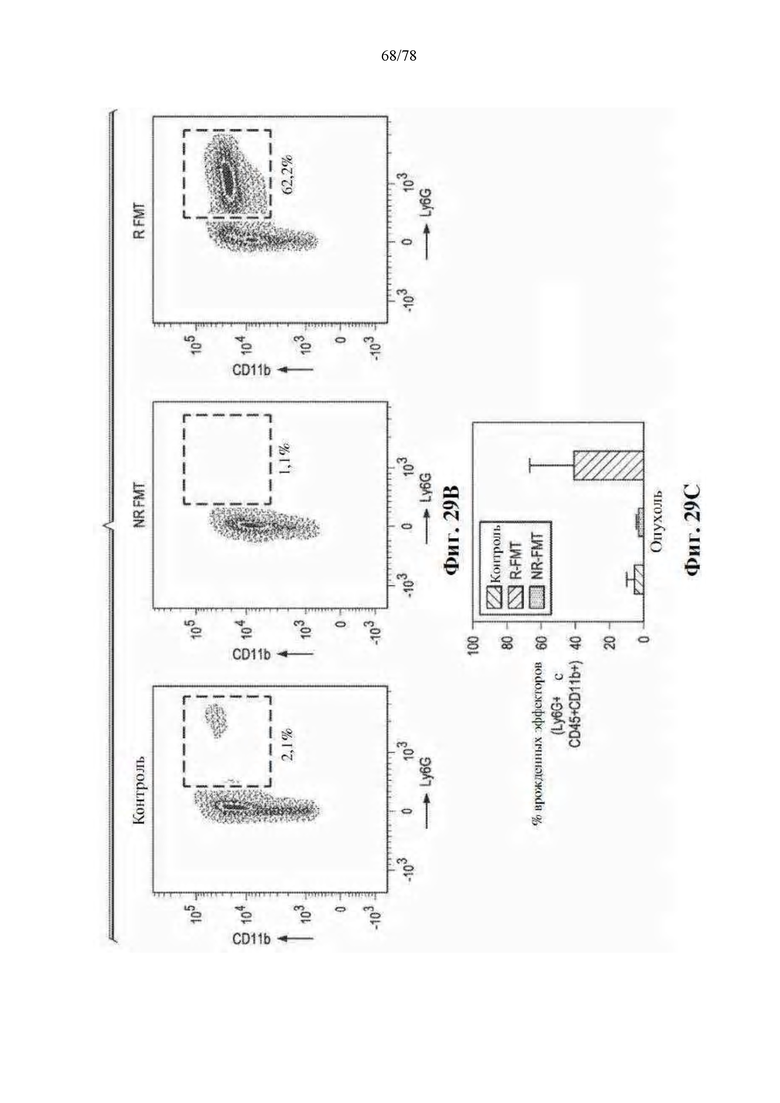
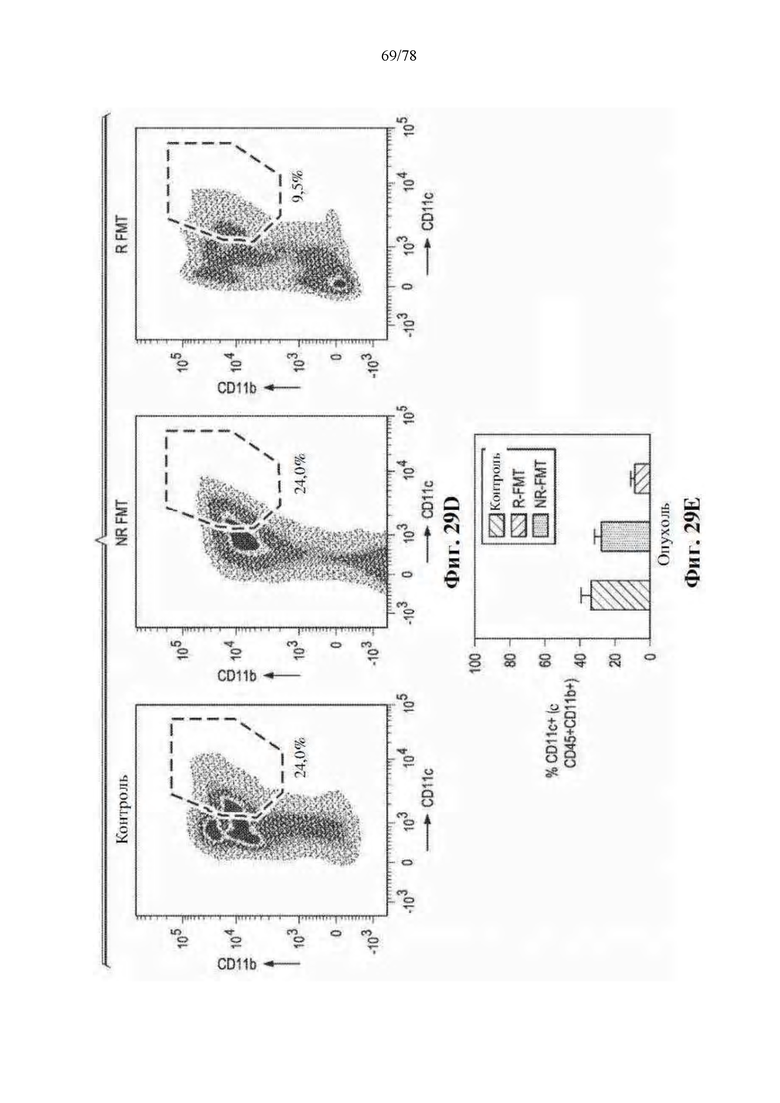
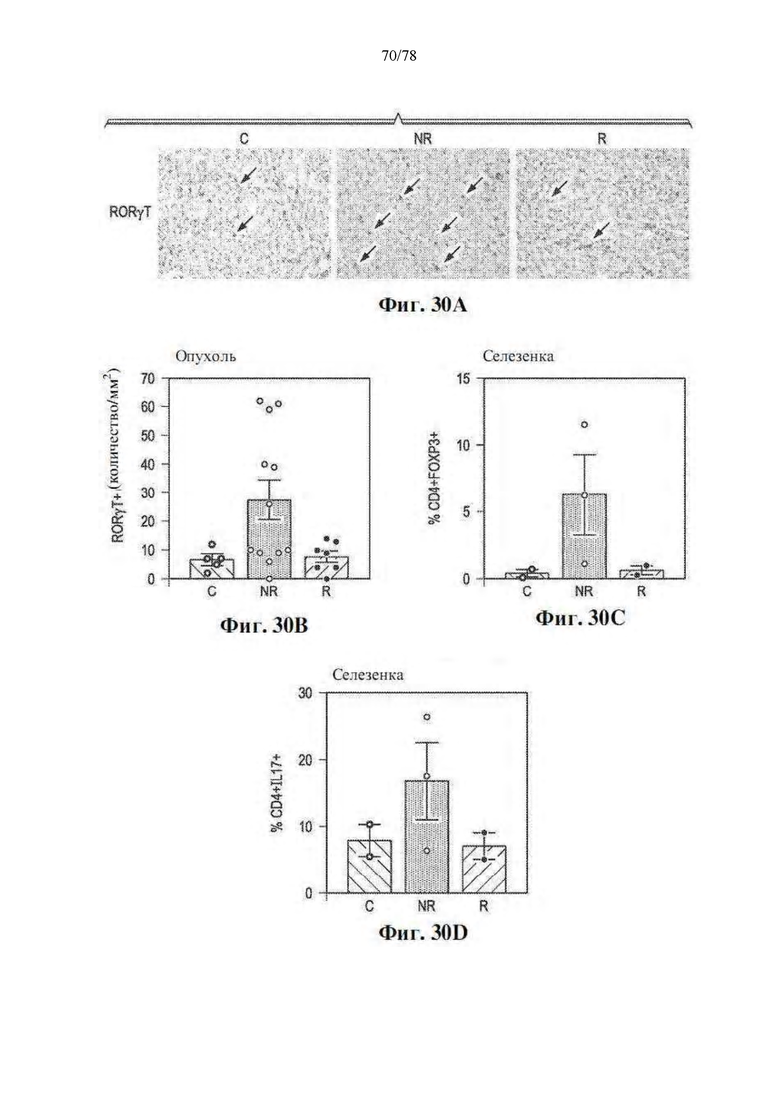
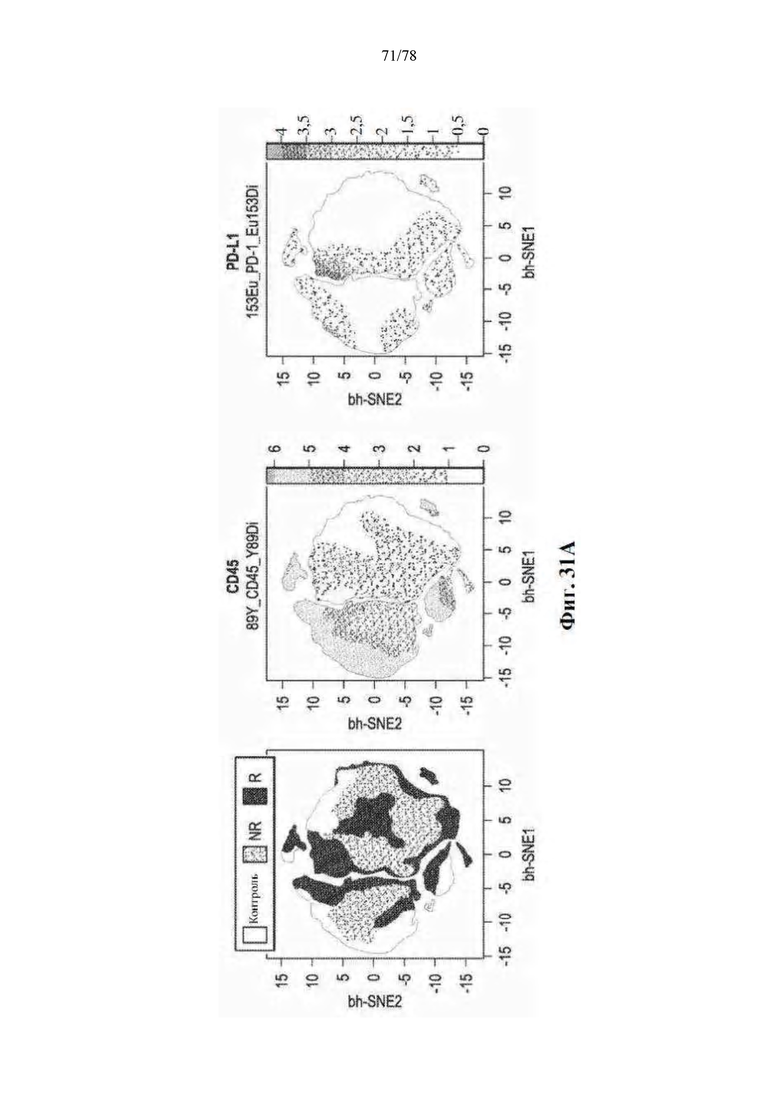
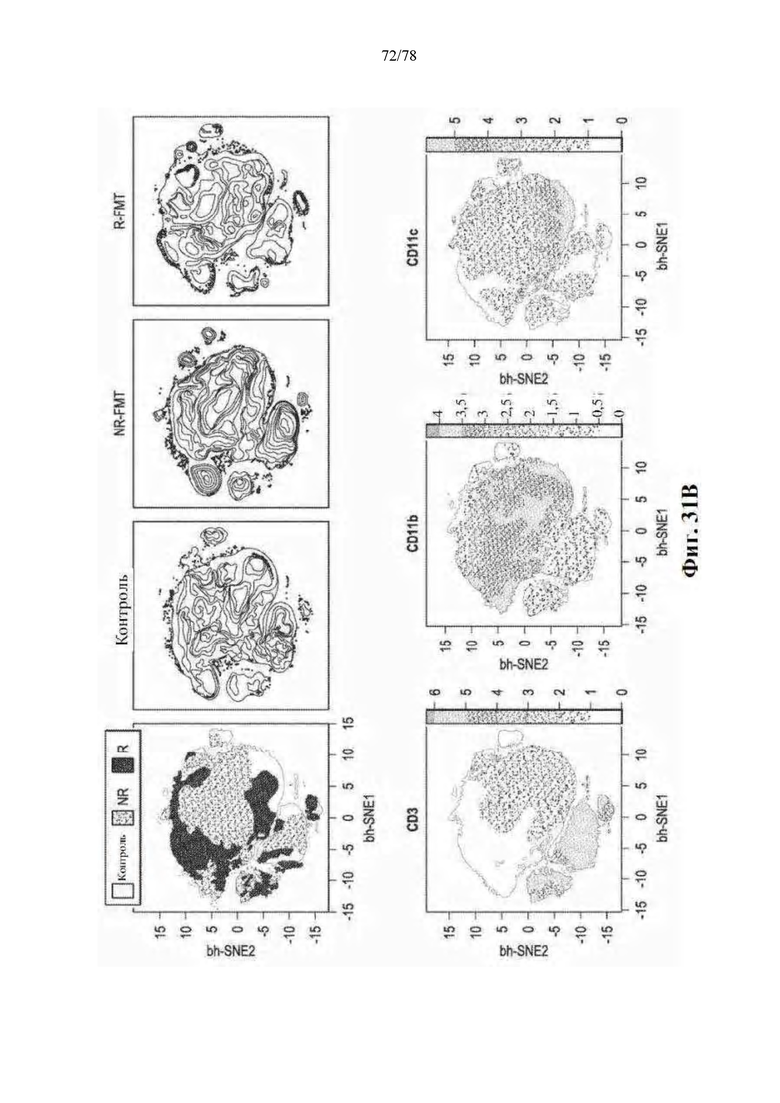
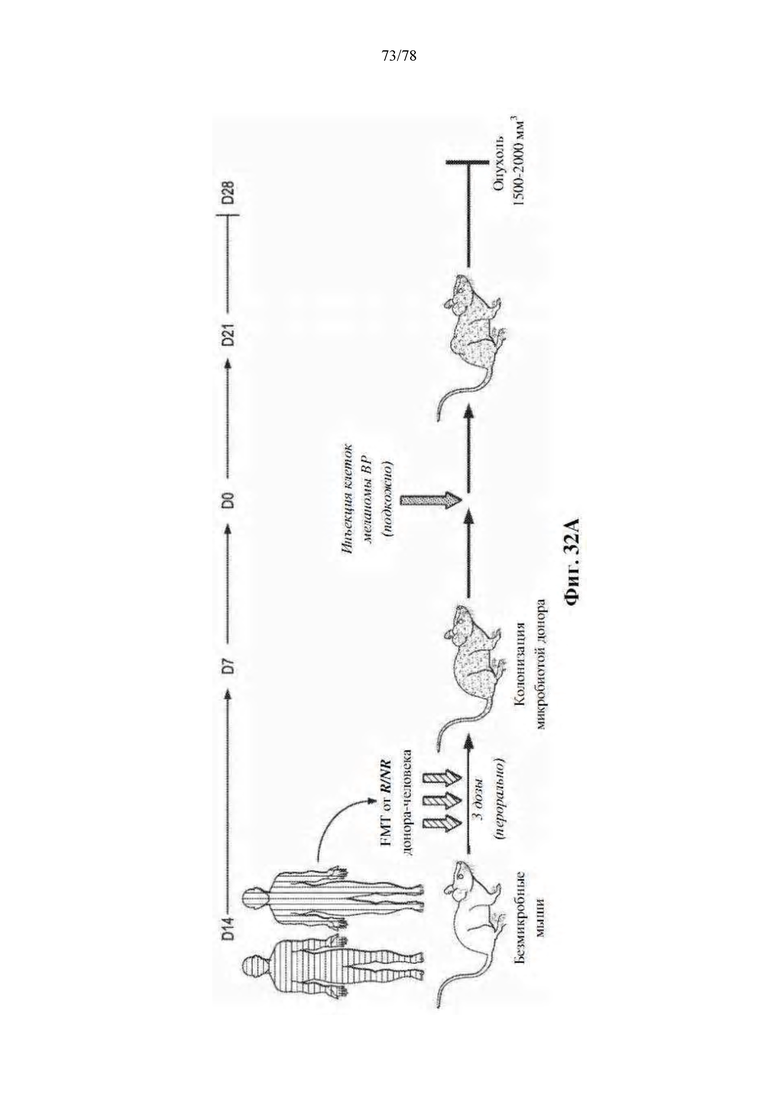
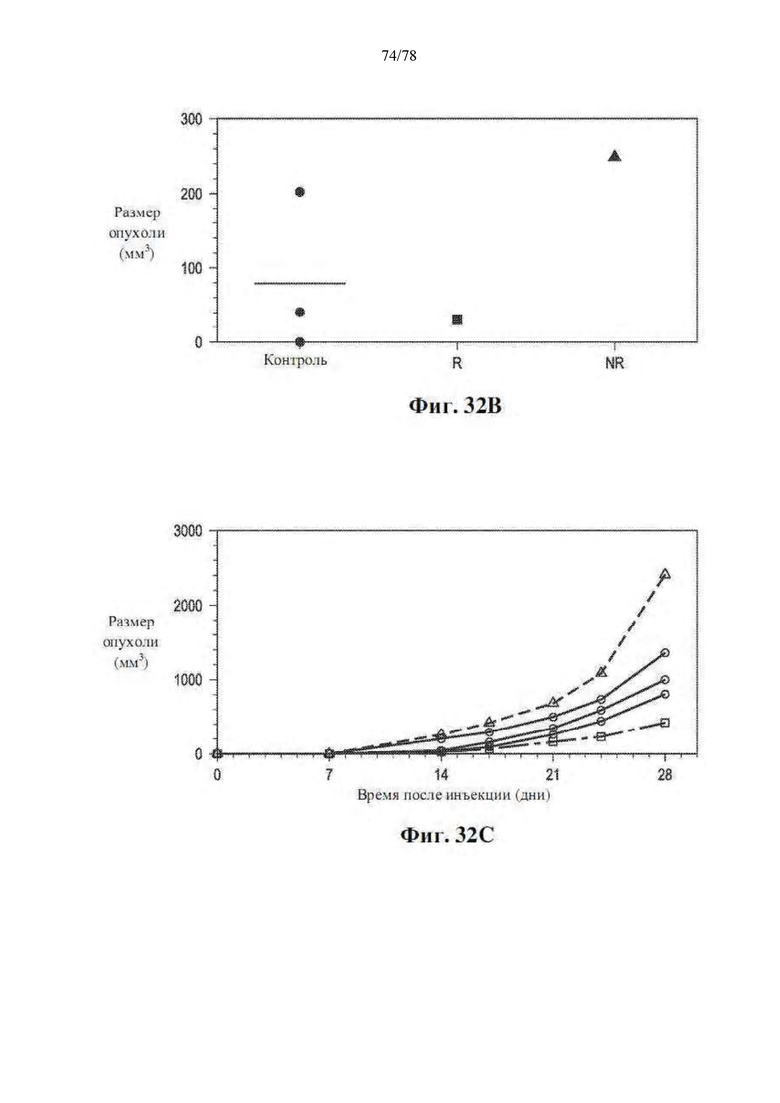

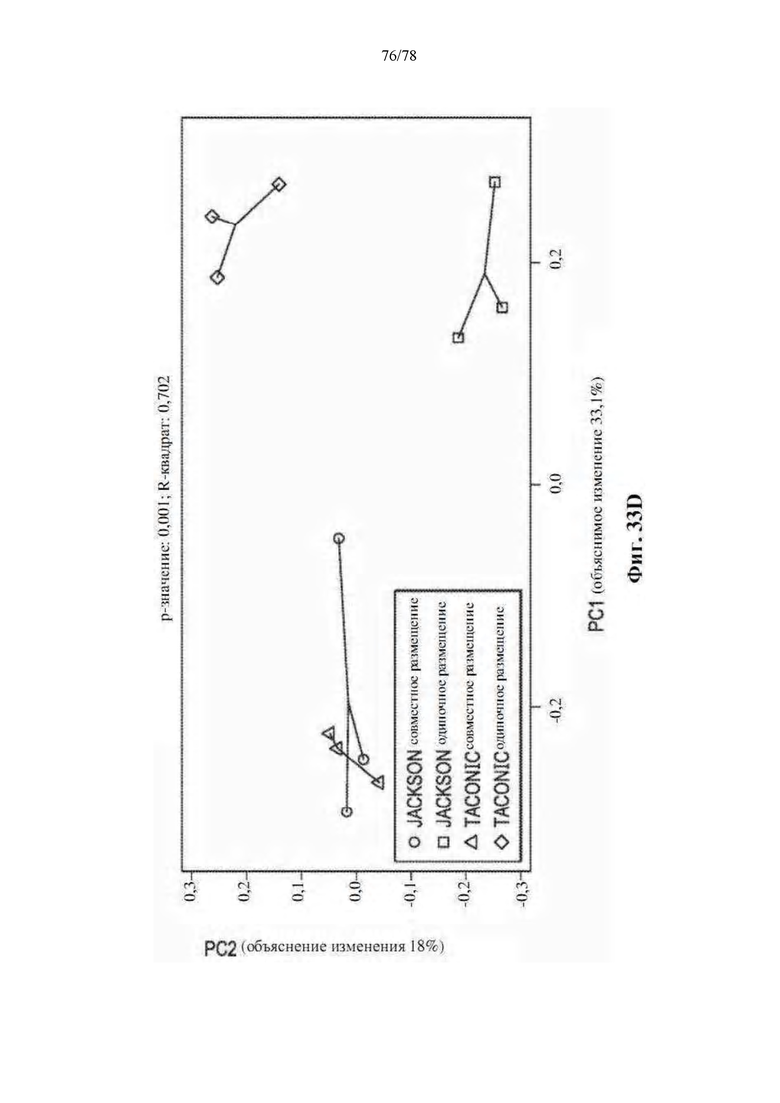


Группа изобретений относится к лечению рака. Раскрыто применение композиции для лечения или профилактики рака кожи у субъекта, где указанная композиция содержит по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae, где (а) выделенная или очищенная популяция бактерий принадлежит к одному или более видам, подвидам или бактериальным штаммам, выбранным из группы, состоящей из видов в таблице 1 с индексом преимущественной представленности (ei) более чем 0,5, или (b) выделенная или очищенная популяция бактерий выбрана из видов в таблице 2, обозначенных статусом ответа, отвечающего на лечение (R). Также раскрыты применение композиции, содержащей ингибитор иммунных контрольных точек, для лечения или профилактики рака кожи у субъекта, способ лечения или профилактики рака кожи, способы прогнозирования ответа на ингибитор иммунных контрольных точек. Группа изобретений обеспечивает модуляцию микробиома для повышения эффективности блокады иммунных контрольных точек. 5 н. и 14 з.п. ф-лы, 34 ил., 10 табл., 5 пр.
1. Применение композиции для лечения или профилактики рака кожи у субъекта, где указанная композиция содержит по меньшей мере одну выделенную или очищенную популяцию бактерий, принадлежащих к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae, где (а) выделенная или очищенная популяция бактерий принадлежит к одному или более видам, подвидам или бактериальным штаммам, выбранным из группы, состоящей из видов в таблице 1 с индексом преимущественной представленности (ei) более чем 0,5, или (b) выделенная или очищенная популяция бактерий выбрана из видов в таблице 2, обозначенных статусом ответа, отвечающего на лечение (R).
2. Применение по п. 1, в котором каждая из популяций бактерий присутствует в композиции в концентрации по меньшей мере 103 КОЕ.
3. Применение по п. 1 или 2, в котором по меньшей мере одна выделенная или очищенная популяция бактерий представлена в виде бактериальных спор.
4. Применение по п. 1 или 2, в котором по меньшей мере одна выделенная или очищенная популяция бактерий принадлежит к семейству Ruminococcaceae или к семейству Clostridiaceae.
5. Применение по п. 4, в котором популяция бактерий принадлежит к роду Ruminococcus или к роду Faecalibacterium.
6. Применение по п. 5, в котором популяция бактерий принадлежит к виду Ruminococcus bromii или Faecalibacterium prausnitzii.
7. Применение по п. 1 или 2, в котором по меньшей мере одна выделенная или очищенная популяция бактерий представляет собой вид, подвид или бактериальный штамм, содержащий последовательность гена 16S рРНК, по меньшей мере на 80% идентичную последовательности SEQ ID NO: 1-876.
8. Применение по п. 1 или 2, в котором по меньшей мере одна выделенная или очищенная популяция бактерий принадлежит к видам, подвидам или штаммам, содержащим нуклеотидные последовательности с идентичностью, составляющей по меньшей мере 80% по отношению к последовательностям группы совместно представленных генов (CAG), выбранным из группы, состоящей из SEQ ID NO: 877-926, SEQ ID NO: 927-976, SEQ ID NO: 977-1026, SEQ ID NO: 1027-1076, SEQ ID NO: 1077-1126, SEQ ID NO: 1127-1176, SEQ ID NO: 1177-1226, SEQ ID NO: 1227-1276, SEQ ID NO: 1277-1326, SEQ ID NO: 1327-1376, SEQ ID NO: 1377-1426, SEQ ID NO: 1427-1476, SEQ ID NO: 1477-1526, SEQ ID NO: 1527-1576, SEQ ID NO: 1577-1626, SEQ ID NO: 1627-1676, SEQ ID NO: 1677-1726, SEQ ID NO: 1727-1776, SEQ ID NO: 1777-1826, SEQ ID NO: 1827-1876, SEQ ID NO: 1877-1926, SEQ ID NO: 1927-1976, SEQ ID NO: 1977-2026, SEQ ID NO: 2027-2076, SEQ ID NO: 2077-2126, SEQ ID NO: 2127-2176, SEQ ID NO: 2177-2226, SEQ ID NO: 2227-2276, SEQ ID NO: 2277-2326, SEQ ID NO: 2327-2376, SEQ ID NO: 2377-2426, SEQ ID NO: 2427-2476, SEQ ID NO: 2477-2526, SEQ ID NO: 2527-2576, SEQ ID NO: 2577-2626 и SEQ ID NO: 2627-2676.
9. Применение по любому из пп. 1-8, причем композиция составлена для пероральной доставки.
10. Применение композиции, содержащей ингибитор иммунных контрольных точек, для лечения или профилактики рака кожи у субъекта, причем установлено, что субъект характеризуется наличием в микробиоме кишечника одной или более бактериальных популяций, принадлежащих к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae, где (а) выделенная или очищенная популяция бактерий принадлежат к одному или более видам, подвидам или бактериальным штаммам, выбранным из группы, состоящей из видов в таблице 1 с индексом преимущественной представленности (ei) более чем 0,5, или (b) выделенная или очищенная популяция бактерий выбрана из видов в таблице 2, обозначенных статусом ответа, отвечающего на лечение (R).
11. Способ лечения или профилактики рака кожи, содержащий применение ингибитора иммунных контрольных точек, в котором установлено, что субъект характеризуется благоприятным микробиологическим профилем в микробиоме кишечника, который определяется как имеющий одну или более бактерий, выбранных из группы, состоящей из видов, представленных в таблице 1, с индексом преимущественной представленности (ei) более чем 0,5 в кишечном микробиоме; или одну или более видов бактерий в таблице 2, обозначенных статусом ответа, отвечающего на лечение (R) в кишечном микробиоме, и причем одна или более бактерий, выбранных из группы, состоящей из видов, представленных в таблице 1, с индексом преимущественной представленности (ei) более чем 0,5 в кишечном микробиоме; или одна или более видов бактерий в таблице 2, обозначенных статусом ответа, отвечающего на лечение (R) в кишечном микробиоме, принадлежат к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae.
12. Способ лечения по п. 11, при котором рак представляет собой рак кожи, выбранный из базально-клеточного рака кожи, плоскоклеточного рака кожи, меланомы, возвышающейся дерматофибросаркомы, карциномы из клеток Меркеля, саркомы Капоши, кератоакантомы, опухолей веретенообразных клеток, карцином сальных желез, микрокистозной карциномы придатков, болезни Педжета молочной железы, атипичной фиброксантомы, лейомиосаркомы или ангиосаркомы.
13. Способ лечения по любому из пп. 11 или 12, при котором композиция, содержащая ингибитор иммунных контрольных точек, подходит для введения с по меньшей мере одним дополнительным противораковым лечением.
14. Способ лечения по п. 13, где по меньшей мере одно противораковое лечение представляет собой по меньшей мере один дополнительный ингибитор иммунных контрольных точек и/или по меньшей мере одно дополнительное противораковое лечение.
15. Способ прогнозирования ответа на ингибитор иммунных контрольных точек, включающий обнаружение микробиологического профиля в образце фекалий, полученном от пациента, характеризующегося наличием рака, причем ответ на ингибитор иммунных контрольных точек является благоприятным, если микробиологический профиль содержит одну или более бактериальных популяций, принадлежащих к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae, где (а) выделенная или очищенная популяция бактерий принадлежат к одному или более видам, подвидам или бактериальным штаммам, выбранным из группы, состоящей из видов в таблице 1 с индексом преимущественной представленности (ei) более чем 0,5, или (b) выделенная или очищенная популяция бактерий выбрана из видов в таблице 2, обозначенных статусом ответа, отвечающего на лечение (R).
16. Способ прогнозирования ответа на ингибитор иммунных контрольных точек, включающий определение микробиологического профиля в образце, полученном от пациента, характеризующегося наличием рака, причем, если микробиологический профиль характеризуется одной или более бактериями, выбранными из группы, состоящей из видов, представленных в таблице 1, с индексом преимущественной представленности (ei) более чем 0,5, или одним или более видами бактерий в таблице 2, обозначенные статусом ответа, отвечающего на лечение (R), тогда прогнозируется, что у пациента будет благоприятный ответ на ингибитор иммунных контрольных точек, причем одна или более бактерий, выбранных из группы, состоящей из видов, представленных в таблице 1, с индексом преимущественной представленности (ei) более чем 0,5 в кишечном микробиоме; или одна или более видов бактерий в таблице 2, обозначенных статусом ответа, отвечающего на лечение (R) в кишечном микробиоме, принадлежат к одному или более из семейств Ruminococcaceae, Clostridiaceae, Lachnospiraceae, Micrococcaceae и/или Veilonellaceae.
17. Способ по п. 15 или 16, при котором рак представляет собой рак кожи, выбранный из базально-клеточного рака кожи, плоскоклеточного рака кожи, меланомы, возвышающейся дерматофибросаркомы, карциномы из клеток Меркеля, саркомы Капоши, кератоакантомы, опухолей веретенообразных клеток, карцином сальных желез, микрокистозной карциномы придатков, болезни Педжета молочной железы, атипичной фиброксантомы, лейомиосаркомы или ангиосаркомы.
18. Способ по п. 15 или 16, при котором микробиологический профиль представляет собой кишечный микробиологический профиль.
19. Способ по п. 15 или 16, при котором ингибитор иммунных контрольных точек представляет собой моноклональное антитело к PD1 или моноклональное антитело к CTLA4.
| WO 2015066625 А1, 07.05.2015 | |||
| PITT J | |||
| M.et al | |||
| Resistance Mechanisms to Immune-Checkpoint Blockade in Cancer: Tumor-Intrinsic and -Extrinsic Factors | |||
| Immunity, 2016-06-21,Vol.44, No | |||
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| Украшение для пуговицы, запонки или броши | 1923 |
|
SU1255A1 |
Авторы
Даты
2023-04-04—Публикация
2017-09-27—Подача