ОБЛАСТЬ ТЕХНИКИ
Изобретение относится к манипуляции метаболизмом триптофана с использованием композиций микроорганизмов.
УРОВЕНЬ ТЕХНИКИ
Помимо его применения в синтезе белка, триптофан играет важную роль в ряде путей, которые приводят к продукции, например, серотонина (5-гидрокситриптамина), мелатонина, кинуренинов и триптамина. Триптофан и его метаболиты могут влиять, например, на иммуносупрессию, иммунную функцию, злокачественное новообразование, воспалительное заболевание, функцию эпителиального барьера, перистальтику кишечника, неврологическую функцию и инфекцию.
Было показано, что некоторые продукты триптофанового пути функционируют как агонисты рецепторов ароматических углеводородов (Ahr). Метаболиты включают, например, индол-3-альдегид, индол-3-ацетат, индол-3-пропионовую кислоту, индол, индол-3-ацетальдегид, индолакриловую кислоту, индол-3-ацетонитрил, 6-формилиндоло[3,2-b]карбазол (FICZ) и триптамин. Ahr играет роль в контроле дифференцировки и активности определенных субпопуляций Т-клеток. Сообщается, что он может влиять на адаптивные иммунные ответы, воздействуя как на Т-клетки, так и на антигенпрезентирующие клетки (АПК). Считается, что Ahr участвует в развитии и поддержании CD4+ FoxP3+ регуляторных Т-клеток (Трег), а также FoxP3-ИЛ-10+CD4+Tr1 и индукции клеток Th17.
Триптамин образуется в результате декарбоксилирования триптофана. Триптамин также может оказывать влияние на нервную систему, например, он, как сообщается, влияет на межмышечное нервное сплетение, которое участвует в перистальтике желудочно-кишечного тракта (Takaki et al., Neuroscience 16:223-240, 1985).
Было показано, что микробиомом желудочно-кишечного тракта можно манипулировать, по меньшей мере в глобальном масштабе, путем введения каловой массы пациенту, например, пациенту, страдающему рецидивирующей инфекцией, вызванной С.difficile. Терапевтические манипуляции с эффектами, связанными с триптофаном, как правило, ограничивались химическими манипуляциями с предшественниками метаболитов. Открытия и изобретения заявителей обеспечивают возможность манипулировать определенными функциями более целенаправленно, используя бактерии, полученные из микробиома человека.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Изобретение относится к идентификации видов бактерий, которые могут влиять на (например, увеличивать) уровни триптамина и/или 5-гидрокситриптамина (серотонина). Соответственно, изобретение относится к композициям, содержащим один или более видов бактерий, которые модулируют уровни триптамина и/или 5-гидрокситриптамина, а также к способам модуляции уровней триптамина и/или 5-гидрокситриптамина путем введения таких композиций. В различных вариантах осуществления уровни триптамина или 5-гидрокситриптамина изменяют (например, повышают) по сравнению с уровнями до лечения в соответствии со способом или с использованием композиции согласно изобретению.
Изобретение обеспечивает способы изменения уровней триптамина или 5-гидрокситриптамина у субъекта, включающие введение субъекту жизнеспособной популяции по меньшей мере одного вида бактерии, выбранных из Таблиц 1, 2, 3, 4, 5 или 6. В различных вариантах осуществления уровни триптамина или 5-гидрокситриптамина у субъекта повышают. Например, уровни могут быть повышены по сравнению с предыдущим введением жизнеспособной бактериальной популяции, отмеченной выше.
В некоторых вариантах осуществления способы включают введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Ruminococcus_gnavus (штамм 1), Lachnospiraceae_bacterium_9_1_43BFAA, Eggerthella_sp., Ruminococcus_gnavus (штамм 2), Clostridium_nexile, Lachnospiraceae_bacterium_6_1_63FAA, и Ruminococcus_torques.
В некоторых вариантах осуществления способы включают введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Clostridium ghonii, Flavonifractor plautii, Ruminococcus gnavus Bacteroides ovatus, Bacteroides stercoris, и Clostridium sporogenes.
В некоторых вариантах осуществления способы включают введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Lachnospiraceae_bacterium_2_1_58FAA, Clostridium_aldenense_SC114, Clostridium_citroniae, и Clostridium_clostridioforme.
В некоторых вариантах осуществления способы включают введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Flavonifractor_plautii, Veillonella_parvula, Blautia_sp_CAG_257_SC146, и Clostridium_bolteae.
В некоторых вариантах осуществления способы включают введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Blautia_hansenii, Lachnospiraceae_bacterium_2_1_46FAA, Coprococcus_sp_HPP0048, Collimella_tanakaei, Clostridium_sporogenes, Clostridium_phytofermentans, Clostridium_bifermentans, Staphylococcus_aureus, Lachnospiraceae_bacterium_4_1_37FAA, Clostridium_asparagiforme, Clostridium_lavalense_SC43, и Holdemania_filliformis.
В различных вариантах осуществления вышеизложенного способы включают введение субъекту жизнеспособной популяции по меньшей мере 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 перечисленных видов (с верхним пределом, составляющим количество видов в группах, указанных выше).
Кроме того, в некоторых вариантах осуществления способы включают введение субъекту композиции, содержащей или состоящей из одной или более композиций 1-47 из Таблицы 6.
В некоторых вариантах осуществления способы согласно изобретению можно использовать для субъекта, имеющего заболевание или патологическое состояние, характеризующееся измененными перистальтикой кишечника, агрегацией тромбоцитов, иммунным ответом, функцией сердца или развитием костей.
В некоторых вариантах осуществления у субъекта имеется заболевание или патологическое состояние, выбранное из группы, состоящей из синдрома раздраженного кишечника, воспалительного заболевания кишечника (например, инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита), запора, депрессии, тревожного расстройства, сердечнососудистого заболевания и остеопороза.
Изобретение также обеспечивает фармацевтические составы, содержащие жизнеспособную популяцию по меньшей мере одного вида бактерии, выбранного из Таблицы 1, 2, 3, 4, 5, или 6.
В некоторых вариантах осуществления фармацевтический состав содержит жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Ruminococcus_gnavus (штамм 1), Lachnospiraceae_bacterium_9_1_43BFAA, Eggerthella_sp., Ruminococcus_gnavus (штамм 2), Clostridium_nexile, Lachnospiraceae_bacterium_6_1_63FAA, и Ruminococcus_torques.
В некоторых вариантах осуществления фармацевтический состав содержит жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Clostridium ghonii, Flavonifractor plautii, Ruminococcus gnavus Bacteroides ovatus, Bacteroides stercoris, и Clostridium sporogenes.
В некоторых вариантах осуществления фармацевтический состав содержит жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Lachnospiraceae_bacterium_2_1_58FAA, Clostridium_aldenense_SC114, Clostridium_citroniae, и Clostridium_clostridioforme.
В некоторых вариантах осуществления фармацевтический состав содержит жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Flavonifractor_plautii, Veillonella_parvula,Blautia_sp_CAG_257_SC146, и Clostridium_bolteae.
В некоторых вариантах осуществления фармацевтический состав содержит жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Blautia_hansenii, Lachnospiraceae_bacterium_2_1_46FAA, Coprococcus_sp_HPP0048, Collinsella_tanakaei, Clostridium_sporogenes, Clostridium_phytofermentans, Clostridium_bifermentans, Staphylococcus_aureus, Lachnospiraceae_bacterium_4_1_37FAA, Clostridium_asparagiforme, Clostridium_lavalense_SC43, и Holdemania_filliformis.
В различных вариантах осуществления вышеизложенного фармацевтический состав содержит жизнеспособную популяцию по меньшей мере 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 перечисленных видов (с верхним пределом, составляющим количество видов в группах, указанных выше).
Кроме того, в некоторых вариантах осуществления фармацевтические составы содержат или состоят из одной или более композиций 1-47 из Таблицы 6.
В различных вариантах осуществления фармацевтические составы содержат фармацевтически приемлемый наполнитель, например, как описано в данном документе.
В некоторых вариантах осуществления фармацевтический состав находится в капсуле. Например, фармацевтический состав в некоторых вариантах осуществления может находиться в капсуле с энтеросолюбильной оболочкой.
Изобретение также обеспечивает способы изменения уровней триптамина или 5-гидрокситриптамина у субъекта, причем способы включают введение субъекту эффективного количества фармацевтического состава, как описано в данном документе (см., например, выше).
В некоторых вариантах осуществления уровни триптамина или 5-гидрокситриптамина у субъекта повышаются.
В некоторых вариантах осуществления уровни триптамина или 5-гидрокситриптамина представляют собой уровни триптамина или 5-гидрокситриптамина в кале субъекта.
В некоторых вариантах осуществления уровни триптамина или 5-гидрокситриптамина представляют собой уровни триптамина или 5-гидрокситриптамина в крови, сыворотке, плазме, моче или спинномозговой жидкости (СМЖ) субъекта.
Изобретение дополнительно обеспечивает способы лечения субъекта, имеющего заболевание или патологическое состояние, характеризующееся наличием низких уровней триптамина или 5-гидрокситриптамина, причем способы включают введение субъекту, у которого диагностировано или имеется риск развития заболевания, терапевтически эффективного количества фармацевтического состава, как описано в данном документе (см., например, выше).
В некоторых вариантах осуществления у субъекта имеется заболевание или патологическое состояние, характеризующееся измененными перистальтикой кишечника, агрегацией тромбоцитов, иммунным ответом, функцией сердца или развитием костей.
В некоторых вариантах осуществления у субъекта имеется заболевание или патологическое состояние, выбранное из группы, состоящей из синдрома раздраженного кишечника, воспалительного заболевания кишечника (например, инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита), запора, депрессии, тревожного расстройства, сердечнососудистого заболевания и остеопороза.
Изобретение также предлагает способы повышения уровня или активности регуляторных Т-клеток у субъекта, причем способы включают введение субъекту фармацевтического состава, как описано в данном документе (см., например, выше).
Кроме того, изобретение относится к способам восстановления или улучшения гомеостаза кишечника, или для предотвращения или лечения злокачественного новообразования кишечника или толстой кишки у субъекта, причем способы включают введение субъекту фармацевтического состава, как описано в данном документе (см., например, выше).
Изобретение также обеспечивает композиции, содержащие по меньшей мере два разных вида бактерий, которые в комбинации могут повышать уровни триптамина или 5-гидрокситриптамина по сравнению с уровнем триптамина или 5-гидрокситриптамина, продуцируемых каждым видом отдельно, (i) в присутствии того же уровня триптофана, что и в комбинации, (ii) в течение определенного периода времени в присутствии того же уровня триптофана, что и в комбинации, (iii) при введении в систему in vivo и/или (iv) при введении в модельную систему in vitro.
В некоторых вариантах осуществления композиция представляет собой композицию, содержащую виды бактерий из фармацевтического состава, как описано в данном документе (см., например, выше).
Изобретение также обеспечивает составы или композиции, как указано выше и в других местах данного документа, для применения в изменении уровней триптамина или 5-гидрокситриптамина у субъекта.
В некоторых вариантах осуществления у субъекта имеется заболевание или патологическое состояние, характеризующееся измененными перистальтикой кишечника, агрегацией тромбоцитов, иммунным ответом, функцией сердца или развитием костей.
В некоторых вариантах осуществления у субъекта имеется заболевание или патологическое состояние, выбранное из группы, состоящей из синдрома раздраженного кишечника, воспалительного заболевания кишечника (инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита), запора, депрессии, тревожного расстройства, сердечнососудистого заболевания и остеопороза.
В некоторых вариантах осуществления состав или композиция предназначены для повышения уровня или активности регуляторных Т-клеток у субъекта.
В некоторых вариантах осуществления состав или композиция предназначены для восстановления гомеостаза кишечника, или для предотвращения или лечения злокачественного новообразования кишечника или толстой кишки у субъекта.
Изобретение включает применение композиций и составов, как описано в данном документе, для целей, изложенных в способах, описанных выше и в других местах данного документа, а также для приготовления лекарств для них.
Полное раскрытие каждого патентного документа и научной статьи, упомянутой в данном документе, а также указанных патентных документов и научных статей, цитируемых таким образом, прямо включено в данный документ посредством ссылки для всех целей.
Дополнительные признаки и преимущества изобретения более подробно описаны
ниже.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
На Фиг. 1 представлен график, на котором приведены результаты анализа трех видов, связанных с высокими уровнями триптамина, и уровней триптамина в присутствии одного, двух или трех видов: Lb = Lachnospiraceae_bacterium_9_1_43BFAA; Rg = Ruminococcus gnavus; Rt = Ruminococcus_torques; 0 означает, что вид отсутствует, 1 означает, что вид присутствует. Только Rt: низкие уровни триптамина; только Rg: средние уровни триптамина; 2 из 3 видов: от средних до высоких уровней триптамина; все 3 вида: высокие уровни триптамина; примечание: только 1 образец имеет Lb=1, Rg=0, Rt=0, поэтому он не приведен.
На Фиг. 2 представлен график, на котором приведены результаты Blastp триптофан-декарбоксилазы (SEQ ID NO: 73 и 75) в сравнении с базой данных пангеномов видов микроорганизмов.
На Фиг. 3 представлен график, демонстрирующий идентификацию видов, присутствие которых в исследуемых образцах кала прогнозирует повышенные уровни триптамина у видов (по сравнению с субъектами, у которых эти виды отсутствуют) в двух клинических исследованиях. Обведенные кружком виды указывают на те, чье присутствие независимо прогнозирует более высокие уровни триптамина в двух клинических исследованиях.
На Фиг. 4 представлен набор графиков, демонстрирующих богатство триптамин-модулирующих видов в двух клинических исследованиях SERES-101 (связанных с сохранением ремиссии язвенного колита) и SERES-004 (связанных с предотвращением рецидива инфекции, вызванной С.difficile).
На Фиг. 5 представлен перечень последовательностей 16S рДНК выбранных бактериальных ОТЕ, которые можно использовать в изобретении (SEQ ID NO: 1-72), а также последовательностей некоторых триптофан-декарбоксилаз (SEQ ID NO: 73-75).
ПОДРОБНОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
Заявители идентифицировали виды бактерий в желудочно-кишечном тракте (ЖКТ) человека, которые связаны с присутствием триптамина. Особенности некоторых видов являются неожиданными, например, в некоторых случаях эти виды ранее не были идентифицированы как участвующие в регуляции триптамина. Кроме того, в некоторых случаях комбинации видов проявляют синергизм. Не придерживаясь какой-либо конкретной теории, это указывает на то, что различные виды бактерий занимают разные экологические ниши и/или играют разные роли в регуляции триптамина в желудочно-кишечном тракте.
Заявители также идентифицировали виды бактерий, связанные с повышением уровня 5-гидрокситриптамина (5НТ). 5НТ, или серотонин, представляет собой нейротрансмиттер головного мозга, оказывающий широкое влияние на физиологию организма-хозяина из-за его влияния на перистальтику кишечника и агрегацию тромбоцитов, иммунные ответы, функцию сердца и развитие костей. Приблизительно 95% 5НТ в организме присутствует в кишечнике, и было показано, что кишечный микробном модулирует уровни 5НТ хозяина (Yano et al., Cell 161:264-276, 2015; Ge et al., J. Transl. Med. 15:13, 2017). Нарушение регуляции 5HT было связано с широким спектром заболеваний человека, включая синдром раздраженного кишечника (СРК), депрессию, тревожное расстройство, сердечно-сосудистые заболевания и остеопороз.
Изобретение обеспечивает композиции и способы для применения в лечении заболеваний и патологических состояний, которые может быть полезны при модуляции триптофана и/или метаболита триптофана (например, триптамина), например, в крови или желудочно-кишечном тракте млекопитающего, такого как человек. В некоторых вариантах осуществления уровни триптамина повышают в крови или кишечнике млекопитающего. В некоторых вариантах осуществления уровень триптофана снижают в крови или желудочно-кишечном тракте млекопитающего.
В некоторых вариантах осуществления описание обеспечивает бактерию, содержащую по меньшей мере один ген или генную кассету, кодирующую один или более ферментов для продукции триптамина. В некоторых вариантах осуществления бактерия содержит последовательность генов, кодирующую триптофан-декарбоксилазу (Trp-декарбоксилазу). В некоторых вариантах осуществления описание обеспечивает композиции, содержащие одну или более бактерий, содержащих по меньшей мере один ген или генную кассету, кодирующую один или более ферментов для продукции триптамина (например, триптофан-декарбоксилазу). В некоторых вариантах осуществления изобретение обеспечивает способы применения таких композиций для изменения (например, увеличения) уровней триптамина.
Композиции триптаминов (ТА)
Композиции, полезные в изобретениях, предложенных в данном документе, содержат один или более (например, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12 или более) микроорганизмов, которые могут модулировать, например, увеличивать, уровни метаболита триптофана, например, триптамина или 5-гидрокситриптамина. В некоторых случаях полезный микроорганизм может метаболизировать триптофан до триптамина или 5-гидрокситриптамина. В некоторых случаях микроорганизм может экспрессировать триптофан-декарбоксилазу или другой фермент, например, тирозин-карбоксилазу или фенилаланин-карбоксилазу, которые также могут метаболизировать триптофан до триптамина или 5-гидрокситриптамина. В некоторых вариантах осуществления один или более видов бактерий включены в композицию, и комбинация может увеличить количество триптамина или 5-гидрокситриптамина, например, в культуре или у субъекта по сравнению с уровнем, если в культуре обеспечивают или субъекту предлагается только один или другой вид.
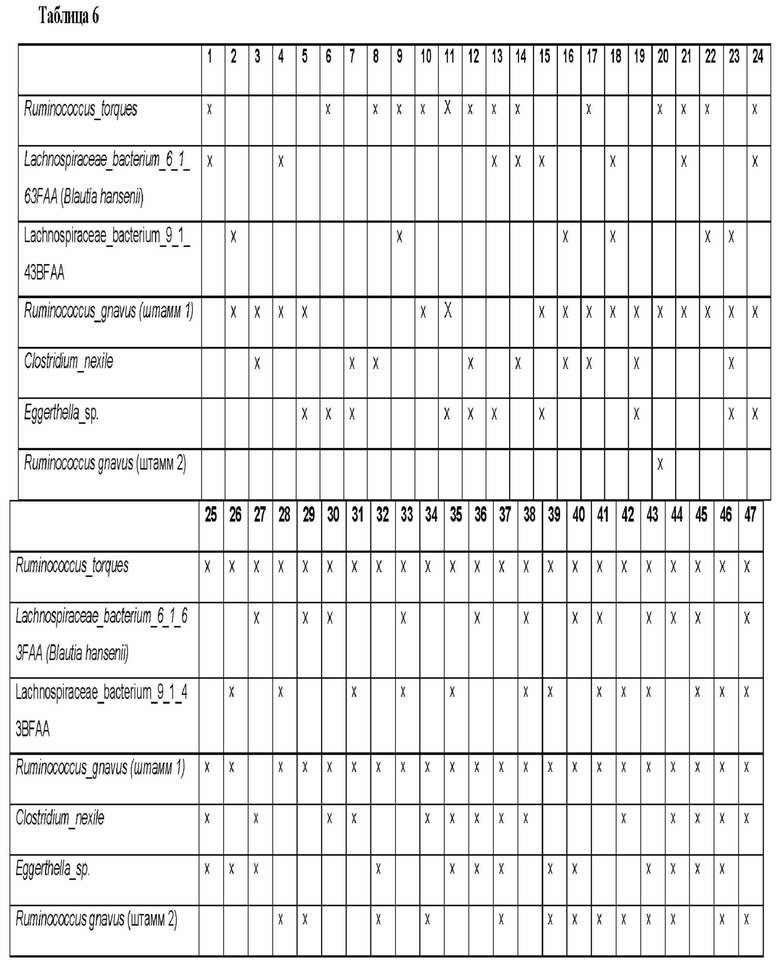
Неограничивающие примеры микроорганизмов, пригодных для применения в предложенных в данном документе изобретениях, включают те, которые перечислены в Таблицах 1-5. Изобретение обеспечивает композиции, содержащие любой из этих микроорганизмов или комбинации, например, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12 или более из этих микроорганизмов, и способы их применения. В отношении комбинаций изобретение обеспечивает композиции, содержащие или состоящие из любого одного или более консорциумов 1-47, как указано в Таблице 6 (композиции 1-47). Изобретение дополнительно включает способы применения этих композиций, как описано в данном документе.
В Таблице 1 приведены неограничивающие примеры видов, которые могут быть включены в композиции согласно изобретению, а также ссылки на последовательности 16S рДНК, которые можно использовать для идентификации этих видов, и дополнительные примеры родственных видов и штаммов. Было обнаружено, что виды, перечисленные в Таблице 1, являются прогностически значимыми для триптамина в двух клинических исследованиях (см., например, Пример 2, Фиг. 3 и Таблицу 9). Композиции согласно изобретению могут содержать 1, 2, 3, 4, 5, 6 или 7 видов, перечисленных в Таблице 1, в любой комбинации друг с другом или с другими видами, перечисленными в данном документе (например, в разных таблицах). Информация о последовательности 16S рДНК для этих и других видов, полезных в композициях и способах согласно изобретению, представлена на Фиг. 5.
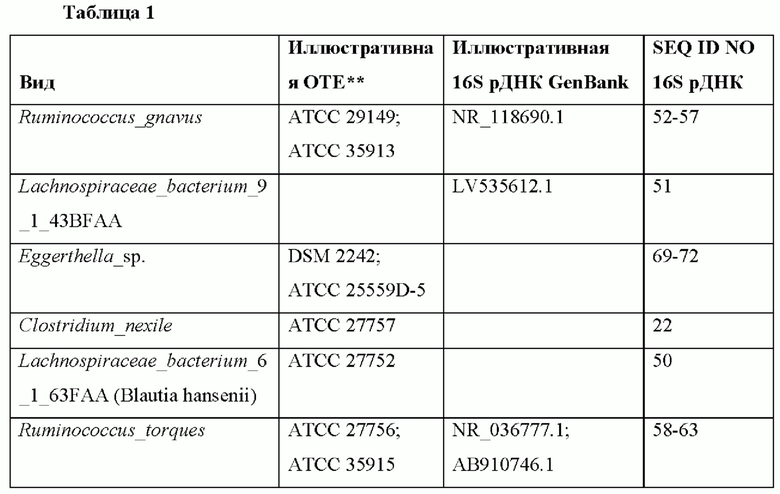

В Таблице 2 приведены дополнительные неограничивающие примеры видов, которые можно использовать в композициях и способах согласно изобретению, а также ссылки на последовательности 16S рДНК, которые можно использовать для идентификации этих видов, и дополнительные примеры родственных видов и штаммов, идентифицированных при помощи скрининга in vitro. Композиции согласно изобретению могут содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или 11 видов, перечисленных в Таблице 2, в любой комбинации друг с другом или с другими видами, перечисленными в данном документе (например, в разных таблицах). Информация о последовательности 16S рДНК для этих и других видов, полезных в композициях и способах согласно изобретению, представлена на Фиг. 5. Эти последовательности можно использовать для идентификации этих и родственных видов и штаммов.
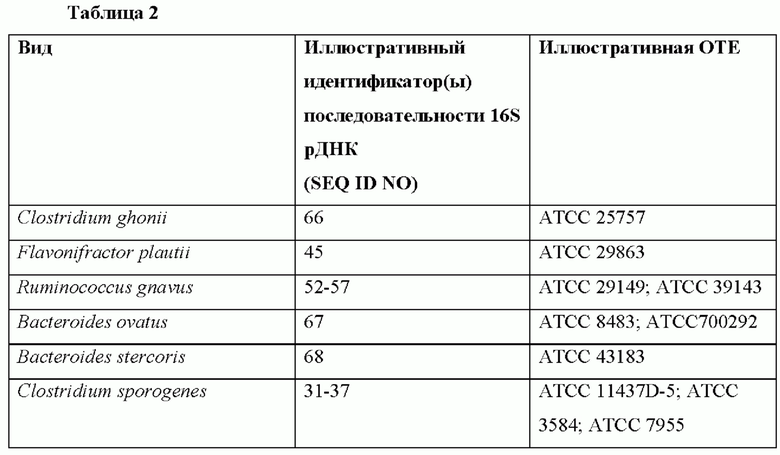
В Таблице 3 приведены дополнительные, неограничивающие примеры видов, которые могут быть использованы в композициях и способах согласно изобретению, а также ссылки на последовательности 16S рДНК, которые можно использовать для идентификации этих видов, и дополнительные примеры родственных видов и штаммов. Композиции согласно изобретению могут содержать 1, 2, 3, или 4 вида, перечисленные в Таблице 3, в любой комбинации друг с другом или с другими видами, перечисленными в данном документе (например, в разных таблицах). Информация о последовательности 16S рДНК для этих и других видов, полезных в композициях и способах согласно изобретению, представлена на Фиг. 5. Эти последовательности можно использовать для идентификации этих и родственных видов и штаммов.
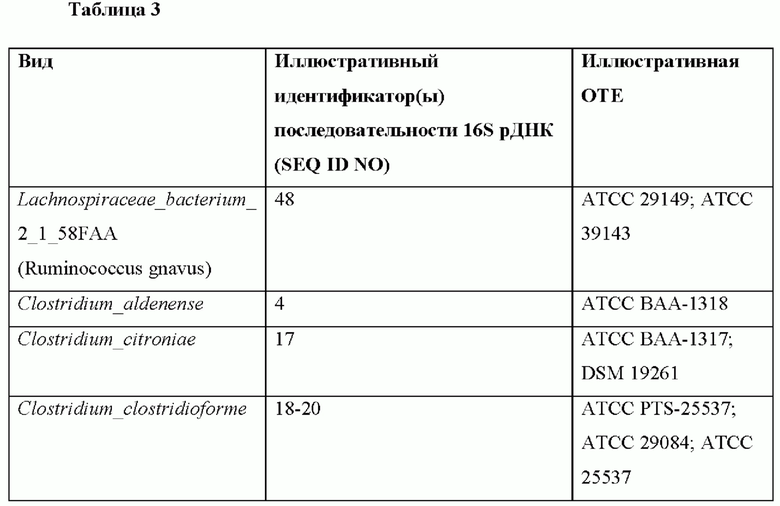
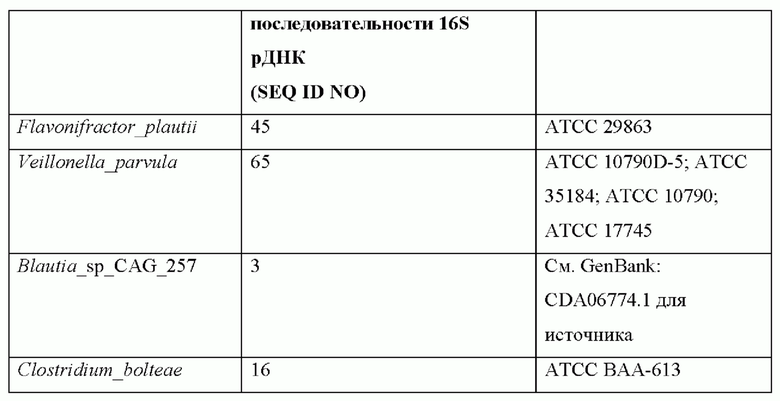
В Таблице 4 приведены дополнительные, неограничивающие примеры видов, которые могут быть использованы в композициях и способах согласно изобретению, а также ссылки на последовательности 16S рДНК, которые можно использовать для идентификации этих видов, и дополнительные примеры родственных видов и штаммов. Композиции согласно изобретению могут содержать 1, 2, 3, или 4 вида, перечисленные в Таблице 4, в любой комбинации друг с другом или с другими видами, перечисленными в данном документе (например, в разных таблицах). Информация о последовательности 16S рДНК для этих и других видов, полезных в композициях и способах согласно изобретению, представлена на Фиг. 5. Эти последовательности можно использовать для идентификации этих и родственных видов и штаммов.

В Таблице 5 приведены дополнительные, неограничивающие примеры видов, которые могут быть использованы в композициях и способах согласно изобретению, а также ссылки на последовательности 16S рДНК, которые можно использовать для идентификации этих видов, и дополнительные примеры родственных видов и штаммов. Композиции согласно изобретению могут содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, или 12 видов, перечисленных в Таблице 5, в любой комбинации друг с другом или с другими видами, перечисленными в данном документе (например, в разных таблицах). Информация о последовательности 16S рДНК для этих и других видов, полезных в композициях и способах согласно изобретению, представлена на Фиг. 5. Эти последовательности можно использовать для идентификации этих и родственных видов и штаммов.
Как отмечено выше, изобретение включает композиции, содержащие комбинации видов бактерий, как описано в данном документе, способы применения этих композиций для изменения (например, повышения) уровней триптамина и способы лечения заболеваний и патологических состояний, при которых изменение (например, повышение) уровней триптамина было бы полезным. Конкретные неограничивающие примеры 47 таких комбинированных композиций приведены в Таблице 6.
Дополнительные микроорганизмы, которые могут быть полезны в изобретениях, представляют собой Clostridium argentinese, Clostridium polymyxa, Erysipelothrix rhusiopathiae, Staphylococcus epidermidis, Staphylococcus haemolyticus, Staphylococcus carnosus, Corynebacterium pyruviciproducens, Staphylococcus pseudintermedius, Corynebacterium glucuronolyticum, Bacillus atrophaeus, Bacillus weihenstephanensis, и непатогенный Bacillus anthracis. Необязательно, один или более из этих дополнительных микроорганизмов могут быть использованы в комбинации с любой из комбинаций, описанных в непосредственно предшествующем абзаце.
В некоторых вариантах осуществления полезный микроорганизм содержит последовательность ДНК, спрогнозированную для кодирования белка, имеющего по меньшей мере 93% идентичности с эталонной последовательностью Trp-декарбоксилазы (полноразмерной последовательностью или триптофан-связывающей областью). В некоторых вариантах осуществления микроорганизм содержит последовательность ДНК, спрогнозированную для кодирования белка, имеющего по меньшей мере 40%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 92%, 93%, 94%, 95%, 98%, 99% или 100% идентичности с эталонной последовательностью Trp-декарбоксилазы. Неограничивающие примеры эталонных последовательностей представляют собой GenBank: EDU35915.1 (SEQ ID NO: 73), GenBank: EDN78222.1 (SEQ ID NO: 74), GenBank ZP_02040762 (SEQ ID NO: 75).
В некоторых вариантах осуществления полезный микроорганизм содержит последовательность 16S рДНК, имеющую идентичность последовательности с одной или более последовательностями 16S рДНК видов, перечисленных в данном документе (например, один или более видов, перечисленных в одной или более Таблицах 1-6; см., например, Фиг. 5 и SEQ ID NO: 1-66). В некоторых вариантах осуществления виды идентифицируют по идентичности последовательностей всей или части последовательности 16S рДНК, например, по меньшей мере 90%, 93%, 95%, 96%, 97%, 98%, 99%, или 100% идентичности. Процент идентичности между эталонной и искомой последовательностью может быть определен с использованием способов, известных в данной области техники. Неограничивающие примеры способов для таких определений представлены ниже. Как используется в данном документе, сходство между двумя нуклеотидными последовательностями описывается параметром «идентичность».
В одном варианте осуществления изобретения, степень идентичности последовательностей между искомой последовательностью и эталонной последовательностью определяют путем 1) выравнивания двух последовательностей с помощью любой подходящей программы выравнивания, используя весовую матрицу по умолчания и штраф за открытие гэпа по умолчанию, 2) идентификации количества точных совпадений, где точное совпадение - это когда программа выравнивания идентифицировала идентичный нуклеотид в двух выровненных последовательностях в данном положении выравнивания и 3) деления числа точных совпадений на длину эталонной последовательности.
В другом варианте осуществления изобретения, степень идентичности последовательностей между искомой последовательностью и эталонной последовательностью определяют путем 1) выравнивания двух последовательностей с помощью любой подходящей программы выравнивания, используя весовую матрицу по умолчания и штраф за открытие гэпа по умолчанию, 2) идентификации количества точных совпадений, где точное совпадение - это когда программа выравнивания идентифицировала идентичный нуклеотид в двух выровненных последовательностях в данном положении выравнивания и 3) деления числа точных совпадений на длину самой длинной из двух последовательностей.
В другом варианте осуществления изобретения степень идентичности последовательностей между искомой последовательностью и эталонной последовательностью определяют путем 1) выравнивания двух последовательностей с помощью любой подходящей программы выравнивания, используя весовую матрицу по умолчания и штраф за открытие гэпа по умолчанию, 2) идентификации количества точных совпадений, где точное совпадение - это когда программа выравнивания идентифицировала идентичную аминокислоту или нуклеотид в двух выровненных последовательностях в данном положении выравнивания и 3) деления числа точных совпадений на «длину выравнивания», где длина выравнивания - это длина всего выравнивания, включая гэпы и выступающие части последовательностей.
Сравнение идентичности последовательностей, как правило, осуществляют с помощью программы сравнения последовательностей. Эти коммерчески или общедоступные компьютерные программы используют сложные алгоритмы сравнения для выравнивания двух или более последовательностей, которые лучше всего отражают эволюционные события, которые могли бы привести к различию(ям) между двумя или более последовательностями. Следовательно, эти алгоритмы работают с системой весов с вознаграждением за выравнивание идентичных или сходных аминокислот и штрафом за вставку гэпов, продление гэпов и выравнивание несходных аминокислот. Система весов алгоритмов сравнения включает:
i) присвоение штрафного балла каждый раз, когда вставляется гэп (штрафной балл
за гэп),
ii) присвоение штрафного балла каждый раз, когда существующий гэп увеличивается на дополнительное положение (дополнительный штрафной балл),
iii) присвоение высоких баллов при выравнивании идентичных аминокислот, и
iv) присвоение вариабельных баллов при выравнивании неидентичных аминокислот.
Как правило, в программе выравнивания для сравнения последовательностей используются значения по умолчанию. Подходящие компьютерные программы, применимые для определения идентичности, включают, например, BLAST (blast.ncbi.nlm.nih.gov).
В варианте осуществления данного изобретения программа выравнивания оптимизирует выравнивание по всей длине выбранных последовательностей, например, полноразмерной последовательности, последовательности V4 или V6 16S рДНК. Например, программа для глобального выравнивания основана на алгоритме Нидлмана-Вунша (Needleman and Wunsch, J. Mol. Biol. 48:443-453, 1970). Неограничивающими примерами таких программ являются программы EMBOSS Needle и EMBOSS Stretcher, доступные по адресу ebi.ac.uk/Tools/psa/.
В одном варианте осуществления последовательности выравниваются с помощью программы для глобального выравнивания, а идентичность последовательностей вычисляют путем определения числа точных совпадений, идентифицированных программой, деленного на «длину выравнивания», где длина выравнивания является длиной всего выравнивания, включая гэпы и выступающие части последовательностей. В дополнительном варианте осуществления в программе для глобального выравнивания используется алгоритм Нидлмана-Вунша, а идентичность последовательностей вычисляют путем определения числа точных совпадений, идентифицированных программой, деленного на «длину выравнивания», где длина выравнивания является длиной всего выравнивания, включая гэпы и выступающие части последовательностей.
В еще одном дополнительном варианте осуществления программу для глобального выравнивания выбирают из группы, состоящей из EMBOSS Needle и EMBOSS Stretcher, а идентичность последовательностей вычисляют путем определения числа точных совпадений, идентифицированных программой, деленного на «длину выравнивания», где длина выравнивания является длиной всего выравнивания, включая гэпы и выступающие части последовательностей.
После того как программное обеспечение произвело выравнивание, можно рассчитать процент (%) сходства и процент идентичности последовательностей.
В некоторых вариантах осуществления по меньшей мере один микроорганизм в композиции может экспрессировать триптофан-декарбоксилазу. В некоторых вариантах осуществления по меньшей мере один микроорганизм в композиции может секретировать триптамин. Способы определения секреции триптамина известны в данной области техники (например, Williams et al., Cell Host Microbe 16:495-503, 2014). В других вариантах осуществления по меньшей мере один микроорганизм в композиции обладает активностью в анализе транзита в ЖКТ (см., например., Yano et al., Cell 161:264-276, 2015).
В некоторых вариантах осуществления композиция ТА содержит от 10е2 до 10е12 (например, 10е8-10е11 или 10е9-10е10) жизнеспособных бактерий и/или спор, которые могут экспрессировать или могут быть индуцированы для экспрессии триптофан-декарбоксилазы.
В некоторых вариантах осуществления композиция ТА может индуцировать повышение уровней триптамина у человека или животного, отличного от человека, например, мыши, крысы, морской свинки, собаки, кошки, свиньи, овцы или примата, отличного от человека.
Как правило, один или более микроорганизмов, полезных в изобретении, являются факультативными или облигатными анаэробами. В некоторых случаях один или более микроорганизмов являются облигатными анаэробами, которые проявляют аэротолерантность. Организмы могут быть способными или образовывать споры (называемые в данном документе «спорообразующими микроорганизмами») или могут быть необразующими споры микроорганизмами. Спорообразующие микроорганизмы могут присутствовать в композиции в основном в виде спор, вегетативных клеток или комбинации спор и вегетативных клеток.
Способы определения триптамина
В некоторых вариантах осуществления изобретения определяют наличие и, в некоторых случаях, уровни триптамина. Триптамин может быть проанализирован с помощью методов, известных в данной области техники. Например, триптамин может быть проанализирован с помощью жидкостной хроматографии в сочетании с тандемной масс-спектрометрией (ЖХ-МС/МС) с использованием тройного квадрупольного масс-спектрометра Thermo TSQ Quantum Мах. Другие подходящие способы включают селективную регистрацию избранных реакций распада нескольких ионов и информационный мониторинг (Sridharan et al., Nature Communications 5:5492, 2014).
Уровни триптамина у субъекта, например, до и после введения композиции ТА, можно анализировать с использованием крови, плазмы, сыворотки, стула, мочи или спинномозговой жидкости.
Триптофан-декарбоксилаза
В некоторых вариантах осуществления композиция содержит по меньшей мере один вид бактерий, кодирующий ген триптофан-декарбоксилазы. Способы идентификации таких генов известны в данной области техники, например, см. Williams et al. (выше). Как описано в примерах, анализ последовательности спрогнозированной структуры белка и сравнение с известными последовательностями триптофан-декарбоксилазы микроорганизмов могут быть использованы для идентификации генов в бактериальном геноме. В некоторых случаях в анализе используют всю последовательность гена декарбоксилазы триптофана и/или последовательность спрогнозированного сайта связывания для триптофана.
Лечение заболевания
В вариантах осуществления изобретения композицию микробиома используют для лечения или предотвращения заболевания или патологического состояния, включая ослабление признака или симптома заболевания у субъекта, нуждающегося в лечении. В некоторых вариантах осуществления способы in silico, in vitro и/или in vivo можно использовать для определения эффективности лечения в изменении уровней триптамина у субъекта.
Триптамин представляет собой агонист Ahr. Сообщается, что активация Ahr может влиять на экспрессию FoxP3 и ИЛ-17 и ослаблять течение колита, снижать риск канцерогенеза, и может оказывать другие эффекты, известные в данной области техники. Описанные в литературе эффекты триптамина включают стимуляцию секреции ионов эпителиальными клетками кишечника и усиление перистальтики кишечника (Bhattarai et al., Cell Host Microbe 23(6):775-785, 2018). Триптамин также был предложен в качестве агента для увеличения количества регуляторных Т-клеток (Трег; US 9028798). Соответственно, композиции, которые могут увеличивать содержание триптамина, полезны, например, для восстановления или улучшения гомеостаза кишечника, лечения колита, лечения или профилактики злокачественного новообразования кишечника и/или улучшения перистальтики желудочно-кишечного тракта. В общем, композиция ТА, которая может повышать уровни триптамина, полезна для модуляции активности рецептора ароматических углеводородов. Композиции ТА также могут быть использованы для увеличения количества Трег.
Композиции согласно изобретению также могут быть использованы для модуляции (например, увеличения) 5-гидрокситриптамина или серотонина. Соответственно, композиции и способы согласно изобретению можно использовать при лечении заболеваний и патологических состояний, связанных с перистальтикой кишечника, гомеостазом кишечника, агрегацией тромбоцитов, иммунными ответами и функцией сердца, а также депрессией, тревожным расстройством, синдромом раздраженного кишечника (СРК), сердечно-сосудистым заболеванием и остеопорозом.
Пациенты
В некоторых вариантах осуществления субъект, подходящий для лечения композицией ТА, идентифицируют как имеющий ненормальный или нежелательно низкий уровень триптамина или 5-гидрокситриптамина, например, в кале, крови, плазме, сыворотке, моче, спинномозговой жидкости (СМЖ) или другой жидкости организма. Эффективное лечение композицией ТА повышает уровень триптамина или 5-гидрокситриптамина в выбранной жидкости организма, ткани или кале. Как правило, повышенный уровень триптамина или 5-гидрокситриптамина связан с обнаруживаемым присутствием по меньшей мере одного вида из композиции ТА в желудочно-кишечном тракте получающего лечение субъекта. В некоторых вариантах осуществления присутствие по меньшей мере одного вида или штамма бактерии из композиции ТА обнаруживают в стуле получающего лечение субъекта. В некоторых вариантах осуществления по меньшей мере один вид или штамм композиции ТА может быть обнаружен по меньшей мере через 1 сутки, 2 суток, 3 суток, 4 суток, 5 суток, 6 суток, 1 неделю, две недели, три недели, 4 недели, 2 месяца, 3 месяца, 4 месяца, 5 месяцев, 6 месяцев, 8 месяцев или 1 год после лечения композицией ТА. Такая персистентность вводимого вида (например, при отсутствии или низком уровне этого вида до начала лечения) называется «приживлением». В некоторых вариантах осуществления вводимый вид может не сохраняться или приживаться в течение ограниченного периода времени; такие виды можно вводить в повторных дозах композиции ТА.
Субъект, получающий лечение в соответствии со способами согласно изобретению, может иметь заболевание или патологическое состояние, характеризующееся измененной перистальтикой кишечника. В различных примерах субъект, получающий лечение в соответствии со способами изобретения, может иметь заболевание или патологическое состояние желудочно-кишечного тракта, выбранное из группы, состоящей, например, из синдрома раздраженного кишечника, воспалительных заболеваний кишечника (например, инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита) и запора (также см. выше).
Определения
«Синергия» относится к эффекту, вызываемому комбинацией, например, разных микроорганизмов (например, разных штаммов, разных видов или разных клад), который превышает ожидаемые аддитивные эффекты компонентов комбинации. Используемые в данном документе термины «синергия» или «синергетические взаимодействия» относятся к кооперации или взаимодействию двух или более микроорганизмов для получения комбинированного эффекта, превышающего сумму их отдельных эффектов. Эффект может быть основан на наблюдениях in vitro или in vivo.
«Терапевтически эффективное количество» композиции ТА, описанной в данном документе, может варьироваться в зависимости от таких факторов, как течение заболевания, возраст, пол и масса индивидуума и способность соединения вызывать желаемый ответ у индивидуума, например, улучшение по меньшей мере одного параметра расстройства, или улучшение по меньшей мере одного симптома расстройства (и, необязательно, эффекта от введения любых дополнительных агентов). Терапевтически эффективное количество также является таким, в котором любые токсичные или вредные эффекты композиции перевешиваются терапевтически благоприятными эффектами. Композицию, как описано в данном документе, обычно вводят в терапевтически эффективном количестве.
Термин «приживление микроорганизмов» или «приживление» относится к приживаемости одного или более штаммов, ОТЕ или видов, вводимых в терапевтическую композицию микроорганизмов, например, бактериальную композицию, в целевой нише, которые отсутствуют, не обнаруживаются или присутствуют в нежелательно низких уровнях у получающего лечение субъекта до начала лечения. Микробы, входящие в состав приживленной экологии, присутствуют в терапевтической композиции микроорганизмов и являются компонентами экологии микроорганизмов у субъекта. Приживление ОТЕ может происходить в течение переходного периода времени или они могут демонстрировать долговременную стабильность в экологии микроорганизмов, которые заселяют субъекта после лечения терапевтической композицией микроорганизмов. Приживление могут быть определено с помощью различных молекулярных технологий, известных в данной области техники, включая, но не ограничиваясь этим, полное метагеномное секвенирование (WMS), ПЦР и количественной ПЦР с праймерами, специфичными к штамму или таксону, или с ДНК-зондами, специфичными к штамму или таксону. С помощью данных полного метагеномного секвенирования (WMS) приживление может быть оценено количественно с использованием геномных вариантов, специфичных к штамму, включая, но не ограничиваясь, однонуклеотидные варианты (SNV). С помощью данных полного метагеномного секвенирования (WMS) приживление штаммов, видов или других таксонов также может быть оценено количественно на основе геномных маркеров, специфичных для штаммов, видов или других таксонов. С помощью данных полного метагеномного секвенирования (WMS) обнаружению приживления также может помочь сборка полного генома. После того, как такие данные получены, приживление может быть оценено статистически на уровне субъекта и/или уровне популяции; на уровне субъекта специфическое для штамма или таксона относительное содержание, относительная распространенность, или обнаружение должно быть больше, чем на исходном уровне или предварительно заданном уровне фонового шума; на уровне популяции специфическое для штамма или таксона относительное содержание, относительная распространенность, или сигнал обнаружения должны быть больше, чем у эталонной группы плацебо или контрольной фоновой популяции или набора данных.
Пределы обнаружения (LOD) могут варьироваться в зависимости, например, от конкретных анализируемых типов образцов (например, образца стула и содержания в нем воды), обнаруживаемых видов и используемых анализов, как понятно в данной области техники. В различных примерах LOD, используемые в изобретении, находятся в диапазоне от около 10е5 до 10е7. В одном конкретном примере применяемый LOD составляет около 1,75е6 (например, 1,75е6).
«Операционная таксономическая единица», «ОТЕ» (или в множественном числе, «ОТЕ») относится к терминальному листу в филогенетическом дереве и определяется последовательностью нуклеиновой кислоты, например, полным геномом или конкретной генетической последовательностью, и всеми последовательностями, которые имеют идентичность последовательности с этой последовательностью нуклеиновой кислоты на уровне видов. В некоторых вариантах осуществления конкретная генетическая последовательность представляет собой последовательность 16S или часть последовательности 16S, такую как вариабельная область, например, область V4. В других вариантах осуществления полные геномы двух объектов секвенируют и сравнивают. В другом варианте осуществления можно генетически сравнивать отдельные области, такие как маркеры мультилокусных последовательностей (MLST), конкретные гены или наборы генов. В вариантах осуществления 16S ОТЕ, которые имеют ≥97% средней идентичности нуклеотидов по всей 16S или вариабельной области последовательности 16S, считаются одной и той же ОТЕ (см., например, Claesson et al., Nucleic Acids Res 38:e200, 2010; Konstantinidis et al., Philos Trans R Soc Lond В Biol Sci 361:1929-1940, 2006). В вариантах осуществления, включающих полный геном, MLST, специфические гены или наборы генов, ОТЕ, которые имеют ≥95% средней идентичности нуклеотидов, считаются одной и той же ОТЕ (см., например, Achtman and Wagner, Nat. Rev. Microbiol. 6:431-440, 2008). В некоторых вариантах осуществления ОТЕ можно различить путем сравнения последовательностей между организмами. Как правило, последовательности с идентичностью последовательностей менее 95% не считаются частью одной и той же ОТЕ. ОТЕ также могут быть характеризованы по любой комбинации нуклеотидных маркеров или генов, в частности высококонсервативных генов (например, генов «домашнего хозяйства»), или их комбинацией. В таком определении характеристик используются, например, данные WGS или полногеномная последовательность. Используемые в данном документе термины «виды» и «ОТЕ» используются взаимозаменяемо, если иное не отличается контекстом.
Составы
Композиции ТА, описанные в данном документе, могут быть получены и введены с использованием способов, известных в данной области техники. Как правило, композиции составляют для пероральной, колоноскопической или назогастральной доставки, хотя может использоваться любой подходящий способ.
Состав ТА может содержать один или более фармацевтических наполнителей, подходящих для приготовления таких составов. В некоторых вариантах осуществления состав представляет собой жидкий состав. В некоторых вариантах осуществления состав, содержащий композицию ТА, может содержать одно или более поверхностно-активных веществ, адъювантов, буферов, антиоксидантов, регуляторов тоничности, загустителей или модификаторов вязкости и тому подобное.
В некоторых вариантах осуществления лечение включает введение фармацевтической композиции ТА в составе, который содержит фармацевтически приемлемый носитель. В некоторых вариантах осуществления наполнитель включает капсулу или другой формат, подходящий для обеспечения композиции ТА в виде лекарственной формы для перорального применения. Когда носитель служит разбавителем, он может быть твердым, полутвердым или жидким материалом, который действует в качестве вспомогательного вещества, носителя или среды для активного ингредиента. Таким образом, составы могут быть в форме таблеток, пилюль, порошков, пастилок, саше, капсул, эликсиров, суспензий, эмульсий, растворов, сиропов, мягких или твердых капсул, суппозиториев или упакованных порошков.
Некоторые примеры подходящих наполнителей включают лактозу, декстрозу, сахарозу, сорбит, маннит, крахмалы, аравийскую камедь, фосфат кальция, альгинаты, трагакант, желатин, силикат кальция, микрокристаллическую целлюлозу, поливинилпирролидон, целлюлозу, воду, сироп, полиэтиленгликоль, глицерин, и метилцеллюлозу. Композиции могут быть составлены так, чтобы обеспечить быстрое, пролонгированное или отсроченное высвобождение активного ингредиента после введения пациенту с использованием процедур, известных в данной области техники.
Композиция ТА может быть составлена в виде единичной лекарственной формы. В общем случае доза составляет от около 10е2 до 10е9 жизнеспособных колониеобразующих единиц (КОЕ). Термин «единичные лекарственные формы» соответствует физически дискретным единицам, пригодным в качестве единичных доз для человека и/или других млекопитающих, причем каждая единичная лекарственная форма содержит заранее определенное количество активного материала, рассчитанное для получения желаемого терапевтического эффекта, в комбинации с подходящим фармацевтическим наполнителем. Дозировка может вводиться в нескольких средствах доставки, например, в нескольких таблетках или капсулах.
Количество и частота введения пациенту композиции ТА будет варьироваться в зависимости от того, что вводится, цели введения, такой как профилактика или терапия, состояния пациента, способа введения и тому подобного. В терапевтических применениях композиции можно вводить пациенту, уже страдающему от заболевания, в количестве, достаточном для излечения или по меньшей мере частичного купирования симптомов заболевания и его осложнений. Эффективные дозы будут зависеть от болезненного состояния, подвергаемого лечению, а также от решения лечащего врача в зависимости от таких факторов, как тяжесть заболевания, возраст, масса и общее состояние пациента и тому подобное.
Дозировка может относиться, например, к общему количеству КОЕ каждого отдельного вида или штамма; или может относиться к общему количеству микроорганизмов в дозе. В данной области техники понятно, что определение количества организмов в дозировке не является точным и может зависеть от способа, используемого для определения количества присутствующих организмов. Например, количество спор в композиции может быть определено с использованием анализа дипиколиновой кислоты. В некоторых случаях количество организмов может быть определено с помощью анализов на основе культуральных приемов. Если присутствуют споры, способность анализов, основанных на методах культивирования, может зависеть от эффективности прорастания спор. Методы количественного определения нуклеиновых кислот могут зависеть от того, достаточно ли снижено содержание или удалены нуклеиновые кислоты нежизнеспособных микроорганизмов.
Эффективные дозы могут быть экстраполированы из кривых доза-ответ, полученных в лабораторных или модельных испытаний на животных системах.
ЭКВИВАЛЕНТЫ
Все технические признаки могут быть отдельно объединены во все возможные комбинации таких признаков.
Изобретение может быть воплощено в других конкретных формах без отклонения от сущности или его существенных характеристик. Таким образом, вышеприведенные варианты осуществления следует рассматривать во всех отношениях как иллюстративные, а не ограничивающие изобретение, описанное в данном документе.
ПРИМЕРЫ
Следующие неограничивающие примеры дополнительно иллюстрируют варианты осуществления изобретений, описанных в данном документе.
Пример 1. Геномный и метаболомный анализ кала человека
Образцы кала человека, полученные от субъектов, участвующих в исследовании II фазы композиции микробиома, состоящей из инкапсулированных очищенных спор эубактерий (clinicaltrials.gov; NCT02437487), предназначенного для оценки безопасности и эффективности композиции микробиома. Образцы были взяты у субъектов, участвовавших в исследовании NCT02437487, включая образцы как от тех субъектов, которые получали лечение, так и от тех, кто получал плацебо. Метаболомный анализ образцов на наличие триптамина проводили в компании Metabolon (Моррисвилл, Северная Каролина). Метаболомные данные включали уровни триптамина на массу стула (пик AUC, пропорциональный концентрации и предполагающий линейную кинетику анализа). Образцы также секвенировали с использованием полного метагеномного секвенирования методом дробовика, а название вида устанавливали на основе видоспецифичных геномных маркеров с использованием базы данных MetaPhlAn2 (Truong et al., Nat. Methods 12:902-903, 2015).
Идентификация взаимосвязей вид-метаболит
Стандартный анализ парных таксономических и метаболических профилей часто включает попарную корреляцию (например, корреляцию Спирмена или Пирсона) между видами и относительным содержанием метаболитов для идентификации видов, относительное содержание которых коррелирует с относительным содержанием метаболитов. Этот тип корреляционного анализа обычно приводит к тому, что большие группы видов коррелируют с большими группами метаболитов, что было замечено как в когортных, так и в интервенционных исследованиях. Это означает, что стандартный корреляционный анализ не позволяет адекватно определить, какие виды действительно механистически вовлечены в выбранную метаболическую функцию.
Для решения этой проблемы заявители использовали новый подход к выявлению специфических взаимосвязей вид-метаболит в парных таксономических и метаболомных профилях. Были проведены вычислительные анализы, которые анализируют взаимосвязь между наличием и уровнем триптамина и наличием отдельных видов бактерий и их комбинаций. Кроме того, были проведены анализы, которые оценивают относительное содержание бактерий и триптамина.
В рамках этого способа была разработана модель, которая может научиться прогнозировать уровни метаболитов в образцах пациентов из профилей видов, определенных в этих образцах. Уровни метаболита прогнозируют на основе изучения лежащих в их основе взаимосвязей вид-метаболит. Наборы данных для обучения были разработаны с использованием подхода перекрестной проверки при помощи методов, известных в данной области техники.
Когда уровень метаболита действительно определяется небольшим количеством видов, такой подход к моделированию восстанавливает разреженный набор взаимосвязей видов и метаболит. Напротив, стандартный корреляционный анализ, который применяется к этим данным, восстанавливает неразреженные взаимосвязи, что препятствует возможности идентифицировать определенные виды, связанные с определенными метаболическими функциями (что необходимо для разработки терапевтических композиций микроорганизмов).
После того как были определены основные взаимосвязи вид-метаболит, была проведена оценка того, а) насколько хорошо идентифицированные виды могут прогнозировать уровни метаболитов in vivo, и b) сколько видов требуется для прогнозирования уровней метаболитов in vivo (то есть много ли или мало видов влияют на изменение уровней метаболитов in vivo). Что касается (а), было обнаружено, что некоторые метаболиты хорошо спрогнозированы по профилям видов и поэтому, вероятно, могут модулироваться консорциумом микроорганизмов, в то время как другие метаболиты не являются таковыми и поэтому, вероятно, для них преобладают внешние факторы или факторы хозяина. Что касается (b), мы обнаружили, что для некоторых метаболитов изменение уровней метаболитов in vivo можно объяснить наличием/отсутствием небольшого числа видов (например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 14, 16, 18 или 20 видов), тогда как для других метаболитов для объяснения уровней метаболитов in vivo требуется присутствие/отсутствие большого количества видов (например, >20-50 видов).
Полученные таким образом данные показали, что триптамин является метаболитом, изменение которого хорошо прогнозируется наличием/отсутствием небольшого числа видов. Таким образом, уровнями триптамина in vivo можно манипулировать путем терапевтического введения небольшого числа видов, связанных с уровнями триптамина. Кроме того, поскольку на уровни триптамина влияют несколько видов бактерий, вполне вероятно, что для обеспечения триптамина терапевтическим консорциумом микроорганизмов можно использовать только ограниченное число видов бактерий.
Пример 2. Виды бактерий, прогнозирующие уровни триптамина in vivo.
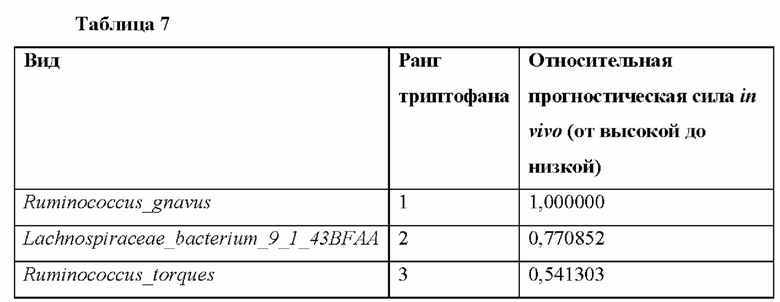
Анализы, связанные с триптамином, описанные в Примере 1, выявили небольшое число видов, присутствие которых было связано с присутствием триптамина в образце кала. В Таблице 7 представлен ранжированный список (из наиболее сильно связанных с наличием триптамина, ранжированных как 1) из трех видов, наиболее сильно связанных с присутствием триптамина; Ruminococcus gnavus, Lachnospiraceae bacterium 9_1_43BFAA, и Ruminococcus torques были тремя видами, связанными с самыми высокими уровнями триптамина.
На Фиг. 1 представлен анализ трех видов, связанных с самыми высокими уровнями триптамина и их относительными уровнями триптамина в присутствии одного, двух или трех видов. Столбики представляют собой средние относительные уровни триптамина, связанные с указанными видами, когда они присутствуют в образце отдельно или в различных комбинациях. Хотя наблюдается тенденция к более высоким уровням триптамина в присутствии большего числа видов, результаты не находятся в зависимости от явного аддитивного эффекта, а являются синергетическими в зависимости от присутствия каждого вида. Эти данные указывают на то, что влияние вида на уровни триптамина также зависит от комбинаторного присутствия/отсутствия других видов.
Эти данные демонстрируют, что такой анализ данных in vivo может выявить комбинации бактерий, которые могут влиять на уровни триптамина в организме субъекта.
Поиск гомологии
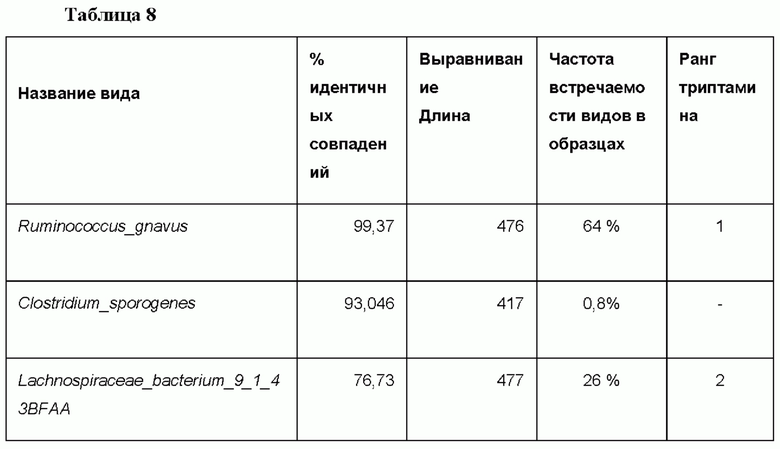
В начальных исследованиях ранее идентифицированные гены триптофан-декарбоксилазы из изолятов Ruminococcus gnavus (идентификационный номер Uniprot, A7B1V0_RUMGN) и Clostridium sporogenes (идентификационный номер Uniprot, J7SZ64_CLOSG) (Williams et al., выше) были использованы в качестве искомых последовательностей для поиска гомологов в других секвенированных бактериальных геномах. Для поиска гомологов с BLASTP была собрана база данных из 1155 бактериальных геномов, связанных с кишечником человека (собранных с использованием информации WGS); пары выровненных остатков оценивали, используя матрицу весов для выравнивания BLOSUM62; были идентифицированы 24 вида, содержащие гомологи белка с Е-значением для выравнивания меньшим чем 1е-100, для каждой из искомых последовательностей (вместе с процентом идентичных совпадений остатков, длиной выравнивания и Е-значением; см. Таблицу 8); среди 24 идентифицированных гомологов процент идентичных остатков составлял 38-99,4%, а длины выравнивания - 407-477. Эти анализы показали удивительные результаты.
Во-первых, из девяти основных видов, связанных с присутствием триптамина, четыре из них, включая Ruminococcus torques (ранг триптамина = 3), не имеют последовательности со значительной гомологией к известной бактериальной триптофан-декарбоксилазе. Эти данные указывают на то, что модуляция уровней триптамина может регулироваться дополнительными ферментами и/или механизмами, помимо тех, которые связаны с известными триптофан-декарбоксилазами.
Во-вторых, для тех видов, которые идентифицированы как имеющие последовательность со значительной гомологией к триптофан-декарбоксилазе, связь этих видов с уровнями триптамина (см. Таблицу 8, ранг триптамина; самый низкий ранг отражает самую высокую связь с триптамином) не соответствует процентной идентичности с «подлинной» бактериальной триптофан-декарбоксилазой.
В-третьих, изучение частоты встречаемости какого-либо отдельного вида показало, что частота встречаемости не является прогностическим фактором для ранга триптамина.
В-четвертых, хотя список видов, идентифицированных как имеющих последовательности с гомологией к триптофан-декарбоксилазе, значительно совпадает со списком видов, прогнозирующих уровни триптамина in vivo, существуют исключения. Например, в репрезентативном геноме R. torques не было обнаружено гомолога последовательности, связанной с триптофан-декарбоксилазой, хотя R. torques занимает третье место по влиянию на уровни триптамина in vivo. Было обнаружено, что Clostridium sporogenes не является прогностическим фактором уровней триптамина in vivo, несмотря на опубликованные данные, указывающие на то, что он несет ген триптофан-декарбоксилазы.
В-пятых, было обнаружено, что ни одна последовательность не имела более чем 30% спрогнозированной идентичности белковой последовательности по длине выравнивания по меньшей мере 100 остатков с опубликованной в литературе последовательностью триптофан-декарбоксилазы С.sporogenes.
В совокупности данные заявителей демонстрируют, что виды, ранее не связанные с присутствием триптамина, могут влиять на продукцию триптамина в желудочно-кишечном тракте и тем самым влиять на уровень триптамина в других тканях.
Кроме того, эти данные демонстрируют, что частота встречаемости видов и степень гомологии с охарактеризованными белками триптофан-декарбоксилазы не связаны с силой взаимосвязей у человека видов in vivo с уровнями триптамина. В совокупности, данные указывают на то, что простое присутствие последовательности триптофан-декарбоксилазы или даже связанной последовательности недостаточно информативно для выбора видов, которые повысят уровни триптамина у субъекта, и представленные в данном документе анализы могут обеспечить эмпирические данные для идентификации видов, влияющих на уровни триптамина, и их применение в композициях для увеличения уровней триптамина у субъекта.
В дальнейших исследованиях при помощи Blastp триптофан-декарбоксилазы в сравнении с более крупной базой данных бактериальных пангеномов (bitbucket.org/biobakery/humann2/wiki/Home; Huang et al., Nucleic Acids Res. D617-24, 2014) выявлены предполагаемые продуценты триптамина (Фиг. 2). В этих исследованиях было идентифицировано семь видов, присутствие которых прогнозирует более высокие концентрации триптамина в двух клинических исследованиях (Фиг. 3; Таблица 9). Пять из семи идентифицированных видов имеют штаммы с идентифицированным гомологом триптофан-декарбоксилазы. Один вид без гомолога (Eggerthella sp.) не имеет связанного генома для оценки.
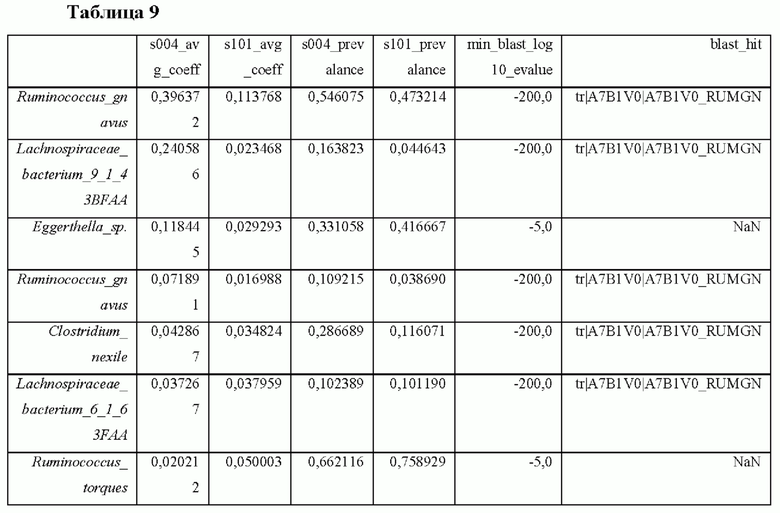
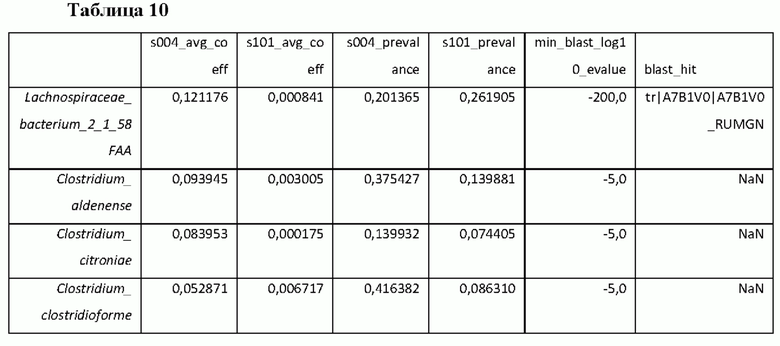
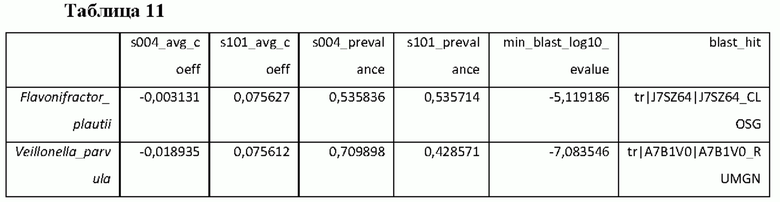
Для Таблиц 9-12 ниже приводятся подробные сведения о заголовках столбцов:
s004_avg_coeff: оценка, указывающая на то, насколько прогностичен вид в отношении уровней триптамина в исследовании SERES-004 (С diff).
s101_avg_coeff: оценка, указывающая на то, насколько прогностичен вид в отношении уровней триптамина в исследовании SERES-101 (язвенный колит).
s004_prevalance: частота встречаемости (относительное содержание в образцах) вида в исследовании SERES-004 (С diff).
s_101_prevalance: частота встречаемости (относительное содержание в образцах) вида в исследовании SERES-101 (язвенный колит).
min_blast_log10_evalue: значимость гомолога для триптофан-декарбоксилазы (чем меньше число, тем лучше, -200 является наименьшим [то есть лучшее совпадение]), -log 10 Е-значения из Blastp.
blast_hit: используемый идентификатор для искомой триптофан-декарбоксилазы.
Четыре дополнительных вида были определены в качестве прогностических для уровней триптамина в клиническом исследовании лечения от С. difficile (Таблица 10).
Четыре дополнительных вида были определены в качестве прогностических для уровней триптамина в клиническом исследовании лечения язвенного колита (Таблица 11).
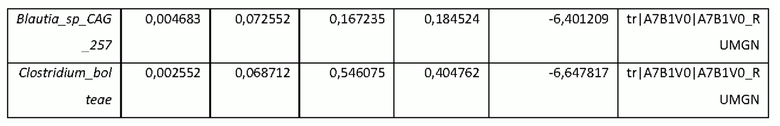
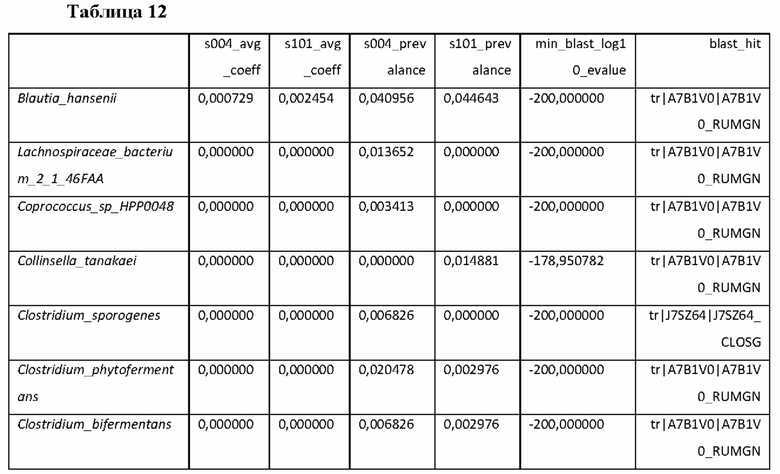
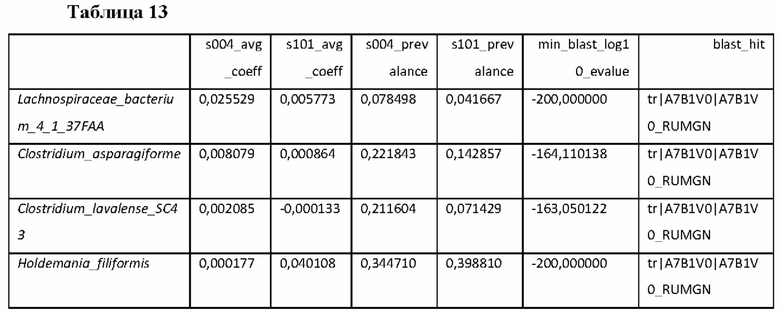
Были идентифицированы другие виды, имеющие гомологию триптофан-декарбоксилазы, но не прогнозирующие уровни триптамина in vivo в исследуемых условиях (Таблицы 12 и 13). Для этих видов гомолог может не обладать активностью в отношении триптофана в качестве субстрата (Williams et al., Cell Host Microbe 16(4):495-503, 2014), или эти виды могут быть незначительно распространены или незначительно встречаться в условиях in vivo.
В Таблице 12 перечислены виды с частотой встречаемости менее 5% в обоих исследованиях (но они были обнаружены по меньшей мере один раз в обоих исследованиях). Эти виды могут оказывать влияние на уровни триптамина in vivo, но их низкая частота встречаемости в этих изученных популяциях означает, что они не могут объяснить существенные различия в уровнях триптамина в изученных популяциях. В Таблице 13 перечислены виды с частотой встречаемости более 5% в обоих исследованиях. Эти виды не объясняют вариации уровней триптамина in vivo, несмотря на их заметную частоту встречаемости в изученных популяциях. Они могут фактически не иметь активности в отношении триптофана в качестве субстрата или могут проявлять вариабельность штамма в отношении активности.

В ходе дополнительных анализов были идентифицированы консорциумы, которые имели комбинации более одного из семи видов, перечисленных в Таблице 1, в клинических исследованиях относительно язвенного колита и С.difficile (Фиг. 4). Эти консорциумы можно использовать, например, в композиции ТА, чтобы влиять на уровни триптамина in vivo в большей степени, чем влияет один отдельный вид.
Пример 2. Обнаружение метаболитов триптофана в супернатантах
Способы
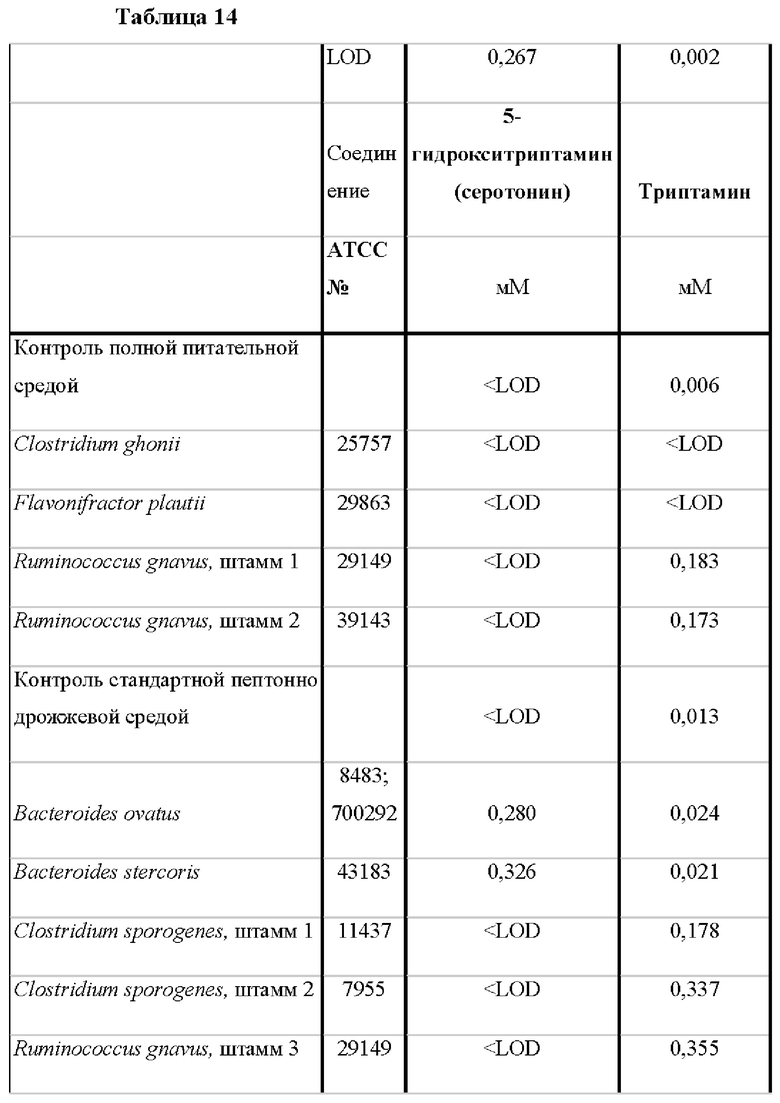
Заявитель протестировал различные виды бактерий на наличие метаболитов триптофана в их супернатантах. Наличие метаболитов триптофана определяли с помощью колориметрического анализа для выявления индольных соединений (реагент на индол, Anaerobe Systems). Супернатанты выбранных штаммов, которые были идентифицированы как продуценты метаболитов триптофана с помощью колориметрического анализа на индол, были дополнительно проанализированы методом ГХ-МС для выявления специфических продуцируемых метаболитов.
Результаты
Было показано, что некоторые виды продуцируют триптамин или 5-гидрокситриптамин (5НТ). В частности, триптамин-продуцирующие виды включают Clostridium sporogenes и Ruminococcus gnavus, два из трех обнаруженных видов коррелируют с присутствием триптамина в образцах кала человека (Таблица 14). 5НТ-продуцирующие виды включают Bacteroides ovatus и Bacteroides stercoris.
В Таблице 14 представлен перечень различных видов бактерий и отдельных метаболитов триптофана, продуцируемых этими видами. <LOD указывает на то, что метаболит был ниже предела обнаружения. Указана фоновая концентрация метаболитов в питательных средах для бактерий. Каталожный номер АТСС указывается для штаммов, полученных из внешних источников, когда это применимо.
Некоторые варианты осуществления изобретения подпадают под действие следующих нумерованных пунктов.
1. Способ изменения уровней триптамина или 5-гидрокситриптамина у субъекта, включающий введение субъекту жизнеспособной популяции по меньшей мере одного вида бактерии, выбранного из Таблицы 1, 2, 3, 4, 5, или 6.
2. Способ по абзацу 1, отличающийся тем, что уровни триптамина или 5-гидрокситриптамина у субъекта повышают.
3. Способ по абзацу 1 или 2, включающий введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Ruminococcus_gnavus (штамм 1), Lachnospiraceae_bacterium_9_1_43BFAA, Eggerthella_sp., Ruminococcus_gnavus (штамм 2), Clostridium_nexile, Lachnospiraceae_bacterium_6_1_63FAA, и Ruminococcus_torques.
4. Способ по абзацу 1 или 2, включающий введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Clostridium ghonii, Flavonifractor plautii, Ruminococcus gnavus Bacteroides ovatus, Bacteroides stercoris, и Clostridium sporogenes.
5. Способ по абзацу 1 или 2, включающий введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Lachnospiraceae_bacterium_2_1_58FAA, Clostridium_aldenense_SC114, Clostridium_citroniae, и Clostridium_clostridioforme.
6. Способ по абзацу 1 или 2, включающий введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Flavonifractor_plautii, Veillonella_parvula, Blautia_sp_CAG_257_SC146, и Clostridium_bolteae.
7. Способ по абзацу 1 или 2, включающий введение субъекту жизнеспособной популяции по меньшей мере двух видов из группы, состоящей из Blautia_hansenii, Lachnospiraceae_bacterium_2_1_46FAA, Coprococcus_sp_HPP0048, Collinsella_tanakaei, Clostridium_sporogenes, Clostridium_phytofermentans, Clostridium_bifermentans, Staphylococcus_aureus, Lachnospiraceae_bacterium_4_1_37FAA, Clostridium_asparagiforme, Clostridium_lavalense_SC43, и Holdemania_filliformis.
8. Способ по любому из абзацев 3-7, включающий введение субъекту жизнеспособной популяции по меньшей мере 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 перечисленных видов.
9. Способ по любому из абзацев 1-8, включающий введение субъекту композиции, содержащей одну или более композиций 1-47 из Таблицы 6.
10. Способ по любому из абзацев 1-9, отличающийся тем, что у субъекта имеется заболевание или патологическое состояние, характеризующееся измененными перистальтикой кишечника, агрегацией тромбоцитов, иммунным ответом, функцией сердца или развитием костей.
11. Способ по любому из абзацев 1-10, отличающийся тем, что у субъекта имеется заболевание или патологическое состояние, выбранное из группы, состоящей из синдрома раздраженного кишечника, воспалительного заболевания кишечника, запора, депрессии, тревожного расстройства, сердечно-сосудистого заболевания и остеопороза.
12. Способ по абзацу 11, отличающийся тем, что воспалительное заболевание кишечника выбирают из группы, состоящей из инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита.
13. Фармацевтический состав, содержащий жизнеспособную популяцию по меньшей мере одного вида бактерии, выбранного из Таблицы 1, 2, 3, 4, 5, или 6.
14. Фармацевтический состав по абзацу 13, содержащий жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Ruminococcus_gnavus (штамм 1), Lachnospiraceae_bacterium_9_1_43BFAA, Eggerthella_sp., Ruminococcus_gnavus (штамм 2), Clostridium_nexile, Lachnospiraceae_bacterium_6_1_63FAA, и Ruminococcus_torques.
15. Фармацевтический состав по абзацу 13, содержащий жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Clostridium ghonii, Flavonifractor plautii, Ruminococcus gnavus Bacteroides ovatus, Bacteroides stercoris, и Clostridium sporogenes.
16. Фармацевтический состав по абзацу 13, содержащий жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Lachnospiraceae_bacterium_2_1_58FAA, Clostridium_aldenense_SC114, Clostridium_citroniae, и Clostridium_clostridioforme.
17. Фармацевтический состав по абзацу 13, содержащий жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Flavonifractor_plautii, Veillonella_parvula, Blautia_sp_CAG_257_SC146, и Clostridium_bolteae.
18. Фармацевтический состав по абзацу 13, содержащий жизнеспособную популяцию по меньшей мере двух видов из группы, состоящей из Blautia_hansenii, Lachnospiraceae_bacterium_2_1_46FAA, Coprococcus_sp_HPP0048, Collinsella_tanakaei, Clostridium_sporogenes, Clostridium_phytofermentans, Clostridium_bifermentans, Staphylococcus_aureus, Lachnospiraceae_bacterium_4_1_37FAA, Clostridium_asparagiforme, Clostridium_lavalense_SC43, и Holdemania_filliformis.
19. Фармацевтический состав по любому из абзацев 14-18, содержащий жизнеспособную популяцию по меньшей мере 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 перечисленных видов.
20. Фармацевтический состав по любому из абзацев 13-19, содержащий введение субъекту композиции, содержащей одну или более композиций 1-47 из Таблицы 6.
21. Фармацевтический состав по любому из абзацев 13-20, содержащий фармацевтически приемлемый наполнитель и/или содержащийся в капсуле (например, капсуле с энтеросолюбильной оболочкой).
22. Способ изменения уровней триптамина или 5-гидрокситриптамина у субъекта, причем способ включает введение субъекту эффективного количества фармацевтического состава по любому из абзацев 13-21.
23. Способ по абзацу 22, отличающийся тем, что уровни триптамина или 5-гидрокситриптамина у субъекта повышают.
24. Способ по абзацу 22 или 23, отличающийся тем, что уровни триптамина или 5-гидрокситриптамина представляют собой уровни триптамина или 5-гидрокситриптамина в кале субъекта.
25. Способ по абзацу 22 или 23, отличающийся тем, что уровни триптамина или 5-гидрокситриптамина представляют собой уровни триптамина или 5-гидрокситриптамина в крови, сыворотке, плазме, моче или спинномозговой жидкости (СМЖ) субъекта.
26. Способ лечения субъекта, имеющего заболевание или патологическое состояние, характеризующееся наличием низких уровней триптамина или 5-гидрокситриптамина, причем способ включает введение субъекту, у которого диагностировано или имеется риск развития заболевания, терапевтически эффективного количества фармацевтического состава по любому из абзацев 13-21.
27. Способ по абзацу 26, отличающийся тем, что у субъекта имеется заболевание или патологическое состояние, характеризующееся измененными перистальтикой кишечника, агрегацией тромбоцитов, иммунным ответом, функцией сердца или развитием костей.
28. Способ по абзацу 26 или 27, отличающийся тем, что у субъекта имеется заболевание или патологическое состояние, выбранное из группы, состоящей из синдрома раздраженного кишечника, воспалительного заболевания кишечника, запора, депрессии, тревожного расстройства, сердечно-сосудистого заболевания и остеопороза.
29. Способ по абзацу 28, отличающийся тем, что воспалительное заболевание кишечника выбирают из группы, состоящей из инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита.
30. Способ повышения уровня или активности регуляторных Т-клеток у субъекта, включающий введение субъекту фармацевтического состава по любому из абзацев 13-21.
31. Способ для восстановления или улучшения гомеостаза кишечника или для профилактики или лечения злокачественного новообразования кишечника или толстой кишки у субъекта, причем способ включает введение субъекту фармацевтического состава по любому из абзацев 13-21.
32. Композиция, содержащая по меньшей мере два разных вида бактерий, которые в комбинации могут повышать уровни триптамина или 5-гидрокситриптамина по сравнению с уровнем триптамина или 5-гидрокситриптамина, продуцируемых каждым видом отдельно, (i) в присутствии того же уровня триптофана, что и в комбинации, (ii) в течение определенного периода времени в присутствии того же уровня триптофана, что и в комбинации, (iii) при введении в систему in vivo или (iv) при введении в модельную систему in vitro.
33. Композиция по абзацу 32, отличающаяся тем, что композицию выбирают из группы, состоящей из композиции, содержащей виды бактерий фармацевтического состава по любому из абзацев 13-21.
34. Состав или композиция по любому из пунктов 13-21, 32 или 33 для применения в изменении уровней триптамина или 5-гидрокситриптамина у субъекта.
35. Состав или композиция по абзацу 34, отличающиеся тем, что у субъекта имеется заболевание или патологическое состояние, характеризующееся измененными перистальтикой кишечника, агрегацией тромбоцитов, иммунным ответом, функцией сердца или развитием костей.
36. Состав или композиция по абзацу 34 или 35, отличающиеся тем, что у субъекта имеется заболевание или патологическое состояние, выбранное из группы, состоящей из синдрома раздраженного кишечника, воспалительного заболевания кишечника, запора, депрессии, тревожного расстройства, сердечно-сосудистого заболевания и остеопороза.
37. Состав или композиция по абзацу 36, отличающиеся тем, что воспалительное заболевание кишечника выбирают из группы, состоящей из инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита.
38. Состав или композиция по абзацу 36, для применения в повышении уровня активности регуляторных Т-клеток у субъекта.
39. Состав или композиция по абзацу 36, для применения в восстановлении гомеостаза кишечника, или для предотвращения или лечения злокачественного новообразования кишечника или толстой кишки у субъекта.
Другие варианты осуществления находятся в пределах объема следующей формулы изобретения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> СЕРЕС ТЕРАПЕУТИКС, ИНК.
< 120> МАНИПУЛЯЦИЯ МЕТАБОЛИЗМОМ ТРИПТАМИНОВ
<130> 51069-021WO2
<150> US 62/567 415
<151> 03.10.2017
<160> 75
<170> PatentIn версия 3.5
<210> 1
<211> 1160
<212> ДНК
<213> Blautia hansenii
<400> 1
tcagagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagca cttatcattg actcttcgga agatttgata tttgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata caggggaata acagttagaa atggctgcta 180
atgccgcata agcgcacagg atcgcatggt ctggtgtgaa aaactgaggt ggtatgagat 240
ggacccgcgt ctgattaggt agttggtggg gtaacggcct accaagccga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccagac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gaagaagtat ctcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggaagagc aagtctgatg tgaaaggctg 600
gggcttaacc ccaggactgc attggaaact gtttttctag agtgccggag aggtaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac ggtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc ggggtgcaaa gcagttcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catctgcctg accgttcctt gaccggagct ttccttcggg 1020
acaggcaaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatccttag tagccagcag ttcggctggg cactctaggg 1140
agactgccgg ggataacccg 1160
<210> 2
<211> 802
<212> ДНК
<213> Blautia hansenii
<400> 2
ggacggtaac tgacgttgag gctcgaaagc gtggggagca aacaggatta gataccctgg 60
tagtccacgc cgtaaacgat gaatactagg tgtcggggtg caaagcagtt cggtgccgca 120
gcaaacgcaa taagtattcc acctggggag tacgttcgca agaatgaaac tcaaaggaat 180
tgacggggac ccgcacaagc ggtggagcat gtggtttaat tcgaagcaac gcgaagaacc 240
ttaccaagtc ttgacatctg cctgaccgtt ccttaaccgg agctttcctt cgggacaggc 300
aagacaggtg gtgcatggtt gtcgtcagct cgtgtcgtga gatgttgggt taagtcccgc 360
aacgagcgca acccctatcc ttagtagcca gcagtccggc tgggcactct agggagactg 420
ccggggataa cccggaggaa ggcggggacg acgtcaaatc atcatgcccc ttatgatttg 480
ggctacacac gtgctacaat ggcgtaaaca aagggaagcg aagcggtgac gcttagcaaa 540
tctcaaaaat aacgtcccag ttcggactgc agtctgcaac tcgactgcac gaagctggaa 600
tcgctagtaa tcgcgaatca gaatgtcgcg gtgaatacgt tcccgggtct tgtacacacc 660
gcccgtcaca ccatgggagt cagtaacgcc cgaagtcagt gacccaacct tatggaggga 720
gctgccgaag gcgggaccga taactggggt gaagtcgtaa caaggtagcc gtatcggaag 780
gtgcggctgg atcacctcct tt 802
<210> 3
<211> 1529
<212> ДНК
<213> Blautia caecimuris
<400> 3
acagagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggggat tattttgaca gagacttcgg ttgaagtcgt tataatccta gtggcggacg 120
ggtgagtaac gcgtgggtaa cctgcctcac actgggggat aacagtcaga aatgactgct 180
aataccgcat aagcgcacag agctgcatgg ctcagtgtga aaaactccgg tggtgtgaga 240
tggacccgcg ttggattagc cagttggcag ggtaacggcc taccaaagcg acgatccata 300
gccggcctga gagggtggac ggccacattg ggactgagac acggcccaga ctcctacggg 360
aggcagcagt ggggaatatt gcacaatggg ggaaaccctg atgcagcgac gccgcgtgaa 420
ggaagaagta tctcggtatg taaacttcta tcagcaggga agaaaatgac ggtacctgac 480
taagaagccc cggctaacta cgtgccagca gccgcggtaa tacgtagggg gcaagcgtta 540
tccggattta ctgggtgtaa agggagcgta gacggagcag caagtctgat gtgaaaggcg 600
ggggctcaac ccccggactg cattggaaac tgttgatctt gagtaccgga gaggtaagcg 660
gaattcctag tgtagcggtg aaatgcgtag atattaggag gaacaccagt ggcgaaggcg 720
gcttactgga cggtaactga cgttgaggct cgaaagcgtg gggagcaaac aggattagat 780
accctggtag tccacgccgt aaacgatgaa tactaggtgt cgggtggcag agccattcgg 840
tgccgcagca aacgcagtaa gtattccacc tggggagtac gttcgcaaga atgaaactca 900
aaggaattga cggggacccg cacaagcggt ggagcatgtg gtttaattcg aagcaacgcg 960
aagaacctta ccaagtcttg acatccctct gaccggtgag taacgtcacc tttccttcgg 1020
gacagaggag acaggtggtg catggttgtc gtcagctcgt gtcgtgagat gttgggttaa 1080
gtcccgcaac gagcgcaacc cctatcctta gtagccagcg gtttggccgg gcactctgag 1140
gagactgcca gggataacct ggaggaaggc ggggatgacg tcaaatcatc atgcccctta 1200
tgatttgggc tacacacgtg ctacaatggc gtaaacaaag ggaagcgaga gtgtgagctt 1260
aagcaaatcc caaaaataac gtcccagttc ggactgcagt ctgcaactcg actgcacgaa 1320
gctggaatcg ctagtaatcg cggatcagaa tgccgcggtg aatacgttcc cgggtcttgt 1380
acacaccgcc cgtcacacca tgggagtcag taacgcccga agtcagtgac cgaaccgaaa 1440
ggacggagct gccgaaggcg ggacggatga ctggggtgaa gtcgtaacaa ggtagccgta 1500
tcggaaggtg cggctggatc acctccttt 1529
<210> 4
<211> 1531
<212> ДНК
<213> Clostridium aldenense
<400> 4
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gaacgaagcg attaggatga agttttcgga tggattcctg gttgactgag tggcggacgg 120
gtgagtaacg cgtggataac ctgcctcaca ctgggggata acagttagaa atggctgcta 180
ataccgcata agcgcacagt accgcatggt acggtgtgaa aaacccaggt ggtgtgagat 240
ggatccgcgt ctgattagcc agttggcggg gtaacggccc accaaagcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggc ggaagcctga tgcagcgacg ccgcgtgagt 420
gaagaagtat ctcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcgacgc aagtctggag tgaaagcccg 600
gggcccaacc ccgggactgc tttggaaact gtgctgctgg agtgcaggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac tgtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat gctaggtgtc ggggggcaaa gcccttcggt 840
gccgccgcta acgcaataag cattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catccccctg accggcgcgt aacggtgccc ttccttcggg 1020
acaggggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ttatccttag tagccagcac gtcgtggtgg gcactctagg 1140
gagactgcca gggataacct ggaggaaggt ggggatgacg tcaaatcatc atgcccctta 1200
tgatttgggc tacacacgtg ctacaatggc gtaaacaaag ggaggcgacc ctgcgaaggt 1260
aagcaaatcc caaaaataac gtcccagttc ggactgtagt ctgcaacccg actacacgaa 1320
gctggaatcg ctagtaatcg cggatcagaa tgccgcggtg aatacgttcc cgggtcttgt 1380
acacaccgcc cgtcacacca tgggagtcag caacgcccga agtcagtgac ccaaccttaa 1440
caggagggag ctgccgaagg cggggcaggt aactggggtg aagtcgtaac aaggtagccg 1500
tatcggaagg tgcggctgga tcacctcctt t 1531
<210> 5
<211> 1527
<212> ДНК
<213> Clostridium asparagiforme
<400> 5
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gaacgaagca ttttagatga agttttcgga tggattctga gatgactgag tggcggacgg 120
gtgagtaaca cgtggataac ctgcctcaca ctgggggaca acagttagaa atgactgcta 180
ataccgcata agcgcacagt accgcatggt acggtgtgaa aaactccggt ggtgtgagat 240
ggatccgcgt ctgattagcc agttggcggg gtaacggccc accaaagcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggc gaaagcctga tgcagcgacg ccgcgtgagt 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gataatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagtctgaag tgaaaaccca 600
gggctcaacc ctgggactgc tttggaaact gtcaagctag agtgcaggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac tgtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgagt gctaggtgtt ggggggcaaa gcccttcggt 840
gccgtcgcaa acgcaataag cactccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catccccctg accggcgtgt aacggcgcct ttccttcggg 1020
acaggggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ttatccttag tagccagcat taagatgggc actctaggga 1140
gactgccagg gacaacctgg aggaaggtgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggcgt aaacaaaggg aagcgaccct gcgaaggtga 1260
gcaaatctca aaaataacgt cccagttcgg actgtagtct gcaacccgac tacacgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtcagca acgcccgaag tcagtgaccc aaccgaaagg 1440
agggagctgc cgaaggcggg gcaggtaact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctccttt 1527
<210> 6
<211> 1499
<212> ДНК
<213> Clostridium bolteae
<400> 6
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aacatatgga agtcgcatga 180
cttttgtatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg aggcagcagt ggggaatatt gcacaatggg 360
cgaaagcctg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taaagctctg 420
tcctcaagga agataatgac ggtacttgag gaggaagccc cggctaacta cgtgccagca 480
gccgcggtaa tacgtagggg gctagcgtta tccggaatta ctgggcgtaa agggtgcgta 540
ggtggttttt taagtcagaa gtgaaaggct acggctcaac cgtagtaagc ttttgaaact 600
agagaacttg agtgcaggag aggagagtag aattcctagt gtagcggtga aatgcgtaga 660
tattaggagg aataccagta gcgaaggcgg ctctctggac tgtaactgac actgaggcac 720
gaaagcgtgg ggagcaaaca ggattagata ccctggtagt ccacgccgta aacgatgagt 780
actaggtgtc gggggttacc cccctcggtg ccgcagctaa cgcattaagt actccgcctg 840
ggaagtacgc tcgcaagagt gaaactcaaa ggaattgacg ggggacccgc acaagtagcg 900
gagcatgtgg tttaattcga agcaacgcga agaaccttac ctaagcttga catcccactg 960
acctctccct aatcggagat tcccttcggg gacagtggtg acaggtggtg catggttgtc 1020
gtcagctcgt gtcgtgagat gttgggttaa gtcccgcaac gagcgcaacc cttgccttta 1080
gttgccagca ttaagttggg cactctagag ggactgccga ggataactcg gaggaaggtg 1140
gggatgacgt caaatcatca tgccccttat gcttagggct acacacgtgc tacaatgggt 1200
ggtacagagg gttgccaagc cgcgaggtgg agctaatccc ttaaagccat tctcagttcg 1260
gattgtaggc tgaaactcgc ctacatgaag ctggagttac tagtaatcgc agatcagaat 1320
gctgcggtga atgcgttccc gggtcttgta cacaccgccc gtcacaccat ggaagttggg 1380
ggcgcccgaa gccggttagc taacctttta ggaagcggcc gtcgaaggtg aaaccaatga 1440
ctggggtgaa gtcgtaacaa ggtagccgta tcggaaggtg cggctggatc acctccttt 1499
<210> 7
<211> 1499
<212> ДНК
<213> Clostridium bifermentans
<400> 7
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aacatatgaa agtcgcatgg 180
cttttgtatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg aggcagcagt ggggaatatt gcacaatggg 360
cgaaagcctg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taaagctctg 420
tcctcaagga agataatgac ggtacttgag gaggaagccc cggctaacta cgtgccagca 480
gccgcggtaa tacgtagggg ggctagcgtt atccggaatt actgggcgta aagggtgcgt 540
aggtggtttt ttaagtcaga agtgaaaggc tacggctcaa ccgtagtaag cttttgaaac 600
tagagaactt gagtgcagga gaggagagta gaattcctag tgtagcggtg aaatgcgtag 660
atattaggag gaataccagt agcgaaggcg gctctctgga ctgtaactga cactgaggca 720
cgaaagcgtg gggagcaaac aggattagat accctggtag tccacgccgt aaacgatgag 780
tactaggtgt cgggggttac ccccctcggt gccgcagcta acgcattaag tactccgcct 840
gggaagtacg ctcgcaagag tgaaactcaa aggaattgac ggggacccgc acaagtagcg 900
gagcatgtgg tttaattcga agcaacgcga agaaccttac ctagcttgac atcccactga 960
cctctcccta atcggagatt tcccttcggg gacagtggtg acaggtggtg catggttgtc 1020
gtcagctcgt gtcgtgagat gttgggttaa gtcccgcaac gagcgcaacc cttgccttta 1080
gttgccagca ttaagttggg cactctagag ggactgccga ggataactcg gaggaaggtg 1140
gggatgacgt caaatcatca tgccccttat gcttagggct acacacgtgc tacaatgggt 1200
ggtacagagg gttgccaagc cgcgaggtgg agctaatccc ttaaagccat tctcagttcg 1260
gattgtaggc tgaaactcgc ctacatgaag ctggagttac tagtaatcgc agatcagaat 1320
gctgcggtga atgcgttccc gggtcttgta cacaccgccc gtcacaccat ggaagttggg 1380
ggcgcccgaa gccggttagc taacctttta ggaagcggcc gtcgaaggtg aaaccaatga 1440
ctggggtgaa gtcgtaacaa ggtagccgta tcggaaggtg cggctggatc acctccttt 1499
<210> 8
<211> 926
<212> ДНК
<213> Clostridium bifermentans
<400> 8
atttgagagt ttgatcctgg ctcaggatga acgctggcgg cgtgcctaac acatgcaagt 60
cgagcgatct cttcggagag agcggcggac gggtgagtaa cgcgtgggta acctgccctg 120
tacacacgga taacataccg aaaggtatac taatacggga taacatacgg aagtcgcatg 180
acttttgtat caaagctccg gcggtacagg atggacccgc gtctgattag ctagttggta 240
aggtaatggc ttaccaaggc aacgatcagt agccgacctg agagggtgat cggccacact 300
ggaactgaga cacggtccag actcctacgg gaggcagcag tggggaatat tgcacaatgg 360
gcgaaagcct gatgcagcaa cgccgcgtga gcgatgaagg ccttcgggtc gtaaagctct 420
gtcctcaagg aagataatga cggtacttga ggaggaagcc ccggctaact acgtgccagc 480
agccgcggta atacgtaggg ggctagcgtt atccggaatt actgggcgta aagggtgcgt 540
aggtggtttt taagtcagaa gtgaaaggct acggctcaac cgtagtaagc ttttgaaact 600
agagaacttg agtgcaggag aggagagtag aattcctagt gtagcggtga aatgcgtaga 660
tattaggagg aataccagta gcgaaggcgg ctctctggac tgtaactgac actgaggcac 720
gaaagcgtgg ggagcaaaca ggattagata ccctggtagt ccacgccgta aacgatgagt 780
actaggtgtc gggggttacc ccctcggtgc cgcagctaac gcattaagta ctccgcctgg 840
gaagtacgct cgcaagagtg aaactcaaag gaattgacgg ggacccgcac aagtagcgga 900
gcatgtggtt taattcgaag caacgc 926
<210> 9
<211> 1497
<212> ДНК
<213> Clostridium bifermentans
<400> 9
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aatatatgaa agtcgcatgg 180
cttttatatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg aggcagcagt ggggaatatt gcacaatggg 360
cgaaagcctg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taagctctgt 420
cctcaaggaa gataatgacg gtacttgagg aggaagcccc ggctaactac gtgccagcag 480
ccgcggtaat acgtaggggg ctagcgttat ccggaattac tgggcgtaaa gggtgcgtag 540
gtggttttta agtcagaagt gaaaggctac ggctcaaccg tagtaagctt ttgaaactag 600
agaacttgag tgcaggagag gagagtagaa ttcctagtgt agcggtgaaa tgcgtagata 660
ttaggaggaa taccagtagc gaaggcggct ctctggactg taactgacac tgaggcacga 720
aagcgtgggg agcaaacagg attagatacc ctggtagtcc acgccgtaaa cgatgagtac 780
taggtgtcgg gggttacccc cctcggtgcc gcagctaacg cattaagtac tccgcctggg 840
aagtacgctc gcaagagtga aactcaaagg aattgacggg gacccgcaca agtagcggag 900
catgtggttt aattcgaagc aacgcgaaga accttaccta agcttgacat cccactgacc 960
tctccctaat cggagatttc ccttcgggga cagtggtgac aggtggtgca tggttgtcgt 1020
cagctcgtgt cgtgagatgt tgggttaagt cccgcaacga gcgcaaccct tgcctttagt 1080
tgccagcatt aagttgggca ctctagaggg actgccgagg ataactcgga ggaaggtggg 1140
gatgacgtca aatcatcatg ccccttatgc ttagggctac acacgtgcta caatgggtgg 1200
tacagagggt tgccaagccg cgaggtggag ctaatccctt aaagccattc tcagttcgga 1260
ttgtaggctg aaactcgcct acatgaagct ggagttacta gtaatcgcag atcagaatgc 1320
tgcggtgaat gcgttcccgg gtcttgtaca caccgcccgt cacaccatgg aagttggggg 1380
cgcccgaagc cggttagcta accttttagg aagcggccgt cgaaggtgaa accaatgact 1440
ggggtgaagt cgtaacaagg tagccgtatc ggaaggtgcg gctggatcac ctccttt 1497
<210> 10
<211> 1503
<212> ДНК
<213> Clostridium bifermentans
<400> 10
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aacatatgaa agtcgcatgg 180
cttttgtatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg aggcagcagt ggggaatatt gcacaatggg 360
cgaaagcctg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taaagctctg 420
tcctcaagga agataatgac ggtacttgag gaggaagccc cggctaacta cgtgccagca 480
gccgcggtaa tacgtagggg gctagcgtta tccggaatta ctgggcgtaa agggtgcgta 540
ggtggttttt caagtcagaa gtgaaaggct acggctcaac cgtagtaagc ttttgaaact 600
agagaacttg agtgcaggag aggagagtag aattcctagt gtagcggtga aatgcgtaga 660
tattaggagg aataccagta gcgaaggcgg ctctctggac tgtaactgac actgaggcac 720
gaaagcgtgg ggagcaaaca ggattagata ccctggtagt ccacgccgta aacgatgagt 780
actaggtgtc gggggttacc cccctcggtc gccgcagcta acgcattaag tactccgcct 840
gggaagtacg ctcgcaagag tagaaactca aaggaattga cggggacccg gccacaagta 900
gcggagcatg tggtttaatt cgaagcaacg cgaagaacct tacctaagct tgacatccca 960
ctgacctctc cctaatcgga gatttccctt cggggacagt ggtgacaggt ggtgcatggt 1020
tgtcgtcagc tcgtgtcgtg agatgttggg ttaagtcccg caacgagcgc aacccttgcc 1080
tttagttgcc agcattaagt tgggcactct agagggactg ccgaggataa ctcggaggaa 1140
ggtggggatg acgtcaaatc atcatgcccc ttatgcttag ggctacacac gtgctacaat 1200
gggtggtaca gagggttgcc aagccgcgag gtggagctaa tcccttaaag ccattctcag 1260
ttcggattgt aggctgaaac tcgcctacat gaagctggag ttactagtaa tcgcagatca 1320
gaatgctgcg gtgaatgcgt tcccgggtct tgtacacacc gcccgtcaca ccatggaagt 1380
tgggggcgcc cgaagccggt tagctaacct tttaggaagc ggccgtcgaa ggtgaaacca 1440
atgactgggg tgaagtcgta acaaggtagc cgtatcggaa ggtgcggctg gatcacctcc 1500
ttt 1503
<210> 11
<211> 1317
<212> ДНК
<213> Clostridium bifermentans
<400> 11
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aacatatgaa agtcgcatgg 180
cttttgtatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg aggcagcagt ggggaatatt gcacaatggg 360
cgaaagcctg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taaagctctg 420
tcctcaagga agataatgac ggtactttga ggaggaagcc ccggctaact acgtgccagc 480
agccgcggta atacgtaggg ggctagcgtt atccggaatt actgggcgta aagggtgcgt 540
aggtggtttt ttaagtcaga agtgaaaggc tacggctcaa ccgtagtaag cttttgaaac 600
tagagaactt gagtgcagga gaggagagta gaattcctag tgtagcggtg aaatgcgtag 660
atattaggag gaataccagt agcgaaggcg gctctctgga ctgtaactga cactgaggca 720
cgaaagcgtg gggagcaaac aggattagat accctggtag tccacgccgt aaacgatgag 780
tactaggtgt cgggggttac ccccctcggt gccgcagcta acgcattaag tactccgcct 840
gggaagtacg ctcgcaagag tgaaactcaa aggaattgac ggggacccgc acaagtagcg 900
gagcatgtgg tttaattcga agcaacgcga agaaccttac ctaagcttga catcccactg 960
acctctccct aatcggagat ttcccttcgg ggacagtggt gacaggtggt gcatggttgt 1020
cgtcagctcg tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgccttt 1080
agttgccagc attaagttgg gcactctaga gggactgccg aggataactc ggaggaaggt 1140
ggggatgacg tcaaatcatc atgcccctta tgcttagggc tacacacgtg ctacaatggg 1200
tggtacagag ggttgccaag ccgcgaggtg gagctaatcc cttaaagcca ttctcagttc 1260
ggattgtagg ctgaaactcg cctacatgaa gctggagtta ctagtaatcg cagatca 1317
<210> 12
<211> 957
<212> ДНК
<213> Clostridium bifermentans
<400> 12
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aatatatgaa agtcgcatgg 180
cttttatatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg aggcagcagt ggggaatatt gcacaatggg 360
cgaaagcctg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taaagctctg 420
tcctcaagga agataatgac ggtacttgag gaggaagccc cggctaacta cgtgccagca 480
gccgcggtaa tacgtagggg gctagcgtta tccggaatta ctgggcgtaa agggtgcgta 540
ggtggttttt taagtcagaa gtgaaaggct acggctcaac cgtagtaagc ttttgaaact 600
agagaacttg agtgcaggag aggagagtag aattcctagt gtagcggtga aatgcgtaga 660
tattaggagg aataccagta gcgaaggcgg ctctctggac tgtaactgac actgaggcac 720
gaaagcgtgg ggagcaaaca ggattagata ccctggtagt ccacgccgta aacgatgagt 780
actaggtgtt gggggttacc cccctcggtg ccgcagctaa cgcattaagt actccgcctg 840
ggaagtacgc tcgcaagagt gaaactcaaa ggaattgacg gggacccgca caagtagcgg 900
agcatgtggt ttaattcgaa gcaacgcgaa gaaccttacc taagcttgac atcccac 957
<210> 13
<211> 1499
<212> ДНК
<213> Clostridium bifermentans
<400> 13
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aacatacgaa agtcgcatgg 180
cttttgtatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg gggcagcagt ggggaatatt gcacaatggg 360
cgaaagactg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taaagctctg 420
tcctcaagga agataatgac ggtacttgag gaggaagccc cggctaacta cgtgccagca 480
gccgcggtaa tacgtagggg gctagcgtta tccggaatta ctgggcgtaa agggtgcgta 540
ggtggttttt taagtcagaa gtgaaaggct acggctcaac cgtagtaagc ttttgaaact 600
agagaacttg agtgcaggag aggagagtag aattcctagt gtagcggtga aatgcgtaga 660
tattaggagg aataccagta gcgaaggcgg ctctctggac tgtaactgac actgaggcac 720
gaaagcgtgg ggagcaaaca ggattagata ccctggtagt ccacgccgta aacgatgagt 780
actaggtgtc gggggttacc cccctcggtg ccgcagctaa cgcattaagt actccgcctg 840
ggaagtacgc tcgcaagagt gaaactcaaa ggaattgacg gggacccgca caagtagcgg 900
agcatgtggt ttaattcgaa gcaacgcgaa gaaccttacc taagcttgac atcccactga 960
cctctcccta atcggagatt tcccttcggg gacagtggtg acaggtggtg catggttgtc 1020
gtcagctcgt gtcgtgagat gttgggttaa gtcccgcaac gagcgcaacc cttgccttta 1080
gttgccagca ttaagttggg cactctagag ggactgccga ggataactcg gaggaaggtg 1140
gggatgacgt caaatcatca tgccccttat gcttagggct acacacgtgc tacaatgggt 1200
ggtacagagg gttgccaagc cgcgaggtgg agctaatccc ttaaagccat tctcagttcg 1260
gattgtaggc tgaaactcgc ctacatgaag ctggagttac tagtaatcgc agatcagaat 1320
gctgcggtga atgcgttccc gggtcttgta cacaccgccc gtcacaccat ggaagttggg 1380
ggcgcccgaa gccggttagc taacctttta ggaagcggcc gtcgaaggtg aaaccaatga 1440
ctggggtgaa gtcgtaacaa ggtagccgta tcggaaggtg cggctggatc acctccttt 1499
<210> 14
<211> 895
<212> ДНК
<213> Clostridium bifermentans
<400> 14
atttgagagt ttgatcctgg ctcaggatga acgctggcgg cgtgcctaac acatgcaagt 60
cgagcgatct cttcggagag agcggcggac gggtgagtaa cgcgtgggta acctgccctg 120
tacacacgga taatataccg aaaggtatac taatacggga taacatacga aagtcgcatg 180
gcttttatat caaagctccg gcggtacagg atggacccgc gtctgattag ctagttggta 240
aggtaatggc ttaccaaggc aacgatcagt agccgacctg agagggtgat cggccacact 300
ggaactgaga cacggtccag actcctacgg gaggcagcag tggggaatat tgcacaatgg 360
gcgaaagcct gatgcagcaa cgccgcgtga gcgatgaagg ccttcgggtc gtaaagctct 420
gtcctcaagg aagataatga cggtacttga ggaggaagcc ccggctaact acgtgccagc 480
agccgcggta atacgtaggg ggctagcgtt atccggaatt actgggcgta aagggtgcgt 540
aggtggtttt ttaagtcaga agtgaaaggc tacggctcaa ccgtagtaag cttttgaaac 600
tagagaactt gagtgcagga gaggagagta gaattcctag tgtagcggtg aaatgcgtag 660
atattaggag gaataccagt agcgaaggcg gctctctgga ctgtaactga cactgaggca 720
cgaaagcgtg gggagcaaac aggattagat accctggtag tccacgccgt aaacgatgag 780
tactaggtgt cgggggttac ccccctcggt gccgcagcta acgcattaag tactccgcct 840
gggaagtacg ctcgcaagag tgaaactcaa aggaattgac ggggacccgc acaag 895
<210> 15
<211> 1498
<212> ДНК
<213> Clostridium bifermentans
<400> 15
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgatctc ttcggagaga gcggcggacg ggtgagtaac gcgtgggtaa cctgccctgt 120
acacacggat aacataccga aaggtatact aatacgggat aatatatgaa agtcgcatgg 180
cttttatatc aaagctccgg cggtacagga tggacccgcg tctgattagc tagttggtaa 240
ggtaatggct taccaaggca acgatcagta gccgacctga gagggtgatc ggccacactg 300
gaactgagac acggtccaga ctcctacggg aggcagcagt ggggaatatt gcacaatggg 360
cgaaagcctg atgcagcaac gccgcgtgag cgatgaaggc cttcgggtcg taaagctctg 420
tcctcaagga agataatgac ggtacttgag gaggaagccc cggctaacta cgtgccagca 480
gccgcggtaa tacgtagggg gctagcgtta tccggaatta ctgggcgtaa agggtgcgta 540
ggtggttttt taagtcagaa gtgaaaggct acggctcaac cgtagtaagc ttttgaaact 600
agagaacttg agtgcaggag aggagagtag aattcctagt gtagcggtga aatgcgtaga 660
tattaggagg aataccagta gcgaaggcgg ctctctggac tgtaactgac actgaggcac 720
gaaagcgtgg ggagcaaaca ggattagata ccctggtagt ccacgccgta aacgatgagt 780
actaggtgtc gggggttacc cccctcggtg ccgcagctaa cgcattaagt actccgcctg 840
ggaagtacgc tcgcaagagt gaaactcaaa ggaattgacg gggaccgcac aagtagcgga 900
gcatgtggtt taattcgaag caacgcgaag aaccttacct aagcttgaca tcccactgac 960
ctctccctaa tcggagattt cccttcgggg acagtggtga caggtggtgc atggttgtcg 1020
tcagctcgtg tcgtgagatg ttgggttaag tcccgcaacg agcgcaaccc ttgcctttag 1080
ttgccagcat taagttgggc actctagagg gactgccgag gataactcgg aggaaggtgg 1140
ggatgacgtc aaatcatcat gccccttatg cttagggcta cacacgtgct acaatgggtg 1200
gtacagaggg ttgccaagcc gcgaggtgga gctaatccct taaagccatt ctcagttcgg 1260
attgtaggct ggaactcgcc tacatgaagc tggagttact agtaatcgca gatcagaatg 1320
ctgcggtgaa tgcgttcccg ggtcttgtac acaccgcccg tcacaccatg gaagttgggg 1380
cgcccgaagc cggtttagct aaccttttag gaagcggccg tcgaaggtga aaccaatgac 1440
tggggtgaag tcgtaacaag gtagccgtat cggaaggtgc ggctggatca cctccttt 1498
<210> 16
<211> 1528
<212> ДНК
<213> Clostridium bolteae
<400> 16
atgagagttt gatcctggct caggatgaac gctggcggcg tgcctaacac atgcaagtcg 60
aacgaagcaa ttgaaggaag ttttcggatg gaattcgatt gactgagtgg cggacgggtg 120
agtaacgcgt ggataacctg cctcacactg ggggataaca gttagaaatg actgctaata 180
ccgcataagc gcacagtacc gcatggtaca gtgtgaaaaa ctccggtggt gtgagatgga 240
tccgcgtctg attagccagt tggcggggta acggcccacc aaagcgacga tcagtagccg 300
acctgagagg gtgaccggcc acattgggac tgagacacgg cccaaactcc tacgggaggc 360
agcagtgggg aatattgcac aatgggcgaa agcctgatgc agcgacgccg cgtgagtgaa 420
gaagtatttc ggtatgtaaa gctctatcag cagggaagaa aatgacggta cctgactaag 480
aagccccggc taactacgtg ccagcagccg cggtaatacg tagggggcaa gcgttatccg 540
gatttactgg gtgtaaaggg agcgtagacg gcgaagcaag tctgaagtga aaacccaggg 600
ctcaaccctg ggactgcttt ggaaactgtt ttgctagagt gtcggagagg taagtggaat 660
tcctagtgta gcggtgaaat gcgtagatat taggaggaac accagtggcg aaggcggctt 720
actggacgat aactgacgtt gaggctcgaa agcgtgggga gcaaacagga ttagataccc 780
tggtagtcca cgccgtaaac gatgaatgct aggtgttggg gggcaaagcc cttcggtgcc 840
gtcgcaaacg cagtaagcat tccacctggg gagtacgttc gcaagaatga aactcaaagg 900
aattgacggg gacccgcaca agcggtggag catgtggttt aattcgaagc aacgcgaaga 960
accttaccaa gtcttgacat cctcttgacc ggcgtgtaac ggcgccttcc cttcggggca 1020
agagagacag gtggtgcatg gttgtcgtca gctcgtgtcg tgagatgttg ggttaagtcc 1080
cgcaacgagc gcaaccctta tccttagtag ccagcaggta aagctgggca ctctagggag 1140
actgccaggg ataacctgga ggaaggtggg gatgacgtca aatcatcatg ccccttatga 1200
tttgggctac acacgtgcta caatggcgta aacaaaggga agcaagacag tgatgtggag 1260
caaatcccaa aaataacgtc ccagttcgga ctgtagtctg caacccgact acacgaagct 1320
ggaatcgcta gtaatcgcga atcagaatgt cgcggtgaat acgttcccgg gtcttgtaca 1380
caccgcccgt cacaccatgg gagtcagcaa cgcccgaagt cagtgaccca actcgcaaga 1440
gagggagctg ccgaaggcgg ggcaggtaac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 17
<211> 1426
<212> ДНК
<213> Clostridium citroniae
<220>
<221> прочий_признак
<222> (1422)..(1426)
<223> N представляет собой A, T, C или G
<400> 17
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gaacgaagca atcagaatga aattttcgga tggatttctg gttgactgag tggcggacgg 120
gtgagtaacg cgtggataac ctgcctcaca ctgggggata acagttagaa atggctgcta 180
ataccgcata agcgcacagt accgcatggt acggtgtgaa aaactccggt ggtgtgagat 240
ggatccgcgt ctgattagtt agttggcggg gtaacggccc accaagacag cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggc gaaagcctga tgcagcgacg ccgcgtgagt 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcgaagc aagtctgaag tgaaaaccca 600
gggctcaacc ctgggactgc tttggaaact gttttgctag agtgtcggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac gataactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat gctaggtgtt ggggggcaaa gcccttcggt 840
gccgtcgcaa acgcaataag cattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catccccctg accggtcagt aaagtgacct ttccttcggg 1020
acaggggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ttatccttag tagccagcag gtaaagctgg gcactctagg 1140
gagactgcca gggataacct ggaggaaggt ggggatgacg tcaaatcatc atgcccctta 1200
tgatttgggc tacacacgtg ctacaatggc gtaaacaaag ggaagcgacc ctgcgaaggc 1260
aagcaaatcc caaaaataac gtcccagttc ggactgtagt ctgcaacccg actacacgaa 1320
gctggaatcg ctagtaatcg cgaatcagaa tgtcgcggtg aatacgttcc cgggtcttgt 1380
acacaccgcc cgtcacacca tgggagtcag caacgcccga annnnn 1426
<210> 18
<211> 1528
<212> ДНК
<213> Clostridium clostridioforme
<400> 18
atgagagttt gatcctggct caggatgaac gctggcggcg tgcctaacac atgcaagtcg 60
aacgaagcaa ttaaaggaag ttttcggacg gaatttgatt gactgagtgg cggacgggtg 120
agtaacgcgt ggataacctg cctcacactg ggggataaca gttagaaatg actgctaata 180
ccgcataagc gcacagtacc gcatggtacg gtgtgaaaaa ctccggtggt gtgagatgga 240
tccgcgtctg attagccagt tggcggggta acggcccacc aaagcgacga tcagtagccg 300
acctgagagg gtgaccggcc acattgggac tgagacacgg cccaaactcc tacgggaggc 360
agcagtgggg aatattgcac aatgggcgaa agcctgatgc agcgacgccg cgtgagtgaa 420
gaagtatttc ggtatgtaaa gctctatcag cagggaagaa aatgacggta cctgactaag 480
aagccccggc taactacgtg ccagcagccg cggtaatacg tagggggcaa gcgttatccg 540
gatttactgg gtgtaaaggg agcgtagacg gcgaagcaag tctgaagtga aaacccaggg 600
ctcaaccctg ggactgcttt ggaaactgtt ttgctagagt gtcggagagg taagtggaat 660
tcctagtgta gcggtgaaat gcgtagatat taggaggaac accagtggcg aaggcggctt 720
actggacgat aactgacgtt gaggctcgaa agcgtgggga gcaaacagga ttagataccc 780
tggtagtcca cgccgtaaac gatgaatgct aggtgttggg gggcaaagcc cttcggtgcc 840
gtcgcaaacg cagtaagcat tccacctggg gagtacgttc gcaagaatga aactcaaagg 900
aattgacggg gacccgcaca agcggtggag catgtggttt aattcgaagc aacgcgaaga 960
accttaccaa gtcttgacat ccttttgacc ggcgtgtaac ggcgccttcc cttcggggca 1020
agagagacag gtggtgcatg gttgtcgtca gctcgtgtcg tgagatgttg ggttaagtcc 1080
cgcaacgagc gcaaccctta tccttagtag ccagcaggta gagctgggca ctctagggag 1140
actgccaggg ataacctgga ggaaggcggg gatgacgtca aatcatcatg ccccttatga 1200
tttgggctac acacgtgcta caatggcgta aacaaaggga agcaagacag tgatgtggag 1260
caaatcccaa aaataacgtc ccagttcgga ctgtagtctg caacccgact acacgaagct 1320
ggaatcgcta gtaatcgcga atcagaatgt cgcggtgaat acgttcccgg gtcttgtaca 1380
caccgcccgt cacaccatgg gagtcagcaa cgcccgaagt cagtgaccca actcgcaaga 1440
gagggagctg ccgaaggcgg ggcaggtaac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 19
<211> 1528
<212> ДНК
<213> Clostridium clostridioforme
<400> 19
atgagagttt gatcctggct caggatgaac gctggcggcg tgcctaacac atgcaagtcg 60
aacgaagcaa ttaaaggaag ttttcggatg gaatttgatt gactgagtgg cggacgggtg 120
agtaacgcgt ggataacctg cctcacactg ggggataaca gttagaaatg actgctaata 180
ccgcataagc gcacagtgcc gcatggtacg gtgtgaaaaa ctccggtggt gtgagatgga 240
tccgcgtctg attagccagt tggcggggta acggcccacc aaagcgacga tcagtagccg 300
acctgagagg gtgaccggcc acattgggac tgagacacgg cccaaactcc tacgggaggc 360
agcagtgggg aatattgcac aatgggcgaa agcctgatgc agcgacgccg cgtgagtgaa 420
gaagtatttc ggtatgtaaa gctctatcag cagggaagaa aatgacggta cctgactaag 480
aagccccggc taactacgtg ccagcagccg cggtaatacg tagggggcaa gcgttatccg 540
gatttactgg gtgtaaaggg agcgtagacg gcgaagcaag tctgaagtga aaacccaggg 600
ctcaaccctg ggactgcttt ggaaactgtt ttgctagagt gtcggagagg taagtggaat 660
tcctagtgta gcggtgaaat gcgtagatat taggaggaac accagtggcg aaggcggctt 720
actggacgat aactgacgtt gaggctcgaa agcgtgggga gcaaacagga ttagataccc 780
tggtagtcca cgccgtaaac gatgaatgct aggtgttggg gggcaaagcc cttcggtgcc 840
gtcgcaaacg cagtaagcat tccacctggg gagtacgttc gcaagaatga aactcaaagg 900
aattgacggg gacccgcaca agcggtggag catgtggttt aattcgaagc aacgcgaaga 960
accttaccaa gtcttgacat cctcttgacc ggcgtgtaac ggcgccttcc cttcggggca 1020
agagagacag gtggtgcatg gttgtcgtca gctcgtgtcg tgagatgttg ggttaagtcc 1080
cgcaacgagc gcaaccctta tccttagtag ccagcaggta gagctgggca ctctagggag 1140
actgccaggg ataacctgga ggaaggtggg gatgacgtca aatcatcatg ccccttatga 1200
tttgggctac acacgtgcta caatggcgta aacaaaggga agcaagacag tgatgtggag 1260
caaatcccaa aaataacgtc ccagttcgga ctgtagtctg caacccgact acacgaagct 1320
ggaatcgcta gtaatcgcga atcagaatgt cgcggtgaat acgttcccgg gtcttgtaca 1380
caccgcccgt cacaccatgg gagtcagcaa cgcccgaagt cagtgaccca actcgcaaga 1440
gagggagctg ccgaaggcgg ggcaggtaac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 20
<211> 1528
<212> ДНК
<213> Clostridium clostridioforme
<400> 20
atgagagttt gatcctggct caggatgaac gctggcggcg tgcctaacac atgcaagtcg 60
aacgaagcaa ttaaaggaag ttttcggatg gaatttgatt gactgagtgg cggacgggtg 120
agtaacgcgt ggataacctg cctcacactg ggggataaca gttagaaatg actgctaata 180
ccgcataagc gcacagtacc gcatggtacg gtgtgaaaaa ctccggtggt gtgagatgga 240
tccgcgtctg attagccagt tggcggggta acggcccacc aaagcgacga tcagtagccg 300
acctgagagg gtgaccggcc acattgggac tgagacacgg cccaaactcc tacgggaggc 360
agcagtgggg aatattgcac aatgggcgaa agcctgatgc agcgacgccg cgtgagtgaa 420
gaagtatttc ggtatgtaaa gctctatcag cagggaagaa aatgacggta cctgactaag 480
aagccccggc taactacgtg ccagcagccg cggtaatacg tagggggcaa gcgttatccg 540
gatttactgg gtgtaaaggg agcgtagacg gcgaagcaag tctgaagtga aaacccaggg 600
ctcaaccctg ggactgcttt ggaaactgtt ttgctagagt gtcggagagg taagtggaat 660
tcctagtgta gcggtgaaat gcgtagatat taggaggaac accagtggcg aaggcggctt 720
actggacgat aactgacgtt gaggctcgaa agcgtgggga gcaaacagga ttagataccc 780
tggtagtcca cgccgtaaac gatgaatgct aggtgttggg gggcaaagcc cttcggtgcc 840
gtcgcaaacg cagtaagcat tccacctggg gagtacgttc gcaagaatga aactcaaagg 900
aattgacggg gacccgcaca agcggtggag catgtggttt aattcgaagc aacgcgaaga 960
accttaccaa gtcttgacat cctcttggcc ggcgtgtaac ggcgccttcc cttcggggca 1020
agagagacag gtggtgcatg gttgtcgtca gctcgtgtcg tgagatgttg ggttaagtcc 1080
cgcaacgagc gcaaccctta tccttagtag ccagcaggta gagctgggca ctctagggag 1140
actgccaggg ataacctgga ggaaggtggg gatgacgtca aatcatcatg ccccttatga 1200
tttgggctac acacgtgcta caatggcgta aacaaaggga agcaagacag tgatgtggag 1260
caaatcccaa aaataacgtc ccagttcgga ctgtagtctg caacccgact acacgaagct 1320
ggaatcgcta gtaatcgcga atcagaatgt cgcggtgaat acgttcccgg gtcttgtaca 1380
caccgcccgt cacaccatgg gagtcagcaa cgcccgaagt cagtgaccca actcgcaaga 1440
gagggagctg ccgaaggcgg ggcaggtaac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 21
<211> 1529
<212> ДНК
<213> Clostridium lavalense
<400> 21
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gaacgaagca tttcagatga agttttcgga tggattctga gatgactgag tggcggacgg 120
gtgagtaaca cgtggataac ctgcctcaca ctgggggaca acagttagaa atgactgcta 180
ataccgcata agcgcacagt accgcatggt acagtgtgaa aaactccggt ggtgtgagat 240
ggatccgcgt ctgattagcc agttggcggg gtaacggccc accaaagcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggc gaaagcctga tgcagcgacg ccgcgtgagt 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gataatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagtctgaag tgaaaaccca 600
gggctcaacc ctgggactgc tttggaaact gtcaagctag agtgcaggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac tgtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgagt gctaggtgtt ggggggcaaa gcccttcggt 840
gccgtcgcaa acgcaataag cactccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catcctcttg accggcgtgt aacggcgcct ttccttcggg 1020
acaagggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ttatccttag tagccagcat taagatgggc actctaggga 1140
gactgccagg gacaacctgg aggaaggtgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggcgt aaacaaaggg aagcgaccct gcgaaggtga 1260
gcaaatctca aaaataacgt cccagttcgg actgtagtct gcaacccgac tacacgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtcagca acgcccgaag tcagtgaccc aactcgcaag 1440
agagggagct gccgaaggcg gggcaggtaa ctggggtgaa gtcgtaacaa ggtagccgta 1500
tcggaaggtg cggctggatc acctccttt 1529
<210> 22
<211> 1524
<212> ДНК
<213> Clostridium nexile
<220>
<221> прочий_признак
<222> (1)..(7)
<223> N представляет собой A, T, C или G
<220>
<221> прочий_признак
<222> (191)..(192)
<223> N представляет собой A, T, C или G
<220>
<221> прочий_признак
<222> (348)..(348)
<223> N представляет собой A, T, C или G
<220>
<221> прочий_признак
<222> (437)..(437)
<223> N представляет собой A, T, C или G
<400> 22
nnnnnnngag atttgatcct ggctcaggat gaacgctggc cggccgtgct tacacatgca 60
gtcgaacgaa gcgcttaaac tggatttctt cggattgaag tttttgctga ctgagtggcg 120
gacgggtgag taacgcgtgg gtaacctgcc tcatacaggg ggataacagt tagaaatgac 180
tgctaatacc nnataagcgc acagtgctgc atggcacagt gtaaaaactc cggtggtatg 240
agatggaccc gcgtctgatt agctagttgg tggggtaacg gcctaccaag gcgacgatca 300
gtagccggcc tgagagggtg aacggccaca ttgggactga gacacggncc aaactcctac 360
gggaggcagc agtggggaat attgcacaat gggggaaacc ctgatgcagc gacgccgcgt 420
gagcgaagaa gtatttnggt atgtaaagct ctatcagcag ggaagaaaat gacggtacct 480
gactaagaag caccggctaa atacgtgcca gcagccgcgg taatacgtag gtgcaagcgt 540
tatccggatt tactgggtgt aaagggagcg tagacggttg tgtaagtctg atgtgaaagc 600
ccggggctca accccggact gcattggaaa ctatgtaact agagtgtcgg agaggtaagc 660
ggaattccta gtgtagcggt gaaatgcgta gatattagga ggaacaccag tggcgaaggc 720
ggcttactgg acgatcactg acgttgaggc tcgaaagcgt ggggagcaaa caggattaga 780
taccctggta gtccacgccg taaacgatga ctactaggtg tcggggagca aagctcttcg 840
gtgccgcagc aaacgcaata agtagtccac ctggggagta cgttcgcaag aatgaaactc 900
aaaggaattg acggggaccc gcacaagcgt ggagcatgtg gtttaattcg agcaacgcga 960
agaccttacc tggtcttgac atcccggtga ccggtccagt aatgggacct ttccttcggg 1020
acacggtgac aggtggtgca tggttgtcgt cagctcgtgt cgtgagatgt tgggttaagt 1080
cccgcaacga gcgcaacccc tatcttcagt agccagcatt taaggtgggc actctggaga 1140
gactgccagg gataacctgg aggaaggtgg ggatgacgtc aaatcatcat gccccttatg 1200
accagggcta cacacgtgct acaatggcgt aaacaaaggg aagcgaacct gtgaggggaa 1260
gcaaatctca aaaataacgt ctcagttcgg attgtagtct gcaactcgac tacatgaagc 1320
tggaatcgct agtaatcgcg aatcagcatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtcagta acgcccgaag tcagtgaccc aaccgtaagg 1440
agggagctgc cgaaggtggg accgataact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctcc 1524
<210> 23
<211> 1527
<212> ДНК
<213> Clostridium phytofermentans
<400> 23
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agtcctcgga tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa catagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
gacccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgtcg gggctatcat ggcttcggtg 840
gcgcagctaa cgcaataagt attccacctg gggagtacgt tcgcaagaat gaaactcaaa 900
ggaattgacg gggacccgca caagcggtgg agcatgtggt ttaattcgaa gcaacgcgaa 960
gaaccttacc aagtcttgac atccctctga caaccgagta acgtcggtct tccttcggga 1020
cagaggtgac aggtggtgca tggttgtcgt cagctcgtgt cgtgagatgt tgggttaagt 1080
cccgcaacga gcgcaacccc tatctttagt agccagcagt tcggctgggc actctagaga 1140
gactgccagg gataacctgg aggaaggcgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggtga ctacaaagag aagcaagcct gcgaggggga 1260
gcaaatctca aaaaggtcat cccagttcgg attgtactct gcaactcgag tacatgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtaggta acgcccgaag tcagtgaccc aaccgtaagg 1440
agggagctgc cgaaggcggg atctataact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctccttt 1527
<210> 24
<211> 1528
<212> ДНК
<213> Clostridium phytofermentans
<400> 24
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agccctcggg tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa tatagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
ggcccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgttg gggtccgata ggacttcggt 840
ggcgcagcta acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catccctctg acaaccgagt aacgtcggtc ttccttcggg 1020
acagaggtga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcag ttcggctggg cactctagag 1140
agactgccag ggataacctg gaggaaggcg gggatgacgt caaatcatca tgccccttat 1200
gatttgggct acacacgtgc tacaatggtg actacaaaga gaagcaagcc tgcgaggggg 1260
agcaaatctc aaaaaggtca tcccagttcg gattgtactc tgcaactcga gtacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtaggt aacgcccgaa gtcagtgacc caaccgtaag 1440
gagggagctg ccgaaggcgg gatctataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 25
<211> 1528
<212> ДНК
<213> Clostridium phytofermentans
<400> 25
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agccctcggg tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa tatagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
ggcccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgttg gggtccgata ggacttcggt 840
ggcgcagcta acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catccctctg acaaccgagt aacgtcggtc ttccttcggg 1020
acagaggtga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcag ttcggctggg cactctagag 1140
agactgccag ggataacctg gaggaaggcg gggatgacgt caaatcatca tgccccttat 1200
gatttgggct acacacgtgc tacaatggtg actacaaaga gaagcaagcc tgcgaggggg 1260
agcaaatctc aaaaaggtca tcccagttcg gattgtactc tgcaactcga gtacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtaggt aacgcccgaa gtcagtgacc caaccgtaag 1440
gagggagctg ccgaaggcgg gatctataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 26
<211> 1527
<212> ДНК
<213> Clostridium phytofermentans
<400> 26
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agtcctcgga tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa tatagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
ggcccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgtcg gggctatcat ggcttcggtg 840
gcgcagctaa cgcaataagt attccacctg gggagtacgt tcgcaagaat gaaactcaaa 900
ggaattgacg gggacccgca caagcggtgg agcatgtggt ttaattcgaa gcaacgcgaa 960
gaaccttacc aagtcttgac atccctctga caaccgagta acgtcggtct tccttcggga 1020
cagaggtgac aggtggtgca tggttgtcgt cagctcgtgt cgtgagatgt tgggttaagt 1080
cccgcaacga gcgcaacccc tatctttagt agccagcagt tcggctgggc actctagaga 1140
gactgccagg gataacctgg aggaaggcgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggtga ctacaaagag aagcaagcct gcgaggggga 1260
gcaaatctca aaaaggtcat cccagttcgg attgtactct gcaactcgag tacatgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtaggta acgcccgaag tcagtgaccc aaccgtaagg 1440
agggagctgc cgaaggcggg atctataact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctccttt 1527
<210> 27
<211> 1527
<212> ДНК
<213> Clostridium phytofermentans
<400> 27
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agtcctcgga tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa tatagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
ggcccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgtcg gggctatcat ggcttcggtg 840
gcgcagctaa cgcaataagt attccacctg gggagtacgt tcgcaagaat gaaactcaaa 900
ggaattgacg gggacccgca caagcggtgg agcatgtggt ttaattcgaa gcaacgcgaa 960
gaaccttacc aagtcttgac atccctctga caaccgagta acgtcggtct tccttcggga 1020
cagaggtgac aggtggtgca tggttgtcgt cagctcgtgt cgtgagatgt tgggttaagt 1080
cccgcaacga gcgcaacccc tatctttagt agccagcagt tcggctgggc actctagaga 1140
gactgccagg gataacctgg aggaaggcgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggtga ctacaaagag aagcaagcct gcgaggggga 1260
gcaaatctca aaaaggtcat cccagttcgg attgtactct gcaactcgag tacatgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtaggta acgcccgaag tcagtgaccc aaccgtaagg 1440
agggagctgc cgaaggcggg atttataact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctccttt 1527
<210> 28
<211> 1527
<212> ДНК
<213> Clostridium phytofermentans
<400> 28
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agtcctcgga tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa catagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
ggcccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgtcg gggctatcat ggcttcggtg 840
gcgcagctaa cgcaataagt attccacctg gggagtacgt tcgcaagaat gaaactcaaa 900
ggaattgacg gggacccgca caagcggtgg agcatgtggt ttaattcgaa gcaacgcgaa 960
gaaccttacc aagtcttgac atccctctga caaccgagta acgtcggtct tccttcggga 1020
cagaggtgac aggtggtgca tggttgtcgt cagctcgtgt cgtgagatgt tgggttaagt 1080
cccgcaacga gcgcaacccc tatctttagt agccagcagt tcggctgggc actctagaga 1140
gactgccagg gataacctgg aggaaggcgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggtga ctacaaagag aagcaagcct gcgaggggga 1260
gcaaatctca aaaaggtcat cccagttcgg attgtactct gcaactcgag tacatgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtaggta acgcccgaag tcagtgaccc aaccgtaagg 1440
agggagctgc cgaaggcggg atttataact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctccttt 1527
<210> 29
<211> 1527
<212> ДНК
<213> Clostridium phytofermentans
<400> 29
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agccctcggg tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa tatagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
ggcccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgtcg gggctatcat ggcttcggtg 840
gcgcagctaa cgcaataagt attccacctg gggagtacgt tcgcaagaat gaaactcaaa 900
ggaattgacg gggacccgca caagcggtgg agcatgtggt ttaattcgaa gcaacgcgaa 960
gaaccttacc aagtcttgac atccctctga caaccgagta acgtcggtct tccttcggga 1020
cagaggtgac aggtggtgca tggttgtcgt cagctcgtgt cgtgagatgt tgggttaagt 1080
cccgcaacga gcgcaacccc tatctttagt agccagcagt tcggctgggc actctagaga 1140
gactgccagg gataacctgg aggaaggcgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggtga ctacaaagag aagcaagcct gcgaggggga 1260
gcaaatctca aaaaggtcat cccagttcgg attgtactct gcaactcgag tacatgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtaggta acgcccgaag tcagtgaccc aaccgtaagg 1440
agggagctgc cgaaggcggg atttataact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctccttt 1527
<210> 30
<211> 1527
<212> ДНК
<213> Clostridium phytofermentans
<400> 30
tttgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacggagtt tttaaatgga agtcctcgga tggaagttta gaaacttagt ggcggacggg 120
tgagtaacgc gtgggtaacc tgcctcatac agggggataa cagtcggaaa cgattgctaa 180
aaccgcataa catagcgaaa ccgcatgatt ttgctatcaa atatttatag gtatgagatg 240
ggcccgcgtc tgattagcta gttggtgggg taatggccta ccaaggcgac gatcagtagc 300
cggcttgaga gagtgaccgg ccacattggg actgagacac ggcccaaact cctacgggag 360
gcagcagtgg ggaatattgg acaatggggg aaaccctgat ccagcgacgc cgcgtgagtg 420
aagaagtatt tcggtatgta aagctctatc agcagggaag ataatgacag tacctgacta 480
agaagccccg gctaactacg tgccagcagc cgcggtaata cgtagggggc aagcgttatc 540
cggatttact gggtgtaaag ggagcgtagg tggtaggtca agtcagatgt gaaagcccag 600
ggctcaaccc tgggactgca tttgaaactg gcttactaga gtgcaggaga ggtaagtgga 660
attcctagtg tagcggtgaa atgcgtagat attaggagga acaccagtgg cgaaggcggc 720
ttactggact gtaactgaca ctgaggctcg aaagcgtggg gagcaaacag gattagatac 780
cctggtagtc cacgccgtaa acgatgaata ctagctgtcg gggctatcat ggcttcggtg 840
gcgcagctaa cgcaataagt attccacctg gggagtacgt tcgcaagaat gaaactcaaa 900
ggaattgacg gggacccgca caagcggtgg agcatgtggt ttaattcgaa gcaacgcgaa 960
gaaccttacc aagtcttgac atccctctga caaccgagta acgtcggtct tccttcggga 1020
cagaggtgac aggtggtgca tggttgtcgt cagctcgtgt cgtgagatgt tgggttaagt 1080
cccgcaacga gcgcaacccc tatctttagt agccagcagt tcggctgggc actctagaga 1140
gactgccagg gataacctgg aggaaggcgg ggatgacgtc aaatcatcat gccccttatg 1200
atttgggcta cacacgtgct acaatggtga ctacaaagag aagcaagcct gcgaggggga 1260
gcaaatctca aaaaggtcat cccagttcgg attgtactct gcaactcgag tacatgaagc 1320
tggaatcgct agtaatcgcg aatcagaatg tcgcggtgaa tacgttcccg ggtcttgtac 1380
acaccgcccg tcacaccatg ggagtaggta acgcccgaag tcagtgaccc aaccgtaagg 1440
agggagctgc cgaaggcggg atctataact ggggtgaagt cgtaacaagg tagccgtatc 1500
ggaaggtgcg gctggatcac ctccttt 1527
<210> 31
<211> 1509
<212> ДНК
<213> Clostridium sporogenes
<400> 31
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgatgaa gcttccttcg ggaagtggat tagcggcgga cgggtgagta acacgtgggt 120
aacctgcctc aaagtggggg atagccttcc gaaaggaaga ttaataccgc ataatataag 180
agaatcgcat gattttttta tcaaagattt attgctttga gatggacccg cggcgcatta 240
gctagttggt aaggtaacgg cttaccaagg caacgatgcg tagccgacct gagagggtga 300
tcggccacat tggaactgag acacggtcca gactcctacg ggaggcagca gtggggaata 360
ttgcgcaatg ggggaaaccc tgacgcagca acgccgcgtg ggtgatgaag gtcttcggat 420
tgtaaagccc tgttttctgg gacgataatg acggtaccag aggaggaagc cacggctaac 480
tacgtgccag cagccgcggt aatacgtagg tggcgagcgt tgtccggatt tactgggcgt 540
aaagggtgcg taggcggatg tttaagtggg atgtgaaatc cccgggctta acctgggggc 600
tgcattccaa actggatatc tagagtgcag gagaggaaag cggaattcct agtgtagcgg 660
tgaaatgcgt agagattagg aagaacacca gtggcgaagg cggctttctg gactgtaact 720
gacgctgagg cacgaaagcg tgggtagcaa acaggattag ataccctggt agtccacgcc 780
gtaaacgatg gatactaggt gtagggggta tcaactcccc ctgtgccgca gttaacacaa 840
taagtatccc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctggac 960
ttgacatccc ttgcatagcc tagagatagg tgaagccctt cggggcaagg agacaggtgg 1020
tgcatggttg tcgtcagctc gtgtcgtgag atgttaggtt aagtcctgca acgagcgcaa 1080
cccttgttat tagttgctac cattaagttg agcactctaa tgagactgcc tgggtaacca 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtccagggc tacacacgtg 1200
ctacaatggt aggtacaata agacgcaaga ccgtgaggtg gagcaaaact tataaaacct 1260
atctcagttc ggattgtagg ctgcaactcg cctacatgaa gctggagttg ctagtaatcg 1320
cgaatcagaa tgtcgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca 1380
tgagagctgg taacacccga agtccgtgag gtaaccgtaa ggagccagcg gccgaaggtg 1440
ggattagtga ttggggtgaa gtcgtaacaa ggtagccgta ggagaacctg cggctggatc 1500
acctccttt 1509
<210> 32
<211> 1509
<212> ДНК
<213> Clostridium sporogenes
<400> 32
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgatgaa gcttccttcg ggaagtggat tagcggcgga cgggtgagta acacgtgggt 120
aacctgcctc aaagtggggg atagccttcc gaaaggaaga ttaataccgc ataacataag 180
agaatcgcat gattttctta tcaaagattt attgctttga gatggacccg cggcgcatta 240
gctagttggt aaggtaacgg cttaccaagg caacgatgcg tagccgacct gagagggtga 300
tcggccacat tggaactgag acacggtcca gactcctacg ggaggcagca gtggggaata 360
ttgcgcaatg ggggaaaccc tgacgcagca acgccgcgtg ggtgatgaag gtcttcggat 420
tgtaaagccc tgttttctgg gacgataatg acggtaccag aggaggaagc cacggctaac 480
tacgtgccag cagccgcggt aatacgtagg tggcgagcgt tgtccggatt tactgggcgt 540
aaagggtgcg taggcggatg tttaagtggg atgtgaaatc cccgggctta acctgggggc 600
tgcattccaa actggatatc tagagtgcag gagaggaaag cggaattcct agtgtagcgg 660
tgaaatgcgt agagattagg aagaacacca gtggcgaagg cggctttctg gactgtaact 720
gacgctgagg cacgaaagcg tgggtagcaa acaggattag ataccctggt agtccacgcc 780
gtaaacgatg gatactaggt gtagggggta tcaactcccc ctgtgccgca gttaacacaa 840
taagtatccc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctggac 960
ttgacatccc ttgcatagcc tagagatagg tgaagccctt cggggcaagg agacaggtgg 1020
tgcatggttg tcgtcagctc gtgtcgtgag atgttaggtt aagtcctgca acgagcgcaa 1080
cccttgttat tagttgctac cattaagttg agcactctaa tgagactgcc tgggtaacca 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtccagggc tacacacgtg 1200
ctacaatggt aggtacaata agacgcaaga ccgtgaggtg gagcaaaact tataaaacct 1260
atctcagttc ggattgtagg ctgcaactcg cctacatgaa gctggagttg ctagtaatcg 1320
cgaatcagaa tgtcgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca 1380
tgagagctgg taacacccga agtccgtgag gtaaccgtaa ggagccagcg gccgaaggtg 1440
ggattagtga ttggggtgaa gtcgtaacaa ggtagccgta ggagaacctg cggctggatc 1500
acctccttt 1509
<210> 33
<211> 1509
<212> ДНК
<213> Clostridium sporogenes
<400> 33
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgatgaa gcttccttcg ggaagtggat tagcggcgga cgggtgagta acacgtgggt 120
aacctgcctc aaagtggggg atagccttcc gaaaggaaga ttaataccgc ataacataag 180
agaatcgcat gattttctta tcaaagattt attgctttga gatggacccg cggcgcatta 240
gctagttggt aaggtaacgg cttaccaagg caacgatgcg tagccgacct gagagggtga 300
tcggccacat tggaactgag acacggtcca gactcctacg ggaggcagca gtggggaata 360
ttgcgcaatg ggggaaaccc tgacgcagca acgccgcgtg ggtgatgaag gtcttcggat 420
tgtaaagccc tgttttctgg gacgataatg acggtaccag aggaggaagc cacggctaac 480
tacgtgccag cagccgcggt aatacgtagg tggcgagcgt tgtccggatt tactgggcgt 540
aaagggtgcg taggcggatg tttaagtggg atgtgaaatc cccgggctta acctgggggc 600
tgcattccaa actggatatc tagagtgcag gagaggaaag cggaattcct agtgtagcgg 660
tgaaatgcgt agagattagg aagaacacca gtggcgaagg cggctttctg gactgtaact 720
gacgctgagg cacgaaagcg tgggtagcaa acaggattag ataccctggt agtccacgcc 780
gtaaacgatg gatactaggt gtagggggta tcaactcccc ctgtgccgca gttaacacaa 840
taagtatccc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctggac 960
ttgacatccc ttgcatagcc tagagatagg tgaagccctt cggggcaagg agacaggtgg 1020
tgcatggttg tcgtcagctc gtgtcgtgag atgttaggtt aagtcctgca acgagcgcaa 1080
cccttgttat tagttgctac cattaagttg agcactctaa tgagactgcc tgggtaacca 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtccagggc tacacacgtg 1200
ctacaatggt aggtacaata agacgcaaga ccgtgaggtg gagcaaaact tataaaacct 1260
atctcagttc ggattgtagg ctgcaactcg cctacatgaa gctggagttg ctagtaatcg 1320
cgaatcagaa tgtcgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca 1380
tgagagctgg taacacccga agtccgtgag gtaaccgtaa ggagccagcg gccgaaggtg 1440
ggattagtga ttggggtgaa gtcgtaacaa ggtagccgta ggagaacctg cggctggatc 1500
acctccttt 1509
<210> 34
<211> 1509
<212> ДНК
<213> Clostridium sporogenes
<400> 34
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgatgaa gcttccttcg ggaagtggat tagcggcgga cgggtgagta acacgtgggt 120
aacctgcctc aaagtggggg atagccttcc gaaaggaaga ttaataccgc ataacataag 180
agaatcgcat gattttctta tcaaagattt attgctttga gatggacccg cggcgcatta 240
gctagttggt aaggtaacgg cttaccaagg caacgatgcg tagccgacct gagagggtga 300
tcggccacat tggaactgag acacggtcca gactcctacg ggaggcagca gtggggaata 360
ttgcgcaatg ggggaaaccc tgacgcagca acgccgcgtg ggtgatgaag gtcttcggat 420
tgtaaagccc tgttttctgg gacgataatg acggtaccag aggaggaagc cacggctaac 480
tacgtgccag cagccgcggt aatacgtagg tggcgagcgt tgtccggatt tactgggcgt 540
aaagggtgcg taggcggatg tttaagtggg atgtgaaatc cccgggctta acctgggggc 600
tgcattccaa actggatatc tagagtgcag gagaggaaag cggaattcct agtgtagcgg 660
tgaaatgcgt agagattagg aagaacacca gtggcgaagg cggctttctg gactgtaact 720
gacgctgagg cacgaaagcg tgggtagcaa acaggattag ataccctggt agtccacgcc 780
gtaaacgatg gatactaggt gtagggggta tcaactcccc ctgtgccgca gttaacacaa 840
taagtatccc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctggac 960
ttgacatccc ttgcatagcc tagagatagg tgaagccctt cggggcaagg agacaggtgg 1020
tgcatggttg tcgtcagctc gtgtcgtgag atgttaggtt aagtcctgca acgagcgcaa 1080
cccttgttat tagttgctac cattaagttg agcactctaa tgagactgcc tgggtaacca 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtccagggc tacacacgtg 1200
ctacaatggt aggtacaata agacgcaaga ccgtgaggtg gagcaaaact tataaaacct 1260
atctcagttc ggattgtagg ctgcaactcg cctacatgaa gctggagttg ctagtaatcg 1320
cgaatcagaa tgtcgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca 1380
tgagagctgg taacacccga agtccgtgag gtaaccgtaa ggagccagcg gccgaaggtg 1440
ggattagtga ttggggtgaa gtcgtaacaa ggtagccgta ggagaacctg cggctggatc 1500
acctccttt 1509
<210> 35
<211> 1509
<212> ДНК
<213> Clostridium sporogenes
<400> 35
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgatgaa gcttccttcg ggaagtggat tagcggcgga cgggtgagta acacgtgggt 120
aacctgcctc aaagtggggg atagccttcc gaaaggaaga ttaataccgc ataacataag 180
agaatcgcat gattttctta tcaaagattt attgctttga gatggacccg cggcgcatta 240
gctagttggt aaggtaacgg cttaccaagg caacgatgcg tagccgacct gagagggtga 300
tcggccacat tggaactgag acacggtcca gactcctacg ggaggcagca gtggggaata 360
ttgcgcaatg ggggaaaccc tgacgcagca acgccgcgtg ggtgatgaag gtcttcggat 420
tgtaaagccc tgttttctgg gacgataatg acggtaccag aggaggaagc cacggctaac 480
tacgtgccag cagccgcggt aatacgtagg tggcgagcgt tgtccggatt tactgggcgt 540
aaagggtgcg taggcggatg tttaagtggg atgtgaaatc cccgggctta acctgggggc 600
tgcattccaa actggatatc tagagtgcag gagaggaaag cggaattcct agtgtagcgg 660
tgaaatgcgt agagattagg aagaacacca gtggcgaagg cggctttctg gactgtaact 720
gacgctgagg cacgaaagcg tgggtagcaa acaggattag ataccctggt agtccacgcc 780
gtaaacgatg gatactaggt gtagggggta tcaactcccc ctgtgccgca gttaacacaa 840
taagtatccc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctggac 960
ttgacatccc ttgcatagcc tagagatagg tgaagccctt cggggcaagg agacaggtgg 1020
tgcatggttg tcgtcagctc gtgtcgtgag atgttaggtt aagtcctgca acgagcgcaa 1080
cccttgttat tagttgctac cattaagttg agcactctaa tgagactgcc tgggtaacca 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtccagggc tacacacgtg 1200
ctacaatggt aggtacaata agacgcaaga ccgtgaggtg gagcaaaact tataaaacct 1260
atctcagttc ggattgtagg ctgcaactcg cctacatgaa gctggagttg ctagtaatcg 1320
cgaatcagaa tgtcgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca 1380
tgagagctgg taacacccga agtccgtgag gtaaccgtaa ggagccagcg gccgaaggtg 1440
ggattagtga ttggggtgaa gtcgtaacaa ggtagccgta ggagaacctg cggctggatc 1500
acctccttt 1509
<210> 36
<211> 1509
<212> ДНК
<213> Clostridium sporogenes
<400> 36
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgatgaa gcttccttcg ggaagtggat tagcggcgga cgggtgagta acacgtgggt 120
aacctgcctc aaagtggggg atagccttcc gaaaggaaga ttaataccgc ataacataag 180
agaatcgcat gattttctta tcaaagattt attgctttga gatggacccg cggcgcatta 240
gctagttggt aaggtaacgg cttaccaagg caacgatgcg tagccgacct gagagggtga 300
tcggccacat tggaactgag acacggtcca gactcctacg ggaggcagca gtggggaata 360
ttgcgcaatg ggggaaaccc tgacgcagca acgccgcgtg ggtgatgaag gtcttcggat 420
tgtaaagccc tgttttctgg gacgataatg acggtaccag aggaggaagc cacggctaac 480
tacgtgccag cagccgcggt aatacgtagg tggcgagcgt tgtccggatt tactgggcgt 540
aaagggtgcg taggcggatg tttaagtggg atgtgaaatc cccgggctta acctgggggc 600
tgcattccaa actggatatc tagagtgcag gagaggaaag cggaattcct agtgtagcgg 660
tgaaatgcgt agagattagg aagaacacca gtggcgaagg cggctttctg gactgtaact 720
gacgctgagg cacgaaagcg tgggtagcaa acaggattag ataccctggt agtccacgcc 780
gtaaacgatg gatactaggt gtagggggta tcaactcccc ctgtgccgca gttaacacaa 840
taagtatccc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctggac 960
ttgacatccc ttgcatagcc tagagatagg tgaagccctt cggggcaagg agacaggtgg 1020
tgcatggttg tcgtcagctc gtgtcgtgag atgttaggtt aagtcctgca acgagcgcaa 1080
cccttgttat tagttgctac cattaagttg agcactctaa tgagactgcc tgggtaacca 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtccagggc tacacacgtg 1200
ctacaatggt aggtacaata agacgcaaga ccgtgaggtg gagcaaaact tataaaacct 1260
atctcagttc ggattgtagg ctgcaactcg cctacatgaa gctggagttg ctagtaatcg 1320
cgaatcagaa tgtcgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca 1380
tgagagctgg taacacccga agtccgtgag gtaaccgtaa ggagccagcg gccgaaggtg 1440
ggattagtga ttggggtgaa gtcgtaacaa ggtagccgta ggagaacctg cggctggatc 1500
acctccttt 1509
<210> 37
<211> 1509
<212> ДНК
<213> Clostridium sporogenes
<400> 37
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgatgaa gcttccttcg ggaagtggat tagcggcgga cgggtgagta acacgtgggt 120
aacctgcctc aaagtggggg atagccttcc gaaaggaaga ttaataccgc ataatataag 180
agaatcgcat gattttttta tcaaagattt attgctttga gatggacccg cggcgcatta 240
gctagttggt aaggtaacgg cttaccaagg caacgatgcg tagccgacct gagagggtga 300
tcggccacat tggaactgag acacggtcca gactcctacg ggaggcagca gtggggaata 360
ttgcgcaatg ggggaaaccc tgacgcagca acgccgcgtg ggtgatgaag gtcttcggat 420
tgtaaagccc tgttttctgg gacgataatg acggtaccag aggaggaagc cacggctaac 480
tacgtgccag cagccgcggt aatacgtagg tggcgagcgt tgtccggatt tactgggcgt 540
aaagggtgcg taggcggatg tttaagtggg atgtgaaatc cccgggctta acctgggggc 600
tgcattccaa actggatatc tagagtgcag gagaggaaag cggaattcct agtgtagcgg 660
tgaaatgcgt agagattagg aagaacacca gtggcgaagg cggctttctg gactgtaact 720
gacgctgagg cacgaaagcg tgggtagcaa acaggattag ataccctggt agtccacgcc 780
gtaaacgatg gatactaggt gtagggggta tcaactcccc ctgtgccgca gttaacacaa 840
taagtatccc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctggac 960
ttgacatccc ttgcatagcc tagagatagg tgaagccctt cggggcaagg agacaggtgg 1020
tgcatggttg tcgtcagctc gtgtcgtgag atgttaggtt aagtcctgca acgagcgcaa 1080
cccttgttat tagttgctac cattaagttg agcactctaa tgagactgcc tgggtaacca 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtccagggc tacacacgtg 1200
ctacaatggt aggtacaata agacgcaaga ccgtgaggtg gagcaaaact tataaaacct 1260
atctcagttc ggattgtagg ctgcaactcg cctacatgaa gctggagttg ctagtaatcg 1320
cgaatcagaa tgtcgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca 1380
tgagagctgg taacacccga agtccgtgag gtaaccgtaa ggagccagcg gccgaaggtg 1440
ggattagtga ttggggtgaa gtcgtaacaa ggtagccgta ggagaacctg cggctggatc 1500
acctccttt 1509
<210> 38
<211> 1504
<212> ДНК
<213> Collinsella tanakaei
<400> 38
cggagagttc gatcctggct caggatgaac gctggcggcg cgcctaacac atgcaagtcg 60
aacggcaccc ctcctcggag ggaagcgagt ggcgaacggc tgagtaacac gtggagaacc 120
tgccccctcc tcggggatag ccgcgggaaa ccgcgggtaa cacccgacga ccccggagcc 180
gcgcatgcgg cgccgggcaa atctccggag ggaggggatg gctccgcggc ccatcaggta 240
gacggcgggg taacggccca ccgtgccgac aacgggtagc cgggttgaga gaccgaccgg 300
ccagattggg actgagacac ggcccagact cctacgggag gcagcagtgg ggaatcttgc 360
gcaatggggg caaccctgac gcagcgacgc cgcgtgcggg acgaaggcct tcgggtcgta 420
aaccgctttc agcagggacg agtcaagacg gtacctgcag aagaagcccc ggctaactac 480
gtgccagcag ccgcggtaat acgtaggggg cgagcgttat ccggattcat tgggcgtaaa 540
gcgcgcgtag gcggccgcgt aggcgggggg tcaaatcccg gggctcaacc ccggtccgcc 600
ccccgaaccc cgcggctcgg gtccggtagg ggagggtgga attcccggtg tagcggtgga 660
atgcgcagat atcgggagga acaccggtgg cgaaggcggc cctctgggcc gagaccgacg 720
ctgaggcgcg aaagctgggg gagcgaacag gattagatac cctggtagtc ccagccgtaa 780
acgatggacg ctaggtgtgg ggggacgatc cctccgtgcc gcagccaacg cattaagcgt 840
cccgcctggg gagtacggcc gcaaggctaa aactcaaagg aattgacggg ggcccgcaca 900
agcagcggag catgtggctt aattcgaagc aacgcgaaga accttaccag ggcttgacat 960
gtgcgtgaag ccggggagac ccggtggccg agaggagcgc acacaggtgg tgcatggctg 1020
tcgtcagctc gtgtcgtgag atgttgggtt aagtcccgca acgagcgcaa cccccgccgc 1080
atgttgccag caggtcaggc tgggcaccca tgcgggaccg ccggcgtcaa gccggaggag 1140
ggcggggacg acgtcaagtc atcatgcccc ttatgccctg ggctgcacac gtgctacaat 1200
ggccggtaca gcgggatgcg atggcgcgag ccggagcgga tccctcaaag ccggccccag 1260
ttcggattgg gggctgcaac ccgcccccat gaagtcggag ttgctagtaa tcgcggatca 1320
gcatgccgcg gtgaatgcgt tcccgggcct tgtacacacc gcccgtcaca ccacccgagt 1380
cgtctgcacc cgaagccgcc ggcccaacct tccgaggggg gaggcgtcga aggtgtggag 1440
ggtgaggggg gtgaagtcgt aacaaggtag ccgtacggga acgtgcggct ggatcacctc 1500
cttt 1504
<210> 39
<211> 1366
<212> ДНК
<213> Coprococcus sp HPP0048
<400> 39
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagca cttgaatgga attcttcgga aggaagctca agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcggtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accggcaagt aatgttgcct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgt 1366
<210> 40
<211> 1528
<212> ДНК
<213> Coprococcus sp HPP0048
<400> 40
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagcg cttgaatgga attcttcgga aggaagctca agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accggcaagt aatgttgcct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgcaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 41
<211> 1366
<212> ДНК
<213> Coprococcus sp HPP0048
<400> 41
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagcg cttgaatgga attcttcgga aggaagctca agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accggcaagt aatgttgcct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgt 1366
<210> 42
<211> 1366
<212> ДНК
<213> Coprococcus sp HPP0048
<400> 42
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagca cttgaatgga attcttcgga aggaagctag agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accggcaagt aatgttgcct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgt 1366
<210> 43
<211> 1434
<212> ДНК
<213> Coprococcus sp HPP0048
<400> 43
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtt 60
gaacgaagcg cttgaatgga attcttcgga aggaagctca agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accggcaagt aatgttgcct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caac 1434
<210> 44
<211> 1366
<212> ДНК
<213> Coprococcus sp HPP0048
<400> 44
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagca cttgaatgga attcttcgga aggaagctag agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accggcaagt aatgttgcct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgt 1366
<210> 45
<211> 1530
<212> ДНК
<213> Flavonifractor plautii
<400> 45
tattgagagt ttgatcctgg ctcaggatga acgctggcgg cgtgcttaac acatgcaagt 60
cgaacggggt gctcatgacg gaggattcgt ccaatggatt gagttaccta gtggcggacg 120
ggtgagtaac gcgtgaggaa cctgccttgg agaggggaat aacactccga aaggagtgct 180
aataccgcat gaagcagttg ggtcgcatgg ctctgactgc caaagattta tcgctctgag 240
atggcctcgc gtctgattag ctagtaggcg gggtaacggc ccacctaggc gacgatcagt 300
agccggactg agaggttgac cggccacatt gggactgaga cacggcccag actcctacgg 360
gaggcagcag tggggaatat tgggcaatgg gcgcaagcct gacccagcaa cgccgcgtga 420
aggaagaagg ctttcgggtt gtaaacttct tttgtcgggg acgaaacaaa tgacggtacc 480
cgacgaataa gccacggcta actacgtgcc agcagccgcg gtaatacgta ggtggcaagc 540
gttatccgga tttactgggt gtaaagggcg tgtaggcggg attgcaagtc agatgtgaaa 600
actgggggct caacctccag cctgcatttg aaactgtagt tcttgagtgc tggagaggca 660
atcggaattc cgtgtgtagc ggtgaaatgc gtagatatac ggaggaacac cagtggcgaa 720
ggcggattgc tggacagtaa ctgacgctga ggcgcgaaag cgtggggagc aaacaggatt 780
agataccctg gtagtccacg ccgtaaacga tggatactag gtgtgggggg tctgaccccc 840
tccgtgccgc agttaacaca ataagtatcc cacctgggga gtacgatcgc aaggttgaaa 900
ctcaaaggaa ttgacggggg cccgcacaag cggtggagta tgtggtttaa ttcgaagcaa 960
cgcgaagaac cttaccaggg cttgacatcc cactaacgag gcagagatgc gttaggtgcc 1020
cttcggggaa agtggagaca ggtggtgcat ggttgtcgtc agctcgtgtc gtgagatgtt 1080
gggttaagtc ccgcaacgag cgcaaccctt attgttagtt gctacgcaag agcactctag 1140
cgagactgcc gttgacaaaa cggaggaagg tggggacgac gtcaaatcat catgcccctt 1200
atgtcctggg ccacacacgt actacaatgg tggttaacag agggaggcaa taccgcgagg 1260
tggagcaaat ccctaaaagc catcccagtt cggattgcag gctgaaaccc gcctgtatga 1320
agttggaatc gctagtaatc gcggatcagc atgccgcggt gaatacgttc ccgggccttg 1380
tacacaccgc ccgtcacacc atgagagtcg ggaacacccg aagtccgtag cctaaccgca 1440
aggagggcgc ggccgaaggt gggttcgata attggggtga agtcgtaaca aggtagccgt 1500
atcggaaggt gcggctggat cacctccttt 1530
<210> 46
<211> 1264
<212> ДНК
<213> Holdemania filiformis
<400> 46
acctaggcga tgatacgtag ccgacctgag agggtgaccg gccacattgg gactgagaca 60
cggcccaaac tcctacggga ggcagcagta gggaattttc ggcaatgggc gaaagcctga 120
ccgagcaacg ccgcgtgagt gaagaaggcc ttcgggttgt aaagctctgt tgtgaaggaa 180
gaacggctca tacagggaat ggtatgggag tgacggtact ttaccagaaa gccacggcta 240
actacgtgcc agcagccgcg gtaatacgta ggtggcgagc gttatccgga attattgggc 300
gtaaagggtg cgcaggcggt ttgttaagtt taaggtgaaa gcgtggggct taaccccata 360
tagccttaga aactgacaga ctagagtaca ggagagggca atggaattcc atgtgtagcg 420
gtaaaatgcg tagatatatg gaggaacacc agtggcgaag gcggttgcct ggcctgtaac 480
tgacgctcat gcacgaaagc gtggggagca aataggatta gataccctag tagtccacgc 540
cgtaaacgat gagaactaag tgttggggaa actcagtgct gcagttaacg caataagttc 600
tccgcctggg gagtatgcac gcaagtgtga aactcaaagg aattgacggg ggcccgcaca 660
agcggtggag tatgtggttt aattcgacgc aacgcgaaga accttaccag gtcttgacat 720
cccctgcaaa gacatagaga tatgttggag gttatcaggg agacaggtgg tgcatggttg 780
tcgtcagctc gtgtcgtgag atgttgggtt aagtcccgca acgagcgcaa cccttgtctt 840
tagttgctaa cattaagttg aggactctag agagactgcc ggtgacaaac cggaggaagg 900
tggggatgac gtcaaatcat catgcccctt atgacctggg ctacacacgt actacaatgg 960
cggatacaac gagaagcaag acagtgatgt ggagcaaaac tcagaaagtc cgtctcagtt 1020
cggattgaag tctgcaaccc gacttcatga agccggaatc gctagtaatc gcggatcagc 1080
atgccgcggt gaatacgttc tcgggccttg tacacaccgc ccgtcaaacc atgagagttg 1140
gcaatacccg aagccggtgg cctaacctcg caagaggagg gagccgtcga aggtagggct 1200
gatgattggg gttaagtcgt aacaaggtat ccctacggga acgtggggat ggatcacctc 1260
cttt 1264
<210> 47
<211> 1529
<212> ДНК
<213> Lachnospiraceae bacterium 2 1 46FAA
<400> 47
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagcg ctttcgattg aatcttcgga ggaaagagga agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atggctgcta 180
ataccgcata agcatacagc accgcatggt gcagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattaggt agttggtggg gtaacggcct accaagccga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gaagaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggaagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgtgactgc tttggaaact gtgcttctag agtgtcggag aggtaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacatcagtg gcgaaggcgg 720
cttactggac gataactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggtacaaa gtacttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctggtcttga catctccctg accggacagt aatgtgtcct ttctttcggg 1020
acagggaaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcaa gtaaagttgg gcactctgga 1140
gagactgcca gggataacct ggaggaaggt ggggacgacg tcaaatcatc atgcccctta 1200
tgaccagggc tacacacgtg ctacaatggc gtaaacaaag ggaagcaagc atgtgagtgt 1260
gagcaaatct caaaaataac gtctcagttc ggattgtagt ctgcaactcg actacatgaa 1320
gctggaatcg ctagtaatcg cgaatcagaa tgtcgcggtg aatacgttcc cgggtcttgt 1380
acacaccgcc cgtcacacca tgggagttgg taacgcccga agtcagtgac ctaaccgaaa 1440
ggaaggagct gccgaaggtg ggactgataa ctggggtgaa gtcgtaacaa ggtagccgta 1500
tcggaaggtg cggctggatc acctccttt 1529
<210> 48
<211> 1529
<212> ДНК
<213> Lachnospiraceae bacterium 2 1 46FAA
<400> 48
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagca ccttgacgga tttcttcgga ttgaagcctt ggtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttggaa acggctgcta 180
ataccgcata agcgcacagt accgcatggt acggtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattaggt agttggtggg gtaacggcct accaagccga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gatgaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagccagatg tgaaagcccg 600
gggctcaacc ccgggactgc atttggaact gtcaggctag agtgtcggag aggaaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
ctttctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctggtcttga catccctctg accgctcttt aatcggagct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcat tttcggatgg gcactctaga 1140
gagactgcca gggataacct ggaggaaggt ggggatgacg tcaaatcatc atgcccctta 1200
tgaccagggc tacacacgtg ctacaatggc gtaaacaaag ggaagcgagc ccgcgagggg 1260
gagcaaatcc caaaaataac gtctcagttc ggattgtagt ctgcaactcg actacatgaa 1320
gctggaatcg ctagtaatcg cgaatcagaa tgtcgcggtg aatacgttcc cgggtcttgt 1380
acacaccgcc cgtcacacca tgggagtcag taacgcccga agtcagtgac ccaaccgcaa 1440
ggagggagct gccgaaggtg ggaccgataa ctggggtgaa gtcgtaacaa ggtagccgta 1500
tcggaaggtg cggctggatc acctccttt 1529
<210> 49
<211> 1528
<212> ДНК
<213> Lachnospiraceae bacterium 4 1 37FAA
<400> 49
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagca cttgaatgga attcttcgga aggaagctca agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accggcaagt aatgttgcct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgcaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 50
<211> 1528
<212> ДНК
<213> Lachnospiraceae bacterium 6 1 63FAA
<400> 50
tcagagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagca cttatcattg actcttcgga agatttgata tttgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata caggggaata acagttagaa atggctgcta 180
atgccgcata agcgcacagg accgcatggt ctggtgtgaa aaactgaggt ggtatgagat 240
ggacccgcgt ctgattaggt agttggtggg gtaacggcct accaagccga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccagac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gaagaagtat ctcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggaagagc aagtctgatg tgaaaggctg 600
gggcttaacc ccaggactgc attggaaact gtttttctag agtgccggag aggtaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac ggtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc ggggtgcaaa gcagttcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac caagtcttga catctgcctg accgttcctt aaccggagct ttccttcggg 1020
acaggcaaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatccttag tagccagcag tccggctggg cactctaggg 1140
agactgccgg ggataacccg gaggaaggcg gggacgacgt caaatcatca tgccccttat 1200
gatttgggct acacacgtgc tacaatggcg taaacaaagg gaagcgaagc ggtgacgctt 1260
agcaaatctc aaaaataacg tcccagttcg gactgcagtc tgcaactcga ctgcacgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccttatg 1440
gagggagctg ccgaaggcgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 51
<211> 1528
<212> ДНК
<213> Lachnospiraceae bacterium 9 1 43BFAA
<400> 51
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagca cttgaatgga attcttcgga aggaagctca agtgactgag tggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agcacacgtg atcgcatgat cgagtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaatggccc accaaggcga cgatcagtag 300
ccggcctgag agggtgaacg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgaag 420
gatgaagtat ttcggtatgt aaacttctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcagtgc aagtctgaag tgaaagcccg 600
gggctcaacc ccgggactgc tttggaaact gtgcagctag agtgtcggag aggcaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttgctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc ggggagcaaa gctcttcggt 840
gccgcagcca acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catccctctg accgctcttt aatcggagct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatcttcag tagccagcgg taaggccggg cactctggag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcagagt cgtgaggccg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgcaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 52
<211> 1528
<212> ДНК
<213> Ruminococcus gnavus
<400> 52
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagca cttttgcgga tttcttcgga ttgaagcaat tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttggaa acggctgcta 180
ataccgcata agcgcacagt accgcatggt accgtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggcct accaaggcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gatgaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagccagatg tgaaagcccg 600
gggctcaacc ccgggactgc atttggaact gtcaggctag agtgtcggag aggaaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
ctttctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctggtcttga catccccctg accgctcttt aatcggagct ttccttcggg 1020
acaggggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcat ttcggatggg cactctagag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gaccagggct acacacgtgc tacaatggcg taaacaaagg gaagcgagcc cgcgaggggg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgcaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 53
<211> 1528
<212> ДНК
<213> Ruminococcus gnavus
<400> 53
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagca ccttgacgga tttcttcgga ttgaagcctt ggtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttggaa acggctgcta 180
ataccgcata agcgcacagt accgcatggt acggtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattaggt agttggtggg gtaacggcct accaagccga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gatgaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagccagatg tgaaagcccg 600
gggctcaacc ccgggactgc atttggaact gtcaggctag agtgtcggag aggaaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
ctttctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctggtcttga catccctctg accgctcttt aatcggagct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcat tttggatggg cactctagag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gatcagggct acacacgtgc tacaatggcg taaacaaagg gaagcgagcc cgcgaggggg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgcaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 54
<211> 1528
<212> ДНК
<213> Ruminococcus gnavus
<400> 54
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagca ccttgacgga tttcttcgga ttgaagcctt ggtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttggaa acggctgcta 180
ataccgcata agcgcacagt accgcatggt acggtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattaggt agttggtggg gtaacggcct accaagccga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gatgaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagccagatg tgaaagcccg 600
gggctcaacc ccgggactgc atttggaact gtcaggctag agtgtcggag aggaaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
ctttctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctggtcttga catccccctg accgctcttt aatcggagct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcat tttggatggg cactctagag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gatcagggct acacacgtgc tacaatggcg taaacaaagg gaagcgagcc cgcgaggggg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgcaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 55
<211> 1242
<212> ДНК
<213> Ruminococcus gnavus
<220>
<221> прочий_признак
<222> (1)..(1)
<223> n представляет собой a, c, g или t
<400> 55
ncgacgatca gtagccgacc tgagagggtg accggccaca ttgggactga gacacggccc 60
aaactcctac gggaggcagc agtggggaat attgcacaat gggggaaacc ctgatgcagc 120
gacgccgcgt gagcgatgaa gtatttcggt atgtaaagct ctatcagcag ggaagaaaat 180
gacggtacct gactaagaag ccccggctaa ctacgtgcca gcagccgcgg taatacgtag 240
ggggcaagcg ttatccggat ttactgggtg taaagggagc gtagacggca tggcaagcca 300
gatgtgaaag cccggggctc aaccccggga ctgcatttgg aactgtcagg ctagagtgtc 360
ggagaggaaa gcggaattcc tagtgtagcg gtgaaatgcg tagatattag gaggaacacc 420
agtggcgaag gcggctttct ggacgatgac tgacgttgag gctcgaaagc gtggggagca 480
aacaggatta gataccctgg tagtccacgc cgtaaacgat gaatactagg tgtcgggtgg 540
caaagccatt cggtgccgca gcaaacgcaa taagtattcc acctggggag tacgttcgca 600
agaatgaaac tcaaaggaat tgacggggac ccgcacaagc ggtggagcat gtggtttaat 660
tcgaagcaac gcgaagaacc ttacctggtc ttgacatccc tctgaccgct ctttaatcgg 720
aactttcctt cgggacagag gagacaggtg gtgcatggtt gtcgtcagct cgtgtcgtga 780
gatgttgggt taagtcccgc aacgagcgca acccctatct ttagtagcca gcattttgga 840
tgggcactct agagagactg ccagggataa cctggaggaa ggtggggatg acgtcaaatc 900
atcatgcccc ttatgaccag ggctacacac gtgctacaat ggcgtaaaca aagggaagcg 960
agcccgcgag ggggagcaaa tcccaaaaat aacgtctcag ttcggattgt agtctgcaac 1020
tcgactacat gaagctggaa tcgctagtaa tcgcgaatca gaatgtcgcg gtgaatacgt 1080
tcccgggtct tgtacacacc gcccgtcaca ccatgggagt cagtaacgcc cgaagtcagt 1140
gacccaaccg caaggaggga gctgccgaag gtgggaccga taactggggt gaagtcgtaa 1200
caaggtagcc gtatcggaag gtgcggctgg atcacctcct tt 1242
<210> 56
<211> 1528
<212> ДНК
<213> Ruminococcus gnavus
<400> 56
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagca ccttgacgga tttcttcgga ttgaagcctt ggtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttggaa acggctgcta 180
ataccgcata agcgcacagt accgcatggt acggtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattaggt agttggtggg gtaacggcct accaagccga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gatgaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagccagatg tgaaagcccg 600
gggctcaacc ccgggactgc atttggaact gtcaggctag agtgtcggag aggaaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
ctttctggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctggtcttga catccctctg accgctcttt aatcggagct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcat ttcggatggg cactctagag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gaccagggct acacacgtgc tacaatggcg taaacaaagg gaagcgagcc cgcgaggggg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgcaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 57
<211> 1528
<212> ДНК
<213> Ruminococcus gnavus
<400> 57
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcttaaca catgcaagtc 60
gagcgaagcg cttttgcgga tttcttcgga ttgaagcaac tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttggaa acggctgcta 180
ataccgcata agcgcacagt accgcatggt accgtgtgaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtggg gtaacggcct accaaggcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gatgaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcccc ggctaactac gtgccagcag ccgcggtaat acgtaggggg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggcatggc aagccagatg tgaaagcccg 600
gggctcaacc ccgggactgc atttggaact gtcaggctag agtgtcggag aggtaagcgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac gatgactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgaat actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tattccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctggtcttga catccctatg accgctcttt aatcggagct ttccttcggg 1020
acagaggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcat tttggatggg cactctagag 1140
agactgccag ggataacctg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gaccagggct acacacgtgc tacaatggcg taaacaaagg gaagcgaacc tgcgaggggg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgtaag 1440
gagggagctg ccgaaggtgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 58
<211> 1529
<212> ДНК
<213> Ruminococcus torques
<400> 58
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgaagca ctttgcttag attcttcgga tgaagaggat tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agaccacagc accgcatggt gcgggggtaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtaag gtaacggctt accaaggcga cgatcagtag 300
ccgacctgag agggtggccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcacc ggctaaatac gtgccagcag ccgcggtaat acgtatggtg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggatgggc aagtctgatg tgaaaacccg 600
gggctcaacc ccgggactgc attggaaact gttcatctag agtgctggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac agtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catcccgctg accggacggt aatgcgtcct tcccttcggg 1020
gcagcggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcgg ccaggccggg cactctagag 1140
agactgccgg ggataacccg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcgagac cgcgaggtgg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgtaag 1440
gagggagctg ccgaaggcgg gaccgataac tgggggtgaa gtcgtaacaa ggtagccgta 1500
tcggaaggtg cggctggatc acctccttt 1529
<210> 59
<211> 1529
<212> ДНК
<213> Ruminococcus torques
<400> 59
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgaagca ctttgcttag attcttcgga tgaagaggat tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agaccacagc accgcatggt gcgggggtaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtaag gtaacggctt accaaggcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcacc ggctaaatac gtgccagcag ccgcggtaat acgtatggtg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggatgggc aagtctgatg tgaaaacccg 600
gggctcaacc ccgggactgc attggaaact gttcatctag agtgctggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac agtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catcccgctg accggacggt aatgcgtcct tcccttcggg 1020
gcagcggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tccccgcaac gagcgcaacc cctatcttta gtagccagcg gccaggccgg gcactctaga 1140
gagactgccg gggataaccc ggaggaaggt ggggatgacg tcaaatcatc atgcccctta 1200
tgagcagggc tacacacgtg ctacaatggc gtaaacaaag ggaagcgaga ccgcgaggtg 1260
gagcaaatcc caaaaataac gtctcagttc ggattgtagt ctgcaactcg actacatgaa 1320
gctggaatcg ctagtaatcg cgaatcagaa tgtcgcggtg aatacgttcc cgggtcttgt 1380
acacaccgcc cgtcacacca tgggagtcag taacgcccga agtcagtgac ccaaccgtaa 1440
ggagggagct gccgaaggcg ggaccgataa ctggggtgaa gtcgtaacaa ggtagccgta 1500
tcggaaggtg cggctggatc acctccttt 1529
<210> 60
<211> 1056
<212> ДНК
<213> Ruminococcus torques
<400> 60
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgaagca ctttgcttag attcttcgga tgaagaggat tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agaccacagc accgcatggt gcgggggtaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtaag gtaacggctt accaaggcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcacc ggctaaatac gtgccagcag ccgcggtaat acgtatggtg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggatgggc aagtctgatg tgaaaacccg 600
gggctcaacc ccgggactgc attggaaact gttcatctag agtgctggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac agtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg ggagcatgtg gtttaattcg aagcaacgcg 960
aagaacctta cctgctcttg acatcccgct gaccggacgg taatgcgtcc ttcccttcgg 1020
gcagcggaga caggtggtgc atggttgtcg tcagct 1056
<210> 61
<211> 1529
<212> ДНК
<213> Ruminococcus torques
<400> 61
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgaagca ctttgcttag attcttcgga tgaagaggat tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agaccacagc accgcatggt gcgggggtaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtaag gtaacggctt accaaggcga cgatcagtag 300
ccgacctgag agggtggccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcacc ggctaaatac gtgccagcag ccgcggtaat acgtatggtg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggatgggc aagtctgatg tgaaaacccg 600
gggctcaacc ccgggactgc attggaaact gttcatctag agtgctggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac agtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catcccgctg accggacggt aatgcgtcct tcccttcggg 1020
gcagcggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcgg ccaggccggg cactctagag 1140
agactgccgg ggataacccg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcgagac cgcgaggtgg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgtaag 1440
gagggagctg ccgaaggcgg gaccgataac tgggggtgaa gtcgtaacaa ggtagccgta 1500
tcggaaggtg cggctggatc acctccttt 1529
<210> 62
<211> 1177
<212> ДНК
<213> Ruminococcus torques
<400> 62
aacgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaaca catgcaagtc 60
gagcgaagca ctttgcttag attcttcgga tgaagaggat tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agaccacagc accgcatggt gcgggggtaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct ggttggtaag gtaacggctt accaaggcga cgatcagtag 300
ccgacctgag agggtgaccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcacc ggctaaatac gtgccagcag ccgcggtaat acgtatggtg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggatgggc aagtctgatg tgaaaacccg 600
gggctcaacc ccgggactgc attggaaact gttcatctag agtgctggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac agtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tagtccacct gggggagtac gttcgcaaga atgaaactca 900
aaggaattga cgggggaccc gcacaagcgg tggagcatgt ggtttaattc gaagcaacgc 960
gaagaacctt acctgctctt gacatcccgc tgaccggacg gtaatgcgtc cttcccttcg 1020
gggcagcgga gacaggtggt gcatggttgt cgtcagctcg tgtcgtgaga tgttgggtta 1080
agtcccgcac gagcgcaacc ctatctttag tagcagcggc caggccgggc actctaaaag 1140
agactgccgg ggataatccc gggaggaagg tggggat 1177
<210> 63
<211> 1528
<212> ДНК
<213> Ruminococcus torques
<400> 63
tacgagagtt tgatcctggc tcaggatgaa cgcgggcggc gtgcctaaca catgcaagtc 60
gagcgaagca ctttgcttag attcttcgga tgaagaggat tgtgactgag cggcggacgg 120
gtgagtaacg cgtgggtaac ctgcctcata cagggggata acagttagaa atgactgcta 180
ataccgcata agaccacagc accgcatggt gcgggggtaa aaactccggt ggtatgagat 240
ggacccgcgt ctgattagct agttggtaag gtaacggctt accaaggcga cgatcagtag 300
ccgacctgag agggtggccg gccacattgg gactgagaca cggcccaaac tcctacggga 360
ggcagcagtg gggaatattg cacaatgggg gaaaccctga tgcagcgacg ccgcgtgagc 420
gaagaagtat ttcggtatgt aaagctctat cagcagggaa gaaaatgacg gtacctgact 480
aagaagcacc ggctaaatac gtgccagcag ccgcggtaat acgtatggtg caagcgttat 540
ccggatttac tgggtgtaaa gggagcgtag acggatgggc aagtctgatg tgaaaacccg 600
gggctcaacc ccgggactgc attggaaact gttcatctag agtgctggag aggtaagtgg 660
aattcctagt gtagcggtga aatgcgtaga tattaggagg aacaccagtg gcgaaggcgg 720
cttactggac agtaactgac gttgaggctc gaaagcgtgg ggagcaaaca ggattagata 780
ccctggtagt ccacgccgta aacgatgact actaggtgtc gggtggcaaa gccattcggt 840
gccgcagcaa acgcaataag tagtccacct ggggagtacg ttcgcaagaa tgaaactcaa 900
aggaattgac ggggacccgc acaagcggtg gagcatgtgg tttaattcga agcaacgcga 960
agaaccttac ctgctcttga catcccgctg accggacggt aatgcgtcct tcccttcggg 1020
gcagcggaga caggtggtgc atggttgtcg tcagctcgtg tcgtgagatg ttgggttaag 1080
tcccgcaacg agcgcaaccc ctatctttag tagccagcgg ccaggccggg cactctagag 1140
agactgccgg ggataacccg gaggaaggtg gggatgacgt caaatcatca tgccccttat 1200
gagcagggct acacacgtgc tacaatggcg taaacaaagg gaagcgagac cgcgaggtgg 1260
agcaaatccc aaaaataacg tctcagttcg gattgtagtc tgcaactcga ctacatgaag 1320
ctggaatcgc tagtaatcgc gaatcagaat gtcgcggtga atacgttccc gggtcttgta 1380
cacaccgccc gtcacaccat gggagtcagt aacgcccgaa gtcagtgacc caaccgtaag 1440
gagggagctg ccgaaggcgg gaccgataac tggggtgaag tcgtaacaag gtagccgtat 1500
cggaaggtgc ggctggatca cctccttt 1528
<210> 64
<211> 1342
<212> ДНК
<213> Staphylococcus aureus
<400> 64
aagacggtct tgctgtcact tatagatgga tccgcgctgc attagctagt tggtaaggta 60
acggcttacc aaggcaacga tgcatagccg acctgagagg gtgatcggcc acactggaac 120
tgagacacgg tccagactcc tacgggaggc agcagtaggg aatcttccgc aatgggcgaa 180
agcctgacgg agcaacgccg cgtgagtgat gaaggtcttc ggatcgtaaa actctgttat 240
tagggaagaa catatgtgta agtaactgtg cacatcttga cggtacctaa tcagaaagcc 300
acggctaact acgtgccagc agccgcggta atacgtaggt ggcaagcgtt atccggaatt 360
attgggcgta aagcgcgcgt aggcggtttt ttaagtctga tgtgaaagcc cacggctcaa 420
ccgtggaggg tcattggaaa ctggaaaact tgagtgcaga agaggaaagt ggaattccat 480
gtgtagcggt gaaatgcgca gagatatgga ggaacaccag tggcgaaggc gactttctgg 540
tctgtaactg acgctgatgt gcgaaagcgt ggggatcaaa caggattaga taccctggta 600
gtccacgccg taaacgatga gtgctaagtg ttagggggtt tccgcccctt agtgctgcag 660
ctaacgcatt aagcactccg cctggggagt acgaccgcaa ggttgaaact caaaggaatt 720
gacggggacc cgcacaagcg gtggagcatg tggtttaatt cgaagcaacg cgaagaacct 780
taccaaatct tgacatcctt tgacaactct agagatagag ccttcccctt cgggggacaa 840
agtgacaggt ggtgcatggt tgtcgtcagc tcgtgtcgtg agatgttggg ttaagtcccg 900
caacgagcgc aacccttaag cttagttgcc atcattaagt tgggcactct aagttgactg 960
ccggtgacaa accggaggaa ggtggggatg acgtcaaatc atcatgcccc ttatgatttg 1020
ggctacacac gtgctacaat ggacaataca aagggcagcg aaaccgcgag gtcaagcaaa 1080
tcccataaag ttgttctcag ttcggattgt agtctgcaac tcgactacat gaagctggaa 1140
tcgctagtaa tcgtagatca gcatgctacg gtgaatacgt tcccgggtct tgtacacacc 1200
gcccgtcaca ccacgagagt ttgtaacacc cgaagccggt ggagtaacct tttaggagct 1260
agccgtcgaa ggtgggacaa atgattgggg tgaagtcgta acaaggtagc cgtatcggaa 1320
ggtgcggctg gatcacctcc tt 1342
<210> 65
<211> 1561
<212> ДНК
<213> Veillonella parvula
<400> 65
ttggagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgaagag cgatggaagc ttgcttctat caatcttagt ggcgaacggg tgagtaacgc 120
gtaatcaacc tgcccttcag agggggacaa cagttggaaa cgactgctaa taccgcatac 180
gatctaatct cggcatcgag gatagatgaa aggtggcctc tacatgtaag ctatcactga 240
aggaggggat tgcgtctgat tagctagttg gaggggtaac ggcccaccaa ggcgatgatc 300
agtagccggt ctgagaggat gaacggccac attgggactg agacacggcc cagactccta 360
cgggaggcag cagtggggaa tcttccgcaa tggacgaaag tctgacggag caacgccgcg 420
tgagtgatga cggccttcgg gttgtaaagc tctgttaatc gggacgaaag gccttcttgc 480
gaacagttag aaggattgac ggtaccggaa tagaaagcca cggctaacta cgtgccagca 540
gccgcggtaa tacgtaggtg gcaagcgttg tccggaatta ttgggcgtaa agcgcgcgca 600
ggcggatcag tcagtctgtc ttaaaagttc ggggcttaac cccgtgatgg gatggaaact 660
gctgatctag agtatcggag aggaaagtgg aattcctagt gtagcggtga aatgcgtaga 720
tattaggaag aacaccagtg gcgaaggcga ctttctggac gaaaactgac gctgaggcgc 780
gaaagccagg ggagcgaacg ggattagata ccccggtagt cctggccgta aacgatgggt 840
actaggtgta ggaggtatcg accccttctg tgccggagtt aacgcaataa gtaccccgcc 900
tggggagtac gaccgcaagg ttgaaactca aaggaattga cgggggcccg cacaagcggt 960
ggagtatgtg gtttaattcg acgcaacgcg aagaacctta ccaggtcttg acattgatgg 1020
acagaactag agatagttcc tcttcttcgg aagccagaaa acaggtggtg cacggttgtc 1080
gtcagctcgt gtcgtgagat gttgggttaa gtcccgcaac gagcgcaacc cctatcttat 1140
gttgccagca cgtaatggtg ggaactcatg agagactgcc gcagacaatg cggaggaagg 1200
cggggatgac gtcaaatcat catgcccctt atgacctggg ctacacacgt actacaatgg 1260
gagttaatag acggaagcga gatcgcgaga tggagcaaac ccgagaaaca ctctctcagt 1320
tcggatcgta ggctgcaact cgcctacgtg aagtcggaat cgctagtaat cgcaggtcag 1380
catactgcgg tgaatacgtt cccgggcctt gtacacaccg cccgtcacac cacgaaagtc 1440
ggaagtgccc aaagccggtg gggtaacctt cgggagccag ccgtctaagg taaagtcgat 1500
gattggggtg aagtcgtaac aaggtagccg tatcggaagg tgcggctgga tcacctcctt 1560
t 1561
<210> 66
<211> 1359
<212> ДНК
<213> Clostridium ghonni
<400> 66
tcgagcgaac ccttcggggt gagcggcgga cgggtgagta acgcgtgggt aacctgccct 60
gtacacacgg ataacatacc gaaaggtatg ctaatacggg ataacatatg agagtcgcat 120
ggcttttgta tcaaagctcc ggcggtacag gatggacccg cgtctgatta gctagttggt 180
aaggtaacgg cttaccaagg caacgatcag tagccgacct gagagggtga tcggccacat 240
tggaactgag acacggtcca aactcctacg ggaggcagca gtggggaata ttgcacaatg 300
ggcgaaagcc tgatgcagca acgccgcgtg agcgatgaag gccttcgggt cgtaaagctc 360
tgtcctcaag gaagataatg acggtacttg aggaggaagc cccggctaac tacgtgccag 420
cagccgcggt aatacgtagg gggctagcgt tatccggaat tactgggcgt aaagggtgcg 480
taggcggtct ttcaagccag aagtgaaagg ctacggctca accgtagtaa gcttttggaa 540
ctgtaggact tgagtgcagg agaggagagt ggaattccta gtgtagcggt gaaatgcgta 600
gatattagga ggaacaccag tagcgaaggc ggctctctgg actgtaactg acgctgaggc 660
acgaaagcgt ggggagcaaa caggattaga taccctggta gtccacgccg taaacgatga 720
gtactaggtg tcgggggtta cccccctcgg tgccgcagct aacgcattaa gtactccgcc 780
tgggaagtac gctcgcaaga gtgaaactca aaggaattga cggggacccg cacaagtagc 840
ggagcatgtg gtttaattcg aagcaacgcg aagaacctta cctaagcttg acatcccatt 900
gacctctccc taatcggaga tttcccttcg gggacagtgg tgacaggtgg tgcatggctg 960
tcgtcagctc gtgtcgtgag atgttggggt aagtcccgca acgagcgcaa cccttgcctt 1020
tagttgccag cattaagttg ggcactctag agggactgcc gaggataact cggaggaagg 1080
tggggatgac gtcaaatcat catgcccctt atgcttaggg ctacacacgt gctacaatgg 1140
gtggtacaga gggttgccaa gccgtgaggt ggagctaatc ccttaaagcc attctcagtt 1200
cggattgtag gctgaaactc gcctacatga agctggagtt actagtaatc gcagatcaga 1260
atgctgcggt gaatgcgttc ccgggtcttg tacacaccgc ccgtcacacc atgggagttg 1320
ggggcgcccg aagccggtta gctaaccttt taggaagcg 1359
<210> 67
<211> 1381
<212> ДНК
<213> Bacteroides ovatus
<220>
<221> прочий_признак
<222> (25)..(25)
<223> N представляет собой A, C, T, G, неизвестное или другое
<400> 67
tcgaggggca gcattttagt ttgcnttgca aactaaagat ggcgaccggc gcacgggtga 60
gtaacacgta tccaacctgc cgataactcg gggatagcct ttcgaaagaa agattaatat 120
ccgatagtat attaaaaccg catggtttta ctattaaaga atttcggtta tcgatgggga 180
tgcgttccat tagtttgttg gcggggtaac ggcccaccaa gactacgatg gataggggtt 240
ctgagaggaa ggtcccccac attggaactg agacacggtc caaactccta cgggaggcag 300
cagtgaggaa tattggtcaa tggacgagag tctgaaccag ccaagtagcg tgaaggatga 360
ctgccctatg ggttgtaaac ttcttttata tgggaataaa gtattccacg tgtggaattt 420
tgtatgtacc atatgaataa ggatcggcta actccgtgcc agcagccgcg gtaatacgga 480
gccgagcgtt atccggattt attgggttta aagggagcgt aggtggattg ttaagtcagt 540
tgtgaaagtt tgcggctcaa ccgtaaaatt gcagttgaaa ctggcagtct tgagtacagt 600
agaggtgggc ggaattcgtg gtgtagcggt gaaatgctta gatatcacga agaactccga 660
ttgcgaaggc agctcactag actgcaactg acactgatgc tcgaaagtgt gggtatcaaa 720
caggattaga taccctggta gtccacacag taaacgatga atactcgctg tttgcgatat 780
acagtaagcg gccaagcgaa agcattaagt attccacctg gggagtacgc cggcaacggt 840
gaaactcaaa ggaattgacg ggggcccgca caagcggagg aacatgtggt ttaattcgat 900
gatacgcgag gaaccttacc cgggcttaaa ttgcatttga ataatctgga aacaggttag 960
ccgcaaggca aatgtgaatt ggctgcatgg ttgtcgtcag ctcgtgccgt gaggtgtcgg 1020
cttaagtgcc ataacgagcg caacccttat ctttagttac taacaggtta tgctgaggac 1080
tctagagaga ctgccgtcgt aagatgtgag gaaggtgggg atgacgtcaa atcagcacgg 1140
cccttacgtc cggggctaca cacgtgttac aatggggggt acagaaggca gctacctggt 1200
gacaggatgc taatcccaaa aacctctctc agttcggatc gaagtctgca acccgacttc 1260
gtgaagctgg attcgctagt aatcgcgcat cagccatggc gcggtgaata cgttcccggg 1320
ccttgtacac accgcccgtc aagccatgaa agccgggggt acctgaagta cgtaaccgca 1380
a 1381
<210> 68
<211> 1261
<212> ДНК
<213> Bacteroides stercoris
<400> 68
atggtatagt tttcccgcat gggataatta ttaaagaatt tcggttgtcg atggggatgc 60
gttccattag gcagttggcg gggtaacggc ccaccaaacc tacgatggat aggggttctg 120
agaggaaggt cccccacatt ggaactgaga cacggtccaa actcatacgg gaggcagcag 180
tgaggaatat tggtcaatgg acgagagtct gaaccagcca agtagcgtga aggatgactg 240
ccctatgggt tgtaaacttc ttttatacgg gaataaagtt agccacgtgt ggttttttgt 300
atgtaccgta tgaataagga tcggctaact ccgtgccagc agccgcggta atacggagga 360
tccgagcgtt atccggattt attgggttta aagggagcgt aggcgggttg ttaagtcagt 420
tgtgaaagtt tgcggctcaa ccgtaaaatt gcagttgata ctggcgacct tgagtgcaac 480
agaggtaggc ggaattcgtg gtgtagcggt gaaatgctta gatatcacga agaactccga 540
ttgcgaaggc agcttactgg attgtaactg acgctgatgc tcgaaagtgt gggtatcaaa 600
caggattaga taccctggta gtccacacag taaacgatga atactcgctg ttggcgatat 660
acggtcagcg gccaagcgaa agcattaagt attccacctg gggagtacgc cggcagcggt 720
gaaactcaaa ggaattgacg ggggcccgca caagcggagg aacatgtggt ttaattcgat 780
gatacgcgag gaaccttacc cgggcttaaa ttgcaactga cggaatcgga aacggttctt 840
tcttcggaca gttgtgaagg tgctgcatgg ttgtcgtcag ctcgtgccgt gaggtgtcgg 900
cttaagtgcc ataacgagcg caacccttac gggtagttac catcaggtta tgctggggac 960
tctacccgga ctgccgtcgt aagatgtgag gaaggtgggg atgacgtcaa atcagcacgg 1020
cccttacgtc cggggctaca cacgtgttac aatggggggt acagaaggca gctacacggc 1080
gacgtggtgc taatcccaaa agcctctctc agttcggatt ggagtctgca acccgactcc 1140
atgaagctgg attcgctagt aatcgcgcat cagccacggc gcggtgaata cgttcccggg 1200
ccttgtacac accgcccgtc aagccatgaa agccgggggt acctgaagta cgtaaccgcg 1260
a 1261
<210> 69
<211> 999
<212> ДНК
<213> Eggerthella lenta
<220>
<221> прочий_признак
<222> (33)..(36)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (57)..(57)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (119)..(119)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (788)..(788)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (848)..(848)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (860)..(860)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (877)..(877)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (877)..(877)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (887)..(887)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (891)..(891)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (899)..(899)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (933)..(944)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (961)..(961)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (963)..(963)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (978)..(978)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (980)..(980)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (983)..(983)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<400> 69
cctcgggcgg acatgaagtg gcgaacgggt gannnncacg tgaccaacct gcccccngct 60
ccgggacaac cttgggaaac cgaggctaat accggatact ccctcccctg ctcctgggng 120
ggtcgggaaa gcccaggcgg agggggatgg ggtcgcggcc cattaggtag taggcggggt 180
aacggcccac ctagcccgcg atgggtagcc gggttgagag accgaccggc cacattggga 240
ctgagatacg gcccagactc ctacgggagg cagcagtggg gaattttgcg caatggggga 300
aaccctgacg cagcaacgcc gcgtgcggga cgacggcctt cgggttgtaa accgctttca 360
gcagggaaga aattcgacgg tacctgcaga agaagctccg gctaactacg tgccagcagc 420
cgcggtaata cgtagggagc gagcgttatc cggattcatt gggcgtaaag agcgcgtagg 480
cggcctctca agcgggatct ctaatccgag ggctcaaccc ccggccggat cccgaactgg 540
gaggctcgag ttcggtagag gcaggcggaa ttcccggtgt agcggtggaa tgcgcagata 600
tcgggaagaa caccgatggc gaaggcagcc tgctgggccg caactgacgc tgaggcgcga 660
aagctagggg agcgaacagg attagatacc ctggtagtcc tagccgtaaa cgatggatac 720
taggtgtggg gggctccgcc ctccgtgccg cagccaacgc attaagtatc ccgcctgggg 780
agtacggncg caaggctaaa actcaaggaa ttgacggggg cccgcacaag cagcggagca 840
tgtggctnaa ttcgaagcan cgcgaagaac cttaccnggg cttgacntgg nacgtgaanc 900
cggggaaacc cggtggccga gaggagcgtc cgnnagtggt gcatggctgt cgtcagctcg 960
ngncgtgaga tgttgggntn agntcccgca acgagcgca 999
<210> 70
<211> 753
<212> ДНК
<213> Eggerthella sp. YY7918
<220>
<221> прочий_признак
<222> (122)..(122)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (197)..(197)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (341)..(341)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (431)..(431)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (508)..(508)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (602)..(602)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (620)..(621)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (623)..(623)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (627)..(627)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (670)..(670)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (676)..(676)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (709)..(709)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (739)..(739)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<400> 70
ctacccttct cttcgggaca accttgggaa accgaggcta ataccgaata ctccggtgag 60
tgcgcatgca cacgccggga aagctttcgc ggagaaggat ggggtcgcgg cccatcaggt 120
anacggcggg gtagcggccc accgtgcttt tgacgggtag ccgggttgag agaccgaccg 180
gccacattgg gactganata cggcccagac tcctacggga ggcagcagtg gggaattttg 240
cgcaatgggg gaaaccctga cgcagcaccg ccgcgtgcgg gatgaaggcc ttcgggtcgt 300
aaaccgcttt cagcagggaa gaaattcgac ggtacctgca naagaagccc cggctaacta 360
cgtgccagca gccgcggtaa tacgtagggg gcgagcgtta tccggattca ttgggcgtaa 420
agcgcgcgta ngcggccgcc taagcgggac ctctaacctt ggggctcaac ctcaagccgg 480
gttccgaact gggtggctcg agtttggnag aggaagatgg aattcccggt gtagcggtgg 540
aatgcgcaga tatcgggaag aacaccgatg gcgaaggcag tcttctgggc tcataactga 600
cnctgaagcg cgaaagctan ngnagcnaac aggattagat accctggtag tcctagccgt 660
aaacgatggn cactangtgt ggggagatac tctttccgtg ccgaagcana cgcattaagt 720
gccccgcctg gggagtacng ccgcaaggct aaa 753
<210> 71
<211> 1346
<212> ДНК
<213> Eggerthella sp. 1_3_56FAA
<220>
<221> прочий_признак
<222> (123)..(123)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<220>
<221> прочий_признак
<222> (1329)..(1329)
<223> N представляют собой каждый независимо A, C, T, G, неизвестное или другое
<400> 71
accgccctcg ggcggacatg aagtggcgaa cgggtgagta acacgtgacc aacctgcccc 60
cctctccggg acaaccttgg gaaaccgagg ctaataccgg atactccctc ccctgctcct 120
ggnggggtcg ggaaagccca ggcggagggg gatggggtcg cggcccatta ggtagtaggc 180
ggggtaacgg cccacctagc ccgcgatggg tagccgggtt gagagaccga ccggccacat 240
tgggactgag atacggccca gactcctacg ggaggcagca gtggggaatt ttgcgcaatg 300
ggggaaaccc tgacgcagca acgccgcgtg cgggacgacg gccttcgggt tgtaaaccgc 360
tttcagcagg gaagaaattc aggcctgcag aagaagctcc ggctaactac gtgccagcag 420
ccgcggtaat acgtagggag cgagcgttat ccggattcat tgggcgtaaa gagcgcgtag 480
gcggcctctc aagcgggatc tctaatccga gggctcaacc cccggccgga tcccgaactg 540
ggaggctcga gttcggtaga ggcaggcgga attcccggtg tagcggtgga atgcgcagat 600
atcgggaaga acaccgatgg cgaaggcagc ctgctgggcc gcaactgacg ctgaggcgcg 660
aaagctaggg gagcgaacag gattagatac cctggtagtc ctagccgtaa acgatggata 720
ctaggtgtgg ggggctccgc cctccgtgcc gcagccaacg cattaagtat cccgcctggg 780
gagtacggcc gcaaggctaa aactcaaagg aattgacggg ggcccgcaca agcagcggag 840
catgtggctt agttcgaagc aacgcgaaga acctcaccag gcctgatccc gaactggaag 900
ccggggaaac ccggtggccg agaggaacgt ccgcaggtgg tgcatggctg tcgtcagctc 960
gtgtcgtgac atgttgggtt aagtcccgca acgagcgcaa cccctgcccc atgttgccag 1020
cattaggttg gggactcatg ggggactgcc ggcgtcaagc cggaggaagg tggggacgac 1080
gtcaagtcat catgcccttt atgccctggg ctgcacacgt gctacaatgg ccggtacaac 1140
gggctgcgag accgcgaggt cgagcgaatc cctcaaagcc ggccccagtt cggatcggag 1200
gctgcaaccc gcctccgtga agtcggagtt gctagtaatc gcggatcagc atgccgcggt 1260
gaatacgttc ccgggccttg tacacaccgc ccgtcacacc acccgagtcg tctgcacccg 1320
aagccgccnt ccgaacccgc aagggg 1346
<210> 72
<211> 1430
<212> ДНК
<213> Eggerthella sp. HGA1
<400> 72
gaacgctggc ggcgtgccta acacatgcaa gtcgaacgat gaaaccgccc tcgggcggac 60
atgaagtggc gaacgggtga gtaacacgtg accaacctgc ccccctctcc gggacaacct 120
tgggaaaccg aggctaatac cggatactcc ctcccctgct cctgcagggg tcgggaaagc 180
ccaggcggag ggggatgggg tcgcggccca ttaggtagta ggcggggtaa cggcccacct 240
agcccgcgat gggtagccgg gttgagagac cgaccggcca cattgggact gagatacggc 300
ccagactcct acgggaggca gcagtgggga attttgcgca atgggggcaa ccctgacgca 360
gcaacgccgc gtgcgggacg acggccttcg ggttgtaaac cgctttcagc agggaagaaa 420
ttcgacggta cctgcagaag aagctccggc taactacgtg ccagcagccg cggtaatacg 480
tagggagcga gcgttatccg gattcattgg gcgtaaagag cgcgtaggcg gcctctcaag 540
cgggatctct aatccgaggg ctcaaccccc ggccggatcc cgaactggga ggctcgagtt 600
cggtagaggc aggcggaatt cccggtgtag cggtggaatg cgcagatatc gggaagaaca 660
ccgatggcga aggcagcctg ctgggccgca actgacgctg aggcgcgaaa gctaggggag 720
cgaacaggat tagataccct ggtagtccta gccgtaaacg atggatacta ggtgtggggg 780
gctccgccct ccgtgccgca gccaacgcat taagtatccc gcctggggag tacggccgca 840
aggctaaaac tcaaaggaat tgacgggggc ccgcacaagc agcggagcat gtggcttaat 900
tcgaagcaac gcgaagaacc ttaccagggc ttgacatgga cgtgaagccg gggaaacccg 960
gtggccgaga ggagcgtccg caggtggtgc atggctgtcg tcagctcgtg tcgtgagatg 1020
ttgggttaag tcccgcaacg agcgcaaccc ctgccccatg ttgccagcat taggttgggg 1080
actcatgggg gactgccggc gtcaagccgg aggaaggtgg ggacgacgtc aagtcatcat 1140
gccctttatg ccctgggctg cacacgtgct acaatggccg gtacaacggg ctgcgagacc 1200
gcgaggtcga gcgaatccct caaagccggc cccagttcgg atcggaggct gcaacccgcc 1260
tccgtgaagt cggagttgct agtaatcgcg gatcagcatg ccgcggtgaa tacgttcccg 1320
ggccttgtac acaccgcccg tcacaccacc cgagtcgtct gcacccgaag ccgccggccg 1380
aacccgcaag gggcggaggc gtcgaaggtg tggagggtaa ggggggtgaa 1430
<210> 73
<211> 417
<212> Белок
<213> Clostridium sporogenes
<400> 73
Met Lys Phe Trp Arg Lys Tyr Thr Gln Gln Glu Met Asp Glu Lys Ile
1 5 10 15
Thr Glu Ser Leu Glu Lys Thr Leu Asn Tyr Asp Asn Thr Lys Thr Ile
20 25 30
Gly Ile Pro Gly Thr Lys Leu Asp Asp Thr Val Phe Tyr Asp Asp His
35 40 45
Ser Phe Val Lys His Ser Pro Tyr Leu Arg Thr Phe Ile Gln Asn Pro
50 55 60
Asn His Ile Gly Cys His Thr Tyr Asp Lys Ala Asp Ile Leu Phe Gly
65 70 75 80
Gly Thr Phe Asp Ile Glu Arg Glu Leu Ile Gln Leu Leu Ala Ile Asp
85 90 95
Val Leu Asn Gly Asn Asp Glu Glu Phe Asp Gly Tyr Val Thr Gln Gly
100 105 110
Gly Thr Glu Ala Asn Ile Gln Ala Met Trp Val Tyr Arg Asn Tyr Phe
115 120 125
Lys Lys Glu Arg Lys Ala Lys His Glu Glu Ile Ala Ile Ile Thr Ser
130 135 140
Ala Asp Thr His Tyr Ser Ala Tyr Lys Gly Ser Asp Leu Leu Asn Ile
145 150 155 160
Asp Ile Ile Lys Val Pro Val Asp Phe Tyr Ser Arg Lys Ile Gln Glu
165 170 175
Asn Thr Leu Asp Ser Ile Val Lys Glu Ala Lys Glu Ile Gly Lys Lys
180 185 190
Tyr Phe Ile Val Ile Ser Asn Met Gly Thr Thr Met Phe Gly Ser Val
195 200 205
Asp Asp Pro Asp Leu Tyr Ala Asn Ile Phe Asp Lys Tyr Asn Leu Glu
210 215 220
Tyr Lys Ile His Val Asp Gly Ala Phe Gly Gly Phe Ile Tyr Pro Ile
225 230 235 240
Asp Asn Lys Glu Cys Lys Thr Asp Phe Ser Asn Lys Asn Val Ser Ser
245 250 255
Ile Thr Leu Asp Gly His Lys Met Leu Gln Ala Pro Tyr Gly Thr Gly
260 265 270
Ile Phe Val Ser Arg Lys Asn Leu Ile His Asn Thr Leu Thr Lys Glu
275 280 285
Ala Thr Tyr Ile Glu Asn Leu Asp Val Thr Leu Ser Gly Ser Arg Ser
290 295 300
Gly Ser Asn Ala Val Ala Ile Trp Met Val Leu Ala Ser Tyr Gly Pro
305 310 315 320
Tyr Gly Trp Met Glu Lys Ile Asn Lys Leu Arg Asn Arg Thr Lys Trp
325 330 335
Leu Cys Lys Gln Leu Asn Asp Met Arg Ile Lys Tyr Tyr Lys Glu Asp
340 345 350
Ser Met Asn Ile Val Thr Ile Glu Glu Gln Tyr Val Asn Lys Glu Ile
355 360 365
Ala Glu Lys Tyr Phe Leu Val Pro Glu Val His Asn Pro Thr Asn Asn
370 375 380
Trp Tyr Lys Ile Val Val Met Glu His Val Glu Leu Asp Ile Leu Asn
385 390 395 400
Ser Leu Val Tyr Asp Leu Arg Lys Phe Asn Lys Glu His Leu Lys Ala
405 410 415
Met
<210> 74
<211> 490
<212> Белок
<213> Ruminococcus gnavus
<400> 74
Met Ser Gln Val Ile Lys Lys Lys Arg Asn Thr Phe Met Ile Gly Thr
1 5 10 15
Glu Tyr Ile Leu Asn Ser Thr Gln Leu Glu Glu Ala Ile Lys Ser Phe
20 25 30
Val His Asp Phe Cys Ala Glu Lys His Glu Ile His Asp Gln Pro Val
35 40 45
Val Val Glu Ala Lys Glu His Gln Glu Asp Lys Ile Lys Gln Ile Lys
50 55 60
Ile Pro Glu Lys Gly Arg Pro Val Asn Glu Val Val Ser Glu Met Met
65 70 75 80
Asn Glu Val Tyr Arg Tyr Arg Gly Asp Ala Asn His Pro Arg Phe Phe
85 90 95
Ser Phe Val Pro Gly Pro Ala Ser Ser Val Ser Trp Leu Gly Asp Ile
100 105 110
Met Thr Ser Ala Tyr Asn Ile His Ala Gly Gly Ser Lys Leu Ala Pro
115 120 125
Met Val Asn Cys Ile Glu Gln Glu Val Leu Lys Trp Leu Ala Lys Gln
130 135 140
Val Gly Phe Thr Glu Asn Pro Gly Gly Val Phe Val Ser Gly Gly Ser
145 150 155 160
Met Ala Asn Ile Thr Ala Leu Thr Ala Ala Arg Asp Asn Lys Leu Thr
165 170 175
Asp Ile Asn Leu His Leu Gly Thr Ala Tyr Ile Ser Asp Gln Thr His
180 185 190
Ser Ser Val Ala Lys Gly Leu Arg Ile Ile Gly Ile Thr Asp Ser Arg
195 200 205
Ile Arg Arg Ile Pro Thr Asn Ser His Phe Gln Met Asp Thr Thr Lys
210 215 220
Leu Glu Glu Ala Ile Glu Thr Asp Lys Lys Ser Gly Tyr Ile Pro Phe
225 230 235 240
Val Val Ile Gly Thr Ala Gly Thr Thr Asn Thr Gly Ser Ile Asp Pro
245 250 255
Leu Thr Glu Ile Ser Ala Leu Cys Lys Lys His Asp Met Trp Phe His
260 265 270
Ile Asp Gly Ala Tyr Gly Ala Ser Val Leu Leu Ser Pro Lys Tyr Lys
275 280 285
Ser Leu Leu Thr Gly Thr Gly Leu Ala Asp Ser Ile Ser Trp Asp Ala
290 295 300
His Lys Trp Leu Phe Gln Thr Tyr Gly Cys Ala Met Val Leu Val Lys
305 310 315 320
Asp Ile Arg Asn Leu Phe His Ser Phe His Val Asn Pro Glu Tyr Leu
325 330 335
Lys Asp Leu Glu Asn Asp Ile Asp Asn Val Asn Thr Trp Asp Ile Gly
340 345 350
Met Glu Leu Thr Arg Pro Ala Arg Gly Leu Lys Leu Trp Leu Thr Leu
355 360 365
Gln Val Leu Gly Ser Asp Leu Ile Gly Ser Ala Ile Glu His Gly Phe
370 375 380
Gln Leu Ala Val Trp Ala Glu Glu Ala Leu Asn Pro Lys Lys Asp Trp
385 390 395 400
Glu Ile Val Ser Pro Ala Gln Met Ala Met Ile Asn Phe Arg Tyr Ala
405 410 415
Pro Lys Asp Leu Thr Lys Glu Glu Gln Asp Ile Leu Asn Glu Lys Ile
420 425 430
Ser His Arg Ile Leu Glu Ser Gly Tyr Ala Ala Ile Phe Thr Thr Val
435 440 445
Leu Asn Gly Lys Thr Val Leu Arg Ile Cys Ala Ile His Pro Glu Ala
450 455 460
Thr Gln Glu Asp Met Gln His Thr Ile Asp Leu Leu Asp Gln Tyr Gly
465 470 475 480
Arg Glu Ile Tyr Thr Glu Met Lys Lys Ala
485 490
<210> 75
<211> 490
<212> Белок
<213> Ruminococcus gnavus
<400> 75
Met Ser Gln Val Ile Lys Lys Lys Arg Asn Thr Phe Met Ile Gly Thr
1 5 10 15
Glu Tyr Ile Leu Asn Ser Thr Gln Leu Glu Glu Ala Ile Lys Ser Phe
20 25 30
Val His Asp Phe Cys Ala Glu Lys His Glu Ile His Asp Gln Pro Val
35 40 45
Val Val Glu Ala Lys Glu His Gln Glu Asp Lys Ile Lys Gln Ile Lys
50 55 60
Ile Pro Glu Lys Gly Arg Pro Val Asn Glu Val Val Ser Glu Met Met
65 70 75 80
Asn Glu Val Tyr Arg Tyr Arg Gly Asp Ala Asn His Pro Arg Phe Phe
85 90 95
Ser Phe Val Pro Gly Pro Ala Ser Ser Val Ser Trp Leu Gly Asp Ile
100 105 110
Met Thr Ser Ala Tyr Asn Ile His Ala Gly Gly Ser Lys Leu Ala Pro
115 120 125
Met Val Asn Cys Ile Glu Gln Glu Val Leu Lys Trp Leu Ala Lys Gln
130 135 140
Val Gly Phe Thr Glu Asn Pro Gly Gly Val Phe Val Ser Gly Gly Ser
145 150 155 160
Met Ala Asn Ile Thr Ala Leu Thr Ala Ala Arg Asp Asn Lys Leu Thr
165 170 175
Asp Ile Asn Leu His Leu Gly Thr Ala Tyr Ile Ser Asp Gln Thr His
180 185 190
Ser Ser Val Ala Lys Gly Leu Arg Ile Ile Gly Ile Thr Asp Ser Arg
195 200 205
Ile Arg Arg Ile Pro Thr Asn Ser His Phe Gln Met Asp Thr Thr Lys
210 215 220
Leu Glu Glu Ala Ile Glu Thr Asp Lys Lys Ser Gly Tyr Ile Pro Phe
225 230 235 240
Val Val Ile Gly Thr Ala Gly Thr Thr Asn Thr Gly Ser Ile Asp Pro
245 250 255
Leu Thr Glu Ile Ser Ala Leu Cys Lys Lys His Asp Met Trp Phe His
260 265 270
Ile Asp Gly Ala Tyr Gly Ala Ser Val Leu Leu Ser Pro Lys Tyr Lys
275 280 285
Ser Leu Leu Thr Gly Thr Gly Leu Ala Asp Ser Ile Ser Trp Asp Ala
290 295 300
His Lys Trp Leu Phe Gln Thr Tyr Gly Cys Ala Met Val Leu Val Lys
305 310 315 320
Asp Ile Arg Asn Leu Phe His Ser Phe His Val Asn Pro Glu Tyr Leu
325 330 335
Lys Asp Leu Glu Asn Asp Ile Asp Asn Val Asn Thr Trp Asp Ile Gly
340 345 350
Met Glu Leu Thr Arg Pro Ala Arg Gly Leu Lys Leu Trp Leu Thr Leu
355 360 365
Gln Val Leu Gly Ser Asp Leu Ile Gly Ser Ala Ile Glu His Gly Phe
370 375 380
Gln Leu Ala Val Trp Ala Glu Glu Ala Leu Asn Pro Lys Lys Asp Trp
385 390 395 400
Glu Ile Val Ser Pro Ala Gln Met Ala Met Ile Asn Phe Arg Tyr Ala
405 410 415
Pro Lys Asp Leu Thr Lys Glu Glu Gln Asp Ile Leu Asn Glu Lys Ile
420 425 430
Ser His Arg Ile Leu Glu Ser Gly Tyr Ala Ala Ile Phe Thr Thr Val
435 440 445
Leu Asn Gly Lys Thr Val Leu Arg Ile Cys Ala Ile His Pro Glu Ala
450 455 460
Thr Gln Glu Asp Met Gln His Thr Ile Asp Leu Leu Asp Gln Tyr Gly
465 470 475 480
Arg Glu Ile Tyr Thr Glu Met Lys Lys Ala
485 490
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ИНСЕКТИЦИДНЫЕ БЕЛКИ | 2017 |
|
RU2765304C2 |
| Штамм Akkermansia muciniphila и его применение | 2019 |
|
RU2792651C2 |
| ВАКЦИНА ПРОТИВ НЕКРОТИЧЕСКОГО ЭНТЕРИТА У ДОМАШНЕЙ ПТИЦЫ | 2018 |
|
RU2788096C2 |
| ШТАММЫ И СПОСОБЫ ДЛЯ ПОЛУЧЕНИЯ БЕЛКА ОДНОКЛЕТОЧНЫХ ОРГАНИЗМОВ ИЛИ БИОМАССЫ | 2020 |
|
RU2806550C1 |
| КОМПЛЕКС, ПРЕПАРАТ И ПРИМЕНЕНИЕ БОТУЛОТОКСИНА ТИПА А CLOSTRIDIUM BOTULINUM | 2020 |
|
RU2783511C1 |
| СПОСОБЫ ПРОИЗВОДСТВА ПРОТЕОЛИТИЧЕСКИ ПРОЦЕССИРОВАННЫХ ПОЛИПЕПТИДОВ | 2019 |
|
RU2727402C1 |
| СПОСОБЫ ПРОИЗВОДСТВА ПРОТЕОЛИТИЧЕСКИ ПРОЦЕССИРОВАННЫХ ПОЛИПЕПТИДОВ | 2018 |
|
RU2719164C1 |
| АНТИТЕЛО ПРОТИВ СТОЛБНЯЧНОГО ТОКСИНА И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2815280C1 |
| ГИБРИДНЫЕ НЕЙРОТОКСИНЫ | 2017 |
|
RU2782382C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ БОРЬБЫ С ФУЗАРИОЗОМ | 2020 |
|
RU2777606C2 |
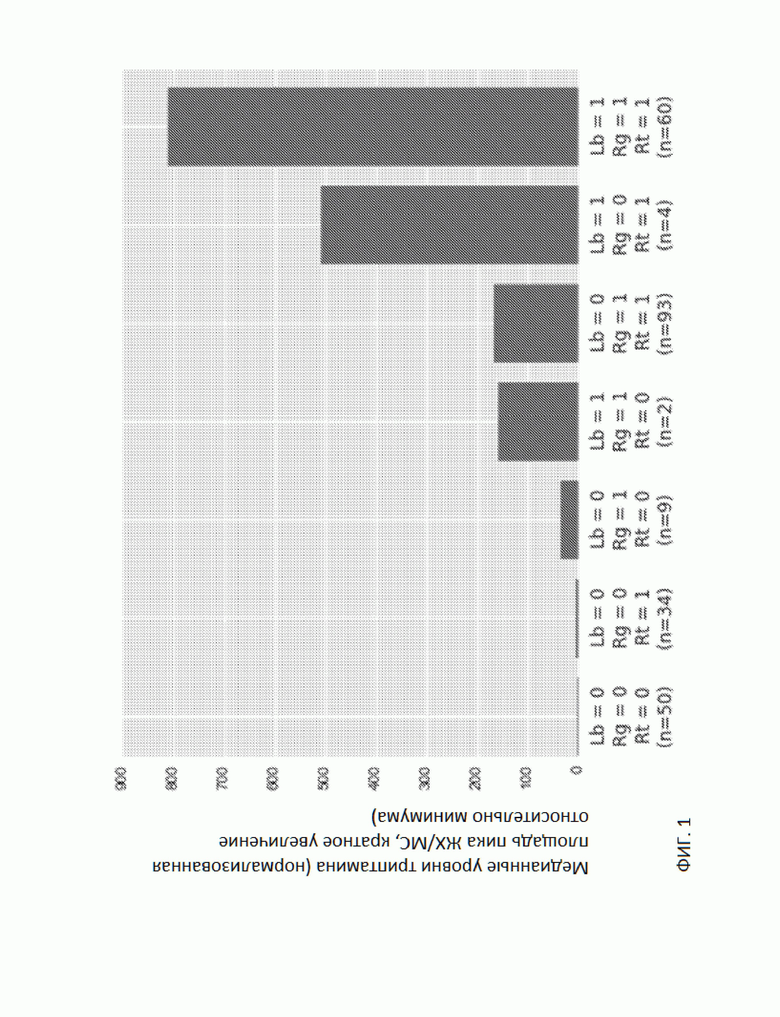
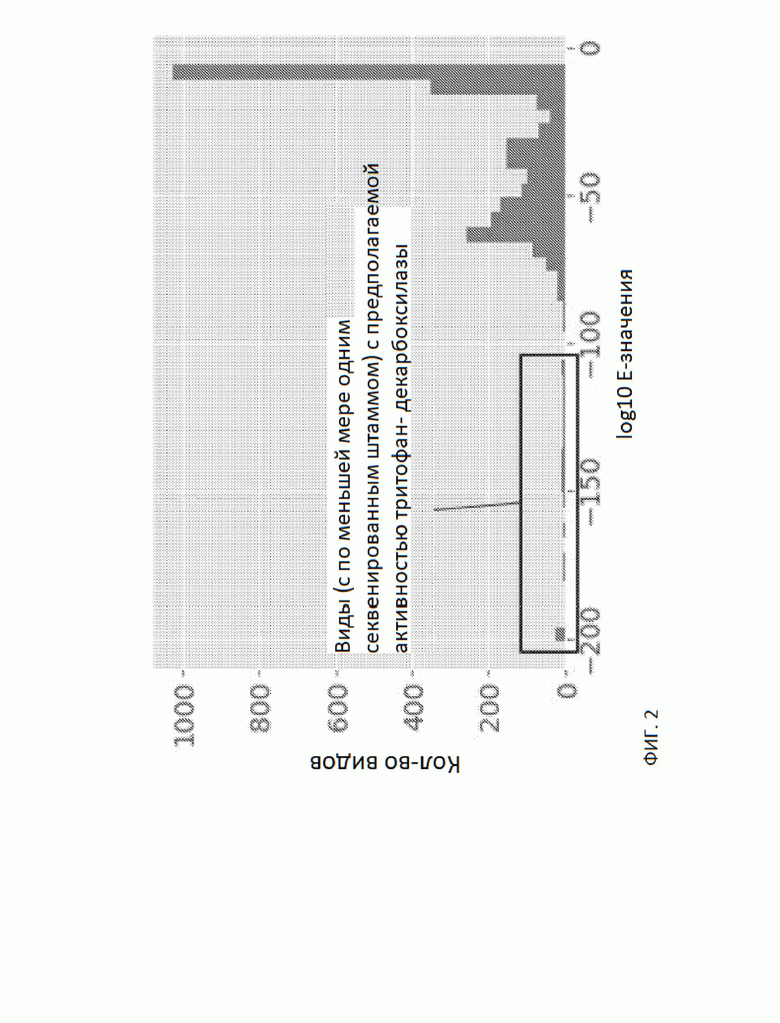

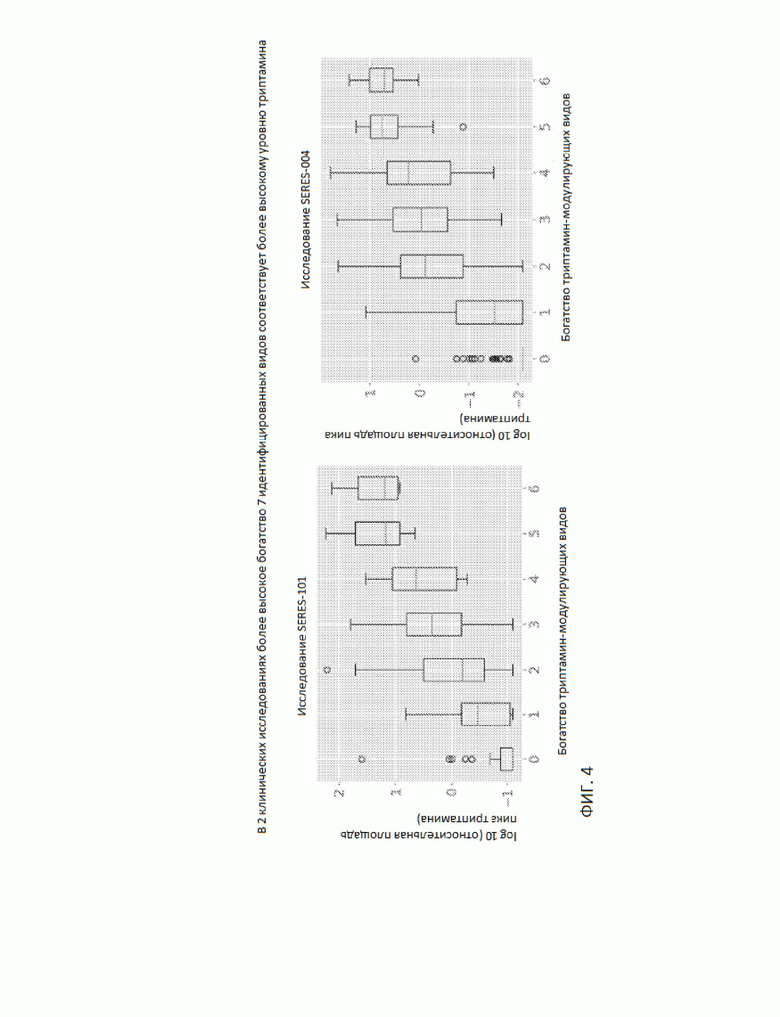

































Приведены композиции и способы изменения уровней триптамина у субъекта. Способ включает введение субъекту фармацевтического состава, который содержит по меньшей мере два вида бактерий, причем первый вид бактерий представляет собой Ruminococcus torques, а второй вид бактерий выбран из: Bacteroides ovatus, Bacteroides stercoris, Blautia sp_CAG_257_SC146, Clostridium aldenense, Clostridium bolteae, Clostridium citroniae, Clostridium clostridioforme, Clostridium nexile, Eggerthella sp., Flavonifractor plautii, Lachnospiraceae bacterium 2_1_58FAA, Lachnospiraceae bacterium 6_1_63FAA, Lachnospiraceae bacterium 9_1_43BFAA, Ruminococcus gnavus, или Veillonella parvula. 18 з.п. ф-лы, 2 пр., 14 табл., 5 ил.
1. Способ лечения субъекта, имеющего заболевание или патологическое состояние, характеризующееся наличием низкого уровня триптамина, низкого уровня 5-гидрокситриптамина или обоих, причем способ включает введение субъекту фармацевтического состава, который содержит по меньшей мере два вида бактерий, причем первый вид бактерий представляет собой Ruminococcus torques, а второй вид бактерий выбран из:
Bacteroides ovatus,
Bacteroides stercoris,
Blautia sp_CAG_257_SC146,
Clostridium aldenense,
Clostridium bolteae,
Clostridium citroniae,
Clostridium clostridioforme,
Clostridium nexile,
Eggerthella sp.,
Flavonifractor plautii,
Lachnospiraceae bacterium 2_1_58FAA,
Lachnospiraceae bacterium 6_1_63FAA,
Lachnospiraceae bacterium 9_1_43BFAA,
Ruminococcus gnavus, или
Veillonella parvula.
2. Способ по п. 1, отличающийся тем, что один или более из по меньшей мере двух видов бактерий способен повышать уровень триптамина, уровень 5-гидрокситриптамина или обоих при введении субъекту.
3. Способ по п. 2, отличающийся тем, что повышение уровня триптамина, уровня 5-гидрокситриптамина или обоих происходит в кале, крови, сыворотке, плазме, моче или спинномозговой жидкости у субъекта.
4. Способ по любому из пп. 1-3, отличающийся тем, что заболевание или патологическое состояние дополнительно характеризуется измененной перистальтикой кишечника, измененной агрегацией тромбоцитов, измененным иммунным ответом, измененной функцией сердца или измененным развитием костей.
5. Способ по любому из пп. 1-4, отличающийся тем, что заболевание или патологическое состояние выбирают из группы, состоящей из синдрома раздраженного кишечника, воспалительного заболевания кишечника, запора, депрессии, тревожного расстройства, сердечно-сосудистого заболевания и остеопороза, злокачественного новообразования кишечника и злокачественного новообразования толстой кишки.
6. Способ по п. 5, отличающийся тем, что воспалительное заболевание кишечника выбирают из группы, состоящей из инфекционного колита, язвенного колита, болезни Крона, ишемического колита, радиационного колита и микроскопического колита.
7. Способ по любому из пп. 1-6, отличающийся тем, что:
(1) Ruminococcus torques содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 58-63;
(2) Ruminococcus gnavus содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 52-57;
(3) Lachnospiraceae bacterium 9_1_43BFAA содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 51;
(4) Eggerthella sp.содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 69-72;
(5) Clostridium nexile содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 22;
(6) Flavonifractor plautii содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 45;
(7) Bacteroides ovatus содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 67;
(8) Bacteroides stercoris содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 68;
(9) Lachnospiraceae bacterium 2_1_58FAA содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 48;
(10) Clostridium aldenense содержит a 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 4;
(11) Clostridium citroniae содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 17;
(12) Clostridium clostridioforme содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 18-20;
(13) Veillonella parvula содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 65;
(14) Blautia sp CAG_257 содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 3;
(15) Clostridium bolteae содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 16; или
(16) Lachnospiraceae bacterium 6_1_63FAA содержит 16S рДНК последовательность, которая по меньшей мере на 95% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 50.
8. Способ по п. 7, отличающийся тем, что:
(1) Ruminococcus torques содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 58-63;
(2) Ruminococcus gnavus (штамм 1) содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 52-57;
(3) Lachnospiraceae bacterium 9_1_43BFAA содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 51;
(4) Eggerthella sp.содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 69-72;
(5) Clostridium nexile содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 22;
(6) Flavonifractor plautii содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 45;
(7) Bacteroides ovatus содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 67;
(9) Bacteroides stercoris содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 68;
(10) Lachnospiraceae bacterium 2_1_58FAA содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 48;
(11) Clostridium aldenense содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 4;
(12) Clostridium citroniae содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 17;
(13) Clostridium clostridioforme содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 18-20;
(14) Veillonella parvula содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 65;
(15) Blautia sp CAG_257 содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 3;
(16) Clostridium bolteae содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 16;
или
(17) Lachnospiraceae bacterium 6_1_63FAA содержит 16S рДНК последовательность, которая по меньшей мере на 96% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 50.
9. Способ по п. 7 или 8, отличающийся тем, что:
(1) Ruminococcus torques содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 58-63;
(2) Ruminococcus gnavus (штамм 1) содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 52-57;
(3) Lachnospiraceae bacterium 9_1_43BFAA содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 51;
(4) Eggerthella sp.содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 69-72;
(5) Clostridium nexile содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 22;
(6) Flavonifractor plautii содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 45;
(7) Bacteroides ovatus содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 67;
(8) Bacteroides stercoris содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 68;
(9) Lachnospiraceae bacterium 2_1_58FAA содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 48;
(10) Clostridium aldenense содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 4;
(11) Clostridium citroniae содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 17;
(12) Clostridium clostridioforme содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 18-20;
(13) Veillonella parvula содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 65;
(14) Blautia sp CAG_257 содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 3;
(15) Clostridium bolteae содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 16; или
(16) Lachnospiraceae bacterium 6_1_63FAA содержит 16S рДНК последовательность, которая по меньшей мере на 97% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 50.
10. Способ по любому из пп. 7-9, отличающийся тем, что:
(1) Ruminococcus torques содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 58-63;
(2) Ruminococcus gnavus (штамм 1) содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 52-57;
(3) Lachnospiraceae bacterium 9_1_43BFAA содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 51;
(4) Eggerthella sp.содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 69-72;
(5) Clostridium nexile содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 22;
(6) Flavonifractor plautii содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 45;
(7) Bacteroides ovatus содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 67;
(8) Bacteroides stercoris содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 68;
(9) Lachnospiraceae bacterium 2_1_58FAA содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 48;
(10) Clostridium aldenense содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 4;
(11) Clostridium citroniae содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 17;
(12) Clostridium clostridioforme содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 18-20;
(13) Veillonella parvula содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 65;
(14) Blautia sp CAG_257 содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 3;
(15) Clostridium bolteae содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 16; или
(16) Lachnospiraceae bacterium 6_1_63FAA содержит 16S рДНК последовательность, которая по меньшей мере на 98% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 50.
11. Способ по любому из пп. 7-10, отличающийся тем, что:
(1) Ruminococcus torques содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 58-63;
(2) Ruminococcus gnavus (штамм 1) содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 52-57;
(3) Lachnospiraceae bacterium 9_1_43BFAA содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 51;
(4) Eggerlla sp. содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 69-72;
(5) Clostridium nexile содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 22;
(6) Flavonifractor plautii содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 45;
(7) Bacteroides ovatus содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 67;
(8) Bacteroides stercoris содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 68;
(9) Lachnospiraceae bacterium 2_1_58FAA содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 48;
(10) Clostridium aldenense содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 4;
(11) Clostridium citroniae содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 17;
(12) Clostridium clostridioforme содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в любой из SEQ ID NOs: 18-20;
(13) Veillonella parvula содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 65;
(14) Blautia sp CAG_257 содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 3;
(15) Clostridium bolteae содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 16; или
(16) Lachnospiraceae bacterium 6_1_63FAA содержит 16S рДНК последовательность, которая по меньшей мере на 99% идентична 16S рДНК последовательности, представленной в SEQ ID NO: 50.
12. Спсоб по любому из пп. 7-11, отличающийся тем, что:
(1) Ruminococcus torques содержит 16S рДНК последовательность, представленную в любой из SEQ ID NOs: 58-63;
(2) Ruminococcus gnavus (штамм 1) содержит 16S рДНК последовательность, представленную в любой из SEQ ID NOs: 52-57;
(3) Lachnospiraceae bacterium 9_1_43BFAA содержит 16S рДНК последовательность, представленную в SEQ ID NO: 51;
(4) Eggerthella sp. содержит 16S рДНК последовательность, представленную в любой из SEQ ID NOs: 69-72;
(5) Clostridium nexile содержит 16S рДНК последовательность, представленную в SEQ ID NO: 22;
(6) Flavonifractorplautii содержит 16S рДНК последовательность, представленную в SEQ ID NO: 45;
(7) Bacteroides ovatus содержит 16S рДНК последовательность, представленную в SEQ ID NO: 67;
(8) Bacteroides stercoris содержит 16S рДНК последовательность, представленную в SEQ ID NO: 68;
(9) Lachnospiraceae bacterium 2_1_58FAA содержит 16S рДНК последовательность, представленную в SEQ ID NO: 48;
(10) Clostridium aldenense содержит 16S рДНК последовательность, представленную в SEQ ID NO: 4;
(11) Clostridium citroniae содержит 16S рДНК последовательность, представленную в SEQ ID NO: 17;
(12) Clostridium clostridioforme содержит 16S рДНК последовательность, представленную в любой из SEQ ID NOs: 18-20;
(13) Veillonellaparvula содержит 16S рДНК последовательность, представленную в SEQ ID NO: 65;
(14) Blautia sp CAG_257 содержит 16S рДНК последовательность, представленную в SEQ ID NO: 3;
(15) Clostridium bolteae содержит 16S рДНК последовательность, представленную в SEQ ID NO: 16; или
(16) Lachnospiraceae bacterium 6_1_63FAA содержит 16S рДНК последовательность, представленную в SEQ ID NO: 50.
13. Способ по любому из пп. 1-12, отличающийся тем, что фармацевтический состав содержит по меньшей мере 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 видов бактерий.
14. Способ по любому из пп. 1-13, отличающийся тем, что фармацевтический состав содержит: (a) Ruminococcus torques and Lachnospiraceae bacterium 6_1_63FAA;
(b) Ruminococcus torques и Eggerthella sp.;
(c) Ruminococcus torques и Clostridium nexile;
(d) Ruminococcus torques и Lachnospiraceae bacterium 9_1_43BFAA;
(e) Ruminococcus torques и Ruminococcus gnavus;
(f) Ruminococcus torques, Ruminococcus gnavus и Eggerthella sp.;
(g) Ruminococcus torques, Clostridium nexile и Eggerthella sp.;
(h) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA и Eggerthella sp.;
(i) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA и Clostridium nexile;
(j) Ruminococcus torques, Ruminococcus gnavus и Clostridium nexile;
(k) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA и Ruminococcus gnavus;
(l) Ruminococcus torques, Lachnospiraceae bacterium 9_1_43BFAA и Ruminococcus gnavus;
(m) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Ruminococcus gnavus и Eggerthella sp.;
(n) Ruminococcus torques, Ruminococcus gnavus, Clostridium nexile и Eggerthella sp.;
(o) Ruminococcus torques, Lachnospiraceae bacterium 9_1_43BFAA, Ruminococcus gnavus и Eggerthella sp.;
(p) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Clostridium nexile и Eggerthella sp.;
(q) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Ruminococcus gnavus и Clostridium nexile;
(r) Ruminococcus torques, Lachnospiraceae bacterium 9_1_43BFAA, Ruminococcus gnavus и Clostridium nexile;
(s) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Lachnospiraceae bacterium 9_1_43BFAA и Ruminococcus gnavus;
(t) Ruminococcus torques, Lachnospiraceae bacterium 9_1_43BFAA, Ruminococcus gnavus, Clostridium nexile и Eggerthella sp.;
(u) Ruminococcus torques, Ruminococcus gnavus, Clostridium nexile, Eggerthella sp.и Ruminococcus gnavus;
(v) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Lachnospiraceae bacterium 9_1_43BFAA, Clostridium nexile и Ruminococcus gnavus;
(w) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Lachnospiraceae bacterium 9_1_43BFAA, Ruminococcus gnavus и Eggerthella sp.;
(x) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Ruminococcus gnavus, Clostridium nexile и Eggerthella sp.;
(y) Ruminococcus torques, Lachnospiraceae bacterium 6_1_63FAA, Lachnospiraceae bacterium 9_1_43BFAA, Ruminococcus gnavus, Clostridium nexile и Eggerthella sp.; или
(z) Ruminococcus torques, Lachnospiraceae bacterium 9_1_43BFAA, Clostridium nexile, Eggerthella sp. и Ruminococcus gnavus.
15. Способ по любому из пп. 1-14, отличающийся тем, что фармацевтический состав дополнительно содержит одно или несколько вспомогательных веществ.
16. Способ по п. 15, отличающийся тем, что одно или несколько вспомогательных веществ содержат глицерин.
17. Способ по любому из пп. 1-16, отличающийся тем, что один или несколько видов бактерий в фармацевтическом составе находятся в форме спор.
18. Способ по любому из пп. 1-17, отличающийся тем, что один или несколько видов бактерий в фармацевтическом составе находятся в вегетативной форме.
19. Способ по любому из пп. 1-18, отличающийся тем, что фармацевтический состав можно вводить субъекту посредством перорального введения.
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
| Токарный резец | 1924 |
|
SU2016A1 |
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
| Achtman and Wagner | |||
| Станок для изготовления деревянных ниточных катушек из цилиндрических, снабженных осевым отверстием, заготовок | 1923 |
|
SU2008A1 |
| Microbial diversity and the genetic nature of microbial species | |||
| Nat | |||
| Rev | |||
| Microbiol | |||
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| BRIANNA B WILLIAMS et al., "Discovery and characterization of gut microbiota decarboxylases that can | |||
Авторы
Даты
2023-04-13—Публикация
2018-10-03—Подача