Область техники, к которой относится изобретение
Настоящее изобретение относится к области биотехнологии, в частности к генетической инженерии, и может быть использовано в различных областях биотехнологии и научных исследованиях (тонкий органический синтез, селективное определение D-Leu, D-Met и D-Phe и др. в сложных биологических системах, включая жидкости и ткани человека). Для достижения указанных целей сконструирована рекомбинантная плазмидная ДНК pNhyDAAO, содержащая модифицированный ген nhydaao, кодирующий оксидазу D-аминокислот бактерий Natronosporangium hydrolyticum ACPA39 (NhyDAAO). Полученная плазмида обеспечивает экспрессию, включенного в нее гена nhydaao в клетках Escherichia coli. Для обеспечения высокой эффективности экспрессии в целевом штамме-продуценте в модифицированном гене nhydaao в качестве стартового кодона используется триплет ATG вместо триплета GTG в исходном гене. В результате трансформации штамма E.coli BL21(DE3) предложенной плазмидой получен новый рекомбинантный штамм-продуцент E.coli BL21(DE3))/pNhyDAAO ВКПМ В-14337, обеспечивающий экспрессию NhyDAAO в активной форме. Оксидазы D-аминокислот находят применение в самых различных областях науки и техники – фармацевтическая промышленность и тонкий органический синтез, определение D-аминокислот как маркеров различных заболеваний человека (включая нейродегенеративные), для определения обсемененности пищи бактериями, как потенциальный антимикробный агент и др.
Уровень техники
Оксидаза D-аминокислот (DAAO, КФ 1.2.1.2) играет важную роль в функционировании как прокариот, так низших (дрожжи и грибы) и высших эукариот (млекопитающие). В бактериях, дрожжах и микроскопических грибах основная роль этого фермента сводится к утилизации экзогенных D-аминокислот (в первую очередь D-Ala) [Tishkov, V. I. & Khoronenkova, S. V. (2005). D-Amino acid oxidase: structure, catalytic mechanism, and practical application. Biochemistry (Mosc.) 70, 40-54, doi:BCM70010051; Pollegioni, L., Piubelli, L., Sacchi, S., Pilone, M. S., & Molla, G. (2007a). Physiological functions of D-amino acid oxidases: from yeast to humans. Cell Mol. Life Sci. 64, 1373-1394, doi:10.1007/s00018-007-6558-4; Takahashi, S., Abe, K., & Kera, Y. (2015). Bacterial d-amino acid oxidases: Recent findings and future perspectives. Bioengineered. 6, 237-241, doi:10.1080/21655979.2015.10529172]. У высших эукариот – позвоночных и особенно млекопитающих, основная роль DAAO заключается в поддержании определенного уровня D-аминокислот, которые являются регуляторами важнейших процессов, в первую очередь нервной деятельности. Например, снижение уровня D-Ser в спинномозговой жидкости за счет повышенной активности DAAO приводит к шизофрении [Chumakov, I., Blumenfeld, M., Guerassimenko, O., et al. (2002). Genetic and physiological data implicating the new human gene G72 and the gene for D-amino acid oxidase in schizophrenia. Proc. Natl. Acad. Sci. U. S. A 99, 13675-13680, doi:10.1073/pnas.182412499; Cheng, Y. J., Lin, C. H., & Lane, H. Y. (2021). D-Amino Acids and pLG72 in Alzheimer's Disease and Schizophrenia. Int. J. Mol. Sci. 22, doi: 10.3390/ijms222010917]. Протекание болезней Альцгеймера и Паркинсона сопровождается повышением уровня D-Ala [Cheng, Y. J., Lin, C. H., & Lane, H. Y. (2021). D-Amino Acids and pLG72 in Alzheimer's Disease and Schizophrenia. Int. J. Mol. Sci. 22, doi: 10.3390/ijms222010917; Pernot, P., Mothet, J. P., Schuvailo, O., Soldatkin, A., Pollegioni, L., Pilone, M., Adeline, M. T., Cespuglio, R., & Marinesco, S. (2008). Characterization of a yeast D-amino acid oxidase microbiosensor for D-serine detection in the central nervous system. Anal. Chem. 80, 1589-1597, doi:10.1021/ac702230w4,5]. Оксидаза D-аминокислот также широко используется на практике [Khoronenkova, S. V. & Tishkov, V. I. (2008). D-amino acid oxidase: physiological role and applications. Biochemistry (Mosc.) 73, 1511-1518, doi: 10.1134/s0006297908130105; Pollegioni, L., Molla, G., Sacchi, S., Rosini, E., Verga, R., & Pilone, M. S. (2008). Properties and applications of microbial D-amino acid oxidases: current state and perspectives. Appl. Microbiol. Biotechnol. 78, 1-16, doi:10.1007/s00253-007-1282-4; Pollegioni, L. & Molla, G. (2011). New biotech applications from evolved D-amino acid oxidases. Trends Biotechnol. 29, 276-283, doi:;10.1016/j.tibtech.2011.01.010 ]. Например, DAAO из дрожжей Trigonopsis variabilis используется в двустадийном биокаталитическом процессе получения 7-аминоцефалоспорановой кислоты (7-АЦК) из цефалоспорина С [Barber MS, Giesecke U, Reichert A, Minas W. 2004. Industrial enzymatic production of cephalosporin-based beta-lactams. Adv. Biochem. Eng Biotechnol. 88:179-215.10; Atroshenko, D. L., Shelomov, M. D., Zarubina, S. A., Negru, N. Y., Golubev, I. V., Savin, S. S., & Tishkov, V. I. (2019). Multipoint TvDAAO Mutants for Cephalosporin C Bioconversion. Int. J. Mol. Sci. 20, doi: 10.3390/ijms20184412]. 7-АЦК является исходным синтоном для производства полусинтетических цефалоспоринов различных поколений. По сравнению с ранее используемым чисто химическим такой процесс позволяет сократить расход органических растворителей в 400 раз. Объем производства 7_АЦК составляет более 10 тыс. тонн в год. Описан биокаталитический способ получения природного гербицида широкого спектра действия-L-глюфозината, в котором на первой стадии используется DAAO [Green B.M., Gradley M.L. Methods For Making L-Glufosinate. US 2017 / 0253897 A1 Published 07.09.2017. filed 28.02.2017]. DAAO используется в фармацевтической промышленности, например, при получении ингибитора ангиотензинпревращаюего фермент Омапатрилата [Patel, R. N. (2001). Enzymatic synthesis of chiral intermediates for Omapatrilat, an antihypertensive drug. Biomol. Eng 17, 167-182.].
Отдельной областью современной аналитической биотехнологии является разработке методов селективного определения различных D-аминокислот в сложных биологических системах. Например, D-Ala является сшивающим агентом в муреиновом слое клеток грам-положительных и грам-отрицательных бактерий. Поэтому наличие в образце D-Ala позволяет определить бактериальное заражение объекта даже если бактерии уже неживые. Однако в первую очередь особое внимание уделяется определению D-аминокислот в тканях и жидкостях человека, поскольку они являются регуляторами важнейших процессов, в первую очередь нервной деятельности. Поскольку основная часть аминокислот в организме находится в виде L-изомера необходимы стереоселективные методы детекции именно D-аминокислот и такая селективность как раз может быть достигнута с помощью оксидаз D-аминокислот [Molla, G., Piubelli, L., Volonte, F., & Pilone, M. S. (2012). Enzymatic detection of D-amino acids. Methods Mol. Biol. 794, 273-289, doi:10.1007/978-1-61779-331-8_18]. Для экспресс анализа и его проведения in situ были разработаны биосенсоры для определения ряда D-0аминокислот [Sacchi, S., Rosini, E., Caldinelli, L., & Pollegioni, L. (2012). Biosensors for D-amino acid detection. Methods Mol. Biol. 794, 313-324, doi:10.1007/978-1-61779-331-8_21]. Однако разработка новых методов и биосенсоров на D-аминокислоты ограничена как свойствами известных оксидаз D-аминокислот, так и их малым выбором. В настоящее время получены и охарактеризовано небольшое количество DAAO - из почек свиньи [Fukui, K., Watanabe, F., Shibata, T., & Miyake, Y. (1987). Molecular cloning and sequence analysis of cDNAs encoding porcine kidney D-amino acid oxidase. Biochemistry 26, 3612-3618, doi:10.1021/bi00386a054], мыши [Tada, M., Fukui, K., Momoi, K., & Miyake, Y. (1990)] Cloning and expression of a cDNA encoding mouse kidney D-amino acid oxidase. Gene 90, 293-297, doi: 10.1016/0378-1119(90)90193-u], человека [Fukui, K. & Miyake, Y. (1992). Molecular cloning and chromosomal localization of a human gene encoding D-amino-acid oxidase. J. Biol. Chem. 267, 18631-18638, doi:S0021-9258(19)37007-3], карпа [Sarower, M. G., Okada, S., & Abe, H. (2003). Molecular characterization of D-amino acid oxidase from common carp Cyprinus carpio and its induction with exogenous free D-alanine. Arch. Biochem. Biophys. 420, 121-129, doi: 10.1016/j.abb.2003.09.035], дрожжей и микроскопических грибов Fusarium solani (FsoDAAO) [Isogai, T., Ono, H., Ishitani, Y., Kojo, H., Ueda, Y., & Kohsaka, M. (1990). Structure and expression of cDNA for D-amino acid oxidase active against cephalosporin C from Fusarium solani. J. Biochem. 108, 1063-1069, doi:10.1093/oxfordjournals.jbchem.a123306], Trigonopsis variabilis (TvaDAAO) [Gonzalez, F. J., Montes, J., Martin, F., Lopez, M. C., Ferminan, E., Catalan, J., Galan, M. A., & Dominguez, A. (1997). Molecular cloning of TvDAO1, a gene encoding a D-amino acid oxidase from Trigonopsis variabilis and its expression in Saccharomyces cerevisiae and Kluyveromyces lactis. Yeast 13, 1399-1408, doi: doi:10.1002/(SICI)1097-0061(199712)13:15<1399], Rhodosporidium toruloides (предыдущее название Rhodotorula gracilis) (RtoDAAO) [Pollegioni, L., Molla, G., Campaner, S., Martegani, E., & Pilone, M. S. (1997). Cloning, sequencing and expression in E. coli of a D-amino acid oxidase cDNA from Rhodotorula gracilis active on cephalosporin C. J. Biotechnol. 58, 115-123, doi: 10.1016/s0168-1656(97)00142-9]. Pichia pastoris (PpaDAAO) [Klompmaker, S. H., Kilic, A., Baerends, R. J., Veenhuis, M., & van der Klei, I. J. (2010). Activation of a peroxisomal Pichia pastoris D-amino acid oxidase, which uses d-alanine as a preferred substrate, depends on pyruvate carboxylase. FEMS Yeast Res. 10, 708-716, doi:10.1111/j.1567-1364.2010.00647.x], Candida boidinii (CboDAAO) [Yurimoto, H., Hasegawa, T., Sakai, Y., & Kato, N. (2001). Characterization and high-level production of D-amino acid oxidase in Candida boidinii. Biosci. Biotechnol. Biochem. 65, 627-633], Rasamsonia emersonii strain YA (RemDAAO) [Shimekake, Y., Furuichi, T., Abe, K., Kera, Y., & Takahashi, S. (2019). A novel thermostable D-amino acid oxidase of the thermophilic fungus Rasamsonia emersonii strain YA. Sci. Rep. 9, 11948, doi:10.1038/s41598-019-48480-y [doi];10.1038/s41598-019-48480-y. ] и бактерий из Rubrobacter xylanophilus (RxyDAAO) [Takahashi, S., Furukawara, M., Omae, K., Tadokoro, N., Saito, Y., Abe, K., & Kera, Y. (2014). A Highly Stable D-Amino Acid Oxidase of the Thermophilic Bacterium Rubrobacter xylanophilus. Appl. Environ. Microbiol. 80, 7219-7229, doi: 10.1128/AEM.02193-14], Streptomyces coelicolor (ScoDAAO) [Saito, Y., Takahashi, S., Kobayashi, M., Abe, K., & Kera, Y. (2014). D-Amino acid oxidase of Streptomyces coelicolor and the effect of D-amino acids on the bacterium. Annals of Microbiology 64, 1167-1177. doi.org/10.1007/s13213-013-0756-0] и Arthrobacter protophormiae (AprDAAO) [Geueke, B., Weckbecker, A., & Hummel, W. (2007). Overproduction and characterization of a recombinant D-amino acid oxidase from Arthrobacter protophormiae. Appl. Microbiol. Biotechnol. 74, 1240-1247, doi:10.1007/s00253-006-0776-9]. Однако, все описанные в литературе DAAO, используемые для детекции D-аминокислот, имеют широкий спектр субстратной специфичности, т.е. проявляют активность со многими D-аминокислотами, что не позволяет обеспечить селективность анализа к определенной аминокислоте. В некоторых случаях, условия анализа позволяют достигнуть определенной селективности за счет выбора фермента с определенной субстратной специфичностью или исходя из особенности условий проведения анализа. Например, DAAO из дрожжей R. gracilis имеет одинаковые каталитические характеристики с D-Ser и D-Ala и селективность определения D-Ser в спинномозговой жидкости достигается только за счет того, что физиологическая концентрация D-Ala в 10 раз ниже, чем концентрация D-Ser [Pernot, P., Mothet, J. P., Schuvailo, O., Soldatkin, A., Pollegioni, L., Pilone, M., Adeline, M. T., Cespuglio, R., & Marinesco, S. (2008). Characterization of a yeast D-amino acid oxidase microbiosensor for D-serine detection in the central nervous system. Anal. Chem. 80, 1589-1597, doi:10.1021/ac702230w]. Однако данный подход покрывает небольшую область необходимых анализов. Проведение экспериментов по изменению спектра субстратной специфичность не позволило решить данную проблему [Sacchi, S., Lorenzi, S., Molla, G., Pilone, M. S., Rossetti, C., & Pollegioni, L. (2002). Engineering the substrate specificity of D-amino-acid oxidase. J. Biol. Chem. 277, 27510-27516, doi:10.1074/jbc.M203946200; Setoyama, C., Nishina, Y., Mizutani, H., Miyahara, I., Hirotsu, K., Kamiya, N., Shiga, K., & Miura, R. (2006). Engineering the substrate specificity of porcine kidney D-amino acid oxidase by mutagenesis of the "active-site lid". J. Biochem. 139, 873-879, doi: 10.1093/jb/mvj094; Komarova, N.V., Golubev, I.V., Khoronenkova, S.V., Chubar', T.A., & Tishkov, V.I. (2012). Engineering of substrate specificity of D-amino acid oxidase from the yeast Trigonopsis variabilis: directed mutagenesis of Phe258 residue. Biochemistry (Mosc.) 77, 1181-1189, doi: 10.1134/S0006297912100100]. В частности, до сих пор не решена проблема энзиматического определения в биологических жидкостях и тканях человека D-Phe, который является биомаркером и потенциальным лекарством в ряде важных нейрональных и метаболитических заболеваний. Например, повышенный уровень D-Phe зарегистрирован в моче беременных женщин с гестационным диабетом [M.P. Lorenzo, D. Dudzik, E. Varas, M. Gibellini, M. Skotnicki, M. Zorawski, W. Zarzycki, F. Pellati, A. García, Optimization and validation of a chiral GC-MS method for the determination of free ᴅ-amino acids ratio in human urine: application to a gestational diabetes mellitus study, J. Pharm. Biomed. Anal. 107 (2015) 480–487, doi: 10.1016/j.jpba.2015.01.015.] Поэтому для определения D-Phe используется очень сложный вариант двумерной высокоэффективной жидкостной хроматографии [Hsiao, S. W., Ishii, C., Furusho, A., Hsieh, C. L., Shimizu, Y., Akita, T., Mita, M., Okamura, T., Konno, R., Ide, T., Lee, C. K., & Hamase, K. (2021). Determination of phenylalanine enantiomers in the plasma and urine of mammals and D-amino acid oxidase deficient rodents using two-dimensional high-performance liquid chromatography. Biochim. Biophys. Acta Proteins Proteom. 1869, 140540, doi: 10.1016/j.bbapap.2020.]. Данный метод является трудозатратным, дорогим и не экспрессным.
Решением данной проблемы является создание рекомбинантных штаммов -продуцентов новых оксидаз D-аминокислот с необходимой субстратной специфичностью. Для выбора пути и способа решения проблемы необходимо учитывать следующие факторы:
1. Фермент должен обладать необходимым спектром субстратной специфичности, т.е должен обеспечивать возможность селективного определения определенной D-аминокислоты (в том числе и D-Phe) в сложных биологических жидкостях и тканях. Литературные данные по субстратной специфичности DAAO из разных источников позволяют сделать вывод, что ферменты из позвоночных не являются перспективными.
2. Для длительного использования фермента он должен иметь одинаковые рН-профили активности и стабильности. В данном случае поиск новых DAAO в дрожжах и микроскопических грибах не является перспективным, поскольку у них максимальная активность наблюдается при рН 8,0 и выше, в то время как оптимальная стабильность находится в районе рН 7,0. Однако в диапазоне рН 6,0-8,0 активность DAAO дрожжей и микроскопических грибов сильно изменяется [Pollegioni, L., Porrini, D., Molla, G., & Pilone, M. S. (2000). Redox potentials and their pH dependence of D-amino-acid oxidase of Rhodotorula gracilis and Trigonopsis variabilis. Eur. J. Biochem. 267, 6624-6632, doi: 10.1046/j.1432-1327.2000.01757.x; Harris, C. M., Pollegioni, L., & Ghisla, S. (2001). pH and kinetic isotope effects in d-amino acid oxidase catalysis. Eur. J. Biochem. 268, 5504-5520, doi: 10.1046/j.1432-1033.2001.02462.x], что не может обеспечить точность и воспроизводимость анализа. Для разработки новых методов анализа наиболее перспективными являются DAAO из бактерий, которые имеют одинаковые рН-оптимумы активности и стабильности [Takahashi, S., Abe, K., & Kera, Y. (2015). Bacterial d-amino acid oxidases: Recent findings and future perspectives. Bioengineered. 6, 237-241, doi: 10.1080/21655979.2015.1052917]. Однако, в настоящее время получены и охарактеризованы всего три бактериальных DAAO и эти три фермента не соответствуют требованиям по спектру субстратной специфичности. Наиболее близкой по спектру субстратной специфичности для анализа D-Leu и D-Phe является DAAO из бактерий Rubrobacter xylanophilus, но он не позволяет обеспечить специфичность в большинстве образцов поскольку активность с D-Ala и D-Ser составляет 76,2 и 61,9% от активности с D-Phe соответственно [Takahashi, S., Furukawara, M., Omae, K., Tadokoro, N., Saito, Y., Abe, K., & Kera, Y. (2014). A Highly Stable D-Amino Acid Oxidase of the Thermophilic Bacterium Rubrobacter xylanophilus. Appl. Environ. Microbiol. 80, 7219-7229, doi: 10.1128/AEM.02193-14]. Поэтому поиск генов новых бактериальных DAAO с другим спектром субстратной специфичности является очень актуальным.
3. Создание рекомбинантного штамма – продуцента бактериальной DAAO. Природные штаммы бактерий не могут быть использованы для получения фермента, поскольку уровень экспрессии бактериальных DAAO в нативных штаммах очень низок. Например, в случае Arthrobacter protophormiae удельная активность в бесклеточном экстракте составляет всего 0,003 ед. на мг белка, что соответствует уровню экспрессии 0,01%, в то время как в рекомбинантных штаммах уровень экспрессии составляет до 30-40% от растворимого белка клетки. Поэтому для обеспечения возможности получения достаточных количеств фермента необходимо использовать методы генной инженерии и создавать рекомбинантный штамм-продуцент. В литературе описано получение рекомбинантных DAAO в различных штаммах-продуцентах. В случае DAAO дрожжей и микроскопических грибов используются как бактерии E.coli [Molla, G., Vegezzi, C., Pilone, M. S., & Pollegioni, L. (1998). Overexpression in Escherichia coli of a recombinant chimeric Rhodotorula gracilis d-amino acid oxidase. Protein Expr. Purif. 14, 289-294, doi: 10.1006/prep.1998.0956; Dib, I., Stanzer, D., & Nidetzky, B. (2007). Trigonopsis variabilis D-amino acid oxidase: control of protein quality and opportunities for biocatalysis through production in Escherichia coli. Appl. Environ. Microbiol. 73, 331-333, doi:10.1128/AEM.01569-06; Эльдаров М.А., Редо В.А., Жгун А.А., Зейналов О.А., Скрябин К.Г. Рекомбинантная плазмида pETTVDAO2, обеспечивающая синтез оксидазы D-аминокислот (DAO) дрожжей Trigonopsis variabilis в клетках Escherichia coli и рекомбинантный штамм Escherichia coli C41(DE3)/pETTvDAO2 – продуцент DAO. Патент RU 2310687, Опубликовано: 20.11.2007, Бюл. № 32, заявка 17.03.2006], так и различные типы дрожжей [Dominguez, A. (1997). Molecular cloning of TvDAO1, a gene encoding a D-amino acid oxidase from Trigonopsis variabilis and its expression in Saccharomyces cerevisiae and Kluyveromyces lactis. Yeast 13, 1399-1408; Abad, S., Nahalka, J., Winkler, M., Bergler, G., Speight, R., Glieder, A., & Nidetzky, B. (2011). High-level expression of Rhodotorula gracilis D-amino acid oxidase in Pichia pastoris. Biotechnol. Lett. 33, 557-563, doi:10.1007/s10529-010-0456-9; Isoai, A., Kimura, H., Reichert, A., Schorgendorfer, K., Nikaido, K., Tohda, H., Giga-Hama, Y., Mutoh, N., & Kumagai, H. (2002). Production of D-amino acid oxidase (DAO) of Trigonopsis variabilis in Schizosaccharomyces pombe and the characterization of biocatalysts prepared with recombinant cells. Biotechnol. Bioeng. 80, 22-32, doi:10.1002/bit.10345; Yurimoto, H., Hasegawa, T., Sakai, Y., & Kato, N. (2001). Characterization and high-level production of D-amino acid oxidase in Candida boidinii. Biosci. Biotechnol. Biochem. 65, 627-633]. В случае бактериальных DAAO в качестве штамма-продуцента используются только клетки E.coli [Takahashi, S., Furukawara, M., Omae, K., Tadokoro, N., Saito, Y., Abe, K., & Kera, Y. (2014). A Highly Stable D-Amino Acid Oxidase of the Thermophilic Bacterium Rubrobacter xylanophilus. Appl. Environ. Microbiol. 80, 7219-7229, doi: 10.1128/AEM.02193-14; Saito, Y., Takahashi, S., Kobayashi, M., Abe, K., & Kera, Y. (2014). D-Amino acid oxidase of Streptomyces coelicolor and the effect of D-amino acids on the bacterium. Ann. Microbiol. 64, 1167-1177. doi.org/10.1007/s13213-013-0756-0; Geueke, B., Weckbecker, A., & Hummel, W. (2007). Overproduction and characterization of a recombinant D-amino acid oxidase from Arthrobacter protophormiae. Appl. Microbiol. Biotechnol. 74, 1240-1247, doi:10.1007/s00253-006-0776-9].
Исходя из вышеизложенного следует, что наиболее близким к заявляемому является штамм-продуцент E.coli рекомбинантной DAAO из бактерий R. xylanophilus [Takahashi, S., Furukawara, M., Omae, K., Tadokoro, N., Saito, Y., Abe, K., & Kera, Y. (2014). A Highly Stable D-Amino Acid Oxidase of the Thermophilic Bacterium Rubrobacter xylanophilus. Appl. Environ. Microbiol. 80, 7219-7229, doi: 10.1128/AEM.02193-14]. Для его получения используется система экспрессии в штамме E.coli BL21(DE3), а плазмида pERDAOnhC, обеспечивающая экспрессию рекомбинантной DAAO в клетках E.coli создана на основе вектора pET15b. Однако данная система экспрессии E.coli BL21(DE3) - pET15b имеет существенные недостатки. Плазмида pET15b, используемая для создания экспрессионного вектора, в качестве маркера селективного отбора клеток с плазмидой содержит ген устойчивости к ампициллину. Известно, что при культивировании рекомбинантного штамма по достижении поглощения культуры клеток на 600 нм величины А600 1 ед. поглощения и более, образующаяся бета-лактамаза полностью разрушает ампициллин и селективное действие антибиотика прекращается. Во-вторых, система экспрессии E.coli BL21(DE3) - pET15b не обеспечивает строгого уровня контроля синтеза целевого белка, т.е. в этой системе идет хоть небольшая, но постоянная базовая экспрессии без всякой индукции. Если белок токсичен, то это приводит к преждевременной гибели клеток. Если нет, то в клетках происходит индукция протеаз, которые могут преждевременно деградировать целевой фермент в процессе культивирования. DAAO из бактерий R. xylanophilus обладает широким рН-оптимумами активности и стабильности в необходимом диапазоне рН 6,0-8,0. Однако существенным недостатком этого фермента является сравнимая активность с D-Ala, D-Ser, D-Pro и D-Phe. Учитывая, что в физиологических жидкостях и тканях млекопитающих уровень первых трех D-аминокислот выше, чем уровень D-Phe, то селективное определение последнего в указанных образцах не представляется возможным. Кроме того, штамм-прототип характеризуется низкой продуктивностью. В результате культивирования штамма-прототипа выход DAAO из R. xylanophilus при измерении активности с D-Leu составляет 1,08 ед. на г сырой биомассы. Однако в прототипе активность RxyDAAO измеряли при 60 °С. С учетом того, что при изменении температуры на 10 градусов скорость реакции изменяется в 2-4 раза, то при 30 °С активность RxyDAAO должна быть минимум в 8 раз ниже, т.е. максимально возможный выход должен составлять ≤ 0,135 ед. на г биомассы.
Таким образом, техническая проблема, решаемая посредством заявляемого изобретения, заключается в необходимости преодоления недостатков, присущих аналогам и прототипу за счет создания рекомбинантного штамма E-coli – продуцента оксидазы D-аминокислот, которая проявляет высокую активность с D-Phe, имеет намного более низкую активность с основными D-аминокислотами в жидкостях и тканях млекопитающих - D-Ala, D-Ser и D-Pro.
Раскрытие изобретения
Техническим результатом заявляемой группы изобретений являются:
1. получение плазмиды pNhyDAAO, которая обеспечивает синтез целевого фермента NhyDAAO в клетках E.coli. За счет наличия в плазмиде гена белка-репрессора laqI осуществляется строгий контроль процесса экспрессии. Отсутствие преждевременной гибели клеток свидетельствует о нетоксичности экспрессируемого фермента. Высокий уровень экспрессии достигается за счет использования высокоэффективного промотора РНК-полимеразы фага Т7.
2. получение штамм продуцент E.coli BL21(DE3)/pNhyDAAO. Данный штамм содержит на хромосоме ген РНК-полимеразы фага Т7, что позволяет использовать полученную плазмиду pNhyDAAO для экспрессии целевого белка. Кроме того, исходный штамм E.coli BL21(DE3), который использовали для получения целевого штамма-продуцента мутантен по основной протеазе клеток E.coli (lonA-), что значительно предотвращает деградацию целевого фермента в процессе культивирования штамма-продуцента. Заявляемый штамм продуцент E.coli BL21(DE3)/pNhyDAAO по своей продуктивности (выход активного фермента на грамм сырой биомассы) минимум в 24 раза превосходит прототип – штамм- E.coli BL21(DE3)/ pERDAOnhC - продуцент окcидазы D-аминокислот бактерий Rubrobacter xylanophilus
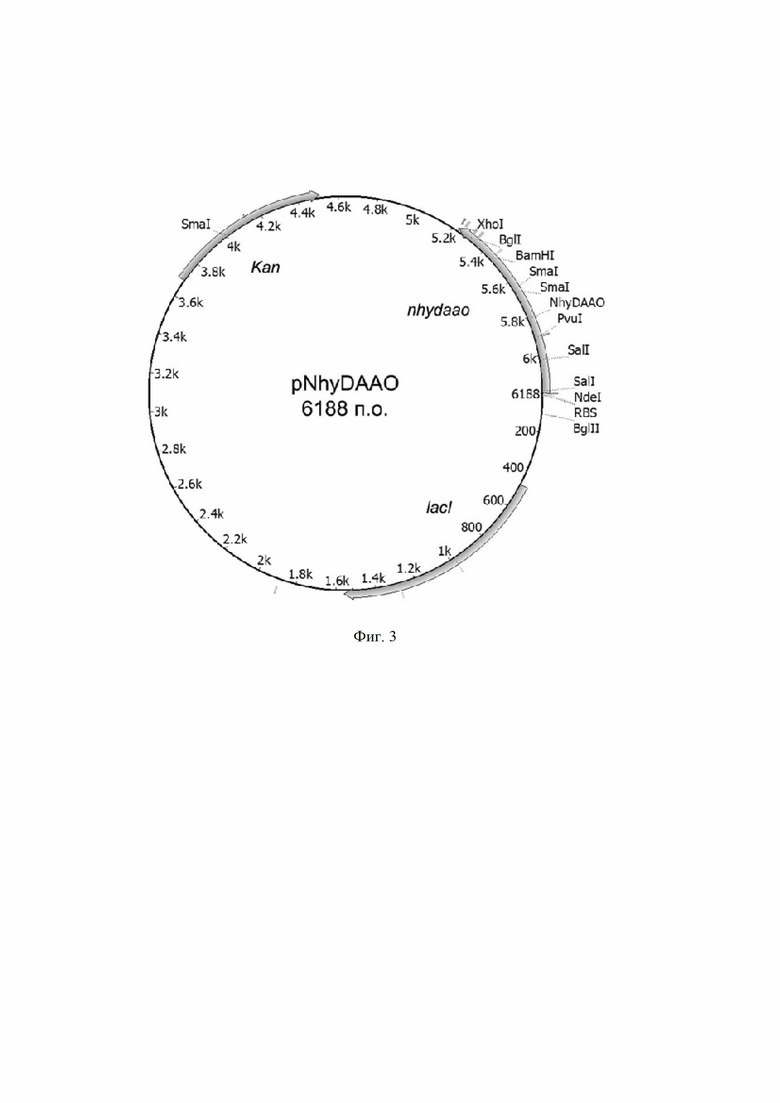
Технический результат достигается рекомбинантной плазмидой pNhyDAAO размером 6188 п.о., содержащей модифицированный ген nhydaao оксидазы D-аминокислот из N. hydrolyticum ACPA39 под контролем T7 промотора. обеспечивающей синтез оксидазы D-аминокислот из N. hydrolyticum ACPA39 (NhyDAAO) в клетках E. coli. Плазмида pNhyDAAO получена путем клонирования по сайтам NdeI/Xho фрагмента размером 958 п.о. (последовательность SEQ ID №3) с геном, кодирующим NhyDAAO (951 п.о.) в плазмиду pET24a(+), расщепленную рестриктазами NdeI и Xho.
Также технический результат достигается рекомбинантным штаммом Escherichia coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 - продуцентом оксидазы D-аминокислот NhyDAAO, полученным путем трансформации штамма Escherichia. coli BL21(DE3) заявляемой плазмидной ДНК, задепонированный во Всероссийской коллекции промышленных микроорганизмов (ВКПМ) по номером В-14337.
Для обеспечения эффективной экспрессии новой DAAO в клетках E.coli ген фермента клонируется в плазмидный вектор под контроль сильного промотора РНК-полимеразы фага Т7. В используемом для клонирования векторе pET24a(+) имеется ген Kan, который в качестве селективного маркера обеспечивает устойчивость к канамицину, а не к ампициллину как в pET15b. Кроме того, исходная плазмида pET24a(+) содержит ген белка-репрессора lac-промотора lacI, который предотвращает неспецифическую базовую экспрессию целевого белка. Многие гены ферментов из экстремофильных микроорганизмов в качестве стартового кодона содержат триплет GTG, который неэффективен при экспрессии в клетках E.coli. Этот вариант наблюдается и в гене новой оксидазы D-аминокислот. В связи с этим, клонируемая в вектор pET24a(+) нуклеотидная последовательность с геном nhydaao содержит замену стартового триплета GTG на ATG.
Краткое описание чертежей
Изобретение поясняется следующими чертежами.
Фиг.1 Выравнивание аминокислотных последовательностей потенциальных DAAO, отобранных после поиска по гомологии в геномах бактерий в базах UniProt (NCBI) и коллекции микроорганизмов МГУ имени М.В. Ломоносова и ФИЦ Биотехнологии РАН. Выравнивание выполнено с помощью программы Clustal X 1.83. Ферменты представлены двумя группами.
1. Оксидазы D-аминокислот бактерий, для которых были известны аминокислотные последовательности
RxyDAAO - Rubrobacter xylanophilus;
ScoDAAO - Streptomyces coelicolor;
AprDAAO - Arthrobacter protophormiae;
2. Новые оксидазы D-аминокислот, найденные по результатам поиска в геномах бактерий
NhyDAAO - Natronosporangium hydrolyticum ACPA39;
GthDAAO - Gandjariella thermophila;
CthDAAO - Chloracidobacterium thermophilum B;
RtaDAAO - Rubrobacter taiwanensis;
RraDAAO - Rubrobacter radiotolerans DSM 5868
NhaDAAO - Natrarchaeobius halalkaliphilus AArcht4
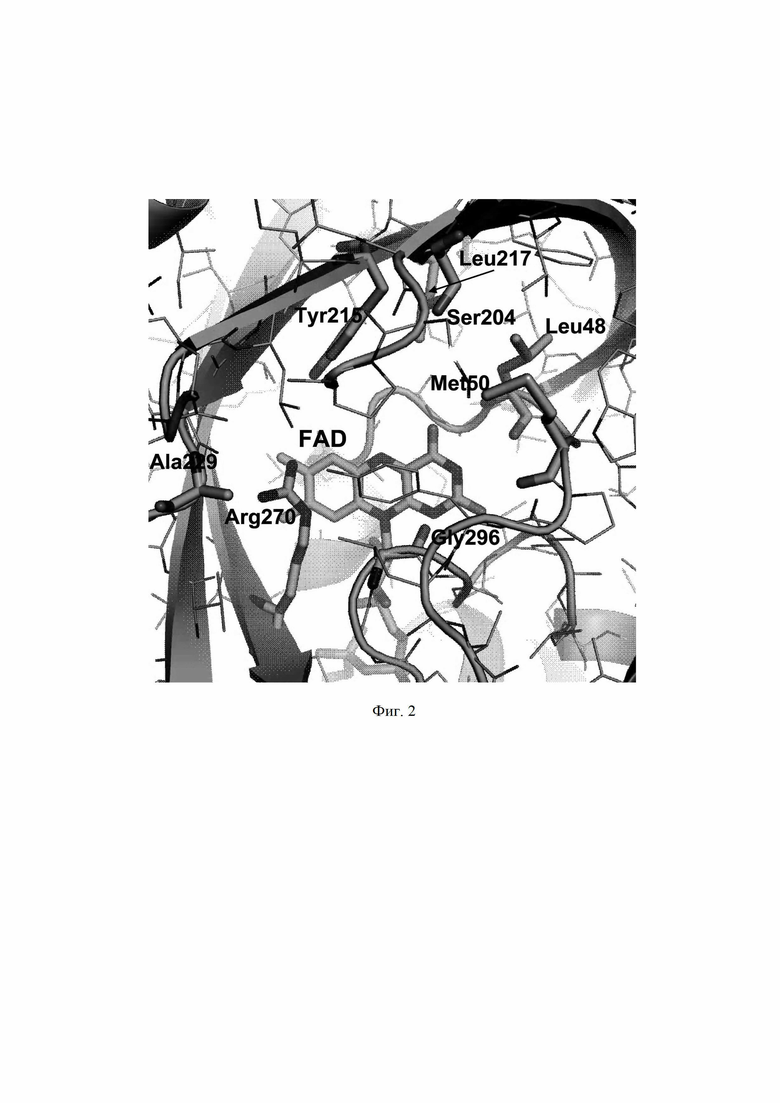
Фиг. 2. Модельная структура активного центра оксидазы D-аминокислот из N. hydrolyticum ACPA39. Структура получена моделированием с использованием алгоритма AjphaFold2.
Фиг. 3. Физическая и генетическая карта плазмидного вектора pNhyDAAO. Обозначены положения индикаторных сайтов рестрикции, рибосом-связывающий участок, ген устойчивости к канамицину (Kan) и ген lac-репрессора (lacI).
Фиг. 4. Последовательность SEQ ID №3.
Осуществление изобретения
С учетом требуемой субстратной специфичности новой DAAO и необходимости создания системы экспрессии (комбинации "экспрессирующий вектор"/"штамм-реципиент") этого фермента достижение поставленных целей было реализовано за счет того, что
1) В результате поиска в бактериальных геномах генов потенциальных DAAO и была отобрана оксидаза D-аминокислот, которая по данным биоинформационно-структурного анализа должна была обладать требуемой субстратной специфичностью.
2) сконструирован экспрессионный вектор, содержащий ген отобранного фермента и который должен обеспечивать экспрессию этого гена в клетках E.coli. В качестве базового вектора для клонирования использовали плазмиду pET24a(+), которая обеспечивает длительное сохранение экспрессионного вектора в клетке в процессе культивирования за счет наличия гена устойчивости к канамицину и которая не допускает неспецифической базовой экспрессии целевого гена за счет наличия в плазмидном векторе гена белка- ингибитора lac- промотора lacI.
3) путем трансформации полученным вектором получается рекомбинантный штамм E.coli – продуцент выбранной DAAO. Проводится культивирование полученного штамма-продуцента для проверки его способности экспрессировать активный рекомбинантный фермент
4) изучаются рН-профиль активности и субстратная специфичность новой DAAO.
I. Выбор целевого фермента проводится в три этапа:
1) первый этап включает биоинформатический поиск в геномах экстремофильных бактерий генов, кодирующих белки, гомологичные бактериальным оксидазам D-аминокислот. Отбирают последовательности с наиболее высоким уровнем гомологии с аминокислотными последовательностями известных бактериальных DAAO.
2) для подтверждения того, что отобранные последовательности являются именно DAAO, а не высокогомологичными глициноксидазами для кандидатных последовательностей были построены модельные структуры; которые затем сравнили с экспериментальными и модельными структурами известных оксидаз D-аминокислот и глициноксидаз. В результате такого анализа из рассмотрения исключаются потенциальные глициноксидазы.
3) далее был проведен анализ структур активных центров, на основании которого был сделан выбор в пользу фермента из бактерий Natronosporangium hydrolyticum ACPA39 (NhyDAAO).
II. Конструирование экспрессирующего вектора включает три этапа:
1) анализ нуклеотидной последовательности гена, кодирующего NhyDAAO, показал, что стартовым кодоном в этом гене является триплет GTG, в то время как в клетках E.coli в качестве стартового кодона используется триплет ATG. Поэтому было принято решение при клонировании в гене nhydaao заменить стартовый кодон GTG на ATG. Для обеспечения эффективной терминации трансляции гена nhydaao в клетках E.coli перед имеющимся в гене стоп-кодоне TAG был добавлен другой стоп-кодон TAA, который среди трех стоп-кодонов наиболее эффективен к клетках E.coli.
2) клонирование модифицированного гена nhydaao проводили с помощью полимеразной цепной реакции с использованием прямого и обратного праймеров (SEQ ID №1 и SEQ ID №2). Для клонирования в экспрессирующий вектор в прямой и обратный праймеры были введены соответственно сайты рестрикции NdeI и XhoI.
3) экспрессирующий вектор pNhyDAAO был получен путем лигирования продукта ПЦР с модифицированным геном nhydaao в плазмидный в вектор pET24a(+) после их обработки рестриктазами Ndei и XhoI. Клонированная последовательность указана как SEQ ID №3 (фиг. 4). Сконструированный вектор pNhyDAAO (Фиг. 3) содержит ген устойчивости к канамицину и ген белка-репрессора lac-промотора.lacI.
Ш. Конструирование рекомбинантного штамма E.coli – продуцента NhyDAAO
Рекомбинантный штамм E. coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 - продуцент NhyDAAO был получен трансформацией компетентных клеток E.coli BL21(DE3) экспрессирующей плазмидой pNhyDAAO. Сконструированный штамм Escherichia coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 продуцировал рекомбинантную NhyDAAO в активной форме.
Штамм E.coli BL21(DE3)/pNhyDAAO депонирован во Всероссийской коллекции промышленных микроорганизмов как E. coli BL21(DE3)/pNhyDAAO ВКПМ В-14337.
Заявляемый штамм E. coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 имеет следующие морфологические и физиолого-биохимические характеристики:
Морфологические признаки: клетки прямые, палочковидные, подвижные, грамотрицателиные, неспорообразующие. При выращивании в течение 24-48 час при температуре 30-37 °С на агаризованных средах (LB, 2YT), содержащих канамицин (25 мкг/мл), колонии гладкие, круглые, блестящие, край ровный.
Состав среды LB, мас.%:
Бакто-триптон – 1
дрожжевой экстракт – 0,5
NaCl – 1,
вода – остальное.
Состав среды 2YT, мас.%:
Бакто-триптон – 1,6
дрожжевой экстракт – 1,0
NaCl – 0,5
вода – остальное.
Физиолого-биохимические признаки: штамм растет при температуре от 15 до 37°С (оптимум 30-37 °С). В качестве источника азота использует органический азот в виде триптона, аминокислот.
Штамм E. coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 синтезирует оксидазу D-аминокислот NhyDAAO после нндукции 0,1 мМ ИПТГ (изопропил-β-D-тиогалактозидом).
Генотипические признаки:
Штамм E. coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 устойчив к канамицину.
Штамм E.coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 содержит на плазмиде pNhyDAAO модифицированный ген оксидазы D-аминокислот (nhydaao) из N. hydrolyticum ACPA39 под контролем T7 промотора.
Условия хранения:
1. В ночную культуру, выращенную на жидкой среде 2YT с канамицином (25 мгк/мл), добавляют стерильный глицерин (до 15%), расфасовывают по 1,5 мл в пластиковые микропробирки и хранят в замороженном виде при -70 °С.
2. Хранят в лиофилизированном виде в ампулах при +4 °C.
3. Штамм может поддерживаться регулярными пересевами (1 раз в 3-4 недели) на агаризованной среде LB или 2YT, содержащей канамицин 25 мкг/мл.
IV. Определение спектра субстратной специфичности
Для определения спектра субстратной специфичности используют препараты бесклеточного экстракта с новой NhyDAAO. Для получения препаратов NhyDAAO штамм E.coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 культивировали при 30 °С на среде 2YT в присутствии канамицина (25 мкг/мл). Индукцию биосинтеза проводили с помощью ИПТГ (0,1 мМ) по достижении поглощения культуральной среды A600 1 ед. поглощения. Культивирование продолжали при 18 °С в течение 48 часов. Клетки осаждали центрифугированием при 5000 об/мин при 4 °С. Далее клетки ресуспендировали в 0,1 мМ фосфатном буфере, pH 8,0 и разрушали на ультразвуковом дезинтеграторе при 4 °С. Клеточный дебрис осаждали центрифугированием при 14000 об/мин, 30 мин, 4 °С. Супернатант использовали для определения активности с набором D-аминокислот (10 мМ) в 0,1 М буфере при рН 6,0, 7,0 и 8,0. Активность определяли пероксидазным методом с о-дианизидином в качестве субстрата пероксидазы.
Таким образом, были получены:
- рекомбинантная плазмида pNhyDAAO размером 6188 п.о., содержащая модифицированный ген nhydaao оксидазы D-аминокислот из N. hydrolyticum ACPA39 под контролем T7 промотора. обеспечивающая синтез оксидазы D-аминокислот из N. hydrolyticum ACPA39 (NhyDAAO) в клетках E. coli. Плазмида pNhyDAAO получена путем клонирования по сайтам NdeI/Xho фрагмента размером 958 п.о. (последовательность SEQ ID №3) с геном, кодирующим NhyDAAO (951 п.о.) в плазмиду pET24a(+), расщепленную рестриктазами NdeI и Xho;
- рекомбинантный штамм Escherichia coli BL21(DE3)/pNhyDAAO - продуцент оксидазы D-аминокислот NhyDAAO, полученный путем трансформации штамма Escherichia. coli BL21(DE3) плазмидной ДНК.
Изобретение поясняется следующими примерами.
Пример 1. Выбор источника новой DAAO на основе биоинформацинно-структурного анализа.
Поиск новых DAAO по гомологии проводили с помощью программы BLASTp (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE=Proteins) против базы данных транслированных белковых последовательностей из геномов экстремофильных бактерий. В качестве основной базы использовали базу UniProt, NCBI. Также проводили поиск в геномах бактерий из коллекции ФИЦ Биотехнологии РАН и МГУ имени М.В.Ломоносова. В качестве референсных использовали аминокислотные последовательности DAAO из бактерий A. protophormiae, R. xylanophilus и S. coelicolor. По результатам поиска отобрали 15 последовательностей, проявляющих наиболее высокую гомологию с референсными последовательностями бактериальных DAAO. Далее проводили экспертную оценку на наличие 5 консервативных для DAAO элементов активного центра – последовательность GxGxxG (кофактор-связывающий домен) и пару Arg-Pro (связывание карбоксильной группы D-аминокислоты). По результатам второго этапа было отобрано 7 последовательностей (фиг. 1), которые удовлетворяли критерию проверки по консервативным остаткам.
Для подтверждения того, что отобранные последовательности относятся к оксидазам D-аминокислот, а не к глициноксидазам были построены модельные структуры с использованием агоритма AlhaaFold2 [Jumper, J. & Hassabis, D. (2022). Protein structure predictions to atomic accuracy with AlphaFold. Nat. Methods 19, 11-12, doi:110.1038/s41592-021-01362-6. В таблице 1 представлены результаты попарного сравнения модельных структур с известными структурами оксидаз D-аминокислот и глициноксидаз. Из таблицы хорошо видно, что модельные структуры отобранных ферментов имеют очень малые различия с известными DAAO (среднеквадратичное отклонение 1 Å или меньше), в то время как среднеквадратичное отклонение со структурами глициноксидаз намного больше (2,3 Å и более).
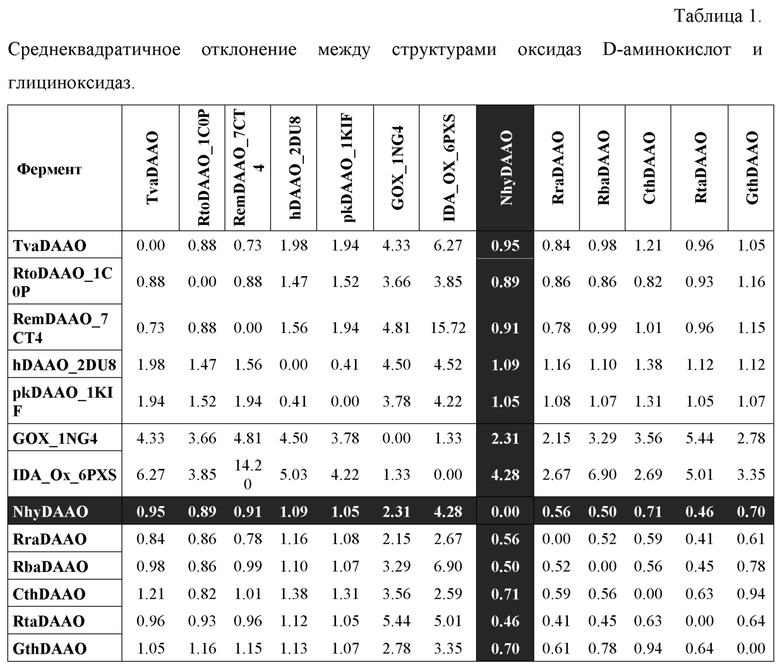
Для финального выбора фермента проводили анализ структуры активного центра новых DAAO. Результаты анализа свидетельствуют, что наиболее перспективной является DAAO из бактерий Natronosporangium hydrolyticum ACPA39. Этот фермент имеет объемный субстрат-связывающий центр, в котором имеется большое количество гидрофобных остатков – Leu48, Tyr215, Leu217, а также Met50 (Фиг. 2), которые должны обеспечивать условия для связывания D-аминокислот с объемной гидрофобной боковой группой – D-Leu, D-Met и D-Phe. Поэтому для создания штамма-продуцента новой DAAO была выбрана NhyDAAO.
Пример 2. Конструирование экспрессионного вектора pNhyDAAO
Экспрессионный вектор pNhyDAAO получали клонированием в плазмиду pET24a(+) фрагмента ДНК с модифицированным геном nhydaao. Фрагмент ДНК с модифицированным геном nhydaao получали с помощью полимеразной цепной реакции (ПЦР). В качестве матрицы использовали геномную ДНК Natronosporangium hydrolyticum ACPA39, которую выделяли из клеток указанных бактерий с помощью набора для выделения геномной ДНК фирмы ThermoFisher Scientific «GeneJET Genomic DNA Purification Kit» согласно протоколу фирмы производителя. Для проведения ПЦР использовали прямой:
ACPA39_DAAO_F_NdeI 5-GGTACGCTGACCCATATGGCTGAGGTCGACGTACTGG-3
и обратный праймеры:
ACPA39_DAAO_R_XhoI 5-GCAGCACTCGAGCTATTACGTGCCGGACGCCAGC-3
В прямой праймер был введен сайт рестрикции NdeI CATATG (указан полужирным шрифтом). Кроме того, в исходном гене nhydaao в качестве стартового кодона используется триплет GTG, который не используется в качестве стартового кодона в клетка E. coli. Поэтому стартовый кодон GTG был заменен на оптимальный для E. coli кодон ATG (выделено подчеркиванием). В обратном праймере для клонирования был введен сайт рестрикции XhoI CTCGAG, а вместо стоп-кодона TAG введен тандем стоп-кодонов TAATAG (выделено подчеркиванием).
ПЦР проводили в тонкостенной пробирке объемом 0,5 мл. Состав реакционной смеси приведен в таблице 2.
Таблица 2.
Состав реакционной смеси для проведения полимеразной цепной реакции
Перед началом ПЦР реакционную смесь без ДНК-полимеразы инкубировали 3 мин при 98 °С, затем в пробирку добавляли фермент и проводили 40 циклов по следующей программе; 98 °С – 15 сек; 56 °С – 20 сек, 72 °С – 1 мин. По окончании ПЦР смесь дополнительно выдерживали 5 мин при 72 °С.
Продукт ПЦР очищали с помощью электрофореза в 1% агарозном геле. Вырезали фрагмент размером 1000 п.о и выделяли ДНК из геля с помощью набора для выделения и очистки ДНК из агарозного геля «PureLink Quick Gel Extraction Kit» (ThermoFisher Scientific).
Очищенный ПЦР фрагмент ДНК и плазмиду pET24a(+) обрабатывают рестриктазами NdeI и XhoI. Условия проведения реакций представлены в таблице 3.
Таблица 3
Условия расщепления продукта ПЦР и плазмиды pET24a(+)ю
Реакцию проводят в пробирке объемом 0,5 мл в течение 1,5 часов при 37 °С. Продукты обработки рестриктазами очищали с помощью электрофореза в 1% агарозном геле. Затем ДНК экстрагировали из геля и очищали с помощью набора для выделения и очистки ДНК из агарозного геля «PureLink Quick Gel Extraction Kit» (ThermoFisher Scientific).
Очищенные продукты рестрикции лигировали при следующих условиях (Таблица 4).
Таблица 4
Условия лигирования продуктов рестрикции ПЦР и плазмиды pET24a(+).
Реакцию лигирования проводили в течение 3 часов при 22°С. Затем по стандартной методике [Sambrook J, Maniatis T, Fritsch EF. 1989. Molecular cloning a laboratory manual Cold Spring Harbor, N.Y: Cold Spring Harbor Laboratory] продуктом реакции лигирования трансформировали компетентные клетки E.coli Dh5α (50 мкл компетентных клеток + 7 мкл лигазной смеси), После теплового шока клетки инкубировали в 1 мл среды LB при 37 °С и затем высевали на агаризованную среду LB, содержащую 25 мкг/мкл канамицина, и инкубировали в течение ночи при 37 °С.
Для выделения плазмид с чашки брали единичную колонию, инкубировали в 4 мл среды LB, содержащую 25 мкг/мкл канамицина, и инкубировали в течение ночи при 37 °С. Полученные клетки осаждали на центрифуге при 5000 об/мин, 4 °С 5 мин и из них выделяли плазмидную ДНК с помощью набора для выделения плазмидной ДНК «PureLink™ HiPure Plasmid Miniprep Kit». Выделение плазмид проводили в трех повторах.
Секвенирование полученных плазмид с помощью стандартных секвенирующих праймеров T7for и T7rev (Novagen) показало, что во всех плазмидах клонирована последовательность ДНК, соответствующая последовательности SEQ ID №3.
Полученная плазмида получила название pNhyDAAO.
Пример 3. Получение рекомбинантного штамма E.coli - продуцента NhyDAAO. Полученной плазмидой pNhyDAAO трансформировали штамм E. coli BL21(DE3) ]F– ompT gal dcm lon hsdSB(rB–mB–) λ(DE3 [lacI lacUV5-T7p07 ind1 sam7 nin5]) [malB+]K-12(λS). Отбор клонов проводили на агаризованной среде LB, содержащую 25 мкг/мкл канамицина.
Для проверки эффективности продуцирования NhyDAAO полученным штаммом с чашки отбирали отдельные колонии и выращивали в 4 мл среды 2YT, содержащей 25 мкг/мкл канамицина, в течение ночи при 30 °С. Затем отбирали 1 мл культуральной жидкости и переносили в колбу с отбойниками объемом 1 л со 100 мл среды 2YT, содержащей 25 мкг/мкл канамицина. Клетки культивировали при 30 °С до поглощения на 600 нм А600 1 ед. поглощения, добавляли индуктор ИПТ до концентрации 0,1 мМ, снижали температуру до 18 °С и культивировали в течение 48 часов. По окончании клетки осаждали центрифугированием при 6000 об/мин в течение 10 мин при 4 °С. Супернатант удаляли, а биомассу ресуспендировали в 0,1 М фосфатном буфере рН 8.0 (10 мас %). Суспензию клеток 20 мл подвергали заморозке - разморозке и затем обрабатывали ультразвуком при постоянном охлаждении 4 °С - 4 цикла 1 мин озвучивания + 4 минуты охлаждения. Клеточный дебрис осаждали центрифугированием при 15000 об/мин в течение 20 мин при 4 °С и определяли активность в бесклеточном экстракте. Активность определяли в 0,1 М фосфатном буфере, рН 8,0, при 30 °С пероксидазным методом в присутствии 10 мМ D-Leu. В качестве субстрата пероксидазы использовали о-дианизидин (10 мг/л). Культивирование проводили в трех повторах. Выход биомассы составил 24±3 г/л среды. Активность в бесклеточном экстракте с D-Leu составила 78 ед. на л среды или 3,25 ед на г сырой биомассы. В случае прототипа – DAAO из R. xylanophilus выход фермента при измерении активности с D-Leu составляет 1,08 ед. на г сырой биомассы. Однако в прототипе активность RxyDAAO измеряли при 60°С. С учетом того, что при изменении температуры на 10 градусов скорость реакции изменяется в 2-4 раза, то при 30 °С активность RxyDAAO должна быть минимум в 8 раз ниже, т.е. максимум 0,135 ед. на г биомассы. Таким образом, полученный рекомбинантный штамм E.coli BL21(DE3)/pNhyDAAO имеет продуктивность по активности минимум в 24 раза выше, чем прототип. Полученный рекомбинантный штамм Escherichia coli BL21(DE3)/pNhyDAAO зарегистрирован во Всероссийской коллекции промышленных микроорганизмов как Escherichia coli BL21(DE3)/pNhyDAAO ВКПМ В-14337.
Пример 4. Субстратная специфичность и рН-зависимость активности NhyDAAO.
В случае прототипа – штамма E.coli – продуцента RxyDAAO данные по субстратной специфичности RxyDAAO приведены для измерений при рН 8,0 [Takahashi, S., Furukawara, M., Omae, K., Tadokoro, N., Saito, Y., Abe, K., & Kera, Y. (2014). A Highly Stable D-Amino Acid Oxidase of the Thermophilic Bacterium Rubrobacter xylanophilus. Appl. Environ. Microbiol. 80, 7219-7229, doi: 10.1128/AEM.02193-14]. Это связано с тем, что у RxyDAAO максимальная активность наблюдается при рН 7,8 – 8,0. Однако известно, что у разных ферментов оптимум рН-активности могут сильно отличаться. Поэтому сначала были определены величины активности NhyDAAO в диапазоне рН 6,0 – 9,0 в 0,1 М буферных растворах - фосфатном буфере (рН 6,0 – 8,0) и буфере трис-HСl (pH 8,0 – 9,0). Активность измеряли с D-Leu (10 мМ) в качестве субстрата минимум в трех повторах. Результаты приведены в таблице 5.
Таблица 5
Зависимости активности оксидаз NhyDAAO и RxyDAAO от рН. среды
*Приведенные результаты являются средним из трех измерений.
**Данные из работы Takahashi, S., Furukawara, M., Omae, K., Tadokoro, N., Saito, Y., Abe, K., & Kera, Y. (2014). A Highly Stable D-Amino Acid Oxidase of the Thermophilic Bacterium Rubrobacter xylanophilus. Appl. Environ. Microbiol. 80, 7219-7229, doi: 10.1128/AEM.02193-14
Как следует из таблицы 5 максимальная активность NhyDAAO наблюдается при рН 8,0, причем эта активность не зависит от природы буфера. Поэтому субстратную специфичность NhyDAAO как и в случае фермента RxyDAAO из прототипа также определяли при рН 8,0 в 0,1 М фосфатном буфере. Активность измеряли в присутствии 10 мМ D-аминокислоты пероксидазным методом с о-дианизидином (10 мг/мл) в качестве субстрата пероксидазы. Результаты измерений представлены в Таблице 6. Для сравнения приведены данные и для RxyDAAO из прототипа. За 100% была принята активность с D-Leu, который является оптимальным субстратом и для RxyDAAO [Takahashi, S., Abe, K., & Kera, Y. (2015). Bacterial d-amino acid oxidases: Recent findings and future perspectives. Bioengineered. 6, 237-241, doi:10.1080/21655979.2015.1052917; Molla G, Piubelli L, Volonte F, Pilone MS. Enzymatic detection of D-amino acids. Methods Mol. Biol. 2012; 794:273-89; PMID:21956570].
Таблица 6
Субстратная специфичность оксидаз NhyDAAO и RxyDAAO
*Приведенные результаты являются средним из трех измерений.
**Данные из работы Takahashi, S., Furukawara, M., Omae, K., Tadokoro, N., Saito, Y., Abe, K., & Kera, Y. (2014). A Highly Stable D-Amino Acid Oxidase of the Thermophilic Bacterium Rubrobacter xylanophilus. Appl. Environ. Microbiol. 80, 7219-7229, doi: 10.1128/AEM.02193-14
*** н.д. – нет данных
Сравнение данных по субстратной специфичности оксидаз NhyDAAO и RxyDAAO в таблице 6 свидетельствует, что:
1) получаемая из сконструированного нами штамма-продуцента оксидаза NhyDAAO имеет активность с D-Phe по отношению к D-Leu в 7 раз выше, чем оксидаза RxyDAAO из прототипа, т.е. определение D-Phe c NhyDAAO возможно при концентрациях D-Leu в 7 раз выше, чем в случае RxyDAAO. Более высокая в 7 раз активность NhyDAAO с D-Phe позволяет снизить предел обнаружения этой D-аминокислотой по сравнению с RxyDAAO в 7 раз.
2) NhyDAAO не проявляет активности с наиболее распространенной в тканях человека D-аминокислотой D-Ser, т.е. детекция D-Phe с помощью NhyDAAO будет специфична в присутствии любых концентраций D-Ser, в то время как такая детекция с помощью RxyDAAO проблематична, поскольку активность RxyDAAO с D-Ser составляет 62% от активности с D-Phe.
3) NhyDAAO также позволяет достаточно селективно определять D-Phe в присутствии D-Pro, поскольку соотношений активностей составляет 7 : 1.
Таким образом можно сделать вывод, что созданный нами рекомбинантный штамм Escherichia coli BL21(DE3)/pNhyDAAO ВКПМ В-14337 - продуцент NhyDAAO, позволяет получить фермент, который имеет гораздо больший биотехнологический и биоаналитический потенциал по сравнению с ферментом RxyDAAO (прототип). С учетом того, что продуктивность по активности с литра среды созданного штамма по синтезу NhyDAAO более, чем в 20 раз выше по сравнению с прототипом, то стоимость получения новой оксидазы NhyDAAO с помощью сконструированного штамма продуцента будет меньше стоимости получения RxyDAAO в прототипе в соответствующее количество раз.
SEQ ID №3:
tatggctgaggtcgacgtactggtcgtaggcgccggggtcagcgggctgaccaccgcggtgtgcctggccgagaccgggcgacgggtgaccgtgcgcaccgccacggagccggcccgcaccacctccgcggtcgccggtgcgctctggatgccctacctggtgcggccggtcgacaaggtcaccgcctggggtgccgccacgctcaccgagctgcgcacgctcgccgaccagccgaccaccggcgtacgccgcaccaacggggttgttctcgccccgaccgcgatcgcgccgccagcctggaccgagacggtggccgcggtgccgtgcccgccggcggaccttccccacgggtacgaggtcgggtggcggttcggttcggtgctggtcgagatgcctatctacctcggctacctggctgaccggctccgcgccgccggcggccggatcgaaccgggcctcgtcaccgacctggccgacgccctcacggtggcgccgctggtggtcaactgcgccgggatcggcgcccgggagctggtccccgacccgggcctgcggccggtccgcggccaggtggtggtggtcgaaaatccgggcatcgacgagttcgtcagcgaacaccccggcgcgtccccctggctgaagtatgtgctgccgcaccgggacactgtggtgctcggcggcaccgccgaaccggaccgcagcgaccccacccccgacccgtcgatcaccgcccggatcctcgccgacagcgtcgagctggtgccggcgctcgccggcgccccggtgctggcggaacgggtggggctgcgcccggcccgccccgaggtccggctggcggtcgaggaccgcccggccggccggatcatccacaactacggccacggcggcggcggggtcacgctctcctggggctgtgcccgcgaagccgccgagctggcgtccggcacgtaatagctc


Изобретение относится к биотехнологии, а именно к рекомбинантному вектору pNhyDAAO для экспрессии оксидазы D-аминокислот бактерий Natronosporangium hydrolyticum ACPA39, и к рекомбинантному штамму Escherichia coli BL21(DE3)/pNhyDAAO, содержащему данный рекомбинантный вектор. Изобретение эффективно для использования в тонком органическом синтезе, селективном определении D-Leu, D-Met и D-Phe и др. в сложных биологических системах, включая жидкости и ткани человека. 2 н.п. ф-лы, 4 ил., 6 табл., 4 пр.
1. Рекомбинантный вектор pNhyDAAO для экспрессии оксидазы D-аминокислот бактерий Natronosporangium hydrolyticum ACPA39 (NhyDAAO), содержащий NdeI-XhoI фрагмент размером 5230 п.о. вектора pET24a(+) и NdeI-XhoI фрагмент размером 958 п.о., включающий нуклеотидную последовательность, соответствующую SEQ ID № 3.
2. Рекомбинантный штамм Escherichia coli BL21(DE3)/pNhyDAAO – продуцент NhyDAAO, полученный путем трансформации штамма Escherichia. coli BL21(DE3) плазмидной ДНК по п.1, задепонированный во Всероссийской коллекции промышленных микроорганизмов (ВКПМ) под номером В-14337.
| LEUCHTENBERGER W | |||
| et al | |||
| Biotechnological production of amino acids and derivatives: current status and prospects, Applied Microbiology and Biotechnology, 2005, vol.69 | |||
| GENCHI G | |||
| An overview on D-amino acids, Amino Acids, 2017 | |||
| POLLEGIONI L | |||
| et al | |||
| Advances in Enzymatic Synthesis of D-Amino Acids, International Journal of Molecular Sciences, |
Авторы
Даты
2023-09-12—Публикация
2022-12-27—Подача