Изобретение относится к области биотехнологии, молекулярно-генетической диагностики, в частности к оценке однонуклеотидного полиморфизма rs12561767 (G>A) гена SERPINE1 мРНК-связывающего белка 1 молекулярно-генетическим методом исследования.
SERBP1 впервые был идентифицирован как белок, связанный с мРНК ингибитора активатора плазминогена типа 1 (PAI-1) [Heaton JH, Dlakic WM, 2001] и регулирующий стабильность транскрипта, тем самым влияя на протромботические эффекты PAI-1, существенно связанные с повышенным риском наиболее распространенных и жизнеугрожающих заболеваний человека - ишемической болезни сердца [Sillen M., Declerck P. J. Targeting PAI-1 in cardiovascular disease: structural insights into PAI-1 functionality and inhibition // Frontiers in Cardiovascular Medicine. - 2020. - Vol. 7. - P. 622473] и мозгового инсульта [Chen R. et al. Plasminogen activator inhibitor links obesity and thrombotic cerebrovascular diseases: The roles of PAI-1 and obesity on stroke // Metabolic brain disease. - 2017. - Vol. 32. - P. 667-673]. Последующие исследования обнаружили, что SERBP1 участвует в передаче сигналов прогестерона посредством взаимодействия с компонентом 1 мембраны рецептора прогестерона (PGRMC1) [Taraborrelli S. Physiology, production and action of progesterone // Acta obstetricia et gynecologica Scandinavica. - 2015. - Vol. 94. - P. 8-16]. В 2020 году соавторами из Японии были открыты шапероноподобные свойства SERBP1 и его возможная роль в нейропротекции [Tsuboyama K. et al. A widespread family of heat-resistant obscure (Hero) proteins protect against protein instability and aggregation // PLoS Biology. - 2020. - Vol. 18. - №. 3. - P. e3000632], что требует проведения молекулярно-генетических исследований в данной области.
Суммируя биологические роли SERBP1, можно предположить его возможную связь с кардио- и цереброваскулярной патологией, заболеваниями женской репродуктивной системы, а также нейродегенеративными заболеванями.
Ген SERBP1 (Gene ID: 26135) локализован на хромосоме 1p31.3. Однонуклеотидный полиморфизм rs12561767, позиция chr1:67421880 (GRCh38.p14) https://www.ncbi.nlm.nih.gov/snp/rs12561767) локализован в интроне и характеризуется заменой G>A,С. Однако, аллель C встречается с частотой <0.000001 в европейских популяциях и также характеризуется низкой частотой в других популяциях мира, в связи с чем именно замена G>A является актуальной для исследований многофакторных болезней человека.
Известен способ анализа генетических вариаций в геноме человека методом секвенирования амплифицированных участков ДНК [Mardis E. R. DNA sequencing technologies: 2006-2016 // Nature protocols. - 2017. - Vol. 12. - №. 2. - P. 213-218]. Недостатками метода являются высокая стоимость оборудования и реагентов, что исключает широкое внедрение метода в экспериментальные исследования, особенно изучение многофакторных заболеваний, которые требуют большого размера выборок для обеспечения высокой мощности исследований.
Известен способ анализа генетических вариаций в геноме человека методом матричноактивированной лазерной десорбционно-ионизационной масс-спектрометрии (MALDI). Метод заключается в том, анализируемая ДНК переносится на подложку, где она кристаллизуется с матрицей. Затем кристаллизованные аналиты переносят, облучают лазером, вызывая десорбцию и ионизацию молекул в вакуумной камере. Положительно заряженные ионы ДНК ускоряются и мигрируют через вакуумную трубку к высокочувствительному детектору с разной скоростью в зависимости от массы ионов, что приводит к различному времени пролета. Используя время пролета отдельных ионизированных ДНК-аналитов, система определяет массу и отображает масс-спектр, идентифицирующий различные генетические мишени [Li D. et al. MALDI-TOF mass spectrometry in clinical analysis and research // ACS Measurement Science Au. - 2022. - Vol. 2. - №. 5. - P. 385-404]. Недостатками метода являются трудоемкость, высокая стоимость оборудования, высокая стоимость эксперимента, наличие высококвалифицированного персонала.
За прототип выбран коммерческий набор по генотипированию rs12561767 (A/G) SERBP1 (Assay ID C___2694738_10; каталожный номер 4351379) компании ThermoFisher. Однако, генотипирование с использованием коммерческих наборов характеризуется высокой стоимостью, а информация о структуре необходимых для проведения ПЦР праймеров и аллель-специфических зондов является закрытой для исследователей, в связи с чем он не может быть воспроизведен при наличии стандартного набора оборудования и реактивов.
Таким образом, существует реальная потребность в создании быстрого, недорогого и легко воспроизводимого способа идентификации полиморфизма rs12561767 (G>A) гена SERBP1, с доступной всем исследователям структурой праймеров и аллель-специфических зондов, который мог бы использоваться в качестве «рутинного» метода генотипирования в любой ПЦР-лаборатории.
Техническим результатом данного изобретения является разработка простого в исполнении и экономически целесообразного способа генотипирования однонуклеотидного полиморфизма rs12561767 (G>A), локализованного в позиции chr1:67421880 (GRCh38.p14) гена SERBP1 (Gene ID:26135) методом полимеразной цепной реакции в режиме «реального времени» с применением аллель-специфических сигнальных зондов, содержащие флуорофоры FAM и ROX.
Технический результат достигается тем, что идентификацию аллельных вариантов rs12561767 (G>A) гена SERBP1 осуществляют с использованием прямого праймера rs12561767 5'-GCATGCCTGTAGTCCCAGTA-3' (SEQ ID NO 1), обратного праймера rs12561767 5'-GTCTCCTGTCTCCCATGCTA-3' (SEQ ID NO 2), rs12561767-G-аллель-специфичного флуоресцентно-меченого зонда 5'-(FAM)CTTGAACTTCAGAATGGCCAAAATC(RTQ1)-3' (SEQ ID NO 3),
rs12561767-A-аллель-специфичного флуоресцентно-меченого зонда 5'-(ROX) CTTGAACTTCAAAATGGCCAAAATC(BHQ2)-3' (SEQ ID NO 4).
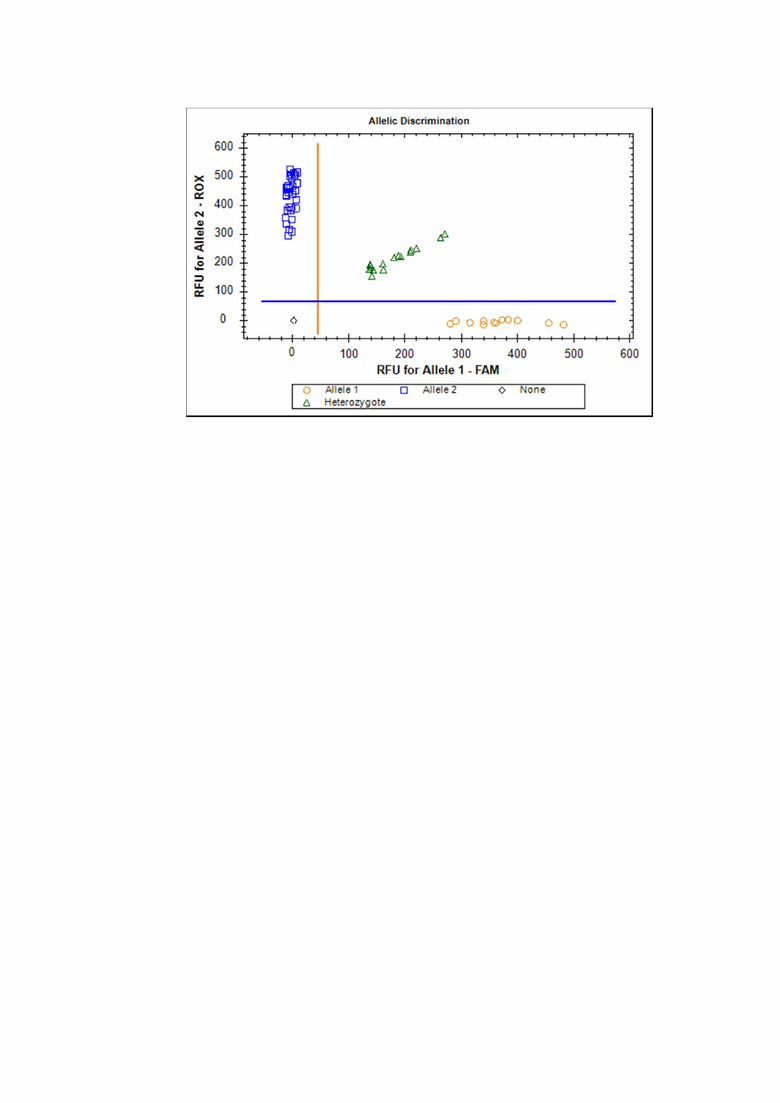
Изобретение поясняется следующей фигурой: дискриминация аллелей по локусу rs12561767 гена SERBP1 при генотипировании методом ПЦР в режиме «реального времени» с применением аллель-специфических флуоресцентных зондов по данным величин RFU (относительные единицы флуоресценции) на амплификаторе CFX96: генотипы rs12561767-G/G показаны оранжевыми кругами, генотипы rs12561767-G/A показаны зелеными треугольниками, генотипы rs12561767-A/A показаны голубыми квадратами; черным ромбом отмечен отрицательный контроль.
Работа над дизайном олигонуклеотидов включала несколько этапов:
1) С применением открытой базы данных Ensembl genome browser 109 [https://www.ensembl.org/index.html] выбран синвенс, фланкирующий искомую однонуклеотидную замену [G/A] rs12561767 SERBP1, и затем с помощью доступного онлайн программного обеспечения Primer3web version 4.1.0 [https://primer3.ut.ee/] подобрана последовательность олигонуклеотидов, используемых для проведения ПЦР-реакции:
прямой общий праймер rs12561767 5'-GCATGCCTGTAGTCCCAGTA-3' (SEQ ID NO 1),
обратный общий праймер rs12561767 5'-GTCTCCTGTCTCCCATGCTA-3' (SEQ ID NO 2).
Размер амплифицируемого в ходе ПЦР фрагмента гена SERBP1 составляет 105 пар нуклеотидов:
(GCATGCCTGTAGTCCCAGTACTCGGGAGGCTGAGGCAGGAGAATCGCTTGAACTTCA[G/A]AATGGCCAAAATCGCGCCACTGCACTCTAGCATGGGAGACAGGAGAC).
2). Для дизайна зондов пользовались практическими рекомендациями [Basu C. (ed.). PCR primer design. - New york : Humana Press, 2015]. В реакции использовались гидролизные зонды. Последовательность зонда подбирали таким образом, чтобы он отжигался на матрицу между прямым и обратным праймерами. Каждый зонд снабжали флуорофором и гасителем флуоресценции, спектр поглощения которого соответствует длинам волн спектра флуорофора. Для гашения флуоресценции FAM пользовались гасителем RTQ1; для гашения флуоресценции ROX - гасителем BHQ2.
На основании изложенных критериев и практических рекомендаций были подобраны зонды со следующей структурой:
rs12561767-G-аллель-специфичный флуоресцентно-меченый зонд 5'-(FAM)CTTGAACTTCAGAATGGCCAAAATC(RTQ1)-3' (SEQ ID NO 3),
rs12561767-A-аллель-специфичный флуоресцентно-меченый зонд 5'-(ROX)CTTGAACTTCAAAATGGCCAAAATC(BHQ2)-3' (SEQ ID NO 4).
3) Изготовление праймеров и зондов осуществлялось в сервисном центре НПК «Синтол», Москва.
4) С помощью практических экспериментов подобраны оптимальные условия для проведения генотипирования, которые включают следующие этапы: 50°C в течение 2 минут, 95°C в течение 10 минут, затем 39 циклов [95°C в течение 10 секунд и 64°C в течение 30 секунд].
5) Разработанный способ был апробирован на 95 образцах ДНК здоровых индивидуумов биобанка НИИ генетической и молекулярной эпидемиологии КГМУ. Генотипирование осуществляли по данным величин RFU (относительные единицы флуоресценции) зондов с флуоресцентными красителями. По результатам генотипирования rs12561767 14 человек (14,7%) оказались гомозиготами по аллелю G (генотип G/G); 42 человека (44,2%) являлись гетерозиготами (генотип G/A), 39 (41,1%) индивидуумов оказались гомозиготами A/A.
6) Валидацию способа проводили методом масс-спектрометрического анализа на геномном времяпролетном масс-спектрометре MassArray analyzer 4 (Agena Bioscience). Результаты обоих способов генотипирования полностью (100% генотипов) совпали. Однако патентуемый способ генотипирования полиморфного локуса rs12561767 (G>A) гена SERBP1 методом ПЦР в режиме «реального времени» с применением аллель-специфических зондов позволяет значительно (на 6 часов) сократить время проведения анализа, а также снижает себестоимость анализа (в 4-5 раз).
Способ осуществляют следующим образом:
1. Выделение ДНК из периферической венозной крови. На первом этапе к 0,5 мл крови добавляли 0,5 мл PBS и центрифугировали 10 мин при 12 тыс. об/мин. Надосадочную жидкость сливали, добавляли 1 мл PBS и вновь центрифугировали при тех же условиях. Надосадочную жидкость сливали, добавляли 200 мкл ТЕ-буфера, пипетировали до растворения осадка и затем последовательно добавляли 10 мкл 1% раствора додецилсульфата натрия SDS и 5 мкл протеиназы К. Пробирки инкубировали в термостате при t=37°C 12 ч. В ходе второго этапа проводили четыре последовательных центрифугирования с фенолом и хлороформом согласно протоколу методики (10 мин, 8 тыс. об/мин), после чего ДНК осаждали ледяным раствором 95% этилового спирта и центрифугировали 10 мин при 14,3 тыс. об/мин. По испарении спирта ДНК растворяли в 100 мкл деионизированной дистиллированной воды. Получаемый раствор ДНК в воде имел чистоту в диапазоне А260/280=1,5-2,0 и среднюю концентрацию около 180-200 нг/мкл.
2. Подготовка образцов ДНК к генотипированию. Качество выделенной ДНК оценивали по степени чистоты и концентрации раствора на спектрофотометре NanoDrop (Thermo Fisher Scientific, США). Все анализируемые образцы ДНК были разведены деионизированной водой до концентрации 15-20 нг/мкл при А260/280=1,5-2,0.
3. Анализ полиморфизма rs12561767 (G>A) гена SERBP1 с помощью полимеразной цепной реакции в реальном времени с использованием аллель-специфических зондов. Для генотипирования использовали два фланкирующих праймера, прямой (SEQ ID NO 1) и обратный (SEQ ID NO 2), а также аллель-специфические зонды: G-аллель-специфичный флуоресцентно-меченый зонд (SEQ ID NO 3),
A-аллель-специфичный флуоресцентно-меченый зонд (SEQ ID NO 4).
ПЦР в «реальном времени» проводили в 25 мл реакционной смеси, содержащей 1,25 ЕД ДНК-полимеразы Hot Start Taq («Биолабмикс», Новосибирск, Россия), 20 нг ДНК, по 10 мкМ каждого праймера, по 5 мкМ каждого зонда, 0.03 мМ каждого dNTP, 1,5 мМ MgCl2; 1хПЦР-буфер [67 мМ Tris-HCl, pH 8,8, 16,6 мМ (NH4)2SO4, 0,01% Tween-20]. Реакция амплификации состояла из стадии нагревания до 50°C в течение 2 минут, 95°C в течение 10 минут, затем 39 циклов [95°C в течение 10 секунд и 64°C в течение 1 минуты].
4. Генотипирование. При проведении ПЦР в амплификаторе с флуоресцентной детекцией (Bio-Rad CFX96 или аналогичном амплификаторе) генотипирование осуществляют по данным величин RFU (относительных единиц флуоресценции). Для rs12561767 (G>A) гена SERBP1 зонд с флуоресцентным красителем FAM соответствует аллелю G, зонд с красителем ROX - аллелю A (фиг.). На фигуре видно четкое разделение образцов на кластеры, где черный ромб соответствуют отрицательному контролю, кластер оранжевых кругов - соответствует зонду с флуоресцентным красителем FAM и позволяет идентифицировать гомозигот G/G. Кластер синих квадратов соответствует зонду с красителем ROX и позволяет идентифицировать гомозигот A/A. Кластер зеленых треугольников соответствует накоплению уровня флуоресценции по обоим зондам и позволяет идентифицировать гетерозигот G/A.
Резюме.
Таким образом, разработан эффективный и недорогой способ для экспресс-идентификации полиморфного варианта rs12561767 (G>A) гена SERBP1 у человека методом ПЦР в режиме «реального времени» с применением аллель-специфических флуоресцентных зондов, который может быть использован в медицине при определении наследственной предрасположенности к развитию заболеваний, ассоциированных с носительством полиморфизмов гена SERBP1, а также в научных целях.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="последовательности
rs1267.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-05-24">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023107906/20(017110)</ApplicationNumberText>
<FilingDate>2023-03-30</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное образовательное учреждение высшего образования
"Курский государственный медицинский университет"
Министерства здравоохранения Российской Федерации,</ApplicantName>
<ApplicantNameLatin>Kursk State Medical
University</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ генотипирования
полиморфного локуса rs12561767 (G>A) гена SERBP1 у человека
методом ПЦР в режиме «реального времени» с применением
аллель-специфических флуоресцентных зондов</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcatgcctgtagtcccagta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcctgtctcccatgcta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttgaacttcagaatggccaaaatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttgaacttcaaaatggccaaaatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области биотехнологии. Описан способ генотипирования полиморфного локуса rs12561767 (G>A) гена SERBP1 у человека методом ПЦР в режиме «реального времени» с применением аллель-специфических флуоресцентных зондов. Способ включает проведение ПЦР с использованием прямого 5'-GCATGCCTGTAGTCCCAGTA-3' и обратного 5'-GTCTCCTGTCTCCCATGCTA-3' праймеров и G-аллель-специфичного флуоресцентно-меченого зонда 5'-(FAM)CTTGAACTTCAGAATGGCCAAAATC(RTQ1)-3' и A-аллель-специфичного флуоресцентно-меченого зонда 5'-(ROX) CTTGAACTTCAAAATGGCCAAAATC(BHQ2)-3'. Изобретение расширяет арсенал способов генотипирования полиморфных вариантов гена SERBP1 для использования при определении наследственной предрасположенности к развитию заболеваний, ассоциированных с носительством полиморфного варианта rs12561767 (G>A) гена SERPINE1 мРНК-связывающего белка 1. 1 ил., 1 пр.
Способ генотипирования полиморфного локуса rs12561767 (G>A) гена SERBP1 у человека методом ПЦР в режиме «реального времени» с применением аллель-специфических флуоресцентных зондов, отличающийся тем, что идентификацию аллельных вариантов rs12561767 (G>A) гена SERBP1 осуществляют с использованием прямого праймера rs12561767 5′-GCATGCCTGTAGTCCCAGTA-3′ (SEQ ID NO 1), обратного праймера rs12561767 5′-GTCTCCTGTCTCCCATGCTA-3′ (SEQ ID NO 2), rs12561767-G-аллель-специфичного флуоресцентно-меченого зонда 5′-(FAM)CTTGAACTTCAGAATGGCCAAAATC(RTQ1)-3′(SEQ ID NO 3),
rs12561767-A-аллель-специфичного флуоресцентно-меченого зонда 5′-(ROX) CTTGAACTTCAAAATGGCCAAAATC(BHQ2)-3′ (SEQ ID NO 4).
| Способ проведения ПЦР с аллель-специфичными зондами для генотипирования крупного рогатого скота по аллелям А и К гена DGAT1 | 2018 |
|
RU2662972C1 |
| Способ проведения ПЦР в режиме реального времени для генотипирования крупного рогатого скота по аллелям 337C/G гена fshr (rs43745234) | 2019 |
|
RU2708559C1 |
| WO 2006110735 A2, 19.10.2006 | |||
| WO 2011056133 A1, 12.05.2011 | |||
| WO 2011106724 A2, 01.09.2011. | |||
Авторы
Даты
2023-09-14—Публикация
2023-03-30—Подача