Изобретение относится к биотехнологии, а именно к набору праймеров для амплификации геномов респираторно-синцитиального вируса (РСВ) А и B для последующего секвенирования.
Респираторно-синцитиальный вирус (РСВ, RSV) - один из главных патогенов среди респираторных вирусов, приводящих к госпитализации младенцев и детей младшего возраста. Для лечения тяжелых случаев заболевания существует эффективная терапия - моноклональные антитела паливизумаб, однако из-за высокой стоимости, их применение ограничено группами населения, проживающими в условиях с хорошо развитой медицинской помощью, где возможна диагностика заболевания и проведение такой дорогостоящей терапии. На данный момент не существует вакцины, предотвращающей инфицирование РСВ, однако, несколько кандидатных вакцинных препаратов, предназначенные, в том числе для вакцинирования маленьких детей, пожилых людей и беременных женщин находятся на разных стадиях клинических исследований. Вероятно, эффективная безопасная вакцина будет доступна в ближайшем будущем.
Для разработки вакцинных и терапевтических препаратов важно наблюдение за эволюцией вируса, вызывающего инфекцию. Использование данных полногеномного секвенирования помогает создать вакцину с наиболее актуальным составом и оценить возможность ее глобального использования. С помощью наблюдения за вирусами методами полногеномного секвенирования возможно отслеживать распространение вариантов вирусов и контролировать изменения генетического кода вируса. Таким образом, данные секвенирования используются для лучшего понимания того, как вирус может повлиять на общественное здоровье. Полногеномное секвенирование респираторно-синцитиального вируса решает важную задачу предоставления данных для создания эффективных препаратов для предупреждения и лечения инфицирования, вызванного РСВ.
Секвенирование генома вируса требует определенного количества ДНК или РНК для достаточного покрытия и достоверности результатов. Обычно содержание вирусных нуклеиновых кислот в образце низкое, преобладают нуклеиновые кислоты инфицированных клеток и других микроорганизмов. Специфическая амплификация вирусных нуклеиновых кислот помогает увеличить количество вирусной ДНК или РНК до уровня, который можно эффективно секвенировать. Таким образом, амплификация позволяет селективно увеличить количество вирусной ДНК или РНК, исключая при этом другие примеси. Требования к методу полногеномной амплификации вирусных нуклеиновых кислот следующие: праймеры, комплементарные наиболее консервативным участкам генома, оптимизированная эффективная ПЦР всех фрагментов генома, отсутствие неспецифической амплификации. В случае соблюдения данных условий вся последовательность генома будет многократно умножена, и такой образец может быть подготовлен для полногеномного секвенирования с использованием реагентов для приготовления библиотек.
Для решения задачи полногеномного секвенирования респираторно-синцитиального вируса известен подход Langedijk A.C. [1], использованный в исследовании генетического разнообразия РСВ «INFORM». Также опубликована панель олигонуклеотидов для полногеномной амплификации в работе Agoti C.N. [2]. При использовании указанных методов, вирусные геномы амплифицируются в виде четырех-шести перекрывающихся фрагментов, смесь которых в дальнейшем используется для приготовления библиотек для секвенирования.
Однако амплифицирование длинных фрагментов ДНК на матрице вирусной РНК не всегда проходит успешно, особенно для первичных клинических образцов (оро- и назофарингеальные мазки) из-за деградации вирусной РНК. Поэтому при работе с заявляемым набором размер амплифицированных фрагментов не превышает 1,5 тысячи п.о., что позволяет эффективно амплифицировать вирусный геном в случае образцов с частично деградированной РНК.
Размер ампликонов заявляемого набора схож с размером ампликонов общеизвестной панели для полногеномной амплификации SARS-CoV-2 «Midnight», что позволяет решить проблему неудобства использования различных протоколов фрагментации для разных ампликонов в ходе приготовления библиотек для секвенирования. Геном РСВ, амплифицированный при помощи данной панели олигонуклеотидов может быть процессирован по широко используемым для SARS-CoV-2 протоколам без дополнительной оптимизации методов для дальнейшего секвенирования на платформах Illumina, Oxford Nanopore, BGI.
Также преимуществом предлагаемой панели является мультиплексная амплификация - амплификация 12 фрагментов происходит в одной пробирке, в то время как в указанных выше методах предлагается амплифицировать каждый фрагмент генома отдельно. Недавно разработанная методика Dong X [3] решает эту проблему, так как авторы показывают эффективную мультиплексную полногеномную амплификацию, однако, протокол сохраняет недостатки описанных выше протоколов из-за амплификации длинных фрагментов.
Также известен набор оптимизированных зондов и праймеров, а также способы их использования для связывания, обнаружения, дифференциации, выделения и секвенирования вирусов гриппа А; гриппа В и респираторно-синцитиального вируса [4].
Также известна группа праймеров для секвенирования следующего поколения (NGS) и набор для выявления возбудителей респираторных вирусов [5]. Группа праймеров состоит из 594 последовательностей праймеров. Согласно изобретению 594 праймера специально разработаны для геномов вирусов гриппа А, гриппа В, синцитиальных вирусов и новых коронавирусов,
Недостатком данного технического решения является сложность реализации такой панели и высокая вероятность неспецифической амплификации, например, человеческих нуклеиновых кислот. При одновременном использовании такого большого количества олигонуклеотидов (594) значительно возрастает количество возможных комбинаций для их неспецифического попарного отжига. В случаях, когда олигонуклеотиды неспецифично отжигаются достаточно близко (продукт может быть амплифицирован в ходе реакции) на нуклеиновых кислотах образца, происходит нецелевая амплификация. Нецелевая амплификация приводит к нецелевому секвенированию, потере части данных и, следовательно, неоптимальной трате средств.
Технической проблемой является необходимость разработки набора олигонуклеотидов для полногеномной амплификации респираторно-синцитиального вируса типов А и В.
Технический результат состоит в обеспечении возможности эффективной высокоспецифичной полногеномной амплификации респираторно-синцитиального вируса типов А и В для последующего секвенирования.
Технический результат достигается тем, что набор олигонуклеотидов для полногеномной амплификации респираторно-синцитиального вируса типов А и В включает
праймеры для выявления респираторно-синцитиального вируса типа А со следующим нуклеотидным составом:
RSVAseq F1 5’-acgcgaaaaaatgcgtacaacaaac-3’ (SEQ ID NO:1)
RSVAseq F2 5’-aacggagcacaggagacagc-3’ (SEQ ID NO:2)
RSVAseq F3 5’-GtATcaTATCtgTcAAcTCaAtAGAtATaGAagtaaccAAaG-3’ (SEQ ID NO:3)
RSVAseq F4 5’-agatctgaacacacttgaaaatataacaaccac-3’ (SEQ ID NO:4)
RSVAseq F5 5’-ccaccacaaaacaacgccaa-3’ (SEQ ID NO:5)
RSVAseq F6 5’-tggagtcagtgtcttaaccagca-3’ (SEQ ID NO:6)
RSVAseq F7 5’-attcgaggtcattgcttgaatggt-3’ (SEQ ID NO:7)
RSVAseq F8 5’-gaacagactactactactaatttacttaaaaagataataagaagagc-3’ (SEQ ID NO:8)
RSVAseq F9 5’-gagtttcggttgcctaaaaaagtggat-3’ (SEQ ID NO:9)
RSVAseq F10 5’-tggtgatcccaacttgttatatcgaagt-3’ (SEQ ID NO:10)
RSVAseq F11 5’-ctttgatactagccctattaatcgcatattaacaga-3’ (SEQ ID NO:11)
RSVAseq F12 5’-cAATAGcacaTCaCTtTatTGcaTgCTtC-3’ (SEQ ID NO:12)
RSVAseq R1 5’-gattagcatcttctgtgattaataacatgccac-3’ (SEQ ID NO:13)
RSVAseq R2 5’-gcTTtCTTtGaTAattgggcTTgtTccc-3’ (SEQ ID NO:14)
RSVAseq R3 5’-tcctgagtaagggatgatttttgcatttg-3’ (SEQ ID NO:15)
RSVAseq R4 5’-ctgcatatgctgcagggtacaaa-3’ (SEQ ID NO:16)
RSVAseq R5 5’-ctatcacagtttcaatgtttgatatgctgca-3’ (SEQ ID NO:17)
RSVAseq R6 5’-tacaagcagtgcatggggtg-3’ (SEQ ID NO:18)
RSVAseq R7 5’-ccagtttattcaatatagcatagactttgacatcact-3’ (SEQ ID NO:19)
RSVAseq R8 5’-atgtgtgacggcatataatttctaggga-3’ (SEQ ID NO:20)
RSVAseq R9 5’-gcctctgtgaggaaatcaggagttc-3’ (SEQ ID NO:21)
RSVAseq R10 5’-caaatgtatctcattaagcttgggtatgagaataattc-3’ (SEQ ID NO:22)
RSVAseq R11 5’-CCtGCtCCtTCaCCTATgAATGc-3’ (SEQ ID NO:23)
RSVAseq R12 5’-acgagaaaaaaagtgtcaaaaactaatatctcgta-3’ (SEQ ID NO:24)
а также включающий праймеры для выявления респираторно-синцитиального вируса типа B со следующим нуклеотидным составом:
RSVBseq F1 5’-acgcgaaaaaatgcgtactacaaact-3’ (SEQ ID NO:25)
RSVBseq F2 5’-ctgctgtcatccagcaaatacact-3’ (SEQ ID NO:26)
RSVBseq F3 5’-ataacatctggcaccaacattatcaatcc-3’ (SEQ ID NO:27)
RSVBseq F4 5’-accatgaagacattcaaccccac-3’ (SEQ ID NO:28)
RSVBseq F5 5’-gcacaaaaccacgtccaaaaaatccac-3’ (SEQ ID NO:29)
RSVBseq F6 5’-tggggtcagtgttttaaccagc-3’ (SEQ ID NO:30)
RSVBseq F7 5’-tcctcgcatcatgctaccca-3’ (SEQ ID NO:31)
RSVBseq F8 5’-gtgatgtaaaggtgtacgccatcttg-3’ (SEQ ID NO:32)
RSVBseq F9 5’-catctgcctaaaaaagtggatcttgaaatga-3’ (SEQ ID NO:33)
RSVBseq F10 5’-accttttttaatcttgatagtatcgatacggc-3’ (SEQ ID NO:34)
RSVBseq F11 5’-gacattgtgtttcaaaattgcataagttttggt-3’ (SEQ ID NO:35)
RSVBseq F12 5’-CAacTtTAcaccACcaCttCacatCa-3’ (SEQ ID NO:36)
RSVBseq R1 5’-gcatcttcagtgattaatagcataccacatag-3’ (SEQ ID NO:37)
RSVBseq R2 5’-gGgtTTTCTtgggtAgtTggcttT-3’ (SEQ ID NO:38)
RSVBseq R3 5’-agggaataattttagcattggtgatagca-3’ (SEQ ID NO:39)
RSVBseq R4 5’-attgttgccacatatactacagggaac-3’ (SEQ ID NO:40)
RSVBseq R5 5’-ctgctgttcttctgctggaattctataac-3’ (SEQ ID NO:41)
RSVBseq R6 5’-gcgacatatttgccccagctg-3’ (SEQ ID NO:42)
RSVBseq R7 5’-ttcatcacctgaattgttgttgggc-3’ (SEQ ID NO:43)
RSVBseq R8 5’-ggcatgtaatttctaggaaaactagtccatattagatc-3’ (SEQ ID NO:44)
RSVBseq R9 5’-agtctggagttctcctataaaagcttcg-3’ (SEQ ID NO:45)
RSVBseq R10 5’-gacatatgtttgtgaattgttccacaactg-3’ (SEQ ID NO:46)
RSVBseq R11 5’-aGGaAGcAtgCAaTAaAgtGAtgcaCta-3’ (SEQ ID NO:47)
RSVBseq R12 5’-acgagaaaaaaagtgtcaaaaactaatgtctcg-3’ (SEQ ID NO:48)
Заявляемый набор олигонуклеотидов для полногеномной амплификации геномов РСВ А и B для последующего секвенирования. В обоих случаях амплифицируются 12 перекрывающихся фрагментов генома, которые вместе составляют полный геном вируса. Длины амплифицирующихся фрагментов РСВ приведены в таблицах 1 и 2.
Из множественного выравнивания 1777 актуальных последовательностей генома RSV A и 1654 последовательностей генома RSV В были получены консенсусы, по котором производилась оценка консервативности участков. Подобранные олигонуклеотиды для полногеномной амплификации комплементарны наиболее консервативным участкам генома, вероятность комплементарности нуклеотидов праймеров консенсусам в большинстве случаев составила >99%
Из-за низкого %GC состава генома РСВ некоторые праймеры имеют GC состав ниже 40%, что в данном случае не влияет на эффективность амплификации, однако влияет на длину праймеров.
Праймеры для амплификации РСВ типа А обеспечивают полногеномную амплификацию всех вариантов РСВ типа А с перекрытием ампликонов в 60-160 оснований (в среднем ≈100 оснований). Длина ампликонов генома РСВ А составляет 1255-1505 п.о. Праймеры для амплификации РСВ типа В обеспечивают полногеномную амплификацию всех вариантов РСВ В с перекрытием ампликонов в 70-190 оснований (в среднем ≈120 оснований). Длина ампликонов генома РСВ В составляет 1283-1567 п.о.
Программа термоциклирования: обратная транскрипция при 45°С - 30 мин, денатурация при 95°С в течение 5 мин, затем 40 циклов при 95°С - 15 с, 60°С - 30 с, 72°С - 30 с. Оптимальное количество олигонуклеотидов на реакцию: 100 nM праймеров. Для амплификации необходимо использовать высокоточную полимеразу.
Заявляемое изобретение поясняется примером.
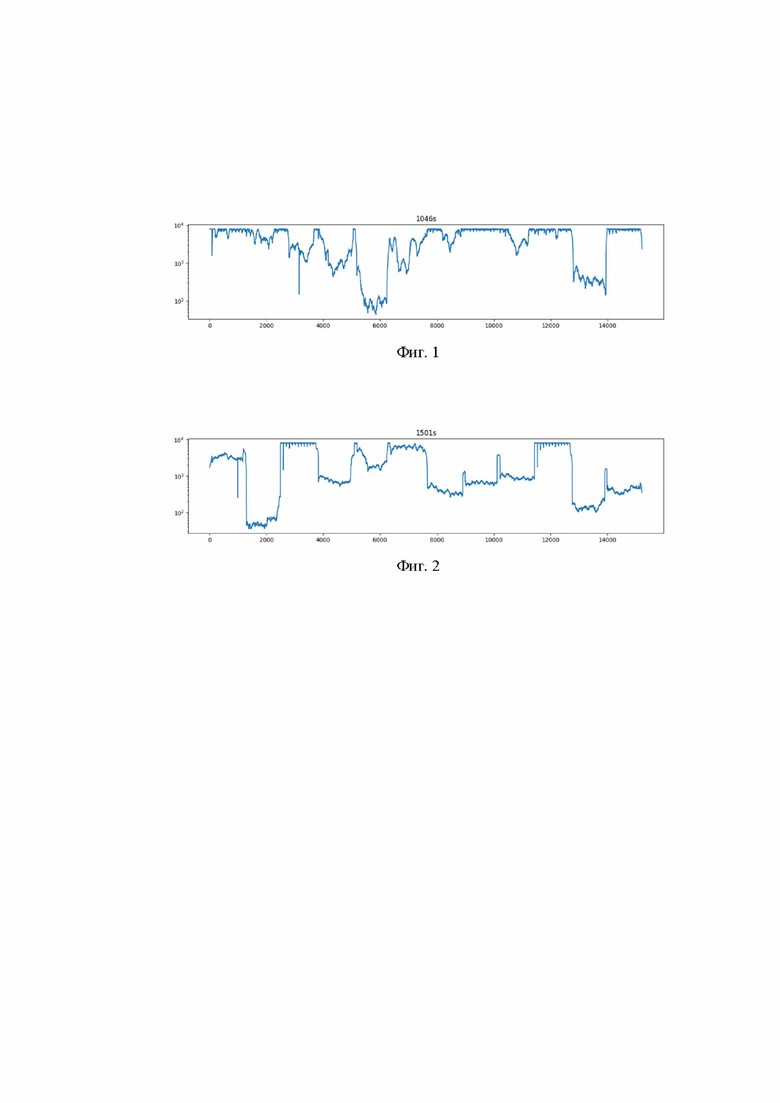
При использовании предлагаемой панели олигонуклеотидов для полногеномной амплификации удается достичь секвенирования всей последовательности генома РСВ с достаточным покрытием всех его частей. Для полногеномной амплификации вируса РСВ А в ПЦР необходимо одновременно использовать праймеры SEQ ID NO:1 - SEQ ID NO:24. Для полногеномной амплификации генома вируса РСВ В необходимо одновременно использовать праймеры SEQ ID NO:25 - SEQ ID NO:48. Успешная амплификация генома РСВ позволяет многократно увеличить содержание в образце ДНК, комплементарной вирусной РНК, что позволяет многократно прочитать геном вируса и уверенно отличать возникшие в геноме мутации от ошибок секвенирования. Однако, для такой работы необходимо отбирать наилучшие по данным ПЦР образцы - с наименьшим Cq, вне зависимости от набора, выбранного для детекции вируса. Пример покрытия генома отсеквенированного образца РСВ А, амплифицированного с помощью предлагаемой панели олигонуклеотидов для полногеномной амплификации представлен на фиг. 1. Пример покрытия генома отсеквенированного образца РСВ В, амплифицированного с помощью предлагаемой панели олигонуклеотидов для полногеномной амплификации представлен на фиг. 2. Из полученных ампликонов может быть приготовлена библиотека для секвенирования с использованием реагентов “Nextera DNA Flex Library Prep Kit” (Illumina) или аналогичных для последующего секвенирования на приборе Illumina - например, на платформе MiSeq.
Список литературы
1. Langedijk, A. C., Lebbink, R. J., Naaktgeboren, C., Evers, A., Viveen, M. C., Greenough, A., Heikkinen, T., Stein, R. T., Richmond, P., Martinón-Torres, F., Nunes, M., Hosoya, M., Keller, C., Bauck, M., Cohen, R., Papenburg, J., Pernica, J., Hennus, M. P., Jin, H., ... Bont, L. J. (2020). Global molecular diversity of RSV - the "INFORM RSV" study. BMC Infectious Diseases, 20(1), [450]. https://doi.org/10.1186/s12879-020-05175-4.
2. Agoti, C. N., Otieno, J. R., Munywoki, P. K., Mwihuri, A. G., Cane, P. A., Nokes, D. J., Kellam, P., & Cotten, M. (2015). Local evolutionary patterns of human respiratory syncytial virus derived from whole-genome sequencing. Journal of virology, 89(7), 3444-3454. https://doi.org/10.1128/JVI.03391-14.
3. Xiaomin Dong, Yi-Mo Deng, Ammar Aziz, Paul Whitney, Julia Clark, Patrick Harris, Catherine Bautista, Anna-Maria Costa, Gregory Waller, Andrew J Daley, Megan Wieringa, Tony Korman, Ian G. Barr, A simplified, amplicon-based method for whole genome sequencing of human respiratory syncytial viruses, Journal of Clinical Virology, Volume 161, 2023, 105423, ISSN 1386-6532, https://doi.org/10.1016/j.jcv.2023.105423.
4. Оптимизированные зонды и праймеры, а также методы их использования для связывания, обнаружения, дифференциации, выделения и секвенирования гриппа A; грипп B и респираторно-синцитиального вируса: международная заявка WO2014066749, опубл. 01.05.2014.
5. Группа праймеров для секвенирования следующего поколения (NGS) и набор для выявления возбудителей респираторных вирусов: заявка CN113151598, Китайская Народная Республика, заявл. 20.04.2021, опубл. 23.07.2021.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2 и олигонуклеотидные праймеры для его реализации | 2021 |
|
RU2762759C1 |
| СПОСОБ ДИФФЕРЕНЦИАЛЬНОЙ ДИАГНОСТИКИ РЕСПИРАТОРНЫХ ВИРУСНЫХ ИНФЕКЦИЙ МЕТОДОМ МУЛЬТИПЛЕКСНОЙ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ И ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2010 |
|
RU2460803C2 |
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавируса человека 2019-nCoV методом ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2020 |
|
RU2734300C1 |
| Панель праймеров для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа | 2024 |
|
RU2833687C1 |
| Набор олигодезоксирибонуклетидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавирусов человека SARS и 2019-nCoV методом ОТ-ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2020 |
|
RU2733665C1 |
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации РНК JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией | 2023 |
|
RU2818960C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| Способ секвенирования экзонов гена HLA-DQA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2833819C1 |
| Способ секвенирования экзонов гена HLA-DPA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2837865C1 |
| Олигонуклеотиды для определения мутации S:delVYY143-145 SARS-CoV-2 | 2022 |
|
RU2795016C1 |
Изобретение относится к биотехнологии, а именно к набору праймеров для амплификации геномов респираторно-синцитиального вируса (РСВ) А и B для последующего секвенирования. Предложен набор, включающий 24 праймера для выявления респираторно-синцитиального вируса типа А и 24 праймера для выявления респираторно-синцитиального вируса типа В. Технический результат состоит в обеспечении возможности эффективной высокоспецифичной полногеномной амплификации респираторно-синцитиального вируса типов А и В для последующего секвенирования. 2 ил., 2 табл.
Набор олигонуклеотидов для полногеномной амплификации респираторно-синцитиального вируса типов А и В, включающий
праймеры для выявления респираторно-синцитиального вируса типа А со следующим нуклеотидным составом:
RSVAseq F1 5’-acgcgaaaaaatgcgtacaacaaac-3’ (SEQ ID NO:1)
RSVAseq F2 5’-aacggagcacaggagacagc-3’ (SEQ ID NO:2)
RSVAseq F3 5’-GtATcaTATCtgTcAAcTCaAtAGAtATaGAagtaaccAAaG-3’ (SEQ ID NO:3)
RSVAseq F4 5’-agatctgaacacacttgaaaatataacaaccac-3’ (SEQ ID NO:4)
RSVAseq F5 5’-ccaccacaaaacaacgccaa-3’ (SEQ ID NO:5)
RSVAseq F6 5’-tggagtcagtgtcttaaccagca-3’ (SEQ ID NO:6)
RSVAseq F7 5’-attcgaggtcattgcttgaatggt-3’ (SEQ ID NO:7)
RSVAseq F8 5’-gaacagactactactactaatttacttaaaaagataataagaagagc-3’ (SEQ ID NO:8)
RSVAseq F9 5’-gagtttcggttgcctaaaaaagtggat-3’ (SEQ ID NO:9)
RSVAseq F10 5’-tggtgatcccaacttgttatatcgaagt-3’ (SEQ ID NO:10)
RSVAseq F11 5’-ctttgatactagccctattaatcgcatattaacaga-3’ (SEQ ID NO:11)
RSVAseq F12 5’-cAATAGcacaTCaCTtTatTGcaTgCTtC-3’ (SEQ ID NO:12)
RSVAseq R1 5’-gattagcatcttctgtgattaataacatgccac-3’ (SEQ ID NO:13)
RSVAseq R2 5’-gcTTtCTTtGaTAattgggcTTgtTccc-3’ (SEQ ID NO:14)
RSVAseq R3 5’-tcctgagtaagggatgatttttgcatttg-3’ (SEQ ID NO:15)
RSVAseq R4 5’-ctgcatatgctgcagggtacaaa-3’ (SEQ ID NO:16)
RSVAseq R5 5’-ctatcacagtttcaatgtttgatatgctgca-3’ (SEQ ID NO:17)
RSVAseq R6 5’-tacaagcagtgcatggggtg-3’ (SEQ ID NO:18)
RSVAseq R7 5’-ccagtttattcaatatagcatagactttgacatcact-3’ (SEQ ID NO:19)
RSVAseq R8 5’-atgtgtgacggcatataatttctaggga-3’ (SEQ ID NO:20)
RSVAseq R9 5’-gcctctgtgaggaaatcaggagttc-3’ (SEQ ID NO:21)
RSVAseq R10 5’-caaatgtatctcattaagcttgggtatgagaataattc-3’ (SEQ ID NO:22)
RSVAseq R11 5’-CCtGCtCCtTCaCCTATgAATGc-3’ (SEQ ID NO:23)
RSVAseq R12 5’-acgagaaaaaaagtgtcaaaaactaatatctcgta-3’ (SEQ ID NO:24),
а также включающий праймеры для выявления респираторно-синцитиального вируса типа B со следующим нуклеотидным составом:
RSVBseq F1 5’-acgcgaaaaaatgcgtactacaaact-3’ (SEQ ID NO:25)
RSVBseq F2 5’-ctgctgtcatccagcaaatacact-3’ (SEQ ID NO:26)
RSVBseq F3 5’-ataacatctggcaccaacattatcaatcc-3’ (SEQ ID NO:27)
RSVBseq F4 5’-accatgaagacattcaaccccac-3’ (SEQ ID NO:28)
RSVBseq F5 5’-gcacaaaaccacgtccaaaaaatccac-3’ (SEQ ID NO:29)
RSVBseq F6 5’-tggggtcagtgttttaaccagc-3’ (SEQ ID NO:30)
RSVBseq F7 5’-tcctcgcatcatgctaccca-3’ (SEQ ID NO:31)
RSVBseq F8 5’-gtgatgtaaaggtgtacgccatcttg-3’ (SEQ ID NO:32)
RSVBseq F9 5’-catctgcctaaaaaagtggatcttgaaatga-3’ (SEQ ID NO:33)
RSVBseq F10 5’-accttttttaatcttgatagtatcgatacggc-3’ (SEQ ID NO:34)
RSVBseq F11 5’-gacattgtgtttcaaaattgcataagttttggt-3’ (SEQ ID NO:35)
RSVBseq F12 5’-CAacTtTAcaccACcaCttCacatCa-3’ (SEQ ID NO:36)
RSVBseq R1 5’-gcatcttcagtgattaatagcataccacatag-3’ (SEQ ID NO:37)
RSVBseq R2 5’-gGgtTTTCTtgggtAgtTggcttT-3’ (SEQ ID NO:38)
RSVBseq R3 5’-agggaataattttagcattggtgatagca-3’ (SEQ ID NO:39)
RSVBseq R4 5’-attgttgccacatatactacagggaac-3’ (SEQ ID NO:40)
RSVBseq R5 5’-ctgctgttcttctgctggaattctataac-3’ (SEQ ID NO:41)
RSVBseq R6 5’-gcgacatatttgccccagctg-3’ (SEQ ID NO:42)
RSVBseq R7 5’-ttcatcacctgaattgttgttgggc-3’ (SEQ ID NO:43)
RSVBseq R8 5’-ggcatgtaatttctaggaaaactagtccatattagatc-3’ (SEQ ID NO:44)
RSVBseq R9 5’-agtctggagttctcctataaaagcttcg-3’ (SEQ ID NO:45)
RSVBseq R10 5’-gacatatgtttgtgaattgttccacaactg-3’ (SEQ ID NO:46)
RSVBseq R11 5’-aGGaAGcAtgCAaTAaAgtGAtgcaCta-3’ (SEQ ID NO:47)
RSVBseq R12 5’-acgagaaaaaaagtgtcaaaaactaatgtctcg-3’ (SEQ ID NO:48).
| СПОСОБ ДИФФЕРЕНЦИАЛЬНОЙ ДИАГНОСТИКИ РЕСПИРАТОРНЫХ ВИРУСНЫХ ИНФЕКЦИЙ МЕТОДОМ МУЛЬТИПЛЕКСНОЙ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ И ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2010 |
|
RU2460803C2 |
| CN 107058619 A, 18.08.2017 | |||
| RU 2021120509 A, 23.01.2023. | |||
Авторы
Даты
2024-01-15—Публикация
2023-06-30—Подача