Изобретение относится к области биотехнологии и может быть использовано в диагностике для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа.
Известен набор праймеров для определения вируса гриппа типа А, в том числе патогенных для человека субтипов вируса H1, H3, H5, H7, вируса гриппа типа В, вируса парагриппа типа 2, респираторно-синцитиального вируса и аденовируса (Мультиплексное выявление респираторных возбудителей: патент US 8232058, Соединенные Штаты Америки, заявка US 16129107, заявл. 22.01.2007, опубл. 31.07.2012), используемые для подтверждения присутствия или отсутствия патогена в образце. Причем праймеры и зонды разработаны и оптимизированы для использования в мультиплексном анализе Luminex на основе ПЦР.
Также известен набор олигонуклеотидов для определения вирусов, вызывающих у человека острые респираторные заболевания (Идентификация микроорганизмов, вызывающих острые инфекции дыхательных путей: патент EP 1115885, Европейская патентная организация, заявка EP 99948833, заявл. 22.09.1999, опубл. 20.08.2008). В частности, набор олигонуклеотидов предназначен для определения методом ОТ-ПЦР следующих вирусов: респираторно-синцитиальный вирус, вирус парагриппа типа 1 и 2, энтеровирус, вирус гриппа типа А и В, аденовирус, M. pneumonia, C. pneumonia.
Также известен набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации рнк и дифференциации вируса парагриппа человека 1, 2, 3 и 4 типов (Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации рнк и дифференциации вируса парагриппа человека 1, 2, 3 и 4 типов: патент RU 2515911, Российская Федерация, заявка RU 2012156694, заявл. 25.12.2012, опубл. 20.05.2014). Представленный набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых ДНК-зондов содержит четыре пары внутренних и четыре пары внешних олигонуклеотидов, обладающих активностью прямого и обратного праймеров в полимеразной цепной реакции, а также четыре флуоресцентно-меченых ДНК-зонда.
Однако на сегодняшний день не известна панель, позволяющая осуществлять полногеномное секвенирование вирусов парагриппа человека 1 и 2 типа. Вышеприведенные патенты относятся к ПЦР в режиме реального времени, и они позволяют только детектировать наличие патогенов, они не предназначены для выявления данных о последовательностях полных геномов вирусов.
Технической проблемой является необходимость разработки панели для эффективного полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа.
Технический результат состоит в обеспечении возможности эффективного полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа.
Технический результат достигается тем, что панель праймеров для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа включает праймеры для секвенирования вируса парагриппа 1 типа:
прямой праймер hPIV1-F1 - ACCAAACAAGAGRAAAAACTTGTTT (SEQ NO:1);
обратный праймер hPIV1-R1 - CATGAACTGGGTCTCTGAGTATACATATA (SEQ NO:2);
прямой праймер hPIV1-F2 - TTACATAAGAGATGCAGGATTAGCATC (SEQ NO:3);
обратный праймер hPIV1-R2 - CGACGTCRAGGAGTCCGATG (SEQ NO:4);
прямой праймер hPIV1-F3 - CAGCATACACGAAACCAACCTT (SEQ NO:5);
обратный праймер hPIV1-R3 - AATTCATGTTGTGATTTCATTACACC (SEQ NO:6);
прямой праймер hPIV1-F4 - CCGTYAAAGAACGAAGAGCC (SEQ NO:7);
обратный праймер hPIV1-R4 - TGAGRGGGAGAGGTTCTACTGT (SEQ NO:8);
прямой праймер hPIV1-F5 - TCYACAATTTCAACCAGCAATC (SEQ NO:9);
обратный праймер hPIV1-R5 - GCTGCCCAGATGACTAGATTCAT (SEQ NO:10);
прямой праймер hPIV1-F6 - ATTGAGAAGATGAAGYTAATATTCTCTCT (SEQ NO:11);
обратный праймер hPIV1-R6 - GCTAGTGCAATTCCYGCAGTTATC (SEQ NO:12);
прямой праймер hPIV1-F7 - GATCTTCAGGAATCCCTGATAACA (SEQ NO:13);
обратный праймер hPIV1-R7 - GGACCCACTTTGATGATTTGATT (SEQ NO:14);
прямой праймер hPIV1-F8 - CATTCATAAATGGTGGTGTRGTAGCTA (SEQ NO:15);
обратный праймер hPIV1-R8 - CCYGTTTGTTGCATGACTTCTCTAT (SEQ NO:16);
прямой праймер hPIV1-F9 - TGACAGTATCCTCCGTGAACGA (SEQ NO:17);
обратный праймер hPIV1-R9 - GAGTGCCAACCTGARGATCTAGTATATA (SEQ NO:18);
прямой праймер hPIV1-F10 - TGCAATGATGCTCTTAAGATAACTTG (SEQ NO:19);
обратный праймер hPIV1-R10 - AAAATCGGATAAGGTTCAAAAGTGTA (SEQ NO:20);
прямой праймер hPIV1-F11 - TGCTAGAYATCAATCAACCTTATGATT (SEQ NO:21);
обратный праймер hPIV1-R11 - TTGCATGACACTCATATAGTGTCTTTAGTAT (SEQ NO:22);
прямой праймер hPIV1-F12 - TCATTGATAAAGATTTTCAGAGAGACAT (SEQ NO:23);
обратный праймер hPIV1-R12 - CTCATTACATCTTTGACCAAACAATG (SEQ NO:24)
прямой праймер hPIV1-F13 - AAATATGAATAAGTGCAATTCAAATGG (SEQ NO:25);
обратный праймер hPIV1-R13 - CTGGTTCTTGATTCATCACACGA (SEQ NO:26);
прямой праймер hPIV1-F14 - GCAATATTGATYCCRGCTAATATAGG (SEQ NO:27);
обратный праймер hPIV1-R14 - GTCGATACAGGAGTGAGTAACTTCAAA (SEQ NO:28);
прямой праймер hPIV1-F15 - TGTTAAGAACTTAAGCAAGCCGG (SEQ NO:29);
обратный праймер hPIV1-R15 - CACGCATGAAAAGATCAACAGAGTA (SEQ NO:30);
прямой праймер hPIV1-F16 - AGACACATCACATGCAGTCYTAAAAGT (SEQ NO:31);
обратный праймер hPIV1-R16 - AAAACCTTGACCCTTTGACATAAACT (SEQ NO:32);
прямой праймер hPIV1-F17 - GCRGGTGCAATGCTGTCTTGT (SEQ NO:33);
обратный праймер hPIV1-R17 - AATCTTGTAGACATAGACAATGCTATCCA (SEQ NO:34);
прямой праймер hPIV1-F18 - AAGCATTACAAATCTTCGGATTTGA (SEQ NO:35);
обратный праймер hPIV1-R18 - ACCAGACAAGAGTTTAAGAAATATCGATAT (SEQ NO:36),
а также включает праймеры для секвенирования вируса парагриппа 2 типа:
прямой праймер hPIV2-F1 - ACCAAGGGGAGAATTAGATGGC (SEQ NO:37);
обратный праймер hPIV2-R1 - ATGGGTCCTAGACTCTGATARTGTAGC (SEQ NO:38);
прямой праймер hPIV2-F2 - CYATGGTGGGAGACATTGGCA (SEQ NO:39);
обратный праймер hPIV2-R2 - GCTCAGTGGTGTATGTTGGTTCC (SEQ NO:40);
прямой праймер hPIV2-F3 - AADCATAGGCCCGGACGG (SEQ NO:41);
обратный праймер hPIV2-R3 - GGATTCRGGCTTTCGTGTGATC (SEQ NO:42);
прямой праймер hPIV2-F4 - YTCCAGTAGTAATTGCYGGTCC (SEQ NO:43);
обратный праймер hPIV2-R4 - ATAYTCAAATCCAGCTTGTAGCTTTG (SEQ NO:44);
прямой праймер hPIV2-F5 - AAGACATCAAGCCAGAGRGAGGA (SEQ NO:45);
обратный праймер hPIV2-R5 - AATAAAGCTAGCRCCACCATCAGT (SEQ NO:46);
прямой праймер hPIV2-F6 - AATGATAGTATGCATCTTTGTTATGTACACT (SEQ NO:47);
обратный праймер hPIV2-R6 - CGCTTRAGAAAGTTCCCGATAATA (SEQ NO:48);
прямой праймер hPIV2-F7 - GTGACACCRAACTCTGTATTYTGTAG (SEQ NO:49);
обратный праймер hPIV2-R7 - GGCAGTTCGGAAAATGATTCTA (SEQ NO:50);
прямой праймер hPIV2-F8 - TGAYACAGCTTAATCCRCTCAACAT (SEQ NO:51);
обратный праймер hPIV2-R8 - GGAGTTWCCGGCACARGTTATG (SEQ NO:52);
прямой праймер hPIV2-F9 - GRAGCGGGATCTATCAYCTAGGC (SEQ NO:53);
обратный праймер hPIV2-R9 - GCGGGAAGTGCCCTAGTAATAAG (SEQ NO:54);
прямой праймер hPIV2-F10 - AAGATTATRATATAGGCCAGAATGGC (SEQ NO:55);
обратный праймер hPIV2-R10 - CRATTAAATCTGGAGAAAGGTTGGA (SEQ NO:56);
прямой праймер hPIV2-F11 - TGGGTGTCTACAACTTAAAGATCCAG (SEQ NO:57);
обратный праймер hPIV2-R11 - ATGTATCATCAGGATCTGTGAGGTATG (SEQ NO:58);
прямой праймер hPIV2-F12 - TCATTGCTTACTATGAGTCAAATTGG (SEQ NO:59);
обратный праймер hPIV2-R12 - CGATATTGCGGTTAAATAATCTGC (SEQ NO:60);
прямой праймер hPIV2-F13 - TCYATTCGTCAACTCACATATGATC (SEQ NO:61);
обратный праймер hPIV2-R13 - CRATATCAAGTGCATCATTCCAGT (SEQ NO:62);
прямой праймер hPIV2-F14 - AAGAAGAGTTGCATCAATGGCATA (SEQ NO:63);
обратный праймер hPIV2-R14 - GCAAGAACTTGTTTAACYCCCCA (SEQ NO:64);
прямой праймер hPIV2-F15 - AGACGTGCAATGAATCTTGATATTATC (SEQ NO:65);
обратный праймер hPIV2-R15 - GTYGATTCGAGATCTATATGRACAAG (SEQ NO:66);
прямой праймер hPIV2-F16 - WGCAGTTACRGACTTATCRACAAAGGA (SEQ NO:67);
обратный праймер hPIV2-R16 - ACCAAGGGGAAAATCAATATGTTTT (SEQ NO:68).
Предложенная панель праймеров обеспечивает возможность полногеномного секвенирования ампликонов вируса парагриппа человека 1 и 2 типов.
Заявляемая панель праймеров была разработана следующим образом.
Набор из 18 пар праймеров для секвенирования вируса парагриппа человека 1-го типа был разработан с использованием инструмента primalscheme (https://primalscheme.com) для покрытия всего вирусного генома. Дизайн праймеров был основан на геномах консенсусных последовательностей, полученных в результате множественных выравниваний полных 975 геномов hPIV1, доступных в NCBI. Каждая пара праймеров покрывает примерно 1 т.п.н. генома с перекрытием ампликонов примерно на 200 п.н. Были приготовлены пулы праймеров, где каждый праймер составлял 100 мкМ. Эти два пула праймеров содержат по 9 пар праймеров каждый, которые генерируют перекрывающиеся ампликоны с нечетными или четными номерами. Данный набор приведен в таблице 1.
Таблица 1 - набор праймеров для секвенирования вируса парагриппа человека 1-го типа
Для каждого образца парагриппа генерировали две отдельные мультиплексные ПЦР-реакции (пулы 1 и 2) с использованием набора для одностадийной методики производства «Биолабмикс БиоМастер ОТ-ПЦР-Премиум (2×)» («Биолабмикс®», Россия) по указанию производителя. В каждую реакционную лунку по 16 мкл реакционной смеси + 4 мкл РНК. Параметры реакций представлены в таблицах 2 и 3.
Таблица 2 - параметры ПЦР (пул 1)
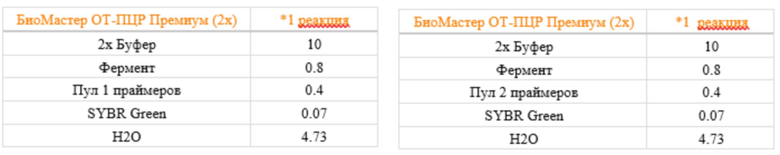
Таблица 3 - параметры ПЦР (пул 2)

Условия проведения ПЦР-амплификации приведены в таблице 4.
Таблица 4 - условия проведения ПЦР-амплификации
Чтобы оценить успешность амплификации, использовали SYBR Green, интеркалирующий чувствительный краситель для обнаружения двухцепочечной ДНК. Поэтому в ходе ПЦР по мере накопления продукта амплификации сигнал флуоресценции увеличивается, образуя кривую сигмовидной формы.
Набор из 18 пар праймеров для секвенирования вируса парагриппа человека 2-го типа был разработан с использованием инструмента primalscheme (https://primalscheme.com) для покрытия всего вирусного генома. Дизайн праймеров был основан на геномах консенсусных последовательностей, полученных в результате множественных выравниваний полных 693 геномов hPIV2, доступных в NCBI. Каждая пара праймеров покрывает примерно 1 т.п.н. генома с перекрытием ампликонов примерно на 200 п.н. Были приготовлены пулы праймеров, где каждый праймер составлял 100 мкМ. Эти два пула праймеров содержат по 8 пар праймеров каждый, которые генерируют перекрывающиеся ампликоны с нечетными или четными номерами. Данный набор приведен в таблице 5.
Таблица 5 - набор праймеров для секвенирования вируса парагриппа человека 2-го типа
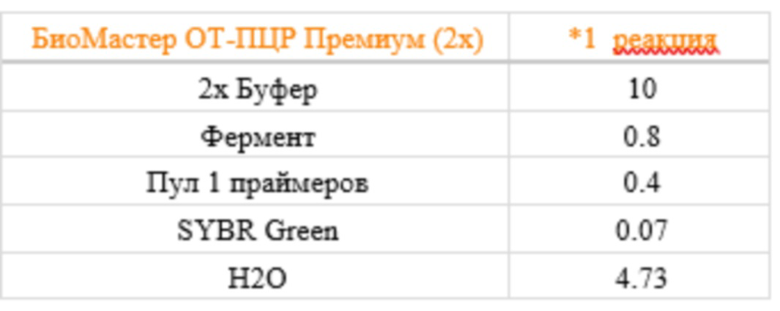
Для каждого образца парагриппа генерировали две отдельные мультиплексные ПЦР-реакции (пулы 1 и 2) с использованием набора для одностадийной методики производства «Биолабмикс БиоМастер ОТ-ПЦР-Премиум (2×)» («Биолабмикс®», Россия) по указанию производителя. В каждую реакционную лунку по 16 мкл реакционной смеси + 4 мкл РНК. Параметры реакций представлены в таблицах 6 и 7.
Таблица 6 - параметры ПЦР (пул 1)
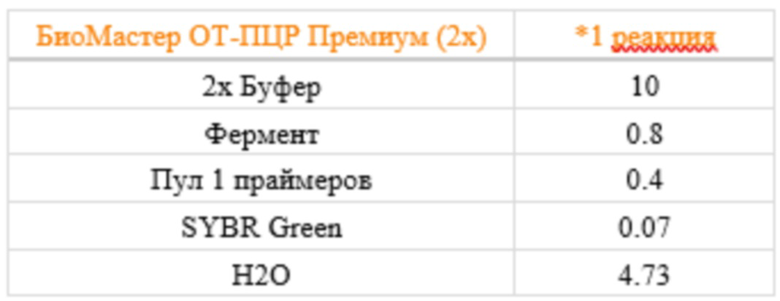
Таблица 7 - параметры ПЦР (пул 2)
Условия проведения ПЦР-амплификации приведены в таблице 8.
Таблица 8 - условия проведения ПЦР-амплификации
Чтобы оценить успешность амплификации, использовали SYBR Green, интеркалирующий чувствительный краситель для обнаружения двухцепочечной ДНК. Поэтому в ходе ПЦР по мере накопления продукта амплификации сигнал флуоресценции увеличивается, образуя кривую сигмовидной формы.
Заявляемое изобретение поясняется примерами.
Пример 1. Покрытие генома вируса парагриппа человека 1 типа
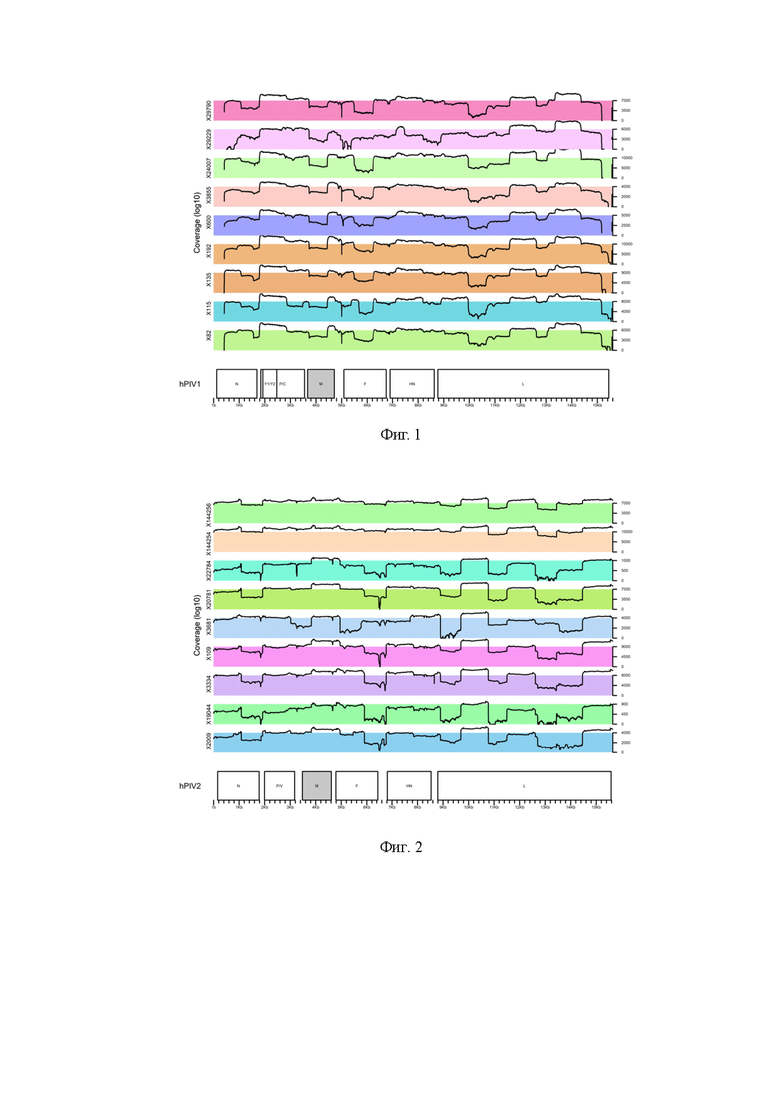
Разработанные панели праймеров были протестированы на клинических образцах, содержащих РНК вирусов парагриппа человека, по данным скрининга с использованием набора реагентов АмплиСенс «ОРВИ-скрин». Для тестирования были отобраны образцы с пороговым значением цикла (Ct) <25. Дизайн набора мозаичных ампликонов привел к равномерному покрытию последовательностей консенсусных геномов hPIV1, что привело к получению высококачественных и полных геномов с минимальными пробелами в секвенировании. График, иллюстрирующий покрытие генома приведен на 1 фиг.
При объеме данных 300 тысяч чтений на образец разработанные панели демонстрируют медианный охват, позволяющий установить консенсусную последовательность генома. Процент закрытых нолем позиций в тестовых выборках был очень низким. Результаты также приведены в таблице 9.
Таблица 9 - покрытие генома
Пример 2. Покрытие генома вируса парагриппа человека 2 типа
Разработанные панели праймеров были протестированы на клинических образцах, содержащих РНК вирусов парагриппа человека, по данным скрининга с использованием набора реагентов АмплиСенс «ОРВИ-скрин». Для тестирования были отобраны образцы с пороговым значением цикла (Ct) <25. Дизайн набора мозаичных ампликонов привел к равномерному покрытию последовательностей консенсусных геномов hPIV2, что привело к получению высококачественных и полных геномов с минимальными пробелами в секвенировании. График, иллюстрирующий покрытие генома приведен на 2 фиг.
При объеме данных 300 тысяч чтений на образец разработанные панели демонстрируют медианный охват, позволяющий установить консенсусную последовательность генома. Процент закрытых нолем позиций в тестовых выборках был очень низким. Результаты также приведены в таблице 10.
Таблица 10 - покрытие генома
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="Последовательность.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-07-17">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024120035</ApplicationNumberText>
<FilingDate>2024-07-17</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>24NII01</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024120035</ApplicationNumberText>
<FilingDate>2024-07-17</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение «Научно-исследовательский институт гриппа имени
А.А. Смородинцева» Министерства здравоохранения Российской
Федерации</ApplicantName>
<ApplicantNameLatin>Smorodintsev Research Institute of
Influenza</ApplicantNameLatin>
<InventionTitle languageCode="ru">Панель праймеров для
полногеномного секвенирования вирусов парагриппа человека 1 и 2
типа</InventionTitle>
<SequenceTotalQuantity>68</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accaaacaagagraaaaacttgttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catgaactgggtctctgagtatacatata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttacataagagatgcaggattagcatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgacgtcraggagtccgatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagcatacacgaaaccaacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aattcatgttgtgatttcattacacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgtyaaagaacgaagagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagrgggagaggttctactgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcyacaatttcaaccagcaatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgcccagatgactagattcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attgagaagatgaagytaatattctctct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctagtgcaattccygcagttatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatcttcaggaatccctgataaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggacccactttgatgatttgatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattcataaatggtggtgtrgtagcta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q149">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccygtttgttgcatgacttctctat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgacagtatcctccgtgaacga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q151">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtgccaacctgargatctagtatata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q152">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcaatgatgctcttaagataacttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q153">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaatcggataaggttcaaaagtgta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgctagayatcaatcaaccttatgatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q155">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgcatgacactcatatagtgtctttagtat</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q156">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcattgataaagattttcagagagacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q157">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcattacatctttgaccaaacaatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaatatgaataagtgcaattcaaatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q159">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggttcttgattcatcacacga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q160">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaatattgatyccrgctaatatagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q161">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgatacaggagtgagtaacttcaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q162">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgttaagaacttaagcaagccgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q163">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacgcatgaaaagatcaacagagta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q164">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agacacatcacatgcagtcytaaaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q165">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaaccttgaccctttgacataaact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q166">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcrggtgcaatgctgtcttgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q167">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcttgtagacatagacaatgctatcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q168">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagcattacaaatcttcggatttga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q169">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accagacaagagtttaagaaatatcgatat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q170">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accaaggggagaattagatggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q171">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggtcctagactctgatartgtagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q172">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cyatggtgggagacattggca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q173">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctcagtggtgtatgttggttcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aadcataggcccggacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q175">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggattcrggctttcgtgtgatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q176">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ytccagtagtaattgcyggtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q177">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataytcaaatccagcttgtagctttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagacatcaagccagagrgagga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q179">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aataaagctagcrccaccatcagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q180">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatgatagtatgcatctttgttatgtacact</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q181">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcttragaaagttcccgataata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgacaccraactctgtattytgtag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q183">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcagttcggaaaatgattcta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q184">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgayacagcttaatccrctcaacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q185">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggagttwccggcacargttatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q186">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gragcgggatctatcayctaggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q187">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgggaagtgccctagtaataag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q188">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagattatratataggccagaatggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q189">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>crattaaatctggagaaaggttgga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q190">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgggtgtctacaacttaaagatccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q191">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtatcatcaggatctgtgaggtatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q192">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcattgcttactatgagtcaaattgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q193">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgatattgcggttaaataatctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q194">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcyattcgtcaactcacatatgatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q195">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cratatcaagtgcatcattccagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q196">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagaagagttgcatcaatggcata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q197">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaagaacttgtttaacycccca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="65">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q198">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agacgtgcaatgaatcttgatattatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="66">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q199">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtygattcgagatctatatgracaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="67">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q200">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>wgcagttacrgacttatcracaaagga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="68">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q201">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accaaggggaaaatcaatatgtttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| Способ определения генотипа ротавирусов первой и второй геногрупп и реассортантов между ними методом мультиплексной ПЦР | 2024 |
|
RU2833073C1 |
| Панель праймеров для секвенирования сезонных коронавирусов человека hCoV-HKU1, hCoV-NL63, hCoV-OC43, hCoV-229E | 2024 |
|
RU2837888C1 |
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Способ анализа крови ВИЧ-инфицированных пациентов | 2024 |
|
RU2830447C1 |
| Способ мультиплексной идентификации 32 генетических маркеров льна | 2022 |
|
RU2804939C1 |
| Способ пробоподготовки образцов ДНК вируса гепатита В для полногеномного секвенирования и олигонуклеотидные праймеры для его реализации | 2023 |
|
RU2818585C1 |
| Способ выявления рекомбинантных форм вируса гепатита C и мутаций лекарственной устойчивости вируса гепатита С к препаратам прямого противовирусного действия методом полимеразной цепной реакции с последующим секвенированием | 2023 |
|
RU2824565C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ГРИБОВ ВИДОВОГО КОМПЛЕКСА COLLETOTRICHUM ACUTATUM | 2022 |
|
RU2807352C1 |
Изобретение относится к области биотехнологии. Описана панель праймеров для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа, включающая праймеры для секвенирования вируса парагриппа 1 типа и праймеры для секвенирования вируса парагриппа 2 типа. Технический результат состоит в обеспечении возможности полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа. 2 ил., 10 табл., 2 пр.
Панель праймеров для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа, включающая праймеры для секвенирования вируса парагриппа 1 типа:
прямой праймер hPIV1-F1 – ACCAAACAAGAGRAAAAACTTGTTT (SEQ NO:1);
обратный праймер hPIV1-R1 – CATGAACTGGGTCTCTGAGTATACATATA (SEQ NO:2);
прямой праймер hPIV1-F2 – TTACATAAGAGATGCAGGATTAGCATC (SEQ NO:3);
обратный праймер hPIV1-R2 – CGACGTCRAGGAGTCCGATG (SEQ NO:4);
прямой праймер hPIV1-F3 – CAGCATACACGAAACCAACCTT (SEQ NO:5);
обратный праймер hPIV1-R3 – AATTCATGTTGTGATTTCATTACACC (SEQ NO:6);
прямой праймер hPIV1-F4 – CCGTYAAAGAACGAAGAGCC (SEQ NO:7);
обратный праймер hPIV1-R4 – TGAGRGGGAGAGGTTCTACTGT (SEQ NO:8);
прямой праймер hPIV1-F5 – TCYACAATTTCAACCAGCAATC (SEQ NO:9);
обратный праймер hPIV1-R5 – GCTGCCCAGATGACTAGATTCAT (SEQ NO:10);
прямой праймер hPIV1-F6 – ATTGAGAAGATGAAGYTAATATTCTCTCT (SEQ NO:11);
обратный праймер hPIV1-R6 – GCTAGTGCAATTCCYGCAGTTATC (SEQ NO:12);
прямой праймер hPIV1-F7 – GATCTTCAGGAATCCCTGATAACA (SEQ NO:13);
обратный праймер hPIV1-R7 – GGACCCACTTTGATGATTTGATT (SEQ NO:14);
прямой праймер hPIV1-F8 – CATTCATAAATGGTGGTGTRGTAGCTA (SEQ NO:15);
обратный праймер hPIV1-R8 – CCYGTTTGTTGCATGACTTCTCTAT (SEQ NO:16);
прямой праймер hPIV1-F9 – TGACAGTATCCTCCGTGAACGA (SEQ NO:17);
обратный праймер hPIV1-R9 – GAGTGCCAACCTGARGATCTAGTATATA (SEQ NO:18);
прямой праймер hPIV1-F10 – TGCAATGATGCTCTTAAGATAACTTG (SEQ NO:19);
обратный праймер hPIV1-R10 – AAAATCGGATAAGGTTCAAAAGTGTA (SEQ NO:20);
прямой праймер hPIV1-F11 – TGCTAGAYATCAATCAACCTTATGATT (SEQ NO:21);
обратный праймер hPIV1-R11 – TTGCATGACACTCATATAGTGTCTTTAGTAT (SEQ NO:22);
прямой праймер hPIV1-F12 – TCATTGATAAAGATTTTCAGAGAGACAT (SEQ NO:23);
обратный праймер hPIV1-R12 – CTCATTACATCTTTGACCAAACAATG (SEQ NO:24)
прямой праймер hPIV1-F13 – AAATATGAATAAGTGCAATTCAAATGG (SEQ NO:25);
обратный праймер hPIV1-R13 – CTGGTTCTTGATTCATCACACGA (SEQ NO:26);
прямой праймер hPIV1-F14 – GCAATATTGATYCCRGCTAATATAGG (SEQ NO:27);
обратный праймер hPIV1-R14 – GTCGATACAGGAGTGAGTAACTTCAAA (SEQ NO:28);
прямой праймер hPIV1-F15 – TGTTAAGAACTTAAGCAAGCCGG (SEQ NO:29);
обратный праймер hPIV1-R15 – CACGCATGAAAAGATCAACAGAGTA (SEQ NO:30);
прямой праймер hPIV1-F16 – AGACACATCACATGCAGTCYTAAAAGT (SEQ NO:31);
обратный праймер hPIV1-R16 – AAAACCTTGACCCTTTGACATAAACT (SEQ NO:32);
прямой праймер hPIV1-F17 – GCRGGTGCAATGCTGTCTTGT (SEQ NO:33);
обратный праймер hPIV1-R17 – AATCTTGTAGACATAGACAATGCTATCCA (SEQ NO:34);
прямой праймер hPIV1-F18 – AAGCATTACAAATCTTCGGATTTGA (SEQ NO:35);
обратный праймер hPIV1-R18 – ACCAGACAAGAGTTTAAGAAATATCGATAT (SEQ NO:36),
а также включающая праймеры для секвенирования вируса парагриппа 2 типа:
прямой праймер hPIV2-F1 – ACCAAGGGGAGAATTAGATGGC (SEQ NO:37);
обратный праймер hPIV2-R1 – ATGGGTCCTAGACTCTGATARTGTAGC (SEQ NO:38);
прямой праймер hPIV2-F2 – CYATGGTGGGAGACATTGGCA (SEQ NO:39);
обратный праймер hPIV2-R2 – GCTCAGTGGTGTATGTTGGTTCC (SEQ NO:40);
прямой праймер hPIV2-F3 – AADCATAGGCCCGGACGG (SEQ NO:41);
обратный праймер hPIV2-R3 – GGATTCRGGCTTTCGTGTGATC (SEQ NO:42);
прямой праймер hPIV2-F4 – YTCCAGTAGTAATTGCYGGTCC (SEQ NO:43);
обратный праймер hPIV2-R4 – ATAYTCAAATCCAGCTTGTAGCTTTG (SEQ NO:44);
прямой праймер hPIV2-F5 – AAGACATCAAGCCAGAGRGAGGA (SEQ NO:45);
обратный праймер hPIV2-R5 – AATAAAGCTAGCRCCACCATCAGT (SEQ NO:46);
прямой праймер hPIV2-F6 – AATGATAGTATGCATCTTTGTTATGTACACT (SEQ NO:47);
обратный праймер hPIV2-R6 – CGCTTRAGAAAGTTCCCGATAATA (SEQ NO:48);
прямой праймер hPIV2-F7 – GTGACACCRAACTCTGTATTYTGTAG (SEQ NO:49);
обратный праймер hPIV2-R7 – GGCAGTTCGGAAAATGATTCTA (SEQ NO:50);
прямой праймер hPIV2-F8 – TGAYACAGCTTAATCCRCTCAACAT (SEQ NO:51);
обратный праймер hPIV2-R8 – GGAGTTWCCGGCACARGTTATG (SEQ NO:52);
прямой праймер hPIV2-F9 – GRAGCGGGATCTATCAYCTAGGC (SEQ NO:53);
обратный праймер hPIV2-R9 – GCGGGAAGTGCCCTAGTAATAAG (SEQ NO:54);
прямой праймер hPIV2-F10 – AAGATTATRATATAGGCCAGAATGGC (SEQ NO:55);
обратный праймер hPIV2-R10 – CRATTAAATCTGGAGAAAGGTTGGA (SEQ NO:56);
прямой праймер hPIV2-F11 – TGGGTGTCTACAACTTAAAGATCCAG (SEQ NO:57);
обратный праймер hPIV2-R11 – ATGTATCATCAGGATCTGTGAGGTATG (SEQ NO:58);
прямой праймер hPIV2-F12 – TCATTGCTTACTATGAGTCAAATTGG (SEQ NO:59);
обратный праймер hPIV2-R12 – CGATATTGCGGTTAAATAATCTGC (SEQ NO:60);
прямой праймер hPIV2-F13 – TCYATTCGTCAACTCACATATGATC (SEQ NO:61);
обратный праймер hPIV2-R13 – CRATATCAAGTGCATCATTCCAGT (SEQ NO:62);
прямой праймер hPIV2-F14 – AAGAAGAGTTGCATCAATGGCATA (SEQ NO:63);
обратный праймер hPIV2-R14 – GCAAGAACTTGTTTAACYCCCCA (SEQ NO:64);
прямой праймер hPIV2-F15 – AGACGTGCAATGAATCTTGATATTATC (SEQ NO:65);
обратный праймер hPIV2-R15 – GTYGATTCGAGATCTATATGRACAAG (SEQ NO:66);
прямой праймер hPIV2-F16 – WGCAGTTACRGACTTATCRACAAAGGA (SEQ NO:67);
обратный праймер hPIV2-R16 – ACCAAGGGGAAAATCAATATGTTTT (SEQ NO:68).
| CN 110484655 A, 22.11.2019 | |||
| CN 108239676 A, 03.07.2018 | |||
| НАБОР ОЛИГОДЕЗОКСИРИБОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНЫХ ЗОНДОВ ДЛЯ ИДЕНТИФИКАЦИИ РНК И ДИФФЕРЕНЦИАЦИИ ВИРУСА ПАРАГРИППА ЧЕЛОВЕКА 1, 2, 3 И 4 ТИПОВ | 2012 |
|
RU2515911C1 |
Авторы
Даты
2025-01-28—Публикация
2024-07-17—Подача