Область техники
[0001] Настоящее изобретение относится к бактериям, относящимся к роду Bifidobacterium, классифицированным как Bifidobacterium pseudocatenulatum.
Уровень техники
[0002] Полагается, что одной из причин влияния пробиотиков на физиологическую функцию хозяина является то, что пробиотики метаболизируют источники углеводов, остающиеся в пищеварительном тракте, и продуцируют короткоцепочечные жирные кислоты, такие как молочная кислота и уксусная кислота. Результаты предшествующих исследований показали, что геномы видов бифидобактерий, доминирующих в кишечном тракте взрослых людей, содержат больше генов, участвующих в метаболизме неперевариваемых полисахаридов растительного происхождения, в частности, ксиланов, содержащихся в большом количестве в рационе взрослых, по сравнению с геномами видов бифидобактерий, выделенных у детей грудного возраста.
[0003] Таким образом, предполагается, что штаммы Bifidobacterium, обладающие высокой способностью утилизировать неперевариваемые полисахариды, будут расти в кишечнике взрослых субъектов и иметь высокую способность продуцировать органические кислоты, и, следовательно, могут стать многообещающим кандидатом в пробиотики для взрослых.
[0004] Ранее было известно, что бифидобактерии способны утилизировать неперевариваемые полисахариды, например, штаммы Bifidobacterium breve (непатентный документ 1) и штаммы Bifidobacterium bifidum (непатентный документ 2) способны утилизировать крахмал, и штаммы Bifidobacterium pseudocaten) способны утилизировать арабиногалактан.
[0005] Однако до сих пор не были известны бифидобактерии, способные расщеплять связи между ксилозными единицами ксиланов, которые представляют собой типичные неперевариваемые полисахариды, содержащиеся в рационе.
Список цитированных ссылок
Непатентная литература
[0006] Непатентный документ 1: BMC Genomics (2014) 15: 170.
Непатентный документ 2: Environmental Microbiology (2015) 17(7), 2515-2531.
Непатентный документ 3: Japanese Journal of Food Microbiology, Vol. 24 No.4 Page 163-170.
Сущность изобретения
Техническая проблема
[0007] Настоящее изобретение относится к получению нового штамма Bifidobacterium, способного утилизировать ксиланы.
Техническое решение проблемы
[0008] Авторы настоящего изобретения провели обширные исследования и в результате выделили и идентифицировали новый штамм микроорганизмов, способный эффективно утилизировать ксиланы из кишечника человека.
[0009] В частности, настоящее изобретение относится к следующим пунктам 1-9.
1. Бактерии, относящиеся к роду Bifidobacterium, содержащие ген ксиланазы в геноме.
2. Бактерии, относящиеся к роду Bifidobacterium по п.1, где ген ксиланазы представляет собой полинуклеотид, состоящий из нуклеотидной последовательности, показанной в SEQ ID NO: 1, или полинуклеотид, имеющий 70% идентичность или более с данной нуклеотидной последовательностью, и кодирующий белок, обладающий ксиланазной активностью.
3. Бактерии, относящиеся к роду Bifidobacterium по п.1 или 2, которые обладают способностью утилизировать ксиланы.
4. Бактерии, относящиеся к роду Bifidobacterium по п.3, где ксиланы представляют собой ксилан и арабиноксилан.
5. Бактерии, относящиеся к роду Bifidobacterium по любому из пп.1-4, которые обладают способностью утилизировать крахмал.
6. Бактерии, относящиеся к роду Bifidobacterium по любому из пп.1-5, где бактерии, относящиеся к роду Bifidobacterium, классифицируются как Bifidobacterium pseudocatenulatum.
7. Bifidobacterium pseudocatenulatum YIT11027 (NITE ВР-02928), Bifidobacterium pseudocatenulatum YIT11055 (NITE ВР-02929), Bifidobacterium pseudocatenulatum YIT11057 (NITE ВР-02930), Bifidobacterium pseudocatenulatum YIT11952 (NITE ВР-02931), Bifidobacterium pseudocatenulatum YIT11954 (NITE ВР-02932), Bifidobacterium pseudocatenulatum YIT12989 (NITE BP-02933) или близкородственный им штамм бактерий.
8. Пищевой продукт или напиток, содержащий бактерии, относящиеся к роду Bifidobacterium по любому из пп.1-7.
9. Пищевой продукт или напиток по п.8, который представляет собой кисломолочный продукт или напиток.
Преимущественные эффекты изобретения
[0010] Новые бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, обладают способностью расщеплять и утилизировать ксиланы, содержащиеся в большом количестве в рационах взрослых субъектов, в частности арабиноксилан. Таким образом, предполагается, что Bifidobacterium pseudocatenulatum по настоящему изобретению будут расти в кишечнике взрослых субъектов и иметь высокую способность продуцировать органические кислоты, и могут использоваться для лекарственных продуктов, пищевых продуктов и т.п. в качестве пробиотиков для взрослых субъектов.
Краткое описание фигур
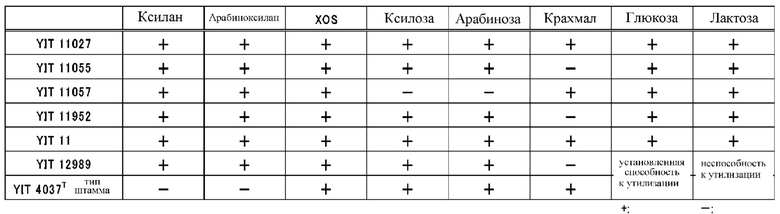
[0011] На фиг. 1 показана активность штамма YIT11057 расщеплять связи между ксилозными единицами.
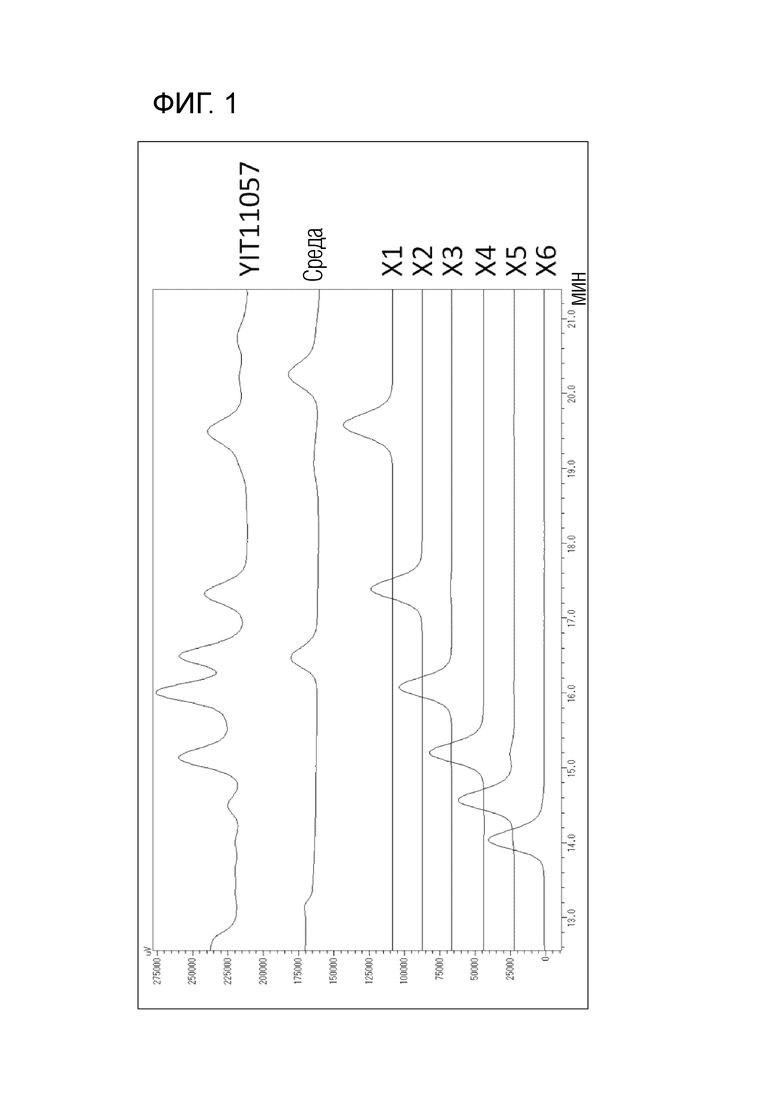
На фиг. 2 показана устойчивость штамма YIT11057 к кислоте/желчным кислотам.
Описание вариантов осуществления
[0012] Тип бактерий, относящихся к роду Bifidobacterium по настоящему изобретению, особым образом не ограничивается, при условии, что бактерии содержат ген ксиланазы в геноме. Типом бактерий, относящихся к роду Bifidobacterium, может быть любой, например, из Bifidobacterium pseudocatenulatum, Bifidobacterium breve, Bifidobacterium longum (B. longum), Bifidobacterium bifidum (B. bifidum), Bifidobacterium animalis (B. animalis), Bifidobacterium suis (B. suis), Bifidobacterium infantis (B. infantis), Bifidobacterium adolescentis (B. adolescentis), Bifidobacterium catenulatum (B. catenulatum), Bifidobacterium lactis (B. lactis), Bifidobacterium globosum (B. globosum), Bifidobacterium angulatum (B. angulatum) и Bifidobacterium dentium (B. dentium), и предпочтительно представляет собой Bifidobacterium pseudocatenulatum.
Бифидобактерии, содержащие ген ксиланазы в геноме, до сих пор не были известны, и бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, являются новыми бактериями.
[0013] Термин «ген ксиланазы» относится к гену, кодирующему ксиланазу, классифицированную как GH10 (эндо-1,4-бета-ксиланаза A; xynA), и «ген ксиланазы», содержащийся в бактериях, относящихся к роду Bifidobacterium по настоящему изобретению, в геноме, в частности, включает полинуклеотид, состоящий из нуклеотидной последовательности, показанной в SEQ ID NO: 1, или полинуклеотид, имеющий 70% идентичность или более, предпочтительно 90% или более, более предпочтительно 95% или более, еще более предпочтительно 98% или более, еще более предпочтительно 99% или более с данной нуклеотидной последовательностью, и кодирует белок, обладающий ксиланазной активностью.
В данном случае, полинуклеотид, состоящий из нуклеотидной последовательности, показанной в SEQ ID NO: 1, относится к гену ксиланазы, содержащемуся в нижеописанном штамме YIT11057.
[0014] Идентичность нуклеотидной последовательности представляет собой величину, полученную выравниванием двух нуклеотидных последовательностей, которые требуется сравнить, таким образом, чтобы две последовательности нуклеиновых кислот максимально соответствовали друг другу, делением числа совпадающих оснований на общее число оснований и выражением частного в процентах. Специалисты в данной области могут соответствующим образом установить параметры программного обеспечения, такого как BLAST, ClustalX или Genetyx, для определения идентичности нуклеотидных последовательностей.
Термин «ксиланазная активность» означает активность гидролиза связей между ксилозными единицами ксиланов с использованием ксиланов в качестве субстрата с образованием ксилоолигосахаридов и ксилозы (гидролизующая активность на ксилане).
В данном случае, примеры ксиланов включают ксилан, арабиноксилан, глюкуроноксилан, глюкуроноарабиноксилан и ацетилксилан, и предпочтительны ксилан и арабиноксилан.
Таким образом, другими словами, бактерии, относящиеся к роду Bifidobacterium, содержащие ген ксиланазы в своем геноме по настоящему изобретению, представляют собой бактерии, относящиеся к роду Bifidobacterium, обладающие способностью расщеплять связи между ксилозными единицами ксиланов.
[0015] В рамках настоящего изобретения, выражение «обладающие способностью утилизировать ксиланы» означает способность расти с использованием ксиланов в качестве источников углерода. В частности, это выражение означает, например, способность расти в среде, содержащей только ксиланы в качестве источников углерода. Например, это выражение означает, что при культивировании в течение 72 ч в среде, содержащей ксиланы в качестве единственного источника углеводов, мутность составляет, например, 0,1 или выше пропорционально увеличению OD600. Мутность предпочтительно составляет 0,3 или выше, более предпочтительно 0,4 или выше пропорционально увеличению OD600. Когда изменение мутности невозможно измерить за счет того, что среда, содержащая ксиланы, имеет большое количество нерастворимых фракций, или по другим причинам, то выражение означает, что количество короткоцепочечных жирных кислот, образовавшихся в культуральном супернатанте после 72-ч культивирования, например, общее количество образовавшейся молочной кислоты, уксусной кислоты и муравьиной кислоты, составляет 10 мМ или более. Количество образовавшихся короткоцепочечных жирных кислот предпочтительно составляет в целом 20 мМ или выше, более предпочтительно 40 мМ или выше.
[0016] Среду, которая используется в анализе способности утилизировать ксиланы, можно получить добавлением ксиланов в среду, не содержащую источников углеводов, и по составу среда, не содержащая источников углеводов, может представлять собой по составу жидкую среду M-ILS, пептон-дрожжевую среду (PY) или аналогичные.
Количество ксиланов, добавленных в среду, предпочтительно составляет от 0,01 до 10 мас. %, более предпочтительно от 0,05 до 5 мас. %, еще более предпочтительно от 0,1 до 1 мас. % в среде. Желательно, чтобы в качестве контроля использовались бактерии, относящиеся к представляющему интерес роду Bifidobacterium, которые культивируют в среде, не содержащей источников углеводов, с последующим определением значений OD.
[0017] Как описано выше, бактерии, относящиеся к роду Bifidobacterium, содержащие ген ксиланазы в геноме по настоящему изобретению, обладают способностью расщеплять и утилизировать ксиланы и, следовательно, могут способствовать обеспечению короткоцепочечных жирных кислот, таких как уксусная кислота, молочная кислота и муравьиная кислота, образовавшихся при деградации ксиланов.
[0018] Бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, можно получить скринингом бактерий, относящихся к роду Bifidobacterium, находящихся, например, в кишечнике людей (например, взрослых людей или детей грудного возраста), предпочтительно Bifidobacterium pseudocatenulatum, с использованием в качестве показателя наличия гена ксиланазы или вышеуказанной способности утилизировать ксиланы. Например, из Bifidobacterium pseudocatenulatum можно выбрать 16 штаммов, приведенных в примерах ниже, и еще шесть штаммов YIT11027, YIT11055, YIT11057, YIT11952, YIT11954 и YIT12989. Бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, не ограничиваются ими и включают бактериальные штаммы, которые биологически и генетически близки к ним, при условии, что бактерии, относящиеся к роду Bifidobacterium, содержат ген ксиланазы в геноме. Бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, могут представлять собой природный штамм, находящийся в природе, вариант природного штамма или генетически модифицированный вид, при условии, что они содержат ген ксиланазы в своем геноме.
[0019] Как показано ниже, штамм YIT11027, штамм YIT11055, штамм YIT11057, штамм YIT11952, штамм YIT11954 и штамм YIT12989 были депонированы в депозитарии патентованных микроорганизмов Национального института технологии и оценки (№122, Kazusakamatari 2-5-8, Kisarazu-shi, Chiba) 25 марта 2019 г.
Bifidobacterium pseudocatenulatum YIT11027 (NITE BP-02928),
Bifidobacterium pseudocatenulatum YIT11055 (NITE BP-02929),
Bifidobacterium pseudocatenulatum YIT11057 (NITE BP-02930),
Bifidobacterium pseudocatenulatum YIT11952 (NITE BP-02931),
Bifidobacterium pseudocatenulatum YIT11954 (NITE BP-02932) и
Bifidobacterium pseudocatenulatum YIT12989 (NITE BP-02933).
Из них штамм YIT11027, штамм YIT11057, штамм YIT11954 или близкородственные им штаммы бактерий являются предпочтительными с точки зрения их способности утилизировать ксиланы и, кроме того, с точки зрения того, что они также обладают способностью к утилизации крахмала в комбинации. Кроме того, штамм YIT11057 или близкородственные ему бактериальные штаммы более предпочтительны с точки зрения того, что они обладают устойчивостью к кислоте/желчным кислотам, и также обладают свойствами, заключающимися в том, что в геноме не обнаруживается лизогенный фаг.
В данном случае, термин «способность к утилизации крахмала» означает способность расти с использованием крахмала в качестве источника углерода. Термин «устойчивость к кислоте/желчным кислотам» означает сохранение жизнеспособности после непрерывного воздействия искусственного желудочного сока и искусственной желчной кислоты. Термин «отсутствие лизогенного фага» означает, что область, имеющая высокую гомологию с присутствующим фагом, не была обнаружена в последовательности генома с использованием программы PHASTER, которая является веб-сервером по поиску фаговых областей в геноме (http://phaster.ca/).
[0020] Термин «близкородственные бактериальные штаммы» означает бактериальные штаммы, имеющие идентичные последовательности в анализе мультилокусных последовательностей (MLSA), используемого для оценки множества последовательностей генов домашнего хозяйства.
В частности, сообщалось, что полноразмерные последовательности субъединицы B ДНК-гиразы (gyrB), 50S рибосомного белка L2 (rplB), амидофосфорибозилтрансферазы (purF), ДНК-направленной РНК-полимеразы субъединицы бета (rpoB), АТФ-зависимой протеолитической субъединицы протеазы Clp (ClpP), фактора элонгации G (fusA) и изолейцин-тРНК-лигазы (ileS), являются генами, подходящими для MLSA-анализа бифидобактерий (International Journal of Systematic and Evolutionary Microbiology (2006), 56, 2783-2792), все гены ассоциируются с каждым бактериальным штаммом, и совпадение последовательности между сравниваемыми штаммами анализируется с использованием такой программы, как VSEARCH или BLAST. Когда идентичность последовательностей составляет 90% или более, предпочтительно 95% или более, более предпочтительно 98% или более, еще более предпочтительно 99% или более, то можно определить, что бактериальные штаммы являются близкородственными видами.
Нуклеотидные последовательности генов gyrB, rplB, purF, rpoB, clpC, fusA и ileS в штамме YIT11057 приведены в списке последовательностей (gyrB: SEQ ID NO: 2, rplB: SEQ ID NO: 3, purF: SEQ ID NO: 4, rpoB: SEQ ID NO: 5, clpC: SEQ ID NO: 6, fusA: SEQ ID NO: 7 и ileS: SEQ ID NO: 8).
[0021] Форма применения бактерий, относящихся к роду Bifidobacterium по настоящему изобретению, особым образом не ограничивается, и можно использовать лиофилизированные бактерии или культивированный продукт, содержащий эти бактерии. В любой из форм бактерии предпочтительно находятся в жизнеспособном состоянии.
[0022] Бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, также можно смешать с твердым или жидким фармацевтическим нетоксичным носителем и использовать в форме обычного фармацевтического препарата. Примеры таких препаратов включают твердые препараты, такие как таблетки, гранулы, порошки и капсулы, жидкие препараты, такие как растворы, суспензии и эмульсии, и лиофилизированные препараты. Данные препараты можно приготовить обычными способами, используемыми для изготовления препаратов. Примеры фармацевтического нетоксичного носителя включают глюкозу, лактозу, сахарозу, крахмал, маннит, декстрин, глицериды жирных кислот, полиэтиленгликоль, гидроксиэтилкрахмал, этиленгликоль, сложные эфиры полиоксиэтиленсорбитана и жирных кислот, аминокислоты, желатин, альбумин, воду и физиологический раствор. При необходимости также могут быть добавлены обычные добавки, такие как стабилизаторы, смачивающие агенты, эмульгаторы, связующие агенты, агенты, обеспечивающие тоничность, и эксципиенты.
[0023] Бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, можно приготовить не только в виде вышеуказанных препаратов, но также добавить в пищевой продукт или напиток для применения. При добавлении в пищевой продукт или напиток бактерии, относящиеся к роду Bifidobacterium, могут быть включены непосредственно или вместе с различными питательными ингредиентами. В частности, при добавлении в пищевой продукт или напиток бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, можно приготовить в форме, подходящей для употребления в пищу, т.е. в гранулированной форме, форме в виде частиц, таблетки, капсулы, пасты или подобной форме, с использованием обычных средств с соответствующими добавками, применяемыми для пищевого продукта или напитков, которые можно добавить в различные продукты питания, например переработанные мясные продукты, такие как ветчина и колбаса, переработанные морепродукты, такие как «пюре» из рыбы с белым мясом (камабоко) и продукт из рыбной пасты наподобие длинных котлеток (чикува), хлеб, кондитерские изделия, масло и сухое молоко, или которые можно добавить в напитки, такие как вода, фруктовый сок, молоко, безалкогольные напитки и чайные напитки. Пищевой продукт или напиток включает корма для животных.
[0024] Кроме того, в качестве пищевого продукта или напитка подходящими для использования являются кисломолочный продукт или напиток, или ферментированный пищевой продукт или напиток, такой как кисломолочный продукт на основе сои, ферментированный фруктовый сок и ферментированный овощной сок, которые содержат бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению в жизнеспособном состоянии, и, в частности, предпочтительно использовать кисломолочные продукты или напитки. Кисломолочный пищевой продукт или напиток можно приготовить в соответствии с обычным способом, и, например, при получении кисломолочного продукта бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, инокулируют и культивируют в стерилизованной молочной среде отдельно или вместе с другими микроорганизмами, и культивированный продукт подвергают обработке гомогенизацией для получения кисломолочной основы. Затем добавляют и перемешивают отдельно приготовленный раствор сиропа, смесь гомогенизируют с помощью гомогенизатора или подобного устройства, и дополнительно добавляют вкусовое вещество для получения конечного продукта. Полученный таким образом кисломолочный пищевой продукт или напиток также может представлять собой продукт в любой форме, такой как продукт без добавок, не содержащий сиропа (подсластителя), продукт мягкого типа, продукт с фруктовым вкусом, в твердой форме или жидкой форме.
[0025] К таким кисломолочным продуктам или напиткам могут быть добавлены необязательные ингредиенты, включающие подсластители, такие как сироп, эмульгаторы, загустители (стабилизаторы) и различные витамины. Можно добавить следующее: в качестве сиропа могут быть добавлены сахариды, такие как глюкоза, сахароза, фруктоза, высокофруктозный кукурузный сироп, сироп глюкозы, палатиноза, трегалоза, лактоза, ксилоза, галактоолигосахарид (GOS), ксилоолигосахарид (XOS), арабиноксилоолигосахарид (AXOS), ксилан, арабиноксилан, арабиноолигосахарид (AOS), арабинан, мальтоза, мед и патока; сахарные спирты, такие как сорбит, ксилит, эритрит, лактит, палатинит, сироп с пониженным содержанием сахара и сироп с пониженным содержанием мальтозы; и интенсивные подсластители, такие как аспартам, тауматин, сукралоза, ацесульфам К и стевия; эмульгаторы, такие как сложные эфиры глицерина и жирных кислот, сложные эфиры полиглицерина и жирных кислот, сложные эфиры сорбитана и жирных кислот и лецитин; и загустители (стабилизаторы), такие как агар, желатин, каррагинан, гуаровая камедь, ксантановая камедь, пектин, камедь рожкового дерева, геллановая камедь, карбоксиметилцеллюлоза, соевый полисахарид и альгинат пропиленгликоля. Кроме того, можно добавить: витамины, такие как витамин A, витамин B, витамин C и витамин E; минеральные вещества, такие как кальций, магний, цинк, железо и марганец; подкислители, такие как лимонная кислота, молочная кислота, уксусная кислота, яблочная кислота, винная кислота и глюконовая кислота; молочные жиры, такие как сливки, масло и сметана; вкусовые вещества на основе йогурта, ягод, апельсина, китайской айвы, периллы, цитрусовых, яблока, мяты, винограда, абрикоса, груши, заварного крема, персика, дыни, банана, тропических фруктов, трав, черного чая, кофе и т.п.; растительные экстракты, экстракты коричневого сахара и тому подобное.
[0026] Для производства кисломолочного пищевого продукта или напитка можно также использовать в комбинации микроорганизмы, отличные от бактерий, относящихся к роду Bifidobacterium по настоящему изобретению. Примеры таких микроорганизмов включают бактерии, относящиеся к роду Lactobacillus, такие как Lactobacillus casei, Lactobacillus acidophilus (L. acidophilus), Lactobacillus plantarum (L. plantarum), Lactobacillus buchneri (L. buchneri), Lactobacillus gallinarlovum (L.) (L. amylovorus), Lactobacillus brevis (L. brevis), Lactobacillus rhamnosus (L. rhamnosus), Lactobacillus kefir (L. kefir), Lactobacillus paracasei (L. paracasei), Lactobacillus crispatus (L. crispatus), Lactobacillus. zeae), Lactobacillus helveticus (L. helveticus), Lactobacillus salivalius (L. salivalius), Lactobacillus gasseri (L. gasseri), Lactobacillus fermentum (L. fermentum), Lactobacillus reuteri (L. reuteri), Lactobacillus delbacillus. подвид bulgaricus (L. delbrueckii подвид bulgaricus), подвид Lactobacillus delbrueckii. delbrueckii (L. delbrueckii подвид delbrueckii) и Lactobacillus johnsonii (L. johnsonii), бактерии, относящиеся к роду Streptococcus, такие как Streptococcus thermophilus, бактерии, относящиеся к роду Lactococcus, такие как Lactococcus lactis подвид lactis (Lactococcus lactis подвид lactis), Lactococcus lactis подвид cremoris (Lactococcus lactis subsp. cremoris), бактерии, относящиеся к роду Enterococcus, такие как Enterococcus faecalis и Enterococcus faecium (E. faecium), бактерии, относящиеся к роду Bacillus, такие как Bacillus subtilis, и дрожжи, принадлежащие к роду Saccharomyses и Torulaspora такие как Saccharomyses cerevisiae, Torulaspora delbrueckii и Candida kefyr. Предпочтительно, чтобы одна или более бактерий, выбранных из группы, состоящей из бактерий, относящихся к роду Lactobacillus, Streptococcus и Lactococcus, использовались в сочетании с бактериями, относящимися к роду Bifidobacterium по настоящему изобретению, для производства кисломолочных пищевых продуктов или напитков, поскольку они очень приятны на вкус, что может способствовать приему таких продуктов питания или напитков.
[0027] Когда используются бактерии, относящиеся к роду Bifidobacterium по настоящему изобретению, то их доза строго не ограничивается, и их подходящая доза составляет предпочтительно от 105 КОЕ до 1013 КОЕ, особенно предпочтительно от 108 КОЕ до 1012 КОЕ в сутки в пересчете на количество жизнеспособных бактерий.
Примеры
[0028] Пример 1: скрининг бактерий, относящихся к роду Bifidobacterium, обладающих способностью утилизировать ксиланы
i) Использованные бактериальные штаммы
Объектами скрининга были 53 штамма, выделенные из кала человека и идентифицированные как относящиеся к Bifidobacterium pseudocatenulatum.
[0029] ii) Субстрат
Для анализа использовали ксилан (полученный из овса), арабиноксилан (полученный из пшеницы) и ксилоолигосахарид (XOS), промышленно полученный ферментативной обработкой ксилана. Результаты анализа ксилана и арабиноксилана с использованием ВЭЖХ не показали присутствия моносахарида (данные не приведены). Кроме того, был проведен анализ на ксилозу и арабинозу, которые являются составляющими моносахаридами ксилана и арабиноксилана, соответственно, и в качестве контролей для подтверждения роста также был анализирован крахмал, который является типичным неперевариваемым полисахаридом, и глюкоза, которая является составляющим моносахаридом крахмала, и лактоза. После приготовления 1% суспензии каждого субстрата, ксилан, арабиноксилан и крахмал подвергали стерилизации в автоклаве, и другие субстраты подвергали стерилизации фильтрованием с использованием 0,22 мкм фильтра.
[0030] iii) Тестирование способности к утилизации
Для предотвращения подавления роста бактерий за счет снижения pH, связанного с ростом, использовали среду m-PY, содержащую 100 мМ PIPES. 1% раствор субстрата смешивали с равным количеством концентрированной в два раза среды m-PY, хранившейся при охлаждении в атмосфере, заполненной азотом, до использования, и смесь использовали для культурального теста (конечная концентрация: 0,5%).
Замороженную бактериальную жидкость инокулировали в модифицированный агар GAM, и затем культивировали в течение 2-3 суток, и полученные таким образом колонии субкультивировали в модифицированной жидкой среде GAM, содержащей 1% глюкозы/лактозы. При субкультивировании старались снять большое количество колоний. Стационарное культивирование проводили в течение ночи при 37°C, и на следующее утро бактериальную жидкость в количестве 5% снова пересевали в ту же свежую жидкую среду. Бактериальную жидкость культивировали в течение примерно 6-9 ч до достижения логарифмической фазы роста, и затем измеряли мутность. 100 мкл культурального раствора центрифугировали, супернатант удаляли и затем добавляли среду m-PY, не содержащую источника углеводов, для обеспечения OD600 0,2 для суспендирования гранул. 198 мкл каждой среды вносили в 96-луночный планшет, добавляли 2 мкл бактериальной жидкости, затем покрывали 50 мкл минерального масла, и бактерии культивировали при 37°C в микропланшетном ридере PowerWave 340 (BIOTEC CO., Ltd.).
Мутность измеряли каждые 30 мин, и проводили мониторинг мутности в течение 60 ч после начала культивирования. Все вышеописанные операции культивирования проводили в анаэробном перчаточном боксе. Через 60 ч отбирали часть культурального раствора, депротеинизировали и затем подвергали анализу ВЭЖХ для измерения количества образовавшихся короткоцепочечных жирных кислот. Когда большое количество нерастворимых частиц присутствовало в среде m-PY, содержащей ксилан, то способность к утилизации определяли на основе наличия или отсутствия образования короткоцепочечных жирных кислот, поскольку невозможно было подтвердить рост бактериальных клеток только с помощью измерения мутности. Короткоцепочечные жирные кислоты в культуральном супернатанте после культивирования в течение 60 ч анализировали с использованием ВЭЖХ для подтверждения того, что общее количество продуцированной молочной кислоты, уксусной кислоты и муравьиной кислоты составляло 10 мМ или более. В табл. 1 показано количество штаммов, которые были способны использовать каждый из различных субстратов.
[0031]
[0032] Способность утилизировать ксилоолигосахариды была показана у всех бактериальных штаммов B. pseudocatenulatum, однако способность утилизировать ксиланы с более длинной цепочкой варьировалась среди бактериальных штаммов. 16 штаммов, которые утилизировали ксилан и арабиноксилан, скринировали как бактерии, относящиеся к роду Bifidobacterium, обладающие способностью утилизировать ксиланы.
[0033] Бактериальные штаммы, относящиеся к роду Bifidobacterium, обладающие способностью утилизировать ксиланы, культивировали в жидкой среде с ксиланом в качестве единственного источника углеводов, и олигосахариды в культуральном супернатанте на более ранней фазе логарифмического роста анализировали с использованием ВЭЖХ. Результаты показали, что были продуцированы ксилоза и ксилоолигосахариды. Полученные данные свидетельствуют о том, что бактерии по настоящему изобретению обладают активностью расщепления связей между ксилозными единицами с образованием ксилозы и ксилоолигосахаридов. На фиг. 1 в качестве иллюстративного примера приведены результаты анализа с использованием штамма YIT11057, который является одним из 16 штаммов. Символы от X1 до X6 на фиг. 1 обозначают ксилозу и ксилоолигосахариды, имеющие соответствующее число ксилозных единиц, и можно видеть, что ксилоза и ксилоолигосахариды, которые отсутствовали в среде, появлялись при культивировании штамма YIT11057.
[0034] Пример 2: свойства шести штаммов YIT11027, YIT11055, YIT11057, YIT11952, YIT11954 и YIT12989
Способность к утилизации шести штаммов YIT11027, YIT11055, YIT11057, YIT11952, YIT11954 и YIT12989, выбранных из вышеуказанных 16 штаммов, обладающих способностью утилизировать ксиланы, показана ниже. Для трех бактериальных штаммов YIT11027, YIT11057 и YIT11954 можно было подтвердить способность к утилизации крахмала, а также способность к утилизации ксилана (табл. 2).
[0035]
[0036] Пример 3: геномный анализ
Геномную ДНК экстрагировали из бактериальных клеток, и черновую геномную последовательность определяли с использованием секвенатора нового поколения Miseq (Illumina, Inc.). Область гена была прогнозирована, и затем был проведен поиск по базе данных генов углевод-активных ферментов dbCAN на предмет всех аминокислотных последовательностей для извлечения генов углевод-активных ферментов. Результаты поиска генов, идентичных по способности утилизировать ксиланы и распределению, показали, что все штаммы, имеющие ген эндо-1,4-бета-ксиланазы А (xynA), относящейся к GH10, способны утилизировать ксиланы. Ни один из штаммов, не содержащих этот ген, не был способен утилизировать ксиланы.
[0037] Пример 4: свойства YIT11057
Для YIT11057 оценивали устойчивость к кислоте/желчным кислотам, проводили исчерпывающий поиск патогенных факторов и обнаружение лизогенного фага в геноме.
1) Устойчивость к кислотам/желчным кислотам
i) Состав искусственного сока
Состав искусственного желудочного сока (pH 3,6):
Пепсин - 40 мг/л
Протеозный пептон - 5 г/л
Желудочный муцин - 1,5 г/л
NaCl - 5 г/л
NaHCO3 - 3 г/л
KH2PO4 - 1 г/л
3,6 N HCl, для доведения pH до 3,6
Искусственный кишечный сок:
NaCl - 5 г/л
KCl - 1 г/л
NaHCO3 - 3 г/л
3 M Na2CO3, для доведения pH до 8,0
[0038] ii) 0,1 мл бактериальной жидкости, культивированной в течение ночи в жидкой среде mGAM, содержащей 1% глюкозы/лактозы, добавляли к 2 мл искусственного желудочного сока, и смесь инкубировали при 37°C в течение 1 ч. Затем добавляли 5,5 мл искусственного кишечного сока и 0,5 мл 16% желчной кислоты, и смесь выдерживали при 37°C в течение 1 ч. До и после кислотной обработки и после обработки желчной кислотой бактериальную жидкость собирали и разбавляли физиологическим раствором, затем 50 мкл разбавленной жидкости наносили на агар mGAM и измеряли КОЕ. Результаты показали, что уровень жизнеспособности составил примерно 0,1% даже после обработки желчной кислотой (фиг. 2). Поскольку уровень КОЕ бактерий без устойчивости после каждой описанной обработки был равен или меньше нижнего предела определения, то было показано, что YIT11057 имеет относительно высокую устойчивость к кислоте/желчным кислотам.
[0039] 2) Всесторонний поиск на патогенные факторы
Черновую геномную последовательность вводили на веб-сервер PATRIC (https://www.patricbrc.org/) (Wattam et al., 2017), и было исследовано наличие или отсутствие генов, имеющих высокую гомологию с последовательностями в базе данных факторов вирулентности. В результате ген патогенного фактора не был обнаружен.
[0040] 3) Обнаружение лизогенных фагов в геноме
Для получения информации о геноме бактериальных штаммов была предпринята попытка обнаружить лизогенные фаги, зарегистрированные в базе данных фагов, с помощью веб-сервера PHASTER (http://phaster.ca/) (Arndt et al., 2016). В результате лизогенные фаги не были обнаружены.
--->
Список последовательностей
<110> KABUSHIKI KAISHA YAKULT HONSHA
<120> НОВЫЙ РОД БАКТЕРИЙ BIFIDOBACTERIUM, ОБЛАДАЮЩИХ ВЫСОКОЙ СПОСОБНОСТЬЮ
УТИЛИЗИРОВАТЬ ПИЩЕВЫЕ ПОЛИСАХАРИДЫ
<130> YK0113
<150> JP 2019-065395
<151> 2019-03-29
<160> 8
<170> PatentIn version 3.5
<210> 1
<211> 2592
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 1
gtgaaggagg tgcgagcgaa cgggttcggg ggcagagtag ctgccgaacc gaacggcccg 60
atacgcgggc tacgcaagga atacaccatt atcaatgagg aaagtgatgg atacacattg 120
ttcaatacaa ccgatgaacg tggggcgtcc aagcgccgtg gcatcgtcgc ggcgcttgcg 180
gccgtagcca tgctggtgcc gctggcattc gctccgtcgg ctaccgccgc ggaaggcgac 240
tactccggtg gcatcaaggg cgagtacaac gctctgggca tcgccgccgg tgtggcgacc 300
gaagcctata gcggtctcaa tagccaggaa cggcaggata cggtgcttgc caacttcaac 360
cagatcactc cggaaaactc gatgaagccg gaaggctggt acgacggcaa tcatgagttc 420
gccatgtccg ataacacccg gagcctgttg aagttcgcca gcgacaacaa catccggatc 480
tacggccacg ttctggtgtg gcactcccag acgcccgact ggttcttcca agcggacgaa 540
tggtgtgagg acgccaataa caccgccggt gtcaccagct gcccgctggc cgacaaggcc 600
acgatgcagg agcgtcagcg tcagcacatc gagaacgtcg ccaaggcgat ctccgatgag 660
ttcggtccgt tcggcagcga cacgaatccc gtcgtggcgt tcgatgtcgt caacgaaacc 720
gtgaacgaca ccgacaatcc ggacaccaac ggcatgcgca attcgctgtg gtaccagacc 780
tacgacggtg aggactacat ctatgacgcg ttcgagaatg cgaacacgta cttcaacgag 840
gtgtacgccg atccttcgga cgatcatccg gtgacgttgt tcatcaacga ttacaacacc 900
gagcagtcgg gtaagcgcac ccggtatgct gccctgctgg accgtatggt tgccgaaggc 960
gtgccgttcg acggcatcgg ccaccagttc catgtgacgt tgaccacggc gacctcgaac 1020
ctggaggccg cgctgaccga catggaacgc ttcaacaaga agcaggccat caccgaactg 1080
gacgtgacga ccggaacccc ggtcacggag gccaagctca tcgaacaggg ccagtactac 1140
tatgaagtaa atcagatcat ccaccgtcac gctgcgcagc tgttctccgt tacggtgtgg 1200
ggcttgatcg acggactgtc ctggcgtagt ggcgaaggcg ctccgctgct gttcgacgac 1260
aacctggaga agaagccggc gtacatcggc tacatcggtg atagcgccaa ccttcccgag 1320
ccgttgaaga gcatgaacgc attcaaggat gacgccgtgg gcatcgactc ggcgcttccc 1380
ggtaccgtgg ccgagtccgg cgcgtcctct ccgtgggaac gtctttcgct ggtcgagatg 1440
accccgtctg cgaatggcgc cgtttccggc tcgttcaatg tctattggaa ggacggctct 1500
ctggtcgtct acgcggatgt cgccgatgtc agcgcggcgg atgacgacac cgtcaccgtg 1560
cgtgtgggtg acgccgagta tacgatcggc cgcaacggtg tgaccggcgg cgagggtgtg 1620
caggccaacg tcgtttcgtc tgatgccgga tacgaagtcg tggccgatat cccatacacc 1680
ggtgcagaga aggacatcgt cgagatgaac gtcatcgcga cggattccgc caccacggag 1740
accagcgcgt ggagcacgaa cgacaccggc gccgtcacgc tggccgagcc gctgagctac 1800
acggaagccg tgaaggttcc cgccgacgcc caagctccgg tcgttgacgc cgacccgtcg 1860
gatcccgtct gggcggaagc caacgaggtt cccgtggata aggtgaccgc cgccacgcct 1920
tcccccgagg cgaccgctac cgccaagacc ctgtggtcgg acggcaagct gtatgtcctc 1980
atggaagtga ccgacgcgga catcgacctg accaactcga atccgtggga gaaggactcc 2040
gtggaggtat acatcgaccg cggcaacacc aagagcggcc agtacaccaa cgacatccag 2100
cagattcgcg tgtccgccga tggcgcggag ctgagcttcg gctccggcgc gtcggaggat 2160
gtccagaagt ccatggtcca gaccgccggc acgatcgtcg atggcggcta tgtcgtcgag 2220
atggccatcg atctgggaac ggctgaggcc ggcaccttcg aaggtgtcga cttccagatc 2280
aacgacgcga agaacggtgc ccgaatcggc atccgcaact gggccgatcc gaccggcgcc 2340
ggctatcaga cggcgtccca ttggggcgtg ctgcgtctgc tggccgattc ctccgaaacc 2400
gagacccccg gcggcgagga gacccctggg gggggtaccg acaagcctgg cgacgagaag 2460
ccgcagcctt ccgacgatgc tgacaacgac gacaagatgc cgcagaccgg ttccgcggtc 2520
atcggagtcg ccgtggtggc gttgctgctg gttgccgccg gatgcgggct ggtcatcgct 2580
cggcgtcgat ga 2592
<210> 2
<211> 2100
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 2
gtggcagacc ttgatgcgga ttcgctgaac gaatcagctg aaaacacgca ggaagatcag 60
atccagaacg atcagctcga agatgcgcag ctcgatgaca ccttggctcc ggagcattac 120
gacgcttccg atctgagggt tctggaaggc cttgaggcgg tgcgaatccg cccgggcatg 180
tacatcggct ccaccggccc ccgaggcttg caccacctgg tgtacgagat cgtcgacaac 240
tccgtcgatg aggcgctggc aggctacgct tcgcacatcg aagtgacgat tctgccggac 300
ggcggcgtgc gcgtggtgga tgacggccga ggcattccgg tcgacgaagt gccgggcgaa 360
ggcgtttccg gcgtggaaac cgtgatgacc aagctgcatg ccggcggcaa gttcggtggc 420
ggcggctatg cggtttccgg cggtctgcac ggcgtgggca tctccgtggt gaacgcgctg 480
tcgacccgcg tcgacatcga agtgcgccga cagggcctcc actggaccca gacctacatc 540
gaccagaagc cgaccgcacg tcttgccaag ggcgagccga tgggcgaaga cgagtccacc 600
ggcacttccg tgaccttctg ggcagacggc gcgatcttcg aaaccaccac ctacgatttc 660
gagaccctgc gcaaccgctt ccagcagatg gcgttcctca acaaggggct gaaactgtcc 720
ctgaccgatt tgcgcgaacc ggatcaggcc ggagacgaag tcgcgggcga aagcgacgac 780
aatgccgaac cgaagcatca gacggtcacc taccaataca acgacggcat caaggattat 840
gtcgattact tggtgaagtc ccgcaaggca acccccgtcg aaccggacgt gatcgatttc 900
gaagcggaag acctgaaaat cggcatttcc gcggaaatcg ccatgcaatg gaccaccgca 960
tactccgaag cggtgcacac cttcgccaac accatctcca ccaccgaagg cggtacccac 1020
gaagaaggct tccgcgccgc gctgacctcg ctcgtcaacc gttacgcacg tgaaaagaac 1080
atcctcaaag acaaggatga gaacctctcc ggcgacgacg tgcgcgaagg cctgaccgca 1140
gtggtgtcgg tgaaactcac caccccgcag ttcgaaggcc agaccaagac caagctcggc 1200
aattccgaag cgaaaacctt cgtacagcgt gtgatgaccg acaagctcgg cgactggttc 1260
gattcgcatc ctagcgaggc caagaacatc atccagaagg ccattgaagc ctcccgtgcg 1320
cgtcttgcag ccaagaaggc acgtgagaat acccgccgta agtcaatctt cgaatctgcg 1380
ggcatgccag acaaactgaa ggattgccag tccaacaatc ctgaagaatg cgaactgttc 1440
atcgtggagg gcgactccgc aggcggctcc gcaattcagg gacgcaaccc gatcacgcag 1500
gccatcctgc cgttgcgagg caaaatcctt aacaccgagc gtgcaagcct cgaccgcatg 1560
atgaagtccg aaaccatcga atcgctgatc accgcggtcg gcggcggcta tggcgaggat 1620
ttcgacctga acaaagtccg ctaccacaag gtcatcatca tggccgatgc cgatgtggac 1680
ggcgcgcata tcgcaaccct gaatctgacg ctgttcttcc gctacatgcg cccgatgatc 1740
accgctggtt acgtgtatgt ggccatgccg ccgctgtacc gactcaagtg gaccaaaggc 1800
gcgcacgact tcgtgtacac cgacgccgaa cgcgaccgcg tgctcgccga aggcaagtcc 1860
gccggccgtc agctgccgaa gggcgaaggc atccagcgtt acaagggtct gggtgaaatg 1920
agctaccagg aactgtggga aaccaccatg gatcctgacc accgcattct gaagcaggtg 1980
cagatcgaag acgcggccgc agccgacgaa accttctcca tgctcatggg agacgaagtc 2040
gaacctcgcc gtctgttcat ccagcgcaac gcccgcaacg tcagctggat cgacgcgtag 2100
<210> 3
<211> 831
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 3
atggctatcc gcgtttataa gccgacgact gcaggccgcc gtaacgcgtc cgtctcggac 60
ttctccgagc ttacgcgttc tacgcctgag aagtcgctgg ttcgcaaact cagcaagact 120
ggcggtcgta actcttacgg ccgtatgacc tcccgtcatc gtggcggcgg tcacaagcgc 180
cagtaccgtc tcatcgactt caagcgttgg gacaaggacg gcgtgccggc aacggtcgct 240
cacatcgagt acgacccgaa ccgttccgcc cgcatcgcac tgctgcacta cgcagacggt 300
gagaagcgct acatcatcgc tccggaaggt atcaagcagg gtgacgtcat cgagaccggc 360
gcccaggctg atatcaagcc gggcaacaac ctgccgctgc gcaacatccc gactggtacc 420
gtggtgcacg caatcgagct ccgcccgctg ggtggcgcca agatcgcccg ctccgctggt 480
gcagccgtgc agctcgtcgc taaggatggc gcttacgccc agctgcgtat gccgtccgga 540
gaaatccgca acgtggatgc tcgctgccgc gcaaccgtcg gtgaggtcgg caactccgat 600
cacgccaaca tccagctcgg taaggcaggt cgtgcacgtt ggatgggcaa gcgcccgatc 660
acccgtggtg aatccatgaa ccctgtcgat cacccgcacg gcggtcgtac ccgcggtggc 720
aagccgccgg tttctccgtg gggcaagggc gaggttcgta cccgccgtcc gaagaaggct 780
tcgaacaaga tgattgttcg tcgtcgcccg aatggtaaga accgtaagta a 831
<210> 4
<211> 1515
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 4
ttgtcagctg aactcgaaga catccacgag gaatgcggta tttttggcgt ttggggtcac 60
ccggacgccg cccgactcac ctacttcggc ctgcacgcgc tgcagcatcg tggtcaggaa 120
ggcgccggca ttgtctcgaa cgataacggt catctcatcg gtcatcgcgg aacgggtctg 180
ttgacccaag tgttcggcga cgaacgcgaa atcgaacgcc tgaagggaga caaggcgatc 240
ggccatgtgc gttacgccac tgccggttca ggcggcaccg acaacatcca gccattcatc 300
ttccgcttcc atgacggcga tatggccttg tgtcacaatg gcaatctgac gaactgcccg 360
actctgaggc gtcagttgga agacgaaggc gcaatcttcc actccaattc cgacaccgaa 420
gtgttgatgc acctgttgcg ccgctccaca cagcgtacgt tcatggacaa gctcaaggaa 480
gcgctcaaca ccgtgcatgg cggtttcgcc tacctgatca tgaccgaaga cgccatgatc 540
ggcgcgcttg atccgaacgg tttccgcccg ctttccctcg gcaagatgaa aaacggcgct 600
tatgtgctcg cttccgaaac ctgcgcgctt gacgttgtcg gcgccgaatt ggtgcgcaat 660
attcgcccgg gcgaaatcgt ggttatcgac gatcacggct acaagatcgt gcagtacacc 720
aatcagaccc agttggccat ctgctcgatg gaattcatct acttcgcacg ccccgattcc 780
gacatttacg gcgtaaacgt gcattccgcc cgcaagcgta tgggtgcgcg tctggcccaa 840
gaatcgccgg tcgatgccga tatggtcatt ggcgtgccga attcctcgct gtccgcggcc 900
tccggctatg cggaagaagc tggtctgcca aacgagatgg gcctgatcaa gaaccagtat 960
gtggctcgaa ccttcatcca gccgacgcag gaactgcgcg agcagggcgt gcgcatgaag 1020
ctgtccgccg tgcgcggcgt ggtcaaaggc aagcgcgtga tcgttatcga cgattccatc 1080
gtgcgcggta ccacgtccaa gcgcatcgtg cagctgctga aggaagctgg cgccgccgaa 1140
gtgcacatgc gtatcagctc gccgccgctg aagtacccgt gcttctacgg catcgacatc 1200
tccaccacca aggagctcat cgccgcaaag aagtccgttg aagaaattcg cgatttcatt 1260
ggcgccgatt ccttggcgtt cctgtcactc gacggattgg tcgaatctat cggcctgggt 1320
gcggacgccc cgtacggcgg tctgtgcgtg gcctacttca atggcgacta cccgaccgca 1380
ctcgacgatt atgaaagtga ttttttgaag tccctcaccc cagaagatcg cgtgcgtctg 1440
cctgaattcg ccttgtataa gagcaagtac gagggcaacg aatacaccac tacttcatcc 1500
caagaagaac actga 1515
<210> 5
<211> 3561
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 5
ttggctgagg ctacgacgaa caccaccacc atcatcgcac gcgccgacca gcacgatatt 60
gatctgcaca aggcgtcgga ccgtgtgaac ttcggctcca tccgcgagcc cattgatgta 120
ccctacctgt tgggtgtaca gactgacagc ttcgactggc tcatcggcaa cgagcgctgg 180
cagaagcgtg tcgaggagga tctggcgaac ggcaccaaca ccgtgcccca cacctccggt 240
cttgatgagg tcttccagga gatctcaccg atcgagaact tcgctcagac catgagcctg 300
accttctccg atccgtattt cgaagaaccg cgccacaccg tgcaggagtg caaggagaag 360
gattacacct actccgcgcc gctgtatgtg aacgctgaat tcgaaaacgg cgacaccggc 420
gaaatcaagt cccagaccgt gttcatgggc gatttcccgc tgcagacccc gcacggcacc 480
ttcatcatcg gtggtaccga gcgagtgatc gtgtctcagc tggtgcgttc tccgggcgtg 540
tacttcgacc gcagccagga tcgcacttcc gacaaggaag tcttcggcgc gaagatcatc 600
ccgagccgtg gcgcatggct tgagttcgag atcgacaagc gtgacgtgct tggcgtgcgc 660
gtggaccgca agcgcaagca gtccgccatc gtgttcctca tggccatcgg catgaccaag 720
gatgagatcg ccgatgcgtt cagggattac ccgctggtta tggacgcgct tgccaaggag 780
accgtgcaga cccaggacga ggctctgacc gatctgtacc gcaagatccg cccggccgac 840
accccgacgc cggaagctgg ccgcaacctg ctggattcct tctacttcaa caccaagcgt 900
tacgatctgg cccgcgttgg ccgttacaag atcaaccgca agcttggtct ggaaaaggat 960
gtcaacgatc gcagcctgtc tcgcgaagac atcatctcca ccatcaagta cctggtcacc 1020
ctgcatgccg gcgacaccaa gttcccgggc aagcgtgacg gccaggatgt ggatctgcgc 1080
gtggacgtcg acgatatcga tcatttcggc aaccgtcgta tccgccaggt cggcgaactg 1140
atccagaacc agctgcgtac cggtctgagc cgtatggagc gtgtggtccg cgaacgtatg 1200
accacccagg atccggaggc catcaccccg cagtccctga tcaacattcg tccggtgaac 1260
gccaccatca aggagttctt cggaacttcc cagctgtcgc agttcatgga tcagaacaac 1320
ccgttggcag gcgtcaccaa caagcgtcgt ctgtccgctc tgggccctgg cggtctgtct 1380
cgtgaccgcg catccatgga agtgcgagac gtgcacccgt cccacttcgg ccgtatgtgc 1440
ccgatcgaat ctcctgaagg tccgaacatc ggtcttatcg gttctctggc aaccttcggc 1500
cgcatcaatc cgttcggctt catcgagacc ccgtaccgta aggtcatcaa cggccatgtg 1560
accgacgagg tcgaatacat gaccgcagac cgcgatgccg agcacgtgat cgcacaggcc 1620
aaccaggaac tcgacgagaa cggcaacttc gtcaagaagc aggctcttgc ccgagtcggc 1680
gaagaagaag cagtcgatgt gcccgtcagc tccgttgact acatggacgt ttccccgcgt 1740
cagatggttt ccgtcggcgc ctccctgatt ccgttcctgg agcacgatga gggccaccga 1800
gcgctgatgg gtaccaacat gcagcgtcag gcagtgccgc tgatcgaatc cgagcgcccg 1860
ctggtgggca ccggtgccga atggcgtgcg gccgtcgatt ccggcgatgt cattctggct 1920
gagaagccgg gtgtggtgac gtatgtttcc gccgacatca tccgtgtgat gaacgacgac 1980
ggcaccacca gctcctacaa gctggccaag ttcctgcgtt ccaaccagac cacctgctac 2040
aaccaggttc cgctgatcca cgacggcgaa cgtgtggaag ccggtaccgt gcttgccgat 2100
ggtccggcca cccagaaggg cgaaatggca ctgggcaaga acctgctcat cgccttcatg 2160
ccgtggaacg gctacaacta cgaggatgct gtgatcatct cccagcgtct cgtgcaggac 2220
gacaccctga gctccatcca catcgaggaa tatgagatcg acgcccgcga aaccaagctg 2280
ggtgccgaag agatcacccg cgatctgccg aacgtcggcg aggacgcggt ggccaacctc 2340
gacgagcgtg gcatcattcg catcggtgcc gaggtcgaag ccggcgacat tctggtgggc 2400
aaggtcaccc cgaagggcga gaccgagctg actccggaag agcgtctgct ccgcgccatc 2460
ttcggcgaga agagccgcga ggtgcgtgac acctcgctgc gcgtgcctca cggcgaaacc 2520
ggtacggtga tcgccgtcaa ggagatcact cgcgaggatg ccgaggaaga cggcgacgag 2580
ctgccgaacg gcgtgaacca gatgatccgc gtctacatcg cgcagcatcg taagatcacc 2640
cagggagaca agctgtccgg ccgccacggc aacaagggtg ttatctcccg cattctgccg 2700
gaagaggata tgccgttcct tgccgacggt actccggtcg acatcatgct gaacccgctg 2760
ggcgtgcctt ctcgaatgaa ccttggccag gtgctggaac tgcacttggg ctggatcgcg 2820
cacgccggct gggacatctc ccttgatccg gatgccgaag ccgcttggaa gaagtacatt 2880
ccgcagggcg ccgaaaaggg cgagccgggc actccggtgg caaccccggt gttcgacggc 2940
gttcgtccgg aaaccatcaa gggcctgctg tcctgcaccc ttccggatcg cgacggcaac 3000
aagctggtcg gcgacgacgg caaggctgtg ctgttcgacg gccgtaccgg cgaaccgtat 3060
ccgaagccga tctccgttgg ctacatgtac atgctgaagc tgcaccacct agtcgacgac 3120
aagatccacg cgcgttccac tggcccgtac tccatgatca cccagcagcc gttgggcggt 3180
aaggctcagt tcggtggcca gcgtttcggc gagatggaag tgtgggccct cgaggcctat 3240
ggcgccgcct acacgctgca tgaaatgatg accaccaagt ccgatgacgt cgacggccgc 3300
gtgcgtgtct acggcgccat cgtgaagggc gacaacctgc cgccggcagg cattccggaa 3360
tccttcaagg tgctgcttaa ggaaatgcag tccctgtcgc tgaacgtcga agtgctcaac 3420
gccgaaggcg tggccatcga catgaaggac gaggacgacg atccgtctac ttcctccgac 3480
gatttgggct tcaacattgg cgcacgtcct gacgcggccg ccaaggaaga ccaggttgtg 3540
gaagaacctg aattccagtg a 3561
<210> 6
<211> 2586
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 6
atgttcgaac ggtttaccga ccgtgcgcgg cgcgtgatcg tgctggcgca ggaagaggct 60
cgtaccctcc agcacaacta catcggtacc gagcatctgc tgcttggcct gattcgtgag 120
ggtgacggcg tcgcagccaa ggcgctggcc tcaaagggtg tgaccttgga tgacacccgc 180
aaacaggtcg aagagatgat cggtaagggg aatgccacgc cgaacggcca cattcccttc 240
accccgcatg ccaggcaggt gttggagctg tcgctgcgtg aggcgttgca gctggggcac 300
agctatatcg gcaccgagca tatcctgctc ggtctgattc atgaaggcga aggcgtcggc 360
acccaggtgc tgatcaagat ggacgtcaat cttggcgagc tgcgcagcgc caccatcgac 420
ctgatccgtg gcaattccgg tgatggcaag ggcgatggca agggcgattt ggccaatgcc 480
ggtggcgtgc aggatcgtcg caaccagacc ggttccgcga ttcttgacca gttcggccgc 540
aatctgaccg ctgaagctgc cgccggcaag cttgatccgg tgattgggcg ttcgagtgag 600
atcgagcgtg tgatggtggt gctgagccgc cgtaccaaga acaatccggt gctgatcggc 660
gagccgggtg tcggcaagac ggccgtggtc gaaggcttgg cgcagaagat caatgccggc 720
gacgttccgg agacgctgaa gggcaagcag gtctattcgt tggatctggg atccatggtg 780
gctggttcgc ggtatcgtgg cgatttcgag gagcgtctga agaaggtgct caaggagatc 840
aagacccgcg gcgacatcgt gctgttcatc gatgagatcc ataccattgt gggcgctggt 900
tccgcggatg gcgctcttgg cgcttccgac atgttgaagc caatgctcgc gcgaggtgag 960
ctgcagacca ttggcgccac caccaccgac gagtaccgta agtacatcga gaaggatgcc 1020
gctttggaac gtcgtttcca gcccatccag gtgcatgagc cgagcattgc cgaaaccatc 1080
gagatcctta agggcttgcg ttcgcgttac gagaaccatc accatgtgac gatcaccgat 1140
ggtgcgttgc aggccgcggc cgatctgtcg agccgttaca ttcaggaccg tcatctgcca 1200
gacaaggcca tcgatctgat cgatgaggcc ggtgcgcgtc tgcgcatccg tcgtctgacc 1260
gctccgccgg agcttaagga acttgacacg aagatcgcca agcttgccga agagaaggac 1320
caggccatca agggccagga cttcgaaaag gccgctgagc tgcgtgataa gcaggagaag 1380
ctggaagccg aacgcaagca gaaggaatct tcgtggcgtg agggcgaatc cgatgtgaag 1440
atggttgtgg atgaggatgt catcgccgaa gtgatttccc agaccaccgg cattccggtg 1500
ttcaagctca cccaggccga gtccaagaag ctcatgacca tggaaagcga gctgcacaag 1560
cgcatcatcg gccaggacga ggccgtgtcc gcgttgagcc gctccatccg tcgtgctcgt 1620
gttggtttga aggatccgaa gcgtccgtcc ggttcgttta tcttcgctgg ccccaccggc 1680
gtcggtaaga ccgagctggc caagacgctc gccgaattcc tgtttgacga cgaggacgca 1740
ctgattcgtg tcgacatgtc tgaattctcc gagaaatatg cggcttcgcg cctatttggt 1800
gctcctccgg gatacgtcgg ttacgaagag ggcggcgaac tcaccgagaa ggttcgtcgc 1860
aagccgttct ccgtggtgct gttcgatgag atcgaaaagg cccatccgga tatcttcaac 1920
acgctgctgc aggtgcttga tgacggtcat ctgactgatg gccagggccg caaggtggac 1980
ttcaagaaca ccatcatcat tctgaccacc aatcttggta cgcgagacat cgccaaggcc 2040
gccaacaccg gcttcaactt gggtgccaac accgagtcga gctaccagcg catgaaggat 2100
caggtttccg cggagctgaa gcagcagttc cgtccggaat tcctgaaccg tctggacgac 2160
atcatcgtgt tcaagcagct caccgagccg caagtgcgtc agatcgtcga tcttgacgtc 2220
aagcagctca atgatcgctt gttcgatcgc cacatgtccc tcgagcttac cgacgctgcc 2280
aaggatctgc tcgcgcagaa gggcttcgac ccgctgctgg gcgcgcgtcc gttgcgtcgc 2340
gtgattcagc gcgacgtcga agacgccatt tcggagaaga tcctgatggg cgaactcgaa 2400
gatggccagc gcgtgaaggt cgatgcggaa ggcgaaggca tcctgggcga gttcaccttt 2460
accggtgagg cgttcgaaga gccaaatacg gagcctgctg aaggtgaagt cgcagctgaa 2520
accgaggcac ctgccgaatc gacggaatcg acagaactta cggaatccgc agaatctgtt 2580
gaataa 2586
<210> 7
<211> 2130
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 7
atggcactag acgtgctcaa tgatctcaac cagatccgta acatcggcat catggctcac 60
atcgatgccg gtaagaccac taccaccgaa cgtatcctgt actacaccgg caagaactac 120
aagatcggcg agacccatga cggtgcctcg acgatggact tcatggctca ggagcaggaa 180
cgcggcatca ccatccagtc cgctgcaacc acttgcttct ggaaccgtca gacccatgac 240
gagaagcaga agttccagat caacatcatc gataccccag gccacgtgga cttcacggcc 300
gaggtggagc gctccctgcg cgtgctcgat ggtgccgttg ccgtgttcga cggcaaggaa 360
ggtgtggagc cgcagtccga gaccgtgtgg cgtcaggctg acaagtacgg cgttccgcgt 420
atctgcttca tcaacaagat ggataagctc ggcgctgact tctactactc cgtcgacacc 480
atcaaaacca agctgggcgc gaccccgctt gtcgtgcagc tgccgatcgg cgctgagaac 540
gacttcgctg gcgttgtcga tctgattcgt atgaaggctt acgtctggaa cgacgtttcc 600
ggcgacatgg gcgctcacta cgacactacc gacatccccg ccgacctgca ggacaaggcc 660
gagcagtatc gtgcagagct gctcgaccag gtcgcagaat ccgacgaaga gctgcttgag 720
aagtatctcg agtccggcga actgactgag gacgagatcc gtagcggcat ccgtaagctc 780
accattaacc gtgaagccta cccggtgctc tgcggctccg ccttcaagga caagggtgtt 840
cagccgatgc tggacgccgt cgtcgactac ctgccgagcc cggaggacgt tccgtccatc 900
gtcggtttcg atcctaagga cgaatccatc gagatcgatc gcaagccgac caccgatgat 960
ccgttctctg ccctggtctt caagatctct acccacccgt tctacggcaa gctcgtgttc 1020
gtgcgcgtct actccggcgc cgtcaccccg ggcgacaccg tgcttgactc caccaagggc 1080
aagaaggaac gcgtcggcaa gatcttccag atgcacgccg acaaggagaa cccggtcgat 1140
gccgccgaag ccggcaacat ctacaccttc gtgggcctga agaacatcac caccggtgac 1200
accctgtgcg acgaaaaggc gcctatctcc ctcgaatcca tgaccttccc ggatccggtg 1260
atcgaggtgg ccgtggagcc gaagaccaag gccgatcagg agaagatgag catcgctctg 1320
gcgaagctgt ccgacgaaga tccgaccttc caggtgaaga ccgacgaaga gtccggccag 1380
accctgatct ccggcatggg cgagctgcag ctcgacatca tcgtcgaccg tatgcgtcgt 1440
gaattcaagg tggagtgcaa cgtgggtaac ccgcaggttg cataccgtga gacgatccgc 1500
aaggccgtca tgaaccagga atacacgcac aagaagcaga ccggtggttc cggccagttc 1560
gcaaaggtct tgatgaactt cgagccgctc aacaccgaag agggcgagac ctacgagttc 1620
gtcaacgagg tcaccggtgg ccacatcacc aaggaattca ttccttccat cgatgctggt 1680
gtgcaggaag ccatggaatc cggcgtgctc gccggcttcc cggtggttgg cgtcaaggct 1740
accgtcactg acggccaggt ccacgacgtc gattcctccg aaatggcctt caagatcgca 1800
ggttccatgt gcttcaagga agctgctccg aaggccaagc cggtcatcct cgagccgatc 1860
atggccgtgg aagtgcgtac tccggaagag tacatgggcg acgtgatggg cgatatcaac 1920
gcccgtcgtg gttccatcca gtccatgacc gactccaccg gtgtcaaggt catcgatgcc 1980
aaggttccgc tgtccgaaat gttcggctac atcggcgacc ttcgctccaa gacccagggc 2040
cgcgcaatgt tcaccatgca gatggactcc tacgctgagg ttccgaagaa cgtctccgag 2100
gagatcatca aggcccagcg cggcgagtga 2130
<210> 8
<211> 3312
<212> ДНК
<213> Bifidobacterium pseudocatenulatum
<400> 8
gtgagcgaaa acaacgtgta tccgaaggca gctgcgggtg agcagagcgc caatgtagcg 60
ccgaacccca gtttcccgaa gctggaagaa tccgtccttg attattggga gaaggacgac 120
actttccaga agtccattga acgccgtcct tccggcgatc acagccagaa cgaattcgtc 180
ttcttcgacg gaccgccgtt cgccaacggc ctgccgcact acggccacct gctgaccggc 240
tacgccaagg atgtgattcc gcgctaccag accatgaagg gccacaaggt caatcgtgtg 300
ttcggctggg atacgcacgg tctgccggcc gagcttgaag cccagaagga actgggcatc 360
gactcggtcg atcaggtcaa ggaaatgggc atcgacaagt tcaacgacgc ttgccgcgcc 420
tccgtgctca agtacaccaa cgagtggaag gactacgtgc accgtcaggc acgttgggtc 480
gatttcgagc atggctacaa gacgctgaac attccataca tggaatccgt gatgtgggcg 540
ttcaagcagc tgtacgacaa gggcctcgca tatcagggct accgcgtgct gccgtactgt 600
ccgaaggatc agacgccgct ttccgcgcac gagctgcgca tggacgccga cgtgtatcag 660
gatcgtcagg acaccaccgt gtccgtggcc gtgaagctgc gcgacgagga agacgcctat 720
gcggtgttct ggaccaccac gccgtggacc gttcccacca acttcgccat cgtggtcggc 780
gcagacatcg actatgtgga agtgcgtccg accgaaggca agttcgcggg caagaagttc 840
tacctgggca agccactgct cggctcctac gccaaggaac ttggcgacaa ctatgagatc 900
gtgcgcgaac tcaagggtgc cgagatggaa ggctggcgct actacccggt gttcccgtac 960
ttcgcaggcg acgagaacgc cgtcgaaggc aaggttccgg gtccggaagg ctatcagatc 1020
ttcaccgccg actatgtcga taccgttgag ggtaccggtc tcgtgcacca ggctccgtac 1080
ggcgaggacg atatgaacac gctcaacgcc aagggcatca agagcgttga cgtgctcgac 1140
gcgggctgca agttcaccgc gctgtgcccg gactacgagg gcatgtacgt gttcgatgcg 1200
aacaagccga ttctgcgtaa cctgcgtgcc ggtgacggtc ctctggagcg tattccggaa 1260
gatcagcgcg cgatcctttt ccaggagaag agctacgtgc actcctaccc gcactgctgg 1320
cgttgcgcca cgccgctcat ctacaagccg gtgagctcgt ggttcgtgtc cgtcaccaag 1380
atcaaggatc gtctgctcga actgaaccag gaaatcaact ggattccggg caatgtgaag 1440
gacggccagt tcggcaagtg gctggccaac gcccgtgact ggtcgatctc ccgcaaccgt 1500
ttctggggtt cgccgattcc ggtgtgggtt tccgatgatc cgaagtatcc acgcgttgac 1560
gtgtacggtt cgttggacga gctgaaggct gattttggcg attatccgcg cgaccatgag 1620
ggcaatgtca acatgcaccg cccgtacatc gacgagctga cccgcgtcaa cccggacgat 1680
ccgaccggca agagccacat gcaccgcatc accgacgtga tggactgctg gttcgaatcc 1740
ggttccatga gcttcgccca gtaccattac ccgttcgaaa acaaggaaac gttcgaacag 1800
catttcccgt gcgattacat cgtggaatac atcggccaga cccgtggctg gttctacgtg 1860
cagcacatca tggcgaccgc actgttcgac aagccggcgt tcaagaacgt gatctgccat 1920
ggcatcgtgc ttggctccga cggccagaag atgtcgaagc atctgcgcaa ctacccggat 1980
gtgaatggag tgttcaacga tttcggctcc gacgccatgc gctggttcct catgagctcg 2040
ccgatcctgc gcggcggcaa cctgatcgtg accgccgatg gcattcgcga caccgtgcgt 2100
caggtcatgc tgccagtgtg gagttcgtac tacttcttca ccctgtatgc gaacgccgcc 2160
aataatggcg ccggcttcga tgctcgtaca ttgcgtgccg atgaggtcgc cgcactgccg 2220
gaaatggatc gttacctgct ggcgcgtacc cgccgactga tcgagaagac gcaaagctcg 2280
ctggacaact tcctgatctc tgacgcatgc gaagccgtgt ccgatttcat tgacatgctc 2340
accaactggt atatccgcaa caatcgtgac cgtttctgga atgaggatgc gaacgcgttc 2400
aacacgctgt acaccgtgct tgaagcattc atgcgcgtga tcgcaccgct tgctccgatg 2460
gaagcggaag cagtgtggcg tggtctgacc ggtggcgaat ccgtgcactt ggccgactgg 2520
ccgttccttg ccgacgagca gaccggagag gcaacagaac tgggccgcgt gcttgttgac 2580
gatccggctc tggttgatgc gatggagaag gtgcgcgagg tcgtgtccgg cactctgtct 2640
atgcgtaaga caaagcagat ccgtgtgcgc cagccgctgt ccaagctgac cgtggtggtg 2700
gagaacactg ccgccgtggc cgcatacgac gagattctga aatccgaact gaacgtgaag 2760
aacgttgaat tgtgcacgct tgaagatgcc gaagcccaag gcttgaagat catcaacgag 2820
ctgcgtgtga acgctcgcgt ggcaggcaag cgcctgcgca aggacgtgca gttcgccatc 2880
aaggcttcca agtccggcgc atggcatgtg aacgccgaag gcgctccggt atgtgagact 2940
ccgaacggtg agatcgtgct tgaggaaggc gaatatgagc tgatcaacag cgtggaggag 3000
aagaacgccg aagaggccgc caactctgta agtgctgcac tgccgaccgg tggcttcgtg 3060
atcctcgaca ccgagctgaa cgacgatctg atcgcagaag gctatgcccg cgacgtgatc 3120
cgcgccgtgc aggacgcccg caaggccgcc gatctgcaga tctccgaccg tatcgcactg 3180
aagctcgttg tgcccgccga agacgtggcc aaggtcgaac agttcaagga acttgtctcc 3240
tccgagacgc tcgccacttc cttcgaagtg actgccggcg atgagctgaa cgttgaggtt 3300
gccaaggcct ga 3312
<---
Изобретение относится к биотехнологии. Предложена генетически модифицированная бактерия Bifidobacterium, обладающая способностью ассимилировать ксиланы, где указанная бактерия имеет ген ксиланазы в геноме. Также предложены штаммы Bifidobacterium pseudocatenulatum, обладающие способностью ассимилировать ксиланы, а также предложены пищевой продукт и напиток, содержащие указанные бактерии. Изобретение обеспечивает эффективную утилизацию ксилана бактериями. 9 н. и 5 з.п. ф-лы, 2 ил., 2 табл., 4 пр.
1. Генетически модифицированная Bifidobacterium бактерия, обладающая способностью ассимилировать ксиланы, где указанная бактерия имеет ген ксиланазы в геноме, где ген ксиланазы представляет собой полинуклеотид, состоящий из нуклеотидной последовательности, показанной в SEQ ID NO: 1, или полинуклеотид, имеющий 90% идентичность или более с данной нуклеотидной последовательностью и кодирующий белок, обладающий ксиланазной активностью.
2. Бактерия, относящаяся к роду Bifidobacterium по п.1, где ксиланы представляют собой ксилан и арабиноксилан.
3. Бактерия, относящаяся к роду Bifidobacterium по п.1, которая обладает способностью ассимилировать крахмал.
4. Бактерия, относящаяся к роду Bifidobacterium по п.1, где бактерия, относящаяся к роду Bifidobacterium, классифицируется как Bifidobacterium pseudocatenulatum.
5. Bifidobacterium pseudocatenulatum YIT11027 NITE ВР-02928, обладающая способностью ассимилировать ксиланы, где указанная Bifidobacterium pseudocatenulatum депонирована в Депозитарии патентованных микроорганизмов Национального института технологии и оценки от 25 марта 2019.
6. Bifidobacterium pseudocatenulatum YIT11055 NITE ВР-02929, обладающая способностью ассимилировать ксиланы, где указанная Bifidobacterium pseudocatenulatum депонирована в Депозитарии патентованных микроорганизмов Национального института технологии и оценки от 25 марта 2019.
7. Bifidobacterium pseudocatenulatum YIT11057 NITE ВР-02930, обладающая способностью ассимилировать ксиланы, где указанная Bifidobacterium pseudocatenulatum депонирована в Депозитарии патентованных микроорганизмов Национального института технологии и оценки от 25 марта 2019.
8. Bifidobacterium pseudocatenulatum YIT11952 NITE ВР-02931, обладающая способностью ассимилировать ксиланы, где указанная Bifidobacterium pseudocatenulatum депонирована в Депозитарии патентованных микроорганизмов Национального института технологии и оценки от 25 марта 2019.
9. Bifidobacterium pseudocatenulatum YIT11954 NITE ВР-02932, обладающая способностью ассимилировать ксиланы, где указанная Bifidobacterium pseudocatenulatum депонирована в Депозитарии патентованных микроорганизмов Национального института технологии и оценки от 25 марта 2019.
10. Bifidobacterium pseudocatenulatum YIT12989 NITE BP-02933, обладающая способностью ассимилировать ксиланы, где указанная Bifidobacterium pseudocatenulatum депонирована в Депозитарии патентованных микроорганизмов Национального института технологии и оценки от 25 марта 2019.
11. Пищевой продукт, содержащий бактерию, относящуюся к роду Bifidobacterium по пп.1 или 5-10.
12. Пищевой продукт по п.11, который представляет собой кисломолочный продукт.
13. Напиток, содержащий бактерию, относящуюся к роду Bifidobacterium по пп.1 или 5-10.
14. Напиток по п.13, который представляет собой напиток, полученный путём ферментации.
| US 20120288585 A1, 15.11.2012 | |||
| MORITA H | |||
| et al | |||
| Способ предохранения железных ванн от разъедания цинком | 1928 |
|
SU15439A1 |
| Genome Announcements, 2015, 3(2), e00255-15 doi:10.1128/genomea.00255-15 | |||
| Паровой двигатель | 1928 |
|
SU12327A1 |
| Способ предохранения железных ванн от разъедания цинком | 1928 |
|
SU15439A1 |
Авторы
Даты
2024-10-30—Публикация
2020-03-27—Подача