Область техники
Настоящее изобретение относится к микроорганизму, продуцирующему L-аргинин, у которого ослаблена активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, и к способу получения L-аргинина.
Предшествующий уровень техники
L-аргинин используют в лекарственных препаратах, таких как стимулятор функции печени, стимулятор работы мозга и комплексный аминокислотный препарат, и в последнее время привлекает внимание к пищевым продуктам, таким как добавка к рыбному пирогу, добавка к напиткам для здоровья и заменитель соли для пациентов с гипертонией. Постоянно проводятся исследования по использованию микроорганизмов для получения промышленно применимого аргинина в высоких концентрациях, и были сделаны отчеты о способах использования мутантных штаммов, полученных из микроорганизмов рода Вrevibacterium или Corynebacterium, которые представляют собой штаммы, продуцирующие глутамат, и о способах применения штаммов, продуцирующих аминокислоты, с улучшенным ростом посредством слияния клеток, и подобные (US 8034602 В2).
Техническая задача
В одном аспекте настоящего изобретения предлагается рекомбинантный микроорганизм рода Corynebacterium, продуцирующий L-аргинин, способ получения L-аргинина с его использованием и его применение.
Техническое решение
В соответствии с одним аспектом настоящего изобретения предлагается рекомбинантный микроорганизм рода Corynebacterium, продуцирующий L-аргинин, у которого активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, ослаблена.
В соответствии с другим аспектом настоящего изобретения предлагается способ получения L-аргинина, включающий культивирование рекомбинантного микроорганизма рода Corynebacterium в среде, у которого активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, ослаблена.
Полезные эффекты
Микроорганизм рода Corynebacterium, у которого активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, ослаблена по настоящему изобретению, может продуцировать L-аргинин с высоким выходом и, таким образом, может выгодно использоваться для промышленного производства.
Подробное описание изобретения
Настоящее изобретение будет конкретно описано следующим образом. Каждое описание и воплощение, раскрытые в настоящем изобретении, также можно использовать для других описаний и воплощений. То есть все комбинации различных элементов, раскрытых в настоящем изобретении, попадают в объем настоящего изобретения. Кроме того, объем настоящего изобретения не ограничивается приведенным ниже конкретным описанием. Кроме того, по всему полному описанию предлагаются ссылки на многие статьи и патентные документы и цитаты из них. Описания цитируемых статей и патентных документов полностью включены в настоящий документ посредством ссылок и уровень области техники, к которой относится настоящее изобретение, и подробности настоящего изобретения объясняются более четко.
В соответствии с одним аспектом настоящего изобретения предлагается рекомбинантный микроорганизм рода Corynebacterium, в котором активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, ослаблена.
Белок по настоящему изобретению может иметь или включать аминокислотную последовательность SEQ ID NO: 1, или может состоять или по существу состоять из этой аминокислотной последовательности.
В настоящем изобретении белок, включающий аминокислотную последовательность SEQ ID NO: 1, может представлять собой эндогенный белок микроорганизма по настоящему изобретению, но без ограничения этим.
Аминокислотная последовательность SEQ ID NO: 1 может быть получена из известной базы данных GeneBank Национального института здравоохранения США (NIH GenBank). В настоящем изобретении аминокислотная последовательность SEQ ID NO: 1 может включать аминокислотную последовательность, имеющую по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,5%, 99,7%, или 99,9% гомологию или идентичность с последовательностью, указанной в SEQ ID NO: 1. Было бы очевидно, что любой белок, который имеет аминокислотную последовательность с делецией, модификацией, заменой, консервативной заменой или добавлением на своем участке, также можно включать в диапазон настоящего изобретения до тех пор, пока аминокислотная последовательность имеет же такую гомологию или идентичность и проявляет эффективность, соответствующую белку по настоящему изобретению.
Например, аминокислотная последовательность может включать аминокислотную последовательность, имеющую добавление или делецию, встречающуюся в природе мутацию, «молчащую» мутацию или консервативную замену на N-конце, С-конце и/или внутри аминокислотной последовательности, которая не изменяет функции белка по настоящему изобретению.
При использовании здесь термин «консервативная замена» относится к замене одной аминокислоты другой аминокислотой, которая имеет сходные структурные и/или химические свойства. Белок может иметь, например, одну или более консервативных замен, сохраняя, однако, при этом одну или более биологических активностей. Такая аминокислотная замена, как правило, может быть произведена на основе сходства полярности, заряда, растворимости, гидрофобности, гидрофильности и/или амфипатической природы остатков. Например, среди электрически заряженных аминокислот положительно заряженные (основные) аминокислоты включают аргинин, лизин и гистидин; и отрицательно заряженные (кислые) аминокислоты включают глутаминовую кислоту и аспарагиновую кислоту. Среди незаряженных аминокислот неполярные аминокислоты включают глицин, аланин, валин, лейцин, изолейцин, метионин, фенилаланин, триптофан и пролин; полярные или гидрофильные аминокислоты включают серии, треонин, цистеин, тирозин, аспарагин и глутамин. Среди аминокислот ароматические аминокислоты включают фенилаланин, триптофан и тирозин.
При использовании здесь термин «гомология» или «идентичность» относится к степени сходства между двумя данными аминокислотными последовательностями или последовательностями оснований нуклеиновых кислот и может выражаться в процентах. Термины «гомология» и «идентичность» часто можно использовать взаимозаменяемо.
Гомология последовательностей или идентичность консервативных полинуклеотидов или белков может быть определена посредством стандартных алгоритмов выравнивания и можно совместно использовать штрафы за пробелы по умолчанию, установленные используемой программой. По существу, все гомологичные или идентичные последовательности или их части как обычно могут гибридизоваться друг с другом в умеренных или очень жестких условиях. Было бы очевидно, что гибридизация также включает в себя гибридизацию с полинуклеотидом, содержащим обычные кодоны или кодоны с учетом вырождения кодонов в полинуклеотиде.
Гомология, сходство или идентичность между любыми двумя полинуклеотидными или белковыми последовательностями может быть определена с использованием известного компьютерного алгоритма, такого как программа "FASTA", с использованием параметров по умолчанию, как указано в литературе (Pearson et al., (1988) [Proc. Natl. Acad. Sci. USA 85]: 2444). Альтернативно, это можно определять, используя алгоритм Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443-453), который выполняют в программе Нидлмана из пакета Европейского открытого комплекса программ для молекулярной биологии (EMBOSS) (Rice etal., 2000, Trends Genet. 16: 276-277) (версия 5.0.0 или последующие версии) (включающего пакет программ GCG (Devereux, J., et al, Nucleic Acids Research 12: 387 (1984)), BLASTP, BLASTN, FASTA (Atschul, [S.] [F.,] [ET AL, J MOLEC BIOL 215]: 403 (1990); Guide to Huge Computers, Martin J. Bishop, [ED.,] Academic Press, San Diego,1994 и [CARILLO ETA/.] (1988) SIAM J Applied Math 48: 1073). Например, гомологию, сходство или идентичность можно определять, используя BLAST информационной базы данных Национального центра биотехнологической информации или ClustalW.
Гомологию, сходство или идентичность между полинуклеотидами или белками можно определять путем сравнения информации о последовательностях с использованием компьютерной программы GAP, например, Needleman et al, (1970), J. Mol Biol. 48:443, как известно из Smith and Waterman, Adv. Appl. Math (1981) 2:482. Вкратце, программа GAP определяет сходство как количество выровненных символов (т.е. нуклеотидов или аминокислот), которые похожи, разделенное на общее количество символов в более короткой из двух последовательностей. Параметры по умолчанию для программы GAP могут включать: (1) двоичную матрицу сравнения (содержащую значение 1 для идентичности и 0 для неидентичности) и взвешенную матрицу сравнения Gribskov et al. (1986) Nucl. Acids Res. 14:6745, которые описаны в Schwartz and Dayhoff, eds., "Atlas of Protein Sequence and Structure", National Biomedical Research Foundation, pp.353-358 (1979) (или матрицу замещения EDNAFULL (EMBOSS-версия NCBI NUC4.4); (2) штраф 3,0 за каждый пробел и дополнительный штраф 0,10 за каждый символ в каждом пробеле (или штраф за открытие пробела 10 и штраф за расширение пробела 0,5); и (3) отсутствие штрафа за конечные пробелы.
В настоящем изобретении полинуклеотид, кодирующий белок, включающий аминокислотную последовательность SEQ ID NO: 1, может быть назван геном NCgl1469.
При использовании здесь термин «полинуклеотид» относится к полимеру, состоящему из нуклеотидной цепочки, вытянутой вдоль посредством ковалентных связей нуклеотидных мономеров, который представляет собой цепь ДНК или РНК конкретной длины и, более конкретно, фрагмент полинуклеотида, кодирующий белок.
Полинуклеотид, кодирующий белок по настоящему изобретению, может включать последовательность оснований нуклеиновой кислоты, кодирующую аминокислотную последовательность, указанную в SEQ ID NO: 1. В качестве примера, полинуклеотид по настоящему изобретению может иметь или включать последовательность оснований нуклеиновой кислоты SEQ ID NO: 2. Альтернативно, полинуклеотид по настоящему изобретению может состоять или по существу состоять из последовательности оснований нуклеиновой кислоты с SEQ ID NO: 2.
В полинуклеотиде по настоящему изобретению можно сделать различные модификации в кодирующей области до тех пор, пока аминокислотная последовательность белка по настоящему изобретению не будет изменяться с учетом вырождения кодонов или кодонов, предпочтительных в организме, который предназначен для экспрессии белка по настоящему изобретению. Конкретно, полинуклеотид по настоящему изобретению может иметь или включать последовательность оснований нуклеиновой кислоты, имеющую по меньшей мере 70%, по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97% или по меньшей мере 98%, но менее чем на 100% гомологию или идентичность с последовательностью нуклеиновой кислоты SEQ ID NO: 2, или может состоять или по существу состоять из нуклеотидной последовательности, имеющей по меньшей мере 70%, по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90%, по крайней мере 95%, по крайней мере 96%, по крайней мере 97% или по крайней мере 98%, но менее 100% гомологию или идентичность последовательности оснований нуклеиновой кислоты SEQ ID NO: 2, но без ограничения этим.
Кроме того, полинуклеотид по настоящему изобретению может включать, без ограничения, последовательность, которая может гидридизоваться, в строгих условиях, с зондом, который может быть получен из известной последовательности гена, например, последовательности, комплементарной части или всей полинуклеотидной последовательности по настоящему изобретению. Термин «строгие условия» относится к условиям, которые обеспечивают возможность специфической гибридизации между полинуклеотидами. Конкретно эти условия подробно описаны в литературе (смотреть J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; and F. M. Ausubel et at, Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York, 9.50-9.51, 11.7-11.8). Примеры жестких условий могут включать условия, при которых полинуклеотиды имеющие более высокую гомологию или идентичность, например, полинуклеотиды, имеющие по меньшей мере 70%, по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% гомологии или идентичности, гибридизуются друг с другом, но полинуклеотиды, имеющие более низкую гомологию или идентичность, не гибридизуются друг с другом; или условия промывки типичной Саузерн-гибридизации, то есть условия промывки один раз, или два, или три раза, при концентрации солей и температуре, соответствующих 1×SSC, 0,1% SDS (додецилсульфат натрия) при 60°С, конкретно 0,1×SSC (раствор цитрата и хлорида натрия), 0,1% SDS при 60°С, или, более конкретно, 0,1×SSC, 0,1% SDS при 60°C. SSC, 0,1% SDS при 68°С.
Для гибридизации требуется, чтобы две нуклеиновые кислоты имели комплементарные последовательности, хотя в зависимости от строгости гибридизации возможны ошибочные спаривания между основаниями. Термин «комплементарный» используют для описания взаимосвязи между нуклеотидными основаниями, которые могут гибридизоваться друг с другом. Например, в отношении ДНК, аденин комплементарен тимину и цитозин комплементарен гуанину. Соответственно, полинуклеотид по настоящему изобретению может также включать выделенные фрагменты нуклеиновых кислот, комплементарные полной последовательности, а также сходные по существу последовательности оснований нуклеиновых кислот.
Конкретно, полинуклеотиды, имеющие гомологию или идентичность с полинуклеотидом по настоящему изобретению, можно обнаруживать посредством применения условий гибридизации, включающих стадию гибридизации при температуре Тm 55°С и использование описанных выше условий. Кроме того, значение Тm может быть равным 60°С, 63°С или 65°С, но без ограничения этим, и может соответствующим образом корректироваться специалистом в данной области техники согласно цели.
Подходящая степень жесткости для гибридизации полинуклеотидов зависит от длины полинуклеотид а и степени его комплементарности, и их переменные параметры хорошо известны в данной области техники (e.g., Sambrook et al., supra).
При использовании здесь термин «микроорганизм (или штамм)» охватывает все микроорганизмы дикого типа или естественным или искусственным путем генетически модифицированные микроорганизмы, относится к микроорганизму, у которого конкретный механизм ослабляют или усиливают за счет вставки экзогенного гена или усиления или инактивации активности эндогенного гена, и может представлять собой микроорганизм, включающий целевой белок или генетическую модификацию для продуцирования белка или продукта.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, в котором белок, включающий аминокислотную последовательность SEQ ID NO: 1 по настоящему изобретению, ослабляют, или полинуклеотид, кодирующий белок, удаляют; или микроорганизм, который был генетически модифицирован (например, рекомбинантный микроорганизм) посредством вектора чтобы ослабить белок, включающий аминокислотную последовательность SEQ ID NO: 1 по настоящему изобретению или удалить полинуклеотид, кодирующий белок, но не ограничиваясь этим.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, имеющий способность продуцировать L-аргинин.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, имеющий повышенную способность продуцировать L-аргинин по сравнению с родительским штаммом.
Микроорганизмом по настоящему изобретению может представлять собой микроорганизм, полученный посредством придания способности продуцировать L-аргинин родительскому штамму (например, родительскому штамму, от природы имеющему способность продуцировать L-аргинин или не имеющему способность продуцировать L-аргинин) посредством ослабления белка, включающего аминокислотную последовательность SEQ ID NO: 1 по настоящему изобретению или делеции полинуклеотида, кодирующего белок, но без ограничения этим.
Например, рекомбинантный микроорганизм по настоящему изобретению может представлять собой штамм или микроорганизм, в котором белок, включающий аминокислотную последовательность SEQ ID NO: 1 по настоящему изобретению, ослаблен, или полинуклеотид, кодирующий белок, удален, посредством трансформации с помощью вектора, для того, чтобы ослабить белок, включающий аминокислотную последовательность SEQ ID NO: 1 по настоящему изобретению или удалить полинуклеотид, кодирующий белок, где белок, включающий аминокислотную последовательность SEQ ID NO: 1, ослаблен, или полинуклеотид, кодирующий белок, удален в нативном микроорганизме дикого типа или микроорганизме, продуцирующем L-аргинин, способность микроорганизма продуцировать L-аргинин повышают по сравнению с нативным микроорганизмом дикого типа или микроорганизмом, в котором белок, включающий аминокислотную последовательность SEQ ID NO: 1 или полинуклеотид, кодирующий белок, не модифицируют, но и не ограничивается этим.
Например, немодифицированный микроорганизм или родительский штамм в качестве штамма сравнения для сравнения увеличения способности продуцировать L-аргинин может представлять собой штамм АТСС13869 или Corynebacterium glutamicum KССМ10741Р (van der Rest et al, Appl. MicrobiolBiotechnol 52:541-545, 1999) или штамм, в котором в Corynebacterium glutamicum дикого типа АТСС 13869 удален ген argR (например, Corynebacterium glutamicum CJ1R), но без ограничения этим.
В качестве примера, рекомбинантный штамм с повышенной продуцирующей способностью может иметь способность продуцировать L-аргинин, которую повышают, по меньшей мере, примерно на 1%, в частности по меньшей мере примерно на 1%, по меньшей мере примерно на 2,5%, по меньшей мере примерно на 5%, по меньшей мере примерно на 6%, по меньшей мере примерно на 7%, по меньшей мере примерно 8%, по меньшей мере примерно 9%, по меньшей мере примерно 10%, по меньшей мере примерно 10,5%, по меньшей мере примерно 11%, по меньшей мере примерно 11,5%, по меньшей мере примерно 12,5%, по меньшей мере примерно 13%, по меньшей мере примерно 13,5%, или по меньшей мере примерно 14% (верхняя граница конкретно не ограничена, и верхняя граница может представлять собой, например, не более 200%, не более примерно 150%, не более примерно 100%, не более примерно 50%, не более примерно 40%, не более примерно 30% или не более примерно 25%), по сравнению с таковой у родительского штамма до модификации или немодифицированного микроорганизма, но способность продуцировать L-аргинин не ограничивается этим до тех пор, пока она имеет нарастание на положительное значение (+) по сравнению с продуцирующей способностью родительского штамма до модификации или немодифицированного микроорганизма. В качестве другого примера, рекомбинантный штамм с увеличенной продуцирующей способностью может иметь способность продуцировать L-аргинин, которую увеличивают по меньшей мере примерно в 1,1 раза, по меньшей мере примерно в 1,12 раза, по меньшей мере примерно в 1,13 раза или по меньшей мере примерно в 1,14 раза (верхняя граница конкретно не ограничена, и верхняя граница может представлять собой, например, не более примерно в 10 раз, не более примерно в 5 раз, не более примерно в 3 раза или не более примерно в 2 раза) по сравнению с таковым у родительского штамма до модификации или немодифицированного микроорганизма, но способность продуцировать L-аргинин этим не ограничивается. При использовании здесь термин «примерно» относится к диапазону, охватывающему все из ±0,5, ±0,4, ±0,3, ±0,2, ±0,1 и тому подобные, и таким образом охватывает все значения в диапазоне, эквивалентном или сходном с указанными после этого термина, но не ограничивается ими.
При использовании здесь термин «немодифицированный микроорганизм» может относиться к штамму дикого типа или нативному штамму как таковому или к штамму до трансформации в результате генетической мутации, вызванной естественными или искусственными факторами, не исключая штаммы, включающие мутации, которые могут возникать в микроорганизмах естественным образом. Например, немодифицированный микроорганизм может обозначать микроорганизм до ослабления белка, включающего аминокислотную последовательность SEQ ID NO: 1 в данном документе, или делеции полинуклеотида, кодирующего белок. «Немодифицированный микроорганизм» можно использовать взаимозаменяемо со «штаммом до модификации», «микроорганизмом до модификации», «немутировавшим штаммом», «немодифицированный штаммом», «немутировавшим микроорганизмом» или «эталонным микроорганизмом».
В качестве еще одного примера по настоящему изобретению микроорганизм рода Corynebacterium по настоящему изобретению может представлять собой Corynebacterium glutamicum, Corynebacterium crudilactis, Corynebacterium deserti, Corynebacterium efficiens, Corynebacterium callunae, Corynebacterium stationis, Corynebacterium singulare, Corynebacterium halotolerans, Corynebacterium striatum, Corynebacterium ammoniagenes, Corynebacterium pollutisoli, Corynebacterium imitans, Corynebacterium testudinoris или Corynebacterium flavescens.
Конкретно, рекомбинантный микроорганизм по настоящему изобретению может представлять собой микроорганизм, в котором весь или часть полинуклеотида, кодирующего белок, включающий аминокислотную последовательность SEQ ID NO: 1, удаляют.
Кроме того, микроорганизм по настоящему изобретению может представлять собой микроорганизм, у которого дополнительно ослабляют активность репрессора аргинина (ArgR). Конкретно, микроорганизм по настоящему изобретению может представлять собой микроорганизм, у которого дополнительно удаляют весь ген argR или его часть.
ArgR может включать полипептид, указанный в аминокислотной последовательности SEQ ID NO: 13. Ген argR может включать полинуклеотид, указанный в последовательности оснований нуклеиновой кислоты SEQ ID NO: 14, но без ограничения этим.
В качестве примера, микроорганизм по настоящему изобретению может представлять собой штамм, в котором ген NCgl1469 удален в Corynebacterium glutamicum KССМ10741Р (van der Rest et al, Appl. Microbiol Biotechnol 52:541-545, 1999), или штамм, в котором в Corynebacterium glutamicum CJ1R удален ген NCgl1469.
Кроме того, микроорганизм по настоящему изобретению может включать субъединицу F орнитинкарбамоилтрансферазы (ArgF), полинуклеотид, кодирующий ArgF или ген argF. ArgF по настоящему изобретению может состоять из аминокислотной последовательности, указанной в SEQ ID NO: 15. Ген argF по настоящему изобретению может состоять из последовательности оснований нуклеиновой кислоты, указанной в SEQ ID NO: 16.
При использовании здесь термин «ослабление» белка имеет концепцию, охватывающую все снижения активности или отсутствие активности по сравнению с присущей белку активностью. Ослабление можно использовать взаимозаменяемо с инактивацией, дефицитом, понижающей регуляцией, уменьшением, редуцированием, аттенуацией или подобным.
Ослабление может также включать в себя: случай, когда снижают или элиминируют активность самого белка по сравнению с активностью белка, которой обладал исходный микроорганизм, вследствие мутации или чего-либо подобного у полинуклеотида, кодирующего белок; случай, когда снижают всю активность и/или концентрацию (уровень экспрессии) белка в клетке по сравнению с нативным штаммом вследствие ингибирования экспрессии гена полинуклеотида, кодирующего этот белок; или ингибирования трансляции в белок; случай, когда экспрессия полинуклеотида не достигается; и/или случай, когда белок не имеет активность, несмотря на экспрессию полинуклеотида. Термин «присущая активность» относится к активности конкретного белка, которой первоначально обладал родительский штамм до модификации, или к активности микроорганизма дикого типа или немодифицированного микроорганизма, когда признак изменяется вследствие генетической мутации, вызванной естественным или искусственным фактором. Этот термин можно использовать взаимозаменяемо с «активностью до модификации». «Инактивация», «дефицит», «снижение», «понижающая регуляция», «редуцирование» или «аттенуация» активности белка по сравнению с его присущей активностью означает, что активность белка снижают по сравнению с активностью конкретного белка, которой исходно обладал родительский штамм до трансформации или немодифицированный микроорганизм.
Ослабление активности белка можно выполнять посредством любого способа, известного в данной области, но без ограничения этим, и можно достигать посредством применения различных способов, хорошо известных в данной области техники (например Nakashima N et al., Bacterial cellular engineering by genome editing and gene silencing, hit J Mol Sci. 2014; 15(2):2773-2793; and Sambrook et al. Molecular Cloning 2012).
Конкретно, ослабление белка по настоящему изобретению может быть достигнуто посредством:
1) удаления всего или части гена, кодирующего белок;
2) модификации области контроля экспрессии (или последовательности контроля экспрессии) таким образом, чтобы снизить экспрессию гена, кодирующего белок;
3) модификации аминокислотной последовательности (например, делеция/замена/добавление по меньшей мере одной аминокислоты в аминокислотной последовательности), составляющей белок, таким образом, чтобы элиминировать или ослабить активность белка;
4) модификации последовательности гена, кодирующего белок, для того чтобы элиминировать или ослабить активность белка (например, делеция/замена/добавление по меньшей мере одного основания нуклеиновой кислоты в последовательность оснований нуклеиновой кислоты гена белка для того, чтобы кодировать белок, модифицированный чтобы элиминировать или ослаблять активность белка);
5) модификации последовательности оснований нуклеиновой кислоты, кодирующей инициирующий кодон или 5'-UTR-участок транскрипта гена, кодирующего белок;
6) введения антисмыслового олигонуклеотида (например антисмысловой РНК), комплементарно связывающегося с транскриптом гена, кодирующего полипептид;
7) добавления последовательности, комплементарной последовательности Шайна-Дальгарно гена, кодирующего белок, в начало последовательности Шайна-Дальгарно таким образом, чтобы формировать вторичную структуру, которая делает невозможным присоединение рибосом;
8) добавления промотора, который подлежит обратной транскрипции, к З'-концу открытой рамки считывания (ОРС) последовательности гена, кодирующего белок (инженерия обратной транскрипции, RTE); или
9) комбинации из двух или более из (1)-(8) выше, но не ограниченных ими.
Например, они описаны следующим образом.
Удаление всего или части гена, кодирующего белок, указанный в пункте (1) выше, может представлять собой элиминирование всего полинуклеотида, кодирующего эндогенный целевой белок в хромосоме, замену полинуклеотидом с делециями некоторых нуклеотидов или замену маркерным геном.
Модификация области контроля экспрессии (или последовательности контроля экспрессии), по пункту 2 выше, может представлять собой индуцирование мутации в области контроля экспрессии (или последовательности контроля экспрессии) посредством делеции, вставки, неконсервативной или консервативной замены, или их комбинации, или замены последовательностью, имеющей более ослабленную активность. Область контроля экспрессии включает в себя промотор, операторную последовательность, последовательность для кодирования сайта связывания с рибосомой и последовательность для контроля терминации транскрипции и трансляции, но без ограничения этим.
Модификация последовательности оснований нуклеиновой кислоты, кодирующей инициирующий кодон или 5'-UTR-участок транскрипта гена, кодирующего белок по пункту 3 выше, может представлять собой, например, замену на другой инициирующий кодон, имеющий более низкую скорость экспрессии белка, кодирующую последовательность оснований нуклеиновой кислоты, а не на эндогенный инициирующий кодон, но без ограничения этим.
Модификация аминокислотной последовательности или полинуклеотидной последовательности в пунктах 4 и 5 выше может представлять собой индуцирование мутации в последовательности посредством делеции, вставки, неконсервативной или консервативной замены или их комбинации в аминокислотной последовательности белка или полинуклеотидной последовательности, кодирующей белок, таким образом, чтобы ослабить активность белка, или замены аминокислотной последовательностью или полинуклеотидной последовательностью, модифицированной для дальнейшего ослабления активности, или аминокислотной последовательностью или полинуклеотидной последовательностью, модифицированными для отсутствия активности, но не ограничивающимися этим. Например, экспрессия гена может быть ингибирована или ослаблена посредством введения мутации в полинуклеотидную последовательность с образованием терминирующего кодона.
Введение антисмыслового олигонуклеотида (например антисмысловой РНК), комплементарно связывающегося с транскриптом гена, кодирующего белок, указанный в пункте 6 выше, может быть описано, например, в литературе [Weintraub, Н. et al.,Antisense-RNA as a molecular tool for genetic analysis, Reviews - Trends in Genetics, Vol. 1(1) 1986].
Добавление последовательности, комплементарной последовательности Шайна-Дальгарно гена, кодирующего белок, выше последовательности Шайна-Дальгарно, чтобы образовать вторичную структуру, которая делает невозможным присоединение рибосом по п. 7 выше, может приводить к невозможности трансляции мРНК или снижению ее скорости.
Добавление промотора, который подлежит обратной транскрипции, к 3'-концу открытой рамки считывания (ОРС) последовательности гена, кодирующей белок (инженерия обратной транскрипции, RTE) по пункту 8 выше, может привести к созданию антисмыслового нуклеотида, комплементарного транскрипту гена, кодирующего полипептид, чтобы так образом ослабить активность белка.
При использовании здесь термин «усиление» активности белка относится к повышению активности белка по сравнению с его собственной активностью. Термин «усиление» можно использовать взаимозаменяемо с такими терминами, как активация, повышающая регуляция, сверхэкспрессия, повышение и тому подобное. Конкретно, активация, усиление, повышающая регуляция, сверхэкспрессия и повышение могут включать в себя проявление активности, которой обладали изначально, или проявление активности, которая была улучшена по сравнению с собственной активностью или активностью до модификации. «Собственная активность» относится к активности конкретного белка, которой изначально обладал родительский штамм до трансформации или немодифицированный микроорганизм, когда признак изменяется вследствие генетической мутации, вызванной естественным или искусственным фактором. Этот термин можно использовать взаимозаменяемо с «активностью до модификации». «Усиление», «повышающая регуляция», «сверхэкспрессия» или «повышение» активности белка по сравнению с его собственной активностью обозначает активность и/или концентрацию (уровень экспрессии) конкретного полипептида, которым изначально обладал родительский штамм до трансформации или немодифицированный микроорганизм.
Усиление может быть достигнуто посредством введения экзогенного белка или повышения активности и/или концентрации (уровня экспрессии) эндогенного белка. Усиление активности белка можно подтверждать путем повышения степени активности или уровня экспрессии соответствующего белка или повышения количества продукта, выделяемого из соответствующего белка.
Усиление активности белка может быть достигнуто посредством применения различных способов, хорошо известных в данной области техники, и эти способы не ограничивают до тех пор, пока способы могут усиливать активность целевого белка по сравнению с активностью микроорганизма до трансформации. Конкретно, усиление активности белка может быть достигнуто посредством использования генной инженерии и/или белковой инженерии, хорошо известных специалисту в данной области техники, которые представляют собой рутинные способы в молекулярной биологии, но не ограничиваются ими (e.g., Sitnicka et al. Functional Analysis of Genes. Advances in Cell Biology. 2010, Vol.2. 1-16; and Sambrook et al. Molecular Cloning 2012).
Конкретно, повышение эффективности белка по настоящему изобретению может быть достигнуто посредством:
1) увеличения числа внутриклеточных копий полинуклеотида, кодирующего белок;
2) замены области контроля экспрессии на хромосоме, кодирующей белок, на последовательность с более высокой активностью;
3) модификации последовательности оснований нуклеиновой кислоты, кодирующей инициирующий кодон или 5'-UTR-участок транскрипта гена, кодирующего белок;
4) модификации аминокислотной последовательности белка таким образом, чтобы повысить активность белка;
5) модификации полинуклеотидной последовательности, кодирующей белок, чтобы усилить активность белка (например, модификации полинуклеотидной последовательности гена белка, чтобы кодировать белок, модифицированный для усиления активности белка);
6) введения экзогенного белка, проявляющего активность данного белка, или экзогенного полинуклеотида, кодирующего его;
7) оптимизации кодона полинуклеотида, кодирующего белок;
8) модификации или химической модификации экспонированного участка, выбранного путем анализа третичной структуры белка; или
9) комбинации из двух или более вариантов, выбранных из пп. (1)-(8) выше, но этим конкретно не ограничивается.
Более конкретное описание приводится ниже.
Увеличение числа внутриклеточных копий полинуклеотида, кодирующего белок, по п. 1 выше, может быть достигнуто посредством введения в клетку-хозяина вектора, функционально связанного с полинуклеотидом, кодирующим соответствующий белок, и способного реплицироваться и функционировать независимо от хозяина. Альтернативно, увеличение можно достигать посредством введения по меньшей мере одной копии полинуклеотида, кодирующего соответствующий белок, в хромосому клетки-хозяина. Введение в хромосому можно выполнять посредством введения в клетку-хозяина вектора, способного вставлять полинуклеотид в хромосому в клетке-хозяине, но без ограничения этим. Вектор представляет собой то, что описано выше.
Замена области контроля экспрессии (или последовательности контроля экспрессии) на хромосоме, кодирующей белок, на последовательность с более высокой активностью по пункту 2 выше, может представлять собой, например, индуцирование мутации в последовательности посредством делеции, вставки, неконсервативной или консервативной замены или их комбинации для дополнительного усиления активности области контроля экспрессии, или замену последовательностью, имеющей более сильную активность. Область контроля экспрессии может включать в себя, но конкретно не ограничивается ими, промотор, операторную последовательность, последовательность, кодирующую сайт связывания с рибосомой, последовательность, контролирующую терминацию транскрипции и трансляции, и тому подобное. Например, замена может представлять собой замену исходного промотора на сильный промотор, но без ограничения этим.
Известные примеры более сильного промотора могут включать промоторы от CJ1 до CJ7 (US 7662943 В2), промотор lac, промотор trp, промотор trc, промотор tac, промотор PR фага лямбда, промотор PL, промотор tet, промотор gapA, промотор SPL7, промотор SPL13 (sm3) (US 10584338 В2), промотор O2 (US 10273491 В2), промотор tkt, промотор уссА и тому подобное, но не ограничиваются этим.
Модификация последовательности оснований нуклеиновой кислоты, кодирующей инициирующий кодон или 5'-UTR-участок генного транскрипта, кодирующего белок, по п. 3 выше, может представлять собой, например, замену на последовательность оснований нуклеиновой кислоты, кодирующую не собственный инициирующий кодон, а другой инициирующий кодон, имеющий более низкую скорость экспрессии из белка, но без ограничения этим.
Модификация аминокислотной последовательности или полинуклеотидной последовательности в пп. 4 и 5 выше может представлять собой индуцирование мутации в последовательности посредством делеции, вставки, неконсервативной или консервативной замены или их комбинации в аминокислотной последовательности белка или полинуклеотидной последовательности, кодирующей белок, чтобы усилить активность белка, или замену на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную для дальнейшего усиления активности, или аминокислотную последовательность или полинуклеотидную последовательность, модифицированную для повышения активности белка, но не ограничиваются этим. Замену можно выполнять путем встраивания полинуклеотида в хромосому посредством гомологичной рекомбинации, но без ограничения этим. Вектор, используемый в данном документе, может дополнительно включать селективный маркер для идентификации встраивания в хромосому. Селективный маркер представляет собой такой, как описано выше.
Введение экзогенного полипептида, проявляющего активность полипептида по п. 6 выше, может представлять собой введение в клетку-хозяина экзогенного полинуклеотида, кодирующего белок, проявляющий такую же/сходную активность, что и этот белок. Экзогенный полинуклеотид не ограничивают происхождением или последовательностью до тех пор, пока экзогенный полинуклеотид проявляет ту же активность, что и белок. Введение можно выполнять посредством любого известного способа трансформации, который соответствующим образом выбирается специалистом в данной области техники, и введенный полинуклеотид экспрессируется в клетке-хозяине, и, таким образом, продуцируют белок и его активность может быть повышена.
Оптимизация кодона полинуклеотида, кодирующего белок по п. 7, может представлять собой оптимизацию кодона эндогенного полинуклеотида, чтобы повышать его транскрипцию или трансляцию в клетке-хозяине, или оптимизацию кодона экзогенного полинуклеотида, чтобы выполнить оптимизированную транскрипцию или трансляцию в клетке-хозяине.
Модификация или химическая модификация экспонированного участка, выбранного в результате анализа третичной структуры белка в пункте 8 выше, может представлять собой, например, модификацию или химическую модификацию экспонированного участка, подлежащего модификации или химической модификации путем сравнения информации о последовательности белка, подлежащего анализу, с базой данных, в которой хранится информация с последовательностях существующих белков, для определения белка-кандидата матрицы в соответствии со сходством последовательностей и идентификации структуры на основе определенного кандидата.
Усиление активности белка может означать, что активность, концентрация или уровень экспрессии соответствующего белка повышают относительно активности или концентрации белка, экспрессируемого в микробном штамме дикого типа или микробном штамме до модификации, или что количество продукта, полученного из соответствующего белка, увеличивается, но без ограничения этим.
При использовании здесь термин «вектор» относится к конструкции ДНК, которая включает в себя последовательность нуклеотидных оснований полинуклеотида, кодирующего целевой белок, и функционально связанную с подходящим участком контроля экспрессии (или последовательностью контроля экспрессии), таким образом, что целевой полипептид может экспрессироваться в подходящем хозяине. Область контроля экспрессии может включать промотор, способный инициировать транскрипцию, любую операторную последовательность для контроля такой транскрипции, последовательность для кодирования подходящего сайта связывания мРНК с рибосомой и последовательность для контроля терминации транскрипции и трансляции. Вектор после трансформации в подходящую клетку-хозяина может реплицироваться или функционировать независимо от генома хозяина или может быть интегрирован в сам геном.
Вектор, используемый в настоящем изобретении, конкретно не ограничивают и можно использовать любой вектор, известный в данной области техники. Примеры вектора, который обычно используют, могут включать нативные или рекомбинантные плазмиды, космиды, вирусы и бактериофаги. Например, pWE15, М13, MBL3, MBL4, IХII, ASHII, АРII, t10, t11, Charon4A и Charon21A можно использовать в качестве фаговых векторов или космидных векторов, и векторы на основе pDZ, pBR, pUC, pBluescriptll, pGEM и pTZ В pCL и pET можно использовать в качестве плазмидных векторов. Конкретно, можно использовать векторы pDZ, pDC, pdc2, pACYC177, pACYC184, pCL, pECCG117, pUC19, pBR322, pMW118 и рСС1ВАС.
В качестве примера, полинуклеотид, кодирующий целевой полипептид, может быть вставлен в хромосому посредством вектора для хромосомной вставки в хромосому. Вставку полинуклеотида в хромосому можно выполнять, используя любой способ, известный в данной области техники, например, гомологичную рекомбинацию, но без ограничения этим. Вектор может дополнительно включать селективный маркер для исследования вставки или невставки в хромосому. Селективный маркер используют для отбора клеток, трансформированных вектором, то есть для идентификации вставки или невставки целевой молекулы нуклеиновой кислоты, и можно использовать маркеры, придающие селектируемый фенотип, такой как устойчивость к лекарственным средствам, ауксотрофия, устойчивость к цитотоксическим лекарствам или экспрессия поверхностных полипептидов. В условиях обработки селективными агентами только клетки, экспрессирующие селективные маркеры, могут выживать или проявлять другие фенотипические признаки, что делает возможным отбор трансформированных клеток.
При использовании здесь термин «трансформация» означает, что вектор, содержащий полинуклеотид, кодирующий целевой полипептид, вводят в клетку-хозяина или микроорганизм для обеспечения возможности экспрессии полипептида, кодируемого этим полинуклеотидом, в клетке-хозяине. Примеры трансформированного полинуклеотида могут включать любой полипептид пока он может экспрессироваться в клетке-хозяине, независимо от того, вставлен ли он и локализован ли в хромосоме клетки-хозяина или расположен вне хромосомы. Кроме того, полинуклеотид включает ДНК и/или РНК, кодирующие целевой полипептид. Полинуклеотид можно вводить в любой форме, пока полинуклеотид может быть введен и экспрессирован в клетке-хозяине. Например, полинуклеотид можно вводить в виде экспрессионной кассеты, которая представляет собой генную конструкцию, содержащую все факторы, необходимые для самоэкспрессии. Экспрессионная кассета, как правило, может включать промотор, функционально связанный с полинуклеотидом, сигнал терминации транскрипции, сайт связывания с рибосомой и сигнал окончания трансляции. Экспрессионная кассета может быть в форме самореплицирующегося вектора экспрессии. Кроме того, полинуклеотид можно вводить в клетку-хозяина таким, как он есть, для функционального связывания с последовательностью, необходимой для экспрессии в клетке-хозяине, но без ограничения этим.
Приведенный выше термин «функционально связанный» относится к функциональной связи между промоторной последовательностью для инициации и опосредования транскрипции полинуклеотида, кодирующего белок-мишень по настоящему изобретению, и полинуклеотидной последовательностью.
В микроорганизме по настоящему изобретению модификация всего или части полинуклеотида может быть индуцирована посредством (а) гомологичной рекомбинации с использованием вектора для встраивания хромосом в хромосому или редактирования генома с использованием модифицированной нуклеазы (например, CRISPR-Cas9) и/или (б) обработки светом, например, ультрафиолетовыми лучами и радиоактивными лучами и/или химическими веществами, не ограничиваясь ими. Модификация всего гена или его части может включать способ с использованием технологии рекомбинантной ДНК. Например, весь ген или его часть можно делетировать посредством введения нуклеотидной последовательности или вектора, который содержит нуклеотидную последовательность, гомологичную гену-мишени, в микроорганизм для индуцирования гомологичной рекомбинации. Введенная нуклеотидная последовательность или вектор может содержать доминирующий селективный маркер, но без ограничения этим.
В соответствии с еще одним аспектом настоящего изобретения предлагается способ получения L-аргинина, включающий культивирование в среде рекомбинантного микроорганизма рода Corynebacterium, в котором активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, ослаблена.
Белок, включающий аминокислотную последовательность SEQ ID NO: 1, ослабление, микроорганизм и тому подобные, представляют собой такие, как описано в других аспектах.
Микроорганизм рода Corynebacterium может представлять собой Corynebacterium glutamicum, но без ограничения этим.
В микроорганизме активность аргининового репрессора (ArgR) можно дополнительно ослаблять, или ген argR можно дополнительно делетировать, но этим не ограничивается. Ослабление и делеция описаны в других аспектах.
При использовании здесь термин «культивирование» относится к выращиванию микроорганизма рода Corynebacterium по настоящему изобретению в условиях окружающей среды, подобранных соответствующим образом. Этап культивирования по настоящему изобретению можно выполнять в соответствии с подходящими средами или условиями культивирования, известными специалистам в данной области техники. Стадия культивирования может легко регулироваться специалистом в данной области техники в соответствии с выбранным штаммом. Конкретно, культивирование можно выполнять периодическим, непрерывным и/или периодическим с подпиткой способами, но без ограничения этим.
При использовании здесь термин «среда» относится к смеси, содержащей в качестве основных ингредиентов питательные вещества, необходимые для культивирования микроорганизма по настоящему изобретению, где среда обеспечивает питательные вещества, факторы роста и тому подобное, включая воду, необходимую для выживания и роста. Конкретно, среда и другие условия культивирования для культивирования микроорганизма рода Corynebacterium по настоящему изобретению могут представлять собой любую среду, которую используют для обычного культивирования микроорганизмов, без особых ограничений. Однако микроорганизм рода Corynebacterium по настоящему изобретению можно культивировать в обычной среде, содержащей подходящие источники углерода, источники азота, источники фосфора, неорганические соединения, аминокислоты и/или витамины, в аэробных условиях, при этом регулируются температура, рН и тому подобное.
Конкретно, культуральные среды для микроорганизмов рода Corynebacterium можно найти в литературе ["Manual of Methods for General Bacteriology" by the American Society for Bacteriology (Washington D.C., USA, 1981].
В настоящем изобретении примеры источников углерода могут включать: углеводы, такие как глюкоза, сахароза, лактоза, фруктоза, сахароза и мальтоза; сахарные спирты, такие как маннит и сорбит; органические кислоты, такие как пировиноградная кислота, молочная кислота и лимонная кислота; и аминокислоты, такие как глутаминовая кислота, метионин и лизин. Кроме того, можно использовать натуральные органические источники питательных веществ, такие как гидролизаты крахмала, меласса, сырая меласса, рисовые отруби, маниока, багасса и жидкий кукурузный экстракт, и, в частности, можно использовать углеводы, такие как глюкоза и стерильная предварительно обработанная меласса (то есть меласса, превращенная в восстановленные сахара), и соответствующие количество других источников углерода можно использовать без ограничений. Эти источники углерода можно использовать по отдельности или в комбинации из двух или более, но не ограничиваются этим.
Примеры источников азота могут включать: неорганические источники азота, такие как аммиак, сульфат аммония, хлорид аммония, ацетат аммония, фосфат аммония, карбонат аммония и нитрат аммония; аминокислоты, такие как глутаминовая кислота, метионин и глутамин; и органические источники азота, такие как пептон, NZ-амин, мясные экстракты, дрожжевые экстракты, солодовые экстракты, кукурузный отвар, гидролизаты казеина, рыба или продукты ее разложения, обезжиренный соевый жмых или продукты его разложения. Эти источники азота можно использовать по отдельности или в комбинации двух или более из них, но не ограничиваются ими.
Примеры источников фосфора могут включать одноосновный фосфат калия, двухосновный фосфат калия и соответствующие им содержащие натрий соли. Примеры неорганических соединений могут включать хлорид натрия, хлорид кальция, хлорид железа, сульфат магния, сульфат железа, сульфат марганца, карбонат кальция и тому подобное, и, кроме того, могут включать аминокислоты, витамины и/или подходящие предшественники. Эти составляющие ингредиенты или предшественники можно добавлять в среду периодическим или непрерывным способом. Однако примеры источников фосфора этим не ограничиваются.
рН среды можно регулировать во время культивирования микроорганизма рода Corynebacterium, описанного в настоящем изобретении путем добавления соответствующим образом в среду соединений, таких как гидроксид аммония, гидроксид калия, аммиак, фосфорная кислота и серная кислота. Кроме того, для подавления образования пены во время культивирования можно добавлять пеногаситель, такой как сложный полигликоле вый эфир жирной кислоты. Дополнительно, кислород или кислородсодержащий газ можно вводить в среду для поддержания аэробного состояния среды, или можно вводить азот, водород или газообразный диоксид углерода, без введения газа для поддержания анаэробного или неаэробного состояния среды, но не ограничиваясь этим.
В настоящем описании температуру культивирования можно поддерживать от 20°С до 45°С и, конкретно, от 25°С до 40°С, и культивирование можно выполнять в течение примерно от 10 до 160 часов, но без ограничения этим.
L-аргинин, полученный посредством культивирования по настоящему изобретению, может высвобождаться в среду или может оставаться в клетках.
Способ получения L-аргинина по настоящему изобретению может дополнительно включать получение микроорганизма рода Corynebacterium по настоящему изобретению, приготовление среды для культивирования микроорганизма или комбинацию этих стадий (независимо от порядка, в любом порядке), например, перед стадией культивирования.
Способ получения L-аргинина по настоящему изобретению может дополнительно включать извлечение L-аргинина из среды, полученной в результате культивирования (среды, в которой выполняли культивирование), или из микроорганизма рода Corynebacterium в среде, полученной в результате культивирования. Способ по настоящему изобретению может дополнительно включать стадию извлечения после стадии культивирования.
Извлечение может представлять собой сбор целевого L-аргинина с использованием соответствующего способа, известного в данной области техники, в соответствии со способом культивирования микроорганизма по настоящему изобретению, например, периодического, непрерывного или периодического с подпиткой типа культивирования. Примеры способа могут включать центрифугирование, фильтрацию, обработку агентом, осаждающим кристаллизованный белок (высаливание), экстракцию, разрушение ультразвуком, ультрафильтрацию, диализ, различные виды хроматографии, такие как хроматография на молекулярных ситах (гель-фильтрация), адсорбционная хроматография, ионообменная хроматография и аффинная хроматография, ВЭЖХ (высокоэффективная жидкостная хроматография) и комбинацию этих способов, и целевой L-аргинин можно извлекать из среды или микроорганизма путем использования подходящего способа, известного в данной области техники.
Способ получения L-аргинина по настоящему изобретению может дополнительно включать стадию очистки. Очистку можно выполнять с использованием подходящего способа, известного в данной области техники. В одном примере, когда способ получения L-аргинина по настоящему изобретению включает в себя как стадию извлечения, так и стадию очистки, стадию извлечения и стадию очистки можно выполнять непрерывно или с перерывами независимо от порядка, или можно выполнять одновременно или объединять в одну стадию, но не ограничиваются этим.
Согласно еще одному аспекту настоящего изобретения предлагается композиция для получения L-аргинина, где композиция содержит: микроорганизм рода Corynebacterium, в котором активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, ослаблена; среду, в которой микроорганизм был культивирован, или их комбинацию.
Композиция по настоящему изобретению может дополнительно содержать любой подходящий эксципиент, который обычно используется в композициях для получения аминокислот, и примеры эксципиента могут включать консервант, смачивающий агент, диспергирующий агент, суспендирующий агент, буфер, стабилизирующий агент или изотонический агент, но не ограничиваясь этим.
В композиции по настоящему изобретению белок, включающий аминокислотную последовательность SEQ ID NO: 1, ослабление, микроорганизм, культивирование, среда и тому подобные представляют собой такие, как описано в других аспектах.
Согласно еще одному аспекту настоящего изобретения предлагается способ получения микроорганизма рода Corynebacterium, где способ включает ослабление активности белка, включающего аминокислотную последовательность SEQ ID NO: 1.
Согласно еще одному аспекту настоящего изобретения предлагается применение микроорганизма рода Corynebacterium для получения L-аргинина, в котором активность белка, включающего аминокислотную последовательность SEQ ID NO: 1, ослаблена.
В способе получения микроорганизма рода Corynebacterium и применения микроорганизма рода Corynebacterium для получения L-аргинина белок, включающий аминокислотную последовательность SEQ ID NO: 1, ослабление, микроорганизм и тому подобные представляют собой такие, как описано в других аспектах.
Вариант осуществления изобретения
Далее настоящее изобретение будет подробно описано со ссылкой на Примеры. Однако эти Примеры представляют собой только предпочтительные воплощения для иллюстрации настоящего изобретения и, следовательно, не предназначены для ограничения объема прав настоящего изобретения. При этом, технические вопросы, не описанные здесь, могут быть достаточно поняты и легко реализованы специалистом в данной области техники или аналогичных настоящему изобретению областях техники.
Пример 1: Создание библиотеки случайных мутантов с использованием транспозона и скрининга
Для получения штаммов, имеющих повышенную способность продуцировать L-аргинин, Corynebacterium glutamicum KССМ10741Р (US 8034602 В2) в качестве родительского штамма трансформировали с помощью плазмиды, полученной с использованием набора Transposome™ EZ-Tn5 <R6Kyori/KAN-2>Tnp (Epicentre), посредством электроимпульсного способа (приложение Microbial. Biotechnol. (1999) 52:541-545), и затем трансформанты высевали на комплексную питательную среду, содержащую канамицин (25 мг/л), в результате чего было получено примерно 20000 колоний.
Комплексная среда для чашек (рН 7,0)
Глюкоза 10 г, пептон 10 г, мясной экстракт 5 г, дрожжевой экстракт 5 г, сердечно-мозговая вытяжка 18,5 г, NaCl 2,5 г, мочевина, 2 г, сорбит 91 г, агар 20 г (из расчета на 1 литр дистиллированной воды)
Семь лучших мутантных штаммов с улучшенной способностью продуцировать L-аргинин по сравнению со штаммом Corynebacterium glutamicum KССМ10741Р в качестве родительского штамма отбирали с использованием способа с нингидрином (Moore, S., Stein, W. Н., Photometric ninhydrin method for use in the chromatography of amino acids. J. Biol. Chem. 1948, 176, 367-388).
Пример 2: Анализ способности к продуцированию L-аргинина у выбранных случайным образом мутантных штаммов
Для окончательного отбора штаммы с воспроизводимо повышенной способностью к продуцированию L-аргинина из семи мутантных штаммов, отобранных в Примере 1, выполняли культивирование в колбе с использованием следующей среды. После завершения культивирования концентрацию L-аргинина в культуре анализировали посредством ВЭЖХ. Концентрации L-аргинина, продуцированного каждым из мутантных штаммов, приведены в Таблице 1 ниже.
Среда для продуцирования (рН 7,2)
Глюкоза 60 г, сульфат аммония 45 г, гептагидрат сульфата магния 2 г, одноосновный фосфат калия 2 г, хлорид аммония 10 г, биотин 0,01 мг, тиамин-НСl 0,1 мг, пантотенат кальция 2 мг, рикотинамид 3 мг, сульфат железа 10 мг, сульфат марганца 10 мг, сульфат цинка 0,02 мг, сульфат меди 0,5 мг, карбонат кальция 30 г (из расчета на 1 л дистиллированной воды).
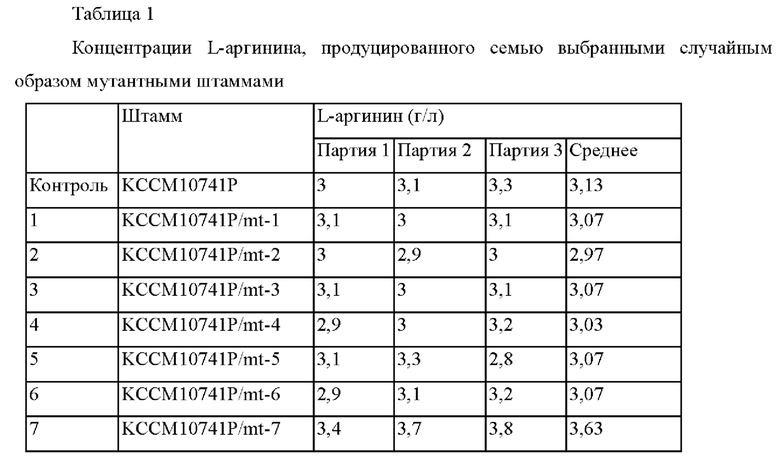
Из десяти отобранных мутантных штаммов окончательно выбрали KCCM10741P/mt-7 в качестве штамма со значительно улучшенной способностью продуцировать L-аргинин.
Пример 3. Установление причин повышенной способности продуцировать L-аргинин у окончательно выбранного штамма
Идентифицировали ген, инактивированный случайной вставкой транспозона в KCCM10741P/mt-7 из Примера 2.
Конкретно, геномную ДНК выделяли из KCCM10741P/mt-7, расщепляли и затем лигировали, и продукт лигирования трансформировали в Е. coli DH5a. Трансформанты высевали на твердую среду LB, содержащую канамицин (25 мг/л). После отбора 20 трансформированных колоний получали плазмиды, содержащие неизвестную часть гена, и их последовательности оснований нуклеиновых кислот анализировали с использованием праймера 1 (SEQ ID NO: 3) и праймера 2 (SEQ ID NO: 4) из набора Transposome™ EZ-Tn5 <R6Kγori/KAN-2> Тпр. В результате было идентифицировано, что ген, включающий нуклеотидную последовательность SEQ ID NO: 2, был инактивирован на основе последовательности оснований нуклеиновой кислоты, представленной в NIH GenBankU.S. National Institutes of Health.
В результате быстрого поиска SEQ ID NO: 2 Corynebacterium glutamicum дикого типа АТСС 13869, эту последовательность идентифицировали как TQuNCgl1469.
Пример 4: Конструирование рекомбинантного вектора для делеции гена NCgl1469
Для повторной идентификации влияния инактивации гена NCgl1469 на выработку L-аргинина сконструировали рекомбинантный вектор для делеции гена NCgl1469 на хромосоме штамма рода Corynebacterium.
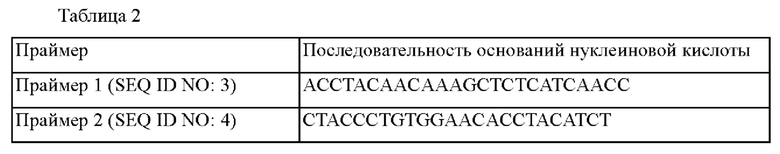
Для получения фрагментов для делеции гена синтезировали праймеры с 3 по 6, которые приведены в Таблице 3.

Для конструирования вектора, имеющего делецию области ОРС на основе SEQ ID NO: 2 выполняли ПЦР с использованием гДНК Corynebacterium glutamicum АТСС 13869 дикого типа в качестве матрицы вместе с парой праймеров SEQ ID NOS: 5 и 6 и парой праймеров SEQ ID NOS: 7 и 8. ПЦР выполняли в условиях денатурации при 95°С в течение 5 минут, 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 30 секунд и затем полимеризации при 72°С в течение 5 минут. Снова выполняли перекрывающуюся ПЦР, используя смесь двух фрагментов, полученных выше, в качестве матрицы, вместе с парой праймеров с SEQ ID NOS: 5 и 8, получая таким образом фрагменты. ПЦР выполняли в условиях денатурации при 94°С в течение 5 минут, 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и затем полимеризации при 72°С в течение 5 минут. Вектор pDCM2 (KR 10-2020-0136813) обрабатывали с помощью SmaI, и полученные таким образом продукты ПЦР подвергали клонированию сливанием. Клонирование сливанием выполняли с использованием набора для клонирования m-Fusion® HD (Clontech). Плазмиду, полученную в результате клонирования, назвали pDCM2-ΔNCgl1469.
Пример 5: Получение штамма с делегированным геном NCgl1469 и оценка способности продуцировать L-аргинин - 1
Corynebacterium glutamicum KССМ10741Р, который представляет собой штамм, продуцирующий L-аргинин, трансформировали с pDCM2-ΔNCgl1469 посредством гомологичной рекомбинации на хромосоме (van der Rest et al., Appl. Microbiol Biotechnol 52:541-545, 1999).
После этого трансформанты подвергали вторичной рекомбинации на чашке Петри с твердой средой, содержащей 4% сахарозы. Трансформированные штаммы Corynebacterium glutamicum после завершения вторичной рекомбинации подвергали ПЦР с использованием праймеров 3 и 6 для идентификации штамма, в котором делетирован ген SEQ ID NO: 2. Рекомбинантный штамм был назван Corynebacterium glutamicum KССМ10741Р-ΔNCgl1469.
Для анализа продуцирующей способности L-аргинина полученной Corynebacterium glutamicum KCCM10741P-ΔNCgl1469 штамм культивировали совместно с родительским штаммом Corynebacterium glutamicum KССМ10741Р следующим способом.
Родительский штамм Corynebacterium glutamicum KССМ10741Р и Corynebacterium glutamicum KCCM10741P-ΔNCgl1469 инокулировали каждый в 250 мл колбу с угловыми перегородками, содержащую 25 мл следующей среды для посева, и культивировали при встряхивании при 200 оборотах в минуту при 30°С в течение 20 часов. Затем 1 мл посевной культуры инокулировали в 250 мл колбу с угловыми перегородками, содержащую 24 мл среды для продуцирования и культивировали при встряхивании со скоростью 200 об/мин при 30°С в течение 72 часов. Составы среды для посева и среды для продуцирования были следующими.
Среда для посева (рН 7,2)
Глюкоза 20 г, сульфат аммония 45 г, гептагидрат сульфата магния 2 г, одноосновный фосфат калия 2 г, хлорид аммония 10 г, биотин 0,01 мг, тиамин-НСl 0,1 мг, пантотенат кальция 2 мг, никотинамид 3 мг, сульфат железа 10 мг, сульфат марганца 10 мг, сульфат цинка 0,02 мг, медный купорос 0,5 мг (из расчета на 1 л дистиллированной воды).
Среда для продуцирования (рН 7,2)
Глюкоза 60 г, сульфат аммония 45 г, гептагидрат сульфата магния 2 г, одноосновный фосфат калия 2 г, хлорид аммония 10 г, биотин 0,01 мг, тиамин-НСl 0,1 мг, пантотенат кальция 2 мг, никотинамид 3 мг, сульфат железа 10 мг, сульфат марганца 10 мг, сульфат цинка 0,02 мг, сульфат меди 0,5 мг, карбонат кальция 30 г (из расчета на 1 л дистиллированной воды).
После завершения культивирования способность продуцировать L-аргинин определяли посредством ВЭЖХ (Waters 2478), и результаты приведены в Таблице 4 ниже.
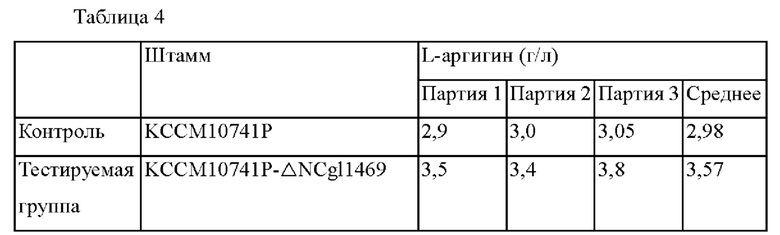
В результате, способность продуцировать L-аргинин у KCCM10741P-ΔNCgl1469 увеличилась в среднем на 19,8% по сравнению с родительским штаммом.
Пример 6: Получение штамма с делегированным геном NCgl1469 и оценка способности продуцировать L-аргинин - 2
Определяли, проявлял ли другой штамм Corynebacterium glutamicum, продуцирующий L-аргинин, тот же эффект, как в Примере 5.
Конкретно, штамм, продуцирующий L-аргинин, получали посредством введения мутации одного типа (делеция гена argR, далее - ΔargR) в штамм Corynebacterium glutamicum дикого типа (АТСС 13869).
Рекомбинантный вектор для ΔargR сконструировали таким же способом, как в способе построения вектора Примера 4, и праймеры, использованные для построения вектора, показаны в Таблице 5.

Плазмиду в которую был вставлен ген-мишень, отбирали посредством ПЦР, и затем плазмиду назвали pDCM2-ΔargR. Штамм Corynebacterium glutamicum дикого типа АТСС13869 трансформировали с помощью pDCM2-ΔargR посредством такого же способа, как в Примере 5. Штаммы-трансформанты подвергали ПЦР с использованием праймеров 7 и 10 для идентификации штамма, в хромосоме которого делетирован argR, и штамм назвали Corynebacterium glutamicum CJ1R. С Corynebacterium glutamicum CJ1R позволили подвергнуться такому же способу, как в Примере 5 для получения штамма, в котором делетирован ген NCgl1469, и штамм назвали CJ1R-ΔNCgl1469.
CJ1R-ΔNCgl1469 культивировали посредством того же способа, что и в Примере 5. После завершения культивирования способность продуцировать L-аргинин определяли посредством ВЭЖХ (Waters 2478), и результаты представлены в Таблице 6 ниже.
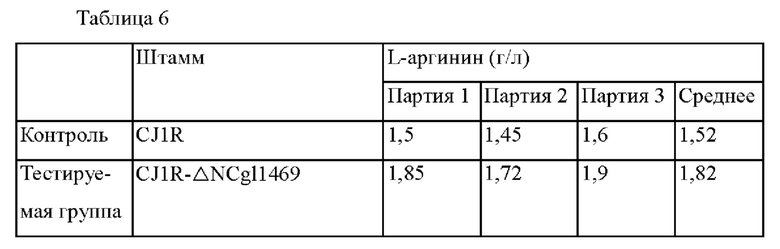
В результате CJlR-ΔNCgl1469 показал среднее увеличение способности продуцировать L-аргинин на 19,7% по сравнению с родительским штаммом CJ1R.
Как указано выше, специалист в данной области техники, к которой относится настоящее раскрытие, сможет понять, что настоящее изобретение может быть воплощено в других конкретных формах без отступления от его технического характера или существенных характеристик. Таким образом, воплощения, описанные выше, следует рассматривать как иллюстрирующие и не ограничивающие настоящее изобретение. Объем настоящего изобретения следует понимать так, что все изменения или модификация, вытекающие из определений и объемов формулы изобретения и их эквивалентов, попадают в объем заявки.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="en"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="P24220RU-st26-RU.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2024-05-06">
<ApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>PCT/KR2022/015551</ApplicationNumberText>
<FilingDate>2022-10-14</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>P24220RU</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2021-0137890</ApplicationNumberText>
<FilingDate>2021-10-15</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">СИДЖЕЙ ЧЕИЛДЖЕДАНГ КОРПОРЕЙШН
</ApplicantName>
<ApplicantNameLatin>CJ CHEILJEDANG CORPORATION</ApplicantNameLatin>
<InventionTitle languageCode="ru">Микроорганизм рода
Corynebacterium, продуцирующий L-аргинин, и способ получения
L-аргинина c применением этого микроорганизма</InventionTitle>
<SequenceTotalQuantity>16</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>203</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..203</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>NCgl1469 amino acid
sequence</INSDQualifier_value>
<NonEnglishQualifier_value>аминокислотная последовательность
NCgl1469</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..203</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSPTVLPATQADFPKIVDVLVEAFANDPAFLRWIPQPDPGSAKLRALFE
LQIEKQYAVAGNIDVARDSEGEIVGVALWDRPDGNHSAKDQAAILPRLVSIFGIKAAQVAWTDLSSARFH
PKFPHWYLYTVATSSSARGTGVGSALLNHGIARAGDEAIYLEATSTRAAQLYNRLGFVPLGYIPSDDDGT
PELAMWKPPAMPTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>612</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..612</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>NCgl1469 nucleotide
sequence</INSDQualifier_value>
<NonEnglishQualifier_value>нуклеотидная последовательность
NCgl1469</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..612</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgagtcccaccgttttgcctgctacacaagctgacttccctaagatcg
tcgatgttctggttgaagcattcgccaacgatccagcatttttacgatggatcccgcagccggaccccgg
ttcagcaaagcttcgagcacttttcgaactgcagattgagaagcagtatgcagtggcgggaaatattgat
gtcgcgcgtgattctgagggagaaatcgtcggcgtcgcgttatgggatcggccagatggtaatcacagtg
ccaaagatcaagcagcgatactcccccggctcgtctccattttcgggatcaaggctgcgcaggtggcgtg
gacggatttgagttcggctcgtttccaccccaaattcccccattggtacctctacaccgtggcaacatct
agttctgcccgtggaacgggtgttggcagtgcgcttcttaatcacggaatcgctcgcgcgggtgatgaag
ctatctatttggaggcgacgtcgactcgtgcggctcaactatataaccgtctgggatttgtgcccttggg
ttatatcccctcagatgatgatggcactcctgaactggcgatgtggaaaccgccagcgatgccaactgtt
taa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q5">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 1</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctacaacaaagctctcatcaacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 2</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
2</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctaccctgtggaacacctacatct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 3</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
3</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgagctcggtaccctcagtgctccaactgcttg</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 4</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
4</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttcagggggataccagaatagcgaaatgaaaag</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 5</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
5</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggtatccccctgaaggcgatttaaggggtcga</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 6</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
6</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctctagaggatccccgcaggggagtttgaaaaac</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 7</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
7</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgagctcggtacccagcaggccttaagggtaag</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 8</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
8</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggggcgctgtcttacctcggctggtgggccagc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 9</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
9</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtaagacagcgcccctagttcaaggcttgttaatc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer 10</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
10</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctctagaggatccccaccgttgaactgcttgccag</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>171</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..171</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>argR amino acid
sequence</INSDQualifier_value>
<NonEnglishQualifier_value>аминокислотная последовательность
argR</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..171</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSLGSTPSTPENLNPVTRTARQALILQILDKQKVTSQVQLSELLLDEGI
DITQATLSRDLDELGARKVRPDGGRAYYAVGPVDSIAREDLRGPSEKLRRMLDELLVSTDHSGNIAMLRT
PPGAAQYLASFIDRVGLKEVVGTIAGDDTVFVLARDPLTGKELGELLSGRTT</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>516</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..516</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>argR nucleotide
sequence</INSDQualifier_value>
<NonEnglishQualifier_value>нуклеотидная последовательность
argR</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..516</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtcccttggctcaaccccgtcaacaccggaaaacttaaatcccgtga
ctcgcactgcacgccaagctctcattttgcagattttggacaaacaaaaagtcaccagccaggtacaact
gtctgaattgctgctggatgaaggcatcgatatcacccaggccaccttgtcccgagatctcgatgaactc
ggtgcacgcaaggttcgccccgatgggggacgcgcctactacgcggtcggcccagtagatagcatcgccc
gcgaagatctccggggtccgtcggagaagctgcgccgcatgcttgatgaactgctggtttctacagatca
ttccggcaacatcgcgatgctgcgcaccccgccgggagctgcccagtacctggcaagtttcatcgatagg
gtggggctgaaagaagtcgttggcaccatcgctggcgatgacaccgttttcgttctcgcccgtgatccgc
tcacaggtaaagaactaggtgaattactcagcgggcgcaccacttaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>319</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table> <INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..319</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>argF amino acid
sequence</INSDQualifier_value>
<NonEnglishQualifier_value>аминокислотная последовательность
argF</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..319</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MTSQPQVRHFLADDDLTPAEQAEVLTLAAKLKAAPFSERPLEGPKSVAV
LFDKTSTRTRFSFDAGIAHLGGHAIVVDSGSSQMGKGETLQDTAAVLSRYVEAIVWRTYAHSNFHAMAET
STVPLVNSLSDDLHPCQILADLQTIVENLSPEEGPAGLKGKKAVYLGDGDNNMANSYMIGFATAGMDISI
IAPEGFQPRAEFVERAEKRGQETGAKVVVTDSLDEVAGADVVITDTWVSMGMENDGIDRTTPFVPYQVND
EVMAKANDGAIFLHCLPAYRGKEVAASVIDGPASKVFDEAENRLHAQKALLVWLLAHQPR</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>960</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..960</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>argF nucleotide
sequence</INSDQualifier_value>
<NonEnglishQualifier_value>нуклеотидная последовательность
argF</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..960</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgacttcacaaccacaggttcgccatttcctggctgatgatgatctca
cccctgcagagcaggcagaggttttgaccctagccgcaaagctcaaggcagcgccgttttcggagcgtcc
actcgagggaccaaagtccgttgcagttctttttgataagacttcaactcgtactcgcttctccttcgac
gcgggcatcgctcatttgggtggacatgccatcgtcgtggattccggcagctcacagatgggtaagggcg
agaccctgcaggacaccgcagctgtattgtcccgctacgtggaagcaattgtgtggcgcacctacgcaca
cagcaatttccacgccatggcggagacgtccactgtgccactggtgaactccttgtccgatgatctgcac
ccatgccagattctggctgatctgcagaccatcgtggaaaacctcagccctgaagaaggcccagcaggcc
ttaagggtaagaaggctgtgtacctgggcgatggcgacaacaacatggccaactcctacatgattggctt
tgccaccgcgggcatggatatctccatcatcgctcctgaagggttccagcctcgtgcggaattcgtggag
cgcgcggaaaagcgtggccaggaaaccggcgcgaaggttgttgtcaccgacagcctcgacgaggttgccg
gcgccgatgttgtcatcaccgatacctgggtatccatgggtatggaaaacgacggcatcgatcgcaccac
acctttcgttccctaccaggtcaacgatgaggtcatggcgaaagctaacgacggcgccatcttcctgcac
tgccttcctgcctaccgcggcaaagaagtggcagcctccgtgattgatggaccagcgtccaaagttttcg
atgaagcagaaaaccgcctccacgctcagaaagcactgctggtgtggctgctggcccaccagccgaggta
a</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии. Предложено применение рекомбинантного микроорганизма рода Corynebacterium, у которого полинуклеотид, кодирующий белок, включающий аминокислотную последовательность SEQ ID NO: 1, удален, для получения L-аргинина. Также предложен способ получения L-аргинина, включающий культивирование в среде рекомбинантного микроорганизма рода Corynebacterium, у которого полинуклеотид, кодирующий белок, включающий аминокислотную последовательность SEQ ID NO: 1, удален. Изобретение обеспечивает продуцирование L-аргинина с высоким выходом. 2 н. и 5 з.п. ф-лы, 6 табл., 6 пр.
1. Применение рекомбинантного микроорганизма рода Corynebacterium, у которого полинуклеотид, кодирующий белок, включающий аминокислотную последовательность SEQ ID NO: 1, удален, для получения L-аргинина.
2. Применение по п. 1, где микроорганизм имеет повышенную способность продуцировать L-аргинин по сравнению с родительским штаммом, в котором активность белка, включающего аминокислотную последовательность SEQ ID NO:1, не удалена.
3. Применение по п. 1, где микроорганизм представляет собой Corynebacterium glutamicum.
4. Применение по п. 1, где активность аргининового репрессора дополнительно ослаблена.
5. Способ получения L-аргинина, включающий культивирование в среде рекомбинантного микроорганизма рода Corynebacterium, у которого полинуклеотид, кодирующий белок, включающий аминокислотную последовательность SEQ ID NO: 1, удален.
6. Способ по п. 5, где микроорганизм представляет собой Corynebacterium glutamicum.
7. Способ по п. 5, где в микроорганизме рода Corynebacterium активность аргининового репрессора дополнительно ослаблена.
| Микроорганизм рода Corynebacterium, продуцирующий L-аргинин, и способ получения L-аргинина с использованием этого микроорганизма | 2018 |
|
RU2716573C1 |
| РЕКОМБИНАНТНЫЙ МИКРООРГАНИЗМ, ОБЛАДАЮЩИЙ ПОВЫШЕННОЙ СПОСОБНОСТЬЮ ПРОДУЦИРОВАТЬ ПУТРЕСЦИН, И СПОСОБ ПОЛУЧЕНИЯ ПУТРЕСЦИНА С ПРИМЕНЕНИЕМ ЭТОГО МИКРООРГАНИЗМА | 2013 |
|
RU2604806C2 |
| База данных NCBI Reference Sequence: WP_060564582.1, 13.05.2018 | |||
| MULTISPECIES: GNAT family N-acetyltransferase [Corynebacterium] | |||
| Найдено по адресу: https://www.ncbi.nlm.nih.gov/protein/983387768?sat=54&satkey=10627576 Дата обращения 11.10.2024 | |||
| US 20130023016 A1, 24.01.2013. | |||
Авторы
Даты
2025-04-03—Публикация
2022-10-14—Подача