ОБЛАСТЬ ТЕХНИКИ
Настоящее раскрытие относится к варианту LysE, микроорганизму рода Corynebacterium, содержащему данный вариант, и способу получения L-аргинина с его применением.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
L-аргинин используется в медицинских целях, как, например, в стимуляторах функции печени, стимуляторах функции мозга, сложных аминокислотных препаратах и т.д., и он представляет собой вещество, которое недавно привлекло внимание в качестве пищи, как, например, добавки в рыбный пирог, добавки в оздоровительные напитки, замены солей для пациентов с высоким кровяным давлением и т.д.
Продолжаются исследования для применения микроорганизмов для продуцирования высоких концентраций промышленно применимого L-аргинина, и имелись сообщения о способе применения вариантов штаммов, происходящих от микроорганизмов рода Brevibacterium или Corynebacterium, которые представляют собой штаммы, продуцирующие глутамат, способе применения штамма, продуцирующего аминокислоты, рост которого улучшается посредством слияния клеток и т.д.
Тем не менее, хотя и было сообщение о том, что lysE микроорганизма рода Corynebacterium, который представляет собой экспортер лизина, также экспортирует L-аргинин (Bellmann A, et al, Microbiology, 147:1765-1774, 2001), требуется исследование для дополнительного увеличения продукции L-аргинина.
ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Техническая проблема
Согласно настоящему раскрытию предложен вариант LysE.
Техническое решение
Целью настоящего раскрытия является предложение варианта LysE, в котором любая одна или более чем одна аминокислота из аминокислот, соответствующих положениям 50, 153 и 215 в SEQ ID NO: 1, заменена другой аминокислотой.
Другой целью настоящего раскрытия является предложение полинуклеотида, кодирующего вариант LysE по настоящему раскрытию.
Еще одной другой целью настоящего раскрытия является предложение микроорганизма, содержащего вариант LysE по настоящему раскрытию, или полинуклеотид, кодирующий данный вариант.
Еще одной другой целью настоящего раскрытия является предложение способа продуцирования L-аргинина, включающего стадию культивирования микроорганизма по настоящему раскрытию.
Полезные эффекты
Выход продукции L-аргинина микроорганизмов может быть увеличен с использованием варианта по настоящему раскрытию.
Подробное описание предпочтительных воплощений
Настоящее раскрытие будет подробно описано следующим образом. Тем временем, каждое описание и воплощение, раскрытое в данном раскрытии, также можно применять к другим описаниям и воплощениям. То есть, все комбинации разных элементов, раскрытых в данном раскрытии, попадают в пределы объема настоящего раскрытия. Кроме того, объем настоящего раскрытия не ограничивается конкретным описанием, описанным ниже. Кроме того, во всем данном описании изобретения дается ссылка на и цитируется целый ряд статей и патентных документов. Раскрытия процитированных статей и патентных документов включаются сюда посредством ссылки во всей их полноте для дальнейшего прояснения уровня и объема объекта, к которому относится настоящее раскрытие.
Согласно одному аспекту настоящего раскрытия предложен вариант LysE, в котором любая одна или более чем одна аминокислота из аминокислот, соответствующих положениям 50, 153 и 215 в SEQ ID NO: 1, заменена другой аминокислотой.
Термин «LysE» в том виде, как он здесь используется, относится к белку-экспортеру лизина, кодируемому геном LysE.
В частности, LysE может представлять собой встречающийся в природе полипептид или полипептид дикого типа, может представлять собой его зрелый полипептид или может включать его вариант или его функциональный фрагмент, но может быть включен любой без ограничения, при условии, что он может представлять собой родительский вариант варианта LysE по настоящему раскрытию.
Последовательность LysE по настоящему раскрытию может быть доступна в GenBank NCBI (Национальный институт биотехнологической информации) и т.д., который представляет собой известную базу данных.
В одном воплощении LysE по настоящему раскрытию может происходить из рода Corynebacterium и, например, может происходить из Corynebacterium glutamicum, Corynebacterium suranareeae, Corynebacterium callunae, Corynebacterium deserti, Corynebacterium crudilactis, Corynebacterium efficiens, Corynebacterium pacaense, но не ограничиваясь ими.
В одном воплощении LysE по настоящему раскрытию может представлять собой полипептид SEQ ID NO: 1. В одном воплощении LysE по настоящему раскрытию может представлять собой полипептид, имеющий примерно 60%-ную или большую идентичность последовательности с полипептидом SEQ ID NO: 1, и в белок LysE может быть включен любой полипептид без ограничения, при условии, что он имеет активность, идентичную или соответствующую активности полипептида, состоящего из аминокислотной последовательности SEQ ID NO: 1.
Например, LysE может представлять собой белок, происходящий из рода Corynebacterium, имеющий по меньшей мере примерно 65%-ную, примерно 66%-ную, примерно 67%-ную, примерно 68%-ную, примерно 69%-ную, примерно 70%-ную, примерно 71%-ную, примерно 72%-ную, примерно 73%-ную, примерно 74%-ную, примерно 75%-ную, примерно 76%-ную, примерно 77%-ную, примерно 78%-ную, примерно 79%-ную, примерно 80%-ную, примерно 81%-ную, примерно 82%-ную, примерно 83%-ную, примерно 84%-ную, примерно 85%-ную, примерно 86%-ную, примерно 87%-ную, примерно 88%-ную, примерно 89%-ную, примерно 90%-ную, примерно 91%-ную, примерно 92%-ную, примерно 93%-ную, примерно 94%-ную, примерно 95%-ную, примерно 96%-ную, примерно 97%-ную, примерно 98%-ную, примерно 99%-ную, примерно 99,1%-ную или примерно 99,5%-ную или большую идентичность с SEQ ID NO: 1. Его примеры могут включать белки с №доступа NCBI

В любом воплощении вышеописанных воплощений LysE по настоящему раскрытию может иметь или включать аминокислотную последовательность SEQ ID NO: 1 или может по существу состоять из данной аминокислотной последовательности, но он может включать полипептиды/белки, имеющие такую же активность без ограничения.
Кроме того, очевидно, что в пределы объема полипептида/белка, подлежащего мутации, по настоящему раскрытию также может быть включен любой полипептид/белок, имеющий аминокислотную последовательность, имеющую делецию, модификацию, замену или присоединение в части данной последовательности, при условии, что он имеет такую гомологию или идентичность и демонстрирует активность экспорта L-лизина.
Термин «гомология» или «идентичность» в том виде, как он здесь используется, относится к степени сходства между двумя данными аминокислотными последовательностями или нуклеотидными последовательностями, и она может быть выражена в виде процентной доли. Термины «гомология и идентичность» часто можно использовать взаимозаменяемо.
Гомологию или идентичность последовательности консервативных полинуклеотидов или полипептидов можно определять посредством стандартного алгоритма выравнивания, и совместно можно использовать штрафы за пробел, устанавливаемые по умолчанию программой, подлежащей применению. По существу гомологичные или идентичные последовательности обычно могут гибридизоваться с друг другом целиком или частично при умеренно жестких или очень жестких условиях. Очевидно то, что гибридизация также включает гибридизацию с полинуклеотидом, содержащим обычные кодоны или кодоны с учетом вырожденности кодонов в по линукл е отид е.
Имеют ли любые две последовательности по линукл е отид а или полипептида гомологию, сходство или идентичность, можно определять, например, посредством известного компьютерного алгоритма, такого как программа «FASTA» с использованием параметров по умолчанию, как в Pearson et al (1988) [Proc. Natl. Acad. Sci. USA 85]: 2444. В качестве альтернативы, их можно определять с использованием алгоритма Needleman-Wunsch (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443-453), как осуществляется в программе Needleman пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000, Trends Genet. 16: 276-277) (версия 5.0.0 или более поздняя) (включая программный пакет GCG (Devereux, J., et al, Nucleic Acids Research 12: 387 (1984)), BLASTP, BLASTN, FASTA (Atschul, [S.] [F.,] [ET AL, J MOLEC BIOL 215]: 403 (1990); Guide to Huge Computers, Martin J. Bishop, [ED.,] Academic Press, San Diego,1994 и [CARILLO ET AL.](1988) SIAM J Applied Math 48: 1073). Например, гомологию, сходство или идентичность можно определять с использованием BLAST или ClustalW Национального центра биотехнологической информации.
Гомологию, сходство или идентичность между полинуклеотидами или полипептидами можно определять посредством сравнения информации о последовательности с использованием компьютерной программы GAP, как вводится, например, Needleman et al. (1970), J Mol Biol. 48:443, как раскрыто Smith and Waterman, Adv. Appl. Math (1981) 2:482. Вкратце, программа GAP определяет гомологию, сходство или идентичность как значение, полученное делением числа аналогично выровненных символов (т.е. нуклеотидов или аминокислот) на общее число символов в более короткой из двух последовательностей. Параметры по умолчанию программы GAP могут включать: (1) матрицу бинарных сравнений (содержащую значение 1 для идентичности и значение 0 для неидентичности) и матрицу взвешенных сравнений Gribskov et al (1986) Nucl. Acids Res. 14: 6745, как раскрыто в Schwartz and Dayhoff, eds., Atlas Of Protein Sequence And Structure, National Biomedical Research Foundation, pp.353-358 (1979) (или матрица замен EDNAFULL (EMBOSS версии NCBI NUC4.4)); (2) штраф 3,0 для каждого пробела и дополнительный штраф 0,10 для каждого символа в каждом пробеле (или штраф за открытие пробела 10 и штраф за расширение пробела 0,5); и (3) отсутствие штрафа за концевые пробелы.
Термин «вариант» в том виде, как он здесь используется, относится к полипептиду, который имеет отличную аминокислотную последовательность от последовательности варианта до модификации посредством консервативной замены и/или модификации одной или более чем одной аминокислоты, но сохраняет функции или свойства. Такой вариант обычно можно идентифицировать посредством осуществления модификации одной или более чем одной аминокислоты аминокислотной последовательности полипептида и оценки свойств модифицированного полипептида. Другими словами, способность варианта может увеличиваться, оставаться неизменной или снижаться по сравнению со способностью полипептида перед изменением. Кроме того, некоторые варианты могут включать варианты, в которых одна или более чем одна часть, такая как N-концевая лидерная последовательность или трансмембранный домен, были удалены. Другие варианты могут включать варианты, в которых из зрелого белка была удалена часть N-и/или С-конца. Термин «вариант» можно использовать взаимозаменяемо с такими терминами, как модификация, модифицированный полипептид, модифицированный белок, мутант, мутеин и дивергент, и не ограничивается ими при условии, что он представляет собой термин, используемый со значением изменения.
Кроме того, вариант может включать делеции или присоединения аминокислот, которые имеют минимальное влияние на свойства и вторичную структуру полипептида. Например, с N-концом варианта может быть конъюгирована сигнальная (или лидерная) последовательность, которая котрансляционно или посттрансляционно участвует в транслокации белка. Вариант может быть конъюгирован с другими последовательностями или линкерами таким образом, чтобы быть идентифицированным, очищенным или с интезиро ванным.
Вариант LysE, предложенный в настоящем раскрытии, может представлять собой полипептид, включающий аминокислотную последовательность, в которой любая одна или более чем одна аминокислота из аминокислот, соответствующих положениям 50, 153 и 215 в SEQ ID NO: 1, заменена другой аминокислотой.
Например, вариант LysE по настоящему раскрытию может представлять собой вариант, включающий замену другой аминокислотой любой одной или более чем одной аминокислоты из аминокислот, соответствующих положениям 50, 153 и 215 от N-конца в SEQ ID NO: 1 в любом полипептиде или белке, имеющем активность экспорта лизина.
Термин «другая аминокислота» не ограничивается, при условии, что она представляет собой отличную аминокислоту от аминокислоты перед заменой. Тем временем, в настоящем раскрытии при выражении «конкретная аминокислота заменена» очевидно то, что данная аминокислота заменена аминокислотой, которая отличается от данной аминокислоты перед заменой, если в противном случае не описывается, что она заменена другой аминокислотой.
В одном воплощении относительно варианта по настоящему раскрытию в аминокислотной последовательности SEQ ID NO: 1, которая представляет собой эталонный белок, аминокислота, соответствующая положению 50, может представлять собой аминокислоту, отличную от фенилаланина, и/или аминокислота, соответствующая положению 153, может представлять собой аминокислоту, отличную от аспарагина, и/или аминокислота, соответствующая положению 215, может представлять собой аминокислоту, отличную от аспарагина.
В любом воплощении вышеописанных воплощений в отношении варианта аминокислота, соответствующая положению 50 в SEQ ID NO: 1, может быть выбрана из глицина, аланина, валина, лейцина, изолейцина, метионина, триптофана, пролина, серина, треонина, цистеина, тирозина, аспарагина, глутамина, аспарагиновой кислоты, глутаминовой кислоты, лизина, гистидина и аргинина. Например, аминокислота, соответствующая положению 50 в SEQ ID NO: 1 варианта, может представлять собой неполярную аминокислоту, выбранную из глицина, аланина, валина, лейцина, изолейцина, метионина, триптофана и пролина. Например, аминокислота, соответствующая положению 50 в SEQ ID NO: 1 варианта, может представлять собой разветвленную аминокислоту, выбранную из валина, лейцина и изолейцина, но не ограничиваясь ими.
В любом воплощении вышеописанных воплощений в отношении варианта аминокислота, соответствующая положению 153 в SEQ ID NO: 1, может быть выбрана из глицина, аланина, валина, лейцина, изолейцина, метионина, фенилаланина, триптофана, пролина, серина, треонина, цистеина, тирозина, глутамина, аспарагиновой кислоты, глутаминовой кислоты, лизина, гистидина и аргинина. Например, аминокислота, соответствующая положению 153 в SEQ ID NO: 1 варианта, может представлять собой полярную аминокислоту, выбранную из серина, треонина, цистеина, тирозина и глутамина. Например, аминокислота, соответствующая положению 153 в SEQ ID NO: 1 варианта, может представлять собой серии, треонин или глутамин, но не ограничиваясь ими.
В любом воплощении вышеописанных воплощений в отношении варианта аминокислота, соответствующая положению 215 в SEQ ID NO: 1, может быть выбрана из глицина, аланина, валина, лейцина, изолейцина, метионина, фенилаланина, триптофана, пролина, серина, треонина, цистеина, тирозина, глутамина, аспарагиновой кислоты, глутаминовой кислоты, лизина, гистидина и аргинина. Например, аминокислота, соответствующая положению 215 в SEQ ID NO: 1 варианта, может представлять собой полярную аминокислоту, выбранную из серина, треонина, цистеина, тирозина и глутамина. Например, аминокислота, соответствующая положению 215 в SEQ ID NO: 1 варианта, может представлять собой серии, треонин или глутамин, но не ограничиваясь ими.
В любом воплощении вышеописанных воплощений вариант по настоящему раскрытию может иметь замену любой одной или более чем одной аминокислоты аминокислотных остатков в положениях 50, 153 и 215 в аминокислотной последовательности SEQ ID NO: 1 другой аминокислотой. В любом воплощении вышеописанных воплощений вариант по настоящему раскрытию может иметь замену любой аминокислоты аминокислотных остатков в положениях 50, 153 и 215 в аминокислотной последовательности SEQ ID NO: 1 другой аминокислотой. Аминокислоты после замены аминокислотного остатка в положении 50, 153 и 215 в аминокислотной последовательности SEQ ID NO: 1 могут быть такими же, как и аминокислота после замены аминокислотного остатка, соответствующего положению 50, 153 или 215 в аминокислотной последовательности SEQ ID NO: 1.
В любом воплощении вышеописанных воплощений вариант по настоящему раскрытию может иметь замену любой одной или более чем одной аминокислоты аминокислотных остатков в положениях 50, 153 и 215 в аминокислотной последовательности SEQ ID NO: 1 другой аминокислотой и может иметь по меньшей мере 60%-ную, 65%-ную, 66%-ную, 67%-ную, 68%-ную, 69%-ную, 70%-ную, 71%-ную, 72%-ную, 73%-ную, 74%-ную, 75%-ную, 76%-ную, 77%-ную, 78%-ную, 79%-ную, 80%-ную, 81%-ную, 82%-ную, 83%-ную, 84%-ную, 85%-ную, 86%-ную, 87%-ную, 88%-ную, 89%-ную, 90%-ную, 91%-ную, 92%-ную, 93%-ную, 94%-ную, 95%-ную, 96%-ную, 97%-ную, 98%-ную или более, или менее, чем 100%-ную идентичность с SEQ ID NO: 1.
В любом воплощении вышеописанных воплощений вариант по настоящему раскрытию может состоять из аминокислотной последовательности, выбранной из SEQ ID NO: 27-35, или может иметь или включать аминокислотную последовательность, имеющую по меньшей мере 60%-ную, 65%-ную, 66%-ную, 67%-ную, 68%-ную, 69%-ную, 70%-ную, 71%-ную, 72%-ную, 73%-ную, 74%-ную, 75%-ную, 76%-ную, 77%-ную, 78%-ную, 79%-ную, 80%-ную, 81%-ную, 82%-ную, 83%-ную, 84%-ную, 85%-ную, 86%-ную, 87%-ную, 88%-ную, 89%-ную, 90%-ную, 91%-ную, 92%-ную, 93%-ную, 94%-ную, 95%-ную, 96%-ную, 97%-ную, 98%-ную, 99%-ную, 99,1%-ную, 99,5%-ную, 99,8%-ную или более, или 100%-ную гомологию или идентичность с ними, или может по существу состоять из данной аминокислотной последовательности, но не ограничивается ей.
Также очевидно то, что вариант, имеющий аминокислотную последовательность, имеющую делецию, модификацию, замену, консервативную замену или присоединение некоторой последовательности, также попадает в пределы объема настоящего раскрытия, при условии, что данная аминокислотная последовательность имеет такую гомологию или идентичность и демонстрирует эффективность, соответствующую эффективности варианта по настоящему раскрытию.
Их примеры включают варианты, имеющие присоединение или делецию последовательности, которые не изменяют функцию варианта по настоящему раскрытию, на N-конце, С-конце аминокислотной последовательности и/или внутри аминокислотной последовательности, встречающуюся в природе мутацию, молчащую мутацию или консервативную замену.
Термин «консервативная замена» означает замену одной аминокислоты другой аминокислотой, имеющей аналогичные структурные и/или химические свойства. Такая замена аминокислоты обычно может происходить на основе сходства в полярности, заряде, растворимости, гидрофобности, гидрофильности и/или амфипатической природы остатков. Обычно консервативная замена может мало влиять или не влияет на активность белков или полипептидов.
Термин «соответствующий» в том виде, как он здесь используется, относится к аминокислотным остаткам в положениях, перечисленных в полипептиде, или к аминокислотным остаткам, которые являются аналогичными, идентичными или гомологичными остаткам, перечисленным в полипептиде. Идентификация аминокислоты в соответствующем положении может определять конкретную аминокислоту в последовательности, которая относится к конкретной последовательности. Термин «соответствующая область» в том виде, как он здесь используется, обычно относится к аналогичному или соответствующему положению в родственном белке или эталонном белке.
Например, произвольную аминокислотную последовательность выравнивают с SEQ ID NO: 1 и, на основе этого, каждый аминокислотный остаток данной аминокислотной последовательности может быть пронумерован со ссылкой на числовое положение аминокислотного остатка, соответствующего аминокислотному остатку SEQ ID NO: 1. Например, алгоритм выравнивания последовательности, как описано в настоящем раскрытии, может определять положение аминокислоты или положение, в котором случается модификация, такая как замена, вставка или делеция, посредством сравнения с аминокислотой в запрашиваемой последовательности (также именуемой «эталонная последовательность»).
Для таких выравниваний, например, можно использовать алгоритм Needleman-Wunsch (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443-453), программу Needleman пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000), Trends Genet. 16: 276-277) и т.д., но не ограничиваясь ими, и можно подходящим образом использовать программу выравнивания последовательности, алгоритм попарного сравнения последовательностей и т.д., которые известны в данной области.
Согласно другому аспекту настоящего раскрытия предложен полинуклеотид, кодирующий вариант по настоящему раскрытию.
Термин «полинуклеотид» в том виде, как он здесь используется, представляет собой нить ДНК или РНК, имеющую определенную или большую длину, в виде полимера нуклеотидов, в котором мономеры нуклеотидов соединяются в длинную цепь ковалентными связями.
В полинуклеотиде по настоящему раскрытию можно делать разные модификации в кодирующей области при условии, что не изменяется аминокислотная последовательность, учитывая вырожденность кодонов или предпочтительные кодоны в организмах, которые предназначены для экспрессии белка.
В любом воплощении вышеописанных воплощений может быть доступен полинуклеотид по настоящему раскрытию из последовательности lysE в GenBank NCBI и т.д., которая представляет собой известную базу данных.
В частности, полинуклеотид, кодирующий вариант белка LysE по настоящему раскрытию, может иметь или включать нуклеотидную последовательность, имеющую 70%-ную или более, 75%-ную или более, 80%-ную или более, 85%-ную или более, 90%-ную или более, 95%-ную или более, 96%-ную или более, 97%-ную или большую, 98%-ную или более и менее, чем 100%-ную гомологию или идентичность с последовательностью SEQ ID NO: 2, или может состоять или по существу состоит из нуклеотидной последовательности, имеющей 70%-ную или более, 75%-ную или более, 80%-ную или более, 85%-ную или более, 90%-ную или более, 95%-ную или более, 96%-ную или более, 97%-ную или более, 98%-ную или более и меньше, чем 100%-ную гомологию или идентичность с последовательностью SEQ ID NO: 2, но не ограничивается ей. В данном отношении в последовательности, имеющей гомологию или идентичность, кодон, кодирующий аминокислоту, соответствующую положению 50 в SEQ ID NO: 1, может представлять собой один из кодонов, кодирующих аминокислоты, отличные от фенилаланина, и/или кодон, кодирующий аминокислоту, соответствующую положению 153, может представлять собой один из кодонов, кодирующих аминокислоты, отличные от аспарагина, и/или кодон, кодирующий аминокислоту, соответствующую положению 215, может представлять собой один из кодонов (например, SEQ ID NO: 36-38), кодирующих аминокислоты, отличные от аспарагина, но не ограничиваясь ими.
Кроме того, полинуклеотид по настоящему раскрытию может включать зонд, который можно получать из последовательности известного гена, например, последовательности без ограничения при условии, что она представляет собой последовательность, которая способна к гибридизации с комплементарной последовательностью, со всей или с частью полинуклеотидной последовательности по настоящему раскрытию при жестких условиях. Термин «жесткие условия» означает условия, которые обеспечивают специфичную гибридизацию между полинуклеотидами. Данные условия конкретно описываются в документах (см. J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York, 9.50-9.51, 11.7-11.8). Их примеры включают условия, при которых по линуклеотиды, имеющие более высокую гомологию или идентичность, т.е. по линуклеотиды, имеющие 70%-ную или более, 75%-ную или более, 80%-ную или более, 85%-ную или более, 90%-ную или более, 95%-ную или более, 96%-ную или более, 97%-ную или более, 98%-ную или более или 99%-ную или более гомологию или идентичность гибридизуются друг с другом, тогда как полинуклеотиды, имеющие меньшую гомологию или идентичность не гибридизуются друг с другом, или условия, при которых промывка осуществляется один раз, конкретно от двух до трех раз при концентрации соли и температуре, эквивалентным 60°С, lx SSC (раствор хлорида и цитрата натрия), 0,1% SDS (додецилсульфат натрия), конкретно при 60°С, 0,1х SSC, 0,1% SDS, более конкретно при 68°С, 0,lx SSC, 0,1% SDS, которые представляют собой условия промывки для обычной саузерн-гибридизации.
Для гибридизации требуется, чтобы две нуклеиновые кислоты имели комплементарные последовательности, хотя несоответствия между основаниями и допускаются, в зависимости от жесткости гибридизации. Термин «комплементарный» используется для описания связи между нуклеотидными основаниями, способными гибридизоваться друг с другом. Например, в отношении ДНК, аденин является комплементарным тимину, а цитозин - комплементарным гуанину. Следовательно, полинуклеотид по настоящему раскрытию также может включать по существу аналогичные последовательности нуклеиновой кислоты, а также выделенные фрагменты нуклеиновой кислоты, которые являются комплементарными всей последовательности.
В частности, полинуклеотид, имеющий гомологию или идентичность с полинуклеотидом по настоящему раскрытию, может быть выявлен с использованием условий гибридизации, включающих стадию гибридизации при значении Tm 55°С и вышеописанных условиях. Кроме того, значение Tm может составлять 60°С, 63°С или 65°С, но не ограничиваясь ими, и может подходящим образом корректироваться специалистами в данной области согласно цели.
Подходящая жесткость для гибридизации полинуклеотида зависит от длины и степени комплементарности полинуклеотида, и данные переменные хорошо известны в данной области (например, J. Sambrook et al., выше).
Согласно еще одному другому аспекту настоящего раскрытия предложен вектор, включающий полинуклеотид, кодирующий вариант LysE. Данный вариант LysE и кодирующий его полинуклеотид являются такими же, как описано выше.
В одном воплощении данный вектор может представлять собой экспрессионный вектор для осуществления экспрессии полинуклеотида в клетке-хозяине, но не ограничивается им.
«Вектор» по настоящему раскрытию может включать конструкцию ДНК, включающую последовательность полинуклеотида, кодирующую интересующий полипептид, который связан функциональным образом с подходящей областью регуляции экспрессии (или последовательностью контроля экспрессии) таким образом, что интересующий полипептид может экспрессироваться в подходящем хозяине. Область регуляции экспрессии может включать промотор, способный инициировать транскрипцию, любую последовательность оператора для осуществления контроля транскрипции, последовательность, кодирующую подходящий сайт связывания мРНК с рибосомой, и последовательность, контролирующую терминацию транскрипции и трансляции. Данный вектор можно трансформировать в подходящую клетку-хозяина, и затем он может реплицироваться или функционировать независимо от генома хозяина, или может сам интегрироваться в геном.
Вектор, используемый в настоящем раскрытии, конкретно не ограничивается, но можно использовать любой вектор, известный в данной области. Примеры обычно используемых векторов включают природные или рекомбинантные плазмиды, космиды, вирусы и бактериофаги. Например, в качестве фагового вектора или космидного вектора можно использовать pWE15, М13, MBL3, MBL4, IXII, ASHII, АРП, t10, t11, Charon4A, Charon21A и т.д., а в качестве плазмидого вектора можно использовать систему pDZ, систему pBR, систему pUC, систему pBluescript II, систему pGEM, систему pTZ, систему pCL, систему рЕТ и т.д. В частности, можно использовать вектор pDZ, pDC, pDCM2, pACYC177, pACYC184, pCL, pECCG117, pUC19, pBR322, pMW118, pCClBAC и т.д.
Например, полинуклеотид, кодирующий интересующий полипептид, может быть вставлен в хромосому посредством вектора для внутриклеточной вставки в хромосому. Вставку полинуклеотида в хромосому можно осуществлять любым способом, известным в данной области, например, гомологичной рекомбинацией, но не ограничиваясь ей. Данный вектор может дополнительно включать селективный маркер для подтверждения вставки в хромосому. Селективный маркер служит для отбора клеток, трансформированных векторами, т.е. для подтверждения вставки интересующей молекулы нуклеиновой кислоты, и можно использовать маркеры, которые придают селектируемые фенотипы, такие как устойчивость к лекарственному средству, ауксотрофия, устойчивость к цитотоксическим средствам или экспрессия поверхностных полипептидов. В среде, обработанной селективным агентом, выживают или демонстрируют другие фенотипические признаки только клетки, экспрессирующие данный селективный маркер, и, таким образом, можно отбирать трансформированные клетки.
Термин «трансформация» в том виде, как он здесь используется, означает то, что вектор, включающий полинуклеотид, кодирующий целевой полипептид, вводится в клетку-хозяина или в микроорганизм таким образом, что полипептид, кодируемый данным полинуклеотидом, может экспрессироваться в данной клетке-хозяине. Трансформированный полинуклеотид может локализоваться, независимо от положения, либо вставленным в хромосому клетки-хозяина, либо локализованным вне хромосомы, при условии, что он может экспрессироваться в данной клетке-хозяине. Данный полинуклеотид включает ДНК и/или РНК, кодирующую интересующий полипептид. Данный полинуклеотид может быть введен в любой форме, при условии, что он может быть введен в клетку-хозяина и может экспрессироваться. Например, данный полинуклеотид может быть введен в клетку-хозяина в форме экспрессионной кассеты, которая представляет собой генетическую конструкцию, содержащую все элементы, требующиеся для автономной экспрессии. Данная экспрессионная кассета обычно может включать промотор, связанный функциональным образом с полинуклеотидом, сигнал терминации транскрипции, сайт связывания рибосомы и сигнал терминации трансляции. Экспрессионная кассета может находиться в форме экспрессионного вектора, способного к автономной репликации. Кроме того, данный полинуклеотид может вводиться в клетку-хозяина в его собственной форме и может функциональным образом связываться с последовательностью, требующейся для экспрессии в клетке-хозяине, но не ограничиваясь ей.
Кроме того, термин «связанный функциональным образом» означает то, что последовательность полинуклеотида функционально связана с последовательностью промотора, которая инициирует и опосредует транскрипцию полинуклеотида, кодирующего вариант по настоящему раскрытию.
Согласно еще одному другому аспекту настоящего раскрытия предложен микроорганизм, включающий вариант LysE или полинуклеотид, кодирующий данный вариант.Вариант LysE и кодирующий его полинуклеотид, являются такими, как описано выше.
Микроорганизм по настоящему раскрытию может включать любой один или более чем один LysE варианта по настоящему раскрытию, кодирующий его полинуклеотид и вектор, включающий данный полинуклеотид.
Термин «микроорганизм (или штамм)» в том виде, как он здесь используется, включает все микроорганизмы дикого типа или естественно или искусственно генетически модифицированные микроорганизмы, и он может представлять собой микроорганизм, в котором ослаблен или усилен специфический механизм из-за вставки чужеродного гена или усиления активности, или инактивации эндогенного гена, и он может представлять собой микроорганизм, включающий генетическую модификацию, для продукции интересующего полипептида, белка или продукта.
Микроорганизм по настоящему раскрытию может представлять собой микроорганизм, включающий любой один или более чем один вариант по настоящему раскрытию, полинуклеотид, кодирующий вариант по настоящему раскрытию, и вектор, включающий полинуклеотид, кодирующий вариант по настоящему раскрытию; микроорганизм, модифицированный для экспрессии варианта по настоящему раскрытию, или полинуклеотида, кодирующего вариант по настоящему раскрытию; микроорганизм (например, рекомбинантный штамм), экспрессирующий вариант по настоящему раскрытию, или полинуклеотид, кодирующий вариант по настоящему раскрытию; или микроорганизм (например, рекомбинантный штамм), имеющий активность варианта по настоящему раскрытию, но не ограничивается ими.
Микроорганизм по настоящему раскрытию может иметь способность к осуществлению экспорта и/или продукции L-аргинина.
Микроорганизм по настоящему раскрытию может представлять собой микроорганизм, экспрессирующий в природе белок LysE или имеющий способность к осуществлению экспорта и/или продукции аргинина, или микроорганизм, в котором вариант по настоящему раскрытию или полинуклеотид, кодирующий данный вариант (или вектор, включающий данный полинуклеотид), вводится в родительский штамм, который не имеет белка LysE или способности к осуществлению экспорта и/или продукции аргинина, и/или в котором родительскому штамму придается способность к осуществлению экспорта и/или продукции аргинина, но не ограничивается ими. Например, микроорганизм по настоящему раскрытию может представлять собой микроорганизм, в который вводится вариант LysE по настоящему раскрытию, и, таким образом, увеличивается способность к экспорту и/или продукции L-аргинина, но не ограничивается им.
Например, штамм по настоящему раскрытию представляет собой клетку или микроорганизм, который трансформирован полинуклеотидом по настоящему раскрытию или вектором, включающим полинуклеотид, кодирующий вариант по настоящему раскрытию, для экспрессии варианта по настоящему раскрытию. Относительно объектов настоящего раскрытия, штамм по настоящему раскрытию может включать любой микроорганизм, который включает вариант по настоящему раскрытию и способен к экспорту или продукции L-аргинина. Например, штамм по настоящему раскрытию может представлять собой рекомбинантный штамм, в котором полинуклеотид, кодирующий вариант по настоящему раскрытию, вводится в природный микроорганизм дикого типа или в микроорганизм, продуцирующий L-аргинин, для экспрессии варианта LysE и для наличия повышенной способности к осуществлению экспорта и/или продукции L-аргинина. Рекомбинантный штамм, имеющий повышенную способность к осуществлению экспорта и/или продукции L-аргинина, может представлять собой микроорганизм, имеющий повышенную способность к экспорту и/или продукции L-аргинина по сравнению с природным микроорганизмом дикого типа или микроорганизмом, в котором LysE не является модифицированным (т.е. микроорганизмом, экспрессирующим LysE дикого типа (SEQ ID NO: 1), или микроорганизмом, который не экспрессирует модифицированный белок LysE), но не ограничивается ими.
Термин «немодифицированный микроорганизм» в том виде, как он здесь используется, не исключает штаммы, включающие мутацию, которая может встречаться в микроорганизмах в природе, и может представлять собой сам штамм дикого типа или природный штамм перед изменением признака посредством генетического изменения из-за природных или искусственных факторов. Например, немодифицированный микроорганизм может представлять собой штамм, в котором вариант LysE, описанный в настоящем описании изобретения, не вводится или еще не был введен. Термин «немодифицированный микроорганизм» можно использовать взаимозаменяемо с терминами «штамм перед модификацией», «микроорганизм перед модификацией», «неизмененный штамм», «немодифицированный штамм», «неизмененный микроорганизм» или «эталонный микроорганизм».
В одном воплощении микроорганизм по настоящему раскрытию может представлять собой микроорганизм рода Corynebacterium. Например, микроорганизм по настоящему раскрытию может представлять собой Corynebacterium glutamicum, Corynebacterium stationis, Corynebacterium crudilactis, Corynebacterium deserti, Corynebacterium efficiens, Corynebacterium callunae, Corynebacterium singulare, Corynebacterium halotoleram, Corynebacterium striatum, Corynebacterium ammoniagenes, Corynebacterium pollutisoli, Corynebacterium imitans, Corynebacterium testudinoris или Corynebacterium flavescens, в частности, Corynebacterium glutamicum, но не ограничиваясь ими.
Микроорганизм, экспортирующий и/или продуцирующий L-аргинин по настоящему раскрытию, может отличаться тем, что он включает вариант LysE, раскрытый в данном описании изобретения, имея, посредством этого, усиленную способность к осуществлению продукции и/или экспорта L-аргинина, как это желательно.
В любом воплощении вышеописанных воплощений микроорганизм, продуцирующий и/или экспортирующий L-аргинин, или микроорганизм, имеющий способность к осуществлению продукции и/или экспорта L-аргинина, в настоящем раскрытии может представлять собой микроорганизм, в котором некоторые из генов, участвующих в пути биосинтеза L-аргинина, усиливаются или ослабляются, или некоторые из генов, участвующих в пути деградации L-аргинина, усиливаются или ослабляются.
В любом воплощении вышеописанных воплощений данный микроорганизм может представлять собой микроорганизм, в котором некоторые из генов, участвующих в пути биосинтеза L-аргинина, усиливаются или ослабляются. Примеры ферментов биосинтеза L-аргинина включают N-ацетилглутамилфосфатредуктазу (argC), орнитинацетилтрансферазу (argJ), N-ацетилглутаматкиназу (argB), ацетилорнитинтрансаминазу (argD), орнитинкарбамоилтрансферазу (argF), синтазу аргининоянтарной кислоты (argG), лиазу аргининоянтарной кислоты (argH) и карбамоилфосфатсинтазу (carAB). Биосинтетические ферменты аргинина, присутствующие на опероне Arg (argCJBDFRGH), могут регулироваться репрессором аргинина, кодируемым argR (J Bacteriol. 2002Dec;184(23):6602-14.). Следовательно, способность к продуцированию L-аргинина может быть предоставлена или усилена делецией или уменьшением активности репрессора аргинина (argR) (US2002-0045223) и/или осуществлением экспрессии одного или более чем одного гена из генов, связанных с биосинтезом. Например, репрессор аргинина может представлять собой полипептид, представленный аминокислотной последовательностью SEQ ID NO: 39, но не ограничивающийся им.
Термин «уменьшение» активности полипептида в том виде, как он здесь используется, имеет идею охватывания всего из уменьшения активности или отсутствия активности по сравнению с эндогенной активностью. Термин «уменьшение» можно использовать взаимозаменяемо с инактивацией, недостаточностью, понижающей регуляцией, уменьшением, снижением, ослаблением или тому подобным.
Уменьшение также может включать случай, где активность самого полипептида снижается или устраняется по сравнению с активностью полипептида, которой обладает исходный микроорганизм, из-за мутации и т.д. полинуклеотида, кодирующего данный полипептид, случай, где общая активность и/или концентрация (уровень экспрессии) полипептидов в клетке ниже по сравнению с природным штаммом из-за ингибирования экспрессии гена полинуклеотида, кодирующего полипептид, или ингибирования трансляции в полипептид, случай, где экспрессия полинуклеотида не осуществляется и/или случай, где полипептид не имеет активности, несмотря на экспрессию полинуклеотида. Термин «эндогенная активность» относится к активности конкретного полипептида, которой исходно обладал родительский штамм перед модификацией или микроорганизм дикого типа или немодифицированный микроорганизм при трансформации генетической мутацией, вызванной природными или искусственными факторами. Данный термин можно использовать взаимозаменяемо с термином «активность перед модификацией». «Инактивация, недостаточность, уменьшение, понижающая регуляция, снижение или ослабление» активности полипептида по сравнению с его эндогенной активностью означают то, что активность данного полипептида снижается по сравнению с активностью конкретного полипептида, которой исходно обладал родительский штамм перед трансформацией или немодифицированный микроорганизм.
Уменьшение активности полипептида может осуществляться любым способом, известным в данной области, но не ограничиваясь им, и может достигаться применением разных способов, хорошо известных в данной области (например, Nakashima N et al., Bacterial cellular engineering by genome editing and gene silencing. Int J Mol Sci. 2014;15(2):2773-2793, Sambrook et al. Molecular Cloning 2012 и т.д.).
В частности, уменьшение активности полипептида по настоящему раскрытию может достигаться посредством следующего:
1) делеция всего или части гена, кодирующего полипептид;
2) модификация области контроля экспрессии (или последовательности контроля экспрессии) для уменьшения экспрессии гена, кодирующего полипептид;
3) модификация аминокислотной последовательности, составляющей полипептид, для устранения или уменьшения активности полипептида (например, делеция/замена/присоединение одной или более чем одной аминокислоты в аминокислотной последовательности);
4) модификация последовательности гена, кодирующего полипептид, для устранения или ослабления активности данного полипептида (например, делеция/замена/присоединение одного или более чем одного нуклеотида в нуклеотидной последовательности гена полипептида для кодирования полипептида, который модифицирован для устранения или уменьшения активности данного полипептида);
5) модификация нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR область (5'-нетранслируемая область) транскрипта гена, кодирующего данный полипептид;
6) введение антисмыслового олигонуклеотида (например, антисмысловой РНК), комплементарно связывающегося с транскриптом гена, кодирующего полипептид;
7) добавление последовательности, комплементарной последовательности Шайна-Дальгарно гена, кодирующего данный полипептид, выше последовательности Шайна-Дальгарно для того, чтобы образовать вторичную структуру, которая делает невозможным присоединение рибосом;
8) добавление промотора, который подлежит транскрипции в противоположном направлении относительно З'-конца открытой рамки считывания (ORF) последовательности гена, кодирующего полипептид (инженерия транскрипции в противоположном направлении, RTE); или
9) комбинация двух или более чем двух, выбранных из (1)-(8), но, в частности, не ограничивается ими.
Например:
1) Делецией части или всего гена, кодирующего полипептид, может быть устранение всего полинуклеотида, кодирующего эндогенный целевой белок в хромосоме, замена полинуклеотидом, имеющим делецию некоторых нуклеотидов, или замена маркерным геном.
Кроме того, 2) модификацией области контроля экспрессии (или последовательности контроля экспрессии) может быть мутация в области контроля экспрессии (или последовательности контроля экспрессии) посредством делеции, вставки, неконсервативной или консервативной замены, или их комбинации, или замена последовательностью, имеющей меньшую активность. Область контроля экспрессии включает промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, и последовательность, контролирующую терминацию транскрипции и трансляции, но не ограничивается ими.
Кроме того, 3) и 4) модификацией аминокислотной последовательности или последовательности полинуклеотида может быть мутация в данной последовательности посредством делеции, вставки, неконсервативной или консервативной замены или их комбинации в аминокислотной последовательности полипептида или последовательности полинуклеотида, кодирующей данный полипептид, или замена аминокислотной последовательностью или последовательностью полинуклеотида, модифицированной так, чтобы иметь более слабую активность, или аминокислотной последовательностью, или последовательностью полинуклеотида, улучшенной для того, чтобы не иметь активности, таким образом, чтобы уменьшать активность данного полипептида, но не ограничиваясь ими. Например, экспрессию гена можно ингибировать или ослаблять посредством введения мутации в последовательность полинуклеотида и образования терминирующего кодона, но не ограничиваясь ей.
Кроме того, 5) модификацией нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR область транскрипта гена, кодирующего полипептид, может быть, например, замена нуклеотидной последовательностью, кодирующей скорее не эндогенный инициирующий кодон, а другой инициирующий кодон, имеющий меньшую скорость экспрессии полипептида, но не ограничивается ей.
6) Для введения антисмыслового олигонуклеотида (например, антисмысловой РНК), комплементарно связывающегося с транскриптом гена, кодирующего полипептид, можно сделать ссылку, например, на литературу [Weintraub, Н. et al., Antisense-RNA as a molecular tool for genetic analysis, Reviews-Trends in Genetics, Vol. 1(1) 1986].
7) Добавление последовательности, комплементарной последовательности Шайна-Дальгарно гена, кодирующего полипептид, выше последовательности Шайна-Дальгарно таким образом, чтобы образовать вторичную структуру, которая делает невозможным присоединение рибосом, что может делать невозможной трансляцию мРНК или уменьшать ее скорость.
8) Добавление промотора, который подлежит транскрипции в противоположном направлении относительно 3'-конца открытой рамки считывания (ORF) последовательности гена, кодирующего полипептид (инженерия транскрипции в противоположном направлении, RTE), может создавать антисмысловой нуклеотид, комплементарный транскрипту гена, кодирующего полипептид, с уменьшением активности данного полипептида.
Термин «усиление» активности полипептида в том виде, как он здесь используется, означает то, что активность полипептида увеличивается по сравнению с эндогенной активностью. Термин «усиление» можно использовать взаимозаменяемо с такими терминами, как активация, повышающая регуляция, сверхэкспрессия и увеличение, и т.д. Здесь активация, усиление, повышающая регуляция, сверхэкспрессия и увеличение могут включать как демонстрирование активности, которой исходно не обладали, так и демонстрирование улучшенной активности по сравнению с эндогенной активностью или активностью перед модификацией. Термин «эндогенная активность» означает активность конкретного полипептида, которой исходно обладал родительский штамм перед изменением признака или немодифицированный микроорганизм, при изменении признака посредством генетического изменения из-за природных или искусственных факторов. Данный термин можно использовать взаимозаменяемо с термином «активность перед модификацией». Тот факт, что активность полипептида «усиливается», «подвергается повышающей регуляции», «сверхэкспрессируется» или «увеличивается» по сравнению с эндогенной активностью означает то, что активность полипептида улучшается по сравнению с активностью и/или концентрацией (уровнем экспрессии) конкретного полипептида, которым исходно обладал родительский штамм перед изменением признака или немодифицированный микроорганизм.
Усиление может достигаться посредством введения чужеродного полипептида или усиления эндогенной активности и/или концентрации (уровня экспрессии) данного полипептида. Усиление активности полипептида может быть подтверждено увеличением уровня активности и уровня экспрессии соответствующего полипептида или количества продукта, продуцированного из соответствующего полипептида.
Для усиления активности полипептида можно применять разные способы, хорошо известные в данной области, и данный способ не ограничивается, при условии, что активность интересующего полипептида может усиливаться по сравнению с активностью микроорганизма перед модификацией. В частности, можно использовать генную инженерию и/или белковую инженерию, хорошо известные специалистам в данной области, которые представляют собой традиционные способы молекулярной биологии, но данный способ не ограничивается ими (например, Sitnicka et al. Functional Analysis of Genes. Advances in Cell Biology. 2010, Vol. 2. 1-16, Sambrook et al. Molecular Cloning 2012 и т.д.).
В частности, усиление активности полипептида по настоящему раскрытию может представлять собой:
1) увеличение числа внутриклеточных копий полинуклеотида, кодирующего полипептид;
2) замену регуляторной области экспрессии гена на хромосоме, кодирующей полипептид, последовательностью, демонстрирующей сильную активность;
3) модификацию нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR область транскрипта гена, кодирующего полипептид;
4) модификацию аминокислотной последовательности полипептида для увеличения активности данного полипептида;
5) модификацию последовательности полинуклеотида, кодирующего полипептид, для усиления активности данного полипептида (например, модификацию последовательности полинуклеотида гена полипептида для кодирования полипептида, который был модифицирован для усиления активности данного полипептида);
6) введение чужеродного полипептида, демонстрирующего активность данного полипептида, или чужеродного полинуклеотида, кодирующего данный полипептид;
7) оптимизацию кодонов полинуклеотида, кодирующего полипептид;
8) анализ третичной структуры полипептида для отбора и модификации или химической модификации экспонированного сайта; или
9) комбинацию двух или более чем двух, выбранных из (1)-(8), но конкретно не ограничиваясь ими.
Более конкретно:
1) Увеличение числа внутриклеточных копий полинуклеотида, кодирующего полипептид, может быть достигнуто посредством введения в клетку-хозяина вектора, который может реплицироваться и функционировать независимо от хозяина, и с которым полинуклеотид, кодирующий соответствующий полипептид, связан функциональным образом. В качестве альтернативы, увеличение может быть достигнуто введением одной копии или двух или более чем двух копий полинуклеотида, кодирующего соответствующий полипептид, в хромосому клетки-хозяина. Введение в хромосому можно осуществлять введением вектора, способного вставлять полинуклеотид в хромосому клетки-хозяина, в клетку-хозяина, но не ограничивается им. Данный вектор является таким, как описано выше.
2) Замена регуляторной области экспрессии гена (или последовательности контроля экспрессии) на хромосоме, кодирующей полипептид, последовательностью, демонстрирующей сильную активность, может представлять собой, например, появление изменения в последовательности из-за делеции, вставки, неконсервативной или консервативной замены, или их комбинации, или замену последовательностью, демонстрирующей более сильную активность, таким образом, что активность регуляторной области экспрессии дополнительно усиливается. Регуляторная область экспрессии может включать промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, последовательность, контролирующую терминацию транскрипции и трансляции, и т.д., но конкретно не ограничивается ими. Например, замена может представлять собой замену исходного промотора сильным промотором, но не ограничивается ей.
Примеры известных сильных промоторов включают промоторы CJ1-CJ7 (патент США №US 7662943 В2), промотор lac, промотор trp, промотор trc, промотор tac, промотор PR фага лямбда, промотор PL, промотор tet, промотор gapA, промотор SPL7, промотор SPL13 (sm3) (патент США №US 10584338 В2), промотор O2 (патент США №US 10273491 В2), промотор tkt, промотор уссА и т.д., но не ограничиваются ими.
3) Модификация нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR область транскрипта гена, кодирующуего полипептид, может представлять собой, например, замену последовательностью оснований, кодирующей другой инициирующий кодон, имеющий более высокую скорость экспрессии полипептида по сравнению с эндогенным инициирующим кодоном, но не ограничивается ей.
4) и 5) Модификация аминокислотной последовательности или последовательности полинуклеотида может представлять собой появление изменения в последовательности из-за делеции, вставки, неконсервативной или консервативной замены аминокислотной последовательности полипептида или последовательности полинуклеотида, кодирующей данный полипептид, или их комбинацию или замену аминокислотной последовательностью или последовательностью полинуклеотида, модифицированной для демонстрации более сильной активности, или аминокислотной последовательностью или последовательностью полинуклеотида, модифицированной для того, чтобы быть более активной, таким образом, что активность данного полипептида усиливается, но не ограничиваясь ими. Данную замену можно конкретно осуществлять посредством вставки полинуклеотида в хромосому гомологичной рекомбинацией, но не ограничиваясь ей. Используемый здесь вектор может дополнительно включать селективный маркер для подтверждения вставки в хромосому. Селективный маркер является таким, как описано выше.
6) Введение чужеродного полинуклеотида, демонстрирующего активность полипептида, может представлять собой введение в клетку-хозяина чужеродного полинуклеотида, кодирующего полипептид, демонстрирующий идентичную или аналогичную данному полипептиду активность. Нет ограничения по его происхождению или последовательности при условии, что данный чужеродный полинуклеотид демонстрирует идентичную или аналогичную данному полипептиду активность. Способ, используемый при введении, можно осуществлять посредством подходящего выбора способа трансформации, известного специалистам в данной области. Поскольку введенный полинуклеотид экспрессируется в клетке-хозяине, может продуцироваться полипептид и может увеличиваться его активность.
7) Оптимизация кодонов полинуклеотида, кодирующего полипептид, может представлять собой оптимизацию кодонов эндогенного полинуклеотида таким образом, чтобы увеличивать транскрипцию или трансляцию в клетке-хозяине, или оптимизацию кодонов чужеродного полинуклеотида таким образом, чтобы осуществлять оптимизированную транскрипцию и трансляцию в клетке-хозяине.
8) Анализ третичной структуры полипептида для выбора и модификации или химическая модификация экспонированного сайта могут осуществляться, например, для определения шаблонного белка-кандидата согласно степени сходства последовательности посредством сравнения информации по последовательности полипептида, подлежащего анализу, с хранилищем информации по последовательностям известных белков базы данных для подтверждения структуры на основе этого и для модификации или химической модификации экспонированного сайта, подлежащего модификации или химической модификации.
Такое усиление активности полипептида может представлять собой увеличение активности или концентрации, или уровня экспрессии соответствующего полипептида на основе активности или концентрации полипептида, экспрессируемого в микробном штамме дикого типа или в микробном штамме перед модификацией, или увеличение количества продукта, продуцируемого от соответствующего полипептида, но не ограничивается ими.
В микроорганизме по настоящему раскрытию частичная или полная модификация полинуклеотида может индуцироваться (а) гомологичной рекомбинацией с использованием вектора для вставки в хромосому в микроорганизме или редактированием генома с использованием генетически модифицированной нуклеазы (например, CRISPR-Cas9), и/или (б) обработкой светом, таким как ультрафиолетовые лучи и излучение, и/или химическими агентами, но не ограничиваясь ими. Способ осуществления модификации части или всего гена может включать способ с использованием технологии рекомбинантной ДНК. Например, посредством введения в микроорганизм нуклеотидной последовательности или вектора, содержащего нуклеотидную последовательность, гомологичную интересующему гену, для вызова гомологичной рекомбинации можно удалить часть гена или весь данный ген. Нуклеотидная последовательность или вектор, подлежащий введению может включать доминантный селективный маркер, но не ограничиваясь им.
Согласно еще одному другому аспекту настоящего раскрытия предложен способ продуцирования L-аргинина, включающий стадию культивирования микроорганизма, содержащего вариант LysE или кодирующий его полинуклеотид.
Вариант LysE, кодирующий его полинуклеотид, включающий его микроорганизм и L-аргинин являются такими, как описано выше.
Способ продуцирования L-аргинина по настоящему раскрытию может включать стадию культивирования в среде микроорганизма, содержащего описанный здесь вариант LysE или кодирующий его полинуклеотид, или вектор, включающий данный полинуклеотид.
Термин «культивирование» в том виде, как он здесь используется, означает выращивание микроорганизма по настоящему раскрытию при условиях среды, контролируемых подходящим образом. Способ культивирования по настоящему раскрытию можно осуществлять в соответствии с подходящей средой и условиями культивирования, известными в данной области. Такой способ культивирования может легко корректироваться и использоваться специалистами в данной области согласно выбранному штамму. В частности, культивирование может быть периодического типа, непрерывного типа и/или типа с подпиткой, но не ограничивается ими.
Термин «среда» в том виде, как он здесь используется, означает смешанное вещество, содержащее питательные вещества, требующиеся для культивирования микроорганизма по настоящему раскрытию в качестве главного компонента, и данная среда поставляет питательные вещества, факторы роста и т.д., включая воду, которые являются незаменимыми для выживания и развития. В частности, в качестве среды и других условий культивирования, используемых для культивирования микроорганизма по настоящему раскрытию, можно использовать любые без конкретного ограничения, при условии, что она представляет собой среду, используемую для обычного культивирования микроорганизмов. Микроорганизм по настоящему раскрытию можно культивировать в обычной среде, содержащей правильные источники углерода, источники азота, источники фосфора, неорганические соединения, аминокислоты и/или витамины, и т.д., при одновременном осуществлении контроля температуры, рН и т.д. при аэробных условиях.
Например, культуральную среду для штамма рода Corynebacterium можно найти в документе ["Manual of Methods for General Bacteriology" by the American Society for Bacteriology (Washington, D.C., USA, 1981)].
В настоящем раскрытии источники углерода включают углеводы, такие как глюкоза, сахароза, лактоза, фруктоза, сахароза, мальтоза и т.д.; сахароспирты, такие как маннит, сорбит и т.д.; органические кислоты, такие как пировиноградная кислота, молочная кислота, лимонная кислота и т.д.; аминокислоты, такие как глутаминовая кислота, метионин, лизин и т.д. Кроме того, можно использовать природные органические питательные вещества, такие как гидролизат крахмала, мелассу, сырую мелассу, рисовые отруби, маниок, остаток сахарного тростника и жидкий кукурузный экстракт. В частности, можно использовать углеводы, такие как глюкоза и стерилизованные пред обработанные мелассы (т.е. мелассы, превращенные в восстанавливающий сахар), и можно использовать подходящие количества других источников углерода разными способами без ограничения. Данные источники углерода можно использовать одиночно или в комбинации с двумя или более чем двумя, но не ограничиваясь ими.
В качестве источников азота можно использовать неорганические источники азота, такие как аммиак, сульфат аммония, хлорид аммония, ацетат аммония, фосфат аммония, карбонат аммония, нитрат аммония и т.д.; и органические источники азота, такие как аминокислоты, такие как глутаминовая кислота, метионин, глутамин и т.д., пептон, NZ-амин, мясной экстракт, дрожжевой экстракт, солодовый экстракт, жидкий кукурузный экстракт, гидролизат казеина, рыба или продукты ее разложения и обезжиренный соевый жмых или продукты его разложения. Данные источники азота можно использовать одиночно или в комбинации двух или более чем двух, но не ограничиваясь ими.
Источники фосфора могут включать монокалия фосфат, дикалия фосфат или соответствующие им натрийсодержащие соли. В качестве неорганических соединений можно использовать хлорид натрия, хлорид кальция, хлорид железа, сульфат магния, сульфат железа, сульфат марганца, карбонат кальция и т.д. Кроме того, могут быть включены аминокислоты, витамины и/или подходящие предшественники и т.д. Данные компоненты или предшественники можно добавлять в среду порционно или непрерывно, но способ добавления не ограничивается ими.
Кроме того, во время культивирования микроорганизма по настоящему раскрытию рН среды можно корректировать добавлением в данную среду правильным способом таких соединений, как гидроксид аммония, гидроксид калия, аммиак, фосфорная кислота, серная кислота и т.д. Кроме того, во время культивирования пенообразование можно подавлять посредством применения пеногасителя, такого как сложный полигликолевый эфир жирной кислоты. Кроме того, в среду можно инъецировать кислород или кислородсодержащий газ для поддержания аэробного состояния данной среды, или газ можно не инъецировать, или можно инъецировать газообразный азот, водород или диоксид углерода для того, чтобы поддерживать анаэробное и микроаэробное состояния, но способ поддержания данного состояния не ограничивается ими.
При культивировании по настоящему раскрытию можно поддерживать температуру культивирования от 20°С до 45°С, в частности, от 25°С до 40°С, и данный штамм можно культивировать в течение примерно от 10 до 160 часов, но условия культивирования не ограничиваются ими.
L-аргинин, продуцируемый посредством культивирования по настоящему раскрытию, может секретироваться в среду или может оставаться в клетках.
Способ продуцирования L-аргинина по настоящему раскрытию может дополнительно включать стадии получения микроорганизма по настоящему раскрытию, приготовления среды для культивирования данного микроорганизма или их комбинацию (в любом порядке), например, перед стадией культивирования.
Способ продуцирования L-аргинина по настоящему раскрытию может дополнительно включать стадию выделения L-аргинина из среды в соответствии с культивированием (среда, подвергнувшаяся воздействию культуры) или из микроорганизма. После стадии культивирования может быть дополнительно включена стадия выделения.
Выделение может служить для сбора интересующего L-аргинина посредством подходящего способа, известного в данной области, согласно способу культивирования микроорганизма по настоящему раскрытию, например, способу периодического, непрерывного культивирования или культивирования с подпиткой. Например, можно использовать центрифугирование, фильтрование, обработку осадителем кристаллизованного белка (высаливание), экстракцию, обработку ультразвуком, ультрафильтрацию, диализ, разные виды хроматографии, такие как хроматография на молекулярных ситах (гель-фильтрация), адсорбционная хроматография, ионообменная хроматография и аффинная хроматография, и т.д., ВЭЖХ (высокоэффективная жидкостная хроматография) или их комбинацию. Интересующий L-аргинин можно выделять из среды или микроорганизма посредством подходящего способа, известного в данной области.
Способ продуцирования L-аргинина по настоящему раскрытию может дополнительно включать стадию очистки. Очистку можно осуществлять посредством подходящего способа, известного в данной области. В одном примере, когда способ продуцирования L-аргинина по настоящему раскрытию включает и стадию выделения, и стадию очистки, данные стадии выделения и стадии очистки можно осуществлять непрерывно или прерывисто, независимо от порядка, или можно осуществлять одновременно, или посредством объединения в одну стадию, но не ограничиваясь ими.
В способе по настоящему раскрытию вариант LysE, полинуклеотид, вектор, микроорганизм/штамм и т.д. являются такими, как описано в других аспектах.
Согласно еще одному другому аспекту настоящего раскрытия предложена композиция для продуцирования L-аргинина, включающая вариант LysE по настоящему раскрытию, полинуклеотид, кодирующий данный вариант, вектор, включающий данный полинуклеотид, или микроорганизм, содержащий полинуклеотид по настоящему раскрытию; его культуральная среда или их комбинация.
Композиция по настоящему раскрытию может дополнительно включать произвольные подходящие эксципиенты, которые обычно используются в композициях для продуцирования L-аргинина. Такие эксципиенты могут представлять собой, например, консервант, увлажнитель, диспергирующий агент, суспендирующий агент, буферизующий агент, стабилизатор или изотоничный агент и т.д., но не ограничиваются ими.
Относительно композиции по настоящему раскрытию вариант LysE, полинуклеотид, вектор, микроорганизм/штамм, среда и т.д. являются такими, как описано в других аспектах.
Согласно еще одному другому аспекту настоящего раскрытия предложен способ увеличения способности микроорганизма к осуществлению экспорта и/или продукции L-аргинина или способ предоставления микроорганизму способности к осуществлению экспорта и/или продукции L-аргинина, включающий стадию осуществления модификации данного микроорганизма для экспрессии варианта LysE по настоящему раскрытию.
Относительно способа увеличения способности микроорганизма по настоящему раскрытию к осуществлению продукции L-аргинина, вариант, полинуклеотид, вектор, микроорганизм/штамм и т.д. являются такими, как описано в других аспектах.
Способ осуществления модификации микроорганизма может включать стадию введения в данный микроорганизм варианта LysE, кодирующего его полинуклеотида или рекомбинантного вектора, содержащего данный полинуклеотид. Однако данный способ модификации не ограничивается им, и можно использовать без ограничения способы, известные в данной области, включая способ, раскрытый в данном документе.
Согласно еще одному другому аспекту настоящего раскрытия предложено применение варианта LysE по настоящему раскрытию в осуществлении экспорта L-аргинина.
Согласно еще одному другому аспекту настоящего раскрытия предложено применение микроорганизма, содержащего один или более чем один вариант LysE по настоящему раскрытию, полинуклеотида, кодирующего данный вариант, и вектора, содержащего данный полинуклеотид, в осуществлении продукции L-аргинина.
Вариант LysE, полинуклеотид, вектор, микроорганизм являются такими, как описано в других аспектах.
Способ осуществления изобретения
Ниже настоящее раскрытие будет более подробно описано со ссылкой на Примеры и Экспериментальные примеры. Однако данные Примеры и Экспериментальные примеры служат лишь для иллюстрирования настоящего раскрытия, и объем настоящего раскрытия не предназначен для того, чтобы ограничиваться данными Примерами и Экспериментальными примерами.
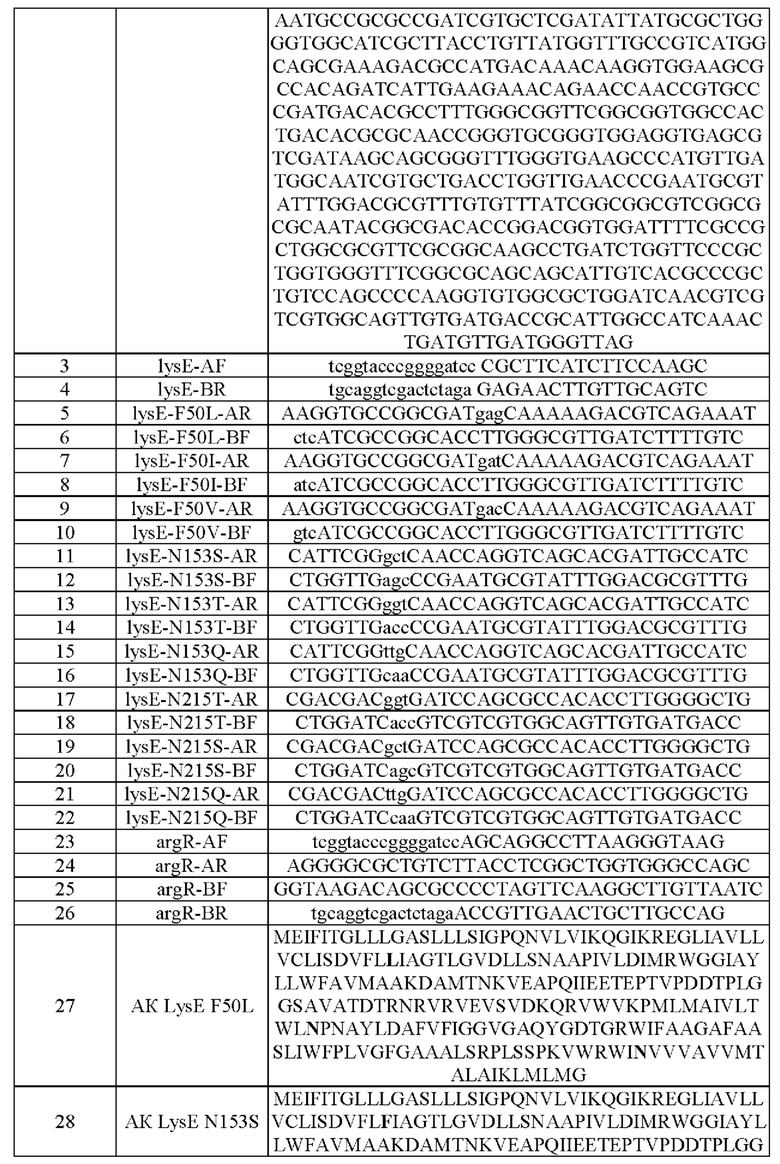
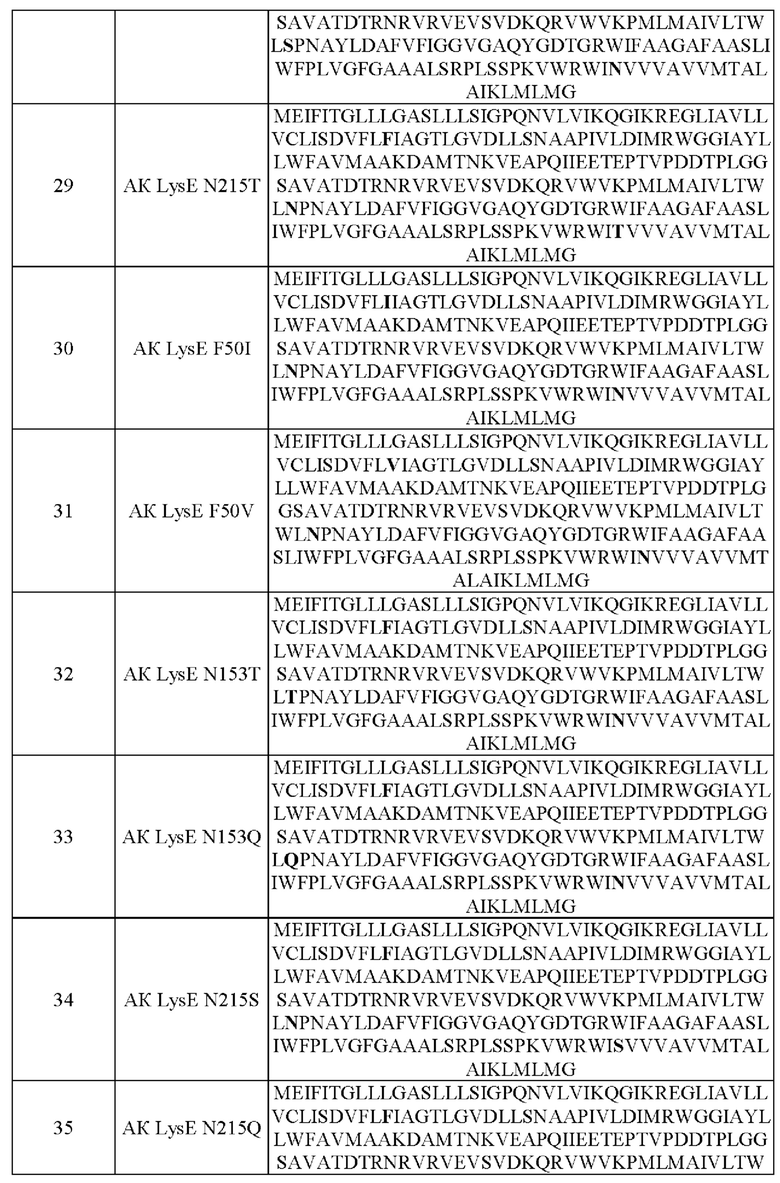
Пример 1. Получение разных вариантов, имеющих замену аминокислотных остатков в положениях 50,153 и 215 LysE
Авторы настоящего изобретения определили то, что аминокислотные остатки в положениях 50, 153 и 215 в SEQ ID NO: 1 являются важными положениями для активности LysE, и получили варианты, в которых соответствующие аминокислотные остатки были заменены другой аминокислотой. В частности, геномную ДНК Corynebacterium glutamicum АТСС13869 использовали в качестве матрицы для проведения ПЦР (полимеразная цепная реакция) с использованием пар праймеров из Таблицы 1 (SEQ ID NO: 3 и 5, SEQ ID NO: 4 и 6, SEQ ID NO: 3 и 7, SEQ ID NO: 4 и 8, SEQ ID NO: 3 и 9, SEQ ID NO: 4 и 10, SEQ ID NO: 3 и 11, SEQ ID NO: 4 и 12, SEQ ID NO: 3 и 13, SEQ ID NO: 4 и 14, SEQ ID NO: 3 и 15, SEQ ID NO: 4 и 16, SEQ ID NO: 3 и 17, SEQ ID NO: 4 и 18, SEQ ID NO: 3 и 19, SEQ ID NO: 4 и 20, SEQ ID NO: 3 и 21, SEQ ID NO: 4 и 22) и ПЦР с перекрывающимися праймерами с использованием пары праймеров SEQ ID NO: 3 и 4, получая, посредством этого, фрагменты гомологичной рекомбинации, каждый из которых имеет мутантную последовательность LysE-F50L, F50I, F50V, N153S, N153T, N153Q, N215T, N215S или N215Q. В это время проводили ПЦР-реакцию посредством 30 циклов, состоящих из денатурации при 95°С в течение 30 секунд; отжига при 55°С в течение 30 секунд и удлинения при 72°С в течение 1 минуты. Затем вектор pDCM2 (публикация корейского патента №10-2020-0136813), который не реплицируется в Corynebacterium glutamicum, и каждый фрагмент, амплифицированный ПЦР, обрабатывали BamHI и Xbal, которые представляют собой рестрикционные ферменты для введения в хромосому, и затем лигировали с использованием ДНК-лигазы. Затем каждый продукт трансформировали в Е. coli DH5α и высаживали на твердую среду LB, содержащую канамицин (25 мг/л). 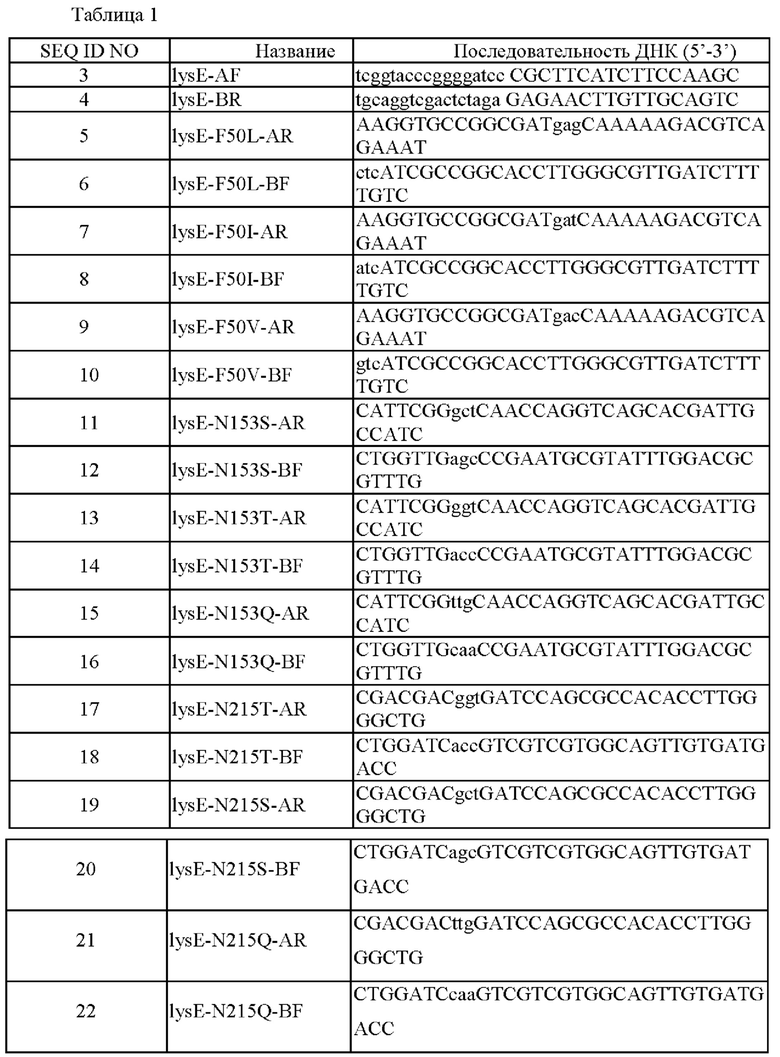
Колонии, трансформированные плазмидой, в которую был вставлен целевой ген, отбирали посредством ПЦР, и затем каждую плазмиду получали с использованием способа выделения плазмиды и называли pDCM2-lysE(F50L), pDCM2-lysE(F50I), pDCM2-lysE(F50V), PDCM2-lysE(N153S), PDCM2-lysE(N153T), PDCM2-lysE(N153Q), pDCM2-lysE(N215T), PDCM2-lysE(N215S), PDCM2-lysE(N215Q) соответственно.
Пример 2. Получение штамма с введенным вариантом LysE, происходящим от Corynebacterium glutamicum КССМ10741Р, и оценка способности к продуцированию L-аргинина
Для того, чтобы ввести мутацию LysE в Corynebacterium glutamicum КССМ10741Р (US 8034602 В2), который представляет собой штамм, продуцирующий L-аргинин, проводили трансформацию с использованием полученной рекомбинантной плазмиды (van der Rest et al., Appl Microbiol Biotechnol 52:541-545, 1999). Затем проводили вторичную рекомбинацию на чашке с твердой средой, содержащей 4% сахарозы, и проводили ПЦР на трансформанте, у которого вторичная рекомбинация была завершена с использованием пары праймеров (SEQ ID NO: 3 и 4) для идентификации введения каждой мутации в ген LysE на хромосоме. В это время проводили ПЦР-реакцию посредством 30 циклов, состоящих из денатурации при 95°С в течение 30 секунд; отжига при 55°С в течение 30 секунд и удлинения при 72°С в течение 1 минуты. Отобранные из него штаммы с введенным вариантом LysE называли KCCM10741P-lysE(F50L), KCCM10741P-lysE(F50I), KCCM10741P-lysE(F50V), KCCM10741P-lysE(N153S), KCCM10741P-lysE(N153T), KCCM10741P-lysE(N153Q), KCCM10741P-lysE(N215T), KCCM10741P-lysE(N215S), KCCM10741P-lysE(N215Q) соответственно.
Для анализа способности к продуцированию L-аргинина штаммов с введенным вариантом LysE их культивировали с родительским штаммом - штаммом Corynebacterium glutamicum КССМ10741Р следующим способом соответственно.
Родительский штамм Corynebacterium glutamicum КССМ10741Р и полученные выше штаммы с вариантом LysE инокулировали в 250 мл колбу с угловыми перегородками, содержащую 25 мл описанной ниже посевной среды, соответственно, и культивировали при 30°С со встряхиванием при 200 об./мин в течение 20 часов. Затем 1 мл посевной культуры инокулировали в 250 мл колбу с угловыми перегородками, содержащую 24 мл продукционной среды и культивировали при 30°С со встряхиванием при 200 об./мин в течение 72 часов. Составы посевной среды и продукционной среды являются следующими.
Посевная среда (рН 7,2)
20 г глюкозы, 45 г сульфата аммония, 2 г сульфата магния гептагидрата, 2 г одноосновного фосфата калия, 10 г хлорида аммония, 0,01 мг биотина, 0,1 мг тиамина-НС1, 2 мг пантотената кальция, 3 мг никотинамида, 10 мг сульфата железа(П), 0,02 мг сульфата цинка, 0,5 мг сульфата меди (на основе 1 литра дистиллированной воды).
Продукционная среда (рН 7,2)
60 г глюкозы, 45 г сульфата аммония, 2 г сульфата магния гептагидрата, 2 г одноосновного фосфата калия, 10 г хлорида аммония, 0,01 мг биотина, 0,1 мг тиамина-НС1, 2 мг пантотената кальция, 3 мг никотинамида, 10 мг сульфата железа, 10 мг сульфата марганца, 0,02 мг сульфата цинка, 0,5 мг сульфата меди, 30 г карбоната кальция (на основе 1 литра дистиллированной воды).
После завершения культивирования способность к продуцированию L-аргинина измеряли посредством ВЭЖХ (Waters 2478) (Таблица 2).
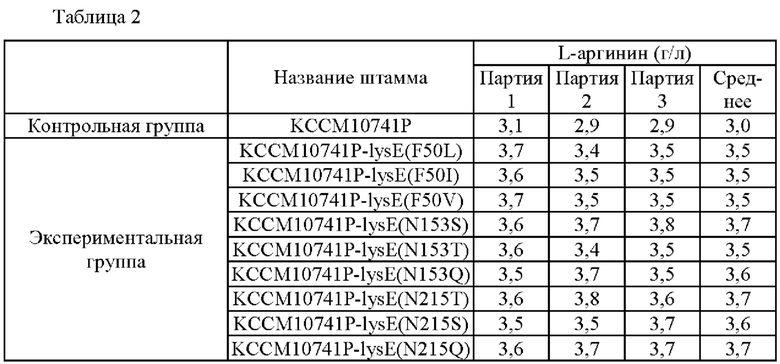
В результате подтвердили то, что все штаммы с введенным вариантом LysE демонстрировали 20%-ное увеличение способности к продуцированию L-аргинина по сравнению с родительским штаммом.
Пример 3. Получение штамма с введенным вариантом LysE, происходящим от Corynebacterium glutamicum CJ1R, имеющего способность к продуцированию L-аргинина, и оценка способности к продуцированию L-аргинина
Следующим образом подтвердили имели ли также или нет другие штаммы Corynebacterium glutamicum, продуцирующие L-аргинин, такое же влияние, как и в Примере 2.
Штамм Corynebacterium glutamicum, имеющий способность к продуцированию L-аргинина, получали посредством введения одного вида мутации (AargR) в штамм дикого типа (АТСС13869). Описывая подробно, конструировали рекомбинантный вектор для делеции гена argR (SEQ ID NO: 40) таким же способом, как и в Примере 1, и праймеры, использованные для конструирования данного вектора, показаны в Таблице 3.
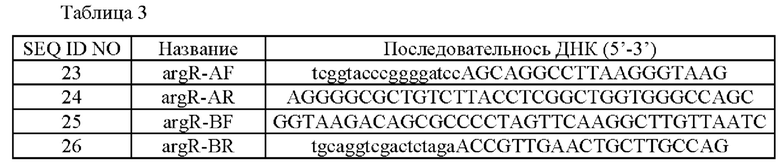
Плазмиду, в которую вставляли желаемый фрагмент, отбирали посредством ПЦР, и данную плазмиду называли pDCM2-AargR. Штамм Corynebacterium glutamicum дикого типа трансформировали с использованием pDCM2-AargR таким же способом, как и в Примере 2, и трансформанта подвергали ПЦР с использованием пары праймеров (SEQ ID NO: 23 и 26) для идентификации штамма с делецией argR на хромосоме, который называли CJ1R.
Варианты штамма получали посредством введения каждого из девяти вариантов LysE в CJ1R таким же способом, как и в Примере 2, и каждый называли CJlR-lysE(F50L), CJlR-lysE(FSOI), CJlR-lysE(F50V), CJlR-lysE(N153S), CJlR-lysE(N153T), CJ1R-lysE(N153Q), CJlR-lysE(N215T), CJlR-lysE(N215S), CJlR-lysE(N215Q).
Для анализа способности к продукции L-аргинина происходящих от CJ1R штаммов с введенным вариантом LysE данные штаммы культивировали также, как и в Примере 2, и после завершения культивирования способность к продуцированию L-аргинина измеряли посредством ВЭЖХ (Waters 2478) (Таблица 4).
Таблица 4
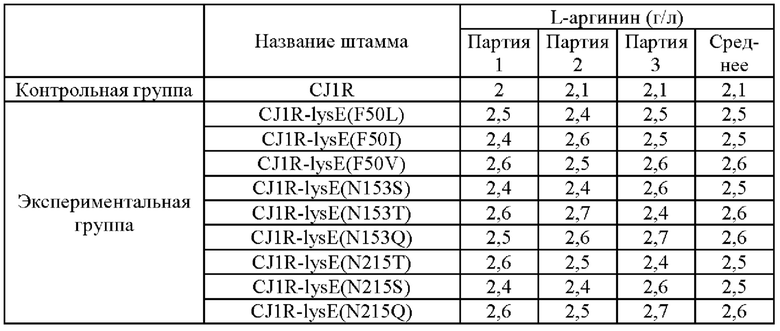
В результате подтвердили то, что все штаммы с введенным вариантом LysE демонстрировали 19%-ное увеличение способности к продуцированию L-аргинина по сравнению с родительским штаммом.
На основе приведенного выше описания специалистам в данной области будет понятно то, что настоящее раскрытие можно осуществлять в другой конкретной форме без изменения его технической сущности или важных характеристик. В данном отношении следует понимать то, что приведенное выше воплощение не является ограничивающим, но иллюстративным во всех аспектах. Объем данного раскрытия определяется приложенной формулой изобретения, а не предшествующим ей описанием, и, следовательно, подразумевается то, что все изменения и модификации, которые попадают в пределы границ формулы изобретения или эквивалентов таких границ, охватываются данной формулой изобретения



--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="en"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="P24193RU-st26-RU.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2024-05-06">
<ApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>PCT/KR2022/016438</ApplicationNumberText>
<FilingDate>2022-10-26</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>P24193RU</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2021-0143660</ApplicationNumberText>
<FilingDate>2021-10-26</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">СИДЖЕЙ ЧЕИЛДЖЕДАНГ КОРПОРЕЙШН
</ApplicantName>
<ApplicantNameLatin>CJ CHEILJEDANG CORPORATION</ApplicantNameLatin>
<InventionTitle languageCode="ru">Вариант LysE и способ получения
L-аргинина с его применением</InventionTitle>
<SequenceTotalQuantity>40</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium sp.</INSDQualifier_value>
<NonEnglishQualifier_value>Виды рода
Corynebacterium</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
FIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLNPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWINVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>702</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium sp.</INSDQualifier_value>
<NonEnglishQualifier_value>Виды рода
Corynebacterium</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaaatcttcattacaggtctgcttttgggggccagtcttttgctgt
ccatcggaccgcagaatgtactggtgattaaacaaggaattaagcgcgaaggactcattgcggttcttct
cgtgtgtttaatttctgacgtctttttgttcatcgccggcaccttgggcgttgatcttttgtccaatgcc
gcgccgatcgtgctcgatattatgcgctggggtggcatcgcttacctgttatggtttgccgtcatggcag
cgaaagacgccatgacaaacaaggtggaagcgccacagatcattgaagaaacagaaccaaccgtgcccga
tgacacgcctttgggcggttcggcggtggccactgacacgcgcaaccgggtgcgggtggaggtgagcgtc
gataagcagcgggtttgggtgaagcccatgttgatggcaatcgtgctgacctggttgaacccgaatgcgt
atttggacgcgtttgtgtttatcggcggcgtcggcgcgcaatacggcgacaccggacggtggattttcgc
cgctggcgcgttcgcggcaagcctgatctggttcccgctggtgggtttcggcgcagcagcattgtcacgc
ccgctgtccagccccaaggtgtggcgctggatcaacgtcgtcgtggcagttgtgatgaccgcattggcca
tcaaactgatgttgatgggttag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-AF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-AF
</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcggtacccggggatcccgcttcatcttccaagc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q5">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-BR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-BR</NonEnglishQualifier
_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcaggtcgactctagagagaacttgttgcagtc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-F50L-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-F50L-AR</NonEnglishQual
ifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaggtgccggcgatgagcaaaaagacgtcagaaat</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-F50L-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-F50L-BF</NonEnglishQual
ifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcatcgccggcaccttgggcgttgatcttttgtc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-F50I-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-F50I-AR</NonEnglishQual
ifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaggtgccggcgatgatcaaaaagacgtcagaaat</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-F50I-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-F50I-BF</NonEnglishQual
ifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcatcgccggcaccttgggcgttgatcttttgtc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-F50V-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-F50V-AR</NonEnglishQual
ifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaggtgccggcgatgaccaaaaagacgtcagaaat</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-F50V-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-F50V-BF</NonEnglishQual
ifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcatcgccggcaccttgggcgttgatcttttgtc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N153S-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N153S-AR</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattcgggctcaaccaggtcagcacgattgccatc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N153S-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N153S-BF</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggttgagcccgaatgcgtatttggacgcgtttg</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N153T-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N153T-AR</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattcggggtcaaccaggtcagcacgattgccatc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N153T-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N153T-BF</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggttgaccccgaatgcgtatttggacgcgtttg</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N153Q-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N153Q-AR</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattcggttgcaaccaggtcagcacgattgccatc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N153Q-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N153Q-BF</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggttgcaaccgaatgcgtatttggacgcgtttg</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N215T-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N215T-AR</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgacgacggtgatccagcgccacaccttggggctg</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N215T-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N215T-BF</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggatcaccgtcgtcgtggcagttgtgatgacc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N215S-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N215S-AR</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgacgacgctgatccagcgccacaccttggggctg</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N215S-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N215S-BF</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggatcagcgtcgtcgtggcagttgtgatgacc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N215Q-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N215Q-AR</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgacgacttggatccagcgccacaccttggggctg</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_lysE-N215Q-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_lysE-N215Q-BF</NonEnglishQua
lifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggatccaagtcgtcgtggcagttgtgatgacc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_argR-AF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_argR-AF</NonEnglishQualifier
_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcggtacccggggatccagcaggccttaagggtaag</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q45">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_argR-AR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_argR-AR</NonEnglishQualifier
_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggggcgctgtcttacctcggctggtgggccagc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q47">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_argR-BF</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_argR-BF</NonEnglishQualifier
_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtaagacagcgcccctagttcaaggcttgttaatc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>37</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q49">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer_argR-BR</INSDQualifier_value>
<NonEnglishQualifier_value>праймер_argR-BR</NonEnglishQualifier
_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcaggtcgactctagaaccgttgaactgcttgccag</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q51">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE F50L</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
LIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLNPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWINVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q53">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N153S</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
FIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLSPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWINVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q55">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N215T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
FIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLNPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWITVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q57">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE F50I</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
IIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLNPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWINVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q59">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE F50V</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
VIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLNPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWINVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q61">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N153T</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
FIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLTPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWINVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q63">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N153Q</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
FIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLQPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWINVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q65">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N215S</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q66">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
FIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLNPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWISVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>233</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q67">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N215Q</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..233</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEIFITGLLLGASLLLSIGPQNVLVIKQGIKREGLIAVLLVCLISDVFL
FIAGTLGVDLLSNAAPIVLDIMRWGGIAYLLWFAVMAAKDAMTNKVEAPQIIEETEPTVPDDTPLGGSAV
ATDTRNRVRVEVSVDKQRVWVKPMLMAIVLTWLNPNAYLDAFVFIGGVGAQYGDTGRWIFAAGAFAASLI
WFPLVGFGAAALSRPLSSPKVWRWIQVVVAVVMTALAIKLMLMG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>702</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q69">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE F50L nt</INSDQualifier_value>
<NonEnglishQualifier_value>LysE F50L
нт</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaaatcttcattacaggtctgcttttgggggccagtcttttgctgt
ccatcggaccgcagaatgtactggtgattaaacaaggaattaagcgcgaaggactcattgcggttcttct
cgtgtgtttaatttctgacgtctttttgctcatcgccggcaccttgggcgttgatcttttgtccaatgcc
gcgccgatcgtgctcgatattatgcgctggggtggcatcgcttacctgttatggtttgccgtcatggcag
cgaaagacgccatgacaaacaaggtggaagcgccacagatcattgaagaaacagaaccaaccgtgcccga
tgacacgcctttgggcggttcggcggtggccactgacacgcgcaaccgggtgcgggtggaggtgagcgtc
gataagcagcgggtttgggtgaagcccatgttgatggcaatcgtgctgacctggttgaacccgaatgcgt
atttggacgcgtttgtgtttatcggcggcgtcggcgcgcaatacggcgacaccggacggtggattttcgc
cgctggcgcgttcgcggcaagcctgatctggttcccgctggtgggtttcggcgcagcagcattgtcacgc
ccgctgtccagccccaaggtgtggcgctggatcaacgtcgtcgtggcagttgtgatgaccgcattggcca
tcaaactgatgttgatgggttag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>702</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q71">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N153S nt</INSDQualifier_value>
<NonEnglishQualifier_value>LysE N153S
нт</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q72">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaaatcttcattacaggtctgcttttgggggccagtcttttgctgt
ccatcggaccgcagaatgtactggtgattaaacaaggaattaagcgcgaaggactcattgcggttcttct
cgtgtgtttaatttctgacgtctttttgttcatcgccggcaccttgggcgttgatcttttgtccaatgcc
gcgccgatcgtgctcgatattatgcgctggggtggcatcgcttacctgttatggtttgccgtcatggcag
cgaaagacgccatgacaaacaaggtggaagcgccacagatcattgaagaaacagaaccaaccgtgcccga
tgacacgcctttgggcggttcggcggtggccactgacacgcgcaaccgggtgcgggtggaggtgagcgtc
gataagcagcgggtttgggtgaagcccatgttgatggcaatcgtgctgacctggttgagcccgaatgcgt
atttggacgcgtttgtgtttatcggcggcgtcggcgcgcaatacggcgacaccggacggtggattttcgc
cgctggcgcgttcgcggcaagcctgatctggttcccgctggtgggtttcggcgcagcagcattgtcacgc
ccgctgtccagccccaaggtgtggcgctggatcaacgtcgtcgtggcagttgtgatgaccgcattggcca
tcaaactgatgttgatgggttag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>702</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q73">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LysE N215T nt</INSDQualifier_value>
<NonEnglishQualifier_value>LysE N215T
нт</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaaatcttcattacaggtctgcttttgggggccagtcttttgctgt
ccatcggaccgcagaatgtactggtgattaaacaaggaattaagcgcgaaggactcattgcggttcttct
cgtgtgtttaatttctgacgtctttttgttcatcgccggcaccttgggcgttgatcttttgtccaatgcc
gcgccgatcgtgctcgatattatgcgctggggtggcatcgcttacctgttatggtttgccgtcatggcag
cgaaagacgccatgacaaacaaggtggaagcgccacagatcattgaagaaacagaaccaaccgtgcccga
tgacacgcctttgggcggttcggcggtggccactgacacgcgcaaccgggtgcgggtggaggtgagcgtc
gataagcagcgggtttgggtgaagcccatgttgatggcaatcgtgctgacctggttgaacccgaatgcgt
atttggacgcgtttgtgtttatcggcggcgtcggcgcgcaatacggcgacaccggacggtggattttcgc
cgctggcgcgttcgcggcaagcctgatctggttcccgctggtgggtttcggcgcagcagcattgtcacgc
ccgctgtccagccccaaggtgtggcgctggatcaccgtcgtcgtggcagttgtgatgaccgcattggcca
tcaaactgatgttgatgggttag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>171</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..171</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q75">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium sp.</INSDQualifier_value>
<NonEnglishQualifier_value>Виды рода
Corynebacterium</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSLGSTPSTPENLNPVTRTARQALILQILDKQKVTSQVQLSELLLDEGI
DITQATLSRDLDELGARKVRPDGGRAYYAVGPVDSIAREDLRGPSEKLRRMLDELLVSTDHSGNIAMLRT
PPGAAQYLASFIDRVGLKEVVGTIAGDDTVFVLARDPLTGKELGELLSGRTT</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>516</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..516</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Corynebacterium sp.</INSDQualifier_value>
<NonEnglishQualifier_value>Виды рода
Corynebacterium</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtcccttggctcaaccccgtcaacaccggaaaacttaaatcccgtga
ctcgcactgcacgccaagctctcattttgcagattttggacaaacaaaaagtcaccagccaggtacaact
gtctgaattgctgctggatgaaggcatcgatatcacccaggccaccttgtcccgagatctcgatgaactc
ggtgcacgcaaggttcgccccgatgggggacgcgcctactacgcggtcggcccagtagatagcatcgccc
gcgaagatctccggggtccgtcggagaagctgcgccgcatgcttgatgaactgctggtttctacagatca
ttccggcaacatcgcgatgctgcgcaccccgccgggagctgcccagtacctggcaagtttcatcgatagg
gtggggctgaaagaagtcgttggcaccatcgctggcgatgacaccgttttcgttctcgcccgtgatccgc
tcacaggtaaagaactaggtgaattactcagcgggcgcaccacttaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии. Предложен полипептид, вовлеченный в продуцирование L-аргинина, имеющий 80%-ную или более идентичность последовательности с SEQ ID NO: 1, и в котором любая одна или более чем одна аминокислота из аминокислот, соответствующих положениям 50, 153 и 215 в SEQ ID NO: 1, заменена другой аминокислотой, где аминокислота, соответствующая положению 50, заменена валином, лейцином или изолейцином, аминокислота, соответствующая положению 153, заменена серином, треонином или глутамином, аминокислота, соответствующая положению 215, заменена серином, треонином или глутамином. Также предложены полинуклеотид, кодирующий указанный полипептид, микроорганизм Corynebacterium для продуцирования L-аргинина, содержащий указанный полипептид или полинуклеотид, кодирующий указанный полипептид, и способ продуцирования L-аргинина. Изобретение позволяет увеличить выход L-аргинина. 4 н. и 4 з.п. ф-лы, 5 табл., 3 пр.
1. Полипептид, вовлеченный в продуцирование L-аргинина, имеющий 80%-ную или более идентичность последовательности с SEQ ID NO: 1, и в котором любая одна или более чем одна аминокислота из аминокислот, соответствующих положениям 50, 153 и 215 в SEQ ID NO: 1, заменена другой аминокислотой,
где аминокислота, соответствующая положению 50, заменена валином, лейцином или изолейцином,
аминокислота, соответствующая положению 153, заменена серином, треонином или глутамином,
аминокислота, соответствующая положению 215, заменена серином, треонином или глутамином.
2. Полипептид по п. 1, где данный полипептид происходит из рода Corynebacterium.
3. Полипептид по п. 1, в котором любая одна аминокислота из аминокислотных остатков в положениях 50, 153 и 215 в аминокислотной последовательности SEQ ID NO: 1 заменена другой аминокислотой,
где аминокислота, соответствующая положению 50, заменена валином, лейцином или изолейцином,
аминокислота, соответствующая положению 153, заменена серином, треонином или глутамином,
аминокислота, соответствующая положению 215, заменена серином, треонином или глутамином.
4. Полипептид по п. 1, вовлеченный в продуцирование L-аргинина, имеющий 90%-ную или более идентичность последовательности с SEQ ID NO: 1.
5. Полинуклеотид, кодирующий полипептид по любому из пп. 1-4.
6. Микроорганизм рода Corynebacterium для продуцирования L-аргинина, содержащий полипептид, вовлеченный в продуцирование L-аргинина, в котором любая одна или более чем одна аминокислота из аминокислот, соответствующих положениям 50, 153 и 215 в SEQ ID NO: 1, заменена другой аминокислотой,
где аминокислота, соответствующая положению 50, заменена валином, лейцином или изолейцином,
аминокислота, соответствующая положению 153, заменена серином, треонином или глутамином,
аминокислота, соответствующая положению 215, заменена серином, треонином или глутамином;
или полинуклеотид, кодирующий полипептид, вовлеченный в продуцирование L-аргинина.
7. Микроорганизм по п. 6, представляющий собой Corynebacterium glutamicum.
8. Способ продуцирования L-аргинина, включающий стадию культивирования микроорганизма рода Corynebacterium, содержащего полипептид, вовлеченный в продуцирование L-аргинина, в котором любая одна или более чем одна аминокислота из аминокислот, соответствующих положениям 50, 153 и 215 в SEQ ID NO: 1, заменена другой аминокислотой,
где аминокислота, соответствующая положению 50, заменена валином, лейцином или изолейцином,
аминокислота, соответствующая положению 153, заменена серином, треонином или глутамином,
аминокислота, соответствующая положению 215, заменена серином, треонином или глутамином;
или полинуклеотид, кодирующий полипептид, вовлеченный в продуцирование L-аргинина.
| База данных NCBI Reference Sequence: WP_003854734.1, 29.04.2021 | |||
| MULTISPECIES: L-lysine exporter [Corynebacterium] | |||
| Найдено по адресу: https://www.ncbi.nlm.nih.gov/protein/489951427?sat=58&satkey=110570110 Дата обращения 25.11.2024 | |||
| KR 102139806 B1, 30.07.2020 | |||
| US 7439038 B2, 21.10.2008 | |||
| СПОСОБ МИКРОБИОЛОГИЧЕСКОГО ПОЛУЧЕНИЯ АМИНОКИСЛОТ ПУТЕМ ПОВЫШЕННОЙ АКТИВНОСТИ ЭКСПОРТ-НОСИТЕЛЕЙ | 1996 |
|
RU2225883C2 |
Авторы
Даты
2025-05-20—Публикация
2022-10-26—Подача