Область техники, к которой относится изобретение
Настоящее изобретение в целом относится к молекулярной биологии. Более конкретно, настоящее изобретение относится к материалам и способам получения нуклеотидных последовательностей, которые являются характерными для ДНК данного источника (например, генома).
Уровень техники
Благодаря глобальным способам анализа генома ученым удалось понять причины и взаимосвязи в патофизиологии рака и других заболеваний или состояний с генетическим компонентом. Такие способы включают кариотипирование, определение плоидности, сравнительную геномную гибридизацию (CGH), анализ репрезентативных различий (RDA) (см., например, патент США №5436142) и анализ геномных репрезентативных выборок (WO 99/23256, опубликованную 14 мая 1999). Обычно такие способы включают либо использование зондов для подробного исследования экспрессии конкретных генов, либо изучение изменений в самом геноме.
Используя олигонуклеотидные матрицы, эти способы могут использоваться для получения высокоэффективного изображения генетических изменений в клетках. Однако эти способы требуют данных о последовательностях конкретных зондов. В частности, эта необходимость ограничивает применение кДНК матрицы, поскольку такие матрицы детально исследуют только ограниченный набор генов. Их применение также ограничено в случае скрининга всего генома, поскольку большинство олигонуклеотидов, разработанных для матрицы, могут быть не представлены в исследуемой популяции, что приводит к недостаточному или неэффективному анализу.
Сущность изобретения
Настоящее изобретение относится к композиции и способам, которые могут использоваться для исследования популяций молекул нуклеиновых кислот. Такие композиции и способы могут использоваться для анализа сложных геномов (например, геномов млекопитающих), необязательно, совместно с технологией микроматриц. В настоящем изобретении охарактеризовано множество по меньшей мере 100 молекул нуклеиновых кислот (А), где (а) каждая молекула нуклеиновой кислоты специфически гибридизуется с последовательностью генома, состоящей по меньшей мере из Z пар оснований; и (b) по меньшей мере Р% указанного количества молекул нуклеиновых кислот: (i) имеют длину по меньшей мере К нуклеотидов; (ii) специфически гибридизуются по меньшей мере с одной молекулой нуклеиновой кислоты, присутствующей или предсказанной, что она присутствует, в репрезентативной выборке, полученной из указанного генома, где указанная репрезентативная выборка имеет не более R% сложность указанного генома; и (iii) не более чем Х точных совпадений L1 нуклеотидов с указанным геномом (или указанной репрезентативной выборкой) и не менее Y точных совпадений L1 нуклеотидов с указанным геномом (или указанной репрезентативной выборкой); и (В), где (а) Z≥1×108; (b) 300≥K≥30; (c) 70≥R≥0,001; (d) P≥90-R; (e) целое число, ближайшее к (log4(Z)+2)≥L1≥целого числа, ближайшего к log4(Z); (f) Х представляет собой целое число, ближайшее к D1×(K-L1+1); (g) Y представляет собой целое число, ближайшее к D2×(K-L1+1); (h) 1,5≥D1≥1; (i) 1≥D2≥0,5.
В некоторых вариантах осуществления (1) множество молекул нуклеиновых кислот содержит по меньшей мере 500; 1000; 2500; 5000; 10000; 25000; 50000; 85000; 190000; 350000; или 550000 молекул нуклеиновых кислот; (2) Z равно по меньшей мере 3×108, 1×109, 1×1010 или 1×1011; (3) R равно 0,001, 1, 2, 4, 10, 15, 20, 30, 40, 50 или 70; (4) Р не зависит от R и равно по меньшей мере 70, 80, 90, 95, 97 или 99; (5) D1 равно 1; (6) L1 равно 15, 16, 17, 18, 19, 20, 21, 22, 23 или 24; (7) Р равно 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100; и/или (8) К равно 40, 50, 60, 70, 80, 90, 100, 110, 120, 140, 160, 180, 200 или 250. В некоторых вариантах осуществления идентичность молекулы нуклеиновой кислоты, которая специфически гибридизуется с другой молекулой нуклеиновой кислоты, составляет по меньшей мере 90% последовательности по сравнению с последовательностью такой же длины другой молекулы нуклеиновой кислоты. В дополнительных вариантах осуществления последовательность идентична по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или по меньшей мере на 100%.
В некоторых вариантах осуществления каждая из указанного Р% указанного множества молекул нуклеиновых кислот дополнительно имеет не более А точных совпадений L2 и не менее В точных совпадений L2 нуклеотидов с указанным геномом, где (а) L1>L2≥целое число ближайшее к log4(Z)-3; (b) А представляет собой целое число, ближайшее к D3×((K-L2+1)×(Z/4L 2)); (c) В представляет собой целое число, ближайшее к D4×((K-L2+1)×(Z/4L 2)); (d) 4≥D3≥1; (е) 1≥D4≥0,5.
Репрезентативная выборка ДНК-популяции может быть получена расщеплением по специфичным последовательностям, например, осуществляемым рестрикционной эндонуклеазой. Репрезентативная выборка также может быть получена из другой репрезентативной выборки. То есть полученная репрезентативная выборка является сложной репрезентативной выборкой.
Молекулы нуклеиновых кислот по настоящему изобретению могут быть идентифицированы способом, предусматривающим: (а) расщепление указанного генома in silico рестрикционным ферментом с получением множества предсказанных молекул нуклеиновых кислот; (b) получение виртуальной репрезентативной выборки указанного генома путем идентификации предсказанных молекул нуклеиновых кислот, длина каждой из которых составляет 200-1200 пар оснований включительно, где сложность виртуальной репрезентативной выборки указанного генома составляет 0,001%-70% включительно; (с) осуществление отбора олигонуклеотида с длиной 30-300 нуклеотидов включительно, и по меньшей мере с 90% идентичностью в отношении последовательности предсказанной молекулы нуклеиновой кислоты (b); (d) расчет сложности указанной виртуальной репрезентативной выборки относительно указанного генома; (е) идентификацию всех участков нуклеотидов L1, встречающихся в указанном олигонуклеотиде; и (f) подтверждение того, что частота встречаемости каждого из указанных участков в указанном геноме удовлетворяет различным заранее заданным требованиям.
Молекулы нуклеиновых кислот по настоящему изобретению могут использоваться в качестве зондов для анализа образца ДНК. Эти зонды могут быть иммобилизованы на поверхности твердой фазы, в том числе на полутвердую поверхность. Твердая фаза включает в себя, без ограничений, нейлоновые мембраны, нитроцеллюлозные мембраны, стеклянные пластинки и микросферы (например, парамагнитные микробусины). В некоторых вариантах осуществления положение молекул нуклеиновых кислот на указанной твердой фазе является известным, например, как такое, которое используют в формате микроматрицы. В настоящем изобретении также описан способ анализа образца нуклеиновой кислоты (например, геномной репрезентативной выборки), где указанный способ предусматривает (а) гибридизацию образца с нуклеотидными зондами по настоящему изобретению; и (b) определение с какой из указанного множества молекул нуклеиновых кислот гибридизуется указанный образец.
В настоящем изобретении также описан способ анализа различия числа копий геномной последовательности двух геномов, где указанный способ предусматривает: (а) получение двух меченых репрезентативных выборок для визуализации, где каждый набор фрагментов получен из соответствующего генома с помощью по меньшей мере одного и того же рестрикционного фермента; (b) приведение в контакт этих двух репрезентативных выборок с нуклеотидными зондами по настоящему изобретению для того, чтобы обеспечить возможность гибридизации между репрезентативными выборками и зондами; (с) анализ уровня гибридизации двух репрезентативных выборок с набором зондов, где разница указанных уровней и числа зондов из набора указывает на различие числа копий двух геномов относительно геномной последовательности, на которую нацелен указанный элемент. В некоторых вариантах осуществления репрезентативные выборки отличаются тем, что являются мечеными; и/или тем, что приведение в контакт двух репрезентативных выборок осуществляют одновременно.
Настоящее изобретение дополнительно относится к способу сравнения уровня метилирования геномной последовательности двух геномов, где указанный способ предусматривает получение двух меченых репрезентативных выборок для визуализации фрагментов соответствующих геномов, где каждую репрезентативную выборку получают способом, чувствительным к метилированию. Например, первую репрезентативную выборку первого генома получают с использованием первого рестрикционного фермента, а вторую репрезентативную выборку второго генома получают с использованием второго рестрикционного фермента, где указанный первый и второй рестрикционные ферменты «узнают» один и тот же сайт рестрикции, но один является чувствительным к метилированию, а другой нет. Последовательности, содержащие метил-С, также могут быть химически расщеплены после получения репрезентативной выборки при помощи рестрикционного фермента, нечувствительного к метилированию, таким образом, что репрезентативная выборка, полученная из метилированного генома, будет отличаться от репрезентативной выборки, полученной из неметилированного генома. Затем две репрезентативные выборки приводят в контакт с зондами по настоящему изобретению для того, чтобы обеспечить возможность гибридизации репрезентативной выборки и зондов. Затем проводят анализ гибридизации двух репрезентативных выборок с зондами, где различие уровня гибридизации между репрезентативными выборками относительно конкретного зонда указывает на различие уровня метилирования между двумя геномами относительно последовательности генома, на которую нацелен указанный зонд.
Аналогичные способы также могут использоваться для анализа полиморфизма сложного генома, как показано ниже.
Некоторые варианты осуществления по настоящему изобретению относятся к алгоритму для точной и эффективной визуализации и подсчета числа «слов» в геноме. Такой алгоритм, иногда в настоящем описании называемый механизмом поиска или mer-механизмом, использует преобразование генома (например, преобразование Барроуза-Уиллера (Burrows-Wheeler)) и вспомогательную структуру данных для подсчета количества конкретных «слов» в геноме. «Слово» означает нуклеотидную последовательность определенной длины.
В общем случае, механизм производит поиск конкретного «слова» путем поиска последней «буквы слова». Затем он продолжает поиск «буквы», непосредственно предшествующей последней «букве». Если первая непосредственно предшествующая «буква» найдена, то затем производит поиск второй «буквы», непосредственно предшествующей последней «букве слова», и так далее до тех пор, пока не будет найдено все «слово». Если предшествующие «буквы» не были найдены, то делается вывод о том, что «слово» в геноме отсутствует. Если первая «буква слова» найдена, то частота ее встречаемости равна частоте встречаемости этого конкретного «слова».
Преимущество этого конкретного алгоритма состоит в его использовании для реализации нескольких конкретных применений, включая изучение генома, что обсуждается ниже.
Другие отличительные признаки и преимущества настоящего изобретения будут очевидными из нижеприведенных чертежей, подробного описания и формулы изобретения.
Краткое описание чертежей
На фиг.1А-1D показаны предсказанные информационные данные и точность измерений с помощью матрицы c использованием микроматриц, содержащих 10000 олигонуклеотидов. На фиг.1А показаны результаты, где гибридизованные образцы являются репрезентативной выборкой, полученной с использованием BglII, и репрезентативная выборка, полученная с использованием BglII, повторно рестриктированных по сайту расщепления HindIII. Ось Y (Среднее отношение) представляет собой среднее измеренное отношение двух гибридизаций, репрезентативной выборки, подвергшихся повторной рестрикции, и нормальной репрезентативной выборки, приведенное на графике в логарифмическом масштабе. Ось Х (Индекс) представляет собой условный индекс, построенный таким образом, что зонды, полученные из фрагментов, определенных как имеющие фрагменты с внутренним сайтом HindIII, находятся с правой стороны. На фиг.1В показана воспроизводимость продублированных экспериментов для получения среднего отношения на фиг.1А. Ось Y (отношение Эксп.1) представляет собой измеренное отношение эксперимента 1 и ось Х (отношение Эксп.2) представляет собой измеренное отношение эксперимента 2. Обе оси изображены на графике в логарифмическом масштабе. На фиг.1С изображено нормализованное отношение по оси Y как функция интенсивности образца, который не является повторно рестриктированным, по оси Х. Как отношение, так и интенсивность изображены на графике в логарифмическом масштабе. На фиг.1D представлены данные, полученные путем моделирования. Ось Х (Индекс) представляет собой условный индекс. Зонды, в группах 600, обнаруживают увеличение числа копий, слева направо. Зонды, примыкающие со стороны 600, обнаруживают нормальное число копий. Ось Y (Среднее отношение) представляет собой среднее отношение, приведенное на графике в логарифмическом масштабе.
На фиг.2А1-2А3, 2В1-2В3 и 2С1-2С3 показаны геномные профили для образца первичного рака молочной железы (CHTN159), с анеуплоидными ядрами, в сравнении с диплоидными ядрами от того же пациента (фиг.2А1-2А3), линии клеток рака молочной железы, в сравнении с эталоном нормальной мужской особи (фиг. 2В1-2В3), и нормальной мужской особи с эталоном нормальной мужской особи (фиг. 2С1-2С3), с использованием печатной матрицы 10К (фиг.2А1, 2В1 и 2С1) и с использованием фотопечатной матрицы 85К (фиг.2А2, 2В2 и 2С2). В каждом случае (фиг.2А1, 2В1 и 2С1 и фиг.2А2, 2В2 и 2С2) ось Y представляет собой среднее отношение, а ось Х (Ген-индекс) представляет собой индекс, который графически представляет расположение зондов в том геномном порядке, в котором они представлены в хромосомах, и дает возможность визуализировать весь геном от хромосомы 1 до Y. На фиг.2А3, 2В3 и 2С3 показано соответствие отношений, измеренных для зондов-«братьев», представленных в микроматрицах 10К и 85К. Ось Y представляет собой измеренное отношение для микроматрицы 10К, а ось Х представляет собой измеренное отношение для микроматрицы 85К.
На фиг.3А-3D показано несколько хромосом с изменяющимися флуктуациями числа копий в случае анализа клеточной линии опухоли SK-BR-3 в качестве сравнения с нормальным эталоном. Ось Y (Среднее отношение) представляет среднее отношение двух гибридизаций в логарифмическом масштабе. Ось Х (Ген-индекс) представляет собой индекс геномных координат. На фиг.3А представлены флуктуации числа копий, идентифицированных для хромосомы 5, на фиг.3В - для хромосомы 8, на фиг.3С - для хромосомы 17 и на фиг.3D - для хромосомы Х.
На фиг.4А-4D показана средняя сегментация, вычисленная в случае анализа SK-BR-3 в сравнении с нормальным эталоном (фиг.4А и фиг.4В) и CHNT159 (фиг.4С и фиг.4D). На фиг.4А-4D ось Y соответствует значению среднего сегмента для каждого зонда в логарифмическом масштабе. На фиг.4А и фиг.4С каждая из осей Х (индекс среднего сегмента) приведена с возрастающим значением своей назначенной средней сегмента. На фиг.4В и фиг.4D ось Х (Ген-индекс) представляет собой геномный индекс, который, как описано выше, представляет весь геном последовательно. Изображенные наверху данные среднего сегмента представляют собой сетку числа копий, экстраполированную из данных матрицы с использованием формул в данном описании (горизонтальные линии). Вычисленное число копий для каждой горизонтальной линии находится справа от сетки.
На фиг.5А-5D по оси Y (Среднее отношение SK-BR-3) изображено среднее отношение двух гибридизаций SK-BR-3 в сравнении с нормальным эталоном в логарифмическом масштабе. Ось Х (Ген-индекс) представляет собой геномный индекс. На фиг.5А показан участок Х хромосомы с участком потери. Изображенное поверх измеренного отношения матрицы представляет собой вычисленное значение сегментации. На фиг.5В показан участок хромосомы 8 (c-myc, расположенный с правой стороны от центра графика) в случае результатов для SK-BR-3 в сравнении с нормальным эталоном. Изображенные поверх данные представляют собой значения сегментации для SK-BR-3 в сравнении с нормальным эталоном с диагональной штриховкой и значения сегментации для первичной опухоли CHTN159 с вертикальной штриховкой. На фиг.5С показано нарушение в хромосоме 5, показывающее разрешающую способность 85К в сравнении с матрицей 10К. Результаты получены в случае SK-BR-3 в сравнении с нормальным эталоном. Незаштрихованные кружки соответствуют результатам для печатной матрицы 10К, а заштрихованные кружки соответствуют результатам для фотопечатной матрицы 85К. Горизонтальные линии соответствуют оценке числа копий, исходя из моделирования значений средних сегментов. На фиг.5D показано сравнение SK-BR-3 с нормальным эталоном, изображающим область гомозиготной делеции в хромосоме 19. Значение среднего сегмента изображено в виде белой линии, а сетка представляет собой оценки числа копий, как описано выше.
На фиг.6А-6D показаны результаты в случае нормы в сравнении с нормой, идентичные изображенным на фиг.2С2 за исключением того, что синглетные зонды отфильтрованы, как описано в тексте. На фиг.6В показано последовательное сравнение экспериментов для небольшого участка хромосомы 4. Ось Y представляет собой среднее отношение в логарифмическом масштабе. Ось Х представляет собой Ген-индекс. Заштрихованные (85К) и незаштрихованные (10К) кружки получены в случае сравнения SK-BR-3 с нормой. Пустые треугольники соответствует сравнению пигмея с нормальным эталоном. На фиг.6С показано нарушение, обнаруженное в нормальной популяции в хромосоме 6. Заштрихованные кружки нанесены исходя из среднего отношения в случае анализа пигмея по отношению к нормальному эталону. Линия с вертикальной штриховкой соответствует значению среднего сегмента для пигмея в сравнении с нормальным эталоном. Линия с диагональной штриховкой соответствует значению среднего сегмента для SK-BR-3 в сравнении с нормальным эталоном. Линия с крестообразной штриховкой соответствует значению сегмента в сравнении с первичной опухолью (анеуплоидной по отношению к диплоидной CHNT159). На фиг.6D показан участок хромосомы 2. Данные, показанные кружками, получены из сравнения SK-BR-3 с нормальным эталоном. Линия среднего сегмента для этого сравнения показана с вертикальной штриховкой. Линия среднего сегмента для сравнения пигмея с нормальным эталоном показана с диагональной штриховкой, а в случае первичной опухоли CHNT159 - с крестообразной штриховкой. Для фиг.6С и 6D вычисленное число копий для горизонтальных линий приведено справа от диаграммы.
На фиг.7 показана блок-схема иллюстративной системы согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.8 показана блок-схема последовательности операций иллюстративного предварительного этапа обработки для выполнения точных подсчетов «слов» согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.9А и 9В показана блок-схема последовательности операций иллюстративного алгоритма подсчета «слов» согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.10А и 10В показан иллюстративный пример алгоритма подсчета «слов» по фиг.9А и 9В согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.11 показан иллюстративный массив суффиксов, имеющий положения координат, соответствующие координатам генома согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.12А показано графическое представление переменных и структур данных, используемых в алгоритме, согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.12В показано представление в виде псевдокода алгоритма согласно некоторым вариантам осуществления настоящего изобретения.
Описание изобретения
Настоящее изобретение относится к олигонуклеотидным зондам для анализа репрезентативной выборки ДНК-популяции (например, генома, хромосомы или смеси ДНК). Олигонуклеотидные зонды могут использоваться в растворе или могут быть иммобилизованы на твердой (включая полутвердую) поверхности, такой как матрица или микробусины (например, Lechner et al., Curr. Opin. Chem. Biol., 6:31-38 (2001); Kwok, Annu. Rev. Genomics Human Genet., 2:235-58 (2001); Aebersold et al., Nature, 422:198-207 (2003); и в патентах США 6355431 и 6429027). Репрезентативная выборка является репродуцируемым образцом ДНК-популяции, где результирующая ДНК обычно имеет новый формат или уменьшенную сложность или и то и другое (Lisitsyn et al., Science 258:946-51 (1993); Lucito et al., Proc. Natl. Acad. Sci. USA, 92:151-5(1998)). Например, репрезентативная выборка генома может состоять из последовательностей ДНК, которые получены только из небольшой части генома и, в основном, не имеют повторяющихся последовательностей. Анализ геномной репрезентативной выборки может выявить изменения в геноме, включая мутации, такие как делеции, амплификации, хромосомные перестройки и полиморфизмы. При проведении анализа в клинических целях с помощью него можно обеспечить понимание молекулярной основы заболевания, а также он может быть полезен для диагностики и лечения такого заболевания.
Композиции олигонуклеотидов по настоящему изобретению могут использоваться для гибридизации с репрезентативной выборкой ДНК-источника, где данные гибридизации обрабатывают для получения генетических профилей ДНК-источника (например, генетических нарушений и полиморфизмов, связанных с заболеваниями). Может быть предпочтительным, чтобы репрезентативные выборки (или в настоящем описании «тестируемая репрезентативная выборка») и по меньшей мере некоторую фракцию олигонуклеотидных зондов в композициях получают из одного и того же вида. Может быть использована ДНК любого вида, включая виды млекопитающих (например, ДНК свиньи, мыши, крысы, примата (например, человека), собаки и кошки), виды рыб, виды рептилий, виды растений и виды микроорганизмов.
I. ОЛИГОНУКЛЕОТИДНЫЕ ЗОНДЫ
Олигонуклеотидные зонды по настоящему изобретению, предпочтительно, разработаны с помощью виртуальной репрезентативной выборки ДНК-источника, такого как геномная ДНК указанного индивида. Репрезентативная выборка генома с получением фрагментов обычно, но не всегда, приводит в результате к упрощению его сложности. Сложность репрезентативной выборки соответствует сложности фракции генома, которая в нем представлена. Один из способов вычисления сложности представляет собой деление количества нуклеотидов во фрагменте на количество нуклеотидов в геноме. Сложность геномной репрезентативной выборки может находиться в пределах от менее 1% до 95% всего генома. Там, где используется ДНК с относительно простым геномом, репрезентативная выборка может иметь 100% сложность общего генома, например, репрезентативная выборка может быть получена путем рестрикционного расщепления всей ДНК без амплификации. Репрезентативная выборка, связанная с настоящим изобретением, обычно имеет сложность между 0,001% и 70%. Уменьшение сложности обеспечивает требуемые кинетические характеристики гибридизации.
«Реальное» рестриктирование ДНК с получением репрезентативной выборки включает в себя лабораторные процедуры («влажные работы»), с помощью которых получают репрезентативную выборку ДНК. «Виртуальное» рестриктирование с получением репрезентативной выборки, с другой стороны, имеет преимущество в том, что секвенированы полные геномы, например человеческий геном. С помощью вычислительного анализа доступных геномных последовательностей можно легко разработать большое количество олигонуклеотидных зондов, которые гибридизуются с картированными областями генома и имеют минимальную степень перекрывания последовательностей с остальным геномом.
В качестве примера для разработки набора олигонуклеотидных зондов для генетического анализа человека можно in silicо (т.е., виртуально) провести расщепление человеческого генома путем определения местоположения всех сайтов расщепления выбранной рестрикционной эндонуклеазы в секвенированном геноме. Затем можно провести анализ полученных фрагментов для идентификации тех, которые находятся в требуемой области (например, 200-1200 п.о., 100-400 п.о. и 400-600 п.о.), которые могут быть амплифицированы при помощи, например, ПЦР. Такие фрагменты определены в настоящем описании как «предсказанные, что они существуют» в репрезентативной выборке. Рестрикционная эндонуклеаза может быть выбрана исходя из сложности требуемой репрезентативной выборки. Например, рестрикционные эндонуклеазы, которые разрезают редко, например, такие как эндонуклеазы, которые «узнают» заданные последовательности из 6 п.о. или 8 п.о., будут продуцировать репрезентативную выборку более низкой сложности, в то время как рестрикционные эндонуклеазы, которые разрезают часто, например, такие как эндонуклеазы, которые «узнают» заданные последовательности из 4 п.о., будут продуцировать репрезентативные выборки более высокой сложности. Кроме того, факторы, такие как содержание G/C анализируемого генома, будут оказывать воздействие на частоту расщепления конкретными рестрикционными эндонуклеазами и, следовательно, влиять на выбор рестрикционных эндонуклеаз. Обычно используются надежные рестрикционные эндонуклеазы, которые не демонстрируют сильной активности. В качестве альтернативы также может применяться расщепление исходя из уровня метилирования заданного сайта, например, путем использования рестрикционного фермента, чувствительного к метилированию, или другого фермента, такого как McrBC, который «узнает» в ДНК метилированные цитозины.
Последовательности всех расщепленных фрагментов требуемой области (например, 200-1200 п.о., 100-400 п.о. и 400-600 п.о.) анализируют при помощи компьютера, где области некоторых из таких фрагментов, которые составляют по меньшей мере около 30 п.о. в длину и имеют минимальную гомологию с оставшимся геномом, могут быть выбраны как реперезентативные олигонуклеотидные зонды для генома человека. В примере 1 и секции IV ниже дополнительно проиллюстрированы способы идентификации олигонуклеотидов настоящего изобретения.
Олигонуклеотиды по настоящему изобретению могут быть длиной от примерно 30 нуклеотидов до примерно 1200 нуклеотидов. Точная длина выбранных олигонуклеотидов будет зависеть от предполагаемого использования, например, размера ДНК-источника, из которого получена репрезентативная выборка, и от того используются ли они как компоненты матрицы. Обычно олигонуклеотиды имеют длину более по меньшей мере 35 нуклеотидов, например по меньшей мере 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95 или 100 нуклеотидов, но они также могут быть короче, например, длиной 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 и 30 нуклеотидов. Обычно олигонуклеотиды имеют длину не более 600 нуклеотидов, например, не более 550, 500, 450, 400, 350, 300, 250, 200 или 150 нуклеотидов. Как очевидно специалистам в данной области техники, длина олигонуклеотидов будет зависеть от характеристик анализируемого генома, например, сложности и количества повторяющихся последовательностей.
II. МАТРИЦЫ ОЛИГОНУКЛЕОТИДОВ
Олигонуклеотидные зонды по настоящему изобретению могут использоваться в формате матрицы. Матрица содержит твердую подложку с зондами нуклеиновых кислот, прикрепленными к ней в определенных координатах или адресах. Каждый адрес содержит либо большое число копий единичного ДНК-зонда, либо смесь различных ДНК-зондов. Матрица нуклеиновых кислот, также называемая «микроматрицей» или «чипом», в общем случае описана, например, в патенте США 6361947 и ссылках, цитируемых в нем. Авторы назвали генетический анализ, использующий новые матрицы, «анализом с помощью микроматрицы репрезентативных олигонуклеотидов» (ROMA) или анализ, где расщепление зависит от метилирования в заданном сайте, «анализ с помощью микроматрицы олигонуклеотидов с визуализацией метилирования» («MOMA»).
Для производства микроматрицы по настоящему изобретению предварительно синтезированные олигонуклеотиды прикрепляют к твердой подложке, которая может быть изготовлена из стекла, пластика (например, полипропилена или нейлона), полиакриламида, нитроцеллюлозы или других материалов, и может быть пористой или непористой. В одном из способов прикрепление нуклеиновых кислот к поверхности происходит с помощью печати на стеклянные плашки, как в общем случае описано у Schena et al., Science 270:467-70 (1995); DeRisi et al., Nature Gen. 14:457-60 (1996); Shalon et al., Genome Res. 6:639-45 (1996); и Schena et al., Proc. Natl. Acad. Sci. USA, 93:10539-1286 (1995). Для матриц с низкой плотностью также могут использоваться дот-блоты на нейлоновой гибридизационной мембране. См., например, Sambrook et al., Molecular Cloning - A Laboratory Manual (2nd Ed.), vol.1-3, Cold Spring Harbor Laboratory, Cold Spring Harbor, New York, 1989.
В другом способе для изготовления микроматриц используют фотолитографические (или «фотопечатные») технологии для синтеза олигонуклеотидов непосредственно на подложке матрицы, т.е. in sutu. См. Fodor et al., Science 251:767-73 (1991); Pease et al., Proc. Natl. Acad. Sci. USA, 91:5022-6 (1994); Lipschutz et al., Nat. Genet., 21 (1 Suppl):20-46 (1999); Nuwaysir et al., Genome Res., 12(11):1749-55 (2002); Albert et al., Nucl. Acids Res., 31(7):e35 (2003); и патенты США 5578832, 5556752 и 5510270. Также могут использоваться другие способы быстрого синтеза и осаждения определенных олигонуклеотидов. См., например, Blanchard et al., Biosensors & Bioelectronics, 11:687-90 (1996); и Maskos and Southern, Nucl. Acids Res., 20:1679-1684.
Матрицы по настоящему изобретению обычно содержат по меньшей мере 100 (например, по меньшей мере 500, 1000, 5000 или 10000) олигонуклеотидных зондов, и могут содержать намного большее количество зондов, например, 25000, 50000, 75000, 85000, 100000, 200000, 250000, 500000 или 700000 зондов. Матрицы по настоящему изобретению обычно содержат не более 700000 зондов. Однако они могут содержать больше, например, более 800000, 900000 или 1000000 зондов. В некоторых вариантах осуществления матрицы представляют собой матрицы с высокой плотностью, с плотностями, превышающими примерно 60 различных зондов на 1 см2. Олигонуклеотиды в матрицах могут быть однонитевыми или двухнитевыми. Для простоты производства и использования матриц олигонуклеотидные зонды по настоящему изобретению могут быть модифицированы, например, путем введения в зонды пептидильной структуры и нуклеотид-аналогов.
III. ТЕСТИРУЕМЫЕ РЕПРЕЗЕНТАТИВНЫЕ ВЫБОРКИ
Олигонуклеотидные матрицы по настоящему изобретению могут использоваться для изучения любого образца нуклеиновой кислоты по выбору. Например, образец может быть библиотекой ДНК, библиотекой геномной ДНК или препаратом РНК. В других вариантах осуществления матрицы по настоящему изобретению могут использоваться для изучения образцов ДНК, которые являются репрезентативными выборками (или «тестируемыми репрезентативными выборками») популяции сложной ДНК, такой как геном более высокоразвитого организма.
Репрезентативные выборки и способы их получения описаны, например, у Lisitsyn et al., Proc. Natl. Acad. Sci. USA, 92:151(1995); Lucito et al., Proc. Natl. Acad. Sci. USA, 95:4487-4492(1998) и WO 99/23256. Один из подходов в создании репрезентативной выборки включает воспроизводимое расщепление ДНК-популяции на фрагменты. Воспроизводимое расщепление обычно выполняется расщеплением одной или несколькими рестрикционными эндонуклеазами (например, DpnI или BglII) или ферментом(ферментами), который выполняет расщепление в конкретных метилированных сайтах (например, McrBC), но может быть использован любой способ, который воспроизводимо расщепляет ДНК. Полученные фрагменты ДНК связывают с адаптером олигонуклеотидов. Затем эти фрагменты амплифицируют, например, с помощью полимеразной цепной реакции («ПЦР») или лигазной цепной реакции, используя праймеры, комплементарные адаптерам. Амплифицированные фрагменты представляют собой подгруппу начальной ДНК-популяции. Благодаря этапу амплификации репрезентативные выборки могут быть получены из очень небольших количеств начального материала (например, 5 нг ДНК). Репрезентативный дифференциальный анализ («RDA»), как описано у Lisitsyn et al., Science 258:946-51(1993) и патентах США 5436142 и 5501946, может быть использован для удаления любых известных нежелательных последовательностей из репрезентативной выборки, включая повторяющиеся последовательности.
Начальная популяция ДНК может представлять собой большие молекулы ДНК, такие как геном организма или его часть (например, хромосома или ее участок). Авторы называют репрезентативные выборки таких ДНК-популяций, как хромосомные или геномные репрезентативные выборки соответственно. Начальные ДНК-популяции могут быть получены, например, из образцов больных тканей, таких как образцы биопсии опухоли, образцов нормальных тканей, клеточных линий опухолей, нормальных клеточных линий, клеток, сохраненных в качестве фиксированных образцов, образцов аутопсии, образцов судебной экспертизы, образцов палео-ДНК, микропрепарированных образцов тканей, изолированных ядер, изолированных хромосом или участков хромосом и фракционированных клеток или образцов тканей. Также могут быть получены репрезентативные выборки из репрезентативной выборки (или «сложные репрезентативные выборки»). Сложные репрезентативные выборки могут использоваться для скрининга полиморфизма. См., например, WO 99/23256.
Для сравнительного анализа репрезентативных выборок двух источников ДНК, такого как сравнение геномных фрагментов из нормальной клетки с геномными фрагментами из раковой клетки или клетки с другим заболеванием, предпочтительным может быть параллельное получение двух репрезентативных выборок, например, изоляцией начальной ДНК из двух клеток в одно и то же время и одним и тем же способом, получением репрезентативных выборок из одинакового количества начальной ДНК и амплификацией фрагментов ДНК в одно и то же время в одинаковых условиях в одном и том же термальном циклере. Также предпочтительным может быть то, что нормальную клетку и больную клетку берут от одного и того же индивида, хотя возможно получение «нормальной» геномной ДНК путем комбинации, например, ДНК обоих родителей данного индивида.
Сложность репрезентативных выборок обычно бывает ниже сложности начальной ДНК-популяции, поскольку существуют последовательности, присутствующие в начальной популяции, которые не присутствуют в репрезентативной выборке. Сложность репрезентативной выборки связана с частотой разрезания рестрикционными эндонуклеазами в конкретной начальной популяции. Разрезания с большей частотой приводят к более сложной репрезентативной выборке. Поскольку фрагменты между 200-1200 парами оснований предпочтительно амплифицируют с помощью ПЦР в обычных условиях, могут быть получены наборы фрагментов высокой сложности путем расщепления начальной ДНК таким образом, что большинство фрагментов будут длиной 200-1200 пар оснований. Напротив, репрезентативные выборки низкой сложности могут быть получены расщеплением молекулы ДНК таким образом, чтобы меньшее количество фрагментов будут длиной 200-1200 пар оснований. Например, расщепление геномной ДНК человека с помощью DpnII может привести к набору фрагментов, имеющему около 70% сложности всего человеческого генома. С другой стороны, расщепление путем разрезания с меньшей частотой, такое как расщепление BamHI или BglII, может привести к репрезентативной выборке, имеющей только около 2% сложности человеческого генома. Репрезентативные выборки высокой сложности пригодны, например, для определения числа копий гена, картирования делеций, определения потери гетерозиготности, сравнительной геномной гибридизации и архивирования ДНК. Обычно репрезентативные выборки низкой сложности полезны для тех же целей, но дают лучшие кинетические характеристики гибридизации, чем репрезентативные выборки высокой сложности.
Сложность репрезентативной выборки может быть дополнительно уточнена путем использования более одного рестрикционного фермента для получения фрагментов перед лигированием с адаптерами, и/или путем использования одного или нескольких дополнительных рестрикционных ферментов для расщепления подгруппы фрагментов после лигирования с адаптерами, таким образом, укорачивая полученные нуклеотидные фрагменты из этих репрезентативных выборок фрагментов. Любой рестрикционный фермент, включая рестрикционные ферменты, чувствительные к метилированию, может быть использован для получения репрезентативной выборки для анализа, как описано в настоящем описании.
Сложность репрезентативной выборки также может быть уточнена с помощью выбора адаптеров, используемых для амплификации. Например, то, какие адаптеры используются, может влиять на размеры элементов репрезентативной выборки. Когда оба конца расщепленных фрагментов сшиты с идентичными адаптерами, формирование «шпильки» между адаптерами в одиночных нитях конкурирует с праймерами отжига, таким образом, ингибируя амплификацию с помощью ПЦР. См. Lukyanov et al., Anal. Biochem., 229:198-202 (1995). Амплификация более коротких фрагментов, по-видимому, ингибируется сильнее, поскольку в более коротких фрагментах адаптеры расположены ближе друг к другу, приводя к более высокой эффективной локальной концентрации сшитых адаптеров и тем самым к более высокому взаимодействию. Адаптеры, которые формируют «шпильки» из приблизительно 29 пар оснований, предоставляют возможность для амплификации фрагментов размером в диапазоне 200-1200 пар оснований. Адаптеры, которые формируют более короткие «шпильки», например из 24 пар оснований, исключают ингибирование некоторых более коротких фрагментов, способствуя получению более мелких продуктов ПЦР амплификации и, следовательно, репрезентативной выборке с измененной сложностью.
IV. ГИБРИДИЗАЦИЯ ОБРАЗЦОВ НУКЛЕИНОВЫХ КИСЛОТ В МАТРИЦАХ
Микроматрицы по настоящему изобретению обычно гибридизуются с образцами однонитевых нуклеиновых кислот в растворе. Поскольку потенциальный сигнал гибридизации может отличаться от сайта к сайту в гибридизационной камере, матрица зондов предпочтительно может использоваться в качестве устройства для сравнения сигналов, измеряющего соотношение гибридизации между двумя по-разному меченными пробами (образцами), которые являются тщательно перемешанными и, следовательно, находятся в одних и тех же условиях гибридизации. Обычно используются по две пробы из тестируемых (например, расщепленных) и контрольных (например, нерасщепленных) ячеек соответственно.
Образцы, предназначенные для гибридизации на микроматрице, например вышеописанные тестируемые репрезентативные выборки, могут быть меченными для визуализации любым способом, известным специалистам в данной области техники. В некоторых вариантах осуществления образцы метят флуоресцентыми агентами, например, меткой случайных праймеров или ник-трансляции. Если образец является репрезентативной выборкой, то он может быть помечен во время этапа амплификации путем введения в реакцию меченых нуклеотидов. Флуоресцентная метка может быть, например, нуклеотидом, соединенным с лиссамином, или нуклеотидным аналогом, соединенным с флуоресцеином. В некоторых вариантах осуществления используются два по-разному меченных образца (например, один помечен лиссамином, а другой флуоросцеином). В некоторых вариантах осуществления образцы являются немеченными.
Условия гибридизации и промывки выбирают таким образом, чтобы молекулы нуклеиновых кислот в образце специфически связывались с комплементарными олигонуклеотидами на матрице. Матрицы, содержащие двухнитевые олигонуклеотиды, обычно подвергают денатурации для того, чтобы получить однонитевые нуклеотиды до взаимодействия с образцом. Оптимальные условия гибридизации будут зависеть от длины и типа (например, РНК или ДНК) олигонуклеотидного зонда и нуклеиновых кислот образца.
Гибридизацию на матрице по настоящему изобретению можно обнаружить любым способом, известным специалистам в данной области техники. В некоторых вариантах осуществления гибридизацию флуоресцентно-меченных нуклеотидов образца детектируют с помощью лазерного сканера. В некоторых вариантах осуществления гибридизацию меченных или немеченных нуклеотидов образца детектируют с помощью измерения их массы. Если используются две различные флуоресцентные метки, сканер может представлять собой такой сканнер, который способен регистрировать флуоресценцию при более одной длине волны, соответствующей длине волны каждой флуоресцентной метки, обычно одновременно или почти одновременно.
V. ИЛЛЮСТРАТИВНОЕ ИСПОЛЬЗОВАНИЕ ОЛИГОНУКЛЕОТИДНЫХ ЗОНДОВ
Олигонуклеотидные зонды по настоящему изобретению могут использоваться для визуализации и количественного определения изменений в количестве копий или уровня метилирования специфических последовательностей в геноме. Когда репрезентативные выборки, полученные из множества образцов ДНК, гибридизуют с одинаковыми олигонуклеотидными зондами, относительная интенсивность гибридизации между двумя образцами и конкретным зондом указывает на относительное число копий или уровень метилирования последовательности, соответствующей такому зонду в двух образцах. Геномы, например, обычно содержат либо избыточные копии конкретных последовательностей в результате амплификации, либо меньше копий конкретной последовательности, либо не содержат их в результате делеции конкретных участков. Такие способы могут использоваться, например, для анализа различий числа копий или уровня метилирования последовательностей в эталонном образце и образцах пациента, где амплификация, делеция или уровень метилирования конкретных последовательностей связаны, например, с предрасположенностью, развитием или стадией определенных заболеваний, включая, например, рак, неврологическое заболевание (например, аутизм), диабет, кардиологические заболевания и воспалительные заболевания (например, аутоимунные заболевания).
Кроме того, может быть получена позиционная информация при изменении числа копий или уровня метилирования в геноме, поскольку известны последовательности в геноме, к которым олигонуклеотидные зонды по настоящему изобретению являются комплементарными. Если олигонуклеотидные зонды разработаны для частой гибридизации с последовательностью генома, и образец является репрезентативной выборкой высокой сложности, возможно точное картирование участков амплификации, делеции или уровня метилирования генома. Таким образом, настоящее изобретение может быть использовано для идентификации отдельных генов, которые могут влиять на предрасположенность, развитие или стадию конкретных заболеваний. Такие гены могут быть онкогенами и генами-супрессорами опухолей, в зависимости от того является ли последовательность амплифицированной, делетированной или метилированной/неметилированной в раковом геноме относительно контрольного генома соответственно.
Олигонуклеотидные зонды по настоящему изобретению также могут использоваться для идентификации полиморфных сайтов, включая единичные нуклеотидные полиморфизмы (SNP) как в одном индивиде, так и между индивидами. Такие полиморфизмы являются обычными, и до 2-3% олигонуклеотидных зондов проявляют полиморфное поведение даже между «нормальными» индивидами. Выявляемые полиморфизмы могут быть результатом потери или увеличения фрагментов рестрикционных эндонуклеаз, например, вследствие точечных мутаций, делеций, геномных перестановок или генных конверсий, распространяющихся на гетерозиготные полиморфизмы, там где они присутствуют или отсутствуют в репрезентативной выборке. Например, расщепление нуклеотидной последовательности рестрикционным ферментом может привести в результате к одному большому (т.е. нерасщепленному) или двум небольшим фрагментам, в зависимости от того, есть ли сайт рестрикции. Известно, что такой полиморфный рестрикционный сайт существует в тестируемом геноме, если олигонуклеотидные зонды детектируют один или оба небольших фрагмента в тестируемой репрезентативной выборке.
Аналогично геномные перестановки, включая транслокации, инсерции, инверсии и делеции, могут в результате привести к получению новых фрагментов рестрикционных эндонуклеаз, охватывающих по меньшей мере часть перестроек. Некоторые из таких новых фрагментов могут быть амплифицируемыми и, следовательно, присутствовать в репрезентативной выборке генома с перестановками, но отсутствовать в контрольной репрезентативной выборке. Напротив, геномные перестановки могут привести к потере фрагмента репрезентативной выборки. В любом случае разница в тестируемых и контрольных репрезентативных выборка при гибридизации с некоторыми зондами подтверждает, что геномные перестановки могут происходить в тестируемом геноме относительно контрольного генома. Путем анализа последовательностей этих зондов и местоположений этих зондов в контрольном геноме можно получить информацию о генетических перестановках, включая тип перестановок и участки соединений перестановок.
Возможность анализа числа копий и других полиморфизмов специфических последовательностей индивида и между индивидами имеет многочисленные возможности для применения, которые очевидны специалистам в данной области техники. Это могут быть, без ограничений, идентификация индивидов, например, для судебной экспертизы и установления отцовства; скрещивание растений или животных; обнаружение полиморфизма, который генетически связан с наследственными признаками, включая предсказание благоприятного или отрицательного ответа на лекарственный препарат; диагностика; и идентификации и стратификации пациентов в клинических исследованиях.
VI. ИЛЛЮСТРАТИВНЫЙ МЕХАНИЗМ ПОИСКА
Ниже описан алгоритм, который может быть использован для получения вышеуказанных олигонуклеотидных зондов. Очевидно, что нижеследующее описание не предназначено для демонстрации того, что этот алгоритм предназначен только для получения таких зондов. Следует также принять во внимание, что этот алгоритм имеет другие приложения, чем получение нуклеотидных зондов по настоящему изобретению. Некоторые такие приложения рассмотрены в настоящем описании.
Этот алгоритм, иногда называемый в настоящем описании механизмом поиска или mer-механизмом, использует преобразование генома (например, преобразование Барроуза-Уиллера) и вспомогательную структуру данных для подсчета количества конкретного «слова» в геноме. «Слово» означает нуклеотидную последовательность любой длины.
В общем случае, с помощью механизма производится поиск конкретного «слова», сначала выполняя поиск последней «буквы слова». Затем он переходит к поиску «буквы», непосредственно предшествующей последней «букве». Если первая непосредственно предшествующая «буква» найдена, производится поиск второй «буквы», непосредственно предшествующей последней «букве слова», и т.д. до тех пор, пока не будет найдено «слово». Если далее следующие предшествующие «буквы» отсутствуют, механизм делает вывод, что данное «слово» отсутствует в геноме.
Преимущество такого конкретного алгоритма состоит в его использовании для осуществления нескольких конкретных применений, включая исследования генома, как описано выше. Одним из применений механизма поиска является его использование для аннотирования нуклеотидной последовательности, такой как геном. В частности, геном может быть аннотирован с использованием подстрок конкретной длины, которые существуют в геноме. Затем с помощью механизма поиска можно произвести подсчет частоты встречаемости подстрок конкретной длины в геноме. Эти частоты встречаемости являются показателем уникальности конкретной подстроки, где более низкие частоты встречаемости соответствуют более высокой степени уникальности по сравнению с более высокой частотой встречаемости.
Разработка зонда является другим конкретным приложением, преимущество которого усиливается при использовании механизма поиска. Способность механизма поиска к выполнению быстрого подсчета количества конкретного «слова» в геноме является особенно полезной при разработке зондов, которые являются уникальными и гибридизуются со специфичным участком ДНК с минимальным уровнем перекрестной гибридизации. При использовании механизма поиска может быть минимизирована вероятная перекрестная гибридизация путем наложения требований, чтобы зонд состоял из составляющих сегментов, которые являются уникальными и удовлетворяют некоторым жестким условиям, таким как низкая частота встречаемости «слов» или нулевая частота встречаемости «слов» во всем геноме.
Другим приложением механизма поиска является визуализация различий между двумя геномами. Например, по мере развития проекта «Геном человека», картируются и становятся общедоступными новые сегменты генома. Используя механизм поиска и зонды, которые разработаны исходя из другой версии того же самого генома, можно определить, какое количество этих зондов может быть применено к новой версии генома.
Еще одним приложением, где может быть использован механизм поиска, является проверка существования конкретного «слова» в геноме. Может быть желательным произвести поиск «слов», которые не появляются в геноме, с тем чтобы существовал небольшой шанс того, что данное «слово» будет гибридизоваться с участком генома. Такие «слова» могут быть генерированы случайным образом в соответствии с заданным набором критериев. Если «слово» обнаружено, его комплемент также направляется в поисковый механизм для определения, появляется ли он в геноме. Если и «слово», и его комплемент не появляются в геноме, известно, что оба эти «слова» будут гибридизоваться друг с другом, а не с геномом.
А. ОПИСАНИЕ СИСТЕМЫ
Механизм поиска и его приложения могут выполняться согласно настоящему описанию с использованием иллюстративной системы 700, показанной на фиг.7. Система 700 может включать в себя компьютер 710, оборудование 730 пользовательского интрефейса, интернет 740 и необязательное лабораторное оборудование (не показано). Система 700 может включать в себя множество компьютеров 710 и комплектов оборудования 730 пользовательского интерфейса, но на фиг.7 показано только по одному из них во избежание усложнения чертежа. Компьютер 710 показан соединенным с оборудованием 730 пользовательского интерфейса и интернетом 740 через линии 790 связи.
Компьютер 710 может включать в себя схему, такую как процессор 712, базу 714 данных (например, жесткий диск), память 716 (например, оперативное запоминающее устройство) и привод 718 со сменным носителем (например, привод гибкого диска, привод CD-ROM или привод DVD). Такая схема может быть использована для передачи данных к оборудованию 730 пользовательского интерфейса, от него и/или обмена данными между оборудованием 730 пользовательского интерфейса и Интернетом 740. Компьютер 710 может инициировать способы настоящего изобретения, отвечая на данные ввода пользователя через оборудование 730 пользовательского интерфейса. Компьютер 710 также может предоставлять информацию пользователю через оборудование 730 пользовательского интерфейса в соответствии с результатами, полученными при работе механизма поиска.
База 714 данных хранит информацию, которая снабжает поисковый механизм данными. Более конкретно, база 714 данных может включать в себя последовательность генома или конкретной части генома. Настоящее изобретение может использовать информацию о геноме, сохраненную в базе 714 данных, для конструирования массива суффиксов, который также может быть сохранен в базе 714 данных. Массив суффиксов представляет собой структуру данных, которая генерируется при подготовке построения преобразования генома или его части. Репрезентативные данные генома могут быть получены, например, из читаемого носителя (например, гибкого диска, CD-ROM или DVD), доступ к которым может выполняться через накопитель 718 со сменным носителем. В качестве альтернативы данные генома могут быть получены через интернет 740, где данные передаются от сервера, расположенного, например, в исследовательском учреждении (например, Национальном Институте Здоровья или университете). Если это необходимо, база 714 данных может обновляться новыми данными генома по мере того, как они становятся доступными.
Обычно количество данных, представляющих массив суффиксов, существенно больше количества данных, представляющих геном. Следовательно, база 714 данных может быть более подходящей для хранения массива суффиксов, чем память 712, поскольку база данных может сохранять больший объем данных, чем память.
Оборудование 730 пользовательского интерфейса позволяет пользователю вводить в компьютер 710 команды через устройство 732 ввода. Устройство 732 ввода может быть любым подходящим устройством, таким как обычная клавиатура, беспроводная клавиатура, мышь, сенсорная панель, трекбол, консоль с речевым управлением или любая комбинация таких устройств. Устройство ввода 732 может, например, позволять пользователю вводить команды для определения частоты встречаемости «слова» конкретного «слова» или выполнения статистического анализа потенциальных зондов. Пользователь может отслеживать процесс обработки в системе 700 на устройстве 734 отображения. Устройство 734 отображения может быть монитором компьютера, телевизором, плоскопанельным телевизором, жидкокристаллическим дисплеем, электронно-лучевой трубкой (ЭЛТ) или любым другим подходящим устройством.
Линии 790 связи могут быть любыми подходящими линиями связи, такими как кабельное соединение, проводное соединение, оптоволоконное соединение, инфракрасное соединение, шинное соединение, соединение «Bluetooth», аналоговое коммуникационное соединение, цифровое коммуникационное соединение или любая комбинация таких соединений. Линии 790 связи выполнены с возможностью передачи данных между компьютером 710, оборудованием 730 пользовательского интерфейса и интернетом 740.
В системе 700 может быть предусмотрено лабораторное оборудование для того, чтобы результаты, полученные с помощью механизма поиска, могли быть сразу применены в экспериментах и наоборот.
Преимущество механизма поиска заключается в том, что способы подсчета точных совпадений «слов» могут происходить в памяти (например, памяти 716) компьютера. Это обеспечивает чрезвычайно быстрое и эффективное исследование генома на точные совпадения «слов». Отсутствует необходимость в доступе к базе данных (например, жесткому диску). Такая необходимость может существенно снизить производительность механизма поиска. Способы, используемые для подсчета точных совпадений «слов» имеют 100% точность.
В. МАССИВ СУФФИКСОВ, ПРЕОБРАЗОВАНИЕ БАРРОУЗА-УИЛЛЕРА И «БУКВЫ-ГРАНИЦЫ»
Приведенная на фиг.8 иллюстративная блок-схема 800 последовательности операций показывает этапы подготовки генома для использования в механизме поиска согласно принципам настоящего изобретения. Блок-схема 800 последовательности операций использует способы для построения структуры данных массива суффиксов, которая обеспечивает основу для получения преобразования конкретного генома. Такое преобразование обеспечивает основу для механизма поиска по настоящему изобретению, где механизм поиска может быстро подсчитать количество появлений конкретного «слова» (например, «слова», имеющего длину 15, 21, 70 или 80 «букв»). На этапе 810 получают нуклеотидную последовательность, такую как геном или часть генома. Геном может быть упорядочен в виде строки «букв», имеющей длину N нуклеотидов, где N означает общее количество нуклеотидов в строке «букв», представляющих геном.
Геном, предоставленный на этапе 810, может быть получен из любого организма или он может быть получен случайным образом. Например, может быть получен полностью известный геном человека или может быть получена часть генома человека (например, часть генома, представляющая хромосому или участок хромосомы). Если это необходимо, может быть получен нечеловеческий геном, такой как геном вируса, бактерии, одноклеточного или многоклеточного организма, включая дрожжи, растения и животных, таких как ящерицы, рыбы и млекопитающие (например, мыши, крысы и приматы, кроме человека).
На этапе 820 геном подвергают процессу преобразования, который реорганизует расположение нуклеотидов генома согласно заданному лексикографическому порядку. Преобразование использует такие же составляющие буквы (например, A, C, G и T), которые появляются в геноме, но эти буквы расположены в другом порядке. В одном из вариантов осуществления геном подвергают известному преобразованию, называемому преобразованием Барроуза-Уиллера. Преобразование Барроуза-Уиллера может быть получено из массива суффиксов. Согласно настоящему изобретению массив суффиксов представляет собой матрицу N×N, представляющую все циклические пермутации генома, где пермутации располагают согласно заданным критериям (например, алфавитному, цифровому и т.п.). Преимущественно преобразование Барроуза-Уиллера представляет собой отсортированную матрицу N×N циклических пермутаций. Таким образом, если механизм поиска по настоящему изобретению производит поиск с помощью преобразования Барроуза-Уиллера, он путем расширения производит поиск по массиву суффиксов, который путем дополнительного расширения производит поиск по исходной строке, представляющей геном.
Совокупность последовательностей генома может включать неопределенную «букву» дополнительно к A, C, G и T, таким образом, увеличивая алфавит генома до пяти «букв». Такая неопределенная «буква», обычно называемая N, как правило, используется, если неизвестен нуклеотид в конкретном положении последовательности нуклеиновых кислот.
Поскольку преобразование Барроуза-Уиллера представляет собой отсортированный массив суффиксов, отсутствует необходимость доступа к массиву суффиксов при проведении поиска для конкретной строки «букв». Предпочтительно, преобразование сохраняют в памяти, где поисковые функции могут быть выполнены гораздо быстрее, чем при хранении преобразования на жестком диске. Кроме того, поскольку количество данных, содержащихся в массиве суффиксов, может быть существенным, возможно, что массив суффиксов должен храниться на жестком диске, а не в более быстрой оперативной памяти (например, оперативное запоминающее устройство в компьютере). Например, размер массива суффиксов для генома человека составляет порядка двенадцати гигабайт. Если такой массив сохранить в памяти, стоимость машины, имеющей двенадцать гигабайт памяти, будет намного превышать стоимость машины, имеющей, например, три гигабайта памяти. Следовательно, одним из преимуществ механизма поиска является отсутствие необходимости в дорогостоящих и интенсивно использующих память машинах, поскольку преобразование представляет собой сжатую версию отсортированного массива суффиксов.
Поскольку согласно настоящему изобретению массив суффиксов не является необходимым при выполнении поиска «слова», полезно описать, каким образом получают такие массивы с тем, чтобы показать взаимосвязь между преобразованием и массивом. Массив суффиксов можно построить сначала путем получения циклических пермутаций нуклеотидной последовательности. Например, в Таблице 1 показаны циклические пермутации генома «AGACAGTCAT$», где «$» предусмотрен для маркировки конца строки генома.
После получения циклических пермутаций, строки сортируют согласно заданным критериям для получения конкретного лексикографического порядка (например, алфавитного лексикографического порядка). Например, в Таблице 2 показано размещение пермутаций, приведенных в Таблице 1, в алфавитном порядке под заголовком «Отсортированный массив».
После сортировки циклических пермутаций можно получить преобразование генома, беря последнюю букву каждой строки отсортированного массива. Такие буквы представлены в столбце, озаглавленном «Преобразование», указывающем, что преобразование генома «AGACAGTCAT$» представляет собой «TG$CCATAAAG».
В одном из вариантов осуществления массив суффиксов генома, такого как геном человека, может быть построен с использованием параллельной поразрядной сортировки, используя 16-узловой кластер. При использовании этого подхода геном подразделяют на Х (например, 100) подстрок одинакового размера, где каждая покрывает около семи нуклеотидов, где Х представляет собой заданное число. Смещения в геноме (т.е. координата «генома») в каждой подстроке назначаются одной из 57 ячеек «префикса» согласно 7-mer (7 нуклеотидам) каждого смещения. Смещения в каждой ячейке «префикса» сортируют, основываясь на последовательности, следующей после 7-mer префикса, таким образом создавая массив суффиксов.
На этапе 830 вычисляют различные статистики для получения вспомогательной структуры данных, которая может включать в себя структуру данных «букв-границ» (буквы, находящаяся на границе строки), структуру данных К-интервалов и структуру данных подсчета «слов». «Буквы-границы» указывают, сколько адениновых, цитозиновых, гуаниновых и тимидиновых нуклеотидов находится в преобразовании. Например, используя геном Таблиц 1 и 2, «буквы-границы» для A, C, G и T составляют 4, 2, 2 и 2 соответственно.
«Буквы-границы» могут использоваться для определения границ областей в преобразовании, которые соответствуют конкретным «буквам», находящимся впереди каждой строки отсортированного массива суффиксов. Например, область с определенными границами для нуклеотида А включает в себя каждую строку массива суффиксов, которая начинается с А. В Таблице 2 показано, что строки 1-4 отсортированного массива начинаются с А. Таким образом, четыре строки соответствуют «буквам-границам», вычисленным для А. В Таблице 2 показано, что строки 5-6 начинаются с С, которые соответствуют «буквам-границам», вычисленным для С. Аналогично, блок G соответствует строкам 7 и 8, а блок Т - строкам 9 и 10 преобразования.
На этапе 830 можно также генерировать К-интервалы для каждого количества К «букв» в преобразовании, где К представляет собой заданное число. К-интервалы могут использоваться для сохранения промежуточной суммы для каждого нуклеотида, когда они появляются в преобразовании. Эти К-интервалы могут использоваться в случае механизма поиска по настоящему изобретению для ускорения процесса подсчета, который обсуждается ниже в связи с фиг.3 и фиг.4. В частности, использование К-интервала позволяет достигать в случае механизма поиска большего быстродействия и использовать меньший объем, чем обычные способы подсчета «слов», особенно применительно к нуклеотидным последовательностям, превышающим в длину четыре миллиона «букв».
Нижеследующий пример дополнительно объясняет, каким образом преобразование сводится в таблицу с использованием К-интервалов. Предположим, что преобразование имеет десять «букв» ACGTCAGTCA, и с помощью К-интервалов сохраняются каждые пять «букв». В первом интервале К-интервал включает в себя одну А, две C, одну G и одну T. Во втором интервале (например, десятая «буква») К-интервал включает в себя сведение в таблицу всех нуклеотидов, которые присутствовали в преобразовании до сих пор. Второй К-интервал включает в себя три A, три C, две G и две T.
На этапе 840 строку Барроуза-Уиллера сжимают согласно заданному отношению сжатия. Предпочтительно строку сжимают, используя отношение сжатия 3 к 1. То есть для каждых трех «букв» строку сжимают в одну «букву» (например, 3000 «букв» сожмутся до 1000 «букв»). Специалистам в данной области техники следует принять во внимание, что может быть использовано сжатие четыре к одному или пять к одному. Строка может быть сжата с использованием схемы сжатия, основанной на словаре, где один из 125 отдельных единичных байтовых кодов представляет собой один из каждых 53 возможных трех подстрок "букв" (например, ААА, ААС, …, ТТТ). Более конкретно, преобразование разделяется на подстроки из трех «букв», и каждую подстроку сжимают согласно схеме сжатия на основании словаря. Например, если подстрока из трех «букв» представляет собой ААА, это может быть эквивалентно байту 0 словарной схемы сжатия. Аналогично, если подстрока представляет собой ТТТ, это может быть эквивалентно байту 124 словарной схемы сжатия.
Структура данных подсчета «слов» может быть получена для содействия механизму поиска в процессе подсчета путем предоставления быстрого доступа к поисковой таблице для быстрой идентификации частоты встречаемости конкретной буквы в сжатом байте. Это является преимуществом, поскольку позволяет механизму поиска выполнять операции подсчета в преобразовании, в то время как оно находится в сжатом состоянии. Однако отмечено, что возможно байт должен быть декомпрессирован для того, чтобы механизм поиска завершил подсчет количества конкретной «буквы» в пределах области поиска. В среднем, обнаружено, что байт сжатого преобразования декомпрессируется в двух третях случаев в процессе выполнения механизмом поиска этапа подсчета «букв».
После сжатия преобразования оно готово для использования в механизме поиска настоящего изобретения. В частности, может быть сделан запрос сжатого преобразования Барроуза-Уиллера для определения местоположения и подсчета количества каждого конкретного «слова», содержащегося в геноме.
С. АЛГОРИТМ ПОДСЧЕТА «СЛОВ»
На фиг.9 показана упрощенная блок-схема последовательности иллюстративных этапов подсчета количества конкретных «слов» в данном геноме согласно принципам mer-механизма. На этапе 910 получают сжатое преобразование генома и вспомогательную структуру данных. Сжатое преобразование генома и вспомогательная структура данных могут быть получены, например, из последовательности операций, показанных на фиг.8. На этапе 914 получают заданный образец конкретной длины (например, ACG…G). Образец предпочтительно представляет собой строку нуклеотидов, поиск которой в преобразовании генома осуществляет механизм поиска.
После ввода заданного образца механизм поиска начинает процесс итеративного поиска для определения наличия образца. Если образец существует, механизм поиска быстро и точно выдает количество его появлений. На этапе 918 итеративный процесс начинается с определения (или переопределения) области поиска, которая определяет границы области положений «букв» в пределах преобразования. Область поиска описывает границы блока «букв», начиная с положения Х и заканчивая положением Y сжатого преобразования. Такая область поиска (или блок) потенциально содержит все количество заданного образца. Область поиска определяется с использованием заданных критериев, таких как конкретная «буква» заданного образца, «буквы-границы» и другие данные. Более подробное объяснение того, каким образом определяется область поиска, обсуждается в связи с описанием, сопровождающим фиг.10.
На этапе 920 определяется, сколько раз появляется следующая предшествующая «буква» заданного образца в области поиска. На этапе 922, если частота встречаемости обрабатываемой «буквы» равна нулю, то заданный образец отсутствует, и процесс завершается (этап 924). Если в ограниченной области обнаруживается по меньшей мере одна «буква», процесс переходит к этапу 926. На этапе 926 определяют, является ли предшествующая «буква» первой «буквой» в заданном образце. Если это так, то процесс переходит к этапу 928, на котором выдают частоту встречаемости, полученную на этапе 920, и процесс завершается. Если предшествующая «буква» не является первой «буквой» заданного образца, то процесс возвращается к этапу 918, поскольку еще не определено, существует или нет заданный образец в геноме. На этапе 918 механизм поиска переопределяет заданные критерии.
Более конкретно, область поиска переопределяют, используя приведенные ниже уравнения 1 и 2:
где А представляет собой начальное положение предшествующей «буквы» согласно «буквам-границам», Z представляет собой частоту встречаемости предшествующей «буквы» в преобразовании до момента определения текущей области поиска, и М представляет собой частоту встречаемости предшествующей «буквы» в текущей области поиска.
Переопределенная область поиска также потенциально содержит все появления заданного образца, но заново определенная область поиска дополнительно ограничивает положения «букв», поиск которых необходимо произвести на этапе 920. После определения новой области поиска процесс переходит к этапу 920, на котором подсчитывают следующую предшествующую «букву» (т.е. «букву», предшествующую последней «букве», использованной на предыдущем этапе 920) заданного образца в пределах заново определенной области поиска. Такой цикл может повторяться столько раз, сколько это необходимо для обнаружения первой «буквы» заданного образца, и, следовательно, выполнения подсчета «слов». Если одну из предшествующих «букв» в области поиска не находят, делают вывод, что такой образец отсутствует в геноме.
На фиг.10А-В показан пример вышеприведенного алгоритма подсчета «слов». Этот пример использует иллюстративный геном (AGACAGTCAT$), массив суффиксов, преобразование Барроуза-Уиллера (TG$CCATAAAG) и «буквы-границы», ранее описанные в связи с Таблицами 1 и 2. В этом примере предполагается, что пользователь хочет определить, сколько раз слово «СAG» появляется в геноме.
На фиг.10А процесс начинается с определения границ блока G, поскольку G представляет собой последнюю «букву» в слове «CAG». Как показано, блок G начинается с положения 7 и заканчивается в положении 8 преобразования Барроуза-Уиллера. Эти положения получены из «букв-границ». После определения границ блока G механизм выполняет поиск и подсчитывает количество A, следующей предшествующей «буквы» «CAG», находящихся в блоке G. На фиг.10А показано, что в блоке G появляются две A, таким образом, указывая, что геном содержит два «AG».
Если необходимо, К-интервалы могут использоваться для облегчения этапа подсчета количества конкретной «буквы» в области поиска (например, подсчета количества A в блоке G), и также могут использоваться для подсчета количества появлений конкретной буквы перед областью поиска. Для выполнения этого этапа подсчета выполняют подсчет конкретной «буквы», начиная с заданного положения (например, начального положения) и продвигаясь к следующему самому близкому положению, которое представляет собой множество К. Преимущество использования К-интервалов в механизме поиска заключается в том, что время, которое затрачивается для определения количества конкретного «слова» в геноме, является линейным относительно К-интервалов, размера искомого «слова» и времени, требуемого для доступа к различным адресам памяти. Таким образом, размер генома не является фактором при определении частоты встречаемости «слова», если только размер сжатого преобразования и структура данных К-интервала не являются слишком большими для размещения их в памяти (например, оперативной памяти). В одном из вариантов осуществления К может быть задано равным 300 «буквам» или эквивалентно 100 сжатым байтам. При такой конфигурации максимальное количество подсчетов, которое необходимо выполнить, не превышает К/2.
Если необходимо, подинтервалы размера К∧ в каждом К-интервале могут использоваться для сохранения промежуточной суммы для каждой «буквы», появляющегося в конкретном К-интервале. Если размер К ограничен таким образом, что он должен быть, например, меньше 28, то результаты подсчетов каждой «буквы» в каждом К-интервале могут быть записаны с использованием одного байта. Это обеспечивает увеличение плотности индекса подсчета на коэффициент К/К∧ при увеличении требований к объему для подсчетов К-интервалов только на коэффициент [(К/К∧)/4]. Такие ограничения подинтервалов и размеров были использованы при помощи вспомогательной структуры данных, которая была применена в этом алгоритме. В зависимости от выбора К и К∧ достигалось от трех- до пятикратного увеличение скорости исполнения запроса при сохранении требований к памяти менее двух гигабайт для генома человека.
Для дальнейшего ускорения процесса подсчета может быть использована структура данных подсчета «слов». Необходимо отметить, что используемая схема сжатия представляет собой схему сжатия 3:1, где байты 0-124 сжимают до «AAA» - «TTT» соответственно. Структура подсчета «слов» представляет собой двумерный массив, который может быть представлен в виде матрицы из 125 строк и 5 столбцов. Каждая строка соответствует одной из сжатых словарных записей, а каждый столбец соответствует каждой букве алфавита генома, от А до Т. Ниже объясняется при помощи примера, каким образом может использоваться структура подсчета «слов» и К-интервалы для выполнения операций подсчета.
Например, предположим, что механизм поиска находится в процессе определения количества A, которые появляются до области поиска. Используя структуру подсчетов с помощью К-интервалов, описанную выше, механизм может «перескакивать» по меньшей мере в пределах по меньшей мере 50 байт от текущего начального положения области поиска за один просмотр. Далее, предположим, что начальное положение указывает на третью «Т» в сжатом «ATT» (байте), который представляет собой 49-й байт данного интервала. Для каждых 48 предшествующих байтов сам байт может использоваться в качестве номера строки в структуре данных подсчета «слов», а интересующая буква «A» представляет номер столбца. Используя эту информацию в качестве координат для получения доступа к массиву подсчета «слов», структура данных подсчета «слов» предоставляет количество «А», находящееся в этом сжатом байте. Следовательно, для определения количества появлений A до начала области поиска необходимо выполнить доступ к структуре подсчетов «слов» 48 раз. Кроме того, 49-й байт возможно должен быть декомпрессирован для проверки первых двух букв «АТ» байта «ATT».
Таким образом, если структура данных подсчета «слов» объединена со структурой данных К-интервалов, этап подсчета любого количества «букв» требует только К/6+1 просмотров таблицы, плюс два сравнения «букв», в худшем случае.
Как показано на фиг.10, затем механизм поиска определяет границы блока AG в преобразовании для того, чтобы знать, где производить поиск следующего предшествующей «буквы». Границы блока AG определяют путем добавления количества предшествований А блоку G в преобразовании к первому положению, где блок А начинается в преобразовании. В этом примере только одно А появляется перед блоком G. Следовательно, используя уравнение 1, приведенное выше, где А равно 1 и Z равно 1, для блока AG получают начальное положение 2. Конечное положение AG получают с помощью уравнения 2, приведенного выше, где М равно 2 (количество A, обнаруженных в блоке G). Уравнение 2 дает конечное положение 3 блока AG, как показано на фиг.10В.
После нахождения блока AG механизм поиска подсчитывает в нем количество С. Такой подсчет дает количество CAG, которое встречается в геноме, поскольку С представляет собой первую «букву» слова «CAG». Таким образом, механизм поиска выдает частоту встречаемости «слова», равную единице.
На фиг.11 показаны иллюстративный геном с координатами положений и отсортированный массив суффиксов с координатами положений, соответствующими координатам положений генома. То есть первая «буква» в каждой строке массива суффиксов соответствует одному из «букв» в геноме. Например, вторая строка массива имеет координату положения, равную 2, которая соответствует положению два в геноме. Таким образом, координаты положений массива суффиксов коррелируют с координатами положений в геноме.
Если необходимо, массив суффиксов может быть использован для определения местоположения координаты положения конкретного «слова». Например, если производится поиск координаты положения «CAG», то осуществляется доступ к массиву суффиксов фиг.11, который указывает, что CAG начинается с положения 3. Однако как упоминалось выше, для получения доступа к массиву суффиксов необходима затрата времени, поскольку процесс требует доступа к жесткому диску. Следовательно, желательно получить координаты «слова» только с помощью выполнения доступа к памяти. Это может быть достигнуто путем назначения заранее выбранных координат массива суффиксов для преобразования, таким образом, давая возможность алгоритму определения координаты местоположения использовать преобразование для определения начальной координаты местоположения конкретного «слова».
Такой алгоритм определения координаты местоположения объяснен с помощью примера. Предположим, что циклическая часть массива суффиксов представляет собой преобразование генома и что только координаты 3 и 7 были вынесены за преобразование из массива суффиксов. Дополнительно предположим, что необходимо найти координаты ТС. (Необходимо отметить, что, если бы преобразование имело координаты, соответствующие G, которая связана с ТС, то координаты ТС были бы известными без выполнения повторной сортировки для осуществления алгоритма определения координаты местоположения). Известно, что ТС связано с последней G в преобразовании. Начиная с этой G алгоритм определяет количество предшествований G. В этом случае имеется одна предшествующая G.
Структура данных «букв-границ» и количество предшествований G используются для определения того, какая «буква» предшествует этому конкретному G. Используя «буквы-границы», известно, что блок G начинается с положения 7. Поскольку существует одна предшествующая G, алгоритм добавляет это значение к 7 и получает 8. Таким образом, А, которая соответствует строке массива суффиксов, которая начинается с GT, представляет собой «букву», которая предшествует вышеупомянутой G. Это завершает одну итерацию алгоритма нахождения координаты местоположения. В общем, эта итерация повторяется до тех пор, пока в преобразовании не достигается координата (например, 3 или 7). После достижения координаты количество итераций добавляют к координате, и полученная сумма является реальной начальной координатой положения требуемого «слова» (например, ТС).
Продолжая процесс итерации, известно, что две A предшествуют А, связанной со строкой массива суффиксов, начиная с GT. Используя «буквы-границы» и количество предшествующих А, алгоритм останавливается на С, связанной с массивом суффиксов, начинающимся с AGT. Поскольку отсутствуют С, предшествующие этой конкретной С, то алгоритм останавливается на А, связанной со строкой массива суффиксов, начинающейся с CAG. Поскольку это А имеет координату положения (например, 3), может быть определено действительное положение «слова» ТС путем прибавления 3 (координаты положения этой А) к количеству итераций, которое в данном примере равно 3, что дает в результате координату положения, равную 6. Таким образом, в исходном геноме ТС начинается с координаты положения 6.
D. ПРИМЕНЕНИЯ МЕХАНИЗМА ПОИСКА
Теперь, поскольку были описаны особенности работы механизма поиска, могут быть обсуждены конкретные применения данного механизма. Одно из применений механизма поиска заключается в том, что он может быть использован для аннотирования генома (или нуклеотидной последовательности другого типа). В частности, геном может быть аннотирован с использованием подстрок конкретной длины, которые существуют в геноме. Затем механизм поиска может выполнить подсчет количества подстрок конкретной длины в геноме. Такое определение частоты встречаемости предоставляет признак уникальности конкретной подстроки, где более низкие частоты встречаемости указывают на более высокую степень уникальности в отличие от более высокой частоты встречаемости.
Если необходимо, любая область генома, или весь геном, может быть аннотирован, исходя из частот его составляющих «mer». Термин «mer» представляет собой еще один термин для «слова» или подстроки конкретной длины. Таким образом, если геном или его часть аннотируются, аннотирование осуществляют, основываясь на mer конкретной длины (например, длинах mer 15, 18, 21 и 24). Вне зависимости от аннотируемой длины mer подсчитывают каждый mer такой длины, который существует в геноме. Например, если длина mer равна 15, механизм поиска будет определять частоту встречаемости «слова» для первого 15-mer и каждого последующего 15-mer, появляющегося после него. Каждый последующий 15-mer перекрывает предыдущий 15-mer «слова» на одну «букву». То есть «буквы» 1-15 составляют 15-mer, «буквы» 2-16 составляют другой 15-mer, «буквы» 3-17 составляют следующий 15-mer и т.д. Это гарантирует, что каждый аннотированный 15-mer определяется частотой встречаемости «слова» таким образом, что частота встречаемости «слова» представляет собой количество этого конкретного 15-mer во всем геноме.
Разработка зонда упрощается при использовании механизма поиска. Способность механизма быстро подсчитывать количество конкретного «слова» в геноме является полезной при разработке зондов, которые являются уникальными и гибридизуются со специфическим участком ДНК с минимальной перекрестной гибридизацией. Путем использования механизма поиска потенциальная перекрестная гибридизация может быть минимизирована путем выбора зонда-кандидата, который содержит меньшие mer, которые являются уникальными, соответствуют некоторым условиям жесткости, таким как наличие низкой частоты встречаемости «слова» или отсутствие «слова» во всем геноме. Уникальное «слово» может быть конкретной строкой нуклеотидов, которая имеет менее заданной величины частоту встречаемости «слов» (например, значения подсчета «слов» меньше 2, 5, 10, 25, 50 или 100) или отсутствие частоты встречаемости «слов» (например, нулевые частоты встречаемости «слов») в геноме или его части.
Более конкретно, зонды-кандидаты получают исходя из набора заданных критериев, таких как требование того, чтобы кандидаты имели длину L1, а также требование того, чтобы кандидаты имели заданную частоту встречаемости «слова» (например, зонд-кандидат, имеющий частоту встречаемости «слова», равную 1). Кроме того, заданные критерии также могут требовать, чтобы обратный комплемент кандидата имел заданную частоту встречаемости «слова» (например, один). После получения кандидатов их подвергают проверке на дополнительные заданные критерии для определения того, какие кандидаты являются подходящими для использования в качестве зондов. Такие дополнительные критерии используются для того, чтобы отфильтровать кандидатов исходя из составляющих их подобластей (т.е. mer некоторой длины, содержащихся в зонде-кандидате). Например, критерий фильтрования может требовать, чтобы mer длиной L2, где L2 меньше L1, имели частоту встречаемости «слов», которая минимизирована относительно других зондов-кандидатов. Таким образом, существует связь между критериями, используемыми при нахождении зондов, - связь между «жесткими» условиями (например, при которых каждый кандидат является уникальным относительно генома) и «мягкими» условиями (например, при которых частота встречаемости составляющих mer минимизирована).
Одним из способов удовлетворения «жестким условиям» является получение кандидатов, исходя из результатов ранее выполненного аннотирования. Используя информацию частоты встречаемости «слова», могут быть выбраны кандидаты из участков генома, имеющие низкие концентрации встречаемости «слов» (например, это является предпочтительным для получения кандидатов, имеющих минимальное среднее значение частоты встречаемости «слов» заранее определенной длины, среднее геометрическое значение частоты встречаемости «слов» заранее определенной длины, значение моды частоты встречаемости «слов» заранее определенной длины, минимизированное максимальное значение частоты встречаемости «слов» заранее определенной длины, суммарное общее значение частоты встречаемости «слов» заранее определенной длины, значение произведения частоты встречаемости «слов» заранее определенной длины, строки максимальной длины конкретного нуклеотида или их комбинации).
Для удовлетворения «мягкому» условию кандидаты могут быть аннотированы согласно заданным критериям, таким как частота встречаемости 15-mer, частота встречаемости 17-mer и др. Данные, полученные из аннотирования, анализируются для определения того, является ли кандидат достаточно уникальным для того, чтобы он был использован в качестве зонда. Кандидат может быть выбран в качестве зонда, если, например, он имеет самую низкую сумму частоты встречаемости 15-mer из всех кандидатов. Другие критерии, такие как минимальные появления композиционных отклонений (например, длинные строки конкретного нуклеотида), могут использоваться для определения, какой зонд является наилучшим. После применения критериев к каждому кандидату отбирают одного или нескольких кандидатов в качестве подходящих зондов.
Другим применением механизма поиска является определение отличий одного генома от другого. Например, по мере развития проекта «Геном человека», картируются и становятся общедоступными новые сегменты генома. Используя механизм поиска и зонды, которые разработаны для другой версии такого же генома, можно определить, насколько эти зонды могут быть применимы к новой версии генома.
Другим применением, где может быть использован механизм поиска, является проверка того, существует ли конкретное «слово» в геноме. Это может быть необходимым для поиска «слов», которые не появляются в геноме, поэтому существует небольшая вероятность того, что «слово» будет гибридизоваться с секцией генома. Такие «слова» могут быть случайно генерированы согласно заданному набору критериев. Если «слово» найдено, его комплемент также вводится в механизм поиска для определения того, присутствует ли он в геноме. Если «слово» и комплемент отсутствуют в геноме, существует минимальная вероятность того, что это «слово» и его комплемент будут гибридизоваться с геномом. Такие негибридизующиеся зонды могут использоваться в гибридизации как читаемые штрих-коды и в контролях гибридизационных матриц и могут быть добавлены к зондам нуклеиновых кислот с целью усиления сигналов гибридизации через формирование сети.
Одним из способов минимизации вероятности гибридизации является минимизация частоты составляющих mer конкретного «слова». То есть предпочтительным является получение зондов, которые имеют столько длин составляющих mer, чтобы иметь частоты встречаемости «слов», равные нулю. Например, предположим, что несколько 20-mer олигонуклеотидов получены с той целью, чтобы они не гибридизовались с геномом человека. Далее предположим, что каждый 20-mer аннотируют для каждого из его составляющих перекрывающихся 19-mer, 18-mer, 17-mer, 16-mer, вплоть до, например, 6-mer. Теоретически, наиболее желательный 20-mer предпочтительно будет иметь нулевые частоты встречаемости «слов» для mer каждой длины. В частности, зонд, который имеет минимальную вероятность гибридизации, предпочтительно имеет как можно больше нулевой частоты встречаемости mer при уменьшении длин mer, насколько это возможно (например, требуемый зонд может иметь нулевые частоты встречаемости «слов» для длин mer 19, 18, 17, 16, 15, 14 и 13). Таким образом, если один зонд имеет нулевые частоты встречаемости своих составляющих 15 и 14-mer, его гибридизация с геномом менее вероятна, чем в случае зонда, который имеет нулевые частоты встречаемости своих составляющих 15-mer, но имеет частоты встречаемости составляющих его 14-mer, равные одному или более. Таким образом, первый из упомянутых зондов имеет меньшую вероятность гибридизации, чем второй зонд, поскольку он не имеет ни одного 14-mer, который соответствует секциям генома.
Негибридизующиеся олигонуклеотиды могут быть получены с использованием составляющих mer конкретного mer, который имеет нулевую или малую частоту встречаемости «слова». Например, если конкретный 20-mer имеет 13-mer, который имеет нулевую частоту встречаемости «слова», то такой 13-mer может быть использован для получения олигонуклеотидов, которые возможно не существуют в геноме (например, два из таких 13-mer можно соединить друг с другом для получения уникального 26-mer).
В лабораторных условиях, например, «слово» с нулевой частотой встречаемости и его комплемент с нулевой частотой встречаемости (негибридизующиеся олигонуклеотиды) могут быть прикреплены к (гибридизующемуся) зонду или «слову-мишени». Выражаясь абстрактно, «слова» представляют собой «плечи», которые прикрепляют к «телу» (т.е. зонду). Если гибридизация начинается, то «слова» («плечи») гибридизуются только друг с другом, в то время как зонды гибридизуются с геномом. Поскольку «слова» («плечи») обычно несут детектируемый материал (например, флуоресцентную метку), самогибридизация помогает отличить местоположение зондов в геноме относительно гибридизационного фона. Таким образом, самогибридизация плеч служит для усиления видимости зонда, который гибридизуется с геномом.
Негибридизующиеся олигонуклеотиды также могут использоваться в качестве зондов (tag) для однозначной идентификации конкретной последовательности среди огромной популяции других последовательностей. Негибридизующиеся олигонуклеотиды могут быть прикреплены к известной последовательности, таким образом присоединяя зонд или осуществляя мечение конкретной последовательность.
В другом примере несколько различающихся последовательностей ДНК могут быть сцеплены для формирования простого генома (например, полученного на этапе 810 на фиг.8). Такой сцепленный геном полезен, например, если требуется сконструировать зонд, который визуализирует конкретный патоген (например, вирус) в образце человеческой крови. Сцепленный геном необходим, поскольку ДНК, экстрагированная из человеческой крови, содержит не только ДНК человека, но также ДНК других источников, таких как патоген. Следовательно, для того, чтобы зонд эффективно визуализировал патоген в человеческой крови, он не должен давать перекрестную гибридизацию с геномом человека.
В случае, если патогенный зонд не является полностью уникальным относительно другого генома в образце ткани (например, у пациента обнаружены его геном и геномы других микроорганизмов), может возникнуть необходимость в сравнении частоты встречаемости «слова» для зонда в геноме патогена с частотами встречаемости «слова» для зонда в других геномах. В случае этого подхода могут потребоваться два механизма поиска - один для интересующего патогена, а другой для комбинации других геномов. Необходимо отметить, что при применении подхода сдвоенного механизма поиска, преимуществом может быть разработка зондов, которые имеют высокие частоты встречаемости mer в геноме патогена при условии, что частоты встречаемости зонда в других геномах в образце ткани несоразмерно низки.
VII. ПРИМЕРЫ
Нижеследующие примеры представлены только с целью иллюстрации. Они не предназначены для ограничения объема настоящего изобретения, раскрытого в настоящем описании.
Пример 1 - Выбор олигонуклеотидов, комплементарных репрезентативным выборкам
В этом примере иллюстрируется идентификация олигонуклеотидных зондов, которые комплементарны репрезентативной выборке генома человека, полученной с использованием BglII. Аналогичные подходы могут использоваться для разработки олигонуклеотидов, комплементарных любой популяции нуклеиновых кислот, последовательности которых известны или предсказаны. Используя опубликованные сведения о последовательности генома человека, авторы осуществили расщепление BglII in silicо генома человека с помощью установления местонахождения всех сайтов рестрикции BglII в опубликованных сведениях. Далее авторы отбирали все последовательности фрагментов после расщепления BglII, длина которых составляла от 200 до 1200 пар оснований. Затем проводили анализ последовательности фрагментов, используя алгоритм, изложенный в настоящем описании. Этот алгоритм (также называемый «mer-механизмом») может быть использован для определения числа копий любой данной олигонуклеотидной последовательности в любом секвенированном геноме. Такое число копий также называется «частотой встречаемости слова» олигонуклеотидной последовательности в геноме.
Авторы аннотировали каждый расщепленный BglII фрагмент с частотой встречаемости «слов» их составляющих, перекрывающихся 15- и 21-mer (т.е. олигонуклеотидов, имеющих 15 или 21 нуклеотидов), используя mer-механизм и те же самые сведения о геноме человека. Для этого, получали in silicо для каждого фрагмента каждой составляющей, перекрывающий 70-mer олигонуклеотид (например, фрагмент из 100 пар оснований будет иметь 31 таких 70-mer). Для каждого такого 70-mer фрагмента определяли следующие признаки, как описано ниже: максимальную частоту встречаемости 21-mer (или максимальную частоту встречаемости 18-mer), среднее арифметическое частоты встречаемости 15-mer, процентное содержание G/C и количество каждого основания и наибольший отрезок, составленный из любого одного основания.
Для определения максимальной частоты встречаемости 21-mer каждый 70-mer разделяли на перекрывающиеся 21-mer и сравнивали каждый из этих 21-mer со всеми 21-mer последовательностями в геноме. Отбрасывали все 70-mer, у которых максимальная частота встречаемости 21-mer превышала 1, т.е. у которых 21-mer последовательность была на 100% комплементарна более чем одной 21-mer последовательности в геноме. Это был начальный набор 70-mer зондов.
Далее оптимизировали набор 70-mer зондов путем удаления зондов с содержанием GC ниже 30% или выше 70%, с отрезком из А/Т, превышающим 6 оснований, или отрезком из G/C, превышающим 4 основания. Из оставшихся 70-mer отбирали для каждого полученного с использованием BglII фрагмента один (или более) 70-mer, который имел отношение GC/AT, наиболее близкое к таковому в геноме в целом. Далее проводили анализ каждого выбранного таким образом 70-mer путем определения частоты встречаемости «слова» в геноме для каждой составляющей 70-mer, перекрывающей 15-mer. Выбирали 70-mer, которые имели наиболее низкое среднее значение частоты встречаемости 15-mer.
В качестве окончательной проверки на общую уникальность оптимальные 70-mer зонды для каждого полученного с использованием BglII фрагмента сравнивали со всем геномом, используя программное обеспечение BLAST. Использовали параметры по умолчанию, исключая фильтрацию последовательностей низкой сложности, которая не выполнялась. Любой 70-mer зонд с любой степенью гомологии на 50% или более к любой последовательности, отличной от него самого, удалялся.
Алгоритм mer-механизма обеспечивает строгость, гибкость и простоту в процессе разработки зонда. Способность быстро определять частоту встречаемости «слова» для «слов» всех размеров позволяет разрабатывать количественно ограниченные критерии способом, аналогичным реальной гибридизации. Частоты встречаемости «слов» могут рассматриваться как количественная мера степени, с которой последовательности относятся к двум или нескольким наборам полинуклеотидов. Например, небольшой зонд «AGT» может рассматриваться как набор, содержащий шесть отдельных «слов», а именно «A», «G», «T», «AG», «GT» и «AGT». Если бы такой зонд аннотировали с частотой встречаемости «слова» для всех «слов» всех размеров, обнаружилось бы, что количество каждого слова в первом наборе, который представляет собой зонд «AGT», было бы в большой степени затенено их количеством во втором наборе, т.е. геноме из трех миллиардов нуклеотидов.
Эта взаимосвязь может быть выражена как отношение X/Y, где Х представляет собой сумму частоты встречаемости для всех составляющих «слов» зонда относительно указанного зонда, а Y представляет собой сумму частоты встречаемости для всех аналогичных «слов» в геноме. При выборе 70-mer зонда, который гибридизуется с заданной последовательностью с минимальной перекрестной гибридизацией, отношение X/Y можно максимизировать, где максимальное значение X/Y для зондов, полученных из геномной последовательности, равно 1. Способ с выбором для аннотации только двух длин «слов» является одним из многих возможных способов достижения такой цели.
В случае, если в интересующей области генома не могут быть найдены уникальные зонды, существует возможность применения неуникальных зондов для получения чистых измерений относительного различия числа копий или просто количества вещества. Затем задача распространяется на сравнение трех наборов «слов»: зонда, интересующей охватывающей области и генома. Допустим Z представляет сумму всех значений частоты встречаемости «слов» зонда относительно охватывающей области. Предположим, что Х и Y также являются суммой всех частот встречаемости «слов» зонда относительно зонда и генома соответственно. Целью является максимизация значения выражения, (X/Y)/(X/Z), или просто, (Z/Y). Другими словами, можно обнаружить зонды, которые являются специфичными для области независимо от общего числа копий. Такой конкретный случай может быть обобщен для включения любого обстоятельства, при котором отбирают зонды для «узнавания» одной конкретной последовательности из многих при помощи гибридизации. Дополнительным примером является распознавание ДНК одного организма на фоне ДНК многих других организмов.
Другое применение такой парадигмы заключается в минимизации элементов набора. Авторы разработали зонды, которые действовали в качестве контролей гибридизации в экспериментах на микроматрицах. Такие зонды представляли собой контроли в том смысле, что они были предназначены для гибридизации только с теми фрагментами ДНК, которые с равной вероятностью узнавались любым другим зондом. В этом случае целью была просто разработка зонда, где Y был настолько близок к нулю, насколько это возможно. Такой зонд также может быть полезен, например, в качестве уникальных идентификаторов, считываемых при гибридизации, или добавок к другим последовательностям нуклеиновых кислот для усиления сигнала гибридизации посредством формирования сети.
Дополнительно к суммам и средним арифметическим значениям частот встречаемости «слов» могут использоваться многие другие статистики, включая, например, дисперсии частоты встречаемости «слов» зонда для «слов» конкретного размера. Такая дисперсия может служить в качестве быстрого предварительного фильтра для отбора зондов, которые должны существовать в определенном количестве копий. Максимум частоты встречаемости «слова» для конкретного размера «слова» может быть принят в качестве указания на наименее удовлетворяющий возможный результат гибридизации для уникального, при прочих равных, зонда. Такие количественные меры являются идеальными для быстрого определения пригодности гибридизационного зонда относительно других кандидатов. По существу алгоритм mer-механизма может упростить процесс отбора зонда до однократного сканирования интересующих последовательностей.
Один из наборов зондов, которые были разработаны, состоял из 85000 70-mer, обладающих средней частотой встречаемости 18-mer относительно генома человека 1,2 со стандартным отклонением 0,8. Среднее вычисляли по набору всех 18-mer всех объединенных зондов. По сравнению с предшествующим уровнем техники, в частности с опубликованным набором примерно из 23000 70-mer матрицы зондов, среднее значение частоты встречаемости 18-mer для всех объединенных зондов составляло 1,9 со стандартным отклонением 14,8. Следовательно, такой набор зондов был наибольшим из двух с коэффициентом 4 и был более уникальным с коэффициентом 18. Набор из 85000 зондов в этом примере был выбран, исходя из комбинации ограничения уникального 21-mer и ограничения минимизированной частоты встречаемости совокупности 15-mer, как описано выше. Преимуществом было увеличение уверенности в том, что зонды, которые эмпирически подтвердили хорошую работоспособность, просто не гибридизовались с большой гетерогенной популяцией фрагментов ДНК, и, таким образом, не увеличивали ее сигнал. Это также иллюстрирует точность, с которой были разработаны наборы зондов, удовлетворяющие точно определенным критериям, таким как чрезвычайно малое стандартное отклонение от заданного среднего значения частоты встречаемости «слова».
Пример 2 - Получение матрицы
Были использованы два способа для получения микроматриц, содержащих олигонуклеотидные зонды, разработанные согласно примеру 1. В случае первого из них, способ «печать», авторы приобрели около 10000 нуклеотидов, полученных с помощью методов твердофазной химии, и напечатали их иглами на стеклянной поверхности. В частности, использовали Cartesian PixSys 5500 (Genetic Mycrosystems) для размещения коллекции зондов на слайдах в виде матрицы, используя конфигурацию 4х4 игл. Размер каждой напечатанной матрицы составлял примерно 2 см2. Матрицы были напечатаны на промышленно изготовленных силиконизированных слайдах (Corning® ultraGAPSTM #40015). Для устройства изготовления матрицы использовали иглы от Majer Precision.
Во втором способе, способ «фотопечать», олигонуклеотиды были синтезированы NimbleGenTM Systems, Inc. непосредственно на силиконовой поверхности, используя метод лазерной фотохимии. Для приблизительно 700000 уникальных 70-mer олигонуклеотидов сначала проводили анализ «рабочих характеристик» путем размещения их в виде матрицы на восьми чипах и гибридизации их с репрезентативной выборкой, полученной с использованием BglII, и репрезентативной выборкой, полученной с использованием BglII, повторно рестриктированных EcoR1, геномной ДНК из нормальной особи мужского пола J.Doe. Отобрали 85000 олигонуклеотидов, с помощью которых получали наиболее сильный сигнал, и разместили их на одном чипе.
В обоих способах разместили нуклеотиды случайным образом для минимизации возможности того, что артефакты, связанные с геометрией, во время гибридизации матрицы могли бы быть некорректно интерпретированы как нарушение генома. В следующих примерах описаны результаты, полученные с 10К матрицами, изготовленными способом печати, и 85К матрицами, изготовленными способом фотопечати.
Пример 3 - Получение и мечение тестируемых репрезентативных выборок
Для некоторых экспериментов, изложенных в настоящем описании, для получения репрезентативных выборок выбрали BglII. BglII обладает характеристиками, подходящими для этих конкретных экспериментов: он является устойчивым ферментом; его сайт расщепления не подвергается CpG метилированию; он оставляет выступающий конец из четырех оснований; и его сайты расщепления имеют приемлемо равномерное распределение в геноме человека. Репрезентативные выборка, полученные с использованием BglII, составлены из коротких фрагментов, обычно менее чем 1200 пар оснований. Авторы оценили, что таких фрагментов примерно 200000, которые составляют около 2,5% генома человека, со средним расстоянием между ними 17 т.п.о.
Во всех экспериментах, приведенных в настоящем описании, использовали сравнительную гибридизацию репрезентативных выборок, полученных параллельно. ДНК двух образцов, которые сравнивали, получали в одно и то же время, и репрезентативные выборки получали из образца с одинаковой концентрацией, используя одинаковые протокол, реагенты и термальный циклер. Это должно было уменьшить вероятный «шум», создаваемый вариациями, имеющими место при амплификации ПЦР.
Получали с использованием BglII репрезентативные выборки человеческой геномной ДНК, как ранее описано у Lucito et al., 1998, см. выше. Кратко, расщепляли 3-10 нг человеческой геномной ДНК с использованием BglII в условиях, предложенных изготовителем. Очищали гидролизат экстракцией фенолом и преципитацией этанолом в присутствии 10 мкг тРНК. Ресуспендировали осадок в 30 мкл 1Х буфера для Т4 ДНК лигазы с 444 пМ каждого адаптера (RBgl24 и RB1gl2; Lucito R. and M. Vigler. 2003. «Preparation of target DNA». In «Microarray-based Representational Analysis of DNA Copy Number» (eds. D.Bowtell & J. Sambrook), р. 386-393. Cold Spring Harbor Press, Cold Spring Harbor, NY). Помещали реакционную смесь в заранее нагретый до 55°С тепловой блок, который затем помещали на лед приблизительно на 1 час до тех пор, пока температура не понижалась до 15°С. Затем добавляли 400 единиц Т4 ДНК лигазы и инкубировали реакционную смесь при 15°С в течение 12-18 часов.
Добавляли 1/40 лигированного материала, 20 мкл 5Х буфера ПЦР [335 мМ Tris-HCl, рН 8,8; 20 мМ MgCl2; 80 мМ (NH4)2SO4; 50 мМ β-меркаптоэтанола и 0,5 мг/мл BSA], 2'-дидеоксинуклеозид 5'-трифосфата до конечной концентрации 0,32 мМ, адаптор RBgl24 до конечной концентрации 0,6 мкМ, 1,25 Ед. Taq полимеразы и водy в пробирки на 250 мкл, доводя объем до 100 мкл. Пробирки помещали в термоциклер MJ Research TETRADTM, заранее нагретый до 72°С. Затем проводили амплификацию следующим образом: один цикл при 72°С в течение 5 мин, а затем 20 циклов по 1 минуте при 95°С и 3 минуты при 72°С и затем 10 минут при 72°С. Очищали репрезентативные выборки (т.е. продукты ПЦР) экстракцией в смеси фенол:хлороформ и преципитацией этанолом, затем ресуспендировали в ТЕ (рН 8) и определяли концентрацию ДНК.
Для некоторых экспериментов получали укороченные репрезентативные выборки расщепления дополнительными рестрикционными эндонуклеазами для расщепления тех фрагментов, которые содержали их сайты рестрикции. В этих случаях расщепляли смесь лигирования второй рестрикционной эндонуклеазой непосредственно перед этапом амплификации. В экспериментах, описанных ниже, укороченные репрезентативные выборки, полученные с использованием BglII, получали с использованием HindIII.
Метили фрагменты в репрезентативных выборках, помещая ДНК в 0,2 мл пробирки ПЦР. Добавляли 10 мкл праймеров из набора для мечения Amersham-Pharmacia MegaprimeTM и перемешивали их тщательно с ДНК. Доводили водой объем до 100 мкл. Помещали пробирки в прибор MJ Research TETRADTM при 100°С на 5 мин, помещали на лед на 5 мин и добавляли 20 мкл буфера для мечения из набора для мечения Amersham-Pharmacia MegaprimeTM, 10 мкл метки (Cy3TM-dCTP или Cy5TM-dCTP) и 1 мкл фрагмента New England BioLabs® Klenow. Пробирки инкубировали при 37°С в течение двух часов, объединяли меченные образцы (Cy3TM и Cy5TM) в одну пробирку Eppendorf® и затем добавляли 50 мкл 1 мкг/мкл ДНК Human Cot 1, 10 мкл 10 мг/мл тРНК исходной культуры дрожжей и 80 мкл Low TE (3 мМ Tris-HCl pH 7,4, 0,2 мМ ЭДТА). Загружали образец на фильтр Centricon® и центрифугировали в течение 10 мин при 12600 об/мин. Удаляли фильтрат, а фильтр промывали 450 мкл Low TE. Повторяли центрифугирование и промывку ТЕ дважды. Собирали меченый образец, обращая колонку Centricon® в новую пробирку и центрифугируя в течение 2 мин при 12600 об/мин. Переносили меченный образец в 200 мкл пробирку ПЦР и доводили объем с помощью Low TE до 10 мкл.
Кроме того, для некоторых экспериментов фрагментированные ДНК выделяли из первичной раковой клетки яичника и из нормального образца с McrBC и лигировали линкерами и амплифицировали, как описано выше.
Пример 4 - Гибридизация тестируемых репрезентативных выборок с матрицами
Осуществляли УФ-поперечную сшивку олигонуклеотидных зондов со слайдом с использованием набора Stratagen® Stratalinker® при 300 мДж, поворачивая слайд на 180 градусов, удерживая слайд в одном и том же положении при сшивке, и повторяли обработку. Слайды промывали в течение 2 мин в 0,1% SDS, 2 мин в воде Milli-Q®, 5 минут в кипящей воде Milli-Q® и, наконец, в охлажденном льдом 95% этаноле без бензола. Слайды сушили путем их размещения на металлическом штативе и вращением их в течение 5 мин при 75 об/мин. Предварительно гибридизировали и печатали микроматрицы путем помещения их в Сoplin jar или другую камеру для обработки слайдов, добавления буфера предгибридизации (25% деионизированного формамида, 5Х SSC и 0,1% SDS) и предварительного нагрева камеры до 61°С в течение двух часов и затем промывали их в воде Milli-Q® в течение 10 сек. Снова сушили слайды путем размещения их в металлическом штативе для слайдов и вращая в течение 5 мин при 75 об/мин. Фотопечатные матрицы NimbleGenTM не требовали УФ-сшивки и предварительной гибридизации.
Добавляли 25 мкл гибридизационного раствора к 10 мкл меченого образца, полученного, как в примере 3, и перемешивали. Для печатных слайдов в качестве гибридизационного раствора использовали 25% формамид, 5Х SSC и 0,1% SDS. Для фотопечатных матриц NimbleGemTM в качестве гибридизационного раствора использовали 50% формамид, 5Х SSC и 0,1% SDS. Образцы денатурировали в MJ ResearchTM TETRADTM при 95°С в течение 5 минут и затем инкубировали при 37°С в течение 30 минут. Образцы центрифугировали и наносили их с помощью пипетки на слайд, снабженный приподнятым покровным стеклом (lifter slip), и инкубировали его в печи для гибридизации (такой как печь Boekel InSlide OutTM), установленной на 58°С для печатных матриц или на 42°С для фотопечатных матриц NimbleGenTM, в течение 14-16 часов.
После гибридизации слайды промывали следующим образом: быстро в 0,2% SDS/0,2X SSC для удаления покровного стекла; 1 минуту в 0,2% SDS/0,2X SSC, 30 секунд в 0,2X SSC; и 30 секунд в 0,05XSSC. Слайды сушили так же, как описано выше, путем размещения на планшете и вращения при 75 об/мин в течение 5 минут. Затем слайды сразу сканировали.
Слайды сканировали с использованием сканера Axon GenePix® 4000 В, установленного на размер пикселя 10 микрон для печатных матриц и 5 микрон для фотопечатных матриц. Количественное определение интенсивности матриц проводили с использованием программного обеспечения GenePixTM Pro 4.0 и импортировали данные в S-PLUS® для дальнейшего анализа. Вычисляли отношение между двумя сигналами в эксперименте, используя измеренные интенсивности без удаления фона. Данные нормализовали, используя основанный на интенсивности алгоритм LOWESS (робастная локально взвешенная регрессия) фитирования кривой, аналогичный алгоритму, описанному у Yang et al., Nucl. Acids Res., 30:e15-15(2002). Усредняли данные, полученные из экспериментов с обращенными цветами, и отображали их, как представлено на фигурах.
Пример 5 - Рабочие характеристики и валидизация матриц
Как описано выше в примере 1, авторы должны были бы получить возможность предсказывать, основываясь на опубликованной последовательности генома человека, какие олигонуклеотидные зонды с какими репрезентативными выборками могут гибридизоваться. Для подтверждения этого протестировали 10К печатные матрицы, изготовленные авторами, путем гибридизации их с полученным с использованием BlgII репрезентативных выборок ДНК генома нормального человека, меченной одним флуоресцентным красителем, и с полученным с использованием BlgII репрезентативных выборок, повторно рестриктированных с помощью HindIII, той же самой ДНК, меченной другим флуоресцентным красителем.
На фиг.1 показаны результаты для полученного с использованием BlgII репрезентативных выборок, повторно рестриктированных с помощью HindIII. На фиг.1А по оси Y графически показано отношение интенсивности гибридизации каждого зонда. Каждый эксперимент проводили с обращением цвета, и среднее геометрическое отношение из отдельных экспериментов изображали на графике. Зонды, предсказанные для обнаружения фрагментов как в полных, так и подвергшихся повторной рестрикции репрезентативных выборок, действительно гибридизовались с обоими видами фрагментов (фиг.1А; слева). Таких зондов было примерно 8000. В случае последовательностей, для которых было предсказано, что они не будут визуализировать фрагменты в повторно рестриктированной репрезентативной выборке, это действительно не происходило (фиг.1А, справа). Таких зондов было примерно 1800. Эти результаты подтверждают то, что: (1) профиль рестрикции характерных фрагментов выборки был предсказан правильно, (2) олигонуклеотиды были размещены в виде матрицы корректно и (3) олигонуклеотиды обнаруживали предсказанные фрагменты с допустимым сигналом интенсивности. На фиг.1В показана согласованность между отношениями экспериментов с обращенным цветом. Эти данные подтверждают репродуцируемость матриц, изготовленных авторами.
Очень небольшое количество олигонуклеотидных зондов не гибридизовалось с заданными фрагментами в репрезентативных выборках, как было предсказано. Например, из 8000 зондов, предсказанных как гибридизирующиеся с фрагментами, нерасщепленными HindIII, оказалось, что примерно 16 гибридизовалось с фрагментами, полученными с использованием BglII, которые фактически были расщеплены. Это могло произойти вследствие несоответствия между образцами авторов и опубликованной последовательностью генома человека, что могло быть результатом полиморфизма или ошибок секвенирования. Однако данные настоящего описания показывают, что опубликованная последовательность генома человека является в достаточной степени достоверной для разработки зондов для характерных олигонуклеотидных микроматриц.
Пример 6 - Глобальный анализ геномов опухолей
Олигонуклеотидные матрицы по настоящему изобретению легко обнаруживают крупные нарушения генома, независимо от того, являются ли они делециями или амплификациями. На фиг.2А1-А3, 2В1-В3 и 2С1-С3 показаны данные гибридизации матриц для трех сравнений генома: на фиг.2А1-А3 сравнивают анеуплоидные клетки рака молочной железы с нормальными диплоидными клетками из той же биопсии (CHTN159) (репрезентативные выборки двух образцов были получены из примерно 100 нг ДНК, каждую выделяли из ядер анеуплоидной и диплодной фракций, разделенных поточной цитометрией); на фиг.2В1-В3 сравнивают линию клеток рака молочной железы (SK-BR-3), полученную от пациента неизвестной этнической принадлежности, с неродственной нормальной особью мужского пола J.Doe (смешанного происхождения из Европы и Африки; см. пример 2); и на фиг.2С1-С3 сравнивают клетки из другой нормальной особи мужского пола (африканского пигмея) с тем же самым J.Doe. В каждом случае гибридизацию образцов проводили дважды, с обращением цвета, и отношение среднего геометрического (в логарифмическом масштабе) наносили на график относительно порядка в геноме нуклеотидных зондов. Увеличенное число копий (амплификация) указано отношением выше 1, а уменьшенное число копий (делеция) отношением меньше 1. Данные, показанные на фиг.2А1, 2В1 и 2С1, получали с помощью печатных матриц 10К. Данные, показанные на фиг.2А2, 2В2 и 2С2, получали с помощью фотопечатных матриц 85К.
Для раковых геномов профили были отчетливыми. Профили двух линий рака молочной железы отличались, но каждый показывал большие участки амплификации и делеции в геноме (фиг.2А1-А2 и 2В1-В2). Напротив, профиль норма-норма был по существу плоским, указывая на отсутствие крупномасштабных амплификаций или делеций между этими геномами (фиг.2С1-С2). Эти данные подтверждают, что олигонуклеотидные матрицы по настоящему изобретению могут визуализировать крупные геномные изменения.
Результаты также указывают на то, что существует большое количество олигонуклеотидных зондов, детектирующих минорные потери и усиления во всех трех геномах (двух раковых геномах и геноме африканской мужской особи). Такие потери и усиления показаны в виде отдельных точек на фиг.2А1-А2, 2В1-В2 и 2С1-С2, и проявляются на фиг.2С2 (сравнение норма-норма) в виде «оболочки» или зоны зондов, которые достигают отношений 0,5 и 2,0 по всему геному. Такие потери и усиления, по-видимому, являются результатом гетерозиготного полиморфизма BglII между приведенными образцами.
Кроме того, сравнение печатного формата 10К с фотопечатным форматом 85К ясно показывает, что, хотя они имеют различную разрешающую способность, оба зафиксировали аналогичный вид крупномасштабных геномных особенностей. Зонды называли «братьями», если они были комплементарны одному и тому же фрагменту, полученному с использованием BglII. Братья необязательно должны были быть перекрывающимися последовательностями, хотя они могли перекрываться до половины свой длины, или могли быть комплементарными по всей свой длине. На фиг.2А3, 2В3 и 2С3 графически показаны отношения олигонуклеотидов-братьев в случае 10К (ось Y) к отношениям их олигонуклеотидов-братьев в случае 85К (ось Х). Существовало более 7000 зондов братьев. Наблюдалось значимое согласие между отношениями зондами-братьями в двух способах для всех трех экспериментов, вопреки тем фактам, что последовательности зондов отличались в способах, что их образцы размещения в матрице были различными, что различались условия гибридизации и различались поверхности матриц. Эти данные подтверждают воспроизводимость результатов, полученных с использованием матриц, содержащих олигонуклеотиды согласно настоящему изобретению.
Кроме того, с помощью анализа МОМА репрезентативных выборок, полученных путем расщепления с помощью McrBC, были установлены участки генома с различным уровнем метилирования в геномах раковых клеток и нормальных клеток. Нормализация различий в числе копий в этих участках с использованием репрезентативной выборки, полученной с использованием BglII, подтвердила, что наблюдаемое различие во многих таких сайтах было результатом различия в уровне метилирования, а не в количестве копий.
Пример 7 - Автоматизированная сегментация и анализ всего генома
Авторы также проводили анализ данных из более мелких участков генома для картирования вариаций, отмеченных в примере 6. Например, анализировали данные из одной хромосомы во время использования алгоритма статистической сегментации, который разделяет данные отношений для зондов на сегменты с одинаковым средним после учета вариации (названного циклической двоичной сегментацией (CBS); см. Olshen and Venkatraman, Change-Point Analysis of Array-Based Comparative Genomic Hybridization Data, Alexandria, VA, American Statistical Association, 2002). Алгоритм рекуррентно идентифицирует наилучшую возможную сегментацию каждой хромосомы, отклоняя или принимая каждое предлагаемое разделение, основываясь на вероятности того, что разница в средних значениях могла возникнуть случайно. Такая вероятность определяется способом рандомизации. Вследствие его непараматерической природы алгоритм не позволял идентифицировать отклонения, узнаваемые меньше чем тремя зондами.
На фиг.3А-3D показан результат анализа четырех хромосом (хромосомы 5, 8, 17 и X на фиг.3А-3D соответственно) линии раковых клеток SK-BR-3 с использованием матрицы 85К. Авторы наблюдали аналогичные профили сегментации и средние значения для сегментов, когда использовали данные из матрицы 10К. Дополнительный анализ данных позволил определить уровень плоидности клеток.
После сегментации для каждого нуклеотида назначали среднее отношение сегмента, с которым оно связано, и отображали на графике средние отношения в отсортированном порядке. Эти данные показаны на графике для раковых геномов CHTN159 (фиг.4А) и SK-BR-3 (фиг.4С). На фигурах показано, что средние отношения для сегментов в пределах каждого генома разбиты на подгруппы с основными и второстепенными горизонтальными участками похожих значений. Авторы вывели число копий таких участков, основываясь на подсчете и знании хода анализа о том, что CHTN159 является суб-триплоидным, а SK-BR-3 - тетраплоидным. Если бы каждый образец был примерно моноклональным, то два основных горизонтальных участка в CHTN159 представляли бы собой две или три копии на клетку, а основные горизонтальные участки SK-BR-3 представляли бы собой три или четыре копии на клетку.
Авторы использовали число копий, вычисленное для основных горизонтальных участков, для нахождения плоидности и SN для каждого эксперимента. Авторы использовали уравнение:
RM=(RT×SN+1)/(SN+1),
где RM представляет собой среднее измеренное отношение, RT представляет собой истинное отношение, а SN представляет собой экспериментально полученное значение, описывающее шум «специфический к неспецифическому». Выбирали RM как среднее значение зондов для сегментов на горизонтальном участке и устанавливали RТ в СN/P, где СN представляет собой истинное число копий, полученное из горизонтального участка, а Р представляет собой плоидность генома опухоли. Такая комбинация дает два уравнения и два неизвестных, Р и SN. Для эксперимента с CHTN159 (фиг.4А) вычислили плоидность Р, которая была равна 2,60, а SN - 1,13. Для эксперимента с SK-BR-3 (фиг.4С) вычислили, что Р было равно 3,93, а SN - 1,21.
Также использовали уравнение для вычисления того, какие средние отношения могли бы быть предсказаны для более высоких и низких количеств копий. Авторы отметили такие предсказанные значения на соответствующих графиках, от нуля до 12 количеств копий, горизонтальными линиями, формирующими «сетку количеств копий». Назначенные средние значения в сегментах для зондов отображали в геномном порядке, с наложенной сеткой ожидаемых количеств копий, на фиг.4В и 4D. Сетка количеств копий исключительно хорошо соответствовала второстепенным горизонтальным участкам данных, особенно для больших количеств копий.
Пример 8 - Анализ мелкомасштабных геномных нарушений.
Авторы также анализировали данные для определения точных точечных нарушений в отдельных хромосомах, которые имели амплификации или делеции. Анализ показал, что матрицы по настоящему изобретению могут использоваться для идентификации геномных нарушений с разрешением отдельных генов. Соответственно данные, полученные из экспериментов с матрицами, могут использоваться для предсказания влияния аберраций в конкретных генах на превращение нормальной клетки в раковую клетку.
Сначала проводили анализ участка нарушения в хромосоме Х, см.фиг.3D. SK-BR-3 клетки, которые получили из женской особи, сравнивали с клетками неродственной мужской особи. Ожидали, что зонды в хромосоме Х будут иметь повышенные отношения. Это имело место почти на всем длинном плече хромосомы Х. Но в середине Xq13.3 существовало четкое нарушение в числе копий на участке, охватывающем 27 т.п.о., и отношения, близкие к единице, наблюдали для остатка хромосомы (фиг.5А). Таким образом, существовала возможность определить границы генетических нарушений с помощью данных матрицы путем сегментации. Наблюдали множество других примеров резкого изменения числа копий, что должно приводить к повреждению генов.
Существовало от трех до четырех узких областей амплификации в SK-BR-3 геноме, каждая из которых содержала два или меньше гена, среди которых находились трансмембранные рецепторы.
Затем проводили анализ данных для хромосомы 8 (фиг.3В), которая имела множество аберраций, включая широкие отдельные участки амплификации (фиг.5В). Самый крайний правый пик представлял собой отрезок приблизительно в один миллион п.о., составленный из тридцати семи зондов (координаты зондов 45099-45138, координаты генома June 126815070-128207342). Кроме того, он содержал единичный хорошо описанный ген, c-myc.
В SK-BR-3 существовал второй широкий пик, находящийся слева от пика c-myc и под графиком (фиг.5В). Этот широкий пик имел широкое плечо на своей правой стороне (координаты зондов 44994-45051, координаты генома June 123976563-125564705), с очень узким пиком в его средней части. Авторы наложили на него данные сегментации из ракового генома, CHTN159, который имел даже более широкий пик, охватывающий c-myc (координаты зондов 44996-45131, координаты генома June 124073565-127828283). Пик в CHTN159 также охватывал плечо второго SK-BR-3 пика (фиг.5В). Таким образом, плечо могло содержать кандидаты онкогенов, которые заслуживали внимания. В этом участке, в узком пике, обнаружили TRC8, мишень транслокаций, имеющих отношение к наследуемому раку почек (Gemmill et al., Proc. Natl. Acad. Sci. USA, 95:9572-7(1998)). Эти результаты иллюстрируют значение координирующих данных из множества геномов, и необходимость в автоматизированных способах анализа множества наборов данных.
Авторы также анализировали узкую делецию на хромосоме 5. На фиг.5С показаны результаты объединенных анализов с помощью 10К (пустые кружки) и 85К (заполненные кружки), наложенные на сетку числа копий. Делеции были очевидны в случае обоих разрешений 10К и 85К (координаты зондов 29496-29540, координаты генома June 14231414-15591226), но границы были более отчетливыми при разрешении 85К. Этот участок содержал TRIO, белок, имеющий домен GEF, домен SH3 и домен серинтреонинкиназы (Lin and Greenberg, Cell, 101:230-42 (2000)); ANKH, трансмембранный белок (Nurnberg et al., Nat. Genet., 28:37-41(2001)); и FBXL, компонент пути дегадрации белка, обусловленной убиквитин-лигазой (Ilyin et al., Genomics 67:40-47 (2000)).
Наконец, авторы анализировали участок гомозиготной потери на хромосоме 19, влияющей на кластер белков «цинковые пальцы» (фиг.5D; координаты зондов 77142-77198, координаты генома June 21893948-24955961). Некоторые из этих генов могут кодировать факторы транскрипции, делеции которых могут играть роль в генезисе опухоли. Авторы наблюдали избыток узких гемизиготных и гомозиготных нарушений, некоторые из которых могли быть свойственны нормальным вариантам. См. пример 9.
Пример 9 - Исследование вариаций «нормального» генома
Авторы также использовали олигонуклеотидные матрицы и способы по настоящему изобретению для анализа вариаций числа копий между двумя нормальными геномами и наблюдали различия, получающиеся в результате полиморфных вариаций. Такой анализ является важным, например, в ситуациях, где образцы раковой ДНК не могут быть сопоставлены с нормальной ДНК, и неродственная нормальная ДНК используется в качестве эталона, поскольку наблюдаемые различия могут быть результатом полиморфных вариаций. Такая вариация может быть двух видов, точечная вариация последовательности такого вида, что создает или разрушает фрагмент, полученный с использованием BglII, например, SNP, или реальные флуктуации числа копий присутствуют в пуле человеческих генов. Первое из вышеперечисленного имеет ограниченное влияние на анализ, использующий матрицы настоящего изобретения, поскольку это продуцирует рассеянный «шум», который в значительной степени может быть отфильтрован статистическими средствами.
На фиг.6А (объединенные данные из наборов данных 10К и 85К) показано, что алгоритм мягкой фильтрации (если отношение имело наибольшее отклонение из четырех соседних, его заменяли отношением, наиболее близким отношением из двух соседних) может минимизировать влияние точечной вариации последовательности и визуализировать случаи, где существует реальная вариация числа копий. Облако рассеянных полиморфизмов, присутствующее в образце, не подвергнутом фильтрации (например, фиг.С2), увеличивается при таком представлении данных, выявляя неслучайные кластеры отклоняющихся отношений зондов, включая крупномасштабные геномные различия между нормальными индивидами.
Полиморфная вариация рассеянного множества также может быть отфильтрована с помощью последовательных сравнений экспериментов. Например, на фиг.6В показаны данные из SK-BR-3 в сравнении с нормальным донором, J.Doe, в случае 85К отношения отображены заполненными кружками, а в случае 10К - пустыми кружками. На одном и том же графике отобразили отношения J.Doe в сравнении с другой нормой, ДНК африканского пигмея, в зеленых треугольниках. Можно видеть три зонда с экстремальными отношениями в гибридизации нормальной SK-BR-3-норма, которые могут быть идентифицированы как полиморфизмы путем сравнения с гибридизацией между двумя нормальными индивидами. Наиболее простоя интерпретацией этих данных заключается в том, что J.Doe имеет +/+, пигмей имеет +/-, а SK-BR-3 имеет -/-, где + означает присутствие небольшого фрагмента BglII (по всей вероятности SNP в сайте BglII). Обычно парные сравнения трех геномов позволяют интерпретировать аллельный статус. Таким образом, такие виды данных являются особенно полезными в случае, если злокачественный геном не может быть сопоставлен с нормальным.
Однако полиморфизм в количестве копий создает различного рода проблемы. На фиг.6А показаны большие различия по областям в количестве копий при сравнении норма-норма. К этим данным был применен анализ сегментации и было идентифицировано множество участков, которые демонстрировали измененное число копий между двумя нормальными индивидами. Наблюдали около дюжины вариантных участков в любом сравнении норма-норма. Они простирались в длину от ста тысяч пар оснований до более чем миллиона пар оснований, могли встретиться в любом месте, но наиболее часто наблюдались вблизи теломер и центромер и часто охватывали известные гены.
Тщательное исследование двух таких участков показано на фиг.6С и фиг.6D, с обозначением отношений в виде соединенных кружков, и значениями сегментации в виде сетки. На фиг.6С аномальный участок составлял 135 т.п.о. на хромосоме 6р21 (координаты зондов 32518-32524, координаты генома June 35669083-35804705), и охватывал три известных гена. На фиг.6D участок составлял 620 т.п.о., участок из хромосомы 2р11 (координаты зондов 9927-9952, координаты генома June 88787694-89385815), который содержал несколько вариабельных участков тяжелой цепи.
Анализировали влияние вариации норма-норма на интерпретацию данных рак-норма. На фиг.6С и 6D представлено наложение значения сегментации из анализа SK-BR-3 с диагональной и вертикальной штриховкой соответственно. Сетка числа копий для SK-BR-3 показана в виде сетки. На фиг.6С показан участок в SK-BR-3, который можно было бы назвать делецией в сравнении с нормой. В SK-BR-3 есть боковые участки с количеством копий, которое авторы оценили как две копии на клетку, и в пределах этого участка число копий уменьшалось до одной. Но такой же участок появлялся при сравнении ДНК пигмея с нормальной ДНК. На фиг.6D наблюдается аналогичное состояние хромосомы 2р11. На фиг.6D также представлены данные сегментации в случае опухоли. Такой участок также очевидно является аномальным.
Пример 10 - Аннотация части генома
Нижеследующие примеры предназначены для иллюстрации использования механизма поиска. Подходящие модификации и адаптации описанных условий и параметров, обычно встречающихся в данной области техники, которые очевидны специалистам в данной области техники, находятся в пределах объема и сущности настоящего изобретения.
Механизм поиска по настоящему изобретению может быть использован для выполнения вычислений на геноме или на подгруппах генома (например, хромосоме). При выполнении таких вычислений обнаруживаются некоторые участки, имеющие высокие частоты встречаемости «слов», которые не детектируются инструментами поиска, такими как Repeat Masker. Показано, что база данных повторов, используемая Repeat Masker, не включает в себя повторы, которые являются специфичными для участка или специфичными для хромосомы. Используя механизм поиска, описанный выше, в секции VII, такие повторы находятся легко, поскольку вычисление точного совпадения может формировать основу для алгебры множеств генома. В частности, подмножество генома может быть превращено в строки преобразования, которые оцениваются для обнаружения повторов, специфичных для хромосомы.
Строка преобразования из хромосомы 1 аннотировалась частотой встречаемости «слов» в ней самой и во всем геноме. Поиск выполняли с тем, чтобы найти смежные участки хромосомы 1 по меньшей мере 100 п.о. в длину с максимальной частотой встречаемости 18-mer, где обнаружены точные соответствия, так, чтобы они были получены в основном из хромосомы 1. Такие участки были легко обнаружены ранжированием по длине от 100 п.о до 35 т.п.о. Концентрируя внимание на этом участке, обнаружили что его mer-топография представляла собой практически ступенчатую функцию, составленную из более коротких последовательностей, каждая из которых имела характерную частоту встречаемости и длину. Участки, специфичные для хромосомы, содержащие один из таких характерных участков, собирали и быстро идентифицировали семейство последовательностей, специфичных для хромосомы 1. Участок, специфичный для хромосомы 1, отобрали с помощью идентификации 18-mer, частота встречаемости которых для хромосомы 1 превышала 90% их частоты встречаемости во всем геноме, такие 18-mer свели вместе для создания повтора, специфичного для хромосомы. Кроме того, расстояние между 18-mer, которые были сведены, не должно было превышать 100 п.о. Обнаружили по меньшей мере один раз, что такой повтор был аннотирован как перекрывающее ген RefSeq (номер регистрации NM_015383), с большим количеством экзонов, которые совместно кодировали большую предсказанную белковую последовательность, имеющую низкую гомологию с миозином.
Такой же процесс, с помощью которого идентифицировали повторы, специфичные для хромосомы, может быть применен для нахождения повторяющихся ДНК во всем геноме, включая те, которые не являются узнаваемыми с помощью Repeat Masker или других программ.
Пример 11 - Разработка зондов с использованием mer-механизма
Вышеупомянутый механизм поиска может быть использован для разработки зондов. Обычно зонды являются полезными из-за их способности специфично гибридизоваться с комплементарной ДНК, и, следовательно, одной из основных задач при разработке зондов является минимизация перекрестной гибридизации. Предыдущие приложения для разработки зондов использовали маскирование повторов для исключения из рассмотрения участков повторов. Такой подход является проблематичным, поскольку он не обеспечивает защиту от участков, которые являются повторяющимися, таких как специфичные для хромосомы повторы, и он исключает «повторяющиеся» участки, которые являются уникальными.
Хотя правила гибридизации между неполностью совпадающими последовательностями не являются достаточно понятными, в данной области техники известно, что зонды, которые имеют точные «небольшие» совпадения с множеством участков генома, предпочтительно должны быть исключены. Ранее для применения зонда отбирали зонды, которые минимизировали совокупное количество точных совпадений 12-mer, но для геномных зондов такие способы не являются адекватными. Во-первых, не ясно, имеют ли точные совпадения 12-mer какое-либо влияние на гибридизацию в нормальных жестких условиях отжига. Частоты встречаемости 12-mer не предсказывают гомологию, не говоря уже об уникальности в геноме. Фактически сравнение частоты встречаемости 15-mer со средней геометрической частоты встречаемости для составляющих 12-mer дало низкую корреляцию между двумя последовательностями, которые по существу являются уникальными.
Обычный протокол для разработки зондов, применяющий mer-механизм, описан ниже. Во-первых, геном аннотируют согласно конкретной длине mer, так что обнаруживаются достаточно длинные строки уникальности (т.е. зонды-кандидаты). Во-вторых, такие зонды-кандидаты аннотируют с использованием по меньшей мере одного mer заранее определенной длины, предпочтительно длины, короче, чем длина mer, используемого для нахождения зондов-кандидатов. Один из зондов-кандидатов выбирают в качестве зонда, исходя из минимальной совокупности частоты встречаемости mer-заданных более коротких длин.
Следуя вышеупомянутому протоколу, 70-mer зонды-кандидаты отбирали из небольших фрагментов BglII, используя данные уникальности, полученные из частоты встречаемости 21-mer. В таких зондах-кандидатах были отобраны 70-mer с наиболее низкой суммой частот встречаемости 15-mer, с ограничением примерно 900. Дополнительные критерии, которые исключали отрезки единичных нуклеотидов и сильные смещения композиции оснований, также применяли для облегчения определения, какой зонд-кандидат выбрать. Отобранные зонды синтезировали и печатали на стекле для тестирования их работы в условиях гибридизации на микроматрице. Обнаружили, что по существу все зонды работали при определенных критериях рабочих характеристик и выше. Более точно, процент успешных попыток от примерно 70% до примерно 98% достигался с зондами, разработанными с использованием вышеупомянутого протокола, где успех определяли, как получение существенного (например, большого) отношения сигнал/шум.
BLAST использовали для тестирования того, являются ли выбранные зонды уникальными в конкретной опубликованной последовательности генома. 30000 таких зондов протестировали с использованием параметров по умолчанию для MegaBLAST (фильтрация элементарной последовательности была отключена). Обнаружили, что более 99% выбранных зондов были уникальными в геноме.
Пример 12 - Представление алгоритма в виде псевдокода
Для дополнительной иллюстрации того, каким образом алгоритм может быть применен для выполнения функции подсчета «слов», обратимся к фиг.12А и 12В. На фиг.12А графически определены переменные и структуры данных, используемые алгоритмом, а на фиг.12В показано представление алгоритма в виде псевдокода. Как упомянуто выше в секции VII, преобразование может быть использовано в качестве навигационного инструмента для «виртуального» словаря генома или массива суффиксов. В простейшем случае, предположим, что необходимо определить, появляется ли подстрока в геноме, и если это так, с каким числом копий. В таком случае, предположим, что подстрока представляет собой единичную «букву» «X». Все появления Х могут быть просмотрены в словаре в виде блока (например, области поиска), где Fx и Lx являются индексами первого и последнего появления Х. Fx и Lx могут быть получены из структуры данных «букв-границ». Размер таких блоков (например, области поиска) составляет kx=Lx-Fx+1, и также представляет собой количество Х. Необходимо отметить, что такое количество может быть определено путем подсчета количества Х в преобразовании.
В более сложном случае, таком, при котором должны быть подсчитаны «слова», состоящие из двух или более «букв», необходимо определять Fx, Lx и kx для каждой «буквы» Х в геноме. Другими словами, Fx и Lx для каждой «буквы» Х хранят в структуре данных, называемой «буква-граница». После получения структуры данных «буква-граница» алгоритм может возобновить подсчет количества конкретных «слов», Z, находящихся в геноме. Предположим W представляет собой суффикс Z, W существует в геноме, и известны «буквы-границы» для W (например, Fw и Lw, как показано на фиг.12А). Затем необходимо определить, существует ли XW в виде подстроки, где Х является знаком, предшествующим W в Z. Кроме того, должны быть определены индексы начала и конца (например, Fxw и Lxw) блока XW.
Только если Х появляется в преобразовании между Fw и Lw, то XW существует в виде подстроки в геноме. Более того, количество Х в «блоке W» преобразования, указанном в виде kxw, является частотой встречаемости «слова» подстроки XW в геноме. Индексы начала и конца XW могут быть определены с использованием: 1) Fxw=Fx+bxw; и 2) Lxw=Fxw-kxw-1, где bxw равно количеству «слов», начиная с X в словаре генома, которое появляется перед XW. bxw может быть определено подсчетом количества Х, которые появляются перед блоком W преобразования.
Эта процедура повторяется несколько раз, прибавляя одну «букву» в суффиксе за один раз, останавливаясь, если данный суффикс отсутствует в словаре генома. Если суффикс W охватывает все «слово», Z, kw равно количеству Z в строке генома. Эта процедура описана в виде псевдокода, как показано на фиг.12В. В соответствии с фиг.12В Z представляет собой строку длины N, составленную из «букв» из алфавита генома, и структура данных «букв-границ» содержит индексы первого и последнего появлений в словаре генома для каждой «буквы» в алфавите генома.
Если не определено иначе, все технические и научные термины, используемые в настоящем описании, имеют те же самые значения, как они обычно понимаются специалистами в данной области техники, к которым относится настоящее изобретение. Все публикации и другие ссылки, упомянутые в настоящем описании, включены здесь в качестве ссылок в полном объеме. В случае противоречия преимущество имеет настоящее описание, включая определение. Материалы, способы и примеры приведены только с целью иллюстрации, а не предназначены для ограничения. Во всем описании слово «содержать», или его варианты, такие как «содержит» или «содержащий», следует понимать для простоты включения целого числа или группы целых чисел, но не исключения любого другого целого числа или группы целых чисел.
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР ЗОНДОВ ДЛЯ АНАЛИЗА ОБРАЗЦОВ ДНК И СПОСОБЫ ИХ ИСПОЛЬЗОВАНИЯ | 2016 |
|
RU2753883C2 |
| УЛАВЛИВАНИЕ И ХАРАКТЕРИСТИКА СОВМЕСТНО ЛОКАЛИЗОВАННОГО ХРОМАТИНА | 2006 |
|
RU2478716C2 |
| СПОСОБ ОБНАРУЖЕНИЯ ТРАНСФОРМАНТА СОИ рDAB9582.814.19.1 | 2012 |
|
RU2622079C2 |
| МАРКЕРЫ, СЦЕПЛЕННЫЕ С УСТОЙЧИВОСТЬЮ РАСТЕНИЙ СОИ К SCN | 2011 |
|
RU2620973C2 |
| СПОСОБЫ СЕКВЕНИРОВАНИЯ ТРЕХМЕРНОЙ СТРУКТУРЫ ИССЛЕДУЕМОЙ ОБЛАСТИ ГЕНОМА | 2011 |
|
RU2603082C2 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНЫЙ АНАЛИЗ ТРАНСГЕННЫХ ГРАНИЦ | 2012 |
|
RU2603265C2 |
| НАБОР ИЗ ДВУХ ПРАЙМЕРОВ ДЛЯ АМПЛИФИКАЦИИ НУКЛЕИНОВОЙ КИСЛОТЫ ВИРУСА ГЕПАТИТА А, СПОСОБ ОБНАРУЖЕНИЯ ВИРУСА ГЕПАТИТА А С ЕГО ИСПОЛЬЗОВАНИЕМ (ВАРИАНТЫ) И НАБОР ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА ГЕПАТИТА А | 2003 |
|
RU2406762C2 |
| МУЛЬТИПЛЕКСНОЕ ДЕТЕКТИРОВАНИЕ НУКЛЕИНОВЫХ КИСЛОТ | 2014 |
|
RU2733887C2 |
| РЕЗИСТЕНТНЫЙ К НАСЕКОМЫМ И УСТОЙЧИВЫЙ К ГЕРБИЦИДАМ ТРАНСФОРМАНТ СОИ рDAB9582.816.15.1 | 2013 |
|
RU2650626C2 |
| ТРАНСГЕННЫЙ ОБЪЕКТ СОИ MON 87708 И СПОСОБЫ ЕГО ПРИМЕНЕНИЯ | 2010 |
|
RU2624025C2 |

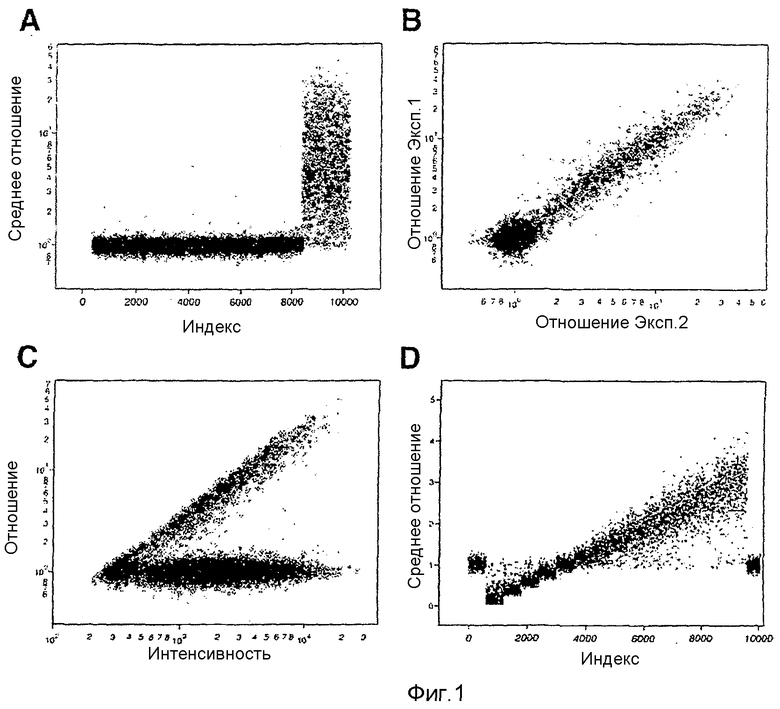
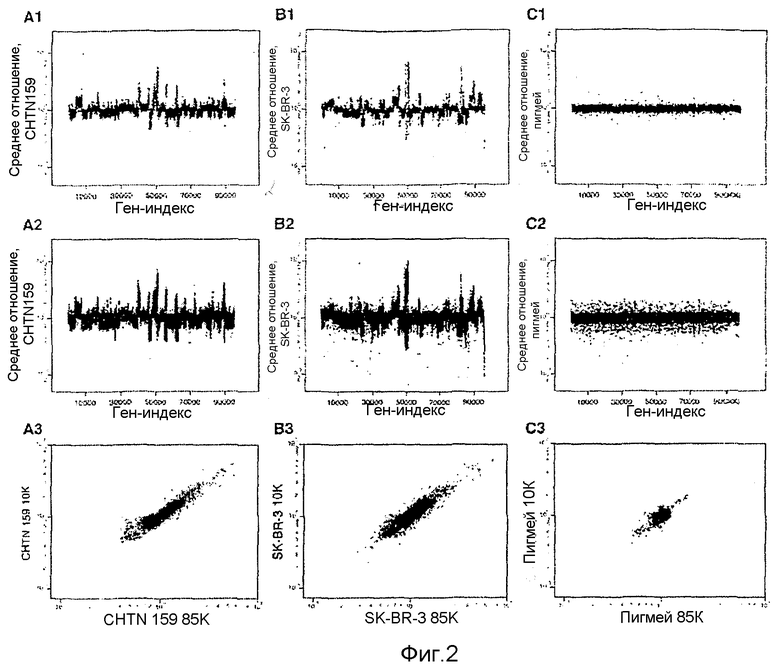
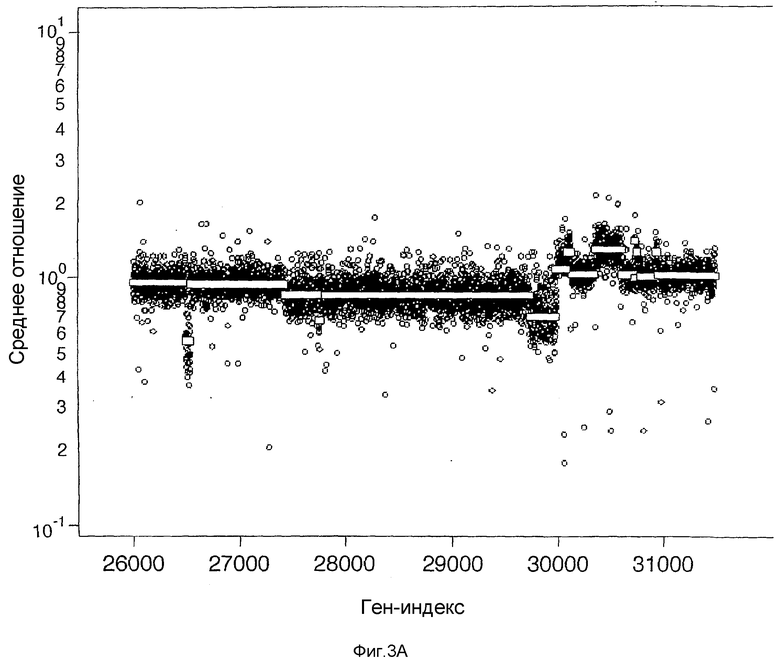
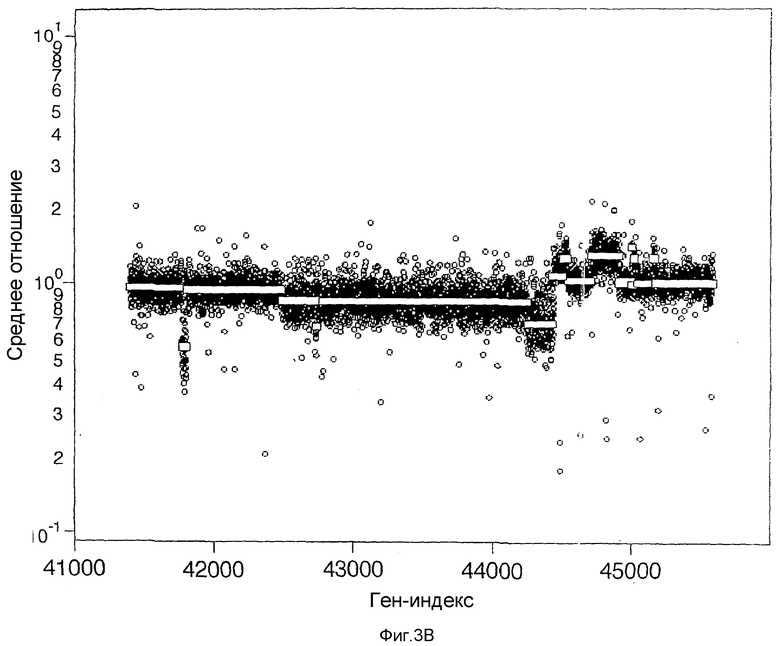
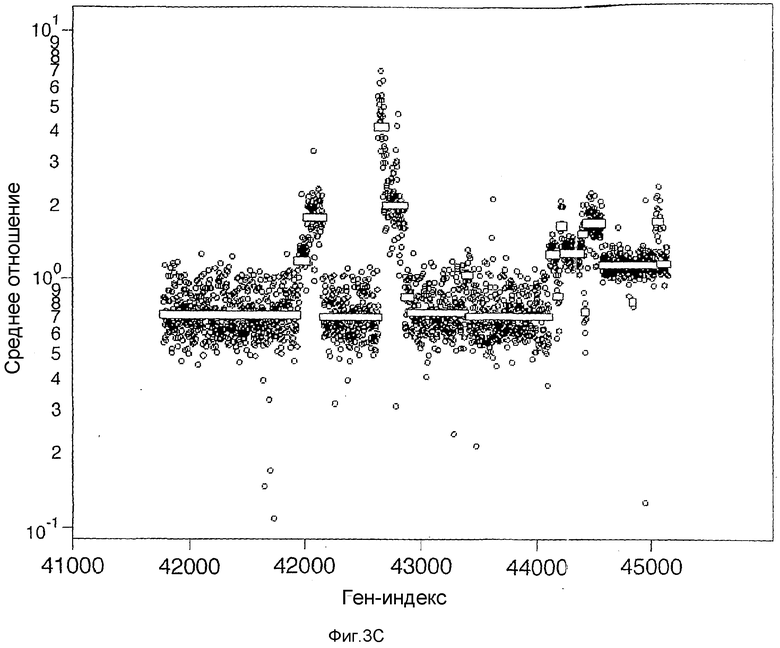
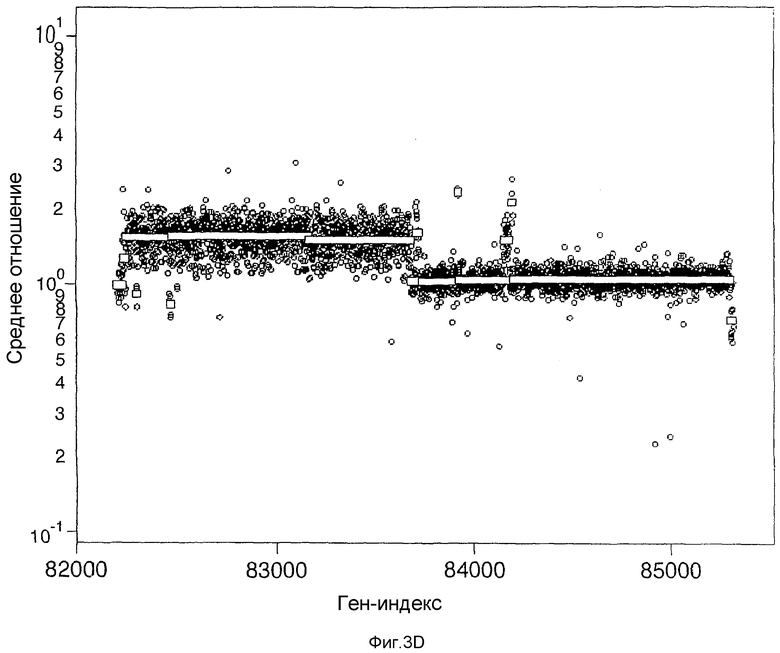
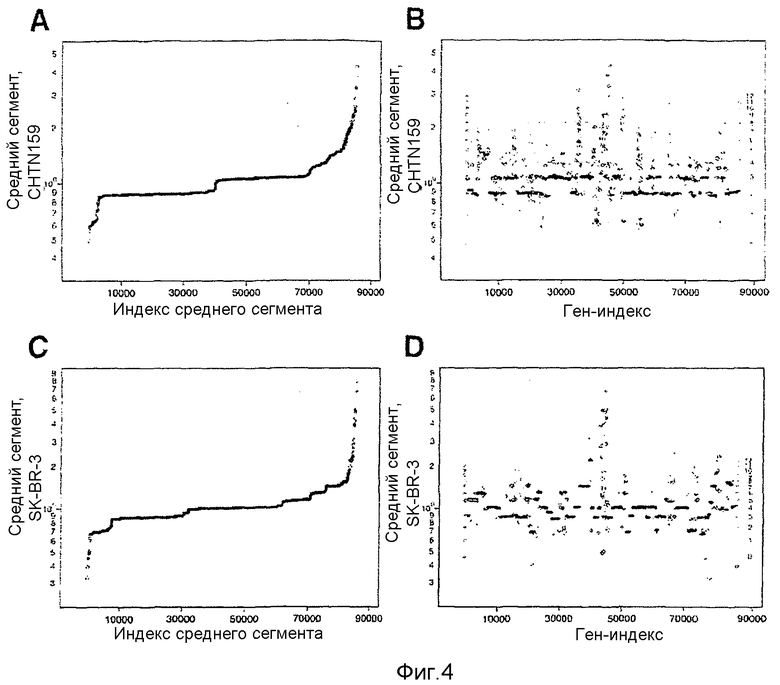
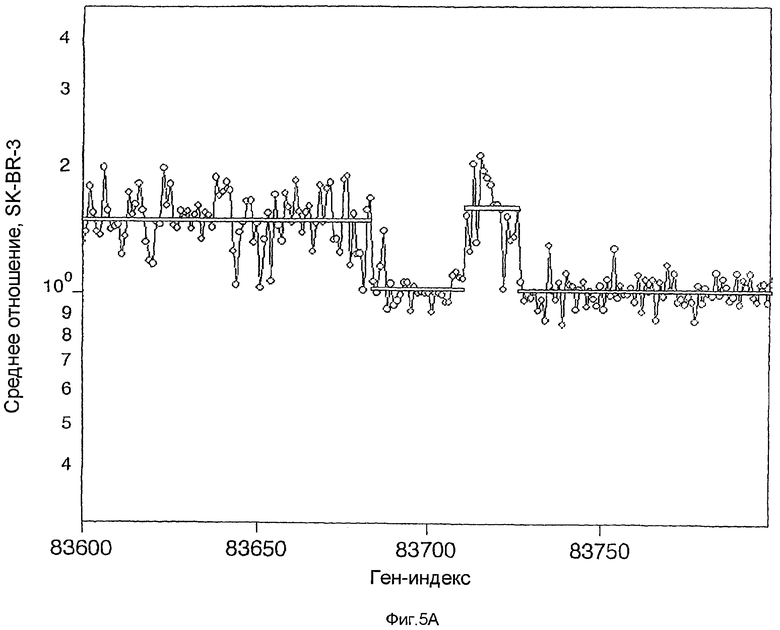
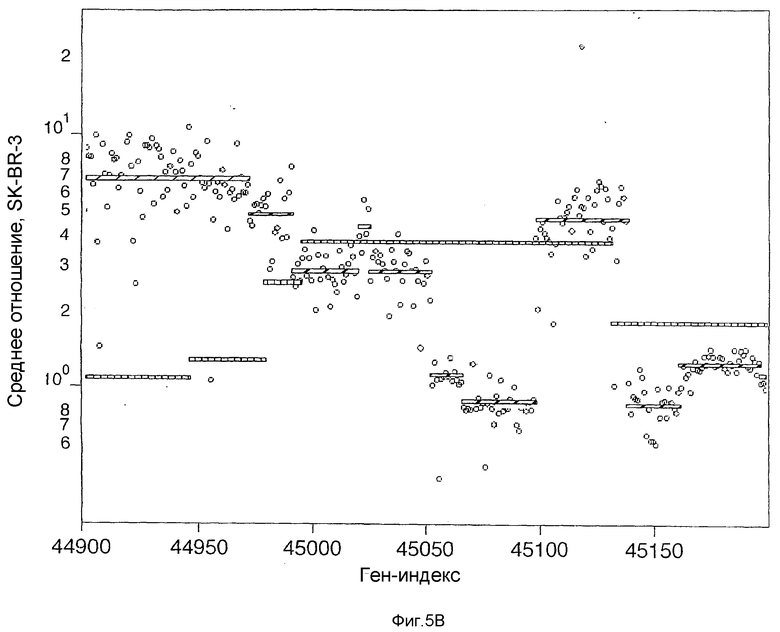
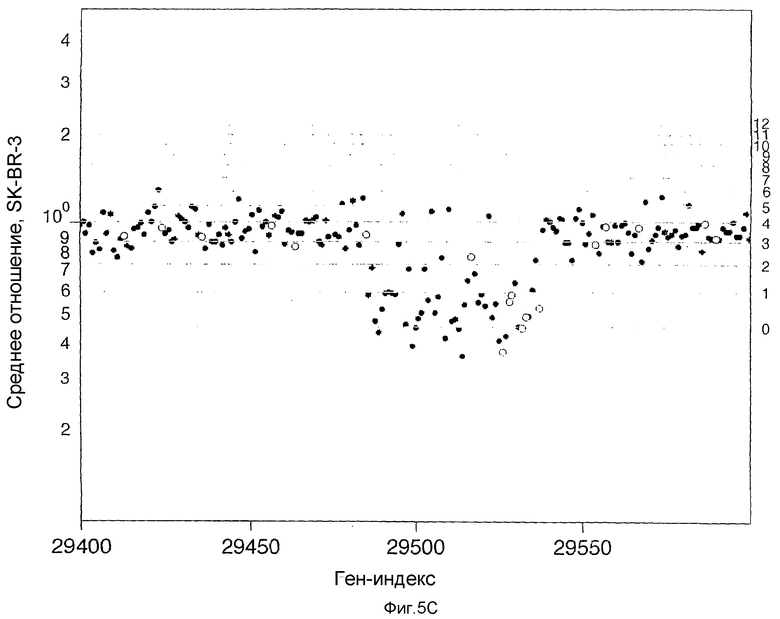
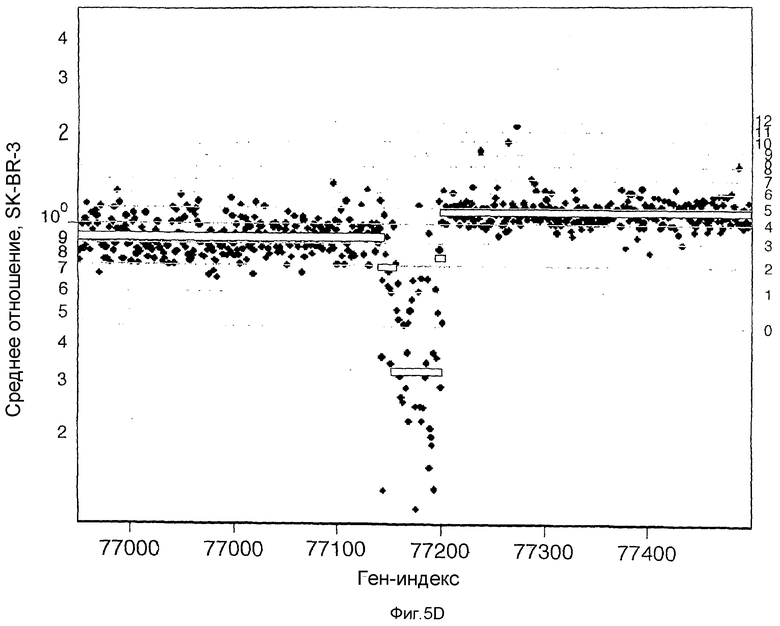
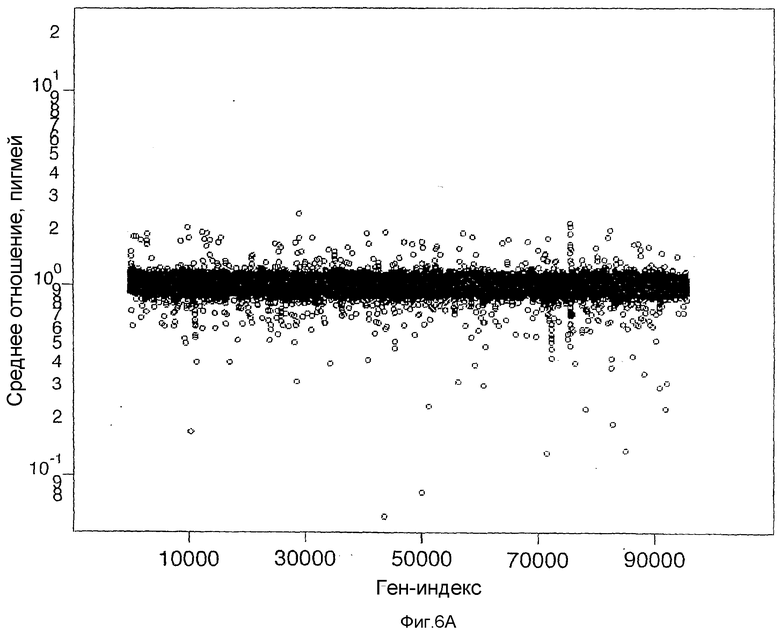
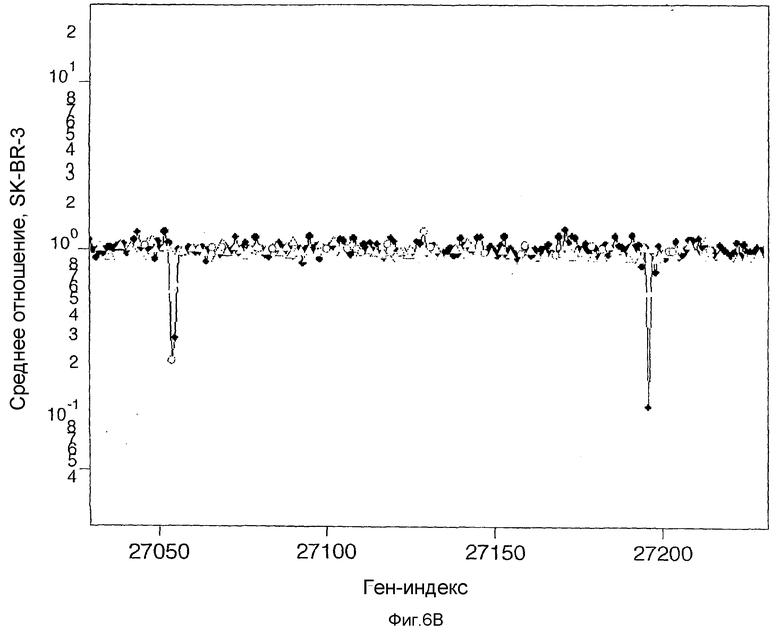
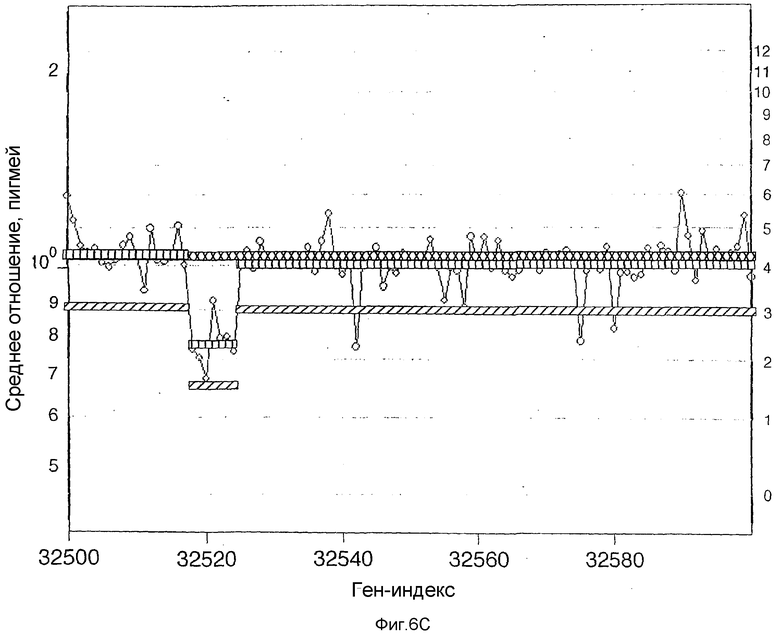
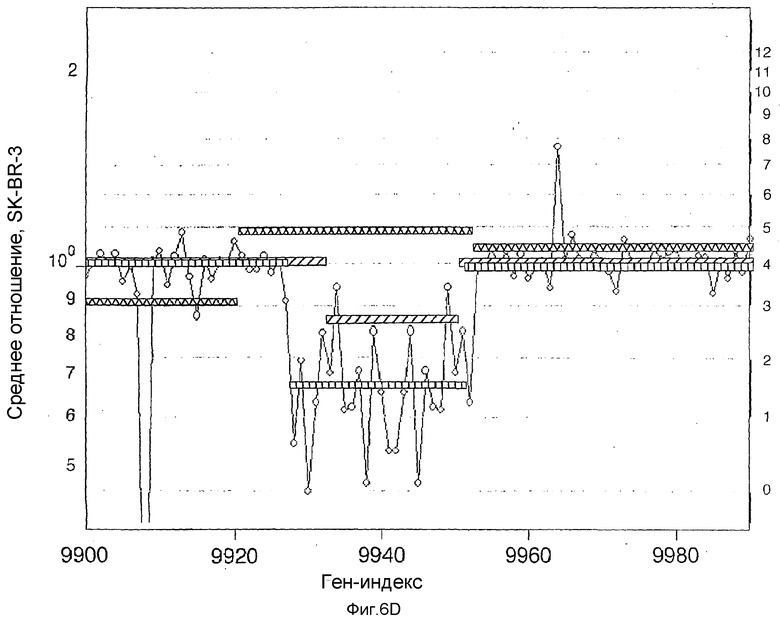
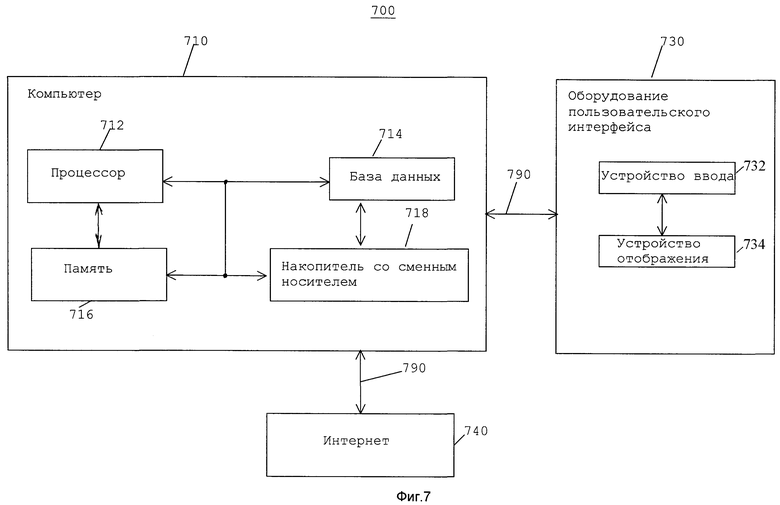
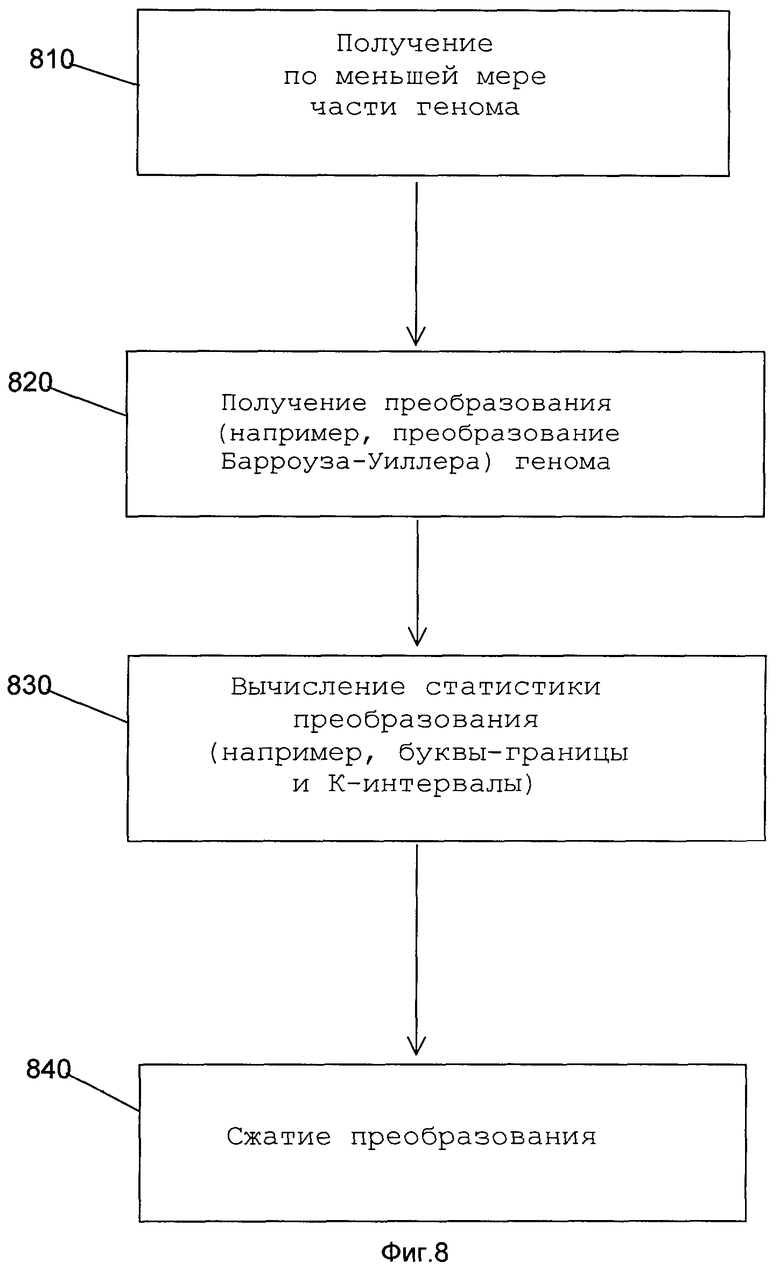
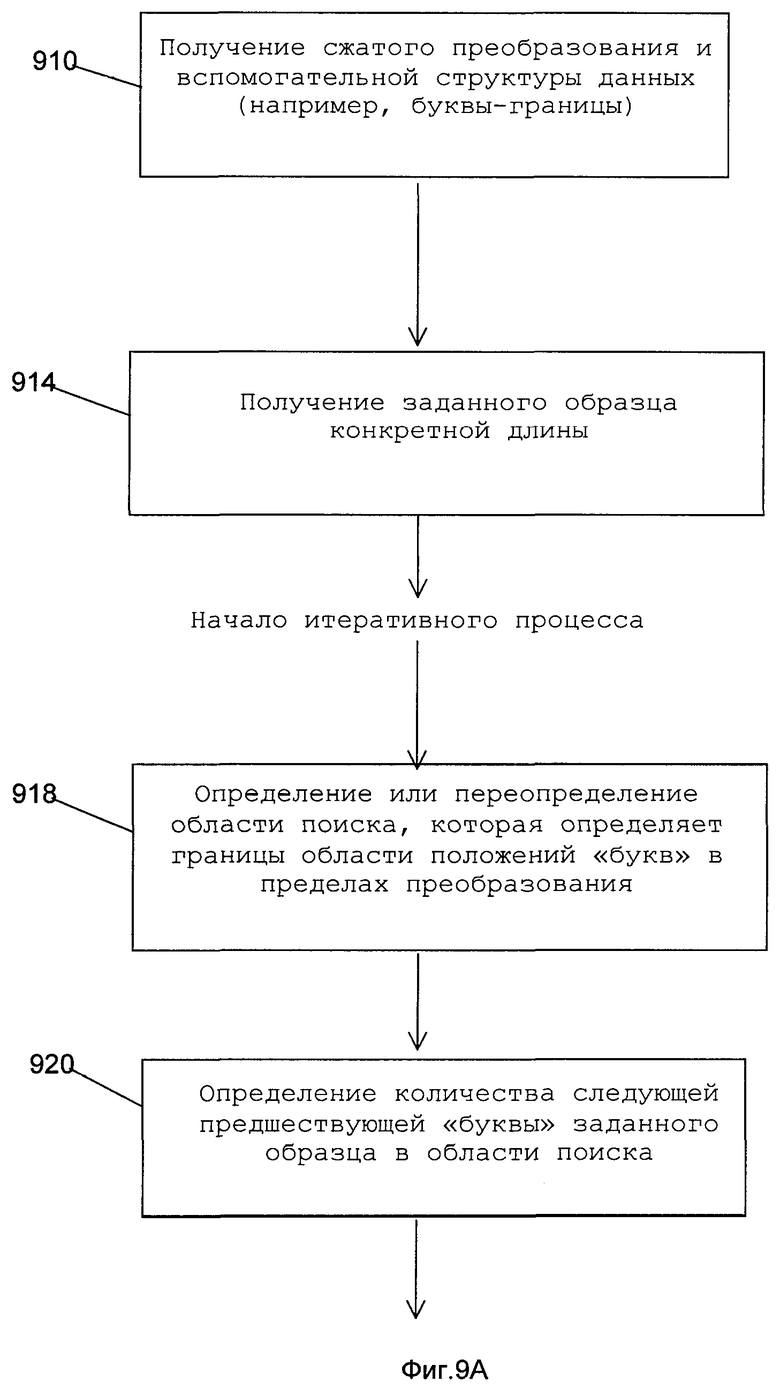
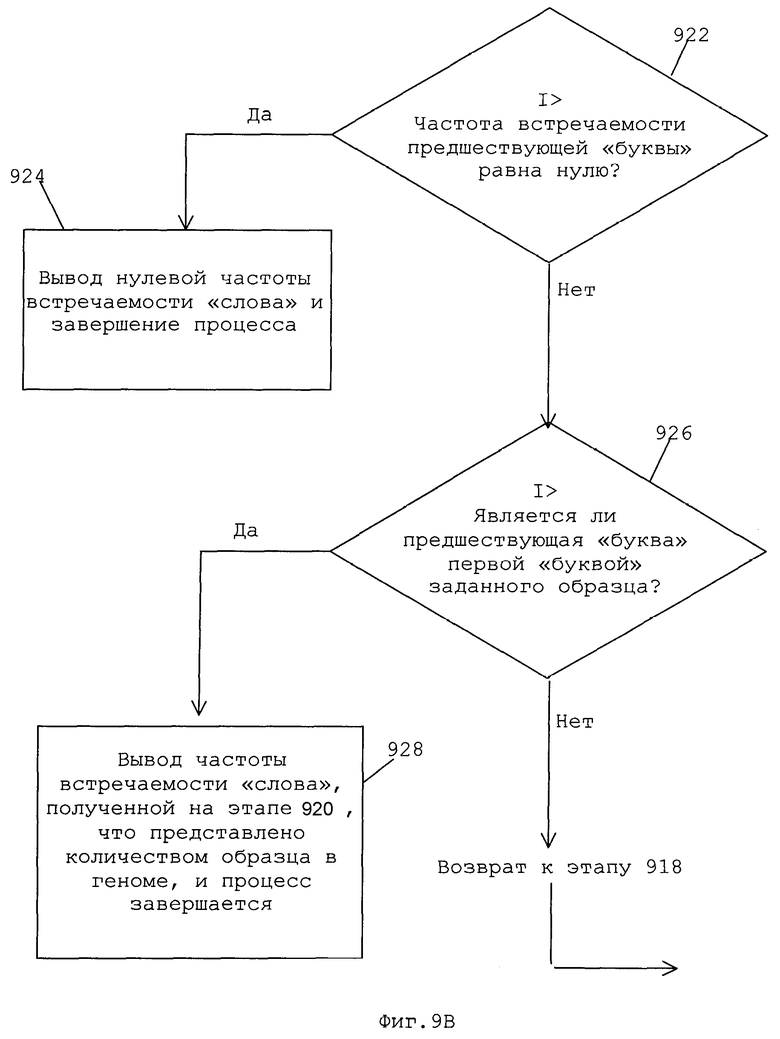
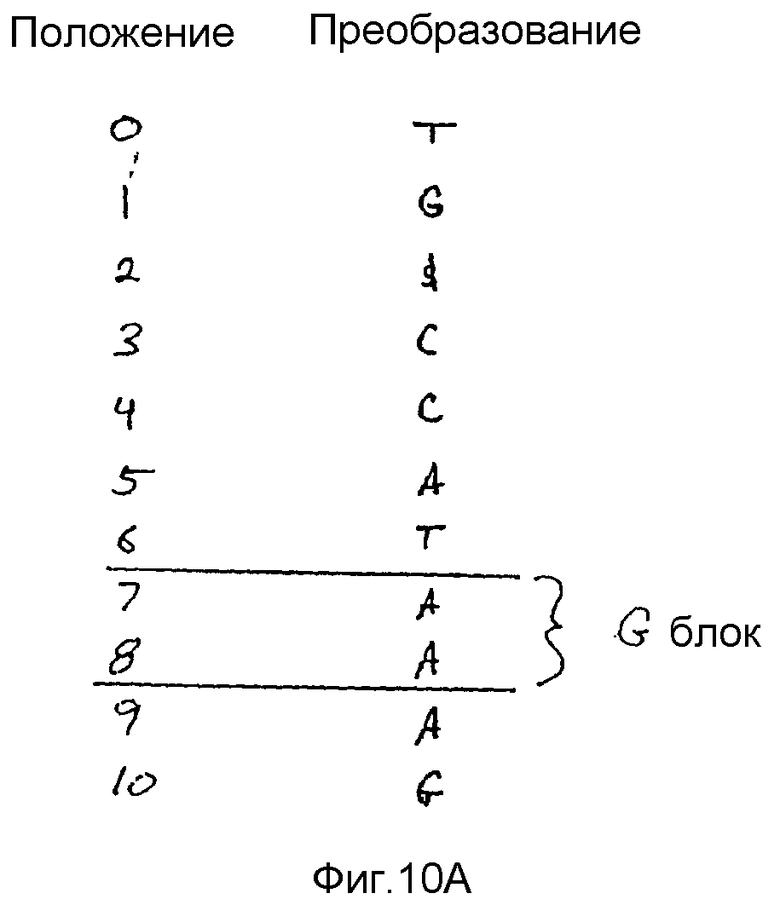
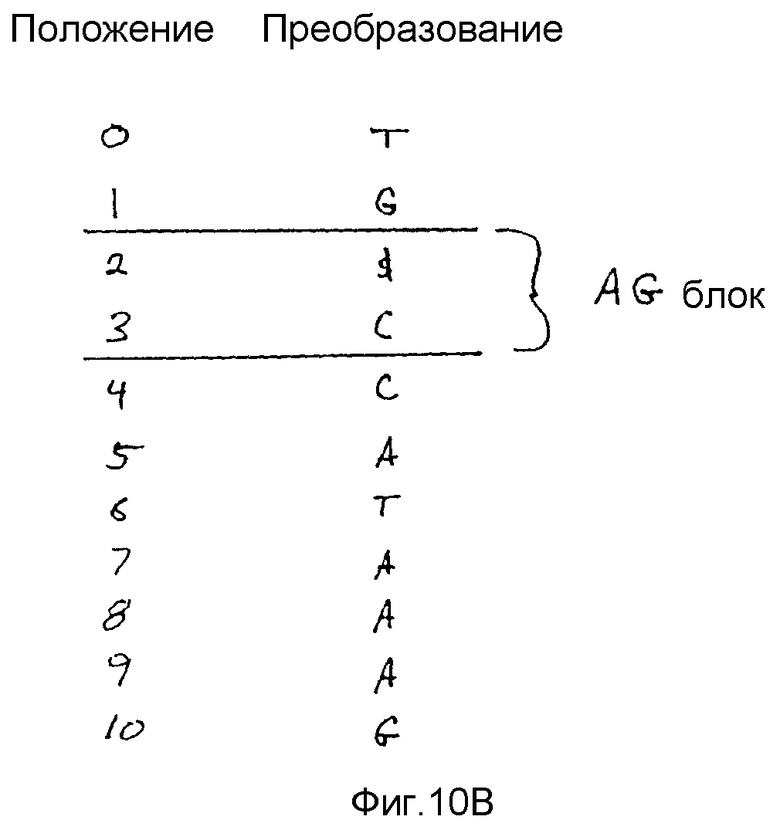
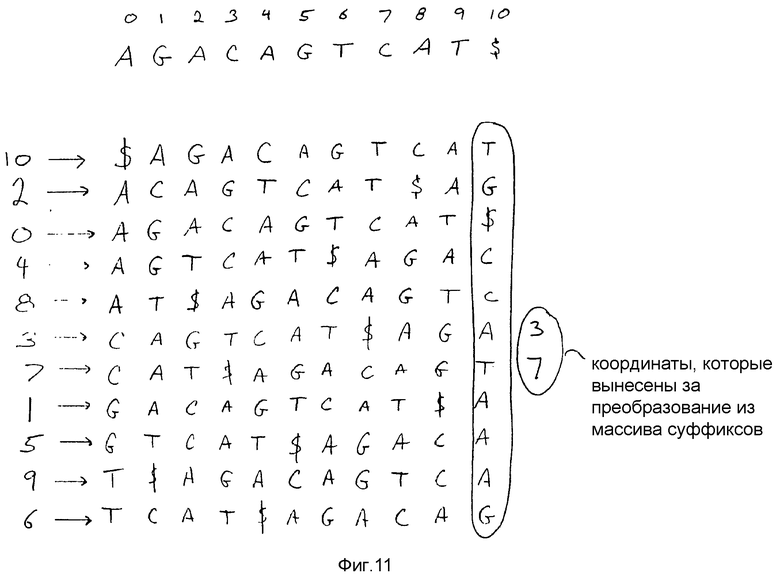
Изобретение относится к олигонуклеотидным зондам и их композициям, которые могут использоваться для анализа репрезентативной выборки генома. Предлагается алгоритм, использующий преобразование генома и вспомогательную структуру данных для быстрого и точного подсчета количества конкретного нуклеотидного мотива в геноме. Подобный алгоритм может быть использован для идентификации олигонуклеотидных зондов по настоящему изобретению. Также предоставляются системы и способы для анализа различий в числе копий нуклеотидных последовательностей и сравнения уровня метилирования между двумя геномами. Изобретение предлагает упрощенный способ анализа генома за счет виртуального представления, с минимальным риском перекрестной гибридизации и возможностью избежать завышенного или заниженного представления некоторых последовательностей при анализе генома. 8 н. и 63 з.п. ф-лы, 12 ил., 2 табл.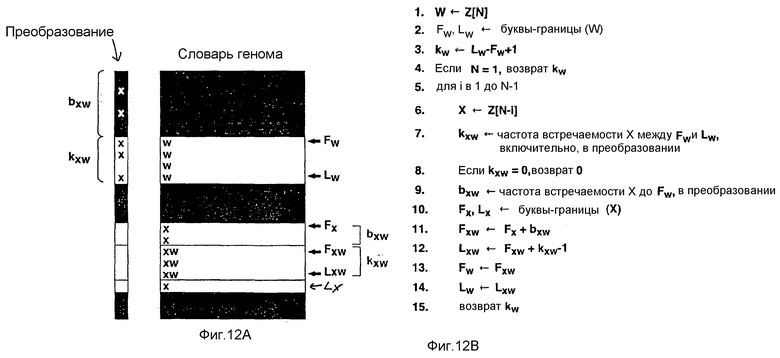
1. Композиция, содержащая множество зондов нуклеиновых кислот для анализа репрезентативной выборки генома, где
(a) указанное множество состоит из N зондов нуклеиновых кислот;
(b) каждый зонд указанного множества зондов нуклеиновых кислот имеет нуклеотидную последовательность, которая специфически гибридизуется с последовательностью генома из Z пар оснований;
(c) каждый зонд указанного множества имеет нуклеотидную последовательность, отличающуюся от нуклеотидной последовательности другого зонда указанного множества; и
(d) по меньшей мере Р% зондов указанного множества зондов нуклеиновых кислот
(i) имеет длину К нуклеотидов;
(ii) специфически гибридизуется по меньшей мере с одной молекулой нуклеиновой кислоты, присутствующей или присутствие которой предсказано, в репрезентативной выборке, полученной из указанного генома, причем действительная или виртуальная репрезентативная выборка имеет не более R% сложности указанного генома, и каждый отличный зонд по меньшей мере Р% указанного множества специфически гибридизуется с отличной молекулой нуклеиновой кислоты, присутствующей или теоретически определенной, в репрезентативной выборке указанного генома; и
(iii) включает от Y до X молекул, которые содержат точные совпадения L1 нуклеотидов с указанным геномом; и
где (A) N≥500;
(B) Z≥1·108;
(C) 300≥К≥30;
(D) 70%≥R≥0,001%;
(E) Р%=((((N·(R%/100%))+(3·сигма))/N)·100;
(F) сигма представляет собой квадратный корень из (N·(R%/100%)+(1-(R%/100)));
(G) целое число, ближайшее к (log4(Z)+2)≥L1≥целое число, ближайшее к log4(Z);
(H) X представляет собой целое число, ближайшее к D1·(K-L1+1);
(I) Y представляет собой целое число, ближайшее к D2·(K-L1+1);
(J) 1,5≥D1≥1; и
(К) 1≥D2≥0,5.
2. Композиция по п.1, где N равен по меньшей мере 85000 зондам нуклеиновых кислот.
3. Композиция по п.1, где Р% составляет по меньшей мере 70%.
4. Композиция по п.1, где Z составляет по меньшей мере 1·109.
5. Композиция по п.1, где геномом является геном млекопитающего.
6. Композиция по п.5, где геномом является геном человека.
7. Композиция по п.1, где R% составляет от 0,001 до 2,5%.
8. Композиция по п.1, где Р% составляет 70-80%.
9. Композиция по п.1, где D1 равно 1.
10. Композиция по п.1, где D2 равно 0,5.
11. Композиция по п.1, где L1 составляет 15, 16, 17, 18, 19, 20, 21, 22, 23 или 24.
12. Композиция по п.1, где
каждый зонд указанного Р% множества зондов нуклеиновых кислот дополнительно включает от В до А зондов, которые содержат точные совпадения L2 нуклеотидов с указанным геномом; и где
(a) L1>L2≥целое число, ближайшее к log4(Z)-3;
(b) А представляет собой целое число, ближайшее к
D3·((K-L2+1)·(Z/4L 2));
(c) В представляет собой целое число, ближайшее к
D4·((K-L2+1)·(Z/4L 2));
(d) 4≥D3≥1; и
(е) 1>D4≥0,5.
13. Композиция по п.12, где D3≤3, 2 или 1,5.
14. Композиция по п.1, где указанные Р% множества зондов нуклеиновых кислот по меньшей мере на 90% идентичны последовательности по меньшей мере одной молекулы нуклеиновой кислоты, находящейся или теоретически определенной, в указанной репрезентативной выборке, и каждая отличная молекула имеет по крайней мере 90% идентичность с отличной молекулой нуклеиновой кислоты, присутствующей или присутствие которой теоретически определено, в репрезентативной выборке.
15. Композиция по п.1, где К равно 40-70.
16. Композиция по п.1, где указанная репрезентативная выборка получена с помощью расщепления по специфичным последовательностям указанного генома.
17. Композиция по п.16, где расщепление по специфичным последовательностям осуществляют рестрикционной эндонуклеазой.
18. Композиция по п.1, где указанная репрезентативная выборка является сложной выборкой двух или более последовательных репрезентативных выборок, каждая из которых получена с помощью различных рестрикционных эндонуклеаз.
19. Композиция по п.1, где указанное множество зондов нуклеиновых кислот иммобилизуют на поверхности твердой фазы.
20. Композиция по п.1, где твердую фазу выбирают из группы, состоящей из нейлоновой мембраны, нитроцеллюлозной мембраны, стеклянного слайда и микросферы.
21. Композиция по п.1, где положения указанного множества зондов нуклеиновых кислот на указанной твердой фазе являются известными.
22. Композиция по п.1, где указанное множество зондов нуклеиновых кислот находится на микроматрице.
23. Композиция по п.1, где указанное множество зондов нуклеиновых кислот иммобилизовано на микросферах микроматрицы.
24. Композиция, содержащая множество зондов нуклеиновых кислот для анализа репрезентативной выборки генома, где
(a) указанное множество состоит по меньшей мере из 100 зондов нуклеиновых кислот;
(b) каждый зонд указанного множества зондов нуклеиновых кислот имеет нуклеотидную последовательность, которая по меньшей мере на 90% идентична последовательности в геноме из по меньшей мере Z пар оснований; и
(c) каждый зонд указанного множества имеет нуклеотидную последовательность, отличающуюся от нуклеотидной последовательности другого зонда указанного множества и по меньшей мере на 90% идентичен отличной последовательности генома; и
(d) по меньшей мере Р% указанного множества зондов нуклеиновых кислот
(i) имеет длину К нуклеотидов;
(ii) по меньшей мере на 90% идентичен последовательности с по меньшей мере одной молекулой нуклеиновой кислоты, находящейся или теоретически определенной, в репрезентативной выборке, полученной из указанного генома, где репрезентативная выборка имеет не более R% сложности указанного генома, и каждый отличный зонд по меньшей мере Р% указанного множества по меньшей мере на 90% идентичен отличной последовательности молекулы нуклеиновой кислоты присутствующей или теоретически определенной в репрезентативной выборке генома; и
(iii) включает от Y до Х зондов, которые содержат точные совпадения L1 нуклеотидов с указанной репрезентативной выборкой; и
где (A) Z≥1·108;
(B) 300≥К≥30;
(C) 70%≥R%≥0,001%;
(D) P%≥90-R%;
(Е) целое число, ближайшее к (log4((Z·R%)/100)+2)≥L1≥целое число, ближайшее к log4((Z·R%)/100);
(F) Х представляет собой целое число, ближайшее к D1·(K-L1+1);
(G) Y представляет собой целое число, ближайшее к D2·(K-L1+1);
(H)1,5≥D1≥1; и
(I) 1>D2≥0,5.
25. Композиция по п.24, содержащая по меньшей мере 85000 нуклеиновых кислот.
26. Композиция по п.24, где Z составляет по меньшей мере 1·109.
27. Композиция по п.24, где геномом является геном млекопитающего.
28. Композиция по п.27, где геномом является геном человека.
29. Композиция по п.24, где R% составляет от 0,001 до 2,5%.
30. Композиция по п.24, где Р% составляет 70-80%.
31. Композиция по п.24, где D1 равно 1.
32. Композиция по п.24, где D2 равно 0,5.
33. Композиция по п.24, где L1 составляет 15, 16, 17, 18, 19, 20, 21, 22, 23 или 24.
34. Композиция по п.24, где
каждый зонд указанного Р% множества зондов нуклеиновых кислот дополнительно включает от В до А зондов, которые содержат точные совпадения L2 нуклеотидов с указанным геномом; и
где (a) L1>L2≥целое число, ближайшее к log4(Z)-3;
(b) А представляет собой целое число, ближайшее к
D3·((K-L2+1)·(Z/4L 2));
(c) В представляет собой целое число, ближайшее к
D4·((K-L2+1)·(Z/4L 2));
(d) 4≥D3≥1; и
(е) 1>D4≥0,5.
35. Композиция по п.34, где D3≤3, 2 или 1,5.
36. Композиция по п.24, где указанные Р% множества зондов нуклеиновых кислот по меньшей мере на 90% идентичны последовательности по меньшей мере одной молекулы нуклеиновой кислоты, находящейся или теоретически определенной, в указанной репрезентативной выборке, и каждая отличная молекула имеет по крайней мере 90% идентичность с отличной молекулой нуклеиновой кислоты, присутствующей или присутствие которой теоретически определенно, в репрезентативной выборке.
37. Композиция по п.24, где К равно 40-70.
38. Композиция по п.24, где указанная репрезентативная выборка получена с помощью расщепления по специфичным последовательностям указанного генома.
39. Композиция по п.38, где расщепление по специфичным последовательностям осуществляют рестрикционной эндонуклеазой.
40. Композиция по п.24, где указанная репрезентативная выборка является сложной выборкой двух или более последовательных репрезентативных выборок, каждая из которых получена с помощью различных рестриктивных эндонуклеаз.
41. Композиция по п.24, где указанное множество зондов нуклеиновых кислот иммобилизуют на поверхности твердой фазы.
42. Композиция по п.24, где твердую фазу выбирают из группы, состоящей из нейлоновой мембраны, нитроцеллюлозной мембраны, стеклянного слайда и микросферы.
43. Композиция по п.24, где положения указанного множества зондов нуклеиновых кислот на указанной твердой фазе являются известными.
44. Композиция по п.24, где указанное множество зондов нуклеиновых кислот находится на микроматрице.
45. Композиция по п.24, где указанное множество зондов нуклеиновых кислот иммобилизовано на микросферах микроматрицы.
46. Способ анализа образца нуклеиновой кислоты, где указанный способ включает
(a) гибридизацию образца нуклеиновой кислоты с множеством зондов нуклеиновых кислот композиции по любому из пп.1-45; и
(b) определение, с какой из указанного множества зондов нуклеиновых кислот гибридизуется указанный образец.
47. Способ по п.46, где указанный образец является репрезентативной выборкой.
48. Способ по п.46, где указанное множество зондов нуклеиновых кислот иммобилизуют на поверхности твердой фазы.
49. Способ по п.48, где твердую фазу выбирают из группы, состоящей из нейлоновой мембраны, нитроцеллюлозной мембраны, стеклянного слайда и микросферы.
50. Способ по п.48, где положения указанного множества зондов нуклеиновых кислот на указанной твердой фазе являются известными.
51. Способ по п.50, где указанное множество зондов нуклеиновых кислот находится на микроматрице.
52. Способ по п.48, где указанное множество зондов нуклеиновых кислот иммобилизовано на микросферах микроматрицы.
53. Способ анализа между вариантами геномных последовательностей двух генов, где указанный способ включает
(a) получение первого генома и второго генома;
(b) получение меченных с возможностью детектирования репрезентативных выборок каждого генома, используя по меньшей мере один идентичный рестрикционный фермент;
(c) приведение в контакт указанных репрезентативных выборок и множества зондов нуклеиновых кислот по любому из пп.1-45 для возможности гибридизации репрезентативных выборок и указанного множества зондов нуклеиновых кислот; и
(d) сравнение уровней гибридизации одного или нескольких членов указанного множества зондов с указанными репрезентативными выборками, где разница указанных уровней указывает на различие числа копий между двумя геномами относительно геномной последовательности, выявляемой указанным членом(ами).
54. Способ по п.53, где две репрезентативные выборки являются меченными с возможностью их детекции.
55. Способ по п.54, где указанные репрезентативные выборки одновременно приводят в контакт с указанным множеством зондов нуклеиновых кислот.
56. Способ сравнения уровня метилирования геномной последовательности двух геномов, где указанный способ включает
(a) получение первого генома и второго генома;
(b) получение меченных с возможностью детектирования репрезентативных выборок каждого генома с использованием по меньшей мере одного идентичного фермента, где указанные репрезентативные выборки получают способом, чувствительным к метилированию;
(c) приведение в контакт указанных репрезентативных выборок с множеством зондов нуклеиновых кислот по любому из пп.1-45 для возможности гибридизации репрезентативных выборок и указанного множества зондов нуклеиновых кислот; и
(d) сравнение уровней гибридизации одного или несколько членов указанного множества зондов с указанными репрезентативными выборками, где разница указанных уровней указывает на различие в уровне метилирования двух геномов относительно геномной последовательности, выявляемой указанным членом(ами).
57. Способ по п.56, где указанный способ, чувствительный к метилированию, включает получение первой репрезентативной выборки с использованием первого рестрикционного фермента и второй репрезентативной выборки с использованием второго рестрикционного фермента, где указанные первый и второй рестрикционные ферменты распознают один и тот же рестрикционный сайт, но один фермент является чувствительным к метилированию, а другой - нет.
58. Способ по п.56, где указанный способ, чувствительный к метилированию, включает химическое расщепление метил-С последовательностей после создания репрезентативной выборки с помощью рестрикционного фермента нечувствительного к метилированию, так что репрезентативная выборка, полученная из метилированного генома отличается от репрезентативной выборки, полученной из неметилированного генома.
59. Способ получения олигонуклеотидного зонда, который
(a) имеет длину К нуклеотидов;
(b) по меньшей мере на 90% идентичен последовательности по меньшей мере одной молекулы нуклеиновой кислоты, находящейся или теоретически определенной, в репрезентативной выборке, полученной из генома по меньшей мере из Z пар оснований; и
(c) включает от Y до Х зондов, которые содержат точные совпадения L1 нуклеотидов с указанным геномом, где
(i) Z≥1·108;
(ii) 300≥K≥30;
(iii) целое число, ближайшее к (log4(Z)+2)>L1>целое число, ближайшее к log4(Z);
(iv) X представляет собой целое число, ближайшее к D1·(K-L1+1);
(v) Y представляет собой целое число, ближайшее к D2·(K-L1+1);
(vi) 1,5≥D1≥1; и
(vii) 1>D2≥0,5;
на основании информации, полученной способом, который включает
(A) расщепление указанного генома in silico рестрикционным ферментом с получением множества предсказанных молекул нуклеиновых кислот;
(B) получение репрезентативной выборки указанного генома путем идентификации предсказанных молекул нуклеиновых кислот, каждая из которых имеет длину 200-1200 пар оснований включительно;
(C) выбор олигонуклеотидного зонда длиной 30-300 нуклеотидов включительно, и который по меньшей мере на 90% идентичен последовательности предсказанной молекулы нуклеиновой кислоты (В);
(D) идентификацию всех отрезков нуклеотидов L1, встречающихся в указанном олигонуклеотиде; и
(Е) подтверждение того, что число каждого из указанных отрезков, встречающихся в указанном геноме, удовлетворяет требованиям (С).
60. Способ по п.59, где этап (Е) включает
получение сжатого преобразования указанного генома;
получение вспомогательной структуры данных, которая включает в себя информацию об указанном геноме; и
определение количества встречаемости «слова», подсчитанного для L1 нуклеотидов с использованием сжатого преобразования и вспомогательной структуры данных.
61. Способ по п.59, где этап (Е) включает
получение сжатого преобразования указанного генома;
осуществление итераций по каждому нуклеотиду указанного отрезка L1 нуклеотидов, начиная с последнего нуклеотида и продвигаясь к первому нуклеотиду, одна «буква» на одну итерацию, где нуклеотид, соответствующий конкретной итерации, сохраняют в виде индексного нуклеотида, при этом осуществлении итераций дополнительно проводят
определение области поиска, которая определяет границы непрерывного ряда нуклеотидов в указанном преобразовании;
подсчет количества нуклеотида, предшествующего указанному индексному нуклеотиду, в указанной области поиска; и
где указанное осуществление итераций прекращается, если не встречается нуклеотид, предшествующий указанному индексному нуклеотиду в указанной области поиска; и
вывод значения частоты встречаемости первого нуклеотида указанного отрезка L1 нуклеотидов, где это значение является эквивалентным значению частоты встречаемости указанного отрезка L1 нуклеотидов в указанном геноме.
62. Способ по п.61, дополнительно предусматривающий
получение вспомогательной структуры данных, где вспомогательная структура данных содержит
структуру данных К-интервалов, которая поддерживает промежуточную сумму каждого нуклеотида, который появлялся в указанном преобразовании вплоть до и включая конкретное заданное местоположение в указанном сжатом преобразовании; и
структуру данных подсчета «слов», которая обеспечивает быстрый доступ для поиска в сжатом преобразовании; и
при этом указанный подсчет и указанное определение выполняют с использованием указанной вспомогательной структуры данных и указанного сжатого преобразования.
63. Способ по п.62, где указанное преобразование остается сжатым, в то время как происходит указанный подсчет.
64. Способ по п.62, где указанное сжатое преобразование сжимают таким образом, чтобы каждые три «буквы» несжатого состояния преобразования сжимались, формируя байт, и где указанный подсчет распаковывает не более одного такого байта во время одной из указанных итераций.
65. Способ по п.60, где указанный геном содержит по меньшей мере три миллиарда «букв».
66. Способ по п.60, где указанное сжатое преобразование представляет собой преобразование генома Барроуза-Уиллера.
67. Способ по п.60, дополнительно предусматривающий получение данных, которые основаны на указанном преобразовании, где указанное определение включает использование указанных данных и индексного нуклеотида для определения указанной области поиска.
68. Способ по п.60, дополнительно предусматривающий
получение данных, которые основаны на указанном преобразовании; и определение частоты встречаемости предшествующего нуклеотида, где указанная частота встречаемости предшествующего нуклеотида представляет собой количество нуклеотида, предшествующего индексному нуклеотиду в указанном преобразовании до начала указанной области поиска;
где указанное определение включает использование указанных данных, указанного индексного нуклеотида и указанной частоты встречаемости предшествующего нуклеотида для определения указанной области поиска.
69. Способ по п.68, где указанную частоту встречаемости предшествующего нуклеотида получают, используя К-интервалы, где указанные К-интервалы хранят в заданных местоположениях в указанном преобразовании и поддерживают промежуточную сумму каждого нуклеотида, который появляется в указанном преобразовании вплоть до и включая конкретное заданное местоположение.
70. Множество олигонуклеотидных зондов для анализа репрезентативной выборки генома, каждый из которых получен способом по п.59, где указанное множество содержит по меньшей мере 500 зондов.
71. Множество олигонуклеотидных зондов для анализа репрезентативной выборки генома, каждый из которых получен способом по п.59, где указанное множество содержит по меньшей мере 85000 зондов.
| US 5846719, 08.12.1998 | |||
| US 2003054346, 20.03.2003 | |||
| US 2003077607, 24.04.2003 | |||
| СПОСОБ И УСТРОЙСТВО ДЛЯ ОБЕСПЕЧЕНИЯ КАЧЕСТВА УСЛУГИ СВЯЗИ ДЛЯ МОБИЛЬНОГО ТЕРМИНАЛА | 2004 |
|
RU2361373C2 |
| RU 94033149 A1, 27.07.1996 | |||
| LI F | |||
| ET AL.: "Selection of optimal DNA oligos for gene expression arrays", Bioinformatics, 2001, v.17, n.11, p.1067-1076 | |||
| FERRAGINA P | |||
| ET AL.: "An experimental study of compressed index", Information sciences, 2001, | |||
Авторы
Даты
2010-05-27—Публикация
2004-05-21—Подача