ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к способу детекции микроорганизма, присутствующего в продуктах питания или клинических образцах, а также к набору для детекции микроорганизма. Более конкретно, настоящее изобретение относится к способу и набору, с помощью которых можно избирательно детектировать живые клетки микроорганизма, присутствующие в продуктах питания или клинических образцах.
УРОВЕНЬ ТЕХНИКИ
Для определения количества живых бактерий в пищевых продуктах, клинических образцах или окружающей среде обычно используют метод культивирования на чашках. Однако при использовании этого метода для получения результата требуется приблизительно два дня.
Вследствие усовершенствования методов стерилизации и обработки пищевых продуктов увеличивается потребность в способах, позволяющих различить живое и мертвое состояние микроорганизмов, присутствующих в тестируемых образцах, даже в тех случаях, когда количество клеток является предельно низким. В частности, в областях санитарной инспекции пищевых продуктов и клинического тестирования предпринимаются попытки разработать способ быстрой детекции бактерий, позволяющий определить присутствие или отсутствие бактерий и их количество, путем амплификации методом ПЦР генов, специфичных для бактерий, с получением такого их количества, которое можно визуально обнаружить. Однако при использовании в качестве матрицы бактериальной ДНК также детектируется шум, обусловленный мертвыми клетками, изначально присутствующими в тестируемом образце, и, следовательно, положительный результат, полученный методом ПЦР, не позволяет сделать однозначный вывод о наличии живых бактерий. Поэтому в настоящее время метод ПЦР не находит широкого применения в областях санитарной инспекции пищевых продуктов и клинического тестирования, хотя он является высокочувствительным и быстрым методом.
В настоящее время предпринимаются попытки разработать способ детекции и количественного определения только живых клеток в тестируемом образце путем получения кДНК методом ПЦР с использованием обратной транскриптазы, мРНК в качестве матрицы и праймеров, специфичных для разных бактерий. Однако в таком способе обратная транскрипция мРНК мертвых клеток не ингибируется и, если в тестируемом образце присутствует 104 кое/мл, или 104 кое/г, или больше мертвых клеток, детектируется шум, обусловленный присутствием мертвых клеток. Следовательно, этот способ не позволяет однозначно проводить различие живого и мертвого состояния.
Способ, позволяющий различать живое и мертвое состояние микроорганизмов, таких как бактерии, с помощью метода ПЦР, описан в патентных документах 1 и 2. Однако способы дифференциальной детекции живого и мертвого состояний микроорганизмов, таких как бактерии, с помощью метода ПЦР имеют указанные ниже проблемы.
Для иллюстрации способа, раскрытого в патентном документе 1, приводятся примеры детекции мертвых клеток в пищевых продуктах, подвергавшихся высокотемпературной стерилизации в течение длительного времени, 100°C в течение 10-30 минут, и микроорганизмов, присутствующих в пищевых продуктах, подвергавшихся стерилизации этанолом или формальдегидом. Однако в действительности такие методы, особенно последний, не используются для пастеризации пищевых продуктов. Более того, не приведены примеры детекции только живых организмов в продуктах, подвергавшихся методам пастеризации, широко использующимся в настоящее время в пищевой промышленности, которые включают низкотемпературную длительную пастеризацию (пастеризация LTLT), высокотемпературную кратковременную пастеризацию (пастеризация HTST) или пастеризацию при сверхвысокой температуре (пастеризация UHT), а также детекции только живых конкретных патогенных бактерий в клинических образцах пациентов с инфекционным заболеванием, получающих антибиотики. Кроме того, в случае тестируемого образца пищевого продукта или клинического образца, содержащего мертвые клетки в концентрации 104 кое/мл или выше, количество конечных продуктов амплификации ПЦР, полученных из мертвых клеток, превышает предел чувствительности способа патентного документа 1, и, следовательно, нельзя определить, от живых или от мертвых клеток получен положительный ответ при анализе тестируемого образца методом ПЦР.
Затем в патентном документе 2 раскрывается способ, позволяющий дифференциально детектировать живые и мертвые клетки, который основан на том, что молярное соотношение РНК/ДНК в мертвых клетках ниже, чем в живых. Этот способ проводят следующим образом: экстрагируют общую РНК, с помощью реакции обратной транскрипции получают комплементарную ей ДНК, затем проводят ПЦР, чтобы рассчитать значение Ct, и определяют молярную концентрацию РНК, используя полученную заранее калибровочную кривую. Отдельно методом ПЦР амплифицируют участок хромосомальной ДНК, соответствующий упомянутой РНК, с получением значения Ct, рассчитывают с помощью калибровочной кривой концентрацию хромосомальной ДНК и определяют молярное соотношение РНК/ДНК. То есть для осуществления вышеуказанного способа требуется проведение сложной экстракции общей РНК, двух стадий реакции обратной транскрипции и ПЦР. Следовательно, с точки зрения количественных показателей и затрат времени этот способ хуже обычного метода ПЦР с использованием ДНК в качестве матрицы. Кроме того, в живых клетках РНК продуцируется постоянно, тогда как в мертвых клетках РНК разлагается на ранней стадии. Следовательно, указанному способу не хватает стабильности. Кроме того, в пищевом продукте или клиническом образце, содержащем большое количество мертвых клеток, с помощью этого способа можно определить живые клетки, только если их количество составляет 1/10 от количества мертвых клеток. Таким образом, этот способ нежелательно использовать в областях санитарной инспекции пищевых продуктов и клинического тестирования, где требуются скорость, высокая чувствительность и точность.
Патентный документ 1: Международная патентная заявка, не рассмотренная публикация в Японии № 2000-530118.
Патентный документ 2: Международная патентная публикация WO2002/052034, брошюра.
ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Задачи, решаемые изобретением
Целью настоящего изобретения является создание способа селективной детекции живых клеток (жизнеспособных и пригодных к культивированию) микроорганизма, присутствующего в пищевом продукте или клиническом образце, на фоне мертвых или поврежденных клеток (поврежденные клетки, другими словами, называют жизнеспособные, но не пригодные к культивированию клетки (клетки VNC)), то есть создание способа быстрой детекции, альтернативного методу культивирования, но превосходящего его по характеристикам, а также набор, используемый для проведения указанного способа.
Способы решения задач
Авторы настоящего изобретения провели тщательные исследования по созданию способа детекции живых и мертвых клеток, применимому к разным методам стерилизации, который обладает высокой чувствительностью, подходит для санитарной инспекции пищевых продуктов и, кроме того, позволяет детектировать конкретные патогенные бактерии у пациентов с инфекционным заболеванием в больницах или клинических центрах. В результате авторы обнаружили, что способ дифференциальной детекции живых и поврежденных клеток микроорганизма в тестируемом образце путем обработки такого образца ядом топоизомеразы и/или ядом ДНК-гиразы или путем обработки тестируемого образца моноазидом этидия, облучения видимым светом и обработки ядом топоизомеразы и/или ядом ДНК-гиразы, отличным от моноазида этидия, позволяет селективно амплифицировать хромосомную ДНК живых клеток методом ПЦР, и создали быстрый способ, являющийся альтернативным методу культивирования. Таким образом, авторы осуществили настоящее изобретение.
Таким образом, настоящее изобретение относится к способу детекции живых клеток микроорганизма в тестируемом образце, который включает следующие стадии:
a) стадию обработки тестируемого образца ядом топоизомеразы и/или ядом ДНК-гиразы,
b) стадию экстракции ДНК из тестируемого образца и амплификацию мишеневого участка экстрагированной ДНК методом ПЦР и
c) стадию анализа продукта амплификации.
В предпочтительном варианте осуществления вышеуказанного способа продукт амплификации анализируют с помощью стандартной кривой, отражающей взаимоотношение количества микроорганизма и продукта амплификации, полученной с использованием стандартных образцов микроорганизма.
В предпочтительном варианте осуществления вышеуказанного способа ПЦР проводят в режиме реального времени с одновременным анализом продукта.
В предпочтительном варианте осуществления вышеуказанного способа тестируемый образец может представлять собой молоко, молочный продукт, пищевой продукт, полученный с использованием молока или молочного продукта в качестве сырья, образец крови, образец мочи, образец спинномозговой жидкости, образец синовиальной жидкости и образец плевральной жидкости.
В предпочтительном варианте осуществления вышеуказанного способа микроорганизм представляет собой бактерию.
В предпочтительном варианте осуществления вышеуказанного способа мишеневый участок представляет собой ген рРНК 23S. В этом варианте осуществления ПЦР предпочтительно проводят с использованием набора праймеров, содержащего праймеры SEQ ID NO: 1 и 2, или набора праймеров, содержащего праймеры SEQ ID NO: 3 и 4.
В предпочтительном варианте осуществления вышеуказанного способа микроорганизм представляет собой патогенную бактерию. В этом варианте осуществления мишеневый участок предпочтительно представляет собой патогенный ген. Кроме того, в этом варианте осуществления ПЦР предпочтительно проводят с использованием набора праймеров, содержащего праймеры SEQ ID NO: 7 и 8.
В предпочтительном варианте осуществления вышеуказанного способа яд топоизомеразы выбран из амсакрина, камптотецина, доксорубицина, эллиптицина, этопозида, митоксантрона, саинтопина, топотекана и CP-115953.
В предпочтительном варианте осуществления вышеуказанного способа яд ДНК-гиразы выбран из ципрофлоксацина, офлоксацина, эноксацина, пефлоксацина, флероксацина, норфлоксацина, налидиксовой кислоты, оксолиновой кислоты и пиромидиновой кислоты.
В предпочтительном варианте осуществления вышеуказанного способа яд топоизомеразы и/или яд ДНК-гиразы представляет собой моноазид этидия, а способ включает стадию облучения тестируемого образца, к которому добавлен моноазид этидия, видимым светом.
В предпочтительном варианте осуществления вышеуказанного способа тестируемый образец обрабатывают моноазидом этидия и ядом топоизомеразы и/или ядом ДНК-гиразы, отличным от моноазида этидия.
В предпочтительном варианте осуществления вышеуказанного способа перед вышеупомянутой стадией a) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
Также настоящее изобретение относится к набору для детекции живых клеток микроорганизма в тестируемом образце методом ПЦР, который содержит следующие компоненты: яд топоизомеразы и/или яд ДНК-гиразы и праймеры для амплификации мишеневого участка ДНК микроорганизма, детектируемого методом ПЦР.
В предпочтительном варианте осуществления вышеуказанный набор содержит топоизомеразу и/или ДНК-гиразу.
В предпочтительном варианте осуществления вышеуказанного набора яд топоизомеразы выбран из амсакрина, камптотецина, доксорубицина, эллиптицина, этопозида, митоксантрона, саинтопина, топотекана и CP-115953.
В предпочтительном варианте осуществления вышеуказанного набора яд ДНК-гиразы выбран из ципрофлоксацина, офлоксацина, эноксацина, пефлоксацина, флероксацина, норфлоксацина, налидиксовой кислоты, оксолиновой кислоты и пиромидиновой кислоты.
В предпочтительном варианте осуществления вышеуказанный набор содержит моноазид этидия и яд топоизомеразы и/или яд ДНК-гиразы, отличный от моноазида этидия.
В предпочтительном варианте осуществления вышеуказанного набора упомянутые праймеры включают праймеры SEQ ID NO: 1 и 2 или праймеры SEQ ID NO: 3 и 4.
В другом предпочтительном варианте осуществления вышеуказанного набора упомянутые праймеры включают праймеры SEQ ID NO: 7 и 8.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[Фиг. 1] Фотографии электрофореза, демонстрирующие влияние внутриклеточной ДНКазы поврежденных клеток на хромосомную ДНК Listeria (поврежденные клетки), влияние амсакрина на хромосомную ДНК Listeria (поврежденные клетки) и влияние амсакрина на хромосомную ДНК Listeria (живые клетки):
Non: необработанные.
Амсакрин(-): амсакрин не добавляют,
Амсакрин(+): амсакрин добавляют,
1: Инкубация при 30°C в течение 24 часов,
2: Инкубация при 30°C в течение 48 часов,
3: Инкубация при 30°C в течение 72 часов.
[Фиг. 2] Фотографии электрофореза, демонстрирующие влияние внутриклеточной ДНКазы поврежденных клеток на хромосомную ДНК Enterobacter (поврежденные клетки):
Non: необработанные.
1: Инкубация при 37°C в течение 24 часов,
2: Инкубация при 37°C в течение 48 часов,
3: Инкубация при 37°C в течение 72 часов.
[Фиг. 3] Фотографии электрофореза, демонстрирующие влияние ципрофлоксацина на хромосомную ДНК Enterobacter (живые клетки и поврежденные клетки):
Non: необработанные.
1: Инкубация при 37°C в течение 1,5 часов,
2: Инкубация при 37°C в течение 3,5 часов,
3: Инкубация при 37°C в течение 5 часов,
4: Инкубация при 37°C в течение 72 часов.
[Фиг. 4] Фотографии электрофореза, демонстрирующие влияние внутриклеточной ДНКазы поврежденных клеток на хромосомную ДНК Listeria (поврежденные клетки):
Non: необработанные.
1: Инкубация при 30°C в течение 24 часов,
2: Инкубация при 30°C в течение 48 часов,
3: Инкубация при 30°C в течение 72 часов.
[Фиг. 5] Фотографии электрофореза, демонстрирующие влияние ципрофлоксацина на хромосомную ДНК Listeria (живые клетки и поврежденные клетки):
Non: необработанные.
1: Инкубация при 30°C в течение 1,5 часов,
2: Инкубация при 30°C в течение 3,5 часов,
3: Инкубация при 30°C в течение 5 часов,
4: Инкубация при 30°C в течение 72 часов.
[Фиг. 6] Кривая амплификации методом ПЦР в режиме реального времени с использованием в качестве матрицы гена рРНК 23S Enterobacter (живые клетки), обработанного ципрофлоксацином (полутоновая фотография).
[Фиг. 7] TM-график (кривая плавления) продукта амплификации методом ПЦР в режиме реального времени с использованием в качестве матрицы гена рРНК 23S Enterobacter (живые клетки), обработанного ципрофлоксацином (полутоновая фотография).
[Фиг. 8] Кривая амплификации методом ПЦР в режиме реального времени с использованием в качестве матрицы гена рРНК 23S Enterobacter (поврежденные клетки), обработанного ципрофлоксацином (полутоновая фотография).
[Фиг. 9] TM-график (кривая плавления) продукта амплификации методом ПЦР в режиме реального времени с использованием в качестве матрицы гена рРНК 23S Enterobacter (поврежденные клетки), обработанного ципрофлоксацином (полутоновая фотография).
[Фиг. 10] Кривая амплификации методом ПЦР в режиме реального времени с использованием в качестве матрицы гена рРНК 23S Listeria (живые клетки), обработанного ципрофлоксацином (полутоновая фотография).
[Фиг. 11] Кривая амплификации методом ПЦР в режиме реального времени с использованием в качестве матрицы гена рРНК 23S Listeria (поврежденные клетки), обработанного ципрофлоксацином (полутоновая фотография).
[Фиг. 12] График зависимости температуры клеточной суспензии, содержащейся в микропробирке, от времени погружения микропробирки в кипящую воду.
[Фиг. 13] Фотографии электрофореза продуктов амплификации генов методом ПЦР с использованием в качестве матрицы генов рРНК 16S семи видов бактерий (живые клетки и поврежденные клетки), обработанных или не обработанных EMA, которые для каждой бактерии показаны в следующем порядке: суспензия живых клеток, не обработанных EMA, суспензия живых клеток, обработанных EMA, суспензия поврежденных клеток, не обработанных EMA, и суспензия поврежденных клеток, обработанных EMA. Такой же порядок используется на фиг. 14 и 15.
[Фиг. 14] Фотографии электрофореза продуктов амплификации генов методом ПЦР с использованием в качестве матрицы генов рРНК 23S четырех видов бактерий (живые клетки и поврежденные клетки), обработанных или не обработанных EMA.
[Фиг. 15] Фотографии электрофореза продуктов амплификации генов методом ПЦР с использованием в качестве матрицы генов рРНК 23S Listeria (живые клетки и поврежденные клетки), обработанных или не обработанных EMA.
[Фиг. 16] Фотографии электрофореза продуктов амплификации генов методом ПЦР с использованием в качестве матрицы гена hlyA Listeria (живые клетки и поврежденные клетки), обработанного или не обработанного EMA и затем обработанного ядом ДНК-гиразы или ядом топоизомеразы:
Non: необработанные,
E: EMA,
1: EMA/ципрофлоксацин,
2: EMA/камптотецин,
3: EMA/этопозид,
4: EMA/эллиптицин,
5: EMA/митоксантрон,
6: EMA/амсакрин.
[Фиг. 17] Фотографии электрофореза продуктов амплификации генов методом ПЦР с использованием в качестве матрицы гена рРНК 16S Listeria (живые клетки и поврежденные клетки), обработанного или не обработанного EMA и затем обработанного ядом ДНК-гиразы или ядом топоизомеразы:
Non: необработанные,
E: EMA,
1: EMA/ципрофлоксацин,
2: EMA/камптотецин,
3: EMA/этопозид,
4: EMA/эллиптицин,
5: EMA/митоксантрон,
6: EMA/амсакрин.
[Фиг. 18] Фотографии электрофореза продуктов амплификации генов методом ПЦР с использованием в качестве матрицы гена рРНК 23S Listeria (живые клетки и поврежденные клетки), обработанного или не обработанного EMA и затем обработанного ядом ДНК-гиразы или ядом топоизомеразы:
Non: необработанные,
E: EMA,
1: EMA/ципрофлоксацин,
2: EMA/камптотецин,
3: EMA/этопозид,
4: EMA/эллиптицин,
5: EMA/митоксантрон,
6: EMA/амсакрин.
НАИЛУЧШИЙ СПОСОБ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Далее приводится подробное описание предпочтительных вариантов осуществления настоящего изобретения. Тем не менее, настоящее изобретение не ограничивается нижеследующими предпочтительными вариантами осуществления, которые можно свободно модифицировать в пределах объема формулы изобретения. В этом описании, если не указано иначе, проценты приводятся в виде массового отношения.
<1> Способ по настоящему изобретению
Способ по настоящему изобретению представляет собой способ детекции живых клеток микроорганизма в тестируемом образце, который включает следующие стадии:
a) стадию обработки тестируемого образца ядом топоизомеразы и/или ядом ДНК-гиразы,
b) стадию экстракции ДНК из тестируемого образца и амплификации мишеневого участка экстрагированной ДНК методом ПЦР и
с) стадию анализа продукта амплификации.
В описании под "тестируемым образцом" понимают объект, в котором детектируют живые клетки микроорганизма, этот термин не имеет конкретных ограничений при условии, что присутствие живых клеток можно детектировать путем амплификации конкретного участка хромосомной ДНК методом ПЦР. Примеры включают пищевые продукты, такие как молоко, молочные продукты, пищевые продукты, полученные с использованием молока или молочных продуктов в качестве сырья, образцы крови, образцы мочи, образцы спинномозговой жидкости, образцы синовиальной жидкости, образцы плевральной жидкости и тому подобное. Молоко, молочные продукты, пищевые продукты, полученные с использованием молока или молочных продуктов в качестве сырья, являются особенно предпочтительными. В настоящем изобретении тестируемый образец может представлять собой один из вышеуказанных продуктов и биологических образцов как есть, или он может быть получен путем разбавления или концентрирования одного из вышеуказанных продуктов и биологических образцов или путем обработки одного из вышеуказанных продуктов и биологических образцов отличной от обработки по способу по настоящему изобретению. Примеры обработки включают тепловую обработку, фильтрацию, центрифугирование и тому подобное.
"Микроорганизм" представляет собой объект, который можно детектировать способом по настоящему изобретению, этот термин не имеет конкретных ограничений при условии, что его можно детектировать методом ПЦР и что яд топоизомеразы и яд ДНК-гиразы действуют на живые клетки микроорганизма иначе, чем на мертвые и поврежденные клетки микроорганизма. Предпочтительные примеры включают бактерии, гифомицеты, дрожжи и тому подобное. Бактерии включают как грамположительные, так и грамотрицательные бактерии. Примеры грамположительных бактерий включают бактерии Staphylococcus, такие как Staphylococcus epidermidis, бактерии Streptococcus, бактерии Listeria, такие как Listeria monocytogenes, бактерии Bacillus, такие как Bacillus cereus, бактерии Mycobacterium и тому подобное. Примеры грамотрицательных бактерий включают кишечные бактерии, например бактерии Escherichia, такие как Escherichia coli, бактерии Enterobacter, такие как Enterobacter sakazakii, бактерии Citrobacter, такие как Citrobacter koseri, бактерии Klebsiella, такие как Klebsiella oxytoca, а также бактерии Salmonella, бактерии Vibrio, бактерии Pseudomonas и тому подобное.
В настоящем изобретении термин "живая клетка" относится к клетке, которая способна пролиферировать и обладает метаболической активностью микроорганизма (состояние живой и пригодной к культивированию клетки) при культивировании в обычных условиях и которая практически не имеет повреждений клеточной стенки. Примером упомянутой выше метаболической активности может служить АТФ-активность, эстеразная активность и др.
Термин "мертвая клетка" относится к клетке, которая не способна пролиферировать и не обладает метаболической активностью микроорганизма (состояние мертвой клетки), даже если ее культивируют в оптимальных условиях. Кроме того, стенка такой клетки имеет значительные повреждения, хотя ее структура в целом сохраняется, и ядерный краситель, который обладает плохой проникающей способностью, такой как иодид пропидия, может проникать через клеточную стенку.
Термин "поврежденная клетка" (поврежденная клетка или живая, но не пригодная к культивированию клетка) относится к клетке, которая плохо пролиферирует, даже если ее культивируют в оптимальных условиях, поскольку она имеет повреждения, вызванные искусственными факторами или воздействием окружающей среды, и обладает более низкой метаболической активностью, чем живая клетка, но сохраняет значительный уровень метаболической активности в отличие от мертвой клетки.
Детекции бактерий, находящихся в состоянии поврежденной клетки, путем тепловой обработки среднего уровня или введения антибиотиков уделяется большое внимание, особенно в областях санитарной инспекции пищевых продуктов и клинического тестирования, и настоящее изобретение относится к способу детекции микроорганизма, который позволяет не только детектировать живые клетки, но и отличать живые клетки от мертвых или поврежденных клеток.
Единицей измерения количества живых, поврежденных и мертвых клеток является число клеток/мл. Число живых клеток можно приблизительно определить как число колоний (кое/мл (колониеобразующих единиц/мл)), образованных при культивировании клеток в оптимальных условиях на подходящей культуральной среде. Стандартный образец поврежденных клеток можно получить путем тепловой обработки суспензии живых клеток, например путем тепловой обработки в кипящей воде. В таком образце число поврежденных клеток можно приблизительно определить как число кое/мл в суспензии живых клеток до тепловой обработки. Хотя время тепловой обработки в кипящей воде, необходимое для получения поврежденных клеток, изменяется в зависимости от типа микроорганизма, поврежденные клетки бактерий, описанных в примерах, можно получить, например, путем тепловой обработки в течение приблизительно 50 секунд. Кроме того, стандартный образец поврежденных клеток можно получить путем обработки антибиотиком. В этом случае число поврежденных клеток можно приблизительно определить как число колоний (кое/мл), образованных при культивировании клеток в оптимальных условиях на подходящей культуральной среде после удаления антибиотика из суспензии живых клеток путем измерения коэффициента пропускания видимого света (длина волны: 600 нм), то есть мутности, и сравнения полученного значения с мутностью суспензии живых клеток с известной плотностью живых клеток.
Способ по настоящему изобретению предназначен для детекции живых клеток, а клетки, отличающиеся от живых, могут быть поврежденными или мертвыми клетками.
В настоящем изобретении под "детекцией живых клеток" понимают определение присутствия или отсутствия, а также количества живых клеток в тестируемом образце. Количество живых клеток не ограничивается абсолютным значением и может представлять собой относительное количество, полученное при сравнении с контрольным образцом.
Далее способ по настоящему изобретению описывается постадийно.
(1) Стадия a)
Тестируемый образец обрабатывают ядом топоизомеразы и/или ядом ДНК-гиразы.
В настоящем изобретении используют яд топоизомеразы и яд ДНК-гиразы, которые не ингибируют активность топоизомеразы и ДНК-гиразы, соответственно, связанную с расщеплением ДНК, однако ингибируют повторное лигирование ДНК или увеличивают прямую скорость расщепления ДНК. Предпочтительно, яд топоизомеразы и яд ДНК-гиразы представляют собой яды, которые способны связываться с хромосомной ДНК микроорганизма путем ковалентного присоединения, встраиваться в хромосомную ДНК и связываться с ней путем ковалентного присоединения при облучении видимым светом, просто встраиваться в хромосомную ДНК или образовывать комплекс с топоизомеразой или ДНК-гиразой.
Можно использовать и яд топоизомеразы, и яд ДНК-гиразы или один из них.
Предпочтительно, яд топоизомеразы и яд ДНК-гиразы представляют собой яды, которые оказывают различное действие на живые клетки и поврежденные клетки, мертвые клетки, соматические клетки, такие как бычьи лейкоциты, лейкоциты, тромбоциты и тому подобное, более конкретно, они представляют собой яды, которые легче проникают через клеточные стенки поврежденных и мертвых клеток и через клеточные мембраны соматических клеток, таких как бычьи лейкоциты, лейкоциты, тромбоциты и тому подобное, чем через клеточные стенки живых клеток.
Примеры ядов топоизомеразы включают амсакрин, камптотецин, доксорубицин, эллиптицин, этопозид, митоксантрон, саинтопин, топотекан, CP-115953 и тому подобное. Можно использовать один вид яда топоизомеразы или сочетание двух или более видов данного яда.
Примеры яда ДНК-гиразы включают ципрофлоксацин, офлоксацин, эноксацин, пефлоксацин, флероксацин, норфлоксацин, налидиксовую кислоту, оксолиновую кислоту, пиромидиновую кислоту и тому подобное. Можно использовать независимо один вид яда ДНК-гиразы или сочетание двух или более видов.
Перед проведением обработки ядом топоизомеразы или ядом ДНК-гиразы подбирают подходящие условия. Например, чтобы подобрать условия, позволяющие легко отличать живые клетки от мертвых и поврежденных клеток, к суспензиям живых клеток и мертвых или поврежденных клеток детектируемого микроорганизма добавляют яд топоизомеразы или яд ДНК-гиразы в разных концентрациях, инкубируют в течение разных промежутков времени, затем клетки собирают центрифугированием или другим способом и анализируют методом ПЦР. Затем, чтобы подобрать условия, позволяющие легко отличать живые клетки детектируемого микроорганизма от соматических клеток, таких как бычьи лейкоциты, тромбоциты и тому подобное, к суспензиям живых клеток и вышеуказанных других клеток добавляют яд топоизомеразы в разных концентрациях, инкубируют в течение заранее установленного времени, затем живые клетки и вышеуказанные другие клетки собирают центрифугированием или другим способом и анализируют методом ПЦР. Примеры таких условий конкретно включают в случае амсакрина - конечную концентрацию от 1 до 100 мкг/мл, температуру от 25 до 37°C и время обработки от 5 минут до 48 часов, в случае эллиптицина - конечную концентрацию от 0,05 до 5 мкг/мл, температуру от 25 до 37°C и время обработки от 10 минут до 48 часов, в случае камптотецина - конечную концентрацию от 1 до 100 мкг/мл, температуру от 25 до 37°C и время обработки от 10 минут до 48 часов, в случае ципрофлоксацина - конечную концентрацию от 0,4 до 40 мкг/мл, температуру от 25 до 37°C и время обработки от 10 минут до 48 часов, в случае этопозида - конечную концентрацию от 1 до 100 мкг/мл, температуру от 25 до 37°C и время обработки от 5 минут до 48 часов и в случае митоксантрона - конечную концентрацию от 0,1 до 10 мкг/мл, температуру от 25 до 37°C и время обработки от 10 минут до 48 часов. Обработку тестируемого образца в заранее определенных условиях завершают путем выведения яда разбавлением и/или путем разделения компонентов смеси центрифугированием или другим способом.
Вышеупомянутые яд топоизомеразы и яд ДНК-гиразы легче проникают через стенки поврежденных и мертвых клеток, чем через стенки живых клеток. Следовательно, считается, что при обработке в течение указанных выше промежутков времени яды практически не проникают через стенки живых клеток, но проникают через стенки поврежденных клеток, мертвых клеток и живых соматических клеток, как мертвых клеток. Считается также, что они могут проникать даже в живые соматические клетки, поскольку у них нет клеточных стенок, а есть только клеточные мембраны. Установлено, что в результате яд топоизомеразы или яд ДНК-гиразы проникает в мертвые соматические клетки, мертвые бактерии и поврежденные бактерии, затем беспорядочно связывается с хромосомной ДНК путем ковалентного присоединения, встраивается в ДНК или образует комплекс с топоизомеразой и затем ингибирует повторное лигирование ДНК под действием топоизомеразы II или топоизомеразы I в соматических клетках, или топоизомеразы IV или топоизомераз I и III, или ДНК-гиразы в мертвых или поврежденные клетках или увеличивает прямую скорость расщепления ДНК, вызывая фрагментацию хромосомной ДНК.
Поскольку хромосомная ДНК в поврежденных и мертвых клетках в отличие от живых клеток преимущественно находится во фрагментированном состоянии, мишеневый участок хромосомной ДНК живых клеток можно амплифицировать методом ПЦР, тогда как расщепление мишеневого участка в поврежденных или мертвых клетках приводит к ингибированию амплификации методом ПЦР. Кроме того, при использовании, например, амсакрина или камптотецина происходит поперечная сшивка, которая также приводит к ингибированию амплификации методом ПЦР. Следовательно, метод ПЦР позволяет избирательно детектировать живые клетки на фоне поврежденных или мертвых клеток.
В мертвых клетках активность внутриклеточной топоизомеразы и/или ДНК-гиразы может отсутствовать. Кроме того, в поврежденных клетках активность указанных ферментов может быть снижена или может отсутствовать. Поэтому в предпочтительном варианте осуществления настоящего изобретения тестируемый образец перед стадией a) обрабатывают топоизомеразой и/или ДНК-гиразой (стадия d)). Подробное описание стадии d) приведено ниже.
В другом предпочтительном варианте осуществления настоящего изобретения яд топоизомеразы или яд ДНК-гиразы представляет собой моноазид этидия, а способ включает стадию облучения тестируемого образца видимым светом после добавления моноазида этидия. Моноазид этидия (EMA) легче проникает через клеточные стенки поврежденных или мертвых клеток, чем через стенки живых клеток микроорганизмов. Следовательно, считается, что EMA практически не проникает через клеточные стенки живых бактерий, но проникает через клеточные стенки поврежденных и мертвых бактерий, а также через клеточные мембраны соматических клеток, как мертвых клеток. Если лейкоциты и тромбоциты в крови представляют собой живые клетки, то EMA легче проникает через клеточные мембраны в стерильной воде или в гипотоническом солевом растворе. EMA проникает в соматические клетки, как мертвые клетки, поврежденные бактерии и мертвые бактерии, беспорядочно встраивается в хромосомную ДНК, затем под действием видимого света встроенный EMA превращается в нитрен и связывается с хромосомной ДНК путем ковалентного присоединения. Установлено, что после этого EMA ингибирует повторное лигирование ДНК под действием топоизомеразы II в соматических клетках, топоизомеразы IV или ДНК-гиразы в поврежденных или мертвых бактериях, вызывая фрагментацию хромосомной ДНК.
Перед обработкой EMA подбирают подходящие условия. Например, чтобы подобрать условия, позволяющие легко отличать живые клетки от поврежденных клеток, к суспензиям живых клеток и мертвых или поврежденных клеток детектируемого микроорганизма добавляют EMA в разных концентрациях, инкубируют в течение разных промежутков времени, облучают видимым светом и затем клетки собирают центрифугированием или другим способом и анализируют методом ПЦР.
Предпочтительные условия облучения видимым светом также подбирают путем проведения эксперимента, подобного описанному выше, с использованием разных периодов облучения. А именно, обработку EMA предпочтительно проводят с использованием конечной концентрации от 0,5 до 100 мкг/мл при температуре от 4 до 10°C в течение от 5 минут до 48 часов. Кроме того, обработку EMA предпочтительно проводят с использованием защиты от света. Видимый свет предпочтительно используют в диапазоне от 500 до 700 нм. Конкретные примеры условий облучения видимым светом включают облучение мощностью от 100 до 750 Вт в течение от 5 минут до 2 часов на расстоянии от 10 до 50 см от тестируемого образца. Облучение видимым светом предпочтительно проводят при низкой температуре, например при охлаждении образца на льду.
В особенно предпочтительном варианте осуществления настоящего изобретения тестируемый образец подвергают обработке EMA, облучению видимым светом и обработке ядом топоизомеразы и/или ядом ДНК-гиразы, отличным от EMA. В данном случае порядок обработки EMA, облучения видимым светом и обработки ядом топоизомеразы и/или ядом ДНК-гиразы, отличным от EMA, конкретно не ограничивается, причем указанные обработки можно проводить одновременно.
(2) Стадия b)
ДНК экстрагируют из тестируемого образца, обработанного по способу стадии a), после чего мишеневый участок экстрагированной ДНК амплифицируют методом ПЦР (White T.J. et al., Trends Genet., 5, 185 (1989)).
Способ экстракции ДНК из тестируемого образца конкретно не ограничивается при условии, что экстрагированную ДНК можно будет использовать в качестве матрицы для ПЦР, причем экстракцию можно проводить с помощью традиционных способов экстракции ДНК микроорганизмов.
Способ экстракции ДНК описан, например, в Maniatis T., Fritsch E.F., Sambrook J. "Molecular Cloning: A Laboratory Manual", 3rd edn., Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 2001.
В настоящем изобретении "мишеневый участок" конкретно не ограничивается при условии, что он представляет собой участок хромосомной ДНК, который можно амплифицировать методом ПЦР с использованием праймеров по настоящему изобретению, и позволяет проводить детекцию микроорганизма, этот участок выбирают в зависимости от поставленной задачи. Например, если в тестируемом образце присутствуют клетки, отличные от детектируемого микроорганизма, то мишеневый участок, предпочтительно, содержит последовательность, специфичную для микроорганизма, являющегося объектом детекции. Кроме того, в зависимости от поставленной задачи мишеневый участок может содержать последовательность, общую для нескольких видов микроорганизмов. Мишеневый участок может состоять из одного участка или из двух или более участков. Используя набор праймеров, подходящий для мишеневого участка детектируемого микроорганизма, и набор праймеров, подходящий для хромосомных ДНК широкого ряда микроорганизмов, можно одновременно определить число живых клеток детектируемого микроорганизма и число живых клеток широкого ряда микроорганизмов. Длина мишеневого участка обычно составляет, например, от 80 до 3000 нуклеотидов, предпочтительно, от 900 до 3000 нуклеотидов, особенно предпочтительно, от 2000 до 3000 нуклеотидов. Конкретные примеры включают ген рРНК 16S и ген рРНК 23S. Из них предпочтительным является ген рРНК 23S.
Праймеры, используемые для ПЦР, конкретно не ограничиваются при условии, что они обеспечивают специфическую амплификацию вышеуказанного мишеневого участка. Конкретные примеры праймеров, подходящих для гена рРНК 23S, включают набор праймеров, имеющих последовательности SEQ ID NO: 1 и 2, и набор праймеров, имеющих последовательности SEQ ID NO: 3 и 4. Примеры праймеров, подходящих для гена рРНК 16S, включают набор праймеров, имеющих последовательности SEQ ID NO: 5 и 6.
Если детектируемый микроорганизм представляет собой патогенную бактерию, примеры мишеневого участка включают патогенный ген. Примеры патогенного гена включают ген листериолизина O (hlyA) бактерии Listeria, ген энтеротоксина и ген invA бактерии Salmonella, ген веротоксина патогенной E. coli 0-157, ген MMS бактерии Enterobacter (Enterobacter sakazakii), ген энтеротоксина Staphylococcus aureus, ген цереулида (токсин, вызывающий рвоту) и ген энтеротоксина Bacillus cereus, гены разных токсинов Clostridium botulinum и тому подобное. Примеры праймеров, подходящих для патогенного гена, включают набор праймеров, имеющих последовательности SEQ ID NO: 7 и 8.
Используя праймеры, подходящие для двух или более видов микроорганизмов, в тестируемом образце можно детектировать живые клетки двух или более видов микроорганизмов. Кроме того, если используются праймеры, специфичные для конкретной бактерии, в тестируемом образце можно детектировать живые клетки данной бактерии.
Для проведения ПЦР можно подобрать подходящие условия, которые конкретно не ограничиваются при условии, что они обеспечивают специфическую амплификацию в соответствии с принципами ПЦР.
В варианте осуществления, который среди прочих вариантов осуществления настоящего изобретения включает обработку EMA и обработку видимым светом, применение длинного мишеневого участка, например содержащего 2000 или более нуклеотидов, позволяет эффективно детектировать живые клетки с использованием только обработки EMA и облучения видимым светом. Наоборот, в случае короткого мишеневого участка, например состоящего из 200 или менее нуклеотидов, предпочтительно наряду с обработкой EMA и облучением видимым светом проводить обработку ядом топоизомеразы и/или ядом ДНК-гиразы, отличным от EMA.
(3) Стадия c)
Затем анализируют продукт амплификации ПЦР. Способ анализа конкретно не ограничивается при условии, что он позволяет осуществлять детекцию или количественное определение продукта амплификации ПЦР, его примеры включают электрофорез, ПЦР в режиме реального времени (Nogva et al., Application of 5'-nuclease PCR for quantitative detection of Listeria monocytogenes in pure cultures and water, skim milk and unpasteurized whole milk, Appl. Environ. Microbiol., vol. 66, 2000, pp.4266-4271; Nogva et al., Application of the 5'-nuclease PCR assay in evaluation and development of methods for quantitative detection of Campylobacter jejuni, Appl. Environ. Microbiol., vol. 66, 2000, pp.4029-4036) и тому подобное. С помощью электрофореза можно определить количество и размер продукта амплификации ПЦР. С помощью ПЦР в режиме реального времени можно быстро определить количество продукта амплификации ПЦР. В случае применения ПЦР в режиме реального времени в силу того, что изменения интенсивности флуоресценции в 1-10 циклах амплификации, как правило, соответствуют уровню шума и практически равны нулю, их можно считать значениями контрольных образцов, не содержащих продуктов амплификации. Значение интенсивности флуоресценции, полученное путем вычисления стандартного отклонения SD для изменения интенсивности флуоресценции в 1-10 циклах амплификации и умножения стандартного отклонения на 10, определяют как пороговое значение. Номер первого цикла ПЦР, в котором изменение интенсивности флуоресценции превышает пороговое значение, называют значением порогового цикла (значение Ct). Следовательно, чем больше исходное количество ДНК-матрицы в растворе для ПЦР, тем меньше значение Ct, и, наоборот, чем меньше исходное количество ДНК-матрицы в растворе для ПЦР, тем больше значение Ct. Кроме того, при одинаковом количестве матрицы ДНК повышенная встречаемость расщепления мишеневого участка ПЦР в матрице приводит к увеличению значения Ct для ПЦР данного участка.
Присутствие или отсутствие продукта амплификации также можно определить путем анализа кривой плавления (TM) продукта амплификации.
Все вышеуказанные способы также можно использовать для оптимизации разных условий, используемых в способе по настоящему изобретению.
Если живые клетки детектируют способом по настоящему изобретению, то точность определения присутствия или отсутствия и количества живых клеток в анализе продукта амплификации ПЦР можно повысить путем применения стандартной кривой, отражающей зависимость количества продукта амплификации от количества микроорганизма, которую получают с использованием стандартных образцов идентифицированного микроорганизма. Хотя стандартная кривая может быть получена заранее, предпочтительно используют стандартную кривую, полученную путем одновременного проведения стадий способа по настоящему изобретению для стандартных образцов и тестируемого образца. Более того, если взаимозависимость между количеством микроорганизма и количеством ДНК установлена заранее, то ДНК, выделенную из микроорганизма, также можно использовать в качестве стандартного образца.
(4) Стадия d)
Как указано выше, внутриклеточная активность топоизомеразы и/или ДНК-гиразы в мертвых клетках может отсутствовать, и расщепление хромосомной ДНК не наблюдается даже после обработки ядом топоизомеразы или ядом ДНК-гиразы. В данном случае, если тестируемый образец перед проведением стадии a) обрабатывают топоизомеразой и/или ДНК-гиразой, происходит расщепление ДНК, характерное для мертвых клеток, и, следовательно, амплификация мишеневого участка методом ПЦР может ингибироваться.
Можно использовать и топоизомеразу, и ДНК-гиразу или одну из них. Кроме того, одновременно можно использовать один, два или более видов каждого фермента.
Конкретные примеры условий реакций, протекающих с участием топоизомеразы или ДНК-гиразы, включают условия, приведенные ниже в примерах отнесения. Однако обычно эти реакции проводят следующим образом: из пищевого продукта или клинического образца, такого как кровь, получают супернатант, содержащий микроорганизмы, который центрифугируют при 4°C и 14000 g в течение 10 минут, супернатант удаляют, к остатку добавляют 1 мл буфера для расщепления ДНК (10 мM буфер Tris-HCl, pH 7,9, 50 мM хлорид калия, 50 мM хлорид натрия, 5 мM хлорид магния, 0,01 мM EDTA, 2,5% глицерин), к полученной смеси добавляют топоизомеразу или ДНК-гиразу до конечной концентрации 1-50 мM и затем АТФ до конечной концентрации 1-50 мM, после чего реакционную смесь оставляют при 30-37°C на 5-30 минут. Поскольку для функционирования топоизомеразы и ДНК-гиразы требуется АТФ, к тестируемому образцу, обрабатываемому данными ферментами, добавляют АТФ или систему синтеза АТФ.
<3> Набор по настоящему изобретению
Набор по настоящему изобретению представляет собой набор для детекции живых клеток микроорганизма в тестируемом образце методом ПЦР, он содержит яд топоизомеразы и/или яд ДНК-гиразы и праймеры для амплификации мишеневого участка ДНК микроорганизма, детектируемого методом ПЦР.
В вышеуказанный набор входят яд топоизомеразы и яд ДНК-гиразы, описанные для способа по настоящему изобретению.
В предпочтительном варианте осуществления набор по настоящему изобретению содержит EMA и другой яд топоизомеразы и/или яд ДНК-гиразы, отличный от EMA, как яда топоизомеразы и/или яда ДНК-гиразы.
Помимо вышеуказанных компонентов набор по настоящему изобретению содержит топоизомеразу и/или ДНК-гиразу.
Набор по настоящему изобретению может дополнительно содержать разбавитель, раствор для реакции с участием топоизомеразы и/или ДНК-гиразы, инструкцию с описанием способа по настоящему изобретению и тому подобное.
ПРИМЕРЫ
Далее настоящее изобретение описано более подробно с помощью нижеследующих примеров. Однако настоящее изобретение не ограничивается указанными примерами.
[Пример 1]
Живые клетки и поврежденные клетки микроорганизма обрабатывают ядом топоизомеразы и определяют степень расщепления каждой хромосомной ДНК.
1. Получение образцов
1-1) Получение суспензий живых и поврежденных клеток
Listeria monocytogenes (Listeria monocytogenes JCM 2873, в дальнейшем называемая также "Listeria"), которая представляет собой грамположительную бактерию, культивируют при 30°C с использованием бульона BHI, 40 мл культуральной среды, содержащей клетки в логарифмической фазе роста, центрифугируют при 4°C и 8000 g в течение 15 минут, после чего супернатант удаляют. К клеткам добавляют 40 мл физиологического раствора, смесь перемешивают с достаточной интенсивностью и снова центрифугируют при охлаждении в тех же условиях, супернатант удаляют, к клеткам добавляют 10 мл физиологического раствора и получают суспензию живых клеток. Число живых клеток, измеренное на стандартной культуральной агаровой среде, в суспензии составляет 1,2×109 кое/мл.
Затем 1 мл вышеуказанной суспензии живых клеток помещают в микропробирку объемом 1,5 мл, пробирку погружают в кипящую воду на 50 секунд и быстро охлаждают водой со льдом, получая суспензию поврежденных клеток. Считается, что такая суспензия содержит небольшое количество живых и мертвых клеток, но в основном она состоит из поврежденных клеток, поэтому клетки суспензии описывают как "поврежденные". Кроме того, способ по настоящему изобретению изначально предназначен для детекции живых клеток, а клетки микроорганизма, отличающиеся от живых клеток, могут представлять собой поврежденные или мертвые клетки. Вышеуказанное относится и к другим бактериям, указанным ниже.
1-2) Обработка ядом топоизомеразы
К 9 мл свежеполученного бульона BHI (число как живых, так и поврежденных клеток в среде составляет 1,2×108 кое/мл) добавляют 1 мл каждой из полученных ранее суспензий живых и поврежденных клеток Listeria (1,2×109 кое/мл, соответственно) и затем к среде добавляют 100 мкл раствора амсакрина в ДМСО с концентрацией 5 мг/мл. Конечная концентрация амсакрина составляет 50 мкг/мл, а конечная концентрация ДМСО составляет 1%. Затем клетки каждого типа инкубируют при 30°C в течение 24 часов, 48 часов или 72 часов.
Для исследования влияния активной ДНКазы, остающейся в поврежденных клетках, на хромосомную ДНК поврежденных клеток, 1 мл суспензии поврежденных клеток добавляют к 9 мл свежеполученного бульона BHI, затем к среде добавляют 100 мкл ДМСО вместо вышеуказанного раствора амсакрина (конечная концентрация ДМСО: 1%) и клетки инкубируют при 30°C в течение 72 часов. В качестве контролей используют суспензию живых клеток и суспензию поврежденных клеток в объеме 1 мл каждая.
1-3) Экстракция ДНК
Каждую суспензию центрифугируют при 4°C и 8000 g в течение 15 минут, после чего супернатант полностью удаляют. К осадку добавляют 0,5 мл 5 мM раствора EDTA и 20 мкл полученного заранее раствора ахромопептидазы (Wako Pure Chemical Industries, номер по каталогу: 014-09661) с концентрацией 5 мг/мл, содержащего 10 мM водный раствор NaCl, после чего смесь оставляют при 50°C на 30 минут. Затем к смеси добавляют 0,5 мл 10 мM буфера Tris-HCl (pH 8,0), 20 мкл протеинкиназы K, 1250 ед./мл (Sigma, E.C. 3.4.21.64), 400 мкл полученного заранее раствора SDS в стерильной воде с концентрацией 10% (мас./об.), после чего реакцию оставляют протекать в течение ночи при 50°C.
Каждую обработанную суспензию помещают в микропробирки объемом 2 мл так, чтобы суспензия занимала половину объема пробирки, добавляют 0,5 мл смеси 1M буфер Tris-HCl (pH 8,0)/насыщенный фенол и осторожно перемешивают в течение 15 минут. Затем добавляют 0,5 мл хлороформа и смесь осторожно перемешивают в течение 5 минут. Смесь центрифугируют при 4°C и 6000 g в течение 10 минут, верхний водный слой переносят в новую микропробирку объемом 2 мл, добавляют 70 мкл 3M натрий-ацетатного буфера (pH 5,2) и 1,21 мл 99,5% холодного этанола, после чего смесь осторожно перемешивают. Затем смесь центрифугируют при 4°C и 15000 g в течение 10 минут, супернатант удаляют и остаток промывают 0,4 мл 70% холодного этанола. К осадку добавляют 0,5 мл буфера TE (10 мM буфер Tris-HCl, 1 мM EDTA·2Na) и затем смесь оставляют на ночь при 4°C для растворения ДНК.
К вышеуказанному раствору ДНК добавляют 5 мкл заранее полученного раствора РНКазы (Sigma, E.C. 3.1.27.5) в стерильной воде с концентрацией 10 мг/мл и смесь инкубируют при 37°C в течение 1 часа. Затем добавляют 0,25 мл смеси фенол/хлороформ (1/1) и осторожно перемешивают в течение 10 минут, после чего добавляют 0,25 мл хлороформа и осторожно перемешивают в течение 5 минут. Смесь центрифугируют при 4°C и 6000 g в течение 10 минут, верхний водный слой переносят в новую микропробирку объемом 2 мл, добавляют 50 мкл 3M натрий-ацетатного водного раствора и 1 мл 99,5% холодного этанола и смесь осторожно перемешивают. Затем смесь центрифугируют при 4°C и 15000 g в течение 10 минут, супернатант удаляют, остаток промывают 0,4 мл 70% холодного этанола и осадок сушат (вышеописанную процедуру также называют "обработка РНКазой").
К сухому осадку добавляют 125 мкл буфера TE, смесь оставляют на ночь при 4°C для растворения ДНК и получают экстрагированную ДНК. Значения поглощения раствора очищенной ДНК измеряют при 260 нм и 280 нм (OD260, OD280, значение OD260 раствора ДНК 50 мкг/мл равно 1,0, длина ячейки: 1 см), концентрацию ДНК рассчитывают исходя из значения OD260, а чистоту очищенной ДНК определяют по отношению OD260/OD280.
2. Результаты тестирования
Хромосомную ДНК, экстрагированную на стадии 1-3), подвергают электрофорезу с использованием 0,8% агарозного геля. После завершения электрофореза агарозный гель на 20 минут погружают в водный раствор бромида этидия с концентрацией 1 мкг/мл, затем дважды промывают водой, очищенной на ионообменной колонке, и определяют степень расщепления хромосомной ДНК с помощью прибора для УФ-просвечивания (длина волны: 254 нм).
Вышеуказанную экстрагированную ДНК анализируют методом электрофореза. Результаты приведены на фиг. 1. Фиг. 1 демонстрирует влияние ДНКазы, содержащейся в поврежденных клетках, на хромосомную ДНК Listeria (поврежденные клетки) и влияние амсакрина на хромосомную ДНК Listeria (живые и поврежденные клетки).
В результате обнаружено, что если к суспензии поврежденных клеток Listeria не добавляют амсакрин и клетки инкубируют в течение периода времени до 72 часов, то для самого длинного фрагмента ДНК, остающегося в лунке, и длинного фрагмента ДНК, расположенного около 19329 п.о., значимые различия не наблюдаются, следовательно, хромосомная ДНК поврежденных клеток Listeria плохо расщепляется ДНКазой, активность которой сохраняется в поврежденных клетках. Однако если к поврежденным клеткам добавляют амсакрин и клетки инкубируют в течение периода времени до 72 часов, количество самого длинного фрагмента ДНК, остающегося в лунке, явно уменьшается, следовательно, можно предположить, что состояние, в котором происходит расщепление хромосомной ДНК, поддерживается амсакрином при участии активной ДНК-гиразы или топоизомеразы, остающейся в поврежденных клетках Listeria.
Кроме того, если амсакрин добавляют к суспензии живых клеток Listeria и клетки инкубируют в течение периода времени до 72 часов, количество самого длинного фрагмента ДНК, остающегося в лунке, и длинного фрагмента ДНК, расположенного около 19329 п.о., временно уменьшается в течение 24 часов, а при продолжении инкубации количество обоих фрагментов ДНК увеличивается пропорционально времени инкубации. Полученный результат позволяет предположить, что амсакрин проникает через стенки живых клеток спустя 24 часа и обеспечивает состояние, в котором происходит расщепление хромосомной ДНК в живых клетках, при участии ДНК-гиразы или топоизомеразы, и оставшиеся живые клетки Listeria слабо или незначительно подвергаются действию амсакрина, которое усиливается во время второй инкубации.
Амсакрин представляет собой желтое, в высокой степени гидрофобное вещество. Следовательно, после короткого времени инкубации, от 1 до 30 минут, он плохо проникает через неповрежденные клеточные стенки высокогидрофильных живых клеток, и поэтому осадок живых клеток имеет белый цвет. Однако, поскольку поврежденные клетки имеют поврежденные клеточные стенки и повышенную гидрофобность, амсакрин может проникать через стенки поврежденных клеток, и поэтому осадок поврежденных клеток окрашен в желтый цвет. Данный пример подтверждает, что и в живых, и в поврежденных клетках амсакрин при проникновении через клеточные стенки обеспечивает состояние, в котором повсюду происходит расщепление хромосомной ДНК, при участии внутриклеточной ДНК-гиразы и/или топоизомеразы. Однако если амсакрин действует в течение короткого промежутка времени, хромосомы поврежденных клеток подвергаются мягкому, но селективному расщеплению случайным образом, и, следовательно, дополнительная обработка клеток в течение короткого промежутка времени ядом топоизомеразы, таким как амсакрин (например, дополнительная обработка после обработки ядом топоизомеразы, таким как EMA), позволяет с помощью ПЦР отличать живые клетки от поврежденных или мертвых клеток с высокой чувствительностью.
[Пример 2]
Анализ гена рРНК 23S проводят методом ПЦР с использованием хромосомной ДНК живых и поврежденных клеток микроорганизмов, обработанных ядом ДНК-гиразы.
1. Получение образцов
1-1) Получение суспензий живых и поврежденных клеток
Enterobacter sakazakii (штамм Enterobacter sakazakii ATCC 51329, в дальнейшем называемый также "Enterobacter"), который представляет собой грамотрицательную бактерию, культивируют при 37°C с использованием бульона BHI, 40 мл культуральной среды, содержащей клетки в логарифмической фазе роста, центрифугируют при 4°C и 8000 g в течение 15 минут, после чего супернатант удаляют. К клеткам добавляют 40 мл физиологического раствора, смесь перемешивают с достаточной интенсивностью и снова центрифугируют при охлаждении в тех же условиях, супернатант удаляют, к клеткам добавляют 10 мл физиологического раствора и получают суспензию живых клеток. Число живых клеток, измеренное на стандартной культуральной агаровой среде, в данной суспензии составляет 4,4×108 кое/мл.
Затем 1 мл вышеуказанной суспензии живых клеток помещают в микропробирку объемом 1,5 мл, пробирку погружают в кипящую воду на 50 секунд и быстро охлаждают водой со льдом, получая суспензию поврежденных клеток.
Суспензию живых клеток и суспензию поврежденных клеток Listeria monocytogenes JCM 2873 получают по способу примера 1. Число живых клеток в суспензии живых клеток составляет 4,0×108 кое/мл.
1-2) обработка ядом ДНК-гиразы (ципрофлоксацином)
К 9 мл свежеполученного бульона BHI (число живых и поврежденных клеток Enterobacter в среде составляет 4,4×107 кое/мл и 4,4×107 кое/мл соответственно, а число живых и поврежденных клеток Listeria в среде составляет 4,0×107 кое/мл и 4,0×107 кое/мл соответственно) добавляют 1 мл каждой из суспензий Enterobacter (живых и поврежденных клеток) и Listeria (живых и поврежденных клеток), после чего к среде добавляют 2 мл раствора ципрофлоксацина (130 мкг/мл в физиологическом растворе).
Для исследования влияния активной ДНКазы, остающейся в поврежденных клетках, на хромосомную ДНК поврежденных клеток 1 мл каждой из суспензий поврежденных клеток Enterobacter и Listeria добавляют к 9 мл свежеполученного бульона BHI и затем вместо вышеуказанного раствора ципрофлоксацина добавляют 2 мл физиологического раствора.
Затем получают суспензии Enterobacter (живых и поврежденных клеток), которые культивируют при 37°C в течение 1 часа и 30 минут, 3 часов и 30 минут, 5 часов и 72 часов. Затем получают суспензии Listeria (живых и поврежденных клеток), которые культивируют при 37°C в течение 1 часа и 30 минут, 3 часов и 30 минут, 5 часов и 72 часов. Кроме того, в качестве контролей, соответствующих времени культивирования 0 часов, используют суспензии живых и поврежденных клеток Enterobacter и Listeria, в которые не добавляют ципрофлоксацин.
Все суспензии центрифугируют при 4°C и 8000 g в течение 15 минут, супернатант полностью удаляют и собирают осадок. ДНК Enterobacter и Listeria экстрагируют с помощью нижеследующих способов.
1-3) Экстракция ДНК
ДНК Enterobacter экстрагируют следующим способом.
К осадку добавляют 0,5 мл 10 мM буфера Tris-HCl (pH 8,0), 10 мкл протеинкиназы K, 1250 ед./мл (Sigma, E.C. 3.4.21.64) и 200 мкл полученного заранее раствора SDS в стерильной воде с концентрацией 10% (мас./об.), после чего реакцию оставляют протекать в течение ночи при 50°C. К обработанной суспензии добавляют 0,5 мл смеси 1M буфер Tris-HCl (pH 8,0)/насыщенный фенол и осторожно перемешивают в течение 15 минут. Затем добавляют 0,5 мл хлороформа и смесь осторожно перемешивают в течение 5 минут. Смесь центрифугируют при 4°C и 6000 g в течение 10 минут, верхний водный слой переносят в новую микропробирку объемом 2 мл, добавляют 70 мкл 3M натрий-ацетатного буфера (pH 5,2) и 1,29 мл 99,5% холодного этанола, после чего смесь осторожно перемешивают. Затем смесь центрифугируют при 4°C и 15000 g в течение 10 минут, супернатант удаляют и остаток промывают 0,4 мл 70% холодного этанола. К осадку добавляют 0,5 мл буфера TE (10 мM буфер Tris-HCl, 1 мM EDTA·2Na) и затем смесь оставляют на ночь при 4°C для растворения ДНК.
К вышеуказанному раствору ДНК добавляют 5 мкл заранее полученного раствора РНКазы (Sigma, E.C. 3.1.27.5) в стерильной воде с концентрацией 10 мг/мл и смесь инкубируют при 37°C в течение 1 часа. Затем добавляют 0,25 мл смеси фенол/хлороформ (1/1) и осторожно перемешивают в течение 10 минут, после чего добавляют 0,25 мл хлороформа и осторожно перемешивают в течение 5 минут. Смесь центрифугируют при 4°C и 6000 g в течение 10 минут, верхний водный слой переносят в новую микропробирку объемом 2 мл, добавляют 50 мкл 3M натрий-ацетатного водного раствора и 1 мл 99,5% холодного этанола и осторожно перемешивают. Затем смесь центрифугируют при 4°C и 15000 g в течение 10 минут, супернатант удаляют, остаток промывают 0,4 мл 70% холодного этанола и осадок сушат (вышеописанную процедуру также называют "обработка РНКазой").
К сухому осадку добавляют 125 мкл буфера TE, смесь оставляют на ночь при 4°C для растворения ДНК и получают экстрагированную ДНК. Значения поглощения раствора очищенной ДНК измеряют при 260 нм и 280 нм (OD260, OD280, значение OD260 раствора ДНК 50 мкг/мл равно 1,0, длина ячейки: 1 см), концентрацию ДНК рассчитывают исходя из значения OD260, а чистоту очищенной ДНК определяют по отношению OD260/OD280.
ДНК Listeria экстрагируют по способу примера 1, стадия 1-3) Экстракция ДНК.
1-4) Электрофорез продуктов амплификации методом ПЦР
Хромосомную ДНК, экстрагированную на стадии 1-3), подвергают электрофорезу с использованием 0,8% агарозного геля. После завершения электрофореза агарозный гель на 20 минут погружают в водный раствор бромида этидия с концентрацией 1 мкг/мл, затем дважды промывают водой, очищенной на ионообменной колонке, и определяют степень расщепления хромосомной ДНК с помощью прибора для УФ-просвечивания (длина волны: 254 нм).
2. Способ тестирования (ПЦР с использованием в качестве матрицы гена рРНК 23S и электрофорез)
2-1) Получение рабочей смеси ПЦР
Получают рабочую смесь (общий объем: 50 мкл), которая содержит следующие компоненты:
Ex-Taq (Takara Shuzo, номер по каталогу: RR001B): 0,25 мкл
10× буфер Ex-Taq (Takara Shuzo, номер по каталогу: RR001B): 5 мкл
смесь dNTP (Takara Shuzo, номер по каталогу: RR001B): 4 мкл
5 пмоль/мкл SEQ ID NO: 1 (23S-F) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 2 (23S-R) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 3 (23S-MF) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 4 (23S-MR) ДНК: 2,5 мкл
2× SYBA Green (BMA, номер по каталогу: 50513): 10 мкл
стерильная вода: 15,75 мкл
матрица ДНК (15 нг/мкл): 10 мкл
Для амплификации практически всего участка рРНК 23S используют праймеры с последовательностями SEQ ID NO: 1 и 2, которые обеспечивают амплификацию фрагмента размером приблизительно 2840 п.о., в основном, из грамотрицательных бактерий. Праймеры SEQ ID NO: 3 и 4, соответствующие центральному участку рРНК 23S, обеспечивают амплификацию фрагмента размером приблизительно 900 п.о. из грамположительных бактерий (а также из грамотрицательных бактерий).
2-2) Профиль термических циклов ПЦР для амплификации гена рРНК 23S
Профиль термических циклов ПЦР для амплификации гена рРНК 23S Enterobacter показан в таблице 1.
Профиль термических циклов ПЦР для амплификации гена рРНК 23S Listeria показан в таблице 2.
Все растворы ДНК, полученные на стадии 1-3), разбавляют до 15 нг/мкл буфером TE и 10 мкл разбавленного раствора используют в качестве матрицы ДНК на стадии 2-1). То есть в 50 мкл реакционной смеси ПЦР содержится 150 нг матрицы ДНК. В качестве отрицательного контроля используют 10 мкл буфера TE.
ПЦР и TM-анализ (анализ температуры плавления) продукта амплификации проводят в соответствии с упомянутым выше профилем термических циклов ПЦР, показанным в пункте 2-2), с помощью прибора для проведения ПЦР в режиме реального времени i Cycler (Biorad, номер модели: iQ). Пороговое значение (граничное значение) ПЦР в режиме реального времени определяют путем умножения стандартного отклонения SD количества флуоресценции, полученного с использованием SYBA Green в течение 0-10 циклов, на 10. В дальнейшем в каждой кинетической кривой амплификации методом ПЦР в режиме реального времени число циклов, превышающее пороговое значение, обозначается как "значение Ct".
3. Результаты тестирования
Результаты тестирования данного примера приведены на фиг. 2-11. На фиг. 2 показано влияние внутриклеточной ДНКазы на хромосомную ДНК Enterobacter (поврежденные клетки). На фиг. 3 показано влияние ципрофлоксацина на хромосомную ДНК той же бактерии (живые и поврежденные клетки). На фиг. 4 показано влияние внутриклеточной ДНКазы поврежденных клеток на хромосомную ДНК Listeria (поврежденные клетки). На фиг. 5 показано влияние ципрофлоксацина на хромосомную ДНК той же бактерии (живые и поврежденные клетки).
Кроме того, на фиг. 6 показана кинетическая кривая амплификации методом ПЦР в режиме реального времени с использованием в качестве матрицы гена рРНК 23S живых клеток Enterobacter, обработанных ципрофлоксацином. На фиг. 7 показаны результаты анализа кривой TM продукта амплификации, указанного на фиг. 6. Подобным образом, кинетическая кривая ПЦР в режиме реального времени и результат анализа кривой TM для поврежденных клеток той же бактерии, обработанных тем же средством, показаны на фиг. 8 и 9 соответственно. Кроме того, кинетическая кривая ПЦР в режиме реального времени для живых и поврежденных клеток Listeria, обработанных тем же средством, показана на фиг. 10 и 11 соответственно.
Результаты, приведенные на фиг. 2, показывают, что хромосомная ДНК поврежденных клеток незначительно расщепляется ДНКазой, остающейся в поврежденных клетках Enterobacter в течение 72 часов. Однако, как показано на фиг. 3, даже если ципрофлоксацин действует на поврежденные клетки в течение 1,5-5 часов, расщепление ДНК происходит в гораздо более высокой степени, чем под действием вышеупомянутой ДНКазы. В случае живых клеток количество длинного фрагмента хромосомной ДНК размером приблизительно 19329 п.о., наоборот, увеличивается, свидетельствуя о том, что кратковременное воздействие ципрофлоксацина приводит к более интенсивному расщеплению хромосомной ДНК под действием бактериальной ДНК-гиразы в поврежденных клетках по сравнению с живыми клетками.
Как показано на фиг. 4, в поврежденных клетках Listeria расщепление длинного фрагмента хромосомной ДНК размером приблизительно 19329 п.о. под действием внутриклеточной ДНКазы происходит в незначительной степени даже спустя 72 часа. Однако, как показано на фиг. 5, воздействие ципрофлоксацина на поврежденные клетки в течение 72 часов приводит к гораздо более интенсивному расщеплению ДНК под действием вышеупомянутой ДНКазы. Можно сделать вывод, что на живые клетки ципрофлоксацин оказывает такое же действие и влияние.
Если воздействие ципрофлоксацина продолжается в течение 72 часов, образование продуктов ПЦР заметно подавляется как для живых, так и для поврежденных клеток Enterobacter. Однако, как показывают вычисления, Ct (живые клетки, воздействие ципрофлоксацина в течение 72 часов) - Ct (живые клетки, отсутствие ципрофлоксацина) = 13, а Ct (поврежденные клетки, воздействие ципрофлоксацина в течение 72 часов) - Ct (поврежденные клетки, отсутствие ципрофлоксацина) = 17, образование продуктов ПЦР сильнее подавляется в случае поврежденных клеток. То есть, если ПЦР завершают после 27 циклов, в случае живых клеток результат является положительным, а в случае поврежденных клеток - отрицательным. Таким образом, полученные данные подтверждают, что использование ципрофлоксацина позволяет различать живые и поврежденные клетки. Также в случае Listeria, если ПЦР завершают после 20 циклов, в случае живых клеток результат является положительным, а в случае поврежденных клеток - отрицательным, как показано на фиг. 10 и фиг. 11 соответственно. Таким образом, полученные данные подтверждают, что использование лекарственного средства позволяет различать живые и поврежденные клетки.
Анализ кривых TM продуктов амплификации методом ПЦР хромосомной ДНК живых и поврежденных клеток Enterobacter sakazakii, обработанных ципрофлоксацином в течение 72 часов, приведенных на фиг. 7 и 9, показывает, что продукт амплификации ПЦР в случае живых клеток соответствует гену рРНК 23S, используемому в качестве матрицы, тогда как продукт амплификации ПЦР в случае поврежденных клеток не соответствует целевому продукту амплификации. Полагают, что в случае поврежденных клеток продукт амплификации ПЦР представляет собой фрагмент гена рРНК 23S, образовавшийся в результате нескольких расщеплений гена рРНК 23S матричной ДНК. Как описано выше, полученные данные показывают, что живые и поврежденные клетки можно дифференциально детектировать методом ПЦР после обработки ципрофлоксацином.
[Пример 3]
Анализ хромосомной ДНК живых и поврежденных клеток микроорганизмов, обработанных EMA, проводят методом ПЦР с использованием в качестве матрицы гена рРНК 16S или гена рРНК 23S.
1. Получение образцов
1-1) Получение суспензий грамотрицательных бактерий (живых и поврежденных клеток)
Каждую из Escherichia coli DH5or (в дальнейшем называемая также "Escherichia coli"), Citrobacter koseri (Citrobacter koseri JCM 1658, в дальнейшем называемая также "Citrobacter"), Salmonella enteritidis (Salmonella enteritidis HD 604, в дальнейшем называемая также "Salmonella") и Klebsiella oxytoca (Klebsiella oxytoca JCM 1665, в дальнейшем называемая также "Klebsiella") культивируют при 37°C с использованием бульона BHI, 40 мл культуральной среды, содержащей клетки в логарифмической фазе роста, центрифугируют при 4°C и 8000 g в течение 15 минут, после чего супернатант удаляют. К клеткам добавляют 40 мл физиологического раствора, смесь перемешивают с достаточной интенсивностью и снова центрифугируют при охлаждении в тех же условиях, супернатант удаляют, к клеткам добавляют 10 мл физиологического раствора и получают суспензию живых клеток. Число живых клеток в полученных суспензиях составляет для Escherichia coli: 3,2×108 кое/мл, для Citrobacter: 6,7×107 кое/мл, для Salmonella: 1,9×108 кое/мл и для Klebsiella: 4,8×108 кое/мл.
Затем 1 мл каждой из вышеуказанных суспензий живых клеток помещают в микропробирки объемом 1,5 мл, пробирки погружают в кипящую воду на 50 секунд и быстро охлаждают водой со льдом, получая суспензии поврежденных клеток.
1-2) Получение суспензий грамположительных бактерий (живых и поврежденных клеток)
Bacillus cereus (Bacillus cereus JCM 2152, в дальнейшем называемая также "Bacillus") и Staphylococcus epidermidis (Staphylococcus epidermidis KD, в дальнейшем называемая также "Staphylococcus") культивируют при 37°C, а Listeria monocytogenes (Listeria monocytogenes JCM 2873, в дальнейшем называемая также "Listeria") культивируют при 30°C, соответственно, в бульоне BHI, 40 мл каждой культуральной среды, содержащей клетки в логарифмической фазе роста (трофозоиты в случае Bacillus), центрифугируют при 4°C и 8000 g в течение 15 минут, после чего супернатант удаляют. Затем к клеткам добавляют 40 мл физиологического раствора, смесь перемешивают с достаточной интенсивностью и снова центрифугируют при охлаждении в тех же условиях, супернатант удаляют, к клеткам добавляют 10 мл физиологического раствора и получают суспензию живых клеток. Число живых клеток в полученных суспензиях составляет для Bacillus: 3,0×107 кое/мл, для Listeria: 1,3×108 кое/мл и для Staphylococcus: 1,1×106 кое/мл.
Затем 1 мл каждой из вышеуказанных суспензий живых клеток помещают в микропробирки объемом 1,5 мл, пробирки погружают в кипящую воду на 50 секунд и быстро охлаждают водой со льдом, получая суспензии поврежденных клеток.
2. Способ тестирования
2-1) Стадии обработки EMA и облучения видимым светом
10 мкл раствора EMA с концентрацией 1000 мкг/мл, полученного путем растворения EMA (Sigma, номер по каталогу: E2028) в стерильной воде и фильтрации раствора через микрофильтр 0,45 мкм, добавляют к каждой суспензии грамотрицательных бактерий (живые и поврежденные клетки) и грамположительных бактерий (живые и поврежденные клетки) в объеме 1 мл, после чего смесь оставляют при 4°C на 30 минут, защищая ее от света. Затем суспензию помещают на лед и в течение 10 минут облучают видимым светом мощностью 500 Вт с помощью лампы (FLOOD PRF, 100 В, 500 Вт, Iwasaki Electric Co., Ltd.), расположенной на расстоянии 20 см от суспензии. Описанные выше стадии добавления раствора EMA и облучения видимым светом также можно назвать "обработкой EMA". Отдельно к 1 мл каждой суспензии грамотрицательных бактерий (живые и поврежденные клетки) и грамположительных бактерий (живые и поврежденные клетки) вместо раствора EMA добавляют 10 мкл стерильной воды, после чего смесь подвергают описанной выше обработке EMA.
2-2) Экстракция ДНК
Микропробирки, содержащие живые и поврежденные клетки грамотрицательных и грамположительных бактерий (в каждом случае обработанных и не обработанных EMA), центрифугируют при 4°C и 15000 g в течение 10 минут. В каждую микропробирку добавляют 990 мкл физиологического раствора, после чего смесь перемешивают с достаточной интенсивностью. Затем весь объем смеси переносят в микропробирку объемом 2 мл и центрифугируют при 4°C и 15000 g в течение 10 минут, после чего супернатант удаляют. Затем с помощью способа примера 1, стадия 1-3) Экстракция ДНК, проводят экстракцию, определение концентрации и чистоты ДНК.
3. ПЦР с использованием в качестве матрицы гена рРНК 16S или гена рРНК 23S
3-1) Получение рабочей смеси ПЦР
Получают рабочую смесь (общий объем: 50 мкл), которая содержит следующие компоненты:
Ex-Taq (Takara Shuzo, номер по каталогу: RR001B): 0,25 мкл
10× буфер Ex-Taq (Takara Shuzo, номер по каталогу: RR001B): 5 мкл
смесь dNTP (Takara Shuzo, номер по каталогу: RR001B): 4 мкл
5 пмоль/мкл SEQ ID NO: 1 (23S-F) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 2 (23S-R) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 3 (23S-MF) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 4 (23S-MR) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 5 (16S-F) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 6 (16S-R) ДНК: 2,5 мкл
2× SYBA Green (BMA, номер по каталогу: 50513): 10 мкл
стерильная вода: 15,75 мкл
матрица ДНК (15 нг/мкл): 10 мкл
3-2) Профиль термических циклов ПЦР для амплификации гена рРНК 16S
Профиль термических циклов ПЦР для амплификации гена рРНК 16S для всех бактерий показан в таблице 3.
3-3) Профиль термических циклов ПЦР для амплификации гена рРНК 23S
Профиль термических циклов ПЦР для амплификации гена рРНК 23S для всех бактерий показан в таблице 4 (грамотрицательные бактерии) или в таблице 5 (грамположительные бактерии).
3-4) ПЦР
Все растворы ДНК, полученные на стадии 1-3), разбавляют до 15 нг/мкл буфером TE и 10 мкл разбавленного раствора используют в качестве матрицы ДНК на стадии 2-1). То есть в 50 мкл реакционной смеси ПЦР содержится 150 нг матрицы ДНК. В качестве отрицательного контроля используют 10 мкл буфера TE.
ПЦР и TM-анализ (анализ температуры плавления) продукта амплификации проводят в соответствии с упомянутым выше профилем термических циклов ПЦР, показанным в пункте 3-2) или 3-3), с помощью прибора для проведения ПЦР в режиме реального времени i Cycler (Biorad, номер модели: iQ). Пороговое значение (граничное значение) и значение Ct для ПЦР в режиме реального времени определяют, как и в примере 2, стадия 2-3).
3-5) Электрофорез в агарозном геле
Используя агарозу Seakem GTG (FMC, номер по каталогу: 50070) и буфер TAE (4,84 г/л Tris, 1,142 мл/л уксусной кислоты, 0,149 г/л EDTA·2Na), получают 0,8% агарозный гель, в качестве маркеров используют гидролизат X-EcoT14I (Takara Shuzo, код: 3401) и ДНК Ladder 100 п.о. (Takara Shuzo, Code: 3407A). Для каждой грамотрицательной и грамположительной бактерии 10 мкл раствора ПЦР помещают в лунку и подвергают электрофорезу. Когда бромфеноловый синий (BPB) мигрирует приблизительно на 90% геля, электрофорез останавливают.
Гель, в котором проводят электрофорез, на 20 минут погружают в водный раствор бромида этидия с концентрацией 1 мкг/мл, затем дважды промывают водой, очищенной на ионообменной колонке, и затем с помощью прибора для УФ-просвечивания (254 нм) наблюдают продукт амплификации ПЦР.
4. Результаты тестирования
На фиг. 12 показано изменение температуры бактериальной суспензии с течением времени при погружении микропробирки объемом 1,5 мл, содержащей 1 мл бактериальной суспензии, в кипящую воду. Из зависимости, показанной на фиг. 12, можно сделать вывод, что тепловая обработка путем погружения в кипящую воду на 50 секунд, используемая для получения суспензий поврежденных клеток, является немного более интенсивной тепловой обработкой, чем высокотемпературная кратковременная пастеризация, проводимая при 72-75°C в течение 15-16 секунд (пастеризация HTST), и, следовательно, соответствует такой тепловой обработке, как пастеризация при сверхвысокой температуре (пастеризация UHT).
Результаты дифференциальной детекции живых и поврежденных клеток с использованием гена рРНК 16S в качестве матрицы приведены на фиг. 13, а результаты дифференциальной детекции живых и поврежденных клеток с использованием гена рРНК 23S в качестве матрицы приведены на фиг. 14 и 15 соответственно. На фиг. 13-15 растворы ПЦР для каждой бактерии загружают на гель в следующем порядке: суспензии не обработанных EMA живых клеток, суспензии обработанных EMA живых клеток, суспензии не обработанных EMA поврежденных клеток и суспензии обработанных EMA поврежденных клеток.
Как показано на фиг. 13, если в качестве матрицы используют ген рРНК 16S, продукты амплификации ПЦР для EMA-обработанных поврежденных клеток Citrobacter и Klebsiella не обнаруживаются, следовательно, можно четко различить живые и поврежденные клетки. В случае EMA-обработанных поврежденных клеток других бактерий продукты амплификации присутствуют, и, следовательно, нельзя однозначно различить живые и поврежденные клетки.
С другой стороны, результаты, приведенные на фиг. 14 и 15, показывают, что, если в качестве матрицы используют ген рРНК 23S, живые и поврежденные клетки можно четко различить после обработки EMA как в случае грамотрицательных бактерий, так и в случае грамположительных бактерий. Если для проведения ПЦР используют тестируемый образец, содержащий бактериальные поврежденные клетки на уровне 108 кое/мл как фон образца и 150 нг матрицы ДНК в объеме 50 мкл, ПЦР поврежденных клеток полностью подавляется, тогда как ПЦР живых клеток практически не подавляется. Кроме того, также показано, что ПЦР также может полностью ингибироваться в случае поврежденных клеток, содержащихся в коровьем молоке в количестве от 105 до 107 кое/мл, что позволяет детектировать живые клетки в низкой концентрации. Следовательно, этот способ можно использовать для скрининга общих бактерий в области санитарной инспекции пищевых продуктов. Кроме того, у пациентов, страдающих от сепсиса и нарушения функции печени, в крови может наблюдаться высокая концентрация, 104 кое/мл или выше, живых и поврежденных клеток, в этом случае с помощью способа по настоящему изобретению можно детектировать только живые клетки.
[Пример 4]
Патогенную бактерию обрабатывают EMA и ядом топоизомеразы или ядом ДНК-гиразы, после чего живые и поврежденные клетки дифференциально детектируют методом ПЦР с использованием в качестве матрицы патогенного гена.
1. Получение бактериальной культуры (живые клетки)
Суспензии живых и поврежденных клеток Listeria (Listeria monocytogenes JCM 2873) получают по способу примера 1. Число живых клеток в суспензии живых клеток составляет 1,3×105 кое/мл.
2. Способ тестирования
2-1) Стадии обработки EMA и облучения видимым светом
Полученные ранее суспензии живых и поврежденных клеток Listeria подвергают обработке EMA и облучению видимым светом по способу примера 3. EMA, по всей вероятности, проникают в клетки грамположительных бактерий, которые не имеют внешней мембраны, даже если клетки являются живыми и имеют неповрежденные клеточные стенки. Поэтому время инкубации суспензии при 4°C в отсутствие света после добавления EMA уменьшают до 5 минут, и время облучения видимым светом тоже уменьшают до 5 минут. Кроме того, к суспензиям живых и поврежденных клеток вместо раствора EMA добавляют 10 мкл стерильной воды, после чего проводят указанные выше обработки.
2-2) Обработка ядом топоизомеразы или ядом ДНК-гиразы
После завершения вышеуказанных стадий обработки EMA и облучения видимым светом микропробирки, содержащие обработанные суспензии, центрифугируют при 4°C и 15000 g в течение 10 минут, после чего супернатант удаляют. К клеткам добавляют 1 мл физиологического раствора, смесь перемешивают с достаточной интенсивностью и центрифугируют в тех же условиях. Супернатант удаляют, к клеткам добавляют 1 мл физиологического раствора и смесь перемешивают с достаточной интенсивностью. Как описано выше, 1 пробирка содержит 1 мл суспензии живых клеток, не обработанных EMA, 7 пробирок содержат 1 мл суспензии живых клеток, обработанных EMA, 1 пробирка содержит 1 мл суспензии поврежденных клеток, не обработанных EMA, и 7 пробирок содержат 1 мл суспензии поврежденных клеток, обработанных EMA.
Суспензии живых и поврежденных клеток, обработанных EMA, подразделяют на 6 групп, каждая из которых включает 1 пробирку с суспензией живых клеток и 1 пробирку с суспензией поврежденных клеток. К суспензиям первой группы добавляют 8 мкл раствора ципрофлоксацина (0,5 мг/мл, в физиологическом растворе), который представляет собой яд ДНК-гиразы, к суспензиям второй группы добавляют 10 мкл раствора камптотецина (1 мг/мл, в диметилсульфоксиде), который представляет собой яд топоизомеразы, к суспензиям третьей группы добавляют 10 мкл раствора этопозида (1 мг/мл, в диметилсульфоксиде), который представляет собой яд топоизомеразы, к суспензиям четвертой группы добавляют 5 мкл раствора эллиптицина (0,1 мг/мл, в диметилсульфоксиде), который представляет собой яд топоизомеразы, к суспензиям пятой группы добавляют 10 мкл раствора митоксантрона (0,1 мг/мл, в диметилсульфоксиде), который представляет собой яд топоизомеразы, и к суспензиям шестой группы добавляют 10 мкл раствора амсакрина (1 мг/мл, в диметилсульфоксиде), который представляет собой яд топоизомеразы, соответственно. Каждый образец инкубируют при 30°C в течение 30 минут, затем образец полностью переносят в микропробирку объемом 2 мл и центрифугируют при 4°C и 15000 g в течение 10 минут, после чего супернатант удаляют.
2-3) Стадия экстракции ДНК
ДНК экстрагируют из всех полученных ранее образцов по способу примера 1, стадия 1-3) Экстракция ДНК.
3. ПЦР с использованием в качестве матриц разных генов Listeria
3-1) Амплификация патогенного гена Listeria, гена листериолицина О (hlyA)
3-1-1) Получение рабочей смеси ПЦР
Получают рабочую смесь (общий объем: 50 мкл), которая содержит следующие компоненты:
Ex-Taq (Takara Shuzo, номер по каталогу: RR001B): 0,25 мкл
10× буфер Ex-Taq (Takara Shuzo, номер по каталогу: RR001B): 5 мкл
смесь dNTP (Takara Shuzo, номер по каталогу: RR001B): 4 мкл
5 пмоль/мкл SEQ ID NO: 7 (hlyA-F) ДНК: 2,5 мкл
5 пмоль/мкл SEQ ID NO: 8 (hlyA-R) ДНК: 2,5 мкл
2× SYBA Green (BMA, номер по каталогу: 50513): 10 мкл
стерильная вода: 15,75 мкл
матрица ДНК (15 нг/мкл): 10 мкл
3-1-2) Профиль термических циклов ПЦР для амплификации гена hlyA
3-1-3) ПЦР
Все растворы ДНК, полученные на описанной выше стадии 2-3), разбавляют до 15 нг/мкл буфером TE и 10 мкл разбавленного раствора используют в качестве матрицы ДНК на описанной выше стадии 3-1-1). То есть в 50 мкл реакционной смеси ПЦР содержится 150 нг матрицы ДНК. В качестве отрицательного контроля используют 10 мкл буфера TE.
ПЦР проводят в соответствии с упомянутым выше профилем термических циклов ПЦР, показанным выше в пункте 3-1-2), с помощью прибора для проведения ПЦР в режиме реального времени i Cycler (Biorad, номер модели: iQ).
3-2) Амплификация генов рРНК 16S и рРНК 23S
3-2-1) Получение рабочей смеси ПЦР
Рабочую смесь (общий объем: 50 мкл) получают по способу примера 3, стадия 3-1) Получение рабочей смеси ПЦР.
3-2-2) Профиль термических циклов ПЦР для амплификации гена рРНК 16S
Используют "профиль термических циклов ПЦР для амплификации гена 16S рРНК", приведенный в примере 3, стадия 3-2) (таблица 3).
3-2-3) Профиль термических циклов ПЦР для амплификации гена рРНК 23S грамположительной бактерии
Используют "профиль термических циклов ПЦР для амплификации гена рРНК 23S грамположительной бактерии", приведенный в примере 3, стадия 3-3) (таблица 5).
3-2-4) ПЦР
Все растворы ДНК, полученные на стадии 2-3), разбавляют до 15 нг/мкл буфером TE и 10 мкл разбавленного раствора используют в качестве матрицы ДНК на описанной выше стадии 3-1-1) или 3-2-1). То есть в 50 мкл реакционной смеси ПЦР содержится 150 нг матрицы ДНК. В качестве отрицательного контроля используют 10 мкл буфера TE.
ПЦР проводят в соответствии с упомянутым выше профилем термических циклов ПЦР, показанным выше в пункте 3-2-2) или 3-2-3), с помощью прибора для проведения ПЦР в режиме реального времени i Cycler (Biorad, номер модели: iQ).
3-3) Электрофорез продукта амплификации ПЦР в агарозном геле
Электрофорез проводят по способу примера 2, стадия 1-4) Электрофорез продукта амплификации ПЦР. Для детекции продукта амплификации гена hlyA используют 3% агарозный гель.
4. Результаты тестирования
Результаты, полученные с использованием в качестве матрицы гена hlyA, показаны на фиг. 16, а результаты, полученные с использованием в качестве матрицы гена рРНК 16S и гена рРНК 23S, показаны на фиг. 17 и 18 соответственно.
Результаты, приведенные на фиг. 16 и 17, показывают, что при использовании в качестве матрицы генов hlyA и рРНК 16S амплификация ДНК поврежденных клеток после обработки EMA подавляется лишь в незначительной степени. Однако если наряду с обработкой EMA проводят обработку ядом топоизомеразы или ядом ДНК-гиразы, амплификация ДНК поврежденных клеток подавляется сильнее. В частности, после применения этопозида, митоксантрона или амсакрина наряду с EMA, при использовании гена hlyA в качестве матрицы, и после применения камптотецина, этопозида, эллиптицина или амсакрина наряду с EMA, при использовании гена рРНК 16S в качестве матрицы, амплификация ДНК поврежденных клеток подавляется в значительной степени.
Кроме того, результаты, приведенные на фиг. 18, показывают, что при использовании в качестве матрицы гена рРНК 23S ПЦР живых клеток также значительно подавляется после обработки EMA и другим ядом топоизомеразы или ядом ДНК-гиразы.
Чтобы быстро провести дифференциальную детекцию живых, поврежденных и мертвых клеток конкретной патогенной бактерии, в качестве матрицы предпочтительно использовать более короткий участок гена, например размером 100-200 п.о., обеспечивающий повышенную специфичность к патогенному гену. Полагают, что, если мишеневый участок имеет указанную выше небольшую длину, он не расщепляется, и его амплификация происходит, даже если EMA проникает через клеточные стенки поврежденных клеток и вызывает расщепление ДНК, и в результате подавление ПЦР не происходит. С другой стороны, даже если в качестве матрицы используется очень короткий ген hlyA размером 113 п.о., после обработки EMA в сочетании с ядом топоизомеразы или ядом ДНК-гиразы ПЦР поврежденных клеток Listeria с концентрацией 108 кое/мл подавляется практически полностью, тогда как ПЦР живых клеток не подавляется.
Полагают, что это происходит, например, в результате того, что яд топоизомеразы вызывает случайную поперечную сшивку ДНК в положениях, отличающихся от положений, в которых происходит поперечная сшивка под действием EMA, ингибируя повторное лигирование расщепленных участков и повторное лигирование под действием еще активной внутриклеточной ДНК-гиразы бактериальных топоизомераз I, III и IV в поврежденных клетках, что приводит к повсеместному расщеплению ДНК в более значительной степени, чем после обработки одним EMA, причем расщепляется даже короткий мишеневый ген размером 100-200 п.о.
Следовательно, можно осуществить достоверную дифференциальную детекцию живых, поврежденных или мертвых клеток конкретной патогенной бактерии в пищевом продукте или различных клинических образцах, содержащих фоновые поврежденные клетки конкретной патогенной бактерии в высокой концентрации. Однако, чтобы ингибировать образование продукта амплификации ПЦР в случае мертвых клеток, предпочтительно заранее добавить АТФ и Mg2+, а также топоизомеразу или ДНК-гиразу (фермент), поскольку топоизомераза или ДНК-гираза в мертвых клетках в отличие от поврежденных клеток может находиться в инактивированном состоянии.
Например, в мокроте туберкулезного пациента, получающего противотуберкулезное средство, на средней или поздней стадии лечения концентрация поврежденных клеток Mycobacterium tuberculosis в результате действия указанного средства составляет от 108 до 109 кое/мл. И даже в данном случае способ по настоящему изобретению позволяет детектировать только живые клетки.
[Пример отнесения 1] Получение тестируемых образцов из пищевых продуктов
Способ получения одного из тестируемых образцов, подходящих для применения в способе по настоящему изобретению, описан ниже с использованием в качестве пищевого продукта коровьего молока, зараженного микроорганизмом.
К коровьему молоку добавляют раствор EDTA с получением конечной концентрации 1-5 мM, предпочтительно 2 мM, и твин 80 с получением конечной концентрации 0,1-0,5%, предпочтительно 0,1%, после чего смесь центрифугируют при 4°C и 10000 g в течение 10 минут. Желательно добавить липазу (Sigma, E.C. 3.1.1.3) с получением конечной концентрации 10-20 ед./мл, после чего реакционную смесь оставляют стоять при 30-37°C в течение периода от 30 минут до 1 часа, затем добавляют протеиназу K (Sigma, E.C. 3.4.21.64) с получением конечной концентрации 20 ед./мл и инкубируют смесь в течение периода от 30 минут до 1 часа. Поверхностный липидный слой и средний водный слой удаляют и собирают осадок. Осадок содержит бактерии и соматические клетки, такие как эпителиальные клетки молочной железы и бычьи лейкоциты, а если смесь центрифугируют при 10000 g или больше, он может дополнительно содержать продукты деградации мицеллярных белков (продукты неполной деградации мицеллярного казеина), образовавшиеся в результате неполного разложения под действием протеиназы K. Предположительно, продукты неполной деградации мицеллярного казеина состоят из высокогидрофобных субмицелл α,β-казеина.
К осадку добавляют физиологический раствор, объем которого равен исходному объему смеси, с получением суспензии, которую центрифугируют при 4°C и 100 g в течение 5 минут, после чего собирают супернатант, содержащий микроорганизмы и т.п.
[Пример отнесения 2] Получение тестируемых образцов из крови
(1)
К гепаринизированной крови добавляют равный объем физиологического раствора, после чего смесь центрифугируют при 4°C и 10000 g в течение 5 минут, супернатант удаляют, а осадок собирают. Осадок содержит бактерии, тромбоциты, мононуклеарные клетки, такие как моноциты и лимфоциты, гранулоциты и эритроциты.
[Пример отнесения 3] Получение тестируемых образцов из крови
(2)
К гепаринизированной крови добавляют равный объем физиологического раствора, после чего смесь центрифугируют при 4°C и 100 g в течение 5 минут, чтобы разделить смесь на плазму и клетки крови (мононуклеарные клетки, такие как моноциты и лимфоциты, гранулоциты и эритроциты), после чего собирают плазму, содержащую микроорганизмы.
[Пример отнесения 4] Получение тестируемых образцов из крови
(3)
К гепаринизированной крови добавляют равный объем физиологического раствора. В стерильную пробирку вначале добавляют фиколл-пак [Amersham Bioscience, 5,7 г/100 мл фиколла 400, 9 г/100 мл диатризоата натрия, плотность: 1,077 г/мл], объем которого равен объему разбавленной в два раза гепаринизированной крови, и затем на него медленно наслаивают вышеуказанную разбавленную в два раза гепаринизированную кровь, держа пробирку в наклоненном состоянии. Затем слои центрифугируют при 4°C и 100 g в течение 5 минут, после чего собирают супернатант, содержащий микроорганизмы. Предпочтительно, перед наслаиванием разбавленной в два раза гепаринизированной крови на фиколл-пак добавляют раствор липазы (Sigma, E.C. 3.1.1.3) до конечной концентрации 10-20 ед./мл, затем добавляют раствор дезоксирибонуклеазы I (Sigma, E.C. 3.1.21.1), 10-50 ед./мл, реакционную смесь инкубируют при 30-37°C в течение периода от 30 минут до 1 часа, после чего добавляют протеиназу K (Sigma, E.C. 3.4.21.64) до конечной концентрации 10-20 ед./мл, и реакционную смесь инкубируют при 30-37°C в течение периода от 30 минут до 1 часа.
[Пример отнесения 5] Получение тестируемых образцов из крови
(4)
В стерильную пробирку сначала добавляют Monopoly™ [Amersham Bioscience, смесь фиколла и метризоата, плотность: 1,115 г/мл], объем которого равен 1/2 объема гепаринизированной крови, и затем на него медленно наслаивают гепаринизированную кровь, держа пробирку в наклоненном состоянии. Затем слои центрифугируют при 4°C и 100 g в течение 5 минут, после чего собирают супернатант, содержащий бактерии.
Промышленное применение
Способ по настоящему изобретению позволяет селективно детектировать живые клетки (жизнеспособное и пригодное к культивированию состояние) микроорганизма, присутствующего в пищевых продуктах или клинических образцах, на фоне мертвых клеток (мертвое состояние) и поврежденных клеток (жизнеспособное, но не пригодное к культивированию состояние), способ является альтернативой методу культивирования, но превосходит его по характеристикам.
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Morinaga Milk Industry Co., Ltd.
<120> Способ детекции микроорганизма и набор для детекции микроорганизма
<130> OP-C5307-PCT
<160> 8
<170> PatentIn version 3.1
<210> 1
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> праймер 23S-F
<400> 1
cagtcagagg cgatgaagga cgtgc 25
<210> 2
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> праймер 23S-R
<400> 2
ccggttagct caacccatcg ctgcg 25
<210> 3
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> праймер 23S-MF
<400> 3
accaggattt tggcttagaa g 21
<210> 4
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> праймер 23S-MR
<400> 4
cacttacccc gacaaggaat 20
<210> 5
<211> 16
<212> ДНК
<213> Искусственная
<220>
<223> праймер 16S-F
<400> 5
agtttgatcc tggctc 16
<210> 6
<211> 16
<212> ДНК
<213> Искусственная
<220>
<223> праймер 16S-R
<400> 6
ggctaccttg ttacga 16
<210> 7
<211> 19
<212> ДНК
<213> Искусственная
<220>
<223> праймер hlyA-F
<400> 7
tgcaagtcct aagacgcca 19
<210> 8
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> праймер hlyA-R
<400> 8
cactgcatctccgtggtatactaa 24
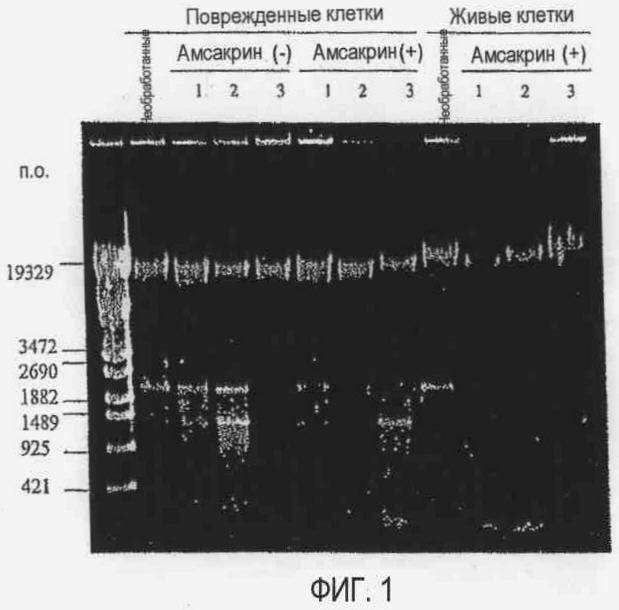



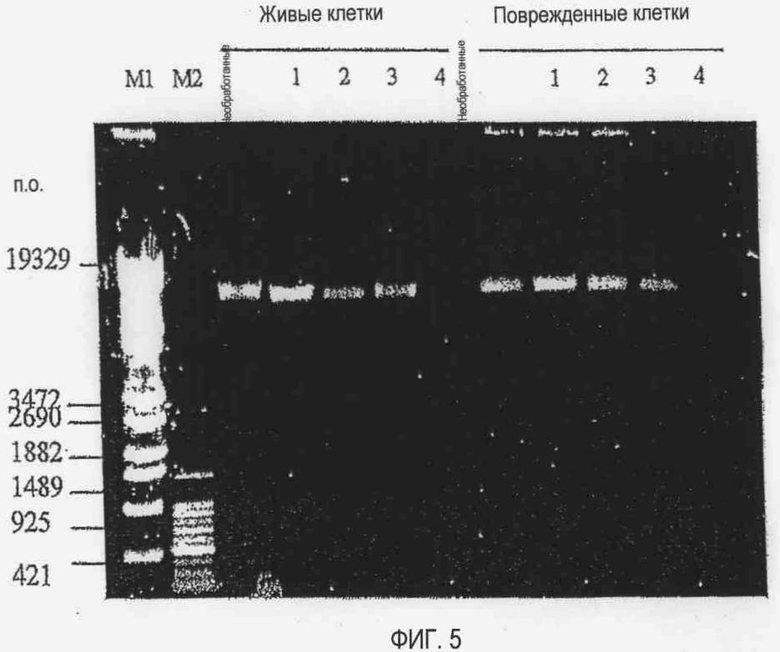
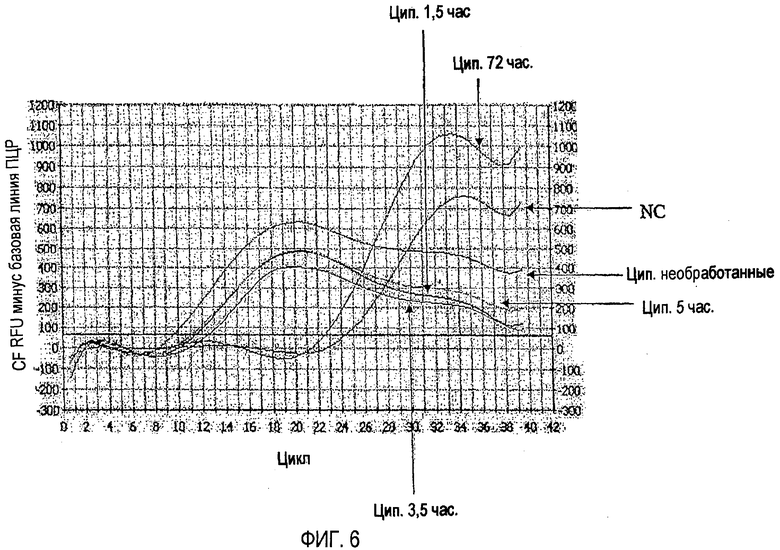
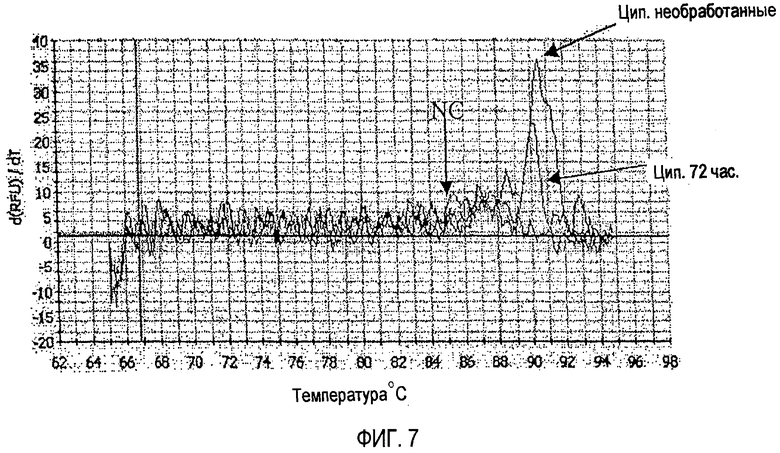

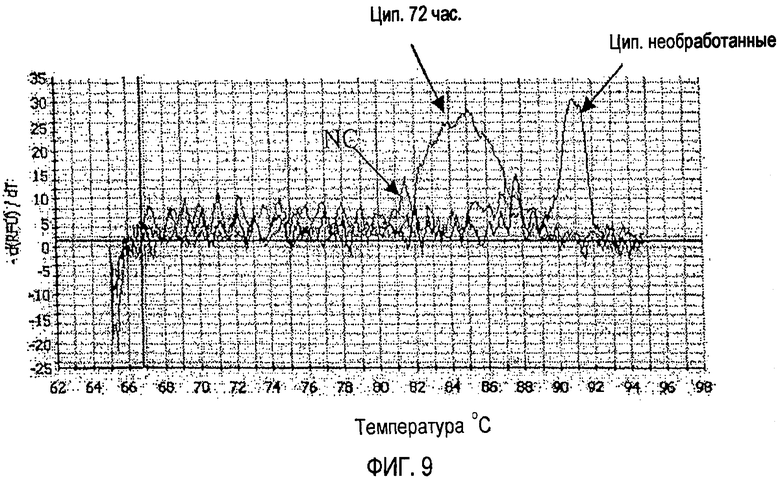

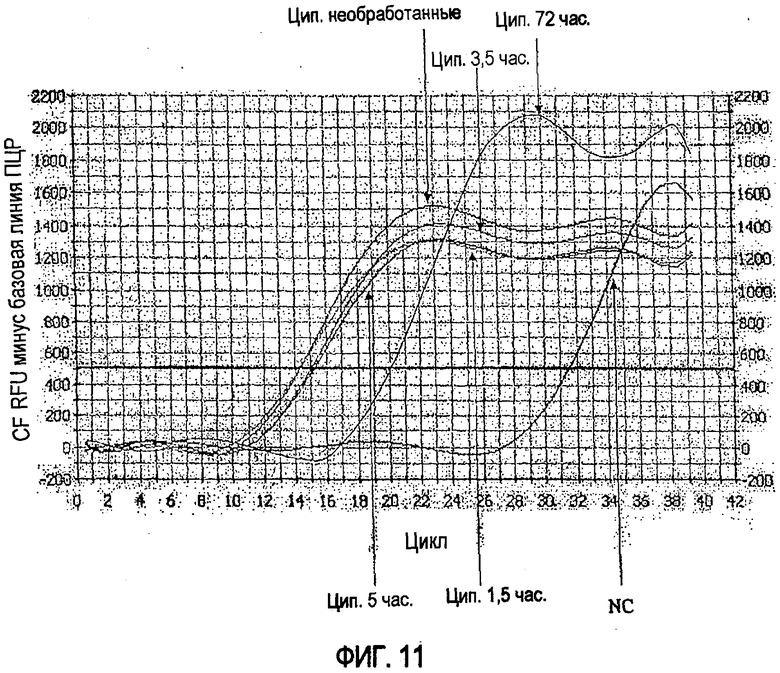
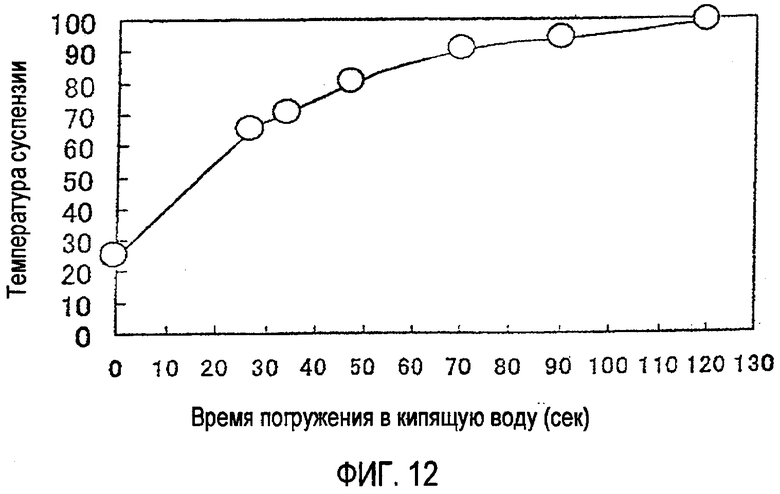
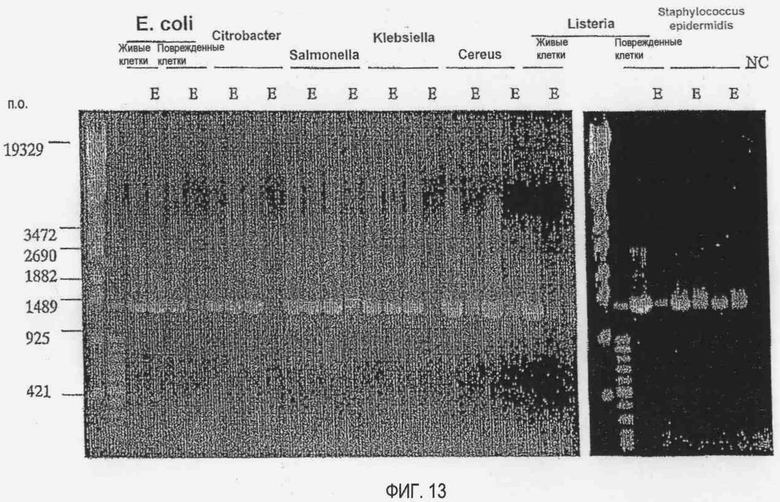
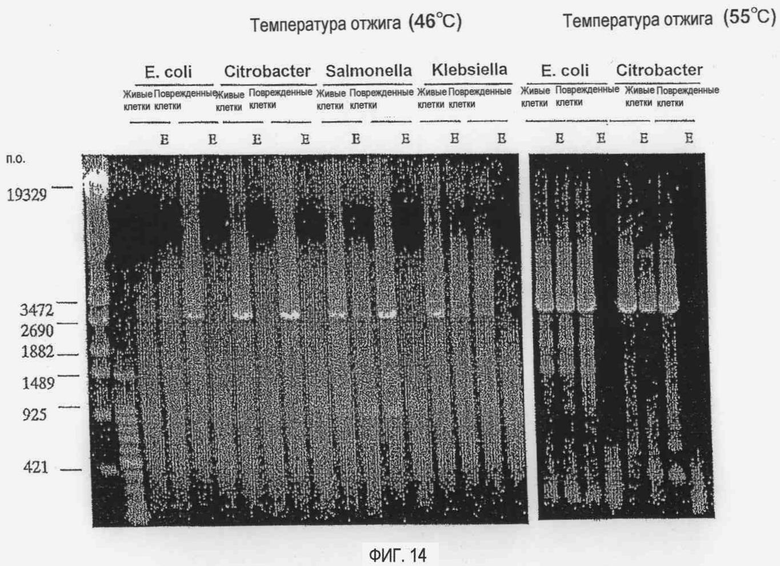



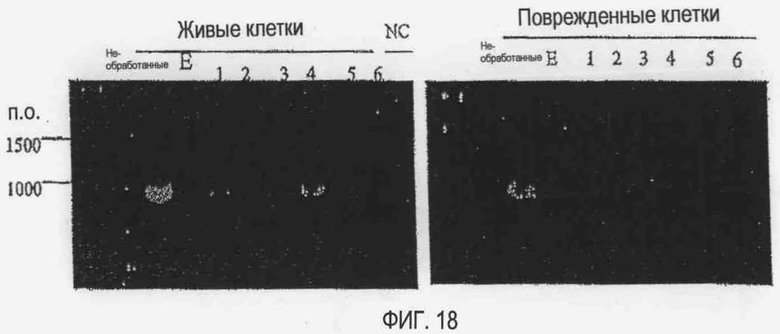
Изобретение относится к биотехнологии и представляет собой способ детекции живых клеток микроорганизма путем дифференцирования живых клеток от мертвых клеток или поврежденных клеток в тестируемом образце. Способ предусматривает стадию обработки тестируемого образца ядом топоизомеразы или ядом ДНК-гиразы, стадию экстракции ДНК из тестируемого образца и амплификацию мишеневого участка экстрагированной ДНК методом ПНР, где длина мишеневого участка составляет от 900 до 3000 нуклеотидов, и стадию анализа продукта амплификации. Изобретение позволяет определять наличие живых организмов с высокой степенью точности. 4 н. и 48 з.п. ф-лы, 18 ил., 6 табл.
1. Способ детекции живых клеток микроорганизма путем дифференцирования живых клеток от мертвых клеток или поврежденных клеток в тестируемом образце, который включает следующие стадии:
a) стадию обработки тестируемого образца ядом топоизомеразы или ядом ДНК-гиразы,
b) стадию экстракции ДНК из тестируемого образца и амплификацию мишеневого участка экстрагированной ДНК методом ПЦР, где длина мишеневого участка составляет от 900 до 3000 нуклеотидов, и
c) стадию анализа продукта амплификации.
2. Способ детекции живых клеток микроорганизма путем дифференцирования живых клеток от мертвых клеток или поврежденных клеток в тестируемом образце, который включает следующие стадии:
a) стадию обработки тестируемого образца ядом топоизомеразы и ядом ДНК-гиразы,
b) стадию экстракции ДНК из тестируемого образца и амплификацию мишеневого участка экстрагированной ДНК методом ПЦР, где длина мишеневого участка составляет от 100 до 3000 нуклеотидов, и
c) стадию анализа продукта амплификации.
3. Способ по п.1, где продукт амплификации анализируют с помощью стандартной кривой, отражающей зависимость количества продукта амплификации от количества микроорганизма, которую получают с использованием стандартных образцов микроорганизма.
4. Способ по п.2, где продукт амплификации анализируют с помощью стандартной кривой, отражающей зависимость количества продукта амплификации от количества микроорганизма, которую получают с использованием стандартных образцов микроорганизма.
5. Способ по п.3, где ПЦР проводят в режиме реального времени одновременно с анализом продукта амплификации.
6. Способ по п.4, где ПЦР проводят в режиме реального времени одновременно с анализом продукта амплификации.
7. Способ по любому из пп.1-6, где тестируемый образец представляет собой молоко, молочный продукт, пищевой продукт, полученный с использованием молока или молочного продукта в качестве сырья, образец крови, образец мочи, образец спинномозговой жидкости, образец синовиальной жидкости и образец плевральной жидкости.
8. Способ по любому из пп.1-6, где микроорганизм представляет собой бактерию.
9. Способ по п.8, где мишеневый участок представляет собой ген pPHK 23S.
10. Способ по п.9, где ПЦР проводят с использованием набора праймеров, содержащего праймеры SEQ ID NO: 1 и 2, или набора праймеров, содержащего праймеры SEQ ID NO: 3 и 4.
11. Способ по любому из пп.1-6, где микроорганизм представляет собой патогенную бактерию.
12. Способ по п.11, где мишеневый участок предпочтительно представляет собой патогенный ген.
13. Способ по п.12, где ПЦР предпочтительно проводят с использованием набора праймеров, содержащего праймеры SEQ ID NO:7 и 8.
14. Способ по любому из пп.1-6, 9-10 и 12, 13, где яд топоизомеразы выбран из амсакрина, камптотецина, доксорубицина, эллиптицина, этопозида, митоксантрона, саинтопина, топотекана и СР-115953.
15. Способ по любому из пп.1-6, 9-10 и 12, 13, где яд ДНК-гиразы выбран из ципрофлоксацина, офлоксацина, эноксацина, пефлоксацина, флероксацина, норфлоксацина, налидиксовой кислоты, оксолиновой кислоты и пиромидиновой кислоты.
16. Способ по любому из пп.1-6, 9-10 и 12, 13, где яд топоизомеразы и/или яд ДНК-гиразы представляет собой моноазид этидия, а способ включает стадию облучения тестируемого образца, к которому добавлен моноазид этидия, видимым светом.
17. Способ по п.16, где тестируемый образец обрабатывают моноазидом этидия и ядом топоизомеразы и/или ядом ДНК-гиразы, отличным от моноазида этидия.
18. Способ по любому из пп.1-6, 9-10 и 12 и 13, где перед стадией а) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
19. Набор для детекции живых клеток микроорганизма путем дифференцирования живых клеток от мертвых клеток или поврежденных клеток в тестируемом образце и детекцию живых клеток методом ПЦР, который содержит следующие компоненты:
яд топоизомеразы или яд ДНК-гиразы и праймеры для амплификации мишеневого участка ДНК микроорганизма, детектируемого методом ПЦР, где длина мишеневого участка составляет от 900 до 3000 нуклеотидов.
20. Набор для детекции живых клеток микроорганизма путем дифференцирования живых клеток от мертвых клеток или поврежденных клеток в тестируемом образце и детекцию живых клеток методом ПЦР, который содержит следующие компоненты:
яд топоизомеразы и яд ДНК-гиразы и праймеры для амплификации мишеневого участка ДНК микроорганизма, детектируемого методом ПЦР, где длина мишеневого участка составляет от 100 до 3000 нуклеотидов.
21. Набор по п.19, который дополнительно содержит топоизомеразу и/или ДНК-гиразу.
22. Набор по п.20, который дополнительно содержит топоизомеразу и/или ДНК-гиразу.
23. Набор по любому из пп.19-22, где яд топоизомеразы выбран из амсакрина, камптотецина, доксорубицина, эллиптицина, этопозида, митоксантрона, саинтопина, топотекана и СР-115953.
24. Набор по любому из пп.19-22, где яд ДНК-гиразы выбран из ципрофлоксацина, офлоксацина, эноксацина, пефлоксацина, флероксацина, норфлоксацина, налидиксовой кислоты, оксолиновой кислоты и пиромидиновой кислоты.
25. Набор по любому из пп.19-22, который содержит моноазид этидия и яд топоизомеразы и/или яд ДНК-гиразы, отличный от моноазида этидия.
26. Набор по любому из пп.19-22, где набор праймеров содержит праймеры SEQ ID NO: 1 и 2 или праймеры SEQ ID NO: 3 и 4.
27. Набор по любому из пп.19-22, где набор праймеров содержит праймеры SEQ ID NO: 7 и 8.
28. Способ по п.7, где микроорганизм представляет собой бактерию.
29. Способ по п.7, где микроорганизм представляет собой патогенную бактерию.
30. Способ по п.7, где яд топоизомеразы выбран из амсакрина, камптотецина, доксорубицина, эллиптицина, этопозида, митоксантрона, саинтопина, топотекана и СР-115953.
31. Способ по п.8, где яд топоизомеразы выбран из амсакрина, камптотецина, доксорубицина, эллиптицина, этопозида, митоксантрона, саинтопина, топотекана и СР-115953.
32. Способ по п.11, где яд топоизомеразы выбран из амсакрина, камптотецина, доксорубицина, эллиптицина, этопозида, митоксантрона, саинтопина, топотекана и СР-115953.
33. Способ по п.7, где яд ДНК-гиразы выбран из ципрофлоксацина, офлоксацина, эноксацина, пефлоксацина, флероксацина, норфлоксацина, налидиксовой кислоты, оксолиновой кислоты и пиромидиновой кислоты.
34. Способ по п.8, где яд ДНК-гиразы выбран из ципрофлоксацина, офлоксацина, эноксацина, пефлоксацина, флероксацина, норфлоксацина, налидиксовой кислоты, оксолиновой кислоты и пиромидиновой кислоты.
35. Способ по п.11, где яд ДНК-гиразы выбран из ципрофлоксацина, офлоксацина, эноксацина, пефлоксацина, флероксацина, норфлоксацина, налидиксовой кислоты, оксолиновой кислоты и пиромидиновой кислоты.
36. Способ по п.7, где яд топоизомеразы и/или яд ДНК-гиразы представляет собой моноазид этидия, а способ включает стадию облучения тестируемого образца, к которому добавлен моноазид этидия, видимым светом.
37. Способ по п.8, где яд топоизомеразы и/или яд ДНК-гиразы представляет собой моноазид этидия, а способ включает стадию облучения тестируемого образца, к которому добавлен моноазид этидия, видимым светом.
38. Способ по п.11, где яд топоизомеразы и/или яд ДНК-гиразы представляет собой моноазид этидия, а способ включает стадию облучения тестируемого образца, к которому добавлен моноазид этидия, видимым светом.
39. Способ по п.7, где перед стадией а) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
40. Способ по п.8, где перед стадией а) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
41. Способ по п.11, где перед стадией а) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
42. Способ по п.14, где перед стадией а) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
43. Способ по п.15, где перед стадией а) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
44. Способ по п.16, где перед стадией а) проводят:
d) стадию обработки тестируемого образца топоизомеразой и/или ДНК-гиразой.
45. Набор по п.23, который содержит моноазид этидия и яд топоизомеразы и/или яд ДНК-гиразы, отличный от моноазида этидия.
46. Набор по п.24, который содержит моноазид этидия и яд топоизомеразы и/или яд ДНК-гиразы, отличный от моноазида этидия.
47. Набор по п.23, где набор праймеров содержит праймеры SEQ ID NO: 1 и 2 или праймеры SEQ ID NO: 3 и 4.
48. Набор по п.24, где набор праймеров содержит праймеры SEQ ID NO: 1 и 2 или праймеры SEQ ID NO: 3 и 4.
49. Набор по п.25, где набор праймеров содержит праймеры SEQ ID NO: 1 и 2 или праймеры SEQ ID NO: 3 и 4.
50. Набор по п.23, где набор праймеров содержит праймеры SEQ ID NO: 7 и 8.
51. Набор по п.24, где набор праймеров содержит праймеры SEQ ID NO: 7 и 8.
52. Набор по п.25, где набор праймеров содержит праймеры SEQ ID NO: 7 и 8.
| RUDI К et.al | |||
| Detection of viable and dead Listeria monocytogenes on gouda-like cheeses by real-time PCR, Lett Appl Microbiol | |||
| Способ обработки целлюлозных материалов, с целью тонкого измельчения или переведения в коллоидальный раствор | 1923 |
|
SU2005A1 |
| RUDI К et.al | |||
| Use of ethidium monoazide and PCR in combination for quantification of viable and dead cells in complex samples, Appl Environ Microbiol 2005, 71(2), p.1018-24 | |||
| ВСЕСОЮЗНАЯ | 0 |
|
SU372261A1 |
Авторы
Даты
2010-07-27—Публикация
2006-02-17—Подача