Изобретение относится к области биотехнологии, ветеринарной микробиологии и генетической инженерии и может быть использовано для идентификации и детекции генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам бактерий семейства Enterobacteriaceae (вид Escherichia coli и Shigella spp. - детекция гена csrB, и Salmonella enterica - детекция гена гена invA) методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме «реального времени» и способу их применения.
Фторхинолоны - препараты широкого спектра действия, наиболее выраженной активностью обладают в отношении грамотрицательных бактерий, главным образом семейства Enterobacteriaceae (Escherichia coli, Proteus spp., Klebsiella spp., Shigella spp., Salmonella spp., Enterobacter spp., Serratia marcescens, Citrobacter spp.).
Фторированные хинолоны отличаются широким спектром антимикробного действия, высокой бактерицидной активностью и хорошей фармакокинетикой, что позволяет применять их для лечения инфекций различной локализации. Наличие у ряда препаратов лекарственных форм для внутривенного введения и приема внутрь в сочетании с высокой биодоступностью последних позволяет проводить ступенчатую терапию препаратом однородной группы.
В ветеринарии применение фторхинолонов возможно у разных видов животных при широком диапазоне патологий: у жвачных - при острых заболеваниях дыхательных путей, инфекциях, вызываемые кишечной палочкой, сальмонеллой, микоплазмами, мастит, метрит, конъюнктивит.У свиней - при лечении инфекций, вызываемых Mycoplasma hyopneumoniae, Actinobacillus pleuropneumoniae, E. coli и Pasteurella multocida. У лошадей - для инфекций, устойчивых к препаратам первой линии, не рекомендуется для молодняка (может приводить к эрозии хрящей). У собак и кошек - при простатите, мастите, рините, пиодермии, отите, инфекциях ран, перитоните, остеомиелите, инфекции мягких тканей. Не рекомендуется для животных моложе 8 месяцев (или 18 месяцев для крупных собак) во избежание повреждения суставов.
Учитывая широкий спектр использования фторхинолонов у разных видов животных и для лечения бактериальных болезней человека, идентификация устойчивых штаммов является важным критерием как в реализации лечебного процесса, так и осуществления эпизоотического мониторинга.
В современной клинической практике можно выделить несколько механизмов резистентности, приводящих к крайне серьезным социально-экономическим последствиям. К таким механизмам относятся устойчивость к β-лактамам среди грамположительных и грамотрицательных бактерий, связанная с продукцией β-лактамаз; устойчивость к гликопептидам среди Enterococcus spp.; устойчивость к фторхинолонам среди грамположительных и грамотрицательных бактерий; устойчивость к макролидам среди Streptococcus spp.[1].
Мишени фторхинолонов - ДНК-гираза, топоизомераза IV - находятся внутри клеток, поэтому для того, чтобы связаться с мишенями грамнегативных бактерий, фторхинолоны должны транспортироваться через две мембраны - цитоплазматическую и наружную. Если транслокация фторхинолонов через цитоплазматическую мембрану не вызывает затруднений, то проникновение через наружную мембрану, содержащую плотно расположенные липополисахариды (ЛПС), возможно только через специфические порины. Для того чтобы понизить эффективность фторхинолонов, бактерии используют относительно простые эффлюкс-помпы, локализованные исключительно в цитоплазматической мембране и обеспечивающие откачку антибиотика из цитоплазмы в периплазму со скоростью, превышающей диффузию фторхинолона в обратном направлении. Такой механизм характерен для большинства грамнегативных бактерий в отношении антибиотиков, мишени которых находятся в цитоплазматическом пространстве (фторхинолоны, макролиды, тетрациклины, хлорамфеникол) [2,3].
Гены семейства qnr кодируют белки, способные связываться с комплексом ДНК/ДНК-гираза или ДНК/топоизомераза IV и препятствовать взаимодействию фторхинолонов с данным ферментом. Таким образом, обеспечивается резистентность энтеробактерий к этому классу антибактериальных средств по механизму «защита мишени». Локализация qnrS и qnrB на плазмидах способствует их горизонтальному переносу между микроорганизмами, в том числе таксономически и экологически отдаленными. Наиболее распространенными вариантами qnr-белков среди бактерий семейства Enterobacteriaceae являются qnrS и qnrB. В качестве дополнительного внутреннего контроля в разработанных методиках предусмотрена идентификация бактерий, часто представленных в анализируемых микробиологических пробах: E.coli и S.enterica. В качестве генетических маркеров для идентификации E.coli и S.enterica выбраны фрагменты хромосомных генов csrB и invA соответственно. Подобраны последовательности гена invA для 26 сероваров Salmonella enterica subsp.Enterica (Agona, Anatum, Blockley. Bovismorbificans, Brandenburg, Bredeney, Corvallis, Derby, Enteritidis, Hadar, Heidelberg, Indiana, Infantis, Kentucky, Manhattan, Mbandaka, Minnesota, Montevideo, Muenchen, Newport, ParatyphiB, Schwarzengrund, Stanley, Thompson. Typhimurium, Virchow), контаминацию которыми выявляли в пищевых продуктах. В силу высокой консервативности гена csrB разработанная методика позволяет выявлять с той же эффективностью ДНК бактерий рода Shigella (S.sonnei, S.dysenteriae, S.boydii, S.flexneri), и с чуть меньшей эффективностью - ДНК бактерий из рода Escherichia (E.marmotae и E.albertii).
Существуют различные методы идентификации и детекции генетических детерминант резистентности, основная часть которых основана на применении технологии ДНК-чипов RU 2636457 C2, RU 2562866 C1, RU 2685188 C2, в свою очередь использование ПЦР в режиме «реального времени» в разработке такого рода методик используют все чаще.
Наиболее близкое по существу к заявленному изобретению является WO 2017095271 A1, предусматривающий способ молекулярно-генетической детекции устойчивости микобактерий туберкулеза к противотуберкулезным препаратам второго ряда (фторхинолонам, аминогликозидам и капреомицину), используемый в медицине. Заявленное изобретение относится к области ветеринарии и предусматривает применение предлагаемых олигонуклеотидных последовательностей для молекулярно-генетической идентификации и детекции генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам бактерий семейства Enterobacteriaceae (вид Escherichia coli,и Shigella spp.- детекция гена csrB, и Salmonella enterica - детекция гена гена invA) методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме «реального времени» и способу их применения. Изобретение не предназначено для выявления генов кластеров qnrA, qnrC, qnrD, qnrVC, qnrE.
Целью настоящего изобретения является разработка синтетических олигонуклеотидных последовательностей для ПЦР в «реальном времени», используемой с целью выявления фрагментов генов устойчивости к фторхинолонам qnrS и qnrB из образцов продовольственного сырья, пищевых продуктов, от животных (фекалии), из объектов окружающей среды (смывы с клеток, стен, оборудования; образцы фекалий, подстилка и др.), без этапа выделения бактериальных изолятов, а также из чистых бактериальных культур с детекцией бактерий семейства Enterobacteriaceae (вид Escherichia coli,и Shigella spp.- детекция гена csrB, и Salmonella enterica - детекция гена гена invA).
Заявленный технический результат достигается конструированием набора специфичных олигонуклеотидных последовательностей для идентификации генетических детерминант резистентности к фторхинолонам qnrS и qnrB и бактерий семейства Enterobacteriaceae (вид Escherichia coli,и Shigella spp.- идентификация гена csrB, и Salmonella enterica -идентификация гена гена invA) в биологических образцах методом ПЦР в режиме реального времени. Аналитическая чувствительность методики с использованием набора олигонуклеотидов для идентификации и детекции генетической детерминанты резистентности к фторхинолонам qnrB- 1,75×104 копий/мл, qnrS, invA и csrB 1×104 коп/мл. Специфичность набора олигонуклеотидов для идентификации и детекции генетических детерминант резистентности к фторхинолонам qnrS и qnrB и бактерий семейства Enterobacteriaceae (вид Escherichia coli,и Shigella spp.- идентификация гена csrB, и Salmonella enterica -идентификация гена гена invA) при исследовании бактериальных культур контрольной панели составляет 100%.
Используют реакционную смесь следующего состава: смесь 1 (qnrS+invA): 10 мм3 ДНК-матрицы, 10 мм3 ПЦР-смеси-1 (6 пмоль каждого из праймеров qnrS-F, qnrS-R, invA-F, invA-R, 3 пмоль каждого зонда qnrS-Z и invA-Z, 2,5 мм3 раствора дНТФ с концентрацией каждого нуклеотида 1,76 ммоль/дм3, деионизованная вода), 0,5 мм3 Taq ДНК полимеразы, 5 мм3 ПЦР-буфера с магнием; а так же детектирование флуоресцентного сигнала непосредственно в процессе реакции амплификации на канале Orange/Yellow ПЦР в режиме реального времени проводится с использованием следующей программы амплификации: 15 мин при 95°С; 15 с при 95°С, 45 с при 62°С (35 циклов с детекцией флуоресцентного сигнала), смесь 2 (qnrB+csrB): 10 мм3 ДНК-матрицы, 10 мм3 ПЦР-смеси-1 (6 пмоль каждого из праймеров qnrB-F, qnrB-R, csrB-F, csrB -R, 3 пмоль каждого зонда qnrB-Z и csrB -Z, 2,5 мм3 раствора дНТФ с концентрацией каждого нуклеотида 1,76 ммоль/дм3, деионизованная вода), 0,5 мм3 Taq ДНК полимеразы, 5 мм3 ПЦР-буфера с магнием; а так же детектирование флуоресцентного сигнала непосредственно в процессе реакции амплификации на канале Red / Yellow ПЦР в режиме «реального времени» проводится с использованием следующей программы амплификации: 15 мин при 95°С; 15 с при 95°С, 45 с при 62°С (35 циклов с детекцией флуоресцентного сигнала). Результаты амплификации в режиме реального времени оценивают с помощью программного обеспечения, при этом результаты интерпретируют на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на уровне 0,05 пороговой линии.
Используют специальный оригинальный набор олигонуклеотидных праймеров и зондов для выявления фрагментов генов устойчивости к фторхинолонам qnrS и qnrB, а также идентификацию и детекцию микроорганизмов семейства Enterobacteriaceae (вид Escherichia coli,и Shigella spp.- детекция гена csrB, и Salmonella enterica - детекция гена гена invA, имеющий следующий нуклеотидный состав: qnrS (qnrS-F-GCCTACAGGGTGCCAATTTA, qnrS-R-CAATACCCAGTGCTTCGAGAAT, qnrS-Z ROX-CACGCCGAACTCGACGGTTTAGAT-BHQ2) и qnrB (qnrB-F-CGTTCAGTGGTTCAGATCTCTC, qnrB-R-CTAAGTCACCCAACTCCGAAT, qnrB-Z-Cy5-AATGTGTGAAGTTTGCTGCTCGCC-BHQ2), а так же гена csrB для выявления ДНК Escherichia coli: csrB-F-GGTGTGAGCAGGAAGCAATA; csrB-R-CGCAGCATTCCAGCTACTT; csrB-Z; R6G-TCAAGGATGAGCAGGGAGCAACAA-BHQ1) и гена invA для выявления ДНК Salmonella enterica: (invA-F-GCACATAAAGATCTTGTCCTCCT; invA-R-CACTGACTTGCTATCTGCTATCTC; invA-Z-R6G-ACGTCTGTCGATGTCCGTCGATTT- BHQ1).
Для выбора и анализа ДНК последовательностей генов резистентности были использованы базы данных ResFinder, Arg-ANNOT, CARD и NCBI BARRGD, а также последовательности геномов различных грамположительных и грамотрицательных бактерий, которые были ранее получены в ФГБУ «ВГНКИ» методом полногеномного секвенирования. Подбор праймеров производили на онлайн-ресурсе «PrimerQuest Tool» (IDT), анализировалось не менее пяти наборов олигонуклеотидов для выявления каждого целевого гена. Для анализа множественного выравнивания последовательностей использовали программу Ugene и алгоритм Clustal omega. Для выбора оптимальных параметров структурных характеристик наборов праймеров использовали программы PCR Primer Stats и Oligo Analysis Tool, оценивали оптимальную температуру отжига, возможность образования димеров и «шпилек». Критерии для выбора праймеров были следующие: средняя длина - 16-25 нуклеотидов; GC состав 35-60%; температура отжига от 50°C до 72°C, разница в температуре отжига в паре праймеров не должна превышать 6°C; вторичная структура (образование шпилек): по минимуму; димеры: нежелательны, необходимо избегать само- и взаимокомплементарности между 3’-концами; минимум G/C на 3’-конце праймеров (не более трех из пяти последних нуклеотидов). Специфичность праймеров оценивали при помощи онлайн-ресурса «Primer-Blast», расположенном на сайте NCBI.
Преимуществом данного изобретения является экспресс-выявление генов резистентности, к антибиотикам, наиболее часто применяемых в ветеринарной практике и являющихся критически важным для клинической медицины.
Сущность изобретения заключается в том, что при проведении полимеразной цепной реакции в «режиме реального времени», используют специальный оригинальный набор олигонуклеотидных праймеров и зондов для идентификации и детекции фрагментов генов устойчивости к фторхинолонам qnrS, qnrB и бактерий семейства Enterobacteriaceae (вид Escherichia coli,и Shigella spp.- детекция гена csrB, и Salmonella enterica - детекция гена гена invA.
В ПЦР используют специфичные праймеры и зонды, меченые флуоресцентными красителями, спектральные характеристики зондов позволяют разбить четыре ПЦР на 2 группы, каждая из которых проводится в отдельных пробирках.
Способ идентификации и детекции генетических детерминант резистентности qnrS и qnrB и бактерий (вид Escherichia coli, и Shigella spp.- детекция гена csrB, и Salmonella enterica - детекция гена гена invA, включает выделение ДНК из биологических образцов и чистых культур, проведение ПЦР-РТ с синтетическими олигонуклеотидными праймерами, при этом используют предложенный набор олигонуклеотидных праймеров и зондов.
Олигонуклеотиды, входящие в набор, отличаются от известных из уровня техники тем, что имеют оригинальные олигонуклеотидные последовательности и позволяют специфическим образом детектировать и выявлять гены qnrS и qnrB, по которым осуществляется определение устойчивости анализируемого образца к фторхинолонам т.е. набор олигонуклеотидов является видоспецифичным. В качестве дополнительного внутреннего контроля предусмотрено выявление бактерий, часто представленных в анализируемых пробах: вид Escherichia coli,и Shigella spp.- детекция гена csrB, и Salmonella enterica - детекция гена гена invA.
В качестве генетических маркеров для выявления E.coli, Shigella spp.и S.enterica выбраны фрагменты хромосомных генов csrB и invA соответственно. Ген csrB кодирует малую некодирующую РНК, которая связываясь с белком CsrA, образует рибопротеин, регулирующий синтез гликогена. Продукт гена invA относится к факторам вирулентности сальмонелл, он необходим для инвазии бактерий через эпителиальные клетки кишечника.
Материалом для исследования могут быть образцы биологического материала, полученные из продовольственного сырья, пищевых продуктов, от животных (фекалии), из объектов окружающей среды (смывы с клеток, стен, оборудования; образцы фекалий, подстилка и др.), без этапа выделения бактериальных изолятов, а также чистые бактериальные культуры.
Отбор проб, выделение, идентификация бактерий семейства Enterobacteriaceae и получение чистой бульонной культуры изолятов проводят по стандартным методикам в соответствии с утвержденными нормативными документами. Отбор проб смывов с тушек, клеток, стен, оборудования и др. проводят с использованием велюр-тампонов eSwab-system. Фекалии и образцы подстилки собирают в пластиковые контейнеры для сбора биоматериала с завинчивающейся крышкой.
Для подготовки анализируемой пробы бактериального изолята отбирают дозатором 1,0 см3 жидкой бульонной культуры бактерий семейства Enterobacteriaceae в предварительно промаркированную одноразовую полипропиленовую пробирку вместимостью 1,5 см3. Центрифугируют на микроцентрифуге в течение 5 мин при 8000 об/мин. Затем удаляют надосадочную жидкость. К полученному осадку добавляют 1,0 см3 той же бульонной культуры и центрифугируют в течение 5 мин при 8000 об/мин. Надосадочную жидкость удаляют. Полученный осадок анализируемой пробы в закрытых промаркированных пробирках передают на выделение ДНК.
Для подготовки анализируемой пробы смывов с тушек, клеток, стен, оборудования и др. велюр-тампон eSwab-системы с биоматериалом помещают в одноразовую полипропиленовую пробирку вместимостью 1,5 см3 с изотоническим 0,9% раствором хлорида натрия (600 мм3). Тщательно перемешивают на вортексе. Для выделения ДНК использовали 100 мм3 полученного раствора.
Анализируемые пробы фекалий или пробы подстилки, отобранные в пластиковые контейнеры, готовят в виде суспензии в стерильном изотоническом 0,9% растворе хлорида натрия. В промаркированную одноразовую полипропиленовую пробирку вместимостью 1,5 см3 шпателем помещают пробу фекалий или подстилки до нижней риски, затем вносят 600 мм3 раствора натрия хлорида. Тщательно перемешивают встряхиванием. Суспензию центрифугируют при 400g в течение 2 мин. Для выделения ДНК используют 100 мм3 верхней фракции суспензии, отобранной при помощи дозатора с наконечником с фильтром.
Выделение ДНК из анализируемой пробы, осуществляют с помощью набора реагентов «АмплиПрайм ДНК-сорб-В» (ФБУН ЦНИИЭ) или «PureLink Genomic DNA» (Invitrogen), или «QIAamp DNA Mini Kit» (Qiagen) в соответствии с инструкцией производителя. В качестве отрицательного контроля выделения (Kв) используют 100 мм3 воды деионизованной.
Для приготовления мастермикса «ПЦР-смесь-1» в отдельных пробирках смешивают из расчета на одну реакцию следующие реагенты: растворы прямых и обратных ПЦР-праймеров и зондов в необходимой концентрации, 0,2 мм3 смеси дНТФ (25 ммоль/дм3) и воды деионизованной до 5 мм3. Смесь с полимеразой для выявления целевых генов готовили в отдельной пробирке (из расчета на каждую реакцию с учетом контролей выделения и ПЦР), вносят по 10 мм3 «ПЦР-смеси-1», 0,5 мм3 ДНК-полимеразы, 5 мм3 ПЦР-буфера с магнием. Затем вносят по 15 мм3 приготовленной смеси с полимеразой в тонкостенные пробирки и по 10 мм3 исследуемых или контрольных проб. Запускают на амплификаторе RotorGene соответствующую программу термоциклирования (табл.1-2).
При идентификации генов устойчивости к фторхинолонам qnrS и qnrB и генов-маркеров бактерий E.coli, Shigella spp.и S.enterica результаты интерпретируются в качественном формате на основании значения Ct. В образце обнаружен ген устойчивости к фторхинолонам из кластера qnrB (qnrS), если по каналу Red (Orange) определено среднее значение порогового цикла C ≤ 32, в образце обнаружена ДНК E.coli, Shigella spp.(ДНК S.enterica), если по каналу Yellow определено среднее значение порогового цикла Ct ≤ 30. Для отрицательных контролей выделения ДНК (Кв) и/или для отрицательных контролей ПЦР (К-) на любом из каналов появление любого значения Ct недопустимо. Для положительного контроля амплификации контрольное значение правильности реакции Ct≤27.
Пример 1 Проверка специфичности набора синтетических олигонуклеотидных последовательностей для идентификации и детекции генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам бактерий семейства Enterobacteriaceae, методом ПЦР с детекцией в режиме «реального времени» и способа их применения
Для подтверждения специфичности методики готовят контрольную панель, содержащую ДНК, выделенную из различных чистых бактериальных культур коллекции ФГБУ «ВГНКИ». В качестве контрольных используют изоляты, которые охарактеризованы полногеномно, с указанием на присутствие или отсутствие каких-либо из таргетных генов: qnrS1 (или любой ген из субкластера qnrS), qnrB2 (или любой ген из субкластера qnrB), а также отсутствие или присутствие других qnr-подобных генов, результаты представлены в таблице 1.
пп
амплификации фрагмента гена
с геном qnrB19
с геном qnrB19
с геном qnrS1
в gyrA/parC
к фторхинолонам
к фторхинолонам
В рамках предложенной панели система детекции генов резистентности показала 100% специфичность: наблюдается амплификация только фрагментов генов qnrS, qnrB, а также генов внутреннего контроля invA, csrB. Предлагаемый способ полностью удовлетворяют критерию специфичности.
Пример 2 Оценка аналитической чувствительности набора олигонуклеотидов синтетических олигонуклеотидных последовательностей для идентификации и детекции генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам бактерий семейства Enterobacteriaceae (вид Escherichia coli,и Shigella spp.- детекция участка гена csrB, и Salmonella enterica - детекция участка гена invA), методом ПЦР с детекцией в режиме «реального времени» и способа их применения. Для определения аналитической чувствительности ПЦР использовали растворы плазмидной ДНК c известной концентрацией, содержащие соответствующие клонированные фрагменты генов qnrB и qnrS. Приготовили серии десятикратных разведений плазмидной ДНК в ТЕ буфере для каждого гена в отдельности. Расчет числа копий плазмид (N) в растворе проводили с использованием формулы (1):
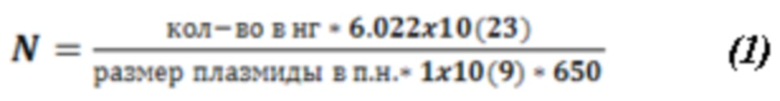
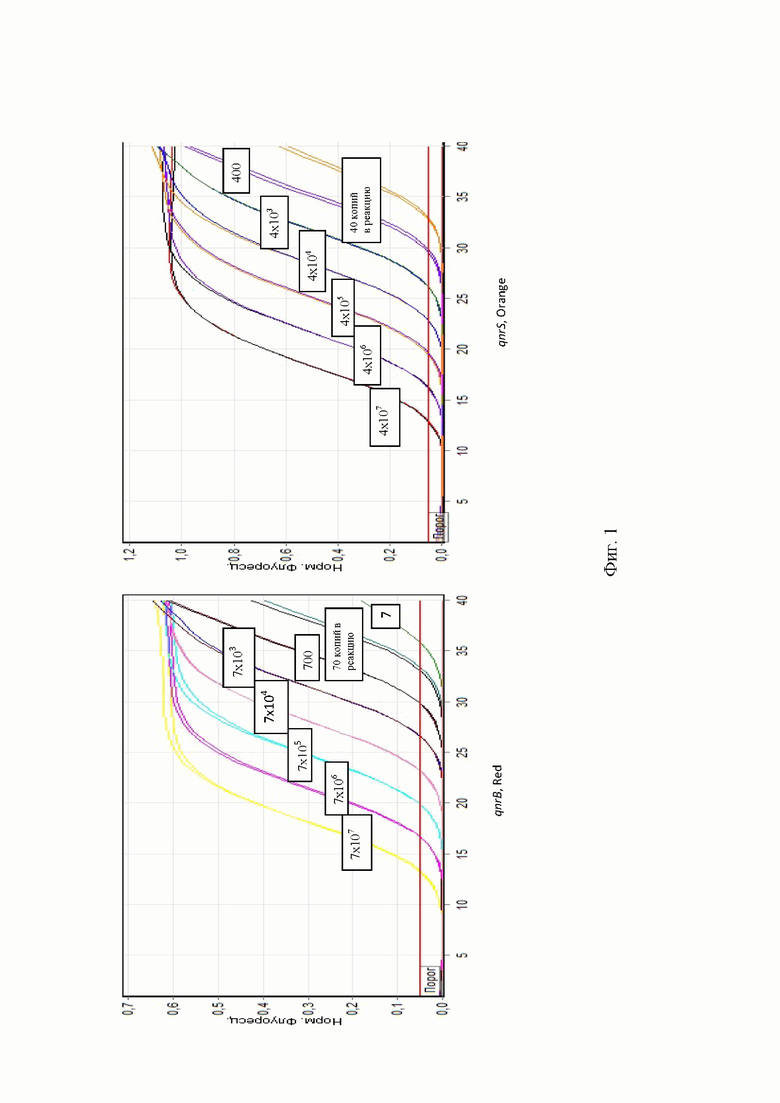
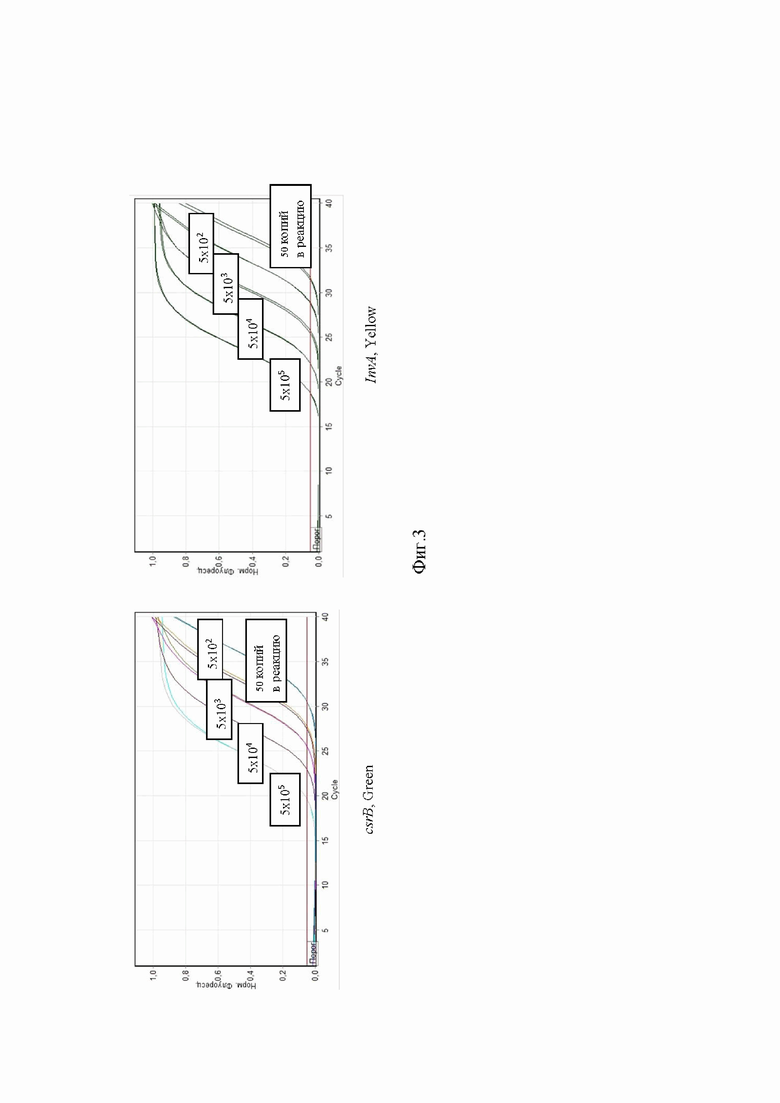
Результаты амплификации фрагментов генов qnrB и qnrS представлены на рисунке 1. Определение чувствительности ПЦР для детекции qnrB и qnrS (плазмидная ДНК). Результаты амплификации фрагментов генов csrB и invA представлены на рисунке 3.
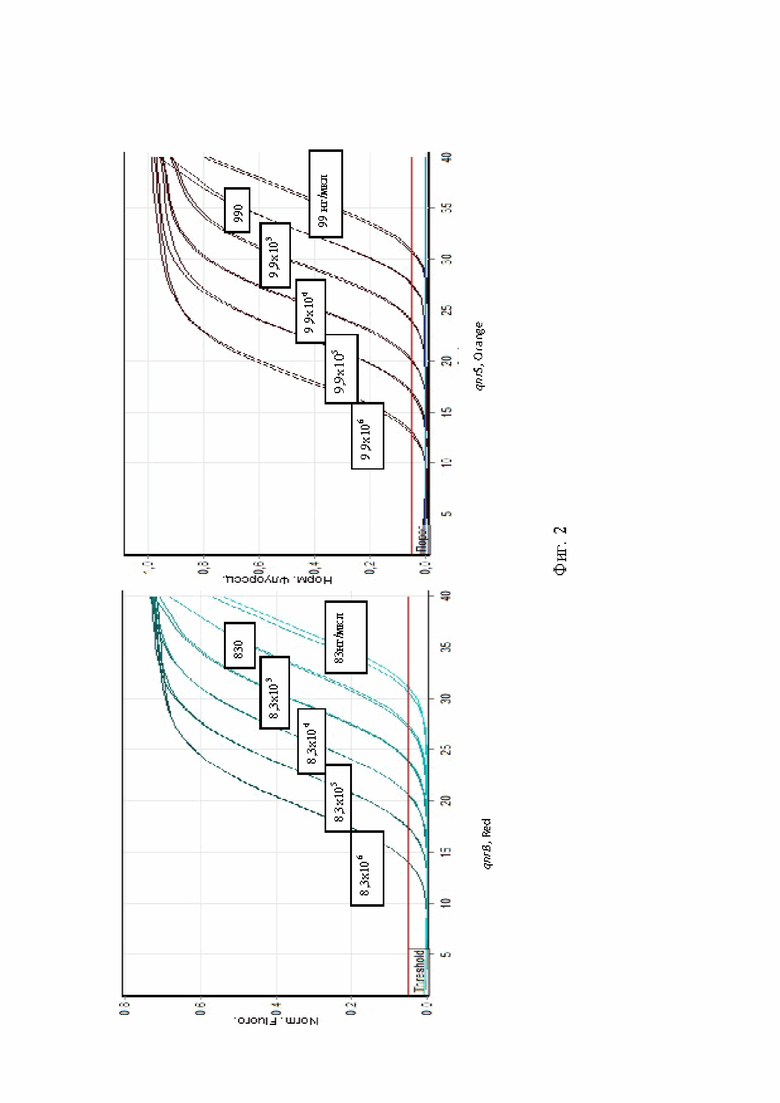
Также для оценки аналитической чувствительности ПЦР используют серийные десятикратные разведения геномной ДНК в ТЕ-буфере, выделенной из чистых культур изолятов бактерий контрольной панели S.enterica, E.coli, Shigella spp.которые охарактеризованы полногеномно и имели гены qnrB, qnrS. Количество геномных копий рассчитывали, исходя из размера генома S.enterica - 4,80 млн.п.н., E.coli - 5,12 млн.п.н. Для каждого разведения проводили ПЦР в двух повторах. Начальная концентрация геномной ДНК S.enterica составляла ~8,3*106 копий в ПЦР, а E.coli - ~9,9*106 копий в ПЦР (при внесении 10 мкл раствора ДНК на реакцию объемом 25 мкл).
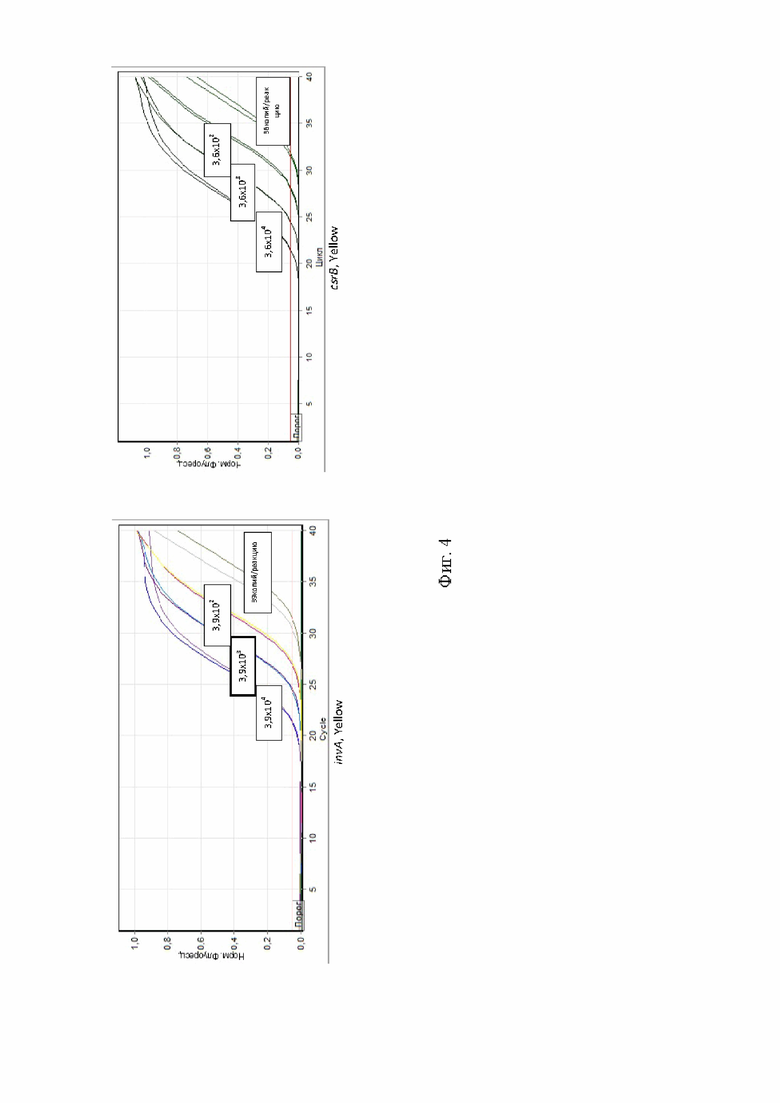
Результаты амплификации фрагментов генов qnrB и qnrS в ПЦР с геномной ДНК представлены на рисунке 2. Определение аналитической чувствительности ПЦР для детекции генов qnrB и qnrS в геномной ДНК бактерий семейства Enterobacteriaceae. Результаты амплификации фрагментов генов csrB и invA в ПЦР с геномной ДНК представлены на рисунке 4.
С целью достоверного установления порога чувствительности методик проводят определение вариабельности результатов определения количества копий целевой ДНК на пределе чувствительности.
Для этого проводят расчет относительного стандартного отклонения RSD. Используют растворы плазмидной ДНК, содержащие клонированные фрагменты генов qnrS, qnrB, csrB и invA. Для каждого гена в отдельности готовят серии десятикратных разведений плазмидной ДНК в ТЕ буфере. Ставят по 10 ПЦР реакций, в которых присутствует от 50 до 200 копий ДНК соответствующих генов в ПЦР. Полученные результаты используют для расчета RSD по формуле [2]. В качестве предела чувствительности выбирают то количество копий, при которой RSD не превышает 25%.

где SD - стандартное отклонение.
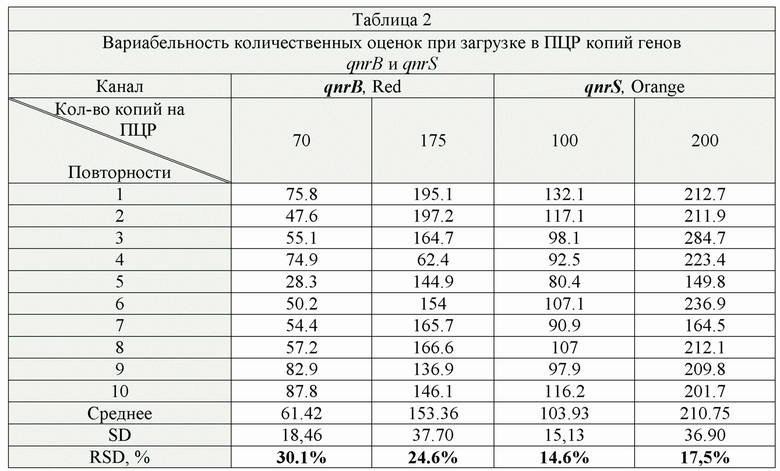
Предел аналитической чувствительности ПЦР с учетом вариабельности (RSD) менее 25% составляет для детекции гена qnrB 175 копий/реакцию или 1,75*104 копий/мл, для детекции генов qnrS - 100 копий/реакцию или 104 копий/мл. Разработанный способ соответствует принятым параметрам чувствительности ПЦР.
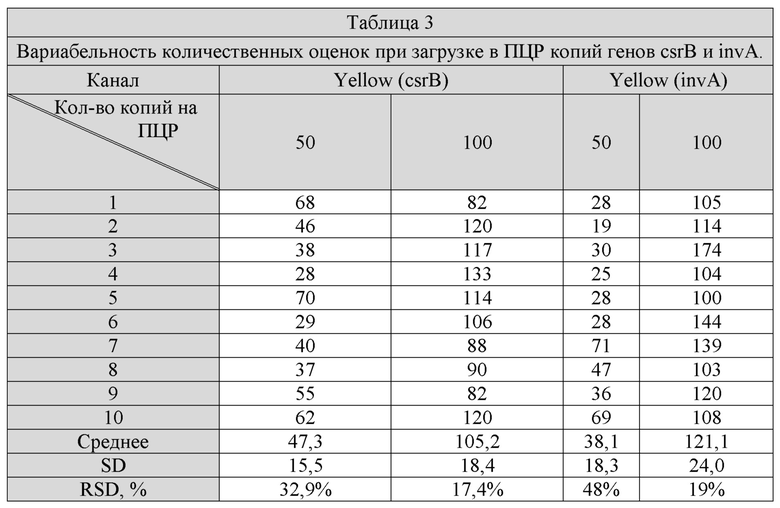
Предел аналитической чувствительности ПЦР с учетом вариабельности (RSD) менее 25% составляет для детекции участка гена csrB и invA - 100 копий/реакцию или 104 копий/мл. Разработанный способ соответствует принятым параметрам чувствительности ПЦР.
Литературные источники
1. Сидоренко С. В. и соавт.Молекулярные механизмы устойчивости грамотрицательных бактерий семейства Enterobacteriaceae к цефалоспориновым антибиотикам. Антибиотики и химиотерапия 2011; 49(3):6-16
2. А.С. Павлова, Ю.А. Бочарова, К.В. Кулешов, А.Т. Подколзин, И.В. Чеботарь, Молекулярные детерминанты резистентности Salmonella enterica к антибиотикам, Журнал микробиологии, эпидемиологии и иммунобиологии, Том 98, №6 (2021) doi.org/10.36233/0372-9311-140
3. Doma AO, Popescu R, Mituletu M, Muntean D, Dégi J, Boldea MV, Radulov I, Dumitrescu E, Muselin F, Puvača N, Cristina RT. Comparative Evaluation of qnrA, qnrB, and qnrS Genes in Enterobacteriaceae Ciprofloxacin-Resistant Cases, in Swine Units and a Hospital from Western Romania. Antibiotics (Basel). 2020 Oct 14;9(10):698. doi: 10.3390/antibiotics9100698. PMID: 33066610; PMCID: PMC7602382.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Набор
синтетических олигонуклеотидных последовательностей для идентификации
и детекции генов qnrS и qnrB.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2022-10-18">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022120606</ApplicationNumberText>
<FilingDate>2022-07-27</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>12345</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022120606</ApplicationNumberText>
<FilingDate>2022-07-27</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение "Всероссийский государственный центр
качества и стандартизации лекарственных средств для животных и
кормов" ФГБУ "ВГНКИ", Federal state budgetary
institution "The russian state center for animal feed and drug
standardization and quality" (FGBU
"VGNKI")</ApplicantName>
<ApplicantNameLatin>FGBU "VGNKI"</ApplicantNameLatin>
<InventorName languageCode="ru">Киш Леонид Карольевич</InventorName>
<InventorNameLatin>Kish Leonid Karolievich</InventorNameLatin>
<InventionTitle languageCode="ru">Набор синтетических
олигонуклеидных последовательностей для идентификации и детекции
генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам
бактерий семейства Enterobacteriaceae, методом ПЦР с детекцией в
режиме "реального времени" и способ их
применения</InventionTitle>
<SequenceTotalQuantity>12</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcctacagggtgccaattta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caatacccagtgcttcgagaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacgccgaactcgacggtttagat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgttcagtggttcagatctctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctaagtcacccaactccgaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatgtgtgaagtttgctgctcgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtgtgagcaggaagcaata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcagcattccagctactt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaaggatgagcagggagcaacaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Salmonella enterica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcacataaagatcttgtcctcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Salmonella enterica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cactgacttgctatctgctatctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Salmonella enterica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgtctgtcgatgtccgtcgattt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
аналитической чувствительности ПЦР использовали растворы плазмидной ДНК c известной концентрацией, содержащие соответствующие клонированные фрагменты генов qnrB и qnrS. Приготовили серии десятикратных разведений плазмидной ДНК в ТЕ буфере для каждого гена в отдельности. Расчет числа копий плазмид (N) в растворе проводили с использованием формулы (1):
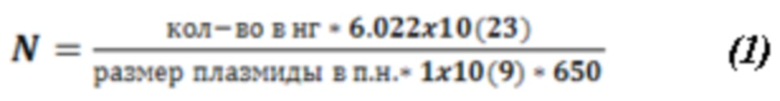
Результаты амплификации фрагментов генов qnrB и qnrS представлены на Фиг 1. Определение чувствительности ПЦР для детекции qnrB и qnrS (плазмидная ДНК). Результаты амплификации фрагментов генов csrB и invA представлены на Фиг 3.
Также для оценки аналитической чувствительности ПЦР используют серийные десятикратные разведения геномной ДНК в ТЕ-буфере, выделенной из чистых культур изолятов бактерий контрольной панели S.enterica, E.coli, Shigella spp.которые охарактеризованы полногеномно и имели гены qnrB, qnrS. Количество геномных копий рассчитывали, исходя из размера генома S.enterica - 4,80 млн.п.н., E.coli - 5,12 млн.п.н. Для каждого разведения проводили ПЦР в двух повторах. Начальная концентрация геномной ДНК S.enterica составляла ~8,3*106 копий в ПЦР, а E.coli - ~9,9*106 копий в ПЦР (при внесении 10 мкл раствора ДНК на реакцию объемом 25 мкл).
Результаты амплификации фрагментов генов qnrB и qnrS в ПЦР с геномной ДНК представлены на Фиг 2. Определение аналитической чувствительности ПЦР для детекции генов qnrB и qnrS в геномной ДНК бактерий семейства Enterobacteriaceae. Результаты амплификации фрагментов генов csrB и invA в ПЦР с геномной ДНК представлены на Фиг.4.
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР ОЛИГОНУКЛЕОТИДОВ ДЛЯ ВЫЯВЛЕНИЯ ГЕНОВ УСТОЙЧИВОСТИ К ТЕТРАЦИКЛИНАМ ИЗ ГРУППЫ tet У БАКТЕРИЙ МЕТОДОМ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ "РЕАЛЬНОГО ВРЕМЕНИ" | 2022 |
|
RU2794156C1 |
| СПОСОБ ОБНАРУЖЕНИЯ ГЕНОВ УСТОЙЧИВОСТИ К АМИНОГЛИКОЗИДАМ ИЗ ГРУППЫ aadA У БАКТЕРИЙ ЖИВОТНОГО ПРОИСХОЖДЕНИЯ МЕТОДОМ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ «РЕАЛЬНОГО ВРЕМЕНИ» | 2023 |
|
RU2816522C1 |
| Олигонуклеотидные праймеры и зонд для выявления фрагмента гена 23S рРНК бактерий семейства Enterococcaceae | 2023 |
|
RU2824466C1 |
| Способ оценки генов токсинов Clostridium perfringens и Clostridioides difficile в консервированных объемистых кормах для крупного рогатого скота | 2024 |
|
RU2841390C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ ДЛЯ ВЫЯВЛЕНИЯ ДНК ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННОГО АТЛАНТИЧЕСКОГО ЛОСОСЯ МЕТОДОМ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2023 |
|
RU2808662C1 |
| ПОЛИРЕЗИСТЕНТНЫЙ ШТАММ БАКТЕРИЙ ESCHERICHIA COLI ДЛЯ ОПРЕДЕЛЕНИЯ БАКТЕРИЦИДНОГО ДЕЙСТВИЯ АНТИБАКТЕРИАЛЬНЫХ ПРЕПАРАТОВ В ВЕТЕРИНАРИИ | 2022 |
|
RU2797383C1 |
| Способ идентификации патогенных бактерий в пищевых субстратах с использованием высокопроизводительного секвенирования | 2018 |
|
RU2712527C2 |
| ШТАММ БАКТЕРИЙ SALMONELLA ENTERICA SBSP. ENTERICA SEROVAR KENTUCKY B-9045 МЕЖДУНАРОДНОГО ПОЛИРЕЗИСТЕНТНОГО КЛОНА SALMONELLA KENTUCKY ST198, ИСПОЛЬЗУЕМЫЙ В КАЧЕСТВЕ КОНТРОЛЬНОГО ШТАММА ДЛЯ ФЕНОТИПИЧЕСКИХ И МОЛЕКУЛЯРНЫХ ИССЛЕДОВАНИЙ ПРИ ДИАГНОСТИКЕ САЛЬМОНЕЛЛЕЗОВ | 2020 |
|
RU2744203C1 |
| Технология для детекции биозараженности нефтегазовых объектов | 2023 |
|
RU2812465C1 |
| Тест-система и способ обнаружения специфических фрагментов нуклеиновых кислот 16 патогенов с использованием изотермической реакции амплификации | 2023 |
|
RU2810751C1 |
Изобретение относится к области биотехнологии, в частности к набору олигонуклеотидов для идентификации и детекции генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам бактерий семейства Enterobacteriaceae. Также раскрывается применение данного набора для идентификации и детекции генов qnrS и qnrB методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Изобретение эффективно для экспресс-выявления генов резистентности к фторхинолонам qnrS и qnrB - антибиотикам, наиболее часто применяемым в ветеринарной практике. 2 н.п. ф-лы, 4 ил., 3 табл., 2 пр.
1. Набор синтетических олигонуклеотидных последовательностей для идентификации и детекции генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам бактерий семейства Enterobacteriaceae методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме «реального времени», содержащий праймеры и флуоресцентно-меченые зонды для выявления фрагментов плазмидных генов резистентности к фторхинолонам имеющие следующий состав:
qnrS-F- GCCTACAGGGTGCCAATTTA,
qnrS-R- CAATACCCAGTGCTTCGAGAAT,
qnrS-Z- ROX-CACGCCGAACTCGACGGTTTAGAT-BHQ2
qnrB–F CGTTCAGTGGTTCAGATCTCTC,
qnrB–R- CTAAGTCACCCAACTCCGAAT,
qnrB–Z- Cy5-AATGTGTGAAGTTTGCTGCTCGCC-BHQ2,
сsrB-F- GGTGTGAGCAGGAAGCAATA;
сsrB-R- CGCAGCATTCCAGCTACTT;
сsrB-Z- R6G-TCAAGGATGAGCAGGGAGCAACAA-BHQ1;
invA-F- GCACATAAAGATCTTGTCCTCCT;
invA-R- CACTGACTTGCTATCTGCTATCTC;
invA-Z- R6G–ACGTCTGTCGATGTCCGTCGATTT– BHQ1.
2. Способ идентификации и детекции генов qnrS и qnrB, обеспечивающих устойчивость к фторхинолонам бактерий семейства Enterobacteriaceae методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме «реального времени», включающий выделение ДНК из образцов биологического материала или бактериальных культур, постановку полимеразной цепной реакции с использованием набора олигонуклеотидных праймеров и зондов по пункту 1, реакционной смеси следующего состава: смесь 1 qnrS+ invA: 10 мм3 ДНК-матрицы, 10 мм3 ПЦР-смеси-1, включающей 6 пмоль каждого из праймеров qnrS-F, qnrS-R, invA-F, invA-R, 3 пмоль каждого зонда qnrS-Z и invA-Z , 2,5 мм3 раствора дНТФ с концентрацией каждого нуклеотида 1,76 ммоль/дм3, деионизованную воду, 0,5 мм3 Taq ДНК полимеразы, 5 мм3 ПЦР-буфера с магнием; детектирование флуоресцентного сигнала непосредственно в процессе реакции амплификации на канале Orange/Yellow ПЦР в режиме «реального времени» проводят с использованием следующей программы амплификации: 15 мин при 95°С; 15 с при 95°С, 45 с при 62°С - 35 циклов с детекцией флуоресцентного сигнала, смесь 2 qnrB+ csrB: 10 мм3 ДНК-матрицы, 10 мм3 ПЦР-смеси-1, включающей 6 пмоль каждого из праймеров qnrB-F, qnrB-R, csrB-F, csrB -R, 3 пмоль каждого зонда qnrB-Z и csrB -Z , 2,5 мм3 раствора дНТФ с концентрацией каждого нуклеотида 1,76 ммоль/дм3, деионизованную воду, 0,5 мм3 Taq ДНК полимеразы, 5 мм3 ПЦР-буфера с магнием; а так же детектирование флуоресцентного сигнала непосредственно в процессе реакции амплификации на канале Red / Yellow ПЦР в режиме «реального времени» проводят с использованием следующей программы амплификации: 15 мин при 95°С; 15 с при 95°С, 45 с при 62°С - 35 циклов с детекцией флуоресцентного сигнала, при этом результаты амплификации в режиме «реального времени» оценивают с помощью программного обеспечения, при этом результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на уровне 0,05 пороговой линии, в случае обнаружения гена invA идентифицируют микроорганизм Salmonella enterica, гена сsrB - Escherichia coli, в случае обнаружения генов qnrS и qnrB, кодирующих экранирующие белки, делают вывод об устойчивости анализируемого штамма к фторхинолонам.
| CN104560981 A, 29.04.2015 | |||
| CN110408712 A, 05.11.2019 | |||
| CN102952875 A, 06.03.2013 | |||
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ДЕТЕКЦИИ УСТОЙЧИВОСТИ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗА К ПРОТИВОТУБЕРКУЛЕЗНЫМ ПРЕПАРАТАМ ВТОРОГО РЯДА (ФТОРХИНОЛОНАМ, АМИНОГЛИКОЗИДАМ И КАПРЕОМИЦИНУ) | 2015 |
|
RU2633507C2 |
| THOMAS GUILLARD et al., Rapid detection of qnr and qepA plasmid-mediated quinolone resistance genes using real-time PCR, Diagn Microbiol Infect Dis, 2011, Vol | |||
| Деревянный торцевой шкив | 1922 |
|
SU70A1 |
| Прибор для измерения угла наклона | 1921 |
|
SU253A1 |
Авторы
Даты
2023-12-27—Публикация
2022-07-27—Подача