Область техники, к которой относится изобретение
Настоящее изобретение относится к молекулярной биологии, медицине, включая фтизиатрию, и может быть использовано для лабораторного определения микобактерий туберкулеза, относящихся к кластеру Beijing B0/W148.
Уровень техники
Среди генетических семейств, выделяемых внутри вида Mycobacterium tuberculosis, для России особое значение имеют представители генотипа Beijing, встречающиеся более чем у половины больных туберкулезом. Данное семейство было впервые описано в 1992-1994 годах в Пекинском регионе Китая. В свою очередь кластер В0/W148 является наиболее распространенным в России вариантом генотипа Beijing [Mokrousov I. et al. Mycobacterium tuberculosis population in northwestern Russia: an update from Russian-EU/Latvian border region // PLoS One. 2012;7(7):e41318]. Представители этого кластера преобладают у больных с тяжелыми формами туберкулеза, демонстрируют высокую вирулентность [Вишневский Б.И. и др. Вирулентность микобактерий туберкулеза // Проблемы туберкулеза. - 2002а. - №10. - С. 33-36] и трансмиссивность [Mokrousov I et al. Mycobacterium tuberculosis Beijing genotype in Russia: in search of informative variable-number tandem-repeat loci // J Clin Microbiol. 2008 Nov;46(11):3576-84], ассоциацию с лекарственной устойчивостью [Toungoussova O. et al. Spread of drug-resistant Mycobacterium tuberculosis strains of the Beijing genotype in the Archangel Oblast, Russia // J. Clin. Microbiol. - 2002. - Vol. 40. - P. 1930-1937]. Текущая эпидемия туберкулеза в России в значительной степени связана с активным распространением резистентных штаммов семейства В0/W148 в популяции, иммунизированной БЦЖ. Таким образом, своевременное выявление штаммов этого кластера является важной составляющей диагностики туберкулеза и имеет существенное значение для выбора адекватного лечения.
На сегодняшний день для молекулярно-генетического типирования микобактерий разработано около десятка различных технологий, однако наибольшее распространение получили три: сполиготипирование (spoligotyping), анализ числа тандемных повторов в различных локусах генома (от англ.Variable Number of Tandem Repeats, VNTR) и анализ полиморфизма длины рестрикционных фрагментов IS6110 (от англ. Restriction Fragment Length Polymorphisms IS6110, IS6110-RFLP).
Метод сполиготипирования (spoligotyping, от англ. Spacer Oligonucleotide Typing) заключается в определении структуры генетического региона M. tuberculosis (DR-регион), состоящего из нескольких десятков повторяющихся последовательностей и уникальных по своему нуклеотидному составу промежутков (спейсеров) между ними [Kamerbeek J. et al. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology // J Clin Microbiol. 1997 Apr;35(4):907-14]. Метод требует проведения полимеразной цепной реакции (ПЦР) и последующей ДНК-гибридизации с нанесенными на мембрану уникальными спейсерными последовательностями в специальном приборе (миниблоттере) и выявлением сигналов хемилюминесценции на светочувствительной пленке. Данный метод позволяет определить генотип Beijing, однако, не позволяет устанавливать принадлежность к кластеру B0/W148.
Еще один метод типирования, основанный на амплификационных технологиях - анализ числа тандемных повторов. Данный метод базируется на оценке длины продукта ПЦР-реакции (минисателлитный анализ, англ. Variable Number of Tandem Repeats Typing, VNTR-typing). Наиболее характерный паттерн для кластера B0/W148 по 24 локусному типированию (s154, s580, s960, s1644, s2059, s2531, s2687, s2996, s3007, s3192, s4348, s802, s2165, s2461, s577, s2163, s4052, s4156, s424, s1955, s2347, s2401, s3171, s3690) представлен в виде 244233352644425173353723, где цифрами обозначено количество повторов в определенном локусе [Mokrousov I. Insights into the Origin, Emergence, and Current Spread of a Successful Russian Clone of Mycobacterium tuberculosis // Clin. Microbiol. Rev 2013 Apr; 26(2): 342-360]. Однако уже сейчас появляются данные, что данный паттерн может видоизменяться, и тем самым данный метод определения представителей кластера B0/W148 малопригоден к использованию [Mokrousov I. Insights into the Origin, Emergence, and Current Spread of a Successful Russian Clone of Mycobacterium tuberculosis // Clin. Microbiol. Rev 2013 Apr; 26(2): 342-360].
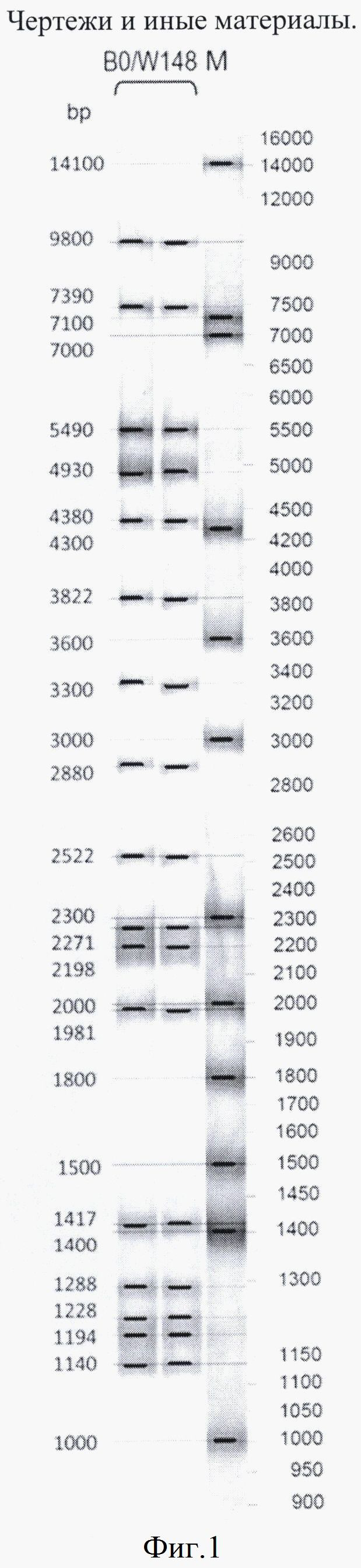
В настоящее время для обнаружения штаммов семейства В0/W148 используют метод типирования IS6110-RFLP (от англ. Restriction Fragment Length Polymorphisms IS6110), основанный на гибридизационном анализе полиморфизма длин рестрикционных фрагментов ДНК, содержащих инсерционный элемент IS6110. Данный метод является аналогом предложенного изобретения. Подход был предложен в 1993 году van Embden и его коллегами [Van Embden J. et al. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology// J. Clin. Microbiol. -1993. -Vol. 31. -P. 406-409]. Метод базируется на определении количества и локализации в геноме M. tuberculosis небольшого (около 1350 п.о.) мобильного генетического элемента - инсерционной последовательности IS6110, имеющей сайт рестрикции PvuII [Thierry D. et al. IS6110, an IS-like element of Mycobacterium tuberculosis complex // NAR. -1990. -Vol. 18(1). -P. 188]. Фрагменты IS6110 распределяются в кольцевой хромосоме M. tuberculosis хаотичным образом, а их число может варьировать от 0 до 26 копий на геном. В процессе обработки рестриктазой PvuII геномная ДНК M. tuberculosis распадается на несколько фрагментов, количество и длина которых зависит от числа IS6110 элементов и их локализации в геноме МТБ. Фрагменты разделяются при помощи гель-электрофореза и визуализируются методом Саузерн-блот-гибридизации с мечеными зондами, комплементарными последовательности IS6110. Таким образом, для каждого штамма получается уникальный гибридизационный паттерн. Паттерн, характерный для представителей кластера B0/W148, представлен на Фигуре 1. Однако метод обладает рядом серьезных недостатков, затрудняющих его широкое применение в лабораторной практике. Ряд проблем связан со сложностью самой методики, необходимостью использовать большое количество бактериальной массы, длительностью процедуры анализа (до 40 дней). Как и многие другие гель-электрофоретические методики анализа, IS6110-RFLP типирование характеризуется сложностью обработки результатов.
Задачей предлагаемого изобретения является разработка способа быстрой и однозначной детекции М. tuberculosis генотипа Beijing В0/W148.
Раскрытие изобретения
Задача идентификации микобактерий туберкулеза кластера Beijing В0/W148 достигается посредством применения полимеразной цепной реакции для амплификации фрагментов ДНК M. tuberculosis с праймерами P1 5'-GTGTTGTACATTGGGCATCG-3', P2 5'-GGTGTACATATCGAAGCTCG-3' и P3 5'-GCTCGACGAAGTGAGATTGC-3', последовательности которых представлены SEQ ID NO: 1, 2, 3, соответственно, в режиме амплификации 98°С - 10 мин, 30 циклов: 98°С - 30 с, 64°С - 30 с, 72°С - 50 с, 72°С - 10 мин, 4°С, разделением продуктов амплификации электрофорезом в 2% агарозном геле с получением фрагмента размером 1021 пн для штаммов Beijing В0/W148 или размером 320 пн - для штаммов, не относящихся к данному семейству.
Для постановки реакции амплификации используются праймеры, подобранные в ходе анализа результатов полногеномного секвенирования 40 штаммов различных генетических семейств, эндемичных для России (GenBank № SRA061654). Амплификация проводится в одной пробирке с использованием праймеров P1, P2 и P3, последовательности которых представлены SEQ ID NO: 1, 2, 3, соответственно. Сущность изобретения поясняется чертежами.
Краткое описание чертежей
Фигура 1. Характерный профиль IS6110-RFLP штаммов, относящихся к кластеру Beijing B0/W148. В качестве маркера выбран профиль рестрикции лабораторного штамма 14323.
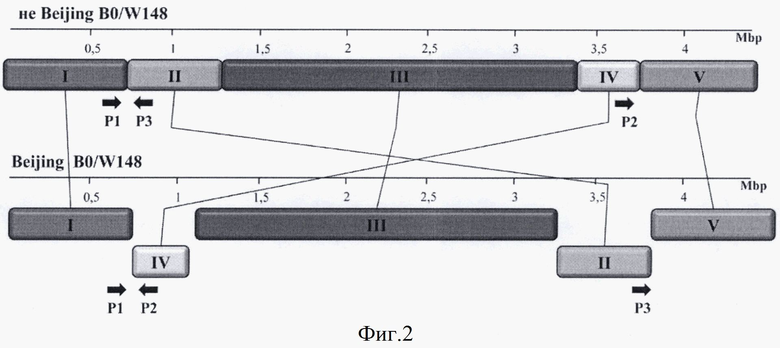
Фигура 2. Схема ПЦР и расположения праймеров Р1, Р2 и Р3. Блоками обозначены участки генома. Стрелками указаны положения праймеров. Блоки II и IV у представителей кластера Beijing B0/W148 инвертированы и переставлены.
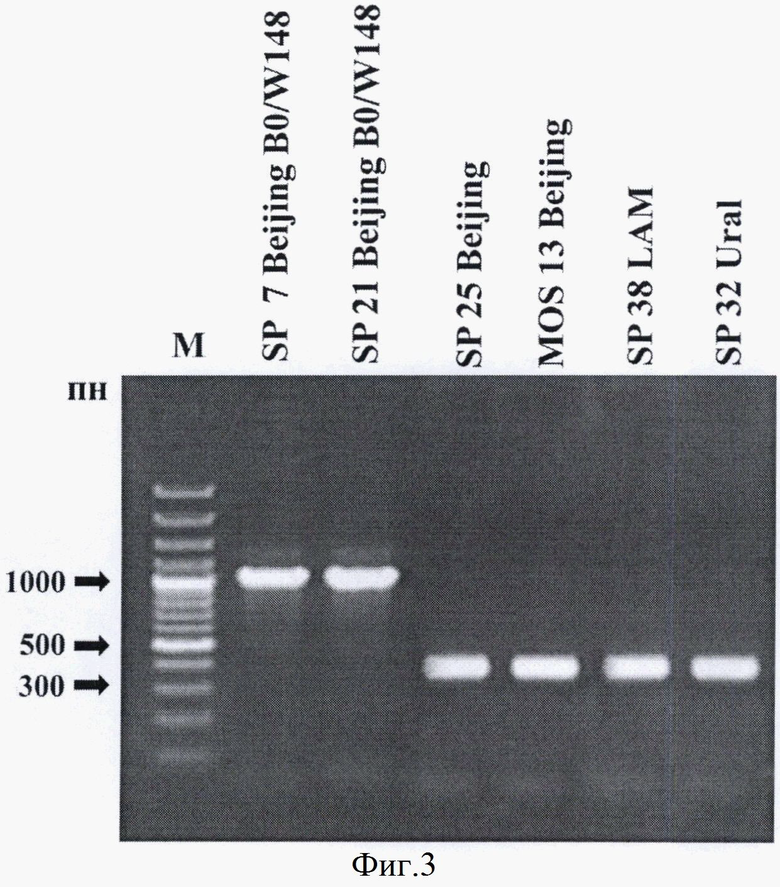
Фигура 3. Примеры электрофореза продуктов амплификации штаммов микобактерий туберкулеза кластера Beijing B0/W148, Beijing и других генетических семейств. М, маркер молекулярных весов «100 bp DNA ladder».
Осуществление изобретения
1) Выделение геномной ДНК
Выделение тотальной геномной ДНК M. tuberculosis проводят по методу van Embden с соавторами [van Embden J. et al. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology // J Clin Microbiol., 1993. 31(2): p. 406-9.]. Полученные в процессе культивирования на питательной среде Lowenstein-Jensen клетки M. tuberculosis собирают микробиологическими петлями в пробирки 1.5 мл (типа Eppendorf), содержащими 500 мкл TE буфер. Тепловую инактивацию клеток проводят при 90°С в течение 30 минут. Полученную суспензию центрифугируют при 6000 g в течение 3 минут с последующим удалением супернатанта. К осадку добавляют 100 мкл 20 мг/мл лизоцима с последующей инкубацией 1 час при 37°С. Далее добавляют 150 мкл раствора, содержащего 10% додецилсульфатнатрия (SDS))(140 мкл) и протеиназу К (10 мг/мл)(10 мкл). Перемешивают встряхиванием и инкубируют при 65°С один час. Далее в пробирку добавляют 100 мкл 5M NaCl. В последующем добавляют 1 объем фенол/хдороформа (соотношение 1:1), интенсивно перемешивают и центрифугируют 10 мин при 14 900 g. Верхнюю фазу переносят в чистую пробирку, не затрагивая нижнюю фазу и интерфазу. К водной фазе добавляют 1,5 объема 96% этилового спирта (-20°С) и аккуратно перемешивают. Инкубируют в течение ночи при -20°С или 1 час при -70°С. В дальнейшем проводят осаждение ДНК центрифугированием 15 мин при 14 900 g. К осадку добавляют 400 мкл 70% этилового спирта (-20 °С). Центрифугируют 5 минут при 14 900 g. Удаляют супернатант и растворяют полученную ДНК в 30 мкл ТЕ буфера. Дальнейшее хранение образцов хромосомной ДНК проводят при -20º С. Полученную ДНК используют для постановки ПЦР.
2) Амплификация фрагментов генома M. tuberculosis
Материал для амплификации - чистая ДНК. Амплификацию фрагментов ДНК проводят методом ПЦР с использованием трех праймеров: P1 GTGTTGTACATTGGGCATCG, P2 GGTGTACATATCGAAGCTCG и P3 GCTCGACGAAGTGAGATTGC. В качестве контрольной используют ДНК типичного штамма Beijing В0/W148. Очищенную ДНК (0.5 мкл) добавляют к смеси ПЦР (конечный объем 25 мкл), содержащей 66 мМ Tris-HCl (pH = 9.0); 16.6 мМ (NH4)2SO4; 2.5 мМ MgCl2, по 250 мкМ каждого дезоксирибонуклеозидтрифосфатов (дНТФ), 1 Ед Taq-полимеразы («ЛИТЕХ», Россия) и по 20 пмоль праймеров Р1, Р2 и Р3. Реакцию проводят в амплификаторе DNA Engine Tetrad 2 (MJ Research, USA) при условиях, адаптированных для максимального выхода ПЦР - продукта: 98°С - 10 мин, 30 циклов: 98°С - 30 с, 64°С - 30 с, 72°С - 50 с, 72°С - 10 мин, 4°С - хранение. Продукты ПЦР (5 мкл) разделяют электрофорезом в 1,5% агарозном геле и визуализируют на УФ-трансиллюминаторе.
Указанные праймеры были подобраны исходя из результатов полногеномного секвенирования 40 эндемичных для России штаммов M. tuberculosis (GenBank № SRA061654). Способ устроен таким образом, что при наличии в реакционной смеси образца с генотипом Beijing B0/W148 срабатывают праймеры P1 и P2. При этом праймер P3 не участвует в амплификации. В свою очередь при наличии в системе представителей других генотипов амплификация проходит с праймеров P1 и P3 (Фигура 2).
Для оценки специфичности выбранных праймеров проводили сравнительный анализ нуклеотидных последовательностей с базой данных GenBank с помощью программы BLAST. Было установлено отсутствие перекрестных реакций при использовании этих праймеров с ДНК других видов микроорганизмов и человека.
3) Оценка результатов
При наличии в образце штамма Beijing В0/W148 ПЦР-продукт представляет собой яркую полосу, представленную амплифицированным фрагментом ДНК размером 1021 пн, в случае любого другого генотипа получается фрагмент длиной 320 пн (Фигура 3). Определение длин фрагментов проводят путем сравнения с маркером молекулярных весов (например, 100bp ladder) или непосредственно путем сравнения с профилем амплифицированных фрагментов контрольного штамма генотипа Beijing В0/W148.
Апробацию предлагаемого способа обнаружения штаммов M. tuberculosis, относящихся к кластеру Beijing B0/W148, проводили на коллекции из 60 образцов геномной ДНК штаммов M. tuberculosis, выделенной в Санкт-Петербургском научно-исследовательском институте эпидемиологии и микробиологии им. Пастера и Московском городском научно-практическом центре борьбы с туберкулезом. Для представленной коллекции было проведено сполиготипирование, IS6110-RFLP типирование и типирование описываемым способом. Согласно сполиготипированию 35 образцов относились к генотипу Beijing. Из них 18 образцов, согласно IS6110-RFLP типированию, были идентифицированы как Beijing B0/W148. Для тех же 18 образцов, тестируемых с использованием предложенного способа, в ходе ПЦР была идентифицирована полоса в области 1021 пн маркера, что соответствует выявлению кластера Beijing B0/W148. Для 42 образцов ДНК была идентифицирована полоса размером 320 пн, что согласуется с результатами, полученными с использованием классических методов типирования. Данный метод имеет ряд существенных преимуществ, таких как быстрота выполнения и возможность одновременного анализа большой коллекции образцов, однозначность интерпретации результатов, простое оборудование для постановки стандартной ПЦР и гель-электрофореза. Разработанный способ является достаточно доступным в условиях специализированной лаборатории и занимает менее суток, что существенно быстрее по сравнению c методом IS6110-RFLP. Таким образом, предлагаемый способ обеспечивает высокую чувствительность и специфичность определения штаммов микобактерий туберкулеза генотипа Beijing B0/W148.
Перечень последовательностей
SEQUENCE LISTING
<110> Федеральное государственное бюджетное учреждение науки
"Научно-исследовательский институт физико-химической медицины
Федерального медико-биологического агентства"
<120> Способ обнаружения микобактерий туберкулёза генетического
кластера Beijing B0/W148
<130>
<140>
<141> 2013-07-03
<160> 3
<170> PatentIn version 3.5
<210> 1
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer P1 for ampliification of Mycobacterium tuberculosis genome fragment
<400> 1
gtgttgtaca ttgggcatcg 20
<210> 2
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer P2 for ampliification of Mycobacterium tuberculosis genome fragment
<400> 2
ggtgtacata tcgaagctcg 20
<210> 3
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer P3 for ampliification of Mycobacterium tuberculosis genome fragment
<400> 3
gctcgacgaa gtgagattgc 20
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления микобактерий туберкулеза Центрально-Азиатского эпидемического кластера генотипа Beijing | 2019 |
|
RU2735415C1 |
| Способ выявления микобактерий туберкулеза генотипа Beijing В0-кластер в формате реального времени | 2017 |
|
RU2684314C2 |
| СПОСОБ ВЫЯВЛЕНИЯ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗА ГЕНОТИПА BEIJING | 2008 |
|
RU2405836C2 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 14717-15-кластер | 2022 |
|
RU2812351C1 |
| СПОСОБ ВЫЯВЛЕНИЯ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗА ГЕНОТИПА ВЕIJING В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2011 |
|
RU2528866C2 |
| Способ детекции изолятов Mycobacterium tuberculosis Beijing 94-32-кластера в формате реального времени | 2017 |
|
RU2689800C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ШТАММОВ Mycobacterium tuberculosis | 2013 |
|
RU2547598C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 1071-32-кластер в формате реального времени | 2021 |
|
RU2768021C1 |
| Способ детекции филогенетических сублиний генотипа Beijing Mycobacterium tuberculosis в формате реального времени | 2020 |
|
RU2743365C1 |
Изобретение относится к биотехнологии, а именно к способу идентификации микобактерий туберкулеза кластера Beijing B0/W148. Применяют полимеразную цепную реакцию для амплификации фрагментов ДНК М. tuberculosis с подобранными праймерами P1, Р2 и Р3, последовательности которых представлены SEQ ID NO 1, 2, 3, соответственно, в режиме амплификации 98°C - 10 мин, 30 циклов: 98°C - 30 с, 64°C - 30 с, 72°C - 50 с, 72°C - 10 мин, 4°C. Проводят разделение продуктов амплификации электрофорезом в 2% агарозном геле. В случае получения фрагмента размером 1021 пн определяют штамм микобактерий туберкулеза кластера Beijing B0/W148. Предложенное изобретение позволяет быстро и с высокой специфичностью идентифицировать микобактерии туберкулеза кластера Beijing B0/W148. 3 ил.
Способ идентификации микобактерий туберкулеза кластера Beijing B0/W148, отличающийся тем, что применяют полимеразную цепную реакцию для амплификации фрагментов ДНК М. tuberculosis с подобранными праймерами P1, Р2 и Р3, последовательности которых представлены SEQ ID NO 1, 2, 3, соответственно, в режиме амплификации 98°C - 10 мин, 30 циклов: 98°C - 30 с, 64°C - 30 с, 72°C - 50 с, 72°C - 10 мин, 4°C, разделение продуктов амплификации проводят электрофорезом в 2% агарозном геле, в случае получения фрагмента размером 1021 пн определяют штамм микобактерий туберкулеза кластера Beijing B0/W148.
| MOKROUSOV I., Insights into the origin, emergence, and current spread of a successful Russian clone of Mycobacterium tuberculosis, Clin Microbiol Rev, 2013, v.26, no.2, p | |||
| Трепальная машина для обработки лубовых растений | 1923 |
|
SU342A1 |
| MOKROUSOV I | |||
| ET AL., Russian "successful" clone B0/W148 of Mycobacterium tuberculosis Beijing genotype: a multiplex PCR assay for rapid detection and global screening, J | |||
Авторы
Даты
2015-05-27—Публикация
2013-07-04—Подача