Изобретение относится к медицине, а именно к фтизиатрии, и может быть использовано для лабораторного определения микобактерий туберкулеза, относящихся к высокорезистентному генетическому варианту Mycobacterium tuberculosis Beijing 1071-32-кластер.
Μ. tuberculosis является строго клональным видом, подразделяемым на филогенетические линии, которые, в свою очередь, разделяют на менее крупные сублинии, генетические семейства и компактные клональные кластеры генетически близкородственных штаммов (Gagneux, 2018). В настоящее время при снижении заболеваемости туберкулезом в России имеет место нарастание доли множественно/широко-лекарственно-устойчивых (МЛУ/ШЛУ) штаммов. Несмотря на усилия по борьбе с туберкулезом (ТБ), продолжают циркулировать эпидемические и мультирезистентные штаммы, которые демонстрируют свою повышенную адаптивность, остаются высокотрансмиссивными и эволюционируют в сторону развития широкой лекарственной устойчивости. Кроме того, в рамках малоизученных сублиний возбудителя могут возникать новые резистентные или высокотрансмиссивные генетические варианты.
Восточно-Азиатская линия М. tuberculosis является одной из наиболее распространенных, а генетическое семейство Beijing (также называемое Пекинским генотипом микобактерий туберкулеза) - наиболее известным ее представителем. Генетическое семейство Beijing разделяют на крупные сублинии древних и современных штаммов (Mokrousov et al., 2002). Штаммы современной сублинии Beijing преобладают во всем мире, а древние сублинии представляют гетерогенную популяцию клонов и составляют заметную долю популяции М. tuberculosis только в некоторых странах, прежде всего в Японии и Корее, реже встречаются в Китае и Вьетнаме и совсем редко в других странах (Shamputa et al., 2010; Wada et al., 2009 Mokrousov et al., 2005; Luo et al., 2015; Kremer et al., 2005; Maeda et al., 2014). Например, в России, где генотип Beijing составляет от 35 до 65% от общей популяции М. tuberculosis, древние пекинские штаммы были обнаружены только в 5% популяции Beijing на северо-западе России в исследовании 20 лет назад (Mokrousov et al., 2002). Недавно штаммы древней сублинии генотипа Beijing были выявлены в относительно высокой доле (14% популяции Beijing) в Омской области Западной Сибири и представляли два кластера по номенклатуре MIRU-VNTRplus.org - меньший 14717-15 и более крупный 1071-32 (Mokrousov et al., 2019). Изучение данных по локусам VNTR продемонстрировало их присутствие в других регионах России и позволило предположить некоторую филогеографическую связь с Восточной Азией. Анализ данных по полногеномному секвенированию (англ. whole genome sequencing [WGS]) российских и зарубежных штаммов древней сублинии генотипа Beijing позволил оценить филогенетическое положение данных кластеров и определить их мутации лекарственной устойчивости. В исследование было включено 184 генома М. tuberculosis ранней древней сублинии генотипа Beijing, характеризуемой делецией RD207 и аллелем дикого типа кодона mutT4 48 (Фиг. 1). В результате проведенного филогенетического анализа геномных данных российские изоляты с профилем по 24 локусам MIRU-VNTR Beijing 1071-32 (согласно номенклатуре ресурса MIRU-VNTRplus.org) также сформировали отдельный монофилетический кластер на глобальном древе. При этом изоляты были от больных как из Азиатской, так и Европейской России, все были МЛУ и почти все были пред-ШЛУ или ШЛУ. Все изоляты имели характерный профиль мутаций множественной лекарственной устойчивости (KatG Ser315Thr, KatG Ile335Val, RpoB Ser450Leu, RpoC Asp485Asn, EmbB Gln497Arg, and RpsL Lys43 Arg), который дополнен в большинстве изолятов различными мутациями в генах рпсА, gyrA, rrs, приводящими к генотипу пред-ШЛУ/ШЛУ (Mokrousov et al., 2020).
Циркуляция этих резистентных изолятов с одинаковой комбинацией мутаций устойчивости в отдаленных районах России подчеркивает важность их тщательного мониторинга ввиду потенциально более широкого распространения таких высокорезистентных штаммов. Таким образом, разработка быстрого и простого способа детекции эпидемиологически и клинически значимого варианта М. tuberculosis - Beijing 1071-32 - является актуальной задачей программы контроля туберкулеза в России и мире.
Известно определение штаммов Beijing 1071-32-кластера на основании анализа 24 локусов VNTR с последующим сравнением с ресурсом www.MIRU-VNTRplus.org (Mokrousov et al., 2019). Этот подход ограничен трудоемкостью выполнения анализа, требующего проведения 24 реакций ПЦР с последующим гель-электрофорезом или, при использовании капиллярного секвенатора, ограничен стоимостью оборудования и также достаточно трудоемкий, как и любой подход на основании мультилокусного VNTR-типирования.
Проведенный нами биоинформационный и филогенетический анализ данных полногеномного секвенирования 184 геномов М. tuberculosis ранней древней сублинии генотипа Beijing, полученных нами или извлеченных из международных баз данных (National Center for Biotechnology Information и European Nucleotide Archive) позволил выявить однонуклеотидный синонимичный полиморфизм (SNP) в гене Rv0144 (позиция в гене 222С>Т, кодон 74 GCC>GCT Ala-Ala, что соответствует позиции 170505 С>Т в полном геноме референтного штамма H37Rv NC_000962.3) характерный и уникальный для филогенетической ветви, содержащей только штаммы Beijing 1071-32-кластера. Эта мутация была выявлена только в геномах изолятов Beijing 1071-32 и для ее детекции нами были сконструированы праймеры и флуоресцентно меченые зонды и разработан способ детекции с использованием полимеразной цепной реакции в формате реального времени (Фиг. 2).
Задачей предлагаемого изобретения является разработка способа быстрой и надежной детекции микобактерий туберкулеза резистентного российского генотипа Mycobacterium tuberculosis Beijing 1071-32.
Поставленная задача достигается выявлением специфической мутации в гене Rv0144 в позиции 222С>Т с помощью ПЦР в формате реального времени для дискриминации дикого и мутантного аллелей с использованием олигонуклеотидных праймеров 170505F 5'-CCAACGGTAGGTACCAAGC и 170505R 5'-GCTTCCGAGTCTCATCTGCT и флуоресцентно-меченых зондов на мутантный аллель Τ 170505Tmut 5'-[FAM]GTTCAATGTCGCTCACGGC[T-LNA]G[BHQ1] и дикий аллель С 170505Cwt 5'-[R6G]GTTCAATGTCGCTCACGGC[C-LNA]G[BHQ1], с оценкой результатов ПЦР между 20-35 циклами путем регистрации сигнала флуоресценции по FAM-каналу с длиной волны 510 нм судят о принадлежности штамма к генотипу М. tuberculosis Beijing 1071-32-кластер, а при регистрации экспоненциального роста сигнала флуоресценции по НЕХ-каналу с длиной волны 555 нм судят о принадлежности штамма М. tuberculosis к любому другому генотипу.
Преимущества предлагаемого способа: 1. быстрота (меньше одного дня от момента выделения ДНК); 2. простая и однозначная интерпретация результатов; 3. возможность быстрого анализа больших коллекций штаммов М. tuberculosis для оценки их принадлежности к кластеру Beijing 1071-32 с диагностической целью и при популяционных исследованиях, 4. Возможность выявления контаминации и микс-инфекции.
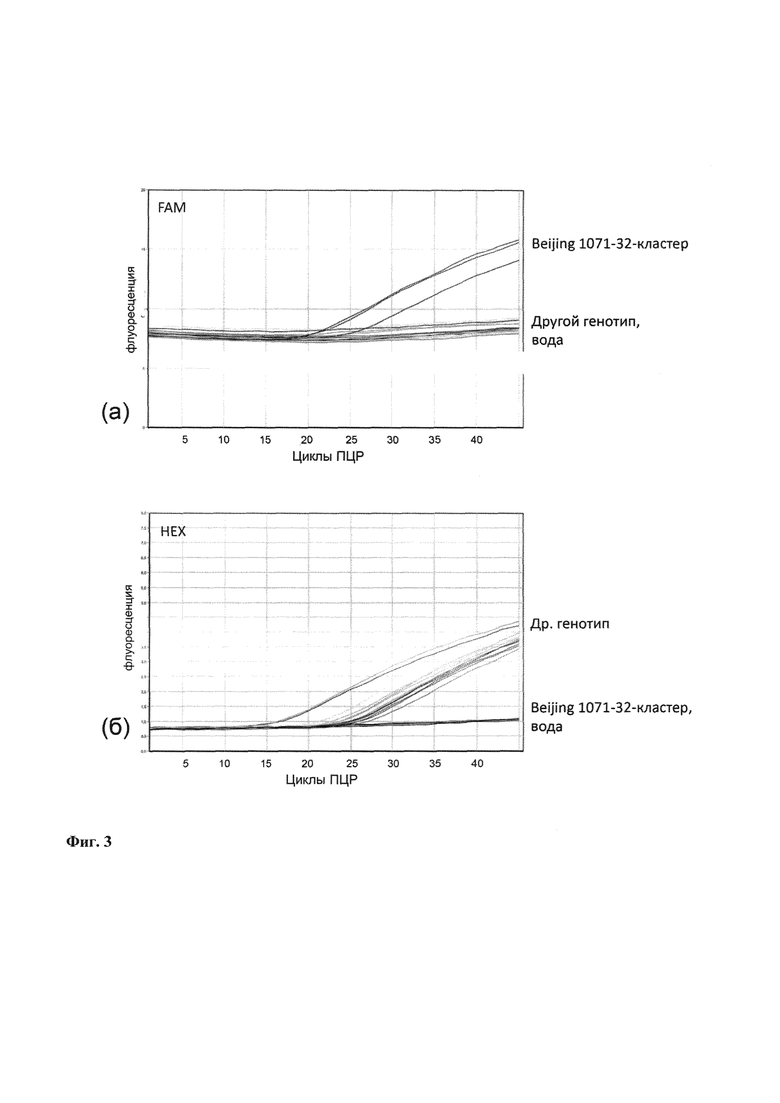
Изобретение поясняется чертежами, где Фиг. 1. Схема эволюции генотипа Beijing Mycobacterium tuberculosis. Фиг. 2 - Схема ПЦР (не в масштабе), где тонкие стрелки -позиции праймеров и зондов в геноме референс-штамма H37Rv; а широкая стрелка обозначает позицию нуклеотидной замены С>Т в гене Rv0144 в позиция 222 (кодон 74). Wt - алелль дикого типа, MUT - мутантный аллель, FAM и HEX - флуорофоры. Фиг. 3 - накопление сигнала флуоресценции: (а) по специфическому (FAM) каналу детекции, на М. tuberculosis Beijing 1071-32-кластер и (б) контрольному (HEX) каналу детекции, на штамм другого генотипа М. tuberculosis. Вода служит отрицательным контрольным образцом.
Способ осуществляется следующим образом. Выделение ДНК из культуры М. tuberculosis, выращенной на среде Левенштейна-Йенсена, проводят по van Embden et al. [1993]: суспендируют 1 стандартную бактериологическую петлю культуры в 400 мкл буфера ТЕ x1 и инкубируют 20 мин при 85°С. Дальнейшую обработку проводят с использованием лизоцима, протеиназы К, додецилсульфата натрия и цетилтриметиламмонийбромида. Полученный клеточный лизат обрабатывают смесью фенол-хлороформ-изоамиловый спирт (25:24:1), центрифугируют, осаждают изопропанолом, промывают 70% этанолом, осадок высушивают и растворяют в 30-50 мкл ТЕ x1.
Были использованы следующие сконструированные нами праймеры и зонды для ПЦР в реальном времени (Фиг. 2) для проведения реакции ПЦР и детекции флуоресценции по двум каналам в одной пробирке одновременно: 170505F 5'-CCAACGGTAGGTACCAAGC, 170505R 5'-GCTTCCGAGTCTCATCTGCT, 170505Tmut 5'-[FAM]GTTCAATGTCGCTCACGGC[T-LNA]G[BHQ1] и 170505Cwt 5'-[R6G]GTTCAATGTCGCTCACGGC[C-LNA]G[BHQ1]. Для детекции штамма генотипа Beijing 1071-32-кластер служил зонд 170505Tmut, детекцию сигнала проводили по FAM-каналу детекции (длина волны 510 нм) (Фиг. 3а). Для детекции штамма любого другого генотипа М. tuberculosis служил зонд WT; детекцию сигнала проводили по НЕХ-каналу (длина волны 555 нм) (Фиг. 3б).
Очищенная ДНК (0.1-0.5 мкл) добавляется к смеси ПЦР (конечный объем 30 мкл) содержащей 1,5 mM MgCl2, 1 U Taq ДНК полимеразы для ПЦР в реальном времени в режиме «горячего старта», 200 μΜ каждого из дНТФ, праймеры и зонды (по 5 пмоль каждый). ПЦР проводили в термоциклере RotorGene6000 (Corbette Research) в следующих условиях: 95°С, 5 мин; далее 45 циклов 95°С, 30 с; 60°С, 10 с, 72°С, 20 с. Считывание сигнала флуоресценции - при 60°С по каналам 510 и 555 нм.
Оценка результатов.
Полученные данные - кривые накопления флуоресцентного сигнала между 20-35 циклом по двум каналам - анализируют с помощью программного обеспечения прибора, используемого для проведения ПЦР в формате реального времени. По каналу FAM (длина волны 510 нм) - регистрируется накопление продукта амплификации фрагмента ДНК, специфического для генотипа М. tuberculosis Beijing 1071-32-кластер (Фиг. 3а), по каналу HEX (длина волны 555 нм) - фрагмента ДНК, специфического для любого другого генотипа М. tuberculosis (Фиг. 3б).
При отсутствии сигнала флуоресценции по обоим каналам (510 и 555 нм) делается вывод о недостаточном количестве и/или качестве ДНК или ингибировании реакции ПЦР и невозможности какого-либо вывода о наличии или отсутствии ДНК штамма Beijing 1071-32-кластера в образце.
Обязательная амплификация одного или другого продукта ПЦР является контролем качества: при отсутствии сигнала судят о деградированной ДНК или ингибировании реакции, при наличии сигнала по обоим каналам делают вывод о микс-образце (контаминация на каком-либо из этапов исследования или выделение двух штаммов от одного больного).
Способ был апробирован на коллекциях ДНК клинических изолятов М. tuberculosis из коллекции ДНК лаборатории молекулярной эпидемиологии и эволюционной генетики ФБУН НИИЭМ им. Пастера, представляющих страны с различными популяциями возбудителя и различной долей штаммов Beijing и Beijing 1071-32-кластер, что показано на примерах ниже. Все изоляты были ранее генотипированы методом мультилокусного VNTR-анализа, позволяющим точно определить принадлежность штамма к VNTR-кластеру.
Пример 1. Анализ ДНК изолятов М. tuberculosis выделенных в областях Северо-Западного федерального округа России, региона с низкой долей древней сублинии генотипа Beijing в популяции М. tuberculosis (5% в исследовании 2002 года, Mokrousov et al., 2002).
Всего было проверено 24 образца ДНК изолятов М. tuberculosis генотипа Beijing, выделенных от больных туберкулезным спондилитом или туберкулезом легких, находившихся на лечении в Санкт-Петербургском НИИ фтизиопульмонологии и областных противотуберкулезных диспансерах Вологды, Пскова, Коми, Карелии и Калининграда в 2009-2019 годах. Все образцы ДНК М. tuberculosis были охарактеризованы методом 24-MIRU-VNTR типирования (Supply et al., 2006) с последующим сравнением с базой данных MIRU-VNTRplus.org. Далее ДНК всех штаммов была протестирована предлагаемым способом ПЦР в формате реального времени для выявления штаммов Beijing 1071-32-кластера. В результате проведения ПЦР было установлено, что 17 изолятов относились к Beijing 1071-32-кластеру, что полностью соответствовало результатам 24-MIRU-VNTR анализа.
Пример 2. Анализ ДНК изолятов М. tuberculosis выделенных в ряде областей Сибири, в том числе в Омской области, для которой характерна достаточно заметная циркуляция кластера Beijing 1071-32 (до 10% от всех штаммов Beijing [Mokrousov et al., 2019]).
Изучено 124 изолята Μ. tuberculosis разных субтипов генотипа Beijing от больных туберкулезом легких из Якутии, Бурятии и Омской области. Все образцы ДНК М. tuberculosis были охарактеризованы методом сполиготипирования (Kamerbeek et al., 1997) и 24-MIRU-VNTR типирования (Supply et al., 2006) с последующим сравнением с базой данных MIRU-VNTRplus.org. Далее ДНК всех штаммов была протестирована предлагаемым способом ПЦР в формате реального времени для выявления штаммов Beijing 1071-32-кластера. В результате проведения ПЦР было установлено, что 38 изолятов относились к Beijing 1071-32-кластеру, что полностью соответствовало результатам 24-MIRU-VNTR анализа. Все результаты были однозначные и микс-образцов выявлено не было.
Пример 3. Анализ ДНК изолятов М. tuberculosis из Китая, Вьетнама и Японии, стран с достаточно высокой долей штаммов древних сублиний генотипа Beijing (Shamputa et al., 2010, Wada et al., 2009; Yin et al., 2016).
Изучено 123 изолятов Μ. tuberculosis генотипа Beijing от больных туберкулезом легких из северного Китая (Пекин и соседние провинции, 23 изолята), Вьетнама (Ханой и Хошимин, 35 изолятов) и Японии (Окинава, 65 изолятов), ранее охарактеризованных нами методами сполиготипирования и MIRU-VNTR типирования по 24 локусам.
Все образцы ДНК М. tuberculosis были охарактеризованы методом сполиготипирования (Kamerbeek et al., 1997) и 24-MIRU-VNTR типирования (Supply et al., 2006) с последующим сравнением с базой данных MIRU-VNTRplus.org. Далее ДНК всех штаммов была протестирована предлагаемым способом ПЦР в формате реального времени для выявления штаммов Beijing 1071-32-кластера. В результате проведения ПЦР, изолятов Beijing 1071-32-кластера выявлено не было, что полностью соответствовало результатам 24-MIRU-VNTR анализа.
СПИСОК ЛИТЕРАТУРЫ
Gagneux S. Ecology and evolution of Mycobacterium tuberculosis. Nat Rev Microbiol. 2018; 16(4):202-213.
Kamerbeek J, Schouls L, Kolk A, van Agterveld M, van Soolingen D, Kuijper S, et al.
Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol. 1997;35:907-14.
Kremer K, Glynn JR, Lillebaek T, Niemann S, Kurepina NE, Kreiswirth BN, et al. Definition of the Beijing/W lineage of Mycobacterium tuberculosis on the basis of genetic markers. J Clin Microbiol. 2004;42:4040-9.
Luo T, Comas I, Luo D, Lu B, Wu J, Wei L, et al. Southern East Asian origin and coexpansion of Mycobacterium tuberculosis Beijing family with Han Chinese. Proc Natl Acad Sci USA. 2015;112:8136-41.
Maeda S, Hang NT, Lien LT, Thuong PH, Hung NV, Hoang NP, et al. Mycobacterium tuberculosis strains spreading in Hanoi, Vietnam: Beijing sublineages, genotypes, drug susceptibility patterns, and host factors. Tuberculosis (Edinb). 2014;94:649-56.
Mokrousov I, Vyazovaya A, Pasechnik O, Gerasimova A, Dymova M, Chernyaeva E, et al.
Early ancient sublineages of Mycobacterium tuberculosis Beijing genotype: unexpected clues from phylogenomics of the pathogen and human history. Clin Microbiol Infect. 2019;25:1039.el-1039.e6.
Mokrousov I, Ly HM, Otten T, Lan NN, Vyshnevskyi B, Hoffher S, et al. Origin and primary ispersal of the Mycobacterium tuberculosis Beijing genotype: clues from human phylogeography. Genome Res. 2005;15:1357-64.
Mokrousov I, Narvskaya O, Otten T, Vyazovaya A, Limeschenko E, Steklova L, et al.
Phylogenetic reconstruction within Mycobacterium tuberculosis Beijing genotype in northwestern Russia. Res. Microbiol. 2002; 153: 629-37
Mokrousov I, Sinkov V, Vyazovaya A, Pasechnik O, Solovieva N, Khromova P, Zhuravlev V, Ogarkov O. Genomic signatures of drug resistance in highly resistant Mycobacterium tuberculosis strains of the early ancient sublineage of Beijing genotype in Russia. Int J Antimicrob Agents. 2020 Aug;56(2): 106036. doi: 10.1016/j.ijantimicag.2020.106036.
Shamputa 1С, Lee J, Allix-Béguec C, Cho EJ, Lee JI, Rajan V, et al. Genetic diversity of Mycobacterium tuberculosis isolates from a tertiary care tuberculosis hospital in South Korea. J Clin Microbiol. 2010;48:387-94.
Supply P, Allix C, Lesjean S, Cardoso-Oelemann M, Rüsch-Gerdes S, Willery E, et al. Proposal for standardization of optimized mycobacterial interspersed repetitive unit-variable-number tandem repeat typing of Mycobacterium tuberculosis. J Clin Microbiol. 2006;44:4498-510.
van Embden J, Cave M, Crawford J, Dale JW, Eisenach KD, Gicquel B, et al. Strain identification on Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J. Clin. Microbiol. 1993;31:406-409.
Wada Т., Iwamoto Т., Maeda S. Genetic diversity of the Mycobacterium tuberculosis Beijing family in East Asia revealed through refined population structure analysis. FEMS Microbiol Lett, 2009, 291: 35-43.
Yin QQ, Liu HC, Jiao WW, Li QJ, Han R, Tian JL, et al. Evolutionary History and Ongoing Transmission of Phylogenetic Sublineages of Mycobacterium tuberculosis Beijing Genotype in China. Sci Rep.2016;6:34353.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ детекции филогенетических сублиний генотипа Beijing Mycobacterium tuberculosis в формате реального времени | 2020 |
|
RU2743365C1 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 14717-15-кластер методом полимеразной цепной реакции в формате реального времени | 2023 |
|
RU2839699C2 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 14717-15-кластер | 2022 |
|
RU2812351C1 |
| Способ детекции изолятов Mycobacterium tuberculosis Beijing 94-32-кластера в формате реального времени | 2017 |
|
RU2689800C1 |
| Способ выявления микобактерий туберкулеза Центрально-Азиатского эпидемического кластера генотипа Beijing | 2019 |
|
RU2735415C1 |
| Способ детекции S-генотипа Mycobacterium tuberculosis | 2024 |
|
RU2832725C1 |
| Способ выявления микобактерий туберкулеза генотипа Beijing В0-кластер в формате реального времени | 2017 |
|
RU2684314C2 |
| СПОСОБ ВЫЯВЛЕНИЯ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗА ГЕНОТИПА ВЕIJING В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2011 |
|
RU2528866C2 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| (4-(3-гидроксифенил)пиперазин-1-ил)(5-нитрофуран-2-ил)метанон, обладающий противотуберкулезной активностью в отношении возбудителя туберкулеза с множественной лекарственной устойчивостью, и способ его получения | 2021 |
|
RU2784399C1 |

Изобретение относится к биотехнологии, медицине, а именно к фтизиатрии, и может быть использовано для лабораторного определения микобактерий туберкулеза, относящихся к генотипу Beijing 1071-32-кластер. Выявляют специфическую для микобактерий туберкулеза генотипа Beijing 1071-32-кластер однонуклеотидную замену C>Т в гене Rv0144 в позиции 222 с помощью ПЦР в формате реального времени с использованием специфических флуоресцентно-меченных зондов для дискриминации дикого и мутантного аллелей Rv0144 222С/Т. По каналу флуоресценции FAM (510 нм) регистрируется накопление продукта амплификации фрагмента ДНК, специфического для Beijing 1071-32-кластер, по каналу HEX (555 нм) - фрагмента ДНК, специфического для штамма любого другого генотипа М. tuberculosis. При наличии в образце ДНК штамма Beijing 1071-32-кластер между 20-35 циклами ПЦР происходит экспоненциальный рост сигнала флуоресценции по каналу детекции FAM и при этом отсутствует сигнал флуоресценции по контрольному каналу детекции HEX. Изобретение позволяет выявить штаммы этого генотипа, что является важной составляющей диагностики туберкулеза и выбора адекватного лечения. 3 ил., 3 пр.
Способ определения микобактерий туберкулеза генотипа Beijing 1071-32-кластер в формате реального времени, отличающийся тем, что выявляют наличие нуклеотидной замены C>Т в гене Rv0144 в позиции 222C>Т с помощью ПЦР в формате реального времени с использованием олигонуклеотидных праймеров 170505F 5'-CCAACGGTAGGTACCAAGC и 170505R 5'-GCTTCCGAGTCTCATCTGCT и флуоресцентно-меченных зондов на мутантный аллель Τ 170505Tmut 5'-[FAM]GTTCAATGTCGCTCACGGC[T-LNA]G[BHQ1] и дикий аллель С 170505Cwt 5'-[R6G]GTTCAATGTCGCTCACGGC[C-LNA]G[BHQ1], с оценкой результатов ПЦР между 20-35 циклами путем регистрации сигнала флуоресценции по FAM-каналу с длиной волны 510 нм судят о принадлежности штамма к генотипу М. tuberculosis Beijing 1071-32-кластер, а при регистрации экспоненциального роста сигнала флуоресценции по НЕХ-каналу с длиной волны 555 нм судят о принадлежности штамма к любому другому генотипу М. tuberculosis.
| ШУР К.В | |||
| и др | |||
| Генотипирование клинических изолятов Mycobacterium tuberculosis, выделенных в Московском регионе, методом MIRU-VNTR | |||
| Вестник РГМУ, 1, 2017, с.48-51, найдено в интернет 28.09.2021, адрес сайта: |
Авторы
Даты
2022-03-23—Публикация
2021-03-31—Подача