Область техники, к которой относится изобретение
Изобретение относится к медицине, фтизиатрии и медицинской микробиологии и может быть использовано для идентификации клинически значимых штаммов Mycobacterium tuberculosis, в том числе с множественной и широкой лекарственной устойчивостью, а также с измененной вирулентностью при работе с пациентами.
Уровень техники
Начиная с 90-х годов прошлого века, в мире наблюдается резкий рост заболеваемости туберкулезом. По данным ВОЗ, на 2011 г. отмечено 8.7 млн. новых случаев заболевания. В структуре смертности от инфекционных и паразитарных заболеваний в России доля умерших от туберкулеза составляет 70%.
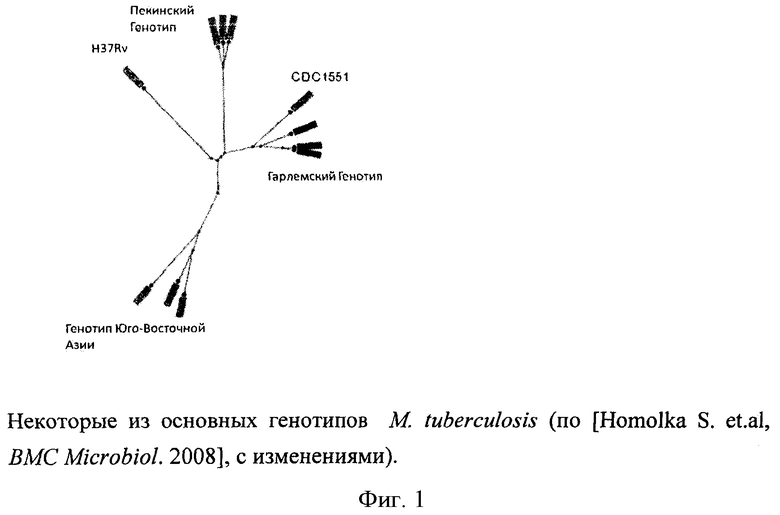
Согласно современной классификации, возбудитель туберкулеза М.tuberculosis подразделяется на несколько генотипов, основными из которых являются следующие (Рис.1):
Генотип Beijing. К данному генотипу относятся штаммы кластеров R220 (типичный представитель М.tuberculosis R1207) и R86 (М.tuberculosis X122), происходящие из Западной Африки, штаммы CCDC5079; CCDC5180 и штаммы широко распространившегося по всему миру кластера W. Являются наиболее часто встречающимся генотипом в мире [Ioerger Т.R. et al. The non-clonality of drug resistance in Beijing genotype isolates of Mycobacterium tuberculosis from the Western Cape of South Africa. BMC Genomics. 2010. 11:670].
Генотип EAI. Данная группа штаммов широко распространена в Юго-Восточной Азии, островах Тихого Океана, в США (Сан-Франциско). [Brudey K. Mycobacterium tuberculosis complex genetic diversity: mining the fourth international spoligotyping database (SpolDB4) for classification, population genetics and epidemiology. BMC Microbiol. 2006. 6(6):23].
Генотип Ural. Широко распространенная на юге и юго-востоке Российской Федерации группа [Mokrousov I. The quiet and controversial: Ural family of Mycobacterium tuberculosis. Infect Genet Evol. 2012 12(4):619-29]. Штаммы, относящиеся к данному генотипу, были обнаружены в Абхазии, Грузии, Индии и Иране. [Ilina E.N. et al. Comparative Genomic Analysis of Mycobacterium tuberculosis Drug Resistant Strains from Russia. PLoS One. 2013. 8(2): e56577].
Генотип LAM. Представляет собой разнородную группу, распространенную повсеместно в мире. Является преобладающим генотипом на территории Южной Америки [Bazira J. et al., Genetic diversity of Mycobacterium tuberculosis in Mbarara, South Western Uganda. Afr Health Sci. 2010. (4):306-3 II]. В России распространен кластер 9 генотипа LAM (LAM9).
Генотип F15/LAM4/KZN. Данный генотип принадлежит к генотипу LAM, и при сполиготипировании определяется как LAM4. Но при помощи RFLP-типирования он выделяется достаточно четко в отдельную группу. К данному генотипу относится группа высоковирулентных штаммов с множественной и широкой лекарственной устойчивостью распространенных на Юге и Юго-Востоке Африки [Loerger T.R. et al. Genome analysis of multi- and extensively-drug resistant tuberculosis from KwaZulu-Natal, South Africa. PLoS ONE. 2009. (4)11].
Генотип SMI-049. К генотипу SMI-049 относят эндемичные для скандинавских стран, устойчивые к изониазиду штаммы ВТВ 05-559, S96-129 и ВТВ05-552 [Sandegren L. et al. Genomic stability over 9 years of an isoniazid resistant Mycobacterium tuberculosis outbreak strain in Sweden. PLoS One. 2011. 6(1): el6647]. Отличаются высоким генетическим сходством и стабильностью.
Генотип Haarlem. Штаммы данной группы распространены в Африке, но в последние годы все чаще встречаются в Аргентине, Бразилии, странах Карибского бассейна и США.
Генотип Т. Данный кластер, наряду с генотипами LAM и Haarlem, принадлежит к т.н. Евро-Американской линии. Чрезвычайно разнороден по своему составу. В данную группу входит референсный штамм H37Rv [Gagneux S., Deriemer K., Van Т. et al. Variable host-pathogen compatibility in Mycobacterium tuberculosis. Proc Natl Acad Sci USA. 2006. 103:2869-2873].
Ряду штаммов, характерных для центральной и южной Африки, в частности генотипу F15/LAM4/KZN, присуща большая вирулентность и, по сравнению с европейскими штаммами, склонность к образованию MDR-форм [Chihota V.N. et al. Population structure of multi- and extensively drug-resistant Mycobacterium tuberculosis strains in South Africa. J. Clin. Microbio. 2012. 50(3):995-1002]. Подобное также характерно для представителей пекинского (Beiling) генотипа, широко распространенного не только в Юго-Восточной Азии, но в последние годы повсеместно в мире [Chakraborty P. et al. Drug resistant clinical isolates of Mycobacterium tuberculosis from different genotypes exhibit differential host responses in THP-1 cells. PLoS One. 2013. 8(5): e62966] [Lasunskaia E. et al. Emerging multidrug resistant Mycobacterium tuberculosis strains of the Beijing genotype circulating in Russia express a pattern of biological properties associated with enhanced virulence. Microbes and Infection. 2010. 12:467-475] [Parwati I. et al. Possible underlying mechanisms for successful emergence of the Mycobacterium tuberculosis Beijing genotype strains. Lancet Infect Dis. 2010. 10:103-111] [Devaux I. et al. Clusters of multidrug-resistant Mycobacterium tuberculosis cases, Europe. Emerg Infect Dis. 2009. 15(7):1052-1060] [Von Groll A. et al. Fitness of Mycobacterium tuberculosis strains of the W-Beijing and Non- W-Beijing genotype. PLoS One 2010. 5: e10191].
В связи с этим для разработки и назначения правильной схемы лечения большое значение имеет генотипирование микобактерий. В настоящее время для идентификации микобактерий применяются следующие методы:
1. Сполиготипирование - основной стандартный метод генотипирования микобактерий. В основе метода лежит полиморфизм хромосомного DR-локуса (от англ. direct repeat - прямой повтор) [Kamerbeek J., et al. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol. 1997. 35:907-14]. Данный метод требует проведения полимеразной цепной реакции (ПЦР) и последующей ДНК-гибридизации с иммобилизованными на мембране зондами в специальном приборе (миниблоттере) и выявлением сигналов хемилюминесценции на светочувствительной пленке. Метод сполиготипирования позволяет идентифицировать несколько наиболее значимых в эпидемиологическом отношении генотипов: Beijing, Haarlem, LAM, EAI и т.д. В последние годы также выделяют генотип Ural [Ilina E.N. et al. Comparative genomic analysis of Mycobacterium tuberculosis drug resistant strains from Russia. PLoS One. 2013. 8(2): e56577].
Метод имеет ряд преимуществ: высокая специфичность, экономичность (в т.ч. возможность использовать гибридизационные мембраны до 20 раз) и в настоящее время считается классическим методом штаммовой дифференцировки. Создана международная база данных по сполиготипам микобактерий туберкулезного комплекса SpolDB4 (www.pasteur.fr/ip/easysite/pasteur/fr).
Недостатком данного метода является его трудоемкость, потребность в специальном оборудовании и программном обеспечении, что делает его неприменимым для использования в практических бактериологических лабораториях.
2. Метод RFLP-типирования. Данный метод основан на анализе полиморфизма длин рестрикционных фрагментов, получающихся при расщеплении геномной ДНК по специфическим сайтам эндонуклеазами рестрикции [van Embden, J.D.A. et al. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J. Clin. Microbiol. 1993. 31:(406-409)]. Размеры и взаимное расположение рестрикционных фрагментов ДНК после их электрофоретического разделения определяются путем гибридизации по Саузерну. RFLP - метод имеет несколько модификаций, но наиболее распространенным маркером является последовательность транспозона IS6110. Метод обладает высокой разрешающей способностью.
Недостатком данного метода является значительная подвижность и способность IS6110-элементов к геномным транспозициям, а также потребность в значительном количестве ДНК, для выделения которой необходимо большое количество биомассы М.tuberculosis.
3. Методы, основанные на IS6110-амплификации:
- Гнездовая (вложенная) и полугнездовая IS6110-ПЦР (Hemi-nested PCR).
Данный метод позволяет повысить чувствительность реакции амплификации за счет последовательного применения двух пар праймеров - внешней и внутренней [Miyazaki Y. et al. Nested polymerase chain reaction for detection of Mycobacterium tuberculosis in clinical samples. Clin. Microbiol. 1993. 31(8):2228-2232].
- Двойная ПЦР повторяющихся элементов (double - repetitive - element PCR).
Метод основан на амплификации и анализе последовательности участков между двумя повторяющимися участками ДНК, которыми чаще всего выступают IS6110-элементы, а также PGRS-последовательности [Friedman C.R et al. Double repetitive element PCR method for subtyping Mycobacterium tuberculosis clinical isolates. J. Clin. Microbiol. 1995. 33:1064-1069].
- Инвертированная IS6110-ПЦР.
Данный метод используется в случае, если в необходимой для анализа последовательности изестен небольшой участок, и особенно полезен, если требуется определить последовательность после инсерции [Plikaytis В.В. et al. Rapid, amplification-based fingerprinting of Mycobacterium tuberculosis. J. Gen. Microbiol. 1993. 139:1537-1542].
Недостатком этих методов является значительная подвижность и способность IS6110-элементов к геномным транспозициям, приводящая к изменению характерного для штамма рисунка генотипирования спустя 3-5 лет. Кроме того, данные методы типирования не всегда оказываются пригодным для идентификации штаммов микобактерий туберкулезного комплекса с малым (не более 5) числом копий IS6110 или с полным их отсутствием.
4. Метод количества тандемных повторов (variable number of tandem repeats, VNTR) [Iwamoto T. et al. Hypervariable loci that enhance the discriminatory ability of newly proposed 15-loci and 24-loci variable-number tandem repeat typing method on Mycobacterium tuberculosis strains predominated by the Beijing family. FEMS Microbiol. Lett. 2007. 272:282-283]. В качестве маркеров используются геномные локусы М.tuberculosis, содержащие консервативные тандемные повторы (exact tandem repeats; ETR). Их количество в каждом локусе варьирует у различных штаммов. Методика обладает высокой дифференцирующей способностью. Одним из усовершенствований данного метода является т.н. MIRU-VNTR. В основе метода лежит амплификация локусов, содержащих распределенные по геному микобактериальные повторяющиеся элементы (mycobacterial interspersed repetitive units, MIRU).
Недостатками всех указанных методов являются: сложность и/или неоднозначная интерпретация полученных результатов.
Раскрытие изобретения
Задачей данного изобретения является разработка метода универсальной и быстрой молекулярно-генетической идентификации основных генотипов штаммов М.tuberculosis с использованием генетического материала изолята.
В заявленном изобретении предложен метод идентификации, основанный на однонуклеотидных полиморфизмах генов систем токсин-антитоксин II типа у М.tuberculosis.
Системы токсин-антитоксин (ТА) II типа широко распространены у М.tuberculosis и представляют собой модуль из двух генов, располагающихся друг за другом (иногда перекрываясь) и образуя оперон [Yamaguchi Y. et al. Toxin-antitoxin systems in bacteria and archaea. Annu Rev Genet. 2011. 45:61-79] [Прозоров А.А., Даниленко В.Н. Системы "токсин-антитоксин" у бактерий: инструмент апоптоза или модуляторы метаболизма? Микробиология. 2010. Т.79. №2. С.147-159] [Sala A. et al. Toxin-antitoxin loci in Mycobacterium tuberculosis. In: Gerdes K., editor. Prokaryotic toxin-antitoxins. Berlin: Springer. 2013. P.295-314]. В настоящее время у М.tuberculosis обнаружено более 75 пар систем-антитоксин.
Основные функции генов систем токсин-антитоксин у М.tuberculosis включают в себя контроль роста клетки в неблагоприятных условиях, переход клеток в покоящееся состояние, апоптоз и др. [Ramage H.R. et al. Comprehensive functional analysis of Mycobacterium tuberculosis toxin-antitoxin systems: implications for pathogenesis, stress responses, and evolution. PLoS Genet. 2009. 5: e1000767].
Предлагаемый нами метод типирования имеет ряд преимуществ по сравнению с имеющимися аналогами - использование функциональных регуляторных генов, важных для толерантности, персистенции и вирулентности М.tuberculosis, а также универсальность (возможность использования при мультилокусном секвенировании, ПЦР-РВ, ПЦР), простота в исполнении, надежность, небольшое число генов, быстрота.
1. Биоинформатический анализ наличия генов систем токсин-антитоксин II типа у штаммов M.tuberculosis
Авторами настоящего изобретения был проведен анализ in silico 75 пар генов систем токсин-антитоксин в 22 «complete»- и 138 «draft»-геномах секвенированных (по данным на ноябрь 2013 г.) штаммов М.tuberculosis с использованием баз данных: National Center for Biotechnology Information (NCBI) (http://www.ncbi.nlm.nih.gov/), UniProt (http://www.uniprot.org/), Tuberculist (http://tuberculist.epfl.ch/index.html), Pathosystems Resours Integration Center (http://patricbrc.org/portal/portal/patric/Taxon?cType=taxon&cId=1763), TADB (http://bioinfo-mml.sjtu.edu.cn/TADB/). Сравнительный анализ последовательностей обнаруженных генов проводили с использованием программ BioEdit (http://www.mbio.ncsu.edu/BioEdit/bioedit), LALIGN (http://www.ch.embnet.org/software/ LALIGN_form.html), Blast (http://blast.ncbi.nlm.nih.gov/) и CLUSTALW (www.ch.embnet.org/software/ClustalW.html).
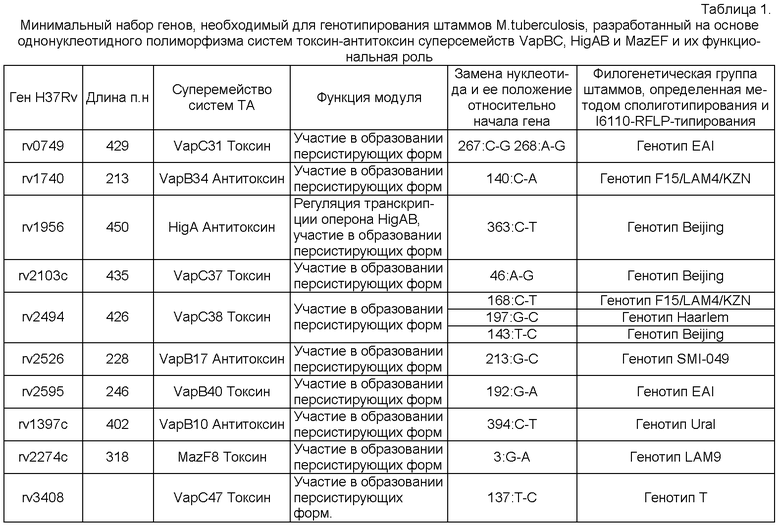
Было установлено, что геномы всех штаммов M.tuberculosis содержат 75 пар ТА-модулей, принадлежащих к 5 суперсемействам - VapBC, RelBE, MazEF, ParDE, HigAB, a также новые системы, обозначенные как novel, которые к настоящему моменту не могут быть однозначно классифицированы. К суперсемейству VapBC из них принадлежат 48, к RelBE - 4, к MazEF - 9, к ParDE - 2, к HigAB - 1, к novel - 11. При анализе были найдено значительное число snp-полиморфизмов, представляющих собой миссенс- и сеймсенс-замены, а также делеции и вставки отдельных нуклеотидов, приводящие к сдвигу рамки считывания. Из обнаруженных snp-полиморфизмов 29 относились к миссенс-мутациям, 19 - к сеймсенс-мутациям, 3 - к мутациям сдвига рамки считывания. Во избежание возможных неточностей, связанных с ошибками секвенирования, учитывались snp-полиморфизмы, повторяющиеся у нескольких штаммов. Для дальнейшего анализа были первоначально отобраны 8 генов, степень полиморфизма которых была достаточно высока и удовлетворяла целям настоящего изобретения (Таблица 1).
2. Биоинформатический анализ snp-полиморфизмов выбранных генов у штаммов M.tuberculosis, относящихся к разным генотипам
2.1. Анализ полиморфизмов в геномах штаммов M.tuberculosis из зарубежных коллекций
Был осуществлен анализ in silico нуклеотидных последовательностей выбранных генов с использованием программы Blast. Для анализа были использованы доступные в базе данных геномы M.tuberculosis с известной генотипической принадлежностью.
При анализе snp-полиморфизмов выбранных генов у всех штаммов, относящихся к генотипу Beijing, были выявлены следующие нуклеотидные замены:
- для гена rv2103c (токсин VapC37) характерна замена A46→G46;
- для гена rv1956 (токсин HigA) характерна замена C363→T363;
- для гена rv2494 (токсин VapC38) характерна замена T143→C143.
Анализ полиморфизма выбранных генов у штаммов генотипа EAI показал наличие характерных для данного генотипа замен:
- в гене rv0749 (токсин Vap31) обнаружены две замены C267→G267 и A268→G268;
- в гене rv2595 (антитоксин VapB40) обнаружена замена G192→A192.
Анализ полиморфизма выбранных генов у штаммов генотипа F15/LAM4/KZN показал наличие двух характерных для данного генотипа замен:
- в гене rv2494 (токсин VapC38) обнаружена замена C168→T168;
- в гене rv1740 (антитоксин VapB34)обнаружена замена C140→A140.
Анализ полиморфизма выбранных генов у штаммов генотипа SMI-049 показал наличие характерной замены G213→C213 в гене rv2526 (антитоксин VapB17).
Анализ полиморфизма выбранных генов у штаммов генотипа Haarlem показал наличие замены G197→С197 в гене rv2494 (токсин VapC38).
Анализ полиморфизма выбранных генов у штаммов генотипа Т выявил наличие полиморфизма T137→C137 в гене rv3408 (токсин VapC47).
Анализ штаммов генотипа LAM показал очень низкую степень полиморфизма не только по выбранным генам, но также по всем 150 генам систем ТА. Выделить данный генотип на основании полиморфизма выбранных десяти генов можно путем детекции полиморфизма по гену токсина VapC47 (rv3408): наличие в гене в 137-м положении тимина и отсутствии других полиморфизмов, маркирующих остальные генотипы.
Полученные результаты представлены в таблице 1.
2.2. Анализ полиморфизмов в геномах штаммов M.tuberculosis из российских коллекций
Проведен анализ in silico выбранных генов систем токсин-антитоксин в геномах 38 эпидемиологически значимых штаммов, выделенных у пациентов на территории Российской Федерации [http://www.ncbi.nlm.nih.gov/sra/?term=SRA061654]. Принадлежность штаммов к различным генотипам (Уральский, пекинский и LAM9) была установлена перед депонированием методом сполиготипирования. К уральскому генотипу принадлежат семь штаммов: MOS9; SP24; SP25; SP28; SP31; SP32; SP33.
В ходе работы было установлено, что штаммы уральского генотипа не имеют полиморфизмов по анализируемым генам. Был проведен анализ ряда других генов систем ТА суперсемейства VapBC. Результатом работы стало обнаружение snp-полиморфизма в гене токсина VapC10 (rv1397c) замены C394→T394. Данная замена присутствует у всех представителей уральского генотипа и отсутствует у штаммов, принадлежащих к другим филогенетическим группам.
К генотипу LAM9 относятся 9 штаммов: MOS2, MOS7, SP34, SP35, SP36, SP37, SP38. Полиморфизмов в системах токсин-антитоксин суперсемейства VapBC не было выявлено. При анализе генов других систем токсин-антитоксин, обнаружена замена G3→A3 в гене токсина MazF8 (rv2274c). Замена присутствует у всех штаммов данного генотипа.
3. Разработка олигонуклеотидов для идентификации основных генотипов M.tuberculosis
Основным требованием к данному набору праймеров является их универсальность и высокая специфичность. Для выбора праймеров учтены требования к длине фрагмента ДНК в случае применения различных методик секвенирования.
Для мультилокусного секвенирования, в соответствии с характеристиками системы (http://roche-applied-science.ru/assets/files/GS_Junior.pdf), максимальная длина анализируемого фрагмента не должна превышать 500 п.н., средняя длина - 400 п.н. При использовании метода PCR-real time длина амплифицируемого фрагмента не должна превышать 200 п.н. Исходя из этих требований, подбор праймеров осуществлялся в интервале 100-150 нуклеотидов от точки замены. Для удобства применения при мультилокусном секвенировании все праймеры должны иметь сходную температуру отжига (64°).
На основании сравнительного анализа последовательностей выбранных генов разработан набор из 10 пар олигонуклеотидов для идентификации основных генотипов М.tuberculosis: Rv0749N-Rv0749C, Rv2103cN-Rv2103cC, Rv1740N-Rv1740C, Rv1956N-Rv1956C, Rv2526N-Rv2526C, Rv2529N-Rv2529C, Rv2494N-Rv2494C, Rv1397cN-Rv1397cC, Rv2274cN-Rv2274cC, Rv3408N-Rv3408C представленный в таблице 2.
На последовательностях 1-10 (см. перечень последовательностей) представлен сравнительный анализ нуклеотидных последовательностей выбранных генов с локализацией всех олигонуклеотидов для генотипирования. Все выявленные полиморфизмы выделены красным цветом.
Таким образом:
1) Метод генотипирования Mycobacterium tuberculosis по настоящему изобретению основан на однонуклеотидном полиморфизме функциональных регуляторных генов систем токсин-антитоксин суперсемейств VapBC, MazEF и HigAB.
2) Предлагаемый метод предусматривает проведение амплификации с геномной ДНК с использованием предлагаемого набора 10 пар олигонуклеотидов с последующим секвенированием наработанных фрагментов.
3) Предлагаемый метод позволяет точно и быстро проводить генотипирование микобактерий в режиме ПЦР, ПЦР в реальном времени, мультилокусной ПЦР с последующим секвенированием, в том числе в системе MiSeq («Illumina», США) и GS Junior («Roche», Швейцария).
Осуществление изобретения
Культуру микобактерий выращивают в жидкой питательной среде LB при 37°С в аэрируемых условиях в термостатируемом шейкере-инкубаторе Multitron («Infors») при 250 об/мин в течение 18 часов, после чего выделяют геномную ДНК.
Для этого клетки из 20 мл ночной культуры осаждают центрифугированием при 4000g в течение 10 мин и ресуспендируют в 10 мл буфера (10 мМ Трис-HCl, 10 мМ ЭДТА-Na2, рН 8.0). Полученную суспензию центрифугируют в тех же условиях и осадок ресуспендируют в 500 мкл выше указанного буфера, затем суспензию переносят в 2-мл центрифужную пробирку и добавляют 50 мкл хлороформа. Смесь энергично встряхивают с помощью вортекса (5 раз по 10 сек), вносят 100 мкл раствора лизоцима (60 мг/мл) и инкубируют 30 мин при 37°С. Добавляют 6 мкл РНКазы А (10 мг/мл) и инкубируют еще 30 мин при 37°С. Для лизиса клеток к суспензии добавляют 200 мкл 10% SDS и 200 мкл 5М NaCl, осторожно перемешивают и инкубируют 16 ч при 65°С. Полученный лизат клеток остужают до комнатной температуры, добавляют 1 мл смеси фенол/хлороформ (1:1) и перемешивают путем переворачивания пробирки в течение 5 мин до состояния гомогенной эмульсии. Затем смесь центрифугируют при 12000g в течение 15 мин. Водную фазу, содержащую ДНК, отбирают в новую 1.5-мл пробирку и смешивают с 600 мкл изопропанола. Смесь инкубируют 30 мин при комнатной температуре и центрифугируют при 12000g в течение 20 мин. Осадок ДНК трижды промывают порциями по 0.5 мл 75% этанола с центрифугированием по 5 мин при 12000g и растворяют в 100 мкл воды.
При выборе для генотипирования режима ПЦР амплификацию ДНК проводят с использованием набора High Fidelity PCR Enzyme Mix («Fermentas») на приборе «Терцик» («ДНК-технология»). Состав смеси для ПЦР (на 100 мкл): 10 мкл 10×ПЦР буфера (10Х High Fidelity PCR Buffer with 15 mM MgCl2), 10 мкл смеси 2 mM ΣdNTPs (либо 2 мкл 10 mM ΣdNTPs), 5 мкл DMSO, 0.3 мкг геномной ДНК и 1 мкл (1.25 и) фермента High Fidelity PCR Enzyme Mix. Олигонуклеотидные праймеры добавляют в концентрации 20 пмоль на 100 мкл смеси.
Параметры ПЦР реакции: 95°С в течение 5 мин (лизис клеток и денатурация геномной ДНК); затем 35 циклов амплификации - 94°С - 1 мин (денатурация), 64°С в течение 0.5 мин (отжиг олигонуклеотидов), 72°С - 40 сек (достройка (элонгация) цепи); финальная элонгация фрагментов при 72°С - 10 мин, хранение при 4°С.
Результаты амплификации учитывают путем анализа продуктов амплификации исследуемых образцов методом электрофореза в 1% агарозном геле. В пробирки с исследуемыми образцами после завершения амплификации вносят аккуратно под масло 1/5 объема раствора 6Х DNA Loading Dye («Fermentas»), перемешивают и 10 мкл полученного образца вносят в лунки агарозного геля. Электрофорез проводят в камере для горизонтального электрофореза "SE-2" (Компания «Хеликон») с источником питания "Эльф-4" («ДНК-технология») при напряжении 120 вольт в течение 60 мин. Результаты электрофореза учитывают в ультрафиолетовом свете с длинной волны 254 нм на трансиллюминаторе TCP-20 MC («Vilber Lourmat», Франция). В качестве контроля размера полученного фрагмента используют ДНК маркер GeneRuler™ 50+ п.н. («Fermentas»).
Полученные целевые фрагменты выделяют из ПЦР-смеси с использованием набора QIAquick® PCR Purification Kit («Qiagen») или GeneJET™ PCR Purification Kit («Fermentas») и секвенируют для выявления полиморфизмов, соответствующих генотипам штаммов М.tuberculosis.
Предлагаемый метод идентификации можно использовать для генотипирования в режиме ПЦР в реальном времени, мультилокусной ПЦР с последующим секвенированием.
Оценка результатов
Исследуемый штамм относится к генотипу Beijing в случае выявления полиморфизмов A46→G46 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2103cN-Rv2103cC; C363→T363 при использовании олигонуклеотидов Rv1956N-Rv1956C и T143→C143 при использовании олигонуклеотидов Rv2494N-Rv2494C.
Исследуемый штамм относится к генотипу EAI в случае выявления полиморфизмов C267→G267 и A268→G268 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv0749N-Rv0749C и G192→A192 при использовании олигонуклеотидов Rv2595N-Rv2595C.
Исследуемый штамм относится к генотипу Ural в случае выявления полиморфизма C394→T394 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv1397N-Rv1397C.
Исследуемый штамм относится к генотипу LAM9 в случае выявления полиморфизма G3→А3 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2274N-Rv2274C.
Исследуемый штамм относится к генотипу LAM в случае выявления полиморфизма C137→Т137 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv3408N-Rv3408C и при отсутствии других полиморфизмов.
Исследуемый штамм относится к генотипу F15/LAM4/KZN в случае выявления полиморфизмов C168→Т168 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2494N-Rv2494C и C140→A140 при использовании олигонуклеотидов Rv1740N-Rv1740C.
Исследуемый штамм относится к генотипу SMI-049 в случае выявления полиморфизма G213→C213 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2526N-Rv2526C.
Исследуемый штамм относится к генотипу Haarlem в случае выявления полиморфизма G197→C197 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2494N-Rv2494C.
Исследуемый штамм относится к генотипу Т в случае выявления полиморфизмов T137→C137 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv3408N-Rv3408C.
Примеры идентификации по настоящему изобретению
Пример 1.
Наработка фрагментов с геномной ДНК контрольного секвенированного штамма M.tuberculosis H37Rv с использованием сконструированных олигонуклеотидов.
Для проверки правильности конструирования олигонуклеотидов была проведена амплификация с геномной ДНК типового штамма M.tuberculosis H37Rv по методике, описанной выше. Анализ ПЦР продуктов в 1% агарозном геле показал, что в процессе реакции получены 10 ампликонов ожидаемой длины. Секвенирование выделенных фрагментов и анализ in silico показали 100% идентичность с соответствующими участками генома штамма H37Rv.
Таким образом, предлагаемый набор олигонуклеотидов позволяет селективно отбирать фрагменты для последующего выявления полиморфизмов, соответствующих разным генотипам M.tuberculosis.
Пример 2.
Генотипирование изолятов M.tuberculosis из российских коллекций с применением предлагаемого метода идентификации.
Для анализа использованы 18 образцов геномной ДНК M.tuberculosis, полученной из коллекции Центрального научно-исследовательского института эпидемиологии (г. Москва). Образцы выделены из мокроты пациентов, больных туберкулезом.
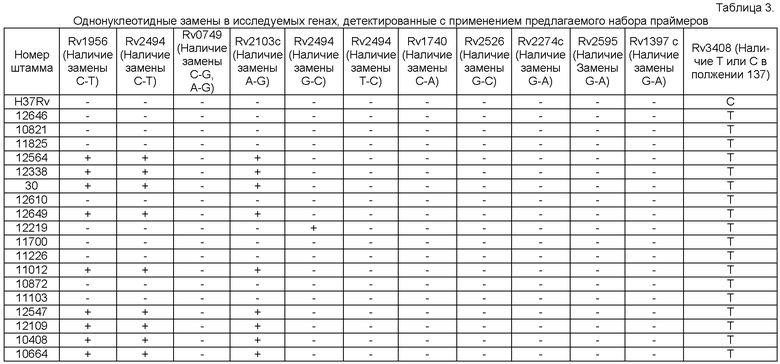
Генотипирование проводили путем амплификации с использованием предложенного набора праймеров. Результаты секвенирования полученных фрагментов показали, что 9 штаммов из исследованного набора принадлежат к пекинскому генотипу, 1 штамм к генотипу Haarlem и 8 штаммов - к генотипу LAM (Таблица 3). Таким образом, результаты совпали с данными генотипирования, полученными методом сполиготипирования. Результаты также совпадают с эпидемиологическими данными по распространению на территории РФ различных генотипов [Mokrousov I. et al. Mycobacterium tuberculosis population in northwestern Russia: an update from Russian-EU/Latvian border region. PLoS One. 2012. 7(7): e41318].
Пример 3.
Генотипирование изолятов M.tuberculosis из российских коллекций с помощью коммерческих наборов для штаммовой идентификации.
Для анализа использованы 18 образцов геномной ДНК, полученной из коллекции Центрального научно-исследовательского института эпидемиологии.
В работе был использованы набор АМПЛИТУБ-Beijing («Синтол», Россия), позволяющий определить принадлежность штамма к пекинскому генотипу. По результатам ПЦР-РТ, проведенной с набором АМПЛИТУБ-Beijing, из 18 исследованных штаммов 9 принадлежат к пекинскому генотипу, что совпадает с генотипированием по предлагаемому методу идентификации.
Пример 4.
Биоинформатический анализ секвенированных геномов российских штаммов M.tuberculosis, представленных в базе данных SRA.
С использованием разработанных праймеров для 10 генов систем ТА с использованием программы BLAST проанализировано 38 секвенированных геномов M.tuberculosis российского происхождения, депонированных в базе данных SRA (http://www.ncbi.nlm.nih.gov/sra), относящихся к 3 генотипам: Beijing, Ural и LAM. Генотипы всех штаммов, установленные с использованием предлагаемых нами генов, полностью идентичны генотипам, заявленным авторами представленных в базе штаммов.
Пример 5.
Биоинформатическое генотипирование M.tuberculosis из базы данных секвенированных геномов, изолированных в Панаме.
С использованием разработанных праймеров для 10 генов систем ТА проведен анализ 67 draft-геномов лекарственно-устойчивых штаммов Mycobacterium tuberculosis, изолированных в Панаме. [Lanzas F. et al. Multidrug-resistant tuberculosis in Panama is driven by clonal expansion of a multidrug-resistant Mycobacterium tuberculosis strain related to the KZN extensively drug-resistant M.tuberculosis strain from South Africa., J Clin Microbiol, 2013. 51(10):3277-85]. Перед депонированием штаммов в базу данных NCBI (http://www.ncbi.nlm.nih.gov/genome/) были определены их сполиготипы.
Штаммы PanR0605 и PanR0606 имели следующие snp-полиморфизмы, на основании которых относятся к генотипу Beijing:
- в гене rv2103c (токсин VapC37) обнаружена замена A46→G46;
- в гене rv1956 (токсин HigA) характерна замена C363→T363;
- в гене rv2494 (токсин VapC38) характерна замена T143→C143.
На основании сполиготипирования данные штаммы также отнесены к линии PGG-1, которой соответствует генотипу Beijing [Rahim Z. et al. Assessment of population structure and major circulating phylogeographical clades of Mycobacterium tuberculosis complex in Bangladesh suggests a high prevalence of a specific subclade of ancient M.tuberculosis genotypes. J Clin Microbiol. 2007. 45(11):3791-3794].
В гене токсина VapC38 (rv2494) у штаммов PanR0907, PanR0902, PanR0801, PanR0206, PanR0315, PanR0301, PanR0305, PanR0316 была обнаружена замена G197→C197, соответствующая генотипу Haarlem.
У штамма PanR0908 присутствует замена C137→T137 в гене rv3408, характерная для генотипа Т.
У штаммов PanR0202, PanR0208 и PanR0309 в гене токсина Vap31 (rv0749) обнаружены две замены C267→G267 и A268→G268, но замены в гене rv2595, характерной для EAI (включая линию филлипин), обнаружено не было. На основании сполиготипирования данные штаммы отнесены к кластеру Т. Можно сделать предположение, что PanR0202, PanR0208 и PanR0309 занимают промежуточное положение между Т и подгруппой филлипин генотипа EAI.
У штаммов PanR1006, PanR1101, PanR0201, PanR0203, PanR0209, PanR0207, PanR0206, PanR0205, PanR0315, PanR0304, PanR0306, PanR0307, PanR0308, PanR0311, PanR0313, PanR0314, PanR0317, PanR0401, PanR0402, PanR0403, PanR0404, PanR0405, PanR0407, PanR0409, PanR0410, PanR0411, PanR0412, PanR0611, PanR0610, PanR0609, PanR0607, PanR0604, PanR0603, PanR0602, PanR0601, PanR0505, PanR0503, PanR0501, PanR1007, PanR0909, PanR0906, PanR0904, PanR0903, PanR0805, PanR0804, PanR0803, PanR0708, PanR0707, PanR0703 и PanR0702 в гене rv3408 был выявлен полиморфизм C137→T137, а другие полиморфизмы отсутствуют, это позволяет отнести их к генотипу LAM.
Таким образом, предлагаемый способ обеспечивает высокую чувствительность генотипирования Mycobacterium tuberculosis.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ОБНАРУЖЕНИЯ ДНК ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА С ОДНОВРЕМЕННЫМ УСТАНОВЛЕНИЕМ ЕГО ГЕНОТИПА И ОПРЕДЕЛЕНИЕМ ГЕНЕТИЧЕСКИХ ДЕТЕРМИНАНТ МНОЖЕСТВЕННОЙ И ШИРОКОЙ ЛЕКАРСТВЕННОЙ УСТОЙЧИВОСТИ, ОЛИГОНУКЛЕОТИДНЫЙ МИКРОЧИП, НАБОР ПРАЙМЕРОВ И НАБОР ОЛИГОНУКЛЕОТИДНЫХ ЗОНДОВ, ИСПОЛЬЗУЕМЫЕ В СПОСОБЕ | 2014 |
|
RU2562866C1 |
| Использование аллельных вариантов генов pks15 и pe_pgrs54 в качестве маркера повышенной вирулентности Mycobacterium tuberculosis в отношении людей с пониженным иммунитетом | 2019 |
|
RU2737622C1 |
| Способ выявления микобактерий туберкулеза Центрально-Азиатского эпидемического кластера генотипа Beijing | 2019 |
|
RU2735415C1 |
| СПОСОБ ДИАГНОСТИКИ ЧУВСТВИТЕЛЬНОСТИ ШТАММОВ Mycobacterium tuberculosis К ФТОРХИНОЛОНАМ ПО ГЕНУ gyr B | 2010 |
|
RU2439162C1 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 14717-15-кластер | 2022 |
|
RU2812351C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Способ детекции филогенетических сублиний генотипа Beijing Mycobacterium tuberculosis в формате реального времени | 2020 |
|
RU2743365C1 |
| Способ детекции изолятов Mycobacterium tuberculosis Beijing 94-32-кластера в формате реального времени | 2017 |
|
RU2689800C1 |
| Способ детекции S-генотипа Mycobacterium tuberculosis | 2024 |
|
RU2832725C1 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 1071-32-кластер в формате реального времени | 2021 |
|
RU2768021C1 |







Изобретение относится к области медицины, фтизиатрии и медицинской микробиологии и касается способа генотипирования штаммов Mycobacterium tuberculosis. Охарактеризованный способ основан на однонуклеотидном полиморфизме генов систем токсин-антитоксин II типа суперсемейств VapBC, HigAB и MazEF и характеризуется тем, что для идентификации проводят амплификацию с геномной ДНК с использованием набора олигонуклеотидных праймеров для 10 генов систем токсин-анатоксин. ПЦР продукты анализируют в агарозном геле, размер полученного фрагмента определяют с помощью ДНК-маркера, выделяют целевые фрагменты из ПЦР-смеси или агарозного геля и секвенируют их с целью обнаружения соответствующих полиморфизмов. Предложенное изобретение позволяет точно и быстро проводить генотипирование микобактерий в режиме ПЦР и может быть использовано для идентификации клинически значимых штаммов M. tuberculosis, в том числе с множественной и широкой лекарственной устойчивостью, а также с измененной вирулентностью при работе с пациентами. 1 ил., 3 табл., 5 пр.
Способ генотипирования штаммов Mycobacterium tuberculosis, отличающийся от известных тем, что основан на однонуклеотидном полиморфизме функциональных регуляторных генов систем токсин-антитоксин II типа суперсемейств VapBC, HigAB и MazEF, и характеризующийся тем, что для идентификации проводят амплификацию с геномной ДНК с использованием набора олигонуклеотидов для 10 генов систем токсин-антитоксин, представленных в таблице 2, причем ПЦР продукты анализируют в агарозном геле, размер полученного фрагмента определяют с помощью ДНК-маркера, затем выделяют целевые фрагменты из ПЦР-смеси или агарозного геля и секвенируют их с целью обнаружения соответствующих полиморфизмов; исследуемый штамм относится к генотипу Beijing в случае выявления полиморфизмов: A46→G46 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2103cN-Rv2103cC, C363→T363 при использовании олигонуклеотидов Rv1956N-Rv1956C, T143→C143 при использовании олигонуклеотидов Rv2494N-Rv2494C; исследуемый штамм относится к генотипу EAI в случае выявления полиморфизмов: C267→G267 и A268→G268 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv0749N-Rv0749C и G192→A192 при использовании олигонуклеотидов Rv2595N-Rv2595C; исследуемый штамм относится к генотипу Ural в случае выявления полиморфизма C394→T394 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv1397N-Rv1397C; исследуемый штамм относится к генотипу LAM9 в случае выявления полиморфизма G3→А3 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2274N-Rv2274C; исследуемый штамм относится к генотипу F15/LAM4/KZN в случае выявления полиморфизма C168→T168 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2494N-Rv2494C и C140→A140 при использовании олигонуклеотидов Rv1740N-Rv1740C; исследуемый штамм относится к генотипу SMI-049 в случае выявления полиморфизма G213→C213 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2526N-Rv2526C; исследуемый штамм относится к генотипу Haarlem в случае выявления полиморфизма G197→C197 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv2494N-Rv2494C; исследуемый штамм относится к генотипу Т в случае выявления полиморфизма Т137→C137 при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv3408-Rv3408C; исследуемый штамм относится к генотипу LAM в случае выявления наличия T137 в сочетании с отсутствием полиморфизмов по остальным генам при секвенировании фрагмента, наработанного при использовании олигонуклеотидов Rv3408-Rv3408C.
| СПОСОБ ГЕНЕТИЧЕСКОГО ТИПИРОВАНИЯ ШТАММОВ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗНОГО КОМПЛЕКСА | 2003 |
|
RU2237090C1 |
| СПОСОБ ВЫЯВЛЕНИЯ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗА ГЕНОТИПА BEIJING | 2008 |
|
RU2405836C2 |
| US 20110105531 A1, 05.05.2011 | |||
| Jia-Ru Chang et al., Clonal Expansion of Both Modern and Ancient Genotypes of Mycobacterium tuberculosis in Southern Taiwan, PLOS ONE, August 2012, Volume 7, Issue 8, e43018-e43018 | |||
Авторы
Даты
2015-04-10—Публикация
2013-12-12—Подача