Изобретение относится к молекулярной биологии, генетической инженерии, медицине и предназначено для типирования вируса гриппа А. Предлагаемый способ типирования вируса гриппа основан на проведении двухэтапной полимеразной цепной реакции (ПЦР) кДНК вируса с последующей гибридизацией меченных биотином ампликонов с ДНК-микрочипом, содержащим соответствующие дискриминирующие гибридизационные зонды. Использование этого способа позволяет определять все 9 субтипов гена нейраминидазы и 14 субтипов гена гемагглютинина вируса гриппа А.
Грипп А является одним из наиболее распространенных инфекционных заболеваний. За последнее столетие вирус гриппа А являлся причиной пандемий гриппа в 1918-1920, 1957-1958, 1968-1969, 2009 годах (Potter CW, Jennings R.A definition for influenza pandemics based on historical records. 2011. J. Infect. V.63. P.252-259). Кроме того, локальные вспышки заболевания неоднократно фиксировались в различных регионах мира. Возможность определения субтипа вируса гриппа на ранних стадиях развития пандемии или эпидемии является важным для предотвращения распространения инфекции и уменьшения последствий заболевания (Chin, J., Koh, G. and Lee, D.-Y. How necessary is a fast testkit for mitigation of pandemic flu? 2010. J.R. Soc. Interface. V.7. P.1033-1047, Dhanasekaran Vijaykrishna, Gavin J.D. Smith, Oliver G. Pybus, Huachen Zhu, Samir Bhatt, Leo L.M. Poon, Steven Riley, Justin Bahl, Siu K. Ma, Chung L. Cheung, Ranawaka A.P.M. Perera, Honglin Chen, Kennedy F. Shortridge, Richard J. Webby, Robert G. Webster, Yi Guan, J.S. Malik Peiris. Long-term evolution and transmission dynamics of swine influenza A virus. 2011. Nature. V.473. P.519-522.).
Для типирования вируса гриппа А используются различные варианты микрочипов. Для этой цели используются как секвенирующие (Baochuan Lin, Kate M. Blaney, Anthony P. Malanoski, Adam G. Ligler, Joel M. Schnur, David Metzgar, Kevin L. Russell, and David A. Stenger. Using a Resequencing Microarray as a Multiple Respiratory Pathogen Detection Assay. 2007. J. Clin. Microbiol. V.45. P.443-452., Liu RH, Lodes MJ, Nguyen Т, Siuda Т, Slota M, Fuji HS, McShea A. Validation of a fully integrated microfluidic array device for influenza A subtype identification and sequencing. 2006. Anal. Chem. V.78. P.4184-4193., Michael J. Lodes, Dominic Suciu, Mark Elliott, Axel G. Stover, Marty Ross, Marcelo Caraballo, Kim Dix, James Crye, Richard J. Webby, Wanda J. Lyon, David L. Danley, and Andrew McShea. Use of Semiconductor-Based Oligonucleotide Microarrays for Influenza A Virus Subtype Identification and Sequencing. 2006. J. Clin. Microbiol. V.44, P.1209-1218.),
так и гибридизационные микрочипы [Dawson, E.D., C.L. Moore, J.A. Smagala, D.M. Dankbar, M. Mehlmann, M.B. Townsend, C.B. Smith, N.J. Cox. MChip: A tool for influenza surveillance. 2006. Anal. Chem. V.78. P.7610-7615., Jiping Li, Shu Chen, and David H. Evans. Typing and Subtyping Influenza Virus Using DNA Microarrays and Multiplex Reverse Transcriptase PCR. 2001. J Clin Microbiol. V.39: P.696-704., Astrid Gall, Bernd Hoffmann, Timm Harder, Christian Grund, Dirk Höper, and Martin Beer. Design and Validation of a Microarray for Detection, Hemagglutinin Subtyping, and Pathotyping of Avian Influenza Viruses. 2009. J. Clin. Microbiol. V.47. P.327-334., Srikumar Sengupta, Kenji Onodera, Alexander Lai, and Ulrich Melcher. Molecular Detection and Identification of Influenza Viruses by Oligonucleotide Microarray Hybridization. 2003. J. Clin. Microbiol. V.41. P.4542-4550., Jiping Li, Shu Chen, and David H. Evans. Typing and Subtyping Influenza Virus Using DNA Microarrays and Multiplex Reverse Transcriptase PCR. 2001. Clin. Microbiol. V.39 P.696-704., Zhang F., Zou M., Li J., Xue Q. Cytometric microsphere array for subtyping avian influenza virus. 2011. Viral Immunol. V.24 P.403-407.).
Гибридизационные микрочипы, описанные в вышеприведенных работах, в основном не являлись универсальными и предназначались для определения только части субтипов нейраминидазы и гемагглютинина. Универсальные микрочипы, способные типировать практически все возможные субтипы вируса гриппа А, малочисленны.
Известен микрочип (Michael J. Lodes, Dominic Suciu, Mark Elliott, Axel G. Stover, Marty Ross, Marcelo Caraballo, Kim Dix, James Crye, Richard J. Webby, Wanda J. Lyon, David L. Danley, and Andrew McShea. Use of Semiconductor-Based Oligonucleotide Microarrays for Influenza A Virus Subtype Identification and Sequencing. 2006. J. Clin. Microbiol. V.44, P.1209-1218) типирования всех подтипов гена гемагглютинина (за исключением субтипа H16) и всех 9 подтипов нейраминидазы вируса гриппа А. Типирование осуществляется с помощью секвенирующего микрочипа, содержащего 12000 типирующих зондов.
Создание такого рода микрочипов требует специального оборудования - микрочиповых синтезаторов, которые не являются коммерчески доступным оборудованием (не представлены на рынке приборов). Стоимость такого рода микрочипов, из-за большого количества типирующих зондов, высока и достигает нескольких сотен долларов (300-400 долларов). Типирование вируса гриппа с помощью этого секвенирующего микрочипа является экономически затратным процессом.
Типирование вируса гриппа А с помощью гибридизационного микрочипа (патент РФ №2470076. Способ выбора ДНК-зондов для микрочиповой диагностики, биочип и способ типирования гена нейраминидазы и гемагглютинина вируса гриппа А) является значительно более экономичным процессом, поскольку стоимость самого микрочипа в десятки раз меньше (10-12 долларов). Сам микрочип изготавливается методом контактной или бесконтактной печати из заранее синтезированных олигонуклеотидов. Общее число зондов, необходимых для типирования вируса гриппа, в этом микрочипе значительно меньше и составляет в сумме 600 штук. Этот микрочип универсален и позволяет типировать вирус гриппа А независимо от организма хозяина и географического распространения.
Этот биочип и метод типирования вируса гриппа, его использующий, имеет также ряд недостатков.
Следует сказать, что полноразмерный вариант микрочипа важен при первоначальном тестировании рассчитанных олигонуклеотидных зондов, но может оказаться излишне объемным для проведения рутинных анализов. Кроме того, при наработке ампликона используются достаточно дорогие флуоресцентные нуклеозидтрифосфаты или меченные флуоресцентной меткой праймеры, а для визуализации картины гибридизации - специальные флуоресцентные сканеры. Уменьшение вклада этих двух параметров в стоимость анализа делает микрочип коммерчески гораздо более привлекательным.
Раскрытие изобретения. Задачей настоящего изобретения является создание нового более экономичного способа типирования вируса гриппа А для устранения недостатков известных методов.
Эта задача решается новым способом обнаружения гибридизации кДНК вируса гриппа с типирующими зондами и уменьшением числа спотов микрочипа. В заявленном методе предложено использовать для анализа гибридизации биотиновую метку, введенную на стадии амплификации кДНК мишени, и использовать споты, содержащие одновременно в своем составе все зонды, выявляющие определенный субтип генов гемагглютина или нейраминидазы. Метод включает также способы регистрации и интерпретации результатов анализа.
Сущность изобретения
Сущность изобретения заключается в новом способе выявления гибридизации на микрочипе и возможности типирования кДНК вируса гриппа с помощью спотов, содержащих смесь всех возможных зондов, для обнаружения определенного субтипа вируса гриппа А.
Отнесение анализируемого изолята гриппа к определенному субтипу гемагглютинина или нейраминидазы с использованием полноразмерного варианта универсального микрочипа (микрочип первого поколения) проводится путем определения средней флуоресценции спотов, характерной для определенного субтипа. Источником ложноположительных результатов может являться высокий уровень фоновой флуоресценции (в этом случае число значимых спотов для всех подтипов может оказаться близким по значению), либо слишком высокая флуоресценция одного из спотов (тогда средняя флуоресценция спотов будет слишком высока, но при этом количество значимых спотов будет мало).
Проанализировав полученные данные, мы пришли к выводу, что, исключив зонды, которые дают или могут давать ложноположительные результаты, можно создать микрочип существенно меньшего размера.
В целом, из полных наборов для типирования нейраминидазы, приведенных в патенте РФ №2470076, были исключены зонды следующей структуры:
N1 #7 GCAAGTGCTTGTCATGATGGC
N1 #11 AAATACGGCAATGGTGTTTGG
N1 #15 ATAGGGTACATATGCAGTGGG
N4 #7 TGTTCCTGTTACCCAAGTGGG
N5 #14 TTTCAGGGAGGTCACATTGAA
N7 #5 GAATGCTCCTGTTACGGACAC
N7 #15 GAATGCTCCTGTTATGGACAC
В результате число зондов для определения субтипа нейраминидазы N1 уменьшилось с 22 до 19, N4 - с 19 до 18, N5 - с 20 до 19, N7 - с 19 до 17.
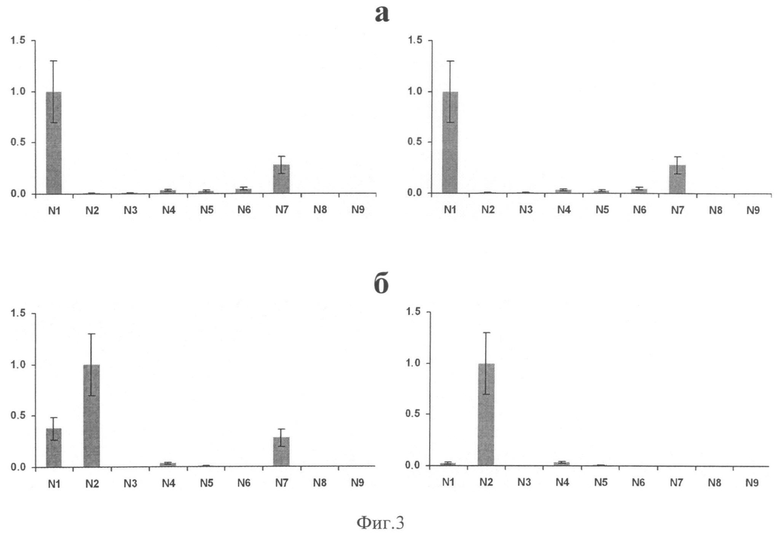
Вместе с тем, исключение этих зондов из расчета не сказывается на результатах анализа изолятов с нейраминидазой N1 или N7, что видно на примере анализа изолята вируса сезонного гриппа H1N1 (Фиг.3а). Слева приведена гистограмма, рассчитанная с использованием всех спотов, справа - с исключением 3-х спотов для субтипа N1 и 2-х - для субтипа N7. При анализе субтипов, отличных от N1 или N7, исключение при построении гистограммы указанных выше слотов повышало достоверность типирования, что показано на примере изолята H2N2 на Фиг.3б.
Аналогичным образом была проведено тестирование зондов, типирующих гемагглютинин. Ложноположительно флуоресцирующих спотов в этом случае не обнаружено. Но анализ показал, что один из зондов, используемый для типирования гемагглютинина H16 (5′-GAAAGAGGATTGTTTGGTGCA-3′, #17, перекрывается 6-ю основаниями с внутренним праймером. Этот зонд также был исключен из используемого набора.
Принципиальная схема типирования гена нейраминидазы вируса гриппа А на микрочипе включает следующие стадии:
1. Из образца слизистой ткани выделяют суммарную РНК.
2. С помощью универсального праймера
5′-GACTAATACGACTCACTATAGGGAGCAAAAGCAGG-3′, специфичного к РНК вируса гриппа А, получают кДНК.
3. С помощью праймеров 5′-CTATAGGGAGCAAAAGCAGGAGT-3′ и 5′-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3′, специфичных к гену нейраминидазы, из кДНК путем проведения ПЦР получают полноразмерный ампликон гена нейраминидазы вируса гриппа А.
4. Полученный ампликон реамплифицируют в асимметричном режиме с праймерами:
5′-CTATAGGGAGCAAAAGCAGGAGT-3′,
5′-TAACAGGA(G/A)CA(C/T)TCCTC(A/G)TA(G/A)TG-3′
и 5′-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3′,
5′-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3′ и метят путем введения метки в его состав.
5. На следующей стадии проводят гибридизацию полученных ампликонов с дискриминирующими зондами, входящими в состав разработанного микрочипа.
6. Анализ результатов гибридизации проводят с помощью сканера микрочипов и программы Scanarray Express. Результаты анализа представляются в графической форме с помощью программы Microsoft Office Excel.
Принципиальная схема типирования гена гемагглютинина вируса гриппа А на микрочипе включает следующие стадии:
1. Из образца слизистой ткани выделяют суммарную РНК.
2. С помощью универсального праймера 5′-GACTAATACGACTCACTATAGGGAGCAAAAGCAGG-3′, специфичного к РНК вируса гриппа А, получают кДНК.
3. С помощью праймеров 5′-GACTCACTATAGGGAGCAAAAGCAGGGG-3′ и 5′-GTGACACTATAGAAGTAGAAACAAGGGTGTTTT-3′, специфичных к гену гемагглютинина, из кДНК путем проведения ПЦР получают полноразмерный ампликон гена гемагглютинина вируса гриппа А.
4. Полученный ампликон реамплифицируют в асимметричном режиме с праймерами:
5′-GACTCACTATAGGGAGCAAAAGCAGGGG-3′,
5′-TCWATRAANCCNGCDATDGCHCC-3′,
5′-GGDGCHATHGCNGGNTTYATWGA-3′,
5′-GGTGACACTATAGAAGTAGAAACAAGGGTGTTTT-3′ и метят путем введения флуоресцентной метки в его состав.
5. На следующей стадии проводят гибридизацию полученных ампликонов с дискриминирующими зондами, входящими в состав разработанного микрочипа.
6. Анализ результатов гибридизации проводят с помощью сканера микрочипов и программы Scanarray Express. Результаты анализа представляются в графической форме с помощью программы Microsoft Office Excel.
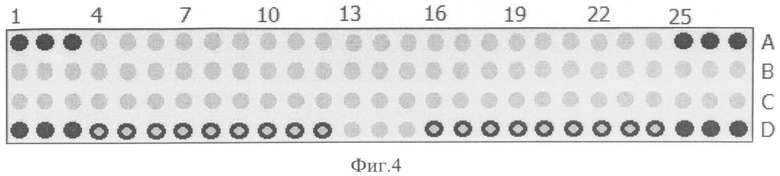
Нами установлено, что можно значительно уменьшить размеры микрочипа путем расположения в одном споте сразу всех зондов, типирующих один и тот же субтип вируса гриппа. При подготовке растворов смеси олигонуклеотидных зондов для печати они смешивались в эквимолярных количествах, так чтобы суммарная концентрация составляла 70 нмоль/мл. Микрочип представляет собой набор спотов, в который, кроме типирующих зондов на нейраминидазу, гемагглютинин и матриксный белок (в трех повторах) включаются маркерные споты, содержащие октатимидилат с красителем Tamra (12 повторов), и споты с печатью гибридизационного буфера для оценки фоновой флуоресценции (отрицательный контроль, 18 повторов). Общее число спотов составило 108. Дизайн микрочипа размером 27×4 приведен на Фиг.4.
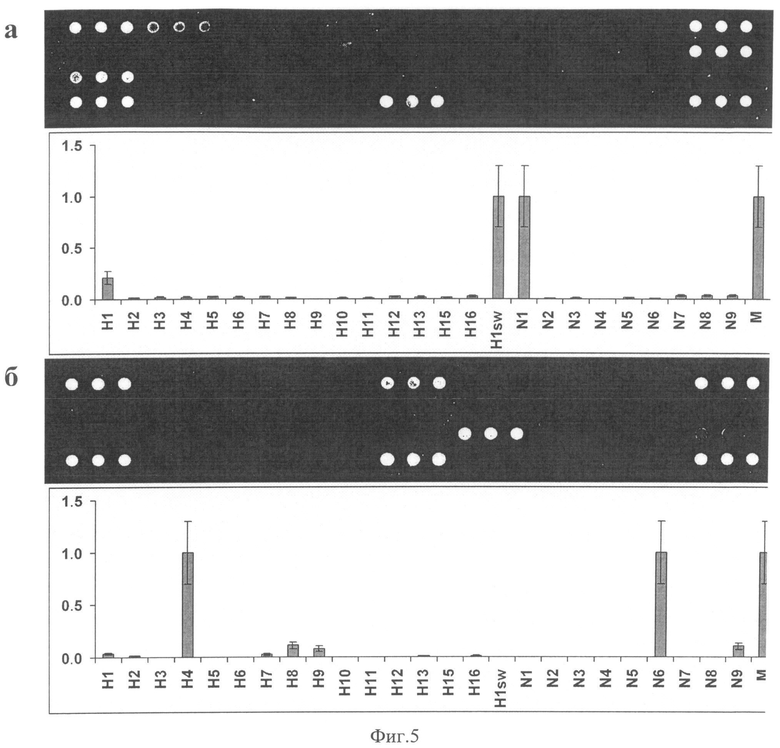
На Фиг.5 в качестве примера приведены гибридизационные картины, полученные при анализе изолятов гриппа А субтипов H1N1 (пандемического 2009) и H4N6 соответственно. Во всех случаях интенсивность флуоресценции спотов матриксного белка была достаточно высока (более чем в 10 раз превышала флуоресценцию спотов отрицательного контроля), что позволяет отнести анализируемые образцы к вирусу гриппа А. Как видно из Фиг.5, при отнесении изолята к гриппу 2009 также наблюдается незначительная флуоресценция спотов гемагглютинина, соответствующих сезонному гриппу H1N1, что является результатом высокой гомологии нуклеотидных последовательностей сезонного и свиного гриппа. Тем не менее, отличия в уровнях флуоресценции достаточно велики, и с учетом возможной ошибки отнесение изолята к свиному гриппу H1N1 является однозначным. Таким образом, представленный универсальный микрочип с минимальным количеством спотов способен давать такую же информацию, что и полноразмерный микрочип.
Использование флуоресценции для анализа результатов гибридизации аналитического образца на микрочипе дает хорошие результаты, но является довольно дорогим процессом, эксплуатирующим дорогое оборудование. С целью нахождения альтернативного и более простого варианта для анализа картины гибридизации нами был рассмотрен подход, при котором для мечения в процессе амплификации кДНК клинического образца используется биотин. После проведения гибридизации слайд обрабатывается раствором конъюгата стрептавидина с коллоидным золотом, в результате чего в качестве метки в структуру ДНК вводится золото. При дальнейшей обработке слайда солями серебра в восстановительных условиях происходит выпадение серебра в районе локализации ДНК, несущей золото. Образующаяся бляшка (серебряное зеркало) может регистрироваться как визуально, так и с использованием обычного бытового планшетного сканера, стоимость которого в тысячи раз меньше флуоресцентного сканера. Этот вариант является хорошей альтернативой визуализации картины гибридизации с использованием флуоресцентной метки. Для введения биотиновой метки в структуру ДНК в процессе амплификации можно использовать два метода: вносить праймеры, несущие на 5′-конце остаток биотина, или применять биотинилированный нуклеозидтрифосфат. В первом случае эффективность амплификации выше за счет использования немодифицированных трифосфатов, во втором случае наработка ампликона несколько затруднена, но при этом увеличивается количество молекул биотина, включенных в структуру ДНК.
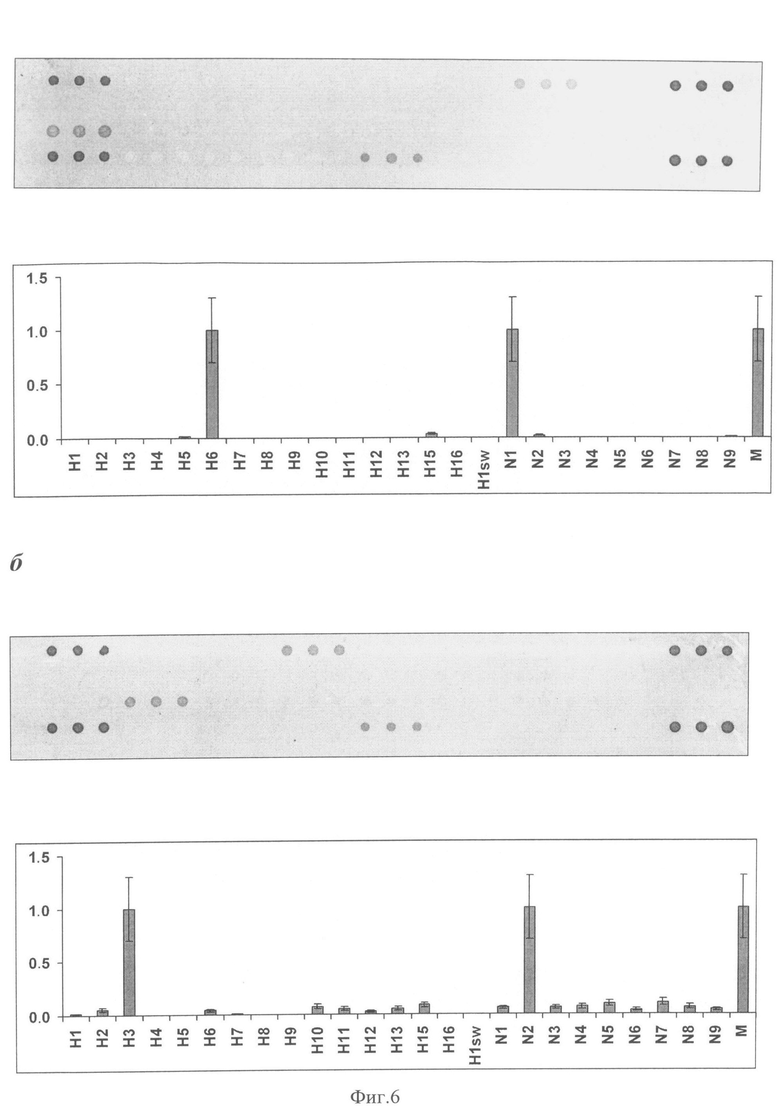
Нами было проведено сравнение картин гибридизации флуоресцентно-меченных и биотинилированных ампликонов. При амплификации в одинаковых условиях использовалось эквимолярное количество праймеров, содержащих на 5′-конце либо флуоресцентную метку TAMRA, либо биотин. Таким образом, предполагалось, что количество ампликонов, несущих каждую из меток, одинаково. Для гибридизации на микрочипе использовали равные объемы каждого из ампликонов. Слайд или сканировали на флуоресцентном сканере, или проявляли изображение, используя систему стрептавидин-коллоидное золото - соли серебра, и сканировали с помощью планшетного сканера, соответственно. При использовании неочищенного ампликона, меченного TAMRA, уровень фоновой флуоресценции был довольно высок, что затрудняло количественную оценку результатов. В то же время использование неочищенного ампликона с биотином позволяло получить качественные картины гибридизации (Фиг.6). Можно сделать вывод, что использование в качестве метки биотина упрощает как процедуру подготовки образца, так и сканирование и является хорошей альтернативой флуоресцентому мечению.
Сопоставительный анализ с прототипом позволяет сделать вывод, что заявленный способ отличается от прототипа по нескольким параметрам.
К преимуществам разработанного микрочипа и способа типирования вируса гриппа относится:
1. Малый размер чипа. Если при использовании полноразмерного варианта универсального микрочипа для определения нейраминидазы, гемагглютинина и матриксного белка необходимо напечатать около около 600 спотов, то данный вариант требует всего 27 спотов: 17 - для определения субтипа гемагглютинина, включая зонды на свиной пандемический грипп 2009 года, 9 - для определения субтипа нейраминидазы и один для определения гена матриксного белка.
2. Уменьшение геометрических размеров самого микромассива позволяет расположить большее их число на слайде, что повышает статистическую достоверность получаемых результатов.
3. Повышение чувствительности метода, поскольку ДНК из всего объема анализируемого раствора локализуется 27 спотов, а не распределяться между 600 спотами.
4. Микромассив столь малого размера может использоваться в микрофлюидных системах для детектирования вируса гриппа А и определения его субтипа.
5. Использование нефлуоресцентной метки (биотина) позволяет значительно снизить затраты на сканирующее оборудование для регистрации результатов проведения анализа.
Таким образом, предложенный микрочип второго поколения для типирования вируса гриппа А по достоверности результатов аналогичный полноразмерному микрочипу. При этом методика визуализации картины гибридизации аналитических образцов с использованием нефлуоресцентной метки и стандартного планшетного сканера является значительно менее затратной.
Изобретение иллюстрируется следующими графическими материалами:
Фиг.1. Таблица 1. Структура олигонуклеотидных зондов для выявления серотипов нейраминидазы вируса гриппа А.
Фиг.2. Таблица 2. Структура олигонуклеотидных зондов для выявления серотипов гемагглютинина вируса гриппа А.
Фиг.3. Гистограммы, полученные при типировании нейраминидазы изолятов гриппа H1N1_(A/TEXAS/36/91 Х-113) (а) и H2N2 (A/Chicken/Novosibirsk region/326/05) (б). Приведена средняя флуоресценция спотов. Гистограммы слева отвечают анализу полного набора спотов, справа - рассчитанные с исключением трех спотов для субтипа N1 и двух спотов - для субтипа N7.
Фиг.4. Дизайн микрочипа размером 27×4 спота. (А1-A3; А25-А27; D1-D3; D25-D27) - маркер; (D4-D12; D16-D24) - буфер; (А4-А6) - H1; (А7-А9) - Н2; (А10-А12) - Н3; (А13-А15) - Н4; (А16-А18) - Н5; (А19-А21) - Н6; (А22-А24) - Н7; (В1-В3) - Н8; (В4-В6) - Н9; (В7-В9) - Н10; (В10-В12) - H11; (В13-В15) - Н12; (В16-В18) - Н13; (В19-В21) - Н15; (В22-В24) Н16; (В25-В27) - H1sw; (C1-С3) - N1; (С4-С6) - N2; (С7-С9) - N3; (С10-С12) - N4; (C13-C15) - N5; (C16-C18) - N6; (C19-C21) - N7; (C22-C24) - N8; (C25-C27) - N9; (D13-D15) - M.
H1-Н16 - споты, содержащие олигонуклеотидные зонды, типирующие определенные субтипы гена гемагглютинина, споты, содержащие олигонуклеотидные зонды, типирующие определенные субтипы гена нейраминидазы N1-N9, M - споты, содержащие олигонуклеотидные зонды, типирующие ген матриксного белка вируса гриппа А.
Фиг.5. Гибридизационные картины и гистограммы, полученные при определении субтипа изолятов гриппа Pandemic flu H1N1 (A/Ekaterinburg /01/2009) (а) и H4N6 (A/Domestic Duck/Novosibirsk region/41/05) (б). Приведена средняя флуоресценция спотов. M - ген матриксного белка гриппа А.
Фиг.6. Гибридизационные картины и гистограммы, полученные с использованием ампликонов, меченных биотином, при определении субтипа изолятов гриппа A/mallard/Irkutsk/165/08 (H6N1) (а и A/Aichi/2/68(H3N2) (б). Приведено среднее значение сигнала для каждого субтипа. M - ген матриксного белка гриппа А.
Для лучшего понимания сущности изобретения ниже приведены примеры его конкретного выполнения.
Пример 1. Изготовление микрочипов для типирования генов нейраминидазы геммаглютинина вируса гриппа А
Печать микрочипов осуществляли на стеклянных, содержащих изотиоцианатную группу слайдах методом контактной печати на споттере BioOdyssey Calligrapher miniarrayer («Bio-Rad») одним пином. Каждый слайд содержал 4 эквивалентных микромассивов зондов (микроэрреея), пригодных для одновременной гибридизации 4 образцов. Средний диаметр спотов (ячеек микрочипа с иммобилизованными зондами) был ~ 360 µm, расстояние между спотами - 380 µm.
Микромассив состоял из 4 строк и помимо типирующих зондов содержал также маркерные олигонуклеотиды (dT)8-TAMRA или (dT)8-BIOTIN (Фиг.4).
При подготовке растворов смеси олигонуклеотидных зондов для печати они смешивались в эквимолярных количествах, так чтобы суммарная концентрация составляла 70 нмоль/мл. Микрочип представляет собой набор спотов, в который кроме типирующих зондов на нейраминидазу, гемагглютинин и матриксный белок (в трех повторах) включаются маркерные споты, содержащие октатимидилат с красителем Tamra (12 повторов), и споты с печатью гибридизационного буфера для оценки фоновой флуоресценции (отрицательный контроль, 18 повторов). Общее число спотов составило 108.
В каждую ячейку планшета при печати типирующих зондов добавлял 60 мкл раствора А (135 мМ NaHCO3) и 20 мкл раствора зондов (С=280 нмоль/мл). При печати маркеров использовали 80 мкл 6 нМ раствора Tamra (dT8)~NH2 в 100 мкл раствора А. При печати буфера использовали 60 мкл раствора А и 10 мкл воды.
Пример 2. Синтез кДНК вируса гриппа А
Геномную РНК выделяли из 140 мкл клеточной культуры (титр вируса 105-107 pfu/мл) с использованием набора QIAquick Viral RNA Kit (QIAGEN, Valencia, Ca).
Обратную транскрипцию проводили следующим образом: смесь, содержащую 1 мкл праймера 5′-GACTAATACGACTCACTATAGGGAGCAAAAGCAGG-3′ (20 мкМ), 9 мкл РНК (из экстрагированного объема 80 мкл) и 2 мкл dNTP (10 мМ), нагревали 10 мин при 65°C, охлаждали во льду. Добавляли 4 мкл 5х кДНК буфера, 1 мкл 0.1М DTT, 1 мкл РНКзина (40ед), 1 мкл воды, 1 мкл обратной транскриптазы ThermoScript (Invitrogen, Carlsbad, CA). Смесь центрифугировали и инкубировали при 55°C - 5 мин, 60°C - 55 мин.
Пример 3. Получение ампликонов гена нейраминидазы для анализа
Для амплификации фрагментов гена нейраминидазы использовали следующие пары праймеров - внешний и внутренний:
NA_F1 5′-CTATAGGGAGCAAAAGCAGGAGT-3′ и
NA_R1 5′-TAACAGGA(G/A)CA(C/T)TCCTC(A/G)TA(G/A)TG-3′;
NA_F2 5′-CA(C/T)TA(C/T)GAGGA(G/A)TG(C/T)TCCTGTTA-3′ и
NA_R2 5′-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3′.
Для получения преимущественно одноцепочечной ДНК проводили ПЦР в асимметричном режиме в двух вариантах.
Вариант 1: состав реакционной смеси (50 мкл): буфер для ПЦР «Qiagen» (1.5 мМ MgCl2, KCl, (NH4)2SO4, Трис-HCl, pH 8.7), MgCl2 - 1.5 мМ, 200 нМ dATP, dCTP dGTP, 70 нМ dTTP, 100 нМ dUTP-Cy5, 80 нМ прямого праймера, 800 нМ обратного праймера, 1.25 ед. Hot Start Taq-полимеразы («Qiagen») и 2 мкл раствора ДНК. ПЦР проводили в термоциклере iCycler («BioRad»), используя следующий протокол: 95°C - 15 мин, затем 35 циклов 94°C - 30 сек, 52°C-55°C - 30 сек, 72°C - 1 мин 10 сек.
Вариант 2: состав реакционной смеси (50 мкл): буфер для ПЦР «Qiagen» (1.5 мМ MgCl2, KCl, (NH4)2SO4, Трис-HCl, pH 8.7), MgCl2 - 1.5 мМ, 200 нМ dNTP, 80 нМ прямого праймера, 800 нМ обратного праймера, несущего на 5′-конце биотин или TAMRA, 1.25 ед. Hot Start Taq-полимеразы («Qiagen») и 2 мкл раствора ДНК. ПЦР проводили в термоциклере iCycler («BioRad»), используя следующий протокол: 95°C - 15 мин, затем 35 циклов 94°C - 30 сек, 52°C-55°C - 30 сек, 72°C - 1 мин 10 сек.
Контроль наработки ампликона осуществляли электрофорезом в 1% агарозном геле с окрашиванием этидий бромидом. Флуоресцентно-меченные ампликоны очищали от избытка меченых трифосфатов на гель-фильтрующих колонках с носителем Sephadex G-50, высушивали досуха на вакуумной центрифуге при 60° и использовали для гибридизации. В варианте ПЦР с использованием меченых праймеров очистку ампликонов не проводили.
Пример 4. Получение ампликонов гена гемагглютинина для анализа
Для амплификации фрагментов гена гемагглютинина использовали следующие пары праймеров - внешний и внутренний:
HA_F1: 5′-GACTCACTATAGGGAGCAAAAGCAGGGG-3′
HA_R1: 5′-TCWATRAANCCNGCDATDGCHCC-3′
HA_F2: 5′-GGDGCHATHGCNGGNTTYATWGA-3′
HA_R1: 5′-GGTGACACTATAGAAGTAGAAACAAGGGTGTTTT-3′
Для получения преимущественно одноцепочечной ДНК проводили ПЦР в асимметричном режиме в двух вариантах.
Вариант 1: состав реакционной смеси (50 мкл): буфер для ПЦР «Qiagen» (1.5 мМ MgCl2, KCl, (NH4)2SO4, Трис-HCl, pH 8.7), MgCl2 - 1.5 мМ, 200 нМ dATP, dCTP dGTP, 70 нМ dTTP, 100 нМ dUTP-Cy5, 80 нМ прямого праймера, 800 нМ обратного праймера, 1.25 ед. Hot Start Taq-полимеразы («Qiagen») и 2 мкл раствора ДНК. ПЦР проводили в термоциклере iCycler («BioRad»), используя следующий протокол: 95°C - 15 мин, затем 35 циклов 94°C - 30 сек, 52°C-55°C - 30 сек, 72°C - 1 мин 10 сек.
Вариант 2: состав реакционной смеси (50 мкл): буфер для ПЦР «Qiagen» (1.5 мМ MgCl2, KCl, (NH4)2SO4, Трис-HCl, pH 8.7), MgCl2 - 1.5 мМ, 200 нМ dNTP, 80 нМ прямого праймера, 800 нМ обратного праймера, несущего на 5′-конце биотин или TAMRA, 1.25 ед. Hot Start Taq-полимеразы («Qiagen») и 2 мкл раствора ДНК. ПЦР проводили в термоциклере iCycler («BioRad»), используя следующий протокол: 95°C - 15 мин, затем 35 циклов 94°C - 30 сек, 52°C-55°C - 30 сек, 72°C - 1 мин 10 сек.
Контроль наработки ампликона осуществляли электрофорезом в 1% агарозном геле с окрашиванием этидий бромидом. Флуоресцентно-меченные ампликоны очищали от избытка меченых трифосфатов на гель-фильтрующих колонках с носителем Sephadex G-50, высушивали досуха на вакуумной центрифуге при 60° и использовали для гибридизации. В варианте ПЦР с использованием меченых праймеров очистку ампликонов не проводили.
Пример 5. Гибридизация анализируемых флуоресцентных ампликонов
Высушенный образец ампликона растворяли в 10 мкл воды, добавляли 10 мкл 2х гибридизационного буфера (1х гибридизационный буфер: 6х SSC, 5х раствор Денхардта, 0.1% Tween 20) и прогревали перед нанесением на слайд 2 мин при 97°C, охлаждали во льду. Гибридизацию проводили в гибридизационной камере фирмы ArrayIt, Sunnyvale при температуре 55-60°C в течение 2 час. Затем слайд последовательно отмывали в растворах 6х SSC, 2x SSC+0,1% SDS, 2x SSC и 1x SSC, центрифугировали и сканировали на сканере Scan Array Express 2.0 («Perkin Elmer») при длинах волны возбуждающего лазера λ=543 нм и λ=633 нм. Изображение анализировали с использованием программы ScanArray Express, входящих в математическое обеспечение сканера.
Пример 6. Гибридизация анализируемых ампликонов, содержащих биотиновую метку
Высушенный образец ампликона растворяли в 10 мкл воды, добавляли 10 мкл 2x гибридизационного буфера (1x гибридизационный буфер: 6х SSC, 5х раствор Денхардта, 0.1% Tween 20) и прогревали перед нанесением на слайд 2 мин при 97°C, охлаждали во льду. Гибридизацию проводили в гибридизационной камере фирмы ArrayIt, Sunnyvale при температуре 60°C в течение 2 час. Затем слайд последовательно отмывали в растворах 2x SSC+0,1% SDS, 1x SSC и центрифугировали.
На каждый микромассив наносили раствор, содержащий Gold Labbeled Streptavidin и Gold Streptavidin Dilute Buffer (Gold Labbeled Streptavidin System, KPL, США) в соотношении 1:5. Слайд накрывали покровным стеклом, оставляли на 30 мин при комнатной температуре, затем отмывали последовательно 2xSSC+0,1% SDS; 1xSSC+0,1% SDS и центрифугировали. На каждый микромассив наносили проявляющий раствор (Sigma, США), накрывали покровным стеклом и оставляли на 15 мин при комнатной температуре до проявления. Покровное стекло убирали, слайд промывали дистиллированной водой и центрифугировали. Слайд сканировали на планшетном сканере Epson Perfection V500 Photo.
Пример 7. Анализ результатов гибридизации
Сканирование слайда проводили на сканере ScanArray Express 2.0 (Perkin Elmer) последовательно с использованием двух возбуждающих лазеров с длинами волн λ1=543 нм для Су3 и λ2=633 нм для Су5. Изображение, полученное по каналу возбуждения флуоресценции с длиной волны λ2, служило показателем качества печати чипа, а интегральная величина флуоресценции каждого спота рассматривалась в качестве нормировочной для флуоресценции того же спота, измеренной по каналу возбуждения с λ1. Анализ изображения осуществляли с использованием программы Scanarray Express, входящих в математическое обеспечение сканера. Для i-го спота нормированную флуоресценцию рассчитывали по формуле:
Ii=Ii(Су3)/Ii(Су5),
где Ii(Cy3)=Fi(Су3)-Bi(Су3), Fi(Су3) - интегральная интенсивность флуоресценции в пределах спота, Bi(Су3) - средняя фоновая интенсивность флуоресценции в окружении спота. Аналогичным образом рассчитывали величину Ii(Су5).
Для анализа результатов гибридизации рассчитывали среднюю флуоресценцию спотов по формуле:
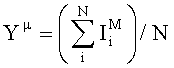
где µ - подтип нейраминидазы,
i - номер спота,
N - число слотов на чипе, используемых для определения подтипа нейраминидазы.
Субтип определяли по столбцу гистограммы с максимальным значением средней флуоресценции спотов. Указанием на однозначность отнесения является отсутствие перекрывания интервалов ошибок субтипов. Интервал ошибок для каждого из субтипов рассчитывался по формуле:
Sµ=SxYµ,
где Sx - относительная ошибка флуоресценции.
Относительная ошибка измерения значения флуоресценции определялась исходя из вариации в интенсивности флуоресценции маркерных спотов субэррея (xi, расположены в левой и правой колонках микрочипа):
Sx=σ/Xcp,
где 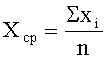
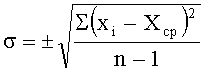
xi - флуоресценция i-го маркерного спота,
n - число маркерных спотов.
Результаты анализа представляются в графической форме с помощью программы Microsoft Office Excel.
Аналогичным образом анализируются результаты гибридизации с использованием биотиновой метки, за исключением того, что сканирование проводят с помощью планшетного сканера, например, Epson Perfection V500 Photo. Для анализа также используется программа Scanarray Express.
Во всех проанализированных случаях типирование генов гемагглютинина и нейраминидазы вируса гриппа А с помощью заявляемого микрочипа осуществлялось корректно.













Изобретение относится к молекулярной биологии, генетической инженерии, медицине и предназначено для типирования вируса гриппа А. Предлагаемый способ типирования вируса гриппа основан на проведении двухэтапной полимеразной цепной реакции (ПЦР) кДНК вируса с последующей гибридизацией меченных биотином ампликонов с ДНК-микрочипом, содержащим соответствующие дискриминирующие гибридизационные зонды. Использование этого способа позволяет определять все 9 субтипов гена нейраминидазы и 14 субтипов гена гемагглютинина вируса гриппа А. 2 н.п. ф-лы, 6 ил., 7 пр.
1. Биочип для типирования вируса гриппа А, содержащий типирующие гибридизационные зонды для генов гемагглютинина и нейраминидазы, ковалентно присоединенные непосредственно к стеклянной микроматрице, имеющие структуру дискриминирующих ДНК-полинуклеотидов, приведенных в Таблице 1 и Таблице 2, отличающийся тем, что все типирующие зонды каждого отдельного вирусного субтипа иммобилизуются в пределах одного спота микрочипа, имеющего размер 27×4 спота и содержащего в спотах (А1 - A3; А25 - А27; D1 - D3; D25 - D27) - маркер; (D4 - D12; D16 - D24) - буфер; (А4 - А6) - типирующие зонды для субтипа H1; (А7 - А9) - типирующие зонды для субтипа Н2; (А10 - А12) - типирующие зонды для субтипа Н3; (А13 - А15) - типирующие зонды для субтипа Н4; (А16 - А18) - типирующие зонды для субтипа Н5; (А 19 - А21) - типирующие зонды для субтипа Н6; (А22 - А24) - типирующие зонды для субтипа Н7; (В1 - В3) - типирующие зонды для субтипа Н8; (В4 - В6) - типирующие зонды для субтипа Н9; (В7 - В9) - типирующие зонды для субтипа Н10; (В10 - В12) - типирующие зонды для субтипа Н11; (В13 - В15) - типирующие зонды для субтипа Η12; (В 16 - В18) - типирующие зонды для субтипа Н13; (В19 - В21) - типирующие зонды для субтипа HI5; (В22 - В24) - типирующие зонды для субтипа H16; (В25 - В27) - типирующие зонды для субтипа Hlsw; (С1 - С3) - типирующие зонды для субтипа N1; (С4 - С6) - типирующие зонды для субтипа N2; (С7 - С9) - типирующие зонды для субтипа N3; (С10 - С12) - типирующие зонды для субтипа N4; (С13 - С15) - типирующие зонды для субтипа N5; (С16 - С18) - типирующие зонды для субтипа N6; (С19 - С21) - типирующие зонды для субтипа N7; (С22 - С24) - типирующие зонды для субтипа N8; (С25 - С27) - типирующие зонды для субтипа N9; (D13 - D15) - типирующие зонды для гена М в соответствии с Фиг. 4.
2. Способ типирования вируса гриппа А, включающий выделение суммарной РНК из природного образца, проведение ОТ-ПЦР для амплификации сегментов генома вируса гриппа, кодирующих гены нейраминидазы и гемагглютинина в асимметричном режиме с использованием специфичных олигонуклеотидов-праймеров для получения меченных преимущественно одноцепочечных ДНК, проведение ДНК-ДНК гибридизации полученных ампликонов с дискриминирующими зондами, входящими в состав биочипа, регистрацию результатов гибридизации меченого образца с помощью регистрирующего устройства и интерпретацию полученных результатов, отличающийся тем, что в качестве метки используют биотин, в качестве биочипа - микроматрицу, охарактеризованную в п. 1, в качестве регистрирующего устройства - планшетный сканер, а интерпретацию полученных результатов проводят с помощью сравнительного анализа интенсивности окраски спотов, дискриминирующих различные субтипы вируса гриппа А.
| СПОСОБ ВЫБОРА ДНК-ЗОНДОВ ДЛЯ МИКРОЧИПОВОЙ ДИАГНОСТИКИ, БИОЧИП И СПОСОБ ТИПИРОВАНИЯ ГЕНА НЕЙРАМИНИДАЗЫ И ГЕМАГГЛЮТИНИНА ВИРУСА ГРИППА А | 2010 |
|
RU2470076C2 |
| WERNERSSON R | |||
| et al., "Oligo Wiz 2.0 - integrating sequence feature annotation into the design of microarray probes", Nucleic Acids Res | |||
| Способ обработки целлюлозных материалов, с целью тонкого измельчения или переведения в коллоидальный раствор | 1923 |
|
SU2005A1 |
| RYABININ V.A | |||
| et al., "Microarray assay for detection and discrimination of Orthopoxvirus species, J Med Virol | |||
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ ВИРУСОВ ГРИППА | 2004 |
|
RU2290444C2 |
Авторы
Даты
2015-08-20—Публикация
2013-05-06—Подача