Изобретение относится к области молекулярной биологии и может быть использовано для выявления генов-мишеней для диагностики и терапии лейкозов человека.
Лейкозы занимают особое место среди злокачественных заболеваний человека и животных и характеризуются повышенным содержанием бластных клеток, которые, с одной стороны, не выполняют нормальные функции зрелых клеток крови, а с другой, за счет активной пролиферации, вытесняют нормальные ростки кроветворения.
Значительную часть лейкозов человека составляют острые миелоидные лейкозы (ОМЛ). Их возникновение связано с нарушением развития клеток-предшественников миелоидных, эритроидных, мегакариоцитарных и моноцитарных клеточных линий. Полагают, что лейкозы имеют клональную природу, т.е. злокачественные бластные клетки являются потомками одной единственной трансформированной клетки, возникающей в результате нескольких, а возможно, даже одной мутации. Считается, что процесс злокачественного перерождения является многостадийным - активация онкогенов в результате мутаций приводит к дальнейшей активации целого каскада генов и, как следствие, к дезорганизации строго слаженных механизмов, отвечающих за развитие кроветворных клеток и нормальное кроветворение. До сих пор не известно, в какой мере и какие именно гены активируются вследствие возникновения уже известных "ключевых" мутаций. Всё это существенно осложняет диагностику лейкозов и выбор эффективных схем лечения.
В настоящее время основными средствами терапии лейкозов являются химиотерапия, лучевая терапия и трансплантация костного мозга. Однако первые два способа сами по себе является довольно болезненными, далеко не всегда эффективными и нередко приводят к рецидивам заболевания, проявляющимся, как правило, в ещё более острой форме, а последний является крайне дорогим и сложным, что, прежде всего, связано с поиском донора костного мозга. Кроме всего трансплантация костного мозга также несёт целый ряд опасностей для здоровья пациента, к основным из которых относят случаи отторжения трансплантата. Именно поэтому поиск новых эффективных мишеней и подходов для диагностики и лечения лейкозов является крайне актуальным направлением исследований учёных всего мира.
Однако возможности поиска новых эффективных мишеней ограничены несовершенством существующих методов и их высокой стоимостью.
Проблема ещё больше обостряется тем, что до сих пор неизвестно какие ещё гены, ответственные за рост и дифференцировку кроветворных клеток, и в какой степени вовлечены в процесс злокачественного перерождения.
Для большинства видов опухолей установлены лишь некоторые из онкогенов, участвующих в канцерогенезе. Это затрудняет разработку и выбор терапевтических подходов, и в частности, терапевтических целей -генов-мишеней и их белковых продуктов.
В патенте RU 2161309
(международная публикация WO 95/25813) описан ген MTS, соматические мутации этого гена в зародышевой линии и способ выявления предрасположенности к злокачественным опухолям. Также описано применение выявленных мутаций для диагностики предрасположенности к -различным формам злокачественных заболеваний, в частности, лейкозам, и лечению заболеваний, при которых произошла мутация в гене MTS, включая генотерапию, заменяющую белковую терапию и использование белковых миметиков.
В патенте RU 2240125
в качестве гена-мишени для лечения Ph+ лейкозов описан онкоген BCR/ABL, и олигонуклеотидная генотерапия Ph+ лейкозов путем обработки суспендизооных культур Ph+ клеток антисмысловыми олигонуклеотидами, длиною 16-26 нуклеотидных звеньев, специфичными к мРНК онкогена BCR/ABL в сочетании с антисмысловыми олигонуклеотидами к мРНК других генов, блокирующих апоптоз Ph+ клеток, отличных от онкогена BCR/ABL.
В патенте RU 2286798
описан способ идентификации хромосомных транслокаций, приводящих к развитию злокачественных заболеваний крови с использованием олигонуклеотидного биологического микрочипа путем гибридизации с олигонуклеотидным микрочипом химерного амплифицированного флуоресцентно меченного продукта, состоящего из последовательностей генов, участвующих в определенной хромосомной транслокации, ассоциированной с лейкозом.
В евразийском патенте ЕА 003637
(международная публикация WO 00/05406) описаны новые способы идентификации биомолекул лигандов и мишеней путем включения случайных нуклеотидных последовательностей в
каркас, состоящий из модулятора активности фермента, трансформации существенно идентичных клеток, полученной таким образом конструкцией и скрининга трансформированных клеток для идентификации клеток, в которых был изменен предварительно выбранный фенотипический признак.
В евразийском патенте ЕА 006512
описаны способы определения генов, детерминирующих синтез диагностически важных белков, определения точечных мутаций, делеций, вставок, инверсий и транслокаций в нуклеиновых кислотах, определения уровня экспрессии генов; определения гомо- и гетерозиготного состояний мутаций основанные на применении методов гибридизации, ПЦР и ее модификаций на микрочипе.
В международной публикации WO 2004/067778
описано выявление дифференциально экспрессирумых генов при лимфогранулематозе, последовательностей этих генов и их применение в качестве маркеров для лимфогранулематоза. Генные последовательности, представленные в изобретении, дифференциально экспрессируются при лимфогранулематозе.
В международной публикации WO 2005/080601
описаны способы генетического анализа для классификации, диагностики и прогнозирования острого миелолейкоза.
В международной публикации WO 2010/111712
описан способ идентификации РНКи мишеней и применение РНКи для рациональной терапии лейкозов, резистентных к химиотерапии. Также представлена мышиная модель, используемая для определения эффективности shPHK in vivo для снижения выживаемости злокачественных клеток, резистентных к химиотерапии.
В международной публикации WO 2009/055907
описана активность не рецепторной тирозинкиназы онкобелков слитого гена Bcr/Abl, которая представляет собой ключевые факторы, ответственные за развитие и прогрессирование Ph+ хронического миелолейкоза и Ph+ острого лимфобластного лейкоза. Также описан пептидный ингибитор Bcr/Abl тирозинкиназы, который индуцирует апоптоз в различных трансформированных клетках и ингибирует Bcr/Abl киназу и, опосредованно, ряд нижележащих мишеней (CrkL, STATS, с-Мус).
В международной публикации WO 02/42482
описаны функциональные лентивирусные векторы на основе химерного гена вируса лейкоза мышей и
вируса иммунодефицита кошачьих, которые используются для генной терапии.
В международной публикации WO 2007/050706
описано применение дифференциального метилирования и гибридизации для идентификации новых маркеров метилирования и профилей метилирования для гематопоэтических злокачественных опухолей, лейкозов, лимфом и т.д., а также маркеров для диагностики, прогноза и мониторинга лечения.
Применение маркеров метилирования генома для идентификации новых генов-мишеней для диагностики и лечения острого миелолейкоза также описано в международной публикации WO 2007/016668.
Гены-мишени и продукты их экспрессии, идентифицированные на основании профиля их экспрессии до и после лечения острого лимфобластного лейкоза, описаны в международной заявке WO 03/087315, изучение профиля экспрессии генов для диагностики и подбора лечения больных лейкозом, описаны в WO 03/083140, способы генотипирования клеток при остром лейкозе ct(llq23)/MLL, и изучение профиля экспрессии генов, а также наборы и системы для их осуществления описаны в WO 2006/048266, при остром промиелоцитарном лейкозе - в WO 2006/048263.
Идентификация новой генной транслокации (3р21, 5q33) при миелолейкозе у людей описана в международной публикации WO 2007/075933, идентификация гена сdс2-зависимой киназы, ассоциированной с лейкозом у людей, описана в WO 00/12719; идентификация лейкоцит-специфичного гена Sp 140 и связанного с ним белка, и его применение в генной терапии для лечения злокачественных заболеваний, а также его применение в качестве диагностического и прогностического маркера, описано в WO 98/14569; идентификация гена MCL-1, ассоциированного с миелоидным лейкозом, а также способы диагностики и лечения с использованием нуклеотидных и полипептидных последовательностей mcl-1, описаны в WO 94/29330; ферментативная молекула РНК, которая специфически расщепляет мРНК, кодируемую геном mdr-1, описана в WO 93/23057; идентификация гена MTS и мутаций этого гена, а также применение в диагностике и прогнозировании, в частности, лейкозов, описано в WO 95/25429.
Перспективным современным подходом исследования функциональной активности генов, в том числе и активированных онкогенов, является РНК-интерференция, основанная на подавлении экспрессии генов
на посттранскрипционном уровне с помощью коротких дуплексов siPHK (small interfering RNA, малые интерферирующие РНК) длиной 21-23 нуклеотидов с выступающими 2-3 нуклеотидами на 3'-концах. Эффективным методом введения интерферирующих РНК в клетки является использование рекомбинантных ретро- и лентивирусных векторов, направляющих в трансдуцированных клетках синтез шпилечных РНК (shPHK) предшественников малых интерферирующих РНК. siPHK образуются из shPHK под действием клеточного белка Дайсера (Dicer), обладающего рибонуклеазной активностью. Шпилечные конструкции могут быть успешно применены для подавления экспрессии генов, принимающих участие в развитии вирусных инфекций, и некоторых активированных онкогенов. Представляется возможным применить этот подход для подавления экспрессии онкогенов, выявляемых при острых миелоидных лейкозах, которые с большой вероятностью могут являться ключевыми факторами развития ОМЛ.
Такие подходы и конструкции малых шпилечных РНК описаны, например, в публикациях китайских патентных заявок CN 102747083, CN 103320444
и в китайском патенте CN 20130925,
и в непатентной литературе, например, у BantounasL, Phylactoul L.A., Uney J.B. 2004. RNA interference and the use of small interfering RNA to study gene function in mammalian systems J. Mol. Endocrinol. 33, 545-557, Agrawal N., Dasaradhi P.V., Mohmmed Α., Malhotra P., Bhatnagar R.K., Mukherjee S.K. 2003. RNA interference: biology, mechanism, and applications. Microbiol. Mol. Biol. Rev. 67, 657-685, в статье Вильгельма А.Э., Чумакова СП., Прасолова B.C. 2006 Интерференция РНК: биология и перспективы применения в биомедицине и биотехнологии. Мол биол. 40(3), 387-403.
Однако, несмотря на активное выявление новых генов-мишеней для диагностики и терапии лейкозов человека и создание конструкций малых шпилечных РНК для РНК-интерференции, проблема поиска новых эффективных мишеней для терапевтического воздействия и разработки новых способов диагностики и лечения лейкозов по-прежнему остается актуальной.
Предлагаемый способ основан на подавлении активности активированного онкогена AML-ETO методом РНК-интерференции в злокачественных кроветворных клетках, полученных от больных лейкозом с использованием конструкции малой шпилечной РНК, для встраивания в лентивирусный вектор, по сайтам рестрикции BamHI и EcoRI или Hpal и
Xhol, кодируемой нуклеотидной последовательностью двухцепочечной ДНК, представляющей собой:
для встраивания по сайтам рестрикции BamHI и EcoRI
shAEl SLP -смысловая
5'-р-
gatccgCCTCGAAATCGTACTGAGActtcctgcaaTCTCAGTACGATTTCGAGGttt ttg-3'
shAE 1 SLP-антисмысловая 5'-p-
aattcaaaaaCCTCGAAATCGTACTGAGAttgcaggaagTCTCAGTACGATTTCGA GGcg-3'
или для встраивания по сайтам рестрикции Hpal и Xhol
shAE 1 GO-смысловая
5'-р-
aacgCCTCGAAATCGTACTGAGActtcctgcaaTCTCAGTACGATTTCGAGGttttt с-3'
shAE 1 GO-антисмысловая 5'-р-
tcgagaaaaaCCTCGAAATCGTACTGAGAttgcaggaagTCTCAGTACGATTTCGA GGcgtt-3'
Этот способ позволяет путём ингибирования активности конкретного онкогена, определить спектр изменения экспрессии генов, ответственных за рост и дифференцировку кроветворных клеток, и обнаружить те из них, изменение активности которых напрямую связано с активацией данного онкогена. Кроме того, данный способ позволяет определить, в какой мере воздействие на такие гены-потенциальные мишени для диагностики и терапии лейкозов оказывает влияние на способность злокачественных клеток к неконтролируемому росту.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Структура препаратов экспрессирующих векторов, необходимых для конструирования препаратов лентивирусных частиц, направляющих синтез shPHK и подавление активированного онкогена AMLI-ETOB злокачественных кроветворных клетках.
Интеграцию последовательности, кодирующей shPHK, в геном клетки-хозяина, могут обеспечить лентивирусные векторы, долговременно подавляя экспрессию гена-мишени. Выбор лентивирусных векторов обусловлен также способностью рекомбинантных лентивирусов заражать широкий спектр клеток, в том числе клетки крови. Это может позволить использовать такие векторы для изучения влияния подавления экспрессии онкогенов на восстановление нормальной дифференцировки клеток крови, а также для исследования генов, экспрессия которых будет изменяться при подавлении активности активированных онкогенов AML1-ETO, которые могут быть перспективными терапевтическими и диагностическими мишенями в случае ОМЛ.
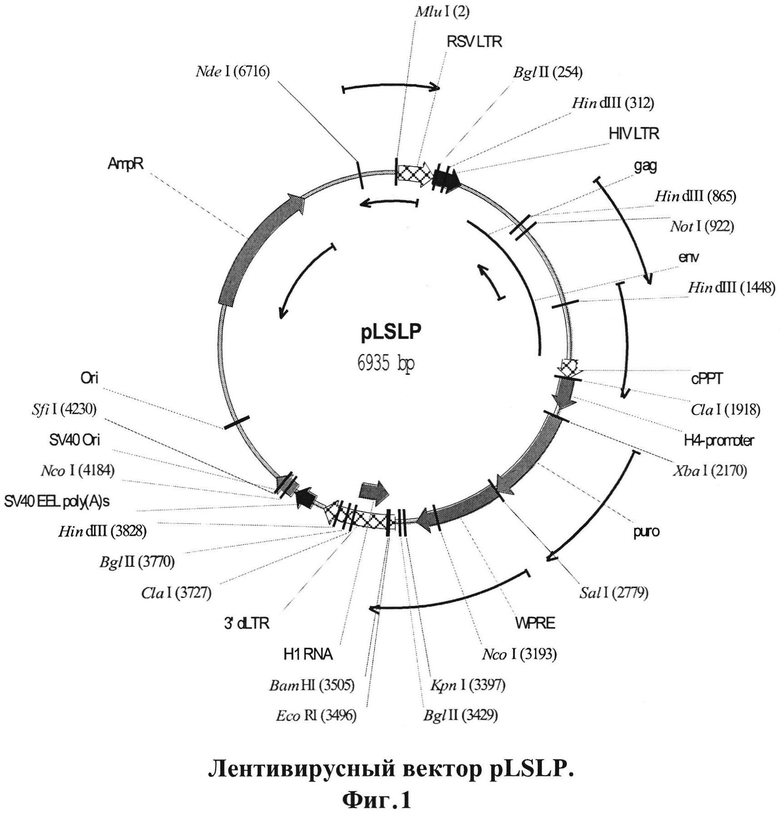
Такие векторы были созданы на базе вектора pLSLP, предназначенного для переноса и экспрессии малых шпилечных РНК в клетках млекопитающих. Вектор позволяет осуществлять встраивание последовательности, кодирующей шпилечную РНК по сайтам рестрикции BamHI и EcoRI. В данном случае экспрессия лентивирусной шпилечной конструкции находится под контролем промотора полимеразы III HI. Помимо этого в составе вектора имеется последовательность, гена устойчивости к пуромицину, наличие которого позволяет осуществлять селекцию клеток, трансдуцированных с помощью данной лентивирусной конструкции. Авторами изобретения был осуществлён дизайн последовательностей, кодирующих shPHK шпилечные структуры -предшественники siPHK:
AML1 -ЕТО-смысловая
5'-р-
gatccgCCTCGAAATCGTACTGAGActtcctgcaaTCTCAGTACGATTTCGAGGttt ttg-3'
AML 1 -ЕТО-антисмысловая
5'-р-
aattcaaaaaCCTCGAAATCGTACTGAGAttgcaggaagTCTCAGTACGATTTCGA GGcg-3'
Эти последовательности могут быть клонированы в лентивирусный вектор. Основой для таких генетических конструкций будет служить лентивирусный вектор pLSLP (Фигура 1).
Основные характеристики вектора pLSLP:
Вектор является самоинактивирующимся (SIN) за счет делеции в U3 области 3'концевого LTR.
Вектор содержит ген резистентности к пуромицину.
Вектор содержит генетические элементы сРРТ и WPRE.
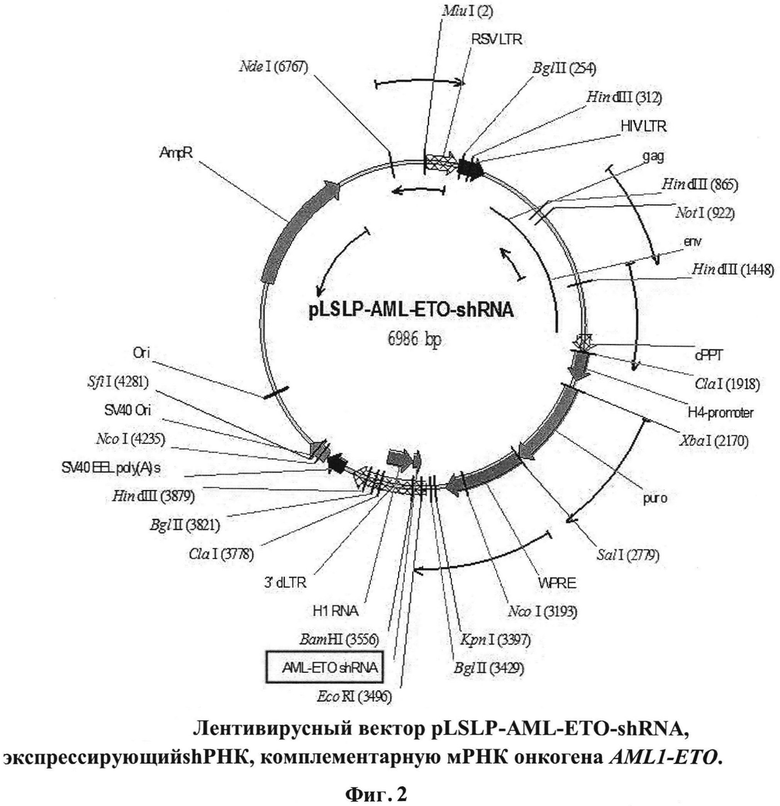
Клонирование проводили с помощью стандартных методов генной инженерии. На Фигуре 2
представлена карта рестрикции сконструированного лентивирусного вектора pLSLP-AML-ETO-shRNA, несущего шпилечную структуру, специфичную в отношении места слияния AML1-ETO. Аналогичным образом может быть сконструирован лентивирусный вектор pLSLP-SCR-shRNA, несущий последовательность, кодирующую shRNA-SCR, не имеющую гомологии с мРНК человека и мыши, используемый в качестве отрицательного контроля.
SCR - смысловая
5'-р-
gatccgCAAGTCTCGTATGTAGTGGcttcctgtcaCCACTACATACGAGACTTGtttt tg-3'
SCR- антисмысловая 5'-р-
aattcaaaaaCAAGTCTCGTATGTAGTGGtgacaggaagCCACTACATACGAGACT TGcg-3'
sh-PHK - экспрессионная кассета в U3 области 3'dLTR. (Экспрессионная кассета находится под контролем РНК-полимеразы Ill-зависимого промотора HI.). shPHK, экспрессирующая последовательность ДНК, клонируется в вектор по сайтам рестрикции BamHI и EcoRI.
ПРИМЕРЫ
ПРИМЕР 1
Анализ подавления экспрессии онкогенов, в перевиваемых кроветворных клетках, трансдуцированных лентивирусным вектором, направляющим синтез shPHK, который опосредует подавление одного из активированных онкогенов
Для анализа подавления экспрессии онкогенов, в перевиваемых кроветворных клетках, трансдуцированных лентивирусным вектором, направляющим синтез shPHK, который опосредует подавление активированных онкогенов AML1-ETO, гиперэкспрессия которого является одним из важнейших факторов, способствующих развитию лейкоза, используют метод ОТ-ПЦР (полимеразная ценная реакция, сопряжённая с обратной транскрипцией), модифицированный метод "гнёздной" ОТ-ПЦР и метод ПЦР в реальном времени (Real-timePCR).
Для этого сначала производят разморозку лабораторных образцов злокачественных перевиваемых клеток человека, полученных от больного лейкозом. Для этого ампулы с образцами клеток, извлекают из сосуда с жидким азотом и помещают в термостат 37°С на 3 минуты, после этого суспензию клеток переносят в полипропиленовые пробирки, содержащие 6 мл среды RPMI и 20% FCS, суспендируют и центрифугируют 6 мин со скоростью 800 об/мин. После этого супернатант отбирают, а осадок ресуспендируют в 2 мл среды RPMI 1640, содержащей 20% эмбриональной сыворотки, 4 мМ L-глутамина, стрептомицин/пенициллин в концентрации 100 мкг/мл и 100 ед/мл, соответственно, рН 6,8-7,0, культивирование осуществляют при температуре 37°С в атмосфере 5% СО2. Культивирование клеток осуществляют в 6-ти луночных планшетах. Через три дня после размораживания к клетки в лунке ресуспендируют, после чего, добавляют 3 мл свежей среды RPMI 1640, после чего опять ресуспендируют и переносят в чашку Петри с площадью поверхности 6 см2. Ещё через 3-4 дня 5 мл суспензии клеток переносят в 10 см2чашку Петри и добавляют 7 мл свежей среды RPMI 1640. Через 4-5 дней осуществляют подсчёт количества клеток. Для расчёта количества клеток используют камеру Нойбауэра. Для этого 10 мкл клеточной суспензии вносят под покровное стекло камеры. Считают количество клеток в четырёх больших квадратах, после чего концентрацию клеток в суспензии определяют по формуле: C=N* 10000/4, где С-количество клеток в 1 мл суспензии, а N - количество клеток в четырёх квадратах.
Таким образом, осуществляют наращивание и подготовку к анализу перевиваемых клеток человека, в которых осуществляется синтез shPHK конструкций (shPHK AML1-ETO, shPHK AML5'), направляющих подавление экспрессии онкогена AML1-ETO, и один образец клеток, в которых происходит синтез неспецифической конструкции shSCR, не приводящей к подавлению какого-либо из генов.
Выделение тотальной РНК из культур клеток, в которых осуществляется синтез shPHK - предшественников siPHK, специфичных в отношении онкогена AML1-ЕТО, и в которых происходит синтез неспецифической конструкции shSCR, осуществляют с помощью тризола. Для этого около 5 млн клеток суспензионной культуры клеток суспендируют и переносят в пробирки. Пробирки с клетками центрифугируют 6 минут при 1000g. Затем удаляют супернатант и добавляют 1 мл lxPBS, суспендируют и снова центрифугируют при прежних условиях, после чего снова удаляют супернатант.
Для выделения используют следующие реактивы:
-Тризол (Invitrogen)
-Хлороформ (Applied Biosystems)
-Изопропиловый спирт
-75% этанол (в ДЭПК воде)
-3Мацетат Na (рН= 5,5) (Applied Biosystems)
-mQ вода
Выделение тотальной РНК осуществляют в следующей последовательности:
Рабочее место обрабатывают ингибиторами РНКаз. К клеткам добавляют тризол (необходимое количество рассчитывают, исходя из соотношения 1 мл тризола на 5-10 млн. клеток). Клетки инкубируют в тризоле в течение 5 минут при комнатной температуре. Лизат переносят в новые пробирки и добавляют хлороформ из расчета 0,2 мл на 1 мл тризола, плотно закрывают пробирку и встряхивают рукой 15 секунд, после чего инкубируют смесь 2-3 минуты при комнатной температуре. Далее пробы центрифугируют 15 минут при 12000g в центрифуге с охлаждением (4°С). В результате центрифугирования смесь разделяется на нижнюю хлороформ фенольную фазу красного цвета, интерфазу белого цвета и бесцветную водную фазу. РНК находится в верхней водной фазе. Водную фазу переносят в чистую пробирку. Добавляют изопропиловый спирт из расчета 0,5 мл на 1 мл тризола и инкубируют 10 минут при комнатной температуре. Затем пробы центрифугируют 10 минут при 12000g в центрифуге с охлаждением (4°С). На дне пробирки виден светлый гелеподобный осадок. Промывают осадок 1 мл 75% ледяного этанола, разбивая осадок на вортексе. После отмывки пробы центрифугируют 10 минут при 12000g в центрифуге с охлаждением (4°С). Промывку проводят дважды. После этого спирт удаляют и высушивают осадок на воздухе. Осадок растворяют в 50 мкл mQ (из расчета 1 мкг/мкл).
Количество и чистоту выделенной РНК определяют с помощью спектрофотометра NanoDrop, определяя поглощение при 230, 260, 280 нм. Количество РНК оценивают по значению А260, т.к. 1 А260=40 мкг РНК/мл. Чистоту образца оценивают по отношению А260/А280 (должно быть в пределах 1,8 - 2,0), степень загрязнения низкомолекулярными органическими соединениями - по А260/А230 (в пределах 1,5 - 2,0). Качество выделенной РНК определяют с помощью электрофореза в 1% агарозном геле. Оценивают присутствие двух полос, соответствующих 28S и 18S рибосомальных РНК, а также отсутствие неспецифических полос и ярко-выраженного "шмера".
Далее к раствору РНК добавляют натрия ацетат и этиловый спирт (1/10 и 2,5 объема раствора РНК соответственно). Смесь перемешивают на вортексе, и затем инкубируют в течение 30 минут при 20 °С. Далее смесь центрифугируют в течение 15 мин при 10°С, 12000 g. Супернатант удаляют, к осадку добавляют 1 мл охлажденного 75% этилового спирта. Полученную смесь перемешивают на вортексе, и затем центрифугируют в течение 15 мин при 10°С, 12000 g. Супернатант удаляют, осадок высушивают на воздухе в течение 10-15 минут. К сухому осадку добавляют mQ воду. Объем воды рассчитывают, исходя из исходного количества РНК (мкг) и желаемой итоговой концентрации. В дальнейшей работе используют растворы РНК с концентрацией 1 мкг/мкл.
ОТ-ПЦР - обратная транскрипция, сопряженная с полимеразной цепной реакцией.
Для синтеза кДНК использовали тотальную РНК клеток (полученную, как описано выше). Выделенную РНК переосаждают и растворяют в воде до концентрации 1 мкг/мл. Смешивают 1 мкл растворенной РНК, 1 мкл поли-Т праймеров (5 пкмоль/мкл), 4 мкл обработанной ДЭПК воды и инкубируют
смесь на водяной бане при 70°С в течение 5 минут. Далее, охлаждают смесь на льду в течение 10 минут. После однократного центрифугирования для осаждения капель конденсата в каждую пробу добавляют 4 мкл пятикратного буфера для обратной транскриптазы (Promega), 2 мкл смеси dNTP (10 мМ, Promega), 1 мкл Rnasine (Promega), 1 мкл обратной транскриптазы MLV (Promega) и 6 мкл воды, обработанной ДЭПК. Перемешивают на вортексе и инкубируют на водяной бане при 42°С в течение 1 часа. Далее пробы охлаждают, доводят водой mQ, до конечного объема 50 мкл. Пробы кДНК хранят при -20°С.
Анализ полученной кДНК методом модифицированной "гнездной" ОТ-ПЦР используют для идентификации в полученной из клеток Kasumi-1 тотальной кДНК онкогена AML-ETO, используя модифицированный метод гнездной ПЦР с двумя парами праймеров, специфичными к данному гену ("внешние" - для 1-й стадии, и "внутренние" - для 2-й стадии). Обе стадии гнездной ПЦР.
Амплификацию проводили в следующих условиях, 1 цикл: денатурация 94°С - 30 секунд, отжиг - 30 секунд, синтез - 72°С, 1 минута. Температуру отжига определяли для каждой пары праймеров.
Для определения уровня экспрессии мРНК гена AMLl-ЕТО на
первой и второй стадиях соответственно оптимальное число циклов составляет 13 и 15 циклов.
Характеристики праймеров, использованных для анализа уровня: экспрессии интересующих генов, приведены в Таблице 1:

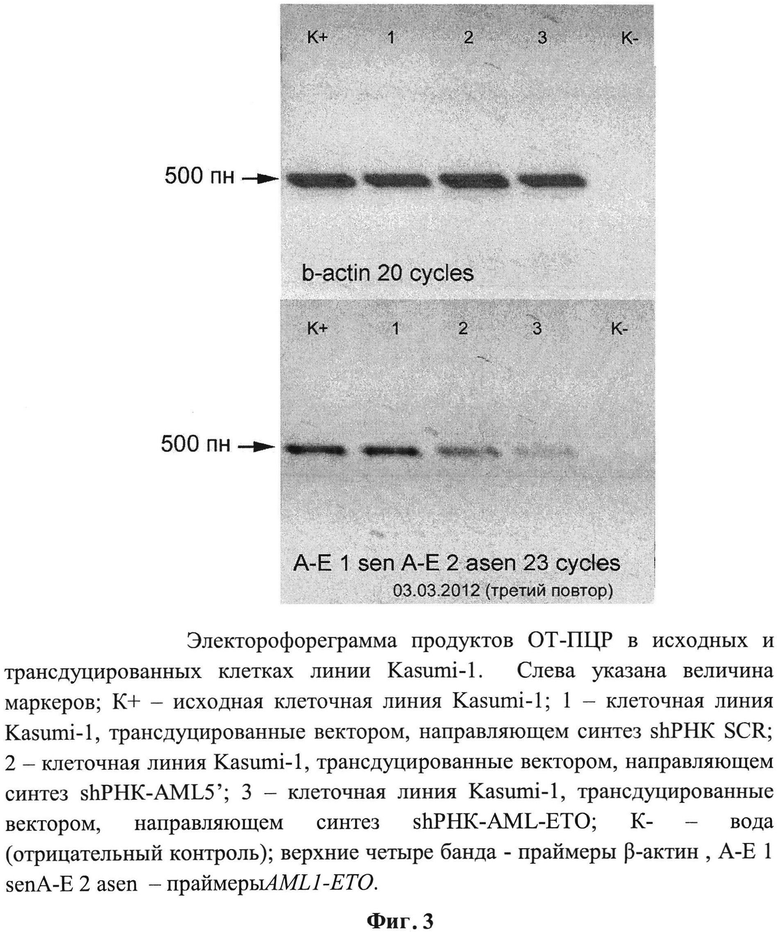
Было показано, что в клетках линии Kasumi-1, трансдуцированных лентивирусными векторами, направляющими синтез shPHK -предшественников siPHK, специфических в отношении активированного онкогена AML1-ETO осуществляется подавление экспрессии указанного онкогена, являющегося ключевым в злокачественном перерождении кроветворной ткани и ответственным за дифференцировку и в определённой степени рост клеток миелоидного ряда. На Фигуре 3
приведена электрофореграмма, иллюстрирующая значительное снижение количества мРНК AML1-ETO в клетках линии Kasumi-1, по сравнению с контрольными клетками, трансдуцированными лентивирусными векторами, направляющими синтез неспецифической в отношении какого либо из генов человека shPHK SCR.
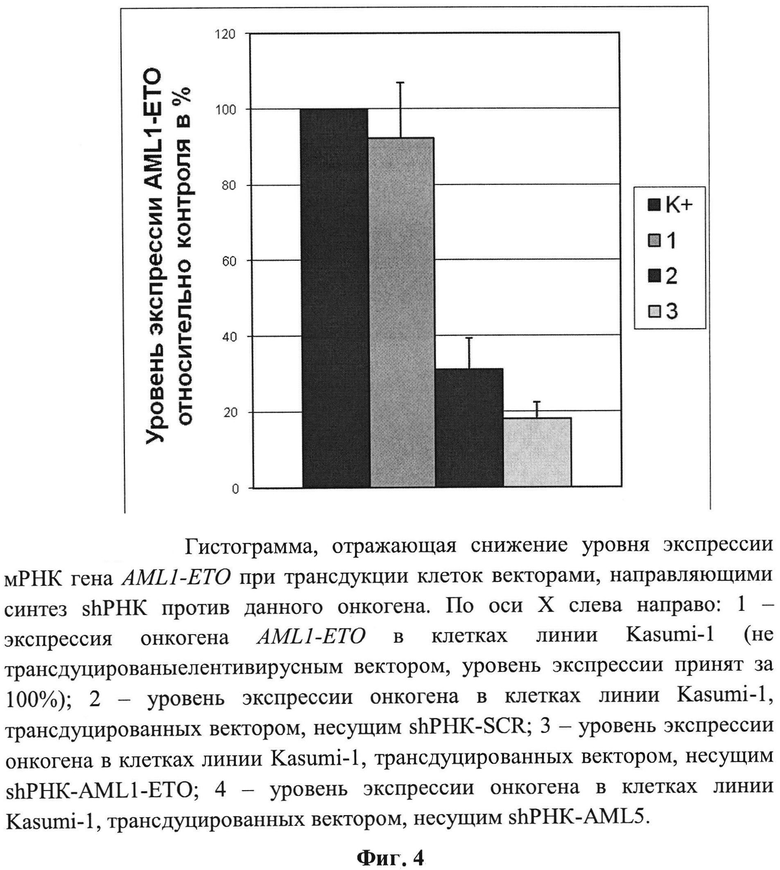
На гистограмме (Фигура 4)
представлены средние значения, полученные из трёх независимых экспериментов.
Синтез шпилечных конструкций shPHK-AMLl-ETO или shPHK-AML5' приводит к снижению уровня экспрессии AML1-ETO в 3 и 5 раз соответственно по сравнению с контролем. В качестве контроля были использованы клетки Kasumi-1, трансдуцированные лентивирусным вектором, направляющем синтез shPHK SCR, не имеющей гомологии с мРНК человека и мыши.
ПРИМЕР 2
Анализ экспрессии генов-мишеней, активность которых контролируется активированными онкогенами
Функциональный эффект, оказываемый подавлением экспрессии активированного онкогена AML1-ETO. В качестве оценочного критерия выбрана скорость роста клеток линии Kasumi-1. Было проведено сравнение скорости роста клеток, трансдуцированных лентивирусным вектором, направляющим специфические шпилечные конструкции, специфические в отношении исследуемого онкогена по сравнению с контрольным образцом клеток, трансдуцированных лентивирусным вектором, направляющем синтез неспецифической shPHK SCR.
Исходные клетки линии Kasumi-1, трансдуцированные shPHK SCR, shPHK AML5' или shPHK-AMLl-ETO, высевают в 96 луночные планшеты в концентрациях 4000, 2000, 1000, .500, 250 и 125 клеток на лунку в 100 мкл среды. Эксперимент осуществляют в трёх повторах. Клетки выращивают на
стандартной среде RPMI 1640, содержащей 20% эмбриональной сыворотки, 4 мМ L-глутамина, стрептомицин/пенициллин в концентрации 100 мкг/мл и 100 ед/мл, соответственно, рН 6,8-7,0 при температуре 37°С в атмосфере 5% СО2. В течение 12 дней после посадки, каждые 2 дня, клетки в лунках ресуспендируют и отбирают по 5 мкл для подсчёта количества клеток в каждой лунке. На протяжении 12 дней контролируют количество среды в каждой лунке на уровне 100 мкл.
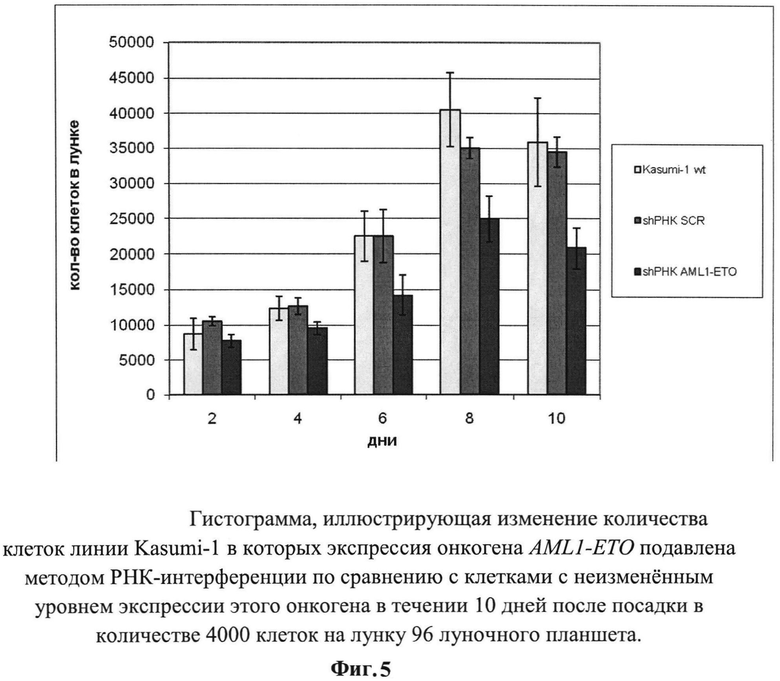
На Фигуре 5
приведена диаграмма, иллюстрирующая изменение количества клеток в лунках 96 луночного планшета в течение 10 дней при начальной плотности посадки 4000 клеток на лунку. Представленные результаты получены из трёх независимых экспериментов (р<0,05).
Видно, что скорость пролиферации клеток Kasumi-1, в которых экспрессия эндогенного AML1-ETO подавлена с помощью shPHK-AMLl-ЕТО, значительно ниже, по сравнению с исходными клетками Kasumi-1 или с клетками, в которых происходит синтез shPHK-SCR, не специфичной в отношении какой либо из мРНК человека или мыши.
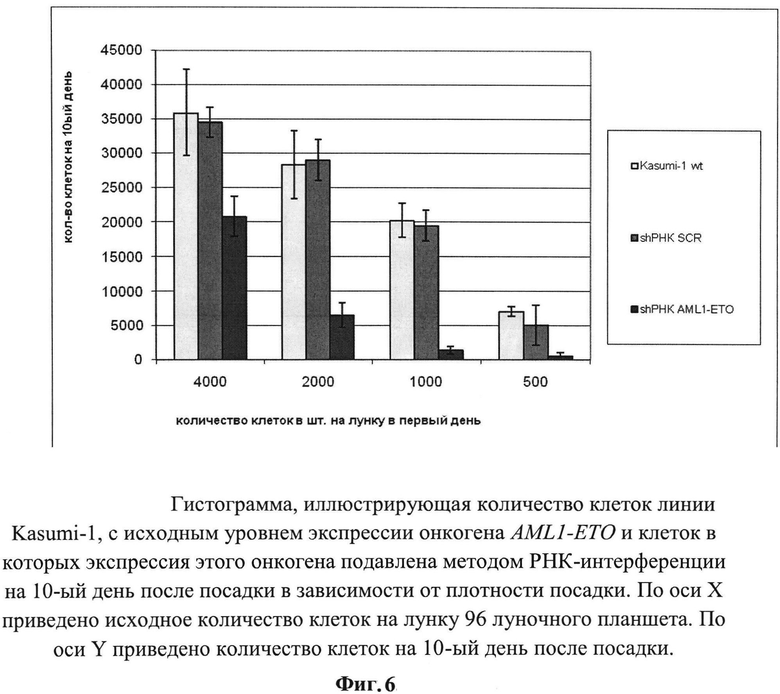
На Фигуре 6
представлена гистограмма, иллюстрирующая изменение количества клеток в лунках на десятый день после посадки в зависимости от плотности посадки. Представленные результаты получены из трёх независимых экспериментов (р<0,05).
Видно, что при посадке по 4000 на лунку клеток Kasumi-1, в которых экспрессия AML1-ETO подавлена, их количество в 1,7 раз ниже, чем клеток, в которых уровень экспрессии онкогена не был изменён. При плотности посадки 2000 шт на лунку и 1000 шт на лунку эти соотношения составили 4,5 и 14 раз. Показано, что при плотности посадки клеток Kasumi-1 с подавленной экспрессией AML1-ETO ниже 1000 шт на лунку, их рост совсем не происходит, в то время как клетки с неизменённым уровнем экспрессии онкогена способны расти при относительно редкой плотности посадки в 500 шт. на лунку и даже 250 шт на лунку.
Приведённые результаты свидетельствуют о том, что подавление экспрессии онкогена AML1-ETO в клетках Kasumi-1 приводит к значительному снижению их скорости пролиферации, а также значительно снижает их способность давать клоны и расти при низких плотностях посадки относительно исходных клеток Kasumi-1 с неизменённым уровнем экспрессии онкогена AML1-ETO.
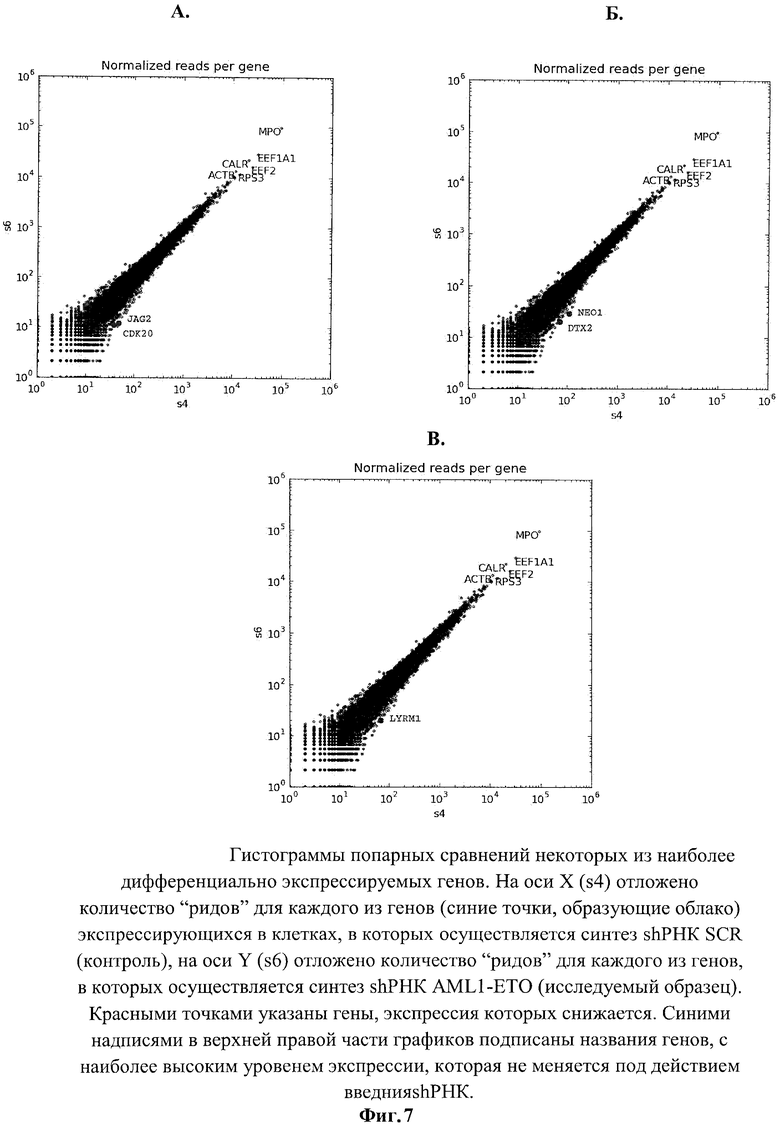
Для поиска генов-мишеней, находящихся под контролем активированного онкогена AML1-ETO, которые могут быть потенциальными генами-мишенями при разработке новых подходов для диагностики и терапии ОМЛ, был использован метод глубокого секвенирования на базе платформы Illumina. Для этого осуществляют выделение мРНК из клеток, трансдуцированных лентивирусными векторами, направляющими синтез shPHK, и синтез цепи кДНК, как описано выше. Анализ полученных образцов кДНК осуществляют с помощью метода глубокого секвенирования на базе платформы Illumina. Для каждого гена был выбран один наиболее представленный транскрипт. Для сортировки генов по уровню дифференциальной экспрессии использовали стандартное отклонение нормализованной экспрессии. Нормализация была осуществлена так, чтобы сумма попаданий для каждого образца была равна этой сумме в контрольном образце (кДНК из клеток, трансдуцированных лентивирусным вектором, направляющим синтез shPHK SCR). Гены были отсортированы по уровню дифференциальной экспрессии.
На Фигуре 7
(А,Б,В) приведены примеры гистограмм попарных сравнений некоторых из наиболее дифференциально экспрессируемых генов, экспрессия которых изменяется в результате подавления активированного онкогена AML1 -ЕТО.
В Таблице 2
представлен перечень всех генов, экспрессия которых меняется при подавлении активированного онкогена AML1-ETO в клетках линии Kasumi-1. В таблице представлено 5 столбцов, первый из которых содержит идентификационный номер гена, во втором столбце приведено краткое наименование гена, в третьем столбце приведено количество "ридов" для данного гена в клетках, трансдуцированных лентивирусным вектором, направляющем синтез shPHK SCR, в четвёртом столбце количество "ридов" для данного гена в клетках, трансдуцированных лентивирусным вектором, направляющем синтез shPHK AML1-ETO, в пятом столбце приведено отношение числа "ридов" из третьего столбца на число "ридов" из четвёртого столбца для данного гена. Это соотношение иллюстрирует снижение уровня экспрессии обнаруженного гена относительно контроля (клеток, трансдуцированных лентивирусным вектором, направляющем синтез shPHK SCR) при подавлении активированного онкогена AML1-ETO.
Таблица 2
ID
ген
shSCR
shAE
shSCR/shAE
NM 144708
ANKAR
28
1
28,0
NM 182689
EFNA4
28
1
28,0
NM 153631
HOXA3
27
1
27,0
NM 004981
KCNJ4
21
1
21,0
NM 000606
C8G
19
1
19,0
NM 001171136 1
ZBTB12
18
1
18,0
NM 004943
DMWD
33
16,5
NR 039856
MIR4707
16
1
16,0
NM 003890
FCGBP
15
1
15,0
NM 025211
GKAP1
15
1
15,0
NM 021135
RPS6KA2
15
1
15,0
NM 005775
SP6
15
1
15,0
NM 001166339
SPDYE5
13
1
13,0
NM 001023561
ZNF774
13
1
13,0
NM 001101426
ISPD
25
12,5
NM 001003674
C18orf1
12
1
12,0
NM 016210
C3orf18
12
1
12,0
NM 033510
DISP2
24
12,0
NR 026771
DKFZP434L187
12
1
12,0
NM 001167857
PPP1R12B
12
1
12,0
NM 130795
RGS3
24
2
12,0
NM 001636
SLC26A1
24
2
12,0
NM 017575
SMPD3
12
1
12,0
NM 033394
TAP1
24
2
12,0
NM 006297
YAF2
36
3
12,0
NM 001122962
SIRPB2
22
2
11,0
NM 198216
SNTA1
43
4
10,8
NM 001975
EN02
10
1
10,0
NR 027051
FLJ39582
10
1
10,0
NR 004390
SNORD110
10
1
10,0
NR 000021
SNORD5
10
1
10,0
NM 032786
ZDHHC1
10
1
10,0
NM 001010880
ZNF782
31
3
10,3
NM 001195605
ZNRD1
10
1
10,0
NM 005683
GPR55
30
3
10,0
NM 021184 4
C6orf47
19
2
9,5
NM 001142966
GREB1L
19
2
9,5
NM 198850
PHLDB3
19
2
9,5
NR 037918
PRH1-PRR4
19
2
9,5
NM 001161440
PTPRH
19
2
9,5
NM 017836
SLC44A2
19
2
9,5
NM 138448
ACYP2
28
3
9,3
NM 152240
ZMAT5
46
5
9,2
NM 033031
CCNB3
63
7
9,0
NR 024009
CN5H6.4
9
1
9,0
NM 000106
CYP2D6
18
2
9,0
NM 207305
FOXD4
18
2
9,0
NM 001004056
GRK4
9
1
9,0
NM 172193
KLHDC1
9
1
9,0
NM 203397
MBLAC1
9
1
9,0
NR 039901
MIR4746
9
1
9,0
NR 030368
MIR638
9
1
9,0
NM 024302
MMP28
9
1
9,0
NM 001204295
MUC1
9
1
9,0
NM 020904
PLEKHA4
9
1
9,0
NM 020780
PTCHD2
18
2
9,0
NM 000355
TCTEX1D4
9
1
9,0
NM 003969
UBE2Q2P2
9
1
9,0
NM 001001974
PLEKHA1
34
4
8,5
NR 037428
MIR3655
25
3
8,3
NM 001033082
MYCL1
25
3
8,3
NM 014686
KIAA0355
42
5
8,4
NM 022842
CDCP1
16
2
8,0
NM 032727
INA
33
4
8,3
NR 031576
MIR762
16
2
8,0
NM_007162
TFPI
16
2
8,0
NM 024807
TRIM16L
16
2
8,0
NM 001199161
USP6
33
4
8,3
NM 017715
ZNF319
66
8
8,3
NM 145271
ZNF701
16
2
8,0
NM 024906
SCD5
24
3
8,0
NM 001197129
SYS1-DBNDD2
24
3
8,0
NM 000407
GP1BB
31
4
7,8
NM 173201
ATP2A1
15
2
7,5
NR 027129
C21orf67
15
2
7,5
NM 001098808
C9orf129
30
4
7,5
NM 001242369
C9orf7
15
2
7,5
NM 015557
CHD5
15
2
7,5
NM 001166346
MDFIC
37
5
7,4
NR 033851
MGC39372
15
2
7,5
NM 020201
NT5M
30
4
7,5
NM 022463
NXN
15
2
7,5
NM 006418
OLFM4
15
2
7,5
NM 017439
PION
15
2
7,5
NM 002702
POU6F1
60
8
7,5
NM 153336
PSTK
22
3
7,3
NM 134428
RFX3
15
2
7,5
NM 014496
RPS6KA6
15
2
7,5
NR 003010
SCARNA12
15
2
7,5
NR 002588
SNORA51
15
2
7,5
NM 016639
TNFSF14
15
2
7,5
NM 001173513
TYMP
22
3
7,3
NM 001102664
EPN2
81
11
7,4
NM 033120
NKD2
72
10
7,2
NM 007122
USP18
36
5
7,2
NM 001141936
C4orf48
28
4
7,0
NM 001040113
MYH11
49
7
7,0
NR 028330
C15orf37
21
3
7,0
NM 001039508
SIRPG
21
3
7,0
NM 001080523
ARRDC5
13
2
6,5
NM 152792
ASPRV1
13
2
6,5
NM 001012279
C6orf174
13
2
6,5
NM 017525
CDC42BPG
13
2
6,5
NR 003529
CDKN2B-AS1
13
2
6,5
NM 033641
COL4A6
13
2
6,5
NM 001144962 1
NFKBIL1
13
2
6,5
NM 001017989
OPA3
13
2
6,5
NM 194248
OTOF
13
2
6,5
NM 178518
TMEM107
27
4
6,8
NM 138396
MARCH9
46
7
6,6
NR 027861
ZNF664-FAM101A
33
5
6,6
NR 038190 3
AGER
19
3
6,3
NM 001145951
TIPARP-AS1
19
3
6,3
NM 001040697
UGT3A2
19
3
6,3
NM 022553 2
VSIG10L
19
3
6,3
NM 014322
OPN3
25
4
6,3
NM 001775
CD38
82
13
6,3
NM 133463
AMZ1
31
5
6,2
NM 002542
OGG1
31
5
6,2
NM 181843
NUDT8
100
16
6,3
NM 001164688
RD3
37
6
6,2
NM 207340
ZEB1-AS1
37
6
6,2
NM 016466
ANKRD39
43
7
6,1
NM 004043 1
ASMT
12
2
6,0
NM 005172
ATOH1
12
2
6,0
NM 145178
ATOH7
12
2
6,0
NM 001008223
C1QL4
12
2
6,0
NM 007337
DLEC1
12
2
6,0
NR 002834
DUSP5P
12
2
6,0
NM 023933
FAM173A
24
4
6,0
NM 182759
FAM19A3
12
2
6,0
NM 005803 2
FLOT1
30
5
6,0
NM 005309
GPT
24
4
6,0
NM 014234 5
HSD17B8
18
3
6,0
NM 212551
LYSMD1
24
4
6,0
NM 020932
MAGEE1
12
2
6,0
NM 018670
MESP1
12
2
6,0
NM 000253
MTTP
12
2
6,0
NM 024927
PLEKHH3
12
2
6,0
NM 080923
PTPRC
36
6
6,0
NM 032709
PYROXD2
12
2
6,0
NM 000334
SCN4A
12
2
6,0
NM 006080
SEMA3A
12
2
6,0
NM 003975
SH2D2A
18
3
6,0
NM 138356
SHF
12
2
6,0
NM 139157
ST7-AS2
12
2
6,0
NM 003807
TNNI3
12
2
6,0
NM 001142646
TPSB2
18
3
6,0
NM 001080461
UPP1
12
2
6,0
NM 001128833
ZBTB42
24
4
6,0
NM 021915
ZNF703
12
2
6,0
NM 198540
B3GNT8
106
18
5,9
NM 000054
AVPR2
34
6
5,7
NM 003587 1
DHX16
34
6
5,7
NM 032090
PCDHGA10
34
6
5,7
NM 001172668
ZNF688
34
6
5,7
NR 026811 1
AGSK1
57
10
5,7
NR 026680
MGC57346
28
5
5,6
NM 133475
ANKRD24
22
4
5,5
NM 001855
COL15A1
22
4
5,5
NM 145315
LACE1
22
4
5,5
NR 037421
MIR3648
90
16
5,6
NM 020979
SH2B2
22
4
5,5
NM 032296
FLYWCH1
94
17
5,5
NM 001887
CRYBB1
16
3
5,3
NM 004409
DMPK
33
6
5,5
NR 033968
GUSBP9
16
3
5,3
NR 036751
HSP90AA6P
16
3
5,3
NM 032452
JPH4
16
3
5,3
NR 028135
KCNMB3
16
3
5,3
NR 039802
MIR4658
16
3
5,3
NM 001083
PDE5A
16
3
5,3
NM 178314
RILPL1
16
3
5,3
NM 000934
SERPINF2
33
6
5,5
NM 024676
SH3D21
33
6
5,5
NM 017844
ANKMY1
60
11
5,5
NM 032370
ZNF469
120
22
5,5
NM 001134876
C14orf80
130
24
5,4
NR 002936
TOM1L2
70
13
5,4
NM 181598
ATL1
27
5
5,4
NM 001083537
FAM86B1
27
5
5,4
NM 007085
FSTL1
27
5
5,4
NM 002183 1
IL3RA
27
5
5,4
NM 001105199
TMEM180
54
10
5,4
NM 000099
CST3
183
34
5,4
NM 080387
CLEC4D
37
7
5,3
NR 024071
PLCD1
37
7
5,3
NM 003794
SOBP
37
7
5,3
NM 032731
TXNRD3
75
14
5,4
NM 001134870 2
KIAA1949
121
23
5,3
NM 002910
RENBP
142
27
5,3
NR 026711 1
ASMTL-AS1
21
4
5,3
NM 130851
BMP4
21
4
5,3
NR 026906 1
C17orf69
10
2
5,0
NM 001374
DNASE1L2
10
2
5,0
NM 017820
EXD3
10
2
5,0
NM 013445
GAD1
21
4
5,3
NM 003516
HIST2H2AA3
10
2
5,0
NM 001242525 2
HLA-DPA1
10
2
5,0
NM 002192
INHBA
10
2
5,0
NM 178863
KCTD13
52
10
5,2
NM 001199659
LETM2
21
4
5,3
NM 002315
LM01
10
2
5,0
NM 001161573
MAFF
21
4
5,3
NM 130385
MRVI1
10
2
5,0
NR 033343
PSCA
10
2
5,0
NM 001024938
SLC2A14
10
2
5,0
NM 001082968
TOMM20L
10
2
5,0
NM 001009958
ZNF658B
21
4
5,3
NM 001159293
ZNF75A
10
2
5,0
NM 024876
ADCK4
93
18
5,2
NM 024785
FAM124B
46
9
5,1
NM 032777
GPR124
108
21
5,1
NM 001098531
RAPGEF3
36
7
5,1
NM 031232
NECAB3
61
12
5,1
NM 130759
GIMAP1
25
5
5,0
NM 018013
SORBS3
25
5
5,0
NM 080678
UBE2Q2P1
25
5
5,0
NM 005773
ZNF296
51
10
5,1
NM 001010877 3
ZNF321P
66
13
5,1
NM 021630
PDLIM2
96
19
5,1
NM 001136528
SERPINE2
55
11
5,0
NM 152783
D2HGDH
115
23
5,0
NM 007053
CD160
15
3
5,0
NM 145019
FAM124A
15
3
5,0
NM 024642
GALNT12
15
3
5,0
NM 145910
NEK11
15
3
5,0
NM 016223
PACSIN3
75
15
5,0
NM 001242319
PNMA6D
15
3
5,0
NM 030907
RSG1
30
6
5,0
NM 001161331
ST20
15
3
5,0
NM 024786
ZDHHC24
148
30
4,9
NM 021047
ZNF256
49
10
4,9
NM 020807
ZNF323
49
10
4,9
NM 001170820
IFITM10
34
7
4,9
NM 014055
IFT81
34
7
4,9
NM 024681
KCTD17
34
7
4,9
NM 145232
CTU1
54
11
4,9
NM 014694
ADAMTSL2
19
4
4,8
NM 181506
LRRC70
19
4
4,8
NR 026808
NCRNA00321
39
8
4,9
NM 000290
PGAM2
19
4
4,8
NM 003709
KLF7
43
9
4,8
NM 001145784
MEF2BNB
67
14
4,8
NM 014787
DNAJC6
24
5
4,8
NM 139245
PPM1L
24
5
4,8
NM 004159 5
PSMB8
48
10
4,8
NM 001098612
SIGLEC14
24
5
4,8
NM 006470
TRIM22
48
10
4,8
NR 026575
GK3P
28
6
4,7
NM 005994
TBX3
61
13
4,7
NM 001964
EGR1
33
7
4,7
NM 001101391
LING03
66
14
4,7
NM 001143821
PLEKHA5
33
7
4,7
NM 182907
PRDM1
33
7
4,7
NM 002904
RDBP
33
7
4,7
NM 020740
ANKFY1
37
8
4,6
NM 001185101
CD22
42
9
4,7
NM 012446
SSH1
46
10
4,6
NM 004163
RAB27B
199
43
4,6
NM 001242416
WDR25
51
11
4,6
NM 004029
IRF7
96
21
4,6
NM 001010887
ACER2
22
5
4,4
NM 138430
ADPRHL1
27
6
4,5
NM 001190724 4
ATAT1
9
2
4,5
NM 001141972
ATPBD4
22
5
4,4
NM 174896
C1orf162
9
2
4,5
NR 026784
C6orf164
9
2
4,5
NM012298
CAND2
9
2
4,5
NM 000732
CD3D
13
3
4,3
NM 139158
CDK15
13
3
4,3
NM 153221
CILP2
9
2
4,5
NM 206833
CTXN1
9
2
4,5
NM 013974 6
DDAH2
18
4
4,5
NR 040063
DKFZP564C196
9
2
4,5
NR 026855
DKFZP686I15217
18
4
4,5
NM 018163
DNAJC17
13
3
4,3
NM 032281
ELAVL3
9
2
4,5
NM 199280
FAM179A
22
5
4,4
NM 001164379
FAM180B
9
2
4,5
NM 001099338
FAM22A
31
7
4,4
NM 017556
FBLIM1
9
2
4,5
NM 145032
FBXL13
13
3
4,3
NR 024101
FLJ35776
9
2
4,5
NM 148899
FOXP2
13
3
4,3
NM 181703
GJA5
9
2
4,5
NM 033177
GPANK1
45
10
4,5
NM 006149
LGALS4
9
2
4,5
NM 001015002
LLGL2
9
2
4,5
NM 005572
LMNA
45
10
4,5
NM 023948
MOSPD3
22
5
4,4
NM 004540
NCAM2
9
2
4,5
NM 201539
NDRG2
9
2
4,5
NM 005386
NNAT
9
2
4,5
NM 006172
NPPA
9
2
4,5
NM 018930
PCDHB10
9
2
4,5
NM 001200053
PMEL
9
2
4,5
NM 014906
PPM1E
9
2
4,5
NM 006663
PPP1R13L
9
2
4,5
NM 004283
RAB3D
22
5
4,4
NM 173587
RCOR2
9
2
4,5
NM 001036
RYR3
9
2
4,5
NM 001204204
SEC14L2
18
4
4,5
NM 004155
SERPINB9
36
8
4,5
NM 003041
SLC7A10
9
2
4,5
NR 004380
SNORD32A
9
2
4,5
NM 201441
TEX22
9
2
4,5
NR 037646
TNFRSF12A
18
4
4,5
NM 001024843
TOB2P1
58
13
4,5
NM 021138
TRAF3IP2-AS1
22
5
4,4
NM 133462
TTC16
9
2
4,5
NM 001039783
TYR03P
9
2
4,5
NM 198129
LAMA3
159
36
4,4
NM 001171133
FAM3A
132
30
4,4
NM 005072
SLC12A4
57
13
4,4
NM 015177
DTX4
52
12
4,3
NM 144596
TTYH3
135
31
4,4
NM 001013693
LDLRAD2
178
41
4,3
NM 015916
CALHM2
117
27
4,3
NM 001002837
INPP5J
34
8
4,3
NM 020418
PCBP4
34
8
4,3
NM 016044
FAHD2A
64
15
4,3
NM 004753
DHRS3
94
22
4,3
NM 006820
IFI44L
30
7
4,3
NM 001135704
ACBD4
25
6
4,2
NR 002710
ALOX12P2
25
6
4,2
NM 014417
BBC3
25
6
4,2
NM 001178135
HDHD1
25
6
4,2
NM 002207
ITGA9
51
12
4,3
NM 001100425
KIAA0895
51
12
4,3
NR 030407
MIR671
51
12
4,3
NM 020229
PRDM11
25
6
4,2
NM 001199577
GIMAP1-GIMAP5
123
29
4,2
NM 012458
TIMM8A
160
38
4,2
NM 018132
CENPQ
88
21
4,2
NM 006889
CD86
21
5
4,2
NM 017803
DUS2L
63
15
4,2
NM 138690
GRIN3B
21
5
4,2
NM 024042
METRN
21
5
4,2
NM 001195628
MLLT10
42
10
4,2
NR 003109
TRO
21
5
4,2
NM 005685
GTF2IRD1
163
39
4,2
NM 006332
IFI30
79
19
4,2
NM 012225
NUBP2
196
47
4,2
NM 021996
GBGT1
58
14
4,1
NM 001135005
DNAJB5
37
9
4,1
NM 021641
ADAM 12
16
4
4,0
NR 027833
APOL3
16
4
4,0
NM 198545
C1orf187
16
4
4,0
NM 014143
CD274
16
4
4,0
NM 001111319
CLDN22
16
4
4,0
NM 152536
FGD5
16
4
4,0
NR 038275
FLJ31813
16
4
4,0
NM 001517
GTF2H4
16
4
4,0
NR 027386
GUSBP3
16
4
4,0
NM 006896
HOXA7
16
4
4,0
NM 005931 3
MICB
16
4
4,0
NM 007243 4
NRM
16
4
4,0
NM 001101401
SBK2
16
4
4,0
NM 001037
SCN1B
16
4
4,0
NM 004858
SLC6A6
49
12
4,1
NM 001199760
ST5
16
4
4,0
NM 198517
TBC1D19
16
4
4,0
NM 015023
WNK2
49
12
4,1
NM .014699
ZNF653
16
4
4,0
NM 007155
ZSCAN12P1
16
4
4,0
NM 002528
NTHL1
61
15
4,1
NM 199285
PRR19
61
15
4,1
NM 002535
OAS2
45
11
4,1
NM 003820
TNIK
73
18
4,1
NM 053050
MRPL53
159
39
4,1
NM 012420
IFIT5
28
7
4,0
NM 018689
KIAA1199
28
7
4,0
NM 001142299
SRCRB4D
28
7
4,0
NM 033093
TRIM71
28
7
4,0
NM 001190810 1
AGAP9
40
10
4,0
NM 004267
CHST2
40
10
4,0
NR 024034
KIAA0664L3
81
20
4,1
NM 001204872
NPEPL1
202
50
4,0
NM 001017530
PRR5
40
10
4,0
NM 001128635 1
RIMBP3B
40
10
4,0
NM 145159
JAG2
52
13
4,0
NM 018384
GIMAP5
117
29
4.0
NM 006602
TCN2
64
16
4,0
NM 199165
ADSSL1
12
3
4,0
NM 005582
CD 180
24
6
4,0
NM 206918
DEGS2
12
3
4,0
NM_ 174899
FBX036
12
3
4,0
NM 005897
IPP
24
6
4,0
NR 024061
LOH12CR2
12
3
4,0
NM 002356
MARCKS
24
6
4,0
NR 024322
MSL3P1
36
9
4,0
NM 006212
PFKFB2
84
21
4,0
NM 002632
PGF
12
3
4,0
NM 032882
PNMA6A
12
3
4,0
NM 002800 2
PSMB9
12
3
4,0
NM 144770
RBM11
12
3
4,0
NM 017452
STK19
12
3
4,0
NM 172209 4
TAPBPL
60
15
4,0
NM 006858
TMEM102
108
27
4,0
NM 001145635
TRIM47
12
3
4,0
NM 003443
ZBTB22
24
6
4,0
NM 001170640
CDK20
43
11
3,9
NM 007220
CA5B
75
19
3,9
NM 032421
CLIP2
75
19
3,9
NM 032836
FIZ1
75
19
3,9
NM 001146175
ZNF462
31
8
3,9
NM 001042601
TTC15
177
45
3,9
NM 001014979
C16orf93
51
13
3,9
NM 152320
ZNF649
51
13
3,9
NM 003507
FZD7
160
41
3,9
NM 001039651 5
C6orf26
19
5
3,8
NM 032961
PCDH10
19
5
3,8
NM 001145199
C12orf75
27
7
3,9
NM 001223
CASP1
27
7
3,9
NM 001128613
NUDT22
61
16
3,8
NM 002648
PIM1
61
16
3,8
NM 003830
SIGLEC5
96
25
3,8
NM 001206665
IGSF8
138
36
3,8
NM 020817
KIAA1407
34
9
3,8
NM 005275 2
GNL1
76
20
3,8
NM 007283
MGLL
84
22
3,8
NM 001172624
NE01
126
33
3,8
NM 031909
C1QTNF4
91
24
3,8
NM 003484
HMGA2
49
13
3,8
NM 001102594
DTX2
79
21
3,8
NM 001031827 1
BOLA2
87
23
3,8
NM 001039182 1
BOLA2B
87
23
3,8
NM 000755
CRAT
132
35
3,8
NM 153697
ANKRD44
15
4
3,8
NM 020811
CARNS1
15
4
3,8
NM 013246
CLCF1
15
4
3,8
NM 001882
CRHBP
15
4
3,8
NM 005232
EPHA1
90
24
3,8
NM 145059
FUK
60
16
3,8
NM 022041
GAN
67
18
3,7
NM 002048
GAS1
15
4
3,8
NR 036632
GOLGA2B
22
6
3,7
NM 001004439
ITGA11
15
4
3,8
NM 139248
LIPH
15
4
3,8
NM 001025101
MBP
75
20
3,8
NR 026902
MGC16142
15
4
3,8
NR 040072
NDRG4
22
6
3,7
NM 012387
PADI4
15
4
3,8
NM 001195263
PDZD7
45
12
3,8
NM 001080492
PRSS54
15
4
3,8
NM 213613
SLC29A4
15
4
3,8
NM 000455
STK32C
15
4
3,8
NM 001144923
TTC28-AS1
37
10
3,7
NM 198310
TUBB2A
52
14
3,7
NM 001039886
ZNF837
22
6
3,7
NM 001128302
LYRM1
78
21
3,7
NM 001129979
SYNE1
70
19
3,7
NM 001114185
MVK
48
13
3,7
NM 000920
PC
154
42
3,7
NM 001122674
ABCD3
33
9
3,7
NM 001173539
BAN Ρ
33
9
3,7
NM 199248
CACNB1
33
9
3,7
NM 052843
OBSCN
99
27
3,7
NM 194250
ZNF816-ZNF321P
66
18
3,7
NM 032290
ANKRD32
124
34
3,6
NM 014726
TBX2
58
16
3,6
NM 031213
FAM108A1
135
37
3,6
NM 001005855
ATP8B2
25
7
3,6
NM 173550
C9orf93
51
14
3,6
NM 006531
IFT88
25
7
3,6
NM 178496
MB21D2
51
14
3,6
NR 037429
MIR3656
25
7
3,6
NM 031430
RILP
51
14
3,6
NM 080861
SPTBN4
25
7
3,6
NM 001025616
ARHGAP24
43
12
3,6
NM 058195
CDKN2A
217
60
3,6
NR 033742
FKSG43
43
12
3,6
NM 018485
GPR77
43
12
3,6
NM 003259
ICAM5
61
17
3,6
NM 006995
BTN2A2
97
27
3,6
NM 004110
FDXR
97
27
3,6
NM 138420
AHNAK2
126
35
3,6
NM 152326
ANKRD9
36
10
3,6
NM 003568
ANXA9
54
15
3,6
NM 004925
AQP3
18
5
3,6
NM 032266
C2orf16
18
5
3,6
NM 016243
CYB5R1
36
10
3,6
NM 001136265
IFF02
54
15
3,6
NM 014994
MAPKBP1
54
15
3,6
NR 031592
MIR1181
18
5
3,6
NM 207127
PAOX
18
5
3,6
NM 015149
RGL1
18
5
3,6
NM 001080496
RGP1
54
15
3,6
NR 002312
RPPH1
36
10
3,6
NM 015278
SASH1
72
20
3,6
NM 001099435
SPINT1
72
20
3,6
NM 003608
GPR65
118
33
3,6
NM 004269
MED27
82
23
3,6
NM 024108
TREML2
82
23
3,6
NM 001170957
SMO
64
18
3,6
NM 002663
PLD2
46
13
3,5
NM 001242788
BRF1
150
42
3,6
NM_003560
PLA2G6
103
29
3,6
NM 005481
MED16
349
98
3,6
NM_003916
AP1S2
28
8
3,5
NM 004952
EFNA3
28
8
3,5
NM_014872
ZCWPW1
57
16
3,6
NM 033375
MY01C
39
11
3,5
NM 001171091
UEVLD
49
14
3,5
NM 000584
IL8
172
49
3,5
NM 000154
GALK1
274
78
3,5
NM 001145128
AKD1
10
3
3,3
NM 001109938 6
C6orf136
10
3
3,3
NM 006566
CD226
10
3
3,3
NR 023386
CROCCP3
10
3
3,3
NR 037860 2
EGFL8
10
3
3,3
NM 025193
HSD3B7
10
3
3,3
NM 003866
INPP4B
31
9
3,4
NR 031598
MIR1229
10
3
3,3
NR 036199
MIR4315-1
10
3
3,3
NR 036271
MIR4315-2
10
3
3,3
NR 030369
MIR639
10
3
3,3
NM 001114132
NBEAL1
52
15
3,5
NM 178140
PDZD2
10
3
3,3
NM 001190728
RALGPS1
10
3
3,3
NR 033396
SLC22A20
10
3
3,3
NM 001199693
SLC4A8
10
3
3,3
NM 032038
SPSB2
42
12
3,5
NM 033278
TRIM39
21
6
3,5
NM 021253 5
TRIM39-RPP21
10
3
3,3
NM 152736
ZNF200
129
37
3,5
NM 001204299
ZNF668
76
22
3,5
NM 020703
AMIG01
66
19
3,5
NM 024816
RABEP2
66
19
3,5
NM 001134492
HS2ST1
121
35
3,5
NM 001170881
GPR137
45
13
3,5
NM 006929 3
SKIV2L
45
13
3,5
NR 002451
ABCA11P
103
30
3,4
NM 004913
C16orf7
103
30
3,4
NM 152742
GPC2
34
10
3,4
NM 004761 3
RGL2
34
10
3,4
NM 000048
ASL
93
27
3,4
NM 001204240
TBC1D16
151
44
3,4
NM 001200001
NOTCH2
327
95
3,4
NM 001042432
CLN3
58
17
3,4
NM 001172780
LRRC34
58
17
3,4
NM 130786
A1BG
24
7
3,4
NM 014885
ANAPC10
48
14
3,4
NM 001723
DST
24
7
3,4
NM 004463
FGD1
48
14
3,4
NM 022107 4
GPSM3
24
7
3,4
NM 001160166
HRH4
24
7
3,4
NM 000428
LTBP2
24
7
3,4
NR 003189
SYTL3
24
7
3,4
NM 007261
CD300A
123
36
3,4
NM 058180
C21orf58
150
44
3,4
NM 014675
CROCC
112
33
3,4
NM 006519
DYNLT1
75
22
3,4
NM 002926
RGS12
265
78
3,4
NM 002333
LRP3
295
87
3,4
NM 152504
C20orf196
78
23
3,4
NM 005246
FER
78
23
3,4
NM 001173474 1
ASMTL
91
27
3,4
NM 145691
ATPAF2
67
20
3,4
NM 001012502
C9orf117
13
4
3,3
NM 024705
DHRS12
27
8
3,4
NR 034098
EML2
54
16
3,4
NM 001001794
FAM116B
13
4
3,3
NM 032301
FBXW9
40
12
3,3
NR 015369
FLJ42709
13
4
3,3
NM 207647
FSD1L
40
12
3,3
NM 174942
GAS2L3
13
4
3,3
NM 178231
ICA1L
13
4
3,3
NM 002204
ITGA3
27
8
3,4
NM 153487
MDGA1
13
4
3,3
NM 175058
PLEKHA7
13
4
3,3
NM 181808
POLN
13
4
3,3
NM 181882
PRX
40
12
3,3
NM 032167
RUNDC2A
13
4
3,3
NR 033801
SLFNL1
13
4
3,3
NM 178509
SUN1
67
20
3,4
NM 001042463
TMEM92
27
8
3,4
NM 031434
TNFRSF11A
13
4
3,3
NM 015516
TSPAN15
67
20
3,4
NM 007191
WNT11
13
4
3,3
NM 145288
ZNF311
13
4
3,3
NM 001243241
ZNF345
27
8
3,4
NM 020928
ZYG11A
13
4
3,3
NM 001099220
ZNHIT2
124
37
3,4
NM 014806
RUSC2
181
54
3,4
NM 001164115
CASS4
70
21
3,3
NM 173540
FUT11
130
39
3,3
NM 001033045
GPR155
43
13
3,3
NM 053053
TAF13
43
13
3,3
NM 001720
BMP8B
60
18
3,3
NM 152759
LRRC43
30
9
3,3
NM 015016
MAST3
120
36
3,3
NM 024578
OCEL1
60
18
3,3
NR 024525
FAM96B
166
50
3,3
NM 033340
CASP7
106
32
3,3
NM 015157
PHLDB1
123
37
3,3
NM 016148
SHANK1
142
43
3,3
NM 001142501
MON1A
129
39
3,3
NM 080622
ABHD16B
33
10
3,3
NM 016584
IL23A
16
5
3,2
NM 001010844
IRAK1BP1
16
5
3,2
NM 001040624
NCALD
33
10
3,3
NM 017699
SIDT1
33
10
3,3
NM 213618
ST6GALNAC2
16
5
3,2
NR 002331
STAC
16
5
3,2
NM 001198526
TCHH
16
5
3,2
NM 052883
TXNRD3NB
16
5
3,2
NM 001164267 3
WDR53
132
40
3,3
NM 001214906
ZNF484
33
10
3,3
NM 006646
WBP11P1
102
31
3,3
NM 016481
C9orf156
85
26
3,3
NM 201446
EGFL7
447
136
3,3
NM 001159391
GUK1
190
58
3,3
NM 146387
MRPL4
279
85
3,3
NM 198475
FAM171A2
88
27
3,3
NM 019121
LRRC68
88
27
3,3
NM 001321
CSRP2
36
11
3,3
NM 134324
TBC1D10A
36
11
3,3
NM 007108
TCF7L2
72
22
3,3
NM 001145845
ROB01
219
67
3,3
NM 024585
ARMC7
130
40
3,3
NM 006123
IDS
75
23
3,3
NM 172251
MRPL54
189
58
3,3
NR 033488 5
ABHD16A
19
6
3,2
NM 052854
CREB3L1
19
6
3,2
NM 004137
KCNMB1
19
6
3,2
NM 004140
LLGL1
97
30
3,2
NM 058169
LOH12CR1
39
12
3,3
NM 002461
MVD
78
24
3,3
NM 019080
NDFIP2
19
6
3,2
NM 001135750
PSMG4
19
6
3,2
NR 024284
ZFHX2
19
6
3,2
NM 005529
HSPG2
844
260
3,2
NM 001003692
ZNF133
100
31
3,2
NM 183241
C9orf142
285
88
3,2
NM 018110
DOK4
187
58
3,2
NM 017679
BCAS3
126
39
3,2
NM 001064
TLCD1
42
13
3,2
NM 175630
DNMT3A
64
20
3,2
NM 001002913
PTRH1
64
20
3,2
NM 024591
CHMP6
222
69
3,2
NR 027271
C19orf23
22
7
3,1
NM 001080849
DNLZ
48
15
3,2
NM 014449
GPR162
48
15
3,2
NM 006423
RABAC1
48
15
3,2
NM 020145
SH3GLB2
121
38
3,2
NM 015681
B9D1
25
8
3,1
NM 024731
KLHL36
127
40
3,2
NM 178817
M RAP
25
8
3,1
NM 001144978
MTHFD2L
25
8
3,1
NM 016606
REEP2
25
8
3,1
NM 014442
SIGLEC8
25
8
3,1
NM 001168247
SMAGP
25
8
3,1
NM 005928
MFGE8
181
57
3,2
NM 022489
INF2
312
98
3,2
NM 001031714
INF2
312
98
3,2
NM 001198897
ACY1
105
33
3,2
NR 037505
MIR3940
54
17
3,2
NM 213598
ZNF552
54
17
3,2
NM 053064
GNG2
333
105
3,2
NM 001199580
SUV420H2
139
44
3,2
NM 001166209
SYS1
196
62
3,2
NM 015407
ABHD14A
85
27
3,1
NM 130781
RAB24
85
27
3,1
NM 018383
WDR46
57
18
3,2
NM 145249
IFI27L1
60
19
3,2
NM 032335
PHF6
91
29
3,1
NM 001008486
SLC44A1
91
29
3,1
NM 024832
RIN3
246
78
3,2
NM 001164638
ENDOV
31
10
3,1
NR 027712
PIP5K1P1
31
10
3,1
NM 018363
RNLS
63
20
3,2
NM 003239
THAP2
31
10
3,1
NM 014008
CCDC22
129
41
3,1
NM 001100418
C19orf60
97
31
3,1
NM 001733
C1R
97
31
3,1
NM 003098
SNX33
66
21
3,1
NM 006427
SIVA1
232
74
3,1
NM 001040134
PALM
166
53
3,1
NM 030945
C1QTNF3
34
11
3,1
NM 080750
DPH3P1
34
11
3,1
NM 001163213
FGFR3
34
11
3,1
NM 153835
GPR113
34
11
3,1
NM 001144925
MX1
34
11
3,1
NR 033799 2
NSFP1
34
11
3,1
NM 014596 2
ZP3
138
44
3,1
NM 005711
EDIL3
106
34
3,1
NM 033400
ZFP41
147
47
3,1
NM 001159846
MPND
78
25
3,1
NM 012121
CDC42EP4
196
63
3,1
NM 173674
DCBLD1
40
13
3,1
NM 022772
EPS8L2
40
13
3,1
NM 001099280
HEATR7A
40
13
3,1
NM 016619
PLAC8
81
26
3,1
NM 004920
AATK
124
40
3,1
NM 031454
SELO
168
54
3,1
NM 032626
RBBP6
127
41
3,1
NM 001198559
DNAJC27
87
28
3,1
NM 006233
POLR2I
43
14
3,1
NM 022903
CCDC71
267
86
3,1
NM 001001662
ZNF787
273
88
3,1
NM 033513
C19orf20
46
15
3,1
NM 203339
CLU
96
31
3,1
NR 003672
SNHG7
618
200
3,1
NM 003965
CCRL2
52
17
3,1
NM 030943
AMN
55
18
3,1
NM 013382
POMT2
55
18
3,1
NM 000101
CYBA
345
112
3,1
NM 016194
GNB5
58
19
3,1
NM 001040126
PQLC2
58
19
3,1
NM 203374
ZNF792
58
19
3,1
NM 001003677
C11orf49
61
20
3,1
NM 032775
KLHL22
123
40
3,1
NM 001006643
SLC25A29
255
83
3,1
NM 001102367
C14orf159
258
84
3,1
NM 198988
LENG9
129
42
3,1
NM 005583
LYL1
1443
470
3,1
NM 138559
BCL11A
132
43
3,1
NM 006848
CCDC85B
531
173
3,1
NM 144589
COMTD1
67
22
3,0
NM 173628
DNAH17
82
27
3,0
NM 005608
PTPRCAP
262
86
3,0
NM 016113
TSHZ3
109
36
3,0
NM 001199694
SLC5A2
115
38
3,0
NM 017723
C9orf167
534
176
3,0
NM 002335
LRP5
486
161
3,0
NM 006675
TSPO
771
256
3,0
NM 001039705
TRPM2
493
164
3,0
NM 181814
ABHD12B
15
5
3,0
NM 080658
ACY3
12
4
3,0
NM 001107
ACYP1
24
8
3,0
NM 025008
ADAMTSL4
33
11
3,0
NR 037186
APITD1-CORT
36
12
3,0
NM 021822
APOBEC3G
240
80
3,0
NM 033515
ARHGAP18
48
16
3,0
NM 032131
ARMC2
15
5
3,0
NM 004655
AXIN2
18
6
3,0
NR 037658
BLOC1S1-RDH5
111
37
3,0
NR 026566
BMS1P1
12
4
3,0
NR 003611
BMS1P5
12
4
3,0
NM 001145033
C11orf96
12
4
3,0
NM 032829
C12orf34
12
4
3,0
NM 001017923
C14orf28
15
5
3,0
NM 001012984
C16orf86
15
5
3,0
NM 152914
C17orf103
36
12
3,0
NM 024323
C19orf57
30
10
3,0
NM 032261
C21orf56
24
8
3,0
NM 001013717
C5orf56
18
6
3,0
NM 001747
CAPG
48
16
3,0
NM 004055
CAPN5
27
9
3,0
NM 001130726
CCDC149
27
9
3,0
NM 001105564 1
CCHCR1
9
3
3,0
NM 006779
CDC42EP2
12
4
3,0
NM 001819
CHGB
36
12
3,0
NM 052999
CMTM1
12
4
3,0
NM 006314
CNKSR1
15
5
3,0
NM 001171174
CX3CR1
12
4
3,0
NM 019074
DLL4
9
3
3,0
NM 001934
DLX4
12
4
3,0
NM 017833
DNAJC28
9
3
3,0
NR 036554
DPY19L2P3
9
3
3,0
NM 024712
ELM03
27
9
3,0
NM 007046
EMILIN1
15
5
3,0
NM 207582
ERVFRD-1
18
6
3,0
NM_001105517
FAM58BP
9
3
3,0
NM__014808
FARP2
69
23
3,0
NM 033449
FCHSD1
177
59
3,0
NR 033904
FLJ39639
12
4
3,0
NM 001459
FLT3LG
15
5
3,0
NM 022158
FN3K
18
6
3,0
NR 034096
FONG
24
8
3,0
NM 005252
FOS
33
11
3,0
NM 005438
FOSL1
15
5
3,0
NM 003923
FOXH1
18
6
3,0
NM 001112808
FPGT-TNNI3K
12
4
3,0
NM 139285
GAS2L2
9
3
3,0
NM 021954
GJA3
18
6
3,0
NR 003945
GVINP1
9
3
3,0
NR 027716
H2AFJ
45
15
3,0
NM 019111 6
HLA-DRA
9
3
3,0
NM 178582
HM13
114
38
3,0
NM 001098213
HRH1
24
8
3,0
NM 003853
IL18RAP
15
5
3,0
NM 005101
ISG15
57
19
3,0
NM 002291
LAMB1
39
13
3,0
NM 001003679
LEPR
9
3
3,0
NM 030891
LRRC3
54
18
3,0
NM 002348
LY9
30
10
3,0
NM 181644
MFSD4
18
6
3,0
NR 003682
MGC70870
9
3
3,0
NR 002208
MRPL42P5
9
3
3,0
NM 178170
NEK8
51
17
3,0
NM 002507
NGFR
12
4
3,0
NM 004387
NKX2-5
12
4
3,0
NM 002593
PCOLCE
9
3
3,0
NM 148172
PEMT
12
4
3,0
NM 004813
PEX16
66
22
3,0
NM 001185181 3
PFDN6
27
9
3,0
NM 058237
PPP4R4
12
4
3,0
NR 037861 5
PPT2-EGFL8
18
6
3,0
NM 001160168
PRR5L
72
24
3,0
NM 003978
PSTPIP1
36
12
3,0
NM 152882
PTK7
27
9
3,0
NM 001013658
PTX4
15
5
3,0
NM 007023
RAPGEF4
18
6
3,0
NM 017805
RAS IP 1
12
4
3,0
NM 001204513
RBAK-LOC389458
15
5
3,0
NM 006871
RIPK3
18
6
3,0
NR 003051
RMRP
12
4
3,0
NM 152470
RNF165
12
4
3,0
NR 026982
RPL23AP82
48
16
3,0
NM 005226
S1PR3
39
13
3,0
NR 003023
SCARNA2
12
4
3,0
NM 003020
SCG5
12
4
3,0
NM 020796
SEMA6A
15
5
3,0
NR 003032
SNORA40
30
10
3,0
NR 002973
SNORA57
18
6
3,0
NM 004710
SYNPO
30
10
3,0
NM 033542
SYT11
30
10
3,0
NM 001199107
TBC1D29
12
4
3,0
NM 080647
TBX21
9
3
3,0
NM 007170
TEX9
9
3
3,0
NM 001097599
TMEM221
36
12
3,0
NM 033227
TRIM26
66
22
3,0
NM 001160389
TRPV1
42
14
3,0
NM 001145338 3
ZBTB26
66
22
3,0
NM 005453 1
ZBTB37
18
6
3,0
NR 024091
ZFYVE28
21
7
3,0
NM 198087
ZNF221
30
10
3,0
NM 001039654
ZNF571
21
7
3,0
NM 173658
ZNF665
12
4
3,0
NM 021216
ZNF713
21
7
3,0
NM 181877
ZSCAN21
162
54
3,0
NM 001006642
SLC25A6
2143
718
3,0
NM 020158
EXOSC5
211
71
3,0
NM 014516
CNOT3
193
65
3,0
NM 000932
PLCB3
369
124
3,0
NM 203458
NOTCH2NL
178
60
3,0
NM 001171907
CXorf40A
130
44
3,0
NM 024812
BAALC
124
42
3,0
NM 017797
BTBD2
483
163
3,0
NM 001039842
C17orf90
240
81
3,0
NM 001012759
CTU2
103
35
2,9
NM 001003791
ERLIN2
100
34
2,9
NM 176792
MRPL43
100
34
2,9
NM 024618
NLRX1
277
94
2,9
NM 013304
ZDHHC12
159
54
2,9
NM 004421
DVL1
232
79
2,9
NM 033397
ITPRIP
153
52
2,9
NM 022092
CHTF18
223
76
2,9
NM 001014811
ME3
73
25
2,9
NM 004808
NMT2
73
25
2,9
NM 012230
POMZP3
73
25
2,9
NM 025076
VARS
73
25
2,9
NM 033046
RTKN
144
49
2,9
NM 001172669
ZNF672
211
72
2,9
NM 018686
CMAS
205
70
2,9
NM 003839
TNFRSF14
67
23
2,9
NM 001142651
NEURL1B
132
45
2,9
NM 002856
PVRL2
196
67
2,9
NM 003562
SLC25A11
243
83
2,9
NM 006951
TANC1
120
41
2,9
NM 000201
ICAM1
237
81
2,9
NM 001170538
DZIP1L
58
20
2,9
NM 032305
POLR3GL
231
79
2,9
NM 000029
AGT
111
38
2,9
NM 001044264
MSMP
55
19
2,9
NM 001160116
C15orf40
52
18
2,9
NM 006653
FRS3
52
18
2,9
NR 026787
LMLN
52
18
2,9
NM 020855
ZNF497
52
18
2,9
NM 033054
MY01G
1294
445
2,9
NM 001005910
IP6K2
142
49
2,9
NR 039964
MIR4800
46
16
2,9
NM 019020
TBC1D24
183
63
2,9
NM 144998
STX10
133
46
2,9
NM 005516
HLA-E
307
106
2,9
NM 005575
LNPEP
217
75
2,9
NM 152519
C2orf67
127
44
2,9
NM 138790
PLD4
787
272
2,9
NM 004672
MAP3K6
40
14
2,9
NM 016354
SLITRK5
321
111
2,9
NM 024668
ANKHD1
312
108
2,9
NM 001204880
UBE2F
78
27
2,9
NM 001164623
EIF2C2
349
121
2,9
NM 001006622
WDR34
228
79
2,9
NM 006478
GAS2L1
37
13
2,8
NR 024154
NUDT7
75
26
2,9
NM 145291
ZC3H10
150
52
2,9
NM 001007523 1
F8A2
147
51
2,9
NM 001007524 1
F8A3
147
51
2,9
NM 001110215
CENPM
109
38
2,9
NM 001039548
KLHL35
109
38
2,9
NM 030665
RAM
219
76
2,9
NM 001010919
FAM26F
106
37
2,9
NM 201265
UBR2
247
86
2,9
NM 001039481
ETNK1
34
12
2,8
NM 001988
EVPL
448
156
2,9
NM 003024
ITSN1
34
12
2,8
NM 198439
KBTBD3
34
12
2,8
NM 001142627
SEC61A2
34
12
2,8
NM 001004351
SPDYE6
34
12
2,8
NM 024100
WDR20
103
36
2,9
NM 002751
MAPK11
273
95
2,9
NM 032341
DDI2
169
59
2,9
NR 039900
MIR4745
100
35
2,9
NM 001172702
SLC38A7
100
35
2,9
NM 012100
DNPEP
232
81
2,9
NM 001143915
FAM76A
66
23
2,9
NM 172208 3
TAPBP
163
57
2,9
NR 039872
MIR4721
97
34
2,9
NM 004424
E4F1
129
45
2,9
NM 001164603
ASXL1
31
11
2,8
NM 031300
MXD3
94
33
2,8
NR 026963
TTC 8
31
11
2,8
NM 182587
UNCX
31
11
2,8
NM 022362
MMS19
217
76
2,9
NM 020342
SLC39A3
432
151
2,9
NM 032306
ALKBH7
240
84
2,9
NM. 002746
MAPK3
120
42
2,9
NM 024083
ASPSCR1
228
80
2,9
NM 024296
CCDC28B
114
40
2,9
NM 001134759
CRYZ
85
30
2,8
NM 001024948
FNBP1L
85
30
2,8
NM_015660
GIMAP2
285
100
2,9
NM 002263
KIFC1
142
50
2,8
NM 001146210
SPNS1
85
30
2,8
NM 001017964
YIF1B
171
60
2,9
NM 012197
RABGAP1
253
89
2,8
NM 032591
SLC04A1
253
89
2,8
NM 005569
LIMK2
196
69
2,8
NM 007097
CLTB
222
78
2,8
NM 002415
MIF
936
329
2,8
NM 181336
LEMD2
247
87
2,8
NM 001013436
MPST
355
125
2,8
NM 018477
ACTR10
136
48
2,8
NM 020719
PRR12
298
105
2,8
NM 175871
C19orf39
54
19
2,8
NR 024348
FBXL19-AS1
54
19
2,8
NM 005668
STAP1
108
38
2,8
NM 001174158
ZFP1
162
57
2,8
NM 006454
MXD4
640
226
2,8
NM 014272
ADAMTS7
25
9
2,8
NM 020785
CC2D2A
25
9
2,8
NM 024553
CCDC132
25
9
2,8
NM 032309
CHCHD5
25
9
2,8
NM 176782
FAM151A
76
27
2,8
NM 022368
PJA1
76
27
2,8
NM 001017369
SC4MOL
51
18
2,8
NM 022968
SERF1A
25
9
2,8
NM 001178087 1
SERF1B
25
9
2,8
NR 000018
SNORD68
25
9
2,8
NM 013351
TCEA2
25
9
2,8
NM 001136053
TPST2
76
27
2,8
NM 199282
ARHGAP27
226
80
2,8
NM 016073
HDGFRP3
147
52
2,8
NM 006236
POU3F3
73
26
2,8
NM 032271
TRAPPC1
147
52
2,8
NM 022060
ABHD4
1122
397
2,8
NM 001128917
TOR2A
195
69
2,8
NM 152441
FBXL14
169
60
2,8
NR 027308
MEF2BNB-MEF2B
48
17
2,8
NM 203473
PORCN
70
25
2,8
NM 019848
SLC10A3
163
58
2,8
NM 001199173
MLST8
186
66
2,8
NR 026962
TTC39C
93
33
2,8
NM 005597
NFIC
205
73
2,8
NM 014603
CDR2L
90
32
2,8
NR 002174
CMAHP
22
8
2,8
NM 001114137
EPB49
22
8
2,8
NM 019013
FAM64A
112
40
2,8
NM 012227 1
GTPBP6
45
16
2,8
NM 014181
HSPC159
45
16
2,8
NM 002373
MAPI A
22
8
2,8
NM 004556
NFKBIE
247
88
2,8
NM 001130102
NR1H3
22
8
2,8
NM 001164721
PTAFR
22
8
2,8
NM 021033
RAP2A
22
8
2,8
NM 023074
ZNF655
112
40
2,8
NM 003775
S1PR4
492
175
2,8
NM 003088
FSCN1
685
244
2,8
NM 001203264
IL17RC
87
31
2,8
NM 003793
CTSF
213
76
2,8
NM 020856
TSNARE1
106
38
2,8
NM 032898
CEP19
42
15
2,8
NR 037909
ARHGAP19-SLIT1
103
37
2,8
NM 001197330
POLE2
61
22
2,8
NM 201400
FAM86A
181
65
2,8
NM 202494
GIPC1
159
57
2,8
NM 022810
SLC25A25
198
71
2,8
NM 001110792
MECP2
276
99
2,8
NM 133450
ANKS3
39
14
2,8
NM 001725
BPI
19
7
2,7
NM 152343
C17orf46
19
7
2,7
NM 178868
CMTM8
78
28
2,8
NM 021958
HLX
19
7
2,7
NR 027145
LIMS3-LOC440895
19
7
2,7
NM 002332
LRP1
19
7
2,7
NM 001080401
PPM1N
19
7
2,7
NM 001204103 1
PPT2
19
7
2,7
NM 002931
RING1
58
21
2,8
NR 002789
SEPHS1P
19
7
2,7
NR 003033
SNORD88B
19
7
2,7
NM 001168316
ULBP2
19
7
2,7
NM 001206802
TPRA1
400
144
2,8
NM 205767
C19orf70
114
41
2,8
NM 018273
TMEM160
75
27
2,8
NM 005452 2
WDR67
205
74
2,8
NM 130791
XAB2
411
148
2,8
NM 001025108
AFF3
55
20
2,8
NM 005740
DNAL4
55
20
2,8
NM 002626
PFKL
1123
405
2,8
NM 001042600
MAP4K1
183
66
2,8
NM 000052
ATP7A
36
13
2,8
NM 175709
CBX7
72
26
2,8
NR 037775
MIA-RAB4B
36
13
2,8
NM 005861
STX16-NPEPL1
465
168
2,8
NM 001207056
DNMT3B
124
45
2,8
NM 001202557
CD44
616
223
2,8
NM 004639 5
BAG6
105
38
2,8
NM 152485
C1orf74
52
19
2,7
NM 001823
СКВ
525
190
2,8
NR 027063
NCRNA00116
52
19
2,7
NM 205842
NCKAP1
121
44
2,8
NM 017728
TMEM115
328
119
2,8
NM 004765
BCL7C
69
25
2,8
NR 003024
EIF3IP1
171
62
2,8
NM 198900
FMNL3
204
74
2,8
NM 015321
CRTC1
237
86
2,8
NM 001048172
MUTYH
151
55
2,7
NM 206967
C16orf74
16
6
2,7
NM 021267
CERS1
33
12
2,8
NM 138481
CHADL
16
6
2,7
NM 001492
GDF1
33
12
2,8
NR 003504
GUSBP2
16
6
2,7
NM 005345
HSPA1A
49
18
2,7
NM 032123
KIRREL2
16
6
2,7
NR 037408
MIR3614
16
6
2,7
NR 027775
PMS2P5
33
12
2,8
NM 001198952
RNF103
66
24
2,8
NM 001172109
SENP8
16
6
2,7
NM 018656
SLC38A6
16
6
2,7
NR 034178
SSC5D
16
6
2,7
NM 000593 4
TAP2
33
12
2,8
NM 018073
TRIM8
264
96
2,8
NM 145294
WIF1
16
6
2,7
NM 001160407
TMED1
327
119
2,7
NM 018140
CEP72
291
106
2,7
NM 004285
H6PD
387
141
2,7
NM 138961
ESAM
126
46
2,7
NR 033322
NSUN5P1
126
46
2,7
NM 001130848
PITPNM1
391
143
2,7
NM 007255
B4GALT7
109
40
2,7
NM 017828
COMMD4
219
80
2,7
NM 015094
HIC2
219
80
2,7
NM 006666
RUVBL2
561
205
2,7
NM 152916
EMR2
46
17
2,7
NM 020704
FAM40B
46
17
2,7
NM 001127396
SUGP1
279
102
2,7
NM 015356
SCRIB
478
175
2,7
NM 018340
CPPED1
169
62
2,7
NM 173793
C22orf39
123
45
2,7
NR 037425
MIR3652
123
45
2,7
NM 004204
PIGQ
76
28
2,7
NM 020722
KIAA1211
286
105
2,7
NR 037846 1
MSH5-C6orf26
30
11
2,7
NM 001171906
NUDT16
30
11
2,7
NM 007280
OIP5
60
22
2,7
NM 001135610
PRDM2
30
11
2,7
NR 027662
SLC25A14
30
11
2,7
NM 001032292
ZNF187
90
33
2,7
NM 001687
ATP5D
223
82
2,7
NM 001080826
SGK223
133
49
2,7
NM 001171888
FGFR10P2
103
38
2,7
NM 152299
NCAPH2
427
157
2,7
NM 032402
PCDHGC3
73
27
2,7
NM 001005362
DNM2
1083
398
2,7
NM 031215
CABLES2
117
43
2,7
NM 203370
C3orf54
394
145
2,7
NM 001077685
AGAP7
43
16
2,7
NM 001007237
IGSF3
87
32
2,7
NM 000442
PECAM1
43
16
2,7
NM 001122764
PPOX
87
32
2,7
NR 002818
RNF126P1
43
16
2,7
NM 016504
MRPL27
285
105
2,7
NM 206901
RTN2
57
21
2,7
NM 001024
RPS21
469
173
2,7
NM 020812
DOCK6
198
73
2,7
NM 012181
FKBP8
1068
394
2,7
NM 182984
TRNAU1AP
84
31
2,7
NM 015603
CCDC9
97
36
2,7
NM 006339
HMG20B
400
148
2,7
NR 024332
POMGNT1
165
61
2,7
NM 009590
AOC2
13
5
2,6
NM 001130414
APBA2
13
5
2,6
NM 004052
BNIP3
67
25
2,7
NM 024650
C11orf80
27
10
2,7
NR 029406
FLJ43681
13
5
2,6
NM 000238
KCNH2
13
5
2,6
NM 001194958
KCNJ18
27
10
2,7
NM 033402
LRRCC1
67
25
2,7
NM 020142
NDUFA4L2
94
35
2,7
NM 022141
PARVG
270
100
2,7
NM 144579
SFXN5
13
5
2,6
NM 013356
SLC16A8
27
10
2,7
NM 003172
SYCE1L
13
5
2,6
NM 001195227
TRPT1
108
40
2,7
NM 003312
TTC14
67
25
2,7
NM 016030
TTC23
40
15
2,7
NM 001184801
UBTD1
13
5
2,6
NM 007130
ZNF414
54
20
2,7
NM 001039140
C20orf27
429
159
2,7
NM 139355
MATK
402
149
2,7
NM 024037
C1orf135
267
99
2,7
NM 006913 6
RNF5
91
34
2,7
NM 021222
PRUNE
169
63
2,7
NR 024063
ZSCAN20
78
29
2,7
NM 152727
CPNE2
363
135
2,7
NM 025256 6
EHMT2
64
24
2,7
NM 005343
H RAS
129
48
2,7
NM 032477
MRPL41
64
24
2,7
NM 001145441
UBE2D1
129
48
2,7
NM 002401
МАРЗКЗ
682
254
2,7
NM 001085365
MZT2A
268
100
2,7
NM 001001976
ATE1
102
38
2,7
NM 005346 3
HSPA1B
51
19
2,7
NM 024112
C9orf16
177
66
2,7
NM 002028
FNTB
88
33
2,7
NM 013265
C11orf2
892
333
2,7
NM 024872
DOK3
313
117
2,7
NM 020445
ACTR3B
75
28
2,7
NM 006020
ALKBH1
75
28
2,7
NM 001193529
CCT6B
37
14
2,6
NM 001018112
FANCA
75
28
2,7
NM 001199162
USP40
112
42
2,7
NM 000033
ABCD1
174
65
2,7
NM 012200
B3GAT3
174
65
2,7
NM 145030
C7orf47
235
88
2,7
NM 014386
PKD2L2
61
23
2,7
NM 001190737
NFIB
85
32
2,7
NM 012454
TICAM1
85
32
2,7
NM 153497
TADA1
133
50
2,7
NM 006384
CIB1
301
113
2,7
NM 001190727
C9orf103
72
27
2,7
NM 152361
EID2B
24
9
2,7
NM 024519
FAM65A
72
27
2,7
NM 001144829
FM05
24
9
2,7
NR 024206
NCRNA00152
120
45
2,7
NM 004822
NTN1
168
63
2,7
NM 181515
MRPL21
250
94
2,7
NM 003149
STARD8
189
71
2,7
NM 022104
PCIF1
364
137
2,7
NM 001242758 5
H LA-A
2173
817
2,7
NM 181718
ASPHD1
58
22
2,6
NM 001083314
CHMP1A
409
154
2,7
NM 020888
KIAA1522
58
22
2,6
NM 001164507
NEB
58
22
2,6
NM 014700
RAB11FIP3
58
22
2,6
NM 001143784
FES
840
316
2,7
NM 004260
RECQL4
778
293
2,7
NM 004938
DAPK1
382
144
2,7
NM 016274
PLEKH01
379
143
2,7
NM 031209
QTRT1
172
65
2,6
NM 001080821
ZNF808
34
13
2,6
NM 012477
WBSCR16
331
125
2,6
NM 002586 5
PBX2
148
56
2,6
NM 080546
SLC45A3
148
56
2,6
NM 017607
PPP1R12C
490
185
2,6
NM 138471
C11orf84
193
73
2,6
NM 001037339
PDE4B
79
30
2,6
NM 001164760
PRKAR1B
159
60
2,7
NM 000958
PTGER4
238
90
2,6
NM 002997
SDC1
79
30
2,6
NM 145003
TSPAN7
79
30
2,6
NM 001136200
C10orf32
124
47
2,6
NM 173852
KRTCAP2
124
47
2,6
NM 175573
ADRM1
1014
383
2,6
NM 173575
STMN3
529
200
2,6
NM 025057
C14orf45
45
17
2,6
NM 021083
XYLB
90
34
2,6
NM 004217
AURKB
211
80
2,6
NM 133261
GIPC3
55
21
2,6
NR 024116
RASA4P
55
21
2,6
NM 001078650
TMEM141
166
63
2,6
NM 144695
BROX
121
46
2,6
NM 001146696
KDM4C
66
25
2,6
NM 001013663
PTRHD1
66
25
2,6
NM 015670
SENP3
483
183
2,6
NM 198723
TCEB2
208
79
2,6
NM 032189
ATP11A
535
203
2,6
NM 173091
NFATC2
153
58
2,6
NM 004073
PLK3
87
33
2,6
NM 004470
FKBP2
97
37
2,6
NM 017884
PINX1
118
45
2,6
NM 017631
DDX60
129
49
2,6
NM_024105
ALG12
150
57
2,6
NM 052951
DNTTIP1
171
65
2.6
NM 024029
ΖΑΚ
811
309
2,6
NM 001013619
AGPHD1
10
4
2,5
NM 019046
ANKRD16
52
20
2,6
NM 001143778
ASAP3
10
4
2,5
NM 174983
C19orf28
199
76
2,6
NM 001146310
C1orf86
21
8
2,6
NR 003545
C5orf44
31
12
2,6
NM 018465
C9orf46
52
20
2,6
NM 024111
CHAC1
10
4
2,5
NM 014693
ECE2
136
52
2,6
NM 001145053
EFCAB5
10
4
2,5
NM 001137610
FAM86B2
10
4
2,5
NM 022110
FKBPL
10
4
2,5
NR 028038
GATS
63
24
2,6
NM 001142853
HES6
10
4
2,5
NM 138284
IL17D
21
8
2,6
NM 138433
KLHDC7B
21
8
2,6
NM 006992
LRRC23
10
4
2,5
NM 001012755
MCART6
10
4
2,5
NM 172166 7
MSH5
10
4
2,5
NM 025198
MTERFD3
63
24
2,6
NM 020884
MYH7B
10
4
2,5
NR 024260
NECAP1
126
48
2,6
NM 002514
NOV
10
4
2,5
NM 001145939
PAFAH1B3
199
76
2,6
NM 003498
SNORA4
31
12
2,6
NM 080744
SSBP2
10
4
2,5
NM 000660
TGM1
10
4
2,5
NM 001190919
THY1
21
8
2,6
NM 003250
TIAM2
10
4
2,5
NM 020431
TMEM67
10
4
2,5
NM 001142301
TMEM79
63
24
2,6
NM 177556
TRPC1
31
12
2,6
NM 016653
ZBED1
157
60
2,6
NM 031486
ZNF486
42
16
2,6
NM 016536
ZNF585B
10
4
2,5
NM 022744
C16orf58
364
139
2,6
NM 019103
ZMYM3
270
103
2,6
NM 006441
MTHFS
123
47
2,6
NM 175875
SIX5
112
43
2,6
NM 001040450
FAM63B
102
39
2,6
NM 001105205
RUSC1
285
109
2,6
NM 001009991
TAB1
91
35
2,6
NM 001080495
TNS1
91
35
2,6
NM 032809
FAM73B
243
93
2,6
NM 152527
SLC16A14
151
58
2,6
NM 153483
IL17RE
70
27
2,6
NM 144638
TMEM55B
211
81
2,6
NM 020328
ACVR1B
391
150
2,6
NM 152400
C4orf32
130
50
2,6
NM 001136108
R3HCC1
370
142
2,6
NR 037192
ABHD14A-ACY1
169
65
2,6
NM 153688
ZFP91-CNTF
586
225
2,6
NM 001008566
TRABD
466
179
2,6
NM 173487
C4orf33
49
19
2,6
NR 026914
MGC16275
49
19
2,6
NM 207351
PRRT3
99
38
2,6
NM 001197181
TUBGCP6
495
190
2,6
NM 145914
ZSWIM4
99
38
2,6
NM 001142298
SRBD1
414
159
2,6
NM 001116
ADCY9
88
34
2,6
NM 000211
ITGB2
442
170
2,6
NM 017825
ADPRHL2
255
98
2,6
NM 025065
RPF1
322
124
2,6
NR 003582
ATP8B5P
78
30
2,6
NM 177938
P4HTM
78
30
2,6
NM 013299
SAC3D1
117
45
2,6
NM 015000
STOX1
39
15
2,6
NM 004626
WWOX
39
15
2,6
NM 004429
EFNB1
67
26
2,6
NM 032641
SPSB3
135
52
2,6
NM 173467
MCAT
163
63
2,6
NM 001005377
PLAUR
96
37
2,6
NM 016260
IKZF2
153
59
2,6
NM 001206682
ATF7
28
11
2,5
NM 001206945
CSPG5
28
11
2,5
NM 020340
KIAA1244
28
11
2,5
NM 003550
MAD1L1
199
77
2,6
NM 031417
MARK4
85
33
2,6
NM 018328
MBD5
28
11
2,5
NM 021959 2
PPP1R11
28
11
2,5
NM 017999
RNF31
228
88
2,6
NM 052978
TRMT12
57
22
2,6
NM 138425
C12orf57
321
124
2,6
NM 001145312
ETV3
75
29
2,6
NM 018727
TRPV2
121
47
2,6
NM 152892
LRWD1
168
65
2,6
NM 015710
GLTSCR2
969
375
2,6
NR 026586
EFCAB2
46
18
2,6
NM 207370
GPR153
46
18
2,6
NM 032701
SYDE2
93
36
2,6
NM 001024683
ZNF69
46
18
2,6
NM 018998
FBXW5
648
251
2,6
NM 002691
POLD1
619
240
2,6
NM 018316
KLHL26
129
50
2,6
NM 021620
PRDM13
64
25
2,6
NM 001040457
RHBDD2
258
100
2,6
NM 203385
RNH1
810
314
2,6
NM_139027
ADAMTS13
82
32
2,6
NM 021922
FANCE
100
39
2,6
NM 003338
UBE2M
219
85
2,6
NM 003977
AIP
496
193
2,6
NM 207354
ANKRD13D
252
98
2,6
NM 024827
HDAC11
18
7
2,6
NM 001036645
HHLA3
18
7
2,6
NM 015133
MAPK8IP3
234
91
2,6
NR 030366
MIR636
18
7
2,6
NM 138383
MTSS1L
288
112
2,6
NM 012339
TSPAN9
18
7
2,6
NM 002741
PKN1
2068
805
2,6
NM 001105079
FBRS
187
73
2,6
NM 032214
SLA2
133
52
2,6
NM 001344
DAD1
759
296
2,6
NM 001144872
CCDC42B
166
65
2,6
NR 026868
ANKRD19P
481
188
2,6
NM 001433
ERN1
105
41
2,6
NM 001760
CCND3
148
58
2,6
NM 033446
FAM125B
148
58
2,6
NM 003926
MBD3
508
199
2,6
NM 007074
COR01A
1731
678
2,6
NM 016162
ING4
145
57
2,5
NM 002532
NUP88
393
154
2,6
NM 001136540
APOL1
51
20
2,6
NM 152854
CD40
25
10
2,5
NM 138782
FCH02
102
40
2,6
NM 001166271
SPDYA
25
10
2,5
NM 001195082
TFEB
25
10
2,5
NM 153028
ZNF775
51
20
2,6
NM 007317
KIF22
558
219
2,5
NM 001004309
ZNF777
295
116
2,5
NM 080669
SLC4A1AP
270
106
2,5
NM 018263
ASXL2
1270
499
2,5
NM 001168303
KLHL13
84
33
2,5
NM 003321
TXNRD2
84
33
2,5
NM 003190 1
TARBP2
226
89
2,5
NM 030587
B4GALT2
259
102
2,5
NM 001136197
FZR1
249
98
2,5
NM 017566
KLHDC4
124
49
2,5
NM 001243014
ABCB9
33
13
2,5
NM 001099407
C20orf12
66
26
2,5
NM 173589
DNHD1
66
26
2,5
NR 036049 1
MIR1184-1
99
39
2,5
NR 036259 1
MIR1184-2
99
39
2,5
NR 036260 1
MIR1184-3
99
39
2,5
NM 001170944
PNMA6C
33
13
2,5
NM 002831
PTPN6
429
169
2,5
NM 002935
RNASE3
33
13
2,5
NR 003129 7
RNF5P1
198
78
2,5
NM 001143676
SGK1
33
13
2,5
NM 001040657
TTC26
33
13
2,5
NM 005706
TSTA3
271
107
2,5
NM 001034023
DMAP1
106
42
2,5
NM 002434
MPG
106
42
2,5
NM 004755
RPS6KA5
106
42
2,5
NM 207121
FAM110A
180
71
2,5
NM 001080530
SNX4
375
148
2,5
NM 003755
EIF3G
676
267
2,5
NM 001130057
EEF1D
2553
1008
2,5
NM 001961
EEF2
36334
14350
2,5
NM 017806
LIME1
40
16
2,5
NM 002198
IRF1
217
86
2,5
NM 001013632
TESK2
88
35
2,5
NM 001487
BLOC1S1
136
54
2,5
NM 001199654
PMF1
273
108
2,5
NM 005189
CBX2
945
374
2,5
NM 022338
C11orf24
144
57
2,5
NM 006613
GRAP
48
19
2,5
NM 025188
TRIM5
48
19
2,5
NM 020226
PRDM8
535
212
2,5
NM 014203
AP2A1
447
177
2,5
NM 017749
AMBRA1
358
142
2,5
NR 024174
MKNK1
159
63
2,5
NM 001202545
CUX1
381
151
2,5
NM 001032281
TGFB1
603
239
2,5
NM 032304
HAGHL
55
22
2,5
NM 001945
HBEGF
55
22
2,5
NM 134425
SLC2A11
55
22
2,5
NM 001199859
ANGPT1
252
100
2,5
NM 001008703
C6orf1
63
25
2,5
NM 032928
TMEM159
126
50
2,5
NM 001997
FAU
2299
913
2,5
NM 004670
PAPSS2
70
28
2,5
NR 033329
KLHL7
78
31
2,5
NM 001008528
MXRA7
78
31
2,5
NR 033257 3
XRCC1
156
62
2,5
NM 181661
VPS13B
561
223
2,5
NR 027696
FLJ39653
4696
1867
2,5
NM 001199455 4
BRD2
171
68
2,5
NM 032932
RAB11FIP4
171
68
2,5
NM 001161565
TNRC18
1747
695
2,5
NM 015060
AVL9
178
71
2,5
NM 005108
YDJC
462
184
2,5
NM 194460
RNF126
246
98
2,5
NM 031937
TBC1D10C
130
52
2,5
NM 001012727
AGPAT2
549
219
2,5
NM 033315
RASL10B
190
76
2,5
NM 001003690
MAD2L1BP
198
79
2,5
NM 007165
SF3A2
468
187
2,5
NM 001037328
STK11
588
235
2,5
NM 139057
ADAMTS17
22
9
2,4
NM 006066
AKR1A1
547
219
2,5
NM 000056
BCKDHB
90
36
2,5
NM 153025
C16orf55
15
6
2,5
NM 001007125
C20orf201
15
6
2,5
NM 032340
C6orf125
75
30
2,5
NM 020311
CXCR7
15
6
2,5
NM 001029955
DCAF4L1
15
6
2,5
NM_001345
DGKA
52
21
2,5
NR 002605
DLEU1
37
15
2,5
NM 004417
DUSP1
45
18
2,5
NM 004447
EPS8
22
9
2,4
NM 000161
GCH1
52
21
2,5
NR 027028
GUSBP1
15
6
2,5
NM 001145805
IRGM
15
6
2,5
NM_003573
LTBP4
150
60
2,5
NM 014641 5
MDC1
67
27
2,5
NR 036523
MICA
22
9
2,4
NR 039895
MIR4741
22
9
2,4
NM 017677
MTMR8
22
9
2,4
NM 001143988
NBPF6
22
9
2,4
NM 001242865
PRMT2
135
54
2,5
NM 016154
RAB4B
37
15
2,5
NM 013347
RPA4
15
6
2,5
NR 002981
SNORD104
52
21
2,5
NM 025250
TUBB3
30
12
2,5
NM 018671
UNC80
15
6
2,5
NM 006295 2
VARS2
15
6
2,5
NM 001007101
ZNF492
30
12
2,5
NM 022752
ZNF593
112
45
2,5
NM 015871
ZNF613
15
6
2,5
NM 001033026
C19orf6
874
350
2,5
NM 015965
NDUFA13
574
230
2,5
NM 133374
ZNF641
259
104
2,5
NM 006377
UNC45A
711
285
2,5
NM 033086
FGD3
207
83
2,5
NM 015649
IRF2BP1
399
160
2,5
NM 023078
PYCRL
162
65
2,5
NR 027954
TK1
2118
850
2,5
NM 017450
BAIAP2
301
121
2,5
NM 003859
DPMI
226
91
2,5
NM 172218
SPATA13
204
82
2,5
NM 001042461
TRAPPC6A
94
38
2,5
NR 033429
ABTB1
181
73
2,5
NM 018067
MAP7D1
355
143
2,5
NM 016034
MRPS2
529
213
2,5
NM 199227
METAP1D
87
35
2,5
NM 147161
ACOT11
159
64
2,5
NM 001790
CDC25C
79
32
2,5
NM 032779
CCDC142
151
61
2,5
NM 001129819
CYB5R3
439
177
2,5
NM 015104
ATG2A
360
145
2,5
NM 024623
OGFOD2
144
58
2,5
NM 152286
PNPLA7
72
29
2,5
NM 138352
SAMD1
144
58
2,5
NM 001001410
C16orf42
136
55
2,5
NM 004581
RABGGTA
136
55
2,5
NM 199330
HOMER2
337
136
2,5
NM 002714
PPP1R10
64
26
2,5
NM 018112
TMEM41B
64
26
2,5
NM 181842 2
ZBTB17
193
78
2,5
NM 004689
MTA1
630
254
2,5
NM 024407
NDUFS7
121
49
2,5
NM 001173408
OBSL1
300
121
2,5
NM 138414
CCDC101
57
23
2,5
NM 004668
MGAM
57
23
2,5
NM 152222
RELT
399
161
2,5
NM 001128847
SMG6
228
92
2,5
NM 153271
SNX8
171
69
2,5
NM 013359
ZNF254
171
69
2,5
NM 001141973
ATP13A2
163
66
2,5
NM 017670
OTUB1
483
195
2,5
NM 001145815
AMDHD2
106
43
2,5
NM 030930
UNK
304
123
2,5
NM 014895
KIAA1009
49
20
2,5
NM 015901
NUDT13
49
20
2,5
NM 006287
TGFB3
99
40
2,5
NM 001100598
ZNF71
99
40
2,5
NM 024658
IP04
339
137
2,5
NM 057749
CCNE2
232
94
2,5
NM 001206951
SLC16A3
309
125
2,5
NM 001800
CDKN2D
42
17
2,5
NM 145177
DHRSX
42
17
2,5
NM 203434
IER5L
42
17
2,5
NM 016332
SEPX1
84
34
2,5
NM 031219
HDHD3
118
48
2,5
NM 020422
TMEM177
118
48
2,5
NM 001288 6
CLIC1
271
110
2,5
NM 001127365
C7orf46
76
31
2,5
NM 144606
FLCN
76
31
2,5
NR 033354
ZNF550
76
31
2,5
NM 152279
ZNF598
444
180
2,5
NM 013976
GCDH
283
115
2,5
NR 015380
A1BG-AS1
34
14
2,4
NM 013385
CYTH4
69
28
2,5
NM 002061
GCLM
103
42
2,5
NM 182647
OPRL1
103
42
2,5
NM 016580
PCDH12
34
14
2,4
NM 214710
PRSS57
1242
504
2,5
NM 001031618
SPDYE3
34
14
2,4
NM 001199662
PMF1-BGLAP
130
53
2,5
NM 002162
ICAM3
733
298
2,5
NM 021805
SIGIRR
157
64
2,5
NM 014855
KIAA0415
342
139
2,5
NM 001165877
ATP5L2
61
25
2,4
NM 020664
DECR2
123
50
2,5
NM 014046 4
MRPS18B
61
25
2,4
NM 003482
MLL2
3522
1432
2,5
NM 004445
EPHB6
204
83
2,5
NM 000992
RPL29
7063
2874
2,5
NM 173545
APLF
27
11
2,5
NM 001003398
BICD1
27
11
2,5
NM 001135751
DERL3
27
11
2,5
NM 022760
FAM113A
297
121
2,5
NR 003579
FRG1B
135
55
2,5
NM 001190825
MAFIP
27
11
2,5
NM 002412
MGMT
108
44
2,5
NM 020654
SENP7
162
66
2,5
NM 016072
GOLT1B
235
96
2,4
NM 001032383
PQBP1
208
85
2,4
NM 006014
LAGE3
127
52
2,4
NM 001136191
KANK2
328
134
2,4
NM 015953
NOSIP
174
71
2,5
NM 001319
CSNK1G2
669
273
2,5
NM 004838
HOMER3
220
90
2,4
NM 054035
UNC13B
73
30
2,4
NM 001029891
PGAM4
139
57
2,4
NM 005892
FMNL1
942
385
2,4
NM 152482
C19orf25
205
84
2,4
NM 000263
NAGLU
283
116
2,4
NM 007209
RPL35
1309
536
2,4
NM 152431
PIWIL4
85
35
2,4
NM 175872
ZNF804A
190
78
2,4
NR 038257
TRPM4
432
177
2,4
NM 001166664
CD244
163
67
2,4
NM 001773
CD34
1857
761
2,4
NM 138353
DCAF15
358
147
2,4
NM 001040031
CD37
495
203
2,4
NM 152426
APOBEC3D
39
16
2,4
NR 026997
C22orf34
78
32
2,4
NM 001168648
CNKSR2
39
16
2,4
NM 016144
COMMD10
39
16
2,4
NM 152511
DUSP18
19
8
2,4
NM 000173
GP1BA
39
16
2,4
NM 001003795
GTF2IRD2B
39
16
2,4
NR 003675
GUSBP5
117
48
2,4
NM 013320
HCFC2
78
32
2,4
NM 018012
KIF26B
19
8
2,4
NR 036154
MIR3187
58
24
2,4
NM 018728
MY05C
19
8
2,4
NM 177533
NUDT14
78
32
2,4
NM 017893
SEMA4G
19
8
2,4
NM 001122752
SERPINI1
19
8
2,4
NM 003114
SPATC1
19
8
2,4
NM 052874
STX8
19
8
2,4
NM 018252
TMEM22
19
8
2,4
NM 001136044
TMX2-CTNND1
97
40
2,4
NM 173555
TYW1B
58
24
2,4
NM 001142864
FAM38A
2185
897
2,4
NM 016129
COPS4
168
69
2,4
NM 080627
KIAA0889
168
69
2,4
NM 207005
USP19
762
313
2,4
NM 031917
ANGPTL6
219
90
2,4
NM 020442
VASP
328
135
2,4
NM 001184751
ZNF41
109
45
2,4
NM 003310
TST
180
74
2,4
NM 001022
RPS19
3846
1582
2,4
NM 001862
COX5B
493
203
2,4
NM 001136180
HSBP1L1
70
29
2,4
NM 175744
RHOC
282
116
2,4
NM 015994
ATP6V1D
121
50
2,4
NM_016337
EVL
121
50
2,4
NM 001134430
TOX
243
100
2,4
NM 144716
CCDC12
102
42
2,4
NM 001129885
CPSF4L
51
21
2,4
NM 001144958
EFCAB4B
102
42
2,4
NM 016093
RPL26L1
51
21
2,4
NR 002450
SNRNP25
133
55
2,4
NM 001039841
ARHGAP11B
82
34
2,4
NM 001105515
ABCC4
196
81
2,4
NM 017857
ST20-MTHFS
114
47
2,4
NM 003036
SKI
861
355
2,4
NM 002168
IDH2
1494
616
2,4
NM 001002796
MCTP1
145
60
2,4
NM 015125
CIC
354
146
2,4
NM 145253
FAM100A
63
26
2,4
NM 001144891
MBIP
63
26
2,4
NM 015178
RHOBTB2
94
39
2,4
NM 020175
DUS3L
363
150
2,4
NM 006455
LEPREL4
256
106
2,4
NM 001127257
SLC39A14
280
116
2,4
NM 024419
PGS1
585
242
2,4
NM 032208
ANTXR1
217
90
2,4
NM 001178
ARNTL
43
18
2,4
NR 028324
FLJ45445
43
18
2,4
NM 004127
GPS1
957
396
2,4
NR 024053 1
HCG18
43
18
2,4
NM 001201573
NUBPL
43
18
2,4
NR 034108
TRAIP
174
72
2,4
NM 032799
ZDHHC8
130
54
2,4
NM 003575
ZNF3
174
72
2,4
NM 004604
STXBP2
360
149
2,4
NM 001163922
WBP1
229
95
2,4
NM 001145636
C1orf228
922
382
2,4
NM 177980
CDH26
99
41
2,4
NM 207368
FAM195B
198
82
2,4
NM 001171
ABCC6
55
23
2,4
NM 006869
ADAP1
111
46
2,4
NM 006411 1
AGPAT1
55
23
2,4
NM 024097
C1orf50
55
23
2,4
NM 014648
DZIP3
111
46
2,4
NM 025241
UBXN8
55
23
2,4
NM 001696
ATP6V1E1
658
273
2,4
NM 012290
TMED8
67
28
2,4
NM 001040110
NRF1
159
66
2,4
NM 005694
COX17
91
38
2,4
NM 001160172
NAT1
91
38
2,4
NM 001018
RPS15
1273
529
2,4
NM 025209
EPC1
310
129
2,4
NM 001030001
RPS29
681
283
2,4
NM 002018
FLU
813
338
2,4
NM 006639
CYSLTR1
163
68
2,4
NM 004579
MAP4K2
163
68
2,4
NM 022727
TRMT61A
175
73
2,4
NM 058182
FAM165B
187
78
2,4
NM 006026
H1FX
187
78
2,4
NM 001190991
CNPY2
199
83
2,4
NM 003670
BHLHE40
223
93
2,4
NM 012292
HMHA1
1194
497
2,4
NM 014711
CCP110
319
133
2,4
NM 194326
RPS19BP1
331
138
2,4
NM 024669
ANKRD55
12
5
2,4
NM 001039083
ARL17B
24
10
2,4
NM 001670
ARVCF
36
15
2,4
NM 006994
BTN3A3
60
25
2,4
NM 001010863
C10orf128
12
5
2,4
NM 018186
C1orf112
60
25
2,4
NM 206921
C6orf204
12
5
2,4
NM 004196
CDKL1
24
10
2,4
NM 182853
CREM
36
15
2,4
NM 001249
ENTPD5
48
20
2,4
NM 001142800
EYS
12
5
2,4
NM 001122834
H HAT
36
15
2,4
NM 001243195
INPP5F
12
5
2,4
NM 000214
JAG1
12
5
2,4
NM 005560
LAMA5
36
15
2,4
NM 024316
LENG1
72
30
2,4
NM 001130090
LRRC48
12
5
2,4
NM 003010
MAP2K4
132
55
2,4
NM 014975
MAST1
12
5
2,4
NM 025176
NINL
120
50
2,4
NM 014323
PATZ1
540
225
2,4
NR 027694
PP2D1
48
20
2,4
NM 033215
PPP1R3F
12
5
2,4
NM 024070
PVRIG
12
5
2,4
NM 020436
SALL4
24
10
2,4
NM 017789
SEMA4C
36
15
2,4
NM 001024939
SLC2A9
24
10
2,4
NM 032354
TMEM134
12
5
2,4
NM 018056
TMEM42
120
50
2,4
NM 001037330
TRIM23
36
15
2,4
NM 001039111
TRIM9
24
10
2,4
NM 138466
ZNF846
24
10
2,4
NM 002229
JUNB
457
191
2,4
NM 001006947
UNC119
136
57
2,4
NM 001013699
H3F3C
795
332
2,4
NM 012398
PIP5K1C
261
109
2,4
NM 001145023
SCRN2
249
104
2,4
NM 001130029
RELL2
237
99
2,4
NM 145647
WDR7
165
69
2,4
NM 001134433
AZI2
282
118
2,4
NM 001032364
GGT1
64
27
2,4
NM 174940
TMEM86B
64
27
2,4
NM 002254
KIF3C
246
103
2,4
NM 002841
PTPRG
298
125
2,4
NM 013379
DPP7
948
397
2,4
NM 178495
ITPRIPL1
117
49
2,4
NM 001031723
DNAJB14
169
71
2,4
NM 006742
PSKH1
339
142
2,4
NM 001033551
TOMM40
339
142
2,4
NM 001421
ELF4
823
345
2,4
NM 006012
CLPP
262
110
2,4
NM 002126
HLF
52
22
2,4
NM 139032
MAPK7
52
22
2,4
NM 052818
N4BP2L1
52
22
2,4
NM 133373
PLCD3
52
22
2,4
NR 027861 1
ZNF660
105
44
2,4
NM 145245
EVI5L
145
61
2,4
NM 016368
ISYNA1
145
61
2,4
NR 036556
ANKRD54
133
56
2,4
NM 004900
APOBEC3B
81
34
2,4
NM 178830
C19orf47
81
34
2,4
NM 001042493
C6orf162
81
34
2,4
NM 006668
CYP46A1
40
17
2,4
NM 020116
FSTL5
40
17
2,4
NM 177417
KLC3
40
17
2,4
NM 144593
RHEBL1
40
17
2,4
NM 001042631
SDHAF1
121
51
2,4
NM 030899
ZNF34
40
17
2,4
NM 000177
GSN
2115
888
2,4
NM 001002877
THRA
300
126
2,4
NM 014297
ETHE1
109
46
2,4
NM 001100599
ZNF710
397
167
2,4
NM 004329
BMPR1A
178
75
2,4
NM 033358
CASP8
138
58
2,4
NM 005144
HR
138
58
2,4
NM 017514
PLXNA3
69
29
2,4
NM 001031628
SMAP1
166
70
2,4
NM 001029882
AHDC1
126
53
2,4
NM 016108
AIG1
126
53
2,4
NM 001001795
C8orf82
211
89
2,4
NR 002595
RPL31P11
354
149
2,4
NR 027995
ANKRD20A9P
57
24
2,4
NM 206895
C2orf82
28
12
2,3
NM 144611
CYB5D2
85
36
2,4
NM 001134945
GHRL
28
12
2,3
NM 153832
GPR161
28
12
2,3
NM 001166292
PTCH2
28
12
2,3
NM 170774
RASSF2
57
24
2,4
NM 033070
CECR5
550
232
2,4
NM 000956
PTGER2
130
55
2,4
NR 026660
RPL19P12
946
399
2,4
NM 001201472
COR07
204
86
2,4
NM 006039
MRC2
4384
1849
2,4
NM 018190
BBS7
73
31
2,4
NM 001130917
LILRA2
808
341
2,4
NM 020461
TXNDC17
73
31
2,4
NM 014171
CRIPT
118
50
2,4
NM 006284
TAF5
118
50
2,4
NM 005273
GNB2
756
319
2,4
NM 031287
SF3B5
417
176
2,4
NM 207174
ABCG1
45
19
2,4
NM 013375
ABT1
225
95
2,4
NR 024573
GHDC
135
57
2,4
NM 001142450
SPNS3
45
19
2,4
NR 015370
NCRNA00219
511
216
2,4
NM 030628
INTS5
331
140
2,4
NM 006257
PRKCQ
151
64
2,4
NM 033428
C9orf123
106
45
2,4
NM 001003897
MANBAL
106
45
2,4
NM 005645
TAGAP
213
90
2,4
NM 080626
BRI3BP
274
116
2,4
NM 052834
WDTC1
459
194
2,4
NM 001242476
ZNF362
229
97
2,4
NM 014343
CLDN15
61
26
2,3
NM 001190844
TMEM38B
295
125
2,4
NM 001144013
RGPD3
156
66
2,4
NM 018364
RSBN1
312
132
2,4
NM 001146154
PTGR2
94
40
2,4
NM 022566
MESDC1
205
87
2,4
NM 203352
PDLIM7
111
47
2,4
NM 002801
PSMB10
144
61
2,4
NM 001030
RPS27
144
61
2,4
NM 001001928
PPARA
193
82
2,4
NM 007346
OGFR
424
180
2,4
NM 020179
C11orf75
33
14
2,4
NM 001195125
C16orf95
16
7
2,3
NM 001145252
CFP
99
42
2,4
NM 001202558
CHURC1-FNTB
198
84
2,4
NM 014780
CUL7
181
77
2,4
NM 018351
FGD6
16
7
2,3
NM 002203
ITGA2
16
7
2,3
NM 032534
KRBA1
214
91
2,4
NM 024507
KREMEN2
16
7
2,3
NM 006337
MCRS1
330
140
2,4
NM 182980
OSGIN1
16
7
2,3
NM 001243136
PELI3
49
21
2,3
NM 001128602
RASGRP1
33
14
2,4
NR 037612
SEPT5-GP1BB
412
175
2,4
NM 001164771
SLC7A5P2
16
7
2,3
NR 002594
SLC9A7P1
16
7
2,3
NM 020705
TBC1D3P1-DHX40P1
16
7
2,3
NM 006510 3
TRIM3
16
7
2,3
NM 001008485
SLC41A3
235
100
2,4
NM 006640
SEPT9
1521
646
2,4
NM 001077624
ZNF865
186
79
2,4
NM 182687
PKMYT1
273
116
2,4
NM 021144
PSIP1
360
153
2,4
NM 001202403
ASB6
327
139
2,4
NM 001110556
FLNA
2794
1188
2,4
NM 001093772
KIT
7143
3110
2,3
NM 024050
DDA1
244
104
2,3
NM 001004356
FGFRL1
70
30
2,3
NM 000213
ITGB4
70
30
2,3
NM 032876
JUB
70
30
2,3
NM 024109
METTL22
70
30
2,3
NM 001097604 3
XK
759
323
2,3
NM 201453 1
CBWD3
108
46
2,3
NM 006494
ERF
561
239
2,3
NM 002117 2
HLA-C
237
101
2,3
NM 012072
CD93
91
39
2,3
NR 027634
FAM35B2
91
39
2,3
NM 020924
ZBTB38
91
39
2,3
NM 014729
TP53I3
166
71
2,3
NM 001165
BIRC3
37
16
2,3
NM 024713
C15orf29
37
16
2,3
NM 018948
ERRFI1
37
16
2,3
NM 007270
FKBP9
37
16
2,3
NM 031485
GRWD1
375
160
2,3
NR 024448
GUSBP11
75
32
2,3
NM 015202
KIAA0556
37
16
2,3
NM 016199
LSM7
337
144
2,3
NM 004546
NDUFB2
562
240
2,3
NM 032349
NUDT16L1
225
96
2,3
NM 001080419
USE1
225
96
2,3
NM 018260
ZNF707
112
48
2,3
NM 015374
SURF1
246
105
2,3
NM 030928
CDT1
1192
509
2,3
NM 001166167
NEK6
342
146
2,3
NM 006598
SLC12A7
304
130
2,3
NM 014061
MAGEH1
96
41
2,3
NM 014045
LRP10
501
214
2,3
NM 032036
IFI27L2
154
66
2,3
NM 021228
SCAF1
463
198
2,3
NM 018667
SNAP47
213
91
2,3
NM 198476
C19orf54
117
50
2,3
NM 018079
SRGAP2P2
58
25
2,3
NM 006369
LRRC41
493
211
2,3
NM 004085
TIPIN
159
68
2,3
NM 181597
USF1
238
102
2,3
NM 018641
CHST12
180
77
2,3
NM 022730
COPS7B
280
120
2,3
NM 004704
RRP9
264
113
2,3
NM 001021
RPS17
2503
1072
2,3
NM 001199057 1
RPS17L
2503
1072
2,3
NM 001031716
OBFC2A
205
88
2,3
NM 001199119
TRIM41
289
124
2,3
NM 001085461
CTNND1
63
27
2,3
NR 027468 1
FLJ39739
42
18
2,3
NM 001144869
LIPT2
21
9
2,3
NR 024402
MGC23284
21
9
2,3
NR 033882
MLK7-AS1
21
9
2,3
NM 002513
NME3
63
27
2,3
NM 001077497 6
PRR3
21
9
2,3
NR 037945
STX4
63
27
2,3
NM 152493
ZNF408
126
54
2,3
NM 001142577
ZNF784
84
36
2,3
NM 013321
SOLH
256
110
2,3
NM 004225
MFHAS1
450
193
2,3
NM 198526
ZNF737
193
83
2,3
NM 002908
REL
151
65
2,3
NM 022483
C5orf28
328
141
2,3
NM 052813
CARD9
109
47
2,3
NM 001164440
ANKRD33B
333
143
2,3
NM 004807
HS6ST1
312
134
2,3
NM 005148
UNC13D
1003
431
2,3
NM 173680
ZNF780A
67
29
2,3
NM 178129 1
P2RY8
949
408
2,3
NM 203282
ZNF282
591
254
2,3
NM 004418
DUSP2
114
49
2,3
NM 003655
CBX4
937
403
2,3
NM 000117
EMD
367
158
2,3
NM 016404
TRMT2A
507
218
2,3
NM 001347
DGKQ
186
80
2,3
NM 007207
DUSP10
46
20
2,3
NM 002428
MMP15
46
20
2,3
NM 145018
C11orf82
165
71
2,3
NM 000714
TSSC1
381
164
2,3
NM 032636
PSRC1
216
93
2,3
NM 213725
RPLP1
3439
1481
2,3
NM 001195215
DENND1B
220
95
2,3
NM 014606
HERC3
220
95
2,3
NM 020761
RPTOR
763
329
2,3
NM 144994
ANKRD23
51
22
2,3
NM 001199055
C16orf5
331
143
2,3
NM 017766
CASZ1
76
33
2,3
NM 001195422
GTPBP3
255
110
2,3
NM 001566
INPP4A
76
33
2,3
NM 175061
JAZF1
25
11
2,3
NM 033343
LHX4
25
11
2,3
NM 021177 1
LSM2
25
11
2,3
NM 016734
PAX5
51
22
2,3
NM 015324
RRP8
204
88
2,3
NM 006247
PPP5C
361
156
2,3
NM 003091
SNX29
157
68
2,3
NM 020971
SQSTM1
1422
614
2,3
NM 007371
BRD3
796
344
2,3
NM 015263
DMXL2
213
92
2,3
NM 001039132
ICAM4
106
46
2,3
NM 016558
SCAND1
213
92
2,3
NM 001334
CTSO
81
35
2,3
NM 018314
UGDH
81
35
2,3
NM 001166621
TRAPPC5
298
129
2,3
NM 005197
FOXN3
1911
826
2,3
NM 001204451
CCPG1
465
201
2,3
NM 005229
ELK1
465
201
2,3
NM 024329
EFHD2
333
144
2,3
NM 021012
KCNJ12
55
24
2,3
NM 030583
MATN2
111
48
2,3
NM 002833
PTPN9
448
194
2,3
NM 020428
SLC46A1
85
37
2,3
NM 001031721
ZNF618
256
111
2,3
NM 006397
RNASEH2A
372
161
2,3
NM 001134368
SLC7A2
201
87
2,3
NM 030813
CLPB
231
100
2,3
NM 021724
NR1D1
115
50
2,3
NM 017907
LAMTOR1
376
163
2,3
NM 133638
ADAMTS19
30
13
2,3
NR 027398
GFOD2
30
13
2,3
NM 001040616
LINS
60
26
2,3
NM 001178102
LOX
30
13
2,3
NM 032348
MXRA8
30
13
2,3
NM 153449
SLC30A1
120
52
2,3
NM 007237
SPAG1
30
13
2,3
NM 032737
LMNB2
1806
783
2,3
NM 001008395
C7orf59
214
93
2,3
NM 024757
EHMT1
678
294
2,3
NR 033338
C17orf70
438
190
2,3
NM 015949
GET4
283
123
2,3
NM 018216
PANK4
94
41
2,3
NM 001145909
TAOK2
189
82
2,3
NR 027882
ΒΑΧ
159
69
2,3
NM 014315
KLHDC2
159
69
2,3
NM 021825
CCDC90B
193
84
2,3
NM 001137551
LRRFIP1
129
56
2,3
NM 001184746
PAFAH1B2
64
28
2,3
NM 020895
GRAMD1A
357
155
2,3
NM 001014763
ETFB
228
99
2,3
NR 027179
NCRNA00188
654
284
2,3
NM 001024809
RARA
133
58
2,3
NM 021194
SLC38A10
1495
650
2,3
NM 001001852
PIM3
409
178
2,3
NM 001009
RPS5
3457
1503
2,3
NM 001135919
SLC4A2
720
313
2,3
NM 001193329
C9orf3
34
15
2,3
NM 001193466
KIAA1267
379
165
2,3
NR 039918
MIR4761
34
15
2,3
NM 004536
NAIP
34
15
2,3
NM 020144
PAPOLB
34
15
2,3
NM 138374
ZNF862
172
75
2,3
NM 001172566
MYD88
427
186
2,3
NR 024603
MPDU1
250
109
2,3
NM 018174
MAPI S
432
188
2,3
NM 022047
DEF6
579
252
2,3
NM 004563
PCK2
622
271
2,3
NM 001040118
ARAP1
882
384
2,3
NM 004701
CCNB2
441
192
2,3
NM 000734
CD247
73
32
2,3
NM 004448
ERBB2
73
32
2,3
NM 001034845
GALNTL6
73
32
2,3
NM 006545
NPRL2
186
81
2,3
NM 199249
C19orf48
1198
522
2,3
NM 000991
RPL28
3604
1570
2,3
NM 001008388
CISD2
268
117
2,3
NR 002796
AKR7A2P1
39
17
2,3
NM 001124767
C3orf78
39
17
2,3
NM 133328
DEDD2
78
34
2,3
NR 003260
DNM1P46
39
17
2,3
NM 022664
ECM1
195
85
2,3
NM 031904
FRMD8
390
170
2,3
NM 002269
KPNA5
39
17
2,3
NR 026052
MGC2752
156
68
2,3
NM 198513
PHF20L1
199
87
2,3
NM 001946
DUSP6
646
282
2,3
NM 001130410
ACAA1
243
106
2,3
NM 198402
PTPLB
165
72
2,3
NM 001099456
NPW
291
127
2,3
NM 006268
DPF2
543
237
2,3
NM 005194
CEBPB
304
133
2,3
NM 018390
PLCXD1
130
57
2,3
NM 015241
MICAL3
400
175
2,3
NM 006923
SDF2
270
118
2,3
NM 181050
AXIN1
274
120
2,3
NM 022153
C10orf54
91
40
2,3
NM 001193375
NDUFA11
231
101
2,3
NM 001199865
KIF16B
139
61
2,3
NM 001184898
PHF8
187
82
2,3
NM 020421
ADCK1
48
21
2,3
NM 005165
ALDOC
48
21
2,3
NM 207315
CMPK2
96
42
2,3
NM 012401
PLXNB2
96
42
2,3
NM 001242534
MFSD11
244
107
2,3
NR 033344
SLC39A10
297
130
2,3
NM 001013
RPS9
3367
1474
2,3
NM 006739
MCM5
1455
637
2,3
NM 022719
DGCR14
100
44
2,3
NM 024641
MANEA
100
44
2,3
NM 001171946
SUN2
1612
706
2,3
NM 181532
ERAS
253
111
2,3
NM 006404
PROCR
406
178
2,3
NM 000485
APRT
511
224
2,3
NM 001173988
C9orf86
1132
496
2,3
NM 015957
APIP
157
69
2,3
NM 001196
BID
630
276
2,3
NM 001217
CA11
52
23
2,3
NM 001810
CENPB
1156
507
2,3
NM 014047
C19orf53
333
146
2,3
NM 021210
TRAPPC4
223
98
2,3
NM 020801
ARRDC3
280
123
2,3
NR 003558
WDR18
337
148
2,3
NM 020914
RNF213
2818
1236
2,3
NM 001242821
DEF8
57
25
2,3
NM 020899
ZBTB49
114
50
2,3
NM 032789
PARP10
522
229
2,3
NR 028451
BCAT2
175
77
2,3
NM 198401
ANKRD46
118
52
2,3
NM 080655
C9orf30
298
131
2,3
NM 014960
ARSG
61
27
2,3
NR 030770
CCM2
307
135
2,3
NM 173502
PRSS36
61
27
2,3
NM 001100814
TMEM62
123
54
2,3
NM_023072
ZXDC
246
108
2,3
NM 001002247
ANAPC11
576
253
2,3
NM 001199254
SGOL1
66
29
2,3
NM 001010927
TIMM13
396
174
2,3
NM 001207009
ZNF543
66
29
2,3
NM_001079804
GAA
1342
590
2,3
NM 001031680
RUNX3
339
149
2,3
NM 001017928
CCDC58
136
60
2,3
NM 024989
PGAP1
136
60
2,3
NM 032901
C12orf62
343
151
2,3
NM 031280
MRPS15
207
91
2,3
NM 005735
ACTR1B
484
213
2,3
NM 080605
B3GALT6
423
186
2,3
NM 001381
DOK1
1128
496
2,3
NM 020205
OTUD7B
70
31
2,3
NM 001130160
STUB1
366
161
2,3
NM 130468
CHST14
75
33
2,3
NM 001113347
ECE1
234
103
2,3
NM 032522
ZBTB4
313
138
2,3
NM 138384
MTG1
79
35
2,3
NM 015148
PASK
159
70
2,3
NM 012268
PLD3
495
218
2,3
NM 001110354
ZSCAN2
247
109
2,3
NM 020862
LRFN1
88
39
2,3
NM 021991
JUP
1075
474
2,3
NM 001203262
NDUFC2-KCTD14
363
160
2,3
NR 034109
TRAF7
456
201
2,3
NM 015909
NBAS
465
205
2,3
NM 172389
NFATC1
186
82
2,3
NM 001143994
RASSF7
93
41
2,3
NM 080704
TSC22D4
664
293
2,3
NM 001143905
C12orf65
190
84
2,3
NM 001243283
ALCAM
106
47
2,3
NM 002972
SBF1
1096
484
2,3
NM 015159
FAM168A
111
49
2,3
NM 001136498
CISD3
337
149
2,3
NM 001145396
ALDH16A1
226
100
2,3
NM 212554
METTL10
115
51
2,3
NM 001003828
PARVB
267
118
2,3
NM 022839
MRPS11
276
122
2,3
NM 024036
LRFN4
147
65
2,3
NM 033257
DGCR6L
165
73
2,3
NM 006532
ELL
330
146
2,3
NR 033339
C4orf42
169
75
2,3
NM 003595
TRAF2
169
75
2,3
NM 033110
A2LD1
178
79
2,3
NM 173082
SHPRH
178
79
2,3
NM 001376
DYNC1H1
2713
1201
2,3
NM 001080542
FBF1
196
87
2,3
NM 004638 1
PRRC2A
397
176
2,3
NM 002360
MAFK
237
105
2,3
NM 033452
TRIM68
246
109
2,3
NM 144679
C17orf56
250
111
2,3
NM 019037
EXOSC4
268
119
2,3
NM 001130072
EPN1
349
155
2,3
NM 213601
TMEM104
453
201
2,3
NM 182565
FAM100B
942
418
2,3
NM 015594
TBCD
529
235
2,3
NM 021940
SMARCA4
925
411
2,3
NM 001142653
AARSD1
9
4
2,3
NM 032592
ACCS
63
28
2,3
NM 032955 5
AIF1
27
12
2,3
NM 170726
ALDH4A1
49
22
2,2
NM 001164443
ANKRD31
9
4
2,3
NM 019087
ARL15
27
12
2,3
NM 018179
ATF7IP
400
178
2,2
NM 178326
ATG4B
108
48
2,3
NM 001164782
ATXN3
225
100
2,3
NM 001136537
BTBD19
9
4
2,3
NR 015404
C12orf47
175
78
2,2
NM 001031726
C19orf12
112
50
2,2
NM 001135580
C19orf71
27
12
2,3
NR 027790
C21orf34
9
4
2,3
NM 182597
C7orf53
13
6
2,2
NR 023390
C9orf130
9
4
2,3
NM 032310
C9orf89
27
12
2,3
NM 001218
CA12
13
6
2,2
NM 024565
CCNJL
49
22
2,2
NM 174892
CD300LB
9
4
2,3
NM 004364
СЕВРА
220
98
2,2
NM 054113
CIB3
18
8
2,3
NM 001031617
COX19
337
150
2,2
NM 001348
DAPK3
198
88
2,3
NM 198083
DHRS4L2
99
44
2,3
NR 023383
DTX2P1-UPK3BP1-PMS2P11
18
8
2,3
NM 004444
EPHB4
85
38
2,2
NM 005236
ERCC4
67
30
2,2
NM 033266
ERN2
13
6
2,2
NM 016046
EXOSC1
45
20
2,3
NM 016049
FAM158A
40
18
2,2
NM 207334
FAM43B
9
4
2,3
NR 024254
FAM86FP
18
8
2,3
NM 001080472
FITM2
13
6
2,2
NM 001145777
FKBP5
229
102
2,2
NR 037596
FLJ14186
27
12
2,3
NR 033839
FLJ39051
9
4
2,3
NM 000148
FUT1
22
10
2,2
NM 003505
FZD1
31
14
2,2
NM 024637
GAL3ST4
9
4
2,3
NM 006143
GPR19
9
4
2,3
NM 001080423
GRIP2
9
4
2,3
NR 002932
GSTM2P1
63
28
2,3
NM 173497
HECTD2
27
12
2,3
NM 001114380
ITGAL
9
4
2,3
NM 014505
KCNMB4
13
6
2,2
NM 001100915
KCTD19
9
4
2,3
NM 001170331
LANCL3
13
6
2,2
NM__001014989
LAT
9
4
2,3
NM 153486
LDHD
13
6
2,2
NM 012163
LRRC29
9
4
2,3
NM 001146082
MBOAT7
193
86
2,2
NM 015078
MCF2L2
9
4
2,3
NR 031589
MIR1178
9
4
2,3
NM 001040000
MLLT4
247
110
2,2
NM 019556
MOSPD1
90
40
2,3
NM 030811
MRPS26
306
136
2,3
NM 000265
NCF1
9
4
2,3
NR 040116
NSF
85
38
2,2
NM 007224
NXPH4
9
4
2,3
NM 030758
OSBP2
9
4
2,3
NM 007365
PADI2
139
62
2,2
NM 001037281
PARD6A
54
24
2,3
NM 001111307
PDE4A
130
58
2,2
NM 005391
PDK3
22
10
2,2
NM 152305
POGLUT1
445
198
2,2
NM 000941
POR
139
62
2,2
NM 020440
PTGFRN
9
4
2,3
NM 002830
PTPN4
27
12
2,3
NM 206827
RASL11A
9
4
2,3
NM 001195303
RGS5
9
4
2,3
NM 173557
RNF152
9
4
2,3
NM 170769
RNF39
9
4
2,3
NM 015077
SARM1
63
28
2,3
NM 014654
SDC3
9
4
2,3
NM 004186
SEMA3F
49
22
2,2
NM 015559
SETBP1
13
6
2,2
NM 001001713
SH3BGR
27
12
2,3
NM 031469
SH3BGRL2
9
4
2,3
NM 001177549
SIGLEC6
31
14
2,2
NM 001242366
SLC46A3
45
20
2,3
NM 001128848
SMCR7
45
20
2,3
NR 024543
SNN
40
18
2,2
NR 024542
SNORA32
9
4
2,3
NR 003078
SNORD35A
9
4
2,3
NM 005632
SP140
9
4
2,3
NM 001146316
SPTBN1
2479
1102
2,2
NM 003765
STX1B
9
4
2,3
NM 001165030
TMEM53
9
4
2,3
NM 024956
TMEM63C
27
12
2,3
NR 026678
TMEM80
171
76
2,3
NM 145274
TMUB1
175
78
2,2
NM 004412
TRIM16
31
14
2,2
NM 020972
ZGLP1
27
12
2,3
NM 001029976
ZNF165
18
8
2,3
NM 003801
GPAA1
784
349
2,2
NM 001006623
WDR33
532
237
2,2
NM 006456
ST8SIA4
348
155
2,2
NM 015894
STRA13
348
155
2,2
NM 001242395
TAC01
330
147
2,2
NM 030577
TMEM206
294
131
2,2
NM 207585
IFNAR2
262
117
2,2
NM 022151
MOAP1
249
111
2,2
NM 212535
PRKCB
249
111
2,2
NM 022061
MRPL17
471
210
2,2
NM 021818
SAV1
235
105
2,2
NM 080656
CDKN2AIPNL
231
103
2,2
NM 005631
SMYD2
208
93
2,2
NM 002975
CLEC11A
2970
1325
2,2
NM 030812
ACTL8
535
239
2,2
NM 199054
MKNK2
703
314
2,2
NM 024762
ZNF574
331
148
2,2
NM 001142557
HMMR
327
146
2,2
NM 000973
RPL8
7986
3566
2,2
NM 014595
NT5C
313
140
2,2
NM 006435
IFITM2
468
209
2,2
NM 030767
AKNA
618
276
2,2
NM 001166139
LCORL
145
65
2,2
NM 030935
TSKU
145
65
2,2
NM 032595
PPP1R9B
1879
840
2,2
NM 139280
ORMDL3
523
234
2,2
NM 015255
UBXN6
364
163
2,2
NM 001130978
DYSF
241
108
2,2
NM 152592
C14orf49
118
53
2,2
NM 001172666
RAB40C
118
53
2,2
NM 138769
RHOT2
237
106
2,2
NM 198088
ZNF253
114
51
2,2
NM 032608
MY018B
219
98
2,2
NM 016373
XAGE1A
109
49
2,2
NM 001097592 3
XAGE1B
109
49
2,2
NM 001097596 3
XAGE1C
109
49
2,2
NM 001097597 3
XAGE1D
109
49
2,2
NM 020411 3
XAGE1E
109
49
2,2
NR 033953
P2RX7
214
96
2,2
NR 003068
SNRNP70
534
239
2,2
NM 001099627
FAM54B
205
92
2,2
NM 014216
ITPK1
511
229
2,2
NM 025029
MZT2B
306
137
2,2
NM 012086
GTF3C3
507
227
2,2
NM 032790
ORAM
196
88
2,2
NM 001098614
PUS7L
196
88
2,2
NR 002924
TBKBP1
585
262
2,2
NM 022337
RAB38
96
43
2,2
NM 012088
PGLS
187
84
2,2
NM 001784
CD97
462
207
2,2
NM 001024382
HMBS
183
82
2,2
NM 001113755
TYSND1
357
160
2,2
NM 020725
ATXN7L1
265
119
2,2
NR 004847 1
UBL7
169
76
2,2
NM 015254
KIF13B
252
113
2,2
NM 004381 2
ATF6B
82
37
2,2
NM 001163023
PLK1S1
325
146
2,2
NM 004957
FPGS
568
255
2,2
NM 003325
HIRA
486
218
2,2
NM 033240
PML
642
288
2,2
NM 000681
ADRA2A
78
35
2,2
NM 198531
ATP9B
156
70
2,2
NM 015192
PLCB1
78
35
2,2
NR 003661
UBE2S
303
136
2,2
NR 026890
RPSAP9
666
299
2,2
NM 006133
DAGLA
147
66
2,2
NM 022117
TSSC4
294
132
2,2
NM 001172659
ZFYVE9
73
33
2,2
NM 001002836
ZNF799
73
33
2,2
NM 005993
TBX1
285
128
2,2
NM 001134431
TP53I13
285
128
2,2
NM 018218
UXS1
211
95
2,2
NR 026844
ANKRD36BP1
69
31
2,2
NM 031910
C1QTNF6
69
31
2,2
NM 014571
HEYL
138
62
2,2
NR 033688
PEF1
207
93
2,2
NM 006747
SIPA1
474
213
2,2
NM 018166
FAM176B
64
29
2,2
NM 183421
FBX025
64
29
2,2
NM 005537
ING1
129
58
2,2
NM 001164811
PET117
253
114
2,2
NM 198264
FAM189B
189
85
2,2
NM 002390
ADAM 11
60
27
2,2
NM 019007
ARMCX6
180
81
2,2
NM 007098
CLTCL1
60
27
2,2
NM 002757
MAP2K5
60
27
2,2
NM 003046
SLC9A7
60
27
2,2
NM 013241
FHOD1
402
181
2,2
NM 030783
PTDSS2
171
77
2,2
NM 130847
AMOTL1
508
229
2,2
NM 013291
CPSF1
1498
675
2,2
NM 138418
FAM195A
55
25
2,2
NM 023007
JMJD4
222
100
2,2
NM 182958
KAT8
111
50
2,2
NM 003730
RNASET2
495
223
2,2
NM 182526
TMEM39B
162
73
2,2
NM 016567
BCCiP
213
96
2,2
NR 037849
INO80B-WBP1
315
142
2,2
NM 001014432
AKT1
727
328
2,2
NM 000465
BARD1
102
46
2,2
NM 001001520
HDGFRP2
306
138
2,2
NM 014714
IFT140
51
23
2,2
NM 198970
AES
705
318
2,2
NM 015292
ESYT1
1494
674
2,2
NM 016219
MAN1B1
747
337
2,2
NM 153690
FAM43A
148
67
2,2
NM 032718
MFSD9
97
44
2,2
NM 001130677
C17orf96
241
109
2,2
NM 001105247
ARMC5
144
65
2,2
NM 001099271
POC5
144
65
2,2
NM 004422
DVL2
190
86
2,2
NM 001199196
ARMC6
664
300
2,2
NM 000980
RPL18A
1599
722
2,2
NM 144581
C14orf149
93
42
2,2
NR 002786
CIDECP
46
21
2,2
NM 031435
THOC5
232
105
2,2
NM 030576
LIMD2
1251
565
2,2
NM 015106
RAD54L2
316
143
2,2
NM 000487
ARSA
135
61
2,2
NM 001169109
SC02
270
122
2,2
NM 014205
ZNRF2
135
61
2,2
NM 001666
ARHGAP4
1033
467
2,2
NM 012345
NUFIP1
88
40
2,2
NM 001150
ANPEP
792
358
2,2
NM 004091
E2F2
261
118
2,2
NM 014698
TMEM64
130
59
2,2
NM 001145462
YIPF2
345
156
2,2
NM 001037161
ACOT1
214
97
2,2
NM 005137
DGCR2
471
213
2,2
NM 173832
ZFYVE1
256
116
2,2
NM 001163323
CCDC120
42
19
2,2
NM 032622
LNX1
42
19
2,2
NM 001001290
SLC35E3
84
38
2,2
NM 182633
ZNF749
42
19
2,2
NR 026717 5
STK38L
327
148
2,2
NM 001111039
ZNF192
435
197
2,2
NR 037874
ZMAT3
355
161
2,2
NM 079834
SCAMP4
196
89
2,2
NM 015285
WDR90
117
53
2,2
NR 029417
SHMT2
1935
877
2,2
NM 024546
RNF219
417
189
2,2
NM 032854
COR06
75
34
2,2
NM 002224
ITPR3
37
17
2,2
NM 002736
PRKAR2B
75
34
2,2
NM 001142532
SH2D3C
37
17
2,2
NM 022553 1
WASF3
262
119
2,2
NM 003902
FUBP1
408
185
2,2
NM 005190
CCNC
258
117
2,2
NM 015229
KIAA0664
699
317
2,2
NM 212533
ABCA2
145
66
2,2
NM 020937
FANCM
145
66
2,2
NM 001011
RPS7
3222
1462
2,2
NM 001146284
TCFL5
178
81
2,2
NM 007104
RPL10A
2448
1111
2,2
NM 001145839
MTFR1
141
64
2,2
NM 198486
RPL7L1
564
256
2,2
NM 005720
ARPC1B
1824
828
2,2
NM 152424
FAM123B
456
207
2,2
NM 006086
TU FM
1575
715
2,2
NM 001025604
ARRDC2
522
237
2,2
NM 004505
VAC 14
174
79
2,2
NM 014602
PIK3R4
409
186
2,2
NM 001142503
STAU1
508
231
2,2
NM 033071
SYNJ2BP
169
77
2,2
NM 080820
DTD1
400
182
2,2
NM 145004
ADAM32
33
15
2,2
NM 138701
C7orf11
264
120
2,2
NM 001145268
FAM185A
33
15
2,2
NM 001177702
IFT27
33
15
2,2
NM 001134774
KLC2
66
30
2,2
NM 014067
MACROD1
66
30
2,2
NM 018049
PLEKHJ1
330
150
2,2
NM 175886
PRPS1L1
33
15
2,2
NM 001039573
SEC14L1
264
120
2,2
NM 006949
STXBP4
33
15
2,2
NM 001078651
TMEM143
33
15
2,2
NM 001167734
VNN1
33
15
2,2
NM 003447
ZNF175
132
60
2,2
NM 145201
NAPRT1
259
118
2,2
NM 012455
PSD4
580
264
2,2
NM 001106
ACVR2B
321
146
2,2
NM 033418
METTL18
127
58
2,2
NR 033814
DLST
477
217
2,2
NM 052852
ZNF496
378
172
2,2
NM 152331
ACOT4
61
28
2,2
NM 003887
ASAP2
61
28
2,2
NM 001201335
C2orf3
123
56
2,2
NM 005211
CSF1R
61
28
2,2
NM 001001660
LYRM5
61
28
2,2
NM 005952
MT1X
61
28
2,2
NM 001130082
PLXNB1
61
28
2,2
NM 198155
C21orf33
838
382
2,2
NM 001172681
ZNF646
327
149
2,2
NM 001243130
RPL13
10660
4858
2,2
NM 006912
RIT1
147
67
2,2
NM 013373
ZFAT
147
67
2,2
NM 015074
KIF1B
175
80
2,2
NM 032504
UNC93B1
640
292
2,2
NR 027856
CLK1
522
238
2,2
NM 001918
DBT
318
145
2,2
NM 001130725
PSMB5
460
210
2,2
NM 001552
IGFBP4
1429
652
2,2
NM 017850
C1orf109
199
91
2,2
NM 024821
CCDC134
28
13
2,2
NM 007186
CEP250
741
338
2,2
NM 018217
EDEM2
399
182
2,2
NM 133509
RAD51B
28
13
2,2
NR 002309
RPS26P11
28
13
2,2
NM 001098509
SGSM2
199
91
2,2
NM 001195267
SSH3
28
13
2,2
NM 001168215
TMEM99
57
26
2,2
NM 001242475
ZNF382
57
26
2,2
NM 198458
ZNF519
28
13
2,2
NM 006218
PIK3CA
195
89
2,2
NM 005934
MLLT1
1141
521
2,2
NM 002695
POLR2E
609
278
2,2
NM 021224
ZNF48
138
63
2,2
NM 024840
ZNF627
385
176
2,2
NM 138346
KIAA2013
766
350
2,2
NM 001190980
YIF1A
219
100
2,2
NM 005586
MDFI
300
137
2,2
NM 002501
NFIX
705
322
2,2
NM 145265
CCDC127
162
74
2,2
NM 005113
GOLGA5
243
111
2,2
NM 001130970
NELF
162
74
2,2
NM 001077493
NFKB2
81
37
2,2
NM 006509
RELB
81
37
2,2
NM 001098
AC02
700
320
2,2
NM 001136158
OTUD5
376
172
2,2
NM 003846
PEX11B
214
98
2,2
NR 024022
SETMAR
133
61
2,2
NM 001040273
UBASH3B
1116
510
2,2
NM 003449 7
TRIM28
2671
1221
2,2
NM 152721
DOK6
52
24
2,2
NM 001040427
HAG H
52
24
2,2
NM 001198989
NFS1
105
48
2,2
NM 001131026
PEX5
105
48
2,2
NR 037673
SERF2-C150RF63
210
96
2,2
NM 174914
UHRF1BP1L
105
48
2,2
NM 001202473
ZNF845
157
72
2,2
NM 001166237
GSDMD
706
323
2,2
NM 004995
MMP14
778
356
2,2
NM 183075
CYP2U1
205
94
2,2
NM 016025
METTL9
358
164
2,2
NM 001037171
ACOT9
76
35
2,2
NM 001193321
IKBKE
306
140
2,2
NM 000363
TNRC6B
483
221
2,2
NM 001134473
KIAA0182
660
302
2,2
NM 016360
TAF10
277
127
2,2
NM 001193307
SAMD9
378
173
2,2
NM 002883
RANGAP1
1057
484
2,2
NM 007323
ZHX1
201
92
2,2
NM 032184
SYNGR2
1728
791
2,2
NM 024555
FBXL6
124
57
2,2
NM 153687
IKBIP
124
57
2,2
NM 001242827
SIRT5
124
57
2,2
NM 020197
SNAPC2
124
57
2,2
NM 138463
TLK1
517
237
2,2
NM 024596
MCPH1
220
101
2,2
NM 138804
C2orf65
264
121
2,2
NM 024793
CLUAP1
24
11
2,2
NM 019609
CPXM1
48
22
2,2
NM 173537
GTF2IRD2
48
22
2,2
NM 022070
HEATR6
360
165
2,2
NM 001134435
IQCG
24
11
2,2
NM 173546
KLHDC8B
72
33
2,2
NM 016027
LACTB2
216
99
2,2
NM 078628
MSL3
72
33
2,2
NM 002135
NR4A1
24
11
2,2
NM 013239
PPP2R3B
24
11
2,2
NM 022370
ROB03
72
33
2,2
NM 182756
SPDYE2
24
11
2,2
NM 001142634
SPDYE2L
24
11
2,2
NM 001242783 7
TRIM27
24
11
2,2
NM 017890
VPS52
24
11
2,2
NM 017984
ZDHHC11
24
11
2,2
NM 145238
ZSCAN5A
24
11
2,2
NM 001127183
CFLAR
379
174
2,2
NM 001077261
NCOR2
1134
520
2,2
NM 001002878
THOP1
519
238
2,2
NM 017858
TKT
2287
1049
2,2
NM 001114618
MGAT1
1153
529
2,2
NM 199287
CCDC137
658
302
2,2
NM 138392
SHKBP1
327
150
2,2
NM 006184
NUCB1
630
289
2,2
NM 001083330
ZNF16
115
53
2,2
NM 022156
DUS1L
847
389
2,2
NM 145267
C6orf57
91
42
2,2
NM 001205029
MDM1
91
42
2,2
NM 001024678
LRRC24
159
73
2,2
NM 001100122
SBN02
318
146
2,2
NM 001164234
DDHD2
67
31
2,2
NR 036432
HERC2P3
67
31
2,2
NM 001243247
NPRL3
135
62
2,2
NM 144568
TMEM63A
135
62
2,2
NM 080732
EGLN2
535
246
2,2
NM 001005920
JMJD8
357
164
2,2
NR 003662
RPSAP58
10575
4858
2,2
NM 001184970
PACSIN2
468
215
2,2
NM 004215
EBAG9
111
51
2,2
NM 024303
ZSWIM6
111
51
2,2
NM 001009939
SEPT5
376
173
2,2
NR 038123
PSMA3
198
91
2,2
NM 014298
QPRT
198
91
2,2
NM 030912
TRMT112
570
262
2,2
NM 001105576
ANKRD58
43
20
2,2
NM 194441
BTN3A1
87
40
2,2
NM 001206879
CTDSP1
174
80
2,2
NM 019005
MIOS
304
140
2,2
NM 001174085
POLL
87
40
2,2
NM 004881
TPM2
43
20
2,2
NM 001199119 4
TRIM45
87
40
2,2
NM 006958
ZNF174
43
20
2,2
NM .002419
MAP3K11
628
289
2,2
NM 006114
TOMM5
387
178
2,2
NM 006901
MY09A
256
118
2,2
NM 016538
SIRT7
276
127
2,2
NM 017457
CYTH2
315
145
2,2
NM 021976 3
RXRB
63
29
2,2
NM 001042462
TRDMT1
63
29
2,2
NM 007113
TCP11L1
145
67
2,2
NM 015318
ARHGEF18
810
373
2,2
NM 020959
AN08
82
38
2,2
NM 005479
FRAT1
82
38
2,2
NM 000359
THAP6
82
38
2,2
NM 018210
CARKD
267
123
2,2
NM 004047
ATP6V0B
553
255
2,2
NM 001122956
DBNL
922
425
2,2
NM 001103167
ZKSCAN1
1024
472
2,2
NM 024661
CCDC51
306
141
2,2
NM 138443
HAUS1
204
94
2,2
NM 001029989
KIAA0101
588
271
2,2
NR 026702
GLYCTK
160
74
2,2
NM 001165417
SLC25A13
199
92
2,2
NM 052853
ADCK2
238
110
2,2
NM 178557
NAT8L
516
238
2,2
NR 037653
ST3GAL2
258
119
2,2
NR 037791
RAB4B-EGLN2
555
256
2,2
NM 003743
NCOA1
316
146
2,2
NM 004958
MTOR
730
337
2,2
NM 001127397
ERLEC1
433
200
2,2
NM 001122772
AGAP2
570
263
2,2
NM 138795
ARL8A
175
81
2,2
NM 001183
ATP6AP1
858
396
2,2
NM 000397
CYBB
19
9
2,1
NM 201563
FCGR2C
19
9
2,1
NM 020400
LPAR5
19
9
2,1
NM 001159644
MCTP2
58
27
2,1
NM 001145785
MEF2B
19
9
2,1
NM 176796
P2RY6
39
18
2,2
NR 029383
PAN3-AS1
19
9
2,1
NM 001127692
PCCA
136
63
2,2
NM 002601
PDE6D
78
36
2,2
NM 014172
PHPT1
234
108
2,2
NM 002690
POLB
39
18
2,2
NM 001199771
RDH5
19
9
2,1
NM 001037442
RUFY3
39
18
2,2
NM 024583
SCRN3
175
81
2,2
NM 001145541
TEAD4
78
36
2,2
NM 025246
TMEM229B
19
9
2,1
NM 017955
CDCA4
385
178
2,2
NM 007311
TSPYL2
327
151
2,2
NM 153717
EVC
268
124
2,2
NM 002013
FKBP3
229
106
2,2
NM 021626
SCPEP1
381
176
2,2
NM 138639
BCL2L12
151
70
2,2
NM 014284
NCDN
415
192
2,2
NM 022460
HS1BP3
132
61
2,2
NM 002473
MYH9
5848
2703
2,2
NM 001080432
FTO
337
156
2,2
NM 033416
IMP4
225
104
2,2
NM 003089
SNRPB
1345
622
2,2
NM 001032282
KLF10
205
95
2,2
NM 003025
SH3GL1
298
138
2,2
NM 018380
DDX28
259
120
2,2
NM 006059
LAMC3
426
197
2,2
NM 001012329
CTNNBIP1
166
77
2,2
NR 002182
NACAP1
333
154
2,2
NM 022648
TOM1
166
77
2,2
NM 006185
NUMA1
2640
1221
2,2
NM 001137601
ZBTB5
387
179
2,2
NM 006916
RPE
201
93
2,2
NM 003824
FADD
382
177
2,2
NM 016458 1
FAM203A
127
59
2,2
NM 001136015
ANXA2
181
84
2,2
NM 015531
C2CD3
181
84
2,2
NM 001004720
NCK2
363
168
2,2
NM 022750
PARP12
235
109
2,2
Таким образом, был проведён анализ подавления экспрессии онкогенов в перевиваемых кроветворных клетках, трансдуцированных лентивирусным вектором, направляющим синтез shPHK, который опосредует подавление активированного онкогена AML1-ETO. Было показано, что трансдукция клеток линии Kasumi-1 с помощью лентивирусных векторов, направляющих синтез специфических shPHK, предшественников siPHK, комплементарных в отношении онкогена AML1-ETO, приводит к десятикратному снижению уровня экспрессии указанного онкогена.
С помощью применённого подхода с использованием метода глубокого секвенирования на платформе Illumina было идентифицировано около 2500 тысяч генов, экспрессия которых меняется при подавлении экспрессии активированного онкогена AML1-ETO. Из представленного списка генов представляется возможным выбрать гены, которые непосредственно могут быть прямым или косвенным образом вовлечены в процессы, связанные с развитием острого миелоидного лейкоза. Исследование функции таких генов с помощью предлагаемого способа предоставляет возможность исследовать обнаруженные гены на предмет их использования в качестве генов-мишеней для диагностики и терапии ОМЛ.