Изобретение относится к области молекулярной биологии, биотехнологии и медицины, и может быть использовано в молекулярно-генетической диагностике онкологических заболеваний и для оценки эффективности противораковой терапии, а именно для анализа метилирования генов-онкомаркеров колоректального рака в образцах биологического материала.
Перспективным инструментом молекулярно-генетической диагностики и мониторинга онкологических заболеваний является эпигенетическая диагностика, то есть идентификация болезни путем выявления эпигенетических маркеров. Эпигенетические маркеры представляют собой химические группы, которые присоединяются к геномной ДНК человека (метилирование цитозиновых оснований с образованием 5-метилцитозина, 5mC) либо к ассоциированным с ней гистоновым комплексам и оказывают влияние на функциональную активность генов. Наиболее распространенным эпигенетическим нарушением при канцерогенезе является локальное гиперметилирование отдельных участков ДНК, так называемых CpG-островков, в регуляторных областях генов-онкосупрессоров, которые в норме лишены метальных групп. В настоящее время показано, что подобное аберрантное метилирование характерно для начальных стадий большинства ненаследственных (спорадических) форм рака, которые в среднем составляют более 90% всех случаев злокачественных новообразований.
Для осуществления эпигенетической диагностики злокачественных новообразований необходимо решить два ключевых вопроса:
а) какой метод детекции следует использовать для определения статуса метилирования генов;
б) метилирование регуляторных участков каких генов ассоциировано с конкретным онкологическим заболеванием?
Для решения первого вопроса, а именно для анализа статуса метилирования генов, применяется ряд методов, которые можно разделить на четыре группы.
Первая группа методов основана на бисульфитной конверсии ДНК, влекущей изменения ее нуклеотидного состава. В дальнейшем нужный участок конвертированного генома может быть амплифицирован в ПЦР и секвенирован для определения всех метилированных цитозиновых остатков. Если исследователя интересует статус метилирования конкретного CG-динуклеотида, то используются более простые способы анализа. Так, возможно проведение метилчувствительной ПЦР (другой употребляемый термин - «метилспецифическая ПЦР»), в которой проводятся две реакции с использованием праймеров, подобранных для случаев прохождения или непрохождения конверсии в исследуемом динуклеотиде. Другим широко используемым способом определения метилирования является обработка конвертированной ДНК эндонуклеазами рестрикции (метод COBRA, основанный на появлении или исчезновении сайтов узнавания ферментов в измененной ДНК). К недостаткам описанных методов относятся существенная деградация и значительные потери нуклеиновых кислот при проведении бисульфитной конверсии, что делает проблематичным анализ малого количества материала, трудоемкость, дороговизна исследования и значительные затраты времени [Umer M, Herceg Z. Deciphering the epigenetic code: an overview of DNA methylation analysis methods // Antioxid Redox Signal. - 2013. - V. 18. - P. 1972-1986].
Вторая группа методов включает в себя процедуры гидролиза ДНК метилчувствительными эндонуклеазами. Анализ статуса метилирования генов основан на том, что данные рестриктазы не способны распознавать и расщеплять последовательности, содержащие метилированные CG-сайты (например, фермент HpaII, узнающий и гидролизующий лишь неметилированный тетрануклеотид CCGG). Результаты реакции гидролиза могут быть проанализированы с помощью ПЦР с фланкирующими праймерами. Существенным ограничением для широкого применения этих методов является относительно небольшое число известных эндонуклеаз рестрикции, распознающих последовательности, содержащие CG-динуклеотиды, и при этом чувствительных к метилированию цитозиновых остатков в них [Shen L, Waterland RA. Methods of DNA methylation analysis // Curr Opin Clin Nutr Metab Care. - 2007. - V. 10. - P. 576-581]. Данный подход также не позволяет выявлять частично метилированную ДНК, тогда как именно такой паттерн метилирования CpG-островков характерен для самых ранних стадий канцерогенеза. Кроме того, использование данных методов предъявляет более высокие требования как к чистоте анализируемой ДНК, так и к качеству проведения исследования на всех стадиях, поскольку вывод о наличии метилирования в этих методах делается при отсутствии гидролиза ДНК метилчувствительными эндонуклеазами рестрикции. Однако недогидролиз ДНК может происходить в случаях наличия загрязнений или нарушений условий анализа. Это приводит к ложным выводам о наличии метилирования и как следствие к ложно-положительному диагнозу.
Третья группа методов базируется на использовании метил-связывающих белков (methyl binding domain containing proteins, MBD-белки) для изоляции метилированной ДНК и ее последующей амплификации. Известна, например, технология MethylMeter, разработанная специалистами американской компании RiboMed (патент Канады №2724343, опубл. 15.05.2008 г.), позволяющая достигать высоких показателей чувствительности и специфичности. Данная технология используется производителями некоторых тест-систем, например, G-CIMP DecisionDX американской компании Castle Biosciences. Для защиты данной тест-системы подана заявка США №2014272986, опубликованная 18.09.2014 г. Тем не менее, подобные методы остаются весьма трудоемкими, а для четкого количественного анализа результатов требуется сложное дорогостоящее оборудование.
Четвертая группа методов базируется на использовании сайт-специфических метилзависимых ДНК эндонуклеаз. После их открытия и начала коммерческого производства их препаратов появилась возможность использования этого нового класса ферментов в эпигенетических исследованиях. В отличие от обычных эндонуклеаз рестрикции, метилзависимые ферменты узнают только метилированные последовательности ДНК, что делает их еще одним удобным инструментом изучения статуса метилирования генов. В число ферментов данного класса входят производимые в России компанией ООО «СибЭнзайм» эндонуклеазы GlaI, BisI, BlsI, MteI и ряд других [http://russia.sibenzyme.com/products/m2_type].
Благодаря совпадению последовательности ДНК, являющейся продуктом метилирования de novo, и субстратной специфичности фермента GlaI исследователями ООО «СибЭнзайм» был разработан метод, названный GLAD-ПЦР (GlaI hydrolysis and Ligation Adapter Dependent PCR), который позволяет определять наличие сайтов R(5mC)GY в исследуемом участке ДНК [Кузнецов В. В. с соавт. Способ определения нуклеотидной последовательности Pu(5mC)GPy в заданном положении протяженной ДНК // Патент РФ №2525710 С1, опубл. 20.08.2014, Бюл. № 23]. Метод включает гидролиз ДНК ферментом GlaI, присоединение специфического адаптера к концам гидролизованной ДНК и последующую реакцию ПЦР в режиме реального времени с праймеров, окружающих целевой участок ДНК. Метод позволяет определить наличие одного или нескольких сайтов Pu(5mC)GPy внутри изучаемого фрагмента ДНК. Таким образом, метод GLAD-ПЦР может быть использован для определения статуса метилирования регуляторных участков генов.
Данный метод обладает рядом преимуществ, заключающихся в простоте выполнения, способности выявлять лишь метилированные сайты в сложных смесях ДНК и возможности количественно оценивать их число. Следует также отметить, что метод обладает значительной универсальностью и может быть применен для анализа любых сложных смесей ДНК, например, ДНК из опухолей, содержащей нуклеиновые кислоты не только из малигнантных, но и из нормальных клеток (клетки сосудов, соединительной ткани и т.д.). Апробация GLAD-ПЦР показала, что метод обладает высокой чувствительностью, позволяя детектировать метилированные сайты в сильноразбавленных растворах.
Указанный метод является универсальным методом детекции метилирования сайтов R(5mC)GY, позволяющим сформировать и описать конкретную панель генов, специфичную, например, для колоректального рака. Но в самом описании метода специфичные для различных видов онкологии панели генов отсутствуют.
Рак толстого кишечника и прямой кишки (колоректальный рак) - один из наиболее часто встречающихся видов злокачественных новообразований. В развитых странах мира ежегодно диагностируется более миллиона случаев этого заболевания, а летальность от рака этой локализации составляет более 33% даже в странах с высоким уровнем онкологической помощи [Walther A. et al. Genetic prognostic and predictive markers in colorectal cancer // Nat. Rev. Cancer. - 2009. - V. 9. - P. 489-499]. Как и в случае большинства других онкологических заболеваний, выявление данного вида рака на ранних стадиях во многом определяет успешность лечения, ввиду чего развитие чувствительных молекулярно-генетических, в том числе эпигенетических, методов диагностики колоректального рака несомненно является актуальной задачей. Однако начальные стадии патологии внутренних органов зачастую не сопровождаются определяемыми при наружном осмотре аномалиями или ощущаемыми пациентами нарушениями функций организма, что делает раннюю диагностику в этих случаях крайне затруднительной. Таким образом, использование биохимических и генетических анализов для диагностики колоректального рака является предпочтительным способом выявления заболевания, тем более, что такая диагностика возможна еще на бессимптомной стадии [Gyparaki MT, Basdra EK, Papavassiliou AG. DNA methylation biomarkers as diagnostic and prognostic tools in colorectal cancer // J Mol Med (Berl). - 2013. - V. 91. - P. 1249-1256].
Для разработки соответствующей диагностической системы необходимо найти ответ на второй вопрос: метилирование регуляторных участков каких именно генов ассоциировано с развитием колоректального рака?
Одна из первых масштабных работ по поиску маркерных генов для эпигенетической диагностики колоректального рака была проведена в 2008 году сотрудниками компании Epigenomics AG (Германия) [Lofton-Day C et al. DNA methylation biomarkers for blood-based colorectal cancer screening // Clin Chem. - 2008. - V. 54. - P. 414-423]. На первом этапе работы были использованы метилчувствительные ферменты рестрикции для анализа более 600 кандидатных фрагментов геномной ДНК, выделенной из опухолей и нормальной ткани. Были выбраны 56 участков ДНК, которые достоверно различались по степени метилирования в здоровых и малигнантных клетках. Далее эти участки дополнительно проверялись на микрочипах и в ПЦР в реальном времени. Это позволило сократить число генов, наиболее пригодных для использования в качестве эпигенетических маркеров колоректального рака, до семи (SEPT9, NGFR, TMEFF2, ALX4, EYA4, FOXL2, SIX6).
В дальнейшем большое число авторов опубликовало результаты своих исследований по поиску генов-онкомаркеров для эпигенетической диагностики колоректального рака, проведенных различными методами, включая такие современные подходы, как применение ДНК-микрочипов (например, серии Infinium HumanMethylation, Illumina, США), ограниченное (RRBS, Reduced Representation Bisulfite Sequencing) и полногеномное бисульфитное секвенирование (WGBS, Whole Genome Bisulphite Sequencing) секвенирующими системами нового поколения (Next Generation Sequencing, NGS) [Reinders J. et al. // Epigenomics. - 2010. - V. 2. - P. 209-220; Kaneda A., Yagi K. // Cancer Sci. - 2011. - V. 102. - P. 18-24; Kibriya M.G. et al. // BMC Med Genomics. - 2011. - 4:50; Kim Y.H. et al. // Ann Surg Oncol. - 2011. - V. 18. - P. 2338-2347; Gu H. et al. // Nat Protoc. - 2011. - V. 6. - P. 468-481; Yi J.M. et al. // Tumour Biol. - 2012. - V. 33. - P. 363-372; Hinoue T. et al. // Genome Res. - 2012. - V. 22. - P. 271-282; Lange C.P. et al. // PLoS One. - 2012. - 7:e50266; Li H. et al. // Oncol Rep. - 2012. - V. 28. - P. 99-104; Roperch J.P. et al. // BMC Cancer. - 2013. - P. 13-566; Naumov V.A. // Epigenetics. - 2013. - V. 8. - P. 921-934; Chen Y.A. et al. // Epigenetics. - 2013. - V. 8. - P. 203-209; Harper K.N. et al. // Cancer Epidemiol Biomarkers Prev. - 2013. - V. 22. - P. 1052-1060; Mitchell S.M. et al. // BMC Cancer. - 2014. - P. 14-54; Melson J. et al. // Int J Cancer. - 2014. - V. 134. - P. 2656-2662; Papadia C. et al. // Inflamm Bowel Dis. - 2014. - V. 20. - P. 271-277].
Некоторые из обнаруженных генов-онкомаркеров уже применяются в коммерческих тест-системах для диагностики колоректального рака. В 2008-2012 годах в компании Epigenomics AG (Германия) была разработана и запатентована тест-система «Epi proColon kit 2.0» для диагностики колоректального рака (международная заявка №WO2005001142, опубл. 06.01.2005 г.; заявка США №2014179563, опубл. 26.06.2014 г.) на основе выявления специфического эпигенетического маркера SEPT9 в периферической крови [Heichman KA. Blood-based testing for colorectal cancer screening // Mol Diagn Ther. 2014. - V. 18. - P. 127-135]. Метилирование другого гена (VIM, виментин) в малигнантных клетках послужило основой для создания тест-системы «ColoGuard assay» от компании LabCorp (США) [Lao VV, Grady WM. Epigenetics and colorectal cancer // Nat Rev Gastroenterol Hepatol. - 2011. - V. 8. - P. 686-700]. Однако подобные моно-тест-системы не предназначены для постановки окончательного диагноза, и их показатели могут служить лишь дополнительными индикаторами развития или отсутствия заболевания.
В совокупности полученные исследователями данные свидетельствуют о ненадежности диагностики, построенной на анализе одного гена и, соответственно, об актуальности разработки и внедрения тест-систем, основанных на определении статуса метилирования ДНК регуляторных областей ряда генов-онкомаркеров, позволяющих проводить диагностику более надежно и точно. Известна работа Lind с соавт., создавших диагностический набор для анализа метилирования шести генов: CNRIP1, FBN1, INA, MAL, SNCA и SPG20 [Lind G.E. et al. Identification of an epigenetic biomarker panel with high sensitivity and specificity for colorectal cancer and adenomas // Mol Cancer 2011. - V 10. - P. 85].
Наиболее близким аналогом (прототипом) является способ определения диагноза рака у индивидуума с подозрением на колоректальный рак (Международная заявка №WO2014062218, МПК C12Q1/68, опубл. 24.04.2014 г.), включающий: получение образца от индивидуума с подозрением на рак; гидролиз высокоочищенной геномной ДНК из исследуемого биоматериала с использованием бисульфитной конверсии ДНК; определения наличия или отсутствия высокого уровня экспрессии генов (определение уровня метилирования регуляторных областей генов-онкомаркеров с использованием цифровой ПЦР) по отношению к нормальному базовому стандарту для одной диагностической панели, включающей следующие биомаркеры: TFPI2, THBD, C9ORF50, ADHFEl, FGF12, PTPRT, ZNF568, KIAA1026, SFMBT2, и / или AUTS2; и диагностики рака при наличии высокого уровня экспрессии по отношению к нормальному базовому стандарту по меньшей мере одного биомаркера.
Недостатком известных аналогов и прототипа является использование бисульфитной конверсии ДНК, для которой характерны трудоемкость, дороговизна исследования и значительные затраты времени, а также деградация и значительные потери нуклеиновых кислот, что ведет к снижению точности и делает проблематичным анализ малого количества материала.
Техническим результатом предлагаемого изобретения является упрощение и повышение точности способа определения метилирования сайтов RCGY регуляторных областей генов-онкомаркеров колоректального рака и расширение его функциональных возможностей.
Указанный технический результат достигается разработанным набором олигонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации злокачественного поражения толстого кишечника и прямой кишки посредством анализа в образцах ДНК метилирования сайтов RCGY в заданных положениях регуляторных областей генов-онкомаркеров методом GLAD-ПЦР, имеющих структуру, приведенную в приложении (список последовательностей) и таблице 1:
Таблица 1
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ADHFE1 (5'→3'):
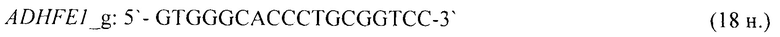
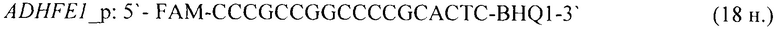
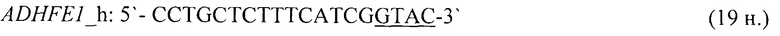
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ALX4 (5'→3'):
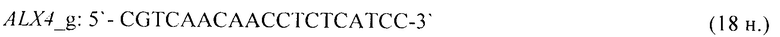
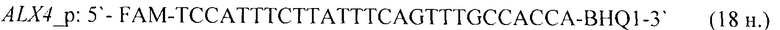
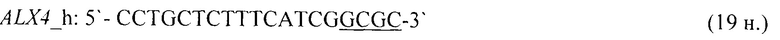
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера CNRIP1 (5'→3'):
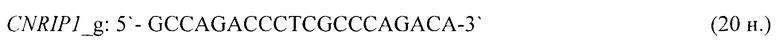

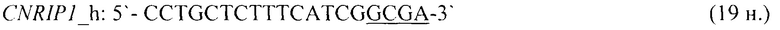
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера EID3 (5'→3'):
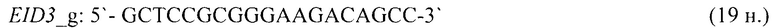
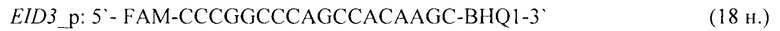
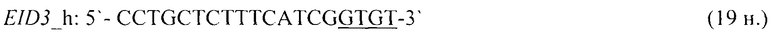
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ELMO1 (5'→3'):

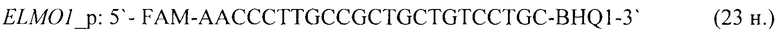

- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ESR1 (5'→3'):
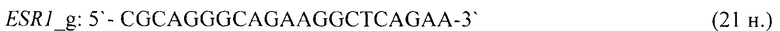
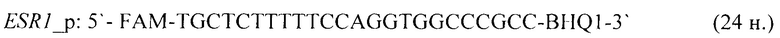
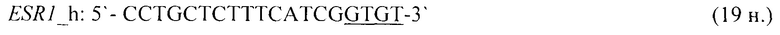
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(1) (5'→3'):
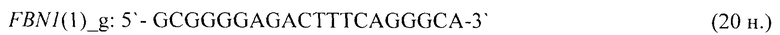
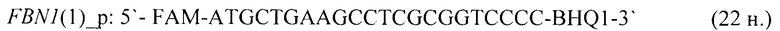
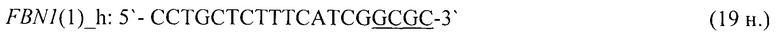
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(2) (5'→3'):
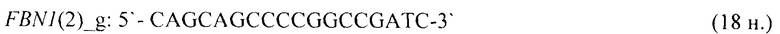
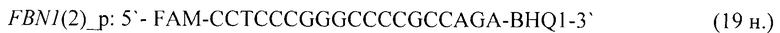
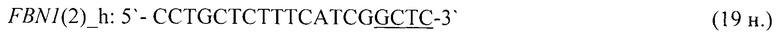
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(3.1) (5'→3'):
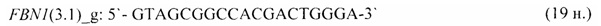
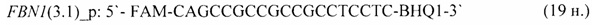
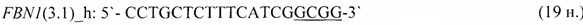
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(3.3) (5'→3'):
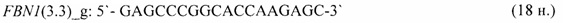
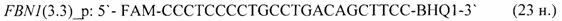
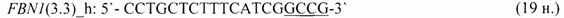
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера HLTF (5'→3'):
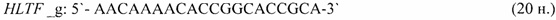
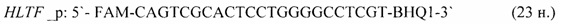
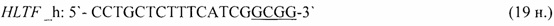
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера LAMA1 (5'→3'):
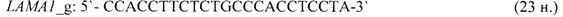
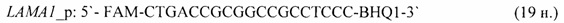
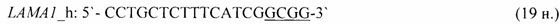
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера NEUROG1 (5'→3'):

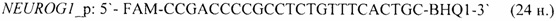
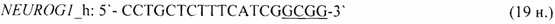
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера NGFR (5'→3'):
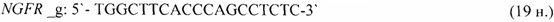
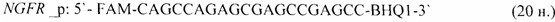
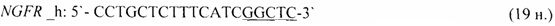
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера RARB (5'→3'):
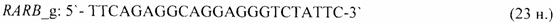
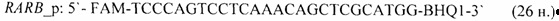
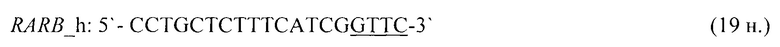
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера RXRG (5'→3'):
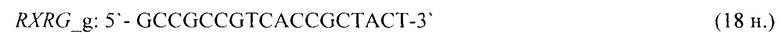
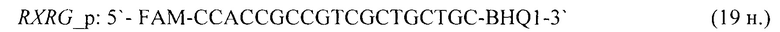
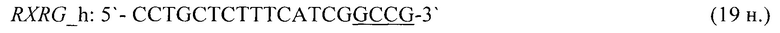
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера RYR2 (5'→3'):
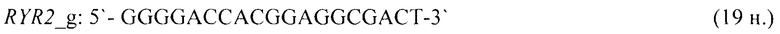
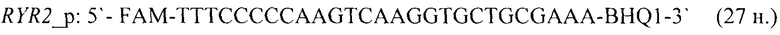
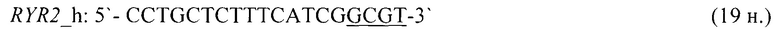
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SDC2 (5'→3'):
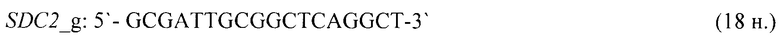
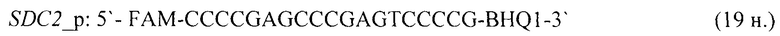

- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SEPT9 (5'→3'):
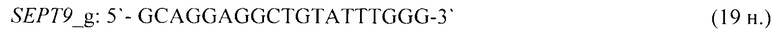
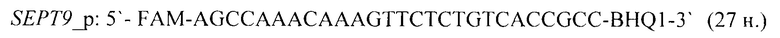
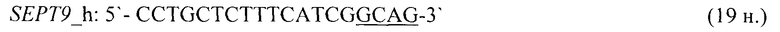
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SFRP2 (5'→3'):
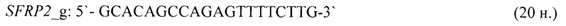
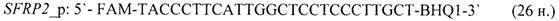
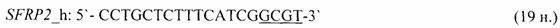
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SOCS3 (5'→3'):
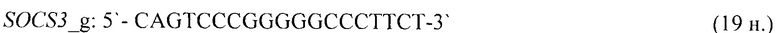
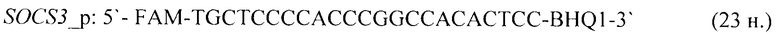
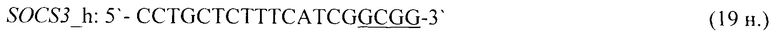
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SOX17 (5'→3'):
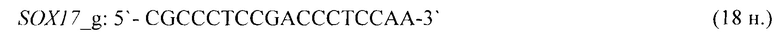
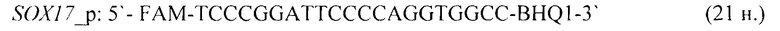
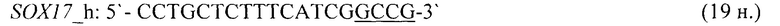
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера THBD (5'→3'):
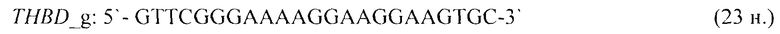
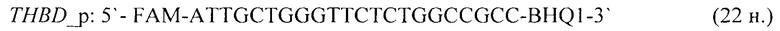

- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера TMEFF2 (5'→3'):
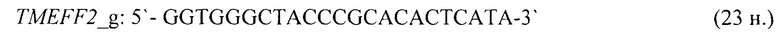
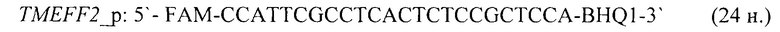
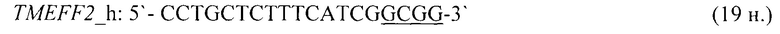
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера UCHL1 (5'→3'):
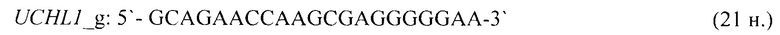
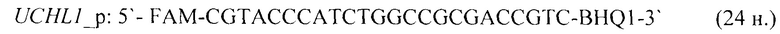
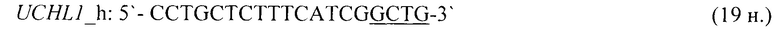
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера VIM (5'→3'):
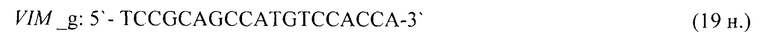
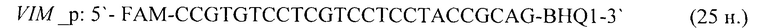
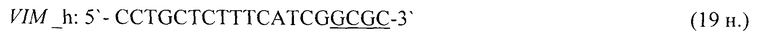
где g - геномный праймер (genome); р - флуоресцентно-меченый зонд (zond); h - гибридный праймер (hybrid); FAM - флуоресцеин; BHQ1 - гаситель флуоресценции (Black Hole Quencher 1).
Учет результатов анализа проводится методом гибридизационно-флуоресцентной детекции продуктов ПЦР-амплификации в режиме реального времени.
Указанный технический результат достигается также созданием способа идентификации злокачественного поражения толстого кишечника и прямой кишки посредством анализа в образцах ДНК метилирования сайтов RCGY в заданных положениях регуляторных областей генов-онкомаркеров методом GLAD-ПЦР, включающем формирование панели генов-онкомаркеров колоректального рака: ADHFE1, ALX4, CNRIP1, EID3, ELMO1, ESR1, FBN1, HLTF, LAMA1, NEUROG1, NGFR, RARB, RXRG, RYR2, SDC2, SEPT9, SFRP2, SOCS3, SOX17, THBD, TMEFF2, UCHL1 и VIM путем отбора генов-онкомаркеров колоректального рака при биоинформационном анализе и рассмотрения нуклеотидных последовательностей предположительных регуляторных участков этих генов, выявление располагающихся в них CpG-островков, анализ первичной структуры CpG-островков и их соответствие определенным критериям выбора. В регуляторной области (промоторе или первом экзоне) этих генов обычно присутствуют несколько сайтов RCGY, метилирование которых может вызывать подавление экспрессии гена при колоректальном раке. Поэтому далее на образцах ДНК из опухолевого материала или клеточных линий осуществляют отбор наиболее метилированных сайтов с помощью GLAD-ПЦР-анализа, которые в могут использоваться для диагностики и/или прогнозирования течения колоректального рака.
Далее осуществляют гидролиз высокоочищенной геномной ДНК из исследуемого биоматериала метилзависимой сайт-специфической ДНК-эндонуклеазой GlaI, лигирование универсального олигонуклеотидного адаптера  , где р - фосфат с последующей амплификацией в реальном времени с использованием геномных праймеров и зондов, комплементарных последовательностям регуляторных областей указанных генов в исследуемой ДНК, и гибридных праймеров, 3'-концы которых комплементарны 5'-концам ДНК в заданных местах гидролиза GlaI, а оставшаяся часть комплементарнаадаптерной последовательности, и делают заключение о наличии последовательности R(5mC)GY, где 5mC - 5-метилцитозин, R-А или G, Y-Т или С в регуляторных областях указанных генов по появлению флуоресцентного сигнала для определения статуса метилирования сайтов в клинических образцах методом GLAD-ПЦР-анализа и на основе информации о статусе метилирования одного или нескольких сайтов делают вывод о наличии или отсутствия колоректального рака.
, где р - фосфат с последующей амплификацией в реальном времени с использованием геномных праймеров и зондов, комплементарных последовательностям регуляторных областей указанных генов в исследуемой ДНК, и гибридных праймеров, 3'-концы которых комплементарны 5'-концам ДНК в заданных местах гидролиза GlaI, а оставшаяся часть комплементарнаадаптерной последовательности, и делают заключение о наличии последовательности R(5mC)GY, где 5mC - 5-метилцитозин, R-А или G, Y-Т или С в регуляторных областях указанных генов по появлению флуоресцентного сигнала для определения статуса метилирования сайтов в клинических образцах методом GLAD-ПЦР-анализа и на основе информации о статусе метилирования одного или нескольких сайтов делают вывод о наличии или отсутствия колоректального рака.
Структура адаптера подбирается таким образом, чтобы его нуклеотидная последовательность была уникальна для генома человека, оптимальная по GC составу и защищена от образования самокомплементарных структур.
Причем, при реализации способа используют вышеуказанный набор праймеров и зондов, представленных в Приложении (список последовательностей).
Изобретение иллюстрируется следующим графическим материалом, представленным на фигурах 1-3. На фиг. 1 представлен пример поиска наиболее часто метилированного сайта RCGY на выбранном участке регуляторной области гена FBN1(3.3). Как видно из фиг. 1, таким оказался третий сайт в ампликоне, которому соответствует гибридный праймер 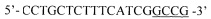 .
.
На панели А представлен фрагмент нуклеотидной последовательности; показаны участки связывания геномного праймера, зонда и геном-специфичных 3'-тетрануклеотидов гибридных праймеров. На панели Б приведены кривые накопления флуоресценции в ходе ПЦР с использованием различных гибридных праймеров. Заглавными буквами «GCGG» обозначен выбранный гибридный праймер, соответствующий точке гидролиза ДНК по наиболее часто метилированному третьему сайту RCGY в ампликоне.
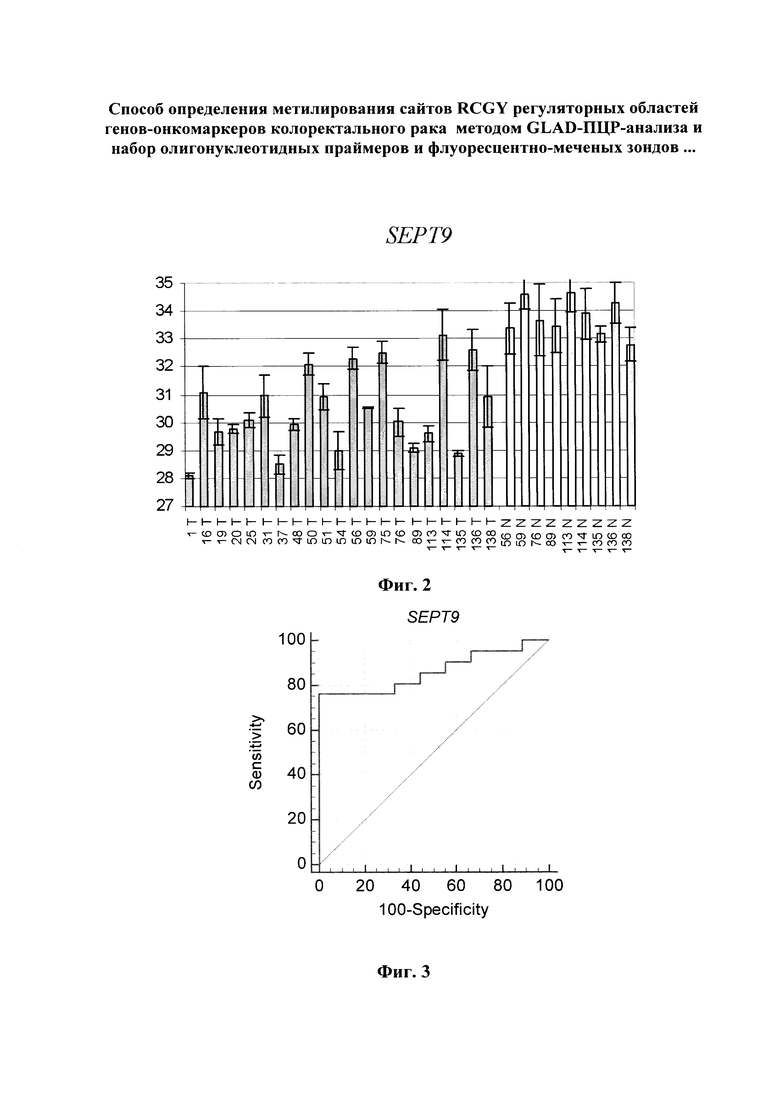
На фиг. 2: представлен результат GLAD-ПЦР-анализа отобранного сайта RCGY в регуляторной области гена SEPT9 в клинических образцах. Погоризонтальной оси отображены номера клинических образцов, где T – соответствует опухолевому образцу, а N – здоровому. По вертикальной оси приведено усредненное значение Cq для каждого образца с указанием доверительного интервала.
На фиг. 3 приведена ROC (Receiver Operating Characteristic) кривая, полученная в результате ROC анализа результатов GLAD-ПЦР-анализа статуса метилирования RCGY сайта-маркера колоректального рака гена SEPT9. Параметры ROC-анализа приведены в таблице 4. ROC-анализ является стандартным методом оценки диагностической ценности тестов.
Сущность изобретения поясняется следующими примерами 1-3, реализующими способ определения метилирования сайтов RCGY регуляторных областей генов-онкомаркеров колоректального рака методом GLAD-ПЦР-анализа, а также состав олигонуклеотидных праймеров и флуоресцентно-меченых зондов для осуществления указанного способа.
Пример 1. Разработка специфических праймеров и флуоресцентно-меченых зондов на участки CpG-островков регуляторных областей генов-онкомаркеров колоректального рака.
Изучение опубликованных в литературе данных по метилированию генов при онкопатологиях нижних отделов кишечника, данных RRBS-секвенирования (Reduced Representation Bisulfite Sequencing, бисульфитное секвенирование ограниченных выборок локусов) в проекте ENCODE/HudsonAlpha и последующий биоинформационный анализ нуклеотидных последовательностей регуляторных участков описанных ранее генов позволил выбрать кандидатную комбинацию участков CpG-островков регуляторных областей генов-онкомаркеров колоректального рака, удовлетворяющих критериям анализа методом GLAD-ПЦР (табл. 2).
На первом этапе работы был проведен анализ литературных данных последних лет по метилированию генов при онкопатологиях нижних отделов кишечника, данных RRBS-секвенирования (Reduced Representation Bisulfite Sequencing, бисульфитное секвенирование ограниченных выборок локусов) в проекте ENCODE/HudsonAlpha и сформирован список из 57 генов-онкомаркеров, метилирование которых происходит с высокой частотой в ДНК клеток колоректального рака. Этот список включает в себя следующие гены: SEPT9, SYNE1, FGF12, GAD2, HAND1, CACNA1G, CNRIP1, TLX2, LOX, LAMA1, GAS7, SLC6A15, BOLL, TFPI2, SOCS3, TRH, MDFI, C1orf70, RUNX3, TMEFF2, SFRP2, WNT5A, IGFBP7, TFPI2, FOXE1, NPY, WIF1, CIDEB, RIC3, KCNQ5, COL4A1, SPG20, SND1, PENK, CDO1, THBD, EDIL3, DUSP26, UCHL1, STOX2, ELMO1, SLC30A10, IGF2, ESR1, EYA4, NALCN, FLI1, RXRg, RYR2, PRKAR1B, BEND5, ITGA4, ADHFE1, NGFR, GATA4, NEYROG1, FBN1. При выборе генов для данного списка учитывалось также количество проанализированных проб в описанных авторами статей экспериментах, так как достоверные данные могут быть получены лишь при большом числе образцов ДНК в выборке. Большое значение также придавалось такому описанному авторами работ свойству, как возможность выявления метилирования гена на ранних стадиях заболевания, хотя, к сожалению, число публикаций, в которых рассматривалась зависимость метилирования гена от стадии онкопатологии, очень незначительно.
На втором этапе работы были рассмотрены нуклеотидные последовательности предположительных регуляторных участков этих 57 генов и выявлены CpG-островки, располагающиеся в них. Первичные структуры CpG-островков были проанализированы на их соответствие критериям выбора:
1) Наличие данных из опубликованных работ, показывающих высокую частоту метилирования регуляторного участка гена в исследованных выборках образцов ДНК от больных колоректальным раком при низкой частоте метилирования данного участка у здоровых доноров или в прилегающих нормальных тканях толстой и прямой кишки онкопациентов.
2) Присутствие СpG-островка в области начала гена (или в районе точки старта кодирования альтернативной изоформы белка - продукта гена), что в большинстве случаев служит свидетельством регуляции активности данного гена путем метилирования этой области.
3) Присутствие потенциального сайта узнавания эндонуклеазы GlaI (RCGY) в нуклеотидной последовательности на выбранном участке.
4) Достаточное расстояние (50 п.н. и более) между двумя потенциальными сайтами узнавания эндонуклеазы GlaI для одновременного отжига на этом участке флуоресцентно-меченого зонда и геномного праймера и возможность подбора для этого участка праймера и зонда, не подверженных теоретической кросс-гибридизации между собой и с последовательностью адаптера.
5) Значение G+C состава на кандидатном участке, не превышающее 80%, что вызвано ограничениями системы амплификации ДНК in vitro, в которой используется HotStart Taq ДНК-полимераза.
6) Уникальность нуклеотидной последовательности кандидатного участка анализа (невхождение его в число повторяющихся элементов генома) во избежание неспецифического отжига подобранного геномного праймера в других областях генома, что может привести к значительному снижению эффективности амплификации и затруднению детекции результатов при проведении ПЦР в реальном времени.
В результате проведенного биоинформационного анализа регуляторных участков каждого из перечисленных генов для проведения экспериментального исследования были отобраны 26 областей из данных участков (табл.2), которые удовлетворяют всем требуемым критериям проведения анализа с помощью метода GLAD-ПЦР, и подобраны специфические праймеры и зонды на выбранные участки регуляторных областей генов-онкомаркеров (табл.3).
Таблица 2.
Участки регуляторных областей генов-онкомаркеров перспективные для GLAD-ПЦР-анализа.
Используемые в работе последовательности регуляторных участков генов по версии генома человека GRCh37/hg19 были получены из международной базы данных GenBank. Для анализа и расчетов праймеров и зондов, изучения их специфичности использовали семейство компьютерных программ Vector NTI 11.5 (Invitrogen, США) и онлайн интернет-ресурсы базы данных Национального центра биотехнологической информации США (Blast NCBI, http://blast.ncbi.nlm.nih.gov/Blast.cgi).
Таблица 3.
Cпецифические праймеры и зонды на участки регуляторных областей генов-онкомаркеров.
p: FAM-CCCGCCGGCCCCGCACTC-BHQ1
r: GGTCGCGTACTTGCTGAGGCA
p: FAM-TCCATTTCTTATTTCAGTTTGCCACCA-BHQ1
r: GGACTCTGGTTTCTAAGATCAG
p: FAM-CGAGGCCCGGCAGGTCCCCC-BHQ1
r: CGTCATTAGGCTGGATGCGCA
p: FAM-CCCGGCCCAGCCACAAGC-BHQ1
r: TTTGAAAAAGAATAGCTGTCCCCTGA
p: FAM-AACCCTTGCCGCTGCTGTCCTGC-BHQ1
r: CGCCAGCCCAGGAAACTTTAC
p: FAM-TGCTCTTTTTCCAGGTGGCCCGCC-BHQ1
r: CGGGACATGCGCTGCGTC
p: FAM-ATGCTGAAGCCTCGCGGTCCCC-BHQ1
r: CACGGGTTGGGCTTGGGA
p: FAM-CCTCCCGGGCCCCGCCAGA-BHQ1
r: GGGTACTTTGCGCCGCGCTC
p: FAM-CAGCCGCCGCCGCCTCCTC-BHQ1
r: CCGGCTGCACCCACTGGA
p: FAM-CCCTCCCCTGCCTGACAGCTTCC-BHQ1
r: CACCGGGGCTGGAGCTGC
p: FAM-CAGTCGCACTCCTGGGGCCTCGT-BHQ1
r: GTTCTTCCCCGCCCCCAA
p: FAM-CTGACCGCGGCCGCCTCCC -BHQ1
r: CCGCCACCCAGACCCCTC
p: FAM-CCGACCCCGCCTCTGTTTCACTGC-BHQ1
r: CCGTAATTACCGCCGGCCAATC
p: FAM-CAGCCAGAGCGAGCCGAGCC-BHQ1
r: TCCAGCTCGGTCCGCTTTG
p: FAM-TCCCAGTCCTCAAACAGCTCGCATGG-BHQ1
r: GGTTCCCAGAAAGATCCCAAGTTC
p: FAM-CCACCGCCGTCGCTGCTGC-BHQ1
r: GTGCCACCCGGTAGGGACC
p: FAM-TTTCCCCCAAGTCAAGGTGCTGCGAAA-BHQ1
r: CGGGGGTGATGGTGCAGGA
p: FAM-CCCCGAGCCCGAGTCCCCG-BHQ1
r: GGGAGTGCAGAAACCAACAAGTGA
p: FAM-AGCCAAACAAAGTTCTCTGTCACCGCC-BHQ1
r: CGCTGCCGTTTAACCCTTG
p: FAM-TACCCTTCATTGGCTCCTCCCTTGCT-BHQ1
r: AGGCTTCTCTGTTTGTTGTTTAAAG
p: FAM-TGCTCCCCACCCGGCCACACTCC-BHQ1
r: CAAGGGCGCAGCGTGGGA
p: FAM-TCCCGGATTCCCCAGGTGGCC-BHQ1
r: CAGTTCAGGGCCAAGGGTGCT
p: FAM-ATTGCTGGGTTCTCTGGCCGCC-BHQ1
r: TTACTCATCCCGGCGAGGTGA
p: FAM-CCATTCGCCTCACTCTCCGCTCCA-BHQ1
r: CGCCGACTCGCCCCTCTC
p: FAM-CGTACCCATCTGGCCGCGACCGTC-BHQ1
r: GGGGCCCGGCCGTACCAC
p: FAM-CCGTGTCCTCGTCCTCCTACCGCAG-BHQ1
r: GCTGCCCAGGCTGTAGGTGC
*Примечание: g – геномный праймер; p – флуоресцентно-меченый зонд; r – дополнительный геномный праймер (reverse), впоследствии заменяемый в GLAD-ПЦР гибридным праймером (пример 2); FAM – флуоресцеин; BHQ1 – гаситель флуоресценции (Black Hole Quencher 1).
Реакцию ПЦР в режиме реального времени проводили в объеме 20 мкл в трех повторах на детектирующем амплификаторе CXF-96 («Bio-Rad», США) по универсальной для всех амплифицируемых участков программе: 95°С 3 мин, далее 5 циклов без детекции: 95°С 10 сек, 61°С 15 сек, 72°С 20 сек; затем 50 циклов с детекцией по каналу FAM на стадии отжига: 95°С 10 сек, 61°С 15 сек, 72°С 20 сек. Концентрации реакционных компонентов ПЦР были аналогичны указанным в примере 2. В экспериментах использовали лейкоцитарную ДНК здорового донора, полученную и очищенную, как описано ранее [Абдурашитов М.А. с соавт. Рестрикционный анализ ДНК человека – новые возможности в ДНК-диагностике. Медицинская генетика. 2007. - Т. 6. - С. 29-36]. Pезультаты амплификации соответствовали ожидаемым. Последовательности разработанных праймеров и зондов представлены в таблице 3. Впоследствии, при проведении GLAD-ПЦР, один из праймеров, обозначенный в таблице как «r» (reverse), заменяли на отобранный гибридный праймер (пример 2).
Пример 2. Поиск метилированных сайтов RCGY для GLAD-ПЦР-анализа
Изучен статус метилирования сайтов RCGY в пределах выбранных участков ДНК с целью выбора оптимальных сайтов для дальнейшего GLAD-ПЦР-анализа клинических образцов. Для поиска перспективных сайтов R(5mC)GY в пределах выбранных для анализа участков регуляторных областей генов-онкомаркеров использовали препарат ДНК из клеток колоректального рака линии SW837 (линия клеток аденокарциномы ободочной кишки), выделенный известным методом [Sambrook J., Russel D. Molecular cloning: a laboratory manual. 3rd ed. – New York: Cold Spring Harbor Laboratory Press, 2001. – 2222 p.]. Поиск проводили методом подбора гибридных праймеров под близлежащие сайты RCGY на протяжении не более 400 п.о. от положения геномного праймера. Использовали методологию, структуры олигонуклеотидного адаптера и гибридных праймеров согласно известному способу [Кузнецов В. В. с соавт. Способ определения нуклеотидной последовательности Pu(5mC)GPy в заданном положении протяженной ДНК // Патент РФ №2525710 С1, опубл. 20.08.2014, Бюл. № 23].
Структура гибридного праймера представляет собой 19-звенную последовательность следующего состава: 5’-CCTGCTCTTTCATCGgYNN-3’, где подчеркиванием выделена 15-звенная часть праймера, комплементарная универсальному олигонуклеотидному адаптеру, а gYNN – 4-звенник, комплементарный геномной последовательности в точке гидролиза ДНК метилзависимой эндонуклеазой GlaI. Данная структура предполагает 32 варианта гибридных праймеров, соответствующих различным точкам гидролиза ДНК по сайту R(5mC)GY.
К 45 нг препарата ДНК добавляли метилзависимую сайт-специфическую ДНК-эндонуклеазу GlaI в количестве 1,5 е.а. и компоненты буфера для достижения следующего состава в объеме 21,7 мкл: 20 мМ Tris-SO4, pH 8.0, 4 мМ MgCl2, 1 мМ ß-меркаптоэтанол, 100 нг/мкл БСА. Гидролиз проводили в течение 30 мин при 30 С с последующей инактивацией GlaI при 65 С в течение 20 мин.
Далее в к GlaI-гидролизату добавляли АТФ до конечной концентрации 0,5 мМ, ß-меркаптоэтанол до 6,8 мМ, универсальный олигонуклеотидный адаптер 5’-CCTGCTCTTTCATCG-3’/3’-p-GGACGAGAAAGTAGC-p-5’, до конечной концентрации 0,5 мкМ и 1000 е.а. высокоактивной T4 ДНК-лигазы. Реакцию лигирования адаптера проводили в 30 мкл реакционной смеси в течение 15 мин при 25 С, затем лигазу термоинактивировали при 65 С в течение 20 мин.
Для проведения ПЦР в режиме реального времени к лигазной смеси добавляли следующие компоненты до достижения итоговых концентраций в конечном объеме, равном 60 мкл: 50 мМ Tris-SO4, pH 9,0, 30 мМ KCl, 10 мМ сульфата аммония, 3 мМ MgCl2, 0,2 мМ смеси дезоксирибонуклеозидтрифосфатов, 0,01 % Tween-20, смесь геномного праймера, гибридного праймера и зонда по 0,4 мкМ каждого и 0,05 е.а./мкл Hot Start ДНК-полимеразы. Для повышения эффективности амплификации GC-богатых участков ДНК использовали стабилизатор. Полученную реакционную смесь (60 мкл) разделяли на три равные части.
Реакцию ПЦР в режиме реального времени проводили в объеме 20 мкл в трех повторах на детектирующем амплификаторе CXF-96 («Bio-Rad», США) по программе: 95°С 3 мин, далее 5 циклов без детекции: 95°С 10 сек, 61°С 15 сек, 72°С 20 сек; затем 50 циклов с детекцией (канал FAM) на стадии отжига: 95°С 10 сек, 61°С 15 сек, 72°С 20 сек. Результаты анализировали при помощи программного обеспечения амплификатора CXF-96 и интерпретировали на основании наличия (или отсутствия) пересечения кривых флуоресценции образцов с пороговыми линиями, что соответствовало наличию или отсутствию значения порогового цикла «Cq» в соответствующей графе таблицы результатов программы амплификатора.
На фиг. 1 представлен пример поиска наиболее часто метилированного сайта RCGY на выбранном участке регуляторной области гена FBN1(3.3). Выбор наиболее метилированного сайта RCGY и соответствующего ему гибридного праймера для GLAD-ПЦР анализа в регуляторной области гена FBN1. (a) Фрагмент нуклеотидной последоваательности регуляторного участка гена FBN1. Показаны геномные праймеры, флуоресцентные зонды, GlaI специфичные сайты (RCGY) и четырехнуклеотидная последовательность соответствующего гибридного праймера. Гибридные праймеры, соответствующие наиболее метилированным сайтам RCGY показаны заглавными буквами. Координаты участка в хромосоме даны в соответствии с геномной сборкой GRCh38/hg38. (b) Кривые ПЦР в реальном времени полученные с применением различных гибридных праймеров в GLAD-ПЦР-анализе регуляторного участка гена FBN1 (3.1).
Как видно из фиг. 1, таким оказался третий сайт в ампликоне, которому соответствует гибридный праймер 5’- CCTGCTCTTTCATCGGCCG -3’.
Результаты поиска наиболее метилированных сайтов RCGY из регуляторных областей генов ADHFE1, ALX4, CNRIP1, EID3, ELMO1, ESR1, FBN1, HLTF, LAMA1, NEUROG1, NGFR, RARB, RXRG, RYR2, SDC2, SEPT9, SFRP2, SOCS3, SOX17, THBD, TMEFF2, UCHL1 и VIM, перспективных для GLAD-ПЦР-анализа клинических образцов и идентификации колоректального рака приведены в Таблице 1.
Пример 3. GLAD-ПЦР-анализ статуса метилирования генов-онкомаркеров колоректального рака
Образцы операционного материала опухолей (n = 21) и нормальной слизистой кишечника (n = 9) от больных колоректальным раком были получены из ФГУП Северский биофизический научный центр ФМБА России (г. Северск, Томская обл., РФ). Для GLAD-ПЦР-анализа статуса метилирования сайтов RCGY в заданных положениях участка ДНК определенного гена использовали препараты геномных ДНК, выделенные из клинических образцов известным методом фенол-хлороформной экстракции [Sambrook J., Russel D. Molecular cloning: a laboratory manual. 3rd ed. – New York: Cold Spring Harbor Laboratory Press, 2001. – 2222 p.], прошедшие обработку РНК-азой А для очистки от примесей РНК и фрагментацию редкощепящей эндонуклеазой рестрикции TaqI для более полного гидролиза ДНК. Концентрации образцов ДНК (нг/мкл) определяли спектрофотометрически. Реакцию GLAD-ПЦР проводили, как описано в примере 2. В реакцию брали 9 нг ДНК, ПЦР проводили в трех повторах для каждого образца (по 3 нг ДНК, т.е. по ~103 копий генома на реакцию). Один из геномных праймеров в ПЦР заменяли на подобранный, как описано в примере 2, т.е. гибридный праймер.
Результаты проведенного анализа для гена SEPT9 представлены на фиг. 2: результаты GLAD-ПЦР-анализа отобранного сайта RCGY в регуляторной области гена SEPT9 в клинических образцах. По горизонтальной оси отображены номера клинических образцов, где T – соответствует опухолевому образцу, а N – здоровому. По вертикальной оси приведено усредненное значение Cq для каждого образца с указанием доверительного интервала. В соответствии с полученными значениями Cq для каждого цикла экспериментов для оценки чувствительности и специфичности каждого маркера были построены ROC кривые и оценена площадь под кривой [E. R. DeLong, D. M. DeLong, and D. L. Clarke-Pearson, “Comparing the areas under two or more correlated receiver operating characteristic curves: a nonparametric approach,” Biometrics, vol. 44, no. 3, pp. 837–845, 1988]. Результаты оценки приведены в таблице 4, а соответствующие кривые на рисунке фиг.3 - ROC кривая для GLAD-ПЦР-анализа статуса метилирования RCGY сайта-маркера колоректального рака для гена SEPT9.
В ходе ROC-анализа рассчитывался оптимальный пороговый цикл отсечения «Ctопт», при котором суммарное значение чувствительности и специфичности было максимальным. Анализируемый образец считали положительным, если среднее значение «Ct» для образца было меньше или равно «Ctопт». Анализируемый образец считали отрицательным, если среднее значение «Ct» для образца превышало «Ctопт».
Таблица 4
Оценка диагностической ценности отобранного сайта RCGY в регуляторной области гена-онкомаркера колоректального рака SEPT9 методом GLAD-ПЦР-анализа.
%
%
(стандартное отклонение)
Таким образом, в примере 3 показана возможность успешного применения метода GLAD-ПЦР для идентификации злокачественного поражения толстого кишечника и прямой кишки с использованием разработанного набора олигонуклеотидных праймеров и флуоресцентно-меченых зондов для анализа методом GLAD-ПЦР статуса метилирования заданных сайтов RCGY в регуляторных областях генов-онкомаркеров колоректального рака.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ОПРЕДЕЛЕНИЯ МЕТИЛИРОВАНИЯ САЙТОВ PuCGPy РЕГУЛЯТОРНЫХ ОБЛАСТЕЙ ГЕНОВ-ОНКОМАРКЕРОВ КОЛОРЕКТАЛЬНОГО РАКА МЕТОДОМ GLAD-ПЦР-АНАЛИЗА И ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ И ФЛУОРЕСЦЕНТНО-МЕЧЕНЫЕ ЗОНДЫ ДЛЯ ОСУЩЕСТВЛЕНИЯ УКАЗАННОГО СПОСОБА | 2015 |
|
RU2596404C1 |
| Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для выявления эпигенетических маркеров рака шейки матки | 2021 |
|
RU2760573C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ ВОЗРАСТА ЧЕЛОВЕКА ПО ДНК | 2021 |
|
RU2797018C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ Pu(5mC)GPy В ЗАДАННОМ ПОЛОЖЕНИИ ПРОТЯЖЕННОЙ ДНК | 2013 |
|
RU2525710C1 |
| Способ обнаружения и анализа метилирования геномных участков ДНК в биологических образцах перифирической крови больных неходжкинской лимфомой | 2022 |
|
RU2804962C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ R(5mC)GY В ЗАДАННОМ ПОЛОЖЕНИИ ПРОТЯЖЕННОЙ ДНК | 2015 |
|
RU2587631C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ ГИПЕРМЕТИЛИРОВАННЫХ CpG ОСТРОВКОВ В ОБЛАСТИ ГЕНОВ-СУПРЕССОРОВ ОПУХОЛЕВОГО РОСТА В ДНК ЧЕЛОВЕКА | 2009 |
|
RU2413773C1 |
| ОПУХОЛЕВЫЙ МАРКЕР, РЕАГЕНТ ДЛЯ ВЫЯВЛЕНИЯ МЕТИЛИРОВАНИЯ, НАБОР И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2770928C1 |
| ОПУХОЛЕВЫЙ МАРКЕР, РЕАГЕНТ ДЛЯ ВЫЯВЛЕНИЯ МЕТИЛИРОВАНИЯ, НАБОР И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2775177C1 |
| СПОСОБ КАРТИРОВАНИЯ ПОЛОЖЕНИЙ РЯДА МЕТИЛИРОВАННЫХ НУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ Pu(5mC)GPy В ПРОТЯЖЕННОЙ ДНК ДЛЯ ПОСТРОЕНИЯ ЭПИГЕНЕТИЧЕСКОГО ПРОФИЛЯ И ВЫЯВЛЕНИЯ АНОМАЛЬНО МЕТИЛИРОВАННЫХ УЧАСТКОВ ДНК | 2015 |
|
RU2586502C1 |







Изобретение относится к области биохимии. Описана группа изобретений, включающая набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации злокачественного поражения толстого кишечника и прямой кишки посредством анализа в образцах ДНК метелирования сайтов RCGY в заданных положениях регуляторных областей генов-онкомаркеров методом GLAD-ПЦР и способ идентификации злокачественного поражения толстого кишечника и прямой кишки посредством анализа в образцах ДНК метилирования сайтов RCGY в заданных положениях регуляторных областей генов-онкомаркеров методом GLAD-ПЦР. Указанный способ включает формирование панели генов-онкомаркеров колоректального рака: ADHFE1, ALX4, CNRIP1, EID3, ELMO1, ESR1, FBN1, HLTF, LAMA1, NEUROG1, NGFR, RARB, RXRG, RYR2, SDC2, SEPT9, SFRP2, SOCS3, SOX17, THBD, TMEFF2, UCHL1 и VIM. Изобретение расширяет арсенал олигонуклеотидных праймеров и флуоресцентно-меченных праймеров. 2 н.п. ф-лы., 3 ил., 4 табл., 3 пр., 1 прил.
1. Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации злокачественного поражения толстого кишечника и прямой кишки посредством анализа в образцах ДНК метилирования сайтов RCGY в заданных положениях регуляторных областей генов-онкомаркеров методом GLAD-ПЦР, имеющих следующую структуру:
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ADHFE1 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ALX4 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера CNRIP1 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера EID3 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ELMO1 (5'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера ESR1 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(1) (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(2) (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(3.1) (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера FBN1(3.3) (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера HLTF(5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера LAMA1 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера NEUROG1 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера NGFR (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера RARB (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера RXRG (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера RYR2 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SDC2 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SEPT9 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SFRP2 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SOCS3 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера SOX17 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера THBD (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера TMEFF2 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера UCHL1 (5'→3'):
- для специфических праймеров и зонда на участки CpG-островков регуляторных областей гена-онкомаркера VIM (5'→3'):
где g - геномный праймер (genome); р - флуоресцентно-меченый зонд (zond); h - гибридный праймер (hybrid); FAM - флуоресцеин; BHQ1 - гаситель флуоресценции (Black Hole Quencher 1).
2. Способ идентификации злокачественного поражения толстого кишечника и прямой кишки посредством анализа в образцах ДНК метилирования сайтов RCGY в заданных положениях регуляторных областей генов-онкомаркеров методом GLAD-ПЦР, включающий формирование панели генов-онкомаркеров колоректального рака: ADHFE1, ALX4, CNRIP1, EID3, ELMO1, ESR1, FBN1, HLTF, LAMA1, NEUROG1, NGFR, RARB, RXRG, RYR2, SDC2, SEPT9, SFRP2, SOCS3, SOX17, THBD, TMEFF2, UCHL1 и VIM; гидролиз высокоочищенной геномной ДНК из исследуемого биоматериала метилзависимой сайт-специфической ДНК-эндонуклеазой GlaI, лигирование универсального олигонуклеотидного адаптера 5'-CCTGCTCTTTCATCG-3'/3'-p-GGACGAGAAAGTAGC-p-5', где р - фосфат с последующей амплификацией в реальном времени с использованием геномных праймеров и зондов, комплементарных последовательностям регуляторных областей указанных генов в исследуемой ДНК, и гибридных праймеров, 3'-концы которых комплементарны 5'-концам ДНК в заданных местах гидролиза GlaI, а оставшаяся часть комплементарна адаптерной последовательности, и делают заключение о наличии последовательности R(5mC)GY, где 5mC - 5-метилцитозин, R-А или G, Y-Т или С в регуляторных областях указанных генов по появлению флуоресцентного сигнала для определения статуса метилирования сайтов в клинических образцах методом GLAD-ПЦР-анализа и на основе информации о статусе метилирования одного или нескольких сайтов делают вывод о наличии или отсутствии колоректального рака, причем при реализации способа используют набор праймеров и зондов по п. 1.
| WO 2014062218 А1, 24.04.2014 | |||
| Топочная решетка для сжигания фрезерованного торфа | 1928 |
|
SU13634A1 |
| US 20080221056 А1, 11.09.2008 | |||
| EP 2886659 В1, 24.06.2015 | |||
| US 20070026393 А1, 01.02.2007 | |||
| US 20080064029 А1, 13.03.2008 | |||
| RU 2013132761 A, 27.01.2015. | |||
Авторы
Даты
2017-09-11—Публикация
2016-07-21—Подача