Изобретение относится к медицине, молекулярно-биологическим исследованиям в области выявления онкологических заболеваний и может быть использовано для ранней неинвазивной диагностики рака шейки матки.
Рак шейки матки (далее - РШМ) является одним из наиболее распространенных онкозаболеваний у женщин во всем мире и занимает 4-е место по смертности. Согласно данным ВОЗ, в мире от РШМ умирает практически каждая вторая женщина. В РФ ежегодно фиксируется 22 новых случая заболевания на каждые 100 000 женщин, при этом у каждой 2-й обнаруживаются предраковые состояния (HSIL). Современные методы позволяют диагностировать заболевание в 78% случаев лишь на 3 стадии.
Раннее выявление РШМ, согласно Приказу МинЗдрава РФ от 24.04.2018 г. №186 «Об утверждении Концепции предиктивной, превентивной и персонализированной медицины», позволит ускорить переход к персонализированной медицине и сформировать индивидуализированный подход к лечению заболевания.
Современное состояние существующей в настоящее время в Российской Федерации программы скрининга, включенной в медосмотр, показывает недостаточную эффективность по обнаружению РШМ. Выделяют две системы скрининга - организованный (систематический) и неорганизованный (спорадический). Обе системы содержат следующие инструменты диагностики: цитологические исследования (включая PAP-тест), ПЦР-анализ на наличие вируса папилломы человека высокого онкогенного риска, ИФА-анализ на онкомаркеры, кольпоскопия и биопсия. Данные методы исследования имеют ряд существенных недостатков - субъективность в оценке результатов, требуется высокая квалификация врача и лаборанта, а также невозможность диагностики злокачественных поражений, локализованных в области цервикального канала, составляющих большинство патологий шейки матки, технические ошибки, связанные с нарушением техники получения мазков; всё это понижает уровень эффективности проведения скрининговых программ. Также возможны случаи расхождения результатов цитологического исследования и прицельной биопсии, обусловленные погрешностями забора материала (объём, взятие биопсии не из той зоны), эндоцервикальное расположение пораженного эпителия.
Непосредственно диагноз РШМ ставится только после проведения гистологического исследования биопсий, т.к. проведение цитологических и биохимических исследований недостаточно для верификации.
Биопсия является инвазивным методом исследования и достаточно неприятной и болезненной процедурой, которую может выполнить только опытный врач-гинеколог, поэтому гистологическое исследование невозможно использовать в качестве скринингового исследования.
В последние годы в РФ проведено достаточно много исследований, посвящённых диагностике РШМ с использованием молекулярно-генетических методов, несмотря на это наборов для молекулярно-генетических исследований для ранней диагностики рака шейки матки не существует.
Среди множества исследований, связанных с использованием генетических маркеров злокачественных образований, наиболее актуальным является определение уровня метилирования отдельных генов. Метилирование генов представляет собой модификацию молекулы ДНК без изменения нуклеотидной последовательности путём присоединения метильной группы к цитозину в составе CpG-динуклеотида в позиции С5 цитозинового кольца. Метилирование в промоторной зоне гена приводит к супрессии соответствующего гена. Изменение экспрессии генов в результате их метилирования может обуславливать развитие онкологических заболеваний. В таких случаях в клетках опухоли тотальное гипометилирование ДНК сочетается с локальным гиперметилированием ДНК промоторов генов, связанных с регуляцией клеточного цикла и вызывающих нестабильность генома [Залетаев Д.В. и др. // Экологическая генетика. - 2011. - Т. 9. - №3; Татаркова Е.А. // Вестник Адыгейского государственного университета. Серия 4: Естественно-математические и технические науки. - 2014].
Уровень метилирования генов может быть определен разными способами, основанными на применении химической модификации цитозиновых оснований, метилчувствительных рестриктаз, реакции иммунопреципитации метилированной ДНК, а также различные комбинации вышеуказанных подходов [Скрябин Н.А. и др. // Сибирский онкологический журнал. - 2013. - №6].
Наиболее перспективным методом, определяющим наличие метилированных участков ДНК, является метод, основанный на использовании сайт-специфических метилзависимых эндонуклеаз, которые узнают только метилированные последовательности ДНК.
Метод, основывающийся на совпадении последовательности ДНК, являющейся продуктом метилирования de novo и субстратной специфичности фермента GlaI и позволяющий определять наличие и количество копий сайта 5'-R(5mC)GY-3' в исследуемой точке генома, называется GLAD-ПЦР (GlaI hydrolysis and Ligation Adapter Dependent PCR) [Кузнецов В.В. с соавт. Способ определения нуклеотидной последовательности Pu(5mC)GPu в заданном положении протяженной ДНК // Патент РФ №2525710 С1, опубл. 20.08.2014, Бюл. №23]. Данный метод состоит из трёх последовательных этапов - гидролиз исследуемой ДНК с помощью GlaI, лигирование универсального адаптера и реакцию ПЦР в режиме реального времени и является универсальным методом определения уровня метилирования сайтов R(5mC)GY, позволяющим сформировать и описать конкретную панель генов, специфичную для различных типов рака, но при этом в описании метода специфичные для различных видов онкологии панели генов отсутствуют.
В последние десятилетия увеличилось количество исследований, посвящённых изучению уровня метилирования генов промоторных регионов генов-онкомаркеров РШМ. В 2018 году был разработан GYN-TEST, основанный на определении уровня метилирования панели генов (ASTN1, DLX1, ITGA4, RXFP3, SOX17 и ZNF671) с помощью бисульфитной обработки без процедуры выделения ДНК. Биологический материал, используемый при анализе - соскобы из цервикального канала [https://www.oncgnostics.com/gyntect-cervical-cancer/information-for-diagnostics-laboratories/test-principle/?lang=en].
В компании Qiagen существует набор для коммерческого использования QIAsure Methylation Test Kit (FAM19A4/mir124-2DNA methylation test), направленный на определение уровня метилирования промоторных участков генов FAM19A4 и mir124-2 у женщин, имеющих положительный ВПЧ-статус. Исследуемый биоматериал - соскобы из цервикального канала.
Недостатком вышеперечисленных известных аналогов является использование метода бисульфитной конверсии ДНК, для которой характерны трудоемкость, дороговизна исследования, значительные временные затраты, деградация ДНК и значительные потери ДНК, что приводит к снижению точности и чувствительности.
Результат предлагаемого нами изобретения - создание набора олигонуклеотидных праймеров и флуоресцентно-меченых зондов для определения уровня метилирования эпигенетических маркеров, обуславливающих возникновение РШМ методом GLAD- ПЦР.
Указанный технический результат достигается за счёт решения следующих задач:
- проведение биоинформационного анализа с целью отбора эпигенетических маркеров РШМ,
- создание набора олигонуклеотидных праймеров и флуоресцентно-меченых зондов для определения уровня метилирования промоторных участков эпигенетических маркеров с помощью метода GLAD-ПЦР, учёт результатов анализа проводится методом гибридизационно-флуоресцентной детекции продуктов ПЦР-амплификации в режиме реального времени.
Сущность изобретения поясняется следующими примерами
Пример 1. Разработка специфических праймеров и флуоресцентно-меченых зондов на участки CpG - островков промоторных областей генов-онкомаркеров и проведение анализа методом GLAD-ПЦР для определения уровня метилирования промоторных участков генов-онкомаркеров.
В качестве эпигенетических маркеров предложены промоторные области генов CADM1, MAL, PAX1, DAPK1, FAM19A4 и PRDM14, т.к. анализ уровня гиперметилирования участков этих генов позволяет определить как предраковые состояния, так и рак на разных этапах развития развития [Luttmer R., et al. // British journal of cancer. - 2016. - Vol.115; Feng C., et al. // International Journal o Genmics. - 2018; Wang X., et al. // Frontiers Genetics. - 2018. - Vol. 9; Fang C., et al. // Molecular Genetics and Genomic Medicine. - 2019. - Vol. 7. - Issue 3; Dankai W., et al.. // Plos One. - 2019] благодаря чему будет возможна ранняя диагностика РШМ (1-2 стадия).
Биоинформационный анализ генов-онкомаркеров CADM1, MAL, PAX1, DAPK1, FAM19A4 и PRDM14 показал наличие в их промоторной области CpG-островков, в которых присутствуют несколько сайтов RCGY, метилирование которых может вызывать подавление экспрессии гена при РШМ. При определении метилированных участков эпигенетических маркеров использовались ресурсы NCBI, а также доступные для анализа отечественные и зарубежные публикации. В результате были выбраны следующие фрагменты, удовлетворяющие условиям специфичности для дальнейшего получения праймеров (табл. 1).
Таблица 1. Выбранные фрагменты генов для проведения GLAD-ПЦР анализа
сайта RCGY
начала сайта
RCGY
Подбор структур праймеров и флуоресцентных зондов для анализа метилирования выбранных сайтов проводили, используя программное обеспечение Oligo 4.0. Олигонуклеотидные структуры подбирались по референсной последовательности генома человека (сборка GRCh38.p7). Поскольку геномы реальных людей отличаются от референсной («усредненной») последовательности, то, чтобы минимизировать вероятность несоответствия подобранных структур исследуемым образцам ДНК, их дополнительно проверяли по базе данных «dbSNP» на содержание часто встречающихся однонуклеотидных замен. При этом допустимым содержанием однонуклеотидных замен в подобранных структурах считалась частота не более 1% при единственной замене в каждой олигонуклеотидной структуре. Каждая из структур также была проверена на уникальность в геноме человека с помощью веб-сервиса BLAST (https://blast.ncbi.nlm.nih.gov).
Для подбора гибридного праймера была использована методология, а также структуры олигонуклеотидного адаптера и гибридных праймеров согласно известному способу [Кузнецов В.В. с соавт. Способ определения нуклеотидной последовательности Pu(5mC)Gpu в заданном положении протяженной последовательности ДНК // Патент РФ №2525710 С1, опубл. 20.08.2014, Бюл. №23].
Структура гибридной праймера представляет собой 19-звенную последовательность следующего состава: 5’ - CCTGCTCTTTCATCCgYNN-3’, где подчеркиванием выделена 15-звенная часть праймера, комплементарная универсальному олигонуклеотидному адаптеру, а gYNN - участок, комплементарный геномной последовательности в точке гидролиза ДНК метилзависимой эндонуклеазой GlaI. Данная структура предполагает наличие 32 варианта гибридных праймеров, соответствующих различным точкам гидролиза ДНК по сайту R(5mC)GY.
Последовательности подобранных праймеров и флуоресцентных зондов приведены в таблице 2.
Таблица 2. Последовательности олигонуклеотидных праймеров и флуоресцентно-меченых зондов
праймера (длина в нуклеотидах)
SEQ ID NO: 2
SEQ ID NO: 3
5' FAM-CCCGCCCCCTGGAGCCCG-BHQ1 3'(18)
5' CGGTCTGATACAGCGATTGCTATAAACAT 3'(29)
SEQ ID NO: 5
SEQ ID NO: 6
5' FAM-CCCAGCTGTGCCGGCCCCA-BHQ1 3'(19)
5' CCTGCTCTTTCATCGGTCT 3'(19)
SEQ ID NO: 8
SEQ ID NO: 9
5' FAM-TGCCCACTCCAAGCCAGACCCAGG-BHQ1 3'(24)
5' TGGGTCCCCAGACAGCTCGG 3'(20)
SEQ ID NO: 11
SEQ ID NO: 12
5' FAM-CGGCGCGGCCCACCCACC-BHQ1 3'(18)
5' GCGGCCAATGGGGACCCT 3'(18)
SEQ ID NO: 14
SEQ ID NO: 15
5' FAM-ACGTGGGCCGAGTTTTCTCCTCTCTCA-BHQ1 3'(27)
5' GCCCGGCCCCAGCTGTATT 3'(19)
SEQ ID NO: 17
SEQ ID NO: 18
5' CGGTCCCGGGATGGCTCT 3'(18)
5' CCTGCTCTTTCATCGGCGG 3'(19)
Специфичность всех подобранных праймеров была подтверждена путём проверки в Nucleotide BLAST (https://blast.ncbi.nlm.nih.gov). Температура плавления, а также вероятность образования димеров между олигонуклеотидами проверялись в OligoAnalyzer 3.1 (https://eu.idtdna.com/calc/analyzer).
Ход работы с применением набора синтетических олигонуклеотидов и флуоресцентно-меченых зондов для определения уровня метилирования промоторных участков ДНК состоит из следующих этапов:
1. Забор и подготовка биологического материала - цервикальных соскобов;
2. Выделение ДНК из биологических образцов с использованием набора для выделения ДНК (например, «Проба-НК», производитель «ДНК-Технология»);
Препараты выделенной ДНК должны обладать чистотой А260/280 от 1,4 до 1,8; А260/230 от 0,4 до 0,6 концентрации нуклеиновых кислот должны быть от 80 до 250 нг/мкл;
3. Проведение анализа по определению уровня метилирования промоторных участков выбранных генов-онкомаркеров с использованием набора для GLAD-ПЦР анализа (производитель ООО «Сибэнзайм»);
3.1. Подготовка ДНК и гидролиз включает в себя подготовку пробирок для ПЦР (200 мкл) по количеству анализируемых образцов; смешивание в каждой пробирке 14 мкл H2O и 1 мкл выделенной ДНК; подготовка общей реакционной смеси для гидролиза ДНК (H2O, 10х SE TMN буфера, ДМСО, БСА); добавление смеси в пробирки с образцами ДНК и последующее инкубирование в течение 30 минут при 30°С;
3.2. Лигирование (сшивка) фрагментов ДНК с универсальным адаптером с помощью ДНК-лигазы включает в себя подготовку общей реакционной смеси для гидролиза ДНК (H2O, универсальный адаптер, АТФ, Т4 ДНК-лигаза); добавление данной смеси в каждую пробирку и последующее инкубирование в течение 15 мин. при 25°С;
3.3. ПЦР в режиме реального времени включает в себя подготовку ПЦР-смеси для исследуемого сайта R(5mC)GY (H2O, 10х SE GLAD-буфер, MgCl2, смесь трифосфатов, БСА, смесь праймеров и SP Taq ДНК Полимераза); добавление подготовленной ПЦР-смеси в пробирки с ДНК; перенос полученной реакционной смеси в объёме 20 мкл в лунки на ПЦР-планшете; центрифугирование планшета и постановка в амплификатор для проведения ПЦР в режиме реального времени по следующей схеме:
- 3 мин. при 95°C;
- 5 «слепых» циклов: 95°C - 10 сек., 63°C- 20 сек., 72°C - 5 сек.;
- 40 циклов: 95°C - 10 сек., 63°C - 20 сек. (с детекцией флуоресцентного сигнала по каналу FAM), 72°C - 5 сек.;
4. Определение значений Cq для всех анализируемых образцов, по которой устанавливают копийность и определяют процент метилирования выбранных сайтов, специфичных для рака шейки матки (фиг.1).
Пример 2. GLAD-ПЦР-анализ определения уровня метилирования генов-онкомаркеров рака шейки матки
Для проведения GLAD-ПЦР-анализа были использованы образцы цервикальных соскобов, полученных от больных РШМ (n=12) и здоровых людей (n=5). Из образцов была выделена ДНК с помощью набора «Экстракция 100» (производитель «Вектор-Бест», Россия). Концентрация образцов была определена с помощью спектрофотометра Nano Drop 2000.
Препараты выделенной ДНК обладали чистотой А260/280 от 1,4 до 1,8; А260/230 от 0,4 до 0,6. Концентрации нуклеиновых кислот составляли от 80 до 250 нг/мкл.
Реакцию GLAD-ПЦР проводили как описано в примере 1. В качестве положительного контрольного образца была использована геномная ДНК Hela.
Полученные результаты представлены в таблице 3.
Таблица 3. Результаты анализа образцов цервикальных соскобов на наличие метилирования промоторных областей генов-онкомаркеров
Примечание: «РШМ» - рак шейки матки; «*» - диагноз ставится на основании гистологического заключения; «+» - наличие метилированных сайтов в промоторной области генов.
Анализируемый образец считают положительным, когда кривая накопления флуоресценции имела характерную экспоненциальную фазу роста и значения Ct составляет меньше либо равно 35, что означает наличие метилированных сайтов в промоторной области генов-онкомаркеров. Анализируемый образец считают отрицательным, когда кривая накопления флуоресценции отсутствует, либо имеет значение Ct больше или равно 35 - в промоторной области отсутствуют метилированные сайты.
Наличие или отсутствие метилированных сайтов в эпигенетических маркерах РШМ дает представление о статусе метилирования гена и может быть использовано в качестве дополнительного инструмента для постановки диагноза РШМ на ранних стадиях.
Использование заявляемого решения позволяет достигать технический результат: создание набора олигонуклеотидных праймеров и флуоресцентно-меченых зондов для определения уровня метилирования эпигенетических маркеров, вызывающих РШМ методом GLAD- ПЦР.
Основными преимуществами предлагаемого способа являются:
• использование доступного ПЦР-оборудования,
• время проведения анализа - 2-4 часа;
• простота использования - 3 последовательных этапа в одной пробирке;
• высокая чувствительность - потери ДНК минимальны;
• высокая специфичность - за счёт использования высокоспецифичных праймеров и зондов.
Технический результат достигнут путём проведения биоинформационного анализа с целью отбора эпигенетических маркеров РШМ и создания набора олигонуклеотидных праймеров и флуоресцентно-меченых зондов для определения уровня метилирования промоторных участков эпигенетических маркеров с помощью метода GLAD-ПЦР, учёт результатов анализа проводится методом гибридизационно-флуоресцентной детекции продуктов ПЦР-амплификации в режиме реального времени.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Общество с ограниченной ответственностью «Эс Джи»
<120> Набор олигонуклеотидных праймеров и флуоресцентно-меченых
зондов для выявления эпигенетических маркеров рака шейки матки.
<212> DNA
<213> Homo sapiens
<223> для специфических праймеров и зондов на участки CpG-островков
промоторных областей генов-онкомаркеров CADM1
<210> 1
<211> 19
<400> 5'-CCTGCTCTTTCATCGGTGC-3'
<210> 2
<211> 18
<400> 5'-FAM-CCCGCCCCCTGGAGCCCG-BHQ1-3'
<210> 3
<211> 29
<400> 5'-CGGTCTGATACAGCGATTGCTATAAACAT-3'
<223> для специфических праймеров и зондов на участки CpG-островков
промоторных областей генов-онкомаркеров MAL
<210> 4
<211> 18
<400> 5'-CCGACCTGCCCCAGCACC-3'
<210> 5
<211> 19
<400> 5'-FAM-CCCAGCTGTGCCGGCCCCA-BHQ1-3'
<210> 6
<211> 19
<400> 5'-CCTGCTCTTTCATCGGTCT-3'
<223> для специфических праймеров и зондов на участки CpG-островков
промоторных областей генов-онкомаркеров PAX1
<210> 7
<211> 19
<400> 5'-CCTGCTCTTTCATCGGCCC-3'
<210> 8
<211> 24
<400> 5'-FAM-TGCCCACTCCAAGCCAGACCCAGG-BHQ1-3'
<210> 9
<211> 20
<400> 5'-TGGGTCCCCAGACAGCTCGG-3'
<223> для специфических праймеров и зондов на участки CpG-островков
промоторных областей генов-онкомаркеров DAPK1
<210> 10
<211> 19
<400> 5'-CCTGCTCTTTCATCGGCAG-3'
<210> 11
<211> 18
<400> 5'-FAM-CGGCGCGGCCCACCCACC-BHQ1-3' (18 н)
<210> 12
<211> 18
<400> 5'-GCGGCCAATGGGGACCCT-3' (18 н)
<223> для специфических праймеров и зондов на участки CpG-островков
промоторных областей генов-онкомаркеров TAFA4 (FAM19A4)
<210> 13
<211> 19
<400> 5'-CCTGCTCTTTCATCGGTGT-3'
<210> 14
<211> 27
<400> 5'-FAM-ACGTGGGCCGAGTTTTCTCCTCTCTCA-BHQ1-3'
<210> 15
<211> 19
<400> 5'-GCCCGGCCCCAGCTGTATT-3'
<223> для специфических праймеров и зондов на участки
CpG-островков промоторных областей генов-онкомаркеров PRDM14
<210> 16
<211> 16
<400> 5'-FAM-CCAGGTTCTGCGGGCTGCTCTCC-BHQ1-3' (23 н)
<210> 17
<211> 18
<400> 5'-CGGTCCCGGGATGGCTCT-3'
<210> 18
<211> 19
<400> 5'-CCTGCTCTTTCATCGGCGG-3'
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ОПРЕДЕЛЕНИЯ МЕТИЛИРОВАНИЯ САЙТОВ PuCGPy РЕГУЛЯТОРНЫХ ОБЛАСТЕЙ ГЕНОВ-ОНКОМАРКЕРОВ КОЛОРЕКТАЛЬНОГО РАКА МЕТОДОМ GLAD-ПЦР-АНАЛИЗА И ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ И ФЛУОРЕСЦЕНТНО-МЕЧЕНЫЕ ЗОНДЫ ДЛЯ ОСУЩЕСТВЛЕНИЯ УКАЗАННОГО СПОСОБА | 2015 |
|
RU2596404C1 |
| Способ определения метилирования сайтов PuCGPy регуляторных областей генов-онкомаркеров колоректального рака методом GLAD-ПЦР-анализа и набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для осуществления указанного способа | 2016 |
|
RU2630669C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ ВОЗРАСТА ЧЕЛОВЕКА ПО ДНК | 2021 |
|
RU2797018C1 |
| БИОМАРКЕРЫ МЕТИЛИРОВАНИЯ ДНК ДЛЯ РАННЕГО ВЫЯВЛЕНИЯ РАКА ШЕЙКИ МАТКИ | 2020 |
|
RU2817569C2 |
| Набор олигонуклеотидных праймеров и зондов для определения аллей полиморфизма rs55986091 и способ его применения | 2022 |
|
RU2804110C1 |
| Набор для определения копийности гена АРР в геноме человека | 2021 |
|
RU2789799C1 |
| Олигонуклеотиды для определения 12FAB серотипа Streptococcus pneumoniae | 2021 |
|
RU2795021C1 |
| Набор олигонуклеотидных праймеров и флуоресцентно-меченого зонда в формате TaqMan для выявления вирусов рода Flavivirus методом ПЦР в режиме реального времени | 2022 |
|
RU2808520C1 |
| Набор для выявления коронавируса SARS-CoV-2 | 2021 |
|
RU2765497C1 |
| СОСТАВ, НАБОР И ПРИМЕНЕНИЕ ДЛЯ ВЫЯВЛЕНИЯ КОЛОРЕКТАЛЬНОГО РАКА | 2021 |
|
RU2836003C2 |
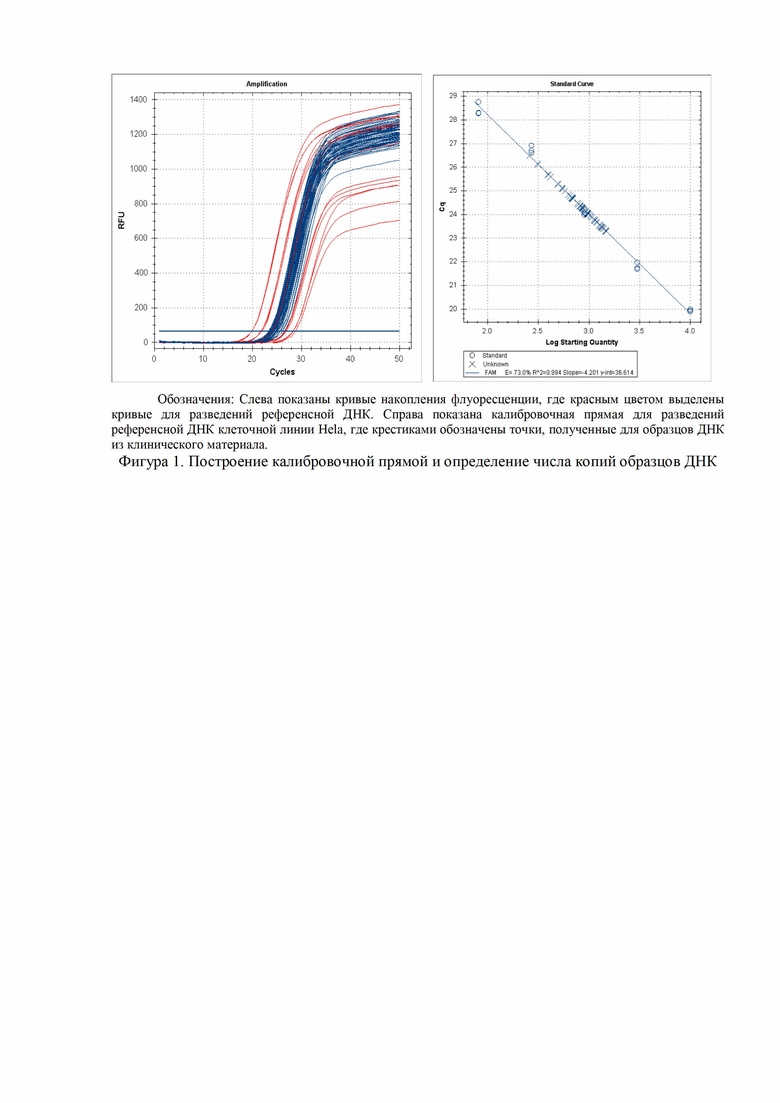
Изобретение относится к области медицинской биотехнологии. Для решения поставленной задачи определяют метилированные сайты RCGY промоторных областей генов-онкомаркеров CADM1, MAL, PAX1, DAPK1, FAM19A4, PRDM14 с помощью метода GLAD-ПЦР и используют специально разработанные олигонуклеотидные праймеры и зонды для определения этих участков. Использование заявляемого решения позволяет достигать технический результат: создание набора олигонуклеотидных праймеров и флуоресцентно-меченых зондов для определения уровня метилирования эпигенетических маркеров, обуславливающих возникновение рака шейки матки, методом GLAD-ПЦР. 3 табл., 1 ил.
Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для определения уровня метилирования промоторных областей генов-онкомаркеров для ранней неинвазивной диагностики рака шейки матки с помощью GLAD-ПЦР-анализа, в образцах ДНК, имеющих следующую структуру:
праймера (длина в нуклеотидах)
SEQ ID NO: 2
SEQ ID NO: 3
5' FAM-CCCGCCCCCTGGAGCCCG-BHQ1 3'(18)
5' CGGTCTGATACAGCGATTGCTATAAACAT 3'(29)
SEQ ID NO: 5
SEQ ID NO: 6
5' FAM-CCCAGCTGTGCCGGCCCCA-BHQ1 3'(19)
5' CCTGCTCTTTCATCGGTCT 3'(19)
SEQ ID NO: 8
SEQ ID NO: 9
5' FAM-TGCCCACTCCAAGCCAGACCCAGG-BHQ1 3'(24)
5' TGGGTCCCCAGACAGCTCGG 3'(20)
SEQ ID NO: 11
SEQ ID NO: 12
5' FAM-CGGCGCGGCCCACCCACC-BHQ1 3'(18)
5' GCGGCCAATGGGGACCCT 3'(18)
SEQ ID NO: 14
SEQ ID NO: 15
5' FAM-ACGTGGGCCGAGTTTTCTCCTCTCTCA-BHQ1 3'(27)
5' GCCCGGCCCCAGCTGTATT 3'(19)
SEQ ID NO: 17
SEQ ID NO: 18
5' CGGTCCCGGGATGGCTCT 3'(18)
5' CCTGCTCTTTCATCGGCGG 3'(19)
| Приспособление для зажима листового металла при загибании его краев под прессом | 1929 |
|
SU21100A1 |
| ЗАЛЕТАЕВ Л.В | |||
| и др | |||
| Аномалии метилирования в процессах канцерогенеза: поиск новых генов, разработка методов и систем ДНК-маркеров для диагностик | |||
| Экологическая генетика человека | |||
| Способ приготовления лака | 1924 |
|
SU2011A1 |
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Прибор с двумя призмами | 1917 |
|
SU27A1 |
| LUTTMER R | |||
| et al | |||
| Способ изготовления электрических сопротивлений посредством осаждения слоя проводника на поверхности изолятора | 1921 |
|
SU19A1 |
Авторы
Даты
2021-11-29—Публикация
2021-01-20—Подача