Перекрестные ссылки на смежные заявки
Настоящая заявка испрашивает преимущество по предварительной заявке на патент США № 61/726,489, поданной 14 ноября 2012 г., и предварительной заявке на патент США № 61/644,294, поданной 8 мая 2012 г., полные описания которых полностью включены в настоящий документ путем ссылки для всех целей.
Предпосылки создания настоящего изобретения
Область применения изобретения
Настоящее описание относится по существу к количественному высокопроизводительному секвенированию ДНК, кодирующей рецепторы адаптивной иммунной системы (например, ДНК, кодирующей рецепторы Т-клеток (TCR) и иммуноглобулины (IG) в мультиплексных реакциях амплификации нуклеиновых кислот. В частности, описанные в настоящем документе композиции и способы решают проблему нежелательных искажений результатов количественного определения последовательностей, кодирующих рецепторы адаптивной иммунной системы, которые могут возникать вследствие систематического избыточного и/или недостаточного использования конкретных олигонуклеотидных праймеров в мультиплексной амплификации ДНК.
Описание области применения изобретения
Адаптивная иммунная система применяет несколько стратегий наработки достаточно разнообразного для обнаружения совокупности потенциальных патогенов репертуара рецепторов антигенов T- и B-клеток, т. е. рецепторов адаптивной иммунной системы. Способность T-клеток обнаруживать совокупность антигенов, ассоциированных с различными типами рака или инфекционными организмами, обеспечивается их антигенными рецепторами T-клеток (TCR), которые представляют собой гетеродимеры α (альфа)-цепи из локуса TCRA и β (бета)-цепи из локуса TCRB или гетеродимеры γ (гамма)-цепи из локуса TCRG и δ (дельта)-цепи из локуса TCRD. Составляющие эти цепи белки кодируются ДНК, которая в лимфоидных клетках применяет уникальный механизм перестройки для генерации широкого разнообразия TCR. Данный мультисубъединичный рецептор иммунного распознавания ассоциируется с комплексом CD3 и связывается с пептидами, которые представлены или белками главного комплекса гистосовместимости (MHC) класса I, или белками MHC класса II на поверхности антигенпредставляющих клеток (АПК). Связывание TCR с пептидом-антигеном на АПК — это центральное для активации T-клетки событие, которое происходит в иммунологическом синапсе в точке контакта T-клетки и АПК.
Каждый пептид TCR содержит вариабельные участки, определяющие комплементарность (CDR), а также каркасные участки (FR) и константный участок. Разнообразие последовательностей αβT-клеток в основном определяется аминокислотной последовательностью петель третьего участка, определяющего комплементарность (CDR3), вариабельных доменов α- и β-цепей, причем разнообразие является следствие рекомбинации сегментов генов, ответственных за вариабельность (Vβ), разнообразие (Dβ) и объединение (Jβ), в локусе β-цепи, а также между аналогичными Vα- и Jα-сегментами генов в локусе α-цепи соответственно. Существование множества таких сегментов генов в TCR локусах α- и β-цепи позволяет кодировать большое число различных последовательностей CDR3. Разнообразие последовательностей CDR3 дополнительно повышается путем независимого добавления и делеции нуклеотидов в участках соединений Vβ-Dβ, Dβ-Jβ и Vα-Jα при перестройке гена TCR. В этом смысле иммунокомпетентность является следствием разнообразия TCR.
Гетеродимер γδ TCR отличается от αβ TCR тем, что он кодирует рецептор, который тесно взаимодействует с врожденной иммунной системой и распознает антиген независимо от HLA. TCRγδ экспрессируется на ранних стадиях развития и имеет специализированное анатомическое распределение, уникальную специфичность к патогенам и малым молекулам, а также широкий диапазон врожденных и адаптивных клеточных взаимодействий. Уже на ранних стадиях онтогенеза устанавливается систематически отклоняющийся характер экспрессии V- и J-сегментов TCRγ. Следовательно, разнообразие репертуара TCRγ в зрелых тканях является следствием обширного периферического размножения, вызванного стимуляцией присутствующими в окружающей среде патогенами и токсичными молекулами.
Иммуноглобулины (Ig или IG), также называемые в настоящем документе рецепторами B-клеток (BCR), представляют собой белки, экспрессируемые B-клетками, которые состоят из четырех полипептидных цепей, двух тяжелых цепей (H-цепей) из локуса IGH и двух легких цепей (L-цепей) из локуса IGK или IGL, которые формируют структуру H2L2. Каждая H- и L-цепь содержит три участка, определяющих комплементарность (CDR), участвующих в распознавании антигена, а также каркасные участки и константный домен, аналогичный TCR. H-цепи в Ig изначально экспрессируются как мембранно-связанные изоформы с использованием экзонов константного участка IGM или IGD, но после обнаружения антигена константный участок может сменить класс на несколько дополнительных изотипов, включая IGG, IGE и IGA. Как и в случае TCR, разнообразие интактных Ig у индивидуума в основном определяется гипервариабельным участком, определяющим комплементарность (CDR). Аналогично TCRB, домен CDR3 H-цепей создается путем комбинаторного соединения сегментов генов VH, DH и JH. Разнообразие последовательностей гипервариабельного домена дополнительно повышается путем независимого добавления и делеции нуклеотидов в участках соединений VH-DH, DH-JH и VH-JH при перестройке гена Ig. В отличие от TCR, разнообразие последовательностей Ig дополнительно повышается путем соматической гипермутации (SHM) в перестроенном гене IG после первичного распознавания антигена интактной B-клеткой. Процесс SHM не ограничен CDR3 и поэтому может приводить к изменениям последовательности зародышевой линии в каркасных участках CDR1 и CDR2, а также в соматически перестроенном CDR3.
Поскольку адаптивная иммунная система функционирует частично путем клонального размножения клеток, которые экспрессируют уникальные TCR или BCR, точное измерение изменений общего содержания каждого из клонов T- или B-клеток важно для понимания динамики адаптивного иммунного ответа. Например, у здорового человека имеется несколько миллионов уникальных перестроенных цепей TCRβ, каждую из которых несут от сотен до тысяч клональных T-клеток из приблизительно триллиона T-клеток у здорового индивидуума. Опираясь на достижения в области высокопроизводительного секвенирования, недавно сформировалась новая область молекулярной иммунологии для профилирования большого репертуара TCR и BCR. Композиции и способы секвенирования генных последовательностей перестроенных рецепторов адаптивной иммунной системы и определения клонотипа рецептора адаптивной иммунной системы описаны в заявке на патент США № 13/217,126; заявке на патент США № 12/794,507; публикациях PCT/US2011/026373 и PCT/US2011/049012, все из которых включены в настоящий документ путем ссылки.
В настоящее время применяются несколько различных стратегий высокопроизводительного количественного секвенирования нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы, и эти стратегии можно различить, например, по подходу, который используется при амплификации участков, кодирующих CDR3, и по выбору объекта секвенирования - геномной ДНК (гДНК) или матричной РНК (мРНК).
Секвенирование мРНК потенциально проще, чем секвенирование гДНК, поскольку события сплайсинга мРНК удаляют интрон между J- и C-сегментами. Это позволяет проводить амплификацию рецепторов адаптивной иммунной системы (например, TCR или Ig) с разными V- и J-участками, используя обычный 3’ ПЦР-праймер в C-участке. Для каждой TCRβ, например, все тринадцать J-сегментов имеют длину менее 60 пар оснований (п. о.). Поэтому события сплайсинга дают идентичные полинуклеотидные последовательности, кодирующие константные участки TCRβ (независимо от того, какие последовательности V и J используются), в пределах менее 100 п. о. от перестроенного соединения VDJ. Затем сплайсированную мРНК можно транскрибировать обратно в комплементарную ДНК (кДНК), используя poly-dT-праймеры, комплементарные poly-A-хвосту мРНК, случайные малые праймеры (обычно гексамеры или нонамеры) или специфические для C-сегмента олигонуклеотиды. Это должно позволить сгенерировать не имеющую систематических ошибок библиотеку TCR кДНК (поскольку все кДНК праймируются одним и тем же олигонуклеотидом, будь то poly-dT, случайный гексамер или специфический для C-сегмента олигонуклеотид), которую затем можно секвенировать для получения информации о V- и J-сегментах, используемых в каждой перестройке, а также о конкретной последовательности CDR3. Для такого секвенирования можно использовать технологию прочтения одиночных длинных фрагментов, охватывающих CDR3 («ПЦР длинных фрагментов»), или же вместо этого можно использовать широкую сборку более длинных последовательностей, используя фрагментированные библиотеки и прочтение более производительных и более коротких последовательностей.
Однако, поскольку каждая клетка потенциально экспрессирует разные количества мРНК TCR, довольно сложно интерпретировать попытки количественного определения в образце числа клеток, которые экспрессируют конкретный перестроенный TCR (или Ig), на основе секвенирования мРНК. Например, активированные in vitro T-клетки имеют в 10–100 раз больше мРНК на клетку, чем T-клетки в фазе покоя. В настоящее время имеется очень ограниченная информация по относительным количествам мРНК TCR в T-клетках в разных функциональных состояниях, поэтому количественное определение общего содержания мРНК не обязательно позволит точно определить число клеток, несущих каждый клональный вариант перестройки TCR.
С другой стороны, большинство T-клеток имеют один продуктивно перестроенный TCRα и один продуктивно перестроенный ген TCRβ (или два перестроенных TCRγ и TCRδ), а большинство B-клеток имеют один продуктивно перестроенный ген тяжелой цепи Ig и один продуктивно перестроенный ген легкой цепи Ig (IGK или IGL). Таким образом, количественное определение в образце геномной ДНК, кодирующей TCR или BCR, должно непосредственно соотноситься с числом T-клеток и B-клеток в образце соответственно. Предпочтительно, чтобы геномное секвенирование полинуклеотидов, кодирующих любую одну или более цепей рецептора адаптивной иммунной системы, предполагало одинаково эффективную амплификацию всех из множества возможных перестроенных последовательностей CDR3, присутствующих в образце, содержащем ДНК из лимфоидных клеток субъекта, с последующим количественным секвенированием для получения количественного значения относительного содержания каждого перестроенного клонотипа CDR3.
Однако при использовании таких подходов возникают трудности, связанные со сложностью достижения одинаковой эффективности амплификации и секвенирования для каждого перестроенного клона при использовании мультиплексной ПЦР. Например, в TCRB каждый клон применяет одну из 54 возможных зародышевых линий, кодирующих V-участок, и один из 13 возможных генов, кодирующих J-участок. Чтобы сгенерировать разнообразный репертуар рецепторов адаптивной иммунной системы, разнообразной должна быть последовательность ДНК V- и J-сегментов. Данное разнообразие последовательностей делает невозможным создание одной универсальной последовательности праймера, который одинаково аффинно гибридизируется со всеми V-сегментами (или J-сегментами) и создает сложные образцы ДНК, в которых точному определению содержащихся в них множеств различных последовательностей препятствуют технические ограничения одновременного количественного определения множества типов молекул с использованием мультиплексной амплификации и высокопроизводительного секвенирования.
Один или более факторов могут приводить к возникновению артефактов, которые искажают корреляцию между выходными данными секвенирования и числом копий клонотипа на входе, ставя под сомнение возможность получения надежных количественных данных при использовании стратегий секвенирования, основанных на мультиплексной амплификации набора матриц гена TCRβ с большим разнообразием. Данные артефакты зачастую возникают из-за неравномерного использования разнообразных праймеров на стадии мультиплексной амплификации. Такое использование одного или более олигонуклеотидных праймеров с систематической ошибкой в мультиплексной реакции с разнообразными матрицами амплификации может быть функцией дифференциальной кинетики отжига, вызванной одним или более из различий в нуклеотидном составе матриц и/или олигонуклеотидных праймеров, длине матрицы и/или праймера, конкретной используемой полимеразе, температур проведения реакции амплификации (например, температуры отжига, элонгации и/или денатурации) и/или других факторах (см., например, Kanagawa, 2003 г., J. Biosci. Bioeng. 96:317; Day et al., 1996 г., Hum. Mol. Genet. 5:2039; Ogino et al., 2002 г., J. Mol. Diagnost. 4:185; Barnard et al., 1998 г., Biotechniques 25:684; Aird et al., 2011 г., Genome Biol. 12:R18).
Несомненно, сохраняется потребность в улучшении композиций и способов точного количественного определения разнообразия последовательностей ДНК, кодирующих рецепторы адаптивной иммунной системы, в сложных образцах таким образом, чтобы предотвратить искажение результатов, такое как ошибочное заключение об избытке или недостатке индивидуальных последовательностей, вызванное систематическими ошибками при амплификации конкретных матриц в наборе олигонуклеотидных праймеров, используемых для мультиплексной амплификации популяции ДНК со сложной матрицей. Описанные в настоящем документе варианты осуществления направлены на удовлетворение данной потребности и обеспечивают другие связанные с ней преимущества.
Изложение сущности изобретения
Композиция для стандартизации эффективности амплификации набора олигонуклеотидных праймеров, который способен проводить амплификацию перестроенных молекул нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы в биологическом образце, содержащем перестроенные молекулы нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего, причем каждый рецептор адаптивной иммунной системы содержит вариабельный участок и соединительный участок, причем композиция содержит множество матричных олигонуклеотидов, имеющих множество олигонуклеотидных последовательностей общей формулы: 5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ [I], причем (а) V представляет собой полинуклеотид, содержащий по меньшей мере 20 и не более 1000 последовательных нуклеотидов генной последовательности, кодирующей вариабельный (V) участок рецептора адаптивной иммунной системы или его комплемент, а каждый V-полинуклеотид содержит уникальную олигонуклеотидную последовательность; (b) J представляет собой полинуклеотид, содержащий по меньшей мере 15 и не более 600 последовательных нуклеотидов генной последовательности, кодирующей соединительный (J) участок рецептора адаптивной иммунной системы или его комплемент, а каждый J-полинуклеотид содержит уникальную олигонуклеотидную последовательность; (c) U1 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) первой универсальной последовательности олигонуклеотида-адаптера и (ii) первой специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5’ и находится в нем относительно первой универсальной последовательности олигонуклеотида-адаптера; (d) U2 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) второй универсальной последовательности олигонуклеотида-адаптера и (ii) второй специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5’ и находится в нем относительно второй универсальной последовательности олигонуклеотида-адаптера; (e) каждый из B1, B2, B3 и B4 независимо либо отсутствует, либо содержит олигонуклеотид, B, который содержит последовательность штрихкода из 3–25 последовательных нуклеотидов, причем каждый B1, B2, B3 и B4 содержит олигонуклеотидную последовательность, которая уникально идентифицирует в качестве спаренной комбинации (i) уникальную олигонуклеотидную последовательность V из (а) и (ii) уникальную олигонуклеотидную последовательность J из (b); (f) R либо отсутствует, либо содержит сайт распознавания рестрикционного фермента, который содержит олигонуклеотидную последовательность, отсутствующую в (a)–(e), и причем: (g) множество матричных олигонуклеотидов содержит по меньшей мере a или по меньшей мере b уникальных олигонуклеотидных последовательностей в зависимости от того, какое значение больше, причем a представляет собой количество уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой количество уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта, и композиция содержит по меньшей мере один матричный олигонуклеотид для каждого уникального V-полинуклеотида и по меньшей мере один матричный олигонуклеотид для каждого уникального J-полинуклеотида.
В одном варианте осуществления a составляет от 1 до максимального числа сегментов V-гена в геноме субъекта-млекопитающего. В другом варианте осуществления b составляет от 1 до максимального числа сегментов J-гена в геноме субъекта-млекопитающего. В других вариантах осуществления a равно 1 или b равно 1.
В некоторых других вариантах осуществления множество матричных олигонуклеотидов содержит по меньшей мере (a x b) уникальных олигонуклеотидных последовательностей, где a представляет собой число уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы субъекта-млекопитающего, b представляет собой число уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы субъекта-млекопитающего, а композиция содержит по меньшей мере один матричный олигонуклеотид для каждой возможной комбинации сегмента гена, кодирующего V-участок, и сегмента гена, кодирующего J-участок. В одном варианте осуществления J содержит константный участок генной последовательности, кодирующей J-участок рецептора адаптивной иммунной системы.
В другом варианте осуществления рецептор адаптивной иммунной системы выбран из группы, состоящей из TCRB, TCRG, TCRA, TCRD, IGH, IGK и IGL. В некоторых вариантах полинуклеотид V из (а) кодирует полипептид V-участка рецептора TCRB, TCRG, TCRA, TCRD, IGH, IGK или IGL. В других вариантах J-полинуклеотид из (b) кодирует полипептид J-участка рецептора TCRB, TCRG, TCRA, TCRD, IGH, IGK или IGL.
В некоторых вариантах осуществления между V и B2 имеется стоп-кодон.
В одном варианте осуществления каждый матричный олигонуклеотид во множестве матричных олигонуклеотидов присутствует в по существу эквимолярном количестве. В другом варианте множество матричных олигонуклеотидов имеет множество последовательностей общей формулы (I), которая выбрана из: (1) множества олигонуклеотидных последовательностей общей формулы (I), в которой V- и J-полинуклеотиды имеют последовательности TCRB V и J, представленные в по меньшей мере одном наборе из 68 TCRB V и J SEQ ID NO. на Фиг. 5a–5l как TCRB V/J набор 1, TCRB V/J набор 2, TCRB V/J набор 3, TCRB V/J набор 4, TCRB V/J набор 5, TCRB V/J набор 6, TCRB V/J набор 7, TCRB V/J набор 8, TCRB V/J набор 9, TCRB V/J набор 10, TCRB V/J набор 11, TCRB V/J набор 12 и TCRB V/J набор 13; (2) множества олигонуклеотидных последовательностей общей формулы (I), в которой V- и J-полинуклеотиды имеют последовательности TCRG V и J, представленные в по меньшей мере одном наборе из 14 TCRG V и J SEQ ID NO. на Фиг. 6a и 6b как TCRG V/J набор 1, TCRG V/J набор 2, TCRG V/J набор 3, TCRG V/J набор 4 и TCRG V/J набор 5; (3) множества олигонуклеотидных последовательностей общей формулы (I), в которой V- и J-полинуклеотиды имеют последовательности IGH V и J, представленные в по меньшей мере одном наборе из 127 IGH V и J SEQ ID NO. на Фиг. 7a–7m как IGH V/J набор 1, IGH V/J набор 2, IGH V/J набор 3, IGH V/J набор 4, IGH V/J набор 5, IGH V/J набор 6, IGH V/J набор 7, IGH V/J набор 8 и IGH V/J набор 9; (4) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 3157–4014; (5) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 4015–4084; (6) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 4085–5200; (7) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 5579–5821; (8) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 5822–6066; и (9) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 6067–6191.
В некоторых вариантах осуществления V представляет собой полинуклеотид, содержащий по меньшей мере 30, 60, 90, 120, 150, 180 или 210 последовательных нуклеотидов генной последовательности, кодирующей V-участок рецептора адаптивной иммунной системы или его комплемент. В другом варианте осуществления V представляет собой полинуклеотид, содержащий не более 900, 800, 700, 600 или 500 последовательных нуклеотидов генной последовательности, кодирующей V-участок рецептора адаптивной иммунной системы или его комплемент.
В других вариантах осуществления J представляет собой полинуклеотид, содержащий по меньшей мере 16–30, 31–60, 61–90, 91–120 или 120–150 последовательных нуклеотидов генной последовательности, кодирующей J-участок рецептора адаптивной иммунной системы или его комплемент. В другом варианте осуществления J представляет собой полинуклеотид, содержащий не более 500, 400, 300, или 200 последовательных нуклеотидов генной последовательности, кодирующей J-участок рецептора адаптивной иммунной системы или его комплемент.
В некоторых вариантах осуществления каждый матричный олигонуклеотид имеет длину менее 1000, 900, 800, 700, 600, 500, 400, 300 или 200 нуклеотидов.
В других вариантах осуществления композиция включает набор олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенных молекул нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы, содержащий множество a’ уникальных олигонуклеотидных праймеров для V-сегмента и множество b’ уникальных олигонуклеотидных праймеров для J-сегмента. В некоторых вариантах осуществления a’ составляет от 1 до максимального числа сегментов V-гена в геноме млекопитающего, а b’ составляет от 1 до максимального числа сегментов J-гена в геноме млекопитающего. В одном варианте осуществления a’ равно a. В другом варианте осуществления b’ равно b.
В еще одном варианте осуществления каждый олигонуклеотидный праймер V-сегмента и каждый олигонуклеотидный праймер J-сегмента в наборе олигонуклеотидных праймеров способен гибридизироваться специфически с по меньшей мере одним матричным олигонуклеотидом во множестве матричных олигонуклеотидов. В других вариантах осуществления каждый олигонуклеотидный праймер для V-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному сегменту гена, кодирующего V-участок рецептора адаптивной иммунной системы. В другом варианте осуществления каждый олигонуклеотидный праймер для J-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному сегменту гена, кодирующего J-участок рецептора адаптивной иммунной системы.
В других вариантах осуществления композиция содержит по меньшей мере один матричный олигонуклеотид, имеющий олигонуклеотидную последовательность общей формулы (I), с которым может специфически гибридизироваться каждый олигонуклеотидный праймер V-сегмента, и по меньшей мере один матричный олигонуклеотид, имеющий олигонуклеотидную последовательность общей формулы (I), с которой может специфически гибридизироваться каждый олигонуклеотидный праймер J-сегмента.
Настоящее изобретение содержит способ определения неравномерного потенциала амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров, который способен выполнять амплификацию перестроенных молекул нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы в биологическом образце, содержащем перестроенные молекулы нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего. Способ включает следующие стадии: (а) амплификация композиции, как описано в настоящем документе, в мультиплексной ПЦР-реакции для получения множества амплифицированных матричных олигонуклеотидов; (b) секвенирование указанного множества амплифицированных матричных олигонуклеотидов для определения для каждого уникального матричного олигонуклеотида, содержащего указанное множество, (i) матричной олигонуклеотидной последовательности и (ii) частоты вхождения указанной матричной олигонуклеотидной последовательности; и (c) сравнения частоты вхождения каждой из указанных матричных олигонуклеотидных последовательностей с ожидаемым распределением, причем указанное ожидаемое распределение основано на заданных молярных соотношениях для указанного множества матричных олигонуклеотидов, составляющих указанную композицию, и причем отклонение указанной частоты вхождения указанных матричных олигонуклеотидных последовательностей от указанного ожидаемого распределения указывает на неравномерный потенциал амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров для амплификации.
В одном варианте осуществления заданные молярные соотношения являются эквимолярными. В другом варианте осуществления ожидаемое распределение содержит равномерный уровень амплификации для указанного набора матричных олигонуклеотидов, амплифицируемых указанным набором олигонуклеотидных праймеров. В еще одном варианте осуществления каждая молекула амплифицируемой матричной нуклеиновой кислоты имеет длину менее 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100, 90, 80 или 70 нуклеотидов.
Способ включает стадии, содержащие для каждого члена набора олигонуклеотидных праймеров, демонстрирующих неравномерный относительно ожидаемого распределения потенциал амплификации, коррекцию относительного представления данного члена набора олигонуклеотидных праймеров в наборе олигонуклеотидных праймеров для амплификации. В одном варианте осуществления коррекция содержит увеличение относительного представления члена в наборе олигонуклеотидных праймеров, посредством этого корректируя неравномерный потенциал амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров. В другом варианте осуществления коррекция содержит уменьшение относительного представления данного члена в наборе олигонуклеотидных праймеров, посредством этого корректируя неравномерный потенциал амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров.
В других вариантах осуществления набор олигонуклеотидных праймеров не включает в себя олигонуклеотидные праймеры, которые специфически гибридизируются с псевдогеном или орфоном V-участка или с псевдогеном или орфоном J-участка.
Способ также включает стадии, содержащие для каждого члена набора олигонуклеотидных праймеров для амплификации, демонстрирующих неравномерный относительно ожидаемого распределения потенциал амплификации, расчет пропорционально увеличенной или сниженной частоты вхождения молекул амплифицированных матричных нуклеиновых кислот, амплификация которых обеспечивается указанным членом, посредством чего корректируя неравномерный потенциал амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров.
Настоящее изобретение включает способ количественного определения множества перестроенных молекул нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы в биологическом образце, содержащем перестроенные молекулы нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего, причем каждый рецептор адаптивной иммунной системы содержит вариабельный (V) участок и соединительный (J) участок, причем способ включает: (А) амплификацию перестроенных молекул нуклеиновых кислот в мультиплексной полимеразной цепной реакции (ПЦР), которая содержит: (1) перестроенные молекулы нуклеиновых кислот из биологического образца, содержащего лимфоидные клетки субъекта-млекопитающего; (2) композицию, как описано в настоящем документе, в которой в известном количестве присутствует каждый из множества матричных олигонуклеотидов, имеющих уникальную олигонуклеотидную последовательность; (3) набор олигонуклеотидных праймеров для амплификации, который способен проводить амплификацию перестроенных молекул нуклеиновых кислот, кодирующих один или множество рецепторов адаптивной иммунной системы из биологического образца.
В некоторых вариантах осуществления набор праймеров содержит: (а) в по существу эквимолярных количествах — множество олигонуклеотидных праймеров для V-сегмента, каждый из которых независимо способен специфически гибридизироваться с по меньшей мере одним полинуклеотидом, кодирующим полипептид V-участка рецептора адаптивной иммунной системы, или с его комплементом, причем каждый праймер для V-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному функциональному сегменту гена, кодирующего V-участок рецептора адаптивной иммунной системы, и причем множество праймеров для V-сегмента специфически гибридизируется с по существу всеми функциональными сегментами гена, кодирующего V-участок рецептора адаптивной иммунной системы, которые присутствуют в композиции, и (b) в по существу эквимолярных количествах — множество олигонуклеотидных праймеров для J-сегмента, каждый из которых независимо способен специфически гибридизироваться с по меньшей мере одним полинуклеотидом, кодирующим полипептид J-участка рецептора адаптивной иммунной системы, или с его комплементом, причем каждый праймер для J-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному функциональному сегменту гена, кодирующего J-участок рецептора адаптивной иммунной системы, и причем множество праймеров для J-сегментов специфически гибридизируется с по существу всеми функциональными сегментами гена, кодирующего J-участок рецептора адаптивной иммунной системы, которые присутствуют в композиции.
В другом варианте осуществления олигонуклеотидные праймеры для V-сегмента и J-сегмента способны обеспечить в указанной мультиплексной полимеразной цепной реакции (ПЦР) амплификацию (i) по существу всех матричных олигонуклеотидов в композиции для получения множества амплифицированных матричных олигонуклеотидов, причем указанное множество амплифицированных матричных молекул нуклеиновых кислот достаточно для количественного определения разнообразия матричных олигонуклеотидов в композиции, и (ii) по существу всех перестроенных молекул нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы в биологическом образце, для получения множества амплифицированных перестроенных молекул ДНК, причем указанное множество амплифицированных перестроенных молекул нуклеиновых кислот достаточно для количественного определения разнообразия перестроенных молекул нуклеиновых кислот в ДНК из биологического образца.
В одном варианте осуществления каждая амплифицированная молекула нуклеиновой кислоты во множестве амплифицированных матричных олигонуклеотидов и во множестве амплифицированных перестроенных молекул нуклеиновых кислот имеет длину менее 1000 нуклеотидов; (B) количественное секвенирование указанных амплифицированных матричных олигонуклеотидов и указанных амплифицированных перестроенных молекул нуклеиновых кислот для количественного определения (i) количества матричного продукта амплифицированных матричных олигонуклеотидов, которые содержат по меньшей мере одну олигонуклеотидную последовательность штрихкода, и (ii) количества перестроенного продукта амплифицированных перестроенных молекул нуклеиновых кислот, в которых отсутствует олигонуклеотидная последовательность штрихкода; (C) вычисление коэффициента амплификации путем деления количества матричного продукта из (B) (i) на известное количество каждого из множества матричных олигонуклеотидов, имеющих уникальную олигонуклеотидную последовательность из (A) (2); (D) деление количества перестроенного продукта из (B) (ii) на коэффициент амплификации, вычисленный в (C), для количественного определения количества уникальных перестроенных молекул нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы, в образце.
В других вариантах осуществления количественно определенное количество уникальных перестроенных молекул нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы, в образце представляет собой количество уникальных геномных матриц B-клеток или уникальных геномных матриц T-клеток в образце.
Настоящее изобретение включает способ вычисления среднего коэффициента амплификации в мультиплексном ПЦР-анализе, содержащий получение биологического образца, который содержит перестроенные молекулы нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего; приведение указанного образца в контакт с известным количеством матричных олигонуклеотидов, составляющих композицию, как описано в настоящем документе; амплификацию матричных олигонуклеотидов и перестроенных молекул нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего в мультиплексной ПЦР-реакции для получения множества амплифицированных матричных олигонуклеотидов и множества амплифицированных перестроенных молекул нуклеиновых кислот; секвенирование указанного множества амплифицированных матричных олигонуклеотидов для определения для каждого уникального матричного олигонуклеотида, содержащего указанное множество, (i) матричной олигонуклеотидной последовательности и (ii) частоты вхождения указанной матричной олигонуклеотидной последовательности; и определение среднего коэффициента амплификации для указанной мультиплексной ПЦР-реакции на основе среднего количества копий указанного множества амплифицированных матричных олигонуклеотидов и указанного известного количества указанных матричных олигонуклеотидов.
Способ также включает секвенирование указанного множества амплифицированных перестроенных молекул нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего для определения для каждой уникальной перестроенной молекулы нуклеиновой кислоты, составляющей указанное множество, (i) последовательности перестроенной молекулы нуклеиновой кислоты и (ii) количества вхождений указанной последовательности перестроенной молекулы нуклеиновой кислоты; и определение количества лимфоидных клеток в указанном образце на основе среднего коэффициента амплификации для указанной мультиплексной ПЦР-реакции и указанного количества вхождений указанных перестроенных молекул нуклеиновых кислот.
В других вариантах осуществления способ включает определение числа лимфоидных клеток в указанном образце путем получения суммы количества вхождений каждой из указанных амплифицированных последовательностей перестроенных молекул нуклеиновых кислот и деления указанной суммы на указанный средний коэффициент амплификации. В некоторых вариантах осуществления известное количество составляет одну копию для каждого из указанных матричных олигонуклеотидов. В одном варианте осуществления 100 < a < 500. В другом варианте осуществления 100 < b < 500.
Предложен способ коррекции систематической ошибки амплификации в мультиплексной реакции ПЦР-амплификации для количественного определения перестроенных молекул нуклеиновых кислот, кодирующих один или множество рецепторов адаптивной иммунной системы, в биологическом образце, который содержит перестроенные молекулы нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего, содержащий: (a) приведение указанного образца в контакт с композицией, описанной в настоящем документе, для получения образца с добавленным известным количеством матриц, причем указанные матрицы и указанные перестроенные молекулы нуклеиновых кислот содержат соответственно последовательности V- и J-участков; (b) амплификацию указанного образца с добавленным известным количеством матриц в мультиплексной ПЦР-реакции для получения множества амплифицированных матричных олигонуклеотидов и множества амплифицированных перестроенных молекул нуклеиновых кислот, кодирующих множество рецепторов адаптивной иммунной системы; (c) секвенирование указанного множества амплифицированных матричных олигонуклеотидов для определения для каждого уникального матричного олигонуклеотида, составляющего указанное множество, (i) матричной олигонуклеотидной последовательности и (ii) частоты вхождения указанной матричной олигонуклеотидной последовательности; (d) секвенирование указанного множества амплифицированных перестроенных молекул нуклеиновых кислот, кодирующих один или множество рецепторов адаптивной иммунной системы, для определения для каждой уникальной перестроенной молекулы нуклеиновой кислоты, кодирующей указанное множество рецепторов адаптивной иммунной системы, составляющей указанное множество, (i) последовательности перестроенной молекулы нуклеиновой кислоты и (ii) частоты вхождения указанной последовательности перестроенной молекулы нуклеиновой кислоты; (e) сравнение частоты вхождения указанных матричных олигонуклеотидных последовательностей с ожидаемым распределением, причем указанное ожидаемое распределение основано на заданных молярных соотношениях для указанного множества матричных олигонуклеотидов, составляющих указанную композицию, и причем отклонение указанной частоты появления указанных матричных олигонуклеотидных последовательностей от указанного ожидаемого распределения указывает на неравномерный потенциал амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров для амплификации; (f) создание набора поправочных коэффициентов для набора последовательностей матричных молекул и перестроенных молекул нуклеиновых кислот, амплифицируемых с использованием указанных членов набора олигонуклеотидных праймеров для амплификации, имеющих указанный неравномерный потенциал амплификации нуклеиновых кислот, причем указанный набор поправочных коэффициентов обеспечивает коррекцию систематических ошибок амплификации в указанной мультиплексной ПЦР-реакции; и (g) необязательно применение указанного набора поправочных коэффициентов к указанным частотам вхождения указанных последовательностей перестроенных молекул нуклеиновых кислот для коррекции систематических ошибок амплификации в указанной мультиплексной ПЦР-реакции.
Настоящее изобретение содержит набор, содержащий реагенты, содержащие композицию, содержащую множество матричных олигонуклеотидов и набор олигонуклеотидных праймеров, как описано в настоящем документе; инструкции по определению неравномерного потенциала амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенных молекул нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы в биологическом образце, содержащем перестроенные молекулы нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего.
В другом варианте осуществления набор содержит инструкции для проведения коррекции для одного или более членов набора олигонуклеотидных праймеров, имеющего неравномерный потенциал амплификации нуклеиновых кислот.
В другом варианте осуществления набор содержит инструкции для количественного определения количества уникальных перестроенных молекул нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы, в образце.
Данные и другие аспекты вариантов осуществления, описанных в настоящем документе, станут понятны после изучения следующего подробного описания и приложенных рисунков. Все патенты США, публикации заявок на патенты США, заявки на патенты США, зарубежные патенты, зарубежные заявки на патенты и непатентные публикации, упоминаемые в настоящей спецификации и/или перечисленные в справочном листе заявки, считаются полностью включенными в настоящий документ путем ссылки, как если бы каждую из них включили индивидуально. Аспекты и варианты осуществления настоящего изобретения при необходимости можно модифицировать для включения концепций различных патентов, заявок и публикаций для получения дополнительных вариантов осуществления.
Краткое описание нескольких видов рисунков
Данные и другие признаки, аспекты и преимущества настоящего изобретения станут более понятны в отношении изучения приведенного ниже описания и сопровождающих рисунков.
На Фиг. 1 показана принципиальная схема примера матричного олигонуклеотида для использования при стандартизации эффективности амплификации набора олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенных ДНК, кодирующих рецептор адаптивной иммунной системы (TCR или BCR). U1, U2 - универсальные олигонуклеотиды-адаптеры; B1-4 - олигонуклеотиды штрихкода; V - олигонуклеотид вариабельного участка; J - олигонуклеотид соединительного участка; R - сайт распознавания рестрикционного фермента; S - необязательный стоп-кодон.
На Фиг. 2 показаны постамплификационные частоты последовательностей, кодирующих индивидуальные сегменты TCRB V-гена, полученные при амплификации с использованием стандартизованной композиции олигонуклеотидных матриц (эквимолярный пул матриц, представленных в SEQ ID NO: 872–1560), используя эквимолярный (нескорректированный) пул из 52 праймеров для ПЦР (SEQ ID NO: 1753–1804), и количественном секвенировании на секвенаторе ДНК Illumina HiSeq™. Вычисленные частоты в отсутствие систематической ошибки составляли 0,0188.
На Фиг. 3 показаны результаты количественного секвенирования после перекрестной амплификации матричных олигонуклеотидов с использованием праймеров, специфических для V-участка TCRB. По оси Y указаны индивидуальные праймеры для амплификации (SEQ ID NO: 1753-1804), которые присутствовали в каждой отдельной реакции амплификации в удвоенной молярной концентрации (2X) по отношению к другим праймерам из того же набора праймеров, для амплификации с использованием стандартизованной композиции олигонуклеотидных матриц (эквимолярный пул матриц, представленных в SEQ ID NO: 872–1560); ось X не размечена, но точки данных представлены в том же порядке, что и по оси Y, причем по оси X представлены соответствующие амплифицированные матрицы V-гена по результатам идентификации с использованием количественного секвенирования. Черными квадратами показано отсутствие изменений в степени амплификации соответствующим праймером, присутствующим в двойной (2X) концентрации по отношению к эквимолярным концентрациям всех других праймеров; а белыми квадратами показано 10-кратное усиление амплификации; серыми квадратами показаны промежуточные степени (в градациях серого) амплификации от нуля до 10 крат. Диагональная линия из белых квадратов показывает, что двойная (2X) концентрация заданного праймера приводит к приблизительно 10-кратному усилению амплификации соответствующей матрицы для большинства праймеров. Белые квадраты вне диагонали указывают на несоответствующие матрицы, с которыми определенные праймеры были способны к отжигу и амплификации.
На Фиг. 4 показаны постамплификационные частоты последовательностей индивидуальных сегментов TCRB V-гена, полученные при амплификации с использованием стандартизованной композиции олигонуклеотидных матриц (эквимолярный пул матриц, представленных в SEQ ID NO: 872–1560), используя эквимолярные концентрации всех членов из набора праймеров для амплификации TCRB (SEQ ID NO: 1753–1804) до коррекции систематической ошибки использования праймера (черные полоски, все праймеры для V-участков присутствуют в эквимолярных концентрациях) и используя тот же набор праймеров (SEQ ID NO: 1753–1804) после коррекции концентраций множества индивидуальных праймеров для компенсации систематической ошибки (серые полоски, концентрации высокоэффективных праймеров были снижены, а концентрации низкоэффективных праймеров были повышены, см. таблицу 6). Постамплификационные частоты определяли путем количественного секвенирования на секвенаторе ДНК Illumina HiSeq™.
На Фиг. 5a–5l показаны наборы TCRB V/J (68 V + 13 J) для использования в матричных композициях, которые содержат множество олигонуклеотидных последовательностей общей формулы 5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ [I], для применения при стандартизации эффективности амплификации набора олигонуклеотидных праймеров, который способен проводить амплификацию перестроенных ДНК, кодирующих один или множество полипептидов β-цепи рецептора T-клетки человека (TCRB).
На Фиг. 6a и 6b показаны наборы TCRG V/J (14 V + 5 J) для использования в матричных композициях, которые содержат множество олигонуклеотидных последовательностей общей формулы 5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ [I], для применения при стандартизации эффективности амплификации набора олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенных ДНК, кодирующих один или множество полипептидов γ-цепи рецептора T-клетки человека (TCRG).
На Фиг. 7a–7m показаны наборы IGH V/J (127 V + 9 J) для использования в матричных композициях, которые содержат множество олигонуклеотидных последовательностей общей формулы 5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ [I], для использования при стандартизации эффективности амплификации набора олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенных ДНК, кодирующих один или множество полипептидов тяжелой цепи иммуноглобулинов человека (IGH).
На Фиг. 8 показаны результаты вычисления коэффициента амплификации для каждой пары VJ в матричной композиции, добавляемой в мультиплексную ПЦР для амплификации последовательностей IGH, и последующего усреднения коэффициента амплификации по всем синтетическим матрицам для оценки кратности покрытия последовательностей по всем молекулам синтетических матриц.
На Фиг. 9 показан график числа B-клеток, оцененного с использованием композиции синтетических матриц и коэффициента амплификации, как описано в настоящем документе, в зависимости от известного числа B-клеток, использованных в качестве источника матриц естественных ДНК.
На Фиг. 10 показано число повтора при секвенировании до проведения амплификации с использованием ПЦР для каждой из 1116 молекул для контроля систематической ошибки по IGH VJ и 243 молекул для контроля систематической ошибки по IGH DJ.
На Фиг. 11 показаны итерации TCRB-праймера для синтетических матриц TCRB VJ, представленные в зависимости от относительной систематической ошибки амплификации.
На Фиг. 12 показаны итерации IGH-праймера для синтетических матриц IGH VJ, представленные в зависимости от относительной систематической ошибки амплификации.
На Фиг. 13 показана относительная систематическая ошибка амплификации для 27 синтетических матриц IGH DJ для V-гена.
На Фиг. 14a–d показаны итерации TCRG-праймера для 55 синтетических матриц TCRG VJ. Относительные систематические ошибки амплификации для праймеров TCRG VJ определяли до проведения химической коррекции на контроль систематической ошибки (Фиг. 14a), первой итерации химической коррекции (Фиг. 14b), второй итерации химической коррекции (Фиг. 14c) и итоговой итерации химической коррекции (Фиг. 14d).
Подробное описание настоящего изобретения
В определенных вариантах осуществления настоящего изобретения и как описано в настоящем документе, предложены композиции и способы, которые можно использовать для надежного количественного определения больших и структурно разнообразных популяций перестроенных генов, кодирующих рецепторы адаптивной иммунной системы, такие как иммуноглобулины (Ig) и/или T-клеточные рецепторы (TCR). Данные перестроенные гены могут присутствовать в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта или биологического источника, включая человеческого индивида.
В настоящем документе термин «перестроенная молекула нуклеиновой кислоты» может включать в себя любую молекулу геномной ДНК, кДНК или мРНК, полученную непосредственно или опосредованно из клеток лимфоидной линии, которая включает последовательности, кодирующие перестроенный рецептор адаптивной иммунной системы.
В настоящем документе описаны неожиданно преимущественные подходы для стандартизации и калибровки сложных наборов олигонуклеотидных праймеров, которые используются в мультиплексных реакциях амплификации нуклеиновых кислот для получения популяции амплифицированных перестроенных молекул ДНК из биологического образца, содержащего перестроенные гены, кодирующие рецепторы адаптивной иммунной системы, до количественного высокопроизводительного секвенирования таких амплифицированных продуктов. Мультиплексная амплификация и высокопроизводительное секвенирование перестроенных последовательностей ДНК, кодирующих TCR и BCR (IG), описаны, например, в работах Robins et al., 2009 г., Blood 114, 4099; Robins et al., 2010 г., Sci. Translat. Med. 2:47ra64; Robins et al., 2011 г., J. Immunol. Meth. doi:10.1016/j.jim.2011.09. 001; Sherwood et al. 2011 г., Sci. Translat. Med. 3:90ra61; заявке на патент США № 13/217,126 (публикации США № 2012/0058902), заявке на патент США № 12/794,507 (публикации США № 2010/0330571), международных заявках WO/2010/151416, WO/2011/106738 (PCT/US2011/026373), WO2012/027503 (PCT/US2011/049012), заявке на патент США № 61/550,311 и заявке на патент США № 61/569,118; соответственно, описания данных документов включены путем ссылки и могут быть выполнены с возможностью использования в соответствии с вариантами осуществления, описанными в настоящем документе.
Вкратце и в соответствии с теорией, не имеющей ограничительного характера, настоящие композиции и способы устраняют неточности, которые могут возникать в используемых в настоящее время способах, в которых для количественного определения разнообразия генов TCR и BCR используют секвенирование продуктов мультиплексной амплификации нуклеиновых кислот. Чтобы охватить огромное разнообразие матричных последовательностей генов TCR и BCR, которые могут присутствовать в биологическом образце, используемые в мультиплексных реакциях амплификации наборы олигонуклеотидных праймеров, как правило, содержат широкое разнообразие длин последовательностей и нуклеотидных композиций (например, содержание GC). Следовательно, в заданном наборе условий проведения реакции амплификации эффективность, с которой разные праймеры гибридизируются и поддерживают амплификацию своих распознаваемых матричных последовательностей, могут значительно различаться, приводя к неравномерному использованию разных праймеров, что приводит к искусственным систематическим ошибкам в относительном количественном представлении разных продуктов амплификации.
Например, относительно избыточное использование некоторых высокоэффективных праймеров приводит к завышенному представлению определенных продуктов амплификации, а относительно недостаточное использование некоторых других низкоэффективных праймеров приводит к заниженному представлению других определенных продуктов амплификации. Затем количественное определение относительного количества каждого типа матрицы, присутствующего в образце, содержащем ДНК из лимфоидных клеток, которое достигается путем секвенирования продуктов амплификации, может привести к получению ошибочной информации в отношении фактического относительного представления различных типов матриц в образце до проведения амплификации. Например, в пилотных исследованиях было обнаружено, что проведение мультиплексной ПЦР с использованием набора олигонуклеотидных праймеров, выполненных с возможностью амплификации последовательности каждого возможного гена вариабельного (V) участка TCRB человека из матриц ДНК лимфоидных клеток человека, не приводило к равномерной амплификации сегментов TCRB V-гена. Вместо этого для некоторых сегментов V-генов наблюдалась относительно избыточная амплификация (для приблизительно 10% всех последовательностей), а для других сегментов V-генов наблюдалась относительно недостаточная амплификация (представляющая приблизительно 4 x 10-3% всех последовательностей); см. также, например, Фиг. 2.
Для решения проблемы такой систематической ошибки в использовании субпопуляций праймеров для амплификации в настоящем изобретении впервые предложена матричная композиция и способ стандартизации эффективности амплификации членов набора олигонуклеотидных праймеров, в котором набор праймеров способен проводить амплификацию перестроенной ДНК, кодирующей множество рецепторов адаптивной иммунной системы (TCR или Ig) в биологическом образце, содержащем ДНК из лимфоидных клеток. Матричная композиция содержит множество разнообразных матричных олигонуклеотидов общей формулы (I), более подробно описанной в настоящем документе:
5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ (I).
Составляющие матричные олигонуклеотиды, из которых состоит матричная композиция, обеспечивают разнообразие в отношении нуклеотидных последовательностей индивидуальных матричных олигонуклеотидов. Таким образом, индивидуальные матричные олигонуклеотиды могут различаться по нуклеотидной последовательности в силу значительной вариабельности последовательностей среди большого числа возможных полинуклеотидов вариабельного (V) и соединительного (J) участков TCR или BCR. Последовательности индивидуальных типов матричных олигонуклеотидов также могут отличаться друг от друга в силу различий в последовательностях олигонуклеотидов U1, U2, B (B1, B2, B3 и B4) и R, которые включены в конкретную матрицу в рамках разнообразного множества матриц.
В некоторых вариантах осуществления олигонуклеотиды штрихкода B (B1, B2, B3 и B4) могут независимо и необязательно содержать олигонуклеотидную последовательность штрихкода, причем последовательность штрихкода выбрана для уникальной идентификации конкретной спаренной комбинации конкретной уникальной олигонуклеотидной последовательности V и конкретной уникальной олигонуклеотидной последовательности J. Преимуществом является то, что относительное расположение олигонуклеотидов штрихкода B1 и B4 и универсальных адаптеров позволяет быстро идентифицировать и количественно определять продукты амплификации заданного уникального матричного олигонуклеотида по результатам прочтения коротких последовательностей и по результатам секвенирования со склейкой концов на автоматических секвенаторах ДНК (например, Illumina HiSeq™ или Illumina MiSEQ®, или GeneAnalyzer™-2, Illumina Corp., г. Сан-Диего, штат Калифорния, США). В частности, данные и связанные варианты осуществления позволяют проводить быстрое высокопроизводительное определение конкретных комбинаций последовательностей V и J, присутствующих в продукте амплификации, чтобы посредством этого охарактеризовать относительную эффективность амплификации для каждого специфического для V-участка праймера и каждого специфического для J-участка праймера, которые могут присутствовать в наборе праймеров, способном проводить амплификацию ДНК, кодирующей перестроенные TCR или BCR, в образце. Проверку идентичностей и/или количеств продуктов амплификации можно проводить с использованием прочтения более длинных последовательностей, необязательно включая прочтение последовательностей, доходящих до B2.
При использовании каждый матричный олигонуклеотид во множестве матричных олигонуклеотидов присутствует в по существу эквимолярном количестве, что в определенных предпочтительных вариантах осуществления включает препараты, в которых молярные концентрации всех олигонуклеотидов находятся в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 процентов относительно друг друга. В других определенных предпочтительных вариантах осуществления, предложенных в настоящем документе, матричные олигонуклеотиды считаются присутствующими в по существу эквимолярных количествах, когда молярные концентрации всех олигонуклеотидов находятся в пределах одного порядка друг от друга, включая препараты, в которых наибольшая молярная концентрация, которую может иметь любой заданный уникальный тип матричного олигонуклеотида, не более чем на 1000, 900, 800, 700, 600, 500, 440, 400, 350, 300, 250, 200, 150, 100, 90, 80, 70, 60, 50, 40 или 30 процентов превышает молярную концентрацию, в которой присутствует уникальный тип матричного олигонуклеотида, имеющего самую низкую концентрацию в композиции.
Аналогичным образом в определенных вариантах осуществления, описанных в настоящем документе, предусмотрены наборы олигонуклеотидных праймеров для амплификации, в которых праймеры-компоненты могут быть предусмотрены в по существу эквимолярных количествах. Как также описано в настоящем документе, в соответствии с другими определенными вариантами осуществления концентрацию одного или более праймеров в наборе праймеров можно целенаправленно скорректировать таким образом, что определенные праймеры не будут присутствовать в эквимолярных количествах или в по существу эквимолярных количествах.
Описанную в настоящем документе матричную композицию в предпочтительных вариантах осуществления можно применять в качестве матрицы для амплификации нуклеиновых кислот (например, с использованием ПЦР) для характеризации набора олигонуклеотидных праймеров, такого как сложные наборы олигонуклеотидных праймеров для V-сегмента и J-сегмента, которые можно использовать для мультиплексной амплификации перестроенных генов TCR или Ig, например, набора праймеров, предложенного в настоящем документе, или любого из наборов праймеров, описанных в публикациях Robins et al., 2009 г., Blood 114, 4099; Robins et al., 2010 г., Sci. Translat. Med. 2:47ra64; Robins et al., 2011 г., J. Immunol. Meth. doi:10.1016/j.jim.2011.09. 001; Sherwood et al. 2011 г., Sci. Translat. Med. 3:90ra61; заявке на патент США № 13/217,126 (публикации США № 2012/0058902), заявке на патент США № 12/794,507 (публикации США № 2010/0330571), международных заявках WO/2010/151416, WO/2011/106738 (PCT/US2011/026373), WO2012/027503 (PCT/US2011/049012), заявке на патент США № 61/550,311 и заявке на патент США № 61/569,118 или т. п.
Предпочтительно все матрицы в матричной композиции для стандартизации эффективности амплификации, которая описана в настоящем документе и которая содержит множество матричных олигонуклеотидов, имеющих разнообразные последовательности и общую структуру общей формулы (I), представляют собой олигонуклеотиды по существу идентичной длины. Без стремления к ограничению какой-либо теорией, по существу считается, что в реакции амплификации нуклеиновых кислот, такой как полимеразная цепная реакция (ПЦР), длина ДНК матрицы может влиять на эффективность амплификации олигонуклеотидных праймеров путем воздействия на кинетику взаимодействий между праймерами и молекулами ДНК матрицы, с которыми праймеры гибридизируются с помощью специфического определяемого нуклеотидной последовательностью механизма гибридизации на основе комплементарности нуклеотидных оснований. Считается, что более длинные матрицы по существу работают менее эффективно, чем относительно более короткие матрицы. В определенных вариантах осуществления описанные в настоящем документе матричные композиции для стандартизации эффективности амплификации набора олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенных ДНК, кодирующих множество TCR или BCR, содержит множество матричных олигонуклеотидов общей формулы (I), как представлено в настоящем документе, причем матричные олигонуклеотиды имеют идентичную длину или по существу идентичную длину, которая не превышает 1000, 950, 900, 850, 800, 750, 700, 650, 600, 550, 500, 450, 400, 350, 300, 250, 200, 150 или 100 нуклеотидов, включая все промежуточные целочисленные значения.
Соответственно, для снижения, устранения или сведения к минимуму потенциального вклада в нежелательные систематические ошибки при использовании олигонуклеотидных праймеров в процессе мультиплексной амплификации в описанных в настоящем документе предпочтительных вариантах осуществления может применяться множество матричных олигонуклеотидов, причем все матричные олигонуклеотиды во множестве матричных олигонуклеотидов, включающем разнообразие последовательностей, имеют по существу идентичную длину. Множество матричных олигонуклеотидов может иметь по существу идентичную длину, если все (например, 100%) или большая часть (например, более 50%) таких олигонуклеотидов в матричной композиции представляют собой олигонуклеотиды, каждый из которых имеет точно такое же число нуклеотидов, или если один или более матричных нуклеотидов в матричной композиции могут различаться по длине не более чем на 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 60, 70, 80, 90 или 100 нуклеотидов. После изучения настоящего описания должно быть понятно, что даже в ситуациях, когда не все матричные олигонуклеотиды имеют точно такую же длину, описанные в настоящем документе композиции и способы все же можно применять для определения и необязательно для коррекции неравномерного потенциала амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров для амплификации.
В соответствии с определенными описанными в настоящем документе вариантами осуществления (i) каждый матричный олигонуклеотид в описанной в настоящем документе матричной композиции присутствует в по существу эквимолярном количестве, (ii) набор олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенной ДНК, кодирующей множество рецепторов адаптивной иммунной системы, содержит множество олигонуклеотидных праймеров для V-участка, которые присутствуют в по существу эквимолярных количествах, (iii) набор олигонуклеотидных праймеров, которые способны проводить амплификацию перестроенной ДНК, кодирующей множество рецепторов адаптивной иммунной системы, содержит множество олигонуклеотидных праймеров для J-участка, которые присутствуют в по существу эквимолярных количествах, и (iv) амплификация масштабируется линейно по количеству исходных матриц заданной последовательности.
Таким образом, можно рассчитать ожидаемый выход продуктов амплификации для каждой матрицы и условно назначить ему теоретическое значение уровня равномерной амплификации 100%. После обеспечения амплификации набором праймеров последовательностей матричных олигонуклеотидов в реакции амплификации любое статистически значимое отклонение от значительной эквивалентности, которое наблюдается среди относительных пропорций различных продуктов амплификации, указывает на наличие систематической ошибки (т. е. неравной эффективности) в использовании праймера в процессе амплификации. Иными словами, количественные различия в относительных количествах разных полученных продуктов амплификации указывают на то, что не все праймеры в наборе праймеров провели амплификацию своих соответствующих матриц со сравнимой эффективностью. В определенных вариантах осуществления предусмотрено присвоение диапазона допусков выше и ниже теоретического 100%-ного выхода, так что любое значение уровня амплификации в пределах диапазона допусков можно считать по существу эквивалентностью.
В таких определенных вариантах осуществления диапазон выходов продуктов амплификации можно считать по существу эквивалентным, когда все выходы продуктов находятся в пределах одного порядка величины (например, различаются менее чем в десять раз). В других таких определенных вариантах осуществления диапазон выходов продуктов амплификации можно считать по существу эквивалентным, когда выходы продуктов отличаются друг от друга не более чем в девять раз, восемь раз, семь раз, шесть раз, пять раз, четыре раза или три раза. В других определенных вариантах осуществления выходы продуктов, которые можно считать находящимися в пределах приемлемого диапазона допусков, могут быть больше или меньше рассчитанного 100%-ного выхода на величину вплоть до 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 30, 40, 50, 60, 100 или 200%.
Поскольку способ включает определение нуклеотидной последовательности каждого продукта амплификации с использованием известных техник как части процесса количественного определения, можно идентифицировать праймер (-ы), ответственный (-е) за амплификацию каждого уникального (как определено последовательностью) продукта, и соответственно скорректировать их относительное (-ые) количество (-а) в наборе праймеров (например, повысить или понизить статистически значимым образом). Концентрации избыточно эффективных праймеров в наборе праймеров можно снизить по отношению к концентрациям других праймеров таким образом, чтобы уровень специфической амплификации такими праймерами матриц в описанной в настоящем документе матричной композиции был по существу эквивалентен уровню амплификации, обеспечиваемому большинством праймеров, которые обеспечивают теоретически равномерный уровень амплификации или которые обеспечивают уровень амплификации в пределах приемлемого диапазона допусков. Концентрации низкоэффективных праймеров в наборе праймеров можно повысить по отношению к концентрациям других праймеров таким образом, чтобы уровень специфической амплификации такими праймерами матриц в описанной в настоящем документе матричной композиции был по существу эквивалентен уровню амплификации, обеспечиваемому большинством праймеров, которые обеспечивают теоретически равномерный уровень амплификации или которые обеспечивают уровень амплификации в пределах приемлемого диапазона допусков.
Таким образом и как описано в настоящем документе, в настоящем документе предложена матричная композиция для стандартизации эффективности амплификации набора олигонуклеотидных праймеров, который выполнен с возможностью амплификации кодирующих последовательностей для полного репертуара заданной цепи TCR или Ig, способ определения неравномерной эффективности амплификации («неравномерного потенциала амплификации») среди членов такого набора праймеров и способ коррекции такого неравномерного потенциала амплификации. Используя описанную в настоящем документе матричную композицию в качестве стандарта, с помощью которого можно осуществлять калибровку наборов олигонуклеотидных праймеров, и в конкретных вариантах осуществления, в которых каждый матричный олигонуклеотид присутствует в по существу эквимолярном количестве, так что концентрации индивидуальных праймеров можно скорректировать с получением по существу равномерной амплификации структурно разнообразного типа продуктов амплификации, настоящее описание таким образом эффективно преодолевает описанные выше проблемы, связанные с систематической ошибкой в эффективности индивидуальных праймеров.
Используя представленные в настоящем документе композиции и способы, можно идентифицировать индивидуальные праймеры как имеющие неравномерный потенциал амплификации на основе стимуляции ими неравномерной амплификации, которая проявляется как повышенная (например, статистически значимо большая) или пониженная (например, статистически значимо меньшая) амплификация конкретных матричных олигонуклеотидов относительно равномерного уровня амплификации, несмотря на наличие в реакции амплификации (i) всех матричных олигонуклеотидов в по существу эквимолярных количествах по отношению друг к другу, (ii) всех праймеров для V-сегмента в по существу эквимолярных количествах по отношению друг к другу и (iii) всех праймеров для J-сегмента в по существу эквимолярных количествах по отношению друг к другу.
Затем относительные концентрации таких праймеров можно понизить или повысить для получения полного модифицированного набора праймеров, в котором все праймеры не присутствуют в по существу эквимолярных количествах по отношению друг к другу для компенсации соответственно повышенного или пониженного уровня амплификации относительно равномерного уровня амплификации. Затем набор праймеров можно повторно протестировать на его способность проводить амплификацию всех последовательностей в описанной в настоящем документе матричной композиции на равномерном уровне амплификации или в пределах допустимого диапазона допусков.
Процесс тестирования модифицированных наборов праймеров на их способность проводить амплификацию описанной в настоящем документе матричной композиции, в которой все матричные олигонуклеотиды присутствуют в по существу эквимолярных количествах по отношению друг к другу, можно повторять итерационно до тех пор, пока все продукты не будут амплифицироваться на равномерном уровне амплификации или в пределах допустимого диапазона допусков. Такой процесс с использованием описанной в настоящем документе матричной композиции позволяет стандартизовать эффективность амплификации набора олигонуклеотидных праймеров, причем набор праймеров способен проводить амплификацию продуктивно перестроенной ДНК, кодирующей один или множество рецепторов адаптивной иммунной системы в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта.
Дополнительно или альтернативно в соответствии с настоящим описанием можно определить, демонстрирует ли любая конкретная пара олигонуклеотидных праймеров для амплификации неравномерный потенциал амплификации, такой как повышенная или пониженная амплификация матричной композиции, по отношению к равномерному уровню амплификации, демонстрируемому большинством олигонуклеотидных праймеров для амплификации, и затем можно использовать нормирующий поправочный коэффициент для вычисления соответственно пропорционально пониженной или повышенной частоты вхождения продуктов амплификации, которая обеспечивается каждой такой парой праймеров для амплификации. Таким образом, матричные композиции настоящего описания в определенных вариантах осуществления обеспечивают способ коррекции неравномерности потенциала амплификации нуклеиновых кислот среди членов набора олигонуклеотидных праймеров для амплификации.
Преимуществом является то, что такие определенные варианты осуществления могут допускать проведение коррекции, калибровки, стандартизации, нормализации и т. п. данных, полученных вследствие событий неравномерной амплификации. Таким образом, варианты осуществления настоящего изобретения позволяют выполнять коррекцию неточностей данных, таких как возможные следствия использования олигонуклеотидных праймеров с систематической ошибкой без необходимости итерационной коррекции концентраций одного или более праймеров для амплификации и повтора стадий амплификации описанных в настоящем документе матричных композиций. Таким образом можно получить преимущества в отношении эффективности, поскольку можно избежать повтора стадий количественного секвенирования продуктов амплификации. Однако другие определенные рассматриваемые варианты осуществления могут применять такой итерационный подход.
Соответственно, а также как описано в настоящем документе, в настоящем документе предложена матричная композиция для стандартизации эффективности амплификации набора олигонуклеотидных праймеров вместе со способами использования такой матричной композиции для определения неравномерного потенциала амплификации нуклеиновых кислот (например, систематической ошибки) среди индивидуальных членов набора олигонуклеотидных праймеров. Также в настоящем документе описаны способы коррекции таких неравномерных потенциалов амплификации нуклеиновых кислот (например, систематических ошибок) среди членов набора олигонуклеотидных праймеров. В данных и связанных вариантах осуществления используется ранее незамеченное преимущество, достигаемое путем калибровки сложных наборов олигонуклеотидных праймеров для компенсации нежелательных систематических ошибок амплификации, используя матричную композицию для стандартизации эффективности амплификации, имеющую описанные в настоящем документе признаки, и которое может использоваться для повышения точности количественного определения конкретных клонотипических последовательностей ДНК, кодирующих TCR и/или Ig, по сравнению с ранее описанными методологиями.
Как также указано выше и описано в других разделах настоящего документа, до настоящего описания существовали неудовлетворительные и трудноразличимые несоответствия между (i) фактическим количественным распределением матричной ДНК, кодирующей перестроенные рецепторы адаптивной иммунной системы, имеющие уникальные последовательности в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта, и (ii) относительным представлением продуктов амплификации нуклеиновых кислот для таких матриц по результатам мультиплексной амплификации с использованием сложного набора олигонуклеотидных праймеров для амплификации с возможностью амплификации по существу всех продуктивно перестроенных генов рецепторов адаптивной иммунной системы в образце. Например, вследствие гетерогенности как популяции матриц, так и набора праймеров для амплификации, а также как показано в настоящем документе, могут быть распространены значительные различия в эффективности амплификации для различных праймеров для амплификации, что приводит к значительному искажению относительных пропорций продуктов амплификации, полученных и количественно секвенированных по результатам проведения реакции амплификации.
Матрицы и праймеры
Таким образом, в соответствии с определенными предпочтительными вариантами осуществления предложена матричная композиция для стандартизации эффективности амплификации набора олигонуклеотидных праймеров, который способен проводить амплификацию перестроенных молекул ДНК (что в определенных вариантах осуществления может относиться к продуктивно перестроенным ДНК, но в других определенных вариантах осуществления не должно быть этим ограничено), кодирующих один или множество рецепторов адаптивной иммунной системы в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта, причем матричная композиция содержит множество матричных олигонуклеотидов общей формулы (I):
5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ (I),
как представлено в настоящем документе. В некоторых предпочтительных вариантах осуществления каждый матричный олигонуклеотид во множестве матричных олигонуклеотидов присутствует в по существу эквимолярном количестве, что в определенных вариантах осуществления, а также как отмечено выше, может относиться к композиции, в которой каждый из матричных олигонуклеотидов присутствует в эквимолярной концентрации или в молярной концентрации, которая отличается от эквимолярной не более чем на 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 50, 60, 70, 80, 90, 100 или 200% в пересчете на количество молей, и что в других определенных вариантах осуществления может относиться к композиции, в которой все матричные олигонуклеотиды присутствуют в молярных концентрациях, которые находятся в пределах одного порядка величины друг от друга. Множество матриц может содержать по меньшей мере 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1100 или более отдельных типов олигонуклеотидов, каждый из которых имеет отдельную нуклеотидную последовательность, включая все промежуточные целочисленные значения.
Таким образом, описанная в настоящем документе матричная композиция содержит множество матричных олигонуклеотидов общей формулы:
5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ [I],
причем вкратце и как более подробно описано в других разделах настоящего документа, в соответствии с некоторыми предпочтительными вариантами осуществления:
V представляет собой полинуклеотид, содержащий по меньшей мере 20, 30, 60, 90, 120, 150, 180 или 210 и не более 1000, 900, 800, 700, 600 или 500 последовательных нуклеотидов генной последовательности, кодирующей вариабельный (V) участок рецептора адаптивной иммунной системы или его комплемент, и в каждой из множества последовательностей матричных олигонуклеотидов V содержит уникальную олигонуклеотидную последовательность;
J представляет собой полинуклеотид, содержащий по меньшей мере 15–30, 31–60, 61–90, 91–120 или 120–150 и не более 600, 500, 400, 300 или 200 последовательных нуклеотидов генной последовательности, кодирующей соединительный (J) участок рецептора адаптивной иммунной системы или его комплемент, и в каждой из множества последовательностей матричных олигонуклеотидов J содержит уникальную олигонуклеотидную последовательность;
каждый из U1 и U2 либо отсутствует, либо содержит олигонуклеотид, независимо имеющий последовательность, выбранную из (i) универсальной последовательности олигонуклеотида-адаптера и (ii) специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5’ и находится в нем относительно универсальной последовательности олигонуклеотида-адаптера;
каждый из B1, B2, B3 и B4 независимо либо отсутствует, либо содержит олигонуклеотид B, который содержит олигонуклеотидную последовательность штрихкода из 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 последовательных нуклеотидов (включая все промежуточные целочисленные значения), причем в каждой из множества последовательностей матричных олигонуклеотидов B содержит уникальную олигонуклеотидную последовательность, которая уникально идентифицирует или идентифицирует в качестве спаренной комбинации (i) уникальную олигонуклеотидную последовательность V матричного олигонуклеотида и (ii) уникальную олигонуклеотидную последовательность J матричного олигонуклеотида; и
R либо отсутствует, либо содержит сайт распознавания рестрикционного фермента, который содержит олигонуклеотидную последовательность, отсутствующую в V, J, U1, U2, B1, B2, B3 и B4.
В некоторых вариантах осуществления композиция матричных олигонуклеотидов содержит дополнительные некодирующие или случайные олигонуклеотиды. Данные олигонуклеотиды могут быть вставлены в различные секции между или в компонентах общей формулы I (5’-U1-B1-V-B2-R-B3-J-B4-U2-3’) и могут иметь различные длины.
В одном варианте осуществления a составляет от 1 до максимального числа сегментов V-гена в геноме субъекта-млекопитающего. В другом варианте осуществления b составляет от 1 до максимального числа сегментов J-гена в геноме субъекта-млекопитающего. В других вариантах осуществления a равно 1 или b равно 1. В некоторых вариантах осуществления a может находиться в диапазоне от 1 сегмента V-гена до 54 сегментов V-гена для TCRA, 1–76 сегментов V-гена для TCRB, 1–15 сегментов V-гена для TCRG, 1–7 сегментов V-гена для TCRD, 1–165 сегментов V-гена для IGH, 1–111 сегментов V-гена для IGK или 1–79 сегментов V-гена для IGL. В вариантах осуществления b может находиться в диапазоне от 1 сегмента J-гена до 61 сегмента J-гена для TCRA, 1–14 сегментов J-гена для TCRB, 1–5 сегментов J-гена для TCRG, 1–4 сегментов J-гена для TCRD, 1–9 сегментов J-гена для IGH, 1–5 сегментов J-гена для IGK или 1–11 сегментов J-гена для IGL.
В приведенной ниже таблице перечислено количество сегментов V-гена (a) и сегментов J-гена (b) для каждого локуса рецептора адаптивной иммунной системы человека, включая функциональные V- и J-сегменты.
* Полное количество генов вариабельных и соединительных сегментов.
** Количество генов вариабельных и соединительных сегментов с по меньшей мере одним функциональным аллелем.
В некоторых вариантах осуществления J-полинуклеотид содержит по меньшей мере 15–30, 31–60, 61–90, 91–120 или 120–150 и не более 600, 500, 400, 300 или 200 последовательных нуклеотидов из константного J-участка рецептора адаптивной иммунной системы или комплементарный ему полинуклеотид.
В определенных вариантах осуществления множество матричных олигонуклеотидов содержит по меньшей мере (a x b) уникальных олигонуклеотидных последовательностей, где a представляет собой количество уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой количество уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта, и композиция содержит по меньшей мере один матричный олигонуклеотид для каждой возможной комбинации сегмента гена, кодирующего V-участок, и сегмента гена, кодирующего J-участок.
Однако рассмотренное в настоящем документе изобретение не предусматривает такого ограничения, и в определенных вариантах осуществления может преимущественно использоваться по существу меньшее число матричных олигонуклеотидов. В данных и связанных вариантах осуществления если a представляет собой число уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой число уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта, минимальное число уникальных олигонуклеотидных последовательностей, из которых состоит множество матричных олигонуклеотидов, может определяться большим значением из a и b, при условии что каждая уникальная последовательность V-полинуклеотида и каждая уникальная последовательность J-полинуклеотида присутствует в по меньшей мере одном матричном олигонуклеотиде в матричной композиции. Таким образом, в соответствии с определенными связанными вариантами осуществления матричная композиция может содержать по меньшей мере один матричный олигонуклеотид для каждого уникального V-полинуклеотида, например, который включает один из каждого уникального V-полинуклеотида в соответствии с общей формулой (I), и по меньшей мере один матричный олигонуклеотид для каждого уникального J-полинуклеотида, например, который включает один из каждого уникального J-полинуклеотида в соответствии с общей формулой (I).
В других определенных вариантах осуществления матричная композиция содержит по меньшей мере один матричный олигонуклеотид, с которым может гибридизироваться каждый олигонуклеотидный праймер для амплификации из набора праймеров для амплификации.
То есть в некоторых вариантах осуществления матричная композиция содержит по меньшей мере один матричный олигонуклеотид, имеющий олигонуклеотидную последовательность общей формулы (I), с которым может специфически гибридизироваться каждый олигонуклеотидный праймер V-сегмента, и по меньшей мере один матричный олигонуклеотид, имеющий олигонуклеотидную последовательность общей формулы (I), с которым может специфически гибридизироваться каждый олигонуклеотидный праймер J-сегмента.
В соответствии с такими вариантами осуществления набор олигонуклеотидных праймеров, который способен проводить амплификацию перестроенной ДНК, кодирующей один или множество рецепторов адаптивной иммунной системы, содержит множество a’ уникальных олигонуклеотидных праймеров V-сегмента и множество b’ уникальных олигонуклеотидных праймеров J-сегмента. Множество a’ уникальных олигонуклеотидных праймеров V-сегмента, в котором каждый способен независимо гибридизироваться или специфически гибридизироваться с по меньшей мере одним полинуклеотидом, кодирующим полипептид V-участка рецептора адаптивной иммунной системы, или его комплементом, причем каждый праймер V-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному сегменту гена, кодирующего V-участок рецептора адаптивной иммунной системы. Множество b’ олигонуклеотидных праймеров J-сегмента, каждый из которых способен независимо гибридизироваться или специфически гибридизироваться с по меньшей мере одним полинуклеотидом, кодирующим полипептид J-участка рецептора адаптивной иммунной системы, или его комплементом, причем каждый праймер J-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному сегменту гена, кодирующего J-участок рецептора адаптивной иммунной системы.
В некоторых вариантах осуществления a’ равно a (описано выше для матричных олигонуклеотидов). В других вариантах осуществления b’ равно b (описано выше для матричных олигонуклеотидов).
Таким образом, в определенных вариантах осуществления, а также как описано в других разделах настоящего документа, настоящую матричную композицию можно использовать в реакциях амплификации с праймерами для амплификации, которые выполнены с возможностью амплификации всех перестроенных генных последовательностей, кодирующих рецепторы адаптивной иммунной системы, включая неэкспрессируемые, тогда как в других определенных вариантах осуществления матричные композиции и праймеры для амплификации могут быть выполнены таким образом, чтобы не давать продуктов амплификации перестроенных генов, которые не экспрессируются (например, псевдогены, орфоны). Следовательно, следует понимать, что в определенных вариантах осуществления может быть желательна амплификация только подмножества генов, кодирующих перестроенные рецепторы адаптивной иммунной системы, так что можно создать и применять подмножества праймеров для амплификации для проведения амплификации только интересующих перестроенных последовательностей V-J. Соответственно, в данных и связанных вариантах осуществления можно использовать описанную в настоящем документе матричную композицию, содержащую только интересующее подмножество перестроенных последовательностей V-J, при условии что матричная композиция содержит по меньшей мере один матричный олигонуклеотид, с которым может гибридизироваться каждый олигонуклеотидный праймер для амплификации из набора праймеров для амплификации. Таким образом, фактическое число матричных олигонуклеотидов в матричной композиции может значительно отличаться в зависимости от предполагаемого для использования набора праймеров для амплификации.
Например, в определенных связанных вариантах осуществления множество матричных олигонуклеотидов в матричной композиции может иметь множество последовательностей общей формулы (I), выбранных из (1) множества олигонуклеотидных последовательностей общей формулы (I), в которых V- и J-полинуклеотиды имеют последовательности TCRB V и J, представленные в по меньшей мере одном наборе из 68 TCRB V и J SEQ ID NO, соответственно, что представлено на Фиг. 5a–5l как TCRB V/J набор 1, TCRB V/J набор 2, TCRB V/J набор 3, TCRB V/J набор 4, TCRB V/J набор 5, TCRB V/J набор 6, TCRB V/J набор 7, TCRB V/J набор 8, TCRB V/J набор 9, TCRB V/J набор 10, TCRB V/J набор 11, TCRB V/J набор 12 и TCRB V/J набор 13; (2) множества олигонуклеотидных последовательностей общей формулы (I), в которых V- и J-полинуклеотиды имеют последовательности TCRG V и J, представленные в по меньшей мере одном наборе из 14 TCRG V и J SEQ ID NO, соответственно, что представлено на Фиг. 6 как TCRG V/J набор 1, TCRG V/J набор 2, TCRG V/J набор 3, TCRG V/J набор 4 и TCRG V/J набор 5; и (3) множества олигонуклеотидных последовательностей общей формулы (I), в которых V- и J-полинуклеотиды имеют последовательности IGH V и J, представленные в по меньшей мере одном наборе из 127 IGH V и J SEQ ID NO, соответственно, что представлено на Фиг. 7 как IGH V/J набор 1, IGH V/J набор 2, IGH V/J набор 3, IGH V/J набор 4, IGH V/J набор 5, IGH V/J набор 6, IGH V/J набор 7, IGH V/J набор 8 и IGH V/J набор 9.
В определенных вариантах осуществления V представляет собой полинуклеотидную последовательность, которая кодирует по меньшей мере 10–70 последовательных аминокислот V-участка рецептора адаптивной иммунной системы, или ее комплемент; J представляет собой полинуклеотидную последовательность, которая кодирует по меньшей мере 5–30 последовательных аминокислот J-участка рецептора адаптивной иммунной системы, или ее комплемент; каждый из U1 и U2 либо отсутствует, либо содержит олигонуклеотид, содержащий нуклеотидную последовательность, выбранную из (i) универсальной последовательности олигонуклеотида-адаптера и (ii) специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5’ и находится в нем относительно универсальной последовательности олигонуклеотида-адаптера; каждый из B1, B2, B3 и B4 независимо либо отсутствует, либо представляет собой олигонуклеотид B, который содержит олигонуклеотидную последовательность штрихкода из 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 последовательных нуклеотидов, причем в каждой из множества олигонуклеотидных последовательностей B содержит уникальную олигонуклеотидную последовательность, которая уникально идентифицирует в качестве спаренной комбинации (i) уникальную олигонуклеотидную последовательность V и (ii) уникальную олигонуклеотидную последовательность J; и R либо отсутствует, либо содержит сайт распознавания рестрикционного фермента, который содержит олигонуклеотидную последовательность, отсутствующую в V, J, U1, U2, B1, B2, B3 и B4. В определенных предпочтительных вариантах осуществления множество матричных олигонуклеотидов содержит по меньшей мере либо a, либо b уникальных олигонуклеотидных последовательностей, где a представляет собой количество уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой количество уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта, и композиция содержит множество матричных олигонуклеотидов, которые содержат по меньшей мере количество, равное большему из a и b, уникальных матричных олигонуклеотидных последовательностей, при условии что в данное множество входит по меньшей мере один V-полинуклеотид, соответствующий каждому сегменту гена, кодирующего V-участок, и по меньшей мере один J-полинуклеотид, соответствующий каждому сегменту гена, кодирующего J-участок.
Известно большое количество генных последовательностей вариабельного (V) участка и соединительного (J) участка рецепторов адаптивной иммунной системы в виде нуклеотидных и/или аминокислотных последовательностей, включая неперестроенные последовательности геномной ДНК локусов TCR и Ig и продуктивно перестроенные последовательности ДНК в таких локусах и последовательности кодируемых ими продуктов, а также включая псевдогены в данных локусах и включая связанные орфоны. См., например, заявку на патент США № 13/217,126; заявку на патент США № 12/794,507; PCT/US2011/026373; PCT/US2011/049012. Данные и другие последовательности, известные в данной области, можно использовать в соответствии с настоящим описанием для создания и производства матричных олигонуклеотидов для включения в описанную в настоящем документе матричную композицию для стандартизации эффективности амплификации набора олигонуклеотидных праймеров, а также для создания и производства набора олигонуклеотидных праймеров, который способен проводить амплификацию перестроенных молекул ДНК, кодирующих полипептидные цепи TCR или Ig, причем перестроенные ДНК могут присутствовать в биологическом образце, содержащем ДНК лимфоидных клеток.
В формуле (I) V представляет собой полинуклеотидную последовательность из по меньшей мере 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 210, 220, 230, 240, 250, 260, 270, 280, 290, 300, 310, 320, 330, 340, 350, 360, 370, 380, 390, 400 или 450 и не более чем 1000, 900, 800, 700, 600 или 500 последовательных нуклеотидов генной последовательности вариабельного (V) участка рецептора адаптивной иммунной системы (например, TCR или BCR) или комплементарного ему полинуклеотида, и в каждой из множества олигонуклеотидных последовательностей V содержит уникальную олигонуклеотидную последовательность. Геномные последовательности для генов V-участка TCR и BCR человека и других видов известны и доступны в публичных базах данных, таких как Genbank; генные последовательности V-участка включают в себя полинуклеотидные последовательности, которые кодируют продукты экспрессируемых генов перестроенных TCR и BCR, а также включают в себя полинуклеотидные последовательности псевдогенов, идентифицированных в локусах V-участка. Разнообразные последовательности V-полинуклеотида, которые можно встроить в описанные в настоящем документе матрицы общей формулы (I), могут различаться в широких пределах по длине, нуклеотидной композиции (например, содержанию GC) и фактической линейной полинуклеотидной последовательности, а также, как известно, включают в себя «горячие точки», или гипервариабельные участки, которые показывают особое разнообразие последовательности.
Полинуклеотид V в общей формуле (I) (или комплементарный ему полинуклеотид) включает последовательности, с которыми могут специфически гибридизироваться члены наборов олигонуклеотидных праймеров, специфических для генов TCR или BCR. Наборы праймеров, которые способны проводить амплификацию перестроенных ДНК, кодирующих множество TCR или BCR, описаны, например, в заявке на патент США № 13/217,126; заявке на патент США № 12/794,507; документах PCT/US2011/026373; или PCT/US2011/049012; или т. п.; или, как описано в настоящем документе, они могут быть выполнены с возможностью включения олигонуклеотидных последовательностей, которые могут специфически гибридизироваться с каждым уникальным V-геном и каждым J-геном в конкретном локусе гена TCR или BCR (например, TCR α, β, γ или δ, или IgH μ, γ, δ, α или ε, или IgL κ или λ). Например, в качестве иллюстрации и не предполагая ограничительного характера, олигонуклеотидный праймер из набора олигонуклеотидных праймеров для амплификации, который способен проводить амплификацию перестроенной ДНК, кодирующей один или множество TCR или BCR, как правило, включает в себя нуклеотидную последовательности из 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 последовательных нуклеотидов или более и может специфически гибридизироваться с комплементарной последовательностью из 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 последовательных нуклеотидов V- или J-полинуклеотида, как представлено в настоящем документе. В определенных вариантах осуществления праймеры могут содержать по меньшей мере 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, и в определенных вариантах осуществления праймеры могут содержать последовательности из не более чем 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 последовательных нуклеотидов. Также явным образом предусмотрены праймеры и сайты отжига с праймерами другой длины, как описано в настоящем документе.
Вся полинуклеотидная последовательность каждого V-полинуклеотида в общей формуле (I) может, но не должна, состоять исключительно из последовательных нуклеотидов из каждого отдельного V-гена. Например, а также в соответствии с определенными вариантами осуществления, в описанной в настоящем документе матричной композиции каждый V-полинуклеотид формулы (I) должен иметь лишь по меньшей мере участок, содержащий уникальную олигонуклеотидную последовательность V, присутствующую в одном V-гене, с которой может специфически гибридизироваться один праймер V-участка из набора праймеров. Таким образом, V-полинуклеотид формулы (I) может содержать всю или любую заданную часть (например, по меньшей мере 15, 20, 30, 60, 90, 120, 150, 180 или 210 последовательных нуклеотидов или любое промежуточное целочисленное значение) встречающейся в естественных условиях последовательности V-гена (включая последовательность V-псевдогена) такой длины, чтобы в ней находился по меньшей мере один уникальный участок олигонуклеотидной последовательности V (сайт отжига праймера), который не включен ни в один другой матричный V-полинуклеотид.
В определенных вариантах осуществления может быть предпочтительно, чтобы множество V-полинуклеотидов, которые присутствуют в описанной в настоящем документе матричной композиции, имели длины, моделирующие полные длины известных встречающихся в естественных условиях нуклеотидных последовательностей V-генов, даже если конкретные нуклеотидные последовательности матричного V-участка и встречающегося в естественных условиях V-гена различаются. Длины V-участка в описанных в настоящем документе матрицах могут отличаться от длин встречающихся в естественных условиях последовательностей V-гена не более чем на 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 процентов.
Таким образом, V-полинуклеотид формулы (I) в определенных вариантах осуществления может содержать нуклеотидную последовательность, имеющую длину, такую же или аналогичную длине типичного V-гена от его старт-кодона до участка, кодирующего CDR3, и может, но не должна, включать в себя нуклеотидную последовательность, кодирующую участок CDR3. Нуклеотидные последовательности и длины таких последовательностей, кодирующих CDR3, могут значительно различаться, а для их характеризации используют несколько разных схем нумерации (например, Lefranc, 1999 г., The Immunologist 7:132; Kabat et al., 1991 г., In: Sequences of Proteins of Immunological Interest, NIH Publication 91–3242; Chothia et al., 1987 г., J. Mol. Biol. 196:901; Chothia et al., 1989 г., Nature 342:877; Al-Lazikani et al., 1997 г., J. Mol. Biol. 273:927; см. также, например, Rock et al., 1994 г., J. Exp. Med. 179:323; Saada et al., 2007 г., Immunol. Cell Biol. 85:323).
Вкратце, участок CDR3, как правило, охватывает часть полипептида, направленную от высококонсервативного остатка цистеина (кодируемого тринуклеотидным кодоном TGY; Y = T или C) в V-сегменте к высококонсервативному остатку фенилаланина (кодируемого кодоном TTY) в J-сегменте TCR или к высококонсервативному триптофану (кодируемого кодоном TGG) в IGH. Более 90% естественных продуктивных перестроек в локусе TCRB имеют определяемую по данному критерию длину участка, кодирующего CDR3, в диапазоне от 24 до 54 нуклеотидов, чему соответствуют от 9 до 17 закодированных аминокислот. Для любого заданного локуса TCR или BCR длины CDR3 описанных в настоящем документе синтетических матричных олигонуклеотидов должны находиться в том же диапазоне, что и 95% встречающихся в естественных условиях перестроек. Таким образом, например, в описанной в настоящем документе матричной композиции для стандартизации эффективности амплификации набора олигонуклеотидных праймеров, который способен проводить амплификацию перестроенных молекул ДНК, кодирующих множество полипептидов TCRB, длина кодирующей CDR3 части V-полинуклеотида может иметь длину от 24 до 54 нуклеотидов, включая все промежуточные целочисленные значения. Описанные выше схемы нумерации для кодирующих CDR3 участков обозначают положения консервативных кодонов цистеина, фенилаланина и триптофана, и данные схемы нумерации также можно применять к псевдогенам, в которых один или более кодонов, кодирующих данные консервативные аминокислоты, можно заменить на кодон, кодирующий другую аминокислоту. Для псевдогенов, в которых не используются данные консервативные аминокислоты, длину CDR3 можно определять относительно соответствующего положения, в котором находился бы такой консервативный остаток в отсутствие замены, в соответствии с одной из указанных выше установленных схем нумерации положения последовательности CDR3.
Также в определенных вариантах осуществления может быть предпочтительно, чтобы множество V-полинуклеотидов, которые присутствуют в описанной в настоящем документе матричной композиции, имели нуклеотидные композиции (например, процентную долю содержания GC), моделирующие полные нуклеотидные композиции известных встречающихся в естественных условиях последовательностей V-гена, даже если конкретные нуклеотидные последовательности различаются. Нуклеотидные композиции V-участка у такой матрицы могут отличаться от нуклеотидных композиций встречающихся в естественных условиях последовательностей V-гена не более чем на 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 процентов. Необязательно и в соответствии с определенными вариантами осуществления V-полинуклеотид описанного в настоящем документе матричного олигонуклеотида включает стоп-кодон у 3’-конца V в общей формуле (I) или поблизости от него.
В формуле (I) J представляет собой полинуклеотид, содержащий по меньшей мере 15–30, 31–60, 61–90, 91–120 или 120–150, но не более 600, 500, 400, 300 или 200 последовательных нуклеотидов генной последовательности, кодирующей соединительный (J) участок рецептора адаптивной иммунной системы или его комплемент, и в каждой из множества олигонуклеотидных последовательностей J содержит уникальную олигонуклеотидную последовательность.
Полинуклеотид J в общей формуле (I) (или его комплемент) включает последовательности, с которыми могут специфически гибридизироваться члены наборов олигонуклеотидных праймеров, специфических для генов TCR или BCR. Наборы праймеров, которые способны проводить амплификацию перестроенных ДНК, кодирующих множество TCR или BCR, описаны, например, в заявке на патент США № 13/217,126; заявке на патент США № 12/794,507; документах PCT/US2011/026373; или PCT/US2011/049012; или т. п.; или, как описано в настоящем документе, они могут быть выполнены с возможностью включения олигонуклеотидных последовательностей, которые могут специфически гибридизироваться с каждым уникальным V-геном и каждым уникальным J-геном в конкретном локусе генов TCR или BCR (например, TCR α, β, γ или δ, или IgH μ, γ, δ, α или ε, или IgL κ или λ).
Полная полинуклеотидная последовательность каждого J-полинуклеотида в общей формуле (I) может, но не должна состоять исключительно из последовательных нуклеотидов из каждого отдельного J-гена. Например, а также в соответствии с определенными вариантами осуществления в описанной в настоящем документе матричной композиции каждый J-полинуклеотид формулы (I) должен иметь только по меньшей мере участок, содержащий уникальную олигонуклеотидную последовательность J, присутствующую в одном J-гене, с которой может специфически гибридизироваться один праймер V-участка в наборе праймеров. Таким образом, V-полинуклеотид формулы (I) может содержать всю или любую заданную часть (например, по меньшей мере 15, 20, 30, 60, 90, 120, 150, 180 или 210 последовательных нуклеотидов или любое промежуточное целочисленное значение) встречающейся в естественных условиях последовательности V-гена (включая последовательность V-псевдогена) такой длины, чтобы в ней находился по меньшей мере один уникальный участок олигонуклеотидной последовательности V (сайт отжига праймера), который не присутствует ни в одном другом матричном J-полинуклеотиде.
В определенных вариантах осуществления может быть предпочтительно, чтобы множество J-полинуклеотидов, которые присутствуют в описанной в настоящем документе матричной композиции, имели длины, моделирующие полные длины известных встречающихся в естественных условиях нуклеотидных последовательностей J-гена, даже если конкретные нуклеотидные последовательности J-участка матрицы и для встречающегося в естественных условиях J-гена различаются. Длины J-участка у описанных в настоящем документе матриц могут отличаться от длин встречающихся в естественных условиях последовательностей J-гена не более чем на 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 процентов.
Таким образом, J-полинуклеотид формулы (I) в определенных вариантах осуществления может содержать нуклеотидную последовательность с длиной, такой же или аналогичной длине типичного встречающегося в естественных условиях J-гена, и может, но не должна включать в себя нуклеотидную последовательность, кодирующую участок CDR3, как описано выше.
Геномные последовательности для генов J-участка TCR и BCR человека и других видов известны и доступны в публичных базах данных, таких как Genbank; генные последовательности J-участка включают в себя полинуклеотидные последовательности, которые кодируют продукты экспрессируемых и неэкспрессируемых генов перестроенных TCR и BCR. Разнообразные последовательности J-полинуклеотида, которые можно использовать в описанных в настоящем документе матрицах общей формулы (I), могут различаться в широких пределах по длине, нуклеотидной композиции (например, содержанию GC) и фактической линейной полинуклеотидной последовательности.
Специалист сможет выбрать альтернативные описанным в настоящем документе последовательности V и J для использования при создании описанных в настоящем документе матричных олигонуклеотидов и/или олигонуклеотидных праймеров для V-сегмента и J-сегмента на основе представленного в настоящем документе описания и накопленных в данной области знаний в отношении опубликованных последовательностей для кодирующих V- и J-участки сегментов генов для каждой субъединицы рецепторов TCR и Ig. Ссылки на записи в Genbank для последовательностей рецепторов адаптивной иммунной системы человека включают в себя: TCRα: (TCRA/D): NC_000014.8 (chr14:22090057..23021075); TCRβ: (TCRB): NC_000007.13 (chr7:141998851..142510972); TCRγ: (TCRG): NC_000007.13 (chr7:38279625..38407656); тяжелая цепь иммуноглобулина, IgH (IGH): NC_000014.8 (chr14: 106032614..107288051); легкая цепь иммуноглобулина-каппа, IgLκ (IGK): NC_000002.11 (chr2: 89156874..90274235); и легкая цепь иммуноглобулина-лямбда, IgLλ (IGL): NC_000022.10 (chr22: 22380474..23265085). Ссылки на записи в Genbank для последовательностей локусов рецепторов адаптивной иммунной системы мыши включают в себя: TCRβ: (TCRB): NC_000072.5 (chr6: 40841295..41508370), и тяжелая цепь иммуноглобулина, IgH (IGH): NC_000078.5 (chr12:114496979..117248165).
Анализы при создании матрицы и праймера и выбор целевого сайта можно проводить, например, с использованием программного пакета для анализа праймеров OLIGO и/или программного пакета BLASTN 2.0.5 (Altschul et al., Nucleic Acids Res. 1997 г., 25(17):3389–402) или других аналогичных доступных в данной области программ.
Соответственно, на основе представленного в настоящем документе описания, а также с учетом данных известных генных последовательностей рецепторов адаптивной иммунной системы и методологий создания праймеров для включения в матричные нуклеотиды настоящего изобретения специалисты в данной области могут создать множество полинуклеотидных последовательностей, специфических для V-участка и специфических для J-участка олигонуклеотидов, каждый из которых независимо содержит олигонуклеотидные последовательности, уникальные для заданного V-гена и J-гена соответственно. Аналогичным образом, исходя из представленного в настоящем документе описания и с учетом известных генных последовательностей рецепторов адаптивной иммунной системы, специалисты в данной области также могут создать набор праймеров, содержащий множество специфических для V-участка и специфических для J-участка олигонуклеотидных праймеров, каждый из которых способен независимо гибридизироваться с конкретной последовательностью, уникальной для заданного V- и J-гена соответственно, посредством чего множество праймеров может проводить амплификацию по существу всех V-генов и по существу всех J-генов в заданном локусе, кодирующем рецепторы адаптивной иммунной системы (например, локус TCR или IgH человека). Такие наборы праймеров в мультиплексной (например, с использованием множества пар прямых и обратных праймеров) ПЦР позволяют генерировать продукты амплификации, имеющие первый конец, который кодируется перестроенным сегментом гена, кодирующего V-участок, и второй конец, который кодируется перестроенным сегментом гена, кодирующего J-участок.
Как правило, в определенных вариантах осуществления такие продукты амплификации могут включать в себя кодирующую CDR3 последовательность, хотя настоящее изобретение не предполагает такого ограничения и предусматривает также продукты амплификации, которые не включают в себя кодирующую CDR3 последовательность. Праймеры можно предпочтительно выполнить с возможностью получения продуктов амплификации, имеющих достаточные части последовательностей V и J и/или последовательностей штрихкода V-J (B), как описано в настоящем документе, так, чтобы путем секвенирования продуктов (ампликонов) было возможно идентифицировать на основе последовательностей, уникальных для каждого сегмента гена, (i) конкретный V-ген и (ii) конкретный J-ген, поблизости от которого произошла перестройка V-гена, для получения гена, кодирующего функциональный рецептор адаптивной иммунной системы. Как правило, в предпочтительных вариантах осуществления продукты ПЦР-амплификации будут иметь размер не более 600 пар оснований, что в соответствии с не имеющей ограничительного характера теорией исключит появление продуктов амплификации неперестроенных генов рецепторов адаптивной иммунной системы. В других определенных предпочтительных вариантах осуществления продукты амплификации будут иметь размер не более 500, 400, 300, 250, 200, 150, 125, 100, 90, 80, 70, 60, 50, 40, 30 или 20 пар оснований, так чтобы обеспечить преимущество быстрого высокопроизводительного количественного определения ампликонов с заданными последовательностями по результатам прочтения коротких последовательностей.
В определенных предпочтительных вариантах осуществления множество матричных олигонуклеотидов содержит по меньшей мере a или по меньшей мере b уникальных олигонуклеотидных последовательностей в зависимости от того, какое значение больше, где a представляет собой число уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой число уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта, и композиция содержит по меньшей мере один матричный олигонуклеотид для каждого уникального V-полинуклеотида и по меньшей мере один матричный олигонуклеотид для каждого уникального J-полинуклеотида. Следует понимать, что, поскольку матричные олигонуклеотиды имеют множество олигонуклеотидных последовательностей общей формулы (I), которая включает V-полинуклеотид и также включает J-полинуклеотид, матричная композиция может содержать менее (a x b) уникальных олигонуклеотидных последовательностей, но будет содержать по меньшей мере большее из a и b количество уникальных олигонуклеотидных последовательностей. Соответственно, композиция может обеспечить по меньшей мере одно вхождение каждой уникальной последовательности V-полинуклеотида и по меньшей мере одно вхождение каждой уникальной последовательности J-полинуклеотида, где в некоторых случаях по меньшей мере одно вхождение конкретного уникального V-полинуклеотида будет обеспечиваться в том же матричном олигонуклеотиде, в котором будет обеспечиваться по меньшей мере одно вхождение конкретного уникального J-полинуклеотида. Таким образом, например, фраза «по меньшей мере один матричный олигонуклеотид для каждого уникального V-полинуклеотида и по меньшей мере один матричный олигонуклеотид для каждого уникального J-полинуклеотида» в определенных случаях может относиться к одному матричному олигонуклеотиду, в котором присутствует один уникальный V-полинуклеотид и один уникальный J-полинуклеотид.
Как также описано в других разделах настоящего документа, в других определенных предпочтительных вариантах осуществления матричная композиция содержит по меньшей мере один матричный олигонуклеотид, с которым может гибридизироваться каждый олигонуклеотидный праймер для амплификации из набора праймеров для амплификации. Таким образом, композиция может содержать менее a или b уникальных последовательностей, например, когда набор праймеров для амплификации может не включать в себя уникальный праймер для каждой возможной последовательности V и/или J.
Следует отметить, что в определенных вариантах осуществления предусмотрена матричная композиция для стандартизации эффективности амплификации набора олигонуклеотидных праймеров, который способен проводить амплификацию продуктивно перестроенных молекул ДНК, кодирующих один или множество рецепторов адаптивной иммунной системы в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта, как представлено в настоящем документе, причем матричная композиция содержит множество матричных олигонуклеотидов, имеющих множество олигонуклеотидных последовательностей общей формулы 5’-U1-B1-V-B2-R-B3-J-B4-U2-3’ (I), как описано в настоящем документе. В соответствии с данными и связанными вариантами осуществления, а также как описано в других разделах настоящего документа, набор олигонуклеотидных праймеров для амплификации, который способен проводить амплификацию продуктивно перестроенных молекул ДНК, может исключать любые олигонуклеотидные праймеры, которые специфически гибридизируются с псевдогеном или орфоном V-участка или с псевдогеном или орфоном J-участка. Таким образом, в таких вариантах осуществления матричная композиция будет желательно исключать матричные олигонуклеотиды общей формулы (I), в которой уникальные олигонуклеотидные последовательности V и/или уникальные олигонуклеотидные последовательности J представляют собой последовательности, которые являются уникальными для псевдогена или орфона V-участка или для псевдогена или орфона J-участка соответственно.
Пример матричной композиции для TCRB, содержащей 858 различных матричных олигонуклеотидов, представлен в списке последовательностей в SEQ ID NO: 3157–4014. Другой пример матричной композиции для TCRB, содержащей 871 различный матричный олигонуклеотид, представлен в списке последовательностей в SEQ ID NO: 1–871. Другой пример матричной композиции для TCRB, содержащей 689 различных матричных олигонуклеотидов, представлен в списке последовательностей в SEQ ID NO: 872–1560.
Пример матричной композиции для TCRG, содержащей 70 различных матричных олигонуклеотидов, представлен в списке последовательностей в SEQ ID NO: 4015–4084. Пример матричной композиции для TCRG, содержащей 70 различных матричных олигонуклеотидов, также представлен в списке последовательностей в SEQ ID NO: 1561–1630.
Пример матричной композиции для IGH, содержащей 1116 различных матричных олигонуклеотидов, представлен в списке последовательностей в SEQ ID NO: 4085–5200. Пример матричной композиции для IGH, содержащей 1116 различных матричных олигонуклеотидов, также представлен в списке последовательностей в SEQ ID NO: 1805–2920.
В настоящем документе также описаны примеры наборов V- и J-полинуклеотидов для включения в описанные в настоящем документе матричные олигонуклеотиды, имеющие множество олигонуклеотидных последовательностей общей формулы (I). Для TCRB множество матричных олигонуклеотидов может иметь множество олигонуклеотидных последовательностей общей формулы (I), в которой V- и J-полинуклеотиды имеют последовательности TCRB V и J, представленные в по меньшей мере одном наборе из 68 TCRB V и J SEQ ID NO соответственно, что указано на Фиг. 5 как TCRB V/J набор 1, TCRB V/J набор 2, TCRB V/J набор 3, TCRB V/J набор 4, TCRB V/J набор 5, TCRB V/J набор 6, TCRB V/J набор 7, TCRB V/J набор 8, TCRB V/J набор 9, TCRB V/J набор 10, TCRB V/J набор 11, TCRB V/J набор 12 и TCRB V/J набор 13.
Для TCRG множество матричных олигонуклеотидов может иметь множество олигонуклеотидных последовательностей общей формулы (I), в которой V- и J-полинуклеотиды имеют последовательности TCRG V и J, представленные в по меньшей мере одном наборе из 14 TCRG V и J SEQ ID NO соответственно, что указано на Фиг. 6 как TCRG V/J набор 1, TCRG V/J набор 2, TCRG V/J набор 3, TCRG V/J набор 4 и TCRG V/J набор 5.
Для IGH множество матричных олигонуклеотидов может иметь множество олигонуклеотидных последовательностей общей формулы (I), в которой V- и J-полинуклеотиды имеют последовательности IGH V и J, представленные в по меньшей мере одном наборе из 127 IGH V и J SEQ ID NO соответственно, что указано на Фиг. 7 как IGH V/J набор 1, IGH V/J набор 2, IGH V/J набор 3, IGH V/J набор 4, IGH V/J набор 5, IGH V/J набор 6, IGH V/J набор 7, IGH V/J набор 8 и IGH V/J набор 9.
ПРАЙМЕРЫ
В соответствии с настоящим описанием предложены олигонуклеотидные праймеры в наборе олигонуклеотидных праймеров, который содержит множество праймеров V-сегмента и множества праймеров J-сегмента, где набор праймеров способен проводить амплификацию перестроенных молекул ДНК, кодирующих рецепторы адаптивной иммунной системы в биологическом образце, содержащем ДНК лимфоидных клеток. Подходящие наборы праймеров известны в данной области и описаны в настоящем документе, например, наборы праймеров в заявке на патент США № 13/217,126; заявке на патент США № 12/794,507; документах PCT/US2011/026373; или PCT/US2011/049012; или т. п.; или представленные в таблице 1. В определенных вариантах осуществления набор праймеров выполнен с возможностью включения в себя множества праймеров, специфических для последовательности V, которое включает для каждого уникального гена V-участка (включая псевдогены) в образце по меньшей мере один праймер, который может специфически гибридизироваться с уникальной последовательностью V-участка; а для каждого уникального гена J-участка в образце — по меньшей мере один праймер, который может специфически гибридизироваться с уникальной последовательностью J-участка.
Создание праймеров можно проводить с использованием стандартных методологий, учитывая известные геномные последовательности для TCR и BCR. Соответственно, набор праймеров предпочтительно способен проводить амплификацию каждой возможной комбинации V-J, которая может быть результатом перестроек ДНК в локусе TCR или BCR. Как также описано ниже, в определенных вариантах осуществления предусмотрены наборы праймеров, в которых один или более V-праймеров могут быть способны специфически гибридизироваться с «уникальной» последовательностью, которая может присутствовать одновременно в двух или более V-участках, но которая не является общей для всех V-участков, и/или в которых один или более J-праймеров могут быть способны специфически гибридизироваться с «уникальной» последовательностью, которая может присутствовать одновременно в двух или более J-участках, но которая не является общей для всех J-участков.
В конкретных вариантах осуществления олигонуклеотидные праймеры для использования в композициях и способах, описанных в настоящем документе, могут содержать или состоять из нуклеиновой кислоты с длиной по меньшей мере приблизительно 15 нуклеотидов, которая имеет ту же последовательность или комплементарна последовательности из 15 последовательных нуклеотидов из целевого V- или J-сегмента (т. е. части геномного полинуклеотида, кодирующего полипептид V-участка или J-участка). В определенных вариантах осуществления также могут использоваться более длинные праймеры, например, имеющие длину приблизительно 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 45 или 50 нуклеотидов, которые имеют ту же последовательность или комплементарны последовательности последовательных нуклеотидов из сегмента полинуклеотида, кодирующего целевой V-участок или J-участок. В настоящем документе также предусмотрены все промежуточные длины описываемых в настоящее время олигонуклеотидных праймеров. Как будет понятно специалистам, праймеры могут иметь дополнительные добавленные последовательности (например, нуклеотиды, которые могут не совпадать или не быть комплементарны сегменту полинуклеотида, кодирующего целевой V- или J-участок), такие как сайты обнаружения рестрикционного фермента, последовательности адаптера для секвенирования, последовательности штрихкода и т. п. (см., например, последовательности праймеров, представленные в таблицах и списках последовательностей, включенных в настоящий документ). Следовательно, праймеры могут иметь большую длину, такую как приблизительно 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 80, 85, 90, 95, 100 или более нуклеотидов или более, в зависимости от конкретного использования или потребности.
Также в определенных вариантах осуществления предусмотрено использование вариантов олигонуклеотидного праймера для V-сегмента или J-сегмента рецептора адаптивной иммунной системы, который может обладать высокой степенью идентичности последовательности с олигонуклеотидными праймерами, для которых в настоящем документе представлены нуклеотидные последовательности, включая представленные в списке последовательностей. Таким образом, в данных и связанных вариантах осуществления варианты олигонуклеотидных праймеров для V-сегмента или J-сегмента рецептора адаптивной иммунной системы могут обладать значительной идентичностью с последовательностями олигонуклеотидных праймеров для V-сегмента или J-сегмента рецептора адаптивной иммунной системы, описанных в настоящем документе, например, степень идентичности последовательностей таких вариантов олигонуклеотидных праймеров составляет по меньшей мере 70%, предпочтительно по меньшей мере 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% или более по сравнению с эталонной полинуклеотидной последовательностью, такой как последовательности олигонуклеотидных праймеров, описанные в настоящем документе, по результатам анализа с использованием описанных в настоящем документе способов (например, анализа в программном пакете BLAST с использованием стандартных параметров). Специалисту в данной области будет понятно, что данные значения можно надлежащим образом корректировать для определения соответствующей способности варианта олигонуклеотидного праймера к отжигу с полинуклеотидом, кодирующим сегмент рецептора адаптивной иммунной системы, принимая во внимание вырожденность кодонов, размещение рамки считывания и т. п.
Как правило, варианты олигонуклеотидного праймера будут содержать одну или более замен, добавлений, делеций и/или вставок, предпочтительно таким образом, чтобы способность к отжигу варианта олигонуклеотида по существу не снижалась по сравнению с последовательностью олигонуклеотидного праймера для V-сегмента или J-сегмента рецептора адаптивной иммунной системы, которая явным образом представлена в настоящем документе.
В таблице 1 представлен не имеющий ограничительного характера пример набора олигонуклеотидных праймеров, который способен проводить амплификацию продуктивно перестроенных молекул ДНК, кодирующих β-цепи TCR (TCRB) в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта. В данном наборе праймеров праймеры для J-сегмента имеют значительную степень гомологии последовательности и, следовательно, допускают перекрестное праймирование для более чем одной целевой последовательности J-полинуклеотида, однако праймеры для V-сегмента выполнены с возможностью специфического отжига с целевыми последовательностями внутри CDR2-участка в V и, следовательно, уникальны для каждого V-сегмента. Однако существует исключение в случае нескольких праймеров для V, где последовательности внутри одного семейства близкородственных целевых генов являются идентичными (например, V6-2 и V6-3 являются идентичными на уровне нуклеотидов на протяжении всей кодирующей последовательности V-сегмента и, следовательно, могут иметь один праймер TRB2V6-2/3).
Таким образом, следует понимать, что в определенных вариантах осуществления можно получить преимущество уменьшенного количества различных матричных олигонуклеотидов в матричной композиции и/или количества различных олигонуклеотидных праймеров в наборе праймеров путем создания матриц и/или праймеров для использования определенных известных элементов сходства в последовательностях V и/или J. Таким образом, в данных и связанных вариантах осуществления «уникальные» олигонуклеотидные последовательности, как описано в настоящем документе, могут включать в себя конкретные последовательности V-полинуклеотида, которые одновременно присутствуют в 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 различных матричных олигонуклеотидах, и/или конкретные последовательности J-полинуклеотида, которые одновременно присутствуют в 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12 или 13 различных матричных олигонуклеотидах, где такие матрицы отличаются друг от друга по последовательности, отличной от общих последовательностей V и/или J.
В соответствии с определенными представленными в настоящем документе вариантами осуществления может быть полезно уменьшить (например, снизить статистически значимым образом) систематическую ошибку амплификации матриц, такую как неравномерный потенциал амплификации нуклеиновых кислот среди членов набора праймеров для амплификации, который может быть следствием неравных показателей эффективности праймеров (например, неодинакового использования праймеров) только для ограниченного подмножества всех встречающихся в естественных условиях V- и J-генов. Например, при анализах иммунного репертуара TCR или BCR, участвующего в иммунном ответе, будь то ответ на конкретный антиген, как в вакцине, или на ткань, как при аутоиммунном заболевании, представлять интерес могут лишь продуктивные перестройки TCR или IG. В таких обстоятельствах может быть экономически выгодно идентифицировать и корректировать неравномерный потенциал амплификации нуклеиновых кислот только для тех праймеров V- и J-сегмента, которые участвуют в амплификации продуктивно перестроенных ДНК, кодирующих TCR или BCR, и исключить попытки корректировать неравномерный потенциал амплификации для псевдогенов и орфонов (т. е. сегментов, кодирующих V-участок TCR или BCR, которые были дублированы на другие хромосомы).
Например, в локусе IGH человека в базе данных ImmunoGeneTics (IMGT) (M.-P. LeFranc, Университет Монпелье, г. Монпелье, Франция; www.imgt.org) аннотированы 165 генов V-сегмента, из которых 26 представляют собой орфоны на других хромосомах, а 139 находятся в локусе IGH на хромосоме 14. Из 139 V-сегментов в локусе IGH 51 имеет по меньшей мере один функциональный аллель, в то время как 6 представляют собой открытые рамки считывания (ORF), в которых отсутствует по меньшей мере один высококонсервативный аминокислотный остаток, а 81 представляют собой псевдогены. Псевдогены могут включать в себя V-сегменты, которые содержат внутрирамочный стоп-кодон внутри кодирующей V-сегмент последовательности, сдвиг рамки считывания между стартовым кодоном и кодирующей CDR3 последовательностью, одну или более вставок элементов повтора и делеций критических участков, таких как первый экзон или сигнальная последовательность рекомбинации (RSS). Следовательно, для характеризации функциональных перестроек IGH в образце без затрат времени и средств на характеризацию псевдогенов и/или орфонов предусмотрено использование подмножества описанных в настоящем документе синтетических матричных олигонуклеотидов, которое выполнено с возможностью включения только тех V-сегментов, которые участвуют в функциональной перестройке для кодирования TCR или BCR, без необходимости в синтезе или калибровке праймеров для амплификации и матричных олигонуклеотидов, специфических для последовательностей псевдогенов. Преимуществом является то, что таким образом достигаются более высокие показатели эффективности в отношении, помимо прочего, затрат времени и средств.
Таблица 1. Пример набора олигонуклеотидных праймеров (ПЦР-праймеры hsTCRB)
В определенных вариантах осуществления олигонуклеотидные праймеры для V-сегмента и J-сегмента, как описано в настоящем документе, выполнены с возможностью включения нуклеотидных последовательностей таким образом, чтобы в последовательности продукта амплификации перестроенного гена рецептора адаптивной иммунной системы (TCR или Ig) содержалась достаточная информация для уникальной идентификации как конкретного V-гена, так и конкретного J-гена, которые породили данный продукт амплификации в локусе перестроенного рецептора адаптивной иммунной системы (например, по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 пар оснований из последовательности до сигнальной последовательности рекомбинации (RSS) V-гена, предпочтительно по меньшей мере приблизительно 22, 24, 26, 28, 30, 32, 34, 35, 36, 37, 38, 39 или 40 пар оснований из последовательности до сигнальной последовательности рекомбинации (RSS) V-гена, и в определенных предпочтительных вариантах осуществления более 40 пар оснований из последовательности до сигнальной последовательности рекомбинации (RSS) V-гена, и по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 пар оснований после последовательности RSS J-гена, предпочтительно по меньшей мере приблизительно 22, 24, 26, 28 или 30 пар оснований после последовательности RSS J-гена, а в некоторых определенных вариантах осуществления более 30 пар оснований после последовательности RSS J-гена).
Данный элемент отличается от описанных в данной области олигонуклеотидных праймеров для амплификации последовательностей гена, кодирующего TCR или Ig, которые в основном полагаются на реакцию амплификации только для простого обнаружения наличия или отсутствия продуктов соответствующих размеров для V- и J-сегментов (например, наличие в продуктах ПЦР-реакции ампликона конкретного размера указывает на наличие V- или J-сегмента, но не могут дать последовательность продукта ПЦР-амплификации и, следовательно, не могут подтвердить его идентичность, что является распространенной практикой при спектротипировании).
Олигонуклеотиды (например, праймеры) можно получить любым подходящим способом, включая прямой химический синтез таким способом, как фосфотриэфирный способ, описанный в работе Narang et al., 1979 г., Meth. Enzymol. 68:90–99; фосфодиэфирный способ, описанный в работе Brown et al., 1979 г., Meth. Enzymol. 68:109–151; диэтилфосфорамидитный способ, описанный в работе Beaucage et al., 1981 г., Tetrahedron Lett. 22:1859–1862; и способ синтеза на твердой подложке, описанный в патенте США № 4,458,066, каждый из которых включен в настоящий документ путем ссылки. Обзор способов синтеза конъюгатов олигонуклеотидов и модифицированных нуклеотидов представлен в работе Goodchild, 1990 г., Bioconjugate Chemistry 1(3): 165–187, включенной в настоящий документ путем ссылки.
В настоящем документе термин «праймер» относится к олигонуклеотиду, способному в подходящих условиях функционировать в качестве точки инициации синтеза ДНК. Такие условия включают в себя условия, при которых синтез продукта достройки праймера, комплементарного цепи нуклеиновой кислоты, индуцируется при наличии четырех различных нуклеозидтрифосфатов и агента для достройки (например, ДНК-полимеразы или обратной транскриптазы) в соответствующем буферном растворе и при подходящей температуре.
Праймер предпочтительно представляет собой одноцепочечную ДНК. Соответствующая длина праймера зависит от предполагаемого назначения праймера, но, как правило, находится в диапазоне от 6 до 50 нуклеотидов, или, в определенных вариантах осуществления, 15–35 нуклеотидов. Короткие молекулы праймеров по существу требуют более низких температур для образования достаточно устойчивых гибридных комплексов с матрицей. Праймер не обязательно должен отражать точную последовательность матричной нуклеиновой кислоты, но должен быть в достаточной степени комплементарным для гибридизации с матрицей. Создание подходящих праймеров для амплификации заданной целевой последовательности хорошо известна в данной области и описана в литературе, процитированной в настоящем документе.
Как описано в настоящем документе, праймеры могут включать дополнительные элементы, позволяющие обнаружение или иммобилизацию праймера, но не изменяют основного свойства праймера, т. е. его функционирования в качестве точки инициации синтеза ДНК. Например, праймеры могут содержать дополнительную последовательность нуклеиновой кислоты на 5'-конце, которая не гибридизируется с целевой нуклеиновой кислотой, но облегчает клонирование, обнаружение или секвенирование амплифицированного продукта. Участок праймера, который в достаточной степени комплементарен матрице для гибридизации, в настоящем документе называется участком гибридизации.
При использовании в настоящем документе праймер является «специфическим» для целевой последовательности, если при его использовании в реакции амплификации в достаточно жестко контролируемых условиях праймер гибридизируется в основном с целевой нуклеиновой кислотой. Как правило, праймер специфичен для целевой последовательности, если устойчивость дуплекса праймер-мишень превышает устойчивость дуплекса, образуемого между праймером и любой другой присутствующей в образце последовательностью. Специалисту в данной области будет понятно, что на специфичность праймера влияют различные факторы, такие как солевые условия и нуклеотидная композиция праймера, а также расположение мест нарушения комплементарности, и что во многих случаях потребуется выполнение стандартной экспериментальной проверки для подтверждения специфичности праймера. Можно выбрать такие условия гибридизации, что праймер сможет образовывать устойчивые дуплексы только с целевой последовательностью. Таким образом, использование специфических для целевой последовательности праймеров в достаточно жестко контролируемых условиях амплификации позволяет обеспечить селективную амплификацию тех целевых последовательностей, которые содержат сайты связывания праймера с целевой последовательностью.
В конкретных вариантах осуществления праймеры для использования в способах, описанных в настоящем документе, могут содержать или состоять из нуклеиновой кислоты длиной по меньшей мере приблизительно 15 нуклеотидов, которая имеет ту же последовательность или комплементарна последовательности из 15 последовательных нуклеотидов из целевого V- или J-сегмента. В определенных вариантах осуществления также можно использовать праймеры большей длины, например, имеющие длину приблизительно 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 45 или 50 нуклеотидов, которые имеют ту же последовательность или комплементарны последовательности последовательных нуклеотидов целевого V- или J-сегмента. В рамках настоящего документа также предусмотрены все промежуточные длины указанных выше праймеров. Как будет понятно специалистам, праймеры могут иметь дополнительные добавленные последовательности (например, нуклеотиды, которые могут не совпадать с или не быть комплементарны целевому V- или J-сегменту), такие как сайты обнаружения рестрикционных ферментов, последовательности адаптера для секвенирования, последовательности штрихкода и т. п. (см., например, последовательности праймеров, представленные в таблицах и списках последовательностей, включенных в настоящий документ). Следовательно, праймеры могут иметь и большую длину, такую как 55, 56, 57, 58, 59, 60, 65, 70, 75 нуклеотидов или более, в зависимости от конкретного использования или потребности. Например, в одном варианте осуществления как прямой, так и обратный праймеры модифицированы на 5'-конце универсальной последовательностью прямого праймера, совместимой с ДНК-секвенатором.
Также в определенных вариантах осуществления предусмотрено использование вариантов олигонуклеотидного праймера для V-сегмента или J-сегмента рецептора адаптивной иммунной системы, который может обладать высокой степенью идентичности последовательности с олигонуклеотидными праймерами, для которых в настоящем документе представлены нуклеотидные последовательности, включая представленные в списке последовательностей. Таким образом, в данных и связанных вариантах осуществления варианты олигонуклеотидных праймеров для V-сегмента или J-сегмента рецептора адаптивной иммунной системы могут обладать значительной идентичностью с последовательностями олигонуклеотидных праймеров для V-сегмента или J-сегмента рецептора адаптивной иммунной системы, описанных в настоящем документе, например, степень идентичности последовательностей таких вариантов олигонуклеотидных праймеров составляет по меньшей мере 70%, предпочтительно по меньшей мере 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% или более по сравнению с эталонной полинуклеотидной последовательностью, такой как последовательности олигонуклеотидных праймеров, описанные в настоящем документе, по результатам анализа с использованием описанных в настоящем документе способов (например, анализа в программном пакете BLAST с использованием стандартных параметров). Специалисту в данной области будет понятно, что данные значения можно надлежащим образом корректировать для определения соответствующей способности варианта олигонуклеотидного праймера к отжигу с полинуклеотидом, кодирующим сегмент рецептора адаптивной иммунной системы, принимая во внимание вырожденность кодонов, размещение рамки считывания и т. п.
Как правило, варианты олигонуклеотидного праймера будут содержать одну или более замен, добавлений, делеций и/или вставок, предпочтительно таким образом, чтобы способность к отжигу варианта олигонуклеотида по существу не снижалась по сравнению с последовательностью олигонуклеотидного праймера для V-сегмента или J-сегмента рецептора адаптивной иммунной системы, которая явным образом представлена в настоящем документе. Как также указано в других разделах настоящего документа, в предпочтительных вариантах осуществления олигонуклеотидные праймеры для V-сегмента и J-сегмента рецептора адаптивной иммунной системы выполнены с возможностью ведения амплификации перестроенной последовательности TCR или IGH, которая включает участок, кодирующий CDR3.
В соответствии с определенными предусмотренными в настоящем документе вариантами осуществления праймеры для использования в способах мультиплексной ПЦР, представленных в настоящем описании, могут быть функционально блокированы для предотвращения неспецифического праймирования последовательностей, отличных от последовательностей T- или B-клетки. Например, праймеры можно блокировать путем химических модификаций, как описано в публикации заявки на патент США № US2010/0167353. В соответствии с определенными описанными в настоящем документе вариантами осуществления использование таких блокированных праймеров в настоящих мультиплексных ПЦР-реакциях включает праймеры, которые могут иметь неактивную конфигурацию, в которой репликация ДНК (т. е. достройка праймера) заблокирована, и активированную конфигурацию, в которой протекает репликация ДНК. Неактивная конфигурация праймера имеет место либо когда праймер находится в одноцепочечном состоянии, либо когда праймер специфически гибридизировался с интересующей целевой последовательностью ДНК, но достройка праймера остается заблокированной химическим фрагментом, связанным в или поблизости от 3'-конца праймера.
Активированная конфигурация праймера имеет место, когда праймер специфически гибридизировался с интересующей целевой последовательностью ДНК и затем был подвергнут обработке РНКазой H или другим расщепляющим агентом для удаления блокирующей 3'-конец группы, посредством этого позволяя ферменту (например, ДНК-полимеразе) катализировать достройку праймера в реакции амплификации. Без стремления к ограничению какой-либо теорией считается, что кинетика гибридизации таких праймеров аналогична реакции второго порядка и, следовательно, зависит от концентрации последовательности T-клеточного или B-клеточного гена в смеси. Блокированные праймеры сводят к минимуму неспецифические реакции, требуя гибридизации с целевой последовательностью и последующего расщепления до запуска процесса достройки праймера. При неправильной гибридизации праймера с последовательностью, которая связана с желаемой целевой последовательностью, но отличается от нее наличием одного или более некомплементарных нуклеотидов, что приводит к ошибкам комплементарности оснований, расщепление праймера ингибируется, в особенности при наличии ошибки комплементарности, находящейся в или поблизости от сайта расщепления. Данная стратегия повышения достоверности амплификации снижает частоту ложного праймирования в таких местах и посредством этого повышает специфичность реакции. Как будет понятно специалисту, условия реакции, в частности, концентрация РНКазы H и продолжительность гибридизации и достройки в каждом цикле, можно оптимизировать для получения максимального различия в показателях эффективности расщепления между высокоэффективным расщеплением праймера в случае, когда он правильно гибридизировался со своей истинной целевой последовательностью, и низкоэффективным расщеплением праймера при наличии ошибки комплементарности между праймером и матричной последовательностью, с которой он мог гибридизироваться не полностью.
Как описано в патенте № US2010/0167353, в данной области известен ряд блокирующих групп, которые можно разместить в или поблизости от 3'-конца олигонуклеотида (например, праймера) для предотвращения достройки. Праймер или другой олигонуклеотид можно модифицировать по 3'-концевому нуклеотиду для предотвращения или ингибирования инициации синтеза ДНК путем, например, добавления 3'-дезоксирибонуклеотидного остатка (например, кордицепина), 2',3'-дидезоксирибонуклеотидного остатка, ненуклеотидных связей или проведения алкан-диольных модификаций (патент США № 5,554,516). Алкан-диольные модификации, которые можно использовать для ингибирования или блокирования достройки праймера, также описаны в работе Wilk et al., (1990 г., Nucleic Acids Res. 18 (8):2065) и в патенте Arnold et al. (патент США № 6,031,091). Дополнительные примеры подходящих блокирующих групп включают в себя 3'-гидроксильные замены (например, 3'-фосфат, 3'-трифосфат или 3'-фосфатные диэфиры со спиртами, такими как 3-гидроксипропил), 2'3'-циклический фосфат, 2'-гидроксильные замены концевого основания РНК (например, фосфатные или стерически затрудненные группы, такие как триизопропилсилил (TIPS) или трет-бутилдиметилсилил (TBDMS)). 2'-Алкилсилильные группы, такие как TIPS и TBDMS, замещенные на 3'-конце олигонуклеотида, описаны в заявке на патент США (Laikhter et al.) № 11/686,894, которая включена в настоящий документ путем ссылки. Для блокирования достройки праймера на 3'-концевом остатке олигонуклеотида также можно ввести объемные заместители.
В определенных вариантах осуществления олигонуклеотид может содержать домен расщепления, расположенный до (например, в 5’ относительно) блокирующей группы, используемой для ингибирования достройки праймера. Например, домен расщепления может представлять собой домен расщепления РНКазой H, или домен расщепления может представлять собой домен расщепления РНКазой H2, содержащий один остаток РНК, или олигонуклеотидный праймер может содержать замену основания РНК на один или более альтернативных нуклеозидов. Дополнительные примеры доменов расщепления описаны в патенте № US2010/0167353.
Таким образом, в системе мультиплексной ПЦР можно использовать 40, 45, 50, 55, 60, 65, 70, 75, 80, 85 или более прямых праймеров, причем каждый прямой праймер комплементарен одному функциональному V-сегменту TCR или Ig или небольшому семейству функциональных V-сегментов TCR или Ig, например, сегменту Vβ TCR (см., например, праймеры TCRBV, как показано в таблице 1 в SEQ ID NO: 1644–1695), и, например, тринадцать обратных праймеров, каждый из которых специфичен для J-сегмента TCR или Ig, такого как Jβ-сегмент TCR (см., например, праймеры TCRBJ в таблице 1, SEQ ID NO: 1631–1643). В другом варианте осуществления в мультиплексной ПЦР-реакции можно использовать четыре прямых праймера, каждый из которых специфичен для одного или более функциональных V-сегментов TCRγ, и четыре обратных праймера, каждый из которых специфичен для одного или более J-сегментов TCRγ. В другом варианте осуществления в мультиплексной ПЦР-реакции можно использовать 84 прямых праймера, каждый из которых специфичен для одного или более функциональных V-сегментов, и шесть обратных праймеров, каждый из которых специфичен для одного или более J-сегментов.
Условия термоциклирования могут соответствовать способам, известным специалистам в данной области. Например, при работе с амплификатором PCR Express™ (Hybaid, г. Эшфорд, Великобритания) можно использовать следующие условия термоциклирования: 1 цикл при 95 °C в течение 15 минут, от 25 до 40 циклов при 94 °C в течение 30 секунд, 59 °C в течение 30 секунд и 72 °C в течение 1 минуты с последующим одним циклом при 72 °C в течение 10 минут. Как будет понятно специалистам в данной области, условия термоциклирования можно оптимизировать, например, путем изменения температур отжига, продолжительности отжига, числа циклов и продолжительности достройки. Как будет понятно специалистам в данной области, количество используемых праймеров и других реагентов для ПЦР, а также параметры ПЦР (например, температуру отжига, продолжительности достройки и число циклов), можно оптимизировать для получения желаемой эффективности ПЦР-амплификации.
Альтернативно в определенных связанных вариантах осуществления, также предусмотренных в настоящем документе, для количественного определения числа целевых геномов в образце можно использовать способы «цифровой ПЦР», не прибегая к стандартной кривой. В цифровой ПЦР параллельно проводят ПЦР-реакцию для одного образца во множестве из более 100 микрокювет или капель так, что в каждой капле амплификация либо проходит (например, наработка продукта амплификации подтверждает наличие по меньшей мере одной матричной молекулы в данной микрокювете или капле), либо не проходит (подтверждение отсутствия матрицы в данной микрокювете или капле). Путем простого подсчета положительных микрокювет можно непосредственно подсчитать число целевых геномов, присутствующих во входном образце.
В способах цифровой ПЦР, как правило, используют считывание в конечной точке вместо получения сигнала традиционной количественной ПЦР, который измеряют после каждого цикла в реакции термоциклирования (см., например, Pekin et al., 2011 г., Lab. Chip 11(13):2156; Zhong et al., 2011 г., Lab. Chip 11(13):2167; Tewhey et al., 2009 г., Nature Biotechnol. 27:1025; 2010 г., Nature Biotechnol. 28:178; Vogelstein and Kinzler, 1999 г., Proc. Natl. Acad. Sci. USA 96:9236–41; Pohl and Shih, 2004 г., Expert Rev. Mol. Diagn. 4(1);41–7, 2004). По сравнению с традиционной ПЦР цифровая ПЦР обладает следующими преимуществами: (1) отсутствие необходимости полагаться на эталоны или стандарты, (2) возможность получения желаемой точности путем увеличения полного числа репликатов ПЦР, (3) высокая стойкость к ингибиторам, (4) способность к анализу сложных смесей и (5) генерация линейного по количеству присутствующих в образце копий ответа, что позволяет обнаруживать малые изменения в числе копий. Соответственно, любые из описанных в настоящем документе композиций (например, матричные композиции и наборы олигонуклеотидных праймеров, специфических для генов рецептора адаптивной иммунной системы) и способов можно выполнить с возможностью использования в такой методологии цифровой ПЦР, например, на системах ABI QuantStudio™ 12K Flex System (Life Technologies, г. Карлсбад, штат Калифорния, США), QX100™ Droplet Digital™ PCR System (BioRad, г. Геркулес, штат Калифорния, США), QuantaLife™ Digital PCR System (BioRad, г. Геркулес, штат Калифорния, США) или RainDance™ Microdroplet Digital PCR System (RainDance Technologies, г. Лексингтон, штат Массачуссетс, США).
АДАПТЕРЫ
Описанные в настоящем документе матричные олигонуклеотиды общей формулы (I) в определенных вариантах осуществления также могут содержать первую (U1) и вторую (U2) универсальные последовательности олигонуклеотида-адаптера или могут не содержать одну или обе из U1 и U2. Таким образом, U1 может либо отсутствовать, либо содержать олигонуклеотид, имеющий последовательность, выбранную из (i) первой универсальной последовательности олигонуклеотида-адаптера и (ii) первой специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5’ и находится в нем относительно первой универсальной последовательности олигонуклеотида-адаптера, и U2 может либо отсутствовать, либо содержать олигонуклеотид, имеющий последовательность, выбранную из (i) второй универсальной последовательности олигонуклеотида-адаптера и (ii) второй специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5’ и находится в нем относительно второй универсальной последовательности олигонуклеотида-адаптера.
Например, U1 и/или U2 могут содержать универсальные последовательности олигонуклеотида-адаптера и/или специфические для используемой секвенирующей платформы олигонуклеотидные последовательности, которые специфичны для используемой технологии секвенирования одиночной молекулы, например, систем HiSeq™, GeneAnalyzer™-2 (GA-2) (Illumina, Inc., г. Сан-Диего, штат Калифорния, США), или другого подходящего комплекса оборудования, реагентов и программного обеспечения для секвенирования. Включение таких специфических для используемой секвенирующей платформы последовательностей адаптера позволяет проводить прямое количественное секвенирование описанной в настоящем документе матричной композиции, которая содержит множество различных матричных олигонуклеотидов общей формулы (I), с использованием технологии секвенирования нуклеотидов, такой как HiSeq™ или GA2 или эквивалент. Следовательно, данный элемент обеспечивает преимущество качественной и количественной характеризации матричной композиции.
В частности, способность непосредственно секвенировать все компоненты матричной композиции обеспечивает возможность проверки того, что каждый матричный нуклеотид во множестве матричных олигонуклеотидов присутствует в по существу эквимолярном количестве. Например, можно создать набор описываемых в настоящее время матричных олигонуклеотидов, имеющих универсальные последовательности адаптера на обоих концах, так что последовательности адаптера можно будет использовать для дополнительного введения специфических для используемой секвенирующей платформы олигонуклеотидов с каждого конца каждой матрицы.
Без стремления к ограничению какой-либо теорией, специфические для используемой секвенирующей платформы олигонуклеотиды можно добавить на концы таких модифицированных матриц, используя 5’- (5’-последовательность специфического для используемой секвенирующей платформы универсального адаптера-1-3’) и 3’- (5’-последовательность специфического для используемой секвенирующей платформы универсального адаптера-2-3’) олигонуклеотиды лишь в двух циклах денатурации, отжига и достройки, так что относительное представление в матричной композиции каждого из составляющих ее матричных олигонуклеотидов количественно не изменяется. Рядом с последовательностями адаптера размещают уникальные идентифицирующие последовательности (например, последовательности штрихкода B, содержащие уникальные олигонуклеотидные последовательности V и B, которые ассоциированы с и таким образом идентифицируют индивидуальные V- и J-участки, соответственно, как описано в настоящем документе), посредством чего позволяя провести количественное секвенирование путем прочтения коротких последовательностей, чтобы охарактеризовать популяцию матриц по критерию относительного количества каждой присутствующей уникальной матричной последовательности.
Если такое прямое количественное секвенирование указывает на то, что один или более конкретных олигонуклеотидов могут быть избыточно или недостаточно представлены в препарате матричной композиции, можно провести соответствующую коррекцию матричной композиции для получения матричной композиции, в которой все олигонуклеотиды присутствуют в по существу эквимолярных количествах. Затем такую матричную композицию, в которой все олигонуклеотиды присутствуют в по существу эквимолярных количествах, можно использовать в качестве стандарта для калибровки наборов праймеров для амплификации, как в описанных в настоящем документе способах, для определения и коррекции неравномерного потенциала амплификации среди членов набора праймеров.
Помимо последовательностей адаптера, описанных в примерах и включенных в примеры матричных последовательностей в списке последовательностей (например, на 5’- и 3’-концах в SEQ ID NO: 1–1630), специалисты в данной области на основе представленного в настоящем документе описания предложат и другие олигонуклеотидные последовательности, которые можно использовать в качестве универсальных последовательностей адаптера, включая выбор последовательностей олигонуклеотида-адаптера, которые отличны от последовательностей, представленных в других частях описанных в настоящем документе матриц. Не имеющие ограничительного характера примеры дополнительных последовательностей адаптера представлены в таблице 2 и указаны в SEQ ID NO: 1710–1731.
Таблица 2. Примеры последовательностей адаптера
ШТРИХКОДЫ
Как описано в настоящем документе, в определенных вариантах осуществления предусмотрено создание матричных олигонуклеотидных последовательностей с включением коротких сигнатурных последовательностей, позволяющих однозначно идентифицировать матричную последовательность, и, следовательно, по меньшей мере один праймер, ответственный за амплификацию данной матрицы, без необходимости в секвенировании всего продукта амплификации. В описанных в настоящем документе матричных олигонуклеотидах общей формулы (I) B1, B2, B3 и B4 каждый независимо либо отсутствует, либо каждый содержит олигонуклеотид B, который содержит олигонуклеотидную последовательность штрихкода из 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 или более последовательных нуклеотидов (включая все промежуточные целочисленные значения), причем в каждой из множества матричных олигонуклеотидных последовательностей B содержит уникальную олигонуклеотидную последовательность, которая уникальным образом идентифицирует в качестве спаренной комбинации (i) уникальную олигонуклеотидную последовательность V матричного олигонуклеотида и (ii) уникальную олигонуклеотидную последовательность J матричного олигонуклеотида.
Таким образом, например, матричные олигонуклеотиды, имеющие идентифицирующие последовательности штрихкода, могут обеспечивать прочтения относительно коротких последовательностей продуктов амплификации, такие как прочтения последовательностей штрихкода длиной не более 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100, 90, 80, 70, 60, 55, 50, 45, 40, 35, 30, 25, 20, 19, 18, 17, 16, 15, 14, 13, 12, 11, 10, 9, 8, 7, 6, 5, 4 или менее нуклеотидов, с последующим сопоставлением информации по данной последовательности штрихкода с ассоциированными с ними последовательностями V и J, которые встроены в матрицу, имеющую штрихкод как часть конфигурации матрицы. При таком подходе может быть возможно проводить одновременное частичное секвенирование большого числа разных продуктов амплификации с использованием высокопроизводительного параллельного секвенирования для идентификации праймеров, ответственных за систематическую ошибку амплификации, в сложном наборе праймеров.
Примеры штрихкодов могут содержать первый олигонуклеотид штрихкода из 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или 16 нуклеотидов, который уникальным образом идентифицирует каждый V-полинуклеотид в матрице, и второй олигонуклеотид штрихкода из 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или 16 нуклеотидов, который уникальным образом идентифицирует каждый J-полинуклеотид в матрице, для получения штрихкодов длиной 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32 нуклеотидов, соответственно, но данные и связанные варианты осуществления не предполагают такого ограничения. Олигонуклеотиды штрихкода могут содержать олигонуклеотидные последовательности любой длины, при условии получения минимальной длины штрихкода, которая исключает возможность вхождения заданной последовательности штрихкода в двух или более матричных олигонуклеотидах, имеющих отличные в остальном последовательности (например, последовательности V и J).
Таким образом, минимальная длина штрихкода, необходимая для предотвращения такой избыточности среди штрихкодов, используемых для уникальной идентификации различных пар последовательности V-J, составляет X нуклеотидов, где 4x превышает число различных типов матриц, которые необходимо дифференцировать на основе наличия неидентичных последовательностей. Например, для набора из 871 матричного олигонуклеотида, представленного в настоящем документе как SEQ ID NO: 1–871, минимальная длина штрихвода составила бы пять нуклеотидов, что позволяет теоретически иметь всего 1024 (т. е. более 871) возможных различных пентануклеотидных последовательностей. На практике длины прочтения олигонуклеотидной последовательности штрихкода могут быть ограничены только пределами длины прочтения олигонуклеотидной последовательности используемого для секвенирования прибора. Для определенных вариантов осуществления различные олигонуклеотиды штрихкода, которые будут отличать индивидуальные типы матричных олигонуклеотидов, должны иметь по меньшей мере два нарушения комплементарности нуклеотидов (например, минимальное расстояние Хэмминга, равное 2) при выравнивании для получения максимального числа нуклеотидов, которые комплементарны в конкретных положениях в олигонуклеотидных последовательностях штрихкода.
В предпочтительных вариантах осуществления для каждого отдельного типа матричного олигонуклеотида, имеющего уникальную последовательность в матричной композиции общей формулы (I), B1, B2, B3 и B4 будут идентичны.
Специалистам в данной области будут знакомы способы создания, синтеза и встраивания в больший по размерам олигонуклеотидный или полинуклеотидный конструкт олигонуклеотидных последовательностей штрихкода, например, по меньшей мере 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 200, 300, 300, 500 или более последовательных нуклеотидов, включая все промежуточные целочисленные значения. Не имеющие ограничительного характера примеры стратегий идентификации на основе конфигурации и реализации олигонуклеотидной последовательности штрихкода описаны, например, в работах de Carcer et al., 2011 г., Adv. Env. Microbiol. 77:6310; Parameswaran et al., 2007 г., Nucl. Ac. Res. 35(19):330; Roh et al., 2010 г., Trends Biotechnol. 28:291.
Как правило, штрихкоды помещают в матрицы в местах, где они в естественных условиях не встречаются, т. е. штрихкоды содержат нуклеотидные последовательности, которые отличны от любых встречающихся в естественных условиях олигонуклеотидных последовательностей, которые могут встречаться вблизи последовательностей, рядом с которыми расположены штрихкоды (например, последовательности V и/или J). В соответствии с определенными описанными в настоящем документе вариантами осуществления такие последовательности штрихкода можно включать как элементы B1, B2 и/или B3 описанного в настоящем документе матричного олигонуклеотида общей формулы (I). Соответственно, определенные из описанных в настоящем документе матричных олигонуклеотидов общей формулы (I) в определенных вариантах осуществления также могут содержать один, два или все три штрихкода B1, B2 и B3, тогда как в других определенных вариантах осуществления некоторые или все из данных штрихкодов могут отсутствовать. В определенных вариантах осуществления все последовательности штрихкода будут иметь идентичное или аналогичное содержание GC (например, будут различаться по содержанию GC не более чем на 20% или не более чем на 19, 18, 17, 16, 15, 14, 13, 12, 11 или 10%).
В матричных композициях в соответствии с определенными описанными в настоящем документе вариантами осуществления содержащий штрихкод элемент B (например, B1, B2, B3 и/или B4) содержит олигонуклеотидную последовательность, которая уникальным образом идентифицирует одну спаренную комбинацию V-J. Необязательно и в определенных вариантах осуществления содержащий штрихкод элемент B также может включать в себя случайный нуклеотид или случайную полинуклеотидную последовательность из по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 45, 50, 55, 60, 70, 80, 90, 100, 200, 300, 300, 500 или более последовательных нуклеотидов, расположенных до и/или после конкретной последовательности штрихкода, которая уникальным образом идентифицирует каждую конкретную спаренную комбинацию V-J. При их наличии как до, так и после конкретной последовательности штрихкода случайный нуклеотид или случайная полинуклеотидная последовательность независимы друг от друга, то есть они могут, но не должны содержать такой же нуклеотид или такую же полинуклеотидную последовательность.
САЙТЫ РЕСТРИКЦИОННОГО ФЕРМЕНТА
В соответствии с определенными описанными в настоящем документе вариантами осуществления матричный олигонуклеотид может содержать сайт распознавания рестрикционной эндонуклеазы (RE), который расположен между последовательностями V и J и больше нигде не встречается в матричной олигонуклеотидной последовательности. сайт распознавания RE может быть необязательно расположен рядом с сайтом штрихкода, который идентифицирует последовательность V-участка. Сайт RE можно включить для достижения любого числа целей, включая, без ограничений, в качестве структурного элемента, который можно использовать для селективного разрушения матриц путем приведения их в контакт с соответствующим рестрикционным ферментом. Может быть желательно селективно разрушить настоящие матричные олигонуклеотиды путем приведения их в контакт с подходящей RE, например, для удаления матричных олигонуклеотидов из других композиций, в которые их могли целенаправленно или случайно ввести. Альтернативно сайт RE можно эффективно использовать в процессе секвенирования матричных олигонуклеотидов в матричной композиции и/или в качестве маркера положения в матричной олигонуклеотидной последовательности независимо от того, производится ли его расщепление рестрикционным ферментом. Примером сайта RE является олигонуклеотидный мотив GTCGAC, который распознается рестрикционным ферментом Sal I. В данной области известно множество дополнительных рестрикционных ферментов и их соответствующих последовательностей сайтов обнаружения RE, и такие ферменты доступны в продаже (например, New England Biolabs, г. Беверли, штат Массачусетс, США). Они включают в себя, например, EcoRI (GAATTC) и SphI (GCATGC). Специалистам в данной области известно, что любой из множества таких сайтов обнаружения RE можно встроить в конкретные варианты осуществления описанных в настоящем документе матричных нуклеотидов.
Секвенирование
Секвенирование можно проводить с использованием любой из различных доступных аппаратных платформ и систем для высокопроизводительного секвенирования одиночных молекул. Примеры систем секвенирования включают в себя системы, основанные на принципе «секвенирование путем синтеза», такие как Illumina Genome Analyzer и связанные аппаратные платформы (Illumina, Inc., г. Сан-Диего, штат Калифорния, США), Helicos Genetic Analysis System (Helicos BioSciences Corp., г. Кембридж, штат Массачусетс, США), Pacific Biosciences PacBio RS (Pacific Biosciences, г. Менло-Парк, штат Калифорния, США) или другие системы, имеющие аналогичные возможности. Секвенирование проводят с использованием набора олигонуклеотидов для секвенирования, которые гибридизируются с определенным участком в молекулах амплифицированной ДНК. Олигонуклеотиды для секвенирования выполнены таким образом, чтобы сегменты гена, кодирующего V и J, можно было уникальным образом идентифицировать по генерируемым последовательностям, опираясь на настоящее описание, а также с учетом известных генных последовательностей рецепторов адаптивной иммунной системы, которые включены в публично доступные базы данных. См., например, заявку на патент США № 13/217,126; заявку на патент США № 12/794,507; документы PCT/US2011/026373; или PCT/US2011/049012. Примеры праймеров для секвенирования J-участка TCRB представлены в таблице 3.
Таблица 3. Праймеры для секвенирования J TCRB
Термин «ген» означает сегмент ДНК, участвующий в продукции полипептидной цепи, такой как весь или часть полипептида TCR или Ig (например, полипептид, содержащий CDR3); он включает в себя участки, предшествующие и следующие за кодирующим участком («лидерная» и «трейлерная» последовательности), а также промежуточные последовательности (интроны) между индивидуальными кодирующими сегментами (экзонами). Он также может включать в себя регуляторные элементы (например, промоторы, энхансеры, сайты связывания репрессора и т. п.) и может включать в себя сигнальные последовательности рекомбинации (RSS), как описано в настоящем документе.
Нуклеиновые кислоты настоящих вариантов осуществления, также называемые в настоящем документе полинуклеотидами, могут быть представлены в форме РНК или в форме ДНК, причем ДНК включает кДНК, геномную ДНК и синтетическую ДНК. ДНК может быть двухцепочечной или одноцепочечной, и в случае одноцепочечной ДНК она может представлять собой кодирующую цепь или некодирующую (антисмысловую) цепь. Кодирующая последовательность, которая кодирует TCR, иммуноглобулин или их участок (например, V-участок, D-сегмент, J-участок, C-участок и т. д.) для использования в соответствии с настоящими вариантами осуществления, может быть идентична кодирующей последовательности, известной в данной области, для любых заданных участков генов или полипептидных доменов TCR или иммуноглобулина (например, доменов V-участка, доменов CDR3 и т. д.), или может представлять собой другую кодирующую последовательность, которая в результате избыточности или вырожденности генетического кода кодирует тот же участок или полипептид TCR или иммуноглобулина.
В определенных вариантах осуществления каждый из амплифицированных сегментов гена, кодирующего J-участок, может иметь уникальную определяемую последовательностью идентифицирующую метку из 2, 3, 4, 5, 6, 7, 8, 9, 10 или приблизительно 15, 20 или более нуклеотидов, расположенных в определенном положении относительно сайта RSS. Например, можно использовать метку из четырех оснований в кодирующем Jβ-участок сегменте амплифицируемых участков, кодирующих CDR3 TCRβ, в положениях с +11 по +14 после сайта RSS. Однако данные и связанные варианты осуществления не требуют такого ограничения и также предусматривают другие относительно короткие определяемые нуклеотидной последовательностью идентифицирующие метки, которые можно обнаружить в сегментах гена, кодирующего J-участок, и определять на основе их положений относительно сайта RSS. Они могут различаться между разными локусами, кодирующими рецепторы адаптивной иммунной системы.
Сигнальная последовательность рекомбинации (RSS) состоит из двух консервативных последовательностей (гептамера 5'-CACAGTG-3' и нонамера 5'-ACAAAAACC-3'), разделенных разделителем из либо 12 +/- 1 п. о. (12-signal), либо 23 +/- 1 п. о. (23-signal). Был идентифицирован ряд важных для рекомбинации положений нуклеотидов, включая динуклеотид CA в положении один и два гептамера; кроме того, была показана значительная предпочтительность нуклеотида C в положении три гептамера, а также нуклеотида A в положениях 5, 6, 7 нонамера. (Ramsden et. al 1994 г.; Akamatsu et. al. 1994 г.; Hesse et. al. 1989 г.). Мутации по другим нуклеотидам имеют минимальные или нестабильные эффекты. Разделитель, хоть он и является более вариабельным, также влияет на рекомбинацию, а также было показано, что однонуклеотидные замены значительно влияют на эффективность рекомбинации (см. Fanning et. al. 1996 г., Larijani et. al 1999 г.; Nadel et. al. 1998 г.). Были описаны критерии идентификации полинуклеотидных последовательностей RSS, имеющих значительно различающуюся эффективность рекомбинации (см. Ramsden et. al 1994 г.; Akamatsu et. al. 1994 г.; Hesse et. al. 1989 г. и Cowell et. al. 1994 г.). Соответственно, олигонуклеотиды для секвенирования могут гибридизироваться рядом с меткой из четырех оснований в сегментах амплифицированных генов, кодирующих J-участок, в положениях с +11 по +14 после сайта RSS. Например, олигонуклеотиды для секвенирования для TCRB могут быть выполнены с возможностью отжига с консенсусным мотивом нуклеотидов, находящихся непосредственно после данной «метки», так что первые четыре основания при прочтении последовательности уникальным образом идентифицируют сегмент гена, кодирующего J-участок (см., например, WO/2012/027503).
Средняя длина участка, кодирующего CDR3, для TCR, определяемого как нуклеотиды, кодирующие полипептид TCR между вторым консервативным цистеином V-сегмента и консервативным фенилаланином J-сегмента, составляет 35 +/- 3 нуклеотида. Соответственно и в определенных вариантах осуществления ПЦР-амплификация с использованием олигонуклеотидных праймеров V-сегмента с олигонуклеотидными праймерами J-сегмента, которые начинаются с метки J-сегмента конкретного J-участка TCR или IgH (например, TCR Jβ, TCR Jγ или IgH JH, как описано в настоящем документе), будет практически всегда захватывать полное соединение V-D-J при прочтении 50 пар оснований. Средняя длина участка CDR3 IgH, определяемого как нуклеотиды между консервативным цистеином в V-сегменте и консервативным фенилаланином в J-сегменте, в меньшей степени ограничена, чем локус TCRβ, но, как правило, будет составлять от приблизительно 10 до приблизительно 70 нуклеотидов. Соответственно и в определенных вариантах осуществления ПЦР-амплификация с использованием олигонуклеотидных праймеров V-сегмента с олигонуклеотидными праймерами J-сегмента, которые начинаются с метки J-сегмента IgH, будет захватывать полное соединение V-D-J при прочтении 100 пар оснований.
ПЦР-праймеры, которые гибридизируются с и поддерживают достройку полинуклеотида на матричных последовательностях с нарушением комплементарности, называют смешанными праймерами. В определенных вариантах осуществления обратные ПЦР-праймеры J-сегмента TCR и Ig могут быть выполнены с возможностью сведения к минимуму перекрывания с олигонуклеотидами для секвенирования, чтобы свести к минимуму смешанное праймирование в контексте мультиплексной ПЦР. В одном варианте осуществления обратные праймеры J-сегмента TCR и Ig можно закрепить на 3'-конце путем отжига с консенсусным мотивом сайта сплайсинга с минимальным перекрыванием с праймерами для секвенирования. По существу праймеры V- и J-сегментов TCR и Ig можно выбрать для функционирования в ПЦР при воспроизводимых температурах отжига, используя известные программы для создания и анализа последовательности/праймера с параметрами по умолчанию.
Для реакции секвенирования примеры праймеров для секвенирования IGHJ охватывают три нуклеотида в консервативных последовательностях CAG, как описано в № WO/2012/027503.
ОБРАЗЦЫ
Субъект или биологический источник, из которого можно получить тестируемый биологический образец, может представлять собой человека или отличное от человека животное, либо трансгенный, клонированный или тканеинженерный (включая путем использования стволовых клеток) организм. В определенных предпочтительных вариантах осуществления настоящего изобретения может быть известно, что у субъекта или биологического источника есть или может предполагаться наличие риска присутствия циркулирующей или солидной опухоли или другого злокачественного состояния, или аутоиммунного заболевания, или воспалительного состояния, а в определенных предпочтительных вариантах осуществления настоящего изобретения может быть известно, что у субъекта или биологического источника отсутствует такое заболевание или риск его наличия.
В определенных предпочтительных вариантах осуществления предусмотрен человеческий индивид или биологический источник, который представляет собой субъекта-человека, такого как пациент, у которого был диагностирован рак или установлено наличие риска развития или приобретения рака в соответствии с принятыми в данной области диагностическими критериями, такими как критерии, которые используются в Национальном институте рака США (г. Бетесда, штат Мэриленд, США) или которые описаны в руководствах DeVita, Hellman, and Rosenberg's Cancer: Principles and Practice of Oncology (2008 г., Lippincott, Williams and Wilkins, Philadelphia/ Ovid, г. Нью-Йорк); Pizzo and Poplack, Principles and Practice of Pediatric Oncology (четвертое издание, 2001 г., Lippincott, Williams and Wilkins, Philadelphia/ Ovid, г. Нью-Йорк); и Vogelstein and Kinzler, The Genetic Basis of Human Cancer (второе издание, 2002 г., McGraw Hill Professional, г. Нью-Йорк); в определенных вариантах осуществления предусмотрен человеческий субъект, у которого в соответствии с данными критериями установлено отсутствие риска наличия, развития или приобретения рака.
В других определенных вариантах осуществления предусмотрен не относящийся к человеку субъект или биологический источник, например, нечеловеческий индивид, такой как макака, шимпанзе, горилла, зеленая мартышка, орангутан, бабуин или другой нечеловеческий индивид, включая таких не относящихся к человеку субъектов, которые могут быть известны специалистам как модели для доклинических исследований, включая модели для доклинических исследований солидных опухолей и/или других типов рака. В других определенных вариантах осуществления предусмотрен не относящийся к человеку субъект, который представляет собой млекопитающее, например, мышь, крысу, кролика, свинью, овцу, лошадь, быка, козла, песчанку, хомяка, морскую свинку или другое млекопитающее; многие такие млекопитающие могут представлять собой субъекты, которые известны специалистам как модели для доклинических исследований определенных болезней или расстройств, включая циркулирующие или солидные опухоли и/или другие типы рака (см., например, Talmadge et al., 2007 г., Am. J. Pathol. 170:793; Kerbel, 2003 г., Canc. Biol. Therap. 2(4 Suppl 1):S134; Man et al., 2007 г., Canc. Met. Rev. 26:737; Cespedes et al., 2006 г., Clin. Transl. Oncol. 8:318). Однако диапазон вариантов осуществления не предполагает такого ограничения, так что предусмотрены также другие варианты осуществления, в которых субъект или биологический источник может представлять собой немлекопитающее позвоночное, например, другое высшее позвоночное, либо один из видов птиц, амфибий или рептилий, либо другой субъект или биологический источник.
Биологические образцы можно получить путем забора образца крови, биопсии образца, эксплантата ткани, культуры органа, биологической жидкости или любого другого препарата ткани или клеток у субъекта или биологического источника. Предпочтительно образец содержит ДНК из лимфоидных клеток субъекта или биологического источника, которая, в качестве иллюстрации и без ограничения, может содержать перестроенные ДНК из одного или более локусов TCR или BCR. В определенных вариантах осуществления тестируемый биологический образец можно получить из твердой ткани (например, солидной опухоли), например, путем хирургической резекции, пункционной биопсии или с помощью других средств получения тестируемого биологического образца, который содержит смесь клеток.
В соответствии с определенными вариантами осуществления может быть желательно выделить лимфоидные клетки (например, T-клетки и/или B-клетки) в соответствии с любой из большого числа разработанных методологий, где выделенные лимфоидные клетки представляют собой клетки, которые были извлечены или отделены от ткани, среды или окружения, в котором они встречаются в естественных условиях. Таким образом, можно получить B-клетки и T-клетки из биологического образца, такие как из различных образцов ткани и биологической жидкости, включая костный мозг, тимус, лимфатические железы, лимфатические узлы, периферические ткани и кровь, однако наиболее простой доступ можно получить к периферической крови. Из любой периферической ткани можно взять образцы для определения наличия B- и T-клеток; следовательно, она предусмотрена для использования в способах, описанных в настоящем документе. Ткани и биологические жидкости, из которых можно получить клетки адаптивной иммунной системы, включают в себя, без ограничений, кожу, эпителиальные ткани, толстую кишку, селезенку, слизистый секрет, слизистые оболочки полости рта, слизистые оболочки желудочно-кишечного тракта, слизистые оболочки влагалища или влагалищный секрет, ткань шейки матки, нервные узлы, слюну, спинномозговую жидкость (СМЖ), костный мозг, кровь пуповины, сыворотку, серозную жидкость, плазму, лимфу, мочу, жидкость брюшной полости, плевральную жидкость, перикардиальную жидкость, перитонеальную жидкость, абдоминальную жидкость, культуральную среду, кондиционированную культуральную среду или смывную жидкость. В определенных вариантах осуществления клетки адаптивной иммунной системы можно выделить из аферезного образца. Образцы периферической крови можно получить путем проведения флеботомии у субъектов. Мононуклеарные клетки периферической крови (МКПК) выделяют известными специалистам в данной области техниками, например, путем разделения в градиенте плотности Ficoll-Hypaque®. В определенных вариантах осуществления для анализа используют цельные клетки МКПК.
Для экстракции нуклеиновой кислоты из клеток можно экстрагировать общую геномную ДНК, используя известные специалистам способы и/или доступные в продаже наборы, например, с использованием мини-набора для извлечения ДНК из образцов крови QIAamp® DNA blood Mini Kit (QIAGEN®). Приблизительная масса одного гаплоидного генома составляет 3 пг. Предпочтительно для анализа используют по меньшей мере от 100000 до 200000 клеток, т. е. приблизительно от 0,6 до 1,2 мкг ДНК из диплоидных T- или B-клеток. При использовании в качестве источника клеток МКПК число T-клеток можно оценить как приблизительно 30% от общего числа клеток. Число B-клеток также можно оценить как приблизительно 30% от общего числа клеток в препарате МКПК.
Локусы гена Ig и TCR содержат множество различных сегментов генов для вариабельных (V), обеспечивающих разнообразие (D) и соединительных (J) участков, которые подвергаются процессам перестройки на ранних стадиях лимфоидного дифференцирования. Последовательности сегментов V-, D- и J-генов Ig и TCR известны в данной области и доступны в публичных базах данных, таких как GENBANK. Перестройки V-D-J опосредованы ферментативным комплексом рекомбиназы, в котором ключевую роль играют белки RAG1 и RAG2, распознающие и разделяющие ДНК по сигнальным последовательностям рекомбинации (RSS), которые находятся после сегментов V-гена, с обеих сторон сегментов D-гена и до сегментов J-гена. Неправильные RSS снижают или даже полностью предотвращают перестройку. Сигнальная последовательность рекомбинации (RSS) включает две консенсусные последовательности (гептамер 5'-CACAGTG-3' и нонамер 5'-ACAAAAACC-3'), разделенные разделителем из либо 12 +/- 1 п. о. (12-signal), либо 23 +/- 1 п. о. (23-signal). На 3'-конце V-сегмента и D-сегмента последовательность RSS представляет собой гептамер (CACAGTG)-разделитель-нонамер (ACAAAAACC). На 5'-конце J-сегмента и D-сегмента последовательность RSS представляет собой нонамер (GGTTTTTGT)-разделитель-гептамер (CACTGTG) со значительными вариациями в последовательности гептамера и нонамера для каждого конкретного сегмента гена.
Был идентифицирован ряд важных для рекомбинации положений нуклеотидов, включая динуклеотид CA в положении один и два гептамера; кроме того, была показана значительная предпочтительность нуклеотида C в положении три гептамера, а также нуклеотида A в положениях 5, 6, 7 нонамера. (см. Ramsden et. al 1994 г., Nucl. Ac. Res. 22:1785; Akamatsu et. al. 1994 г., J. Immunol. 153:4520; Hesse et. al. 1989 г., Genes Dev. 3:1053). Мутации по другим нуклеотидам имеют минимальные или нестабильные эффекты. Разделитель, хоть он и является более вариабельным, также влияет на рекомбинацию, а также было показано, что однонуклеотидные замены значительно влияют на эффективность рекомбинации (см. Fanning et. al. 1996 г., Cell. Immunol. Immumnopath. 79:1, Larijani et. al 1999 г., Nucl. Ac. Res. 27:2304; Nadel et. al. 1998 г., J. Immunol. 161:6068; Nadel et al., 1998 г., J. Exp. Med. 187:1495). Были описаны критерии для идентификации полинуклеотидных последовательностей RSS, имеющих значительно различающуюся эффективность рекомбинации (см. Ramsden et. al 1994 г., Nucl. Ac. Res. 22:1785; Akamatsu et. al. 1994 г., J. Immunol. 153:4520; Hesse et. al. 1989 г., Genes Dev. 3:1053, и Lee et al., 2003 г., PLoS 1(1):E1).
Процесс перестройки генов тяжелой цепи Ig (IgH), TCR-бета (TCRB) и TCR-дельта (TCRD) по существу начинается с перестройки от D к J с последующей перестройкой от V к D-J, в то время как для генов Ig-каппа (IgK), Ig-лямбда (IgL), TCR-альфа (TCRA) и TCR-гамма (TCRG) происходят прямые перестройки от V к J. Последовательности между перестраиваемыми сегментами генов по существу удаляются в форме кольцевого продукта вырезания, также называемого эксцизионным кольцом TCR (TREC) или эксцизионным кольцом B-клеточного рецептора (BREC).
Множество различных комбинаций сегментов V-, D- и J-гена представляют так называемый комбинаторный репертуар, который оценивается как ~2 x 106 для молекул Ig, ~3 x 106 для молекул TCRαβ и ~ 5 x 103 для молекул TCRγδ. В сайтах соединения сегментов V-, D- и J-гена в процессе перестройки происходит делеция и случайная вставка нуклеотидов, что приводит к соединительным участкам с большим разнообразием, которые вносят значительный вклад в создание полного репертуара молекул Ig и TCR, оцениваемого как > 1012 возможных аминокислотных последовательностей.
Зрелые B-лимфоциты дополнительно расширяют свой репертуар Ig после распознавания антигена в зародышевых центрах посредством соматической гипермутации - процесса, приводящего созреванию аффинности молекул Ig. Процесс соматической гипермутации концентрируется на экзоне V- (D-) J-генов тяжелой цепи IgH и легкой цепи Ig и главным образом генерирует однонуклеотидные мутации, а иногда также вставки или делеции нуклеотидов. Прошедшие процесс соматической мутации гены Ig также, как правило, находятся в зрелых B-клеточных злокачественных новообразованиях.
В определенных описанных в настоящем документе вариантах осуществления праймеры V-сегмента и J-сегмента можно использовать в ПЦР-реакции для амплификации перестроенных участков ДНК, кодирующих CDR3, в TCR или BCR, в тестируемом биологическом образце, причем каждый функциональный сегмент гена, кодирующего V-участок TCR или Ig, содержит сигнальную последовательность рекомбинации (RSS) V-гена, а каждый функциональный сегмент гена, кодирующего J-участок TCR или Ig, содержит RSS J-гена. В данных и связанных вариантах осуществления каждая амплифицируемая молекула перестроенной ДНК может содержать (i) по меньшей мере приблизительно 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 (включая все промежуточные целочисленные значения) или более последовательных нуклеотидов смысловой цепи сегмента гена, кодирующего V-участок TCR или Ig, где по меньшей мере приблизительно 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или более последовательных нуклеотидов расположены в 5’ относительно RSS V-гена, и/или каждая амплифицируемая молекула перестроенной ДНК может содержать (ii) по меньшей мере приблизительно 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500 (включая все промежуточные целочисленные значения) или более последовательных нуклеотидов смысловой цепи сегмента гена, кодирующего J-участок TCR или Ig, где по меньшей мере приблизительно 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500 или более последовательных нуклеотидов расположены в 3’ относительно RSS J-гена.
Определение коэффициента амплификации
В дополнение к использованию описанных в настоящем документе матричных композиций для стандартизации эффективности амплификации наборов олигонуклеотидных праймеров для амплификации, как описано в настоящем документе, в других определенных вариантах осуществления предусмотрено использование матричной композиции для определения коэффициентов амплификации для оценки количества перестроенных последовательностей, кодирующих рецепторы адаптивной иммунной системы, в образце. Данные и связанные варианты осуществления можно использовать для количественного определения числа кодирующих последовательностей рецепторов адаптивной иммунной системы в образце ДНК, полученном из лимфоидных клеток, включая лимфоидные клетки, которые присутствуют в смеси клеток, содержащей клетки, в которых кодирующая рецептор адаптивной иммунной системы ДНК прошла процесс перестройки ДНК, но где образец также содержит ДНК из клеток, в которых такая перестройка не происходила (например, клетки, отличные от лимфоидных, незрелые клетки, мезенхимальные клетки, раковые клетки и т. д.).
Полное число различных членов заданного класса рецепторов адаптивной иммунной системы (например, TCR или IG) у субъекта можно оценить с помощью мультиплексной ПЦР, используя исчерпывающий набор праймеров для амплификации V-J с последующим количественным секвенированием продуктов амплификации. Мультиплексная амплификация и высокопроизводительное секвенирование перестроенных последовательностей ДНК, кодирующих TCR и BCR (IG), описаны, например, в работах Robins et al., 2009 г., Blood 114, 4099; Robins et al., 2010 г., Sci. Translat. Med. 2:47ra64; Robins et al., 2011 г., J. Immunol. Meth. doi:10.1016/j.jim.2011.09. 001; Sherwood et al. 2011 г., Sci. Translat. Med. 3:90ra61; заявке на патент США № 13/217,126 (публикации США № 2012/0058902), заявке на патент США № 12/794,507 (публикации США № 2010/0330571), международных заявках WO/2010/151416, WO/2011/106738 (PCT/US2011/026373), WO2012/027503 (PCT/US2011/049012), заявке на патент США № 61/550,311 и заявке на патент США № 61/569,118.
В данную методологию, как правило, включена выборка ДНК из субпопуляции лимфоидных клеток, таких как лимфоидные клетки, присутствующие в образце крови, который, как известно, также содержит ядерные клетки, в которых отсутствует перестроенная ДНК, кодирующая TCR или IG. Настоящие композиции и способы могут позволить повысить точность и достоверность при определении количества перестроенных молекул ДНК, кодирующих TCR и IG, в таком образце. Как описано в настоящем документе, например, добавление известного количества описанной в настоящем документе матричной композиции в образец ДНК создает внутренний стандарт амплификации матриц для оценки относительной эффективности членов набора олигонуклеотидных праймеров, которые присутствуют в наборе праймеров для мультиплексной амплификации. Оценивая таким образом продукты амплификации описанной в настоящем документе искусственной матричной композиции, добавляемой в известных количествах в реакцию амплификации, можно определить коэффициент амплификации (например, мультипликативный, нормирующий, масштабирующий или геометрический коэффициент и т. д.) для набора олигонуклеотидных праймеров для амплификации, который затем можно использовать для вычисления количества естественных ДНК матриц в образце.
В качестве другого примера данные и связанные варианты осуществления позволяют количественно определить остаточную минимальную болезнь (ОМБ) при лимфоме или лейкемии путем количественного обнаружения перестроенных ДНК, кодирующих TCR или IG, в образцах, полученных из смешанных препаратов лимфоидных и нелимфоидных клеток, включая клетки персистирующей лимфомы или лейкемии. В способах предшествующего уровня техники ОМБ определяли как число обнаруживаемых злокачественных клеток в виде доли от полного количества клеток в образце. В отличие от этого настоящие способы позволяют выполнить оценку общего числа клеток в образце с перестроенной ДНК, кодирующей TCR или IG, так что можно количественно определить долю злокачественных клеток (например, имеющих конкретную перестройку TCR или IG, такую как клонотип) относительно таких перестроенных клеток вместо доли относительно всех клеток. Без стремления к ограничению какой-либо теорией считается, что, так как представление всех перестроенных клеток в образце клинического материала субъекта, имеющего или предположительно имеющего ОМБ, как правило, является очень низким, настоящие способы позволят значительно повысить чувствительность, с которой можно обнаруживать ОМБ, включая повышение такой чувствительности путем улучшения отношения сигнал/шум.
Таким образом, в определенных вариантах осуществления предложен способ количественного определения перестроенных молекул ДНК, кодирующих один или множество рецепторов адаптивной иммунной системы в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта, причем каждый рецептор адаптивной иммунной системы содержит вариабельный участок и соединительный участок. Вкратце, способ включает следующие стадии:
(A) в мультиплексной реакции амплификации с использованием описанного в настоящем документе набора олигонуклеотидных праймеров для амплификации, который способен проводить амплификацию по существу всех комбинаций, кодирующих V-J для заданного рецептора адаптивной иммунной системы, проведение амплификации ДНК из образца, к которому добавлено известное количество описанной в настоящем документе матричной композиции для стандартизации эффективности амплификации, для получения продуктов амплификации;
(B) количественное секвенирование продуктов амплификации из (A) для количественного определения (i) продуктов амплификации матриц, которые представляют собой продукты амплификации описанной в настоящем документе матричной композиции и могут быть идентифицированы, поскольку они содержат по меньшей мере одну олигонуклеотидную последовательность штрихкода, и (ii) продуктов амплификации перестроенных последовательностей ДНК, кодирующих рецепторы адаптивной иммунной системы, в образце, которые могут быть идентифицированы, поскольку они содержат конкретные последовательности V и J, но не содержат олигонуклеотидной последовательности штрихкода;
(C) вычисление коэффициента амплификации на основе количественной информации, полученной на стадии (B); и
(D) использование коэффициента амплификации из (C) для определения количества уникальных молекул ДНК, кодирующих рецепторы адаптивной иммунной системы, в образце путем вычисления.
Без стремления к ограничению какой-либо теорией в соответствии с данными и связанными способами измеряют количество перестроенных молекул ДНК, кодирующих TCR или IG, которые анализируют в мультиплексной реакции амплификации. Для этого определяют значение покрытия последовательностей, например, количество прочтений в выходном сигнале последовательности, определяемых для каждой входной молекулы (матрицы), и усредняют их по всем различным присутствующим матричным олигонуклеотидам для получения среднего значения покрытия последовательностей. Разделив (i) количество прочтений, полученных для заданной последовательности, на (ii) среднее значение покрытия последовательностей, можно вычислить количество перестроенных молекул, присутствующих в качестве матриц в начале реакции амплификации.
Таким образом, например, для вычисления значения покрытия последовательностей для каждой ПЦР-амплификации добавляют известное количество набора синтетических молекул описанной в настоящем документе матричной композиции, причем синтетические матрицы имеют базовую структуру формулы (I) 5’ U-B1-V-B2-R-(B3)-J-B4-U 3’, где каждый V представляет собой сегмент из 300 пар оснований, имеющий последовательность, соответствующую последовательности V-гена TCR или IG, а J представляет собой сегмент из 100 пар оснований, имеющий последовательность, соответствующую последовательности J-гена TCR или IG. B2 представляет собой уникальную олигонуклеотидную последовательность штрихкода, которая уникальным образом идентифицирует каждую пару VJ и которая также дифференцирует продукты амплификации синтетических ДНК матриц (которые содержат последовательность штрихкода) от продуктов амплификации встречающихся в естественных условиях биологических молекул ДНК матриц, которые поступили в образец из ДНК лимфоидных клеток (в которых будет отсутствовать последовательность штрихкода). В данном примере B3 формулы (I) отсутствует. После проведения ПЦР-амплификации и секвенирования подсчитывают количества каждой секвенированной синтетической молекулы (т. е. продуктов амплификации, содержащих последовательность штрихкода). Затем вычисляют покрытие последовательности для синтетических молекул на основе известного количества исходных молекул синтетических матриц, добавленных в реакцию амплификации.
Например, в реакцию амплификации можно добавить пул из 5000 синтетических содержащих штрихкод матричных молекул, содержащий по 4–5 копий для каждой из 1100 уникальных последовательностей синтетических матричных олигонуклеотидов (представляющих каждую возможную пару VJ). Если продукты амплификации включают в себя 50 000 последовательностей, которые соответствуют синтетическим матричным молекулам, получают значение покрытия последовательностей 10X, а коэффициент амплификации - 10. Затем для оценки количества естественных перестроенных по VDJ матричных молекул в ДНК, полученной из образца, количество продуктов амплификации естественных матриц (т. е. продуктов амплификации без какой-либо последовательности штрихкода) делят на коэффициент амплификации. Поскольку в данном примере 5000 синтетических молекул представляют собой сложный пул из 1100 молекул, представляющих каждую пару VJ, для повышения точности можно индивидуально вычислить коэффициент амплификации для каждой пары VJ. Затем коэффициент амплификации можно усреднить по всем синтетическим молекулам (Фиг. 8). Точность и устойчивость способа показаны на Фиг. 9, а более подробное описание представлено ниже в примере 5.
Альтернативный вариант осуществления идентичен описанному выше и ниже в данном разделе, за исключением отличия в использовании подмножества общего пула синтетических матричных молекул таким образом, что в образец добавляется не более 1 копии подмножества различных матричных молекул. Применение хорошо известных специалистам в данной области статистических способов Пуассона позволяет определить необходимое для добавления количество матрицы на основе известных свойств пула (например, полного количества отдельных последовательностей и концентрации матричных молекул). Например, в реакцию амплификации добавляют 200–500 матричных молекул таким образом, что в среднем в пуле присутствует не более одной копии каждого из подмножества матричных молекул.
Соответственно в данных вариантах осуществления способ включает: (А) амплификацию ДНК в мультиплексной полимеразной цепной реакции (ПЦР), которая содержит: (1) ДНК из биологического образца, содержащего лимфоидные клетки субъекта; (2) матричную композицию по п. 1 формулы изобретения, в которой в известном количестве присутствует каждый из множества матричных олигонуклеотидов, имеющих уникальную олигонуклеотидную последовательность; (3) набор олигонуклеотидных праймеров для амплификации, который способен проводить амплификацию перестроенных ДНК, кодирующих один или множество рецепторов адаптивной иммунной системы в ДНК из биологического образца, причем набор праймеров содержит: (а) в по существу эквимолярных количествах — множество олигонуклеотидных праймеров V-сегмента, каждый из которых способен независимо специфически гибридизироваться с по меньшей мере одним полинуклеотидом, кодирующим полипептид V-участка рецептора адаптивной иммунной системы, или с его комплементом, представленным в настоящем документе, где каждый праймер V-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному функциональному сегменту гена, кодирующего V-участок рецептора адаптивной иммунной системы, и где множество праймеров V-сегмента специфически гибридизируется с по существу всеми функциональными сегментами гена, кодирующего V-участок рецептора адаптивной иммунной системы, которые присутствуют в матричной композиции, и (b) в по существу эквимолярных количествах - множество олигонуклеотидных праймеров J-сегмента, каждый из которых способен независимо специфически гибридизироваться с по меньшей мере одним полинуклеотидом, кодирующим полипептид J-участка рецептора адаптивной иммунной системы, или с его комплементом, представленным в настоящем документе, где каждый праймер J-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному функциональному сегменту гена, кодирующего J-участок рецептора адаптивной иммунной системы, и где множество праймеров J-сегмента специфически гибридизируется с по существу всеми функциональными сегментами гена, кодирующего J-участок рецептора адаптивной иммунной системы, которые присутствуют в матричной композиции, причем олигонуклеотидные праймеры V-сегмента и J-сегмента могут обеспечить в указанной мультиплексной полимеразной цепной реакции (ПЦР) амплификацию (i) по существу всех матричных олигонуклеотидов в композиции для получения множества амплифицированных матричных молекул ДНК, причем указанное множество амплифицированных матричных молекул ДНК достаточно для количественного определения разнообразия матричных олигонуклеотидов в матричной композиции, и (ii) по существу всех перестроенных молекул ДНК, кодирующих рецепторы адаптивной иммунной системы в биологическом образце, для получения множества амплифицированных перестроенных молекул ДНК, причем указанное множество амплифицированных перестроенных молекул ДНК достаточно для количественного определения разнообразия перестроенных молекул ДНК в ДНК из данного биологического образца, и где каждая амплифицированная молекула ДНК из множества амплифицированных матричных молекул ДНК и из множества амплифицированных перестроенных молекул ДНК имеет длину менее чем 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100, 90, 80 или 70 нуклеотидов;
(B) количественное секвенирование всей или достаточной части каждой из указанных амплифицированных матричных молекул ДНК и каждой из указанных амплифицированных перестроенных молекул ДНК для количественного определения (i) количества матричного продукта амплифицированных матричных молекул ДНК, которые содержат по меньшей мере одну олигонуклеотидную последовательность штрихкода, и (ii) количества перестроенного продукта амплифицированных перестроенных молекул ДНК, в которых отсутствует олигонуклеотидная последовательность штрихкода;
(С) вычисление коэффициента амплификации путем деления количества матричного продукта из (B) (i) на известное количество каждого из множества матричных олигонуклеотидов, имеющих уникальную олигонуклеотидную последовательность из (A) (2); и
(D) деление количества перестроенного продукта из (B) (ii) на коэффициент амплификации, вычисленный в (C), для количественного определения содержания молекул уникальных молекул ДНК, кодирующих рецепторы адаптивной иммунной системы, в образце.
Предусмотренные варианты осуществления не предполагают ограничения описанным выше способом, так что из представленного в настоящем документе описания специалист сможет предложить возможные для применения варианты. Например, в альтернативном подходе можно не использовать описанную в настоящем документе композицию из синтетических матриц в качестве контрольной матрицы, добавляемой в известном количестве при проведении мультиплексной ПЦР-амплификации образца ДНК, содержащего перестроенные ДНК, кодирующие TCR и/или IG лимфоидных клеток, а также неперестроенные ДНК. Вместо этого в соответствии с одним таким альтернативным подходом для реакции амплификации с использованием V- и J-праймеров для амплификации можно добавить известный набор олигонуклеотидных праймеров для амплификации, которые обеспечивают амплификацию отдельного высококонсервативного участка геномной последовательности. Данные геномные контрольные праймеры могут проводить амплификацию каждого присутствующего в образце ДНК генома независимо от того, содержит ли он перестроенные последовательности, кодирующие TCR и/или IG, тогда как праймеры V и J могут давать продукты амплификации только для геномов с перестроенным участком VDJ. Соотношение между данными двумя классами молекул продуктов амплификации позволяет оценить общее количество B-клеточных геномов в образце.
Применение на практике определенных вариантов осуществления настоящего изобретения потребует привлечения, если конкретно не указано обратное, традиционных способов из области микробиологии, молекулярной биологии, биохимии, молекулярной генетики, клеточной биологии, вирусологии и иммунологии, хорошо известных специалистам в данной области, и ниже в целях иллюстрации даны ссылки на некоторые из них. Такие методики подробно описаны в литературе. См., например, руководства Sambrook, et al., Molecular Cloning: A Laboratory Manual (3-е изд., 2001 г.); Sambrook, et al., Molecular Cloning: A Laboratory Manual (2-е изд., 1989 г.); Maniatis et al., Molecular Cloning: A Laboratory Manual (1982 г.); Ausubel et al., Current Protocols in Molecular Biology (John Wiley and Sons, обновление: июль 2008 г.); Short Protocols in Molecular Biology: A Compendium of Methods from Current Protocols in Molecular Biology, Greene Pub. Associates and Wiley-Interscience; Glover, DNA Cloning: A Practical Approach, тома I и II (IRL Press, Oxford Univ. Press USA, 1985 г.); Current Protocols in Immunology (ред.: John E. Coligan, Ada M. Kruisbeek, David H. Margulies, Ethan M. Shevach, Warren Strober, 2001 г., John Wiley & Sons, г. Нью-Йорк, штат Нью-Йорк, США); Real-Time PCR: Current Technology and Applications, ред. Julie Logan, Kirstin Edwards and Nick Saunders, 2009 г., Caister Academic Press, г. Норфолк, Великобритания; Anand, Techniques for the Analysis of Complex Genomes, (Academic Press, г. Нью-Йорк, 1992 г.); Guthrie and Fink, Guide to Yeast Genetics and Molecular Biology (Academic Press, г. Нью-Йорк, 1991 г.); Oligonucleotide Synthesis (ред. N. Gait, 1984 г.); Nucleic Acid Hybridization (ред. B. Hames & S. Higgins, 1985 г.); Transcription and Translation (ред. B. Hames & S. Higgins, 1984 г.); Animal Cell Culture (ред. R. Freshney, 1986 г.); Perbal, A Practical Guide to Molecular Cloning (1984 г.); Next-Generation Genome Sequencing (Janitz, 2008 г., Wiley-VCH); PCR Protocols (Methods in Molecular Biology) (ред. Park, 3-е изд., 2010 г., Humana Press); Immobilized Cells And Enzymes (IRL Press, 1986 г.); монография Methods In Enzymology (Academic Press, Inc., г. Нью-Йорк); Gene Transfer Vectors For Mammalian Cells (ред. J. H. Miller and M. P. Calos, 1987 г., Cold Spring Harbor Laboratory); Harlow and Lane, Antibodies, (Cold Spring Harbor Laboratory Press, Cold Spring Harbor, г. Нью-Йорк, 1998 г.); Immunochemical Methods In Cell And Molecular Biology (ред. Mayer and Walker, Academic Press, г. Лондон, 1987 г.); Handbook Of Experimental Immunology, тома I–IV (ред. D. M. Weir and CC Blackwell, 1986 г.); Riott, Essential Immunology, 6-е изд., (Blackwell Scientific Publications, г. Оксфорд, 1988 г.); Embryonic Stem Cells: Methods and Protocols (Methods in Molecular Biology) (ред. Kurstad Turksen, 2002 г.); Embryonic Stem Cell Protocols: том I: Isolation and Characterization (Methods in Molecular Biology) (ред. Kurstad Turksen, 2006 г.); Embryonic Stem Cell Protocols: том II: Differentiation Models (Methods in Molecular Biology) (ред. Kurstad Turksen, 2006 г.); Human Embryonic Stem Cell Protocols (Methods in Molecular Biology) (ред. Kursad Turksen, 2006 г.); Mesenchymal Stem Cells: Methods and Protocols (Methods in Molecular Biology) (ред. Darwin J. Prockop, Donald G. Phinney, and Bruce A. Bunnell, 2008 г.); Hematopoietic Stem Cell Protocols (Methods in Molecular Medicine) (ред. Christopher A. Klug, and Craig T. Jordan, 2001 г.); Hematopoietic Stem Cell Protocols (Methods in Molecular Biology) (ред. Kevin D. Bunting, 2008 г.) Neural Stem Cells: Methods and Protocols (Methods in Molecular Biology) (ред. Leslie P. Weiner, 2008 г.).
Если не дано конкретных определений, вся используемая в данном случае номенклатура и все лабораторные процедуры и методики из областей молекулярной биологии, аналитической химии, синтетической органической химии и медицинской и фармацевтической химии, описанные в настоящем документе, хорошо известны и общеприняты среди специалистов в данной области. Для рекомбинантных технологий, молекулярно-биологических, микробиологических и химических синтезов, химических анализов, получения, подготовки лекарственных форм, введения препаратов и терапии пациентов можно использовать стандартные техники.
Если контекст не требует иного, в тексте настоящей спецификации и формуле изобретения слово «содержать» и его варианты, такие как «содержит» и «содержащий», следует толковать в открытом, включающем смысле, то есть как «включая, без ограничений». Под фразой «состоящий из» подразумевается включение и, как правило, ограничение тем, что следует после фразы «состоящий из». Под фразой «состоящий по существу из» подразумевается включение любых перечисленных после данной фразы элементов и ограничение другими элементами, которые не входят в противоречие с или не вносят вклад в активность или действие, установленные в описании для перечисленных элементов. Таким образом, фраза «состоящий по существу из» указывает на то, что перечисленные элементы являются необходимыми или обязательными, но что другие элементы при этом не требуются и могут присутствовать или отсутствовать в зависимости от того, влияют ли они на активность или действие перечисленных элементов.
В данной спецификации и в приложенной формуле изобретения формы единственного числа включают в себя ссылки на множественное число, если иное явно не предусмотрено контекстом. При использовании в настоящем документе в конкретных вариантах осуществления термин «приблизительно», предшествующий численному значению, указывает на значение плюс или минус диапазон 5%, 6%, 7%, 8% или 9%. В других вариантах осуществления термин «приблизительно», предшествующий численному значению, указывает на значение плюс или минус диапазон 10%, 11%, 12%, 13% или 14%. В других вариантах осуществления термин «приблизительно», предшествующий численному значению, указывает на значение плюс или минус диапазон 15%, 16%, 17%, 18%, 19% или 20%.
В тексте данной спецификации ссылка на «один вариант осуществления», «вариант осуществления» или «аспект» означает, что конкретный элемент, структура или характеристика, описанные в связи с вариантом осуществления, включены по меньшей мере в один вариант осуществления настоящего изобретения. Таким образом, все фразы «в одном варианте осуществления» или «в варианте осуществления», появляющиеся в различных местах данной спецификации, не обязательно относятся к одному варианту осуществления. Более того, конкретные элементы, структуры или характеристики можно комбинировать любым подходящим способом в одном или более вариантах осуществления.
ПРИМЕРЫ
ПРИМЕР 1. СОЗДАНИЕ МАТРИЧНЫХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ КАЛИБРОВКИ КОНТРОЛЯ СИСТЕМАТИЧЕСКОЙ ОШИБКИ ПРАЙМЕРА ДЛЯ АМПЛИФИКАЦИИ
В данном и последующих примерах применяются стандартные молекулярно-биологические и биохимические материалы и методологии, включая техники, описанные, например, в руководствах Sambrook, et al., Molecular Cloning: A Laboratory Manual (3-е изд., 2001 г.); Sambrook, et al., Molecular Cloning: A Laboratory Manual (2-е изд., 1989 г.); Maniatis et al., Molecular Cloning: A Laboratory Manual (1982 г.); Ausubel et al., Current Protocols in Molecular Biology (John Wiley and Sons, обновление: июль 2008 г.); Short Protocols in Molecular Biology: A Compendium of Methods from Current Protocols in Molecular Biology, Greene Pub. Associates and Wiley-Interscience; Glover, DNA Cloning: A Practical Approach, тома I и II (IRL Press, Oxford Univ. Press USA, 1985 г.); Anand, Techniques for the Analysis of Complex Genomes, (Academic Press, г. Нью-Йорк, 1992 г.); Oligonucleotide Synthesis (ред. N. Gait, 1984 г.); Nucleic Acid Hybridization (ред. B. Hames & S. Higgins, 1985 г.); Transcription and Translation (ред. B. Hames & S. Higgins, 1984 г.); Perbal, A Practical Guide to Molecular Cloning (1984 г.); Next-Generation Genome Sequencing (Janitz, 2008 г., Wiley-VCH); PCR Protocols (Methods in Molecular Biology) (ред. Park, 3-е изд., 2010 г., Humana Press).
Был создан набор матричных олигонуклеотидов в виде двухцепочечных ДНК (дцДНК) в качестве калибровочного стандарта для использования в качестве контрольной матрицы, имитирующей все возможные комбинации V/J в установленном локусе рецептора адаптивной иммунной системы (TCR или BCR). Для каждого локуса TCR и BCR человека был составлен список известных геномных последовательностей V-сегмента в 5' RSS и список известных геномных J-сегментов в 3' RSS. Для простоты интерпретации в настоящем документе приведены последовательности кодирующей цепи дцДНК матрицы в соответствии с соглашением, по которому направление от 5’- к 3’-концу читается слева направо.
На Фиг. 1 представлено схематическое изображение общей структуры матричных олигонуклеотидов. Для использования при перекрестной проверке идентичности каждого уникального матричного олигонуклеотида во множестве контекстов в каждую матрицу встраивали разные олигонуклеотиды штрихкода (B) из 16 п. о., которые уникальным образом идентифицировали полинуклеотид V-сегмента матрицы по первым 8 п. о. штрихкода и J-сегмента матрицы по вторым 8 п. о. штрихкода. Копии данного штрихкода встраивали трижды: (B3) между внешним адаптером (U2) и последовательностью J-сегмента (J), так чтобы короткое одностороннее прочтение с использованием стандартных праймеров Illumina или Ion позволяло идентифицировать уникальную комбинацию последовательностей V и J в каждом матричном олигонуклеотиде, (B2) между V- и J-сегментами, так чтобы стандартная стратегия секвенирования (например, Illumina GA-2, или HiSeq™, или MiSEQ®) захватила уникальную комбинацию последовательностей V и J в каждом матричном олигонуклеотиде, и (B3) между V-сегментом и другим внешним адаптером (U1), так чтобы при необходимости короткое парное двухстороннее прочтение могло подтвердить идентичность уникальной комбинации последовательностей V и J в каждом матричном олигонуклеотиде.
Как показано на Фиг. 1, матричные олигонуклеотидные последовательности начинались с последовательности адаптера (U1), которая способна обеспечить встраивание специфических для используемой секвенирующей платформы коротких олигонуклеотидных последовательностей на концах молекулы. В данном примере использовали адаптеры Illumina Nextera™, однако следует отметить, что по существу любая пара устойчивых праймеров для ПЦР будет работать так же хорошо. В качестве примера адаптера на конце U1 со стороны V-сегмента добавили олигонуклеотидную последовательность GCCTTGCCAGCCCGCTCAG [SEQ ID NO: 1746] (Фиг. 1) для поддержания совместимости с адаптером Nextera™ Illumina (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) (CAAGCAGAAGACGGCATACGAGATCGGTCTGCCTTGCCAGCCCGCTCAG) [SEQ ID NO: 1747] для добавления стандартного олигонуклеотида Illumina, который был совместим с проточными ячейками Illumina как для одностороннего, так и для парного двухстороннего секвенирования.
Непосредственно после (в 3’ относительно) U1 находилась первая копия (B1) олигонуклеотида штрихкода ACACACGTGACACTCT [SEQ ID NO: 1748]. Затем в матричный олигонуклеотид встраивали отрезок фиксированной длины последовательности V-сегмента, причем все матрицы в наборе матриц заканчивались на заданное количество оснований до естественной RSS, чтобы имитировать перестройку естественного гена TCR или BCR, имеющую фиксированное количество делеций оснований у V-сегмента. В данном примере исходно удаляли нуль оснований перед RSS. Затем для получения максимальной распознаваемости данных последовательностей все полинуклеотидные последовательности V-сегмента обрезали для удаления частичных кодонов рядом с RSS, так чтобы остаточные последовательности V-сегмента были в рамке со стартовым кодоном. В качестве обеспечивающих разнообразие последовательностей V-сегмента выступали последовательности, показанные в примерах наборов матричных олигонуклеотидов, представленных в списке последовательностей (например, набор V-сегментов TCRB в последовательностях формулы (I) набора матричных олигонуклеотидов TCRB в SEQ ID NO: 1–871; отдельный набор V-сегментов TCRB в последовательностях формулы (I) набора матричных олигонуклеотидов TCRB в SEQ ID NO: 872–1560; набор V-сегментов TCRG в последовательностях формулы (I) набора матричных олигонуклеотидов TCRG в SEQ ID NO: 1561–1630); один пример V-полинуклеотида имел следующую последовательность:
TCTTATTTTCATAGGCTCCATGGATACTGGAATTACCCAGACACCAAAATACCTGGTCACAGCAATGGGGAGTAAAAGGACAATGAAACGTGAGCATCTGGGACATGATTCTATGTATTGGTACAGACAGAAAGCTAAGAAATCCCTGGAGTTCATGTTTTACTACAACTGTAAGGAATTCATTGAAAACAAGACTGTGCCAAATCACTTCACACCTGAATGCCCTGACAGCTCTCGCTTATACCTTCATGTGGTCGCACTGCAGCAAGAAGACTCAGCTGCGTATCTCTGCACCAGCAG [SEQ ID NO: 1749].
В каждый матричный олигонуклеотид встраивали стоп-кодон TGA в рамке на 3’-конце последовательности V-полинуклеотида, чтобы гарантировать, что матричные олигонуклеотидные последовательности не будут рассматриваться как релевантные в том случае, если они контаминировали биологический образец. После стоп-кодона, между V-сегментом и J-сегментом, где обычно находилась бы NDN, встраивали вторую копию последовательности штрихкода B2 (SEQ ID NO: 1748), идентифицирующую V/J. Затем встраивали последовательность сайта распознавания рестрикционного фермента Sal1 (R) GTCGAC; данную последовательность выбрали на основе того, что она в естественных условиях не присутствует ни в одной из геномных последовательностей V- или J-сегмента TCRB, обеспечивая при необходимости возможность специфического разрушения синтетической матрицы, либо для использования в качестве информационного маркера для идентификации синтетических последовательностей. Сайт B3 в данной версии матрицы является пустым.
J-полинуклеотид (J) встраивали как фрагмент фиксированной длины последовательности из сегмента J-гена, отмеряемый от находящейся на фиксированном количестве оснований от естественной RSS точки для имитации естественной перестройки, и в данном примере направленный в интрон J-C. В данном примере из J-сегмента удалили нуль оснований, однако в других конфигурациях матричного олигонуклеотида использовали делецию 5 п. о. для освобождения пространства под штрихкод VJ (B2) в месте слияния V-J, сохраняя общую длину J-сегмента в естественном диапазоне. Пример J-полинуклеотида имел следующую последовательность:
ACTGAAGCTTTCTTTGGACAAGGCACCAGACTCACAGTTGTAGGTAAGACATTTTTCAGGTTCTTTTGCAGATCCGTCACAGGGAAAAGTGGGTCCACAG [SEQ ID NO: 1750].
После полинуклеотида J-сегмента находилась третья копия (B4) олигонуклеотида штрихкода для идентификации V/J (SEQ ID NO: 1748). В примере матричной олигонуклеотидной последовательности последовательность заканчивается второй последовательностью адаптера (U2), которая способна обеспечить встраивание специфических для используемой секвенирующей платформы последовательностей на концах молекулы. Как указано выше, на конце U2 со стороны J-сегмента использовали совместимый с Nextera™ адаптер (CTGATGGCGCGAGGGAGGC) [SEQ ID NO: 1751] для использования с адаптером Nextera™ Illumina (AATGATACGGCGACCACCGAGATCTACACGCCTCCCTCGCGCCATCAG) [SEQ ID NO: 1752] и для обеспечения добавления стандартного олигонуклеотида для секвенирования Illumina, который совместим с проточными ячейками либо для одностороннего, либо для парного двухстороннего секвенирования.
Получили примеры наборов матричных олигонуклеотидов TCRB и TCRG в соответствии с настоящим описанием, которые имели нуклеотидные последовательности, представленные в SEQ ID NO: 1–1630. Наборы матричных нуклеотидов, имеющих последовательности, представленные в SEQ ID NO: 1–871 и 1561–1630, были синтезированы на заказ на основе информации по конфигурации последовательности, описанной в настоящем документе, в компании Integrated DNA Technologies, Inc. (г. Коралвилл, штат Айова, США) с использованием химической системы gBlocks™ Gene Fragments. Набор матричных нуклеотидов, имеющих последовательности, представленные в SEQ ID NO: 872–1560, генерировали с использованием подхода тайлинга при ПЦР, описанного в примере 2.
Матричные олигонуклеотиды TCRB (SEQ ID NO: 1–871). Используя последовательности V- и J-полинуклеотидов TCRB человека, создали набор из 871 матричного олигонуклеотида общей формулы (I) (в которой B3 отсутствует):
5’-U1-B1-V-B2-R-(B3)-J-B4-U2-3’ (I).
Каждый матричный олигонуклеотид состоял из молекулы ДНК размером 495 пар оснований. Последовательности смысловых цепей представлены как SEQ ID NO: 1–871.
Принципиальная схема, на которой представлена конфигурация данного набора матриц, показана на Фиг. 1. По соглашению конфигурация олигонуклеотида на схеме показана в направлении от 5’ к 3’ (слева направо). «V-сегмент» представляет собой последовательность гена, кодирующего вариабельный (V) участок рецептора адаптивной иммунной системы или его комплемент. «J-сегмент» представляет собой последовательность гена, кодирующего соединительный (J) участок рецептора адаптивной иммунной системы или его комплемент, представленный в настоящем документе. U1 и U2 представляют собой первую и вторую универсальные последовательности олигонуклеотида-адаптера, соответственно, которые могут необязательно дополнительно содержать первую и вторую специфические для используемой секвенирующей платформы олигонуклеотидные последовательности, соответственно, которые связаны с и расположены в 5’ относительно первой и второй универсальных последовательностей олигонуклеотида-адаптера. B1, B2 и B4 представляют собой олигонуклеотидные последовательности штрихкода, каждая из которых содержит олигонуклеотидную последовательность штрихкода, содержащую уникальную олигонуклеотидную последовательность, которая уникальным образом идентифицирует в качестве спаренной комбинации (i) уникальную последовательность V-сегмента и (ii) уникальную последовательность J-сегмента; в данном примере B3 отсутствует.
S представляет собой необязательный стоп-кодон, который может находиться в рамке или за пределами рамки на 3’-конце V. R представляет собой необязательный сайт распознавания рестрикционного фермента. В SEQ ID NO: 1-871 адаптеры U1 и U2 включали 19-меры, как описано выше (SEQ ID NO: 1746 и 1751 соответственно), а все идентифицирующие (V+J) последовательности штрихкода (B1, B2, B4) имели длину 16 нуклеотидов и включали стоп-кодон TGA и сайт распознавания рестрикционного фермента Sal1 (GTCGAC).
Матричные олигонуклеотиды TCRB (SEQ ID NO: 872–1560). Был создан второй набор из 689 матричных олигонуклеотидов, в котором в соответствии с общей формулой (I) V и J содержали последовательности V- и J-полинуклеотидов TCRB человека, соответственно, U1 и U2 независимо содержали отдельные сайты обнаружения рестрикционного фермента (R1 и R3), а B1, B3 и B4 независимо отсутствовали, давая общую формулу (II):
R1-V-B2-R2-J-R3 (II),
где B2 представлял собой идентификатор штрихкода из 8 нуклеотидов (например, последовательность штрихкода, представленную в таблице 7); R1, R2 и R3 представляли собой сайты обнаружения рестрикционного фермента EcoR1 (GAATTC), Sal1 (GTCGAC) и Sph1 (GCATGC) соответственно; а V и J представляли собой полинуклеотиды V-участка и J-участка, соответственно, как описано в настоящем документе. Каждый матричный олигонуклеотид состоял из молекулы ДНК размером 239 пар оснований. Последовательности смысловых цепей представлены как SEQ ID NO: 872–1560.
Матричные олигонуклеотиды TCRG (SEQ ID NO: 1561–1630). При использовании последовательности V- и J-полинуклеотидов TCRG человека был создан третий набор из 70 матричных олигонуклеотидов общей формулы (I). Каждый матричный олигонуклеотид состоял из молекулы ДНК размером 495 пар оснований. Последовательности смысловых цепей представлены как SEQ ID NO: 1561–1630. Подробная информация о 70-олигонуклеотидном наборе матриц TCRG (SEQ ID NO: 1561–1630) является типичной и представляет собой следующее.
На основании ранее определенных геномных последовательностей было показано, что локус TCRG человека содержал 14 Vγ-сегментов, каждый из которых имел последовательность RSS и, следовательно, рассматривался как компетентный в отношении перестройки. Данные 14 Vγ-сегментов включали шесть сегментов гена с известной экспрессией, три V-сегмента, которые были классифицированы как имеющие открытые рамки считывания, и пять V-псевдогенов. Vγ-сегменты гена были связаны с пятью Jγ-сегментами гена. Чтобы включить все возможные комбинации генов V+J для 14 V- и 5 J-сегментов, были созданы 70 (5 x 14) матриц, представляющих все возможные комбинации VJ. Каждая матрица соответствовала общей формуле (I) (5’-U1-B1-V-B2-R-(B3)-J-B4-U2-3’) (Фиг. 1) и, таким образом, включала девять секций: универсальный адаптер (U1) размером 19 пар оснований (п. о.), нуклеотидную метку размером 16 п. о., уникальным образом идентифицирующую каждую спаренную комбинацию сегментов V-гена и J-гена (B1), фрагмент специфической последовательности V-гена (V) длиной 300 п. о., стоп-кодон из 3 п. о. (S), другую копию нуклеотидной метки размером 16 п. о. (B2), метку слияния размером 6 п. о., присутствующую во всех молекулах (R), пустое место для B3, фрагмент специфической последовательности J-гена (J) длиной 100 п. о., третью копию нуклеотидной метки размером 16 п. о. (B4) и универсальную последовательность адаптера размером 19 п. о. (U2).
Каждую из 70 матриц (SEQ ID NO: 1561–1630) амплифицировали индивидуально с использованием олигонуклеотидных праймеров (таблица 4; SEQ ID NO: 1732–1745), выполненных с возможностью отжига с универсальными последовательностями адаптера (U1, U2).
Таблица 4. Праймеры для амплификации TCRG
Полученные концентрации каждого продукта амплификации матричного олигонуклеотида определяли количественно с использованием системы для капиллярного электрофореза LabChip GX™ (Caliper Life Sciences, Inc., г. Хопкингтон, штат Массачусетс, США) в соответствии с инструкциями производителя. Частоты вхождения каждой из 70 возможных комбинаций V-J, определенные по результатам секвенирования штрихкодов B1, представлены в таблице 5. 70 амплифицированных препаратов матричных олигонуклеотидов нормировали на стандартную концентрацию и затем смешивали.
Для проверки того, что все 70 матричных олигонуклеотидов присутствовали в по существу эквимолярных концентрациях, пул секвенировали с использованием платформы для секвенирования Illumina HiSeq™ в соответствии с рекомендациями производителя. Вкратце, для встраивания специфических для используемой секвенирующей платформы олигонуклеотидных последовательностей в смешанные матричные олигонуклеотиды создали праймеры с хвостами, которые гибридизировались с универсальными сайтами праймирования (U1, U2) и имели хвосты последовательности адаптера Illumina Nextera™ на 5’-концах. Затем провели ПЦР-реакцию из семи циклов для отжига адаптеров Illumina с матричными олигонуклеотидами. Затем смесь продуктов ПЦР-реакции очистили с использованием гранул Agencourt® AMPure® XP (Beckman Coulter, Inc., г. Фуллертон, штат Калифорния, США) в рекомендованных производителем условиях. Секвенировали по 60 п. о. продуктов ПЦР-реакции с использованием секвенатора Illumina HiSEQ™ (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и проанализировали результаты, оценив частоту вхождения каждой молекулярной метки штрихкода из 16 п. о. (B1).
По расчетам, по существу эквимолярный препарат для набора из 70 различных матричных олигонуклеотидов должен содержать приблизительно 1,4% каждого члена набора, а пороговый допуск для всех типов составлял плюс или минус десять раз по частоте (0,14–14%). Количественное секвенирование показало, что 70 типов модифицированных адаптером матричных олигонуклеотидов в исходном пуле не были представлены равномерно.
Соответственно, проводили коррекцию концентраций индивидуальных матричных олигонуклеотидов и повторную итерацию стадий количественного секвенирования до тех пор, пока каждая молекула не была представлена в пределах порогового допуска по концентрации (0,14–14%).
Таблица 5. Относительное представление (число вхождений указанной комбинации V-J) продуктов амплификации для каждой пары VJ TCRG (14 V x 5 J) в пуле матриц до амплификации
ПРИМЕР 2. ОБНАРУЖЕНИЕ СИСТЕМАТИЧЕСКОЙ ОШИБКИ АМПЛИФИКАЦИИ V-ГЕНА TCRB
В данном примере описано, как набор из 689 матричных олигонуклеотидов TCRB человека общей формулы (I) был собран вместе с помощью тайлинга четырех одноцепочечных олигонуклеотидов длиной 50–90 нуклеотидов каждый для генерации набора матриц, содержащего мишени для гибридизации для всех возможных комбинаций V-J в наборе олигонуклеотидных праймеров, который способен проводить амплификацию последовательностей TCRB человека. Затем набор матричных олигонуклеотидов использовали для характеризации относительной эффективности амплификации набора праймеров для амплификации TCRB V и J.
Синтезировали набор из 689 матричных олигонуклеотидов для TCRB, содержащий полинуклеотидные последовательности, представляющие все возможные продуктивно перестроенные комбинации V и J для цепей TCRB человека с помощью тайлинга четырех одноцепочечных ДНК праймеров вместе в стандартной ПЦР-реакции. Вкратце, создали по два фрагмента из 90 п. о. (один в «прямой» ориентации и один в «обратной» ориентации) для каждого сегмента V-гена TCRB, по одному фрагменту из 90 п. о. (в «обратной» ориентации) для каждого сегмента J-гена TCRB, а также молекулу-линкер (прямой) из 50 п. о. для связывания вместе фрагментов V- и J-гена. Всего создали 52 прямых фрагмента для V и 52 обратных фрагмента для V, 13 обратных фрагментов для J и 689 молекул-линкеров. Два фрагмента по 90 п. о. (один прямой и один обратный), которые соответствовали каждому из сегментов V-гена, имели комплементарную перекрывающуюся последовательность длиной 39 п. о. Один конец каждого обратного фрагмента для V имел комплементарную последовательность длиной 25 п. о., которая перекрывалась с молекулой-линкером длиной 50 п. о. Оставшиеся 25 п. о. в каждой из молекул-линкеров представляли собой последовательность, которая комплементарно перекрывалась с одним концом молекулы для J. Молекулы создавали таким образом, чтобы комплементарные последовательности гибридизировались друг с другом и образовывали двухцепочечную ДНК, с которой Taq-полимераза может связаться и ферментативно достроить молекулу.
В каждой ПЦР-реакции для сборки таких уложенных с помощью тайлинга молекул использовали мастер-микс для проведения мультиплексной ПЦР QIAGEN (QIAGEN, номер по каталогу 206145, Qiagen, г. Валенсия, штат Калифорния, США), 10%-ный Q-раствор (QIAGEN) и четыре одноцепочечные олигонуклеотидные последовательности (две TCRB V, одну TCRB J и линкер, как описано выше). Две внешние молекулы (одну прямую для V и одну обратную для J) добавляли в итоговой концентрации 1 мкМ каждая, тогда как две внутренние молекулы (одну обратную для V и один прямой линкер) добавляли в итоговой концентрации 0,01 мкМ каждая. Использовали следующие настройки термоциклера: 95 °C в течение 15 минут, затем 35 циклов при 94 °C в течение 30 секунд, 59 °C в течение 30 секунд и 72 °C в течение 1 минуты с последующим 1 циклом при 72 °C в течение 10 минут. После синтеза молекулы количественно определяли с помощью системы для капиллярного электрофореза LabChip GX™ (Caliper Life Sciences, Inc., г. Хопкингтон, штат Массачусетс, США) в соответствии с инструкциями производителя и рассчитывали концентрацию (в нг/мкл) в каждой полученной полосе с помощью программного пакета Caliper LabChip GX.
Нуклеотидные последовательности для полученного набора из 689 матричных олигонуклеотидов для TCRB представлены в SEQ ID NO: 872–1560. В SEQ ID NO: 872–1560 каждую различную последовательность V-участка идентифицировали по уникальной последовательности штрихкода из восьми нуклеотидов, как показано в таблице 7. Все 689 матриц нормализовали до стандартной концентрации 25 нг/мкл и затем смешали. Полученный пул использовали для проведения описанных в настоящем документе анализов TCRB для обнаружения смещенного (неравномерного) использования праймеров для амплификации TCRB в ходе амплификации набора из 689 матричных олигонуклеотидов (SEQ ID NO: 872–1560).
Каждая из 689 матриц присутствовала в пуле матричных олигонуклеотидов в концентрации, максимально экспериментально возможно близкой к эквимолярной, и пул использовали в качестве матрицы для ПЦР-реакции амплификации TCRB, используя эквимолярную смесь из 52 праймеров для V-участка TCRB, которые включали совместимую с адаптером Illumina последовательность (SEQ ID NO: 1753–1804, таблица 6), и эквимолярную смесь из 13 праймеров для J-участка TCRB (SEQ ID NO: 1631–1643, таблица 1). Члены пула из 689 матриц амплифицировали с использованием эквимолярного пула из 52 праймеров TCRB VβF (прямых) («пул VF») и эквимолярного пула из 13 праймеров TCRB JβR (обратных) («пул JR»), как показано в таблице 1 (SEQ ID NO: 1631–1695). Полимеразные цепные реакции (ПЦР) (50 мкл каждая) готовили с 1,0 мкМ пула VF (22 нМ для каждого уникального праймера TCRB VβF), 1,0 мкМ пула JR (77 нМ для каждого уникального праймера TCRB JβR), 1 мкМ мастер-микса QIAGEN для мультиплексной ПЦР (QIAGEN, номер по каталогу 206145, Qiagen, г. Валенсия, штат Калифорния, США), 10%-ным Q-раствором (QIAGEN) и 16 нг/мкл геномной ДНК (гДНК). Использовали следующие настройки термоциклирования для термоциклера C100 (Bio-Rad Laboratories, г. Геркулес, штат Калифорния, США): один цикл при 95 °C в течение 15 минут, 25-40 циклов при 94 °C в течение 30 секунд, 59 °C в течение 30 секунд и 72 °C в течение 1 минуты с последующим одним циклом при 72 °C в течение 10 минут. Для анализа миллионов перестроенных локусов TCRβ CDR3 ПЦР для каждой библиотеки проводили в 12–20 лунках. Как указано выше, праймеры для V и J включали хвост, который соответствовал и был совместим с адаптерами Illumina для секвенирования.
Продукты амплификации количественно секвенировали на секвенаторе Illumina HiSeq™. Для каждой молекулы продукта секвенировали участок размером 60 пар оснований, используя стандартные праймеры для секвенирования J (таблица 3), начиная с молекул J. Частоты вхождения каждой последовательности TCRB в продуктах реакции показаны на Фиг. 2, из которой очевидно, что не все последовательности TCRB были амплифицированы в сравнимой степени.
Таблица 6. Праймеры для амплификации TCRB
Таблица 7. Последовательности штрихкода, используемые для идентификации V-участков TCRB в SEQ ID NO: 872–1560
Используя данные, полученные для создания Фиг. 2, как описано выше, оценили способность к перекрестной амплификации (способности проводить амплификацию сегмента V-гена, отличного от того, для которого был конкретно создан праймер на основе комплементарности последовательности для отжига) для каждого праймера для амплификации, выполненного с возможностью отжига с конкретным сегментом V-гена. Приготовили 52 независимых пула праймеров для амплификации, где каждый пул праймеров содержал 51 из 52 праймеров V-участка TCRB из таблицы 6, смешанных в эквимолярных концентрациях, а 52-й праймер V-участка TCRB присутствовал в пуле в молярной концентрации, вдвое превышающей концентрацию других из 51 праймера. Отдельный пул праймеров для амплификации готовили так, что для каждого из 52 праймеров для V-участка имелся один пул, в котором один праймер присутствовал в молярной концентрации, вдвое превышающей концентрацию остальных праймеров, что позволило получить 52 уникальных пула праймеров. Затем подготовили 52 отдельные реакции амплификации, по одной для каждого уникального пула праймеров для амплификации, используя в каждой реакции описанный выше набор из 689 матричных олигонуклеотидов (SEQ ID NO: 872–1560). Матричные олигонуклеотиды присутствовали в эквимолярных концентрациях относительно друг друга. Амплификацию и секвенирование проводили с использованием описанных выше условий. Результаты показаны на Фиг. 3.
На Фиг. 3 черными квадратами указано отсутствие изменений в степени амплификации с праймером, специфическим для соответствующего указанного V-участка TCRB, присутствующим в двойной концентрации по отношению к эквимолярным концентрациям всех других праймеров; а белыми квадратами указано 10-кратное усиление амплификации; серыми квадратами указаны промежуточные степени (в градациях серого) амплификации от нуля до 10-кратной степени. Диагональная линия указывает на то, что удвоение молярной концентрации для заданного праймера приводило к приблизительно 10-кратному усилению амплификации соответствующего матричного олигонуклеотида, имеющего специфическую целевую последовательность для отжига, в случае большинства протестированных праймеров для V-участка TCRB. Белые квадраты вне диагонали указывали на несоответствующие матрицы, с которыми определенные праймеры были способны к отжигу и амплификации.
Если один или более праймеров демонстрировали потенциал амплификации, который был значительно выше или ниже приемлемого диапазона потенциала амплификации (например, указанного диапазона равномерного потенциала амплификации), проводили дополнительную коррекцию концентраций индивидуальных олигонуклеотидов праймеров и повторение стадий амплификации матриц и количественного секвенирования до тех пор, пока каждый тип молекул продукта не начинал присутствовать в продукте в требуемом диапазоне, что указывало на коррекцию неравномерности потенциала амплификации среди праймеров в наборе праймеров для амплификации.
Соответственно, концентрации праймеров корректировали, как указано в таблице 6, для определения того, можно ли уменьшить степень серьезности систематической ошибки амплификации, показанной на Фиг. 2 и 3, путем увеличения или снижения относительного присутствия высокоэффективных и низкоэффективных праймеров для амплификации соответственно. В мультиплексной ПЦР с использованием скорректированного набора праймеров последовательности праймеров для V-гена были такими же (последовательности, указанные в таблице 6), однако относительные концентрации каждого праймера либо увеличивали, если праймер проводил амплификацию своей матрицы с недостаточной эффективностью (Фиг. 3), либо уменьшали, если праймер проводил амплификацию своей матрицы с избыточной эффективностью (Фиг. 3). Затем скорректированную смесь праймеров для амплификации использовали в ПЦР для амплификации матричной композиции, содержащей в эквимолярных количествах набор из 689 матричных олигонуклеотидов (SEQ ID NO: 872–1560), которые использовали для получения данных, представленных на Фиг. 2 и 3.
Амплификацию и количественное секвенирование провели, как описано выше, результаты показаны на Фиг. 4, на которой представлено сравнение частоты, с которой был получен каждый амплифицированный продукт, содержащий указанную последовательность V-участка, когда все праймеры для амплификации присутствовали в эквимолярных концентрациях (черные полоски), с частотой, с которой был получен каждый продукт после коррекции концентраций праймеров для амплификации (серые полоски) до концентраций, указанных в таблице 6.
Дополнительные последовательности праймеров для hs-TCRB указаны в SEQ ID NO: 6192–6264.
ПРИМЕР 3. КОРРЕКЦИЯ НЕРАВНОМЕРНОГО ПОТЕНЦИАЛА АМПЛИФИКАЦИИ (СИСТЕМАТИЧЕСКОЙ ОШИБКИ ПЦР) В НАБОРАХ ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ АМПЛИФИКАЦИИ TCR
Обеспечивающие разнообразие праймеры для амплификации TCR выполнены с возможностью амплификации каждой возможной комбинации перестроенных сегментов V- и J-гена TCR в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта. Препарат, содержащий эквимолярные концентрации обеспечивающих разнообразие праймеров для амплификации, используют в мультиплексной ПЦР для амплификации обеспечивающей разнообразие матричной композиции, которая содержит эквимолярные концентрации специфических для TCR матричных олигонуклеотидов в соответствии с формулой (I), причем по меньшей мере одна матрица представляет каждую возможную комбинацию V-J для локуса TCR. Продукты амплификации количественно секвенируют и получают частоту вхождения каждой последовательности продукта с уникальной V-J из частоты вхождения каждой последовательности молекулярного штрихкода из 16 п. о. (B в формуле (I)), которая уникальным образом идентифицирует каждую комбинацию V-J.
В случае TCRG проводят амплификацию матричных олигонуклеотидов для TCRG (SEQ ID NO: 1561–1630), используя праймеры, специфические для V и J TCRG (SEQ ID NO: 1732–1745, таблица 4). Независимость праймера для J от соответствующих спаренных праймеров для V идентифицируют путем отдельной амплификации каждого из восьми праймеров, специфических для сегмента V-гена TCRG, с пулом из пяти праймеров, специфических для сегмента J-гена. Продукты амплификации количественно секвенируют на секвенирующей платформе Illumina HiSeq™, и частота вхождения внутренних последовательностей штрихкода из 16 п. о. (B), которые уникальным образом идентифицируют конкретные комбинации V-J, позволяет количественно определить каждую пару V-J. Независимость праймера для V от соответствующих спаренных праймеров для J идентифицируют путем проведения обратной реакции, т. е. путем независимой амплификации каждого из пяти праймеров для сегмента J-гена TCRG с пулом из восьми праймеров, специфических для сегмента V-гена.
Для проверки возможности перекрестной амплификации праймеров для V или праймеров для J TCRG (например, проверки неспецифической амплификации праймерами, специфическими для заданного сегмента гена, например, для проверки того, способен ли праймер для V, разработанный с возможностью специфической амплификации сегментов V7 TCRG, проводить амплификацию сегментов V-гена как TCRG V6, так и TCRG V7) готовят независимые пулы праймеров, которые содержат эквимолярные концентрации всех праймеров, кроме одного, а затем отсутствующий праймер добавляют в пул в молярной концентрации, вдвое превышающей концентрацию всех других праймеров. Затем праймеры используют для амплификации матричной композиции, которая содержит множество матричных олигонуклеотидов общей формулы (I), как описано в настоящем документе, с использованием последовательностей V- и J-генов TCRG, соответственно, в V- и J-полинуклеотидах формулы (I). Используя количественное секвенирование, идентифицируют любую одну или более матриц, которые избыточно представлены среди продуктов амплификации, когда один праймер для амплификации присутствует в пуле праймеров в концентрации, вдвое превышающей концентрацию всех других праймеров. Затем смесь праймеров корректируют, увеличивая или уменьшая относительные концентрации одного или более праймеров, чтобы в процессе итерации получить частоты амплификации в пределах приемлемых количественных допусков. Скорректированную таким образом смесь праймеров рассматривают как скорректированную для снижения неравномерности потенциала амплификации среди членов набора праймеров.
Для определения того, демонстрирует ли скорректированная смесь праймеров свободный от систематической ошибки потенциал амплификации при использовании для амплификации перестроенных матричных ДНК TCR в биологическом образце из лимфоидных клеток субъекта, готовят искусственные матричные композиции, как описано в настоящем документе, содержащие все пары VJ, присутствующие с аналогичными частотами, а также с варьируемыми соотношениями относительного представления определенных пар VJ. Каждый тип матричного препарата отдельно тестируют в качестве матрицы для амплификации с набором праймеров для амплификации, который был скорректирован для снижения неравномерности потенциала амплификации среди определенных членов набора праймеров. Количественное определение последовательностей продуктов амплификации позволяет идентифицировать, что относительное количественное представление конкретных последовательностей в матричном препарате отражается в относительном количественном представлении конкретных последовательностей среди продуктов амплификации.
В качестве альтернативы описанному выше итерационному процессу или в дополнение к таким итерационным стадиям амплификации с последующим количественным секвенированием систематическую ошибку амплификации можно скорректировать численно. В соответствии с данным расчетным подходом исходная частота каждого типа последовательностей матричного олигонуклеотида в синтезированной матричной композиции известна. Частоту каждого из данных типов последовательностей матричного олигонуклеотида среди продуктов амплификации, полученных в результате ПЦР-амплификации, определяют путем количественного секвенирования. Различие между относительными частотами последовательностей матричного олигонуклеотида до ПЦР-амплификации и их частотами после ПЦР-амплификации представляет собой «систематическую ошибку ПЦР». Данное различие представляет собой систематическую ошибку амплификации, внесенную в процессе амплификации, например, вследствие разной эффективности амплификации среди разных праймеров для амплификации.
После количественного определения для каждой известной последовательности матричного олигонуклеотида систематическую ошибку ПЦР для каждого праймера используют для расчета коэффициента систематической ошибки амплификации (нормирующего коэффициента), в соответствии с который выполняют коррекцию наблюдаемой частоты для каждого продукта амплификации для отражения фактической частоты соответствующей матричной последовательности в матричной композиции. Если по результатам эмпирического определения с использованием описанной в настоящем документе матричной композиции систематическая ошибка ПЦР для набора праймеров для амплификации находится в пределах коэффициента 10, то систематическую ошибку можно скорректировать численно для продуктов амплификации, полученных при использовании того же набора праймеров для амплификации при амплификации образца ДНК неизвестной композиции. Посредством этого достигается повышение точности количественного определения разных типов матриц в образце ДНК.
Поскольку эмпирическое тестирование праймеров для V и J показало их независимость, коэффициент систематической ошибки амплификации можно получить независимо для каждого типа V и каждого типа J, и необходимости в коэффициенте амплификации для каждой пары типа VJ нет. Соответственно, для каждого типа V и типа J получают коэффициент систематической ошибки амплификации, используя описанную в настоящем документе матричную композицию. В настоящем способе частоты последовательностей V- и J-генов в матричной композиции известны (или могут быть рассчитаны на основе информации о концентрациях каждого типа матричного олигонуклеотида в синтезированной матричной композиции) до ПЦР-амплификации. После ПЦР-амплификации выполняют количественное секвенирование для обнаружения частоты каждой последовательности сегмента V- и J-гена в продуктах амплификации. Для каждой последовательности различие в частоте сегмента гена представляет собой систематическую ошибку амплификации.
Частота начального продукта/частота конечного продукта = коэффициент систематической ошибки амплификации.
Коэффициенты систематической ошибки амплификации рассчитывают для каждого сегмента V-гена и каждого сегмента J-гена. Рассчитанные таким образом коэффициенты амплификации можно применять к образцам, у которых исходная частота V- и J-генов неизвестна.
В смешанной матричной популяции (такой как сложный образец ДНК, полученный из биологического источника, содержащего ДНК из лимфоидных клеток, которые, предположительно, содержат перестроенные ДНК, кодирующие рецепторы адаптивной иммунной системы, или сложный образец ДНК, который дополнительно содержит ДНК из других клеток, где такой перестройки не происходит), если исходная частота каждого сегмента V- и J-генов неизвестна, для коррекции остаточной систематической ошибки ПЦР-амплификации можно использовать вычисленные коэффициенты амплификации для набора праймеров, который был предварительно охарактеризован с использованием матричных композиций, описанных в настоящем документе. Для каждого типа молекул секвенированных продуктов амплификации используемые молекулой V- и J-гены определяют на основе сходства последовательностей. Для коррекции систематической ошибки амплификации количество появлений молекулы при секвенировании умножают на скорректированные коэффициенты амплификации как для V, так и для J. Полученное число для последовательности представляет собой численно «нормированный» набор.
ПРИМЕР 4. СОЗДАНИЕ ДОПОЛНИТЕЛЬНЫХ МАТРИЧНЫХ КОМПОЗИЦИЙ
В соответствии с по существу описанными выше методологиями создали и получили дополнительные матричные композиции.
V- и J-полинуклеотиды. Для включения в описанное в настоящем документе множество матричных олигонуклеотидов создали последовательности V- и J-полинуклеотидов TCRB, которые собраны в наборы из 68 последовательностей TCRB V и J SEQ ID NO соответственно, как показано на Фиг. 5a–5l как TCRB V/J набор 1, TCRB V/J набор 2, TCRB V/J набор 3, TCRB V/J набор 4, TCRB V/J набор 5, TCRB V/J набор 6, TCRB V/J набор 7, TCRB V/J набор 8, TCRB V/J набор 9, TCRB V/J набор 10, TCRB V/J набор 11, TCRB V/J набор 12 и TCRB V/J набор 13.
Для включения в описанное в настоящем документе множество матричных олигонуклеотидов создали последовательности V- и J-полинуклеотидов TCRG, которые собраны в наборы из 14 последовательностей TCRG V и J SEQ ID NO соответственно, как показано на Фиг. 6a–6b как TCRG V/J набор 1, TCRG V/J набор 2, TCRG V/J набор 3, TCRG V/J набор 4 и TCRG V/J набор 5.
Для включения в описанное в настоящем документе множество матричных олигонуклеотидов создали последовательности полинуклеотидов IGH V и J, которые собраны в наборы из 127 IGH V и J SEQ ID NO соответственно, как показано на Фиг. 7a–7m как IGH V/J набор 1, IGH V/J набор 2, IGH V/J набор 3, IGH V/J набор 4, IGH V/J набор 5, IGH V/J набор 6, IGH V/J набор 7, IGH V/J набор 8 и IGH V/J набор 9.
Матричные композиции. Приготовили матричную композицию для стандартизации эффективности амплификации наборов праймеров для амплификации TCRB. Композиция содержала множество матричных олигонуклеотидов, имеющих множество олигонуклеотидных последовательностей общей формулы (I). Матричная композиция TCRB, содержащая 858 различных матричных олигонуклеотидов, представлена в списке последовательностей в SEQ ID NO: 3157-4014.
Приготовили матричную композицию для стандартизации эффективности амплификации наборов праймеров для амплификации TCRG. Композиция содержала множество матричных олигонуклеотидов, имеющих множество олигонуклеотидных последовательностей общей формулы (I). Матричная композиция для TCRG, содержащая 70 различных матричных олигонуклеотидов, представлена в списке последовательностей в SEQ ID NO: 4015-4084.
Приготовили матричную композицию для стандартизации эффективности амплификации наборов праймеров для амплификации IGH. Композиция содержала множество матричных олигонуклеотидов, имеющих множество олигонуклеотидных последовательностей общей формулы (I). Матричная композиция для IGH, содержащая 1116 различных матричных олигонуклеотидов, представлена в списке последовательностей в SEQ ID NO: 4085-5200. Матричная композиция для полинуклеотида IGH, содержащая набор из 1116 матричных олигонуклеотидов, также представлена в списке последовательностей в SEQ ID NO: 1805–2920.
ПРИМЕР 5. ИСПОЛЬЗОВАНИЕ МАТРИЧНОЙ КОМПОЗИЦИИ ДЛЯ ОПРЕДЕЛЕНИЯ КОЭФФИЦИЕНТА АМПЛИФИКАЦИИ
В данном примере описано количественное определение перестроенных молекул ДНК, кодирующих множество молекул IG, с использованием описанной в настоящем документе композиции матричных олигонуклеотидов в качестве добавляемой в известном количестве синтетической матрицы, при мультиплексной ПЦР-амплификации образца ДНК, содержащего ДНК B-клеток и фибробластов.
Биологическая матричная ДНК. В качестве источников матричной ДНК использовали восемь биологических образцов, причем каждый биологический образец содержал одно и то же количество общей геномной ДНК (гДНК), равное 300 нг, но разные соотношения (i) ДНК, экстрагированной из B-клеток, и (ii) ДНК, экстрагированной из клеток фибробластов человека, - типа клеток, в котором не происходит перестройка генов, кодирующих IG и TCR. Образцы содержали 0, 0,07, 0,3, 1, 4, 18, 75 или 300 нг B-клеточной гДНК и количество гДНК фибробластов, доводящее количество гДНК в каждом препарате до 300 нг. Каждый образец готовили в четырех повторностях.
Синтетическая матричная ДНК. Для каждой ПЦР-реакции (ниже) добавили по 5000 молекул (4–5 молекул из каждой последовательности) из композиции олигонуклеотидных матриц, содержащей пул из 1116 синтетических матричных олигонуклеотидных молекул IGH (SEQ ID NO: 4085–5200). Матричная композиция для полинуклеотида IGH, содержащая набор из 1116 матричных олигонуклеотидов, также представлена в списке последовательностей как SEQ ID NO: 1805–2920.
ПЦР-реакция. Для ПЦР-реакции использовали мастер-микс QIAGEN для мультиплексной ПЦР QIAGEN Multiplex Plus™ PCR (QIAGEN, номер по каталогу 206152, Qiagen, г. Валенсия, штат Калифорния, США), 10%-ный Q-раствор (QIAGEN) и 300 нг биологической матричной ДНК (описанной выше). Объединенные праймеры для амплификации добавили в таком количестве, что итоговая реакция содержала общую концентрацию прямых праймеров 2 мкМ и общую концентрацию обратных праймеров 2 мкМ. Прямые праймеры (SEQ ID NO: 5201–5286) включали 86 праймеров, имеющих на 3’-конце сегмент длиной приблизительно 20 п. о., который гибридизировался с кодирующей V-сегмент IGH последовательностью, а на 5’-конце — универсальный праймер pGEXf длиной приблизительно 20 п. о. Обратные праймеры (SEQ ID NO: 5287–5293) включали агрегат из специфических для J-сегмента праймеров, имеющих на 3’-конце сегмент длиной приблизительно 20 п. о., который гибридизировался с кодирующей J-сегмент IGH последовательностью, а на 5’-конце праймеров для J находился универсальный праймер pGEXr. Использовали следующие настройки термоциклирования для термоциклера C100 (Bio-Rad Laboratories, г. Геркулес, штат Калифорния, США): один цикл при 95 °C в течение 10 минут, 30 циклов при 94 °C в течение 30 секунд, 63 °C в течение 30 секунд и 72 °C в течение 1 минуты с последующим одним циклом при 72 °C в течение 10 минут. Каждую реакцию проводили в четырех повторностях.
Для секвенирования на концы продуктов ПЦР-реакции встроили адаптеры Illumina (Illumina Inc., г. Сан-Диего, штат Калифорния, США), которые также включали метку длиной 8 п. о. и случайный набор нуклеотидов длиной 6 п. о., используя ПЦР-реакцию из 7 циклов. Использовали описанные выше реагенты и условия ПЦР, за исключением условий термоциклирования, которые составили: 95 °C в течение 5 минут, затем 7 циклов при 95 °C в течение 30 секунд, 68 °C в течение 90 секунд и 72 °C в течение 30 секунд. После термоциклирования реакции выдерживали при 72 °C в течение 10 минут, и праймеры представляли собой праймеры для наращивания адаптера Illumina (SEQ ID NO: 5387–5578). Образцы секвенировали на секвенаторе Illumina MiSEQ™ с использованием праймера Illumina_PE_RD2.
Результаты. Получили данные секвенирования для каждого образца и идентифицировали продукты амплификации синтетических матриц по наличию в них олигонуклеотидной последовательности штрихкода. Для каждого образца количество матричных продуктов разделили на количество различных уникальных последовательностей синтетических матричных олигонуклеотидов (1116), получив коэффициент амплификации для образца. Затем полное количество продуктов амплификации биологических матриц для каждого образца разделили на коэффициент амплификации для вычисления количества перестроенных биологических матричных молекул (например, рекомбинаций VDJ) в исходной реакции амплификации в качестве оценки количества уникальных матриц B-клеточного генома. Построили зависимость средних значений со стандартными отклонениями от известного количества перестроенных биологических матричных молекул, исходя из количества входного B-клеточного материала (Фиг. 9). На Фиг. 9 точки представляют собой средние коэффициенты амплификации, а линии показывают стандартное отклонение по четырем повторностям. Использование коэффициентов амплификации, рассчитанных так, как описано в настоящем документе, для оценки количества перестроенных по VJ молекул, кодирующих IG (в качестве оценки количества B-клеток) дало результаты определения, которые согласовывались с известным количеством B-клеток вплоть до по меньшей мере 100 B-клеток на входе. Значения оцененных коэффициентов амплификации и наблюдаемый коэффициент амплификации показали высокую корреляцию (Фиг. 9, R2 = 0,9988).
ПРИМЕР 6. МАТРИЦЫ ДЛЯ КОНТРОЛЯ СИСТЕМАТИЧЕСКОЙ ОШИБКИ IgH, IgL, и IgK
Матричные олигонуклеотиды для VJ IgH
В одном варианте осуществления создали и проанализировали матричные олигонуклеотиды для VJ IgH. Используя полинуклеотидные последовательности IgH V и J человека, создали набор из 1134 матричных олигонуклеотидов общей формулы (I). Каждый матричный олигонуклеотид состоял из молекулы ДНК размером 495 пар оснований. Конструкции набора из 1134 олигонуклеотидов из матриц IgH типичны и были следующими.
На основании ранее определенных геномных последовательностей было показано, что локус IgH человека содержал 126 Vh-сегментов, каждый из которых имел последовательность RSS и поэтому рассматривался как компетентный в отношении перестройки. Данные 126 Vh-сегментов включали 52 сегмента генов с известной экспрессией, пять V-сегментов, которые были классифицированы как имеющие открытые рамки считывания, и 69 V-псевдогенов. Сегменты Vh-генов были связаны с 9 сегментами Jh-генов. Чтобы включить все возможные комбинации генов V+J для 126 V-сегментов и 9 J-сегментов, создали 1134 (9 x 126) матрицы, представляющие все возможные комбинации VJ. Каждая матрица соответствовала общей формуле (I) (5’-U1-B1-V-B2-R-J-B4-U2-3’) (Фиг. 1) и, таким образом, включала девять секций: универсальный адаптер (U1) размером 19 пар оснований (п. о.), нуклеотидную метку размером 16 п. о., уникальным образом идентифицирующую каждую спаренную комбинацию сегментов V-гена и J-гена (B1), фрагмент специфической последовательности V-гена (V) длиной 300 п. о., стоп-кодон из 3 п. о. (S), вторую копию нуклеотидной метки размером 16 п. о. (B2), метку слияния размером 6 п. о., присутствующую во всех молекулах (R), пустое место в качестве B3, фрагмент специфической последовательности J-гена (J) длиной 100 п. о., третью копию нуклеотидной метки размером 16 п. о. (B4) и универсальную последовательность адаптера размером 19 п. о. (U2). Два V-сегмента представляли собой нуклеотиды, идентичные другим двум V-сегментам, и, таким образом, не были заказаны. Это уменьшило число включенных сегментов с 1134 до 1116. Матричная композиция для IGH, содержащая 1116 различных матричных олигонуклеотидов, представлена в списке последовательностей в SEQ ID NO: 4085–5200.
Каждую из 1116 матриц амплифицировали индивидуально с использованием олигонуклеотидных праймеров, выполненных с возможностью отжига с универсальными последовательностями адаптера (U1, U2). Данные олигонуклеотидные последовательности могут представлять собой любой универсальный праймер. Для данного приложения использовали универсальный праймер с кодом Nextera.
Таблица 8. Универсальные последовательности праймеров, включенных в матрицы для контроля систематической ошибки
Последовательности универсальных праймеров могут гибридизироваться с любой последовательностью праймера, описанной в настоящем документе. Примеры праймеров для ПЦР, включающие последовательность универсального праймера, показаны ниже.
Таблица 9. Примеры праймеров для ПЦР для IGH с универсальными последовательностями (выделены жирным шрифтом и подчеркиванием)
Полученные концентрации каждого продукта амплификации матричного олигонуклеотида определяли количественно с использованием системы для капиллярного электрофореза LabChip GX™ (Caliper Life Sciences, Inc., г. Хопкингтон, штат Массачусетс, США) в соответствии с инструкциями производителя. 1116 амплифицированных препаратов матричных олигонуклеотидов нормировали на стандартную концентрацию и затем смешали.
Для проверки того, что все 1116 матричных олигонуклеотидов присутствовали в по существу эквимолярных концентрациях, пул секвенировали с использованием платформы для секвенирования Illumina MiSeq™ в соответствии с рекомендациями производителя. Для встраивания специфических для используемой секвенирующей платформы олигонуклеотидных последовательностей в смешанные матричные олигонуклеотиды создали праймеры с хвостами, которые гибридизировались с универсальными сайтами праймирования (U1, U2) и имели хвосты последовательности адаптера Illumina™ на 5’-концах. Затем провели ПЦР-реакцию из семи циклов для отжига адаптеров Illumina с матричными олигонуклеотидами. Затем смесь продуктов ПЦР-реакции очистили с использованием гранул Agencourt® AMPure® XP (Beckman Coulter, Inc., г. Фуллертон, штат Калифорния, США) в рекомендованных производителем условиях. Секвенировали первые 29 п. о. продуктов ПЦР-реакции с использованием секвенатора Illumina MiSEQ™ (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и проанализировали результаты, оценив частоту каждой молекулярной метки штрихкода из 16 п. о. (B1).
По расчетам, по существу эквимолярный препарат для набора из 1116 различных матричных олигонуклеотидов должен содержать приблизительно 0,09% каждого члена набора, а желательный пороговый допуск для всех типов продуктов составлял плюс или минус десять раз по частоте (0,009%–0,9%). Количественное секвенирование показало, что 1116 типов модифицированных адаптером матричных олигонуклеотидов в исходном пуле не были представлены равномерно.
Соответственно, проводили коррекцию концентраций индивидуальных матричных олигонуклеотидов и повторную итерацию стадий количественного секвенирования до тех пор, пока каждая молекула не была представлена в пределах допустимого порогового допуска по концентрации (0,009–0,9%).
Матричные олигонуклеотиды для DJ IgH
В другом варианте осуществления создали и проанализировали матричные олигонуклеотиды для DJ IgH. Используя полинуклеотидные последовательности IgH D и J человека, создали набор из 243 матричных олигонуклеотидов общей формулы (I). Каждый матричный олигонуклеотид состоял из молекулы ДНК размером 382 пары оснований. Последовательности матричных олигонуклеотидов для DJ IgH представлены в SEQ ID NO: 5579–5821. Конструкции набора из 243 олигонуклеотидов из матриц IgH типичны и были следующими.
На основании ранее определенных геномных последовательностей было показано, что локус IgH человека содержал 27 Dh-сегментов. Указанные 27 сегментов Dh-генов были связаны с 9 сегментами Jh-генов. Чтобы включить все возможные комбинации генов D+J для 27 D- и 9 J-сегментов, создали 243 (9 x 27) матрицы, представляющие все возможные комбинации DJ. Каждая матрица соответствовала общей формуле (I) (5’-U1-B1-V-B2-R-J-B4-U2-3’) (Фиг. 1) и, таким образом, включала девять секций: универсальный адаптер (U1) размером 19 пар оснований (п. о.), нуклеотидную метку размером 16 п. о., уникальным образом идентифицирующую каждую спаренную комбинацию сегментов D-гена и J-гена (B1). Однако для данных молекул фрагмент специфической последовательности V-гена (V) длиной 300 п. о. заменили на фрагмент специфической последовательности D-гена длиной 182 п. о. Данный сегмент включал как экзонные, так и интронные сегменты нуклеотидов. Как и другие молекулы, они включали стоп-кодон из 3 пар оснований (п. о.) (S), вторую копию нуклеотидной метки размером 16 п. о. (B2), метку слияния размером 6 п. о., присутствующую во всех молекулах (R), пустое место в качестве B3, фрагмент специфической последовательности J-гена (J) длиной 100 п. о., третью копию нуклеотидной метки размером 16 п. о. (B4) и универсальную последовательность адаптера размером 19 п. о. (U2).
Каждую из 243 матриц (SEQ ID NO: 5579–5821) амплифицировали индивидуально с использованием олигонуклеотидных праймеров, выполненных с возможностью отжига с универсальными последовательностями адаптера (U1, U2; см. таблицу 8). Данные олигонуклеотидные последовательности могут представлять собой любой универсальный праймер; для данного применения использовали универсальный праймер с кодом Nextera.
Примеры праймеров для ПЦР со включенными универсальными последовательностями адаптера представлены в таблице 10.
Таблица 10. Примеры праймеров для ПЦР для DJ IgH с универсальными последовательностями (выделены жирным шрифтом и подчеркиванием)
Полученные концентрации каждого продукта амплификации матричного олигонуклеотида определяли количественно с использованием системы для капиллярного электрофореза LabChip GX™ (Caliper Life Sciences, Inc., г. Хопкингтон, штат Массачусетс, США) в соответствии с инструкциями производителя. 243 амплифицированных препарата матричных олигонуклеотидов нормировали на стандартную концентрацию и затем смешали.
Для проверки того, что все 243 матричных олигонуклеотида присутствовали в по существу эквимолярных концентрациях, пул секвенировали с использованием платформы для секвенирования Illumina MiSeq™ в соответствии с рекомендациями производителя. Для встраивания специфических для используемой секвенирующей платформы олигонуклеотидных последовательностей в смешанные матричные олигонуклеотиды создали праймеры с хвостами, которые гибридизировались с универсальными сайтами праймирования (U1, U2) и имели хвосты последовательности адаптера Illumina™ на 5’-концах. Затем провели ПЦР-реакцию из семи циклов для отжига адаптеров Illumina с матричными олигонуклеотидами. Затем смесь продуктов ПЦР-реакции очистили с использованием гранул Agencourt® AMPure® XP (Beckman Coulter, Inc., г. Фуллертон, штат Калифорния, США) в рекомендованных производителем условиях. Секвенировали первые 29 п. о. продуктов ПЦР-реакции с использованием секвенатора Illumina MiSEQ™ (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и проанализировали результаты, оценив частоту каждой молекулярной метки штрихкода из 16 п. о. (B1).
По расчетам, по существу эквимолярный препарат для набора из 243 различных матричных олигонуклеотидов должен содержать приблизительно 0,4% каждого члена набора, а желательный пороговый допуск для всех типов продуктов составлял плюс или минус десять раз по частоте (0,04%–4,0%). Количественное секвенирование показало, что 243 типа модифицированных адаптером матричных олигонуклеотидов в исходном пуле не были представлены равномерно.
Соответственно, проводили коррекцию концентрации индивидуальных матричных олигонуклеотидов и повторную итерацию стадий количественного секвенирования до тех пор, пока каждая молекула не была представлена в пределах допустимого порогового допуска по концентрации (0,04–4,0%). После нормализации данный набор скомбинировали с набором из 1116 олигонуклеотидов для контроля систематической ошибки по IgH VJ, получив пул из 1359 матриц.
На Фиг. 10 показаны результаты для числа повторов при секвенировании до проведения ПЦР-амплификации для каждой из 1116 молекул для контроля систематической ошибки по IGH VJ и 243 молекул для контроля систематической ошибки по IGH DJ. По оси X расположены индивидуальные молекулы для контроля систематической ошибки. Набор включал 1116 молекул для контроля систематической ошибки по IGH VJ и 243 молекулы для контроля систематической ошибки по IGH DJ, итого 1359 г-блоков. По оси Y отложены числа повторов при секвенировании для каждого индивидуального г-блока. Данный расчет позволяет выполнить количественную характеризацию композиции по представлению каждой пары VJ до амплификации. Данные использовали для оценки изменения частоты в образце до и после ПЦР-амплификации для вычисления систематической ошибки амплификации, вносимой праймерами.
Матричные олигонуклеотиды для VJ IgL
В другом варианте осуществления создали и проанализировали матричные олигонуклеотиды для VJ IgL. Используя полинуклеотидные последовательности IgL V и J человека, разработали набор из 245 матричных олигонуклеотидов общей формулы (I). Каждый матричный олигонуклеотид состоял из молекулы ДНК размером 495 пар оснований. Матричные олигонуклеотиды для IgL представлены как SEQ ID NO: 5822–6066. Конструкции набора из 245 олигонуклеотидов из матриц IgL типичны и были следующими.
На основании ранее определенных геномных последовательностей было показано, что локус IgL человека содержал 75 VL-сегментов, каждый из которых имел последовательность RSS и, следовательно, рассматривался как компетентный в отношении перестройки. Данные VL-сегменты включали 33 сегмента генов с известной экспрессией, 5 V-сегментов, которые были классифицированы как имеющие открытые рамки считывания, и 37 V-псевдогенов. Сегменты VL-генов были связаны с 6 сегментами JL-генов. Чтобы включить все возможные функциональные и экспрессируемые комбинации генов V+J для 33 функциональных V-сегментов и 6 J-сегментов, создали 204 (6 x 33) матрицы, представляющие все возможные комбинации VJ. Кроме того, два из V-псевдогенов также находились под вопросом; создали дополнительные 12 (2 x 6) матриц VJ, что позволило получить всего 216 матриц. Каждая матрица соответствовала общей формуле (I) (5’-U1-B1-V-B2-R-J-B4-U2-3’) (Фиг. 1) и, таким образом, включала девять секций: универсальный адаптер (U1) размером 19 пар оснований (п. о.), нуклеотидную метку размером 16 п. о., уникальным образом идентифицирующую каждую спаренную комбинацию сегментов V-гена и J-гена (B1), фрагмент специфической последовательности V-гена (V) длиной 300 п. о., стоп-кодон из 3 п. о. (S), вторую копию нуклеотидной метки размером 16 п. о. (B2), метку слияния размером 6 п. о., присутствующую во всех молекулах (R), пустое место в качестве B3, фрагмент специфической последовательности J-гена (J) длиной 100 п. о., третью копию нуклеотидной метки размером 16 п. о. (B4) и универсальную последовательность адаптера размером 19 п. о. (U2).
Каждую из 216 матриц амплифицировали индивидуально с использованием олигонуклеотидных праймеров, выполненных с возможностью отжига с универсальными последовательностями адаптера (U1, U2). Данные олигонуклеотидные последовательности могут представлять собой любой универсальный праймер; для данного применения использовали универсальный праймер с кодом Nextera.
Полученные концентрации каждого продукта амплификации матричного олигонуклеотида определяли количественно с использованием системы для капиллярного электрофореза LabChip GX™ (Caliper Life Sciences, Inc., г. Хопкингтон, штат Массачусетс, США) в соответствии с инструкциями производителя. 216 амплифицированных препаратов матричных олигонуклеотидов нормировали на стандартную концентрацию и затем смешали.
Для проверки того, что все 216 матричных олигонуклеотидов присутствовали в по существу эквимолярных концентрациях, пул секвенировали с использованием платформы для секвенирования Illumina MiSeq™ в соответствии с рекомендациями производителя. Для встраивания специфических для используемой секвенирующей платформы олигонуклеотидных последовательностей в смешанные матричные олигонуклеотиды создали праймеры с хвостами, которые гибридизировались с универсальными сайтами праймирования (U1, U2) и имели хвосты последовательности адаптера Illumina™ на 5’-концах. Затем провели ПЦР-реакцию из семи циклов для отжига адаптеров Illumina с матричными олигонуклеотидами. Затем смесь продуктов ПЦР-реакции очистили с использованием гранул Agencourt® AMPure® XP (Beckman Coulter, Inc., г. Фуллертон, штат Калифорния, США) в рекомендованных производителем условиях. Секвенировали первые 29 п. о. продуктов ПЦР-реакции с использованием секвенатора Illumina MiSEQ™ (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и проанализировали результаты, оценив частоту каждой молекулярной метки штрихкода из 16 п. о. (B1).
По расчетам, по существу эквимолярный препарат для набора из 216 различных матричных олигонуклеотидов должен содержать приблизительно 0,46% каждого члена набора, а желательный пороговый допуск для всех типов продуктов составлял плюс или минус десять раз по частоте (0,046–4,6%). Количественное секвенирование показало, что 216 типов модифицированных адаптером матричных олигонуклеотидов в исходном пуле были представлены равномерно.
Матричные олигонуклеотиды для VJ IgK
В одном варианте осуществления создали и проанализировали матричные олигонуклеотиды для VJ IgK. Используя полинуклеотидные последовательности IgK V и J человека, создали набор из 560 матричных олигонуклеотидов общей формулы (I). Каждый матричный олигонуклеотид состоял из молекулы ДНК размером 495 пар оснований. Примеры матричных олигонуклеотидов для IgK представлены в SEQ ID NO: 6067-6191. Конструкции набора из 560 олигонуклеотидов из матриц IgK типичны и были следующими.
На основе ранее определенных геномных последовательностей было показано, что локус IgK человека содержал 112 Vk-сегментов, каждый из которых имел последовательность RSS и, следовательно, рассматривался как компетентный в отношении перестройки. Данные 112 Vk-сегментов включали 46 сегментов генов с известной экспрессией, 8 V-сегментов, которые были классифицированы как имеющие открытые рамки считывания, и 50 V-псевдогенов. Для данной цепи IgK анализировали только экспрессируемые перестройки VJ IgK. Гены, классифицированные как псевдогены и открытые рамки считывания, были исключены. Сегменты Vk-генов были связаны с пятью сегментами Jk-генов. Это дало 230 перестроек VJ-генов (46 x 5). Чтобы включить все возможные функциональные комбинации V+J-генов для 46 функциональных V-сегментов и 5 J-сегментов, создали 230 (5 x 46) матриц, представляющих все возможные комбинации VJ. Каждая матрица соответствовала общей формуле (I) (5’-U1-B1-V-B2-R-J-B4-U2-3’) (Фиг. 1) и, таким образом, включала девять секций: универсальный адаптер (U1) размером 19 пар оснований (п. о.), нуклеотидную метку размером 16 п. о., уникальным образом идентифицирующую каждую спаренную комбинацию сегментов V-гена и J-гена (B1), фрагмент специфической последовательности V-гена (V) длиной 300 п. о., стоп-кодон из 3 п. о. (S), вторую копию нуклеотидной метки размером 16 п. о. (B2), метку слияния размером 6 п. о., присутствующую во всех молекулах (R), пустое место в качестве B3, фрагмент специфической последовательности J-гена (J) длиной 100 п. о., третью копию нуклеотидной метки размером 16 п. о. (B4) и универсальную последовательность адаптера размером 19 п. о. (U2).
Каждую из 230 матриц амплифицировали индивидуально с использованием олигонуклеотидных праймеров, выполненных с возможностью отжига с универсальными последовательностями адаптера (U1, U2). Данные олигонуклеотидные последовательности могут представлять собой любой универсальный праймер; для данного применения использовали универсальный праймер с кодом Nextera.
Полученные концентрации каждого продукта амплификации матричного олигонуклеотида определяли количественно с использованием системы для капиллярного электрофореза LabChip GX™ (Caliper Life Sciences, Inc., г. Хопкингтон, штат Массачусетс, США) в соответствии с инструкциями производителя. 230 амплифицированных препаратов матричных олигонуклеотидов нормировали на стандартную концентрацию и затем смешали.
Для проверки того, что все 230 матричных олигонуклеотидов присутствовали в по существу эквимолярных концентрациях, пул секвенировали с использованием платформы для секвенирования Illumina MiSeq™ в соответствии с рекомендациями производителя. Вкратце, для встраивания специфических для используемой секвенирующей платформы олигонуклеотидных последовательностей в смешанные матричные олигонуклеотиды создали праймеры с хвостами, которые гибридизировались с универсальными сайтами праймирования (U1, U2) и имели хвосты последовательности адаптера Illumina™ на 5’-концах. Затем провели ПЦР-реакцию из семи циклов для отжига адаптеров Illumina с матричными олигонуклеотидами. Затем смесь продуктов ПЦР-реакции очистили с использованием гранул Agencourt® AMPure® XP (Beckman Coulter, Inc., г. Фуллертон, штат Калифорния, США) в рекомендованных производителем условиях. Секвенировали первые 29 п. о. продуктов ПЦР-реакции с использованием секвенатора Illumina MiSEQ™ (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и проанализировали результаты, оценив частоту каждой молекулярной метки штрихкода из 16 п. о. (B1).
По расчетам, по существу эквимолярный препарат для набора из 230 различных матричных олигонуклеотидов должен содержать приблизительно 0,4% каждого члена набора, а желательный пороговый допуск для всех типов продуктов составлял плюс или минус десять раз по частоте (4,0%–0,04%). Количественное секвенирование показало, что 230 типов модифицированных адаптером матричных олигонуклеотидов в исходном пуле были представлены равномерно.
ПРИМЕР 7. КОМБИНИРОВАННЫЕ АНАЛИЗЫ
Комбинированный анализ для IgH DJ и IgH VJ
В некоторых вариантах осуществления желательно проводить совместную амплификацию и секвенирование перестроенных цепей IgH VDJ CDR3 и перестроенных цепей IgH DJ. Для создания пула матриц для проведения комбинированного анализа цепей IgH DJ и IgH VJ с использованием матриц для DJ IgH и VJ IgH. После смешивания итоговый пул включает 1116 матриц для VJ и 243 матриц для DJ, что составляет в итоге 1359 индивидуальных матриц. Матричная композиция для VJ IgH, содержащая 1116 различных матричных олигонуклеотидов, представлена в списке последовательностей в SEQ ID NO: 4085–5200. Последовательности матричных олигонуклеотидов для DJ IgH представлены в SEQ ID NO: 5579–5821.
Для проверки того, что все 1359 матричных олигонуклеотидов присутствовали в по существу эквимолярных концентрациях, пул секвенировали с использованием платформы для секвенирования Illumina MiSeq™ в соответствии с рекомендациями производителя. Для встраивания специфических для используемой секвенирующей платформы олигонуклеотидных последовательностей в смешанные матричные олигонуклеотиды создали праймеры с хвостами, которые гибридизировались с универсальными сайтами праймирования (U1, U2) и имели хвосты последовательности адаптера Illumina™ на 5’-концах. Затем провели ПЦР-реакцию из семи циклов для отжига адаптеров Illumina с матричными олигонуклеотидами. Затем смесь продуктов ПЦР-реакции очистили с использованием гранул Agencourt® AMPure® XP (Beckman Coulter, Inc., г. Фуллертон, штат Калифорния, США) в рекомендованных производителем условиях. Секвенировали первые 29 п. о. продуктов ПЦР-реакции с использованием секвенатора Illumina MiSEQ™ (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и проанализировали результаты, оценив частоту каждой молекулярной метки штрихкода из 16 п. о. (B1).
По расчетам, по существу эквимолярный препарат для набора из 1359 различных матричных олигонуклеотидов должен содержать приблизительно 0,073% каждого члена набора, а желательный пороговый допуск для всех типов продуктов составлял плюс или минус десять раз по частоте (0,73%–0,0073%). Количественное секвенирование показало, что 1359 типов модифицированных адаптером матричных олигонуклеотидов в исходном пуле были представлены равномерно.
Комбинированный анализ для IgL и IgK
В других вариантах осуществления желательно проводить совместную амплификацию и секвенирование перестроенных цепей IgL и IgK CDR3. Для создания пула матриц для проведения комбинированного анализа для IgL и IgK (комбинировали матрицы для IgL и IgK). После смешивания итоговый пул включает 216 матриц для IgL и 230 матриц для IgK, что в итоге составляет 446 индивидуальных матриц. Матричные олигонуклеотиды для IgL представлены как SEQ ID NO: 5822–6066.
Для проверки того, что все 446 матричных олигонуклеотидов присутствовали в по существу эквимолярных концентрациях, пул секвенировали с использованием платформы для секвенирования Illumina MiSeq™ в соответствии с рекомендациями производителя. Вкратце, для встраивания специфических для используемой секвенирующей платформы олигонуклеотидных последовательностей в смешанные матричные олигонуклеотиды создали праймеры с хвостами, которые гибридизировались с универсальными сайтами праймирования (U1, U2) и имели хвосты последовательности адаптера Illumina™ на 5’-концах. Затем провели ПЦР-реакцию из семи циклов для отжига адаптеров Illumina с матричными олигонуклеотидами. Затем смесь продуктов ПЦР-реакции очистили с использованием гранул Agencourt® AMPure® XP (Beckman Coulter, Inc., г. Фуллертон, штат Калифорния, США) в рекомендованных производителем условиях. Секвенировали первые 29 п. о. продуктов ПЦР-реакции с использованием секвенатора Illumina MiSEQ™ (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и проанализировали результаты, оценив частоту каждой молекулярной метки штрихкода из 16 п. о. (B1).
По расчетам, по существу эквимолярный препарат для набора из 446 различных матричных олигонуклеотидов должен содержать приблизительно 0,22% каждого члена набора, а желательный пороговый допуск для всех типов продуктов составлял плюс или минус десять раз по частоте (2,2%–0,022%). Количественное секвенирование показало, что 446 типов модифицированных адаптером матричных олигонуклеотидов в исходном пуле были представлены равномерно.
ПРИМЕР 8. КОРРЕКЦИЯ НЕРАВНОМЕРНОГО ПОТЕНЦИАЛА АМПЛИФИКАЦИИ (СИСТЕМАТИЧЕСКОЙ ОШИБКИ ПЦР) В НАБОРЕ ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ АМПЛИФИКАЦИИ IGH
Обеспечивающие разнообразие праймеры для амплификации IgH выполнили с возможностью амплификации каждой возможной комбинации перестроенных сегментов V- и J-гена IgH в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта. Препарат, содержащий эквимолярные концентрации обеспечивающих разнообразие праймеров для амплификации, использовали в мультиплексной ПЦР для амплификации обеспечивающей разнообразие матричной композиции, которая содержит эквимолярные концентрации специфических для IgH матричных олигонуклеотидов в соответствии с формулой (I), причем по меньшей мере одна матрица представляет каждую возможную комбинацию V-J для локуса IgH. Продукты амплификации количественно секвенировали и получили частоту вхождения каждой последовательности продукта с уникальным V-J из частоты вхождения каждой последовательности молекулярного штрихкода из 16 п. о. (B в формуле (I)), которая уникальным образом идентифицировала каждую комбинацию V-J.
Мультиплексная ПЦР-реакция выполнялась с возможностью амплификации всех возможных перестроек V- и J-гена локуса IgH в соответствии с аннотацией коллаборации IMGT. См. Yousfi Monod M, Giudicelli V, Chaume D, Lefranc. MP. IMGT/JunctionAnalysis: the first tool for the analysis of the immunoglobulin and T cell receptor complex V-J and V-D-J JUNCTIONs. Bioinformatics. 2004;20(доп. 1):i379–i385. Локус включал 126 уникальных V-генов; 52 функциональных гена, 6 предполагаемых открытых рамок считывания с отсутствующими критическими для функционирования аминокислотами и 69 псевдогенов; а также 9 J-генов, 6 функциональных генов и 3 псевдогена. Целевые последовательности для отжига с праймером для некоторых V-сегментов были идентичны, что позволило проводить амплификацию всех 126 V-сегментов с использованием 86 уникальных прямых праймеров. Аналогичным образом 7 уникальных обратных праймеров гибридизировались со всеми 9 J-генами. В качестве базы для оценки систематической ошибки провели амплификацию пула из 1116 матриц с использованием эквимолярного пула из 86 прямых праймеров для V (VF; специфических для V-генов) и эквимолярного пула из 7 обратных праймеров для J (JR; специфических для J-генов).
Полимеразные цепные реакции (ПЦР) (25 мкл каждая) готовили с 2,0 мкМ пула VF, 2,0 мкМ пула JR (Integrated DNA Technologies), 1 мкМ мастер-микса QIAGEN для мультиплексной ПЦР QIAGEN Multiplex Plus PCR (Qiagen, г. Валенсия, штат Калифорния, США), 10%-ным Q-раствором (QIAGEN) и 200000 целевых молекул из смеси с синтетическим репертуаром IgH. Использовали следующие настройки термоциклирования для термоциклера C100 (Bio-Rad Laboratories, г. Геркулес, штат Калифорния, США): один цикл при 95 °C в течение 6 минут, 31 цикл при 95 °C в течение 30 секунд, 64 °C в течение 120 секунд и 72 °C в течение 90 секунд с последующим одним циклом при 72 °C в течение 3 минут. Для всех экспериментов каждую ПЦР проводили в трех повторностях.
После первичной оценки систематической ошибки провели эксперименты по определению характеристик амплификации всех индивидуальных праймеров. Для определения специфичности праймеров VF и JR приготовили 86 смесей, содержащих один праймер VF и все праймеры JR, а также 7 смесей, содержащих один праймер JR и все праймеры VF. Данные наборы праймеров использовали для амплификации синтетической матрицы и полученные библиотеки секвенировали для измерения специфичности каждого праймера для целевых сегментов V- или J-гена, а также для идентификации случаев нецелевого праймирования. Провели эксперименты по титрованию с использованием пулов с двукратными и четырехкратными концентрациями каждого индивидуального праймера VF или JF при эквимолярном содержании всех других праймеров (например, 2x IgHV1-01+ эквимолярные количества всех других праймеров VF и JR) для определения масштабирующих коэффициентов, связывающих концентрацию праймера с наблюдаемой частотой матрицы.
Оптимизация смеси праймеров
Используя масштабирующие коэффициенты, полученные при титровании праймеров по одному, разработали альтернативные смеси праймеров, в которых для сведения к минимуму систематической ошибки амплификации праймеры комбинировали в неравных концентрациях. Затем уточненные смеси праймеров использовали для амплификации пула матриц и измерения остаточной систематической ошибки амплификации. Данный процесс повторяли итерационно, повышая или понижая концентрацию каждого праймера соответственно на основе того, было ли представление матриц, амплифицируемых данным праймером, недостаточным или избыточным на предыдущем этапе результатов. На каждой стадии данного итерационного процесса определяли общую степень систематической ошибки амплификации, рассчитывая показатели для динамического диапазона (максимальная систематическая ошибка/минимальная систематическая ошибка) и суммы квадратов (SS, рассчитывается по значениям log (систематическая ошибка)) и итерационно повторяя процесс коррекции концентраций праймеров до тех пор, пока улучшение между итерациями не становилось минимальным. Для оценки устойчивости результатов итоговой оптимизации смеси праймеров и масштабирующих коэффициентов для отклонений от эквимолярных входных концентраций матриц использовали очень неоднородную смесь стандартных матриц для IgH для определения воздействия на результат секвенирования. Итоговая смесь была значительно лучше, чем эквимолярная смесь.
ПРИМЕР 9. КОРРЕКЦИЯ НЕРАВНОМЕРНОГО ПОТЕНЦИАЛА АМПЛИФИКАЦИИ (СИСТЕМАТИЧЕСКОЙ ОШИБКИ ПЦР) В НАБОРЕ ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ АМПЛИФИКАЦИИ TCRB
Обеспечивающие разнообразие праймеры для амплификации TCRB выполнили с возможностью амплификации каждой возможной комбинации перестроенных сегментов V- и J-гена TCRB в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта. Препарат, содержащий эквимолярные концентрации обеспечивающих разнообразие праймеров для амплификации, использовали в мультиплексной ПЦР для амплификации обеспечивающей разнообразие матричной композиции, которая содержит эквимолярные концентрации специфических для TCRB матричных олигонуклеотидов в соответствии с формулой (I), причем по меньшей мере одна матрица представляет каждую возможную комбинацию V-J для локуса TCRB. Продукты амплификации количественно секвенировали и получили частоту вхождения каждой последовательности продукта с уникальным V-J из частоты вхождения каждой последовательности молекулярного штрихкода из 16 п. о. (B в формуле (I)), которая уникальным образом идентифицирует каждую комбинацию V-J.
Мультиплексная ПЦР-реакция выполнялась с возможностью амплификации всех возможных перестроек V- и J-гена локуса TCRB в соответствии с аннотацией коллаборации IMGT. См. Yousfi Monod M, Giudicelli V, Chaume D, Lefranc. MP. IMGT/JunctionAnalysis: the first tool for the analysis of the immunoglobulin and T cell receptor complex V-J and V-D-J JUNCTIONs. Bioinformatics. 2004; 20 (доп. 1):1379–1385. Локус включал 67 уникальных V-генов. Целевые последовательности для отжига с праймером для некоторых V-сегментов были идентичны, что позволило проводить амплификацию всех 67 V-сегментов с использованием 60 уникальных прямых праймеров. Для J-локуса 13 уникальных обратных праймеров гибридизировались с 13 J-генами. В качестве базы для оценки систематической ошибки провели амплификацию пула из 868 матриц с использованием эквимолярного пула из 60 прямых праймеров для V (VF; специфических для V-генов) и эквимолярного пула из 13 обратных праймеров для J (JR; специфических для J-генов). Полимеразные цепные реакции (ПЦР) (25 мкл каждая) готовили с 3,0 мкМ пула VF, 3,0 мкМ пула JR (Integrated DNA Technologies), 1 мкМ мастер-микса QIAGEN для мультиплексной ПЦР QIAGEN Multiplex Plus PCR (Qiagen, г. Валенсия, штат Калифорния, США), 10%-ным Q-раствором (QIAGEN) и 200000 целевых молекул из смеси с синтетическим репертуаром TCRB. Использовали следующие настройки термоциклирования для термоциклера C100 (Bio-Rad Laboratories, г. Геркулес, штат Калифорния, США): один цикл при 95 °C в течение 5 минут, 31 цикл при 95 °C в течение 30 секунд, 62 °C в течение 90 секунд и 72 °C в течение 90 секунд с последующим одним циклом при 72 °C в течение 3 минут. Для всех экспериментов каждую ПЦР проводили в трех повторностях.
После первичной оценки систематической ошибки провели эксперименты по определению характеристик амплификации всех индивидуальных праймеров. Для определения специфичности праймеров VF и JR приготовили 60 смесей, содержащих один праймер VF и все праймеры JR, а также 13 смесей, содержащих один праймер JR и все праймеры VF. Данные наборы праймеров использовали для амплификации синтетической матрицы и полученные библиотеки секвенировали для измерения специфичности каждого праймера для целевых сегментов V- или J-гена, а также для идентификации случаев нецелевого праймирования. Провели эксперименты по титрованию с использованием пулов с двукратными и четырехкратными концентрациями каждого индивидуального праймера VF или JF при эквимолярном содержании всех других праймеров (например, 2x TCRBV07-6 + эквимолярные количества всех других праймеров VF и JR) для определения масштабирующих коэффициентов, связывающих концентрацию праймера с наблюдаемой частотой матрицы.
Оптимизация смеси праймеров
Используя масштабирующие коэффициенты, полученные при титровании праймеров по одному, разработали альтернативные смеси праймеров, в которых для сведения к минимуму систематической ошибки амплификации праймеры комбинировали в неравных концентрациях. Затем уточненные смеси праймеров использовали для амплификации пула матриц и измерения остаточной систематической ошибки амплификации. Данный процесс повторяли итерационно, повышая или понижая концентрацию каждого праймера соответственно на основе того, было ли представление матриц, амплифицируемых данным праймером, недостаточным или избыточным на предыдущем этапе результатов. На каждой стадии данного итерационного процесса определяли общую степень систематической ошибки амплификации, рассчитывая показатели для динамического диапазона (максимальная систематическая ошибка/минимальная систематическая ошибка) и суммы квадратов (SS, рассчитывается по значениям log (систематическая ошибка)) и итерационно повторяя процесс коррекции концентраций праймеров до тех пор, пока улучшение между итерациями не становилось минимальным. Итоговая смесь оказалась по существу лучше, чем эквимолярная смесь праймеров.
На Фиг. 11 показаны итерации TCRB-праймера для синтетических матриц TCRB VJ, представленные в зависимости от относительной систематической ошибки амплификации. Определили относительную систематическую ошибку амплификации для 858 синтетических матриц TCRB VJ до химической коррекции для контроля систематической ошибки (эквимолярные праймеры (черный)), после химической коррекции (оптимизированные праймеры (темно-серый)) и после химической и расчетной коррекции (после расчетной коррекции (светло-серый)). Эквимолярные праймеры давали динамический диапазон 264, межквартильный размах 0,841 и сумму квадратов (логарифмический показатель систематической ошибки) 132. Оптимизированные праймеры давали динамический диапазон 147, межквартильный размах 0,581 и сумму квадратов (логарифмический показатель систематической ошибки) 50,7. Скорректированные праймеры (после расчетной коррекции) давали динамический диапазон 90,8, межквартильный размах 0,248 и сумму квадратов (логарифмический показатель систематической ошибки) 12,8.
ПРИМЕР 10. КОРРЕКЦИЯ НЕРАВНОМЕРНОГО ПОТЕНЦИАЛА АМПЛИФИКАЦИИ (СИСТЕМАТИЧЕСКОЙ ОШИБКИ ПЦР) В КОМБИНИРОВАННЫХ НАБОРАХ ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ АМПЛИФИКАЦИИ VJ И DJ IGH
Обеспечивающие разнообразие праймеры для амплификации IgH выполнили с возможностью амплификации каждой возможной комбинации перестроенных сегментов V- и J-гена IgH и сегментов D- и J-гена IgH в биологическом образце, содержащем ДНК из лимфоидных клеток субъекта. Препарат, содержащий эквимолярные количества обеспечивающих разнообразие праймеров для амплификации, использовали в мультиплексной ПЦР для амплификации обеспечивающей разнообразие матричной композиции, которая содержит эквимолярные концентрации специфических для IgH матричных олигонуклеотидов в соответствии с формулой (I), причем по меньшей мере одна матрица представляет каждую возможную комбинацию V-J для локуса IgH и каждую возможную комбинацию D-J для локуса IgH. Продукты амплификации количественно секвенировали и получили частоту вхождения каждой последовательности продукта с уникальными V-J и D-J из частоты вхождения каждой последовательности молекулярного штрихкода из 16 п. о. (B в формуле (I)), которая уникальным образом идентифицирует каждую комбинацию V-J и D-J.
Мультиплексная ПЦР-реакция выполнялась с возможностью амплификации всех возможных перестроек V- и J-гена и всех возможных перестроек D- и J-гена локуса IgH в соответствии с аннотацией коллаборации IMGT. Локус включал 126 уникальных V-генов; 52 функциональных гена, 6 предполагаемых открытых рамок считывания с отсутствующими критическими для функционирования аминокислотами и 69 псевдогенов; а также 9 J-генов, 6 функциональных генов и 3 псевдогена. Локус также включал 27 уникальных D-генов. Целевые последовательности для отжига с праймером для некоторых V-сегментов были идентичны, что позволило проводить амплификацию всех 126 V-сегментов с использованием 86 уникальных прямых праймеров. Аналогичным образом 7 уникальных обратных праймеров гибридизировались со всеми 9 J-генами. Для анализа D-J создали праймеры, выполненные с возможностью отжига с перестроенными стеблями -DJ. В процессе развития B-клетки в обоих аллелях происходит перестройка между сегментами D-гена и J-гена, что приводит к двум стеблям -DJ. Стебель -DJ включает J-ген, один N-участок и D-ген. После перестроек DJ V-ген одного из двух аллелей перестраивается со стеблем –DJ для кодирования CDR3-участка гена (VnDnJ). Для амплификации стебля -DJ 27 уникальных праймеров выполнили с возможностью отжига с каждым конкретным D-геном в интронном участке перед экзоном D-гена. Данные сегменты, присутствующие в стебле -DJ, вырезаются после рекомбинации V с –DJ. Однако праймеры для J повторно не создавали; для анализа DJ использовали те же праймеры для J, что и для анализа VJ.
В качестве базы для оценки систематической ошибки провели амплификацию пула из 1359 матриц с использованием оптимизированного (смесь 2-1) пула из 86 прямых праймеров для V (VF; специфических для V-генов), 27 прямых праймеров для D (DF; специфических для D-генов) и эквимолярного пула из 7 обратных праймеров для J (JR; специфических для J-генов). Полимеразные цепные реакции (ПЦР) (25 мкл каждая) готовили с 1,0 мкМ пула VF, 1,0 мкМ пула DF и 2,0 мкМ пула JR (Integrated DNA Technologies), 1x мастер-микса QIAGEN для мультиплексной ПЦР QIAGEN Multiplex Plus PCR (Qiagen, г. Валенсия, штат Калифорния, США), 10%-ным Q-раствором (QIAGEN) и 200 000 целевых молекул из смеси с синтетическим репертуаром VJ и DJ IgH. Использовали следующие настройки термоциклирования для термоциклера C100 (Bio-Rad Laboratories, г. Геркулес, штат Калифорния, США): один цикл при 95 °C в течение 6 минут, 31 цикл при 95 °C в течение 30 секунд, 64 °C в течение 120 секунд и 72 °C в течение 90 секунд с последующим одним циклом при 72 °C в течение 3 минут. Для всех экспериментов каждую ПЦР проводили в трех повторностях.
После первичной оценки систематической ошибки провели эксперименты по определению характеристик амплификации всех индивидуальных праймеров. Для определения специфичности праймеров DF и JR приготовили 27 смесей, содержащих один праймер DF и все праймеры JR и идентифицированную ранее оптимизированный пул праймеров VF, а также 7 смесей, содержащих один праймер JR и все праймеры VF и DF. Данные наборы праймеров использовали для амплификации синтетической матрицы и полученные библиотеки секвенировали для измерения специфичности каждого праймера для целевых сегментов V-, D- или J-гена, а также для идентификации случаев нецелевого праймирования.
Провели эксперименты по титрованию с использованием пулов с двукратными и четырехкратными концентрациями каждого индивидуального праймера DF или JF при эквимолярном содержании всех других праймеров, включая оптимизированную смесь праймеров VF (например, 2x gHD2-08 + эквимолярные количества всех других праймеров DF, оптимизированную смесь праймеров VF и праймеры JR) для определения масштабирующих коэффициентов, связывающих концентрацию праймера с наблюдаемой частотой матрицы.
Оптимизация смеси праймеров
Используя тест на перекрестную амплификацию, идентифицировали праймеры DF как ведущие перекрестную амплификацию. Удалили 12 из праймеров DF, получив итоговый пул из 15 праймеров DF. Используя масштабирующие коэффициенты, полученные при титровании праймеров по одному, разработали альтернативные смеси праймеров, в которых для сведения к минимуму систематической ошибки амплификации праймеры комбинировали в неравных концентрациях. Затем уточненные смеси праймеров использовали для амплификации пула матриц и измерения остаточной систематической ошибки амплификации. Данный процесс повторяли итерационно, повышая или понижая концентрацию каждого праймера соответственно на основе того, было ли представление матриц, амплифицируемых данным праймером, недостаточным или избыточным на предыдущем этапе результатов. На каждой стадии данного итерационного процесса определяли общую степень систематической ошибки амплификации, рассчитывая показатели для динамического диапазона (максимальная систематическая ошибка/минимальная систематическая ошибка) и суммы квадратов (SS, рассчитывается по значениям log (систематическая ошибка)) и итерационно повторяя процесс коррекции концентраций праймеров до тех пор, пока улучшение между итерациями не становилось минимальным. Итоговая смесь праймеров давала по существу меньшую систематическую ошибку, связанную с праймерами, чем эквимолярная смесь праймеров.
На Фиг. 12 показаны итерации IGH-праймера для синтетических матриц IGH VJ, представленные в зависимости от относительной систематической ошибки амплификации. Определили относительную систематическую ошибку амплификации для 1116 синтетических матриц IGH VJ до химической коррекции для контроля систематической ошибки (эквимолярные праймеры (черный)), после химической коррекции (оптимизированные праймеры (темно-серый)) и после химической и расчетной коррекции (после расчетной коррекции (светло-серый)). Эквимолярные праймеры давали динамический диапазон 1130, межквартильный размах 0,991 и сумму квадратов (логарифмический показатель систематической ошибки) 233. Оптимизированные праймеры давали динамический диапазон 129, межквартильный размах 0,732 и сумму квадратов (логарифмический показатель систематической ошибки) 88,2. Праймеры после расчетной коррекции давали динамический диапазон 76,9, межквартильный размах 0,545 и сумму квадратов (логарифмический показатель систематической ошибки) 37,9.
На Фиг. 13 показана относительная систематическая ошибка амплификации для 27 синтетических матриц IGH DJ для V-гена. Относительная систематическая ошибка амплификации для сегмента V-гена показана для трех итераций праймеров: 1) до химической коррекции для контроля систематической ошибки (черный), 2) после первой итерации химической коррекции (белый) 3) и после второй итерации химической коррекции (светло-серый).
ПРИМЕР 11. ИТЕРАЦИИ ПРАЙМЕРОВ ДЛЯ VJ TCRG
В других вариантах осуществления во множестве итераций праймеров тестировали праймеры для VJ TCRG на относительную систематическую ошибку амплификации. На Фиг. 14a–d показаны итерации TCRG-праймера для 55 синтетических матриц TCRG VJ. Относительные систематические ошибки амплификации для праймеров TCRG VJ определяли до химической коррекции на контроль систематической ошибки (Фиг. 14a), после первой итерации химической коррекции (Фиг. 14b), после второй итерации химической коррекции (фиг. 14c) и после итоговой итерации химической коррекции (Фиг. 14d).
ПРИМЕР 12. АЛЬТЕРНАТИВНЫЙ СПОСОБ КОНТРОЛЯ СИСТЕМАТИЧЕСКОЙ ОШИБКИ И ВВЕДЕНИЯ ИЗВЕСТНОГО КОЛИЧЕСТВА СТАНДАРТА
В других вариантах осуществления можно использовать альтернативные способы определения систематической ошибки амплификации. Две основные цели такого способа заключаются в следующем: (1) устранить систематическую ошибку амплификации при мультиплексной ПЦР-амплификации генов BCR или TCR и (2) оценить долю B-клеток или T-клеток в исходной матрице.
Способ включает получение набора клеток, содержащих ДНК или кДНК (мРНК), экстрагированную из образца, содержащего B-клетки и/или T-клетки. Для содержащего клетки образца проводят экстракцию ДНК, используя стандартные способы в данной области.
Экстрагированную ДНК разделяют на множество частей и вносят в разные лунки для ПЦР. В некоторых вариантах осуществления используют одну лунку до полной емкости, или можно использовать тысячи лунок. В одном варианте осуществления для ПЦР используют 188 лунок (два 96-луночных планшета). Количество матриц TCR или BCR на лунку должно быть малым, так чтобы вероятность наличия в одной лунке множества молекул одного и того же клонотипа была малой.
Затем способ включает амплификацию ДНК отдельно в каждой лунке с использованием того же мультиплексного набора праймеров. Для этого можно использовать набор праймеров, описанный в настоящем документе. Как описано выше, для амплифицированных молекул используется способ кодирования с использованием штрихкода с той же последовательностью штрихкода в каждой лунке. Например, каждая лунка получает свой собственный штрихкод.
Затем молекулы секвенируют на высокопроизводительной секвенирующей системе с достаточным количеством амплифицированных последовательностей BCR или TCR для идентификации по последовательности используемых V- и J-цепей, а также последовательности штрихкода.
Каждая лунка имеет среднее число копий. Поскольку каждый клонотип появляется в лунке один раз, количество данной матрицы относительно среднего представляет собой систематическую ошибку для данной комбинации V-J. Поскольку систематические ошибки по V и J независимы, для определения систематических ошибок необходимы не все комбинации V-J. Затем данные относительные систематические ошибки используют либо для повторного создания праймеров, которые обеспечивают либо сильно заниженную или завышенную амплификацию, либо для титрования концентрации праймеров для усиления или ослабления амплификации. Полный описанный процесс повторяют с новой партией праймеров и продолжают итерационно для продолжения снижения систематической ошибки.
После любого цикла итерации для устранения систематической ошибки можно применить набор расчетных коэффициентов (для относительных коэффициентов амплификации). Систематическую ошибку можно уменьшить путем изменения праймеров и/или расчетной коррекции.
Способ включает вычисление доли ядросодержаших клеток из аналогичного анализа. Для каждой лунки идентифицируют каждый клонотип и определяют число прочтений последовательности для каждого клона. В некоторых вариантах осуществления число матриц на лунку не должно быть малым. Число прочтений для каждого клона корректируют с помощью коэффициентов контроля систематической ошибки, как описано выше.
Строят гистограмму скорректированных чисел прочтений, и на диаграмме имеется основная мода (коэффициент амплификации). Данную моду идентифицируют визуально (на основе идентификации первого большого пика), или путем взятия преобразования Фурье, или иными известными способами.
Полное скорректированное число прочтений для каждой лунки делят на коэффициент амплификации для данной лунки. Полученное значение представляет собой оценочное число геномных матриц TCR или BCR, исходно находившихся в лунке. Полное число BCR или TCR в образце представляет собой сумму значений по всем лункам. До ПЦР измеряют полное число геномов в каждой лунке. Это можно сделать нанокапельным способом или другими известными способами для определения количества ДНК. Измеренный вес ДНК делят на вес двухцепочечного генома (например, ~6,2 пикограмм у человека).
Доля B-клеток или T-клеток в образце равна полному количеству BCR или TCR в образце, деленному на полное количество двухцепочечных молекул ДНК, добавленных к реакции. Результат нуждается в небольшой коррекции, поскольку в небольшой доле T-клеток происходит перестройка обоих аллелей. Данный поправочный коэффициент составляет приблизительно 15% для альфа бета T-клеток, 10% для B-клеток. Для гамма дельта T-клеток перестройка обоих аллелей происходит практически во всех клетках, так что коэффициент коррекции равен двум.
Данные дополнительные способы позволяют определить систематическую ошибку амплификации при мультиплексной ПЦР-амплификации генов BCR или TCR и могут использоваться для оценки доли B-клеток или T-клеток в исходной матрице.
Различные описанные выше варианты осуществления можно комбинировать для получения дополнительных вариантов осуществления. Все патенты США, публикации заявок на патенты США, заявки на патенты США, зарубежные патенты, зарубежные заявки на патенты и непатентные публикации, на которые даны ссылки в настоящем описании и/или которые перечислены в справочном листе настоящей заявки, считаются полностью включенными в настоящий документ путем ссылки. Аспекты вариантов осуществления при необходимости можно модифицировать для использования концепций из различных патентов, заявок и публикаций для получения дополнительных вариантов осуществления.
Данные и другие изменения можно вносить в варианты осуществления в свете представленного выше подробного описания. В общем в следующей формуле изобретения используемые термины не должны толковаться как ограничивающие формулу изобретения конкретными вариантами осуществления, описанными в настоящей спецификации и формуле изобретения, но должны считаться включающими все возможные варианты осуществления вместе с полным объемом их эквивалентов, которые входят в такую формулу изобретения. Соответственно, формула изобретения не ограничена настоящим описанием.
Неформальный список последовательностей
Последовательности для контроля систематической ошибки для hs-IgH-DJ (243 последовательности)
Последовательности для контроля систематической ошибки для hs-IGL
Последовательности для контроля систематической ошибки для hs-IgK
Последовательности праймеров для hs-TCRB-P10
Последовательности праймеров для hs-IGH-D
Последовательности праймеров для IGK и IGL
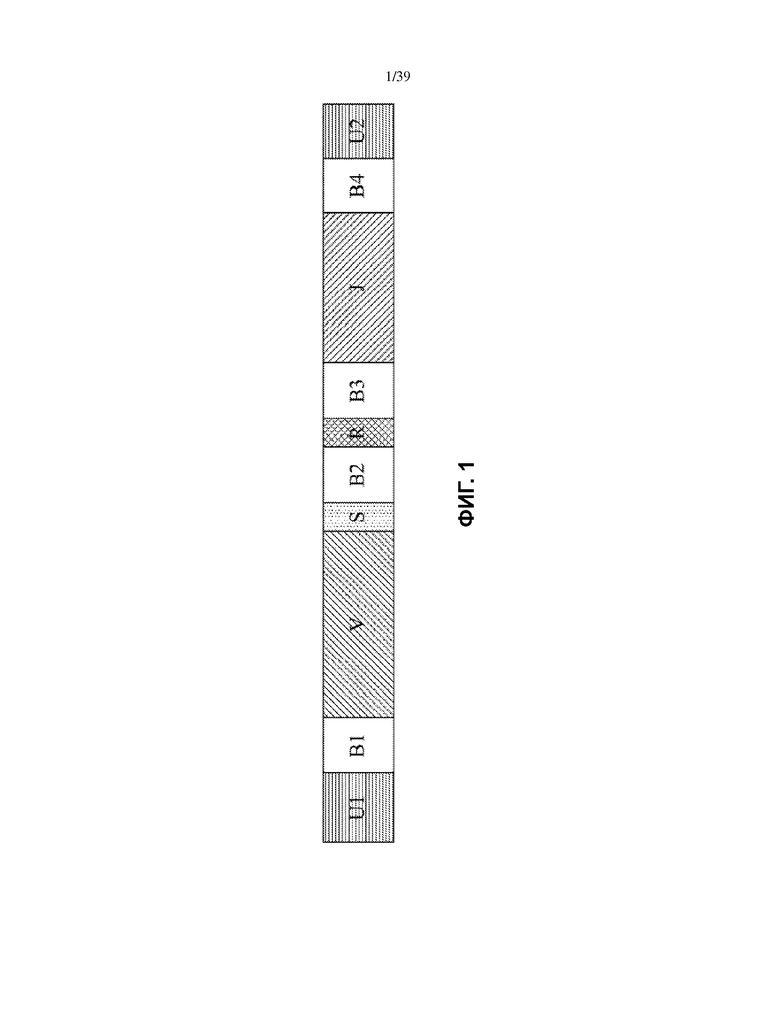
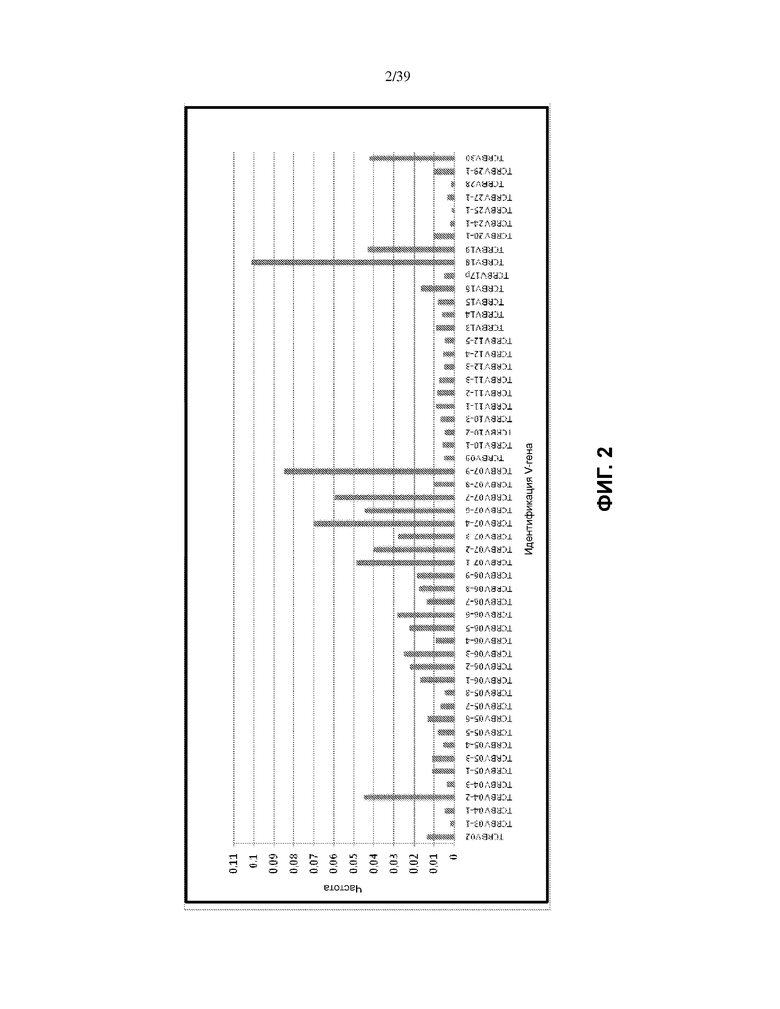
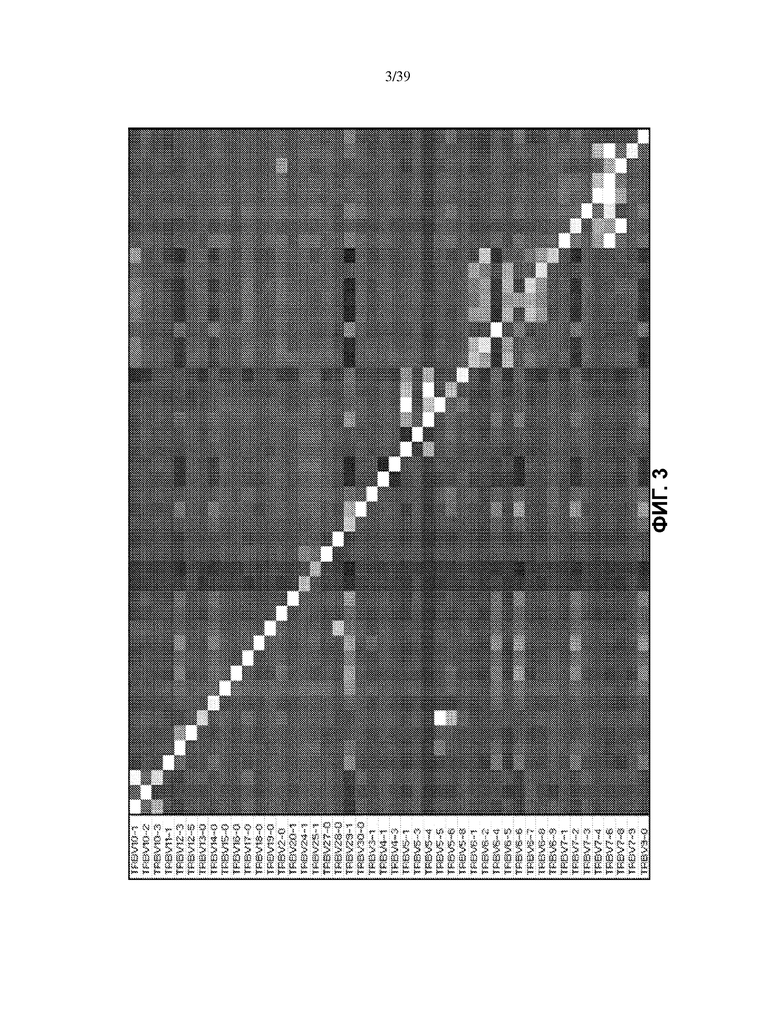
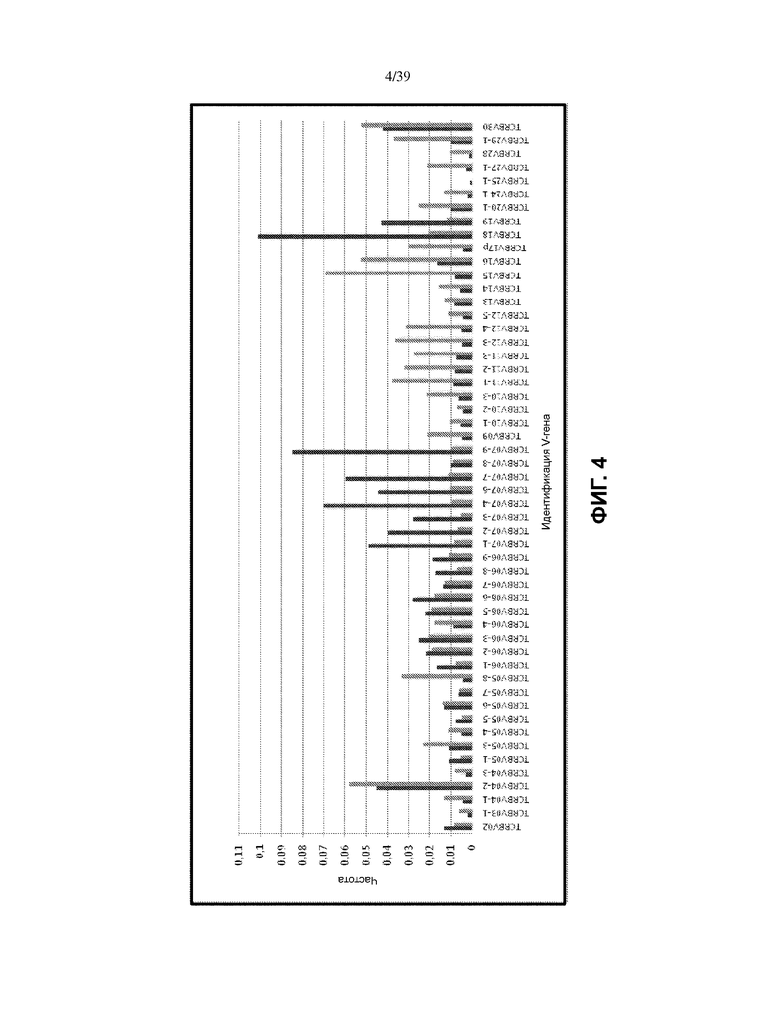
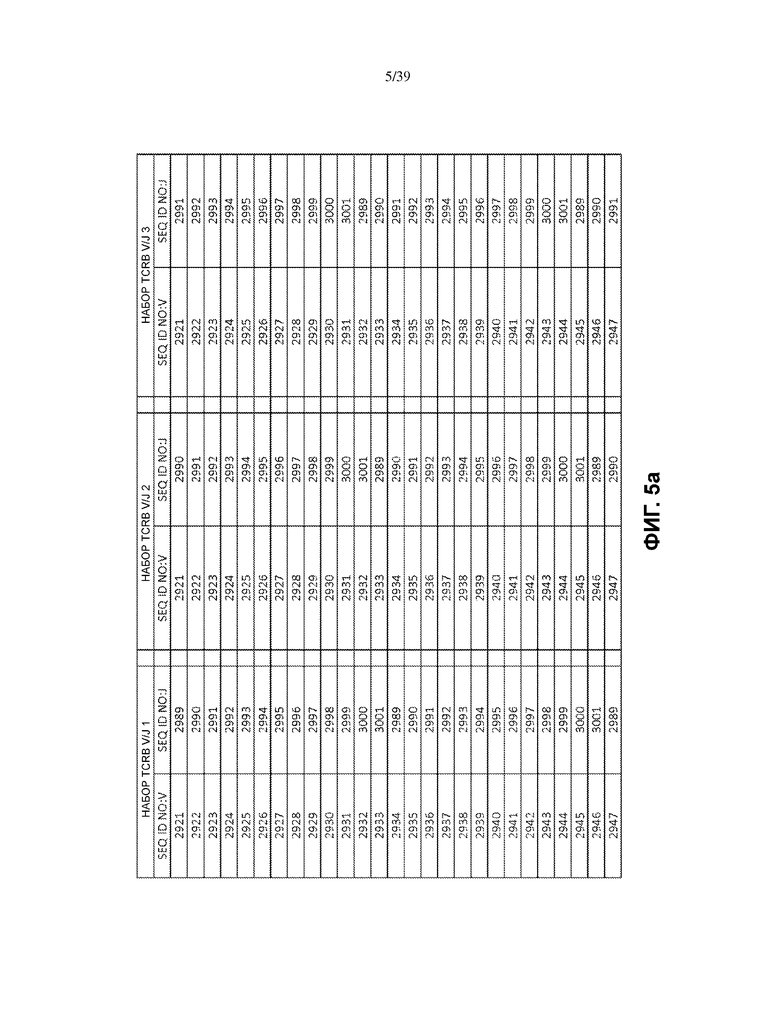
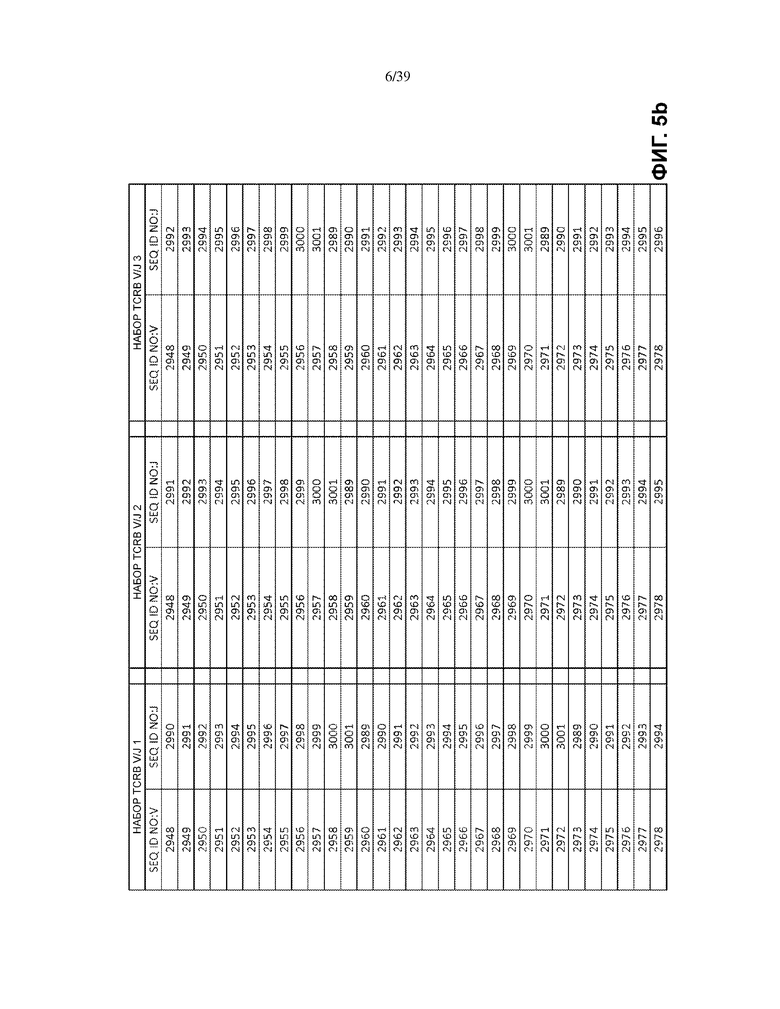
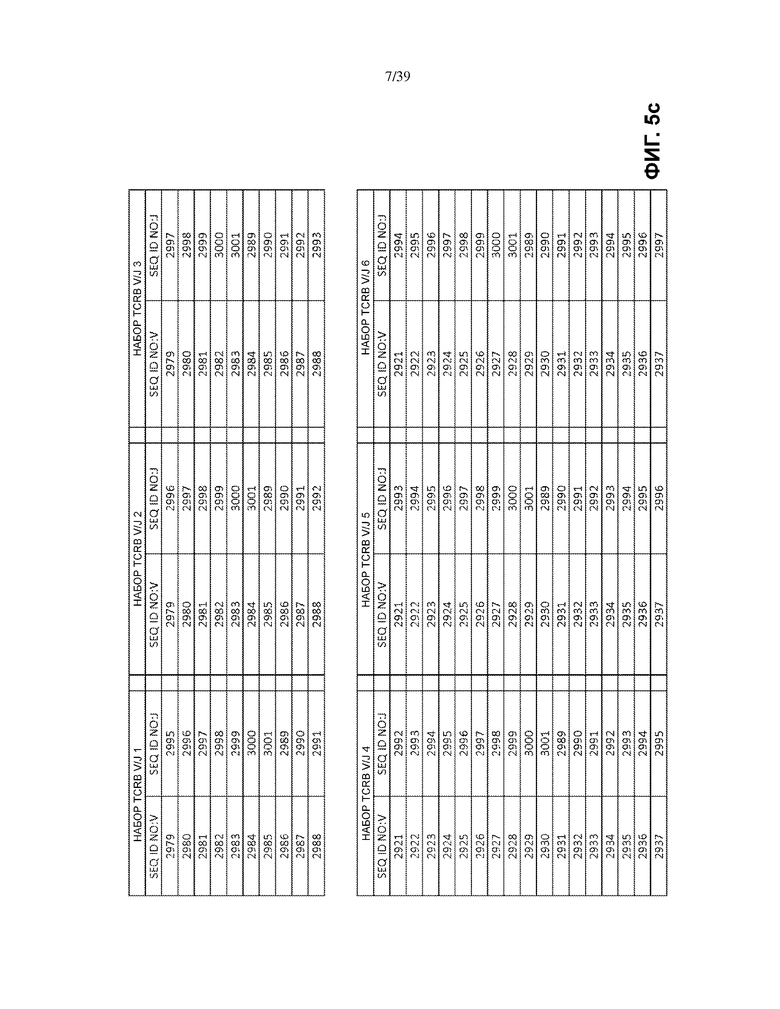
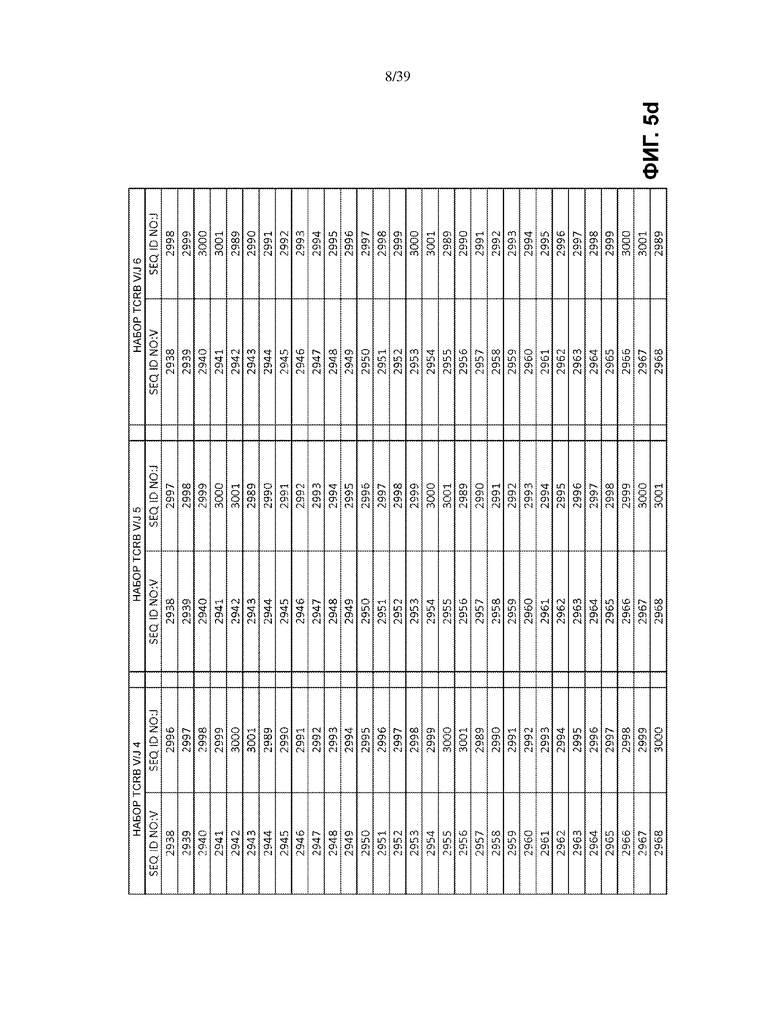
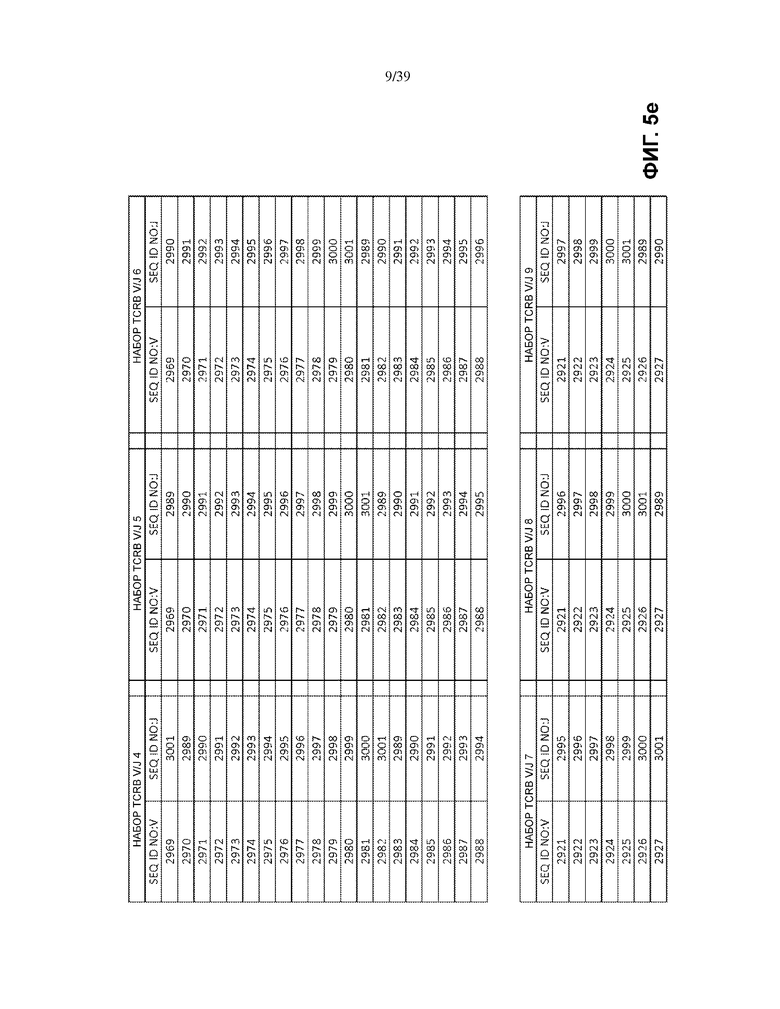
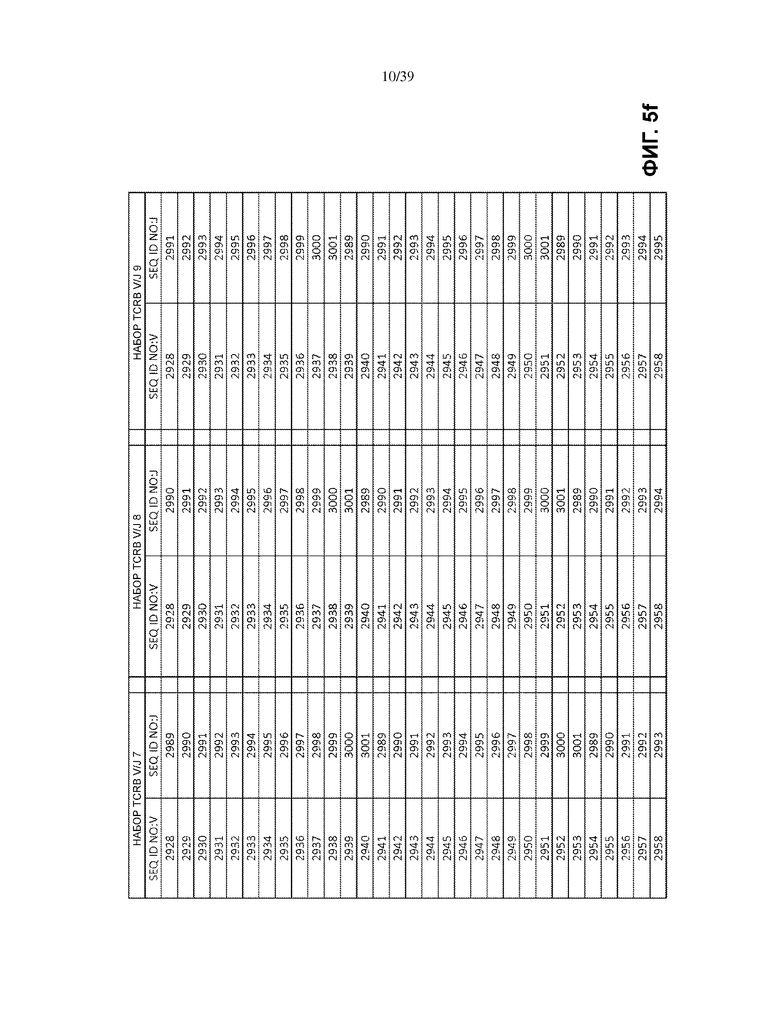
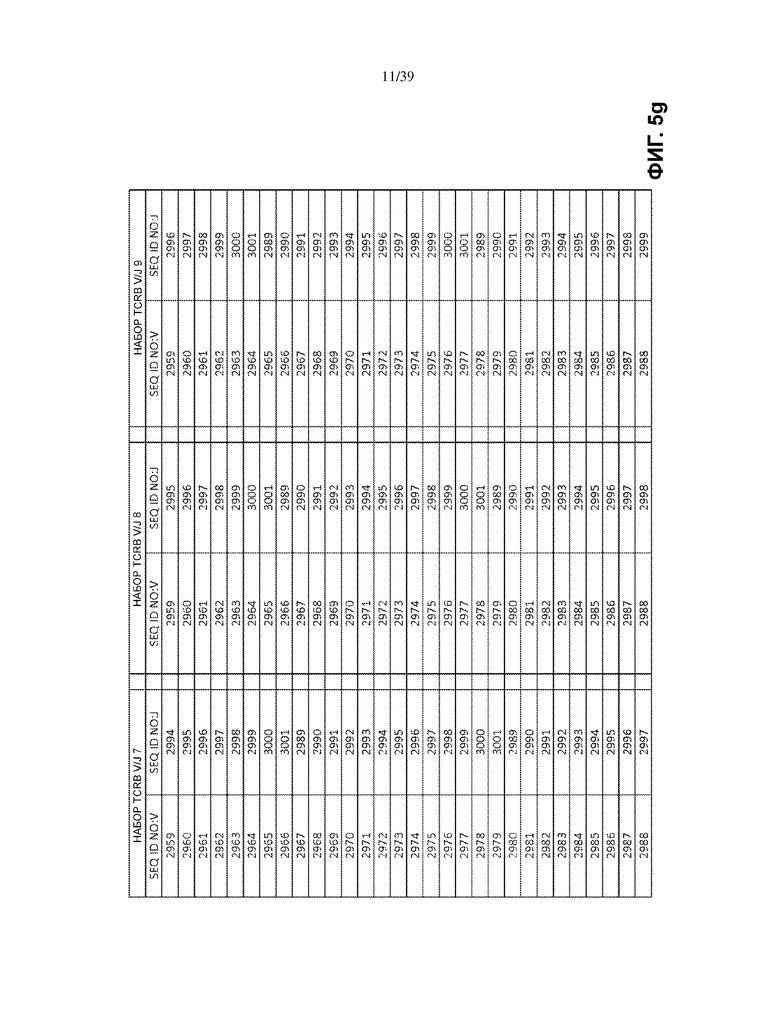
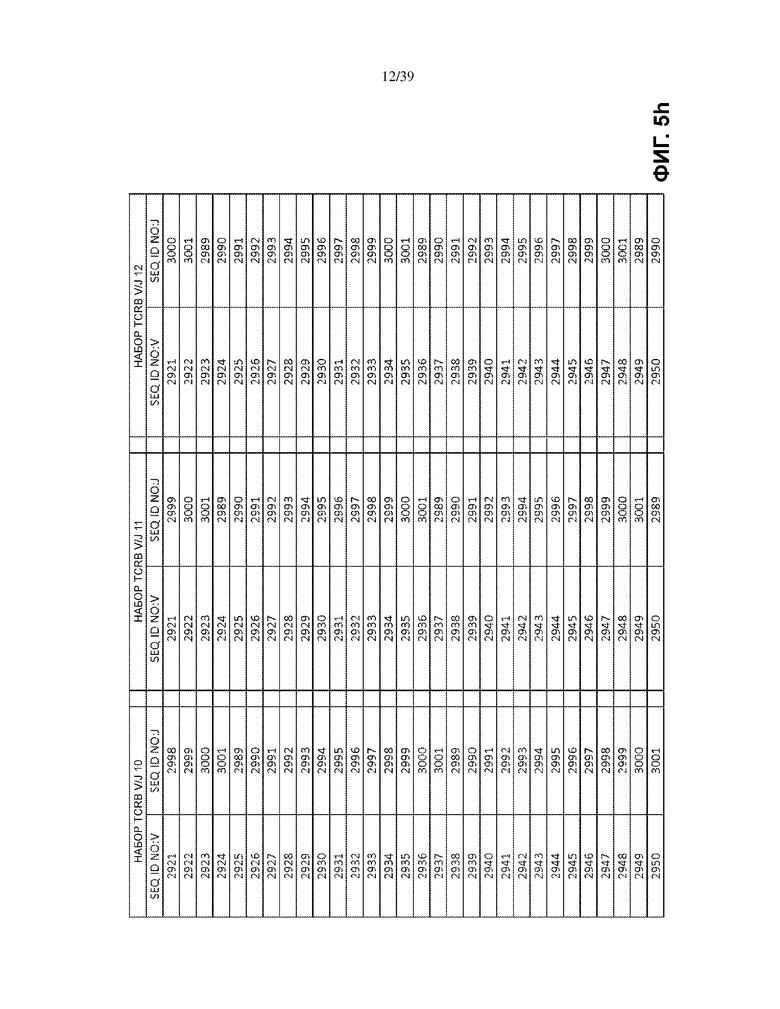
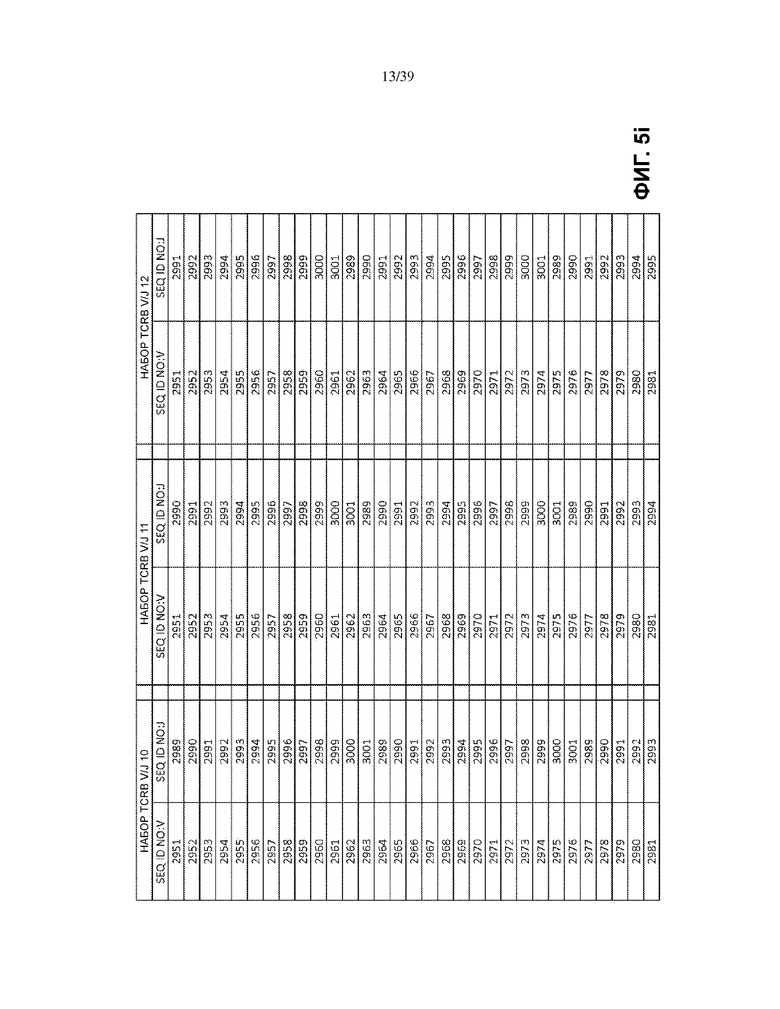
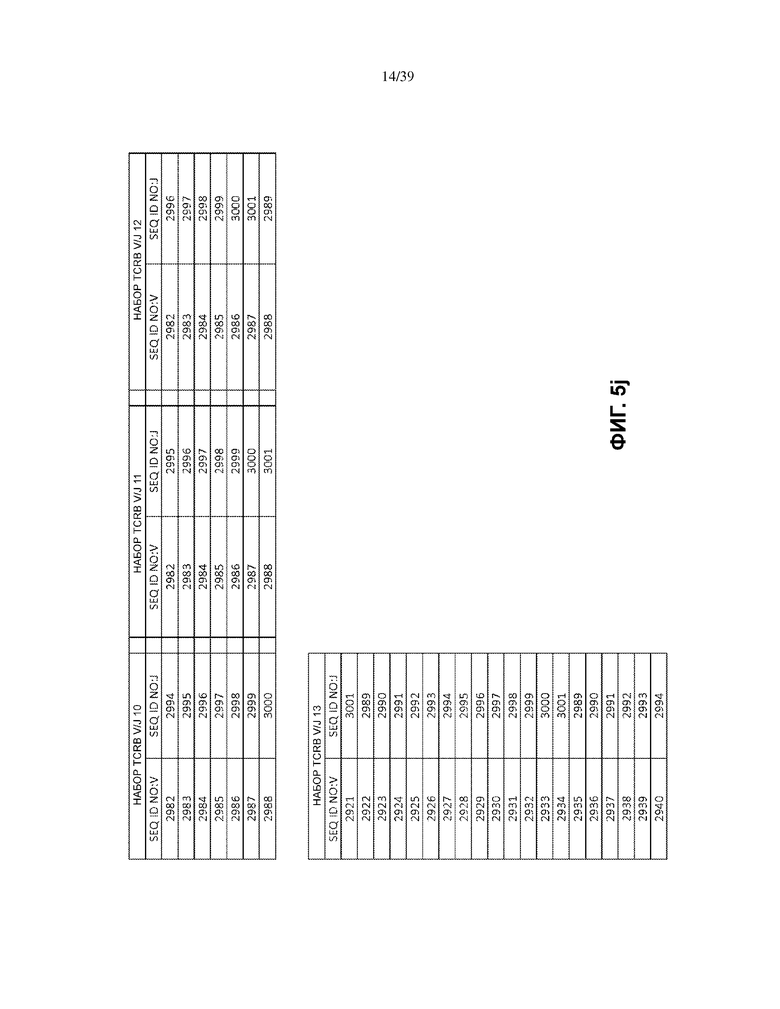
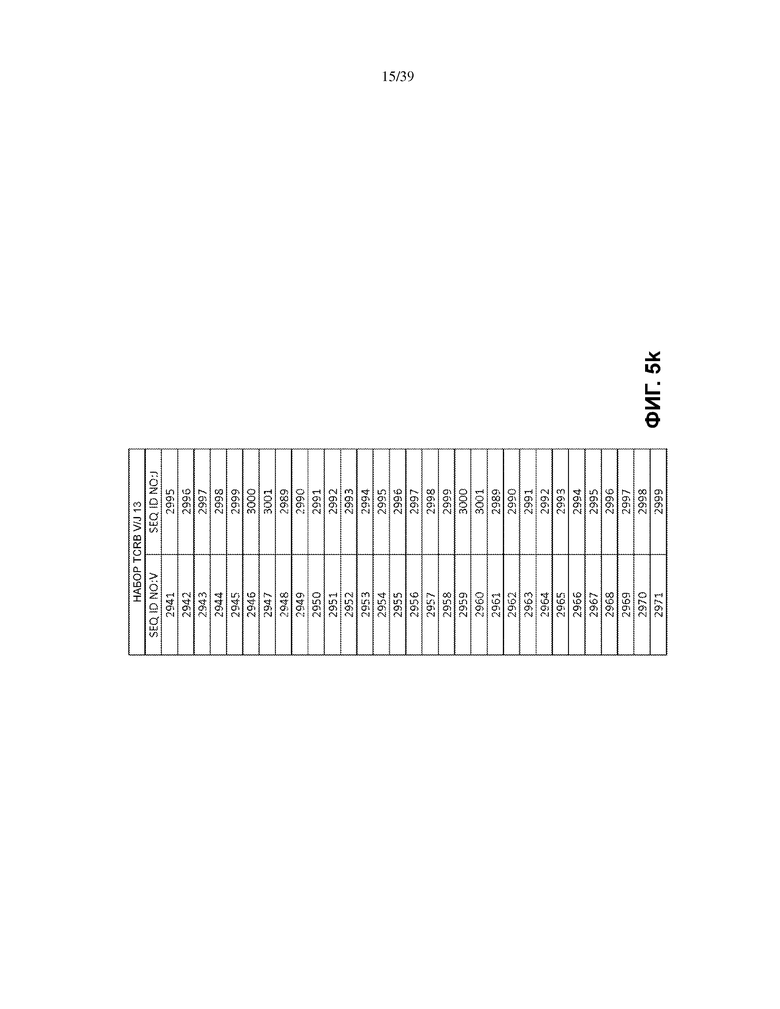
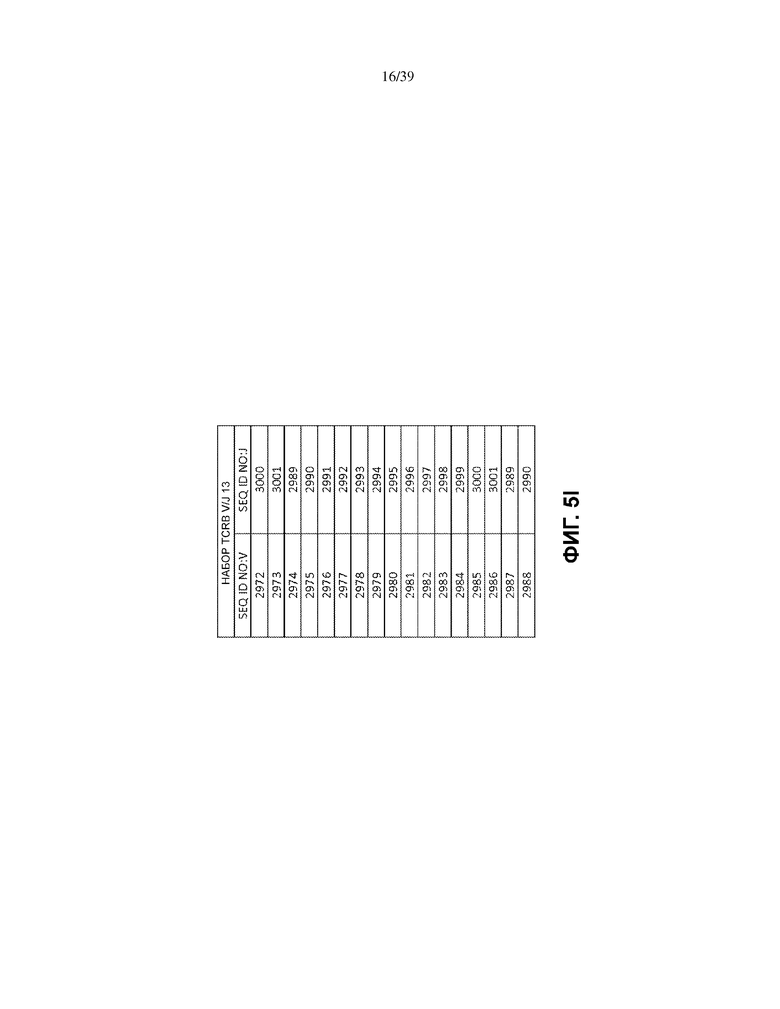
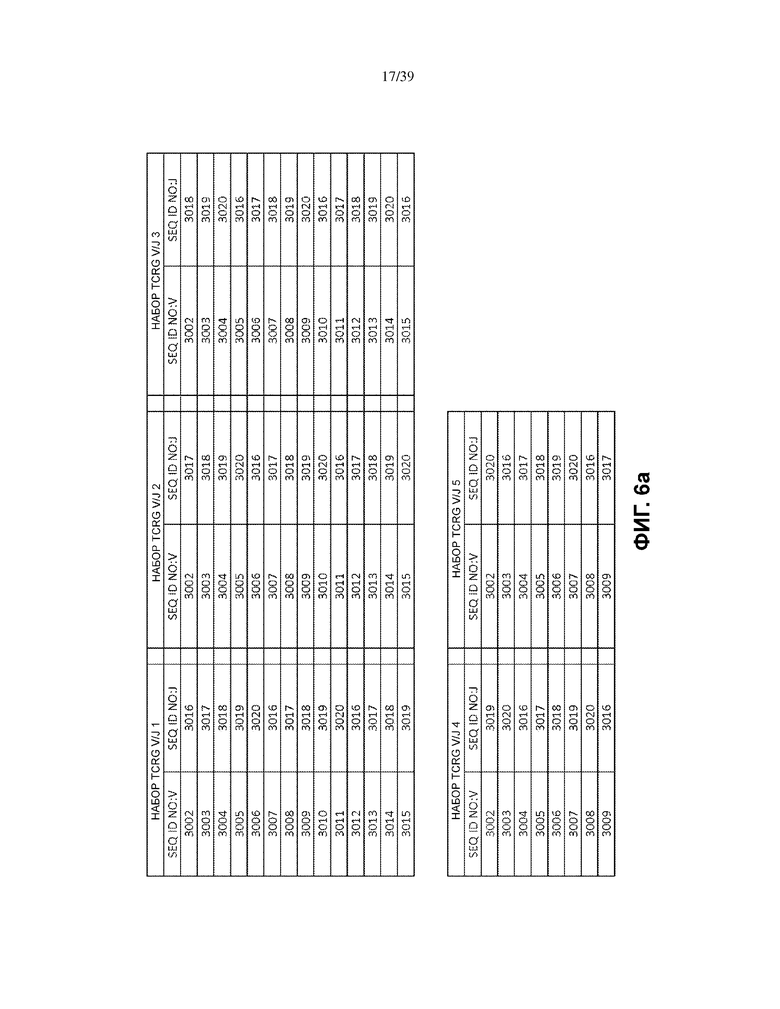
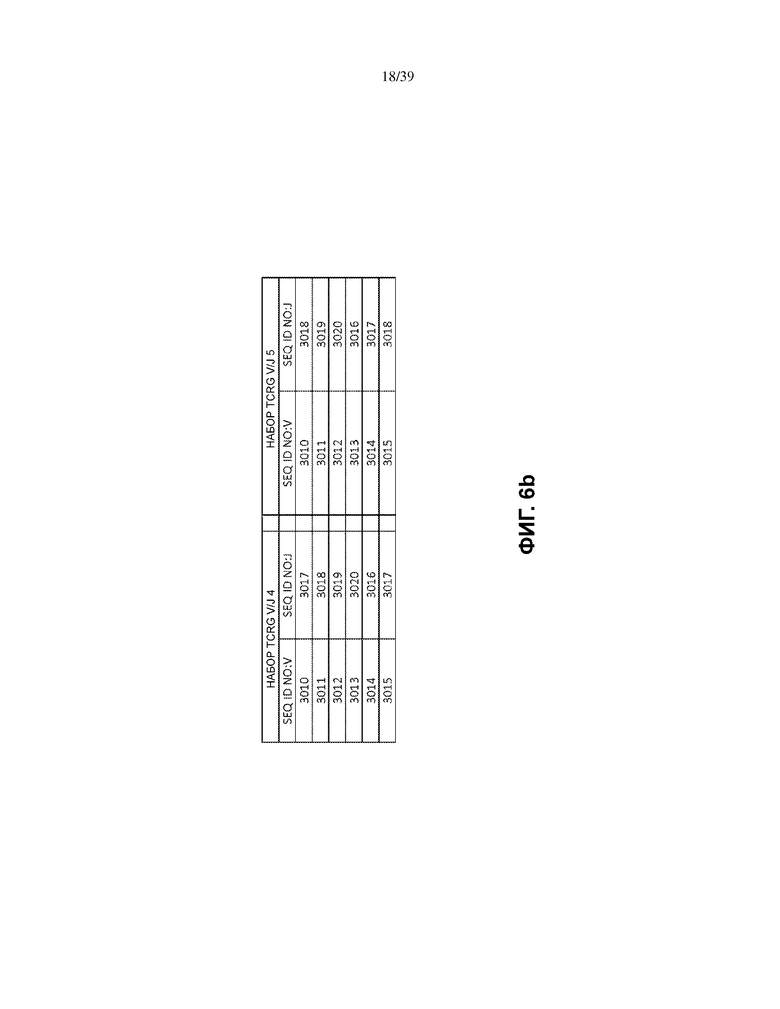

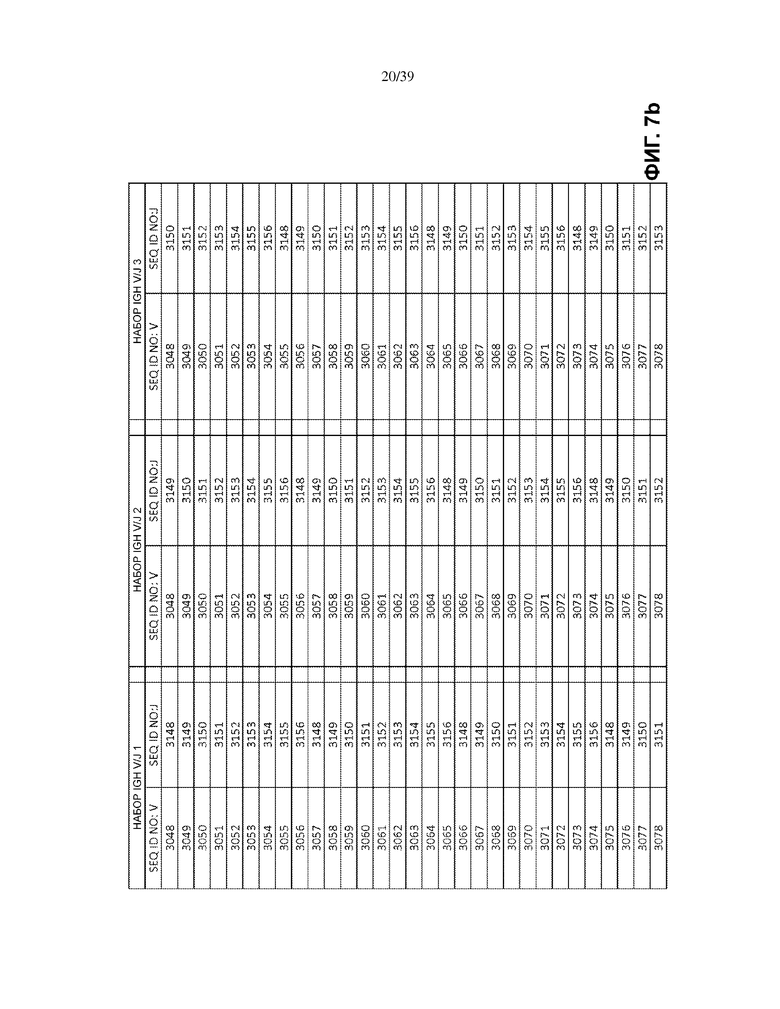
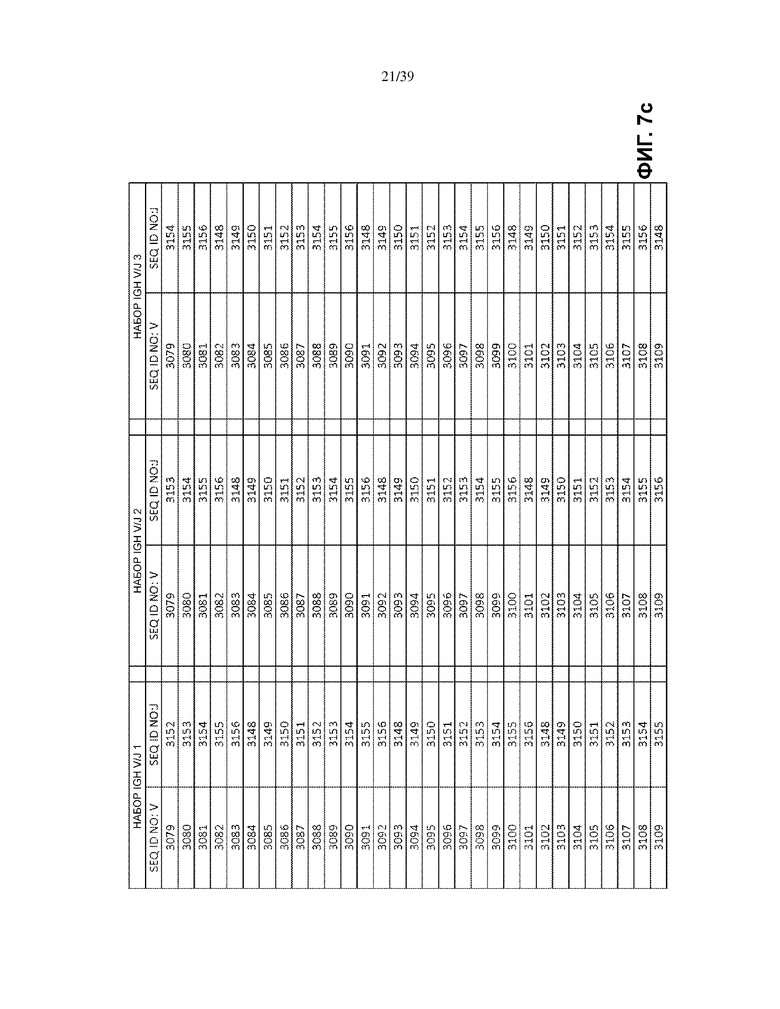
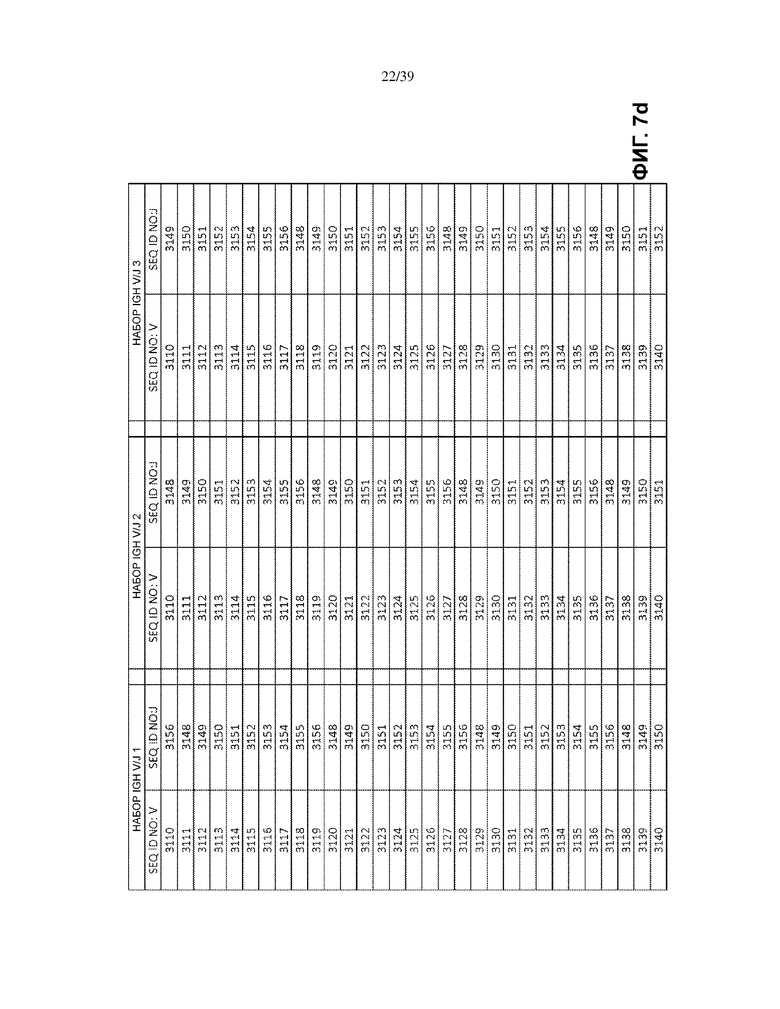
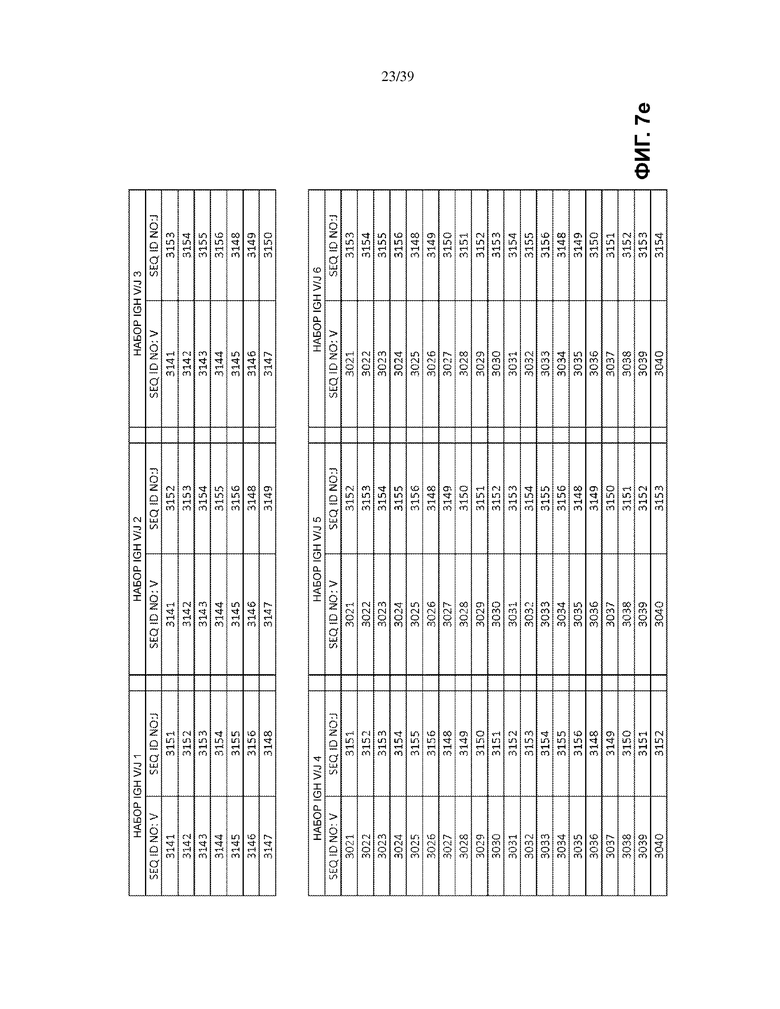
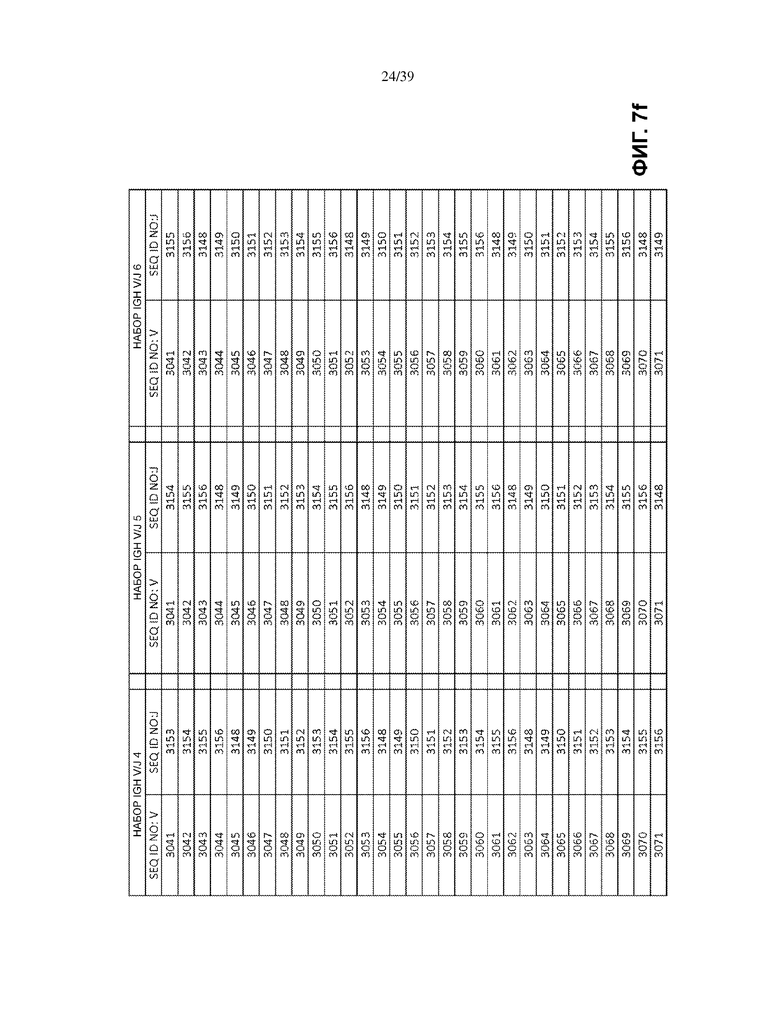
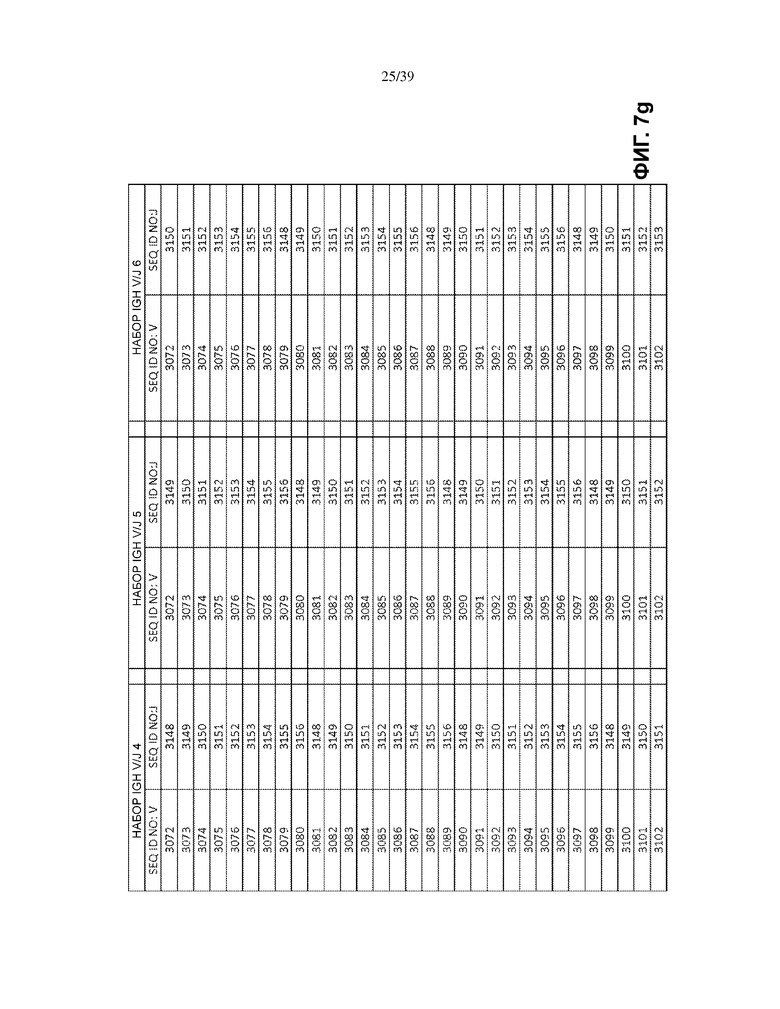
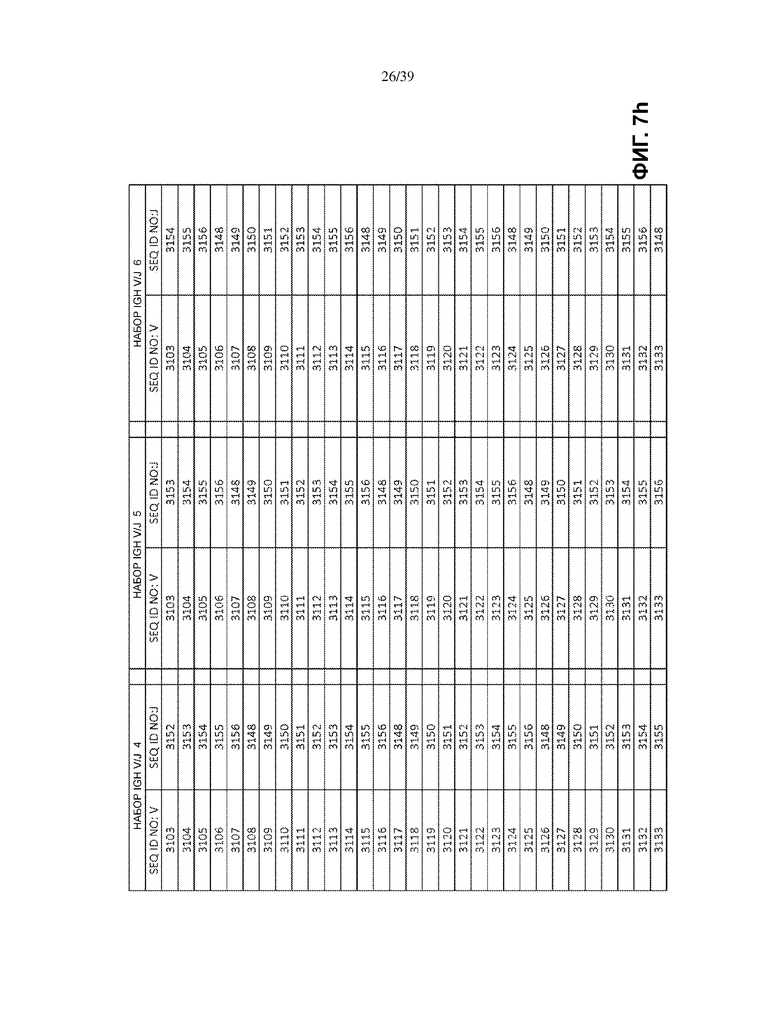

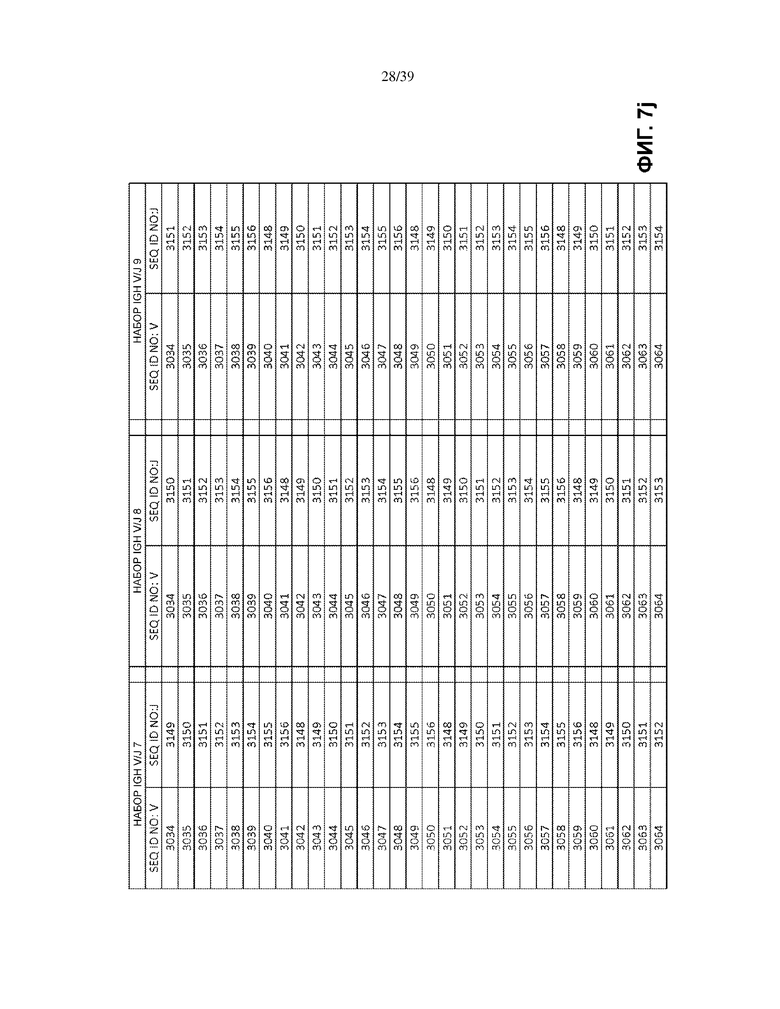
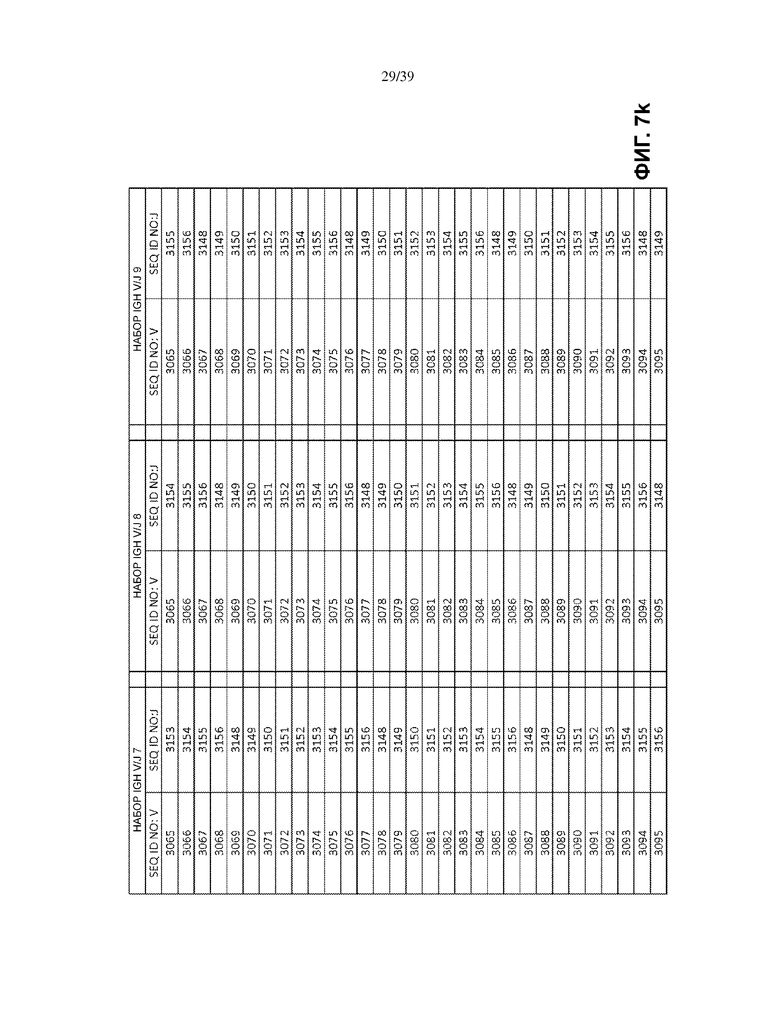
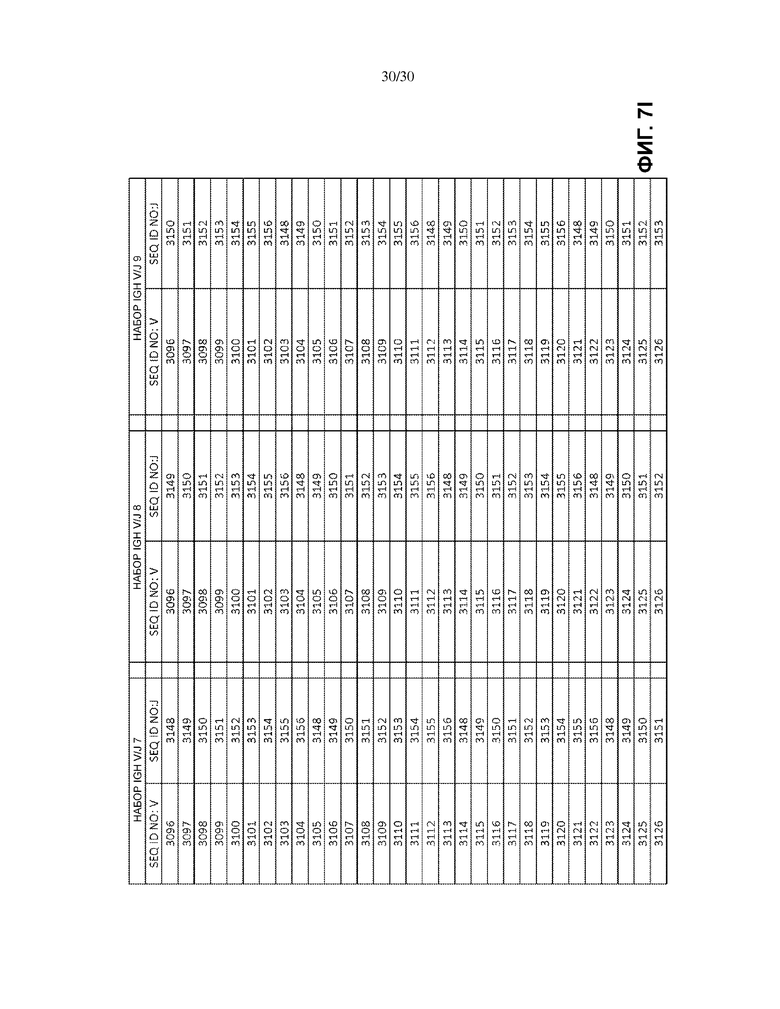
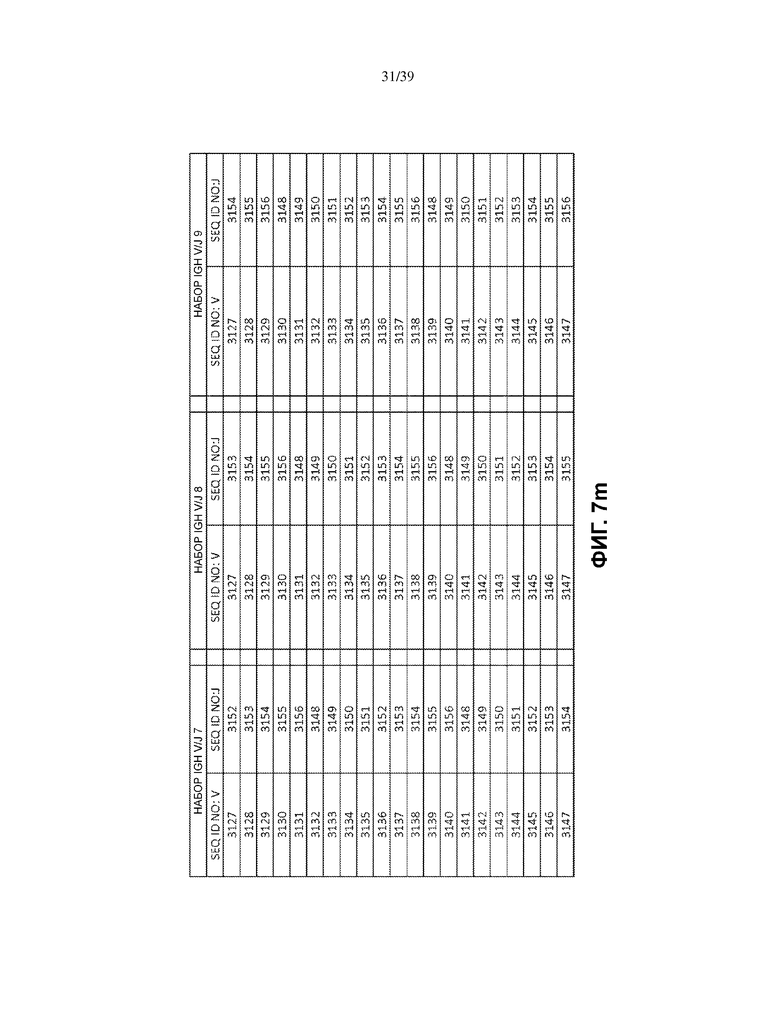
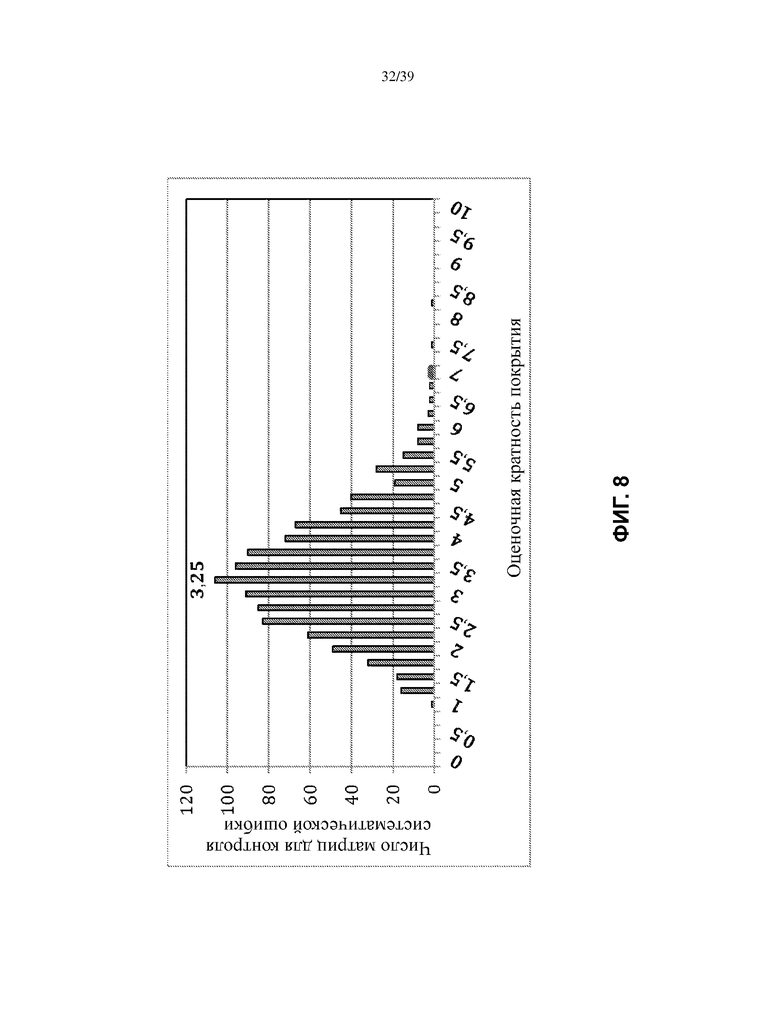
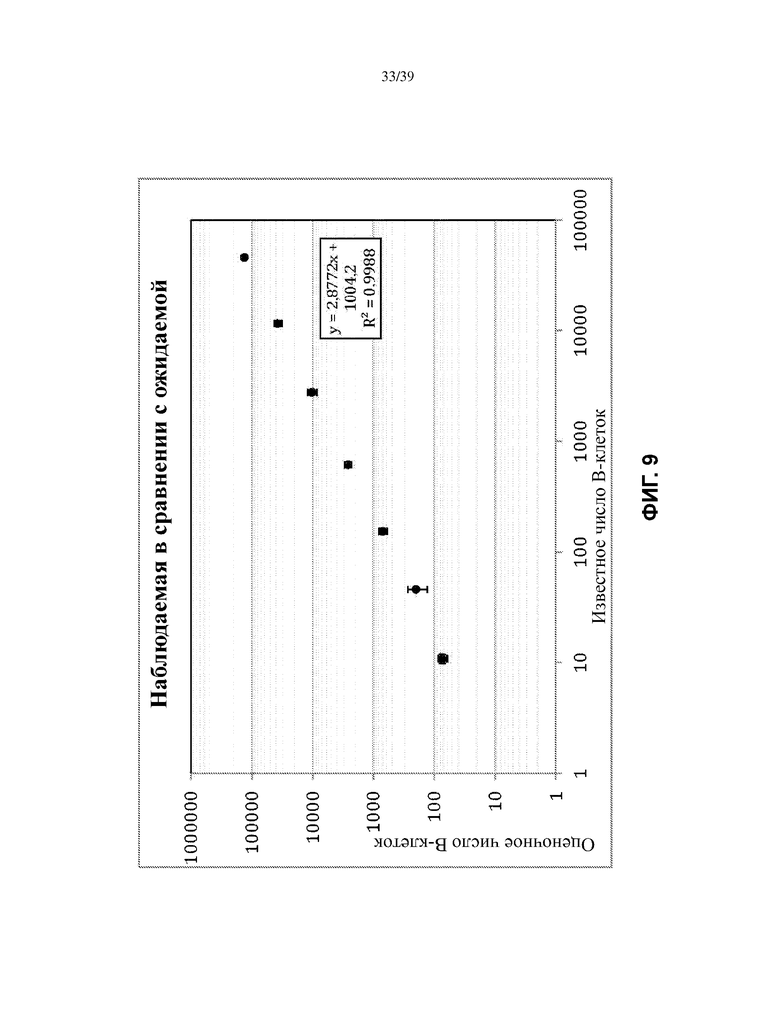
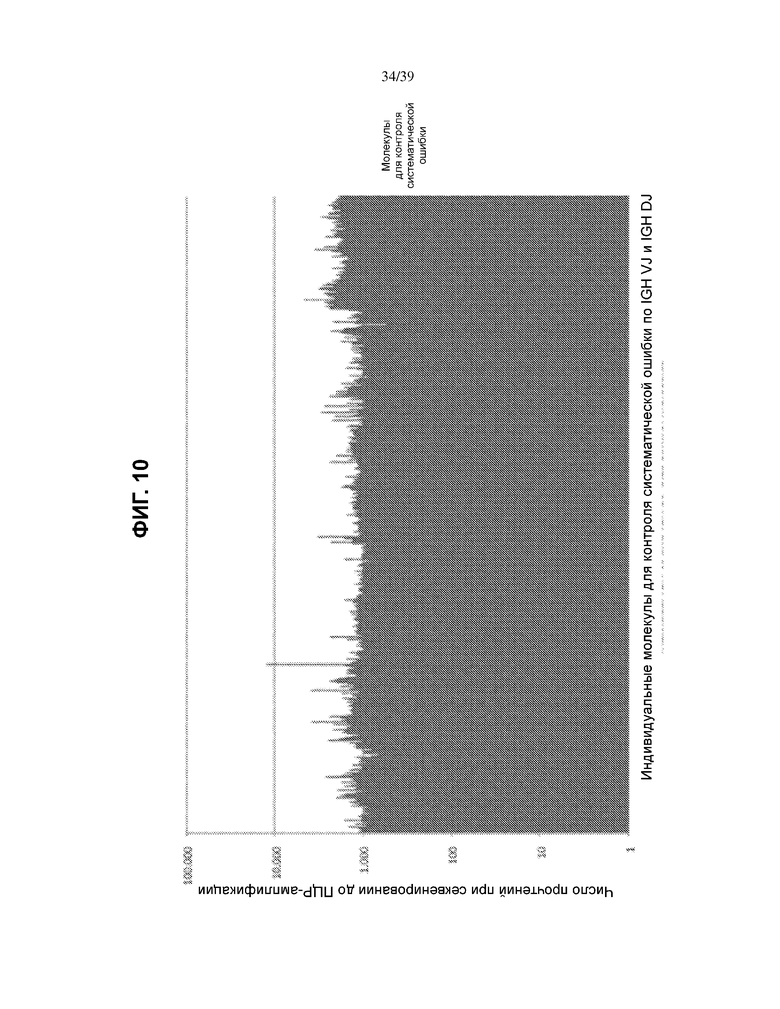
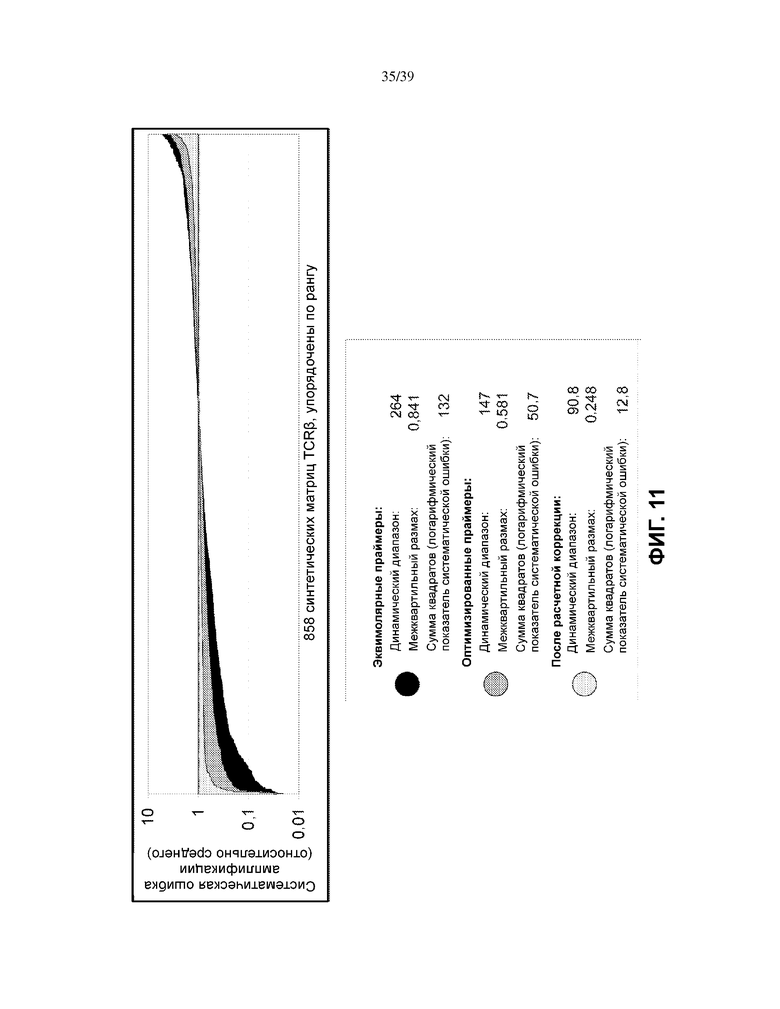
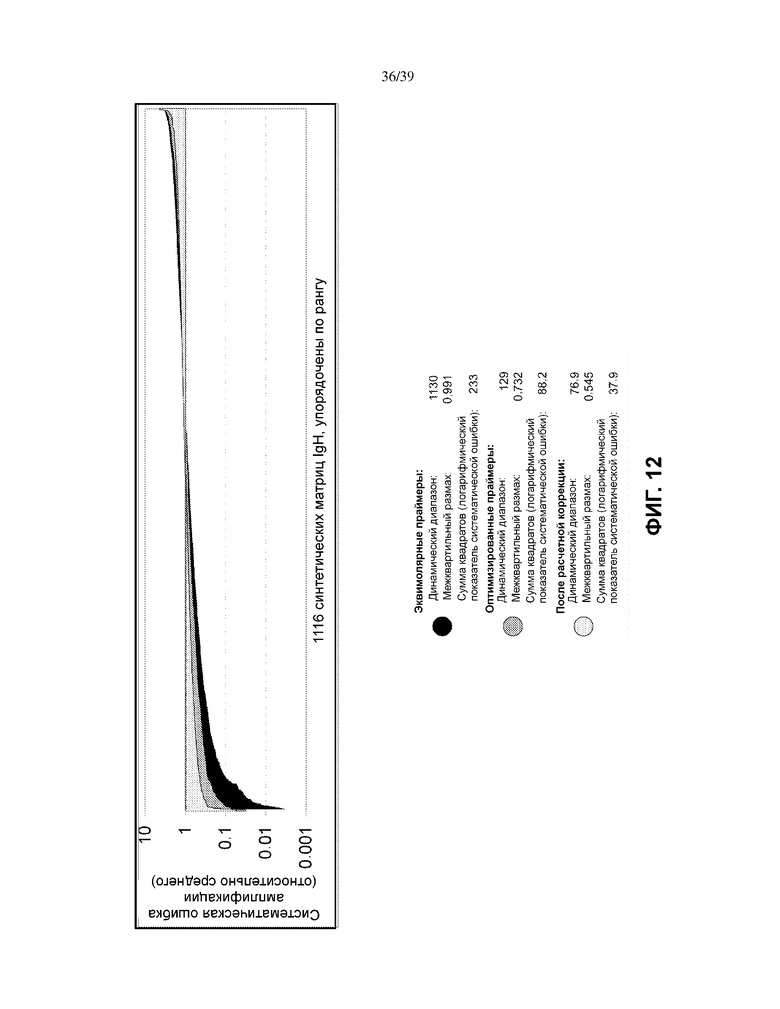
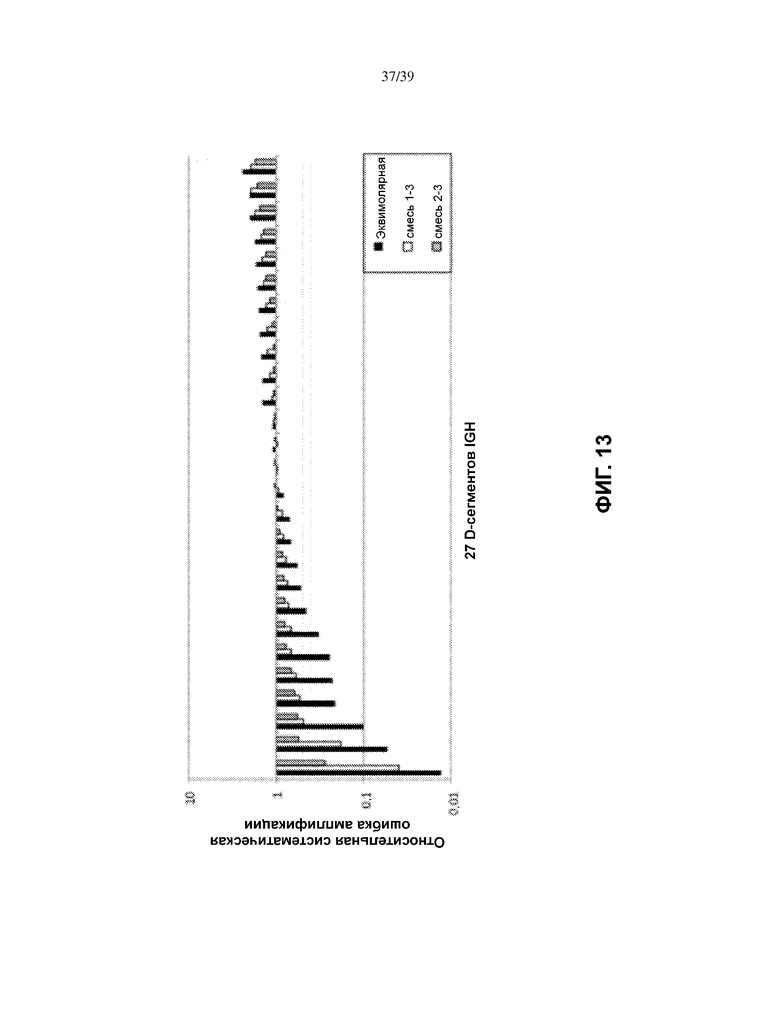
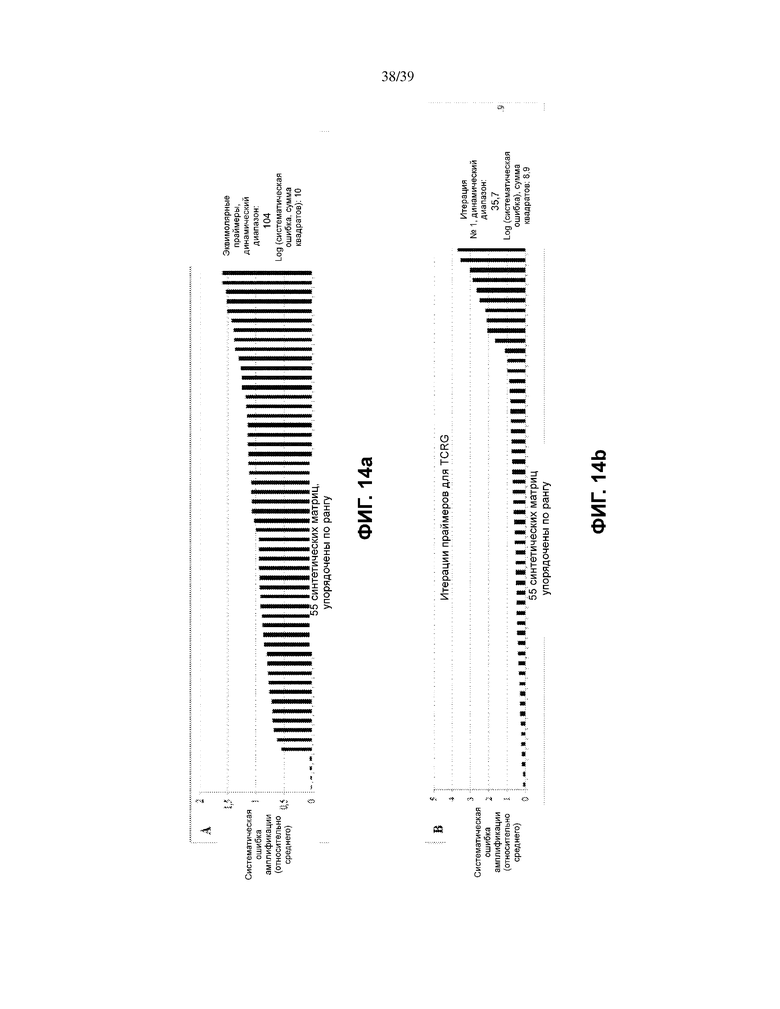
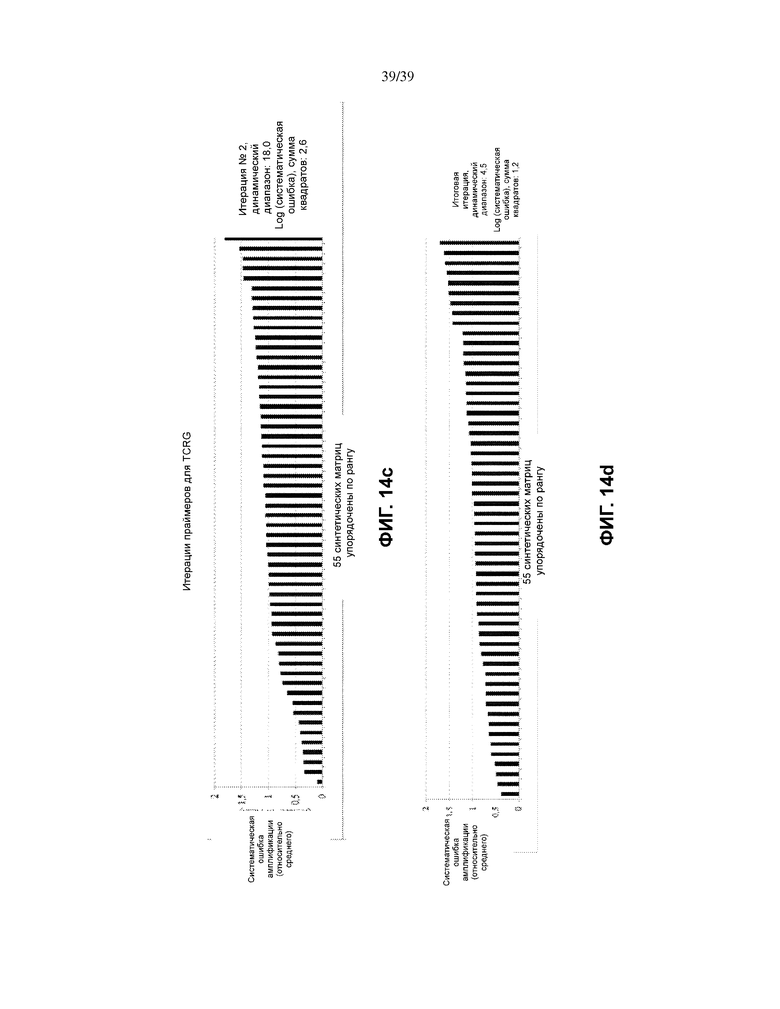
Изобретение относится к биотехнологии. Описана композиция для стандартизации эффективности амплификации набора олигонуклеотидных праймеров для амплификации перестроенных последовательностей нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы в биологическом образце, полученном из лимфоидных клеток субъекта-млекопитающего, причем каждый рецептор адаптивной иммунной системы содержит вариабельный участок и соединительный участок, причем композиция содержит: множество синтетических матричных олигонуклеотидов, причем каждый синтетический матричный олигонуклеотид имеет известную концентрацию до амплификации и олигонуклеотидную последовательность общей формулы:
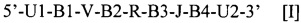 , причем: (а) V представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 20 и не более 1000 последовательных нуклеотидов генной последовательности, кодирующей вариабельный (V) участок рецептора адаптивной иммунной системы или его комплемент, и каждый V содержит уникальную олигонуклеотидную последовательность V-участка; (b) J представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 15 и не более 600 последовательных нуклеотидов генной последовательности, кодирующей соединительный (J) участок рецептора адаптивной иммунной системы или его комплемент, и каждый J содержит уникальную олигонуклеотидную последовательность J-участка; (с) U1 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) первой универсальной последовательности олигонуклеотида-адаптера и (ii) первой специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5' и находится в нем относительно первой универсальной последовательности олигонуклеотида-адаптера; (d) U2 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) второй универсальной последовательности олигонуклеотида-адаптера и (ii) второй специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5' и находится в нем относительно второй универсальной последовательности олигонуклеотида-адаптера; (e) присутствует по меньшей мере один из B1, В2, В3 и В4 и каждый из B1, В2, В3 и В4 содержит олигонуклеотид В, содержащий последовательность штрихкода из 3-25 последовательных нуклеотидов, которая уникальным образом идентифицирует в качестве спаренной комбинации, (i) уникальную олигонуклеотидную последовательность V-участка из (а) и (ii) уникальную олигонуклеотидную последовательность J-участка из (b); (f) R либо отсутствует, либо содержит сайт распознавания рестрикционного фермента, который содержит олигонуклеотидную последовательность, отсутствующую в (а)-(е); и причем: (g) множество синтетических матричных олигонуклеотидов содержит количество по меньшей мере а или по меньшей мере b уникальных олигонуклеотидных последовательностей в зависимости от того, какое значение больше, причем а представляет собой количество уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой количество уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта, и композиция содержит по меньшей мере один синтетический матричный олигонуклеотид для каждой уникальной олигонуклеотидной последовательности V-участка и по меньшей мере один синтетический матричный олигонуклеотид для каждой уникальной олигонуклеотидной последовательности J-участка. Также заявлен способ количественного определения множества перестроенных молекул нуклеиновых кислот, кодирующих один или множество рецепторов адаптивной иммунной системы в биологическом образце. 2 н. и 25 з.п. ф-лы, 14 ил., 17 табл., 12 пр.
, причем: (а) V представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 20 и не более 1000 последовательных нуклеотидов генной последовательности, кодирующей вариабельный (V) участок рецептора адаптивной иммунной системы или его комплемент, и каждый V содержит уникальную олигонуклеотидную последовательность V-участка; (b) J представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 15 и не более 600 последовательных нуклеотидов генной последовательности, кодирующей соединительный (J) участок рецептора адаптивной иммунной системы или его комплемент, и каждый J содержит уникальную олигонуклеотидную последовательность J-участка; (с) U1 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) первой универсальной последовательности олигонуклеотида-адаптера и (ii) первой специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5' и находится в нем относительно первой универсальной последовательности олигонуклеотида-адаптера; (d) U2 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) второй универсальной последовательности олигонуклеотида-адаптера и (ii) второй специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5' и находится в нем относительно второй универсальной последовательности олигонуклеотида-адаптера; (e) присутствует по меньшей мере один из B1, В2, В3 и В4 и каждый из B1, В2, В3 и В4 содержит олигонуклеотид В, содержащий последовательность штрихкода из 3-25 последовательных нуклеотидов, которая уникальным образом идентифицирует в качестве спаренной комбинации, (i) уникальную олигонуклеотидную последовательность V-участка из (а) и (ii) уникальную олигонуклеотидную последовательность J-участка из (b); (f) R либо отсутствует, либо содержит сайт распознавания рестрикционного фермента, который содержит олигонуклеотидную последовательность, отсутствующую в (а)-(е); и причем: (g) множество синтетических матричных олигонуклеотидов содержит количество по меньшей мере а или по меньшей мере b уникальных олигонуклеотидных последовательностей в зависимости от того, какое значение больше, причем а представляет собой количество уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой количество уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта, и композиция содержит по меньшей мере один синтетический матричный олигонуклеотид для каждой уникальной олигонуклеотидной последовательности V-участка и по меньшей мере один синтетический матричный олигонуклеотид для каждой уникальной олигонуклеотидной последовательности J-участка. Также заявлен способ количественного определения множества перестроенных молекул нуклеиновых кислот, кодирующих один или множество рецепторов адаптивной иммунной системы в биологическом образце. 2 н. и 25 з.п. ф-лы, 14 ил., 17 табл., 12 пр.
1. Композиция для стандартизации эффективности амплификации набора олигонуклеотидных праймеров для амплификации перестроенных последовательностей нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы в биологическом образце, полученном из лимфоидных клеток субъекта-млекопитающего, причем каждый рецептор адаптивной иммунной системы содержит вариабельный участок и соединительный участок, причем композиция содержит:
множество синтетических матричных олигонуклеотидов, причем каждый синтетический матричный олигонуклеотид имеет известную концентрацию до амплификации и олигонуклеотидную последовательность общей формулы:
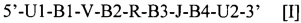 ,
,
причем:
(а) V представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 20 и не более 1000 последовательных нуклеотидов генной последовательности, кодирующей вариабельный (V) участок рецептора адаптивной иммунной системы или его комплемент, и каждый V содержит уникальную олигонуклеотидную последовательность V-участка;
(b) J представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 15 и не более 600 последовательных нуклеотидов генной последовательности, кодирующей соединительный (J) участок рецептора адаптивной иммунной системы или его комплемент, и каждый J содержит уникальную олигонуклеотидную последовательность J-участка;
(с) U1 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) первой универсальной последовательности олигонуклеотида-адаптера и (ii) первой специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5' и находится в нем относительно первой универсальной последовательности олигонуклеотида-адаптера;
(d) U2 либо отсутствует, либо содержит олигонуклеотидную последовательность, выбранную из (i) второй универсальной последовательности олигонуклеотида-адаптера и (ii) второй специфической для секвенирующей платформы олигонуклеотидной последовательности, которая связана с положением 5' и находится в нем относительно второй универсальной последовательности олигонуклеотида-адаптера;
(e) присутствует по меньшей мере один из B1, В2, В3 и В4 и каждый из B1, В2, В3 и В4 содержит олигонуклеотид В, содержащий последовательность штрихкода из 3-25 последовательных нуклеотидов, которая уникальным образом идентифицирует в качестве спаренной комбинации, (i) уникальную олигонуклеотидную последовательность V-участка из (а) и (ii) уникальную олигонуклеотидную последовательность J-участка из (b);
(f) R либо отсутствует, либо содержит сайт распознавания рестрикционного фермента, который содержит олигонуклеотидную последовательность, отсутствующую в (а)-(е);
и причем:
(g) множество синтетических матричных олигонуклеотидов содержит количество по меньшей мере а или по меньшей мере b уникальных олигонуклеотидных последовательностей в зависимости от того, какое значение больше, причем а представляет собой количество уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта, а b представляет собой количество уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта,
и композиция содержит по меньшей мере один синтетический матричный олигонуклеотид для каждой уникальной олигонуклеотидной последовательности V-участка и по меньшей мере один синтетический матричный олигонуклеотид для каждой уникальной олигонуклеотидной последовательности J-участка.
2. Композиция по п. 1, в которой a находится в диапазоне от 1 до максимального количества сегментов V-гена в геноме млекопитающего субъекта.
3. Композиция по любому из пп. 1 или 2, в которой b находится в диапазоне от 1 до максимального количества сегментов J-гена в геноме млекопитающего субъекта.
4. Композиция по п. 1, в которой а равно 1 или b равно 1.
5. Композиция по п. 1, в которой множество матричных олигонуклеотидов содержит по меньшей мере (а×b) уникальных олигонуклеотидных последовательностей, причем a представляет собой количество уникальных сегментов гена, кодирующего V-участок рецептора адаптивной иммунной системы у субъекта-млекопитающего, а b представляет собой количество уникальных сегментов гена, кодирующего J-участок рецептора адаптивной иммунной системы у субъекта-млекопитающего, и композиция содержит по меньшей мере один матричный олигонуклеотид для каждой возможной комбинации сегмента гена, кодирующего V-участок, и сегмента гена, кодирующего J-участок.
6. Композиция по п. 1, в которой J содержит олигонуклеотидную последовательность, содержащую константный участок генной последовательности, кодирующей J-участок рецептора адаптивной иммунной системы.
7. Композиция по п. 1, в которой рецептор адаптивной иммунной системы выбран из группы, состоящей из TCRB, TCRG, TCRA, TCRD, IGH, IGK и IGL.
8. Композиция по п. 1, в которой олигонуклеотидная последовательность V из (a) кодирует полипептид V-участка рецептора TCRB, TCRG, TCRA, TCRD, IGH, IGK или IGL.
9. Композиция по п. 1, в которой олигонуклеотидная последовательность J из (b) кодирует полипептид J-участка рецептора TCRB, TCRG, TCRA, TCRD, IGH, IGK или IGL.
10. Композиция по п. 1, дополнительно содержащая последовательность стоп-кодона между V и В2.
11. Композиция по п. 1, в которой каждый синтетический матричный олигонуклеотид во множестве синтетических матричных олигонуклеотидов присутствует в эквимолярном количестве.
12. Композиция по п. 1, в которой множество синтетических матричных олигонуклеотидов имеет множество последовательностей общей формулы (I), которая выбрана из:
(1) множества олигонуклеотидных последовательностей общей формулы (I), в которой олигонуклеотидные последовательности V и J имеют последовательности TCRB V и J, представленные в по меньшей мере одном наборе из 68 TCRB V и J SEQ ID NO, где
набор 1 содержит следующие последовательности TCRB V/J: 2921/2989, 2922/2990, 2923/2991, 2924/2992, 2925/2993, 2926/2994, 2927/2995, 2928/2996, 2929/2997, 2930/2998, 2931/2999, 2932/3000, 2933/3001, 2934/2989, 2935/2990, 2936/2991, 2937/2992, 2938/2993, 2939/2994, 2940/2995, 2941/2996, 2942/2997, 2943/2998, 2944/2999, 2945/3000, 2946/3001, 2947/2989, 2948/2990, 2949/2991, 2950/2992, 2951/2993, 2952/2994, 2953/2995, 2954/2996, 2955/2997, 2956/2998, 2957/2999, 2958/3000, 2959/3001, 2960/2989, 2961/2990, 2962/2991, 2963/2992, 2964/2993, 2965/2994, 2966/2995, 2967/2996, 2968/2997, 2969/2998, 2970/2999, 2971/3000, 2972/3001, 2973/2989, 2974/2990, 2975/2991, 2976/2992, 2977/2993, 2978/2994, 2979/2995, 2980/2996, 2981/2997, 2982/2998, 2983/2999, 2984/3000, 2985/3001, 2986/2989, 2987/2990, 2988/2991;
набор 2 содержит следующие последовательности TCRB V/J: 2921/2990, 2922/2991, 2923/2992, 2924/2993, 2925/2994, 2926/2995, 2927/2996, 2928/2997, 2929/2998, 2930/2999, 2931/3000, 2932/3001, 2933/2989, 2934/2990, 2935/2991, 2936/2992, 2937/2993, 2938/2994, 2939/2995, 2940/2996, 2941/2997, 2942/2998, 2943/2999, 2944/3000, 2945/3001, 2946/2989, 2947/2990, 2948/2991, 2949/2992, 2950/2993, 2951/2994, 2952/2995, 2953/2996, 2954/2997, 2955/2998, 2956/2999, 2957/3000, 2958/3001, 2959/2989, 2960/2990, 2961/2991, 2962/2992, 2963/2993, 2964/2994, 2965/2995, 2966/2996, 2967/2997, 2968/2998, 2969/2999, 2970/3000, 2971/3001, 2972/2989, 2973/2990, 2974/2991, 2975/2992, 2976/2993, 2977/2994, 2978/2995, 2979/2996, 2980/2997, 2981/2998, 2982/2999, 2983/3000, 2984/3001, 2985/2989, 2986/2990, 2987/2991, 2988/2992;
набор 3 содержит следующие последовательности TCRB V/J: 2921/2991, 2922/2992, 2923/2993, 2924/2994, 2925/2995, 2926/2996, 2927/2997, 2928/2998, 2929/2999, 2930/3000, 2931/3001, 2932/2989, 2933/2990, 2934/2991, 2935/2992, 2936/2993, 2937/2994, 2938/2995, 2939/2996, 2940/2997, 2941/2998, 2942/2999, 2943/3000, 2944/3001, 2945/2989, 2946/2990, 2947/2991, 2948/2992, 2949/2993, 2950/2994, 2951/2995, 2952/2996, 2953/2997, 2954/2998, 2955/2999, 2956/3000, 2957/3001, 2958/2989, 2959/2990, 2960/2991, 2961/2992, 2962/2993, 2963/2994, 2964/2995, 2965/2996, 2966/2997, 2967/2998, 2968/2999, 2969/3000, 2970/3001, 2971/2989, 2972/2990, 2973/2991, 2974/2992, 2975/2993, 2976/2994, 2977/2995, 2978/2996, 2979/2997, 2980/2998, 2981/2999, 2982/3000, 2983/3001, 2984/2989, 2985/2990, 2986/2991, 2987/2992, 2988/2993;
набор 4 содержит следующие последовательности TCRB V/J: 2921/2992, 2922/2993, 2923/2994, 2924/2995, 2925/2996, 2926/2997, 2927/2998, 2928/2999, 2929/3000, 2930/3001, 2931/2989, 2932/2990, 2933/2991, 2934/2992, 2935/2993, 2936/2994, 2937/2995, 2938/2996, 2939/2997, 2940/2998, 2941/2999, 2942/3000, 2943/3001, 2944/2989, 2945/2990, 2946/2991, 2947/2992, 2948/2993, 2949/2994, 2950/2995, 2951/2996, 2952/2997, 2953/2998, 2954/2999, 2955/3000, 2956/3001, 2957/2989, 2958/2990, 2959/2991, 2960/2992, 2961/2993, 2962/2994, 2963/2995, 2964/2996, 2965/2997, 2966/2998, 2967/2999, 2968/3000, 2969/3001, 2970/2989, 2971/2990, 2972/2991, 2973/2992, 2974/2993, 2975/2994, 2976/2995, 2977/2996, 2978/2997, 2979/2998, 2980/2999, 2981/3000, 2982/3001, 2983/2989, 2984/2990, 2985/2991, 2986/2992, 2987/2993, 2988/2994;
набор 5 содержит следующие последовательности TCRB V/J: 2921/2993, 2922/2994, 2923/2995, 2924/2996, 2925/2997, 2926/2998, 2927/2999, 2928/3000, 2929/3001, 2930/2989, 2931/2990, 2932/2991, 2933/2992, 2934/2993, 2935/2994, 2936/2995, 2937/2996, 2938/2997, 2939/2998, 2940/2999, 2941/3000, 2942/3001, 2943/2989, 2944/2990, 2945/2991, 2946/2992, 2947/2993, 2948/2994, 2949/2995, 2950/2996, 2951/2997, 2952/2998, 2953/2999, 2954/3000, 2955/3001, 2956/2989, 2957/2990, 2958/2991, 2959/2992, 2960/2993, 2961/2994, 2962/2995, 2963/2996, 2964/2997, 2965/2998, 2966/2999, 2967/3000, 2968/3001, 2969/2989, 2970/2990, 2971/2991, 2972/2992, 2973/2993, 2974/2994, 2975/2995, 2976/2996, 2977/2997, 2978/2998, 2979/2999, 2980/3000, 2981/3001, 2982/2989, 2983/2990, 2984/2991, 2985/2992, 2986/2993, 2987/2994, 2988/2995;
набор 6 содержит следующие последовательности TCRB V/J: 2921/2994, 2922/2995, 2923/2996, 2924/2997, 2925/2998, 2926/2999, 2927/3000, 2928/3001, 2929/2989, 2930/2990, 2931/2991, 2932/2992, 2933/2993, 2934/2994, 2935/2995, 2936/2996, 2937/2997, 2938/2998, 2939/2999, 2940/3000, 2941/3001, 2942/2989, 2943/2990, 2944/2991, 2945/2992, 2946/2993, 2947/2994, 2948/2995, 2949/2996, 2950/2997, 2951/2998, 2952/2999, 2953/3000, 2954/3001, 2955/2989, 2956/2990, 2957/2991, 2958/2992, 2959/2993, 2960/2994, 2961/2995, 2962/2996, 2963/2997, 2964/2998, 2965/2999, 2966/3000, 2967/3001, 2968/2989, 2969/2990, 2970/2991, 2971/2992, 2972/2993, 2973/2994, 2974/2995, 2975/2996, 2976/2997, 2977/2998, 2978/2999, 2979/3000, 2980/3001, 2981/2989, 2982/2990, 2983/2991, 2984/2992, 2985/2993, 2986/2994, 2987/2995, 2988/2996;
набор 7 содержит следующие последовательности TCRB V/J: 2921/2995, 2922/2996, 2923/2997, 2924/2998, 2925/2999, 2926/3000, 2927/3001, 2928/2989, 2929/2990, 2930/2991, 2931/2992, 2932/2993, 2933/2994, 2934/2995, 2935/2996, 2936/2997, 2937/2998, 2938/2999, 2939/3000, 2940/3001, 2941/2989, 2942/2990, 2943/2991, 2944/2992, 2945/2993, 2946/2994, 2947/2995, 2948/2996, 2949/2997, 2950/2998, 2951/2999, 2952/3000, 2953/3001, 2954/2989, 2955/2990, 2956/2991, 2957/2992, 2958/2993, 2959/2994, 2960/2995, 2961/2996, 2962/2997, 2963/2998, 2964/2999, 2965/3000, 2966/3001, 2967/2989, 2968/2990, 2969/2991, 2970/2992, 2971/2993, 2972/2994, 2973/2995, 2974/2996, 2975/2997, 2976/2998, 2977/2999, 2978/3000, 2979/3001, 2980/2989, 2981/2990, 2982/2991, 2983/2992, 2984/2993, 2985/2994, 2986/2995, 2987/2996, 2988/2997;
набор 8 содержит следующие последовательности TCRB V/J: 2921/2996, 2922/2997, 2923/2998, 2924/2999, 2925/3000, 2926/3001, 2927/2989, 2928/2990, 2929/2991, 2930/2992, 2931/2993, 2932/2994, 2933/2995, 2934/2996, 2935/2997, 2936/2998, 2937/2999, 2938/3000, 2939/3001, 2940/2989, 2941/2990, 2942/2991, 2943/2992, 2944/2993, 2945/2994, 2946/2995, 2947/2996, 2948/2997, 2949/2998, 2950/2999, 2951/3000, 2952/3001, 2953/2989, 2954/2990, 2955/2991, 2956/2992, 2957/2993, 2958/2994, 2959/2995, 2960/2996, 2961/2997, 2962/2998, 2963/2999, 2964/3000, 2965/3001, 2966/2989, 2967/2990, 2968/2991, 2969/2992, 2970/2993, 2971/2994, 2972/2995, 2973/2996, 2974/2997, 2975/2998, 2976/2999, 2977/3000, 2978/3001, 2979/2989, 2980/2990, 2981/2991, 2982/2992, 2983/2993, 2984/2994, 2985/2995, 2986/2996, 2987/2997, 2988/2998;
набор 9 содержит следующие последовательности TCRB V/J: 2921/2997, 2922/2998, 2923/2999, 2924/3000, 2925/3001, 2926/2989, 2927/2990, 2928/2991, 2929/2992, 2930/2993, 2931/2994, 2932/2995, 2933/2996, 2934/2997, 2935/2998, 2936/2999, 2937/3000, 2938/3001, 2939/2989, 2940/2990, 2941/2991, 2942/2992, 2943/2993, 2944/2994, 2945/2995, 2946/2996, 2947/2997, 2948/2998, 2949/2999, 2950/3000, 2951/3001, 2952/2989, 2953/2990, 2954/2991, 2955/2992, 2956/2993, 2957/2994, 2958/2995, 2959/2996, 2960/2997, 2961/2998, 2962/2999, 2963/3000, 2964/3001, 2965/2989, 2966/2990, 2967/2991, 2968/2992, 2969/2993, 2970/2994, 2971/2995, 2972/2996, 2973/2997, 2974/2998, 2975/2999, 2976/3000, 2977/3001, 2978/2989, 2979/2990, 2980/2991, 2981/2992, 2982/2993, 2983/2994, 2984/2995, 2985/2996, 2986/2997, 2987/2998, 2988/2999;
набор 10 содержит следующие последовательности TCRB V/J: 2921/2998, 2922/2999, 2923/3000, 2924/3001, 2925/2989, 2926/2990, 2927/2991, 2928/2992, 2929/2993, 2930/2994, 2931/2995, 2932/2996, 2933/2997, 2934/2998, 2935/2999, 2936/3000, 2937/3001, 2938/2989, 2939/2990, 2940/2991, 2941/2992, 2942/2993, 2943/2994, 2944/2995, 2945/2996, 2946/2997, 2947/2998, 2948/2999, 2949/3000, 2950/3001, 2951/2989, 2952/2990, 2953/2991, 2954/2992, 2955/2993, 2956/2994, 2957/2995, 2958/2996, 2959/2997, 2960/2998, 2961/2999, 2962/3000, 2963/3001, 2964/2989, 2965/2990, 2966/2991, 2967/2992, 2968/2993, 2969/2994, 2970/2995, 2971/2996, 2972/2997, 2973/2998, 2974/2999, 2975/3000, 2976/3001, 2977/2989, 2978/2990, 2979/2991, 2980/2992, 2981/2993, 2982/2994, 2983/2995, 2984/2996, 2985/2997, 2986/2998, 2987/2999, 2988/3000;
набор 11 содержит следующие последовательности TCRB V/J: 2921/2999, 2922/3000, 2923/3001, 2924/2989, 2925/2990, 2926/2991, 2927/2992, 2928/2993, 2929/2994, 2930/2995, 2931/2996, 2932/2997, 2933/2998, 2934/2999, 2935/3000, 2936/3001, 2937/2989, 2938/2990, 2939/2991, 2940/2992, 2941/2993, 2942/2994, 2943/2995, 2944/2996, 2945/2997, 2946/2998, 2947/2999, 2948/3000, 2949/3001, 2950/2989, 2951/2990, 2952/2991, 2953/2992, 2954/2993, 2955/2994, 2956/2995, 2957/2996, 2958/2997, 2959/2998, 2960/2999, 2961/3000, 2962/3001, 2963/2989, 2964/2990, 2965/2991, 2966/2992, 2967/2993, 2968/2994, 2969/2995, 2970/2996, 2971/2997, 2972/2998, 2973/2999, 2974/3000, 2975/3001, 2976/2989, 2977/2990, 2978/2991, 2979/2992, 2980/2993, 2981/2994, 2982/2995, 2983/2996, 2984/2997, 2985/2998, 2986/2999, 2987/3000, 2988/3001;
набор 12 содержит следующие последовательности TCRB V/J: 2921/3000, 2922/3001, 2923/2989, 2924/2990, 2925/2991, 2926/2992, 2927/2993, 2928/2994, 2929/2995, 2930/2996, 2931/2997, 2932/2998, 2933/2999, 2934/3000, 2935/3001, 2936/2989, 2937/2990, 2938/2991, 2939/2992, 2940/2993, 2941/2994, 2942/2995, 2943/2996, 2944/2997, 2945/2998, 2946/2999, 2947/3000, 2948/3001, 2949/2989, 2950/2990, 2951/2991, 2952/2992, 2953/2993, 2954/2994, 2955/2995, 2956/2996, 2957/2997, 2958/2998, 2959/2999, 2960/3000, 2961/3001, 2962/2989, 2963/2990, 2964/2991, 2965/2992, 2966/2993, 2967/2994, 2968/2995, 2969/2996, 2970/2997, 2971/2998, 2972/2999, 2973/3000, 2974/3001, 2975/2989, 2976/2990, 2977/2991, 2978/2992, 2979/2993, 2980/2994, 2981/2995, 2982/2996, 2983/2997, 2984/2998, 2985/2999, 2986/3000, 2987/3001, 2988/2989; и
набор 13 содержит следующие последовательности TCRB V/J: 2921/3001, 2922/2989, 2923/2990, 2924/2991, 2925/2992, 2926/2993, 2927/2994, 2928/2995, 2929/2996, 2930/2997, 2931/2998, 2932/2999, 2933/3000, 2934/3001, 2935/2989, 2936/2990, 2937/2991, 2938/2992, 2939/2993, 2940/2994, 2941/2995, 2942/2996, 2943/2997, 2944/2998, 2945/2999, 2946/3000, 2947/3001, 2948/2989, 2949/2990, 2950/2991, 2951/2992, 2952/2993, 2953/2994, 2954/2995, 2955/2996, 2956/2997, 2957/2998, 2958/2999, 2959/3000, 2960/3001, 2961/2989, 2962/2990, 2963/2991, 2964/2992, 2965/2993, 2966/2994, 2967/2995, 2968/2996, 2969/2997, 2970/2998, 2971/2999, 2972/3000, 2973/3001, 2974/2989, 2975/2990, 2976/2991, 2977/2992, 2978/2993, 2979/2994, 2980/2995, 2981/2996, 2982/2997, 2983/2998, 2984/2999, 2985/3000, 2986/3001, 2987/2989, 2988/2990;
(2) множества олигонуклеотидных последовательностей общей формулы (I), в которой олигонуклеотидные последовательности V и J имеют последовательности TCRG V и J, представленные в по меньшей мере одном наборе из 14 TCRG V и J SEQ ID NO, причем
набор 1 содержит следующие последовательности TCRG V/J: 3002/3016, 3003/3017, 3004/3018, 3005/3019, 3006/3020, 3007/3016, 3008/3017, 3009/3018, 3010/3019, 3011/3020, 3012/3016, 3013/3017, 3014/3018, 3015/3019;
набор 2 содержит следующие последовательности TCRG V/J: 3002/3017, 3003/3018, 3004/3019, 3005/3020, 3006/3016, 3007/3017, 3008/3018, 3009/3019, 3010/3020, 3011/3016, 3012/3017, 3013/3018, 3014/3019, 3015/3020;
набор 3 содержит следующие последовательности TCRG V/J: 3002/3018, 3003/3019, 3004/3020, 3005/3016, 3006/3017, 3007/3018, 3008/3019, 3009/3020, 3010/3016, 3011/3017, 3012/3018, 3013/3019, 3014/3020, 3015/3016;
набор 4 содержит следующие последовательности TCRG V/J: 3002/3019, 3003/3020, 3004/3016, 3005/3017, 3006/3018, 3007/3019, 3008/3020, 3009/3016, 3010/3017, 3011/3018, 3012/3019, 3013/3020, 3014/3016, 3015/3017; и
набор 5 содержит следующие последовательности TCRG V/J: 3002/3020, 3003/3016, 3004/3017, 3005/3018, 3006/3019, 3007/3020, 3008/3016, 3009/3017, 3010/3018, 3011/3019, 3012/3020, 3013/3016, 3014/3017, 3015/3018;
(3) множества олигонуклеотидных последовательностей общей формулы (I), в которой олигонуклеотидные последовательности V и J имеют последовательности IGH V и J, представленные в по меньшей мере одном наборе из 127 IGH V и J SEQ ID NO, причем
набор 1 содержит следующие последовательности IGH V/J: 3021/3148, 3022/3149, 3023/3150, 3024/3151, 3025/3152, 3026/3153, 3027/3154, 3028/3155, 3029/3156, 3030/3148, 3031/3149, 3032/3150, 3033/3151, 3034/3152, 3035/3153, 3036/3154, 3037/3155, 3038/3156, 3039/3148, 3040/3149, 3041/3150, 3042/3151, 3043/3152, 3044/3153, 3045/3154, 3046/3155, 3047/3156, 3048/3148, 3049/3149, 3050/3150, 3051/3151, 3052/3152, 3053/3153, 3054/3154, 3055/3155, 3056/3156, 3057/3148, 3058/3149, 3059/3150, 3060/3151, 3061/3152, 3062/3153, 3063/3154, 3064/3155, 3065/3156, 3066/3148, 3067/3149, 3068/3150, 3069/3151, 3070/3152, 3071/3153, 3072/3154, 3073/3155, 3074/3156, 3075/3148, 3076/3149, 3077/3150, 3078/3151, 3079/3152, 3080/3153, 3081/3154, 3082/3155, 3083/3156, 3084/3148, 3085/3149, 3086/3150, 3087/3151, 3088/3152, 3089/3153, 3090/3154, 3091/3155, 3092/3156, 3093/3148, 3094/3149, 3095/3150, 3096/3151, 3097/3152, 3098/3153, 3099/3154, 3100/3155, 3101/3156, 3102/3148, 3103/3149, 3104/3150, 3105/3151, 3106/3152, 3107/3153, 3108/3154, 3109/3155, 3110/3156, 3111/3148, 3112/3149, 3113/3150, 3114/3151, 3115/3152, 3116/3153, 3117/3154, 3118/3155, 3119/3156, 3120/3148, 3121/3149, 3122/3150, 3123/3151, 3124/3152, 3125/3153, 3126/3154, 3127/3155, 3128/3156, 3129/3148, 3130/3149, 3131/3150, 3132/3151, 3133/3152, 3134/3153, 3135/3154, 3136/3155, 3137/3156, 3138/3148, 3139/3149, 3140/3150, 3141/3151, 3142/3152, 3143/3153, 3144/3154, 3145/3155, 3146/3156, 3147/3148;
набор 2 содержит следующие последовательности IGH V/J: 3021/3149, 3022/3150, 3023/3151, 3024/3152, 3025/3153, 3026/3154, 3027/3155, 3028/3156, 3029/3148, 3030/3149, 3031/3150, 3032/3151, 3033/3152, 3034/3153, 3035/3154, 3036/3155, 3037/3156, 3038/3148, 3039/3149, 3040/3150, 3041/3151, 3042/3152, 3043/3153, 3044/3154, 3045/3155, 3046/3156, 3047/3148, 3048/3149, 3049/3150, 3050/3151, 3051/3152, 3052/3153, 3053/3154, 3054/3155, 3055/3156, 3056/3148, 3057/3149, 3058/3150, 3059/3151, 3060/3152, 3061/3153, 3062/3154, 3063/3155, 3064/3156, 3065/3148, 3066/3149, 3067/3150, 3068/3151, 3069/3152, 3070/3153, 3071/3154, 3072/3155, 3073/3156, 3074/3148, 3075/3149, 3076/3150, 3077/3151, 3078/3152, 3079/3153, 3080/3154, 3081/3155, 3082/3156, 3083/3148, 3084/3149, 3085/3150, 3086/3151, 3087/3152, 3088/3153, 3089/3154, 3090/3155, 3091/3156, 3092/3148, 3093/3149, 3094/3150, 3095/3151, 3096/3152, 3097/3153, 3098/3154, 3099/3155, 3100/3156, 3101/3148, 3102/3149, 3103/3150, 3104/3151, 3105/3152, 3106/3153, 3107/3154, 3108/3155, 3109/3156, 3110/3148, 3111/3149, 3112/3150, 3113/3151, 3114/3152, 3115/3153, 3116/3154, 3117/3155, 3118/3156, 3119/3148, 3120/3149, 3121/3150, 3122/3151, 3123/3152, 3124/3153, 3125/3154, 3126/3155, 3127/3156, 3128/3148, 3129/3149, 3130/3150, 3131/3151, 3132/3152, 3133/3153, 3134/3154, 3135/3155, 3136/3156, 3137/3148, 3138/3149, 3139/3150, 3140/3151, 3141/3152, 3142/3153, 3143/3154, 3144/3155, 3145/3156, 3146/3148, 3147/3149;
набор 3 содержит следующие последовательности IGH V/J: 3021/3150, 3022/3151, 3023/3152, 3024/3153, 3025/3154, 3026/3155, 3027/3156, 3028/3148, 3029/3149, 3030/3150, 3031/3151, 3032/3152, 3033/3153, 3034/3154, 3035/3155, 3036/3156, 3037/3148, 3038/3149, 3039/3150, 3040/3151, 3041/3152, 3042/3153, 3043/3154, 3044/3155, 3045/3156, 3046/3148, 3047/3149, 3048/3150, 3049/3151, 3050/3152, 3051/3153, 3052/3154, 3053/3155, 3054/3156, 3055/3148, 3056/3149, 3057/3150, 3058/3151, 3059/3152, 3060/3153, 3061/3154, 3062/3155, 3063/3156, 3064/3148, 3065/3149, 3066/3150, 3067/3151, 3068/3152, 3069/3153, 3070/3154, 3071/3155, 3072/3156, 3073/3148, 3074/3149, 3075/3150, 3076/3151, 3077/3152, 3078/3153, 3079/3154, 3080/3155, 3081/3156, 3082/3148, 3083/3149, 3084/3150, 3085/3151, 3086/3152, 3087/3153, 3088/3154, 3089/3155, 3090/3156, 3091/3148, 3092/3149, 3093/3150, 3094/3151, 3095/3152, 3096/3153, 3097/3154, 3098/3155, 3099/3156, 3100/3148, 3101/3149, 3102/3150, 3103/3151, 3104/3152, 3105/3153, 3106/3154, 3107/3155, 3108/3156, 3109/3148, 3110/3149, 3111/3150, 3112/3151, 3113/3152, 3114/3153, 3115/3154, 3116/3155, 3117/3156, 3118/3148, 3119/3149, 3120/3150, 3121/3151, 3122/3152, 3123/3153, 3124/3154, 3125/3155, 3126/3156, 3127/3148, 3128/3149, 3129/3150, 3130/3151, 3131/3152, 3132/3153, 3133/3154, 3134/3155, 3135/3156, 3136/3148, 3137/3149, 3138/3150, 3139/3151, 3140/3152, 3141/3153, 3142/3154, 3143/3155, 3144/3156, 3145/3148, 3146/3149, 3147/3150;
набор 4 содержит следующие последовательности IGH V/J: 3021/3151, 3022/3152, 3023/3153, 3024/3154, 3025/3155, 3026/3156, 3027/3148, 3028/3149, 3029/3150, 3030/3151, 3031/3152, 3032/3153, 3033/3154, 3034/3155, 3035/3156, 3036/3148, 3037/3149, 3038/3150, 3039/3151, 3040/3152, 3041/3153, 3042/3154, 3043/3155, 3044/3156, 3045/3148, 3046/3149, 3047/3150, 3048/3151, 3049/3152, 3050/3153, 3051/3154, 3052/3155, 3053/3156, 3054/3148, 3055/3149, 3056/3150, 3057/3151, 3058/3152, 3059/3153, 3060/3154, 3061/3155, 3062/3156, 3063/3148, 3064/3149, 3065/3150, 3066/3151, 3067/3152, 3068/3153, 3069/3154, 3070/3155, 3071/3156, 3072/3148, 3073/3149, 3074/3150, 3075/3151, 3076/3152, 3077/3153, 3078/3154, 3079/3155, 3080/3156, 3081/3148, 3082/3149, 3083/3150, 3084/3151, 3085/3152, 3086/3153, 3087/3154, 3088/3155, 3089/3156, 3090/3148, 3091/3149, 3092/3150, 3093/3151, 3094/3152, 3095/3153, 3096/3154, 3097/3155, 3098/3156, 3099/3148, 3100/3149, 3101/3150, 3102/3151, 3103/3152, 3104/3153, 3105/3154, 3106/3155, 3107/3156, 3108/3148, 3109/3149, 3110/3150, 3111/3151, 3112/3152, 3113/3153, 3114/3154, 3115/3155, 3116/3156, 3117/3148, 3118/3149, 3119/3150, 3120/3151, 3121/3152, 3122/3153, 3123/3154, 3124/3155, 3125/3156, 3126/3148, 3127/3149, 3128/3150, 3129/3151, 3130/3152, 3131/3153, 3132/3154, 3133/3155, 3134/3156, 3135/3148, 3136/3149, 3137/3150, 3138/3151, 3139/3152, 3140/3153, 3141/3154, 3142/3155, 3143/3156, 3144/3148, 3145/3149, 3146/3150, 3147/3151;
набор 5 содержит следующие последовательности IGH V/J: 3021/3152, 3022/3153, 3023/3154, 3024/3155, 3025/3156, 3026/3148, 3027/3149, 3028/3150, 3029/3151, 3030/3152, 3031/3153, 3032/3154, 3033/3155, 3034/3156, 3035/3148, 3036/3149, 3037/3150, 3038/3151, 3039/3152, 3040/3153, 3041/3154, 3042/3155, 3043/3156, 3044/3148, 3045/3149, 3046/3150, 3047/3151, 3048/3152, 3049/3153, 3050/3154, 3051/3155, 3052/3156, 3053/3148, 3054/3149, 3055/3150, 3056/3151, 3057/3152, 3058/3153, 3059/3154, 3060/3155, 3061/3156, 3062/3148, 3063/3149, 3064/3150, 3065/3151, 3066/3152, 3067/3153, 3068/3154, 3069/3155, 3070/3156, 3071/3148, 3072/3149, 3073/3150, 3074/3151, 3075/3152, 3076/3153, 3077/3154, 3078/3155, 3079/3156, 3080/3148, 3081/3149, 3082/3150, 3083/3151, 3084/3152, 3085/3153, 3086/3154, 3087/3155, 3088/3156, 3089/3148, 3090/3149, 3091/3150, 3092/3151, 3093/3152, 3094/3153, 3095/3154, 3096/3155, 3097/3156, 3098/3148, 3099/3149, 3100/3150, 3101/3151, 3102/3152, 3103/3153, 3104/3154, 3105/3155, 3106/3156, 3107/3148, 3108/3149, 3109/3150, 3110/3151, 3111/3152, 3112/3153, 3113/3154, 3114/3155, 3115/3156, 3116/3148, 3117/3149, 3118/3150, 3119/3151, 3120/3152, 3121/3153, 3122/3154, 3123/3155, 3124/3156, 3125/3148, 3126/3149, 3127/3150, 3128/3151, 3129/3152, 3130/3153, 3131/3154, 3132/3155, 3133/3156, 3134/3148, 3135/3149, 3136/3150, 3137/3151, 3138/3152, 3139/3153, 3140/3154, 3141/3155, 3142/3156, 3143/3148, 3144/3149, 3145/3150, 3146/3151, 3147/3152;
набор 6 содержит следующие последовательности IGH V/J: 3021/3153, 3022/3154, 3023/3155, 3024/3156, 3025/3148, 3026/3149, 3027/3150, 3028/3151, 3029/3152, 3030/3153, 3031/3154, 3032/3155, 3033/3156, 3034/3148, 3035/3149, 3036/3150, 3037/3151, 3038/3152, 3039/3153, 3040/3154, 3041/3155, 3042/3156, 3043/3148, 3044/3149, 3045/3150, 3046/3151, 3047/3152, 3048/3153, 3049/3154, 3050/3155, 3051/3156, 3052/3148, 3053/3149, 3054/3150, 3055/3151, 3056/3152, 3057/3153, 3058/3154, 3059/3155, 3060/3156, 3061/3148, 3062/3149, 3063/3150, 3064/3151, 3065/3152, 3066/3153, 3067/3154, 3068/3155, 3069/3156, 3070/3148, 3071/3149, 3072/3150, 3073/3151, 3074/3152, 3075/3153, 3076/3154, 3077/3155, 3078/3156, 3079/3148, 3080/3149, 3081/3150, 3082/3151, 3083/3152, 3084/3153, 3085/3154, 3086/3155, 3087/3156, 3088/3148, 3089/3149, 3090/3150, 3091/3151, 3092/3152, 3093/3153, 3094/3154, 3095/3155, 3096/3156, 3097/3148, 3098/3149, 3099/3150, 3100/3151, 3101/3152, 3102/3153, 3103/3154, 3104/3155, 3105/3156, 3106/3148, 3107/3149, 3108/3150, 3109/3151, 3110/3152, 3111/3153, 3112/3154, 3113/3155, 3114/3156, 3115/3148, 3116/3149, 3117/3150, 3118/3151, 3119/3152, 3120/3153, 3121/3154, 3122/3155, 3123/3156, 3124/3148, 3125/3149, 3126/3150, 3127/3151, 3128/3152, 3129/3153, 3130/3154, 3131/3155, 3132/3156, 3133/3148, 3134/3149, 3135/3150, 3136/3151, 3137/3152, 3138/3153, 3139/3154, 3140/3155, 3141/3156, 3142/3148, 3143/3149, 3144/3150, 3145/3151, 3146/3152, 3147/3153;
набор 7 содержит следующие последовательности IGH V/J: 3021/3154, 3022/3155, 3023/3156, 3024/3148, 3025/3149, 3026/3150, 3027/3151, 3028/3152, 3029/3153, 3030/3154, 3031/3155, 3032/3156, 3033/3148, 3034/3149, 3035/3150, 3036/3151, 3037/3152, 3038/3153, 3039/3154, 3040/3155, 3041/3156, 3042/3148, 3043/3149, 3044/3150, 3045/3151, 3046/3152, 3047/3153, 3048/3154, 3049/3155, 3050/3156, 3051/3148, 3052/3149, 3053/3150, 3054/3151, 3055/3152, 3056/3153, 3057/3154, 3058/3155, 3059/3156, 3060/3148, 3061/3149, 3062/3150, 3063/3151, 3064/3152, 3065/3153, 3066/3154, 3067/3155, 3068/3156, 3069/3148, 3070/3149, 3071/3150, 3072/3151, 3073/3152, 3074/3153, 3075/3154, 3076/3155, 3077/3156, 3078/3148, 3079/3149, 3080/3150, 3081/3151, 3082/3152, 3083/3153, 3084/3154, 3085/3155, 3086/3156, 3087/3148, 3088/3149, 3089/3150, 3090/3151, 3091/3152, 3092/3153, 3093/3154, 3094/3155, 3095/3156, 3096/3148, 3097/3149, 3098/3150, 3099/3151, 3100/3152, 3101/3153, 3102/3154, 3103/3155, 3104/3156, 3105/3148, 3106/3149, 3107/3150, 3108/3151, 3109/3152, 3110/3153, 3111/3154, 3112/3155, 3113/3156, 3114/3148, 3115/3149, 3116/3150, 3117/3151, 3118/3152, 3119/3153, 3120/3154, 3121/3155, 3122/3156, 3123/3148, 3124/3149, 3125/3150, 3126/3151, 3127/3152, 3128/3153, 3129/3154, 3130/3155, 3131/3156, 3132/3148, 3133/3149, 3134/3150, 3135/3151, 3136/3152, 3137/3153, 3138/3154, 3139/3155, 3140/3156, 3141/3148, 3142/3149, 3143/3150, 3144/3151, 3145/3152, 3146/3153, 3147/3154;
набор 8 содержит следующие последовательности IGH V/J: 3021/3155, 3022/3156, 3023/3148, 3024/3149, 3025/3150, 3026/3151, 3027/3152, 3028/3153, 3029/3154, 3030/3155, 3031/3156, 3032/3148, 3033/3149, 3034/3150, 3035/3151, 3036/3152, 3037/3153, 3038/3154, 3039/3155, 3040/3156, 3041/3148, 3042/3149, 3043/3150, 3044/3151, 3045/3152, 3046/3153, 3047/3154, 3048/3155, 3049/3156, 3050/3148, 3051/3149, 3052/3150, 3053/3151, 3054/3152, 3055/3153, 3056/3154, 3057/3155, 3058/3156, 3059/3148, 3060/3149, 3061/3150, 3062/3151, 3063/3152, 3064/3153, 3065/3154, 3066/3155, 3067/3156, 3068/3148, 3069/3149, 3070/3150, 3071/3151, 3072/3152, 3073/3153, 3074/3154, 3075/3155, 3076/3156, 3077/3148, 3078/3149, 3079/3150, 3080/3151, 3081/3152, 3082/3153, 3083/3154, 3084/3155, 3085/3156, 3086/3148, 3087/3149, 3088/3150, 3089/3151, 3090/3152, 3091/3153, 3092/3154, 3093/3155, 3094/3156, 3095/3148, 3096/3149, 3097/3150, 3098/3151, 3099/3152, 3100/3153, 3101/3154, 3102/3155, 3103/3156, 3104/3148, 3105/3149, 3106/3150, 3107/3151, 3108/3152, 3109/3153, 3110/3154, 3111/3155, 3112/3156, 3113/3148, 3114/3149, 3115/3150, 3116/3151, 3117/3152, 3118/3153, 3119/3154, 3120/3155, 3121/3156, 3122/3148, 3123/3149, 3124/3150, 3125/3151, 3126/3152, 3127/3153, 3128/3154, 3129/3155, 3130/3156, 3131/3148, 3132/3149, 3133/3150, 3134/3151, 3135/3152, 3136/3153, 3137/3154, 3138/3155, 3139/3156, 3140/3148, 3141/3149, 3142/3150, 3143/3151, 3144/3152, 3145/3153, 3146/3154, 3147/3155; и
набор 9 содержит следующие последовательности IGH V/J: 3021/3156, 3022/3148, 3023/3149, 3024/3150, 3025/3151, 3026/3152, 3027/3153, 3028/3154, 3029/3155, 3030/3156, 3031/3148, 3032/3149, 3033/3150, 3034/3151, 3035/3152, 3036/3153, 3037/3154, 3038/3155, 3039/3156, 3040/3148, 3041/3149, 3042/3150, 3043/3151, 3044/3152, 3045/3153, 3046/3154, 3047/3155, 3048/3156, 3049/3148, 3050/3149, 3051/3150, 3052/3151, 3053/3152, 3054/3153, 3055/3154, 3056/3155, 3057/3156, 3058/3148, 3059/3149, 3060/3150, 3061/3151, 3062/3152, 3063/3153, 3064/3154, 3065/3155, 3066/3156, 3067/3148, 3068/3149, 3069/3150, 3070/3151, 3071/3152, 3072/3153, 3073/3154, 3074/3155, 3075/3156, 3076/3148, 3077/3149, 3078/3150, 3079/3151, 3080/3152, 3081/3153, 3082/3154, 3083/3155, 3084/3156, 3085/3148, 3086/3149, 3087/3150, 3088/3151, 3089/3152, 3090/3153, 3091/3154, 3092/3155, 3093/3156, 3094/3148, 3095/3149, 3096/3150, 3097/3151, 3098/3152, 3099/3153, 3100/3154, 3101/3155, 3102/3156, 3103/3148, 3104/3149, 3105/3150, 3106/3151, 3107/3152, 3108/3153, 3109/3154, 3110/3155, 3111/3156, 3112/3148, 3113/3149, 3114/3150, 3115/3151, 3116/3152, 3117/3153, 3118/3154, 3119/3155, 3120/3156, 3121/3148, 3122/3149, 3123/3150, 3124/3151, 3125/3152, 3126/3153, 3127/3154, 3128/3155, 3129/3156, 3130/3148, 3131/3149, 3132/3150, 3133/3151, 3134/3152, 3135/3153, 3136/3154, 3137/3155, 3138/3156, 3139/3148, 3140/3149, 3141/3150, 3142/3151, 3143/3152, 3144/3153, 3145/3154, 3146/3155, 3147/3156;
(4) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 3157-4014;
(5) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 4015-4084;
(6) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 4085-5200;
(7) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 5579-5821;
(8) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 5822-6066; и
(9) множества олигонуклеотидных последовательностей общей формулы (I), представленных в SEQ ID NO: 6067-6191.
13. Композиция по п. 1, в которой V представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 30, 60, 90, 120, 150, 180 или 210 последовательных нуклеотидов генной последовательности, кодирующей V-участок рецептора адаптивной иммунной системы или его комплемент.
14. Композиция по п. 1, в которой V представляет собой олигонуклеотидную последовательность, содержащую не более 900, 800, 700, 600 или 500 последовательных нуклеотидов генной последовательности, кодирующей V-участок рецептора адаптивной иммунной системы или его комплемент.
15. Композиция по п. 1, в которой J представляет собой олигонуклеотидную последовательность, содержащую по меньшей мере 16-30, 31-60, 61-90, 91-120 или 120-150 последовательных нуклеотидов генной последовательности, кодирующей J-участок рецептора адаптивной иммунной системы или его комплемент.
16. Композиция по п. 1, в которой J представляет собой олигонуклеотидную последовательность, содержащую не более 500, 400, 300 или 200 последовательных нуклеотидов генной последовательности, кодирующей J-участок рецептора адаптивной иммунной системы или его комплемент.
17. Композиция по п. 1, в которой каждый синтетический матричный олигонуклеотид имеет длину менее 1000, 900, 800, 700, 600, 500, 400, 300 или 200 нуклеотидов.
18. Композиция по п. 1, дополнительно содержащая: набор олигонуклеотидных праймеров, который способен проводить амплификацию перестроенных молекул нуклеиновых кислот, кодирующих один или более рецепторов адаптивной иммунной системы, содержащий множество a' уникальных олигонуклеотидных праймеров для V-сегмента и множество b' уникальных олигонуклеотидных праймеров для J-сегмента.
19. Композиция по п. 18, в которой а' находится в диапазоне от 1 до максимального числа сегментов V-гена в геноме млекопитающего, а b' находится в диапазоне от 1 до максимального числа сегментов J-гена в геноме млекопитающего.
20. Композиция по п. 19, в которой a' равно а.
21. Композиция по п. 19, в которой b' равно b.
22. Композиция по любому из пп. 18-21, в которой каждый олигонуклеотидный праймер для V-сегмента и каждый олигонуклеотидный праймер для J-сегмента в наборе олигонуклеотидных праймеров способен специфически гибридизироваться с по меньшей мере одним матричным олигонуклеотидом из множества матричных олигонуклеотидов.
23. Композиция по любому из пп. 18-21, в которой каждый олигонуклеотидный праймер для V-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному сегменту гена, кодирующего V-участок рецептора адаптивной иммунной системы.
24. Композиция по любому из пп. 18-21, в которой каждый олигонуклеотидный праймер для J-сегмента содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному сегменту гена, кодирующего J-участок рецептора адаптивной иммунной системы.
25. Композиция по любому из пп. 18-21, в которой композиция содержит по меньшей мере один синтетический матричный олигонуклеотид, имеющий олигонуклеотидную последовательность общей формулы (I), с которым может специфически гибридизироваться каждый олигонуклеотидный праймер для V-сегмента, и по меньшей мере один матричный олигонуклеотид, имеющий олигонуклеотидную последовательность общей формулы (I), с которым может специфически гибридизироваться каждый олигонуклеотидный праймер для J-сегмента.
26. Способ количественного определения множества перестроенных молекул нуклеиновых кислот, кодирующих один или множество рецепторов адаптивной иммунной системы в биологическом образце, содержащем перестроенные молекулы нуклеиновых кислот из лимфоидных клеток субъекта-млекопитающего, причем каждый рецептор адаптивной иммунной системы содержит вариабельный (V) участок и соединительный (J) участок, причем способ включает:
(А) амплификацию перестроенных молекул нуклеиновых кислот в одиночной мультиплексной полимеразной цепной реакции (ПЦР), которая содержит:
(1) перестроенные молекулы нуклеиновых кислот, полученные из биологического образца, содержащего лимфоидные клетки субъекта-млекопитающего;
(2) композицию, содержащую указанное множество синтетических матричных олигонуклеотидов и указанный набор олигонуклеотидных праймеров по любому из пп. 18-25, в которой в известном количестве присутствует каждый из множества матричных олигонуклеотидов, имеющих уникальную олигонуклеотидную последовательность;
(3) набор олигонуклеотидных праймеров для амплификации, проводящий амплификацию перестроенных молекул нуклеиновых кислот, кодирующих один или множество рецепторов адаптивной иммунной системы из биологического образца, причем набор праймеров содержит:
(a) в по существу эквимолярных количествах множество олигонуклеотидных праймеров для V-сегментов, каждый из которых независимо специфически гибридизируется с по меньшей мере одним полинуклеотидом, кодирующим полипептид V-участка рецептора адаптивной иммунной системы, или с его комплементом, причем каждый праймер для V-сегментов содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному функциональному сегменту гена, кодирующего V-участок рецептора адаптивной иммунной системы, и причем множество праймеров для V-сегментов специфически гибридизируется с по существу всеми функциональными сегментами гена, кодирующего V-участок рецептора адаптивной иммунной системы, которые присутствуют в композиции, и
(b) в по существу эквимолярных количествах множество олигонуклеотидных праймеров для J-сегментов, каждый из которых независимо специфически гибридизируется с по меньшей мере одним полинуклеотидом, кодирующим полипептид J-участка рецептора адаптивной иммунной системы, или с его комплементом, причем каждый праймер для J-сегментов содержит нуклеотидную последовательность из по меньшей мере 15 последовательных нуклеотидов, которая комплементарна по меньшей мере одному функциональному сегменту гена, кодирующего J-участок рецептора адаптивной иммунной системы, и причем множество праймеров для J-сегментов специфически гибридизируется с по существу всеми функциональными сегментами гена, кодирующего J-участок рецептора адаптивной иммунной системы, которые присутствуют в композиции,
причем олигонуклеотидные праймеры для V-сегментов и J-сегментов проводят в указанной одиночной мультиплексной полимеразной цепной реакции (ПЦР) амплификацию (i) по существу всех синтетических матричных олигонуклеотидов в композиции для получения множества амплифицированных синтетических матричных олигонуклеотидов, причем указанное множество амплифицированных синтетических матричных молекул нуклеиновых кислот достаточно для количественного определения разнообразия синтетических матричных олигонуклеотидов в композиции, и (ii) по существу всех перестроенных молекул нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы в биологическом образце, для получения множества амплифицированных перестроенных молекул ДНК для количественного определения разнообразия перестроенных молекул нуклеиновых кислот в ДНК из биологического образца;
и причем каждая амплифицированная молекула нуклеиновой кислоты во множестве амплифицированных синтетических матричных олигонуклеотидов и множестве амплифицированных перестроенных молекул нуклеиновых кислот имеет длину менее 1000 нуклеотидов;
(B) количественное секвенирование указанных амплифицированных синтетических матричных олигонуклеотидов и указанных амплифицированных перестроенных молекул нуклеиновых кислот для количественного определения (i) количества матричного продукта амплифицированных синтетических матричных олигонуклеотидов, которые содержат по меньшей мере одну олигонуклеотидную последовательность штрихкода, и (ii) количества перестроенного продукта амплифицированных перестроенных молекул нуклеиновых кислот, в которых отсутствует олигонуклеотидная последовательность штрихкода;
(C) вычисление коэффициента амплификации путем деления количества матричного продукта из (B)(i) на известное количество каждого из множества синтетических матричных олигонуклеотидов, имеющих уникальную олигонуклеотидную последовательность из (A)(2); и
(D) деление количества перестроенного продукта из (B)(ii) на коэффициент амплификации, вычисленный в (С), для количественного определения содержания уникальных перестроенных молекул нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы, в образце.
27. Способ по п. 26, в котором указанное количественно определенное содержание уникальных перестроенных молекул нуклеиновых кислот, кодирующих рецепторы адаптивной иммунной системы, в образце представляет собой количество уникальных геномных матриц В-клеток или уникальных геномных матриц Т-клеток в образце.
| WO 2010151416 A1, 29.12.2010 | |||
| УСТРОЙСТВО ДЛЯ ИЗМЕРЕНИЯ ПЛОТНОСТИ ЖИДКОСТИ | 2010 |
|
RU2418287C1 |
| Барабанная коллекторная обмотка для компенсированного асинхронного двигателя | 1926 |
|
SU7958A1 |
Авторы
Даты
2017-09-26—Публикация
2013-05-08—Подача