Настоящее изобретение относится к способам прогнозирования резистентности к инфекционному некрозу поджелудочной железы лососевых рыб, более конкретно, изобретение относится к прогнозированию такой резистентности путем анализа ДНК-полиморфизма.
Инфекционный некроз поджелудочной железы (IPN) является одной из наиболее серьезных угроз при разведении лососевых рыб во всем мире. Причиной заболевания является водный бирнавирус, вызывающий некроз клеток поджелудочной железы и клеток печени, что приводит к летаргии и внезапной смерти. Вирус широко распространен в природе, но, по-видимому, как-либо значительно не влияет на диких лососевых. В условиях аквакультуры болезнь вызывает смертность как на стадии мальков, когда рыба по-прежнему живет в пресной воде, так и на стадии постсмолта, вскоре после перевода в морскую воду. В отрасли потери из-за IPN оценочно составляют 8% в течение фазы пресной воды и 5% в течение морской фазы.
Лососевая промышленность в общем делится на несколько направлений, соответствующих различным этапам жизни рыб: производители яиц продают оплодотворенные яйца производителям смолта, которые предоставляют готовую для соленой воды рыбу (смолт) выращивающим производителям. Для каждого направления целесообразно отбирать яйца или рыбу, имеющие резистентность к заболеваниям выше среднего. Разводящие лососевых компании ведут непрерывные программы селекции рыбы, направленные на улучшение аквакультурных запасов в отношении резистентности к заболеваниям, и были разработаны протоколы для тестирования резистентности рыбы к нескольким конкретным заболеваниям. Эти тесты на заболеваемость использовались для отбора рыбы в качестве производителей, которые обладают резистентностью к интересующим заболеваниям выше среднего. Стандартные тесты включают тестирование с помощью контролируемого заражения сиблингов кандидатов в производители. Эта методика, однако, затруднена тем, что инфицированная рыба не может использоваться в качестве производителей. Поэтому приходится прибегать к случайному отбору (нетестированных) животных из семей тестируемых рыб, которые показывали лучшие результаты в тестах на заболеваемость (так называемый семейный отбор).
Поэтому существует потребность в альтернативных методологиях для анализа резистентности животных к инфекционному некрозу поджелудочной железы; в частности, методологиях, которые позволяют прямой анализ резистентности индивидуума к инфекционному некрозу поджелудочной железы, при этом сохраняя возможность использования проверенного животного в качестве производителя.
Автором настоящего изобретения после обширных экспериментов было установлено, что можно прогнозировать резистентность к инфекционному некрозу поджелудочной железы у лососевых рыб путем анализа одного или нескольких ДНК-полиморфизмов (тем самым, удовлетворяя вышеуказанную потребность).
Соответственно, в первом аспекте настоящего изобретения предложен способ прогнозирования резистентности к инфекционному некрозу поджелудочной железы у лосося, при этом способ включает определение аллелей, присутствующих в ДНК-полиморфизме у лосося, и прогнозирование, является или нет лосось резистентным к инфекционному некрозу поджелудочной железы, на основе определения аллелей.
Автором изобретения было обнаружено, что ДНК-полиморфизмы по настоящему изобретению могут быть представлены в любой из двух форм, т.е. полиморфизмы имеют два аллеля. Один аллель может быть охарактеризован как прогнозирующий резистентность к инфекционному некрозу поджелудочной железы (т.е. аллель резистентности), а другой как являющийся предиктором восприимчивости к инфекционному некрозу поджелудочной железы (т.е. аллель восприимчивости). Лососевые рыбы являются диплоидными организмами и поэтому имеют по две копии полиморфизмов по настоящему изобретению (по одной копии в каждом наборе хромосом). Стадия определения аллелей в способе по первому аспекту настоящего изобретения поэтому включает в себя стадию анализа ДНК-полиморфизма, представленного в каждом наборе хромосом, для того чтобы определить, является ли каждая присутствующая копия ДНК-полиморфизма аллелем резистентности или аллелем восприимчивости. Когда с помощью способа по настоящему изобретению определяют, что лосось имеет две копии аллеля резистентности для ДНК-полиморфизма (т.е. лосось является гомозиготным по аллелю резистентности), прогнозируется, что лосось имеет резистентность к инфекционному некрозу поджелудочной железы. И наоборот, когда с помощью способа по настоящему изобретению определяют, что лосось имеет две копии аллеля восприимчивости для ДНК-полиморфизма (т.е. является гомозиготным по аллелю восприимчивости), прогнозируется, что лосось не имеет резистентности к инфекционному некрозу поджелудочной железы. Можно сделать вывод, что лосось, для которого с помощью способа по настоящему изобретению предсказана резистентность к инфекционному некрозу поджелудочной железы, имеет выше нормы вероятность наличия резистентности к инфекционному некрозу поджелудочной железы. Напротив, можно сделать вывод, что лосось, для которого с помощью способа по настоящему изобретению предсказано отсутствие резистентности к инфекционному некрозу поджелудочной железы, имеет ниже нормы риск развития резистентности к инфекционному некрозу поджелудочной железы. Когда с помощью способа по настоящему изобретению определяют, что лосось имеет одну копию аллеля резистентности для ДНК-полиморфизма и одну копию аллеля восприимчивости для ДНК-полиморфизма (т.е. является гетерозиготным), для лосося не будет прогнозироваться в соответствии с настоящим изобретением, что он имеет резистентность к инфекционному некрозу поджелудочной железы. Однако для этого лосося будет прогнозироваться, что он имеет больший шанс быть резистентным к инфекционному некрозу поджелудочной железы, чем лосось с двумя копиями аллеля восприимчивости. В дальнейшем, такой лосось будет упоминаться как имеющий половинную резистентность к инфекционному некрозу поджелудочной железы.
Рассматриваемым ДНК-полиморфизмом может быть любой из нескольких ДНК-полиморфизмов, которые, как было найдено автором изобретения, имеют это прогностическое свойство. Все эти ДНК-полиморфизмы расположены на 26-й хромосоме. ДНК-полиморфизмы связана своим общим признаком прогнозирования резистентности к IPN. Способность ДНК-полиморфизмов прогнозировать резистентность к IPN может быть определена количественно с помощью r2-статистики, которая будет описана ниже. Все ДНК-полиморфизмы имеют общую характеристику, что r2-статистика больше 0,3. ДНК-полиморфизм может быть мультинуклеотидным полиморфизмом (т.е. не-SNP-полиморфизмом), однонуклеотидным полиморфизмом, инсерционной мутацией или делеционной мутацией. Каждый тип ДНК-полиморфизма, приведенного выше, рассматривается индивидуально как часть настоящего изобретения для стадии определения в способах по настоящему изобретению.
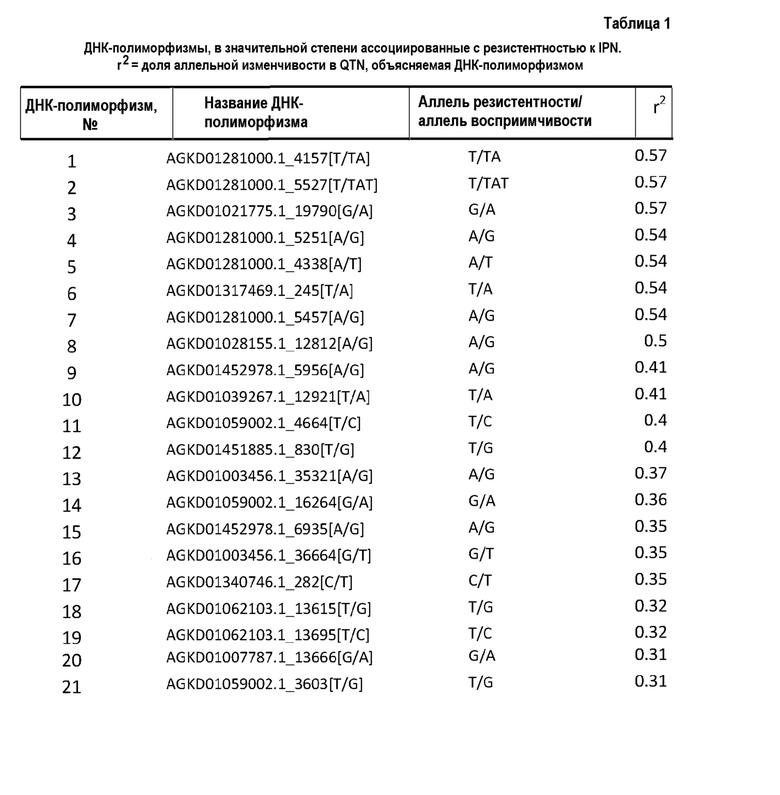
ДНК-полиморфизм может быть выбран из любого из ДНК-полиморфизмов, представленных в таблице 1. Каждый из ДНК-полиморфизмов, представленных в таблице 1, рассматривается индивидуально как часть настоящего изобретения.
ДНК-полиморфизмы, описанные в данной заявке, указаны относительно полной геномной последовательности Salmo salar, опубликованной в Genebank под номером доступа AGKD00000000 (версия AGKD00000000.1 GI: 354459050). Более конкретно, каждый ДНК-полиморфизм в настоящей заявке получил свое название, описанное в данном документе, из следующего: номера Genbank, следующего за ним подчеркивания ('_') и следующего за ним положения ДНК-полиморфизма в последовательности GenBank, со следующими за ним квадратными скобками, включающими эталонный аллель (указывается первым) и альтернативный аллель (указывается вторым). Эталонным аллелем является аллель, указанный в эталонной последовательности.
Например, ДНК-полиморфизмом может быть:
AGKD01281000.1_4157[T/TA];
AGKD01281000.1_5527[T/TAT];
AGKD01021775.1_19790[G/A];
AGKD01281000.1_5251[A/G] или;
AGKD01281000.1_4338[A/T].
Каждый из вышеуказанных ДНК-полиморфизмов рассматривается индивидуально как часть настоящего изобретения.
В способе могут использоваться два ДНК-полиморфизма. Когда способ используется с двумя ДНК-полиморфизмами, два ДНК-полиморфизма составляют одно целое, далее именуемое гаплотипом. Каждый гаплотип может иметь четыре различных аллеля, что соответствует четырем различным комбинациям аллелей ДНК-полиморфизма в индивидуальных ДНК-полиморфизмах (например, если гаплотип состоит из одного ДНК-полиморфизма с аллелями А и Т и одного ДНК-полиморфизма с аллелями Т и G, четырьмя возможными гаплотипными аллелями являются A-T, A-G, T-T и T-G). Каждый из этих четырех аллелей будет являться либо аллелем резистентности, либо аллелем восприимчивости, аналогично способу с единственным ДНК-полиморфизмом, изложенному выше. Таким образом, в гипотетическом случае гаплотипа, имеющего четыре аллеля, A-T, A-G, T-T и T-G, может быть, что все A-T, A-G и T-T являются аллелями резистентности, тогда как T-G является аллелем восприимчивости. В этом случае, животное, имеющее одну копию A-T аллеля и одну копию A-G аллеля, будет резистентно к IPN, животное, имеющее одну копию A-T аллеля и одну копию T-G аллеля будет полурезистентным, тогда как животное, имеющее две копии T-G будет восприимчивым.
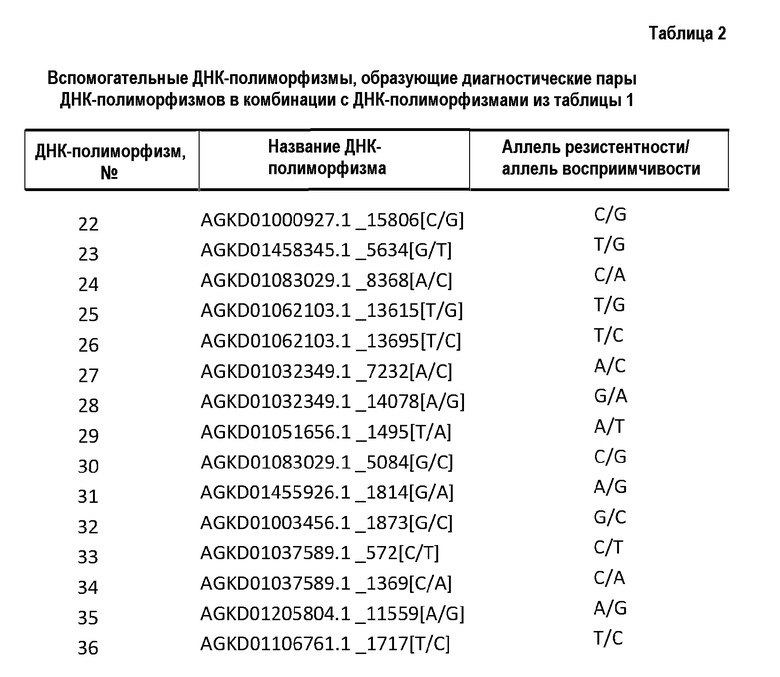
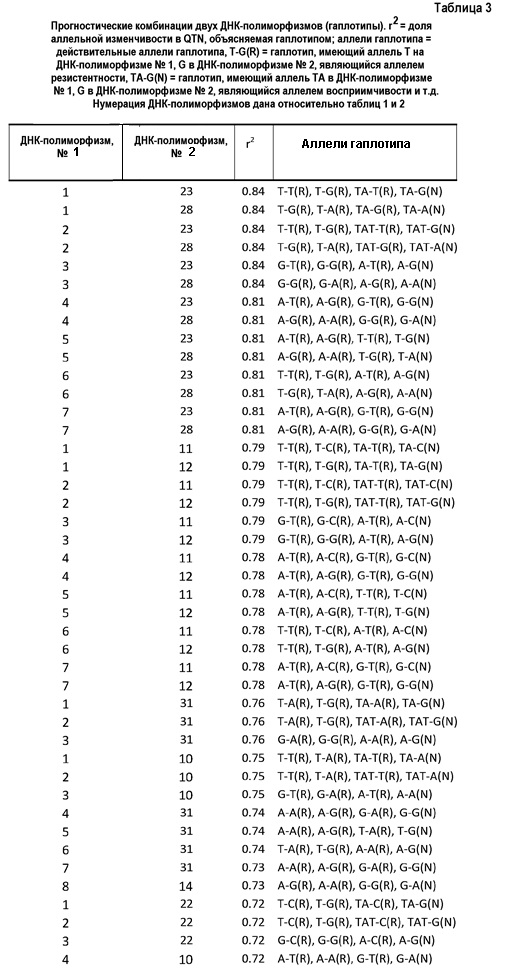
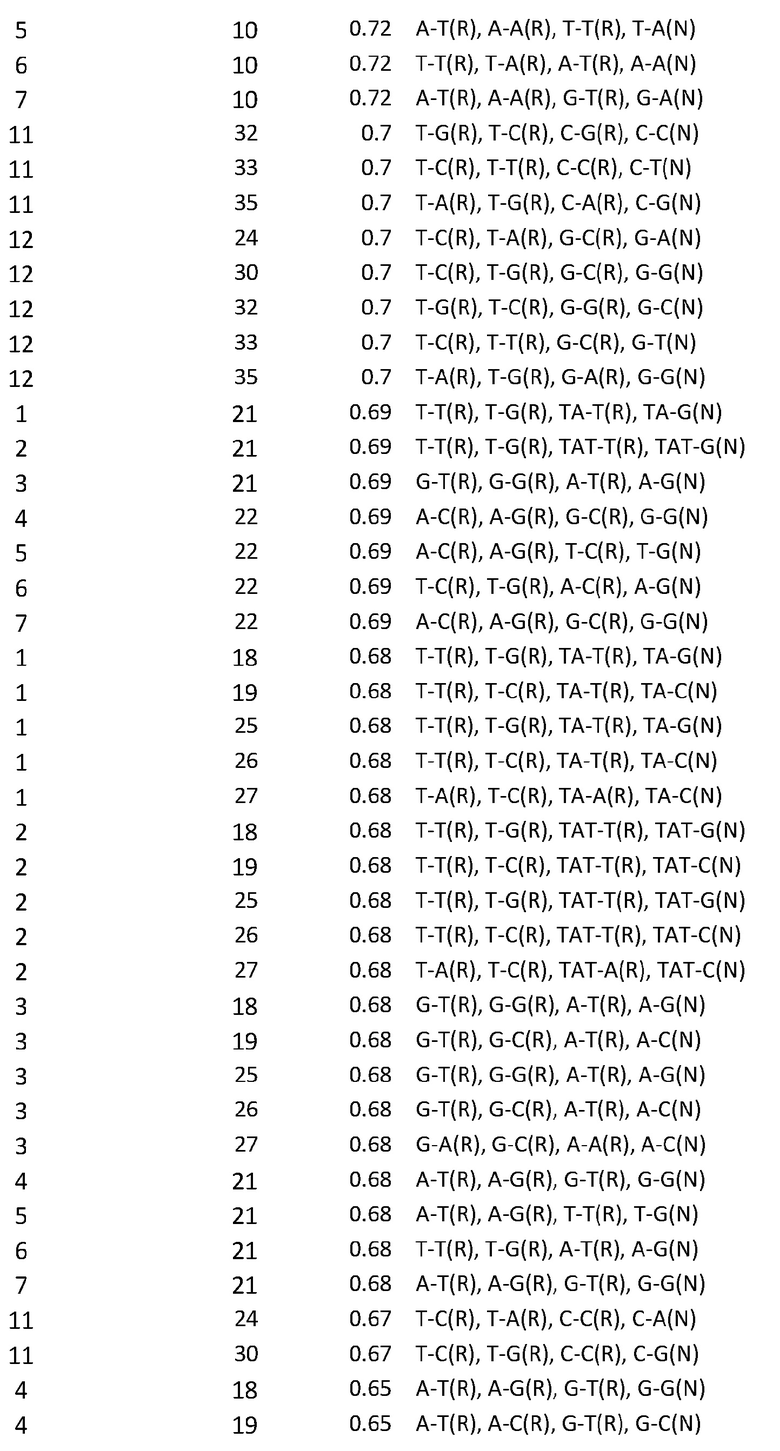
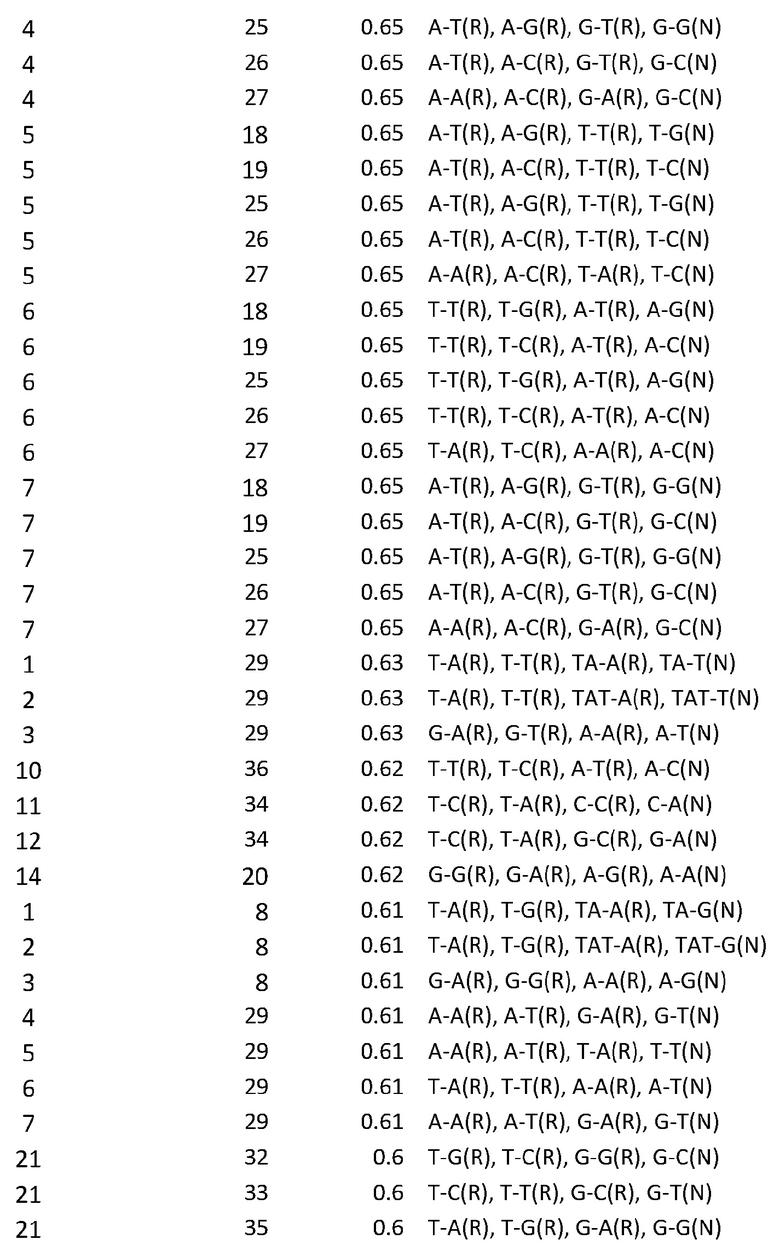
Автором изобретения было обнаружено большое количество таких гаплотипов, то есть комбинаций двух ДНК-полиморфизмов, которые являются мощными предикторами резистентности к IPN, более мощными, чем одиночные ДНК-полиморфизмы. Для каждого из этих гаплотипов автором изобретения было определено, какие аллели являются аллелями резистентности, и какие аллели являются аллелями восприимчивости. Парами ДНК-полиморфизмов, которые составляют прогностические гаплотипы, является либо любая комбинация ДНК-полиморфизмов из приведенных в таблице 1, либо они представляют собой любые комбинации одного ДНК-полиморфизма из таблицы 1 с одним ДНК-полиморфизмом из таблицы 2. Все прогностические гаплотипы приведены в таблице 3, где ДНК-полиморфизмы обозначены номерами относительно таблицы 1 и таблицы 2. Каждая из пар ДНК-полиморфизмов рассматривается для применения индивидуально как часть настоящего изобретения. Все пары ДНК-полиморфизмов имеют общую характеристику, что их r2-значение (которое будет описано ниже), больше 0,6.
Следовательно, настоящее изобретение может, поэтому, относиться к способу, который дополнительно включает стадию определения аллеля, присутствующего в дополнительном ДНК-полиморфизме, и прогнозированию, является или нет лосось резистентным к инфекционному некрозу поджелудочной железы, основанному на определении аллелей в обоих ДНК-полиморфизмах.
Например, способ по настоящему изобретению может включать в себя определение аллелей, присутствующих в ДНК-полиморфизме AGKD01458345.1_5634[G/T] и в дополнительном ДНК-полиморфизме AGKD01021775.1_19790[G/A], и прогнозирование, является или нет лосось резистентным к инфекционному некрозу поджелудочной железы, основанное на определении аллелей в обоих полиморфизмах ДНК.
Когда гаплотипы из двух ДНК-полиморфизмов, а не индивидуальные ДНК-полиморфизмы, используются для прогнозирования резистентности, аллели гаплотипа во-первых должны быть определены в тестируемой рыбе, другими словами, должно быть определено, какие аллели в индивидуальном ДНК-полиморфизме расположены на одинаковых хромосомах. Это может быть сделано с помощью компьютерных программ, таких как PHASE (http://stephenslab.uchicago.edu/software.html#phase), хотя для большинства животных аллели гаплотипа будут очевидны (например, если животное имеет две копии А-аллеля в одном ДНК-полиморфизме, и одну копию Т и одну копию G в другом ДНК-полиморфизме, только две конфигурации аллелей в гаплотипе возможны, а именно, A-G+A-T).
Когда используется гаплотип из двух ДНК-полиморфизмов, а не одиночный ДНК-полиморфизм, тест становится более предиктивным по сравнению с тем, когда используется только один ДНК-полиморфизм.
Способ может включать в себя анализ более чем двух полиморфизмов ДНК. Например, способ по настоящему изобретению может включать в себя определение более двух полиморфизмов, где по меньшей мере один из полиморфизмов приведен в таблице 1 и/или по меньшей мере два полиморфизма приведены в виде пары в таблице 3.
Способ может быть применен к атлантическому лососю (т.е. Salmo salar).
Стадия определения присутствия или отсутствия у лосося может быть осуществлена на образце, взятом от лосося. Образцом может быть любой образец, на котором возможен анализ нуклеотидного материала, что будет легко понятно специалисту в данной области техники. Во избежание сомнений, образцом может быть образец мышечной ткани, образец крови, образец печени и/или отрезанная часть плавника.
Специалист в данной области будет хорошо осведомлен обо всех доступных способах, обеспечивающих возможность тестирования на присутствие или отсутствие ДНК-полиморфизма. Например, способ может включать анализ последовательности подлежащего тестированию лосося. В альтернативном варианте, способ может включать в себя однонуклеотидное удлинение фрагментов ДНК, оканчивающихся на полиморфном сайте (например, IPLEX-анализы от Sequenom и Infinium-анализы от Illumina), аллель-специфический ПЦР (например, анализы SNPtype от Fluidigm или KASPar от KBiosciences) или конкурентную гибридизацию зондов, комплементарных различным аллелям (например, TaqMan-анализ от Applied Biosystems).
Следовательно, в дополнительном аспекте настоящего изобретения предложен гибридизационный зонд, который является специфичным для одного или нескольких вышеуказанных ДНК-полиморфизмов.
ДНК в месте ДНК-полиморфизмов и в их окружении может быть экстраполирована из названий, присвоенных ДНК-полиморфизмам в таблице 1 и таблице 2. Кроме того, последовательности ДНК в месте ДНК-полиморфизмов и в их окружении можно найти в таблице 4. Гибридизационные зонды, которые являются избирательными в отношении этих последовательностей ДНК, могут образовывать часть настоящего изобретения.
Лосось, который согласно прогнозу имеет резистентность к инфекционному некрозу поджелудочной железы в соответствии с первым аспектом настоящего изобретения, более вероятно, чем в норме продуцирует потомство, которое имеет более высокий, чем в нормальный шанс обладать резистентностью к инфекционному некрозу поджелудочной железы. Следовательно, в дополнительном аспекте настоящего изобретения предложен способ отбора лосося для использования в качестве производителей, в котором лосось отбирают на основе прогнозирования с помощью способа, заявленного в первом аспекте настоящего изобретения, чтобы получить резистентность к инфекционному некрозу поджелудочной железы.
С другой стороны, лосось, который по прогнозу с помощью способа по первому аспекту настоящего изобретения не обладает резистентностью к инфекционному некрозу поджелудочной железы, не будет отобран в качестве производителя.
Настоящее изобретение также относится к выделенному полинуклеотиду, содержащему один или несколько из индивидуальных ДНК-полиморфизмов, выбранных из группы, приведенной в таблице 1, расположенному внутри участка генома лососевых рыб. Примеры последовательностей для таких выделенных полинуклеотидов можно найти в таблице 4.
Термины «аллель гаплотипа» и «аллель ДНК-полиморфизма» принимают свое обычное значение, которое будет хорошо понятно специалисту в данной области техники. Тем не менее, во избежание сомнений, «аллель ДНК-полиморфизма» может означать одну из двух различных нуклеотидных последовательностей в месте ДНК-полиморфизма по настоящему изобретению (при этом один аллель является «аллелем резистентности», а другой - «аллелем восприимчивости»). Тем не менее, во избежание сомнений, «аллель гаплотипа» может означать одну из четырех возможных пар аллелей ДНК-полиморфизма по настоящему изобретению.
или:
… «аллель гаплотипа» может означать любую возможную уникальную комбинацию аллелей для этого гаплотипа, т.е. любую уникальную комбинацию одного аллеля из каждого из ДНК-полиморфизмов, образующих гаплотип (в контексте гаплотипов, состоящих из двух би-аллельных ДНК-полиморфизмов, возможны четыре такие комбинации).
Настоящее изобретение далее будет описано на примерах со ссылкой на прилагаемые чертежи, в которых:
На фигуре 1 показан график, иллюстрирующий выживаемость лосося при инфекционном некрозе поджелудочной железы, - тест на заболеваемость.
На фигуре 2 показан график, иллюстрирующий совокупную смертность при заражении в садке лосося стандартным вирусным изолятом C-1244.
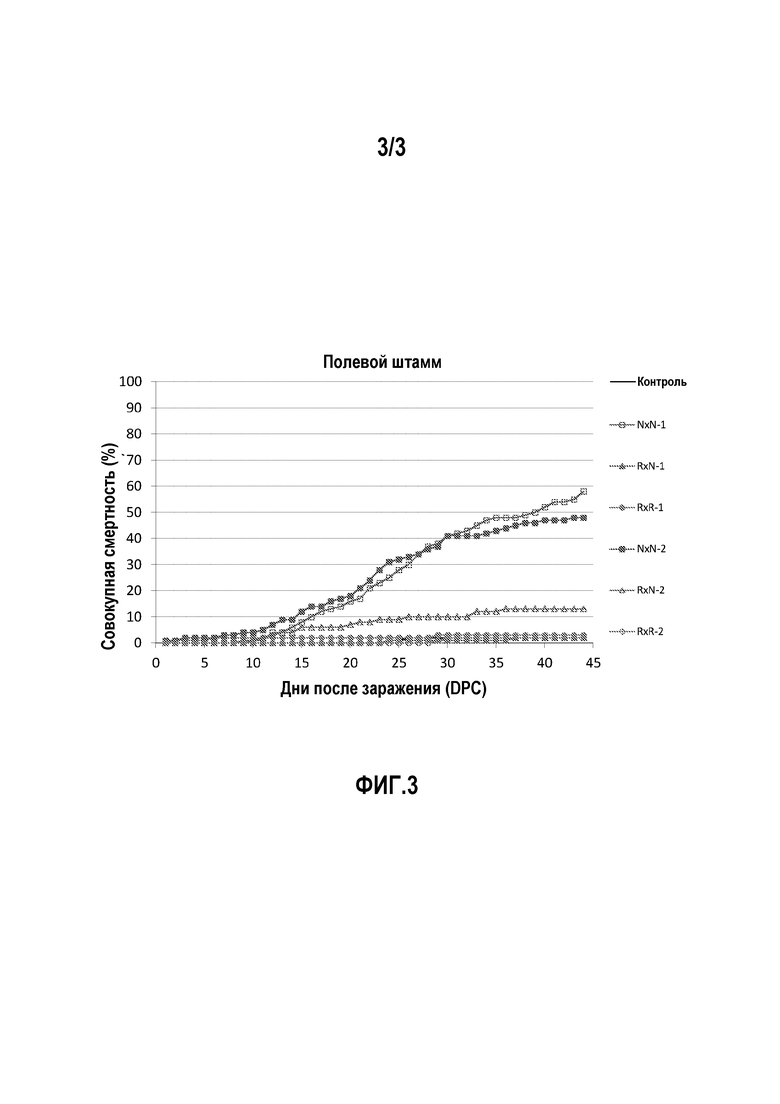
На фигуре 3 показан график, иллюстрирующий совокупную смертность при заражении в садке лосося Норвежским полевым штаммом вируса IPN.
1. Отбор тестируемых животных
Сорок пять экземпляров атлантического лосося из центрального питомника Aqua Gen в Норвегии (отобранных из родителей классов 2005 и 2008 года) были выбраны для массивного параллельного секвенирования (Illumina Hi Seq 2000). Весь лосось в центральном питомнике был получен от лосося, взятого из норвежских рек.
У атлантического лосося локусы количественных признаков (QTL) были связаны с резистентностью к IPN (Moen et al. 2009). Недавно было опубликовано, что три отдельных ДНК-полиморфизма связаны с QTL (Houston et al. 2012), но теста для определения того, являются или нет отдельные животные резистентными или восприимчивыми, представлено не было. QTL расположен на хромосоме 26.
Авторы изобретения предполагают, что вышеупомянутый QTL резистентности к IPN обусловлен первопричиной, но неизвестной мутацией в гене или другом функциональном элементе ДНК. Эта неизвестная мутация в дальнейшем будет называться нуклеотидом количественных признаков (QTN). Кроме того, предполагается, что QTN имеет два аллеля; один аллель, который дает повышенную резистентность (аллель резистентности, Q), и один аллель, который дает пониженную резистентность (аллель восприимчивости, q).
Четыреста пятьдесят четыре полносиблинговые группы мальков атлантического лосося были заражены в отдельных садках вскоре после начала кормления (протоколы для стандартного теста на выживаемость можно найти в Moen et al. 2009. Каждая полносиблинговая группа состояла из 103 рыб (в среднем), и образцы тканей собирали у 10 первых умерших в группе, а также у 10 выживших (или у 10 последних умерших), после чего ДНК выделяли с помощью набора DNAeasy от QIAGEN (Qiagen, Венло, Нидерланды). У 206 отобранных полносиблинноговых групп пораженное и выжившее потомство генотипировали по трем микросателлитным маркерам, расположенным в районе QTL резистентности к IPN; Alu333, Ssa0384BSFU/ii и Ssa0285BSFU, после чего определяли фазу сцепления между аллелями трех микросателлитов у каждого картируемого родителя с использованием наблюдаемой косегрегации аллелей от родителей к потомству (генотипирование микросателлитных маркеров обсуждается более подробно в Moen et al. 2009). Это генотипирование было осуществлено в итеративном режиме, так что, в итоге, почти все полносиблинговые группы, которые, вероятно, имели по меньшей мере одного QTN-гетерозиготного родителя (см. ниже), были генотипированы. Критерий хи-квадрат был применен для того, чтобы проверить совместное наследование трех-микросателлитного гаплотипа и восприимчивого/резистентного фенотипа, выявив тем самым, 110 QTN-гетерозиготных родителей. С использованием данных об этих QTN-гетерозиготных родителях была создана таблица, связывающая аллели в трех-микросателлитном гаплотипе с QTN-аллелями. (Если было обнаружено, что трех-микросателлитный аллель связан как с Q, так и с q, только наиболее распространенную фазу сцепления вносили в таблицу). Эту таблицу впоследствии использовали для экстраполяции генотипов в QTN для картирования родителей, являющихся QTN-гомозиготными, а также для других животных из центрального питомника Aqua Gen. Для последующего полногеномного секвенирования были отобраны выявленные таким образом двадцать два животных из Aqua Gen, которые имеют QTN-генотип QQ (т.е. который, как ожидается, обеспечивает хорошую резистентность к IPN), а также аналогичным образом, найденные 23 животных из Aqua Gen, имеющих qq-генотип (т.е. который, как ожидается, обеспечивает плохую резистентность к IPN). Эти наборы из 22 и 23 животных были объединены таким образом, чтобы свести к минимуму родство между животными в группе, за счет максимального разнообразия трех-микросателлитных аллелей в каждой группе.
2. Создание сборки эталонной последовательности ДНК для QTL-области
QTL-область была определена как область между SNP ESTNV_31602_808 и GCR_cBin30387_Ctg1_91 на карте сцепления SNP атлантического лосося (Lien et al. 2011). Клоны бактериальных искусственных хромосом (BAC), соответствующие этим SNP, были выделены из существующей BAC-библиотеки (Торсен et al. 2004). Исходя из физической карты, созданной для этой библиотеки (www.asalbase.org), было определено минимальное количество перекрывающихся клонов для этого участка, составляющее 31 ВАС-клон (таблица 5). Геномную ДНК атлантического лосося (т.е. вставку) выделяли для каждого BAC. Для каждого образца ДНК ВАС была создана индивидуально меченная библиотека концевых пар (со средним размером вставки 350 п.о.), после чего образцы были секвенированы в мультиплексном режиме на HiSeq2000 (Illumina Inc., Сан-Диего, США) до средней глубины покрытия гаплоидного генома, составляющей приблизительно 800Х. После удаления остаточных адаптерных последовательностей, отбрасывания слишком коротких прочтенных последовательностей, обрезки концов плохо прочтенных последовательностей и согласования с данными секвенирования концевых пар, была выполнена de novo сборка для каждого BAC с использованием программы «clc_novo_assemble» из пакета CLC Assemble Cell (CLC Bio, Орхус, Дания). Затем использовали Phrap версии 1.090518 (http://phrap.org.) для сборки последовательностей индивидуальных ВАС-контигов в набор контигов, охватывающих все ВАС. Наконец, контиги из этой сборки были объединены в один непрерывный геномный скаффолд путем выравнивания его со скаффолдами из предварительной версии последовательности генома атлантического лосося (который был получен в лаборатории автора с помощью программного обеспечения Celera Assembler на основе данных из первых 27 серий последовательностей, поданных в рамках проекта секвенирования в NCBI Trace Archive).
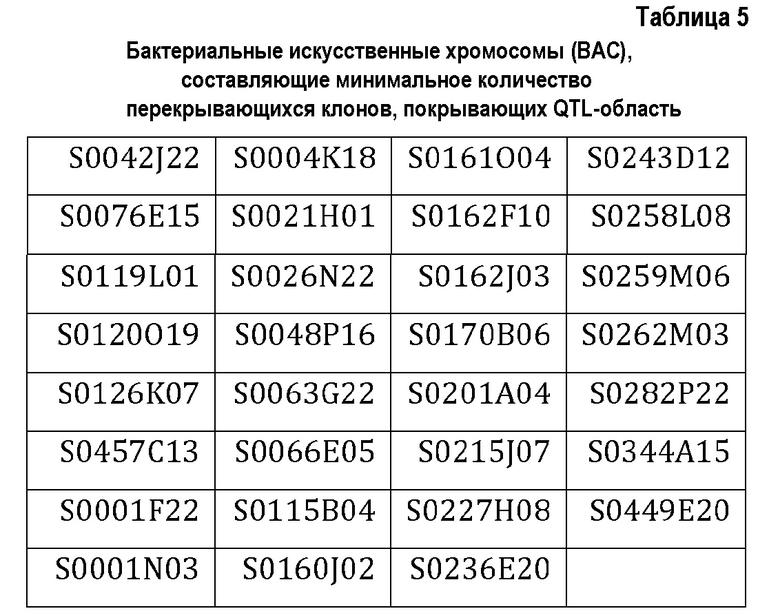
3. Открытие ДНК-полиморфизмов, прогнозирующих IPN
Геномы указанных выше 23 QQ-животных и 22 qq-животных были просеквенированы с использованием технологии HiSeq2000 от Illumina. Прежде чем образцы объединяли для секвенирования, для каждого образца были созданы индивидуально меченые библиотеки концевых пар. В общей сложности было получено 264×109 нуклеотидных последовательностей, что соответствует для каждого животного 2-кратному покрытию гаплоидного генома. Последовательности были собраны на вышеуказанной эталонной последовательности QTL-области с помощью программ «clc_ref_assemble_long» и «clc_ref_assemble» из пакета CLC Assemble Cell, дающих две сборки, соответствующие двум группам QTN-генотипа. В попытке свести к минимуму картирование последовательностей из гомологичных хромосом были предусмотрены совпадающая доля длины последовательности 0,9 и минимальный порог подобия 0,98. Обнаружение SNP проводили на этих отдельных сборках с использованием программы «find_variations» из пакета CLC Assemble Cell, позволяющей как минимум однонуклеотидное отличие от эталонной последовательности. Точный тест Фишера был использован для того, чтобы проверить независимость между QTN-генотипом (т.е. сборкой) и (SNP/InDel)-аллелями (однонуклеотидного/инсерционно-делеционного полиморфизма). SNP с наиболее значимой статистикой из этого точного теста были генотипированы у 110 QTN-гетерозиготных животных, указанных выше, а также у протестированного на выживаемость потомства этих животных, и точный тест Фишера был проведен для того, чтобы проверить независимое наследование SNP-аллелей и QTN-аллелей. Также для каждого SNP рассчитывали коэффициент корреляции (r2) между аллелями в SNP и в QTN, меру степени неравновесного сцепления (LD) между локусами, используя функцию «LD» модуля «genetics» в статистическом программном пакете R. SNP был определен как пригодный для прогнозирования резистентности к IPN, если он имел значение r2 выше 0,3 (это является общим допущением среди генетиков, см., например, Shifman et al., Human Molecular Genetics 2003). В контексте настоящего изобретения, значением r2 является доля аллельной изменчивости QTL, объясняемая прогностическим ДНК-полиморфизмом. Например, если r2=0,5, в два раза больше животных необходимо генотипировать для прогностического ДНК-полиморфизма относительно гипотетического случая, когда прогностическими ДНК-полиморфизмами является сам QTN.
SNP, идентифицированные как наиболее сильно коррелирующие с резистентностью к IPN, представлены в таблице 1.
4. Открытие гаплотипов из двух ДНК-полиморфизмов, прогнозирующих резистентность к IPN
Генотипы ДНК-полиморфизмов у QTN-гетерозиготных родителей и их протестированного на выживаемость потомства, описанные выше, были также использованы для того, чтобы найти комбинации 2 ДНК-полиморфизмов, которые лучше прогнозировали резистентность к IPN, чем наилучшие прогностические индивидуальные ДНК-полиморфизмы. ДНК-полиморфизмы были объединены во всех возможных парных комбинациях, и для каждого гаплотипа, состоящего из двух ДНК-полиморфизмов, были определены аллели гаплотипа для каждого QTN-гетерозиготного родителя, и точный тест Фишера был использован для того, чтобы проверить независимость между аллелем гаплотипа и QTN-аллелями. Коэффициент корреляции (r2) между аллельными состояниями в 2ХSNP-гаплотипе и в QTN рассчитывали, сначала картируя 2ХSNP-гаплотип до двухаллельной системы путем замены названия каждого аллеля на название QTN-аллеля, с которым исследуемый аллель 2ХSNP-гаплотипа был преимущественно связан (таблица 3), с последующим расчетом r2 с использованием функции «LD» модуля «genetics» в статистическом программном пакете R.
Гаплотипами, прогнозирующими резистентность к IPN, которые были определены таким образом, были либо комбинации двух ДНК-полиморфизмов из таблицы 1, либо они являлись комбинациями одного ДНК-полиморфизма из таблицы 1 и одного ДНК-полиморфизма из таблицы 2. Таблица 3 содержит все комбинации найденных ДНК-полиморфизмов, которые имеют значение r2 выше 0,60. Таблица 3 также содержит сами аллели гаплотипов, найденные для соответствующих прогностических гаплотипов, а также классификацию (резистентный относительно восприимчивого) этих аллелей гаплотипов.
Таблица 4 содержит ДНК-последовательности ДНК-полиморфизмов. Эти последовательности также могут быть выведены на основании названий ДНК-полиморфизмов, указанных выше.

5. Доказательство эффекта отбора с помощью SNP
Испытание заражением 1: эксперимент был проведен для того, чтобы проверить эффект от внедрения ДНК-теста на основе SNP-гаплотипа, описанного выше (1-4): С использованием ДНК-теста на основе гаплотипа (с парой маркеров 3+23, таблица 3) из популяции производителей в Aqua Gen были отобраны 4 восприимчивых самца, 6 резистентных самцов, 6 восприимчивых самок и 4 резистентных самки. Всех самцов скрещивали со всеми самками, получая четыре группы R×R, R×N, N×R и N×N; R×R, состоящую из потомков резистентных самцов и резистентных самок, R×N, состоящую из потомков резистентных самцов и восприимчивых самок, N×R, состоящую из потомков восприимчивых самцов и резистентных самок, и N×N, состоящую из потомков восприимчивых самцов и восприимчивых самок. Группы были доставлены в службы для проведения проверочных тестов (VESO Vikan, Намсос, Норвегия) при среднем размере 0,2 г (мальки перед началом откорма), начинали откорм в течение 1 дня после прибытия, акклиматизировали в соответствии со стандартной рабочей процедурой (SOP) S-2023, ухаживали и осуществляли наблюдение ежедневно в соответствии с S-2002 и S-2004. Мертвую рыбу собирали каждый день в соответствии с S-2000 и регистрировали смертность. Параметры окружающей среды регистрировали каждый день. Каждую из групп R×R, R×N, N×R и N×N тестировали в двух садках с последующей моделью заражения в воде (S-1079). Каждый садок содержал 100 мальков из соответствующей группы. Использовали свежий (т.е. не замороженный) инфекционный вирус некроза поджелудочной железы на основе изолята серотипа SP 1, пассаж j.no. V-1244 (норвежский полевой изолят от 2001 года); выращивание и титрование вируса осуществлялось в Норвежской школе ветеринарных наук (Осло, Норвегия). В качестве контроля были включены два дополнительных садка, содержащие псевдозараженную рыбу из всех четырех групп. Результаты представлены на фигуре 1.
Фигура 1: смертность при тестировании на заражение IPN, проведенном на четырех различных группах рыб, полученных способом, описанным в этой заявке. Согласно тесту вся рыба в группе N×N была восприимчивой, вся рыба в группах N×R и R×N была полурезистентной, тогда как вся рыба в группе R×R была резистентной. Рыба в контрольной группе была псевдозараженной, так что смертность в этой группе представляла собой ожидаемую смертность в отсутствие вируса.
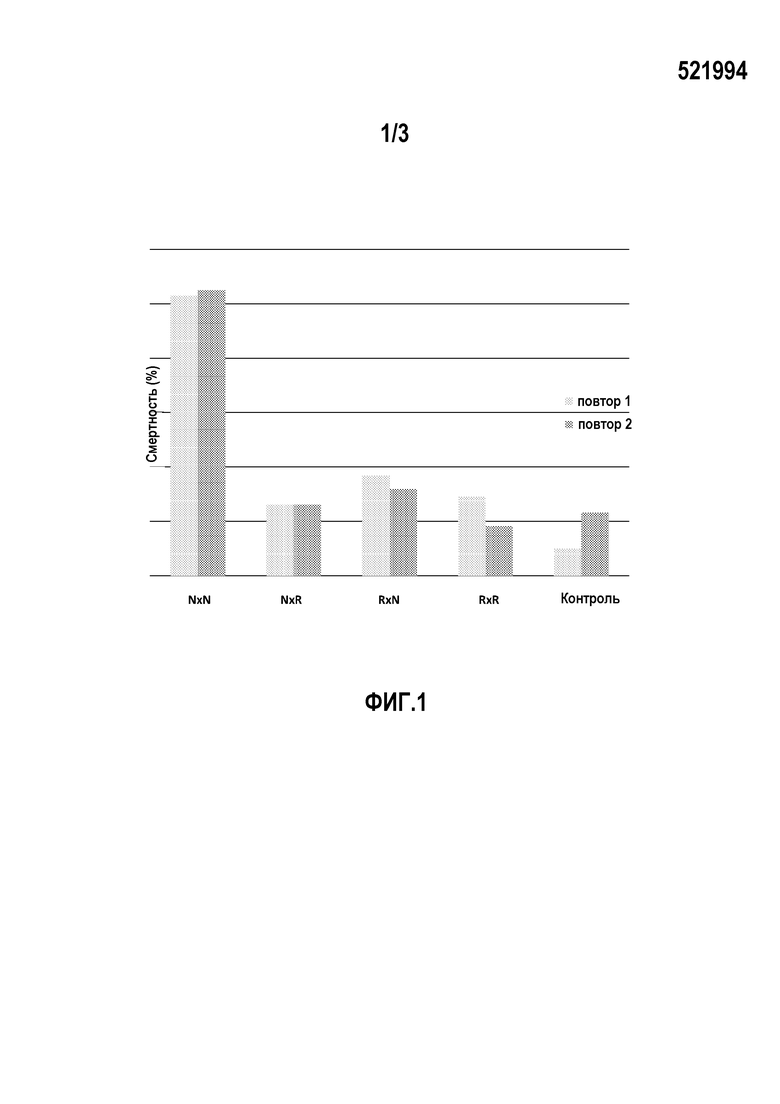
Испытание заражением 2: эксперимент был проведен для того, чтобы сравнить смертность от стандартного вирусного изолята V-1244, выделенного в 2001 году, со смертностью от норвежского полевого штамма, выделенного в 2012 году в инкубаторной службе, страдающей от связанной с IPN смертности. С использованием ДНК-теста на основе гаплотипа (с парой маркеров 3+23, таблица 3) из популяции производителей в Aqua Gen были отобраны 6 восприимчивых самцов, 5 резистентных самцов, 6 восприимчивых самок и 6 резистентных самок. Всех самцов скрещивали со всеми самками, получая три группы R×R, R×N и N×N; R×R, состоящую из потомков резистентных самцов и резистентных самок, R×N, состоящую из потомков резистентных самцов и восприимчивых самок, а также из потомков восприимчивых самцов и резистентных самок, и N×N, состоящую из потомков восприимчивых самцов и восприимчивых самок. Группы были доставлены в службы для проведения проверочных тестов (VESO Vikan, Намсос, Норвегия) при среднем размере 0,2 г (мальки перед началом откорма), начинали откорм в течение 1 дня после прибытия, акклиматизировали в соответствии со стандартной рабочей процедурой (SOP) S-2023, ухаживали и осуществляли наблюдение ежедневно в соответствии с S-2002 и S-2004. Мертвую рыбу собирали каждый день в соответствии с S-2000 и регистрировали смертность. Параметры окружающей среды регистрировали каждый день. Каждую из групп R×R, R×N и N×N тестировали в двух параллельных садках для каждого штамма вируса (V-1244 и полевой штамм) с последующей моделью заражения в воде (S-1079). Каждый садок содержал 100 мальков из соответствующей группы. Штамм V-1244, выделенный в 2001 году, был получен в Норвежской школе ветеринарных наук (Осло, Норвегия), тогда как полевой штамм размножали и титровали в компании Vaxxinova Norway. Оба вирусных изолята хранили охлажденными до заражения. В качестве отрицательного контроля включали один садок, содержащий псевдозараженную рыбу всех трех генотипов. Испытания прекращали через 45 дней после заражения, и результаты приведены на фигурах 2 и 3. Результаты показывают, что рыба R×R (как было определено с помощью способов по настоящему изобретению) является полностью резистентной к обоим штаммам IPNV.
Фигура 2.
Совокупная смертность после заражения в воде мальков атлантического лосося с различными IPN-QTL-генотипами хорошо известным эталонным изолятом IPNV, V-1244 (фигура 2) или полевым штаммом, выделенным в инкубаторной службе в 2012 году (фигура 3).
6. Сравнение известных SNP с SNP по настоящему изобретению
Houston et al. (2012) обнаружили однонуклеотидные полиморфизмы (SNP), которые якобы были ассоциированы с резистентностью к IPN. В этой статье они выявили два SNP (названные Ssa0139ECIG и RAD_HT01), которые согласно публикации имели очень сильную ассоциацию с резистентностью к IPN. SNP Ssa0139ECIG впервые был опубликован в статье Moen et al., но в этом исследовании не было опубликовано никакой ассоциации с резистентностью к IPN. RAD_HT01 впервые был опубликован группой Houston et al. (2012).
SNP RAD_HT01 был независимо идентифицирован автором заявки как часть основанного на секвенировании скрининга на ДНК-полиморфизмы, ассоциированные с резистентностью к IPN, обсуждаемой выше. Однако было найдено, что ожидаемая ассоциация с резистентностью к IPN являлась слишком слабой, чтобы служить основанием для дополнительного тестирования с помощью генотипирования; p-значение (уровень достоверности) для SNP составляло 0,0199, тогда как все SNP, отобранные автором заявки для тестирования с помощью генотипирования, имели р-значения ниже 0,005.
SNP Ssa0139ECIG не был независимо обнаружен автором заявки, поскольку данный SNP не входил в эталонную последовательность ДНК, используемую в проводимом автором заявки поиске SNP, ассоциированных с IPN. Вместо этого, ассоциация между этим SNP и резистентностью к IPN была протестирована автором заявки путем генотипирования родителей зараженной IPN рыбы с последующим статистическим тестированием эффекта SNP-генотипов у этих родителей на уровень смертности у их потомков (таким же образом, как описанный выше для настоящего изобретения). Набор данных состоял из 285 полносиблинговых групп с зарегистрированным уровнем смертности и генотипированных родителей. SNP AGKD01021775.1_19790[G/A], приведенный в таблице 1, был включен в анализ в качестве положительного контроля.
Ассоциация между SNP и резистентностью к IPN была протестирована с использованием линейной модели (один SNP за один раз):
y=1μ+(Zs+Zd)u+pb+e
где y представляет собой вектор уровней смертности для всех полносиблинговых групп, μ является общим средним, u представляет собой вектор случайных аддитивных генетических эффектов родителей, Zs и Zd являются матрицами встречаемости у отцов и матерей, p является вектором копий SNP-аллеля у родителей (0-4) для каждой полносиблинговой группы, b является случайным коэффициентом регрессии, ассоциированным с числом родительских SNP-аллелей, и е представляет собой вектор случайных остатков. Кроме того, u~N(0,A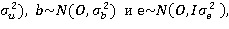 где А представляет собой матрицу отношений числительных для родителей,
где А представляет собой матрицу отношений числительных для родителей, 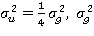 , является общей аддитивной генетической (полигенной) дисперсией,
, является общей аддитивной генетической (полигенной) дисперсией, 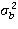 является дисперсией случайного коэффициента регрессии, и
является дисперсией случайного коэффициента регрессии, и 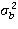 является остаточной дисперсией уровней смертности полносиблинговых групп.
является остаточной дисперсией уровней смертности полносиблинговых групп.
Компоненты дисперсии оценивали для всех случайных эффектов (аддитивного генетического отцовского и материнского, случайной регрессии эффекта SNP и остатка) с использованием методологии REML и программного пакета DMU (Madsen and Jensen 2008). Для проверки значимости SNP, полную модель сравнивали с упрощенной моделью без случайной регрессии числа родительских SNP-аллелей, используя тест отношения правдоподобия.
Было найдено, что SNP Ssa0139ECIG не оказывало достоверного эффекта на смертность от IPN (p-значение = 0,64), тогда как SNP AGKD01021775.1_19790[G/A] был весьма достоверным (p-значение = 2,86-18).
В статье Houston et al. (2012), SNP Ssa0139ECIG и RAD_HT01 представлены, как имеющие сильные (и приблизительно равные) эффекты на резистентность к IPN. Описанные выше результаты указывают на то, что ДНК-полиморфизм, описанный Houston et al. (2012), имеет маленький эффект или вообще не оказывает эффекта на резистентность к IPN у протестированной популяции, тогда как ДНК-полиморфизм, описанный в настоящей заявке, имеет сильные и весьма достоверные эффекты.
Ссылки
Houston R.D., Haley C.S., Hamilton A., Guy D.R., Tinch A.E., Taggart J.B., McAndrew B.J., Bishop S.C. (2008) Major quantitative trait loci affect resistance to infectious pancreatic necrosis in Atlantic salmon (Salmo salar). Genetics 178: 1109-15.
Houston R.D., Davey J.W., Bishop S.C., Lowe N.R., Mota-Velasco J.C. et al. (2012) Characterisation of QTL-linked and genome-wide restriction site-associated DNA (RAD) markers in farmed Atlantic salmon. BMC Genomics 13: 244.
Lien S., Gidskehaug L., Moen T., Hayes B.J., Berg P.R., Davidson W.S., Omholt S.W., Kent M.P. (2011) A dense SNP-based linkage map for Atlantic salmon (Salmo salar) reveals extended chromosome homeologies and striking differences in sex-specific recombination patterns. BMC Genomics 12: 615.
Madsen and Jensen (2008) DMU: a user's guide. A package for analysing multivariate mixed models, version 6, release 5.0. University of Aarhus, Tjele, Denmark.
Moen T., Hayes B., Baranski M., Berg P.R., Kjøglum S., Koop B.F., Davidson W.S., Omholt S.W., Lien S. (2008) A linkage map of the Atlantic salmon (Salmo salar) based on EST-derived SNP markers. BMC Genomics 9: 223.
Moen T., Baranski M., Sonesson A.K., Kjøglum S. (2009) Confirmation and fine-mapping of a major QTL for resistance to infectious pancreatic necrosis in Atlantic salmon (Salmo salar): population-level associations between markers and trait. BMC Genomics 10: 368.
Shifman S., Kuypers J., Kokoris M., Yakir B., Darvasi A. (2003) Linkage diseuilibrium patterns of the human genome across populations. Human Molecular Genetics 12: 771-776.
Thorsen J., Zhu B., Frengen E., Osoegawa K., de Jong, P.J., Koop B.F., Davidson W.S., Hoyheim B. (2005) A highly redundant BAC library of Atlantic salmon (Salmo salar): an important tool for salmon projects. BMC Genomics 6: 50.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ПРОГНОЗИРОВАНИЯ РЕЗИСТЕНТНОСТИ | 2015 |
|
RU2754039C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| ГЕНЕТИЧЕСКИЙ ЛОКУС, АССОЦИИРОВАННЫЙ С ГНИЛЬЮ КОРНЯ И СТЕБЛЯ, ОБУСЛОВЛЕННОЙ PHYTOPHTHORA, У СОИ | 2016 |
|
RU2748688C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM4 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2718584C2 |
| СПОСОБ ИДЕНТИФИКАЦИИ СВИНЕЙ, РЕЗИСТЕНТНЫХ К КИШЕЧНЫМ ЗАБОЛЕВАНИЯМ, АССОЦИИРОВАННЫМ С Е.Сoli, СПОСОБ РАЗВЕДЕНИЯ СВИНЕЙ, РЕЗИСТЕНТНЫХ К КИШЕЧНЫМ ЗАБОЛЕВАНИЯМ, АССОЦИИРОВАННЫМ С E.Coli, ВЫДЕЛЕННАЯ ДНК-МОЛЕКУЛА (ВАРИАНТЫ), МОЛЕКУЛЯРНЫЙ АНАЛИЗ ДЛЯ ОБНАРУЖЕНИЯ РЕЦЕПТОРОВ Е.Сoli F-18 У СВИНЕЙ И НАБОР ДЛЯ ОБНАРУЖЕНИЯ РЕЦЕПТОРОВ Е.Сoli F-18 | 1998 |
|
RU2204609C2 |
| РАСТЕНИЯ САЛАТ-ЛАТУК, ИМЕЮЩИЕ РЕЗИСТЕНТНОСТЬ ПРОТИВ NASONOVIA RIBISNIGRI БИОТИПА 1 | 2015 |
|
RU2731639C2 |
| УЛУЧШЕННЫЙ СПОСОБ И НАБОР ДЛЯ ОПРЕДЕЛЕНИЯ ТЯЖЕСТИ И ПРОГРЕССИРОВАНИЯ ПЕРИОДОНТАЛЬНОГО ЗАБОЛЕВАНИЯ | 2011 |
|
RU2664431C2 |
| РАСТЕНИЯ ОГУРЦА, УСТОЙЧИВЫЕ К ЗАБОЛЕВАНИЯМ | 2006 |
|
RU2418405C2 |
| ОБНАРУЖЕНИЕ МУТАЦИЙ И ПЛОИДНОСТИ В ХРОМОСОМНЫХ СЕГМЕНТАХ | 2015 |
|
RU2717641C2 |
Группа изобретений относится к биотехнологии, в частности к способу прогнозирования резистентности к инфекционному некрозу поджелудочной железы у лосося и способу отбора лосося для использования в качестве производителя. Определяют аллели, присутствующие в ДНК-полиморфизме у лосося, причем ДНК-полиморфизм выбран из группы, представленной в таблице 1. Лосося-производителя отбирают на основе прогнозирования вышеуказанным способом. Изобретения обеспечивают прямой анализ резистентности лосося к инфекционному некрозу поджелудочной железы, при этом сохраняя возможность использования проверенного животного в качестве производителя. 2 н. и 7 з.п. ф-лы, 3 ил., 5 табл.
1. Способ прогнозирования резистентности к инфекционному некрозу поджелудочной железы у лосося, причем способ включает определение аллелей, присутствующих в ДНК-полиморфизме у лосося, и прогнозирование, является или нет лосось резистентным к инфекционному некрозу поджелудочной железы, на основе определения аллелей, где ДНК-полиморфизм выбран из группы, представленной в таблице 1.
2. Способ по п.1, в котором способность ДНК-полиморфизмов прогнозировать резистентность к инфекционному панкреатическому некрозу может быть количественно оценена как имеющих r2-статистику выше 0,3.
3. Способ по п.1 или 2, в котором ДНК-полиморфизм выбран из любого из нижеследующих: AGKD01281000.1_4157[T/TA], AGKD01281000.1_5527[T/TAT], AGKD01021775.1_19790[G/A], AGKD01281000.1_5251[A/G] и AGKD01281000.1_4338[A/T].
4. Способ по п.1 или 2, в котором определяют аллель, присутствующий в дополнительном ДНК-полиморфизме, и прогноз, является или нет лосось резистентным к инфекционному некрозу поджелудочной железы, основанный на определении аллелей в обоих ДНК-полиморфизмах.
5. Способ по п.4, в котором ДНК-полиморфизм и дополнительный ДНК-полиморфизм выбраны из любой из пар, приведенных в таблице 3.
6. Способ по п.1 или 2, в котором ДНК-полиморфизм(ы) расположен(ы) на хромосоме 26.
7. Способ по п.1 или 2, в котором лососем является атлантический лосось.
8. Способ по п.1 или 2, в котором стадию определения аллелей, присутствующих в ДНК-полиморфизме у лосося, выполняют на образце мышечной ткани, образце крови, образце печени и/или отрезанной части плавника.
9. Способ отбора лосося для использования в качестве производителя, в котором лосося отбирают на основе прогнозирования способом по любому из пп.1-7, что этот лосось будет иметь резистентность к инфекционному некрозу поджелудочной железы.
| GHEYAS A | |||
| A | |||
| et al | |||
| "Segregation of infectious pancreatic necrosis resistance QTL in the early life cycle of Atlantic Salmon (Salmo salar)", Animal Genetics, 2010, v.41, no.5, p.531-536 | |||
| HOUSTON R | |||
| D | |||
| et al | |||
| "Characterisation of QTL-linked and genome-wide restriction site-associated DNA (RAD) markers in farmed Atlantic salmon", BMC Genomics, 15 June 2012, DOI: 10.1186/1471-2164-13-244 | |||
| MOEN T | |||
| et al | |||
| "Confirmation and fine-mapping of a major QTL for resistance to infectious pancreatic necrosis in Atlantic salmon (Salmo salar): population-level associations between markers and trait", BMC GENOMICS, 2009, v.10, no.1, p.368, DOI: 10.1186/1471-2164-10-368 | |||
| MOEN T | |||
| Топка с несколькими решетками для твердого топлива | 1918 |
|
SU8A1 |
| WO 2004024956 A1, 25.03.2004 | |||
| СПОСОБ ПРОГНОЗИРОВАНИЯ РИСКА ВОЗНИКНОВЕНИЯ, КЛИНИЧЕСКОГО ТЕЧЕНИЯ И ИСХОДА ОСТРОГО ИДИОПАТИЧЕСКОГО ПАНКРЕАТИТА | 2006 |
|
RU2310848C1 |
Авторы
Даты
2018-07-11—Публикация
2013-07-08—Подача