Область изобретения
Настоящее изобретение относится, главным образом, к полиморфизмам, и, в частности, к однонуклеотидным полиморфизмам (SNP), ассоциированным с повышенной резистентностью радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN). В частности, настоящее изобретение относится к способам прогнозирования повышенной резистентности радужной форели к инфекционному некрозу поджелудочной железы (IPN) и к способам селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы. Кроме того, настоящее изобретение относится к радужной форели, клеткам радужной форели и к их популяциям, имеющим по меньшей мере один аллель, например, по меньшей мере два аллеля, сообщающих резистентность к IPN ("аллель резистентности к IPN") в их геноме, а также к молекулам нуклеиновых кислот, содержащим нуклеотидные последовательности, ассоциированные с SNP по настоящему изобретению.
Уровень техники, к которому относится изобретение
Инфекционный некроз поджелудочной железы (IPN) представляет собой вирусное заболевание, вызывающее высокую смертность при разведении радужной форели в Норвегии и во всем мире. Заболевание вызывается вирусом IPN (IPNV), классифицируемым как водный биРНК-вирус, вызывающий некроз клеток поджелудочной железы и клеток печени, что приводит к летаргии и внезапной гибели.
Компании по разведению, такие как AquaGen AS, проводят непрерывные программы селекции рыб, нацеленные на улучшение исходных аквакультур в отношении резистентности к заболеванию, и были разработаны протоколы для исследования резистентности рыб к нескольким конкретным заболеваниям. Эти испытания с заражением используют для селекции рыб в качестве производителей, которые обладают резистентностью выше средней к рассматриваемым заболеваниям. Общепринятые испытания вовлекают испытание с контролируемым заражением сибсов кандидатов для разведения. Однако этой методологии препятствует тот факт, что инфицированную рыбу нельзя использовать в качестве производителя (родителей следующего поколения). Таким образом, необходимо прибегать к выбору случайных (неисследованных) животных из семей исследованных рыб, которые продемонстрировали наилучшие результаты в испытании с заражением (так называемая семейная селекция).
Таким образом, существует потребность в усовершенствованных методологиях оценки резистентности радужной форели к инфекционному некрозу поджелудочной железы (IPN), в частности, методологиях, которые позволяют прямой анализ и селекцию особей, резистентных к IPN, при сохранении возможности использования исследованной рыбы в качестве производителя.
Сущность изобретения
В первом аспекте настоящее изобретение относится к способу прогнозирования резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN). В частности, настоящее изобретение относится к способу прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN), причем способ включает:
определение присутствия по меньшей мере одного (например, по меньшей мере двух) аллеля, сообщающего резистентность к IPN ("аллель резистентности к IPN"), в геноме (например, на хромосоме 1 генома) указанной радужной форели.
Согласно определенным вариантам осуществления, настоящее изобретение относится к способу прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN), причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229 посредством 1-5, например, 1-2, нуклеотидных замен.
Радужная форель имеет увеличенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно определенным другим вариантам осуществления, настоящее изобретение относится к способу прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN), причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267, chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX_89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX_89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX_89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
В следующем аспекте настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы. В частности, настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, причем способ включает:
определение присутствия по меньшей мере одного аллеля, сообщающего резистентность к IPN ("аллель резистентности к IPN") в геноме (например, на хромосоме 1 генома) указанной радужной форели; и
отбор указанной радужной форели как имеющей увеличенную резистентность, когда присутствует по меньшей мере один аллель резистентности к IPN.
Согласно конкретным вариантам осуществления, настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно другим конкретным вариантам осуществления, настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267, chr1-7515539, chr1-7108873, chr1-6864558, chr1-7186663, chr1-6730531, chr1-27891953, AX-89953259, chr1-6740481, chr1-6770611, chr1-7412807, chr1-7360179, chr1-7411803, chr1-7431445, chr1-7433199, chr1-7441254, chr1-7441877, chr1-7533570, chr1-6834898, chr1-6730142, chr1_6746052, chr1-6794061, chr1-7399212, chr1-7442637, chr1-7358019, chr1-7709828, chr1-7598090, chr1-7626471, chr1-7598743, chr1-7670293, chr1-7670561, chr1-7647634, chr1-7356089, chr1-8109044, chr1-10439048, chr1-8142346, chr1-8092208, chr1-8138683, chr1-8139206, chr1-8139744, chr1-8140789, chr1-8141687, chr1-8154917, chr1-7454708, chr1-7504847, chr1-7505686, chr1-7505817, chr1-8202031, chr1-8228173, chr1-8309469, chr1-8163977, chr1-27786931, chr1-8194629, chr1-7505259, chr1-8474659, chr1-8282602, chr1-8306806, chr1-8341618, chr1-8343786, chr1-8345836, chr1-8350569, chr1-8402403, AX-89962103, chr1-8279302, chr1-8334901, chr1-7561600, AX-89956272, chr1-7938827, chr1-10810229, chr1-11007071 и chr1-10884171, и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
В следующем аспекте настоящее изобретение относится к радужной форели, такой как изолированная радужная форель, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы. В частности, настоящее изобретение относится к радужной форели или к ее потомству, содержащим в их геноме (например, на хромосоме 1 их генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, настоящее изобретение относится к радужной форели или ее потомству, таким как изолированная радужная форель или ее потомство, которые содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к радужной форели или ее потомству, которые содержат в их геноме по меньшей мере один аллель, сообщающий резистентность к IPN, получаемым способом, включающим стадии:
генотипирования форели, отбора особей, имеющих по меньшей мере один аллель, предпочтительно два аллеля, сообщающих резистентность к IPN ("аллель резистентности к IPN"); и скрещивания особей таким образом, чтобы по меньшей мере одна особь в каждой скрещенной паре имела два аллеля, сообщающих резистентность к IPN.
Согласно определенным вариантам осуществления у радужной форели или ее потомства, получаемых этим способом, по меньшей мере один аллель резистентности к IPN может представлять собой аллель по меньшей мере одного однонуклеотидного полиморфизма (SNP). Кроме того, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1. Кроме того, радужная форель или ее потомство могут содержать в их геноме по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299 и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к популяции радужной форели, такой как изолированная популяция, причем каждая особь в популяции имеет повышенную резистентность к инфекционному некрозу поджелудочной железы. В частности, настоящее изобретение относится к популяции радужной форели, причем каждая особь в популяции содержит в ее геноме по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, настоящее изобретение относится к популяции радужной форели, такой как изолированная популяция радужной форели, причем каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к популяции радужной форели, содержащей в ее геноме по меньшей мере один аллель, сообщающий резистентность к IPN, получаемой посредством способа, включающего стадии:
генотипирования форели, отбора особей, имеющих по меньшей мере один аллель, предпочтительно два аллеля, сообщающих резистентность к IPN ("аллель резистентности к IPN"); и скрещивания особей таким образом, чтобы по меньшей мере одна особь в каждой скрещенной паре имела два аллеля, сообщающих резистентность к IPN.
Согласно определенным вариантам осуществления в популяции радужной форели, получаемой этим способом, по меньшей мере один аллель резистентности к IPN может представлять собой аллель по меньшей мере одного однонуклеотидного полиморфизма (SNP). Кроме того, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1. Кроме того, популяция радужной форели может содержать в ее геноме по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299 и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к клетке радужной форели, такой как выделенная клетка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, настоящее изобретение относится к клетке радужной форели, такой как выделенная клетка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к популяции клеток радужной форели, такой как выделенная популяция клеток радужной форели, причем каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, настоящее изобретение относится популяции клеток радужной форели, такой как выделенная популяция клеток радужной форели, причем каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к икринке радужной форели, такой как выделенная икринка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, настоящее изобретение относится к икринке радужной форели, такой как выделенная икринка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к популяции икринок радужной форели, такой как выделенная популяция икринок радужной форели, причем каждая индивидуальная икринка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, настоящее изобретение относится к популяции икринок радужной форели, такой как выделенная популяция икринок радужной форели, причем каждая индивидуальная икринка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
В следующем аспекте настоящее изобретение относится к молекуле нуклеиновой кислоты, такой как выделенная молекула нуклеиновой кислоты, содержащая по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
Согласно определенным вариантам осуществления, настоящее изобретение относится к молекуле нуклеиновой кислоты, такой как выделенная молекула нуклеиновой кислоты, которая содержит по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79, 80, 230, 231 и 232 b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79, 80, 230, 231 и 232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
В следующем аспекте настоящее изобретение относится к олигонуклеотиду, такому как выделенный олигонуклеотид, содержащий по меньшей мере 10 последовательно расположенных нуклеотидов, таких как по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательность выбранный из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарная указанному олигонуклеотиду.
Краткое описание чертежей
Фиг.1. "Манхэттенский график" по результатам полногеномного исследования ассоциаций (GWAS), в котором проводился поиск SNP, ассоциированных с резистентностью к IPN у радужной форели. SNP, распределенные по геному радужной форели, исследовали в отношении их ассоциации с резистентностью к IPN, и, следовательно, в отношении их способности прогнозировать резистентность к IPN. Каждая точка данных соответствует одному индивидуальному SNP, каждый SNP исследовали индивидуально. Положение SNP (ось x) соответствует их положению на генетической карте для женских особей. Горизонтальная линия указывает на уровень значимости, соответствующий частоте ложноположительных результатов (α) 0,05, когда нуль-гипотеза предполагает, что ни один из SNP не ассоциирован с резистентностью к IPN, и с использованием поправки Бонферрони для внесения поправки на тот факт, что было исследовано (приблизительно) 50000 SNP. Поправка Бонферрони в этом случае является высококонсервативной, поскольку она предполагает, что все тесты (SNP) являются независимыми, но они такими не являются. На оси y нанесены на график SNP в соответствии с обратным значением логарифма с основанием 10 их значений p. Как проиллюстрировано на фигуре, SNP, наиболее ассоциированные с резистентностью к IPN, расположены на хромосоме 1.
Фиг.2. Уровни значимости для SNP, расположенных на хромосоме 1 радужной форели, которые исследовали в отношении их ассоциации с резистентностью к IPN. SNP упорядочены в соответствии с их положением на генетической карте (точнее, на генетической карте не основе рекомбинаций, встречающихся у самок радужной форели). cM=сантиморган, стандартная единица измерения генетического расстояния; -log10(значение p)=отрицательное значение логарифмов с основанием 10 значений p для SNP.
Фиг.3. Уровни значимости для SNP, полученных в исследовании, идентифицирующем дополнительные SNP, ассоциированные с резистентностью к IPN (пример 3). Новые и уже известные SNP на хромосоме 1 исследовали в отношении их ассоциации с резистентностью к IPN. Величины на оси x представляют собой положения в парах оснований для SNP вдоль эталонной последовательности ДНК хромосомы 1 радужной форели, величины на оси y представляют собой отрицательное значение логарифма с основанием 10 значений p.
Подробное описание изобретения
Если в настоящем описании не определено конкретно, все использованные технические и научные термины обладают тем же значением, которое обычно подразумевает специалист в области генетики, биохимии и молекулярной биологии.
Все способы и материалы, сходные или эквивалентные способам и материалам, описанным в настоящем описании, можно использовать при применении на практике или исследовании настоящего изобретения, причем подходящие способы и материалы описаны в настоящем описании. Все публикации и патентные заявки, патенты и другие ссылки, упомянутые в настоящем описании, включены в настоящее описание в качестве ссылок в полном объеме. В случае противоречия, настоящее описание, включая определения, будет иметь приоритет.
Полиморфизмы и аллель(и) резистентности к IPN по изобретению
Авторы настоящего изобретения идентифицировали локус количественных признаков (QTL), ответственный за значительную часть генетического варьирования резистентности к IPN у радужной форели. Более конкретно, авторы настоящего изобретения идентифицировали полиморфизмы, и, в частности, однонуклеотидные полиморфизмы (SNP), в геноме, более конкретно на хромосоме 1, радужной форели, которые ассоциированы с увеличенной резистентностью рыбы к инфекционному некрозу поджелудочной железы (IPN). Конкретные детали, касающиеся однонуклеотидных полиморфизмов по изобретению, представлены в таблице 1 ниже. Соответствующие нуклеотидные последовательности, включающие SNP (в положении 36), показаны в таблице 2.
Полиморфизмы по изобретению могут присутствовать в любой из двух форм, т.е. полиморфизмы имеют два аллеля. Один аллель может быть охарактеризован как аллель, сообщающий резистентность к инфекционному некрозу поджелудочной железы. Это означает, что радужная форель, имеющая такой аллель в положении полиморфизма, подробно описанного в настоящем описании, демонстрирует повышенную резистентность к IPN. Этот аллель обозначают в настоящем описании как "аллель резистентности к IPN". Соответствующий аллель резистентности к IPN для каждого из однонуклеотидных полиморфизмов по изобретению указан в таблице 1 ниже. Таким образом, аллель резистентности к IPN в соответствии с настоящим изобретением можно использовать для прогнозирования увеличенной резистентности радужной форели к инфекционному некрозу поджелудочной железы. Аллель резистентности к IPN в соответствии с настоящим изобретением также можно использовать для селекции радужной форели, имеющей увеличенную резистентность к инфекционному некрозу поджелудочной железы. Другой аллель может быть охарактеризован как аллель, который не сообщает увеличенную резистентность к инфекционному некрозу поджелудочной железы. Такой аллель обозначают в настоящем описании как "аллель не резистентности к IPN".
Радужная форель представляет собой диплоидный, в некоторых случаях триплоидный, организм, и, таким образом, она обладает по меньшей мере двумя копиями полиморфизмов по изобретению (по одной копии на каждой копии хромосомы 1).
Как продемонстрировано в настоящем описании, если по меньшей мере один аллель полиморфизма, и, более конкретно, SNP, представляет собой соответствующий аллель резистентности к IPN, тогда радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы по сравнению с радужной форелью, у которой оба аллеля представляют собой аллель не резистентности к IPN (т.е. такая радужная форель является гомозиготной по аллелю не резистентности к IPN). В значительном большинстве случаев резистентность к инфекционному некрозу поджелудочной железы еще более возрастает, если оба аллеля полиморфизма, и, более конкретно SNP, представляют собой соответствующий аллель резистентности к IPN (такая радужная форель является гомозиготной по аллелю резистентности к IPN). Такое дальнейшее повышение, например, наблюдают для SNP AX-89929954 (SNP #1), AX-89918280 (SNP #2) и chr1_7515539 (SNP # 160), которые являются наиболее статистически значимыми SNP, ассоциированными с IPN (см. таблицу 3).
Полиморфизм по изобретению может представлять собой любой из нескольких полиморфизмов, ассоциированных с повышенной резистентностью радужной форели к инфекционному некрозу поджелудочной железы. В частности, полиморфизм по изобретению представляет собой полиморфизм, расположенный на хромосоме 1 радужной форели (согласно номенклатуре Palti et al. (2011)), т.е. полиморфизм, обнаруженный на хромосоме 1, исходя из анализа генетического сцепления, флуоресцентной гибридизации in situ (FISH) или любого другого способа, который приписывает ДНК-полиморфизмы их соответствующим хромосомам.
Полиморфизм по изобретению может представлять собой любой полиморфизм, включая однонуклеотидный полиморфизм, расположенный в любой из геномных последовательностей радужной форели, приведенных в столбце под названием "контиг GenBank" в таблице 1.
Полиморфизм по изобретению может представлять собой любой полиморфизм, включая однонуклеотидный полиморфизм, расположенный в геномной последовательности радужной форели, имеющей ID GenBank FR904293.1.
Полиморфизм по изобретению может представлять собой любой полиморфизм, включая однонуклеотидный полиморфизм, генетическое расстояние которого от SNP AX-89929954 является меньшим или равным 10 сантиморган. В рамках настоящего изобретения, генетическое расстояния оценивают на основе события рекомбинации у самки радужной форели, но не на основе событий рекомбинации у самца радужной форели. Специалисту в данной области известно, как оценивать расстояния на генетической карте, а также то, какие данные требуются для этой оценки.
Полиморфизм по изобретению может представлять собой любой полиморфизм, включая однонуклеотидный полиморфизм, который находится в прочном неравновесии по сцеплению (LD) с SNP AX-89929954. В рамках настоящего изобретения, полиморфизмы определяют как находящиеся в высоком LD, если квадрат коэффициента корреляции между двумя локусами (r2, наиболее часто используемая мера LD) равен или превышает 0,5. Специалисту в данной области известно, как оценивать r2, а также какие данные требуются для этой оценки.
Полиморфизм по изобретению может представлять собой по меньшей мере один из однонуклеотидных полиморфизмов, приведенных в таблице 1. Таким образом, согласно определенным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, приведенных в таблице 1. Каждый из SNP, приведенных в таблице 1, рассматривается как индивидуально описанный в качестве части настоящего изобретения.
Таблица 1: SNP, ассоциированные с повышенной резистентностью к IPN. A=аденин, G=гуанин; C=цитозин, T=тимин. ID Affymetix представляет собой уникальный идентификатор, присвоенный каждому SNP в Affymetrix, являющемся поставщиком коммерческого анализа для генотипирования, который включает многие из SNP, приведенных в таблице; ID Affymerix служит в качестве ссылки на дополнительные детали, касающиеся SNP, предоставленные в файле, который может быть загружен с http://www.affymetrix.com/estore/. Контиг GenBank представляет собой название контига ДНК GenBank (последовательность генома радужной форели), где расположен SNP, и положение представляет собой положение SNP в контиге. dbSNP ss-no.(ss#) представляет собой заявочный номер SNP в базе данных однонуклеотидных полиморфизмов (dbSNP) NCBI (National Center for Biotechnology Information); соответствующий ссылочный номер SNP (rs#) может быть извлечен из NCBI.
#
NCBI dbSNP ss-no. в таблице 1 выше указывает на эталонную последовательность и положение SNP в этой эталонной последовательности. Специалисты в данной области могут без труда идентифицировать эталонную последовательность и положение SNP с использованием заявочного номера dbSNP ss.
Таблица 2: Нуклеотидная последовательность, содержащая SNP. [Аллель резистентности к IPN/аллель не резистентности к IPN] указывает на полиморфный участок, включающий аллельные варианты.
#
7515539
В столбце таблицы 2, обозначенном "нуклеотидная последовательность, содержащая SNP," представлена эталонная нуклеотидная последовательность для идентификации SNP в геноме радужной форели. Каждая из последовательностей SEQ ID NO: 1-78 и SEQ ID NO: 160-229 представляет собой полиморфную последовательность, включающую полиморфный участок. "Полиморфная последовательность" представляет собой нуклеотидную последовательность, включающую полиморфный участок, в котором находится SNP. Квалифицированный специалист можно использовать всю или только часть полиморфной последовательности, фланкирующей полиморфный участок, для идентификации SNP в геноме радужной форели.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267, chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX-89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX-89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
Согласно другим конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317 и AX-89966423.
Согласно другим конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945 и AX-89934682.
Согласно другим конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой AX-89929954 или AX-89918280.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой AX-89929954.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой AX-89918280.
Согласно следующим конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из группы, состоящей из: chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044 и chr1 10439048.
Согласно следующему конкретному варианту осуществления, по меньшей мере один SNP по изобретению выбран из группы, состоящей из: chr1_7515539, chr1_7108873 и chr1_6864558.
Согласно определенным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, соответствующих положению 36 полиморфных последовательностей, указанных в любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, соответствующих положению 36 полиморфных последовательностей, указанных в любой из SEQ ID NO: 1-34.
Согласно следующим другим конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, соответствующих положению 36 полиморфных последовательностей, указанных в любой из SEQ ID NO: 160-193.
Согласно следующим другим конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, соответствующих положению 36 полиморфных последовательностей, указанных в любой из SEQ ID NO: 1-18.
Согласно следующим другим конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, соответствующих положению 36 полиморфных последовательностей, указанных в любой из SEQ ID NO: 160-162.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, соответствующих положению 36 полиморфных последовательностей, указанных в SEQ ID NO: 1 или SEQ ID NO: 2.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой SNP, определяемый положением 36 полиморфной последовательности, указанной в SEQ ID NO: 1.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой SNP, определяемый положением 36 полиморфной последовательности, указанной в SEQ ID NO: 2.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP по изобретению выбран из SNP, соответствующих положению 36 полиморфных последовательностей, указанных в SEQ ID NO: 230, SEQ ID NO: 231 и SEQ ID NO: 232.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой SNP, определяемый положением 36 полиморфной последовательности, указанной в SEQ ID NO: 230.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой SNP, определяемый положением 36 полиморфной последовательности, указанной в SEQ ID NO: 231.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP по изобретению представляет собой SNP, определяемый положением 36 полиморфной последовательности, указанной в SEQ ID NO: 232.
Понятно, что представленное выше описание полиморфизмов по изобретению, и, в частности, SNP и аллеля(ей) резистентности к IPN применимо к следующим аспектам.
Способы по изобретению
В одном аспекте настоящее изобретение относится к способу прогнозирования повышенной резистентности форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN). В частности, настоящее изобретение относится к способу прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN), причем способ включает:
определение присутствия по меньшей мере одного аллеля, сообщающего резистентность к IPN ("аллель резистентности к IPN") в геноме (например, на хромосоме 1 генома) указанной радужной форели.
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1. Каждый из SNP, приведенных в таблице 1, рассматривается как индивидуально описанный в качестве части настоящего изобретения.
Согласно определенным вариантам осуществления, настоящее изобретение относится к способу прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN), причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229 посредством 1-5, например, 1-2, нуклеотидных замен.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1 (и повторен в таблице 2).
Согласно конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-34 и 160-193, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-34 и 160-193 посредством 1-5, например, 1-2, нуклеотидных замен.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно другим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, присутствующих в полиморфном участке по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-18 и 160-162, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-18 и 160-162 посредством 1-5, например, 1-2, нуклеотидных замен.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 1 или SEQ ID NO: 2, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 1 или SEQ ID NO: 2 посредством 1-5, например, 1-2, нуклеотидных замен;
где присутствие цитозина в положении, соответствующем положению 36 SEQ ID NO: 1, или присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 2, указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно следующим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 160, SEQ ID NO: 161 или SEQ ID NO: 162, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 160, SEQ ID NO: 161 или SEQ ID 162 посредством 1-5, например, 1-2, нуклеотидных замен;
где присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 160, присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 161, или присутствие цитозина в положении, соответствующем положению 36 SEQ ID NO: 162, указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 1, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 1 посредством 1-5, например, 1-2, нуклеотидных замен;
где присутствие цитозина в положении, соответствующем положению 36 SEQ ID NO: 1, указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 2, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 2 посредством 1-5, например, 1-2, нуклеотидных замен;
где присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 2 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно следующим более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 160, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 160 посредством 1-5, например, 1-2, нуклеотидных замен;
где присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 160 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно следующим более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 161, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 161 посредством 1-5, например, 1-2, нуклеотидных замен;
где присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 161 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно следующим более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 162, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 162 посредством 1-5, например, 1-2, нуклеотидных замен;
где присутствие цитозина в положении, соответствующем положению 36 SEQ ID NO: 162, указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно определенным другим вариантам осуществления, настоящее изобретение относится к способу прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN), причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085 AX-89925267, chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX-89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX-89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317 и AX-89966423.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945 и AX-89934682.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280;
где присутствие цитозина в положении AX-89929954 или гуанина в положении AX-89918280 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой AX-89929954;
где присутствие цитозина в положении AX-89929954 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой AX-89918280;
где присутствие гуанина в положении AX-89918280 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно следующим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044 и chr1 10439048.
Радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой chr1_7515539, chr1_7108873 или chr1_6864558, где присутствие гуанина в положении chr1_7515539, гуанина в положении chr1_7108873 или цитозина в положении chr1_6864558 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно следующим более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой chr1_7515539;
где присутствие гуанина в положении chr1_7515539 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой chr1_7108873;
где присутствие гуанина в положении chr1_7108873 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой chr1_6864558;
где присутствие цитозина в положении chr1_6864558 указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
Способы прогнозирования повышенной резистентности радужной форели к IPN могут вовлекать определение идентичности нуклеотида, присутствующего по меньшей мере в одном аллеле более чем одного SNP, например, по меньшей мере двух, по меньшей мере трех или по меньшей мере 4 SNP. Затем прогнозирование может быть основано на присутствии аллелей резистентности к IPN для проанализированных SNP. Например, можно проводить генотипирование по меньшей мере SNP AX-89929954 (SNP #1) и AX-89918280 (SNP #2). Также можно проводить генотипирование по меньшей мере SNP AX-89929954 (SNP #1), AX-89918280 (SNP #2) и AX-89938309 (SNP #3). Также можно проводить генотипирование по меньшей мере SNP AX-89929954 (SNP #1), AX-89918280 (SNP #2), AX-89938309 (SNP #3), AX-89960828 (SNP #4) и chr_1 7515539 (SNP #160).
В следующем аспекте настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы. В частности, настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, причем способ включает:
определение присутствия по меньшей мере одного (например, по меньшей мере двух) аллеля, сообщающего резистентность к IPN ("аллель резистентности к IPN") в геноме (например, на хромосоме 1 генома) указанной радужной форели; и
отбор указанной радужной форели как имеющей увеличенную резистентность, когда присутствует по меньшей мере один аллель резистентности к IPN.
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно определенным вариантам осуществления, настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-34, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-34 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно следующим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 160-193, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 160-193 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-18, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-18 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно следующим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 160-162, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 160-162 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 1 или SEQ ID NO: 2, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 1 или SEQ ID NO: 2 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда цитозин присутствует в положении, соответствующем положению 36 SEQ ID NO: 1, или гуанин присутствует в положении, соответствующем положению 36 SEQ ID NO: 2.
Согласно следующим более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 160, SEQ ID NO: 161 или SEQ ID NO: 162, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 160, SEQ ID NO: 161 или SEQ ID NO: 162 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда гуанин присутствует в положении, соответствующем положению 36 SEQ ID NO: 160, гуанин присутствует в положении, соответствующем положению 36 SEQ ID NO: 161, или цитозин присутствует в положении, соответствующем положению 36 SEQ ID NO: 162.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 1, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 1 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда цитозин присутствует в положении, соответствующем положению 36 SEQ ID NO: 1.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 1 или SEQ ID NO: 2, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 2 посредством 1-5, например, 1-2, нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда гуанин присутствует в положении, соответствующем положению 36 SEQ ID NO: 2.
Согласно другим определенным вариантам осуществления, настоящее изобретение относится к способу селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267; и chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX-89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX-89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317 и AX-89966423; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно другим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945 и AX-89934682; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой аллель резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно следующим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: 7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044 и chr1 10439048, и отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой аллель резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно следующим другим конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP выбран из группы, состоящей из: chr1 7515539, chr1_7108873 и chr1_6864558,
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой аллель резистентности к IPN соответствующего SNP. Аллель резистентности к IPN для каждого SNP указан в таблице 1.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда цитозин присутствует в положении AX-89929954 или гуанин присутствует в положении AX-89918280.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой AX-89929954; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда цитозин присутствует в положении AX-89929954.
Согласно более конкретным вариантам осуществления, способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой AX-89918280; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда гуанин присутствует в положении AX-89918280.
Согласно следующим более конкретным вариантам осуществления, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме (например, на хромосоме 1 генома) указанной радужной форели, причем указанный по меньшей мере один SNP представляет собой chr1_7515539, chr1_7108873 или chr1_6864558; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда гуанин присутствует в положении chr1_7515539, гуанин присутствует в положении chr1_7108873 или цитозин присутствует в положении chr1_6864558.
Способы селекции радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, могут вовлекать определение идентичности нуклеотида по меньшей мере одного аллеля более чем одного, например, по меньшей мере двух, по меньшей мере трех или по меньшей мере 4 SNP. Затем селекция может быть основана на присутствии аллелей резистентности к IPN для проанализированных SNP. Например, можно генотипировать по меньшей мере SNP AX-89929954 (SNP #1) и AX-89918280 (SNP #2). Также можно генотипировать по меньшей мере SNP AX-89929954 (SNP #1), AX-89918280 (SNP #2) и AX-89938309 (SNP #3). Также можно генотипировать по меньшей мере SNP AX-89929954 (SNP #1), AX-89918280 (SNP #2), AX-89938309 (SNP #4), AX-89960828 (SNP #4) и chr1_7515539 (SNP#160).
В данной области известны многочисленные способы определения идентичности нуклеотида аллеля, присутствующего в полиморфном участке. Например, определение может вовлекать анализ последовательности радужной форели, подлежащей исследованию, с использованием, например, традиционных методологий анализа последовательности (например, "способ дидезокси-опосредуемой терминации цепи", также известный как "способ Сэнгера" (Sanger, F., et al., J. Molec. Biol. 94: 441 (1975); Prober et al. Science 238: 336-340 (1987)) и "способ химической деградации", также известный как "способ Максама-Гилберта" (Maxam, A. M., et al., Proc. Natl. Acad. Sci. (U. S. A.) 74: 560 (1977). Альтернативно определение может вовлекать удлинение на одно основание ДНК-олинуклеотидов, оканчивающихся на полиморфном участке (например, анализ iPLEX от Sequenom (San Diego, США) и анализ Infinium от Illumina (San Diego, США), анализ аллелеспецифического лигирования (например, технология Axiom от Affymetrix (San Diego, США), аллелеспецифическая ПЦР (например, анализ SNPtype от Fluidigm (San Francisco) или анализ KASP от LGC Genomics (Teddington, Великобритания)), или конкурентная гибридизация зондов, комплементарных различным аллелям (например, анализ TaqMan от Applied Biosystems (Foster City, США)).
Способы выявления аллельного варьирования также рассмотрены в Nollau et al., Clin. Chem. 43, 1114-1120, 1997; и в стандартных справочниках, например, "Laboratory Protocols for Mutation Detection", Ed. by U. Landegren, Oxford University Press, 1996, и "PCR", 2nd Edition by Newton & Graham, BIOS Scientific Publishers Limited, 1997.
Для анализа SNP, например, может быть пригодным использование олигонуклеотидов, специфических к альтернативным аллелям SNP. Такие олигонуклеотиды, которые выявляют варьирование на один нуклеотид в последовательностях-мишенях, могут быть обозначены такими терминами, как "аллелеспецифические олигонуклеотиды", "аллелеспецифические зонды" или "аллелеспецифические праймеры". Конструирование и применение аллелеспецифических зондов для анализа полиморфизмов описано, например, в Mutation Detection A Practical Approach, ed. Cotton et al. Oxford University Press, 1998; Saiki et al., Nature 324, 163-166 (1986); Dattagupta, EP235726; и Saiki, WO 89/11548.
Радужная форель по изобретению
В следующем аспекте настоящее изобретение относится к радужной форели, такой как изолированная радужная форель, обладающая повышенной резистентностью к инфекционному некрозу поджелудочной железы. В частности, настоящее изобретение относится к радужной форели или к ее потомству, содержащим в их геноме (например, на хромосоме 1 их генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267, chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX-89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX-89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317 и AX-89966423.
Согласно другим конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945 и AX-89934682.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89918280.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1 7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044 и chr1 10439048.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_7515539, chr1_7108873 и chr1_6864558.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_7515539.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_7108873.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_6864558.
Согласно определенным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156, и 230-299 и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-112, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-112 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно другим конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-96, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-96 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно другим более конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 79, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 79 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно другим более конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно следующим конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-263, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-263 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно следующим более конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно другим более конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 230, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 230 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно другим более конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 231, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 231 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно другим более конкретным вариантам осуществления, радужная форель или ее потомство, такие как изолированная радужная форель или ее потомство, содержат в их геноме (например, на хромосоме 1 их генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 232, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, радужная форель представляет собой самку.
Согласно определенным другим вариантам осуществления, радужная форель представляет собой самца.
Согласно определенным вариантам осуществления, радужная форель или ее потомство получают способом в соответствии с настоящим изобретением.
В следующем аспекте настоящего изобретения, радужная форель или ее потомство содержат в их геноме по меньшей мере один аллель, сообщающий резистентность к IPN, и их получают способом, включающим стадии:
a) генотипирования форели,
b) отбора особей, имеющих по меньшей мере один аллель, предпочтительно два аллеля, сообщающих резистентность к IPN ("аллель резистентности к IPN"); и
c) скрещивания особей таким образом, чтобы по меньшей мере одна особь в каждой скрещенной паре имела два аллеля, сообщающих резистентность к IPN.
Согласно определенным вариантам осуществления скрещивание на c) также можно проводить таким образом, чтобы каждая скрещиваемая пара имела два аллеля, сообщающих резистентность к IPN, или чтобы каждая скрещиваемая пара имела один аллель, сообщающий резистентность к IPN.
Согласно определенным вариантам осуществления радужной форели или ее потомства, получаемых посредством способа, по меньшей мере один аллель резистентности к IPN может представлять собой аллель по меньшей мере одного однонуклеотидного полиморфизма (SNP).
Согласно следующим определенным вариантам осуществления радужной форели или ее потомства, получаемых посредством способа, по меньшей мере один SNP может быть выбран из SNP, приведенных в таблице 1.
Согласно более определенным вариантам осуществления, радужная форель или ее потомство, получаемые посредством способа, содержат в их геноме по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, радужная форель представляет собой самку.
Согласно определенным другим вариантам осуществления, радужная форель представляет собой самца.
В следующем аспекте настоящее изобретение относится к популяции радужной форели, такой как изолированная популяция, причем каждая особь в популяции имеет повышенную резистентность к инфекционному некрозу поджелудочной железы. В частности, настоящее изобретение относится к популяции радужной форели, причем каждая особь в популяции содержит в ее геноме по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267, chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX-89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX-89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317 и AX-89966423.
Согласно другим конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945 и AX-89934682.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89918280.
Согласно следующим конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы chr1 7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044 и chr1 10439048.
Согласно следующим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1 7515539, chr1_7108873 и chr1_6864558.
Согласно определенным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156, и 230-299 и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-112, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 81-112 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно другим конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-96, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-96 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 79, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 79 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно следующим конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-263, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-263, посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 230, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 230 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 231, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 231 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 232, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, популяция радужной форели представляет собой популяцию самок радужной форели.
Согласно определенным вариантам осуществления, популяция радужной форели представляет собой популяцию самцов радужной форели.
Согласно определенным вариантам осуществления, популяция радужной форели представляет собой популяцию самцов и самок радужной форели.
В одном следующем аспекте настоящего изобретения, популяция радужной форели может содержать в ее геноме по меньшей мере один аллель, сообщающий резистентность к IPN, и ее получают способом, включающим стадии:
a) генотипирования форели,
b) селекции особей, имеющих по меньшей мере один аллель, предпочтительно два аллеля, сообщающих резистентность к IPN ("аллель резистентности к IPN"); и
c) скрещивания особей таким образом, чтобы по меньшей мере одна особь в каждой скрещенной паре имела два аллеля, сообщающих резистентность к IPN.
Согласно определенным вариантам осуществления скрещивание на c) также можно проводить таким образом, чтобы каждая скрещиваемая пара имела два аллеля, сообщающих резистентность к IPN, или чтобы каждая скрещиваемая пара имела один аллель, сообщающий резистентность к IPN.
Согласно определенным вариантам осуществления популяции радужной форели, получаемой посредством способа, по меньшей мере один аллель резистентности к IPN может представлять собой аллель по меньшей мере одного однонуклеотидного полиморфизма (SNP).
Согласно следующим определенным вариантам осуществления популяции радужной форели, получаемой посредством способа, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно следующим более определенным вариантам осуществления, популяция, получаемая посредством способа, содержит в ее геноме по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299 и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, популяция радужной форели представляет собой популяцию самок радужной форели.
Согласно определенным вариантам осуществления, популяция радужной форели представляет собой популяцию самцов радужной форели.
Согласно определенным вариантам осуществления, популяция радужной форели представляет собой популяцию самцов и самок радужной форели.
В следующем аспекте настоящее изобретение относится к клетке радужной форели, такой как выделенная клетка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267, chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX-89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX-89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317 и AX-89966423.
Согласно другим конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945 и AX-89934682.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89918280.
Согласно определенным вариантам осуществления, настоящее изобретение относится к клетке радужной форели, такой как выделенная клетка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно конкретным вариантам осуществления, клетка радужной форели, такая как выделенная клетка радужной форели, содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-112, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-112 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно другим конкретным вариантам осуществления, клетка радужной форели, такая как выделенная клетка радужной форели, содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-96, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-96 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, клетка радужной форели, такая как выделенная клетка радужной форели, содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, клетка радужной форели, такая как выделенная клетка радужной форели, содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 79, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 79 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, клетка радужной форели, такая как выделенная клетка радужной форели, содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно следующим конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-263, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-263 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 230, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 230 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 231, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 231 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 232, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, клетка радужной форели представляет собой гамету.
Согласно конкретным вариантам осуществления, клетка радужной форели представляет собой икринку, такую как икринка на стадии глазка.
Согласно более конкретным вариантам осуществления, икринка является неоплодотворенной.
Согласно другим более конкретным вариантам осуществления, икринка является оплодотворенной.
Согласно конкретным вариантам осуществления, клетка радужной форели представляет собой спермий.
Согласно определенным другим вариантам осуществления, клетка радужной форели представляет собой соматическую клетку.
Согласно определенным вариантам осуществления, клетка радужной форели выделена из радужной форели по изобретению.
Согласно конкретным вариантам осуществления, клетка радужной форели выделена из самки радужной форели по изобретению.
Согласно конкретным вариантам осуществления, клетка радужной форели выделена из самца радужной форели по изобретению.
В следующем аспекте настоящее изобретение относится к популяции клеток радужной форели, такой как выделенная популяция клеток радужной форели, причем каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317, AX-89966423, AX-89933348, AX-89969315, AX-89919958, AX-89968417, AX-89946851, AX-89976917, AX-89945446, AX-89919457, AX-89973597, AX-89938138, AX-89971866, AX-89958882, AX-89961273, AX-89944901, AX-89919465, AX-89959425, AX-89917102, AX-89959281, AX-89916766, AX-89920507, AX-89957370, AX-89934009, AX-89929663, AX-89952300, AX-89916572, AX-89946911, AX-89974593, AX-89927158, AX-89970383, AX-89965404, AX-89955634, AX-89932926, AX-89941493, AX-89943031, AX-89957682, AX-89960611, AX-89950199, AX-89928407, AX-89962035, AX-89931951, AX-89976536, AX-89916801, AX-89929085, AX-89925267, chr1_7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044, chr1_10439048, chr1_8142346, chr1_8092208, chr1_8138683, chr1_8139206, chr1_8139744, chr1_8140789, chr1_8141687, chr1_8154917, chr1_7454708, chr1_7504847, chr1_7505686, chr1_7505817, chr1_8202031, chr1_8228173, chr1_8309469, chr1_8163977, chr1_27786931, chr1_8194629, chr1_7505259, chr1_8474659, chr1_8282602, chr1_8306806, chr1_8341618, chr1_8343786, chr1_8345836, chr1_8350569, chr1_8402403, AX-89962103, chr1_8279302, chr1_8334901, chr1_7561600, AX-89956272, chr1_7938827, chr1_10810229, chr1_11007071 и chr1_10884171.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945, AX-89934682, AX-89951942, AX-89937020, AX-89924837, AX-89958601, AX-89923477, AX-89959350, AX-89929482, AX-89937712, AX-89949602, AX-89925103, AX-89938051, AX-89924174, AX-89936461, AX-89916703, AX-89935317 и AX-89966423.
Согласно другим конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы, состоящей из: AX-89929954, AX-89918280, AX-89938309, AX-89960828, AX-89930342, AX-89928530, AX-89949788, AX-89928131, AX-89949832, AX-89916790, AX-89973719, AX-89962023, AX-89921280, AX-89931666, AX-89921585, AX-89953905, AX-89952945 и AX-89934682.
Согласно более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89918280.
Согласно следующим конкретным вариантам осуществления, по меньшей мере один SNP выбран из группы: chr1 7515539, chr1_7108873, chr1_6864558, chr1_7186663, chr1_6730531, chr1_27891953, AX-89953259, chr1_6740481, chr1_6770611, chr1_7412807, chr1_7360179, chr1_7411803, chr1_7431445, chr1_7433199, chr1_7441254, chr1_7441877, chr1_7533570, chr1_6834898, chr1_6730142, chr1_6746052, chr1_6794061, chr1_7399212, chr1_7442637, chr1_7358019, chr1_7709828, chr1_7598090, chr1_7626471, chr1_7598743, chr1_7670293, chr1_7670561, chr1_7647634, chr1_7356089, chr1_8109044 и chr1_10439048.
Согласно следующим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_7515539, chr1_7108873 и chr1_6864558.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_7515539.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_7108873.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1_6864558.
Согласно определенным вариантам осуществления, настоящее изобретение относится к популяции клеток радужной форели, такой как выделенная популяция клеток радужной форели, причем каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156, и 230-299 и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно конкретным вариантам осуществления, популяция клеток радужной форели, такая как выделенная популяция клеток радужной форели, представляет собой популяцию, где каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-112, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-112 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно другим конкретным вариантам осуществления, популяция клеток радужной форели, такая как выделенная популяция клеток радужной форели, представляет собой популяцию, где каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-96, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-96 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция клеток радужной форели, такая как выделенная популяция клеток радужной форели, представляет собой популяцию, где каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция клеток радужной форели, такая как выделенная популяция клеток радужной форели, представляет собой популяцию, где каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 79, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 79 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция клеток радужной форели, такая как выделенная популяция клеток радужной форели, представляет собой популяцию, где каждая индивидуальная клетка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно следующим конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-263, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-263 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 230, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 230 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 231, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 231 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно более конкретным вариантам осуществления, популяция радужной форели, такая как изолированная популяция радужной форели, представляет собой популяцию, где каждая особь в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 232, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, клетки указанной популяции представляют собой гаметы.
Согласно конкретным вариантам осуществления, клетки указанной популяции представляют собой икринки, такие как икринки на стадии глазка.
Согласно более конкретным вариантам осуществления, икринки являются неоплодотворенными.
Согласно другим более конкретным вариантам осуществления, икринки являются оплодотворенными.
Согласно другим более конкретным вариантам осуществления, популяция клеток радужной форели представляет собой смешанную популяцию оплодотворенных и неоплодотворенных икринок.
Согласно другим конкретным вариантам осуществления клетки указанной популяции представляют собой спермии.
Согласно определенным другим вариантам осуществления, клетки указанной популяции представляют собой соматические клетки.
Согласно определенным вариантам осуществления, популяция клеток радужной форели выделена из радужной форели по изобретению.
Согласно конкретным вариантам осуществления, популяция клеток радужной форели выделена из самки радужной форели по изобретению.
Согласно конкретным вариантам осуществления, популяция клеток радужной форели выделена из самца радужной форели по изобретению.
В конкретном аспекте настоящее изобретение относится к икринке радужной форели, такой как выделенная икринка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280.
Согласно определенным вариантам осуществления, настоящее изобретение относится к икринке радужной форели, такой как выделенная икринка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно конкретным вариантам осуществления, настоящее изобретение относится к икринке радужной форели, такой как выделенная икринка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно другим более конкретным вариантам осуществления, по меньшей мере один SNP представляет собой chr1 7515539, chr1_7108873 или chr1_6864558.
Согласно конкретным вариантам осуществления, настоящее изобретение относится к икринке радужной форели, такой как выделенная икринка радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, икринка радужной форели является неоплодотворенной.
Согласно определенным другим вариантам осуществления, икринка радужной форели является оплодотворенной.
Согласно конкретным вариантам осуществления, икринка радужной форели представляет собой икринку на стадии глазка.
Согласно определенным вариантам осуществления, икринка радужной форели выделена из самки радужной форели по изобретению.
В следующем аспекте настоящее изобретение относится к популяции икринок радужной форели, такой как выделенная популяция икринок радужной форели, причем каждая индивидуальная икринка популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере один аллель, сообщающий резистентность к IPN ("аллель резистентности к IPN").
Согласно определенным вариантам осуществления, по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного полиморфизма, такого как по меньшей мере один однонуклеотидный полиморфизм (SNP).
Согласно определенным вариантам осуществления, по меньшей мере один SNP выбран из SNP, приведенных в таблице 1.
Согласно конкретным вариантам осуществления, по меньшей мере один SNP представляет собой AX-89929954 или AX-89918280.
Согласно следующим вариантам осуществления, по меньшей мере один SNP представляет собой chr1 7515539, chr1_7108873 или chr1_6864558.
Согласно определенным вариантам осуществления, настоящее изобретение относится к популяции икринок радужной форели, такой как выделенная популяция икринок радужной форели, которая содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены не находятся в положении 36 указанной происходящей последовательности.
Согласно конкретным вариантам осуществления, настоящее изобретение относится к популяции икринок радужной форели, такой как выделенная популяция икринок радужной форели, причем каждая индивидуальная икринка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно конкретным вариантам осуществления, настоящее изобретение относится к популяции икринок радужной форели, такой как выделенная популяция икринок радужной форели, причем каждая индивидуальная икринка в популяции содержит в ее геноме (например, на хромосоме 1 ее генома) по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности.
Согласно определенным вариантам осуществления, популяция икринок радужной форели является неоплодотворенной.
Согласно определенным другим вариантам осуществления, популяция икринок радужной форели является оплодотворенной.
Согласно определенным другим вариантам осуществления, популяция икринок радужной форели представляет собой популяцию икринок на стадии глазка.
Согласно определенным вариантам осуществления, популяция икринок радужной форели выделена из самки радужной форели по изобретению.
Молекулы нуклеиновых кислот по изобретению
В следующем аспекте настоящее изобретение относится к молекуле нуклеиновой кислоты, такой как выделенная молекула нуклеиновой кислоты. Более конкретно, настоящее изобретение относится к нуклеиновой кислоте, такой как выделенная нуклеиновая кислота, содержащая по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
Согласно определенным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-112, b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-112 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
Согласно определенным другим вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-96, b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-96 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
Согласно конкретным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и SEQ ID NO: 80, b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
Согласно более конкретным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит нуклеотидную последовательность, указанную в SEQ ID NO: 79, или нуклеотидную последовательность, происходящую из SEQ ID NO: 79 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, или последовательности, комплементарные им.
Согласно более конкретным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит нуклеотидную последовательность, указанную в SEQ ID NO: 80, или нуклеотидную последовательность, происходящую из SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, или последовательности, комплементарные им.
Согласно определенным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-263, b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-263 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
Согласно конкретным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит по меньшей мере одну нуклеотидную последовательность, выбранную из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, и c) последовательностей, комплементарных a) и b).
Согласно более конкретным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит нуклеотидную последовательность, указанную в SEQ ID NO: 230, или нуклеотидную последовательность, происходящую из SEQ ID NO: 230 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, или последовательности, комплементарные им.
Согласно более конкретным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит нуклеотидную последовательность, указанную в SEQ ID NO: 231 или нуклеотидную последовательность, происходящую из SEQ ID NO: 231 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, или последовательности, комплементарные им.
Согласно более конкретным вариантам осуществления, молекула нуклеиновой кислоты, такая как выделенная молекула нуклеиновой кислоты, содержит нуклеотидную последовательность, указанную в SEQ ID NO: 232 или нуклеотидную последовательность, происходящую из SEQ ID NO: 232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности, или последовательности, комплементарные им.
Молекула нуклеиновой кислоты может иметь длину по меньшей мере 71 нуклеотид, такую как по меньшей мере 75 нуклеотидов или по меньшей мере 100 нуклеотидов.
Согласно определенным вариантам осуществления, нуклеиновая кислота имеет длину от 71 нуклеотида до 400 нуклеотидов, такую как от 71 нуклеотида до 200 нуклеотидов или от 71 до 100 нуклеотидов.
В следующем аспекте настоящее изобретение относится к олигонуклеотиду, такому как выделенный олигонуклеотид. Более конкретно, настоящее изобретение относится к олигонуклеотиду, такому как выделенный олигонуклеотид, содержащий по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-156 и 230-299, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и 156 и 230-299 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно определенным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-112, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и 112 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно определенным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79-96, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79-96 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно конкретным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 79 и 80, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 79 и 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно более конкретным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 79, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 79 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно другим более конкретным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 80 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно определенным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-263, и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230 и 263 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно конкретным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидных последовательностей, указанных в SEQ ID NO: 230-232 и b) нуклеотидных последовательностей, происходящих из любой из SEQ ID NO: 230-232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно более конкретным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 230, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 230 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно другим более конкретным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 231 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно другим более конкретным вариантам осуществления, олигонуклеотид содержит по меньшей мере 10 последовательно расположенных нуклеотидов, например, по меньшей мере 16 последовательно расположенных нуклеотидов, нуклеотидной последовательности, выбранной из группы, состоящей из a) нуклеотидной последовательности, указанной в SEQ ID NO: 80, и b) нуклеотидных последовательностей, происходящих из SEQ ID NO: 232 посредством 1-5, например, 1-2, нуклеотидных замен, при условии, что указанные нуклеотидные замены находятся не в положении 36 указанной происходящей последовательности; где указанные по меньшей мере 10 последовательно расположенных нуклеотидов включают нуклеотид в положении 36 a) или b); или последовательность, комплементарную указанному олигонуклеотиду.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину по меньшей мере 10 нуклеотидов, например, по меньшей мере 16 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину по меньшей мере 16 нуклеотиды, например, по меньшей мере 20 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину по меньшей мере 20 нуклеотидов, например, по меньшей мере 25 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину 10-200 нуклеотидов, такую как 10-150 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину 10-100 нуклеотидов, такую как 10-70 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину 16-100 нуклеотидов, такую как 16-70 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину 10-50 нуклеотидов, такую как 10-40 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину 16-50 нуклеотидов, такую как 16-40 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину 10-30 нуклеотидов, такую как 8-25 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, имеет длину 16-30 нуклеотидов, такую как 16-25 нуклеотидов.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, представляет собой праймер, такой как праймер для ПЦР.
Согласно определенным вариантам осуществления, олигонуклеотид или последовательность, комплементарная ему, представляет собой зонд, такой как зонд для гибридизации.
Согласно определенным вариантам осуществления, настоящее изобретение относится к последовательности, комплементарной олигонуклеотиду, указанному выше. Такую комплементарную последовательность можно использовать в качестве зонда, такого как зонд для гибридизации.
Зонд или праймер в соответствии с настоящим изобретением может иметь связанную с ним поддающуюся обнаружению метку или репортерную молекулу. Типичные метки включают радиоактивные изотопы, субстраты ферментов, кофакторы, лиганды, хемилюминесцентные или флуоресцентные агенты, гаптены и ферменты. Способы мечения и рекомендации по выбору меток, подходящих для различных целей, описаны, например, в Sambrook et al. (Molecular Cloning, A Laboratory Manual, CSHL, New York, 1989) и Ausubel et al. (Current Protocols in Molecular Biology, John Wiley & Sons, New York, 1998). В качестве конкретного примера, зонд или праймер может включать один флуорофор, такой как акцепторный флуорофор или донорный флуорофор. Такой флуорофор может быть связан с 5'- или 3'-концом зонда/праймера.
Зонды обычно имеют длину по меньшей мере 15 нуклеотидов, например, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18, по меньшей мере 19, по меньшей мере 20, по меньшей мере 25, по меньшей мере 30, по меньшей мере 35, по меньшей мере 40, по меньшей мере 45, по меньшей мере 50, по меньшей мере 55, по меньшей мере 60, по меньшей мере 65, по меньшей мере 70, или более последовательно расположенных нуклеотидов, комплементарных молекуле нуклеиновой кислоты, являющейся мишенью, например, 20-70 нуклеотидов, 20-60 нуклеотидов, 20-50 нуклеотидов, 20-40 нуклеотидов или 20-30 нуклеотидов.
Праймеры являются более короткими. Олигонуклеотид, используемый в качестве праймера, может иметь длину по меньшей мере 10 нуклеотидов. Специфичность праймера возрастает с увеличением его длины. Таким образом, например, праймер, который включает 30 последовательно расположенных нуклеотидов, будет гибридизоваться с последовательностью-мишенью с более высокой специфичностью, чем соответствующий праймер только из 15 нуклеотидов. Таким образом, для достижения более высокой специфичности, праймеры по изобретению имеют длину по меньшей мере 15 нуклеотидов, например, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18, по меньшей мере 19, по меньшей мере 20, по меньшей мере 25, по меньшей мере 30, по меньшей мере 35, по меньшей мере 40, по меньшей мере 45, по меньшей мере 50, по меньшей мере 55, по меньшей мере 60, по меньшей мере 65, по меньшей мере 70, или более последовательно расположенных нуклеотидов, комплементарных молекуле нуклеиновой кислоты, являющейся мишенью, например, 15-70 нуклеотидов, 15-60 нуклеотидов, 15-50 нуклеотидов, 15-40 нуклеотидов или 15-30 нуклеотидов. Пары праймеров можно использовать для амплификации последовательностей нуклеиновых кислот, например, посредством PCT, ПЦР в реальном времени или других способов амплификации нуклеиновых кислот, известных в данной области.
Подтверждение результатов, лежащих в основе настоящего изобретения
Для подтверждения ассоциации между резистентностью к IPN и аллелями четырех полиморфизмов по изобретению проводили два испытания с заражением. Испытания проводили в двух емкостях объемом 100 литров, и в каждой емкости группу особей радужной форели исследовали в отношении резистентности к одному из двух штаммов вируса IPN. Эти два штамма представляли собой 1) штамм (AGT11-2) серотипа Sp, выделенный из радужной форели, выращенной в воде Норвежского моря; этот же штамм использовали, когда впервые было осуществлено изобретение, и 2) штамм серотипа Wb был выделен при вспышке у радужной форели в Чили. Подтверждающий эксперимент подтвердил, что существует статистически значимая ассоциация между резистентностью к IPN и аллелями всех четырех исследованных полиморфизмов. Более того, ассоциация также была достоверной, когда штамм вируса IPN, использованный при первоначальном открытии изобретения (штамм серотипа Sp) заменяли другим штаммом (серотипа Wb, West Buxon). Следовательно, ассоциация между ДНК-полиморфизмами и резистентностью к IPN является воспроизводимой и независимой от штамма вируса.
Четыре полиморфизма, исследованных в подтверждающем эксперименте, были репрезентативными для всех полиморфизмов по изобретению. Остальные полиморфизмы по изобретению не исследовали прямо. Однако, поскольку все полиморфизмы по изобретению являются маркерами одного и того же локуса количественных признаков (QTL), можно сделать заключение, что любые другие полиморфизмы по изобретению прошли бы подтверждающее испытание.
Естественным и необходимым следствием этих открытий является то, что ДНК-полиморфизмы по настоящему изобретению можно использовать для получения радужной форели с повышенной резистентностью к IPN. Результаты этого подтверждающего исследования представлены в примерах 2 и 3.
Определенные определения
Как используют в рамках изобретения, "повышенная резистентность" к инфекционному некрозу поджелудочной железы означает, что особь, имеющая повышенную резистентность, имеет более высокую вероятность выжить при вспышке IPN, чем случайная особь (при той же вспышке), с которой она сравнима. Две особи являются сравнимыми, если они являются, с точки зрения дискриминирующих факторов, за исключением генотипа в SNP, который используют для прогнозирования резистентности к IPN, случайными представителями одной и той же популяции радужной форели. Вспышка IPN представляет собой состояние, при котором живая радужная форель подвергается воздействию IPN таким образом, что некоторые особи становятся инфицированными и распространяют вирус (что приводит к распространению заболевания). Вспышка может представлять собой, например, непреднамеренную вспышку вируса у радужной форели, выращиваемой в емкости или в пруду со свежей водой, непреднамеренную вспышку в садке форели, выращиваемой в морской воде, или контролируемую вспышку, индуцированную в качестве части лабораторного эксперимента. Испытания с заражением IPN, описанные в настоящем описании (испытания с заражением 1, 2, 3 и 4), являются примерами лабораторных экспериментов, которые определяют уровень выживаемости при вспышках IPN.
Как используют в рамках изобретения, "аллель резистентности к IPN" представляет собой аллель, сообщающий повышенную резистентность к инфекционному некрозу поджелудочной железы. Это означает, что радужная форель, имеющая такой аллель в положении полиморфизма, описанного в настоящем описании, демонстрирует повышенную резистентность к IPN. "Аллель резистентности к IPN" может соответствовать однонуклеотидному полиморфизму, который можно использовать для обнаружения или определения степени резистентности к IPN.
Как используют в рамках изобретения, "полиморфизм" представляет собой варьирование геномной последовательности. В частности, полиморфизм представляет собой положение в геноме, где обычно встречаются различные аллельные варианты между особями популяции или между особами различных популяций. Полиморфизм может представлять собой однонуклеотидное различие, присутствующее в локусе, или он может представлять собой вставку или делецию одного или нескольких нуклеотидов в некотором положении гена.
Как используют в рамках изобретения, "однонуклеотидный полиморфизм" или "SNP" относится к полиморфизму одного основания (нуклеотида) в последовательности ДНК среди особей в популяции. По существу, однонуклеотидный полиморфизм характеризуется присутствием в популяции одного или двух, трех или четырех различных нуклеотидов (т.е. аденин, цитозин, гуанин или тимин), как правило, менее чем всех четырех нуклеотидов, в конкретном локусе в геноме, таком как геном радужной форели.
Как используют в рамках изобретения, "полиморфная последовательность" относится к нуклеотидной последовательности, включающей полиморфный участок, в котором встречается SNP или другой тип полиморфизма.
Как используют в рамках изобретения, "полиморфный участок" представляет собой локус или положение в данной последовательности, в котором встречается расхождение. Предпочтительные полиморфные участки имеют по меньшей мере два аллеля, каждый из которых встречается с частотой более 1% и более предпочтительно более 10%. Специалистам в данной области будет понятно, что молекулы нуклеиновых кислот могут представлять собой двухцепочечные молекулы, и что указание на конкретный участок на одной цепи также относится к соответствующему участку на комплементарной цепи. При определении полиморфного участка или аллеля, указание на аденин, тимин, цитозин или гуанин в конкретном участке на одной цепи молекулы нуклеиновой кислоты также определяет тимин, аденин, гуанин или цитозин (соответственно) в соответствующем участке на комплементарной цепи нуклеиновой кислоты.
В рамках настоящего изобретения, когда полиморфизм указан как имеющий конкретный аллель, понятно, что этот конкретный аллель комбинируется с последовательностью, приведенной для данного полиморфизма. Например, когда указывается, что гуанин является аллелем резистентности SNP AX-89929954 (SNP #1), тогда понятно, что аллель резистентности AX-89929954 содержит гуаниновый нуклеотид в полиморфном участке, определенном в таблице 2, когда ДНК считывают в направлении, определенном в таблице 2. Иными словами, как указано в таблице 2, форма резистентности последовательности ДНК AX-89929954 (с фланкирующей последовательностью) представляет собой GAAAGAAACAGTGATAGGCTTTTAGTGAGCACATACATTTGACACACAGTTGTGTGAAAACAAAGCATGTG (полиморфный участок подчеркнут) при считывании в направлении, определенном в таблице 2. При считывании в противоположном направлении последовательность AX-89929954 (с фланкирующей последовательностью) представляет собой CACATGCTTTGTTTTCACACAACTGTGTGTCAAATGTATGTGCTCACTAAAAGCCTATCACTGTTTCTTTC (полиморфный участок подчеркнут). Хотя используют только одно направление, когда определяют аллели резистентности к IPN и аллели не резистентности к IPN в настоящем описании, два направления считывания являются эквивалентными.
Как используют в рамках изобретения, "образец", такой как биологический образец, который включает молекулы нуклеиновых кислот, представляет собой образец, полученный из радужной форели, включая, но не ограничиваясь ими, клетки, ткань и жидкости организма.
Как используют в рамках изобретения, "олигонуклеотид" представляет собой множество связанных нуклеотидов, которые соединены нативными фосфодиэфирными связями, как правило, длиной от 8 до 300 нуклеотидов.
Как используют в рамках изобретения, "зонды" и "праймер" представляют собой выделенные олигонуклеотиды длиной по меньшей мере 8 нуклеотидов, например, по меньшей мере 10 нуклеотидов, способные гибридизоваться с нуклеиновой кислотой-мишенью.
Как используют в рамках изобретения, "выделенный" означает, что организм или биологический компонент, такой как клетка, популяция клеток или молекула нуклеиновой кислоты, отделен от его естественного окружения.
Как используют в рамках изобретения, "генетическое сцепление" относится к тенденции полиморфизмов, расположенных вблизи друг от друга на хромосоме, наследоваться совместно в ходе мейоза. Таким образом, полиморфизмы, расположенные вблизи друг от друга на одной и той же хромосоме, называют генетически сцепленными. Аллели в двух таких генетически сцепленных локусах наследуются совместно (от родителей к потомкам) более часто, чем не совместно. Допустим, например, что существует два полиморфизма: полиморфизм A, имеющий аллели A1 и A2, и полиморфизм B, имеющий аллели B1 и B2. Далее, допустим, что данная радужная форель имеет все аллели A1, A2, B1 и B2 (иными словами, эта радужная форель является гетерозиготной как по маркеру A, так и по маркеру B). Если у этой конкретной радужной форели аллели A1 и B1 расположены на одной копии хромосомы, тогда аллели A1 и B1 передаются совместно потомству радужной форели чаще, чем не передаются.
Как используют в рамках изобретения, "генетический анализ сцепления" относится к статистической методике, где исследуют данные о генотипе, полученные для набора животных, включающих родителей и их потомков, для исследования присутствия генетического сцепления между полиморфизмами. Генетический анализ сцепления можно использовать для отнесения полиморфизмов к хромосомам при условии, что анализ включает полиморфизмы, которые уже были отнесены к хромосоме, с использованием, например, флуоресцентной гибридизации in situ.
Как используют в рамках изобретения "Флуоресцентная гибридизация in situ" или "FISH" относится к способу, который обнаруживает присутствие или отсутствие конкретных последовательностей ДНК на хромосомах. FISH можно использовать для отнесения известных ДНК-полиморфизмов к хромосомам.
"Сантиморган" представляет собой единицу измерения, используемую для описания генетических расстояний, где генетическое расстояние является мерой степени, с которой два полиморфизма генетически сцеплены.
Неравновесие по сцеплению (LD) или, точнее, неравновесие по сцеплению гаметической фазы, используют для описания совместного наследования аллелей генетически сцепленных полиморфизмов на уровне популяции. Допустим, например, что два полиморфизма расположены на одной хромосоме: полиморфизм A, имеющий аллели A1 и A2, и полиморфизм B, имеющий аллели B1 и B2. Все копии рассматриваемой хромосомы содержат комбинацию аллелей в двух локусах (т.е. гаплотипы), и существует четыре возможных гаплотипа: A1-B1, A1-B2, A2-B1 и A2-B2. Два локуса называют имеющими LD друг с другом, если количество гаплотипов A1-B1 и A2-B2 в популяции значимо больше или значимо меньше, чем количество гаплотипов A1-B2 и A2-B1.
Когда в настоящем описании указан числовой предел, его конечные точки включены. Также, все величины и поддиапазоны в числовом пределе или диапазоне прямо включены, если явно не указано иное.
После описания в общем настоящего изобретения, может быть достигнуто дальнейшее понимание с помощью определенных конкретных примеров, которые предоставлены в настоящем описании только для иллюстрации и не предназначены для ограничения, если нет иных указаний.
Примеры
Пример 1: Идентификация однонуклеотидных полиморфизмов, прогнозирующих IPN
Проводили два испытания с заражением для исследования резистентности мальков радужной форели к IPN. Получение и выращивание групп семей, а также подготовку к заражению проводили, как описано ранее в Wetten et al., 2011.
Первое испытание (заражение 1) проводили с целью найти оптимальный изолят вируса для заражения IPN радужной форели. Исследовали два отдельных штамма вируса: штамм V-1244, который является вирулентным у атлантического лосося, и другой штамм, выделенный из морской воды, в которой выращивали радужную форель в Норвегии (серотип Sp, AGTT11-2). Оба штамма исследовали в трех экземплярах емкостей, каждая из которых содержала 100 рыб, происходящих из десяти отдельных семейств форели. Взятие образцов погибших или умирающих особей проводили каждые сутки. Испытание завершали через 38 суток после начала. Штамм лосося вызвал общую смертность 20%, в то время как штамм радужной форели был значительно более вирулентным, вызвав общую смертность 85%.
Штамм вируса, выделенный из радужной форели (AGTT11-2), использовали во втором заражении (заражение 2). Целью этого исследования была идентификация SNP, которые являются диагностическими для уровня резистентности к IPN у отдельной радужной форели, т.е. SNP, ассоциированные с резистентностью к IPN. В испытание было включено пятьдесят различных семейств радужной форели, каждое из которых было представлено отдельной емкостью из 200 мальков (средняя масса малька=0,2 грамма). Все семейства заражали в емкости посредством добавления объема супернатанта вируса, соответствующего конечной концентрации вируса приблизительно 106 TCID50/мл воды. Взятие образцов погибших или умирающих особей проводили каждые сутки. Всех рыб, погибших во время испытания, а также всех выживших рыб, собранных при завершении испытания через 40 суток после заражения, замораживали при -18°C для анализа ДНК. Испытание завершилось через 48 суток после начала.
После заражения 2 в анализ было включено 8683 животных; эти животные включали 46 полностью сибсовых групп, происходящих из 29 мужских родительских особей и 25 женских родительских особей. Количество полностью сибсовых групп на мужскую родительскую особь находилось в диапазоне от 1 до 3, как и количество полностью сибсовых групп на женскую родительскую особь.
Общий уровень смертности в испытании заражения 2 составлял 93%. В полностью сибсовых группах уровень смертности варьировался от 67,8% до 99,5%. Из 8683 особей, подвергнутых испытанию заражением, 1723 было прогенотипировано. Эти 1723 животных включали (в среднем) 19 ранних смертей и 19 выживших или поздних смертей из каждой из 46 полностью сибсовых групп. Здесь, ранняя смертность представляла собой первую рыбу, погибшую в ее соответствующей полностью сибсовой группе, за исключением особей, которые погибли до 13 суток испытания с заражением 1 (было предположено, что несколько смертей, произошедших до 13 суток не являлись следствием IPN). Поздняя смертность представляла собой особей, которые погибли, или погибли последними, в их соответствующих полностью сибсовых группах. Умерших особей, которые продемонстрировали признаки неспособности поддерживаться на твердом корме, не генотипировали; это идентифицировали, исходя из их малого размера и отсутствия красных пигментов (поступающих из их корма) в их плавниках.
ДНК экстрагировали из хвостового плавника животных, подлежащих генотипированию, с использованием стандартного способа (набор DNAeasy 96 от QIAGEN (Venlo, Нидерланды)).
1723 животных генотипировали с использованием матрицы для генотипирования форели Axiom®, чипа с SNP, содержащего 57501 однонуклеотидный полиморфизм (SNP) в 96-луночном формате. Этот SNP-чип был разработан AquaGen в сотрудничестве с Департаментом сельского хозяйства США (USDA) и Affymetrix, и он коммерчески доступен от Affymetrix (San Diego, США). Генотипирование проводили с использованием собственной платформы Affymetrix Axiom в соответствии с руководством пользователя Axiom® 2.0 Assay Automated Workflow User Guide (http://media.affymetrix.com/support/downloads/manuals/axiom_2_assay_auto_workflow_user_guide.pdf).
Исходя из исходных данных, предоставленных с помощью оборудования Axiom, генотипам были присвоены названия с использованием программного обеспечения Affymetrix PowerTools (http://www.affymetrix.com/estore/partners_programs/programs/developer/tools/powertools.affx).
Анализ и интерпретацию исходных данных проводили в соответствии с Best Practices Workflow, предоставленным Affymetrix (http://www.affymetrix.com/estore/partners_programs/programs/developer/tools/powertools.affx). SNP и животных, имеющих параметры качества ниже параметров по умолчанию, предоставленных в Best Practices Workflow, не рассматривали для дальнейшего анализа.
SNP исследовали индивидуально в отношении ассоциации с резистентностью к IPN, определяемой как время до смерти (или конец испытания для выживших) в испытании с заражением. Испытание проводили с помощью отношения правдоподобия, сравнивающего линейную смешанную модель, включающую случайный эффект семейства (включая полигенные эффекты) и данный SNP с базовой моделью, игнорирующей эффект SNP:
H0: y=1μ+Zu+e
H1: y=1μ+Zu+Mg+e
где y представляет собой вектор фенотипов времени до смерти особей с известными генотипами для данного локуса SNP, μ представляет собой фиксированный эффект общего среднего значения, 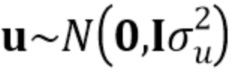 представляет собой вектор случайных эффектов семейств, Z представляет собой матрицу встречаемости, связывающую особей с семействами,
представляет собой вектор случайных эффектов семейств, Z представляет собой матрицу встречаемости, связывающую особей с семействами, 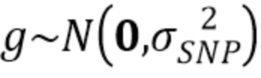 представляет собой эффект замещения аллелей конкретного SNP, M представляет собой матрицу генотипов (с генотипами, кодируемыми 0, 1 и 2 для первой гомозиготы, гетерозиготы и другой гомозиготы) и
представляет собой эффект замещения аллелей конкретного SNP, M представляет собой матрицу генотипов (с генотипами, кодируемыми 0, 1 и 2 для первой гомозиготы, гетерозиготы и другой гомозиготы) и 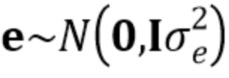 представляет собой вектор случайных остатков. Ассоциированные компоненты дисперсии и отношение правдоподобия для двух моделей оценивали с помощью программного обеспечения DMU (Madsen & Jensen, 2013), с использованием методологии ограниченной максимальной вероятности (REML). Вероятности REML для иерархических моделей сравнимы, только когда фиксированные части двух моделей являются идентичными, и, таким образом эффект замещения SNP был определен как случайный.
представляет собой вектор случайных остатков. Ассоциированные компоненты дисперсии и отношение правдоподобия для двух моделей оценивали с помощью программного обеспечения DMU (Madsen & Jensen, 2013), с использованием методологии ограниченной максимальной вероятности (REML). Вероятности REML для иерархических моделей сравнимы, только когда фиксированные части двух моделей являются идентичными, и, таким образом эффект замещения SNP был определен как случайный.
Тестирование отношения правдоподобия проводили следующим образом:
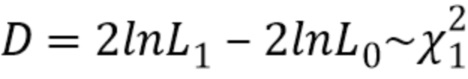
где 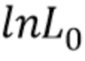 и
и 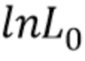 представляют собой логарифмические вероятности REML для моделей H0 и H1, соответственно. Тестирование отношения правдоподобия проводили локус за локусом с использованием методик параллельного вычисления.
представляют собой логарифмические вероятности REML для моделей H0 и H1, соответственно. Тестирование отношения правдоподобия проводили локус за локусом с использованием методик параллельного вычисления.
Для внесения поправки на множественность тестирования очень строгим образом, пороговое значение для утверждения о значимости в тестировании ассоциации между генотипами SNP и резистентностью к IPN делили на 50000 (приблизительное количество высококачественных полиморфных SNP), т.е. использовали поправку Бонферрони. Таким образом, пороговая величина для значения p 0,05 в эксперименте преобразовывалась в пороговую величину для значения p 10-6 для каждого индивидуального SNP. Иными словами, нуль-гипотеза (H0) утверждала, что в исследуемом материале не обнаружится QTL для резистентности к IPN, альтернативная гипотеза утверждала, что в исследуемом материале существует по меньшей мере один QTL для резистентности к IPN, вероятность выявления по меньшей мере одного QTL составляла только 0,05 (5%), если нуль-гипотеза была справедливой, и индивидуальный SNP требовал значения p ниже 10-6, чтобы соответствовать утверждаемой значимости в эксперименте.
Карты сцепления получали с использованием программного обеспечения Lep-MAP (Rastas et. al. 2013). Первоначально, SNP помещали в группы сцепления посредством двухточечного анализа с использованием модуля "SeparateChromosomes", определяющего пороговое значение LOD 110 (lodLimit=110), вместе с параметрами missingLimit=5, achiasmaticMeiosis=0, dataZTolerance=2, malePrior=0,1, femalePrior=0,1 dataTolerance=0,05 sizeLimit=20 (см. опции программы для полного описания параметров для этой и следующих стадий). Затем несцепленные SNP добавляли в каждую группу с использованием модуля "JoinSingles", определяющего пороговое значение LOD 30 (lodLimit=30) и требующего минимального различия LOD 10 между областями предполагаемых групп сцепления (lodDifference=10), вместе с параметрами achiasmaticMeiosis=0, dataZTolerance=2, malePrior=0,1, femalePrior=0,1, dataTolerance=0,05. Упорядочивание SNP в каждой группе первоначально проводили с использованием модуля "OrderMarkers2" (четыре повторения), с параметрами missingLimit=5, achiasmaticMeiosis=0, nonNearIdenticalLimit=2 0,01, missingClusteringLimit=0,01, hammingClusteringLimit=0,001, filterIdenticalSubset=25 2, dataZTolerance=2, initError=0,005, initRecombination=0,0001 0,001, alpha=1, MAFLimit=0,05, informativeFamilyLimit=3. После первоначального упорядочивания удаляли маркеры с частотами ошибок, превышающими 0,01. Конечную оценку этого скорректированного порядка SNP проводили с использованием "OrderMarkers2" (четыре повторения) и указания "improveOrder=1" в дополнение к тем же параметрам, которые использовались для первоначального упорядочивания. Номера хромосом соотносили с полученными группами сцепления в соответствии с Phillips et al. (2006). Получали карты сцепления самцов и самок, исходя из событий рекомбинации, наблюдаемых для самцов и самок, соответственно.
Последовательности SNP, т.е. последовательности ДНК размером 71 п.н., центрированные по SNP, выравнивали против эталонной последовательности генома радужной форели (Berthelot et al. 2014; справочный id последовательности в GenBank: CCAF010000000). Для этого использовали BLAST+ (Altschul et al. 1990, Camacho et al. 2008) с параметрами ожидание=0,1, оценка соответствия=1, оценка несоответствия=-2, штраф за внесение пропуска=0, штраф за продолжение пропуска=0. Для каждой последовательности из 71 п.н. использовали две входных последовательности, по одной для каждого варианта (аллеля) SNP. Подпоследовательность CCAF010000000, имеющая наиболее высокий показатель BLAST, принималась как подпоследовательность, содержащая SNP при условии, что было не более двух несоответствий между подпоследовательностью и наиболее соответствующей из двух последовательностей размером 71 п.н., соответствующих каждому SNP.
Результаты
Среди 57501 SNP, исследованных в отношении ассоциации с резистентностью к IPN, пять SNP удовлетворили требованию наличия значения p менее 10-6, которое требовалось для утверждения от статистической значимости во всем эксперименте. Как можно видеть из фиг.1, все из этих пяти SNP расположены на одной и той же хромосоме, а именно, на хромосоме 1, в соответствии с номенклатурой Palti et al. (2011). Более того, как можно видеть на фиг.1, хромосома 1 содержала большую часть SNP, которые были индивидуально, но не во всем эксперименте, значимыми (здесь определяются как SNP, имеющие значения p менее 0,01). Как можно видеть из фиг.2, SNP на хромосоме 1, наиболее высоко ассоциированные с резистентностью к IPN, были локализованы в подобласти хромосомы, с наиболее значимыми SNP в центре. Кластеризация значимых SNP в относительно узкой области хромосомы строго указывает на то, что SNP, значимо ассоциированные с IPN, являются маркерами одного и того же QTL. Восемьдесят два SNP были индивидуально или во всем эксперименте значимыми в тестировании ассоциации с резистентностью к IPN, причем все они также были расположены на хромосоме 1. Выравнивание последовательностей ДНК, относящихся к этим SNP, против последовательности генома радужной форели, доступной в GenBank (Bertheloet et al. 2014; справочный id последовательности в GenBank: CCAF010000000) показало, что SNP располагались в ограниченном количестве контигов или остовов генома (таблица 1).
Ожидается, что для любого значимого SNP, особи радужной форели, имеющие различные генотипы SNP, будут отличаться между собой с точки зрения резистентности к IPN. Например, для наиболее значимого SNP - SNP, имеющего идентификатор SNP Affymetrix AX-89929954 (SNP #1, таблица 1), ожидается, что группы форели, гомозиготные по аллелю, сообщающему относительную резистентность к IPN, будут иметь средние показатели выживаемости 45% в условиях, сходных с условиями испытания с заражением 1 (с учетом только особей, которые были прогенотипированы). Напротив, ожидается, что группы форели, гомозиготные по аллелю, не сообщающему относительную резистентность к IPN, будут иметь средние показатели выживаемости 17% в сходных условиях (с учетом только особей, которые были прогенотипированы), в то время как ожидается, что группы особей, гетерозиготных по этому SNP, в сходных условиях будут иметь средние показатели выживаемости 36% (с учетом только особей, которые были прогенотипированы) (см. таблицу 3). Таким образом, SNP AX-89929954 можно использовать в качестве инструмента для прогнозирования уровня резистентности к IPN любой особи. Здесь, уровень резистентности определяют как уровень относительной резистентности, что означает, что особь будет тем больше резитентной к IPN, чем больше копий аллеля резистентности к IPN особь имеет в положении AX-89929954. Точнее, ожидается, что особь, имеющая одну копию аллеля резистентности к IPN (который представляет собой цитозин) будет более резистентной к IPN, чем особь, не имеющая аллеля резистентности к IPN в положении AX-89929954, учитывая, что другие детерминанты резистентности особи к IPN являются сходными у двух особей. Аналогично, ожидается, что особь, имеющая две копии аллеля резистентности к IPN в положении AX-89929954, будет более резистентной к IPN, чем особь, которая имеет одну копию аллеля резистентности к IPN в положении AX-89929954, с учетом, что другие детерминанты резистентности особи к IPN являются сходными у двух особей. Таким образом, генотипы в положении AX-89929954 можно использовать для прогнозирования резистентности к IPN изолированной радужной форели и популяции радужной форели. Также, поскольку более вероятно, что особь передаст (ее потомству) копию аллеля резистентности к IPN в положении AX-89929954, чем больше копий аллеля резистентности к IPN она имеет, генотипы в положении AX-89929954 также можно использовать для прогнозирования уровня резистентности к IPN у потомства особи. Путем отбора животных, которые содержат одну или две копии аллеля резистентности к IPN в положении AX-89929954, в качестве родителей, можно проводить селекцию более высокой степени резистентности к IPN в следующем поколении.
Другие SNP, которые представляют собой индивидуально или во всем эксперименте значимые SNP, представленные в таблице 1, обладают общей с AX-89929954 способностью прогнозировать уровни резистентности к IPN, как можно видеть из таблицы 1 и таблицы 3. Более того, эти SNP можно использовать в комбинации, например, в комбинациях по два SNP, для получения еще более мощных прогнозирующих инструментов.
Таблица 3: Показатели выживаемости в группах рыб среди прогенотипированных рыб испытания с заражением 2. Каждая группа состоит из всех прогенотипированных рыб, имеющих рассматриваемый генотип в рассматриваемом SNP. R=аллель резистентности к IPN; A=аллель не резистентности к IPN; AA. AR и RR=три возможных генотипа в любом конкретном SNP; NA=не применимо (поскольку особи не имели рассматриваемый генотип в рассматриваемом SNP). Показатели выживаемости представляют собой средние показатели выживаемости (±стандартная ошибка) в группе животных, имеющей рассматриваемый генотип в рассматриваемом SNP.
Пример 2: Получение радужной форели с повышенной резистентностью к IPN
Проводят взятие образца ткани от каждого потенциального родителя, т.е. от каждой радужной форели, которая является родителем-кандидатом для создания указанной форели с повышенной резистентностью к IPN. Взятие образцов тканей можно проводить с использованием любого из нескольких доступных способов неинвазивного взятия образцов из живой форели. Например, образец может представлять собой фрагмент жирового плавника форели, вырезанный с использованием ножниц или скальпеля, или он может представлять собой биоптат мышечной ткани, полученный с использованием дерматома, такого как 3,0-мм дерматом с поршнем (BPP-30F) от Brymill (Basingstoke, Великобритания). Образец также может представлять собой несколько чешуек, полученных с использованием пинцета. После взятия образцов, образцы тканей следует сразу заморозить и хранить в замороженном состоянии до экстракции ДНК, или альтернативно их можно помещать в этанол для длительного хранения в морозильной камере. Образцы чешуек перед хранением можно сушить на фрагменте бумаги. В момент взятия образцов потенциальных родителей можно физически маркировать с использованием, например, пассивных встраиваемых передатчиков (PIT). Физическое маркирование может облегчать последующее извлечение особой, отобранных с использованием способа.
ДНК экстрагируют из образца ткани с использованием любого из нескольких доступных способов экстракции высококачественной ДНК из образцов форели. Например, ДНК можно экстрагировать с использованием набора DNAeasy от QIAGEN (Venlo, Нидерланды), в соответствии с протоколом, предоставляемым с этим набором.
Экстрагированную ДНК генотипируют в отношении по меньшей мере одного из однонуклеотидных полиморфизмов (SNP), указанных в таблице 1. Например, экстрагированную ДНК можно генотипировать с использованием SNP AX-89929954 (SNP #1). Генотипирование можно проводить с использованием любого общеизвестного способа генотипирования SNP. Например, генотипирование можно проводить с использованием протокола iPlex® на системе MassARRAY® от Sequenom (San Diego, США). Для генотипирования SNP AX-89929954 с использованием протокола iPlex можно использовать следующие праймеры:
Прямой праймер для ПЦР: ACGTTGGATGTCCACAGTCCACATGCTTTG (SEQ ID NO: 157),
Обратный праймер для ПЦР: ACGTTGGATGGGAAAGAAACAGTGATAGGC (SEQ ID NO: 158),
Праймер для удлинения: CACACAACTGTGTGTCAAAT (SEQ ID NO: 159).
Все другие экспериментальные параметры соответствуют протоколу iPlex. Протокол iPlex можно применять в мультиплексном формате для нескольких SNP, и в этом случае экспериментальные параметры, в том числе последовательности праймеров, можно корректировать в соответствии со свойствами других SNP в мультиплексе. Эту коррекцию проводят с использованием программного обеспечения Assay Design Suite от Sequenom (https://ww.mysequenom.com/Tools).
Исходные данные генотипирования iPlex обрабатывают с использованием программного обеспечения Typer от Sequenom. Генотипированные образцы распределяют на три отдельных и строго определенных кластера в соответствии с тремя генотипами при условии, что все три генотипа присутствуют в прогенотипированных образцах.
С использованием стадий, указанных выше, можно обнаружить, что часть прогенотипированной форели имеет две копии аллеля цитозина (C), в то время другая часть может иметь две копии аллеля аденина (A). Другая часть может иметь одну копию каждого аллеля (AC). В качестве родителей будут выбирать родителей, имеющих две копии C (т.е. имеющие генотип CC). Все потомство этих родителей будет гомозиготным по аллелю C в SNP AX-89929954, что означает, что оно будет гомозиготным (CC) по аллелю, ассоциированному с повышенной резистентностью к IPN. В условиях, сходных с условиями, использованными в эксперименте для испытания с заражением 2, описанного в примере 1 выше, ожидается, что такие (CC) животные будут иметь показатель выживаемости 45%, в то время как животные, происходящие от случайным образом отобранных родителей, будут иметь ожидаемый показатель выживаемости 26%.
Если не будет обнаружено особей, имеющих генотип CC, в качестве родителей можно выбирать особей с генотипом AC. Также ожидается, что, если бы родители-кандидаты (т.е. прогенотипированные животные) представляли собой случайную подгруппу популяции, из которой они происходят, также ожидается, что использование этих животных AC в качестве родителей приведет к потомству с повышенной резистентностью к IPN.
Способ можно применять с использованием любого из SNP, приведенных в таблице 1. Способ также можно применять с использованием комбинации двух или более SNP. Например, можно прогенотипировать SNP AX-89929954 и AX-89918280 (SNP #2), и использовать в качестве родителей особей, имеющих генотип CC в положении AX-89929954 и генотип GG в положении AX-89918280.
После идентификации родителей с использованием этого способа, этих родителей извлекают путем отсортировывания их из емкости, в которую они помещены (обычно проводимого путем перемещения каждой рыбы в другую емкость). Получают потомство и оплодотворенные яйца выводят с использованием стандартных способов культивирования водных организмов.
Пример 3: Эксперименты для подтверждения результатов, лежащих в основе изобретения
Проводили два дополнительных испытания с заражением (испытания с заражением 3 и 4) для подтверждения ассоциации между резистентностью к IPN и аллелями ДНК-полиморфизмов по изобретению. Испытания проводили в двух емкостях объемом 100 литров, и в каждой емкости группу особей радужной форели исследовали в отношении резистентности к одному из двух штаммов вируса IPN. Эти два штамма представляли собой 1) штамм (AGT11-2) серотипа Sp, выделенный из выращиваемой в Норвежском море радужной форели; этот же штамм использовали в примере 1, и 2) штамм серотипа Wb, выделенный при вспышке IPN у радужной форели в Чили. Каждая емкость содержала приблизительно 12 мальков радужной форели из каждой из 133 полностью сибсовых групп. В обоих испытаниях использовали одинаковый набор полностью сибсовых групп. Испытание проводили через 1 неделю после первого кормления (т.е. после перехода на твердый корм). Рыб акклиматизировали и первоначально кормили в области испытания. В начале испытаний объем воды уменьшали до ½ исходного объема, и в этом время в каждую емкость добавляли 100 мл соответствующего изолята вируса для получения конечной концентрации, равной TCID50 105 вирусных частиц на мл воды. Через три часа после добавления вируса объем воды возвращали до уровня до заражения (в ходе этих трех часов поддерживали аэрацию воды). Взятие образцов умерших животных проводили и смертность регистрировали два раза в сутки на протяжении периода испытания. ДНК экстрагировали из исследуемых рыб, от которых были получены образцы, с использованием стандартного протокола. Оба испытания завершали через 28 суток после начала испытания. В этот момент суточные показатели смертности составляли 0,9% (Sp) и 0,19% (Wb), и они снижались. Напротив, на пике кривой смертности суточная смертность составляла 10,1% (Sp) и 1,56% (Wb). Иными словами, в момент завершения испытания кривая выживаемости выравнивалась, и можно обоснованно предположить, что большинство рыб, которые пережили период испытания, выжили бы также, если период испытания был более длительным. Совокупная смертность составляла 70,0 (Sp) и 9,38% (Wb). У всех животных из испытания Sp (1603 животных) и всех умерших животных из испытания Wb (174 животных) проводили взятие образцов и генотипирование в отношении четырех ДНК-полиморфизмов по изобретению. Генотипирование проводили с использованием системы генотипирования iPLEX от Agena Bioscience (San Diego, США) (владельцем системы iPLEX ранее была Sequenom, San Diego, США). Праймеры для ПЦР и удлинения для генотипирования iPLEX конструировали с использованием Assay Design Suite v2.0 (доступного на www.mysequenom.com/Tools), с использованием параметров по умолчанию и все четыре ДНК-полиморфизма генотипировали в одной и той же мультиплексной реакции. Как можно видеть из таблицы 4, частоты аллелей, обозначаемых как аллели резистентности к IPN, были значимо более высокими у выживших особей из испытания Sp, чем у умерших особей из испытания Sp, для всех четырех полиморфизмов. Аналогично, частоты аллелей, обозначаемых как аллели резистентности к IPN, были значимо более высокими у выживших особей из испытания Wb, чем у умерших особей из испытания Wb. Здесь, для каждого полиморфизма статистическую значимость исследовали с использованием логистической регрессии ряда аллелей резистентности к IPN на бинарной основе выживший/не выживший. Таблица 4 содержит значения p для этого испытания для всех четырех полиморфизмов. Для испытания Wb, в котором регистрировали только смертность, генотипы для 1416 выживших особей оценивали посредством допущения, что общие аллельные частоты в испытании Wb были такими же, как и в испытании Sp (обоснованное предположение с учетом того, что эти два испытания по заражению включали животных из одних и тех же семей в одинаковых соотношениях), и далее посредством допущения, что каждый полиморфизм находится в равновесии Харди-Вайнберга.
Подтверждающий эксперимент подтвердил, что существует статистически значимая ассоциация между резистентностью к IPN и аллелями во всех четырех исследованных полиморфизмах. Более того, ассоциация также была достоверной, когда штамм вируса IPN, использованный при первоначальном открытии изобретения (штамм серотипа Sp) заменяли другим штаммом (серотипа Wb, West Buxon). Следовательно, ассоциация между ДНК-полиморфизмами и резистентностью к IPN является воспроизводимой и независимой от штамма вируса.
Естественным и необходимым следствием этих результатов является то, что ДНК-полиморфизмы по изобретению можно использовать для получения радужной форели с повышенной резистентностью к IPN. Например, можно использовать ДНК-полиморфизм AX-89929954 для скрининга количества особей радужной форели. После идентификации одного самца и одной самки, оба из которых являются гомозиготными по аллелю резистентности к IPN (т.е. оба они имеют генотип CC), этих двух животных можно скрещивать и все потомство, полученное в результате скрещивания, будет иметь генотип CC согласно правилам Менделя. Можно ожидать, что эти особи будут более резистентными к IPN, чем случайные (но в остальном сравнимые) особи, полученные из той же популяции радужной форели при условии, что аллель смертности (A в случае AX-89929954) также существует в популяции.
Таблица 4: Результаты эксперимента, подтверждающего ассоциацию между резистентностью к IPN и полиморфизмами по изобретению. Для каждого из четырех полиморфизмов таблица содержит: 1) обозначение аллелей резистентности и смертности (как определено в таблице 1 и в таблице 2), 2) количество животных, имеющих любой из трех возможных генотипов в подгруппах выживших животных после заражения Sp (SP_SURV), смертность So (SP_MORT) и смертность Wb (WB_MORT), 3) значения p после регрессии ряда аллелей резистентности к IPN по бинарному признаку выживший/не выживший.
*Количество WB_SURV оценивали, как описано выше.
Пример 4: Идентификация и исследование дополнительных SNP, ассоциированных с резистентностью к IPN
Двенадцать особей радужной форели из популяции радужной форели AquaGen подвергали полногеномному секвенированию с использованием HiSeq2000 от Illumina (San Diego, США); см. Palti et al. (2015). Считанные последовательности этих 12 животных выравнивали с общедоступной эталонной геномной последовательностью радужной форели (Berthelot et al. 2014), с использованием bowtie2 (Langmead and Salzberg, 2012). Перед выравниванием считанных посредством Illumina последовательностей, подпоследовательности (каркасы и контиги) эталонной последовательности объединяли и упорядочивали посредством совместного выравнивания подпоследовательностей с хромосомными последовательностями атлантического лосося (предоставленных GenBank); эти два вида являются близкородственными и демонстрируют высокую степень синтении. Из выровненных считанных последовательностей идентифицировали SNP с использованием Freebayes (Garrion and Marth, 2012). Набор (предполагаемых) SNP фильтровали в Freebayes с использованием следующей цепи параметров: "--no-indels --no-mnps --no-complex --min-mapping-quality 30 --read-mismatch-limit 2 --read-indel-limit 1". Для каждого отфильтрованного SNP, генотипы у 12 отсеквенированных животных устанавливали с использованием Freebayes. Генотипы сравнивали с генотипами одного из оригинальных SNP по изобретению (AX-89929954), вычисляя для каждого отфильтрованного SNP квадрат коэффициента корреляции между этим SNP и AX-89929954. Квадрат коэффициента корреляции (r2) между двумя ДНК-полиморфизмами является показателем величины неравновесия по сцепления между ДНК-полиморфизмами; чем более высоким является r2, тем более коррелирующими являются генотипы в этих двух ДНК-полиморфизмах. Следует отметить, что высокие уровни r2 в основном наблюдались для ДНК-полиморфизмов, которые находились на расстоянии не более чем на 3 миллиона пар оснований (3 м.п.н.) от AX-89929954, большинство SNP, которые находились на расстоянии не более чем 3 м.п.н. от AX-89929954, удаляли, также как и все SNP, имеющие значение r2 ниже 0,2. Более того, SNP, имеющим значения r2 выше 0,5, отдавали приоритет, также как и SNP, находящимся на расстоянии не более 500 п.н. от генной области (генную область определяли как область, имеющую совпадение в BLASTN, когда BLASTN запускали против наиболее поздней версии базы данных RefSeq-RNA со значением параметров BLASTN по умолчанию). В завершение, была отобрана подгруппа из 500 SNP и было проведено их генотипирование с использованием химической реакции KASP, осуществленной с использованием системы SNPline от LGC Genomics (http://www.lgcgroup.com/products/genotyping-instruments/snpline/#.VkNMKLcvdhE). Генотипирование проводили на том же генетическом материале, как описано в примере 1 (1723 животных из испытания по заражению из испытания по заражению IPN), и ассоциацию между генотипами и резистентностью к IPN исследовали аналогично тому, как описано в примере 1. Индивидуальные SNP, демонстрирующие статистику распределения хи-квадрат более 13,0, были определены как высокоассоциированные с IPN, их можно использовать в качестве инструментов для селекции животных, резистентных к IPN. На фиг.3, нанесен обратный логарифм значений p (H0: генотипы не ассоциированы с резистентностью к IPN, H1: генотипы ассоциированы с резистентностью к IPN) против положений на "физически упорядоченном" эталонном геноме радужной форели для всех ДНК-полиморфизмов, протестированных либо в качестве части эксперимента, описанного в примере 1, либо в качестве части подтверждающего исследования, описанного в настоящем описании. На фиг. иллюстрируется, что все полиморфизмы, наиболее высокоассоциированные с резистентностью к IPN, расположены в узкой области, что означает, что наиболее вероятное положение каузативных ДНК-полиморфизмов, лежащих в основе QTL, относительно хорошо определено, и что любые другие ДНК-полиморфизмы, расположенные в области QTL ("область пика" на графике), если они ассоциированы с IPN, вероятно являются маркерами одной и той же подразумеваемой каузативной мутации.
Определенные ссылки, цитированные в заявке
Altschul SF, Gish W, Miller W, Myers EW, and Lipman DJ (1990) Basic local alignment search tool. J. Mol. Biol. 215:403-410.
Berthelot C, Brunet F, Chalopin D, Juanchich A, Bernard M et al. (2014) The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertabrates. Nature Communiciations 5: 3657.
Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J et al. (2008) BLAST+: architecture and applications. BMC Bioinformatics 10:421.
Garrison E, Marth G. Haplotype-based variant detection from short-read sequencing (2012) arXiv preprint arXiv:1207.3907 [q-bio.GN].
Langmead B, Salzberg SL (2012) Fast gapped-read alignment with Bowtie 2. Nature Methods 9:357-359.
Madsen P, Su G, Labouriau R, and Christensen OF (2010) DMU - A package for analysing multivariate mixed models. Proceedings from the 9th World Congress on Genetics Applied to Livestock Production (WCGALP); http://www.kongressband.de/wcgalp2010/assets/pdf/0732.pdf.
Palti Y, Genet C, Luo MC, Charlet A, Gao G et al. (2011) A first generation integrated map of the rainbow trout genome. BMC Genomics 12:180.
Palti Y, Gao G, Liu S, Kent MP, Lien S, Miller MR, Rexroad CE III, Moen T (2015) The development and characterization of a 57K single nucleotide polymorphism array for rainbow trout. Molecular Ecology Resources 15: 662-672.
Phillips RB, Nichols KM, Dekoning JH, Morasch MR, Keatley KA et al. (2006) Assignment of rainbow trout linkage groups to specific chromosomes. Genetics 174: 1661-1670.
Rastas P, Paulin L, Hanski I, Lehtonen R, and Auvinen P (2013) Lep-MAP: fast and accurate linkage map construction for large SNP datasets. Bioinformatics 29: 3128-34.
Wetten M, Kjøglum S, Fjalestad KT, Skjærvik O, Storset A. (2011) Genetic variation in resistance to infectious pancreatic necrosis in rainbow trout (Onchorhynchus mykiss) after a challenge test. Aquaculture Research 1-7.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> AquaGen AS
<120> СПОСОБ ПРОГНОЗИРОВАНИЯ РЕЗИСТЕНТНОСТИ
<130> P61402282PCT00
<150> 20141382
<151> 2014-11-18
<160> 299
<170> PatentIn version 3.5
<210> 1
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или A
<400> 1
gaaagaaaca gtgataggct tttagtgagc acatanattt gacacacagt tgtgtgaaaa 60
caaagcatgtg 71
<210> 2
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 2
aatatatgcc ttatatcagg atcgctaacc acagancagg attacaattt aatacttgca 60
caatatacata 71
<210> 3
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 3
tccttgtatc gcagaacttt taaatgtttg aatccntctt gatgttatgt gattggtgga 60
ttcaaataagt 71
<210> 4
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 4
gatgcagggt tgcacagaac gttgatgcca gtagtnatgg catggctctc agtacaaact 60
catactgagtg 71
<210> 5
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 5
gaatggcaat taatttcatg ctgaactaac tgaatnaaga aaggaaatga ccccaaccct 60
ggttgcatac t 71
<210> 6
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 6
ctcacattct tcaccttatt ggaatgcatg gaaagncgcc atgggaagct cactgcggtt 60
tcgaacctac g 71
<210> 7
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 7
agtcaaaacc atgaaaaagc tgattttaga atgacntttg taacactctc catgatgacg 60
gttaatagaa g 71
<210> 8
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 8
cgtgtcaata ttggaacgac taaatacgtg aatctntcag gacgggtgaa ctgagcacaa 60
atctagatca t 71
<210> 9
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 9
agtccctccc ttagtggtat caaaccataa ctaatnattt cttcacaaat tatggaacaa 60
aaataaatcc c 71
<210> 10
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 10
aaacggagtg ccgaagactc tgaactcaca gactcnctgc cgaaaaaaac gaaagtaatg 60
tcctcaactc t 71
<210> 11
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 11
tgtaaattca taagtaaaga gaacacctgt ttaagnagag cacattatgc aaaacctcat 60
atggaaaacg t 71
<210> 12
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 12
gcgtggacac atgagggacg ctgtgctccc tgtgtnctcc cagcaacacg aggtaattct 60
gcagaacaac c 71
<210> 13
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 13
aaaggaagaa gaatggtcag gagaggtaag gttggnagga attatgcttt tcaatgatct 60
ggtcctgcaa g 71
<210> 14
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 14
gcaataataa ccattgaaaa atatgctttg ggaatntctc cattctttcc ctagtccaat 60
atgtgttctt t 71
<210> 15
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 15
aggggcggtt agacacatgg gtgtggctag aaatgngggt tggtgacacc cactccttgg 60
cactcgatga t 71
<210> 16
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 16
cagccagctt tcgagtagca gggagaggac agtaantatt gacacagtgt aagcactagg 60
cagcactagg c 71
<210> 17
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 17
caatacaatg aggtgtaaat ggttgaattc actgtnggat aaagactgca ggacaggcca 60
gtaaaacatt t 71
<210> 18
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 18
gtcctctatg cctcctatga gttcttcgag gccatntgca gcgtgagtag ctgcctggac 60
cccatgctgt a 71
<210> 19
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 19
attacttttg aatcacagct tcagcatata gccctngcta tagatacaat tcatacatca 60
agataatgac t 71
<210> 20
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 20
tatagtagat aattgattca aatggcagtt gtattncact tttgtttttc tttacagtgg 60
tcagtgctat t 71
<210> 21
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 21
cacacaaggt agatacacct gcagagcatg tttcgnaaat taataaggta agtctgaata 60
ccaaatactg a 71
<210> 22
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 22
ctgttgttgg ccagattacc atcagtgcag ttgganttca ggccttatct ctgcctcaca 60
caacatcatc t 71
<210> 23
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 23
atgggtcgtg ttcatcaggc agaaaaatga cgtatnatgc cctaatgaac atgaccctgg 60
cattacctag a 71
<210> 24
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 24
gaacccctag gctagatgtt caacctggcc tcaggncaat tctgaagatt tggtacgcaa 60
atatgttcgc c 71
<210> 25
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 25
ctgttcattc tgtctgtttc agttggtgct ctgganagga gaaaagccca cctgctgtga 60
gccccttatt g 71
<210> 26
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 26
tcagcgtcct acagctaaac catacgatga aattanaaca ataaattcag tgtgatatcc 60
gttatggacc a 71
<210> 27
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 27
aggtggcagg aaaaagaata cctccagcca atcgcntgac atctgtccat tcaagctgca 60
gcgaatctga c 71
<210> 28
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 28
cacgtctctc caaaacgttt ccacttactt tcccangaag cctttcccgt tgggctgctc 60
cttcagccac t 71
<210> 29
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 29
tccatagtgg ctaccagccc acatacgcac tgacanaatc acagacagac tgacagacag 60
cagcttgatc a 71
<210> 30
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 30
atttgagaat cagatgcaga agagcaaggt tttccnagcc tgtggctatc ctccatacga 60
ttcaaccacc t 71
<210> 31
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 31
taccgtacag ccctgctaaa ggaggaaaac aagggncatg atggtatgtc ttggggcttc 60
ctcagggccc a 71
<210> 32
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 32
aaacaactct tcaagatgat gagtaacaac caaagncaga aattcccctt aaaataactg 60
aaaggaaaaa g 71
<210> 33
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 33
gtgtttgtaa actggtaatt gaaattgtac tgatancaga tgatgtagaa ataaatgtgt 60
tttgatgtag g 71
<210> 34
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 34
tacagaggag ctatgggctt catcctcatg tacganatct gcaatgaaga gtccttcaac 60
gctgtgcagg a 71
<210> 35
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 35
ggccccatta ttttggcttc ttgtgtagca gacttngtag tgtgtaagga agccttgctg 60
gtcttgcaca g 71
<210> 36
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 36
tctgctgagc tcccctgaaa gactgtgagt cacaanggtc atttatttac cttctctgct 60
tcactcaaca c 71
<210> 37
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 37
actattcctc acatgctaca gaatagctag ggtaanagga tagtaacatt aaccataaca 60
ccaaagctaa t 71
<210> 38
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 38
tccagtccca ctagtttggc tttgaagtcg cggatngtag actcgctctt gtatctcttc 60
tcagtcaggt c 71
<210> 39
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 39
gtaaaggcta gcagaccctg ggaacattcc cctgcnctca gcctctctgc catggaggaa 60
atgctaaaag t 71
<210> 40
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 40
ttttgaacag cacttatctc ttctctccag aggggnatat cacagagcat gaccaaaaag 60
ttagccagct a 71
<210> 41
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 41
aagttgacct cttatgattt tattattggt ttgtgntgca agatgttctg tccaggtttc 60
aacttatagc c 71
<210> 42
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 42
accaccacac ctgcctgagt catgtaagaa gattangcat ggtggatgga ggtgggaaga 60
caattaatgg t 71
<210> 43
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 43
tggtcgtctg agccctatgt agtgaattca aacttncttg tctaagccaa gtatcaacct 60
gcaaacccaa g 71
<210> 44
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 44
tccccttctg tgtgctcaag gtgtgaatat tttatngtta acttacttca ctcgtgtcct 60
gcagttagat g 71
<210> 45
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 45
agcaggcagg ttgagacaag cctgcagggc caatanctgt cactatcata actcaagcca 60
acaataccca a 71
<210> 46
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или A
<400> 46
cttgcttgcc atcacccgtc tggtccaagg gactanggtc aatataacct ccaatcttag 60
taacctacct c 71
<210> 47
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 47
gcagacaccc tgggcagcgt tggagtgatc atctcngcca tcctgatgca gaagtatgac 60
ctgatgatcg c 71
<210> 48
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 48
aactgggcta aaacgatggg acggtgtgcg aaaacnaact aaccctaacc agaaaattgt 60
atgctttgtt t 71
<210> 49
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 49
accaccttca cattaacctt ctccatgaca aaacancccc aagcctgaac agcccctagc 60
cccttccact a 71
<210> 50
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 50
gaagacacaa actcaacaag agcacaacaa cacagnctta aggtactgca attcctgctt 60
attttcataa a 71
<210> 51
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 51
aaatgaaaag cgagaaagga cggaggtatt ttaaanatat ttaccatagt actcaccgaa 60
ggctgcagcc a 71
<210> 52
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 52
gaaattgccc cttgattttg tcagtttagc gatcantata cacaaaataa ttaactaaag 60
gaacaaccat a 71
<210> 53
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 53
aaaccacatg gtcttcctgc aactttgtgc caaatnagta gtttcacaat gaacgttgtg 60
aggtctgcag c 71
<210> 54
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или A
<400> 54
agacacacag cagactagac tgaggatgtg aaccantcct ccacttaatg caaatgcagg 60
gacacattca g 71
<210> 55
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 55
ctattcctgc ttaccgtagt tgaactggct gttggntttc tcacagttga tgatgttgaa 60
gcgatagggc a 71
<210> 56
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 56
ggtgtaagta cagactcttt gaaagcatgc aaatanaagt aaagacactg tcattccttt 60
aaatgttctt g 71
<210> 57
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или A
<400> 57
cttctttatt tgctatgatt attacttaat agtgcngatt gtatttgtca tccgtattga 60
ctgcagaact a 71
<210> 58
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 58
attgttcaag gacattatgc ttgtcctaca tattgncaat ttgatgtcgt tctttaacat 60
ttataattga t 71
<210> 59
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 59
aaaacttctt aagggacaag aaggaagttg aagttngggg tgggctagga agataaagag 60
ttgggggtgt g 71
<210> 60
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 60
accaacacag agatgagacg tgccgagcgc aaggcnacca agaagaagct cccgctgaaa 60
cgagagatgg a 71
<210> 61
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или A
<400> 61
ttaatctaac tcactctcca taacatcaca gaagtngatg tattcgatta taacaagctc 60
agggctgtca t 71
<210> 62
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 62
ccctttacct agaatggtct gcagcgtgat gtcaangtgg ttattttgtc cattgttgcc 60
agtgataagc c 71
<210> 63
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 63
tgcagaatgg acaactgaag agagatatgt cgcacntgag ggaaacaact ccgtgtctag 60
gccttctgaa g 71
<210> 64
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 64
gttagtgaaa gccatttcag ggtaaaccct ccaggncgtc caatgtacca tagaagcaaa 60
acaatgataa t 71
<210> 65
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 65
cccatctgtc agaaccttgc ccacagctgt ttcccnactc aatgaaaaca agctaacatc 60
ctgcaggttg a 71
<210> 66
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 66
ggaatattcg aacggcttgt tgtccaatga gtcggnggcc ttaccaccac aaaccccaag 60
gcctgaggca g 71
<210> 67
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 67
ttaagagagt cacaaacatg aaaaactgtg atagtncaaa gaagatgaac gataggcttg 60
tggatagatt a 71
<210> 68
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 68
tttatttcag catttagccc aatcctgcta agaacngtca gttaatcact aattaggaga 60
atatcaataa a 71
<210> 69
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 69
ctcgaagtaa gaaatgaagc tgcaggtctg caggcngagt gctgtcagtg gaatataata 60
cccttaatag a 71
<210> 70
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 70
gataaggatg caacagattt attttagttt tagatnatgc tttcagactg atttcggctc 60
ttaaaaagat a 71
<210> 71
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 71
tctctgttca atatttagaa taaaaagctg acaaangtca cgtaatggac tggaaacagc 60
agacacatgg c 71
<210> 72
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 72
ctataggtgg atgatatgat atggttgcag ctaganagtg acagctgcct accttgtaag 60
taccacctcg a 71
<210> 73
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 73
gcgtttccag taaaacgacg tcccccttcg ccctanattt aatgagcacg tagtctagat 60
ttttgtttaa c 71
<210> 74
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 74
gcaggttttt gcagaaatca gttgctaata aagttnttct gtaaccattg tataagcagg 60
gtcaccatga c 71
<210> 75
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 75
tttctcttaa tgcatcatcc ttgtgcgaaa tcatgntaag tacacaccgt taaagttagg 60
tgctttgtta c 71
<210> 76
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 76
aaactaatga aaaacacaag agtgcctgca gtaacnctgt actaacgctg tactaacagt 60
acactctcag g 71
<210> 77
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 77
ctgcagcaga tggaactata tctctagtgg ctgtgngtgg aggaggagat gtggtgaaga 60
ctgagcagac a 71
<210> 78
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 78
cagaaaggaa aaatgtgtca aagttctaga tagtgngtgg aaagactcaa acaatgcagt 60
ttggaatgaa g 71
<210> 79
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 79
gaaagaaaca gtgataggct tttagtgagc acatacattt gacacacagt tgtgtgaaaa 60
caaagcatgt g 71
<210> 80
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 80
aatatatgcc ttatatcagg atcgctaacc acagagcagg attacaattt aatacttgca 60
caatatacat a 71
<210> 81
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 81
tccttgtatc gcagaacttt taaatgtttg aatccttctt gatgttatgt gattggtgga 60
ttcaaataag t 71
<210> 82
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 82
gatgcagggt tgcacagaac gttgatgcca gtagttatgg catggctctc agtacaaact 60
catactgagt g 71
<210> 83
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 83
gaatggcaat taatttcatg ctgaactaac tgaatgaaga aaggaaatga ccccaaccct 60
ggttgcatac t 71
<210> 84
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 84
ctcacattct tcaccttatt ggaatgcatg gaaaggcgcc atgggaagct cactgcggtt 60
tcgaacctac g 71
<210> 85
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 85
agtcaaaacc atgaaaaagc tgattttaga atgacgtttg taacactctc catgatgacg 60
gttaatagaa g 71
<210> 86
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 86
cgtgtcaata ttggaacgac taaatacgtg aatctatcag gacgggtgaa ctgagcacaa 60
atctagatca t 71
<210> 87
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 87
agtccctccc ttagtggtat caaaccataa ctaataattt cttcacaaat tatggaacaa 60
aaataaatcc c 71
<210> 88
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 88
aaacggagtg ccgaagactc tgaactcaca gactctctgc cgaaaaaaac gaaagtaatg 60
tcctcaactc t 71
<210> 89
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 89
tgtaaattca taagtaaaga gaacacctgt ttaagaagag cacattatgc aaaacctcat 60
atggaaaacg t 71
<210> 90
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 90
gcgtggacac atgagggacg ctgtgctccc tgtgttctcc cagcaacacg aggtaattct 60
gcagaacaac c 71
<210> 91
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 91
aaaggaagaa gaatggtcag gagaggtaag gttggaagga attatgcttt tcaatgatct 60
ggtcctgcaa g 71
<210> 92
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 92
gcaataataa ccattgaaaa atatgctttg ggaatatctc cattctttcc ctagtccaat 60
atgtgttctt t 71
<210> 93
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 93
aggggcggtt agacacatgg gtgtggctag aaatgagggt tggtgacacc cactccttgg 60
cactcgatga t 71
<210> 94
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 94
cagccagctt tcgagtagca gggagaggac agtaagtatt gacacagtgt aagcactagg 60
cagcactagg c 71
<210> 95
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 95
caatacaatg aggtgtaaat ggttgaattc actgttggat aaagactgca ggacaggcca 60
gtaaaacatt t 71
<210> 96
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 96
gtcctctatg cctcctatga gttcttcgag gccatttgca gcgtgagtag ctgcctggac 60
cccatgctgt a 71
<210> 97
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 97
attacttttg aatcacagct tcagcatata gcccttgcta tagatacaat tcatacatca 60
agataatgac t 71
<210> 98
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 98
tatagtagat aattgattca aatggcagtt gtattacact tttgtttttc tttacagtgg 60
tcagtgctat t 71
<210> 99
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 99
cacacaaggt agatacacct gcagagcatg tttcgaaaat taataaggta agtctgaata 60
ccaaatactg a 71
<210> 100
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 100
ctgttgttgg ccagattacc atcagtgcag ttggagttca ggccttatct ctgcctcaca 60
caacatcatc t 71
<210> 101
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 101
atgggtcgtg ttcatcaggc agaaaaatga cgtataatgc cctaatgaac atgaccctgg 60
cattacctag a 71
<210> 102
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 102
gaacccctag gctagatgtt caacctggcc tcaggtcaat tctgaagatt tggtacgcaa 60
atatgttcgc c 71
<210> 103
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 103
ctgttcattc tgtctgtttc agttggtgct ctggatagga gaaaagccca cctgctgtga 60
gccccttatt g 71
<210> 104
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 104
tcagcgtcct acagctaaac catacgatga aattaaaaca ataaattcag tgtgatatcc 60
gttatggacc a 71
<210> 105
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 105
aggtggcagg aaaaagaata cctccagcca atcgcgtgac atctgtccat tcaagctgca 60
gcgaatctga c 71
<210> 106
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 106
cacgtctctc caaaacgttt ccacttactt tcccaagaag cctttcccgt tgggctgctc 60
cttcagccac t 71
<210> 107
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 107
tccatagtgg ctaccagccc acatacgcac tgacataatc acagacagac tgacagacag 60
cagcttgatc a 71
<210> 108
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 108
atttgagaat cagatgcaga agagcaaggt tttccaagcc tgtggctatc ctccatacga 60
ttcaaccacc t 71
<210> 109
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 109
taccgtacag ccctgctaaa ggaggaaaac aaggggcatg atggtatgtc ttggggcttc 60
ctcagggccc a 71
<210> 110
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 110
aaacaactct tcaagatgat gagtaacaac caaagtcaga aattcccctt aaaataactg 60
aaaggaaaaa g 71
<210> 111
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 111
gtgtttgtaa actggtaatt gaaattgtac tgatatcaga tgatgtagaa ataaatgtgt 60
tttgatgtag g 71
<210> 112
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 112
tacagaggag ctatgggctt catcctcatg tacgacatct gcaatgaaga gtccttcaac 60
gctgtgcagg a 71
<210> 113
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 113
ggccccatta ttttggcttc ttgtgtagca gactttgtag tgtgtaagga agccttgctg 60
gtcttgcaca g 71
<210> 114
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 114
tctgctgagc tcccctgaaa gactgtgagt cacaatggtc atttatttac cttctctgct 60
tcactcaaca c 71
<210> 115
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 115
actattcctc acatgctaca gaatagctag ggtaagagga tagtaacatt aaccataaca 60
ccaaagctaa t 71
<210> 116
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 116
tccagtccca ctagtttggc tttgaagtcg cggatagtag actcgctctt gtatctcttc 60
tcagtcaggt c 71
<210> 117
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 117
gtaaaggcta gcagaccctg ggaacattcc cctgcgctca gcctctctgc catggaggaa 60
atgctaaaag t 71
<210> 118
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 118
ttttgaacag cacttatctc ttctctccag aggggcatat cacagagcat gaccaaaaag 60
ttagccagct a 71
<210> 119
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 119
aagttgacct cttatgattt tattattggt ttgtggtgca agatgttctg tccaggtttc 60
aacttatagc c 71
<210> 120
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 120
accaccacac ctgcctgagt catgtaagaa gattaggcat ggtggatgga ggtgggaaga 60
caattaatgg t 71
<210> 121
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 121
tggtcgtctg agccctatgt agtgaattca aactttcttg tctaagccaa gtatcaacct 60
gcaaacccaa g 71
<210> 122
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 122
tccccttctg tgtgctcaag gtgtgaatat tttattgtta acttacttca ctcgtgtcct 60
gcagttagat g 71
<210> 123
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 123
agcaggcagg ttgagacaag cctgcagggc caatatctgt cactatcata actcaagcca 60
acaataccca a 71
<210> 124
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 124
cttgcttgcc atcacccgtc tggtccaagg gactacggtc aatataacct ccaatcttag 60
taacctacct c 71
<210> 125
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 125
gcagacaccc tgggcagcgt tggagtgatc atctcggcca tcctgatgca gaagtatgac 60
ctgatgatcg c 71
<210> 126
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 126
aactgggcta aaacgatggg acggtgtgcg aaaacaaact aaccctaacc agaaaattgt 60
atgctttgtt t 71
<210> 127
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 127
accaccttca cattaacctt ctccatgaca aaacagcccc aagcctgaac agcccctagc 60
cccttccact a 71
<210> 128
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 128
gaagacacaa actcaacaag agcacaacaa cacaggctta aggtactgca attcctgctt 60
attttcataa a 71
<210> 129
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 129
aaatgaaaag cgagaaagga cggaggtatt ttaaatatat ttaccatagt actcaccgaa 60
ggctgcagcc a 71
<210> 130
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 130
gaaattgccc cttgattttg tcagtttagc gatcagtata cacaaaataa ttaactaaag 60
gaacaaccat a 71
<210> 131
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 131
aaaccacatg gtcttcctgc aactttgtgc caaatgagta gtttcacaat gaacgttgtg 60
aggtctgcag c 71
<210> 132
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 132
agacacacag cagactagac tgaggatgtg aaccattcct ccacttaatg caaatgcagg 60
gacacattca g 71
<210> 133
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 133
ctattcctgc ttaccgtagt tgaactggct gttggatttc tcacagttga tgatgttgaa 60
gcgatagggc a 71
<210> 134
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 134
ggtgtaagta cagactcttt gaaagcatgc aaatagaagt aaagacactg tcattccttt 60
aaatgttctt g 71
<210> 135
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 135
cttctttatt tgctatgatt attacttaat agtgccgatt gtatttgtca tccgtattga 60
ctgcagaact a 71
<210> 136
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 136
attgttcaag gacattatgc ttgtcctaca tattggcaat ttgatgtcgt tctttaacat 60
ttataattga t 71
<210> 137
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 137
aaaacttctt aagggacaag aaggaagttg aagtttgggg tgggctagga agataaagag 60
ttgggggtgt g 71
<210> 138
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 138
accaacacag agatgagacg tgccgagcgc aaggctacca agaagaagct cccgctgaaa 60
cgagagatgg a 71
<210> 139
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 139
ttaatctaac tcactctcca taacatcaca gaagtcgatg tattcgatta taacaagctc 60
agggctgtca t 71
<210> 140
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 140
ccctttacct agaatggtct gcagcgtgat gtcaaagtgg ttattttgtc cattgttgcc 60
agtgataagc c 71
<210> 141
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 141
tgcagaatgg acaactgaag agagatatgt cgcacgtgag ggaaacaact ccgtgtctag 60
gccttctgaa g 71
<210> 142
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 142
gttagtgaaa gccatttcag ggtaaaccct ccaggccgtc caatgtacca tagaagcaaa 60
acaatgataa t 71
<210> 143
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 143
cccatctgtc agaaccttgc ccacagctgt ttccctactc aatgaaaaca agctaacatc 60
ctgcaggttg a 71
<210> 144
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 144
ggaatattcg aacggcttgt tgtccaatga gtcgggggcc ttaccaccac aaaccccaag 60
gcctgaggca g 71
<210> 145
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 145
ttaagagagt cacaaacatg aaaaactgtg atagtacaaa gaagatgaac gataggcttg 60
tggatagatt a 71
<210> 146
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 146
tttatttcag catttagccc aatcctgcta agaaccgtca gttaatcact aattaggaga 60
atatcaataa a 71
<210> 147
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 147
ctcgaagtaa gaaatgaagc tgcaggtctg caggcagagt gctgtcagtg gaatataata 60
cccttaatag a 71
<210> 148
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 148
gataaggatg caacagattt attttagttt tagattatgc tttcagactg atttcggctc 60
ttaaaaagat a 71
<210> 149
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 149
tctctgttca atatttagaa taaaaagctg acaaatgtca cgtaatggac tggaaacagc 60
agacacatgg c 71
<210> 150
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 150
ctataggtgg atgatatgat atggttgcag ctagatagtg acagctgcct accttgtaag 60
taccacctcg a 71
<210> 151
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 151
gcgtttccag taaaacgacg tcccccttcg ccctacattt aatgagcacg tagtctagat 60
ttttgtttaa c 71
<210> 152
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 152
gcaggttttt gcagaaatca gttgctaata aagttattct gtaaccattg tataagcagg 60
gtcaccatga c 71
<210> 153
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 153
tttctcttaa tgcatcatcc ttgtgcgaaa tcatgttaag tacacaccgt taaagttagg 60
tgctttgtta c 71
<210> 154
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 154
aaactaatga aaaacacaag agtgcctgca gtaacgctgt actaacgctg tactaacagt 60
acactctcag g 71
<210> 155
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 155
ctgcagcaga tggaactata tctctagtgg ctgtgggtgg aggaggagat gtggtgaaga 60
ctgagcagac a 71
<210> 156
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 156
cagaaaggaa aaatgtgtca aagttctaga tagtgggtgg aaagactcaa acaatgcagt 60
ttggaatgaa g 71
<210> 157
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для ПЦР
<400> 157
acgttggatg tccacagtcc acatgctttg 30
<210> 158
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для ПЦР
<400> 158
acgttggatg ggaaagaaac agtgataggc 30
<210> 159
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Праймер для удлинения
<400> 159
cacacaactg tgtgtcaaat 20
<210> 160
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 160
ataatttact tttaagattt ctgaccggcc ttgttntttt tgcttatgtg ccattattgc 60
cggctagacc a 71
<210> 161
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 161
taaagaacaa gaaaacagta cacatgcatt aactcnccat gttggtgttg gagaactcga 60
tacagagaca g 71
<210> 162
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 162
ctcatggaga ggcatatctt gtcctatccc cataanggcc acctggtaat gagccgtgaa 60
acactagagc c 71
<210> 163
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 163
ccatttagat tattcaacgg tgaaacatac acatcntgta aattactctc aggtaaccgg 60
acttgatttg t 71
<210> 164
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 164
gtttgtagcc ccatctcact ggcttcttga aagtanaatt tattatgatt gtttaattat 60
aatagtgaat a 71
<210> 165
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 165
atttcatgta ttggccaaca aacgaacttg taggcntacg tgccatggtt gtcacatttt 60
aataaaacat g 71
<210> 166
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 166
cacagttata gcaacactta agtagaatgg aaatgntttc atttaatttt agtcagttgg 60
cattcagttg a 71
<210> 167
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 167
agtctgcaga ccctacccag cctggtctcc caggcngtca cacagcagca cagggacttt 60
ctggatggct t 71
<210> 168
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 168
atttcatgaa cctacacaaa tccagtgtca ggaaancctt ataaactttt gctcatgggt 60
gtggagatgt g 71
<210> 169
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или C
<400> 169
atagggccaa gacagaagac agacatgaaa gtcctnctga cgggcaaaac atacagaccc 60
cacctggaga a 71
<210> 170
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или T
<400> 170
ttcagttcag tcaaactggc tgtcgttggc gctgcnggac tagctggcac attcaatggg 60
aatcgtttgt c 71
<210> 171
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 171
aaaggtcttg atggatattg tgagttatcg gtgtcntaag aaatcgccac ctcgcaaccc 60
atgcgacccc a 71
<210> 172
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 172
actccaaagc caccacagtc tcctccagcc atggtncatc cctccagtag cccaaccaat 60
taccaaacag a 71
<210> 173
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или A
<400> 173
acatgcgaca catggacaga ttaattagat tgggtnacaa cacattgtat tgcaaacatg 60
tgaagctata a 71
<210> 174
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 174
ctctcattcc tcctattcat atgtatatac actggnctag ttagtgttat ggttgttatt 60
cactggcaat a 71
<210> 175
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 175
caaacaaccc tggaagtcaa atcaagaggc aaggcnctgt gtttccttga aagccagagc 60
tgtttgtgtc c 71
<210> 176
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 176
ggaccagtgt ttcatatcct gtggtgagct tcacangtca aatgtgatta atcataattg 60
aaatcaaatt a 71
<210> 177
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 177
aagagaatat ttggaatagc attggcaaat acaccnagtg gggtggagct gcgtcagtag 60
tgcacagcac a 71
<210> 178
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 178
gaaaatactg ttactgtaga atataatagt cataancctc tgatccaaat aattatgcat 60
aggtagtgtt c 71
<210> 179
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 179
ctcaacataa ttaaatacca acaccaatgt aaatcnttct tcagaaacat tgagtaaata 60
tacctttact a 71
<210> 180
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 180
agaaagcagg aagttcaggg gtcaactggg caaggncaat aagaggcatt tctaaccgtg 60
atcctgaacc c 71
<210> 181
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 181
cgaatcaagc caaataaagc ggccacatct caaatntggt cagcctttgg aggagaacga 60
taaacggact t 71
<210> 182
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 182
ccgcagatga catcactaca ctgcctgata cagcanagcg tgctttgcgg tgagttaaaa 60
aaataccatg g 71
<210> 183
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 183
catgagctca agcacatctg cttctttctt cagggnaaaa aaatacaggg atccccaact 60
gcatttgatt t 71
<210> 184
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 184
tgtagtctaa taatgagggg attagtgaaa actttnagtc agacctttgt ctttaaaaca 60
atagatttct g 71
<210> 185
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 185
atgttggcat tgtaggtgtc atagcaacca ggaccnaatc cctgtaccaa acatgtgatt 60
aaaaacatat a 71
<210> 186
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 186
ttacccggct aaggagcgct ttcttcgcac ttggantata atgaaacctc aaactgtctc 60
atttaatatg c 71
<210> 187
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 187
ttgggacagt ttaacgttca cctcaggaat ccacancctt tcattttaag tttattttac 60
ttggcagagc a 71
<210> 188
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или T
<400> 188
caacaatgca acagaaatta gtgtgtgaca aaaatntgaa cggctgcttt gaaaattatt 60
atcaaggcag t 71
<210> 189
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 189
gtgcccttat cttaccgctg atcagtggca acccantagt ttttactaac tgaaaacacc 60
attgacattc t 71
<210> 190
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или A
<400> 190
actgcctggt tatgacacct gaaccctaca gagagngtgg ggctatagtt aaaatttact 60
cccctaaggt t 71
<210> 191
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или G
<400> 191
aggatcccat cccataatga atgggtctag ctatanattt atgaccagtt gttttccggg 60
tttatgacct c 71
<210> 192
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 192
taaatagctt tgtggagtag attatgaatt gtattnatgc catatccact gttctgcaat 60
gactctccat a 71
<210> 193
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 193
accctttgat gtgatttgct tctgagaaac atcatnattt attgatgctt ccattaaagt 60
agcatagatg t 71
<210> 194
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 194
aaatcacagt gcagttatca caaaacatta tcttcngtgt tgtagcctaa ctagactata 60
cagctgtaaa a 71
<210> 195
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 195
aagtttgtac cccaaatttc catttatgga atgganagtt taattgcatt tttggattga 60
tacagtaacc a 71
<210> 196
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или T
<400> 196
gggttatgta taaatcgatg taattattat ttttgnttta aaaggtataa tattgtataa 60
cattgtaata a 71
<210> 197
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 197
gatggcattc actatccttt aacaccacat cgtagntgat gtggcacaaa agcagtgctt 60
aaaaaataaa t 71
<210> 198
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или C
<400> 198
cacacaaaaa ctattagccc atcgttggta tagtgncaaa atgttttaaa tgtcagcaat 60
caaattcaag a 71
<210> 199
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или A
<400> 199
tcagtgacgg ctgtgaacat aaagggtata gttgcnttac tggtccacgt tcaaaaacca 60
gagttgagat t 71
<210> 200
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 200
accaatttta tagtgacaca gaaaaatatc tagatntgat tctcaccaaa gagaccatat 60
tttgaaatag t 71
<210> 201
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 201
ctcgatcttc tcaagtcaag tggccaatta aatatnaatc taaacacaac aatccagttt 60
gactagttgt t 71
<210> 202
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 202
aggacacacg ctgggtgagc aacacacatc cccagncccc ctgagaaatc aggcttctta 60
caaggttata a 71
<210> 203
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 203
ggggcctttg tcacacagaa agagatgaca tcagtngcaa gagaggccat cagtgtgttc 60
aaggactgga a 71
<210> 204
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или A
<400> 204
ggaagtctag ggtggaaggg aggacattgt gcgggncgtt ccaccaattg agtacctttt 60
cagcagtcac t 71
<210> 205
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или T
<400> 205
catctcaaaa ataagttaaa taaataaatt actatngtaa gtgccaaata aagtaacagg 60
gttgaatttt a 71
<210> 206
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 206
tgtagattaa acaacaaagt cagattatct gagccntgtg tgccccaact tcaacaagga 60
gaccgtattg t 71
<210> 207
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 207
ttatcaataa ttataatcaa tgactcacat cttgantatc tacagatgta gacttgtgat 60
tgagctactg t 71
<210> 208
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 208
aacgacctca tactgggccg gaggatctcc ttctangagc tcagggggga aatagggtgt 60
gggaacttct c 71
<210> 209
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или C
<400> 209
aacaatacac tcttgtcact tgcctttact gagaangtcg tggtggacac cagattccca 60
tgtgaaggag a 71
<210> 210
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или G
<400> 210
aagtcattga ccttgctgcc ttggtcgtcc ctctcngtgg tggtgaacac gcgcgttttg 60
gactcctctg t 71
<210> 211
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 211
tgctgaagct ggacaaggag aacgccgtcg accgcncaga gcaggctgag accgacaaga 60
aggcagcaga g 71
<210> 212
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 212
gatcagctgg agaacatcta caaggacaat cccctngtga atctccatta tgccactttt 60
agccaacaac t 71
<210> 213
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 213
tatgagcagc tgaaaaacaa ttaaaatatt tttttncctg tgtttgagga aggggaagag 60
tggacccagg g 71
<210> 214
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или G
<400> 214
atatttcctt cctcacatcc ctggcaatta tagtanaatc tgagccataa caacatgacc 60
tggatagatg a 71
<210> 215
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или A
<400> 215
aaataatggc atgcatttga tattagtgta tgtttnaaaa cattacaggt tacagagaaa 60
ctataaggaa t 71
<210> 216
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 216
acattcaggt aatggtacat tttgtttaat taaacnactt tccatagttt gtggagaaag 60
ggtgtgtact c 71
<210> 217
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 217
ggttttatgc ttgaacattc attttggaat ttccangact gtctctagct gctttaatct 60
tctttcaagg a 71
<210> 218
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 218
tagatgttga gtatatctaa cacttccaga acatcnagtt tagtgctgat gtgtcatttc 60
tgttccaggc a 71
<210> 219
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 219
caatggaacg cctcctcttt ctaataaccc tagtanagtg ccgtcaaatg tcgttgacag 60
atttgagtct t 71
<210> 220
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или A
<400> 220
aaaggatata ttgatgaata tgacctatgt actgtnctac ttaaattcag atagctgttt 60
gttcatgtgt g 71
<210> 221
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 221
gctatattaa ttcagaaatg ccattttctg tcatgnggga aaatatagtt ttacacttat 60
cccagaaaca c 71
<210> 222
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 222
tgtacattgt aaagatggag aaatattgac aaaaanatgt cgtataggct actgtattac 60
ttgatatgtt t 71
<210> 223
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 223
tttaacccag cattgtgaca catttttatt aaatcnagga tgtgcagttt gttttatcca 60
cttcattaat a 71
<210> 224
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 224
aatttgacca atttgtcttc atacatttca gataanctca cgattcttaa gtcatgttgt 60
atttttaccg a 71
<210> 225
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 225
cctgactgaa agcagggcac aatatcagga agttgnatta gccaccatca tggcggtgga 60
aaattgtgct t 71
<210> 226
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> A или G
<400> 226
gttatggtga aagagaagct cagttacgga gcacancagc aaatcctcaa caagccaaac 60
ctgcaagaca a 71
<210> 227
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> T или C
<400> 227
gacatctgga gagctaagga aacaaccaag cctgtnggaa cttctattgg gtgtctctgc 60
tagcagtcca a 71
<210> 228
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> G или T
<400> 228
caataactag aaaaatacat ttcctaaaga aaatgngtgt gcttgcttgc ttgtcttaaa 60
gtatttatgt t 71
<210> 229
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<220>
<221> дополнительный признак
<222> (36)..(36)
<223> C или T
<400> 229
tatcaggaca agctggaact agatagctgg ttatgnaacg ttaactattg ggatcagaaa 60
ctgaactagc t 71
<210> 230
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 230
ataatttact tttaagattt ctgaccggcc ttgttgtttt tgcttatgtg ccattattgc 60
cggctagacc a 71
<210> 231
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 231
taaagaacaa gaaaacagta cacatgcatt aactcgccat gttggtgttg gagaactcga 60
tacagagaca g 71
<210> 232
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 232
ctcatggaga ggcatatctt gtcctatccc cataacggcc acctggtaat gagccgtgaa 60
acactagagc c 71
<210> 233
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 233
ccatttagat tattcaacgg tgaaacatac acatcttgta aattactctc aggtaaccgg 60
acttgatttg t 71
<210> 234
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 234
gtttgtagcc ccatctcact ggcttcttga aagtataatt tattatgatt gtttaattat 60
aatagtgaat a 71
<210> 235
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 235
atttcatgta ttggccaaca aacgaacttg taggcttacg tgccatggtt gtcacatttt 60
aataaaacat g 71
<210> 236
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 236
cacagttata gcaacactta agtagaatgg aaatggtttc atttaatttt agtcagttgg 60
cattcagttg a 71
<210> 237
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 237
agtctgcaga ccctacccag cctggtctcc caggctgtca cacagcagca cagggacttt 60
ctggatggct t 71
<210> 238
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 238
atttcatgaa cctacacaaa tccagtgtca ggaaaccctt ataaactttt gctcatgggt 60
gtggagatgt g 71
<210> 239
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 239
atagggccaa gacagaagac agacatgaaa gtcctgctga cgggcaaaac atacagaccc 60
cacctggaga a 71
<210> 240
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 240
ttcagttcag tcaaactggc tgtcgttggc gctgcaggac tagctggcac attcaatggg 60
aatcgtttgt c 71
<210> 241
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 241
aaaggtcttg atggatattg tgagttatcg gtgtcgtaag aaatcgccac ctcgcaaccc 60
atgcgacccc a 71
<210> 242
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 242
actccaaagc caccacagtc tcctccagcc atggtccatc cctccagtag cccaaccaat 60
taccaaacag a 71
<210> 243
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 243
acatgcgaca catggacaga ttaattagat tgggtcacaa cacattgtat tgcaaacatg 60
tgaagctata a 71
<210> 244
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 244
ctctcattcc tcctattcat atgtatatac actggactag ttagtgttat ggttgttatt 60
cactggcaat a 71
<210> 245
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 245
caaacaaccc tggaagtcaa atcaagaggc aaggcactgt gtttccttga aagccagagc 60
tgtttgtgtc c 71
<210> 246
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 246
ggaccagtgt ttcatatcct gtggtgagct tcacaggtca aatgtgatta atcataattg 60
aaatcaaatt a 71
<210> 247
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 247
aagagaatat ttggaatagc attggcaaat acacctagtg gggtggagct gcgtcagtag 60
tgcacagcac a 71
<210> 248
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 248
gaaaatactg ttactgtaga atataatagt cataatcctc tgatccaaat aattatgcat 60
aggtagtgtt c 71
<210> 249
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 249
ctcaacataa ttaaatacca acaccaatgt aaatcgttct tcagaaacat tgagtaaata 60
tacctttact a 71
<210> 250
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 250
agaaagcagg aagttcaggg gtcaactggg caagggcaat aagaggcatt tctaaccgtg 60
atcctgaacc c 71
<210> 251
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 251
cgaatcaagc caaataaagc ggccacatct caaatttggt cagcctttgg aggagaacga 60
taaacggact t 71
<210> 252
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 252
ccgcagatga catcactaca ctgcctgata cagcaaagcg tgctttgcgg tgagttaaaa 60
aaataccatg g 71
<210> 253
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 253
catgagctca agcacatctg cttctttctt caggggaaaa aaatacaggg atccccaact 60
gcatttgatt t 71
<210> 254
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 254
tgtagtctaa taatgagggg attagtgaaa actttaagtc agacctttgt ctttaaaaca 60
atagatttct g 71
<210> 255
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 255
atgttggcat tgtaggtgtc atagcaacca ggacctaatc cctgtaccaa acatgtgatt 60
aaaaacatat a 71
<210> 256
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 256
ttacccggct aaggagcgct ttcttcgcac ttggagtata atgaaacctc aaactgtctc 60
atttaatatg c 71
<210> 257
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 257
ttgggacagt ttaacgttca cctcaggaat ccacatcctt tcattttaag tttattttac 60
ttggcagagc a 71
<210> 258
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 258
caacaatgca acagaaatta gtgtgtgaca aaaatatgaa cggctgcttt gaaaattatt 60
atcaaggcag t 71
<210> 259
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 259
gtgcccttat cttaccgctg atcagtggca acccattagt ttttactaac tgaaaacacc 60
attgacattc t 71
<210> 260
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 260
actgcctggt tatgacacct gaaccctaca gagagtgtgg ggctatagtt aaaatttact 60
cccctaaggt t 71
<210> 261
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 261
aggatcccat cccataatga atgggtctag ctatacattt atgaccagtt gttttccggg 60
tttatgacct c 71
<210> 262
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 262
taaatagctt tgtggagtag attatgaatt gtattgatgc catatccact gttctgcaat 60
gactctccat a 71
<210> 263
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 263
accctttgat gtgatttgct tctgagaaac atcataattt attgatgctt ccattaaagt 60
agcatagatg t 71
<210> 264
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 264
aaatcacagt gcagttatca caaaacatta tcttctgtgt tgtagcctaa ctagactata 60
cagctgtaaa a 71
<210> 265
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 265
aagtttgtac cccaaatttc catttatgga atggatagtt taattgcatt tttggattga 60
tacagtaacc a 71
<210> 266
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 266
gggttatgta taaatcgatg taattattat ttttgattta aaaggtataa tattgtataa 60
cattgtaata a 71
<210> 267
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 267
gatggcattc actatccttt aacaccacat cgtaggtgat gtggcacaaa agcagtgctt 60
aaaaaataaa t 71
<210> 268
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 268
cacacaaaaa ctattagccc atcgttggta tagtggcaaa atgttttaaa tgtcagcaat 60
caaattcaag a 71
<210> 269
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 269
tcagtgacgg ctgtgaacat aaagggtata gttgctttac tggtccacgt tcaaaaacca 60
gagttgagat t 71
<210> 270
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 270
accaatttta tagtgacaca gaaaaatatc tagatatgat tctcaccaaa gagaccatat 60
tttgaaatag t 71
<210> 271
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 271
ctcgatcttc tcaagtcaag tggccaatta aatatgaatc taaacacaac aatccagttt 60
gactagttgt t 71
<210> 272
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 272
aggacacacg ctgggtgagc aacacacatc cccagtcccc ctgagaaatc aggcttctta 60
caaggttata a 71
<210> 273
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 273
ggggcctttg tcacacagaa agagatgaca tcagttgcaa gagaggccat cagtgtgttc 60
aaggactgga a 71
<210> 274
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 274
ggaagtctag ggtggaaggg aggacattgt gcgggtcgtt ccaccaattg agtacctttt 60
cagcagtcac t 71
<210> 275
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 275
catctcaaaa ataagttaaa taaataaatt actatagtaa gtgccaaata aagtaacagg 60
gttgaatttt a 71
<210> 276
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 276
tgtagattaa acaacaaagt cagattatct gagccttgtg tgccccaact tcaacaagga 60
gaccgtattg t 71
<210> 277
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 277
ttatcaataa ttataatcaa tgactcacat cttgaatatc tacagatgta gacttgtgat 60
tgagctactg t 71
<210> 278
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 278
aacgacctca tactgggccg gaggatctcc ttctatgagc tcagggggga aatagggtgt 60
gggaacttct c 71
<210> 279
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 279
aacaatacac tcttgtcact tgcctttact gagaaagtcg tggtggacac cagattccca 60
tgtgaaggag a 71
<210> 280
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 280
aagtcattga ccttgctgcc ttggtcgtcc ctctccgtgg tggtgaacac gcgcgttttg 60
gactcctctg t 71
<210> 281
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 281
tgctgaagct ggacaaggag aacgccgtcg accgcacaga gcaggctgag accgacaaga 60
aggcagcaga g 71
<210> 282
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 282
gatcagctgg agaacatcta caaggacaat cccctggtga atctccatta tgccactttt 60
agccaacaac t 71
<210> 283
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 283
tatgagcagc tgaaaaacaa ttaaaatatt tttttccctg tgtttgagga aggggaagag 60
tggacccagg g 71
<210> 284
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 284
atatttcctt cctcacatcc ctggcaatta tagtataatc tgagccataa caacatgacc 60
tggatagatg a 71
<210> 285
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 285
aaataatggc atgcatttga tattagtgta tgttttaaaa cattacaggt tacagagaaa 60
ctataaggaa t 71
<210> 286
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 286
acattcaggt aatggtacat tttgtttaat taaacaactt tccatagttt gtggagaaag 60
ggtgtgtact c 71
<210> 287
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 287
ggttttatgc ttgaacattc attttggaat ttccacgact gtctctagct gctttaatct 60
tctttcaagg a 71
<210> 288
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 288
tagatgttga gtatatctaa cacttccaga acatctagtt tagtgctgat gtgtcatttc 60
tgttccaggc a 71
<210> 289
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 289
caatggaacg cctcctcttt ctaataaccc tagtaaagtg ccgtcaaatg tcgttgacag 60
atttgagtct t 71
<210> 290
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 290
aaaggatata ttgatgaata tgacctatgt actgtgctac ttaaattcag atagctgttt 60
gttcatgtgt g 71
<210> 291
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 291
gctatattaa ttcagaaatg ccattttctg tcatgaggga aaatatagtt ttacacttat 60
cccagaaaca c 71
<210> 292
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 292
tgtacattgt aaagatggag aaatattgac aaaaaaatgt cgtataggct actgtattac 60
ttgatatgtt t 71
<210> 293
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 293
tttaacccag cattgtgaca catttttatt aaatcaagga tgtgcagttt gttttatcca 60
cttcattaat a 71
<210> 294
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 294
aatttgacca atttgtcttc atacatttca gataaactca cgattcttaa gtcatgttgt 60
atttttaccg a 71
<210> 295
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 295
cctgactgaa agcagggcac aatatcagga agttgtatta gccaccatca tggcggtgga 60
aaattgtgct t 71
<210> 296
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 296
gttatggtga aagagaagct cagttacgga gcacaacagc aaatcctcaa caagccaaac 60
ctgcaagaca a 71
<210> 297
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 297
gacatctgga gagctaagga aacaaccaag cctgttggaa cttctattgg gtgtctctgc 60
tagcagtcca a 71
<210> 298
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 298
caataactag aaaaatacat ttcctaaaga aaatgggtgt gcttgcttgc ttgtcttaaa 60
gtatttatgt t 71
<210> 299
<211> 71
<212> ДНК
<213> Oncorhynchus mykiss
<400> 299
tatcaggaca agctggaact agatagctgg ttatgcaacg ttaactattg ggatcagaaa 60
ctgaactagc t 71
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM4 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2718584C2 |
| SNP-МАРКЕРЫ И ОТБОР В ОТНОШЕНИИ НИЗКОГО СОДЕРЖАНИЯ КЛЕТЧАТКИ В ПРЕДСТАВИТЕЛЯХ РОДА BRASSICA | 2019 |
|
RU2834673C2 |
| РАСТЕНИЯ ТОМАТОВ С УЛУЧШЕННЫМИ ПРИЗНАКАМИ | 2019 |
|
RU2821992C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ПОЛУЧЕНИЯ УКОРОЧЕННЫХ РАСТЕНИЙ КУКУРУЗЫ | 2016 |
|
RU2745987C2 |
| РАСТЕНИЯ ТОМАТА С УЛУЧШЕННОЙ УСТОЙЧИВОСТЬЮ К БОЛЕЗНЯМ | 2015 |
|
RU2721952C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |
| РАСТЕНИЯ САЛАТ-ЛАТУК, ИМЕЮЩИЕ РЕЗИСТЕНТНОСТЬ ПРОТИВ NASONOVIA RIBISNIGRI БИОТИПА 1 | 2015 |
|
RU2731639C2 |
| Участки генов и гены, ассоциированные с повышенной урожайностью у растений | 2016 |
|
RU2758718C2 |
| ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ (CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2011 |
|
RU2747979C2 |
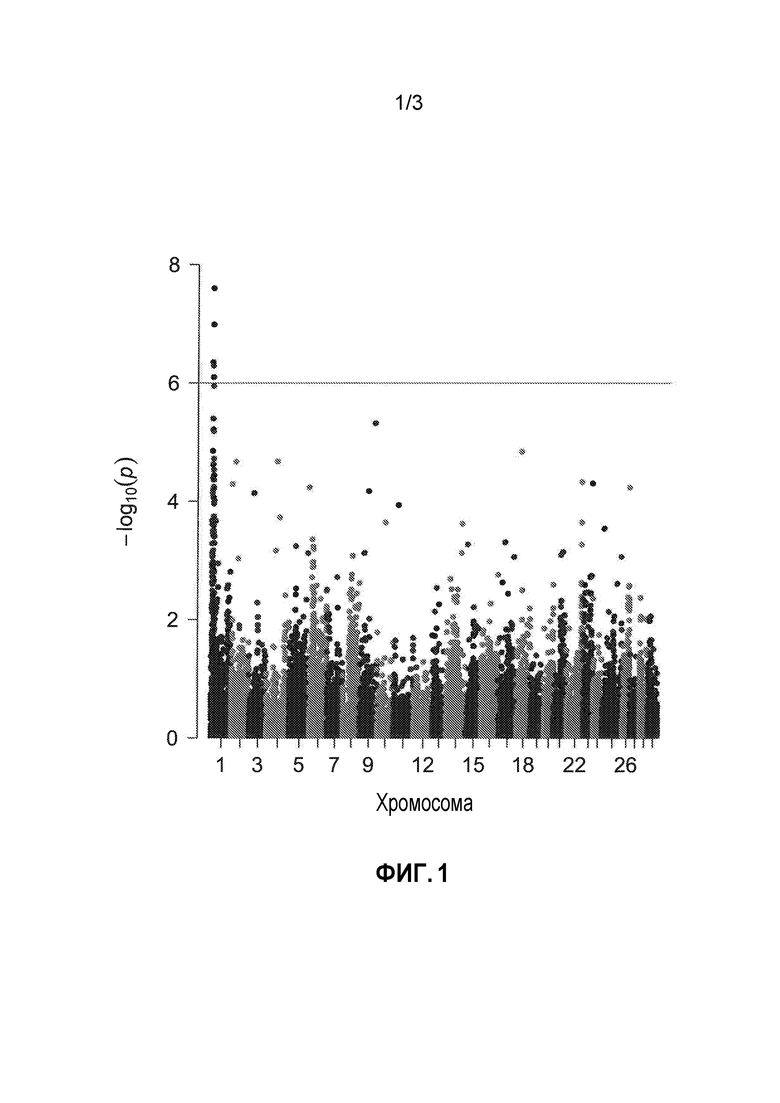
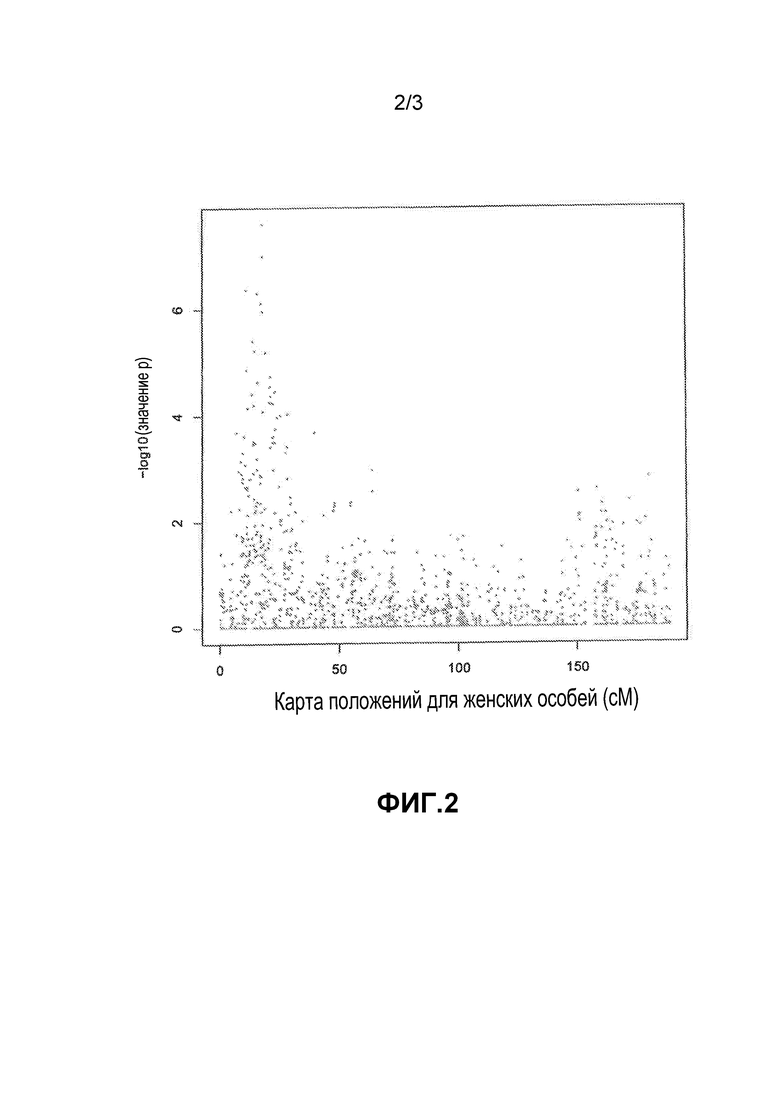
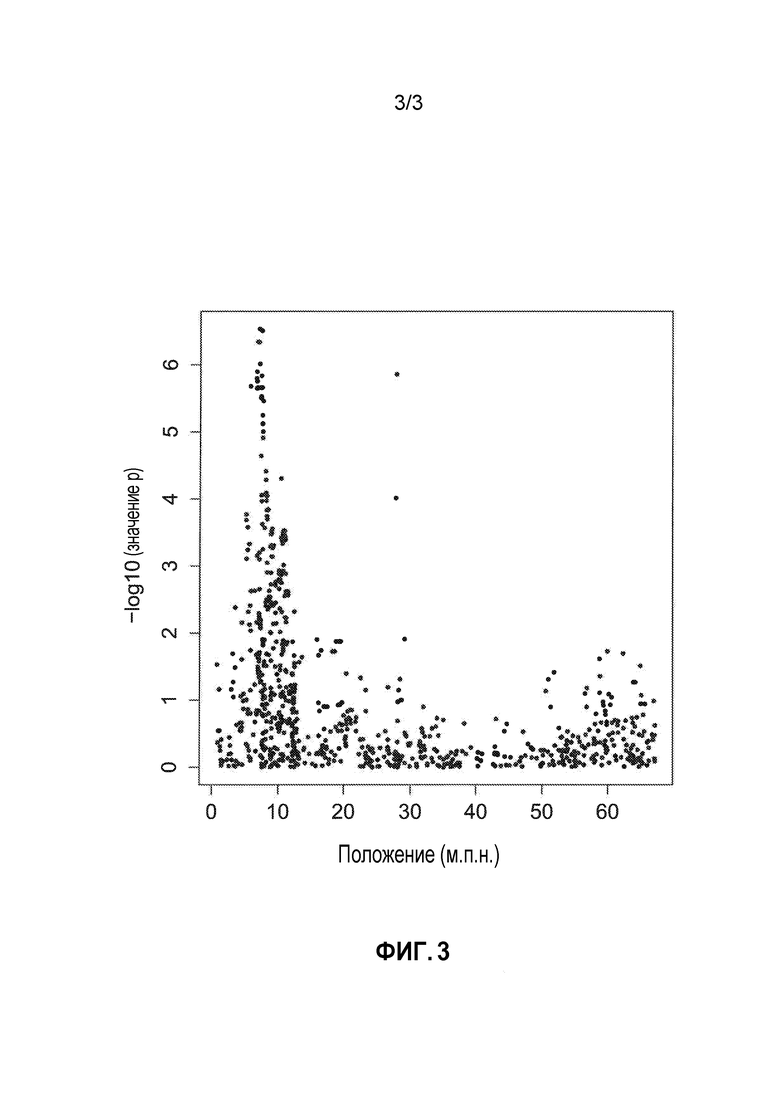
Изобретение относится к области биотехнологии. Описана группа изобретений, включающая способ прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN) и способ отбора радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы. В одном из вариантов реализации изобретения способ прогнозирования повышенной резистентности радужной форели к инфекционному некрозу поджелудочной железы включает в себя определение присутствия по меньшей мере одного аллеля, сообщающего резистентность к IPN ("аллель резистентности к IPN"), в геноме указанной радужной форели, где указанный по меньшей мере один аллель резистентности к IPN представляет собой аллель однонуклеотидного полиморфизма (SNP). Изобретение расширяет арсенал средств для прогнозирования повышенной резистентности радужной форели к инфекционному некрозу поджелудочной железы. 2 н. и 4 з.п. ф-лы, 3 ил., 4 табл., 4 пр.
1. Способ прогнозирования повышенной резистентности радужной форели (Oncorhynchus mykiss) к инфекционному некрозу поджелудочной железы (IPN), причем способ включает:
определение присутствия по меньшей мере одного аллеля, сообщающего резистентность к IPN ("аллель резистентности к IPN"), в геноме указанной радужной форели, где указанный по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного однонуклеотидного полиморфизма (SNP), где указанный по меньшей мере один SNP выбран из SNP, приведенных ниже:
#
2. Способ по п.1, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы, в геноме указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229 посредством 1-5 нуклеотидных замен.
3. Способ по п.1 или 2, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 230, SEQ ID NO: 231 или SEQ ID NO: 232;
или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 1, SEQ ID NO: 2 SEQ ID NO: 230, SEQ ID NO: 231 или SEQ ID NO: 232 посредством 1-5 нуклеотидных замен;
где присутствие цитозина в положении, соответствующем положению 36 SEQ ID NO: 1, присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 2, присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 230, присутствие гуанина в положении, соответствующем положению 36 SEQ ID NO: 231, или присутствие цитозина в положении, соответствующем положению 36 SEQ ID NO: 232, указывает на то, что радужная форель имеет повышенную резистентность к инфекционному некрозу поджелудочной железы.
4. Способ отбора радужной форели, имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, причем способ включает:
определение присутствия по меньшей мере одного аллеля, сообщающего резистентность к IPN ("аллель резистентности к IPN") в геноме указанной радужной форели; и
отбор указанной радужной форели как имеющей увеличенную резистентность, когда присутствует по меньшей мере один аллель резистентности к IPN,
где указанный по меньшей мере один аллель резистентности к IPN представляет собой аллель по меньшей мере одного однонуклеотидного полиморфизма (SNP), где указанный по меньшей мере один SNP выбран из SNP, приведенных ниже
#
5. Способ по п.4, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с повышенной резистентностью к инфекционному некрозу поджелудочной железы, в геноме указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229, или в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из любой из SEQ ID NO: 1-78 и SEQ ID NO: 160-229 посредством 1-5 нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность, когда нуклеотид по меньшей мере одного аллеля представляет собой нуклеотид, соответствующий аллелю резистентности к IPN у SNP.
6. Способ по п.4, причем способ включает:
определение идентичности нуклеотида по меньшей мере одного аллеля, необязательно по меньшей мере двух аллелей, по меньшей мере одного однонуклеотидного полиморфизма (SNP), ассоциированного с инфекционным некрозом поджелудочной железы, в геноме указанной радужной форели, причем указанный по меньшей мере один SNP расположен в указанном геноме в положении, соответствующем положению 36 нуклеотидной последовательности, указанной в SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 160, SEQ ID NO: 161 или SEQ ID NO 162, в положении, соответствующем положению 36 нуклеотидной последовательности, которая происходит из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 160, SEQ ID NO: 161 или SEQ ID NO: 162 посредством 1-5 нуклеотидных замен; и
отбор указанной радужной форели как имеющей повышенную резистентность к инфекционному некрозу поджелудочной железы, когда цитозин присутствует в положении, соответствующем положению 36 SEQ ID NO: 1, гуанин присутствует в положении, соответствующем положению 36 SEQ ID NO: 2, гуанин присутствует в положении, соответствующем положению 36 SEQ ID NO: 230, гуанин присутствует в положении, соответствующем положению 36 SEQ ID NO: 231, или цитозин присутствует в положении, соответствующем положению 36 SEQ ID NO: 232.
| WO 2014006428 A1, 09.01.2014 | |||
| СПОСОБ ИЗГОТОВЛЕНИЯ БЕТОНА | 2020 |
|
RU2766494C1 |
| В.В | |||
| Стаффорд и др | |||
| "Изменения внутренних органов радужной форели при эксперементальном инфекционном некрозе поджелудочной железы лососевых", Патоморфология 636.3: 616.98 Российский ветеринарный журнал, СЛХ, 2013, 4, стр | |||
| Машина для добывания торфа и т.п. | 1922 |
|
SU22A1 |
| Johansen L.H., Sommer A.I | |||
| "Infectious pancreatic necrosis | |||
Авторы
Даты
2021-08-25—Публикация
2015-11-18—Подача