ОБЛАСТЬ ТЕХНИКИ
Изобретение относится к способу обнаружения токсикогенной бактерии Clostridium difficile (C. difficile) путем амплификации ДНК на основе внедрения в цепь. Изобретение также относится к олигонуклеотидам, композициям и наборам, подходящим для применения в настоящем способе, и их применению для обнаружения токсикогенной C. difficile.
УРОВЕНЬ ТЕХНИКИ
Клостридии являются грамположительными спорообразующими анаэробными бактериями. Патогенные виды клостридий вырабатывают белковые токсины, из которых состоит группа крупных клостридиальных цитотоксинов из очень больших токсинов как с высокой токсичностью in vivo, так и с высокой степенью гомологии структуры и последовательностей (von Eichel-Streiber et al., 1996). Токсины А и В бактерии C. difficile являются основной причиной патогенности C. difficile. Длительное время токсин А рассматривали как основной фактор вирулентности, но возрастающее количество доказательств указывает, что фактически токсин В играет главную роль в инфекциях, вызванных C. difficile (Lyras et al., 2009; Carter et al., 2012).
Токсины А и В C. difficile кодируются соответственно генами tcdA и tcdB, и гены расположены в патогенном локусе (PaLoc) размером примерно в 19,6 тыс. оснований. PaLoc содержит также два регуляторных гена, а именно tcdC и tcdR, которые действуют соответственно как отрицательный и положительный регуляторы экспрессии токсинов. Ген tcdE, также включенный в PaLoc, кодирует холиноподобный белок, необходимый для секреции токсинов А и В. В нетоксикогенных штаммах PaLoc заменен последовательностью в 115 п.о. (Braun et al., 1996). Амплификацию ДНК использовали для обнаружения токсикогенных штаммов C. difficile (Wren et al., 1990; McMillin et al., 1991; McMillin et al., 1992).
Способ изотермической амплификации ДНК, основывающийся на применении прямого праймера, обратного праймера и системы внедрения в цепь ДНК, описан в WO 2009/150467.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к обнаружению целевой нуклеотидной последовательности токсикогенной C. difficile, что позволяет установить наличие токсикогенной C. difficile в образце. В способе согласно настоящему изобретению применяют прямой праймер, обратный праймер и внедряющийся в цепь олигонуклеотид, каждый из которых содержит участок, комплементарный указанной целевой нуклеотидной последовательности. Вместе праймеры и внедряющийся в цепь олигонуклеотид обеспечивают процесс амплификации целевой нуклеотидной последовательности. Внедряющийся в цепь олигонуклеотид переводит по меньшей мере часть целевой нуклеотидной последовательности в одноцепочечное состояние, что позволяет связаться прямому праймеру и обратному праймеру, таким образом, делая возможной амплификацию ДНК. Как правило, амплификация проводится в изотермических условиях, не требуя термической денатурации двухцепочечной ДНК.
Если обнаруживают амплификацию целевой нуклеотидной последовательности, то это указывает на присутствие в образце токсикогенного штамма целевой патогенной C. difficile, а не нетоксикогенного, непатогенного штамма C. difficile или другого вида клостридий. Авторы настоящего изобретения продемонстрировали, что способ согласно настоящему изобретению позволяет с высокой специфичностью и чувствительностью обнаруживать различные целевые последовательности токсикогенной C. difficile.
Согласно настоящему изобретению предложен способ обнаружения целевой нуклеотидной последовательности токсикогенной C. difficile в образце, причем указанный способ включает приведение указанного образца в контакт с по меньшей мере одним прямым праймером, по меньшей мере одним обратным праймером и по меньшей мере одним внедряющимся в цепь олигонуклеотидом в условиях, способствующих амплификации указанной целевой нуклеотидной последовательности, где каждый указанный праймер и указанный олигонуклеотид содержит участок, комплементарный указанной целевой нуклеотидной последовательности, и где указанный внедряющийся в цепь олигонуклеотид переводит по меньшей мере часть целевой нуклеотидной последовательности в одноцепочечное состояние, что позволяет связаться указанным прямому праймеру и обратному праймеру.
Дополнительно согласно настоящему изобретению предложены композиция и набор, каждый из которых содержит по меньшей мере два олигонуклеотида, выбранные из (a) прямого праймера, (b) обратного праймера и (c) внедряющегося в цепь олигонуклеотида, где:
- (I) прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 2 или вариант указанной последовательности, обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 3 или вариант указанной последовательност, и внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, содержащим последовательность SEQ ID NO: 4 или вариант указанной последовательности дополнительно содержащим один или более модифицированных нуклеотидов в своей 3'-области; или
- (II) прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 7 или вариант указанной последовательност, обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 8 или вариант указанной последовательност, и внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, содержащим последовательность SEQ ID NO: 9 или вариант указанной последовательности дополнительно содержащим один или более модифицированных нуклеотидов в своей 3'-области.
Дополнительно согласно настоящему изобретению предложено применение прямого праймера, обратного праймера и внедряющегося в цепь олигонуклеотида, каждый из которых как определено в пункте (I) выше или каждый из которых как определено в пункте (II) выше, в способе обнаружения C. difficile.
Дополнительно согласно настоящему изобретению предложен способ диагностики инфекции, вызванной C. difficile, у пациента, включающий осуществление способа обнаружения целевой нуклеотидной последовательности токсикогенной C. difficile согласно настоящему изобретению для образца от указанного пациента.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
На фигуре 1 показано: (А) График амплификации при анализе на ген tcdB. Для детектирования применяли краситель SybrGreen I и измеряли флуоресценцию с помощью прибора для ПЦР в реальном времени. Ось X: время (минуты), ось Y: флуоресценция SybrGreen I (интенсивность флуоресценции, относительные единицы). Верхняя линия: 10000 геномных копий геномной ДНК штамма C. difficile BAA-1382 (630), использованной в качестве матрицы для анализа на ген tcdB способом амплификации на основе внедрения в цепь (SIBA). Нижняя кривая - контроль без матрицы, амплификации не было. (В) Анализ кривой плавления реакции амплификации гена tcdB. Ось X: температура (градусы по Цельсию), ось Y: (-d(флуоресценция)/d(температура), относительные единицы). Анализ кривой плавления ДНК с SybrGreen I после амплификации показал амплификацию единственного специфического ампликона в реакции амплификации гена tcdB с геномной ДНК штамма C. difficile BAA-1382 (630) в качестве матрицы. Контроль без матрицы не показал амплификации. (С) Электрофореграмма положительной и отрицательной реакции амплификации гена tcdB. Ось X: индекс миграции (%, где нижний маркер - это 0%, а верхний маркер - 100%), ось Y: (интенсивность флуоресценции, относительные единицы). Нижняя кривая - 10000 копий геномной ДНК штамма C. difficile BAA-1382 (630), использованной в качестве матрицы при анализа на ген tcdB. Верхняя кривая - контроль без матрицы. Реакции амплификации гена tcdB были проанализированы при помощи системы электрофореза на микрочипе MultiNA. В результате реакции амплификации контроля без матрицы обнаружили только реакционные олигонуклеотиды (прямые и обратные праймеры и внедряющийся олигонуклеотид), тогда как в результате реакции амплификации гена tcdB, в которой в качестве матрицы использовали 10000 копий геномной ДНК С.difficile, обнаружили специфический продукт амплификации.
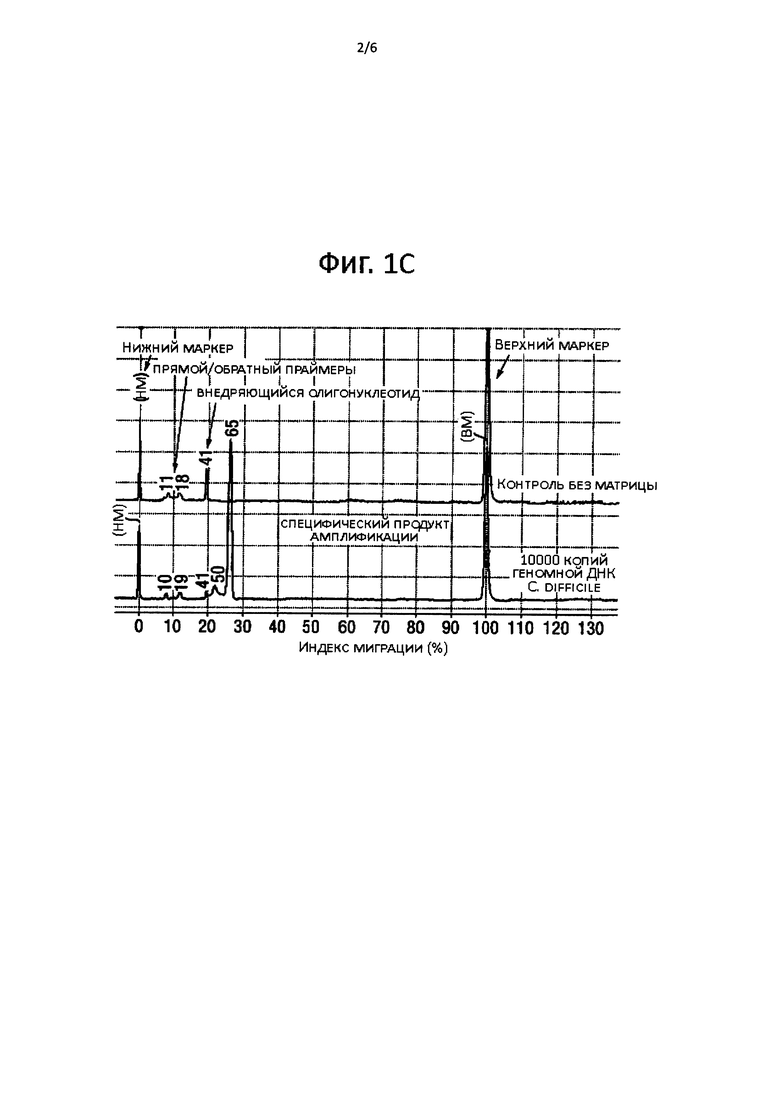
На фигуре 2 показано: (А) Специфичность анализа на ген tcdB. Ось X: время (минуты), ось Y: флуоресценция SybrGreen I (интенсивность флуоресценции, милливольты, мВ). 10000 копий геномной ДНК штамма C. difficile ВАА-1382 (630) и 10000 копий геномной ДНК штамма C. sordellii АТСС 9714 использовали в качестве матриц для реакции амплификации гена tcdB. Была амплифицирована и обнаружена только геномная ДНК C. difficile. Контроль без матрицы не показал амплификации. (В) Чувствительность анализа на ген tcdB. Ось X: время (минуты), ось Y: флуоресценция SybrGreen I (интенсивность флуоресценции, относительные единицы). Чувствительность анализа на ген tcdB определяли с помощью серии разведений геномной ДНК штамма C. difficile ВАА-1382 (630), используемой в качестве матрицы. 10-10000 копий геномной ДНК C. difficile показали амплификацию, измеренную по увеличению флуоресценции SybrGreen I, тогда как в контроле без матрицы и в реакциях отрицательного контроля амплификации не было. Все реакции проводились в присутствии 0,5 нг/мкл ДНК из молок сельди.
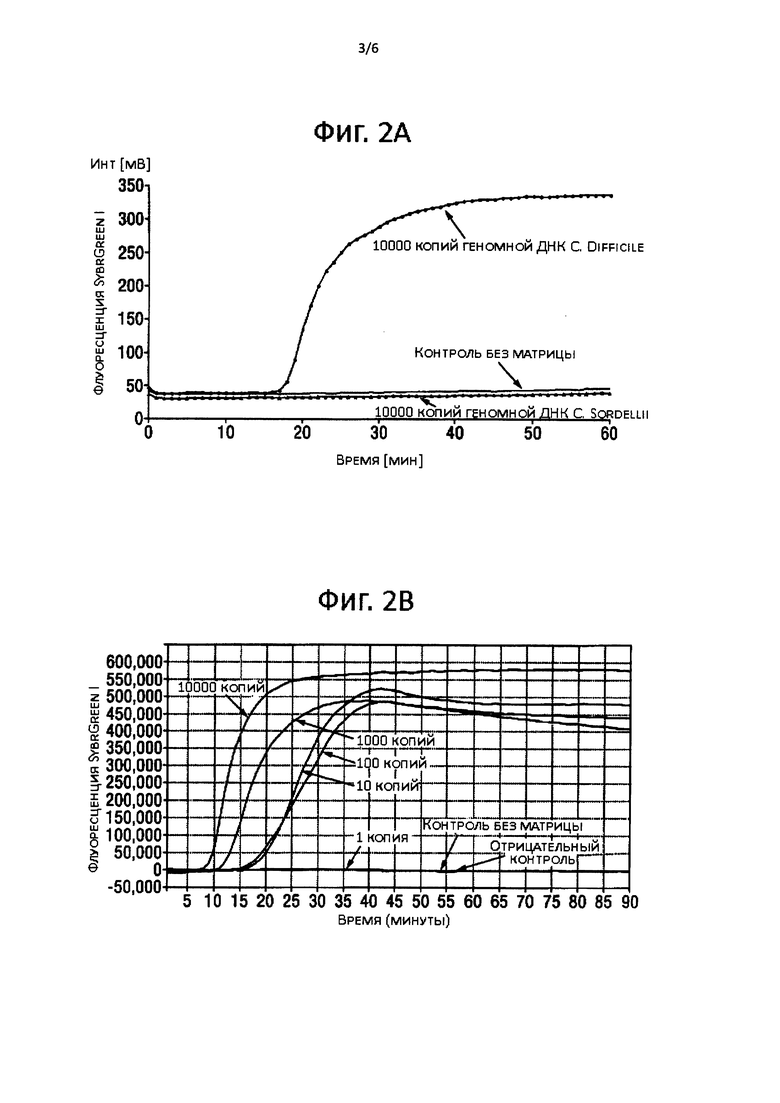
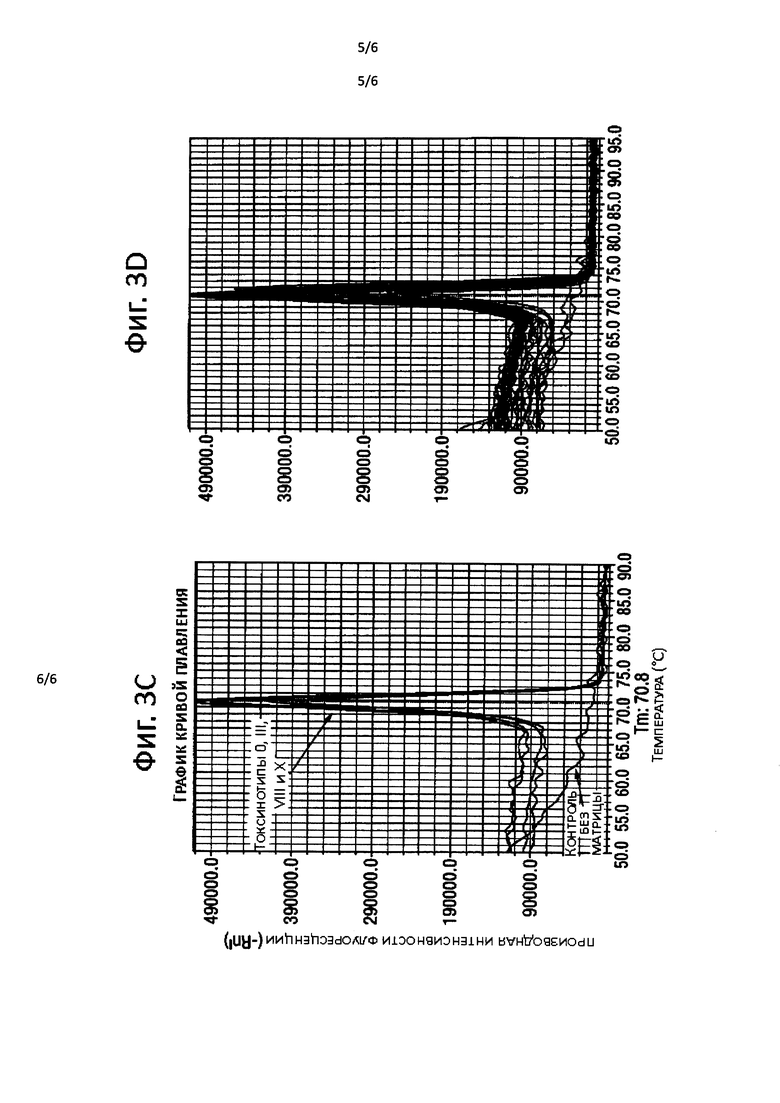
На фигуре 3 показано: (А) Всеобъемлемость анализа на ген tcdB. Ось X: время (минуты), ось Y: флуоресценция SybrGreen I (интенсивность флуоресценции, относительные единицы). 2 нг геномной ДНК, выделенной из чистых культур C. difficile токсинотипов 0, III, VIII и X, использовали в качестве матрицы для анализа на ген tcdB. Контроль без матрицы. Все протестированные токсинотипы дали положительный результат амплификации. (В) Всеобъемлемость анализа на ген tcdB для панели токсинотипов, подробности как в пункте (А) выше, но с использованием 2 нг геномной ДНК, выделенной из чистых культур C. difficile токсинотипов 0, I, II, IIIa, IIIb, IIIc, IV, V, VI, VII, VIII, IX, X, XIa, XIb, XII, XIII, XIV, XV, XVI, XVII, XVIII; XIX, XX, XXI; XXII; XXIII; XXIV, XXV, XXVI, XXVII, XXVIII, XXIX, XXX, XXXI, XXXII и XXXIII. Только два токсинотипа, а именно XIa и XIb, дали отрицательный результат амплификации. (С) Анализ кривой плавления для реакций амплификации гена tcdB с матричной геномной ДНК, выделенной из различных токсинотипов C. difficile, указанных в пункте (А). Ось X: температура (градусы по Цельсию), ось Y: (-d(флуоресценция)/d(температура), относительные единицы). Все токсинотипы C. difficile показали амплификацию единичного ампликона. Контроль без матрицы не показал амплификации. (D) Анализ кривой плавления, подробности как в пункте (С) выше, для панели токсинотипов (В). Анализ кривой плавления также показал отсутствие амплификации для токсинотипов XIa и XIb.
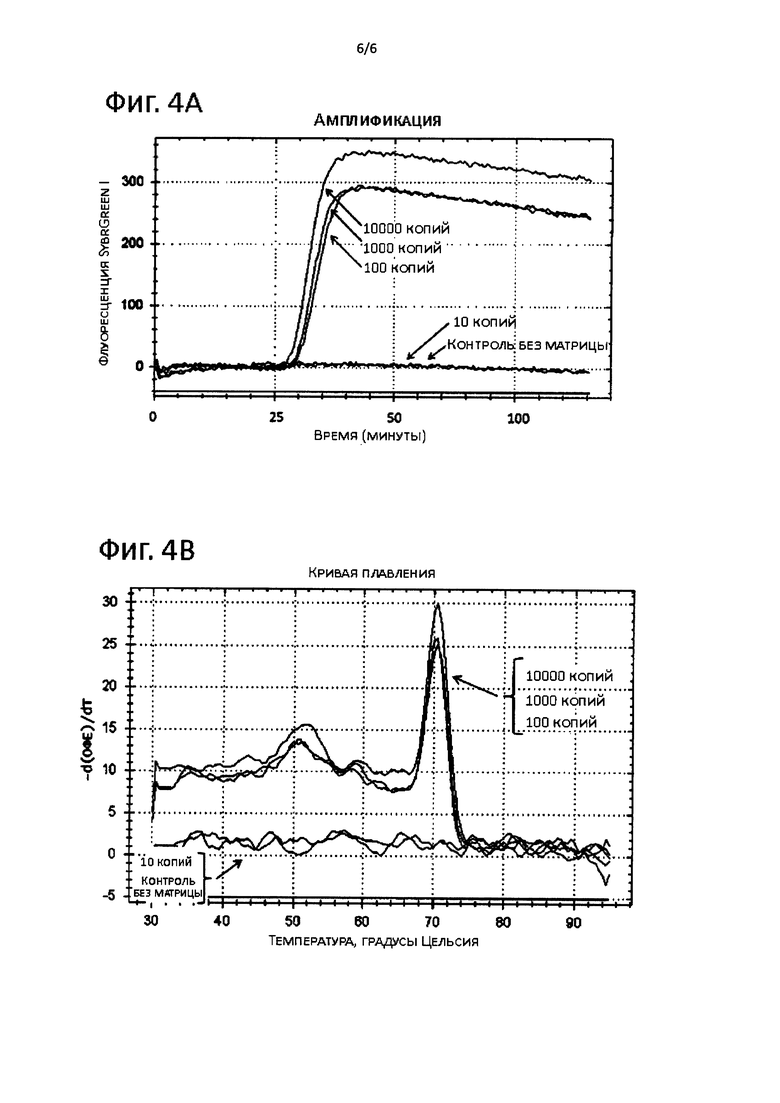
На фигуре 4 показано: (А) График амплификации при анализе на ген tcdA. Для детектирования применяли краситель SybrGreen I и измеряли флуоресценцию с помощью прибора для ПЦР в реальном времени. Ось X: время (минуты), ось Y: флуоресценция SybrGreen I (интенсивность флуоресценции, относительные единицы). Амплификацию наблюдали при 100-10000 копий геномной ДНК штамма С. difficile ВАА-1382 (630) на реакцию и не наблюдали амплификации в присутствии 0-10 копий геномной ДНК С. difficile на реакцию и в контроле без матрицы. (В) Анализ кривой плавления при анализе на ген tcdA. Ось X: температура (градусы по Цельсию), ось Y: (-d(флуоресценция)/d(температура), относительные единицы). Анализ кривой плавления показывает амплификацию единственного специфического ампликона. Контроль без матрицы не показывает амплификации.
ОПИСАНИЕ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQ ID NO: 1 - нуклеотидная последовательность целевого участка гена tcdB.
SEQ ID NO: 2 - нуклеотидная последовательность прямого праймера для гена tcdB.
SEQ ID NO: 3 - нуклеотидная последовательность обратного праймера для гена tcdB.
SEQ ID NO: 4 - нуклеотидная последовательность внедряющегося в цепь олигонуклеотида для гена tcdB.
SEQ ID NO: 5 - нуклеотидная последовательность модифицированного внедряющегося в цепь олигонуклеотида для гена tcdB.
SEQ ID NO: 6 - нуклеотидная последовательность целевого участка гена tcdA.
SEQ ID NO: 7 - нуклеотидная последовательность прямого праймера для гена tcdA.
SEQ ID NO: 8 - нуклеотидная последовательность обратного праймера для гена tcdA.
SEQ ID NO: 9 - нуклеотидная последовательность внедряющегося в цепь олигонуклеотида для гена tcdA.
SEQ ID NO: 10 - нуклеотидная последовательность модифицированного внедряющегося в цепь олигонуклеотида для гена tcdA.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Следует понимать, что различные применения описанных способов могут быть адаптированы под специфические нужды данной области техники. Следует также понимать, что используемая здесь терминология служит только для цели описания частных вариантов реализации изобретения и не предполагает накладывания ограничений. В дополнении, используемые в описании изобретения и в прилагаемой форме изобретения единичные формы слов включают множественность обозначаемых объектов, если содержание четко не предполагает другого. Таким образом, например, ссылка на «полипептид» включает ссылку на два или более таких полипептидов и тому подобное. Все публикации, патенты и заявки на патенты, процитированные здесь выше или ниже, настоящим полностью включены посредством ссылки.
Способ обнаружения токсикогенной C. difficile в образце
Образец
В большинстве случаев образец представляет собой клинический образец, например, образец, полученный от пациента, у которого подозревают наличие или у которого есть инфекция, вызванная С. difficile. Однако может быть использован любой образец при условии, что нуклеиновая кислота может быть получена или выделена из образца. Таким образом, в настоящем изобретении могут быть использованы контрольные образцы специфических штаммов С. difficile или образцы из окружающей среды. Подходящие типы клинических образцов варьируются в зависимости от специфического типа инфекции, присутствующей или подозреваемой на наличие у пациента. Образцом может быть кровь, плазма, сыворотка, моча или образец кала. В предпочтительном варианте реализации образцом является образец кала. Образец кала может быть отобран у пациента с инфекцией желудочно-кишечного тракта. Инфекция может присутствовать у пациента с диареей.
В предпочтительных вариантах реализации образцы отбирают у животных, таких как млекопитающие. В большинстве случаев образцы отбирают у людей, но настоящее изобретение также применимо в общем к домашним животным, домашнему скоту, птицам и рыбам. Например, настоящее изобретение может применяться в ветеринарной или сельскохозяйственной области.
Образец содержит нуклеиновую кислоту, которая может быть ДНК или РНК. Если нуклеиновая кислота присутствует в образце в подходящей форме, позволяющей обнаружение согласно настоящему изобретению, образец можно использовать напрямую. Однако, как правило, нуклеиновую кислоту выделяют, получают или экстрагируют из образца. Способы подготовки образцов, содержащих нуклеиновые кислоты, экстрагирования нуклеиновых кислот и/или очищения нуклеиновых кислот для использования в способах обнаружения нуклеиновых кислот хорошо известны в данной области техники. Можно выделить тотальную нуклеиновую кислоту или можно выделить ДНК и РНК по отдельности.
Как правило, образец подготавливают соответствующим образом, так что нуклеиновая кислота предоставлена в форме, удобной для приведения в контакт с праймерами и внедряющимся в цепь олигонуклеотидом. В случае, когда нуклеиновая кислота - ДНК, ДНК, как правило, предоставлена в двухцепочечной форме. В случае, когда нуклеиновая кислота - РНК, ее, как правило, конвертируют в кДНК с помощью обратной транскриптазы или полимеразы с обратно-транскриптазной активностью. РНК может быть полезна для обнаружения бактерий по причине того, что в бактериальных клетках присутствует очень большое число рибосом, которые эффективно увеличивают концентрацию целевых последовательностей.
Целевая нуклеотидная последовательность
Целевая нуклеотидная последовательность - это участок генома С. difficile (или ампликон), подходящий для применения в специфическом обнаружении токсикогенной С. difficile. Это позволяет высококачественно, однозначно определять наличие токсикогенной С. difficile в образце, даже если присутствуют близкородственные микроорганизмы. Выбор специфических целевых нуклеотидных последовательностей в токсикогенных штаммах специфического патогенна и последующая разработка олигонуклеотидов праймеров и внедряющегося в цепь олигонуклеотида для обнаружения данных последовательностей являются важным аспектом для рассмотрения. Примеры подходящих последовательностей здесь приведены.
Как правило, целевая нуклеотидная последовательность является уникальной для генома С. difficile. Таким образом, целевая нуклеотидная последовательность, как правило, отличается от любой гомологичной нуклеотидной последовательности у родственных видов, например в гомологичных видах клостридий. Как правило, целевая нуклеотидная последовательность содержит несколько несовпадений нуклеотидов с гомологичной нуклеотидной последовательностью у родственного вида. Предпочтительно, чтобы целевая нуклеотидная последовательность не присутствовала у видов клостридий, кроме как у С. difficile, несущей гены для крупных клостридиальных токсинов. Целевая нуклеотидная последовательность предпочтительно не присутствует у С. sordellii и/или С. novyi. Целевая нуклеотидная последовательность, как правило, позволяет осуществлять специфическое обнаружение С. difficile в образце, содержащем С. difficile и С. sordellii и/или С. novyi.
Целевая нуклеотидная последовательность, как правило, обладает хорошей всеобъемлимостью для различных токсинотипов С. difficile, и таким образом, как правило, присутствует и может быть обнаружена у более одного токсинотипа С. difficile. Предпочтительно, чтобы целевая нуклеотидная последовательность охватывала по меньшей мере три, более предпочтительно по меньшей мере пять, по меньшей мере семь, по меньшей мере десять, по меньшей мере пятнадцать, по меньшей мере двадцать, по меньшей мере двадцать пять, по меньшей мере тридцать, по меньшей мере тридцать пять, наиболее оптимально все токсинотипы С. difficile. Токсинотипы С. difficile включают токсинотипы, I, II, IIIa, IIIb, IIIc, IV, V, VI, VII, VIII, IX, X, XIa, XIb, XII, XIII, XIV, XV, XVI, XVII, XVIII; XIX, XX, XXI; XXII; XXIII; XXIV, XXV, XXVI, XXVII, XXVIII, XXIX, XXX, XXXI, XXXII и XXXIII и любые дополнительные токсинотипы, описанные в данной области техники или существующие в природе. Как правило, целевая нуклеотидная последовательность является всеобъемлющей для токсинотипов С. difficile, для которых обнаружена клиническая значимость в расстройствах, связанных с инфекцией, вызванной С. difficile.
Целевая нуклеотидная последовательность, как правило, имеет высокое содержание GC-пар по сравнению со средним содержанием GC-пар в геноме С. difficile, которое составляет 29,1% для референтного штамма С. difficile 630. Целевая нуклеотидная последовательность может содержать GC-пар по меньшей мере 30%, более предпочтительно по меньшей мере 31%, по меньшей мере 32% или по меньшей мере 33%. В случае, когда целевая нуклеотидная последовательность присутствует в гене tcdA или гене tcdB, среднее содержание GC-пар в этих генах составляет 27%, и таким образом, предпочтительные содержания GC-пар, упомянутые выше, также больше по сравнению со средним значением для этих генов. Содержание GC-пар в целевой нуклеотидной последовательности также выбирают с учетом необходимости связывания праймеров и плавления целевой последовательности при применяемых изотермических температурных условиях.
Целевая нуклеотидная последовательность или ампликон имеет достаточную длину для обеспечения специфического обнаружения токсикогенной С. difficile и для гибридизации подходящим образом прямого и обратного праймеров и внедряющегося в цепь олигонуклеотида с различными частями целевой последовательности. Предпочтительно, чтобы ампликон был длиною по меньшей мере 45 нуклеотидов, более предпочтительно по меньшей мере 50, по меньшей мере 55 или по меньшей мере длиною 60 нуклеотидов, как измерено от 5'-сайта связывания прямого праймера до 5'-сайта связывания обратного праймера.
Целевая нуклеотидная последовательность может присутствовать в любой области генома С. difficile при условии, что обладает необходимыми характеристиками для специфического обнаружения токсикогенной С. difficile, как описано выше. Целевая нуклеотидная последовательность может присутствовать в некодирующей области ДНК специфичной для токсикогенной С. difficile или кодирующей области ДНК специфичной для токсикогенной С. difficile. Целевая нуклеотидная последовательность может присутствовать в патогенном локусе С. difficile. Предпочтительно, чтобы целевая нуклеотидная последовательность присутствовала в гене tcdA или гене tcdB С. difficile. Другие подходящие целевые гены могут включать гены tcdC, tcdE, tcdR внутри PaLoc или гены бинарных токсинов С. difficile. Последовательности доступны по перечисленным номерам доступа для полного генома штамма Clostridium difficile 630 (GenBank: АМ180355.1), гена tcdA (АМ180355.1: 795843-803975), гена tcdB (АМ180355.1: 787393-794493).
Целевая нуклеотидная последовательность предпочтительно содержит последовательность SEQ ID NO: 1 или вариант указанной последовательности (для обнаружения гена tcdA) или последовательность SEQ ID NO: 6 или вариант указанной последовательности (для обнаружения гена tcdB). Следует понимать, что целевая нуклеотидная последовательность является дуплексом, содержащим смысловую цепь, представляющую последовательность SEQ ID NO: 1 или вариант указанной последовательности или SEQ ID NO: 6 или вариант указанной последовательности, и комплементарную несмысловую цепь. Прямой и обратные праймеры, применяемые для амплификации целевой нуклеотидной последовательности, связываются с противоположными цепями данного дуплекса.
Целевая нуклеотидная последовательность может содержать встречающиеся в природе варианты последовательности SEQ ID NO: 1 или SEQ ID NO: 6, которые присутствуют у различных токсинотипов референтного штамма С. difficile 630. Встречающиеся в природе варианты последовательности SEQ ID NO: 1 или SEQ ID NO: 6 обнаружены в известных последовательностях различных токсинотипов С. difficile, и, например, соответствующие последовательности у некоторых известных токсинотипов содержат 1, 2 или 3 несовпадений нуклеотидов с последовательностью SEQ ID NO: 1. Еще не секвенированные токсинотипы также могут содержать несовпадения нуклеотидов с последовательностью SEQ ID NO: 1 или SEQ ID NO: 6. Авторы настоящего изобретения с удивлением показали, что способ согласно настоящему изобретению является всеобъемлющим для обнаружения широкого диапазона токсинотипов, даже при наличии таких несовпадений нуклеотидов.
Варианты последовательности SEQ ID NO: 1 или SEQ ID NO: 6 могут содержать участок, который частично или полностью комплементарен по меньшей мере 35 смежным нуклеотидам, более часто по меньшей мере 40, предпочтительно по меньшей мере 45 или по меньшей мере 50 смежным нуклеотидам последовательности SEQ ID NO: 1 или SEQ ID NO: 6. Варианты могут содержать участок, имеющий 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12 или более несовпадений нуклеотидов (замен нуклеотидов) по отношению к участку соответствующей оригинальной целевой последовательности SEQ ID NO: 1 или SEQ ID NO: 6. Таким образом, например, варианты могут содержать участок длиною по меньшей мере 35 нуклеотидов, который имеет 1, 2, 3, 4, 5, или 6 несовпадений нуклеотидов, например, 1-3 или 1-5 несовпадений нуклеотидов, с соответствующим участком по меньшей мере в 35 смежных нуклеотидов соответствующей оригинальной целевой последовательности. Варианты могут содержать участок длиною по меньшей мере 40, 45 или 50 нуклеотидов, который имеет 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10, например, 1-5 или 1-8 несовпадений нуклеотидов с соответствующим участком эквивалентной длины соответствующей исходной целевой последовательности. Любые несовпадения нуклеотидов в последовательности варианта могут быть на расстоянии по меньшей мере 2, по меньшей мере 4, по меньшей мере 5 или по меньшей мере 10 нуклеотидов друг от друга.
Альтернативно варианты могут содержать участок длиною по меньшей мере 35, 40 или 45 нуклеотидов, который полностью комплементарен оригинальной целевой последовательности.
Более предпочтительно, целевая нуклеотидная последовательность содержит последовательность SEQ ID NO: 1 или SEQ ID NO: 6 или состоит из последовательности SEQ ID NO: 1 или SEQ ID NO: 6 и комплементарной антисмысловой цепи.
Более одной целевой нуклеотидной последовательности можно обнаружить посредством способа согласно настоящему изобретению при условии наличия двух и более наборов прямого праймера, обратного праймера и внедряющегося в цепь олигонуклеотида, каждый набор адаптирован для обнаружения разной целевой нуклеотидной последовательности. Например, способ согласно настоящему исследованию может определять оба гена tcdB и tcdA.
Прямой и обратный праймеры
Подходящие прямой и обратный праймеры выбирают на основе интересующей целевой нуклеотидной последовательности и с учетом сайта связывания внедряющегося в цепь олигонуклеотида, который приводит по меньшей мере часть целевой нуклеотидной последовательности в одноцепочечное состояние, позволяющее связываться прямому и обратному праймерам.
Прямой и обратный праймеры содержат последовательность, которая частично или полностью комплементарна целевой последовательности и при необходимости 5' и/или 3'-фланкирующей некомплементарной последовательности. Альтернативно прямой и обратный праймеры могут целиком состоять из последовательности, частично или полностью комплементарной целевой последовательности. Длина последовательности праймера, которая комплементарна целевой последовательности, достаточна для обеспечения специфической гибридизации с целевой нуклеотидной последовательностью. Длина комплементарной последовательности, как правило, составляет по меньшей мере 10 нуклеотидов, более предпочтительно по меньшей мере 15, по меньшей мере 16 или по меньшей мере 17 нуклеотидов. Длина комплементарной последовательности может быть 10-25, 15-25, 10-30 или 15-30 нуклеотидов.
Следует понимать, что упомянутые выше длины последовательностей относятся к частям последовательностей праймеров, которые могут быть частично или полностью комплементарны целевой нуклеотидной последовательности. Между праймерами и целевой последовательностью могут существовать несовпадения нуклеотидов по определенным позициям, вместе с тем все же делая возможным специфическую амплификацию и обнаружение целевой последовательности, в частности, с учетом совместного применения прямого и обратного праймеров и внедряющегося в цепь олигонуклеотида для достижения амплификации. Могут существовать 1, 2, 3, 4 или 5 несовпадений нуклеотидов между комплементарным участком праймера и соответствующим участком целевой последовательности.
Предпочтительно, чтобы разработали праймер, позволяющий специфическое обнаружение токсикогенной С. difficile. Таким образом, праймер, как правило, специфически или селективно гибридизуется с комплементарной последовательностью, обнаруженной только у токсикогенной С. difficile. Однако праймер может также гибридизоваться с другими последовательностями, такими как последовательности, обнаруженные у других видов клостридий, при условии, что при применении в комбинации со вторым праймером и внедряющимся в цепь олигонуклеотидом получают специфическую амплификацию последовательности, обнаруженной только у токсикогенной С. difficile.
Специфическая или селективная гибридизация относится к связыванию праймера только с особой нуклеотидной последовательностью при заданных условиях, когда такая последовательность присутствует в нуклеиновой кислоте в образце, таком как сложная биологическая смесь, включающая тотальную клеточную и чужую ДНК или РНК. Подходящие условия гибридизации известны в данной области техники. Например, смотрите издание Sambrook, Fritsche and Maniatis "Molecular Cloning: A Laboratory Manual", 2nd Ed. Cold Spring Harbor Press (1989), которое настоящим полностью включено посредством ссылки. Подходящие условия гибридизации также представлены в Примерах ниже. Как известно специалистам, подходящие условия гибридизации могут варьироваться в зависимости от длины пробы и состава ее оснований. Гибридизацию, как правило, выполняют при той же температуре, что и амплификацию, и таким образом, она тоже зависит от профиля активности применяемых ферментов полимеразы и рекомбиназы.
Как правило, прямой и обратный праймеры имеют общую длину менее 30 нуклеотидов, более предпочтительно длину менее 25 нуклеотидов, например, длину 15-25 или 15-23 нуклеотидов. Особенно предпочтительно, чтобы применяли праймеры длиною менее 30 нуклеотидов в случаях, когда применяют рекомбиназу для внедрения в цепь. Данные праймеры не способны служить субстратами для рекомбиназ.
Прямой праймер связывается с 5'-областью на одной из цепей дуплекса целевой нуклеотидной последовательности в позиции проксимальной или перекрывающейся с 5'-сайтом связывания внедряющегося в цепь олигонуклеотида. Обратный праймер связывается с 5'-областью противоположной по отношению к прямому праймеру цепи дуплекса целевой нуклеотидной последовательности в позиции проксимальной или перекрывающейся с 3'-сайтом связывания внедряющегося в цепь олигонуклеотида. 5'-сайты связывания прямого и обратного праймера, как правило, находятся на расстоянии по меньшей мере 45 нуклеотидов, более предпочтительно по меньшей мере 50, по меньшей мере 55 или по меньшей мере 60 нуклеотидов друг от друга на дуплексе целевой последовательности.
Прямой и/или обратный праймер может иметь участок последовательности, перекрывающийся с последовательностью внедряющегося в цепь олигонуклеотида. Участок перекрывающихся последовательностей, как правило, составляет 1-8 нуклеотидов в длину и может быть по меньшей мере 5 или по меньшей мере 6 нуклеотидов в длину. Обратный праймер также может иметь участок последовательности длиною 1-8 нуклеотидов, перекрывающийся с последовательностью внедряющегося в цепь олигонуклеотида.
Альтернативно может не существовать перекрывания последовательностей прямого и/или обратного праймера и внедряющегося в цепь олигонуклеотида, при этом праймер связывается в позиции, проксимальной в целевой последовательности по отношению к сайту связывания внедряющегося в цепь олигонуклеотида.
В случае, когда праймер связывается проксимально по отношению к внедряющемуся в цепь олигонуклеотиду, как правило, есть 25 нуклеотидов или менее, более предпочтительно 20 нуклеотидов или менее, 15 нуклеотидов или менее или 10 нуклеотидов или менее между соответствующим сайтом связывания внедряющегося в цепь олигонуклеотида и 5'-концом праймера. Это гарантирует, что праймер способен гибридизоваться с одноцепочечным участком, созданным в результате связывания внедряющегося в цепь олигонуклеотида.
Специфические примеры подходящих прямых и обратных праймеров для связывания с целевыми нуклеотидными последовательностями в генах tcdA и tcdB С. difficile приводятся здесь. Предпочтительными прямым и обратным праймерами для обнаружения целевой последовательности SEQ ID NO: 1 гена tcdB являются праймеры с последовательностями SEQ ID NO: 2 и SEQ ID NO: 3 и вариантами указанных последовательностей. Предпочтительными прямым и обратным праймерами для обнаружения целевой последовательности SEQ ID NO: 6 гена tcdA являются праймеры с последовательностями SEQ ID NO: 7 и SEQ ID NO: 8 и вариантами указанных последовательностей.
Варианты последовательностей SEQ ID NO: 2, 3, 7 и 8 могут быть олигонуклеотидами длиною до 30 нуклеотидов, содержащие участок, который частично или полностью комплементарен по меньшей мере 10 смежным нуклеотидам соответствующей оригинальной последовательности праймера SEQ ID NO: 2, 3, 7 или 8. Предпочтительно, чтобы указанные варианты содержали участок, который частично или полностью комплементарен по меньшей мере 11, 12, 13, 14 или 15 смежным нуклеотидам соответствующей оригинальной последовательности праймера SEQ ID NO: 2, 3, 7 или 8. В случае, когда оригинальная последовательность праймера длиннее 16 нуклеотидов, например, длиною 21 нуклеотид (SEQ ID NO: 2), варианты могут соответственно содержать участок, который частично или полностью комплементарен 16, 17, 18, 19 или 20 смежным нуклеотидам такой последовательности.
Упомянутые выше варианты могут содержать участок, который имеет 1, 2, 3, 4 или 5 несовпадений нуклеотидов (замен нуклеотидов) по отношению к соответствующему участку оригинальной последовательности праймера (и следовательно, целевой последовательности), и тем самым быть частично комплементарным ему. Таким образом, например, варианты могут содержать участок длиною по меньшей мере 10 нуклеотидов, который имеет 1, 2 или 3 несовпадения нуклеотидов, например, 1-2 несовпадения нуклеотидов с соответствующим участком по меньшей мере в 10 смежных нуклеотидов соответствующей оригинальной последовательности праймера. Варианты могут содержать участок длиною по меньшей мере 13, 14 или 15 нуклеотидов, который имеет 1, 2, 3, 4 или 5 несовпадений нуклеотидов, например, 1-3 несовпадения нуклеотидов с соответствующим участком эквивалентной длины в соответствующей оригинальной последовательности праймера. Любые несовпадения нуклеотидов в варианте последовательности праймера могут быть на расстоянии по меньшей мере 2, по меньшей мере 4, по меньшей мере 5 или по меньшей мере 10 нуклеотидов друг от друга.
Альтернативно варианты могут содержать участок длиною по меньшей мере 10, 11, 12, 13, 14 или 15 нуклеотидов, который полностью комплементарен оригинальной последовательности праймера.
Варианты последовательностей SEQ ID NO: 2, 3, 7 и 8 могут также быть олигонуклеотидами длиною до 30 нуклеотидов, которые обладают по меньшей мере 70% идентичности по отношению к соответствующей оригинальной последовательности праймера, предпочтительно по меньшей мере 75%, по меньшей мере 80%, более предпочтительно по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95% идентичности по отношению к последовательности.
Дополнительно варианты праймеров могут содержать 5' или 3'-фланкирующую нуклеотидную последовательность из гена tcdA или tcdB по отношению к области связывания оригинальных праймеров, например, 5-10 нуклеотидов из 5'-фланкирующей области и/или 3'-области. Варианты праймеров могут дополнительно содержать последовательность, не имеющую отношение к целевой последовательности.
Любой прямой или обратный праймер, применяемый в настоящем изобретении, может содержать один или несколько модифицированных нуклеотидов и/или детектируемую метку, например, флуоресцентный краситель.
Внедряющийся в цепь олигонуклеотид
Подходящий внедряющийся в цепь олигонуклеотид выбирают на основе интересующей целевой нуклеотидной последовательности и с учетом сайта связывания прямого и обратного праймеров и требования для внедряющегося в цепь олигонуклеотида приводить целевую нуклеотидную последовательность в одноцепочечное состояние в соответствующих областях, что позволяет связываться прямому праймеру и обратному праймеру.
Внедряющийся в цепь олигонуклеотид содержит последовательность, которая комплементарна целевой последовательности, и при необходимости дополнительной(ым) фланкирующей(им) некомплементарной(ым) последовательности(ям). Длина последовательности, комплементарной целевой последовательности, может быть определена специалистом эмпирически, и она является достаточной для обеспечения эффективного внедрения в цепь в месте целевой нуклеотидной последовательности при необходимости при изотермических условиях. Комплементарная последовательность может содержать РНК-ДНК комплементарное спаривание оснований и модифицированные нуклеотиды. Как правило, длина комплементарной последовательности составляет по меньшей мере 25 или по меньшей мере 27 нуклеотидов, часто по меньшей мере 30 нуклеотидов, например, по меньшей мере 32, по меньшей мере 33 или по меньшей мере 35 нуклеотидов, более предпочтительно по меньшей мере 36, 37, 38, 39 или 40 нуклеотидов или более. Длина комплементарной последовательности может быть 30-50, 32-50, 35-50, 40-50, от 35 до 48, от 35 до 46, от 38 до 45 или от 40 до 45 нуклеотидов.
Следует понимать, что упомянутые выше длины последовательности относятся к части последовательности внедряющегося в цепь олигонуклеотида, которая может быть частично или полностью комплементарна целевой нуклеотидной последовательности. Несовпадения нуклеотидов между внедряющимся в цепь олигонуклеотидом и целевой последовательностью могут существовать по определенным позициям, вместе с тем все же делая возможным специфическую амплификацию и обнаружение целевой последовательности, в частности, с учетом совместного применения прямого и обратного праймеров и внедряющегося в цепь олигонуклеотида для достижения амплификации. Может существовать 1, 2, 3, 4, 5, 6, 7 или 8 несовпадений нуклеотидов между комплементарным участком внедряющегося в цепь олигонуклеотида и соответствующим участком целевой последовательности в зависимости от общей длины комплементарной последовательности.
Предпочтительно, чтобы разработали комплементарную последовательность внедряющегося в цепь олигонуклеотида, позволяющую специфическое обнаружение токсикогенной С. difficile. Таким образом, внедряющийся в цепь олигонуклеотид предпочтительно специфически или селективно гибридизуется с комплементарной последовательностью, обнаруженной только у токсикогенной С. difficile. Однако внедряющийся в цепь олигонуклеотид может также гибридизоваться с другими последовательностями, такими как последовательности, обнаруженные у других видов клостридий, при условии, что при применении в комбинации с праймерами получают специфическую амплификацию последовательности, обнаруженной только у токсикогенной С. difficile.
Комплементарная последовательность внедряющегося в цепь олигонуклеотида гибридизуется с частью целевой последовательности, находящейся между сайтами связывания прямого и обратного праймеров (и, как правило, перекрываясь с одним или более из них). Внедряющийся в цепь олигонуклеотид может иметь участок в 1-8 нуклеотидов, например, участок длиною по меньшей мере 5 или по меньшей мере 6 нуклеотидов, перекрывающийся с прямыми и/или обратными праймерами.
5'-часть комплементарной последовательности внедряющегося в цепь олигонуклеотида, как правило, связывается в пределах 25 нуклеотидов или менее, более предпочтительно 20 нуклеотидов или менее от 5'-границы разрушаемого дуплекса целевой нуклеотидной последовательности (ампликона).
Внедряющийся в цепь олигонуклеотид при необходимости дополнительно содержит участок(ки) последовательности, некомплементарный(ые) целевой последовательности, который(ые) фланкирует(ют) участок комплементарной последовательности. Внедряющийся в цепь олигонуклеотид может содержать некомплементарную 5'-область, которая может быть любой нуклеотидной последовательности. 5'-некомплементарная область, как правило, длиною по меньшей мере 3 нуклеотида, более часто по меньшей мере 6, по меньшей мере 8, предпочтительно по меньшей мере 10, по меньшей мере 12 или по меньшей мере 14 нуклеотидов. 5'-некомплементарная область может содействовать связыванию рекомбиназы. Внедряющийся в цепь олигонуклеотид может содержать 3'-некомплементарную область, как правило, длиною 1-3 нуклеотида, содержащую нуклеотиды, блокирующие удлинение цепи полимеразой, например, invdT.
Внедряющийся в цепь олигонуклеотид, как правило, длиною по меньшей мере 30 нуклеотидов в случаях, когда применяют рекомбиназу совместно с олигонуклеотидом. Внедряющийся в цепь олигонуклеотид предпочтительно длиною по меньшей мере 35, по меньшей мере 40 или по меньшей мере 45 нуклеотидов, более предпочтительно по меньшей мере 50 и может быть длиною по меньшей мере 55 нуклеотидов или больше. Внедряющийся в цепь олигонуклеотид может быть длиною 40-70, 45-70, 45-70, 50-70, 55-70, 45-65, 50-65, 50-60 или 55-65 нуклеотидов.
Как правило, внедряющийся в цепь олигонуклеотид имеет неудлиняемый 3'-конец, такой, что он не может служить субстратом для амплификации ДНК, и целевая последовательность амплифицируется только тогда, когда дополнительное связываются специфические прямой и обратный праймеры. Это предупреждает образование неспецифических продуктов амплификации. Внедряющийся в цепь олигонуклеотид может содержать один, два, три, четыре, пять, шесть, семь, восемь или более модифицированных нуклеотидов в своей 3'-области, например, в 10-15 или 10-20 нуклеотидах от 3'-конца. Внедряющийся в цепь олигонуклеотид может содержать 3'-модификацию 3'-концевого нуклеотида и, возможно, дидезоксинуклеотид или содержать 3'-аминоаллильную группу, 3'-углеродный спейсер, 3'-фосфат, 3'-биотин, 3'-сиалил или 3'-тиол. 3'-нуклеотид может быть нуклеотидом, встроенным в обратной ориентации посредством 3'-3'-связи. Альтернативно или дополнительно 3'-область внедряющегося в цепь олигонуклеотида может содержать нуклеотиды с низкой субстратной ценностью для ДНК-полимеразы, например, нуклеотиды ПНК (пептидной нуклеиновой кислоты), ЗНК (закрытая нуклеиновая кислота), ДНК с 2'-5'-связями или 2'-O-метил-РНК или комбинации их.
В случаях, когда внедряющийся в цепь олигонуклеотид является олигомером ПНК, состоящий целиком из ПНК, такой олигонуклеотид может дестабилизировать и внедряться в ДНК-дуплекс в отсутствии фермента рекомбиназы. Таким образом, при использовании олигонуклеотида ПНК способ согласно настоящему изобретению можно выполнять без фермента рекомбиназы.
Специфические примеры подходящих внедряющихся в цепь олигонуклеотидов для целевой нуклеотидной последовательности в генах tcdA и tcdB С. difficile приводятся здесь. Предпочтительным внедряющимся в цепь олигонуклеотидом для обнаружения целевой последовательности SEQ ID NO: 1 гена tcdB является последовательность SEQ ID NO: 4. Особенно предпочтительным внедряющимся в цепь олигонуклеотидом является модифицированное производное последовательности SEQ ID NO: 4, наиболее предпочтительно последовательность SEQ ID NO: 5. Предпочтительным внедряющимся в цепь олигонуклеотидом для обнаружения целевой последовательности SEQ ID NO: 6 гена tcdA является последовательность SEQ ID NO: 9. Особенно предпочтительным внедряющимся в цепь олигонуклеотидом является модифицированное производное последовательности SEQ ID NO: 9, наиболее предпочтительно последовательность SEQ ID NO: 10.
Как отмечалось выше, предпочтительно, чтобы внедряющийся в цепь олигонуклеотид, применяемый в настоящем изобретении, содержал один или более модифицированных олигонуклеотидов в своей 3'-области, чтобы предотвратить его использование в качестве субстрата для полимеразы. Таким образом модифицированное производное последовательности SEQ ID NO: 4 или 9 может содержать один, два, три, четыре, пять, шесть, семь, восемь или более модифицированных нуклеотидов в своей 3'-области, как правило, в 10-15 или 10-20 нуклеотидах от 3'-конца. Модификации могут быть выбраны любые из рассмотренных выше. Модифицированное производное может быть олигомером ПНК с последовательностью, соответствующей последовательности SEQ ID NO: 4 или 9.
В дополнении к модифицированным производным последовательностей SEQ ID NO: 4 и 9 можно использовать варианты внедряющегося в цепь олигонуклеотида.
Варианты последовательностей SEQ ID NO: 4 и 9 являются, как правило, олигонуклеотидами длиною более 30 нуклеотидов, более предпочтительно по меньшей мере 35, по меньшей мере 40 или длиною по меньшей мере 45 нуклеотидов, содержащими участок, который частично или полностью комплементарен по меньшей мере 30 смежным нуклеотидам соответствующей оригинальной комплементарной целевой последовательности, присутствующей в последовательности SEQ ID NO: 4 или 9. Предпочтительно, чтобы указанные варианты содержали участок, который частично или полностью комплементарен по меньшей мере 32, 35, 37, 40, 42 или 45 смежным нуклеотидам соответствующей оригинальной комплементарной целевой последовательности, присутствующей в последовательности SEQ ID NO: 4 или 9.
Упомянутые выше варианты могут содержать участок, который имеет 1, 2, 3, 4, 5, 6, 7 или 8 несовпадений нуклеотидов (замен нуклеотидов) по отношению к соответствующему комплементарному целевой последовательности участку оригинальной последовательности внедряющегося в цепь олигонуклеотида SEQ ID NO: 4 или 9 (и следовательно, целевой последовательности), и тем самым быть частично комплементарным ему. Таким образом, например, варианты могут содержать участок длиною по меньшей мере 30 нуклеотидов, который имеет 1, 2, 3 или 4, например, 1-4 или 1-3 несовпадений нуклеотидов с соответствующим участком по меньшей мере в 40 смежных нуклеотидов соответствующего оригинального внедряющегося в цепь олигонуклеотида. Варианты могут содержать участок длиною по меньшей мере 35, 40, 42, или 45 нуклеотидов, который имеет 1, 2, 3, 4, 5 или 6, например, 1-5 или 1-3 несовпадений нуклеотидов с соответствующим участком эквивалентной длины в соответствующем оригинальном внедряющемся в цепь олигонуклеотиде. Любые несовпадения нуклеотидов в варианте последовательности внедряющегося в цепь олигонуклеотида могут находиться на расстоянии по меньшей мере 2, по меньшей мере 4, по меньшей мере 5 или по меньшей мере 10 нуклеотидов друг от друга.
Альтернативно варианты могут содержать участок длиною по меньшей мере 32, 35, 37, 40, 42 или 45 нуклеотидов, который полностью комплементарен участку, комплементарному целевой последовательности, оригинального внедряющегося в цепь олигонуклеотида.
Варианты последовательностей SEQ ID NO: 4 и 9 могут также быть олигонуклеотидами длиною более 30 нуклеотидов, содержащими комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности, комплементарной целевой последовательности, соответствующего оригинального внедряющегося в цепь олигонуклеотида, предпочтительно по меньшей мере 75%, по меньшей мере 80%, более предпочтительно по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95% идентичности по отношению к последовательности.
Дополнительно варианты внедряющегося в цепь олигонуклеотида могут содержать дополнительную последовательность, комплементарную 5' или 3'-фланкирующей нуклеотидной последовательности гена tcdA или tcdB по отношению к области связывания оригинального внедряющегося в цепь олигонуклеотида, например, 5-10 или 5-15 нуклеотидов из 5'-фланкирующей области и/или 3'-области.
Оставшаяся последовательность вариантов внедряющегося в цепь олигонуклеотида, как правило, не имеет отношения к целевой последовательности, а также, как правило, не имеет отношения к оригинальному внедряющемуся в цепь олигонуклеотиду.
Варианты внедряющегося в цепь олигонуклеотида дополнительно содержат один или более модифицированных олигонуклеотидов в своей 3'-области, например, два, три, четыре, пять, шесть, семь, восемь или более модифицированных нуклеотидов, которые могут быть в 10-15 или 10-20 нуклеотидах от 3'-конца. Модификации могут быть выбраны любые из рассмотренных ранее.
Внедряющийся в цепь олигонуклеотид согласно настоящему изобретению может дополнительно содержать детектируемую метку, например, флуоресцентную метку.
Амплификация целевой нуклеотидной последовательности
С целью обнаружения целевой нуклеотидной последовательности с нуклеиновой кислотой, выделенной из образца, контактируют прямой и обратный праймеры и внедряющийся в цепь олигонуклеотид в условиях, способствующих амплификации целевой нуклеотидной последовательности.
Такие условия, как правило, включают наличие фермента ДНК-полимеразы. Подходящие условия включают любые условия, применяемые для обеспечения активности ферментов полимеразы, известных в данной области техники.
Условия, как правило, включают наличие всех четырех дНТФ, дАТФ, дТТФ, дЦТФ и дГТФ или их аналогов, подходящих буферных агентов/pH и другие факторы, необходимые для работы ферментов или их стабильности. Условия могут включать наличие детергентов и стабилизирующих агентов. Применяемая температура является, как правило, изотермической, то есть постоянной в ходе процесса амплификации. Применяемая температура, как правило, зависит от природы фермента полимеразы и других ферментных компонентов, а также отражает температуру гибридизации, необходимую для праймеров и внедряющихся в цепь олигонуклеотидов. В случае применения Bsu-полимеразы подходящая температура составляет 40 градусов Цельсия.
Как правило, применяемая полимераза имеет специфическую активность, проявляющуюся в замещении цепей. Термин «замещение цепей» используется здесь для описания способности ДНК-полимеразы при необходимости в сочетании с акцессорными белками замещать комплементарные цепи при встрече с областью двухцепочечной ДНК в ходе синтеза ДНК. Подходящие ДНК-полимеразы включают polI из Е. coli, В. subtilis или В. stearothermophilus и их функциональные фрагменты или варианты и ДНК-полимеразы фагов Т4 и Т7 и их функциональные фрагменты или варианты. Предпочтительной полимеразой является Bsu ДНК-полимераза или ее функциональный фрагмент или вариант.
Условия могут дополнительно включать наличие рекомбиназы. В способе согласно настоящему изобретению можно применять любую рекомбиназную систему. Рекомбиназная система может быть прокариотического или эукариотического происхождения и может быть от бактерий, дрожжей, фагов или млекопитающих. Рекомбиназа может полимеризовать на одноцепочечном олигонуклеотиде в направлении 5'-3' или 3'-5'. Рекомбиназа может быть выделена из фага семейства Myoviridae, например, Т4, Т2, Т6, Rb69, Aeh1, KVP40, фага Acinetobacter 133, фага Aeromonas 65, цианофага P-SSM2, цианофага PSSM4, цианофага S-PM2, Rbl4, Rb32, фага Aeromonas 25, фага Vibrio nt-1, phi-1, Rbl6, Rb43, фага 31, фага 44RR2.8t, Rb49, фага Rb3 или фага LZ2. В предпочтительном варианте реализации применяют рекомбиназу UvsX фага Т4 (номер доступа: Р04529), или ее функциональный вариант или фрагмент. Можно также применять Rad-систему эукариот или recA-Reco-систему Е. coli или других прокариотических систем.
Условия могут дополнительно включать наличие акцессорных белков рекомбиназы, таких как белок, связывающийся с одноцепочечной ДНК (например, gp32, номер доступа Р03695), и белок-грузчик рекомбиназы (например, UvsY, номер доступа NP_049799.2). В предпочтительном варианте реализации условия включают наличие белков gp32, UvsX и UvsY фага Т4.
Рекомбиназа (например, UvsX), и если применяют, белок-грузчик рекомбиназы (например, UvsY) и белок, связывающийся с одноцепочечной ДНК (например, gp32), могут быть нативными, гибридами или мутантными белками из одного или различных источников, принадлежащих фагам семейства Myoviridae. Нативный белок может быть диким типом или естественным вариантом белка.
Условия могут дополнительно включать другие факторы, применяемые для повышения эффективности рекомбиназы, такие как соединения, применяемые для контроля ДНК-взаимодействий, например, пролин, диметилсульфоксид (ДМСО) или краудинг-агенты, которые известны тем, что повышают интенсивность посадок рекомбиназ на ДНК (Lavery Р et. Al JBC 1992, 26713, 9307-9314; WO 2008/035205).
Условия могут также включать наличие системы регенерации АТФ. Специалистам в данной области техники известны различные системы регенерации АТФ, и эти системы включают гликолитические ферменты. Подходящие компоненты системы регенерации АТФ могут включать один или более креатинфосфат, креатинкиназу, миокиназу, пирофосфотазу, сахарозу и сахарозофосфорилазу. Условия могут дополнительно включать наличие АТФ.
Дополнительные компоненты, такие как ионы магния, дитиотреитол (ДТТ) и другие восстанавливающие агенты, соли, бычий сывороточный альбумин (БСА)/полиэтиленгликоль (ПЭГ) или другие краудинг-агенты, также могут быть включены.
Рассмотренные выше различные компоненты, включая праймеры и внедряющийся в цепь олигонуклеотид, могут присутствовать в различных концентрациях с целью обеспечения амплификации ДНК. Специалист может выбрать подходящие рабочие концентрации различных компонентов на практике.
Обнаружение наличия амплифицированной ДНК
Наличие амплифицированной ДНК в результате приведения целевой нуклеотидной последовательности в контакт с праймерами и внедряющимся в цепь олигонуклеотидом в условиях, способствующих амплификации ДНК, можно проконтролировать любым подходящим способом.
Один или оба праймера или внедряющийся в цепь олигонуклеотид может нести метку или другой детектируемый компонент. Можно использовать любую метку или детектируемый компонент. Примеры подходящих меток включают радиоизотопы или флуоресцентные метки и пары флюофора и акцептора для метода флуоресцентного резонансного переноса энергии (FRET). Альтернативно или дополнительно можно использовать один или несколько зондов, обнаруживающих амплифицированную ДНК, которые также несут метку или другой детектируемый компонент. Зонды могут связываться с любым подходящим местом на ампликоне. Зонды, обнаруживающие различные амплифицированные целевые последовательности, могут давать сигнал на различных длинах волн флуоресцентного излучения для обеспечения мультиплексного детектирования. Красители, которые интеркалируют с амплифицированной ДНК, также можно использовать для обнаружения амплифицированной ДНК, например, SYBR Green и тиазол оранжевый.
Обнаружение сигнала от амплифицированной ДНК можно осуществлять на основе любой подходящей системы, включая ПЦР в реальном времени.
Праймеры и олигонуклеотиды
Согласно настоящему изобретению дополнительно предложены праймеры и внедряющиеся в цепь олигонуклеотид, имеющие последовательности SEQ ID NO: 2-5 и 7-10 и варианты указанных последовательностей, в качестве продуктов как таковых и композиции и составы, содержащие указанные праймеры и внедряющиеся в цепь олигонуклеотиды. Праймеры и при необходимости внедряющийся в цепь олигонуклеотид можно применять в любом способе обнаружения токсикогенной С. difficile. Как правило, этим способом является способ амплификации ДНК на основе внедрения в цепь. Однако можно применять любой подходящий способ амплификации ДНК, который позволяет специфически обнаруживать токсикогенную С. difficile. Прямой и обратный праймеры можно применять в способе амплификации ДНК, который не требует применения внедряющегося в цепь олигонуклеотида, например, ПЦР.
Композиции и наборы
Согласно настоящему изобретению также предложены композиции и наборы, содержащие по меньшей мере два олигонуклеотида, выбранные из (a) прямого праймера, (b) обратного праймера и (c) внедряющегося в цепь олигонуклеотида. Прямой праймер, обратный праймер и внедряющийся в цепь олигонуклеотид описаны выше. Композиция или набор могут содержать прямой и обратный праймеры, прямой праймер и внедряющийся в цепь олигонуклеотид или обратный праймер и внедряющийся в цепь олигонуклеотид. Предпочтительно, чтобы композиция или набор содержали прямой праймер, обратный праймер и внедряющийся в цепь олигонуклеотид. Композиция или набор могут подходить для обнаружения С. difficile в соответствии со способом согласно настоящему изобретению или в соответствии с альтернативным способом амплификации ДНК.
В случае, когда композиция или набор подходят для применения для обнаружения целевой нуклеотидной последовательности SEQ ID NO: 1 (ген tcdB), как правило, прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 2 или вариант указанной последовательности, обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 3 или вариант указанной последовательности, и внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, содержащим последовательность SEQ ID NO: 4 или вариант указанной последовательности и дополнительно содержащим один или более модифицированных нуклеотидов в своей 3'-области. Внедряющийся в цепь олигонуклеотид может иметь последовательность SEQ ID NO: 5.
В случае, когда композиция или набор подходят для применения для обнаружения целевой нуклеотидной последовательности SEQ ID NO: 6 (ген tcdA), как правило, прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 7 или вариант указанной последовательности, обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 8 или вариант указанной последовательности, и внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, содержащим последовательность SEQ ID NO: 9 или вариант указанной последовательности и дополнительно содержащим один или более модифицированных нуклеотидов в своей 3'-области. Внедряющийся в цепь олигонуклеотид может иметь последовательность SEQ ID NO: 10.
Композиция или набор могут предоставлять первый комплект олигонуклеотидов, позволяющий обнаруживать целевую нуклеотидную последовательность SEQ ID NO: 1, и дополнительно второй комплект олигонуклеотидов, позволяющий обнаруживать целевую нуклеотидную последовательность SEQ ID NO: 6.
Упомянутая выше композиция может быть, например, раствором, лиофилизатом, суспензией или эмульсией в масляном или водной растворителе.
В упомянутом выше наборе по меньшей мере два олигонуклеотида могут быть представлены в виде смеси или в отдельных емкостях. Набор при необходимости дополнительно содержит инструкцию по применению в соответствии со способом согласно настоящему изобретению. Набор может содержать средства для обнаружения амплифицированной ДНК.
Набор или композиция при необходимости содержит один или более зондов для обнаружения амплифицированной ДНК. Набор или композиция при необходимости содержит одну или более ДНК-полимеразу, рекомбиназу и акцессорные белки рекомбиназы. Предпочтительно, чтобы ДНК-полимераза была Bsu-полимеразой. Предпочтительно, чтобы рекомбиназой была рекомбиназа UvsX бактериофага Т4, при необходимости в сочетании с акцессорными белками рекомбиназы UvsY и gp32. Набор или композиция могут дополнительно содержать дНТФ, подходящие буферы или другие факторы, необходимые для амплификации ДНК в соответствии со способом согласно настоящему изобретению, описанном выше.
Диагностика инфекции, вызванной С. difficile, и медицинское применение
Настоящее изобретение особенно выгодно для медицинской области. Способы обнаружения согласно настоящему изобретению обеспечивают высокоспецифичный анализ, позволяющий определить, содержит ли клинический образец целевую нуклеотидную последовательность из токсикогенной С. difficile. Способ можно применять к целому ряду болезней, связанных с токсикогенной С. difficile. Дополнительно способ можно применять для скрининга переносчиков токсикогенной С. difficile.
Обнаружение наличия или отсутствия токсикогенной С. difficile можно проводить при любой болезни или заболевании, имеющейся или подозреваемой на наличие у пациента. Подобные болезни могут включать те, что вызваны, связаны или обостренны наличием токсикогенной С. difficile. Таким образом, пациент может демонстрировать симптомы, указывающие на присутствие токсикогенной С. difficile, и от пациента можно получить образец, чтобы определить наличие С. difficile и при необходимости также токсинотип С. difficile способом, описанным выше.
Таким образом, согласно настоящему изобретению предложен способ диагностики инфекции, вызванной токсикогенной С. difficile у пациента, включающий обнаружение наличия целевой нуклеотидной последовательности токсикогенной С. difficile согласно настоящему изобретению в образцы от указанного пациента. Способ может дополнительно включать другие этапы идентификации штамма токсикогенной С. difficile, например, по микробиологической культуре из образца, полученного от пациента.
Особенно предпочтительным вариантом реализации настоящего изобретения является идентификация наличия токсикогенной С. difficile у пациентов с инфекцией желудочно-кишечного тракта, особенно, с симптомами диареи.
Таким образом, согласно настоящему изобретению предложен способ диагностики желудочно-кишечных заболеваний, например, диареи, вызванной токсикогенной С. difficile. Способ диагностики может дополнительно включать обнаружение маркеров устойчивости к антибиотикам и маркеров вирулентности. Данный способ обеспечивает резкое улучшение ведения пациента с желудочно-кишечным заболеванием, потому что обеспечивает возможность оптимального терапевтического лечения данного пациента. Вследствие этого, анализ снизит продолжительность госпитализации, частоту повторных госпитализаций и снизит стоимость.
Способ диагностики можно для удобства провести на основе нуклеиновой кислоты, выделенной из образца пациента, что укажет врачам-клиницистам, вызвана ли желудочно-кишечное заболевание заражением токсикогенной С. difficile. Способ диагностики может также предоставлять информацию о токсинотипе и вирулентности С. difficile и об устойчивости С. difficile к каким-либо антибиотикам. В зависимости от результата анализа медицинское лечение можно затем оптимизировать, например, путем применения антибиотиков.
ПРИМЕРЫ
Пример 1. РАЗРАБОТКА АНАЛИЗОВ ГЕНОВ tcdB И tcdA C. difficile
Разработка специфических олигонуклеотидов для обнаружения и амплификации генов of tcdA и tcdB вызывала сложности. Гены tcdA и tcdB показывают высокую степень гомологии не только между собой, но также и с другими крупными клостридиальными токсинами. Цитотоксиновый ген C. sordellii, tcsL, является ближайшим гомологом гена tcdB (Popoff, 1987; Green, 1995). Антитела к токсину tcsL С. sordellii перекрестно реагируют с токсином В С. difficile. Таким образом, ИФА-тесты на тест-полосках для обнаружения токсина В С. difficile обычно дают перекрестную реакцию с токсином tcsL С. sordellii. Разработка специфических олигонуклеотидов была дополнительно затруднена необходимостью подбора специфического включающегося в цепь олигонуклеотида.
Более того, содержание GC-nap в геноме С. difficile низкое (29,1%), что также применимо к гену tcdB, содержание GC-пар в котором 27,4%. Такое низкое содержание GC-пар также вызывало трудности в разработке олигонуклеотидов, подходящих для амплификации ДНК. Праймеры с низким содержанием GC-пар будут иметь низкую температуру плавления (Tm), что дестабилизирует связывание олигонуклеотидов и приводит к низкой эффективности амплификации.
Во-первых, выбирали подходящие участки генов tcdA и tcdB, чтобы сделать их мишенями для обнаружения и амплификации. Отдельные целевые участки гена tcdB (SEQ ID NO: 1) и гена tcdA (SEQ ID NO: 6) показаны ниже. Выбирали целевые участки, 1) специфические для С. difficile, то есть отличающиеся от сильно гомологичных видов клостридий, 2) показывающие хорошую всеобъемлемость для токсинотипов С. difficile и 3) имеющие более высокое содержание GC-пар, чем в среднем по геному С. difficile.
Во-вторых, специфические олигонуклеотиды разрабатывали для амплификации целевых участков. Отдельные олигонуклеотиды, применяемые для обнаружения и амплификации гена tcdB и гена tcdA С. difficile показаны ниже (SEQ ID NO: 2-5 и 7-10).
Пример 2. ПРОВЕРКА АНАЛИЗА НА ГЕН tcdB
Олигонуклеотиды для обнаружения гена tcdB с последовательностями SEQ ID NO: 2, 3 и 5 применяли для амплификации гена tcdB С. difficile путем изотермической амплификации ДНК с внедрением в цепь при 40°С. Флуоресцентный краситель SybrGreen I, связывающийся с ДНК, применяли для обнаружения амплификации и флуоресценцию измеряли либо флуорометром, либо прибором для ПЦР в реальном времени. Реакционная смесь содержала рабочий буфер (10 мМ Трис-ацетата, pH 8, 0,5 мМ ЭДТК, 4 мМ ДТТ, 150 мМ сахарозы, 0,1 мг/мл БСА, 5% ДМСО), 2 мМ АТФ, 5% ПЭГ-1000, 60 мМ ди-(трис) соль креатинфосфата, дНТФ (с дТТФ замененной на дУТФ), 0,0125 единиц/мкл сахарозофосфорилазы, 0,025 единиц/мкл креатинфосфокиназы, 0,0625 единиц/мкл Bsu-полимеразы, 0,1-0,3 мг/мл рекомбиназы UvsX бактериофага Т4, 0,25-0,5 мг/мл белка gp32 бактериофага Т4, 10 мМ ацетата магния и олигонуклеотиды в присутствии или отсутствии матричной ДНК.
Способность концевых праймеров и внедряющегося олигонуклеотида образовывать димеры или самими быть праймерами друг для друга проверяли в отсутствии матричной ДНК (контроль без матрицы). Способность олигонуклеотидов обнаруживать и амплифицировать правильную целевую ДНК проверяли в присутствии геномной ДНК штамма С. difficile ВАА-1382. Присутствие или отсутствие продукта амплификации определяли по увеличению флуоресценции SybrGreen I (Фиг. 1А) и с помощью анализа кривой плавления (Фиг. 1В).
Продукты амплификации дополнительно анализировали системой электрофореза на микрочипе MultiNA (Фиг. 1С). Электрофореграмма положительной реакции амплификации гена tcdB с 10000 копий геномной ДНК С. difficile в качестве матрицы показала выход фрагмента ДНК ожидаемой длины. Этот фрагмент не был обнаружен в реакции амплификации контроля без матрицы.
Пример 3. СПЕЦИФИЧНОСТЬ АНАЛИЗА ДЛЯ С. DIFFICILE
Специфичность анализа на ген tcdB проверяли, используя геномную ДНК штамма С. sordellii АТСС 9714 в качестве матрицы в реакции, описанной в Примере 2. Ген токсина tcsL С. sordellii является ближайшим обнаруженным гомологом гена tcdB. Результаты показаны на Фиг. 2А. Реакция амплификации гена tcdB с 10000 копий геномной ДНК С. sordellii в качестве матричной ДНК не показала амплификации ДНК, тогда как с 10000 копий геномной ДНК штамма С. difficile ВАА-1382 (630) показала четкую амплификацию, измеренную по повышению интенсивности флуоресценции SybrGreen I.
Пример 4. ЧУВСТВИТЕЛЬНОСТЬ ОБНАРУЖЕНИЯ C. DIFFICILE
Чувствительность анализа на ген tcdB проверяли с помощью серии разведений геномной ДНК штамма С. difficile ВАА-1382 (630) и амплификацию измеряли по флуоресценции SybrGreen I и обнаруживали посредством ПЦР в реальном времени. Результаты показаны на Фиг. 2В. 10-10000 копий геномной ДНК С. difficile, применяемой в качестве матрицы, на реакцию показали положительную амплификацию, тогда как контроль без матрицы, отрицательный контроль и реакция с 1 копией геномной ДНК на реакцию не показали амплификации. Реакция отрицательного контроля содержала смесь геномной ДНК, выделенной из Enterobacter aerogenes, Citrobacter sp., Shigella sonnei, Shigella flexneri, Streptococcus agalactiae, Listeria monocytogenes, Eschericia coli, Enterobacter aerogenes, Enterobacter cloacae, Enterococcus faecalis, Citrobacter freundii и Klebsiella pneumoniae, по меньшей мере 1000 копий каждой на реакцию.
Пример 5. ВСЕОБЪЕМЛИМОСТЬ ОБНАРУЖЕНИЯ ТОКСИНОТИПОВ C. DIFFICILE
Всеобъемлемость анализа на ген tcdB проверяли с C. difficile токсинотипов 0, III, VIII и X. Результаты показаны на Фигуре 3А и 3С. 2 нг геномной ДНК на реакцию от каждого токсинотипа применяли как матрицу в реакции амплификации гена tcdB. Все протестированные токсинотипы давали положительный результат амплификации (Фиг. 3А). Более того, анализ кривой плавления указал на амплификацию единственного специфического ампликона (Фиг. 3С).
Всеобъемлемость анализа на ген tcdB была дополнительно протестирована с большей панелью из 37 токсинотипов 0, I, II, IIIa, IIIb, IIIc, IV, V, VI, VII, VIII, IX, X, XIa, XIb, XII, XIII, XIV, XV, XVI, XVII, XVIII; XIX, XX, XXI; XXII; XXIII; XXIV, XXV, XXVI, XXVII, XXVIII, XXIX, XXX, XXXI, XXXII и XXXIII С. difficile. Результаты показаны на Фигурах 3В и 3D. Все токсинотипы, кроме XIa и X1b, итого 35, давали положительный результат амплификации (Фиг. 3В), и последующий за амплификацией анализ кривой плавления положительных реакций амплификации показал амплификацию единственного ампликона (Фиг. 3С). Последующий за амплификацией анализ кривой плавления реакций токсинотипов XIa и XIb также показал отсутствие амплификации. Отрицательный результат амплификации токсинотипов XIa и XIb предсказуем, так как они не производят токсин А или В (Rupnik et al. 2001). Они совсем не обладают геном tcdB, но содержат по меньшей мере некоторые части гена tcdA.
Пример 6. ПРОВЕРКА SIBA-АНАЛИЗА НА ГЕН tcdA
Олигонуклеотиды для обнаружения гена tcdA с последовательностями SEQ ID NO: 7, 8 и 10 применяли для амплификации гена tcdA С. difficile при 40°C с реакционной смесью, описанной в Примере 1. Способность концевых праймеров образовывать димеры или самими быть праймерами друг для друга проверяли в отсутствии матричной ДНК (контроль без матрицы). Способность олигонуклеотидов обнаруживать и амплифицировать правильную целевую ДНК проверяли в присутствии 10-10000 копий геномной ДНК штамма С. difficile ВАА-1382 на реакцию. Присутствие или отсутствие продукта амплификации обнаруживали по увеличению флуоресценции SybrGreen I (Фиг. 4А) и измеряли прибором для ПЦР в реальном времени. Анализ кривой плавления проводили, чтобы подтвердить амплификацию единственного специфического ампликона (Фиг. 4В).
ПОСЛЕДОВАТЕЛЬНОСТИ СОГЛАСНО НАСТОЯЩЕМУ ИЗОБРЕТЕНИЮ
SEQ ID NO: 1
AACCAAAGTGGAGTGTTACAAACAGGTGTATTTAGTACAGAAGATGGATTTAAATATTTTGCCCCA
SEQ ID NO: 2 AACCAAAGTGGAGTGTTACAA
SEQ ID NO: 3 TGGGGCAAAATATTTA
SEQ ID NO: 4 TCCTCCTGTACCTCGTTACAAACAGGTGTATTTAGTACAGAAGATGGATTTAAATA
SEQ ID NO: 5
TCCTCCTGTACCTCGTTACAAACAGGTGTATTTAGTACAGAAGmAmUmGmGmAmUmUmUmAmAmAmUmA/InvdT/
mX = 2'-O-метил-РНК. invdT = инвертированный дТТФ.
SEQ ID NO: 6
ATGGATAGGTGGAGAAGTCAGTGATATTGCTCTTGAATACATAAAACAATGGGCTGATATTAA
SEQ ID NO: 7 ATGGATAGGTGGAGAAGTC
SEQ ID NO: 8 TTAATCTCAGCCCATTG
SEQ ID NO: 9 TCCTCCTGTACCTCAGAAGTCAGTGATATTGCTCTTGAATACATAAAACAATGG
SEQ ID NO: 10
TCCTCCTGTACCTCAGAAGTCAGTGATATTGCTCTTGAATmAmCmAmUmAmAmAmAmCmAmAmUmGmG/InvdT/
mX = 2'-O-метил РНК. invdT = инвертированный дТТФ.
ПЕРЕЧЕНЬ ССЫЛОЧНЫХ ДОКУМЕНТОВ
Braun, V., Hundsberger, Т., Leukel, P., Sauerborn, M. and von Eichel-Streiber, C. (1996) Definition of the single integration site of the pathogenicity locus in Clostridium difficile. Gene 181 (1-2): 29-38
Carter, G.P., Rood, J.I. and Lyras, D. (2012) The role of toxin A and toxin В in the virulence of Clostridium difficile. Trends in Microbiology 20(1): 21-29
Green, G.A., 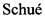 , V. and Monteil, H. (1995) Cloning and characterization of the cytotoxin L-encoding gene of Clostridium sordellii: homology with Clostridium difficile cytotoxin B. Gene 161(1): 57-61
, V. and Monteil, H. (1995) Cloning and characterization of the cytotoxin L-encoding gene of Clostridium sordellii: homology with Clostridium difficile cytotoxin B. Gene 161(1): 57-61
Lyras, D., O'Connor, J., Howarth, P.M., Sambol, S.P., Carter, G.P., Phumoonna, Т., Poon, R., Adams, V., Vedantam, G., Johnson, S., Gerding, D.N., and Rood, J.I. (2009) Toxin В is essential for virulence of Clostridium difficile. Nature 458(7242): 1176-1179
McMillin, D.E., Muldrow, L.L. and Laggette, S.J. (1990) Simultaneous detection of toxin A and toxin В genetic determinants of Clostridium difficile using the multiplex polymerase chain reaction. Canadian journal of microbiology 38(1), 81-83.
McMillin, D.E., Muldrow, L.L., Leggette, S.J., Abdulahi, Y. and Ekanemesang, U.M. (1991) Molecular screening of Clostridium difficile toxins A and В genetic determinants and identification of mutant strains. FEMS Microbiology Letters 62(1):75-80.
Popoff, M.R. (1987) Purification and characterization of Clostridium sordellii lethal toxin and cross-reactivity with Clostridium difficile cytotoxin. Infect. Immun. 55(1): 35-43
Rupnik, M., Brazier, J.S., Duerden, В.I., Grabnar, M. and Stubbs, S.L.J. (2001) Comparison of toxinotyping and PCR ribotyping of Clostridium difficile strains and description of novel toxinotypes Microbiology 147, 439-447.
von Eichel-Streiber, C., Boquet, P., Sauerborn, M. and Thelestam, M. (1996) Large clostridial cytotoxins - a family of glycosyltransferases modifying small GTP-binding proteins. Trends in Microbiology 4: 375-382
Wren, B.W., Clayton, C.L. and Tabaqchali, S. (1990) Nucleotide sequence of Clostridium difficile toxin A gene fragment and detection of toxigenic strains by polymerase chain reaction. FEMS Microbiology Letters 58(1), 1-6
| название | год | авторы | номер документа |
|---|---|---|---|
| ЧЕЛОВЕЧЕСКИЕ ГЕНЕТИЧЕСКИЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С ОТВЕТОМ НА СРЕДСТВА ДЛЯ ЛЕЧЕНИЯ, КОТОРЫЕ ЦЕЛЕНАПРАВЛЕННО ВОЗДЕЙСТВУЮТ НА ТОКСИН B CLOSTRIDIUM DIFFICILE | 2017 |
|
RU2761249C2 |
| СПОСОБ ДЕТЕКТИРОВАНИЯ И ХАРАКТЕРИЗАЦИИ ТОКСИНОГЕННОГО ШТАММА CLOSTRIDIUM DIFFICILE | 2010 |
|
RU2612789C2 |
| ОПРЕДЕЛЕНИЕ НУКЛЕИНОВЫХ КИСЛОТ ПУТЕМ АМПЛИФИКАЦИИ, ОСНОВАННОЙ НА ВСТРАИВАНИИ В ЦЕПЬ | 2014 |
|
RU2694976C1 |
| ЭЛИТНОЕ СОБЫТИЕ ЕЕ-GM3 И СПОСОБЫ И НАБОРЫ ДЛЯ ИДЕНТИФИКАЦИИ ТАКОГО СОБЫТИЯ В БИОЛОГИЧЕСКИХ ОБРАЗЦАХ | 2010 |
|
RU2639530C2 |
| Композиции и способы, имеющие отношение к мутантному токсину из Clostridium Difficile | 2013 |
|
RU2630671C2 |
| РАСТЕНИЯ СОИ, УСТОЙЧИВОЙ К ГЕРБИЦИДАМ, И СПОСОБЫ ИХ ИДЕНТИФИКАЦИИ | 2010 |
|
RU2639520C2 |
| СПОСОБЫ И НАБОРЫ ДЛЯ ВЫЯВЛЕНИЯ МУЧНИСТОЙ РОСЫ | 2016 |
|
RU2720255C1 |
| Vβ-Dβ-Jβ ОЛИГОНУКЛЕОТИД Т-КЛЕТОЧНОГО РЕЦЕПТОРА, ПАРА ПРАЙМЕРОВ, ОЛИГОНУКЛЕОТИДНЫЙ ЗОНД, СПОСОБ ОБНАРУЖЕНИЯ КЛОНА MBP83-99Vβ13.1 Т-КЛЕТОК, ЭКСПРЕССИРУЮЩИХ МОТИВ LGRAGLTY Т-КЛЕТОЧНОГО РЕЦЕПТОРА, НАБОР, СПОСОБ ЛЕЧЕНИЯ АУТОИММУННОГО ЗАБОЛЕВАНИЯ И СПОСОБ ЕГО МОНИТОРИНГА | 2000 |
|
RU2251552C2 |
| ОБЪЕКТ КУКУРУЗЫ MON 87411 | 2013 |
|
RU2648155C2 |
| Способ получения аминокислотных последовательностей, кодирующих наноантитела, нейтрализующие токсин А Clostridioides difficile | 2023 |
|
RU2833930C1 |






Изобретение относится к биотехнологии. Предложен способ обнаружения токсикогенной Clostridium difficile (C. difficile) путем амплификации ДНК на основе внедрения в цепь вместе с олигонуклеотидами, композициями и наборами, подходящими для применения в настоящем способе. Способ включает приведение указанного образца в контакт с по меньшей мере одним прямым праймером, по меньшей мере одним обратным праймером и по меньшей мере одним внедряющимся в цепь олигонуклеотидом в условиях, способствующих амплификации указанной целевой нуклеотидной последовательности. Указанный праймер и указанный внедряющийся в цепь олигонуклеотид содержит участок, комплементарный указанной целевой нуклеотидной последовательности. Указанный внедряющийся в цепь олигонуклеотид переводит по меньшей мере часть целевой нуклеотидной последовательности в одноцепочечное состояние, что позволяет связаться указанным прямому праймеру и обратному праймеру. Техническим результатом настоящего изобретения является обеспечение высокой специфичности и чувствительности способа обнаружения различных целевых последовательностей токсикогенной С. difficile. 8 н. и 11 з.п. ф-лы, 4 ил., 6 пр.
1. Способ обнаружения целевой нуклеотидной последовательности токсикогенной С. difficile в образце, причем указанный способ включает приведение указанного образца в контакт с по меньшей мере одним прямым праймером, по меньшей мере одним обратным праймером и по меньшей мере одним внедряющимся в цепь олигонуклеотидом в условиях, способствующих амплификации указанной целевой нуклеотидной последовательности,
где каждый указанный праймер и указанный внедряющийся в цепь олигонуклеотид содержит участок, комплементарный указанной целевой нуклеотидной последовательности;
где указанный внедряющийся в цепь олигонуклеотид переводит по меньшей мере часть целевой нуклеотидной последовательности в одноцепочечное состояние, что позволяет связаться указанным прямому праймеру и обратному праймеру; и
при этом указанный прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 2 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 2, и при этом указанный обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 3 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 3, и при этом указанный внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, причем указанный олигонуклеотид содержит последовательность SEQ ID NO: 4 или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 4, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 4, комплементарной целевой последовательности, и при этом указанный внедряющийся в цепь олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в 3'-области, делая ее неудлиняемой.
2. Способ по п. 1, отличающийся тем, что указанный вариант прямого праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 2, указанный вариант обратного праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 3, и указанный вариант внедряющегося в цепь олигонуклеотида содержит комплементарный целевой последовательности участок, имеющий 1, 2, 3 или 4 несовпадения с последовательностью SEQ ID NO: 4, комплементарной целевой последовательности.
3. Способ по любому из пп. 1, 2, дополнительно включающий приведение указанного образца в контакт с рекомбиназой.
4. Способ по любому из пп. 1-3, отличающийся тем, что указанный способ осуществляется при изотермических условиях, способствующих амплификации указанной целевой нуклеотидной последовательности.
5. Способ по любому из пп. 1-3, отличающийся тем, что указанный внедряющийся в цепь олигонуклеотид является олигонуклеотидом с последовательностью SEQ ID NO: 5.
6. Способ обнаружения целевой нуклеотидной последовательности токсикогенной С. difficile в образце, причем указанный способ включает приведение указанного образца в контакт с по меньшей мере одним прямым праймером, по меньшей мере одним обратным праймером и по меньшей мере одним внедряющимся в цепь олигонуклеотидом в условиях, способствующих амплификации указанной целевой нуклеотидной последовательности,
где каждый указанный праймер и указанный внедряющийся в цепь олигонуклеотид содержит участок, комплементарный указанной целевой нуклеотидной последовательности;
где указанный внедряющийся в цепь олигонуклеотид переводит по меньшей мере часть целевой нуклеотидной последовательности в одноцепочечное состояние, что позволяет связаться указанным прямому праймеру и обратному праймеру; и
при этом указанный прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 7 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 7, и при этом указанный обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 8 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 8, и при этом указанный внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, содержащим последовательность SEQ ID NO: 9, или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 9, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 9, комплементарной целевой последовательности, и при этом указанный внедряющийся в цепь олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в своей 3'-области, делая ее неудлиняемой.
7. Способ по п. 6, отличающийся тем, что указанный вариант прямого праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 7, указанный вариант обратного праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 8, и указанный вариант внедряющегося в цепь олигонуклеотида содержит комплементарный целевой последовательности участок, имеющий 1, 2, 3 или 4 несовпадения с последовательностью SEQ ID NO: 9, комплементарной целевой последовательности.
8. Способ по любому из пп. 6, 7, дополнительно включающий приведение указанного образца в контакт с рекомбиназой.
9. Способ по любому из пп. 6-8, отличающийся тем, что указанный способ осуществляется при изотермических условиях, способствующих амплификации указанной целевой нуклеотидной последовательности.
10. Способ по любому из пп. 6-8, отличающийся тем, что указанный внедряющийся в цепь олигонуклеотид является олигонуклеотидом с последовательностью SEQ ID NO: 10.
11. Композиция для применения в способе обнаружения токсикогенной С. difficile, содержащая олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 2 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 2; олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 3 или вариант указанной последовательности, имеющий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 3; и олигонуклеотид длиною более 30 нуклеотидов, причем указанный олигонуклеотид содержит последовательность SEQ ID NO: 4 или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который имеет по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 4, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 4, комплементарной целевой последовательности, и при этом указанный олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в 3'-области, делая ее неудлиняемой.
12. Композиция для применения в способе обнаружения токсикогенной С. difficile, содержащая олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 7 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 7; олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 8 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 8; и олигонуклеотид длиною более 30 нуклеотидов, причем указанный олигонуклеотид содержит последовательность SEQ ID NO: 9 или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 9, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 9, комплементарной целевой последовательности, и при этом указанный олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в 3'-области, делая ее неудлиняемой.
13. Набор для применения в способе обнаружения токсикогенной С. difficile, содержащий три олигонуклеотида, причем указанные олигонуклеотиды включают:
олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 2 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 2; олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 3 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 3; и олигонуклеотид длиною более 30 нуклеотидов, причем указанный олигонуклеотид содержит последовательность SEQ ID NO: 4 или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 4, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 4, комплементарной целевой последовательности, и при этом указанный олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в 3'-области, делая ее неудлиняемой.
14. Набор для применения в способе обнаружения токсикогенной С. difficile, содержащий три олигонуклеотида, причем указанные олигонуклеотиды включают:
олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 7 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 7; олигонуклеотид длиною менее 30 нуклеотидов, содержащий последовательность SEQ ID NO: 8 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 8; и олигонуклеотид длиною более 30 нуклеотидов, причем указанный олигонуклеотид содержит последовательность SEQ ID NO: 9 или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 9, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 9, комплементарной целевой последовательности, и при этом указанный олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в 3'-области, делая ее неудлиняемой.
15. Композиция по п. 11 или набор по п. 13, где указанный вариант прямого праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 2, указанный вариант обратного праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 3, и указанный вариант внедряющегося в цепь олигонуклеотида содержит комплементарный целевой последовательности участок, имеющий 1, 2, 3 или 4 несовпадения с последовательностью SEQ ID NO: 4, комплементарной целевой последовательности.
16. Композиция по п. 12 или набор по п. 14, где вариант прямого праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 7, указанный вариант обратного праймера содержит последовательность, имеющую 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 8, и указанный вариант внедряющегося в цепь олигонуклеотида содержит комплементарный целевой последовательности участок, имеющий 1, 2, 3 или 4 несовпадения по отношению к последовательности SEQ ID NO: 9, комплементарной целевой последовательности.
17. Композиция по любому из пп. 11, 12 или набор по любому из пп. 13, 14, дополнительно содержащие ДНК-полимеразу и/или рекомбиназу.
18. Применение прямого праймера, обратного праймера и внедряющегося в цепь олигонуклеотида в способе обнаружения токсикогенной С. difficile, где
(а) указанный прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 2 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 2, и при этом указанный обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 3 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 3, и при этом указанный внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, причем указанный олигонуклеотид содержит последовательность SEQ ID NO: 4 или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 4, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 4, комплементарной целевой последовательности, и при этом указанный внедряющийся в цепь олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в своей 3'-области, делая ее неудлиняемой; или
(b) указанный прямой праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 7 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 7, и при этом указанный обратный праймер является олигонуклеотидом длиною менее 30 нуклеотидов, содержащим последовательность SEQ ID NO: 8 или вариант указанной последовательности, обладающий по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 8, и при этом указанный внедряющийся в цепь олигонуклеотид является олигонуклеотидом длиною более 30 нуклеотидов, причем указанный олигонуклеотид содержит последовательность SEQ ID NO: 9 или представляет собой вариант указанной последовательности, содержащий комплементарный целевой последовательности участок, который обладает по меньшей мере 70% идентичности по отношению к последовательности SEQ ID NO: 9, комплементарной целевой последовательности, или содержащий участок, комплементарный последовательности SEQ ID NO: 9, комплементарной целевой последовательности, и при этом указанный внедряющийся в цепь олигонуклеотид дополнительно содержит один или более модифицированных нуклеотидов в своей 3'-области, делая ее неудлиняемой.
19. Способ диагностики инфекции, вызванной С. difficile, у субъекта, включающий осуществление способа по любому из пп. 1-10, в образце от указанного субъекта.
| WO 2009150467 от 17.12.2009 | |||
| Lalande V | |||
| et al | |||
| Evaluation of a loop-mediated isothermal amplification assay for diagnosis of Clostridium difficile infections //Journal of clinical microbiology | |||
| Способ приготовления лака | 1924 |
|
SU2011A1 |
| - Vol | |||
| Способ смешанной растительной и животной проклейки бумаги | 1922 |
|
SU49A1 |
| - n | |||
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
| - P | |||
| Способ получения безводного сернокислого натрия из глауберовой соли | 1924 |
|
SU2714A1 |
| Лобзин Ю | |||
| В., Захаренко С | |||
| М., Иванов Г | |||
| А | |||
| Современные представления об инфекции Clostridium difficile //Клиническая микробиология и антимикробная химиотерапия | |||
| Топчак-трактор для канатной вспашки | 1923 |
|
SU2002A1 |
| - Т | |||
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
| - n | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| - С | |||
| Мяльно-трепальный станок для обработки тресты лубовых растений | 1922 |
|
SU200A1 |
Авторы
Даты
2018-11-29—Публикация
2014-04-23—Подача