Область техники:
Изобретение относится к области медицины, биотехнологии и генной инженерии, а именно к системе для воздействия на ДНК вируса гепатита В, обеспечивающей ее деградацию и элиминацию из организма пациента, а также к применению такой системы для деградации вирусной ДНК и ее элиминации из клеток пациента, в частности, из гепатоцитов.
Уровень техники:
Вирус гепатита В (ВГВ, HBV), представитель семейства гепаднавирусов (Hepadnaviridae), представляет собой небольшой, оболочечный, двуцепочечный ДНК-содержащий вирус, образующий особую форму ДНК в ядре инфицированных клеток, так называемую кольцевую ковалентно-замкнутую ДНК (ккзДНК), которая, наряду с внутриклеточной ДНК вируса и прегеномной РНК (пгРНК), персистирует в инфицированном гепатоците, присутствуя в нем в форме минихромосомы (эписомы, упакованной в гистоновые белки) (Newbold J.E., Xin H. et al., 1995 [1]). КкзДНК HBV служит матрицей для транскрипции всех вирусных РНК, включая пгРНК, из которой образуется кольцевая частично-двуцепочечная ДНК (кчдДНК), основная форма генома ВГВ в вирионе. HBV – гепатотропный вирус, инфицирует в основном гепатоциты, связываясь с NTCP (Na+-таурохолат ко-транспортирующий пептид), транспортером желчных кислот, который используется HBV для высокоаффинного связывания и проникновения в клетку (Glebe D., Bremer C.M., 2013 [2]; Yan, H., Zhong, G. et al., 2012 [3]), однако показано, что ВГВ также способен персистировать в лимфоцитах, эпителии желчных путей, эндотелии сосудов, а также в клетках поджелудочной железы, почек и костного мозга (Joshi at al., 2018 [4]). Инфицирование HBV может вызвать острый или хронический гепатит B, одно из самых распространенных инфекционных заболеваний в мире. HBV считается «скрытным» вирусом, который слабо распознается внутриклеточной иммунной системой, и может избегать иммунного надзора (Mutz P. et al., 2018 [5]). Таким образом, HBV обычно не индуцирует мощного противовирусного иммунного ответа. Традиционная терапия вирусного гепатита B, включающая применение аналогов нуклеот(з)идов, таких как ламивудин и энтекавир, имеет ограниченный терапевтический потенциал ввиду того, что указанные аналоги нуклеотидов (нуклеозидов) не оказывают действия на вирусную ккзДНК, длительная персистенция которой в клетках печени имеет исход в цирроз печени и/или в гепатоцеллюлярную карциному (Zeisel M.B., Lucifora J. et al., 2015 [6]). Применяемая для лечения вирусного гепатита В интерферонотерапия, включающая использование интерферонов, в том числе, пегилированных, также имеет свои ограничения, поскольку существует ряд противопоказаний к интерферонотерапии (например, декомпенсированный цирроз печени). Кроме того, около 70% пациентов не отвечают на терапию интерферонами (Janssen HL, van Zonneveld M et al., 2005 [7]).
В связи с этим, остается актуальной разработка новых подходов к терапии хронического гепатита B, который позволял бы воздействовать на ккзДНК ВГВ, персистирующую в клетках человека, в частности, в гепатоцитах.
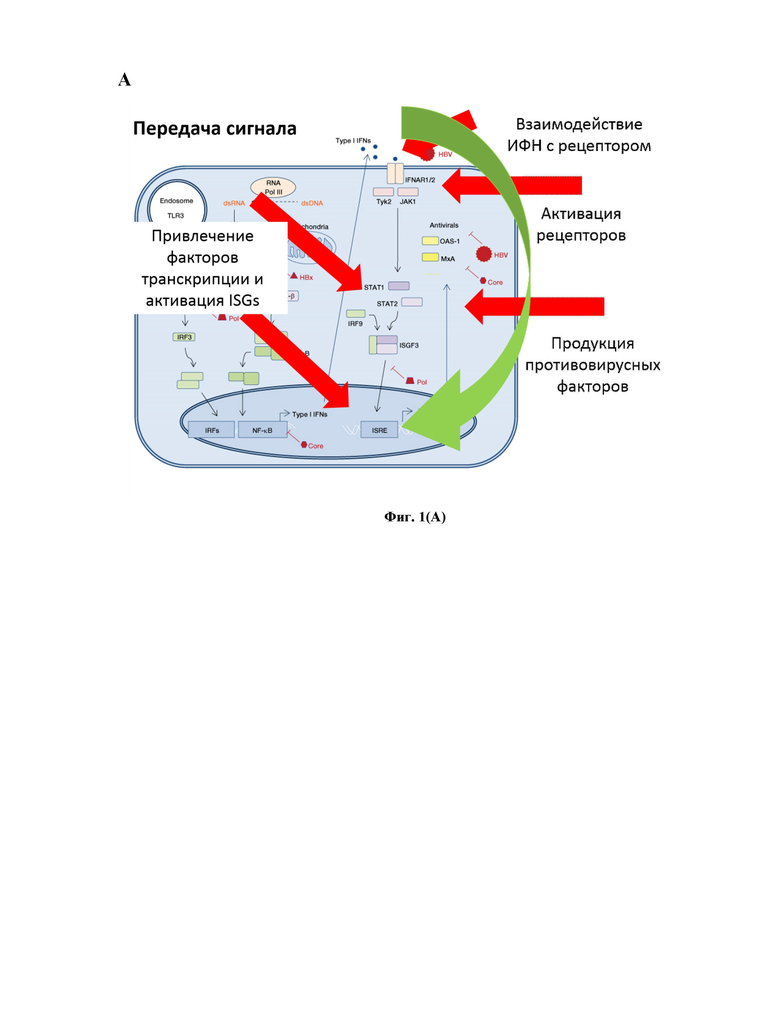
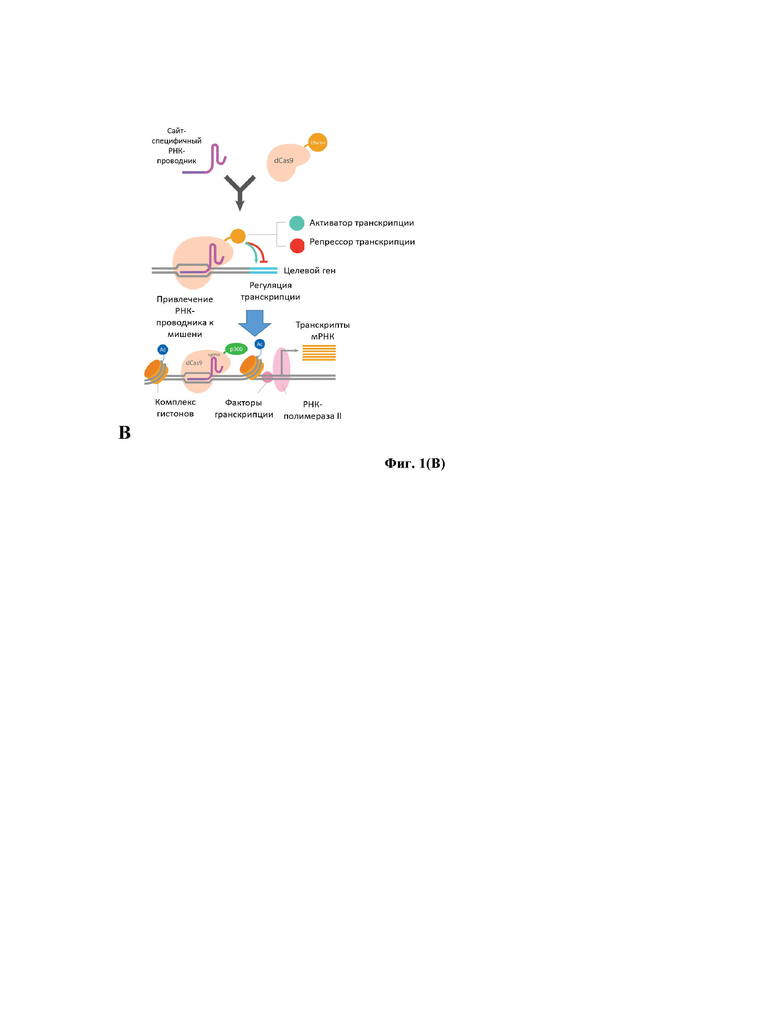
В основе предлагаемой в рамках настоящего изобретения технологии лежат принципы работы системы CRISPR/Cas9 - системы сайт-специфичных ДНК-нуклеаз. В недавних работах CRISPR/Cas9 нуклеазы были генетически видоизменены, что сделало их пригодными для активации транскрипции целевых генов. К наиболее эффективным и специфичным относится подход по использованию химерного Cas9 белка, dCas9, лишенного нуклеазной активности (nuclease-dead), генетически слитого с каталитической субъединицей гистоновой ацетилтрансферазы р300 или доменами активации транскрипции VP16 вируса простого герпеса в виде тетрамера VP64, который коэкспрессируют с комплексом MS2-p65-HSF1 (Konermann S. et al., 2015 [7]). Соответственно, dCas9-p300 или dCas9-VP64/MS2-p65-HSF1 привлекаются к регуляторным элементам целевых генов с помощью РНК-проводника или набора РНК-проводников, специфичных к указанным регуляторным элементам, что приводит к активации транскрипции целевых генов (Hilton I.B. et al., 2015 [9], Рис. 1В). Указанные технологии не имеют ограничений, свойственных для систем гиперэкспрессии кДНК. Ранние поколения систем активации dCas9 приводили к низкой активации транскрипции генов или были вовсе неэффективны (Perez-Pinera P et al., 2013 [10]). На сегодняшний день слитые белки dCas9-p300 и dCas9-VP64/MS2-p65-HSF1 являются самыми действенными активаторами эндогенной транскрипции с уровнями активации транскрипции, варьирующими от 2 до тысячекратной активации в большинстве линиях клеток (Chavez A, Tuttle M et al., 2016 [11]). Уровни активации наиболее высоки в условиях репрессированного эпигенетического контекста, т.е. если базальные уровни экспрессии низкие. Наоборот, активация менее эффективна в случае, если ген экспрессируется на высоком базальном уровне. Большинство интерферон-стимулированных генов (к которым относятся и семейство APOBEC/AID-дезаминаз) слабо активируются в ответ на HBV (Morikawa K, Shimazaki T et al., 2016 [12]). Более того, действие цитокинов, таких как интерфероны, при гепатите В часто блокировано на уровне прохождения сигнала (Рис. 1А) (Christen V, Duong F et al., 2007 [13]). Кроме того, недавно было продемонстрировано, что агонист рецептора лимфотоксина-β также может активировать APOBEC3B и вызывать специфичную деградацию ккзДНК HBV. Тем не менее, его использование при гепатите B осложняется влиянием пути лимфотоксина в развитии гепатоцеллюлярной карциномы, воспаления и индукции апоптоза. (Haybaeck J, Zeller N et al., 2009 [14]). Важно отметить, что эндогенная активация транскрипции с помощью CRISPR/dCas9 высокоспецифична (см. работу [8], Рис. 1D), при этом активация транскрипции с помощью CRISPR/dCas9 систем может обеспечиваться при помощи единой молекулы РНК-проводника и единого слитого белка dCas9-X, где Х – белок-эффектор, например, p300, VP64 и так далее. Тем самым, наблюдаемые при эндогенной активации генов эффекты можно уверенно относить к bona fide эффектам целевых генов. Одними из специфических и прямых эффекторных интерферон-стимулированных генов являются представители семейства цитидиндезаминаз APOBEC/AID, включая APOBEC3A и APOBEC3B, осуществляющих дезаминирование остатков цитидина в вирусной ккзДНК до урацила (Lucifora J. et al., 2014 [20]; Hoopes JI, Cortez LM et al., 2016 [15]). В дополнение к этому, показано, что дезаминазы APOBEC3G (Turelli P, Mangeat B et al., 2004 [16]) и AID (Qiao Y, Han X et al., 2016 [16]) также проявляют анти-HBV активность.
С момента разработки стратегии активации транскрипции с помощью dCas9 в 2013 году Perez-Pinera P et al., 2013 [10] известно о нескольких примерах успешного использования стратегии по активации эндогенной транскрипции с помощью dCas9. В работе [10] авторы продемонстрировали, что факторы вирусной рестрикции в клетках человека можно эффективно индуцировать при помощи систем dCas9. В этой работе также высказано мнение, что новые поколения систем dCas9 открывают совершенно новые перспективы для изучения эффектов индивидуальных генов на репликацию вирусов или на протекание латентной инфекции. В статье Hal P. Bogerd et al., 2015 [18] система dCas9-VP64/MS2-p65-HSF1 была использована для индукции противовирусных эффекторов APOBEC3G и APOBEC3B в клетках человека, инфицированных РНК-содержащим вирусом – вирусом иммунодефицита человека (ВИЧ-1). Однако в работах [10] и [18] не рассматривается воздействие систем, использующих dCas9, на геном ДНК-содержащих вирусов, к которым относится и вирус гепатита В.
В 2017 году Klann T.S. et al. выполнили масштабный скрининг функциональных регуляторных элементов в геноме человека, используя dCas9-KRAB репрессор и dCas9-p300 активатор транскрипции и библиотеки одиночных РНК-проводников для идентификации регуляторных элементов генов интереса в сайтах, гиперчувствительных к ДНКазе I (Klann T.S. et al., 2017 [19]). В дополнение к этому, стратегии по сайт-специфической активации и репрессии транскрипции с использованием dCas9 помогли идентифицировать ранее неизвестные регуляторные элементы генов по всему геному. Кроме того, стратегии по активации эндогенной транскрипции с помощью dCas9 применялись для управления фенотипом раковых клеток (Braun CJ et al., 2016 [20]), определения генов, ответственных за устойчивость к лекарственным препаратам (Carlos le Sage et al., 2017 [21]), а также транскрипционного репрограммирования клеток (Chavez A et al., 2015 [11]).
В работе Lucifora J. et al., 2014 [23] было показано, что транскрипция генов дезаминаз APOBEC3A и APOBEC3B активируется под действием интерферона α, а также при активации рецепторов лимфотоксина β, при этом дезаминазы APOBEC3A и APOBEC3B действуют напрямую на ккзДНК, вызывая дезаминирование остатков цитозина в ккзДНК (см. фигуру 6D на стр. 1227 работы [23], а также комментарий к фигуре 6D на стр. 1226 указанной работы). В то же время, согласно работе [23], дезаминазы APOBEC3A и APOBEC3B не привлекаются к ДНК генома клетки за счет взаимодействия с кор-антигеном вируса (HBcAg). Следовательно, внецелевые эффекты APOBEC3A и APOBEC3B, включающие дезаминирование цитозиновых оснований в ДНК генома человека, а также связанные с этим генотоксические и промутагенные эффекты APOBEC3A и APOBEC3B, не характерны при активации APOBEC3A/3B дезаминаз и инфекции HBV.
Согласно работе [23], рецепторы лимфотоксина β активировали при помощи тетравалентного биспецифического антитела BS1 (Hu X., Zimmerman M.A. et al., 2013 [24]) и бивалентного моноклонального анти-LTβR антитела CBE11 (Lukashev M., LePage D. et al., 2006 [25]). Рецепторы интерферона α в работе [23] активировали при помощи интерферона α. Таким образом, Lucifora J. et al., 2014 [23] не используют для активации транскрипции генов дезаминаз APOBEC3A и APOBEC3B РНК-проводники, специфичные к генам указанных дезаминаз или к регуляторным элементам указанных генов.
Кроме того, прямой анализ генотоксических эффектов APOBEC/AID дезаминаз в работе [23] не проводился. В дополнение к этому, в работе Sajeda Chowdhury et al., 2013 [26] ранее было показано, что дезаминаза AID и урацил-ДНК-гликозилаза UNG действуют сообща, усиливая противовирусные эффекты на ккзДНК, тогда как подавление активности урацил-ДНК-гликозилазы UNG с помощью специфического ингибитора урацил-ДНК-гликозилазы (UGI), согласно данной работе, напротив, полностью аннулировало снижение ккзДНК, вызванное активацией AID. Помимо этого, в работе Sebastien Landry et al., 2011 [27] было показано, что цитидиндезаминаза АРОВЕС3А вызывает повреждение вирусной ДНК за счет активности урацил-ДНК-гликозилазы UNG, основной гликозилазы клетки, которая необходима для распознавания в цепях ДНК остатков урацила, образующихся при дезаминировании остатков цитозина, и их эксцизии, в результате чего происходит деградация вирусной ккз ДНК. Таким образом, активация урацил-ДНК-гликозилазы в дополнение к активации транскрипции дезаминаз APOBEC/AID может усиливать противовирусное действие в отношении вируса гепатита В и приводить к деградации вирусной ккзДНК, персистирующей в инфицированной клетке, в частности, в гепатоците.
В опубликованной патентной заявке США US 2016/0317677 A1, 03.11.2016 (МПК: A61K 38/46, A61K 48/00) [28], поданной от имени The Broad Institute Inc. (US), Massachusetts Institute of Technology (US) и The Rockefeller University (US), описана рассматриваемая в качестве ближайшего аналога заявляемого изобретения система, применимая для воздействия на целевую последовательность нуклеиновой кислоты, например, на целевую последовательность ДНК вируса, такого как вирус гепатита B.
Система [28] включает в себя белок Cas9, предпочтительно, Cas9, лишенный эндонуклеазной активности, т.е. dCas9, не обязательно генетически слитый с доменом активации транскрипции, таким как VP64, и РНК-проводники, специфичные к целевой последовательности нуклеиновой кислоты. В некоторых вариантах осуществления технического решения [28] целевая последовательность может представлять собой высоко консервативный сайт ДНК вируса гепатита B.
Заявка [28] при этом отмечает, что целевая последовательность ДНК, на которую направлены РНК-проводники, может представлять собой последовательность интерферонзависимого гена, например, гена APOBEC 3A, APOBEC 3B и т.д. Молекула РНК-проводника при этом может представлять собой единый РНК-проводник.
Несмотря на высказываемые в заявке [28] утверждения о возможности воздействия на вирусную ккзДНК и обеспечения ее деградации при помощи вышеописанной системы, включающей РНК-проводники, специфичные к генам APOBEC 3A, APOBEC 3B или к иным интерферонзависимым генам, данный документ не приводит примеров воздействия на ккзДНК вируса гепатита В при помощи Cas9 белка, лишенного нуклеолитической активности (dCas9), и РНК-проводников, специфичных к генам дезаминаз APOBEC, то есть способных связываться с указанными генами и привлекать к ним dCas9. При этом, согласно заявке [28], воздействие на ккзДНК вируса гепатита В с обеспечением ее деградации осуществляли при помощи РНК-проводников, специфичных к высоко консервативным участкам генома вируса гепатита В (см. [28], абзацы описания [1299] – [1300]), тогда как возможность воздействия на ккзДНК вируса гепатита B за счет активации генов инфицированных клеток человека, таких как урацил-ДНК-гликозилазы и/или дезаминазы APOBEC/AID, осуществляемой при помощи систем, включающих dCas9 и РНК-проводники, специфичные к генам дезаминаз APOBEC и/или к гену урацил-ДНК-гликозилазы UNG, в заявке [28] не показана. Кроме того, заявка [28] в принципе не упоминает возможность воздействия на ккзДНК вируса гепатита B путем активации генов иных дезаминаз, чем APOBEC3A и APOBEC3B (например, дезаминазы APOBEC3G и APOBEC/AID). Также заявка [28] в принципе не рассматривает возможность воздействия на ккзДНК вируса гепатита B (обеспечения деградации вирусной ккз ДНК) путем активации гена урацил-ДНК-гликозилазы (UNG).
Следовательно, сохраняет свою актуальность задача расширения арсенала средств воздействия на ккзДНК вируса гепатита В, использующих преимущества систем на основе сайт-специфичных ДНК-нуклеаз CRISPR/dCas9, содержащих белок Cas9, лишенный эндонуклеазной активности, т.е. dCas9, и направляющих dCas9 РНК-проводников, специфичных к генам APOBEC/AID дезаминаз и/или урацил-ДНК-гликозилаз UNG, которые бы обеспечивали деградацию вирусной ккзДНК, персистирующей в инфицированной клетке человека, такой как гепатоцит, или интегрированной в геном клетки человека, такой как гепатоцит, и элиминацию ккзДНК из инфицированной клетки, в частности, такой как гепатоцит, что позволит, в свою очередь, предотвратить исход хронической HBV инфекции (хронической персистенции вируса гепатита B в организме пациента) в цирроз печени и/или в гепатоцеллюлярную карциному, и, таким образом, могли бы быть использованы для лечения вирусного гепатита B.
Поставленная выше задача решается созданной в рамках настоящего изобретения системой, включающей:
(i) слитый белок dCas9-Х, где dCas9 представляет собой мутантный белок Cas9, лишенный эндонуклеазной активности, а Х представляет собой эффекторный белок, выбранный из кор-белка ацетилтрансферазы человека p300 или из VP48, VP64, VP160, VP192 и p65HSF1, и
(ii) РНК-проводники, специфичные к регуляторным элементам одного или нескольких генов цитидиндезаминаз человека (APOBEC/AID) и/или гена урацил-ДНК-гликозилазы человека (UNG).
В наиболее предпочтительных вариантах осуществления настоящего изобретения, dCas9, без ограничения, представляет собой мутантный белок Cas9 Streptococcus pyogenes, лишенный эндонуклеазной активности. В других наиболее предпочтительных вариантах осуществления настоящего изобретения, также без ограничения, dCas9 представляет собой мутантный белок Cas9 II типа, лишенный эндонуклеазной активности, например (без ограничения), мутантный Cas 9 II типа из Streptococcus thermophilus, Cas9 Neisseria meningitidis или Cas9 Staphylococcus aureus.
В предпочтительных вариантах осуществления изобретения, РНК-проводники направлены на регуляторные элементы генов APOBEC/AID и/или UNG, а именно, на промоторы и/или энхансеры указанных генов, при этом указанные РНК-проводники образованы первой нуклеотидной последовательностью, специфичной к регуляторному элементу гена APOBEC/AID и/или UNG, такому как промотор и/или энхансер соответствующего гена, то есть способной связываться с указанным регуляторным элементом, и второй нуклеотидной последовательностью, фланкирующей первую нуклеотидную последовательность с 3'-конца и представляющую собой РНК-шпильку, способную направлять белок dCas9-X к указанному регуляторному элементу.
В наиболее предпочтительных вариантах осуществления изобретения, без ограничения, указанная первая нуклеотидная последовательность представляет собой мотив из 19-20 нуклеотидов, комплементарных целевой последовательности регуляторного элемента в геноме клеток человека, тогда как указанная вторая нуклеотидная последовательность представляет собой шпилечную структуру, способную связывать белок dCas9-X и направлять к целевой последовательности, и специфичную для каждого вида организма, из которого был получен белок Cas9 (Streptococcus pyogenes или другой Cas9 белок II типа).
Заявляемая в рамках настоящего изобретения система, охарактеризованная выше, обеспечивает ряд технических результатов, в том числе:
(а) обеспечивает более высокую степень деградации ккзДНК, а также других стадий жизненного цикла вируса гепатита В и более высокую степень элиминации указанных стадий жизненного цикла вируса из инфицированной клетки, в частности, из гепатоцита, при отсутствии внецелевых эффектов;
(б) обеспечивает уменьшение уровня секретируемой вирусной ДНК;
(в) обеспечивает расширение арсенала средств элиминации ккзДНК и других стадий жизненного цикла вируса гепатита B из инфицированной клетки, в которой вирус персистирует, в частности, из инфицированного гепатоцита;
(г) обеспечивает сокращение сроков элиминации ккзДНК и других стадий жизненного цикла вируса гепатита B (в первую очередь, пгРНК) из инфицированной клетки, в частности, из инфицированного гепатоцита и, как следствие, сокращение сроков терапии.
Технические результаты (а) – (г) достигаются за счет того, что предлагаемая в соответствии с настоящим изобретением система активирует собственную систему APOBEC/AID-дезаминаз и урацил-ДНК-гликозилаз клетки человека (гепатоцита или иной клетки), при этом активированные APOBEC/AID-дезаминазы и урацил-ДНК-гликозилазы обеспечивают дезаминирование цитозиновых оснований вирусной ккзДНК и последующую ее деградацию.
Следствием этого становится обеспечиваемая настоящим изобретением элиминация ДНК вируса гепатита B, включая ккзДНК, из инфицированной клетки, в частности, из гепатоцита и, как следствие, возможность предотвратить исход хронической персистенции вируса гепатита B в цирроз печени или в гепатоцеллюлярную карциному.
Также в рамках настоящего изобретения заявляется применение системы, включающей слитый белок dCas9-Х и РНК-проводники, специфичные к регуляторным элементам одного или нескольких генов APOBEC/AID и/или UNG, для активации цитидиндезаминазы APOBEC/AID человека или урацил-ДНК-гликозилазы UNG человека и для APOBEC-опосредованной и UNG-опосредованной деградации ДНК вируса гепатита В гепаднавируса, персистирующей в клетке указанного человека, в частности, в гепатоците; при этом также достигаются, как будет показано в примерах, иллюстрирующих настоящее изобретение, технические результаты (а) – (г).
Список сокращений:
HBV (ВГВ) – вирус гепатита В
A3A – цитидиндезаминаза APOBEC3A
A3B - цитидиндезаминаза APOBEC3B
A3G - цитидиндезаминаза APOBEC3G
AID - цитидиндезаминаза AID
UNG – урацил-ДНК-гликозилаза (Uracil-DNA Glycosylase)
мРНК – матричная РНК
pgRNA (пгРНК) – прегеномная РНК
S-RNA (S-РНК) – РНК HBs-антигена
cccDNA (ккзДНК) – кольцевая ковалентно замкнутая ДНК
NGS – секвенирование нового поколения (Next Generation Sequencing)
HBcAg – кор-антиген вируса гепатита В (Hepatitis B core Antigen)
Краткое описание фигур:
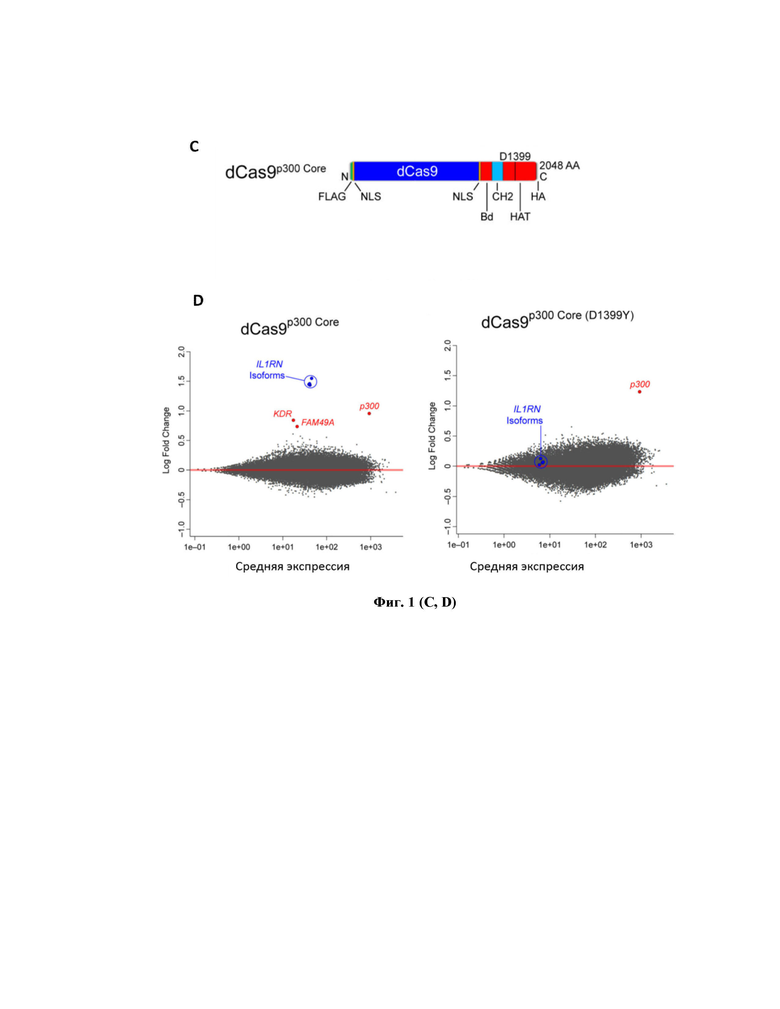
Фигура 1 – Принципиальная схема активации генов АРОВЕС/AID-дезаминаз с помощью систем dCas9-опосредованной активации транскрипции. (А) Преимущества dCas9-опосредованной активации транскрипции перед традиционными методами активации АРОВЕС/AID-дезаминаз с помощью различных цитокинов; (В) Принцип работы dCas9-опосредованной активации транскрипции; (С) Строение активирующего химерного белка dCas9p300; (D) Данные по специфичности действия dCas9p300 и dCas9p300mut с помощью РНК-секвенирования (согласно Hilton IB et al., Nature Biotechnology 2015 [9]).
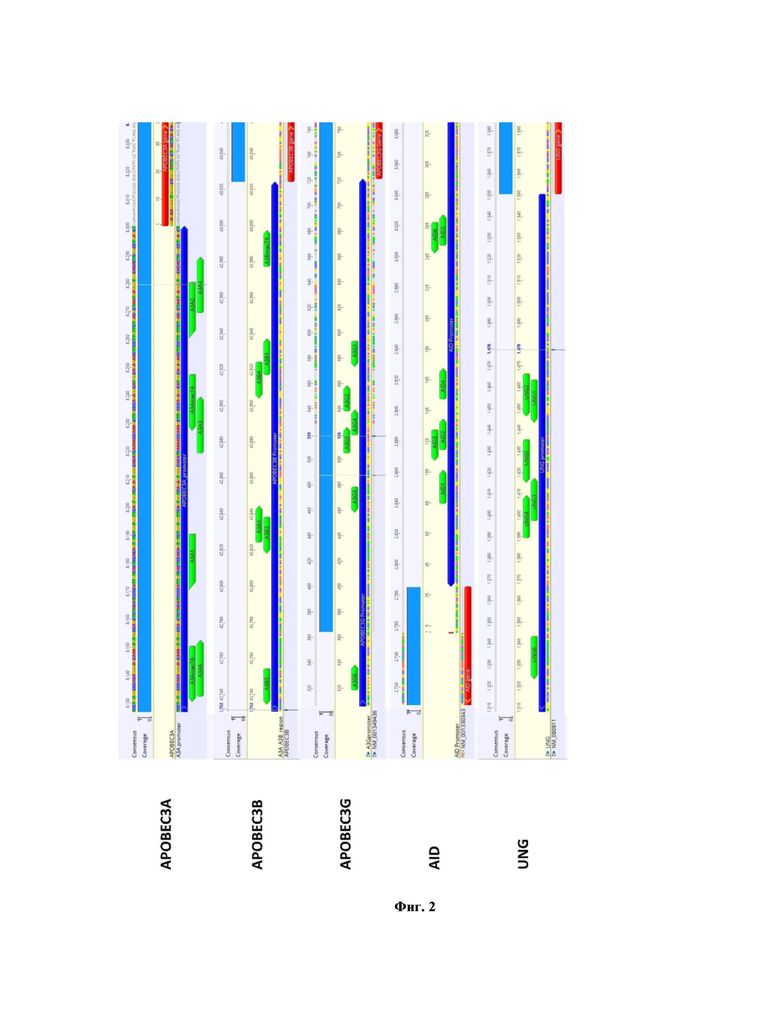
Фигура 2 – РНК-проводники к промоторам генов семейства APOBEC/AID (APOBEC3A, APOBEC3B, APOBEC3G, AID) и UNG. Синим цветом обозначены промоторы генов, красным – гены APOBEC/AID, зеленым – участки посадки РНК-проводников.
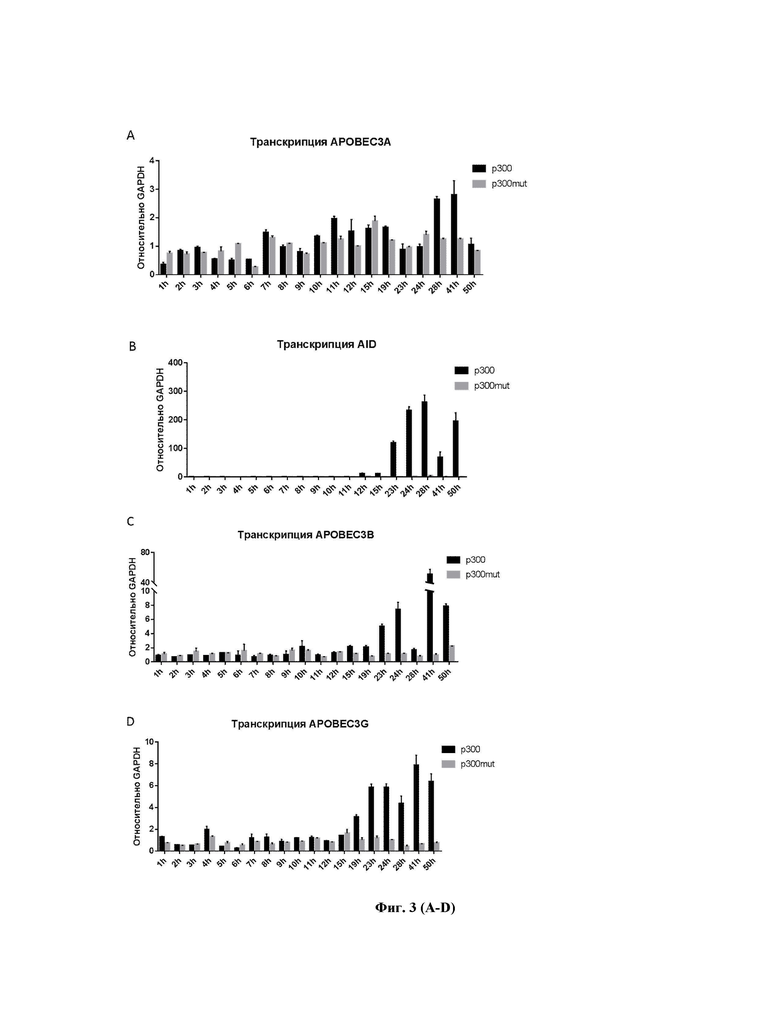
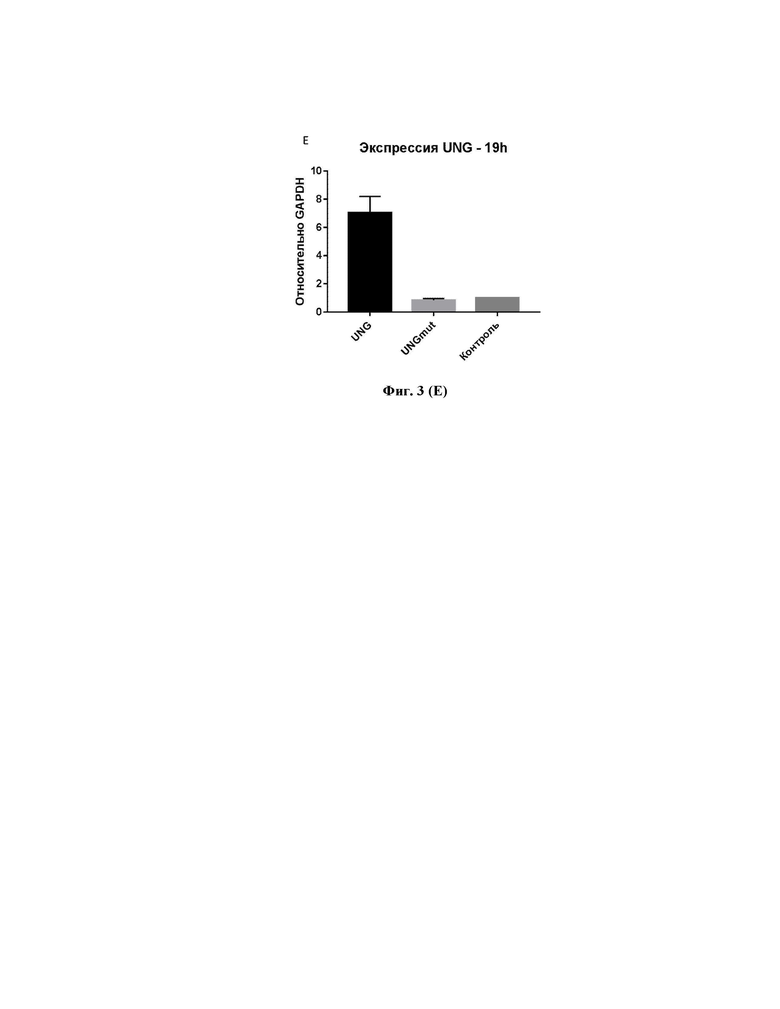
Фигура 3 – Активация транскрипции целевых генов APOBEC/AID и UNG на определенный час после трансфекции. (А-E) Активация транскрипции целевых генов (APOBEC3A, APOBEC3B, APOBEC3G, AID, UNG) при действии dCas9p300 (p300) или в контрольной группе при трансфекции dCas9p300mut (mut). Планки погрешностей соответствуют стандартным отклонениям.
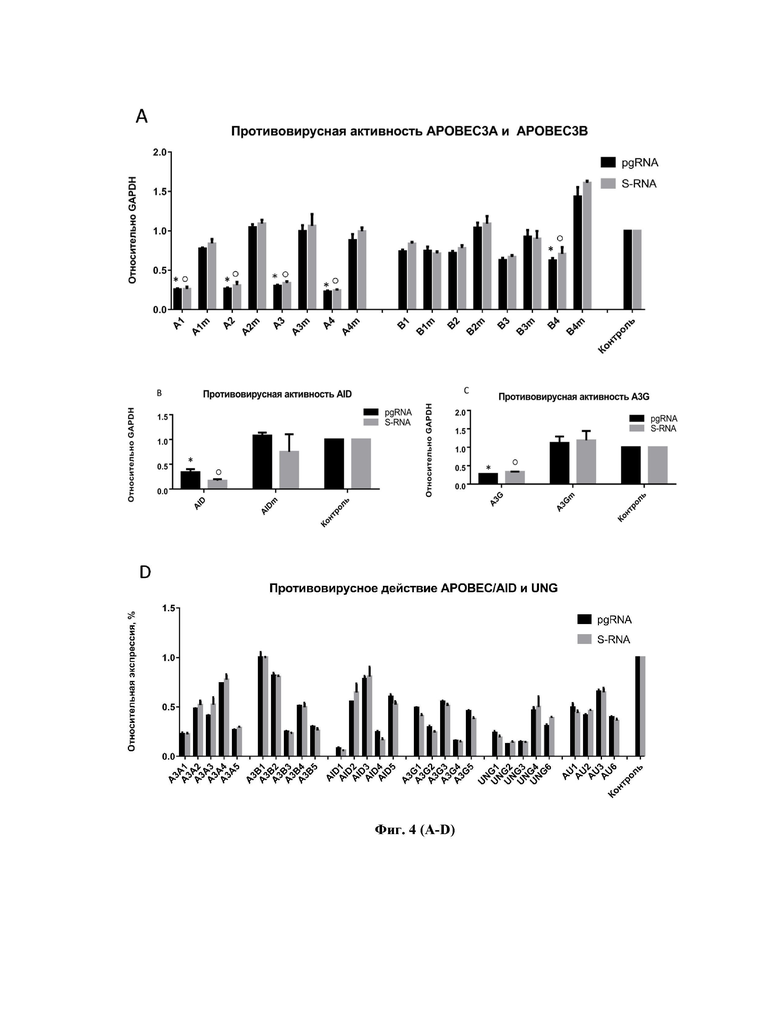
Фигура 4 – Эффекты APOBEC/AID на цикл вируса гепатита B. (А) Противовирусная активность APOBEC3A и APOBEC3B при ко-трансфекции в клетки HepG2; (B) Противовирусное действие AID; (C): APOBEC3G при ко-трансфекции в клетки HepG2; (D) Противовирусное действие APOBEC/AID и UNG при ко-трансфекции в клетки HEK293T (a – APOBEC3A; b – APOBEC3B; aid – AID; g – APOBEC3G; ung – UNG; au – AID и UNG), номера обозначают РНК-проводник, использованный для воздействия на промотор гена; (E) Анализ цитотоксичности APOBEC/AID на клетках HepG2-1.1merHBV и HEK293T. Примечания: pgRNA– прегеномная РНК HBV; S-RNA – S-РНК HBV; mut – dCas9p300mut; *, o – значимые отличия при p<0.05. Планки погрешностей соответствуют стандартным отклонениям.
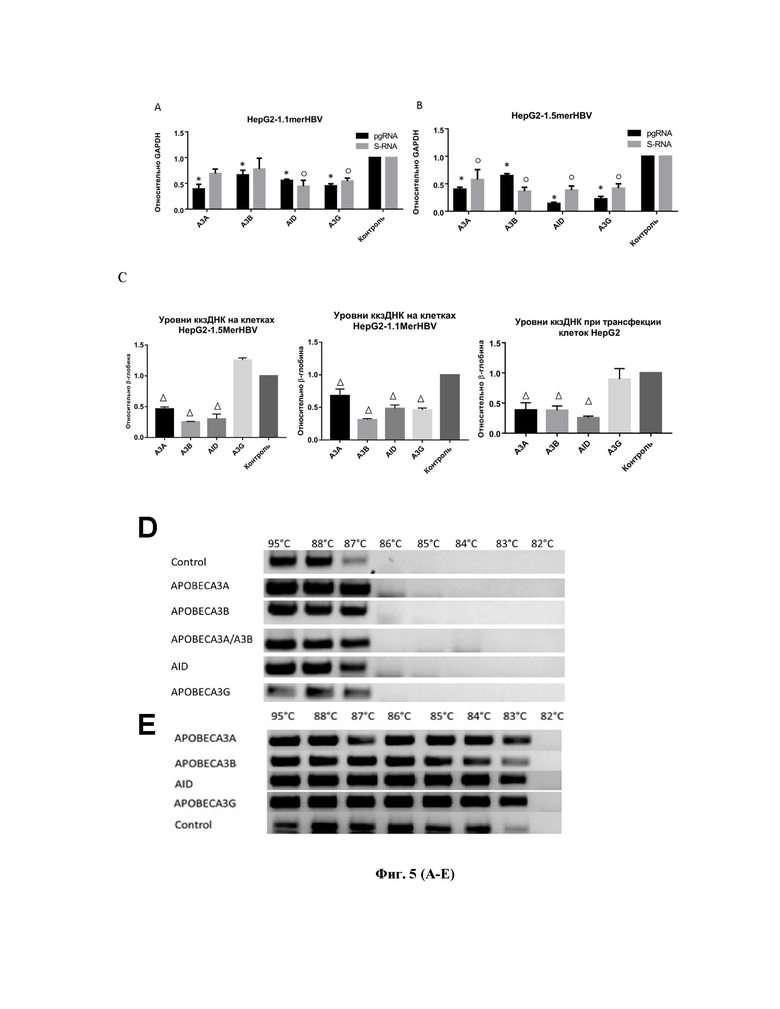
Фигура 5 – Эффекты активации APOBEC/AID-дезаминаз на цикл HBV на стабильных линиях клеток. (A) Действие APOBEC3A (A3A), APOBEC3B (A3B), APOBEC3G (A3G) и AID на транскрипцию HBV (пгРНК, S-RNA) при действии dCas9p300 или мутантной формы dCas9p300mut на клетках HepG2-1.1merHBV и (B) HepG2-1.5merHBV; (C) Изменение уровней ккзДНК при активации APOBEC/AID-дезаминаз на моделях трансфекции HepG2 рекомбинантной ккзДНК, трансфекции HepG2-1.1 и HepG2-1.5, на графиках показаны уровни ккзДНК относительно β-глобина генома; 3D-ПЦР анализ уровней дезаминирования ккзДНК при активации APOBEC/AID-дезаминаз в сравнении с контролем при амплификации с высокоспецифичной Q5-полимеразой (D) или TaqF-полимеразой (E); (F) Результаты NGS-секвенирования CpG-богатого региона ккзДНК после активации A3A (APOBEC3A), A3B (APOBEC3B) или AID в сравнении с контролем (reference) демонстрируют большое число G-to-A и C-to-T замен для A3A и A3B, и большое число G-to-A замен для AID; (G) Крупные делеции (>50 – более 50 нуклеотидов, 10<D<50 – от 10 до 50 нуклеотидов, 3<D<10 – от 3 до 10 нуклеотидов), детектируемые в ккзДНК при активации A3A, A3B и AID. Планки погрешностей соответствуют стандартным отклонениям;(H) Результаты микроэррэй анализа транскриптома клеток HepG2-1.1merHBV после активации APOBEC3A, APOBEC3B, AID и APOBEC3G.
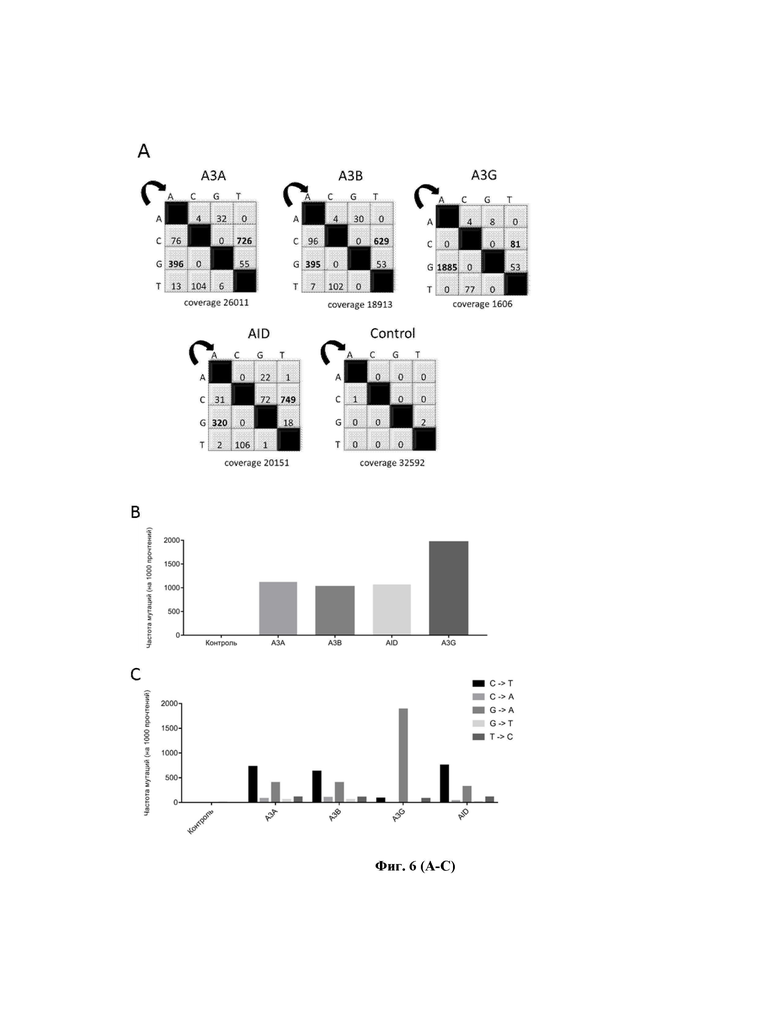
Фигура 6 – Обширное дезаминирование ккзДНК HBV в образцах клеток HepG2-1.1merHBV на 7-е сутки после трансфекции систем активации транскрипции генов APOBEC/AID. (A): Частота мутаций матриц ккзДНК HBV в группах по активации А3А, А3В, A3G, AID, одновременной активации A3A и A3B и в контрольном образце (Control), coverage означает число прочтений образца с качеством прочтений Q>20, данные представлены как частота мутаций на 1000 прочтений образца; (B): Сумма всех замен G->A и C->T в экспериментальных группах на 1000 прочтений; (С): Сравнительный анализ наиболее частых мутаций в экспериментальных группах, цвет колонки обозначает тип мутации.
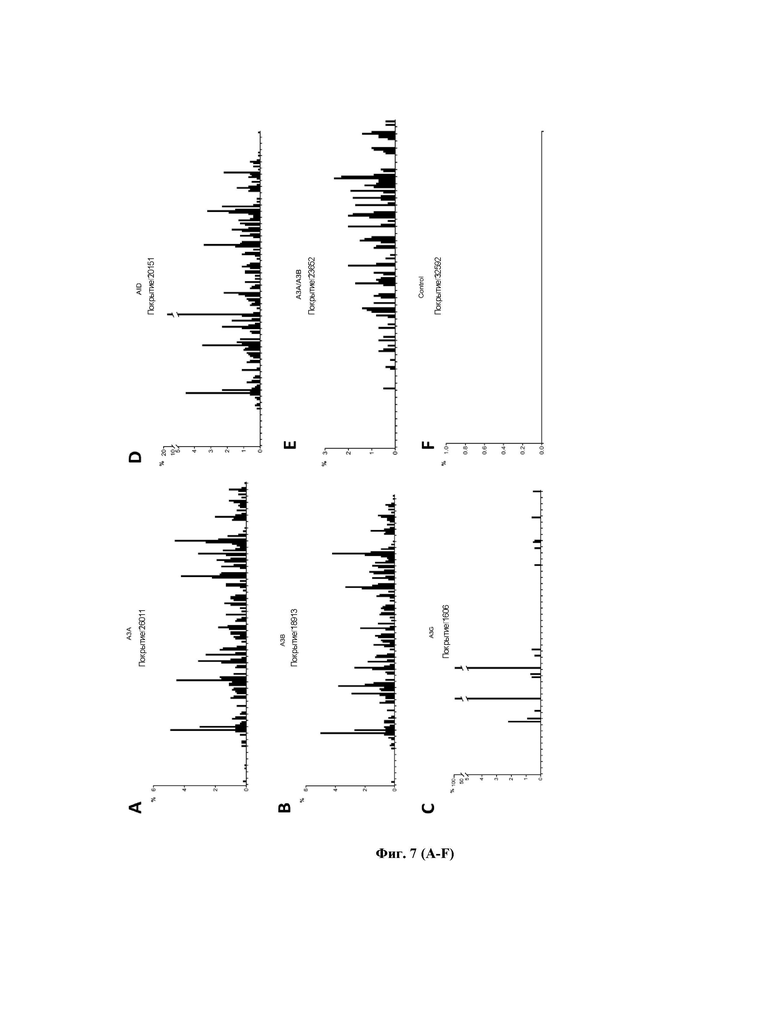
Фигура 7 - Ландшафт дезаминирования CpG-богатого региона ккзДНК HBV в экспериментальных группах после активации генов APOBEC/AID. Совокупная частота замен G->A и C->T (ось ординат) в последовательности ккзДНК HBV длиной 213 нуклеотидов (ось абсцисс).
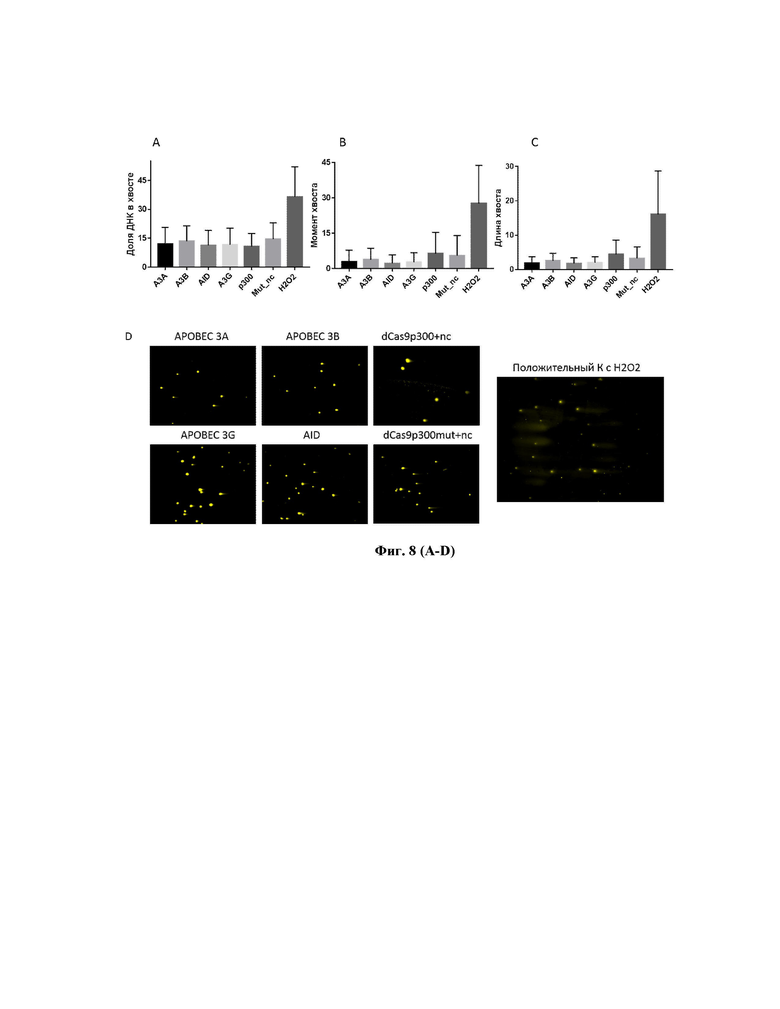
Фигура 8 – Оценка генотоксического действия методом анализа длины комет (Ostling O., Johanson K., 1984 [29]; Tice R., Agurell E. et al., 2000 [30]). Результаты количественного анализа изменения параметра доли ДНК в хвосте кометы (А), момента хвоста (В), длины хвоста (С). (D) Флуоресцентные фотографии комет в группах по активации транскрипции APOBEC3A, APOBEC3B, APOBEC3G, AID, негативных контролях dCas9p300+nc и dCas9p300mut+nc и положительном контроле, обработанном пероксидом водорода (K с Н2О2); Примечание: nc – некодирующий РНК-проводник. Планки погрешностей соответствуют стандартным отклонениям.
Материалы и методы:
Культуры клеток
Клетки гепатомы человека HepG2, HepG2-1.1merHBV и HepG2-1.5merHBV (предоставлены проф. Dieter Glebe, Университет Гиссена, Германия) или HEK293T культивировали в среде DMEM с высоким (4,5 г/л) содержанием глюкозы (Thermo Fisher Scientific), 1% раствора пенициллина/стрептомицина, 1% раствора L-глутамина, 10% фетальной бычьей сыворотки (Gibco) при 370С (5% СО2). Клетки HepG2-1.1merHBV до трансфекции активировали доксициклином (100 нг/мл) в течение 24 часов, после чего клетки дважды промывали фосфатным буфером. Клетки HepG2 или HEK293T ко-трансфицировали плазмидой, кодирующей геном HBV, ПЦР-продуктом, кодирующим РНК-проводник, и плазмидой dCas9p300 или мутантной формой плазмиды dCas9p300mut без ацетилтрансферазной активности (Рис. 1С).
Дизайн РНК-проводников и синтез ПЦР-продуктов
Мишень в геноме ВГВ была подобрана с использованием сервиса Broad Institute Genetic Perturbation Platform (http://portals.broadinstitute.org) и программы Geneious 7.1.9 (https://www.geneious.com) ПЦР-продукт с U6-промотором, кодирующий РНК-проводник, получали при помощи двухэтапного мутагенного ПЦР с высокоточной полимеразой Q5 (New England Biolabs), с проведением амплификации с плазмиды pLX-sgRNA (Addgene plasmid #50662). РНК-проводник содержит мишень в геноме ВГВ и шпильку, необходимую для распознавания белком Streptococcus pyogenes Cas9.
Трансфекция
Клеточные линии HepG2 и HEK293T были ко-трансфицированы с помощью полиэтиленимина плазмидами pCH9-1.1MerHBV и pcDNA-dCas9-p300 Core либо pcDNA-dCas9-p300 Core (D1399Y), а также ПЦР-продуктами, кодирующими РНК-проводники к промоторным регионам целевых генов под контролем промотора U6. Плазмида pCH9-1.1merHBV содержит трансген генома HBV генотипа D субтипа ayw под контролем цитомегаловирусного промотора (предоставлено Dieter Glebe, Гиссен, Германия). pcDNA-dCas9-p300 Core (Addgene #61357) кодирует каталитическую субъединицу ацетилтрансферазы р300 (аминокислоты 1048-1664). pcDNA-dCas9-p300 Core (D1399Y) (Addgene #61358) содержит инактивирующую мутацию, внесение которой приводит к блокированию ацетилтрансферазной активности белка. Клетки линии HepG2-1.1MerHBV и HepG2-1.5 MerHBV рассаживали в 6-луночные планшеты с плотностью 50-60% и трансфицировали плазмидой pcDNA-dCas9-p300 либо pcDNA-dCas9-p300 Core (D1399Y) и ПЦР-продуктами с помощью реагента Lipofectamine3000 по протоколу производителя. Клетки линии HepG2 трансфицировали плазмидами pcDNA-dCas9-p300 либо pcDNA-dCas9-p300 Core (D1399Y), ПЦР-продуктами и рекомбинантной ккзДНК по аналогичному протоколу.
Анализ цитотоксичности методом колориметрии
Изменение пролиферации клеток HepG2-1.1merHBV или HEK293T при активации APOBEC/AID дезаминаз проводили с помощью набора для оценки пролиферации и жизнеспособности по запатентованной технологии восстановления хромогенного субстрата делящимися клетками (Cell cytotoxicity assay, Abcam Biochemicals, ab112118) по протоколу производителя. Клетки рассаживали в 96-луночные планшеты с 30-40% плотностью, трансфицировали клетки HepG2-1.1merHBV с помощью реагента Lipofectamine3000, клетки HEK293T с помощью полиэтиленимина. Клетки HepG2-1.1merHBV или HEK293T промывали на 2 или 1 сутки после трансфекции, соответственно. Анализ цитотоксичности проводили на 3 сутки после трансфекции. Измерение оптического поглощения производили на планшетном спектрофотометре iMark (BioRad).
Выделение нуклеиновых кислот
Выделение нуклеиновых кислот проводили с помощью набора AmpliSens «РИБО-преп» по протоколу производителя, добавляя лизирующий буфер непосредственно к клеткам после удаления среды и двукратного промывания фосфатным буфером. Для выделения прегеномной РНК HBV, нуклеиновые кислоты обрабатывали ДНКазой I типа без РНКазной активности (Thermo Fisher Scientific) в течение 30 мин при 37С0, после чего РНК перевыделяли с помощью набора AmpliSens «РИБО-преп» и проводили постановку обратной транскрипции с помощью набора AmpliSens «РЕВЕРТА-L». Для выделения ккзДНК HBV, ДНК обрабатывали в течение 12 ч ферментом Plasmid-safe ATP-dependent DNase (Epicentre) при 37С0 для разрушения всех форм ДНК, кроме ккзДНК, с последующей инактивацией фермента в течение 30 мин при 70С0.
Полуколичественная ПЦР
Полуколичественный ПЦР-анализ проводили со специфическими праймерами и зондами TaqMan или с интеркалирующим флуоресцентным красителем SybrGreen на прегеномную РНК HBV, S-РНК HBV, мРНК APOBEC3A, APOBEC3B, APOBEC3G, AID, UNG, GAPDH, ДНК ВГВ, β-глобина генома, ккзДНК ВГВ (см. Список праймеров и зондов) на приборах RotorGene6000.
Таблица 1. Праймеры и зонды для анализ противовирусной активности.
(SEQ ID NO: 6)
(SEQ ID NO: 8)
(SEQ ID NO: 9)
(SEQ ID NO: 12)
Таблица 2. Последовательности РНК-проводников к промоторам APOBEC/AID и UNG.
Таблица 3. Последовательности праймеров для 3D-PCR для получения первого ампликона (3DcccDNA) и гнездовая ПЦР (HBxin).
3D-ПЦР
ПЦР-продукты, амплифицированные с ккзДНК HBV c помощью праймеров 3D cccDNA, очищали гель-электрофорезом с последующим выделением очищенной фракции с помощью набора Qiagen Gel Extraction kit. Одинаковая масса очищенных ПЦР-продуктов использовалась для nested-ПЦР с Q5- или TaqF-полимеразой и праймерами HBxin прямой и обратный и декременте температур денатурации (950С-820С). Полученные ампликоны использовались для гель-электрофореза.
Анализ микроэррэй
Анализ генов ответа на повреждение ДНК был проведен с использованием коммерческого набора RT2 Profiler PCR Array Human DNA Damage Signaling Pathway (QIAGEN, №PAHS-029Z) на приборе BioRad CFX96 Touch согласно протоколу производителя. В качестве референсного гена был использован бета-2-микроглобулин. Обработка результатов осуществлялась с использованием онлайн-программного обеспечения (http://saweb2.sabiosciences.com/pcr/arrayanalysis.php).
Секвенирование нового поколения
Ампликоны, полученные при проведении 3D-ПЦР с использованием ДНК-полимеразы Q5 на минимальной возможной температуре (87°С) выделяли с помощью набора Qiagen Gel Extraction Kit и использовали для секвенирования на платформе MiSeq Illumina. Анализ данных секвенирования проводился с использованием программы Geneious 7.1.9.
Анализ генотоксичности методом оценки ДНК-комет
Анализ генотоксичности проводили методом ДНК-комет (Ostling O., Johanson K., 1984 [29]; Tice R., Agurell E. et al., 2000 [30]) с помощью набора "Comet SCGE assay kit", выпускаемого "Enzo Life Sciences". Вкратце, клетки HepG2-1.5merHBV на 5-е сутки после липофекции снимали с планшетов с помощью трипсина, центрифугировали 5 мин при 500хg. В расплавленную LTM агарозу при температуре 370С добавляли по 2,000 клеток, ресуспендировали и наносили на стекло в объеме 75 мкл. Для затвердевания геля, суспензию на стеклах помещали на 10 мин в среду с температурой +40С. Далее стекла выдерживали в лизирующем буфере 45 мин. Вслед за этим, стекла помещались в щелочной раствор с pH>13 на 50 мин. Стекла выдерживали дважды по 5 мин в TBE буфере, после чего помещались в камеру для электрофореза с ТВЕ буфером с параметрами 1 В/см на 50 мин. По завершении электрофореза, стекла помещали в 70% этанол на 5 мин и после этого сушили на воздухе. На стекло помещали 50 мкл красителя SyGreen на 30 мин. Стекла промывали в деионизированной воде и высушивали при температуре +370С. Микроскопию проводили на канале FITC на микроскопе Olympus IX71.
Результаты
Используя инструменты ENSEMBL (http://www.ensembl.org), SwissRegulon (http://swissregulon.unibas.ch), CCTop – CRISPR/Cas9 target online predictor (https://crispr.cos.uni-heidelberg.de/) и BroadInstitute dCas9 activation tool (https://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design-crisprai), мы создали РНК-проводники, мишенью которых являются промоторы каждого из генов APOBEC3A (A3A), APOBEC3B (A3B), APOBEC3G (A3G) и APOBEC/AID, которые, как известно из работы Lucifora J. et al., 2014 [23], являются факторами анти-HBV ответа в клетке, и гена UNG (Рис. 2), усиливающего противовирусный эффект AID и APOBEC3A. Ко-трансфекция клеток HepG2 ПЦР-продуктами с U6-промотором и последовательностью, кодирующей РНК-проводник, и плазмид, кодирующих HBV и dCas9p300 или dCas9p300mut (точковая мутация элиминирует ацетилтрансферазную активность р300), вызывает активацию транскрипции гена А3А уже к 28 часу после трансфекции (Рис. 3А), гена А3В в 57 раз к 41 часу (Рис. 3В), гена А3G в 8 раз к 41 часу (Рис. 3С), АID почти в 300 раз к 28 часу после трансфекции (Рис. 3D), и гена UNG в 7 раз на 19 час после трансфекции клеток (Рис. 3Е).
Активация генов APOBEC/AID и ко-активация AID с UNG оказывает мощное противовирусное действие уже к 3 суткам после трансфекции как на клетках HepG2 (Рис. 4А-С), так и на клетках HEK293T (Рис. 4D). На клетках HepG2 активация А3А вызывает снижение транскрипции HBV (уровней пгРНК и S-RNA) на 80%, А3В на 20-40%, A3G более, чем на 90%, и AID на 60-70%. Вместе с этим, отдельные РНК-проводники для активации A3A, A3B, AID, A3G, UNG и комбинации AID-UNG на клетках HEK293T приводит к снижению транскрипции HBV>90%. Стоит отметить, что наблюдаемое снижение показателей транскрипции вируса не ассоциировано с общим токсическим действием на клетки, что было показано методами анализа цитотоксичности на клетках HepG2-1.1merHBV и HEK293T (Рис. 4E).
Несмотря на мощное действие АРОВЕС/AID-дезаминаз на транскрипцию вируса, изучение их влияния на ккзДНК HBV на моделях ко-трансфекции HepG2 и HEK293T плазмидой pCH-1.1MerHBV невозможно, что связано с особенностями выделения ккзДНК HBV. В этой связи мы провели опыты по липофекции стабильных клеточных линий HepG2-1.1merHBV (Рис. 5А) и HepG2-1.5merHBV, а также по липофекции клеток HepG2 рекомбинантной ккзДНК HBV (Рис. 5В) системами для активации APOBEC/AID-дезаминаз. На указанных клеточных моделях АРОВЕС/AID также вызывают существенное снижение транскрипции HBV, более выраженное на линии клеток с высокоактивной репликацией вируса (HepG2-1.1merHBV: снижение пгРНК и S-RNA>80%), и снижение транскрипции HBV на модели клеток с конститутивной продукцией вируса под промотором дикого типа (HepG2-1.5merHBV) на 40-60%. Далее, мы провели измерение уровней ккзДНК на трех данных моделях (Рис. 5С) и выяснили, что уровни ккзДНК в группах по активации А3А, А3В и AID снижаются на 60-70%, а в группе активации A3G эффект на уровень ккзДНК отмечен только на модели с наиболее активной индуцированной транскрипцией прегеномной РНК HBV (HepG2-1.1MerHBV). Вместе с этим, 3D-ПЦР анализ (Рис. 5D-E) продемонстрировал наличие значительного уровня дезаминирования ккзДНК по CpG-богатому региону во всех экспериментальных группах. Кроме того, проведенный анализ секвенирования нового поколения по CpG-богатому региону ккзДНК (Рис. 5F) выявил значительное количество мутаций G-to-A и C-to-T при активации А3А (25 170 и 26 363, соответственно, при 12 666 проанализированных последовательностей), А3В (26 915 и 18 107, соответственно, при 9303 проанализированных последовательностях), АID (259 390 и практически референсные значения C-to-T замен 1 178, соответственно, при 14 266 проанализированных последовательностях) в сравнении с контролем (3 861 и 579, соответственно, при 16 096 последовательностях). Помимо этого, анализ секвенирования нового поколения выявил большое количество крупных делеций (Рис. 5G) в матрицах ккзДНК, свидетельствующих об активности факторов репарации повреждений ДНК. Кроме того, были проведены дополнительные опыты по анализу дезаминирования CpG-богатого региона в клетках HepG2-1.1merHBV на 7-е сутки после активации APOBEC/AID дезаминаз A3A, A3B, A3G, AID и совместной активации A3A/A3B. Методом NGS секвенирования показано, что во всех экспериментальных группах также резко возрастает частота замен G->A и C->T, характерные для действия АРОВЕС/AID дезаминаз (Рис. 6A-C). При этом совокупная частота мутаций G->A/C->T составляет около 1000 на 1000 прочтений ккзДНК HBV для групп А3А, А3В, AID и А3А/А3В, в то время как для A3G, действующей главным образом на прегеномную РНК HBV, частота замен G->A составляет около 2000, при этом замены C->T практически не встречаются (Рис. 6В). Анализ ландшафта дезаминирования участка ккзДНК HBV (Рис. 7С) выявил, что в группах А3А, А3В и А3А/А3В дезаминирование происходит по всей последовательности практически во всех CpG нуклеотидах с 6 пиками дезаминирования в точках 35 нт, 78 нт, 100 нт, 130 нт и 153 нт. Напротив, при активации AID и A3G были обнаружены т.н. «hot spots» («горячие точки») дезаминирования, т.е. участки преобладающего действия дезаминаз.
Микроэррэй анализ профилей экспрессии факторов репарации (Рис. 5H) выявил значительные изменения в уровнях факторов ответа клетки на повреждение ДНК (наиболее значительная индукция отмечена преимущественно для факторов, которые обеспечивают репарацию дезаминированных нуклеотидов в ДНК): экспрессия EXO1 повышается более чем в 100 раз, MPG в 355.38 раз, TP73 в 84.45 раза (AID), XRCC3 в 20-98 раз; происходит снижение уровня RAD50 в 304.86 раза при активации А3А.
В совокупности, полученные результаты свидетельствуют об эффективной активации экспрессии APOBEC/AID-дезаминаз при помощи dCas9-инструментов, которые вызывают массивное дезаминирование нуклеотидов ккзДНК, активацию факторов репарации повреждений ДНК, вырезание нуклеотидов, исходом которых является деградация и/или гипермутация ккзДНК HBV.
Мы провели оценку влияния активации APOBEC/AID-дезаминаз на повреждение генома в клетках HepG2-1.5merHBV методом оценки комет ДНК (Tice R., Agurell E. et al., 2000 [22]). Чем длиннее хвост кометы и выше в хвосте содержание ДНК, тем выше повреждение генома. Ни по одному из параметров не было выявлено повреждение генома клетки (Рис. 8A-D). Напротив, момент хвоста и длина хвоста снижались в экспериментальных группах в сравнении с контрольными (p300+nc и mut+nc), что говорит о снижении повреждения генома клетки, ассоциированного с генотоксическим действием HBV.
Ссылки
1. Newbold J E, Xin H, Tencza M, Sherman G, Dean J, Bowden S, and Locarnini S. The covalently closed duplex form of the hepadnavirus genome exists in situ as a heterogeneous population of viral minichromosomes. – "Journal of virology", 1995 Jun; 69(6): 3350–3357.
2. Glebe D, Bremer CM. The molecular virology of hepatitis B virus. – "Seminars in liver disease", 2013 May;33(2):103-12.
3. Huan Yan, Guocai Zhong, Guangwei Xu, Wenhui He, Zhiyi Jing, Zhenchao Gao, Yi Huang, Yonghe Qi, Bo Peng, Haimin Wang, Liran Fu, Mei Song, Pan Chen, Wenqing Gao, Bijie Ren, Yinyan Sun, Tao Cai, Xiaofeng Feng, Jianhua Sui, and Wenhui Li. Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus. – "eLife", 2012; 1: e00049.
4. Joshi SS, Coffin CS. Hepatitis B virus lymphotropism: emerging details and challenges. Biotechnol Genet Eng Rev. 2018 May 22:1-13.
5. Mutz P., Metz P., Lempp F.A., Bender S., Qu B., Schöneweis K., Seitz S., Tu T., Restuccia A., Frankish J., Dächert C., Schusser B., Koschny R., Polychronidis G., Schemmer P., Hoffmann K., Baumert T.F., Binder M., Urban S., Bartenschlager R. HBV Bypasses the Innate Immune Response and Does Not Protect HCV From Antiviral Activity of Interferon. – "Gastroenterology", 2018 May; 154(6):1791-1804.
6. Zeisel M.B., Lucifora J., Mason W.S., Sureau C., Beck J., Levrero M., Kann M., Knolle P.A., Benkirane M., Durantel D. et al. Towards an HBV cure: State-of-the-art and unresolved questions—Report of the anrs workshop on HBV cure. – "Gut", 2015; 64:1314–1326.
7. Janssen HL, van Zonneveld M, Senturk H, Zeuzem S, Akarca US, Cakaloglu Y, Simon C, So TM, Gerken G, de Man RA, Niesters HG, Zondervan P, Hansen B, Schalm SW; HBV 99-01 Study Group; Rotterdam Foundation for Liver Research. Pegylated interferon alfa-2b alone or in combination with lamivudine for HBeAg-positive chronic hepatitis B: a randomised trial. - "Lancet", 2005 Jan 8-14;365(9454):123-9
8. Konermann S, Brigham MD., Trevino AE., Joung J, Abudayyeh OO., Barcena C, Hsu PD., Habib N, Gootenberg JS., Nishimasu H, Nureki O, and Zhang F. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. – "Nature", 2015 January 29; 517 (7536): 583–588.
9. Hilton IB, D’Ippolito AM, Vockley CM, Pratiksha I. Thakore, Gregory E. Crawford, Timothy E. Reddy, and Charles A. Gersbach. Epigenome editing by a CRISPR/Cas9-based acetyltransferase activates genes from promoters and enhancers. – "Nature biotechnology", 2015 May; 33(5): 510–517.
10. Perez-Pinera P, Kocak DD, Vockley CM, Adler AF, Kabadi AM, Polstein LR, Thakore PI, Glass KA, Ousterout DG, Leong KW, Guilak F, Crawford GE, Reddy TE, Gersbach CA. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. – "Nature Methods", 2013 Oct; 10(10):973-6.
11. Chavez A, Tuttle M, Pruitt BW, et al. Comparative Analysis of Cas9 Activators Across Multiple Species. - "Nature methods", 2016;13(7):563-567. doi:10.1038/nmeth.3871
12. Morikawa K, Shimazaki T, Takeda R, Izumi T, Umumura M, Sakamoto N. Hepatitis B: progress in understanding chronicity, the innate immune response, and cccDNA protection. - "Annals of Translational Medicine", 2016 Sep;4(18):337.
13. Christen V, Duong F, Bernsmeier C, Sun D, Nassal M, Heim MH. Inhibition of Alpha Interferon Signaling by Hepatitis B Virus. - "Journal of Virology", 2007; 81(1):159-165. doi:10.1128/JVI.01292-06
14. Haybaeck J, Zeller N, Wolf MJ, et al. A lymphotoxin-driven pathway to hepatocellular carcinoma. - "Cancer Cell", 2009; 16:295–308
15. Hoopes JI, Cortez LM, Mertz TM, Malc EP, Mieczkowski PA, Roberts SA. APOBEC3A and APOBEC3B Preferentially Deaminate the Lagging Strand Template during DNA Replication. - "Cell Reports", 2016 Feb 16; 14(6): 1273-1282. doi: 10.1016/j.celrep.2016.01.021
16. Turelli P, Mangeat B, Jost S, Vianin S, Trono D. Inhibition of hepatitis B virus replication by APOBEC3G. - "Science", 2004 Mar 19; 303(5665): 1829.
17. Qiao Y, Han X, Guan G, Wu N, Sun J, Pak V, Liang G. TGF-β triggers HBV cccDNA degradation through AID-dependent deamination. - "FEBS Letters", 2016 Feb; 590(3):419-27. doi: 10.1002/1873-3468.12058
18. Bogerd HP, Kornepati AV, Marshall JB, Kennedy EM, Cullen BR. Specific induction of endogenous viral restriction factors using CRISPR/Cas-derived transcriptional activators. – "Proceedings of the National Academy of Sciences of USA", 2015 Dec 29; 112(52): E7249-56.
19. Klann TS, Black JB, Chellappan M, Safi A, Song L, Hilton IB, Crawford GE, Reddy TE, Gersbach CA. CRISPR–Cas9 epigenome editing enables high-throughput screening for functional regulatory elements in the human genome. – "Nature biotechnology", 2017; 35(6):561-568.
20. Braun CJ, Bruno PM, Horlbeck MA, Gilbert LA, Weissman JS, Hemann MT. Versatile in vivo regulation of tumor phenotypes by dCas9-mediated transcriptional perturbation. - "Proceedings of the National Academy of Sciences of USA", 2016 Jul 5; 113(27):E3892-900.
21. Carlos le Sage, Lawo S, Panicker P, Scales TME, Rahman SA, Little AS, McCarthy NJ, Moore JD, Cross BCS. Dual direction CRISPR transcriptional regulation screening uncovers gene networks driving drug resistance. – "Scientific Reports", 2017; 7: 17693.
22. Chavez A., Scheiman J, Vora S, Pruitt BW, Tuttle M, P R Iyer E, Lin S, Kiani S, Guzman CD, Wiegand DJ, Ter-Ovanesyan D, Braff JL, Davidsohn N, Housden BE, Perrimon N, Weiss R, Aach J, Collins JJ, Church GM. Highly efficient Cas9-mediated transcriptional programming. – "Nature Methods", 2015 Apr; 12(4):326-8.
23. Lucifora J., Xia Y, Reisinger F, Zhang K, Stadler D, Cheng X, Sprinzl MF, Koppensteiner H, Makowska Z, Volz T, Remouchamps C, Chou WM, Thasler WE, Hüser N, Durantel D, Liang TJ, Münk C, Heim MH, Browning JL, Dejardin E, Dandri M, Schindler M, Heikenwalder M, Protzer U. Specific and nonhepatotoxic degradation of nuclear hepatitis B virus cccDNA. – "Science", 2014 Mar 14; 343(6176): 1221-8.
24. Hu X, Zimmerman MA, Bardhan K, Yang D, Waller JL, Liles GB, Lee JR, Pollock R, Lev D, Ware CF, Garber E, Bailly V, Browning JL, and Liu K. Lymphotoxin β receptor mediates caspase-dependent tumor cell apoptosis in vitro and tumor suppression in vivo despite induction of NF-κB activation. – "Carcinogenesis", 2013 May; 34(5): 1105–1114.
25. Lukashev M, LePage D, Wilson C, Bailly V, Garber E, Lukashin A, Ngam-ek A, Zeng W, Allaire N, Perrin S, Xu X, Szeliga K, Wortham K, Kelly R, Bottiglio C, Ding J, Griffith L, Heaney G, Silverio E, Yang W, Jarpe M, Fawell S, Reff M, Carmillo A, Miatkowski K, Amatucci J, Crowell T, Prentice H, Meier W, Violette SM, Mackay F, Yang D, Hoffman R, Browning JL. Targeting the lymphotoxin-beta receptor with agonist antibodies as a potential cancer therapy. – "Cancer research", 2006 Oct 1; 66(19): 9617-24.
26. Chowdhury S., Kitamura K, Simadu M, Koura M, Muramatsu M. Concerted action of activation-induced cytidine deaminase and uracil-DNA glycosylase reduces covalently closed circular DNA of duck hepatitis B virus. – "FEBS letters", Vol 587, Issue 18, 2013, p.3148-3152
27. Landry S., Narvaiza I, Linfesty DC, Weitzman MD. APOBEC3A can activate the DNA damage response and cause cell-cycle arrest. – "EMBO Reports", 2011 May 1; 12(5): 444-450.
28. Опубликованная патентная заявка США US 2016/0317677 A1 «DELIVERY, USE AND THERAPEUTIC APPLICATIONS OF THE CRISPR-CAS SYSTEMS AND COMPOSITIONS FOR HBV AND VIRAL DISEASES AND DISORDERS», заявители: The Broad Institute Inc (US), Massachusetts Institute of Technology (US), The Rockefeller University (US), дата публикации: 03.11.2016, приоритет: 10.06.2016, МПК: A61K 38/46, A61K 48/00.
29. Ostling O, Johanson KJ. Microelectrophoretic study of radiation-induced DNA damage in individual mammalian cells. – "Biochemical and Biophysical Research Communications" 1984; 123: 291–8.
30. Tice RR, Agurell E, Anderson D, Burlinson B. Single cell gel/Comet assay: guidelines for in vitro and in vivo genetic toxicology testing. – "Environmental and Molecular Mutagenesis" 2000; 35: 206–21.
| название | год | авторы | номер документа |
|---|---|---|---|
| РНК-проводники для подавления репликации вируса гепатита B и для элиминации вируса гепатита B из клетки-хозяина | 2017 |
|
RU2652899C1 |
| РНК-проводник St10 для использования в высокоспецифической системе нуклеаз Streptococcus thermophilus CRISPR/Cas9 (StCas9) и применение указанного РНК-проводника и белка StCas9 для подавления экспрессии вируса гепатита B в клетке-хозяине и для элиминации вирусной ДНК из клетки-хозяина | 2018 |
|
RU2694396C1 |
| Универсальный эпигеномный репрессор транскрипции целевых генов в клетках человека | 2020 |
|
RU2748997C1 |
| НОВЫЙ АКТИВАТОР ТРАНСКРИПЦИИ | 2019 |
|
RU2800921C2 |
| МЕЧЕНЫЙ АНТИГЕН Е ГЕПАДНАВИРУСА И ЕГО ПРИМЕНЕНИЕ В СКРИНИНГЕ ПРОТИВОВИРУСНЫХ ВЕЩЕСТВ | 2015 |
|
RU2711750C2 |
| РЕДАКТИРОВАНИЕ ГЕНОВ С ИСПОЛЬЗОВАНИЕМ МОДИФИЦИРОВАННОЙ ДНК С ЗАМКНУТЫМИ КОНЦАМИ (зкДНК) | 2018 |
|
RU2811724C2 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas12 и препарат для выявления ДНК вируса гепатита B в ультранизких концентрациях | 2021 |
|
RU2792891C1 |
| Система CRISPR-Cas12 для выявления ДНК вируса гепатита B в ультранизких концентрациях | 2021 |
|
RU2782700C1 |
| Способ обнаружения ДНК вируса гепатита B в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2021 |
|
RU2782951C1 |
| ЛЕЧЕНИЕ HBV ИНФЕКЦИИ | 2011 |
|
RU2620966C2 |



Группа изобретений относится к области медицины, а именно к биотехнологии и генной инженерии, и может быть использована для получения системы для АРОВЕС/AID-опосредованной и/или UNG-опосредованной деградации ДНК вируса гепатита В. Система для АРОВЕС/AID-опосредованной и/или UNG-опосредованной деградации ДНК вируса гепатита В, включает слитый белок dCas9-Х, где dCas9 представляет собой мутантный белок Cas9, лишенный эндонуклеазной активности, а Х представляет собой эффекторный белок, выбранный из кор-белка ацетилтрансферазы человека p300 или VP48, VP64, VP160, VP192 и p65HSF1, и (ii) РНК-проводники, специфичные к регуляторным элементам одного или нескольких генов цитидиндезаминаз APOBEC/AID человека и/или гена урацил-ДНК-гликозилазы UNG человека, где указанные РНК-проводники используются для привлечения dCas9 к указанным регуляторным элементам. Группа изобретений также относится к применению заявленной системы для АРОВЕС/AID-опосредованной и/или UNG-опосредованной деградации ДНК вируса гепатита В. Использование данной группы изобретения обеспечивает активацию генов цитидиндезаминаз APOBEC/AID и/или урацил-ДНК-гликозилазы UNG человека с обеспечением дезаминирования цитозиновых оснований вирусной ДНК с последующей эксцизией образующихся урациловых остатков, деградацией вирусной ДНК и элиминацией ее из клеток человека, в частности, гепатоцита. 2 н. и 3 з.п. ф-лы, 3 табл., 8 ил.
1. Система для АРОВЕС/AID-опосредованной и/или UNG-опосредованной деградации ДНК вируса гепатита В, включающая:
(i) слитый белок dCas9-Х, где dCas9 представляет собой мутантный белок Cas9, лишенный эндонуклеазной активности, а Х представляет собой эффекторный белок, выбранный из кор-белка ацетилтрансферазы человека p300 или VP48, VP64, VP160, VP192 и p65HSF1, и (ii) РНК-проводники, специфичные к регуляторным элементам одного или нескольких генов цитидиндезаминаз APOBEC/AID человека и/или гена урацил-ДНК-гликозилазы UNG человека, где указанные РНК-проводники используются для привлечения dCas9 к указанным регуляторным элементам.
2. Система по п. 1, где регуляторные элементы генов цитидиндезаминазAPOBEC/AID человека и/или гена урацил-ДНК-гликозилазы UNG человека представляют собой промоторы и/или энхансеры соответствующих генов.
3. Система по п. 1, где dCas9 представляет собой мутантный белок Cas9 Streptococcus pyogenes, лишенный эндонуклеазной активности.
4. Система по п. 1, где dCas9 представляет собой мутантный белок Cas9 системы CRISPR/Cas9 типа II, лишенный эндонуклеазной активности.
5. Применение системы по любому из пп. 1-4 для APOBEC/AID-опосредованной и/или UNG-опосредованной деградации ДНК вируса гепатита В.
6. Применение по п. 5, где ДНК вируса гепатита B представляет собой кольцевую ковалентно-замкнутую ДНК, персистирующую в клетках человека, которые, в частности, могут представлять собой гепатоцит.
| WO 2017184768 A1, 26.10.2017 | |||
| WO 2015089465 A1, 18.06.2015 | |||
| Shuliang Chen et al | |||
| CRISPR-Cas Targeting of Host Genes as an Antiviral Strategy // Viruses | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Arnaud Perrin et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
| Albert W Cheng,1 Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system // Cell Res | |||
| Многоступенчатая активно-реактивная турбина | 1924 |
|
SU2013A1 |
| Зуева А.П | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
Авторы
Даты
2019-10-21—Публикация
2018-06-15—Подача