Область техники
Изобретение относится к области маркерной селекции, и представляет собою молекулярные маркеры для селекции сои по хозяйственно ценным признакам - процентному содержанию белка и масла в семенах и продуктивности.
Уровень техники
Молекулярные маркеры представляют собой последовательность ДНК, которая достаточна уникальна, чтобы охарактеризовать специфический локус генома.
Соя - один из самых динамичных и перспективных в России агрорынков, биологический потенциал которого сейчас почти не ограничен. Одним из ключевых требований времени к селекции сортов сои является проблема качества: содержание белка и масла в семенах, а также повышение продуктивности.
Селекция на качество у бобовых осложняется взаимодействием признаков, зависимостью их от внешней среды и значительным взаимодействием генотип х среда (Kumawat G, et al., QTLomics in Soybean: A Way Forward for Translational Genomics and Breeding. Front Plant Sci. 2016; 7:1852). Значительные отрицательные корреляции были обнаружены между продуктивностью и содержанием белка в семенах сои, а также между содержанием белка и масла, в то время как продуктивность и масло коррелируют положительно (Hwang et al. (2014). A genome wide association study of seed protein and oil content in soybean. BMC Genomics, 15:1). Было также показано, что условия культивирования влияют на эти признаки (Van Warrington et al., 2014. Resource allocation for selection of seed protein and amino acids in soybean. Crop Sci. 54, 963-970. 2).
Использование молекулярных маркеров облегчает селекцию хозяйственно-полезных признаков. Маркеры, тесно сцепленные с казуальным геном, позволяют быстро идентифицировать линии сои, несущие казуальный ген на основании анализа генома. Введение казуальных генов в линии сои тоже облегчается благодаря маркерам.
Разработка молекулярных маркеров требует обнаружения ассоциации между целевыми признаками и генотипами. Два подхода часто используются для выявления таких ассоциаций: (a) QTL анализ со структурированными семействами (Wang, J., et al. Identification and mapping of stable QTL for protein content in soybean seeds. Mol Breeding (2015) 35: 92. doi: 10.1007/s11032-015-0285-6) и (б) полногеномный поиск ассоциаций, который использует преимущества неравновесия сцепления (LD) в разнообразной зародышевой плазме, чтобы выявить ассоциацию между маркерами и казуальными генами (Bandillo NB, et al., Genome-wide Association Mapping of Qualitatively Inherited Traits in a Germplasm Collection. Plant Genome. 2017 Jul; 10(2):10.3835/plantgenome2016.06.0054). В данной заявке для разработки маркеров был использован второй подход.
Множественные платформы генотипирования и типы молекулярных маркеров были использованы для маркерной селекции, включая простые повторы последовательности (SSR), полиморфизмы длины рестрикционных фрагментов (RFLP), амплифицированные полиморфизмы длины рестрикционных фрагментов (AFLP) и однонуклеотидные полиморфизмы (SNP), которые лишены многих недостатков, присущих другим типам и поэтому предпочтительны (Sohn Н-В, et al., (2017) Barcode System for Genetic Identification of Soybean [Glycine max (L.) Merrill] Cultivars Using InDel Markers Specific to Dense Variation Blocks. Front. Plant Sci. 8:520. doi: 10.3389/fpls.2017.00520; Yoon, M. S., et al., (2007) BARCSoySNP23: a panel of 23 selected SNPs for soybean cultivar identification. Theoretical and Applied Genetics, 11,5: 885- 899). В настоящее время, для генотипирования и поиска SNP может использоваться генотипирование путем секвенирования (GBS) (Heim, Crystal B., Gillman, Jason D. (2017) Genotyping-by-Sequencing-Based Investigation of the Genetic Architecture Responsible for a ~Sevenfold Increase in Soybean Seed Stearic Acid (2017) G3: Genes|Genomes|Genetics, 1, 7: 299-308) или полногеномное секвнирование. Функциональные SNP-маркеры разрабатывают на основе известной нуклеотидной последовательности фрагмента ДНК, который содержит SNP, тесно сцепленный с казуальным геном. Использование маркеров SNP основано на амплификации фрагмента ДНК при помощи ПЦР со специфическими праймерами. Последующий анализ продуктов ПЦР может осуществляться разными способами в зависимости от типа маркера. В случае CAPS SNP-маркеров осуществляется гидролиз с помощью эндонуклеаз рестрикции, и продукты гидролиза разделяются с помощью электрофореза в агарозном геле (Шавруков Ю.Н. CAPS-маркеры в биологии растений. Вавиловский журнал генетики и селекции. 2015; 19(2) 205-213). В случае KasPar и TaqMan SNP маркеров для детекции продуктов реакции используется флуоресценция (Ma, Yu,, et al., (2017) Development and validation of breeder-friendly KASPar markers for er1, a powdery mildew resistance gene in pea (Pisum sativum L.) Molecular Breeding, 37, 12, 151).
За истекшее двадцатилетие было обнаружено много участков генома, контролирующих каждый из признаков интереса (Kumawat et al., QTLomics in Soybean: A Way Forward for Translational Genomics and Breeding. Front Plant Sci. 2016; 7:1852. Published 2016 Dec 21. doi: 10.3389/fpls.2016.01852). Однако из-за их частого плейотропного (одновременного) действия на отрицательно коррелированные признаки, а также из-за отсутствия у них большого эффекта (т.е. сильного влияния на признаки) и стабильности (воспроизводимости в разных условиях и в разных популяциях), очень немногие из этих районов были далее использованы или включены в селекционные программы.
Сущность изобретения
Одним из способов решения вышеозначенной проблемы создания сортов/линий сои с улучшенными сельскохозяйственно значимыми признаками является 1) создание панели маркеров, направленной на характеризацию сортов/линий по всем признакам - продуктивности, процентному содержанию белка и масла в семенах - и исключающей существование в одном гаплобоке (участке ДНК в котором однонуклеотидные полиморфизмы (SNP) наследуются вместе из-за неравновеского сцепления) наряду со SNP, оказывающими положительное влияние на один из выше перечисленных признаков, полиморфизмов с отрицательным влиянием на какой-либо из признаков, 2) панель маркеров разрабатывается на основании анализа нуклеотидного разнообразия сортов, используемых в отечественной селекции, для характеризации которых эта панель и будет затем использоваться.
Задача осуществления оценки геномной ценности селекционного материала сои и проведения селекции сои по комплексу хозяйственно-ценных признаков решается путем создания панели маркеров для генотипирования, представляющей собой набор из молекулярных маркеров на основе однонуклеотидных полиморфизмов генома сои. Описанная в данном изобретении панель маркеров разработана на основании анализа нуклеотидного разнообразия сортов, используемых в отечественной селекции. Она отражает аллельное разнообразие в таких сортах и это делает ее применение в отечественной маркерной селекции предпочтительным.
Указанная задача решается путем создания способа отбора сортов/линий сои с по меньшей мере одним признаком, выбранным из следующей группы признаков: повышенное процентное содержание масла, повышенное процентное содержание белка и повышенная продуктивность сорта, включающего следующие стадии:
а) выделяют геномную ДНК из растений сои по меньшей мере двух различных сортов/линий;
б) для каждого из этих сортов определяют последовательность ДНК в по меньшей мере одном релевантном для определяемого признака маркере, выбранном из следующих групп:
для повышенного процентного содержания масла в семенах:
SEQ ID NO:2, SEQ ID NO:5, SEQ ID NO:8, SEQ ID NO:11, SEQ ID NO:13, SEQ ID NO:16, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:27, SEQ ID NO:29, SEQ ID NO:32, SEQ ID NO:35, SEQ ID NO:38, SEQ ID NO:47, SEQ ID NO:48, SEQ ID NO:49, SEQ ID NO:51, SEQ ID NO:57, SEQ ID NO:59, SEQ ID NO:60, SEQ ID NO:61, SEQ ID NO:63, SEQ ID NO:70, SEQ ID NO:71, SEQ ID NO:80, SEQ ID NO:84, SEQ ID NO:86, SEQ ID NO:94, SEQ ID NO:96, SEQ ID NO:97, SEQ ID NO:100, SEQ ID NO:102, SEQ ID NO:103, SEQ ID NO:105, SEQ ID NO:106, SEQ ID NO:109, SEQ ID NO:110, SEQ ID NO:114, SEQ ID NO:121, SEQ ID NO:125, SEQ ID NO:127;
для повышенного процентного содержания белка в семенах:
SEQ ID NO:1, SEQ ID NO:7, SEQ ID NO:15, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:37, SEQ ID NO:43, SEQ ID NO:50, SEQ ID NO:56, SEQ ID NO:72, SEQ ID NO:73, SEQ ID NO:74, SEQ ID NO:75, SEQ ID NO:78, SEQ ID NO:93, SEQ ID NO:98, SEQ ID NO:112, SEQ ID NO:117, SEQ ID NO:118, SEQ ID NO:123, SEQ ID NO:128;
для продуктивности сорта:
SEQ ID NO:3, SEQ ID NO:4, SEQ ID NO:6, SEQ ID NO:9, SEQ ID NO:10, SEQ ID NO:12, SEQ ID NO:14, SEQ ID NO:23, SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:28, SEQ ID NO:30, SEQ ID NO:31, SEQ ID NO:33, SEQ ID NO:34, SEQ ID NO:36, SEQ ID NO:39, SEQ ID NO:40, SEQ ID NO:41, SEQ ID NO:42, SEQ ID NO:44, SEQ ID NO:45, SEQ ID NO:46, SEQ ID NO:52, SEQ ID NO:53, SEQ ID NO:54, SEQ ID NO:55, SEQ ID NO:58, SEQ ID NO:62, SEQ ID NO:64, SEQ ID NO:65, SEQ ID NO:66, SEQ ID NO:67, SEQ ID NO:68, SEQ ID NO:69, SEQ ID NO:76, SEQ ID NO:77, SEQ ID NO:79, SEQ ID NO:81, SEQ ID NO:82, SEQ ID NO:83, SEQ ID NO:85, SEQ ID NO:87, SEQ ID NO:88, SEQ ID NO:89, SEQ ID NO:90, SEQ ID NO:91, SEQ ID NO:92, SEQ ID NO:95, SEQ ID NO:99, SEQ ID NO:101, SEQ ID NO:104, SEQ ID NO:107, SEQ ID NO:108, SEQ ID NO:111, SEQ ID NO:113, SEQ ID NO:115, SEQ ID NO:116, SEQ ID NO:119, SEQ ID NO:120, SEQ ID NO:122, SEQ ID NO:124, SEQ ID NO:126,
при этом для каждого из этих сортов/линий определяют последовательность ДНК в одних и тех же маркерах.
в) для каждого сорта вычисляют параметр процентного содержания масла в семенах, процентного содержания белка в семенах и/или продуктивности сорта/линии, соответствующий определенному в (б) по меньшей мере одному маркеру согласно приведенной в описании SNP-панели Таблицы 1, при этом вычисление для каждого признака производят независимо, определяя значения эффекта маркера (ЭМ) по соответствующим значениям эффектов аллеля для каждого маркера, чья последовательность ДНК была определена и соответствующего данному признаку, по следующим формулам: ЭМ=ЭА1*2 (формула 1) или ЭМ=ЭА2*2 (формула 2) для гомозиготного состояния маркера, и ЭМ=ЭА1+ЭА2 (формула 3), для гетерозиготного состояния маркера, где ЭА1 - эффект аллеля 1, ЭА2- эффект аллеля 2, определенные в соответствии с Таблицей 1, и далее суммируя данные значения по всем маркерам, чья последовательность ДНК была определена и соответствующего данному признаку;
д) сравнивают вычисленные параметры каждого из определяемых признаков, соответствующих определенному в (б) по меньшей мере одному релевантному для определяемого признака маркеру согласно приведенной в описании SNP-панели, для каждого сорта/линии сои, и определяют сорт с желаемыми параметрами для определяемых признаков.
В некоторых вариантах изобретения данный способ характеризуется тем, что для каждого из сортов/линий сои последовательность ДНК маркеров определяют при помощи секвенирования ампликонов.
В некоторых вариантах изобретения данный способ характеризуется тем, что желаемыми параметрами для определяемых признаков являются одновременное повышенное процентное содержание белка и масла в семенах, а также повышенная продуктивность растений сорта/линии.
В других вариантах изобретения желаемыми параметрами для определяемых признаков могут являться повышенное процентное содержание белка в семенах, повышенная продуктивность растений сорта/линии, повышенное содержание белка и масла в семенах, повышенное процентное содержание белка в семенах и повышенная продуктивность или другие комбинации признаков, выбранные из следующей группы признаков: процентное содержание масла в семенах, процентное содержание белка в семенах и продуктивность растений сорта/линии.
Подробное раскрытие изобретения
В описании данного изобретения термины «включает» и «включающий» интерпретируются как означающие «включает, помимо всего прочего». Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из». Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Сортом растений является группа растений, которая определяется по признакам, характеризующим данный генотип или комбинацию генотипов, и отличается от других групп растений того же ботанического таксона одним или несколькими признаками. Сорт может быть представлен одним или несколькими растениями, частью или несколькими частями растения при условии, что такая часть или такие части могут быть использованы для воспроизводства целых растений сорта. Линия - внешне однородная популяция, выравненность которой поддерживается отбором. Термин «линия» применяют к группе особей селекционного материала, отличающихся от остальных характерным признаком или группой признаков и сохраняющих это отличие в течение ряда поколений. Обычно большинство особей линии гомозиготно по генам, определяющим характерные для нее признаки, но не исключено наличие гетерозигот по другим признакам, особенно по количественным. В рамках описания данного изобретения, термины сорт и линия являются взаимозаменяемыми, поскольку раскрываемый в данном изобретении способ отбора на основе панели маркеров применим как различным сортам сои, так и к различным линиям сои. Продуктивность сорта/линии определяют как массу семян растений в граммах, собранных с 1 кв. м площади.
Данное изобретение описывает ДНК-маркеры для определения генотипа и отбора сортов/линий сои, несущих оптимальный набор аллелей локусов генома, ассоциированных с тремя существенными признаками (продуктивность сорта/линии, процентное содержание масла и процентное содержание белка в семенах).
Описанная в данном изобретении панель маркеров создана на основании анализа естественной вариабельности в популяции сортов культурной сои, используемых в отечественной селекции, с помощью полногеномного поиска ассоциаций. Маркеры находятся в участках генома, контролирующих продуктивность (продуктивность сорта/линии), процентное содержание белка и масла в семенах и представляют собою однонуклеотидные полиморфизмы, оказывающие положительный или отрицательный эффект на эти признаки. Решение, предлагаемое в данном изобретении, состоит в создании панели маркеров, позволяющей 1) контролировать участки генома, оказывающие положительное влияние на один из выше перечисленных признаков и не содержащие в том же гаплоблоке полиморфизмы с отрицательным влиянием на любой из признаков, и 2) контролировать участки генома с отрицательным влиянием на продуктивность, процентное содержание белка или масла в семенах.
Возможность контроля участков генома, оказывающих положительное влияние на один из выше перечисленных признаков и не содержащих в том же гаплоблоке полиморфизмы с отрицательным влиянием на любой из признаков исключит ситуацию, когда участки генома, контролирующие, например, продуктивность, могут одновременно определять низкое процентное содержание белка в семенах и, следовательно, не подходить для дальнейшего использования в маркерной селекции. Т.е. такой подход принимает в расчет взаимозависимости между признаками. Дополнительно панель позволяет контролировать участки генома с отрицательным влиянием на продуктивность, процентное содержание белка или масла в семенах и, тем самым, позволяет исключать такие участки при скрещиваниях.
Праймеры для реакции полимеризации разрабатывали не на основе рефересного генома, а на основе последовательностей генотипированных или полногеномно секвенированных линий. Для детекции маркеров растения генотипируют по методу AmpSeq, включаюшем два раунда ПЦР (Shanshan Yang, et al., A next-generation marker genotyping platform (AmpSeq) in heterozygous crops: a case study for marker-assisted selection in grapevine. Hortic Res. 2016; 3: 16002). В первом раунде происходит амплификация маркеров и добавление линкерных последовательностей, а во втором к линкерным последовательностям добавляют уникальные для каждого образца баркоды. Полученный пул ампликонов секвенируется по протоколу Illumina, анализ результатов секвенирования проводится стандартными биоинформатическими методами.
По Таблице 1, содержащей данные о SNP-панели, можно определить ассоциацию маркеров с параметрами следующих признаков: продуктивностью сорта/линии, процентным содержанием белка или масла в семенах, а также для каждого маркера определить соответствующие значения эффектов аллеля. Например, маркер SEQ ID NO:1 соответствует признаку «процентное содержание белка»; поэтому, для вычисления параметра содержания белка можно использовать значения эффектов аллеля для маркера SEQ ID NO:1 (0,192631 или 0, в зависимости от аллелей, присутствующих в ДНК конкретного сорта/линии сои), либо значения эффектов аллеля для маркеров, выбранных из группы: SEQ ID NO:1, SEQ ID NO:7, SEQ ID NO:15, SEQ ID NO:128, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:37, SEQ ID NO:43, SEQ ID NO:50, SEQ ID NO:56, SEQ ID NO:72, SEQ ID NO:73, SEQ ID NO:74, SEQ ID NO:75, SEQ ID NO:78, SEQ ID NO:93, SEQ ID NO:98, SEQ ID NO:112, SEQ ID NO:117, SEQ ID NO:118, SEQ ID NO:123. При этом можно использовать как один маркер, так и любую комбинацию этих маркеров, в том числе одновременно все вышеуказанные маркеры; при использовании комбинации маркеров значения эффектов аллеля, соответствующие этим маркерам, необходимо суммировать для вычисления параметра содержания белка. Для каждого из сортов сои необходимо определить последовательность ДНК в одних и тех же (одинаковых) маркерах.
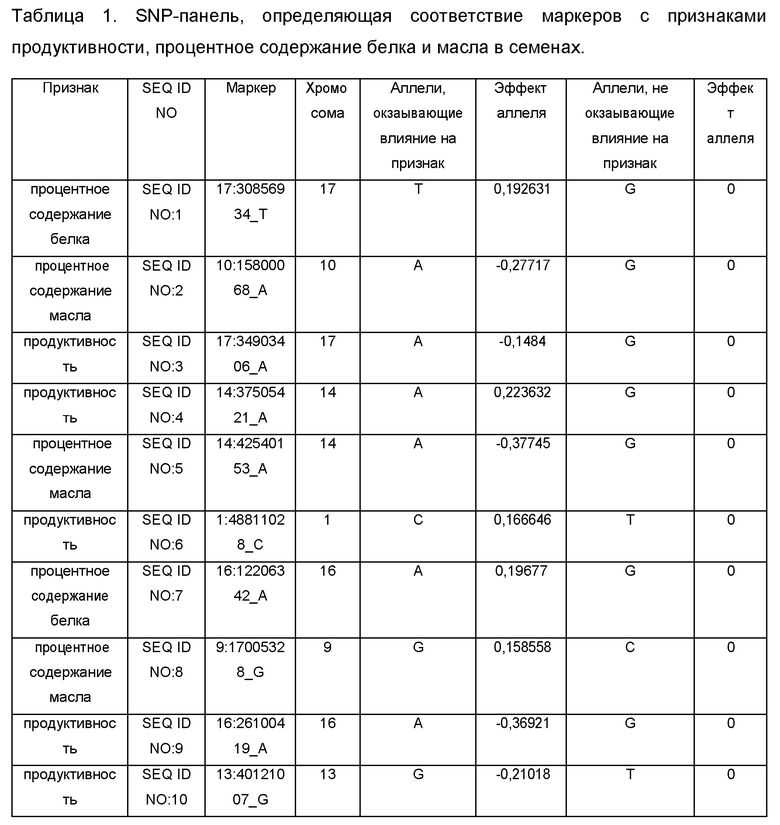
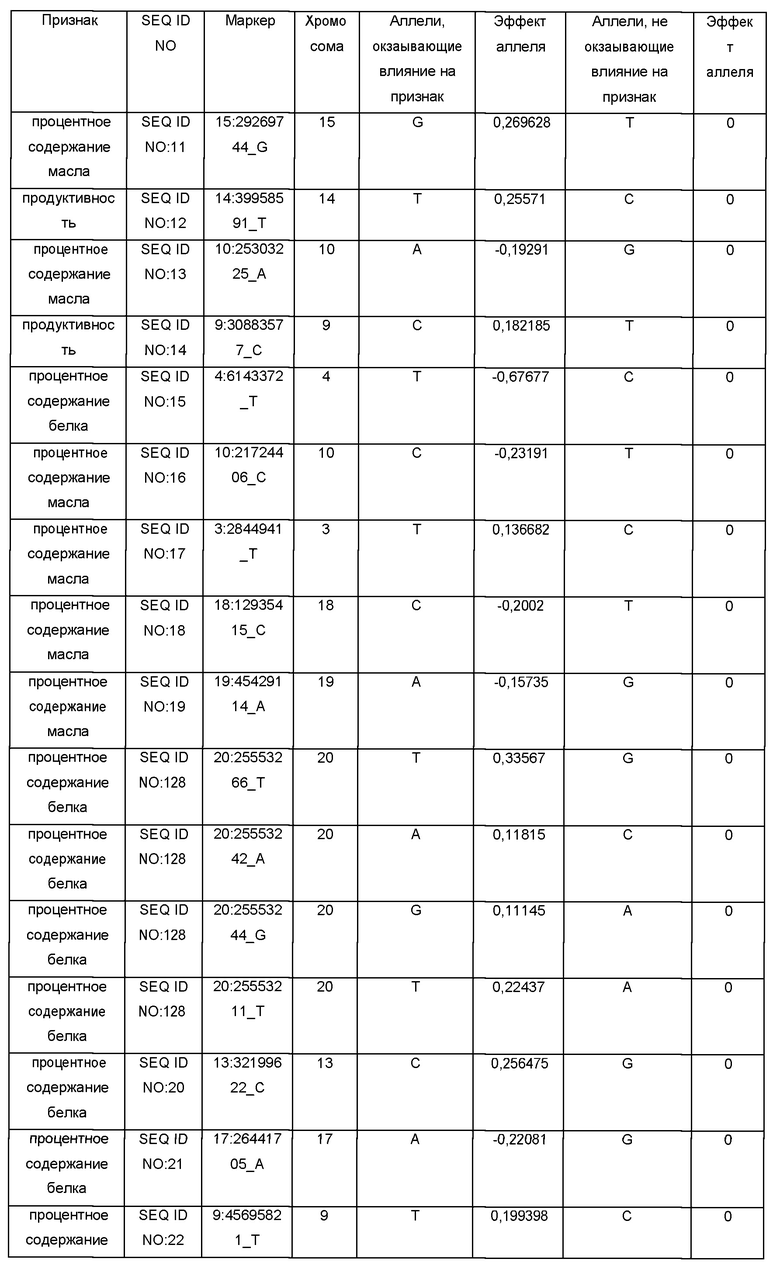
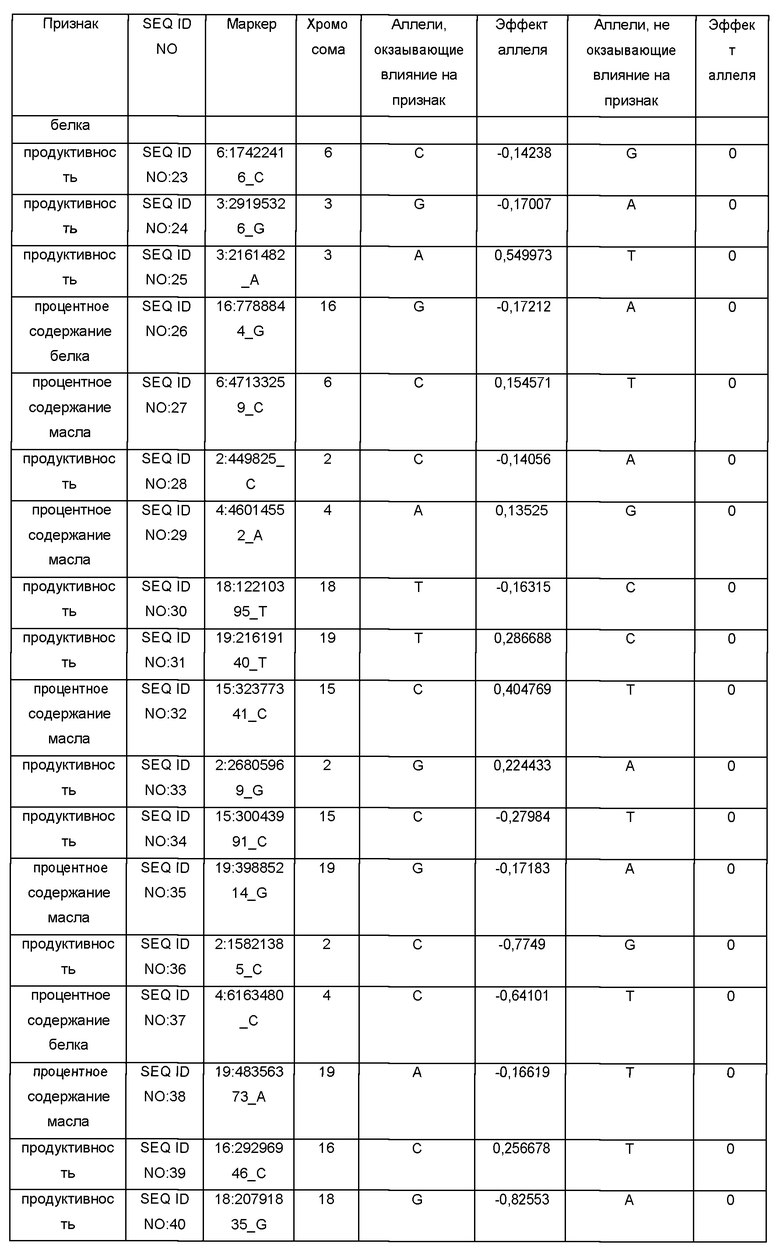
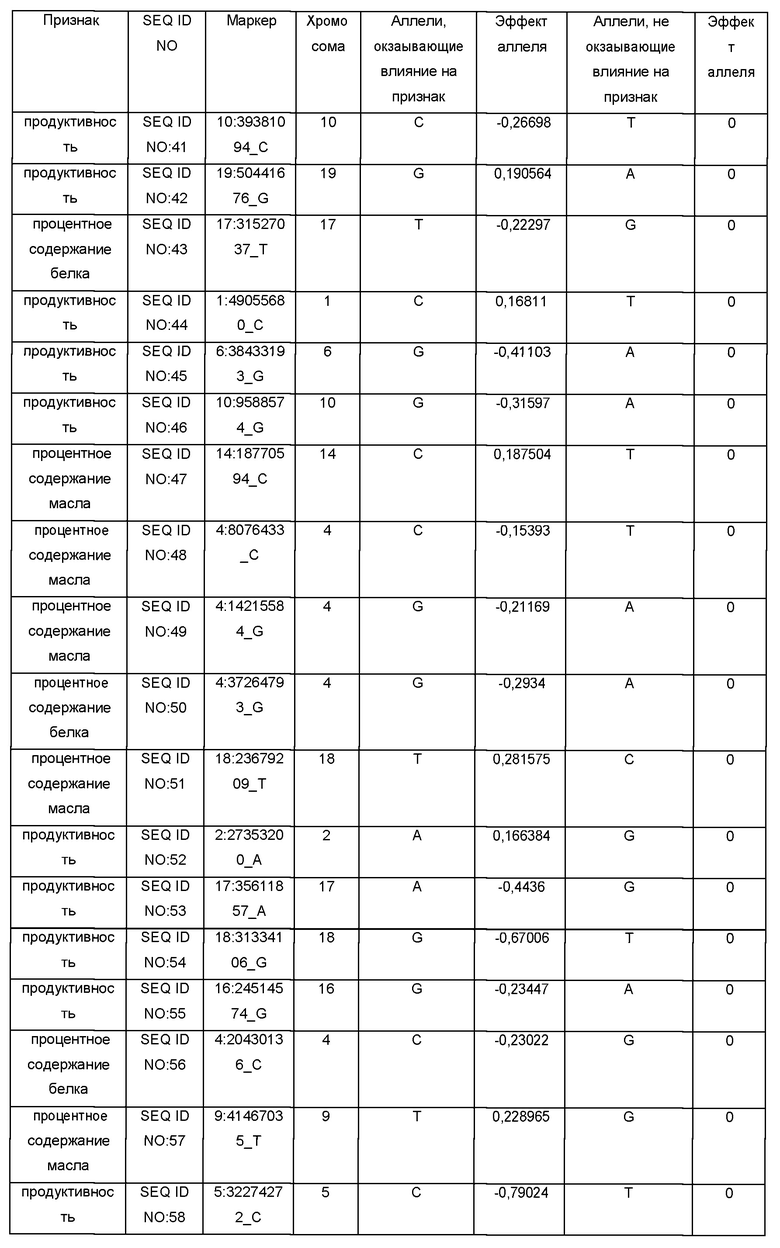
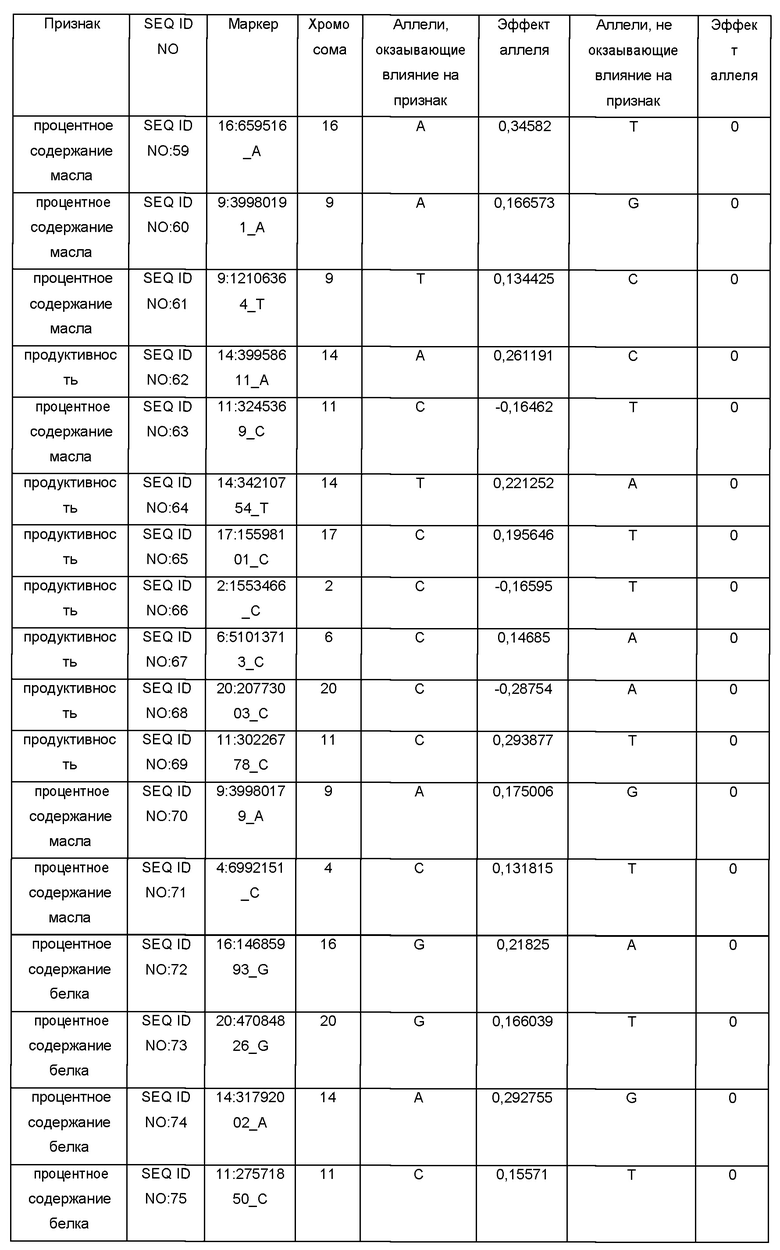
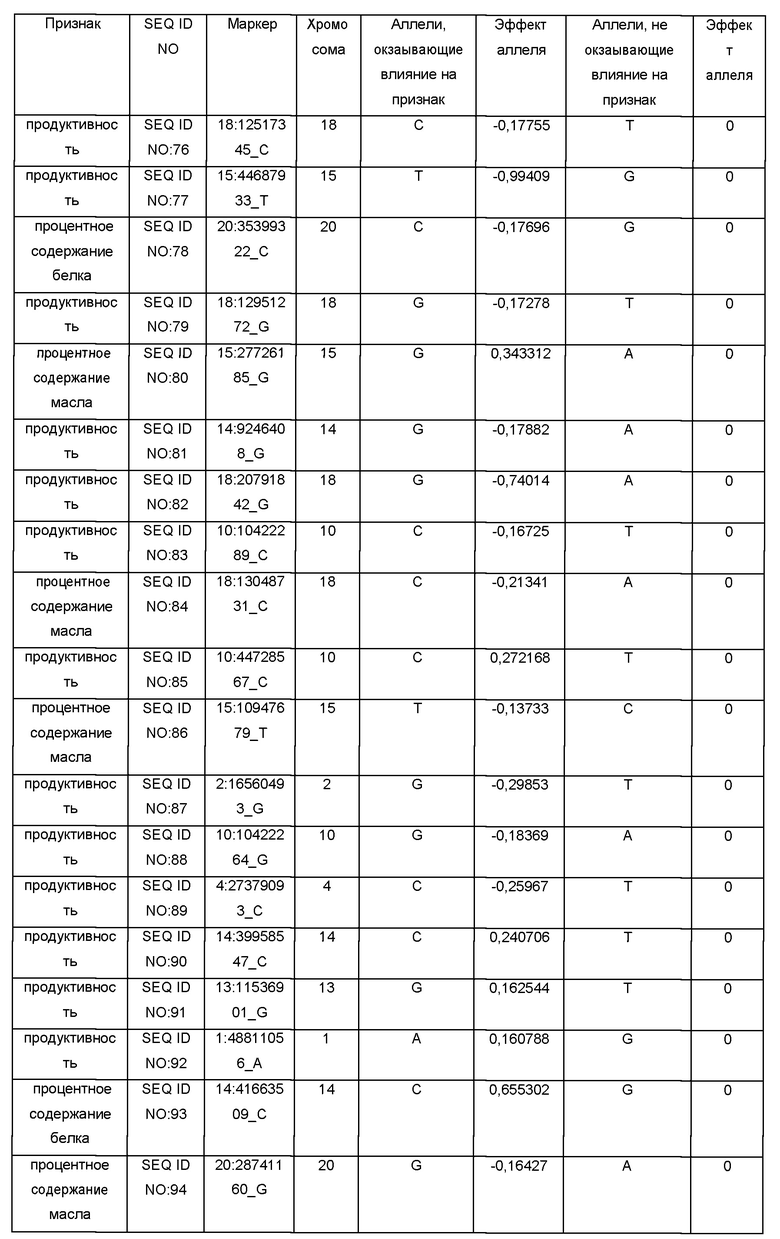
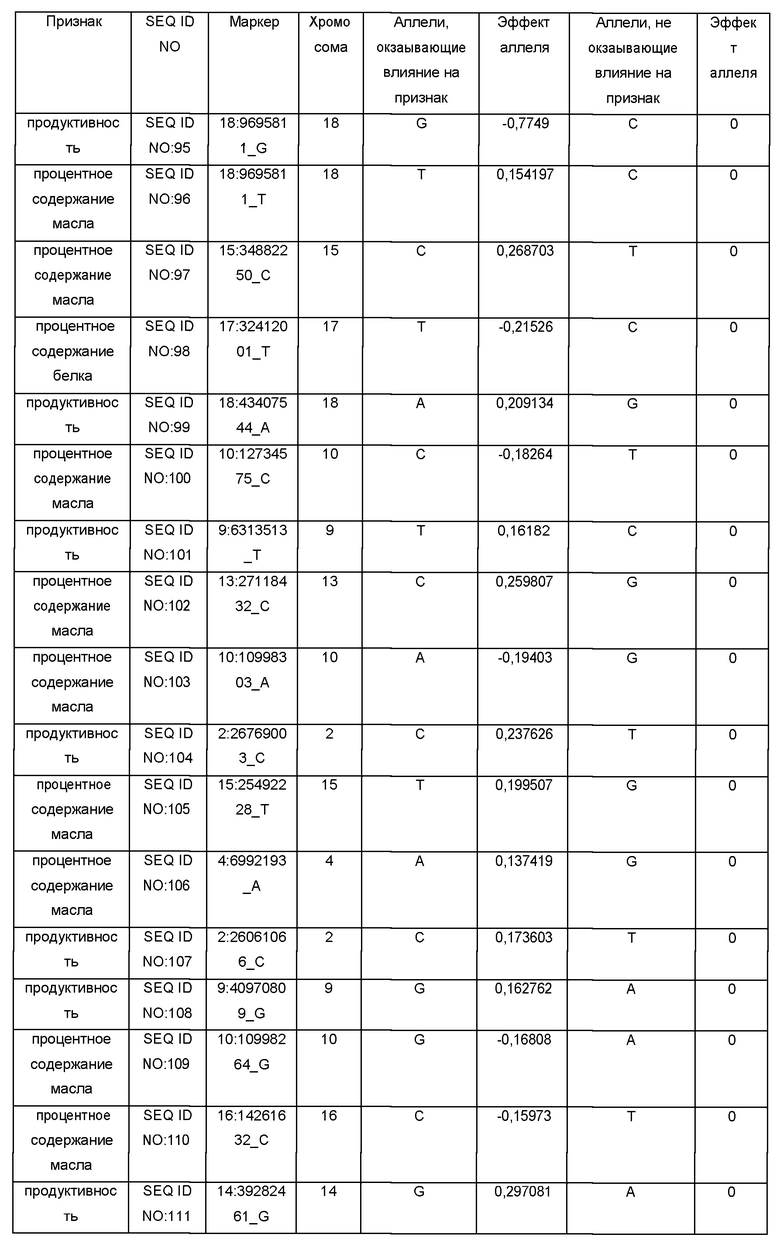
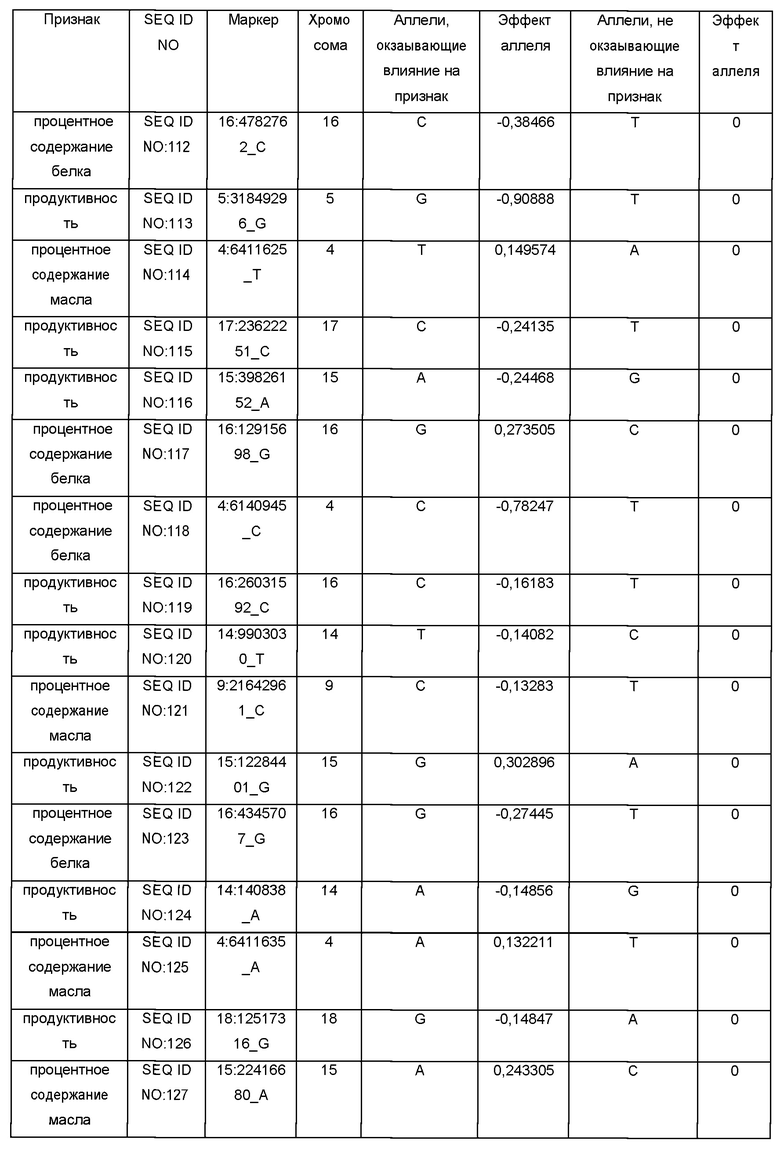
Коэффициенты "Эффект аллеля" были рассчитаны с помощью использованием однолокусной линейной смешанной модели, реализованной в пакетах программ plink (версия 1.9) (Christopher С Chang, et al., Second-generation PLINK: rising to the challenge of larger and richer datasets GigaScience, 4, 1, s13742-015-0047-8) и rMVP (версия 0.99.17) (https://cran.r-project.org/web/packages/rMVP/index.html). Каждый из указанных аллелей для процентного содержания белка, содержания масла в семенах или продуктивности сорта/линии может быть использован для предсказания независимо от других аллелей. Использование сразу нескольких аллелей для процентного содержания белка, масла в семенах или продуктивности одновременно повышает вероятность нахождения сортов/линий с существенно отличными показателями присутствия признака. Поэтому, хотя использование только одного аллеля для предсказания может быть достаточным для обнаружения разницы по присутствию определенного признака, в предпочтительных вариантах изобретения для предсказания используют одновременно или несколько, или все из указанных аллелей для одного признака.
Обозначения маркеров SNP панели в Таблице 1 соответствуют позициям полиморфизма ДНК в геноме сои, при этом авторы использовали версию генома сои Glycine max WM82.a2.v1 хранящуюся на сайте www.soybase.org. В названии каждого маркера закодировано его уникальное положение в указанном выше геноме. Например, для маркера SEQ ID NO:1 в колонке «Маркер» 17 перед двоеточием обозначает хромосому номер 17 из указанного выше генома. Число после двоеточия (в данном случае 30856934) соответствует позиции маркера от начала этой хромосомы. Для однозначной идентификации позиций маркеров на хромосомах сои ниже приведены фрагменты последовательностей геномной ДНК сои, содержащие маркеры SNP из Таблицы 1 (приведены 75 п.н. до каждого маркера и 75 п.н. после каждого маркера, каждый SNP полиморфизм в Таблице 1 указан после нижнего подчеркивания в названии маркера в колонке «Маркер»):
SEQ ID NO:1
AAAAACAAACCAACCACCAGGAGCCATTAACAACGAGAATGAAGCTTACCTTGACGGCA GAAGCAGCACAAATCG[T/G]TGGAGACTCCTCAATGACGACAACAGAAGAACTCAACGACGACGACGCGGAGGAAACTAAGGAAGACGAAGCAAG
SEQ ID NO:2
AAAAATGGTGGTGCAAATAGCAACCAGGCCCACTTGGGCCTTCCAGAAAATTCCTCCA GAAGGCGGTTGCTTCTG[A/G]AGGAAGCAACCTAGCTCGCCTGGGCGAGCTGGGTGGCAAGCTTCTCCCCTATTTTCCTATAAATAGGGGGAGAGG
SEQ ID NO:3
AAAGCAAAGAGAAATACTTGATACAAGGTTGATCTCGTAACAATTGAATTGACAGAGAAGCTTTCGAACCAAGTG[A/G]GAGAAGATTATTGTAGGGGAGAGAGGGGGTGATCATCGTTGGAGGTGCGGTGACTGTTTGGTTTGAGATGGTGGC
SEQ ID NO:4
AAAGCAAGAAAGTTATGGCATTGATTTTAGACTTTTAAATGGTTCATTTTAACCGATAAAAGTAAAAAGGGAGCG[A/G]TAAGGTGTTGGACCTTAGAACGGTTTCAAGCGATTTTCTTGCGGACAAAGCTTGATTTGTGAGTTGATTTTAACT
SEQ ID NO:5
AAATAAAAAACTAAATGATTAGTTTCACACTGAATCATTATCAAATTCCTGCACAACAGTACTAAACTTGTCAAG[A/G]AAAACACAAGGAGATTGTTTTTAACCAAAAAGAGACCGTTTCAACTTACACACCAGCAAGCTTGACTCCTTCTTG
SEQ ID NO:6
AAATTAAATTGAAAGAGAATTTGCAAAAGTTGTTTTCATTTGTCATTTATCATTTTCGCGA ATATTTTATTGTGT[T/C]AGTATCATTGTTCTATTTATTGTATGGATAACTTTTTTTTTTGTGTGTGTGTTATATGATCTAGTAGCGTATGGA
SEQ ID NO:7
AACAACACCAAGGTTCTAGGTGAGGCCCTCTCTCTTCTTTCTCTCTTCTCTTTTTCCTTTATTTTCGTTTTTGCG[G/A]TTCCAGTTCTGACTTTTCGTTTTAGCAATAAAATTTTGTTCTTCAATCTATAATTTCCTTCTCTATTTGATTAAT
SEQ ID NO:8
AACTAACTTTCCAAATGTTTGTCCCCGCAGGAAATGGTTCCGAGAAAGCTTTCCGCAAAAAGATCTAGGATGGAC[C/G]CCGCGGCTGAAGGGACCAGTGCCGCTCCCGAGTTTGATAGCCACTGTTTCAGGAGCACTGAGCATCAGCAACGTT
SEQ ID NO:9
AAGAAGAGGCACAAAGAAGATGGGCAAGCAATGAAGAAGAAGACGTAGGAAGAAGAAGAACCAGGAAGCTGGTCG[G/A]CTTGTTGATGGTTTCTCCAGAGTCTGCTAGGGCTGAGACAGTCCAGTGTTGCGGACCATACGTACGCCTGCAATG
SEQ ID NO:10
AAGAGACTTGAATGCATACTATGTCATAACTCATAAGGTTTACGTTGTAACAGGGTGACCATATAATTAAAGGTT[T/G]ACAAAATCATAGGTGTGCTTATTAGGTCTAATAATGGGGAAGGTGAGAGCTCCGTTTATGGTGCTGCAATAGTAA
SEQ ID NO:11
AAGCATTTCCTCCGTGGATTCCTTCTCTGGGCAAGCTTGCCTGTCTGGGATGGGTTTAGGCAAAGACAACGGCGT[T/G]ATAACTAGCCTGATAAATGCCAAAGGAAATCGTGGGAAGTATGGGTTAGGCTATAAGCCCACTCAGGCAGATATA
SEQ ID NO:12
AAGGTACAAAGCTTGATTAACGCTAGATGGTTGACATTCCAAGAAGATAGTCCGAATGTGAGGACTAATCCACTC[C/T]TCAATCATAAAGGCTTGGCGGTGAATGCAGTGGAAGATTGGGAGCCTCGAGGGCTGAAACAAATAGGGAATGTGT
SEQ ID NO:13
AATTTCAGACCCAATTCACAGCAAATAAACATTTTATTCTGCTTTCAAGCAGTTTTAATCACACAATAAACATTG[G/A]ATTATTGATTTATCATAAACAACTATCAACAAATGAATTCAATGTTCAAATT ACATAGGAAATGTCAAAAATGTT
SEQ ID NO:14
AATTTTGAGTTCTTTTTAGTTACAGATGGAATCAAGAAAAAAAGCTAGAACGAAAGACATCACTTCGTCATCCAT[T/C]TCAACTCGTCCGTCAGTAACTATAGGACAGGATGTCCAAAGCTCACTAACTTTAGTTCCTATGC TACAAAGCTTA
SEQ ID NO:15
ACAATGATGATTTTTTCAATTATTAAATGGAGCTTTTGGGGTTTCTTTCTCGCTGCTTTTGCGTAATTGCTGTGC[C/T]AAACACGCTCTACATATTTACAATAACAAGCTTGGCTGTTTTAACCCAGCCTTGTCTTTAGCATGCAGTGGCATA
SEQ ID NO:16
ACACGAAAAGAGGCTAGGGTCACTACCAATAGGTCATCGTCCTTAACCTAGCCCGGGACAAGCAGAAAGTGCTGT[T/C]GTAAAGCAGCCCAGTATCCTTTGAATTCATAGTAATTTTTACCTTTGTTCGATGCCTAATCTATCCTGGGGGGTC
SEQ ID NO:17
ACAGCTCCCATCCCCACTCCTGAAGCATCGGTCTCCAATGTGAATGATTTGGTGAAATCTGGCAAGGCCAGCACC[C/T]GCGCTGTAGATAGAACCTCCTTGAGTTGGTCAAAGGCCAGTTGAGCTTGGGTTGACCAGTGAAAAGGGGAAAGGG
SEQ ID NO:18
ACCTAGGACAAAATTGACAGAAGCAACCACTTGTCAAGGTTGGAGGGTTTGACAAGCTTGACAAGCCTGCAATGT[T/C]TGCACCTATCGAGGTTACCACTCTCAACATCAGACAATACTTGTCCATACTTGGGGGCTCCAAGAACTCGCGACC
SEQ ID NO:19
ACCTAGTTGTGGACGGAGGTTACAGTGTTAACAATACTGCTTCGGCTGAAGTAGCTTTAGGAAAGTTTTCTGCTG[G/A]TTAAGCCCAAATTGTAAGCTTTCATATTTGAGTTCCTTCGACCAAAAGCCAAAAGGTGTGTTTTCTAGTTTGAAT
SEQ ID NO:20
ACGTTTGGAACGCAAGCTGTGTCCTGGCTAGACCTAATATCCCTTTACCACTTTTGGGTAAGTTGTTTAGAAGCG[G/C]AGTGTCATCAATGGAAGTGAAACCGGAAATAAGACCAGAGAGTGAGAGTGAGGTGTGGGACAGGAACAGGACATC
SEQ ID NO:21
ACTAGGAGAGACTCGACATAAGCATTGCTTACCACTTCCAAAGCTTGGAAGGATATCTCTAAGGATTCTTCTGCG[G/A]CTTCCACATATGGTGTAGAAGAAGGATAACTTACAAGAATGTCTTCCTCCTCGGAGACTATGATTAACTGCCCTT
SEQ ID NO:22
ACTTGGCCAAAAGGCAATCTTTGAACGAGTGCCAACCATTTTCCAAGTCCTTGTCATAGCAAGCACAAAGTGTGC[C^GACCCCAACGTGTCACTTTCTTAAGTTGATTCGTGTGGGCAAATTATCTCTATACGTCCTACAAGTGAAAATGTC
SEQ ID NO:23
ACTTTGTTCAAGTGTTGAGACGAGAGTTAAAATTAGTAGTTCTCCTTTTTAATTCTATTAATCACAACCATTTTG[G/C]TGTAGAAAAAATCCATCCACAGAAACGAAATGAAACTCCAATTGAATGACAGATTCACATTGCATATCCAATTTC
SEQ ID NO:24
AGAAATTTGAACTCTAAAGCATCACCATTCTCCTCTTAAGCACTAAACTCTATCATCCATCTACCCACATTGTCA[A/G]AGATAACTCCCCATATACCCATTTGATCTATAGGTTGTTGGACAAAAGCTACCGTCTACATTAACTTTAATCACC
SEQ ID NO:25
AGAAGATTGGGACCCTATAATGAGATCAACATTTGAAGAATAAGCAACAGACAATATTCTTTCGTTTGGCCAAAT[T/A]GGATCCAGAGGCAAGAAGGCTTCCCCACACCTCAGTACTGAAAGAACAGCAACAACATATTCAACAGAAGGAGGC
SEQ ID NO:26
AGCATGATGGAAAAAGGCCAAATCCGAGTAGGGCTGGTTCGGTTTGGAAAAGCTTAAAGGGAGAAGCACTAGACA[A/G]ATTCCGATAATCGATTAAGAAGGTGGAGCCCCCTTCTTTCACTAGCGAAGATCCCGTCAGCTGGATTTCAAGGGC
SEQ ID NO:27
AGCTGGAGCGACCAAAGTGGGTTGGGGAGACCCCTCTTTCACGTCTCGTTCAAGCTCTCATCTCCTTCAAGCCTT[T/C]CTACTCTGTTCTCAAGCTTGGTGCTAGAAGGGCTTTGATAAGGTTCATCATCATTCATTCCTCACTATCCTAATT
SEQ ID NO:28
AGCTTCTCATCCGCCCCGAGCAGCGCGTCAATGTCATTGTTCCTCTTATCATTCCAGCGCCCATTCCCACCACCA[A/C]TTTCCTTCACATCCTCCCCAGCATTCACCTTGTCTTTCTCTTTACCGATCAGAGCCAAAAACTCGACTGCAACCA
SEQ ID NO:29
AGGTAGCACCCATCCATCAAGAAGCTTCTCCTCTTGAAGGCGTGTAAGGCTAGCCGCTTGAACCAAGGTCAGGGG[G/A]TGCAACGCCTGGACCTCACACCGGATTTCCAAGGTGAGCCCAGAAATAAAACAACTCAGGAGGAACGGCGGAGGA
SEQ ID NO:30
AGTATAAGAATAGATGAAAAAATAACCCAAAAAAGTGAAAAGAAAAAGCGCAGACAAGAAAGCACATCA6AATTC[C/T]GTTAGCAAAGCTTTCCAAGCTCAGCTCTATACTCATTCAACCAAGAAAGCTCTCCAAAATTACCATTGTCTGAAC
SEQ ID NO:31
AGTCAGGGAACTCATCATCCCTCAGCTGGACCCGCCTTTCTCTGAGGAATGACCATCCCTTGATGGTCTCGAAGC[C/T]CTGCTGATGCTCGGCGCTCCGAAATCGGTGGCTGTCATAATCCGTGTTTGGTGGCTGGGCAGCACTGGAGCCTTC
SEQ ID NO:32
AGTTGTACCAATGTTGCAAGTTCCAGGTTAGTGTCCAAACTGAATTTGGATACTAAGCCCCATCCTAGGCCATAT[T/C]GACTTTAGTGGCTTAGTGAAAATGAAGAGGTAAAAGTGACTCAGCAGGTTGAGGTATGTTTCACCATTGGAAGAT
SEQ ID NO:33
AGTTTTAGAAGAACGAAGTCTCTGTGACTCAAGATAATCAATTACCGCATTTACTAATCGAGTAGATTGCATCTA[A/G]AAACTTTAGGAGCTCTTTGTGCTTCTAAATAATTACTTACCACATTTGATAATCGATTATTCCATAACCATAAAA
SEQ ID NO:34
ATAGGGGTGAACTTAGCTATTATAGGGGGTGTGTAGCTAAGCTCTAGCTTCTCATCTCAAGGAGGTGAGCTTAGT[T/C]ATTAGAGAGGTATGTGTAGCTAGGCTCTAACTTCTTTAAGAATCTTCTTAGGGAAACTTCTCAAGGAGGTGAACT
SEQ ID NO:35
ATATTTTTAAGCCATGTCCAGCAGAAAAATATCGCGTCATCCATCAGCTTTTGGCCGTTGAAGCTTTCGTTTGAA[G/A]AGACAATCCTGGTCCTATGATACCATATGCACCAGGTCAAAGAAAGGAATCCACCCAGTTTGCCATCGCTGAAGT
SEQ ID NO:36
ATCAGGCAACCCCTAAGGAAGAATGCATCACACTAGCGTCATTTTACCTCAACGGACCTGCCCTCAGCTGGTTCG[G/C]GTGGTCCTATCGAAAAGCTTTTATCACTACCTGGCCAGGATTCTTGCAGGTGCTGGAGTCAAGATTTGCACCGAT
SEQ ID NO:37
ATCCCAGACTTCTTGAGAGCATCCAAGATACCAGCATCAGCATCAAAAAGCTTCACCTTCTGAATGCCATTGTCT[T/C]TCAGCATCTTCACCACCTTCGACGGTGACAAAGGGTGTGTTGACTGTGTCCCCCAATTAACACCAATCCCACTCA
SEQ ID NO:38
ATCCTTATTTCGATTAAAAGTACTAATTACAACGCAAAACCCTAACTAAAATGTAAGGAGGTGCAGAAAGCTTCT[T/A]CGGAGAGAAAATGACGAGAAATAAAAATATCTTTCCGATTAGTAAAGTGGCGCCATAATCAAAGCGTAATTTCAG
SEQ ID NO:39
ATGAAGGTCGGAATTCTTTTATGCGACATTCATCCAAGTCAAGTACTGTTATATTTTCCATTTTTCCTAATATTT[T/C]AGGAAAACTCTCGAGATTGTGACAACCCGAAAGTTCAAACTGTTCAAGAGAGGTCAACTTCAATGGTGGAAAACT
SEQ ID NO:40
ATTAAAAGCTTTTTTCAATCGCCGATACGAGTGAAAATGTTTCAAAAATCTTCTATGTCTGGTATATACTTGTCA[A/G]AACCTACCCTTCGACGGGTGGCACGAAAAGGCCTCTCGTATGGGCCAAGGGTGCGTCTTCCATCGAATGAAAACG
SEQ ID NO:41
ATTATGGAGAGTGTTGATGTTGTGCCCTACCACCTTCAACTTCCGGCTTCATCAGAGATTCATCCCGTTTTCCAT[T/C]GTTCGCTGTTGAAACCTCACCACAGGGACATTCCCACTAAGATTGCCACCTTACCATCTAAAGCAAGGGACCATC
SEQ ID NO:42
ATTCCTTCTCCTTCTGCCTCTCAACCCAAAAAGACGAATACTTTGCCTAAGCTTTCCATCTCTGTTGAATTGCCA[A/G]CTCACATAAGAAGAACAACCGTCTCGTTGTCACCGCCAATAGCAACACATTGACAAAGGCAAAGAAAAAGACCGA
SEQ ID NO:43
CAAACGTTTGGAAAGTTAATTTACAAGGAAATGCCATTTTAAAGCAAAAACGGCATACTAATCTTTTCGACTTAG[G/T]ACAAACTTGTGCACACATTCCCTAGAAGAAGAACATTTATGAACGTGCATACGCACAAAATATCCTGCTATCTAT
SEQ ID NO:44
CAAGATCAACTTACAAAGTCGGAAAAAAGGCATAAGTCAGACAAGTGTTAGCCTCTCCCAAAAGGGCCCAGGTAT[T/C]AGCGTTACACACCAATCGCACTACAATTATGGAGGAAGCTTTTAATTTGGAGGTACCTATTAGGTTACCCGCGAC
SEQ ID NO:45
CAATGTGCAACCAACTAGATGCAACAAATATACTCAAGCTTCTACGGTCCACTATCCCGCATCACATTTGCTACA[A/G]TAAATGTCTTGTAGCACGGCTAGCCGAACATATGCCCATCTGGTTTGCAATGTCTCATTTTTTTCTTCTTGTGTA
SEQ ID NO:46
CACCATTAGCCACCACAAAGCGCCGTTGTTCTCCGTTAAAACCCCACACCGAGAGGAACCCTTCAACCAAAGCGA[A/G]ATCTTCCAACTTGCCTCGCTGTTTCGGTAGAGAACGAAACCCTAATCTAACCTTTTGTTTTCCTTCAAGGTAACC
SEQ ID NO:47
CACTCCTCGTGACATTGGTATAAGAAGCCGCTTCGACCCATTTGGTGAAATAATCTATCACCACGAGGATGAAGT[T/C]ATGACCATTCGAAGCTTTGGGTTCGATGGCCCCAATGGCATCTATCCCCCACATGGAAAAAGGCCAAGGGGCAGA
SEQ ID NO:48
CAGCCCAGTACGAGTCCTTTCTCGTCCCCGGTCTTATTAGTGCGCAAGCAATATGGCTCATGGCGGTTCTGTGTT[T/C]ACTACAGGGCCCTCAATGCAGTAACGATTAAGGACCAGTTTCCGATTCCTACTATCGACAAACTGTTGGACGAGC
SEQ ID NO:49
CAGGAAACACAGAGAAAAATCCTAAGGAAGAGTGCAAAGCTGTGATGACTAGAAGCAGAATGGCGATTCAAGTAA[A/G]GGAAAGTAGAGCTTGTAGAAGCAAAGCTTCATGATGAATCAAGATTGATTCAAAGATGTTTTGATGATAACAAAG
SEQ ID NO:50
CAGTTAGCGAAATTTCAGGGCGTTACACTATGATACCATAAGGGGATACCGCGGTATCTCAAGGGAACCATCTCA[A/G]GGGGCTCGCATTTCAAGCTTGCTTCTGCACCTTTTTTCTATTGTGACTTTCTGTGATGCTGACTAGGCGTCTGAC
SEQ ID NO:51
CATAGATTGAAGGATGAAGATGTTGTATGTGATATATTTTAGAGTCATCCTGATACAGTCAAATTATCCAATGCC[C/T]GTAATTTGGTATTTTTGATAGATAGTACCAACAAAACAAACAAGTGAAGGCTGCTGTCGCGACCTCTCTTCCGGC
SEQ ID NO:52
CATATCTGCTAAGCCCAAAAGGCCCGCATAGGAAGCAAAGGAGAAAGACACATCGAGACTCAGAGCTCTCTAATG[G/A]ATACATCCAAATCCTAAGCATCTCTAATAGGGGAAACCCTCTTTCTATAGTCATTCCCCCCTTCTCTTTCATTAT
SEQ ID NO:53
CCCAATTCAAAATGTTAAAATTCCAAAGAAGCTTTTGGGCTTAGTGCGCAAGATCGTGCATAGCGGGTACTGCAG[G/A]TTTGATTGGTCCTGCAACTTGCGCTTAGCCGCCAAAGGCTACACTTAGCGACTTAATGCTCATTAACTGCAGTAG
SEQ ID NO:54
CCCACTCAAGCTTGACGCAGTACGCAGAAGTTGAAAGACAATAGGAGCTCTCAAAGTCGCATCTCTCAGGAACTT[T/G]TCTCAAAGCAAAGAAGGGACCTTTGGAAAGAAGTGGAACCTCCAAAGATACAGTTTTAGAAGAAGGTGCACTTAT
SEQ ID NO:55
CCCCTGACGTACAATCAACACTCAAGATGATCCAAGTCTCTCCCACATCCACCATCCTGTAGGGCGGACTACTCA[A/G]CGACACACCTTTGTTGACCGCATCATAGAAGCTTACATACCTCTAGGATGGAAACCACTCAACCTGGAGCGATAT
SEQ ID NO:56
CCGCCATTGTGGTTACAGTGGTTGTTGTGGAGTGTGCGGGATAAGGGAGATGGAGAGTTTTCTGGTGCAGTAGGG[G/C]GCAATTTGAAGCTTATTCTGAATTTGAAGCTTTTTCTGAAGAGAGAAAGGAGAATTTGGGTTTAATTTTGGTTTT
SEQ ID NO:57
CCGTTGTTGTTACCATTTCATTTTAGGGCTTTTTTCTAATCAAAATTGGAAAATAGAATGACTGTGTCTGTGTGG[G/T]TCCTCCCATAAGGTGTTTGTGAAAATGCTACAATGAGGAAAATAGTTGGATAGGGTTTTATTTGTACTTCTATTG
SEQ ID NO:58
CCTCTGCCAAGCGCGTGTTCTTCGTGTCCGCCACGGTGGCGCCGAAGGTGTCCGCCCCGCAGCGGCAGAAGGACT[T/C]GAAGAAGCGCGTGGTTATCACGGGAATGGGGCTCGCGTCGGTGTTCGGGAACGACGTGGAGGGGTACTACGAGAA
SEQ ID NO:59
CCTTTACTCAAAGCAGGAATTAACACTGCATTTGTCAATGACTTTCCTGCTCTCAATGGCCTTGGCGTCTCTGCT[T/A]CACGTGTAGTTATTGAAAAAGATGGATTTTTTCCACTGCACACACATTCAGATGCCACTGAACTAATAATCACAG
SEQ ID NO:60
CCTTTCATAGATAAAGCAATGTTGATGTTCAAAGGACACTTTAAATCCCTTTTTAATCAACTG[G/A]CCTACACTTAGAAAGTTTTGGTCAATGTTAGGTACATAAAGAACATCTGATATTAATTTGATACCTGAACACGTTGAAATTGCAACA
SEQ ID NO:61
CGACGGCTGGTGTGTTTTCGCACCGCACCTTGGCTCTTCATATCAACACCATAAAGCTTGGCTATATCGTCAACC[C/T]CCACTAGGTCAGAGTCCAAATATATCACGTGTTTCACGTCTTCTGGTATGGTATTAGCCAGGTATATTTTGGCGT
SEQ ID NO:62
CGCTAGATGGTTGACATTCCAAGAAGATAGTCCGAATGTGAGGACTAATCCACTCGTCAATCATAAAGGCTTGGC[C/A]GTGAATGCAGTGGAAGATTGGGAGCCTCGAGGGCTGAAACAAATAGGGAATGTGTTAACTTTTAGGTGGTTCATA
SEQ ID NO:63
CGGCTACTACAACACCTACACCGGCGGCATTCCGCCGGAGATCGGAAACTTGTCGGAGCTGGTGAGGCTTGACGT[T/C]GCGTACTGTGCGTTGTCCGGGGAGATTCCGGCGGCGCTTGGGAAGCTTCAGAAGCTGGACACGCTGTTCCTTCAG
SEQ ID NO:64
CGTACGCAGAGAGAAGGAACTTTTCTTTAATTGTGATGAGCGGTTTAGTCGTAGCCATCACTGCTTTTCCAAAAA[A/T]TTTCTACTGATAGCTAATGATGATAAACTTTCACCATAAGAAATGCCACATACTGAAGCTTTTCTGGTTGCACAG
SEQ ID NO:65
CTCGGTGAAGAAAGGGAACGAGTTGTAAAGGAAGAATTTGAGAAATTGGTTAGAGCTGGATTTATTAGAGAAGTT[T/C]CATACAAACATTGTGATGGTCAAGAAATCCAACAGAAAGTGGACGATGTGTGTTGACTACACTGACCTCAACAAA
SEQ ID NO:66
CTCTGTGTTCCATCCTTAAACCCCTTGTGTGGCTGAGCTGCACACTCAGCTGCAAATTTTTTAAGCTTCTGCAGT[T/C]CCTCAGTCTCCCCTCCTACCATAGAAGCATTAGCAACGCACTTTCCATTCTGCACAATGATAACCTACATAAACC
SEQ ID NO:67
CTGATGAATGAGAGTTTCCCAAAATTGCTATTGGATGCCAATCACTGCATTGAATGAGATGGATTCAATTAGTTA[A/C]AGTCGATGCCAGATACAACAAAAATGACCCTCCAAAACTCTTGCTCGACAGAAGCCAATACCAAAATATGGTCTA
SEQ ID NO:68
CTGCCTAAGGAAGGCCGTAATCAGATCCTTCCCGGTGCGGATGCGGGAAGCTTCTAGGTTAGTGTACCAGATGAA[A/C]GCTGCCCTAGCCAAACAATCTTGAAAGAAATGCATCAATAACTTCTCGTCCCTGGAATATGCACCCATCTTTCGA
SEQ ID NO:69
CTTCTCATTCCCTCATTCACACACTCTCTCCCTCATCTCCTCCCTGTTTGCACTCTGCAGAGAGGAGTTCAAGGT[T/C]TTTGTGTGTGAGTTCTGTCTCCATCGACCATCGACCATCTCCAGTCTTTTTACAGCCATCTCTGTTTCTTCCTTC
SEQ ID NO:70
GAAAAGCTTTTACCTTTCATAGATAAAGCAATGTTGATGTTCAAAGGACACTTTAAATCCCTTTTTAATCAACTG[G/A]CCTACACTTAGAAAGTTTTGGTCAATGTTAGGTACATAAAGAACATCTGATATTAATTTGATACCTGAACACGTT
SEQ ID NO:71
GAAGCTTACCTCAGCGGTGGAACAACAAAACGGAGCTGCAGGTCCTCAATGCCGACAACGAACCACCCAATGCGT[T/C]GCAAATGAACCTCCAAGGAAAGGAAATAACGCCAAGAGGAGAAGAAGCTCGAACGTCGTCGCGAAAACAACCAAA
SEQ ID NO:72
GACAACATCATAGTCGGGGGACTCAAGATGTATGTCAATGTCCCAAAGTATGGGAGGGGAAAGAAGAAACTACAA[A/G]AGCACACTAACAGACACGGCACCAAGCCTGAAGGGCATCACACCAAAATGTGTCATCGGAATCCTTCCATGACTT
SEQ ID NO:73
GAGACCGACAATTGTTAGCGATAAGTCACCTGGCCAATATGTTCTTCAACCTGAAAGAGCCCATAGTAACGTTTT[T/G]AAAGCTTGGTGTAGGCCGGGGCCAGGGAAGTTTGGCGATAAGGTCTGAGCTTTACATACACCTATTCTCTTGCAG
SEQ ID NO:74
GAGCAACCACTTCTCCTGATAAGGAATCAGAAAGATCGGCAGCTTCTCCAACTGGGGAATCAGGGGGAGCAGCGG[G/A]TTCTCTAGATGGGGAATCGAATATGATAATAAAAGGTGGAGATGGGGGAACTACTTCAAGCTCAGGAGAAGGTGG
SEQ ID NO:75
GAGTTTCTCCTAAAAATGAAGATGAAGTTCGTCCATCTAACCTGCAACGGTAGTGCCTTCCTGCACCAATGTCGT[T/C]GCCTACCACTATCAACTGGACACCTAACACGCGTCGAAGCACCGCGCCTGGTAAGATATTTCGAATACACTCTGT
SEQ ID NO:76
GATGTAATATTCTCACTATCTATTTAATGTTATTTCTTTGTGTTCATTGCTTCTATCTATGCTTATTTTATATGT[T/C]TGTGGCTTGCTCACCCATTTGTATATGTAGTTAGGCTTTTTAGTATTGGAAAATGCTTTAAAACCTTAAAACTGG
SEQ ID NO:77
GCAAGCTTGTCTCACTAAGCGGGAGTCCACTTTTCACACTCAACACGCAAATTCTCACTCAGCGCAACTTCCTCG[G/T]CACTATAGCTAGCTCAATGGGCAATTTCTCGCTCATTGCAATTCTCTCTCGGGTTGGAATTGTGCTTAGCGCGCC
SEQ ID NO:78
GCCTATAACGTGGTTTCCCATATTTTGTATAGTACGTAATATGTGGTCCCCCCCGTATGGTATGTGTGCCTATCG[G/C]GTCCTTTACAAGGACTAGCTTGAAATCACGCGTTCAATGAACAGTATCTTCACTAGATATGGATGGAAGGACTAT
SEQ ID NO:79
GCCTTTTAGGCTGGCCTTGGATTCACTTAGTCCAGGTAGTCCCTTCAACGCTCCACCAAAGGTTGAAGTTTGTGT[T/G]GGAAGGGCAGTTAATCATAGTATTGGGGGAGGAAGACATCCTTGTAAGTTTCCCTTCTTCTACATCATATATGGA
SEQ ID NO:80
GCTTTCTGCAAAGAGATCTAGGAGGGACGCCGAGGCTGAAGGGACCAGTGCCACCCCCGAGTTTGATAGCCACCA[A/G]TTTAGGAGCGCCGAGCACCAGCAGCGCTTCGAGGCCATCAAAGGAGGGTCGTTCCACAGAGAGAGACGCGTCCAG
SEQ ID NO:81
GCTTTGTGCGCGTGACCTCTTCGACTTTGGGACTTGGAGTTGTACGACAATGCACTCCTTCCCATGGGAACGACA[A/G]CGACGAGGAAGTGACAAACACGAAGACTGTGTTAGGCAATAGGAGATGCGACAAGGTTTTGGGAAAAGAAAACAC
SEQ ID NO:82
GCTTTTTTCAATCGCCGATACGAGTGAAAATGTTTCAAAAATCTTCTATGTCTGGTATATACTTGTCACAACCTA[A/G]CCTTCGACGGGTGGCACGAAAAGGCCTCTCGTATGGGCCAAGGGTGCGTCTTCCATCGAATGAAAACGCGTGGAG
SEQ ID NO:83
GGACCTTGAGAAGTCCAATAGTGCACTGGGTTTTCCGGCATTGATTACAAGTCTCTGCCAATTCTACGGAGTACT[T/C]ATCGCACCGACAAAGCTTATCCAACCTCCCATTAATAGGACATTCACTGAGAAATATTGCATGCCAAGGTAGGCA
SEQ ID NO:84
GGATTTGTGGCGATGATTGTAAGGTCTGACACAAACACAGGTAGTAGAGGAAGGACTTCGTTTATGTTAATTGGA[A/C]GTGAAAGGAGTGACGAGTATAGGTGTAGGAAGGAAGAATTTGTTAGAAGAGACATTGGGACTAGGAAATATGGGT
SEQ ID NO:85
GGGATGCAGATGTTTGGTAAAAATGTTGTCTAAAAGCTTGAGGAGTAGCTGGGTTTATTTATTCAGGGCGTTTGT[T/C]TGTTATTCGTTAAGTAATGCTTGATTTCTCTCTCTTTTGGTTTGGGCCTGCAGGATGAGAATTTGGAACACACTG
SEQ ID NO:86
GGGGTGGCAGTTTTCAGCACCTATGACAACTACAATTGGAGGATTAAAGCTTCTGCTCTTCTCAGACCAGATTTC[C/T]AAGGACTCCTCCGCCACGGGAAAGAACTCTTGATCCACGATCACTGCCACGGCGGATGAATGAACAAGAAGGAAA
SEQ ID NO:87
GTACGATCCCAAACAACAACAGTGCTAAAACATCAATGAATGAAGTCCACTCTCCTTGATCTGCCAAAGCTTTTT[T/G]CTTCTCCTCCAAGTGTTTCCTTGGCATCCCAACCACCCTATTTCTATTTTGTTTCACTTGGTCTAGCTCTTGTGC
SEQ ID NO:88
GTATTGCACCACCGAGACACCCGGTGGACCTTGAGAAGTCCAATAGTGCACTGGGTTTTCCGGCATTGATTACAA[A/G]TCTCTGCCAATTCTACGGAGTACTGATCGCACCGACAAAGCTTATCCAACCTCCCATTAATAGGACATTCACTGA
SEQ ID NO:89
GTCCTCGAAGCGCTGCTGGTGCTCGGCGCTCCTGAAACGGTGGCTATCAAACTCGGGGGCGGCACTGGTCCCTTT[T/C]GTCGCGGCGTCCCTCCTAGATCTCTTTGCGGAAAGCTTTTTCAGAGCCAATTCCTGCGAGGACAAACATTTGGAA
SEQ ID NO:90
GTGTTCTGGGGCACTCTATCGAGTAGCGTGTAGCCCTCAAGCACAAGGTACAAAGCTTGATTAACGCTAGATGGT[T/C]GACATTCCAAGAAGATAGTCCGAATGTGAGGACTAATCCACTCGTCAATCATAAAGGCTTGGCGGTGAATGCAGT
SEQ ID NO:91
GTTGTGGAAGCTTGCTGGACTTCATAACTTTGCCCCACACCCTAATTCGACTGTACATTCCATCCCTAGGTAAAT[T/G]ATGTTGTTGAACTCCCATACGATCTTGATAATCCATTGTCAAACTCCTTCACATTATGATACTCATTATGGACCA
SEQ ID NO:92
GTTGTTTTCATTTGTCATTTATCATTTTCGCGAATATTTTATTGTGTAAGTATCATTGTTCTATTTATTGTATGG[G/A]TAACTTTTTTTTTTGTGTGTGTGTTATATGATCTAGTAGCGTATGGAAAAGA ACCACGTCTGTTGGGTTTGGAAA
SEQ ID NO:93
TAAGTGTCTTTGAGCTTGACGGTCACGATCAACAAAGTACTTTCGACACCTACTATATGTTGATTTGACCAACGG[G/C]ATTATCGGTATGTTGTGATAATCCTTTAATACCTTATTTATACATTCTGAGAGGTTTGTTGTCATGTGGTCATAT
SEQ ID NO:94
TACTCGTCTACAGTGCCTGATATTTTCTTAAGCATTCTAAGTGCTTGTTTGGGCAGCATAAGCTTGATTATCTGA[A/G]CCACGTGATCTCCGGCGAGGGTGTGCAACCAGACCCTTCGAAAATTCAAGCCATACTTGAGTGGCCCCAACCCAG
SEQ ID NO:95
TACTGTGAGTTCCTCGGCGGTGAGCCTCTTAATCAGAGGTCGAGCTGGGGAGGGAACTAGAGAAGAGCTAGTGAC[C/G]GGAGGTGACACCTGTGACTGATGCACAGGTGGTGTTCGAGGTGGTCGACGACGATCTTGGAGTTTTTCCTCTTGA
SEQ ID NO:96
TACTGTGAGTTCCTCGGCGGTGAGCCTCTTAATCAGAGGTCGAGCTGGGGAGGGAACTAGAGAAGAGCTAGTGAC[C/T]GGAGGTGACACCTGTGACTGATGCACAGGTGGTGTTCGAGGTGGTCGACGACGATCTTGGAGTTTTTCCTCTTGA
SEQ ID NO:97
TAGCTAGGCATCTTTGTCCGGAGCTCTACGGAATGGTTTCAGCAAAGAACAGTGGAAGACAAGATGAATACGGGT[T/C]CCCTCTGGTAAACGAAGCTTATATGCTACAGGCCCAATTCTCTCCATGATTTCAAATGGGCCATAAAATTTCTTA
SEQ ID NO:98
TAGTTGTGACCCTGAAGGGGCCTTCCCCATTGGGGTCAAGCTTTTTCACTCGGGGATCTTTTCTGGCTTCACCTC[C/T]GACTCGCCATACAAGGTTACCAAGCTGAAAGGCTTATGGTTGAACCTTTGTATTGTATCTCCTTGCTGCTCGTAG
SEQ ID NO:99
TATATTAACATCGGTTTTTGAAAAAACCAATGTTAAGCTTCATCAACAACATTGGTTTTTATAAAACCGATATTG[G/A]TGTTGAACATATAACATTGATTTTTTTTTTAAATCGATGTTTGTTATCAGAAAACAACATCGGTTTTTGGTAAAA
SEQ ID NO:100
TATCAAGCTTAGTCAAGGAGGTAGGTTGGTGTCGTATTGCTATCATTATCCACCTCTGAAAAACCAACCTTGTCT[T/C]TAATGTTGGGCAATAAAAACAAGCAAATACAGGGGGTAGGTGTAGAAAAAATAGGTATGTGAGTGAAATTGCTGG
SEQ ID NO:101
TATCTTGACAACATTGTGCGGTGTAATATTCAAATAACATTTAGAGTGAAGAGGAAATGGACAAAACACAACTGC[C/T]ACAAGGTCATCGAAGACGCACAAAGAAATGCAAACGCTCACACTATTTTTGAGTTCGATTGAAAGGACAAAAGCT
SEQ ID NO:102
TCAAAAAAGCTTCCACTGTTTTTTTAAGTAAGGAATACGAAAAGAACGGATTGACATAAGCTGAAACTTGTGTAG[G/C]TATAAGAACGGATTTGTGATGCCACTGCAATCTAGTTGATACCTTATAAATTGCTAACATACTCTAAAGAAAAGC
SEQ ID NO:103
TCACAGACCTCATCTTCACGCCAGCACCAACAATCAGAGGGTGGGAACAACTGAGGCAAGGAAGAAGGAAGGCGG[G/A]ACCCACACTGTCACCTCCACACCTACTTAGCCTAAGTCTCAACCGGCCCCACACGGCACCCACCAGTATGCCCAG
SEQ ID NO:104
TCCAGATGTTTGTCTTCGCAGGAAATGGCTCCGAAAAAAGCTTTCCACAAAGCGATCTAGGAAGGACGCCGCGGT[T/C]GAAGGAACTAGCGCTGCTCCCGAGTTTGACAACCACCGTTTCAGGAGCGCTGAGCACCAGCAGCGTTTCGAGGCC
SEQ ID NO:105
TCCCTGAGCTGAACGCGCCTCTCTTTGTGGAACGACCATCCTTTGATGGCCTCAAAGCGCTGTTGGTGCTCGGTG[G/T]TCCAGAAACGGTGACTATCAAACTCGGGGGTGGCACTGGACCCTTCCGTTGCGGCGTCTCTTCTAGACCTCTTCG
SEQ ID NO:106
TCCTCAATGCCGACAACGAACCACCCAATGCGTGGCAAATGAACCTCCAAGGAAAGGAAATAACGCCAAGAGGAG[G/A]AGAAGCTCGAACGTCGTCGCGAAAACAACCAAACAACGCAGGAAAGAGAAATGTACGGAACGCGTGAATAGTGTT
SEQ ID NO:107
TCCTCTTCGGAGTCTATTTCTGTATCAAATGTTGTACCAGTCAATACTCACCCTATGCAAACAAGGTCTAAATCT[T/C]GAATTCACAATCCTTGGTTACATCCTTCTCTGTTTCTTACCCATTCTGAGCCTAAAACTGTAAAACAAGCTTTGG
SEQ ID NO:108
TCGGCTCGACTAGGTTGCTCTCGACCGGGAATTGGCATTTATGTGAAGCCGCGGTCAAAGCCGTTCTAAATGGCA[A/G]GCCGTTTCAGCAGGCTCCTTTGGGCGGCGCCGCAAGCCTGCAGCTCGAGGCTTGTGATATTCGCCACGTGGCAAA
SEQ ID NO:109
TCTACTATGAAAAGTTGGTAGGCTATATGCCCTCTAGCTTCACAGACCTCATCTTCACGCCAGCACCAACAATCA[A/G]AGGGTGGGAACAACTGAGGCAAGGAAGAAGGAAGGCGGCACCCACACTGTCACCTCCACACCTACTTAGCCTAAG
SEQ ID NO:110
TCTCCCCCTATTTATAGCAAAATGGGGGAGAAGCTTGCCACCCAGCTCGCCCAGGCGAGCAAGGTTGCTTCCTCT[T/C]GATTGCTATTTGCACCTCCCTTTTTACTAAGTGCACCCCCTTCTATTTTTTTGGTAATTCTTTTTCCGTAATATT
SEQ ID NO:111
TGAAACGGTGGCTATCAAACTCGGGGGCGGCACTAGTCCCTTCAGCCGCGGCGTCCCTCCTAGATCTCTTTGCGA[A/G]AAGCTTTTTCGTAGCCATTTCCTACGAGGACAAACATTTGGAAAGTTAGTTTACAAGGAAATGCTATTTTAAAGC
SEQ ID NO:112
TGAAGCTTTCTGATGGTTTCTCTATTTTCATCATTTCTTTGGACCAACTCTAACAATTGATGCAGATTGTCCTTT[T/C]TAAGTTTGGTAATTTGATACTCCAGCGTTGACCACATATTGTCAAATTTGAAATTGACCATTGATGGATCTTCAT
SEQ ID NO:113
TGGCCCACTATGGGCACTGGTGGTCACTTCAGTGGCGGTGGCTATGGATTTTTGATGCATAAGTATGGTCTTGCT[T/G]CTGATAATGTAATTGATGCTCACATAGTTGACGTGAAAGGTAACCTTCTTGACAGAAAATCAATGGGTGAGGATC
SEQ ID NO:114
TGGTGGATAATTTGCTGTAGCGAGTAAACACATTGGTTACACGTTGTGGTTTAATGGCTGGCCGTTTTGGTGGTA[A/T]GTCTTCCGTTGTCCACGGAAGAAAGCAAGTCAAACCCATTTTGTCCAAATCCAAAACCGTACTTTAAGCGTTGAA
SEQ ID NO:115
TGTACGCGTCCAGTCTAGTCTCTCTAAGCCCTGAAACCGTAACTATGGCTATGTTCTGAGATTCGTCTTTTCTGT[T/C]GTTCAAAGATCTCTCCGATTCCTTTTCTTTCGAAGTGGCATTGCCCAAAGTCGAATCAAAGGCTTGACAGGGTCT
SEQ ID NO:116
TGTATGTATGTTGCCTTTGTCTTTGCAACGCTCCATACTTGTGGTAATATGAATAGGCCACCGTGATGTAGATCG[G/A]GTAGGAAGCAAGACCTTGATGTAGCAGTAGGATCGTGGTGGTCTTGTAAAAAATGGAATGATGCGGAGTCATCTC
SEQ ID NO:117
TGTCGCAACCTACCCTTCGGCGGGAGGGCGACGCGTGACTCGCGGGTGCGTCTTCCAATAAAGGAATACACACGC[C/G]GTCGCCACCAACGTTTATTTGAGGAAAACGTCAGAAAAACCGGAAAAAACGTGGTCTACAAACATTAAGTGAAAA
SEQ ID NO:118
TTCAAGATGATGCTATTCCTGCTCTTGAAAAGCTTCGGAAGGTATGGACATCCTTTTGTTTAGTGTTTGATTGGT[T/C]TACATTCTAGGGAACACCCTCATCTTCGGTTGTGATATTTTGATGTAAAGGGAATATCACCTTTGATTTATGCAG
SEQ ID NO:119
TTGCATAACTATCATTCATGGAGTGGCACGTACTAGAGACATAGTGCGTTTGGAGAATTGACTAGGGAATTGTAT[T/C]TCTTGAGTTTTGGGGATGACTTTATTGCCTGCCTCTGCTTTGTTGTTGGTCTAAGAATGTTCGACAGAGGTGCTT
SEQ ID NO:120
TTGGGTTGATAGGCCACATCCCTACAATTGTTGTCAACATACTTTTTCATTGTATCTTGAGCCTTCAGAAGCTTC[C/T]GACGTATGGTTTGGAACGTCGCGTCTCTGTCCACCAACAATTCTTGCACTACATCAATCTTAGAAGGCCCCACAA
SEQ ID NO:121
TTGTTATGATCAAAACACCTTGAATCAATCTTGATTCACCATCATGAAGCAATGAAGCTTGCTTCTACAGCAGGT[T/C]GGGATACCTTATGGTTATTCGCACCTTTTGTCAACCAGAGGCAAACGAGCCCGTTGACACGCAAAGAATAATGTC
SEQ ID NO:122
TTTAATGCCAGAGGTTCCACACCTTTGCGCAATACCAGGTGCCCCAGATACTCAACCTGTGGTTGCGCAAAACAA[A/G]ATTTTGTCAGCTTTAAAACAAATTGATTGTCAGACAATACCTGGAAGGCCTTCTCCAGGTGTAGCAAGTGTTCAT
SEQ ID NO:123
TTTCTGGAACTCTTGCAATGTGGTTTGGAAATCTCTTGCAAGCTTAGCATCCTCAATTTTCTTACTAGCCTGGGT[T/G]TAGATGAAGAAAAACTGGGAGGGTTAACTAACATCCAATTCATATTTAGCATTCACAGTTTTCAAGCATAGAAAA
SEQ ID NO:124
TTTGCAAATGCCCAAATCTGCCGACACACAGACAGTTCAGTGTGATTTCTCAAGTATTTAGCATTTAGACAAACG[G/A]ATTTCTAATGCAGTGACTAACTAATTCAAAAAAAACAATGCTCTGAGATGCTTGGTGGCACTTGATGTGAAGTAA
SEQ ID NO:125
TTTGCTGTAGCGAGTAAACACATTGGTTACACGTTGTGGTTTAATGGCTGGCCGTTTTGGTGGTAAGTCTTCCGT[T/A]GTCCACGGAAGAAAGCAAGTCAAACCCATTTTGTCCAAATCCAAAACCGTACTTTAAGCGTTGAATTGAATCAAA
SEQ ID NO:126
TTTGTTGGGGAGTTAATACTGAACATCTTGATGTAATATTCTCACTATCTATTTAATGTTATTTCTTTGTGTTCA[A/G]TGCTTCTATCTATGCTTATTTTATATGTTTGTGGCTTGCTCACCCATTTGTATATGTAGTTAGGCTTTTTAGTAT
SEQ ID NO:127
TTTTGAGCCTCTACAAAAAGATCTCAATATGGAGATCATTGTTGATGTGGTAGCCAAGCTAAGGACTATTGATTC[C/A]AGCTTGAAGAAGGAAAACACTAAGAAGGAATAATACAAGCAAGAAGAGCCTTTTGAGAAAGTAGAAAAGGTTGAA
SEQ ID NO:128
ACGTCATTGCTGACGTCGACAACGTCTTCGAAGGCGAAGCTGCAACTTCG[G/A]CCGTGAAGTCA[T/A]CTTCGTCGCAGGAATTGCGTCCTTCTAAAA[A/C]G[G/A]TGTTTAGAGACCGTCGTAGAT[T/G]GGCCGGGGTCTACGACGACGTCGCCTTTAAAGACGGTTGTGCACCATCGT
Нижеследующие примеры осуществления способа приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Пример 1. Применение SNP-панели для генотипирования образцов сои
Для демонстрации возможности использования SNP-панели для генотипирования растительного материала, были взяты 10 образцов сои с неизвестным генотипом, отличающихся по процентному содержанию масла в семенах от 19,7 до 24,8%. С использованием SNP-панели для генотипирования и геномной селекции сои были определены генотипы 10 образцов сои на платформе Miniseq Illumina. Этапы определения генотипов описаны ниже.
1. На первом этапе была проведена подготовка образцов ДНК сои к дальнейшему анализу, которая включала несколько последовательных этапов:
a. Мультиплексная ПЦР по 10 пар (в одной группе 8 пар) праймеров в 13 группах (всего 128 пар праймеров) заключается в многократном увеличении количества фрагментов, содержащих однонуклеотидные полиморфизмы, одновременно для 10 локусов в одной реакции ПЦР. Для этого была проведена классическая ПЦР-реакция, включающая денатурацию, отжиг (60°С) и элонгацию;
b. Равномерное смешивание продуктов 16-ти мультиплексных ПЦР-реакций для каждого образца ДНК - получение 10-ти пулов;
c. Проведение второй реакции ПЦР для добавления индексных последовательностей к каждой из 10 групп ампликонов. В качестве праймеров используются известные, специально подобранные, последовательности, которые совпадают с последовательностями на 5'-концах ампликонов, получившихся в результате первой ПЦР-реакции. Целью данного этапа является добавление индивидуальных последовательностей (индексов) к каждому ампликону, так как в последующем, при одновременном секвенирование всех образцов, остается возможность отнесения полученных последовательностей ДНК к определенному образцу сои.
d. Очистка каждого из десяти пулов ампликонов от ненужных на последующих этапах солей, неиспользованных нуклеотидов и т.д., с применением силико-колонок (Qiagen) и их перерастворение в 0,1× ТЕ буфере.
е. Для получения библиотеки производится равномерное (по концетрации) смешивание десяти пулов ампликонов. В последующем, библиотека подготавливается и секвенируется согласно протоколам производителя секвенатора, в данном случае - Illumina.
Результаты определения генотипа 10 образцов сои с помощью SNP-панели для генотипирования и геномной селекции сои приведены в Таблице 2.
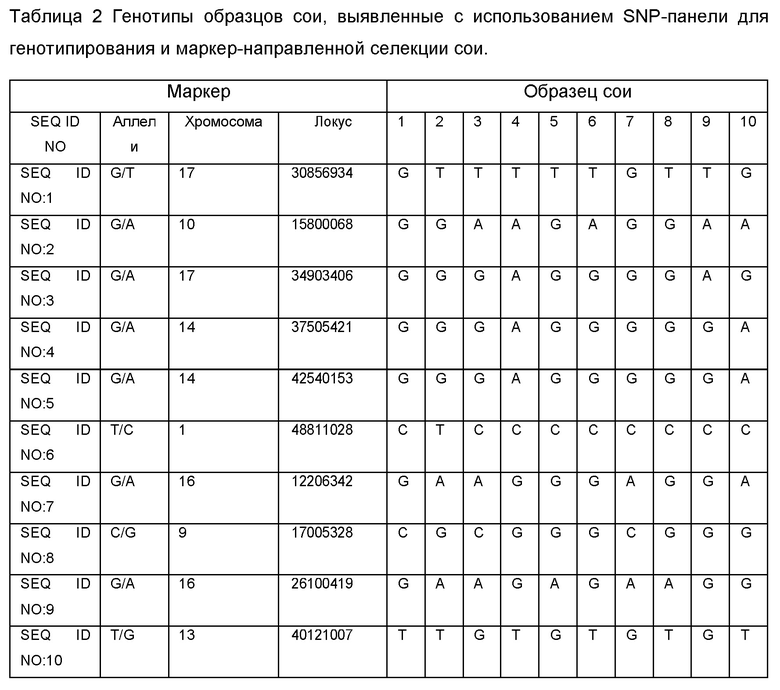
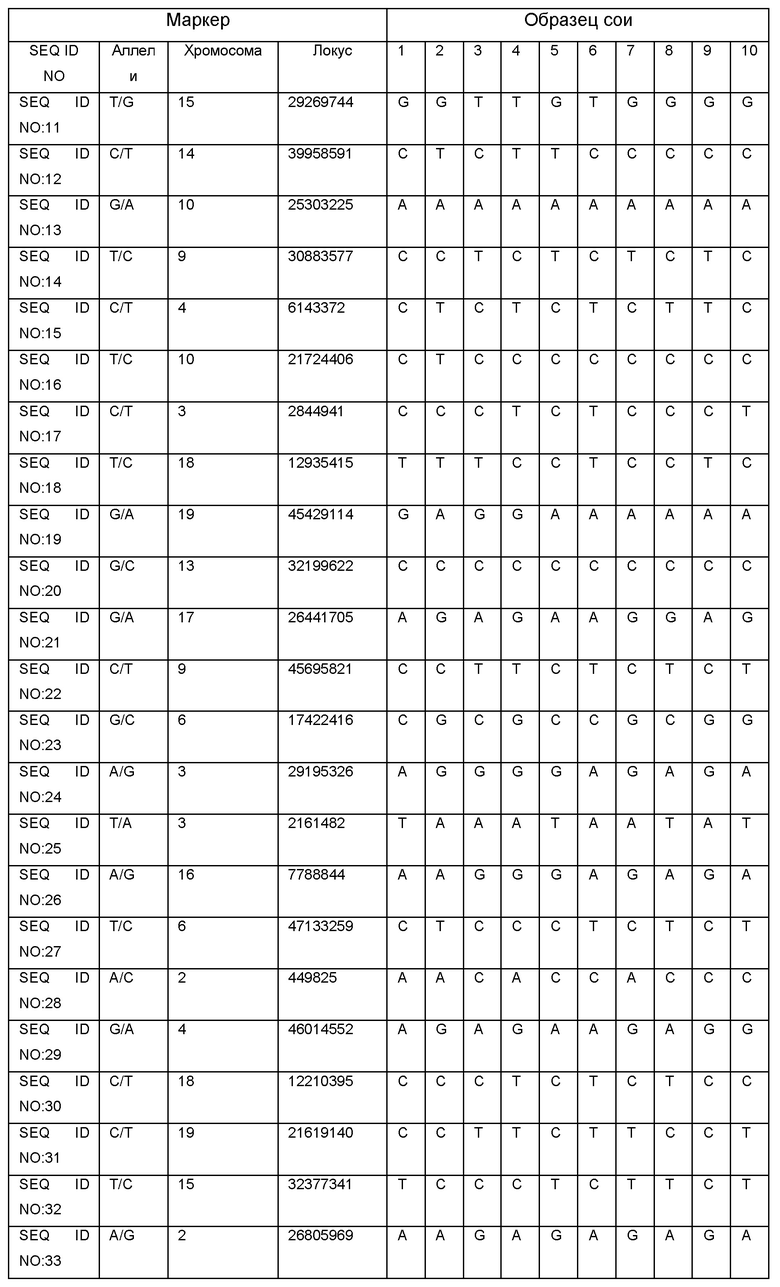
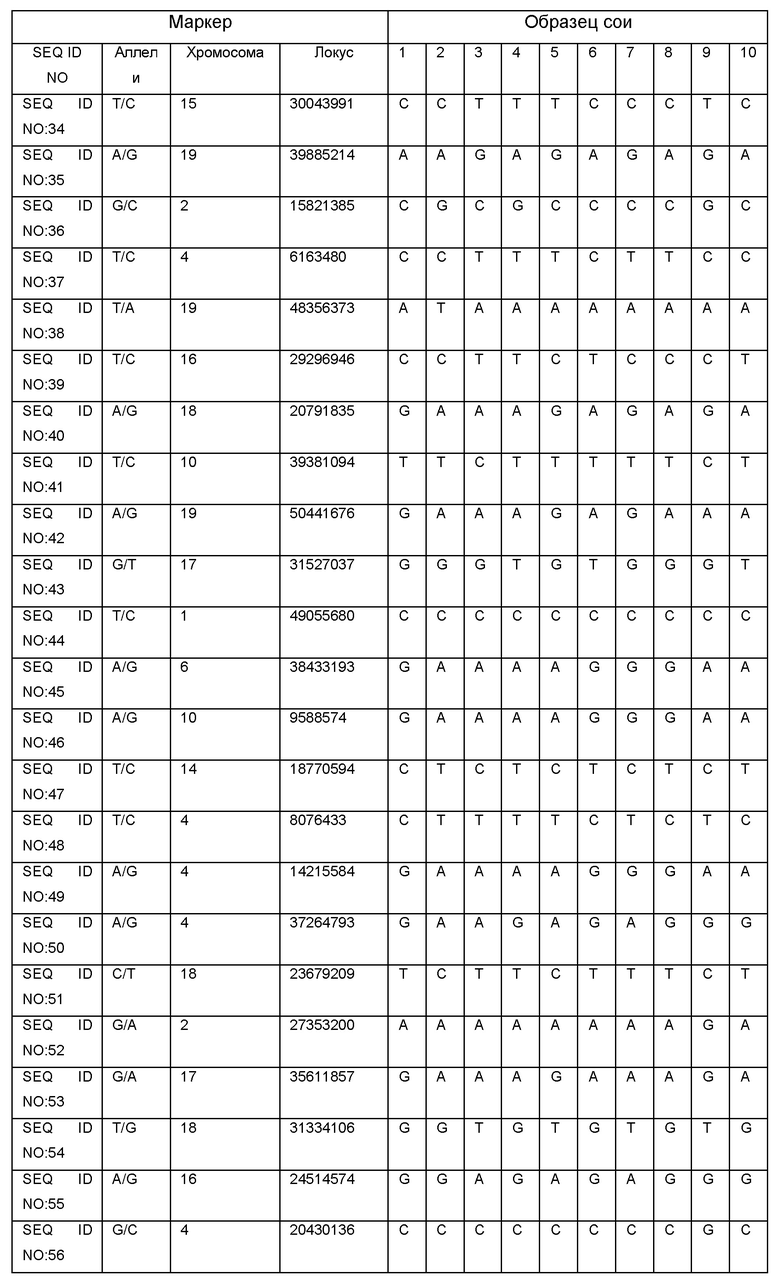
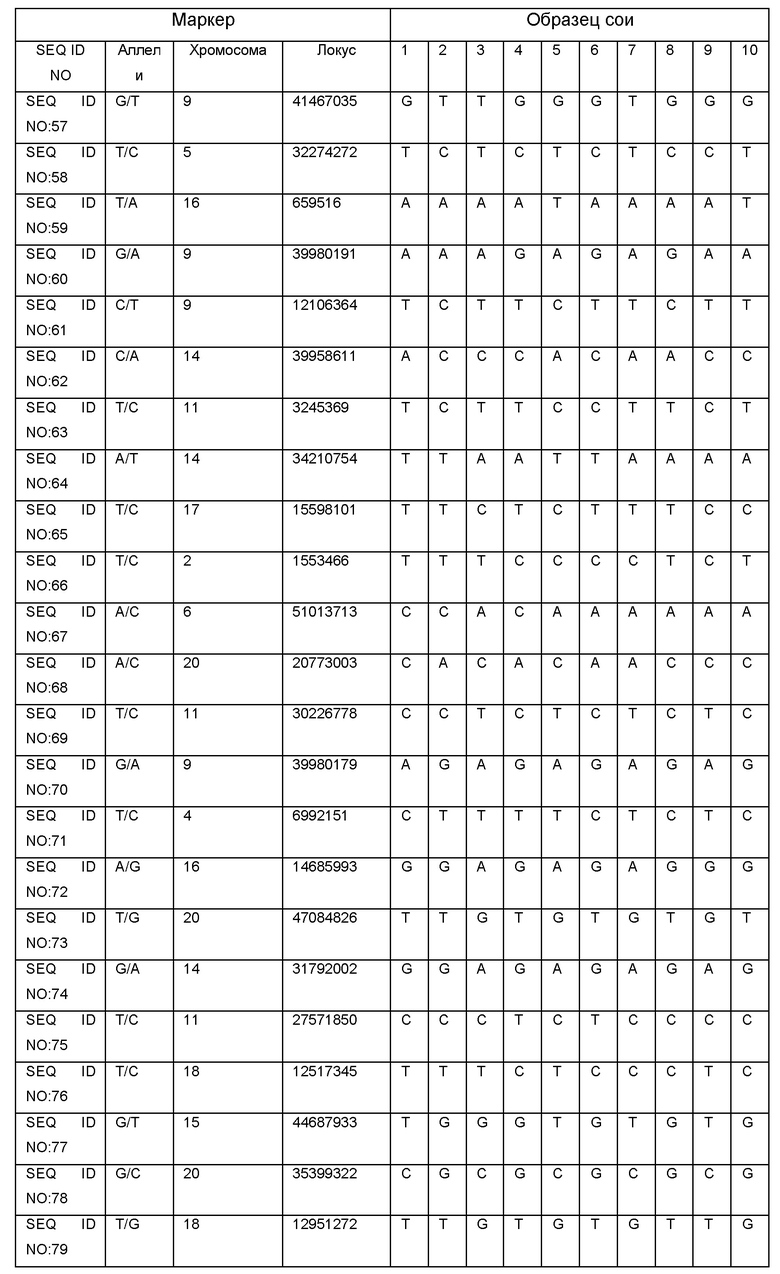
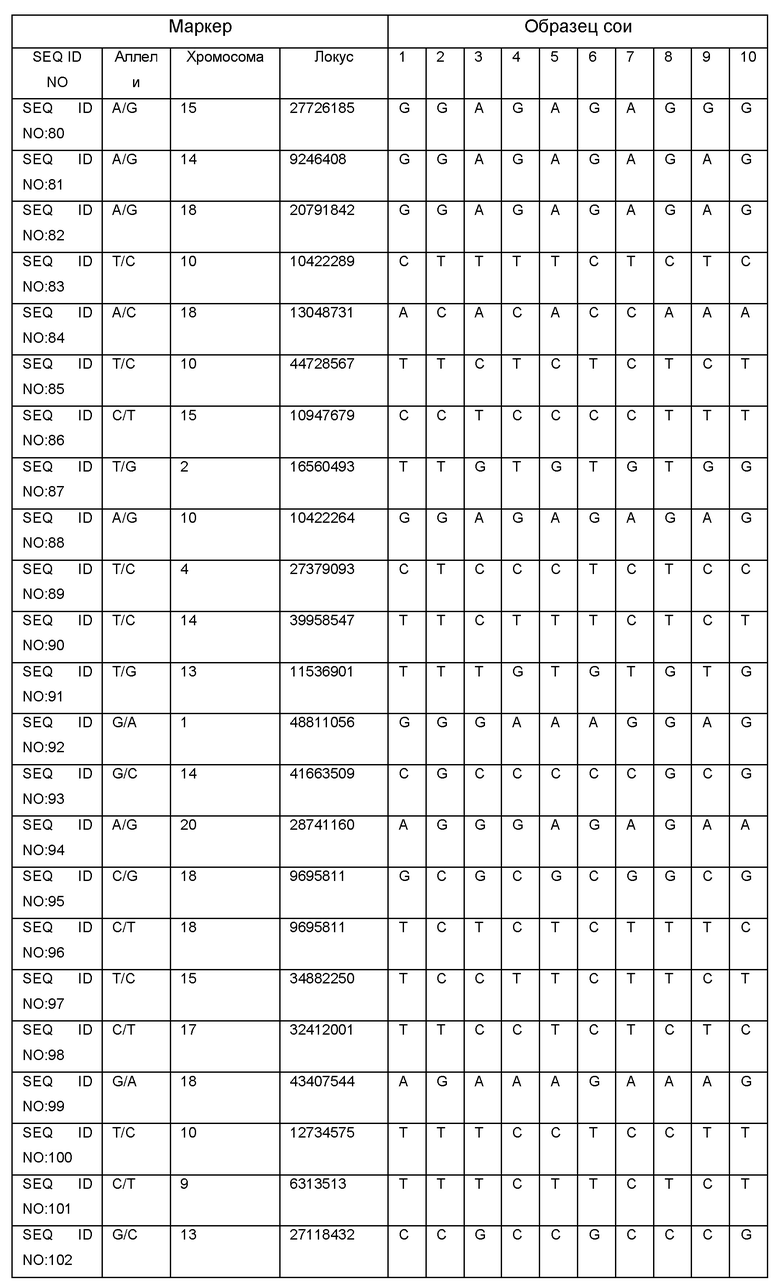
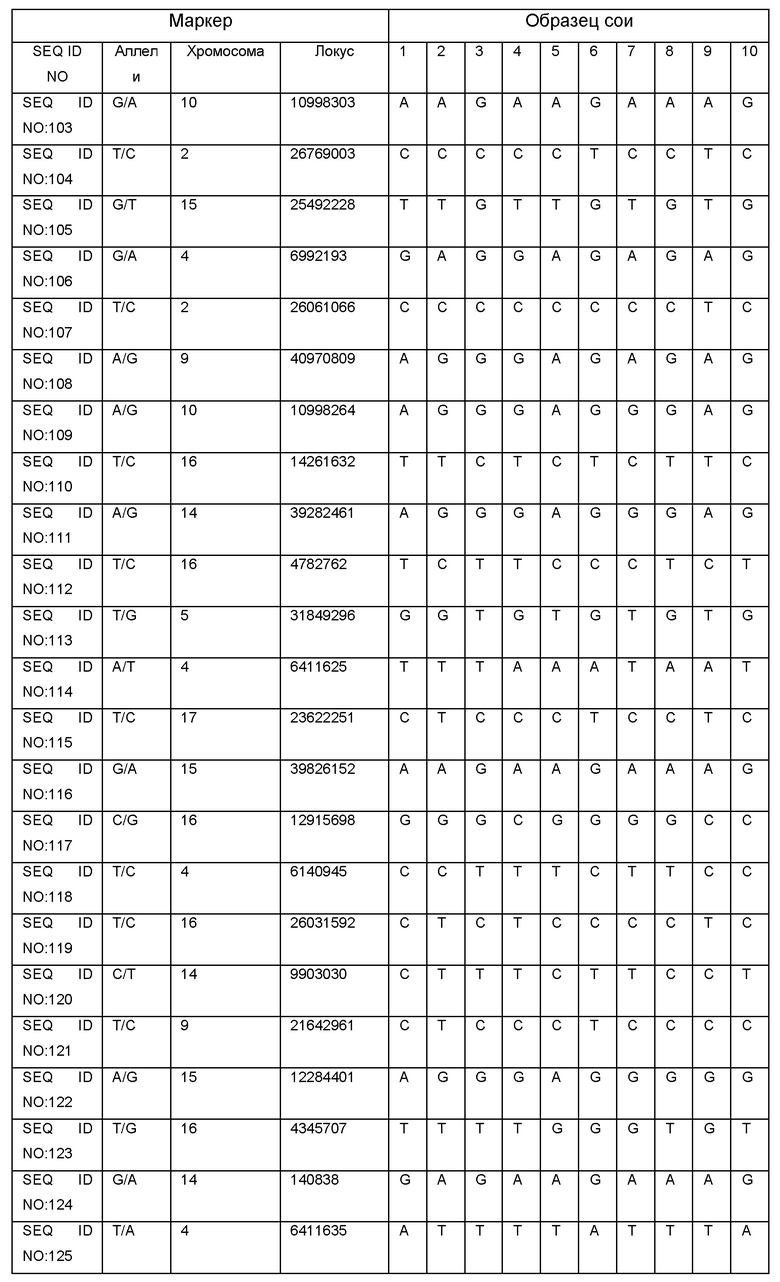
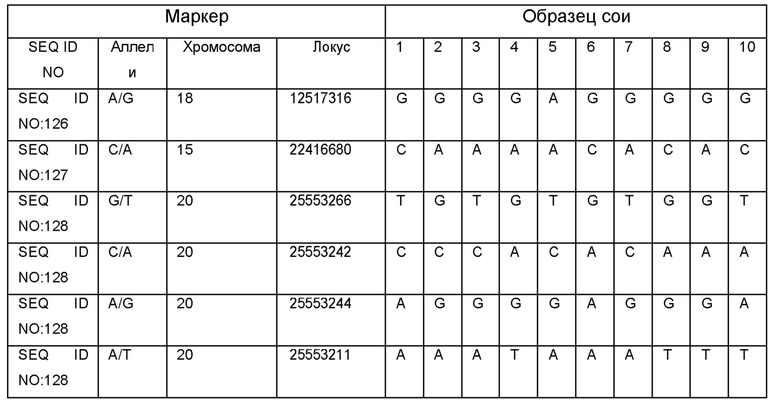
С - обозначают гомозиготу СС,
А - обозначают гомозиготу АА,
Т - обозначают гомозиготу ТТ,
G - обозначают гомозиготу GG,
N - обозначают гетерозиготные состояния маркера.
Полученные результаты показывают возможность успешного применения SNP-панели для определения генотипа селекционного материала сои.
Пример 2. Схема отбора растений сои для селекции по признаку повышенного содержания масла в семенах сои на основе данных генотипирования с использованием SNP-панели.
Определение селекционной ценности сортов включает в себя несколько этапов.
(1) Определение генотипа сорта/линии сои (интересующих релевантных аллелей) и сравнение их с SNP-панелью, раскрытой в настоящем изобретении в Таблице 1, по локусам, ассоциированным с увеличенным процентным содержанием масла в семенах (как пример, генотип образцов 2 и 6, определенный с использованием SNP-панели, приведен в Таблице 3 в столбцах «Генотип образца 2» и «Генотип образца 6»). Суммарную ДНК выделяли с использованием набора Nucleospin Plant Extraction Kit («MacheryNagel», Германия), следуя стандартному протоколу производителя.
(2) Используя значения эффекта аллелей для каждого из маркеров из соответствующего столбца таблицы 3 вычисляли эффект маркера по формуле ЭМ=ЭА1*2 (формула 1) или ЭМ=ЭА2*2 (формула 2) для гомозиготного состояния маркера, и ЭМ=ЭА1+ЭА2 (формула 3), для гетерозиготного состояния маркера, где ЭА1 - эффект аллеля 1, ЭА2 - эффект аллеля 2. Поскольку образцы 2 и 6 являются гомозиготными по всем маркерам, использовали только формулы 1 и 2.
(3) Вычисляли сумму эффектов по всем маркерам, ассоциированным с содержанием масла для образцов 2 и 6. Сумма эффектов маркеров для образца 2 равняется 3,842541792, сумма эффектов для образца 6 равняется 0,743185068.
Таким образом, для селекции по признаку повышенного процентного содержания масла в семенах сои следует отобрать образец 2, как имеющий большее значение суммы эффектов маркеров.
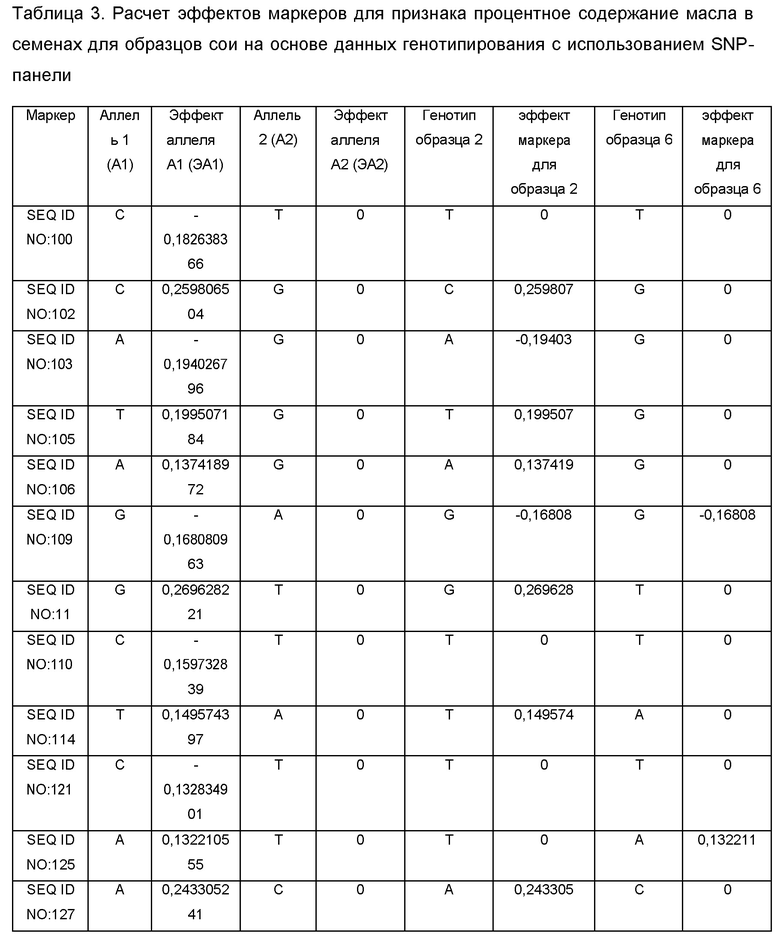
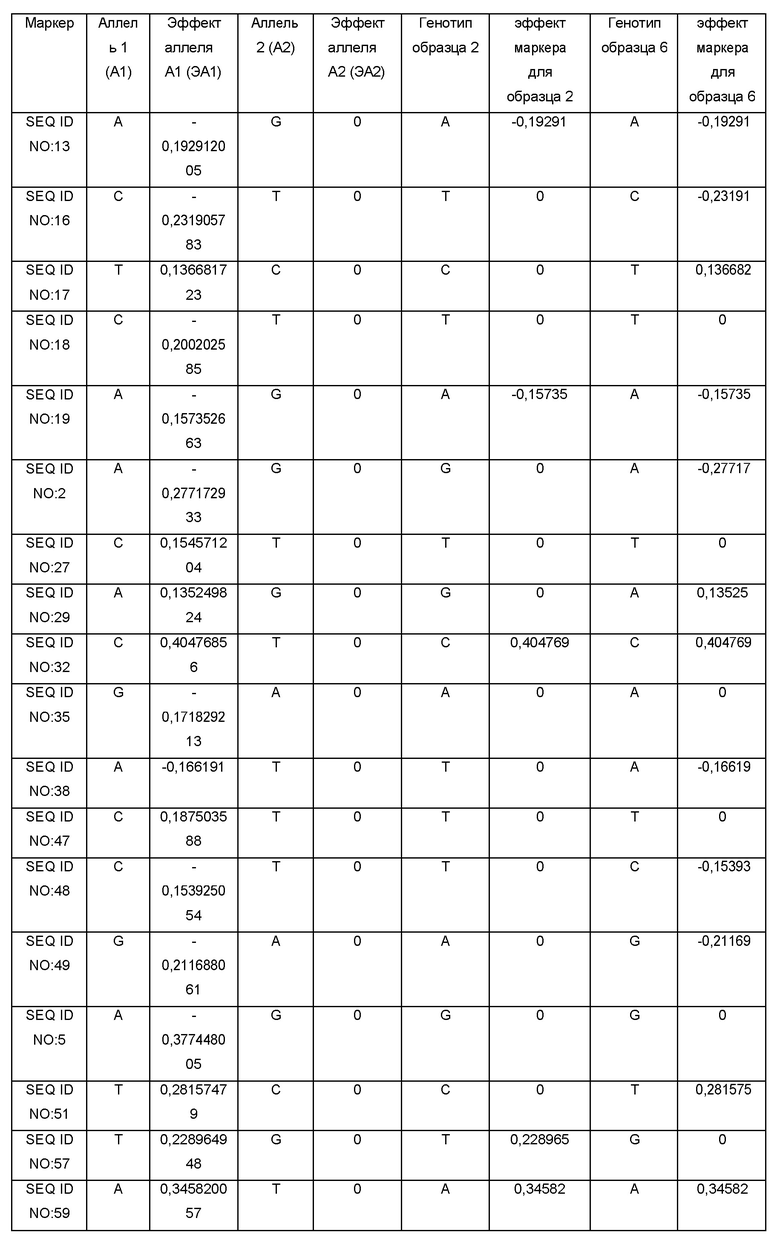
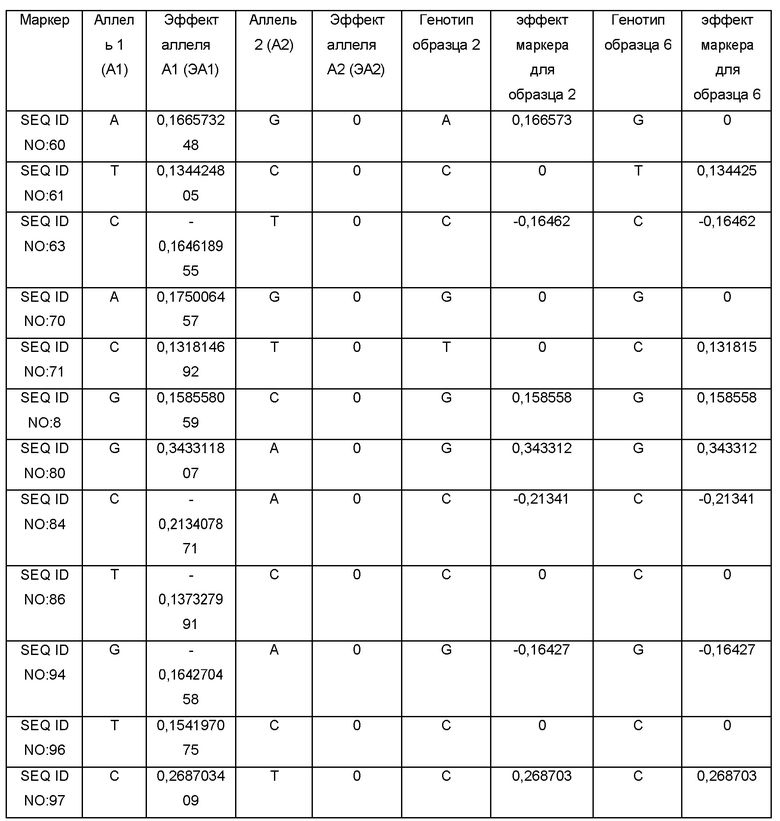
Для проверки правильности предсказания было проведено прямое измерение содержания процентного содержания масла в семенах образцов 2 и 6 с использованием ИК-анализатора Bruker. Полученные значения содержания масла для образца 2 равнялась 23,9, для образца 6 - 21,8.
Таким образом, разработанная SNP-панель может быть использована для селекции сортов/линий сои по процентному содержанию масла, процентному содержанию белка в семенах и по продуктивности.
Несмотря на то, что изобретение описано со ссылкой на раскрываемые варианты воплощения, для специалистов в данной области должно быть очевидно, что конкретные подробно описанные случаи приведены лишь в целях иллюстрирования настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения. Должно быть понятно, что возможно осуществление различных модификаций без отступления от сути настоящего изобретения.
| название | год | авторы | номер документа |
|---|---|---|---|
| SNP-панель для генотипирования и геномной селекции подсолнечника по содержанию жирных кислот в масле семян | 2018 |
|
RU2717642C1 |
| SNP-МАРКЕРЫ И ОТБОР В ОТНОШЕНИИ НИЗКОГО СОДЕРЖАНИЯ КЛЕТЧАТКИ В ПРЕДСТАВИТЕЛЯХ РОДА BRASSICA | 2019 |
|
RU2834673C2 |
| МАРКЕРЫ, СЦЕПЛЕННЫЕ С УСТОЙЧИВОСТЬЮ РАСТЕНИЙ СОИ К SCN | 2011 |
|
RU2620973C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM4 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2718584C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ПОЛУЧЕНИЯ УКОРОЧЕННЫХ РАСТЕНИЙ КУКУРУЗЫ | 2016 |
|
RU2745987C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| ГЕНЕТИЧЕСКИЕ ЛОКУСЫ, СВЯЗАННЫЕ С УСТОЙЧИВОСТЬЮ К ПЫЛЬНОЙ ГОЛОВНЕ У МАИСА | 2009 |
|
RU2562864C2 |
| ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ (CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2011 |
|
RU2603005C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ НИЗКОГО СОДЕРЖАНИЯ ПАЛЬМИТИНОВОЙ КИСЛОТЫ В ПОДСОЛНЕЧНИКЕ (HELIANTHUS ANNUS) И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2013 |
|
RU2670517C2 |
Изобретение относится к области биотехнологии. Изобретение описывает разработанную SNP-панель, представляющую собой набор молекулярных маркеров на основе однонуклеотидных полиморфизмов генома сои, которые позволяют идентифицировать генотип, осуществлять оценку геномной ценности селекционного материала сои и проводить селекцию сои по комплексу хозяйственно-ценных признаков, включающих продуктивность сорта/линии, процентное содержание масла и процентное содержание белка в семенах. 5 з.п. ф-лы, 3 табл.
1. Способ отбора сорта/линии сои, содержащего необходимую совокупность признаков, выбранных из следующей группы признаков: повышенное процентное содержание масла в семенах, повышенное процентное содержание белка в семенах и повышенная продуктивность сорта/линии, включающий следующие стадии:
а) выделяют геномную ДНК из растений по меньшей мере двух различных сортов/линий;
б) для каждого из этих сортов/линий определяют последовательность ДНК в нижеследующих релевантных для определяемой совокупности признаков маркерах, выбранных из следующих групп:
для повышенного процентного содержания масла в семенах:
SEQ ID NO: 2, SEQ ID NO: 5, SEQ ID NO:8, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 32, SEQ ID NO: 35, SEQ ID NO: 38, SEQ ID NO:47, SEQ ID NO: 48, SEQ ID NO:49, SEQ ID NO: 51, SEQ ID NO: 57, SEQ ID NO: 59, SEQ ID NO: 60, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 70, SEQ ID NO:71, SEQ ID NO: 80, SEQ ID NO: 84, SEQ ID NO: 86, SEQ ID NO: 94, SEQ ID NO: 96, SEQ ID NO: 97, SEQ ID NO: 100, SEQ ID NO: 102, SEQ ID NO: 103, SEQ ID NO: 105, SEQ ID NO: 106, SEQ ID NO: 109, SEQ ID NO: 110, SEQ ID NO: 114, SEQ ID NO: 121, SEQ ID NO: 125, SEQ ID NO: 127;
для повышенного процентного содержания белка в семенах:
SEQ ID NO: 1, SEQ ID NO: 7, SEQ ID NO:15, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO:22, SEQ ID NO: 26, SEQ ID NO: 37, SEQ ID NO: 43, SEQ ID NO: 50, SEQ ID NO: 56, SEQ ID NO: 72, SEQ ID NO: 73, SEQ ID NO: 74, SEQ ID NO:75, SEQ ID NO: 78, SEQ ID NO: 93, SEQ ID NO: 98, SEQ ID NO: 112, SEQ ID NO: 117, SEQ ID NO: 118, SEQ ID NO: 123, SEQ ID NO: 128;
для продуктивности сорта/линии:
SEQ ID NO:3, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO:14, SEQ ID NO:23, SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:28, SEQ ID NO: 30, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 36, SEQ ID NO: 39, SEQ ID NO: 40, SEQ ID NO: 41, SEQ ID NO: 42, SEQ ID NO: 44, SEQ ID NO: 45, SEQ ID NO: 46, SEQ ID NO: 52, SEQ ID NO:53, SEQ ID NO: 54, SEQ ID NO: 55, SEQ ID NO: 58, SEQ ID NO:62, SEQ ID NO: 64, SEQ ID NO: 65, SEQ ID NO: 66, SEQ ID NO:67, SEQ ID NO: 68, SEQ ID NO: 69, SEQ ID NO;76, SEQ ID NO: 77, SEQ ID NO: 79, SEQ ID NO: 81, SEQ ID NO: 82, SEQ ID NO: 83, SEQ ID NO: 85, SEQ ID NO: 87, SEQ ID NO: 88, SEQ ID NO: 89, SEQ ID NO: 90, SEQ ID NO: 91, SEQ ID NO:92, SEQ ID NO: 95, SEQ ID NO: 99, SEQ ID NO: 101, SEQ ID NO: 104, SEQ ID NO: 107, SEQ ID NO: 108, SEQ ID NO: 111, SEQ ID NO: 113, SEQ ID NO: 115, SEQ ID NO: 116, SEQ ID NO:119, SEQ ID NO: 120, SEQ ID NO: 122, SEQ ID NO: 124, SEQ ID NO: 126;
в) для каждого сорта/линии вычисляют процентное содержание масла в семенах, процентное содержание белка в семенах и/или продуктивность, соответствующие определенным в (б) маркерам согласно приведенной в описании SNP-панели Таблицы,
1. при этом вычисление для каждого признака производят независимо, определяя значения эффекта маркера (ЭМ) по соответствующим значениям эффектов аллеля для каждого маркера, чья последовательность ДНК была определена и соответствующего данному признаку, по следующим формулам: ЭМ=ЭА1*2 (формула 1) или ЭМ=ЭА2*2 (формула 2) для гомозиготного состояния маркера, и ЭМ=ЭА1+ЭА2 (формула 3), для гетерозиготного состояния маркера, где ЭА1 - эффект аллеля 1, ЭА2 - эффект аллеля 2, определенные в соответствии с Таблицей 1, и далее суммируя данные значения по всем маркерам, чья последовательность ДНК была определена и соответствующего данному признаку;
д) сравнивают вычисленные параметры каждого из определяемых признаков, соответствующих определенным в (б) релевантным для определяемого признака маркерам согласно приведенной в описании SNP-панели, для каждого сорта/линии сои и определяют сорт с желаемыми параметрами для определяемой совокупности признаков.
2. Способ по п. 1, характеризующийся тем, что для отбора сорта/линии сои с повышенным или пониженным процентным содержанием масла в семенах для каждого из сортов сои определяют последовательность ДНК во всех маркерах, выбранных из следующей группы: SEQ ID NO: 2, SEQ ID NO: 5, SEQ ID NO:8, SEQ ID NO: 11, SEQ ID NO:13, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 32, SEQ ID NO: 35, SEQ ID NO: 38, SEQ ID NO: 47, SEQ ID NO:48, SEQ ID NO: 49, SEQ ID NO:51, SEQ ID NO:57, SEQ ID NO:59, SEQ ID NO: 60, SEQ ID NO:61, SEQ ID NO: 63, SEQ ID NO: 70, SEQ ID NO: 71, SEQ ID NO: 80, SEQ ID NO:84, SEQ ID NO: 86, SEQ ID NO: 94, SEQ ID NO:96, SEQ ID NO: 97, SEQ ID NO: 100, SEQ ID NO: 102, SEQ ID NO: 103, SEQ ID NO: 105, SEQ ID NO: 106, SEQ ID NO: 109, SEQ ID NO: 110, SEQ ID NO:114, SEQ ID NO: 121, SEQ ID NO:125, SEQ ID NO: 127.
3. Способ по п. 1, характеризующийся тем, что для отбора сорта/линии сои с повышенным или пониженным процентным содержанием белка в семенах для каждого из сортов сои определяют последовательность ДНК во всех маркерах, выбранных из следующей группы: SEQ ID NO: 1, SEQ ID NO;7, SEQ ID NO: 15, SEQ ID NO: 128, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 26, SEQ ID NO: 37, SEQ ID NO: 43, SEQ ID NO: 50, SEQ ID NO: 56, SEQ ID NO:72, SEQ ID NO: 73, SEQ ID NO: 74, SEQ ID NO:75, SEQ ID NO: 78, SEQ ID NO:93, SEQ ID NO: 98, SEQ ID NO: 112, SEQ ID NO: 117, SEQ ID NO: 118, SEQ ID NO: 123.
4. Способ по п. 1, характеризующийся тем, что для отбора сорта/линии сои с повышенной или пониженной продуктивностью для каждого из сортов/линий сои определяют последовательность ДНК во всех маркерах, выбранных из следующей группы: SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO:14, SEQ ID NO: 23, SEQ ID NO: 24, SEQ SD NO: 25, SEQ ID NO: 28, SEQ ID NO: 30, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 36, SEQ ID NO:39, SEQ ID NO: 40, SEQ ID NO: 41, SEQ ID NO: 42, SEQ ID NO: 44, SEQ ID NO: 45, SEQ ID NO:46, SEQ ID NO: 52, SEQ ID NO.53, SEQ ID NO:54, SEQ ID NO:55, SEQ ID NO: 58, SEQ ID NO: 62, SEQ ID NO: 64, SEQ ID NO:65, SEQ ID NO:66, SEQ ID NO: 67, SEQ ID NO: 68, SEQ ID NO: 69, SEQ ID NO: 76, SEQ ID NO: 77, SEQ ID NO: 79, SEQ ID NO: 81, SEQ ID NO: 82, SEQ ID NO: 83, SEQ ID NO:85, SEQ ID NO: 87, SEQ ID NO: 88, SEQ SD NO: 89, SEQ ID NO: 90, SEQ ID NO: 91, SEQ ID NO: 92, SEQ ID NO: 95, SEQ ID NO: 99, SEQ ID NO: 101, SEQ ID NO: 104, SEQ ID NO: 107, SEQ ID NO: 108, SEQ ID NO: 111, SEQ ID NO: 113, SEQ ID NO: 115, SEQ ID NO: 116, SEQ ID NO: 119, SEQ ID NO: 120, SEQ ID NO: 122, SEQ ID NO: 124, SEQ ID NO: 126.
5. Способ по п. 1, характеризующийся тем, что для каждого из сортов/линий сои последовательность ДНК маркеров определяют при помощи секвенирования ампликонов.
6. Способ по п. 1, характеризующийся тем, что желаемыми параметрами для определяемой совокупности признаков являются одновременное повышенное процентное содержание белка и масла в семенах, а также повышенная продуктивность сорта/линии.
| WANG, J | |||
| et al | |||
| Identification and mapping of stable QTL for protein content in soybean seeds | |||
| Mol Breeding, (2015) 35: 92 | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| АБУГАЛИЕВА С.И | |||
| Генетическое разнообразие сои (Glycine Max (L) Merrill), разнообразие сои, Биотехнология | |||
| Теория и практика, N4, 2013, с.13-19, doi: 10.11134/btp.4.2013.2 | |||
| РАМАЗАНОВА С.А | |||
Авторы
Даты
2021-01-21—Публикация
2019-12-23—Подача