Область техники
Изобретение относится к областям геномики, молекулярной биологии, биоинформатики и селекции растений, в частности относится к селекции семян подсолнечника с помощью разработанной SNP-панели.
Уровень техники
Жирнокислотный состав масла является параметром, определяющим функциональные и технические свойства масла. Высокое содержание олеиновой кислоты определяет большую устойчивость масла к окислению, а также вносит вклад в снижение уровня холестерола в крови и уменьшение риска сердечно-сосудистых заболеваний (Grundy, 1986, Premnath et al., 2016, Fuller et al., 1967). Пониженное содержание линолевой кислоты и повышенное содержание линоленовой кислоты улучшают соотношениие омега-6 к омега-3 в масле, что является важным для уменьшения риска сердечно-сосудистых и воспалительных заболеваний ( and Murphy, 2009). Масло подсолнечника с оптимизированным соотношением омега-6 к омега-3 может найти применение в кондитерской и медицинской промышленности (Rauf et al., 2017). Получение подсолнечника с улучшенным соотношением омега-6 к омега-3 в масле семян представляется особенно важным в РФ, где он является основной масличной культурой.
and Murphy, 2009). Масло подсолнечника с оптимизированным соотношением омега-6 к омега-3 может найти применение в кондитерской и медицинской промышленности (Rauf et al., 2017). Получение подсолнечника с улучшенным соотношением омега-6 к омега-3 в масле семян представляется особенно важным в РФ, где он является основной масличной культурой.
По теме применения молекулярных маркеров в селекции подсолнечника и в области геномной селекции существуют публикации и патенты, однако, большинство из них касаются применения отдельных молекулярных маркеров на единичные хозяйственно-ценные признаки (например, US 2013254927, US 20090258346, WO 2017106274; Hongtrakul V., et al., (1998) A seed specific delta-12 oleate desaturase gene is duplicated, rearranged, and weakly expressed in high oleic acid sunflower lines. Crop Sci 38(5):1245-1249; Lacombe S., Berville A. (2001) A dominant mutation for high oleic acid content in sunflower seed oil is genetically linked to a single oleate-desaturase RFLP locus. Mol Breed 8(2):129-137; Perez-Vich B., Fernandez-Martinez J.M., Grondona M., Knapp S.J., Berry S.T. (2002) Stearoyl-ACP and oleoyl-PC desaturase genes cosegregate with quantitative trait loci underlying high stearic and high oleic acid mutant phenotypes in sunflower. Theor Appl Genet 104(2-3):338-349).
Несмотря на известность ряда способов селекции семян подсолнечника, на сегодняшний день на рынке сохраняется потребность в эффективном методе генотипирования семян подсолнечника для отбора семян с улучшенным жирнокислотным составом масла.
Сущность изобретения
Задачей настоящего изобретения является создание панели маркеров для генотипирования, представляющей собой набор из молекулярных маркеров на основе однонуклеотидных полиморфизмов генома подсолнечника. Данная панель предназначена для осуществления оценки геномной ценности селекционного материала подсолнечника и проведения селекции подсолнечника по комплексу хозяйственно-ценных признаков, включающих содержание жирных кислот 16:2, 18:1, 18:2, 18:3 и 20:2 в масле семян. Маркеры были выявлены в результате полногеномного поиска ассоциаций на основании данных о генотипе и фенотипе примерно 500 линий подсолнечника различного происхождения. Подобная панель маркеров имеет ряд преимуществ. Во-первых, панель маркеров обеспечивает лучший результат прогнозирования, чем единичный маркер. Во-вторых, использование панели позволяет принимать селекционные решения раньше, чем в традиционных селекционных программах, тем самым сокращая время селекции подсолнечника. Наконец, ДНК-маркеры легко измеримы стандартными методами (секвенированием, ПЦР или с помощью микрочипов).
Указанная задача решается путем создания способа отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирных кислот, выбранных из группы: 16:2, 18:1, 18:2, 18:3 и 20:2, включающего следующие стадии: а) выделяют геномную ДНК из семян подсолнечника меньшей мере двух различных сортов; б) для каждого из этих сортов определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из одной из следующих групп: S02_52501989, S06_5246521, S07_90144106, S11_68995900, S13_145305830, S14_109987127, S14_56559549, S14_64205529, S14_65302793, S14_65303987, S14_72096756, S17_18759543, или S01_117769266, S03_35155696, S03_45985345, S03_45985383, S03_57714775, S03_57714809, S08_129069633, или S02_22008907, S12_31930711, S12_36426640, или S01_110639563, S02_172598425, S02_179620148, S02_179872061, S03_167862882, S05_134224005, S05_165389465, S06_24822077, S08_5534412, S08_6242126, S09_112143602, S09_112153552, S09_14502473, S09_54150875, S10_178263586, S10_178337729, S10_178433439, S10_178513648, S10_204069518, S10_209927613, S10_213767877, S10_51538842, S11_82358252, S11_97936654, S12_107524160, S12_112746220, S12_120762889, S12_77297771, S13_105463640, S13_105487148, S13_123854633, S13_123930072, S13_124931273, S13_126530608, S13_133448536, S13_158874709, S13_159575588, S13_159575716, S13_160298625, S13_160341835, S13_162181797, S13_163335561, S13_165620043, S13_165620174, S13_166163943, S13_166236370, S13_39228680, S13_83080014, S13_83080207, S14_145387573, S14_27659348, S14_53314465, S14_53314632, S14_53329907,S14_53423360, S14_53423548, S14_53450243, S14_53450417, S14_53480813, S14_53480947, S14_53551912, S14_53562978, S14_59747903, S14_59866616, S14_59958364, S14_60107013, S14_60321165, S14_60321330, S14_60355581, S14_60429765, S14_60721757, S14_60765247, S14_60814564, S14_60826402, S14_60828070, S14_60952895, S14_61085378, S14_67039039, S14_70475580, S14_81249398, S15_118132600, S15_15985773, S15_54276880, S15_54276984, S15_54277167, S15_79200965, S16_108240559, S17_123498014, S17_158529906, S17_4501092, S17_55725068, или S04_154189714, S05_135016906, S09_49744939, S09_51030763, S10_193542723, S12_117949481, S16_4537999, при этом для каждого из этих сортов определяют последовательность ДНК в одинаковых маркерах; в) для каждого сорта вычисляют параметр содержания жирной кислоты, соответствующей определенному в (б) маркеру согласно приведенной в описании SNP-панели, при этом вычисление производят суммируя соответствующие значения эффектов аллеля, взятые из приведенной в описании SNP-панели, для каждого маркера, чья последовательность ДНК была определена и соответствующего данной жирной кислоте, и суммируя данные значения по всем маркерам, чья последовательность ДНК была определена и соответствующего данной жирной кислоте согласно приведенной в описании SNP-панели; г) сравнивают вычисленные параметры содержания жирной кислоты, соответствующей определенному в (б) маркеру согласно приведенной в описании SNP-панели, для каждого сорта семян подсолнечника, и определяют сорт с наибольшим или наименьшим параметром содержания данной жирной кислоты.
В некоторых вариантах изобретения данный способ характеризуется тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 16:2 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: S02_52501989, S06_5246521, S07_90144106, S11_68995900, S13_145305830, S14_109987127, S14_56559549, S14_64205529, S14_65302793, S14_65303987, S14_72096756, S17_18759543.
В некоторых вариантах изобретения данный способ характеризуется тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 18:1 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: S01_117769266, S03_35155696, S03_45985345, S03_45985383, S03_57714775, S03_57714809, S08_129069633.
В некоторых вариантах изобретения данный способ характеризуется тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 18:2 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: S02_22008907, S12_31930711, S12_36426640.
В некоторых вариантах изобретения данный способ характеризуется тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 18:3 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: S01_110639563, S02_172598425, S02_179620148, S02_179872061, S03_167862882, S05_134224005, S05_165389465, S06_24822077, S08_5534412, S08_6242126, S09_112143602, S09_112153552, S09_14502473, S09_54150875, S10_178263586, S10_178337729, S10_178433439, S10_178513648, S10_204069518, S10_209927613, S10_213767877, S10_51538842, S11_82358252, S11_97936654, S12_107524160, S12_112746220, S12_120762889, S12_77297771, S13_105463640, S13_105487148, S13_123854633, S13_123930072, S13_124931273, S13_126530608, S13_133448536, S13_158874709, S13_159575588, S13_159575716, S13_160298625, S13_160341835, S13_162181797, S13_163335561, S13_165620043, S13_165620174, S13_166163943, S13_166236370, S13_39228680, S13_83080014, S13_83080207, S14_145387573, S14_27659348, S14_53314465, S14_53314632, S14_53329907,S14_53423360, S14_53423548, S14_53450243, S14_53450417, S14_53480813, S14_53480947, S14_53551912, S14_53562978, S14_59747903, S14_59866616, S14_59958364, S14_60107013, S14_60321165, S14_60321330, S14_60355581, S14_60429765, S14_60721757, S14_60765247, S14_60814564, S14_60826402, S14_60828070, S14_60952895, S14_61085378, S14_67039039, S14_70475580, S14_81249398, S15_118132600, S15_15985773, S15_54276880, S15_54276984, S15_54277167, S15_79200965, S16_108240559, S17_123498014, S17_158529906, S17_4501092, S17_55725068.
В некоторых вариантах изобретения данный способ характеризуется тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 20:2 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: S04_154189714, S05_135016906, S09_49744939, S09_51030763, S10_193542723, S12_117949481, S16_4537999.
В предпочтительных вариантах изобретения данный способ характеризуется тем, что для каждого из сортов семян подсолнечника последовательность ДНК маркеров определяют при помощи ДНК-микрочипов.
Особенностью разработанной SNP-панели является то, что входящие в нее маркеры ассоциированы с признаками жирнокислотного состава масла семян подсолнечника. Техническим результатом настоящего изобретения является повышение надежности предсказания содержания определенных жирных кислот в масле семян подсолнечника.
Краткое описание рисунков
Фиг. 1. Этапы подготовки образцов ДНК к генотипированию.
Подробное раскрытие изобретения
В описании данного изобретения термины «включает» и «включающий» интерпретируются как означающие «включает, помимо всего прочего». Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из». Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе. Для обозначения ЖК в описании используется формула N:K, где N обозначает число атомов углерода в цепи (длину цепи), а K - число двойных С=С связей. Обозначения маркеров SNP панели в Таблице 1 соответствуют позициям полиморфизма ДНК в хромосоме ДНК подсолнечника, при этом авторы использовали версию генома подсолнечника HanXRQr1.0, впервые опубликованную в статье (Badouin, Helene et al. "The sunflower genome provides insights into oil metabolism, flowering and Asterid evolution". Nature. 2017 Jun 1;546(7656):148-152), и хранящуюся на сайте https://www.heliagene.org/. Последовательность хромосом доступна по ссылке https://www.heliagene.org/HanXRQ-SUNRISE/HanXRQr1.0-20151230-EGN-r1.2.zip . В названии каждого маркера закодировано его уникальное положение в указанном выше геноме. Например, для маркера S02_52501989, 02 перед нижним подчеркиванием обозначает хромосому номер 2 из указанного выше генома. Число после нижнего подчеркивания (в данном случае 52501989) соответствует позиции маркера от начала этой хромосомы.
Данное изобретение описывает ДНК-маркеры для определения генотипа и отбора растений подсолнечника, несущих оптимальный набор аллелей локусов генома, ассоциированных с содержанием жирных кислот в масле семян.
Определение генотипа растений подсолнечника с помощью SNP-панели может быть использовано в работах по анализу разнообразия, родства, сортовой чистоты образцов подсолнечника. Полученные с помощью SNP-панели данные о генотипе могут быть использованы для выявления образцов несущих набор аллелей, ассоциированных с пониженным, или повышенным содержанием отдельных жирных кислот для использования их в селекции.
Для отбора образцов подсолнечника для селекции на повышенное содержание жирной кислоты необходимо отбирать образцы, несущие комбинации аллелей с максимальным суммарным эффектом, в то время как для отбора образцов подсолнечника для селекции на пониженное содержание жирной кислоты необходимо отбирать образцы, несущие комбинации аллелей с минимальным суммарным эффектом.
Для этого проводится определение генотипа образцов и его сопоставление с SNP-панелью, раскрытой в настоящем изобретении. Используя значения эффекта аллелей для каждого из маркеров для соответствующей жирной кислоты вычисляется эффект маркера по формуле ЭМ=ЭА1*2 (формула 1) или ЭМ=ЭА2*2 (формула 2) для гомозиготного состояния маркера, и ЭМ=ЭА1+ЭА2 (формула 3) для гетерозиготного состояния маркера, где ЭA1 - эффект аллеля 1, ЭА2 - эффект аллеля 2. Вычисляется сумма эффектов по всем маркерам и отбирается образец с максимальным (для селекции на повышенное содержание жирной кислоты), или минимальным (для селекции на пониженное содержание жирной кислоты) суммарным эффектом. Схема отбора растений подсолнечника для селекции по содержанию жирных кислот в масле семян подсолнечника на основе данных генотипирования с использованием SNP-панели раскрыта в примере 2.
Маркеры, используемые в разработанной SNP-панели, были выявлены в результате полногеномного поиска ассоциаций на основании данных о генотипе и фенотипе примерно 500 линий подсолнечника различного происхождения. Суммарную ДНК из семян подсолнечника выделяли с использованием набора Nucleospin Plant Extraction Kit («MacheryNagel», Германия), следуя стандартному протоколу производителя. Генотипирование линий проводилось с использованием RAD-анализа c использованием эндонуклеаз рестрикции HindIII и Nlalll. Библиотеки RAD были секвенированы методами массового параллельного секвенирования при помощи Illumina HiSeq4000. Предварительная обработка сырых чтений была выполнена с использованием программного обеспечения Trimmomatic (версия 0.30) (Bolger, Lohse & Usadel, 2014), после чего в качестве входного сигнала для программы Tassel (версия 5.0) использовали 552 481 747 хороших баркодированных односторонних считываний (Bradbury et al., 2007) с длиной k-мера 65 пар нуклеотилов (п.н.). При выборе вариантов были применены дополнительные опции фильтрации: -mnMAF 0.01, -minMAPQ 10 и -mnQS 20. Для извлечения SNP маркеров использовали программу bcftools (версия 1.9).
Анализ состава жирных кислот в семенах культур был выполнен с использованием ультраэффективной жидкостной хроматографии, совмещенной с масс-спектрометром (Ultra performance liquid chromatography - tandem mass spectrometer (UPLC-MS)). Для экстракции липидов 10 мг (для каждой линии) семян подсолнечника гомогенизировали при температуре не более 10°C с использованием шести шариков из оксида циркония размером 2,8 мм (Bertin Instruments, Франция) в гомогенизаторе Precellys Evolution (Bertin Instruments, Франция), совмещенным с Cryolis, заполненным сухим льдом. Этот протокол основан на протоколе, описанном Giavalisco et al. (2011). Для количественного определения жирных кислот (ЖК) к экстракционной смеси добавляли три внутренних стандарта ЖК, меченных углеродным изотопом 13C (3 мкг каждой ЖК на образец: олеиновая кислота-13C18 (№ 490431, Sigma Aldrich), пальмитиновая кислота-13C16 (№ 605573, Sigma Aldrich) и стеариновая кислота. кислота-13C18 (# 605581, Sigma Aldrich)). Для анализа ЖК экстракты, полученные на предыдущем этапе, были гидролизованы с использованием протокола, описанного у Bromke et al (2015).
Чтобы подготовить образцы для инъекции в систему UPLC-MS, высушенные экстракты сначала ресуспендировали в смеси ацетонитрил / изопропанол (70:30), встряхивали в течение 10 сек и инкубировали в течение 10 минут при 4°С при встряхивании с последующей обработкой ультразвуком в течение 10 минут на льду и центрифугирования в течение 7 минут при 4°С со скоростью 12700 об/мин. После завершения этих процедур готовили конечные разведения ацетонитрил / изопропанол (70:30) во пробирках для масс-спектрометрии. Разделение UPLC осуществляли с использованием колонки C8 Acquity Beh (2,1 мм × 100 мм, размер частиц - 1,7 мкм; Waters, США) и предварительной колонки Acquity BEH C8 1,7 мкм Vanguard (Waters) при 60°C. Для градиента UPLC подвижные фазы состояли из двух растворителей. Были испытаны две разные системы растворителей и градиентов. Конечное разведение образца составляло 5 для буферных систем, описанных ниже:
Растворитель А: 1% 1 М NH4Ac и 0,1% муравьиной кислоты в воде; и растворитель B: ацетонитрил / изопропанол (7:3, 1% 1 М NH4Ac, 0,1% муравьиная кислота) с объемом введения 3 мкл. Применяли следующий профиль градиента: 55% растворителя В, 1 мин; линейный градиент от 55% растворителя В до 80% растворителя В, 3 мин; линейный градиент от 80% растворителя B до 85% растворителя B, 8 мин; линейный градиент от 85% растворителя B до 100% растворителя B, 3 мин. После промывки колонки в течение 4 минут 50 секунд со 100% растворителя B смесь снова доводили до 55% растворителя B, и колонку повторно уравновешивали в течение 4 минут 10 секунд (общее время работы 24,5 минут) со скоростью потока подвижной фазы равной 400 мкл/мин.
Программное обеспечение Xcms использовали для обработки данных UPLC-MS. Оптимальные параметры были выбраны с помощью пакета IPO. Пиковые интенсивности были получены с использованием функции peakTable, реализованной в пакете xcms при помощи метода method='maxint'. В результате был получен набор пиков, содержащий информацию о времени удержания молекул, массе ионов, деленной на заряд (m/z), и интенсивности для каждого образца. Чтобы идентифицировать пики, соответствующие жирным кислотам (ЖК), были сгенерированы все возможные химические формулы, которые соответствуют ЖК с длиной цепи, варьирующей от 10 до 28 атомов углерода (n) и с числом двойных связей, варьирующим от 0 до 6 (k), определяемые как CnH2n-2kO2. Поскольку используемый метод не позволяет различать изомерные ЖК, в дальнейшем в настоящем описании термин «ЖК» будет использоваться для обозначения групп изомеров, имеющих определенные n и k, например, ЖК С18:1 обозначает все жирные кислоты с 18 углеродами в цепи и однократной двойной связью. Были рассчитаны массы всех этих ЖК в депротонированном состоянии, и проанализированы все пики, которые соответствовали рассчитанным массам в пределах 20 ppm (часть на миллион, для двух масс m1 и m2 ppm = abs (m1-m2)/(max(m1,m2)*106)).
Затем вручную были выбраны пики на основе сетевых паттернов. Вкратце, было обнаружено, что увеличение длины цепи ЖК увеличивает время удержания молекул, в то время как добавление двойной связи уменьшает время удержания молекул, и таким образом, пики ЖК образуют сетевой паттерн. Интенсивность всех обнаруженных ЖК была как минимум на три порядка выше в образцах растений, чем в контрольных (пустых) образцах. В результате было выявлено 29 ЖК: C14:0, C15:0, C15:1, C16:0, C16:1, C16:2, C17:0, C17:1, C17:2, C18:0, C18:1, C18:2, C18:3, C19:0, C19:1, C19:2, C20:0, C20:1, C20:2, C21:0, C22:0, C22:1, C23:0, C23:1, C24:0, C24:1, C25:0, C26:0, C28:0. Данные RAD-генотипирования и количественные данные о содержании жирных кислот в масле семян были использованы для полногеномного поиска ассоциаций. Далее, был выполнен статистический анализ с использованием смешанных линейных моделей (MLMs) (Bradbury et al., 2007), реализованный в программном обеспечении Tassel, для выявления ассоциаций, используя MDS (многомерное шкалирование) и матрицы родства в качестве ковариатов. Перед анализом были отфильтрованы многоаллельные варианты и варианты с высокой возможностью ошибки (high missing call rates). Также исключены образцы подсолнечника с большим количеством недостающих данных. Статистически значимые локусы были идентифицированы на основе скорректированных методом Bonferroni p-значений. Локусы, для которых было показано наличие значимых ассоциаций, были отобраны в качестве маркеров. Были выявлены эффекты для аллелей каждого отобранного локуса.
Таким образом, изобретение раскрывает ДНК-маркеры для определения генотипа и отбора растений подсолнечника, несущих оптимальный набор аллелей локусов генома, ассоциированных с содержанием жирных кислот в масле семян. Список отобранных маркеров приведен в Таблице 1. Для использования в качестве маркеров аллельные состояния данных локусов генома могут быть идентифицированы с помощью ПЦР, ПЦР в реальном времени, гибридизации, или прямого секвенирования.
По Таблице 1, содержащей данные о SNP-панели, можно определить соответствие маркеров с типом жирной кислоты, а также для каждого маркера определить соответствующие значения эффектов аллеля. Например, маркер S02_52501989 соответствует жирной кислоте С16:2; поэтому, для вычисления параметра содержания жирной кислоты С16:2 можно использовать значения эффектов аллеля для маркера S02_52501989 (4,27187 или 4,2379, в зависимости от аллелей, присутствующих в ДНК конкретного сорта семян), либо значения эффектов аллеля для маркеров, выбранных из группы: S02_52501989, S06_5246521, S07_90144106, S11_68995900, S13_145305830, S14_109987127, S14_56559549, S14_64205529, S14_65302793, S14_65303987, S14_72096756, S17_18759543. При этом можно использовать как один маркер, так и любую комбинацию этих маркеров, в том числе одновременно все вышеуказанные маркеры; при использовании комбинации маркеров значения эффектов аллеля, соответствующие этим маркерам, необходимо суммировать для вычисления параметра содержания жирной кислоты. Для каждого из сортов подсолнечника необходимо определить последовательность ДНК в одинаковых маркерах.
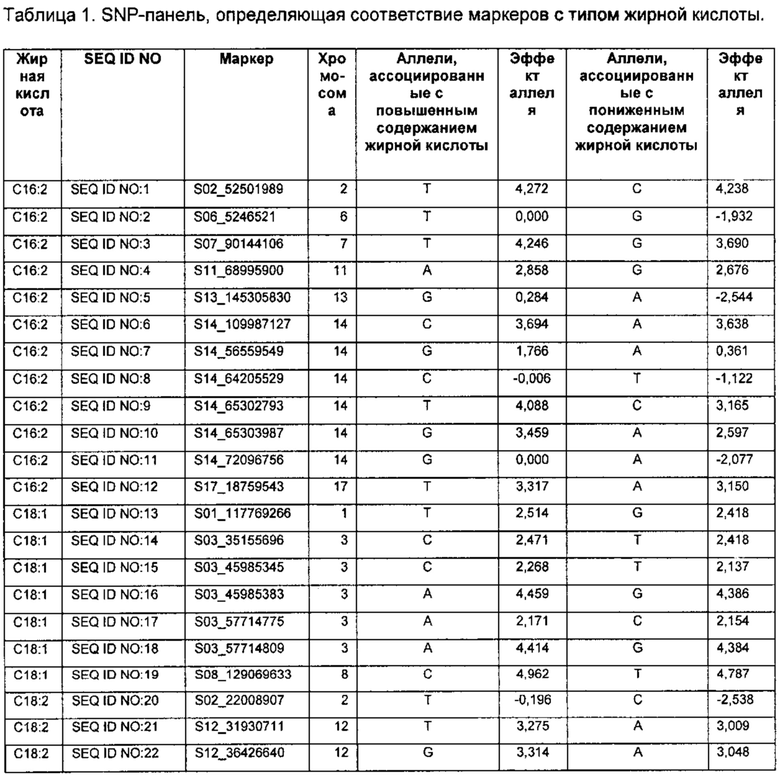
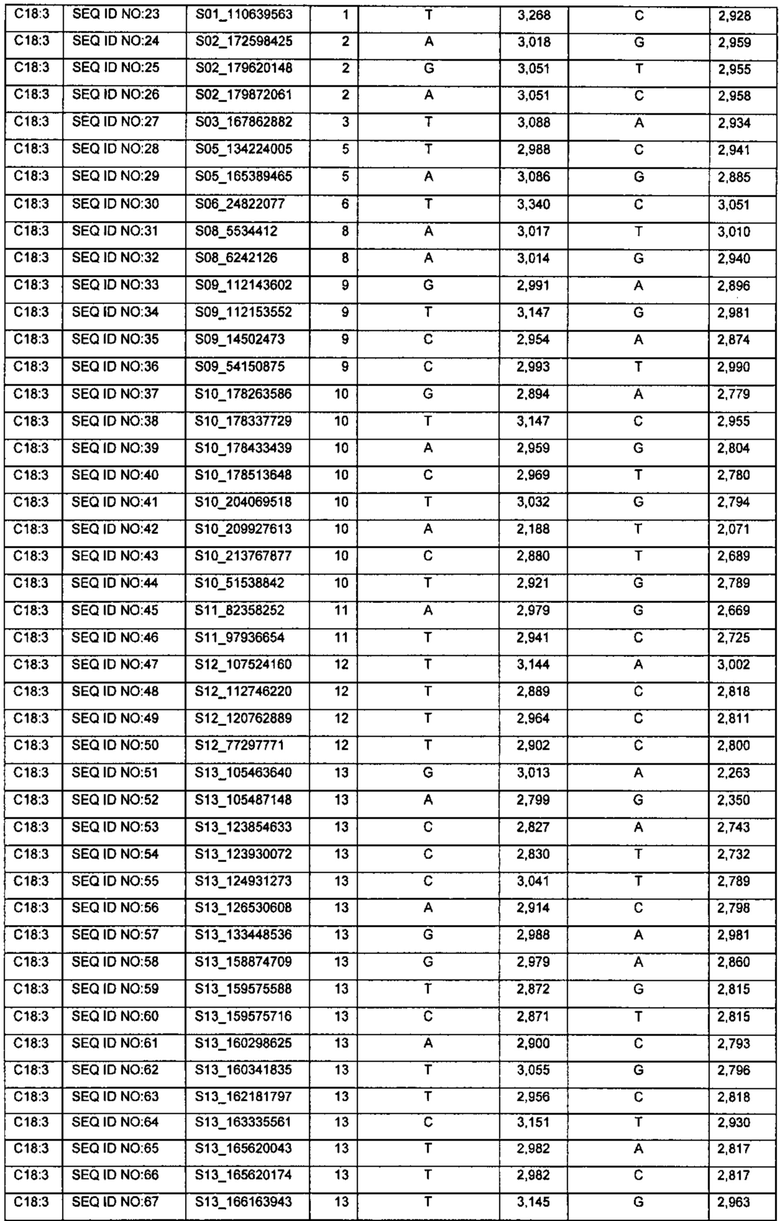
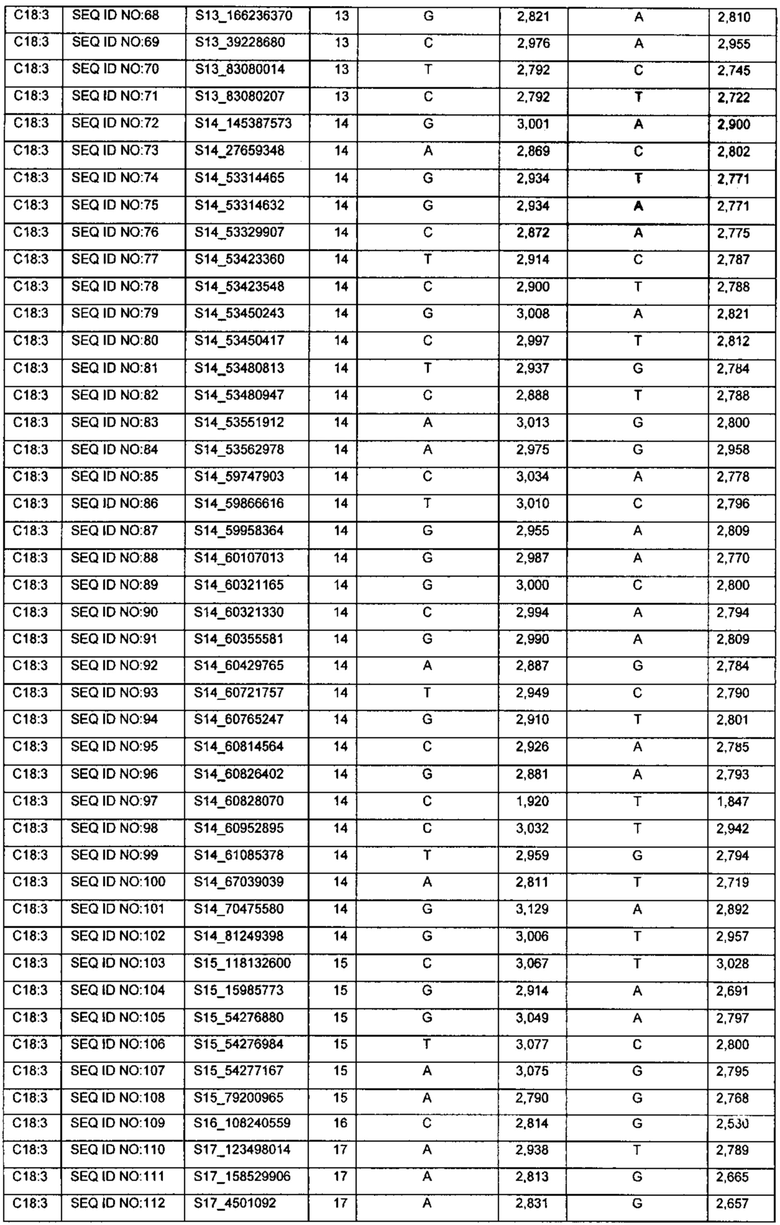
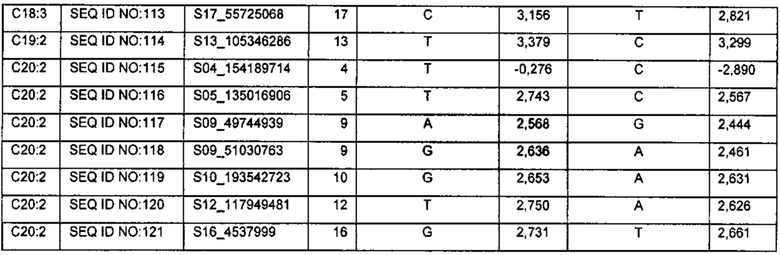
Коэффициенты "Эффект аллеля" были рассчитаны с помощью программы Tassel 5 (Bradbury PJ, Zhang Z, Kroon DE, Casstevens TM, Ramdoss Y, Buckler ES. (2007) TASSEL: Software for association mapping of complex traits in diverse samples. Bioinformatics 23:2633-2635). Каждый из указанных аллелей для одной ЖК может быть использован для предсказания независимо от других аллелей. Использование сразу нескольких аллелей для одной ЖК одновременно повышает вероятность нахождения семян с существенно отличными показателями присутствия этой ЖК. Поэтому, хотя использование только одного аллеля для предсказания может быть достаточным для обнаружения разницы по присутствию определенной ЖК, в предпочтительных вариантах изобретения для предсказания используют одновременно несколько, или все из указанных аллелей для одной ЖК.
Обозначения маркеров SNP панели в Таблице 1 соответствуют позициям полиморфизма ДНК в хромосоме ДНК подсолнечника, при этом авторы использовали версию генома подсолнечника HanXRQr1.0, впервые опубликованную в статье (Badouin, Helene et al., Nature, 2017 Jun 1;546(7656): 148-152), и хранящуюся на сайте https://www.heliagene.org/. В названии каждого маркера закодировано его уникальное положение в указанном выше геноме. Например, для маркера S02_52501989, 02 перед нижним подчеркиванием обозначает хромосому номер 2 из указанного выше генома. Число после нижнего подчеркивания (в данном случае 52501989) соответствует позиции маркера от начала этой хромосомы. Для однозначной идентификации позиций маркеров на хромосомах подсолнечника ниже приведены фрагменты последовательностей геномной ДНК подсолнечника, содержащие маркеры SNP из Таблицы 1 (приведены 150 п. н. до каждого маркера и 150 п. н. после каждого маркера, каждый SNP полиморфизм из Таблицы 1 указан в скобках):
SEQ ID NO: 1

SEQ ID NO: 117

SEQ ID NO: 118

SEQ ID NO: 12

SEQ ID NO: 6

SEQ ID NO: 36

SEQ ID NO: 121

SEQ ID NO: 31

SEQ ID NO: 72

SEQ ID NO: 4

SEQ ID NO: 58


SEQ ID NO: 29

SEQ ID NO: 32

SEQ ID NO: 10

SEQ ID NO: 45

SEQ ID NO: 43

SEQ ID NO: 103

SEQ ID NO: 46

SEQ ID NO: 8
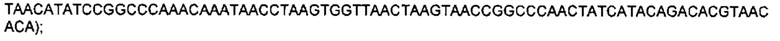
SEQ ID NO: 104
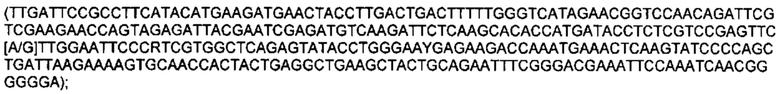
SEQ ID NO: 27

SEQ ID NO: 13

SEQ ID NO: 14

SEQ ID NO: 9

SEQ ID NO: 18

SEQ ID NO: 16
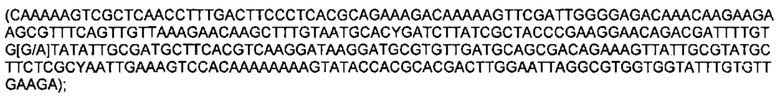
SEQ ID NO: 47

SEQ ID NO: 28

SEQ ID NO: 3

SEQ ID NO: 119

SEQ ID NO: 101
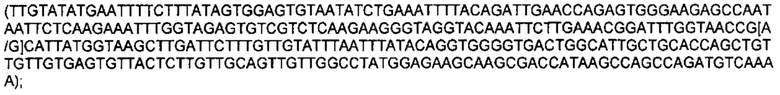
SEQ ID NO: 88

SEQ ID NO: 38

SEQ ID NO: 42

SEQ ID NO: 22

SEQ ID NO: 116

SEQ ID NO: 111

SEQ ID NO: 85

SEQ ID NO: 109

SEQ ID NO: 25
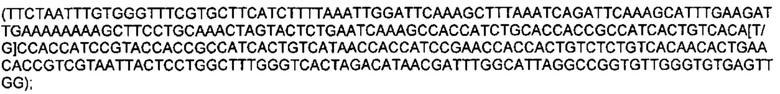
SEQ ID NO: 86

SEQ ID NO: 87

SEQ ID NO: 94

SEQ ID NO: 35

SEQ ID NO: 84

SEQ ID NO: 21

SEQ ID NO: 81

SEQ ID NO: 74

SEQ ID NO: 75

SEQ ID NO: 80

SEQ ID NO: 95

SEQ ID NO: 98

SEQ ID NO: 93

SEQ ID NO: 26

SEQ ID NO: 78
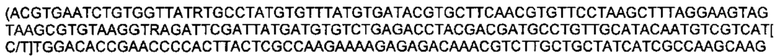

SEQ ID NO: 19

SEQ ID NO: 120

SEQ ID NO: 89

SEQ ID NO: 90

SEQ ID NO: 91

SEQ ID NO: 40

SEQ ID NO: 79

SEQ ID NO: 48
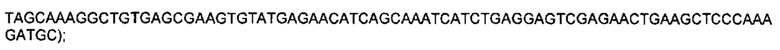
SEQ ID NO: 99

SEQ ID NO: 97

SEQ ID NO: 68

SEQ ID NO: 77

SEQ ID NO: 92

SEQ ID NO: 83

SEQ ID NO: 102
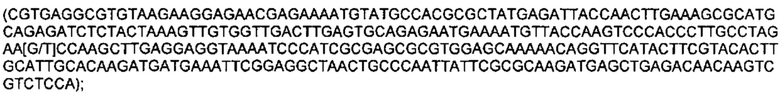
SEQ ID NO: 7

SEQ ID NO: 76

SEQ ID NO: 37

SEQ ID NO: 100

SEQ ID NO: 39

SEQ ID NO: 49

SEQ ID NO: 2

SEQ ID NO: 82

SEQ ID NO: 44
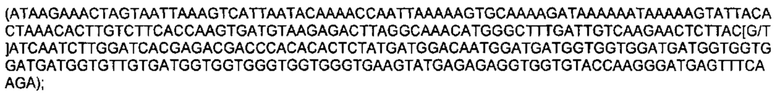
SEQ ID NO: 30

SEQ ID NO: 96
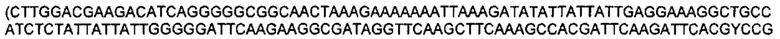

SEQ ID NO: 112

SEQ ID NO: 69

SEQ ID NO: 114

SEQ ID NO: 20

SEQ ID NO: 65

SEQ ID NO: 66
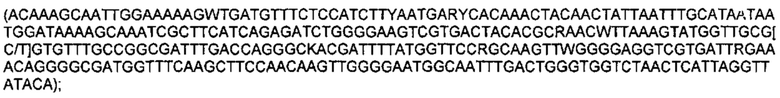
SEQ ID NO: 5

SEQ ID NO: 11

SEQ ID NO: 55

SEQ ID NO: 57

SEQ ID NO: 24
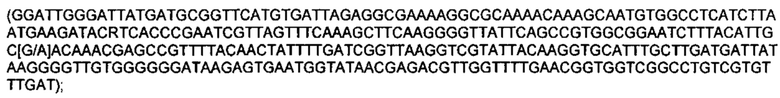
SEQ ID NO: 70

SEQ ID NO: 64

SEQ ID NO: 56

SEQ ID NO: 54

SEQ ID NO: 53

SEQ ID NO: 71
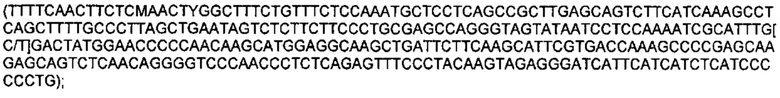
SEQ ID NO: 41

SEQ ID NO: 73

SEQ ID NO: 33
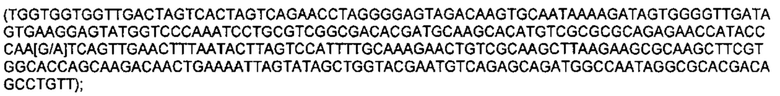
SEQ ID NO: 115

SEQ ID NO: 23

SEQ ID NO: 63

SEQ ID NO: 50

SEQ ID NO: 105

SEQ ID NO: 62
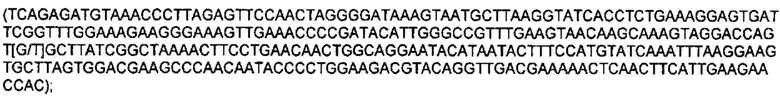
SEQ ID NO: 52

SEQ ID NO: 67
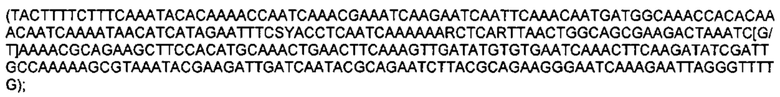
SEQ ID NO: 61

SEQ ID NO: 59

SEQ ID NO: 60

SEQ ID NO: 108

SEQ ID NO: 106

SEQ ID NO: 107

SEQ ID NO: 110

SEQ ID NO: 113

SEQ ID NO: 34
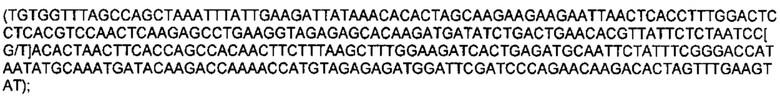
SEQ ID NO: 51

Нижеследующие примеры осуществления способа приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Пример 1. Применение SNP-панели для генотипирования образцов подсолнечника.
Для демонстрации возможности использования SNP-панели для генотипирования растительного материала, были взяты 8 образцов подсолнечника с ранее неизвестным генотипом. С использованием SNP-панели для генотипирования и геномной селекции подсолнечника были определены генотипы 8 образцов подсолнечника на платформе MassArray. Этапы определения генотипов описаны ниже.
1. На первом этапе была проведена подготовка образцов к дальнейшему анализу, которая включала несколько последовательных этапов (Фиг. 1):
• Мультиплексная ПЦР заключается в многократном увеличении количества фрагментов, содержащих однонуклеотидные полиморфизмы. Для этого была проведена классическая ПЦР-реакция, включающая денатурацию, отжиг и элонгацию;
• SAP-реакция проводится с целью удаления не включенных в ампликоны одиночных нуклеотидов (dNTPs) с использованием фермента щелочной фосфатазы (SAP). Для этого сразу после мультиплексной ПЦР к образцам была добавлена фосфатаза, после чего смесь инкубировали 40 мин при 37°С, и инактивировали действие фермента прогреванием при 85°С в течении 15 мин;
• iPlex-реакция заключается во включении модифицированного нуклеотида (ddNTP) в позицию, комплементарную каждому SNP, находящегося в полиморфной позиции, т.о. была получена смесь фрагментов по полиморфным маркерам. Для проведения iPlex-реакции к образцам были добавлены специальный буфер, ДНК-полимераза, нуклеотиды с модифицированной молекулярной массой и праймеры и была проведена амплификация. Затем образцы ДНК были очищены от остатков реакции (соли, ионы, ферменты), которые при анализе на масс-спектрометре вносят шум;
• Перенос образцов на чип (SpectroCHIP Array) для генотипирования методом масс-спектрометрии MALDI-TOF. Чип представлен в виде подложки с матрицей для ионизации.
2. Далее была произведена ионизация образцов. Образцы были помещены в рабочую камеру, будучи равномерно смешаны с большим количеством тонкого слоя кислоты, выполняющего роль матрицы. Матрица поглощает ультрафиолетовый свет (длина волны азотного лазера 337 нм), преобразуя его в тепловую энергию. При этом небольшая часть матрицы (до 100 нм от верхней наружной поверхности анализируемого вещества) нагревается за несколько нано секунд и испаряется вместе с образцом.
Заряженные ионы различных размеров генерируются на слайде образца. Разность потенциалов между скольжением образца и землей обеспечивает движение ионов. Скорость притягиваемых ионов определяется законом сохранения энергии. Поскольку разность потенциалов постоянна по отношению ко всем ионам, ионы с меньшим значением отношения массы к заряду (более легкие ионы) и более высоконагруженные ионы движутся быстрее через дрейфовое пространство, пока не достигнут детектора. Следовательно, время полета иона различается в зависимости от отношения массы к заряду (m/z) иона.
Процесс ионизации и получения масс-спектров 96-луночного спектро-чипа занимает примерно 20 минут. Анализ масс-спектров в реальном времени поводили с помощью программного обеспечения MassARRAY TYPER 4.0 ("Sequenom").
Результаты определения генотипа 8 образцов подсолнечника с помощью SNP-панели для генотипирования и геномной селекции подсолнечника приведены в Таблице 2.
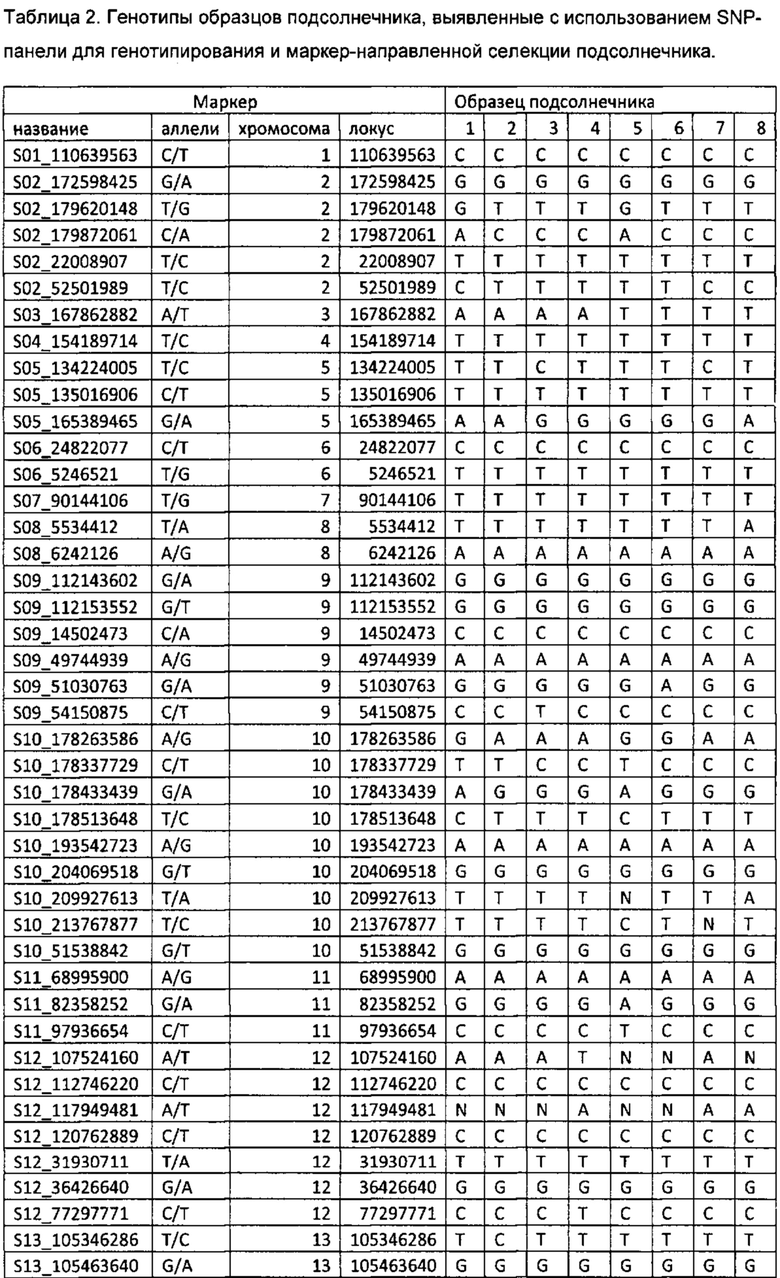
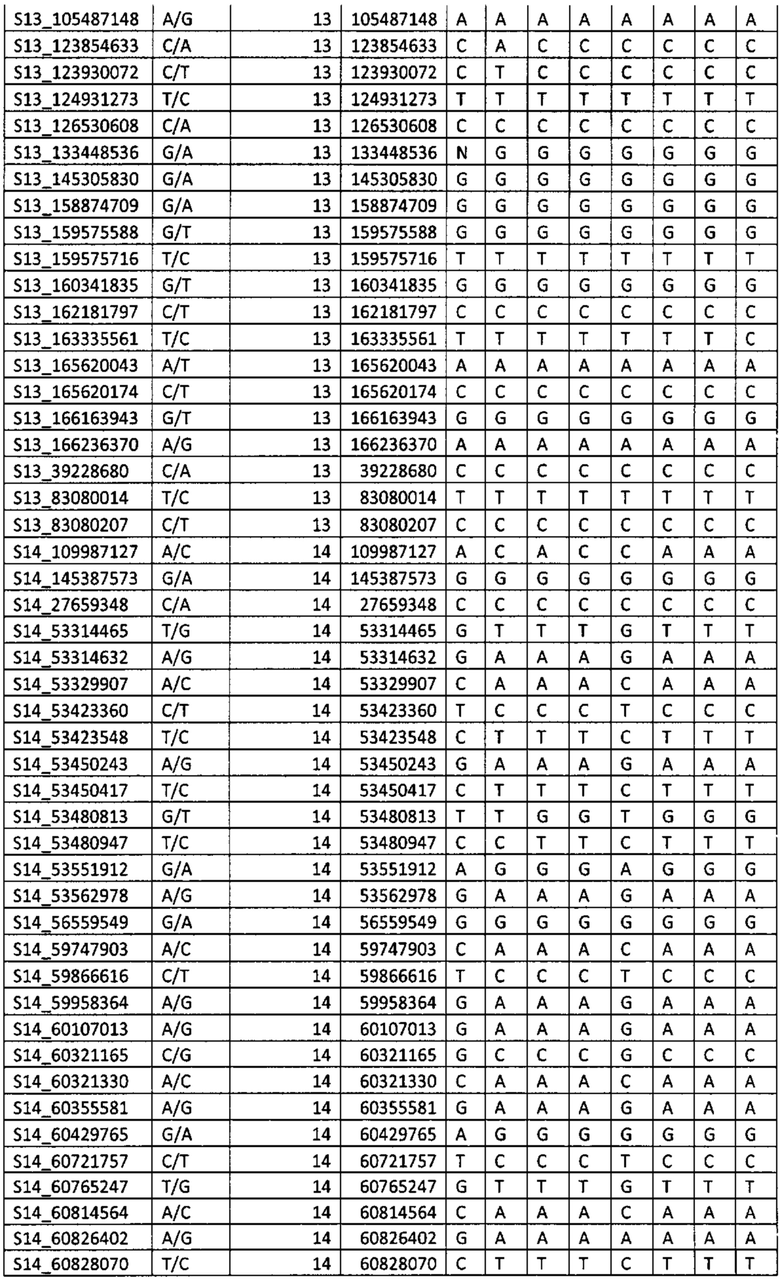
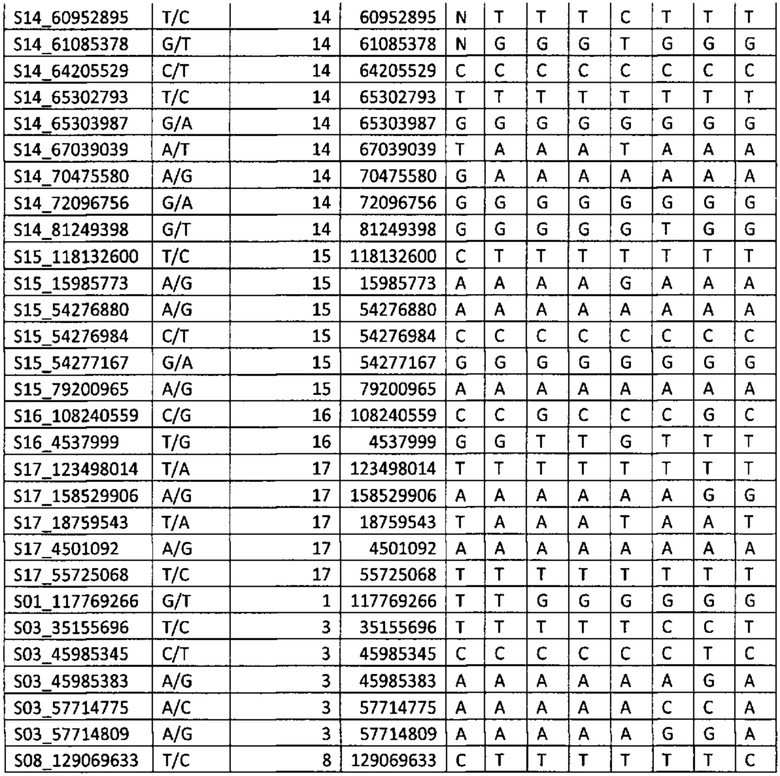
С - обозначают гомозиготу СС,
А - обозначают гомозиготу АА,
Т - обозначают гомозиготу ТТ,
G - обозначают гомозиготу GG,
N - обозначают гетерозиготные состояния маркера.
Полученные результаты показывают возможность успешного применения SNP-панели для определения генотипа селекционного материала подсолнечника.
Пример 2. Схема отбора растений подсолнечника для селекции по признаку повышенного содержания омега-3 линоленовой кислоты в масле семян подсолнечника на основе данных генотипирования с использованием SNP-панели.
Определение селекционной ценности сортов включает в себя несколько этапов.
(1) Определение генотипа семян подсолнечника (интересующих релевантных аллелей) и сравнение их с SNP-панелью, раскрытой в настоящем изобретении, по локусам, ассоциированным с содержанием омега-3 линоленовой кислоты (генотип образцов 5 и 2, определенный с использованием SNP-панели, приведен в таблице 3 в столбцах «Генотип образца 5» и «Генотип образца 2»). Суммарную ДНК выделяли с использованием набора Nucleospin Plant Extraction Kit («MacheryNagel», Германия), следуя стандартному протоколу производителя.
(2) Используя значения эффекта аллелей для каждого из маркеров из соответствующего столбца таблицы 3 вычисляли эффект маркера по формуле ЭМ=ЭА1*2 (формула 1) или ЭМ=ЭА2*2 (формула 2) для гомозиготного состояния маркера, и ЭМ=ЭА1+ЭА2 (формула 3), для гетерозиготного состояния маркера, где ЭА1 - эффект аллеля 1, ЭА2 - эффект аллеля 2. Поскольку образцы 2 и 5 являются гомозиготными по всем маркерам, использовали только формулы 1 и 2.
(3) Вычисляли сумму эффектов по всем маркерам, ассоциированным с содержанием омега-3 линоленовой кислоты для образцов 5 и 2. Сумма эффектов маркеров для образца 5 равняется 520,926, сумма эффектов для образца 2 равняется 510,3483.
Таким образом, для селекции по признаку повышенного содержания омега-3 линоленовой кислоты в масле семян подсолнечника следует отобрать образец 5, как имеющий большее значение суммы эффектов маркеров.
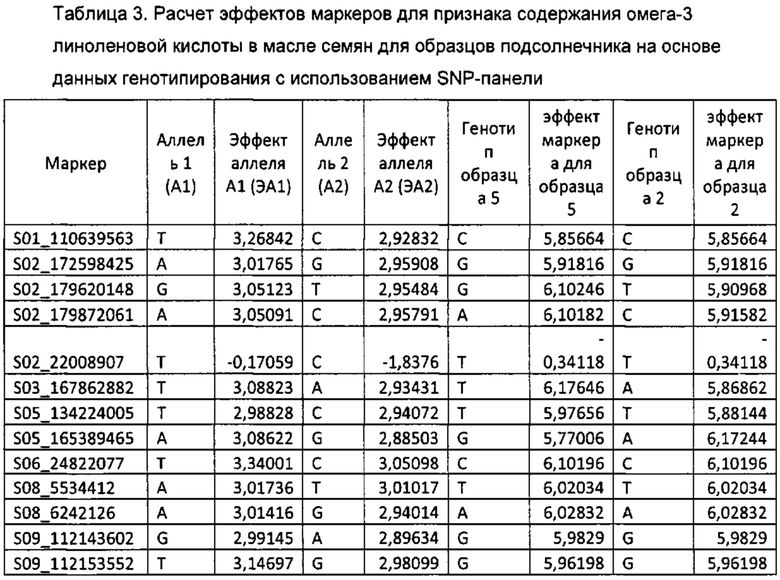
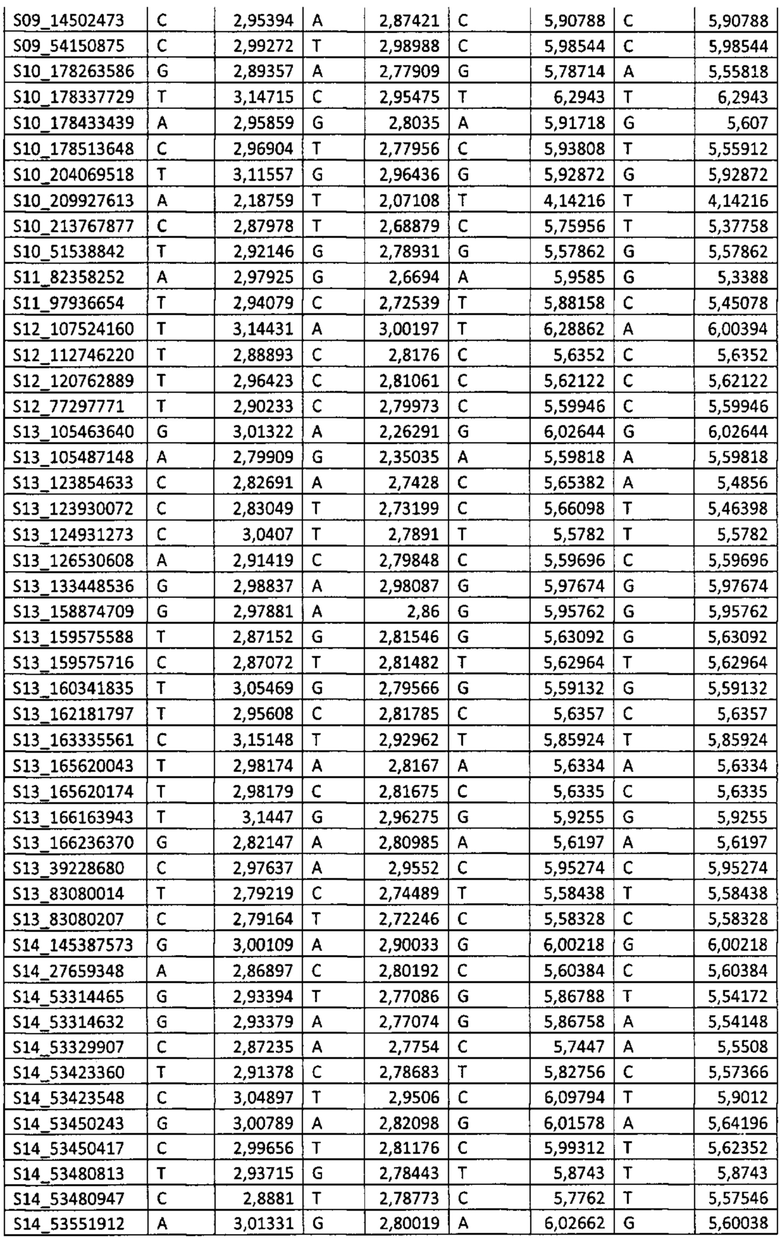
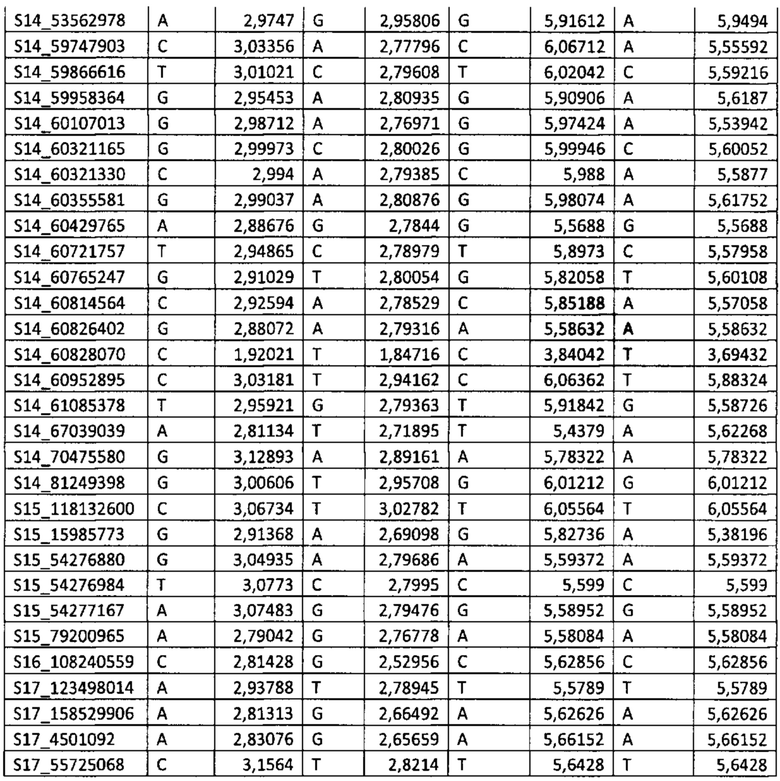
Для проверки правильности предсказания было проведено прямое измерение содержания омега-3 линоленовой кислоты в образцах 2 и 5 в масле семян подсолнечника с использованием UPLC-MS. Для перевода результатов измерения на масс-спектрометре в значения содержания жирной кислоты рассчитывались нормированные на верхнюю квартиль логарифмы интенсивностей. Полученные значения содержания омега-3 линоленовой кислоты для образца 5 равнялась 13.143773502, для образца 2-11.6786155956.
Таким образом, разработанная SNP-панель может быть использована для селекции подсолнечника по жирнокислотному составу семян.
Несмотря на то, что изобретение описано со ссылкой на раскрываемые варианты воплощения, для специалистов в данной области должно быть очевидно, что конкретные подробно описанные случаи приведены лишь в целях иллюстрирования настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения. Должно быть, понятно, что возможно осуществление различных модификаций без отступления от сути настоящего изобретения.
Список литературы:
1. Bolger, A.M.; Lohse, М.; Usadel, В. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics 2014, 30, 2114-2120.
2. Bradbury PJ, Zhang Z, Kroon DE, Casstevens TM, Ramdoss Y, Buckler ES. (2007) TASSEL: Software for association mapping of complex traits in diverse samples. Bioinformatics 23:2633-2635
3. Giavalisco, P. et a/. Elemental formula annotation of polar and lipophilic metabolites using 13C, 15N and 34S isotope labelling, in combination with high-resolution mass spectrometry. Plant J. 68, (2011).
4. Bromke, M.A. et al. Liquid chromatography high-resolution mass spectrometry for fatty acid profiling. Plant J. 81, 529-536 (2015).
5. Zhang, Z.; Ersoz, E.; Lai, C.-Q.; Todhunter, R.J.; Tiwari, H.K.; Gore, M.A.; Bradbury, P.J.; Yu, J.; Arnett, D.K.; Ordovas, J.M.; et al. Mixed linear model approach adapted for genome-wide association studies. Nat. Genet. 2010, 42, 355-360.
6. Grundy, S. M. Comparison of monounsaturated fatty acids and carbohydrates for lowering plasma cholesterol. N. Engl. J. Med. 314, 745-748 (1986).
7. Premnath, A., Narayana, M., Ramakrishnan, C, Kuppusamy, S. & Chockalingam, V. Mapping quantitative trait loci controlling oil content, oleic acid and linoleic acid content in sunflower (Helianthus annuus L.). Mol. Breed. 36, (2016).
8. Fuller, G., Diamond, M. J. & Applewhite, Т. H. High-oleic sanflower oil. Stability and chemical modification. J. Am. Oil Chem. Soc. 44, 264-266
9.  G and Murphy E J, Alpha-linolenic acid and its conversion to longer chain n-3 fatty acids: Benefits for human health and a role in maintaining tissue n-3 fatty acid levels. Prog Lipid Res 48: 355-374 (2009).
G and Murphy E J, Alpha-linolenic acid and its conversion to longer chain n-3 fatty acids: Benefits for human health and a role in maintaining tissue n-3 fatty acid levels. Prog Lipid Res 48: 355-374 (2009).
10. Rauf, S., Jamil, N., Tariq, S. A., Khan, M., Kausar, M., & Kaya, Y. (2017). Progress in modification of sunflower oil to expand its industrial value. Journal of the Science of Food and Agriculture, 97(7), 1997-2006. doi:10.1002/jsfa.8214.
| название | год | авторы | номер документа |
|---|---|---|---|
| Маркеры для маркерной селекции сои по хозяйственно-полезным признакам | 2019 |
|
RU2740798C1 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ НИЗКОГО СОДЕРЖАНИЯ ПАЛЬМИТИНОВОЙ КИСЛОТЫ В ПОДСОЛНЕЧНИКЕ (HELIANTHUS ANNUS) И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2013 |
|
RU2670517C2 |
| ПОДСОЛНЕЧНИК С НИЗКИМ СОДЕРЖАНИЕМ НАСЫЩЕННЫХ ЖИРОВ И СООТВЕТСТВУЮЩИЕ СПОСОБЫ | 2008 |
|
RU2489849C2 |
| ПОДСОЛНЕЧНИК С НИЗКИМ СОДЕРЖАНИЕМ НАСЫЩЕННЫХ ЖИРОВ И СООТВЕТСТВУЮЩИЕ СПОСОБЫ | 2013 |
|
RU2639561C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| Brassica juncea КАЧЕСТВА ОМЕГА-9 | 2009 |
|
RU2557316C2 |
| МАРКЕРЫ, СЦЕПЛЕННЫЕ С УСТОЙЧИВОСТЬЮ РАСТЕНИЙ СОИ К SCN | 2011 |
|
RU2620973C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM4 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2718584C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ПОЛУЧЕНИЯ УКОРОЧЕННЫХ РАСТЕНИЙ КУКУРУЗЫ | 2016 |
|
RU2745987C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |

Изобретение относится к области биохимии, в частности к способу отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирных кислот, выбранных из группы: 16:2, 18:1, 18:2, 18:3 и 20:2. Изобретение позволяет эффективно определять содержание жирных кислот. 6 з.п. ф-лы, 1 ил., 3 табл., 2 пр.
1. Способ отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирных кислот, выбранных из группы: 16:2, 18:1, 18:2, 18:3 и 20:2, включающий следующие стадии:
а) выделяют геномную ДНК из семян подсолнечника по меньшей мере двух различных сортов;
б) для каждого из этих сортов определяют последовательность ДНК в по меньшей мере одном релевантном для типа жирной кислоты маркере, выбранном из следующих групп:
для группы жирных кислот 16:2:
SEQ ID NO:1, SEQ ID NO:2, SEQ ID NO:3, SEQ ID NO:4, SEQ ID NO:5, SEQ ID NO:6, SEQ ID NO:7, SEQ ID NO:8, SEQ ID NO:9, SEQ ID NO:10, SEQ ID NO:11, SEQ ID NO:12;
для группы жирных кислот 18:1:
SEQ ID NO:13, SEQ ID NO:14, SEQ ID NO:15, SEQ ID NO:16, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19;
для группы жирных кислот 18:2:
SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:22
для группы жирных кислот 18:3:
SEQ ID NO:23, SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:30, SEQ ID NO:31, SEQ ID NO:32, SEQ ID NO:33, SEQ ID NO:34, SEQ ID NO:35, SEQ ID NO:36, SEQ ID NO:37, SEQ ID NO:38, SEQ ID NO:39, SEQ ID NO:40, SEQ ID NO:41, SEQ ID NO:42, SEQ ID NO:43, SEQ ID NO:44, SEQ ID NO:45, SEQ ID NO:46, SEQ ID NO:47, SEQ ID NO:48, SEQ ID NO:49, SEQ ID NO:50, SEQ ID NO:51, SEQ ID NO:52, SEQ ID NO:53, SEQ ID NO:54, SEQ ID NO:55, SEQ ID NO:56, SEQ ID NO:57, SEQ ID NO:58, SEQ ID NO:59, SEQ ID NO:60, SEQ ID NO:61, SEQ ID NO:62, SEQ ID NO:63, SEQ ID NO:64, SEQ ID NO:65, SEQ ID NO:66, SEQ ID NO:67, SEQ ID NO:68, SEQ ID NO:69, SEQ ID NO:70, SEQ ID NO:71, SEQ ID NO:72, SEQ ID NO:73, SEQ ID NO:74, SEQ ID NO:75, SEQ ID NO:76, SEQ ID NO:77, SEQ ID NO:78, SEQ ID NO:79, SEQ ID NO:80, SEQ ID NO:81, SEQ ID NO:82, SEQ ID NO:83, SEQ ID NO:84, SEQ ID NO:85, SEQ ID NO:86, SEQ ID NO:87, SEQ ID NO:88, SEQ ID NO:89, SEQ ID NO:90, SEQ ID NO:91, SEQ ID NO:92, SEQ ID NO:93, SEQ ID NO:94, SEQ ID NO:95, SEQ ID NO:96, SEQ ID NO:97, SEQ ID NO:98, SEQ ID NO:99, SEQ ID NO:100, SEQ ID NO:101, SEQ ID NO:102, SEQ ID NO:103, SEQ ID NO:104, SEQ ID NO:105, SEQ ID NO:106, SEQ ID NO:107, SEQ ID NO:108, SEQ ID NO:109, SEQ ID NO:110, SEQ ID NO:111, SEQ ID NO:112, SEQ ID NO:113;
для группы жирных кислот 20:2:
SEQ ID NO:115, SEQ ID NO:116, SEQ ID NO:117, SEQ ID NO:118, SEQ ID NO:119, SEQ ID NO:120, SEQ ID NO:121;
при этом для каждого из этих сортов определяют последовательность ДНК в одинаковых маркерах;
в) для каждого сорта вычисляют параметр содержания жирной кислоты, соответствующей определенному в (б) маркеру согласно приведенной в описании SNP- панели по Таблице 1, при этом вычисление производят определяя значения эффекта маркера (ЭМ) по соответствующим значениям эффектов аллеля для каждого маркера, чья последовательность ДНК была определена, и соответствующего данной жирной кислоте, по следующим формулам: ЭМ=ЭА1*2 (формула 1) или ЭМ=ЭА2*2 (формула 2) для гомозиготного состояния маркера, и ЭМ=ЭА1+ЭА2 (формула 3), для гетерозиготного состояния маркера, где ЭД1 - эффект аллеля 1, ЭА2- эффект аллеля 2, определенные в соответствии с Таблицей 1, и далее суммируя данные значения по всем маркерам, чья последовательность ДНК была определена, и соответствующего данной жирной кислоте согласно приведенной в описании SNP-панели по Таблице 1;
г) сравнивают вычисленные параметры содержания жирной кислоты, соответствующей определенному в (б) маркеру согласно приведенной в описании SNP- панели по Таблице 1, для каждого сорта семян подсолнечника и определяют сорт с наибольшим или наименьшим параметром содержания данной жирной кислоты.
2. Способ по п. 1, характеризующийся тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 16:2 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: SEQ ID NO:1, SEQ ID NO:2, SEQ ID NO:3, SEQ ID NO:4, SEQ ID NO:5, SEQ ID NO:6, SEQ ID NO:7, SEQ ID NO:8, SEQ ID NO:9, SEQ ID NO:10, SEQ ID NO:11, SEQ ID NO:12.
3. Способ по п. 1, характеризующийся тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 18:1 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: SEQ ID NO:13, SEQ ID NO:14, SEQ ID NO:15, SEQ ID NO:16, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19.
4. Способ по п. 1, характеризующийся тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 18:2 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:22.
5. Способ по п. 1, характеризующийся тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 18:3 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: SEQ ID NO:23, SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:30, SEQ ID NO:31, SEQ ID NO:32, SEQ ID NO:33, SEQ ID NO:34, SEQ ID NO:35, SEQ ID NO:36, SEQ ID NO:37, SEQ ID NO:38, SEQ ID NO:39, SEQ ID NO:40, SEQ ID NO:41, SEQ ID NO:42, SEQ ID NO:43, SEQ ID NO:44, SEQ ID NO:45, SEQ ID NO:46, SEQ ID NO:47, SEQ ID NO:48, SEQ ID NO:49, SEQ ID NO:50, SEQ ID NO:51, SEQ ID NO:52, SEQ ID NO:53, SEQ ID NO:54, SEQ ID NO:55, SEQ ID NO:56, SEQ ID NO:57, SEQ ID NO:58, SEQ ID NO:59, SEQ ID NO:60, SEQ ID NO:61, SEQ ID NO:62, SEQ ID NO:63, SEQ ID NO:64, SEQ ID NO:65, SEQ ID NO:66, SEQ ID NO:67, SEQ ID NO:68, SEQ ID NO:69, SEQ ID NO:70, SEQ ID NO:71, SEQ ID NO:72, SEQ ID NO:73, SEQ ID NO:74, SEQ ID NO:75, SEQ ID NO:76, SEQ ID NO:77, SEQ ID NO:78, SEQ ID NO:79, SEQ ID NO:80, SEQ ID NO:81, SEQ ID NO:82, SEQ ID NO:83, SEQ ID NO:84, SEQ ID NO:85, SEQ ID NO:86, SEQ ID NO:87, SEQ ID NO:88, SEQ ID NO:89, SEQ ID NO:90, SEQ ID NO:91, SEQ ID NO:92, SEQ ID NO:93, SEQ ID NO:94, SEQ ID NO:95, SEQ ID NO:96, SEQ ID NO:97, SEQ ID NO:98, SEQ ID NO:99, SEQ ID NO:100, SEQ ID NO:101, SEQ ID NO:102, SEQ ID NO:103, SEQ ID NO:104, SEQ ID NO:105, SEQ ID NO:106, SEQ ID NO:107, SEQ ID NO:108, SEQ ID NO:109, SEQ ID NO:110, SEQ ID NO:111, SEQ ID NO:112, SEQ ID NO:113, SEQ ID NO:114.
6. Способ по п. 1, характеризующийся тем, что для отбора сорта семян подсолнечника с повышенным или пониженным содержанием жирной кислоты с формулой 20:2 для каждого из сортов семян подсолнечника определяют последовательность ДНК в по меньшей мере одном маркере, выбранном из следующей группы: SEQ ID NO:115, SEQ ID NO:116, SEQ ID NO:117, SEQ ID NO:118, SEQ ID NO:119, SEQ ID NO:120, SEQ ID NO:121.
7. Способ по п. 1, характеризующийся тем, что для каждого из сортов семян подсолнечника последовательность ДНК маркеров определяют при помощи ДНК- микрочипов.
| ЯЧМЕНЬ С НИЗКИМ СОДЕРЖАНИЕМ ГОРДЕИНОВ | 2008 |
|
RU2518241C2 |
| НУКЛЕОТИДНЫЕ ПОСЛЕДОВАТЕЛЬНОСТИ, ПОДВЕРГНУТЫЕ МУТАЦИИ С ПОМОЩЬЮ ВСТАВКИ, КОТОРАЯ КОДИРУЕТ УКОРОЧЕННЫЙ БЕЛОК ОЛЕАТ-ДЕСАТУРАЗЫ, БЕЛКИ, СПОСОБЫ И ПРИМЕНЕНИЯ | 2011 |
|
RU2630641C2 |
| WO 2015109230 A1, 23.07.2015 | |||
| US 20060147962 A1, 06.07.2006. | |||
Авторы
Даты
2020-03-24—Публикация
2018-12-26—Подача