Изобретение относится к области биотехнологии, биоинженерии, биофармакологии, может быть использовано в качестве препарата, нейтрализующего летальный токсин возбудителя сибирской язвы, и использоваться в качестве специфической терапии, направленной против сибиреязвенной инфекции.
Сибирская язва является инфекционным заболеванием людей и животных, относящейся ко II группе патогенных биологических агентов. Возбудителем сибирской язвы является аэробная, грамположительная, спорообразующая бактерия Bacillus anthracis [Mock М., Fouet А. // Ann. Rev. Microbiol. - 2001. - Vol. 55. - №. 1. - P. 647-671]. Бактерия сибирской язвы синтезирует термолабильные белки: протективный антиген (ПА) с молекулярной массой 83 кДа и летальный фактор (ЛФ) с молекулярной массой 90 кДа. Каждый из компонентов в отдельности не обладает токсическими свойствами, а в комплексе образуют летальный токсин (ЛТ). Из-за своей иммуногенности ПА является одним из основных компонентов ЛТ. Протективный антиген состоит из четырех структурных доменов. Каждый домен выполняет свои функции. Функция I домена заключается в связывании ПА с ЛФ. Функция II домена заключается в формировании петли, которая в дальнейшем образует катионселективный канал. Домен III отвечает за олигомеризацию антигена. Функция IV домена заключается во взаимодействии с рецептором на поверхности эукариотических клеток, что является первым этапом в механизме проникновения ЛТ в эукариотическую клетку. [Liu S. et al. The receptors that mediate the direct lethality of anthrax toxin //Toxins. - 2013. - T. 5. - №. 1. - C. 1-8.].
Для получения эффективного антитоксического препарата необходимо учитывать ключевые звенья механизма действия токсина. Принимая во внимание механизмы активации сибиреязвенного токсина, становится понятным, что потенциальные антитоксические препараты должны ингибировать: 1) связывание ПА с рецептором клетки; 2) активность внеклеточного фурина, разрезающего ПА на две части массой 20 и 63 кДа; 3) образование олигомерной препоры из мономеров ПА массой 63 кДа; 4) связывание ЛФ с ПА; 5) эндоцитоз токсина; 6) превращение олигомерных ПА препор в истинные поры; 7) транслокацию из эндосом ЛФ; 8) ферментативную активность ЛФ [Turk В. Е. Manipulation of host signalling pathways by anthrax toxins //Biochemical Journal. - 2007. - T. 402. - №. 3. - C. 405-417.].
Одним из резервуаров инфекции является почва. Споры сибирской язвы могут долгие годы храниться в ней, при этом сохранить свою патогенность и жизнеспособность. Оставаясь жизнеспособными в почве долгое время, споры являются потенциальным источником инфекции, в основном для крупнорогатого скота, а человек инфицируется при уходе за больным животным, выделке шерсти, обработке мяса. В связи с этим заражение сибирской язвой возможно следующими путями: при попадании через поврежденные покровы, через слизистую желудочно-кишечного тракта, при вдыхании инфицированной пыли через дыхательные пути. В зависимости от способа попадания возбудителя сибирской язвы выделяют три формы заболевания: кожная, кишечная и легочная.
Самой распространенной формой заболевания является кожная форма. Она легко диагностируется и лечится антибиотиками, поэтому смертность невысока. Кишечная и легочная форма сложно диагностируются, а симптомы заболевания похожи на другие бактериальные инфекции [Burnett J. С.et al. The evolving field of biodefence: therapeutic developments and diagnostics //Nature Reviews Drug Discovery. - 2005. - T. 4. - №. 4. - C. 281-296.]. Применение антибиотиков на поздних стадиях заболевания увеличивает токсический эффект, так как антибиотики активны против самой бактерии, но не способны нейтрализовать ЛТ, и впоследствии больной погибает от токсического шока.
Уже доказана эффективность применения моноклональных антител (МКАт) к различным компонентам ЛТ в терапии сибиреязвенной инфекции на различных этапах заболевания. Наибольшее количество работ связано с получением антител против ПА В. anthracis. Это объясняется тем, что ЛФ без ПА не способен проявлять токсический эффект. Кроме того, именно связывание ПА с рецепторами на поверхности эукариотической клетки (ТЕМ8/ ANTXR1, CMG2/ANTXR2) является первым событием многоэтапного внутриклеточного проникновения токсина В. anthracis. Поэтому препараты, ингибирующие взаимодействие ПА с рецепторами эукариотической клетки, будут, вероятно, проявлять особенно высокую эффективность при терапии на ранних стадиях инфекции [Liu S. et al. The receptors that mediate the direct lethality of anthrax toxin //Toxins. - 2013. - T. 5. - №. 1. - C. 1-8.].
Известно моноклональное антитело Obiltoxaximab (Anthim®, ETI-204) (US 8093360 B2), утвержденное в 2016 г. FDA для профилактики и лечения ингаляционной сибирской язвы. Obiltoxaximab имеет молекулярную массу 145,5 кДа и представляет собой химерное моноклональное антитело IgG1 с легкой цепью к. Obiltoxaximab связывает ПА-компонент токсина В. anthracis [Greig S. L. Obiltoxaximab: first global approval //Drugs. - 2016. - T. 76. - №. 7. - C. 823-830.].
Большинство коммерчески доступных рекомбинантных терапевтических моноклональных антител производится в клетках млекопитающих, в 70% случаев с использованием клеток яичника китайского хомяка (СНО) [Kim J. Y., Kim Y. G., Lee G. M. CHO cells in biotechnology for production of recombinant proteins: current state and further potential //Applied microbiology and biotechnology. - 2012. - T. 93. - №. 3. - C. 917-930.]. Поскольку иммуноглобулины - гетеродимерные полипептидные молекулы, и в лимфоцитах цепи белка транскрибируются с двух разных хромосом, в клетке продуцента транскрибируются либо две отдельные мРНК, либо одна мРНК, на которой происходит синтез двух независимых белков, что достигается внедрением сайта внутренней посадки рибосомы IRES между кодирующими последовательностями LC и НС (или НС и LC). Однако коэкспрессия двух цепей происходит зачастую не сбалансировано. В качестве альтернативы предложено использование так называемых «саморасщепляющихся» вирусных пептидов 2А между цепями иммуноглобулина для их эквимолярной экспрессии в каждой клетке, содержащей экспрессионную кассету [Fang J. et al. Stable antibody expression at therapeutic levels using the 2A peptide //Nature biotechnology. - 2005. - T. 23. - №. 5. - C. 584-590.]. Рибосома, встречая в процессе синтеза белка такие последовательности, делает «проскок» и пептидная связь между предпоследней аминокислотой глицином и последней аминокислотой пролином не образуется. Согласно последним исследованиям, наиболее удачной аминокислотной последовательностью между цепями для экспрессии терапевтических иммуноглобулинов в клетках СНО является RAKRGSGEGRGSLLTCGDVEENPGP [Chng J. et al. Cleavage efficient 2A peptides for high level monoclonal antibody expression in CHO cells //MAbs. - Taylor & Francis, 2015. - T. 7. - №. 2. - C. 403-412.], [Luke G. A., Ryan M. D. Therapeutic applications of the 'NPGP'family of viral 2As //Reviews in medical virology. - 2018. - T. 28. - №. 6. - С.e2001.] Последовательность RAKR является сайтом узнавания протеазой фурином. Пептид EGRGSLLTCGDVEENPGPA является одним из представителей семейства 2А и носит название Т2А, поскольку присущ вирусу Thosea asigna. Короткий глицин-сериновый линкер GSG между сайтом фурина и Т2А помогает эффективнее разделять цепи иммуноглобулина в составе полипротеина-предшественника. В результате на С-конце цепи, стоящей выше по ходу трансляции от последовательности фурин-080-Т2А, остаются только аминокислоты RAK. Нижняя цепь остается нативной.
Оценка токсиннейтрализующей активности МКАт in vitro с использованием перевиваемых клеточных линий J774 АЛ или RAW 264.7, которые представляют собой макрофаги моноцитарного происхождения Mus musculus, - быстрый и дешевый метод, который может быть эффективно использован для проведения скрининговых исследований. Клетки макрофаги являются мишенью для летального токсина возбудителя сибирской язвы [Friedlander А. М. Macrophages are sensitive to anthrax lethal toxin through an acid-dependent process //Journal of Biological Chemistry. - 1986. - T. 261. - №. 16. - C. 7123-7126.]. Однако этот метод не отражает полной картины, происходящей в организме при введении МКАт. В ряде работ было показано, что данные о токсиннейтрализующей активности МКАт, полученные в тестах in vitro, могут не коррелировать с результатами, полученными в экспериментах in vivo [Staats, H.F. In Vitro and In Vivo Characterization of Anthrax Anti-Protective Antigen and Anti-Lethal Factor Monoclonal Antibodies after Passive Transfer in a Mouse Lethal Toxin Challenge Model To Define Correlates of Immunity / H.F. Staats, S.M. Alam, R.M. Scearce, S.M. Kirwan, J.X. Zhang, W.M. Gwinn, B.F. Haynes // Infection and immunity. - 2007. - N. 11. - P. 5443-5452.], [Whiting, G. Development of an in Vitro Potency Assay for Anti-anthrax Lethal Toxin Neutralizing Antibodies / G. Whiting, M. Baker, S. Rijpkema // Toxins. -2012. - N. 4. - P. 28-41.]. В качестве моделей для оценки токсиннейтрализующей активности МКАт могут использоваться мыши, крысы, морские свинки и приматы. Наиболее доступной и дешевой из перечисленных биомоделей является мышиная. Линия мышей BALB/c устойчива к заражению спорами В. anthracis, но прекрасно подходит в качестве модели для оценки токсиннейтрализующей активности МКАт [Moayeri, М. Mouse Susceptibility to Anthrax Lethal Toxin Is Influenced by Genetic Factors in Addition to Those Controlling Macrophage Sensitivity / M. Moayeri, N.W. Martinez, J. Wiggins, H.A. Young, S.H. Leppla // Infect hnmun. - 2004. - Vol.72. N. 8. - P. 4439-4447.]. Применение мышиной модели позволяет проводить оценку фармакокинетики МКАт в организме, подобрать эффективные дозы МКАт, наблюдать за клинической картиной после введения МКАт и ЛТ. Введение МКАт за 24 часа до введения ЛТ дает время для распространения антител во всех тканях хозяина.
Технический результат заключается в получении двух моноклональных антител с одинаковыми вариабельными доменами: мышиного и рекомбинантного, в частности химерного (мышь-человек), - которые способны к высокоаффинному и высокоспецифичному связыванию с протективным антигеном Bacillus anthracis и обладают нейтрализующим действием в отношении летального токсина.
Технический результат достигается тем, что предложен штамм мышиной гибридной клеточной линии Mus musculus 1Е10 - продуцент моноклонального антитела 1Е10, полученной в результате гибридизации мышиной миеломной линии Sp2/0-Ag14 со спленоцитами мыши линии BALB/c, гипериммунной в отношении белка рекомбинантного протективного антигена Bacillus anthracis. Штамм гибридных культивируемых клеток животных Mus musculus (гибридомы), являющийся продуцентом моноклонального антитела 1Е10, депонирован в «ГКПМ-Оболенск» ФБУН ГНЦ «Государственный научный центр прикладной микробиологии и биотехнологии» 02.09.2019 г. под № Н-89.
Технический результат также достигается тем, что предложено мышиное моноклональное антитело 1Е10 к протективному антигену Bacillus anthracis и нейтрализующее летальный токсин Bacillus anthracis, представленное двумя идентичными тяжелыми и двумя идентичными легкими цепями, с известными аминокислотными последовательностями легкой цепи SEQ Ш NO:l и Fd фрагмента тяжелой цепи SEQ ID NO: 2.
Технический результат также достигается тем, что предложено химерное моноклональное антитело xi1E10 к протективному антигену Bacillus anthracis, нейтрализующее летальный токсин Bacillus anthracis, синтезируемое в эукариотических клеточных линиях штаммом клеток ExpiCHO-S, представленное двумя идентичными тяжелыми и двумя идентичными легкими цепями, с аминокислотной последовательностью легкой цепи SEQ ID NO: 3 и с аминокислотной последовательностью тяжелой цепи SEQ ID NO:4, полученное путем замены константных доменов легкой цепи и тяжелой цепи мышиного антитела 1Е10 на аналогичные области иммуноглобулина G человека.
Изобретение иллюстрируют следующие графические материалы:
Фиг. 1. Графическое представление результата выделения и очистки МКАт 1Е10 из асцитической жидкости после электрофоретического разделения в 10%-ом ПААГ. Окраска геля кумасси бриллиантовым синим R-250.
Дорожка 1 - маркер молекулярной массы белков Spectra Multicolor Broad Range Protein Ladder (Thermo Fisher). Дорожка 2 - образец очищенного МКАт 1Е10 (нередуцирующие условия). Дорожка 3 - образец очищенного МКАт 1Е10 с добавлением 2-меркаптоэтанола (редуцирующие условия).
Фиг. 2. Результат электрофоретического разделения рекомбинантных белков, связывающихся с МКАт 1E10 и xi1E10. Окраска геля кумасси бриллиантовым синим R-250.
Дорожка 1 - маркер молекулярной массы белков Spectra Multicolor Broad Range Protein Ladder (Thermo Fisher). Дорожка 2 - рекомбинантный белок ПА. Дорожка 3 -рекомбинантный белок IV домен ПА.
Фиг. 3. Данные иммуноблота по связыванию МКАт 1Е10 и xi1E10 с рекомбинантными белками ПА и IV домен ПА.
Дорожка 1 - связывание МКАт 1Е10 с рекомбинантным ПА. Дорожка 2 -связывание МКАт 1Е10 с рекомбинантным IV доменом ПА. Дорожка 3 - маркер молекулярной массы белков Spectra Multicolor Broad Range Protein Ladder (Thermo Fisher). Дорожка 4 - связывание МКАт xi1E10 с рекомбинантным ПА. Дорожка 5 -связывание МКАт xi1E10 с рекомбинантным IV доменом ПА.
Фиг. 4. Результат определения подкласса и изотипа легкой цепи очищенных МКАт 1Е10 с помощью иммунохроматографического экспресс-теста IsoQuick™ Strips for Mouse Monoclonal Isotyping (Envirologix, США).
Фиг. 5. Кривые зависимости абсорбции при длине волны 492 нм от концентрации антител 1E10/xi1E10 в ИФА. Легенда на рисунке.
Фиг. 6. Праймеры для сборки химерной легкой и тяжелой цепи МКАт (1Е10) с кратким описанием.
Фиг. 7. Графическое представление результата амплификации ДНК, кодирующих различные участки МКАт после электрофоретического разделения в 1,5% агарозном геле. Дорожка 1 - легкая цепь 1Е10. Дорожка 2 - тяжелая цепь 1Е10 (Fd фрагмент). Дорожка 3 - VL домен IE 10. Дорожка 4 - VH домен 1Е10. Дорожка 5 - легкая цепь xi1E10. Дорожка 6 - тяжелая цепь xi1E10 (полностью). Дорожка 7 -. Дорожка 8 -Маркер молекулярной массы ДНК GeneRuler DNA Ladder Mix (Thermo Fisher).
Фиг 8. Графическое представление результата выделения и очистки МКАт xi1E10 из культуральной жидкости после электрофоретического разделения в 10%-ом ПААГ. Окраска геля кумасси бриллиантовым синим R-250.
Дорожка 1 - маркер молекулярной массы белков Spectra Multicolor Broad Range Protein Ladder (Thermo Fisher). Дорожка 2 - образец очищенного МКАт xi1E10 (нередуцирующие условия). Дорожка 3 - образец очищенного МКАт xi1E10 с добавлением 2-меркаптоэтанола (редуцирующие условия).
Фиг. 9. Данные иммуноблота, отражающего взаимодействие моноклонального антитела xi1E10 с антителами против его константных областей.
Дорожка 1 - образец очищенного МКАт xi1E10 после взаимодействия с антителами против к цепи человеческого иммуноглобулина. Дорожка 2 - маркер молекулярной массы белков Spectra Multicolor Broad Range Protein Ladder (Thermo Fisher). Дорожка 3 - образец очищенного МКАт xi1E10 после взаимодействия с антителами против Fc фрагмента человеческого иммуноглобулина G.
Фиг. 10. Термофоретические кривые во всех капиллярах для МКАт 1Е10 и МКАт xi1E10 при взаимодействии с IV доменом ПА.
Фиг. 11. Графики аффинного взаимодействия МКАт 1Е10 и МКАт xi1E10 с белком-мишенью IV доменом ПА.
Фиг. 12. Определение токсиннейтрализующей активности МКАт in vitro в тесте МТТ.
Фиг. 13. Определение токсиннейтрализующей активности МКАт 1Е10 in vivo на модели мышей.
Фиг. 14. Определение токсиннейтрализующей активности МКАт xi1E10 in vivo на модели мышей.
Фиг. 15. Выживаемость мышей в контрольных группах в эксперименте по нейтрализации ЛТ моноклональными антителами in vivo.
Изобретение осуществляют следующим образом.
Мышей линии BALB/c иммунизируют четырехкратно введением рекомбинантного белка ПА (рПА, Seq ID NO: 5) в дозе 100 мкг/мышь с периодичностью в 2 недели в течение 2 месяцев. На третьи сутки после последней иммунизации проводится гибридизация спленоцитов иммунных мышей с партнером для слияния - клетками мышиной миеломной линии Sp2/0-Ag14. Гибридизацию производят по методу Келера и Мильштейна [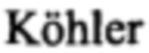 G., Milstein С.Continuous cultures of fused cells secreting antibody of predefined specificity //nature. - 1975. - T. 256. - №. 5517. - C. 495-497.] с модификациями. После гибридизации проводят селекцию, скрининг, клонирование и криоконсервацию гибридных клеток. Полученный моноклональный штамм гибридомы депонируют в коллекцию «ГКПМ-Оболенск» при ФБУН ГНЦ «Государственный научный центр прикладной микробиологии и биотехнологии» под названием «штамм гибридных культивируемых клеток животных Mus musculus 1Е10». Для получения препаративных количеств МКАт 1Е10 гибридомные клетки штамма 1Е10 нарабатывают в асцитной жидкости мышей с последующим выделением из нее иммуноглобулиновой фракции посредством аффинной хроматографии. Готовый белковый продукт валидируют электрофорезом по Лэммли по типовому распределению иммуноглобулина G в полиакриламидном геле до и после воздействия редуцирующего агента в сравнении с маркером молекулярной массы: без восстановления дисульфидных связей молекулярной вес белка представлен монопродуктом с массой 150 кДа, а редуцированная молекула отображается в виде двух полос с массами 25 и 50 кДа (Фиг. 1). Подкласс антитела и изотип легкой цепи определяют в иммунохроматографическом тесте (Фиг. 4). Для создания химерного антитела, именуемого xi1E10, (константная часть от иммуноглобулина IgG1 человека, вариабельная часть от мышинокого МКАт 1Е10) выделают фракцию тотальной РНК штамма гибридных клеток 1Е10, проводят реакцию обратной транскрипции, затем амплифицируют методом 5'-РАСЕ кДНК, кодирующие Fab-фрагмент, по методике (с модификациями), описанной в [Bradbury A. Cloning Hybridoma cDNA by RACE //Antibody Engineering. - Springer, Berlin, Heidelberg, 2010. - C. 15-20.]. Продукты ПЦР реакции (Фиг. 7, дорожки 1 и 2) клонируют в вектор pUC19 и секвенирют. По результатам секвенирования определяют границы вариабельной части и осуществляют «химеризацию» антитела путем аплификации участков VL и VH (Фиг. 7, дорожки 3 и 4) с последующим клонированием ПЦР продуктов в коммерческие вектора CL и CH доменами человеческого IgG1. Получив таким образом гены полноразмерных цепей (Фиг. 7, дорожки 5 и 6) в двух отдельных плазмидах, далее собирают рекомбинантную ДНК, кодирующую молекулу химерного иммуноглобулина путем соединения генов двух цепей с помощью OE-PCR. В итоге получают молекулу ДНК (Фиг. 7, дорожка 7; Seq ID NO: 26) с кодонами следующих полипептидных участков (сверху вниз по ходу транскрипции): тяжелая цепь МКАт xi1E10, сайт узнавания протеазой фурином RAKR, глицин-сериновый линкер GSG, Т2А пептид, легкая цепь МКАт xi1E10 (Seq ID NO: 27). Полученный ПЦР продукт клонируют в плазмидный вектор pcDNA 3.4 для экспрессии в эукариотических клетках и именуют его pcDNA3.4_xi1E10. Данный вектор трансфецируют в клетки яичника китайского хомячка СНО и получают продуцента моноклонального антитела xi1E10. Клетки-продуценты культивируют в бессывороточной среде в колбах Эрленмейера, затем выделяют МКАт из культуральной жидкости с применением концентрирования и аффинной хроматографии. Белковый продукт валидируют электрофоретически подобно очищенному из асцитической жидкости антителу 1Е10 (Фиг. 8), а также показывают взаимодействие белка с антителами к константным человеческим регионам в иммуноблоте (Фиг. 9), что отражает химерное строение рекомбинантного иммуноглобулина. С очищенными моноклональными антителами 1Е10 и xi1E10 проводят ряд анализов: оценку специфичности связывания с антигеном в иммуноблоте (Фиг. 3), оценку количественного взаимодействия с антигеном в ИФА (Фиг. 6), определение равновесной константы диссоциации в методе, основанном на явлении микроскопического термофореза (Фиг. 10 и 11), определение токсиннейтрализующей активности in vitro на клеточной линии J774A.1 в микротесте МТТ (Фиг. 12) и in vivo на модели мышей BALB/c, используя рекомбинантный летальный токсин В. anthracis и получая в результате графики выживаемости животных (Фиг. 13, 14 и 15). Устанавливают, что предложенные антитела в равной степени высокоаффинно связываются с антигеном ПА и обладает токсиннейтрализующей активностью. Другие характерные признаки антител, установленные в процессе их создания, кратко перечислены ниже.
G., Milstein С.Continuous cultures of fused cells secreting antibody of predefined specificity //nature. - 1975. - T. 256. - №. 5517. - C. 495-497.] с модификациями. После гибридизации проводят селекцию, скрининг, клонирование и криоконсервацию гибридных клеток. Полученный моноклональный штамм гибридомы депонируют в коллекцию «ГКПМ-Оболенск» при ФБУН ГНЦ «Государственный научный центр прикладной микробиологии и биотехнологии» под названием «штамм гибридных культивируемых клеток животных Mus musculus 1Е10». Для получения препаративных количеств МКАт 1Е10 гибридомные клетки штамма 1Е10 нарабатывают в асцитной жидкости мышей с последующим выделением из нее иммуноглобулиновой фракции посредством аффинной хроматографии. Готовый белковый продукт валидируют электрофорезом по Лэммли по типовому распределению иммуноглобулина G в полиакриламидном геле до и после воздействия редуцирующего агента в сравнении с маркером молекулярной массы: без восстановления дисульфидных связей молекулярной вес белка представлен монопродуктом с массой 150 кДа, а редуцированная молекула отображается в виде двух полос с массами 25 и 50 кДа (Фиг. 1). Подкласс антитела и изотип легкой цепи определяют в иммунохроматографическом тесте (Фиг. 4). Для создания химерного антитела, именуемого xi1E10, (константная часть от иммуноглобулина IgG1 человека, вариабельная часть от мышинокого МКАт 1Е10) выделают фракцию тотальной РНК штамма гибридных клеток 1Е10, проводят реакцию обратной транскрипции, затем амплифицируют методом 5'-РАСЕ кДНК, кодирующие Fab-фрагмент, по методике (с модификациями), описанной в [Bradbury A. Cloning Hybridoma cDNA by RACE //Antibody Engineering. - Springer, Berlin, Heidelberg, 2010. - C. 15-20.]. Продукты ПЦР реакции (Фиг. 7, дорожки 1 и 2) клонируют в вектор pUC19 и секвенирют. По результатам секвенирования определяют границы вариабельной части и осуществляют «химеризацию» антитела путем аплификации участков VL и VH (Фиг. 7, дорожки 3 и 4) с последующим клонированием ПЦР продуктов в коммерческие вектора CL и CH доменами человеческого IgG1. Получив таким образом гены полноразмерных цепей (Фиг. 7, дорожки 5 и 6) в двух отдельных плазмидах, далее собирают рекомбинантную ДНК, кодирующую молекулу химерного иммуноглобулина путем соединения генов двух цепей с помощью OE-PCR. В итоге получают молекулу ДНК (Фиг. 7, дорожка 7; Seq ID NO: 26) с кодонами следующих полипептидных участков (сверху вниз по ходу транскрипции): тяжелая цепь МКАт xi1E10, сайт узнавания протеазой фурином RAKR, глицин-сериновый линкер GSG, Т2А пептид, легкая цепь МКАт xi1E10 (Seq ID NO: 27). Полученный ПЦР продукт клонируют в плазмидный вектор pcDNA 3.4 для экспрессии в эукариотических клетках и именуют его pcDNA3.4_xi1E10. Данный вектор трансфецируют в клетки яичника китайского хомячка СНО и получают продуцента моноклонального антитела xi1E10. Клетки-продуценты культивируют в бессывороточной среде в колбах Эрленмейера, затем выделяют МКАт из культуральной жидкости с применением концентрирования и аффинной хроматографии. Белковый продукт валидируют электрофоретически подобно очищенному из асцитической жидкости антителу 1Е10 (Фиг. 8), а также показывают взаимодействие белка с антителами к константным человеческим регионам в иммуноблоте (Фиг. 9), что отражает химерное строение рекомбинантного иммуноглобулина. С очищенными моноклональными антителами 1Е10 и xi1E10 проводят ряд анализов: оценку специфичности связывания с антигеном в иммуноблоте (Фиг. 3), оценку количественного взаимодействия с антигеном в ИФА (Фиг. 6), определение равновесной константы диссоциации в методе, основанном на явлении микроскопического термофореза (Фиг. 10 и 11), определение токсиннейтрализующей активности in vitro на клеточной линии J774A.1 в микротесте МТТ (Фиг. 12) и in vivo на модели мышей BALB/c, используя рекомбинантный летальный токсин В. anthracis и получая в результате графики выживаемости животных (Фиг. 13, 14 и 15). Устанавливают, что предложенные антитела в равной степени высокоаффинно связываются с антигеном ПА и обладает токсиннейтрализующей активностью. Другие характерные признаки антител, установленные в процессе их создания, кратко перечислены ниже.
Мышиное МКАт 1Е10:
A) Предоставлено иммуноглобулином IgG1 с легкой цепью к;
Б) Эпитоп локализован в IV домене протективного антигена Bacillus anthracis
B) Kd=9,1564×10-10 М., молекулярный вес = 145 кДа;
Г) Сигнальная последовательность и последовательность легкой цепи Seq ID NO: 1;
Д) Сигнальная последовательность и последовательность Fd-фрагмента тяжелой цепи (вариабельная область и СН1 домен) Seq ID NO: 2;
Е) последовательность вариабельной области легкой цепи Seq ID NO: 9;
Ж) последовательность вариабельной области тяжелой цепи Seq ID NO: 11.
Химерное (мыши-человек) МКАт xi1E10:
A) Предоставлено иммуноглобулином IgG1 с легкой цепью κ;
Б) Эпитоп локализован в IV домене протективного антигена Bacillus anthracis;
B) Kd = 1,2663×10-9 М, молекулярный вес = 147 кДа;
Г) Сигнальная последовательность и последовательность легкой цепи Seq ID NO: 3
Д) Сигнальная последовательность и последовательность тяжелой цепи Seq ID NO: 4;
Для лучшего понимания сущности изобретения ниже следуют примеры его конкретного выполнения.
Пример 1. Получение и культивирование гибридной клеточной линии.
Иммунизация лабораторных животных.
Мышей линии BALB/c весом 18-20 г иммунизируют рекомбинантным белком ПА Bacillus anthracis по следующей схеме.
1) Вводят рекомбинантный белок ПА с концентрацией 1 мг/мл в дозе 100 мкг/мышь с полным адъювантом Фрейнда в объемном соотношении 1:1 подкожно.
2) Через 2 недели вводят рекомбинантный белок ПА с концентрацией 1 мг/мл в дозе 100 мкг/мышь с неполным адъювантом Фрейнда в объемном соотношении 1:1 подкожно.
3) Через 2 недели вводят рекомбинантный белок ПА в дозе 10 мкг/мышь внутрибрюшинно.
Кровь иммунизированных мышей отбирают на третий день после финальной инъекции. Титры специфических антител в полученных сыворотках крови определяют с помощью непрямого твердофазного ИФА.
Гибридизация. Через 3 суток после финальной иммунизации мышей, чьи сыворотки крови показали наилучшие результаты при тестировании в ИФА, умерщвляют методом дислокации шейных позвонков, извлекают селезенки, и проводят процедуру слияния (гибридизации) 1×108 спленоцитов гипериммунной мыши с 1×107 клеток миеломной мышиной линии Sp2/0-Ag14 (АТСС® CRL-1581™). В качестве агента для слияния применяют раствор полиэтиленгликоля в диметилсульфоксиде (Sigma Р7306, США). Слившиеся клетки рассевают в лунки культуральных планшетов с заранее подготовленным фидерным слоем, состоящим из мышиных перитонеальных макрофагов в количестве 1×104 клеток на лунку. Культивирование полученных гибридных клеток осуществляют в СО2-инкубаторе Forma Serias II (Thermo Fisher, США) при температуре 37°С во влажной атмосфере, содержащей 5% СО2, в среде DMEM с 20% эмбриональной телячьей сывороткой, 2 мМ L-глутамина, 100 мкг/мл гентамицина, однократным раствором гипоксантина-аминоптерина-тимидина (HAT) (Gibco 21060-017, Thermo Fisher, США).
Селекция. Селекцию полученных гибридных клеток проводят с использованием однократного раствора гипоксантина-аминоптерина-тимидина (HAT) в течение 3 недель.
Скрининг гибридных клеток. Отбор положительных гибридных клеток, продуцирующих специфичные антитела, осуществляют методом непрямого твердофазного ИФА. Для проведения непрямого варианта ИФА в лунки 96-луночного полистиролового планшета для иммуноферментного анализа вносят по 1 мкг/лунку антигена ПА В. anthracis в фосфатно-солевом буфере (ФСБ) (137 мМ NaCl, 2,7 мМ KCl, 10 мМ Na2HPO4, 1,76 мМ KH2PO4, рН 7.4) и инкубируют при температуре 37°С в течение 1 часа на планшетном орбитальной шейкере (Elmi, Латвия) при скорости вращения платформы 300 об./мин. Трижды отмывают фосфатно-солевым буфером, содержащим 0,5% детергента Tween® 20 (ФСБ-Тв) по 200 мкл. Затем свободные валентности пластика блокируют молоком с массовой долей жира не более 0,5%, внося по 200 мкл на лунку, и инкубируют при температуре 37°С в течение 1 часа при тех же условиях. Трижды отмывают ФСБ-Тв. Подготовленные таким образом планшеты также используют в примерах: 2 и 8. Далее вносят в лунки супернатант культуральной жидкости, в которой выращивали гибридные клетки, в объеме 100 мкл, инкубируют при температуре 37°С в течение 1 часа на шейкере. После этого лунки планшета трижды отмывают ФСБ-Тв, и добавляют в лунки раствор кроличьих антител к целой молекуле IgG мыши, конъюгированных с пероксидазой хрена (Sigma-Aldrich, США), в рабочем разведении 1:10000. Планшет инкубируют при температуре 37°С в течение 1 часа на шейкере, затем 6 раз отмывают буфером ФСБ-Тв. После этого в лунки вносят по 100 мкл субстрат-индикаторного раствора (10 мкл 30% Н2О2 и 8 мг ортофенилендиамина на 10 мл фосфатно - цитратного буфера рН 5.0). Положительную реакцию оценивают по появлению желто-коричневого окрашивания раствора. Реакцию останавливают добавлением в лунки по 50 мкл 4N серной кислоты. Детекцию проводят на планшетном спектрофотометре (Bio-Rad xMark, США) при длине волны 492 нм.
Клонирование. Клонирование гибридных клеток проводят методом лимитирующих разведений в 96-луночных планшетах на фидерном слое, состоящем из перитонеальных макрофагов в количестве 1×104 клеток на лунку. Скрининг клонов проводят методом непрямого твердофазного ИФА, как было описано выше. Скринируют только те лунки, которые содержат моноклональную популяцию клеток. Клоны, показавшие наилучшие результаты в ИФА, тестируют не менее трех раз с интервалом 3-4 дня. Наиболее стабильные клоны гибридных клеток отбирают для дальнейшей работы. Наилучшие и стабильные результаты в ИФА показал клон гибридной клеточной линии 1Е10.
Культивирование. Культивирование клона гибридной клеточной линии 1Е10 ведут в культуральных флаконах Corning® Т-25 и Т-75 при температуре 37°С в атмосфере, содержащей 5% углекислого газа. Средой культивирования является DMEM, содержащая 20% эмбриональной телячьей сыворотки, 2 мМ L-глутамина, 100 мкг/мл гентамицина. Количество клеток при субкультивировании поддерживают в диапазоне между 1×105-1×106 на 1 мл культуральной среды. Обновление среды путем частичной или полной замены производят каждые 2-3 дня.
Выявление контаминации гибридной клеточной линии клона 1Е10. Контаминацию культуры гибридных клеток 1Е10 оценивают визуально и с помощью набора флюоресцентных красителей для определения бактериальной и грибковой контаминации клеточных культур Cell Culture Contamination Detection Kit C-7028 (Thermo Fisher, США) и флюоресцентного красителя для ДНК, подходящего для определения контаминации микоплазмами Hoechst 33258 (Sigma-Aldrich, США). Всю пробоподготовку клеток гибридной линии и культуральной жидкости проводят согласно рекомендациям производителей. Подготовленные образцы рассматривают с помощью инвертированного флуоресцентного микроскопа CKX41 с ртутным осветителем U-RFLT50 (все Olympus, Япония).
Криоконсервация. Замораживание клонов гибридных клеток 1Е10 производят в специальных криопробирках (виалах) объемом 2 мл. В качестве среды для криоконсервации используем фетальную телячью сыворотку с добавлением 10% диметилсульфоксида. Снятие клеток с поверхностей культуральных флаконов осуществляют потряхиванием и пипетированием. Суспензию клеток центрифугируют при 125×g в течение 5 минут, удаляем супернатант и ресуспендируем в 1 мл среды для криоконсервации. Криопробирки охлаждаем до -80°С со скоростью заморозки 1°С/мин. Хранение замороженных культур осуществляем в жидком азоте. При разморозке виал нагревают на водяной бане при 40°С до растапливания льда, содержимое переносят в пробирку со средой DMEM, центрифугируют 5 минут при 200×g. После чего супернатант удаляют, а клеточный осадок высевают и продолжают культивирование.
Культивирование в организме животного. Мышам линии BALB/c в возрасте 18-20 недель внутрибрюшинно вводят по 0,5 мл пристана (Sigma, США), а через 2-3 недели вводят внутрибрюшинно гибридные клетки 1Е10 в пределах от 5×105 до 2×106 в среде DMEM без фетальной телячьей сыворотки. Иммуноасциты появляются на 7-15 сутки, а отбор асцитической жидкости производят на 10-21 сутки в соответствии со скоростью созревания каждого отдельного асцита. Клеточные компоненты асцитической жидкости удаляют центрифугированием (1000 × g 15 мин), преципитируют белковую фракцию асцитической жидкости добавлением насыщенного раствора (NH4)2SO4 в соотношении 1:1, выдерживают при температуре 4°С не менее 12 часов.
Пример 2. Очистка и валидация МКАт 1Е10
Выделение МКАт 1Е10 из асцитической жидкости проводят методом аффинной хроматографии на колонке с сорбентом Protein G сефарозой (Protein G Sepharose 4 FastFlow, GE Healthcare, Великобритания). Для очисти МКАт 1E10 используют хроматографическую систему  Start (GE Healthcare, Швеция).
Start (GE Healthcare, Швеция).
Очистка. Преципитат антител сульфатом аммония осаждают центрифугированием при 10000 х g в течение 20 минут, удаляют супернатант. Осадок растворяют в 50 мл буфера нанесения 50 мМ Tris, 100 мМ NaCl, 0,02% NaN3, рН 8,0. Колонку с полезной емкостью 3 мл, заполненную Protein G сефарозой, уравновешивают буфером нанесения. Производят нанесение образца со скоростью 2 мл/мин на льду. Далее колонку отмывают от не связавшихся компонентов буфером нанесения, и проводят элюцию иммуноглобулинов буфером состава: 100 мМ глицина, 100 мМ NaCl, рН 2.4. Элюат собирают после начала экспоненциального роста величины абсорбции при 280 нм (А280) и заканчивают, когда А280 начинает снижаться по логарифмическому закону (выходить на плато). Уровень рН элюата доводят до 7,5 - 8,0 добавлением 1 М раствора Tris основания. Впоследствии элюат иммуноглобулиновой фракции вносят в коммерчески доступную колонку HiPrep™ 26/10 Desalting (GE Healthcare) емкостью 53 мл, уравновешенную фосфатно-солевым буфером ФСБ. на скорости потока 10 мл/мин. Иммуноглобулиновую фракцию собирают через некоторое время, идентифицируя ее по изменению А280. Для измерения концентрации МКАт проводят спектрофотометрию при А280, затем для расчета концентрации [мг/мл] полученное значение разделяют на коэффициент массовой экстинкции для IgG, равный 1,37. Чистоту полученной иммуноглобулиновой фракции оценивают методом SDS-электрофореза по Лэммли в полиакриламидном геле (ПААГ) в редуцирующих и нередуцирующих условиях (Фиг. 1).
Оценку иммунологической специфичности очищенного МКАт 1Е10 проводят методом иммуноблота (Вестерн-блоттинга) с рекомбинантным ПАВ. anthracis и рекомбинантным IV доменом ПА (Seq ID NO: 7). Элеткрофоретическое разделение антигенов изображено на Фиг. 2. Рекомбинантные белки подвергают электрофорезу в 12% полиакриламидном геле, после чего осуществляют горизонтальный перенос белков из геля на нитроцеллюлозную мембрану Hybond-C Extra (GE Healthcare, Великобритания) с помощью оборудования Trans-Blot® Turbo™ Transfer System (Bio-Rad, США). После завершения переноса мембрану блокируют погружением в обезжиренное молоко с массовой долей жира не более 0,5% и инкубируют в течение 1
4 на орбитальном термостатируемом шейкере при 300 об./мин и температуре 37°С. Далее, после трехкратной промывки буфером ФСБ-Тв, мембрану инкубируют с МКАт 1Е10, разведенным до концентрации 0,1-1 мкг/мл в ФСБ. После трехкратной промывки буфером ФСБ-Тв мембрану инкубируют с антителами к суммарной фракции IgG мыши, конъюгированными с пероксидазой хрена (А9044, Sigma, США) в разведении 1:10000 в буфере ФСБ-Тв в течение 30 минут. Затем мембрану промывают
5 раз в ФСБ-Тв. Визуализацию реакции проводят раствором субстратной смеси на основе диаминобензидина (0,05% диаминобензидина (Sigma, США), 0,015% H2O2 в ФСБ рН 7,4). Реакцию останавливают после отчетливо различимого окрашивания образцов путем погружения мембраны в дистиллированную воду. Как показывает проведенный анализ, результаты которого отражены на Фиг. 3 (дорожки 1 и 2), моноклональное антитело 1Е10 специфически взаимодействует с рекомбинантными белками ПА и IV доменом ПА.
Для оценки количественного взаимодействия очищенного МКАт 1Е10 с белком ПА, в лунки 96-луночного планшета с иммобилизированым антигеном, который подготовлен по методике из Примера 1, вносят методом двукратных разведений антитело в концентрациях 1000, 500, 250, 125, 62,5, 31,25, 15,6, 7,8, 3,9, 2, 1 нг/мл. Инкубируют на термостатируемом шейкере при температуре 37°С в течение 1 часа 300 об./мин. После этого лунки планшета трижды отмывают ФСБ-Тв и добавляют в лунки раствор кроличьих антител к целой молекуле IgG мыши, конъюгированных с пероксидазой хрена в рабочем разведении 1:10 000. Планшет инкубируют при температуре 37°С в течение 1 часа на шейкере, затем 6 раз отмывают буфером ФСБ-Тв. После этого в лунки вносят по 100 мкл субстрат-индикаторного раствора на основе ортофенилендиамина. Положительную реакцию оценивают по появлению желто-коричневого окрашивания раствора. Реакцию останавливают добавлением в лунки по 50 мкл 4N серной кислоты. Детекцию проводят на планшетном спектрофотометре (Bio-Rad xMark, США) при длине волны 492 нм. По результатам данного анализа, отраженного на Фиг. 6 (кривая с квадратными символами), минимально детектируемая концентрация МКАт 1Е10 составила 2 нг/мл, кривая А = f(C) соответствует типовой кривой для восокоаффинных антител в непрямом тведофазном ИФА.
Пример 3. Определение подкласса очищенных моноклональных антител 1Е10.
Для определения используют иммунохроматографический экспресс-тест IsoQuick™ Strips for Mouse Monoclonal Isotyping (Envirologix, США). Перед использованием экспресс-теста очищенные моноклональные антитела разводят до концентрации 0,1-1 мкг/мл в 200 мкл буфера ФСБ в микроцентрифужной пробирке объемом 1,5 мл. После этого в пробирку погружают тест-полоску и оставляют до проявки контрольной полосы.
По результатам иммунохроматографического изотипирования МКАт 1Е10 относятся к мышиным иммуноглобулинам G, подкласс IgG1, изотип легкой цепи к. Результаты отображены на Фиг. 4.
Пример 4. Установление первичной последовательности Fab фрагмента МКАт 1Е10.
Для установления генетической и первичной аминокислотной последовательности участков легкой и тяжелой цепи (LC и Fd фрагмента) МКАт 1Е10 используют методику 5'RACE-метода амплификации кДНК. На первом этапе выделяют тотальную РНК из 2×106-4×106 клеток гибридомы 1Е10 с помощью набора QIAamp RNA Blood Mini Kit (QIAGEN) согласно методике производителя для суспензионной культуры клеток, с включением в протокол этапа расщепления остаточной ДНК непосредственно на колонке с использованием набора RNase-Free DNase Set (QIAGEN). Далее проводят две независимые реакции обратной транскрипции, в каждой реакции задействуют по 1 мкг полученной РНК и реактивы набора RevertAid First Strand cDNA Synthesis Kit (Thermo Fisher). Для синтеза первой цепи кДНК LC иммуноглобулина 1Е10 используют праймер Oligo(dT)18, гибридизующийся с поли(А)-хвостом мРНК. Синтез первой цепи кДНК Fd фрагмента антитела 1Е10 осуществляют с помощью ген-специфического праймера RACEMOG1 (TAT GCA AGG СТТ АСА АСС АСА), комплементарного области мРНК, кодирующей «шарнирный» участок молекулы иммуноглобулина.
Следующим этапом к 3'-концу полученных кДНК присоединяют поли(А)-хвост ферментом концевой дезоксинуклеотидилтрансферазой (TdT). На одну реакцию объемом 10 мкл используют 1 мкл продукта реакции обратной транскрипции, 2 мМ дАТФ и 5 ед. TdT (Thermo Fisher). Смесь инкубируют в течение 5 минут, после чего фермент подвергают термической инактивации: 10 минут при 70°С.
Первую цепь кДНК LC амплифицируют с парой олигонуклеотидных праймеров CKMOsp (СТС ATT ССТ GTT GAA GCT СТТ GAC ААТ GGG) и XSCTnTag (GAC TCG AGT CGA CAT CGA ТТТ ТТТ ТТТ ТТТ ТТТ ТТ). Реакционная смесь (25 мкл) помимо воды, свободной от нуклеиновых кислот и нуклеаз, содержит однократный буфер для Pfu ДНК полимеразы, 1 мМ смеси дезоксинуклеотидтрифосфатов (dNTP), по 5 пкМ каждого праймера, 0,5 мкл продукта реакции полиаденилирования кДНК и 1 ед. Pfu полимеразы (Promega). Амплификацию проводят, используя следующие параметры: предварительная денатурация 95°С в течение 5 мин, далее 40 циклов: 95°С 30 с, 60°С 30 с, 72°С 2 мин, затем финальная элонгация при 72°С в течение 5 минут.
Первую цепь кДНК Fd амплифицируют с парой праймеров MOCG12FOR (СТС ААТ ТТТ СТТ GTC САС СТТ GGT GC) и XSCTnTag. Для приготовления ПЦР смеси (25 мкл) используют воду, свободную от нуклеиновых кислот и нуклеаз, по 5 пкМ каждого праймера, 0,5 мкл продукта реакции полиаденилирования кДНК и смесь Phusion High-Fidelity PCR Master Mix (Thermo Fisher). Амплификацию проводят, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 40 циклов: 98°С 15 с, 40°С 15 с, 72°С 30 с, затем финальная элонгация при 72°С в течение 5 минут.
ПЦР продукты LC и Fd (Фиг. 7, дорожки 1 и 2 соответственно) очищают через электрофорез в агарозном геле при помощи GeneJET Gel Extraction Kit. Очищенные ампликоны в количестве 100 нг подвергают обработке Т4 полинуклеотидкиназой и одновременно гидролизуют эндонуклеазой рестрикции Sail FastDigest™ в буфере FD с добавлением 0,5 мМ АТФ. В результате получают фрагменты ДНК с «липким» и «тупым» концами, готовые к встраиванию в вектор для клонирования pUC19. В свою очередь, плазмиду pUC19 в количестве 100 нг разрезают эндонуклеазами рестрикции Sail и Smal FastDigest™ и обрабатывают щелочной фосфатазой FastAP. Спустя час инкубации при 37°С ферменты в реакциях с плазмидой и вставками инактивируют температурой 75°С в течение 5 минут, затем соединяют по 50 нг плазмиды и вставки, добавляют АТФ до 0,5 мМ и 5 ед. Вейсса Т4 ДНК-лигазы (все реактивы Thermo Fisher). Выдерживают 2-3 часа при комнатной температуре, после чего подвергают фенол-хлороформной экстракции и спиртовой преципитации. Осадки ДНК растворяют в 1 мкл деионизованной воды и используют для электротрансформации компетентных клеток E.coli DH12S. Из позитивных трансформантов, выросших в виде единичных колоний на агаризованной среде LB с добавлением ИПТГ и X-gal с ампициллином, выделяют плазмидную ДНК, которую впоследствии секвенируют по Сэнгеру с праймеров M13/pUC forward - 17-mer (-20) (GTA AAA CGA CGG CCA GT) и M13_Rev (AAC AGC TAT GAC CAT G). В результате определяют первичную аминокислотную последовательность легкой цепи (включая сигнальную последовательность и исключая аминокислотный остаток цистеина на с-конце), обозначенную в Seq ID NO: 1; определяют первичную аминокислотную последовательность Fd-фрагмента тяжелой цепи (включая сигнальную последовательность и исключая часть с-концевых аминокислот в домене CH1), обозначенную в Seq ID NO: 2. Устанавливают следующие границы вариабельных доменов: для легкой цепи Seq ID NO: 8, для тяжелой цепи Seq ID NO: 10. Последовательности участков, определяющих комплементарность антитела, определяют, аннотируя аминокислотные последовательности VL и VH с использованием системы нумерации Кэбота: CDRL-1 - Seq ID NO: 12, CDRL-2 - Seq ID NO: 13, CDRL-3 - Seq ID NO: 14, CDRH-1 - Seq ID NO: 15, CDRH-2 - Seq ID NO: 16, CDRH-3 - Seq ID NO: 17.
Пример 5. Сборка ДНК, кодирующей МКАт xi1E10
Создают плазмидные вектора pSF-CMV-xi1E10_LC и pSF-CMV-xi1E10_HC, содержащие генные конструкции полноразмерных цепей химерного иммуноглобулина xi1E10, на основе векторов pSF-CMV-HuKappa_LC (OG528) и pSF-CMV-HuIgG1_HC (OG527) из набора векторов для инженерии химерных или человеческих иммуноглобулинов Human IgG Vector Set (PP2409, Oxford Genetics). Осуществляют дизайн и синтез олигонуклеотидов 1E10VL_SSL_Nco_F, 1E10VL_BseRI_R 1E10VH_SSH_Nco_F, 1E10VHBseRTR для амплификации Vl- и Ун-кодирующих участков МКАт 1Е10, которые устанавливают по методике примера 4. В состав прямых праймеров вводят сайт рестрикции Ncol и кодоны, определяющие синтез сигнальной последовательности, а обратных - сайт BseRI в определенном положении для «бесшовного соединения» VL-CL и VH-CH. Описание олигонуклеотидов дано в таблице на Фиг. 5. Осуществляют дизайн и синтез олигонуклеотидов HC_F1/2 (Seq ID NO: 22), HC_R1 (Seq ID NO: 23), LC_F1 (Seq ID NO: 24), LC_R1/2 (Seq ID NO: 25) для амплификации полноразмерных цепей химерного МКАт xi1E10 и соединения двух цепей в единый ДНК-конструкт.
Получение химерной легкой цепи путем создания плазмидного вектора pSF-CMV-xi1E10_LC. Составляют амлификационную смесь объемом 25 мкл из Phusion High-Fidelity PCR Master Mix, праймеров 1E10VL_SSL_Nco_F и 1E10VL_BseRI_R (no 5 пкМ каждого), воды для ПЦР. В качестве матрицы используют 10-20 нг плазмидной ДНК, полученной после лигирования pUC19 с ДЦ-кДНК легкой цепи МКАт 1Е10 по примеру 4. Амплификацию проводят, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 25 циклов: 98°С 15 с, 58°С 15 с, 72°С 30 с, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически (Фиг. 7, дорожка 3). После проведения ПЦР ДНК подвергают фенол-хлороформной экстракции и спиртовой преципитации. Далее 100 нг очищенного ПЦР-продукта гидролизуют эндонуклеазами рестрикции Ncol FastDigest и BseRI (NEB) в буфере FD. Плазмидный вектор OG528 в количестве 100 нг гидролизуют рестриктазами Ncol FastDigest и BseRI в буфере FD с добавлением щелочной фосфатазы FastAP. Спустя час инкубации при 37°С ДНК в обеих реакциях подвергают фенол-хлороформной экстракции и спиртовой преципитации. Плазмиду и вставку соединяют в одной реакционной смеси для лигирования с помощью Т4 ДНК-лигазы (Thermo Fisher). Пробирку выдерживают 2-3 часа при комнатной температуре, после чего подвергают фенол-хлороформной экстракции и спиртовой преципитации. Осадки ДНК растворяют в 1 мкл деионизованной воды и используют для электротрансформации компетентных клеток E.coli DH12S. Из позитивных трансформантов, выросших в виде единичных колоний на агаризованной среде LB с добавлением канамицина, выделяют плазмидную ДНК, которую впоследствии секвенируют по Сэнгеру с праймера CMV For.
Получение химерной тяжелой цепи путем создания плазмидного вектора pSF-CMV-xilE10_HC. Составляют амлификационную смесь объемом 25 мкл из Phusion High-Fidelity PCR Master Mix, праймеров lE10VH_SSH_Nco_F и 1E10VH_BseRI_R (no 5 пкМ каждого), воды для ПЦР. В качестве матрицы используют 10-20 нг плазмидной ДНК, полученной после лигирования pUC19 с ДЦ-кДНК тяжелой цепи МКАт 1Е10 по примеру 4. Амплификацию проводят, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 25 циклов: 98°С 15 с, 58°С 15 с, 72°С 30 с, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически (Фиг. 7, дорожка 4). Далее методика получения вектора pSF-CMV-xi1E10_HC не отличается от методики получения pSF-CMV-xi1E10_LC, описанной выше, за исключением использования плазмиды OG527 для клонирования ПЦР-продукта.
Следующим этапом производят сборку рекомбинантной ДНК (Seq ID NO: 26) с открытой рамкой считывания, которая кодирует химерный полипептид (Seq ID NO: 27): тяжелая цепь xi1E10, аминокислотная последовательность RAKRGSGEGRGSLLTCGDVEENPGP, легкая цепь xi1E10. Сначала проводят две отдельные ПЦР с использованием Phusion High-Fidelity PCR Master Mix. Первую реакцию подготавливают с праймерами HC_F1/2 и HC_R1 (по 2 пкМ каждого), а также плазмидой pSF-CMV-xilE10_HC в качестве матрицы (10-20 нг). Вторую - с праймерами LC_F1 и LC_R1/2 (по 2 пкМ каждого) и плазмидой pSF-CMV-xi1E10_LC в качестве матрицы (10-20 нг). Проводят амплификацию, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 18 циклов: 98°С 15 с, 56°С 15 с, 72°С 45 с, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически. Изображение ампликона полноразмерной легкой цепи xi1E10 представлено на Фиг. 7 в дорожке 5. Изображение ампликона полноразмерной тяжелой цепи xi1E10 представлено на Фиг. 7 в дорожке 6. Далее подготавливают реакционную смесь с Phusion High-Fidelity PCR Master Mix в качестве основы, праймерами HC_F1/2 и LC_R1/2 (по 5 пкМ каждого) и по 0,1 мкл продукта из каждой проведенной ранее ПЦР. Для ПЦР используют следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 25 циклов: 98°С 15 с, 56°С 15 с, 72°С 1,5 мин, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически (Фиг. 7, дорожка 7).
После проведения ПЦР ДНК подвергают фенол-хлороформной экстракции и спиртовой преципитации.
Пример 6. Получение плазмидного вектора для экспрессии моноклональных антител xi1E10
ДНК, получаемую по примеру 5, в количестве 1 мкг, вносят в реакционную смесь объемом 50 мкл, состоящую из 1х Taq-буфера с 1,5 мМ MgCl2, 0,2 мМ dATP и 1 ед Taq-полимеразы. Реакцию инкубируют 20 мин при температуре 72°С.Затем производят очистку от фермента на колонке QIAquick PCR Purification Kit (Qiagen) согласно инструкции. Очищенную ДНК клонируют в вектор pcDNA3.4 из набора pcDNA 3.4-ТОРО ТА Cloning Kit (Thermo Fisher) по методу, описанному в инструкции производителя, и смешивают продукт реакции с хемокомпетентными клетками Е. coli One Shot ТОРЮ (Thermo Fisher). Из позитивных трансформантов, выросших в виде единичных колоний на агаризованной среде LB с добавлением канамицина, выделяют плазмидную ДНК набором, обеспечивающим хорошую очистку от эндотоксина Е. coli, например PureLink HiPure Plasmid Maxiprep Kit (Invitrogen). Полученный плазмидный вектор именуют pcDNA3.4_xi1E10.
Пример 7. Экспрессия МКАт xi1E10 в клетках СНО.
Клетки ExpiCHO-S, являющиеся частью набора ExpiCHO Expression System Kit (Gibco, Thermo Fisher), выращивают в среде ExpiCHO Expression Medium в одноразовых колбах Эрленмейера объемом 500 мл с вентилируемой крышкой (Corning) на шейкере Minitron (Infors НТ), режим 125 мин"1 в атмосфере 8% СО2 при 37°С. По достижении плотности 6×106 кл/мл и количества жизнеспособных клеток не менее 95% (оценивают по окрашиванию трипановым синим), проводят трансфекцию клеток плазмидой pcDNA3.4_xi1E10, полученной по примеру 6. В день трансфекции частично или полностью производят замену среды на аналогичную новую. Трансфекцию проводят с использованием реактивов и методики набора ExpiCHO Expression System Kit, а затем клетки выращивают согласно протоколу «Мах Titer Protocols Кратко: в каждую колбу объемом 500 мл, в которой находится 100 мл культуры ExpiCHO, добавляют смесь из 80 мкл плазмидной ДНК pcDNA3.4_xilE10 с концентрацией 1 мкг/мл, 7,7 мл среды OptiPRO SFM и 320 мкл ExpiFectamine СНО Reagent. Культуру растят на шейкере при 32°С в атмосфере 5% СО2. Через 24 часа после трансфекции в колбу добавляют 600 мкл энхансера ExpiCHO Enhancer и 16 мл подкормки ExpiCHO Feed. На 5 сутки после трансфекции добавляют еще 16 мл ExpiCHO Feed. На 14 день культуру центрифугируют 1000 × g в течение 10 минут. Супернатант, содержащий антитела xi1E10, дополнительно фильтруют через мембрану с диаметром пор 0,2 мкм, затем концентрируют десятикратно в ячейке Amicon Stirred Cell (MerckMillipore). При необходимости замораживают -20°С или сразу переходят к очистке по примеру 8.
Пример 8. Очистка и валидация МКАт xi1E10
Выделение МКАт xi1E10 из концентрированной культуральной жидкости проводят методом аффинной хроматографии на колонке с сорбентом Protein G сефарозой с использованием хроматографической системы  Start. Колонку с полезной емкостью 3 мл, заполненную Protein G сефарозой, уравновешивают буфером нанесения (50 мМ Tris, 100 мМ NaCl, 0,02% NaN3, рН 8,0). Производят нанесение образца со скоростью 2 мл/мин на льду. Далее колонку отмывают от не связавшихся компонентов буфером нанесения, и проводят элюцию иммуноглобулинов буфером состава: 100 мМ глицина, 100 мМ NaCl, рН 2.4. Уровень рН элюата доводят до 7,5 - 8,0 добавлением 1 М раствора Tris основания. Впоследствии элюат иммуноглобулиновой фракции вносят в коммерчески доступную колонку HiTrap Desalting (GE Healthcare) емкостью 5 мл, уравновешенную фосфатно-солевым буфером ФСБ на скорости потока 5 мл/мин. Иммуноглобулиновую фракцию собирают через некоторое время, идентифицируя ее по изменению А280. Для измерения концентрации МКАт проводят спектрофотометрию при А280, затем для расчета концентрации [мг/мл] полученное значение разделяют на коэффициент массовой экстинкции для IgG, равный 1,37. Чистоту полученной иммуноглобулиновой фракции оценивают методом SDS-электрофореза по Лэммли в 10% ПААГ в редуцирующих и нередуцирующих условиях. (Фиг. 8). Как правило, выход составляет 25-30 мг белка с 1 литра культуры.
Start. Колонку с полезной емкостью 3 мл, заполненную Protein G сефарозой, уравновешивают буфером нанесения (50 мМ Tris, 100 мМ NaCl, 0,02% NaN3, рН 8,0). Производят нанесение образца со скоростью 2 мл/мин на льду. Далее колонку отмывают от не связавшихся компонентов буфером нанесения, и проводят элюцию иммуноглобулинов буфером состава: 100 мМ глицина, 100 мМ NaCl, рН 2.4. Уровень рН элюата доводят до 7,5 - 8,0 добавлением 1 М раствора Tris основания. Впоследствии элюат иммуноглобулиновой фракции вносят в коммерчески доступную колонку HiTrap Desalting (GE Healthcare) емкостью 5 мл, уравновешенную фосфатно-солевым буфером ФСБ на скорости потока 5 мл/мин. Иммуноглобулиновую фракцию собирают через некоторое время, идентифицируя ее по изменению А280. Для измерения концентрации МКАт проводят спектрофотометрию при А280, затем для расчета концентрации [мг/мл] полученное значение разделяют на коэффициент массовой экстинкции для IgG, равный 1,37. Чистоту полученной иммуноглобулиновой фракции оценивают методом SDS-электрофореза по Лэммли в 10% ПААГ в редуцирующих и нередуцирующих условиях. (Фиг. 8). Как правило, выход составляет 25-30 мг белка с 1 литра культуры.
Наличие человеческого константного региона в МКАт xi1E10 подтверждают методом иммуноблота. Антитело подвергают электрофорезу в 10% ПААГ в нередуцирующих условиях, задействуя две дорожки на геле, после чего осуществляют горизонтальный перенос белка из геля на нитроцеллюлозную мембрану Hybond-C Extra. После завершения переноса мембрану блокируют погружением в обезжиренное молоко с массовой долей жира не более 0,5% и инкубируют в течение 1 ч на орбитальном термостатируемом шейкере при 300 об./мин и температуре 37°С. Далее, после трехкратной промывки буфером ФСБ-Тв, мембрану разрезают и инкубируют в ФСБ с разведенными (согласно паспортным рекомендациям для вестерн-блоттинга) HRP-конъюгированными поликлональными антителами козы: одну дорожку с антителами против к цепи человеческого IgG (Sigma-Aldrich А7164), другую с антителами против Fc фрагмента человеческого IgG (Sigma-Aldrich АО 170). Спустя 1 час инкубации при 37°С 300 об/мин мембраны промывают 5 раз в ФСБ-Тв. Визуализацию реакции проводят раствором субстратной смеси на основе диаминобензидина. В результате проведения данного анализа, появление специфического окрашивания, отраженного на Фиг. 9, рекомбинантное моноклональное антитело xi1E10 в действительности является химерным и содержит человеческую константную часть.
Оценку иммунологической специфичности очищенного МКАт xi1E10 проводят методом иммуноблота с рекомбинантным ПА В. anthracis и рекомбинантным IV доменом ПА. Рекомбинантные белки подвергают электрофорезу в 12% полиакриламидном геле, после чего осуществляют горизонтальный перенос белков из геля на нитроцеллюлозную мембрану Hybond-C Extra. После завершения переноса мембрану блокируют погружением в обезжиренное молоко с массовой долей жира не более 0,5% и инкубируют в течение 1 ч на орбитальном термостатируемом шейкере при 300 об./мин и температуре 37°С. Далее, после трехкратной промывки буфером ФСБ-Тв, мембрану инкубируют с МКАт xi1E10, разведенным до концентрации 0,1-1 мкг/мл в ФСБ. После трехкратной промывки буфером ФСБ-Тв мембрану инкубируют с антителами к Fc фрагменту человеческого IgG (Sigma-Aldrich А0170) в разведении 1:10000 в буфере ФСБ-Тв в течение 30 минут. Затем мембрану промывают 5 раз в ФСБ-Тв. Визуализацию реакции проводят раствором субстратной смеси на основе диаминобензидина (0,05% диаминобензидина (Sigma, США), 0,015% Н2О2 в ФСБ рН 7,4). Реакцию останавливают после отчетливо различимого окрашивания образцов путем погружения мембраны в дистиллированную воду. Как показывает проведенный анализ, результаты которого отражены на Фиг. 3 в дорожках 4 и 5, моноклональное антитело xi1E10 специфически взаимодействует с рекомбинантными белком ПА, а именно с IV доменом ПА.
Для оценки количественного взаимодействия очищенного МКАт xi1E10 с белком ПА, в лунки 96-луночного планшета с иммобилизированым антигеном, который подготовлен по методике из Примера 1, вносят методом двукратных разведений антитело в концентрациях 1000, 500, 250, 125, 62,5, 31,25, 15,6, 7,8, 3,9, 2, 1 нг/мл. Инкубируют на термостатируемом шейкере при температуре 37°С в течение 1 часа 300 об./мин. После этого лунки планшета трижды отмывают ФСБ-Тв и добавляют в лунки раствор антител к Fc фрагменту человеческого IgG (Sigma-Aldrich А0170), конъюгированных с пероксидазой хрена в рабочем разведении 1:10000. Планшет инкубируют при температуре 37°С в течение 1 часа на шейкере, затем 6 раз отмывают буфером ФСБ-Тв. После этого в лунки вносят по 100 мкл субстрат-индикаторного раствора на основе ортофенилендиамина. Положительную реакцию оценивают по появлению желто-коричневого окрашивания раствора. Реакцию останавливают добавлением в лунки по 50 мкл 4N серной кислоты. Детекцию проводят на планшетном спектрофотометре (Bio-Rad xMark, США) при длине волны 492 нм. По результатам данного анализа минимально детектируемая концентрация МКАт xi1E10 составила 2 нг/мл, кривая А = f(C) соответствует типовой кривой для высокоаффинных антител в непрямом тведофазном ИФА.
Пример 9. Определение аффинности
Определение параметров аффинного взаимодействия МКАт 1Е10 и МКАт xi1E10 с целевым антигеном (IV доменом ПА) проводят методом микроскопического термофореза (МСТ) на приборе Monolith NT. 115 Pico (NanoTemper, Германия). В соответствии с инструкцией производителя, при помощи набора Monolith His-Tag Labeling Kit RED-tris-NTA (NanoTemper, Германия) в состав белка IV домена ПА, несущего His-тэг, вносят красную флуоресцентную метку. Флуоресцентно меченый белок IV домен ПА используют в качестве лиганда в концентрации 50 нМ в каждой точке эксперимента. Против лиганда в указанной выше концентрации титруют с двукратным шагом раствор антител (МКАт 1Е10 или МКАт xi1E10) в концентрациях от 400 нм до 12,2 пМ (16 точек). Все разведения белков проводят в растворе ФСБ-Тв. Полученными смесями наполняют 16 капилляров, которые устанавливают в специальную подложку и устанавливают в прибор. Регистрируют флуоресценцию в начальной точке и детекцию флуоресценции в каждом капилляре в процессе МСТ, получают термофоретическую кривую. При помощи встроенного в прибор ПО обрабатывают полученные результаты.
Измеренные равновесные константы диссоциации (KD) для МКАт равны: KD1E10 = 9,1564×10-10 М, KDxi1E10 = 1,2663×10-9 М.
Пример 10. Определение токсиннейтрализующей активности МКАт in vitro.
Анализ проводят на клеточной линии мышиных макрофагов J774A.1 в цитотоксическом тесте МТТ. О нейтрализации летального токсина судят по возрастанию жизнеспособности клеточного монослоя при кратном повышении концентрации антител в присутствии неизменного количества токсина. В качестве среды для культивирования используют среду Игла в модификации Дульбекко (DMEM) с добавлением 2 мМ L-глутамина и 10% инактивированной фетальной бычьей сыворотки (все Gibco, Thermo Fisher, США). Клетки выращивают в 75 см2 культуральных флаконах (Corning, США) в атмосфере 5% СО2 при 37°С, не превышая 70%-80% конфлюэнции. Клетки собирают при помощи 0,05% раствора трипсина-ЭДТА (Панэко, Россия) и готовят суспензию в полной ростовой среде 2×105 жизнеспособных клеток в 1 мл. Количество клеток определят с помощью автоматического счетчика клеток ТС20 (Bio-Rad, США). Состояние клеток оценивают по устойчивости к окрашиванию трипановым синим красителем. Далее в лунки 96-луночного планшета вносят по 90 мкл клеточной суспензии. Планшет оставляют в СО2-инкубаторе (Panasonic, Япония) на 12 часов для прикрепления клеток ко дну лунок. Далее проводят пробоподготовку смесей в фосфотно-солевом буфере рПА + рЛФ + антитело в различных концентрациях. При подготовке проб все компоненты наводят в концентрации, десятикратно превышающей конечную. Пробы инкубируют 30 минут при 37°С, затем вносят в лунки по 10 мкл. Конечные концентрации рПА и рЛФ составляют 4 мкг/мл и 0,8 мкг/мл соответственно. Концентрации антител в анализе составляют 20, 10, 5, 2,5 и 0 мкг/мл, что соответствует молярным соотношениям антитело:рПА 2,5:1, 1,25:1, 0,625:1, 0,3125:1 и 0:1. Для расчета жизнеспособности клеток по показателю оптической плотности дополнительно анализируют жизнеспособность в группе лунок с добавлением 10 мкл чистого ФСБ и принимают ее за 100%. Также в другую группу лунок добавляют по 10 мкл 0,25% тиомерсаля и после проведения теста приравнивают их жизнеспособность к 0%. Дополнительно исследуют цитотоксичность отдельных компонентов смеси, то есть добавляют к клеткам только рПА (4 мкг/мл), только рЛФ (0,8 мкг/мл), или только антитело 1E10/xi1E10 (20 мкг/мл). Планшет помещают в СО2-инкубатор на 4 часа, затем добавляют по 10 мкл раствора МТТ в каждую лунку. Раствор МТТ подготавливают разбавлением готовой навески из набора Vybrant МТТ Cell Proliferation Assay Kit (Thermo Fisher) в 1 мл фосфатно-солевого буфера. Планшет инкубируют еще 4 часа. Затем аккуратно удаляют 85 мкл содержимого лунок и лизируют клетки добавлением 175 мкл диметилсульфоксида и аккуратным пипетированием. Оптическую плотность клеточного лизата измеряют при длине волны 540 нм на планшетном спектрофотометре xMark (Bio-Rad, США).
По кривым зависимости жизнеспособности макрофагов от концентрации МКАт (Фиг. 12) делают вывод о том, что и МКАт 1Е10, и МКАт xi1E10 в МТТ-тесте нейтрализуют действие летального токсина В. anthracis во всех исследуемых соотношениях, однако при избытке антигена нейтрализующая способность химерного xi1E10 немного уступает по сравнению с таковой у мышиного 1Е10.
Пример 11. Определение токсиннейтрализующей активности МКАт in vivo на модели мышей.
Для оценки нейтрализации летального токсина антителами 1Е10 и xi1E10 в живом организме используют мышей линии BALB/c весом 18-20 г. Животных делят на 4 группы: две опытные, по 27 особей в каждой, и три контрольные, по 9 особей в каждой. Мышей из первой опытной группы иммунизируют внутрибрюшинно МКАт 1Е10 в дозах 25, 50, 75 и 100 мкг/мышь в 200 мкл ФСБ по 9 особей на каждую дозу. Мышей второй опытной группы аналогично имммунизируют МКАт xi1E10. Мышам из двух контрольных групп вводят по 100 мкг МКАт 1E10/xi1E10. Через 24 часа всем мышам из опытных групп и третьей контрольной группы (не подвергнутых пассивной иммунизации) внутривенно вводят рекомбинантный летальный токсин В. anthracis. Для приготовления летального токсина рекомбинантные белки ЛФ (рЛФ, Seq ID NO: 6) и ПА смешивают в ФСБ и вводят в ретроорбитальный синус в дозе 50 мкг рПА + 50 мкг рЛФ, что соответствует значению 4LD50. Учет гибели животных проводят раз в сутки в течение 14 дней. По результатам падежа животных строят графики выживаемости, отраженные в Фиг. 13, 14, 15. Сравнивая графики выживаемости мышей в опытных (Фиг. 13, 14) и контрольных (Фиг. 15) группах, можно сделать вывод о том, что исследуемые МКАт обладают нейтрализующим действием in vivo, при этом xi1E10 защищают мышей от гибели эффективнее антитела 1Е10. Мы связываем это с повышенной активацией иммунной системы мышей в ответ на введение химерного антитела, содержащего чужеродную для организма мышей человеческую константную часть иммуноглобулина.
--->
SEQUENCE LISTING
<110> ФБУН ГНЦ ПМБ; FBIS SRCAM&B
<120> Антитело моноклональное мышиное 1E10 и антитело рекомбинантное химерное (мышь-человек) xi1E10, нейтрализующие летальный токсин Bacillus anthracis, и штамм гибридных культивируемых клеток животных Mus musculus 1E10
<130>
<160> 27
<170> BiSSAP 1.3.6
<210> 1
<211> 237
<212> PRT
<213> Mus musculus
<220>
<221> SIGNAL
<222> -1..-19
<400> 1
Met Lys Leu Pro Val Arg Leu Leu Val Leu Met Phe Trp Ile Pro Ala
-15 -10 -5
Ser Ser Ser Asp Val Leu Met Thr Gln Thr Pro Leu Ser Leu Pro Val
-1 1 5 10
Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Ile
15 20 25
Val His Asp Asn Gly Asn Thr Tyr Leu Glu Trp Phe Leu Gln Lys Pro
30 35 40 45
Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
50 55 60
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
65 70 75
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys
80 85 90
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu
95 100 105
Glu Ile Lys Arg Ala Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro
110 115 120 125
Ser Ser Glu Gln Leu Thr Ser Gly Gly Ala Ser Val Val Cys Phe Leu
130 135 140
Asn Asn Phe Tyr Pro Lys Asp Ile Asn Val Lys Trp Lys Ile Asp Gly
145 150 155
Ser Glu Arg Gln Asn Gly Val Leu Asn Ser Trp Thr Asp Gln Asp Ser
160 165 170
Lys Asp Ser Thr Tyr Ser Met Ser Ser Thr Leu Thr Leu Thr Lys Asp
175 180 185
Glu Tyr Glu Arg His Asn Ser Tyr Thr Cys Glu Ala Thr His Lys Thr
190 195 200 205
Ser Thr Ser Pro Ile Val Lys Ser Phe Asn Arg Asn Glu
210 215
<210> 2
<211> 198
<212> PRT
<213> Mus musculus
<220>
<221> SIGNAL
<222> -1..-19
<400> 2
Met Gly Trp Ser Arg Ile Phe Leu Phe Leu Leu Ser Ile Thr Ala Gly
-15 -10 -5
Val His Cys Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys
-1 1 5 10
Pro Gly Ala Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ala Phe
15 20 25
Ser Thr Ser Trp Met Asn Trp Val Lys Gln Arg Pro Gly Gln Gly Leu
30 35 40 45
Glu Trp Ile Gly Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr Asn
50 55 60
Gly Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Asn
65 70 75
Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Val Asp Ser Ala Val
80 85 90
Tyr Ile Cys Val Gly Ala Ile Val Ile Ser Tyr Gly Met Asp Tyr Trp
95 100 105
Gly Gln Gly Thr Ser Val Thr Val Ser Ser Ala Lys Thr Thr Pro Pro
110 115 120 125
Ser Val Tyr Pro Leu Ala Pro Gly Ser Ala Ala Gln Thr Asn Ser Met
130 135 140
Val Thr Leu Gly Cys Leu Val Lys Gly Tyr Phe Pro Glu Pro Val Thr
145 150 155
Val Thr Trp Asn Ser Gly Ser Leu Ser Ser Gly Val His Thr Phe Pro
160 165 170
Ala Val Leu Gln Ser Asp
175
<210> 3
<211> 243
<212> PRT
<213> Artificial Sequence
<220>
<221> SIGNAL
<222> -1..-22
<400> 3
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
-20 -15 -10
Leu Ser Gly Ala Arg Cys Asp Val Leu Met Thr Gln Thr Pro Leu Ser
-5 -1 1 5 10
Leu Pro Val Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser
15 20 25
Gln Ser Ile Val His Asp Asn Gly Asn Thr Tyr Leu Glu Trp Phe Leu
30 35 40
Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn
45 50 55
Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr
60 65 70
Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val
75 80 85 90
Tyr Tyr Cys Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly
95 100 105
Thr Lys Leu Glu Ile Lys Arg Ala Arg Thr Val Ala Ala Pro Ser Val
110 115 120
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
125 130 135
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
140 145 150
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
155 160 165 170
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
175 180 185
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
190 195 200
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
205 210 215
Gly Glu Cys
220
<210> 4
<211> 468
<212> PRT
<213> Artificial Sequence
<220>
<221> SIGNAL
<222> -1..-19
<400> 4
Met Asp Trp Thr Trp Arg Phe Leu Phe Val Val Ala Ala Ala Thr Gly
-15 -10 -5
Val Gln Ser Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys
-1 1 5 10
Pro Gly Ala Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ala Phe
15 20 25
Ser Thr Ser Trp Met Asn Trp Val Lys Gln Arg Pro Gly Gln Gly Leu
30 35 40 45
Glu Trp Ile Gly Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr Asn
50 55 60
Gly Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Asn
65 70 75
Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Val Asp Ser Ala Val
80 85 90
Tyr Ile Cys Val Gly Ala Ile Val Ile Ser Tyr Gly Met Asp Tyr Trp
95 100 105
Gly Gln Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
110 115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
160 165 170
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
175 180 185
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
190 195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
240 245 250
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
255 260 265
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
270 275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
320 325 330
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
335 340 345
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
350 355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
400 405 410
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
415 420 425
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
430 435 440 445
Ser Pro Gly Lys
<210> 5
<211> 779
<212> PRT
<213> Artificial Sequence
<220>
<223> Рекомбинантный
<300>
<301> Belova, E. V., Kolesnikov, A. V., Zakharova, M. Y., Dubiley, S.
A., Dyatlov, I. A., & Shemyakin, I. G.
<302> Special features of immune response to the lethal toxin of
bacillus anthracis
<303> Russian Journal of Bioorganic Chemistry
<304> 34
<305> 5
<306> 571-577
<307> 2008
<400> 5
Met Ala Ser Met Thr Gly Gly His His His His His His Gly Asn Glu
1 5 10 15
Asp Leu Glu Gln Lys Leu Ile Ser Glu Glu Asp Leu Glu Asp Leu Glu
20 25 30
Gln Lys Leu Ile Ser Glu Glu Asp Leu Glu Asp Pro Glu Val Lys Gln
35 40 45
Glu Asn Arg Leu Leu Asn Glu Ser Glu Ser Ser Ser Gln Gly Leu Leu
50 55 60
Gly Tyr Tyr Phe Ser Asp Leu Asn Phe Gln Ala Pro Met Val Val Thr
65 70 75 80
Ser Ser Thr Thr Gly Asp Leu Ser Ile Pro Ser Ser Glu Leu Glu Asn
85 90 95
Ile Pro Ser Glu Asn Gln Tyr Phe Gln Ser Ala Ile Trp Ser Gly Phe
100 105 110
Ile Lys Val Lys Lys Ser Asp Glu Tyr Thr Phe Ala Thr Ser Ala Asp
115 120 125
Asn His Val Thr Met Trp Val Asp Asp Gln Glu Val Ile Asn Lys Ala
130 135 140
Ser Asn Ser Asn Lys Ile Arg Leu Glu Lys Gly Arg Leu Tyr Gln Ile
145 150 155 160
Lys Ile Gln Tyr Gln Arg Glu Asn Pro Thr Glu Lys Gly Leu Asp Phe
165 170 175
Lys Leu Tyr Trp Thr Asp Ser Gln Asn Lys Lys Glu Val Ile Ser Ser
180 185 190
Asp Asn Leu Gln Leu Pro Glu Leu Lys Gln Lys Ser Ser Asn Ser Arg
195 200 205
Lys Lys Arg Ser Thr Ser Ala Gly Pro Thr Val Pro Asp Arg Asp Asn
210 215 220
Asp Gly Ile Pro Asp Ser Leu Glu Val Glu Gly Tyr Thr Val Asp Val
225 230 235 240
Lys Asn Lys Arg Thr Phe Leu Ser Pro Trp Ile Ser Asn Ile His Glu
245 250 255
Lys Lys Gly Leu Thr Lys Tyr Lys Ser Ser Pro Glu Lys Trp Ser Thr
260 265 270
Ala Ser Asp Pro Tyr Ser Asp Phe Glu Lys Val Thr Gly Arg Ile Asp
275 280 285
Lys Asn Val Ser Pro Glu Ala Arg His Pro Leu Val Ala Ala Tyr Pro
290 295 300
Ile Val His Val Asp Met Glu Asn Ile Ile Leu Ser Lys Asn Glu Asp
305 310 315 320
Gln Ser Thr Gln Asn Thr Asp Ser Gln Thr Arg Thr Ile Ser Lys Asn
325 330 335
Thr Ser Thr Ser Arg Thr His Thr Ser Glu Val His Gly Asn Ala Glu
340 345 350
Val His Ala Ser Phe Phe Asp Ile Gly Gly Ser Val Ser Ala Gly Phe
355 360 365
Ser Asn Ser Asn Ser Ser Thr Val Ala Ile Asp His Ser Leu Ser Leu
370 375 380
Ala Gly Glu Arg Thr Trp Ala Glu Thr Met Gly Leu Asn Thr Ala Asp
385 390 395 400
Thr Ala Arg Leu Asn Ala Asn Ile Arg Tyr Val Asn Thr Gly Thr Ala
405 410 415
Pro Ile Tyr Asn Val Leu Pro Thr Thr Ser Leu Val Leu Gly Lys Asn
420 425 430
Gln Thr Leu Ala Thr Ile Lys Ala Lys Glu Asn Gln Leu Ser Gln Ile
435 440 445
Leu Ala Pro Asn Asn Tyr Tyr Pro Ser Lys Asn Leu Ala Pro Ile Ala
450 455 460
Leu Asn Ala Gln Asp Asp Phe Ser Ser Thr Pro Ile Thr Met Asn Tyr
465 470 475 480
Asn Gln Phe Leu Glu Leu Glu Lys Thr Lys Gln Leu Arg Leu Asp Thr
485 490 495
Asp Gln Val Tyr Gly Asn Ile Ala Thr Tyr Asn Phe Glu Asn Gly Arg
500 505 510
Val Arg Val Asp Thr Gly Ser Asn Trp Ser Glu Val Leu Pro Gln Ile
515 520 525
Gln Glu Thr Thr Ala Arg Ile Ile Phe Asn Gly Lys Asp Leu Asn Leu
530 535 540
Val Glu Arg Arg Ile Ala Ala Val Asn Pro Ser Asp Pro Leu Glu Thr
545 550 555 560
Thr Lys Pro Asp Met Thr Leu Lys Glu Ala Leu Lys Ile Ala Phe Gly
565 570 575
Phe Asn Glu Pro Asn Gly Asn Leu Gln Tyr Gln Gly Lys Asp Ile Thr
580 585 590
Glu Phe Asp Phe Asn Phe Asp Gln Gln Thr Ser Gln Asn Ile Lys Asn
595 600 605
Gln Leu Ala Glu Leu Asn Val Thr Asn Ile Tyr Thr Val Leu Asp Lys
610 615 620
Ile Lys Leu Asn Ala Lys Met Asn Ile Leu Ile Arg Asp Lys Arg Phe
625 630 635 640
His Tyr Asp Arg Asn Asn Ile Ala Val Gly Ala Asp Glu Ser Val Val
645 650 655
Lys Glu Ala His Arg Glu Val Ile Asn Ser Ser Thr Glu Gly Leu Leu
660 665 670
Leu Asn Ile Asp Lys Asp Ile Arg Lys Ile Leu Ser Gly Tyr Ile Val
675 680 685
Glu Ile Glu Asp Thr Glu Gly Leu Lys Glu Val Ile Asn Asp Arg Tyr
690 695 700
Asp Met Leu Asn Ile Ser Ser Leu Arg Gln Asp Gly Lys Thr Phe Ile
705 710 715 720
Asp Phe Lys Lys Tyr Asn Asp Lys Leu Pro Leu Tyr Ile Ser Asn Pro
725 730 735
Asn Tyr Lys Val Asn Val Tyr Ala Val Thr Lys Glu Asn Thr Ile Ile
740 745 750
Asn Pro Ser Glu Asn Gly Asp Thr Ser Thr Asn Gly Ile Lys Lys Ile
755 760 765
Leu Ile Phe Ser Lys Lys Gly Tyr Glu Ile Gly
770 775
<210> 6
<211> 809
<212> PRT
<213> Artificial Sequence
<220>
<223> Рекомбинантный
<300>
<301> Zakharova, M. Y., Kuznetsov, N. A., Dubiley, S. A., Kozyr, A. V.,
Fedorova, O. S., Chudakov, D. M., ... & Kolesnikov, A. V.
<302> Substrate recognition of anthrax lethal factor examined by
combinatorial and pre-steady-state kinetic approaches
<303> Journal of Biological Chemistry
<304> 284
<305> 27
<306> 17902-17913
<307> 2009
<400> 6
Met Ala Ser Met Thr Glu Asp Leu Glu Gln Lys Leu Ile Ser Glu Glu
1 5 10 15
Asp Leu Glu Asp Pro His His His His His His Gly Gly Ser Glu Asp
20 25 30
Pro Ala Gly Gly His Gly Asp Val Gly Met His Val Lys Glu Lys Glu
35 40 45
Lys Asn Lys Asp Glu Asn Lys Arg Lys Asp Glu Glu Arg Asn Lys Thr
50 55 60
Gln Glu Glu His Leu Lys Glu Ile Met Lys His Ile Val Lys Ile Glu
65 70 75 80
Val Lys Gly Glu Glu Ala Val Lys Lys Glu Ala Ala Glu Lys Leu Leu
85 90 95
Glu Lys Val Pro Ser Asp Val Leu Glu Met Tyr Lys Ala Ile Gly Gly
100 105 110
Lys Ile Tyr Ile Val Asp Gly Asp Ile Thr Lys His Ile Ser Leu Glu
115 120 125
Ala Leu Ser Glu Asp Lys Lys Lys Ile Lys Asp Ile Tyr Gly Lys Asp
130 135 140
Ala Leu Leu His Glu His Tyr Val Tyr Ala Lys Glu Gly Tyr Glu Pro
145 150 155 160
Val Leu Val Ile Gln Ser Ser Glu Asp Tyr Val Glu Asn Thr Glu Lys
165 170 175
Ala Leu Asn Val Tyr Tyr Glu Ile Gly Lys Ile Leu Ser Arg Asp Ile
180 185 190
Leu Ser Lys Ile Asn Gln Pro Tyr Gln Lys Phe Leu Asp Val Leu Asn
195 200 205
Thr Ile Lys Asn Ala Ser Asp Ser Asp Gly Gln Asp Leu Leu Phe Thr
210 215 220
Asn Gln Leu Lys Glu His Pro Thr Asp Phe Ser Val Glu Phe Leu Glu
225 230 235 240
Gln Asn Ser Asn Glu Val Gln Glu Val Phe Ala Lys Ala Phe Ala Tyr
245 250 255
Tyr Ile Glu Pro Gln His Arg Asp Val Leu Gln Leu Tyr Ala Pro Glu
260 265 270
Ala Phe Asn Tyr Met Asp Lys Phe Asn Glu Gln Glu Ile Asn Leu Ser
275 280 285
Leu Glu Glu Leu Lys Asp Gln Arg Met Met Ala Arg Tyr Glu Lys Trp
290 295 300
Glu Lys Ile Lys Gln His Tyr Gln His Trp Ser Asp Ser Leu Ser Glu
305 310 315 320
Glu Gly Arg Gly Leu Leu Lys Lys Leu Gln Ile Pro Ile Glu Pro Lys
325 330 335
Lys Asp Asp Ile Ile His Ser Leu Ser Gln Glu Glu Thr Glu Leu Leu
340 345 350
Lys Arg Ile Gln Ile Asp Ser Ser Asp Phe Leu Ser Thr Glu Glu Lys
355 360 365
Glu Phe Leu Lys Lys Leu Gln Ile Asp Ile Arg Asp Ser Leu Ser Glu
370 375 380
Glu Glu Lys Glu Leu Leu Asn Arg Ile Gln Val Asp Ser Ser Asn Pro
385 390 395 400
Leu Ser Glu Lys Glu Glu Glu Phe Leu Lys Lys Leu Lys Leu Asp Ile
405 410 415
Gln Pro Tyr Asp Ile Asn Gln Arg Leu Gln Asp Thr Gly Gly Leu Ile
420 425 430
Asp Ser Pro Ser Ile Asn Leu Asp Val Arg Lys Gln Tyr Lys Arg Asp
435 440 445
Ile Gln Asn Ile Asp Ala Leu Leu His Gln Ser Ile Gly Ser Thr Leu
450 455 460
Tyr Asn Lys Ile Tyr Leu Tyr Glu Asn Met Asn Ile Asn Asn Leu Thr
465 470 475 480
Ala Thr Leu Gly Ala Asp Leu Val Asp Ser Thr Asp Asn Thr Lys Ile
485 490 495
Asn Arg Gly Ile Phe Asn Glu Phe Lys Lys Asn Phe Lys Tyr Ser Ile
500 505 510
Ser Ser Asn Tyr Met Ile Val Asp Ile Asn Glu Arg Pro Ala Leu Asp
515 520 525
Asn Glu Arg Leu Lys Trp Arg Ile Gln Leu Ser Pro Asp Thr Gln Ala
530 535 540
Gly Tyr Leu Glu Asn Gly Lys Leu Ile Leu Gln Arg Asn Ile Gly Leu
545 550 555 560
Glu Ile Lys Asp Val Gln Ile Ile Lys Gln Ser Glu Lys Glu Tyr Ile
565 570 575
Arg Ile Asp Ala Lys Val Val Pro Lys Ser Lys Ile Asp Thr Lys Ile
580 585 590
Gln Glu Ala Gln Leu Asn Ile Asn Gln Glu Trp Asn Lys Ala Leu Gly
595 600 605
Leu Pro Lys Tyr Thr Lys Leu Ile Thr Phe Asn Val His Asn Arg Tyr
610 615 620
Ala Ser Asn Ile Val Glu Ser Ala Tyr Leu Ile Leu Asn Glu Trp Lys
625 630 635 640
Asn Asn Ile Gln Ser Asp Leu Ile Lys Lys Val Thr Asn Tyr Leu Val
645 650 655
Asp Gly Asn Gly Arg Phe Val Phe Thr Asp Ile Thr Leu Pro Asn Ile
660 665 670
Ala Glu Gln Tyr Thr His Gln Asp Lys Ile Tyr Glu Gln Val His Ser
675 680 685
Lys Gly Leu Tyr Val Pro Glu Ser Arg Ser Ile Leu Leu His Gly Pro
690 695 700
Ser Lys Gly Val Glu Leu Arg Asn Asp Ser Glu Gly Phe Ile His Glu
705 710 715 720
Phe Gly His Ala Val Asp Asp Tyr Ala Gly Tyr Leu Leu Asp Lys Asn
725 730 735
Gln Ser Asp Leu Val Thr Asn Ser Lys Lys Phe Ile Asp Ile Phe Lys
740 745 750
Glu Glu Gly Ser Asn Leu Thr Ser Tyr Gly Arg Thr Asn Glu Ala Glu
755 760 765
Phe Phe Ala Glu Ala Phe Arg Leu Met His Ser Thr Asp His Ala Glu
770 775 780
Arg Leu Lys Val Gln Lys Asn Ala Pro Lys Thr Phe Gln Phe Ile Asn
785 790 795 800
Asp Gln Ile Lys Phe Ile Ile Asn Ser
805
<210> 7
<211> 158
<212> PRT
<213> Artificial Sequence
<220>
<223> Рекомбинантный
<400> 7
Met Ala Ser Met Thr Gly Gly His His His His His His Gly Asn Glu
1 5 10 15
Asp Pro Phe His Tyr Asp Arg Asn Asn Ile Ala Val Gly Ala Asp Glu
20 25 30
Ser Val Val Lys Glu Ala His Arg Glu Val Ile Asn Ser Ser Thr Glu
35 40 45
Gly Leu Leu Leu Asn Ile Asp Lys Asp Ile Arg Lys Ile Leu Ser Gly
50 55 60
Tyr Ile Val Glu Ile Glu Asp Thr Glu Gly Leu Lys Glu Val Ile Asn
65 70 75 80
Asp Arg Tyr Asp Met Leu Asn Ile Ser Ser Leu Arg Gln Asp Gly Lys
85 90 95
Thr Phe Ile Asp Phe Lys Lys Tyr Asn Asp Lys Leu Pro Leu Tyr Ile
100 105 110
Ser Asn Pro Asn Tyr Lys Val Asn Val Tyr Ala Val Thr Lys Glu Asn
115 120 125
Thr Ile Ile Asn Pro Ser Glu Asn Gly Asp Thr Ser Thr Asn Gly Ile
130 135 140
Lys Lys Ile Leu Ile Phe Ser Lys Lys Gly Tyr Glu Ile Gly
145 150 155
<210> 8
<211> 342
<212> DNA
<213> Mus musculus
<220>
<221> CDS
<222> 1..342
<223> /transl_table=1
<400> 8
gat gtt ttg atg acc caa act cca ctc tcc ctg cct gtc agt ctt gga 48
Asp Val Leu Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
gat caa gcc tcc atc tct tgc aga tct agt cag agc att gta cat gat 96
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Ile Val His Asp
20 25 30
aat gga aac acc tat tta gaa tgg ttc ctg cag aaa cca ggc cag tct 144
Asn Gly Asn Thr Tyr Leu Glu Trp Phe Leu Gln Lys Pro Gly Gln Ser
35 40 45
cca aag ctc ctg atc tac aaa gtt tcc aac cga ttt tct ggg gtc cca 192
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
gac agg ttc agt ggc agt gga tca ggg aca gat ttc aca ctc aag atc 240
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
agc aga gtg gag gct gag gat ctg gga gtt tat tac tgc ttt caa ggt 288
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Phe Gln Gly
85 90 95
tca cat gtt ccg tgg acg ttc ggt gga ggc acc aag ctg gaa atc aaa 336
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
cgg gct 342
Arg Ala
<210> 9
<211> 114
<212> PRT
<213> Mus musculus
<220>
<223> [CDS]:1..342 from SEQ ID NO 8
<400> 9
Asp Val Leu Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Ile Val His Asp
20 25 30
Asn Gly Asn Thr Tyr Leu Glu Trp Phe Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Phe Gln Gly
85 90 95
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Ala
<210> 10
<211> 357
<212> DNA
<213> Mus musculus
<220>
<221> CDS
<222> 1..357
<223> /transl_table=1
<400> 10
cag gtc cag ctg cag cag tct gga cct gag cta gtg aag cct ggg gcc 48
Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
tca gtg aag att tcc tgc aaa gct tct ggt tac gca ttc agt acc tct 96
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ala Phe Ser Thr Ser
20 25 30
tgg atg aac tgg gtg aag cag agg cct gga cag ggt ctt gag tgg att 144
Trp Met Asn Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
gga cgg att tat cct ggt gat gga gat act aac tac aat ggg aag ttc 192
Gly Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr Asn Gly Lys Phe
50 55 60
aag ggc aag gcc aca ctg act gca gac aaa tcc tcc aac aca gcc tac 240
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Asn Thr Ala Tyr
65 70 75 80
atg cag ctc agc agc ctg acc tct gtg gac tct gcg gtc tat atc tgt 288
Met Gln Leu Ser Ser Leu Thr Ser Val Asp Ser Ala Val Tyr Ile Cys
85 90 95
gta ggg gcg ata gta atc tct tat ggt atg gac tac tgg ggt caa gga 336
Val Gly Ala Ile Val Ile Ser Tyr Gly Met Asp Tyr Trp Gly Gln Gly
100 105 110
acc tca gtc acc gtt tcc tca 357
Thr Ser Val Thr Val Ser Ser
115
<210> 11
<211> 119
<212> PRT
<213> Mus musculus
<220>
<223> [CDS]:1..357 from SEQ ID NO 10
<400> 11
Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ala Phe Ser Thr Ser
20 25 30
Trp Met Asn Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr Asn Gly Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Asn Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Val Asp Ser Ala Val Tyr Ile Cys
85 90 95
Val Gly Ala Ile Val Ile Ser Tyr Gly Met Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Ser Val Thr Val Ser Ser
115
<210> 12
<211> 16
<212> PRT
<213> Mus musculus
<400> 12
Arg Ser Ser Gln Ser Ile Val His Asp Asn Gly Asn Thr Tyr Leu Glu
1 5 10 15
<210> 13
<211> 7
<212> PRT
<213> Mus musculus
<400> 13
Lys Val Ser Asn Arg Phe Ser
1 5
<210> 14
<211> 9
<212> PRT
<213> Mus musculus
<400> 14
Phe Gln Gly Ser His Val Pro Trp Thr
1 5
<210> 15
<211> 5
<212> PRT
<213> Mus musculus
<400> 15
Thr Ser Trp Met Asn
1 5
<210> 16
<211> 17
<212> PRT
<213> Mus musculus
<400> 16
Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr Asn Gly Lys Phe Lys
1 5 10 15
Gly
<210> 17
<211> 10
<212> PRT
<213> Mus musculus
<400> 17
Ala Ile Val Ile Ser Tyr Gly Met Asp Tyr
1 5 10
<210> 18
<211> 91
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 18
ataccatgga catgagagtg ccggcccagc tgctgggact cctgctcctg tggctgagcg 60
gcgcaaggtg cgatgttttg atgacccaaa c 91
<210> 19
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 19
tatgaggagg catgcatcta gcccgtttga tttccagc 38
<210> 20
<211> 82
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 20
ataccatgga ctggacgtgg cgctttctct tcgtcgtcgc cgctgccact ggagtccagt 60
cgcaggtcca gctgcagcag tc 82
<210> 21
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 21
tatgaggagg catgcatgct gaggaaacgg tgactgagg 39
<210> 22
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 22
accatggact ggacgtggc 19
<210> 23
<211> 69
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 23
ccgcaggtca gcaggctgcc tctgccctcg ccgctgcctc tcttggctct cttgccaggg 60
ctcagggac 69
<210> 24
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 24
cagaggcagc ctgctgacct gcggcgacgt ggaggagaac cccggcccca tggacatgag 60
agtgccggcc 70
<210> 25
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер
<400> 25
ctagcactcg cccctgttg 19
<210> 26
<211> 2214
<212> DNA
<213> Artificial Sequence
<220>
<221> CDS
<222> 4..2211
<223> /transl_table=1
<400> 26
acc atg gac tgg acg tgg cgc ttt ctc ttc gtc gtc gcc gct gcc act 48
Met Asp Trp Thr Trp Arg Phe Leu Phe Val Val Ala Ala Ala Thr
1 5 10 15
gga gtc cag tcg cag gtc cag ctg cag cag tct gga cct gag cta gtg 96
Gly Val Gln Ser Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val
20 25 30
aag cct ggg gcc tca gtg aag att tcc tgc aaa gct tct ggt tac gca 144
Lys Pro Gly Ala Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ala
35 40 45
ttc agt acc tct tgg atg aac tgg gtg aag cag agg cct gga cag ggt 192
Phe Ser Thr Ser Trp Met Asn Trp Val Lys Gln Arg Pro Gly Gln Gly
50 55 60
ctt gag tgg att gga cgg att tat cct ggt gat gga gat act aac tac 240
Leu Glu Trp Ile Gly Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr
65 70 75
aat ggg aag ttc aag ggc aag gcc aca ctg act gca gac aaa tcc tcc 288
Asn Gly Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser
80 85 90 95
aac aca gcc tac atg cag ctc agc agc ctg acc tct gtg gac tct gcg 336
Asn Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Val Asp Ser Ala
100 105 110
gtc tat atc tgt gta ggg gcg ata gta atc tct tat ggt atg gac tac 384
Val Tyr Ile Cys Val Gly Ala Ile Val Ile Ser Tyr Gly Met Asp Tyr
115 120 125
tgg ggt caa gga acc tca gtc acc gtt tcc tca gcc agc acc aag gga 432
Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
cct agc gtg ttc cct ctg gcc cct tcc tcc aag agc acc agc ggc gga 480
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155
aca gct gcc ctg gga tgc ctg gtg aaa gac tac ttc ccc gag ccc gtg 528
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
160 165 170 175
acc gtg agc tgg aac agc gga gcc ctg aca tcc ggc gtg cac aca ttc 576
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
ccc gcc gtg ctg caa tcc tcc ggc ctg tac agc ctg tcc tcc gtg gtg 624
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
aca gtg cct agc agc agc ctg ggc acc cag acc tac atc tgc aat gtg 672
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
aac cac aag ccc agc aac acc aag gtg gac aag aag gtg gag ccc aag 720
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235
agc tgc gat aaa acc cac acc tgc cct cct tgc cct gct cct gag ctg 768
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
240 245 250 255
ctg gga gga ccc agc gtg ttt ctg ttc ccc ccc aag ccc aaa gac aca 816
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
ctg atg atc tcc agg acc cct gag gtg aca tgc gtg gtg gtg gac gtg 864
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
tcc cac gag gac ccc gaa gtg aag ttc aac tgg tac gtg gat ggc gtg 912
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
gag gtg cac aac gcc aag acc aag ccc agg gaa gag cag tac aac agc 960
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315
acc tac aga gtg gtg tcc gtg ctg acc gtg ctc cac cag gac tgg ctg 1008
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
320 325 330 335
aac ggc aag gag tac aag tgc aag gtg agc aac aag gct ctg ccc gcc 1056
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
ccc atc gag aag acc att tcc aag gcc aag ggc cag ccc agg gaa cct 1104
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
cag gtg tat acc ctg ccc ccc agc agg gac gaa ctc acc aag aac cag 1152
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
gtg tcc ctc acc tgc ctc gtg aag ggc ttt tac ccc agc gac att gcc 1200
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395
gtg gag tgg gag agc aac ggc cag ccc gag aac aac tac aag acc aca 1248
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
400 405 410 415
cct ccc gtg ctg gac agc gac ggc agc ttc ttc ctg tac agc aag ctc 1296
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
acc gtg gac aaa tcc agg tgg cag caa ggc aac gtg ttc tcc tgc agc 1344
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
gtg atg cac gag gcc ctc cac aac cac tac aca cag aag tcc ctg tcc 1392
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
ctg agc cct ggc aag aga gcc aag aga ggc agc ggc gag ggc aga ggc 1440
Leu Ser Pro Gly Lys Arg Ala Lys Arg Gly Ser Gly Glu Gly Arg Gly
465 470 475
agc ctg ctg acc tgc ggc gac gtg gag gag aac ccc ggc ccc atg gac 1488
Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met Asp
480 485 490 495
atg aga gtg ccg gcc cag ctg ctg gga ctc ctg ctc ctg tgg ctg agc 1536
Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp Leu Ser
500 505 510
ggc gca agg tgc gat gtt ttg atg acc caa act cca ctc tcc ctg cct 1584
Gly Ala Arg Cys Asp Val Leu Met Thr Gln Thr Pro Leu Ser Leu Pro
515 520 525
gtc agt ctt gga gat caa gcc tcc atc tct tgc aga tct agt cag agc 1632
Val Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser
530 535 540
att gta cat gat aat gga aac acc tat tta gaa tgg ttc ctg cag aaa 1680
Ile Val His Asp Asn Gly Asn Thr Tyr Leu Glu Trp Phe Leu Gln Lys
545 550 555
cca ggc cag tct cca aag ctc ctg atc tac aaa gtt tcc aac cga ttt 1728
Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe
560 565 570 575
tct ggg gtc cca gac agg ttc agt ggc agt gga tca ggg aca gat ttc 1776
Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
580 585 590
aca ctc aag atc agc aga gtg gag gct gag gat ctg gga gtt tat tac 1824
Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr
595 600 605
tgc ttt caa ggt tca cat gtt ccg tgg acg ttc ggt gga ggc acc aag 1872
Cys Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys
610 615 620
ctg gaa atc aaa cgg gct aga acc gtg gcc gcc ccc agc gtg ttc atc 1920
Leu Glu Ile Lys Arg Ala Arg Thr Val Ala Ala Pro Ser Val Phe Ile
625 630 635
ttc cct ccc agc gac gag cag ctg aag agc ggc acc gcc agc gtg gtg 1968
Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val
640 645 650 655
tgt ctg ctg aac aac ttc tac ccc agg gag gcc aag gtc cag tgg aag 2016
Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys
660 665 670
gtg gac aac gcc ctg cag agc ggc aac agc cag gag agc gtg acc gag 2064
Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu
675 680 685
cag gac agc aag gac agc acc tac agc ctg agc agc acc ctc acc ctg 2112
Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu
690 695 700
agc aag gcc gac tac gag aag cac aag gtg tac gcc tgt gag gtg acc 2160
Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr
705 710 715
cac cag ggc ctg agc agc cct gtg acc aag agc ttc aac agg ggc gag 2208
His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu
720 725 730 735
tgc tag 2214
Cys
<210> 27
<211> 736
<212> PRT
<213> Artificial Sequence
<220>
<223> [CDS]:4..2211 from SEQ ID NO 26
<400> 27
Met Asp Trp Thr Trp Arg Phe Leu Phe Val Val Ala Ala Ala Thr Gly
1 5 10 15
Val Gln Ser Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys
20 25 30
Pro Gly Ala Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ala Phe
35 40 45
Ser Thr Ser Trp Met Asn Trp Val Lys Gln Arg Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr Asn
65 70 75 80
Gly Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Asn
85 90 95
Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Val Asp Ser Ala Val
100 105 110
Tyr Ile Cys Val Gly Ala Ile Val Ile Ser Tyr Gly Met Asp Tyr Trp
115 120 125
Gly Gln Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
130 135 140
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
145 150 155 160
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
165 170 175
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
180 185 190
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
195 200 205
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
210 215 220
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
225 230 235 240
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
245 250 255
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
260 265 270
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
275 280 285
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
290 295 300
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
305 310 315 320
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
325 330 335
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
340 345 350
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
355 360 365
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
370 375 380
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
385 390 395 400
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
405 410 415
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
420 425 430
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
435 440 445
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
450 455 460
Ser Pro Gly Lys Arg Ala Lys Arg Gly Ser Gly Glu Gly Arg Gly Ser
465 470 475 480
Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met Asp Met
485 490 495
Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp Leu Ser Gly
500 505 510
Ala Arg Cys Asp Val Leu Met Thr Gln Thr Pro Leu Ser Leu Pro Val
515 520 525
Ser Leu Gly Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Ile
530 535 540
Val His Asp Asn Gly Asn Thr Tyr Leu Glu Trp Phe Leu Gln Lys Pro
545 550 555 560
Gly Gln Ser Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
565 570 575
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
580 585 590
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys
595 600 605
Phe Gln Gly Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu
610 615 620
Glu Ile Lys Arg Ala Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe
625 630 635 640
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
645 650 655
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
660 665 670
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
675 680 685
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
690 695 700
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
705 710 715 720
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
725 730 735
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Антитела против белка р17 ВИЧ-1 субтипа А | 2019 |
|
RU2727673C1 |
| АНТИТЕЛО К CD73 ЧЕЛОВЕКА | 2017 |
|
RU2754058C2 |
| УНИВЕРСАЛЬНЫЙ АНТИТЕЛООПОСРЕДОВАННЫЙ БИОСЕНСОР | 2016 |
|
RU2746486C2 |
| ШТАММ КЛЕТОК CHO-SE-9/4 - ПРОДУЦЕНТ ХИМЕРНОГО АНТИТЕЛА ПРОТИВ ЭРИТРОПОЭТИНА ЧЕЛОВЕКА И ХИМЕРНОЕ АНТИТЕЛО, ПРОДУЦИРУЕМОЕ ДАННЫМ ШТАММОМ | 2019 |
|
RU2717038C1 |
| АНТИТЕЛО ДЛЯ ЛЕЧЕНИЯ АУТОИММУННЫХ ЗАБОЛЕВАНИЙ | 2017 |
|
RU2781148C2 |
| МАТЕРИАЛЫ И МЕТОДЫ, ИСПОЛЬЗУЕМЫЕ ДЛЯ ЛЕЧЕНИЯ РЕСПИРАТОРНЫХ ЗАБОЛЕВАНИЙ У СОБАК | 2020 |
|
RU2811752C2 |
| АНТИТЕЛА, ИНДУЦИРУЮЩИЕ ИММУННУЮ ТОЛЕРАНТНОСТЬ, ИНДУЦИРОВАННЫЕ ЛИМФОЦИТЫ И ТЕРАПЕВТИЧЕСКИЙ АГЕНТ/СПОСОБ КЛЕТОЧНОЙ ТЕРАПИИ С ИСПОЛЬЗОВАНИЕМ ИНДУЦИРОВАННЫХ ЛИМФОЦИТОВ | 2019 |
|
RU2816592C2 |
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО | 2020 |
|
RU2814713C2 |
| АНТИТЕЛО ПРОТИВ Fn14 ЧЕЛОВЕКА | 2019 |
|
RU2787044C2 |
| СПОСОБ УПРАВЛЕНИЯ РИТМОМ СЕРДЦА И СОКРАЩЕНИЕМ ОТДЕЛЬНЫХ КАРДИОМИОЦИТОВ ПРИ ПОМОЩИ ТЕРМОГЕНЕТИКИ | 2022 |
|
RU2802995C1 |

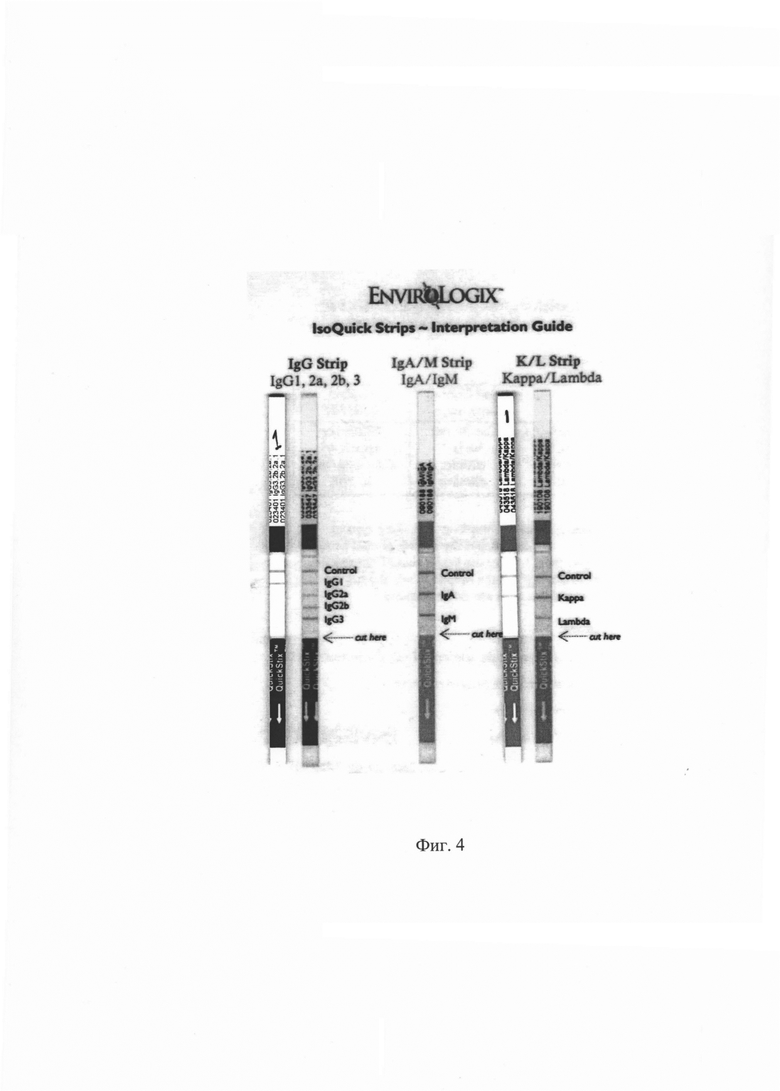
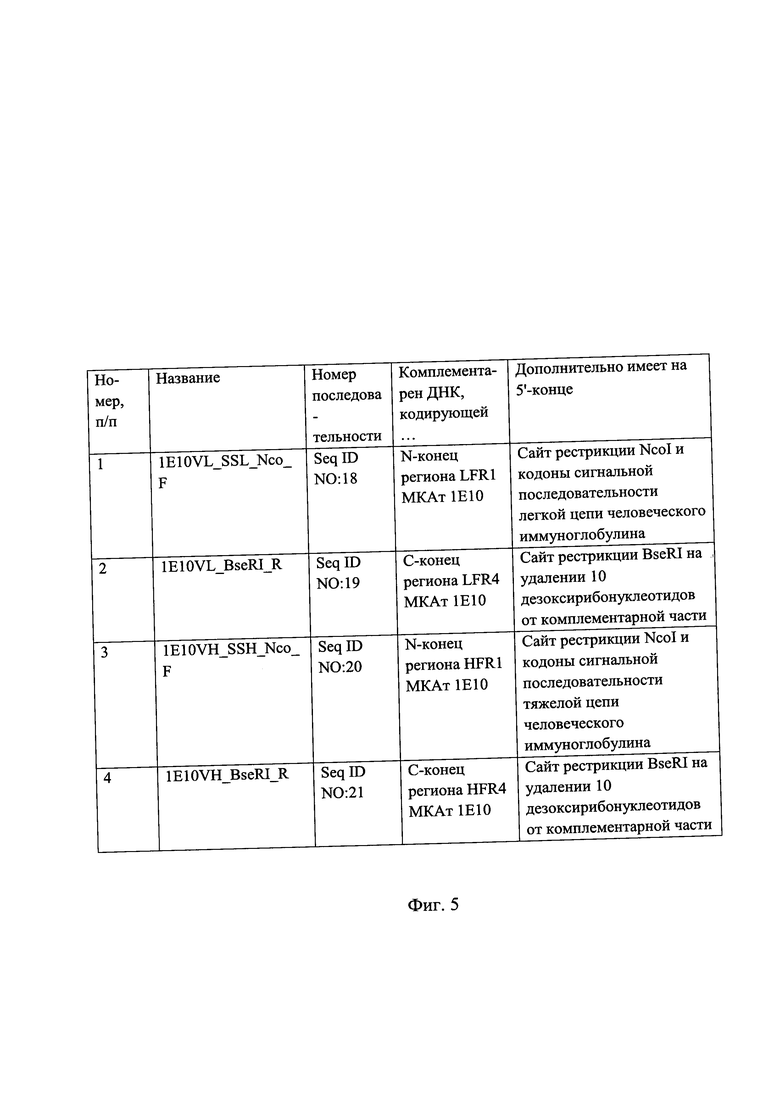
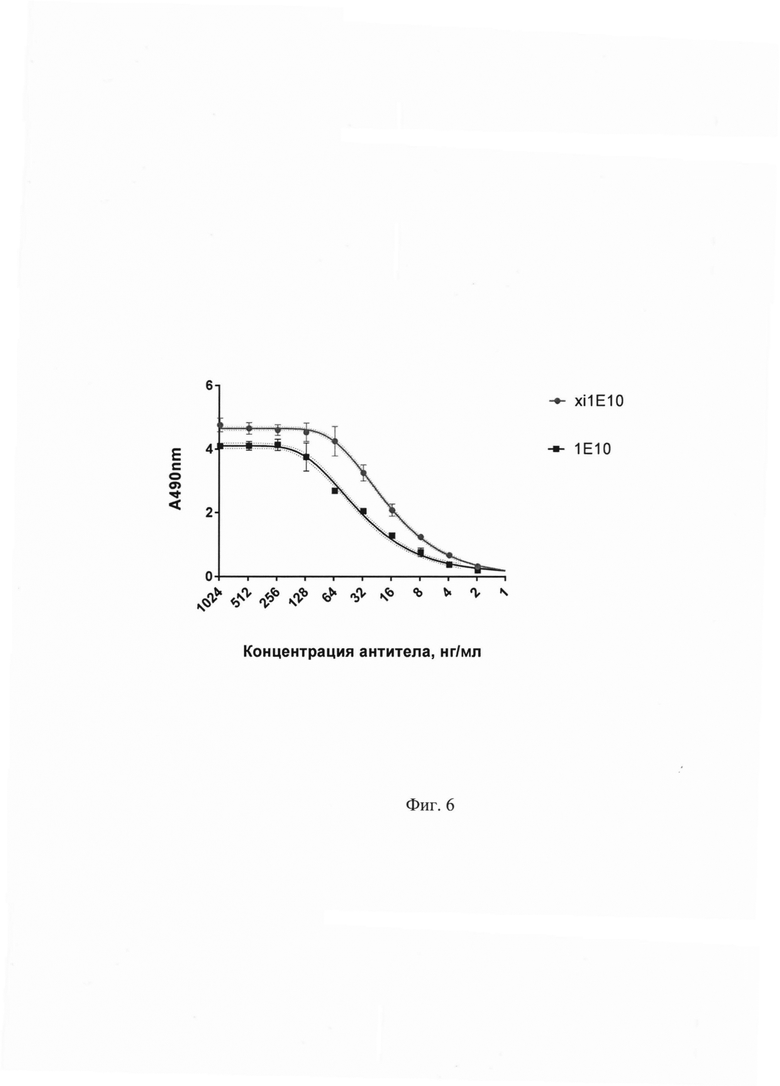
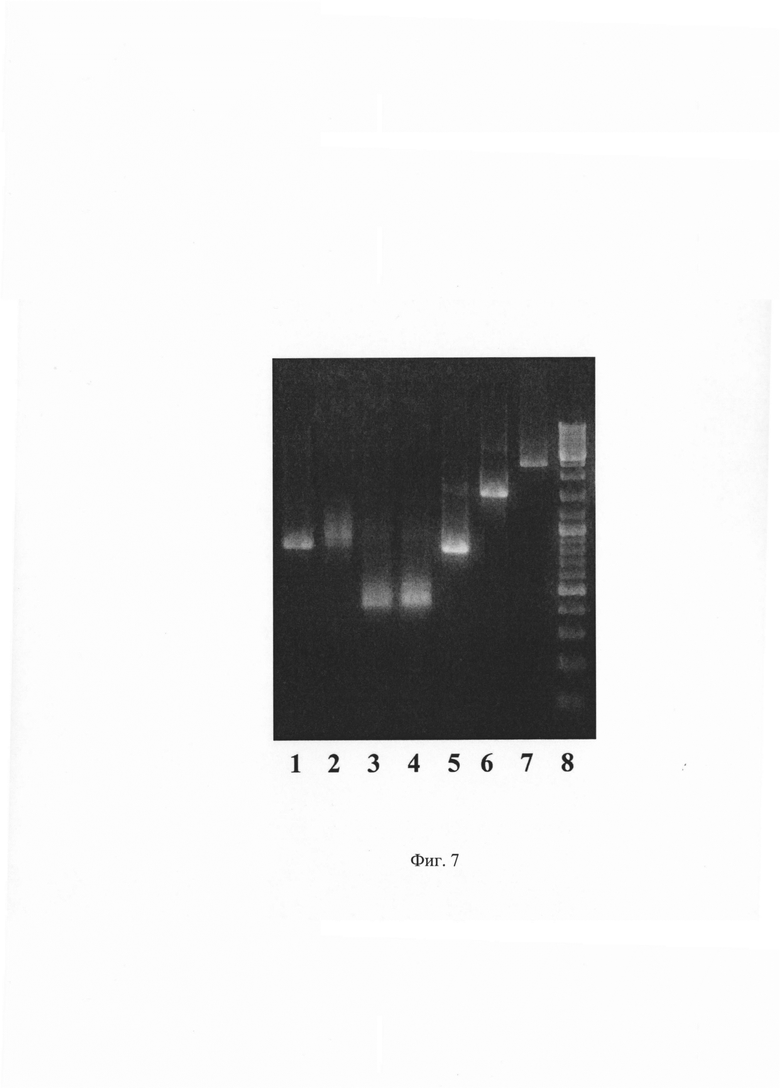
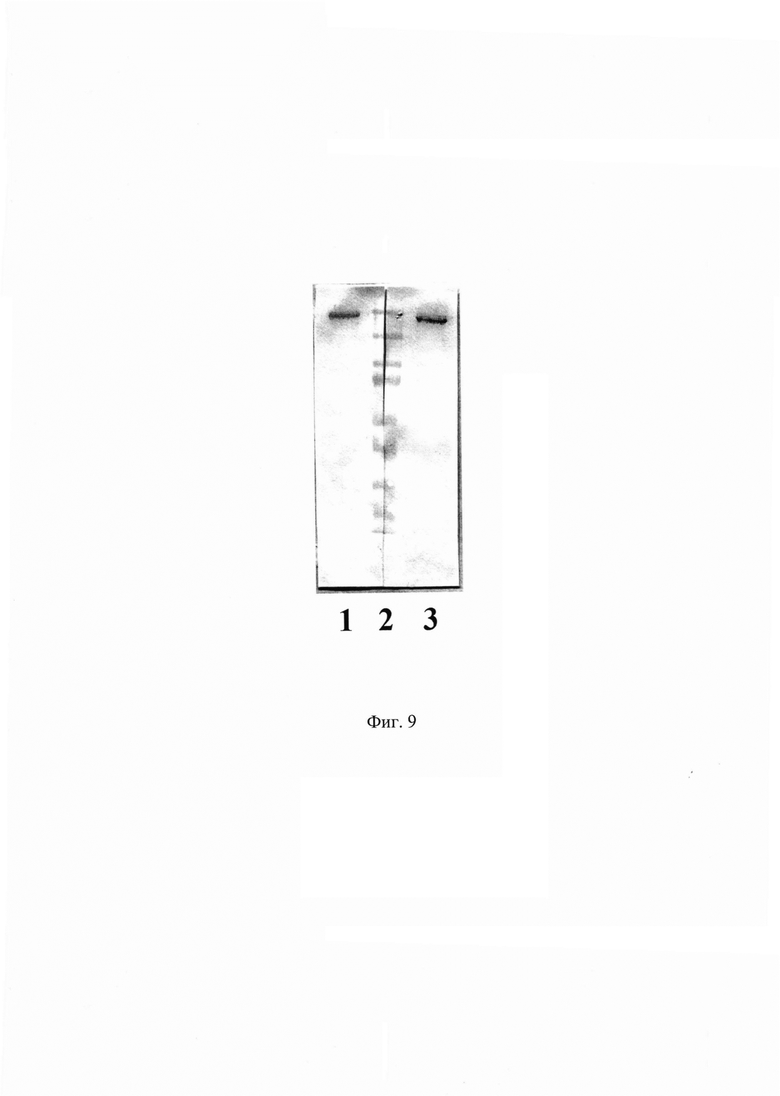
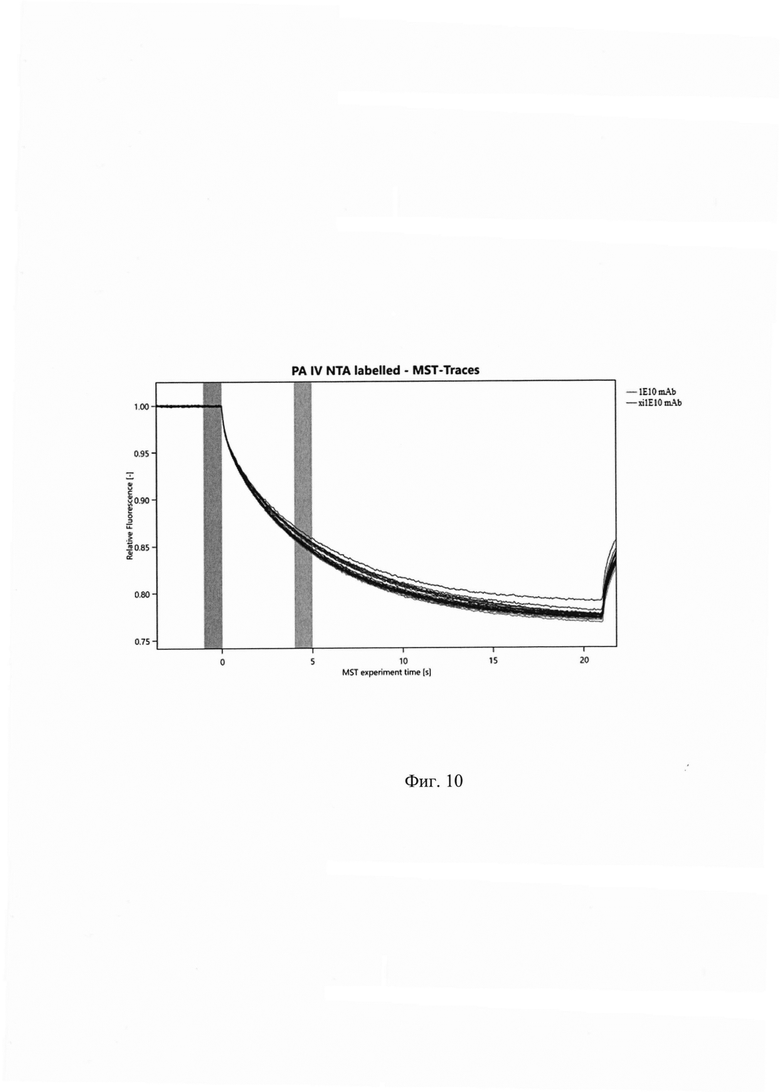


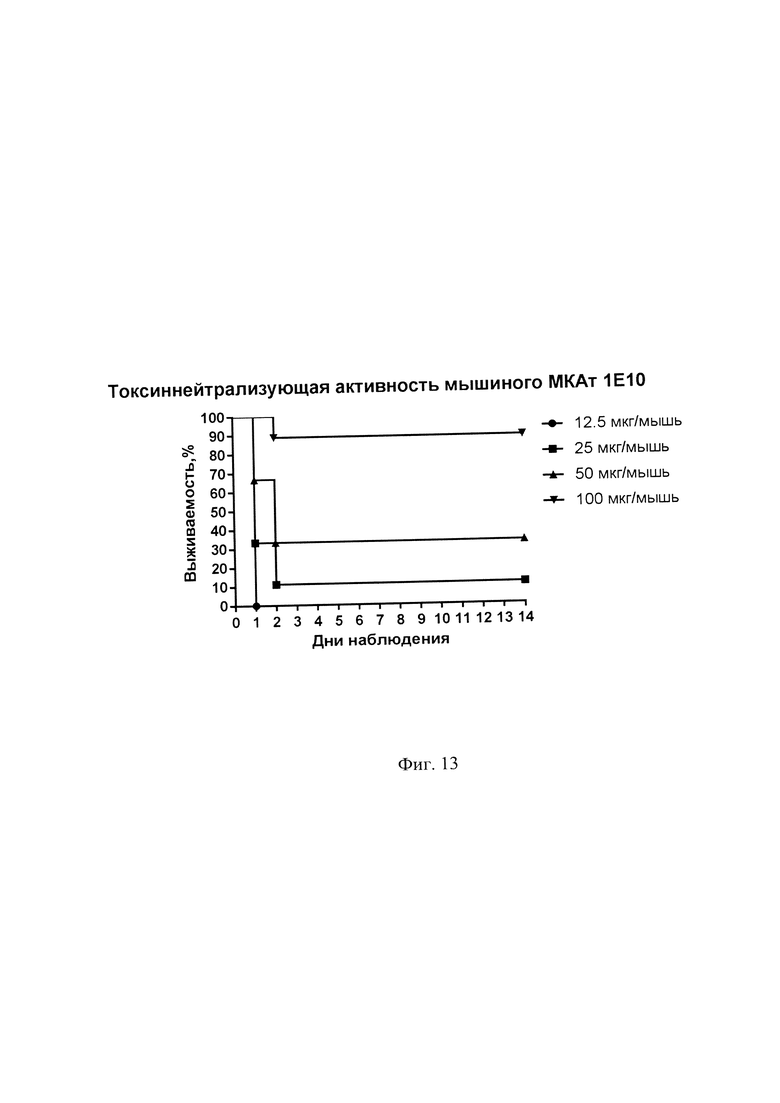
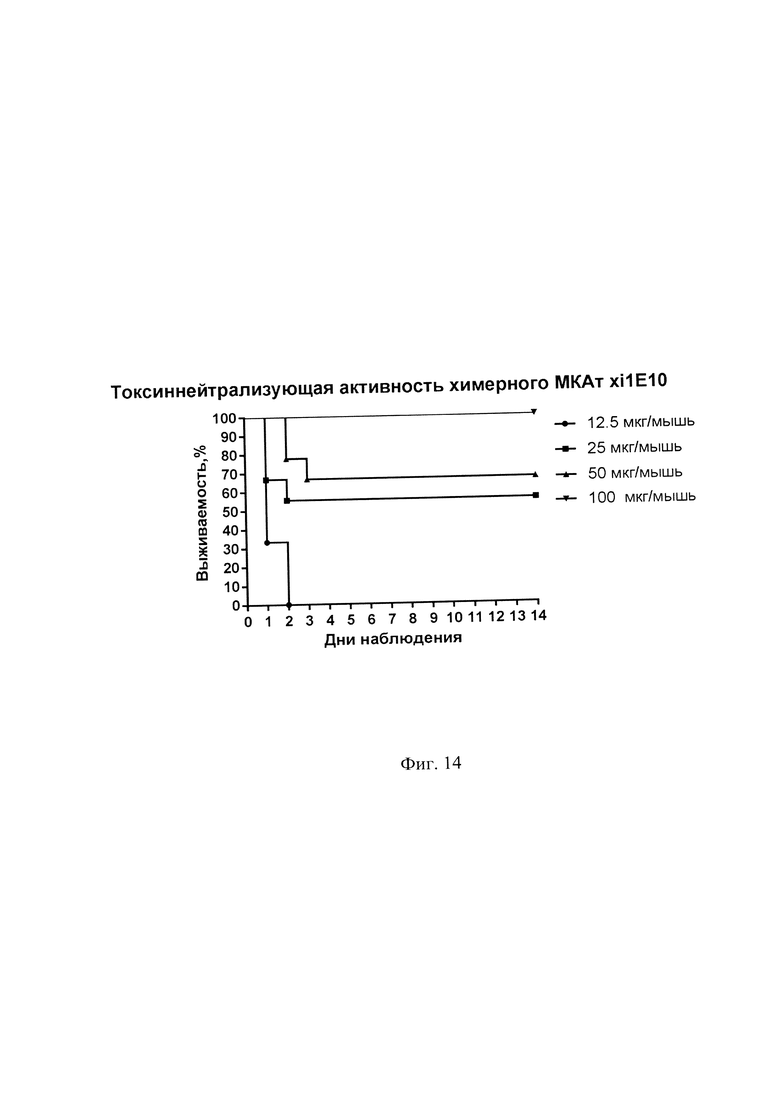
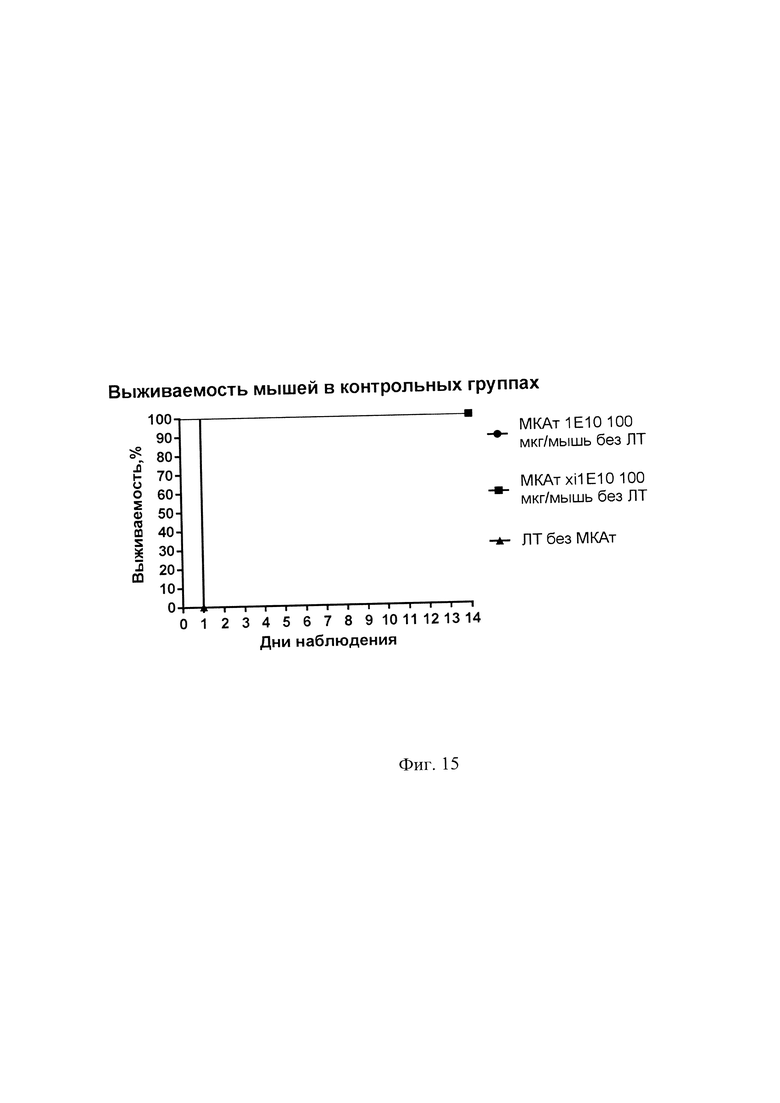
Предложен штамм гибридных культивируемых клеток животных Mus musculus 1E10, продуцент мышиного моноклонального антитела 1Е10, которое специфически связывается с протективным антигеном Bacillus anthracis в области IV домена. Указанный штамм депонирован в «ГКПМ-Оболенск» ФБУН ГНЦ «Государственный научный центр прикладной микробиологии и биотехнологии» под № Н-89. Предложено мышиное моноклональное антитело 1Е10, нейтрализующее летальный токсин Bacillus anthracis, специфически связывающееся с протективным антигеном Bacillus anthracis в области IV домена. Антитело 1Е10 характеризуется константой равновесной диссоциации 9,1564×10-10 М и представлено двумя идентичными тяжелыми и двумя идентичными легкими цепями, образующими молекулу IgG1, с аминокислотными последовательностями легкой цепи SEQ ID NO: 1 и Fd фрагмента тяжелой цепи SEQ ID NO: 2. Также предложено химерное рекомбинантное моноклональное антитело xi1E10, нейтрализующее летальный токсин Bacillus anthracis, полученное путем замены константных доменов легкой цепи и тяжелой цепи вышеуказанного антитела 1Е10 на аналогичные области иммуноглобулина G1 человека, экспрессируемое в эукариотических клетках линии ExpiCHO-S, специфически связывающееся с протективным антигеном Bacillus anthracis в области IV домена. Антитело xi1E1 характеризуется константой равновесной диссоциации 1,2663×10-9 М и представлено двумя идентичными тяжелыми и двумя идентичными легкими цепями с аминокислотными последовательностями легкой цепи SEQ ID NO: 3 и тяжелой цепи SEQ ID NO: 4. Группа изобретений обеспечивает получение антител с высокоаффинным и высокоспецифичным связыванием с протективным антигеном Bacillus anthracis в области IV домена, имеющим нейтрализующее действие в отношении летального токсина Bacillus anthracis. 3 н.п. ф-лы, 15 ил., 11 пр.
1. Штамм гибридных культивируемых клеток животных Mus musculus 1E10, продуцент мышиного моноклонального антитела 1Е10, которое специфически связывается с протективным антигеном Bacillus anthracis в области IV домена, депонированный в «ГКПМ-Оболенск» ФБУН ГНЦ «Государственный научный центр прикладной микробиологии и биотехнологии» под № Н-89.
2. Мышиное моноклональное антитело 1Е10, нейтрализующее летальный токсин Bacillus anthracis, специфически связывающееся с протективным антигеном Bacillus anthracis в области IV домена, также характеризующееся константой равновесной диссоциации 9,1564×10-10 М, представленное двумя идентичными тяжелыми и двумя идентичными легкими цепями, образующими молекулу IgG1, с аминокислотными последовательностями легкой цепи SEQ ID NO: 1 и Fd фрагмента тяжелой цепи SEQ ID NO: 2.
3. Химерное рекомбинантное моноклональное антитело xi1E10, нейтрализующее летальный токсин Bacillus anthracis, полученное путем замены константных доменов легкой цепи и тяжелой цепи мышиного антитела 1Е10 по п. 2 на аналогичные области иммуноглобулина G1 человека, экспрессируемое в эукариотических клетках линии ExpiCHO-S, специфически связывающееся с протективным антигеном Bacillus anthracis в области IV домена, также характеризующееся константой равновесной диссоциации 1,2663×10-9 М, представленное двумя идентичными тяжелыми и двумя идентичными легкими цепями с аминокислотными последовательностями легкой цепи SEQ ID NO: 3 и тяжелой цепи SEQ ID NO: 4.
| МАРЬИН М.А | |||
| И ДР | |||
| Антитела к протективному антигену Вacillus anthracis, обеспечивающие защиту против сибиреязвенных токсинов | |||
| Дальневосточный Журнал Инфекционной Патологии, N37, 2019 г., с | |||
| Дорожная спиртовая кухня | 1918 |
|
SU98A1 |
| Штамм гибридных культивируемых клеток H.sapiens/Mus musculus 8D4E9-Ba-LF-продуцент человеческих моноклональных антител против летального фактора возбудителя сибирской язвы | 2018 |
|
RU2699193C1 |
| РЯБКО А.К | |||
| И ДР | |||
| Оценка механизмов токсиннейтрализующей активности антител, специфичных к протективному антигену Bacillus | |||
Авторы
Даты
2021-03-22—Публикация
2020-07-03—Подача