СВЯЗАННЫЕ ЗАЯВКИ
Согласно настоящей заявке испрашивается приоритет в соответствии с предварительной заявкой №62/323684, поданной 16 апреля 2016 г. под названием «СПОСОБ УСИЛЕНИЯ БИОЛОГИЧЕСКОЙ АКТИВНОСТИ РЕКОМБИНАНТНОГО АДЕНОАССОЦИИРОВАННОГО ВИРУСА, ПРОДУЦИРУЕМОГО БАКУЛОВИРУСНОЙ СИСТЕМОЙ», в соответствии с 35 U.S.С. §119(e), полное содержание которой включено в настоящий документ посредством ссылки.
ГОСУДАРСТВЕННАЯ ПОДДЕРЖКА
Настоящее изобретение было сделано при государственной поддержке Национального института здоровья в соответствии с HL097088. Государство обладает определенными правами на настоящее изобретение.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
Аденоассоциированный вирус (AAV) стал одним из наиболее перспективных вирусных векторов для генной терапии человека. Рекомбинантный аденоассоциированный вирус (rAAV) оценивается в клинических исследованиях по генной терапии гемофилии В, злокачественной меланомы, кистозного фиброма и других заболеваний. Несмотря на то, что продукция rAAV в промышленных масштабах с помощью культур клеток млекопитающих в прошлом была проблематичной, было показано, что системы продукции на основе AAV с использованием клеток насекомых являются эффективной альтернативой. Однако различное клеточное окружение создало проблемы в отношении сборки вирусных частиц некоторых серотипов AAV.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
В настоящем документе предусмотрены композиции и способы, связанные с продукцией rAAV в клетках-хозяевах-продуцентах (например, в клетках насекомых). Биологическая активность (например, инфицирующая способность) частиц rAAV частично зависит от состава капсида вириона, например, стехиометрических соотношений капсидных белков VP1, VP2 и VP3. Некоторые серотипы rAAV (например, AAV5, AAV8 и AAV9), полученные в гетерологичной системе, такой как клетки насекомых (например, клетки Sf9), характеризуются патологическими уровнями экспрессии VP1. При сборке вирусов патологическая экспрессия VP1 приводит к продукции вирусных частиц, состоящих из неправильных соотношений капсидных белков, что затрудняет способность частиц эффективно трансдуцировать клетки. Способы получения пригодных стехиометрических соотношений капсидных белков осложняются результатами, указывающими на то, что избыток VP1 может нарушить способность клетки насекомого эффективно упаковывать вирусную частицу, в то время как низкие уровни VP1 могут приводить к неэффективной трансдукции. Таким образом, для систем продукции rAAV на основе клеток насекомых остается проблемой применение существующих в настоящее время методик для получения повышенных количеств инфицирующих частиц, чтобы при этом отсутствовало отрицательное влияние на сборку частиц.
В способах и композициях, описанных в настоящем документе, предусмотрены рекомбинантные гены VP1, содержащие модифицированную последовательность Козак, ассоциированную с участком инициации трансляции VP1. Рекомбинантные гены VP1, описанные в настоящей заявке, пригодны для продукции инфицирующих частиц rAAV, содержащих ген, представляющий интерес (например, для последующего исследования и/или терапевтических применений). В соответствии с некоторыми вариантами осуществления рекомбинантные гены VP1 в настоящей заявке могут быть использованы для продукции частиц rAAV одного или нескольких серотипов в клетках насекомых. В соответствии с некоторыми вариантами осуществления способы и композиции, описанные в настоящей заявке, могут быть использованы для скрининга и модификации рекомбинантных генов VP1 одного или нескольких серотипов, которые пригодны для продукции частниц rAAV одного или нескольких серотипов в клетках насекомых и/или в других клетках-продуцентах, представляющих интерес (например, в клетках млекопитающих). В соответствии с некоторыми вариантами осуществления рекомбинантные гены одного или нескольких VP1 могут быть пригодными для продукции стабильных и/или инфицирующих частиц rAAV, которые содержат рекомбинантные геномы, представляющие интерес, при сравнительно высоких частотах и/или при сравнительно низких уровнях ошибочно упакованных нуклеиновых кислот.
В соответствии с некоторыми аспектами в настоящем раскрытии предусмотрены композиции и способы, пригодные в продукции rAAV, происходящего из серотипа AAV5. В соответствии с некоторыми аспектами в настоящем раскрытии предусмотрены композиции и способы, пригодные в продукции rAAV, происходящего из серотипа AAV8. В соответствии с еще одними аспектами в настоящем раскрытии предусмотрены композиции и способы, пригодные в продукции rAAV, происходящего из серотипа AAV9
В соответствии с некоторыми аспектами нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 2-11 (Таблица 1). В соответствии с некоторыми вариантами осуществления модифицированная последовательность Козак представляет собой SEQ ID NO: 8 или SEQ ID NO: 9. В соответствии с некоторыми последовательностями капсидные белки VP1, VP2 и VP3 происходят из серотипа AAV5. В соответствии с некоторыми вариантами осуществления капсидные белки VP1, VP2 и/или VP3 представляют собой вариантные капсидные белки AAV5 (например, они кодируются последовательностью, содержащей одну или несколько мутаций, и которая кодирует капсидный белок, имеющий одну или несколько аминокислотных замен относительно соответствующего капсидного белка дикого типа).
В соответствии с некоторыми вариантами осуществления нуклеиновая кислота дополнительно содержит промоторную последовательность. В соответствии с некоторыми вариантами осуществления промоторная последовательность представляет собой промоторную последовательность полиэдрина (polh). В соответствии с некоторыми вариантами осуществления модифицированная последовательность Козак содержит кодон инициации трансляции капсидного белка VP1. В соответствии с некоторыми вариантами осуществления кодон инициации трансляции капсидного белка VP1 представляет собой AUG. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота упакована в вирусную частицу (например, бакуловирусную частицу). В соответствии с некоторыми вариантами осуществления в настоящем раскрытии предусмотрена клетка насекомого, содержащая нуклеиновую кислоту.
В соответствии с некоторыми вариантами осуществления настощяее раскрытие связано со способом продукции rAAV в клетке насекомого, в котором rAAV происходит из серотипа AAV5, причем способ включает (а) трансфекцию клетки насекомого (i) бакуловирусом, содержащим нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV5, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 2-11, (ii) бакуловирусом, содержащим нуклеотидную последовательность, кодирующую белок Rep AAV, (iii) бакуловирусом, содержащим нуклеотидные последовательности двух инвертированных концевых повторов (ITR) AAV, фланкирующие ген, представляющей интерес, функционально связанный с промоторной последовательностью; (b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и (с) извлечение rAAV из клетки насекомого. В соответствии с некоторыми вариантами осуществления клетка насекомого представляет собой клетку Sf9.
В соответствии с некоторыми аспектами нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 13-32 (Таблица 2). В соответствии с некоторыми вариантами осуществления модифицированная последовательность Козак представляет собой SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29, SEQ ID NO: 30, SEQ ID NO: 31 или SEQ ID NO: 32. В соответствии с некоторыми последовательностями капсидные белки VP1, VP2 и VP3 происходят из серотипа AAV8. В соответствии с некоторыми вариантами осуществления капсидные белки VP1, VP2 и/или VP3 представляют собой вариантные капсидные белки AAV8 (например, они кодируются последовательностью, содержащей одну или несколько мутаций, и которая кодирует капсидный белок, имеющий одну или несколько аминокислотных замен относительно соответствующего капсидного белка дикого типа).
В соответствии с некоторыми вариантами осуществления нуклеиновая кислота дополнительно содержит промоторную последовательность. В соответствии с некоторыми вариантами осуществления промоторная последовательность представляет собой промоторную последовательность полиэдрина (polh). В соответствии с некоторыми вариантами осуществления модифицированная последовательность Козак содержит кодон инициации трансляции капсидного белка VP1. В соответствии с некоторыми вариантами осуществления кодон инициации трансляции капсидного белка VP1 представляет собой AUG. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота упакована в вирусную частицу (например, бакуловирусную частицу). В соответствии с некоторыми вариантами осуществления в настоящем раскрытии предусмотрена клетка насекомого, содержащая нуклеиновую кислоту.
В соответствии с некоторыми вариантами осуществления настощяее раскрытие связано со способом продукции rAAV в клетке насекомого, в котором rAAV происходит из серотипа AAV8, причем способ включает (а) трансфекцию клетки насекомого (i) бакуловирусом, содержащим нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV8, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 13-32, (ii) бакуловирусом, содержащим нуклеотидную последовательность, кодирующую белок Rep AAV, (iii) бакуловирусом, содержащим нуклеотидные последовательности двух ITR AAV, фланкирующие ген, представляющей интерес, функционально связанный с промоторной последовательностью; (b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и (с) извлечение rAAV из клетки насекомого. В соответствии с некоторыми вариантами осуществления клетка насекомого представляет собой клетку Sf9.
В соответствии с некоторыми аспектами нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 34-45 (Таблица 3). В соответствии с некоторыми вариантами осуществления модифицированная последовательность Козак представляет собой SEQ ID NO: 35, SEQ ID NO: 36, SEQ ID NO: 38 или SEQ ID NO: 42. В соответствии с некоторыми последовательностями капсидные белки VP1, VP2 и VP3 происходят из серотипа AAV9. В соответствии с некоторыми вариантами осуществления капсидные белки VP1, VP2 и/или VP3 представляют собой вариантные капсидные белки AAV9 (например, они кодируются последовательностью, содержащей одну или несколько мутаций, и которая кодирует капсидный белок, имеющий одну или несколько аминокислотных замен относительно соответствующего капсидного белка дикого типа).
В соответствии с некоторыми вариантами осуществления нуклеиновая кислота дополнительно содержит промоторную последовательность. В соответствии с некоторыми вариантами осуществления промоторная последовательность представляет собой промоторную последовательность полиэдрина (polh). В соответствии с некоторыми вариантами осуществления модифицированная последовательность Козак содержит кодон инициации трансляции капсидного белка VP1. В соответствии с некоторыми вариантами осуществления кодон инициации трансляции капсидного белка VP1 представляет собой AUG. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота упакована в вирусную частицу (например, бакуловирусную частицу). В соответствии с некоторыми вариантами осуществления в настоящем раскрытии предусмотрена клетка насекомого, содержащая нуклеиновую кислоту.
В соответствии с некоторыми вариантами осуществления настоящее раскрытие связано со способом продукции rAAV в клетке насекомого, в котором rAAV происходит из серотипа AAV9, причем способ включает (а) трансфекцию клетки насекомого (i) бакуловирусом, содержащим нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV9, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 34-45, (ii) бакуловирусом, содержащим нуклеотидную последовательность, кодирующую белок Rep AAV, (iii) бакуловирусом, содержащим нуклеотидные последовательности двух ITR AAV, фланкирующие ген, представляющей интерес, функционально связанный с промоторной последовательностью; (b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и (с) извлечение rAAV из клетки насекомого. В соответствии с некоторыми вариантами осуществления клетка насекомого представляет собой клетку Sf9.
В соответствии с некоторыми вариантами осуществления в настоящей заявке предусмотрена библиотека нуклеиновых кислот, содержащая нуклеотидную последовательность, кодирующую модифицированную последовательность Козак, содержащую кодон инициации трансляции капсидного белка VP1 AAV, и вариации нуклеотидных последовательностей из 1-6 нуклеотидов, расположенных непосредственно выше кодона инициации трансляции VP1, и/или вариации нуклеотидных последовательностей из 1-2 нуклеотидов, расположенных непосредственно ниже кодона инициации трансляции VP1. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота содержит XXXXXX(ATG), где (ATG) представляет собой кодон инициации трансляции VP1. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота содержит XXXXXX(AUG), где (AUG) представляет собой кодон инициации трансляции VP1. В соответствии с некоторыми вариантами осуществления X представляет собой, например, любую нуклеотидную последовательность, которая отличается от встречающегося в природе нуклеотида в этом положении в гене VP1 дикого типа в случае серотипа AAV, представляющего интерес.
Эти и другие аспекты описаны более подробно в следующем описании и прилагаемых примерах и фигурах.
Краткое описание чертежей
Следующие чертежи образуют часть настоящего описания и включены, чтобы дополнительно продемонстрировать определенные аспекты настоящего раскрытия, которое можно лучше понять посредством ссылки на один или несколько из этих чертежей в сочетании с подробным описанием конкретных вариантов осуществления, представленных в настоящем документе.
На Фиг. 1 представлена неограничивающая схема вспомогательного нуклеотида для сборки капсида, кодирующего VP1, VP2 и VP3, и кодируемые капсидные белки.
На Фиг. 2А представлен пример вестерн-блоттинга rAAV5, упакованного в клетки Sf9.
На Фиг. 2В представлен пример вестерн-блоттинга rAAV8, упакованного в клетки Sf9.
На Фиг. 2С представлен пример вестерн-блоттинга rAAV9, упакованного в клетки Sf9.
На Фиг. 3 представлена неограничивающая нуклеотидная последовательность консенсусного аттенуированного элемента Козак выше гена, участвующего в образовании капсидного белка VP1, сконструированная для экспрессии в клетках насекомых.
На Фиг. 4A-4D представлены неограничивающие примеры композиций на основе капсидных белков частиц rAAV, продуцируемых в клетках Sf9. На Фиг. 4А представлен неограничивающий пример схемы кассеты экспрессии на основе генов rep и cap. На Фиг. 4В представлен пример прямой связи между экспрессией белка VP1 rAAV5 и его относительной эффективностью участка инициации трансляции (TIS) Козак VP1 на основании вестерн-блоттинга капсидных белков, выделенных из десяти отдельных клеточных линий, включающих стабильно интегрированные кассеты экспрессии на основе гена cap. Относительные эффективности (%) TIS для каждой конструкции на основе гена VP1 капсида представлены ниже под соответствующей линией. На Фиг. 4С представлены примеры композиций на основе капсидных белков rAAV5, очищенные от клеток HEK 293 и Sf9: SDS-белковый гель-анализ дважды очищенного в йодиксаноле rAAV5-GFP, непосредственно визуализируемого с помощью коротковолновой УФ-фотоактивации (технология без содержания красителей, Bio-Rad). (*) обозначает более медленную миграцию полосы, часто наблюдаемой в образцах rAAV5, очищенных от клеток HEK 293, и которая была исключена из количественного анализа VP2. На Фиг. 4D представлены примеры композиций на основе капсидных белков rAAV9, очищенные от клеток HEK 293 и Sf9: Анализ является таким же самым, как и в панели на Фиг. 4С.
На Фиг. 5A-5D обозначен анализ MALDI-TOF стехиометрического состава капсидных белков VP1, VP2 и VP3 AAV5. На Фиг. 5А представлена аминокислотная последовательность (SEQ ID NO: 55) капсида AAV5 с уникальными для VP1 N-концами, обозначенная пунктирной линией, уникальными для VP2 N-концами, обозначенная точечно-пунктирной линией и обычными для VP3 С-концами, обозначенная линией черного цвета. Опускающиеся стрелки указывают на соответствующие белки. Триптические пептиды, выбранные для МС-анализа, подчеркнуты и представлены ниже возле их соответствующих наблюдаемых масс. Триптические пептиды соответствуют SEQ ID NO: 56, 48 и 57, сверху вниз. На Фиг. 5В представлена спектр MALDI-TOF-MS всех трех триптических пептидов rAAV5, расщепленных в H218O. Кольцевой пептид представляет собой один типичный пример из трех проанализированных. На Фиг. 5С представлены два накладывающихся спектра MALDI-TOF MS того же самого триптического пептида TWVLPSYNNHQYR (SEQ ID NO: 48), происходящего из гель-полосы VP1, расщепляемой трипсином, приготовленным в воде 16О, или из гель-полосы VP3, расщепляемой трипсином, приготовленным в воде 18O. Вода 18O включает два атома 18O на С-конце пептида, смещая таким образом массу на 4 атомные единицы массы (а.е.м.). Эти продукты расщепления наносили/анализировали отдельно и спектры накладывались, показывая полное включение 18O в пептид VP3. На Фиг. 5D представлены изотопные «пальцы» того же самого пептида, происходящего из VP1 или VP3 после того, как продукты расщепления смешивали в соотношениях 1:1 с расчетом относительного содержания.
На Фиг. 6A-6F представлен трекинг-анализ наночастиц с размерами и титрами rAAV9 (Фиг. 6А-С) или rAAV5 (Фиг. 6D-F), образованных в клетках HEK 293 (Фиг. 6А, 6D) или клетках Sf9 (Фиг. 6В, 6Е). На Фиг. 6А, 6В, 6С и 6D представлено графическое представление алгоритма корректировки конечного трек-расстояния (FTLA), среднее из трех независимых видеозахватов комплексов rAAV/частиц на основе нанозолота, при этом каждый из них записывали в течение 30 секунд для каждого образца. На Фиг. 6С и 6F представлены рассчитанные соотношения полных/всех частиц для каждого препарата. Количества возле пиков показывают рассчитанные размеры комплексов rAAV/частиц на основе нанозолота. Пики меньшего размера с более крупными диамитерами представляют собой агрегированные димеры и тримеры частиц rAAV. На Фиг. 6С и 6F представлены рассчитанные соотношения ДНК-содержащих частиц AAV к их общему числу в соответствующих исходных культурах вирусов.
На Фиг. 7А-7С представлена экспрессия люциферазы и mApple после инъекции в область полосатого тела rAAV5, происходящего из HEK 293 или Sf9 В8. На Фиг. 7А представлены изображения теплокарты, которые показывают биолюминесцентный (BLI) сигнал, выявляемый через 3 недели после инъекций AAV. На шкале псевдоокрашивания представлена интенсивность света, испускаемого при количестве импульсов. Макс. и Мин. представляют собой максимальное и минимальное количество импульсов соответственно. На Фиг. 7В представлен график, на котором показана интенсивность BLI, выраженная в виде общего количества импульсов, в виде среднего ± SEM (n=4 на группу). Анализ с применением критерия Манна-Уитни показывает значимую разницу между группами (р=0,0286, односторонний критерий). На Фиг. 7С показана флуоресценция mApple в фронтальных срезах головного мозга, выявленная с помощью лазерного сканера с переменным режимом (черное и белое, ч/б изображения) или флуоресцентной микроскопии (цветное изображение).
На Фиг. 8A-8D представлено распределение считываемых фрагментов NGS кассеты rAAV-Bac-UF26 и ее непосредственных соединений. На Фиг. 8А представлен охват считываемых фрагментов Illumina кассеты rAAV5-Bac-UF26, упакованной в клетки HEK 293, численно нормализованных с помощью деления числа считываемых фрагментов в каждом положении нуклеотида на общее число считываемых фрагментов упомянутой последовательности. Графики охвата последовательностей для двойных образцов указаны стрелками в случае, где не использовали ПЦР (без использования ПЦР 1 и без использования ПЦР 2), и в случае использования ПЦР (ПЦР 1 и ПЦР 2). Снижение охвата между кассетой rAAV и прилегающими последовательностями отражает графическое представление ограничений биоинформационного анализа, а не фактическое снижение охвата последовательностей. Представленный ниже график представляет собой аннотированную карту упомянутой кассеты rAAV-Bac-UF26, приведенной к шкале последовательностей в панелях (Фиг. 8А) и (Фиг. 8В). Последовательности, непосредственно прилегающие к кассете rAAV в бактериальной плазмидной ДНУ заштрихованы более светлым оттенком серого. На Фиг. 8В представлен охват считываемых фрагментов Illumina кассеты rAAV5-Bac-UF26, упакованной в клетки Sf9 В8. Последовательности с высокими содержанием GC в кассете rAAV заштрихованы более темным оттенком серого. На Фиг. 8С представлен увеличенный масштаб на основе панелей на Фиг. 8А и Фиг. 8В последовательностей, непосредственно прилегающих к левому ITR rAAV как в клетках HEK 293, так и в клетках Sf9 В8. На Фиг. 8D представлен увеличенный масштаб на основе панелей на Фиг. 8А и Фиг. 8В последовательностей, непосредственно прилегающих к правому ITR rAAV как в клетках HEK 293, так и в клетках Sf9 В8.
На Фиг. 9А-9В представлено распределение SNP, идентифицированных в кассете rAAV5-Bac-UF26, включенной в капсид с помощью клеток HEK 293 по сравнению с клетками Sf9 В8. На Фиг. 9А на графике представлены замены четырех остатков A, G, С и Т при считывании в определенном положении по отношению к эталонной последовательности из базы данных pTR-Bac-UF26. Относительные соотношения SNP (Y-оси) определяли в виде доли от всего числа считываемых фрагментов в определенном положении с заменой к общей глубине секвенирования в том же самом положении. Глубина секвенирования представляет собой сумму фрагментов считывания последовательностей эталонного и альтернативного нуклеотида в определенном положении, за исключением вставок/делеций (indel). Анализ вариантов SNP включал в себя положительный контроль, образец плазмиды pTR-Bac-UF26. Соответственно, нулевой уровень SNP представляет собой остаток, идентичный эталону базы данных, а положительное значение означает вновь приобретенный SNP по отношению к родительской плазмидной последовательности из базы данных. Участки с высоким содержанием GC заштрихованы. На Фиг. 9В представлена аннотированная карта упомянутой кассеты rAAV-Bac-UF26, приведенной к шкале последовательности на Фиг. 9А.
На Фиг. 10А-10В представлены аннотированные графические карты кассет rAAV, упакованных в капсиды rAAV5 и rAAV9.
На Фиг. 11 представлены сканированные профили препаратов на основе rAAV5, разделенных с помощью SDS-PAGE электрофореза (Фиг. 4С). Заштрихованные области указывают на площади под кривыми (AUC), изображенными для количественных оценок. (*) указывает пик, исключенный из анализа.
На Фиг. 12А-12В представлены анализы трансдукции in vitro rAAV-UF50-BC, упакованного в капсиды rAAV5 или rAAV9. На Фиг. 12А представлена флуоресцентно-активируемая клеточная сортировка (FACS) и ее графическая количественная оценка rAAV5-UF50-BC. На Фиг. 12В представлена флуоресцентно-активируемая клеточная сортировка (FACS) и ее графическая количественная оценка rAAV9-UF50-BC.
На Фиг. 13 представлен неограничивающий пример схематической технологической карты получения ДНК для вектора rAAV5 в случае NGS.
На Фиг. 14А-14С представлены распределения библиотек NGS размеров фрагментов ДНК. Анализ библиотек NGS осуществляли с помощью высокочувствительной D5000 ScreenTape. На Фиг. 14А представлены полученные без использования ПЦР библиотеки, секвенированные непосредственно с помощью NGS: 1,2 - rAAV5/Sf9; 3,4 - rAAV5/HEK 293. На Фиг. 14В представлены полученные без использования ПЦР библиотеки, используемые для обогащения с помощью ПЦР с малым числом циклов: 5,6 - rAAV5/Sf9, 7,8 - rAAV5/HEK293. На Фиг. 14С представлены обогащенные ПЦР библиотеки: 9, 10 - rAAV5/Sf9; 11, 12 - rAAV5/HEK 293. Библиотеки 9-12 секвенировали непосредственно с помощью NGS.
На Фиг. 15 представлен неограничивающий пример схематической технологической карты обработки данных NGS.
Подробное раскрытие настоящего изобретения
В соответствии с аспектами настоящей заявки предусмотрены способы и композиции для продукции частиц rAAV в клетках-хозяевах-продуцентах (например, в клетках-продуцентах насекомых). В соответствии с некоторыми аспектами модифицированные последовательности инициации трансляции (TIS, также обозначаемые как последовательности Козак) ассоциированы со старт-кодоном VP1 (например, старт-кодоном ATG/AUG в случае рекомбинантного гена, кодирующего капсидный белок VP1) и включены в рекомбинантную нуклеиновую кислоту, которая кодирует капсидные белки VP1, VP2 и VP3 серотипа AAV, представляющего интерес. В соответствии с некоторыми вариантами осуществления способы и композиции, описанные в настоящей заявке, могут быть использованы для идентификации последовательностей Козак VP1, которые пригодны для продукции частиц эффективного rAAV в клетках-хозяевах-продуцентах (например, частиц rAAV, которые продуцируются с высоким выходом, которые являются инфицирующими, ДНК-содержащими, стабильными и т.д., или любая их комбинация). В соответствии с некоторыми вариантами осуществления частицы rAAV, продуцируемые с помощью способов и композиций, описанных в настоящей заявке, могут содержать ген, представляющий интерес (например, ген терапевтического значения), и могут быть использованы для терапевтических целей.
rAAV широко используется в качестве вектора для генной терапии/доставки ДНК-вакцин, однако продукция в промышленных масштабах инфицирующих rAAV для клинических приложений остается проблематичной, исходя из современной технологии. Капсид AAV состоит из трех капсидных белков, VP1, VP2 и VP3, получаемых в результате альтернативного сплайсинга и различной частоты использования кодона гена одного капсидного белка в геноме AAV. Последовательность VP3 является общей для всех трех сплайс-вариантов, a VP2 и VP1 имеют более длинные N-концевые последовательности, при этом VP1 является самый длинным капсидным белком (как показано в примере на Фиг. 1). По оценкам, соотношение VP1/VP2/VP3 в типичной капсидной частице составляет 1/1/10. Более того, по-видимому, отсутствует определенный стехиометрический состав VP1/VP2/VP3 и сборка является стохастической, поэтому относительное количество VP1/VP2/VP3, которое включено в капсид, зависит главным образом от уровней их относительной экспрессии в клетке-хозяине-продуценте (или в системе продукции in vitro). В композициях и способах, описанных в настоящей заявке, предусмотрены рекомбинантные капсидные гены, имеющие модифицированные последовательности Козак VP1 (например, окружающие кодон инициации трансляции ATG/AUG), которые транслируются в капсидные белки VP1, VP2 и VP3 в клетках-продуцентах в относительных количествах, которые пригодны для продукции инфицирующих частиц rAAV, содержащих рекомбинантные гены, представляющие интерес. В соответствии с некоторыми вариантами осуществления рекомбинантный капсидный ген не имеет соответствующих участков донора и/или акцептора сплайсинга для того, чтобы обеспечить естественный сплайсинг капсидного гена (например, в соответствии с некоторыми вариантами осуществления транскрипты на основе рекомбинантного капсидного гена не сплайсируются соответствующим образом или вообще в клетках-продуцентах). В соответствии с некоторыми вариантами осуществления для повышения ослабленного сканирования рибосом и получения необходимого соотношения VP1/VP2/VP3 на основе капсидного гена канонический кодон AUG, ассоциированный с аттенуированной последовательностью Козак использовали для участка начала трансляции VP1. Частицы rAAV, продуцируемые на основе модифицированного капсидного гена и заключающие в капсид ген, представляющий интерес, можно использовать для исследований, клинического тестирования и терапевтических целей. В соответствии с некоторыми вариантами осуществления модифицированные капсидные гены, описанные в настоящей заявке, могут быть использованы для производства частиц rAAV в промышленных масштабах.
В соответствии с некоторыми аспектами в настоящей заявке предусмотрены способы и композиции для скрининга и оценки конструкций экспрессии на основе генов капсида AAV для идентификации последовательностей, которые пригодны для продуцирования инфицирующих частиц rAAV. В соответствии с некоторыми вариантами осуществления множество различных конструкций экспрессии на основе генов капсида rAAV оценивают в отношении сборки частиц rAAV в клетке-хозяине, представляющей интерес. В соответствии с некоторыми вариантами осуществления различные конструкции экспрессии на основе генов капсида rAAV содержат одинаковый промотор, одинаковую кодирующую последовательность капсида AAV (например, кодирующую капсидные белки VP1, VP2 и VP3 любого серотипа, представляющего интерес, необязательно включающие одну или несколько мутаций капсида), но имеют различные последовательности Козак, ассоциированные с кодоном инициации трансляции VP1 (например, ATG/AUG). Как правило, кодоны инициации трансляции и окружающие последовательности инициации трансляции VP2 и VP3 являются не измененными (например, они сохраняются в виде своих природных последовательностей дикого типа). Однако, в соответствии с некоторыми вариантами осуществления в этих последовательностях могут быть выполнены одна или несколько изменения. В соответствии с некоторыми вариантами осуществления различные последовательности Козак представляют собой вариации последовательности в шести нуклеотидах, расположенных непосредственно выше кодона инициации трансляции VP1. В соответствии с некоторыми вариантами осуществления различные последовательности Козак включают в себя вариации последовательности в двух нуклеотидах, расположенных непосредственно ниже кодона инициации трансляции VP1. Соответственно, в соответствии с некоторыми вариантами осуществления различные последовательности Козак выбирают из библиотеки различных последовательностей Козак, которые представляют собой вариации нуклеотидных последовательностей в одном или нескольких положениях (например, 1, 2, 3, 4, 5 или 6 положениях) в шести нуклеотидах, расположенных непосредственно выше кодона инициации трансляции VP1, и/или одном или нескольких положениях (например, 1 или 2 из положений) в двух нуклеотидах, расположенных непосредственно после кодона инициации трансляции VP1. В соответствии с некоторыми вариантами осуществления различные последовательности Козак могут представлять собой все или подсовокупность из всех возможных вариаций последовательности Козак VP1. В соответствии с некоторыми вариантами осуществления вариации выполнены по отношению к природной последовательности Козак VP1 в случае капсидного гена серотипа AAV (например, серотипа 1, 2, 3, 3В, 4, 5, 6, 7, 8, 9, 10, 11, 12 и др.), который модифицируют. В соответствии с некоторыми вариантами осуществления оценивают различные последовательности Козак, имеющие определенный процент эффективности трансляции (например, по отношению к эталонной эффективности трансляции, например, по отношению к эффективности трансляции, ассоциированной с природной или консенсусной последовательностью Козак). В соответствии с некоторыми вариантами осуществления различные последовательности Козак представляют диапазоны эффективности трансляции между 25% и 50%, например, между 25% и 45%, например, между 30% и 40%, около 25%, около 30%, около 35%, около 40%, около 45%, около 50% или около других значений (например, промежуточных значений между любыми из вышеуказанных). В соответствии с некоторыми вариантами осуществления оценивали все последовательности Козак, имеющие необходимую эффективность трансляции или диапазон эффективностей трансляции. В соответствии с некоторыми вариантами осуществления оценивали подсовокупность последовательностей Козак, имеющих эффективность трансляции в необходимом процентном диапазоне. В соответствии с некоторыми вариантами осуществления оценивали последовательности Козак, представляющие процентные приросты (например, 1-5% приросты, например, приросты около 1%, 2%, 3%, 4% или 5%) в целевом диапазоне эффективностей трансляции. В соответствии с некоторыми вариантами осуществления различные последовательности Козак оценивали с помощью введения рекомбинантных нуклеиновых кислот, при этом каждая из них содержит рекомбинантный капсидный ген, имеющий отличающуюся последовательность Козак (например, ассоциированную со старт-кодоном VP1) в клетках-хозяевах, которые также экспрессируют другие вспомогательные гены (например, Rep и другие вспомогательные гены) и которые также содержат геном рекомбинантного AAV, представляющего интерес (например, контролирующий ген, например, кодирующий выявляемый маркер или ген терапевтического значения, представляющий интерес), фланкированную последовательностями ITR (таким образом, что ген, представляющий интерес, фланкированный последовательностями ITR, может быть упакован в частницы rAAV, содержащие капсидные белки, экспрессируемые на основе рекомбинантного капсидного гена). Клетки-хозяева могут представлять собой клетки любого типа (например, клетки-хозяева млекопитающих, насекомых или другие типы клеток-хозяев). Клетки-хозяева могут экспрессировать другие вспомогательные гены и/или содержать геном rAAV, представляющий интерес, в результате трансфекции плазмидами (например, с помощью одной или нескольких плазмид, которые экспрессируют другие вспомогательные гены, и/или которые содержат геном rAAV, представляющего интерес), трансдукции вирусами (например, с помощью одного или нескольких рекомбинантных вирусов, например, рекомбинантного бакуловируса, аденовируса, вируса герпеса или другого рекомбинантного вируса, имеющего рекомбинантные вирусные геномы, которые экспрессируют другие вспомогательные гены, и/или которые содержат геном rAAV, представляющий интерес), или геномной интеграции (например, с помощью одного или нескольких вспомогательных генов и/или генома rAAV, представляющего интерес). После введения различных конструкций на основе капсидного гена в клетки-хозяева, клетки инкубируют в условиях, при которых продуцируются частицы rAAV, и частицы rAAV выделяют и/или очищают и оценивают. Выделенные и/или очищенные частицы rAAV могут быть оценены на основе одного или нескольких факторов, в том числе без ограничения одного или нескольких из следующего.
a) Выход частиц rAAV. В соответствии с некоторыми вариантами осуществления выбирают аналогичный или более высокий выход, чем в случае эталонного rAAV, например, того же самого же серотипа (например, продуцируемого с помощью эталонной методики, например, в клетках HEK 293 или клетках Sf9).
b) Соотношение белков VP1/VP2/VP3 в частицах rAAV. В соответствии с некоторыми вариантами осуществления выбирают соотношение VP1/VP2/VP3, составляющее >0,5/>0,5/10. Соответственно, в соответствии с некоторыми вариантами осуществления соотношение VP1/VP3 составляет около или >0,5/10, например, около или >0,75/10, например, около или >1/10, например, около ил >1,25/10, например, около или >1,5/10, например, около или >1,75/10, или около 2/10, или любое их промежуточное соотношение. Аналогично, в соответствии с некоторыми вариантами осуществления соотношение VP2/VP3 составляет около или >0,5/10, например, около или >0,75/10, например, около или >1/10, например, около ил >1,25/10, например, около или >1,5/10, например, около или >1,75/10, или около 2/10, или любое их промежуточное соотношение.
c) Соотношение полных/пустых частиц rAAV. В соответствии с некоторыми вариантами осуществления соотношение полных/пустых частиц, которое является аналогичным или большим, чем эталонное соотношение (например, соотношение полных/пустых частиц rAAV, например, того же самого серотипа, продуцируемых с помощью эталонной методики, например, в клетках HEK 293 или клетках Sf9).
d) Эффективность трансдукции частиц rAAV. В соответствии с некоторыми вариантами осуществления выбирают аналогичную или более высокую эффективность трансдукции, чем в случае эталонного rAAV, например, того же самого серотипа (например, продуцируемого с помощью эталонной методики, например, в клетках HEK 293 или клетках Sf9). В соответствии с некоторыми вариантами осуществления эффективность трансдукции оценивают in vitro (например, в клетках, например, клетках HeLa, выращиваемых в культуре). В соответствии с некоторыми вариантами осуществления эффективность трансдукции оценивают in vivo (например, в животной модели, например, в модели на грызунах, такой как мышиной модели).
e) Точность упаковки ДНК в частицы rAAV. В соответствии с некоторыми вариантами осуществления выбирают аналогичную или более высокую точность упаковки ДНК (например, геномной ДНК rAAV по отношению к другой ДНК, например, ДНК хозяина или ДНК, фланкирующей геном rAAV в плазмидном или вирусном вектора в клетке-хозяине), чем в случае эталонного rAAV. В соответствии с некоторыми вариантами осуществления эталонный rAAV может представлять собой rAAV, например, того же самого серотипа, который продуцируется с помощью эталонной методики, например, в клетках HEK 293 или Sf9. В соответствии с некоторыми вариантами осуществления точность упаковки ДНК может быть оценена с помощью определения процента генома, не принадлежащего rAAV, который является упакованным. Это можно определить с помощью любого подходящего способа, например, с помощью секвенирования упакованной ДНК в частицах rAAV (например, с помощью NGS или других методик).
В качестве результата этого оценивания можно выбрать и использовать для продукции rAAV, представляющего интерес, в определенном типе клеток одну или несколько последовательностей Козак (и/или конструкций экспрессии на основе Сар, включающих последовательности Козак, расположенные выше кодона инициации трансляции VP1).
Соответственно, аспекты заявки относятся к способам и композициях, пригодным для продукции rAAV в клетках насекомых. В соответствии с некоторыми аспектами в настоящем раскрытии предусмотрены композиции на основе нуклеиновой кислоты, содержащие нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которых капсидные белки происходят из серотипа AAV9. В соответствии с некоторыми вариантами осуществления капсидные белки происходят из капсидных белков серотипа AAV5. В соответствии с некоторыми вариантами осуществления капсидные белки происходят из капсидных белков серотипа AAV8. В соответствии с некоторыми вариантами осуществления капсидные белки происходят из капсидных белков серотипа AAV9. В соответствии с некоторыми вариантами осуществления нуклеотидная последовательность дополнительно содержит промоторную последовательность. В соответствии с некоторыми вариантами осуществления промотор функционально связан с последовательностью, кодирующей модифицированную последовательность Козак и капсидные белки AAV.
Неограничивающий пример генетической структуры, содержащей нуклеиновую кислоту, предусмотренный в настоящем документе, представлен на Фиг. 1. Нуклеиновая кислота 100 содержит нуклеотидную последовательность, кодирующую модифицированную последовательность Козак 120 и капсидные белки VP1, VP2 и VP3 AAV 110. В AAV дикого типа в клетках млекопитающих капсидные белки транскрибируются в виде одной мРНК, которую можно сплайсировать с образованием двух изоформ мРНК. Такой посттранскрипционный процессинг и ослабленное сканирование рибосом способствует продукции трех отдельных капсидных белков: капсидного белка VP1 AAV 111, капсидного белка VP2 AAV 112, инициируемого с альтернативного кодона инициации ACG, и капсидного белка VP3 AAV 113. Кроме того, в соответствии с некоторыми вариантами осуществления отдельная открытая рамка считывания в той же самой нуклеотидной последовательности кодирует белок, активирующий сборку, 114. В соответствии с некоторыми вариантами осуществления промоторная последовательность 130 функционально связана с нуклеотидной последовательностью. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота содержит энхансер. В соответствии с некоторыми вариантами осуществления нуклеотидная последовательность включает в себя более одного промотора. В соответствии с некоторыми вариантами осуществления кассеты rAAV, упакованные в капсиды на основе rAAV, напоминают таковые, встречающиеся на Фиг. 10A и 10В.
Промотор «функционально связан» с нуклеотидной последовательностью, если промоторная последовательность контролирует и/или регулирует транскрипцию нуклеотидной последовательности. В соответствии с некоторыми вариантами осуществления предпочтительно использовать промоторы, которые являются активными в клетках насекомых. В соответствии с некоторыми вариантами осуществления промотор представляет собой полиэдриновый (polh) промотор. В соответствии с некоторыми вариантами осуществления промотор выбирают из группы, состоящей из р10, р35 и IE-1. Способы и методики для экспрессии чужеродных генов в клетках-хозяевах насекомых известны в данной области. Методика молекулярной разработки и экспрессии полипептидов в клетках насекомых описана, например, в Summers and Smith. 1986. А Manual of Methods for Baculovirus Vectors and Insect Culture Procedures, Texas Agricultural Experimental Station Bull. No. 7555, College Station, Tex.; Luckow. 1991, в Prokop et al., Cloning and Expression of Heterologous Genes in Insect Cells with Baculovirus Vectors' Recombinant DNA Technology and Applications, 97-152; King, L.A. and R.D. Possee, 1992, The baculovirus expression system, Chapman and Hall, United Kingdom; O'Reilly, D.R., L.K. Miller, V.A. Luckow, 1992, Baculovirus Expression Vectors: A Laboratory Manual, New York; W.H. Freeman and Richardson, C.D., 1995, Baculovirus Expression Protocols, Methods in Molecular Biology, volume 39; патенте США №4745051; US 2003148506 и WO 03/074714. Однако может быть использован любой другой промотор (например, любой природный, вариантный, синтетический, химерный, усеченный, конститутивный, индуцибельный, тканеспецифичный, видоспецифиченый и т.д. промотор или комбинации из двух или нескольких из вышеизложенного) в зависимости от типа клетки, используемой в качестве клетки-продуцента (например, насекомого, млекопитающего или другой).
В соответствии с некоторыми вариантами осуществления модифицированная последовательность Козак, описанная в настоящем документе, содержит кодон инициации трансляции капсидного белка VP1. В соответствии с некоторыми вариантами осуществления кодон инициации трансляции представляет собой AUG. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота, описанная в настоящем документе, дополнительно содержит вирусную частицу. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота содержится в вирусной частице (например, заключается в капсид с помощью вирусной частицы). В соответствии с некоторыми вариантами осуществления вирусная частица представляет собой частицу AAV. В соответствии с некоторыми вариантами осуществления вирусная частица представляет собой бакуловирусную частицу.
В соответствии с некоторыми аспектами в настоящем раскрытии предусмотрены способы продукции rAAV в клетке насекомого, включающие стадии (а) трансфекции клетки насекомого: (i) бакуловирусом, содержащим нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 2-11, 13-32 или 33-45, (ii) бакуловирусом, содержащим нуклеотидную последовательность, кодирующую белок Rep AAV, и (iii) бакуловирусом, содержащим нуклеотидные последовательности двух ITR AAV, фланкирующие ген, представляющей интерес, функционально связанный с промоторной последовательностью; (b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и (с) извлечение rAAV из клетки насекомого.
В соответствии с некоторыми вариантами осуществления в настоящем раскрытии предусмотрена клетка насекомого, при этом клетка насекомого содержит нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которой модифицированная последовательность Козак выбрана из SEQ ID NO: 2-11, 13-32 или 33-45. В соответствии с некоторыми вариантами осуществления нуклеиновая кислота интегрирована в геном клетки насекомого. В соответствии с некоторыми вариантами осуществления ген, кодирующий белок Rep, интегрирована в геном клетки насекомого. Таким образом, в соответствии с некоторыми вариантами осуществления в настоящем раскрытии предусмотрен способ продукции rAAV в клетке насекомого, включающий стадии (а) трансфекции клетки насекомого бакуловирусом, содержащим две нуклеотидные последовательности ITR AAV, фланкирующие ген, представляющий интерес, функционально связанный с промоторной последовательностью, при этом геном клетки насекомого содержит нуклеотид, описанный в настоящем документе; (b) культивирования клетки-насекомого в условиях, подходящих для продуцирования rAAV; и (с) извлечения rAAV из клетки насекомого.
Продукция rAAV в клетках насекомых были описана ранее (см., например, публикацию заявки на патент США № US 20120100606, который включен в настоящий документ посредством ссылки во всей своей полноте). Способы и композиции, предусмотренные в настоящем раскрытии, могут быть применены в любой клетке насекомого, которая обеспечивает репликацию AAV и которая поддерживается в культуре. Например, используемая клеточная линия может быть взята из клеточных линий Spodoptera frugiperda, drosophila или клеточных линий москитов (например, клеточных линий, происходящих из Aedes albopictus). В соответствии с некоторыми вариантами осуществления клетка насекомого восприимчива к бакуловирусной инфекции (например, Se301, SeIZD2109, SeUCR1, Sf9, Sf900+, Sf21, BTI-TN-5B1-4, MG-1, Tn368, HzAm1, Ha2302 и Hz2E5). Соответственно, способы и композиции, предусмотренные в настоящем документе, пригодны для продукции rAAV с помощью бакуловирусных векторов экспрессии (BEV).
BEV появились в качестве одной из наиболее разнообразных систем для продукции белка. Помимо основной продукции белка BEV использовали для более сложных задач, таких как синтез гетерологичных многобелковых комплексов и для сборки средств доставки для генной терапии, таких как rAAV. В соответствии с некоторыми вариантами осуществления в последней стратегии используются клетки насекомых, котрансфицированные тремя BEV, каждая из которых предусматривает вспомогательную функциональную возможность. Например, клетки насекомых котрансфицируют первым бакуловирусом, содержащим ген, представляющий интерес, фланкированный последовательностями инвертированных концевых повторов (ITR) AAV, вторым бакуловирусом, содержащим rep AAV, и третьим бакуловирусом, содержащим cap AAV. Ген cap AAV кодирует белок, активирующий сборку (ААР), и вирусные капсидные белки VP1, VP2 и VP3. Капсидные белки VP1, VP2 и VP3 транслируются из того же самого транскрипта, который можно посттранскрипционно сплайсировать в две изоформы мРНК различного размера. Посттранскрипционный сплайсинг в клетках млекопитающих происходит со скоростью, подходящей для получения распределения форм мРНК, которые приведут к экспрессии соответствующего стехиометрического соотношения капсидных белков VP1:VP2:VP3. Кроме того, в клетках насекомых продукция некоторых серотипов (например, AAV5, AAV8 и AAV9) приводит к образованию частиц, которые являются неэффективными в трансдукции клеток. Соответственно композиции и способы пригодны для модификации уровней экспрессии VP1 с повышением продукции rAAV в клетках насекомых или в любых других типах клеток-хозяев, представляющих интерес (например, клетках-хозяевах-продуцентах млекопитающих или клетках другого типа).
В соответствии с некоторыми аспектами в настоящем раскрытии предусмотрены композиции на основе нуклеиновых кислот, содержащие нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которых капсидные белки происходят из серотипа AAV9. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит кодон инициации трансляции капсидного белка VP1 AAV5 и дополнительные нуклеотиды, расположенные выше кодона инициации трансляции. В соответствии с некоторыми вариантами осуществления последовательность Козак дополнительно содержит нуклеотиды, расположенные ниже кодона инициации трансляции. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит нуклеотидную последовательность RNXTXT(ATG)NY (SEQ ID NO: 1), где (ATG) представляет собой кодон инициации трансляции VP1; R представляет собой нуклеотид С или Т; N представляет собой нуклеотид, выбранный из А, Т, G или С; X представляет собой G или Т; и Y представляет собой нуклеотид, выбранный из А, Т или С. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит нуклеотидную последовательность, приведенную в Таблице 1.
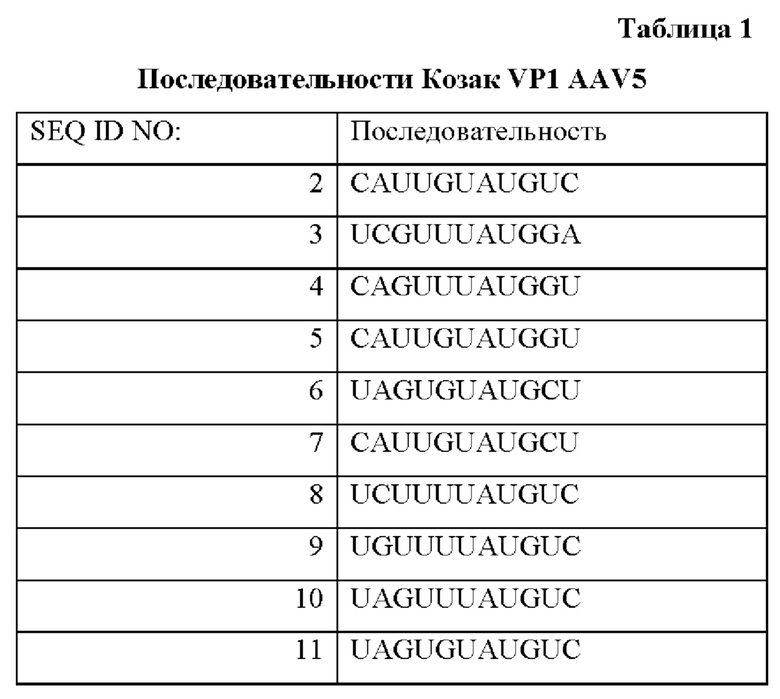
В соответствии с некоторыми аспектами в настоящем раскрытии предусмотрены композиции на основе нуклеиновых кислот, содержащие нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которых капсидные белки происходят из серотипа AAV8. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит кодон инициации трансляции капсидного белка VP1 AAV8 и дополнительные нуклеотиды, расположенные выше кодона инициации трансляции. В соответствии с некоторыми вариантами осуществления последовательность Козак дополнительно содержит нуклеотиды, расположенные ниже кодона инициации трансляции. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит нуклеотидную последовательность YNXRNR(ATG)XZ (SEQ ID NO: 12), где (ATG) представляет собой кодон инициации трансляции VP1; Y представляет собой нуклеотид, выбранный из А, Т или С; N представляет собой нуклеотид, выбранный из А, Т, G или С; X представляет собой нуклеотид G или Т; R представляет собой нуклеотид С или Т; и Z представляет собой нуклеотид G или С. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит нуклеотидную последовательность, приведенную в Таблице 2.
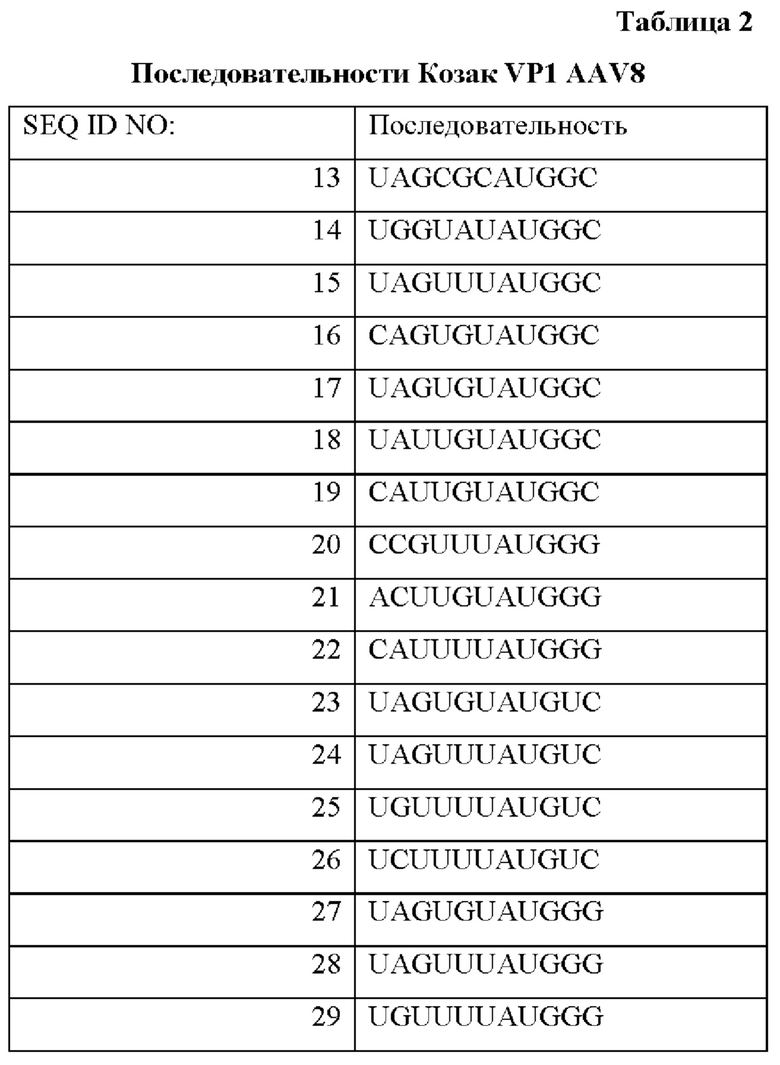
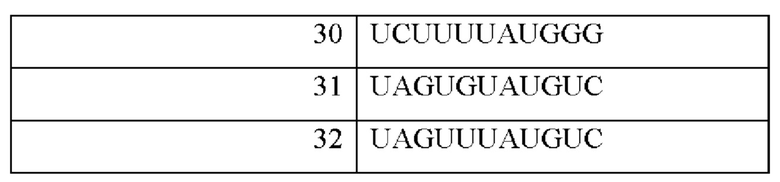
В соответствии с некоторыми аспектами в настоящем раскрытии предусмотрены композиции на основе нуклеиновых кислот, содержащие нуклеотидную последовательность, кодирующую модифицированную последовательность Козак и капсидные белки VP1, VP2 и VP3 AAV, в которых капсидные белки происходят из серотипа AAV9. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит кодон инициации трансляции капсидного белка VP1 AAV9 и дополнительные нуклеотиды, расположенные выше кодона инициации трансляции. В соответствии с некоторыми вариантами осуществления последовательность Козак дополнительно содержит нуклеотиды, расположенные ниже кодона инициации трансляции. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит нуклеотидную последовательность TNXTXT(ATG)XZ (SEQ ID NO: 33), где (ATG) представляет собой кодон инициации трансляции VP1; N представляет собой нуклеотид, выбранный из А, Т, С или G; X представляет собой нуклеотид G или Т; и Z представляет собой нуклеотид G или С. В соответствии с некоторыми вариантами осуществления последовательность Козак содержит нуклеотидную последовательность, приведенную в Таблице 3.
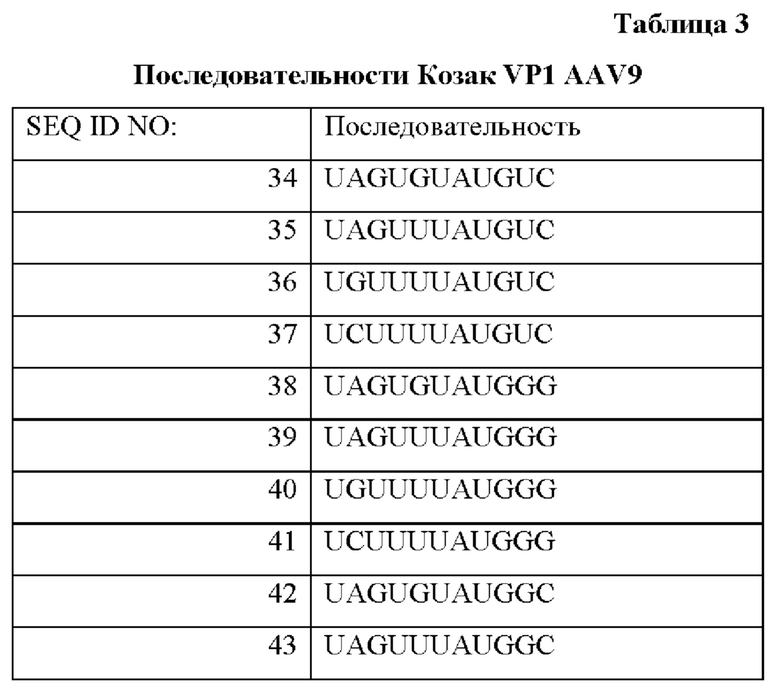

Следует понимать, что можно ссылаться на нуклеотид U или Т в зависимости от того, является ли нуклеиновая кислота, на которую ссылаются, молекулой РНК или ДНК.
Геном AAV дикого типа представляет собой однонитевую дезоксирибонуклеиновую кислоту (ssDNA), как положительно, так и отрицательно направленную. Геном содержит два инвертированных концевых повтора (ITR), по одному на каждом конце нити ДНК, и две открытые рамки считывания (ORF): rep и cap между ITR. ORF rep содержит четыре перекрывающихся гена, кодирующих белки Rep, необходимые для жизненного цикла AAV. ORF cap содержит перекрывающиеся гены, кодирующие капсидные белки: VP1, VP2 и VP3, которые взаимодействуют вместе с образованием вирусного капсида. VP1, VP2 и VP3 транслируются на основе одного транскрипта мРНК, который можно сплайсировать двумя различными путями: можно вырезать либо более длинный либо более короткий интрон, что приводит к образованию двух изоформ мРНК - изоформы мРНК размером ~2,3 кб и ~2,6 кб. Капсид образуется надмолекулярный комплекс примерно из 60 отдельных капсидных белковых субъединиц в безоболочечную Т-1 икосаэдральную решетку, способную защищать геном AAV. Зрелый капсид состоит из VP1, VP2 и VP3 (с молекулярными массами, составляющими примерно 87, 73 и 62 кДа соответственно) в примерном соотношении, составляющем приблизительно 1:1:10. Уровень VP1 по отношению к VP2 и VP3 можно скорректировать, как описано в настоящем документе, в целях получения эффективной продукции инфицирующих частиц в клетках насекомых. В соответствии с некоторыми вариантами осуществления соотношение VP1:VP2:VP3 находится в диапазоне (0,5-2):(0,5-2):10. В соответствии с некоторыми вариантами осуществления соотношение VP1:VP2:VP3 находится в диапазоне (0,5-1,5):(0,5-1,5):10.
Рекомбинантные частицы AAV (rAAV) могут содержать вектор на основе нуклеиновой кислоты, который может содержать как минимум: (а) один или несколько гетерологичных участков нуклеиновой кислоты, содержащих ген, представляющий интерес, кодирующий белок или пептид, представляющие интерес, или РНК, представляющую интерес (например, siRNA или microRNA); и (b) один или несколько участков, содержащих последовательности инвертированных концевых повторов (ITR) (например, последовательности ITR дикого типа или разработанные последовательности ITR), фланкирующие один или несколько участков нуклеиновой кислоты (например, гетерологичные участки нуклеиновой кислоты). В соответствии с некоторыми вариантами осуществления размер вектора на основе нуклеиновой кислоты составляет от 2 кб до 7 кб. В соответствии с некоторыми вариантами осуществления размер вектора на основе нуклеиновой кислоты составляет от 4 кб до 5 кб (например, от 4,2 до 4,7 кб). В соответствии с некоторыми вариантами осуществления вектор на основе нуклеиновой кислоты является кольцевым. В соответствии с некоторыми вариантами осуществления вектор на основе нуклеиновой кислоты является однонитевым. В соответствии с некоторыми вариантами осуществления вектор на основе нуклеиновой кислоты является двунитевым. В соответствии с некоторыми вариантами осуществления вектор на основе двунитевой нуклеиновой кислоты может представлять собой, например, аутокомплементарный вектор, который содержит участок вектора на основе нуклеиновой кислоты, который является комплементарным другому участку вектора на основе нуклеиновой кислоты, инициирующему образование двунитчатости вектора на основе нуклеиновой кислоты.
Последовательности ITR могут происходить из серотипа AAV (например, 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10) или могут происходить из более одного серотипа. Последовательности ITR и плазмиды, содержащие последовательности ITR, известны в данной области и коммерчески доступны (см., например, продукты и услуги, доступные от компании Vector Biolabs, Филадельфия, Пенсильвания; Cellbiolabs, Сан-Диего, Калифорния; Agilent Technologies, Санта-Клара, Калифорния; и Addgene, Кембридж, Массачусетс; и доставка генов в скелетные мышцы приводит к длительной экспрессии и системной доставке терапевтического белка, Kessler PD, Podsakoff GM, Chen X, McQuiston SA, Colosi PC, Matelis LA, Kurtzman GJ, Byrne BJ. Proc Natl Acad Sci USA. 1996 Nov 26; 93(24): 14082-7; и Curtis A. Machida. Methods in Molecular Medicine™. Viral Vectors for Gene Therapy Methods and Protocols. 10.1385/1-59259-304-6:201 © Humana Press Inc. 2003. Chapter 10. Targeted Integration by Adeno-Associated Virus. Matthew D. Weitzman, Samuel M. Young Jr., Toni Cathomen and Richard Jude Samulski; патенты США №№5139941 и 5962313, все из которых включены в настоящий документ посредством ссылки).
В соответствии с некоторыми вариантами осуществления частица rAAV или частица в препарате rAAV может быть любого серотипа AAV, в том числе любого производного или псевдотипа (например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 2/1, 2/5, 2/8, 2/9, 3/1, 3/5, 3/8 или 3/9). Используемый в настоящем документе серотип вирусного вектора на rAAV (например, частицы rAAV) относится к серотипу капсидных белков рекомбинантного вируса. Неограничивающие примеры производных и псевдотипов включают в себя rAAV2/1, rAAV2/5, rAAV2/8, rAAV2/9, гибрид AAV2-AAV3, AAVrh.10, AAVhu.14, AAV3a/3b, AAVrh32.33, AAV-HSC15, AAV-HSC17, AAVhu.37, AAVrh.8, CHt-P6, AAV2.5, AAV6.2, AAV2i8, AAV-HSC15/17, AAVM41, AAV9.45, AAV6(Y445F/Y731F), AAV2.5T, AAV-HAE1/2, AAV клон 32/83, AAVShH10, AAV2 (Y->F), AAV8 (Y733F), AAV2.15, AAV2.4, AAVM41 и AAVr3.45. Такие серотипы и производные/псевдотипы AAV, а также способы продукции таких производных/псевдотипов известны в данной области (см., например, Mol Ther. 2012 Apr; 20(4):699-708. doi: 10.1038/mt.2011.287. Epub 2012 Jan 24. The AAV vector toolkit: poised at the clinical crossroads. Asokan A1, Schaffer DV, Samulski RJ.). В соответствии с некоторыми вариантами осуществления частица rAAV представляет собой псевдотипированную частицу rAAV, которая содержит (а) вектор на основе нуклеиновой кислоты, содержащий ITR от одного серотипа (например, AAV2, AAV3) и (b) капсид, содержащий капсидные белки, происходящие от другого серотипа (например, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9 или AAV10). В соответствии с некоторыми вариантами осуществления способы, описанные в настоящем документе, могут быть использованы для продукции AAV любого серотипа или производного/псевдотипа (например, AAV5, AAV8, AAV9 или другого серотипа или производного/псевдотипа на их основе) в клетках насекомых. Другие способы продукции и использования псевдотипированных векторов rAAV известны в данной области (см., например, Duan et al., J. Virol., 75:7662-7671, 2001; Halbert et al., J. Virol., 74:1524-1532, 2000; Zolotukhin et al., Methods, 28:158-167, 2002; и Auricchio et al., Hum. Molec. Genet., 10:3075-3081, 2001). В соответствии с некоторыми вариантами осуществления одна или нескольких капсидных субъединиц (например, по отношению к соответствующей последовательности дикого типа) могут быть закодированы в рекомбинантном капсидном гене (например, рекомбинантном гене VP1, описанном в настоящей заявке).
Способы продуцирования частиц rAAV и векторов на основе нуклеиновой кислоты описаны в настоящем документе. Другие способы также известны в данной области и коммерчески доступны (см., например, Zolotukhin et al. Production and purification of serotype 1, 2, and 5 recombinant adeno-associated viral vectors. Methods 28 (2002) 158-167; и публикации заявок на патент США №№ US 20070015238 и US 20120322861, которые включены в настоящий документ посредством ссылки; а также плазмиды и наборы от АТСС и Cell Biolabs, Inc.). Например, плазмиду, содержащую вектор на основе нуклеиновой кислоты, можно сочетать с одной или несколькими плазмидами-помощниками, например, которые содержат ген rep (например, кодирующий Rep78, Rep68, Rep52 и Rep40) и ген cap (кодирующий VP1, VP2 и VP3, в том числе модифицированный участок VP3, описанный в настоящем документе), и трансфицировать в линию клеток-продуцентов таким образом, что частица rAAV может быть упакована и затем очищена.
В соответствии с некоторыми вариантами осуществления линия клеток-продуцентов происходит из клетки насекомого. Примеры клеток насекомых включают без ограничения клетки Sf9 (см., например, АТСС® CRL-1711™). Клетка-продуцент может содержать гены rep и/или cap (например, нуклеиновую кислоту, кодирующую VP1, VP2 и VP3 AAV, описанный в настоящем документе), которые кодируют белок Rep и/или Cap для применения в способе, описанном в настоящем документе. В соответствии с некоторыми вариантами осуществления упаковку осуществляют in vitro.
В соответствии с некоторыми вариантами осуществления способа, предусмотренного в настоящем документе, плазмида, содержащая вектор на основе нуклеиновой кислоты, сочетается с одной или несколькими плазмидами-помощниками, например, которые содержат ген rep первого серотипа и ген cap того же самого или другого серотипа, и трансфицируется в линии вспомогательных клеток таким образом, что частица rAAV упаковывается.
В соответствии с некоторыми вариантами осуществления одна или несколько плазмид-помощников включают в себя первую плазмиду-помощника, содержащую ген rep и ген cap, и вторую плазмиду-помощника, содержащую ген E1a, ген E1b, ген Е4, ген Е2а и ген VA. В соответствии с некоторыми вариантами осуществления ген rep представляет собой ген rep, происходящий из AAV2. Плазмиды-помощники и способы получения таких плазмид известны в данной области и коммерчески доступны (см., например, плазмиды pDM, pDG, pDP1rs, pDP2rs, pDP3rs, pDP4rs, pDP5rs, pDP6rs, pDG(R484E/R585E) и pDP8.ape от PlasmidFactory, Билефельд, Германия; другие продукты и услуги от Vector Biolabs, Филадельфия, Пенсильвания; Cellbiolabs, Сан-Диего, Калифорния; Agilent Technologies, Санта-Клара, Калифорния; и Addgene, Кембридж, Массачусетс; рхх6; Grimm et al. (1998), Novel Tools for Production and Purification of Recombinant Adenoassociated Virus Vectors, Human Gene Therapy, Vol. 9, 2745-2760; Kern, A. et al. (2003), Identification of a Heparin-Binding Motif on Adeno-Associated Virus Type 2 Capsids, Journal of Virology, Vol. 77, 11072-11081.; Grimm et al. (2003), Helper Virus-Free, Optically Controllable, and Two-Plasmid-Based Production of Adeno-associated Virus Vectors of Serotypes 1 to 6, Molecular Therapy, Vol. 7, 839-850; Kronenberg et al. (2005), A Conformational Change in the Adeno-Associated Virus Type 2 Capsid Leads to the Exposure of Hidden VP1 N Termini, Journal of Virology, Vol. 79, 5296-5303; и Moullier, P. and Snyder, R.O. (2008), International efforts for recombinant adeno-associated viral vector reference standards, Molecular Therapy, Vol. 16, 1185-1188).
В соответствии с некоторыми вариантами осуществления одна или несколько клеток-хозяев (например, клеток-продуцентов) может иметь одну или несколько вспомогательных функций (например, кодируемых рекомбинантной нуклеиновой кислотой, интегрированной в геном клетки-хозяина, или кодируемых плазмидой или вектором на основе нуклеиновой кислоты в клетке-хозяине). В соответствии с некоторыми вариантами осуществления рекомбинантный капсидный ген (например, ген VP1), содержащий модифицированную последовательность Козак, описанную в настоящей заявке, может быть предусмотрен в плазмиде, вирусном геноме (например, геноме на основе бакуловируса, вируса герпеса, аденовируса или другого вируса) или интегрирован в геном клетки-хозяина (например, клетки млекопитающего или насекомого, например, клетки Sf9).
В соответствии с некоторыми вариантами осуществления ген, представляющий интерес (например, ген, который упакован в частицу rAAV), кодирует фермент, гормон, антитело, рецептор, лиганд или другой белок. В соответствии с некоторыми вариантами осуществления ген, представляющий интерес, кодирует терапевтически пригодный белок. В соответствии с некоторыми вариантами осуществления ген, представляющий интерес, кодирует эталонный или маркерный белок (например, детектируемый маркер, например, GFP). В соответствии с некоторыми вариантами осуществления ген, представляющий интерес, кодирует РНК, например, регуляторную РНК, такую как siRNA или другую регуляторную РНК (например, РНК терапевтического значения). В соответствии с некоторыми вариантами осуществления терапевтическое средство представляет собой полипептид, пептид, антитело или его антигенсвязывающий фрагмент, рибозим, пептид-нуклеиновую кислоту siRNA, RNAi, антисмысловой олигонуклеотид или антисмысловой полинуклеотид. В соответствии с некоторыми вариантами осуществления ген, представляющий интерес, кодирует терапевтический белок. В соответствии с некоторыми вариантами осуществления ген терапевтического значения кодирует антитело, пептитело, фактор роста, фактор свертывания крови, гормон, мембранный белок, цитокин, хемокин, активирующий или ингибиторный пептид, действующий на рецепторы клеточной поверхности или ионные каналы, проникающий в клетку пептид, целенаправленно воздействующий на внутриклеточные процессы, тромболитик, фермент, костные морфогенетические белки, нуклеазу или другой белок, используемый для редактирования генома, белок слияния, содержащий Fc, антикоагулянт, направляющую РНК или другую нуклеиновую кислоту или белок для геномного редактирования. В соответствии с некоторыми вариантами осуществления терапевтический белок является терапевтическим по отношению к лизосомной болезни накопления. В соответствии с некоторыми вариантами осуществления терапевтический белок является терапевтическим по отношению к неврологическому поражению, нейромоторному расстройству, нейроскелетному нарушению или нейромышечному заболеванию. В соответствии с некоторыми вариантами осуществления терапевтический белок является терапевтическим по отношению к мышечному поражению или дистрофии, миопатии или кардиомиопатии. В соответствии с некоторыми вариантами осуществления ген, представляющий интерес, кодирует вакцину (например, вакцину на основе нуклеиновой кислоты или пептидную вакцину).
В соответствии с некоторыми вариантами осуществления ген, представляющий интерес (например, кодирующий терапевтическое средство), функционально связан с промотором. Промотор может представлять собой без ограничения природный промотор, синтетический или рекомбинантный промотор, химерный промотор, усеченный промотор, конститутивный промотор, индуцибельный промотор, видоспецифичный промотор, тканеспецифичный промотор или комбинацию из двух или более из вышеуказанного. В соответствии с некоторыми вариантами осуществления промотор может быть регулируемым (например, уровень экспрессии со стороны промотора может быть повышен или снижен в результате изменения внешнего состояния или в результате добавления или удаления регуляторной молекулы). В соответствии с некоторыми вариантами осуществления ген, представляющий интерес, может быть функционально связан со своим природным промотором или альтернативно с другим промотором.
В соответствии с некоторыми вариантами осуществления композиция, содержащая rAAV, описанная в настоящем документе, может быть использована для лечения субъекта-млекопитающего (например, человека). В соответствии с некоторыми вариантами осуществления субъект имеет рак, сахарный диабет, аутоиммунное заболевание, заболевание почек, сердечно-сосудистое заболевание, заболевание поджелудочной железы, заболевание кишечника, заболевание печени, неврологическое заболевание, нейромышечное заболевание, болезнь Баттена, болезнь Альцгеймера, болезнь Хантингтона, болезнь Паркинсона, заболевание легких, недостаточность α-1-антитрипсина, неврологическое поражение, нейромоторную недостаточность, нейроскелетное нарушение, ишемию, инсульт, лизосомную болезнь накопления, болезнь Помпе, мышечную дистрофию Дюшенна, атаксию Фридрейха, болезнь Канавана, недостаточность декарбоксилазы ароматических L-аминокислот, гемофилию А/В или другое заболевание или любую их комбинацию.
Без дальнейшего детального рассмотрения, считается, что специалист в данной области на основании вышеизложенного описания может использовать настоящее раскрытие в его наиболее полной степени. Таким образом, следующие конкретные варианты осуществления следует рассматривать только в качестве иллюстративных и никоим образом не ограничивающих оставшуюся часть раскрытия. Все публикации, цитируемые в настоящем документе, включены посредством ссылки для целей или предмета изобретения, на которые дается отсылка в настоящем документе.
ПРИМЕРЫ
Пример 1: Модулирование соотношений VP1:VP2:VP3 в стабильных линиях клеточных с помощью аттенуированных последовательностей Козак
Биологическая активность (например, инфицирующая способность) частиц rAAV частично зависит от состава капсида вириона, например, стехиометрических соотношений капсидных белков VP1, VP2 и VP3. Некоторые серотипы rAAV (например, AAV5, AAV8 и AAV9), полученные в гетерологичной системе, такой как клетки насекомых (например, клетки Sf9), характеризуются патологическими уровнями экспрессии VP1. При сборке вирусов патологическая экспрессия VP1 приводит к продукции вирусных частиц, состоящих из неправильных соотношений капсидных белков, что затрудняет способность частиц эффективно трансдуцировать клетки. Способы получения пригодных стехиометрических соотношений капсидных белков осложняются результатами, указывающими на то, что избыток VP1 может нарушить способность клетки насекомого эффективно упаковывать вирусную частицу, в то время как низкие уровни VP1 могут приводить к неэффективной трансдукции. Таким образом, применительно к продукции rAAV в клетках насекомых остается проблемой найти способ повышения выхода инфицирующих частиц, чтобы при этом отсутствовало отрицательно влияние на сборку частиц.
В текущем исследовании стремились улучшить соотношение белков в результате модулирования инициации скорости трансляции VP1 в rAAV, экспрессируемом в клетках насекомых Sf9. В частности, последовательности Козак VP1 в AAV5, AAV8 и AAV9 аттенуировали, в то время как канонический старт-кодон ATG сохраняли константным, и измеряли эффективности трансдукции для оценки собранных частиц.
Совокупность аттенуированных элементов Козак конструировали для AAV5, и создавали нуклеиновые кислоты, кодирующие эти элементы. Кодируемые последовательности Козак представлены в Таблице 4. Было обнаружено, что подсовокупность четырех элементов Козак кодирует капсидные белки с соотношениями, близкими к соотношению VP1:VP2:VP3 AAV5 дикого типа, составляющему 1:1:10, как показано с помощью вестерн-блоттинга, представленного на Фиг. 2А. В случае этих элементов (последовательности Козак AAV5 7-10) соответствующие соотношения (VP1):(VP2):(VP3) находились в диапазонах (0,5-2):(0,5-1):10. Относительные эффективности in vitro последовательностей инициации трансляции (TIS, также обозначаемых как последовательности Козак) измеряли для каждой собранной частицы (Таблица 4). Эти числа соответствуют числам под панелью вестерн-блоттинга на Фиг. 4В. Последовательности Козак AAV5 7 (SEQ ID NO: 8) и 8 (SEQ ID NO: 9) приводили к образованию rAAV, характеризующегося наибольшими эффективностями, и их отбирали для дальнейшего анализа.
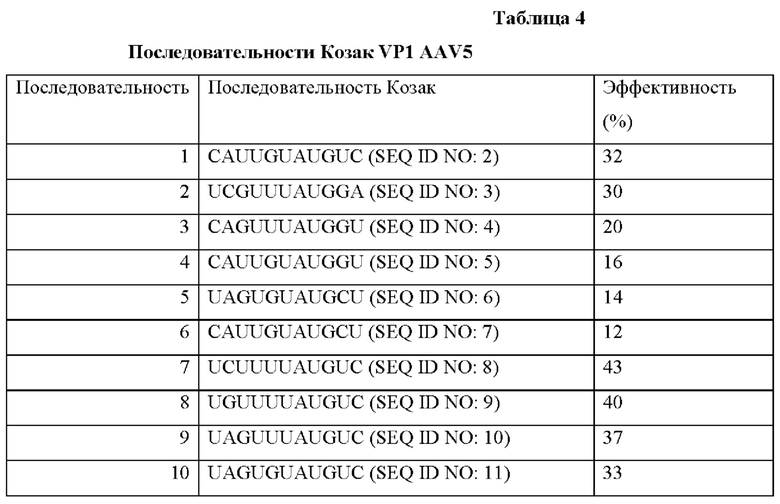
Последовательность Козак AAV5 7 приводила к образованию соответствующего соотношения VP1:VP2:VP3, составляющего 2:1:(8-9). По сравнению с AAV5, выделенными из клеток HEK 293, частицы с последовательностью 7, генерированные в клетках насекомых, характеризовались в 2-кратной эффективностью трансдукции. Последовательность Козак AAV5 8 приводила к образованию соответствующего соотношения VP1:VP2:VP3, составляющего (1-1,5):1:10. По сравнению с AAV5, выделенными из клеток HEK 293, частицы с последовательностью 8 характеризовались в 3-5-кратной эффективностью трансдукции. Было замечено, что выход упакованного в rAAV5 варианта последовательности 8 был примерно в 10 раз больше, чем таковой последовательности 7.
Совокупность аттенуированных элементов Козак конструировали для AAV8, и создавали нуклеиновые кислоты, кодирующие эти элементы. Кодируемые последовательности Козак представлены в Таблице 5.
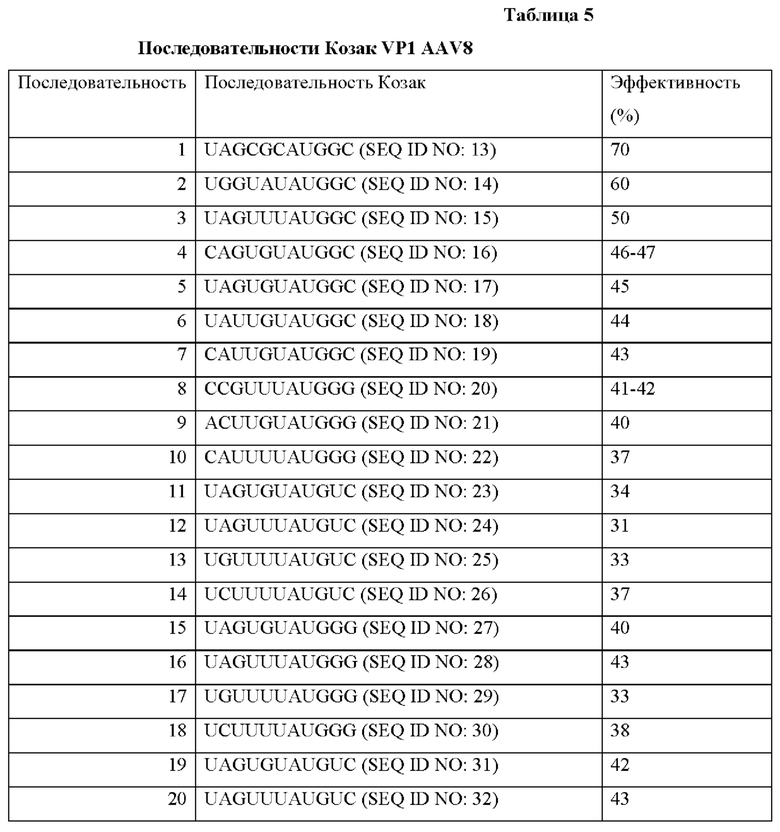
Последовательности Козак AAV8 1-12 характеризовались гиперэкспрессией VP1 и низкой экспрессией VP2 и VP3, показанными с помощью вестерн-блоттинга, представленного на Фиг. 2В. Эти последовательности исключали из последующего исследования. Оставшиеся элементы (последовательности 13-20) контролировали экспрессию VP1:VP2:VP3 в аналогичных соотношениях по отношению к AAV8, выделенному из клеток HEK293.
Совокупность аттенуированных элементов Козак конструировали для AAV9, и создавали нуклеиновые кислоты, кодирующие эти элементы. Кодируемые последовательности Козак представлены в Таблице 6.
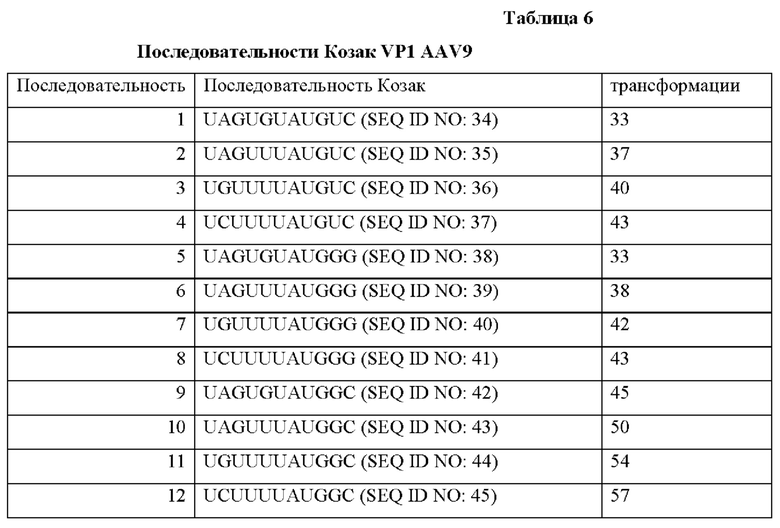
Последовательности Козак AAV9 2 (SEQ ID NO: 35), 3 (SEQ ID NO: 36), 5 (SEQ ID NO: 38) и 9 (SEQ ID NO: 42) контролировали капсиды AAV9 с удовлетворительными соотношениями VP1:VP2:VP3, показанными с помощью вестерн-блоттинга, представленного на Фиг. 2С. Принимая во внимание эксперименты, осуществляемые с каждым серотипом AAV, получали «консенсусный» аттенуированный элемент Козак. Как представлено на Фиг. 3, положение, которое представляет собой -5 (5 нуклеотидов выше от старт-кодона) является наиболее гибким для точной регуляции соотношения экспрессии VP1:VP2:VP3 для определенного капсида, выбранного из серотипов 5, 8 и 9 AAV.
Пример 2: Оценка rAAV
Основным недостатком системы на основе бакуловируса/Sf9 для производства rAAV является то, что большинство происходящих из бакуловирусов серотипов вектора rAAV, за несколькими исключениями, характеризуются измененными составами капсида и более низкими биологическими активностями. Описана новая платформа для продукции на основе клеток насекомых с использованием аттенуированной последовательности и ослабленного сканирования рибосом в целях достижения специфичной в отношении серотипа модуляции стехиометрического соотношения капсидных белков AAV. В качестве примера rAAV5 и rAAV9 продуцировали и всесторонне характеризовали, наряду с происходящими из клеток HEK 293 векторами. Масс-спектрометрический анализ показал увеличение в 3 раза содержания обоих капсидных белков VP1 и VP2 по сравнению с векторами, происходящими из клеток человека. Кроме того, проводили подробный анализ включенной в капсид однонитевой вирусной ДНК с помощью секвенирования следующего поколения и показали 6-кратное снижение коллатерально упакованной контаминирующей ДНК в случае rAAV5, продуцируемого в клетках насекомых. Соответственно, повторно сконструированные rAAV характеризовались значимо более высокими биологическими эффективностями, даже по сравнению с полученными в HEK 293 rAAV, опосредующими в случае rAAV5 в 4 раза более высокую трансдукцию в тканях мозга у мышей. Таким образом, описанная система приводит к образованию векторов rAAV с превосходящей инфицирующей способностью и исключительной чистотой, обеспечивая масштабируемую платформу для продукции векторов степени соответствия GMP.
rAAV широко используется в качестве вектора для генной терапии/доставки ДНК-вакцин, однако продукция в промышленных масштабах высокоинфекционных rAAV для клинических исследований остается проблематичным вопросом. Капсид AAV состоит из трех капсидных белков, VP1, VP2 и VP3, получаемых в результате альтернативного сплайсинга и различной частоты использования кодона гена одного капсидного белка в геноме AAV. Последовательность VP3 является общей для всех трех сплайс-вариантов, а VP2 и VP1 имеют более длинные N-концевые последовательности, при этом VP1 содержит домен фосфолипазы А2, ключевой для вирусной инфекции 1. Точные количества VP1/VP2/VP3 в капсиде неизвестны, однако, по оценкам, составляют 1/1/10, на основе денситометрических анализов капсидных белков, отделенных с помощью SDS-PAGE 2-4. Более того, по-видимому, отсутствует определенный стехиометрический состав VP1/VP2/VP3 и сборка является стохастической, поэтому относительное количество VP1/VP2/VP3, которое включено в капсид, зависит главным образом от уровней их относительной экспрессии 5. Таким образом, конструирование единицы экспрессии капсидных белков в определенной системе продукции rAAV является необходимым для сборки биологически активных векторов для генной терапии.
С этой целью в одной из исходных масштабируемых систем использовали суспензионную культуру клеток насекомых Sf9, ко инфицированных тремя рекомбинантными бакуловирусами, происходящими из мультикапсидного нуклеополиэдровируса A. californica (AcMNPV), кодирующего соответственно кассету на основе трансгенного rAAV, вспомогательные гены rep и cap AAV 6. Большинство серотипов AAV, продуцируемых в этой системе, однако, характеризовались низкими эффективностями трансдукции по сравнению с векторами, происходящими из клеток HEK 293, вследствие неподходящего содержания капсидного белка VP1 и активности его фосфолипазы А2 7-10. Такой недостаток был следствием конструирования вспомогательного вектора с капсидными генами, использующего неканонический кодон инициации трансляции ACG в случае VP1 для индукции ослабленного сканирования рибосом 6. Даже несмотря на то, что в других группах проблему решали в некоторой степени, используя другой кодон инициации трансляции CUG 11 или искусственный интрон 12, по-видимому, в решение этих проблем отсутствовала гибкость, необходимая для специфичной в отношении серотипа корректировки последовательности. Вводили новую систему регуляции относительного состава VP1/VP2/VP3 посредством корректируемого ослабленного сканирования рибосом.
В клетках, происходящих от млекопитающих, контролируемый Р40 транскрипт в AAV подвергается сплайсингу с образованием двух сплайсированных вариантов мРНК, кодирующих капсидные белки VP1 или VP2/VP3 соответственно. Поскольку в системе бакуловирус/Sf9 промотор polh замещен Р40/интронной последовательностью, регуляция с помощью сплайсинга недоступна и используется альтернативная регуляция экспрессии VP1 посредством ослабленного сканирования рибосом. Считается, что консенсусная последовательность 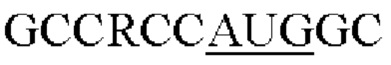 (SEQ ID NO: 49) (R=A или G) является эффективным участком инициации трансляции (TIS) у млекопитающих, также известным как последовательность Козак 13. Любое отклонение от этой последовательности приводило бы к повышению ослабленного сканирования AUG VP1 и инициации трансляции внутрирамочных нижерасположенных кодонов ACG VP2 или AUG VP3, изменяя таким образом стехиметрическое соотношение VP1/VP2/VP3. В текущем исследовании описан подход для рационального модулирования соотношений состава VP1/VP2/VP3 в системе бакуловируса/Sf9 для получения частиц с более высоким содержанием VP1/VP2, приводящим к значимо более высокой биологической активности даже по сравнению с векторами, происходящими из клеток HEK 293.
(SEQ ID NO: 49) (R=A или G) является эффективным участком инициации трансляции (TIS) у млекопитающих, также известным как последовательность Козак 13. Любое отклонение от этой последовательности приводило бы к повышению ослабленного сканирования AUG VP1 и инициации трансляции внутрирамочных нижерасположенных кодонов ACG VP2 или AUG VP3, изменяя таким образом стехиметрическое соотношение VP1/VP2/VP3. В текущем исследовании описан подход для рационального модулирования соотношений состава VP1/VP2/VP3 в системе бакуловируса/Sf9 для получения частиц с более высоким содержанием VP1/VP2, приводящим к значимо более высокой биологической активности даже по сравнению с векторами, происходящими из клеток HEK 293.
Для характеристики такой усовершенствованной платформы для продукции во всей ее полноте анализ секвенирования следующего поколения (NGS) производимой включенной в капсид ДНК проводили с помощью двух способов: традиционной тройной котрансфекцией плазмидами клеток HEK 293 и одиночной инфекцией BEV клеточной линии Sf9, включающей стабильно интегрированные вспомогательные гены rep/cap. Прямой параллельный анализ NGS кассет rAAV, полученных с помощью двух платформ, показал более высокую точность упаковки вирусной ДНК в клетках насекомых, включающих в капсид значительно менее контаминированную ДНК.
Разработка капсидных генов AAV5 и rAAV9. Для повышения ослабленного сканирования рибосом использовали канонический кодон AUG с предшествующими аттенуированными последовательностями Козак. Случайно модифицированные нуклеотиды выше или ниже от AUG не представляли бы собой реалистичного подхода, поскольку множество возможных последовательностей TIS, охватывающих относительную последовательность из восьми остатков, составляет 65536 возможных комбинаций. Более того, по-видимому, консенсусная последовательность Козак отличается для дрожжевых грибов 14, высших растений 15, беспозвоночных 16 или позвоночных 17. Таким образом, одним путем обоснования скрининга аттенуированных TIS было использование эмпирической теплокарты из всех возможных комбинаций TIS млекопитающих, полученных Noderer et al., 18 где всем возможным комбинациям TIS присваивали значения «эффективности инициации» по отношению к консенсусной последовательности Козак.
Выбирали диапазон, составляющий 12-43% эффективности инициации, и исследовали в отношении десяти мутантов, отличающихся 2-4% повышениями. Все десять исследуемых TIS VP1 AAV5 представлены в Таблице 7 и их использовали в контексте cap-экспрессирующих плазмид-помощников (Фиг. 4А) для получения объеденных клеточных линий BSR, описанных ранее 7, и оценки составов их капсида. За несколькими заметными исключениями (например, Фиг. 4В, линия 6), относительное содержание VP1 постепенно снижалось, при этом близко соответствуя снижениям теоретической величины относительных эффективностей TIS. Эта связь поддерживает исходную гипотезу о том, что ослабленное сканирование инициации трансляции можно использовать для модулирования содержания VP1. Для дополнительной идентификации пригодных стехиометрических соотношений VP1/VP2/VP3 определяли инфекционные и общие титры соответствующих десяти векторов rAAV5. Для продукции инфекционного вектора на основе rAAV5 получали кодирующий BEV CBA-контролируемый GFP (Фиг. 10А, pTR-Bac-UF26). Параллельное сравнение показало, что конструкция с 40% относительной эффективностью TIS (Фиг. 4В, линия 2) была более эффективной по сравнению со всеми другими конструкциями, приводящими к наиболее высокому выходу. Таким образом, выбирали такую вспомогательную конструкцию, содержащую определенные гены, включающую аттенуированный TIS, 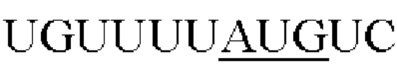 (SEQ ID NO: 9 в Таблице 7), с получением линии клеток-продуцентов. Однако можно также использовать другие последовательности. Аналогичным образом подвергали скринингу двенадцать плазмидных конструкций на основе VP1 AAV9 с аттенуированными последовательностями Козак (Таблица 7). В случае AAV9 особенно пригодной последовательностью была
(SEQ ID NO: 9 в Таблице 7), с получением линии клеток-продуцентов. Однако можно также использовать другие последовательности. Аналогичным образом подвергали скринингу двенадцать плазмидных конструкций на основе VP1 AAV9 с аттенуированными последовательностями Козак (Таблица 7). В случае AAV9 особенно пригодной последовательностью была  (SEQ ID NO: 42), составляющая 45% относительной эффективности TIS. Однако можно также использовать другие последовательности.
(SEQ ID NO: 42), составляющая 45% относительной эффективности TIS. Однако можно также использовать другие последовательности.
Характеристика стабильной клеточной линии rep2/cap5. Отдельные клеточные линии получали с помощью Rep2- и Сар5-экспрессирующих плазмид, не содержащих Rep-связывающих элементов (RBE) 8. Вспомогательный ген Cap5 содержал следующий аттенуированный TIS: 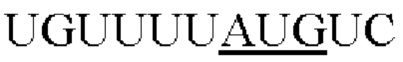 (SEQ ID NO: 9). Пять отдельных клеточных линий размножали и исследовали, как описано раньше7. Одну клеточную линию, получившую название В8, характеризующуюся наиболее высоким выходом rAAV5--UF26, выбирали для дополнительной характеристики. Изучали следующие параметры.
(SEQ ID NO: 9). Пять отдельных клеточных линий размножали и исследовали, как описано раньше7. Одну клеточную линию, получившую название В8, характеризующуюся наиболее высоким выходом rAAV5--UF26, выбирали для дополнительной характеристики. Изучали следующие параметры.
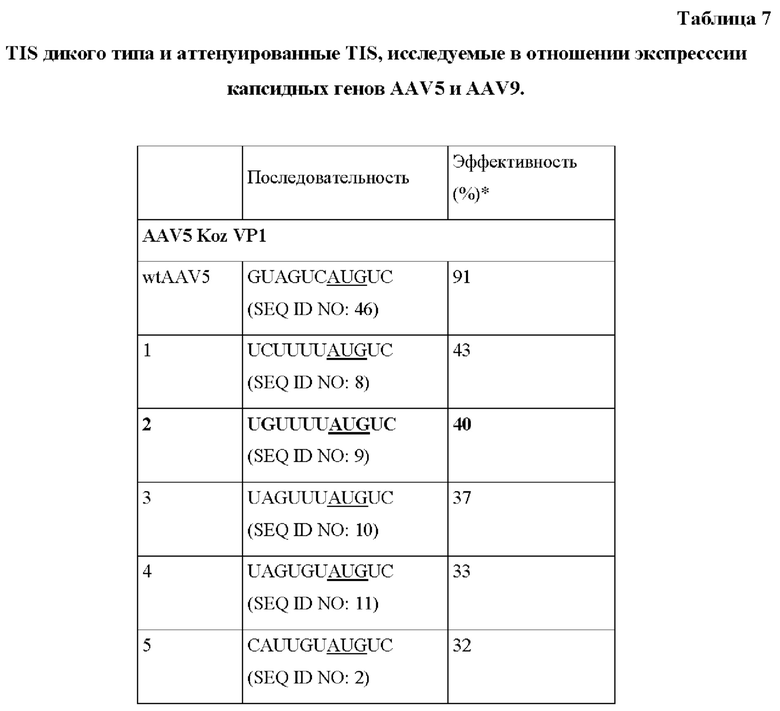
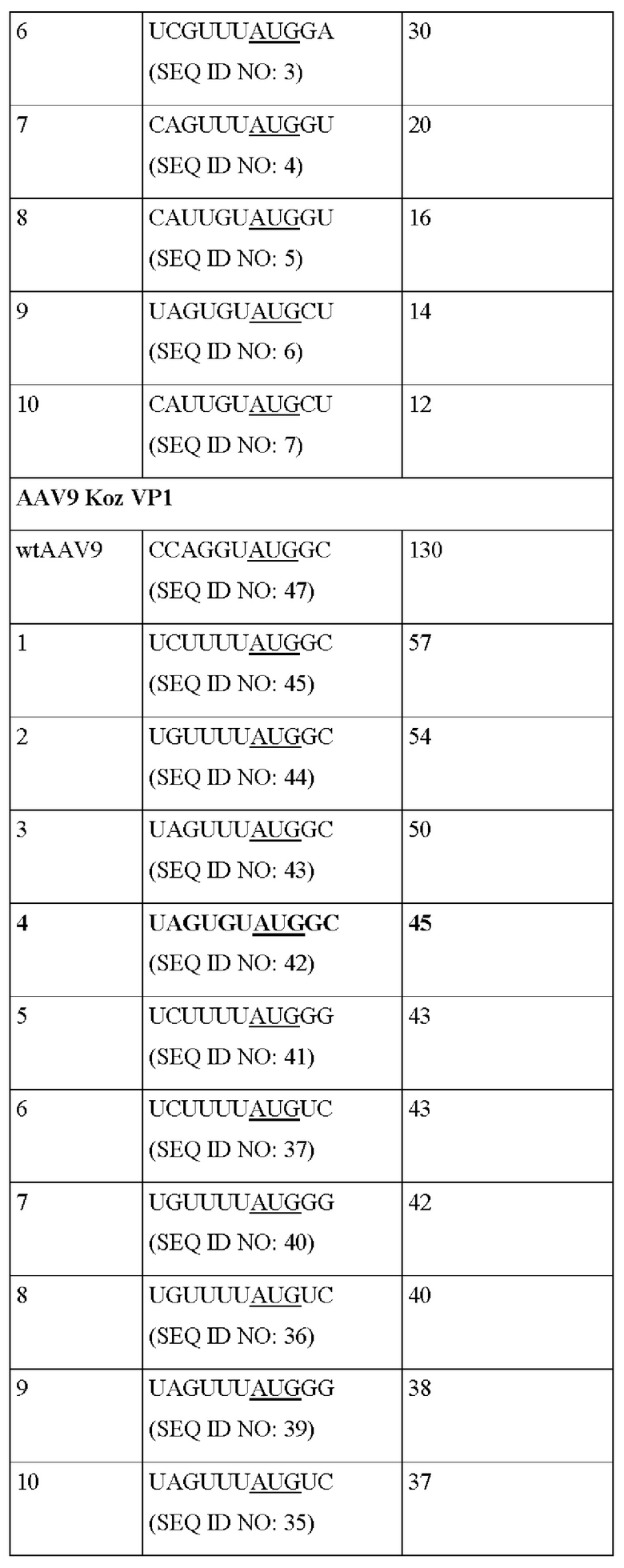
*Оцененные относительные эффективности TIS 18
Некоторые последовательности для каждого серотипа подчеркнуты жирным. Подчеркивание указывает на старт-кодон VP1.
Стехиометрическое соотношение VP1/VP2/VP3. Состав капсида rAAV5-GFP, очищенного из клеток HEK 293 с помощью двойного последовательного градиента в йодиксаноле, отклонялся от теоретического значения, составляющего 1/1/10 в случае VP1/VP2/VP3, таким образом, что он имел меньшее содержание VP1 (Фиг. 4С). Более того, этот капсид включал дополнительный капсидный белок VP2 с немного более высоким значением MW. В отличие от этого, вектор, происходящий из Sf9 В8, состоял из более высоких уровней VP1 и VP2 (Фиг. 4С). В случае rAAV9 состав капсида вектора, происходящего из Sf9, был почти идентичен таковому вектора, образованного в клетках HEK 293 (Фиг. 4D). В отличие от rAAV5, по-видимому, оба вирусных образца включают больше VP1, чем VP2, и также содержат полосы меньшего размера, чем VP3, который мог быть продуктом специфического по отношению к капсиду протеолиза 19.
Для количественного определения точных числовых значений плотности соответстующих капсидных полос в случае rAAV5 наносили на график в виде площадей под кривыми (AUC) (Фиг. 11). Получали следующие значения (средние значения из двух независимых экспериментов, скорректированные на общий знаменатель VP3=10): HEK 293 VP1/VP2/VP3=0,2/0,5/10; Sf9 В8 VP1/VP2/VP3=0,7/1,7/10. Для проверки рассчитанного стехиометрического соотношения состав капсида rAAV5 анализировали с помощью масс-спектрометрии с использованием лазерной десорбции/ионизации в присутствии матрицы время пролета (MALDI-TOF).
MALDI-TOF. VP AAV, составляющие оболочку вириона, имели общие С-концевые участки своих VP3 (Фиг. 5А, показано черным цветом). VP1 (пунктир) и VP2 (штрих-пунктир) уникальные N-концевые участки были сравнительно маленькими по сравнению с общим доменом VP3, что создавало проблему для понимания относительного стехиометрического состава с помощью анализа их уникальных триптических пептидов. Таким образом, подход на основе дифференциального мечения 16O/18O использовали для различения идентичных пептидов, но происходящих из VP1, VP2 или VP3. При использовании стабилизированного тяжелым кислородом стабильного изотопа воды H218O во время расщепления трипсином два атома 18O обменивали в С-концевой карбоксильной группе триптических пептидов, смещая массу на 4 Да 20, 21. Такое смещение между идентичными пептидами, происходящими из двух различных белков (например, VP1 или VP3) способствовало идентификации и количественному определению белков.
После проведения пилотного расщепления три пептида (Фиг. 5А) в участке VP3 выбирали для количественной оценки относительных избытков каждого белка. Полное включение двух атомов 18O подтверждали by отдельным пропусканием образцов VP3/VP1 и VP2/VP3. Затем анализировали полный спектр MALDI всех триптических продуктов расщепления VP3 в H218O (Фиг. 5В). Все из трех пептидов характеризовались сдвигом массы на 4 Да от предполагаемых молекулярных масс пептидов, подтверждая включение двух атомов 18O. Если наблюдаемый сдвиг массы составлял только 2 Да, то либо реакция была неполной, либо происходил обратный обмен в результате неполной инактивации трипсином 20. На Фиг. 5С показано наложение триптического пептида TWVLPSYNNHQYR (SEQ ID NO: 48) либо от VP1 (красная линия в исходных цветных фигурах) либо от VP3 (синяя линия в исходных цветных фигурах), нанесенных отдельно и анализируемых с помощью MALDI-TOF. Отчетливый сдвиг массы на 4 Да в случае образца VP3 доказывал полный обмен атома 18O без доказательства обратного обмена. На Фиг. 5D представлены иллюстративные спектры MALDI-TOF VP3, расщепленных H218O и смешанных 1:1 с VP1, расщепленным в Н216О. Площади пиков интегрировали и использовали для расчета избытка каждого белка.
Анализировали три уникальных пептида (Фиг. 5А) на каждый белок, каждый - в трех повторах пропускания с использованием MALDI-TOF (Таблицы 8 и 9). Это обеспечивало высокую достоверность количественной оценки их относительного избытка 22. Получали следующие значения (среднее значение из трех независимых экспериментов, скорректированные на общий знаменатель VP3=10): HEK 293 VP1/VP2/VP3=0,4/0,5/10; Sf9 В8 VP1/VP2/VP3=1,1/1,7/10. Эти числа заметно согласуются с плотностями AUC, описанными выше.

* м/з представляет собой массу, деленную на зарядовое число.
* м/з представляет собой массу, деленную на зарядовое число.
Соотношение полных/пустых частиц. Этот параметр изучали, чтобы определить, приводит ли какая-то из двух платформ для продукции к образованию более высоких соотношений пустых частиц, потенциального источника нежелательного иммунного ответа и технической проблемы во время очистки векторов. rAAV5 и rAAV9 очищали с помощью одноступенчатой хроматографии в смолах с аффинностью к моноспецифических антителам, AVB в случае rAAV5, и POROS CaptureSelect™ в случае rAAV9. После очистки титры частиц векторных геномов анализировали с помощью QC ПЦР. Титры всех частиц векторов rAAV5 и rAAV9 анализировали с помощью анализа трекинга наночастиц (NTA) капсидов rAAV, оснащенных золотыми частицами. В таком подходе используется электростатическое притяжение между высокорассеивающим материалом, таким как золотые наночастицы и вирусным капсидом. Полученные в результате меченные золотом вирусные частицы рассеивают достаточно света для того, чтобы быть визуализированными и подлежать трекингу с помощью оптической системы (Фиг. 6A-6F), позволяя использовать NTA для измерения размера и концентрации AAV. Интересно, что в то время как средние размеры частиц rAAV9 для обоих платформ были одинаковыми (основной пик 38 нм, Фиг. 6А, 6В), средние размеры rAAV5 из HEK 293 и из Sf9 В8 отличались (48 нм и 42 нм, Фиг. 6D, 6Е). Следует отметить, что эти размеры представляют собой аппроксимации комплексов вирус/частица на основе нанозолота что, в свою очередь, является функциями заряда вирусной частицы.
После расчета соотношения всех частиц к полным частица эти значения в случае rAAV9, происходящего из клеток HEK 293, составляло 2,8, и в случае rAAV9, происходящего из клеток Sf9, составляло 3,1 (Фиг. 6С). Аналогично в случае rAAV5 соотношения также значимо не различались: 14,8 и 15,2 (Фиг. 6F). В целом более низкие соотношения в случае rAAV9, по-видимому, были результатом смещения при очистке при хроматографии со смолой CaptureSelect, что указывало на предпочтительно связывание и обогащение ДНК-содержащих частиц на основе rAAV9 (данные не представлены).
Эффективность трансдукции in vitro. Кассету на основе трансгенного rAAV, включающую гены люциферазы и репортера mApple (Фиг. 10В, pTR-UF50-BC 23) использовали для генерации соответствующего BEV для инфицирования клеточной линии Sf9 В8 в целях исследования эффективности трансдукции. Ту же самую плазмиду использовали для генерации rAAV с помощью тройной контрансфекции клеток HEK 293. Оба вектора rAAV5-Luc-mApple, очищенный и титрованный параллельно, использовали для инфицирования клеточной линии С12, происходящей из HeLa 24. В результате анализа FACS (Фиг. 12А) была обнаружена значимо более высокая (~5-кратная) эффективность трансдукции в случае вектора rAAV5, продуцированного в Sf9. В случае rAAV9 отсутствовала значимая разница между двумя образцами rAAV9 (Фиг. 12В). Данные обоих экспериментов согласуются с содержанием белка.
Эффективность трансдукции in vivo (Фиг. 7). rAAV5-Luc-mApple, продуцируемый в клетках HEK 293 и Sf9 В8, анализировали in vivo в головном мозге (полосатом теле) мышей. Через три недели после инъекции эффективность трансдукции определяли с помощью биолюминесценции (BLI) (Фиг. 7А). Животным, которым вводили AAV5, происходящий из Sf9 В8, характеризовались 4-кратным повышением интенсивности сигнала BLI по сравнению с AAV5, происходящим из HEK 293 (Фиг. 7В, 4,3×105±1,8×105, и 1,1×105±3,5×104 соответственно). Аналогично анализ флуоресценции срезов головного мозга показал более сильный сигнал mApple в образцах, происходящих из Sf9 В8, по сравнению с HEK 293 (Фиг. 7С).
Анализ секвенирования следующего поколения (NGS) векторов rAAV5, продуцируемых в клетках HEK 293 и Sf9. После определения улучшенной системы OneBac для продукции векторов rAAV5 с повышенной активностью проводили сравнительный анализ NGS включенной в капсид ssDNA (pTR-Bac-UF26, Фиг. 10А), полученной с помощью стандартного протокола тройной котрансфекции в клетках HEK 293, или с помощью одной инфекции BEV-UF26 стабильной клеточной линии rep2cap5 Sf9 В8. Целью анализа NGS была характеристика специфичного по отношению к кассете rAAV, а также коллатерально упакованных контаминированных молекул ДНК, таким образом, при этом определяли предпочтительную платформу, отвечающую требованиям продукции векторов rAAV степени соответствия GMP. Все библиотеки NGS получали, секвенировали и анализировали в двух повторностях. Общий технологический процесс представлен на технологической карте (Фиг. 13). Анализировали следующие параметры.
Коллатеральная упаковка контаминирующих последовательностей ДНК
После фильтрования общее количество считываемых фрагментов, которым присваивался индекс, составляло 757433116. После выравнивания считываемых фрагментов с упомянутыми последовательностями, охват в случае кассеты rAAV достигал до 2260074 считываемых фрагментов/нуклеотид (положение нуклеотида 2000). В случае коллатерально упакованных последовательностей охват был значимо ниже: 10781 считываемых фрагментов/нуклеотид (положение нуклеотида 1299, векторный остов), или 6424 считываемых фрагментов/нуклеотид (положение нуклеотида 200, геном AcMNPV).
В случае обоих протоколов продукции большинство считываемых фрагментов представляли собой кассету rAAV-Bac-UF26, которая составляла 96,5% (HEK 293) и 99,4% (Sf9) от всех включенных в капсид последовательностей ДНК (Таблица 10, Фиг. 8А, 8В). Большинство контаминированной ДНК в системе HEK 293 представляло собой плазмидный остов (2,5%), при более низких уровнях вспомогательных последовательностей rep2/cap5 (0,7%) и геномной ДНК человека (0,17%), в то время как контаминанты в препарате Sf9 представляли собой остов шаттл-плазмиды (0,3%) и геном AcMNPV (0,2%). В случае rAAV5, происходящего из Sf9, было обнаружено, что считываемые фрагменты не были гомологичны добавочному рабдовирусу, который недавно выделили из клеточной линии Sf9, полученной из АТСС (Sf-рабдовирус) 25.
Коллатерально упакованные последовательности были представлены в большем избытке с помощью непосредственных соединений кассеты rAAV и ее соответствующих остовов: бактериальной плазмиды в случае клеток HEK 293 и бакуловирусного генома в случае клеток Sf9. Примечательно, что присутствовала значимая разница, по меньшей мере десятикратная, в охвате считываемых фрагментов соединения между двумя системами, при этом клетки HEK 293, по-видимому, включали в капсид с гораздо большими частотами последовательности бактериальных плазмидных остовов, которые являются более удаленными как от левых, так и от правых концевых повторов AAV (Фиг. 8С, 8D). Таким образом, анализ последовательностей контаминирующей ДНК указывало на то, что система OneBac характеризовалась большей точностью и обеспечивала более высокое качество векторов rAAV вследствие чего только 0,6% включенного в капсид векторного генома включала чужеродную ДНК в отличие от 3,5% в случае клеток HEK 293.
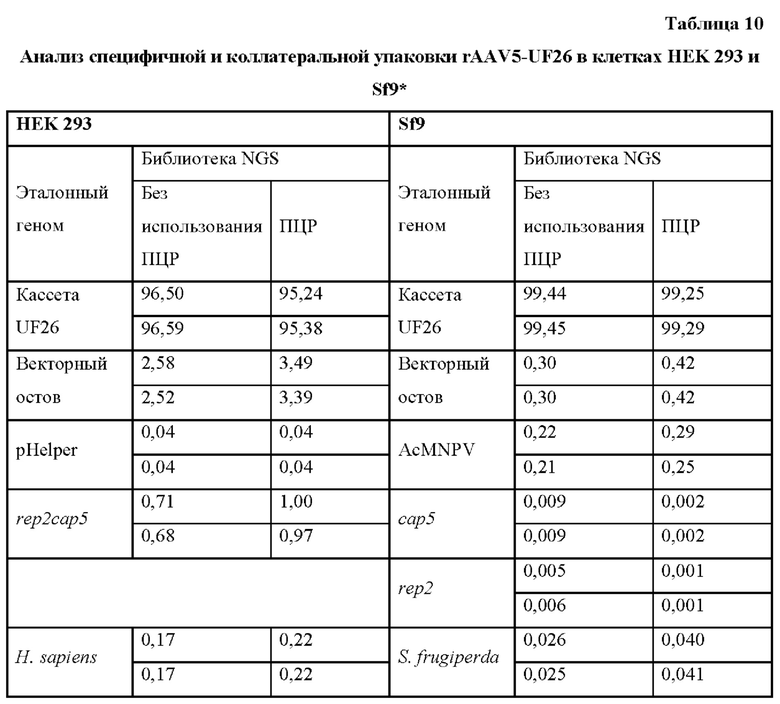
* Относительное содержание ДНК в очищенных вирионах представлено в виде процента от всех последовательностей, идентифицированных с помощью анализа NGS. Все анализы NGS проводили в двух повторностях, показывая каждый эталонный геном в двух параллельных рядах.
Охват генома rAAV. С помощью стандартного протокола, который включал только восемь циклов стадии ПЦР-амплификации в целях генерации библиотек NGS, идентифицировали несколько последовательностей в промоторе СВА, а расположенный ниже интрон характеризовался по меньшей мере в 10 раз меньшим охватом секвенирования по сравнению с оставшейся частью кассеты rAAV. Тщательное исследование выявило, что эти последовательности имеют крайне высокое содержание GC (Фиг. 8А, 8В). Такой сравнительно низкий охват мог отражать более низкое представление этих последовательностей в упакованных вирионах в связи с усечением во время репликации ДНК. Альтернативно это снижение представляло собой артефакт, вводимый получением библиотек NGS, связанное с ПЦР. Чтобы исключить вторую возможность, библиотеки NGS получали непосредственно из очищенной включенной в капсид ДНК rAAV без ПЦР-амплификации. При таком подходе охват рассматриваемых последовательностей восстанавливали до уровней, сравнимых с оставшейся частью кассеты. Таким образом, как клетки HEK 293, так и клетки Sf9 поддерживали упаковку полноразмерных кассет rAAV, при этом практически отсутствовали доказательства незначительного усечения.
Геномная идентичность кассеты rAAV-Bac-UF26 Высокая глубина секвенирования кассеты AAV обеспечивала подробный анализ идентичности упакованных последовательностей ДНК и корреляции со ее соответствующей родительской бактериальной плазмидой. Для снижения вероятности ложных клеток и повышения достоверности анализа однонуклеотидного полиморфизма (SNP) использовали только данные NGS из образцов без использования ПЦР. Варианты SNP для ДНК от обоих вирусных образцов, а также образец положительной контрольной плазмиды, характеризовались очень аналогичными профилями замен (коэффициент корреляции, составляющий 0,75-0,77) (Фиг. 9А). Интересно, что большинство SNP были колокализованы с некодирующими последовательностями, идентифицированными выше в виде участков с высоким содержанием GC (Фиг. 9В): промотора гена β-актина птиц и интронной последовательности. Кроме того, и в соответствии с Lecomte et al. 26, TR AAV характеризовались сравнительно высокой изменчивостью SNP.
Относительно более низкая активность векторов rAAV AAV5 и AAV8, созданных на основе клеток насекомых, ранее была описана Urabe et al. 10 и Kohlbrenner et al. 9 Затем было показано, что много других серотипов AAV, происходящих из OneBac, характеризовались более низкой инфицирующей способностью по сравнению с AAVs, происходящими из 293 27. Обобщенной причиной всех пораженных серотипов была модифицированная последовательность капсидного вспомогательного гена, которая приводила к более низкому содержанию капсидного белка VP1, включающего активность фосфолипазы А2. Как и ожидалось, рекомендованные решения были направлены на нормализацию этой проблемы с помощью другого кодона инициации трансляции CUG 11 или искусственного интрона 8, 12. Даже несмотря на то, что новые разработки способствовали повышению инфицирующей способности в случае некоторых векторов, по-видимому, решения характеризовались отсутствием гибкости для корректировки специфической по отношению к серотипам последовательности.
В настоящем документе описан новый подход для повышения относительного содержания VP1/VP2 капсида. Его осуществляли в результате модификации канонической последовательности Козак, предшествующей старт-кодону AUG VP1. В качестве доказательства принципа, диапазон последовательности Козак исследовали по отношению к серотипам AAV5 и AAV9, в результате чего было показало, что наиболее благоприятными участками инициации трансляции были специфическое по отношению к серотипу продуцирование векторов rAAV, которое превосходило эффективности трансдукции аналогов, происходящих из HEK 293. Однако для описанного подхода требуется тонкое регулирование последовательности Козак VP1 в узком окне относительных эффективностей TIS, которое также меняется в случае различных серотипов. Одной из причин этой изменчивости является то, что соотношения VP1:VP2:VP3 зависят не только от относительной эффективности TIS VP1, но также и от таковых TIS VP2 и VP3, которые также различаются в случае всех серотипов (Таблица 11). Более того, по-видимому, намеренное повышение содержания VP1 выше определенного порогового значения (например, Фиг. 4В, линии 1 и 6) является непродуктивным, поскольку выход вируса резко снижается, как только соотношение VP1:VP2:VP3 слишком смешается от теоретического соотношения 1:1:10. Можно получить «консенсусный» VP1TIS: 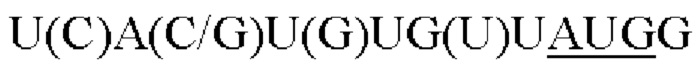 (SEQ ID NO: 50) с пониманием того, что конкретные TIS для определенных применений могут быть идентифицированы эмпирически.
(SEQ ID NO: 50) с пониманием того, что конкретные TIS для определенных применений могут быть идентифицированы эмпирически.
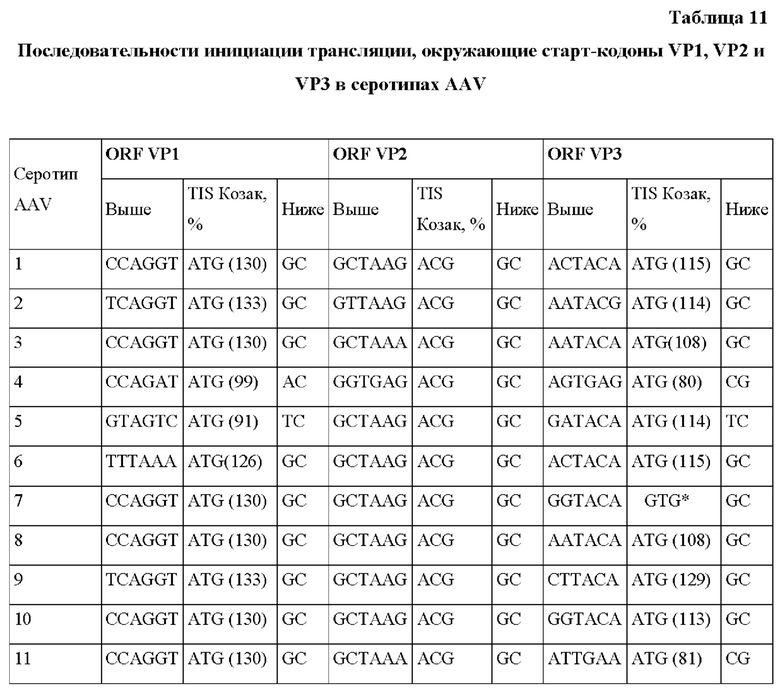
* обозначает предположительный альтернативный кодон инициации VP3 в AAV7, установленный на основании выравнивания последовательностей капсидных генов серотипов AAV. Относительная сила TIS Козак выражается в % ближе к кодону инициации трансляции18
В конструкции rAAV5, выбранной для анализа, количественное значение относительного содержания VP1 повышалось от 0,2-0,4 в векторе, происходящем из HEK 293 (на основе двух независимых анализов), до 0,7-1,1 в клетках Sf9 (например, повышалось в среднем в 3 раза). В случае VP2 эти значения повышались от 0,5 до 1,7, т.е. наблюдалось аналогичное 3-кратное повышение. Сопутствующее повышение VP2 было одним из непредсказуемых эффектов относительного повышения VP1 rAAV5. N-концы как VP1, так и VP2 включают так называемый основной участок 3 (BR3, PKRKKART (SEQ ID NO: 51)), представляющий собой консервативный мотив последовательности ядерной локализации (NLS), который пригоден для того, чтобы AAV доставил свой геном в ядре и затем трансдуцировал клетки 28-30. Таким образом, неудивительно, что 3-кратное повышение каждого VP1 и VP2 повышает выход более инфекционного вируса.
Данные, представленные выше, не включали прямого сравнения rAAV5 и rAAV9, полученных в недавно описанной системе OneBac2.0 8 и текущей системе. Однако можно связать соответствующие капсидные соотношения и сделать заключение, что в случае этих серотипов вновь разработанные вспомогательные гены cap могли значимо улучшать стехиометрическое соотношение как капсида AAV5, так и капсида AAV9, что трансформировалось в более высокоэффективные вирусные векторы.
Другим неожиданным результатом было сходство эффективностей упаковки, проявляемых клетками HEK 293 и Sf9, которые оценивали с помощью косвенного параметра соотношений полных и пустых частиц. С помощью ранее выявленного соотношения кассет экспрессии rep/cap, составляющего 1:2,5, для конструкции стабильной линии клеток-продуцентов 7, аналогичной HEK 293, достигали эффективности упаковки.
Во многих текущих клинических исследованиях определение генетической идентичности исходных культур rAAV, полученных с помощью различных протоколов, становится актуальным вопросом нормативно-правового регулирования. Многие группы сообщали о коллатеральном включении в капсид последовательностей, происходящих в результате упаковки клеток-хозяев 26, 31, 32, остовов бактериальных плазмид-помощников 26, 33, вирусов-помощников 8, 32 и последовательностей rep/cap AAV дикого типа 26, 34. Для оценки генетической идентичности упакованного rAAV проводили анализ NGS включенной в капсид однонитевой вирусной ДНК. В пилотном анализе был показан неодинаковый охват последовательностей кассет в последовательностях с высоким содержанием GC, которые были почти идентичны для обоих платформ. С помощью протокола, в котором не используется ПЦР, было показано, что снижение охвата, по-видимому, происходило в результате ПЦР-индуцированных артефактов во время получения библиотек, а не в результате упаковки усеченных геномов rAAV 35, 36. Точность способов на основе ПЦР в случае получения библиотек NGS ставилась под сомнение несколькими группами, как неподходящих для связанного с AAV анализа 37, особенно в палиндромах с высоким содержанием GC, таких как инвертированные концевые повторы (ITR), или при использовании в отношении к последовательности с высоким содержанием GC 38. Соответственно, анализ NGS препаратов на основе векторов rAAV должен проводиться с помощью соответствующих протоколов.
Анализ генетической идентичности вирусной ДНК, происходящей из обоих платформ, показал отсутствие значимых различий между включенной в капсид ДНК rAAV в клетках насекомых по сравнению с клетками человека. Однако следует отметить факт описанного в настоящем документе отклонения последовательности из пула на основе плазмидной ДНК pTR-Bac-UF26, используемой для трансфекции клеток HEK 293, от последовательностей этой плазмиды в базе данных. Факт такого генетического дрейфа плазмидной ДНК, хотя и неожиданный, можно объяснить следующим механизмом. Плазмиды, содержащие кассеты rAAV, размножаются в штаммах Е. coli, у которых отсутствует рекомбинация, в целях поддержания целостности ITR AAV. У этих штаммов отсутствуют компоненты путей (endA, recB recJ), которые катализируют переупорядочение и удаление нестандартных вторичных и третичных структур. В результате, плазмидная ДНК накапливает ошибочно спаренные основания и точковые мутации, которые иначе восстановились бы с помощью системы исправления ошибок спаривания оснований. Ошибочные спаривания в ДНК при отсутствии репарации приводят к высокой частоте спонтанных мутаций. Таким образом, во время трансфекции мутировавшая плазмидная ДНК несет свою гетеродуплексную «имринтинговую» структуру в ядра клеток-хозяев HEK 293, где она либо репарируется, 39 либо реплицируется, копируя мутацию. Таким образом, пул на основе плазмидной ДНК в протоколе котрансфекции переносит некоторую часть груза вследствие мутагенеза Е. coli в кассеты с заключенными в капсид AAV, что могло быть значительным для некоторых последовательностей с высоким содержанием GC. Интересно, что cDNA репортера GFP, по-видимому, имело минимальное число мутаций (Фиг. 8А), которые могли отражать ее синтетическое «гуманизированное» происхождение 40. В этом отношении расход мотивов CpG из кассет, помимо его связанных с иммуногенностью эффектов 41, мог также способствовать повышению генетической стабильности родительской плазмидной ДНК.
Прямой параллельный анализ NGS кассет rAAV, полученных с помощью двух платформ, неожиданно показал более высокую точность упаковки вирусной ДНК в клетках насекомых, включающих в капсид значительно менее контаминированную ДНК. Такое 6-кратное снижение, по-видимому, является значимым в том случае, если учитывается, что в указаниях FDA отмечается, «что уровень остаточной клеточной ДНК не должно быть ≤10 нг на дозу» fda.gov/downloads/AdvisoryCommittees/CommitteesMeetingMaterials/BloodVaccinesandOtherBiologics/VaccinesandRelatedBiologicalProductsAdvisoryCommittee/UCM319573.pdf).
С учетом текущих клинических исследований с использованием rAAV, в которых дозы достигали до 6×1013 vg/кг 42, 43, вектор, в случае его продукции в клетках HEK 293, включал бы количества контаминирующей ДНК, которые бы превышали таковые в указаниях FDA. В настоящем документе было показано, что векторы rAAV5, продуцируемые с помощью системы OneBac были значимо более инфекционными, и в то же время включали меньшее количество чужеродной ДНК. Таким образом, этот способ продукции повышал терапевтический результат введения вектора в результате снижения его эффективной дозы и повышения безопасности.
Материалы и методы
Следующие неограничивающие материалы и методы использовали в связи с определенными экспериментами, описанными в настоящей заявке. Эти или другие материалы и методы можно использовать в связи с вариантами осуществления, описанными в настоящей заявке.
Продукция rAAV5 и rAAV9 в клетках HEK 293
Как векторы rAAV5, так и векторы rAAV9 продуцировали с помощью процедуры тройной котрансфекции, как описано ранее 44. Плазмиды, несущие кассеты rAAV (rAAV5-Bac-UF26 или rAAV5-UF50-BC, а также rAAV9-Bac-UF26, Фиг. 10А-10В) использовали в комбинации с pHelper и pRep2Cap5 (или pRep2Cap9) в молярном соотношении 1:1:1. Как rAAV5, так и rAAV9 очищали с помощью последовательных двойных циклов центрифугирований с использованием йодиксанола в растворе с градиентом плавучей плотности. В частности, фракцию йодиксанола после первого цикла, содержащую полные частицы, разбавляли 1× PBS-MK буфером в 2,5 раза и использовали вместо стадия с использованием 15% йодиксанола -1 М NaCl в стандартном градиенте 44.
Продукция rAAV5 и rAAV9 в клетках Sf9
Кассеты rAAV Bac-UF26 или UF50-BC включали в остов pFastBac и соответствующие BEV получали в соответствии с руководствами системы Bac-to-Bac. Очищенные от бляшек BEV размножали до Р3, титровали с помощью метода бляшек и использовали для инфицирования стабильных клеточных линий на основе Sf9, несущих rep2cap5 или rep2cap9-индуцибельные вспомогательные гены, как описано ранее 7. При сборе лизаты, образовавшиеся при замораживании и оттаивании, обрабатывали бензоатом и осветляли с помощью высокоскоростного центрифугирования при 20000 g в течение 30 минут. Супернатанты очищали, как описано выше, в случае AAV, полученных на основе 293 HEK.
Одностадийная очистка rAAV5 и rAAV9. rAAV5 очищали из осветленного неочищенного лизата с помощью смолы для высокоэффективной аффинной хроматографии с использованием сефарозы AVB (GE Healthcare), как описано ранее 45. rAAV9 очищали с помощью одностадийной аффинной хроматографии с использованием смолы POROS CAPTURESELECT™ AAV9 (Thermo Fisher) из неочищенного лизата. В частности, осветленный неочищенный лизат наносили в условиях гравитации на колонку, содержащую 0,5 мл смолы, затем колонку промывали 10 объемами колонки 1×PBS, после чего 1 мл буфером для элюирования: 50 мМ цитратный буфер рН 3,0-1 М NaCl. Элюированный rAAV9 незамедлительно нейтрализовали с помощью 0,1 мл 1 М TrisHCl рН 8,4.
Титрование rAAV
Для прямого сравнительного анализа векторов rAAV требовалась точная оценка соответствующих титров вируса. Использовали четыре независимых анализа для титрования rAAV, происходящего из клеток HEK 293 или Sf9: 1) цифровую капельную ПЦР (ddPCR) 46 - для определения эталонного стандарта с помощью цифровой системы ПЦР BioRad QX200; 2) количественную сравнительную ПЦР (QC-PCR) с использованием набора iTaq™ Universal SYBR® Green Supermix (BioRad, 1725121) и системы qPCR BioRad CFX Connect RealTime; 3) протокол на основе PicoGreen 47, и 4) анализ трекинга наночастиц (NTA) с использованием NanoSight 300 (NS-300, Malvern Instruments, Малверн, Великобритания) для количественной оценки тритра и размера частиц rAAV. После определения условий каждого протокола и использования эталонного стандарта, полученного с помощью ddPCR, рассчитанные титры из протоколов 2) и 3) всегда находились в пределах множителя 2, и усреднялись с получением рабочих титров. Вкратце, следовали такие процедуры.
ddPCR. Совокупность разбавлений, в тройных повторностях, в диапазоне 1-103 копий вирусных геномов на реакцию получали с помощью 1× лактатного раствора Рингера (LR) с добавлением 0,05% Pluronic F-68. DdPCR проводили с помощью следующих праймеров: TM_CMV_F 5'-ATAGGGACTTTCCATTGACGTC-3' (SEQ ID NO: 52), TM_CMV_R 5'-TGATACACTTGATGTACTGCCAAG-3' (SEQ ID NO: 53), TM_CMV_Probe FAM 5'-TGGGTGGACTATTTACGGTAAACTGCC-3' BHQ (SEQ ID NO: 54).
QC-PCR. Для получения стандартной кривой кассету на основе трансгенного rAAV-UF26 очищали в гель после расщепления SmaI соответствующей плазмиды pTR-Bac-UF26. Извлеченную ДНК разбавляли до примерной концентрации 1 нг/мкл и точную концентрацию определяли с помощью анализа dsDNA QUBIT. Прямое титрование с использованием QC-PCR высокоочищенных rAAV, продуцируемых в Sf9 или HEK 29, выполняли с набором тех же самых праймеров и с помощью стандартной кривой, полученной с помощью 10-кратных серийных разбавлений эталонного образца rAAV (ddPCR) и очищенной в геле кассеты rAAV (QC-PCR).
Анализ на основе PicoGreen проводили с помощью набора для анализа Quanti-iT PicoGreen dsDNA (Life Technology, P7589), с использованием стандарта ДНК Lambda для калибровки стандартной кривой, как описано 47. Оптическую плотность измеряли с помощью Perkin Elmer 1420 Multilabel Counter Victor V.
NTA. До анализа NTA титры исходных вирусных культур оценивали с помощью PAAG-гель-электрофореза. Зная примерные титры, 50 мкл исходной культуры AAV, разбавленной в буфере LR, добавляли к метящей смеси, содержащей метящий буфер (20 мМ лимонной кислоты, рН 3,5, 0,1% Pluronic F68, 1 мМ NaCl) и наночастиц на основе золота (Sigma, № по кат. 741949). Через 30 минут после инкубирования при RT меченый золотом AAV разбавляли с помощью метящего буфера до конечной концентрации 5×108-3×109 частиц/мл. Метящую смесь без AAV и AAV в метящем буфере без золота использовали в качестве отрицательного контроля. Измерения проводили с помощью инструмента NS-300 со следующими настройками: тип лазера Blue488, уровень фотоаппарата - 15, число кадров - 749, время записи - 30 сек, число записей - 3 на каждую точку данных. По меньшей мере четыре точки данных, генерированные с помощью NS-300, использовали для расчета титра AAV.
MALDI-TOF Полосы VP1, VP2 и VP3 отрезали от SDS-PAAG и готовили для расщепления трипсином. Капсидные белки расщепляли в гелевых слоях трипсином со степенью чистоты для секвенирования от Promega (Мадисон, Висконсин) с использованием рекомендованного производителем протокола. Вкратце, полосы подрезала как можно ближе, чтобы свести к минимуму фоновый полиакриламидный материал. Затем гелевые фрагменты промывали в наночистой H2O в течение 5 минут. Стадию промывания повторяли дважды с последующими двумя циклами обесцвечивания смесью 1:1 об./об. метанол: 50 мМ аммония бикарбонат в течение 10 минут на каждый цикл. Гелевые фрагменты дегидрировали смесью 1:1 об./об. ацетонитрил : 50 мМ аммония бикарбоната. Гелевые фрагменты регидрировали и инкубировали с раствором дитиотреитола (DTT) (25 мМ в 100 мМ аммония бикарбоната) в течение 30 минут перед добавлением 55 мМ йодоацетамида в 100 мМ растворе аммония бикарбоната. Йодоацетамид инкубировали с гелевыми фрагментами в темноте в течение 30 минут. Гелевые фрагменты промывали повторно в двух циклах H2O и дегидрировали с помощью смеси 1:1 об./об. ацетонитрил : 50 мМ аммония бикарбонат. Протеазу вводили в гелевые фрагменты с помощью регидратации в 12 нг/мл трипсина в 0,01% растворе поверхностно-активного вещества ProteaseMAX в течение 5 минут. На гелевый фрагмент затем накладывали 40 мкл смеси 0,01% раствора поверхностно-активного вещества ProteaseMAX: 50 мМ аммония бикарбоната и осторожно смешивали на шейкере в течение 1 часа. Расщепление останавливали в результате добавления 0,5% TFA. МС-анализ либо проводили незамедлительно для получения триптических пептидов высокого качества с минимальным неспецифическим расщеплением, либо образцы замораживали при -80°С до того момента, когда их можно было анализировать. 18O-меченое расщепление выполняли таким же образом, за исключением использования поверхностно-активного вещества ProteaseMAX, и трипсин получали в H218O. В целях предупреждения обратного обмена трипсин инактивировали при 100°С в течение 15 минут. VP3 расщепляли с помощью H218O, в то время как VP1 расщепляли в обычной Н216О; продукты расщепления смешивали в соотношении 1:1 и анализировали с помощью MALDI-TOF. Аналогичный анализ также проводили в случае VP2/VP3.
MALDI-TOF выполняли на масс-спектрометре Bruker Daltonics Microflex LRF (Bruker Daltonics, Бремен, Германия), применяемым в режиме рефлектрона с положительным ионов с использованием лазера N2. Мощность лазера использовали при пороговом уровне, необходимом для генерации сигнала. Инструмент калибровали с помощью стандарта для калибровки пептидов II (Bruker Daltonics), который представлял собой смесь ангиотензина II, ангиотензина I, вещества Р, бомбезина, АСТН clip 1-17, АСТН clip 18-39, соматостатина 28, фрагмента брадикинина 1-7, ренинового субстрата тетрадекапептда свиного с охватываемым диапазоном масс ~700 Да - 3200 Да. α-Циано-4-гидроксикоричную кислоту использовали в качестве матрицы и получали в виде насыщенных растворов в 50% ACN/0,1% TFA (в H2O). Части 1 мкл матрицы и 1 мкл образца тщательно смешивали вместе; 0,5 мкл этого наносили на целевую пластину и сушили.
Анализ трансдукции in vitro. rAAV5-Bac-UF26 и rAAV9-Bac-UF26 анализировали с помощью клеток С12 24, инфицированных rAAV при MOI, составляющем 2000, и ко инфицировали Ad5 при MOI, составляющем 5. Через 48 часов после инфицирования клетки, положительные в отношении флуоресценции mApple, оценивали с помощью флуоресцентно-активируемой клеточной сортировки (FACS).
Инъекции AAV
Все процедуры на животных были одобрены Институциональным комитетом по уходу за животными и их использованию Флоридского университета. Для экспериментов использовали самок BALB/c в возрасте четырех-пяти недель (Jackson Laboratory, Бар-Харбор, Мэн, США).
Все хирургические процедуры выполняли с помощью асептических методик и анестезии изофлураном. Хирургические вмешательства на головном мозге выполняли, как описано ранее 23. Вкратце, после анестезии мышей помещали в стереотактическую рамку (Kopf Instruments, Туюнга, Калифорния), и 2 мкл векторов rAAV5-UF50-BC, происходящих либо из HEK, либо из 8В (4,75×1011 vg/мл), вводили в правую часть полосатого тела (координаты: переднезадний -0,3 мм, латеральный -2,0 мм, дорсовентральный -3,0 мм), с помощью стеклянной микропипетки с внутренним диаметром 30-10 мкм со скоростью 0,5 мкл/минут. Иглу оставляли на месте на 5 минут перед извлечением из головного мозга.
Биолюминесцентная визуализация
Мышей визуализировали, как описано ранее 23. Через 12 минут после интраперитонеальной инъекции D-люциферина (15 мг/мл в PBS, 126 мг люциферина/кг массы тела) получали показатели биолюминесценции из на основе анализа области, представляющей интерес, с помощью системы визуализации Xenogen IVTS Lumia in vivo (PerkinElmer, Уолтем, Массачусетс). Трех мышей визуализировали одновременно в поле зрения, составляющем 25 см. Время визуализации, составляющее 60 с, с группировкой в среде и делением диафрагмы, составляющим 1, использовали для настроек фотоаппарата. Изображения, отображенные в каждой совокупности данных, нормализовали по отношению к соответствующей шкале интенсивности цвета. Данные BLI описывали в виде первичных данных, в виде общего числа импульсов, достигающих детектора прибора с зарядовой связью.
Приготовление и флуоресцентная визуализация образцов головного мозга
Мышей подвергали глубокой анестезии пентобарбиталом (Beuthanasia-D) и перфузировали через восходящую аорту 10 мл солевого раствора, с последующим введением 10 мл охлажденного на льду 4% параформальдегида (PFA) в 0,1 М фосфатном буфере, рН 7,4. Головной мозг удаляли и подвергали последующей фиксации в течение ночи при 4°С в 4% PFA. Коронарные срезы толщиной 60 мкм нарезали на вибратомном столе VT1000 S (Leica Microsystems, Вецлар, Германия). Флуоресценцию mApple анализировали с помощью лазерного сканера с переменным режимом (Typhoon 9200, GE Amersham, Питсбург, Пенсильвания, США) или с помощью инвертированного микроскопа DMI4000 В (Leica Microsystems, Вецлар, Германия).
Анализ NGS
NGS выполняли с помощью UF ICBR Core с использованием инструмента HiSeq 3000 (Illumina, Сан-Диего, Калифорния) и секвенирования спаренных концов. Чтобы показать воспроизводимость некоторых протоколов NGS, все образцы ДНК готовили в двух повторностях. Аналогично все стадии синтеза, секвенирования и биоинформационного анализа библиотеки NGS проводили параллельно и в двух повторностях.
Упомянутая ДНК в случае продукции на основе HEK 293 включала все последовательности ДНК, которые могли потенциально контаминировать исходную культуру rAAV: последовательность генома Н. sapiens (HEK 293, GRCh38.p9, ncbi.nlm.nih.gov/assembly/GCF_000001405.35 или номер доступа RefSeq: GCF 000001405.35); аденовирусной плазмиды-помощника (pHelper, GenBank: AF369965.1); pACGr2c5, кодирующей белки Rep AAV2 и VP AAV5 (rep2cap5), и соответствующие плазмидные остовы. В случае продукции на основе Sf9 анализировали следующие последовательности: геном S. frugiperda (JQCY02.1.fsa_nt.gz, GenBank: JQCY00000000.2); геном AcMNPV (GenBank NC_001623.1); изолят рабдовируса S. frugiperda Sf (GenBank KF947078.1); остов шаттл-плазмиды FastBac и последовательности, кодирующие Rep AAV2 и VP AAV5.
Набор для библиотеки ДНК, полученной без использования ПЦР, ACCEL-NGS® 2S (Swift Biosciences, Анн-Арбор, Мичиган), содержащий опции как без ПЦР, так и с обогащением ПЦР, использовали для получения библиотек NGS. Глубокую обработку ДНКазой очищенных частиц rAAV и синтез двунитевой (ds) ДНК осуществляли согласно протоколу, описанному Lecomte et al., 26 с незначительными модификациями. Вкратце, 4×1011 частиц, содержащих векторные геномы (vg), тщательно обрабатывали экзонуклеазами Baseline ZERO и Plasmid-Safe, с последующей обработкой протеинкиназой K и РНКазой А. ДНК очищали с помощью набора Mag-Bind RxnPure Plus (Omega Bio-Tek, Норкросс, Джорджия) с помощью соотношения гранулы : ДНК, составляющего 2:1. После синтеза второй нити с помощью DNApolI ДНК подвергали ультразвуковой обработке с помощью инструмента Covaris. Использовали следующие настройки для фрагментации ДНК: целевой размер 400 п.о., мощность увеличения пика -175 W, коэффициент заполнения 10%, циклов на импульс 200; время 38 с, уровень воды - 15, температура воды - 7,6-7,8°С, и объем реакции - 47 мкл. Профили фрагментированной входящей dsDNA и синтезированных библиотек NGS представлены на Фиг. 14. Образцы ДНК rAAV, происходящие из клеток Sf9 и HEK 293, характеризовались аналогичными профилями и качеством на каждой стадии во время приготовления библиотеки NGS. На всех стадиях приготовления библиотек качество ДНК контролировали с помощью Таре station (Agilent) и Qubit (Thermo Fisher Scientific). Для синтеза библиотек NGS без использования ПЦР фрагментировали 220 нг dsDNA до целевого размера, составляющего 200 п.о. на входе. Для библиотек с обогащением ПЦР 10% ДНК после стадии 2 лигирования амплифицировали для восьми циклов в соответствии с рекомендациями производителя. В конечном итоге все библиотеки очищали с помощью 1×Mag-Bind гранул.
Биоинформационный анализ
Технологическая карта биоинформационного технологического процесса представлена на Фиг. 15. Файлы Fastq анализировали с использованием специально разработанного открытого программного обеспечения ContaVect v2.0 26, модифицированного для использования в суперкомпьютерном кластере в своей конфигурации по умолчанию. Код ContaVect адаптировали таким образом, чтобы примерно масштабировать выполнение инструментов blastn и bwa для максимальной эффективности и производительности в пакетной среде, контролируемой разработчиком задач. Одно из изменений было связано с ограничением ContaVect, которое проявлялось в том, что запуск blastn и bwa всегда происходил бы с рядом инструкций, равных общему числу ядер ЦП на вычислительном узле. Несмотря на то, что это предположение может быть верным в некоторых случаях при использовании персонального автоматизированного рабочего места в общей вычислительной среде, число инструкций, как правило, должно равняться числу ядер ЦР, требующихся для работы в пакетном режиме, или доступным в виртуальной машине. При других обстоятельствах эффективность упомянутой программы существенно страдает от снижения производительности анализа. Новые модификации все еще позволяют применять старую модель в целях совместимости, если число инструкций устанавливается на нуль в файле конфигурации ContaVect, однако в настоящее время ContaVect может запускать blastn и bwa с определенным числом инструкций. Одна инструкция используется по умолчанию. Кроме того, был добавлен необязательный файл конфигурации, который экспортировали в среду посредством среды MATPLOTLIBRC, которая варьирует с тем, чтобы обеспечить построение графиков в ContaVect с помощью matplotlib без сбоев при запуске на кластерных узлах без среды X11. С учетом вышеуказанных модификаций анализ был успешно завершен на суперкомьютере UF Research Computing HiPerGator, а время работы ContaVect по обработке данных было снижено от нескольких дней до нескольких часов. Все модификации были отправлены в виде запросов на включение GitHub авторам ContaVect в следующие репозитории: github.com/a-slide/ContaVect and github.com/a-slide/pyDNA. Все описанные адаптации были подтверждены и включены в программный код. Варианты SNP извлекали из файлов ВАМ с помощью компьютерной программы MiniCaller, приложения на основе Java, используемого для анализа последовательностей с высокой глубиной охвата (github.com/lindenb/jvarkit/wiki/MiniCaller). Созданные в MiniCaller файлы VCF для библиотек rAAV из HEK 293 или Sf9, а также библиотеки положительного контроля (каждая в двойном экземпляре) выравнивали по отношению к эталонным молекулам GenBank. Парсинг файлов VCF выполняли с помощью скрипта Python, который генерирует файлы CSV (nbviewer.ipython.org/github/a-slide/iPython-Notebook/blob/master/Notebooks/VCF_analysis.ipynb). Файлы VCF и CSV доступны из репозитория Dryad Digital: bio.rc.ufl.edu/secure/zolotukhin/genther/; имя пользователя: «genther», пароль: «KrAkkegZ8F».
Литературные источники
1. Girod, A, Wobus, СЕ, Zadori, Z, Ried, М, Leike, K, Tijssen, Р et al. (2002). The VP1 capsid protein of adeno-associated virus type 2 is carrying a phospholipase A2 domain required for virus infectivity. J Gen Virol 83: 973-978.
2. Rose, JA, Maizel, JV, Jr., Inman, JK & Shatkin, AJ. (1971). Structural proteins of adenovirus-associated viruses. J Virol 8: 766-770.
3. Johnson, FB, Ozer, HL & Hoggan, MD. (1971). Structural proteins of adenovirus-associated virus type 3. J Virol 8: 860-863.
4. Buller, RM & Rose, JA. (1978). Characterization of adenovirus-associated virus-induced polypeptides in KB cells. J Virol 25: 331-338.
5. Snijder, J, van de Waterbeemd, M, Damoc, E, Denisov, E, Grinfeld, D, Bennett, A et al. (2014). Defining the stoichiometry and cargo load of viral and bacterial nanoparticles by Orbitrap mass spectrometry. J Am Chem Soc 136: 7295-7299.
6. Urabe, M, Ding, С & Kotin, RM. (2002). Insect cells as a factory to produce adeno-associated virus type 2 vectors. Hum Gene Ther 13: 1935-1943.
7. Aslanidi, G, Lamb, K & Zolotukhin, S. (2009). An inducible system for highly efficient production of recombinant adeno-associated virus (rAAV) vectors in insect Sf9 cells. Proc Natl Acad Sci USA 106: 5059-5064.
8. Mietzsch, M, Casteleyn, V, Weger, S, Zolotukhin, S & Heilbronn, R. (2015). OneBac 2.0: Sf9 Cell Lines for Production of AAV5 Vectors with Enhanced Infectivity and Minimal Encapsidation of Foreign DNA. Hum Gene Ther 26: 688-697.
9. Kohlbrenner, E, Aslanidi, G, Nash, K, Shklyaev, S, Campbell-Thompson, M, Byrne, BJ et al. (2005). Successful production of pseudotyped rAAV vectors using a modified baculovirus expression system. Mol Ther 12: 1217-1225.
10. Urabe, M, Nakakura, T, Xin, KQ, Obara, Y, Mizukami, H, Kume, A et al. (2006). Scalable generation of high-titer recombinant adeno-associated virus type 5 in insect cells. J Virol 80: 1874-1885.
11. Urabe, M, Ozawa, K, Haast, S & Hermens, W in EP 2311967 A2 20110420 (EN) (Amsterdam Molecular Therapeutics (AMT) B.V., 2015).
12. Chen, H. (2008). hitron splicing-mediated expression of AAV Rep and Cap genes and production of AAV vectors in insect cells. Mol Ther 16: 924-930.
13. Kozak, M. (1999). Initiation of translation in prokaryotes and eukaryotes. Gene 234: 187-208.
14. Cigan, AM & Donahue, TF. (1987). Sequence and structural features associated with translational initiator regions in yeast--a review. Gene 59: 1-18.
15. Joshi, CP, Zhou, H, Huang, X & Chiang, VL. (1997). Context sequences of translation initiation codon in plants. Plant Mol Biol 35: 993-1001.
16. Pesole, G, Gissi, C, Grillo, G, Licciulli, F, Liuni, S & Saccone, C. (2000). Analysis of oligonucleotide AUG start codon context in eukariotic mRNAs. Gene 261: 85-91.
17. Kozak, M. (1987). An analysis of 5'-noncoding sequences from 699 vertebrate messenger RNAs. Nucleic Acids Res 15: 8125-8148.
18. Noderer, WL, Flockhart, RJ, Bhaduri, A, Diaz de Arce, AJ, Zhang, J, Khavari, PA et al. (2014). Quantitative analysis of mammalian translation initiation sites by FACS-seq. Mol Syst Biol 10: 748.
19. Salganik, M, Venkatakrishnan, B, Bennett, A, Lins, B, Yarbrough, J, Muzyczka, N et al. (2012). Evidence for pH-dependent protease activity in the adeno-associated virus capsid. J Virol 86: 11877-11885.
20. Storms, HF, van der Heijden, R, Tjaden, UR & van der Greef, J. (2006). Considerations for proteolytic labeling-optimization of 18O incorporation and prohibition of back-exchange. Rapid Commun Mass Spectrom 20: 3491-3497.
21. Ye, X, Luke, B, Andresson, T & Blonder, J. (2009). 18O stable isotope labeling in MS-based proteomics. Brief Fund Genomic Proteomic 8: 136-144.
22. Qian, WJ, Liu, T, Petyuk, VA, Gritsenko, MA, Petritis, BO, Polpitiya, AD et al. (2009). Large-scale multiplexed quantitative discovery proteomics enabled by the use of an (18)O-labeled "universal" reference sample. J Proteome Res 8: 290-299.
23. Marsic, D, Mendez-Gomez, HR & Zolotukhin, S. (2015). High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Mol Ther Methods Clin Dev 2: 15041.
24. Clark, KR, Voulgaropoulou, F & Johnson, PR. (1996). A stable cell line carrying adenovirus-inducible rep and cap genes allows for infectivity titration of adeno-associated virus vectors. Gene Ther 3: 1124-1132.
25. Ma, H, Galvin, ТА, Glasner, DR, Shaheduzzaman, S & Khan, AS. (2014). Identification of a novel rhabdovirus in Spodoptera frugiperda cell lines. J Virol 88: 6576-6585.
26. Lecomte, E, Tournaire, B, Cogne, B, Dupont, JB, Lindenbaum, P, Martin-Fontaine, M et al. (2015). Advanced Characterization of DNA Molecules in rAAV Vector Preparations by Single-stranded Virus Next-generation Sequencing. Mol Ther Nucleic Acids 4: e260.
27. Mietzsch, M, Grasse, S, Zurawski, C, Weger, S, Bennett, A, Agbandje-McKenna, M et al. (2014). OneBac: platform for scalable and high-titer production of adeno-associated virus serotype 1-12 vectors for gene therapy. Hum Gene Ther 25: 212-222.
28. Grieger, JC, Snowdy, S & Samulski, RJ. (2006). Separate basic region motifs within the adeno-associated virus capsid proteins are essential for infectivity and assembly. J Virol 80: 5199-5210.
29. Sonntag, F, Bleker, S, Leuchs, B, Fischer, R & Kleinschmidt, JA. (2006). Adeno-associated virus type 2 capsids with externalized VP1/VP2 trafficking domains are generated prior to passage through the cytoplasm and are maintained until uncoating occurs in the nucleus. J Virol 80: 11040-11054.
30. Popa-Wagner, R, Sonntag, F, Schmidt, K, King, J & Kleinschmidt, JA. (2012). Nuclear translocation of adeno-associated virus type 2 capsid proteins for virion assembly. J Gen Virol 93: 1887-1898.
31. Allay, JA, Sleep, S, Long, S, Tillman, DM, Clark, R, Carney, G et al. (2011). Good manufacturing practice production of self-complementary serotype 8 adeno-associated viral vector for a hemophilia В clinical trial. Hum Gene Ther 22: 595-604.
32. Ye, GJ, Scotti, MM, Liu, J, Wang, L, Knop, DR & Veres, G. (2011). Clearance and characterization of residual HSV DNA in recombinant adeno-associated virus produced by an HSV complementation system. Gene Ther 18: 135-144.
33. Chadeuf, G, Ciron, C, Moullier, P & Salvetti, A. (2005). Evidence for encapsidation of prokaryotic sequences during recombinant adeno-associated virus production and their in vivo persistence after vector delivery. Mol Ther 12: 744-753.
34. Nony, P, Chadeuf, G, Tessier, J, Moullier, P & Salvetti, A. (2003). Evidence for packaging of rep-cap sequences into adeno-associated virus (AAV) type 2 capsids in the absence of inverted terminal repeats: a model for generation of rep-positive AAV particles. J Virol 77: 776-781.
35. Kapranov, R Chen, L, Dederich, D, Dong, B, He, J, Steinmann, KE et al. (2012). Native molecular state of adeno-associated viral vectors revealed by single-molecule sequencing. Hum Gene Ther 23: 46-55.
36. Wang, Y, Ling, C, Song, L, Wang, L, Aslanidi, GV, Tan, M et al. (2012). Limitations of encapsidation of recombinant self-complementary adeno-associated viral genomes in different serotype capsids and their quantitation. Hum Gene Ther Methods 23: 225-233.
37. Cogne, B, Snyder, R, Lindenbaum, P, Dupont, JB, Redon, R, Moullier, P et al. (2014). NGS library preparation may generate artifactual integration sites of AAV vectors. Nat Med 20: 577-578.
38. Kozarewa, I, Ning, Z, Quail, MA, Sanders, MJ, Berriman, M & Turner, DJ. (2009). Amplification-free Illumina sequencing-library preparation facilitates improved mapping and assembly of (G+C)-biased genomes. Nat Methods 6: 291-295.
39. Folger, KR, Thomas, K & Capecchi, MR. (1985). Efficient correction of mismatched bases in plasmid heteroduplexes injected into cultured mammalian cell nuclei. Mol Cell Biol 5: 70-74.
40. Zolotukhin, S, Potter, M, Hauswirth, WW, Guy, J & Muzyczka, N. (1996). A "humanized" green fluorescent protein cDNA adapted for high-level expression in mammalian cells. J Virol 70: 4646-4654.
41. Faust, SM, Bell, P, Cutler, BJ, Ashley, SN, Zhu, Y, Rabinowitz, JE et al. (2013). CpG-depleted adeno-associated virus vectors evade immune detection. J Clin Invest 123: 2994-3001.
42. Herzog, RW. (2016). A Cure For Hemophilia: the Promise Becomes a Reality. Mol Ther 24: 1503-1504.
43. Dolgin, E. (2016). Early clinical data raise the bar for hemophilia gene therapies. Nat Biotechnol 34: 999-1001.
44. Zolotukhin, S, Byrne, BJ, Mason, E, Zolotukhin, I, Potter, M, Chesnut, K et al. (1999). Recombinant adeno-associated virus purification using novel methods improves infectious titer and yield. Gene Ther 6: 973-985.
45. Smith, RH, Levy, JR & Kotin, RM. (2009). A simplified baculovirus-AAV expression vector system coupled with one-step affinity purification yields high-titer rAAV stocks from insect cells. Mol Ther 17: 1888-1896.
46. Lock, M, Alvira, MR, Chen, SJ & Wilson, JM. (2014). Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Ther Methods 25: 115-125.
47. Piedra, J, Ontiveros, M, Miravet, S, Penalva, C, Monfar, M & Chillon, M. (2015). Development of a rapid, robust, and universal picogreen-based method to titer adeno-associated vectors. Hum Gene Ther Methods 26: 35-42.
ДРУГИЕ ВАРИАНТЫ ОСУЩЕСТВЛЕНИЯ
Все из характеристик, раскрытых в настоящем описании, могут комбинирования в любом сочетании. Каждая характеристика, раскрытая в настоящем описании, может быть заменена альтернативной характеристикой, служащей для выполнения той же самой, эквивалентной или аналогичной цели. Таким образом, если явно не указано иное, то каждая раскрытая характеристика представляет собой только пример характерной последовательности эквивалентных или аналогичных характеристик.
Из приведенного выше описания специалист в данной области может легко установить необходимые характеристики настоящего раскрытия, и не отклоняясь от его смысла и области применения, может выполнить различные изменения и модификации настоящего раскрытия для адаптации его к различному применению и условиям. Таким образом, другие варианты осуществления также находятся в объеме формулы изобретения.
ЭКВИВАЛЕНТЫ
Несмотря на то, что варианты осуществления настоящего изобретения были описаны и проиллюстрированы в настоящем документе, специалисты в данной области легко предусмотрят множество средств и/или структур для осуществления функции и/или получения результатов и/или одного или нескольких преимуществ, описываемых в настоящем документе, и каждое из таких вариаций и/или модификаций описано в объеме настоящего изобретения. В более общем плане специалисты в данной области легко примут во внимание, что все параметры, размеры, материалы и конфигурации, описываемые в настоящем документе, являются неограничивающими, и что фактические параметры, размеры, материалы и/или конфигурации будут зависеть от конкретного применения или применений, для которых используются идеи настоящего изобретения. Специалистам в данной области техники будут понятны многие эквиваленты конкретных вариантов осуществления настоящего изобретения, описанного в настоящем документе, или с помощью проведения только лишь обычных экспериментов они будут способны установить такие эквиваленты. Таким образом, следует понимать, что вышеизложенные варианты осуществления представлены лишь в качестве примера и что в объеме прилагаемой формулы изобретения и эквивалентов к ней варианты осуществления настоящего изобретения могут применяться на практике иначе, чем так, как конкретным образом описано и представлено в формуле изобретения. Варианты осуществления настоящего изобретения направлены на каждую отдельную характеристику, систему, изделие, материал, набор и/или способ, описываемые в настоящем документе. Кроме того, любая комбинация из двух или более таких свойств, систем, изделий, материалов и/или способов в случае, если такие свойства, системы, изделия, материалы, наборы и/или способы не являются взаимно противоречащими, включена в объем настоящего раскрытия.
Все определения, определенные и использованные в настоящем документе, следует понимать в целях контроля за словарными определениями, определениями в документах, включенных посредством ссылки и/или обычных значений определяемых терминов.
Все ссылки, патенты и заявки на патенты, раскрытые в настоящем документе, включены посредством ссылки по отношению к рассматриваемому вопросу, для которого каждое из них цитируется, что в некоторых случаях может охватывать полноту документа.
Формы единственного числа, как используется в настоящем документе в описании и в формуле изобретения, если четко не указано противоположное, необходимо понимать как означающие «по меньшей мере одно».
Фраза «и/или», как используется в настоящем документе в описании и в формуле изобретения, следует понимать как означающую «любое из двух или оба вместе» из элементов таким образом соединенных, например, элементов, которые совместно присутствуют в некоторых случаях и раздельно присутствуют в других случаях. Несколько элементов, приведенных с фразой «и/или», следует подразумевать тем же самым образом, например, что и «один или несколько» из элементов, объединенных таким образом. Могут необязательно присутствовать другие элементы, отличные от элементов, специфическим образом определяемых условием «и/или», связанных или несвязанных с этими элементами, специфическим образом определяемых. Таким образом, в качестве неограничивающего примера отсылка на «А и/или В» при использовании в связи с открытой формулировкой, такой как «содержащий», может относиться в соответствии с одним вариантом осуществления только по отношению к А (при этом необязательно включаются элементы, отличные от В); в соответствии с другим вариантом осуществления только по отношению к В (при этом необязательно включаются элементы, отличные от А); в соответствии с еще одним вариантом осуществления как к А, так и к В (при этом необязательно включаются другие элементы); и т.д.
Как используется в настоящем описании и в формуле изобретения термин «или» необходимо понимать как имеющую тоже самое значение, что и "и/или", как определено выше. Например, при разделении пунктов в перечне фраза «или» или «и/или» будет пониматься как включающая, например, включение по меньшей мере одного, но также включающая более одного, ряда или перечня из элементов, и необязательно дополнительных неприведенных пунктов. Только термины, явно указывающие на противоположное, такие как «только одно из» или «точно одно из» при использовании в формуле изобретения, «состоящий из» будет относиться к включению точно одного элемента из ряда или перечня элементов. Как правило, термин «или», как используется в настоящем документе, будет истолковываться как указывающий на исключающие альтернативы (например, «одно или другое, но не оба») в случае, если ему предшествуют термины исключительности, такие как «либо», «один из», «только один из» или «точно один из». Термин «состоящий по сути из» при использовании в формуле изобретения, будет иметь свое обычное значение, используемое в области патентного права.
Используемую в настоящем документе в описании и в формуле изобретения фразу «по меньшей мере» по отношению к перечню из одного или нескольких элементов необходимо понимать как означающую по меньшей мере один элемент, выбранный из любого одного или нескольких элементов в перечне элементов, но необязательно включающем по меньшей мере одно из каждого и всякого элемента, специфическим образом представленного в перечне элементов, и не исключающем любую комбинацию из элементов в перечне элементов. Это определение также допускает возможность того, что могут необязательно присутствовать элементы, отличные от элементов, специфическим образом определяемых в перечне элементов, к которым относится фраза «по меньшей мере один», вне зависимости от того связаны или не связаны они с этими элементами, специфическим образом определяемыми. Таким образом, в качестве неограничивающего примера «по меньшей мере одно из А и В» (или, что является эквивалентным «по меньшей мере одно из А или В», или, что является эквивалентным «по меньшей мере одно из А и/или В») может относиться в соответствии с одним вариантом осуществления по меньшей мере к одному, необязательно включающему более одного, А, при этом В не присутствует (и необязательно включающему элементы, отличные от В); в соответствии с другим вариантом осуществления по меньшей мере к одному, необязательно включающему более одного, В, при этом А не присутствует (и необязательно включающему элементы, отличные от А); в соответствии с еще одним вариантом осуществления по меньшей мере к одному, необязательно включающему более одного, А, и по меньшей мере к одному, необязательно включающему более одного, В (и необязательно включающему другие элементы); и т.д.
Следует понимать, что, если четко не указано иное, в любых способах, заявляемых в настоящем документе, которые предусматривают одну стадию или действие, порядок стадий или действий способа не обязательно ограничен порядком, при котором стадии или действия способа упомянуты.
В формуле изобретения, а также в описании выше, все переходные фразы, такие как «содержащий», «включающий», «несущий», «имеющий», «содержащий в себе», «включающий в себя», «хранящийся», «состоящий из» и т.п. следует понимать как открытые, т.е., означают включение, но не ограничение. Только переходные фразы «состоящий из» и «состоящий по сути из» будут закрытыми или полузакрытыми переходными фразами соответственно, как изложено в Руководстве по проведению патентной экспертизы Патентного ведомства США, раздел 2111.03. Следует понимать, что также предусмотрены варианты осуществления, описанные в настоящем документе, в которых используется открытая переходная фраза (например, «содержащий»), в альтернативных вариантах осуществления, таких как «состоящий из» и «состоящий по сути из» характеристики, описанной с помощью открытой переходной фразы. Например, если в раскрытии описана «композиция, содержащая А и В», то раскрытие также предусматривает альтернативные варианты осуществления «композиция, состоящая из А и В» и «композиция, состоящая из А и В».
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ЮНИВЕРСИТИ ОФ ФЛОРИДА РИСЕРЧ ФАУНДЭЙШН, ИНКОРПОРЕЙТЕД
<120> СПОСОБЫ УСИЛЕНИЯ БИОЛОГИЧЕСКОЙ АКТИВНОСТИ РЕКОМБИНАНТНОГО
АДЕНОАССОЦИИРОВАННОГО ВИРУСА, ПРОДУЦИРУЕМОГО БАКУЛОВИРУСНОЙ СИСТЕМОЙ
<130> U1197.70082WO00
<140> Еще не присвоен
<141> одновременно с данным документом
<150> US 62/323,684
<151> 2016-04-16
<160> 57
<170> PatentIn version 3.5
<210> 1
<211> 11
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<220>
<221> misc_feature
<222> (1)..(1)
<223> n представляет собой c или t
<220>
<221> misc_feature
<222> (2)..(2)
<223> n представляет собой a, c, g, или t
<220>
<221> misc_feature
<222> (3)..(3)
<223> n представляет собой g или t
<220>
<221> misc_feature
<222> (5)..(5)
<223> n представляет собой g или t
<220>
<221> misc_feature
<222> (10)..(10)
<223> n представляет собой a, c, g, или t
<220>
<221> misc_feature
<222> (11)..(11)
<223> n представляет собой a, c, или t
<400> 1
nnntntatgn n 11
<210> 2
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 2
cauuguaugu c 11
<210> 3
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 3
ucguuuaugg a 11
<210> 4
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 4
caguuuaugg u 11
<210> 5
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 5
cauuguaugg u 11
<210> 6
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 6
uaguguaugc u 11
<210> 7
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 7
cauuguaugc u 11
<210> 8
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 8
ucuuuuaugu c 11
<210> 9
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 9
uguuuuaugu c 11
<210> 10
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 10
uaguuuaugu c 11
<210> 11
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 11
uaguguaugu c 11
<210> 12
<211> 11
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<220>
<221> misc_feature
<222> (1)..(1)
<223> n представляет собой a, c, или t
<220>
<221> misc_feature
<222> (2)..(2)
<223> n представляет собой a, c, g, или t
<220>
<221> misc_feature
<222> (3)..(3)
<223> n представляет собой g или t
<220>
<221> misc_feature
<222> (4)..(4)
<223> n представляет собой c или t
<220>
<221> misc_feature
<222> (5)..(5)
<223> n представляет собой a, c, g, или t
<220>
<221> misc_feature
<222> (6)..(6)
<223> n представляет собой c или t
<220>
<221> misc_feature
<222> (10)..(10)
<223> n представляет собой g или t
<220>
<221> misc_feature
<222> (11)..(11)
<223> n представляет собой c или g
<400> 12
nnnnnnatgn n 11
<210> 13
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 13
uagcgcaugg c 11
<210> 14
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 14
ugguauaugg c 11
<210> 15
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 15
uaguuuaugg c 11
<210> 16
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 16
caguguaugg c 11
<210> 17
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 17
uaguguaugg c 11
<210> 18
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 18
uauuguaugg c 11
<210> 19
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 19
cauuguaugg c 11
<210> 20
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 20
ccguuuaugg g 11
<210> 21
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 21
acuuguaugg g 11
<210> 22
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 22
cauuuuaugg g 11
<210> 23
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 23
uaguguaugu c 11
<210> 24
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 24
uaguuuaugu c 11
<210> 25
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 25
uguuuuaugu c 11
<210> 26
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 26
ucuuuuaugu c 11
<210> 27
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 27
uaguguaugg g 11
<210> 28
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 28
uaguuuaugg g 11
<210> 29
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 29
uguuuuaugg g 11
<210> 30
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 30
ucuuuuaugg g 11
<210> 31
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 31
uaguguaugu c 11
<210> 32
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 32
uaguuuaugu c 11
<210> 33
<211> 11
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<220>
<221> misc_feature
<222> (2)..(2)
<223> n представляет собой a, c, g, или t
<220>
<221> misc_feature
<222> (3)..(3)
<223> n представляет собой g или t
<220>
<221> misc_feature
<222> (5)..(5)
<223> n представляет собой g или t
<220>
<221> misc_feature
<222> (10)..(10)
<223> n представляет собой g или t
<220>
<221> misc_feature
<222> (11)..(11)
<223> n представляет собой c или g
<400> 33
tnntntatgn n 11
<210> 34
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 34
uaguguaugu c 11
<210> 35
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 35
uaguuuaugu c 11
<210> 36
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 36
uguuuuaugu c 11
<210> 37
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 37
ucuuuuaugu c 11
<210> 38
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 38
uaguguaugg g 11
<210> 39
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 39
uaguuuaugg g 11
<210> 40
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 40
uguuuuaugg g 11
<210> 41
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 41
ucuuuuaugg g 11
<210> 42
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 42
uaguguaugg c 11
<210> 43
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 43
uaguuuaugg c 11
<210> 44
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 44
uguuuuaugg c 11
<210> 45
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 45
ucuuuuaugg c 11
<210> 46
<211> 11
<212> РНК
<213> Аденоассоциированный вирус 5
<400> 46
guagucaugu c 11
<210> 47
<211> 11
<212> РНК
<213> Аденоассоциированный вирус 9
<400> 47
ccagguaugg c 11
<210> 48
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 48
Thr Trp Val Leu Pro Ser Tyr Asn Asn His Gln Tyr Arg
1 5 10
<210> 49
<211> 11
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<220>
<221> misc_feature
<222> (4)..(4)
<223> n представляет собой a или g
<400> 49
gccnccaugg c 11
<210> 50
<211> 14
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<220>
<221> misc_feature
<222> (4)..(4)
<223> n представляет собой c или g
<400> 50
ucanuguguu augg 14
<210> 51
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 51
Pro Lys Arg Lys Lys Ala Arg Thr
1 5
<210> 52
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 52
atagggactt tccattgacg tc 22
<210> 53
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 53
tgatacactt gatgtactgc caag 24
<210> 54
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 54
tgggtggact atttacggta aactgcc 27
<210> 55
<211> 724
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 55
Met Ser Phe Val Asp His Pro Pro Asp Trp Leu Glu Glu Val Gly Glu
1 5 10 15
Gly Leu Arg Glu Phe Leu Gly Leu Glu Ala Gly Pro Pro Lys Pro Lys
20 25 30
Pro Asn Gln Gln His Gln Asp Gln Ala Arg Gly Leu Val Leu Pro Gly
35 40 45
Tyr Asn Tyr Leu Gly Pro Gly Asn Gly Leu Asp Arg Gly Glu Pro Val
50 55 60
Asn Arg Ala Asp Glu Val Ala Arg Glu His Asp Ile Ser Tyr Asn Glu
65 70 75 80
Gln Leu Glu Ala Gly Asp Asn Pro Tyr Leu Lys Tyr Asn His Ala Asp
85 90 95
Ala Glu Phe Gln Glu Lys Leu Ala Asp Asp Thr Ser Phe Gly Gly Asn
100 105 110
Leu Gly Lys Ala Val Phe Gln Ala Lys Lys Arg Val Leu Glu Pro Phe
115 120 125
Gly Leu Val Glu Glu Gly Ala Lys Thr Ala Pro Thr Gly Lys Arg Ile
130 135 140
Asp Asp His Phe Pro Lys Arg Lys Lys Ala Arg Thr Glu Glu Asp Ser
145 150 155 160
Lys Pro Ser Thr Ser Ser Asp Ala Glu Ala Gly Pro Ser Gly Ser Gln
165 170 175
Gln Leu Gln Ile Pro Ala Gln Pro Ala Ser Ser Leu Gly Ala Asp Thr
180 185 190
Met Ser Ala Gly Gly Gly Gly Pro Leu Gly Asp Asn Asn Gln Gly Ala
195 200 205
Asp Gly Val Gly Asn Ala Ser Gly Asp Trp His Cys Asp Ser Thr Trp
210 215 220
Met Gly Asp Arg Val Val Thr Lys Ser Thr Arg Thr Trp Val Leu Pro
225 230 235 240
Ser Tyr Asn Asn His Gln Tyr Arg Glu Ile Lys Ser Gly Ser Val Asp
245 250 255
Gly Ser Asn Ala Asn Ala Tyr Phe Gly Tyr Ser Thr Pro Trp Gly Tyr
260 265 270
Phe Asp Phe Asn Arg Phe His Ser His Trp Ser Pro Arg Asp Trp Gln
275 280 285
Arg Leu Ile Asn Asn Tyr Trp Gly Phe Arg Pro Arg Ser Leu Arg Val
290 295 300
Lys Ile Phe Asn Ile Gln Val Lys Glu Val Thr Val Gln Asp Ser Thr
305 310 315 320
Thr Thr Ile Ala Asn Asn Leu Thr Ser Thr Val Gln Val Phe Thr Asp
325 330 335
Asp Asp Tyr Gln Leu Pro Tyr Val Val Gly Asn Gly Thr Glu Gly Cys
340 345 350
Leu Pro Ala Phe Pro Pro Gln Val Phe Thr Leu Pro Gln Tyr Gly Tyr
355 360 365
Ala Thr Leu Asn Arg Asp Asn Thr Glu Asn Pro Thr Glu Arg Ser Ser
370 375 380
Phe Phe Cys Leu Glu Tyr Phe Pro Ser Lys Met Leu Arg Thr Gly Asn
385 390 395 400
Asn Phe Glu Phe Thr Tyr Asn Phe Glu Glu Val Pro Phe His Ser Ser
405 410 415
Phe Ala Pro Ser Gln Asn Leu Phe Lys Leu Ala Asn Pro Leu Val Asp
420 425 430
Gln Tyr Leu Tyr Arg Phe Val Ser Thr Asn Asn Thr Gly Gly Val Gln
435 440 445
Phe Asn Lys Asn Leu Ala Gly Arg Tyr Ala Asn Thr Tyr Lys Asn Trp
450 455 460
Phe Pro Gly Pro Met Gly Arg Thr Gln Gly Trp Asn Leu Gly Ser Gly
465 470 475 480
Val Asn Arg Ala Ser Val Ser Ala Phe Ala Thr Thr Asn Arg Met Glu
485 490 495
Leu Glu Gly Ala Ser Tyr Gln Val Pro Pro Gln Pro Asn Gly Met Thr
500 505 510
Asn Asn Leu Gln Gly Ser Asn Thr Tyr Ala Leu Glu Asn Thr Met Ile
515 520 525
Phe Asn Ser Gln Pro Ala Asn Pro Gly Thr Thr Ala Thr Tyr Leu Glu
530 535 540
Gly Asn Met Leu Ile Thr Ser Glu Ser Glu Thr Gln Pro Val Asn Arg
545 550 555 560
Val Ala Tyr Asn Val Gly Gly Gln Met Ala Thr Asn Asn Gln Ser Ser
565 570 575
Thr Thr Ala Pro Ala Thr Gly Thr Tyr Asn Leu Gln Glu Ile Val Pro
580 585 590
Gly Ser Val Trp Met Glu Arg Asp Val Tyr Leu Gln Gly Pro Ile Trp
595 600 605
Ala Lys Ile Pro Glu Thr Gly Ala His Phe His Pro Ser Pro Ala Met
610 615 620
Gly Gly Phe Gly Leu Lys His Pro Pro Pro Met Met Leu Ile Lys Asn
625 630 635 640
Thr Pro Val Pro Gly Asn Ile Thr Ser Phe Ser Asp Val Pro Val Ser
645 650 655
Ser Phe Ile Thr Gln Tyr Ser Thr Gly Gln Val Thr Val Glu Met Glu
660 665 670
Trp Glu Leu Lys Lys Glu Asn Ser Lys Arg Trp Asn Pro Glu Ile Gln
675 680 685
Tyr Thr Asn Asn Tyr Asn Asp Pro Gln Phe Val Asp Phe Ala Pro Asp
690 695 700
Ser Thr Gly Glu Tyr Arg Thr Thr Arg Pro Ile Gly Thr Arg Tyr Leu
705 710 715 720
Thr Arg Pro Leu
<210> 56
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 56
Leu Ala Asn Pro Leu Val Asp Gln Tyr Leu Tyr Arg
1 5 10
<210> 57
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 57
Ile Pro Glu Thr Gly Ala His Phe His Pro Ser Pro Ala Met Gly Gly
1 5 10 15
Phe Gly Leu Lys
20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НОВЫЕ НАЦЕЛИВАЮЩИЕ НА ПЕЧЕНЬ АДЕНОАССОЦИИРОВАННЫЕ ВИРУСНЫЕ ВЕКТОРЫ | 2019 |
|
RU2793735C2 |
| СПОСОБЫ ВЫЯВЛЕНИЯ AAV | 2017 |
|
RU2771622C2 |
| Выделенный модифицированный белок VP1 капсида аденоассоциированного вируса 5 серотипа (AAV5), капсид и вектор на его основе | 2019 |
|
RU2751592C2 |
| Выделенный модифицированный белок VPI капсида аденоассоциированного вируса 5 серотипа (AAV5), капсид и вектор на его основе | 2021 |
|
RU2820088C1 |
| AAV ВЕКТОРЫ С УСОВЕРШЕНСТВОВАННЫМИ Rep-КОДИРУЮЩИМИ ПОСЛЕДОВАТЕЛЬНОСТЯМИ, ИСПОЛЬЗУЕМЫМИ В СИСТЕМАХ ПРОДУКЦИИ НА ОСНОВЕ КЛЕТОК НАСЕКОМЫХ | 2007 |
|
RU2457252C2 |
| Выделенный модифицированный белок VPI капсида аденоассоциированного вируса 9 серотипа (AAV9), капсид и вектор на его основе | 2021 |
|
RU2825667C2 |
| УСИЛИВАЮЩИЕ АГЕНТЫ ДЛЯ ПОВЫШЕНИЯ ТРАНСФЕКЦИИ КЛЕТОК И/ИЛИ ПРОДУКЦИИ ВЕКТОРА rAAV | 2018 |
|
RU2802520C2 |
| РЕКОМБИНАНТНЫЕ ВИРУСНЫЕ ВЕКТОРЫ С МОДИФИЦИРОВАННЫМ ТРОПИЗМОМ И ПУТИ ИХ ПРИМЕНЕНИЯ ДЛЯ НАЦЕЛЕННОГО ВВЕДЕНИЯ ГЕНЕТИЧЕСКОГО МАТЕРИАЛА В КЛЕТКИ ЧЕЛОВЕКА | 2018 |
|
RU2809246C2 |
| Кодон-оптимизированная нуклеиновая кислота, которая кодирует белок SMN1, и ее применение | 2020 |
|
RU2742837C1 |
| ГЕНОТЕРАПЕВТИЧЕСКИЕ КОНСТРУКЦИИ ДЛЯ ЛЕЧЕНИЯ БОЛЕЗНИ ВИЛЬСОНА | 2020 |
|
RU2807158C2 |
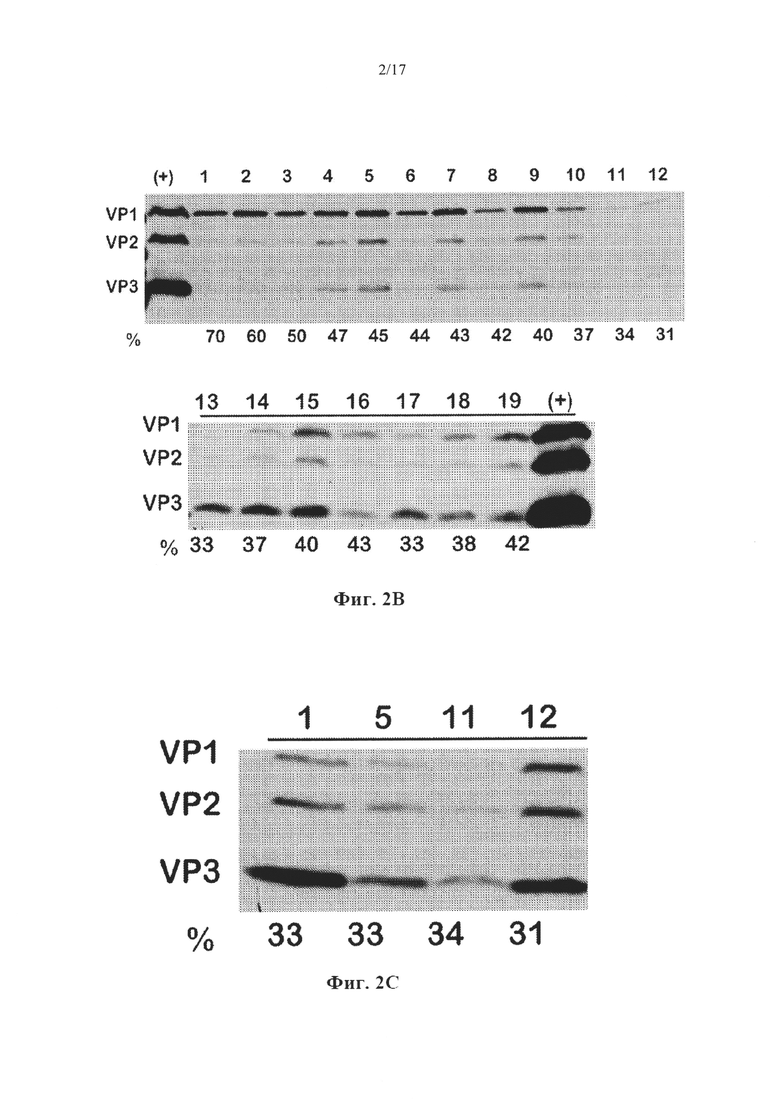
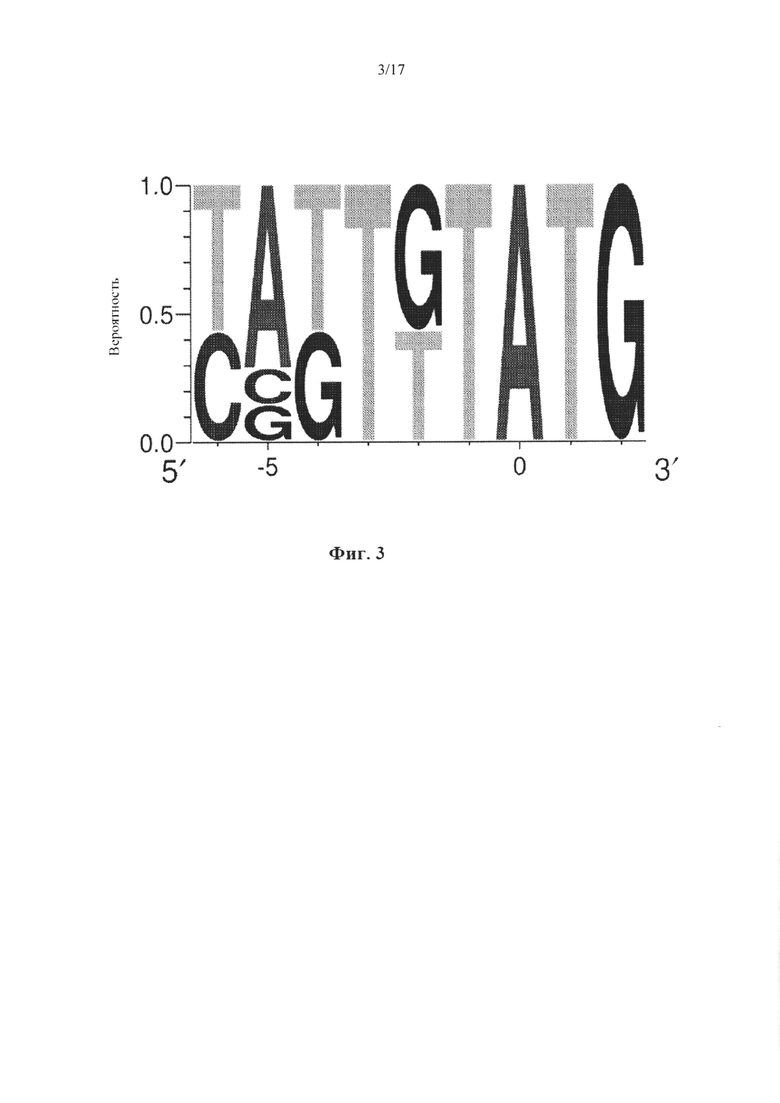
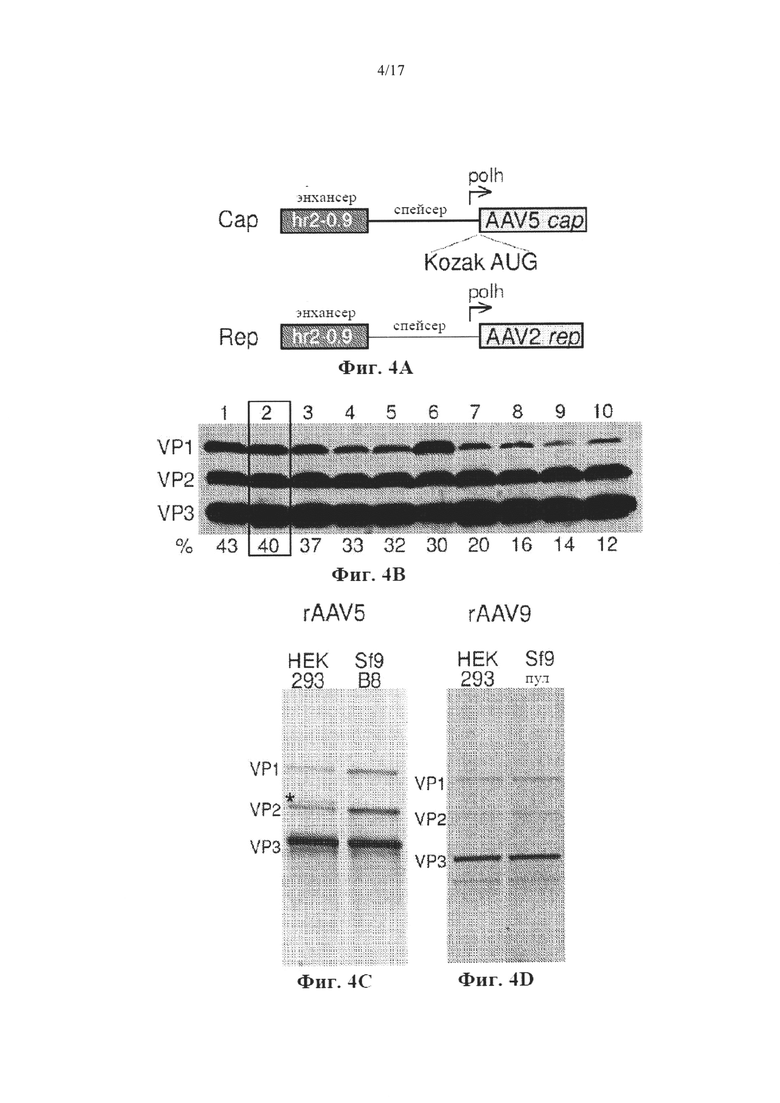
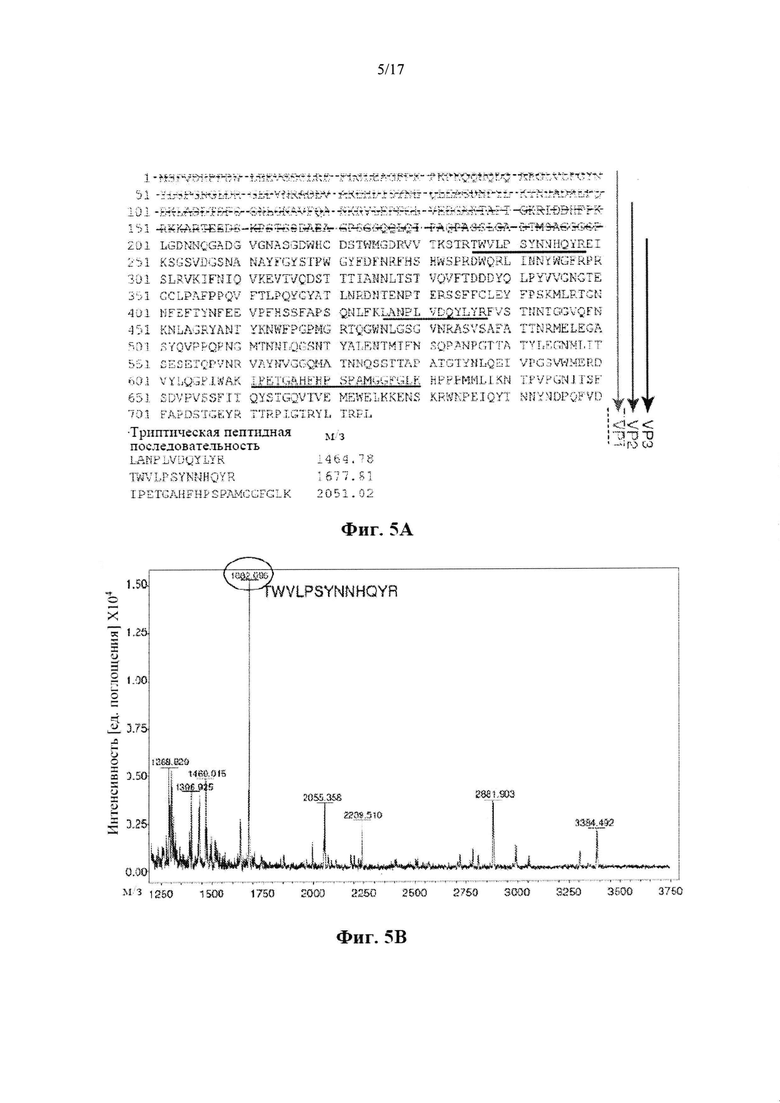
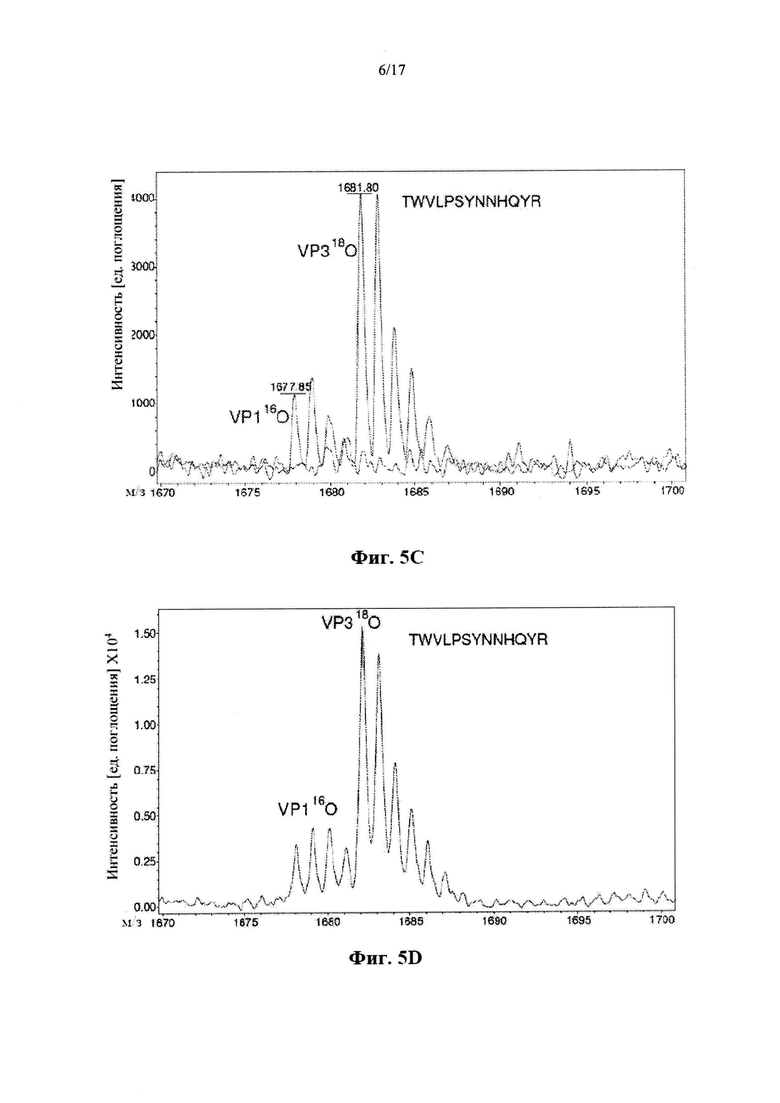
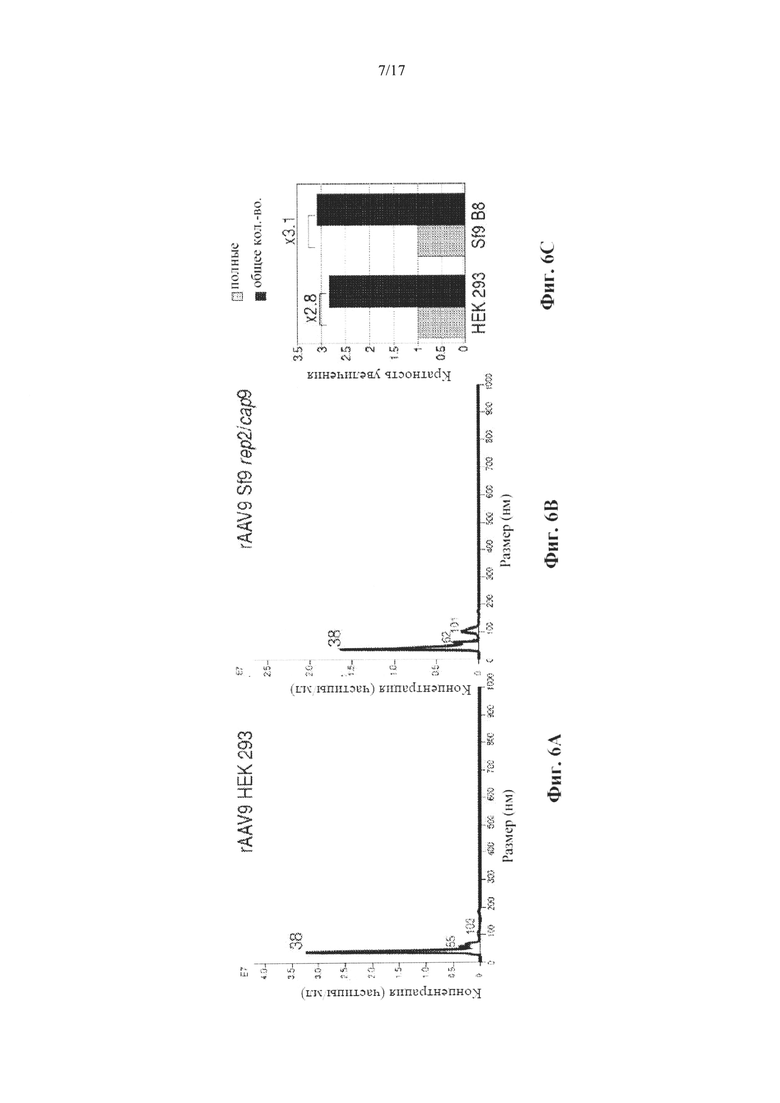
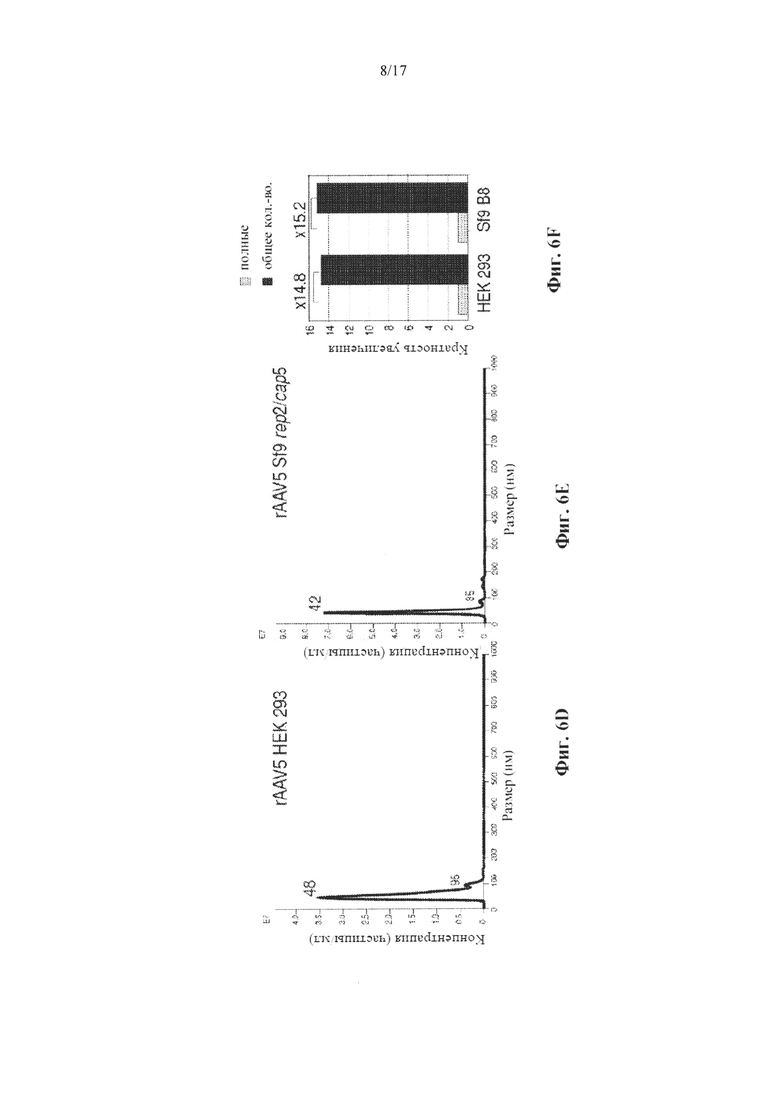
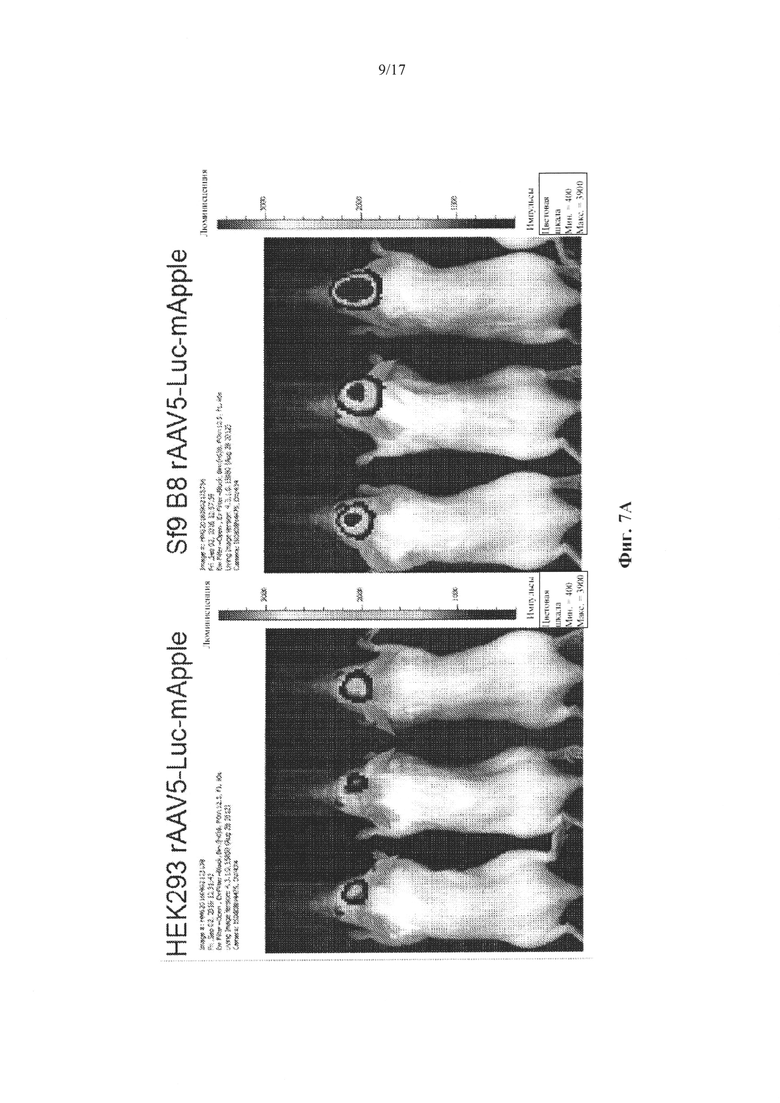
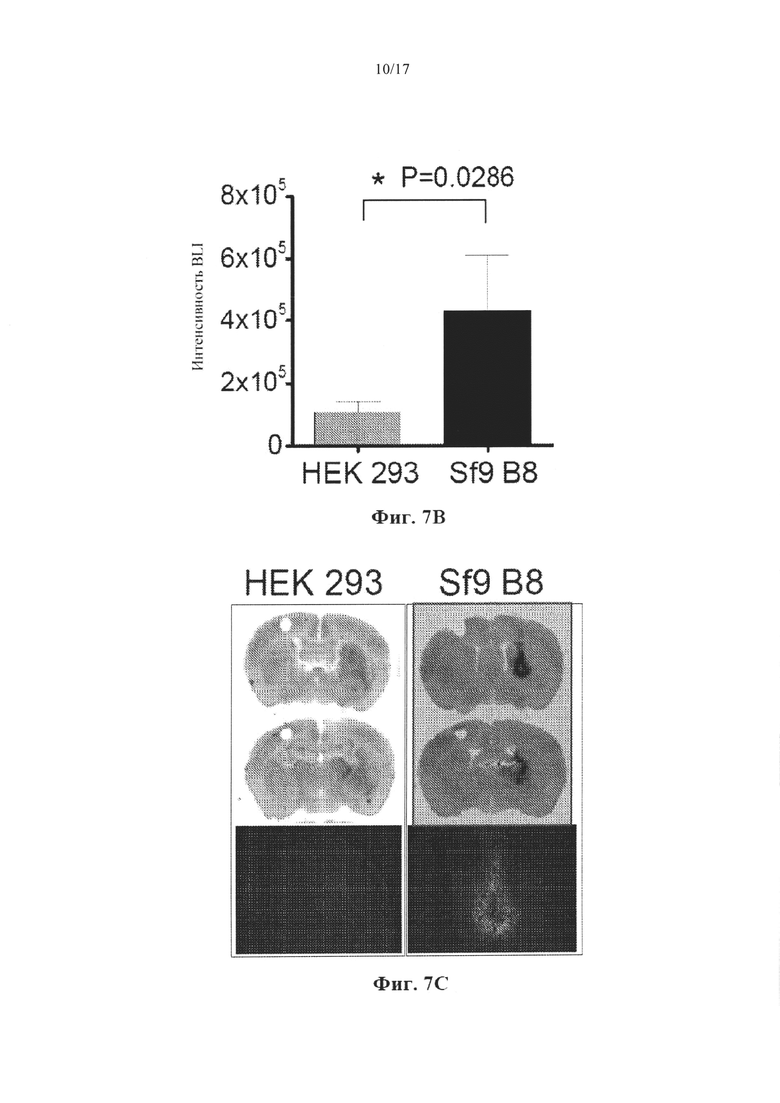
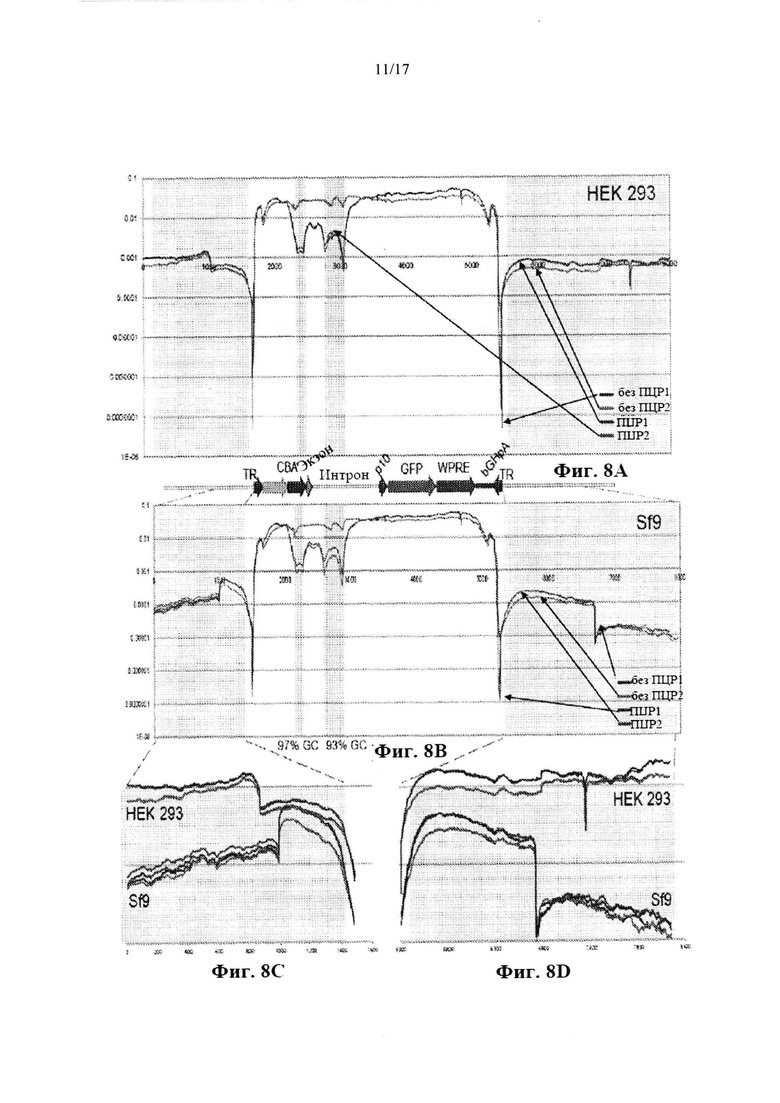
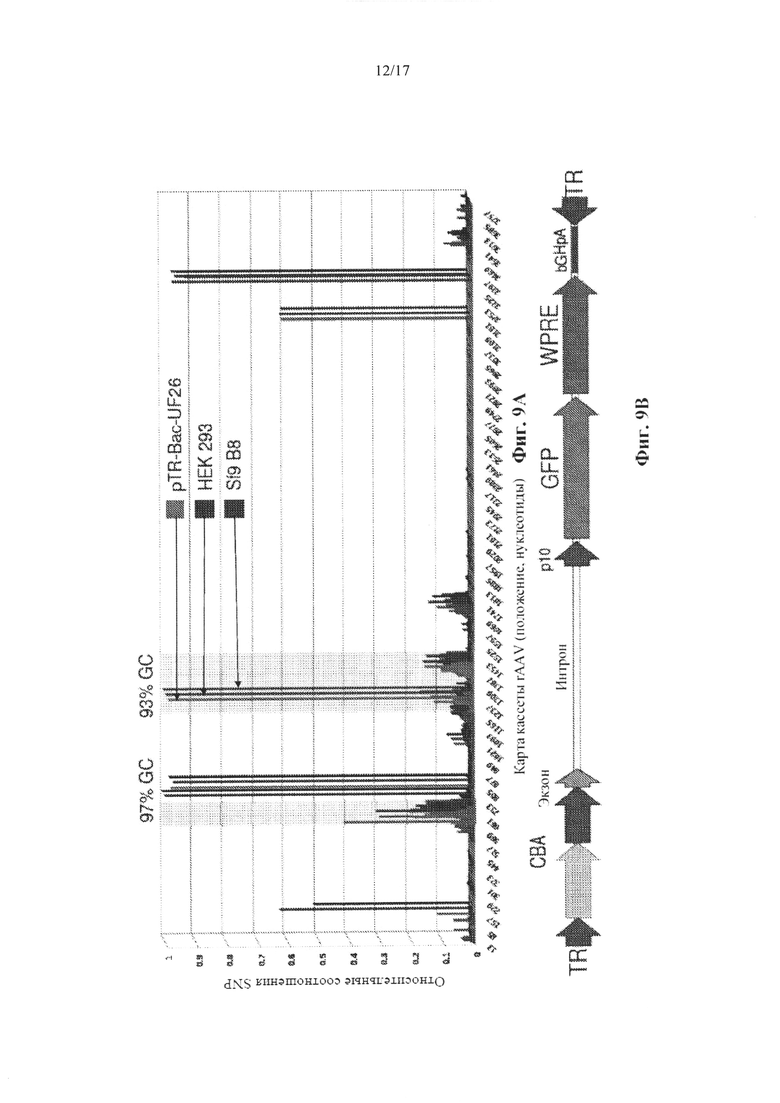
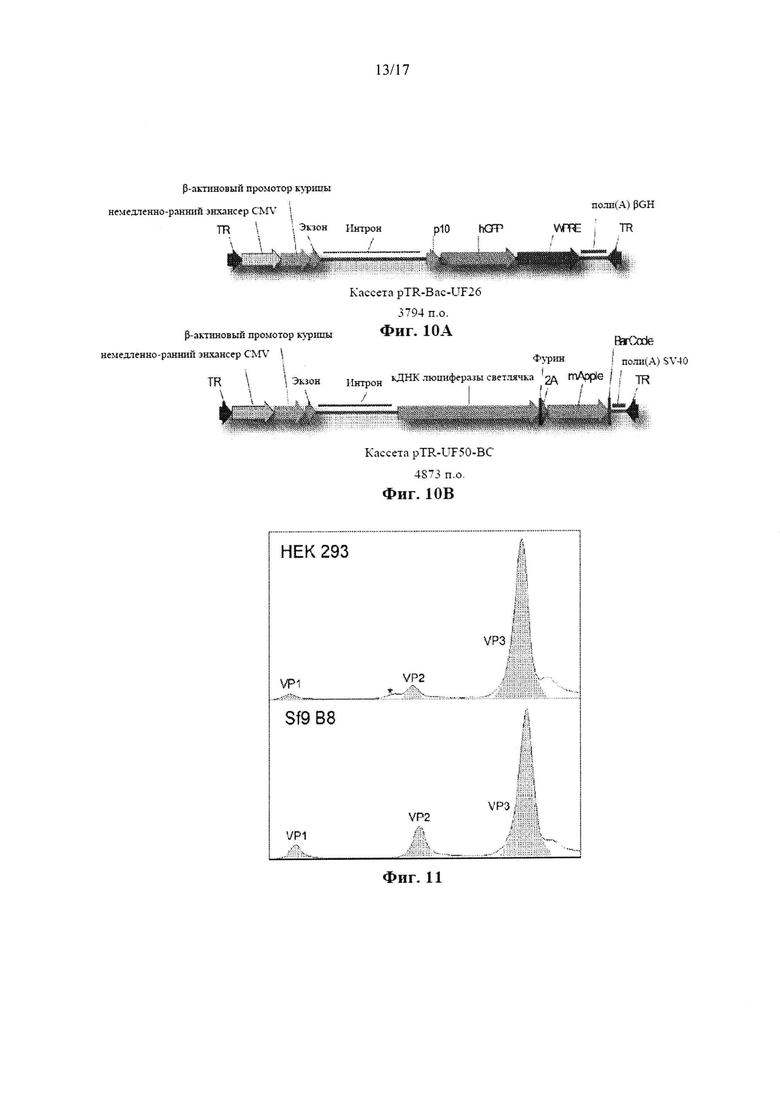
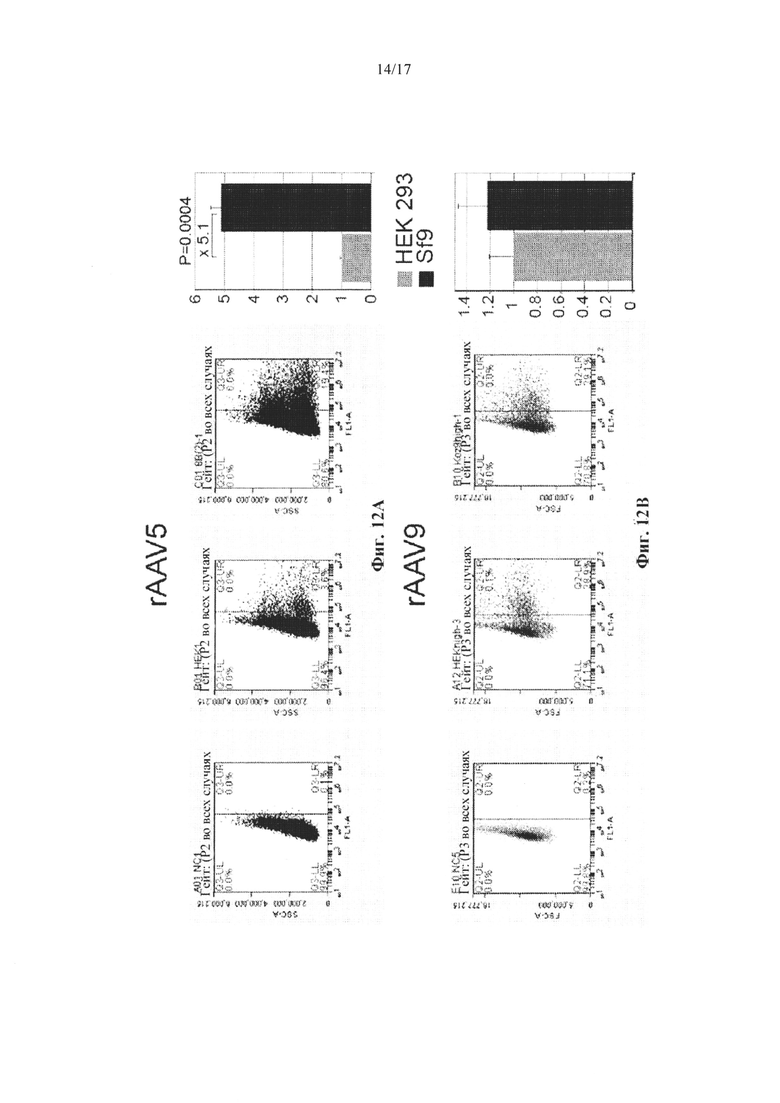
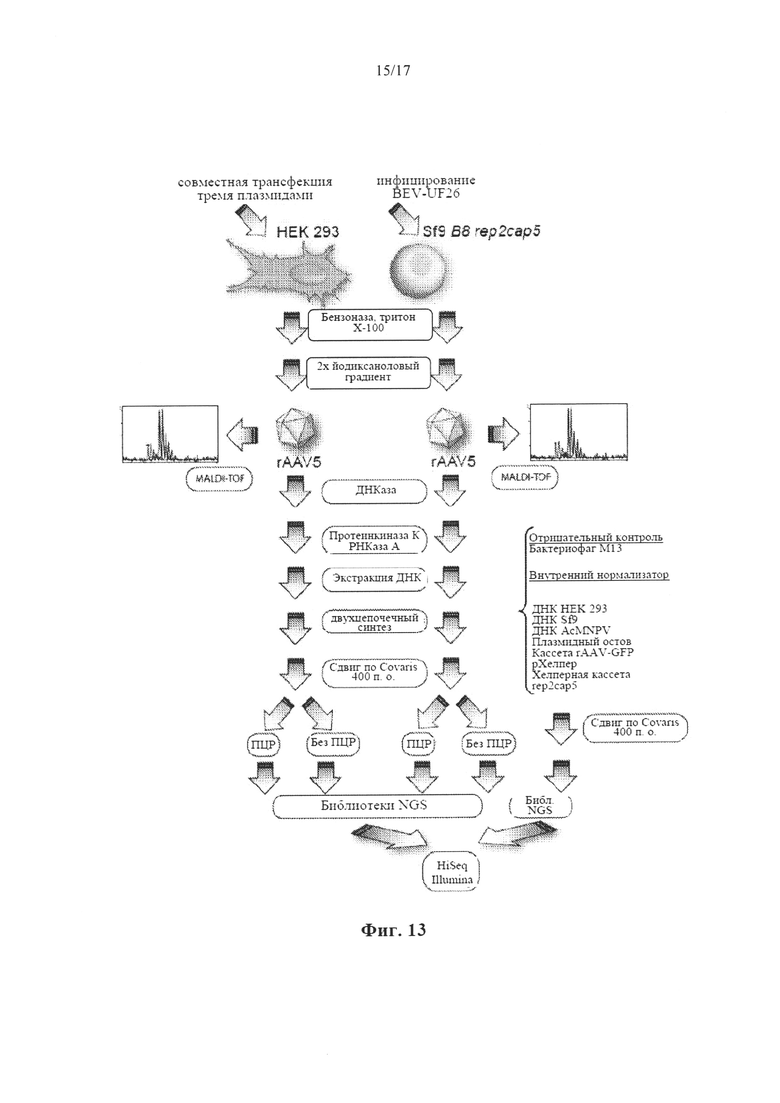
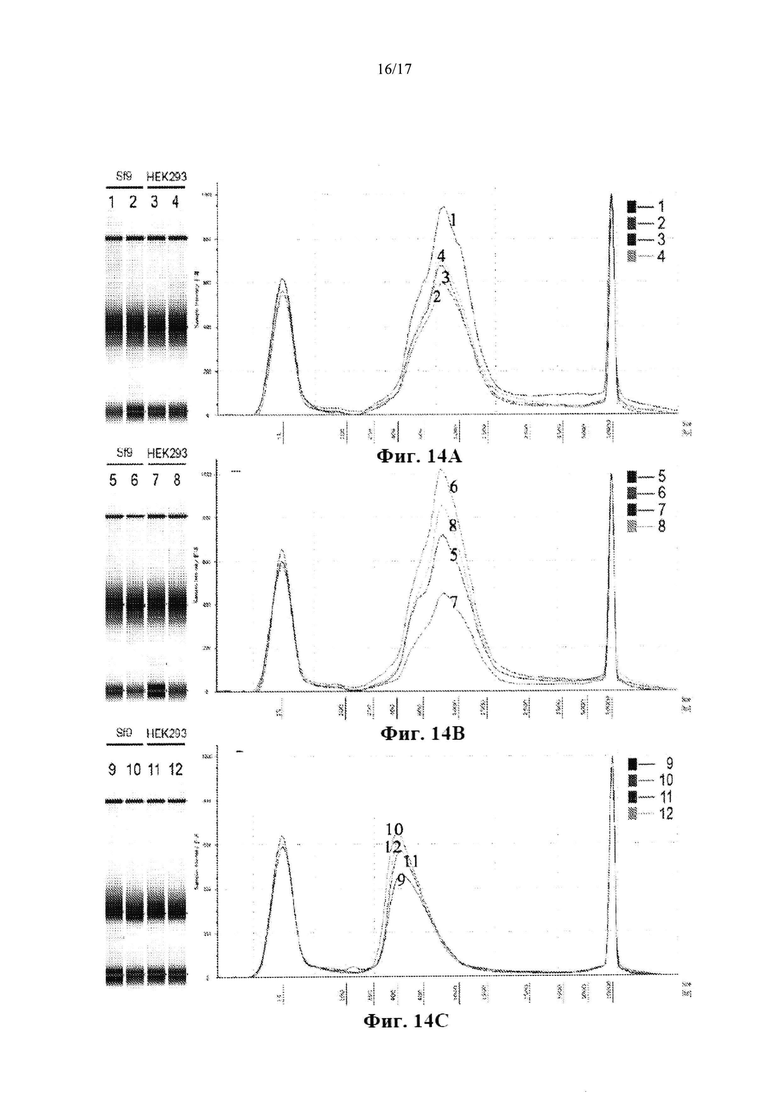
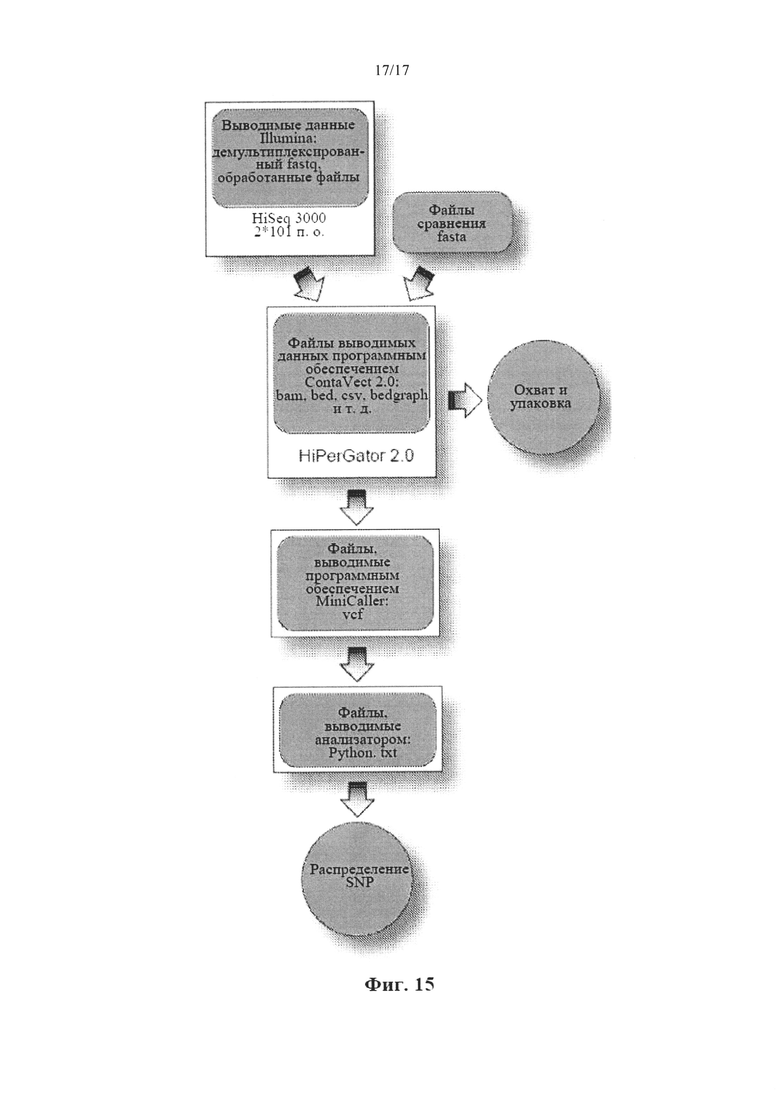
Изобретение относится к биотехнологии. Описаны нуклеиновые кислоты, кодирующие капсидные белки VP1, VP2 и VP3 аденоассоциированного вируса (AAV), содержащие модифицированную последовательность Козак, выбранную из SEQ ID NO: 2-11, SEQ ID NO: 13-32 или SEQ ID NO: 34-45. Также представлены клетки насекомого для получения капсидных белков VP1, VP2 и VP3 аденоассоциированного вируса (AAV), содержащие указанные нуклеиновые кислоты. Кроме того, описаны способы продукции рекомбинантного AAV (rAAV) в клетке насекомого. Изобретение предоставляет векторы AAV с последовательностями инициации трансляции VP1, которые приводят к частицам rAAV с повышенной биологической активностью (например, инфекционность), а также системы продукции на основе AAV для получения этих инфицирующих rAAV частиц. 12 н. и 29 з.п. ф-лы, 15 ил., 11 табл., 2 пр.
1. Нуклеиновая кислота, кодирующая капсидные белки VP1, VP2 и VP3 аденоассоциированного вируса (AAV), причем нуклеиновая кислота содержит модифицированную последовательность Козак, выбранную из SEQ ID NO: 2-11.
2. Нуклеиновая кислота по п. 1, в которой модифицированная последовательность Козак представляет собой SEQ ID NO: 8 или SEQ ID NO: 9.
3. Нуклеиновая кислота по п. 1, в которой капсидные белки VP1, VP2 и VP3 происходят из серотипа AAV5.
4. Нуклеиновая кислота по п. 1 или 3, дополнительно содержащая промоторную последовательность.
5. Нуклеиновая кислота по п. 4, в которой промоторная последовательность представляет собой промоторную последовательность полиэдрина (polh).
6. Нуклеиновая кислота по любому из пп. 1-5, в которой модифицированная последовательность Козак содержит кодон инициации трансляции капсидного белка VP1.
7. Нуклеиновая кислота по п. 6, в которой кодон инициации трансляции капсидного белка VP1 представляет собой AUG.
8. Нуклеиновая кислота по любому из пп. 1-7, в которой нуклеиновая кислота упакована в вирусную частицу, необязательно бакуловирусную частицу.
9. Клетка насекомого для получения капсидных белков VP1, VP2 и VP3 аденоассоциированного вируса (AAV), содержащая нуклеиновую кислоту по любому из пп. 1-8.
10. Клетка насекомого по п. 9, в которой нуклеиновая кислота интегрирована в геном клетки насекомого.
11. Способ продукции рекомбинантного AAV (rAAV) в клетке насекомого, в котором rAAV происходит из серотипа AAV5, причем способ включает:
a) трансфекцию клетки насекомого:
i) бакуловирусом, содержащим нуклеиновую кислоту по п. 1;
ii) бакуловирусом, содержащим нуклеиновую кислоту, кодирующую белок Rep AAV; и
iii) бакуловирусом, содержащим нуклеиновую кислоту, содержащую нуклеотидные последовательности двух инвертированных концевых повторов (ITR) AAV,
фланкирующие ген, представляющий интерес, функционально связанный с промоторной последовательностью;
b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и
c) извлечение rAAV из клетки насекомого.
12. Способ продукции рекомбинантного AAV (rAAV) в клетке насекомого, в котором rAAV происходит из серотипа AAV5, причем способ включает:
а) трансфекцию клетки насекомого по п. 9 или 10 нуклеиновой кислотой,
необязательно в бакуловирусе, содержащей нуклеотидные последовательности двух инвертированных концевых повторов (ITR) AAV, фланкирующие ген, представляющий интерес, функционально связанный с промоторной последовательностью;
b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и
c) извлечение rAAV из клетки насекомого.
13. Способ по п. 11 или 12, в котором клетка насекомого представляет собой клетку Sf9.
14. Нуклеиновая кислота, кодирующая капсидные белки VP1, VP2 и VP3 аденоассоциированного вируса (AAV), причем нуклеиновая кислота содержит модифицированную последовательность Козак, выбранную из SEQ ID NO: 13-32.
15. Нуклеиновая кислота по п. 14, в которой модифицированная последовательность Козак представляет собой SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29, SEQ ID NO: 30, SEQ ID NO: 31 или SEQ ID NO: 32.
16. Нуклеиновая кислота по п. 14, в которой капсидные белки VP1, VP2 и VP3 происходят из серотипа AAV8.
17. Нуклеиновая кислот по п. 14 или 16, дополнительно содержащая промоторную последовательность.
18. Нуклеиновая кислота по п. 17, в которой промоторная последовательность представляет собой промоторную последовательность полиэдрина (polh).
19. Нуклеиновая последовательность по любому из пп. 14-18, в которой модифицированная последовательность Козак содержит кодон индициации трансляции капсидного белка VP1.
20. Нуклеиновая кислота по п. 19, в которой кодон инициации трансляции капсидного белка VP1 представляет собой AUG.
21. Нуклеиновая кислота по любому из пп. 14-20, в которой нуклеиновая кислота упакована в вирусную частицу, необязательно бакуловирусную частицу.
22. Клетка насекомого для получения капсидных белков VP1, VP2 и VP3 аденоассоциированного вируса (AAV), содержащая нуклеиновую кислоту по любому из пп. 14-21.
23. Клетка насекомого по п. 22, в которой нуклеиновая кислота интегрирована в геном клетки насекомого.
24. Способ продукции рекомбинантного AAV (rAAV) в клетке насекомого, в котором rAAV происходит из серотипа AAV8, причем способ включает:
a) трансфекцию клетки насекомого:
i) бакуловирусом, содержащим нуклеиновую кислоту по п. 14;
ii) бакуловирусом, содержащим нуклеиновую кислоту, кодирующую белок
Rep AAV; и
iii) бакуловирусом, содержащим нуклеиновую кислоту, содержащую нуклеотидные последовательности двух инвертированных концевых повторов (ITR) AAV,
фланкирующие ген, представляющий интерес, функционально связанный с промоторной
последовательностью;
b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и
c) извлечение rAAV из клетки насекомого.
25. Способ продукции рекомбинантного AAV (rAAV) в клетке насекомого, в котором rAAV происходит из серотипа AAV8, при этом способ включает:
а) трансфекцию клетки насекомого по п. 22 или 23 нуклеиновой кислотой, необязательно в бакуловирусе, содержащей нуклеотидные последовательности двух инвертированных концевых повторов (ITR) AAV, фланкирующие ген, представляющий интерес, функционально связанный с промоторной последовательностью;
b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и
c) извлечение rAAV из клетки насекомого.
26. Способ по п. 24 или 25, в котором клетка насекомого представляет собой клетку Sf9.
27. Нуклеиновая кислота, кодирующая капсидные белки VP1, VP2 и VP3 аденоассоциированного вируса (AAV), причем нуклеиновая кислота содержит модифицированную последовательность Козак, выбранную из SEQ ID NO: 34-45.
28. Нуклеиновая кислота по п. 27, в которой модифицированная последовательность
Козак представляет собой SEQ ID NO: 35, SEQ ID NO: 36, SEQ ID NO: 38 или SEQ ID NO: 42.
29. Нуклеиновая кислота по п. 27, в которой капсидные белки VP1, VP2 и VP3 происходят из серотипа AAV9.
30. Нуклеиновая кислот по п. 27 или 29, дополнительно содержащая промоторную последовательность.
31. Нуклеиновая кислота по п. 30, в которой промоторная последовательность представляет собой промоторную последовательность полиэдрина (polh).
32. Нуклеиновая последовательность по любому из пп. 27-31, в которой модифицированная последовательность Козак содержит кодон инициации трансляции капсидного белка VP1.
33. Нуклеиновая кислота по п. 32, в которой кодон инициации трансляции капсидного белка VP1 представляет собой AUG.
34. Нуклеиновая кислота по любому из пп. 27-33, в которой нуклеиновая кислота упакована в вирусную частицу, необязательно бакуловирусную частицу.
35. Клетка насекомого для получения капсидных белков VP1, VP2 и VP3 аденоассоциированного вируса (AAV), содержащая нуклеиновую кислоту по любому из пп. 27-34.
36. Клетка насекомого по п. 35, в которой нуклеиновая кислота интегрирована в геном клетки насекомого.
37. Способ продукции рекомбинантного AAV (rAAV) в клетке насекомого, в котором rAAV происходит из серотипа AAV9, причем способ включает:
a) трансфекцию клетки насекомого:
i) бакуловирусом, содержащим нуклеиновую кислоту по п. 27;
ii) бакуловирусом, содержащим нуклеиновую кислоту, кодирующую белок Rep AAV; и
iii) бакуловирусом, содержащим нуклеиновую кислоту, содержащую нуклеотидные последовательности двух инвертированных концевых повторов (ITR) AAV, фланкирующие ген, представляющий интерес, функционально связанный с промоторной последовательностью;
b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и
c) извлечение rAAV из клетки насекомого.
38. Способ продукции рекомбинантного AAV (rAAV) в клетке насекомого, в котором rAAV происходит из серотипа AAV9, причем способ включает:
а) трансфекцию клетки насекомого по п. 35 или 36 нуклеиновой кислотой,
необязательно в бакуловирусе, содержащей нуклеотидные последовательности двух инвертированных концевых повторов (ITR) AAV, фланкирующие ген, представляющий интерес, функционально связанный с промоторной последовательностью;
b) культивирование клетки насекомого в условиях, подходящих для продукции rAAV; и
c) извлечение rAAV из клетки насекомого.
39. Способ по п. 37 или 38, в котором клетка насекомого представляет собой клетку Sf9.
40. Нуклеиновая кислота по п. 1, в которой модифицированная последовательность Козак представляет собой последовательность, представленную в SEQ ID NO: 9.
41. Нуклеиновая кислота по п. 27, в которой модифицированная последовательность
Козак представляет собой последовательность, представленную в SEQ ID NO: 42.
| Прибор для измерения количества бензина в баках | 1930 |
|
SU20969A1 |
| AAV ВЕКТОРЫ С УСОВЕРШЕНСТВОВАННЫМИ Rep-КОДИРУЮЩИМИ ПОСЛЕДОВАТЕЛЬНОСТЯМИ, ИСПОЛЬЗУЕМЫМИ В СИСТЕМАХ ПРОДУКЦИИ НА ОСНОВЕ КЛЕТОК НАСЕКОМЫХ | 2007 |
|
RU2457252C2 |
| US 20090191597 A1, 30.07.2009 | |||
| CN 101287837 B, 18.03.2015 | |||
| CN 103849629 A, 11.06.2014. | |||
Авторы
Даты
2021-12-24—Публикация
2017-04-16—Подача