Изобретение относится к медицине, а именно к фтизиатрии, и может быть использовано в лабораторной диагностике штаммов возбудителя туберкулеза, относящихся к гипервирулентному генотипу Mycobacterium tuberculosis Beijing 14717-15-кластер.
В структуре вида М. tuberculosis выделяют иерархичную филогенетическую структуру с несколькими основными линиями, которые в свою очередь подразделяются на сублинии. Внутри сублиний также выделяют кластеры, объединяющие схожие по патобиологическим признакам и генетически близкородственные штаммы (Gagneux, 2018). Несмотря на тенденцию к снижению уровня заболеваемости туберкулезом, в России продолжают циркулировать лекарственно-устойчивые штаммы. По данным Всемирной организации здравоохранения, Россия входит в тройку стран, на которые приходится 42% случаев заболевания туберкулезом с множественной лекарственной устойчивостью (WHO, 2022).
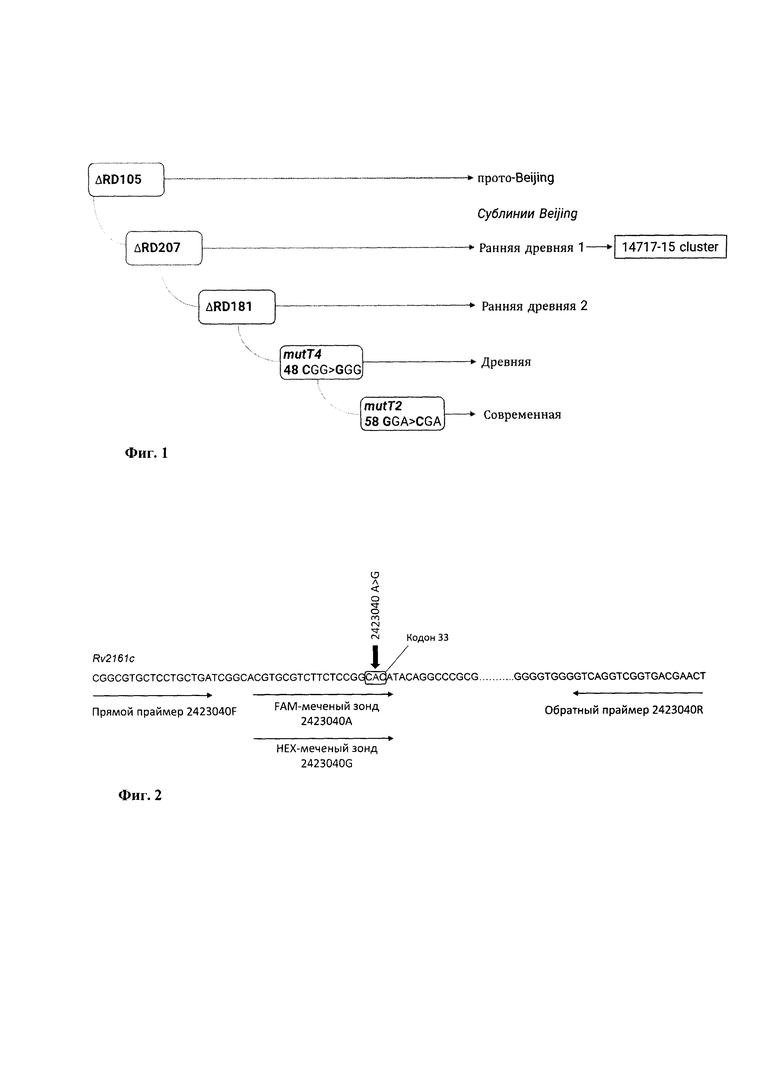
Генетическое семейство Beijing является основным компонентом Восточно-Азиатской линии, преобладающей на территории России. Доля данного генотипа различается в зависимости от региона, но остается в диапазоне 45-60% (Pasechnik 2018, Zhdanova 2022 Umpeleva 2020, Vyazovaya, 2023). В семействе Beijing выделяют древнюю и современную сублинию (Mokrousov et al., 2002; 2005) (Фиг. 1). Штаммы древней сублинии в основном встречаются в азиатских странах (Япония, Корея, реже - Китай и Вьетнам), в то время как современная сублиния распространена по всему миру (Iwamoto et al., 2009; Kang et al., 2010; Luo et al., 2015, Maeda et al., 2014). Генотип Beijing в России особенно опасен тем, что его штаммы ассоциированы с резистентностью ко многим лекарственным препаратам, в том числе и новым антибиотикам, и наиболее известными являются субтипы, входящие в современную сублинию, B0/W148 и САО (Central Asia Outbreak) (Shitikov et al., 2019; Merker et al., 2015, 2020; Mokrousov, 2013). Кроме того, в рамках древней сублинии недавно были обнаружены два новых кластера в азиатской части России (Mokrousov et al., 2019,2020). Среди изолятов из России и Восточной Азии были выявлены штаммы, формирующие отдельные филогенетические кластеры на дендрограмме штаммов генотипа Beijing М. tuberculosis. По номенклатуре сервиса MIRU-VNTRplus.org они относятся к генотипам Beijing 1071-32 и Beijing 14717-15. В ходе проведенного биоинформатического анализа было обнаружено, что полимеразной цепной реакции в формате реального времени все изоляты, относящиеся к кластеру Beijing 14717-15 (профиль VNTR 14717-15 и близкородственные ему профили), обладали множественной лекарственной устойчивостью (МЛУ) и резистентностью к стрептомицину. Часть образцов относилась к профилю резистентности, предшествующей возникновению широкой лекарственной устойчивости (пред-ШЛУ). В некоторых штаммах этого кластера были обнаружены мутации в генах embB, gyrA, pncA, rrs ассоциированных с дополнительной устойчивостью к этамбутолу, фторхинолонам, пиразинамиду и аминогликозидам (Mokrousov et al., 2020). Данный кластер составлял около 16% в структуре популяции М. tuberculosis в Бурятии, также был обнаружен в нескольких других регионах России (Жданова, 2019). Мыши, зараженные штаммом кластера 14717-15, значительно теряли в весе, обладали высокой бактериальной нагрузкой и имели наибольший показатель смертности, в сравнении с другими штаммами в эксперименте (Беспятых и соавт.2019; Vinogradova et al., 2021). Также процент летального исхода заболевания туберкулезом был выше среди больных, зараженных штаммами Beijing 14717-15-кластера в Омской области (Mokrousov et al. 2022).
С учетом вышеизложенного, ранняя детекция и эпидемиологический надзор за распространением высокорезистентного и летального генотипа Beijing 14717-15-кластер является важным и необходимым условием в глобальной борьбе с туберкулезом. Разработка простого в исполнении, быстрого метода детекции клинически значимого генотипа М. tuberculosis Beijing 14717-15 является первостепенной задачей в рамках контроля туберкулеза в регионах России.
Известно определение кластера Beijing 14717-15 с помощью MIRU-VNTR типирования, основанного на определении числа вариабельных тандемных повторов в 24 локусах М. tuberculosis и сравнение с сервисом www.MIRU-VNTRplus.org (Mokrousov et al., 2019). Метод включает в себя постановку 24 ПЦР амплификаций каждого локуса по разным протоколам с последующим анализом с помощью электрофореза в агарозном геле. Также известен метод обнаружения данного штамма с помощью ПЦР-ПДРФ (ПЦР с последующей HhaI-рестрикцией продукта ПЦР и разделением рестриктов в агарозном геле (Мокроусов и соавт., 2023). Выявление штаммов данного кластера также возможно на основе полногеномного секвенирования с последующей биоинформатической обработкой данных и филогенетическим анализом (Mokrousov et al, 2020). Эти способы являются или длительными, трудоемкими (включают несколько этапов эксперимента) или дорогостоящими и, следовательно, неудобными для ежедневного скрининга.
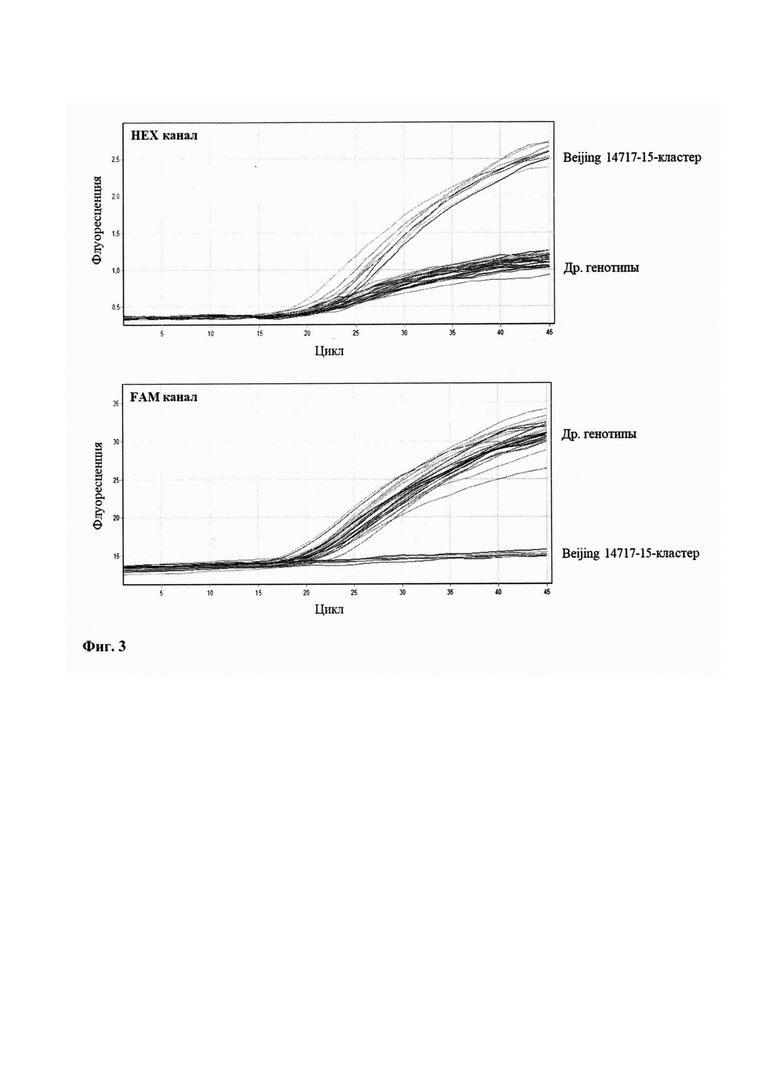
В ходе проведенного нами филогенетического анализа 184 геномов М. tuberculosis ранней древней сублинии генотипа Beijing с использованием биоинформатических программ и международных баз данных (National Center for Biotechnology Information и European Nucleotide Archive) и сравнения с имеющимися данными VNTR-типирования тех же штаммов было установлено, что штаммы кластера Beijing 14717-14 формируют отдельную ветвь на дендрограмме ("Beijing 14717-15-кластер"). Для этой ветви мы установили набор уникальных мутаций (в сравнении с референтным штаммом H37Rv) из которых выбрали нейтральный однонуклеотидный синонимичный полиморфизм (SNP-мутация) в гене Rv2161c (позиция в геноме штамма H37Rv 2423040 A>G, кодон 33 GTG>GCG), являющийся уникальным для штаммов Beijing 14717-15-кластера. Эта мутация встречается только в геномах изолятов Beijing 14717-15, и для ее определения был разработан способ детекции, включающий в себя подбор специфичных праймеров, флуоресцентно-меченых зондов, содержащих модификацию LNA (Locked Nucleic Acid), и протокола проведения ПЦР в реальном времени (Фиг. 2).
Задачей предлагаемого изобретения является разработка способа, который позволит эффективно и быстро определять образцы возбудителя туберкулеза высоколетального и вирулентного российского генотипа Mycobacterium tuberculosis Beijing 14717-15-кластер.
Задача осуществляется определением уникальной мутации в гене Rv2161c в геномной позиции 2423040A>G с помощью ПЦР в реальном времени с дискриминацией дикого и мутантного аллелей однонуклеотидными праймерами 2423040F 5'-CGGCGTGCTCCTGCTGAT и 2423040R 5'-AGTTCGTCACCGACCTGAC, и флуоресцентно-мечеными зондами на дикий аллель 2423040А 5'-[FAM] CGTGCGTCTTCTCCGGC[A-LNA]CAT[BHQ1] и на мутантный аллель 2423040G 5'-[НЕХ] CGTGCGTCTTCTCCGGC[G-LNA]CAT[BHQ1], и последующей оценкой результата ПЦР в 20-35 циклах посредством регистрации экспоненциального роста сигнала флуоресценции по НЕХ-каналу с длиной волны 555 нм и отсутствием сигнала флуоресценции по FAM-каналу с длиной волны 510 нм для принятия решения о принадлежности образца к генотипу М. tuberculosis Beijing 14717-15-кластер.
Техническим результатом от использования заявляемого изобретения является: простота использования метода; легкая и однозначная интерпретация результата; возможность определения возможной контаминации или микс-инфекции.
Изобретение поясняется чертежами. Фиг. 1 - эволюционное древо генотипа Beijing Mycobacterium tuberculosis и положение Beijing 14717-15-кластера на нем. Фиг. 2 -способ детекции, где тонкие стрелки -направление и позиции праймеров и флуоресцентно-меченых зондов в геноме референтного штамма H37Rv, а широкая стрелка указывает на позицию однонуклеотидной замены 2423040A>G (кодон 33) в гене Rv2161c. Фиг. 3 - накопление сигнала флуоресценции: (а) по специфическому (HEX) каналу детекции, на М. tuberculosis Beijing 14717-15 кластер и (б) контрольному (FAM) каналу детекции, на штамм другого генотипа М. tuberculosis.
Способ осуществляется следующим образом.
Выделение ДНК из культуры М. tuberculosis, выращенной на среде Левенштейна-Йенсена, проводят по van Embden et al. (1993): суспендируют 1 стандартную бактериологическую петлю культуры в 400 мкл буфера ТЕ x1 и инкубируют 20 мин при 85°С. Дальнейшую обработку проводят с использованием лизоцима, протеиназы К, додецилсульфата натрия (SDS) и цетилтриметиламмонийбромида (СТАВ). Полученный клеточный лизат обрабатывают смесью фенол-хлороформ-изоамиловый спирт (25:24:1), центрифугируют, осаждают изопропанолом, промывают 70% этанолом, осадок высушивают и растворяют в 40 мкл ТЕ x1. Также возможно выделение ДНК коммерческими наборами, например, "ДНК-Сорб" и "Рибо-Сорб" Амплисенс (Интерлабсервис) или "К-Сорб" (Синтол).
Определение принадлежности изолята к кластеру Beijing 14717-15 производится с помощью сконструированных нами праймеров и флуоресцентных зондов для постановки ПЦР в реальном времени (Фиг. 2) и детекции флуоресценции по двум каналам в одной пробирке одновременно: 2423040F 5'-CGGCGTGCTCCTGCTGAT, 2423040R 5'-AGTTCGTCACCGACCTGAC, 2423040А 5'-[FAM] CGTGCGTCTTCTCCGGC[А-LNA]CAT[BHQ1], 2423040G 5'-[НЕХ] CGTGCGTCTTCTCCGGC[G-LNA]CAT[BHQ1]. Для детекции кластера Beijing 14717-15 служил зонд 2423040G с экспоненциальным ростом флуоресцентного сигнала по НЕХ-каналу (555 нм длина волны) (Фиг. 3а). Для детекции любого другого генотипа М. tuberculosis служил зонд 2423040А с экспоненциальным ростом флуоресцентного сигнала по FAM-каналу (510 нм длина волны) при отсутствии сигнала по НЕХ-каналу (555 нм длина волны) (Фиг. 3б).
Рабочая смесь для ПЦР включает 1,5 mM MgCl2, 1 U Taq ДНК полимеразы для ПЦР в реальном времени в режиме «горячего старта», по 200 μМ каждого из дНТФ, праймеры и зонды (по 5 пмоль каждый) и выделенной ДНК (0,1-0,5 мкл). Конечный объем на одну пробирку составляет 30 мкл. ПЦР проводили в термоциклере RotorGene6000 (Corbette Research) по режиму: 95 С, 10 минут (для активации Taq полимеразы), с последующими 40 циклами - 95°С 20 секунд, 60°С 20 секунд. Считывание сигнала флуоресценции - при 60°С по каналам 510 и 555 нм.
Оценка результатов.
Результатом ПЦР в реальном времени являются кривые накопления флуоресцентного сигнала, оценка которых производится между 20-35 циклами по обоим каналам. Кривые анализируют с помощью программного обеспечения прибора, используемого для ПЦР в реальном времени. По НЕХ-каналу (длина волны 555 нм) регистрируется накопление продукта амплификации, специфического для М. tuberculosis Beijing 14717-15-кластер (Фиг. 3а), а по FAM-каналу (длина волны 510 нм) определяется накопление продукта амплификации, специфического для любого другого генотипа М. tuberculosis (Фиг. 3б).
Наличие и рост сигнала флуоресценции т.е. амплификации продукта ПЦР по первому или второму каналу является необходимым условием для контроля качества реакции. Отсутствие роста сигнала флуоресценции по обоим каналам свидетельствует об ингибировании реакции ПЦР, связанном с недостаточным качеством/количеством ДНК и/или нарушением правил хранения реагентов или неисправности прибора. В таком случае, вывод о наличии или отсутствии ДНК штамма Beijing 14717-15-кластера в образце сделать невозможно. Рост сигнала флуоресценции по обоим каналам свидетельствует о наличии микс-образца (контаминация ДНК на каком-либо этапе исследования или выделение двух штаммов от одного больного).
Данный способ был использован для анализа коллекций ДНК клинических изолятов М. tuberculosis из коллекции ДНК лаборатории молекулярной эпидемиологии и эволюционной генетики ФБУН НИИЭМ им. Пастера, представляющих регионы России и некоторые другие стран с различными популяциями возбудителя и различной долей штаммов Beijing и Beijing 14717-15-кластер, что показано на примерах ниже. Все изоляты были ранее генотипированы методом мультилокусного VNTR-анализа, позволяющим точно определить принадлежность штамма к тому или иному VNTR-кластеру.
Пример 1. Анализ ДНК изолятов М. tuberculosis, выделенных в Западной Сибири, в Омской области, в которой популяция М. tuberculosis характеризуется доминированием штаммов генотипа Beijing (66%) (Pasechnik et al., 2018).
Было исследовано 138 изолятов ДНК М. tuberculosis генотипа Beijing, выделенных от больных туберкулезом, проходивших лечение в областном противотуберкулезном диспансере Омской области в 2020-2021 годах. Было проведено сполиготипирование (Kamerbeek et al., 1997) и MIRU-VNTR типирование (Supply et al., 2006) всех образцов и сравнение с базой MIRU-VNTRplus.org для установления принадлежности к кластеру Beijing 14717-15. Было выявлено 4 образца, относящихся к этому генотипу. Затем на этой же выборке мы протестировали предлагаемый метод детекции Beijing 14717-15 с помощью ПЦР в реальном времени. Было установлено, что 4 образца принадлежали генотипу Beijing 14717-15, что полностью соответствовало результатам MIRU-VNTR типирования.
Пример 2. Анализ ДНК изолятов М. tuberculosis выделенных в Республике Бурятия, для которой характерна заметная (до 16-18%) доля штаммов древней сублинии Beijing и в частности, кластера Beijing 14717-15 (Zhdanova et al., 2022).
Нами были исследованы 265 изолятов от больных с впервые выявленным туберкулезом в Бурятии 2021-2022 гг. Генотипирование проводилось предлагаемым методом полимеразной цепной реакции в реальном времени. Кластер 14717-15, входящий в раннюю древнюю сублинию Beijing, определяли по специфической SNP-мутации 2423040 A>G. В результате проведенной ПЦР были выявлены 47 изолятов, относящихся к генотипу Beijing 14717-15. Все результаты были однозначные и микс-образцов выявлено не было. MIRU-VNTR-типирование по 24 локусам этих 47 изолятов подтвердило их принадлежность кластеру 14717-15.
Пример 3. Анализ ДНК изолятов М. tuberculosis выделенных в Китае и Японии, странах в которых штаммы древних сублинии генотипа Beijing составляют заметную долю популяции возбудителя туберкулеза, 20% в Китае и 70% в Японии (Shamputa et al., 2010, Wada et al., 2009; Yin et al., 2016).
Всего были изучены 123 изолята М. tuberculosis генотипа Beijing, полученные от больных из Окинавы, Япония (65 образцов) и Пекина, Китай (58 изолятов), для которых ранее было проведено MIRU-VNTR типирование и сполиготипирование (Mokrousov et al., 2006; Millet et al., 2012) Затем все ДНК были протестированы предлагаемым способом детекции для выявления образцов Beijing 14717-15 кластера. Было определено, что изоляты 14717-15 кластера в данной выборке отсутствовали, что полностью соответствовало результатам MIRU-VNTR типирования по 24 локусам.
Список литературы
Беспятых Ю.А., Виноградова Т.Н., Маничева О.А., Заболотных Н.В., Догонадзе М.З., Витовская М.Л., Гуляев А.С., Журавлев В.Ю., Шитиков Е.А., Ильина Е.Н. Вирулентность Mycobacterium tuberculosis генотипа Beijing в условиях in vivo // Инфекция и иммунитет.2019. Т. 9, No 1. С.173-182. doi: 10.15789/2220-7619-2019-1-173-182.
Жданова С.Н. Молекулярно-эпидемиологическое исследование распространения основных генотипов возбудителя туберкулеза в Северной Азии. Диссертация на соискание ученой степени доктора медицинских наук. Иркутск. 2018. 276 с.
Мокроусов Мокроусов И.В., Вязовая А.А., Бадлеева М.В., Герасимова А.А., Белопольская О.Б., Машарский А.Э., Костюкова И.В., Жданова С.Н., Мударисова Р.С., Авадэний И., Соловьева Н.С., Найзабаева Д.А., Скиба Ю.А., Журавлев В.Ю., Пасечник О.А., Огарков О.Б. Молекулярная детекция и филогеография гипервирулентного субтипа Mycobacterium tuberculosis Beijing 14717-15 // Инфекция и иммунитет.2023. Т. 13, №1. С.91-99. doi: 10.15789/2220-7619-MDA-2082.
Gagneux S. Ecology and evolution of Mycobacterium tuberculosis. Nat Rev Microbiol. 2018; 16(4):202-213. doi: 10.1038/nrmicro.2018.8.
Iwamoto T, Fujiyama R, Yoshida S, et al. Population structure dynamics of Mycobacterium tuberculosis Beijing strains during past decades in Japan. J Clin Microbiol. 2009;47:3340-3343. http://dx.doi.org/10.1128/JCM.01061 -09.
Kamerbeek J, Schouls L, Kolk A, van Agterveld M, van Soolingen D, Kuijper S, Bunschoten A, Molhuizen H, Shaw R, Goyal M, van Embden J. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol. 1997 Apr;35(4):907-14.
Kang HY, Wada T, Iwamoto T, Maeda S, Murase Y, Kato S, et al. Phylogeographical particularity of the Mycobacterium tuberculosis Beijing family in South Korea based on international comparison with surrounding countries. J Med Microbiol. 2010; 59:1191-7.
Luo T, Comas I, Luo D, Lu B, Wu J, Wei L, Yang C, Liu Q, Gan M, Sun G, Shen X, Liu F, Gagneux S, Mei J, Lan R, Wan K, Gao Q. Southern East Asian origin and coexpansion of Mycobacterium tuberculosis Beijing family with Han Chinese. Proc Natl Acad Sci U S A. 2015 Jun 30; 112(26):8136-41. doi: 10.1073/pnas. 1424063112.
Maeda S, Hijikata M, Hang NTL, Thuong PH, Huan HV, Hoang NP, et al. Genotyping of Mycobacterium tuberculosis spreading in Hanoi, Vietnam using conventional and whole genome sequencing methods. Infect Genet Evol. 2019 Nov 6;78:104107.
Merker M, Barbier M, Cox H, Rasigade JP, Feuerriegel S, Kohl ТА, Diel R, Borrell S, Gagneux S, Nikolayevskyy V, Andres S, Nubel U, Supply P, Wirth T, Niemann S. Compensatory evolution drives multidrug-resistant tuberculosis in Central Asia. Elife. 2018 Oct 30;7:e38200.
Merker M, Blin C, Mona S, Duforet-Frebourg N, Lecher S, Willery E, Blum MG, Riisch-Gerdes S, Mokrousov I, Aleksic E, Allix-Beguec C, Antierens A, Augustynowicz-Kopec E, Ballif M, Barletta F, Beck HP, Barry CE 3rd, Bonnet M, Borroni E, Campos-Herrero I, Cirillo D, Cox H, Crowe S, Crudu V, Diel R, Drobniewski F, Fauville-Dufaux M, Gagneux S, Ghebremichael S, Hanekom M, Hoffher S, Jiao WW, Kalon S, Kohl ТА, Kontsevaya I, Lillebask T, Maeda S, Nikolayevskyy V, Rasmussen M, Rastogi N, Samper S, Sanchez-Padilla E, Savic B, Shamputa 1С, Shen A, Sng LH, Stakenas P, Toit K, Varaine F, Vukovic D, Wahl C, Warren R, Supply P, Niemann S, Wirth T. Evolutionary history and global spread of the Mycobacterium tuberculosis Beijing lineage. Nat Genet. 2015 Mar;47(3):242-9.
Merker M, Rasigade JP, Barbier M, Cox H, Feuerriegel S, Kohl ТА, Shitikov E, Klaos K, Gaudin C, Antoine R, Diel R, Borrell S, Gagneux S, Nikolayevskyy V, Andres S, Crudu V, Supply P, Niemann S, Wirth T. Transcontinental spread and evolution of Mycobacterium tuberculosis W148 European/Russian clade toward extensively drug resistant tuberculosis. Nat Commun. 2022 Aug 30;13(1):5105.
Millet J, Miyagi-Shiohira C, Yamane N, Mokrousov I, Rastogi N. High-resolution MIRU-VNTRs typing reveals the unique nature of Mycobacterium tuberculosis Beijing genotype in Okinawa,
Japan. Infect Genet Evol. 2012 Jun;12(4):637-41. Mokrousov I, Jiao WW, Sun GZ, Liu JW, Valcheva V, Li M, Narvskaya O, Shen AD. Evolution of drug resistance in different sublineages of Mycobacterium tuberculosis Beijing genotype.
Antimicrob Agents Chemother. 2006 Aug; 50(8):2820-3. Mokrousov I, Ly HM, Otten T, Lan NN, Vyshnevskyi B, Hoffher S, Narvskaya O. Origin and primary dispersal of the Mycobacterium tuberculosis Beijing genotype: clues from human phylogeography. Genome Res. 2005 Oct; 15(10): 1357-64. Mokrousov I, Narvskaya O, Otten T, et al. Phylogenetic reconstruction within Mycobacterium tuberculosis Beijing genotype in northwestern Russia. Res Microbiol. 2002;153:629-637.
Mokrousov I, Pasechnik O, Vyazovaya A, Yarusova I, Gerasimova A, Blokh A, Zhuravlev V. Impact of pathobiological diversity of Mycobacterium tuberculosis on clinical features and lethal outcome of tuberculosis. BMC Microbiol. 2022 Feb 8; 22(1):50.
Mokrousov I, Sinkov V, Vyazovaya A, Pasechnik O, Solovieva N, Khromova P, Zhuravlev V, Ogarkov O. Genomic signatures of drug resistance in highly resistant Mycobacterium tuberculosis strains of the early ancient sublineage of Beijing genotype in Russia. Int J Antimicrob Agents. 2020 Aug; 56(2): 106036.
Mokrousov I, Vyazovaya A, Pasechnik O, Gerasimova A, Dymova M, Chernyaeva E, Tatarintseva M, Stasenko V. Early ancient sublineages of Mycobacterium tuberculosis Beijing genotype: unexpected clues from phylogenomics of the pathogen and human history. Clin Microbiol Infect. 2019 Aug; 25(8):1039.el-1039.e6.
Mokrousov I. Insights into the origin, emergence, and current spread of a successful Russian clone of Mycobacterium tuberculosis. Clin Microbiol Rev. 2013 Apr; 26(2):342-60.
Pasechnik О, Vyazovaya A, Vitriv S, Tatarintseva M, Blokh A, Stasenko V, Mokrousov I. Major genotype families and epidemic clones of Mycobacterium tuberculosis in Omsk region, Western Siberia, Russia, marked by a high burden of tuberculosis-HIV coinfection. Tuberculosis (Edinb). 2018 Jan; 108:163-168.
Shamputa IС, Lee J, Allix-Beguec C, Cho EJ, Lee JI, Rajan V, et al. Genetic diversity of Mycobacterium tuberculosis isolates from a tertiary care tuberculosis hospital in South Korea. J Clin Microbiol. 2010;48:387-94. Shitikov E, Kolchenko S, Mokrousov I, Bespyatykh J, Ischenko D, Ilina E, et al. Evolutionary pathway analysis and unified classification of East Asian lineage of Mycobacterium tuberculosis. Sci Rep.2017;7:9227.
Supply, P., et al. Proposal for standardization of optimized mycobacterial interspersed repetitive unit-variable-number tandem repeat typing of Mycobacterium tuberculosis. J. Clin. Microbiol. 44, 4498-4510 (2006)
Umpeleva T, Belousova K, Golubeva L, Boteva T, Morozova I, Vyazovaya A, Mokrousov I, Eremeeva N, Vakhrusheva D. Molecular characteristics of Mycobacterium tuberculosis in the "closed" Russian town with limited population migration. Infect Genet Evol. 2020 Apr;79:104174.
van Embden JD, Cave MD, Crawford JT, Dale JW, Eisenach KD, Gicquel B, Hermans P, Martin C, McAdam R, Shinnick TM, Small P. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J Clin Microbiol. 1993 Feb;31(2):406-9. doi: 10.1128/jcm.31.2.406-409.1993.
Vinogradova T, Dogonadze M, Zabolotnykh N, Badleeva M, Yarusova I, Vyazovaya A, Gerasimova A, Zhdanova S, Vitovskaya M, Solovieva N, Pasechnik O, Ogarkov O, Mokrousov I. Extremely lethal and hypervirulent Mycobacterium tuberculosis strain cluster emerging in Far East, Russia. Emerg Microbes Infect. 2021 Dec; 10(1): 1691-1701.
Vyazovaya A, Gerasimova A, Mudarisova R, Terentieva D, Solovieva N, Zhuravlev V, Mokrousov I. Genetic Diversity and Primary Drug Resistance of Mycobacterium tuberculosis Beijing Genotype Strains in Northwestern Russia. Microorganisms. 2023 Jan 19; 11(2):255.
Wada T, Iwamoto T, Maeda S. Genetic diversity of the Mycobacterium tuberculosis Beijing family in East Asia revealed through refined population structure analysis. FEMS Microbiol Lett. 2009 Feb;291(l):35-43.
World Health Organization. Global tuberculosis report 2022. Geneva: World Health Organization; 2022.
Yin QQ, Liu HC, Jiao WW, Li QJ, Han R, Tian JL, et al. Evolutionary History and Ongoing Transmission of Phylogenetic Sublineages of Mycobacterium tuberculosis Beijing Genotype in China. Sci Rep.2016;6:34353.
Zhdanova S, Mokrousov I, Orlova E, Sinkov V, Ogarkov O. Transborder molecular analysis of drug-resistant tuberculosis in Mongolia and Eastern Siberia, Russia. Transbound Emerg Dis. 2022 Sep; 69(5):el800-el814.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="Beijing14717-15.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2025-01-05">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023120491</ApplicationNumberText>
<FilingDate>2023-08-03</FilingDate>
</ApplicationIdentification>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023120491</ApplicationNumberText>
<FilingDate>2023-08-03</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">P$P5P4P5QP0P;QP=P>P5
P3P>QQ P4P0QQQ P2P5P=P=P>P5 P1QP4P6P5Q P=P>P5 Q QQP5P6P4P5P=P8P5
B+P!P0P=P:Q -P¬P5Q P5QP1Q QP3QP:P8P9
P=P0Q QP=P>-P8QQP;P5P4P>P2P0Q P5P;QQP:P8P9 P8P=QQ P8Q Q Q
Q Q P8P7P8P>P?Q P;QP<P>P=P>P;P>P3P8P8B; P_P8P=P8QQ P5QQQ P2P0
P7P4QP0P2P>P>QQP0P=P5P=P8Q P P>QQP8P9QP:P>P9 P$P5P4P5QP0QP8P8
(P$P_P_P# B+P!P¬P1 PPPP$B; P_P8P=P7P4QP0P2P0 P
P>QQP8P8)</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Institution "St.
Petersburg Research Institute of Phthisiopulmonology" of the
Ministry of Health of the Russian Federation (FSBI "SPb
NIIF" of the Ministry of Health of Russia)</ApplicantNameLatin>
<InventorName languageCode="ru">P_P>P:QP>Q QP>P2 PP3P>QQ
P_P;P0P4P8QP;P0P2P>P2P8Q</InventorName>
<InventorNameLatin>Mokrousov Igor Vladislavovich</InventorNameLatin>
<InventionTitle languageCode="ru">P!P?P>QP>P1 P4P5Q P5P:QP8P8
Mycobacterium tuberculosis P3P5P=P>Q P8P?P0 Beijing
14717-15-P:P;P0QQ P5Q P<P5Q P>P4P>P< P?P>P;P8P<P5QP0P7P=P>P9
QP5P?P=P>P9 QP5P0P:QP8P8 P2 Q P>QP<P0Q P5 QP5P0P;QP=P>P3P>
P2QP5P<P5P=P8</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>PCR_primers</INSDQualifier_name>
<INSDQualifier_value>fwd_name: 2423040F, fwd_seq:
cggcgtgctcctgctgat,rev_name: 2423040R, rev_seq:
agttcgtcaccgacctgac</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggcgtgctcctgctgat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>PCR_primers</INSDQualifier_name>
<INSDQualifier_value>fwd_name: 2423040F, fwd_seq:
cggcgtgctcctgctgat,rev_name: 2423040R, rev_seq:
agttcgtcaccgacctgac</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agttcgtcaccgacctgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>FAM-cytosine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA-adenine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>BHQ1-thymine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgcgtcttctccggcacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>HEX-cytosine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA-guanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>BHQ1-thymine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgcgtcttctccggcgcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 14717-15-кластер | 2022 |
|
RU2812351C1 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 1071-32-кластер в формате реального времени | 2021 |
|
RU2768021C1 |
| Способ детекции филогенетических сублиний генотипа Beijing Mycobacterium tuberculosis в формате реального времени | 2020 |
|
RU2743365C1 |
| Способ детекции изолятов Mycobacterium tuberculosis Beijing 94-32-кластера в формате реального времени | 2017 |
|
RU2689800C1 |
| Способ выявления микобактерий туберкулеза Центрально-Азиатского эпидемического кластера генотипа Beijing | 2019 |
|
RU2735415C1 |
| Способ детекции S-генотипа Mycobacterium tuberculosis | 2024 |
|
RU2832725C1 |
| (4-(3-гидроксифенил)пиперазин-1-ил)(5-нитрофуран-2-ил)метанон, обладающий противотуберкулезной активностью в отношении возбудителя туберкулеза с множественной лекарственной устойчивостью, и способ его получения | 2021 |
|
RU2784399C1 |
| СПОСОБ ВЫЯВЛЕНИЯ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗА ГЕНОТИПА ВЕIJING В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2011 |
|
RU2528866C2 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Способ выявления микобактерий туберкулеза генотипа Beijing В0-кластер в формате реального времени | 2017 |
|
RU2684314C2 |
Изобретение относится к области молекулярной биологии. Описан способ детекции Mycobacterium tuberculosis генотипа Beijing 14717-15-кластер методом полимеразной цепной реакции в формате реального времени, отличающийся тем, что выявляют наличие однонуклеотидной замены A>G в гене Rv2161c в геномной позиции 2423040 с помощью ПЦР в формате реального времени с использованием праймеров и зондов. Техническим результатом изобретения является простота использования метода, легкая и однозначная интерпретация результата, возможность определения возможной контаминации или микс-инфекции. 3 ил., 3 пр.
Способ детекции Mycobacterium tuberculosis генотипа Beijing 14717-15-кластер методом полимеразной цепной реакции в формате реального времени, отличающийся тем, что выявляют наличие однонуклеотидной замены A>G в гене Rv2161c в геномной позиции 2423040 с помощью ПЦР в формате реального времени с использованием праймеров 2423040F 5'-CGGCGTGCTCCTGCTGAT и 2423040R 5'-AGTTCGTCACCGACCTGAC и флуоресцентно-меченых зондов на дикий аллель 2423040А 5'-[FAM] CGTGCGTCTTCTCCGGC[A-LNA]CAT[BHQ1] и на мутантный аллель 2423040G 5'-[HEX] CGTGCGTCTTCTCCGGC[G-LNA]CAT[BHQ1] с оценкой результата ПЦР в 20-35 циклах путем регистрации экспоненциального роста сигнала флуоресценции по НЕХ-каналу с длиной волны 555 нм и отсутствием сигнала флуоресценции по FAM-каналу с длиной волны 510 нм для утверждения о принадлежности образца к генотипу М. tuberculosis Beijing 14717-15-кластер.
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 1071-32-кластер в формате реального времени | 2021 |
|
RU2768021C1 |
| Способ детекции изолятов Mycobacterium tuberculosis Beijing 94-32-кластера в формате реального времени | 2017 |
|
RU2689800C1 |
| Mokrousov I | |||
| et al | |||
| Early ancient sublineages of Mycobacterium tuberculosis Beijing genotype: unexpected clues from phylogenomics of the pathogen and human history, Clinical Microbiology and Infection, 2019, Т | |||
| Видоизменение пишущей машины для тюркско-арабского шрифта | 1923 |
|
SU25A1 |
| Топка с несколькими решетками для твердого топлива | 1918 |
|
SU8A1 |
| РЕГЕНЕРАТИВНЫЙ РАДИО-ПРИЕМНИК | 1924 |
|
SU1039A1 |
Авторы
Даты
2025-05-12—Публикация
2023-08-03—Подача