ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
По этой заявке испрашивается приоритет временной заявки на патент США, серийный номер 62/516324, поданной 7 июня 2017 г., которая включена в настоящее описание посредством ссылки.
ОБЛАСТЬ ТЕХНИКИ
Варианты осуществления настоящего изобретения относятся к секвенированию нуклеиновых кислот. В частности, варианты осуществления способов и композиций по изобретению относятся к получению библиотек бисульфитного секвенирования отдельных клеток и получению данных о последовательностях из них.
УРОВЕНЬ ТЕХНИКИ
Секвенирование отдельных клеток при высоком содержании клеток показало свою эффективность при разделении популяций в сложных тканях посредством транскриптомов, доступности хроматина и мутационных различий. Кроме того, разрешение отдельных клеток дало возможность оценить траектории дифференцировки клеток по геномно-специфическим закономерностям, таким как метилирование ДНК. Метилирование ДНК является ковалентным дополнением к цитозину; метка с типоспецифичностью клеток, которая является субъектом активной модификации в развивающихся тканях. Метилирование ДНК может быть исследовано при разрешении пар оснований с использованием дезаминирующей химической реакции обработки бисульфитом натрия.
Результаты недавних работ дали возможность оптимизировать бисульфитное секвенирование настолько, что индукция отдельных клеток требуется либо для бисульфитного секвенирования при сниженном представительстве отдельных клеток (scRRBS), либо для полногеномного бисульфитного секвенирования отдельных клеток (scWGBS). Однако этим способам не хватает масштабируемости, так как они основаны на деконволюции отдельных клеток посредством создания параллельной и изолированной библиотеки, в которой реакции отдельных клеток осуществляются изолированно. Для каждого секвенирования клетки необходим совершенно новый набор реагентов, что приводит к пропорциональному изменению затрат для каждой дополнительной клетки. Из-за проблем, связанных с бисульфитной конверсией ДНК, не применялось ни одной микроструйной системы на основе капель или чипов для бисульфитного секвенирования отдельных клеток, а также не существует какой-либо теоретически эффективной стратегии с использованием альтернативных платформ.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Изобретение относится к композициям и масштабируемым профилирующим анализам большого количества клеток и метилома отдельных клеток. Полногеномное секвенирование отдельных клеток (scWGBS) улучшили с помощью описанных в настоящем изобретении стратегий комбинаторной индексации отдельных клеток, так что клетки можно обрабатывать в большом количестве, а выход из одной клетки демультиплексировать in silico. В некоторых вариантах осуществления в описанных в настоящем изобретении способах используют встраивание адаптора на основе транспозазы, что приводит к повышению эффективности и намного более высоким скоростям выравнивания по сравнению со способами выхода. Использование транспозазы для добавления одного из двух адапторов секвенирования дает возможность гораздо более эффективно создавать библиотеки с меньшим количеством считываний шума, что приводит к скорости выравнивания ~60% (аналогично скоростям для стратегий с комплексом клеток) по сравнению с 10-30% с использованием способов с отдельной клеткой на одну лунку. Это приводит к более удобному считыванию последовательностей и значительному снижению затрат на часть анализа, включающую секвенирование. Использование стратегий комбинаторной индексации отдельных клеток для получения библиотек бисульфитного секвенирования отдельных клеток продемонстрировано на смеси клеток человека и мыши с минимальной частотой столкновений. Также продемонстрирована успешная деконволюция сочетания трех типов клеток человека и достижения назначения типа клетки с использованием общедоступных данных.
Определения
Используемые в рамках изобретения термины "организм", "пациент" используются взаимозаменяемо и относятся к животным и растениям. Примером животного является млекопитающее, такое как человек.
Используемый в рамках изобретения термин "тип клеток" предназначен для идентификации клеток на основе морфологии, фенотипа, происхождения развития или других известных или распознаваемых отличительных клеточных характеристик. Ряд различных типов клеток может быть получен из отдельного организма (или из одного и того же вида организма). Иллюстративные типы клеток включают без ограничения мочевой пузырь, эпителий поджелудочной железы, альфа поджелудочной железы, бета поджелудочной железы, эндотелий поджелудочной железы, лимфобласт костного мозга, В-лимфобласт костного мозга, макрофаг костного мозга, эритробласт костного мозга, дендрит костного мозга, адипоцит костного мозга, остеоцит костного мозга, хондроцит костного мозга, промиелобласт, мегакариобласт костного мозга, пузырь, В-лимфоцит головного мозга, глиальную клетку головного мозга, нейрон, астроцит мозга, нейроэктодерму, макрофаг мозга, микроглию головного мозга, эпителий головного мозга, кортикальный нейрон, фибробласт мозга, эпителий молочной железы, эпителий прямой кишки, В-лимфоцит прямой кишки, эпителий молочной железы, миоэпителий молочной железы, фибробласт молочной железы, энтероцит прямой кишки, эпителий шейки матки, эпителий яичника, фибробласт яичника, эпителиальный проток молочной железы, эпителий языка, дендритную клетку миндалины, В-лимфоцит миндалины, лимфоцит периферической крови, Т-лимфоцит периферической крови, кожный Т-лимфоцит периферической крови, естественный киллер периферической крови, B-лимфоцит периферической крови, моноцит периферической крови, промиелобласт периферической крови, макрофаг периферической крови, базофил периферической крови, эндотелий печени, тучную клетку печени, эпителий печени, В-лимфоцит печени, эндотелий селезенки, эпителий селезенки, В-лимфоцит селезенки, гепатоцит печени, клетка печени Александра, фибробласт печени, эпителиальную клетку легкого, эпителиальную клетку бронха, фибробласт легкого, В-лимфоцит легкого, шванновскую клетку легкого, плоскоклеточную клетку легкого, макрофаг легкого, остеобласт легкого, нейроэндокринную клетку, альвеолярную клетку легкого, эпителиальную клетку желудка и желудочный фибробласт.
Используемый в рамках изобретения термин "ткань" предназначен для обозначения скопления или агрегации клеток, которые действуют вместе для выполнения одной или нескольких конкретных функций в организме. Клетки могут необязательно быть морфологически сходными. Иллюстративные ткани включают без ограничения глаз, мышцу, кожу, сухожилие, вену, артерию, кровь, сердце, селезенку, лимфатический узел, кость, костный мозг, легкое, бронхи, трахею, кишку, тонкую кишку, заднюю кишку, толстую кишку, прямую кишку, слюнную железу, язык, желчный пузырь, аппендикс, печень, поджелудочную железу, мозг, желудок, кожу, почку, мочеточник, мочевой пузырь, уретру, гонаду, яичко, яичник, матку, маточную трубу, тимус, гипофиз, щитовидную железу, надпочечник или паращитовидную железу. Ткань может происходить из любого из ряда органов человека или другого организма. Ткань может являться здоровой или нездоровой. Примеры нездоровых тканей включают без ограничения различные злокачественные новообразования с аберрантным метилированием, например, злокачественные новообразования в легком, молочной железе, толстой кишке, предстательной железе, носоглотке, желудке, яичках, коже, нервной системе, кости, яичнике, печени, гематологических тканях, поджелудочной железе, матке, почке, лимфоидных тканях и т.д. Злокачественные новообразования могут иметь ряд гистологических подтипов, например, карциномы, аденокарциномы, саркомы, фиброаденокарциномы, нейроэндокринные или недифференцированные.
Используемый в рамках изобретения термин "компартмент" предназначен для обозначения области или объема, которые отделяют или изолируют что-то от других вещей. Иллюстративные компартменты включают без ограничения флаконы, пробирки, лунки, капли, болюсы, микроносители, сосуды, поверхностные элементы, или области, или объемы, разделенные физическими силами, такими как поток жидкости, магнетизм, электрический ток или т.п. В одном варианте осуществления компартмент представляет собой лунку многолуночного планшета, такого как 96- или 384-луночный планшет.
Используемый в рамках изобретения термин "транспосомный комплекс" относится к ферменту интеграции и нуклеиновой кислоте, включая сайт распознавания интеграции. "Транспосомный комплекс" представляет собой функциональный комплекс, образованный транспозазой и сайтом распознавания транспозазы, который способен катализировать реакцию транспозиции (см., например, Gunderson et al., WO 2016/130704). Примеры ферментов интеграции включают без ограничения такие, как интеграза или транспозаза. Примеры сайтов распознавания интеграции включают без ограничения сайт распознавания транспозазы.
Используемый в рамках изобретения термин "нуклеиновая кислота" предназначен для использования в данной области и включает встречающиеся в природе кольцевые нуклеиновые кислоты или их функциональные аналоги. Особенно применяемые функциональные аналоги способны гибридизоваться с нуклеиновой кислотой специфичным к последовательности образом или способны применяться в качестве матрицы для репликации конкретной нуклеотидной последовательности. Природные нуклеиновые кислоты обычно содержат каркас, содержащий сложные фосфодиэфирные связи. Аналоговая структура может иметь альтернативную связь в каркасе, включая любую из множества известных из уровня техники. Природные нуклеиновые кислоты обычно содержат дезоксирибозный сахар (например, найденный в дезоксирибонуклеиновой кислоте (ДНК)) или рибозный сахар (например, найденный в рибонуклеиновой кислоте (РНК)). Нуклеиновая кислота может содержать любой из множества аналогов этих сахарных фрагментов, которые известны из уровня техники. Нуклеиновая кислота может включать нативные или ненативные основания. С данной целью нативная дезоксирибонуклеиновая кислота может содержать одно или несколько оснований, выбранных из группы, состоящей из аденина, тимина, цитозина или гуанина, и рибонуклеиновая кислота может содержать одно или несколько оснований, выбранных из группы, состоящей из урацила, аденина, цитозина или гуанина. Применяемые ненативные основания, которые могут быть включены в нуклеиновую кислоту, известны из уровня техники. Примеры ненативных оснований включают закрытую нуклеиновую кислоту (LNA) и мостиковую нуклеиновую кислоту (BNA). Основания LNA и BNA могут быть встроены в олигонуклеотид ДНК и повышают прочность и специфичность гибридизации олигонуклеотидов. Основания LNA и BNA и их применение известны специалисту в данной области и являются обычными.
Используемый в рамках изобретения термин "мишень", в случае если он используется в отношении нуклеиновой кислоты, предназначен в качестве семантического идентификатора для нуклеиновой кислоты в контексте способа или композиции, изложенных в рамках изобретения, и не обязательно ограничивает структуру или функцию нуклеиновой кислоты сверх того, что явно указано иным образом. Нуклеиновая кислота-мишень может являться практически любой нуклеиновой кислотой известной или неизвестной последовательности. Она может являться, например, фрагментом геномной ДНК или кДНК. Секвенирование может привести к определению последовательности всей или части молекулы-мишени. Мишени можно получить из первичного образца нуклеиновой кислоты, такого как ядро. В одном варианте осуществления мишени могут быть переработаны в матрицы, подходящие для амплификации, путем размещения универсальных последовательностей на концах каждого фрагмента-мишени. Мишени также могут быть получены из образца первичной РНК путем обратной транскрипции в кДНК.
Используемый в рамках изобретения термин "универсальный", в случае если он используется для описания нуклеотидной последовательности, относится к области последовательности, которая является общей для двух или более молекул нуклеиновой кислоты, где молекулы также содержат области последовательности, которые отличаются друг от друга. Универсальная последовательность, которая присутствует в разных членах скопления молекул, может обеспечить возможность захвата нескольких различных нуклеиновых кислот с использованием популяции универсальных нуклеиновых кислот для захвата, например, олигонуклеотидов для захвата, которые комплементарны части универсальной последовательности, например, универсальная последовательность для захвата. Неограничивающие примеры универсальных последовательностей для захвата включают последовательности, которые являются идентичными или комплементарными праймерам P5 и P7. Аналогично, универсальная последовательность, присутствующая в разных членах скопления молекул, может обеспечить возможность репликации или амплификации множества различных нуклеиновых кислот с использованием популяции универсальных праймеров, которые комплементарны части универсальной последовательности, например универсальной якорной последовательности. Следовательно, олигонуклеотид для захвата или универсальный праймер включает в себя последовательность, которая может конкретно гибридизоваться с универсальной последовательностью.
Термины "P5" и "P7" можно использовать в отношении праймеров для амплификации, например, олигонуклеотида для захвата. Термины "P5'" (P5 штрих) и "P7'" (P7 штрих) относятся соответственно к комплементам P5 и P7. Понятно, что любые подходящие праймеры для амплификации можно применять в способах, представленных в рамках изобретения, и что применение P5 и P7 является только иллюстративными вариантами осуществления. Использование амплификационных праймеров, таких как P5 и P7, на проточных ячейках известно в данной области техники, как описано, например, в изобретениях WO 2007/010251, WO 2006/064199, WO 2005/065814, WO 2015/106941, WO 1998/044151 и WO 2000/018957. Например, любой подходящий прямой амплификационный праймер, иммобилизованный или в растворе, можно применять в способах, представленных в рамках изобретения, для гибридизации с комплементарной последовательностью и амплификации последовательности. Аналогично, любой подходящий обратный амплификационный праймер, либо иммобилизованный, либо в растворе, можно применять в способах, представленных в рамках изобретения, для гибридизации с комплементарной последовательностью и амплификации последовательности. Специалист в данной области поймет, как сконструировать и использовать последовательности праймеров, которые подходят для захвата и/или амплификации нуклеиновых кислот, как представлено в рамках изобретения.
Используемый в рамках изобретения термин "праймер" и его производные обычно относятся к любой нуклеиновой кислоте, которая может гибридизоваться с последовательностью-мишенью, представляющей интерес. Как правило, праймер выполняет функцию субстрата, на котором нуклеотиды могут полимеризоваться с помощью полимеразы; однако в некоторых вариантах осуществления праймер может встраиваться в синтезированную нить нуклеиновой кислоты и обеспечивать сайт, с которым другой праймер может гибридизоваться для запуска синтеза новой нити, которая комплементарна синтезированной молекуле нуклеиновой кислоты. Праймер может включать любую комбинацию нуклеотидов или их аналогов. В некоторых вариантах осуществления праймер представляет собой однонитевой олигонуклеотид или полинуклеотид. Термины "полинуклеотид" и "олигонуклеотид" используются в рамках изобретения взаимозаменяемо для обозначения полимерной формы нуклеотидов любой длины и могут включать рибонуклеотиды, дезоксирибонуклеотиды, их аналоги или их смеси. Понятно, что термины включают в качестве эквивалентов аналоги либо ДНК, либо РНК, полученные из нуклеотидных аналогов, и используются к однонитевым (таким как смысловые или антисмысловые) и двухнитевым полинуклеотидам. Используемый в рамках изобретения термин также охватывает кДНК, которая является комплементарной или копийной ДНК, полученной из матрицы РНК, например, с помощью действия обратной транскриптазы. Этот термин относится только к первичной структуре молекулы. Таким образом, этот термин включает трех-, двух- и однонитевую дезоксирибонуклеиновую кислоту ("ДНК"), а также трех-, двух- и однонитевую рибонуклеиновую кислоту ("РНК").
Используемый в рамках изобретения термин "адаптер" и его производные, например универсальный адаптер, обычно относится к любому линейному олигонуклеотиду, который можно лигировать с молекулой нуклеиновой кислоты по настоящему изобретению. В некоторых вариантах осуществления адаптер фактически не комплементарен 3'-концу или 5'-концу любой последовательности-мишени, присутствующей в образце. В некоторых вариантах осуществления подходящие значения длины адаптера находятся в диапазоне, составляющем приблизительно 10-100 нуклеотидов, приблизительно 12-60 нуклеотидов и приблизительно 15-50 нуклеотидов в длину. Обычно адаптер может включать в себя любую комбинацию нуклеотидов и/или нуклеиновых кислот. В некоторых аспектах адаптер может включать в себя одну или несколько расщепляемых групп в одном или нескольких местоположениях. В другом аспекте адаптер может включать в себя последовательность, которая фактически идентична или фактически комплементарна по меньшей мере части праймера, например универсального праймера. В некоторых вариантах осуществления адаптер может включать штрих-код или метку, чтобы помочь с последующим исправлением ошибок, идентификацией или секвенированием. Термины "адаптор" и "адаптер" используются взаимозаменяемо.
Используемый в рамках изобретения термин "каждый" при использовании в отношении скопления элементов предназначен для идентификации компартментного элемента в скоплении, но не обязательно относится к каждому элементу в скоплении, если контекст явно не указывает на иное.
Используемый в рамках изобретения термин "транспорт" относится к движению молекулы через жидкость. Термин может включать пассивный транспорт, такой как движение молекул вдоль градиента их концентрации (например, пассивная диффузия). Термин также может включать активный транспорт, при котором молекулы могут двигаться вдоль градиента концентрации или против градиента концентрации. Таким образом, транспорт может включать использование энергии для перемещения одной или нескольких молекул в требуемом направлении или в требуемое местоположение, такую как сайт амплификации.
Используемые в рамках изобретения термины "амплифицировать", "амплификация" или "реакция амплификации" и их производные обычно относятся к любому действию или способу, при которых по меньшей мере часть молекулы нуклеиновой кислоты реплицируется или копируется в по меньшей мере одну дополнительную молекулу нуклеиновой кислоты. Дополнительная молекула нуклеиновой кислоты необязательно включает последовательность, которая фактически идентична или фактически комплементарна по меньшей мере некоторой части матричной молекулы нуклеиновой кислоты. Матричная молекула нуклеиновой кислоты может быть однонитевой или двухнитевой, а дополнительная молекула нуклеиновой кислоты может независимо быть однонитевой или двухнитевой. Амплификация необязательно включает линейную или экспоненциальную репликацию молекулы нуклеиновой кислоты. В некоторых вариантах осуществления такую амплификацию можно осуществлять с использованием изотермических условий; в других вариантах осуществления такая амплификация может включать термоциклирование. В некоторых вариантах осуществления амплификация представляет собой мультиплексную амплификацию, которая включает одновременную амплификацию совокупности последовательностей-мишеней в отдельной реакции амплификации. В некоторых вариантах осуществления "амплификация" включает амплификацию по меньшей мере некоторой части нуклеиновых кислот на основе ДНК и РНК по отдельности или в комбинации. Реакция амплификации может включать любой из способов амплификации, известных специалисту в данной области. В некоторых вариантах осуществления реакция амплификации включает полимеразную цепную реакцию (ПЦР).
Используемый в рамках изобретения термин "условия амплификации" и его производные обычно относится к условиям, подходящим для амплификации одной или нескольких последовательностей нуклеиновых кислот. Такая амплификация может быть линейной или экспоненциальной. В некоторых вариантах осуществления условия амплификации могут включать изотермические условия или, как альтернатива, могут включать условия термоциклирования или комбинацию изотермических условий и условий термоциклирования. В некоторых вариантах осуществления условия, подходящие для амплификации одной или нескольких последовательностей нуклеиновых кислот, включают условия полимеразной цепной реакции (ПЦР). Как правило, условия амплификации относятся к реакционной смеси, которой достаточно для амплификации нуклеиновых кислот, таких как одна или несколько последовательностей-мишеней, или для амплификации амплифицированной последовательности-мишени, лигированной с одним или несколькими адаптерами, например, лигированной с помощью адаптера амплифицированной последовательности-мишени. Обычно условия амплификации включают катализатор для амплификации или для синтеза нуклеиновой кислоты, например полимеразу; праймер, который обладает некоторой степенью комплементарности с нуклеиновой кислотой, подлежащей амплификации; и нуклеотиды, такие как дезоксирибонуклеотидтрифосфаты (dNTP), для ускорения удлинения праймера после гибридизации с нуклеиновой кислотой. Условия амплификации могут требовать гибридизации или отжига праймера с нуклеиновой кислотой, удлинения праймера и стадии денатурирования, на которой удлиненный праймер отделяется от последовательности нуклеиновой кислоты, подвергающейся амплификации. Как правило, но не обязательно, условия амплификации могут включать термоциклирование; в некоторых вариантах осуществления условия амплификации включают множество циклов, где стадии отжига, удлинения и отделения повторяются. Как правило, условия амплификации включают катионы, такие как Mg2+ или Mn2+, и могут также включать различные модификаторы ионной силы.
Используемый в рамках изобретения термин "повторная амплификация" и его производные обычно относится к любому способу, при котором по меньшей мере часть амплифицированной молекулы нуклеиновой кислоты дополнительно амплифицируется посредством любого подходящего способа амплификации (в некоторых вариантах осуществления упоминаемого как "вторичная" амплификация), получая тем самым повторно амплифицированную молекулу нуклеиновой кислоты. Вторичная амплификация не должна быть идентична первоначальному способу амплификации, при котором была получена амплифицированная молекула нуклеиновой кислоты; также повторно амплифицированная молекула нуклеиновой кислоты не должна быть полностью идентичной или полностью комплементарной амплифицированной молекуле нуклеиновой кислоты; все, что требуется, - это чтобы молекула повторно амплифицированной нуклеиновой кислоты включала по меньшей мере часть амплифицированной молекулы нуклеиновой кислоты или ее комплемента. Например, повторная амплификация может включать использование других условий амплификации и/или других праймеров, включая другие специфические по отношению к мишени праймеры, чем первичная амплификация.
Используемый в рамках изобретения термин "полимеразная цепная реакция" ("ПЦР") относится к способу Mullis из патентов США №№ 4683195 и 4683202, в которых описан способ увеличения концентрации сегмента полинуклеотида в смеси геномной ДНК, представляющего интерес, без клонирования или очистки. Этот способ амплификации полинуклеотида, представляющего интерес, состоит из введения в большом количестве двух олигонуклеотидных праймеров в смесь ДНК, содержащую требуемый полинуклеотид, представляющий интерес, с последующей серией термоциклирования в присутствии ДНК-полимеразы. Два праймера являются комплементарными их соответствующим нитям двухнитевого полинуклеотида, представляющего интерес. Смесь сначала денатурируют при более высокой температуре, а затем праймеры отжигают с комплементарными последовательностями в молекуле полинуклеотида, представляющего интерес. После отжига праймеры удлиняют с помощью полимеразы, чтобы сформировать новую пару комплементарных нитей. Стадии денатурации, отжига праймеров и удлинения полимеразы могут повторяться много раз (что называется термоциклированием) для получения высокой концентрации амплифицированного сегмента требуемого полинуклеотида, представляющего интерес. Длина амплифицированного сегмента требуемого полинуклеотида, представляющего интерес (ампликона), определяется относительными положениями праймеров относительно друг друга, а следовательно эта длина является контролируемым параметром. В силу повторения процесса способ называется "полимеразной цепной реакцией" (далее "ПЦР"). Поскольку требуемые амплифицированные сегменты полинуклеотида, представляющего интерес, становятся преобладающими последовательностями нуклеиновых кислот (с точки зрения концентрации) в смеси, - они называются "амплифицированными с помощью ПЦР". В модификации описанного выше способа молекулы нуклеиновой кислоты-мишени могут быть амплифицированы с помощью ПЦР с использованием совокупности различных пар праймеров, в некоторых случаях одна или несколько пар праймеров на молекулу-мишень нуклеиновой кислоты, представляющую интерес, образуя тем самым мультиплексную реакцию ПЦР.
Используемый в рамках изобретения термин "мультиплексная амплификация" относится к селективной и неслучайной амплификации двух или более последовательностей-мишеней в образце с использованием по меньшей мере одного специфического по отношению к мишени праймера. В некоторых вариантах осуществления мультиплексная амплификация выполняется таким образом, что некоторые или все последовательности-мишени амплифицируются в отдельном реакционном сосуде. "Плексность" или "плекс" данной мультиплексной амплификации обычно относится к числу различных мишень-специфических последовательностей, которые амплифицируются во время этой отдельной мультиплексной амплификации. В некоторых вариантах осуществления плексность может являться приблизительно 12-плексной, 24-плексной, 48-плексной, 96-плексной, 192-плексной, 384-плексной, 768-плексной, 1536-плексной, 3072-плексной, 6144-плексной или выше. Также возможно выявить амплифицированные последовательности-мишени с помощью нескольких различных методологий (например, гель-электрофорез с последующей денситометрией, количественное определение с помощью биоанализатора или количественная ПЦР, гибридизация с меченым зондом; встраивание биотинилированных праймеров с последующим выявлением с помощью конъюгата авидин-фермент; встраивание меченых 32P дезоксинуклеотидтрифосфатов в амплифицированную последовательность-мишень).
Используемый в рамках изобретения термин "амплифицированные последовательности-мишени" и его производные обычно относится к последовательности нуклеиновой кислоты, полученной амплификацией последовательностей-мишеней с использованием мишень-специфических праймеров и способов, описанных в настоящем изобретении. Амплифицированные последовательности-мишени могут быть либо односмысловыми (т.е., положительная нить), либо антисмысловыми (т.е., отрицательная нить) по отношению к последовательностям-мишеням.
Используемые в рамках изобретения термины "лигировать", "лигирование" и их производные обычно относятся к способу ковалентного связывания двух или более молекул вместе, например, ковалентного связывания двух или более молекул нуклеиновой кислоты друг с другом. В некоторых вариантах осуществления лигирование включает присоединение ников между соседними нуклеотидами нуклеиновых кислот. В некоторых вариантах осуществления лигирование включает формирование ковалентной связи между концом первой и концом второй молекулы нуклеиновой кислоты. В некоторых вариантах осуществления лигирование может включать формирование ковалентной связи между 5'-фосфатной группой одной нуклеиновой кислоты и 3'-гидроксильной группой второй нуклеиновой кислоты, формируя тем самым лигированную молекулу нуклеиновой кислоты. Обычно для целей настоящего изобретения амплифицированная последовательность-мишень может быть лигирована с адаптером с получением лигированной с помощью адаптера амплифицированной последовательности-мишени.
Используемый в рамках изобретения термин "лигаза" и его производные, как правило, относится к любому средству, способному катализировать лигирование двух молекул субстрата. В некоторых вариантах осуществления лигаза включает фермент, способный катализировать присоединение ников между соседними нуклеотидами нуклеиновой кислоты. В некоторых вариантах осуществления лигаза включает фермент, способный катализировать образование ковалентной связи между 5'-фосфатом одной молекулы нуклеиновой кислоты и 3'-гидроксилом другой молекулы нуклеиновой кислоты, образуя тем самым лигированную молекулу нуклеиновой кислоты. Подходящие лигазы могут включать без ограничения ДНК-лигазу Т4, РНК-лигазу Т4 и ДНК-лигазу Е. coli.
Используемый в рамках изобретения термин "условия лигирования" и его производные обычно относится к условиям, подходящим для лигирования двух молекул друг с другом. В некоторых вариантах осуществления условия лигирования являются подходящими для заполнения ников или гэпов между нуклеиновыми кислотами. Используемый в рамках изобретения термин "ник или гэп" согласуется с использованием термина в данной области техники. Как правило, ник или гэп можно лигировать в присутствии фермента, такого как лигаза, при подходящей температуре и pH. В некоторых вариантах осуществления лигаза ДНК Т4 может соединять ник между нуклеиновыми кислотами при температуре приблизительно 70-72°С.
Используемый в рамках изобретения термин "проточная ячейка" относится к камере, имеющей твердую поверхность, по которой может протекать один или несколько жидких реагентов. Примеры проточных ячеек и связанных с ними струйных систем и платформ для выявления, которые можно легко использовать в способах по настоящему изобретению, описаны, например, в Bentley et al., Nature 456: 53-59 (2008), WO 04/018497; US 7057026; WO 91/06678; WO 07/123744; US 7329492; US 7211414; US 7315019; US 7405281 и US 2008/0108082, каждый из которых включен в настоящее описание посредством ссылки.
Используемый в рамках изобретения термин "ампликон" в случае применения в отношении нуклеиновой кислоты означает продукт копирования нуклеиновой кислоты, где продукт имеет нуклеотидную последовательность, которая является такой же или комплементарной по меньшей мере части нуклеотидной последовательности нуклеиновой кислоты. Ампликон может быть получен любым из множества способов амплификации, в которых используются нуклеиновая кислота или ее ампликон в качестве матрицы, включая, например, удлинение полимеразы, полимеразную цепную реакцию (ПЦР), рамификацию (RCA), удлинение лигирования или цепную реакцию лигирования. Ампликон может представлять собой молекулу нуклеиновой кислоты, содержащую отдельную копию конкретной нуклеотидной последовательности (например, продукт ПЦР) или несколько копий нуклеотидной последовательности (например, конкатемерный продукт RCA). Первый ампликон нуклеиновой кислоты-мишени, как правило, является комплементарной копией. Последующие ампликоны являются копиями, которые создаются после получения первого ампликона из нуклеиновой кислоты-мишени или из первого ампликона. Последующий ампликон может содержать последовательность, которая фактически комплементарна нуклеиновой кислоте-мишени или фактически идентична нуклеиновой кислоте-мишени.
Используемый в рамках изобретения термин "сайт амплификации" относится к сайту в чипе или на нем, где может быть создания одного или нескольких ампликонов. Сайт амплификации можно дополнительно настроить для содержания, удержания или присоединения по меньшей мере одного ампликона, который создан в сайте.
Используемый в рамках изобретения термин "чип" относится к популяции сайтов, которые можно дифференцировать друг от друга в соответствии с относительным местоположением. Разные молекулы, которые находятся в разных местах чипа, могут отличаться друг от друга в соответствии с местоположением сайтов в чипе. Отдельный сайт чипа может включать одну или несколько молекул определенного типа. Например, сайт может включать отдельную молекулу нуклеиновой кислоты-мишени, имеющую конкретную последовательность, или сайт может включать несколько молекул нуклеиновой кислоты, имеющих одинаковую последовательность (и/или ее комплементарную последовательность). Сайты чипа могут являться разными элементами, расположенными на одном субстрате. Иллюстративные элементы включают без ограничения лунки в субстрате, микроносители (или другие частицы) в субстрате или на нем, выступы на субстрате, края на субстрате или каналы в субстрате. Сайты чипа могут представлять собой отдельные субстраты, каждый из которых содержит различную молекулу. Различные молекулы, прикрепленные к отдельным субстратам, можно идентифицировать в соответствии с расположением субстратов на поверхности, с которой связаны субстраты, или в соответствии с расположением субстратов в жидкости или геле. Иллюстративные чипы, в которых отдельные субстраты расположены на поверхности, включают без ограничения чипы, имеющие микроносители в лунках.
Используемый в рамках изобретения термин "емкость" при использовании в отношении сайта и материала нуклеиновой кислоты означает максимальное количество материала нуклеиновой кислоты, которое может временно занимать сайт. Например, термин может относиться к общему количеству молекул нуклеиновой кислоты, которые могут временно занимать сайт в конкретном состоянии. Также могут применяться другие меры, включая, например, общую массу материала нуклеиновой кислоты или общее количество копий конкретной нуклеотидной последовательности, которая может временно занимать сайт в конкретном состоянии. Как правило, емкость сайта для нуклеиновой кислоты-мишени будет фактически эквивалентна емкости сайта для ампликонов нуклеиновой кислоты-мишени.
Используемый в рамках изобретения термин "средство для захвата" относится к материалу, химическому веществу, молекуле или их фрагменту, которые способны присоединяться, удерживаться или связываться с молекулой-мишенью (например, нуклеиновой кислотой-мишенью). Иллюстративные средства для захвата включают без ограничения нуклеиновую кислоту для захвата (также называемую в рамках изобретения как олигонуклеотид для захвата), которая комплементарна по меньшей мере части нуклеиновой кислоты-мишени, член пары связывания рецептор-лиганд (например, авидин, стрептавидин, биотин, лектин, углевод, белок, связывающий нуклеиновую кислоту, эпитоп, антитело и т.д.), способный связываться с нуклеиновой кислотой-мишенью (или связывающим фрагментом, присоединенным к ней), или химический реагент, способный образовывать ковалентную связь с нуклеиновой кислотой-мишенью (или связывающим фрагментом, присоединенным к ней).
Используемый в рамках изобретения термин "клональная популяция" относится к популяции нуклеиновых кислот, которая является гомогенной по отношению к конкретной нуклеотидной последовательности. Гомогенная последовательность, как правило, имеет длину, составляющую по меньшей мере 10 нуклеотидов, однако может быть даже более длинной, в том числе, например, длину, составляющую по меньшей мере 50, 100, 250, 500 или 1000 нуклеотидов. Клональную популяцию можно получить из отдельной нуклеиновой кислоты-мишени или матричной нуклеиновой кислоты. Как правило, все нуклеиновые кислоты в клональной популяции будут иметь одинаковую нуклеотидную последовательность. Понятно, что небольшое количество мутаций (например, из-за артефактов амплификации) может происходить в клональной популяции без отклонения от клональности.
Используемый в рамках изобретения термин "получение" в контексте композиции, изделия, нуклеиновой кислоты или ядра означает создание композиции, изделия, нуклеиновой кислоты или ядра, приобретение композиции, изделия, нуклеиновой кислоты или ядра или иным образом получение соединения, композиции, изделия или ядра.
Термин "и/или" означает один или все из перечисленных элементов или комбинацию любых двух или более из перечисленных элементов.
Слова "предпочтительный" и "предпочтительно" относятся к вариантам осуществления изобретения, которые могут давать определенные преимущества при определенных обстоятельствах. Однако другие варианты осуществления также могут быть предпочтительными при таких же или других обстоятельствах. Более того, изложение одного или нескольких предпочтительных вариантов осуществления не подразумевает, что другие варианты осуществления не являются применимыми, и не предназначено для исключения других вариантов осуществления из объема настоящего изобретения.
Термины "содержит" и их варианты не имеют ограничивающего значения, когда эти термины появляются в описании и формуле изобретения.
Понятно, что везде, где варианты осуществления описаны в рамках изобретения с помощью понятий "включать", "включает" или "включающий" и т.п., также предусматриваются в остальном аналогичные варианты осуществления, описанные терминами "состоящий из" и/или "состоящий практически из".
Если не указано иное, то "один" и "по меньшей мере один" используются взаимозаменяемо и означают один или более чем один.
Также в рамках изобретения изложение числовых диапазонов по конечным точкам включает все числа, включенные в этот диапазон (например, от 1 до 5 включает 1, 1,5, 2, 2,75, 3, 3,80, 4, 5 и т.д.).
Для любого способа, раскрытого в рамках изобретения, который включает отдельные стадии, стадии могут проводиться в любом возможном порядке. И, в случае необходимости, любая комбинация из двух или более стадий может выполняться одновременно.
Ссылка по всему объему настоящего изобретения на "один вариант осуществления", "вариант осуществления", "определенные варианты осуществления" или "некоторые варианты осуществления" и т.д. означает, что конкретный элемент, конфигурация, композиция или характеристика, описанные в связи с вариантом осуществления, включены в по меньшей мере один вариант осуществления изобретения. Таким образом, появление таких фраз в различных местах по всему объему настоящего изобретения не обязательно относится к одному и тому же варианту осуществления настоящего изобретения. Более того, конкретные элементы, конфигурации, композиции или характеристики могут быть объединены любым подходящим способом в одном или нескольких вариантах осуществления.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Последующее подробное описание иллюстративных вариантов осуществления настоящего изобретения может быть лучше понято при чтении вместе со следующими графическими материалами.
На ФИГ. 1 показана общая блок-схема общего иллюстративного способа комбинаторного индексирования отдельных клеток в соответствии с настоящим изобретением.
На ФИГ. 2 показано схематическое изображение одного из вариантов осуществления способа комбинаторного индексирования отдельных клеток, в целом показанного на ФИГ. 1.
На ФИГ. 3 показано схематическое изображение иллюстративного варианта осуществления молекулы фрагмент-адаптер после линейной амплификации.
На ФИГ. 4 показано схематическое изображение иллюстративного варианта осуществления молекулы фрагмент-адаптер после добавления универсальных адаптеров.
Схематические изображения не обязательно выполнены в масштабе. Одинаковые номера, используемые на фигурах, относятся к одинаковым компонентам, стадиям и т.п. Однако понятно, что использование номера для ссылки на компонент на определенной фигуре не предназначено для ограничения компонента на другой фигуре, обозначенной тем же номером. Кроме того, использование разных номеров для обозначения компонентов не предназначено для указания того, что разные пронумерованные компоненты не могут быть такими же или похожими на другие пронумерованные компоненты.
ПОДРОБНОЕ ОПИСАНИЕ ИЛЛЮСТРАТИВНЫХ ВАРИАНТОВ ОСУЩЕСТВЛЕНИЯ
Предусматриваемый в рамках изобретения способ включает получение изолированных ядер из совокупности клеток (ФИГ. 1, блок 12). Клетки могут быть из любого организма(-ов) и из любого типа клеток или любой ткани организма(-ов). Способ может дополнительно включать диссоциацию клеток (ФИГ. 2, блок i) и/или выделение ядер (ФИГ. 2, блок ii). Способы выделения ядер из клеток известны специалисту в данной области и являются обычными. Количество ядер может составлять по меньшей мере 2. Верхний предел зависит от практических ограничений оборудования (например, многолуночных планшетов), применяемых на других стадиях способа, как описано в рамках изобретения. Например, в одном варианте осуществления число ядер может составлять не более чем 1 000 000 000, не более чем 100 000 000, не более чем 10 000 000, не более чем 1 000 000, не более чем 10 000 или не более чем 1000. Специалист поймет, что молекулы нуклеиновой кислоты в каждом ядре представляют собой всю генетическую систему комплемента организма и являются молекулами геномной ДНК, которые включают как интронные, так и экзонные последовательности, а также некодирующие регуляторные последовательности, такие как последовательности промотора и энхансера.
В одном варианте осуществления ядра включают нуклеосомы, связанные с геномной ДНК. Такие ядра могут применяться в способах, которые не определяют последовательность ДНК всего генома клетки, таких как sciATAC-seq. В другом варианте осуществления изолированные ядра подвергаются условиям, которые обедняют ядра нуклеосом с получением обедненных нуклеосомами ядер (ФИГ. 1, блок 13, и ФИГ. 2, блок ii). Такие ядра могут применяться в способах, направленных на определение всей последовательности геномной ДНК клетки. В одном варианте осуществления условия, применяемые для обеднения нуклеосом, поддерживают целостность выделенных ядер. Способы получения обедненных нуклеосомами ядер известны специалисту (см., например, Vitak et al., 2017, Nature Methods, 14 (3): 302-308). В одном варианте осуществления условия представляют собой химическую обработку, которая включает обработку хаотропным средством, способным нарушать взаимодействия нуклеиновой кислоты с белком. Пример применяемого хаотропного средства включает без ограничения дийодсалицилат лития. В другом варианте осуществления условия представляют собой химическую обработку, которая включает обработку детергентом, способным нарушать взаимодействия нуклеиновой кислоты с белком. Пример полезного детергента включает без ограничения додецилсульфат натрия (SDS). В некоторых вариантах осуществления, в случае если применяется детергент, такой как SDS, клетки, из которых выделены ядра, обрабатывают сшивающим средством перед выделением. Применяемый пример сшивающего средства включает без ограничения формальдегид.
Предусмотренный в рамках изобретения способ включает распределение субпопуляции ядер, таких как обедненные нуклеосомами ядра, в первую совокупность компартментов (ФИГ. 1, блок 14, и ФИГ. 2, левая схема). Число ядер, присутствующих в субпопуляции, а следовательно в каждом компартменте, может составлять по меньшей мере 1. В одном варианте осуществления число ядер, присутствующих в субпопуляции, составляет не более чем 2000. Способы распределения ядер на субпопуляции известны специалисту в данной области и являются обычными. Примеры включают без ограничения сортировку ядер с активированной флуоресценцией (FANS).
Каждый компартмент включает транспосомный комплекс. Транспосомный комплекс, транспозаза, связанная с сайтом распознавания транспозазы, способен вставить сайт распознавания транспозазы в нуклеиновую кислоту-мишень внутри ядра в способе, который иногда называют "тагментацией". В некоторых таких событиях вставки одна нить сайта распознавания транспозазы может быть перенесена в нуклеиновую кислоту-мишень. Такая нить называется "перенесенной нитью". В одном варианте осуществления транспосомный комплекс включает димерную транспозазу, содержащую две субъединицы и две несмежные транспозонные последовательности. В другом варианте осуществления транспозаза включает димерную транспозазу, имеющую две субъединицы, и смежную транспозонную последовательность.
Некоторые варианты осуществления могут включать применение гиперактивной транспозазы Tn5 и сайта распознавания транспозазы типа Tn5 (Goryshin and Reznikoff, J. Biol. Chem., 273:7367 (1998)) или транспозазы MuA и сайт распознавания транспозазы Mu, содержащих концевые последовательности R1 и R2 (Mizuuchi, K., Cell, 35: 785, 1983; Savilahti, H, et al., EMBO J., 14: 4893, 1995). Согласно оптимизации, специалистом в данной области также можно применять мозаичные концевые (ME) последовательности Tn5.
Дополнительные примеры систем транспозиции, которые можно применять с определенными вариантами осуществления композиций и способов, представленных в рамках изобретения, включают Staphylococcus aureus Tn552 (Colegio et al., J. Bacteriol., 183: 2384-8, 2001; Kirby C et al., Mol. Microbiol., 43: 173-86, 2002), Ty1 (Devine & Boeke, Nucleic Acids Res., 22: 3765-72, 1994 и международную публикацию WO 95/23875), транспозон Tn7 (Craig, N L, Science. 271: 1512, 1996; Craig, N L, обзор в: Curr Top Microbiol Immunol., 204:27-48, 1996), Tn/O и IS10 (Kleckner N, et al., Curr Top Microbiol Immunol., 204:49-82, 1996), транспозаза Mariner (Lampe D J, et al., EMBO J., 15: 5470-9, 1996), Tc1 (Plasterk R H, Curr. Topics Microbiol. Immunol., 204: 125-43, 1996), элемент P (Gloor, G B, Methods Mol. Biol., 260: 97-114, 2004), Tn3 (Ichikawa & Ohtsubo, J Biol. Chem. 265:18829-32, 1990), бактериальные инсерционные последовательности (Ohtsubo & Sekine, Curr. Top. Microbiol. Immunol. 204: 1-26, 1996), ретровирусы (Brown, et al., Proc Natl Acad Sci USA, 86:2525-9, 1989), и ретротранспозон дрожжей (Boeke & Corces, Annu Rev Microbiol. 43:403-34, 1989). Дополнительные примеры включают IS5, Tn10, Tn903, IS911 и сконструированные версии ферментов семейства транспозаз (Zhang et al., (2009) PLoS Genet. 5:e1000689. Epub 2009 Oct 16; Wilson C. et al (2007) J. Microbiol. Methods 71:332-5).
Другие примеры интеграз, которые можно применять со способами и композициями, предусмотренными в рамках изобретения, включают ретровирусные интегразы и последовательности распознавания интеграз для таких ретровирусных интеграз, как например, интегразы из ВИЧ-1, ВИЧ-2, SIV, PFV-1, RSV.
Транспозонные последовательности, применяемые в описанных в рамках изобретения способах и композициях, представлены в публикации заявки на патент США № 2012/0208705, публикации заявки на патент США № 2012/0208724 и публикации международной заявки на патент № WO 2012/061832. В некоторых вариантах осуществления транспозонная последовательность включает первый сайт распознавания транспозазы, второй сайт распознавания транспозазы и индекс, присутствующий между двумя сайтами распознавания транспозазы.
Некоторые транспосомные комплексы, применяемые в рамках изобретения, включают транспозазу, содержащую две транспозонные последовательности. В некоторых таких вариантах осуществления две транспозонные последовательности не связаны друг с другом, иными словами транспозонные последовательности являются несмежными друг с другом. Примеры таких транспосом известны из уровня техники (см., например, публикации заявки на патент США № 2010/0120098).
В некоторых вариантах осуществления транспосомный комплекс включает в себя нуклеиновую кислоту транспозонной последовательности, которая связывает две субъединицы транспозазы с образованием "петлевого комплекса" или "петлевой транспосомы". В одном примере транспосома включает димерную транспозазу и транспозонную последовательность. Петлевые комплексы могут обеспечивать возможность того, что транспозоны будут вставлены в ДНК-мишень, сохраняя при этом информацию об упорядочении исходной ДНК-мишени и без фрагментации ДНК-мишени. Понятно, что петлевые структуры могут вставлять требуемые последовательности нуклеиновой кислоты, такие как индексы, в нуклеиновую кислоту-мишень, сохраняя при этом физическую связность нуклеиновой кислоты-мишени. В некоторых вариантах осуществления транспозонная последовательность петлевого транспосомного комплекса может включать в себя сайт фрагментации, так что транспозонную последовательность можно фрагментировать для создания транспосомного комплекса, содержащего две транспозонные последовательности. Такие транспосомные комплексы используются для обеспечения того, чтобы соседние фрагменты ДНК-мишени, в которые встраиваются транспозоны, получали кодовые комбинации, которые можно однозначно собрать на более поздней стадии анализа.
Транспосомный комплекс также включает по меньшей мере одну индексную последовательность, также называемую индексом транспозазы. Индексная последовательность присутствует как часть транспозонной последовательности. В одном варианте осуществления индексная последовательность может присутствовать на перенесенной нити, нити сайта распознавания транспозазы, которая переносится в нуклеиновую кислоту-мишень. Индексная последовательность, также называемая меткой или штрих-кодом, применяется в качестве маркерной характеристики компартмента, в котором присутствовала конкретная нуклеиновая кислота-мишень. Индексная последовательность транспосомного комплекса различна для каждого компартмента. Соответственно, в данном варианте осуществления индекс представляет собой метку последовательности нуклеиновой кислоты, которая прикреплена к каждой из нуклеиновых кислот-мишеней, присутствующих в конкретном компартменте, присутствие которых указывает на идентификацию компартмента или применяется для таковой, в котором присутствовала популяция ядер на данной стадии способа.
Индексная последовательность может иметь длину до 20 нуклеотидов, например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20. Четырехнуклеотидная метка дает возможность мультиплексировать 256 образцов в одном чипе, а метка с шестью основами дает возможность обрабатывать 4096 образцов в одном чипе.
В одном варианте осуществления перенесенная нить также может включать универсальную последовательность, последовательность первого праймера для секвенирования или их комбинацию. Универсальные последовательности и последовательности праймеров для секвенирования описаны в рамках изобретения. Таким образом, в некоторых вариантах осуществления, где перенесенная нить переносится в нуклеиновые кислоты-мишени, нуклеиновые кислоты-мишени включают индекс транспозазы, а также включают в себя универсальную последовательность, последовательность первого праймера для секвенирования или их комбинацию.
В одном варианте осуществления цитозиновые нуклеотиды перенесенной нити метилированы. В другом варианте осуществления нуклеотиды перенесенной нити не содержат цитозин. Такая перенесенная нить и любая последовательность, присутствующая на перенесенной нити, включая индексную последовательность транспозазы, универсальную последовательность и/или последовательность первого праймера для секвенирования, могут называться обедненными цитозином. Использование обедненных цитозином нуклеотидных последовательностей в транспосомном комплексе не оказывает существенного влияния на эффективность транспозазы.
Способ также включает получение индексированных ядер (ФИГ. 1, блок 15, и ФИГ. 2, блок iii). В одном варианте осуществления получение индексированных ядер включает фрагментирование нуклеиновых кислот, присутствующих в субпопуляциях обедненных нуклеосомами ядер (например, нуклеиновых кислот, присутствующих в каждом компартменте), в совокупность фрагментов нуклеиновых кислот. В одном варианте осуществления фрагментация нуклеиновых кислот осуществляется с использованием сайта фрагментации, присутствующего в нуклеиновых кислотах. Как правило, сайты фрагментации встраивают в нуклеиновые кислоты-мишени с использованием транспосомного комплекса. Например, петлевой транспосомный комплекс может включать сайт фрагментации. Сайт фрагментации может использоваться для расщепления физической, но не информационной ассоциации между индексными последовательностями, которые были встроены в нуклеиновую кислоту-мишень. Расщепление может осуществляться биохимическим, химическим или другими способами. В некоторых вариантах осуществления сайт фрагментации может включать нуклеотид или нуклеотидную последовательность, которая может быть фрагментирована различными способами. Примеры сайтов фрагментации включают без ограничения сайт рестрикционной эндонуклеазы, по меньшей мере один рибонуклеотид, расщепляемый с помощью РНКазы, аналоги нуклеотидов, расщепляемые в присутствии определенного химического средства, диоловое сцепление, расщепляемое обработкой периодатом, дисульфидную группу, расщепляемую с химическим восстановителем, расщепляемый фрагмент, который может подвергаться фотохимическому расщеплению, и пептид, расщепляемый ферментом пептидазой или другими подходящими способами (см., например, публикацию заявки на патент США № 2012/0208705, публикации заявки на патент США №№ 2012/0208724 и WO 2012/061832. Результатом фрагментации является популяция индексированных ядер, каждое из которых содержит фрагменты нуклеиновой кислоты, где фрагменты нуклеиновой кислоты включают по меньшей мере однонитевую индексную последовательность, что указывает на конкретный компартмент.
Индексированные ядра из множества компартментов могут быть объединены (ФИГ. 1, блок 16, и ФИГ. 2, схема слева). Например, индексированные ядра от 2 до 96 компартментов (в случае использования 96-луночного планшета) или от 2 до 384 компартментов (в случае использования 384-луночного планшета) объединяют. Субпопуляции этих объединенных в пул индексированных ядер, называемых в рамках изобретения объединенными в пул индексированными ядрами, затем распределяются во вторую совокупность компартментов. Количество ядер, присутствующих в субпопуляции, а следовательно, в каждом компартменте, частично основано на требовании уменьшить столкновения индексов, к которому приводит присутствие двух ядер с одинаковым индексом транспозазы в одном и том же компартменте на данной стадии способа. Количество ядер, присутствующих в субпопуляции в данном варианте осуществления, может составлять от 2 до 30, например, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30. В одном варианте осуществления число ядер, присутствующих в субпопуляции, составляет от 20 до 24, например 22. Способы распределения ядер на субпопуляции известны специалисту в данной области и являются обычными. Примеры включают без ограничения сортировку ядер с активированной флуоресценцией (FANS).
Распределенные индексированные ядра обрабатывают для идентификации метилированных нуклеотидов (ФИГ. 1, блок 17, и ФИГ. 2, блок iv). Метилирование сайтов, таких как динуклеотидные последовательности CpG, можно измерить с использованием любого из ряда методов, применяемых в данной области для анализа таких сайтов. Одним применяемым способом является идентификация метилированных динуклеотидных последовательностей CpG. Идентификацию метилированных динуклеотидных последовательностей CpG определяют с использованием технологий, основанных на превращении цитозина, которые основаны на зависимой от статуса метилирования химической модификации последовательностей CpG в выделенной геномной ДНК или ее фрагментах с последующим анализом последовательности ДНК. Химические реагенты, которые способны различать метилированные и неметилированные динуклеотидные последовательности CpG, включают гидразин, который расщепляет нуклеиновую кислоту, и бисульфит. Обработка бисульфитом с последующим щелочным гидролизом, в частности, превращает неметилированный цитозин в урацил, оставляя 5-метилцитозин немодифицированным, как описано в Olek A., 1996, Nucleic Acids Res. 24:5064-6 или в Frommer et al., 1992, Proc. Natl. Acad. Sci. USA 89:1827-1831. Обработанную бисульфитом ДНК впоследствии можно анализировать молекулярными методами, такими как ПЦР-амплификация, секвенирование и выявление, включая гибридизацию олигонуклеотидов (например, с использованием микрочипов нуклеиновых кислот). В одном варианте осуществления индексированные ядра в каждом компартменте подвергаются воздействию условий для обработки бисульфитом. Обработка нуклеиновых кислот бисульфитом известна специалисту в данной области и является обычной. В одном варианте осуществления обработка бисульфитом превращает неметилированные остатки цитозина динуклеотидов CpG в остатки урацила и оставляет остатки 5-метилцитозина неизмененными. Обработка бисульфитом приводит к получению обработанных бисульфитом фрагментов нуклеиновых кислот.
После получения обработанных бисульфитом фрагментов нуклеиновых кислот, фрагменты модифицируют для включения дополнительных нуклеотидов на одном или обоих концах (ФИГ. 1, блок 18, и ФИГ. 2, блоки v и vi). В одном варианте осуществления модификация включает подвергание обработанных бисульфитом фрагментов нуклеиновых кислот, линейной амплификации с использованием совокупности праймеров. Каждый праймер включает по меньшей мере две области; универсальную нуклеотидную последовательность на 5-'конце и случайную нуклеотидную последовательность на 3'-конце. Универсальная нуклеотидная последовательность идентична в каждом праймере, и в одном варианте осуществления она включает вторую последовательность праймера для секвенирования (также называемую праймером Read 2 на ФИГ. 2 (блок vii). Область случайной нуклеотидной последовательности применяется так, чтобы присутствовал по меньшей мере один праймер, комплементарный каждой последовательности в обработанных бисульфитом фрагментах нуклеиновой кислоты. Количество случайных нуклеотидов, которые можно применять для увеличения вероятности полного охвата до требуемого уровня, может быть определено с помощью обычных способов и может составлять от 6 до 12 случайных нуклеотидов, например 9 случайных нуклеотидов. В одном варианте осуществления количество циклов ограничено не более чем 10 циклами, например, 9 циклов, 8 циклов, 7 циклов, 6 циклов, 5 циклов, 4 цикла, 3 цикла, 2 цикла или 1 цикл. Результатом линейной амплификации являются амплифицированные молекулы фрагмент-адаптер. Пример молекулы фрагмент-адаптер показан на ФИГ. 3. Молекула фрагмент-адаптер 30 включает в себя нуклеотиды, происходящие из перенесенной нити транспосомного комплекса 31 и 32, который включает индекс транспозазы и универсальную последовательность, которую можно применять для амплификации и/или секвенирования. Молекула фрагмент-адаптер также включает нуклеотиды, происходящие из геномной ДНК ядра 33, области случайной нуклеотидной последовательности 34 и универсальной нуклеотидной последовательности 35.
За линейной амплификацией следует реакция экспоненциальной амплификации, такая как ПЦР, для дополнительной модификации концов молекулы фрагмент-адаптер перед иммобилизацией и секвенированием. Эта стадия приводит к индексации молекул фрагмент-адаптер с помощью ПЦР (ФИГ. 1, блок 19). Универсальные последовательности 31, 32 и/или 35, присутствующие на концах молекулы фрагмент-адаптер, можно использовать для связывания универсальных якорных последовательностей, которые могут служить в качестве праймеров и удлиняться в реакции амплификации. Как правило, используют два разных праймера. Один праймер гибридизуется с универсальными последовательностями на 3'-конце одной нити молекулы фрагмент-адаптер, а второй праймер гибридизуется с универсальными последовательностями на 3'-конце другой нити молекулы фрагмент-адаптер. Таким образом, якорная последовательность каждого праймера может быть разной. Каждый из подходящих праймеров может включать в себя дополнительные универсальные последовательности, такие как универсальная последовательность для захвата и другая индексная последовательность. Поскольку каждый праймер может включать индекс, то эта стадия приводит к добавлению одной или двух индексных последовательностей, например, второго и необязательного третьего индекса. Молекулы фрагмент-адаптор, имеющие второй и необязательный третий индексы, называют молекулами фрагмент-адаптер с двойным индексом. Второй и третий индексы могут представляет собой последовательности, обратно комплементарные друг другу, или второй и третий индексы могут иметь последовательности, которые не являются обратно комплементарными друг другу. Эта вторая индексная последовательность и необязательный третий индекс уникальны для каждого компартмента, в которые были помещены распределенные индексированные ядра перед обработкой бисульфитом натрия. Результатом этой ПЦР-амплификации является совокупность или библиотека молекул фрагмент-адаптер, имеющих структуру, подобную или идентичную молекуле фрагмент-адаптер, показанной на ФИГ. 2, блок vii.
В другом варианте осуществления модификация включает подвергание обработанных бисульфитом фрагментов нуклеиновых кислот, условиям, которые приводят к лигированию дополнительных последовательностей с обоими концами фрагментов. В одном варианте осуществления можно применять лигирование тупых концов. В другом варианте осуществления фрагменты получают с отдельными выступающими нуклеотидами, например, с помощью активности определенных типов ДНК-полимеразы, такой как Taq-полимераза или полимераза Кленова exo minus, которая обладает не матричнозависимой терминальной трансферазной активностью, которая добавляет отдельный дезоксинуклеотид, например дезоксиаденозин (A), к 3'-концам обработанных бисульфитом фрагментов нуклеиновых кислот. Такие ферменты можно применять для добавления отдельного нуклеотида "А" к тупому 3'-концу каждой нити фрагментов. Таким образом, "A" можно добавить к 3'-концу каждой нити двухнитевых фрагментов-мишеней путем реакции с Taq или полимеразой Кленова exo minus, в то время как дополнительные последовательности, которые должны быть добавлены к каждому концу фрагмента, могут включать совместимый "липкий" конец "Т", присутствующий на 3'-конце каждой области двухнитевой нуклеиновой кислоты, подлежащей добавлению. Эта концевая модификация также предотвращает самолигирование нуклеиновых кислот, чтобы произошел сдвиг в сторону образования обработанных бисульфитом фрагментов нуклеиновых кислот, фланкированных последовательностями, которые добавляются в данном варианте осуществления.
Фрагментация молекул нуклеиновой кислоты описанными в рамках изобретения способами приводит к образованию фрагментов с гетерогенной смесью тупых и 3'- и 5'-"липких" концов. Поэтому желательно репарировать концы фрагментов с использованием способов или наборов (таких как набор для репарации конца терминатора ДНК Lucigen), известных из уровня техники, с получением концов, которые являются оптимальными для инсерции, например, в тупые сайты векторов для клонирования. В конкретном варианте осуществления концы фрагментов популяции нуклеиновых кислот являются тупыми. Более конкретно, концы фрагментов являются тупыми и фосфорилированными. Фосфатный фрагмент можно ввести посредством ферментативной обработки, например, с использованием полинуклеотидкиназы.
В одном варианте осуществления обработанные бисульфитом фрагменты нуклеиновых кислот обрабатывают сначала с помощью лигирования идентичных универсальных адаптеров (также называемых "ошибочно спаренными адапторами", общие характеристики которых описаны в Gormley et al., US 7741463 и Bignell et al., US 8053192,) к 5'- и 3'-концам обработанных бисульфитом фрагментов нуклеиновых кислот, с образованием молекул фрагмент-адаптер. В одном варианте осуществления универсальный адаптор включает в себя все последовательности, необходимые для секвенирования, включая иммобилизацию молекул фрагмент-адаптер на чипе. Поскольку нуклеиновые кислоты, подлежащие секвенированию, происходят из отдельных клеток, то дополнительная амплификация молекул фрагмент-адаптер способствует достижению достаточного количества молекул фрагмент-адаптер для секвенирования.
В другом варианте осуществления, в случае если универсальный адаптер не включает в себя все последовательности, необходимые для секвенирования, тогда можно использовать стадию ПЦР для дополнительной модификации универсального адаптера, присутствующего в каждой молекуле фрагмент-адаптер перед иммобилизацией и секвенированием. Например, начальную реакцию удлинения праймера осуществляют с использованием универсальной якорной последовательности, комплементарной универсальной последовательности, присутствующей в молекуле фрагмент-адаптер, в которой образуются продукты удлинения, комплементарные обеим нитям каждой отдельной молекулы фрагмент-адаптер. Как правило, ПЦР добавляет дополнительные универсальные последовательности, такие как универсальная последовательность для захвата и еще одна индексная последовательность. Поскольку каждый праймер может включать индекс, - эта стадия приводит к добавлению одной или двух индексных последовательностей, например, второго и необязательного третьего индекса, и индексации молекул фрагмент-адаптер путем лигирования адаптера (ФИГ. 1, блок 19). Полученные молекулы фрагмент-адаптор называют молекулами фрагмент-адаптер с двойным индексом.
После добавления универсальных адаптеров либо с помощью одностадийного способа лигирования универсального адаптора, включающего в себя все последовательности, необходимые для секвенирования, либо с помощью двухстадийного способа лигирования универсального адаптера с последующей ПЦР-амплификацией для дополнительной модификации универсального адаптера, окончательная молекула фрагмент-адаптер будет включать универсальную последовательность для захвата, последовательность второго индекса и последовательность необязательного третьего индекса. Эти индексы аналогичны второму и третьему индексам, описанным при получении фрагмент-адаптеров с двойным индексом посредством линейной амплификации. Второй и третий индексы могут представляет собой последовательности, обратно комплементарные друг другу, или второй и третий индексы могут иметь последовательности, которые не являются обратно комплементарными друг другу. Эти последовательности второго и необязательного третьего индекса являются уникальными для каждого компартмента, в который распределенные индексированные ядра помещали перед обработкой бисульфитом натрия. Результатом добавления универсальных адаптеров к каждому концу является совокупность или библиотека молекул фрагмент-адаптор, имеющих структуру, аналогичную или идентичную молекуле фрагмент-адаптор 40, показанной на ФИГ. 4. Молекула фрагмент-адаптер 40 включает последовательность для захвата 41 и 48, также называемую соответственно 3'-адаптером проточной ячейки (например, P5) и 5'-адаптером проточной ячейки (например, P7'), и индекс 42 и 47, как например i5 и i7. Молекула фрагмент-адаптер 40 также включает нуклеотиды, происходящие из перенесенной нити транспосомного комплекса 43, который включает индекс транспозазы 44 и универсальную последовательность 45, которую можно применять для амплификации и/или секвенирования. Молекула фрагмент-адаптер также включает нуклеотиды, происходящие из геномной ДНК ядра 46.
Полученные молекулы фрагмент-адаптер с двойным индексом совместно обеспечивают библиотеку нуклеиновых кислот, которые можно иммобилизовать, а затем секвенировать. Термин библиотека относится к скоплению фрагментов из отдельных клеток, содержащих известные универсальные последовательности на их 3'- и 5'-концах.
После модификации обработанных бисульфитом фрагментов нуклеиновых кислот, для включения дополнительных нуклеотидов молекулы фрагмент-адаптер с двойным индексом можно подвергнуть условиям, в которых они отбираются на основе заранее определенного диапазона размеров, например, от 150 до 400 нуклеотидов в длину, например от 150 до 300 нуклеотидов. Полученные молекулы фрагмент-адаптер с двойным индексом объединяют в пул и необязательно могут подвергаться способу очистки для повышения чистоты молекул ДНК путем удаления по меньшей мере части не встроенных универсальных адаптеров или праймеров. Можно применять любой подходящий способ очистки, такой как электрофорез, эксклюзионная хроматография по размеру или т.п. В некоторых вариантах осуществления можно применять твердофазные парамагнитные микроносители с обратимой иммобилизацией для отделения требуемых молекул ДНК от неприкрепленных универсальных адаптеров или праймеров и для отбора нуклеиновых кислот в зависимости от размера. Твердофазные парамагнитные микроносители с обратимой иммобилизацией коммерчески доступны от Beckman Coulter (Agencourt AMPure XP), Thermofisher (MagJet), Omega Biotek (Mag-Bind), Promega Beads (Promega) и Kapa Biosystems (Kapa Pure Beads).
Для секвенирования можно получить совокупность молекул фрагмент-адаптер. После объединения в пул молекулы фрагмент-адаптер иммобилизуют и амплифицируют перед секвенированием (ФИГ. 1, блок 20). Способы присоединения молекул фрагмент-адаптер из одного или нескольких источников к субстрату известны из уровня техники. Аналогичным образом, способы амплификации иммобилизованных молекул фрагмент-адаптер включают без ограничения мостиковую амплификацию и кинетическое исключение. Способы иммобилизации и амплификации перед секвенированием описаны, например, в Bignell et al. (US 8053192), Gunderson et al. (WO 2016/130704), Shen et al. (US 8895249) и Pipenburg et al. (US 9309502).
Объединенный в пул образец можно иммобилизовать при подготовке к секвенированию. Секвенирование можно осуществлять в виде множества отдельных молекул или можно амплифицировать перед секвенированием. Амплификацию можно проводить с использованием одного или нескольких иммобилизованных праймеров. Иммобилизованный(-е) праймер(-ы) может(-гут) быть "газоном" на плоской поверхности или на пуле микроносителей. Пул микроносителей может быть выделен в виде эмульсии с отдельным микроносителем в каждом "компартменте" эмульсии. При концентрации только одна матрица на "компартмент" на каждом микроносителе амплифицируется только одна матрица.
Используемый в рамках изобретения термин "твердофазная амплификация" относится к любой реакции амплификации нуклеиновой кислоты, которую осуществляют на твердой подложке или совместно с ней, так что все или часть амплифицированных продуктов иммобилизуются на твердой подложке по мере их образования. В частности, данный термин охватывает твердофазную полимеразную цепную реакцию (твердофазную ПЦР) и твердофазную изотермическую амплификацию, которые являются реакциями, аналогичными стандартной жидкофазной амплификации, за исключением того, что прямой, или обратный, или оба праймера амплификации иммобилизованы на твердой подложке. Твердофазная ПЦР охватывает системы, такие как эмульсии, где один праймер прикреплен к микроносителю, а другой находится в свободном растворе, и при этом происходит образование колоний в твердофазных гелевых матрицах, где один праймер прикреплен к поверхности, а другой находится в свободном растворе.
В некоторых вариантах осуществления твердая подложка содержит узорчатую поверхность. "Узорчатая поверхность" относится к расположению различных областей в открытом слое твердой подложки или на нем. Например, одна или несколько областей могут быть элементами, где присутствуют один или несколько праймеров для амплификации. Элементы могут быть разделены промежуточными областями, где отсутствуют праймеры для амплификации. В некоторых вариантах осуществления матрица может представлять собой формат элементов x-y, представленных в виде строк и столбцов. В некоторых вариантах осуществления матрица может представлять собой повторяющееся расположение элементов и/или промежуточных областей. В некоторых вариантах осуществления матрица может представлять собой случайное расположение элементов и/или промежуточных областей. Иллюстративные узорчатые поверхности, которые можно применять в способах и композициях, изложенных в рамках изобретения, описаны в патентах США № 8778848, 8778849 и 9079148 и публикации США № 2014/0243224.
В некоторых вариантах осуществления твердая подложка включает чип из лунок или углублений на поверхности. Это может быть изготовлено, как общеизвестно из уровня техники, с использованием различных методов, включая без ограничения фотолитографию, методы штамповки, методы формования и методы микротравления. Специалистам в данной области понятно, что применяемый метод будет зависеть от композиции и формы субстрата чипа.
Элементами на узорчатой поверхности могут быть лунки в чипе из лунок (например, микролунки или нанолунки) на стекле, кремнии, пластике или других подходящих твердых подложках с узорчатым ковалентно связанным гелем, таким как поли(N-(5-азидоацетамидилпентил)акриламид-соакриламид) (PAZAM, см., например, публикации США №№ 2013/184796, WO 2016/066586 и WO 2015/002813). В способе создают гелевые прокладки, применяемые для секвенирования, которые могут быть стабильными в течение секвенирования с большим количеством циклов. Ковалентное связывание полимера с лунками помогает поддерживать гель в структурированных элементах в течение всего срока службы структурированного субстрата при различных путях применения. Однако во многих вариантах осуществления гель не обязательно должен быть ковалентно связан с лунками. Например, в некоторых условиях акриламид, не содержащий силан (SFA, см., например, патент США № 8563477, который полностью включен в настоящее описание посредством ссылки), и который ковалентно не прикреплен к какой-либо части структурированного субстрата, можно использовать в качестве гелевого материала.
В конкретных вариантах осуществления структурированный субстрат можно получить путем нанесения узора на твердую подложку с лунками (например, микролунками или нанолунками), покрывая узорчатую подложку гелевым материалом (например, PAZAM, SFA или его химически модифицированными вариантами, такими как азидолизированная версия SFA (азидо-SFA)) и полируя подложку, покрытую гелем, например, посредством химической или механической полировки, удерживая таким образом гель в лунках, но удаляя или инактивируя фактически весь гель из промежуточных областей на поверхности структурированного субстрата между лунками. Праймерные нуклеиновые кислоты можно присоединить к гелевому материалу. Затем раствор молекул фрагмент-адаптер может контактировать с полированным субстратом таким образом, что отдельные молекулы фрагмент-адаптер будут занимать отдельные лунки посредством взаимодействия с праймерами, прикрепленными к гелевому материалу; однако нуклеиновые кислоты-мишени не будут временно занимать промежуточные области из-за отсутствия или неактивности гелевого материала. Амплификация молекул фрагмент-адаптер будет ограничена лунками, поскольку отсутствие или неактивность геля в промежуточных областях предотвращает миграцию растущей колонии нуклеиновых кислот наружу. Процесс может быть удобно осуществлен, будучи масштабируемым и с использованием традиционных способов микро- или нанообработки.
Хотя изобретение охватывает "твердофазные" способы амплификации, в которых иммобилизован только один праймер для амплификации (другой праймер обычно присутствует в свободном растворе), предпочтительно, чтобы твердая подложка была обеспечена иммобилизованными праймерами, как прямым, так и обратным. На практике будет "совокупность" идентичных прямых праймеров и/или "совокупность" идентичных обратных праймеров, иммобилизованных на твердой подложке, поскольку процесс амплификации требует избытка праймеров для поддержания амплификации. Ссылки в рамках изобретения на прямые и обратные праймеры должны интерпретироваться соответственно как охватывающие "совокупность" таких праймеров, если контекст не указывает на иное.
Специалисту в данной области понятно, что в любой данной реакции амплификации требуется по меньшей мере один тип прямого праймера и по меньшей мере один тип обратного праймера, специфических по отношению к амплифицируемой матрице. Однако в некоторых вариантах осуществления прямой и обратный праймеры могут включать специфические по отношению к матрице части идентичной последовательности и могут иметь полностью идентичную нуклеотидную последовательность и структуру (включая любые ненуклеотидные модификации). Другими словами, можно осуществлять твердофазную амплификацию, применяя только один тип праймера, и такие способы с отдельным праймером включены в объем настоящего изобретения. В других вариантах осуществления могут применяться прямой и обратный праймеры, которые содержат идентичные специфические по отношению к матрице последовательности, но которые отличаются некоторыми другими структурными элементами. Например, один тип праймера может содержать ненуклеотидную модификацию, которая отсутствует в другом.
Во всех вариантах осуществления настоящего изобретения праймеры для твердофазной амплификации предпочтительно иммобилизуют путем одноточечного ковалентного присоединения к твердой подложке на 5'-конце праймера или рядом с ним, оставляя специфическую для матрицы часть праймера свободной для отжига с его родственной матрицей и 3'-гидроксильную группу свободной для удлинения праймера. Для данной цели можно применять любые подходящие способы ковалентного присоединения, известные из уровня техники. Выбранный химический состав присоединения будет зависеть от природы твердой подложки и любой применяемой к ней дериватизации или функционализации. Для способствования присоединению сам праймер может включать фрагмент, который может являться ненуклеотидной химической модификацией. В конкретном варианте осуществления праймер может включать серосодержащий нуклеофил, такой как фосфоротиоат или тиофосфат, на 5'-конце. В случае полиакриламидных гидрогелей на твердой подложке этот нуклеофил будет связываться с бромацетамидной группой, присутствующей в гидрогеле. Более конкретным способом присоединения праймеров и матриц к твердой подложке является 5'-фосфоротиоатное присоединение к гидрогелю, состоящему из полимеризованного акриламида и N-(5-бромацетамидилпентил)акриламида (BRAPA), как полностью описано в WO 05/065814.
В определенных вариантах осуществления настоящего изобретения могут применяться твердые подложки, состоящие из инертного субстрата или матрицы (например, стеклянных пластинок, полимерных микроносителей и т.д.), которые были "функционализированы", например, путем нанесения слоя или покрытия из промежуточного материала, содержащего реактивные группы, которые обеспечивают ковалентное присоединение к биомолекулам, таким как полинуклеотиды. Примеры таких подложек включают без ограничения полиакриламидные гидрогели, нанесенные на инертный субстрат, такой как стекло. В таких вариантах осуществления биомолекулы (например, полинуклеотиды) могут быть непосредственно ковалентно присоединены к промежуточному материалу (например, гидрогелю), но сам промежуточный материал может быть нековалентно присоединен к субстрату или матрице (например, стеклянному субстрату). Термин "ковалентное прикрепление к твердой подложке" следует интерпретировать соответствующим образом как охватывающий этот тип расположения.
Объединенные в пул образцы могут быть амплифицированы на микроносителях, где каждый микроноситель содержит прямой и обратный праймеры для амплификации. В конкретном варианте осуществления библиотека молекул фрагмент-адаптер применяется для получения кластерных чипов колоний нуклеиновых кислот, аналогичных тем, которые описаны в публикации США № 2005/0100900, патентах США №№ 7115400, WO 00/18957 и WO 98/44151 путем твердофазной амплификации и, более конкретно, твердофазной изотермической амплификации. Термины "кластер" и "колония" используются в рамках изобретения взаимозаменяемо для обозначения дискретного сайта на твердой подложке, включая совокупность идентичных иммобилизованных нитей нуклеиновой кислоты и совокупность идентичных иммобилизованных комплементарных нитей нуклеиновой кислоты. Термин "кластерный чип" относится к чипу, сформированному из таких кластеров или колоний. В данном контексте термин "чип" не следует понимать как требующий упорядоченного расположения кластеров.
Термин "твердая фаза" или "поверхность" используется для обозначения либо плоского чипа, где праймеры прикреплены к плоской поверхности, например, пластинки из стекла, диоксида кремния или пластмассы, или подобные устройства с проточной ячейкой; микроносители, где один или два праймера прикреплены к микроносителям, и микроносители амплифицированы; или чипа из микроносителей на поверхности после того, как сферы микроносители были амплифицированы.
Кластерные чипы можно получить с использованием либо способа термоциклирования, как описано в WO 98/44151, либо способа, где температура поддерживается как постоянная, а циклы удлинения и денатурации осуществляются с использованием смен реагентов. Такие способы изотермической амплификации описаны в патентных заявках № WO 02/46456 и публикации США № 2008/0009420. Из-за более низких температур, применяемых в изотермическом способе, это особенно предпочтительно.
Понятно, что любую из методологий амплификации, описанных данном документе или общеизвестных из уровня техники, можно использовать с универсальными или мишень-специфическими праймерами для амплификации иммобилизованных фрагментов ДНК. Подходящие способы амплификации включают без ограничения полимеразную цепную реакцию (ПЦР), амплификацию с замещением нитей (SDA), транскрипционно-опосредованную амплификацию (TMA) и амплификацию, основанную на последовательности нуклеиновых кислот (NASBA), как описано в патенте США № 8003354, полностью включенном в настоящее описание посредством ссылки. Указанные выше способы амплификации могут применяться для амплификации одной или нескольких нуклеиновых кислот, представляющих интерес. Например, ПЦР, включая мультиплексную ПЦР, SDA, TMA, NASBA и т.п., можно использовать для амплификации иммобилизованных фрагментов ДНК. В некоторых вариантах осуществления праймеры, направленные конкретно на полинуклеотид, представляющий интерес, включены в реакцию амплификации.
Другие подходящие способы амплификации полинуклеотидов могут включать технологии удлинения и лигирования олигонуклеотидов, рамификации (RCA) (Lizardi et al., Nat. Genet. 19:225-232 (1998)) и лигирования олигонуклеотидных зондов (OLA) (см., в основном, патенты США №№ 7582420, 5185243, 5679524 и 5573907; EP 0320308 B1; EP 0336731 B1; EP 0439182 B1; WO 90/01069; WO 89/12696 и WO 89/09835). Понятно, что эти методики амплификации могут быть разработаны для амплификации иммобилизованных фрагментов ДНК. Например, в некоторых вариантах осуществления способ амплификации может включать реакции амплификации лигированных зондов или лигирования олигонуклеотидных зондов (OLA), которые содержат праймеры, направленные в частности на нуклеиновую кислоту, представляющую интерес. В некоторых вариантах осуществления способ амплификации может включать реакцию лигирования-удлинения праймера, которая включает праймеры, направленные конкретно на нуклеиновую кислоту, представляющую интерес. В качестве неограничивающего примера удлинения праймеров и праймеров лигирования, которые могут быть специально сконструированы для амплификации нуклеиновой кислоты, представляющей интерес, амплификация может предусматривать праймеры, применяемые для анализа GoldenGate (Illumina, Inc., Сан-Диего, Калифорния), как описано, например, в патентах США №№ 7582420 и 7611869.
Иллюстративные способы изотермической амплификации, которые можно применять в способе по настоящему изобретению, включают без ограничения амплификацию с множественным замещением (MDA), как описано, например, в Dean et al., Proc. Natl. Acad. Sci. USA 99:5261-66 (2002) или изотермическую амплификацию нуклеиновых кислот с замещением нитей, описанную, например, в патенте США № 6214587. Другие не основанные на ПЦР способы, которые можно применять в настоящем изобретении, включают, например, амплификацию с замещением нитей (SDA), которая описана, например, в Walker et al., Molecular Methods for Virus Detection, Academic Press, Inc., 1995; патентах США №№ 5455166 и 5130238 и в Walker et al., Nucl. Acids Res. 20:1691-96 (1992), или амплификацию с замещением гиперразветвленных нитей, которая описана, например, в Lage et al., Genome Res. 13:294-307 (2003). Способы изотермической амплификации можно применять с большим фрагментом полимеразы с нить-замещающей активностью Phi 29 или ДНК-полимеразы Bst, 5'->3' экзо- для амплификации случайных праймеров геномной ДНК. Применение этих полимераз дает преимущество их высокой технологичности и нить-замещающей активности. Высокая технологичность дает возможность полимеразам образовывать фрагменты длиной 10-20 т.о. Как указано выше, более мелкие фрагменты можно получить в изотермических условиях с использованием полимераз, характеризующихся низкой технологичностью и нить-замещающей активностью, таких как полимераза Кленова. Дополнительное описание реакций амплификации, условий и компонентов подробно изложено в раскрытии патента США № 7670810.
Другим способом амплификации полинуклеотидов, который применим в настоящем изобретении, является меченая ПЦР, в которой используется популяция двухдоменных праймеров, имеющих константную 5'-область, за которой следует случайная 3'-область, как описано, например, в Grothues et al. Nucleic Acids Res. 21(5):1321-2 (1993). Первые циклы амплификации осуществляют для обеспечения множества инициаций на денатурированной высокой температурой ДНК на основе индивидуальной гибридизации из случайно синтезированной 3'-области. Из-за природы 3'-области предполагается, что сайты инициации являются случайными по всему геному. В дальнейшем несвязанные праймеры можно удалить, и дальнейшая репликация может осуществляться с использованием праймеров, комплементарных константной 5'-области.
В некоторых вариантах осуществления изотермическую амплификацию можно осуществлять с использованием амплификации кинетического исключения (KEA), также называемой амплификацией исключения (ExAmp). Библиотека нуклеиновых кислот по настоящему изобретению может быть создана с использованием способа, который включает стадию применения реагента для амплификации для получения совокупности сайтов амплификации, каждый из которых включает фактически клональную популяцию ампликонов из отдельной нуклеиновой кислоты-мишени, которая занимает сайт. В некоторых вариантах осуществления реакция амплификации продолжается до тех пор, пока не будет получено достаточное количество ампликонов для заполнения емкости соответствующего сайта амплификации. Вследствие этого заполнение уже занятого сайта до такой емкости препятствует присоединению и амплификации нуклеиновых кислот-мишеней в сайте, что приводит таким образом к образованию клональной популяции ампликонов в сайте. В некоторых вариантах осуществления очевидную клональность можно достичь, даже если сайт амплификации не заполнен перед тем, как вторая нуклеиновая кислота-мишень достигнет сайта. В некоторых условиях амплификация первой нуклеиновой кислоты-мишени может продолжаться до того момента, когда будет сделано достаточное количество копий, чтобы эффективно обойти или превзойти получение копий из второй нуклеиновой кислоты-мишени, которая транспортируется в сайт. Например, в варианте осуществления, в котором используется способ мостиковой амплификации на кольцевом элементе диаметром менее чем 500 нм, было определено, что после 14 циклов экспоненциальной амплификации для первой нуклеиновой кислоты-мишени загрязнение второй нуклеиновой кислотой-мишенью на том же сайте приведет к недостаточному количеству загрязняющих ампликонов, чтобы неблагоприятно повлиять на анализ секвенирования путем синтеза на платформе для секвенирования Illumina.
В некоторых вариантах осуществления сайты амплификации в чипе могут быть, но не обязательно, полностью клональными. Вместо этого для некоторых путей применения отдельный сайт амплификации может быть преимущественно заселен ампликонами из первой молекулы фрагмент-адаптер, а также может иметь низкий уровень загрязняющих ампликонов из второй нуклеиновой кислоты-мишени. Чип может содержать один или несколько сайтов амплификации, которые имеют низкий уровень загрязняющих ампликонов, при условии, что уровень загрязнения не оказывает неприемлемого влияния на последующее применение чипа. Например, в случае если чип должен применяться в приложении выявления, приемлемым уровнем загрязнения будет уровень, который не влияет неприемлемым образом на сигнал-шум или разрешение метода выявления. Соответственно, очевидная клональность, как правило, будет соответствовать конкретному использованию или применению чипа, созданного способами, изложенными в рамках изобретения. Иллюстративные уровни загрязнения, которые могут быть приемлемы в отдельном сайте амплификации для конкретных применений, включают без ограничения не более 0,1%, 0,5%, 1%, 5%, 10% или 25% загрязняющих ампликонов. Чип может включать в себя один или несколько сайтов амплификации, имеющих эти иллюстративные уровни загрязняющих ампликонов. Например, до 5%, 10%, 25%, 50%, 75% или даже 100% сайтов амплификации в чипе могут иметь некоторые загрязняющие ампликоны. Понятно, что в чипе или другом скоплении сайтов по меньшей мере 50%, 75%, 80%, 85%, 90%, 95% или 99% или более сайтов могут быть клональными или внешне клональными.
В некоторых вариантах осуществления кинетическое исключение может происходить, в случае если процесс происходит с достаточно высокой скоростью, чтобы эффективно исключать другое событие или процесс из происходящего. Возьмем, к примеру, создание чипа нуклеиновых кислот, в котором сайты чипа случайным образом заняты молекулами фрагмент-адаптер из раствора, а копии молекул фрагмент-адаптер создаются в процессе амплификации, чтобы полностью заполнить каждый из занятых сайтов. В соответствии со способами кинетического исключения по настоящему изобретению способы занятия и амплификации могут протекать одновременно в условиях, когда скорость амплификации превышает скорость занятия. Ввиду этого, относительно быстрая скорость, с которой копии создаются в сайте, который был засеян первой нуклеиновой кислотой-мишенью, будет эффективно исключать занятие сайта для амплификации второй нуклеиновой кислотой. Способы амплификации кинетического исключения могут быть выполнены, как подробно описано в публикации заявки на патент США № 2013/0338042.
В кинетическом исключении может использоваться относительно медленная скорость для инициирования амплификации (например, медленная скорость создания первой копии молекулы фрагмент-адаптер) по сравнению с относительно высокой скоростью для создания последующих копий молекулы фрагмент-адаптер (или первой копии молекулы фрагмент-адаптер). В примере из предыдущего абзаца кинетическое исключение происходит из-за относительно медленной скорости занятия молекулой фрагмент-адаптер (например, относительно медленной диффузии или транспорта) по сравнению с относительно быстрой скоростью, с которой происходит амплификация для заполнения сайта копиями фрагмент-адаптер, предназначенными для занятия. В другом иллюстративном варианте осуществления кинетическое исключение может происходить из-за задержки в формировании первой копии молекулы фрагмент-адаптер, которая заняла сайт (например, замедленной или медленной активации), по сравнению с относительно быстрой скоростью, с которой создаются последующие копии для заполнения сайта. В данном примере отдельный сайт мог быть занят несколькими различными молекулами фрагмент-адаптер (например, несколько молекул фрагмент-адаптер могут присутствовать в каждом сайте перед амплификацией). Однако формирование первой копии для любой данной молекулы фрагмент-адаптер может быть активировано случайным образом, так что средняя скорость формирования первой копии является относительно низкой по сравнению со скоростью, с которой создаются последующие копии. В данном случае, хотя отдельный сайт мог быть занят несколькими различными молекулами фрагмент-адаптер, кинетическое исключение даст возможность амплифицировать только одну из этих молекул фрагмент-адаптер. Более конкретно, после активации первой молекулы фрагмент-адаптер для амплификации сайт быстро заполнится своими копиями, предотвращая тем самым создание копий второй молекулы фрагмент-адаптер на сайте.
Реагент для амплификации может включать дополнительные компоненты, которые способствуют образованию ампликона и в некоторых случаях увеличивают скорость образования ампликона. Примером является рекомбиназа. Рекомбиназа может способствовать образованию ампликона, обеспечивая возможность многократного встраивания/удлинения. Более конкретно, рекомбиназа может способствовать встраиванию молекулы фрагмент-адаптер с помощью полимеразы и удлинению праймера с помощью полимеразы с использованием молекулы фрагмент-адаптер в качестве матрицы для формирования ампликона. Этот способ можно повторять в виде цепной реакции, где ампликоны, полученные из каждого цикла встраивания/удлинения, служат матрицами в следующем цикле. Процесс может происходить быстрее, чем стандартная ПЦР, поскольку цикл денатурации (например, посредством нагревания или химической денатурации) не требуется. Таким образом, амплификацию с использованием рекомбиназы можно осуществлять изотермически. Обычно для способствования амплификации целесообразно включать АТФ или другие нуклеотиды (или в некоторых случаях их негидролизуемые аналоги) в реагент для амплификации с использованием рекомбиназы. Смесь рекомбиназы и однонитевого связывающего белка (SSB) особенно применима, поскольку SSB может дополнительно способствовать амплификации. Иллюстративные составы для амплификации с использованием рекомбиназы включают те, которые продаются коммерчески как наборы TwistAmp от TwistDx (Кембридж, Великобритания). Применяемые компоненты реагента для амплификации с использованием рекомбиназы и условия реакции изложены в US 5223414 и US 7399590.
Другим примером компонента, который можно включать в реагент для амплификации для способствования формирования ампликона и в некоторых случаях для увеличения скорости формирования ампликона, является геликаза. Геликаза может способствовать формированию ампликона путем обеспечения цепной реакции формирования ампликона. Процесс может происходить быстрее, чем стандартная ПЦР, поскольку цикл денатурации (например, посредством нагревания или химической денатурации) не требуется. Таким образом, амплификацию с использованием геликазы можно осуществить изотермически. Смесь геликазы и однонитевого связывающего белка (SSB) особенно применима, поскольку SSB может дополнительно способствовать амплификации. Иллюстративные составы для амплификации с использованием геликазы включают те, которые продаются коммерчески как наборы IsoAmp от Biohelix (Беверли, Массачусетс, США). Кроме того, примеры применимых составов, которые включают белок геликазы, описаны в US 7399590 и US 7829284, каждый из которых включен в настоящее описание посредством ссылки.
Еще одним примером компонента, который можно включить в реагент для амплификации, чтобы способствовать формированию ампликона и в некоторых случаях увеличению скорости формирования ампликона, является связывающий белок точки начала репликации.
После прикрепления молекул фрагмент-адаптер к поверхности определяют последовательность иммобилизованных и амплифицированных молекул фрагмент-адаптер. Секвенирование можно осуществлять с использованием любого подходящего метода секвенирования, и способы определения последовательности иммобилизованных и амплифицированных молекул фрагмент-адаптер, включая повторный синтез нити, известны из уровня техники и описаны, например, в Bignell et al. (US 8053192), Gunderson et al. (WO2016/130704), Shen et al. (US 8895249) и Pipenburg et al. (US 9309502).
Способы, описанные в рамках изобретения, можно применять в сочетании с различными методами секвенирования нуклеиновых кислот. Особенно применимыми методами являются те, где нуклеиновые кислоты присоединены в фиксированных местоположениях в чипе, так что их относительные положения не изменяются, и где чип многократно визуализируется. Особенно применимы варианты осуществления, в которых изображения получены в разных цветовых каналах, например, совпадающих с разными метками, применяемыми для отличения одного типа нуклеотидных оснований от другого. В некоторых вариантах осуществления способ определения нуклеотидной последовательности молекулы фрагмент-адаптер может представлять собой автоматизированный способ. Предпочтительные варианты осуществления включают методы секвенирования путем синтеза ("SBS").
Методы SBS обычно включают ферментативное удлинение растущей нити нуклеиновой кислоты посредством многократного добавления нуклеотидов к матричной нити. В традиционных способах SBS нуклеотиду-мишени может предоставляться отдельный нуклеотидный мономер в присутствии полимеразы при каждой доставке. Однако в описанных в рамках изобретения способах нуклеиновой кислоте-мишени при доставке может доставляться более чем один тип нуклеинового мономера-мишени в присутствии полимеразы.
В одном варианте осуществления нуклеотидный мономер включает закрытые нуклеиновые кислоты (LNA) или мостиковые нуклеиновые кислоты (BNA). В случае получения молекул фрагмент-адаптер с использованием одной или нескольких обедненных цитозином нуклеотидных последовательностей, например, как происходит в случае если обедненные цитозином нуклеотидные последовательности присутствуют в перенесенной нити из транспосомного комплекса, температура плавления нуклеотидного мономера, который гибридизуется с цитозин-обедненной областью, изменяется. Использование LNA или BNA в нуклеотидном мономере увеличивает прочность гибридизации между нуклеотидным мономером и последовательностью праймера для секвенирования, присутствующими на иммобилизованной молекуле фрагмент-адаптер.
SBS может использовать нуклеотидные мономеры, которые содержат терминаторный фрагмент, или те, которые не содержат терминаторных фрагментов. Способы с использованием нуклеотидных мономеров, в которых отсутствуют терминаторы, включают, например, пиросеквенирование и секвенирование с использованием γ-фосфат-меченых нуклеотидов, как изложено более подробно ниже. В способах с использованием нуклеотидных мономеров, в которых отсутствуют терминаторы, количество нуклеотидов, добавляемых в каждом цикле, обычно является переменным и зависит от последовательности матрицы и способа доставки нуклеотидов. Для методов SBS, в которых используются нуклеотидные мономеры, содержащие терминаторный фрагмент, терминатор может быть эффективно необратимым в условиях секвенирования, применяемых в случае традиционного секвенирования по Сенгеру, в котором используются дидезоксинуклеотиды, или терминатор может быть обратимым, как в случае способов секвенирования, разработанных Solexa (сейчас Illumina, Inc.).
В методах SBS могут использоваться нуклеотидные мономеры, которые содержат метку, или те, которые не содержат метки. Соответственно, события встраивания можно обнаружить на основе характеристики метки, такой как флуоресценция метки; характеристики нуклеотидного мономера, такой как молекулярная масса или заряд; побочного продукта встраивания нуклеотида, такого как высвобождение пирофосфата; или т.п. В вариантах осуществления, где два или более разных нуклеотида присутствуют в реагенте для секвенирования, разные нуклеотиды могут отличаться друг от друга, или, как альтернатива, две или более разных метки могут быть неразличимы при применяемых методах выявления. Например, разные нуклеотиды, присутствующие в реагенте для секвенирования, могут иметь разные метки, и их можно различить с использованием подходящей оптики, как описано, например, в способах секвенирования, разработанных Solexa (сейчас Illumina, Inc.).
Предпочтительные варианты осуществления включают методы пиросеквенирования. Пиросеквенирование выявляет высвобождение неорганического пирофосфата (PPi), когда конкретные нуклеотиды встраиваются в растущую нить (Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M. и Nyren, P. (1996) "Real-time DNA sequencing using detection of pyrophosphate release." Analytical Biochemistry 242(1), 84-9; Ronaghi, M. (2001) "Pyrosequencing sheds light on DNA sequencing." Genome Res. 11(1), 3-11; Ronaghi, M., Uhlen, M. и Nyren, P. (1998) "A sequencing method based on real-time pyrophosphate." Science 281(5375), 363; патенты США №№ 6210891; 6258568 и 6274320). При пиросеквенировании высвобождаемый PPi можно выявить по немедленному превращению последнего в аденозинтрифосфат (АТФ) с помощью АТФ-сульфуразы, и уровень полученной АТФ выявляется с помощью фотонов, продуцируемых люциферазой. Нуклеиновые кислоты, подлежащие секвенированию, могут присоединяться к элементам в чипе, и чип можно сфотографировать для захвата хемилюминесцентных сигналов, которые вырабатываются в результате встраивания нуклеотидов в элементы чипа. Изображение можно получить после обработки чипа определенным типом нуклеотидов (например, A, T, C или G). Изображения, полученные после добавления каждого типа нуклеотидов, будут отличаться в зависимости от того, какие элементы в чипе выявлены. Эти различия в изображении отражают различное содержание последовательностей элементов в чипе. Однако относительные местоположения каждого элемента останутся неизменными на изображениях. Изображения можно сохранить, обработать и проанализировать с использованием изложенных в рамках изобретения способов. Например, изображения, полученные после обработки чипа каждым различным типом нуклеотидов, можно обрабатывать таким же образом, как показано в рамках изобретения в качестве примера для изображений, полученных из разных каналов выявления для способов секвенирования на основе обратимых терминаторов.
В другом иллюстративном типе SBS цикличное секвенирование осуществляют путем постадийного добавления обратимых терминаторных нуклеотидов, содержащих, например, метку расщепляемого или фотоотбеливающего красителя, как описано, например, в WO 04/018497 и патенте США № 7057026, раскрытия которых включены в настоящее описание посредством ссылки. Данный подход коммерциализируется Solexa (сейчас Illumina Inc.) и также описан в WO 91/06678 и WO 07/123744. Доступность флуоресцентно-меченых терминаторов, в которых одновременно может быть обращенная терминация и расщепленная флуоресцентная метка, способствует эффективному секвенированию по типу циклической обратимой терминации (CRT). Полимеразы также можно сконструировать для эффективного встраивания и удлинения из этих модифицированных нуклеотидов.
Предпочтительно, в вариантах осуществления секвенирования на основе обратимого терминатора метки фактически не подавляют удлинение в условиях реакции SBS. Однако метки выявления можно удалить, например, путем расщепления или деградации. Изображения могут быть захвачены после встраивания меток в элементы чипа нуклеиновых кислот. В конкретных вариантах осуществления каждый цикл включает одновременную доставку четырех разных типов нуклеотидов в чип, и каждый тип нуклеотидов имеет спектрально отличную метку. Затем можно получить четыре изображения, в каждом из которых используется канал выявления, который является избирательным для одной из четырех разных меток. Как альтернатива, различные типы нуклеотидов можно добавить последовательно, и изображение чипа можно получить между каждой стадией добавления. В таких вариантах осуществления каждое изображение будет демонстрировать признаки нуклеиновой кислоты, которые встраивают нуклеотиды конкретного типа. Различные функции будут присутствовать или отсутствовать на разных изображениях из-за разного содержания последовательности каждого элемента. Однако относительное положение элементов останется неизменным на изображениях. Изображения, полученные с помощью таких способов по типу обратного терминатора-SBS, можно сохранить, обработать и проанализировать, как изложено в рамках изобретения. После стадии захвата изображения метки могут быть удалены и обратимые терминаторы могут быть удалены для последующих циклов добавления и выявления нуклеотидов. Удаление меток после их выявления в определенном цикле и перед последующим циклом может обеспечить преимущество уменьшения фонового сигнала и перекрестных помех между циклами. Примеры применяемых меток и способов удаления изложены ниже.
В конкретных вариантах осуществления некоторые или все нуклеотидные мономеры могут включать обратимые терминаторы. В таких вариантах осуществления обратимые терминаторы/расщепляемые флуорофоры могут включать флуорофоры, связанные с рибозным фрагментом через 3'-сложноэфирную связь (Metzker, Genome Res. 15:1767-1776 (2005)). Другие подходы отделяют химическую реакцию терминатора от расщепления флуоресцентной метки (Ruparel et al., Proc Natl Acad Sci USA 102: 5932-7 (2005)). Ruparel et al. описали разработку обратимых терминаторов, которые применяли небольшую 3'-аллильную группу для блокирования удлинения, но их можно было легко разблокировать краткосрочной обработкой палладиевым катализатором. Флуорофор прикрепляли к основанию с помощью фоторасщепляемого линкера, который легко расщеплялся 30-секундным воздействием длинноволнового УФ-излучения. Таким образом, в качестве расщепляемого линкера можно использовать либо восстановление дисульфида, либо фоторазщепление. Другим подходом к обратимой терминации является применение естественной терминации, которая следует после размещения объемного красителя на dNTP. Наличие заряженного объемного красителя на dNTP может действовать как эффективный терминатор через стерическую и/или электростатическую помехи. Наличие одного события встраивания предотвращает дальнейшие встраивания, пока краситель не удален. Расщепление красителя удаляет флуорофор и эффективно обращает терминацию. Примеры модифицированных нуклеотидов также описаны в патентах США №№ 7427673 и 7057026, раскрытия которых полностью включены в настоящее описание посредством ссылки.
Дополнительные иллюстративные системы и способы SBS, которые можно применять со способами и системами, описанными в рамках изобретения, описаны в публикациях США. №№ 2007/0166705, 2006/0188901, 2006/0240439, 2006/0281109, 2012/0270305 и 2013/0260372, патенте США № 7057026, публикации согласно PCT № WO 05/065814, публикации заявки на патент США № 2005/0100900 и публикации согласно PCT №№ WO 06/064199 и WO 07/010251.
В некоторых вариантах осуществления может применяться выявление четырех разных нуклеотидов с использованием менее чем четырех разных меток. Например, SBS можно выполнять с использованием способов и систем, описанных во включенных материалах публикации США № 2013/0079232. В качестве первого примера пара нуклеотидных типов может быть выявлена на одной и той же длине волны, но различаться на основании разницы в интенсивности для одного члена пары по сравнению с другим или на основании изменения одного члена пары (например, посредством химической модификации, фотохимической модификации или физической модификации), которая вызывает появление или исчезновение видимого сигнала по сравнению с сигналом, выявленным для другого члена пары. В качестве второго примера три из четырех различных типов нуклеотидов можно выявить при определенных условиях, в то время как у четвертого типа нуклеотидов отсутствует метка, которую можно выявить в этих условиях, или минимально выявить в этих условиях (например, минимальное выявление из-за фоновой флуоресценции и т.д.) Встраивание первых трех типов нуклеотидов в нуклеиновую кислоту можно определить на основании наличия их соответствующих сигналов, а встраивание четвертого типа нуклеотидов в нуклеиновую кислоту можно определить на основании отсутствия или минимального выявления какого-либо сигнала. В качестве третьего примера, один тип нуклеотидов может включать метку(-и), которая(-ые) выявляется(-ются) в двух разных каналах, тогда как другие типы нуклеотидов выявляются не более чем в одном из каналов. Вышеупомянутые три иллюстративные конфигурации не считаются взаимоисключающими и могут применяться в различных комбинациях. Иллюстративный вариант осуществления, который объединяет все три примера, представляет собой способ SBS на основе флуоресценции, в котором применяется первый тип нуклеотидов, который выявляется в первом канале (например, dATP, содержащий метку, которая выявляется в первом канале, когда возбуждается первой длиной волны возбуждения), второй тип нуклеотидов, который выявляется во втором канале (например, dCTP, содержащий метку, которая выявляется во втором канале при возбуждении второй длиной волны возбуждения), третий тип нуклеотидов, который выявляется как в первом, так и во втором канале (например, dTTP, содержащий по меньшей мере одну метку, которая выявляется в обоих каналах при возбуждении первой и/или второй длиной волны возбуждения), и четвертый тип нуклеотида, в котором отсутствует метка, который не выявляется или минимально выявляется в любом канале (например, dGTP, не содержащий метки).
Дополнительно, как описано во включенных материалах публикации США № 2013/0079232, данные секвенирования могут быть получены с использованием отдельного канала. В таких так называемых подходах секвенирования с одним красителем первый тип нуклеотида метят, однако метку удаляют после получения первого изображения, а второй тип нуклеотида метят только после получения первого изображения. Третий тип нуклеотидов сохраняет свою метку как на первом, так и на втором изображениях, а четвертый тип нуклеотидов остается немеченым на обоих изображениях.
В некоторых вариантах осуществления может применяться секвенирование методами лигирования. В таких методах ДНК-лигаза используется для встраивания олигонуклеотидов и идентификации встраивания таких олигонуклеотидов. Олигонуклеотиды обычно содержат разные метки, которые коррелируют с идентичностью конкретного нуклеотида в последовательности, с которой гибридизуются олигонуклеотиды. Как и в случае других методов SBS, изображения можно получить после обработки чипа с элементами нуклеиновых кислот мечеными реагентами для секвенирования. Каждое изображение будет демонстрировать элементы нуклеиновых кислот, которые содержат встроенные метки конкретного типа. Различные элементы будут присутствовать или отсутствовать на разных изображениях из-за разного содержания последовательности каждого элемента, однако относительное положение элементов останется неизменным на изображениях. Изображения, полученные с помощью способов секвенирования на основе лигирования, можно сохранить, обработать и проанализировать, как изложено в рамках изобретения. Иллюстративные системы и способы SBS, которые можно использовать со способами и системами, описанными в рамках изобретения, описаны в патентах США №№ 6969488, 6172218 и 6306597.
В некоторых вариантах осуществления может применяться нанопоровое секвенирование (Deamer, D. W. & Akeson, M. "Nanopores and nucleic acids: prospects for ultrarapid sequencing." Trends Biotechnol. 18, 147-151 (2000); Deamer, D. и D. Branton, "Characterization of nucleic acids by nanopore analysis", Acc. Chem. Res. 35:817-825 (2002); Li, J., M. Gershow, D. Stein, E. Brandin, и J. A. Golovchenko, "DNA molecules and configurations in a solid-state nanopore microscope" Nat. Mater. 2:611-615 (2003)). В таких вариантах осуществления молекула фрагмент-адаптер проходит через нанопоры. Нанопора может представлять собой синтетическую пору или биологический мембранный белок, такой как α-гемолизин. Когда молекула фрагмент-адаптер проходит через нанопору, каждую пару оснований можно идентифицировать, измеряя флуктуации электропроводимости поры. (Патент США № 7001792; Soni, G. V. & Meller, "A. Progress toward ultrafast DNA sequencing using solid-state nanopores." Clin. Chem. 53, 1996-2001 (2007); Healy, K. "Nanopore-based single-molecule DNA analysis." Nanomed. 2, 459-481 (2007); Cockroft, S. L., Chu, J., Amorin, M. & Ghadiri, M. R. "A single-molecule nanopore device detects DNA polymerase activity with single-nucleotide resolution." J. Am. Chem. Soc. 130, 818-820 (2008), раскрытия которых полностью включены в настоящее описание посредством ссылки). Данные, полученные в результате нанопорового секвенирования, можно сохранить, обработать и проанализировать, как изложено в рамках изобретения. В частности, данные можно обработать в виде изображения в соответствии с иллюстративной обработкой оптических изображений и других изображений, которая изложена в рамках изобретения.
В некоторых вариантах осуществления могут применяться способы, включающие мониторинг активности ДНК-полимеразы в реальном времени. Встраивания нуклеотидов можно выявить через взаимодействия флуоресцентного резонансного переноса энергии (FRET) между флуорофорсодержащей полимеразой и γ-фосфат-мечеными нуклеотидами, как описано, например, в патентах США №№ 7329492 и 7211414, оба их которых включены в настоящее описание посредством ссылки, или встраивания нуклеотидов можно выявить с помощью волноводов с нулевой модой, как описано, например, в патенте США № 7315019, и с использованием флуоресцентных аналогов нуклеотидов и сконструированных полимераз, как описано, например, в патенте США № 7405281 и публикации США № 2008/0108082. Освещение может быть ограничено объемом в масштабе зептолитра вокруг поверхностно-связанной полимеразы, так что встраивание флуоресцентно меченых нуклеотидов может наблюдаться при низком уровне фона (Levene, M.J. et al. "Zero-mode waveguides for single-molecule analysis at high concentrations." Science 299, 682-686 (2003); Lundquist, P. M. et al. "Parallel confocal detection of single molecules in real time." Opt. Lett. 33, 1026-1028 (2008); Korlach, J. et al. "Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nano structures." Proc. Natl. Acad. Sci. USA 105, 1176-1181 (2008)). Изображения, полученные такими способами, можно сохранить, обработать и проанализировать, как изложено в рамках изобретения.
Некоторые варианты осуществления SBS включают выявление протона, высвобождаемого при встраивании нуклеотида в продукт удлинения. Например, для секвенирования, основанного на обнаружении высвобожденных протонов, можно применять электрический детектор и связанные с ним методы, которые коммерчески доступны от Ion Torrent (Guilford, CT, дочерняя компания Life Technologies), или в способах и системах секвенирования, описанных в публикациях США №№ 2009/0026082; 2009/0127589; 2010/0137143 и 2010/0282617. Способы, изложенные в рамках изобретения для амплификации нуклеиновых кислот-мишеней с использованием кинетического исключения, могут быть легко применены к субстратам, применяемым для выявления протонов. Более конкретно, способы, изложенные в рамках изобретения, можно применять для получения клональных популяций ампликонов, которые используются для выявления протонов.
Вышеуказанные способы SBS можно выгодно реализовать в мультиплексных форматах, так что одновременно манипулируют множеством различных молекул фрагмент-адаптер. В конкретных вариантах осуществления разные молекулы фрагмент-адаптер можно обработать в общем реакционном сосуде или на поверхности конкретного субстрата. Это дает возможность удобно доставлять реагенты для секвенирования, удалять непрореагировавшие реагенты и выявлять события встраивания мультиплексным образом. В вариантах осуществления с использованием поверхностно-связанных нуклеиновых кислот-мишеней молекулы фрагмент-адаптер могут иметь формат чипа. В формате чипа молекулы фрагмент-адаптер, как правило, могут быть связаны с поверхностью пространственно различимым образом. Молекулы фрагмент-адаптер могут быть связаны прямым ковалентным присоединением, присоединением к микроносителю или другой частице или связыванием с полимеразой или другой молекулой, которая прикреплена к поверхности. Чип может включать отдельную копию молекулы фрагмент-адаптер на каждом сайте (также называемую элементом), или множество копий с одинаковой последовательностью могут присутствовать на каждом сайте или элементе. Множество копий можно получить с помощью способов амплификации, таких как мостиковая амплификация или эмульсионная ПЦР, как описано более подробно ниже.
В способах, изложенных в рамках изобретения, могут использоваться чипы, содержащие элементы при любой из ряда плотностей, включая, например, по меньшей мере приблизительно 10 элементов/см2, 100 элементов/см2, 500 элементов/см2, 1000 элементов/см2, 5000 элементов/см2, 10000 элементов/см2, 50000 элементов/см2, 100000 элементов/см2, 1000000 элементов/см2, 5000000 элементов/см2 или выше.
Преимущество способов, изложенных в рамках изобретения, заключается в том, что они предусматривают быстрое и эффективное выявление совокупности см2 одновременно. Соответственно, настоящее изобретение предусматривает интегрированные системы, способные получать и выявлять нуклеиновые кислоты с использованием методов, известных из уровня техники, таких как те, примеры которых приведены выше. Таким образом, интегрированная система по настоящему изобретению может включать струйные компоненты, способные доставлять реагенты для амплификации и/или реагенты для секвенирования к одному или нескольким иммобилизованным фрагментам ДНК, при этом система включает такие компоненты, как насосы, клапаны, резервуары, струйные линии и т.п. Проточную ячейку можно сконфигурировать и/или использовать в интегрированной системе для выявления нуклеиновых кислот-мишеней. Иллюстративные проточные ячейки описаны, например, в публикации США № 2010/0111768 и документе США под серийным № 13/273666. В соответствии с примерами для проточных ячеек один или несколько струйных компонентов интегрированной системы можно применять для способа амплификации и для способа выявления. Принимая вариант секвенирования нуклеиновой кислоты в качестве примера, один или несколько струйных компонентов интегрированной системы можно использовать для способа амплификации, изложенного в рамках изобретения, и для доставки реагентов для секвенирования в способе секвенирования, таком как те, что приведены выше в качестве примера. Как альтернатива, интегрированная система может включать отдельные струйные системы для осуществления способов амплификации и для осуществления способов выявления. Примеры интегрированных систем секвенирования, которые способны создавать амплифицированные нуклеиновые кислоты, а также выявлять последовательность нуклеиновых кислот, включают без ограничения платформу MiSeqTM (Illumina, Inc., Сан-Диего, Калифорния, США) и устройства, описанные в документе США под серийным № 13/273666, включенном в настоящее описание посредством ссылки.
Во время практического осуществления способов, описанных в рамках изобретения, могут быть получены различные композиции. Например, может быть получена молекула фрагмент-адаптер с двойным индексом, включая молекулу фрагмент-адаптер с двойным индексом, имеющую структуру, показанную на ФИГ. 2 блок vii или ФИГ. 4, и композиции, включающие молекулу фрагмент-адаптер с двойным индексом. Может быть получена библиотека секвенирования молекул фрагмент-адаптер с двойным индексом, включая молекулы фрагмент-адаптер с двойным индексом, имеющие структуру, показанную на ФИГ. 2 блок vii или ФИГ. 4, и композиции, включающие библиотеку секвенирования. Такая библиотека секвенирования может быть связана с чипом.
Настоящее изобретение проиллюстрировано следующими примерами. Понятно, что конкретные примеры, материалы, количества и процедуры должны интерпретироваться в широком смысле в соответствии с объемом и сущностью настоящего изобретения, как изложено в рамках изобретения.
Примеры
Реагенты, используемые в примерах
Фосфатно-солевой буферный раствор (PBS, Thermo Fisher, кат. № 10010023)
· 0,25% трипсин (Thermo Fisher, кат. № 15050057)
· Трис (Fisher, кат. № T1503)
· HCl (Fisher, кат. № A144)
· NaCl (Fisher, кат. № M-11624)
· MgCl2 (Sigma, кат. № M8226)
· Igepal® CA-630 (Sigma, I8896)
· Ингибиторы протеаз (Roche, кат. № 11873580001)
· ddH2O PCR-Clean
· Литиевая соль 3,5-дийодсалициловой кислоты (Sigma, кат. № D3635) - только для способа LAND
· Формальдегид (Sigma, кат. № F8775) - только для способа xSDS
· Глицин (Sigma, кат. № G8898) - только для способа xSDS
· Буфер NEBuffer 2.1 (NEB, кат. № B7202) - только для способа xSDS
· SDS (Sigma, кат. № L3771) - только для способа xSDS
· Triton™ X-100 (Sigma, кат. № 9002-93-1) - только для способа xSDS
· DAPI (Thermo Fisher, кат. № D1306)
· TD буфер из набора Nextera® (Illumina, кат. № FC-121-1031)
· 96 индексированных, обедненных по содержанию цитозина транспосом (собранных с помощью опубликованных способов, последовательности показаны в таблице 1)
· 9-нуклеотидный случайный праймер (таблица 2)
· 10 мM смесь dNTP (NEB, кат. № N0447)
· (3'->5' экзо-) полимераза Кленова (Enzymatics, кат. № P7010-LC-L)
· Этанол крепостью 200 пруф-градусов
· Индексированные ПЦР-праймеры i5 и i7 PCR (таблица 3)
· Смесь Kapa HiFi™ HotStart ReadyMix
· Краситель SYBR® Green (FMC BioProducts, кат. № 50513)
· Набор для очистки продуктов ПЦР QIAquick® (Qiagen, кат. № 28104)
· Аналитический набор dsDNA High Sensitivity Qubit® (Thermo Fisher, кат. № Q32851)
· Набор для высокочувствительного биоанализатора (Agilent, кат. № 5067-4626)
· Набор для секвенирования NextSeq (высоко- или среднепроизводительный, на 150 циклов)
· Неметилированная лямбда-ДНК (Promega, кат. № D1521)
· Набор для секвенирования HiSeq® 2500 (Illumina)
· Набор для секвенирования HiSeq® X (Illumina)
· Набор для метилирования ДНК EZ-96 DNA Methylation MagPrep Kit (Zymo Research, кат. № D5040)
· Специализированные LNA-праймеры для секвенирования (таблица 4)
· Полиэтиленгликоль (PEG)
· Микроносители SPRI
Оборудование, используемое в примерах
· 35 мкМ клеточный фильтр (BD Biosciences, кат. № 352235)
· 96-луночный планшет, совместимый с магнитным держателем
· Клеточный сортер Sony SH800 (Sony Biotechnology, кат. № SH800) или другой прибор для FACS, с помощью которого можно выполнять сортировку отдельных ядер по DAPI
· Термоциклер CFX Connect RT Thermal Cycler (Bio-Rad, кат. № 1855200) или другой термоциклер для реакций в режиме реального времени
· Термомиксер
· Флюорометр Qubit® 2.0 (Thermo Fisher, кат. № Q32866)
· Биоанализатор 2100 (Agilent, кат. № G2939A)
· NextSeq® 500 (Illumina, кат. № SY-415-1001-1)
· HiSeq® 2500 (Illumina)
· HiSeq® X (Illumina)
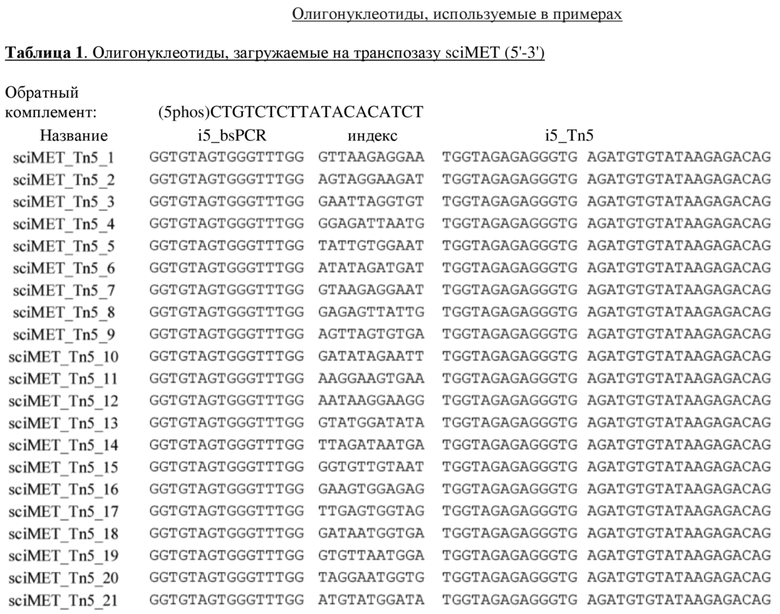





Пример 1
Получение неметилированной контрольной ДНК лямбда
Объединяли сто нанограмм неметилированной ДНК лямбда, 5 мкл 2X буфера TD, 5 мкл буфера NIB (10 мМ Трис-HCl, pH 7,4, 10 ММ NaCl, 3 мМ MgCl2, 0,1% Igepal®, 1x ингибиторы протеаз) и 4 мкл 500 нМ уникально индексированной, обедненной цитозину транспосомы. Смесь инкубировали в течение 20 минут при 55°С, а затем очищали с использованием колонки для ПЦР-очистки QIAquick® и элюировали в 30 мкл EB.
Концентрацию ДНК количественно оценивали с помощью высокочувствительного флуорометра Qubit 2.0 для dsDNA с использованием 2 мкл смеси. Концентрацию разбавляли до 17,95 пг/мкл, что имитировало геномную массу около 5 человеческих клеток.
Пример 2
Получение 18% смеси PEG с микроносителями SPRI
Микроносители Sera-Mag (1 мл) разделяли на аликвоты в 1,5 мл пробирку с низким связыванием, а затем помещали на магнитную стойку до полного осветления супернатанта. Микроносители промывали раствором 500 мкл 10 мМ Трис-HCl, рН 8,0, и раствор удаляли после осветления супернатанта, и эту стадию промывания повторяли в общей сложности четыре раза. Микроносители ресуспендировали в следующей смеси: 18% PEG 8000 (по массе), 1 М NaCl, 10 мМ Трис-HCl, рН 8,0, 1 мМ EDTA, 0,05% Tween-20, инкубированные при комнатной температуре с легким перемешиванием в течение по меньшей мере часа, а затем 18% смесь PEG с микроносителями SPRI хранили при 4°С. Перед применением микроносители оставляли до достижения комнатной температуры.
Пример 3
Получение ядер с использованием литиевой соли 3,5-дийодсалициловой кислоты (LAND) или SDS (xSDS)
A. LAND-способ получения ядер и обеднения нуклеосомами
Если клетки находились в суспензионной клеточной культуре, - культуру аккуратно растирали для разрушения агломератов клеток, клетки осаждали центрифугированием при 500 x g в течение 5 минут при 4°C и промывали 500 мкл охлажденного до температуры льда PBS.
Если клетки находились в культуре адгезивных клеток, - среду аспирировали и клетки промывали 10 мл PBS при 37°C, а затем добавляли достаточное количество 0,25% трипсина при 37°C для покрытия монослоя. После инкубации при 37°С в течение 5 минут или до тех пор, пока 90% клеток не переставали прилипать к поверхности, вносили 37°С среду в соотношении 1:1 для гашения трипсина. Клетки осаждали центрифугированием при 500 x g в течение 5 минут при 4°С, а затем промывали 500 мкл охлажденного до температуры льда PBS.
Клетки либо из суспензионной клеточной культуры, либо из культуры адгезивных клеток осаждали путем центрифугирования при 500 x g в течение 5 минут, а затем ресуспендировали в 200 мкл 12,5 мМ LIS в NIB-буфере (2,5 мкл 1 М LIS+197,5 мкл NIB-буфера). После инкубации на льду в течение 5 минут добавляли 800 мкл NIB-буфера. Клетки аккуратно пропускали через 35 мкМ клеточный фильтр и добавляли 5 мкл DAPI (5 мг/мл).
B. xSDS-способ получения ядер и обеднения нуклеосомами
Если клетки находились в суспензионной клеточной культуре, - среду аккуратно растирали для разрушения агломератов клеток. К 10 мл клеток в среде добавляли 406 мкл 37% формальдегида и инкубировали при комнатной температуре в течение 10 минут с осторожным встряхиванием. К клеткам добавляли восемьсот микролитров 2,5 М глицина и инкубировали на льду в течение 5 минут, а затем центрифугировали при 550 x g в течение 8 минут при 4°C. После промывки посредством 10 мл охлажденного до температуры льда PBS клетки ресуспендировали в 5 мл ледяного NIB (10 мМ TrisHCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2, 0,1% Igepal®, 1x ингибиторов протеаз) и инкубировали на льду в течение 20 минут с осторожным встряхиванием.
Если клетки находились в культуре адгезивных клеток, - среду аспирировали и клетки промывали 10 мл PBS при 37°C, а затем добавляли достаточное количество 0,25% трипсина при 37°C для покрытия монослоя. После инкубации при 37°С в течение 5 минут или до тех пор, пока 90% клеток не переставали прилипать к поверхности, вносили среду 37°С в соотношении 1:1 для гашения трипсина и объем доводили средой до 10 мл. Клетки ресуспендировали в 10 мл среды, и добавляли 406 мкл 37% формальдегида, и инкубировали при комнатной температуре в течение 10 минут с осторожным встряхиванием. К клеткам добавляли восемьсот микролитров 2,5 М глицина и инкубировали на льду в течение 5 минут. Клетки центрифугировали при 550 x g в течение 8 минут при 4°С и промывали посредством 10 мл охлажденного до температуры льда PBS. После ресуспендирования клеток в 5 мл охлажденного до температуры льда NIB их инкубировали на льду в течение 20 минут с осторожным встряхиванием.
Клетки или ядра либо из суспензионной клеточной культуры, либо из культуры адгезивных клеток осаждали центрифугированием при 500 x g в течение 5 минут и промывали посредством 900 мкл 1x буфера NEBuffer 2.1. После центрифугирования при 500 x g в течение 5 минут осадок ресуспендировали в 800 мкл 1x буфера NEBuffer 2.1 с 12 мкл 20% SDS и инкубировали при 42°С с интенсивным встряхиванием в течение 30 минут, а затем добавляли 200 мкл 10% Triton™ X-100 и инкубировали при 42°С с интенсивным встряхиванием в течение 30 минут. Клетки аккуратно пропускали через 35 мкМ клеточный фильтр и добавляли 5 мкл DAPI (5 мг/мл).
Пример 4
Сортировка и тагментация ядер
Планшет для тагментации получали с 10 мкл 1x TD-буфера (для 1 планшета: 500 мкл NIB-буфера+500 мкл TD-буфера) и 2500 отдельных ядер отсортировывали в каждую лунку планшета для тагментации. На этой стадии количество ядер на лунку можно слегка варьировать, пока количество ядер на лунку остается неизменным для всего планшета. Также возможно мультиплексировать разные образцы в разные лунки планшета, так как транспозазный индекс будет сохраняться. Клетки гейтировали в соответствии с фигурой 2. После центрифугирования планшета в течение 5 минут при 500 x g в каждую лунку добавляли 4 мкл 500 нМ обедненной цитозином транспосомы с уникальной индексацией. После герметизации планшет инкубировали при 55°С в течение 15 минут с осторожным встряхиванием. Затем планшет помещали на лед. Все лунки объединяли в пул, а затем пропускали через 35 мкМ клеточное сито. Добавляли пять микролитров DAPI (5 мг/мл).
Пример 5
Вторая сортировка ядер
Для каждой лунки готовили мастер-микс с 5 мкл реагента Zymo Digestion (2,5 мкл буфера M-Digestion, 2,25 мкл H2O и 0,25 мкл протеиназы K). Либо 10, либо 22 отдельных ядра отсортировывали в каждую лунку с использованием наиболее жестких параметров сортировки. Десять отдельных ядер отсортировывали в лунки для применения с неметилированными контрольными реагентами Spike-ins, а в другие лунки отсортировывали 22 клетки. Затем содержимое планшета осаждали центрифугированием при 600 x g в течение 5 минут при 4°C.
Пример 6
Расщепление и бисульфитная конверсия
Приблизительно ~35 пг (2 мкл) неметилированной контрольной ДНК лямбда, заранее обработанной C-обедненной транспосомой, применяли для внесения в лунки с 10 отдельными ядрами. Планшет инкубировали в течение 20 минут при 50°C для расщепления ядер и добавляли 32,5 мкл свежеприготовленного реагента для конверсии Zymo CT Conversion Reagent в соответствии с протоколом производителя. Содержимое лунок перемешивали растиранием и планшет центрифугировали при 600 x g в течение 2 мин. при 4°C. Перед продолжением планшет помещали на термоциклер для осуществления следующих стадий: 98°C в течение 8 минут, 64°C в течение 3,5 часа, затем выдерживали при 4°C в течение менее чем 20 часов. В каждую лунку вносили микроносители Zymo MagBinding (5 мкл), и в каждую лунку вносили 150 мкл буфера M-Binding. После перемешивания содержимого лунок с помощью растирания планшет инкубировали при комнатной температуре в течение 5 минут. Планшет помещали в 96-луночный совместимый магнитный держатель до осветления супернатанта.
Супернатант удаляли и лунки промывали свежим 80% этанолом (по объему) путем i) удаления планшета из магнитного держателя, ii) добавления в каждую лунку 100 мкл 80% этанола, покрывая осадок микроносителей, и iii) помещения планшета обратно на магнитный держатель, а затем удаления супернатанта после его осветления.
Десульфонирование осуществляли путем добавления в каждую лунку 50 мкл буфера для десульфонирования M-Desulphonation Buffer, полного ресуспендирования микроносителей растиранием, инкубирования при комнатной температуре в течение 15 минут и помещения планшета на магнитный держатель, а затем удаления супернатанта после его осветления.
Супернатант удаляли и лунки промывали свежим 80% этанолом (по объему) путем i) удаления планшета из магнитного держателя, ii) добавления в каждую лунку 100 мкл 80% этанола, покрывая осадок микроносителей, и iii) помещения планшета обратно на магнитный держатель, а затем удаления супернатанта после его осветления.
Осадкам микроносителей давали высохнуть в течение ~10 минут до тех пор, пока осадки не начинали заметно растрескиваться.
Осуществляли элюирование путем добавления в каждую лунку 25 мкл буфера для элюирования Zymo M-Elution Buffer, растирания до полной диссоциации осадка и нагревания планшета до 55°C в течение 4 минут.
Пример 7
Линейная амплификация
Весь элюат переносили в планшет, полученный со следующей реакционной смесью на лунку: 16 мкл чистой для ПЦР H2O, 5 мкл 10X буфера NEBuffer 2.1, 2 мкл 10 мМ смеси dNTP и 2 мкл 10 мкМ 9-нуклеотидного случайного праймера.
Линейную амплификацию проводили следующим образом: i) приводили ДНК в состояние однонитевой путем инкубирования при 95°C в течение 45 секунд, затем быстрого охлаждения на льду и выдерживания на льду, ii. добавляли 10 ед. полимеразы Кленова (3'->5' экзо-) в каждую лунку после полного охлаждения и iii) инкубировали планшет при 4°C в течение 5 минут, затем линейно повышали температуру со скоростью +1°C/15⋅с до 37°C, затем охлаждали при 37°C в течение 90 минут.
Стадии i-iii повторяли еще три раза в течение в общей сложности четырех раундов линейной амплификации. Для каждой амплификации в каждую лунку добавляли следующую смесь: 1 мкл 10 мкМ 9-нуклеотидного случайного праймера, 1 мкл 10 мМ dNTP-смеси и 1,25 мкл 4-кратного буфера NEBuffer 2.1. Четыре раунда линейной амплификации обычно значительно увеличивают скорость выравнивания результатов считывания и сложность библиотеки по сравнению с меньшим количеством раундов.
Лунки очищали с помощью полученной 18% смеси PEG с микроносителями SPRI в соотношении 1,1X (концентрация по объему по сравнению с объемом реакционной смеси в лунке), как описано далее. Планшет инкубировали в течение 5 минут при комнатной температуре, помещали на магнитный держатель и удаляли супернатант после его осветления. Осадки микроносителей промывали посредством 50 мкл 80% этанола. Всю оставшуюся жидкость удаляли и осадку микроносителей давали высыхать до начала растрескивания. ДНК элюировали в 21 мкл 10 мМ Трис-Cl (рН 8,5).
Пример 8
Индексирование продуктов ПЦР-реакции
Весь элюат переносили в планшет, полученный со следующей реакционной смесью на лунку: 2 мкл 10 мкМ ПЦР-праймера с индексом i7, 2 мкл 10 мкМ ПЦР-праймера с индексом i5, 25 мкл 2X смеси KAPA HiFi ™ HotStart ReadyMix и 0,5 мкл 100X SYBR® Green I. ПЦР-амплификацию осуществляли на термоциклере в режиме реального времени со следующими циклами: 95°C в течение 2 минут (94°C в течение 80 секунд, 65°C в течение 30 секунд, 72°C в течение 30 секунд), и реакцию останавливали, как только в большинстве лунок наблюдали отклонение измеряемой флуоресценции SYBR® Green. Изгиб плато наблюдали между 16-21 циклами ПЦР для препаратов библиотек.
Пример 9
Очистка и количественная оценка библиотек
Библиотеки очищали в каждой лунке с помощью 18% смеси PEG с микроносителями SPRI в соотношении 0,8X (концентрация по объему по сравнению с объемом реакционной смеси в лунке) согласно описанному далее. Планшет инкубировали в течение 5 минут при комнатной температуре, помещали на магнитный держатель и удаляли супернатант после его осветления. Осадки микроносителей промывали посредством 50 мкл 80% этанола. Всю оставшуюся жидкость удаляли и осадку микроносителей давали высыхать до начала растрескивания. ДНК элюировали в 25 мкл 10 мМ Трис-Cl (рН 8,5).
Библиотеки объединяли в пул, используя 5 мкл из каждой лунки, и 2 мкл использовали для количественного определения концентрации ДНК с помощью флуорометра dsDNA High Sensitivity Qubit® 2.0 согласно протоколу производителя. Результаты считывания Qubit® использовали для разбавления библиотеки до ~4 нг/мкл, и 1 мкл прогоняли на высокочувствительном биоанализаторе High Sensitivity Bioanalyser 2100 согласно протоколу производителя. Затем библиотеку количественно оценивали по диапазону 200 п. о. - 1 т. п. о. для разбавления пула до 1 нМ для секвенирования на Illumina.
Пример 10
Секвенирование
Настраивали NextSeq® 500 для прогона в соответствии с инструкциями производителя для 1 нМ образца, за исключением приведенных далее изменений. Пул библиотек загружали с концентрацией 0,9 пМ и общим объемом 1,5 мл и размещали в положение картриджа 10; вносили специализированные праймеры путем разбавления 9 мкл исходного 100 мкМ праймера 1 для секвенирования в общей сложности 1,5 мл буфера HT1 в положение картриджа 7 и 18 мкл каждого специализированного праймера для индексированного секвенирования при 100 мкМ исходных концентрациях до общего количества 3 мл HT1-буфера в положение картриджа 9; NextSeq® 500 осуществляли в автономном режиме; выбирали специализированный набор параметров для химических реакций SCIseq (Amini et al., 2014, Nat. Genet. 46, 1343-1349); выбирали двойной индекс; вводили соответствующее количество циклов считывания (рекомендуется 150); 10 циклов для индекса 1 и 20 циклов для индекса 2; выбирали пункт "специализированный" для всех считываний и выбирали индексы.
Полное раскрытие всех патентов, патентных заявок и публикаций, а также материалов, доступных в электронном виде (включая, например, записи нуклеотидных последовательностей, например, в GenBank и RefSeq, и записи аминокислотных последовательностей, например, в SwissProt, PIR, PRF, PDB, и транслированные последовательности из аннотированных кодирующих областей в GenBank и RefSeq), упомянутых в рамках изобретения, включены посредством ссылки во всей своей полноте. Дополнительные материалы, на которые приведены ссылки в публикациях (такие как дополнительные таблицы, дополнительные фигуры, дополнительные материалы и способы и/или дополнительные экспериментальные данные), также включены посредством ссылки во всей своей полноте. В случае если существует какое-либо несоответствие между раскрытием настоящей заявки и раскрытием(-ями) любого документа, включенного в настоящее описание посредством ссылки, раскрытие настоящей заявки будет иметь преимущественную силу. Вышеизложенное подробное описание и примеры приведены лишь для ясности понимания. Изложенную в них информацию не следует понимать как необязательные ограничения. Настоящее изобретение не ограничено конкретными показанными и описанными деталями, поскольку в настоящее изобретение будут включаться и вариации, очевидные для специалиста в данной области, которые определяются формулой изобретения.
Если не указано иное, то все числа, выражающие количества компонентов, значения молекулярной массы и т.д., используемые в описании и формуле изобретения, следует понимать как модифицированные во всех случаях термином "приблизительно". Соответственно, если не указано иное, числовые параметры, изложенные в описании и формуле изобретения, являются примерными значениями, которые могут варьировать в зависимости от требуемых свойств, которые необходимо получить с помощью настоящего изобретения. Как минимум, и не как попытка ограничить принципы эквивалентов объемом формулы изобретения, каждый числовой параметр следует рассматривать по меньшей мере в свете числа сообщаемых значащих разрядов и с использованием стандартных методов округления.
Несмотря на то, что числовые диапазоны и параметры, определяющие широкий объем настоящего изобретения, являются примерными, числовые значения, изложенные в конкретных примерах, приведены с максимально возможной точностью. Тем не менее все числовые значения по своей сути содержат диапазон, обязательно вытекающий из стандартного отклонения, обнаруживаемого при их соответствующих измерениях в ходе испытаний.
Все заголовки предназначены для удобства читателя и не должны использоваться для ограничения значения текста, идущего после заголовка, если специально не указано иное.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> OREGON HEALTH & SCIENCE UNIVERSITY
ILLUMINA, INC.
<120> ПОЛНОГЕНОМНЫЕ БИБЛИОТЕКИ ОТДЕЛЬНЫХ КЛЕТОК ДЛЯ БИСУЛЬФИТНОГО
СЕКВЕНИРОВАНИЯ
<130> IP-1584-PCT
<140> PCT/US2018/036078
<141> 2018-06-05
<150> 62/516,324
<151> 2017-06-07
<160> 132
<170> PatentIn версия 3.5
<210> 1
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 1
ctgtctctta tacacatct 19
<210> 2
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 2
ggtgtagtgg gtttgggtta agaggaatgg tagagagggt gagatgtgta taagagacag 60
<210> 3
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 3
ggtgtagtgg gtttggagta ggaagattgg tagagagggt gagatgtgta taagagacag 60
<210> 4
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 4
ggtgtagtgg gtttgggaat taggtgttgg tagagagggt gagatgtgta taagagacag 60
<210> 5
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 5
ggtgtagtgg gtttggggag attaatgtgg tagagagggt gagatgtgta taagagacag 60
<210> 6
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 6
ggtgtagtgg gtttggtatt gtggaattgg tagagagggt gagatgtgta taagagacag 60
<210> 7
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 7
ggtgtagtgg gtttggatat agatgattgg tagagagggt gagatgtgta taagagacag 60
<210> 8
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 8
ggtgtagtgg gtttgggtaa gaggaattgg tagagagggt gagatgtgta taagagacag 60
<210> 9
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 9
ggtgtagtgg gtttgggaga gttattgtgg tagagagggt gagatgtgta taagagacag 60
<210> 10
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 10
ggtgtagtgg gtttggagtt agtgtgatgg tagagagggt gagatgtgta taagagacag 60
<210> 11
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 11
ggtgtagtgg gtttgggata tagaatttgg tagagagggt gagatgtgta taagagacag 60
<210> 12
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 12
ggtgtagtgg gtttggaagg aagtgaatgg tagagagggt gagatgtgta taagagacag 60
<210> 13
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 13
ggtgtagtgg gtttggaata aggaaggtgg tagagagggt gagatgtgta taagagacag 60
<210> 14
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 14
ggtgtagtgg gtttgggtat ggatatatgg tagagagggt gagatgtgta taagagacag 60
<210> 15
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 15
ggtgtagtgg gtttggttag ataatgatgg tagagagggt gagatgtgta taagagacag 60
<210> 16
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 16
ggtgtagtgg gtttggggtg ttgtaattgg tagagagggt gagatgtgta taagagacag 60
<210> 17
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 17
ggtgtagtgg gtttgggaag tggagagtgg tagagagggt gagatgtgta taagagacag 60
<210> 18
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 18
ggtgtagtgg gtttggttga gtggtagtgg tagagagggt gagatgtgta taagagacag 60
<210> 19
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 19
ggtgtagtgg gtttgggata atggtgatgg tagagagggt gagatgtgta taagagacag 60
<210> 20
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 20
ggtgtagtgg gtttgggtgt taatggatgg tagagagggt gagatgtgta taagagacag 60
<210> 21
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 21
ggtgtagtgg gtttggtagg aatggtgtgg tagagagggt gagatgtgta taagagacag 60
<210> 22
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 22
ggtgtagtgg gtttggatgt atggatatgg tagagagggt gagatgtgta taagagacag 60
<210> 23
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 23
ggtgtagtgg gtttggtgat tgttggttgg tagagagggt gagatgtgta taagagacag 60
<210> 24
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 24
ggtgtagtgg gtttggaaga gaattattgg tagagagggt gagatgtgta taagagacag 60
<210> 25
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 25
ggtgtagtgg gtttggaatg gttggtatgg tagagagggt gagatgtgta taagagacag 60
<210> 26
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 26
ggtgtagtgg gtttggggtt aattgagtgg tagagagggt gagatgtgta taagagacag 60
<210> 27
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 27
ggtgtagtgg gtttgggtat aatagtttgg tagagagggt gagatgtgta taagagacag 60
<210> 28
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 28
ggtgtagtgg gtttggttag ttgaatttgg tagagagggt gagatgtgta taagagacag 60
<210> 29
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 29
ggtgtagtgg gtttggttgg tgaaggttgg tagagagggt gagatgtgta taagagacag 60
<210> 30
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 30
ggtgtagtgg gtttggttaa tattgaatgg tagagagggt gagatgtgta taagagacag 60
<210> 31
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 31
ggtgtagtgg gtttgggtta gaattggtgg tagagagggt gagatgtgta taagagacag 60
<210> 32
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 32
ggtgtagtgg gtttgggtta ttaattatgg tagagagggt gagatgtgta taagagacag 60
<210> 33
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 33
ggtgtagtgg gtttgggatt ggtaagatgg tagagagggt gagatgtgta taagagacag 60
<210> 34
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 34
ggtgtagtgg gtttggtgaa gtattgttgg tagagagggt gagatgtgta taagagacag 60
<210> 35
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 35
ggtgtagtgg gtttgggatg gattatgtgg tagagagggt gagatgtgta taagagacag 60
<210> 36
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 36
ggtgtagtgg gtttggatta gtatatttgg tagagagggt gagatgtgta taagagacag 60
<210> 37
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 37
ggtgtagtgg gtttgggtag gtgtggttgg tagagagggt gagatgtgta taagagacag 60
<210> 38
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 38
ggtgtagtgg gtttggagtt gaatgtatgg tagagagggt gagatgtgta taagagacag 60
<210> 39
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 39
ggtgtagtgg gtttggattg tgagatatgg tagagagggt gagatgtgta taagagacag 60
<210> 40
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 40
ggtgtagtgg gtttggttgt ggtgagttgg tagagagggt gagatgtgta taagagacag 60
<210> 41
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 41
ggtgtagtgg gtttggttaa gttggtttgg tagagagggt gagatgtgta taagagacag 60
<210> 42
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 42
ggtgtagtgg gtttggtata ataatattgg tagagagggt gagatgtgta taagagacag 60
<210> 43
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 43
ggtgtagtgg gtttggaagg tatgagttgg tagagagggt gagatgtgta taagagacag 60
<210> 44
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 44
ggtgtagtgg gtttggagga ttataagtgg tagagagggt gagatgtgta taagagacag 60
<210> 45
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 45
ggtgtagtgg gtttggagag ttaggtttgg tagagagggt gagatgtgta taagagacag 60
<210> 46
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 46
ggtgtagtgg gtttggatgg atagtattgg tagagagggt gagatgtgta taagagacag 60
<210> 47
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 47
ggtgtagtgg gtttggatat tatgttgtgg tagagagggt gagatgtgta taagagacag 60
<210> 48
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 48
ggtgtagtgg gtttggggtg gagatagtgg tagagagggt gagatgtgta taagagacag 60
<210> 49
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 49
ggtgtagtgg gtttggtggt ggtagtgtgg tagagagggt gagatgtgta taagagacag 60
<210> 50
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 50
ggtgtagtgg gtttggaggt gagaagttgg tagagagggt gagatgtgta taagagacag 60
<210> 51
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 51
ggtgtagtgg gtttggtagg aggttgttgg tagagagggt gagatgtgta taagagacag 60
<210> 52
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 52
ggtgtagtgg gtttggtgta taggtattgg tagagagggt gagatgtgta taagagacag 60
<210> 53
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 53
ggtgtagtgg gtttggtgtt atgtagatgg tagagagggt gagatgtgta taagagacag 60
<210> 54
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 54
ggtgtagtgg gtttggtgga aggtatgtgg tagagagggt gagatgtgta taagagacag 60
<210> 55
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 55
ggtgtagtgg gtttggaatg taaggagtgg tagagagggt gagatgtgta taagagacag 60
<210> 56
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 56
ggtgtagtgg gtttgggtta tgttaagtgg tagagagggt gagatgtgta taagagacag 60
<210> 57
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 57
ggtgtagtgg gtttggtgtt ataggtgtgg tagagagggt gagatgtgta taagagacag 60
<210> 58
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 58
ggtgtagtgg gtttggaagg agaattgtgg tagagagggt gagatgtgta taagagacag 60
<210> 59
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 59
ggtgtagtgg gtttggagag gtggaagtgg tagagagggt gagatgtgta taagagacag 60
<210> 60
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 60
ggtgtagtgg gtttgggatt aggtgtatgg tagagagggt gagatgtgta taagagacag 60
<210> 61
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 61
ggtgtagtgg gtttggatta tataagatgg tagagagggt gagatgtgta taagagacag 60
<210> 62
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 62
ggtgtagtgg gtttgggaga atatggttgg tagagagggt gagatgtgta taagagacag 60
<210> 63
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 63
ggtgtagtgg gtttggggat tgagaggtgg tagagagggt gagatgtgta taagagacag 60
<210> 64
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 64
ggtgtagtgg gtttggatta tggtggttgg tagagagggt gagatgtgta taagagacag 60
<210> 65
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 65
ggtgtagtgg gtttgggaag gaagttatgg tagagagggt gagatgtgta taagagacag 60
<210> 66
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 66
ggtgtagtgg gtttgggaat atgtaagtgg tagagagggt gagatgtgta taagagacag 60
<210> 67
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 67
ggtgtagtgg gtttggtagt taatatttgg tagagagggt gagatgtgta taagagacag 60
<210> 68
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 68
ggtgtagtgg gtttggtgaa tgaatagtgg tagagagggt gagatgtgta taagagacag 60
<210> 69
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 69
ggtgtagtgg gtttggagga tggattatgg tagagagggt gagatgtgta taagagacag 60
<210> 70
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 70
ggtgtagtgg gtttggaagt gtatagatgg tagagagggt gagatgtgta taagagacag 60
<210> 71
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 71
ggtgtagtgg gtttgggagg ttgaagatgg tagagagggt gagatgtgta taagagacag 60
<210> 72
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 72
ggtgtagtgg gtttggtgtg taataggtgg tagagagggt gagatgtgta taagagacag 60
<210> 73
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 73
ggtgtagtgg gtttggttga ttagagatgg tagagagggt gagatgtgta taagagacag 60
<210> 74
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 74
ggtgtagtgg gtttggtatg tgtgtggtgg tagagagggt gagatgtgta taagagacag 60
<210> 75
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 75
ggtgtagtgg gtttgggaga tgagaattgg tagagagggt gagatgtgta taagagacag 60
<210> 76
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 76
ggtgtagtgg gtttggtggt gaagtgatgg tagagagggt gagatgtgta taagagacag 60
<210> 77
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 77
ggtgtagtgg gtttgggtgg taggatgtgg tagagagggt gagatgtgta taagagacag 60
<210> 78
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 78
ggtgtagtgg gtttggtgta ggtgatatgg tagagagggt gagatgtgta taagagacag 60
<210> 79
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 79
ggtgtagtgg gtttgggtaa ggtgtgatgg tagagagggt gagatgtgta taagagacag 60
<210> 80
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 80
ggtgtagtgg gtttggagaa gagagtgtgg tagagagggt gagatgtgta taagagacag 60
<210> 81
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 81
ggtgtagtgg gtttggggat gttgtattgg tagagagggt gagatgtgta taagagacag 60
<210> 82
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 82
ggtgtagtgg gtttggaagt tatataatgg tagagagggt gagatgtgta taagagacag 60
<210> 83
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 83
ggtgtagtgg gtttggtgga attaagttgg tagagagggt gagatgtgta taagagacag 60
<210> 84
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 84
ggtgtagtgg gtttggtaat gagaggatgg tagagagggt gagatgtgta taagagacag 60
<210> 85
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 85
ggtgtagtgg gtttggataa ttgatggtgg tagagagggt gagatgtgta taagagacag 60
<210> 86
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 86
ggtgtagtgg gtttggtgtg aagagtatgg tagagagggt gagatgtgta taagagacag 60
<210> 87
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 87
ggtgtagtgg gtttgggatg aatatgttgg tagagagggt gagatgtgta taagagacag 60
<210> 88
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 88
ggtgtagtgg gtttggtgag gatagattgg tagagagggt gagatgtgta taagagacag 60
<210> 89
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 89
ggtgtagtgg gtttggatta attagagtgg tagagagggt gagatgtgta taagagacag 60
<210> 90
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 90
ggtgtagtgg gtttggggag agatggatgg tagagagggt gagatgtgta taagagacag 60
<210> 91
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 91
ggtgtagtgg gtttggtaat tgaggaatgg tagagagggt gagatgtgta taagagacag 60
<210> 92
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 92
ggtgtagtgg gtttggttgg aattaattgg tagagagggt gagatgtgta taagagacag 60
<210> 93
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 93
ggtgtagtgg gtttggaatg ttattgttgg tagagagggt gagatgtgta taagagacag 60
<210> 94
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 94
ggtgtagtgg gtttgggtag ttattagtgg tagagagggt gagatgtgta taagagacag 60
<210> 95
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 95
ggtgtagtgg gtttggtata ttgtgagtgg tagagagggt gagatgtgta taagagacag 60
<210> 96
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 96
ggtgtagtgg gtttgggtgt aggatagtgg tagagagggt gagatgtgta taagagacag 60
<210> 97
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
олигонуклеотид"
<400> 97
ggtgtagtgg gtttggagag aagttggtgg tagagagggt gagatgtgta taagagacag 60
<210> 98
<211> 37
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<220>
<221> модифицированное_основание
<222> (29)..(37)
<223> a, c, t, g, неизвестное или другое
<400> 98
ggagttcaga cgtgtgctct tccgatctnn nnnnnnn 37
<210> 99
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 99
caagcagaag acggcatacg agatcaagat gccggtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 100
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 100
caagcagaag acggcatacg agataacgtc tagtgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 101
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 101
caagcagaag acggcatacg agataggtat actcgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 102
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 102
caagcagaag acggcatacg agatttcata ggacgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 103
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 103
caagcagaag acggcatacg agatggaggc ctccgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 104
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 104
caagcagaag acggcatacg agatttcaat ataagtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 105
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 105
caagcagaag acggcatacg agatacgtca tatagtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 106
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 106
caagcagaag acggcatacg agatttgacc aggagtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 107
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 107
caagcagaag acggcatacg agatcggttg cgcggtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 108
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 108
caagcagaag acggcatacg agatcaagga ggtcgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 109
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 109
caagcagaag acggcatacg agatttacga tgaagtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 110
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 110
caagcagaag acggcatacg agatttgctg gcatgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 111
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 111
caagcagaag acggcatacg agataatact cttcgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 112
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 112
caagcagaag acggcatacg agatccaact aaccgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 113
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 113
caagcagaag acggcatacg agattatcct caatgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 114
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 114
caagcagaag acggcatacg agatgccgtc gcgtgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 115
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 115
caagcagaag acggcatacg agatccgctg cttcgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 116
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 116
caagcagaag acggcatacg agattgaccg aatcgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 117
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 117
caagcagaag acggcatacg agatgtctcc agaggtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 118
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 118
caagcagaag acggcatacg agataatgct agtcgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 119
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 119
caagcagaag acggcatacg agatgacgac ctgcgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 120
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 120
caagcagaag acggcatacg agatagagcc agccgtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 121
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 121
caagcagaag acggcatacg agatccaggc cgcagtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 122
<211> 68
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 122
caagcagaag acggcatacg agatcaggta tggagtgact ggagttcaga cgtgtgctct 60
tccgatct 68
<210> 123
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 123
aatgatacgg cgaccaccga gatctacacg tatcatcgag gtgtagtggg tttgg 55
<210> 124
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 124
aatgatacgg cgaccaccga gatctacacc cgcgattatg gtgtagtggg tttgg 55
<210> 125
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 125
aatgatacgg cgaccaccga gatctacaca ttcaggtacg gtgtagtggg tttgg 55
<210> 126
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 126
aatgatacgg cgaccaccga gatctacaca tggaattggg gtgtagtggg tttgg 55
<210> 127
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 127
aatgatacgg cgaccaccga gatctacacg acgaagcgtg gtgtagtggg tttgg 55
<210> 128
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 128
aatgatacgg cgaccaccga gatctacacc ttgcagtagg gtgtagtggg tttgg 55
<210> 129
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 129
aatgatacgg cgaccaccga gatctacacc ttggtaatgg gtgtagtggg tttgg 55
<210> 130
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 130
aatgatacgg cgaccaccga gatctacacc aagtcgaccg gtgtagtggg tttgg 55
<210> 131
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 131
tggtagagag ggtgagatgt gtataagaga tag 33
<210> 132
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: Синтетический
праймер"
<400> 132
ctatctctta tacacatctc accctctcta cca 33
<---

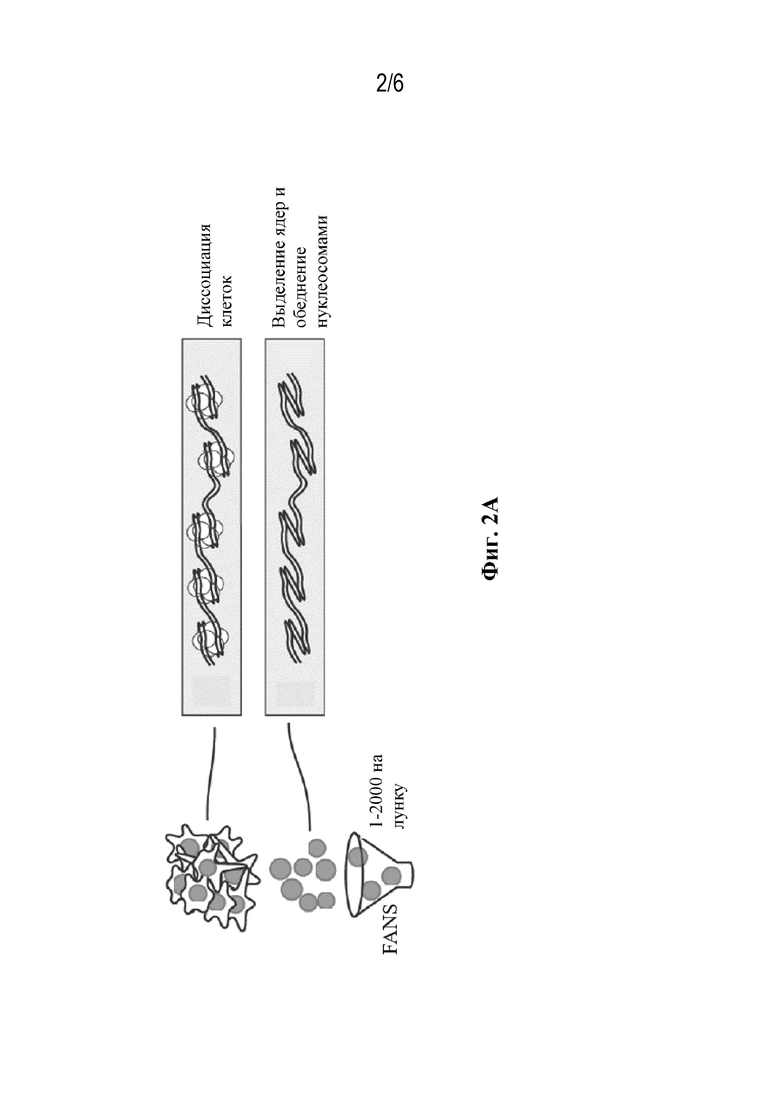




Изобретение относится к биотехнологии. Описаны способы получения библиотек для секвенирования для определения статуса метилирования нуклеиновых кислот из совокупности отдельных клеток. Способы по изобретению объединяют методики комбинаторной индексации с разделением и объединением в пул и методы обработки бисульфитом. Изобретение позволяет быстро, точно и недорогостояще определить характеристики профилей метилирования больших количеств отдельных клеток. 2 н. и 84 з.п. ф-лы, 4 ил., 4 табл., 10 пр.
1. Способ получения библиотеки для секвенирования для определения статуса метилирования нуклеиновых кислот из совокупности отдельных клеток, включающий:
(a) получение выделенных ядер из совокупности клеток;
(b) химическую обработку выделенных ядер с получением обедненных нуклеосомами ядер при сохранении целостности выделенных ядер;
(c) распределение субпопуляций обедненных нуклеосомами ядер в первую совокупность компартментов, содержащих транспосомный комплекс, где транспосомный комплекс в каждом компартменте содержит первую индексную последовательность, которая отличается от первых индексных последовательностей в других компартментах;
(d) фрагментирование нуклеиновых кислот в субпопуляциях обедненных нуклеосомами ядер с получением совокупности фрагментов нуклеиновых кислот и встраивание первых индексных последовательностей в по меньшей мере одну нить фрагментов нуклеиновых кислот с получением индексированных ядер;
(e) объединение индексированных ядер с получением объединенных в пул индексированных ядер;
(f) распределение субпопуляций объединенных в пул индексированных ядер во вторую совокупность компартментов и обработку бисульфитом индексированных ядер с получением обработанных бисульфитом фрагментов нуклеиновых кислот;
(g) амплификацию обработанных бисульфитом фрагментов нуклеиновых кислот в каждом компартменте посредством линейной амплификации с использованием совокупности праймеров, содержащих универсальную нуклеотидную последовательность на 5'-конце и случайную нуклеотидную последовательность на 3'-конце, с получением амплифицированных молекул фрагмент-адаптер;
(h) встраивание второй индексной последовательности в амплифицированные молекулы фрагмент-адаптер с получением молекул фрагмент-адаптер с двойным индексом, где вторая индексная последовательность в каждом компартменте отличается от вторых индексных последовательностей в других компартментах; и
(i) объединение молекул фрагмент-адаптер с двойным индексом с получением тем самым библиотеки для секвенирования для определения статуса метилирования нуклеиновых кислот из совокупности отдельных клеток.
2. Способ по п. 1, в котором химическая обработка включает обработку хаотропным средством, способным нарушать взаимодействия нуклеиновых кислот с белками.
3. Способ по п. 2, в котором хаотропное средство содержит дийодсалицилат лития.
4. Способ по п. 1, в котором химическая обработка включает обработку детергентом, способным нарушать взаимодействия нуклеиновых кислот с белками.
5. Способ по п. 4, в котором детергент включает додецилсульфат натрия (SDS).
6. Способ по п. 5, в котором клетки перед стадией (а) обрабатывают сшивающим средством.
7. Способ по п. 6, в котором сшивающее средство представляет собой формальдегид.
8. Способ по п. 1, в котором распределение на стадиях (с) и (f) осуществляют путем сортировки ядер с активированной флуоресценцией.
9. Способ по п. 1, в котором субпопуляции обедненных нуклеосомами ядер содержат примерно равные количества ядер.
10. Способ по п. 9, в котором субпопуляции обедненных нуклеосомами ядер содержат от 1 до приблизительно 2000 ядер.
11. Способ по п. 1, в котором первая совокупность компартментов представляет собой многолуночный планшет.
12. Способ по п. 11, в котором многолуночный планшет представляет собой 96-луночный планшет или 384-луночный планшет.
13. Способ по п. 1, в котором субпопуляции объединенных в пул индексированных ядер содержат примерно равные количества ядер.
14. Способ по п. 13, в котором субпопуляции объединенных в пул индексированных ядер содержат от 1 до приблизительно 25 ядер.
15. Способ по п. 1, в котором субпопуляции объединенных в пул индексированных ядер включают в по меньшей мере 10 раз меньшее количество ядер, чем субпопуляции обедненных нуклеосомами ядер.
16. Способ по п. 1, в котором субпопуляции объединенных в пул индексированных ядер включают в по меньшей мере 100 раз меньшее количество ядер, чем субпопуляции обедненных нуклеосомами ядер.
17. Способ по п. 1, в котором вторая совокупность компартментов представляет собой многолуночный планшет.
18. Способ по п. 17, в котором многолуночный планшет представляет собой 96-луночный планшет или 384-луночный планшет.
19. Способ по п. 1, в котором каждый из транспосомных комплексов содержит транспозазы и транспозоны, при этом каждый из транспозонов содержит перенесенную нить.
20. Способ по п. 19, в котором перенесенная нить не содержит остатка цитозина.
21. Способ по п. 20, в котором перенесенная нить содержит первую индексную последовательность.
22. Способ по п. 21, в котором перенесенная нить дополнительно содержит первую универсальную последовательность и первую последовательность праймера для секвенирования.
23. Способ по п. 1, в котором обработка бисульфитом превращает неметилированные остатки цитозина динуклеотидов CpG в остатки урацила и оставляет неизмененными остатки 5-метилцитозина.
24. Способ по п. 1, в котором линейная амплификация обработанных бисульфитом фрагментов нуклеиновых кислот предусматривает от 1 до 10 циклов.
25. Способ по п. 1, в котором универсальная нуклеотидная последовательность на 5'-конце праймеров на стадии (g) содержит вторую последовательность праймера для секвенирования.
26. Способ по п. 1, в котором случайная нуклеотидная последовательность на 3'-конце праймеров на стадии (g) состоит из 9 случайных нуклеотидов.
27. Способ по п. 1, в котором встраивание второй индексной последовательности на стадии (h) предусматривает приведение амплифицированных молекул фрагмент-адаптер в каждом компартменте в контакт с первым универсальным праймером и вторым универсальным праймером, каждый из которых содержит индексную последовательность, и осуществление реакции экспоненциальной амплификации.
28. Способ по п. 27, в котором индексная последовательность первого универсального праймера представляет собой последовательность, обратно комплементарную индексной последовательности второго универсального праймера.
29. Способ по п. 27, в котором индексная последовательность первого универсального праймера отличается от последовательности, обратно комплементарной индексной последовательности второго универсального праймера.
30. Способ по п. 27, в котором первый универсальный праймер дополнительно содержит первую последовательность для захвата и первую якорную последовательность, комплементарную универсальной последовательности на 3'-конце амплифицированных молекул фрагмент-адаптер.
31. Способ по п. 30, в котором первая последовательность для захвата содержит последовательность праймера P5.
32. Способ по п. 27, в котором второй универсальный праймер дополнительно содержит вторую последовательность для захвата и вторую якорную последовательность, комплементарную универсальной последовательности на 5'-конце амплифицированных молекул фрагмент-адаптер.
33. Способ по п. 32, в котором вторая последовательность для захвата содержит последовательность, обратно комплементарную последовательности праймера P7.
34. Способ по п. 27, в котором реакция экспоненциальной амплификации включает полимеразную цепную реакцию (ПЦР).
35. Способ по п. 34, в котором ПЦР включает от 15 до 30 циклов.
36. Способ по п. 1, дополнительно включающий обогащение нуклеиновых кислот-мишеней с помощью совокупности олигонуклеотидов для захвата, характеризующихся специфичностью по отношению к нуклеиновым кислотам-мишеням.
37. Способ по п. 36, в котором олигонуклеотиды для захвата иммобилизованы на поверхности твердого субстрата.
38. Способ по п. 36, в котором олигонуклеотиды для захвата содержат первый член универсальной пары связывания, и где второй член пары связывания иммобилизован на поверхности твердого субстрата.
39. Способ по п. 1, дополнительно включающий отбор молекул фрагмент-адаптер с двойным индексом, которые попадают в заранее определенный диапазон размеров.
40. Способ по п. 1, дополнительно включающий секвенирование молекул фрагмент-адаптер с двойным индексом для определения статуса метилирования нуклеиновых кислот из совокупности отдельных клеток.
41. Способ получения библиотеки для секвенирования для определения статуса метилирования нуклеиновых кислот из совокупности отдельных клеток, включающий:
(a) получение выделенных ядер из совокупности клеток;
(b) химическую обработку выделенных ядер с получением обедненных нуклеосомами ядер при сохранении целостности выделенных ядер;
(c) распределение субпопуляций обедненных нуклеосомами ядер в первую совокупность компартментов, содержащих транспосомный комплекс, где транспосомный комплекс в каждом компартменте содержит первую индексную последовательность, которая отличается от первых индексных последовательностей в других компартментах;
(d) фрагментирование нуклеиновых кислот в субпопуляциях обедненных нуклеосомами ядер с получением совокупности фрагментов нуклеиновых кислот и встраивание первых индексных последовательностей в по меньшей мере одну нить фрагментов нуклеиновых кислот с получением индексированных ядер;
(e) объединение индексированных ядер с получением объединенных в пул индексированных ядер;
(f) распределение субпопуляций объединенных в пул индексированных ядер во вторую совокупность компартментов и обработку бисульфитом индексированных ядер с получением обработанных бисульфитом фрагментов нуклеиновых кислот;
(g) лигирование обработанных бисульфитом фрагментов нуклеиновых кислот в каждом компартменте с универсальным адаптером с получением лигированных молекул фрагмент-адаптер;
(h) встраивание второй индексной последовательности в лигированные молекулы фрагмент-адаптер с получением молекул фрагмент-адаптер с двойным индексом, где вторая индексная последовательность в каждом компартменте отличается от вторых индексных последовательностей в других компартментах; и
(i) объединение молекул фрагмент-адаптер с двойным индексом с получением тем самым библиотеки для секвенирования для определения статуса метилирования нуклеиновых кислот из совокупности отдельных клеток.
42. Способ по п. 41, в котором химическая обработка включает обработку хаотропным средством, способным нарушать взаимодействия нуклеиновых кислот с белками.
43. Способ по п. 42, в котором хаотропное средство содержит дийодсалицилат лития.
44. Способ по п. 41, в котором химическая обработка включает обработку детергентом, способным нарушать взаимодействия нуклеиновых кислот с белками.
45. Способ по п. 44, в котором детергент включает додецилсульфат натрия (SDS).
46. Способ по п. 45, в котором клетки перед стадией (а) обрабатывают сшивающим средством.
47. Способ по п. 46, в котором сшивающее средство представляет собой формальдегид.
48. Способ по п. 41, в котором распределение на стадиях (с) и (f) осуществляют путем сортировки ядер с активированной флуоресценцией.
49. Способ по п. 41, в котором субпопуляции обедненных нуклеосомами ядер содержат примерно равные количества ядер.
50. Способ по п. 49, в котором субпопуляции обедненных нуклеосомами ядер содержат от 1 до приблизительно 2000 ядер.
51. Способ по п. 41, в котором первая совокупность компартментов представляет собой многолуночный планшет.
52. Способ по п. 51, в котором многолуночный планшет представляет собой 96-луночный планшет или 384-луночный планшет.
53. Способ по п. 41, в котором субпопуляции объединенных в пул индексированных ядер содержат примерно равные количества ядер.
54. Способ по п. 53, в котором субпопуляции объединенных в пул индексированных ядер содержат от 1 до приблизительно 25 ядер.
55. Способ по п. 41, в котором субпопуляции объединенных в пул индексированных ядер включают в по меньшей мере 10 раз меньшее количество ядер, чем субпопуляции обедненных нуклеосомами ядер.
56. Способ по п. 41, в котором субпопуляции объединенных в пул индексированных ядер включают в по меньшей мере 100 раз меньшее количество ядер, чем субпопуляции обедненных нуклеосомами ядер.
57. Способ по п. 41, в котором вторая совокупность компартментов представляет собой многолуночный планшет.
58. Способ по п. 57, в котором многолуночный планшет представляет собой 96-луночный планшет или 384-луночный планшет.
59. Способ по п. 41, в котором каждый из транспосомных комплексов содержит транспозазы и транспозоны, при этом каждый из транспозонов содержит перенесенную нить.
60. Способ по п. 59, в котором перенесенная нить не содержит остатка цитозина.
61. Способ по п. 60, в котором перенесенная нить содержит первую индексную последовательность.
62. Способ по п. 61, в котором перенесенная нить дополнительно содержит первую универсальную последовательность и первую последовательность праймера для секвенирования.
63. Способ по п. 41, в котором обработка бисульфитом превращает неметилированные остатки цитозина динуклеотидов CpG в остатки урацила и оставляет неизмененными остатки 5-метилцитозина.
64. Способ по п. 41, дополнительно включающий добавление одного или нескольких нуклеотидов к 3'-концам обработанных бисульфитом фрагментов нуклеиновых кислот для создания 3'-"липкого" конца перед лигированием универсального адаптера.
65. Способ по п. 64, в котором добавление одного или нескольких нуклеотидов осуществляют с использованием концевой трансферазы.
66. Способ по п. 64, в котором универсальный адаптер содержит "липкий" конец, который является обратно комплементарным 3'-"липкому" концу обработанных бисульфитом фрагментов нуклеиновых кислот.
67. Способ по п. 41, в котором встраивание второй индексной последовательности на стадии (h) включает приведение молекул фрагмент-адаптер с двойным индексом в каждом компартменте в контакт с первым универсальным праймером и вторым универсальным праймером, каждый из которых содержит индексную последовательность, и осуществление реакции экспоненциальной амплификации.
68. Способ по п. 67, в котором индексная последовательность первого универсального праймера представляет собой последовательность, обратно комплементарную индексной последовательности второго универсального праймера.
69. Способ по п. 67, в котором индексная последовательность первого универсального праймера отличается от последовательности, обратно комплементарной индексной последовательности второго универсального праймера.
70. Способ по п. 67, в котором первый универсальный праймер дополнительно содержит первую последовательность для захвата и первую якорную последовательность, комплементарную универсальной последовательности на 3'-конце молекул фрагмент-адаптер с двойным индексом.
71. Способ по п. 70, в котором первая последовательность для захвата содержит последовательность праймера P5.
72. Способ по п. 67, в котором второй универсальный праймер дополнительно содержит вторую последовательность для захвата и вторую якорную последовательность, комплементарную универсальной последовательности на 5'-конце молекул фрагмент-адаптер с двойным индексом.
73. Способ по п. 72, в котором вторая последовательность для захвата содержит последовательность, обратно комплементарную последовательности праймера P7.
74. Способ по п. 67, в котором реакция экспоненциальной амплификации включает полимеразную цепную реакцию (ПЦР).
75. Способ по п. 74, в котором ПЦР включает от 15 до 30 циклов.
76. Способ по п. 41, дополнительно включающий обогащение нуклеиновых кислот-мишеней с помощью совокупности олигонуклеотидов для захвата, характеризующихся специфичностью по отношению к нуклеиновым кислотам-мишеням.
77. Способ по п. 76, в котором олигонуклеотиды для захвата иммобилизованы на поверхности твердого субстрата.
78. Способ по п. 76, в котором олигонуклеотиды для захвата содержат первый член универсальной пары связывания, и где второй член пары связывания иммобилизован на поверхности твердого субстрата.
79. Способ по п. 41, дополнительно включающий отбор молекул фрагмент-адаптер с двойным индексом, которые попадают в заранее определенный диапазон размеров.
80. Способ по п. 41, дополнительно включающий секвенирование молекул фрагмент-адаптер с двойным индексом для определения статуса метилирования нуклеиновых кислот из совокупности отдельных клеток.
81. Способ по п. 40, дополнительно включающий:
получение поверхности, содержащей совокупность сайтов амплификации,
где сайты амплификации содержат по меньшей мере две популяции присоединенных однонитевых нуклеиновых кислот, имеющих свободный 3'-конец, и
приведение в контакт поверхности, содержащей сайты амплификации, с библиотекой для секвенирования в условиях, подходящих для получения совокупности сайтов амплификации, каждый из которых содержит клональную популяцию ампликонов из отдельной молекулы фрагмент-адаптер с двойным индексом.
82. Способ по п. 81, в котором количество молекул фрагмент-адаптер с двойным индексом превышает количество сайтов амплификации, где молекулы фрагмент-адаптер с двойным индексом имеют доступ посредством текучей среды к сайтам амплификации, и где каждый из сайтов амплификации характеризуется емкостью, соответствующей нескольким молекулам фрагмент-адаптер с двойным индексом в библиотеке для секвенирования.
83. Способ по п. 81, в котором приведение в контакт включает одновременно (i) транспортировку молекул фрагмент-адаптер с двойным индексом к сайтам амплификации со средней скоростью транспортировки и (ii) амплификацию молекул фрагмент-адаптер с двойным индексом, которые находятся в сайтах амплификации, со средней скоростью амплификации, где средняя скорость амплификации превышает среднюю скорость транспортировки.
84. Способ по п. 80, дополнительно включающий:
получение поверхности, содержащей совокупность сайтов амплификации,
где сайты амплификации содержат по меньшей мере две популяции присоединенных однонитевых нуклеиновых кислот, имеющих свободный 3'-конец, и
приведение в контакт поверхности, содержащей сайты амплификации, с библиотекой для секвенирования в условиях, подходящих для получения совокупности сайтов амплификации, каждый из которых содержит клональную популяцию ампликонов из отдельной молекулы фрагмент-адаптер с двойным индексом.
85. Способ по п. 84, в котором количество молекул фрагмент-адаптер с двойным индексом превышает количество сайтов амплификации, где молекулы фрагмент-адаптер с двойным индексом имеют доступ посредством текучей среды к сайтам амплификации, и где каждый из сайтов амплификации характеризуется емкостью, соответствующей нескольким молекулам фрагмент-адаптер с двойным индексом в библиотеке для секвенирования.
86. Способ по п. 84, в котором приведение в контакт включает одновременно (i) транспортировку молекул фрагмент-адаптер с двойным индексом к сайтам амплификации со средней скоростью транспортировки и (ii) амплификацию молекул фрагмент-адаптер с двойным индексом, которые находятся в сайтах амплификации, со средней скоростью амплификации, где средняя скорость амплификации превышает среднюю скорость транспортировки.
| SARAH A VITAK et al., "Sequencing thousands of single-cell genomes with combinatorial indexing", NATURE METHODS, Vol | |||
| Паровоз для отопления неспекающейся каменноугольной мелочью | 1916 |
|
SU14A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| WO 2015026853 A2, 26.02.2015 | |||
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ПОЛУЧЕНИЯ БИБЛИОТЕКИ ПОЛНОРАЗМЕРНЫХ ГЕНОВ, КОДИРУЮЩИХ ИММУНОДОМИНАНТНЫЕ БЕЛКИ Р17 (D11L), Р30 (CP204L), Р54 (E183L), Р60 (CP530R), Р72 (B646L) ВИРУСА АФРИКАНСКОЙ ЧУМЫ СВИНЕЙ | 2014 |
|
RU2571855C2 |
| WO 2015026853 A2, 26.02.2015. | |||
Авторы
Даты
2022-04-22—Публикация
2018-06-05—Подача