Область техники, к которой относится изобретение
Настоящее изобретение относится к области анализа и амплификации нуклеиновых кислот.
Уровень техники
В US 20100273219 A1 описан способ мультипраймерной амплификации для баркодирования целевых нуклеиновых кислот.
В WO 2012/134884 A1 описано баркодирование матричных нуклеиновых кислот в реакции мультиплексной амплификации.
В WO 2013038010 A2 описан способ получения амплифицированной части нуклеиновой кислоты из матрицы нуклеиновой кислоты с использованием олигонуклеотидных праймеров и стопперов для предотвращения смещения нити и считывания полимеразой, который применяется для получения частей нуклеиновой кислоты для секвенирования. Этот способ устраняет ошибки в виде предпочтения одной последовательности при амплификации нуклеиновой кислоты.
В WO 2014071361 A1 описан способ получения нуклеиновых кислот с двойным баркодом с помощью баркодированных адаптерных нуклеиновых кислот.
В US 20140274729 A1 описан способ получения библиотек кДНК с помощью ДНК-полимераз с активностью смещения нити.
В EP 3119886 B1 описан количественный способ получения нуклеиновокислотных продуктов из РНК-матрицы.
В US 2018163201 A1 раскрыт способ обратной транскрипции, в котором к 3'-концу нити кДНК добавляется C-хвост.
В WO 2016138500 A1 описан способ баркодирования нуклеиновых кислот для секвенирования. В качестве молекулярных меток используются стохастические, то есть случайные баркоды.
Молекулярные метки или уникальные молекулярные идентификаторы (UMI), также называемые молекулярными баркодами, были разработаны для идентификации ПЦР-дублей для уменьшения ошибки ПЦР выраженной в виде предпочтения определенных последовательности и для выявления редких мутаций. Присоединение уникальных молекулярных идентификаторов к молекулам РНК перед любой ПЦР-амплификацией при получении библиотеки для секвенирования обеспечивает отличительный идентификатор каждой введенной исходной молекулы. Это дает возможность устранить эффекты ошибок в виде предпочтения определенной последовательности при последующей ПЦР-амплификации, что особенно важно там, где требуется много циклов ПЦР, например, при создании библиотек секвенирования из небольших исходных количеств матрицы, как при исследовании отдельных клеток. После ПЦР предполагается, что молекулы, имеющие одну и ту же последовательность, а также один и тот же UMI, являются идентичными копиями, полученными из одной и той же исходной молекулы (Sena et al., Scientific Reports (2018) 8: 13121).
Сущность изобретения
Целью изобретения является обеспечение усовершенствованного способа создания фрагментов последовательности нуклеиновой кислоты-матрицы, который облегчает выделение и сборку данных фрагментов последовательности в объединенную последовательность, соответствующую последовательности нуклеиновой кислоты-матрицы. Желательное усовершенствование также должно уменьшить ошибки в виде предпочтение определенной последовательности при получении фрагментов и повысить охват фрагментов последовательности по всей длине матрицы, повышая правильность сборки полученной объединенной последовательности.
Соответственно, изобретением предусмотрен способ получения меченых амплификационных фрагментов нуклеиновой кислоты-матрицы, включающий стадии обеспечения такой матрицы нуклеиновой кислоты, отжига по меньшей мере одного олигонуклеотидного праймера с данной матрицей нуклеиновой кислоты, элонгации по меньшей мере одного олигонуклеотидного праймера специфичным для матрицы образом, получая при этом продукт элонгации, причем такая реакция элонгации останавливается при достижении продуктом элонгации 5'-конца матрицы нуклеиновой кислоты или ограничителя (стоппера) элонгации нуклеиновой кислоты, загибридизованного на матрице нуклеиновой кислоты в направлении 3' от продукта элонгации, обеспечения адаптерной нуклеиновой кислоты, содержащей идентификационную последовательность на своем 5'-конце, причем данная идентификационная последовательность не гибридизуется со стоппером элонгации при контакте с ним, и предпочтительно не гибридизуется с матрицей, лигирования адаптерной нуклеиновой кислоты по её 5'-концу с 3'-концом продукта элонгации, получая при этом меченый амплификационный фрагмент.
Изобретением также предусмотрен способ получения меченых амплификационных фрагментов нуклеиновой кислоты-матрицы, включающий стадии обеспечения такой нуклеиновой кислоты-матрицы, отжига по меньшей мере одного олигонуклеотидного праймера с данной нуклеиновой кислотой-матрицей, элонгации по меньшей мере одного олигонуклеотидного праймера специфичным для матрицы образом, получая при этом продукт элонгации, обеспечения адаптерной нуклеиновой кислоты, содержащей идентификационную последовательность, причем такая идентификационная последовательность не гибридизуется с матрицей, лигирования адаптерной нуклеиновой кислоты по её 5'-концу с 3'-концом продукта элонгации, получая при этом меченый амплификационный фрагмент.
Изобретением также предусмотрены наборы, подходящие для выполнения способа. Набор по изобретению может содержать по меньшей мере один олигонуклеотидный праймер, способный гибридизироваться с нуклеиновой кислотой-матрицей и запускать реакцию элонгации на своем 3'-конце, один или несколько стопперов элонгации, способных гибридизироваться с нуклеиновой кислотой-матрицей, предпочтительно способных запускать реакцию элонгации на своем 3'-конце, одну или несколько адаптерных нуклеиновых кислот, содержащих идентификационную последовательность на своем 5'-конце, причем такая идентификационная последовательность не гибридизуется со стоппером элонгации, предпочтительно при этом адаптерная нуклеиновая кислота связывается, гибридизуется или не связывается со стоппером элонгации, обратную транскриптазу и лигазу олигонуклеотидов. Различные компоненты набора могут быть представлены в разных контейнерах типа флаконов.
В нижеследующем подробном описании изложены все аспекты, включая методы и наборы, а также воплощения настоящего изобретения. Т.е. описания способов могут соответственно описывать содержимое наборов. Любые компоненты, описанные в способах, могут входить в состав наборов. Компоненты набора могут использоваться в способах по изобретению.
Раскрытие сущности изобретения
Настоящим изобретением предусмотрен способ получения меченых амплификационных фрагментов нуклеиновой кислоты-матрицы, в котором перед амплификацией этих фрагментов вводится идентификационная последовательность в качестве метки. Матрица нуклеиновой кислоты может присутствовать в нескольких копиях. Согласно изобретению, фрагментация - это процесс, который обычно происходит при амплификации, то есть на основании матрицы заданной длины создается один или несколько (обычно несколько) фрагментов при амплификации частей этой матрицы. Последовательности образовавшихся фрагментов могут перекрываться, когда разных копий матрицы одновременно образуются фрагменты, а праймеры для синтеза этих комплементарных фрагментов нуклеиновой кислоты садятся в различных местах на разных копиях матрицы. Хотя концепции изобретения работают для одного фрагмента на 1 матрицу, но предпочтительно из одной молекулы матрицы образуется много фрагментов, обычно при помощи нескольких праймеров, которые связываются с матрицей в различных местах.
Изобретение улучшает существующие способы путем связывания идентификационной последовательности с образовавшимся фрагментом. Идентификационные последовательности можно вводить с помощью праймера или после элонгации, т.е. синтеза комплементарного фрагмента нуклеиновой кислоты. Тогда идентификационная последовательность вводится путем лигирования продукта элонгации с адаптерной нуклеиновой кислотой. Вне ожидания, реакция лигирования протекает с одноцепочечными идентификационными последовательностями, т.е. части идентификационных последовательностей, содержащие негибридизованный (или “свободный”) 5'-конец, могут лигироваться с 3'-концом продукта элонгации. В реакции лигирования обычно участвует фосфатный остаток, который предпочтительно находится на 5'-конце идентификационной последовательности. Вне ожидания, не требуется, чтобы адаптерная нуклеиновая кислота в зависимости от последовательности матрицы или стоппера при гибридизации находилась поблизости от 3'-конца продукта элонгации (как показано в примерах). Хотя такое соседство и можно обеспечить путем снабжения адаптерной нуклеиновой кислоты комплементарной частью последовательности (ниже, т.е. в 3'-направлении идентификационной последовательности) для гибридизации с олигонуклеотидом, связанным с матрицей (который также именуется здесь стоппером элонгации или просто стоппером и также может служить дополнительным праймером в том случае, если образуется более одного фрагмента на 1 матрицу), однако направленная близость не требуется и может быть результатом ненаправленного простого процесса диффузии. В частности, было показано, что адаптерная нуклеиновая кислота может лигироваться с продуктом элонгации, достигшим 5'-конца матрицы нуклеиновой кислоты, где уже нет других стопперов элонгации. Такая реакция лигирования может доходить до этого конца продукта элонгации непосредственно или же после того, как полимераза добавит один или несколько нематричных нуклеотидов на основе своей терминальной трансферазной активности, которой обладают некоторые полимеразы. Такое лигирование с продуктом элонгации, который соответствует 5'-концу матрицы, имеет некоторые неожиданные и полезные преимущества: оно увеличивает количество фрагментов на 5'-конце матрицы и тем самым существенно повышает охват последовательности, чего лишены способы предшествующего уровня техники. В прежних способах распределение начальных участков фрагментов является постоянным, что приводит к высокому охвату фрагментами средней части матрицы при гораздо меньшем охвате, близком к нулю, на её 3'- и 5'-концах (что зависит от количества копий матрицы, среднего размера фрагментов и длины ридов при секвенировании). Этот эффект на 5'-конце ослабляется способом по изобретению. Кроме того, изобретением также предусмотрены воплощения для повышения охвата и на 3'-конце матрицы.
Амплифицированные фрагменты (образующиеся по одной молекуле фрагмента за 1 реакцию элонгации) обычно подвергаются дальнейшей амплификации, то есть копированию. Это значит, что амплифицируется, то есть копируется и лигированная идентификационная последовательность. Обычно идентификационные последовательности настолько разнообразны, что процесс случайного отбора позволяет однозначно идентифицировать отдельные фрагменты, которые несут одну и ту же последовательность, но происходят из разных копий одной матрицы. Во всех воплощениях изобретения идентификационная последовательность помогает определить, происходят ли копии фрагментов после секвенирования из разных копий матрицы, так как они имеют разные идентификационные последовательности, или же происходят из одной и той же молекулы матрицы и просто являются копиями, полученными при такой дальнейшей амплификации.
В следующем способе предусмотрено получение меченых амплификационных фрагментов нуклеиновой кислоты-матрицы, включающее стадии обеспечения такой нуклеиновой кислоты-матрицы, отжига по меньшей мере одного олигонуклеотидного праймера с данной матрицей нуклеиновой кислоты, элонгации по меньшей мере одного олигонуклеотидного праймера специфичным для матрицы образом, получая при этом продукт элонгации, обеспечения адаптерной нуклеиновой кислоты, содержащей идентификационную последовательность, причем данная идентификационная последовательность не гибридизуется с матрицей, лигирования адаптерной нуклеиновой кислоты предпочтительно на её 5'-конце с 3'-концом продукта элонгации, получая при этом меченый амплификационный фрагмент. Этот способ по сути такой же, что и выше, а также применимы все описанные здесь предпочтительные воплощения, только не используются стопперы. Можно использовать несколько праймеров, возможно и без стопперной функции. Адаптерные нуклеиновые кислоты все еще могут лигироваться с продуктами элонгации посредством процесса диффузии. Для лигирования продукты элонгации еще могут гибридизироваться с матрицей или в виде одной нити. Однако предпочтительно используются стопперы.
Способ по изобретению начинается со стадии обеспечения нуклеиновых кислот-матриц. Молекулы матрицы для использования в способе по изобретению должны быть доступными специалистам. Обычно матрица обеспечивается в виде образца молекул нуклеиновой кислоты. Такие нуклеиновые кислоты-матрицы могут быть выделены из клеток типа эукариотических или прокариотических клеток. В особенно предпочтительных воплощениях матрица представляет собой РНК. Может быть обеспечена тотальная РНК или фракция РНК типа мРНК либо РНК из клеток, обедненная по рРНК. Количество РНК, с которым легко работать, составляет, напр., от 0,1 пг до 500 нг, от 1 пг до 200 нг, от 10 пг до 100 нг или от 0,1 пг до 100 нг РНК, обедненной по рРНК, либо от 0,1 нг до 1000 нг общей РНК. В некоторых воплощениях количество общей РНК может составлять, напр., 10 пг, а количество РНК без рРНК может быть ниже 1 пг. Праймеры, стопперы и адаптеры предпочтительно представляют собой ДНК.
Способ также включает отжиг по меньшей мере одного олигонуклеотидного праймера с нуклеиновой кислотой-матрицей. Олигонуклеотидный праймер представляет собой молекулу олигонуклеотида, предпочтительно ДНК, которая гибридизуется с матрицей и способна примировать реакцию элонгации, что является стандартной практикой в данной области. Олигонуклеотидный праймер (или просто “праймер”) предпочтительно гибридизуется с матрицей по меньшей мере на одном её участке по длине, напр., длиной от 4 нуклеотидов до 30 нуклеотидов (нт). Отжиг происходит путем этой гибридизации. Праймер может содержать часть, которая не гибридизуется с матрицей. Такие дополнительные части могут использоваться для гибридизации с другими олигонуклеотидами и/или для дополнительной амплификации, упомянутой выше, когда амплификационные фрагменты подвергаются дальнейшей амплификации для получения их копий. При этом такие дополнительные части или участки могут содержать последовательность, с которой связываются другие праймеры для этой реакции амплификации/копирования. Такие части также именуются линкерной последовательностью праймера. Линкерная последовательность праймера предпочтительно имеет длину от 4 до 30 нт.
Возвращаясь к основному способу по изобретению, по меньшей мере один олигонуклеотидный праймер подвергается элонгации специфичным для матрицы образом, образуя при этом продукт элонгации (комплементарную последовательность). Такие реакции являются стандартными в данной области и в них обычно используются полимеразы. Если матрицей является РНК, то используется РНК-зависимая полимераза типа обратной транскриптазы. Если матрицей является ДНК, то используется ДНК-зависимая полимераза. Реакция элонгации останавливается, когда она достигнет стоппера элонгации нуклеиновой кислоты, загибридизованного на нуклеиновой кислоте-матрице ниже продукта элонгации, или же когда продукт элонгации достигнет 5'-конца нуклеиновой кислоты-матрицы. Ясно, что когда реакция элонгации достигнет 5'-конца матрицы и тем самым выйдет за пределы матрицы, она остановится. Некоторые полимеразы в этот момент могут добавить один или несколько нематричных нуклеотидов к продукту элонгации, что допустимо и даже может быть полезно при отборе 5'-охватывающих продуктов при анализе последовательности полученных меченых амплифицированных фрагментов. Однако в таком добавлении нематричных нуклеотидов нет необходимости. Реакция элонгации также остановится, когда она достигнет стоппера элонгации нуклеиновой кислоты, загибридизованного на матрице нуклеиновой кислоты ниже продукта элонгации. Такая остановка реакции подробно описана в WO 2013/038010 A2 (включена сюда путем ссылки). В этом WO-документе стоппер элонгации упоминается как “олигонуклеотидный стоппер” или “дополнительный олигонуклеотидный праймер”. В настоящем изобретении применяются такие термины как. стоппер элонгации нуклеиновой кислоты или просто “стоппер элонгации” или же просто “стоппер”. Такой стоппер по изобретению также может быть и праймером и тогда он соответствует “дополнительному олигонуклеотидному праймеру” в WO 2013/038010 A2. По сути, такой стоппер останавливает протекающую перед ним реакцию элонгации (поэтому стоппер находится ниже продукта элонгации), создавая препятствие на матрице. Стоппер отжигается или гибридизуется с матрицей, а реакция элонгации не может его сместить и поэтому прекращается. Дальнейшее продвижение, т.е. смещение стоппера, было бы побочной реакцией. Меры по предотвращению смещения стоппера подробно описаны в WO 2013/038010 A2 и могут применяться в соответствии с изобретением. Вкратце, предпочтительные способы и средства для предотвращения смещения стоппера (вызванного активностью смещения нити) заключаются в использовании стопперов элонгации, содержащих один или несколько модифицированных нуклеотидов, повышающих температуру плавления загибридизованных последовательностей, когда она загибридизовалась с матрицей (части стоппера, которая отжигается/гибридизуется с матрицей). Повышение температуры плавления относится к немодифицированной природной нуклеиновой кислоте типа ДНК или РНК. Такие модификации, напр., как LNA (блокированная нуклеиновая кислота), ZNA (zip-нуклеиновая кислота), 2'-фторнуклеозиды/2'-фторнуклеотиды или PNA (пептидная нуклеиновая кислота). Другие меры включают использование полимеразы, не обладающей активностью смещения нити, или использование интеркаляторов. Предпочтительно модифицируют 1, 2, 3, 4, 5 или 6 нуклеотидов. Предпочтительно модифицированные нуклеотиды находятся на 5'-стороне той части последовательности стоппера, которая гибридизуется с матрицей. Могут быть и другие части стоппера в 5'-направлении, которые не гибридизуются, типа амплификационных последовательностей, которые действуют так же, как описано для олигонуклеотидного праймера, описанного выше для амплификации/копирования в дальнейшей реакции амплификации (“линкерной последовательности праймера”), но в самом деле такая дополнительная часть предпочтительна для связывания/гибридизации с адаптерной нуклеиновой кислотой - см. ниже. Адаптер может связываться/гибридизироваться с “линкерной последовательностью праймера” или с другой частью олигонуклеотидного стоппера. В предпочтительных воплощениях стоппер элонгации, а также предпочтительно олигонуклеотидный праймер содержит один или несколько модифицированных нуклеотидов, повышающих температуру плавления отжигаемой последовательности (линкера) при отжиге с матрицей.
Предпочтительно после реакции элонгации те праймеры и стопперы, которые не связались с матрицей, удаляются на стадии очистки. Т.е. происходит очистка продуктов элонгации, гибридизовавшихся с матрицей, которые сохраняются для дальнейшей обработки. Другие воплощения изобретения выполняются в одном объеме без очистки. Такая очистка может проводиться методами, известными в данной области, напр., иммобилизации матрицы или продуктов элонгации на твердой фазе (напр., на шариках) и отмывки для удаления несвязавшихся праймеров и стопперов. Примером такого метода является твердофазная обратимая иммобилизация (SPRI; DeAngelis et al., Nucleic Acids Research, 1995, 23(22): 4742-4743).
Способ по изобретению включает стадию получения адаптерной нуклеиновой кислоты, содержащей идентификационную последовательность на своем 5'-конце. В состав адаптерной нуклеиновой кислоты также могут входить и другие метки последовательности типа последовательностей для амплификации (амплификационные последовательности). Именно 5'-конец предназначен для лигирования с 3'-концом продукта элонгации для мечения последнего с помощью идентификационной последовательности. Идентификационная последовательность не должна гибридизироваться ни со стоппером элонгации, ни с матрицей. Поэтому она обычно одноцепочечна и не гибридизуется. При этом термин “идентификационная последовательность” применяется для обозначения 5'-концевой части адаптерной нуклеиновой кислоты, которая не гибридизуется и не отжигается, даже если позже для идентификации будут использоваться только части идентификационной последовательности. Другие части адаптерной нуклеиновой кислоты могут образовывать гибриды со стоппером элонгации или отжигаться с ним. Адаптерная нуклеиновая кислота также может содержать комплементарную последовательность праймера, которая служит мишенью для дальнейшей реакции амплификации меченых амплификационных фрагментов, как указано выше (так называемая линкерная последовательность адаптера). Гибридизацию идентификационной последовательности со стоппером элонгации или с матрицей можно предотвратить путем выбора такой последовательности для идентификационной последовательности, которая не имеет комплементарности со стоппером элонгации. Также можно выбрать такую последовательность для идентификационной последовательности, которая не имеет комплементрности с матрицей. Это легко сделать, если известна последовательность матрицы. Если же она не известна, но происходит из биологического источника, то идентификационную последовательность можно выбрать из последовательностей, которые не встречаются или редко встречаются в биологических нуклеиновых кислотах. Такие последовательности известны среди нуклеиновых кислот “spike-in” типа последовательностей ERCC (External RNA Controls Consortium) или последовательностей SIRV (spike-in варианты РНК) (напр., см. ERCC, BMC Genomics 2005, 6:150; Jiang et al., Genome Res. 2011, 21(9): 1543-1551; WO 2016/005524 A1, которые все включены сюда путем ссылки). Если идентификационная последовательность гибридизуется с матрицей в побочной реакции, то это обычно предотвращает лигирование на следующей стадии и тем самым не дает меченых фрагментов, поэтому не отражается как результат. Такие побочные реакции допустимы, но не предпочтительны. Самый легкий и наиболее предпочтительный способ предотвращения отжига идентификационной последовательности (и предпочтительно всей адаптерной нуклеиновой кислоты) с матрицей заключается просто во введении адаптерной нуклеиновой кислоты после реакции элонгации. Матрица после реакции элонгации находится в двухцепочечной форме с продуктами элонгации (а также с праймером и стопперами). В таком виде адаптерная нуклеиновая кислота больше не может связываться с матрицей, так как матрица уже закрыта партнерами по гибридизации. В этом предпочтительном способе идентификационная последовательность даже может иметь такую последовательность, которая комплементарна матрице и способна гибридизироваться с матрицей, но этому препятствует последовательность стадий способа. Таким образом, в этом воплощении нет необходимости учитывать последовательность матрицы.
Наиболее предпочтительный вариант предотвращения отжига идентификационной последовательности на стоппере состоит в том, чтобы части стоппера и части адаптера имели комплементарные друг другу последовательности. Потому что при приближении адаптера к стопперу сначала будут гибридизироваться комплементарные последовательности, а идентификационная последовательность останется одноцепочечной.
Способ по изобретению также включает лигирование адаптерной нуклеиновой кислоты по её 5'-концу с 3'-концом продукта элонгации, при этом образуется меченый амплификационный фрагмент. Лигирование обычно проводится с помощью фермента лигазы. Тип лигазы зависит от природы лигируемых олигонуклеотидов и может быть выбран специалистом. Примеры лигаз включают ДНК-лигазы или РНК-лигазы. Лигаза также может быть РНК-лигазой, особенно такой РНК-лигазой, которая обладает активностью лигирования ДНК типа РНК-лигазы-2 T4. Другие лигазы - это ДНК-лигаза T4, РНК-лигаза-1 T4, ДНК-лигаза I, ДНК-лигаза III, ДНК-лигаза IV, ДНК-лигаза E. coli, ДНК-лигаза амплигаза, усеченная Rnl2, усеченная Rnl2 K227Q, лигаза Thermus scotoductus, РНК-лигаза Methanobacterium thermoautotrophicum, термостабильная App-лигаза (NEB), ДНК-лигаза вируса хлореллы или лигаза SplintR. Лигаза может быть одноцепочечной лигазой или двухцепочечной лигазой. Также возможны комбинации лигаз для различных реакций в одном реакционном объеме, которые должны протекать параллельно, напр., когда присутствуют различные продукты элонгации и/или молекулы адаптерной нуклеиновой кислоты, которые должны лигироваться одновременно. Предпочтительные комбинации: ДНК-лигаза и РНК-лигаза или одноцепочечная лигаза и двухцепочечная лигаза. В лигазной реакции обычно участвует фосфатный остаток, который предпочтительно находится на 5'-конце идентификационной последовательности адаптерной нуклеиновой кислоты. Для лигирования, напр., лигирования аденилированных концов, также можно использовать и другие 5'-ферменты. Их можно лигировать с помощью усеченных лигаз или App-лигаз.
Полученные меченые амплификационные фрагменты после лигирования будут иметь такую структуру от 5'- к 3'-концу: последовательность праймера - последовательность продукта элонгации - последовательность адаптера с идентификационной последовательностью, граничащей с последовательностью продукта элонгации. Последовательность праймера может включать “линкерную последовательность праймера” и/или последовательность адаптера может включать “линкерную последовательность адаптера”. Полученные способом по изобретению продукты, т.е. полученные меченые амплификационные фрагменты предпочтительно подвергаются дальнейшей амплификации. При такой дальнейшей амплификации образуются копии полученных меченых амплификационных фрагментов известными в данной области способами типа ПЦР (полимеразной цепной реакции) или линейной амплификации. В такой дальнейшей амплификации обычно участвуют дополнительные праймеры, которые связываются с мечеными амплификационными фрагментами, предпочтительно на линкерных последовательностях, особенно на линкерных последовательностях, расположенных на концах фрагментов, т.е. в пределах части последовательности праймера и части последовательности адаптера, особенно предпочтительно на 5'-конце последовательности праймера и на 3'-конце последовательности адаптера. Как указано выше в отношении этих праймеров и адаптеров, они могут включать участки с известной последовательностью для связывания таких праймеров при дальнейшей амплификации (“линкерной последовательности праймера” и “линкерной последовательность адаптера”). Эти участки (или “части”) могут быть настолько длинными и специфичными, что не будут связываться с матрицей; они могут быть универсальными сайтами связывания праймеров, т.е. неселективными между различными адаптерами/праймерами, в отличие от идентификационной последовательности, которая предпочтительно является уникальной.
Идентификационная последовательность обеспечивает уникальную метку для амплификационного фрагмента и поэтому также именуется здесь уникальным молекулярным идентификатором (UMI). Идентификационные последовательности могут идентифицировать повторы при дальнейшей амплификации (напр., ПЦР) и снижать эффекты зависимой от последовательности ошибки амплификации, которая выражена в предпочтительной амплификации одной последовательности. В предпочтительных воплощениях идентификационные последовательности представляют собой олигонуклеотиды, главным образом со случайным распределением нуклеотидов в каждом положении, которые лигируют с продуктами элонгации (фрагментами) перед дальнейшей амплификацией. Если идентификационные последовательности распределяются равномерно и их количество значительно превышает количество идентичных продуктов элонгации, то маловероятно, чтобы одна и та же идентификационная последовательность лигировалась с двумя идентичными продуктами элонгации (разными копиями). В этом случае количество различных идентификационных последовательностей после дальнейшей амплификации будет таким же, как и их количество перед дальнейшей амплификацией. Идентификационные последовательности по изобретению также можно использовать и так, как описано для UMI в Sena et al. (Scientific Reports (2018) 8:13121). Всю последовательность или части всей последовательности меченого фрагмента можно рассматривать как “рид” в способах секвенирования следующего поколения и при дальнейшем анализе последовательности. При анализе данных происходит сборка одного или нескольких ридов для получения объединенной последовательности матрицы. Впоследствии анализ данных также может превратиться в количественный анализ молекул и фрагментов матрицы, который может дать представление о том, представлены ли определенные копии матрицы слишком много или недостаточно, что, напр., свидетельствует о различной скорости экспрессии сплайс-вариантов РНК. В предпочтительных воплощениях настоящее изобретение дополнительно включает стадию сборки последовательностей таких амплификационных фрагментов, которые являются уникальными, причем для идентификации уникальных амплификационных фрагментов используются метки. Различные идентификационные последовательности в амплифицированных меченых амплификационных фрагментах идентифицируют уникальные амплификационные фрагменты. Идентификационные последовательности дают возможность дублировать и повторять идентификацию и удаление при сборке или на любой другой стадии анализа данных.
В предпочтительных воплощениях идентификационная последовательность имеет длину от 3 нуклеотидов (нт) и более, предпочтительно от 3 нт до 20 нт, особенно предпочтительно от 4 нт до 15 нт или от 5 нт до 10 нт, как-то 3 нт, 4 нт, 5 нт, 6 нт, 7 нт, 8 нт, 9 нт, 10 нт, 11 нт, 12 нт, 13 нт, 14 нт, 15 нт или больше. Такая длина достаточно мала для легкости работы с ними и эффективных реакций лигирования, но все-таки обеспечивает достаточно большое количество различных идентификационных последовательностей из-за перестановки в них нуклеотидов, обеспечивая требуемую идентификацию одиночных амплифицированных фрагментов, предпочтительно обеспечивая уникальные метки для них.
В предпочтительных воплощениях, когда продукт элонгации достигает 5'-конца матрицы нуклеиновой кислоты, нуклеотидная полимераза получает возможность добавлять нематричные нуклеотиды к продукту элонгации, предпочтительно за счет терминальной трансферазной активности полимеразы, и/или предпочтительно при этом добавляется от 1 до 15 нематричных нуклеотидов по меньшей мере в 70% продуктов элонгации. Как указано выше, такое добавление нематричных нуклеотидов является свойством некоторых полимераз (см. Chen et al., Biotechniques 2001, 30(3): 574-582). Эта активность наиболее выражена у обратных транскриптаз типа обратной транскриптазы M-MLV (вируса лейкемии мышей) или обратной транскриптазы AMV (вируса мозаичности люцерны). Эти нематричные нуклеотиды обычно представляют нуклеотидов любого типа (A, T (U), G, C) и могут появляться случайным образом. Это значит, что продукты элонгации 5'-концов у разных матриц могут иметь одну и ту же последовательность, соответствующую 5'-концу, но затем могут продолжаться различными, явно случайными дополнительными нуклеотидами, которые являются продуктами такого нематричного добавления. Эти различные добавления можно использовать для определения точного положения 5'-конца последовательности матрицы на переходе между повторяющейся матричной последовательностью и случайными нематричными добавлениями. После нематричных нуклеотидов меченого фрагмента следует идентификационная последовательность, которую можно использовать, как описано выше. В случае, если идентификационная последовательность (тоже) является случайной, то нематричные случайные нуклеотиды можно рассматривать как часть идентификационной последовательности. Положение идентификационной последовательности относительно постоянной части адаптерной последовательности однозначно определяет идентификационную последовательность.
В особенно предпочтительных воплощениях обеспечивается множество адаптерных нуклеиновых кислот, которые используются на стадии лигирования. Адаптеры из этого множества могут иметь разные идентификационные последовательности. Это позволяет однозначно идентифицировать адаптеры и полученные фрагменты, с которыми они лигированы. Предпочтительно на стадии лигирования обеспечивается и используется по меньшей мере 10, более предпочтительно по меньшей мере 50 или даже 100 и более или 200 и более адаптерных нуклеиновых кислот с различными идентификационными последовательностями. В особенно предпочтительных воплощениях используется столько же адаптеров с различными идентификационными последовательностями, сколько ожидается различных получаемых фрагментов с такой же последовательностью, а предпочтительно еще больше адаптеров с различными идентификационными последовательностями. Ожидаемое количество копий может основываться на типе образцов, напр., РНК из целых клеток, мРНК из целых клеток (транскриптом), количестве РНК и сложности образцов (сколько намечается различных вариантов транскриптов, что может означать весь транскриптом или же только выбранные гены или транскрипты, как в случае генных панелей) и др.
В особенно предпочтительных воплощениях идентификационная последовательность представляет собой случайную последовательность. “Случайные последовательности” следует понимать как смесь различных последовательностей с высокой дисперсией вследствие случайного синтеза по крайней мере части идентификационной последовательности. Случайные последовательности потенциально охватывают всю область комбинаций для данной последовательности по 4 природным нуклеотидам (A, T (U), G, C). Случайная последовательность может охватывать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 и более нуклеотидов, выбранных случайным образом из A, G, C или T (U). С точки зрения способности к гибридизации последовательностей нуклеотидов T и U используются здесь взаимозаменяемо. Вся область возможных комбинаций для случайной части последовательности равна mn, где m - количество используемых типов нуклеотидов (предпочтительно все четыре из A, G, C, T (U)), а n - количество случайных нуклеотидов. Следовательно, случайный гексамер, в котором представлена каждая возможная последовательность, состоит из 46 = 4096 различных последовательностей. Идентификационная последовательность не должна связываться с матрицей. Во всех случаях, но особенно для случайных идентификационных последовательностей, предпочтительно адаптерную нуклеиновую кислоту добавляют после реакции элонгации. Когда продукт элонгации достигнет стоппера (или конца матрицы) и практически вся матрица будет иметь двухцепочечную форму с продуктами элонгации, то предотвращается связывание адаптерной нуклеиновой кислоты с матрицей.
В дальнейших воплощениях изобретения праймеры и стопперы выбираются так, чтобы они связывались с одной или несколькими конкретными представляющими интерес целевыми последовательностями в нуклеиновой кислоте-матрице (причем стопперы находятся по нисходящей от продуктов элонгации) с тем, чтобы получить последовательность элонгации определенной части матрицы. Такое нацеливание на определенные участки предпочтительно используется для транскриптов (РНК) или генов (гДНК) в качестве матриц. Идентификационные последовательности особенно полезны при использовании в генных панелях. Например, для анализа вариантов последовательности у различных видов матриц типа сплайс-вариантов или других изменчивых последовательностей матриц.
В особенно предпочтительных воплощениях изобретения для всех его воплощений и аспектов стопперы элонгации обладают активностью праймеров и тоже подвергаются элонгации на стадии элонгации. Это значит, что используется более чем один праймер и большая их часть обладает функцией стоппера (т.е. предотвращает смещение - см. выше). Использование нескольких праймеров означает, что матрица дает много образующихся фрагментов, т.е. улучшается охват. Хотя каждый праймер связывается с одной матрицей, они обеспечат полный охват, если разные праймеры связываются с разными участками одной матрицы. Способ по изобретению с использованием множества праймеров (которые предпочтительно также являются стопперами) увеличит охват, так как новый продукт элонгации будет начинаться в том положении на матрице, где как раз остановился предыдущий продукт элонгации. Это дает много фрагментов, покрывающих всю матрицу. Кроме того, это значит, что используются такие стопперы/праймеры (в данном воплощении это синонимы), которые связываются с разными частями молекулы матрицы. В общем, связывание с молекулой матрицы определяется последовательностями отжига праймеров и стопперов. Эти последовательности гибридизуются с матрицей и могут варьироваться для связывания с разными участками матрицы. Предпочтительно используется по меньшей мере 9, по меньшей мере 10, более предпочтительно по меньшей мере 49, по меньшей мере 50, напр., 100 и более или 200 и более стопперов элонгации, которые имеют разные последовательности отжига для посадки на матрицу. Тем самым они потенциально будут гибридизоваться с различными участками нуклеиновой кислоты-матрицы. Предпочтительно последовательность отжига представляет собой случайную последовательность. Случайные последовательности описаны выше в отношении идентификационной последовательности и то же самое относится и к последовательностям отжига праймеров, стопперов и стопперов с функцией праймеров. Предпочтительно случайная последовательность последовательности отжига может охватывать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 и более нуклеотидов, выбранных случайным образом из A, G, C или T (U).
Предпочтительно адаптерная нуклеиновая кислота связывается, гибридизуется или же не связывается со стоппером элонгации. Такая реакция связывания, напр., посредством химической реакции, образования комплекса или гибридизации, облегчает позиционирование адаптерной нуклеиновой кислоты возле 3'-конца вышележащего продукта элонгации, с которым связана идентификационная последовательность, которая сама по себе не гибридизуется со стоппером или матрицей и вне ожидания не требуется для протекания реакции лигирования. Предпочтительно, когда адаптерная нуклеиновая кислота связывается со стоппером элонгации или гибридизуется с ним, то идентификационная последовательность выбирается независимо от последовательности отжига стоппера элонгации для посадки стоппера элонгации на матрицу. И последовательность отжига, и идентификационная последовательность может представлять собой случайную последовательность, предпочтительно независимо друг от друга. Обычно это гарантируется тем, что части нуклеиновой кислоты стоппера и адаптера представляют собой универсальные последовательности, т.е. любой адаптер может связываться с любым стоппером (что предпочтительно для всех воплощений изобретения), а также тем, что адаптерная нуклеиновая кислота не связывается со стоппером, напр., адаптер предоставляется только после реакции элонгации. В другом воплощении или в других частях реакции они не связываются типа когда реакция элонгации достигает 5'-конца матрицы, с которым обычно не гибридизуется стоппер, потому что для стоппера требуется по крайней мере минимальная последовательность отжига на матрице, что сдвигает самое нижнее положение остановки на несколько нуклеотидов вверх от 5'-конца. Адаптер также может лигироваться с продуктом элонгации без связывания или гибридизации со стоппером элонгации. Однако во всех воплощениях предпочтительно, чтобы при лигировании адаптерной нуклеиновой кислоты с продуктом элонгации данный стоппер элонгации и/или продукт элонгации, особенно предпочтительно его 3'-конец, все-таки гибридизировался с матрицей. Также предпочтительно, чтобы адаптерная нуклеиновая кислота гибридизировалась со стоппером элонгации, причем особенно предпочтительно - после реакции элонгации и/или, что особенно предпочтительно, - для лигирования.
В предпочтительных воплощениях способа и набора по изобретению олигонуклеотидный праймер, а предпочтительно, но не обязательно и стоппер элонгации, содержит универсальную амплификационную последовательность (“линкерную последовательность праймера”, см. выше), и/или адаптерная нуклеиновая кислота содержит универсальную амплификационную последовательность адаптера (“линкерную последовательность адаптера”, см. выше). Такая амплификационная последовательность или “линкер” может использоваться для связывания праймеров для дальнейшей амплификации, как уже сказано выше. Универсальная последовательность означает то, что она одинакова для всех праймеров, стопперов или адаптеров, соответственно. Это позволяет одному и тому же типу праймера связываться с этими олигонуклеотидами. В особенно предпочтительных воплощениях универсальная амплификационная последовательность (последовательность линкера) также одинакова для праймеров, стопперов и адаптеров, т.е. праймер для дальнейшей амплификации может равным образом связываться с олигонуклеотидными праймерами, стопперами элонгации и адаптерными нуклеиновыми кислотами. Это упрощает использование, так как для дальнейшей амплификации нужен только один тип праймера. В других воплощениях праймеры, стопперы и адаптеры имеют различные универсальные амплификационные последовательности (линкерные последовательности), т.е. праймер для дальнейшей амплификации может связываться только с олигонуклеотидными праймерами, другой праймер для дальнейшей амплификации может связываться только со стопперами элонгации, а еще другой праймер для дальнейшей амплификации может связываться только с адаптерными нуклеиновыми кислотами. В пределах этих групп праймеры предпочтительно являются универсальными. Это все еще упрощает использование, но обеспечивает лучший контроль, так как праймеры для обоих концов меченого фрагмента будут разными и их можно выбирать специфически.
В предпочтительных воплощениях используется специальный олигонуклеотидный праймер для выбора и гибридизации с выбранной последовательности матрицы, предпочтительно на 3'-конце матрицы. В случае мРНК или любого другого типа РНК, содержащей хвост олиго(A), такой 3'-конец может гибридизироваться с комплементарным олигонуклеотидным праймером, напр., содержащим последовательность отжига олиго(dT), комплементарную данному хвосту олиго(A). Предпочтительно по меньшей мере один олигонуклеотидный праймер содержит последовательность отжига для гибридизации с выбранной последовательностью матрицы, которая может находиться на 3'-конце матрицы или возле него. Такая выбранная последовательность представляет собой любую известную последовательность матрицы типа хвоста олиго(A), но может использоваться и любая другая последовательность, если она известна. Предпочтительно олигонуклеотидный праймер для выбранной последовательности содержит последовательность олиго(dT) для гибридизации с последовательностью олиго(A) в матрице. Предпочтительно такая последовательность олиго(dT) содержит один или несколько якорных 3'-нуклеотидов, отличных от последовательности олиго(dT). Это обеспечивает правильную локализацию и связывание с 5'-концом последовательности олиго(A) матрицы. Якорный нуклеотид будет связываться с ближайший нуклеотидом, кроме A (напр., T, G, C) на матрице рядом с участком олиго(A). Если ближайший нуклеотид не-A неизвестен, то можно использовать смесь олигонуклеотидного праймера с различными якорными праймерами, напр., использовать три олигонуклеотидных праймера, каждый без нуклеотида T (напр., A, G, C) (комплементарных ближайшему не-A (напр., T, G, C) на матрице). В предпочтительных воплощениях используются два якорных нуклеотида. Якорный нуклеотид, ближайший к данному нуклеотиду не-T, может быть выбран из любого типа нуклеотидов (A, T (U), G, C), так как он не граничит с олиго(T). Такой специальный олигонуклеотидный праймер может не быть стоппером и может не содержать последовательность для гибридизации с адаптером, так как они не нужны, если специальный олигонуклеотидный праймер гибридизуется с или рядом с 3'-концом матрицы, а это значит, что вышележащий продукт элонгации не дойдет до этого положения. Конечно, для простоты или единства в производстве праймеров/стопперов такая последовательность и/или стопперная функция может присутствовать.
Предпочтительно реакция лигирования проводится в присутствии краудинг агента (crowding agent). Краудинг агент повышает вероятность взаимодействия адаптера и продукта элонгации друг с другом за счет уменьшения эффективного объема реакции, см. Zimmerman et al., Proc. Natl. Acad. Sci. USA 1983, 80(19):5852-6. Другие краудинг агенты приведены, напр., в US 5,554,730, US 8,017,339 и WO 2013/038010 A2. Предпочтительно краудинг агент представляет собой макромолекулу, полимер или содержащее полимер соединение типа полиалкилгликоля, предпочтительно ПЭГ, октоксинол или Triton X либо полисорбат, предпочтительно Tween. В предпочтительных воплощениях краудинг агент используется в концентрациях от 5% до 35% (об./об.), особенно предпочтительно от 10% до 25% (об./об.). Предпочтительно краудинг агент имеет молекулярную массу от 200 до 35000 г/моль, предпочтительно от 1000 до 10000 г/моль. Особенно предпочтительным является полиалкилгликоль типа PEG, особенно с указанной молекулярной массой. Краудинг агент предпочтительно представлено в наборе по изобретению, предпочтительно в буфере для лигирования.
Другими ингредиентами набора являются такие компоненты, как буферы, соли, кофакторы ферментов и металлы типа Mn2+ и Mg2+ для полимераз и лигаз, растворители, контейнеры.
Настоящим изобретением предусмотрены наборы для выполнения способа по изобретению. Такой набор может включать любые из уже описанных соединений и средств. Предпочтительно набор включает: (i) по меньшей мере один олигонуклеотидный праймер, способный гибридизироваться с матричной нуклеиновой кислотой и запускать реакцию элонгации на своем 3'-конце, (ii) один или несколько стопперов элонгации, способных гибридизироваться с матричной нуклеиновой кислотой, предпочтительно способных запускать реакцию элонгации на своем 3'-конце, (iii) одну или несколько адаптерных нуклеиновых кислот, содержащих идентификационную последовательность на своем 5'-конце, причем такая идентификационная последовательность не гибридизируется со стоппером элонгации, предпочтительно при этом адаптерная нуклеиновая кислота связывается, гибридизуется или же не связывается со стоппером элонгации, (iv) обратную транскриптазу и (v) лигазу олигонуклеотидов, причем (iv) и (v) могут быть необязательными, так как они могут быть доступны во многих лабораториях, независимо от настоящего изобретения. Важными частями являются конструкции адаптеров/стопперов, в частности, идентификационные последовательности в адаптерах. Предпочтительно в наборе представлено множество адаптеров с разными идентификационными последовательностями, как описано выше. Все эти компоненты набора были описаны выше, и любые их предпочтительные воплощения также применимы к наборам. Предпочтительно набор содержит по меньшей мере 10, более предпочтительно по меньшей мере 50 адаптерных нуклеиновых кислот с различными идентификационными последовательностями. Причины такого предпочтительного воплощения были приведены выше. Предпочтительно олигонуклеотидный праймер содержит последовательность отжига для гибридизации с матрицей, которая включает последовательность олиго(dT) для отжига с последовательностью олиго(A) в матрице, при этом предпочтительно такая последовательность олиго(dT) включает один или несколько якорных 3'-нуклеотидов, отличных от последовательности олиго(dT). Набор также может включать твердую фазу для очистки типа гранул, предпочтительно магнитных гранул (см. подробности способа выше, где также сказано о пригодности компонентов набора и воплощениях).
Все предпочтительные воплощения, описанные выше, можно комбинировать. В таком способе применяется случайный праймер (с линкерной последовательностью), который также является стоппером (а также именуется “праймером остановки смещения нити”). После реакции элонгации предпочтительно проводится очистка продуктов элонгации (гибридизованных с матрицей) для удаления несвязанных праймеров и стопперов. Затем адаптеры со своими линкерами и идентификационными последовательностями лигируются с продуктами элонгации. Идентификационная последовательность имеет случайную последовательность длиной предпочтительно от 4 до 12 нт. Одним из предпочтительных вариантов является использование смесей идентификационных последовательностей различной длины, так как лигазы склонны вызывать неравномерность лигирования, отдавая предпочтение некоторым 5'-расположенным нуклеотидам в последнем и предпоследнем положении. Поскольку такая неравномерность может повлиять на качество прочтения при секвенировании, то в таких смесях уравнивают распределение нуклеотидов при секвенировании в районе стыков лигирования. Однако вариабельность идентификационных последовательностей обеспечивает гораздо меньшую неравномерность лигирования, чем любая другая установленная последовательность, и в то же время также служит в качестве UMI (уникального молекулярного идентификатора). Идентификационная последовательность типа UMI позволяет определить, будут ли риды секвенирования, обладающие идентичной последовательностью или идентифицируемые в идентичной позиции в эталонной аннотации, учитывающей незначительные ошибки при секвенировании, происходить от разных молекул матрицы или же из одной молекулы матрицы и просто являются результатом дальнейшей амплификации (ПЦР-дупликации). Адаптер гибридизируется с праймером, если тот присутствует.
Идентификационные последовательности типа UMI также могут отличить реальные SNP (однонуклеотидные полиморфизмы) между индивидами от ошибок (мутаций), внесенных при обратной транскрипции или на ранних циклах ПЦР, которые затем амплифицируются. Все эти возникающие случайно и амплифицируемые ошибки должны иметь одинаковые идентификаторы, тогда как “реальные SNP” в образцах имеют различные другие идентификаторы. Или же можно более надежно определять те события при редактировании РНК, при которых вводятся модифицированные основания, что ведет к неправильному включению и тем самым к ошибкам при RT.
Идентификационные последовательности типа UMI также можно использовать для надежного определения и количественной оценки частоты аллелей в популяциях, молекулярных маркеров и вызывающих болезни мутаций при наследственных заболеваниях. Предпочтительно для этого воплощения используются ДНК-матрицы.
Другая предпочтительная комбинация представляет собой способ по изобретению, в котором по меньшей мере один, а предпочтительно по меньшей мере 9 стопперов элонгации обладают активностью праймеров и подвергаются элонгации на стадии элонгации, а также используются по меньшей мере две, а предпочтительно по меньшей мере 10 адаптерных нуклеиновых кислот, содержащих разные идентификационные последовательности, при этом образуются по меньшей мере два, а предпочтительно по меньшей мере 10 различных меченых фрагментов, которые необязательно подвергаются амплификации, дополнительно включающий сборку тех последовательностей амплификационных фрагментов, которые являются уникальными, причем для идентификации уникальных амплификационных фрагментов используются метки. Для идентификации уникальных амплификационных фрагментов в амплифицируемых меченых фрагментах можно использовать различные метки.
В другом предпочтительном способе используются стопперы с функциями праймеров. Предпочтительно используется множество таких праймеров. В таком способе, без различия между стопперами и праймерами, одно воплощение изобретения можно определить следующим образом: Способ получения меченых амплификационных фрагментов из матричной нуклеиновой кислоты, включающий стадии обеспечения такой нуклеиновой кислоты-матрицы, отжига множества олигонуклеотидных праймеров с данной матричной нуклеиновой кислотой, элонгации олигонуклеотидных праймеров специфичным для матрицы образом, получая при этом множество продуктов элонгации, причем реакции элонгации останавливаются, когда продукты элонгации достигают 5'-конца матричной нуклеиновой кислоты или олигонуклеотидного праймера, загибридизованного на матрице нуклеиновой кислоте ниже такого продукта элонгации, обеспечения множества адаптерных нуклеиновых кислот, содержащих идентификационную последовательность на своих 5'-концах, причем данные идентификационные последовательности не гибридизуются с олигонуклеотидным праймером или с матрицей, лигирования адаптерных нуклеиновых кислот из этого множества по своим 5'-концам с 3'-концом продуктов элонгации, получая при этом множество меченых амплификационных фрагментов. Это предпочтительное воплощение, которое можно комбинировать с любыми конкретно описанными аспектами в формуле изобретения и описанными выше. В этом воплощении все, что описано выше для стопперов, применимо и к праймерам, так как эти праймеры являются стопперами с функцией праймеров. Термин “множество” применяется для олигонуклеотидных праймеров, продуктов элонгации (которые являются результатом элонгации праймеров), адаптерных нуклеиновых кислот и меченых амплификационных фрагментов (которые являются результатом элонгации и лигирования с адаптерами). Как указано, количество некоторых из этих множеств является результатом способа. Количество олигонуклеотидных праймеров и адаптерных нуклеиновых кислот можно выбрать, как описано выше. Такие количества можно выбрать независимо, но предпочтительно они примерно одинаковы для попарного объединения с данным продуктом элонгации. Предпочтительно множество составляет, напр., 10 и более, 50 и более, 100 и более, 200 и более и т.д. Можно использовать много различных олигонуклеотидных праймеров и адаптерных нуклеиновых кислот: чтобы олигонуклеотидные праймеры связывались со множеством различных мест на матрице, чтобы адаптерные нуклеиновые кислоты имели разные идентификационные последовательности, предпочтительно уникальные идентификационные последовательности для меченых амплификационных фрагментов. Хотя в этом воплощении праймеры и стопперы это одно и то же, однако может быть добавлен и специальный праймер, которому не нужна (но он может иметь) функция стоппера, типа праймера, специфичного для 5'-конца, типа праймера, нацеленного на олиго(A), как описано выше.
Далее настоящее изобретение будет описано на следующих фигурах и примерах, но без ограничения настоящего изобретения этими воплощениями.
Краткое описание фигур
Фиг. 1. Схема получения библиотеки коротких кДНК, помеченных UMI-линкером, с помощью праймера со свойствами SDS и частично комплементарного содержащего UMI линкерного олигонуклеотида в общей массе РНК.
a) Общий праймер Pn для остановки смещением нити гибридизуется с транскриптом РНК, а праймер Pn+1 гибридизуется с более вышележащим (5') положением РНК-матрицы, чем праймер Pn. Когда обратная транскриптаза при элонгации Pn достигнет праймера Pn+1, полимеразная реакция остановится по технологии остановки со смещением нити, описанной в WO 2013/038010 A2. Содержащий UMI линкерный олигонуклеотид, охватывающий L2, который комплементарен L1, гибридизуется с праймерами Pn и Pn+1. b) При лигировании продукт элонгации теперь лигируется с UMI, предшествующим нити L2 линкера. Таким же образом создается библиотека кДНК, которая содержит две линкерные последовательности (L1, L2) на концах и содержит уникальные молекулярные идентификаторы. c) Наконец, проводится ПЦР для амплификации этих библиотек.
Фиг. 2. Получение содержащих UMI библиотек.
На фиг. a) представлены библиотеки, полученные способом SDS + лигирования. Лигирование содержащего UMI частично комплементарного адаптера L2 (см. фиг. 1 для справки) может проводиться с помощью либо ss-лигазы, либо ds-лигазы (дорожки 2, 3). В отсутствие лигазы библиотеки не образуются (дорожка 1). После лигирования фрагменты кДНК, содержащие линкеры L1 и L2, амплифицировали методом ПЦР и анализировали. Представлены снимки гелей, полученные при анализе HS DNA Assay на биоанализаторе (Agilent Technologies, Inc.). b) Схема получения содержащих UMI библиотек по методу SDS + лигирования с негибридизирующимися праймерными и адаптерными олигонуклеотидами. В этом случае адаптерный олигонуклеотид L2' не содержит последовательностей, комплементарных праймеру элонгации Pn. c) Снимок геля и электрофореграмма повторных библиотек, полученных с помощью негибридизирующегося праймера элонгации и содержащих UMI адаптерных олигонуклеотидов (SEQ ID No. 10). Снимки получали при анализе HS DNA Assay на биоанализаторе (Agilent Technologies, Inc.).
Фиг. 3. Улучшение охвата 5'-конца транскриптов, достигаемое при лигировании линкеров L2 с кДНК по 5'-концу РНК-матрицы.
a) Схема реакции RT на 5'-конце транскриптов. Без SDS при нижележащих праймерах Pn+1, терминальная дезоксинуклеотидилтрансферазная активность (TdT) RT добавляет нематричные нуклеотиды на 3'-конец кДНК, образуя выступ (overhang). b) Нематричные нуклеотиды могут служить сайтом гибридизации для содержащего L1 праймера Pn+1. В сочетании с частично гибридизированным L2 может происходить лигирование UMI с линкером L2 в двойной цепи. c) С другой стороны, в отсутствие прайминга UMI может лигироваться с линкером L2 в виде одиночной цепи. d) Библиотеки, полученные, как показано схематически на фиг. 3 a-c, секвенировали на установке Illumina NextSeq 500 (однократное прочтение, 75 п.н.). Показаны риды, относящиеся к 5'-концу ERCC-0130 (который присутствует в наборе SIRV 3, Lexogen, кат. № 051.0N). Риды анализировали без отсечения дополнительных и несовпадающих оснований. Нуклеотиды, выделенные серым цветом, соответствуют аннотации ERCC-0130, а нуклеотиды, выделенные черным цветом, получены при нематричном добавлении за счет TdT-активности RT. Ниже представлено 30 репрезентативных последовательностей ридов, полученных для 5'-конца ERCC-0130. Последовательности ридов представлены как SEQ ID NO: от 12 до 42, сверху вниз. e) Улучшение охвата 5'-концов методом SDS/лигирования по сравнению со стандартной методикой. Библиотеки получали по стандартной методике (набор NEBNext® Ultra™ II directional RNA Library Prep Kit for Illumina®, New England Biolabs, кат. № E7760S) или методом SDS/лигирования и секвенировали на Illumina NextSeq 500 (риды парных концов, 150 п.н.). Риды, относящиеся к ERCC-0130, налагали друг на друга и сравнивали с ожидаемым охватом, представленным в виде прямоугольника; слева: стандартная методика получения библиотек РНК, справа: охват, полученный по новой технологии SDS/лигирования.
Фиг. 4. Схема реакции, используемой для улучшения охвата 3'-конца с помощью метода SDS/лигирования и комбинации общих (Pn) и олиго(dT)-праймеров (PdT).
a) Общий праймер Pn гибридизуется с РНК-матрицей в общей массе РНК. Кроме того, присутствующие олиго(dT)-праймеры (PdT) гибридизуются с хвостом поли(A) на 3'-конце полиаденилированных транскриптов. RT будет продолжать элонгацию PdT до тех пор, пока не достигнет нижележащего праймера Pn и прекратит смещение нити. b) При лигировании содержащий UMI линкер L2 будет лигироваться с фрагментами кДНК, охватывающими 3'-конец, образуя L1- и L2-связанные, содержащие UMI библиотеки кДНК, охватывающие 3'-концы транскриптов. c) График охвата тела генов, показывающий повышение охвата 3'-конца транскриптов по всему транскриптому. Получали библиотеки по методике SDS + лигирования с использованием смеси случайных праймеров и олиго(dT)-праймера для синтеза первой нити, как описано в примере 3. Библиотеки секвенировали на установке NextSeq 500 и строили графики охвата тела генов по всему транскриптому в сравнении с описанной ранее методикой SDS + лигирования. d) Пример охвата по эндогенному хозяйственному гену (HSP90) для стандартного метода получения библиотек (верхняя панель) и для методики SDS + лигирования с титрованием олиго-dT (нижняя панель), которая приводит к улучшению охвата 3'-конца.
Фиг. 5. Глобальное улучшение охвата 5'- и 3'-концов транскриптов. Сайты начала транскрипции, т.е. истинные 5'-концы транскриптов, и сайты окончания транскрипции, т.е. истинные 3'-концы транскриптов, разрешаются по методике SDS + лигирования, но не разрешаются при использовании двух типичных традиционных методов получения библиотек. Библиотеки, полученные по методике SDS + лигирования, как показано схематически на фиг. 3 а-с, секвенировали на Illumina NextSeq 500 (парные концы, 150 п.н.). Традиционные библиотеки получали согласно инструкциям производителя, используя либо набор TruSeq Stranded Total RNA Library Prep Human/Mouse/Rat, Illumina, кат. № 20020596 или 20020597 (= обычная 1), либо набор NEBNext® Ultra™ II directional RNA Library Prep Kit for Illumina®, New England Biolabs, кат. № E7760S (= обычная 2). a) Представлены риды, относящиеся к истинным 5'- и 3'-концам обнаруженных ERCC (которые присутствуют в наборе SIRV set 3, Lexogen, кат. № 051.0N). Риды соотносили с внесенными в РНК ERCC с известной последовательностью. Нормализованные значения охвата накопленных картированных ридов для всех обнаруженных ERCC наносили на график по абсолютным положениям нуклеотидов относительно сайтов начала транскрипции (TSS) и окончания транскрипции (TES), отмеченных пунктирными линиями. b) Широкий охват 5'-концов выявляет типичные TSS. Верхняя панель: профиль охвата для gapdh с конденсированной визуализацией интронов, полученный по методике SDS + лигирования или традиционных препаратов библиотек, как описано выше. Риды, относящиеся к gapdh, анализировали без отсечения дополнительных и несовпадающих оснований. Последовательности ридов представлены в виде SEQ ID Nos. от 43 до 67, сверху вниз. Нуклеотиды, выделенные черным цветом, соответствуют аннотации gapdh, а нуклеотиды, выделенные серым цветом, не совпадают или получены при безматричном добавлении за счет TdT-активности RT. Кластеры начальных сайтов, полученные при наложении ридов на 5'-конце транскриптов, можно использовать для повторного аннотирования TSS. Аннотированные и определенные вручную TSS обозначены стрелками на аннотированной консенсусной последовательности, выделенной жирным шрифтом.
Примеры
Пример 1. Лигирование уникальных молекулярных идентификаторов (UMI) с фрагментами первой нити кДНК
Получали библиотеки из универсальной эталонной РНК человека (Agilent Technologies, кат. № 740000), содержащей контрольную смесь SIRV Set 3 spike-in (Lexogen, кат. № 051.0N), в соответствии с инструкциями производителя.
После синтеза кДНК можно лигировать нижележащие праймеры (Pn+1 (L2)), содержащие уникальный молекулярный идентификатор длиной от 2 до 24 нуклеотидов, а предпочтительно от 6 до 12 нуклеотидов, с вновь транскрибированной нитью кДНК в виде гибрида с РНК-матрицей. Обратную транскрипцию проводили, используя олигонуклеотиды, матрицы и условия, описанные в WO 2013/038010 A2. Можно использовать различные лигазы и их комбинации для лигирования олигонуклеотидов типа:
SEQ ID No: 1: (Phos)(5'-NNNNNNAGATCGGAAGAGCACACGTCTGAACTCCAGTCAC-3'(3InvdT)),
SEQ ID No: 2: (Phos)(5'-NNNNNNNNNNAGATCGGAAGAGCACACGTCTGAA-3'(3InvdT)),
SEQ ID No: 3: (Phos)(5'-NNNNNNNNNNAGATCGGAAGAGCGTCGTGTAGGGAAA GAGTG-3'(3InvdT)),
SEQ ID No: 4: (Phos)(5'-NNNNNNNNNNAGATCGGAAGAGCGTCGTGTAGG-3'(3InvdT)),
SEQ ID No: 5: (Phos)(5'-NNNNNNNNNNNAGATCGGAAGAGCGTCGTGTAGGGAAAGAGTG-3'(3InvdT)),
SEQ ID No: 6: (Phos)(5'-NNNNNNNNNNNNAGATCGGAAGAGCGTCGTGTAGGGAAAGAGTG-3'(3InvdT)),
SEQ ID No: 7: (Phos)(5'-NNNNNNNNNNNNAGATCGGAAGAGCGTCGTGTAGG-3'(3InvdT)),
SEQ ID No: 8: (Phos)(5'-+NNNNNNNNNNAGATCGGAAGAGCGTCGTGTAGG-3'(3InvdT)),
SEQ ID No: 9: (Phos)(5'-+NNNNNNNNNNNNAGATCGGAAGAGCGTCGTGTAGG-3'(3InvdT)).
После обратной транскрипции (RT) образцы очищали методом твердофазной обратимой иммобилизации (SPRI) с помощью магнитных гранул (AMPure Beads; Agentcourt) согласно инструкциям производителя. Гибриды кДНК:РНК элюировали в 20 мкл воды или 10 мМ трис, pH 8,0, а затем по 17 мкл супернатантов переносили в новый планшет для ПЦР. Затем проводили реакции лигирования в 60 мкл с 20% PEG-8000, 50 мМ трис-HCl (pH 7,5 при 25°C), 10 мМ MgCl2, 5 мМ DTT, 0,4 мМ АТФ, 0,01% Triton-X100, 50 мкг/мл БСА и 20 единиц лигазы, которая может быть одноцепочечно-специфичной лигазой и/или двухцепочечно-специфичной лигазой. Нелигированные небольшие фрагменты и остающиеся олигонуклеотиды удаляли методом SPRI. Все оставшиеся первичные библиотеки кДНК амплифицировали в реакции ПЦР с помощью высокоточной полимеразы по следующей программе: 30 сек при 98°C, а затем 10-25 циклов ПЦР по 10 сек при 98°C, по 20 сек при 65°C и по 30 сек при 72°C. Затем проводили заключительную элонгацию при 72°C в течение 60 сек. На фиг. 1b представлен общий принцип, лежащий в основе лигирования прошедшей элонгацию кДНК с содержащим UMI линкерным олигонуклеотидом (L2), последовательность которого комплементарна праймеру остановки смещения нити (L1).
На фиг. 2 представлен пример того, что различные лигазы могут выполнять реакцию лигирования содержащего UMI олигонуклеотида и при этом образовывать фрагменты кДНК, содержащие оба линкера для ПЦР и способные амплифицироваться при ПЦР (фиг. 2a, дорожка 2-3). Напротив, контрольный эксперимент без какой-либо лигазы показывает, что библиотеки не могут амплифицироваться, подчеркивая специфичность реакции (фиг. 2a, дорожка 1).
Пример 2. Получение библиотек с использованием негибридизирующихся олигонуклеотидов - инициатора элонгации и адаптера
Получали библиотеки из универсальной эталонной РНК человека (Agilent Technologies, кат. № 740000), содержащей контрольную смесь SIRV Set 3 spike-in (Lexogen, кат. № 051.0N), в соответствии с инструкциями производителя.
Проводили обратную транскрипцию (RT), как описано в Примере 1. После RT образцы очищали методом твердофазной обратимой иммобилизации (SPRI) с помощью магнитных гранул (AMPure Beads; Agentcourt) согласно инструкциям производителя и элюировали гибриды кДНК:РНК в 20 мкл 10 мМ трис, pH 8,0, а затем по 17 мкл супернатантов переносили в новый планшет для ПЦР. Лигирование проводили в условиях, описанных в Примере 1, но использовали адаптерный олигонуклеотид, не содержащий последовательности, комплементарной инициатору элонгации, используемому для прайминга реакции обратной транскрипции. Поэтому адаптерный олигонуклеотид не может гибридизироваться и поэтому не попадает в окрестности вновь образовавшихся 3'-концов продуктов элонгации при рекрутинге (фиг. 2b). Олигонуклеотиды типа SEQ ID No. 10: (Phos)(5'-NNNNNNNNNNNNTGGAATTCTCGGGTGCCAAGG-3'(SpcC3)) не содержат последовательностей, комплементарных инициаторам элонгации. Фрагменты, содержащие обе линкерные последовательности, амплифицировали после очистки, как описано в Примере 1. На фиг. 2c представлены снимки гелей и электрофореграммы для двух повторных библиотек типа SDS + лигирования, полученных с негибридизирующимися олигонуклеотидами - инициаторами элонгации и адаптерами.
Пример 3. Улучшение охвата 5'-концов в результате терминальной трансферазной активности и ss-лигирования UMI-линкера с фрагментами первой нити кДНК
Получали библиотеки из универсальной эталонной РНК человека (Agilent Technologies, кат. № 740000), содержащей контрольную смесь SIRV Set 3 spike-in (Lexogen, кат. № 051.0N), в соответствии с инструкциями производителя.
Синтез первой нити кДНК останавливается на 5'-концах молекул РНК-матрицы. Терминальная трансферазная активность обратных транскриптаз катализирует безматричное добавление нуклеотидов на 3'-конце нити кДНК (фиг. 3a).
Лигирование UMI-линкерных олигонуклеотидов (напр., SEQ IDs 1-9) после обратной транскрипции может протекать в двухцепочном виде (фиг. 3b) и на одноцепочечных выступах (фиг. 3c). После SPRI-очистки и ПЦР-амплификации библиотеки секвенировали на NextSeq 500 в режиме однократного риды либо в режиме парных концов. Риды, относящиеся к 5'-концу ERCC-0130, анализировали без предварительного отсечения несовпадающих нуклеотидов. Риды, охватывающие 5'-конец ERCC-0130, приведены для примера на фиг. 3d. Добавление концевых нуклеотидов и лигирование UMI по прошедшим элонгацию одноцепочечным нитям приводит к улучшению охвата 5'-концов. На фиг. 3e представлено сравнение профилей охвата между обычным препаратом библиотеки RNA-seq и настоящим изобретением. Охват представлен в виде наложения всех выровненных ридов (профиль серого цвета) и сравнивается с ожидаемым равномерным охватом, представленным в виде прямоугольника. В то время, как данные по секвенированию, полученные по стандартным методикам, охватывают 5'- и 3'-концы менее эффективно, что видно по наклонам в сторону обоих концов (фиг. 3e, слева), новая методика дает больше ридов, относящихся к самому 5'-концу транскриптов (фиг. 3e, справа).
Пример 4. Улучшение охвата 3'-конца при титровании олиго(dT)-праймеров для синтеза первой нити
Охват 3'-концов транскриптов можно модифицировать, предпочтительно повысить, используя олиго(dT)-содержащие праймеры первой нити (Pn, содержащие L1), которые добавляют в смесь олигонуклеотидов для SDS со случайным праймингом, которая уже содержит порцию обогащенных T и содержащих только T последовательностей праймеров (типа SEQ ID No. 11: 5'-GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT+TTT TTT TTT TTT TTT TTT+V-3') в соответствии с нормальным распределением случайных нуклеотидов, чтобы повысить охват по 3'-концам. В зависимости от выбранного соотношения между случайными праймерами и поли(dT)-праймерами L1 можно задавать изменение глубины секвенирования на 3'-концевых участках (фиг. 4). Соотношение между случайными праймерами для SDS и специфическими олиго(dT)-праймерами, а также длина праймера и содержание LNA может варьироваться и будет определять степень повышения охвата 3'-концов.
Получали библиотеки методом SDS + лигирования, используя либо только случайные праймеры с остановкой смещения, либо смеси с различным количеством олиго(dT)-праймеров первой нити (SEQ ID No: 11). Полученные библиотеки подвергали секвенированию на NextSeq 500, анализировали данные и составляли графики охвата тела генов по всему транскриптому из картированных ридов с помощью скрипта Python geneBody_coverage, доступного от RSeqC (фиг. 4c). Охват 3'-концов может значительно повышаться при добавлении олиго(dT)-праймеров во время обратной транскрипции.
Кроме того, визуализировали охват генов для примера по эндогенным генам с помощью специального скрипта для оценки охвата отдельных генов. На фиг. 4d представлен охват хозяйственного гена HSP90, полученный по стандартной методике получения библиотек РНК (верхняя панель), с сильно заниженным охватом 5'- и 3'-концов. Напротив, методика SDS + лигирования с титрованием олиго-dT дает улучшение охвата 5'- и 3'-концов (нижняя панель).
Пример 5. Улучшение охвата 5'- и 3'-концов облегчает определение истинных сайтов начала и окончания транскрипции
Получали библиотеки методом SDS + лигирования из истощенной по рибосомной РНК универсальной эталонной РНК человека (Agilent Technologies, кат. № 740000), содержащей контрольную смесь SIRV Set 3 spike-in (Lexogen, кат. № 051.0N), как описано в Примерах 3 и 4. Удаление рибосомной РНК осуществляли с помощью RiboCop (Lexogen, кат. № 037.96) в соответствии с инструкциями производителя. Для сравнения использовали два традиционных метода на одной и той же универсальной эталонной РНК человека, истощенной по рибосомной РНК: набор TruSeq Stranded Total RNA Library Prep Human/Mouse/Rat, Illumina, кат. № 20020596 или 20020597 (= обычный 1) либо набор NEBNext® Ultra™ II directional RNA Library Prep Kit for Illumina®, New England Biolabs, кат. № E7760S (= обычный 2), согласно инструкциям производителя. Полученные библиотеки подвергали секвенированию на NextSeq 500 и анализировали данные. Составляли графики охвата тела гена по всем обнаруженным ERCC из набора SIRV Set 3. На фиг. 5a представлен нормализованный охват по накопленным картированным ридам ERCC для абсолютных положений нуклеотидов относительно известных сайтов начала транскрипции (TSS) и окончания транскрипции (TES), которые оба обозначены пунктирными линиями. Охват на 5'- и 3'-концах значительно повышается для образцов, полученных из библиотек SDS + лигирования, по сравнению с обоими традиционными препаратами библиотек, проявляющими снижение охвата 3'-конца и неточное разрешение 5'-конца.
Кроме того, визуализировали охват генов для примера по эндогенному хозяйственному гену gapdh с помощью специального скрипта для оценки охвата по отдельным генам. На фиг. 5b представлен профиль охвата для gapdh с конденсированной визуализацией интронов. Риды, относящиеся к gapdh (SEQ ID Nos. от 43 до 67), анализировали без отсечения дополнительных и несовпадающих оснований. Нуклеотиды, совпадающие с консенсусной последовательностью (верхний ряд), отмечены черным цветом, а нуклеотиды, отклоняющиеся от аннотированной консенсусной последовательности или полученные при безматричном добавлении, отмечены серым цветом. Исходя из совпадения ридов, наблюдаемого для образцов, полученных из препаратов библиотек SDS + лигирования, можно определить истинные сайты начала транскрипции и заново аннотировать представляющие интерес транскрипты. В примере, представленном на фиг. 5b, TSS был вручную установлен в положении -15 (по отношению к аннотированному положению +1). Точно так же можно заново установить истинные сайты начала и окончания транскрипции и для других представляющих интерес транскриптов, что позволит провести всесторонний анализ полных транскриптов, включая разрешение на уровне отдельных нуклеотидов по истинным TSS для экспериментов по высокопроизводительному NGS. Этого можно добиться, просто используя для получения библиотек метод SDS + лигирования, в отличие от специализированных и более сложных подходов типа методов секвенирования с 5'-захватом (CAGE-Seq) или методов с низкой пропускной способностью типа 5'-RACE (быстрой амплификации концов кДНК).
--->
Перечень последовательностей
SEQUENCE LISTING
<110> LEXOGEN GMBH
<120> Nucleic acid amplification and identification method
<130> R 75980
<150> EP18212743
<151> 2018-12-14
<160> 67
<170> BiSSAP 1.3
<210> 1
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 41
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 1
nnnnnnagat cggaagagca cacgtctgaa ctccagtcac n 41
<210> 2
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 35
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 2
nnnnnnnnnn agatcggaag agcacacgtc tgaan 35
<210> 3
<211> 43
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 43
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 3
nnnnnnnnnn agatcggaag agcgtcgtgt agggaaagag tgn 43
<210> 4
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 34
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 4
nnnnnnnnnn agatcggaag agcgtcgtgt aggn 34
<210> 5
<211> 44
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10,11
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 44
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 5
nnnnnnnnnn nagatcggaa gagcgtcgtg tagggaaaga gtgn 44
<210> 6
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10,11,12
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 45
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 6
nnnnnnnnnn nnagatcgga agagcgtcgt gtagggaaag agtgn 45
<210> 7
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10,11,12
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 36
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 7
nnnnnnnnnn nnagatcgga agagcgtcgt gtaggn 36
<210> 8
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 34
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 8
nnnnnnnnnn agatcggaag agcgtcgtgt aggn 34
<210> 9
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10,11,12
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 36
<223> /mod_base="OTHER"
/note="3' Inverted dT (reverse linkage)"
<400> 9
nnnnnnnnnn nnagatcgga agagcgtcgt gtaggn 36
<210> 10
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<220>
<221> modified_base
<222> 1
<223> /mod_base="OTHER"
/note="5' phosphorylated; a or g or c or t"
<220>
<221> misc_difference
<222> 2,3,4,5,6,7,8,9,10,11,12
<223> /note="a or g or c or t"
<220>
<221> modified_base
<222> 33
<223> /mod_base="OTHER"
/note="g, 3' Spacer C3"
<400> 10
nnnnnnnnnn nntggaattc tcgggtgcca agn 33
<210> 11
<211> 53
<212> DNA
<213> Artificial Sequence
<220>
<223> oligo
<400> 11
gtgactggag ttcagacgtg tgctcttccg atcttttttt tttttttttt ttv 53
<210> 12
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 12
cgatttctaa agggaattcg agctcgcatt ttgaaaattc tatggaagag ctagcatctc 60
tgacgaaaac agcag 75
<210> 13
<211> 68
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 13
cctttgggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgacg 60
aaaaccag 68
<210> 14
<211> 68
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 14
caaaacggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgacg 60
aaaacaac 68
<210> 15
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 15
agtggtggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgacg 60
aaatgc 66
<210> 16
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 16
caaaatggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgacg 60
aaaacagcgt 70
<210> 17
<211> 65
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 17
tcggacggga attcgagctc gcattttgaa aattctatgg aagagctagc atctcttacg 60
aaaac 65
<210> 18
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 18
ggggacggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgaca 60
aaaaca 66
<210> 19
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 19
cccgagggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgacg 60
aaaacggcag aca 73
<210> 20
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 20
aatacaggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgacg 60
aaaacagaga g 71
<210> 21
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 21
caaaatggga attcgagctc gcattttgaa aattctatgg aagagctagc atctctgacg 60
aaaacagcgt 70
<210> 22
<211> 74
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 22
atttggggaa ttcgagctcg cattttgaaa attctatgga agagctagca tctctgacga 60
aaacagcagg cgga 74
<210> 23
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 23
aatggggaat tcgagctcgc attttgaaaa ttctatggaa gagctagcat ctctgacgaa 60
aacagcaatc ggaaa 75
<210> 24
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 24
aaggggaatt cgagctcgca ttttgaaaat tctctggaag agctagcatc tctgacgaaa 60
acagcagaac agaaa 75
<210> 25
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 25
ggggaattcg agctcgcatt ttgaaaatac tatggaagag ctagcatctc tgacgaaaac 60
agcagacgaa aaagt 75
<210> 26
<211> 61
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 26
gggaattcga gctcgcattt tgaaaattct atggaagagc tagcatctct gactactaca 60
g 61
<210> 27
<211> 60
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 27
aagatctcgc attttgaaaa ttctatggaa gagctagcat ctctgacgaa aacagcagaa 60
<210> 28
<211> 74
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 28
cgagctcgca ttttgaaaat tctatggaag agctagcatc tctgacgaaa acagcagacg 60
gaaaaggaga gacc 74
<210> 29
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 29
cgagctcgca ttttgaaaat tctatggaag agctagcatc tctgacgaaa acagcagacg 60
gaaaagtact gacca 75
<210> 30
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 30
cgagctcgca ttttgaaaat tctatggaag agctagcacc tctgacgaaa acagcagacg 60
gaaaaggact gaaaa 75
<210> 31
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 31
cgagctcgca ttttgaaaat tctatggaag agctagcatc tctgacgaaa acagcagacg 60
gaaaagtact gagcc 75
<210> 32
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 32
cgagctcgca ttttgaaaat tctatggaag agctagcacc tctgacgaaa acagcagacg 60
gaaaaggact gaaaa 75
<210> 33
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 33
cgagctcgca ttttgaaaat tctatggaag agctagcatc tctgacgaaa acagcagacg 60
gaaaagtact gactc 75
<210> 34
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 34
cgagctcgca ttttgaaaat tctatggaag agctagcatc tctgacgaaa acagcagacg 60
gaaaagtact gacca 75
<210> 35
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 35
cgagctcgca ttttgaaaat tctatggaag agctagcatc tctgacgaaa acagcagacg 60
gaaaagtaca aaacc 75
<210> 36
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 36
gagctcgcat tttgaaaatt ctatggaaga gctagcatct ctgacgaaaa cagcagacgg 60
aaaagtagct gacca 75
<210> 37
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 37
agctcgcatt ttgaaaattc tatggaagag ctagcatctc tgacgaaaac agcagacgga 60
aaagtactga ccaga 75
<210> 38
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 38
gctcgcattt tgaaaattct atggaagagc tagcatctct gacgaaaaca gcagacggaa 60
aagtacagac ccaac 75
<210> 39
<211> 74
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 39
cgcattttga aaattctatg gaagagctag catctctgac gaaaacagca gacggaaaag 60
tactgaccag ctag 74
<210> 40
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 40
cgcattttga aaattctatg gaagagctag catctctgac gaaaacagca gacggaaaag 60
tactgaccat gca 73
<210> 41
<211> 74
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 41
cgcattttga aaattctatg gaagagctag catctctgac gaaaacagca gacggaaaag 60
tactgaccag ccac 74
<210> 42
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 42
cgcattttga aaattctatg gaagagctag catctctgac gaaaacagca gacggaaaag 60
tactgaccag cat 73
<210> 43
<211> 129
<212> DNA
<213> Artificial Sequence
<220>
<223> consensus sequence
<400> 43
ataaattgag cccgcagcct cccgcttcgc tctctgctcc tcctgttcga cagtcagccg 60
catcttcttt tgcgtcgcca gccgagccac atcgctcaga caccatgggg aaggtgaagg 120
tcggagtca 129
<210> 44
<211> 104
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 44
acgtgtgctc gtcactacct ccccgggtgc tctctgctcc tcctgttcga cagtcagccg 60
catcttcttt tgcgtcgcca gccgagccac atcgctcaga cacc 104
<210> 45
<211> 129
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 45
gaaaattgag cccgcagcct cccgcttcgc tctctgctcc tcctgttcga cagtcagccg 60
catcttcttt tgcgtcgcca gccgagccac atcgctcaga caccatgggg aaggtgaagg 120
tcggagtca 129
<210> 46
<211> 122
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 46
aaaatgcatt agaggaactg taaaaatctg ctcctccgtt cgacagtcag ccgcatcttc 60
ttttgcgtcg ccagccgagc cacatcgctc agacaccatg gggaaggtga aggtcggagt 120
ca 122
<210> 47
<211> 122
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 47
ctagaggaga ttggccaacg agattcactg gactcctgtt cgacagtcag ccgcatcttc 60
ttttgcgtcg ccagccgagc cacatcgctg agacaccatg gggaaggtga aggtcggagt 120
ca 122
<210> 48
<211> 118
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 48
ttttctgaac gctctggccg ctctgctcct cctgttcgac agtcagccgc ctcttcgttt 60
gcgtcgccag ccgagccaca tagctcagac accaagggga aggtgaaggt cggagtca 118
<210> 49
<211> 114
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 49
accgagcacc agctttctcc gacgccggga agtcgcagtc agccgcatct tcttttgcgt 60
cgccagccga gccacatcgc tcagacacca tggggaaggt gaaggtcgga gtca 114
<210> 50
<211> 112
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 50
aacgtgtgct ggctctctgc tcctcctgtt cgacagtcag ccgcatcttc ttttgcgtcg 60
ccagccgagc cacatcgctc agacaccatg gggaaggtga aggtcggagt ca 112
<210> 51
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 51
ttgctttggg ctctctgctc ctcctgttcg acagtcagcc gcatcttctt ttgcgtcgcc 60
agccgagcca catcgctcag acaccatggg gaaggtgaag gtcggagtca 110
<210> 52
<211> 106
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 52
gatgggctct ctgctcctcc tgttcgacag tcagccgcat cttcttttgc gtcgccagcc 60
gagccacatc gctcagacac catggggaag gtgaaggtcg gagtca 106
<210> 53
<211> 106
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 53
atagggctct ctgctcctcc tgttcgacag tcagccgcat cttcttttgc gtcgccagcc 60
gagccacatc gctcagacac catggggaag gcgaaggtcg gagtca 106
<210> 54
<211> 106
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 54
atctggctct ctgctcctcc tgttcgacag tcagccgcat cttcttttgc gtcgccagcc 60
gagccacatc gctgagacac catggggaag gtgaaggtcg gagtca 106
<210> 55
<211> 106
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 55
acgtggctct ctgctcctcc tgttcgacag tcagccgcat cttcttttgc gtcgccagcc 60
gagccacatc gctcagacac catggggaag gtgaaggtcg gagtca 106
<210> 56
<211> 106
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 56
acgtggctct ctgctcctcc tgttcgacag tcagccgcat cttcttttgc gtcgccagcc 60
gagccacatc gctcagacac catggggaag gtgaaggtcg gagtca 106
<210> 57
<211> 97
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 57
ttgcggctct ctgctcctcc tgttcgacag tcagccgcat cttcttttgc gtcgccagcc 60
gagccacatc gctcagacac catggggaag cggaaca 97
<210> 58
<211> 82
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 58
gggggctctc tgctcctcct gttcgacagt cagccgcatc ttcttttgcg tcgccagccg 60
agccacatcg ctcagacccc ac 82
<210> 59
<211> 105
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 59
aatggctctc tgctcctcct gttcgacagt cagccgcatc ttcttttgcg tcgccagccg 60
agccacatcg ctcagacacc atggggaagg tgaaggtcgg agtca 105
<210> 60
<211> 105
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 60
atcggctctc tgctcctcct gttcgacagt cagccgcatc ttcttttgcg tcgccagccg 60
agccacatcg ctcagacacc atggggaagg tgaaggtcgg agtca 105
<210> 61
<211> 105
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 61
attggctctc tgctcctcct gttcgacagt cagccgcatc ttcttttgcg tcgccagccg 60
agccacatcg ctcagacacc atggggaagg tgaaggtcgg agtca 105
<210> 62
<211> 57
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 62
gttggctctc tgctcctcct gttcgacagt cagccgcatc ttcttttgca atcgcca 57
<210> 63
<211> 104
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 63
atggctctct gctcctcctg ttcgacagtc agccgcatct tcttttgcgt cgccagccga 60
gccacatcgc tcagacacca tggggaaggt gaaggtcgga gtca 104
<210> 64
<211> 104
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 64
ttggctctct gctcctcctg ttcgacagtc agccgcatct tcttttgcgt cgccagccga 60
gccacatcgc tcagacacca tggggaaggt gaaggtcgga gtca 104
<210> 65
<211> 82
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 65
ggggctctct gctcctcctg ttcgacagtc agccgcatct tcttttgcgt cgccagccga 60
gccacatcgc tcagaacagc ca 82
<210> 66
<211> 104
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 66
gtggctctct gctcctcctg ttcgacagtc agccgcatct tcctttgcgt cgccagccga 60
gccacatcgc tcagacacca tggggaaggt gaaggtcgga gtca 104
<210> 67
<211> 103
<212> DNA
<213> Artificial Sequence
<220>
<223> sequencing read
<400> 67
gggctctctg ctcctcctgt tcgacagtca gccgcatctt cttttgcgtc gccagccgag 60
ccacatcgct cagacaccat ggggaaggtg aaggtcggag tca 103
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ОПРЕДЕЛЕНИЕ НУКЛЕИНОВЫХ КИСЛОТ ПУТЕМ АМПЛИФИКАЦИИ, ОСНОВАННОЙ НА ВСТРАИВАНИИ В ЦЕПЬ | 2014 |
|
RU2694976C1 |
| Рекомбинантный штамм, продуцирующий L-лизин, способы его конструирования и его применение | 2020 |
|
RU2832108C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ГЕНЕТИЧЕСКИХ ФАКТОРОВ РИСКА РАЗВИТИЯ ДЕМЕНЦИЙ АЛЬЦГЕЙМЕРОВСКОГО ТИПА НА ОСНОВЕ ГИДРОГЕЛЕВОГО МАТРИЧНОГО БИОЧИПА | 2022 |
|
RU2795795C1 |
| МИКРООРГАНИЗМ, СОДЕРЖАЩИЙ ВАРИАНТ LysE, И СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТЫ С ЕГО ИСПОЛЬЗОВАНИЕМ | 2021 |
|
RU2805078C1 |
| НОВЫЙ ПРОМОТОР И СПОСОБ ПОЛУЧЕНИЯ ЖЕЛАЕМОГО ВЕЩЕСТВА С ЕГО ИСПОЛЬЗОВАНИЕМ | 2020 |
|
RU2794946C1 |
| БИБЛИОТЕКИ ДЛЯ СЕКВЕНИРОВАНИЯ НОВОГО ПОКОЛЕНИЯ | 2014 |
|
RU2698125C2 |
| Нуклеиновокислотная последовательность для обнаружения наличия трансгенного трансформанта сои DBN9004 в биологическом образце, набор, содержащий такую последовательность, и способ такого обнаружения | 2017 |
|
RU2743397C2 |
| ГЕНОМНАЯ ИНЖЕНЕРИЯ | 2014 |
|
RU2764757C2 |
| ГЕНОМНАЯ ИНЖЕНЕРИЯ | 2021 |
|
RU2812848C2 |
| ПОЛНОГЕНОМНЫЕ БИБЛИОТЕКИ ОТДЕЛЬНЫХ КЛЕТОК ДЛЯ БИСУЛЬФИТНОГО СЕКВЕНИРОВАНИЯ | 2018 |
|
RU2770879C2 |

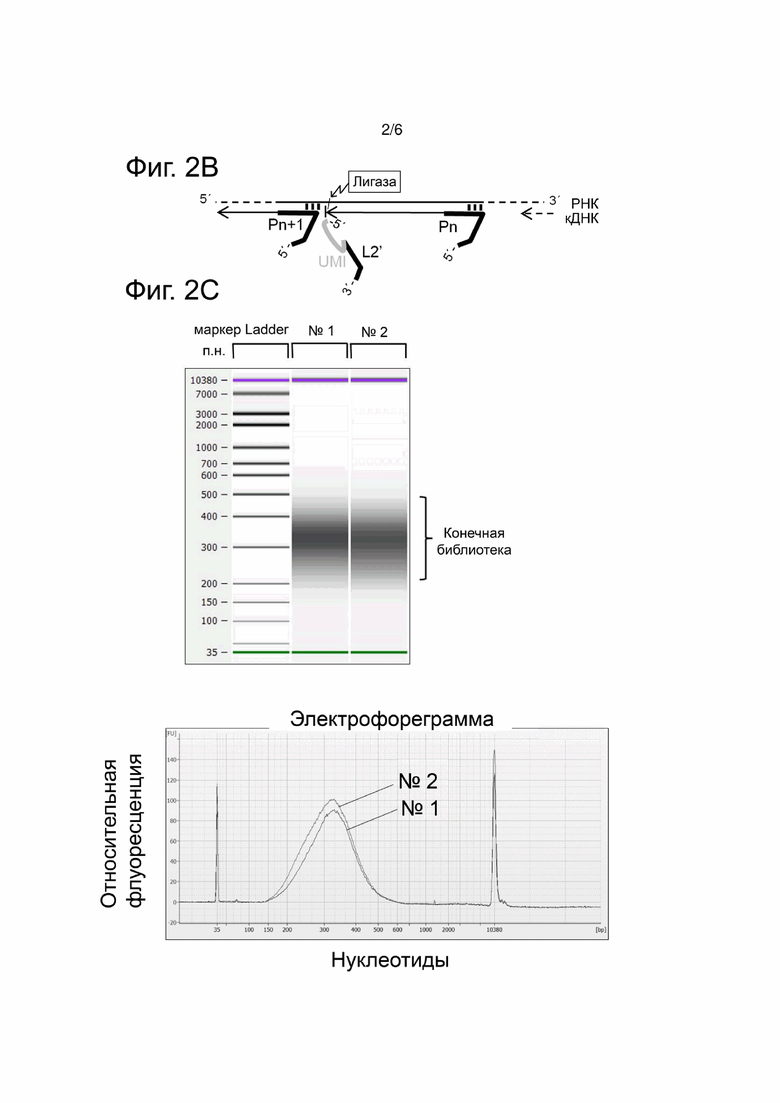




Изобретение относится к биотехнологии. Описан способ получения меченых амплификационных фрагментов нуклеиновой кислоты-матрицы. Обеспечивают матрицу нуклеиновой кислоты. Отжигают по меньшей мере два олигонуклеотидных праймера с матрицей нуклеиновой кислоты, где по меньшей мере один нижележащий праймер представляет собой стоппер элонгации. Осуществляют элонгацию по меньшей мере двух олигонуклеотидных праймеров специфичным для матрицы образом. Реакция элонгации останавливается при достижении продуктом элонгации 5'-конца матрицы нуклеиновой кислоты или олигонуклеотидного праймера как стоппера элонгации нуклеиновой кислоты, загибридизованного на матрице нуклеиновой кислоты ниже продукта элонгации. Обеспечивают множество из по меньшей мере двух адаптерных нуклеиновых кислот, где указанные адаптеры из множества содержат разные идентификационные последовательности на своем 5'-конце, причем данные идентификационные последовательности не гибридизуются с олигонуклеотидным праймером, который является стоппером элонгации, или с матрицей. Лигируют адаптерную нуклеиновую кислоту по её 5'-концу с 3'-концом продукта элонгации, причем для лигирования продукты элонгации гибридизируют с матрицей, получая при этом меченый амплификационный фрагмент. Также описан набор, содержащий по меньшей мере два олигонуклеотидных праймера, способных гибридизироваться с нуклеиновой кислотой-матрицей и запускать реакцию элонгации на своем 3'-конце, где по меньшей мере один праймер представляет собой стоппер элонгации, множество из по меньшей мере двух адаптерных нуклеиновых кислот, где указанные адаптеры из множества содержат разные идентификационные последовательности на своем 5'-конце, причем такие идентификационные последовательности не гибридизуется с олигонуклеотидными праймерами, предпочтительно где адаптерная нуклеиновая кислота связывается, гибридизуется или же не связывается или не гибридизуется с олигонуклеотидным праймером, обратную транскриптазу и лигазу олигонуклеотидов. Изобретение расширяет арсенал средств для амплификации нуклеиновых кислот. 2 н. и 13 з.п. ф-лы, 5 ил., 5 пр.
1. Способ получения меченых амплификационных фрагментов нуклеиновой кислоты-матрицы, включающий стадии:
обеспечения такой матрицы нуклеиновой кислоты,
отжига по меньшей мере двух олигонуклеотидных праймеров с данной матрицей нуклеиновой кислоты, где по меньшей мере один нижележащий праймер представляет собой стоппер элонгации,
элонгации по меньшей мере двух олигонуклеотидных праймеров специфичным для матрицы образом, получая при этом продукт элонгации, причем такая реакция элонгации останавливается при достижении продуктом элонгации (a) 5′-конца матрицы нуклеиновой кислоты или (b) олигонуклеотидного праймера как стоппера элонгации нуклеиновой кислоты, загибридизованного на матрице нуклеиновой кислоты ниже продукта элонгации,
обеспечения множества из по меньшей мере двух адаптерных нуклеиновых кислот, где указанные адаптеры из множества содержат разные идентификационные последовательности на своем 5′-конце, причем данные идентификационные последовательности не гибридизуются с олигонуклеотидным праймером который является стоппером элонгации или с матрицей,
лигирования адаптерной нуклеиновой кислоты по её 5'-концу с 3'-концом продукта элонгации, причем для лигирования продукты элонгации гибридизируют с матрицей, получая при этом меченый амплификационный фрагмент.
2. Способ по п. 1, при этом, когда продукт элонгации достигает 5'-конца матрицы нуклеиновой кислоты, нуклеотидная полимераза получает возможность добавлять нематричные нуклеотиды к продукту элонгации, предпочтительно за счет терминальной трансферазной активности полимеразы, и/или предпочтительно при этом добавляется от 1 до 15 нематричных нуклеотидов по меньшей мере в 70% продуктов элонгации.
3. Способ по п. 1 или 2, при этом на стадии лигирования обеспечивается и используется по меньшей мере 10, более предпочтительно по меньшей мере 50 адаптерных нуклеиновых кислот с различными идентификационными последовательностями.
4. Способ по любому из пп. 1-3, при этом идентификационная последовательность представляет собой случайную последовательность.
5. Способ по любому из пп. 1-4, при этом используется по меньшей мере 9, более предпочтительно по меньшей мере 49 оликонуклеотидных праймеров как стопперов элонгации, которые имеют разные последовательности отжига для гибридизации с матрицей и поэтому могут гибридизоваться с различными участками матрицы нуклеиновой кислоты.
6. Способ по п. 5, при этом последовательность отжига представляет собой случайную последовательность.
7. Способ по любому из пп. 1-6, при этом адаптерная(ые) нуклеиновая(ые) кислота(ы) связывается(ются), гибридизуется(ются) или же не связывается(ются) или не гибридизуется)ются) с олигонуклеотидным праймером(ами) который(ые) является(ются) стоппером(ами) элонгации, а предпочтительно, когда адаптерная(ые) нуклеиновая(ые) кислота(ы) связывается(ются) или гибридизуется(ются) с олигонуклеотидным праймером(ами) который(ые) является(ются) стоппером(ами) элонгации, то идентификационная последовательность не зависит от последовательности отжига с олигонуклеотидного праймера(ов) который(ые) является(ются) стоппером(ами) элонгации для гибридизации праймера(ов) который(ые) является(ются) стоппером(ами) с матрицей.
8. Способ по любому из пп. 1-7, при этом матрица представляет собой РНК, а для элонгации предпочтительно используется обратная транскриптаза.
9. Способ по любому из пп. 1-8, при этом каждый олигонуклеотидный праймер содержит универсальную амплификационную последовательность и/или адаптерная нуклеиновая кислота содержит универсальную амплификационную последовательность адаптера.
10. Способ по любому из пп. 1-9, при этом олигонуклеотидный праймер содержит последовательность отжига для посадки на матрицу, которая включает последовательность олиго(T) для отжига с последовательностью олиго(A) в матрице, причем предпочтительно такая последовательность олиго(T) содержит один или несколько якорных 3'-нуклеотидов, отличных от последовательности олиго(T).
11. Способ по любому из пп. 1-10, при этом реакция лигирования проводится в присутствии краудинг агента, предпочтительно полимера или содержащего полимер соединения типа полиалкилгликоля, предпочтительно ПЭГ, октоксинола или Triton X, или полисорбата, предпочтительно Tween, и/или олигонуклеотидный праймер содержит один или несколько модифицированных нуклеотидов, повышающих температуру плавления отжигаемой последовательности при отжиге с матрицей.
12. Способ по любому из пп. 1-11, при этом используется по меньшей мере две, а предпочтительно по меньшей мере 10 адаптерных нуклеиновых кислот, содержащих разные идентификационные последовательности, при этом образуются по меньшей мере два, а предпочтительно по меньшей мере 10 различных меченых амплифицированных фрагментов, необязательно включающий амплификацию меченых амплифиированных фрагментов, а также включает сборку тех последовательностей амплификационных фрагментов, которые являются уникальными, причем для идентификации уникальных амплификационных фрагментов используются метки.
13. Набор для выполнения способа по любому из пп. 1-12, содержащий:
по меньшей мере два олигонуклеотидных праймера, способных гибридизироваться с нуклеиновой кислотой-матрицей и запускать реакцию элонгации на своем 3′-конце, где по меньшей мере один праймер представляет собой стоппер элонгации,
множество из по меньшей мере двух адаптерных нуклеиновых кислот, где указанные адаптеры из множества содержат разные идентификационные последовательности на своем 5'-конце, причем такие идентификационные последовательности не гибридизуется с олигонуклеотидными праймерами, предпочтительно где адаптерная нуклеиновая кислота связывается, гибридизуется или же не связывается или не гибридизуется с олигонуклеотидным праймером,
обратную транскриптазу и лигазу олигонуклеотидов.
14. Набор по п. 13, содержащий по меньшей мере 10, более предпочтительно по меньшей мере 50 адаптерных нуклеиновых кислот с разными идентификационными последовательностями.
15. Набор по п. 13 или 14, при этом по меньшей мере один олигонуклеотидный праймер содержит последовательность отжига для гибридизации с матрицей, которая включает последовательность олиго(T) для отжига с последовательностью олиго(A) в матрице, причем предпочтительно такая последовательность олиго(T) содержит один или несколько якорных 3′-нуклеотидов, отличных от последовательности олиго(T).
| WO 2013038010 A2, 21.03.2013 | |||
| WO 2014152155 A1, 25.09.2014 | |||
| СПОСОБ АМПЛИФИКАЦИИ НУКЛЕИНОВЫХ КИСЛОТ | 2013 |
|
RU2603253C2 |
Авторы
Даты
2024-01-12—Публикация
2019-12-13—Подача