Область техники, к которой относится изобретение
Изобретение относится к молекулярной биологии, микробиологии и медицине и обеспечивает видоспецифическую идентификацию шести бактериальных и двух вирусных возбудителей пневмонии человека.
Уровень техники
Инфекционная пневмония является одним из наиболее распространенных инфекционных заболеваний и важной причиной смерти, особенно у детей до 5 лет и взрослых старше 65 лет. Основной причиной пневмонии являются вирусные и бактериальные агенты (Musherand D.M., Thorner A.R. Community-acquired pneumonia, The New England Journal of Medicine, 2014; 371 (7): 619-1628). В случае вирусного поражения легочной ткани пневмония может развиться не только как результат первичной вирусной инфекции, но также вследствие присоединения вторичной инфекции бактериальной природы. В ряде случаев наблюдают смешанную инфекцию вирусно-бактериальной этиологии (Aliberti S., Kауе K.S. The changing microbiologic epidemiology of community-acquired pneumonia. Postgrad. Med. 2013; 125 (6): 31-42; Зайцев A.A., Щеголев A.B. Диагностика и лечение тяжелых поражений легких при гриппе A(H1N1/09): практические рекомендации Военно-медицинский журнал. 2016; 337 (3): 39-46; Tarsia P., Aliberti S., Pappalettera М., Blasi F. Mixed community-acquired lower respiratory tract infections. Current Infectious Disease Reports. 2007; 9(1): 14-20). Ряд бактериальных патогенов, способных вызывать воспаление легких достаточно широк (Musherand D.M., Thorner A.R. Community-acquired pneumonia, The New England Journal of Medicine, 2014; 371 (7): 619-1628). Чаще всего в качестве инфекционных агентов выступают внеклеточные бактерии такие, как Streptococcus pneumoniae, Haemophilus influenzae и Staphylococcus aureus. От 1 до 7% тяжелых случаев пневмонии вызывают другие бактерии, например Legionella pneumophila (Cilloniz С., Torres A., Niederman М. et al. Community acquired pneumonia related to intracellular pathogens. Intensive Care Medicine. 2016; 42(9): 1374-1386).
Способность быстро выявлять и идентифицировать различных инфекционных возбудителей является важной задачей при точной постановке диагноза, в первую очередь при сезонных и спорадических вспышках заболевания, но также и в случае появления новых инфекционных агентов или биотерроризме (Yin Y1, Wunderink RG2. MERS, SARS and other coronaviruses as causes of pneumonia. Respirology. 2018; 23(2): 130-137. doi: 10.1111/resp.13196; Zhang Z.W., Zhou Y.M., Zhang Y., Guo Y., Tao S.C., et al. Sensitive detection of SARS coronavirus RNA by a novel asymmetric multiplex nested RT-PCR amplification coupled with oligonucleotide microarray hybridization. Methods Mol Med. 2005; 114: 59-78; Petric M., Comanor L, Petti C.A. Role of the laboratory in diagnosis of influenza during seasonal epidemics and potential pandemics. J Infect Dis. 2006; 194 Suppl 2: S98-110; Lessa F.C., Gould P.L., Pascoe N.. Erdman D.D., Lu X., et al. Health care transmission of a newly emergent adenovirus serotype in health care personnel at a military hospital in Texas, 2007. J Infect Dis 2009; 200: 1759-1765). Правильная идентификация патогена позволяет определить необходимость дополнительных диагностических исследований, выбрать антивирусную или антибактериальную терапию, выбрать методы контроля за течением инфекционного процесса (Mahony J.B., Blackhouse G., Babwah J., Smieja M., Buracond S., et al. Cost Analysis of Multiplex PCR Testing for Diagnosing Respiratory Virus Infections. J Clin Microbiol. 2009; 47(9):2812-7; Falsey A.R., Murata Y., Walsh E.E. Impact of rapid diagnosis on management of adults hospitalized with influenza. Arch Intern Med. 2007; 167: 354-360). Стандартное микробиологическое тестирование требует нескольких дней для первичной идентификации патогенного возбудителя, а некоторые агенты не могут быть выявлены только микробиологическими методами (Hatchette TF, Bastien N, Berry J, Booth TF, Chernesky M, et al. The limitations of point of care testing for pandemic influenza: what clinicians and public health professionals need to know. Can J Public Health. 2009; 100: 204-207).
Микробная этиология инфекционной пневмонии. Являясь инфекционной патологией, пневмония связана с внедрением в респираторный тракт и развитием внутриальвеолярных экссудаций (Чучалин А.Г., Синопальников А.И., Козлов Р.С., и соавт. Внебольничная пневмония у взрослых. Практические рекомендации по диагностике, лечению и профилактике (пособие для врачей). Клин микробиол антимикроб химиотер 2010; 12: 186-225) под воздействием жизнедеятельности патогенных микроорганизмов, среди которых представлены бактерии, вирусы, грибки и простейшие. Внутри большого перечня описанных возбудителей пневмонии можно выделить ряд патогенов, которые наиболее часто являются причиной заболевания.
Лидером среди «типичных» возбудителей (Mccracken, G. Н. (2000). Etiology and treatment of pneumonia) являются бактерии вила Streptococcus pneumoniae. Во всем мире стрептококк признан наиболее распространенным патогеном, который вызывает внебольничную пневмонию, и обычно проявляется острыми симптомами инфекции нижних дыхательных путей. На долю S. pneumoniae приходится 30-50% всех случаев выявления пневмонии установленной этиологии (Welte Т., Torres A., Nathwani D. Clinical and economic burden of community-acquired pneumonia among adults in Europe. Thorax 2010). Пневмококк (S. pneumoniae) имеет несколько факторов вирулентности, наиболее важным из которых является полисахаридная капсула (Mitchell, А. М., & Mitchell, Т. J. (2010). Streptococcus pneumoniae: virulence factors and variation. Clinical Microbiology and Infection, 16(5), 411-418). Различия в составе капсулы S. pneumoniae приводят к существованию различных типов или серотипов (Geno KА, Gilbert GL, Song JY, Skovsted IС, Klugman KP, Jones C, Konradsen HB, Nahm MH. 17 June 2015. Pneumococcal capsules and their types: past, present, and future. Clin Microbiol Rev), часть из которых вовлечены в большинство инвазивных инфекций.
К числу актуальных бактериальных возбудителей также причисляют энтеробактерию Klebsiella pneumoniae, она же клебсиелла пневмонии или палочка Фриндлендера - грамотрицательная, заключенная в капсулу и неподвижная бактерия. Согласно статистике, ее патогенность обуславливается наличием ряда факторов, способных привести к развитию вирулентности и устойчивости к антибиотикам. В качестве основного фактора здесь выделяют полисахаридную капсулу, позволяющую бактериям избегать механизмов защиты против инфекций и ликвидации организмом-носителем (Li, В., Zhao, Y., Liu, С, Chen, Z., & Zhou, D. (2014). Molecular pathogenesis of Klebsiella pneumoniae. Future Microbiology, 9(9), 1071-1081).
Другими нередкими возбудителями пневмонии выступают золотистый стафилококк Staphylococcus aureus и гемофильная палочка Haemophilus influenzae.
Стафилококки, часто колонизирующие кожу и слизистые оболочки людей и животных, обладают способностью быстро приобретать детерминанты устойчивости к противомикробным препаратам. Среди многих видов стафилококков S. aureus демонстрирует самый высокий патогенный потенциал. Особенно важны MRSA штаммы, поскольку во всем мире они являются ведущей причиной инфекций, связанных с оказанием медицинской помощи (Teruyo Ito (2009). Classification of Staphylococcal Cassette Chromosome mec (SCCmec): Guidelines for Reporting Novel SCCmec Elements. Antimicrobial Agents and Chemotherapy, 53(12), 4961-4967).
Штаммы H. influenzae можно разделить на инкапсулированные (с шестью различными серотипами a-f на основе капсулярных полисахаридов) и неинкапсулированные (или нетипируемые с помощью традиционной реакции агглютинации со специальной сывороткой, они же NTHi штаммы). NTHi вызывают широкий спектр внебольничных инфекций слизистой оболочки, включая ВП, у пожилых пациентов, пациентов с сопутствующими респираторными заболеваниями или пациентов с рецидивирующей пневмонией (Forstner, С, Rohde, G., Rupp, J., Schuette, H., Ott, S. R., Hagel, S., … Suttorp, N. (2016). Community-acquired Haemophilus influenzae pneumonia - New insights from the CAPNETZ study. Journal of Infection, 72(5), 554-563).
Среди «атипичных» возбудителей наиболее часто выделяют подвижные палочковидные бактерии вида Legionella pneumophila. Среди многих агентов, обычно вызывающих бактериальную пневмонию у людей, легионеллы не являются частью нормальной человеческой флоры, а приобретаются из источников окружающей среды. Легионеллезная пневмония составляет от 2% до 15% всех внебольничных пневмоний, требующих госпитализации в Европе и Северной Америке (Muder RR, Yu VL, Fang GD. Community-acquired Legionnaires' disease. Semin Respir Infect. 1989 Mar;4(1):32-9).
Проблемой в лечении пневмонии выступает вид Pseudomonas aeruginosa вследствие своей высокой вирулентности и наличия множественных механизмов устойчивости к различным антимикробным средствам (О.Л. Палковский, Л.И. Новогран, А.В. Шершнев проблемы терапии нозокомиальной пневмонии, вызванной P. aeruginosa (обзор литературы) // Проблемы здоровья и экологии. 2010. №3 (25)). Чаще всего развитие устойчивости к противомикробным препаратам у P. aeruginosa объясняется мутациями хромосомных генов, однако описывается также приобретение факторов устойчивости (например, генов, кодирующих β-лактамазы и ферменты, модифицирующие аминогликозиды) посредством горизонтального переноса генов (Rossolini, G. М., and Mantengoli, Е. (2005). Treatment and control of severe infections caused by multiresistant Pseudomonas aeruginosa. Clin. Microbiol. Infect. 11(Suppl. 4), 17-32).
Меньшую актуальность в этиологии пневмонии представляют Mycoplasma pneumoniae и Chlamydophila pneumoniae.
M. pneumoniae является важным патогеном дыхательных путей человека и во время эпидемической активности уступает только S. pneumoniae как наиболее частый этиологический агент ВП. Дети и молодые люди наиболее часто поражаются М. pneumoniae, однако никакая возрастная группа не является защищенной, а также нередко описываются и повторные инфекции. С. pneumoniae является генетически, морфологически и антигенно типичным членом семейства Chlamydiaceae, и представляет собой облигатный внутриклеточный патоген, клеточная стенка которого содержит липополисахариды, похожие на грамотрицательные бактерии. В последние годы данный вид все чаще встречается респираторным патогеном как у детей, гак и у взрослых (L. Barry Seltz MD, …Leslie L. Barton MD, in Kendig & Chernick's Disorders of the Respiratory Tract in Children (Eighth Edition), 2012. Pages 493-505).
Несмотря на распространенность бактериальных инфекций, все большее эпидемиологическое значение приобретают на сегодняшний день исследования, касающиеся пневмонии, вызванной вирусными агентами (Galvan, J. М., Rajas, О., & Aspa, J. (2015). Review of Non-Bacterial Infections in Respiratory Medicine: Viral Pneumonia. Archivos de bronconeumologia, 51(11), 590-597). По оценкам, ежегодно в мире происходит порядка 100 миллионов случаев заражения вирусной пневмонией (Ruuskanen, О.; Lahti, Е.; Jennings, L.C.; Murdoch, D.R. Viral pneumonia. Lancet 2011, 377, 1264-1275).
К вирусам, связанным с внебольничной пневмонией у детей и взрослых, относятся респираторно-синцитиальный вирус человека, риновирусы, гриппы А, В и С, парагрипп, а также коронавирусы разных типов. В условиях пандемии, вызванной SARS-CoV-2, важную роль в сдерживании темпов распространения и развития болезни имеет детекция возбудителя на ранних стадиях и его дифференциация от бактериальных патогенов в сходной клинической картине.
Ниже рассмотрены основные современные подходы к идентификации возбудителей пневмонии.
Этиологическая диагностика пневмонии
Хотя микробиологическая диагностика пневмонии имеет фундаментальное значение для обеспечения надлежащей антибактериальной терапии, позволяя тем самым снизить количество летальных исходов, микробная диагностика пневмонии достигается менее чем в 50% случаев. По этой причине большинству пациентов, поступающих в медицинские учреждения с диагнозом пневмонии, на первом этапе назначается эмпирическая антибактериальная терапия, чтобы избежать задержки в лечении и повышение летальности (Garau, J.; Baquero, F.; Perez-Trallero, E.; Perez, J.L.; Martin-Sanchez, A.M.; Garcia-Rey, C.; Martin-Herrero, J.E.; Dal-Re, R. Factors impacting on length of stay and mortality of community-acquired pneumonia. Clin. Microbiol. Infect. 2008. 14, 322-329).
Текущая диагностика патогенов на сегодняшний день в первую очередь основана на анализе культур респираторных образцов, собранных с использованием таких подходов как аспирация трахеи и бронхоальвеолярный лаваж (БАЛ). После забора материала проводится количественная оценка колониеобразующих единиц и определение чувствительности к антибиотикам для выявления патогенных профилей изолятов (Torres, А., & El-Ebiary, М. (2000). Bronchoscope BAL in the diagnosis of ventilator-associated pneumonia. Chest, 117(4 Suppl 2), 198S-202S). Основным недостатком таких методов является задержка в получении результатов, связанная с необходимостью культивирования, в том числе на средах, содержащих антибиотики.
Другие стратегии для обнаружения микробных агентов, особенно атипичных, связаны с применением молекулярно-биологических методов, обладающих большей чувствительностью, точностью и специфичностью. К таким, прежде всего, относится полимеразная цепная реакция и различные ее модификации, а также серологические тесты и тестирование на антигены в биологических образцах.
Прямое исследование респираторных образцов
Для оказания своевременной помощи и последовательного ведения пациента с пневмонией большое значение имеет микроскопическое исследование респираторных образцов. Идентификация патогенов в высококачественных пробах, собранных непосредственно с места инфекции или из обычно асептического места (например, крови), обеспечивает надежное выявление вероятных возбудителей болезни.
Микроскопическое исследование можно легко применить к жидкостям БАЛ для исследования внеклеточных и внутриклеточных бактерий после окрашивания красителями Май-Грюнвальда Гимза и Грама. Идентификация грибков также может быть получена микроскопически после окрашивания реактивом Шиффа (Bousbia, S., Raoult, D., & La Scola, В. (2013). Pneumonia pathogen detection and microbial interactions in polymicrobial episodes. Future Microbiology, 8(5), 633-660). Другие красители, такие как окраска Циля-Нильсена для микобактерий, используются для идентификации конкретных микроорганизмов. Также микроскопия позволяет провести количественную оценку бактериальной нагрузки путем прямого подсчета, что, принимая во внимания пороговые значения патогенной нагрузки, дает возможность отличить простую колонизацию от легочной инфекции.
К преимуществам данного подхода в определении возбудителя относится его быстрота, позволяющая определять начальные этапы терапии с применением антимикробных препаратов. Среди недостатков - низкие чувствительность и точность.
Культуральный метод исследования
Культуральное исследование, предполагающее посев клинических образцов на селективные и дифференциально-диагностические среды и их последующую идентификацию с помощью различных методов (биохимические тесты, время-пролетная масс-спектрометрия) до настоящего времени остается основным методом диагностики пневмонии, вызванной S. pneumoniae, Н. influenzae, энтеробактериями, S. aureus и P. aeruginosa, и обладает высокой специфичностью (МУК 4.2.189004. Методические указания. Определение чувствительности микоорганизмов к антибактериальным препаратам. Клин, микробиол. и антимикроб, химиотер. 2004; 6 (4): 306-359).
Культуральное исследование респираторных образцов с целью выявления Legionella spp., несмотря на специфичность метода, в рутинной практике не рекомендуется, так как является дорогостоящим и трудоемким, а чувствительность его существенно варьирует в зависимости от исследуемого материала (низкая при исследовании мокроты) и квалификации персонала.
Несмотря на тот факт, что культуральный метод является золотым стандартом в определении возбудителей пневмонии, к его слабым сторонам относятся сложность в осуществлении необходимых для выращивания колоний операций и длительность в получении результатов (от 48 часов), вследствие чего затруднено его применение в случаях заболеваний с высоким темпом развития патологического процесса.
Времяпролетная масс-спектрометрия
В последние десятилетия в качестве сильного современного инструмента микробиологической диагностики выделяется матричная времяпролетная масс-спектрометрия с лазерной десорбцией и ионизацией.
MALDI-TOF MS широко используется в качестве мощного метода анализа для быстрой и высокоточной идентификации микроорганизмов в лабораториях. Данная технология может предоставить альтернативный метод диагностики патогенов, включая грамположительные и грамотрицательные бактерии, грибки и другие микроорганизмы, идентификация которых с помощью классических инструментов порой затруднена. MALDI-TOF MS с минимальными затратами на расходные материалы способен дать воспроизводимые, видоспецифичные паттерны, которые в основном не зависят от культуры, условий роста или выбора среды, что выгодно отличает его от стандартного культурального метода.
Микробные образцы, нанесенные на пластину-мишень, покрываются матричным раствором, который кристаллизуется с образцом и лизирует вегетативные организмы. Трудными для лизиса являются некоторые грамположительные бактерии, микобактерии, дрожжи и плесень, и часто требуют предварительной обработки сильной органической кислотой или механического лизиса. После помещения в прибор образцы ионизируются короткими лазерными импульсами, образуя ионы газовой фазы с минимальной фрагментацией (то есть проводится «мягкой ионизацией») с последующим ускорением частиц в вакууме посредством электрического поля. Время, необходимое каждой частице для достижения детектора зависит от ее массы и заряда. После того, как все обильные белки в образце обнаружены масс-спектрометром, создается спектр, уникальный для анализируемого организма (Dingle, Т.С, & Butler-Wu, S.М. (2013). MALDI-TOF Mass Spectrometry for Microorganism Identification. Clinics in Laboratory Medicine, 33(3), 589-609).
Одним из основных недостатков MALDI-TOF MS является то, что преимущества в отношении быстрой идентификации микроорганизмов ограничены необходимостью выделения чистых культур. Показано также, что подготовка образца и тип используемой среды могут влиять на его воспроизводимость анализа (Seng Р, Rolain JM, Fournier РЕ, La Scola В, Drancourt М, Raoult D. MALDI-TOF-mass spectrometry applications in clinical microbiology. Future Microbiol. 5(11), 1733-1754 (2010)). Серьезным недостатком также является сложность и дороговизна используемого оборудования (Williams TL, Andrzejewski D, Lay JO, Musser SM. Experimental factors affecting the quality Pneumonia pathogen detection & microbial interactions Review 656 Future Microbiol. (2013) 8(5) future science group and reproducibility of MALDI TOF mass spectra obtained from whole bacteria cells. J. Am. Soc. Mass Spectrom. 14(4), 342-351 (2003)).
Молекулярно-биологические методы
Применение молекулярной биологии в лабораториях в качестве рутинного инструмента сильно изменило лабораторные диагностические процедуры. На сегодняшний день молекулярные тесты широко применяются в качестве инструмента для повседневной идентификации микроорганизмов, поскольку дают много преимуществ в микробной диагностике.
Прежде всего с применением данных методов достигается более чувствительное обнаружение патогенов, даже когда те присутствуют в малых количествах, что отличительно выделяет высокоточную идентификация по сравнению с обычным выделением культуры. Во-вторых, молекулярные методы особенно полезны для идентификации некультивируемых или трудно растущих микроорганизмов, к которым относятся представители родов Chlamydia, Legionella, Mycobaterium и Mycoplasma, или тех, что являются строго внутриклеточными (к примеру, респираторные вирусы). Наконец, использование молекулярных подходов в обнаружении патогенов не сопряжено с жизнеспособностью микроорганизмов или атмосферными условиями. Так, с их помощью анаэробные бактерии при респираторных инфекциях удобнее обнаруживать с использованием молекулярно-биологических методов, а не с помощью анализа культур.
В рутинной микробной диагностике особенно широкое применение находят методы, основанные на полимеразной цепной реакции и ее модификациях, часто дополненные последующим секвенированием. Другие методы на основе ПЦР, такие как количественная ПЦР и ПЦР с обратной транскрипцией (ОТ-ПЦР), значительно улучшают диагностику пневмонии, поскольку они являются быстрыми, специфическими и более чувствительными системами обнаружения патогенов.
Полиморфизм длины рестрикционных фрагментов и флуоресцентная гибридизация in situ (или метод FISH) также используются для идентификации бактерий.
Другие молекулярные технологии, такие как биологические чипы и мультиплексная ПЦР, значительно повышают способность этих молекулярных методов одновременно воздействовать на большое количество патогенов, облегчая чувствительное обнаружение и точную идентификацию множества возбудителей при полимикробных инфекциях.
ПЦР как инструмент диагностики
К преимуществам ПЦР как диагностического инструмента, основанного на амплификации определенных фрагментов генома возбудителей, относятся высокая чувствительность, скорость и специфичность. ПЦР-анализ позволяет выявить патологические агенты в различных биологических субстратах как при хронических, так и при острых формах заболевания. Помимо этого, постановка ПЦР позволяет идентифицировать не только типичных возбудителей пневмонии, но и атипичных, а также обладающих резистентностью к антимикробным препаратам, что способствует выбору антибактериальной терапии.
Наиболее частой мишенью амплификации в данном случае выступает последовательность 16S рибосомальной РНК (16S рРНК). Гены, кодирующие данную последовательность, содержат гипервариабельные области, уникальные для различных видов бактерий, по которым последние могут быть идентифицированы.
Тем не менее, для некоторых бактерий последовательность 16S рДНК недостаточно точно определяет видовой уровень. Фактически, некоторые бактерии, различающиеся по фенотипу и морфологии, могут иметь сходные последовательности в рибосомных генах (к примеру, Escherichia и Shigella или некоторые стрептококки). Вследствие этого проводится анализ генотипа на видоспецифичные сегменты: ген gyrB для L. pneumophila, cpsA для S. pneumoniae, SCCmec для метициллинрезистентных стафилококков или внутренние транскрибируемые спейсеры. Благодаря такому уточненному анализу можно успешно отнести патоген к нужным уровням вида, штамма или линии, а также дифференцировать вирулентные патогены от авирулентных.
ДНК-биочипы
Биочипы являются комплексной платформой для одновременного определения нескольких генов и находят применение в научной, клинической и экологической областях благодаря своей способности размещать на весьма компактной поверхности до сотен или тысяч биологических зондов в относительно небольшом пространстве и объеме реакции.
Технология биочипов восходит к традиционным твердофазным анализам вроде дот-блот-гибридизации или иммуноферментных анализов, которые десятилетиями использовались в лабораториях. В твердофазном анализе молекулы прикреплены к твердой подложке и представляют собой так называемые зонды, - с их помощью в образце определяется наличие целевых молекул, вступая во взаимодействие с которыми зонд должен демонстрировать как можно более высокую специфичность и сродство. Зонды могут быть продуктами ПЦР, олигонуклеотидами, плазмидами или бактериальными искусственными хромосомами для анализа геномов и транскриптомов, а также белками, антителами, аптамерами ДНК/РНК или углеводами (Dufva, М. (Ed.). (2009). DNA Microarrays for Biomedical Research. Methods in Molecular Biology).
Молекулярные зонды иммобилизуются на поверхности мембран или пластинок из стекла, пластика, полупроводника или металла, В гелевых биочипах зонды иммобилизуются или в слое полиакриламидного геля толщиной порядка 20 микрон, нанесенного на специально обработанную поверхность стекла, или в полусферических гидрогелевых ячейках с диаметром около 100 микрон (как в чипах, разработанных в Институте молекулярной биологии РАН). Иммобилизация осуществляется за счет образования ковалентных связей при облучении ультрафиолетовым излучением или с помощью химических реагентов. Иммобилизуемые зонды наносятся на поверхность с помощью игольчатых растворов механического робота или с помощью технологи струйного принтера (Биочипы для медицинской диагностики. В.Р. Чечеткин, Д.В. Прокопенко, А.А. Макаров, А.С. Заседателев. Российские нанотехнологии 1 (1-2), 13-28, 2006).
Анализируемые молекулы в растворе метятся с помощью флуоресцентной или радиоактивной метки до или после гибридизации, в следствие чего их можно впоследствии обнаружить по определенному сигналу. Свойства флуоресцентного красителя не должны сильно зависеть от состава анализируемых молекул (А/Т или G/C для ДНК) и температуры раствора. В настоящее время наиболее типичными красителями, использующимися в технологии биочипов, являются флуорофоры Су3 и Су5.
В настоящем изобретении предлагается способ, основанный на мультиплексном ПЦР-анализе с предварительным использованием обратной транскрипции (ОТ) с проведением ферментативных реакций непосредственно на биологическом микрочипе. Способ предназначен для одновременного выявления в исследуемом образце шести бактериальных и двух вирусных возбудителей пневмонии по их ДНК/РНК. Способ в представленном виде не имеет отечественных и зарубежных аналогов и описывается впервые.
Раскрытие сущности изобретения
Целью изобретения является создание способа быстрого выявления в образце возбудителей инфекционной пневмонии человека на биологических микрочипах. В качестве образцов могут быть использованы: клинический материал, лизат клеточной культуры, различные физиологические жидкости (мокрота при откашливании, бронхоальвеолярный лаваж (БАЛ), аспираты из трахеи), соскобы из гортани и носоглотки.
Важным этапом разработки изобретения явился выбор видоспепифичных генетических мишеней и конструирования соответствующих специфичных праймеров.
Изобретение предлагает использование способа мультиплексной ПЦР с обратной транскрипцией (мультиплексная ОТ-ПЦР) для быстрого выявления шести социально значимых бактериальных и двух вирусных возбудителей пневмонии человека на биологических микрочипах. Сконструированные праймеры для мультиплексной ОТ-ПЦР обладают видовой специфичностью к возбудителям. Для каждой пары видоспецифичых праймеров применяется также один или два вложенных праймера, иммобилизованных на твердой подложке (биологическом микрочипе). Именно по удлинению этих праймеров, благодаря ковалентному введению метки, производится детекция результата обнаружения патогена. Введение метки осуществляется путем применения флуоресцентно-меченных трифосфатов дезоксинуклеозидов непосредственно в процессе проведения ферментативных реакций. Таким образом, после проведения ОТ-ПЦР множество меченых молекул оказываются ковалентно связанными с иммобилизованным праймером. Это позволяет проводить «жесткую» отмывку биочипа после проведения реакции, удаляя все непрореагировавшие компоненты, включая не вступившие в реакцию свободные меченые дезоксинуклеозиды. Такой способ существенно повышает чувствительность анализа на биочипах, поскольку избавляет от фонового сигнала при детекции результата.
Чувствительность способа, определенная раститровкой ДНК каждого из анализируемых возбудителей, составляет от 102 до 103 копий геномной ДНК на реакционную пробирку, в том числе при одновременном введении в смесь нескольких матриц.
Данное изобретение обеспечивает способ идентификации шести социально значимых бактериальных возбудителей пневмонии человека: Staphilococcus aureus, Streptococcus pneumoniae, Haemophilus influenza, Legionella pneumophila, Klebsiella pneumoniae, Pseudomonas aeruginosa, а также двух вирусных: SARS-CoV-2 и Грипп А.
Краткое описание фигур и таблиц
Фигура 1. Схема анализа с применением иммобилизованных праймеров. Литерами f и r обозначены прямой и обратный фланкирующие праймеры, литерами r1 и r2 - вложенные иммобилизованные праймеры (содержат аминогруппы на 5'-конце), dU* - меченный цианиновым красителем дезоксиуридин, встраиваемый в процессе удлинения праймера.
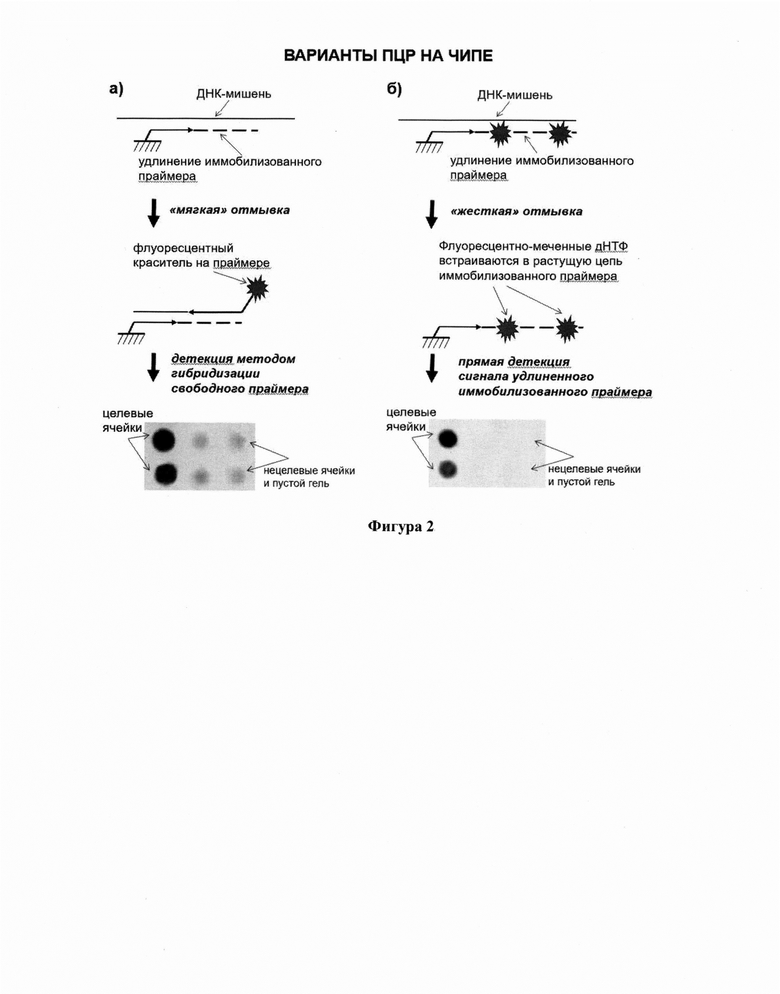
Фигура 2. Варианты ПЦР на чипе (схема): а) стандартный вариант ПЦР с использованием свободного (не иммобилизованного) флуоресцентно-меченного праймера в смеси, б) предлагаемый изобретением вариант ПЦР с использованием флуоресцентно-меченного трифосфата дезоксиуридина.
Фигура 3. Специализированный биологический микрочип для видового определения возбудителей пневмонии методом мультиплексной ОТ-ПЦР. а - Схема: М - флуоресцентный маркер для автоматического наложения сетки при программном обеспечении расчета интенсивности сигнала ячеек; Gel - ячейка пустого геля; Sta - Staphylococcus aureus (здесь и во всех последующих случаях цифры "1" и "2" обозначают использование двух различных праймеров внутри последовательности, фланкируемой краевыми праймерами, литера "а" обозначает использование праймера в концентрации, в 2 раза меньшей, чем в ячейке чипа без литеры - это необходимо для повышения надежности автоматического анализа); Str - Streptococcus pneumoniae, Leg - Legionella pneumophila, Hae - Haemophilus influenzae, Pse - Pseudomonas aeruginosa, Kle - Klebsiella pneumoniae, Cov - SARS-CoV-2, Inf - вирус гриппа A; Conl -внутренний контроль обратной транскрипции (резервный; в настоящее время не используется), Соn2 - внутренний контроль ПЦР. б-и - результаты определения вида (примеры) соответственно: St. aureus, S. pneumoniae, H. influenzae, SARS-CoV-2, вирус гриппа А, K. Pneumoniae, L. Pneumophila, Ps. aeruginosa. Для удобства приведены фото с инвертированными сигналами (негативы).
Осуществление изобретения
Изобретение нацелено на идентификацию шести социально значимых бактериальных возбудителей пневмонии человека: Staphilococcus aureus, Streptococcus pneumoniae, Haemophilus influenza, Legionella pneumophila, Klebsiella pneumoniae, Pseudomonas aeruginosa, а также двух вирусных: SARS-CoV-2 и Грипп А, в различных биологических материалах.
Обработка образца перед проведением анализа осуществляется любым из имеющихся методов выделения ДНК из биологических образцов, например - фенол-хлороформным методом (Molecular Cloning, Volume 3, 3rd edition, by Joseph Sambrook and David W. Russell. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, USA, 2001) или методом с использованием силикатных сорбентов (Cady N.C., Stelick S., Batt C.A. Nucleic acid purification using microfabricated silicon structures. Biosens Bioelectron. 2003 Oct 30:19(1):59-66). но не ограничивается ими.
Подбор олигонуклеотидных праймеров SEQ ID NO: 1-31 (Перечень последовательностей) выполняется на основе систематизации последовательностей, кодирующих видоспецифичные гены возбудителей. Конструирование высокоспецифичных праймеров осуществляется на основе анализа литературных источников и последовательностей генов в базах данных (GenBank, Silva). Далее проводится множественное выравнивание (Multiplex Alignment) последовательностей генов-мишеней. Каждому выравниванию присваивается консенсусная последовательность. Выравнивания проводятся с помощью специализированного программного обеспечения, например BioEdit Sequence Alignment Editor (версия 7.1.9) по алгоритму ClustalW, консенсусная последовательность присваивалась со значением параметра «threshold frequency» равным 100%. Далее, используя соответствующее программное обеспечение, например Oligo v.6.3 (Molecular Biology Insights Inc., США) или Fast PCR (http://www.biocenter.helsinki.fi/bi/ Programs/fastpcr.htm), проводится расчет температуры отжига праймеров и, варьируя их длину, добиваются того, чтобы разброс температур отжига праймеров внутри набора не превышал 3-4°С. При подборе праймеров избегают таких последовательностей, которые способны формировать вторичные структуры типа шпильки с высокими температурами плавления. Специфичность подобранных праймеров проверятся путем поиска по базам данных нуклеотидных последовательностей (GenBank, Silva) с использованием программного обеспечения, поддерживающего алгоритм BLAST (например, www.ncbi.nlm.nih.gov/BLAST)
Включение флуоресцентной метки осуществляется в удлиняющиеся в процессе ферментативных реакций иммобилизованные на биологических микрочипах праймеры, представленные последовательностями SEQ ID NO: 17-31 (Перечень последовательностей). Для этого используются флуоресцентно-меченные трифосфаты дезоксиуридина. Принципиальная схема ферментативного удлинения иммобилизованного праймера с одновременным включением метки представлена на Фигуре 1. Видно, что используются праймеры, фланкирующие регион для идентификации возбудителя, при этом на чипе находятся иммобилизованные «вложенные» праймеры, содержащие аминогруппу. Детекция осуществляется с помощью специализированных флуоресцентных микроскопов для биочипов (чип-детекторов), производимых коммерчески и не составляющих предмет настоящего изобретения. При этом производится полное удаление реакционной смеси после прохождения ОТ-ПЦР и отмывка биологического микрочипа. После этого получаемый флуоресцентный сигнал обусловлен лишь теми флуоресцентными метками, которые встроились в процессе ОТ-ПЦР в последовательности иммобилизованных праймеров (для ферментативного введения метки используется трифосфат дезоксиуридина, в С5-положение пиримидинового цикла которого включен краситель Су5). Преимущество такого метода перед классическим, где используется меченый праймер, заключается в том, что отмывку можно производить «жестко», удаляя все компоненты реакционной смеси. В классическом случае, когда детекция производится путем гибридизации флуоресцентно-меченного праймера, такая отмывка привела бы к полной потере сигнала по причине разрушения водородных связей между флуоресцентно-меченным праймером и иммобилизованным удлиненным праймером (Фигура 2а). Невозможность применения «жесткой» отмывки (кипячения в присутствии детергента, ПАВ) приводит к повышенному нецелевому (фоновому) сигналу из-за оставшихся несовершенных дуплексов флуоресцентно-меченных праймеров и неспецифично сорбировавшихся в гидрогеле флуоресцентно-меченных праймеров. В случае предлагаемого способа производится полное удаление всех компонентов смеси после проведения ферментативных реакций, при этом удаляются и непрореагировавшие флуоресцентно-меченные трифосфаты дезоксиуридина. Таким образом, фоновый сигнал уменьшается вплоть до базового фонового сигнала чип-детектора, тем самым резко увеличивая чувствительность способа (фирура 2б).
Идентификация возбудителя в образце осуществляется с помощью проведения мультиплексной ОТ-ПЦР на биологическом микрочипе. В изобретении применяются биочипы, изготовленные по технологии и ИМБ РАН (Москва, Россия) с иммобилизованными на них оригинальными праймерами для проведения обратной транскрипции и последующей амплификациии ДНК (методом ПЦР) для РНК-содержащих вирусов, либо только амплификации ДНК - для бактериальных возбудителей пневмонии.
Принципиальная схема чипа приведена на Фигуре 3а. Из Фигуры 3а видно, что каждый специфический иммобилизованный праймер имеет четко заданные координаты на биочипе, что позволяет при интерпретации результата точно сопоставлять праймеры, дающие флуоресцентный сигнал, с соответствующими им патогенными агентами. Из той же фигуры видно, что на чипе применен положительный контроль прохождения реакции (ячейки Соn2 и Соn2а), отсутствие сигнала с ячеек которого говорит о том, что идентификация проведена некорректно и определение возбудителя необходимо произвести повторно. На чипе присутствуют резервные ячейки Con1 и Con1а, сигнал с которых в норме отсутствует.
Примеры обнаружения в образце различных возбудителей инфекционной пневмонии представлены на Фигуре 3б-и. Идентификация возбудителя производится визуально в соответствии со схемой расположения специфичных к различным возбудителям иммобилизованных праймеров на биологическом микрочипе (Фигура 3а).
Далее изобретение будет проиллюстрировано примерами, которые предназначены для обеспечения лучшего понимания сущности заявленного изобретения, но не должны рассматриваться как ограничивающие данное изобретение.
Пример 1. Набор специфических олигонуклеотидных праймеров, используемых в способе обнаружения видоспецифичных генов шести бактериальных и двух вирусных возбудителей пневмонии человека.
Праймеры для проведения полимеразной цепной реакции синтезируют на автоматическом синтезаторе 394 DNA/RNA synthesizer (Applied Biosystems, США). Список праймеров приведен в Перечне последовательностей (SEQ ID NO: 1-16).
Пример 2. Выделение ДНК из клинических образцов.
1. В пробирки добавляют 20 мкл универсального сорбента (набор "ДНК-сорб-АМ", ЦНИИ Эпидемиологии Федеральной службы Роспотребнадзора, Россия), после чего вносят по 300 мкл лизирующего раствора.
2. В пробирки с добавленным сорбентом и лизирующим раствором вносят по 100 мкл клинического образца.
3. Содержимое пробирок тщательно перемешивают на вортексе и инкубируют в течении 5 мин при температуре 65°С в термостате. После окончания инкубации пробирки повторно перемешивают на вортексе и оставляют при комнатной температуре на 2 мин.
4. Сорбент осаждают в пробирках с помощью центрифугирования при 10000 об/мин в течение 30 секунд. Не захватывая сорбент, удаляют надосадочную жидкость.
5. К осадку добавляют по 1 мл отмывочного раствора и перемешивают на вортексе до полного ресуспендирования сорбента.
6. Повторяют пункт 5.
7. Пробирки с открытой крышкой помещают в термостат с температурой 65°С на 5-10 мин для подсущивания сорбента.
8. В пробирки добавляют по 100 мкл ТЕ-буфера (10 мМ Трис-НСl, 1 мМ ЭДТА), рН 8,0 для элюции суммарной ДНК и РНК. Перемешивают пробирки на вортексе до полного ресуспендирования сорбента.
9. Центрифугируют пробирки при 12000 об/мин в течении 1 мин. Надосадочную жидкость используют для проведения обратной транскрипции и ПЦР.
Пример 3. Проведение обратной транскрипции и полимеразной цепной реакции для амплификации фрагментов видоспецифичных генов шести бактериальных и двух вирусных возбудителей пневмонии человека.
К 29 мкл реакционной смеси вносят 1 мкл образца, полученного в Примере 2.
Мультиплексная ОТ-ПЦР в иммобилизованной фазе. Для проведения ОТ-ПЦР используют обратную транскриптазу MMLV и другие компонентов набора "PEBEPTA-L" (ФГУН ЦНИИЭ Роспотребнадзора, Россия) и Hot Start Taq-полимеразу ("Thermo Scientific") в буфере, соответствующем набору для Hot Start Taq-полимеразы с добавлением дитиотрейтола.
1. К аликвоте смеси, содержащей природные dNTP (по 400 мкМ каждого) и праймеры (5-10 мкМ прямого и 0.5-1.0 мкМ обратного для каждого из возбудителей (различались для разных пар праймеров в результате проведенной оптимизации)) добавляют в качестве флуоресцентного субстрата полимеразы Cy5-dUTP в концентрации 8 мкМ (Shershov V.E., Lapa S.A., Kuznetsova V.E., Spitsyn M.A., Guseinov Т.О., Polyakov S.A., Stomahin A.A., Zasedatelev A.S., Chudinov A.V. (2017) Comparative study of novel fluorescent cyanine nucleotides: hybridization analysis of labeled PCR products using a biochip.J. Fluoresc. 27, 2001-2016), а также специальную ДНК-матрицу-положительный контроль прохождения реакции.
2. Смесь помещают на чип и герметизируют с помощью Frame-Seal 25 mkl ("Bio-Rad", США).
3. Проводят реакции обратной транскрипции и амплификации (ОТ-ПЦР) на ДНК-амплификаторе для in situ ПЦР TGradient Thermocycler ("Biometra", США). При этом ОТ проводят в течение 30 мин при 42°С, после чего проводят ПЦР в следующих условиях: 95°С в течение 3 мин (начальная денатурация); 36 циклов по 20 с при 95°С, 30 с при 64°С и 40 с при 72°С; завершающая инкубация в течение 5 мин при 72°С.
4. Определяют чувствительность ОТ-ПЦР на чипе раститровкой ДНК/РНК анализируемых образцов в интервале 101-105 копий на реакционный объем (25 мкл). Данный пункт осуществляют только на стадии оптимизации способа.
5. 10 мкл реакционной смеси, полученной после проведения ОТ-ПЦР на биологическом микрочипе, используют для электрофоретического контроля образовавшихся продуктов для каждого из возбудителей.
Пример 4. Проведение электрофоретического разделения продуктов амплификации (только на стадии оптимизации способа).
1. Готовят 1 × ТАЕ-буфер путем пятидесятикратного разведения 50 × ТАЕ буфера (2 М Трис, 1 М уксусная кислота, 50 мМ ЭДТА, рН 8,4) в объеме, достаточном для заполнения электрофорезной камеры и приготовления геля (1000 мл).
2. Добавляют к 1 × ТАЕ буферу агарозу в количестве, необходимом для получения 2% раствора, и нагревают в микроволновой печи до полного расплавления агарозы.
3. Охлаждают смесь до 50°С.
4. Добавляют к раствору агарозы бромистый этидий до конечной концентрации 0,5 мкг/мл и осторожно перемешивают.
5. Полученный раствор выливают в кювету для геля и равномерно распределяют по объему кюветы.
6. В кювету вставляют гребенку в вертикальном положении так, чтобы ее зубцы не доставали до дна примерно 1,5 мм.
7. Оставляют кювету с агарозным гелем на 30 мин при комнатной температуре для застывания геля. Далее, аккуратно удаляют гребенку и помещают кювету с гелем в электрофорезную камеру, содержащую необходимое количество 1 × ТАЕ буфера.
8. По 10 мкл образов, полученных в Примере 3, смешивают с 2 мкл буфера для нанесения (10 мМ Трис-HCl, рН 7.8, 40% глицерин, 40 мМ ЭДТА, 0,01% бромфеноловый синий, 0,01% ксиленцианол) и аккуратно вносили в лунки геля.
9. Проводят электрофорез при градиенте напряженности 1,5 В на 1 см геля в течение 30 мин.
10. Извлекают гель из электрофорезной камеры и просматривают в УФ-свете на транс-иллюминаторе. Результаты документируют путем фотографирования геля и сохранения полученных изображений.
Пример 5. Интерпретация результатов электрофоретического разделения продуктов амплификации.
1. Изображения, полученные в Примере 4 интерпретируют следующим образом. По наличию полосы оптической плотности с длиной в диапазоне 200-400 п.н. в дорожке на агаразном геле делается вывод о наличии соответствующего гена в исследуемом образце, соответствующем конкретному возбудителю.
Пример 6. Интерпретация результатов идентификации шести бактериальных и двух вирусных возбудителей пневмонии методом ОТ-ПЦР на биологическом микрочипе.
1. Считывание флуоресцентного сигнала с чипа после удлинения иммобилизованных праймеров осуществляют с применением анализатора "Чип-детектор" (ИМБ, Россия). Интенсивность полученных сигналов определяют с помощью программного обеспечения "ImaGeWare" ver. 3.50 (ИМБ, Россия).
2. Интерпретацию наличия в образце того или иного возбудителя осуществляют визуально, согласно схеме, представленной на Фигуре 3а.
--->
Перечень последовательностей
<210> 1
<211> 23
<212> cpsB-f1
<213> Streptococcus pneumoniae
<220>
<223> праймер прямой
<400> 1
TTGATGTAGATGACGGTCCCAAG (SEQIDNO: 1)
<210> 2
<211> 23
<212> cpsB-r1
<213> Streptococcus pneumoniae
<220>
<223> праймер обратный
<400> 2
TATATCTCTG CGCCATAAGC AAT (SEQ ID NO: 2)
<210> 3
<211> 23
<212> ebpS-f1
<213> Staphylococcus aureus
<220>
<223> праймер прямой
<400> 3
ACTCGACTGA GGATAAAGCG TCT (SEQ ID NO: 3)
<210> 4
<211> 23
<212> ebpS-r1
<213> Staphylococcus aureus
<220>
<223> праймер обратный
<400> 4
CCTCCAAATA TCGCTAATGC ACC (SEQ ID NO: 4)
<210> 5
<211> 23
<212> fucK-f1
<213> Haemophilus influenzae
<220>
<223> праймер прямой
<400> 5
TGCTCACTCA ACGCTTAACT GGT (SEQ ID NO: 5)
<210> 6
<211> 23
<212> fucK-r1
<213> Haemophilus influenzae
<220>
<223> праймер обратный
<400> 6
TTCTGGGCTA ATGGTGTACG TAA (SEQ ID NO: 6)
<210> 7
<211> 23
<212> oprL-f1
<213> Pseudomonas aeruginosa
<220>
<223> праймер прямой
<400> 7
GCGTGCGATC ACCACCTTCT ACT (SEQ ID NO: 7)
<210> 8
<211> 23
<212> oprL-r1
<213> Искусственная последовательность
<220>
<223> Pseudomonas aeruginosa
<400> 8
TTCTTCAGCT CGACGCGACG GTT (SEQ ID NO: 8)
<210> 9
<211> 22
<212> sidA-f1
<213> Legionella pneumophila
<220>
<223> праймер прямой
<400> 9
TTCCACTGGT GGGTGGGTTT TG (SEQ ID NO: 9)
<210> 10
<211> 23
<212> sidA-r1
<213> Legionella pneumophila
<220>
<223> праймер обратный
<400> 10
TCATGTTGGA GTTCTATGGC ACG (SEQ ID NO: 10)
<210> 11
<211> 23
<212> rmpA-f2
<213> Klebsiella pneumoniae
<220>
<223> праймер прямой
<400> 11
GGACTACCTC TGTTTCATAT TAC (SEQ ID NO: 11)
<210> 12
<211> 20
<212> rmpA-r2
<213> Искусственная последовательность
<220>
<223> Klebsiellapneumoniae
<400> 12
CCCCATTTTT CAGTAGGCAT (SEQ ID NO: 12)
<210> 13
<211> 23
<212> E-forw
<213> SARS-CoV-2
<220>
<223> праймер прямой
<400> 13
TCGTTTCGGA AGAGACAGGT ACG (SEQ ID NO: 13)
<210> 14
<211> 23
<212> E-rev
<213> SARS-CoV-2
<220>
<223> праймер обратный
<400> 14
AAGACTCACG TTAACAATAT TGC (SEQ ID NO: 14)
<210> 15
<211> 24
<212> М2-forw
<213> Influenza A
<220>
<223> праймер прямой
<400> 15
CACGCTCACC GTGCCCAGTG AGCG (SEQ ID NO: 15)
<210> 16
<211> 24
<212> М2-rev
<213> Influenza A
<220>
<223> праймер обратный
<400> 16
TATATGAGGC CCATGCAACT GGCA (SEQ ID NO: 16)
<210> 17
<211> 23
<212> R3
<213> Streptococcus pneumoniae
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 17
NH2-TATATCTCTG CGCCATAAGC AAT (SEQ ID NO: 17)
<210> 18
<211> 23
<212> R5
<213> Streptococcus pneumoniae
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 18
NH2-GCAATGACTAAATCATCTGCCAC (SEQ ID NO: 18)
<210> 19
<211> 23
<212> R1
<213> Staphylococcus aureus
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 19
NH2-CCTCCAAATA TCGCTAATGC ACC (SEQ ID NO: 19)
<210> 20
<211> 26
<212> R2
<213> Staphylococcus aureus
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 20
NH2-GGTAACAATA CTTTGGCCAT GCCACC (SEQ ID NO: 20)
<210> 21
<211> 23
<212> R8
<213> Haemophilus influenzae
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 21
NH2-TTCACCTGCA TAACGCATAG GAG (SEQ ID NO: 21)
<210> 22
<211> 23
<212> R9
<213> Haemophilus influenzae
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 22
NH2-CATAACGCAT AGGAGGGAAA TGG (SEQ ID NO: 22)
<210> 23
<211> 22
<212> R10
<213> Pseudomonas aeruginosa
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 23
NH2-GTAGCGACCG GACGCTCTTT AС (SEQ ID NO: 23)
<210> 24
<211> 22
<212> R11
<213> Pseudomonas aeruginosa
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 24
NH2-GCTCTTTACC ATAGGAAACC AG (SEQ ID NO: 24)
<210> 25
<211> 26
<212> R6
<213> Legionella pneumophila
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 25
NH2-TCATTATATT TATCATTGTT TGGCTC (SEQ ID NO: 25)
<210> 26
<211> 23
<212> R7
<213> Legionella pneumophila
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 26
NH2-ACGTTTCGCT ACAAGATCTA TAA (SEQ ID NO: 26)
<210> 27
<211> 21
<212> R13
<213> Klebsiella pneumoniae
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 27
NH2-GGCGTCAGAT ACAGGACGGC T (SEQ ID NO: 27)
<210> 28
<211> 22
<212> R14
<213> Klebsiella pneumoniae
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 28
NH2-CCACTCCACC GGCAGTGCTCAC (SEQ ID NO: 28)
<210> 29
<211> 23
<212> E-f-n
<213> SARS-CoV-2
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 29
NH2-CTTGCTTTCG TGGTATTCTT GCT (SEQ ID NO: 29)
<210> 30
<211> 23
<212> E-r-n
<213> SARS-CoV-2
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 30
NH2-CACACAATCG AAGCGCAGTA AGG (SEQ ID NO: 30)
<210> 31
<211> 23
<212> INF-F1
<213> InfluenzaA
<220>
<223> праймер иммобилизованный вложенный обратный
<400> 31
NH2-CTCACCGTGC CCAGTGAGCG AGG (SEQ ID NO: 31)
<---


Изобретение относится к молекулярной биологии, микробиологии. Описан способ идентификации генов возбудителей пневмонии человека, шести бактериальных: Staphilococcus aureus, Steptococcus pneumonia, Haemophilus influenza, Legionella pneumophila, Klebsiella pneumonia, Pseudomonas aeruginosa и двух вирусных: SARS-CoV-2, Грипп А, включающий: а) амплификацию фрагментов видоспецифичных генов шести бактериальных и двух вирусных возбудителей пневмонии человека путем проведения обратной транскрипции и полимеразной цепной реакции с набором неиммобилизованных специфических олигонуклеотидных праймеров в реакционном объеме биологического микрочипа, представленных последовательностями SEQ ID NO: 1-16, и иммобилизованных на том же биологическом микрочипе, представленных последовательностями SEQ ID NO: 17-31; б) отмывку биологического микрочипа; в) анализ картины ОТ-ПЦР на биологическом микрочипе с помощью Чип-детектора; г) визуальную интерпретацию результатов анализа. Представлен биологический микрочип, используемый в способе идентификации генов шести бактериальных и двух вирусных возбудителей пневмонии человека по п.1, с гидрогелевыми ячейками с иммобилизованными в них высокоспецифичными праймерами, представленными последовательностями SEQ ID NO: 17-31. Способ обладает высокой скоростью проведения анализа при одновременном обнаружении ряда патогенных агентов, устойчив к перекрестной контаминации благодаря закрытой реакционной камере на биологическом микрочипе, совместим со стандартными in situ амплификаторами, а также с детекторами флуоресцентного сигнала, разработанными для биологических микрочипов (чип-детекторы). 2 н.п. ф-лы, 3 ил., 6 пр.
1. Способ идентификации генов шести социально значимых бактериальных и двух вирусных возбудителей пневмонии человека, включающий:
а) амплификацию фрагментов видоспецифичных генов шести социально значимых бактериальных и двух вирусных возбудителей пневмонии человека путем проведения обратной транскрипции и полимеразной цепной реакции с оригинальным набором специфических олигонуклеотидных праймеров на одном биологическом микрочипе, представленным последовательностями SEQ ID NO: 1-16;
б) отмывку биологического микрочипа;
в) анализ картины ОТ-ПЦР на биологическом микрочипе с помощью Чип-детектора;
г) визуальную интерпретацию результатов анализа.
2. Биочип, используемый в способе идентификации генов шести социально значимых бактериальных и двух вирусных возбудителей пневмонии человека, с гидрогелевыми ячейками с иммобилизованными в них высокоспецифичными праймерами, представленными последовательностями SEQ ID NO: 17-31.
| С | |||
| А | |||
| ЛАПА, Е | |||
| С | |||
| КЛОЧИХИНА и др | |||
| МУЛЬТИПЛЕКСНАЯ ПЦР ДЛЯ ВЫЯВЛЕНИЯ БАКТЕРИАЛЬНЫХ ВОЗБУДИТЕЛЕЙ ИНФЕКЦИОННОЙ ПНЕВМОНИИ, БИООРГАНИЧЕСКАЯ ХИМИЯ, 2020, том 46, номер 5, стр.550-552 | |||
| ДИАГНОСТИКА ИНФЕКЦИОННОГО ЗАБОЛЕВАНИЯ ДЫХАТЕЛЬНЫХ ПУТЕЙ С ИСПОЛЬЗОВАНИЕМ ОБРАЗЦОВ КРОВИ | 2012 |
|
RU2660352C2 |
| WO 2012157750 A1, 22.11.2012 | |||
| Е.А | |||
| КОЛОСКОВА, Б.А | |||
| РАМАЗАНОВА, Современная эпидемиологическая и микробиологическая характеристика пневмококковой | |||
Авторы
Даты
2022-11-29—Публикация
2021-12-06—Подача